p53
The p53 tumor suppressor is a 53 kDa nuclear phosphoprotein of 393 amino acids that is encoded by the TP53 gene (20 kb with 11 exons and 10 introns) and characterized by the presence of several structural and functional domains, including a N-terminus, a central core domain, a C-terminal region, a strongly basic carboxyl-terminal regulatory domain, a nuclear localization signal sequence and three nuclear export signal sequence. The p53 is considered as a major “guardian of genome” for its activities in a wide range of cellular events, including cell-cycle regulation, induction of apoptosis, gene amplification, DNA recombination, chromosomal segregation and cellular senescence.
Produkte für p53
- Bestell-Nr. Artikelname Informationen
-
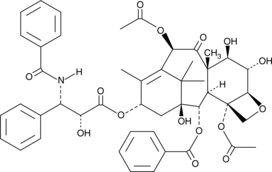
GC42586
6α-hydroxy Paclitaxel
6α-Hydroxy Paclitaxel ist ein primÄrer Metabolit von Paclitaxel. 6α-hydroxy Paclitaxel behÄlt eine zeitabhÄngige Wirkung auf die organischen Anionen-transportierenden Polypeptide 1B1/SLCO1B1 (OATP1B1) mit Ähnlicher Hemmkraft wie Paclitaxel, wÄhrend es keine zeitabhÄngige Hemmung von OATP1B3 mehr zeigte. 6α-Hydroxy-Paclitaxel kann fÜr die Krebsforschung verwendet werden.
-
GC41643
9(Z),11(E),13(E)-Octadecatrienoic Acid
9(Z),11(E),13(E)-Octadecatrienoic Acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.

-
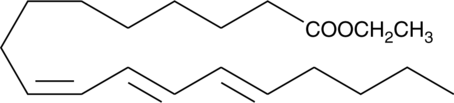
GC40785
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
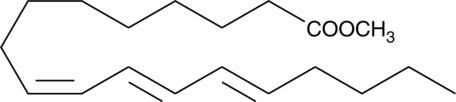
GC40710
9(Z),11(E),13(E)-Octadecatrienoic Acid methyl ester
9Z,11E,13E-octadecatrienoic acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
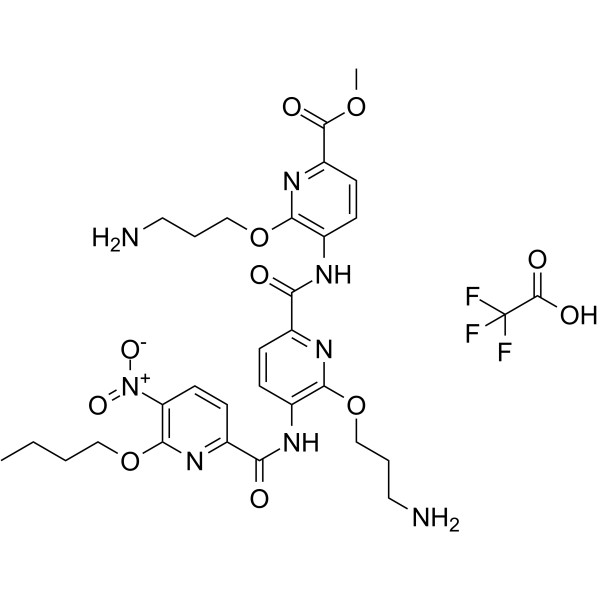
GC65880
ADH-6 TFA
ADH-6 TFA ist eine Tripyridylamid-Verbindung. ADH-6 hebt die Selbstorganisation der aggregationskernbildenden SubdomÄne von mutiertem p53 DBD auf. ADH-6 TFA zielt auf mutierte p53-Aggregate ab und dissoziiert sie in menschlichen Krebszellen, wodurch die TranskriptionsaktivitÄt von p53' wiederhergestellt wird, was zu Zellzyklusarrest und Apoptose fÜhrt. ADH-6 TFA hat das Potenzial fÜr die Erforschung von Krebserkrankungen.
-
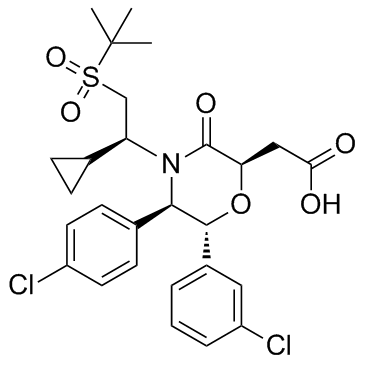
GC33356
AM-8735
AM-8735 ist ein potenter und selektiver MDM2-Inhibitor mit einem IC50 von 25 nM.
-
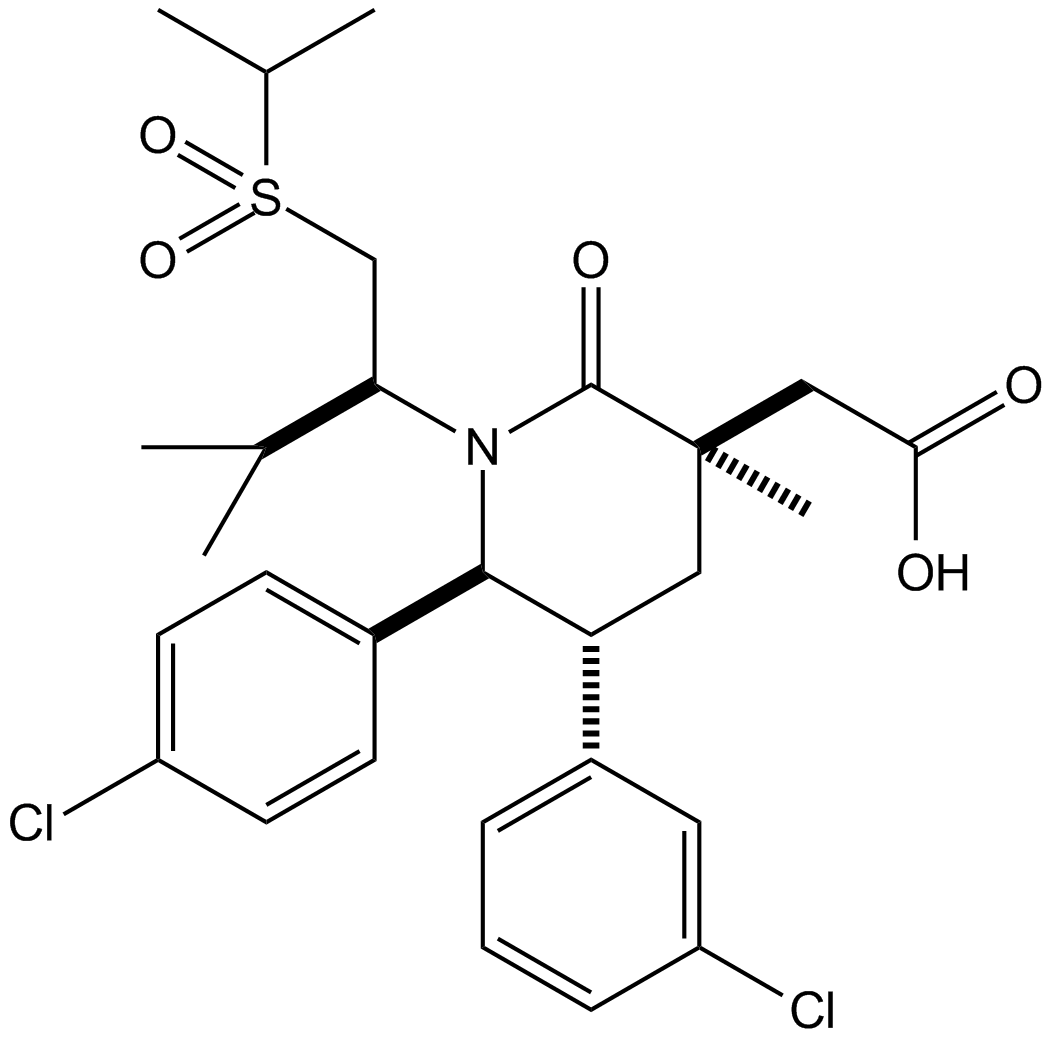
GC15828
AMG232
AMG232 (AMG 232) ist ein potenter, selektiver und oral verfÜgbarer Inhibitor der p53-MDM2-Interaktion mit einem IC50 von 0,6 nM. AMG232 bindet an MDM2 mit einem Kd von 0,045 nM.
-
GC42785
Amifostine (hydrate)
Amifostin (Hydrat) (WR2721-Trihydrat) ist ein Breitspektrum-Zellschutzmittel und ein Strahlenschutzmittel. Amifostin (Hydrat) schÜtzt selektiv normales Gewebe vor SchÄden durch Bestrahlung und Chemotherapie. Amifostin (Hydrat) ist ein potenter Hypoxie-induzierbarer Faktor-α1 (HIF-α1) und p53-Induktor. Amifostin (Hydrat) schÜtzt Zellen vor SchÄden, indem es freie Radikale aus Sauerstoff abfÄngt. Amifostin (Hydrat) reduziert die NierentoxizitÄt und hat eine antiangiogene Wirkung.
-
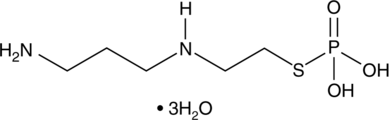
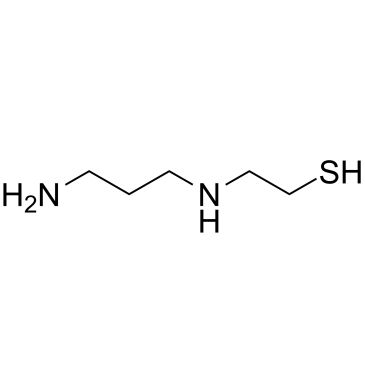
GC61804
Amifostine thiol
Amifostin-Thiol (WR-1065) ist ein aktiver Metabolit des Zytoprotektors Amifostin. Amifostin-Thiol ist ein zytoprotektives Mittel mit radioprotektiven FÄhigkeiten. Amifostin-Thiol aktiviert p53 Über einen JNK-abhÄngigen Signalweg.
-
GC35367
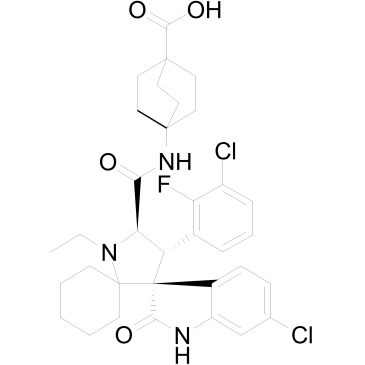
APG-115
APG-115 (APG-115) ist ein oral aktiver MDM2-Protein-Inhibitor, der an das MDM2-Protein mit IC50- und Ki-Werten von 3,8 nM bzw. 1 nM bindet. APG-115 blockiert die Interaktion von MDM2 und p53 und induziert auf p53-abhÄngige Weise Zellzyklusarrest und Apoptose.
-
GC18136
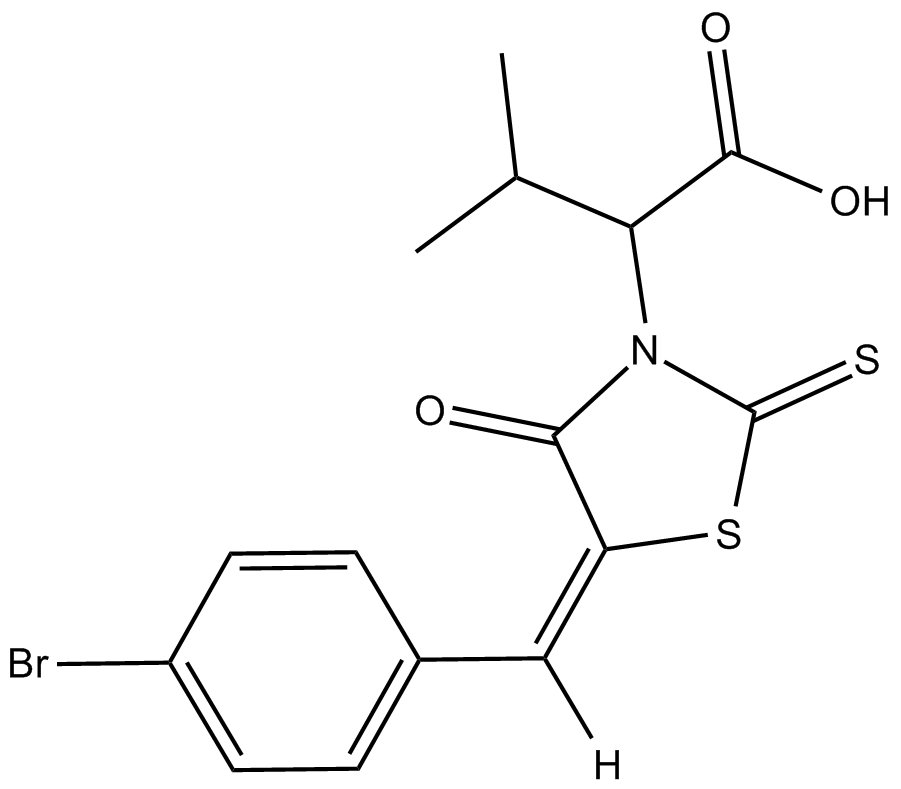
BH3I-1
BH3I-1 ist ein Antagonist der Bcl-2-Familie, der die Bindung des Bak-BH3-Peptids an Bcl-xL mit einem Ki von 2,4 ± 0,2 μM im FP-Assay hemmt. BH3I-1 hat einen Kd von 5,3 μM gegenÜber dem p53/MDM2-Paar.
-
GC35511
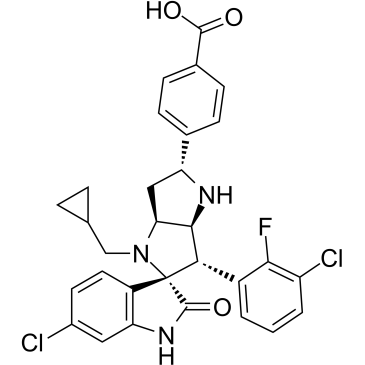
BI-0252
BI-0252 ist ein oral aktiver, selektiver MDM2-p53-Inhibitor mit einem IC50 von 4 nM. BI-0252 kann Tumorregressionen in allen Tieren eines Maus-SJSA-1-Xenotransplantats induzieren, mit gleichzeitiger Induktion der Zielgene des Tumorproteins p53 (TP53) und Apoptosemarkern.
-
GC43189
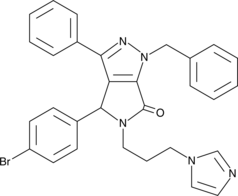
CAY10681
Inactivation of the tumor suppressor p53 commonly coincides with increased signaling through NF-κB in cancer.
-
GC43190
CAY10682
(±)-Nutlin-3 blocks the interaction of p53 with its negative regulator Mdm2 (IC50 = 90 nM), inducing the expression of p53-regulated genes and blocking the growth of tumor xenografts in vivo.
-
GC14634
CBL0137
curaxin that activates p53 and inhibits NF-κB
-
GC15394
CBL0137 (hydrochloride)
CBL0137 (Hydrochlorid) ist ein Inhibitor des Histon-Chaperons FACT. CBL0137 (Hydrochlorid) kann auch p53 aktivieren und hemmt NF-κB mit EC50-Werten von 0,37 bzw. 0,47 µM.
-
GC43239
Chk2 Inhibitor
Chk2-Inhibitor (Verbindung 1) ist ein potenter und selektiver Inhibitor der Checkpoint-Kinase 2 (Chk2) mit IC50-Werten von 13,5 nM und 220,4 nM fÜr Chk2 bzw. Chk1. Der Chk2-Inhibitor kann einen starken, von Ataxia telangiectasia mutated (ATM) abhÄngigen Chk2-vermittelten Strahlenschutzeffekt hervorrufen.
-
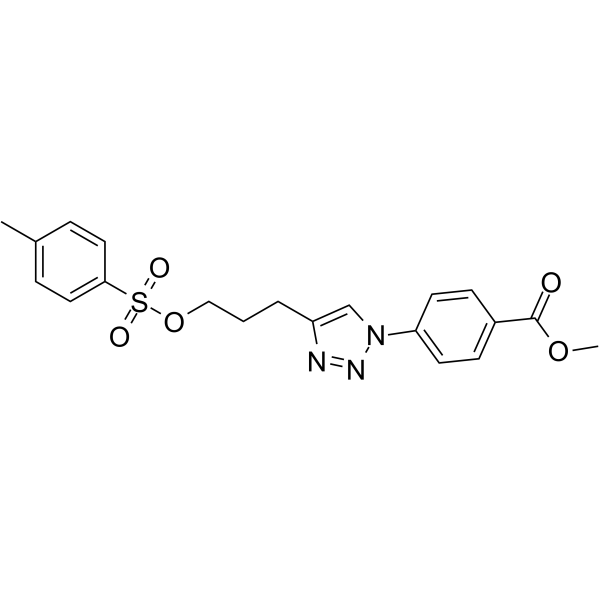
GC64649
Cjoc42
Cjoc42 ist eine Verbindung, die an Gankyrin binden kann. Cjoc42 hemmt die Gankyrin-AktivitÄt dosisabhÄngig. Cjoc42 verhindert die Abnahme des p53-Proteinspiegels, der normalerweise mit hohen Gankyrin-Mengen verbunden ist. Cjoc42 stellt die p53-abhÄngige Transkription und Empfindlichkeit gegenÜber DNA-SchÄden wieder her.
-
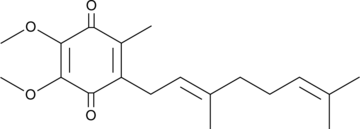
GC43297
Coenzyme Q2
Coenzyme Q10 is a component of the electron transport chain and participates in aerobic cellular respiration, generating energy in the form of ATP.
-
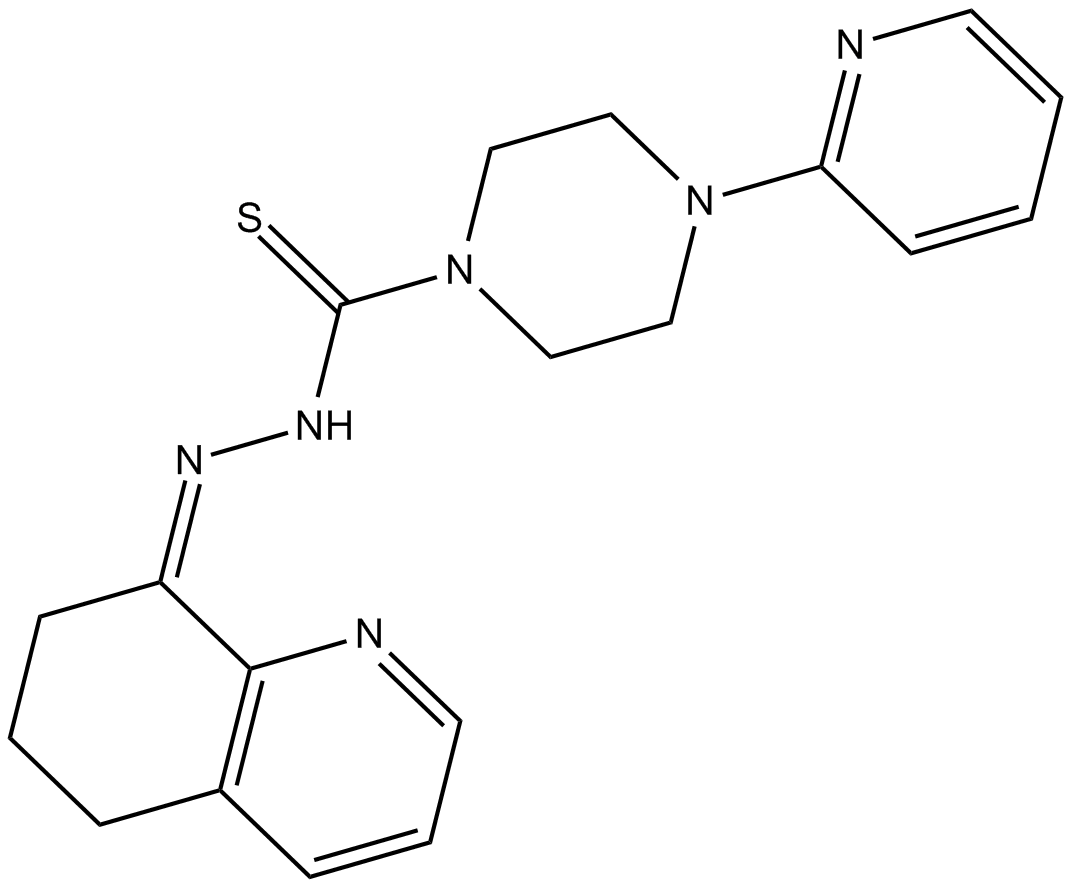
GC15225
COTI-2
COTI-2, ein Anti-Krebs-Medikament mit geringer ToxizitÄt, ist ein oral verfÜgbarer Aktivator der dritten Generation von p53-Mutantenformen. COTI-2 wirkt sowohl durch die Reaktivierung des mutierten p53 als auch durch die Hemmung des PI3K/AKT/mTOR-Signalwegs. COTI-2 induziert Apoptose in mehreren menschlichen Tumorzelllinien. COTI-2 zeigt AntitumoraktivitÄt bei HNSCC durch p53-abhÄngige und -unabhÄngige Mechanismen. COTI-2 wandelt mutiertes p53 in die Wildtyp-Konformation um.
-
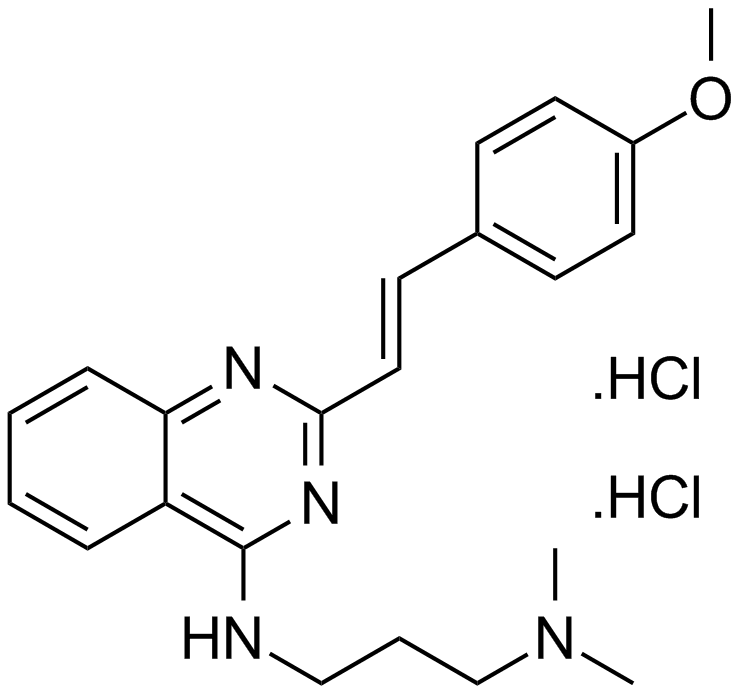
GC15840
CP 31398 dihydrochloride
A p53 stabilizing agent
-
GC32911
CTX1
CTX1 ist ein p53-Aktivator, der die HdmX-vermittelte p53-Repression Überwindet. CTX1 zeigt in einem Maus-Modellsystem fÜr akute myeloische LeukÄmie (AML) eine starke Anti-Krebs-AktivitÄt.
-
GC43408
Deoxycholic Acid (sodium salt hydrate)
DesoxycholsÄure (CholansÄure) Natriumhydrat, eine GallensÄure, ist ein Nebenprodukt des Darmstoffwechsels, das den G-Protein-gekoppelten GallensÄurerezeptor TGR5 aktiviert.
-
GC47187
Deoxycholic Acid-d4
DesoxycholsÄure-d4 ist die mit Deuterium bezeichnete DesoxycholsÄure.
-
GC33384
DPBQ
DPBQ aktiviert p53 und lÖst polyploid-spezifisch Apoptose aus, hemmt aber weder Topoisomerase noch bindet es DNA. DPBQ lÖst die Expression und Phosphorylierung von p53 aus und diese Wirkung ist spezifisch fÜr tetraploide Zellen.
-
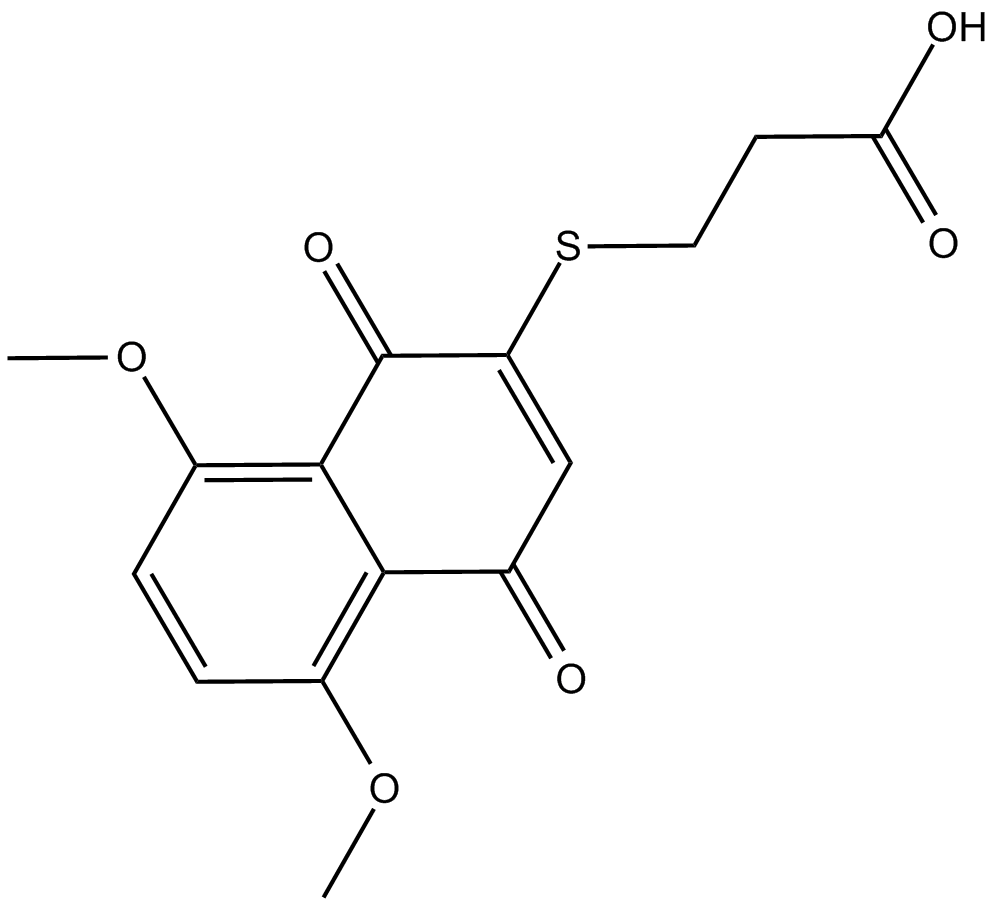
GC15258
GN25
p53-Snail binding Inhibitor
-
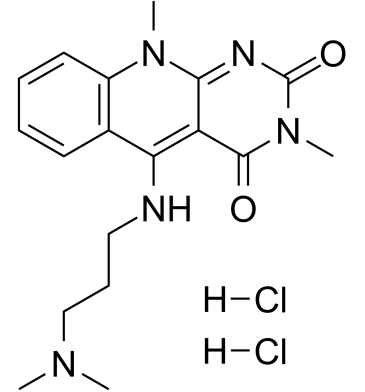
GC61608
HLI373 dihydrochloride
HLI373-Dihydrochlorid ist ein wirksamer Hdm2-Hemmer.
-
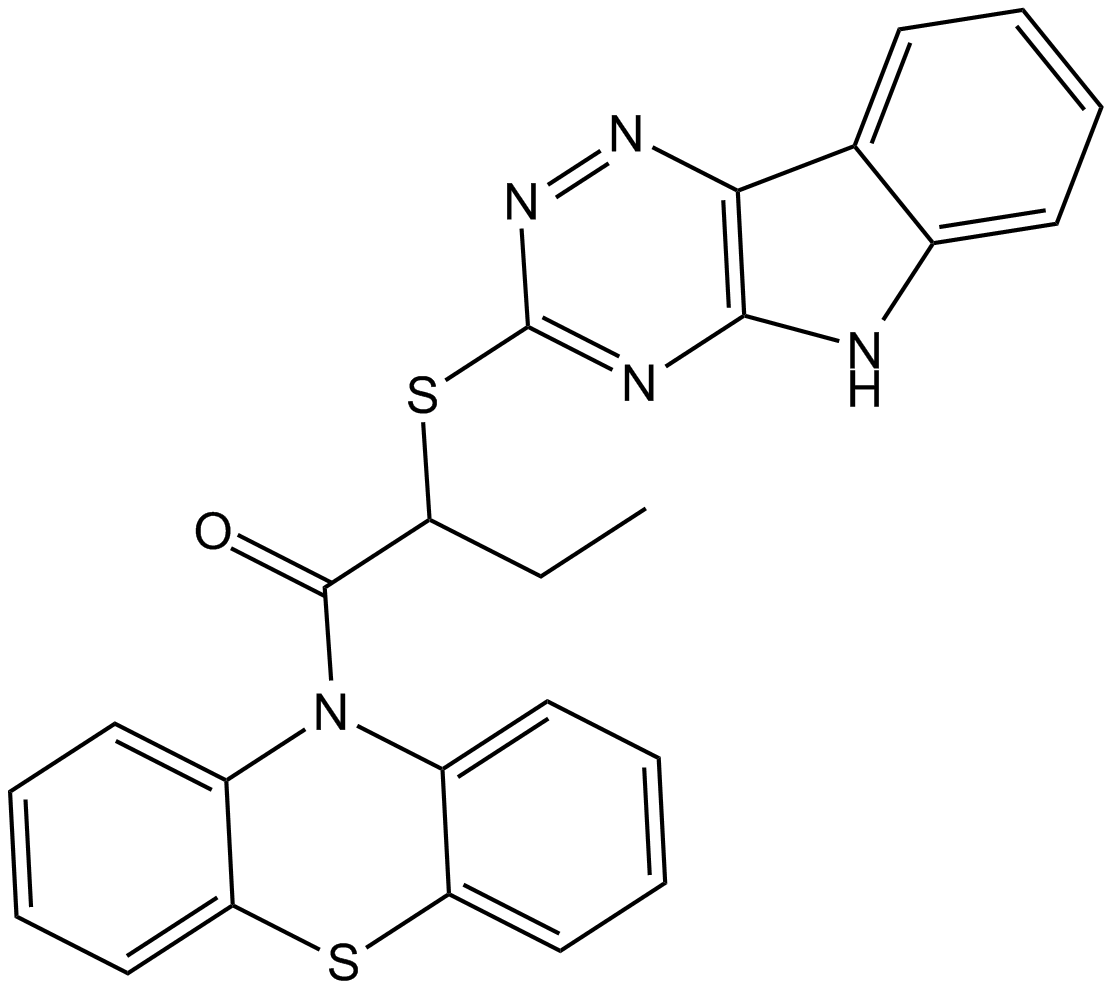
GC14755
Inauhzin
Inauhzin ist ein dualer SirT1/IMPDH2-Inhibitor und fungiert als Aktivator p53, der in der Krebsforschung eingesetzt wird.
-
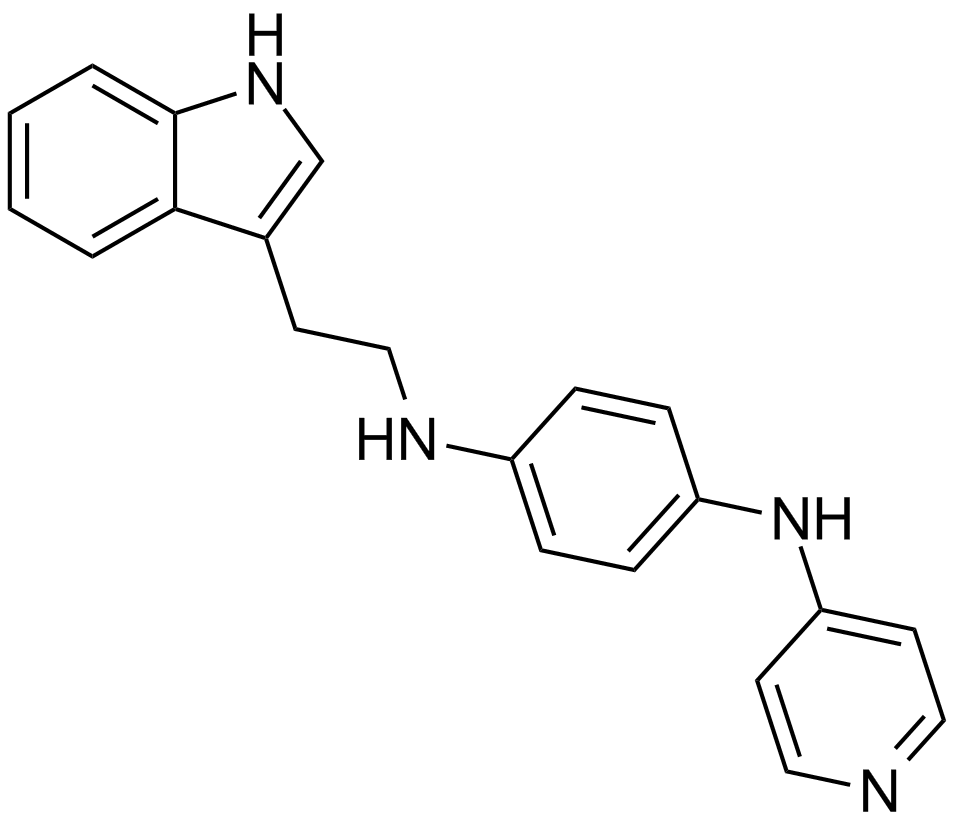
GC12117
JNJ-26854165 (Serdemetan)
An antagonist of MDM2 action
-
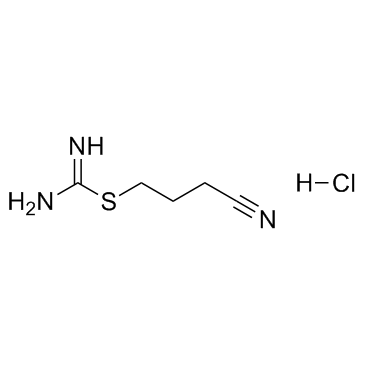
GC32919
Kevetrin hydrochloride (4-Isothioureidobutyronitrile hydrochloride)
Kevetrin-Hydrochlorid (4-Isothioureidobutyronitril-Hydrochlorid) ist ein potenter Aktivator von p53, induziert Apoptose in TP53-Wildtyp- und mutierten akuten myeloischen LeukÄmiezellen. Kevetrin eine bevorzugte zytotoxische AktivitÄt gegen Blastenzellen.
-
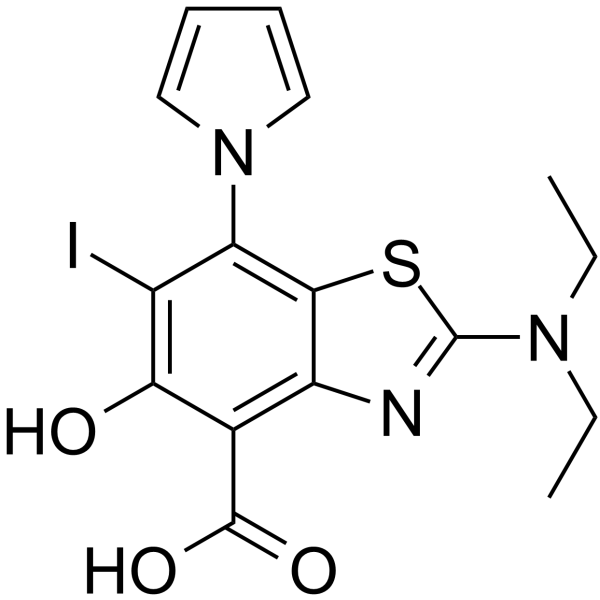
GC62629
MB710
MB710, ein Aminobenzothiazol-Derivat, ist ein Stabilisator der onkogenen p53-Mutation Y220C. MB710 bindet fest an die Y220C-Tasche und stabilisiert p53-Y220C mit einem Kd von 4,1 μM. MB710 zeigt AntikrebsaktivitÄt in p53-Y220C-Zelllinien.
-
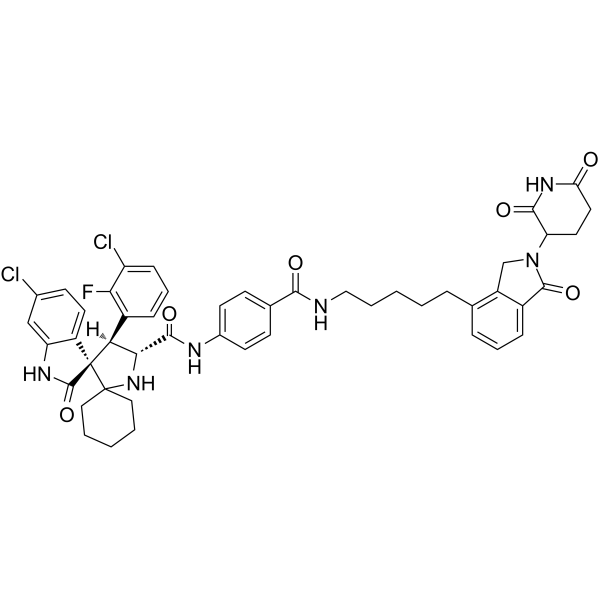
GC62716
MD-222
MD-222 ist der erste hochwirksame PROTAC-Abbaustoff seiner Klasse fÜr MDM2. MD-222 besteht aus Liganden fÜr Cereblon und MDM2. MD-222 induziert den schnellen Abbau des MDM2-Proteins und die Aktivierung von Wildtyp-p53 in Zellen. MD-222 hat Antikrebswirkungen.
-
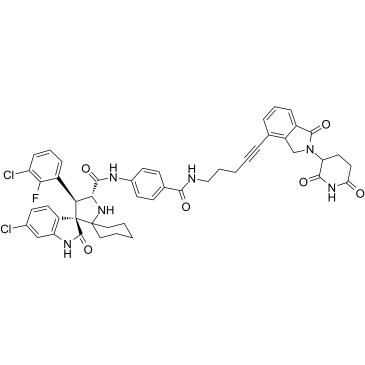
GC38812
MD-224
MD-224 ist ein erstklassiger und hochwirksamer niedermolekularer Human-Maus-Doppelminuten-2-Abbaustoff (MDM2), der auf dem Proteolyse-Targeting-ChimÄren-Konzept (PROTAC) basiert. MD-224 besteht aus Liganden fÜr Cereblon und MDM2. MD-224 induziert einen schnellen Abbau von MDM2 bei Konzentrationen <1 nM in menschlichen LeukÄmiezellen und erreicht einen IC50-Wert von 1,5 nM bei der Hemmung des Wachstums von RS4;11-Zellen. MD-224 hat das Potenzial, eine neue Klasse von Krebsmitteln zu werden.
-
GC62557
MDM2-IN-1
MDM2-IN-1 (Verbindung 30) ist ein synthetischer Inhibitor der MDM2-p53-Interaktion (MDM2) und enthÄlt die trans-(D-)Konfiguration.
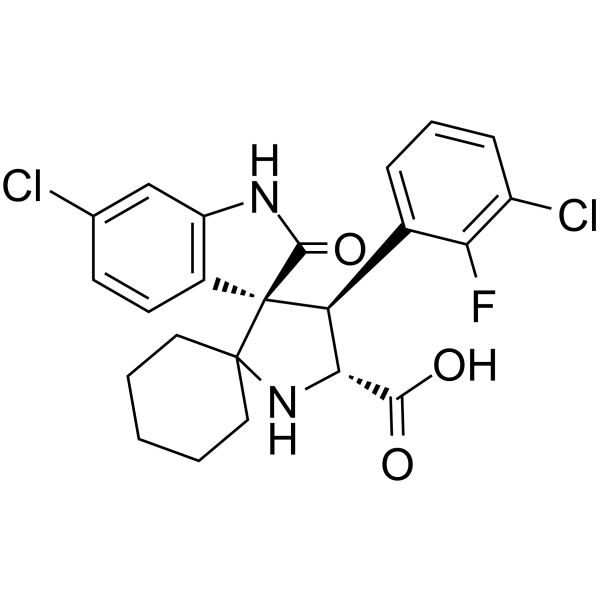
-
GC36605
MI-1061
MI-1061 ist ein potenter, oral bioverfÜgbarer und chemisch stabiler MDM2-Inhibitor (MDM2-p53-Wechselwirkung) (IC50 = 4,4 nM; Ki = 0,16 nM). MI-1061 aktiviert stark p53 und induziert Apoptose im SJSA-1-Xenotransplantat-Tumorgewebe bei MÄusen. Anti-Tumor-AktivitÄt.
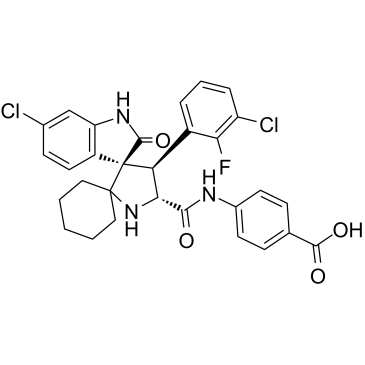
-
GC62598
MI-1061 TFA
MI-1061 TFA ist ein potenter, oral bioverfÜgbarer und chemisch stabiler MDM2-Inhibitor (MDM2-p53-Interaktion) (IC50 = 4,4 nM; Ki = 0,16 nM). MI-1061 TFA aktiviert stark p53 und induziert Apoptose im SJSA-1-Xenotransplantat-Tumorgewebe in MÄusen. Anti-Tumor-AktivitÄt.
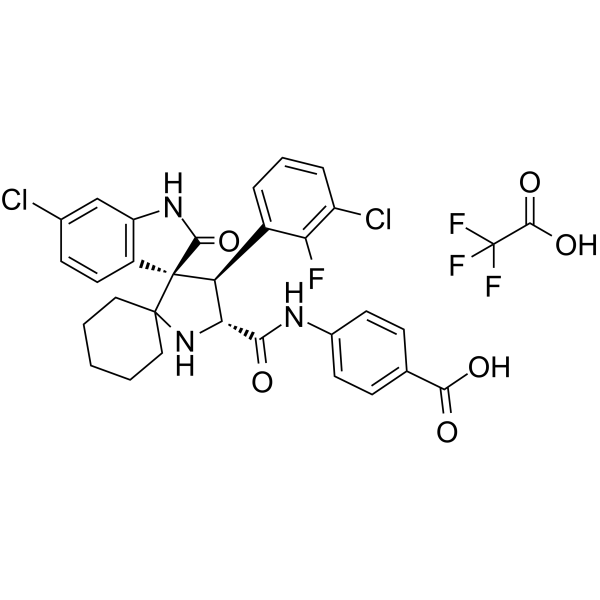
-
GC16296
MI-773
MI-773 ist ein potenter Inhibitor der MDM2-p53-Protein-Protein-Interaktion (PPI) mit hoher BindungsaffinitÄt zu MDM2 (Kd=8,2 nM). MI-773 hat AntitumoraktivitÄt.
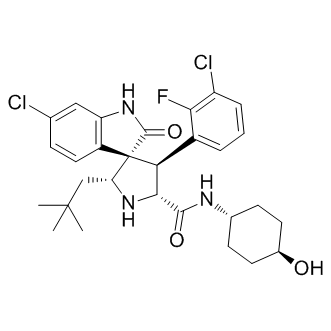
-
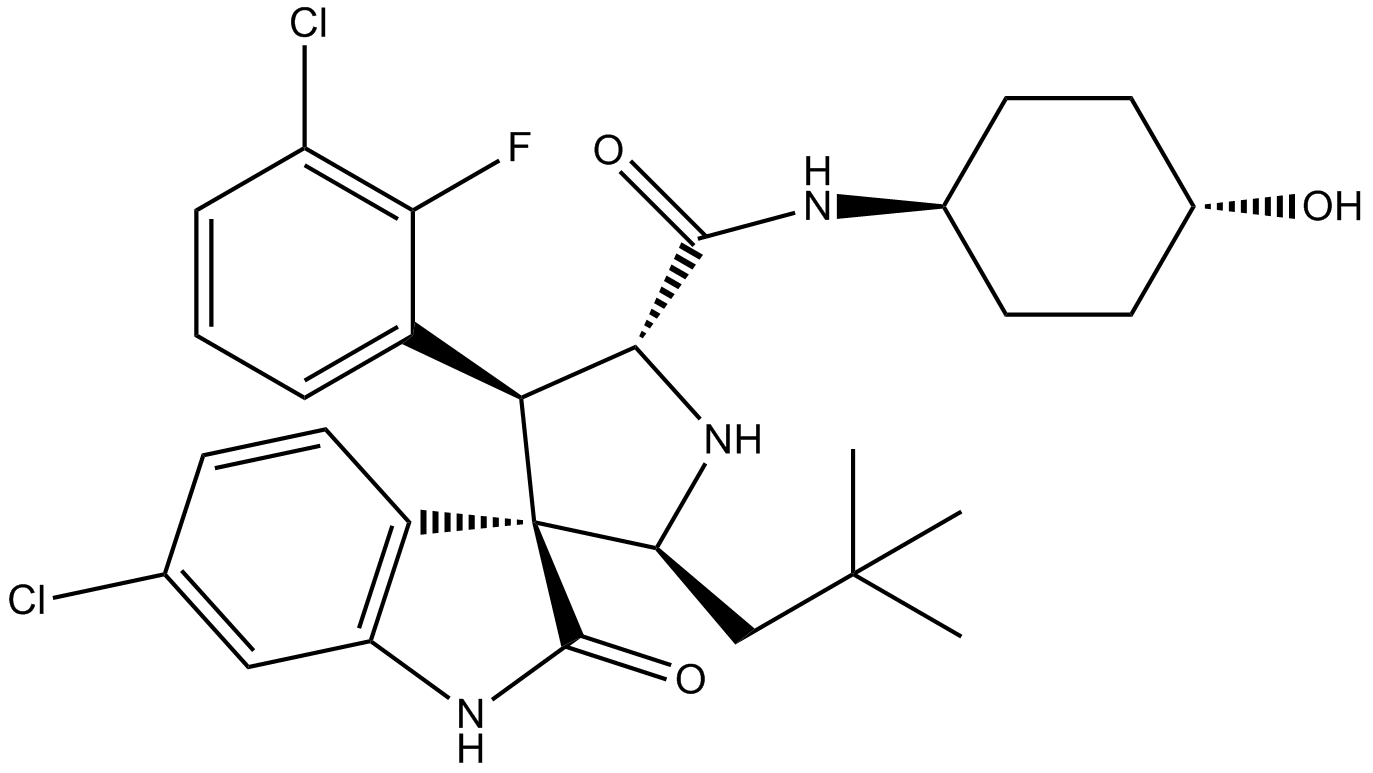
GC11547
MI-773 (SAR405838)
MI-773 (SAR405838) (MI-77301), ein Analogon von MI-773, ist ein hochwirksamer und selektiver MDM2-p53-Interaktionsinhibitor. MI-773 (SAR405838) bindet an MDM2 mit einem Ki von 0,88 nM. MI-773 (SAR405838) induziert Apoptose und hat starke AntitumoraktivitÄt.
-
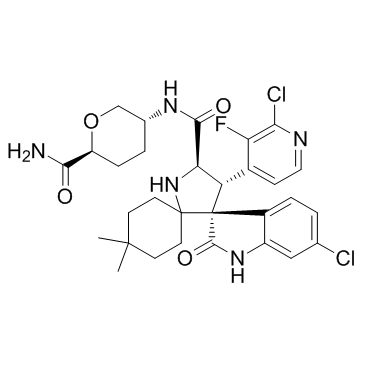
GC32881
Milademetan (DS-3032)
Milademetan (DS-3032) (DS-3032) ist ein spezifischer und oral aktiver MDM2-Inhibitor zur Erforschung von akuter myeloischer LeukÄmie (AML) oder soliden Tumoren. Milademetan (DS-3032) (DS-3032) induziert G1-Zellzyklusarrest, Seneszenz und Apoptose.
-
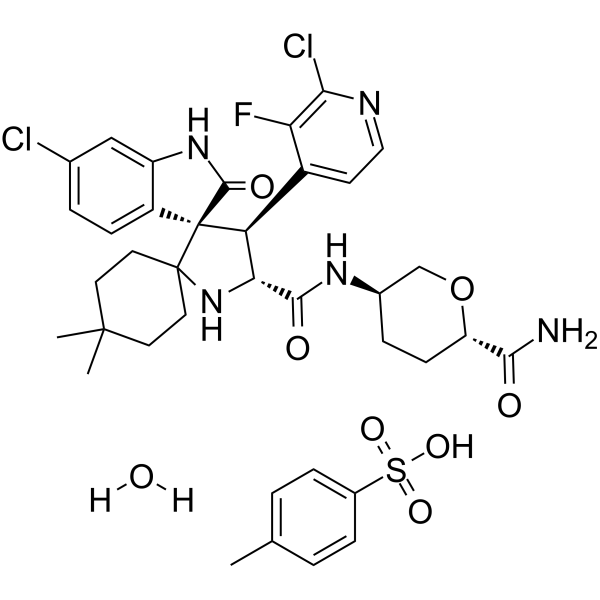
GC62621
Milademetan tosylate hydrate
Milademetan (DS-3032) Tosylathydrat ist ein spezifischer und oral wirksamer MDM2-Inhibitor zur Erforschung der akuten myeloischen LeukÄmie (AML) oder solider Tumoren. Milademetan (DS-3032) Tosylathydrat induziert G1-Zellzyklusarrest, Seneszenz und Apoptose.
-
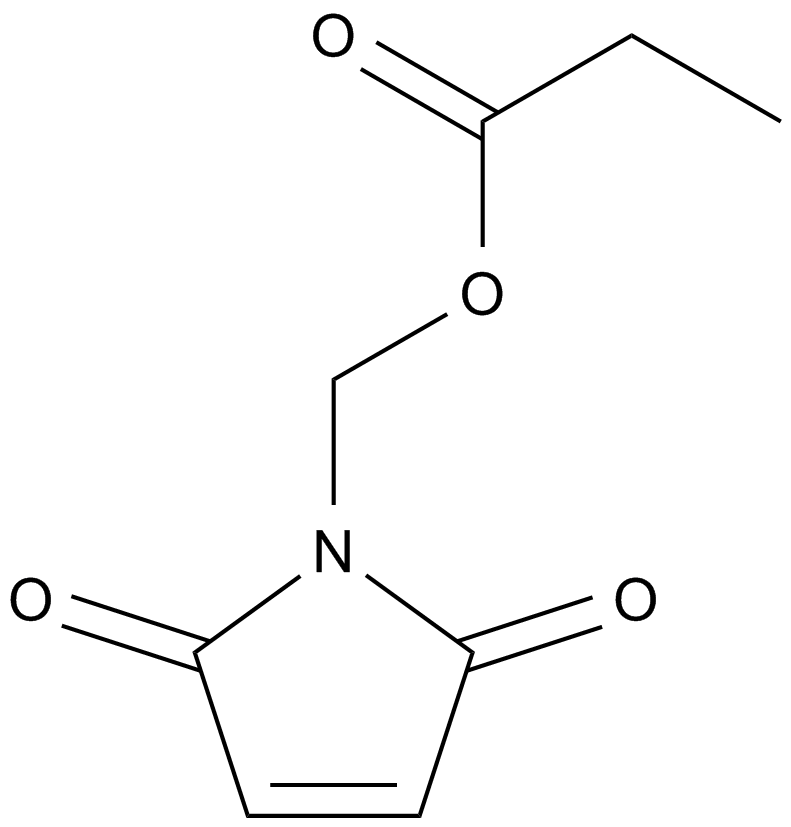
GC12893
MIRA-1
A mutant p53 reactivator
-
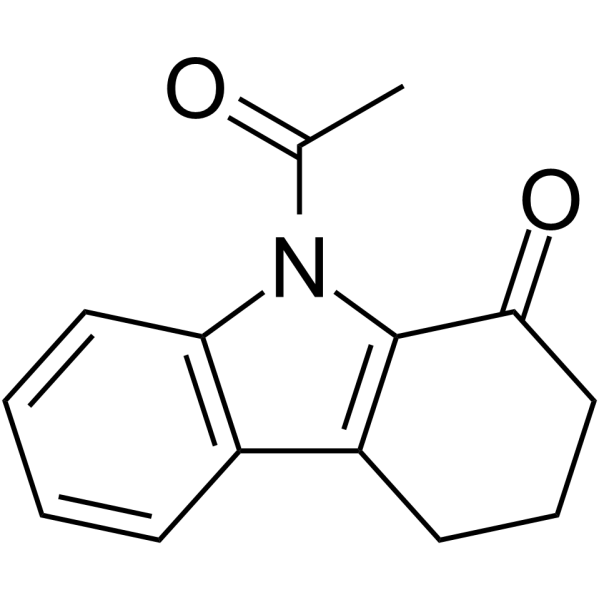
GC62322
MS7972
MS7972 ist ein kleines MolekÜl, das die Assoziation von humanem p53 und CREB-Bindungsprotein blockiert. MS7972 kann diese BRD-Interaktion bei 50 μ fast vollstÄndig blockieren;M.
-
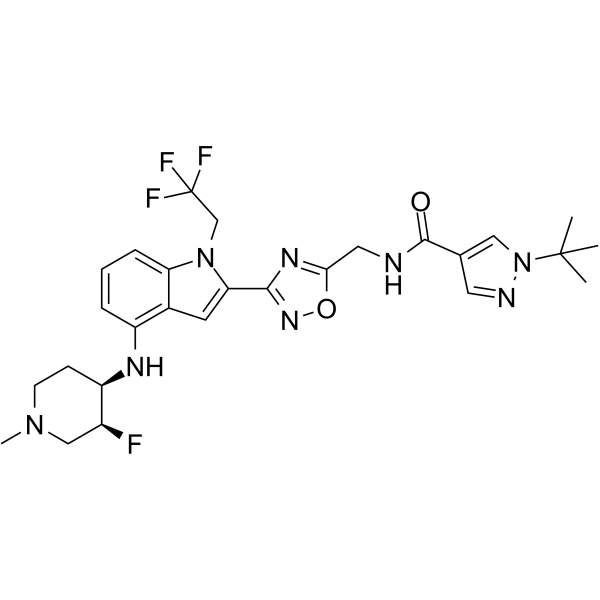
GC68371
Mutant p53 modulator-1
-
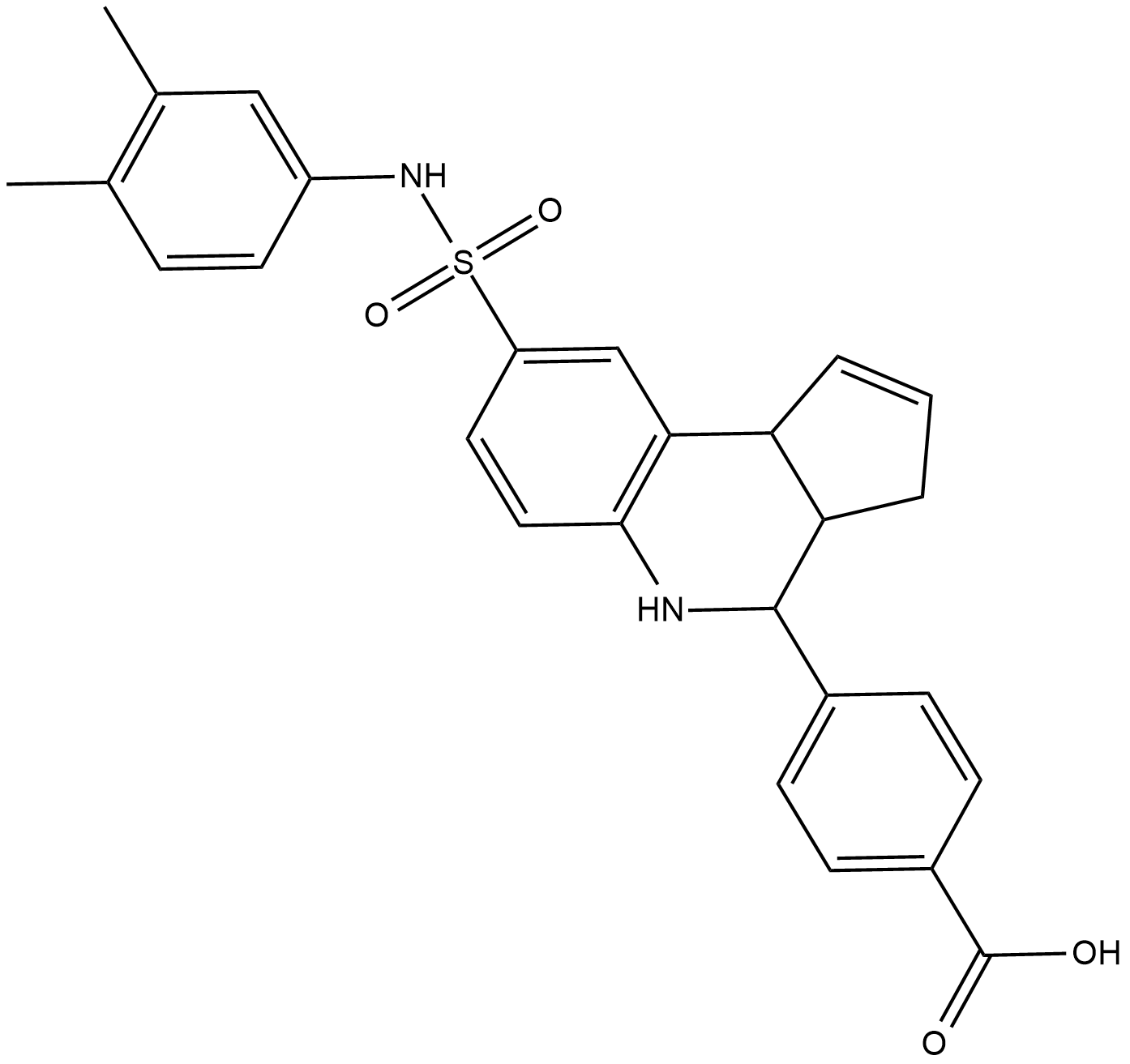
GC19257
MX69
MX69 ist ein Inhibitor von MDM2/XIAP, der zur Krebsbehandlung eingesetzt wird.
-
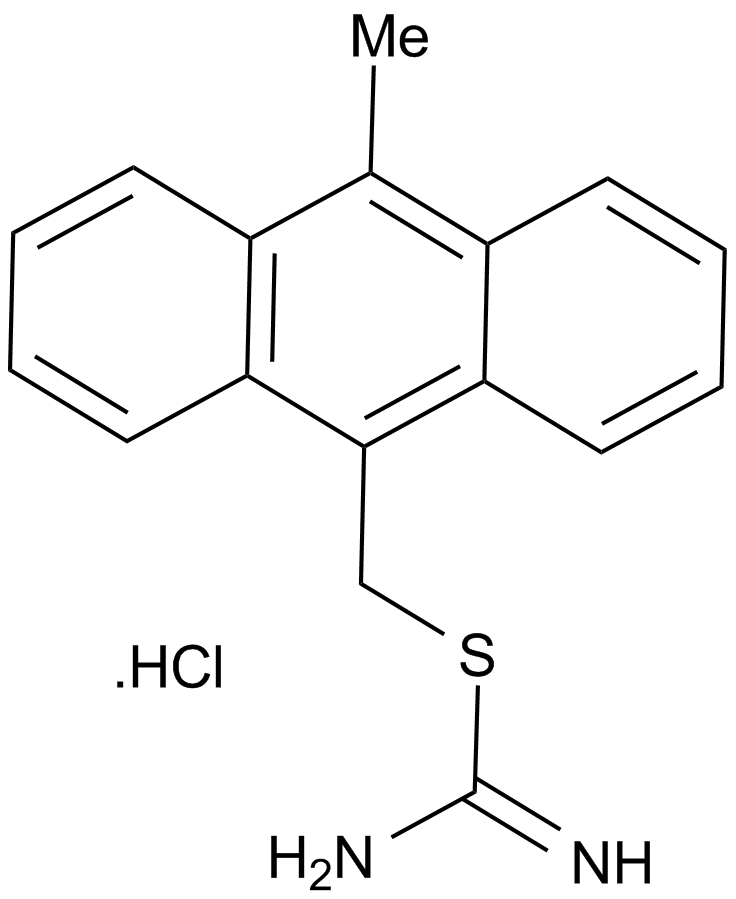
GC15621
NSC 146109 hydrochloride
An activator of p53
-
GC15404
NSC 319726
A p53 reactivator
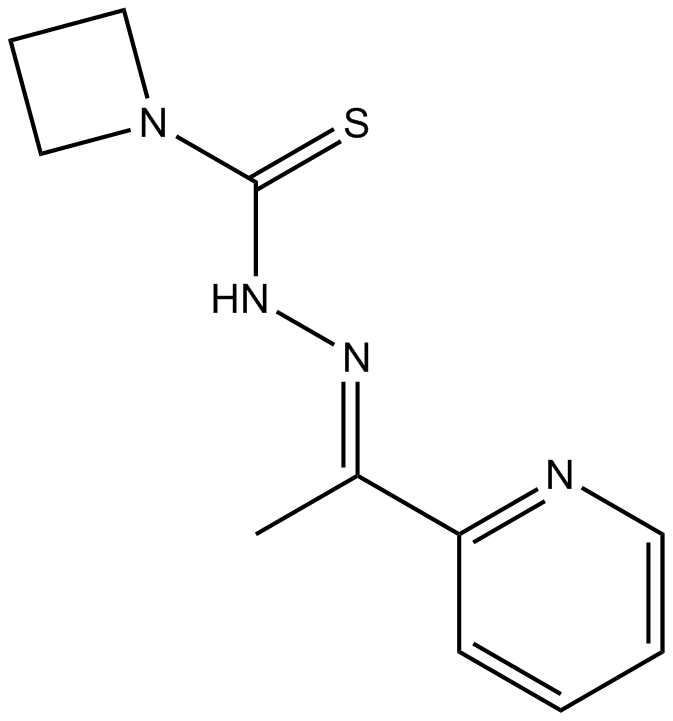
-
GC16151
NSC348884
NSC348884 ist ein Nucleophosmin (NPM)-Inhibitor, der die Oligomerbildung unterbricht und Apoptose induziert, die Zellproliferation mit IC50-Werten von 1,7-4,0 μM in verschiedenen Krebszelllinien hemmt. NSC348884 kann fÜr die Krebsforschung verwendet werden.
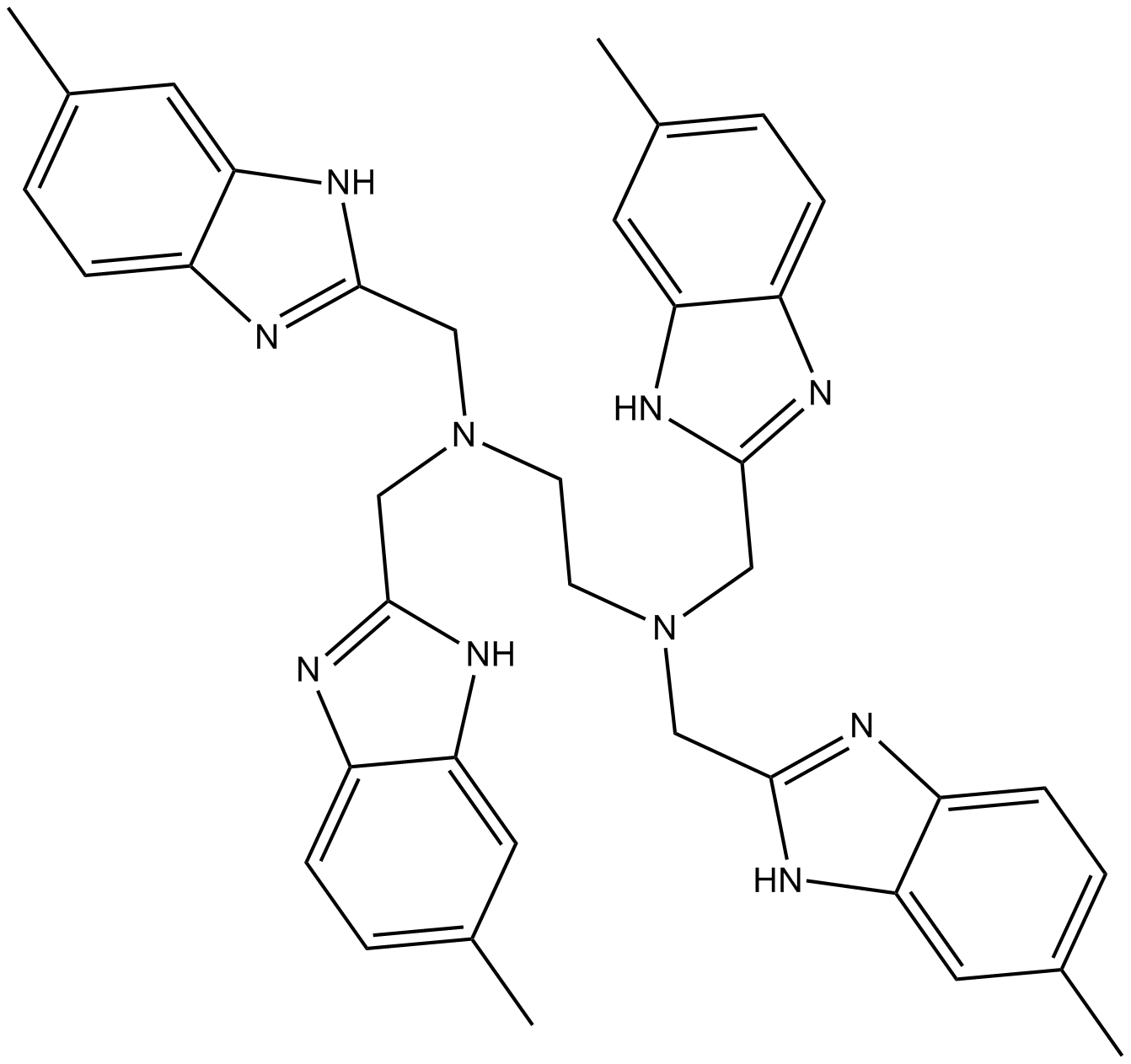
-
GC16179
NSC59984
NSC59984 induziert den Abbau des mutierten p53-Proteins Über MDM2 und den Ubiquitin-Proteasom-Weg. NSC59984 wirkt, indem es auf das GOF-mutierte p53 abzielt und p73 stimuliert, um die SignalÜbertragung des p53-Signalwegs wiederherzustellen.
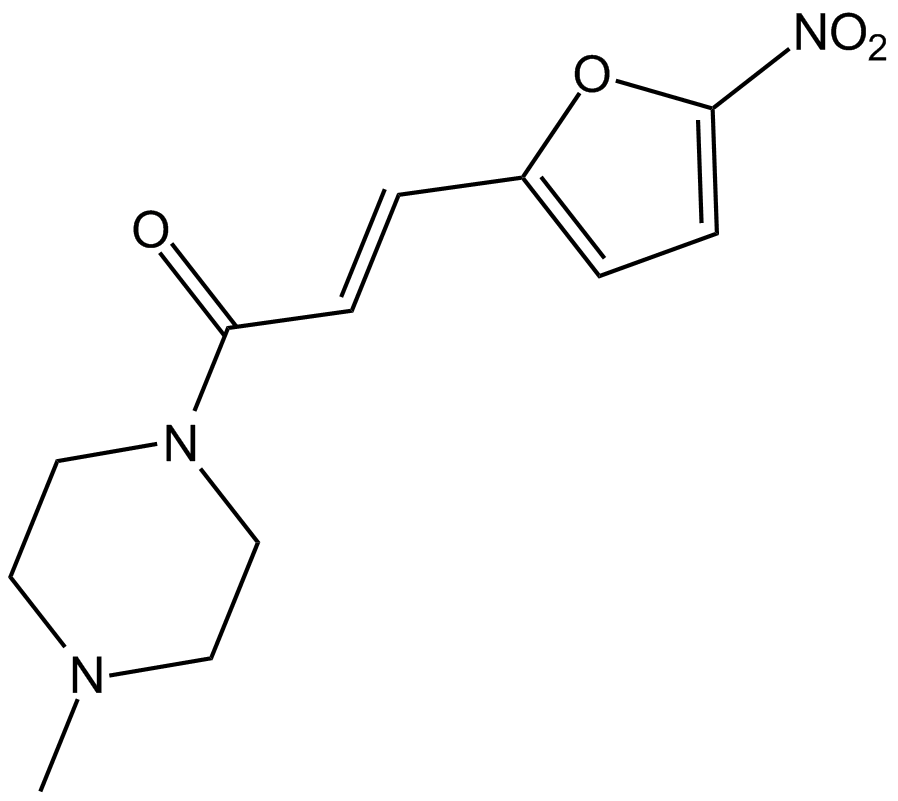
-
GC16051
Nutlin-3
A racemic mixture of (?)-nutlin-3 and (+)-nutlin-3
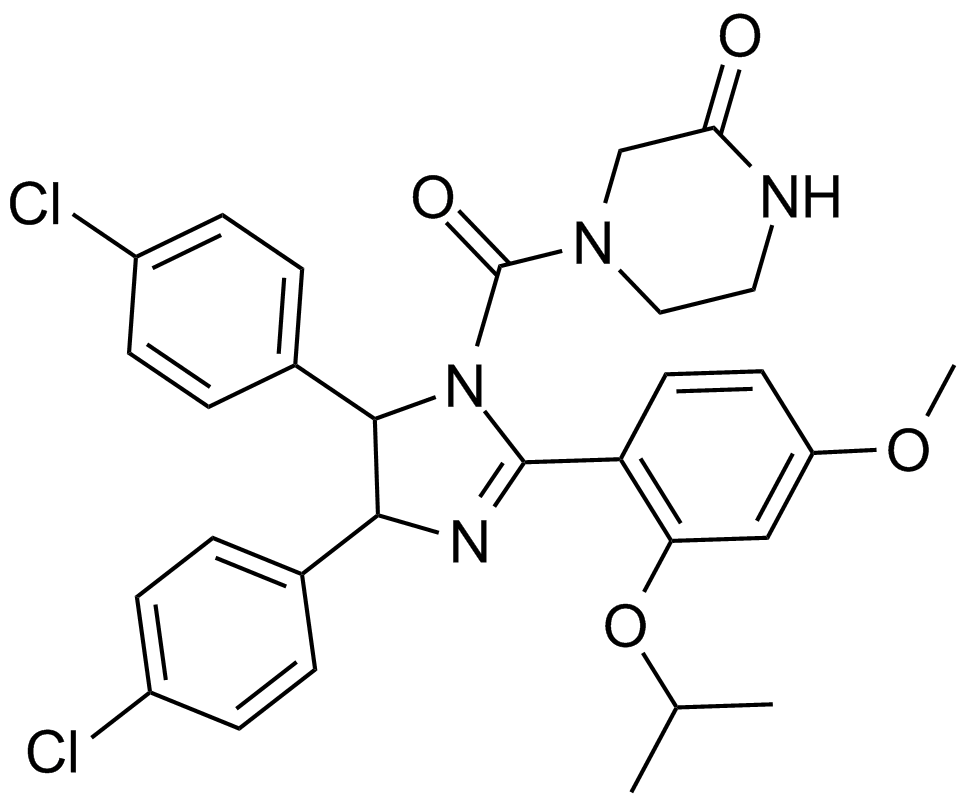
-
GC10470
Nutlin-3a chiral
An inhibitor of the p53-Mdm2 interaction
-
GC12508
Nutlin-3b
Nutlin-3b ist ein p53/MDM2-Inhibitor mit einem IC50 von 13,6 μM. Nutlin-3b bindet 150-mal weniger stark an MDM2 als Nutlin-3a.
-
GC12647
NVP-CGM097
NVP-CGM097 ist ein potenter und selektiver MDM2-Inhibitor mit einem IC50-Wert von 1,7 ± 0,1 nM fÜr hMDM2.
-
GC36785
NVP-CGM097 sulfate
NVP-CGM097-Sulfat ist ein potenter und selektiver MDM2-Inhibitor mit einem IC50-Wert von 1,7 ± 0,1 nM fÜr hMDM2.
-
GC19268
NVP-HDM201
NVP-HDM201 (NVP-HDM201) ist ein potenter, oral bioverfÜgbarer und hochspezifischer p53-MDM2-Interaktionsinhibitor.
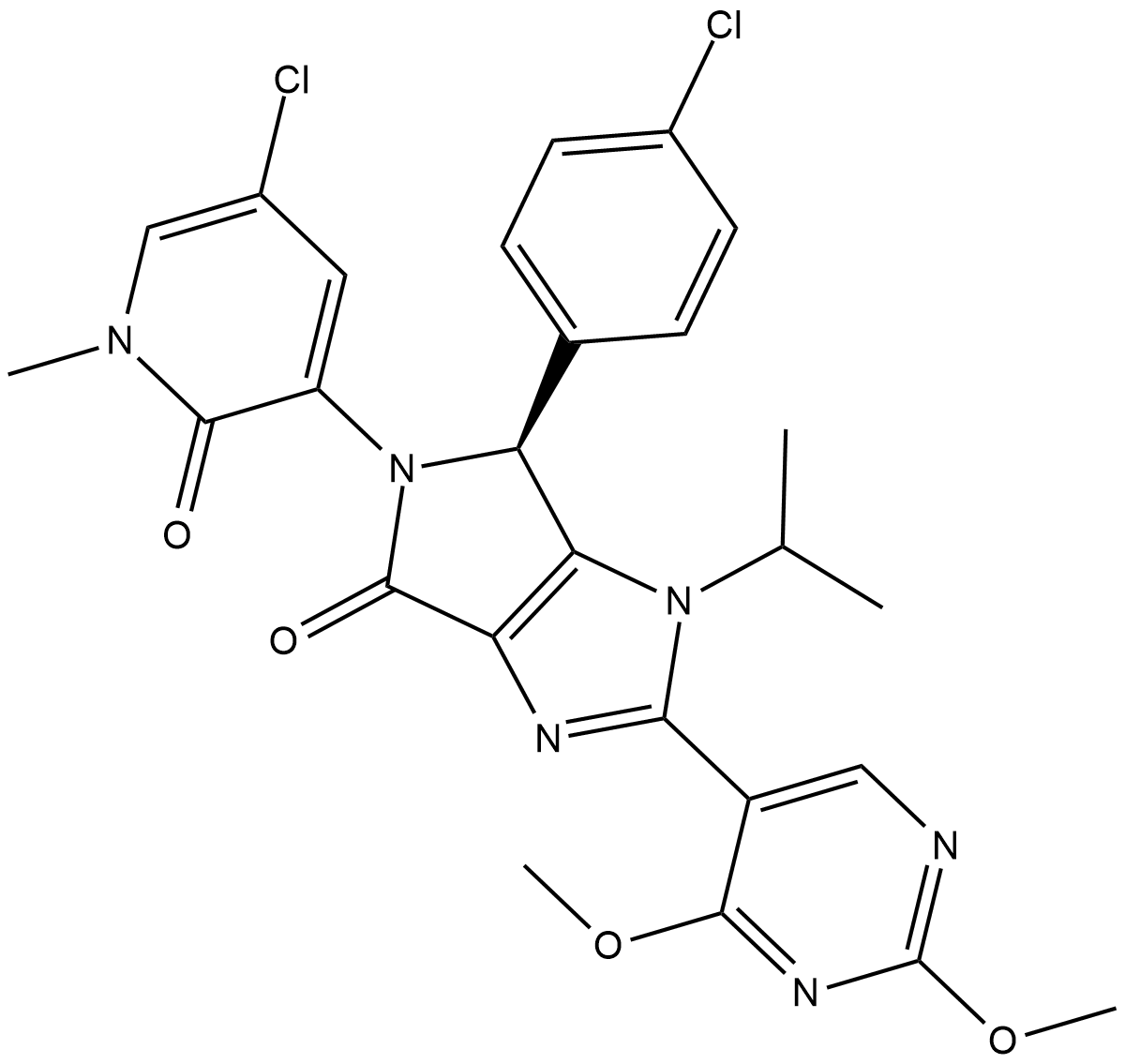
-
GC48652
Olomoucine II
A CDK inhibitor
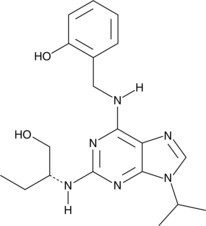
-
GC11711
ONC201
ONC201 (ONC-201) ist ein potenter, oral aktiver und stabiler Tumornekrosefaktor-bezogener Apoptose-induzierender Ligand (TRAIL), der Akt und ERK hemmt, folglich Foxo3a aktiviert und ZelloberflÄchen-TRAIL signifikant induziert. ONC201 kann die Blut-Hirn-Schranke passieren.
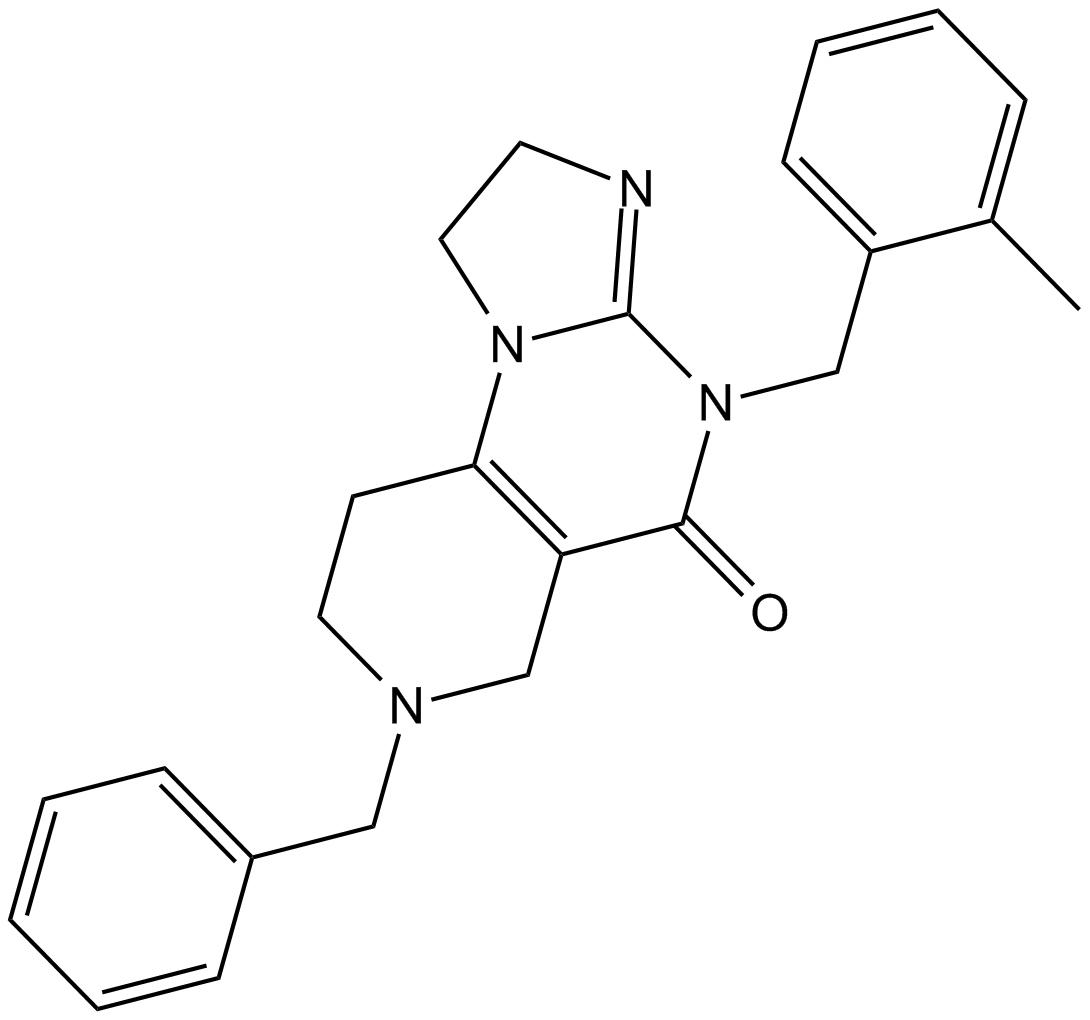
-
GC15613
p-nitro-Cyclic Pifithrin-α
p-nitro-cyclisches Pifithrin-⋱ (PFN-α) ist ein zelldurchlÄssiger p53-Inhibitor in aktiver Form.
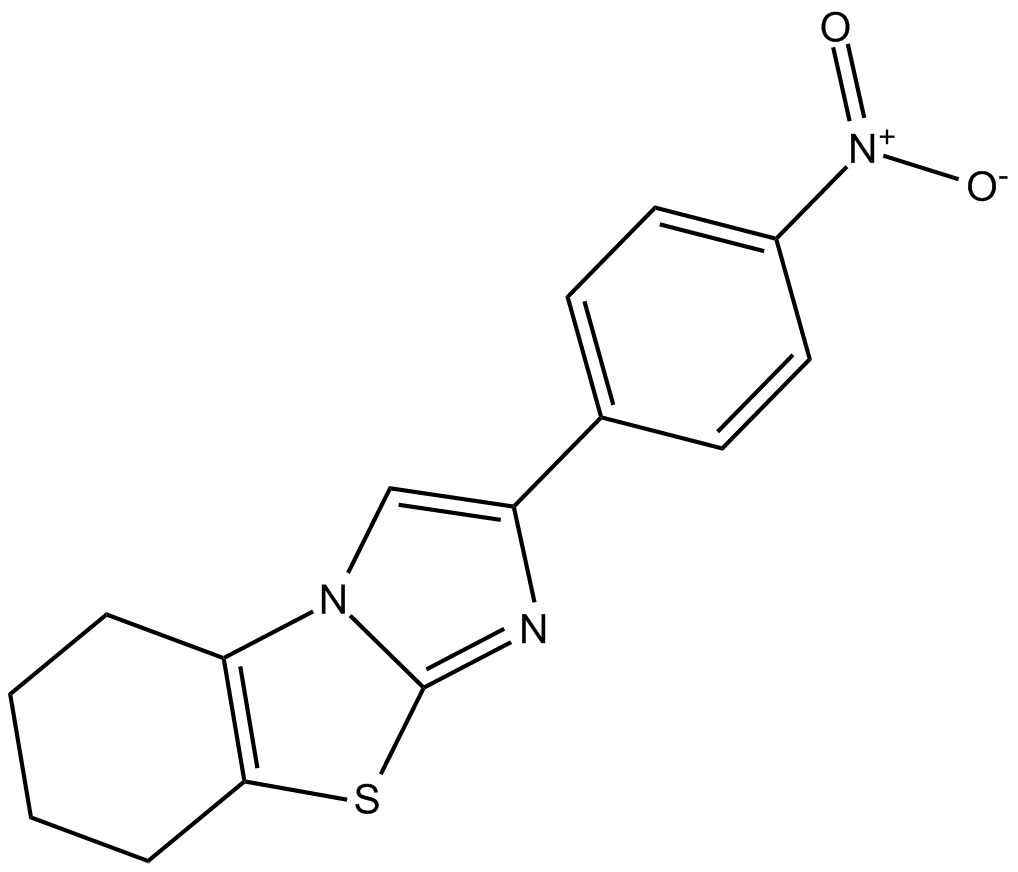
-
GC15812
p-nitro-Pifithrin-α
p-Nitro-Pifithrin-α, ein zelldurchlÄssiges Analogon von Pifithrin-α, ist ein potenter p53-Inhibitor.
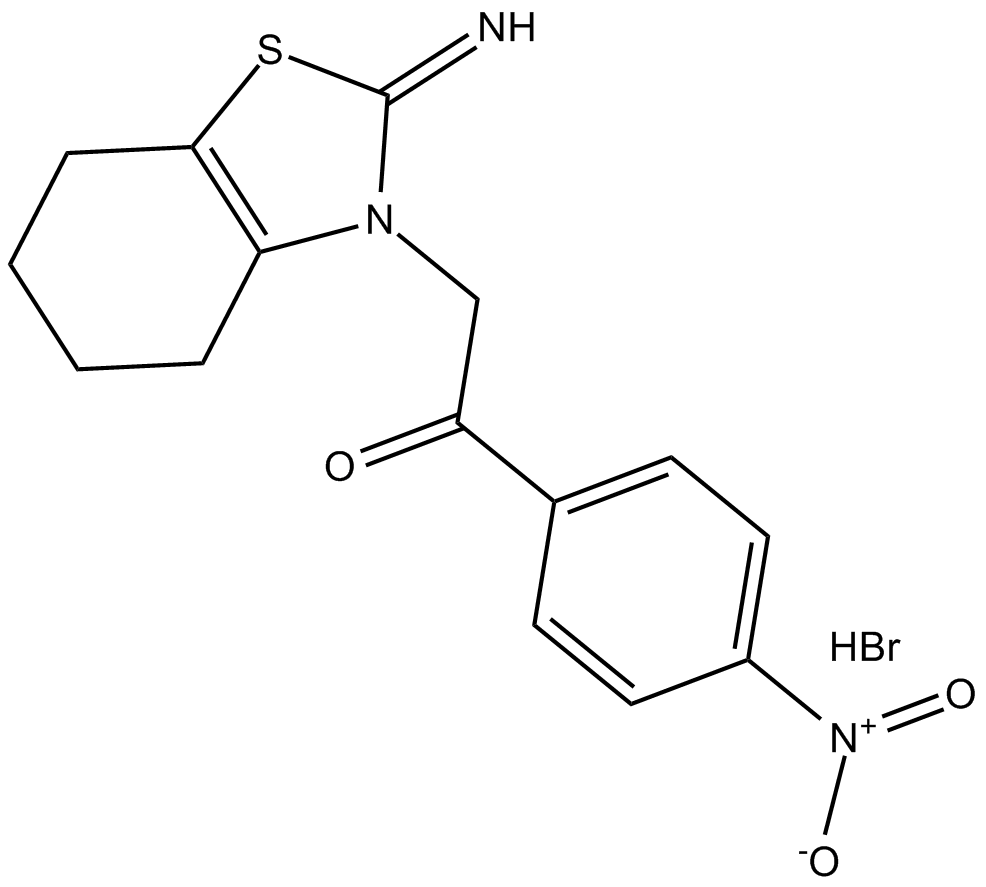
-
GC67765
p53 Activator 5
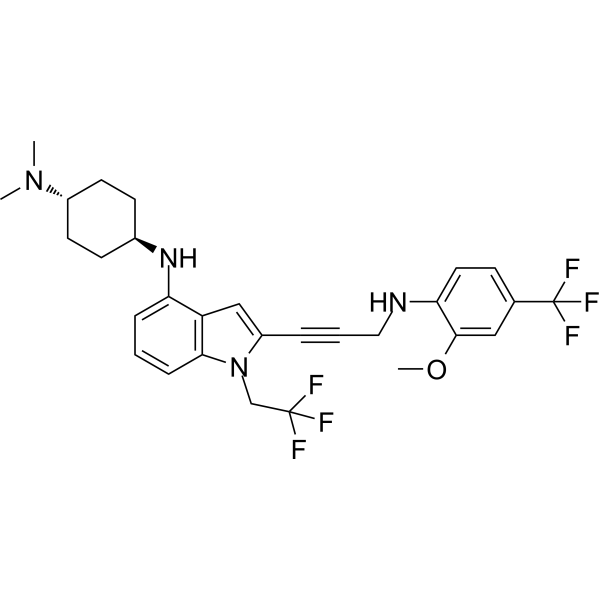
-
GC69648
p53 Activator 7
p53 Activator 7 is a p53 mutation Y220C (MDM-2/p53) activator with an EC50 of 104 nM. It can bind to p53 mutants and restore their ability to bind DNA (WO2022213975A1; Example B-1).
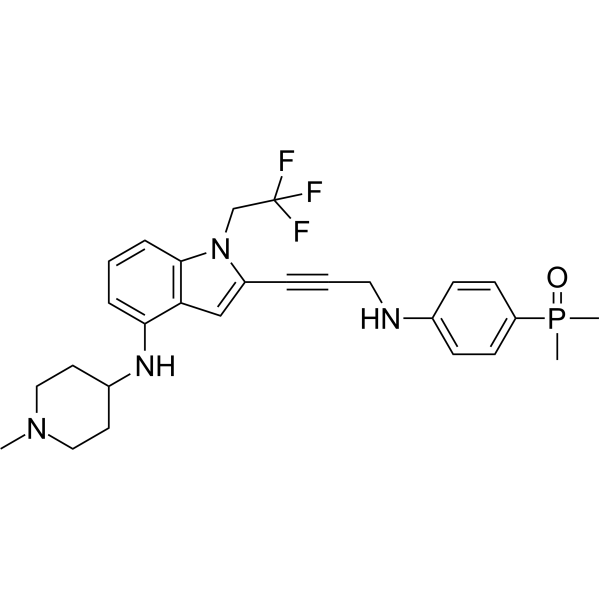
-
GC36835
p53 and MDM2 proteins-interaction-inhibitor chiral
p53- und MDM2-Protein-Interaktionsinhibitor chiral (Verbindung 32) ist ein Inhibitor der Wechselwirkung zwischen p53- und MDM2-Proteinen.
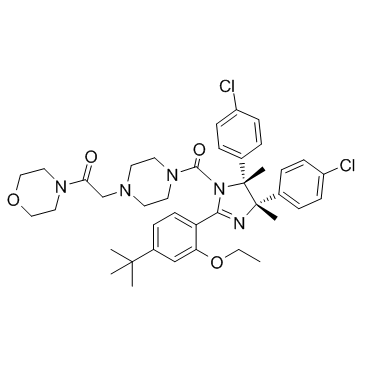
-
GC36836
p53 and MDM2 proteins-interaction-inhibitor dihydrochloride
p53- und MDM2-Protein-Interaktionsinhibitor Dihydrochlorid ist ein Inhibitor der Wechselwirkung zwischen p53- und MDM2-Proteinen.
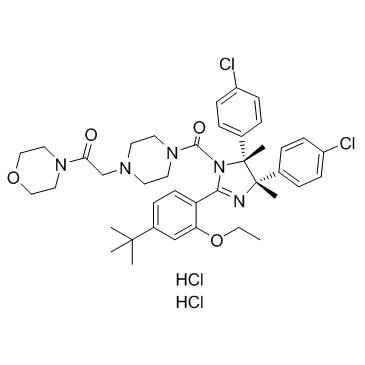
-
GC36837
p53 and MDM2 proteins-interaction-inhibitor racemic
p53- und MDM2-Protein-Interaktionsinhibitor racemisch (Verbindung 2j) ist ein Inhibitor der Wechselwirkung zwischen p53- und MDM2-Proteinen.
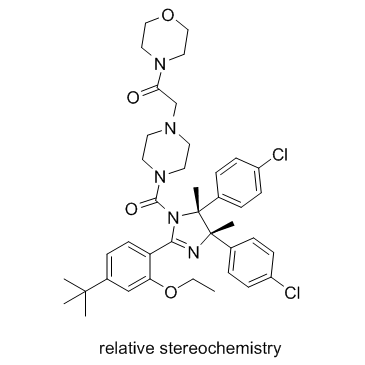
-
GP10036
p53 tumor suppressor fragment
Regulates cell cycle
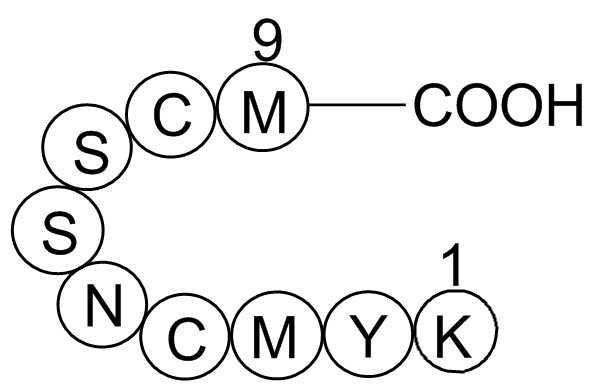
-
GC65961
P53R3
P53R3 ist ein potenter p53-Reaktivator und stellt die sequenzspezifische DNA-Bindung von p53-Hot-Spot-Mutanten wieder her, einschließlich p53R175H, p53R248W und p53R273H. P53R3 induziert p53-abhÄngige antiproliferative Wirkungen mit viel hÖherer SpezifitÄt als PRIMA-1. P53R3 verstÄrkt die Rekrutierung von Wildtyp-p53 und p53M237I zu mehreren Zielgen-Promotoren. P53R3 verstÄrkt stark die mRNA-, Gesamtprotein- und ZelloberflÄchenexpression des Todesrezeptors Todesrezeptor 5 (DR5). P53R3 wird fÜr die Krebsforschung verwendet.
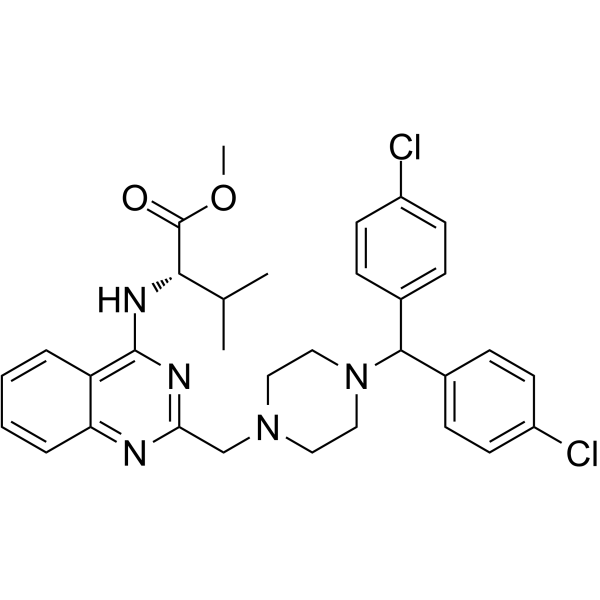
-
GC45758
Paclitaxel octadecanedioate
A prodrug form of paclitaxel
-
GC47853
Paclitaxel-d5
Paclitaxel-d5 ist ein Deuterium-markiertes Paclitaxel. Paclitaxel ist ein natÜrlich vorkommendes antineoplastisches Mittel und stabilisiert die Tubulinpolymerisation.
-
GC12658
PhiKan 083
-
GC36896
PhiKan 083 hydrochloride
PhiKan 083-Hydrochlorid ist ein Carbazol-Derivat, das an die OberflÄchenkavitÄt bindet und Y220C (eine p53-Mutante) mit einer Kd von 167 μM und einer relativen BindungsaffinitÄt (Kd) von 150 μM in Ln229-Zellen stabilisiert.
-
GC10538
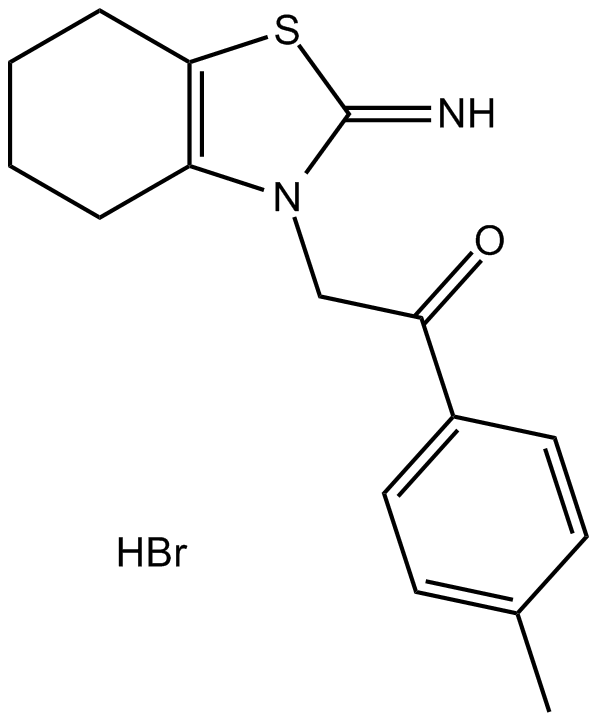
Pifithrin-α (PFTα)
Ein Inaktivator von p53
-
GC17262
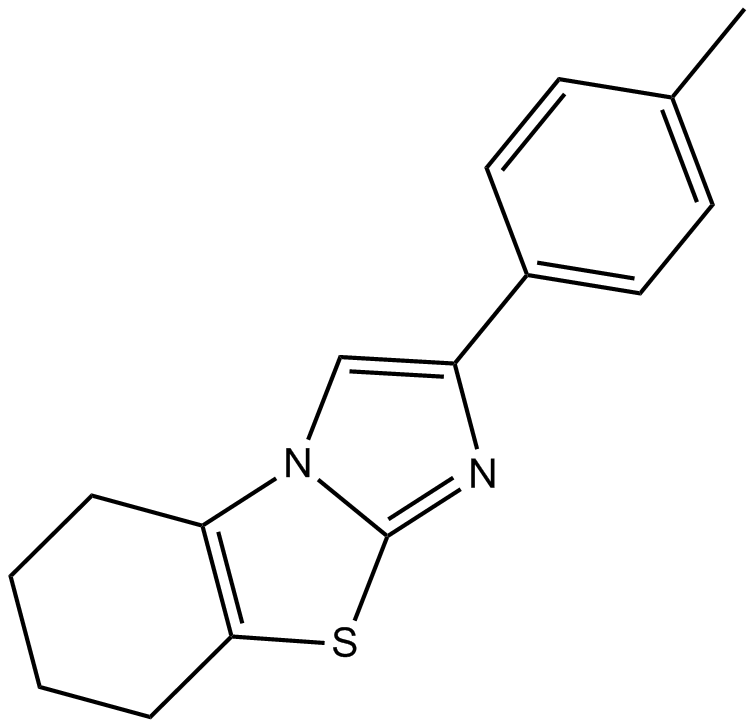
Pifithrin-β
Pifithrin-β (PFT β) ist ein potenter p53-Inhibitor mit einem IC50 von 23 μM.
-
GC10282
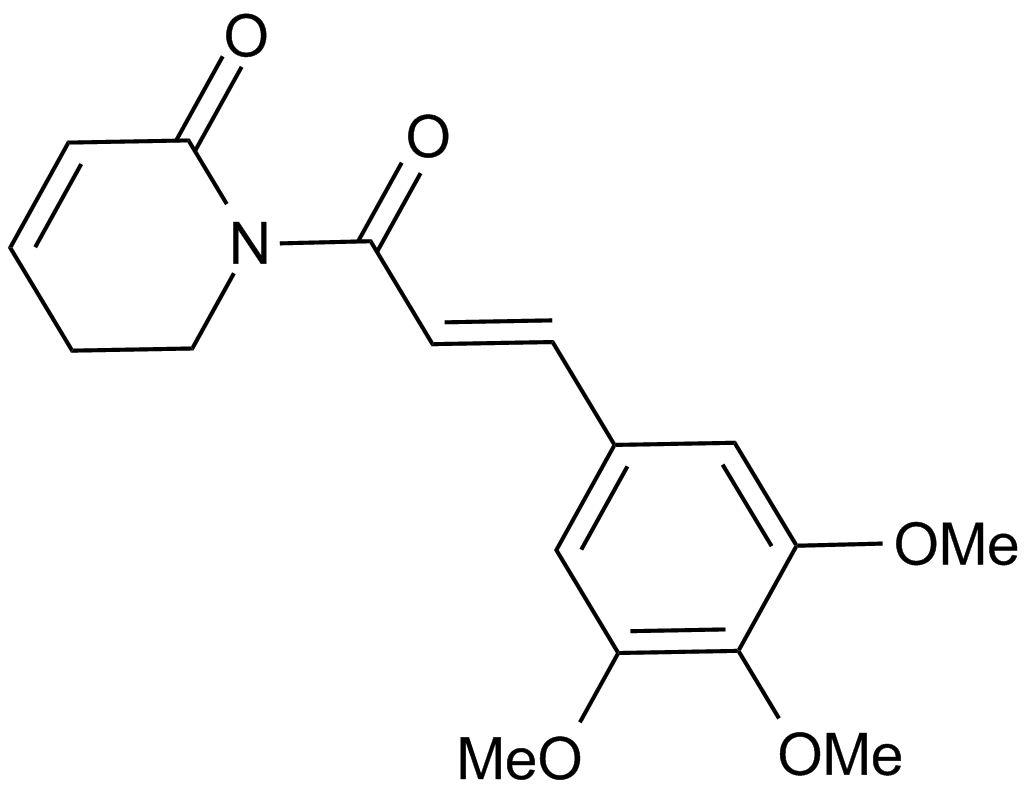
Piperlongumine
An alkaloid with anticancer and antioxidant activities
-
GC32946
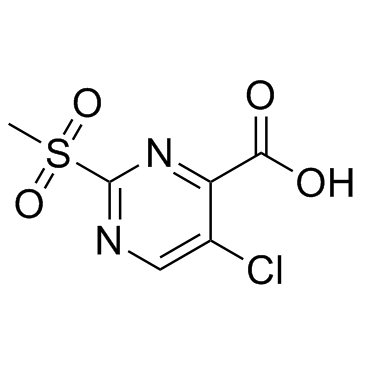
PK11007
PK11007 ist ein milder Thiolalkylator mit AntikrebsaktivitÄt. PK11007 stabilisiert p53 durch selektive Alkylierung von zwei oberflÄchenexponierten Cysteinen, ohne seine DNA-BindungsaktivitÄt zu beeintrÄchtigen. PK11007 induziert den Tod von mutierten p53-Krebszellen, indem es die Werte der reaktiven Sauerstoffspezies (ROS) erhÖht.
-
GC44652
PK7242 (maleate)
The protein p53, often called the 'guardian of the genome,' is a transcription factor that is activated in response to cellular stress (low oxygen levels, heat shock, DNA damage, etc.) and acts to prevent further proliferation of the stressed cell by promoting cell cycle arrest or apoptosis.
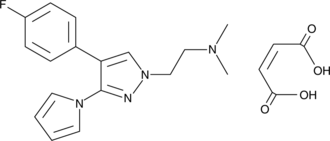
-
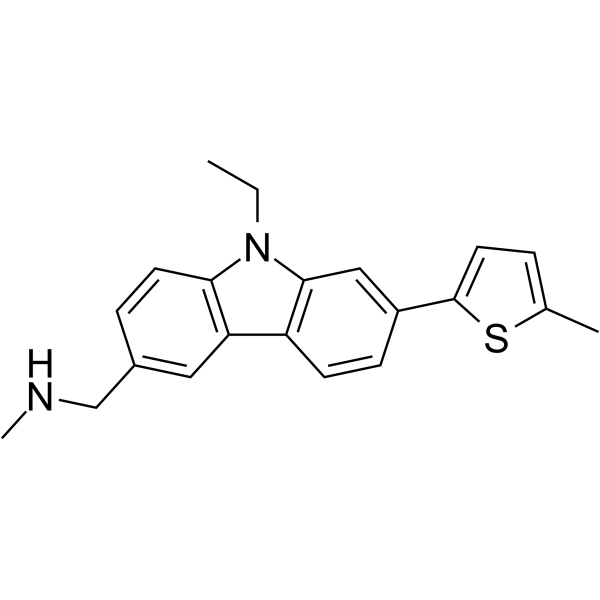
GC64768
PK9327
PK9327 ist ein niedermolekularer Stabilisator, der auf hohlraumerzeugende p53-Krebsmutationen abzielt.
-
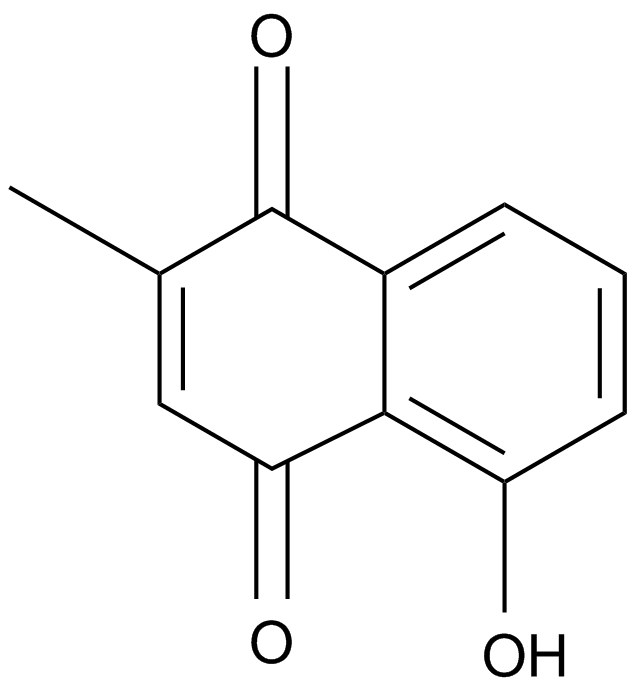
GC10315
Plumbagin
A natural naphthoquinone
-
GC12086
PRIMA-1
A re-activator of the apoptotic function of mutant p53 proteins
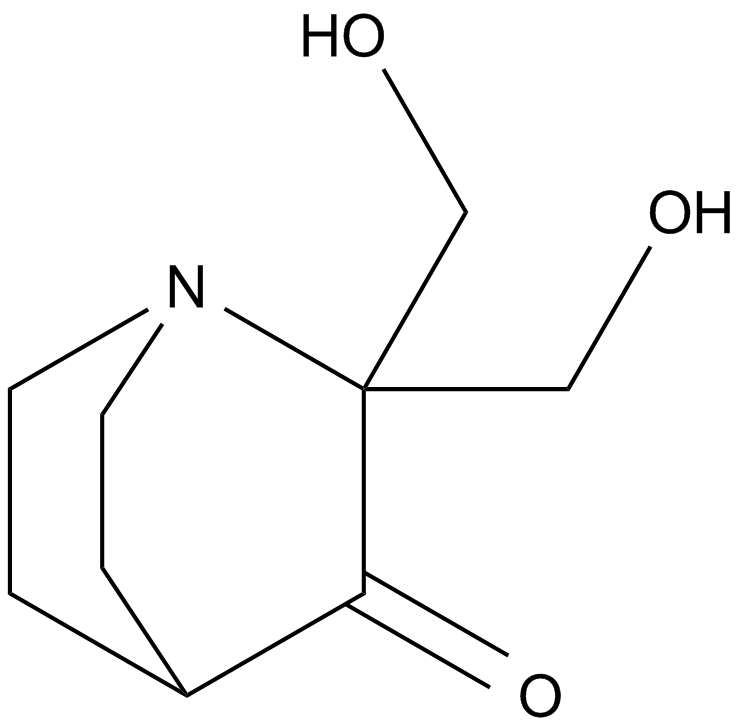
-
GC37010
PROTAC MDM2 Degrader-1
PROTAC MDM2 Degrader-1 ist ein MDM2-Degrader, der auf der PROTAC-Technologie basiert. PROTAC MDM2 Degrader-1 besteht aus einem potenten MDM2-Inhibitor, Linker und dem MDM2-Liganden fÜr E3-Ubiquitin-Ligase.
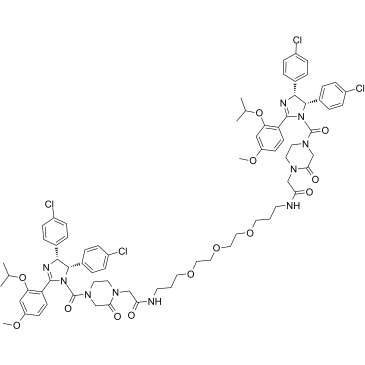
-
GC37011
PROTAC MDM2 Degrader-2
PROTAC MDM2 Degrader-2 ist ein MDM2-Degrader, der auf der PROTAC-Technologie basiert. PROTAC MDM2 Degrader-2 besteht aus einem potenten MDM2-Inhibitor, Linker und dem MDM2-Liganden fÜr E3-Ubiquitin-Ligase.
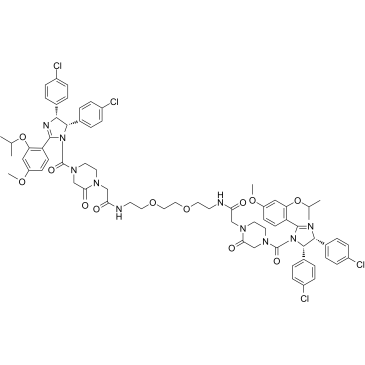
-
GC37012
PROTAC MDM2 Degrader-3
PROTAC MDM2 Degrader-3 ist ein MDM2-Degrader, der auf der PROTAC-Technologie basiert. PROTAC MDM2 Degrader-3 besteht aus einem potenten MDM2-Inhibitor, Linker und dem MDM2-Liganden fÜr E3-Ubiquitin-Ligase.
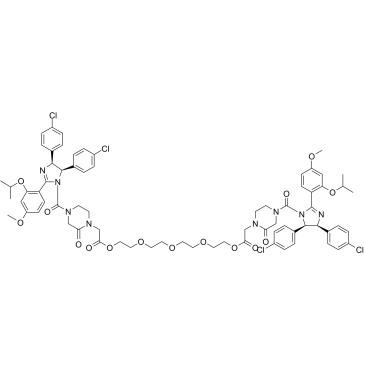
-
GC37013
PROTAC MDM2 Degrader-4
PROTAC MDM2 Degrader-4 ist ein MDM2-Degrader, der auf der PROTAC-Technologie basiert. PROTAC MDM2 Degrader-4 besteht aus einem potenten MDM2-Inhibitor, Linker und dem MDM2-Liganden fÜr E3-Ubiquitin-Ligase.
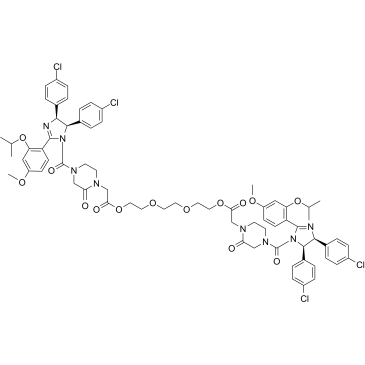
-
GC15946
ReACp53
ReACp53 kÖnnte die p53-Amyloidbildung hemmen und die p53-Funktion in Krebszelllinien retten.
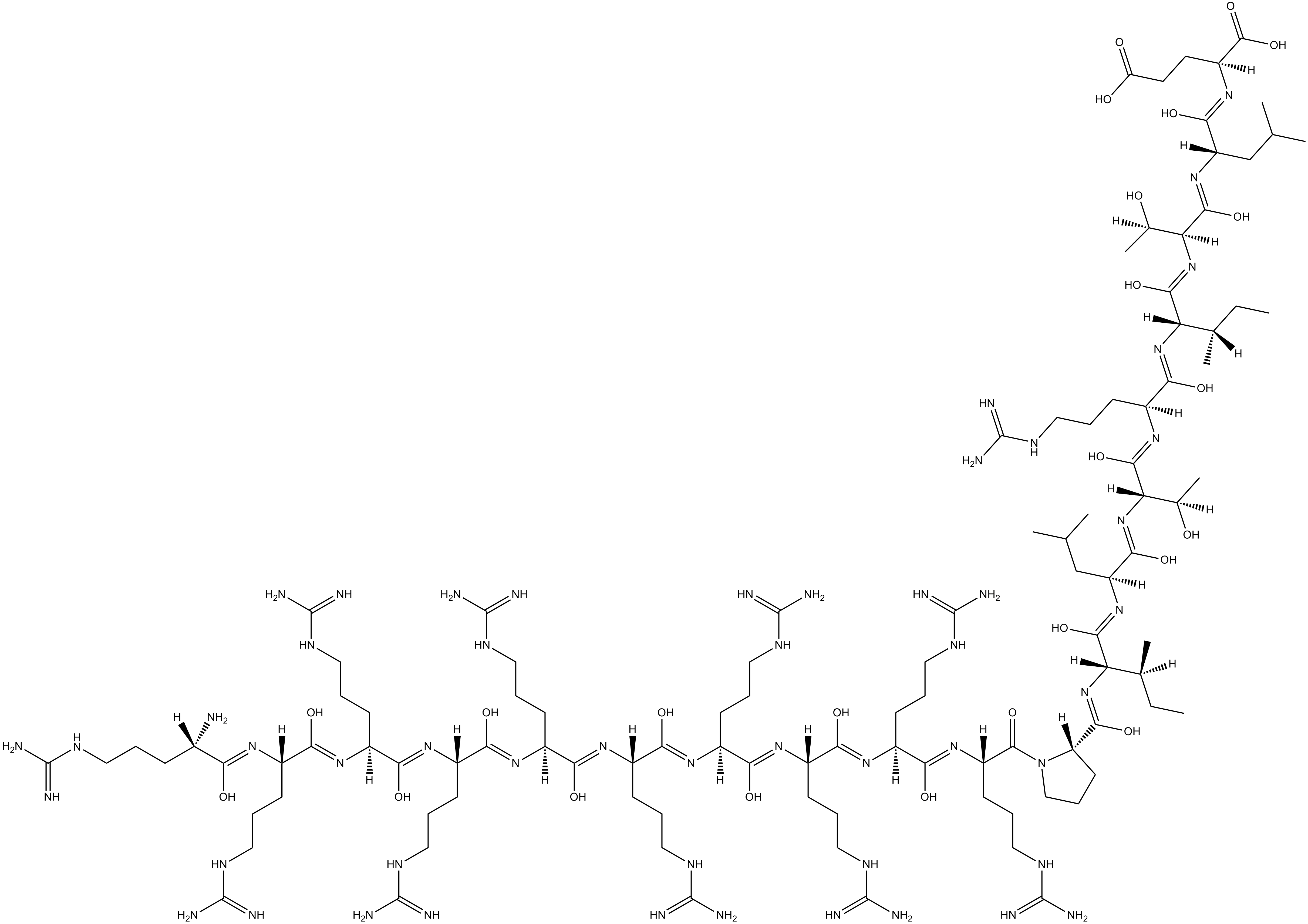
-
GC13019
RG7112
An inhibitor of the MDM2-p53 interaction
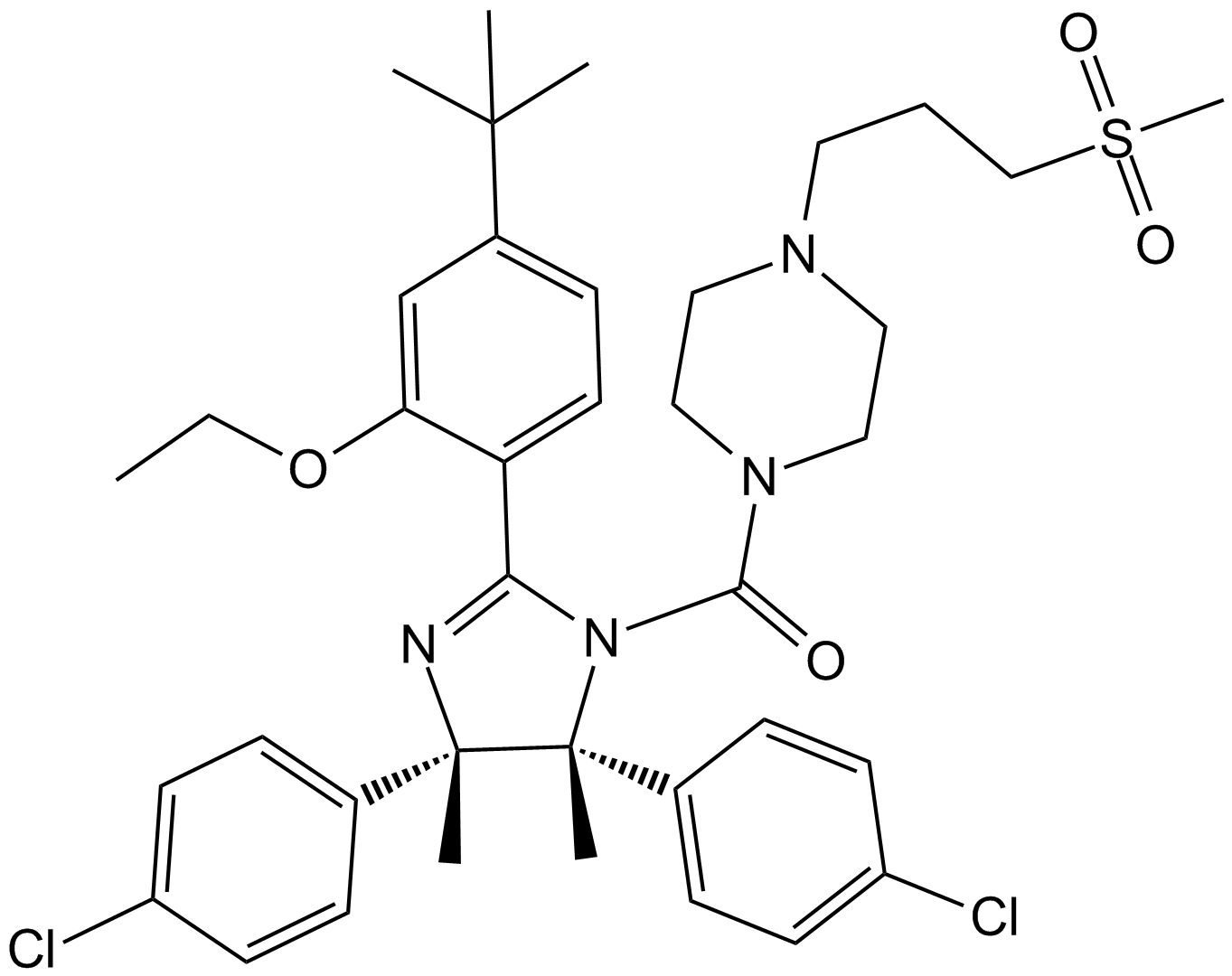
-
GC11594
RG7388
An inhibitor of the MDM2-p53 interaction
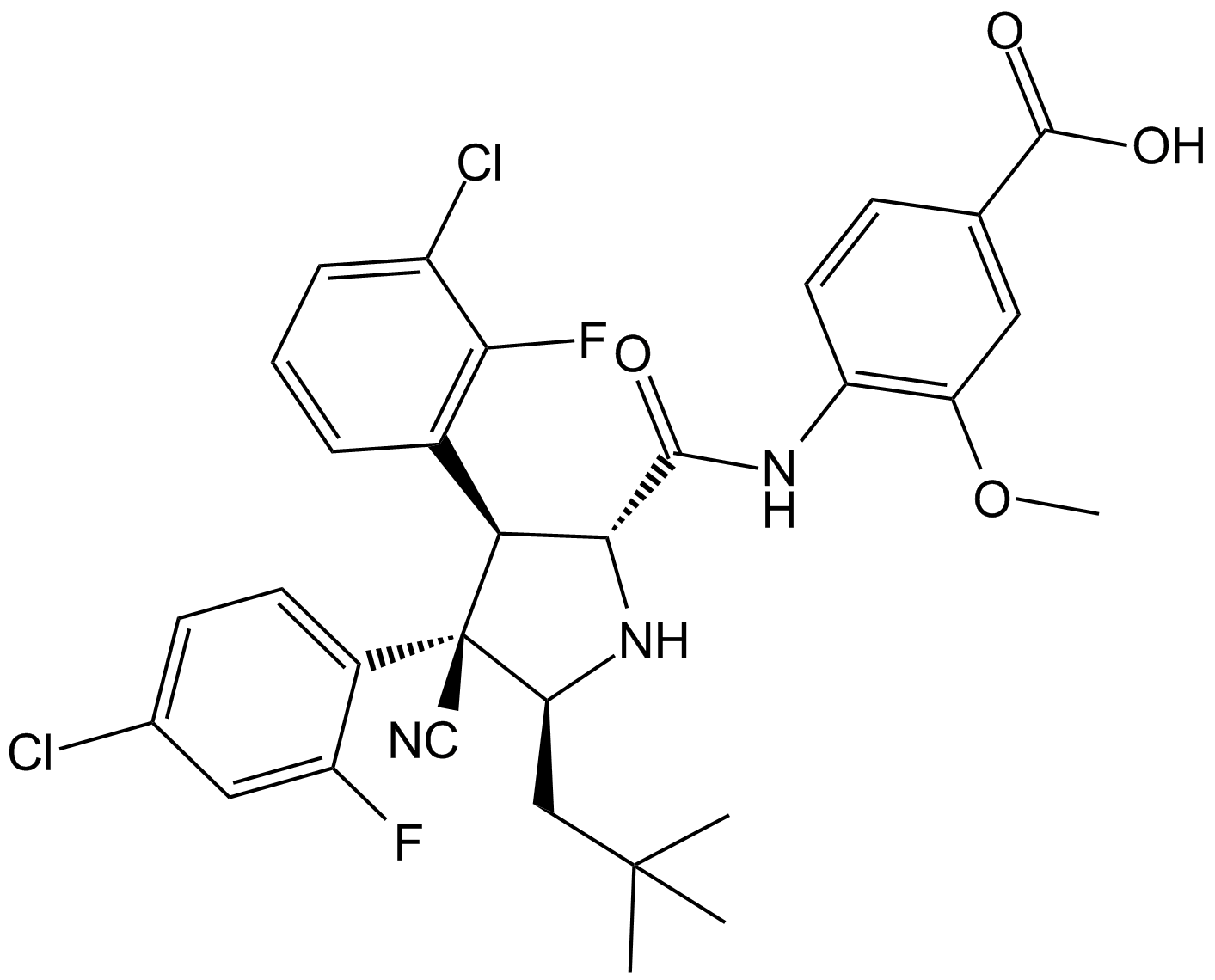
-
GC12793
RITA (NSC 652287)
An inhibitor of the p53-HDM-2 interaction
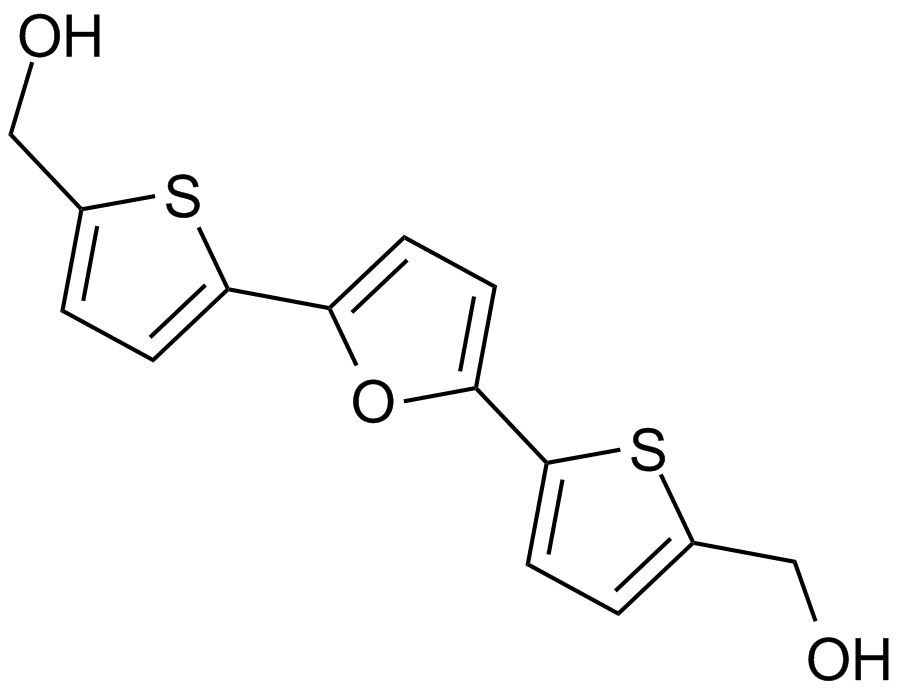
-
GC37549
RO-5963
RO-5963 ist ein dualer p53-MDM2- und p53-MDMX-Inhibitor mit IC50-Werten von ~17 nM bzw. ~24 nM.
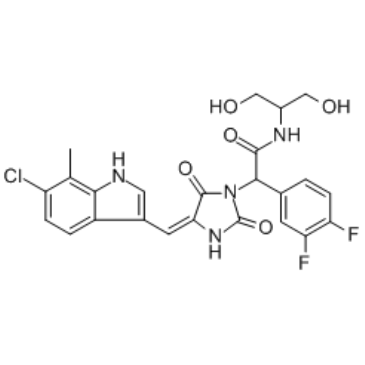
-
GC19312
RO8994
RO8994 ist eine hochwirksame und selektive Serie von niedermolekularen Spiroindolinon-MDM2-Inhibitoren mit IC50 von 5 nM (HTRF-Bindungsassays) und 20 nM (MTT-Proliferationsassays).
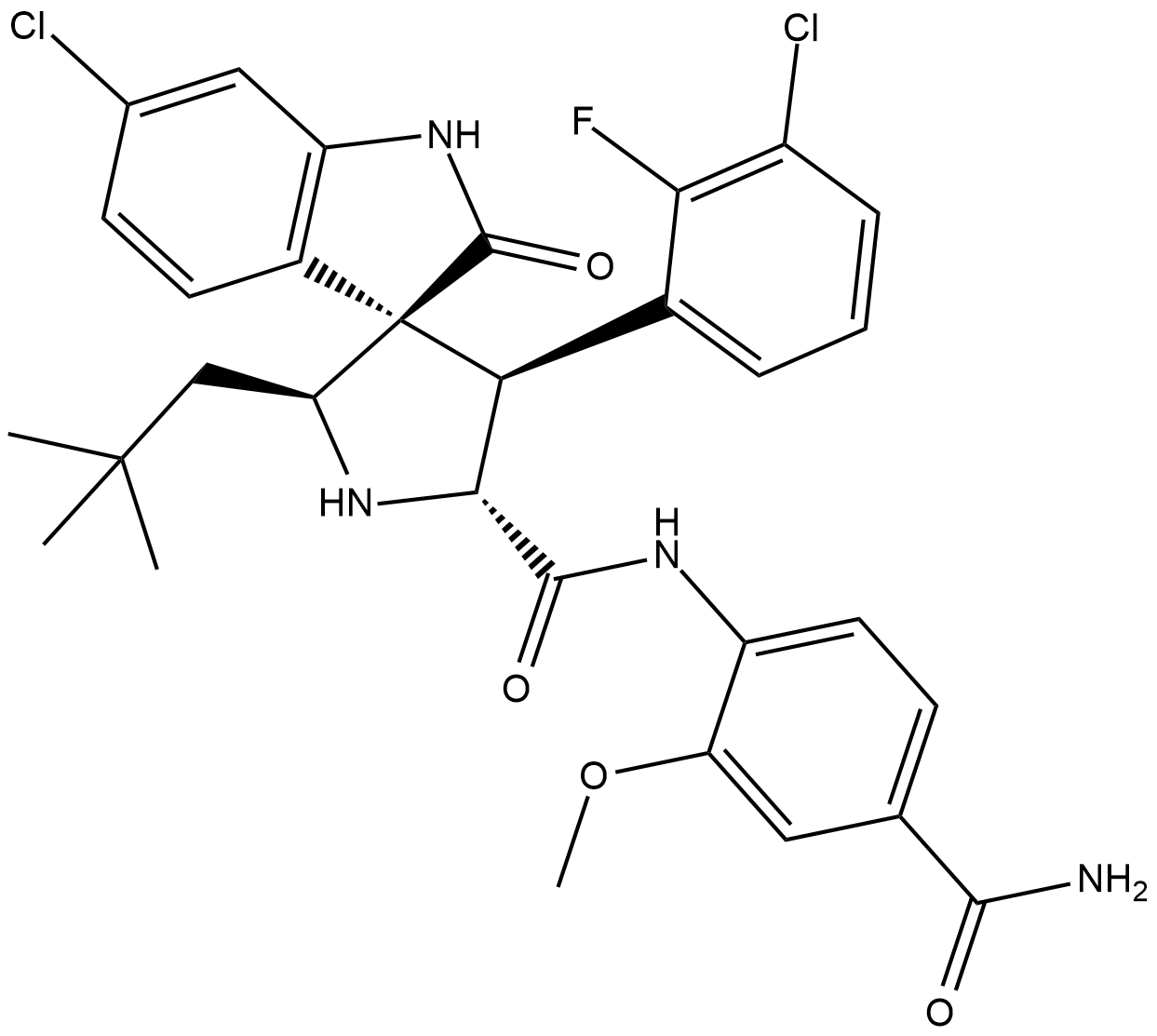
-
GC18624
Roslin-2
Roslin-2 (Benzylhexamethylentetraminbromid) ist ein p53-Reaktivator mit Antikrebswirkung. Roslin-2 bindet FAK, unterbricht die Bindung von FAK und p53.
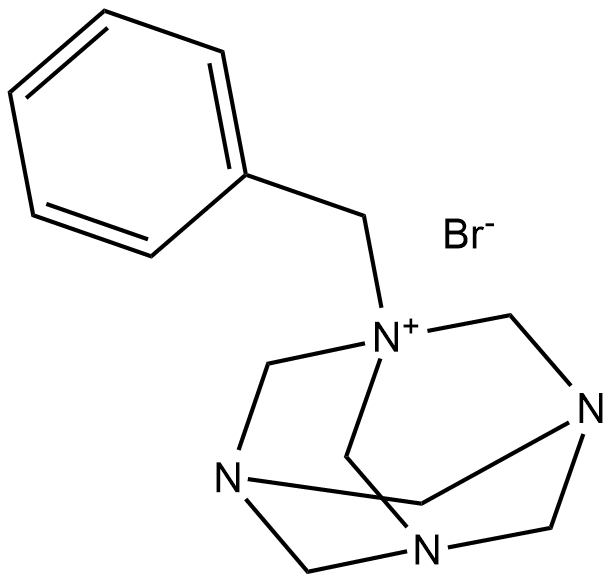
-
GC13590
SJ 172550
A small molecule inhibitor of MDMX
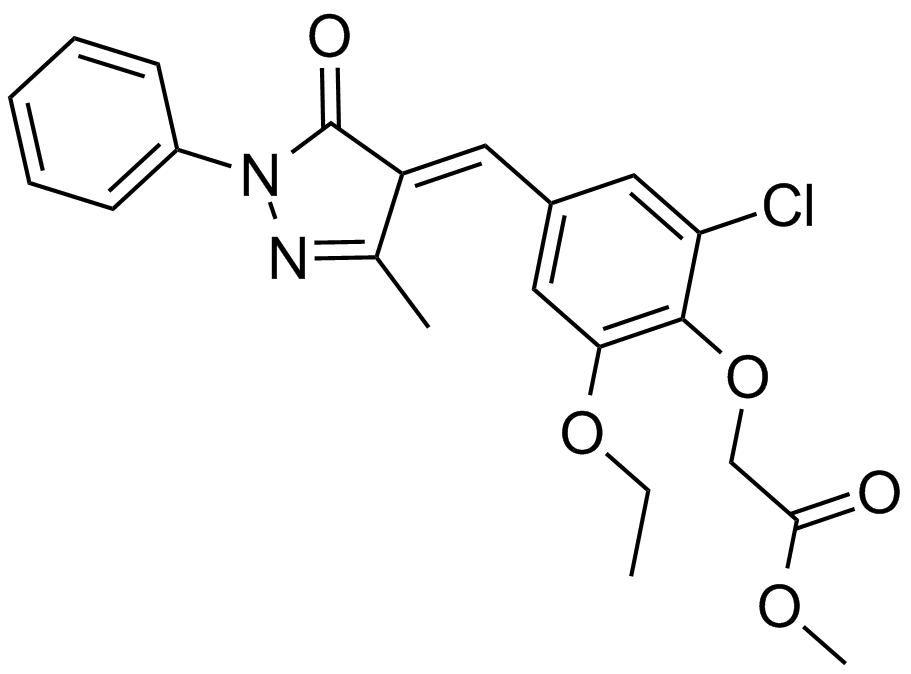
-
GC65284
SLMP53-1
SLMP53-1 ist ein Wildtyp- und mutanter p53-Reaktivator mit vielversprechender AntitumoraktivitÄt. SLMP53-1 vermittelt die Neuprogrammierung des Glukosestoffwechsels in Krebszellen. SLMP53-1 verringert die Angiogenese, verringert die Bildung von EndothelzellschlÄuchen und die Expressionsspiegel des vaskulÄren endothelialen Wachstumsfaktors (VEGF).
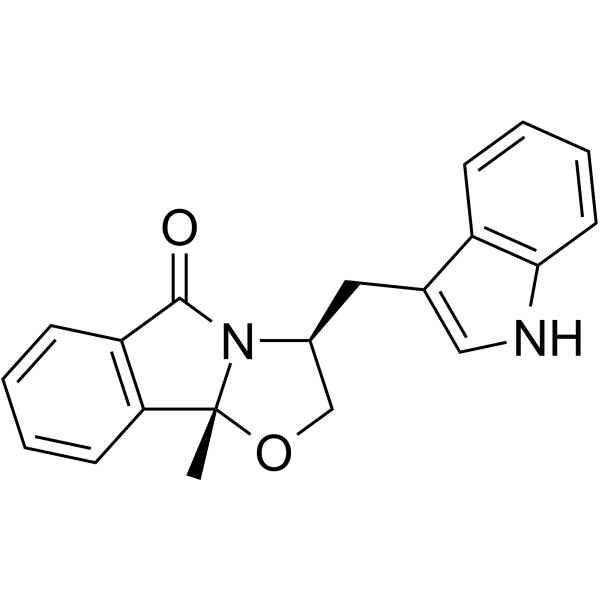
-
GC16371
Solasodine
An alkaloid with diverse biological activities
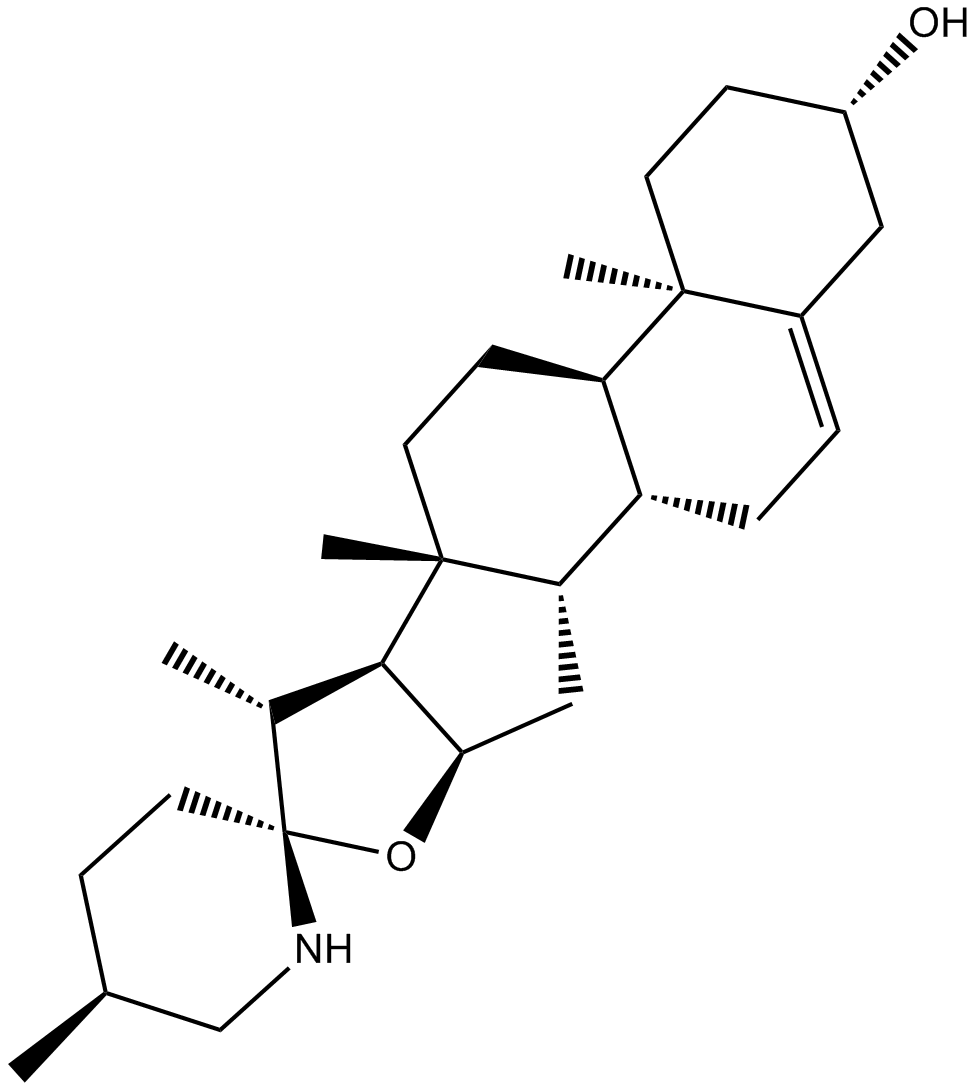
-
GC69968
Sulanemadlin
Sulanemadlin (ALRN-6924) is a potent p53-based peptide macrocycle. Sulanemadlin is an inhibitor of protein-protein interactions between p53-MDM2, p53-MDMX, or p53 and MDM2 and MDMX. Sulanemadlin can be used for cancer research.
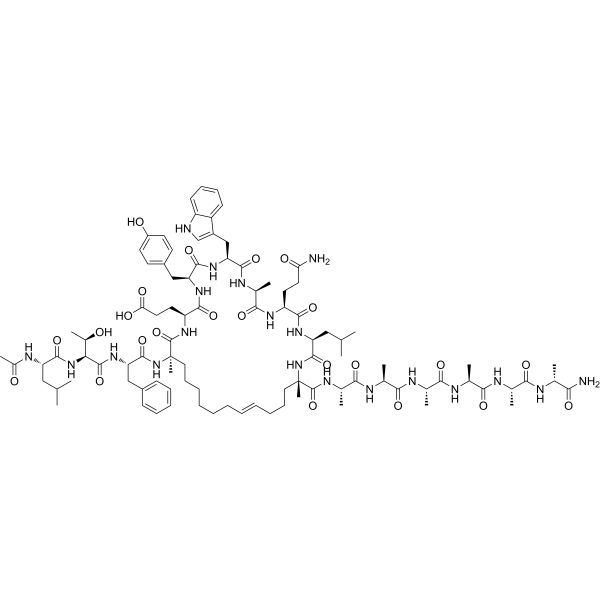
-
GC14165
Tenovin-1
A small molecule activator of p53
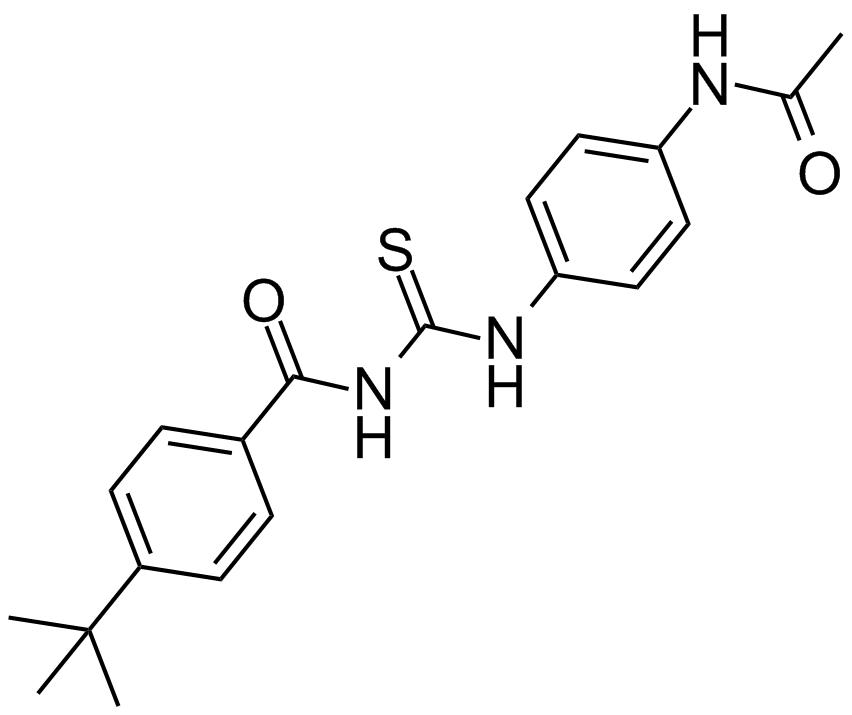
-
GC12337
Tenovin-3
Tenovin-3 ist ein p53-Aktivator.
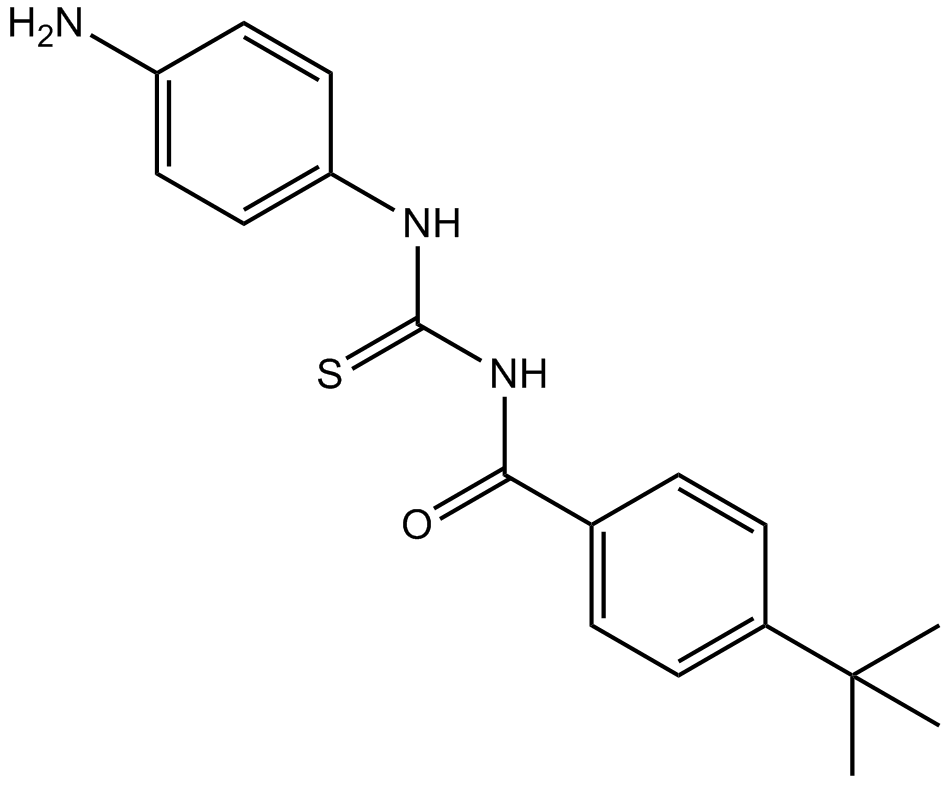
-
GC16436
Tenovin-6
Tenovin-6, ein Analogon von Tenovin-1, ist ein Aktivator der TranskriptionsaktivitÄt von p53. Tenovin-6 hemmt die Protein-Deacetylase-AktivitÄten von gereinigtem humanem SIRT1, SIRT2 und SIRT3 mit IC50-Werten von 21 μM, 10 μM bzw. 67 μM. Tenovin-6 hemmt auch die Dihydroorotat-Dehydrogenase (DHODH).
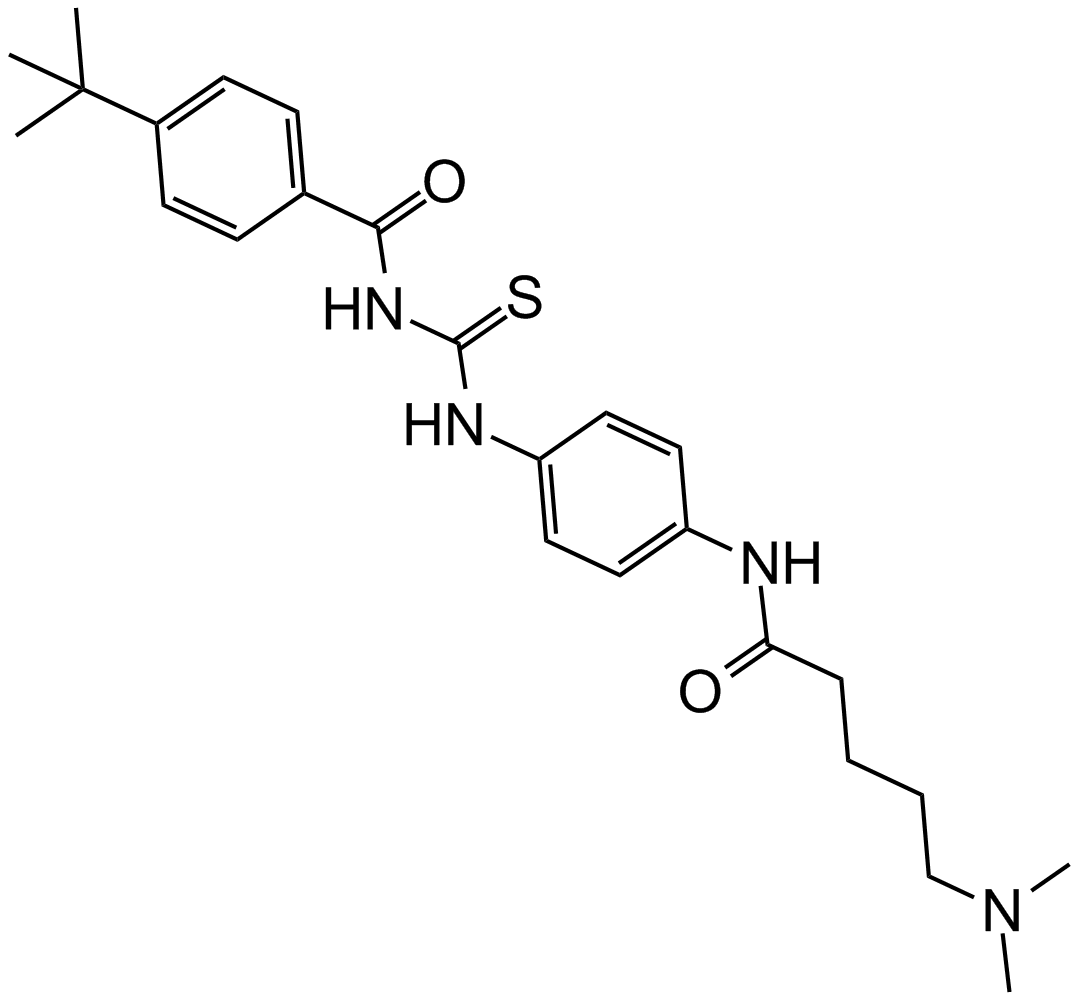
-
GC37761
Tenovin-6 Hydrochloride
Tenovin-6-Hydrochlorid, ein Analogon von Tenovin-1, ist ein Aktivator der TranskriptionsaktivitÄt von p53. Tenovin-6-Hydrochlorid hemmt die Protein-Deacetylase-AktivitÄten von gereinigtem humanem SIRT1, SIRT2 und SIRT3 mit IC50-Werten von 21 μM, 10 μM bzw. 67 μM. Tenovin-6-Hydrochlorid hemmt auch die Dihydroorotat-Dehydrogenase (DHODH).
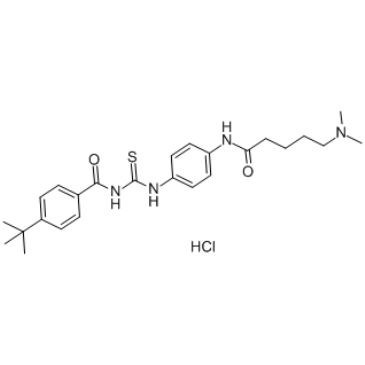
-
GC61352
Triglycidyl isocyanurate
Triglycidylisocyanurat (TGIC; Teroxirone) ist ein Triazentriepoxid mit antiangiogenetischen und antineoplastischen AktivitÄten.
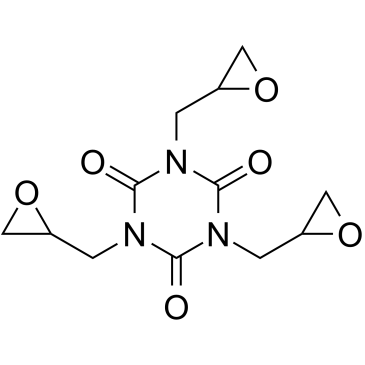
-
GP10028
tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus]
P53 binding protein fragment
![tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus] Chemical Structure tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus] Chemical Structure](/media/struct/GP1/GP10028.png)
-
GC11557
WR 1065
A radioprotective agent
-
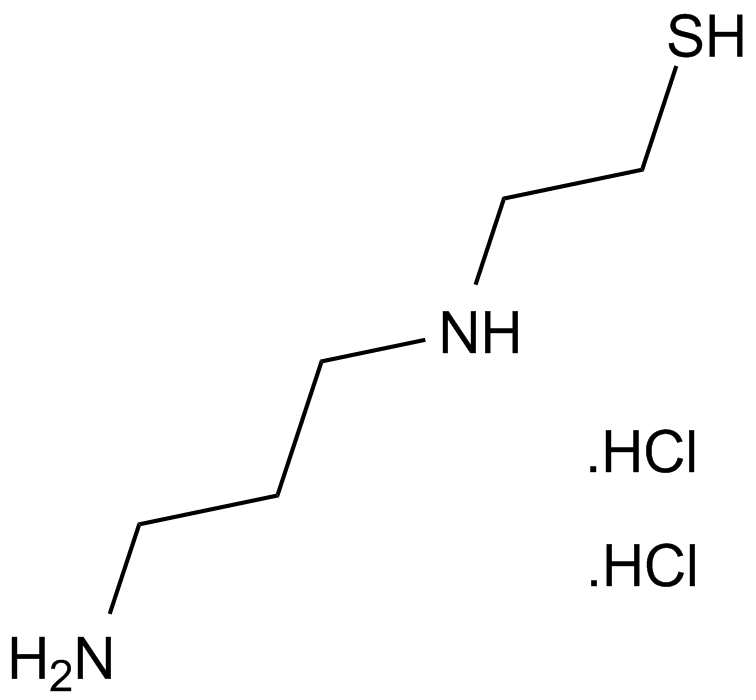
GC19545
WR-1065 dihydrochloride
WR-1065 dihydrochloride can protect normal tissues from the toxic effects of certain cancer drugs and activate p53 through a JNK-dependent signaling pathway.



