DNA Damage/DNA Repair
- MTH1(4)
- PARP(84)
- ATM/ATR(30)
- DNA Alkylating(20)
- DNA Ligases(3)
- DNA Methyltransferase(25)
- DNA-PK(30)
- HDAC(136)
- Nucleoside Antimetabolite/Analogue(140)
- Telomerase(17)
- Topoisomerase(151)
- tankyrase(5)
- Antifolate(38)
- CDK(250)
- Checkpoint Kinase (Chk)(31)
- CRISPR/Cas9(9)
- Deubiquitinase(72)
- DNA Alkylator/Crosslinker(71)
- DNA/RNA Synthesis(400)
- Eukaryotic Initiation Factor (eIF)(23)
- IRE1(23)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(61)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Produkte für DNA Damage/DNA Repair
- Bestell-Nr. Artikelname Informationen
-
GC45353
4-oxo-27-TBDMS Withaferin A
4-oxo-27-TBDMS Withaferin A, ein Withaferin A-Derivat, zeigt starke antiproliferative Wirkungen auf die Tumorzellen. 4-oxo-27-TBDMS Withaferin A induziert die Apoptose von Tumorzellen. 4-Oxo-27-TBDMS Withaferin A ist ein Antikrebsmittel.
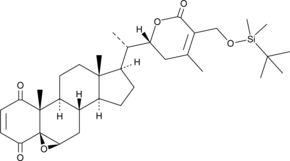
-
GC68557
4-Thio-2'-deoxyuridine
4-Thio-2'-deoxyuridine ist ein Analogon von Purinnukleosiden. Purinnukleosid-Analoga haben eine breite antitumorale Aktivität und zielen auf maligne Lymphsystem-Tumoren ab. Der Mechanismus der Antikrebswirkung in diesem Prozess beruht auf der Hemmung der DNA-Synthese, Induktion von Zellapoptose (Apoptose) usw.
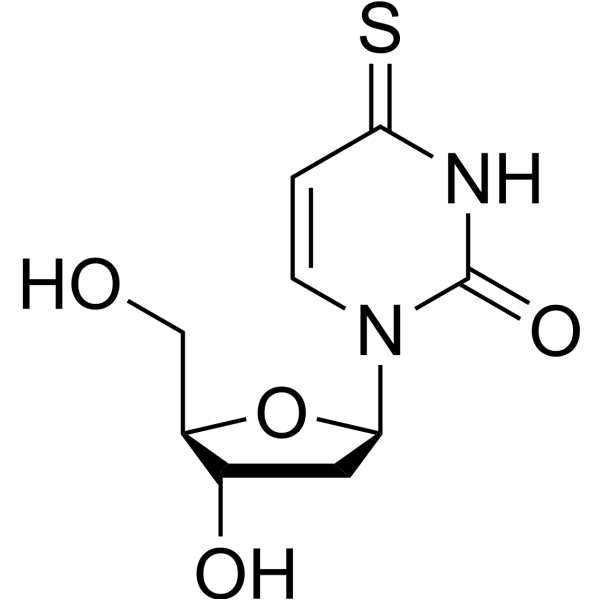
-
GC40477
4-Thiouracil
4-Thiouracil is a site-specific, photoactivatable probe used to detect RNA structures and nucleic acid-nucleic acid contacts.
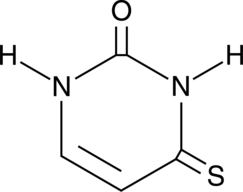
-
GC42474
4-Thiouridine
4-Thiouridin ist ein Ribonukleosid-Analogon, es wird hÄufig in der RNA-Analyse und (m)RNA-Markierung verwendet.
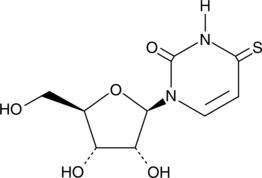
-
GC13104
4E1RCat
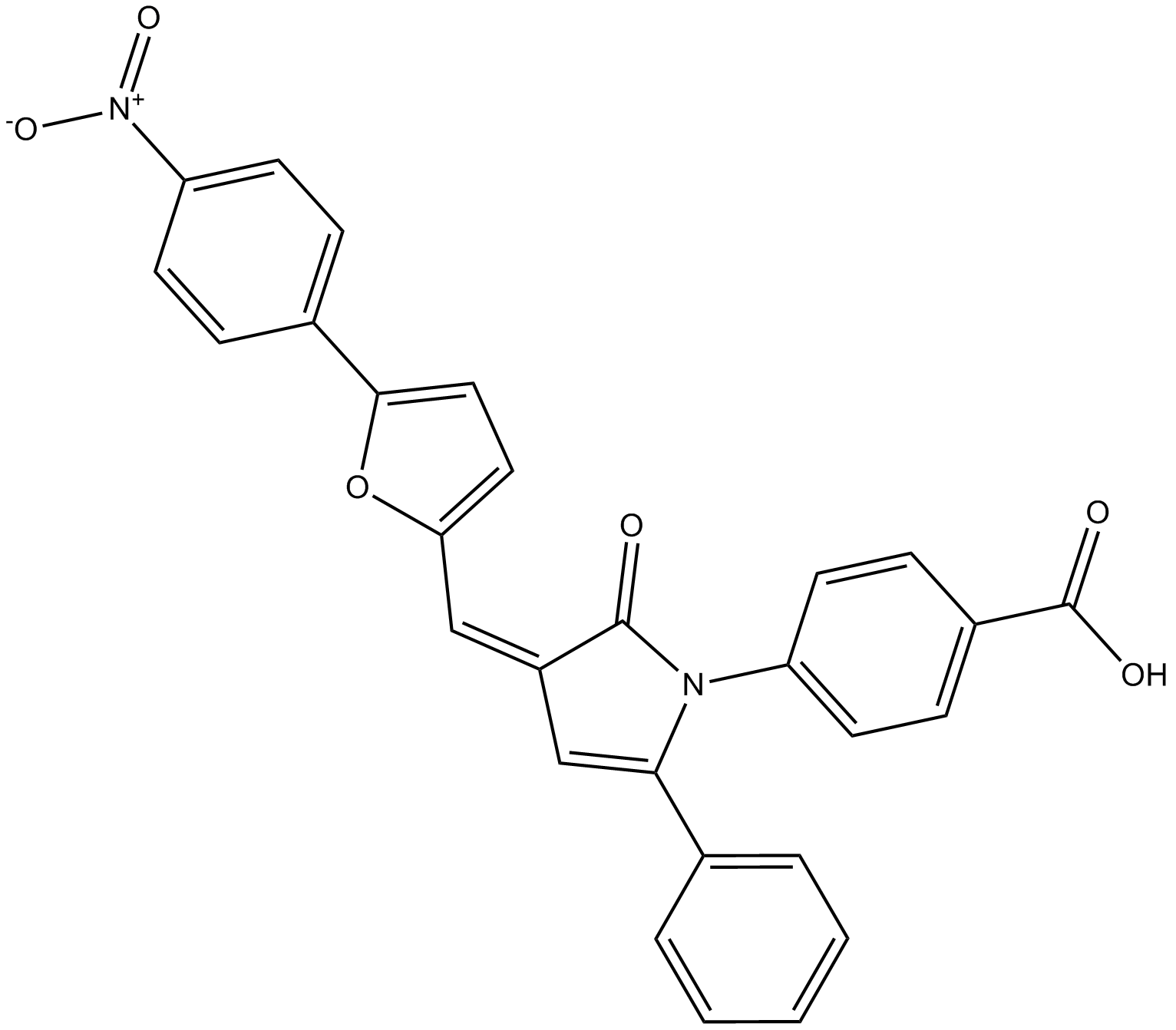
4E1RCat ist ein Inhibitor der Cap-abhÄngigen Translation und hemmt die eIF4E:eIF4GI-Wechselwirkung mit einem IC50 an von ~4 μM.
-
GC30785
4E2RCat
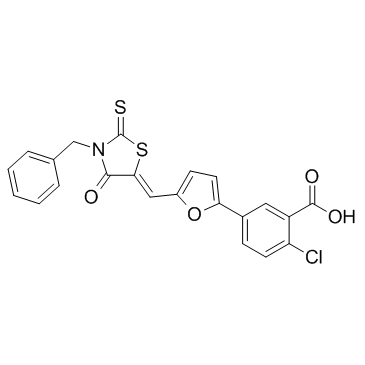
4E2RCat ist ein Inhibitor der eIF4E-eIF4G-Interaktion mit einer IC50 von 13,5 μM.
-
GC10468
4EGI-1
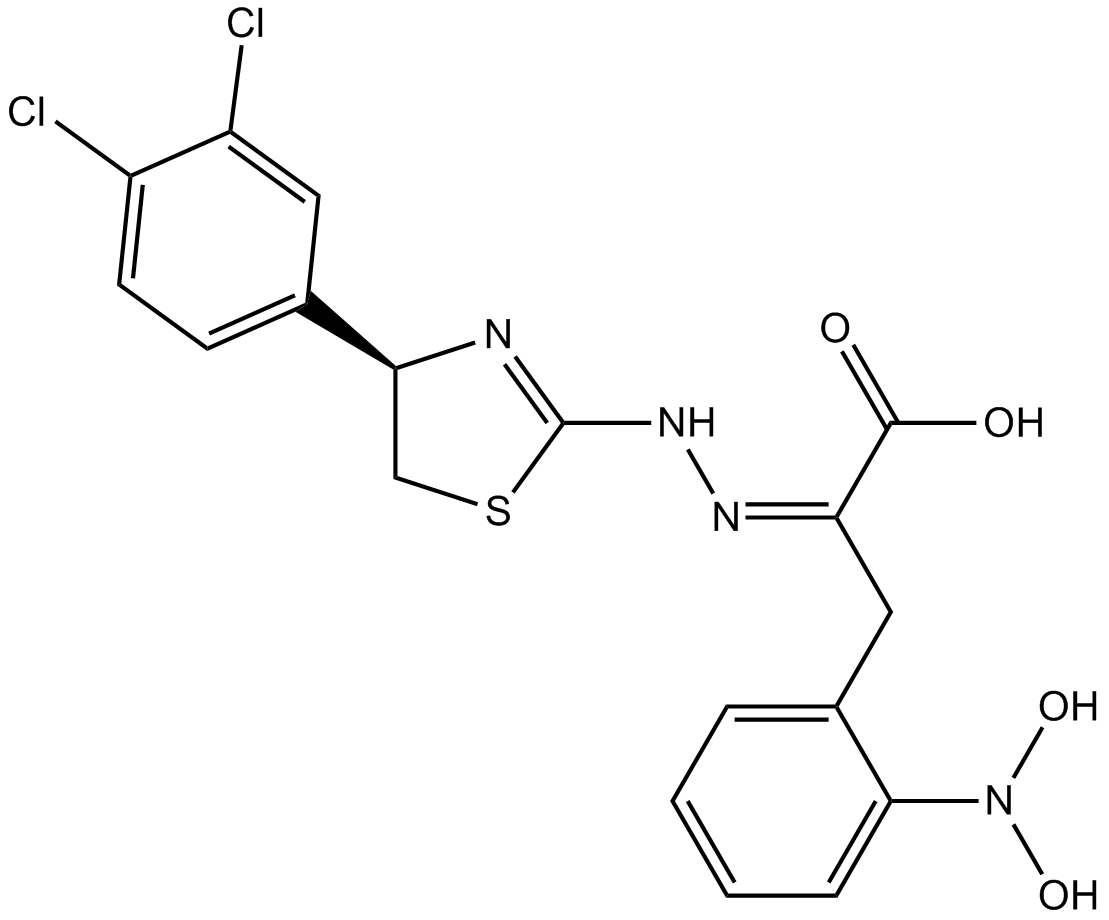
4EGI-1 ist ein Inhibitor der eIF4E/eIF4G-Interaktion mit einer Kd von 25 μM gegen die eIF4E-Bindung.
-
GC12667
4SC-202
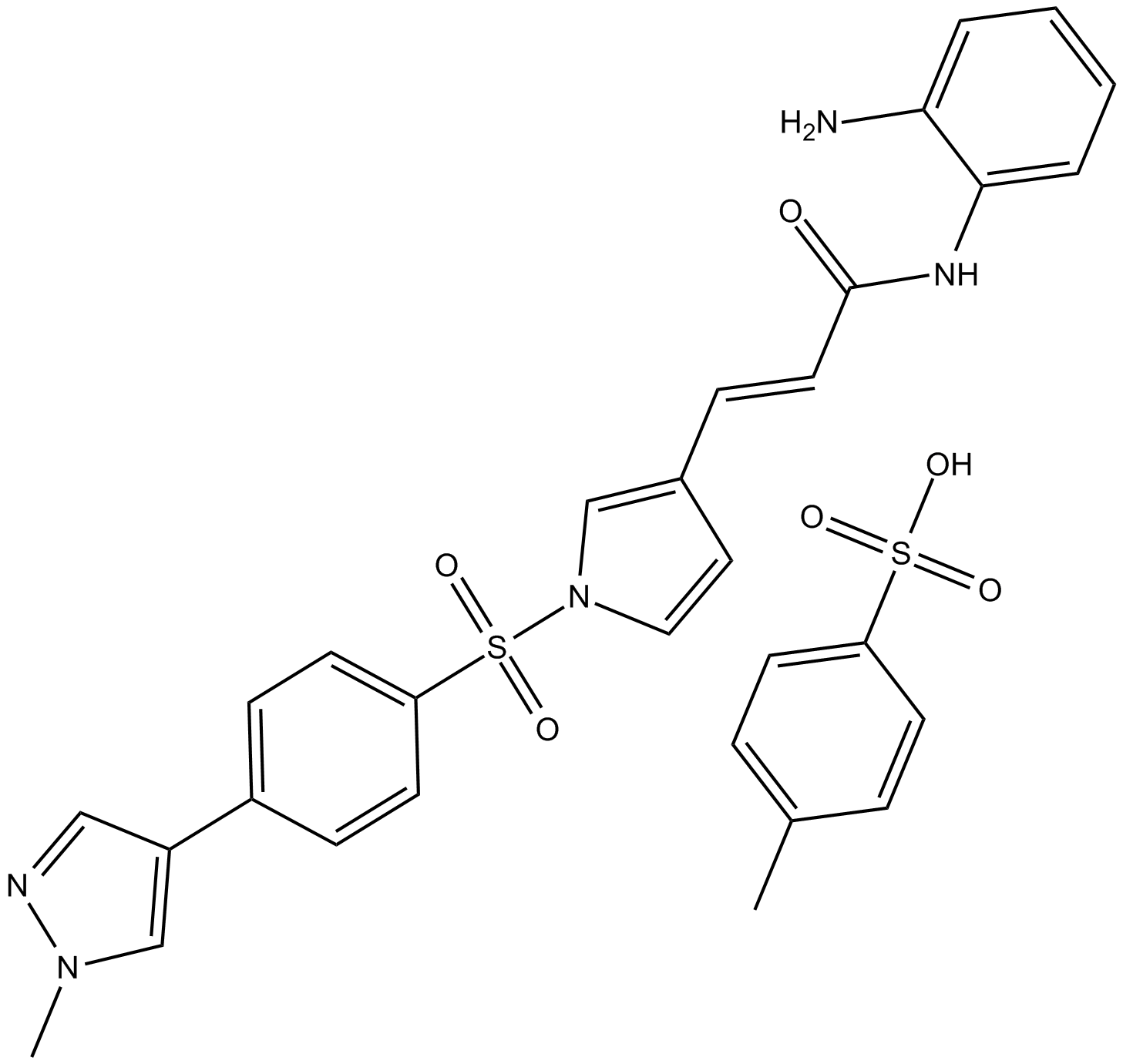
4SC-202 (4SC-202) ist ein selektiver HDAC-Inhibitor der Klasse I mit einem IC50-Wert von 1,20 μM, 1,12 μM und 0,57 ⋼M fÜr HDAC1, HDAC2 bzw. HDAC3. Es zeigt auch eine hemmende AktivitÄt gegen Lysin-spezifische Demethylase 1 (LSD1).
-
GC65520
5'-DMT-3'-TBDMS-ibu-rG
5'-DMT-3'-TBDMS-ibu-rG ist ein modifiziertes Nukleosid.

-
GC65402
5'-O-DMT-2'-O-TBDMS-Bz-rC
5'-O-DMT-&2#39;-O-TBDMS-Bz-rC ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
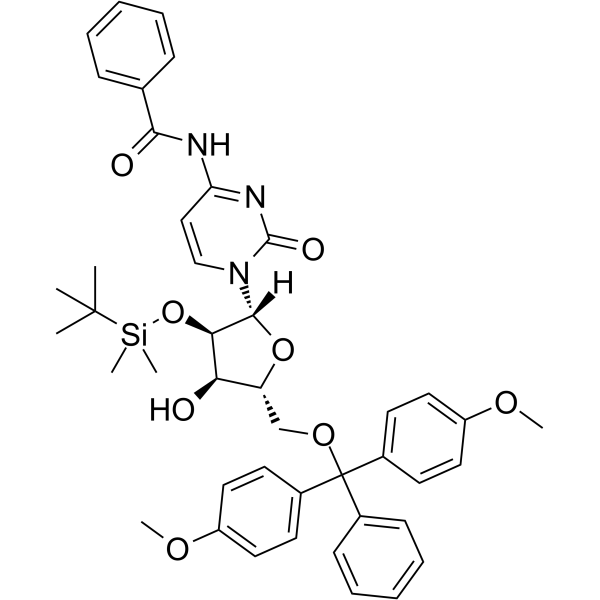
-
GC65164
5'-O-DMT-2'-O-TBDMS-rI
5'-O-DMT-&2#39;-O-TBDMS-rI ist ein modifiziertes Nukleosid.
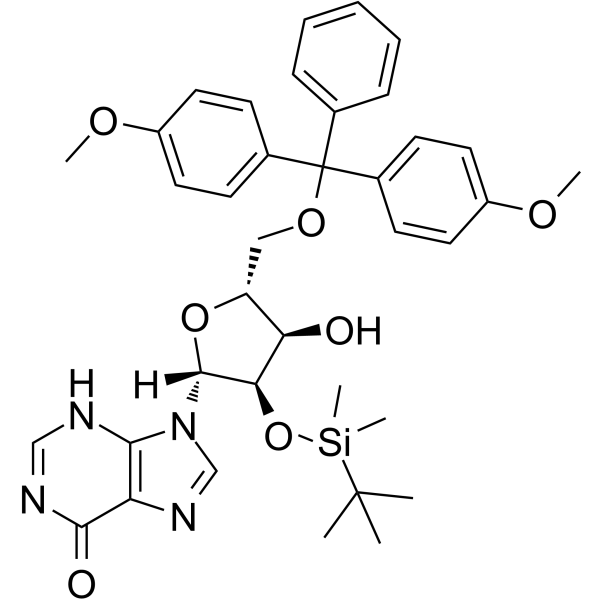
-
GC65396
5'-O-DMT-ibu-dC
5'-O-DMT-ibu-dC kann bei der Synthese von Oligodesoxyribonukleotiden verwendet werden.

-
GC64982
5'-O-DMT-N2-DMF-dG
5'-O-DMT-2'-O-TBDMS-rI ist ein modifiziertes Nucleosid.
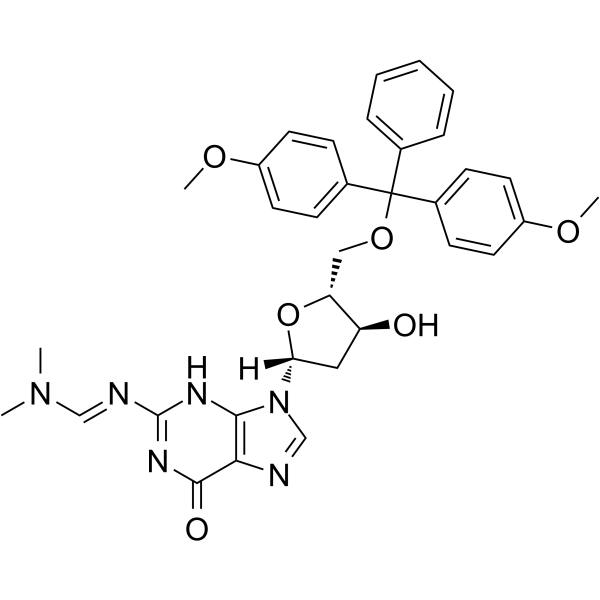
-
GC65482
5'-O-DMT-N6-ibu-dA
5'-O-DMT-N6-ibu-dA kann bei der Synthese von Oligodesoxyribonukleotiden verwendet werden.
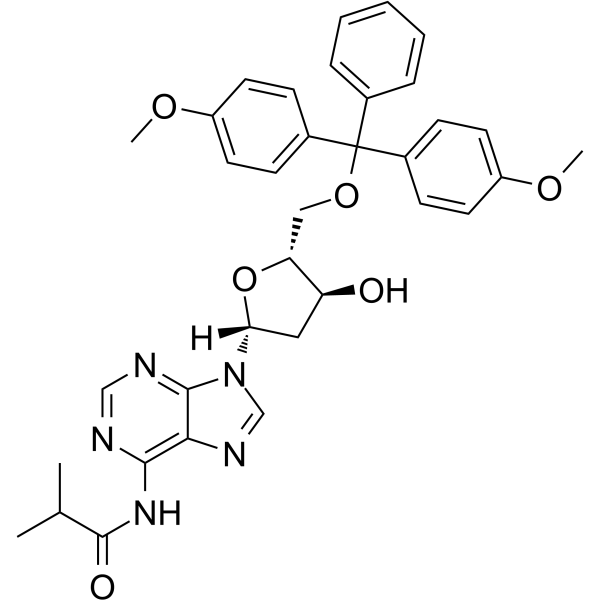
-
GC65562
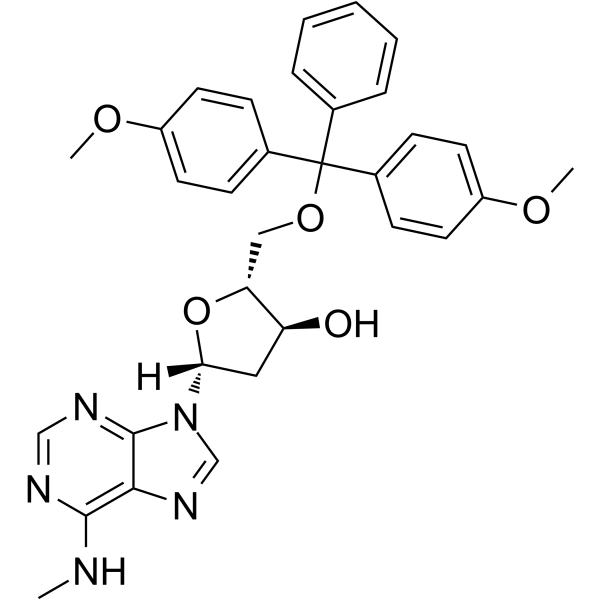
5'-O-DMT-N6-Me-2'-dA
5'-O-DMT-N6-Me-&2#39;-dA ist ein Nukleosid mit Schutz- und Modifikationswirkung.
-
GC65401
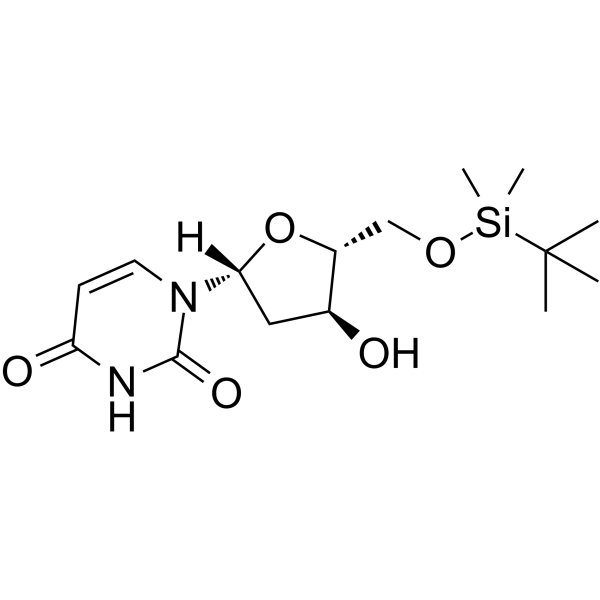
5'-O-TBDMS-dU
5'-O-TBDMS-dU kann bei der Synthese von Oligoribonukleotiden verwendet werden.
-
GC62157
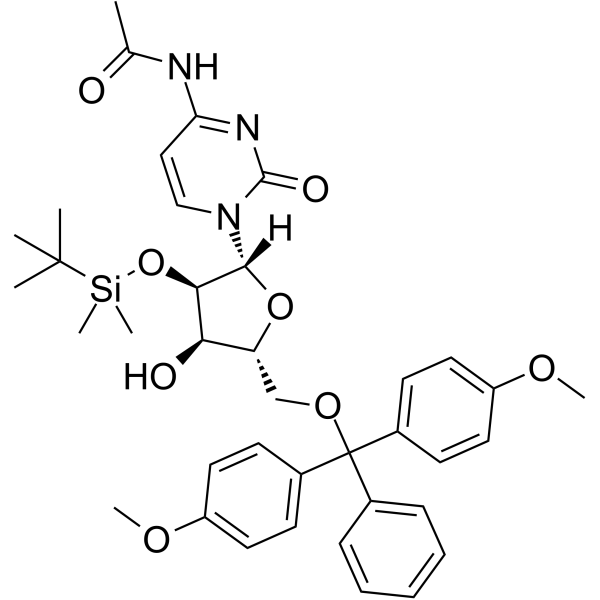
5’-O-DMT-2’-O-TBDMS-Ac-rC
5’-O-DMT-&2rsquo;-O-TBDMS-Ac-rC ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
-
GC62149
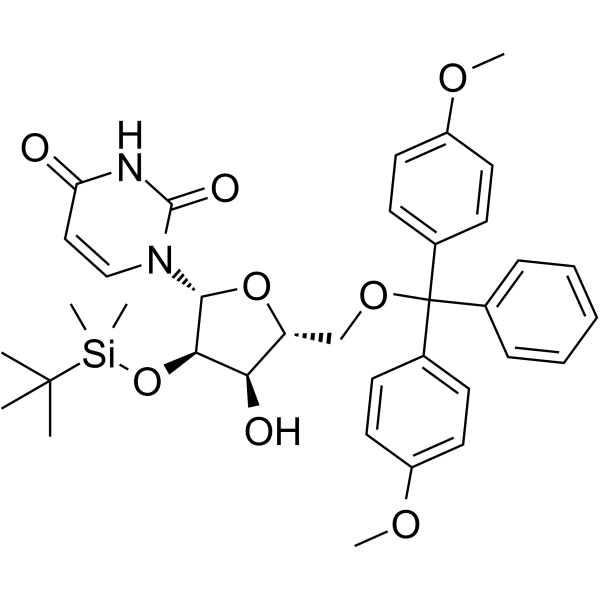
5’-O-DMT-2’-TBDMS-Uridine
5’-O-DMT-&2rsquo;-TBDMS-Uridin ist ein Desoxyribonukleosid, das für die Oligonukleotidsynthese verwendet wird.
-
GC62532
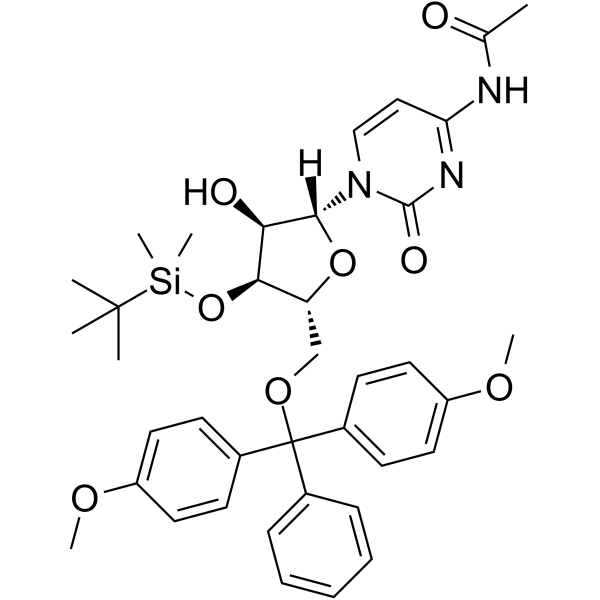
5’-O-DMT-3’-O-TBDMS-Ac-rC
5’-O-DMT-3’-O-TBDMS-Ac-rC ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
-
GC62573
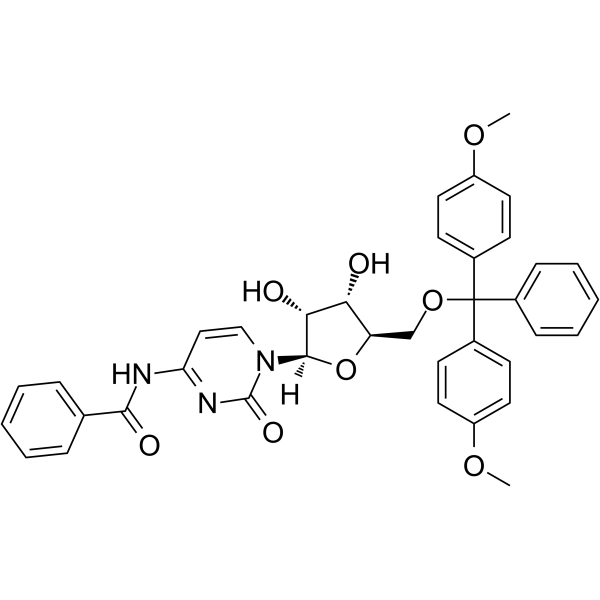
5’-O-DMT-Bz-rC
5'-O-DMT-Bz-Rc ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
-
GC62574
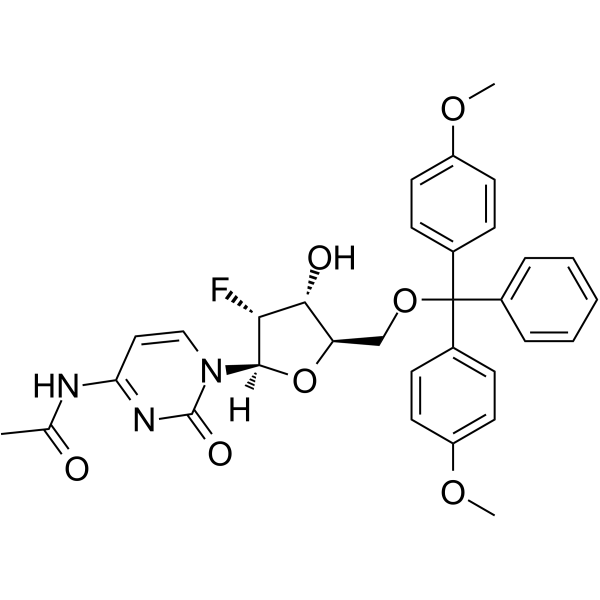
5’-O-DMT-N4-Ac-2’-F-dC
5’-O-DMT-N4-Ac-&2rsquo;-F-dC ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
-
GC62575
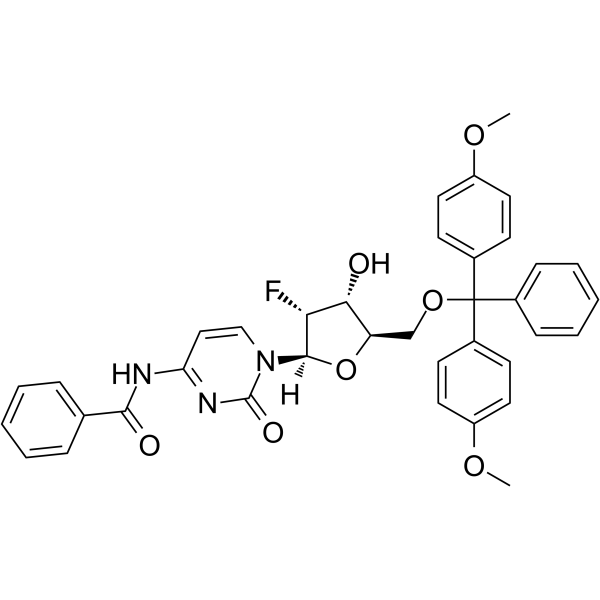
5’-O-DMT-N4-Bz-2’-F-dC
5’-O-DMT-N4-Bz-&2rsquo;-F-dC ist ein Nukleosid mit Schutz- und Modifikationswirkung.
-
GC62576
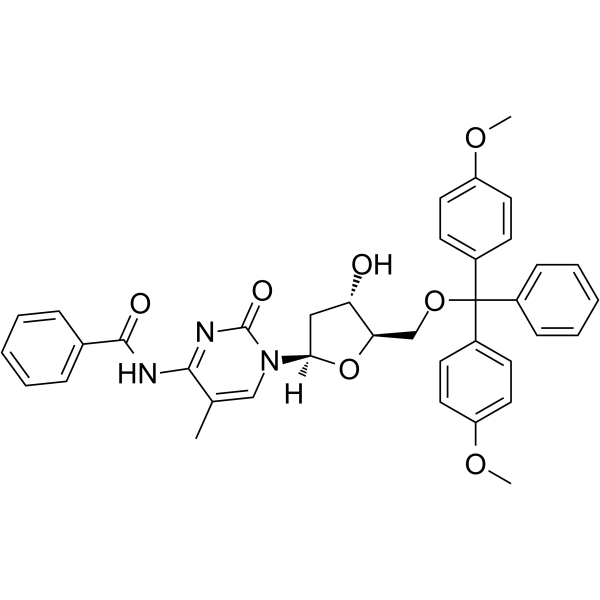
5’-O-DMT-N4-Bz-5-Me-dC
5’-O-DMT-N4-Bz-5-Me-dC ist ein modifiziertes Nukleosid.
-
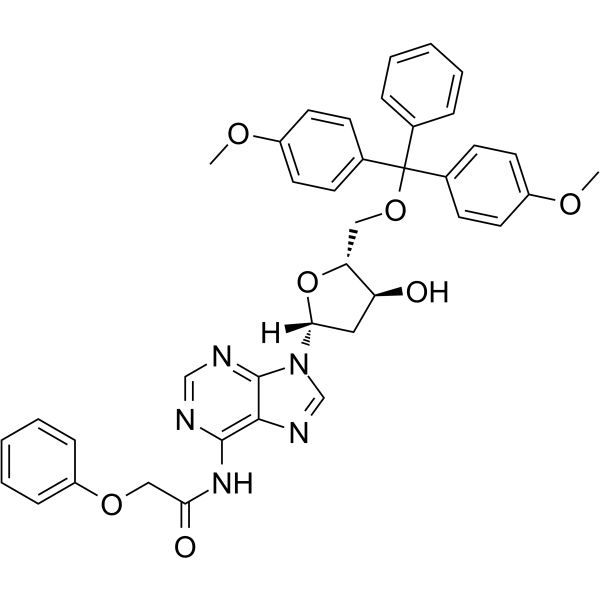
GC62812
5’-O-DMT-PAC-dA
5’-O-DMT-PAC-dA kann bei der Synthese von Oligoribonukleotiden verwendet werden.
-
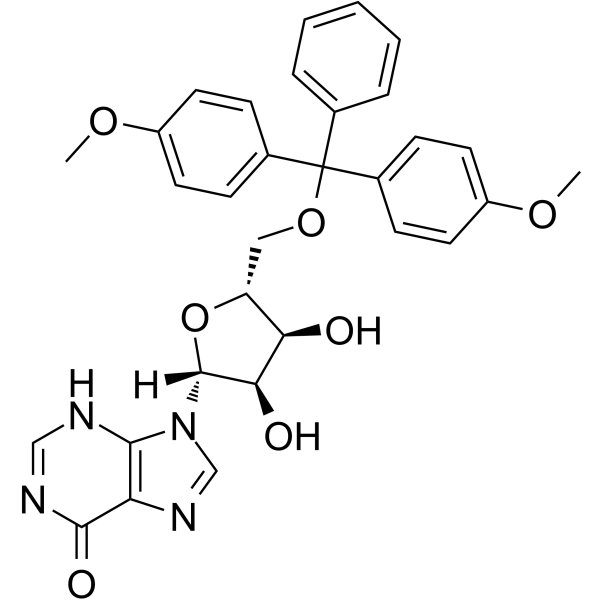
GC62813
5’-O-DMT-rI
5'-O-DMT-Ri kann bei der Synthese von Oligoribonukleotiden verwendet werden.
-
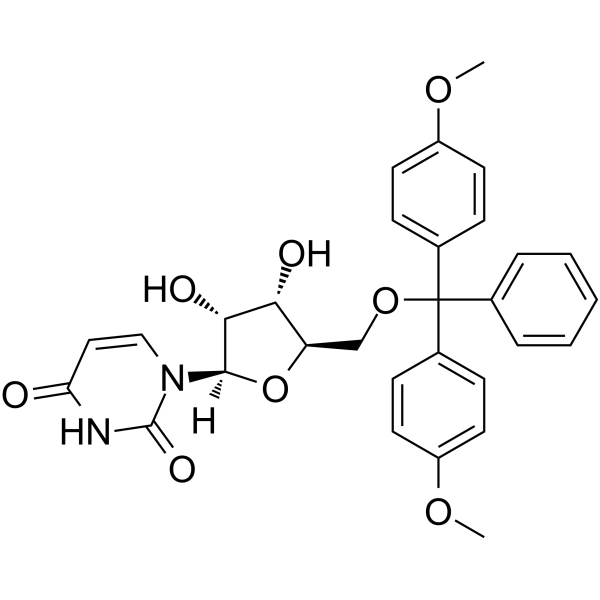
GC62577
5’-O-DMT-rU
5’-O-DMT-rU ist ein modifiziertes Nukleosid und kann zur Synthese von RNA verwendet werden.
-
GC62578
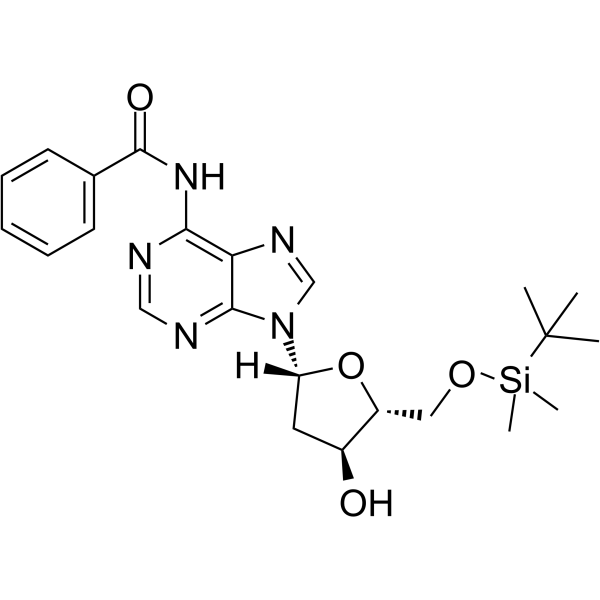
5’-O-TBDMS-Bz-dA
5’-O-TBDMS-Bz-dA ist ein Nukleosid mit schützender und modifizierender Wirkung.
-
GC62579
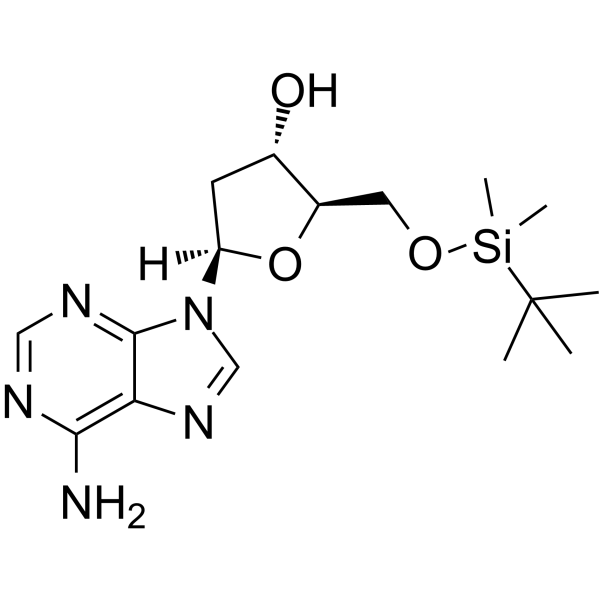
5’-O-TBDMS-dA
5’-O-TBDMS-dA ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
-
GC62580
5’-O-TBDMS-dG
5’-O-TBDMS-dG ist ein modifiziertes Nukleosid.

-
GC62581
5’-O-TBDMS-dT
5’-O-TBDMS-dT ist ein Nukleosid mit Schutz- und Modifikationswirkung.

-
GC40641
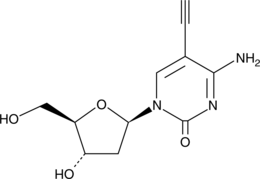
5'-Ethynyl-2'-deoxycytidine
5'-Ethynyl-2'-deoxycytidine (EdC) is a nucleoside analog that inhibits replication of the herpes simplex virus-1 (HSV-1) KOS strain (ID50 = 0.2 μg/mL).
-
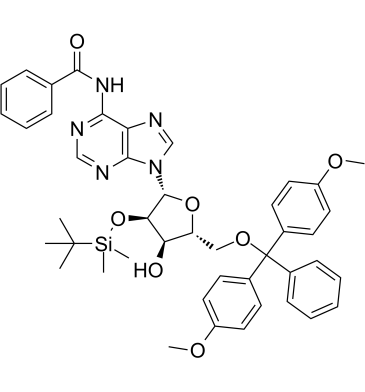
GC61432
5'-O-DMT-2'-O-TBDMS-N-Bz-Adenosine
5'-O-DMT-2'-O-TBDMS-N-Bz-Adenosinist ein Adenosinderivat und kann als Zwischenprodukt fÜr die Oligonukleotidsynthese verwendet werden.
-
GC61726
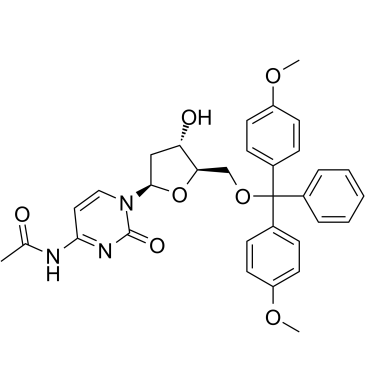
5'-O-DMT-N4-Ac-dC
5'-O-DMT-N4-Ac-dC (N4-Acetyl-2'-desoxy-5'-O-DMT-cytidin, Verbindung 7), ein Desoxynucleosid, kann verwendet werden, um Dodecylphosphoramidit zu synthetisieren, das das Rohprodukt ist Material fÜr die dod‐DNA‐Synthese (amphiphile DNA, die eine interne hydrophobe Region enthÄlt, die aus Dodecyl‐Phosphotriester‐Bindungen besteht).
-
GC48662
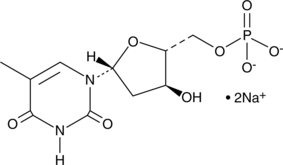
5'-Thymidylic Acid (sodium salt)
5'-ThymidylsÄure (Natriumsalz) ist ein kÖrpereigener Metabolit.
-
GC35150
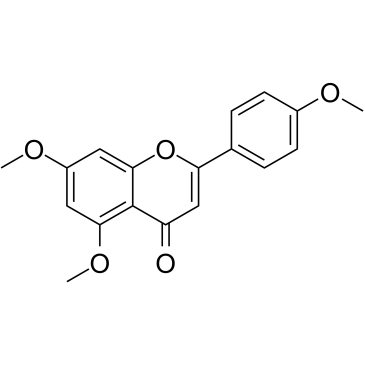
5,7,4'-Trimethoxyflavone
5,7,4'-Trimethoxyflavon wird aus Kaempferia parviflora (KP) isoliert, einer berühmten Heilpflanze aus Thailand. 5,7,4'-Trimethoxyflavon induziert Apoptose, wie durch Inkremente der Sub-G1-Phase, DNA-Fragmentierung, Annexin-V/PI-Färbung, das Bax/Bcl-xL-Verhältnis, proteolytische Aktivierung von Caspase-3 und Abbau von Poly belegt wird (ADP-Ribose)-Polymerase (PARP)-Protein.5,7,4'-Trimethoxyflavon ist bei der konzentrationsabhängigen Hemmung der Proliferation von menschlichen SNU-16-Magenkrebszellen signifikant wirksam.
-
GC68161
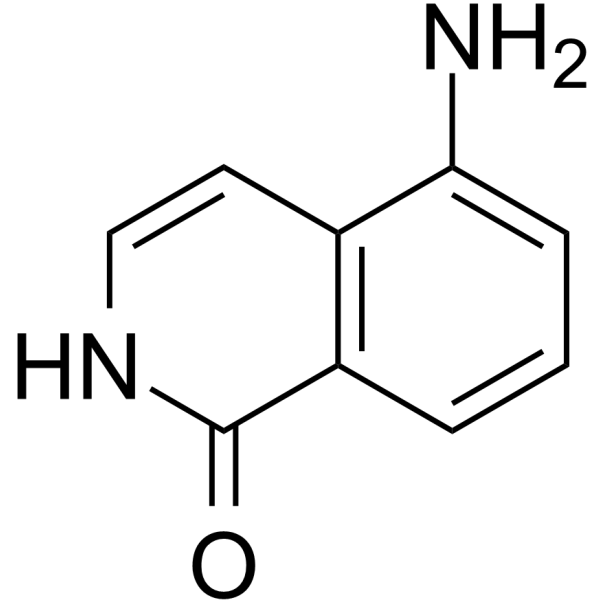
5-AIQ
-
GC64952
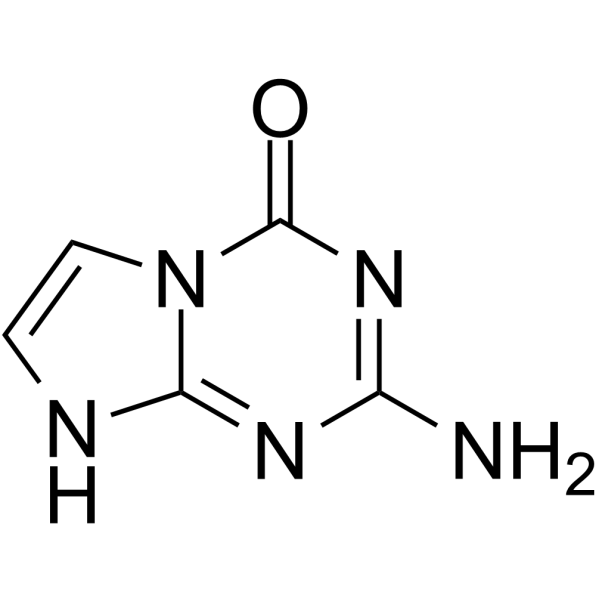
5-Aza-7-deazaguanine
5-Aza-7-Deazaguanin ist ein Substrat fÜr Wildtyp (WT) E. coli-Purin-Nukleosid-Phosphorylase und ihre Ser90Ala-Mutante bei der Synthese basenmodifizierter Nukleoside.
-
GC10946
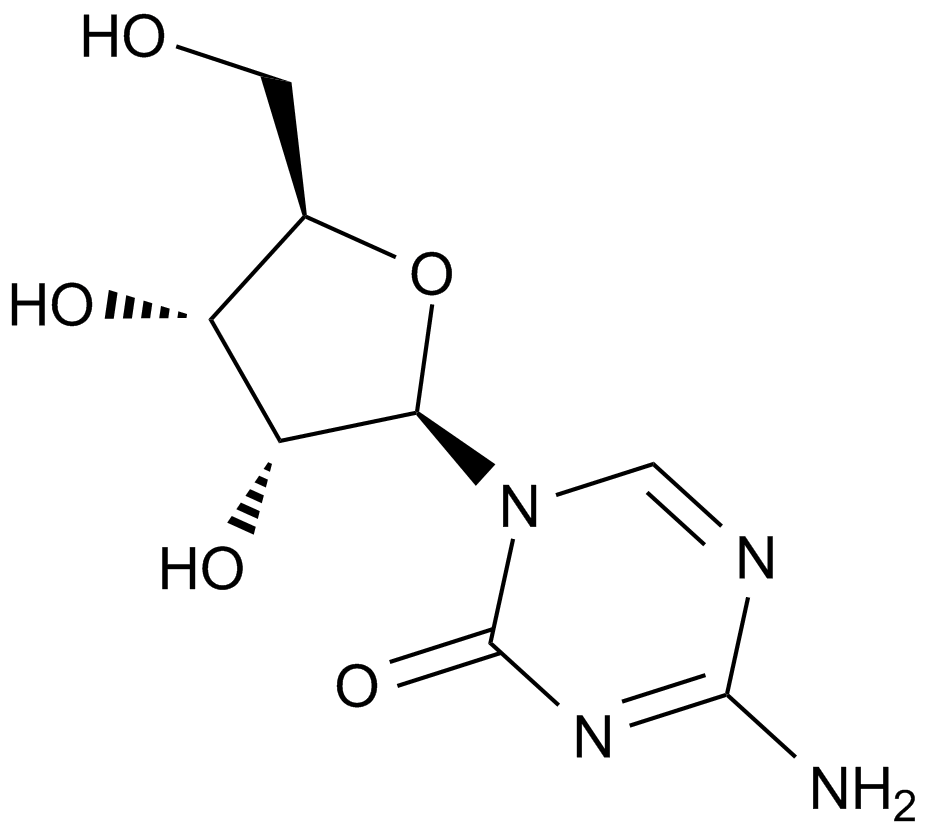
5-Azacytidine
A DNA methyltransferase inhibitor
-
GC11940
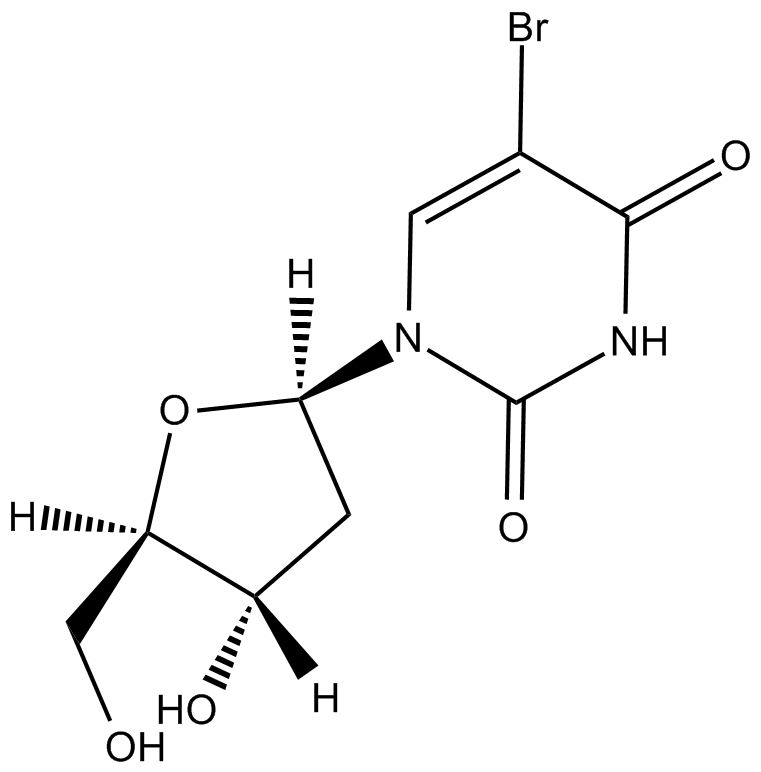
5-BrdU
Synthetisches Thymidin-Analogon
-
GC46681
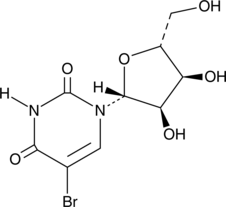
5-Bromouridine
A brominated uridine analog
-
GC49245
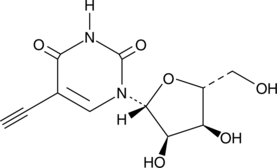
5-Ethynyluridine
5-Ethinyluridin (5-EU) ist ein potentes zellgÄngiges Nukleosid, das zur Markierung neu synthetisierter RNA verwendet werden kann.
-
GC42545
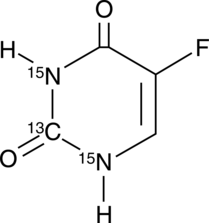
5-Fluorouracil-13C,15N2
5-Fluorouracil-13C,15N2 is intended for use as an internal standard for the quantification of 5-flurouracil by GC- or LC-MS.
-
GC46033
5-Heneicosylresorcinol
An alkylresorcinol

-
GC62389
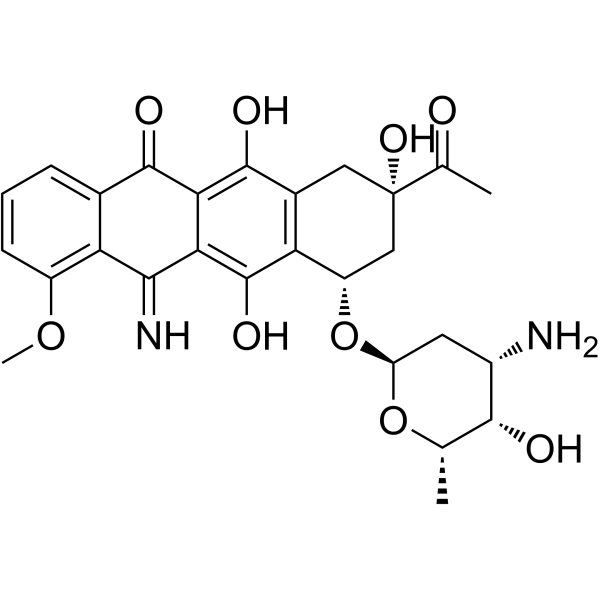
5-Iminodaunorubicin
5-Iminodaunorubicin ist ein Chinon-modifiziertes Anthracyclin, das seine AntitumoraktivitÄt behÄlt. 5-Iminodaunorubicin erzeugt in Krebszellen proteinverdeckte DNA-StrangbrÜche.
-
GC35162
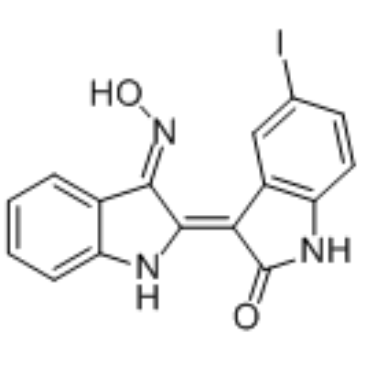
5-Iodo-indirubin-3'-monoxime
5-Iod-Indirubin-3'-Monoxim ist ein potenter GSK-3β-, CDK5/P25- und CDK1/Cyclin-B-Inhibitor, der mit ATP um die Bindung an die katalytische Stelle der Kinase konkurriert, mit IC50-Werten von 9, 20 und 25 nM, beziehungsweise.
-
GC66658
5-Me-dC(Ac) amidite
5-Me-dC(Ac)-Amidit wird zur Synthese von DNA oder RNA verwendet.
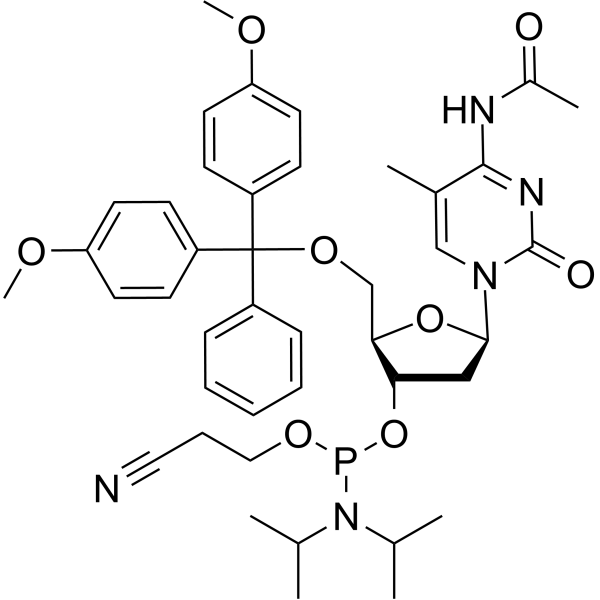
-
GC31187
5-Methoxyflavone
5-Methoxyflavon, das zur Familie der Flavonoide gehÖrt, ist ein DNA-Polymerase-Beta-Inhibitor und ein neuroprotektives Mittel gegen Beta-Amyloid-ToxizitÄt.
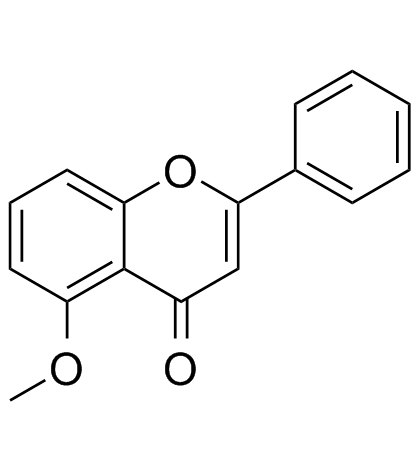
-
GC42562
5-Methyl-2'-deoxycytidine
5-Methyl-2'-desoxycytidin in einzelstrÄngiger DNA kann in cis wirken, um die De-novo-DNA-Methylierung zu signalisieren.
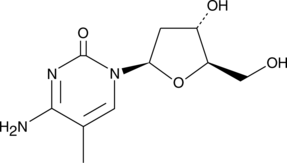
-
GC67671
5-Methylcytidine 5′-triphosphate trisodium
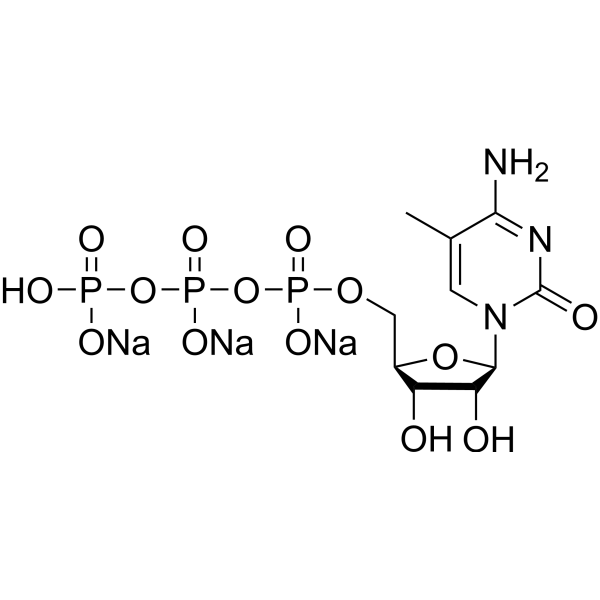
-
GC35166
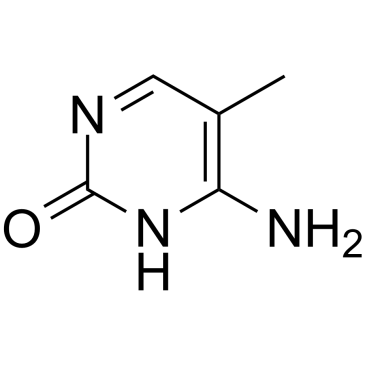
5-Methylcytosine
5-Methylcytosin ist eine gut charakterisierte DNA-Modifikation und kommt auch Überwiegend in zahlreichen nichtkodierenden RNAs sowohl in Prokaryoten als auch in Eukaryoten vor.
-
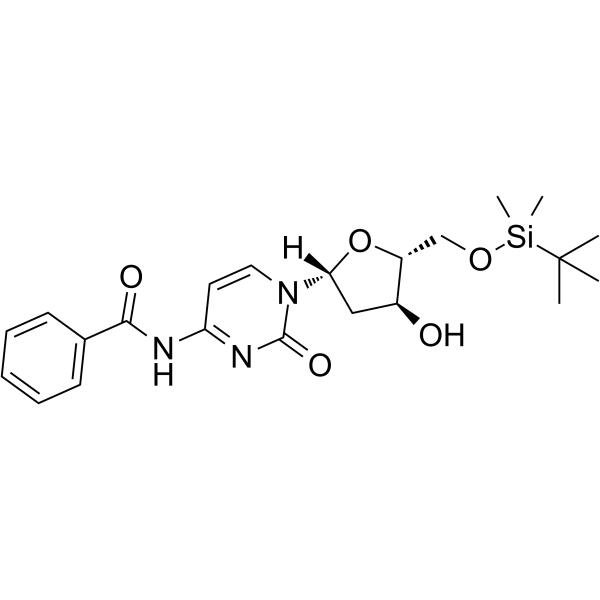
GC62547
5-O-TBDMS-N4-Benzoyl-2-deoxycytidine
5-O-TBDMS-N4-Benzoyl-2-desoxycytidin ist ein modifiziertes Nucleosid.
-
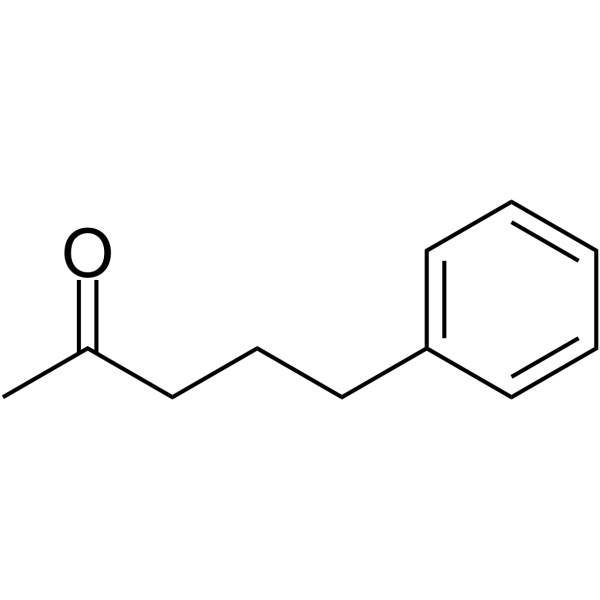
GC66055
5-Phenylpentan-2-one
5-Phenylpentan-2-on ist ein potenter Inhibitor der Histon-Deacetylase (HDACs). 5-Phenylpentan-2-on kann fÜr die Erforschung von StÖrungen des Harnstoffzyklus verwendet werden.
-
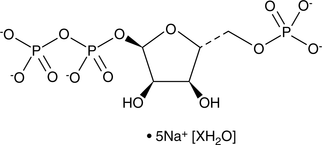
GC46713
5-Phospho-D-ribose 1-diphosphate (sodium salt hydrate)
An intermediate in several biochemical pathways
-
GC46079
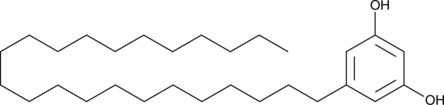
5-Tricosylresorcinol
5-Tricosylresorcin ist das erste Zystenlipid.
-
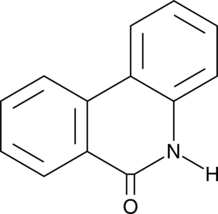
GC45772
6(5H)-Phenanthridinone
An inhibitor of PARP1 and 2
-
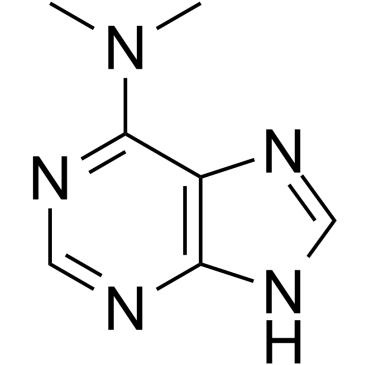
GC60532
6-(Dimethylamino)purine
6-(Dimethylamino)purin ist ein dualer Inhibitor von Proteinkinase und CDK.
-
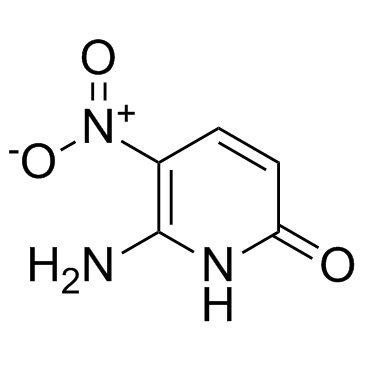
GC30548
6-Amino-5-nitropyridin-2-one
6-Amino-5-nitropyridin-2-on ist eine Pyridinbase und wird als Nukleobase von Hachimoji-DNA verwendet, in der es sich mit 5-Aza-7-Deazaguanin paart.
-
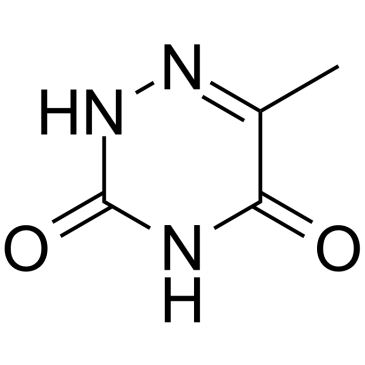
GC60031
6-Azathymine
6-Azathymin, ein 6-Stickstoff-Analogon von Thymin, ist ein potenter D-3-Aminoisobutyrat-Pyruvat-Aminotransferase-Inhibitor.
-
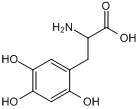
GC50249
6-Hydroxy-DL-DOPA
6-Hydroxy-DL-DOPA ist ein selektiver und wirksamer allosterischer Inhibitor der RAD52-ssDNA-BindungsdomÄne. 6-Hydroxy-DL-DOPA kann fÜr die Krebsforschung eingesetzt werden.
-
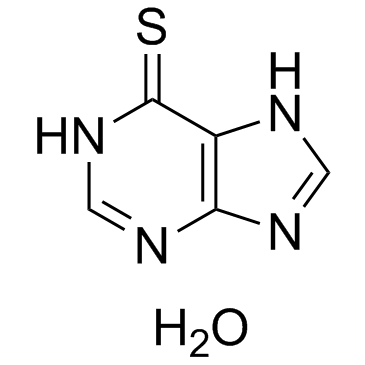
GC32975
6-Mercaptopurine hydrate
6-Mercaptopurin-Hydrat (Mercaptopurin-Hydrat; 6-MP-Hydrat) ist ein Purin-Analogon, das als Antagonist der endogenen Purine wirkt und als antileukÄmisches Mittel und immunsuppressives Medikament weit verbreitet ist.
-
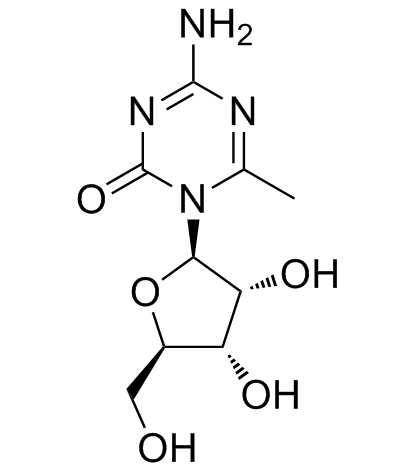
GC35180
6-Methyl-5-azacytidine
6-Methyl-5-azacytidin ist ein potenter DNMT-Inhibitor.
-
GC49235
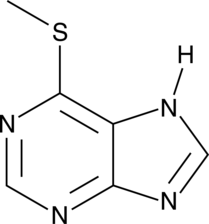
6-Methylmercaptopurine
A metabolite of 6-mercaptopurine
-
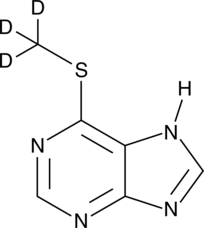
GC49488
6-Methylmercaptopurine-d3
An internal standard for the quantification of 6-MMP
-
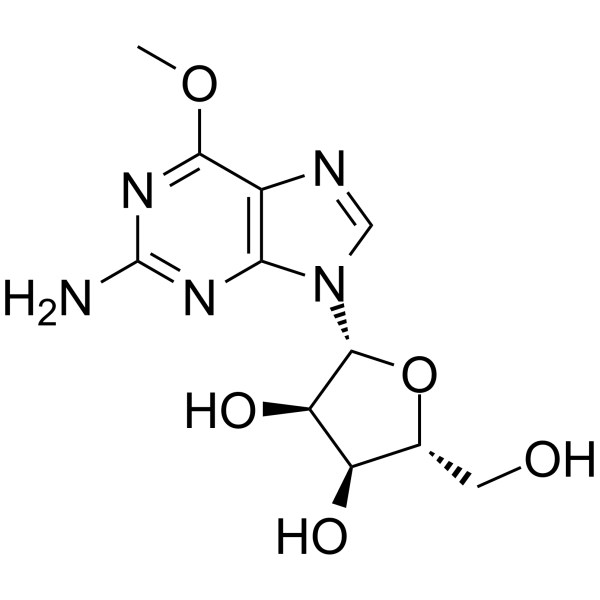
GC65082
6-O-Methyl Guanosine
6-O-Methylguanosin ist ein modifiziertes Nukleosid.
-
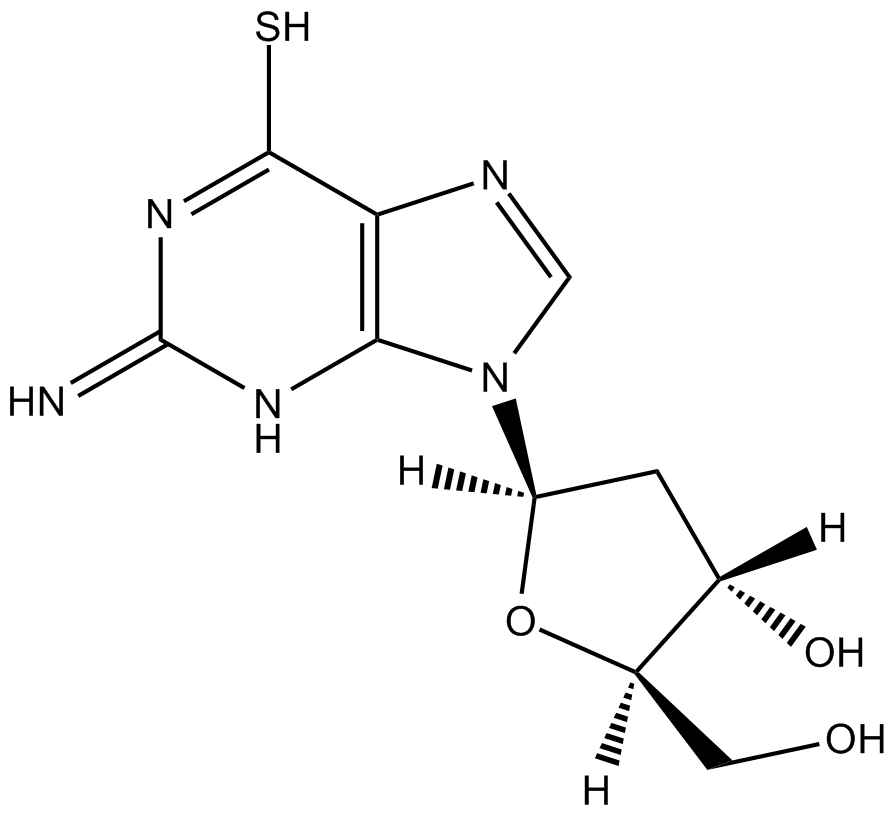
GC12193
6-Thio-dG
6-Thio-dG ist ein Nukleosid-Analogon, das durch Telomerase in de novo-synthetisierte Telomere eingebaut werden kann.
-
GC35171
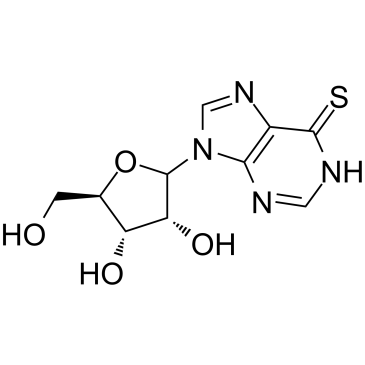
6-Thioinosine
6-Thioinosin (6TI) ist ein Purin-Antimetabolit, wirkt als Anti-Adipogenese-Mittel, reguliert die mRNA-Spiegel von PPAR γ und C/EBPα sowie von PPAR γ-Zielproteinen wie LPL, CD36, aP2 und LXRα herunter.
-
GC39625
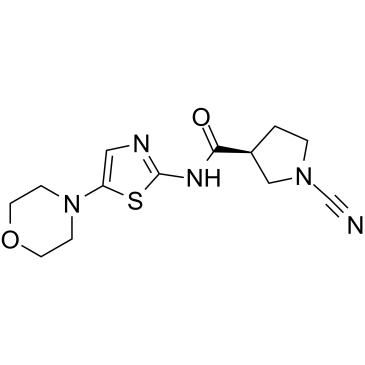
6RK73
6RK73 ist ein kovalenter irreversibler und spezifischer UCHL1-Inhibitor mit einem IC50 von 0,23 μM. 6RK73 zeigt fast keine Hemmung von UCHL3 (IC50 = 236 μM). 6RK73 hemmt spezifisch die UCHL1-AktivitÄt bei Brustkrebs.
-
GC46733
7,12-Dimethylbenz[a]anthracene
7,12-Dimethylbenz[a]anthracen hat als polyzyklischer aromatischer Kohlenwasserstoff (PAK) krebserzeugende Wirkung. 7,12-Dimethylbenz[a]anthracen wird verwendet, um die Tumorbildung in verschiedenen Nagetiermodellen zu induzieren.
![7,12-Dimethylbenz[a]anthracene Chemical Structure 7,12-Dimethylbenz[a]anthracene Chemical Structure](/media/struct/GC4/GC46733.png)
-
GC33577
7-Aminoactinomycin D (7-AAD)
7-Aminoactinomycin D (7-AAD) (7-AAD), ein fluoreszierender DNA-Farbstoff, ist ein potenter RNA-Polymerase-Inhibitor.
-
GC65408
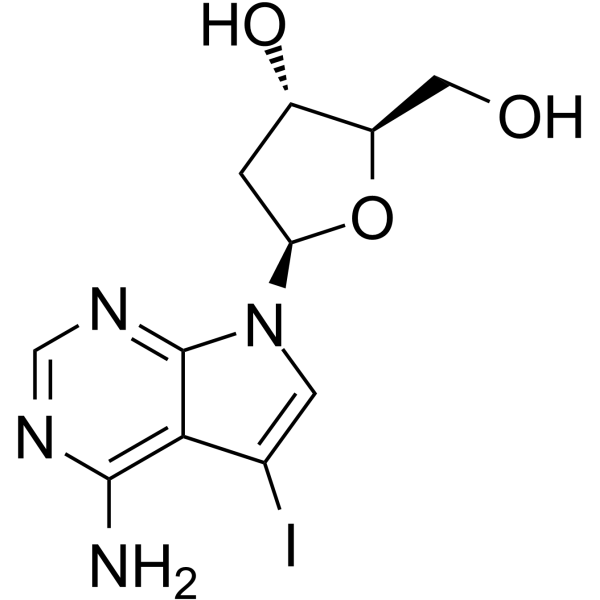
7-Deaza-2'-deoxy-7-iodoadenosine
7-Deaza-&2#39;-Desoxy-7-iodoadenosin ist ein modifiziertes Oligonukleotid, das 7-Deazaadenin enthält.
-
GC66060
7-Iodo-2',3'-dideoxy-7-deaza-guanosine
7-Iod-2',3'-Didesoxy-7-deaza-guanosin ist ein Didesoxynucleosid, das in DNA-Synthese- und Sequenzierungsreaktionen verwendet werden kann.
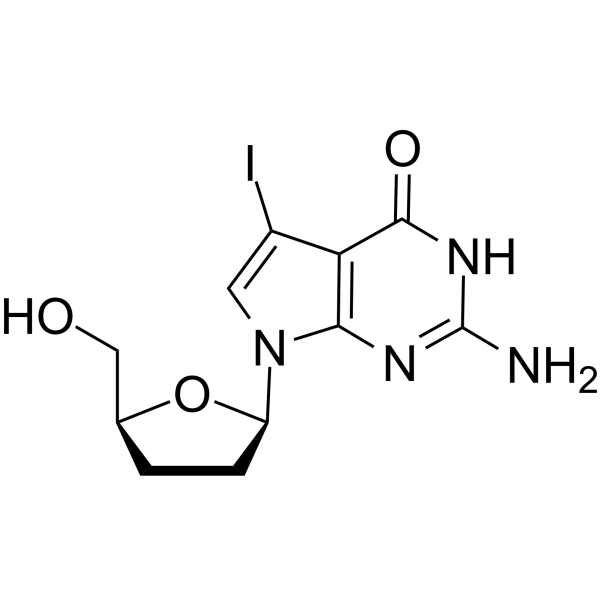
-
GC65675
7-Iodo-7-deaza-2'-deoxyguanosine
7-Iodo-7-deaza-2'-Desoxyguanosin (7-Deaza-7-Iodo-2'-Desoxyguanosin) ist ein Desoxyguanosin-Derivat, das in DNA-Synthese- und Sequenzierungsreaktionen verwendet werden kann.
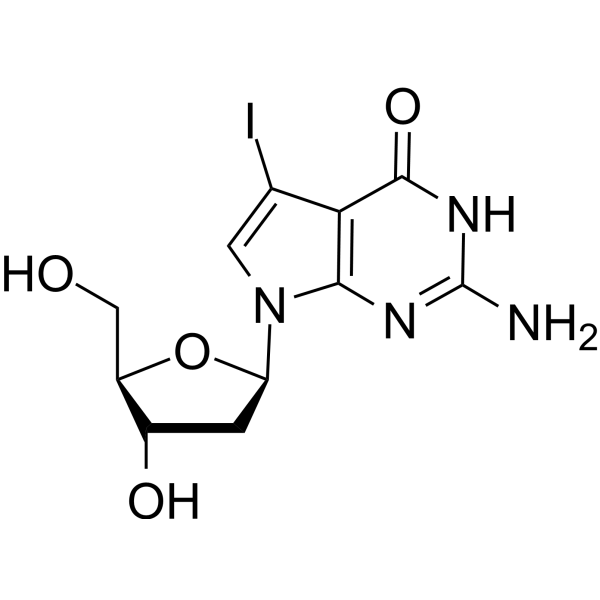
-
GC30531
7-Methylguanosine
7-Methylguanosin ist ein neuartiger cNIIIB-Nukleotidase-Inhibitor mit einem IC50-Wert von 87,8 ± 7,5 μM.
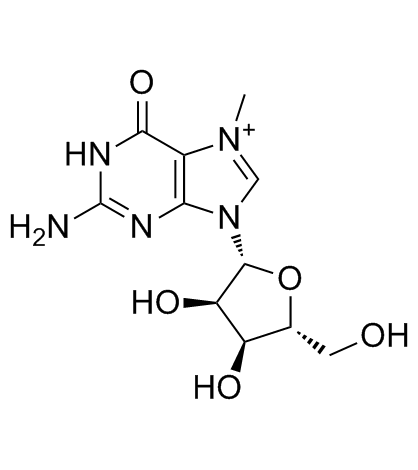
-
GC49561
7-Methylguanosine-d3
An internal standard for the quantification of 7-methylguanosine
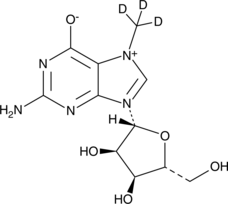
-
GC66058
7-TFA-ap-7-Deaza-dA
7-TFA-ap-7-Deaza-dA ist ein modifiziertes Nukleosid. 7-TFA-ap-7-Deaza-dA kann bei der Synthese von DesoxyribonukleinsÄure oder NukleinsÄure verwendet werden.
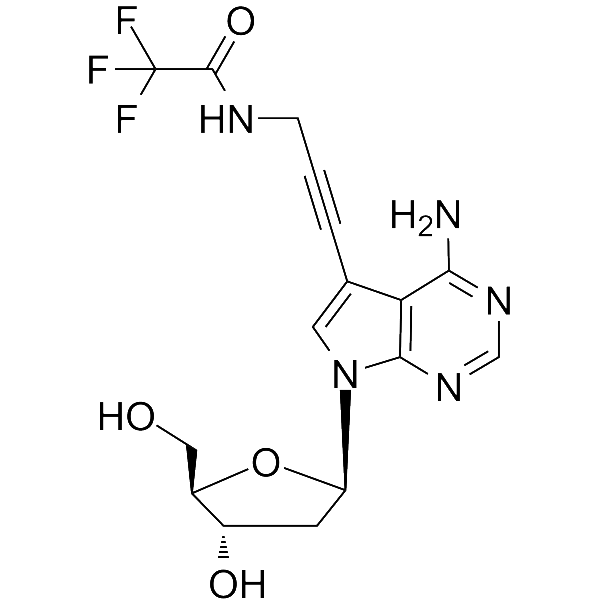
-
GC65525
7-TFA-ap-7-Deaza-dG
5'-O-TBDMS-dG ist ein modifiziertes Nukleosid.
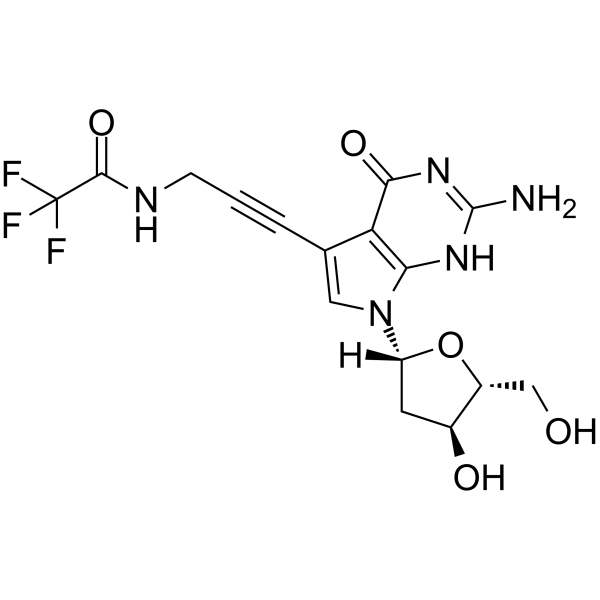
-
GC12777
8-Chloroadenosine
Nucleoside analog, inhibits RNA synthesis
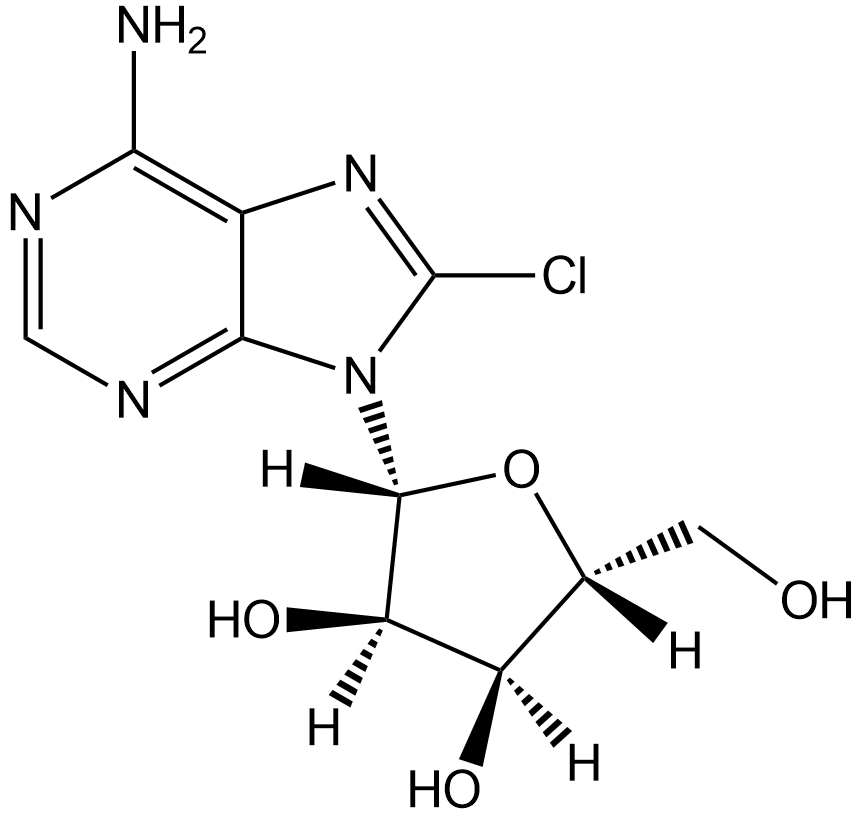
-
GC42627
8-Hydroxyguanine (hydrochloride)
8-Hydroxyguanine is produced by oxidative degradation of DNA by hydroxyl radical.
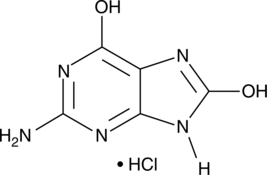
-
GC18426
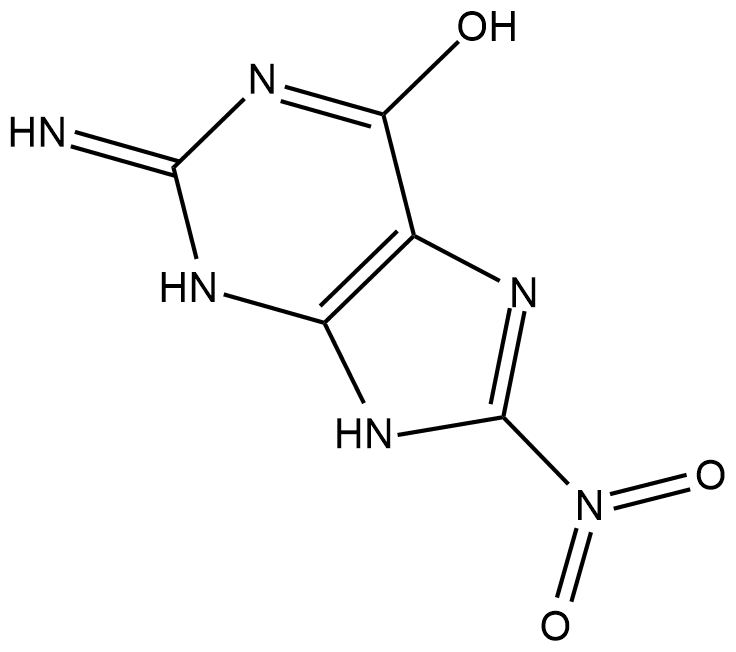
8-Nitroguanine
8-Nitroguanine is a nitrative guanine derivative formed by oxidative damage to the guanine base in DNA by reactive nitrogen species (RNS) during inflammation and in vitro by reaction of DNA with peroxynitrite and other RNS reagents.
-
GC41643
9(Z),11(E),13(E)-Octadecatrienoic Acid
9(Z),11(E),13(E)-Octadecatrienoic Acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.

-
GC11903
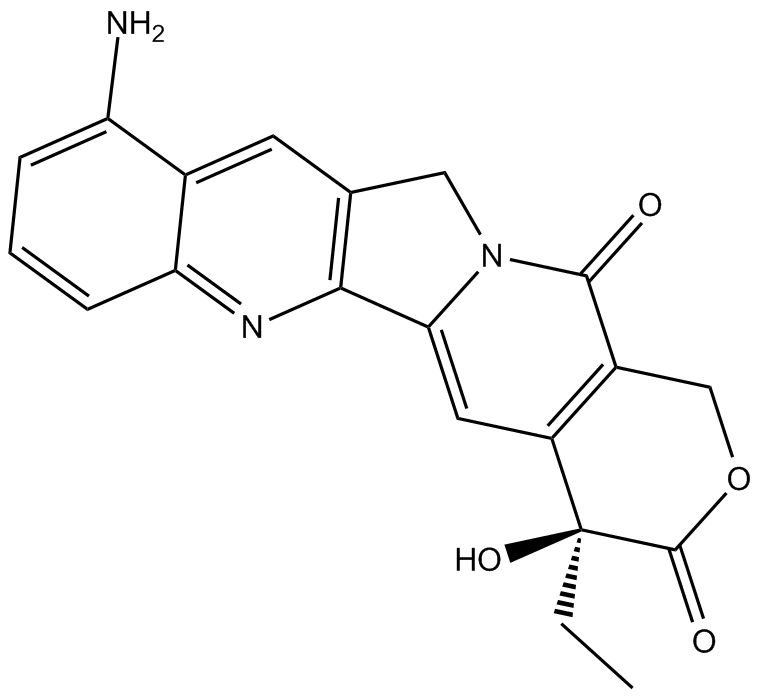
9-amino Camptothecin
9-Amino-Camptothecin (9-Amino-20(S)-Camptothecin) ist ein Topoisomerase-I-Inhibitor mit starker antikarzinogener Wirkung.
-
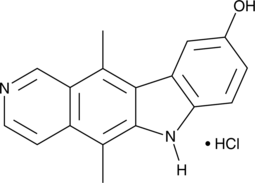
GC45367
9-Hydroxyellipticine (hydrochloride)
9-Hydroxyellipticin (Hydrochlorid) ist ein Inhibitor von Topo II und RyR.
-
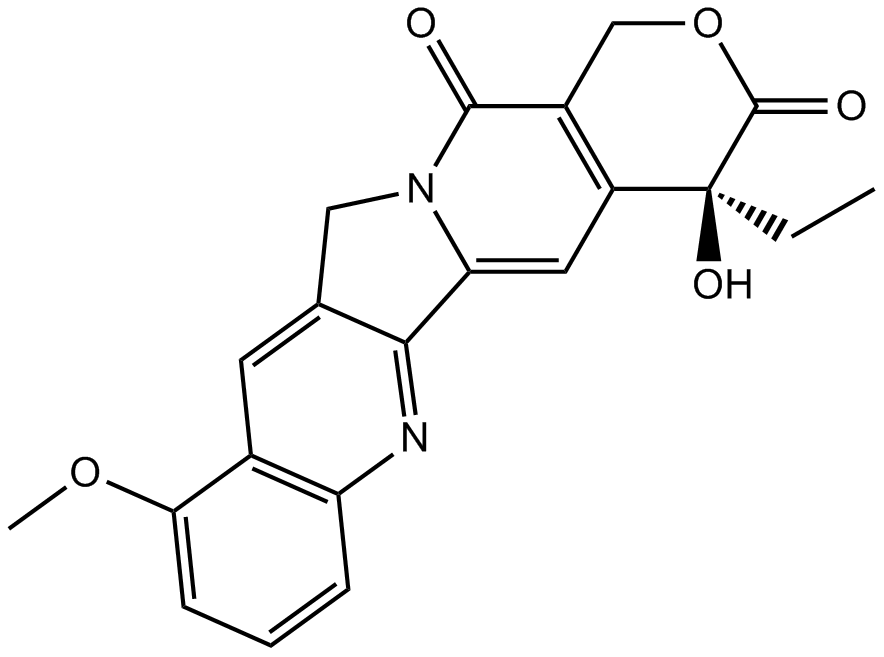
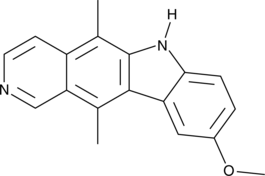
GN10035
9-Methoxycamptothecin
-
GC48840
9-Methoxyellipticine
An alkaloid with anticancer activity
-
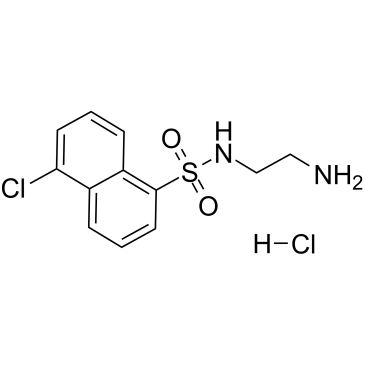
GC60037
A-3 hydrochloride
A-3-Hydrochlorid ist ein potenter, zellgÄngiger, reversibler, ATP-kompetitiver, nicht-selektiver Antagonist verschiedener Kinasen.
-
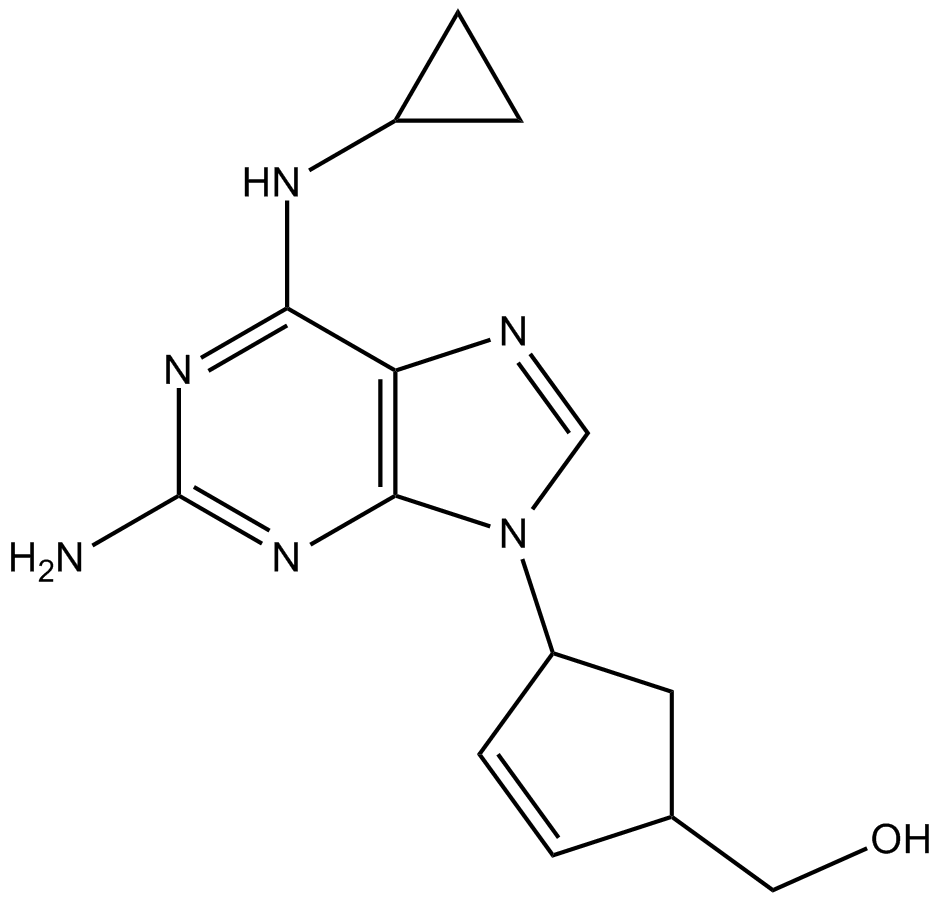
GC13805
Abacavir
-
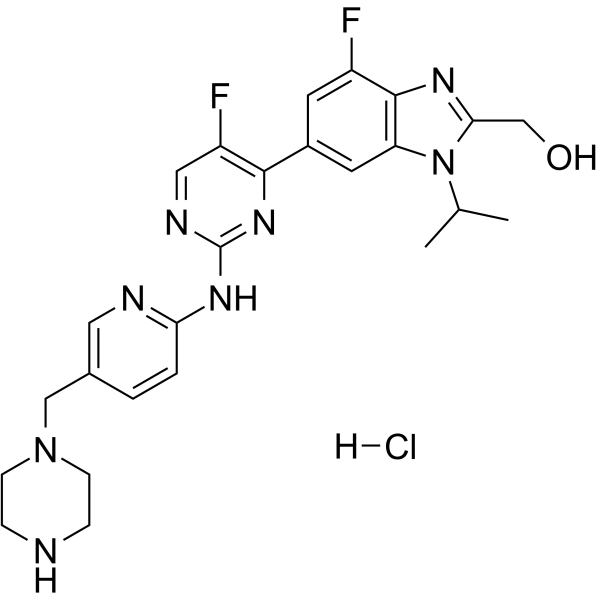
GC63728
Abemaciclib metabolite M18 hydrochloride
Abemaciclib-Metabolit M18 (LSN3106729)-Hydrochlorid, der Metabolit von Abemaciclib, ist ein CDK-Inhibitor mit AntitumoraktivitÄt. Der Abemaciclib-Metabolit M18-Hydrochlorid und ein CRBN-Ligand wurden verwendet, um PROTAC CDK4/6-Abbaumittel zu entwickeln.
-
GC38749
Abemaciclib metabolite M20
Abemaciclib-Metabolit M20 (LSN3106726), der aktive Metabolit von Abemaciclib, ist ein selektiver CDK4/6-Inhibitor zur Behandlung von Krebs.
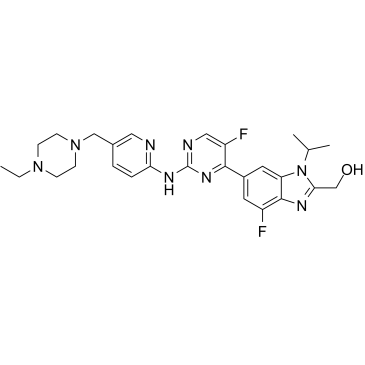
-
GC35218
Abemaciclib Metabolites M2
Abemaciclib Metabolites M2 (LSN2839567) ist ein Metabolit von Abemaciclib und wirkt als potenter CDK4- und CDK6-Inhibitor mit IC50-Werten von 1,2 bzw. 1,3 nM. Anti-Krebs-AktivitÄt.
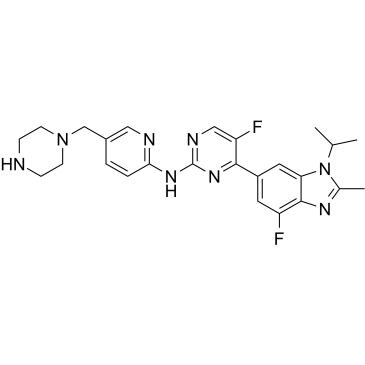
-
GC64280
Abemaciclib-d8
Abemaciclib-d8 (LY2835219-d8) ist das Deuterium mit der Bezeichnung Abemaciclib. Abemaciclib (LY2835219) ist ein selektiver CDK4/6-Inhibitor mit IC50-Werten von 2 nM und 10 nM fÜr CDK4 bzw. CDK6.
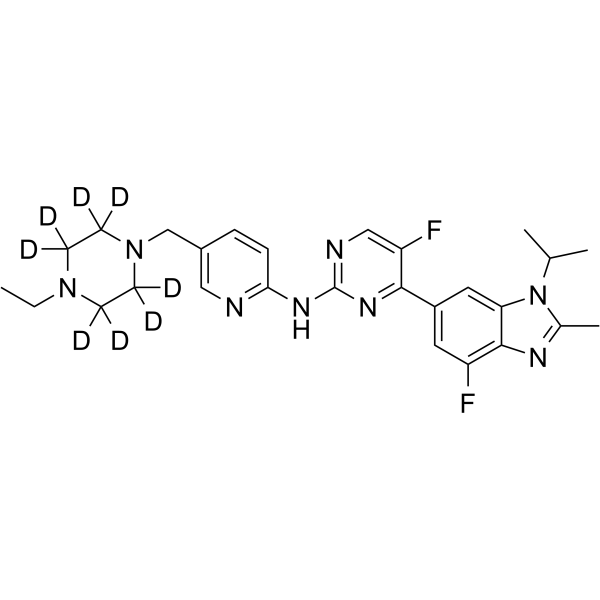
-
GA20605
Ac-Lys-AMC
Ac-Lys-AMC (Hexanamid), auch MAL genannt, ist ein fluoreszierendes Substrat fÜr Histon-Deacetylase-HDACs.

-
GC61763
Ac-rC Phosphoramidite
Ac-rC-Phosphoramidit wird fÜr die Oligoribonukleotid-Phosphorodithioat-Modifikation (PS2-RNA) verwendet.

-
GC34090
Acelarin (NUC-1031)
Acelarin (NUC-1031) (NUC-1031) ist eine ProTide-Transformation und -VerstÄrkung des weit verbreiteten Nukleosid-Analogons Gemcitabin.
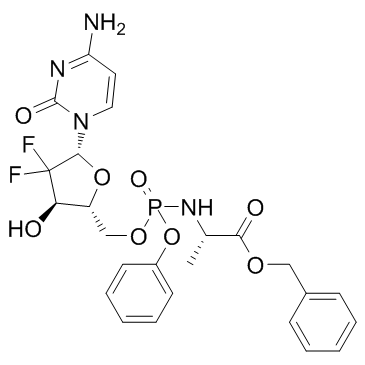
-
GC35238
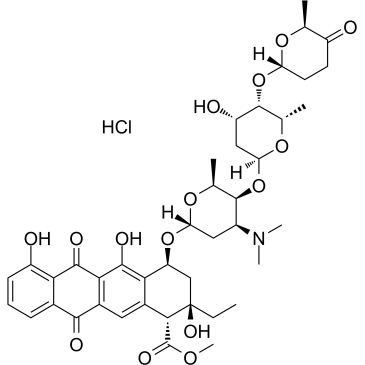
Aclacinomycin A hydrochloride
Alacinomycin A-Hydrochlorid (Aclarubicin-Hydrochlorid), ein fluoreszierendes MolekÜl und der erste beschriebene nicht-peptidische Inhibitor, der eine diskrete SpezifitÄt fÜr die CTRL-AktivitÄt (Chymotrypsin-Ähnlich) des 20S-Proteasoms zeigt. Alacinomycin A-Hydrochlorid ist auch ein dualer Inhibitor der Topoisomerase I und II. Ein wirksames Anthracyclin-Chemotherapeutikum fÜr hÄmatologische Krebsarten und solide Tumore.
-
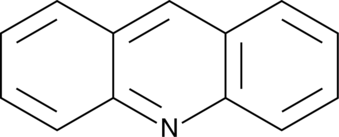
GC49804
Acridine
An azaarene
-
GC16866
Actinomycin D
Ein DNA-interagierender Transkriptionsblocker mit antikarzinogener Aktivität.

-
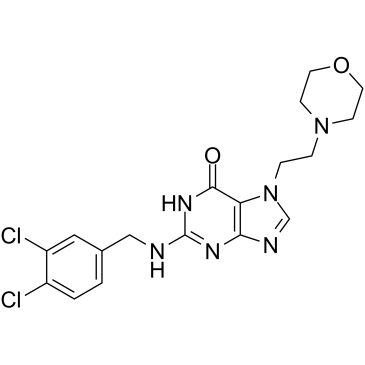
GC35247
ACX-362E
ACX-362E (ACX-362E) ist ein erster oral aktiver DNA-Polymerase IIIC (pol IIIC)-Inhibitor seiner Klasse mit einem Ki von 0,325 μM fÜr die DNA pol IIIC von C.
-
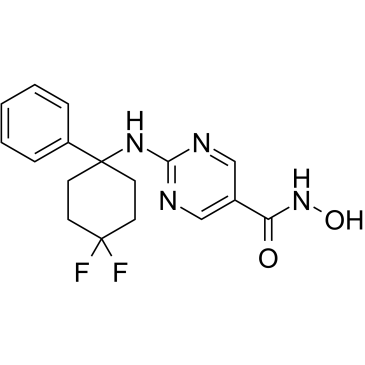
GC34458
ACY-1083
ACY-1083 ist ein selektiver und gehirngängiger HDAC6-Inhibitor mit einer IC50 von 3 nM und ist 260-fach selektiver für HDAC6 als alle anderen Klassen von HDAC-Isoformen. ACY-1083 kehrt die durch Chemotherapie verursachte periphere Neuropathie effektiv um.
-
GC10417
ACY-241
ACY-241 (ACY241) ist ein potenter, oral aktiver und hochselektiver HDAC6-Inhibitor der zweiten Generation mit einem IC50-Wert von 2,6 nM (IC50-Werte von 35 nM, 45 nM, 46 nM und 137 nM fÜr HDAC1, HDAC2, HDAC3 bzw. HDAC8). ). ACY-241 hat Antikrebswirkungen.
-
GC19020
ACY-738
ACY-738 ist ein potenter, selektiver und oral bioverfÜgbarer HDAC6-Inhibitor mit einem IC50 von 1,7 nM; ACY-738 hemmt auch HDAC1, HDAC2 und HDAC3 mit IC50-Werten von 94, 128 und 218 nM.



