DNA Damage/DNA Repair
- MTH1(4)
- PARP(84)
- ATM/ATR(30)
- DNA Alkylating(20)
- DNA Ligases(3)
- DNA Methyltransferase(25)
- DNA-PK(30)
- HDAC(136)
- Nucleoside Antimetabolite/Analogue(140)
- Telomerase(17)
- Topoisomerase(151)
- tankyrase(5)
- Antifolate(38)
- CDK(250)
- Checkpoint Kinase (Chk)(31)
- CRISPR/Cas9(9)
- Deubiquitinase(72)
- DNA Alkylator/Crosslinker(71)
- DNA/RNA Synthesis(400)
- Eukaryotic Initiation Factor (eIF)(23)
- IRE1(23)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(61)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Produkte für DNA Damage/DNA Repair
- Bestell-Nr. Artikelname Informationen
-
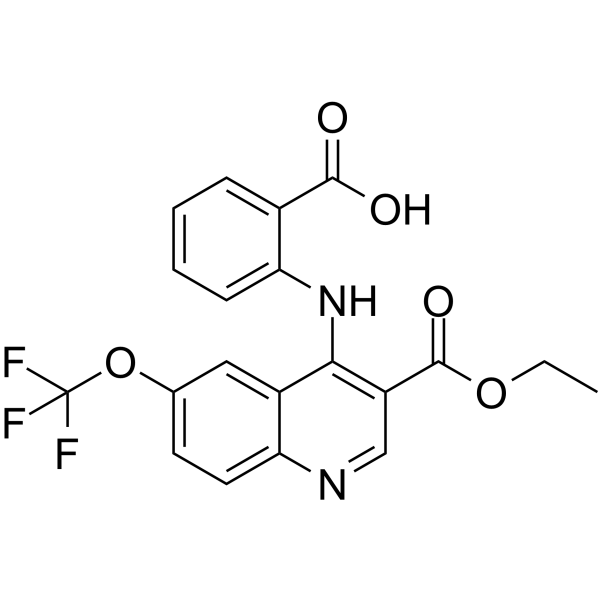
GC30782
ACY-775
ACY-775 ist ein potenter und selektiver Inhibitor der Histon-Deacetylase 6 (HDAC6) mit einem IC50 von 7,5nM.
-
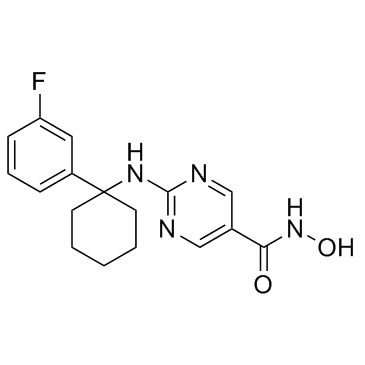
GC30526
ACY-957
ACY-957 ist ein oral aktiver und selektiver Inhibitor von HDAC1 und HDAC2 mit IC50-Werten von 7 nM, 18 nM bzw. 1300 nM gegen HDAC1/2/3 und zeigt keine Hemmung von HDAC4/5/6/7/8 /9.
-
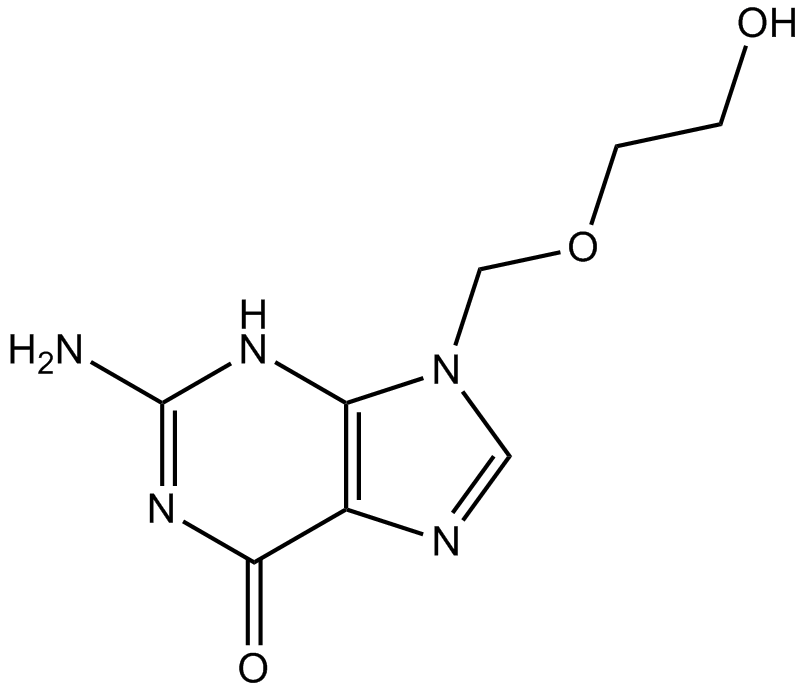
GC17186
Acyclovir
Aciclovir (Aciclovir) ist ein starkes, oral aktives antivirales Mittel.
-
GC13432
Adenine
Adenin (6-Aminopurin), ein Purin, ist eine der vier Nukleobasen in der NukleinsÄure der DNA. Adenin fungiert als chemischer Bestandteil von DNA und RNA. Adenin spielt auch eine wichtige Rolle in der Biochemie, die an der Zellatmung, der Form von ATP und den Cofaktoren (NAD und FAD) und der Proteinsynthese beteiligt ist.

-
GC11825
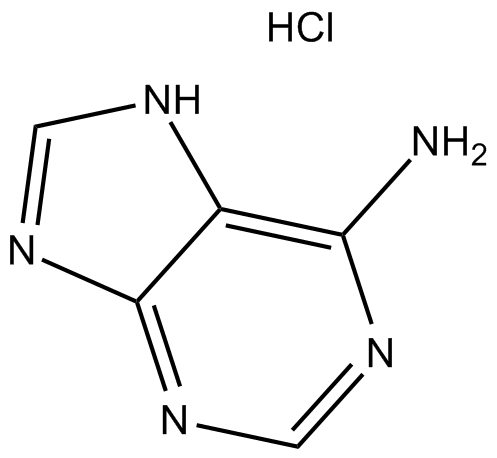
Adenine HCl
Adenin-HCl (6-Aminopurin-Hydrochlorid), ein Purin, ist eine der vier Nukleobasen in der NukleinsÄure der DNA. Adenin-HCl wirkt als chemischer Bestandteil von DNA und RNA. Adenin-HCl spielt auch eine wichtige Rolle in der Biochemie, die an der Zellatmung, der Form von ATP und den Cofaktoren (NAD und FAD) und der Proteinsynthese beteiligt ist.
-
GC17278
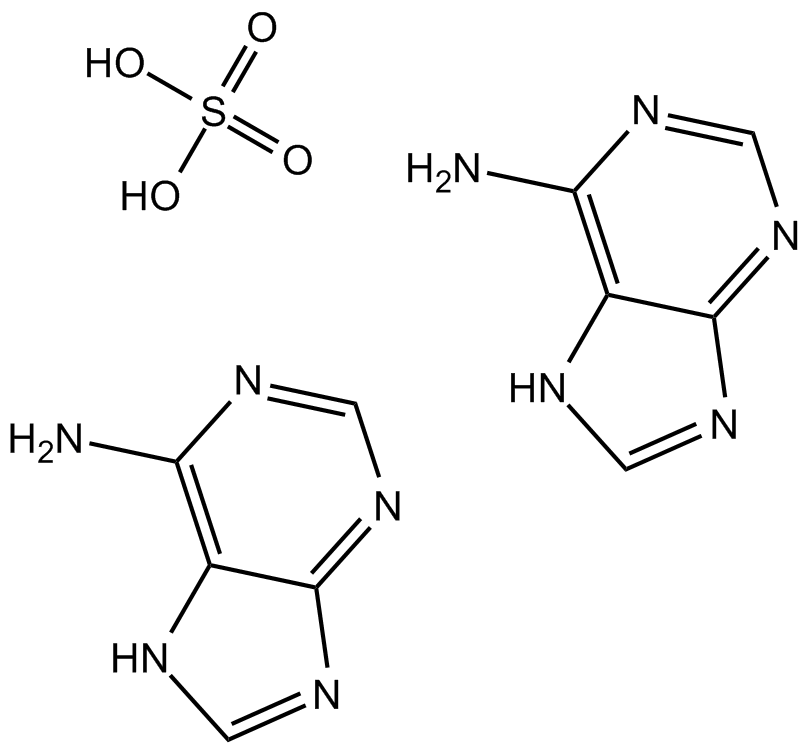
Adenine sulfate
Adeninsulfat (6-Aminopurin-Hemisulfat), ein Purin, ist eine der vier Nukleobasen in der NukleinsÄure der DNA. Adeninsulfat wirkt als chemischer Bestandteil von DNA und RNA. Adeninsulfat spielt auch eine wichtige Rolle in der Biochemie, die an der Zellatmung, der Form von ATP und den Cofaktoren (NAD und FAD) und der Proteinsynthese beteiligt ist.
-
GC67946
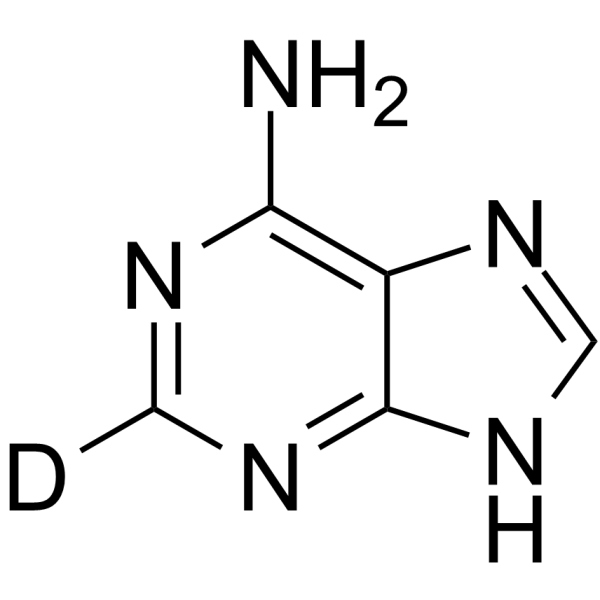
Adenine-d1
-
GC14106
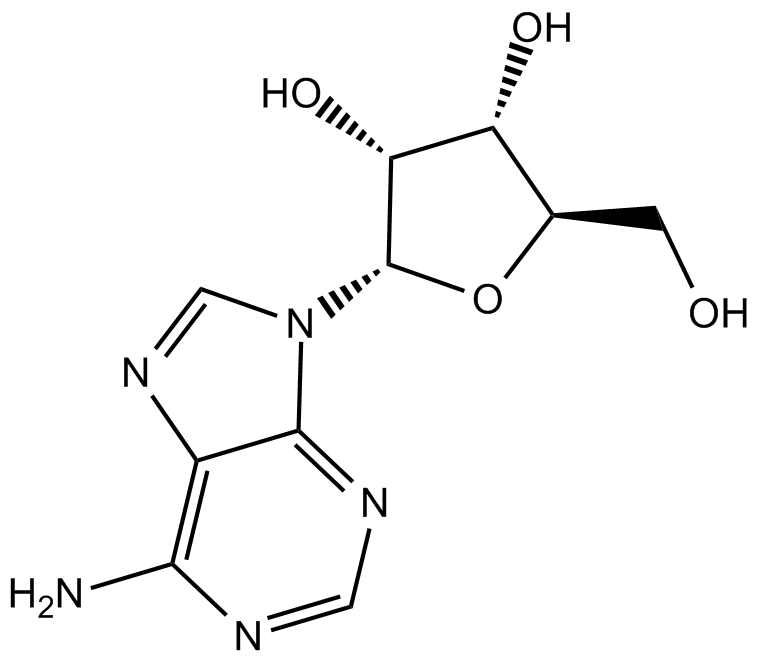
Adenosine
Nukleosid
-
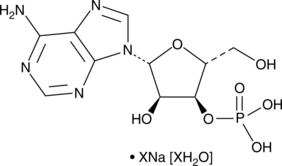
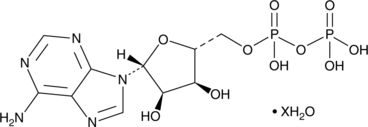
GC49004
Adenosine 3’-monophosphate (sodium salt hydrate)
A nucleotide
-
GC42732
Adenosine 3',5'-diphosphate (sodium salt)
Adenosin 3',5'-Diphosphat (Natriumsalz) ist ein Hydroxysteroid-Sulfotransferase-Inhibitor.
-
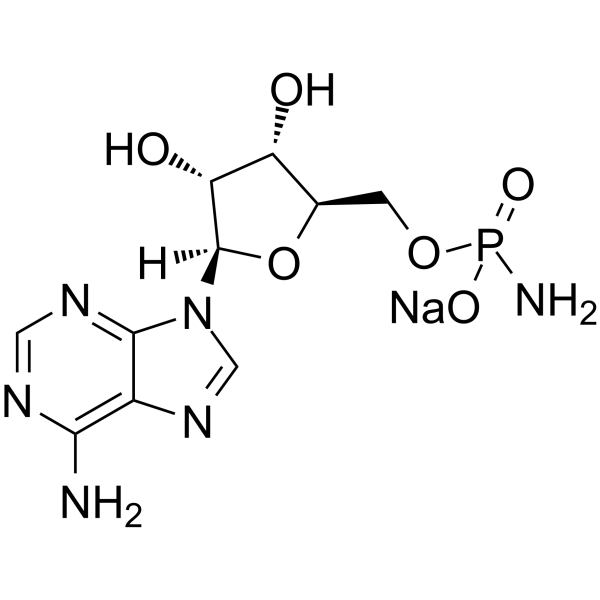
GC65462
Adenosine 5′-monophosphoramidate sodium
Adenosin 5′-monophosphoramidate Natrium ist ein Adenosin-Derivat und kann als Zwischenprodukt für die Nukleotidsynthese verwendet werden.
-
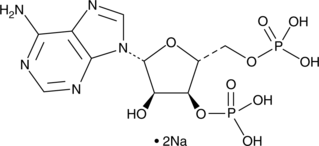
GC49285
Adenosine 5’-methylenediphosphate (hydrate)
An inhibitor of ecto-5’-nucleotidase
-
GC42733
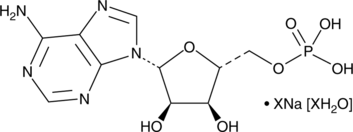
Adenosine 5'-monophosphate (sodium salt hydrate)
Adenosine 5'-monophosphate (AMP) is a central nucleotide with functions in metabolism and cell signaling.
-
GC42734
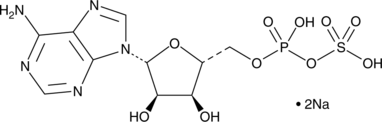
Adenosine 5'-phosphosulfate (sodium salt)
Adenosine 5'-phosphosulfate is an ATP and sulfate competitive inhibitor of ATP sulfurylase in humans, S.
-
GC49231
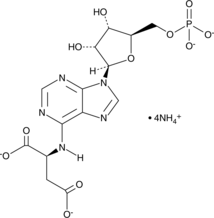
Adenylosuccinic Acid (ammonium salt)
A purine nucleotide and an intermediate in the purine nucleotide cycle
-
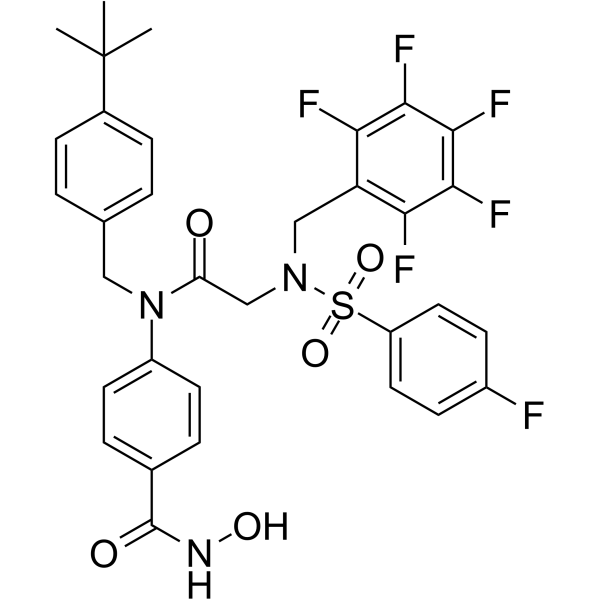
GC65330
AES-135
AES-135, ein auf HydroxamsÄure basierender Pan-HDAC-Inhibitor, verlÄngert das Überleben in einem orthotopen Mausmodell fÜr BauchspeicheldrÜsenkrebs. AES-135 hemmt HDAC3, HDAC6, HDAC8 und HDAC11 mit IC50-Werten im Bereich von 190-1100 nM.
-
GC46810
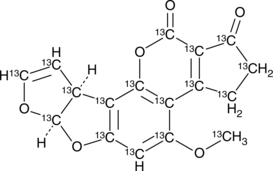
Aflatoxin B1-13C17
An internal standard for the quantification of aflatoxin B1
-
GC62470
AG-636
AG-636 ist ein potenter, reversibler, selektiver und oral aktiver Dihydroorotat-Dehydrogenase (DHODH)-Hemmer mit einem IC50 von 17 nM. AG-636 hat starke Antikrebswirkungen.
-
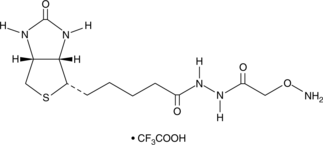
GC42765
Aldehyde Reactive Probe (trifluoroacetate salt)
Die DNA wird kontinuierlich durch endogene und Umweltfaktoren beschädigt, was zur Bildung von abasischen (apurinischen/apyrimidinischen, AP) Stellen führt, die die DNA-Synthese stören.
-
GC14858
Aldoxorubicin
Aldoxorubicin (INNO-206) ist ein albuminbindendes Prodrug von Doxorubicin (DNA-Topoisomerase-II-Inhibitor), das unter sauren Bedingungen aus Albumin freigesetzt wird. Aldoxorubicin (INNO-206) hat starke AntitumoraktivitÄten in verschiedenen Krebszelllinien und in murinen Tumormodellen.
-
GC15841
Alsterpaullone
Alsterpaullon (9-Nitropaullon) ist ein potenter CDK-Inhibitor mit IC50-Werten von 35 nM, 15 nM, 200 nM und 40 nM fÜr CDK1/Cyclin B, CDK2/Cyclin A, CDK2/Cyclin E bzw. CDK5/p35. Alsterpaullon konkurriert auch mit ATP um die Bindung an GSK-3alpha/GSK-3beta mit IC50-Werten von jeweils 4 nM. Alsterpaullon hat AntitumoraktivitÄt und besitzt Potenzial fÜr die Untersuchung von neurodegenerativen und proliferativen Erkrankungen. Alsterpaullon induziert Apoptose in LeukÄmie-Zelllinien.
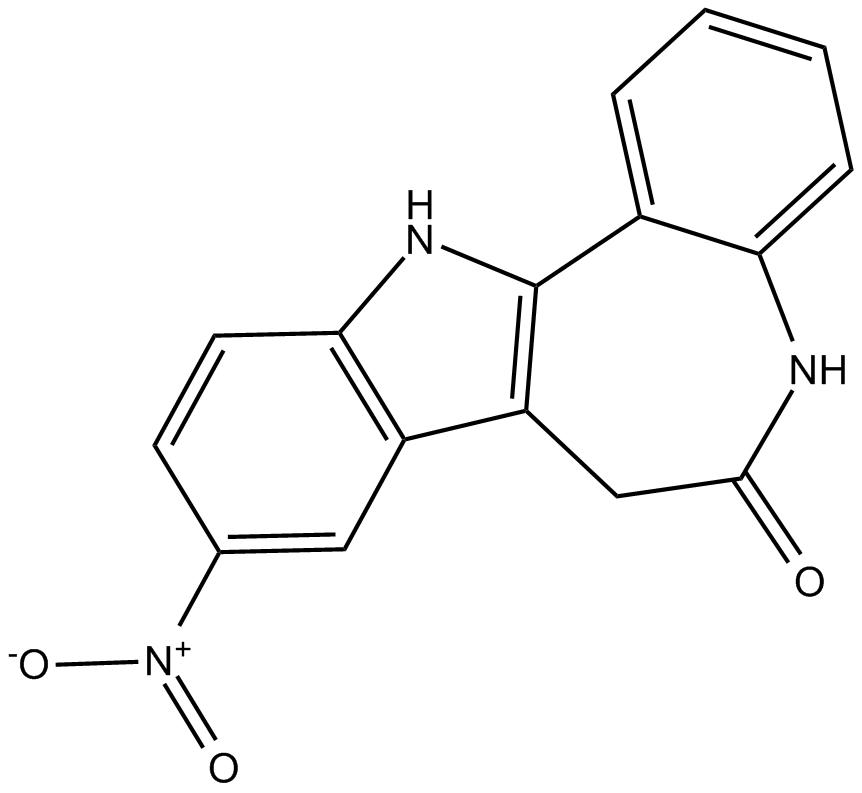
-
GC67984
Alteminostat
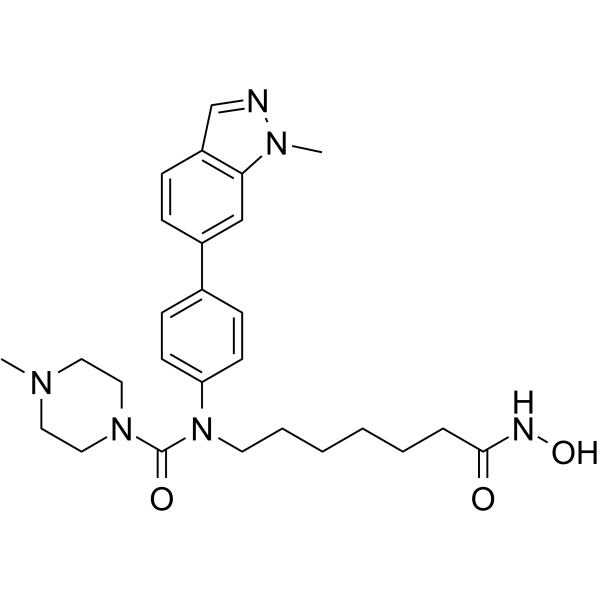
-
GC10779
Alternariol
Alternariol ist ein Mykotoxin, das von Alternaria-Arten produziert wird.
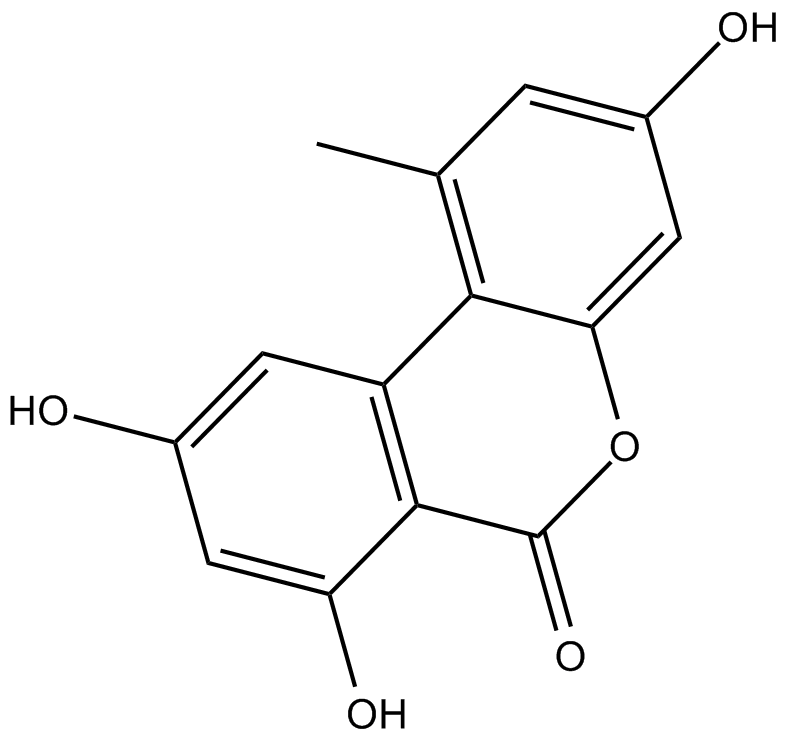
-
GC18437
Alternariol monomethyl ether
Alternariolmonomethylether, isoliert aus den Wurzeln von Anthocleista djalonensis (Loganiaceae), ist ein wichtiger taxonomischer Marker der Pflanzenart.
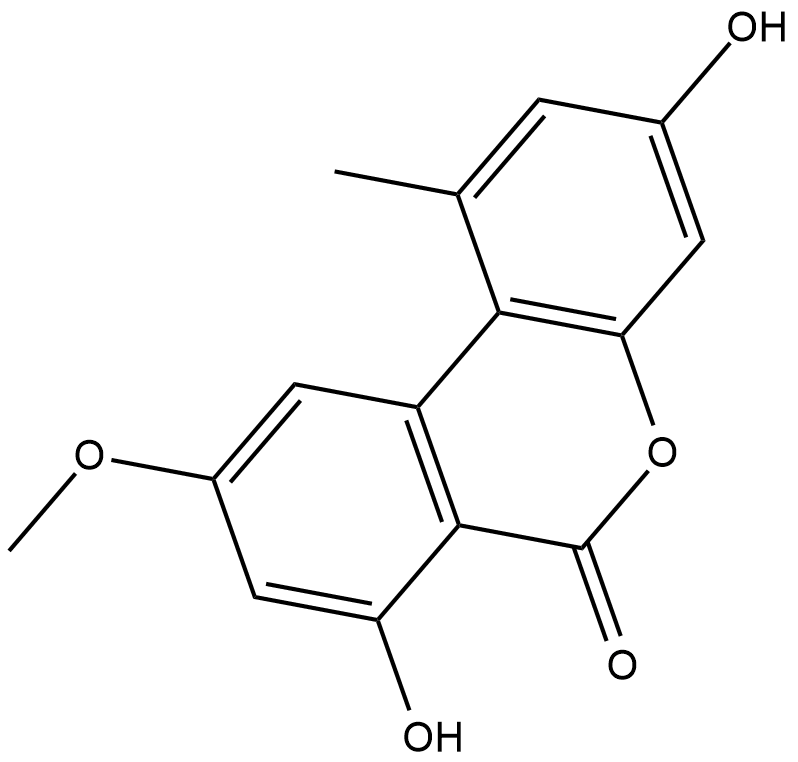
-
GC49039
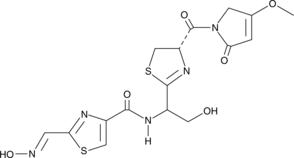
Althiomycin
A thiazole antibiotic
-
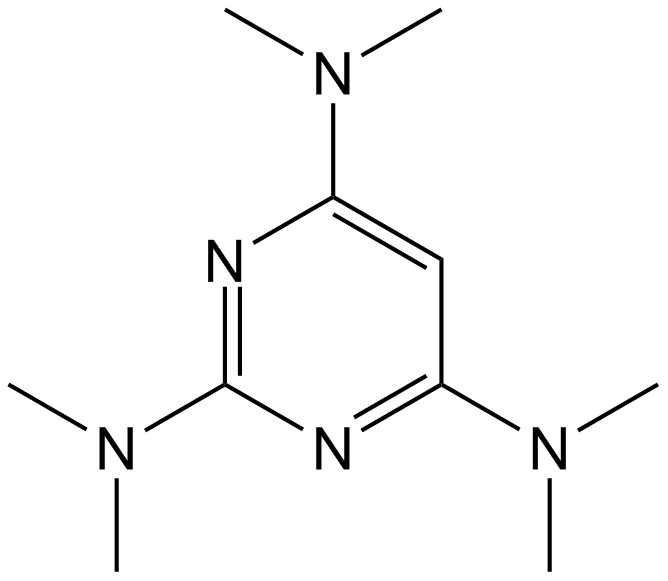
GC14564
Altretamine
Altretamin ist ein alkylierendes antineoplastisches Mittel.
-
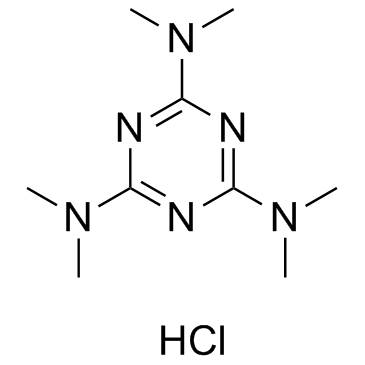
GC35310
Altretamine hydrochloride
Altretaminhydrochlorid ist ein alkylierendes antineoplastisches Mittel.
-
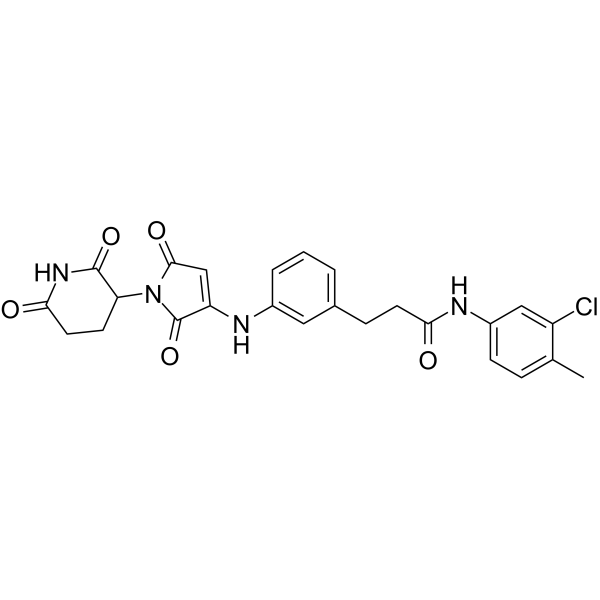
GC64638
ALV1
ALV1 ist ein potenter Abbauer von Ikaros und Helios.
-
GC64818
AMA-37
AMA-37, ein Arylmorpholin-Analogon, ist ein ATP-kompetitiver DNA-PK-Inhibitor mit IC50-Werten von 0,27 μM (DNA-PK), 32 μM (p110α), 3,7 μM (p110β) bzw. 22 μM (p110γ).
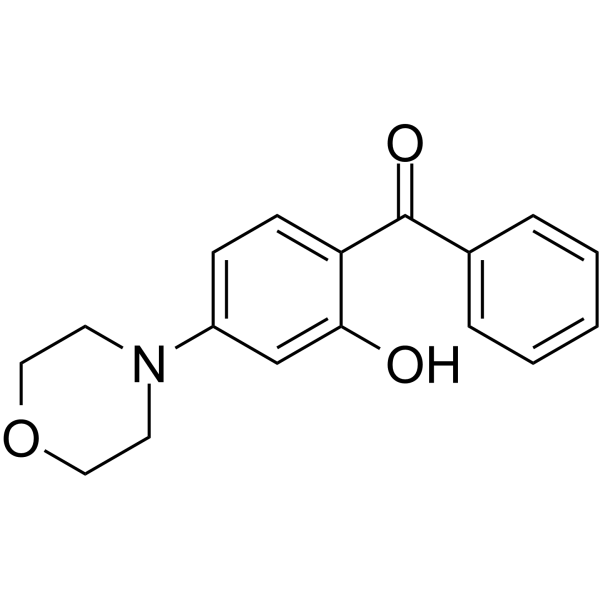
-
GC42783
Ametantrone
Ametantron (NSC 196473) ist ein Antitumormittel, das in DNA interkaliert und einen durch Topoisomerase II (TOP2) vermittelten DNA-Bruch induziert.
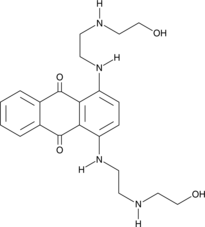
-
GC50366
AMG 18 hydrochloride
AMG 18-Hydrochlorid (AMG-18-Hydrochlorid) ist ein monoselektives IRE1α Inhibitor, der IRE1α allosterisch abschwÄcht; RNase-AktivitÄt mit einem IC50 von 5,9 nM.
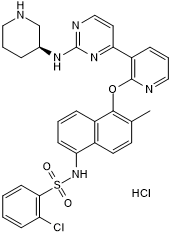
-
GC14899
AMG 548
AMG 548, ein oral aktiver und selektiver p38α-Inhibitor (Ki \u003d 0,5 nM), zeigt eine geringfügige Selektivität gegenüber p38β (Ki \u003d 36 nM) und eine \u003e1000-fache Selektivität gegenüber p38γ und p38δ.
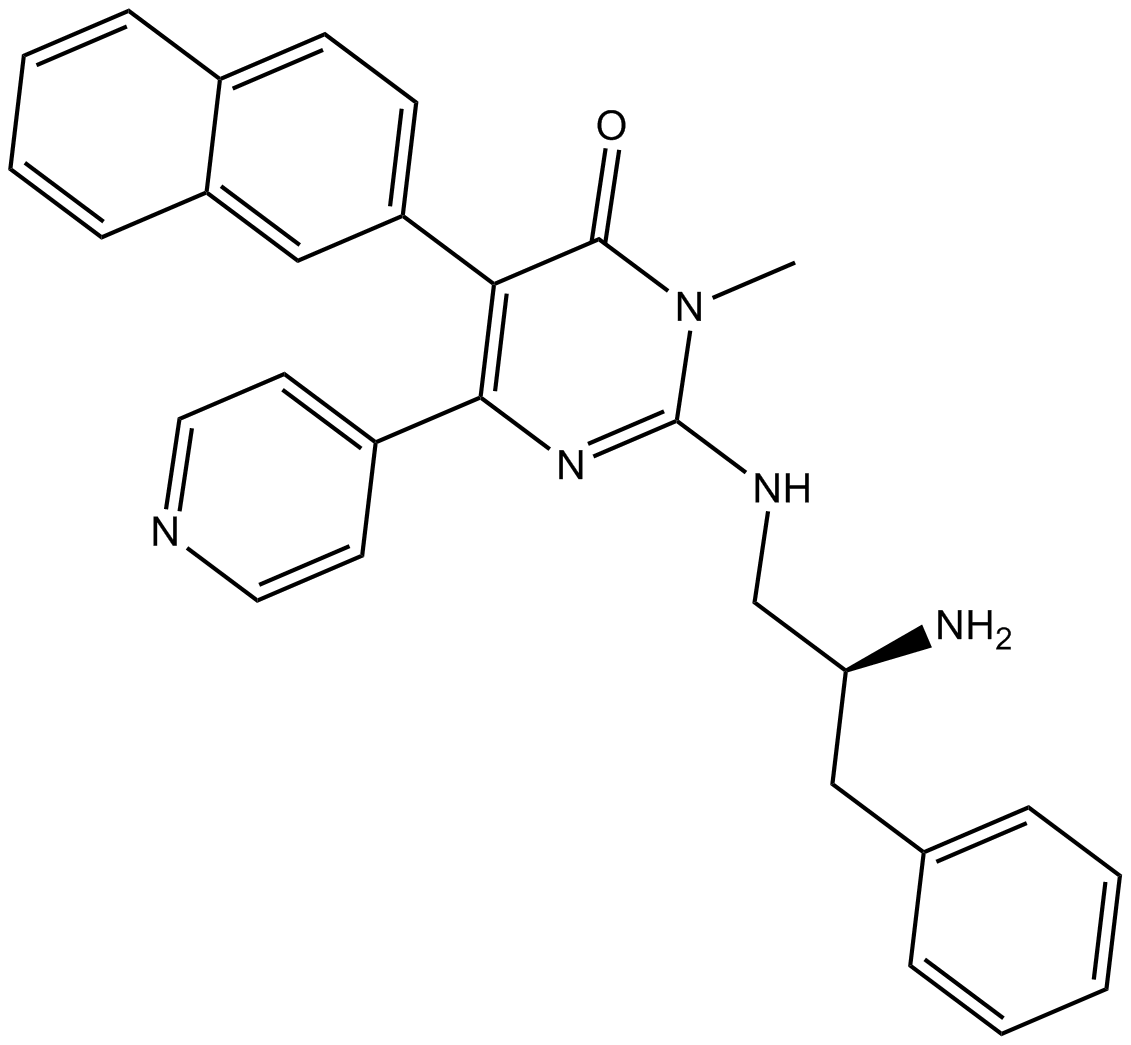
-
GC14974
AMG 925
AMG 925 ist ein potenter, selektiver und oral verfÜgbarer FLT3/CDK4-Doppelinhibitor mit IC50-Werten von 2±1 nM bzw. 3±1 nM.
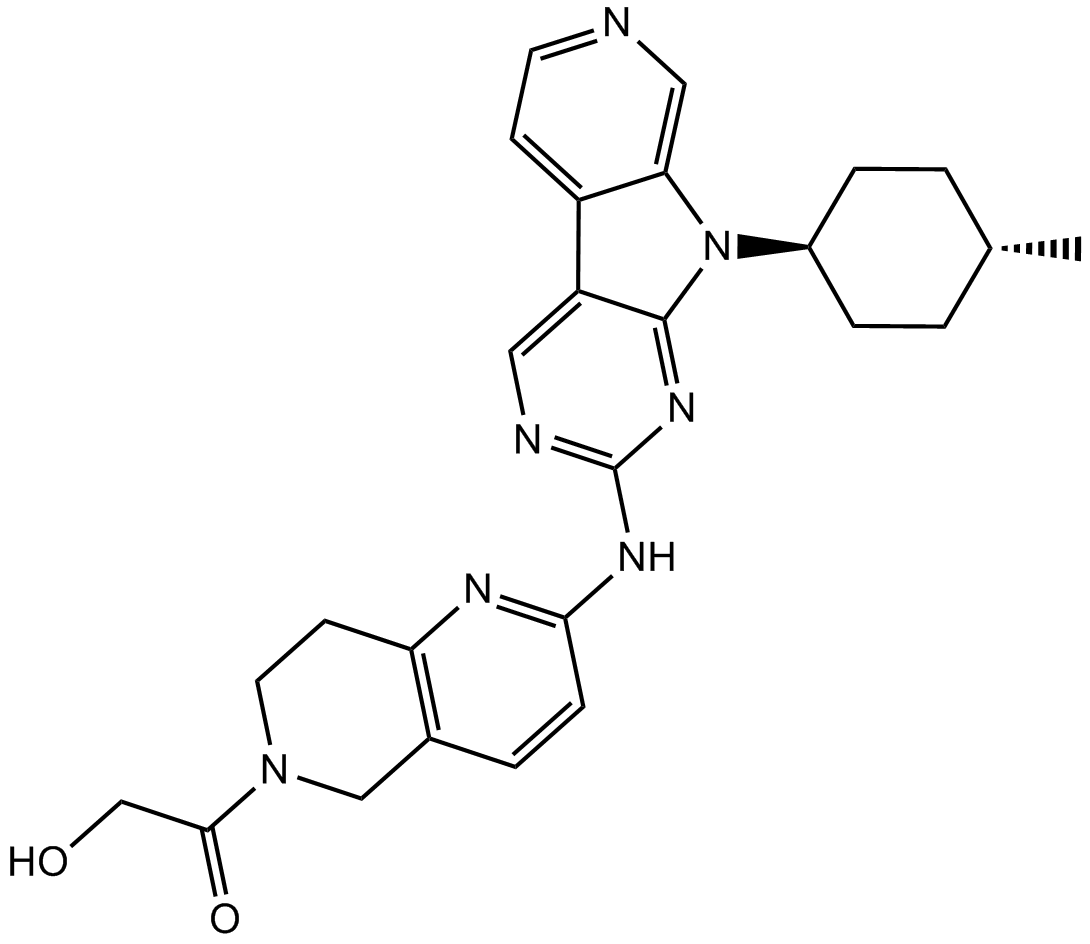
-
GC35315
AMG 925 HCl
AMG 925 HCl ist ein potenter, selektiver und oral verfÜgbarer dualer FLT3/CDK4-Inhibitor mit IC50-Werten von 2±1 nM bzw. 3±1 nM.

-
GC38518
AMG-548 dihydrochloride
AMG-548-Dihydrochlorid, ein oral aktiver und selektiver p38α-Inhibitor (Ki = 0,5 nM), zeigt eine geringfÜgige SelektivitÄt gegenÜber p38β (Ki = 36 nM) und >1000-fach selektiv gegenÜber p38γ und p38δ.
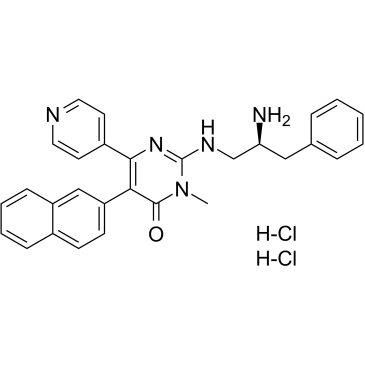
-
GC42789
Aminopterin
Aminopterin (4-AminofolsÄure), das 4-Amino-Derivat der FolsÄure, ist ein FolsÄureantagonist.
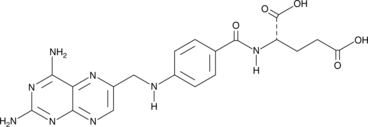
-
GC11019
Amonafide
Amonafide ist ein Topoisomerase-II-Inhibitor und DNA-Interkalator, der apoptotische Signale induziert, indem er die Bindung von Topo II an DNA blockiert.
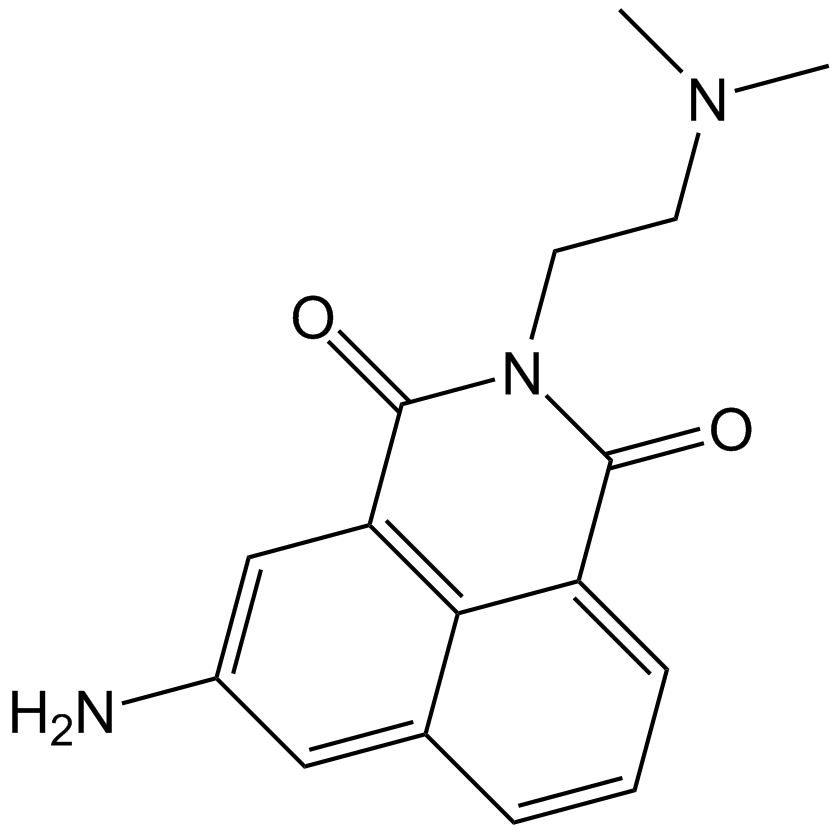
-
GC15644
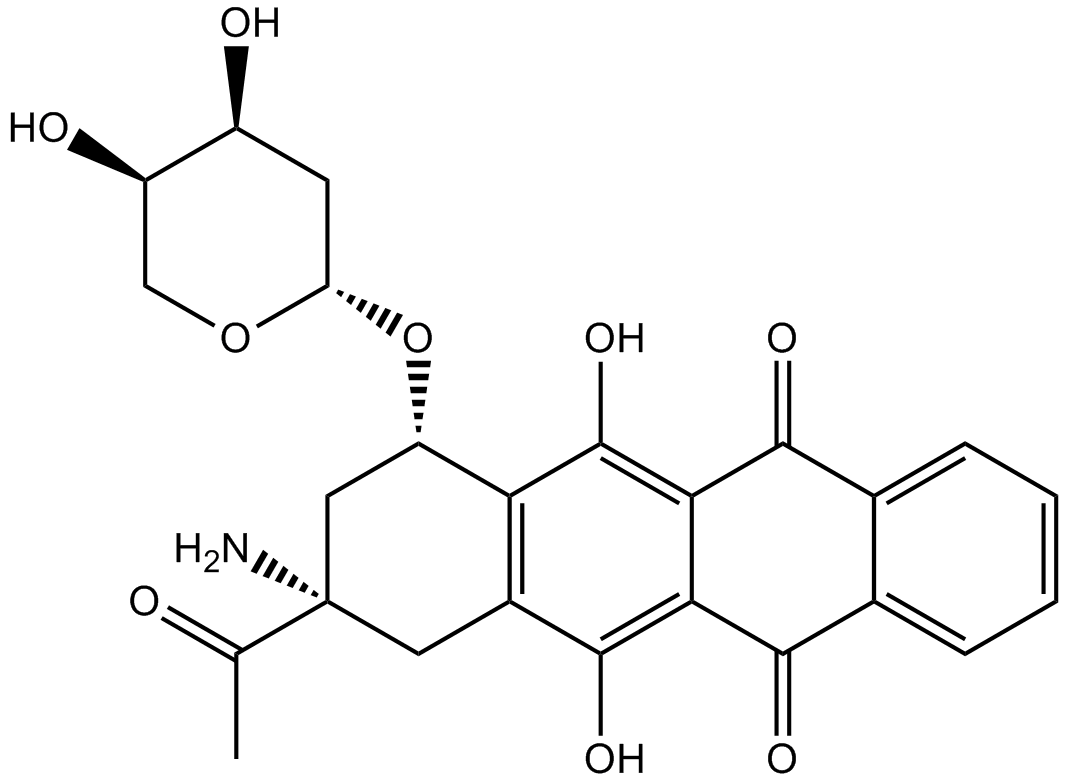
Amrubicin
A synthetic anthracycline antibiotic. It inhibits DNA topoisomerase II. Antineoplastic.
-
GC12326
Amsacrine
Amsacrin (m-AMSA; Acridinylanisidid) ist ein Inhibitor der Topoisomerase II und wirkt als antineoplastisches Mittel, das in die DNA von Tumorzellen interkalieren kann.

-
GC11747
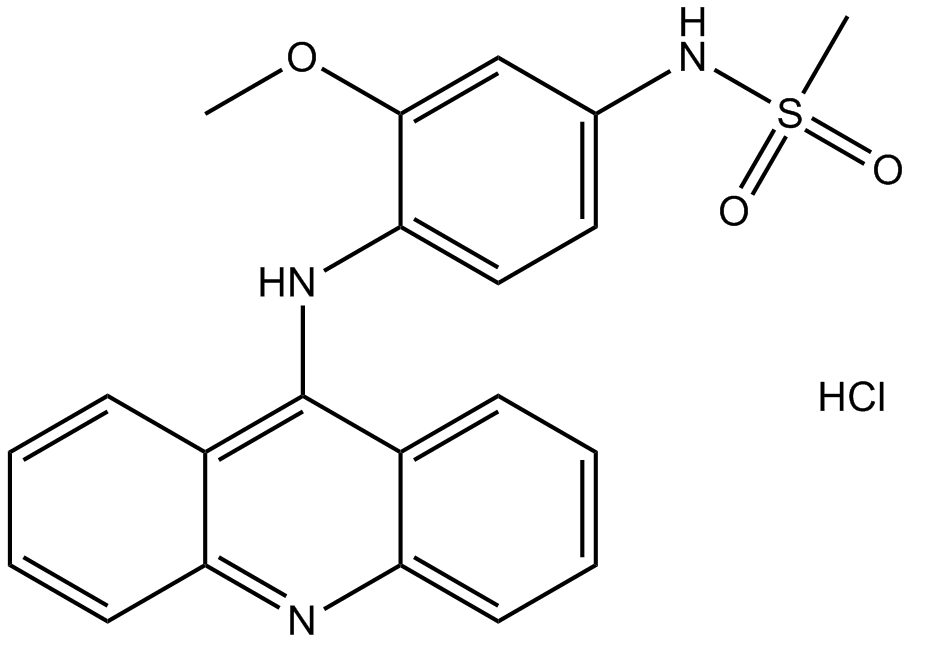
Amsacrine hydrochloride
Amsacrinhydrochlorid (m-AMSA-Hydrochlorid; Acridinylanisidid-Hydrochlorid) ist ein Inhibitor der Topoisomerase II und wirkt als antineoplastisches Mittel, das in die DNA von Tumorzellen interkalieren kann.
-
GC39284
ANI-7
ANI-7 ist ein Aktivator des Aryl-Kohlenwasserstoff-Rezeptor (AhR)-Signalwegs. ANI-7 hemmt das Wachstum mehrerer Krebszellen und hemmt wirksam und selektiv das Wachstum von MCF-7-Brustkrebszellen mit einem GI50 von 0,56 μM. ANI-7 induziert CYP1-metabolisierende Monooxygenasen durch Aktivierung des AhR-Signalwegs und induziert auch DNA-SchÄden, Aktivierung der Checkpoint-Kinase 2 (Chk2), Stillstand des S-Phasen-Zellzyklus und Zelltod in empfindlichen Brustkrebs-Zelllinien.
-
GC11559
Anisomycin
JNK-Agonist, stark und spezifisch.
-
GC68667
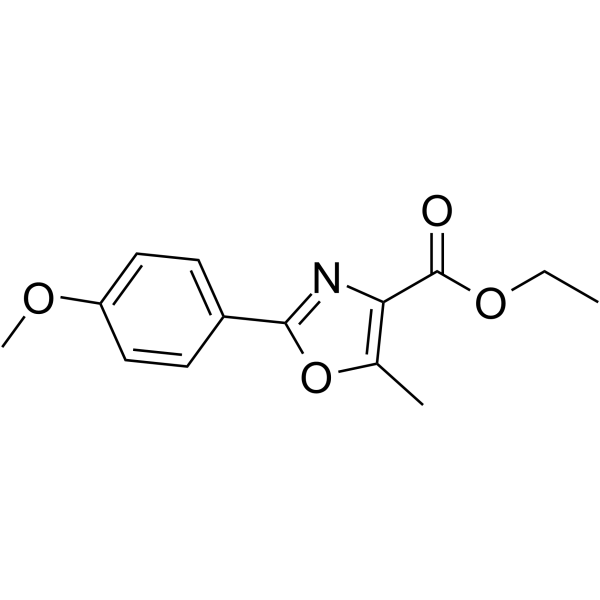
Anticancer agent 73
Antikrebsmittel 73 (Verbindung CIB-3b) ist ein Krebsmedikament, das auf TAR RNA bindendes Protein 2 (TRBP) abzielt und dessen Wechselwirkung mit Dicer zerstört. Antikrebsmittel 73 kann das Expressionsmuster von krebsverursachenden oder tumorunterdrückenden miRNAs neu ausbalancieren. Antikrebsmittel 73 hat eine hemmende Wirkung auf das Wachstum und die Metastasierung von Leberkrebszellen (HCC) in vitro und in vivo.
-
GC40032
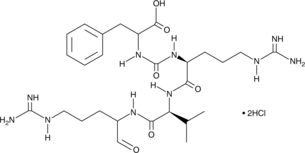
Antipain (hydrochloride)
Antipain (Hydrochlorid) ist ein aus Actinomycetes isolierter Protease-Inhibitor.
-
GC68448
AOH1160
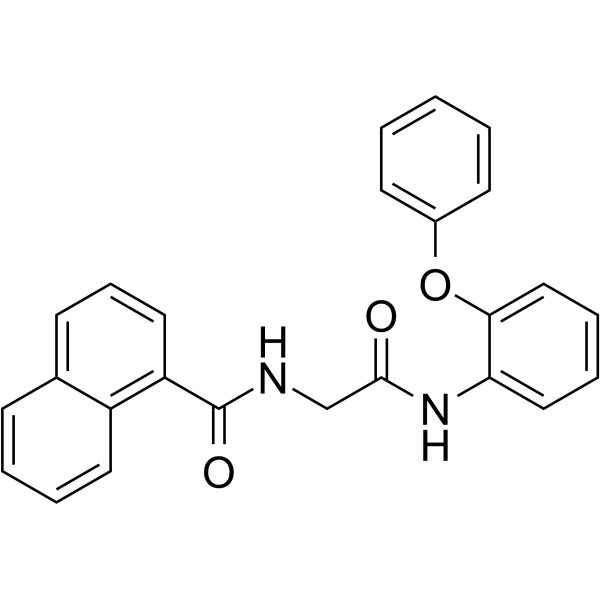
-
GC67876
APE1-IN-1

-
GC17139
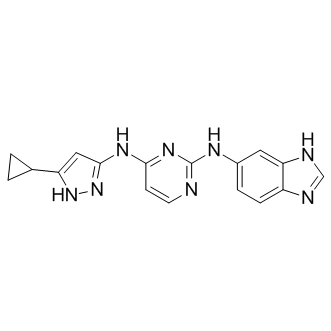
APY29
APY29, ein ATP-kompetitiver Inhibitor, ist ein allosterischer Modulator von IRE1α, der die Autophosphorylierung von IRE1α durch Bindung an die ATP-Bindungstasche mit einem IC50 von 280 nM hemmt. APY29 fungiert als Ligand, der die benachbarte RNase-DomÄne von IRE1α allosterisch aktiviert.
-
GC45385
Ara-G
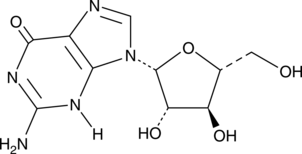
-
GC62425
ARN-21934
ARN-21934 ist ein potenter, hochselektiver Penetrationshemmer der Blut-Hirn-Schranke (BBB) fÜr die humane Topoisomerase II α Über β. ARN-21934 hemmt die DNA-Relaxation mit einem IC50 von 2 μM im Vergleich zum Antikrebsmittel Etoposid (IC50=120 μM). ARN-21934 weist ein gÜnstiges pharmakokinetisches Profil in vivo auf und ist eine vielversprechende Leitsubstanz fÜr die Krebsforschung.
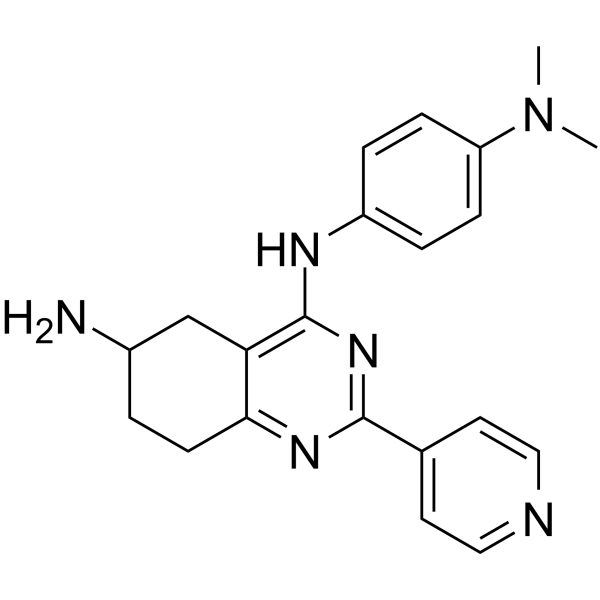
-
GC46880
ARN24139
A topoisomerase II poison
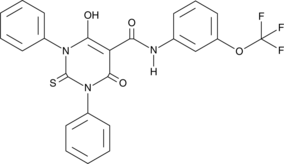
-
GC38736
AS2863619
AS2863619 ermÖglicht die Umwandlung antigenspezifischer Effektor-/GedÄchtnis-T-Zellen in regulatorische Foxp3+-T-Zellen (Treg) zur Behandlung verschiedener immunologischer Erkrankungen.
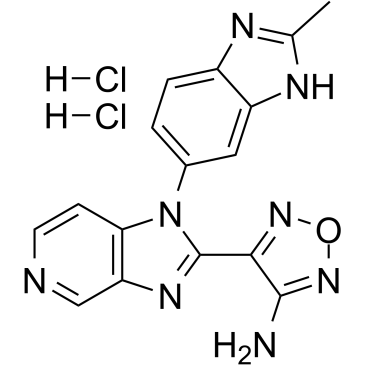
-
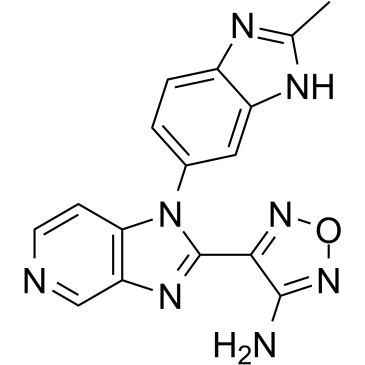
GC38737
AS2863619 free base
Die freie Base AS2863619 ermÖglicht die Umwandlung antigenspezifischer Effektor-/GedÄchtnis-T-Zellen in regulatorische Foxp3+-T-Zellen (Treg) zur Behandlung verschiedener immunologischer Erkrankungen.
-
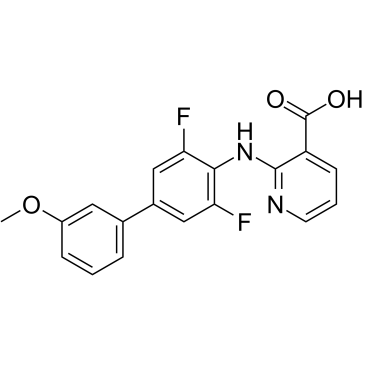
GC38463
ASLAN003
ASLAN003 (ASLAN003) ist ein oral aktiver und potenter Dihydroorotat-Dehydrogenase (DHODH)-Hemmer mit einem IC50-Wert von 35 nM fÜr das menschliche DHODH-Enzym. ASLAN003 hemmt die Proteinsynthese Über die Aktivierung von AP-1-Transkriptionsfaktoren. ASLAN003 induziert Apoptose und verlÄngert das Überleben von Xenograft-MÄusen mit akuter myeloischer LeukÄmie (AML) erheblich.
-
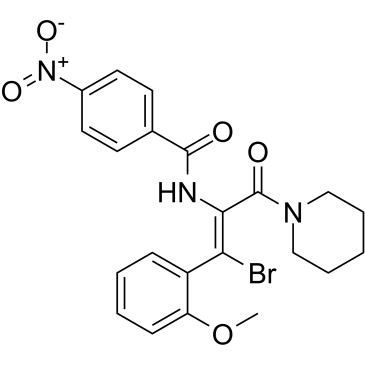
GC61821
AT-130
AT-130, ein Phenylpropenamid-Derivat, ist ein potenter Hepatitis-B-Virus (HBV)-Replikations-Nicht-Nukleosid-Hemmer.
-
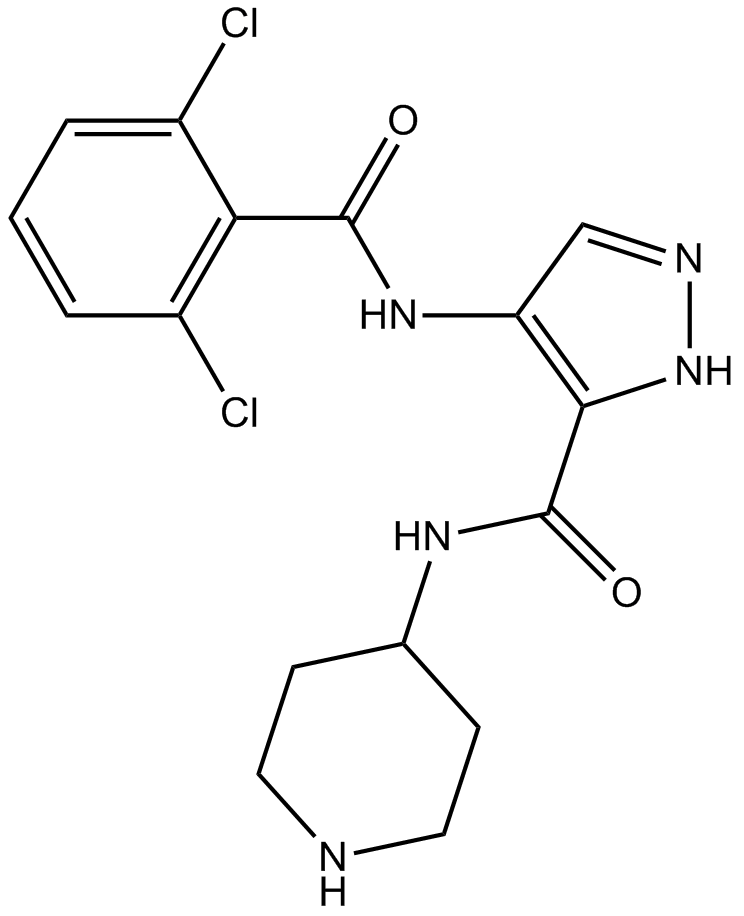
GC15870
AT7519
AT7519 (AT7519M) als potenter Inhibitor von CDKs mit IC50-Werten von 210, 47, 100, 13, 170 und < 10 nM fÜr CDK1, CDK2, CDK4 bis CDK6 bzw. CDK9.
-
GC13998
AT7519 Hydrochloride
A Cdk inhibitor
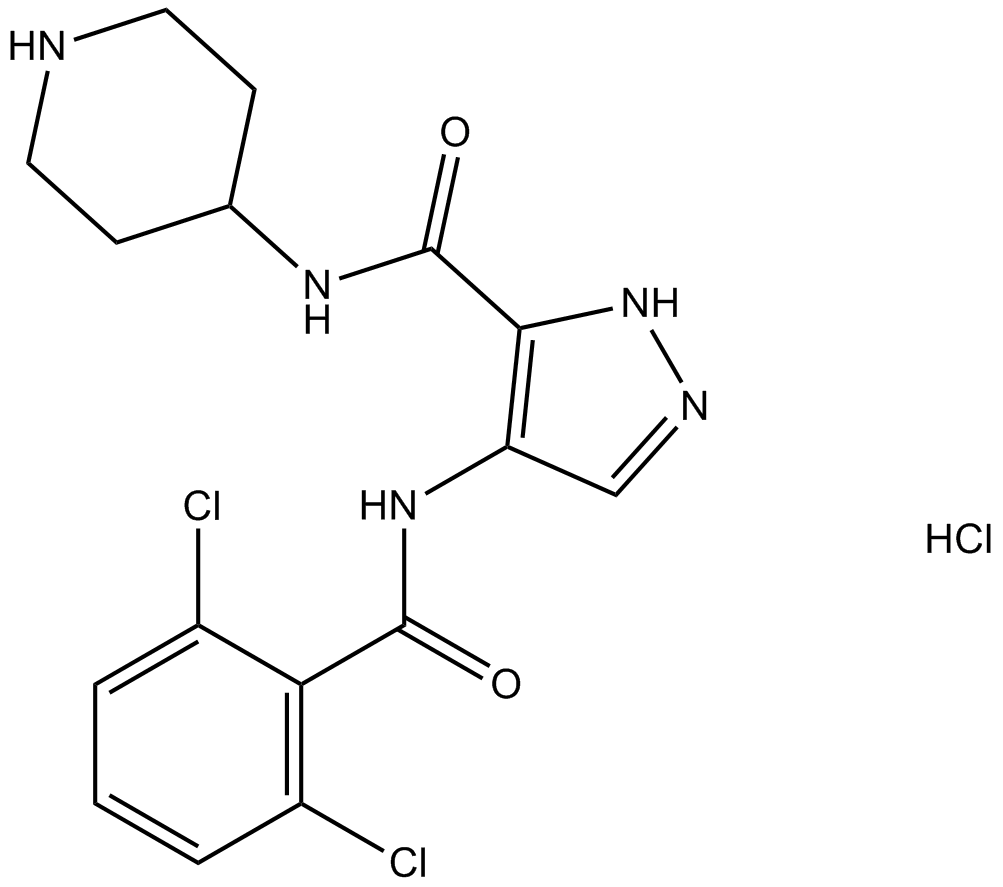
-
GC15597
AT7519 trifluoroacetate
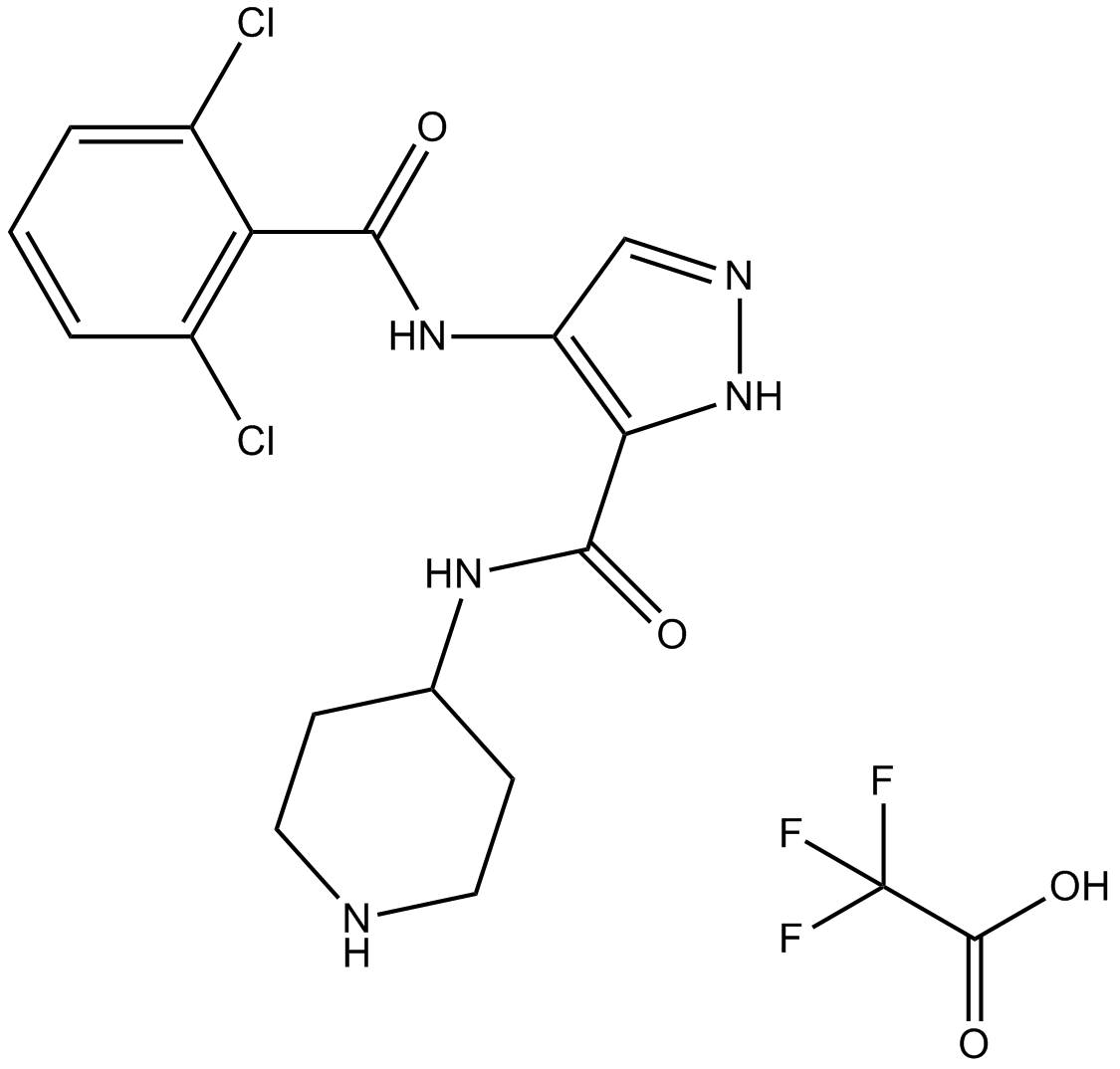
-
GC65327
ATM Inhibitor-5
ATM Inhibitor-5 [Formel (1)] ist ein potenter Inhibitor der Serin/Threonin-Proteinkinase ATM (aus dem Patent WO2022058351A1 entnommen).
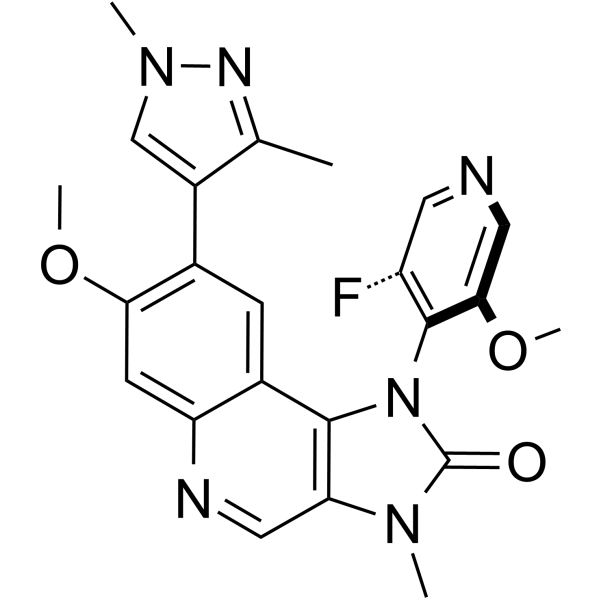
-
GC65514
ATR inhibitor 1
ATR-Inhibitor 1 ist ein aus Patent WO2015187451A1 extrahierter ATR-Inhibitor, Verbindung I-1, hat einen Ki-Wert unter 1 μμ.
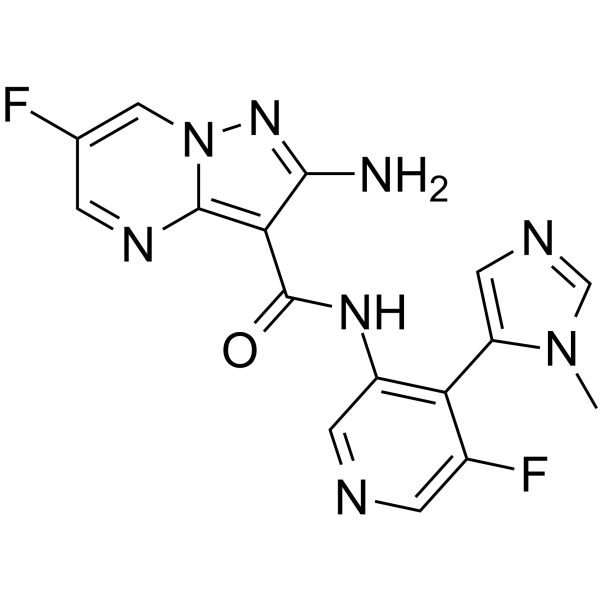
-
GC68707
ATR-IN-4
ATR-IN-4 ist ein wirksamer ATR-Inhibitor. ATR-IN-4 hemmt das Wachstum von menschlichen Prostatakrebszellen DU145 und menschlichen Lungenkrebszellen NCI-H460 mit IC50-Werten von jeweils 130,9 nM und 41,33 nM. (Aus dem Patent CN112142744A, Verbindung 13).
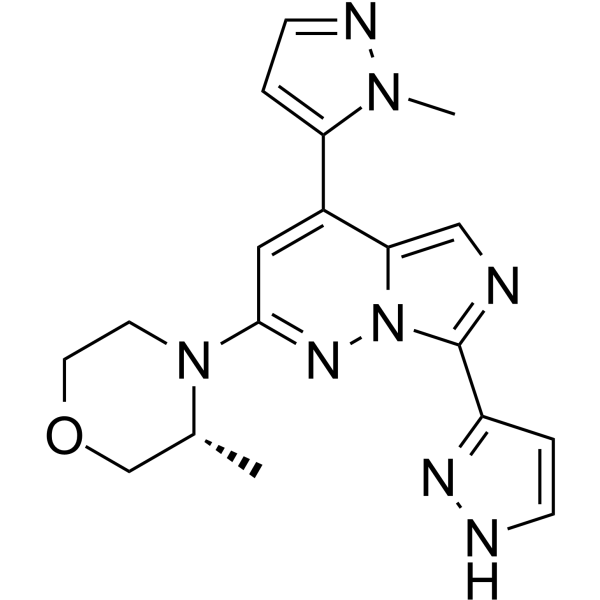
-
GC34422
Atuveciclib (BAY-1143572)
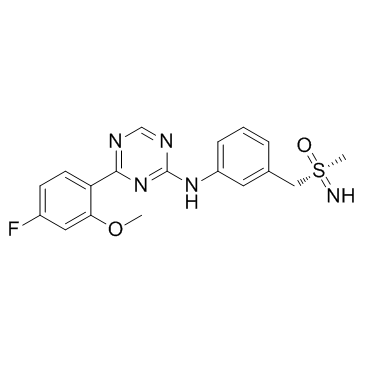
-
GC34059
Atuveciclib Racemate (BAY-1143572 Racemate)
Atuveciclib Racemate (BAY-1143572 Racemate) (BAY-1143572 Racemate) ist die Racematmischung von Atuveciclib. Atuveciclib ist ein potenter und hochselektiver oraler P-TEFb/CDK9-Inhibitor, der CDK9/CycT1 mit einem IC50 von 13 nM unterdrÜckt.
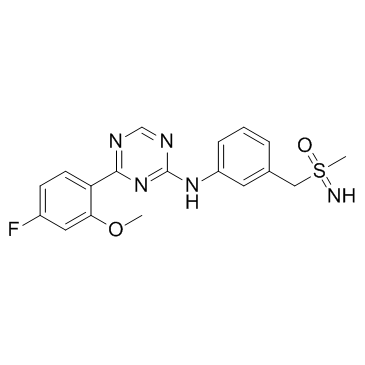
-
GC34421
Atuveciclib S-Enantiomer (BAY-1143572 S-Enantiomer)
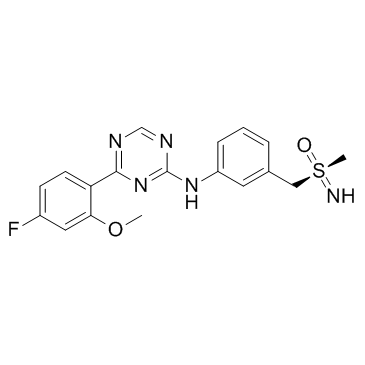
-
GC39699
Aurintricarboxylic acid
AurintricarbonsÄure ist ein nanomolarer, allosterischer Antagonist mit SelektivitÄt gegenÜber αβ-Methylen-ATP-sensitiven P2X1Rs und P2X3Rs, mit IC50s von 8,6 nM und 72,9 nM fÜr rP2X1R bzw. rP2X3R.
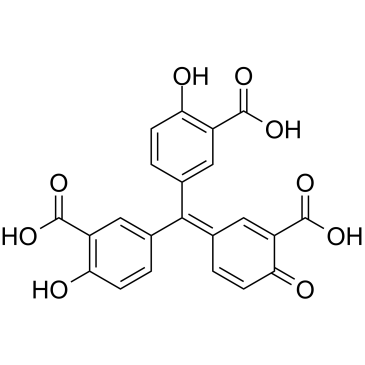
-
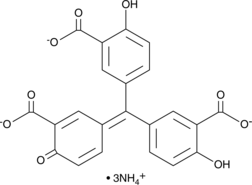
GC46895
Aurintricarboxylic Acid (ammonium salt)
A protein synthesis inhibitor with diverse biological activities
-
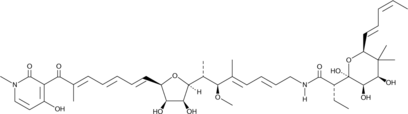
GC40005
Aurodox
Aurodox is a polyketide antibiotic originally isolated from S.
-
GC60062
AV-153 free base
Die freie Base von AV-153, ein 1,4-Dihydropyridin (1,4-DHP)-Derivat, ist ein Antimutagen. Die freie Base von AV-153 interkaliert in einem Einzelstrangbruch in die DNA und reduziert DNA-SchÄden, stimuliert die DNA-Reparatur in menschlichen Zellen in vitro. Die freie Base von AV-153 interagiert mit Thymin und Cytosin und hat einen Einfluss auf die Poly(ADP)ribosylierung. Die freie Base von AV-153 hat Anti-Krebs-AktivitÄt.
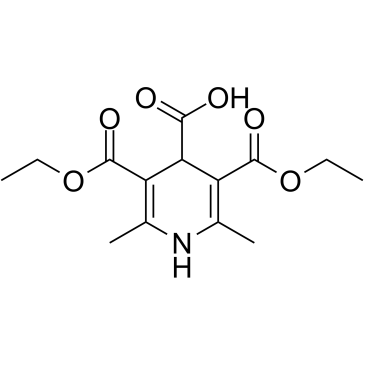
-
GC67960
AVG-233
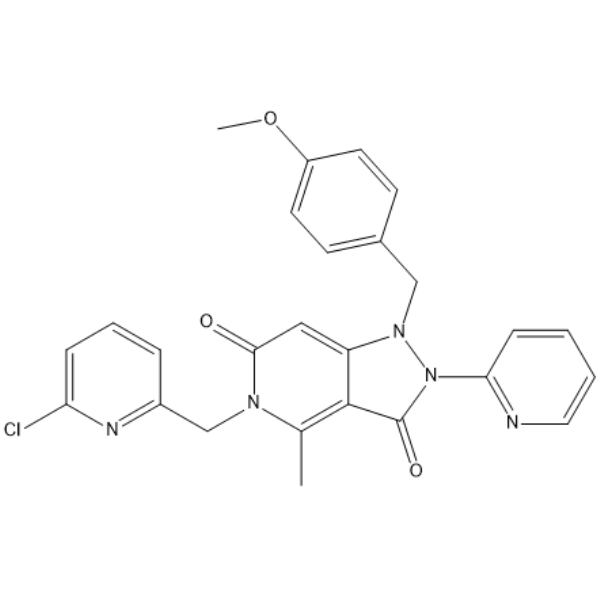
-
GC63449
Avotaciclib
Avotaciclib (BEY1107) ist ein potenter und oral aktiver Inhibitor der Cyclin-abhÄngigen Kinase 1 (CDK1). Avotaciclib kann zur Erforschung von lokal fortgeschrittenem oder metastasiertem BauchspeicheldrÜsenkrebs eingesetzt werden.
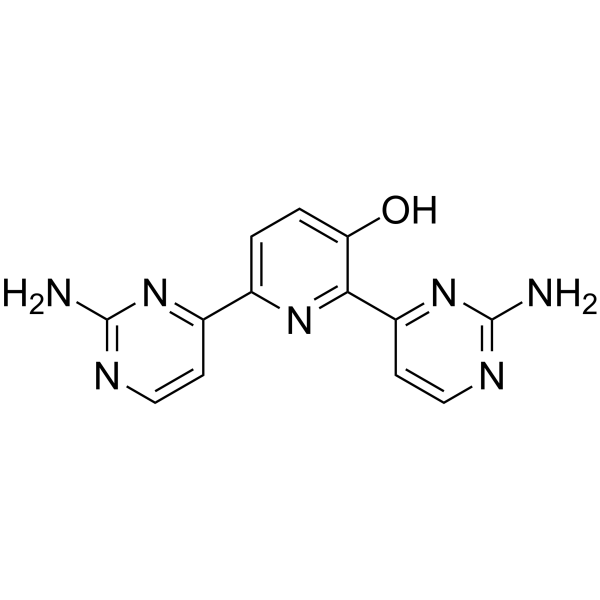
-
GC50424
AZ 5704
Potent and selective ATM kinase inhibitor; orally bioavailable
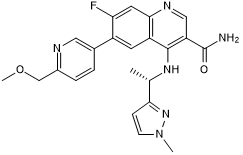
-
GC14949
AZ20
A potent, selective ATR inhibitor
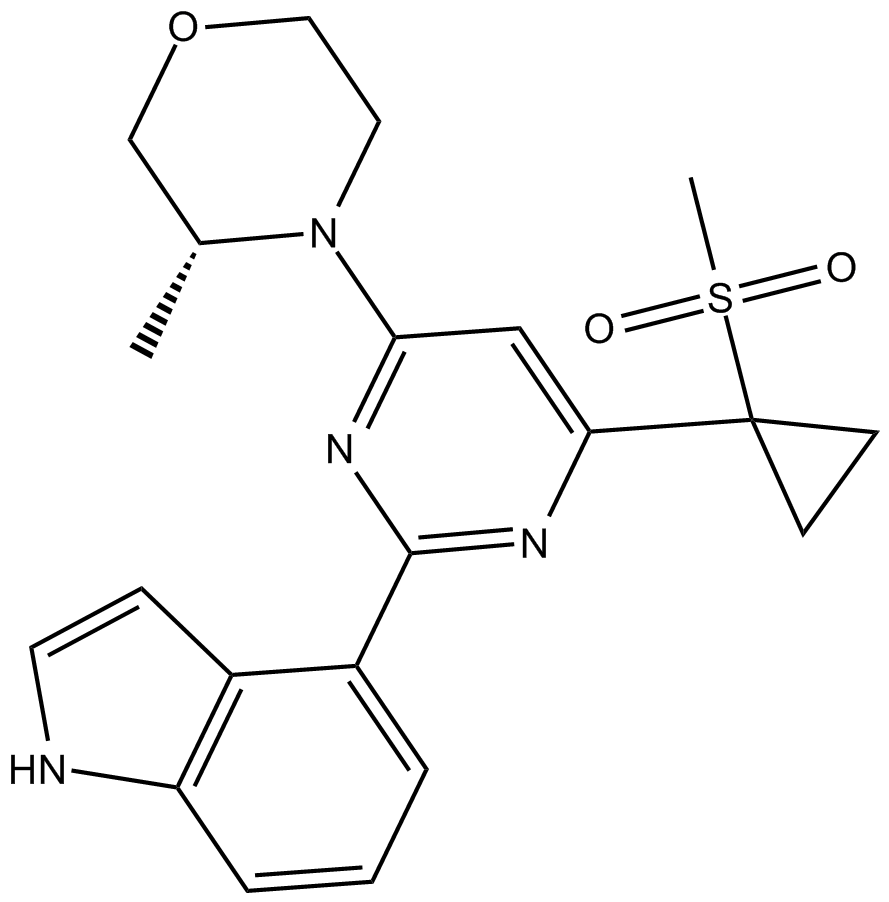
-
GC19047
AZ32
AZ32 ist ein oral bioverfÜgbarer und die Blut-Hirn-Schranke durchdringender ATM-Inhibitor mit einem IC50 von <6,2 nM fÜr das ATM-Enzym und einem IC50 von 0,31 μM fÜr ATM in der Zelle.
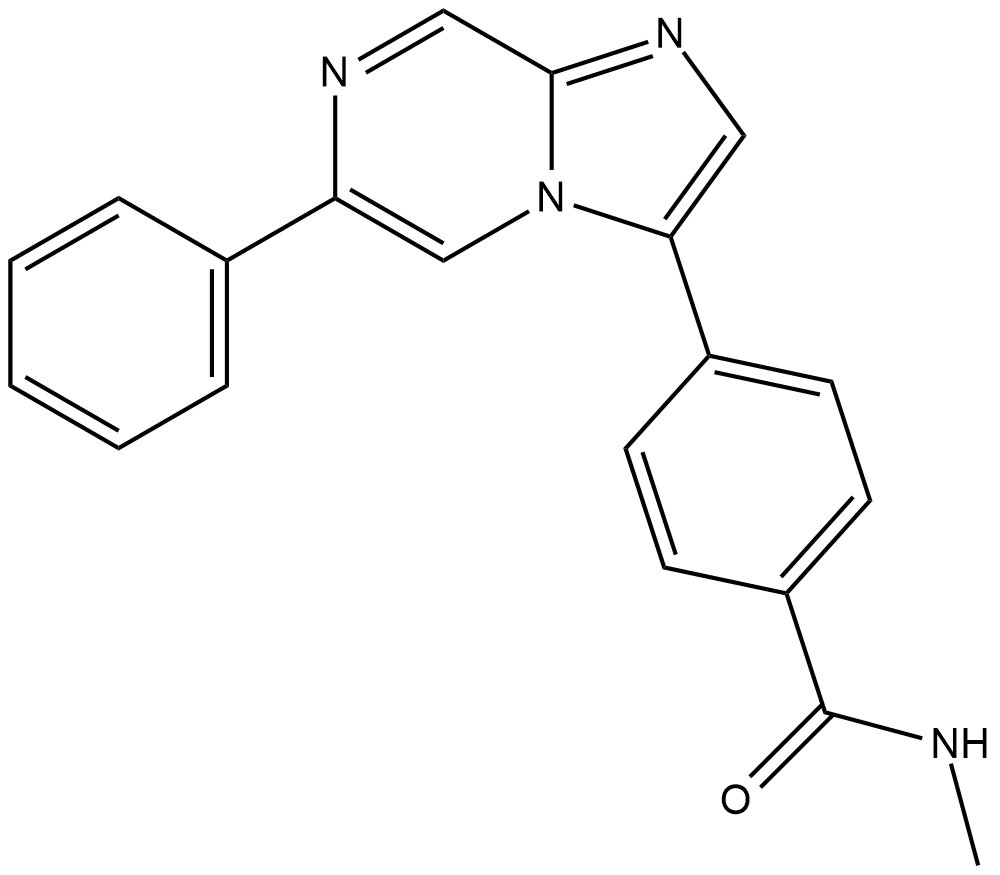
-
GC65899
AZ3391
AZ3391 ist ein potenter Inhibitor von PARP. AZ3391 ist ein Chinoxalin-Derivat. Die PARP-Enzymfamilie spielt eine wichtige Rolle bei einer Reihe von zellulÄren Prozessen, wie Replikation, Rekombination, Chromatin-Umbau und Reparatur von DNA-SchÄden. AZ3391 hat das Potenzial fÜr die Erforschung von Krankheiten und ZustÄnden, die in Geweben des zentralen Nervensystems wie Gehirn und RÜckenmark auftreten (aus Patent WO2021260092A1, Verbindung 23).
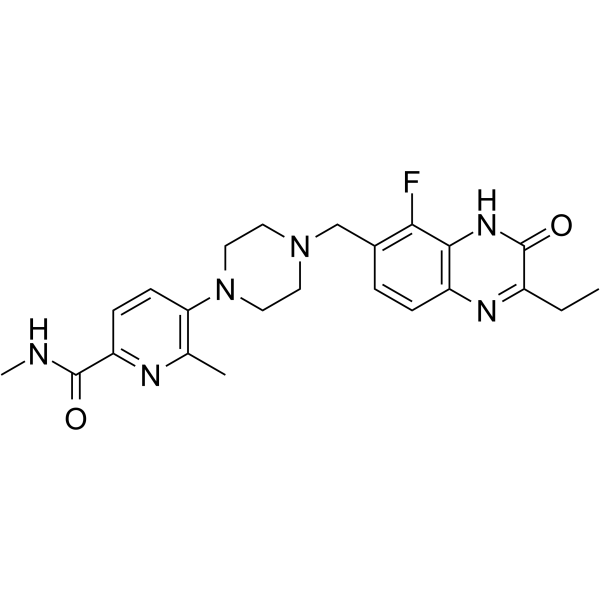
-
GC16725
AZ6102
AZ6102 ist ein potenter dualer TNKS1- und TNKS2-Inhibitor mit IC50-Werten von 3 nM bzw. 1 nM, und alao hat eine 100-fache SelektivitÄt gegenÜber anderen Enzymen der PARP-Familie mit IC50-Werten von 2,0 μM, 0,5 μM und >3 μM fÜr PARP1 , PARP2 bzw. PARP6.
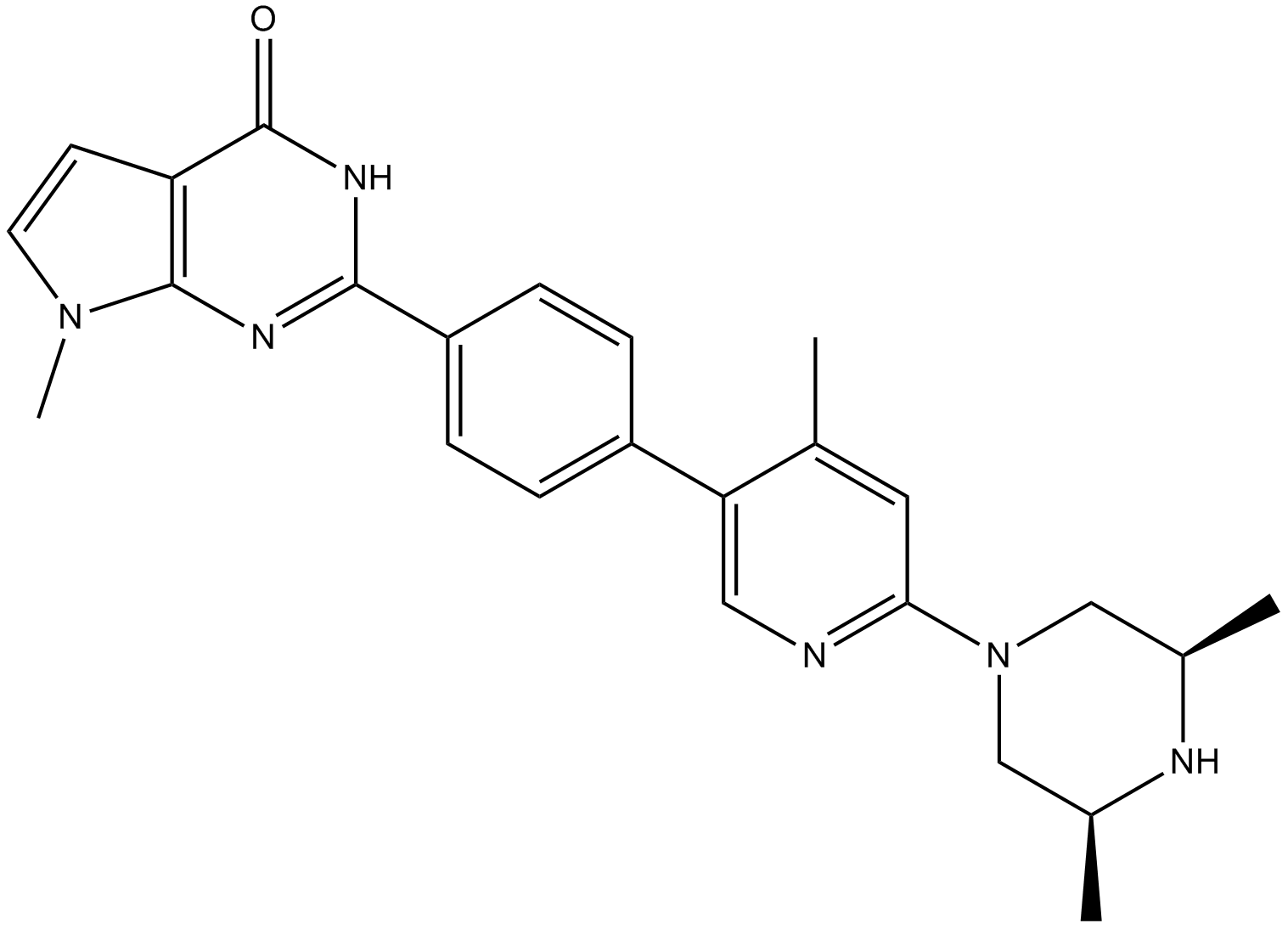
-
GC46900
AZ9482
AZ9482 ist ein dreifacher PARP1/2/6-Inhibitor mit IC50-Werten von 1 nM, 1 nM und 640 nM fÜr PARP1, PARP2 bzw. PARP6.
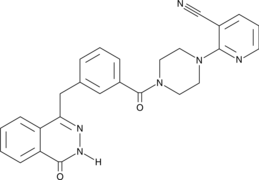
-
GC11843
Azaguanine-8
Azaguanin-8 ist ein Purin-Analogon, das antineoplastische AktivitÄt zeigt. Azaguanin-8 wirkt als Antimetabolit und baut sich leicht in RibonukleinsÄuren ein, stÖrt normale Biosynthesewege und hemmt so das Zellwachstum.
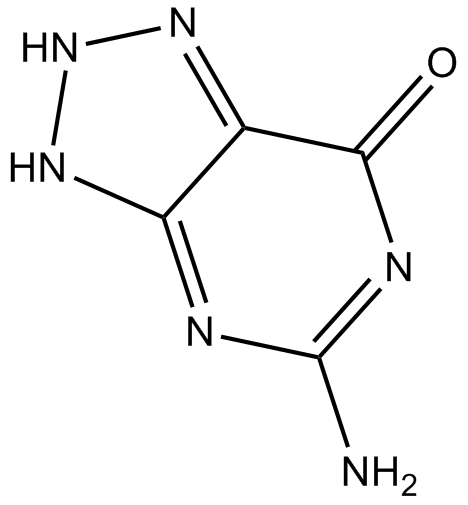
-
GC15033
Azathioprine
Azathioprin (BW 57-322) ist ein oral wirksames Immunsuppressivum.
-
GC50119
AZD 7762 hydrochloride
Potent and selective ATP-competitive inhibitor of Chk1 and Chk2; also enhances CRISPR-Cpf1-mediated genome editing
-
GC12438
AZD-5438
AZD-5438 ist ein potenter CDK1-, CDK2- und CDK9-Inhibitor mit IC50-Werten von 16 nM, 6 nM bzw. 20 nM in zellfreien Assays. AZD-5438 zeigt eine geringere HemmaktivitÄt gegen GSK3β, CDK5 und CDK6.
-
GC64938
AZD-7648
AZD-7648 ist ein potenter, oral aktiver, selektiver DNA-PK-Inhibitor mit einem IC50 von 0,6 nM. AZD-7648 induziert Apoptose und zeigt AntitumoraktivitÄt.
-
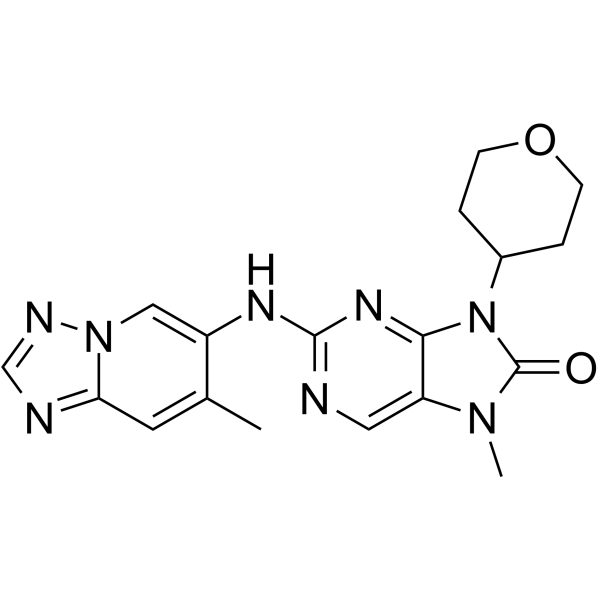
GC11593
AZD0156
AZD0156 ist ein potenter, selektiver und oral aktiver ATM-Inhibitor mit einem IC50 von 0,58 nM. AZD0156 hemmt die ATM-vermittelte SignalÜbertragung, verhindert die Aktivierung von Checkpoints fÜr DNA-SchÄden, unterbricht die Reparatur von DNA-SchÄden und induziert die Apoptose von Tumorzellen.
-
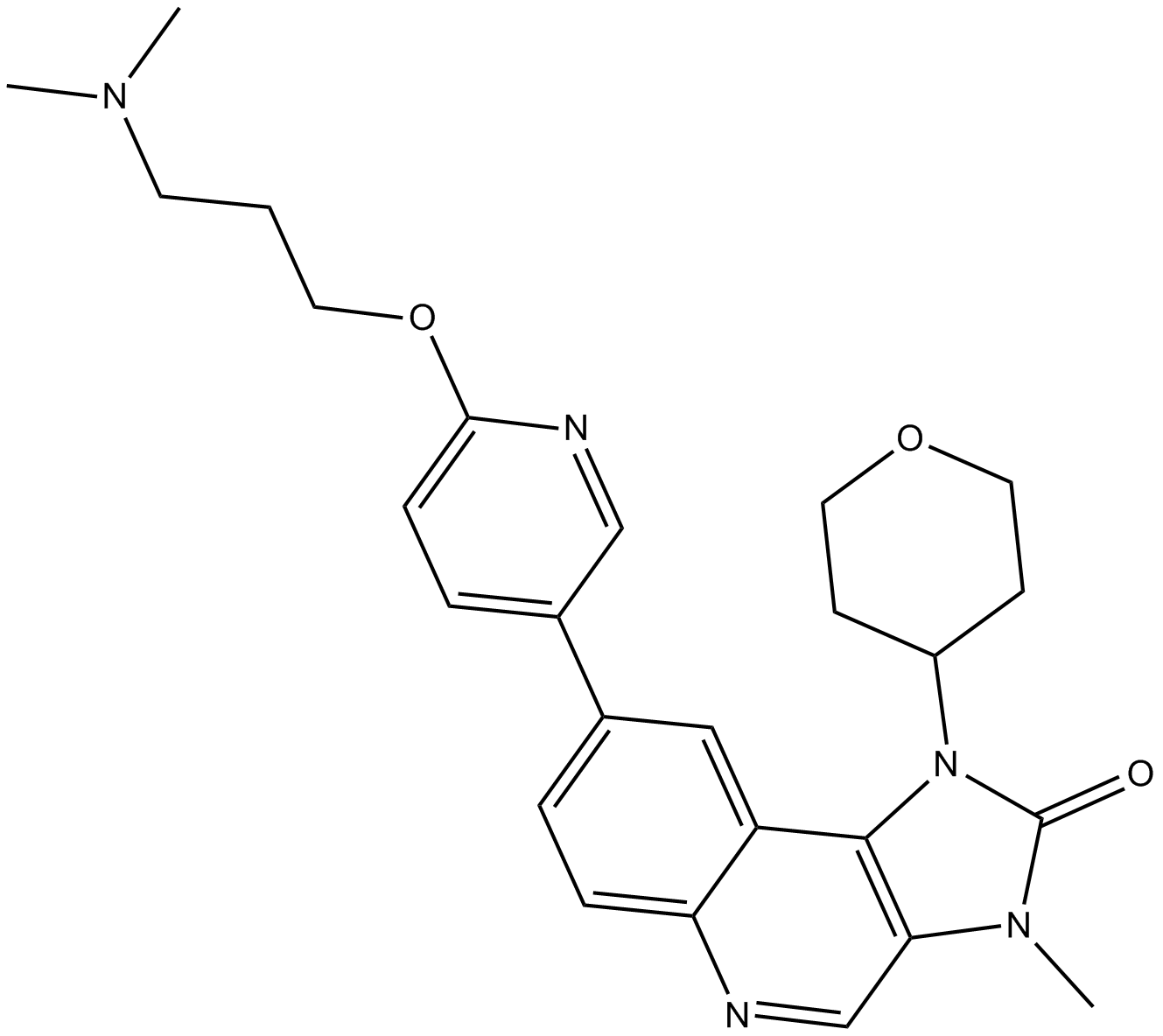
GC19468
AZD1390
AZD1390 ist ein potenter und selektiver ATM-Inhibitor.

-
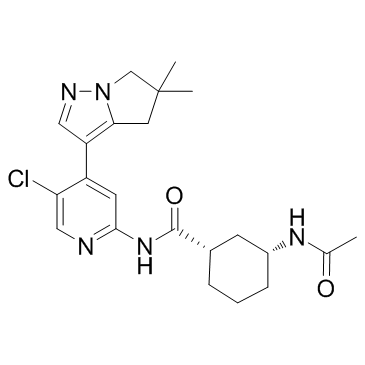
GC32717
AZD4573
AZD4573 ist ein potenter und hochselektiver CDK9-Inhibitor (IC50 von <4 nM), der eine transiente Zielerfassung zur Behandlung hÄmatologischer Malignome ermÖglicht.
-
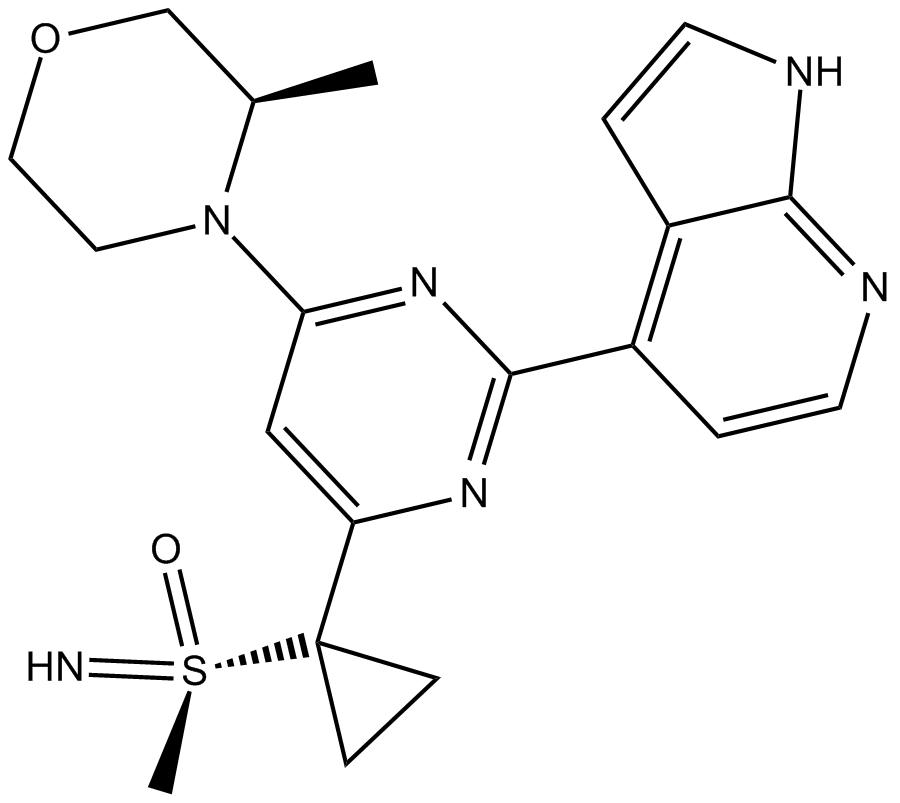
GC16941
AZD6738
AZD6738 (AZD6738) ist ein oral aktiver und bioverfÜgbarer Inhibitor der ATR-Kinase mit einem IC50 von 1 nM.
-
GC10546
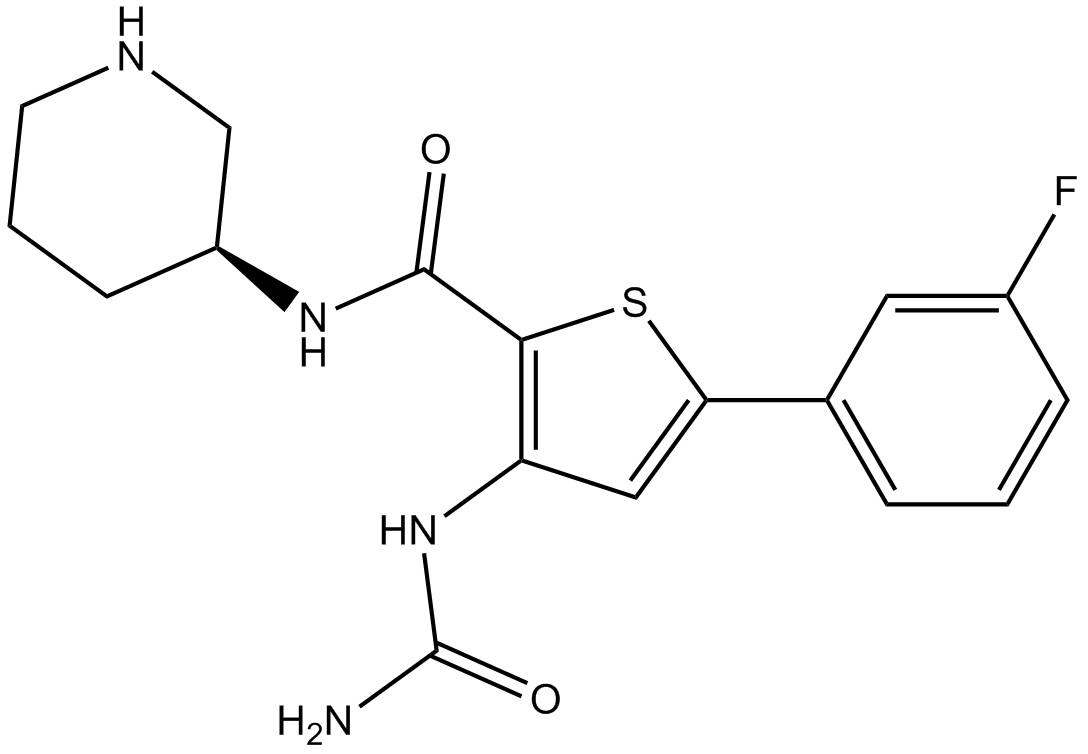
AZD7762
AZD7762 ist ein potenter ATP-kompetitiver Checkpoint-Kinase (Chk)-Inhibitor mit einem IC50 von 5 nM für Chk1.
-
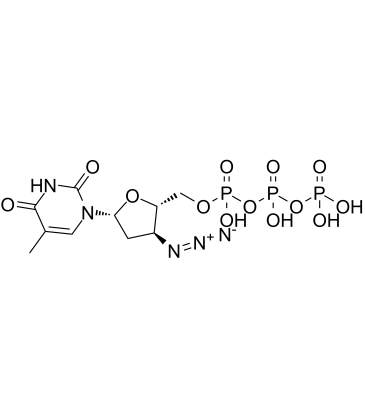
GC60616
AZT triphosphate
AZT-Triphosphat (3'-Azido-3'-desoxythymidin-5'-triphosphat) ist ein aktiver Triphosphat-Metabolit von Zidovudin (AZT).
-
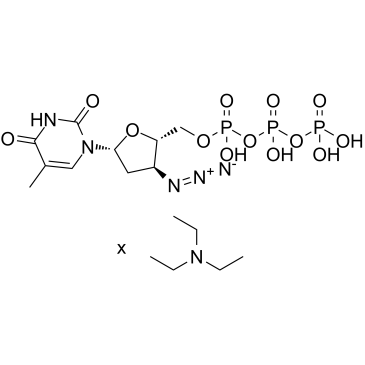
GC60617
AZT triphosphate TEA
AZT-Triphosphat TEA (3'-Azido-3'-desoxythymidin-5'-triphosphat TEA) ist ein aktiver Triphosphat-Metabolit von Zidovudin (AZT).
-
GC33406
B I09
B I09 ist ein IRE-1-RNase-Inhibitor mit einem IC50-Wert von 1230 nM.
-
GC33240
Banoxantrone D12 (AQ4N D12)
Banoxantron D12 (AQ4N D12) ist das mit Deuterium markierte Banoxantron. Banoxantron ist ein neuartiges bioreduktives Mittel, das zu einer stabilen, DNA-affinen Verbindung AQ4 reduziert werden kann, die ein wirksamer Topoisomerase-II-Inhibitor ist.
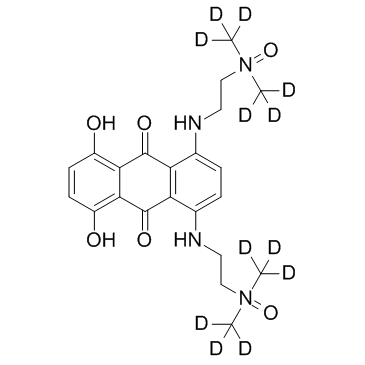
-
GC34169
Banoxantrone D12 dihydrochloride (AQ4N D12 dihydrochloride)
Banoxantron-d12 (AQ4N-d12) Dihydrochlorid ist das mit Deuterium markierte Banoxantron-Dihydrochlorid. Banoxantron ist ein neuartiges bioreduktives Mittel, das zu einer stabilen, DNA-affinen Verbindung AQ4 reduziert werden kann, die ein wirksamer Topoisomerase-II-Inhibitor ist.
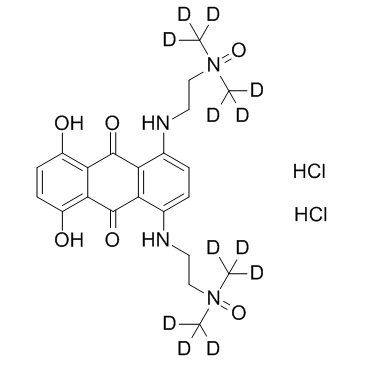
-
GC34085
Banoxantrone dihydrochloride (AQ4N dihydrochloride)
Banoxantron-Dihydrochlorid (AQ4N-Dihydrochlorid) ist ein neuartiges bioreduktives Mittel, das zu einem stabilen, DNA-affinen Verbindung AQ4 reduziert werden kann. AQ4 ist ein potenter Topoisomerase-II-Inhibitor.
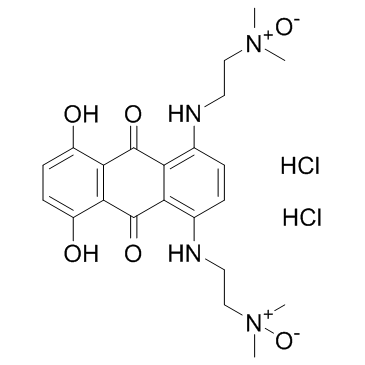
-
GC13035
Bay 11-7821
Ein selektiver und irreversibler NF-κB-Inhibitor
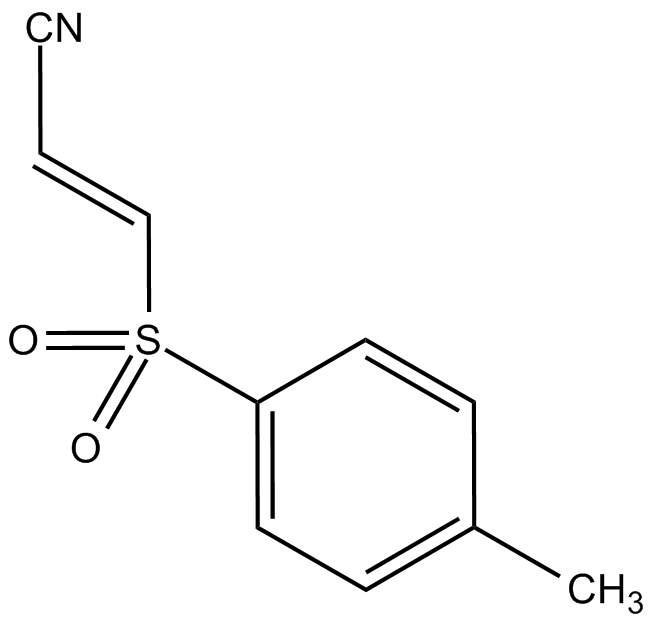
-
GC50656
BAY 707
BAY 707 ist ein substratkompetitiver, hochpotenter und selektiver Inhibitor von MTH1(NUDT1) mit einem IC50 von 2,3 nM. BAY 707 hat ein gutes pharmakokinetisches (PK) Profil im Vergleich zu anderen MTH1-Verbindungen und wird von Mäusen gut vertragen, zeigt jedoch einen deutlichen Mangel an In-vitro- oder In-vivo-Antikrebswirksamkeit.
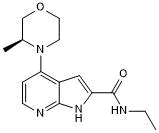
-
GC33420
BAY-1895344
BAY-1895344 (BAY-1895344) ist ein potenter, oral aktiver und selektiver ATR-Inhibitor mit einem IC50 von 7 nM. BAY-1895344 hat Anti-Tumor-AktivitÄt. BAY-1895344 kann zur Erforschung von soliden Tumoren und Lymphomen eingesetzt werden.
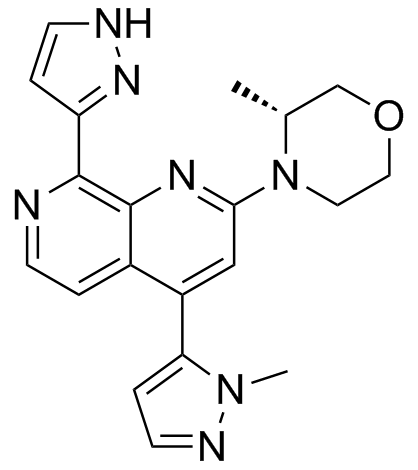
-
GC34374
BAY-1895344 hydrochloride
An ATR inhibitor
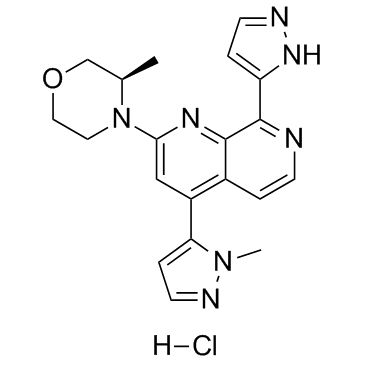
-
GC32838
BAY-2402234
BAY-2402234 ist ein selektiver Dihydroorotat-Dehydrogenase (DHODH)-Hemmer zur Behandlung myeloischer Malignome.
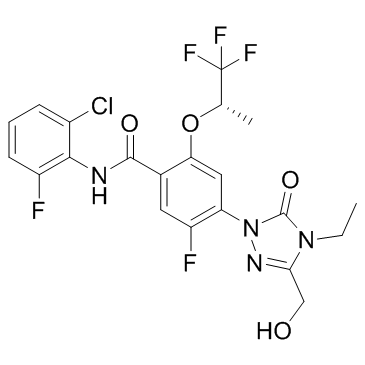
-
GC63763
BAY-8400
BAY-8400 ist ein oral aktiver, potenter und selektiver DNA-abhÄngiger Proteinkinase (DNA-PK)-Inhibitor (IC50=81 nM). BAY-8400 kann fÜr die Krebsforschung eingesetzt werden.
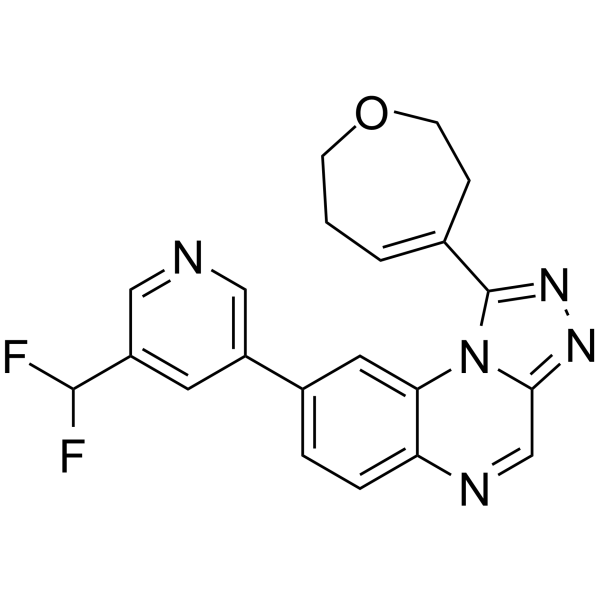
-
GC65259
BC-1471
BC-1471 ist ein STAM-bindendes Protein (STAMBP)-Deubiquitinase-Inhibitor.
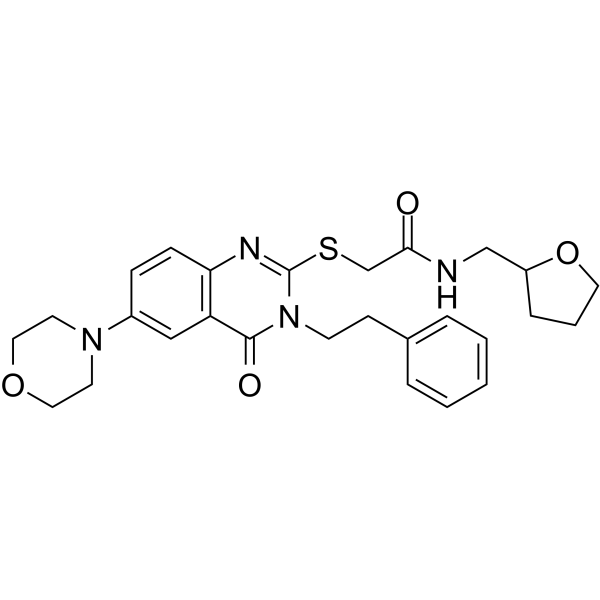
-
GC50572
BC-LI-0186
BC-LI-0186 ist ein potenter und selektiver Inhibitor der Interaktion von Leucyl-tRNA-Synthetase (LRS; LeuRS) und Ras-verwandtem GTP-bindendem Protein D (RagD) (IC50 = 46,11 nM). BC-LI-0186 bindet kompetitiv an die RagD-Interaktionsstelle von LRS (Kd = 42,1 nM) und hat Auswirkungen auf LRS-Vps34, LRS-EPRS, RagB-RagD-Assoziation, mTORC1-Komplexbildung oder die AktivitÄten von 12 Kinasen. BC-LI-0186 kann die AktivitÄt von krebsassoziierten MTOR-Mutanten und das Wachstum von Rapamycin-resistenten Krebszellen wirksam unterdrÜcken. BC-LI-0186 ist ein vielversprechender Wirkstoff fÜr die Lungenkrebsforschung.
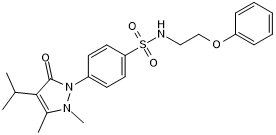
-
GC62863
BCH001
BCH001, ein Chinolinderivat, ist ein spezifischer PAPD5-Hemmer.