DNA/RNA Synthesis
RNA synthesis, which is also called DNA transcription, is a highly selective process. Transcription by RNA polymerase II extends beyond RNA synthesis, towards a more active role in mRNA maturation, surveillance and export to the cytoplasm.
Single-strand breaks are repaired by DNA ligase using the complementary strand of the double helix as a template, with DNA ligase creating the final phosphodiester bond to fully repair the DNA.DNA ligases discriminate against substrates containing RNA strands or mismatched base pairs at positions near the ends of the nickedDNA. Bleomycin (BLM) exerts its genotoxicity by generating free radicals, whichattack C-4′ in the deoxyribose backbone of DNA, leading to opening of the ribose ring and strand breakage; it is an S-independentradiomimetic agent that causes double-strand breaks in DNA.
First strand cDNA is synthesized using random hexamer primers and M-MuLV Reverse Transcriptase (RNase H). Second strand cDNA synthesis is subsequently performed using DNA Polymerase I and RNase H. The remaining overhangs are converted into blunt ends using exonuclease/polymerase activity. After adenylation of the 3′ ends of DNA fragments, NEBNext Adaptor with hairpin loop structure is ligated to prepare the samples for hybridization. Cell cycle and DNA replication are the top two pathways regulated by BET bromodomain inhibition. Cycloheximide blocks the translation of mRNA to protein.
Ziele für DNA/RNA Synthesis
Produkte für DNA/RNA Synthesis
- Bestell-Nr. Artikelname Informationen
-
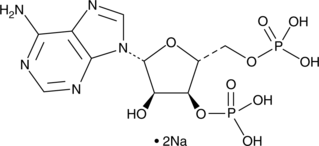
GC38000
β-Boswellic acid
- Boswelliasäure wird aus dem Gummiharz von Boswellia serrate isoliert . β-Boswelliasäure hemmt die Synthese von DNA, RNA und Protein in menschlichen Leukämie-HL-60-Zellen.
-
GC10867
(+)-Aphidicolin
Aphidicolin ((+)-Aphidicolin), ein reversibler Inhibitor der nuklearen DNA-Replikation bei Eukaryoten, kann den Zellzyklus in der prä-S-Phase blockieren[1].
-
GC48635
(-)-Cryptopleurine
An alkaloid with diverse biological activities
-
GC41695
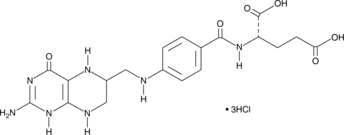
(6R,S)-5,6,7,8-Tetrahydrofolic Acid (hydrochloride)
(6R,S)-5,6,7,8-Tetrahydrofolic acid (THFA), the reduced form of folic acid, serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
-
GC41088
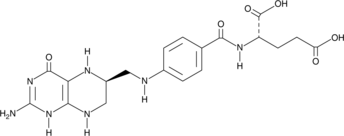
(6S)-Tetrahydrofolic Acid
(6S)-Tetrahydrofolic acid is a diastereomer of tetrahydrofolic acid, a reduced form of folic acid that serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
-
GC41633
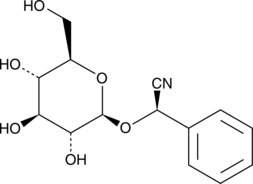
(R)-Prunasin
(R)-Prunasin ist ein Inhibitor der DNA-Polymerase β.
-
GC41859
1,5,7-Triazabicyclo[4.4.0]dec-5-ene
1,5,7-Triazabicyclo[4.4.0]dec-5-ene is a bicyclic guanidine base that can be used as a catalyst for a variety of base-mediated organic transformations, including Michael additions, Wittig reactions, Henry reactions, and transesterification reactions.
![1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure 1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure](/media/struct/GC4/GC41859.png)
-
GC48909
1-Hydroxyanthraquinone
1-Hydroxyanthrachinon, eine natÜrlich vorkommende Verbindung mit oraler Wirkung aus einigen Pflanzen wie Tabebuia avellanedae, weist eine krebserzeugende Wirkung auf.

-
GC49872
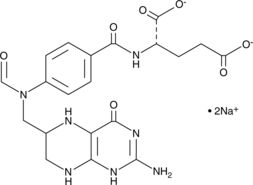
10-Formyltetrahydrofolate (sodium salt) (technical grade)
10-Formyltetrahydrofolat (Natriumsalz) (technische QualitÄt) ist eine Form von TetrahydrofolsÄure, die als Spender von Formylgruppen im Anabolismus wirkt.
-
GC63796
116-9e
116-9e (MAL2-11B) ist ein Hsp70-Co-Chaperon-DNAJA1-Inhibitor.
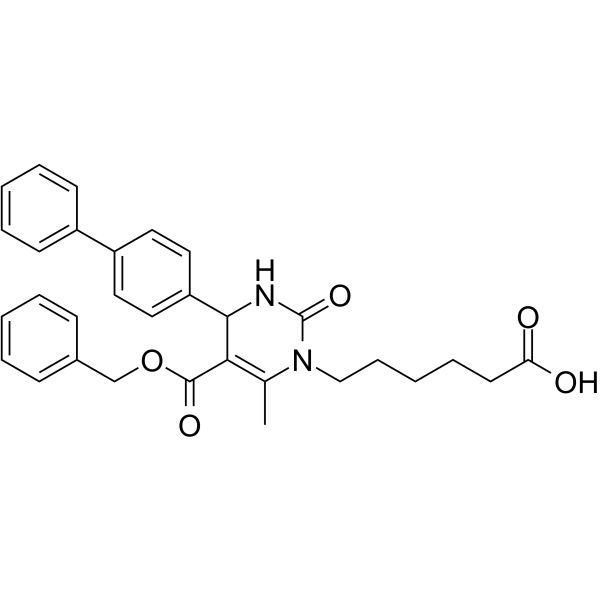
-
GC49759
13C17-Mycophenolic Acid
An internal standard for the quantification of mycophenolic acid
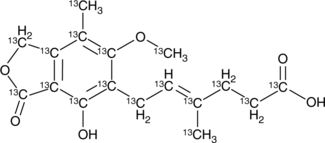
-
GC46474
18-Deoxyherboxidiene
18-Desoxyherboxidien (RQN-18690A) ist ein potenter Angiogenese-Inhibitor.

-
GC65489
2'-F-Bz-dC Phosphoramidite
&2#39;-F-Bz-dC Phosphoramidit kann bei der Synthese von Oligoribonukleotiden verwendet werden.
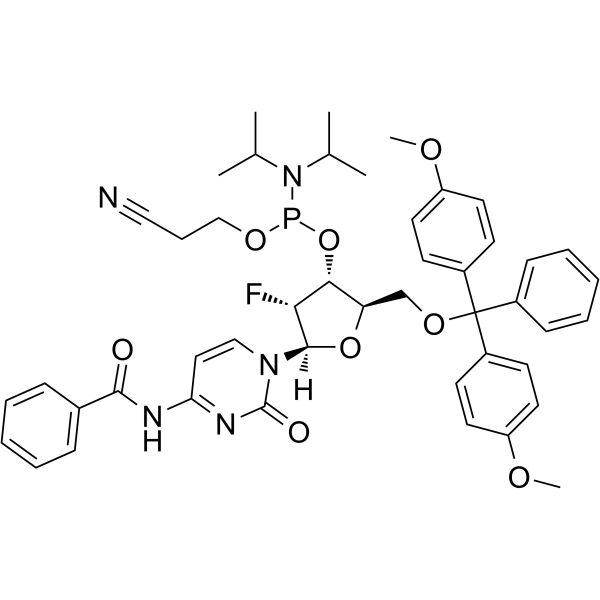
-
GC52183
2'-O-Methyl-5-methyluridine
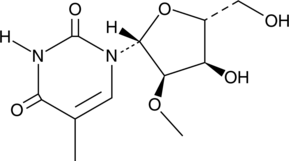
-
GC66713
2'-O-MOE-5-Me-C(Bz)
2'-O-MOE-5-Me-C (Bz) ist ein Nukleotid für die stereoselektive Synthese von Nukleosid-Alkylphosphonaten.
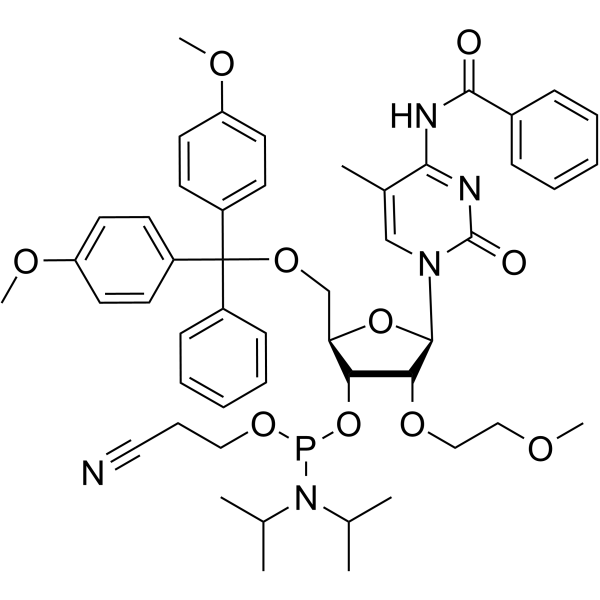
-
GC66657
2'-O-MOE-5-Me-rC
2'-O-MOE-5-Me-rC ist ein Wirkstoff. 2'-O-MOE-5-Me-rC kann für die Oligonukleotidsynthese verwendet werden.
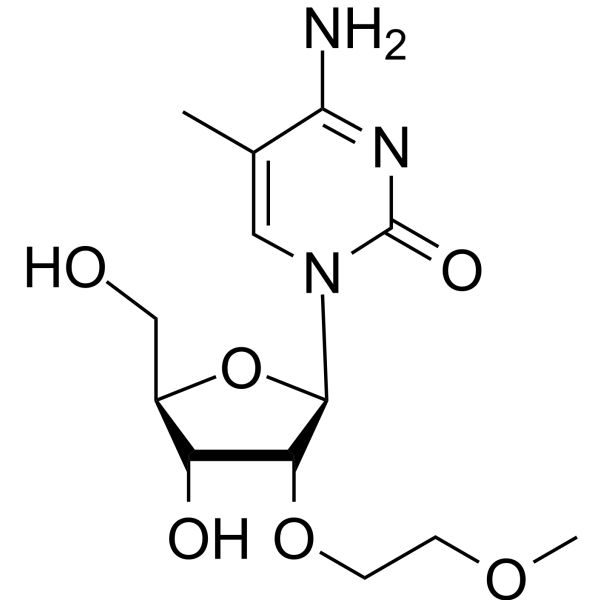
-
GC66091
2'-O-MOE-5-Me-rU
2'-O-MOE-5-Me-rU ist ein Wirkstoff. 2'-O-MOE-5-Me-rU kann zur Oligonukleotidsynthese verwendet werden.
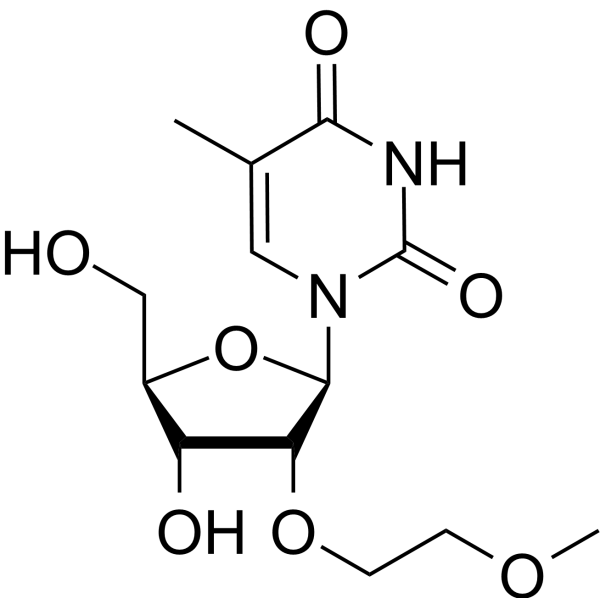
-
GC66656
2'-O-MOE-rC
2'-O-MOE-rC ist ein 2&7#39;-O-MOE-modifiziertes Nucleosid. 2'-O-MOE-rC kann zur Synthese von DNA verwendet werden.
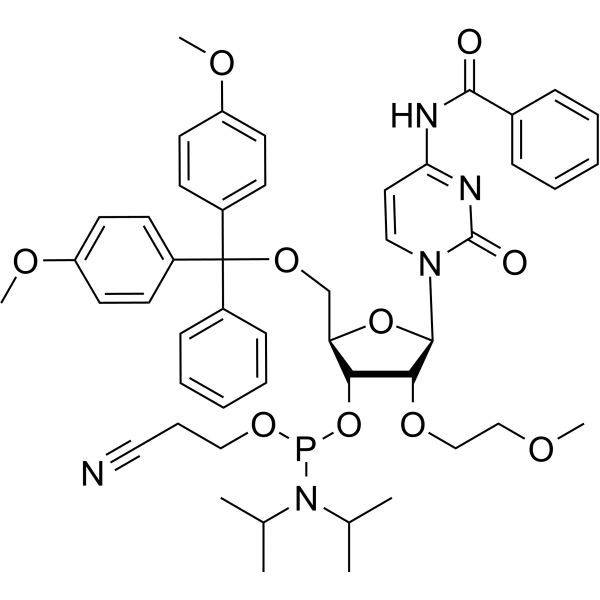
-
GC66652
2'-O-MOE-U
2'-O-MOE-U ist ein Phosphoramidit, das zur Oligonukleotidsynthese verwendet werden kann.

-
GC62530
2’-O-Me-C(Bz) Phosphoramidite
&2rsquo;-O-Me-C(Bz) Phosphoramidit ist ein modifiziertes Phosphoramidit-Monomer, das für die Oligonukleotidsynthese verwendet werden kann.
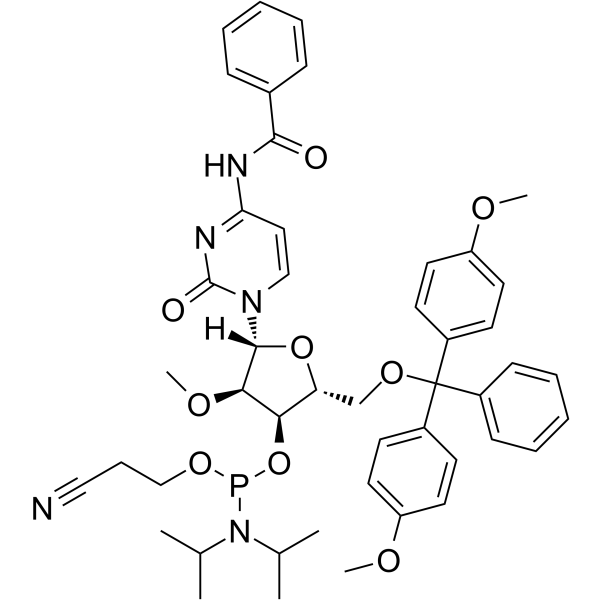
-
GC62529
2’-OMe-A(Bz) Phosphoramidite
&2rsquo;-OMe-A(Bz) Phosphoramidit ist ein modifiziertes Phosphoramidit-Monomer, das für die Oligonukleotidsynthese verwendet werden kann.
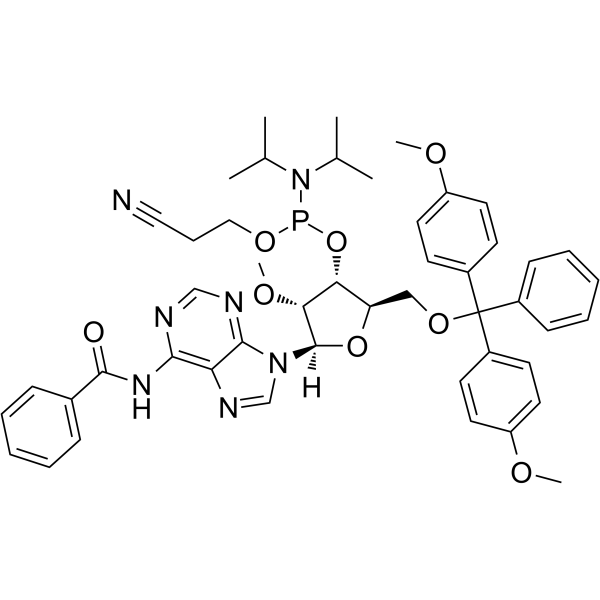
-
GC62531
2’-OMe-G(ibu) Phosphoramidite
&2rsquo;-OMe-G(ibu) Phosphoramidit ist ein modifiziertes Phosphoramidit-Monomer, das für die Oligonukleotidsynthese verwendet werden kann.
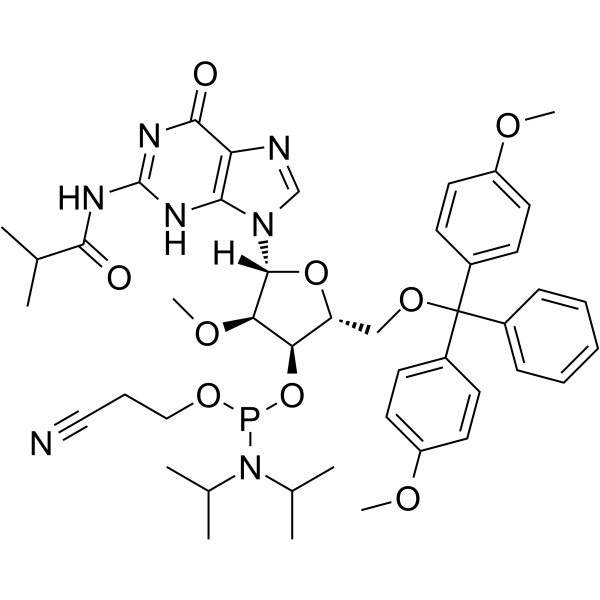
-
GC42061
2',3'-Dideoxyadenosine 5'-triphosphate
2',3'-Dideoxyadenosine 5'-triphosphate (2',3'-Dideoxyadenosine 5'-triphosphate), an active metabolite of 2',3'-dideoxyinosine, is a chain-elongating inhibitor of DNA polymerase.
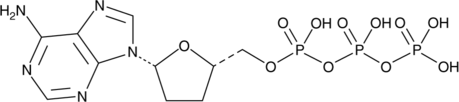
-
GC46519
2',3'-O-Isopropylideneguanosine
A building block
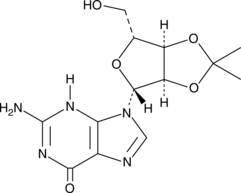
-
GC46540
2'-Deoxyadenosine-5'-triphosphate (sodium salt hydrate)
A purine nucleotide
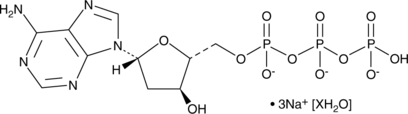
-
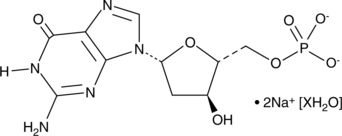
GC48440
2'-Deoxycytidine-5'-triphosphate (sodium salt)
2'-Desoxycytidin-5'-Triphosphat (Natriumsalz) (dCTP-Trinatriumsalz) ist ein Nukleosidtriphosphat, das fÜr die DNA-Synthese verwendet werden kann.
-
GC42151
2'-Deoxyguanosine 5'-monophosphate (sodium salt hydrate)
2'-Deoxyguanosine 5'-monophosphate (dGMP) is used as a substrate of guanylate kinases to generate dGDP, which in turn is phosphorylated to dGTP, a nucleotide precursor used in DNA synthesis.
-
GC42080
2'2'-cGAMP (sodium salt)
2'2'-cGAMP is a synthetic dinucleotide (CDN) that contains non-canonical 2'5'-phosphodiester bonds.
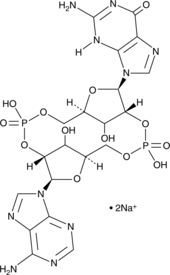
-
GC60459
2,4-D (2,4-Dichlorophenoxyacetic acid)
2,4-D (2,4-DichlorphenoxyessigsÄure) (2,4-D (2,4-DichlorphenoxyessigsÄure)-ichlorphenoxyessigsÄure) ist ein selektives systemisches Herbizid zur BekÄmpfung von breitblÄttrigen UnkrÄutern.
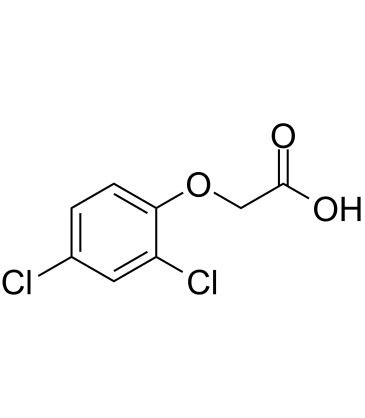
-
GC46524
2,6-Dichloropurine-9-β-D-riboside
A building block
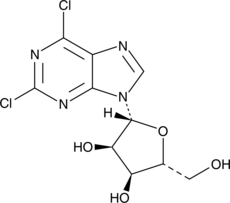
-
GC33496
2-(Methylamino)-1H-purin-6(7H)-one (N2-methylguanine)
2-(Methylamino)-1H-purin-6(7H)-on (N2-Methylguanin) (N2-Methylguanin) ist ein modifiziertes Nukleosid.
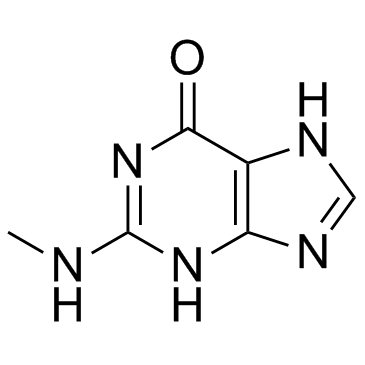
-
GC64983
2-Amino-2'-deoxyadenosine
2-Amino-&2#39;-Desoxyadenosin ist ein Desoxyribonukleosid, das für die Oligonukleotidsynthese verwendet wird.
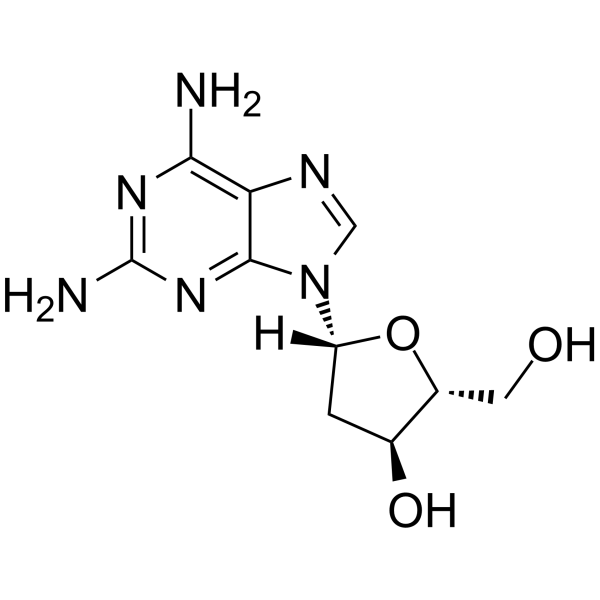
-
GC39527
2-O-Methylcytosine
2-O-Methylcytosin, ein O-alkyliertes Analogon eines DNA-Addukts, ist die beschÄdigte Nukleobase.
-
GC49348
2-Thiocytidine
A modified nucleobase
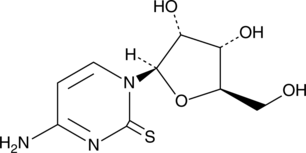
-
GC42197
2-Thiouridine
2-Thiouridine (s2U) is a modified nucleobase found in tRNAs that is known to stabilize U:A pairs and modestly destabilize U:G wobble pairs.
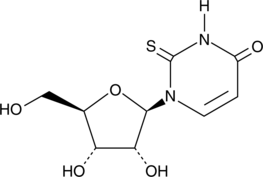
-
GC40618
3',4',7-Trihydroxyisoflavone
3',4',7-Trihydroxyisoflavon, ein Hauptmetabolit von Daidzein, ist ein ATP-kompetitiver Inhibitor von Cot (Tpl2/MAP3K8) und MKK4. 3',4',7-Trihydroxyisoflavon hat krebshemmende, antiangiogene, chemoprotektive und RadikalfÄnger-AktivitÄten.
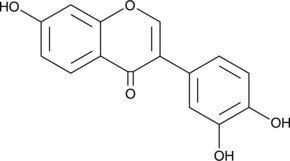
-
GC60023
3'-Deoxyuridine-5'-triphosphate
3'-Desoxyuridin-5'-Triphosphat (3'-dUTP) ist ein Nukleotidanalogon, das die DNA-abhÄngigen RNA-Polymerasen I und II hemmt.
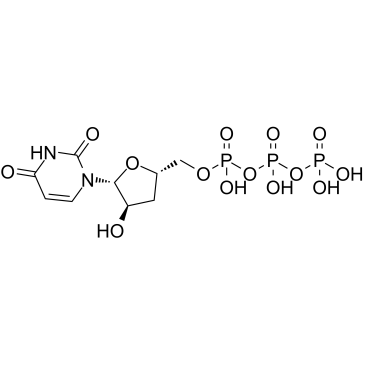
-
GC61862
3'-Deoxyuridine-5'-triphosphate trisodium
3'-Desoxyuridin-5'-Triphosphat-Trinatrium (3'-dUTP-Trinatrium) ist ein Nukleotidanalogon, das die DNA-abhÄngigen RNA-Polymerasen I und II hemmt.
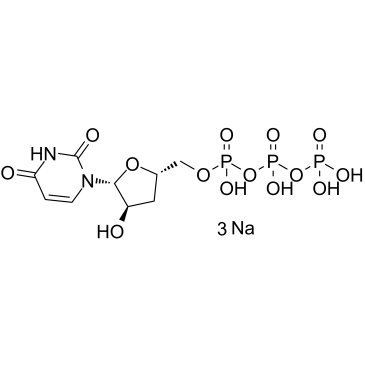
-
GC45332
3'-Dephosphocoenzyme A
An intermediate in the biosynthesis of CoA
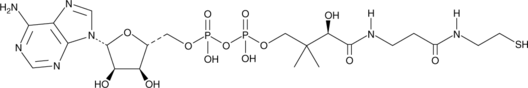
-
GC34452
3,7,4'-Trihydroxyflavone
3,7,4'-Trihydroxyflavon, isoliert aus Rhus javanica Var.
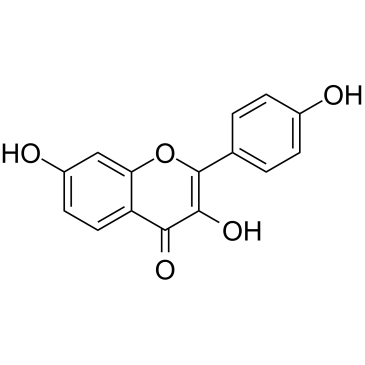
-
GC13510
3-AP
3-AP (PAN-811) ist ein potenter Inhibitor der M2-Untereinheit der Ribonukleotidreduktase (RR) und ein potenter Radiosensibilisator.
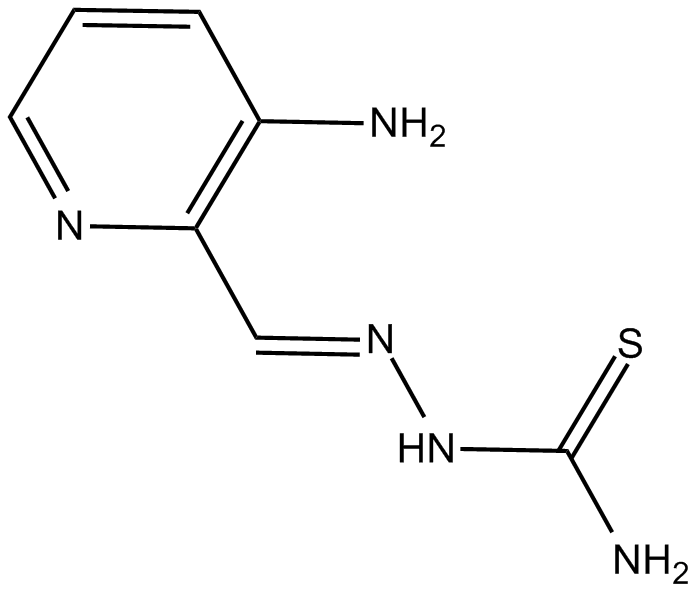
-
GC48457
3-keto Fusidic Acid
An active metabolite of fusidic acid
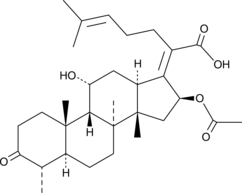
-
GC52391
306-O12B-3
An ionizable cationic lipidoid
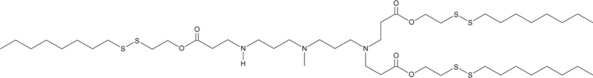
-
GC18853
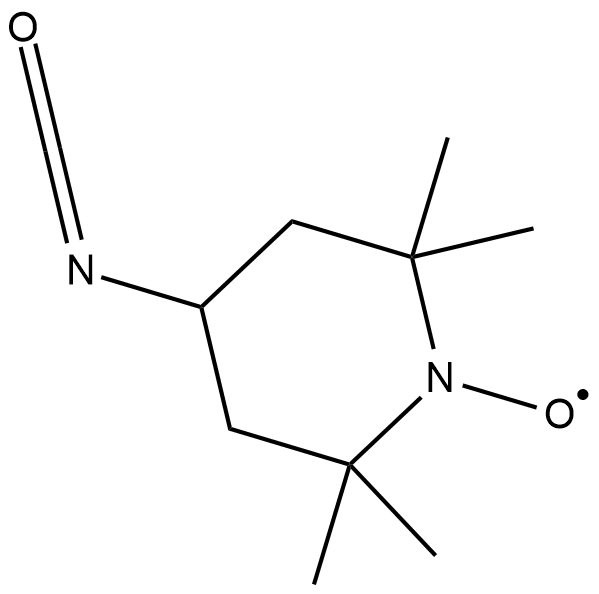
4-isocyanato TEMPO
4-isocyanato TEMPO is a spin labeling reagent used to label the 2'-position in RNA.
-
GC61683
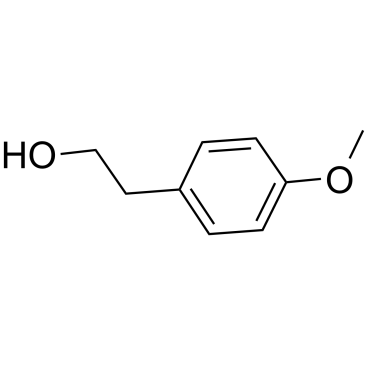
4-Methoxyphenethyl alcohol
4-Methoxyphenethylalkohol, ein aromatischer Alkohol, ist die Hauptkomponente des anisartigen Geruchs, der von A.
-
GC65520
5'-DMT-3'-TBDMS-ibu-rG
5'-DMT-3'-TBDMS-ibu-rG ist ein modifiziertes Nukleosid.

-
GC65402
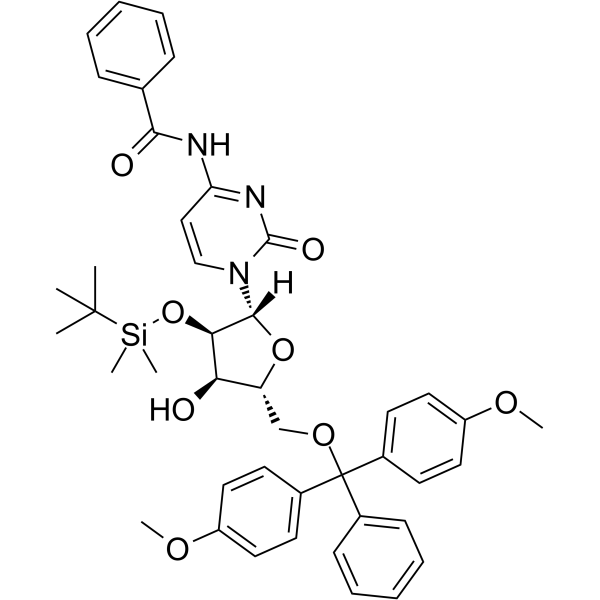
5'-O-DMT-2'-O-TBDMS-Bz-rC
5'-O-DMT-&2#39;-O-TBDMS-Bz-rC ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
-
GC65164
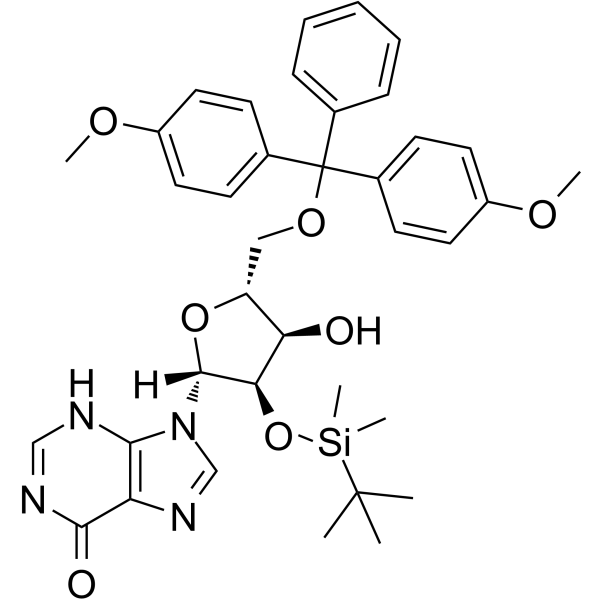
5'-O-DMT-2'-O-TBDMS-rI
5'-O-DMT-&2#39;-O-TBDMS-rI ist ein modifiziertes Nukleosid.
-
GC65396
5'-O-DMT-ibu-dC
5'-O-DMT-ibu-dC kann bei der Synthese von Oligodesoxyribonukleotiden verwendet werden.

-
GC64982
5'-O-DMT-N2-DMF-dG
5'-O-DMT-2'-O-TBDMS-rI ist ein modifiziertes Nucleosid.
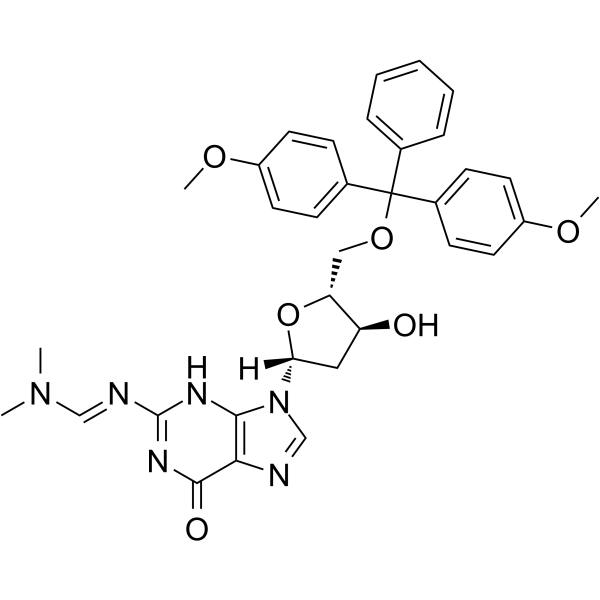
-
GC65482
5'-O-DMT-N6-ibu-dA
5'-O-DMT-N6-ibu-dA kann bei der Synthese von Oligodesoxyribonukleotiden verwendet werden.
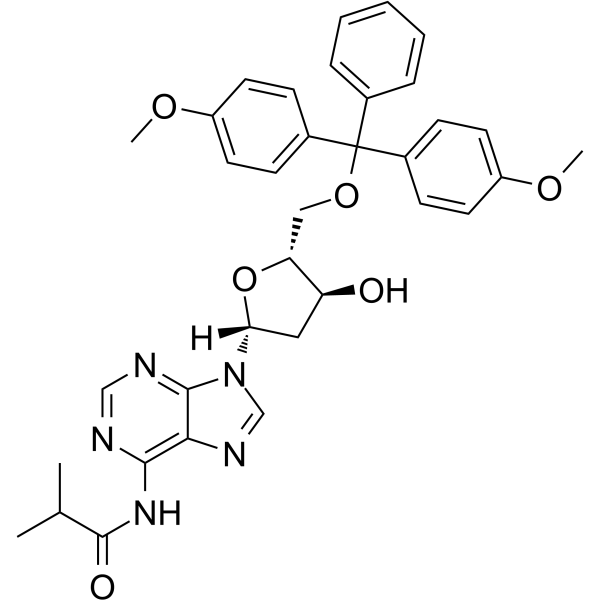
-
GC65562
5'-O-DMT-N6-Me-2'-dA
5'-O-DMT-N6-Me-&2#39;-dA ist ein Nukleosid mit Schutz- und Modifikationswirkung.
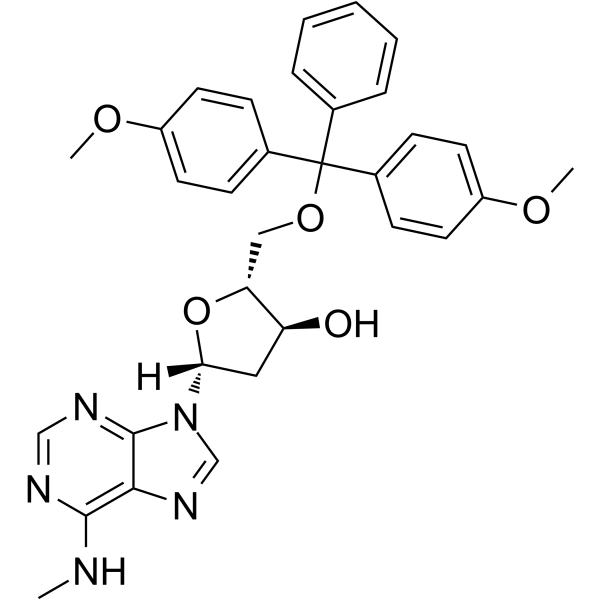
-
GC65401
5'-O-TBDMS-dU
5'-O-TBDMS-dU kann bei der Synthese von Oligoribonukleotiden verwendet werden.
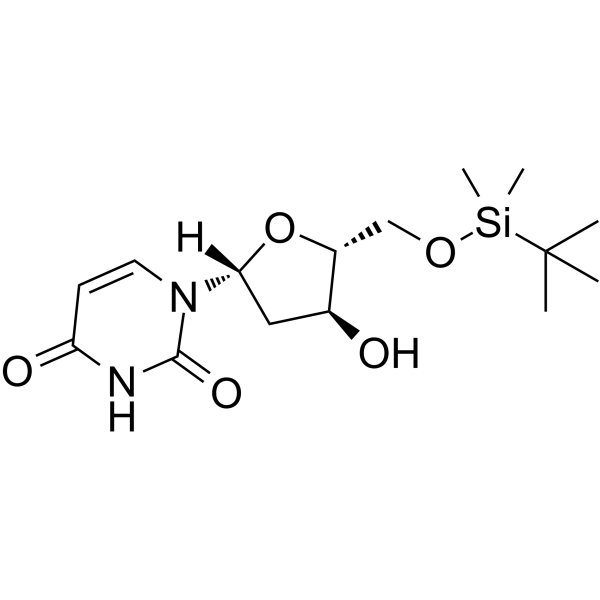
-
GC62157
5’-O-DMT-2’-O-TBDMS-Ac-rC
5’-O-DMT-&2rsquo;-O-TBDMS-Ac-rC ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
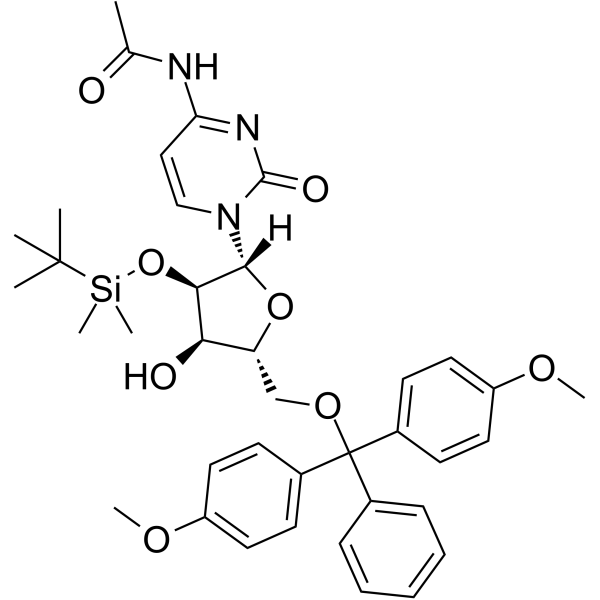
-
GC62149
5’-O-DMT-2’-TBDMS-Uridine
5’-O-DMT-&2rsquo;-TBDMS-Uridin ist ein Desoxyribonukleosid, das für die Oligonukleotidsynthese verwendet wird.
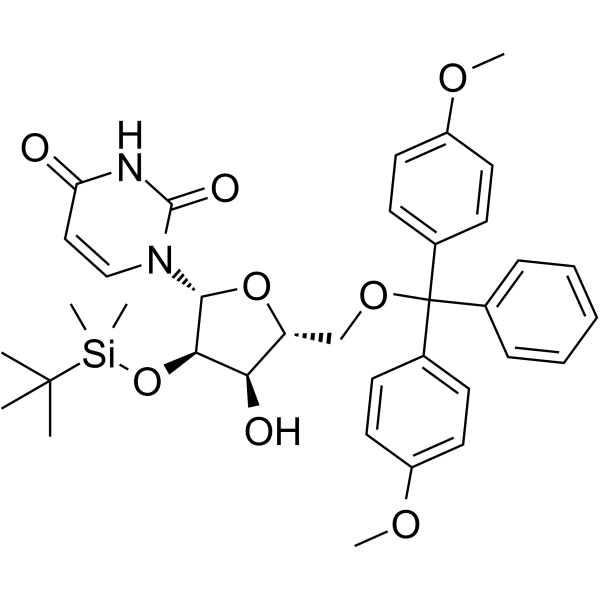
-
GC62532
5’-O-DMT-3’-O-TBDMS-Ac-rC
5’-O-DMT-3’-O-TBDMS-Ac-rC ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
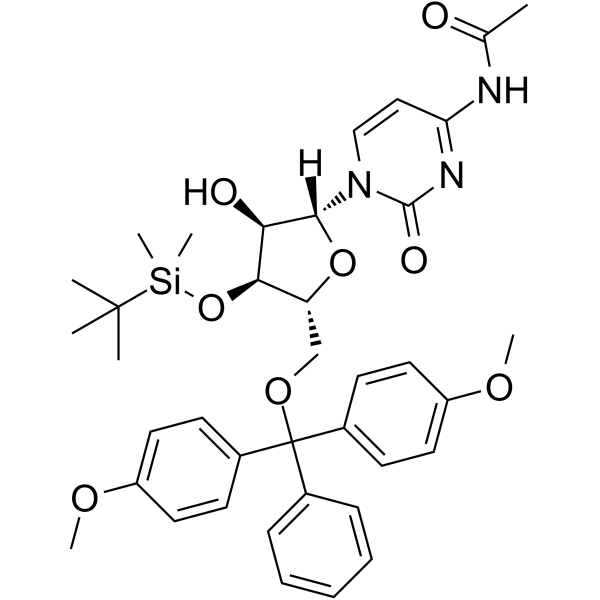
-
GC62573
5’-O-DMT-Bz-rC
5'-O-DMT-Bz-Rc ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
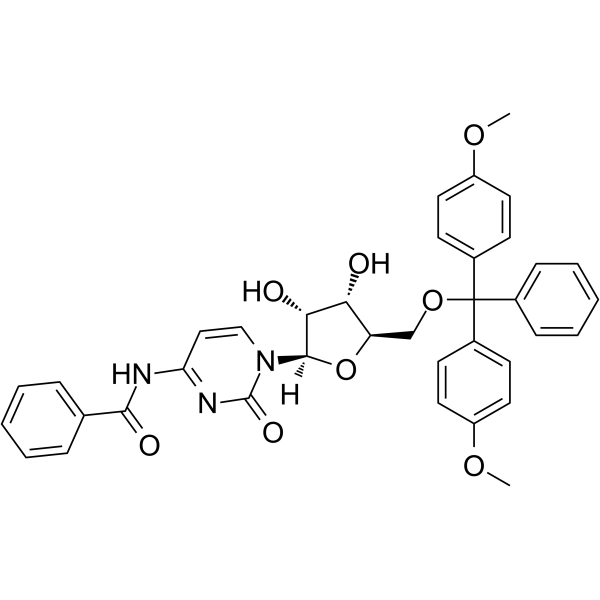
-
GC62574
5’-O-DMT-N4-Ac-2’-F-dC
5’-O-DMT-N4-Ac-&2rsquo;-F-dC ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
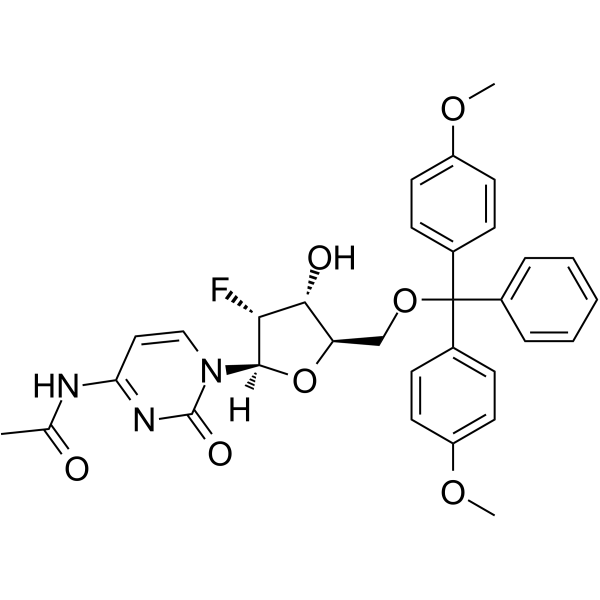
-
GC62575
5’-O-DMT-N4-Bz-2’-F-dC
5’-O-DMT-N4-Bz-&2rsquo;-F-dC ist ein Nukleosid mit Schutz- und Modifikationswirkung.
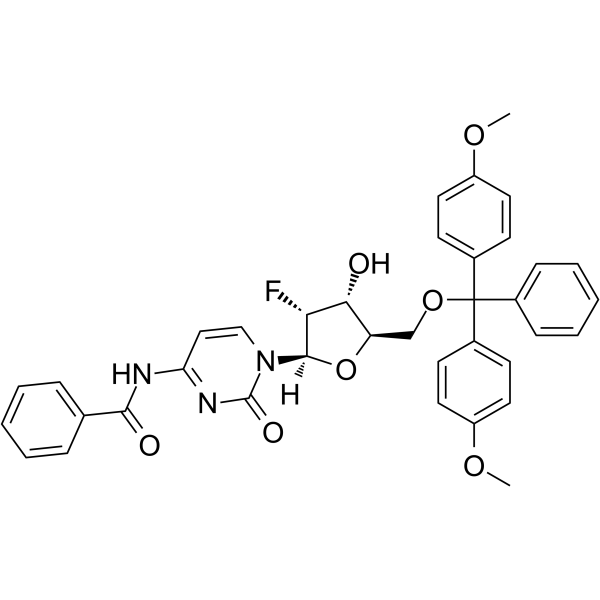
-
GC62576
5’-O-DMT-N4-Bz-5-Me-dC
5’-O-DMT-N4-Bz-5-Me-dC ist ein modifiziertes Nukleosid.
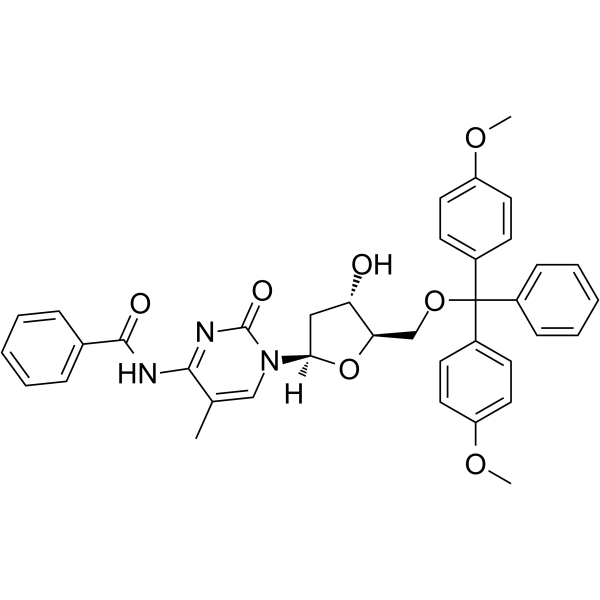
-
GC62812
5’-O-DMT-PAC-dA
5’-O-DMT-PAC-dA kann bei der Synthese von Oligoribonukleotiden verwendet werden.
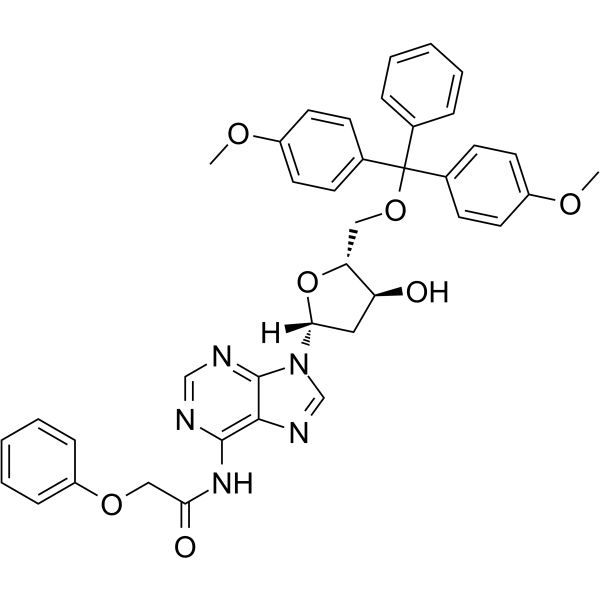
-
GC62813
5’-O-DMT-rI
5'-O-DMT-Ri kann bei der Synthese von Oligoribonukleotiden verwendet werden.
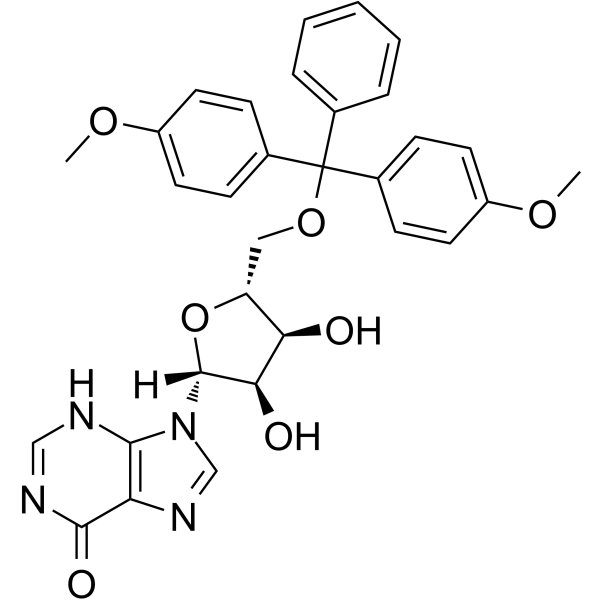
-
GC62577
5’-O-DMT-rU
5’-O-DMT-rU ist ein modifiziertes Nukleosid und kann zur Synthese von RNA verwendet werden.
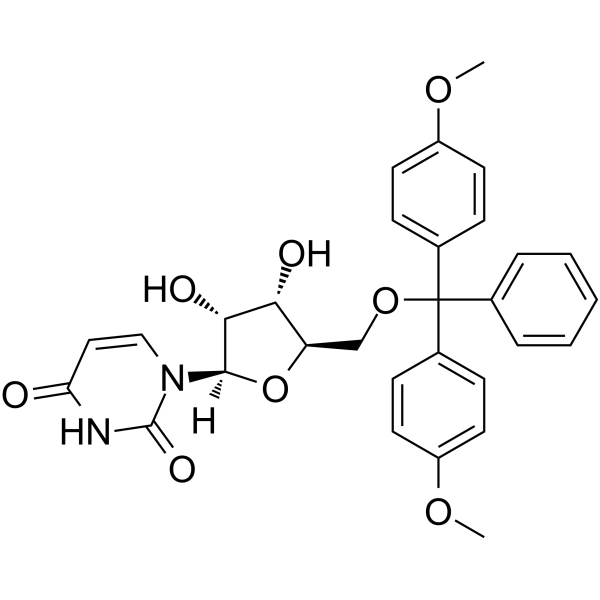
-
GC62578
5’-O-TBDMS-Bz-dA
5’-O-TBDMS-Bz-dA ist ein Nukleosid mit schützender und modifizierender Wirkung.
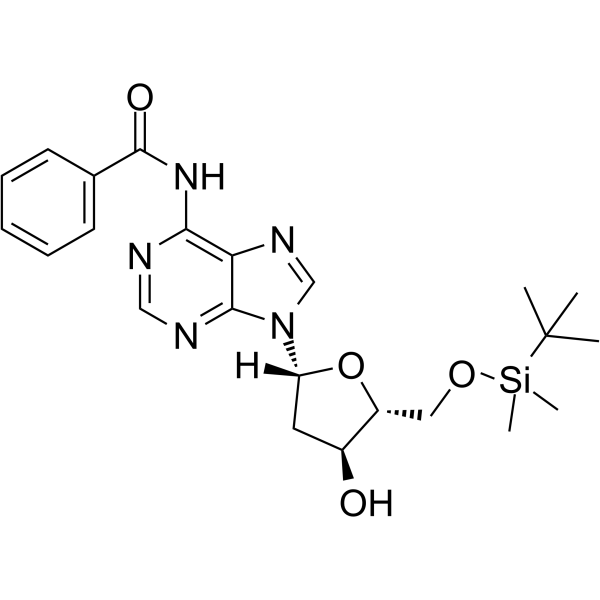
-
GC62579
5’-O-TBDMS-dA
5’-O-TBDMS-dA ist ein modifiziertes Nukleosid und kann zur Synthese von DNA oder RNA verwendet werden.
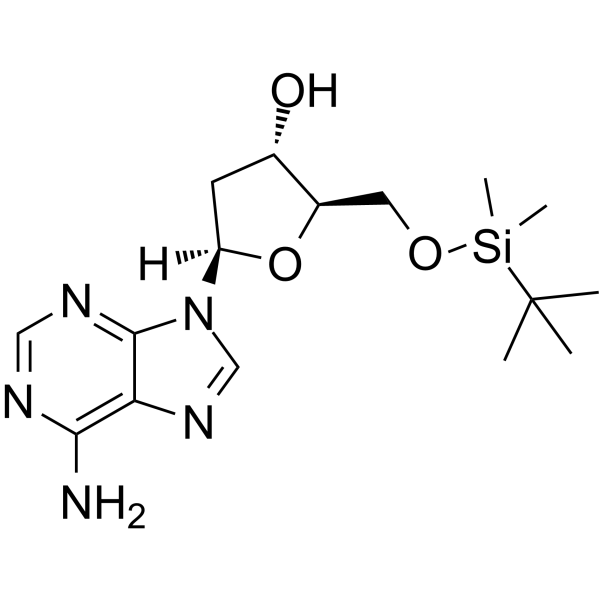
-
GC62580
5’-O-TBDMS-dG
5’-O-TBDMS-dG ist ein modifiziertes Nukleosid.

-
GC62581
5’-O-TBDMS-dT
5’-O-TBDMS-dT ist ein Nukleosid mit Schutz- und Modifikationswirkung.

-
GC40641
5'-Ethynyl-2'-deoxycytidine
5'-Ethynyl-2'-deoxycytidine (EdC) is a nucleoside analog that inhibits replication of the herpes simplex virus-1 (HSV-1) KOS strain (ID50 = 0.2 μg/mL).
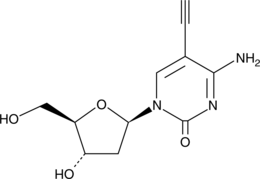
-
GC61726
5'-O-DMT-N4-Ac-dC
5'-O-DMT-N4-Ac-dC (N4-Acetyl-2'-desoxy-5'-O-DMT-cytidin, Verbindung 7), ein Desoxynucleosid, kann verwendet werden, um Dodecylphosphoramidit zu synthetisieren, das das Rohprodukt ist Material fÜr die dod‐DNA‐Synthese (amphiphile DNA, die eine interne hydrophobe Region enthÄlt, die aus Dodecyl‐Phosphotriester‐Bindungen besteht).
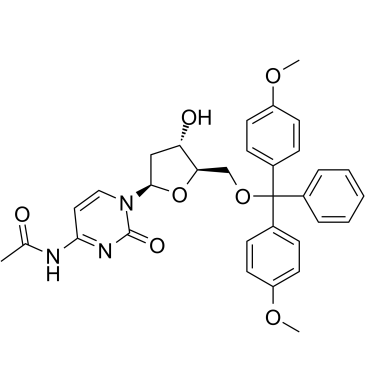
-
GC48662
5'-Thymidylic Acid (sodium salt)
5'-ThymidylsÄure (Natriumsalz) ist ein kÖrpereigener Metabolit.
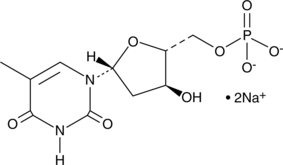
-
GC46681
5-Bromouridine
A brominated uridine analog
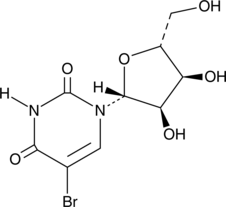
-
GC49245
5-Ethynyluridine
5-Ethinyluridin (5-EU) ist ein potentes zellgÄngiges Nukleosid, das zur Markierung neu synthetisierter RNA verwendet werden kann.
-
GC62389
5-Iminodaunorubicin
5-Iminodaunorubicin ist ein Chinon-modifiziertes Anthracyclin, das seine AntitumoraktivitÄt behÄlt. 5-Iminodaunorubicin erzeugt in Krebszellen proteinverdeckte DNA-StrangbrÜche.
-
GC66658
5-Me-dC(Ac) amidite
5-Me-dC(Ac)-Amidit wird zur Synthese von DNA oder RNA verwendet.
-
GC31187
5-Methoxyflavone
5-Methoxyflavon, das zur Familie der Flavonoide gehÖrt, ist ein DNA-Polymerase-Beta-Inhibitor und ein neuroprotektives Mittel gegen Beta-Amyloid-ToxizitÄt.
-
GC67671
5-Methylcytidine 5′-triphosphate trisodium
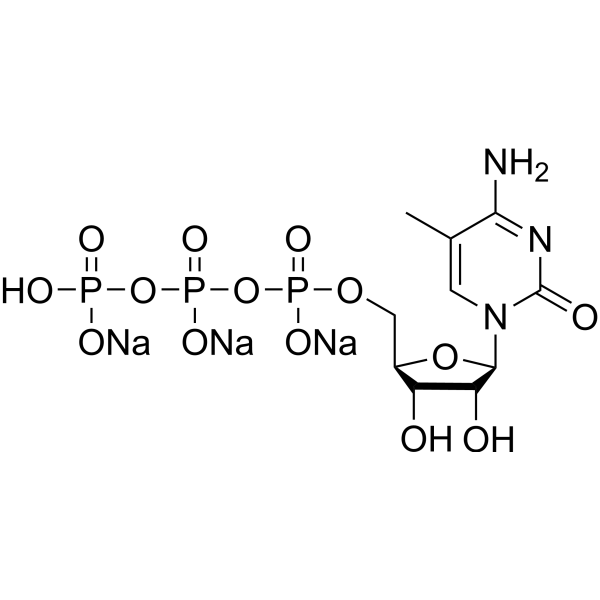
-
GC35166
5-Methylcytosine
5-Methylcytosin ist eine gut charakterisierte DNA-Modifikation und kommt auch Überwiegend in zahlreichen nichtkodierenden RNAs sowohl in Prokaryoten als auch in Eukaryoten vor.
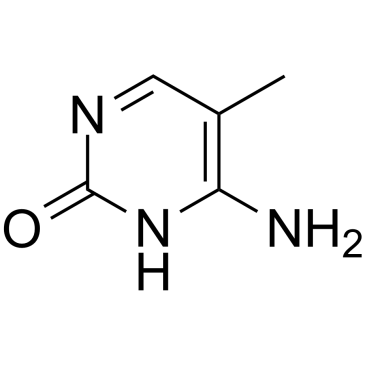
-
GC62547
5-O-TBDMS-N4-Benzoyl-2-deoxycytidine
5-O-TBDMS-N4-Benzoyl-2-desoxycytidin ist ein modifiziertes Nucleosid.
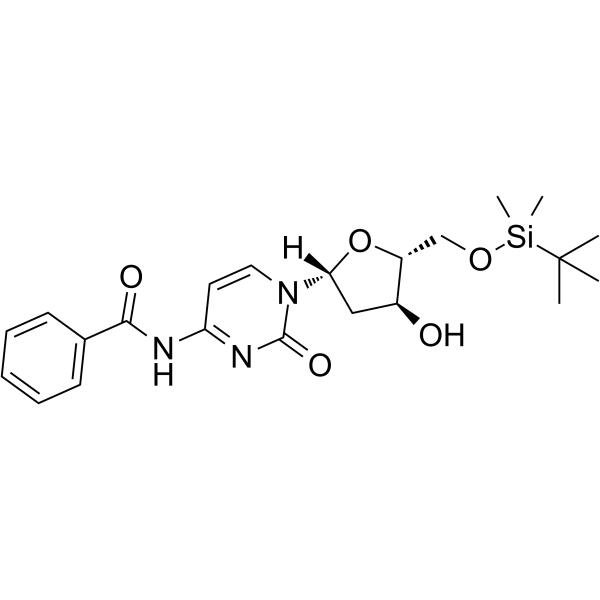
-
GC46713
5-Phospho-D-ribose 1-diphosphate (sodium salt hydrate)
An intermediate in several biochemical pathways
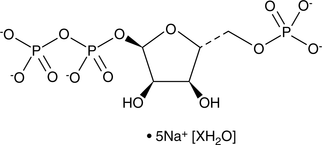
-
GC60031
6-Azathymine
6-Azathymin, ein 6-Stickstoff-Analogon von Thymin, ist ein potenter D-3-Aminoisobutyrat-Pyruvat-Aminotransferase-Inhibitor.
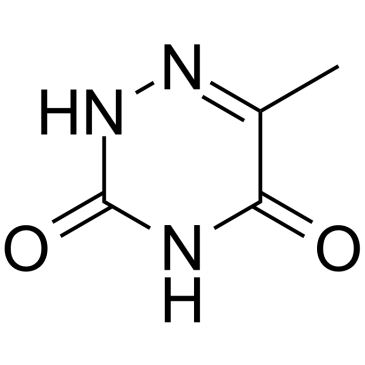
-
GC50249
6-Hydroxy-DL-DOPA
6-Hydroxy-DL-DOPA ist ein selektiver und wirksamer allosterischer Inhibitor der RAD52-ssDNA-BindungsdomÄne. 6-Hydroxy-DL-DOPA kann fÜr die Krebsforschung eingesetzt werden.
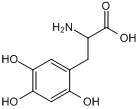
-
GC12193
6-Thio-dG
6-Thio-dG ist ein Nukleosid-Analogon, das durch Telomerase in de novo-synthetisierte Telomere eingebaut werden kann.
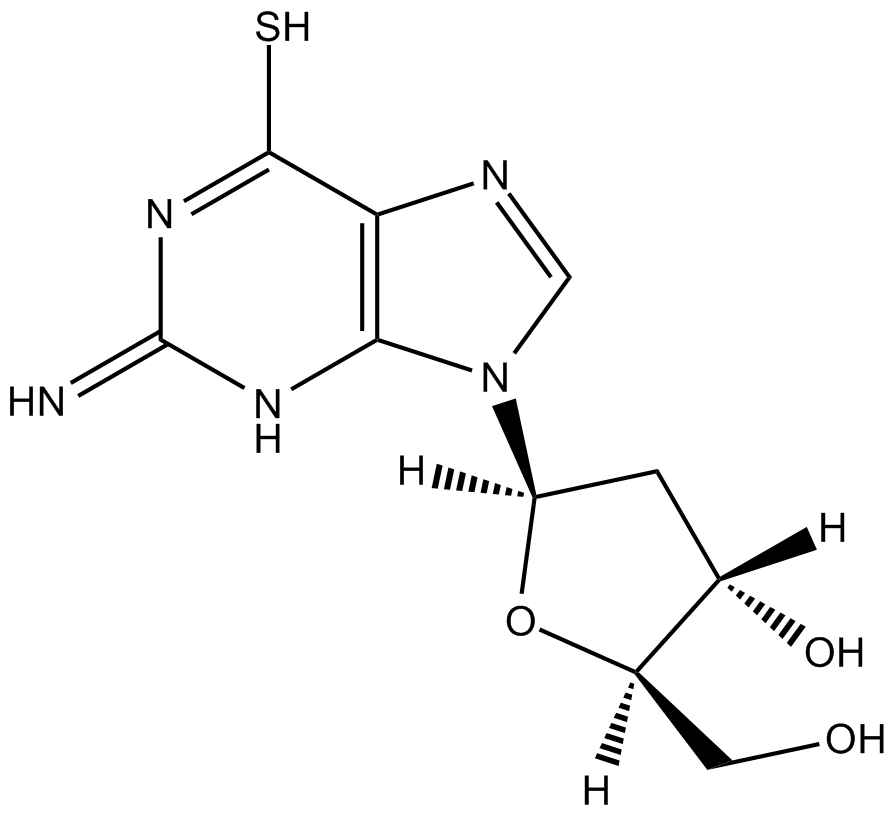
-
GC33577
7-Aminoactinomycin D (7-AAD)
7-Aminoactinomycin D (7-AAD) (7-AAD), ein fluoreszierender DNA-Farbstoff, ist ein potenter RNA-Polymerase-Inhibitor.
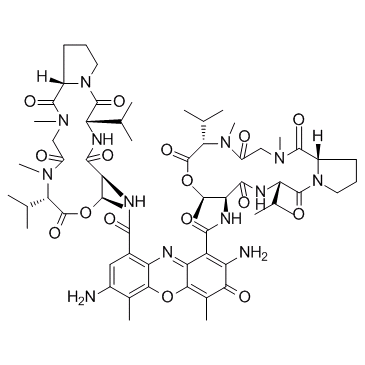
-
GC65408
7-Deaza-2'-deoxy-7-iodoadenosine
7-Deaza-&2#39;-Desoxy-7-iodoadenosin ist ein modifiziertes Oligonukleotid, das 7-Deazaadenin enthält.
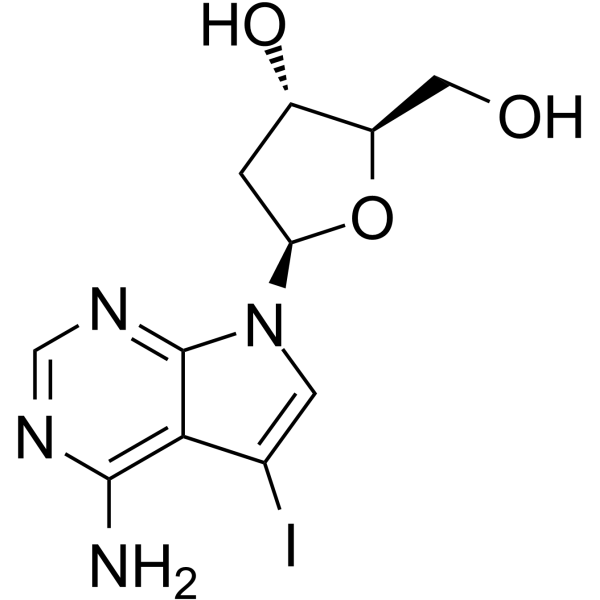
-
GC65675
7-Iodo-7-deaza-2'-deoxyguanosine
7-Iodo-7-deaza-2'-Desoxyguanosin (7-Deaza-7-Iodo-2'-Desoxyguanosin) ist ein Desoxyguanosin-Derivat, das in DNA-Synthese- und Sequenzierungsreaktionen verwendet werden kann.
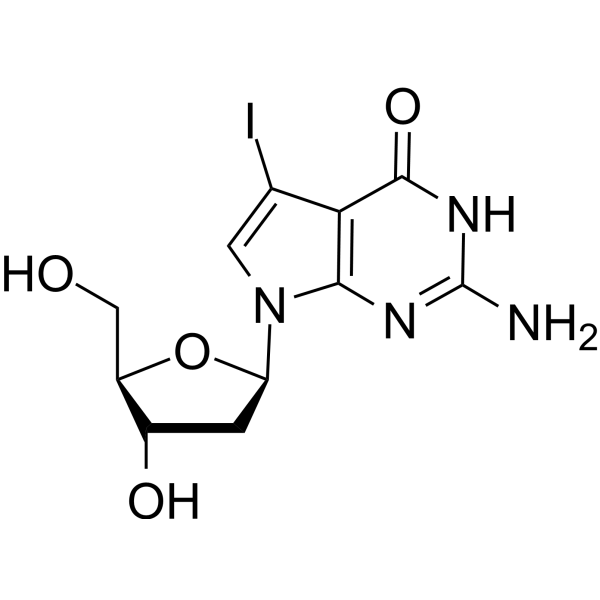
-
GC49561
7-Methylguanosine-d3
An internal standard for the quantification of 7-methylguanosine
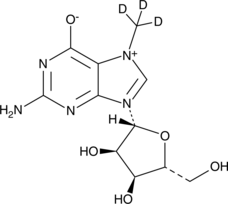
-
GC66058
7-TFA-ap-7-Deaza-dA
7-TFA-ap-7-Deaza-dA ist ein modifiziertes Nukleosid. 7-TFA-ap-7-Deaza-dA kann bei der Synthese von DesoxyribonukleinsÄure oder NukleinsÄure verwendet werden.
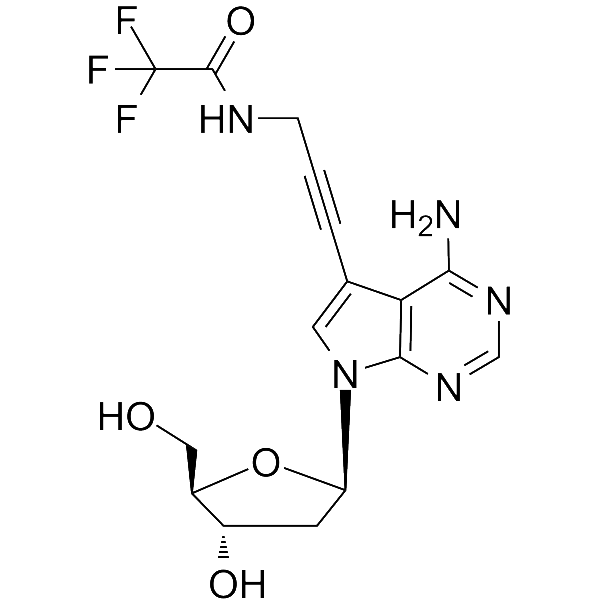
-
GC65525
7-TFA-ap-7-Deaza-dG
5'-O-TBDMS-dG ist ein modifiziertes Nukleosid.
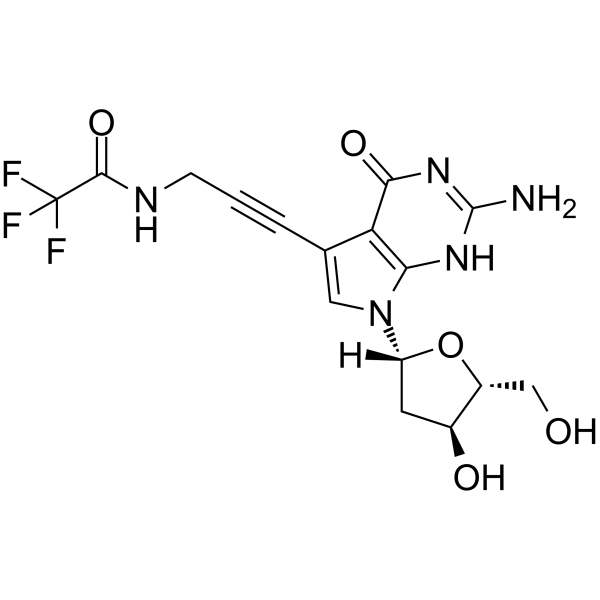
-
GC12777
8-Chloroadenosine
Nucleoside analog, inhibits RNA synthesis
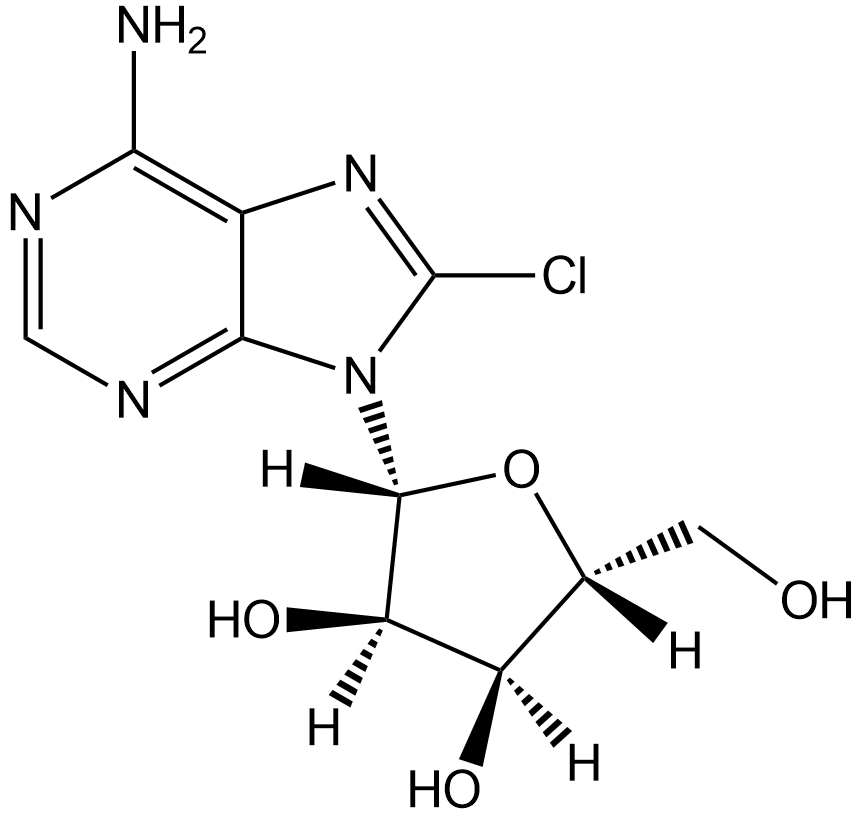
-
GC61763
Ac-rC Phosphoramidite
Ac-rC-Phosphoramidit wird fÜr die Oligoribonukleotid-Phosphorodithioat-Modifikation (PS2-RNA) verwendet.

-
GC34090
Acelarin (NUC-1031)
Acelarin (NUC-1031) (NUC-1031) ist eine ProTide-Transformation und -VerstÄrkung des weit verbreiteten Nukleosid-Analogons Gemcitabin.
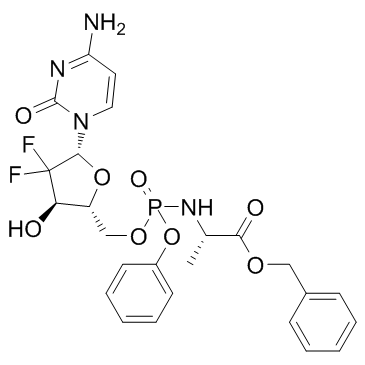
-
GC16866
Actinomycin D
Ein DNA-interagierender Transkriptionsblocker mit antikarzinogener Aktivität.

-
GC35247
ACX-362E
ACX-362E (ACX-362E) ist ein erster oral aktiver DNA-Polymerase IIIC (pol IIIC)-Inhibitor seiner Klasse mit einem Ki von 0,325 μM fÜr die DNA pol IIIC von C.
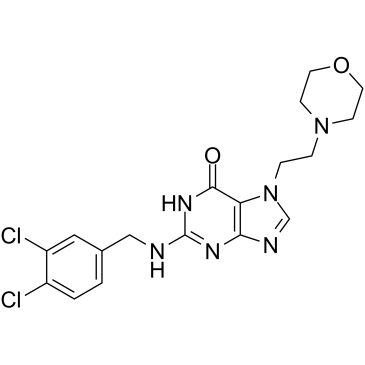
-
GC17186
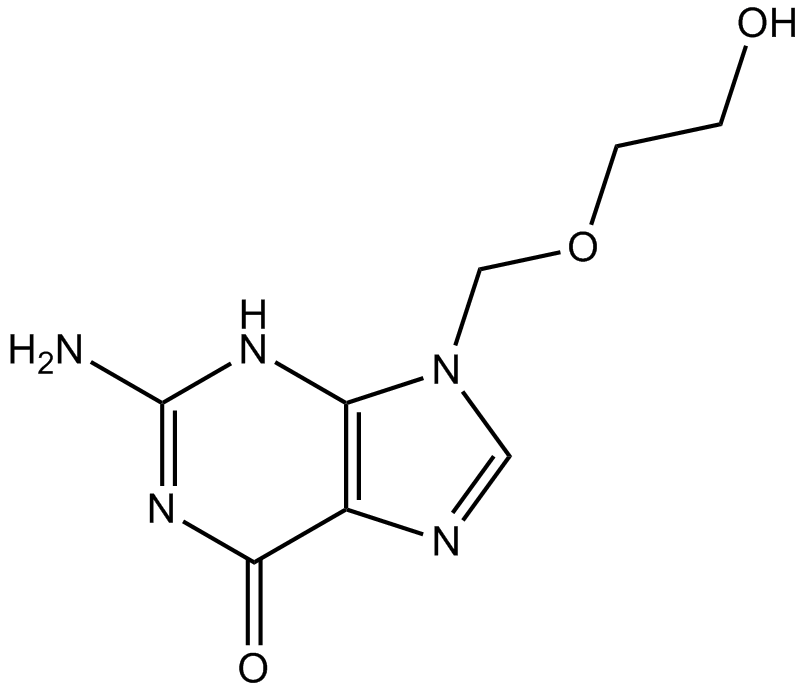
Acyclovir
Aciclovir (Aciclovir) ist ein starkes, oral aktives antivirales Mittel.
-
GC13432
Adenine
Adenin (6-Aminopurin), ein Purin, ist eine der vier Nukleobasen in der NukleinsÄure der DNA. Adenin fungiert als chemischer Bestandteil von DNA und RNA. Adenin spielt auch eine wichtige Rolle in der Biochemie, die an der Zellatmung, der Form von ATP und den Cofaktoren (NAD und FAD) und der Proteinsynthese beteiligt ist.

-
GC11825
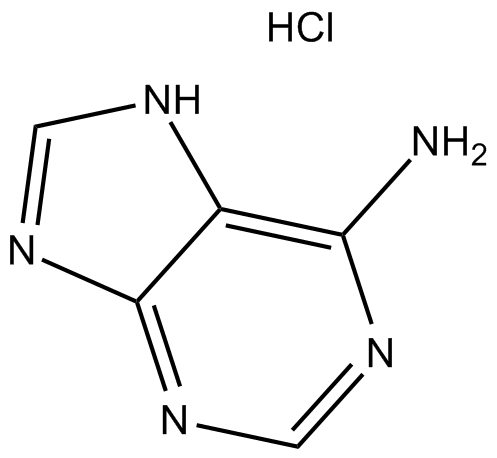
Adenine HCl
Adenin-HCl (6-Aminopurin-Hydrochlorid), ein Purin, ist eine der vier Nukleobasen in der NukleinsÄure der DNA. Adenin-HCl wirkt als chemischer Bestandteil von DNA und RNA. Adenin-HCl spielt auch eine wichtige Rolle in der Biochemie, die an der Zellatmung, der Form von ATP und den Cofaktoren (NAD und FAD) und der Proteinsynthese beteiligt ist.
-
GC17278
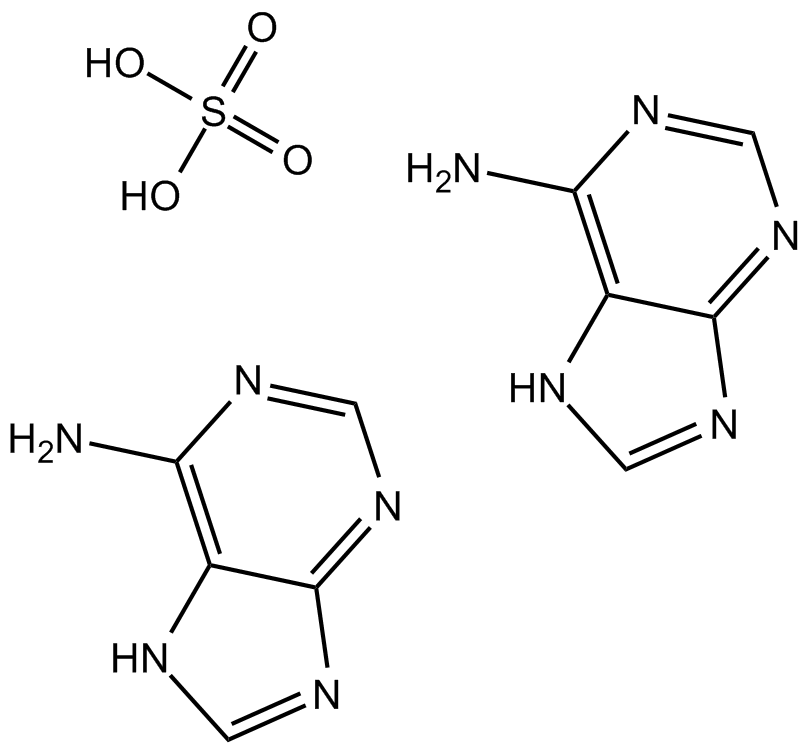
Adenine sulfate
Adeninsulfat (6-Aminopurin-Hemisulfat), ein Purin, ist eine der vier Nukleobasen in der NukleinsÄure der DNA. Adeninsulfat wirkt als chemischer Bestandteil von DNA und RNA. Adeninsulfat spielt auch eine wichtige Rolle in der Biochemie, die an der Zellatmung, der Form von ATP und den Cofaktoren (NAD und FAD) und der Proteinsynthese beteiligt ist.
-
GC67946
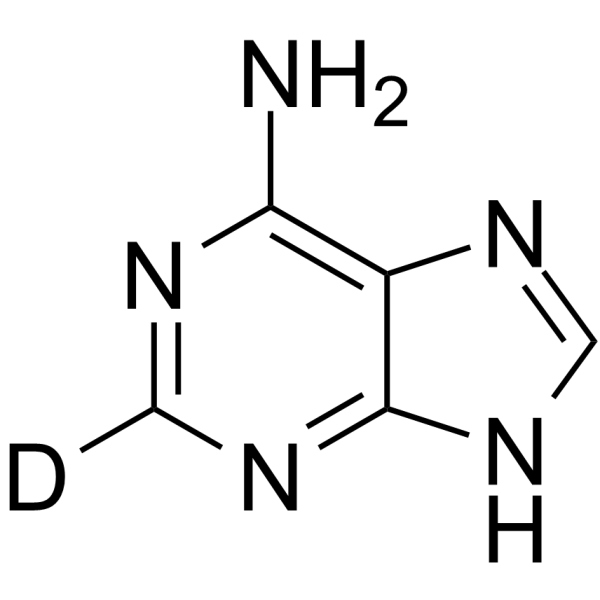
Adenine-d1
-
GC49004
Adenosine 3’-monophosphate (sodium salt hydrate)
A nucleotide
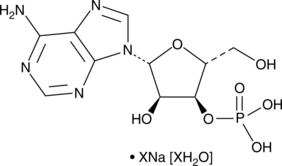
-
GC42732
Adenosine 3',5'-diphosphate (sodium salt)
Adenosin 3',5'-Diphosphat (Natriumsalz) ist ein Hydroxysteroid-Sulfotransferase-Inhibitor.