Histone Methyltransferase
Histone methyltransferases are a group of enzymes that catalyze the methylation of histone lysine and arginine by adding methyl groups to specific histone arginine or lysine residues. Histone methyltransferaes can be classified into 3 classes, including SET domain lysine methyltransferases, non-SET domain lysine methyltransferases and arginine methyltransferases (PRMTs), all of which use S-adenosylmethionine as a cosubstrate for the transfer of the methyl group. Aberrant histone methylation has been associated with a wide range of human cancers (such as hematological malignancies), which leads to the development of novel cancer chemotherapies targeting cancer-associated histone methyltransferases (more than 20 lysine methyltransferases and 9 arginine methyltransferases in humans).
Produkte für Histone Methyltransferase
- Bestell-Nr. Artikelname Informationen
-
GC45908
(R)-BAY-598
An inhibitor of SMYD2
-
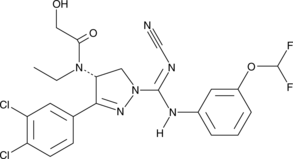
GC11340
(R)-PFI 2 hydrochloride
PFI-2 ((R)-(R)-PFI 2 Hydrochlorid) Hydrochlorid ist eine potente und selektive SET-DomÄne, die den Lysinmethyltransferase 7 (SETD7)-Inhibitor enthÄlt.
-
GC65045
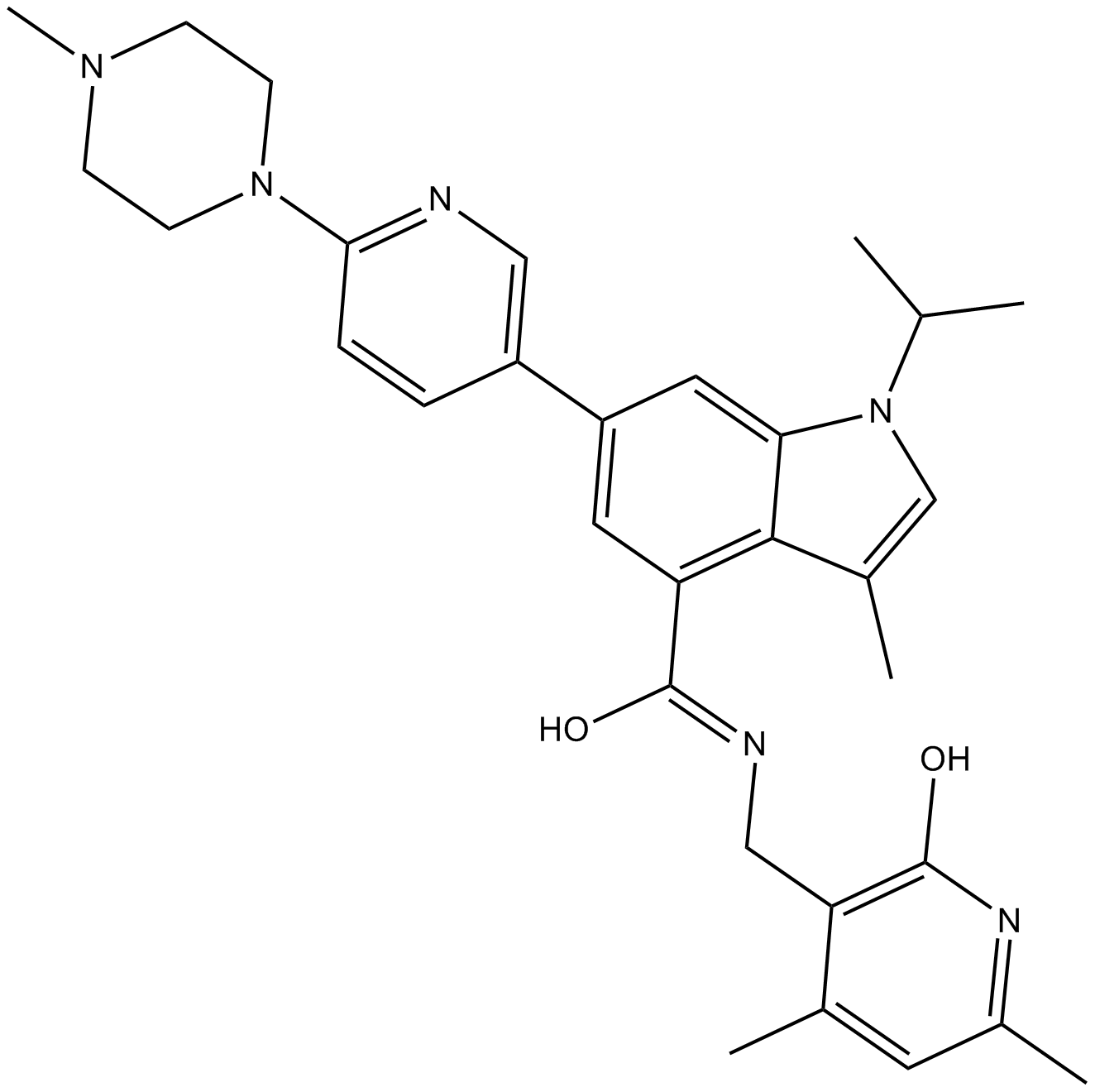
(S)-MRTX-1719
(S)-MRTX-1719 (Beispiel 16-7) ist das S-Enantiomer von MRTX-1719. (S)-MRTX-1719 ist ein PRMT5/MTA-Komplex-Inhibitor mit einem IC50-Wert von 7070 nM.
-
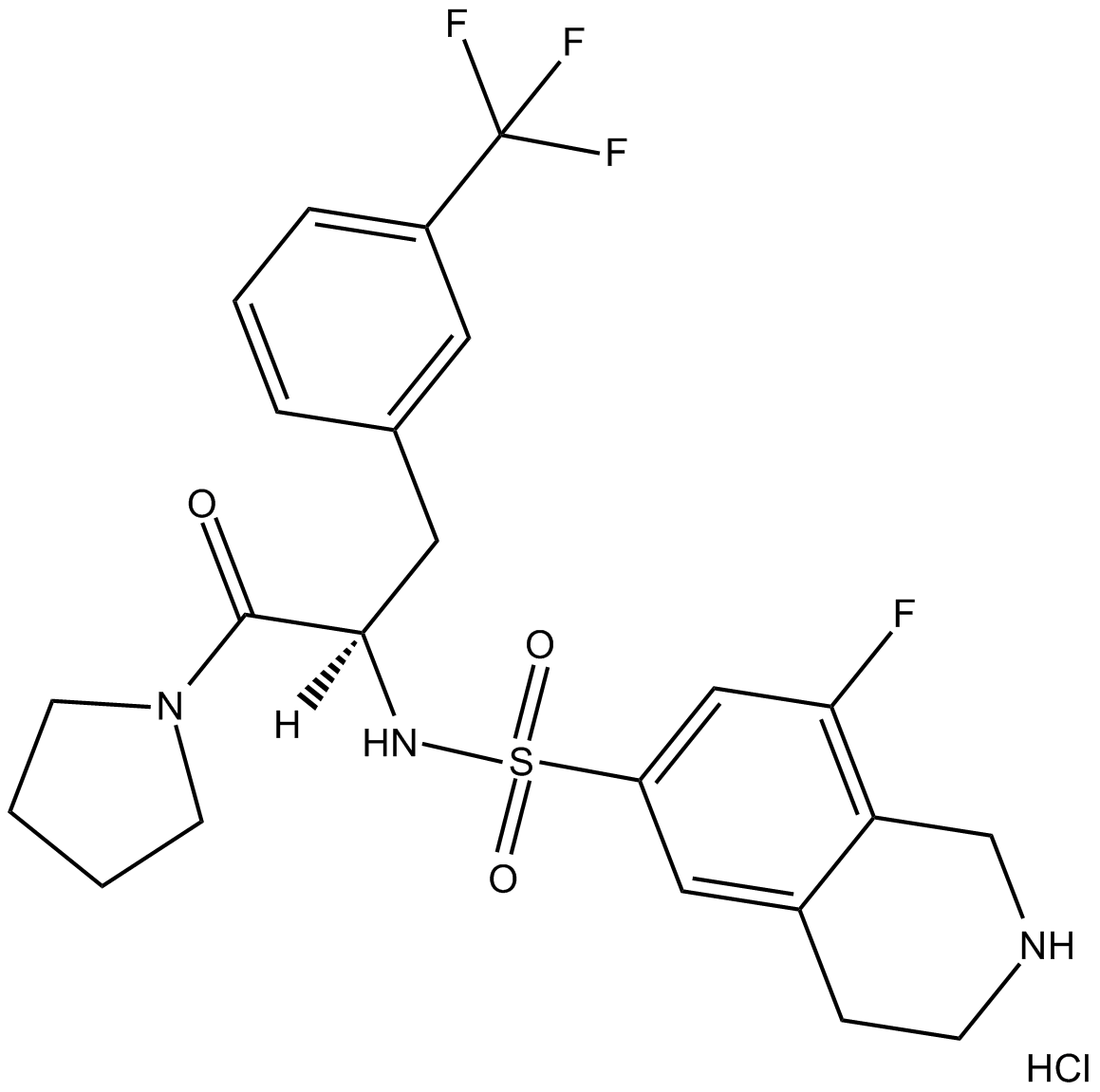
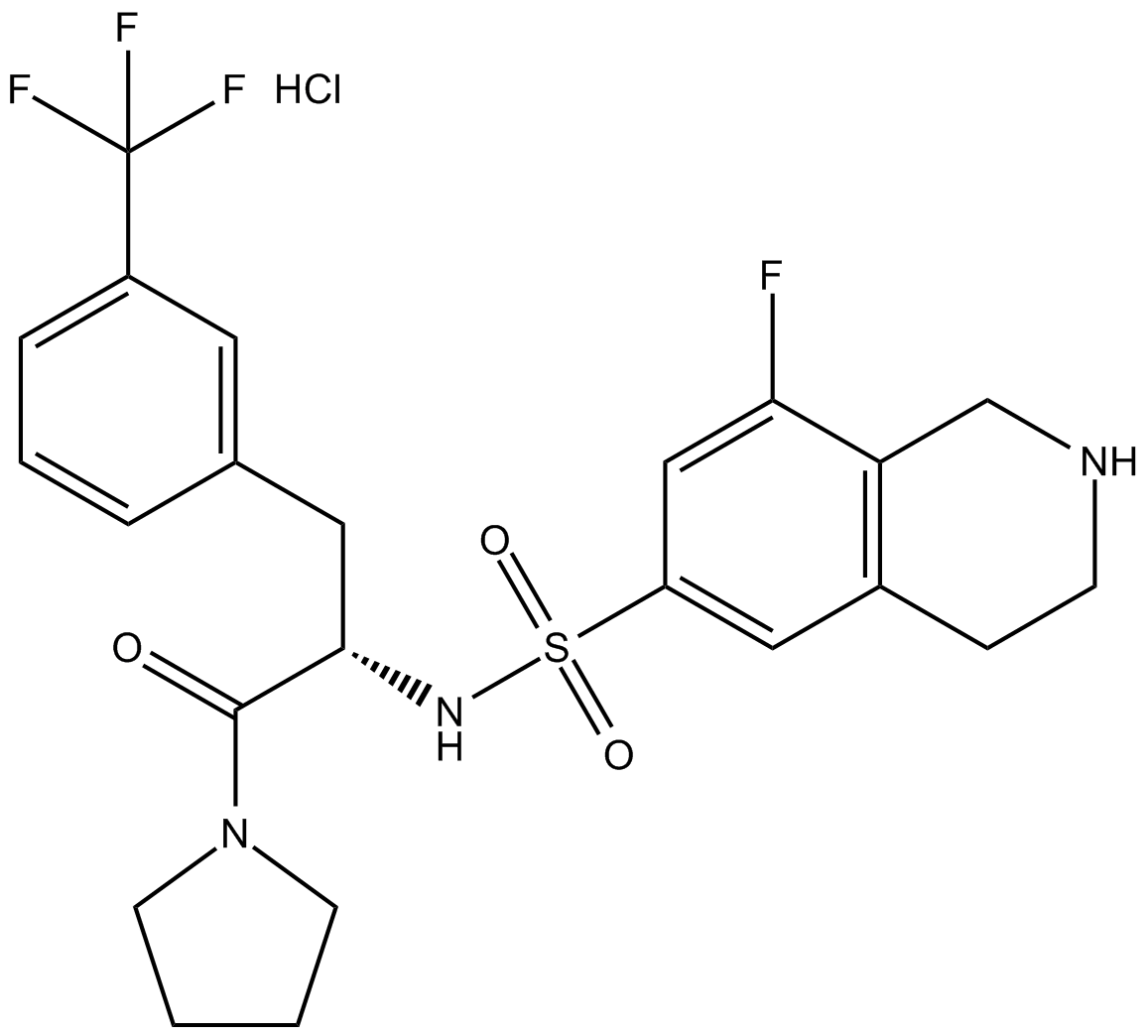
GC13634
(S)-PFI-2 (hydrochloride)
Negative control of (R)-PFI 2 hydrochloride
-
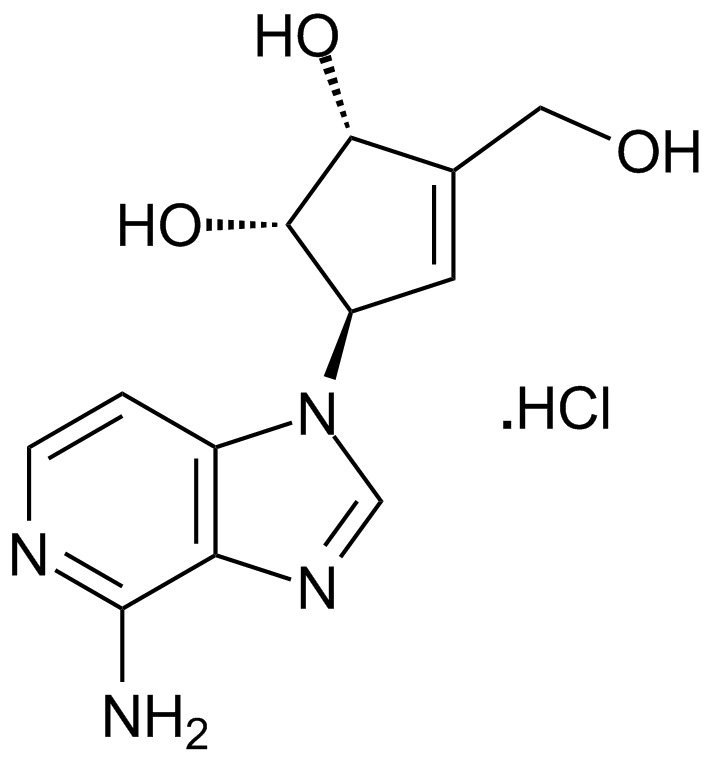
GC17907
3-Deazaneplanocin A (DZNep) hydrochloride
Ein Inhibitor der Lysin-Methyltransferase EZH2.
-
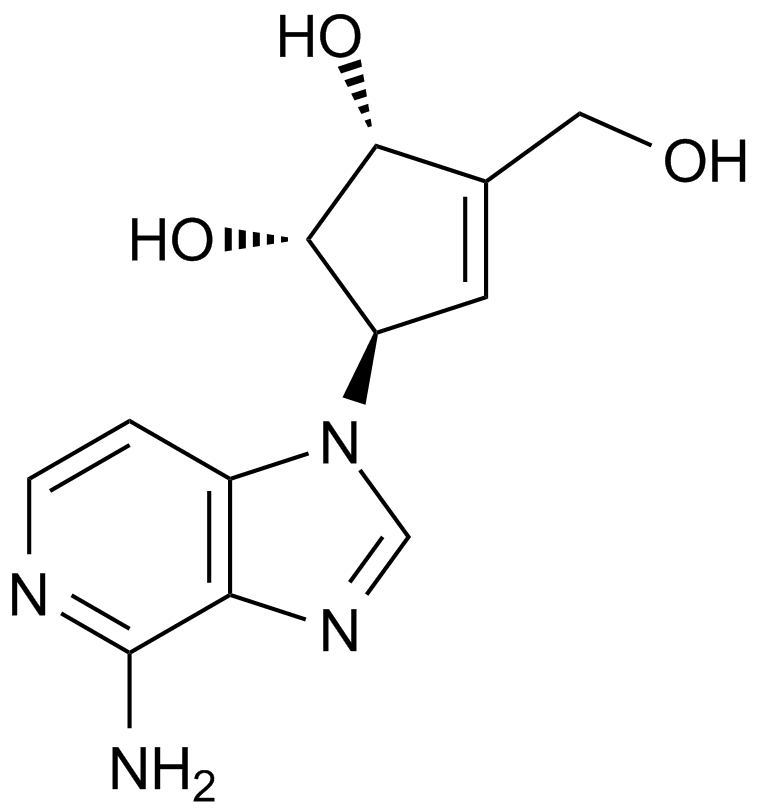
GC13145
3-Deazaneplanocin,DZNep
An inhibitor of lysine methyltransferase EZH2
-
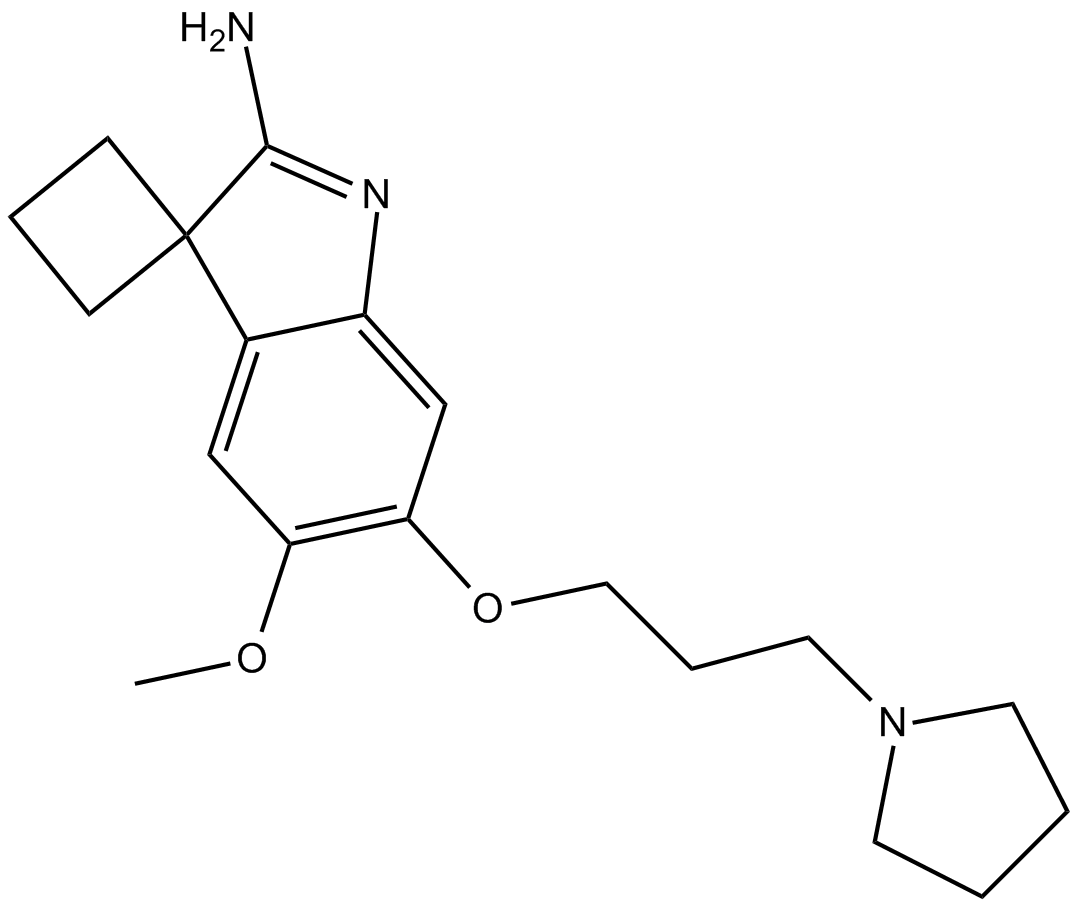
GC16015
A 366
A 366 ist ein potenter, hochselektiver, Peptid-kompetitiver Histon-Methyltransferase-G9a-Inhibitor mit IC50-Werten von 3,3 und 38 nM für G9a bzw. GLP (EHMT1). A 366 zeigt eine \u003e1000-fache Selektivität gegenüber 21 anderen Methyltransferasen. A 366 ist auch ein potenter, nanomolarer Inhibitor der Spindlin1-H3K4me3-Interaktion (IC50\u003d182,6 nM). A 366 zeigt eine hohe Affinität zum menschlichen Histamin-H3-Rezeptor (Ki \u003d 17 nM) und zeigt Subtyp-Selektivität unter Untergruppen der histaminergen und dopaminergen Rezeptorfamilien.
-
GC32861
A-196
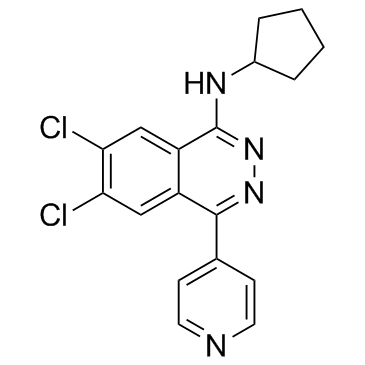
A-196 ist ein potenter und selektiver Inhibitor von SUV420H1 und SUV420H2 mit IC50-Werten von 25 nM bzw. 144 nM. A-196 hemmt SUV4-20 biochemisch substratkompetitiv. A-196 ist die erste chemische Sonde ihrer Klasse von SUV4-20 zur Untersuchung der Rolle von Histon-Methyltransferasen bei der genomischen IntegritÄt.
-
GC33187
A-395 (A395)
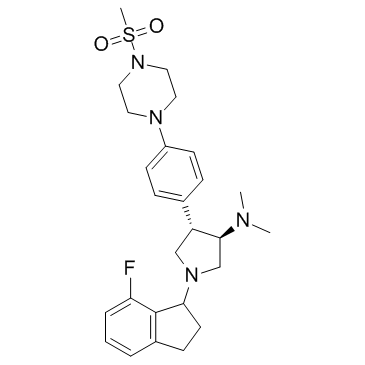
A-395 (A395) ist ein Antagonist der Protein-Protein-Interaktionen des polycomb repressiven Komplexes 2 (PRC2), der den trimeren PRC2-Komplex (EZH2-EED-SUZ12) mit einer IC50 von 18 nM stark hemmt.
-
GC30503
A-893
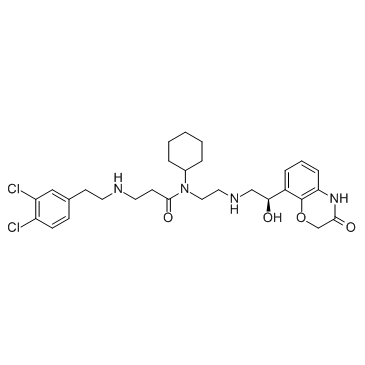
A-893 ist ein zellaktiver Inhibitor der Methyltransferase SMYD2 mit einem IC50 von 2,8 nM.
-
GC16509
Adox
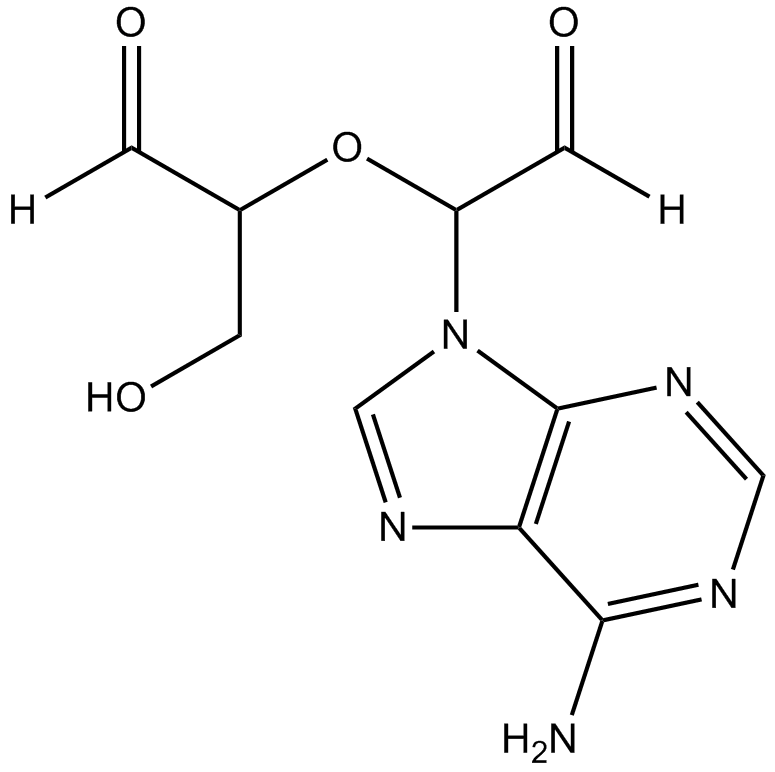
Adox, ein Purin-Nucleosid-Analogon, ist ein potenter Inhibitor der S-Adenosylhomocystein-Hydrolase (SAHH) (Ki=3,3 nM). Adenosindialdehyd weist in vivo eine starke AntitumoraktivitÄt auf und kann fÜr die Krebsforschung verwendet werden.
-
GC17275
AMI-1
A cell permeable inhibitor of PRMTs
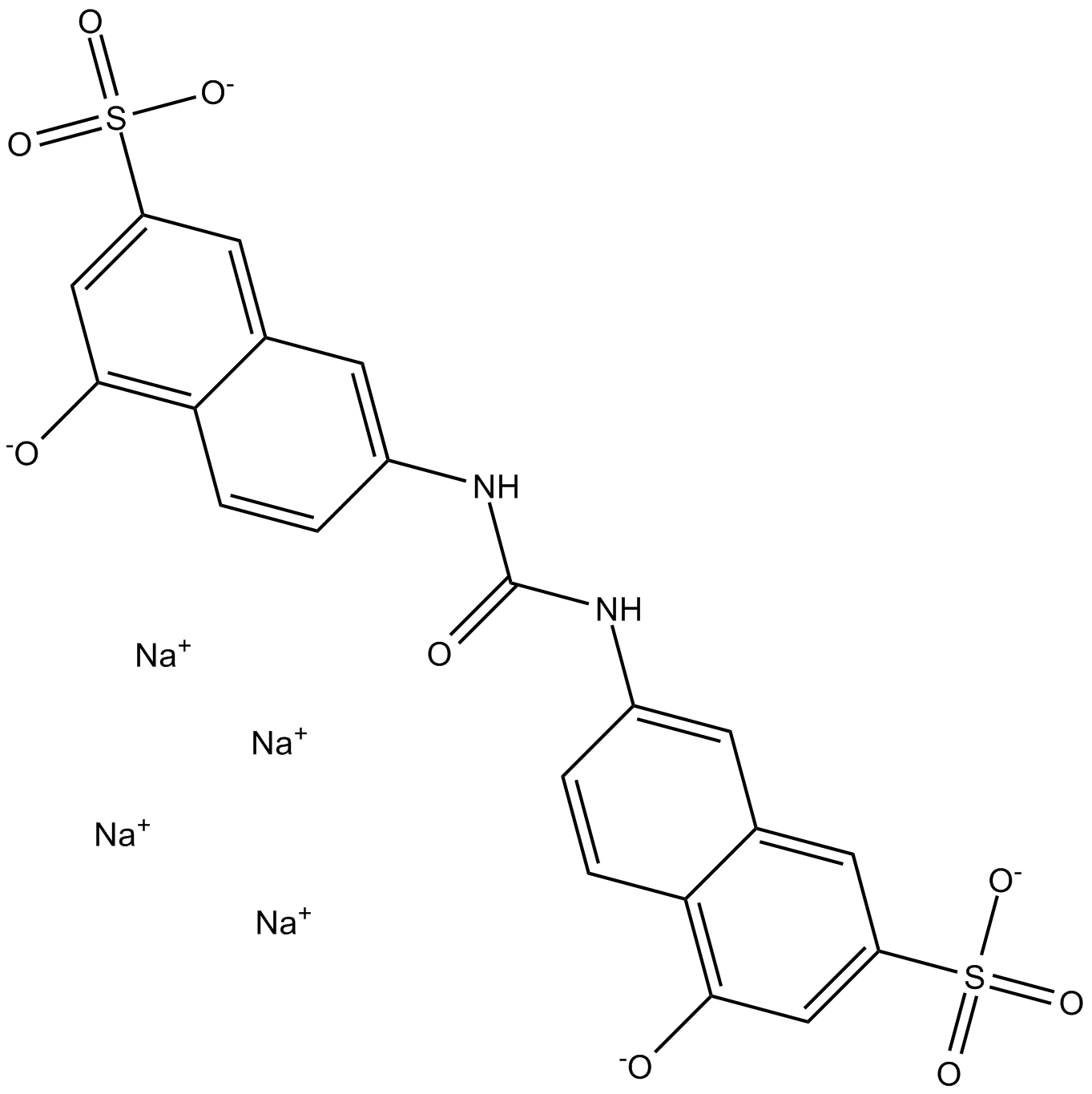
-
GC39840
AMI-1 free acid
Die freie SÄure von AMI-1 ist ein potenter, zellgÄngiger und reversibler Inhibitor von Protein-Arginin-N-Methyltransferasen (PRMTs) mit IC50-Werten von 8,8 μM und 3,0 μM fÜr menschliches PRMT1 bzw. Hefe-Hmt1p. Die freie SÄure von AMI-1 Übt PRMTs hemmende Wirkungen aus, indem sie die Peptid-Substrat-Bindung blockiert.
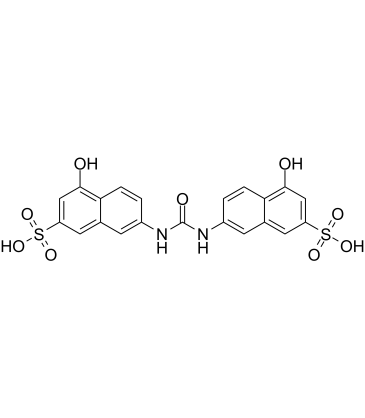
-
GC17546
AMI5
Eosin Y (Dinatrium) ist ein lÖsliches sÄurerotes FarbstoffmolekÜl.
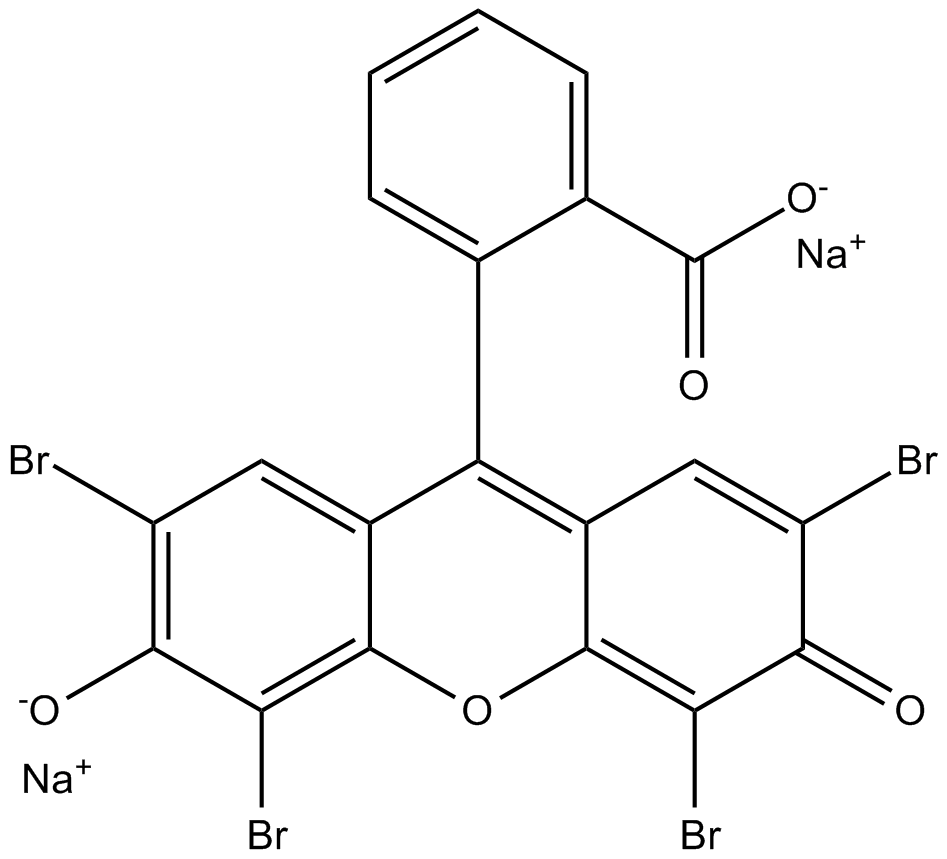
-
GC42790
Amodiaquine
Amodiaquin ist eine Aminoquinolin-Antimalariaverbindung.
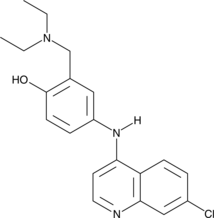
-
GC60579
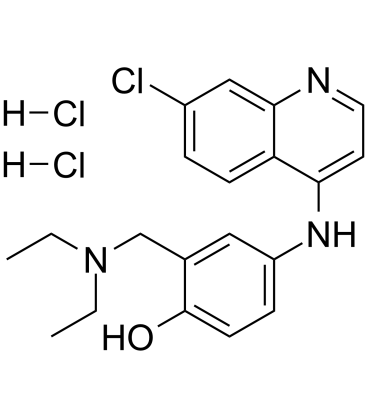
Amodiaquine dihydrochloride
Amodiaquindihydrochlorid (Amodiaquindihydrochlorid), eine 4-Aminochinolin-Klasse von Antimalariamitteln, ist ein potenter und oral aktiver Histamin-N-Methyltransferase-Inhibitor mit einem Ki von 18,6 nM.
-
GC10905
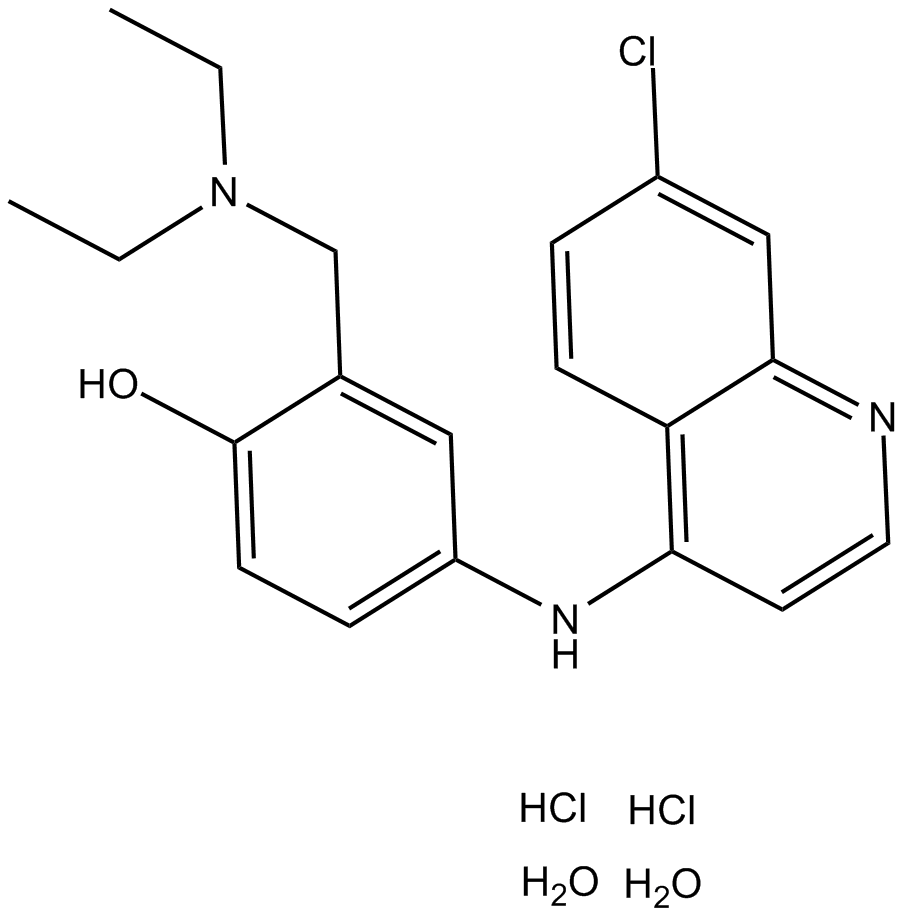
Amodiaquine dihydrochloride dihydrate
Amodiaquin-Dihydrochlorid-Dihydrat (Amodiaquin-Dihydrochlorid-Dihydrat), eine 4-Aminochinolin-Klasse von Antimalariamitteln, ist ein starker und oral aktiver Histamin-N-Methyltransferase-Inhibitor.
-
GC65918
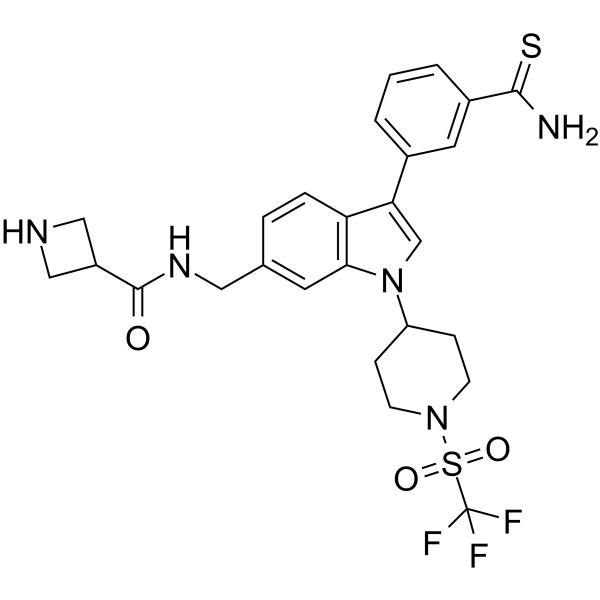
AS-85
AS-85 ist ein potenter ASH1L-Histon-Methyltransferase-Inhibitor (IC50=0,6 μM) mit antileukÄmischer AktivitÄt. AS-85 bindet stark an die ASH1L-SET-DomÄne mit einem Kd-Wert von 0,78&7#956;M.
-
GC62615
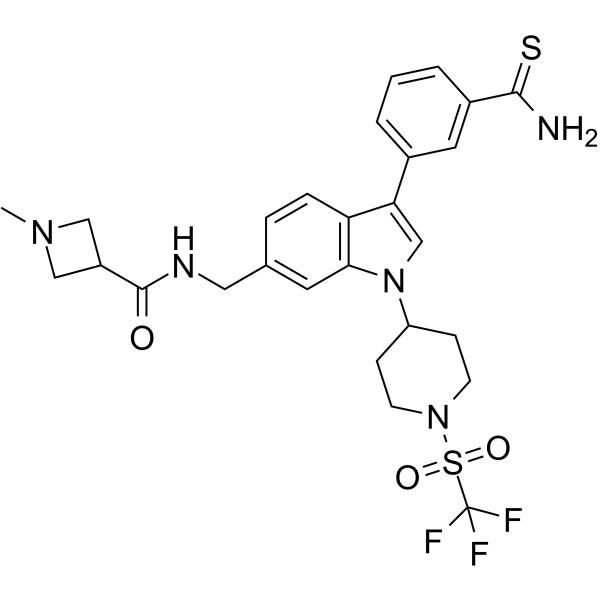
AS-99
AS-99 ist ein erstklassiger, wirksamer und selektiver ASH1L-Histon-Methyltransferase-Inhibitor (IC50=0,79⋼M, Kd=0,89&7#956;M) mit antileukÄmischer AktivitÄt. AS-99 blockiert die Zellproliferation, induziert Apoptose und Differenzierung, reguliert MLL-Fusionszielgene herunter und reduziert die LeukÄmiebelastung in vivo.
-
GC62849
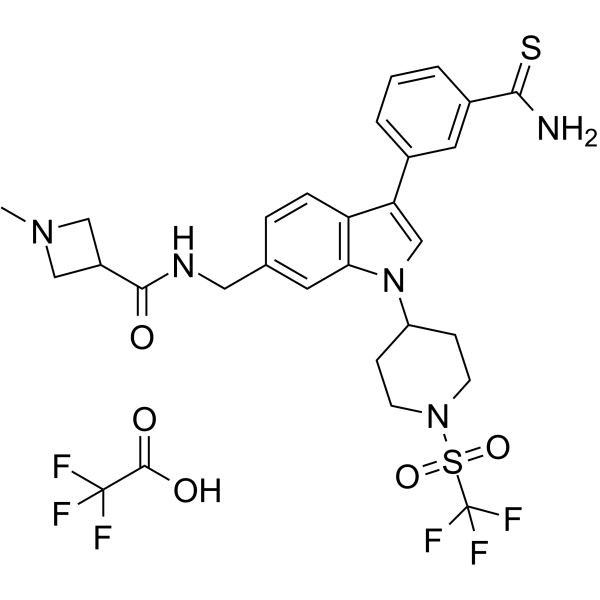
AS-99 TFA
AS-99 TFA ist ein erstklassiger, wirksamer und selektiver ASH1L-Histon-Methyltransferase-Inhibitor (IC50=0,79μM, Kd=0,89μM) mit antileukÄmischer AktivitÄt. AS-99 TFA blockiert die Zellproliferation, induziert Apoptose und Differenzierung, reguliert MLL-Fusionszielgene herunter und reduziert die LeukÄmiebelastung in vivo.
-
GC13744
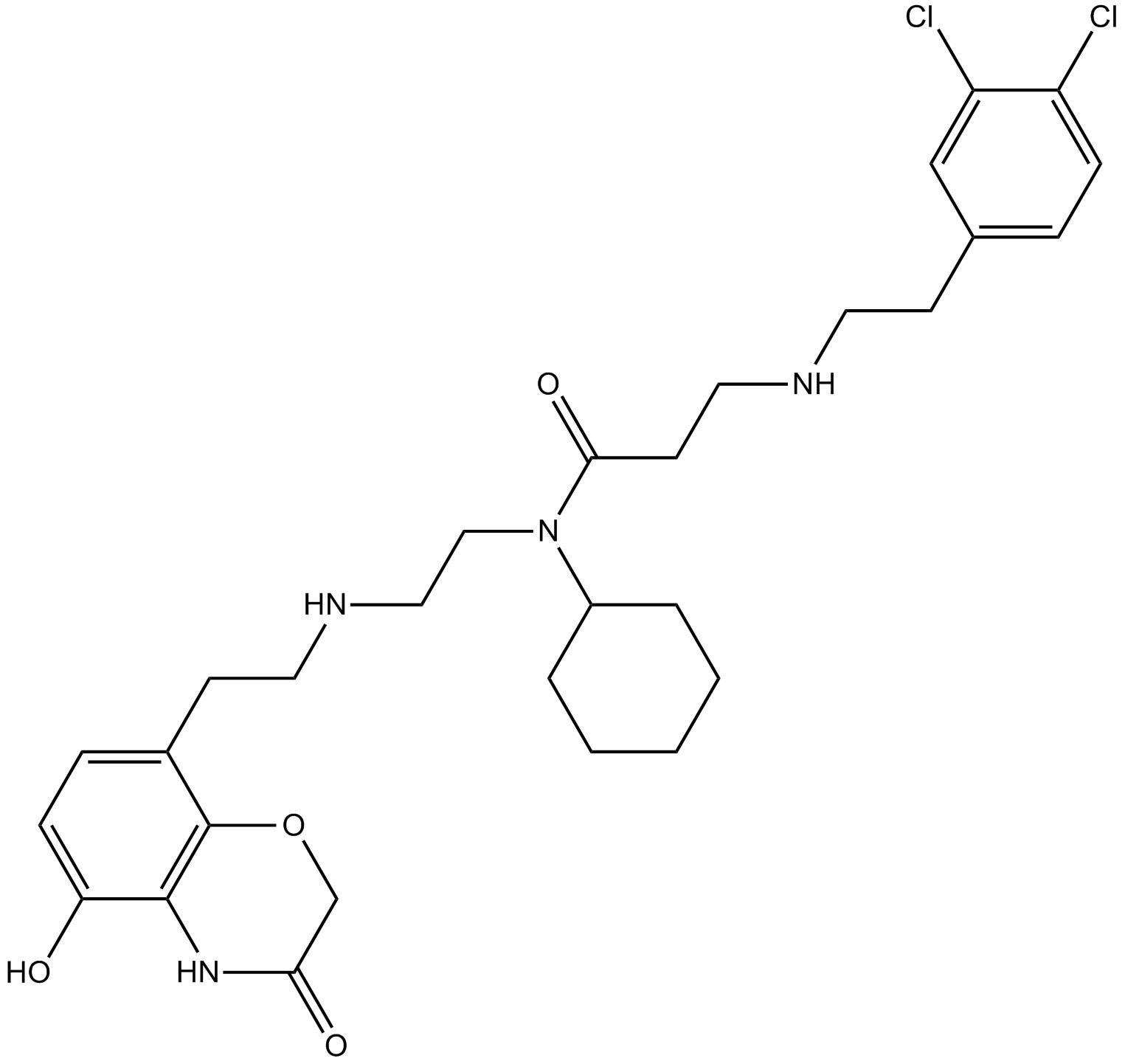
AZ505
AZ505 ist ein potenter und selektiver SMYD2-Inhibitor mit einem IC50 von 0,12 μM.
-
GC13103
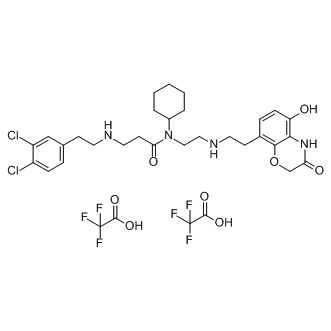
AZ505 ditrifluoroacetate
AZ505-Ditrifluoracetat ist ein potenter und selektiver SMYD2-Inhibitor mit einem IC50-Wert von 0,12 μM.
-
GC64124
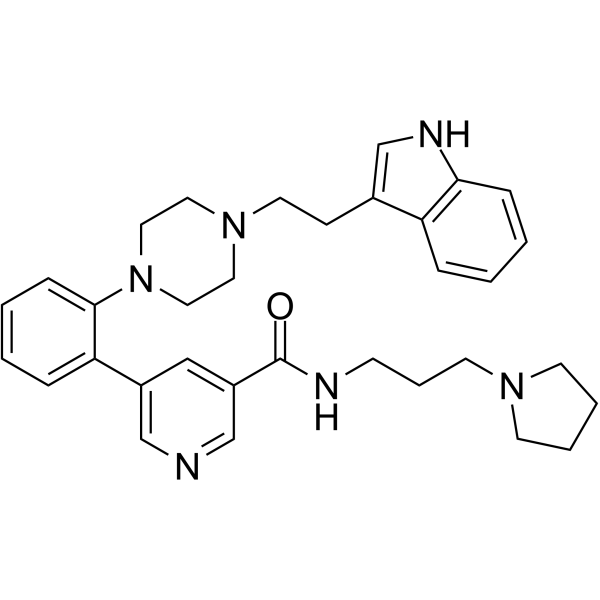
AZ506
AZ506 ist ein potenter SMYD2-Inhibitor mit einem IC50 von 17 nM. AZ506 hemmt die AktivitÄt der SMYD2-Methyltransferase in Zellen, was zu einer Abnahme des SMYD2-vermittelten Methylierungssignals fÜhrt.
-
GC18159
BAY-598
BAY-598 ist ein selektiver niedermolekularer Inhibitor von SMYD2 mit einem IC50 von 27 nM.
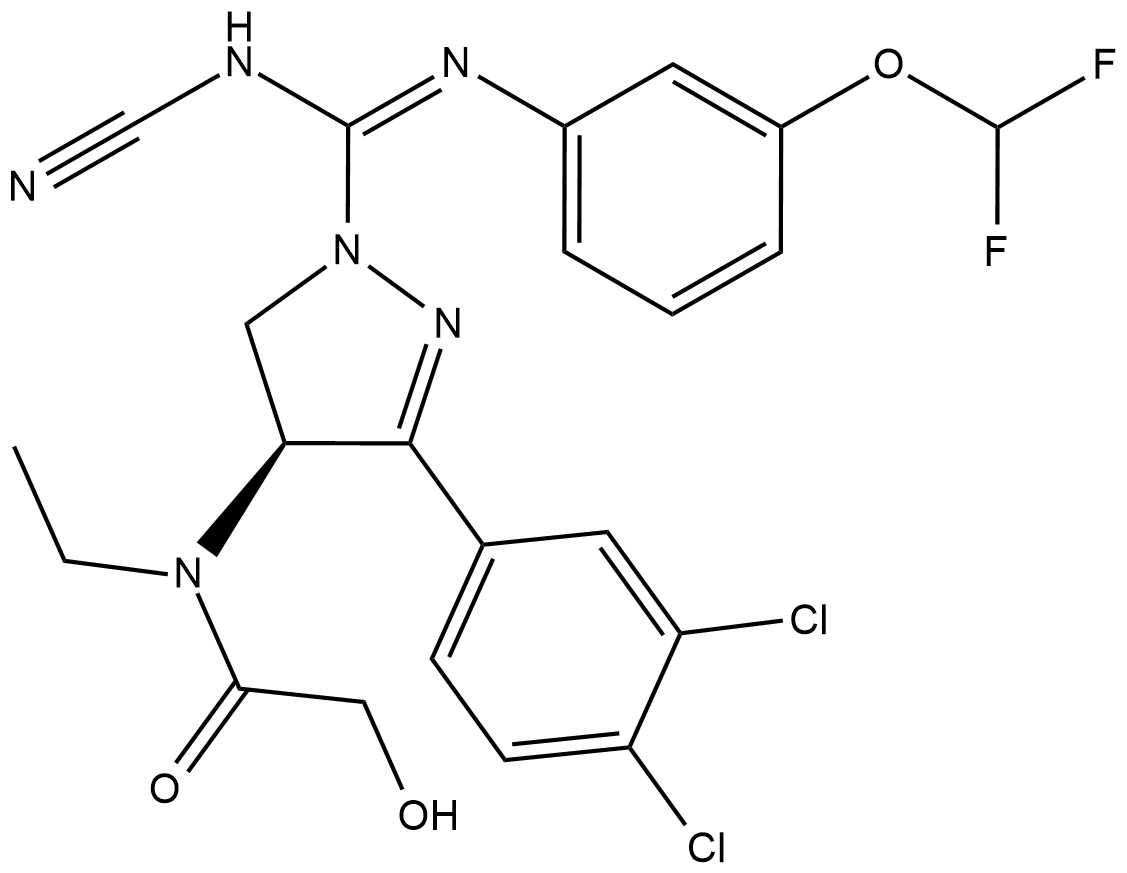
-
GC45389
BAY-6035
BAY-6035 ist ein potenter, selektiver und substratkompetitiver Inhibitor von SMYD3. BAY-6035 hemmt die Methylierung des MEKK2-Peptids mit einem IC50 von 88 nM.
-
GC18161
BCI-121
BCI-121 ist ein SMYD3-Inhibitor, der die Vermehrung von Krebszellen hemmt.
-
GC50540
BI 9321
Nuclear receptor-binding SET domain (NSD) 3 antagonist; selectively binds PWWP1 domain
-
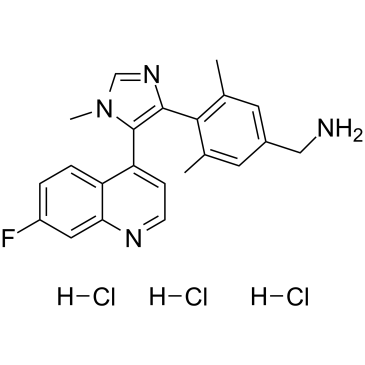
GC39663
BI-9321 trihydrochloride
BI-9321-Trihydrochlorid ist ein potenter, selektiver und zellaktiver Nuklearrezeptor-bindender Antagonist der SET-DomÄne 3 (NSD3)-PWWP1-DomÄne mit einem Kd-Wert von 166 nM. BI-9321-Trihydrochlorid ist gegen NSD2-PWWP1 und NSD3-PWWP2 inaktiv. BI-9321-Trihydrochlorid unterbricht spezifisch Histon-Wechselwirkungen der NSD3-PWWP1-DomÄne mit einem IC50 von 1,2 μM in U2OS-Zellen.
-
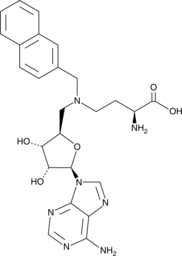
GC48463
Bisubstrate Inhibitor 78
An inhibitor of NNMT
-
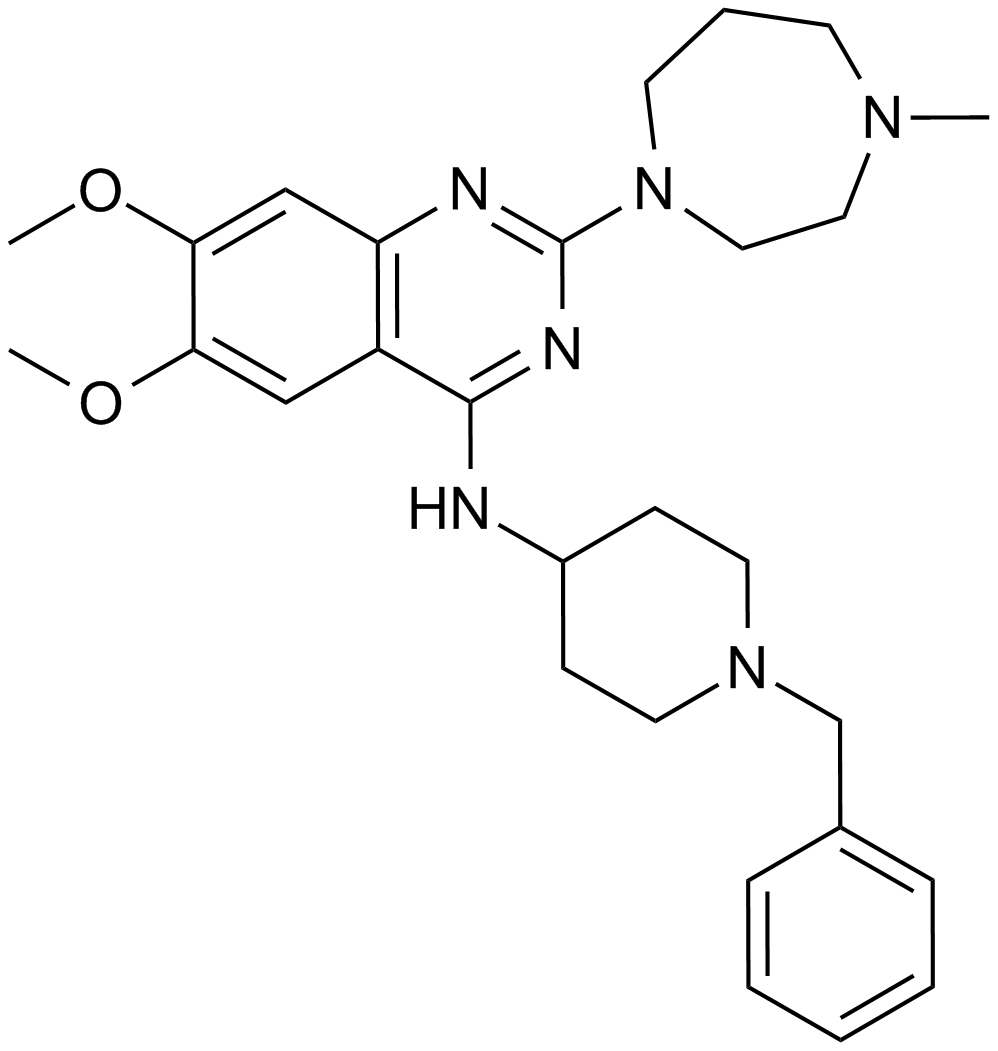
GC12171
BIX 01294
An inhibitor of G9a histone methyltransferase
-
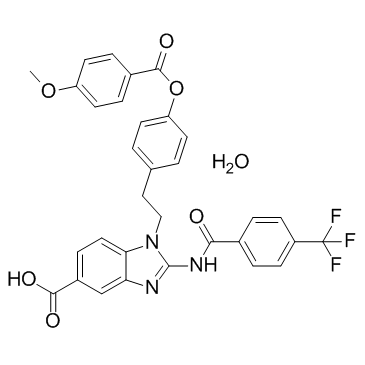
GC33301
BIX-01338 hydrate (BIX01338 hydrate)
BIX-01338-Hydrat (BIX01338-Hydrat) ist ein Histon-Lysin-Methyltransferase-Inhibitor.
-
GC42944
BIX01294 (hydrochloride hydrate)
The methylation of lysine residues on histones plays a central role in determining euchromatin structure and gene expression.
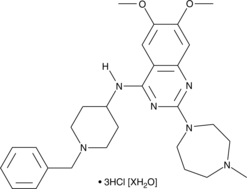
-
GC63731
BRD0639
BRD0639 ist ein erstklassiger Inhibitor der PRMT5-Substratadapter-Wechselwirkung. BRD0639 ist ein PRMT5-Bindungsmotiv (PBM)-kompetitiver Wirkstoff, der Studien zu PBM-abhÄngigen PRMT5-AktivitÄten unterstÜtzen kann.
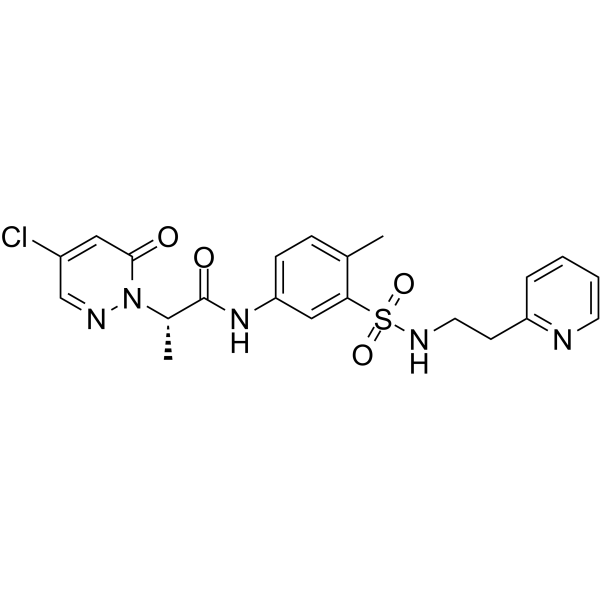
-
GC10259
BRD4770
BRD4770 ist ein Inhibitor der Histon-Methyltransferase G9a. BRD4770 reduziert die Di- und Trimethylierung von Lysin 9 auf Histon H3 (H3K9) mit einem EC50 von 5 μM und hat eine geringere oder geringe Wirkung auf H3K27me3, H3K36me3, H3K4me3 und H3K79me3. BRD4770 kann den Ataxia telangiectasia mutated (ATM) Signalweg aktivieren und Zellalterung induzieren.
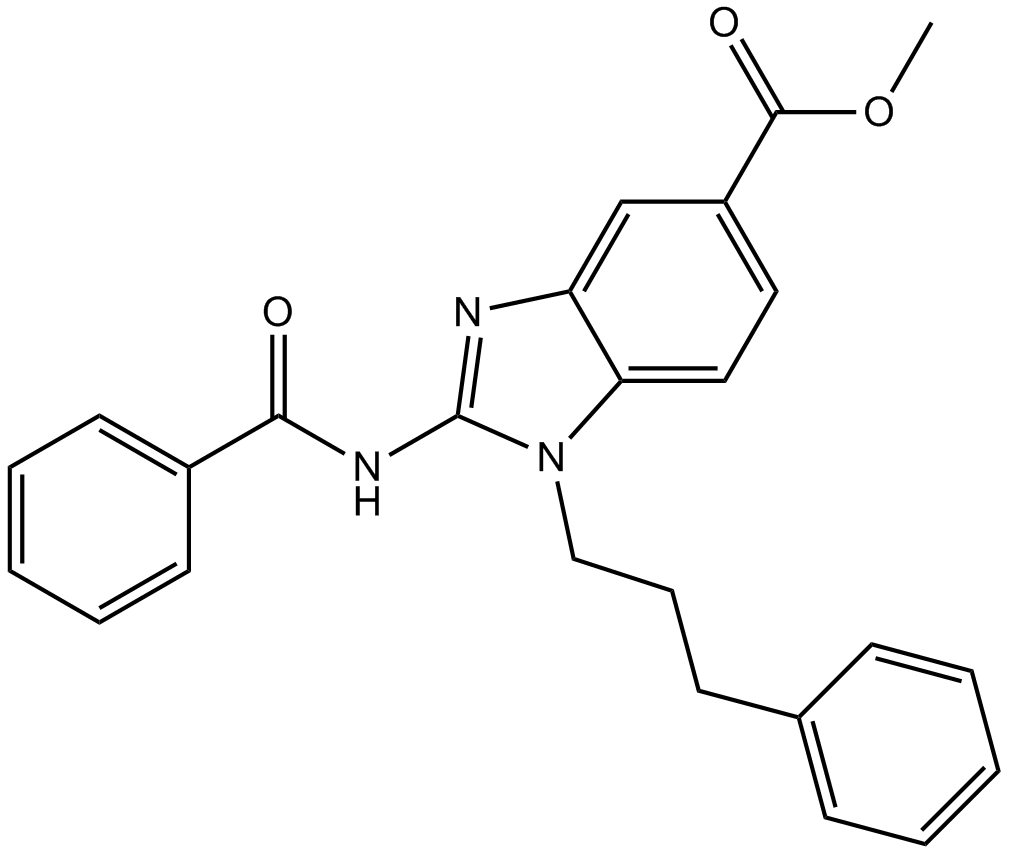
-
GC32988
BRD9539
BRD9539 ist ein Histon-Methyltransferase-G9a-Inhibitor mit einem IC50-Wert von 6,3 μM. BRD9539 hemmt auch die PRC2-AktivitÄt und ist gegen SUV39H1, NSD2 und DNMT1 inaktiv.
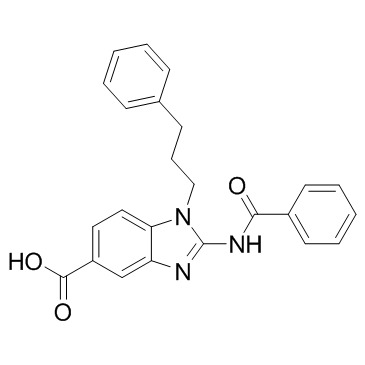
-
GC16130
C 21
Protein arginine methyltransferase 1 (PRMT1) inhibitor

-
GC12005
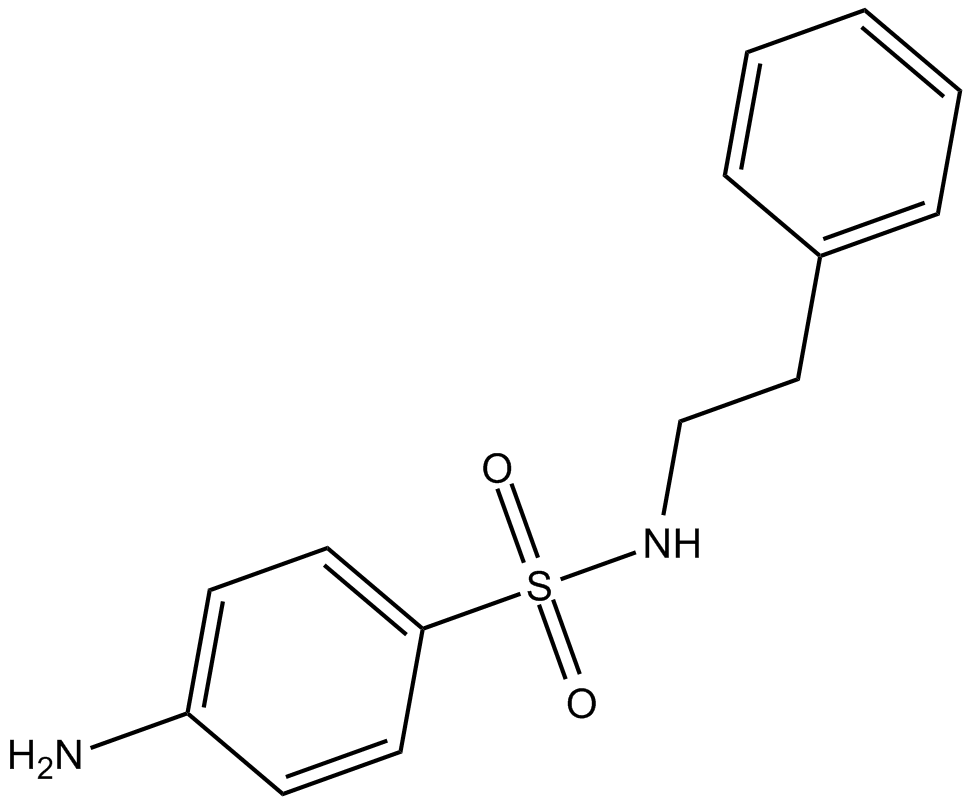
C7280948
C7280948 ist ein selektiver und potenter Protein-Methyltransferase1 (PRMT1)-Inhibitor mit einem IC50-Wert von 12,75 μM.
-
GC34108
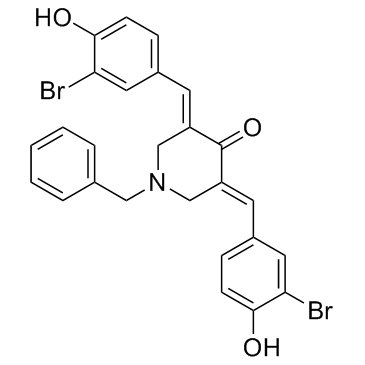
CARM1-IN-1
CARM1-IN-1 ist ein potenter und spezifischer CARM1(Coactivator-associated arginine methyltransferase 1)-Inhibitor mit einem IC50-Wert von 8,6 uM; zeigt eine sehr geringe AktivitÄt gegen PRMT1 und SET7 (IC50 > 600 uM).
-
GC34424
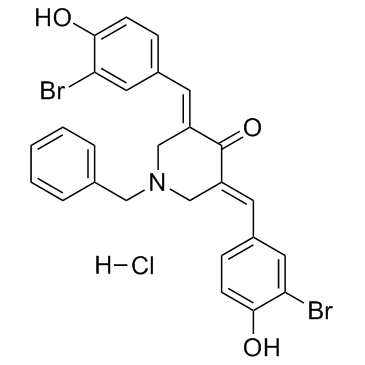
CARM1-IN-1 hydrochloride
CARM1-IN-1-Hydrochlorid ist ein potenter und spezifischer CARM1-Inhibitor (Coactivator-associated arginine methyltransferase 1) mit einem IC50-Wert von 8,6 uM; zeigt eine sehr geringe AktivitÄt gegen PRMT1 und SET7 (IC50 > 600 uM).
-
GC18949
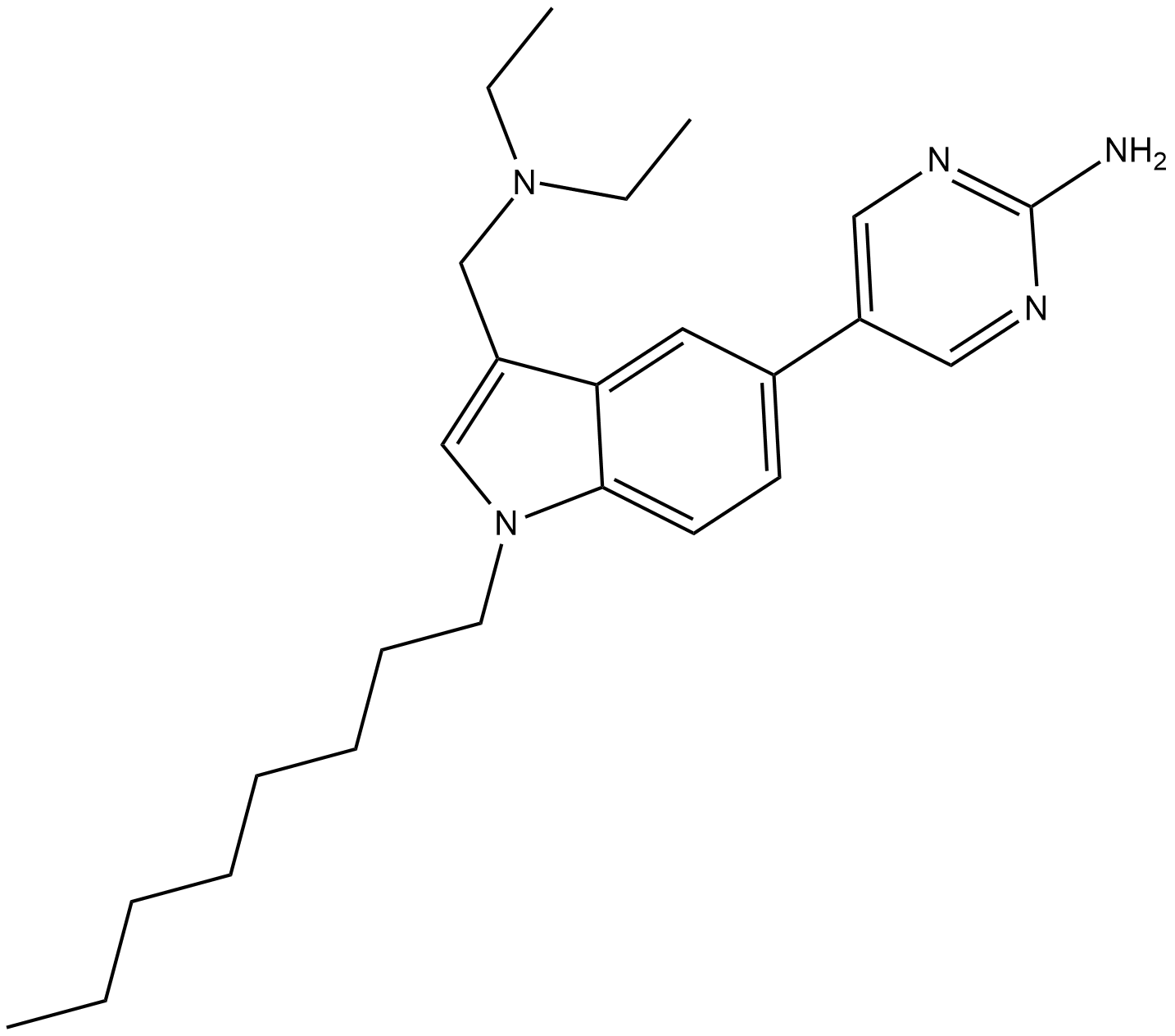
CAY10677
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.
-
GC14936
Chaetocin
Inhibitor of lys9-specific HMTs
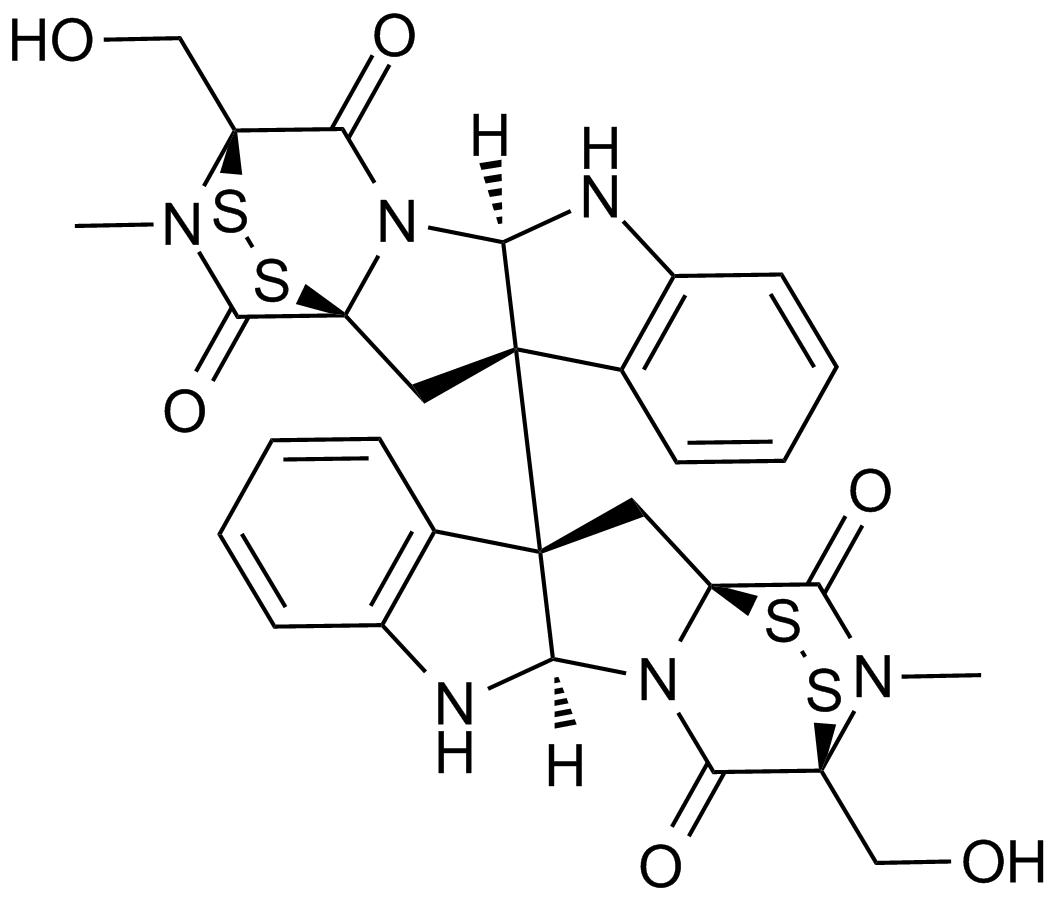
-
GC18577
CID-2818500
An inhibitor of PRMT1
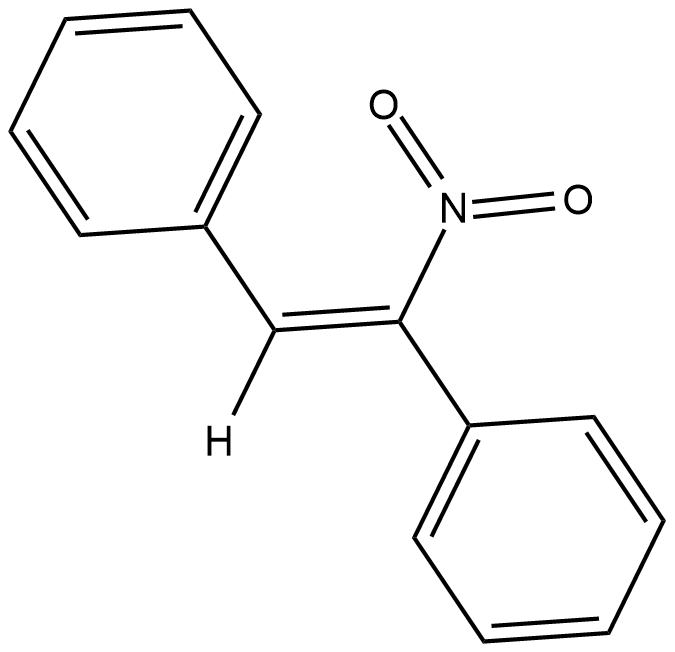
-
GC12367
CM-272
CM-272 ist ein erstklassiger, potenter, selektiver, substratkompetitiver und reversibler dualer G9a/DNA-Methyltransferase (DNMTs)-Inhibitor mit AntitumoraktivitÄten. CM-272 hemmt G9a, DNMT1, DNMT3A, DNMT3B und GLP mit IC50-Werten von 8 nM, 382 nM, 85 nM, 1200 nM bzw. 2 nM. CM-272 hemmt die Zellproliferation und fÖrdert die Apoptose, wodurch IFN-stimulierte Gene und immunogener Zelltod induziert werden.
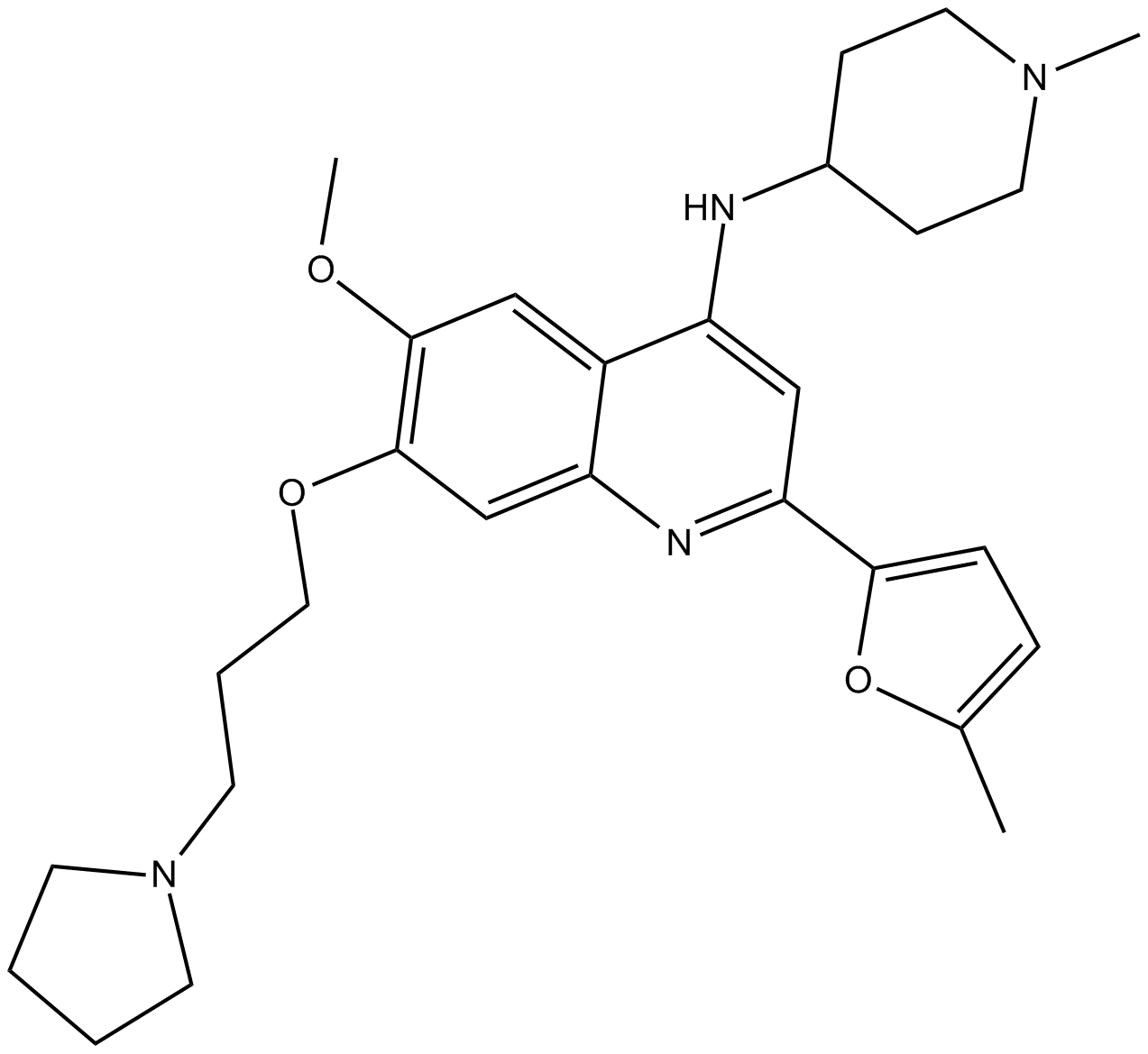
-
GC33320
CM-579
CM-579 ist ein erstklassiger reversibler dualer Inhibitor von G9a und DNMT mit IC50-Werten von 16 nM, 32 nM fÜr G9a bzw. DNMT. Hat eine starke zellulÄre In-vitro-AktivitÄt in einer Vielzahl von Krebszellen.
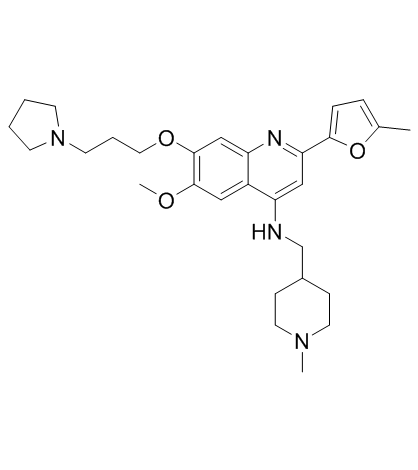
-
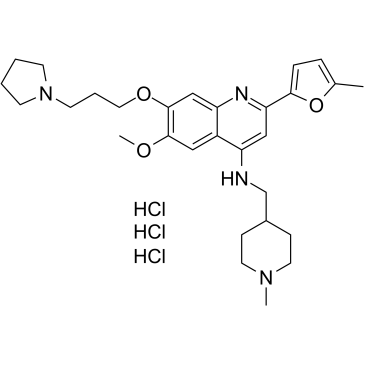
GC35714
CM-579 trihydrochloride
CM-579-Trihydrochlorid ist ein erstklassiger reversibler dualer Inhibitor von G9a und DNMT mit IC50-Werten von 16 nM bzw. 32 nM fÜr G9a und DNMT. Hat eine starke zellulÄre In-vitro-AktivitÄt in einer Vielzahl von Krebszellen.
-
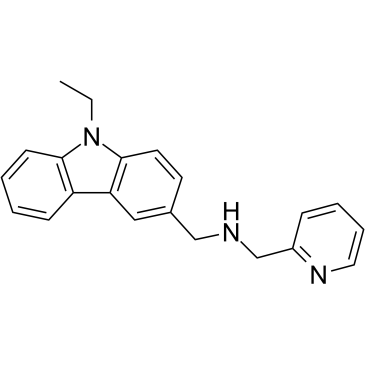
GC39665
CMP-5
CMP-5 ist ein potenter, spezifischer und selektiver PRMT5-Inhibitor, wÄhrend es keine AktivitÄt gegen PRMT1-, PRMT4- und PRMT7-Enzyme zeigt. CMP-5 blockiert selektiv S2Me-H4R3, indem es die PRMT5-Methyltransferase-AktivitÄt auf HistonprÄparaten hemmt. CMP-5 verhindert die vom Epstein-Barr-Virus (EBV) getriebene B-Lymphozyten-Transformation, lÄsst aber normale B-Zellen unbeeinflusst.
-
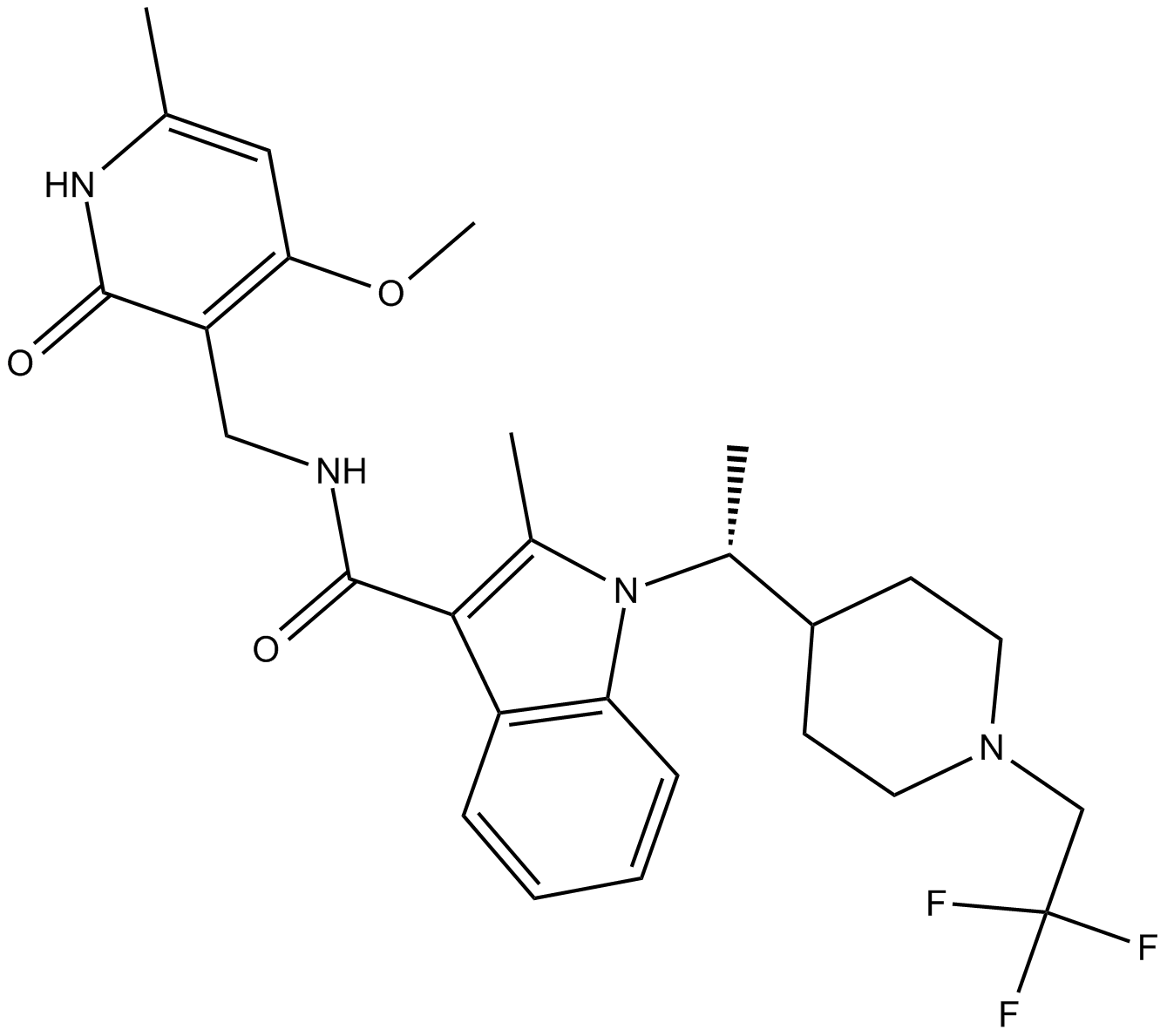
GC16298
CPI-1205
EZH2 inhibitor
-
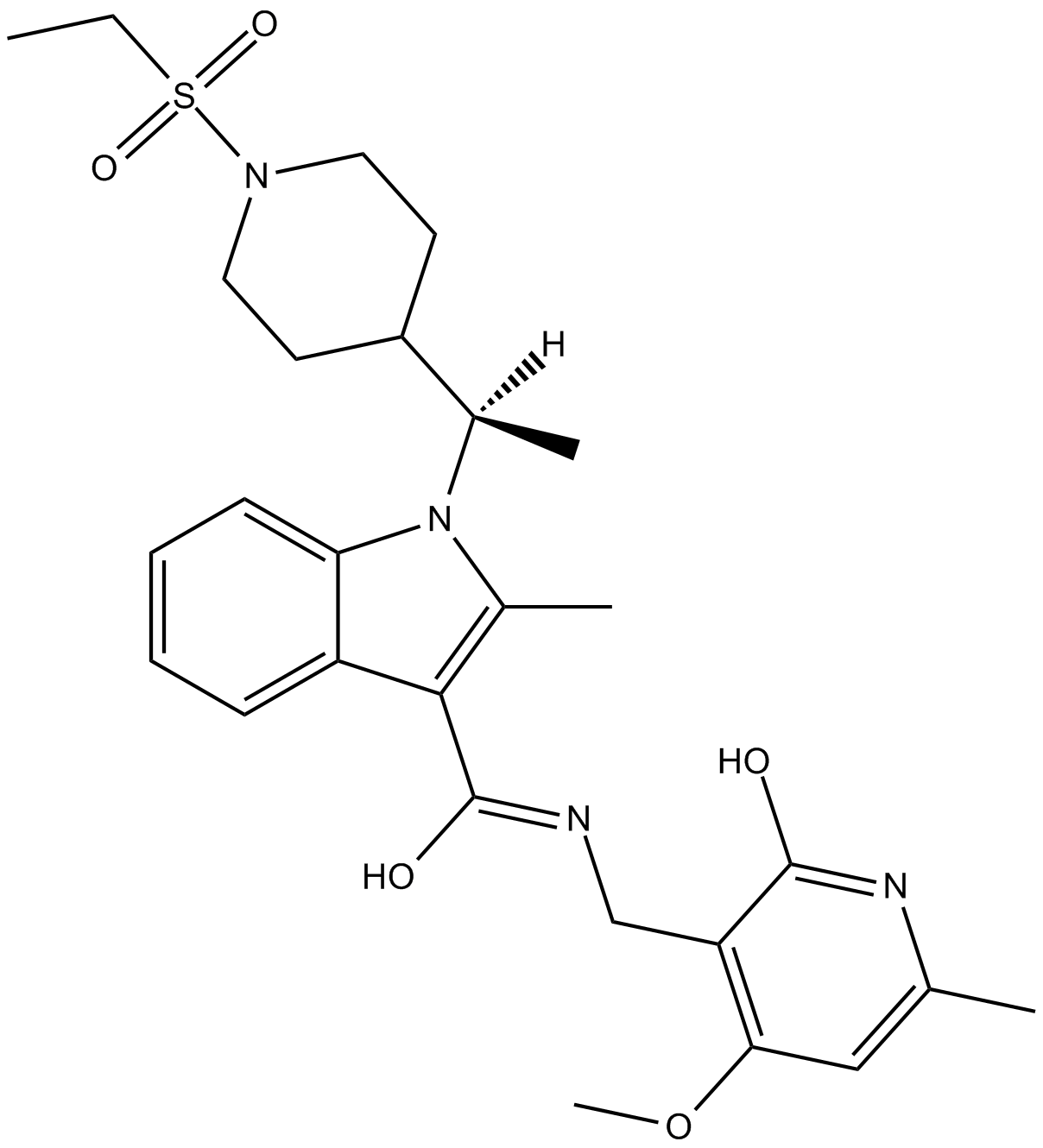
GC16599
CPI-169
EZH2 inhibitor
-
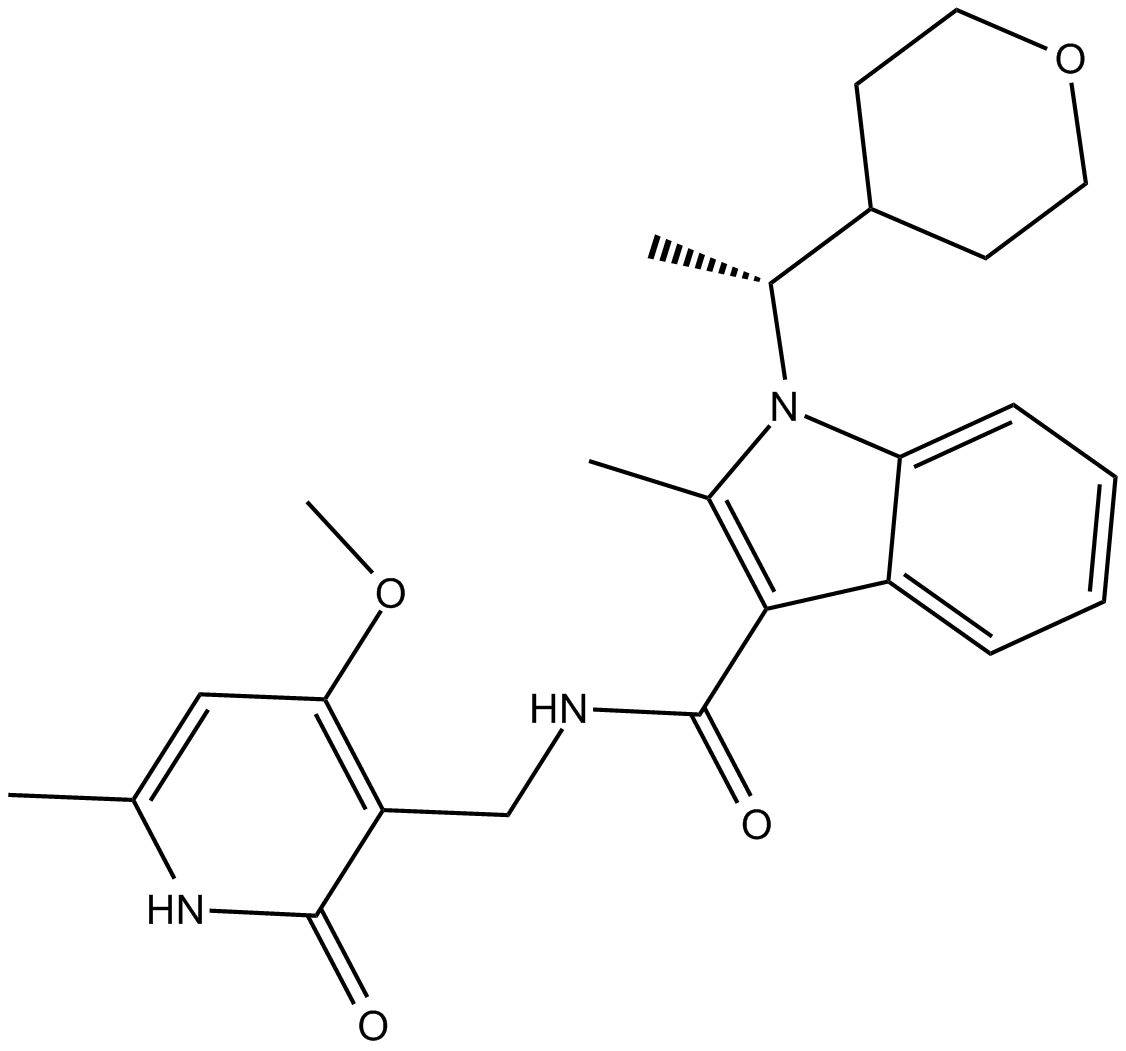
GC10021
CPI-360
EZH2 inhibitor
-
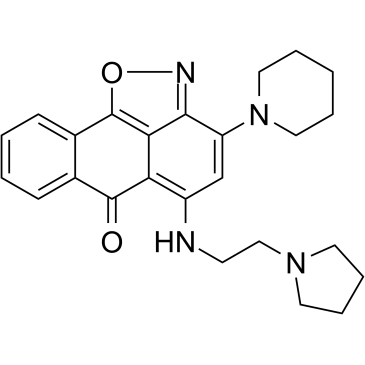
GC35742
CPUY074020
CPUY074020 ist ein potenter und oraler bioverfÜgbarer Inhibitor der Histon-Methyltransferase G9a mit einem IC50 von 2,18 μM. CPUY074020 besitzt antiproliferative AktivitÄt.
-
GC43354
Cysmethynil
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.

-
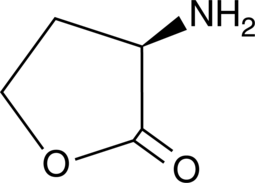
GC48967
D-Homoserine lactone
An enantiomer of L-homoserine lactone
-
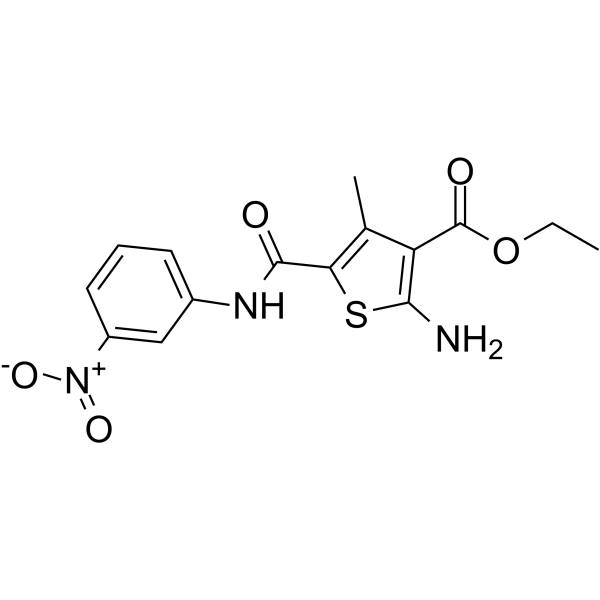
GC65186
DC-S239
DC-S239 ist ein selektiver Histon-Methyltransferase-SET7-Inhibitor mit einem IC50-Wert von 4,59 μM. DC-S239 zeigt auch SelektivitÄt fÜr DNMT1, DOT1L, EZH2, NSD1, SETD8 und G9a. DC-S239 hat AntikrebsaktivitÄt.
-
GC63662
DCLX069
DCLX069 ist ein selektiver Protein-Arginin-Methyltransferase-1 (PRMT1)-Inhibitor mit einem IC50-Wert von 17,9 μM. DCLX069 zeigt weniger AktivitÄt gegenÜber PRMT4 und PRMT6. DCLX069 hat Antikrebswirkungen.
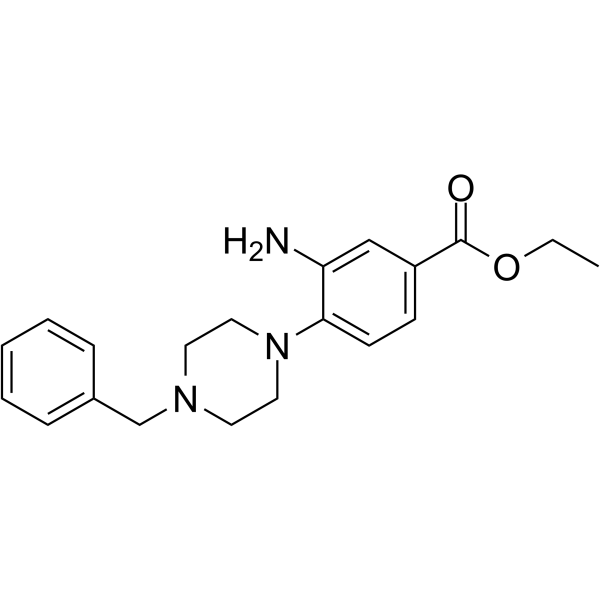
-
GC35816
DC_C66
DC_C66 ist ein zelldurchlÄssiger, selektiver Coaktivator-assoziierter Arginin-Methyltransferase-1 (CARM1)-Inhibitor mit einem IC50 von 1,8 μM. DC_C66 hat eine gute SelektivitÄt fÜr CARM1 gegenÜber PRMT1 (IC50 = 21 μM), PRMT6 (IC50 = 47 μM) und PRMT5.
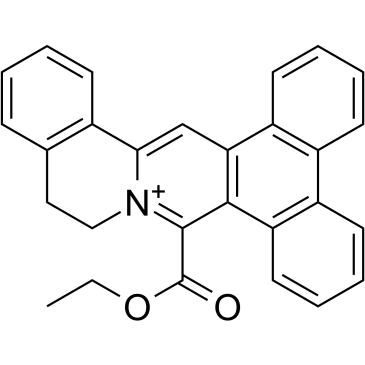
-
GC67863
DDO-2093 dihydrochloride

-
GC33208
Dot1L-IN-1
Dot1L-IN-1 ist ein hochwirksamer, selektiver und strukturell neuer Dot1L-Inhibitor mit einem Ki von 2 pM.
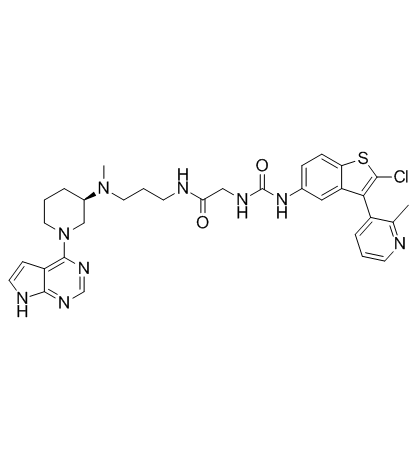
-
GC30530
Dot1L-IN-2
Dot1L-IN-2 ist ein potenter, selektiver und oral bioverfÜgbarer Inhibitor von Dot1L (einer Histon-Methyltransferase) mit einem IC50 und Ki von 0,4 nM bzw. 0,08 nM.
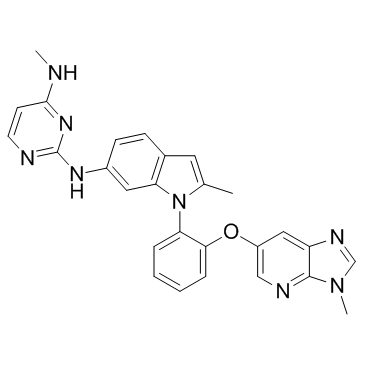
-
GC62613
Dot1L-IN-4
Dot1L-IN-4 ist ein potenter Disruptor des telomeric silencing 1-like protein (DOT1L)-Inhibitors mit einem IC50 SPA DOT1L von 0,11 nM.
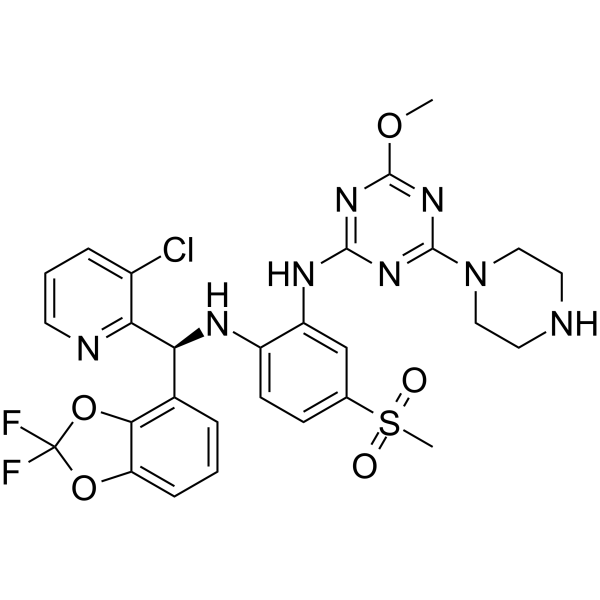
-
GC65962
Dot1L-IN-5
Dot1L-IN-5 ist ein potenter Disruptor des telomeric silencing 1-like protein (DOT1L)-Inhibitors mit einem IC50 SPA DOT1L von 0,17 nM.
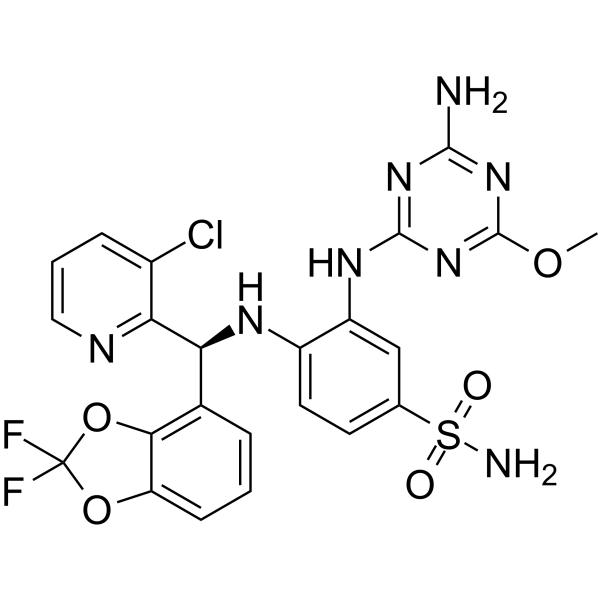
-
GC45927
DS-437
DS-437 ist ein dualer PRMT5/7-Inhibitor (IC50s von PRMT5/7 = 6 μM).
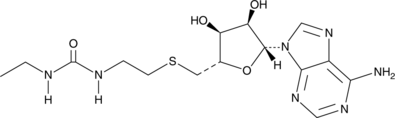
-
GC35914
DW14800
DW14800 ist ein Inhibitor der Protein-Arginin-Methyltransferase 5 (PRMT5) mit einem IC50-Wert von 17 nM. DW14800 reduziert die H4R3me2s-Spiegel und verstÄrkt die Transkription von HNF4α, verÄndert jedoch nicht die PRMT5-Expression. Anti-Krebs-AktivitÄt.
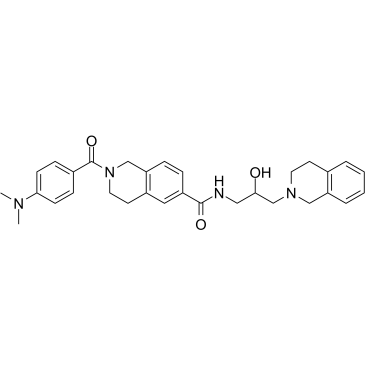
-
GC33220
EBI-2511
EBI-2511 ist ein hochpotenter und oral aktiver EZH2-Inhibitor mit einem IC50-Wert von 6 nM in Pfeffiera-Zelllinien.
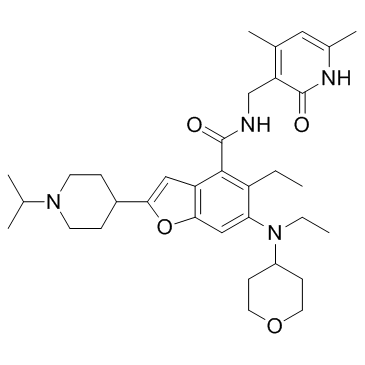
-
GC19130
EED226
EED226 ist ein Polycomb Repressive Complex 2 (PRC2)-Inhibitor, der bei der embryonalen Ektodermentwicklung (EED) an die K27me3-Tasche bindet und im Xenograft-Mausmodell eine starke AntitumoraktivitÄt zeigt. EED226 ist ein potenter, selektiver und oral bioverfÜgbarer EED-Inhibitor. EED226 hemmt PRC2 mit einem IC50 von 23,4 nM, wenn das H3K27me0-Peptid als Substrat in den enzymatischen In-vitro-Assays verwendet wird.
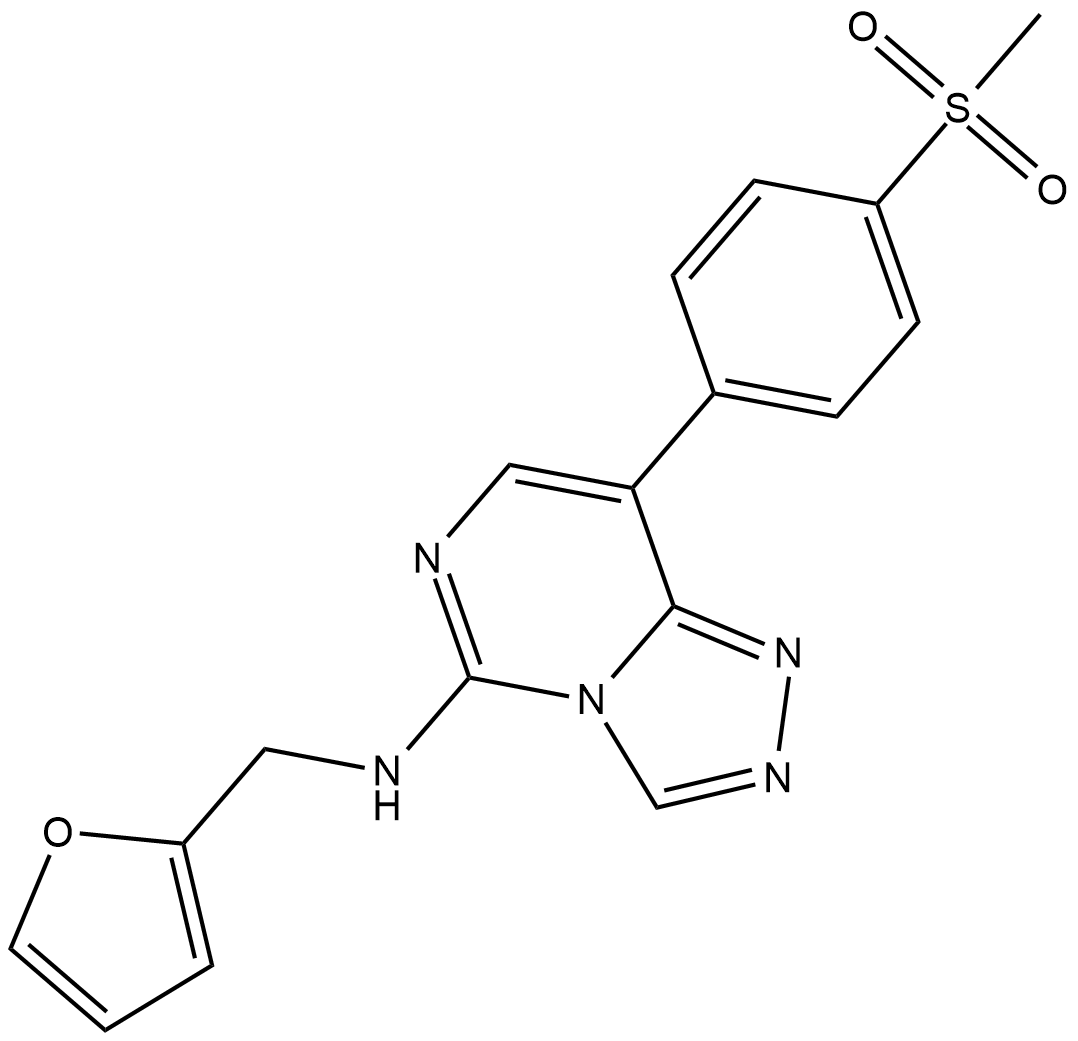
-
GC67934
EEDi-5285
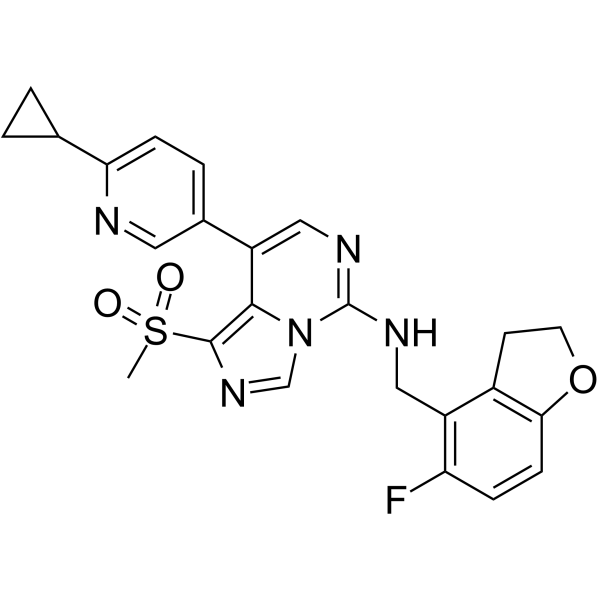
-
GC34567
EHMT2-IN-1
EHMT2-IN-1 ist ein potenter EHMT-Inhibitor mit IC50-Werten von allen < 100 nM fÜr EHMT1-Peptid, EHMT2-Peptid und zellulÄres EHMT2. Wird in der Erforschung von Blutkrankheiten oder Krebs verwendet.
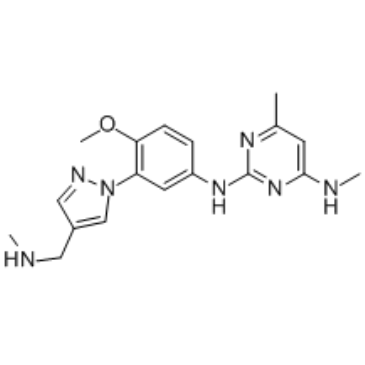
-
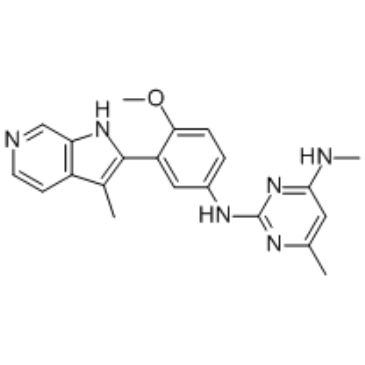
GC34568
EHMT2-IN-2
EHMT2-IN-2 ist ein potenter EHMT-Inhibitor mit IC50-Werten von allen < 100 nM fÜr EHMT1-Peptid, EHMT2-Peptid und zellulÄres EHMT2. Wird in der Erforschung von Blutkrankheiten oder Krebs verwendet.
-
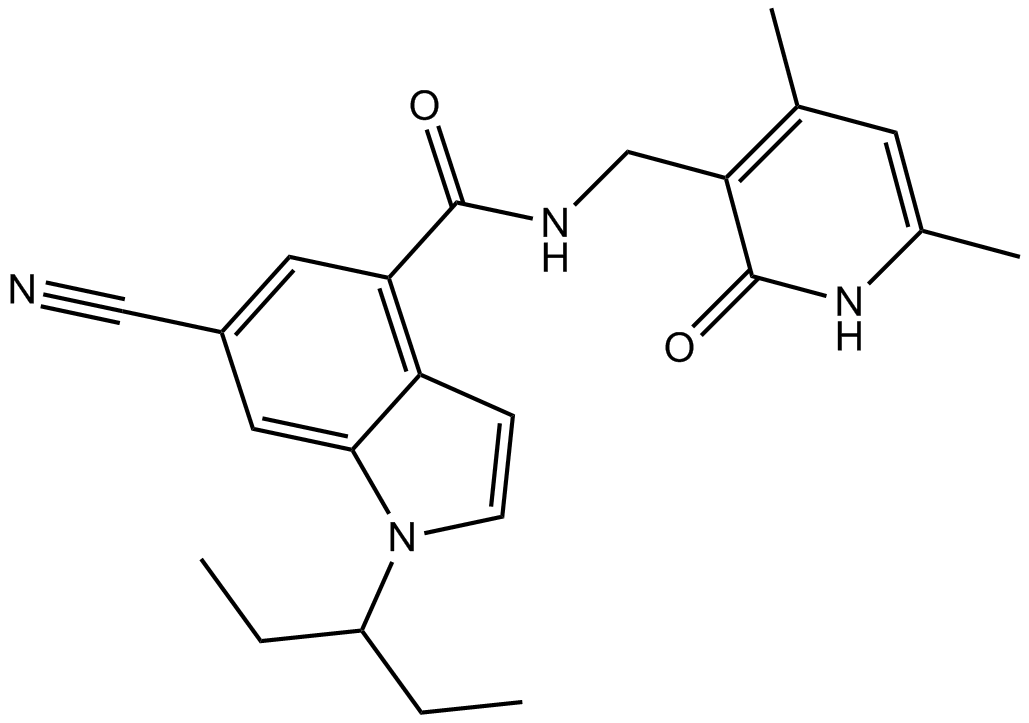
GC14756
EI1
EI1 (KB-145943) ist ein potenter und selektiver EZH2-Inhibitor mit einem IC50-Wert von 15 nM bzw. 13 nM fÜr EZH2 (WT) und EZH2 (Y641F).
-
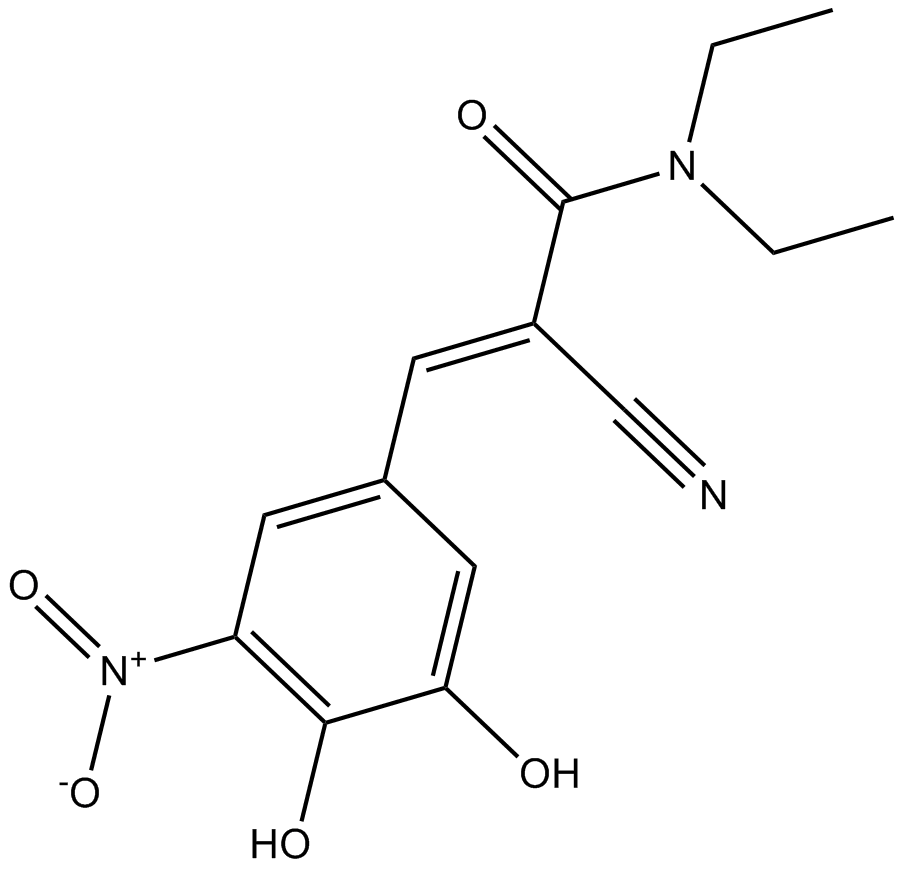
GC17334
Entacapone
A reversible COMT inhibitor
-
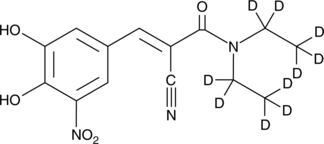
GC47294
Entacapone-d10
Entacapone-d10 ist das Deuterium mit der Bezeichnung Entacapone.
-
GC14062
EPZ-6438
A EZH2 inhibitor,potent and selective
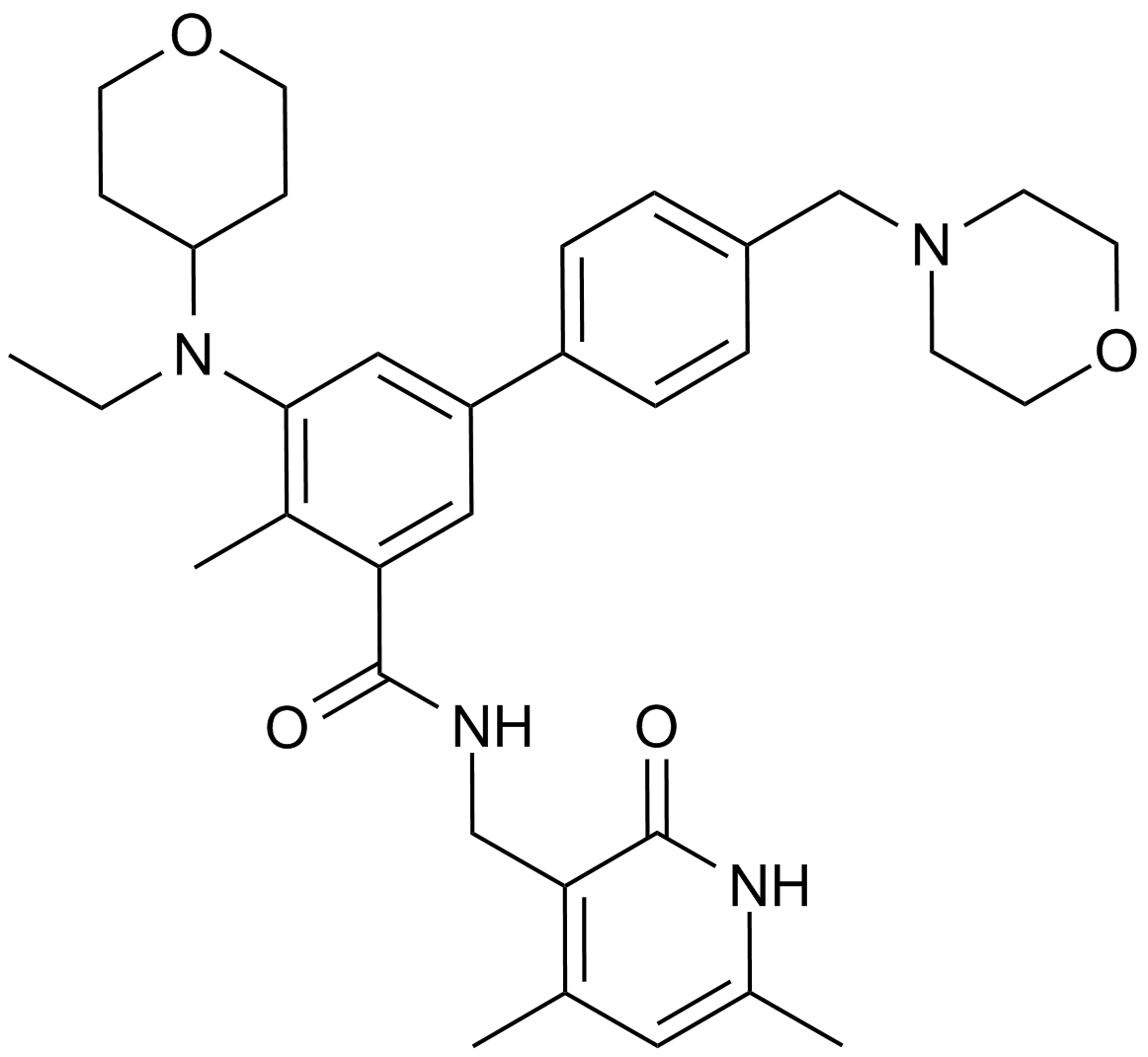
-
GC64343
EPZ-719
EPZ-719 ist ein neuartiger und potenter SETD2-Inhibitor (IC50 = 0,005 μM) mit einer hohen SelektivitÄt gegenÜber anderen Histon-Methyltransferasen.
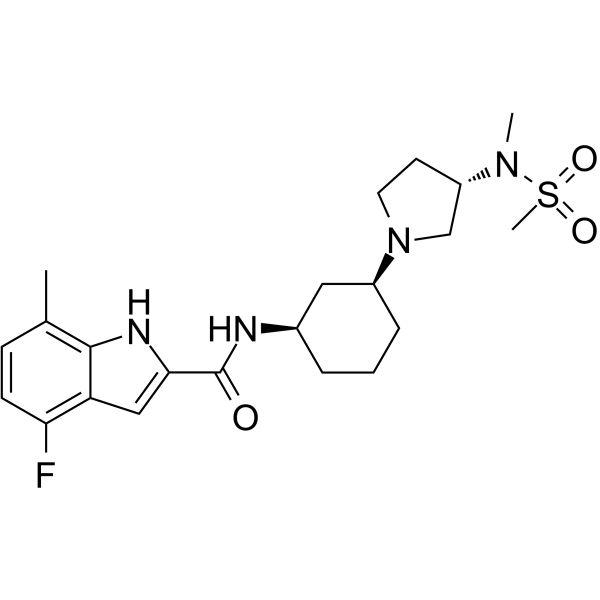
-
GC13383
EPZ004777
A potent inhibitor of DOT1L
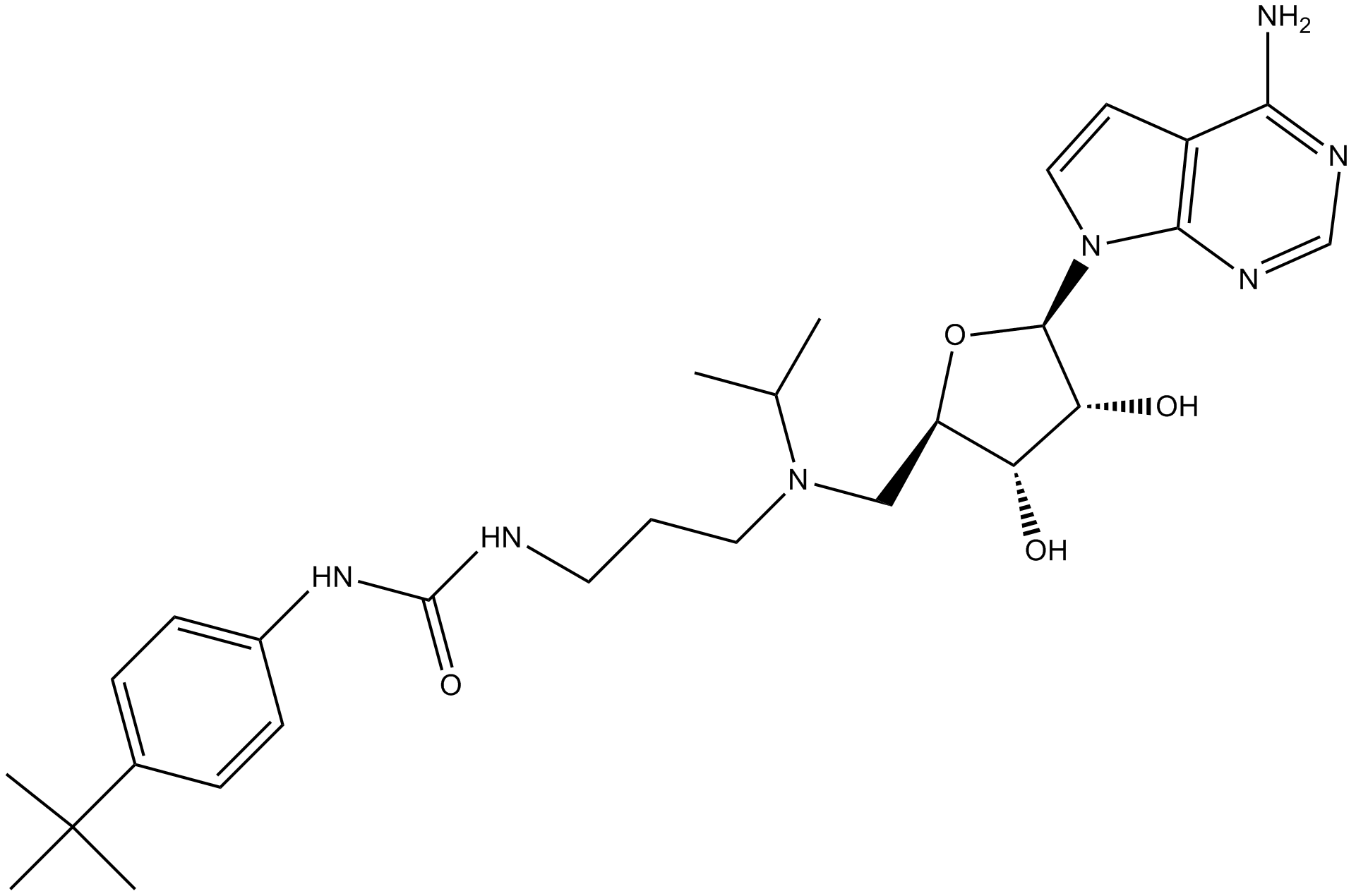
-
GC48980
EPZ004777 (formate)
A potent inhibitor of DOT1L

-
GC15259
EPZ004777 HCl
EPZ004777 HCl ist ein potenter, selektiver DOT1L-Inhibitor mit einem IC50 von 0,4 nM.
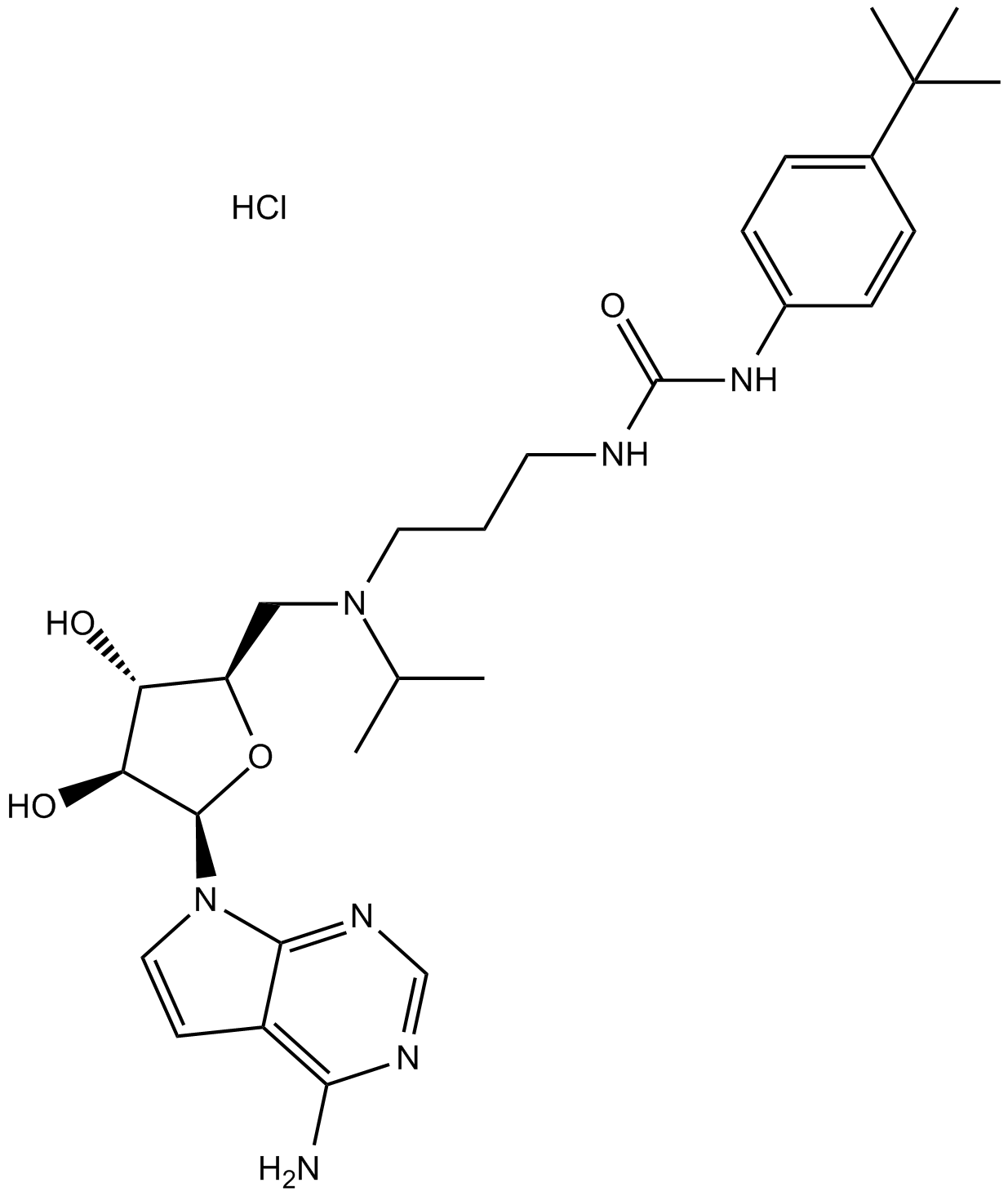
-
GC13878
EPZ005687
A potent, selective inhibitor of EZH2
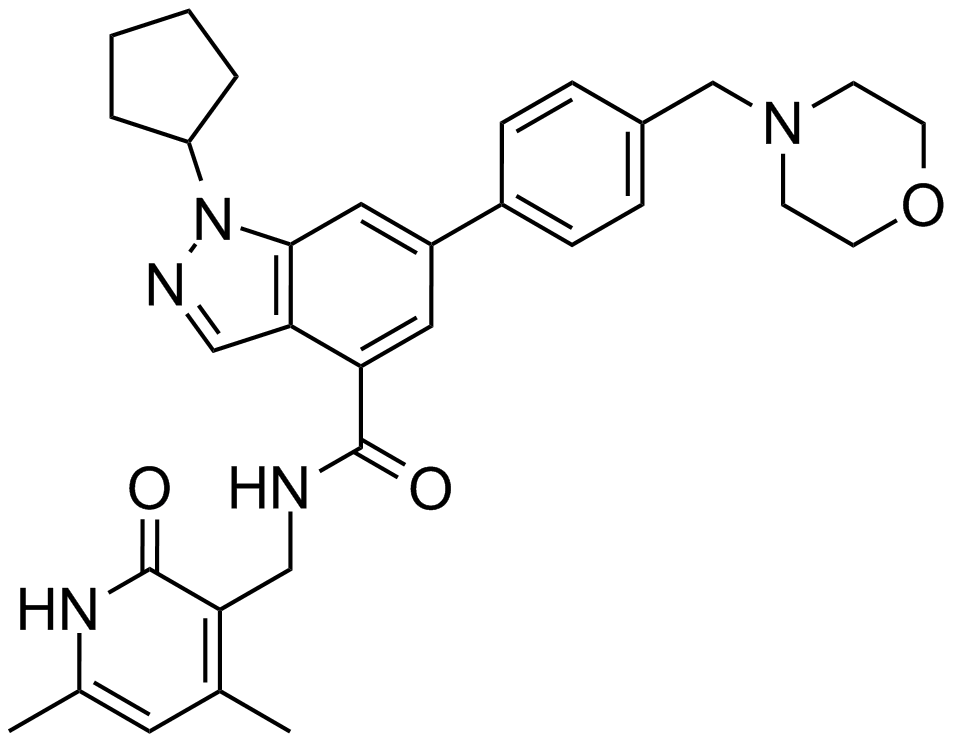
-
GC19141
EPZ011989
EPZ011989 ist ein potenter und oral aktiver Hemmer von Zeste Homolog 2 (EZH2) mit metabolischer StabilitÄt. EPZ011989 hat eine hemmende Hemmung fÜr EZH2 mit einem Ki-Wert von <3 nM. EPZ011989 zeigt eine robuste Methylmarkierungshemmung und AntitumoraktivitÄt. EPZ011989 kann fÜr die Erforschung verschiedener Krebsarten verwendet werden.
-
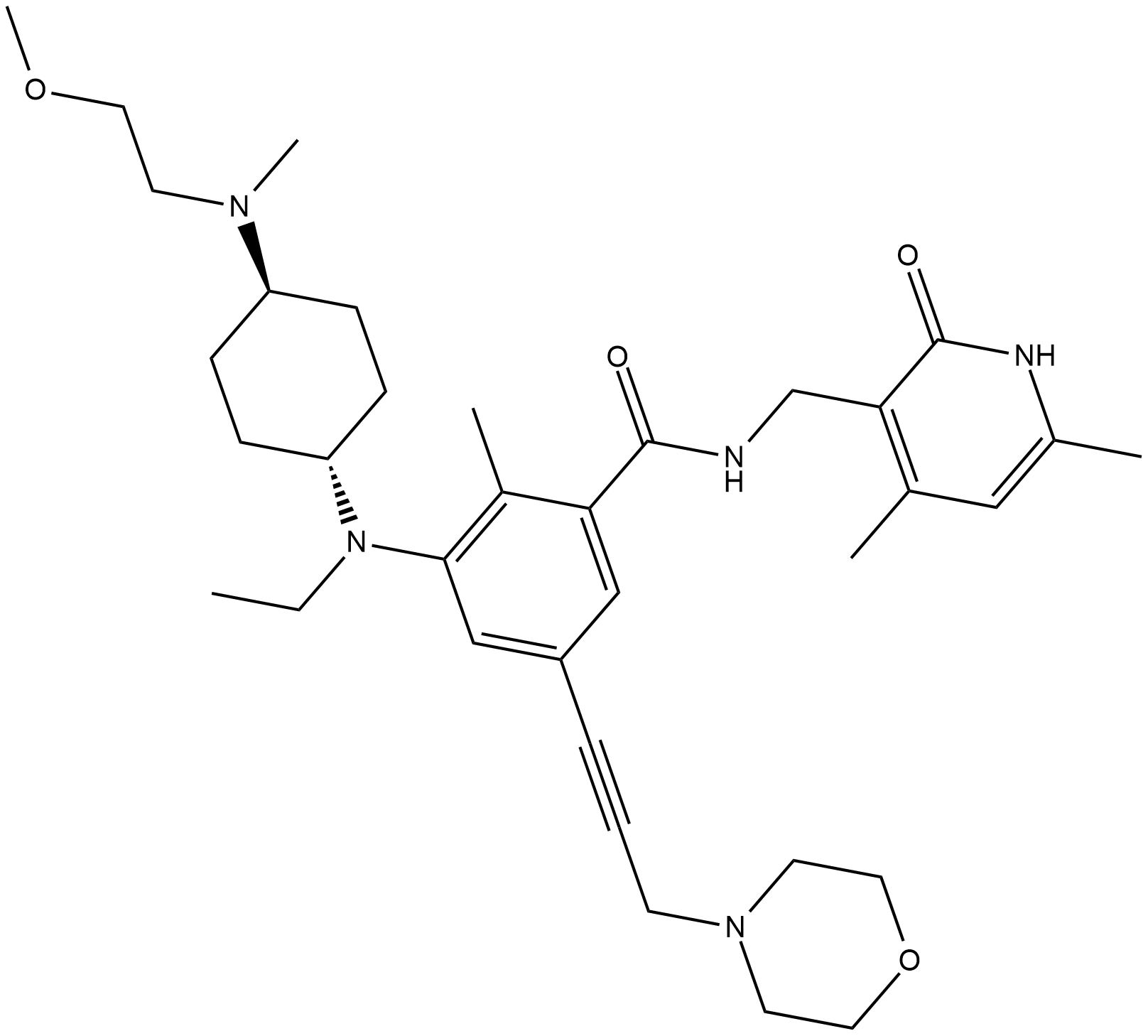
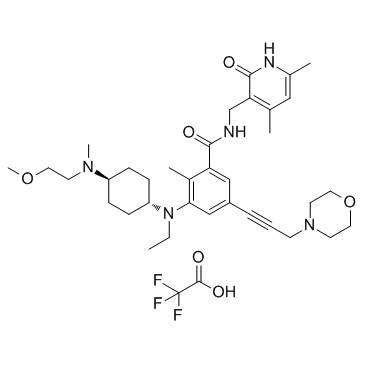
GC34136
EPZ011989 trifluoroacetate (EPZ-011989 trifluoroacetate)
EPZ-011989 Trifluoracetat ist ein potenter und oral aktiver Hemmer von Zeste Homolog 2 (EZH2) mit metabolischer StabilitÄt. EPZ-011989 Trifluoracetat hat eine hemmende Hemmung fÜr EZH2 mit einem Ki-Wert von <3 nM. EPZ-011989 Trifluoracetat zeigt eine robuste Methylmarkierungshemmung und AntitumoraktivitÄt. EPZ-011989 Trifluoracetat kann fÜr die Erforschung verschiedener Krebsarten verwendet werden.
-
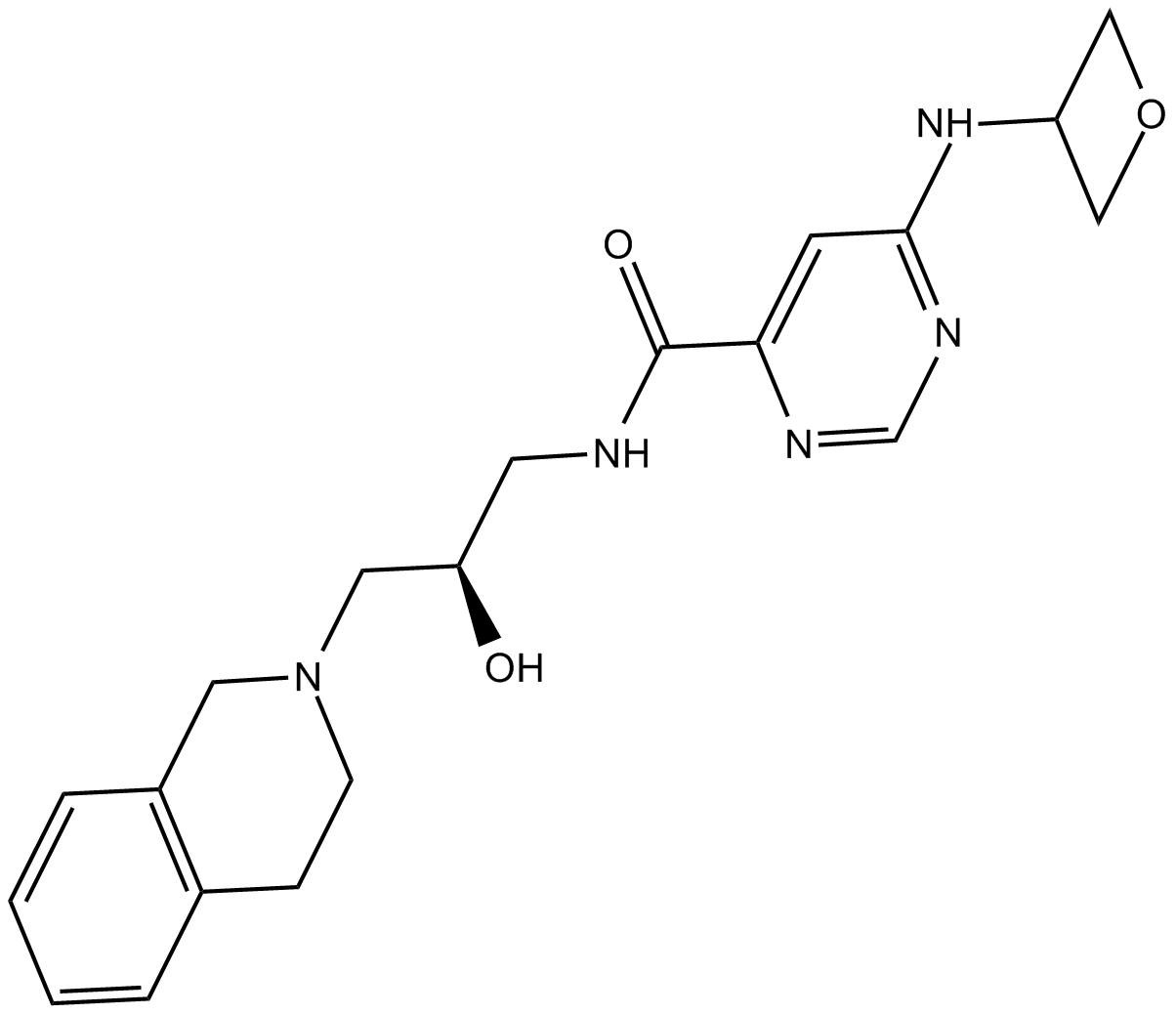
GC15302
EPZ015666
EPZ015666 (GSK3235025) ist ein oral verfÜgbarer Inhibitor von PRMT5 mit einem IC50 von 22 nM.
-
GC15102
EPZ020411
EPZ020411 ist ein selektiver Inhibitor von PRMT6 mit einem IC50 von 10 nM und hat eine >10-fache SelektivitÄt fÜr PRMT6 gegenÜber PRMT1 und PRMT8. EPZ020411 kann fÜr die Krebsforschung verwendet werden.
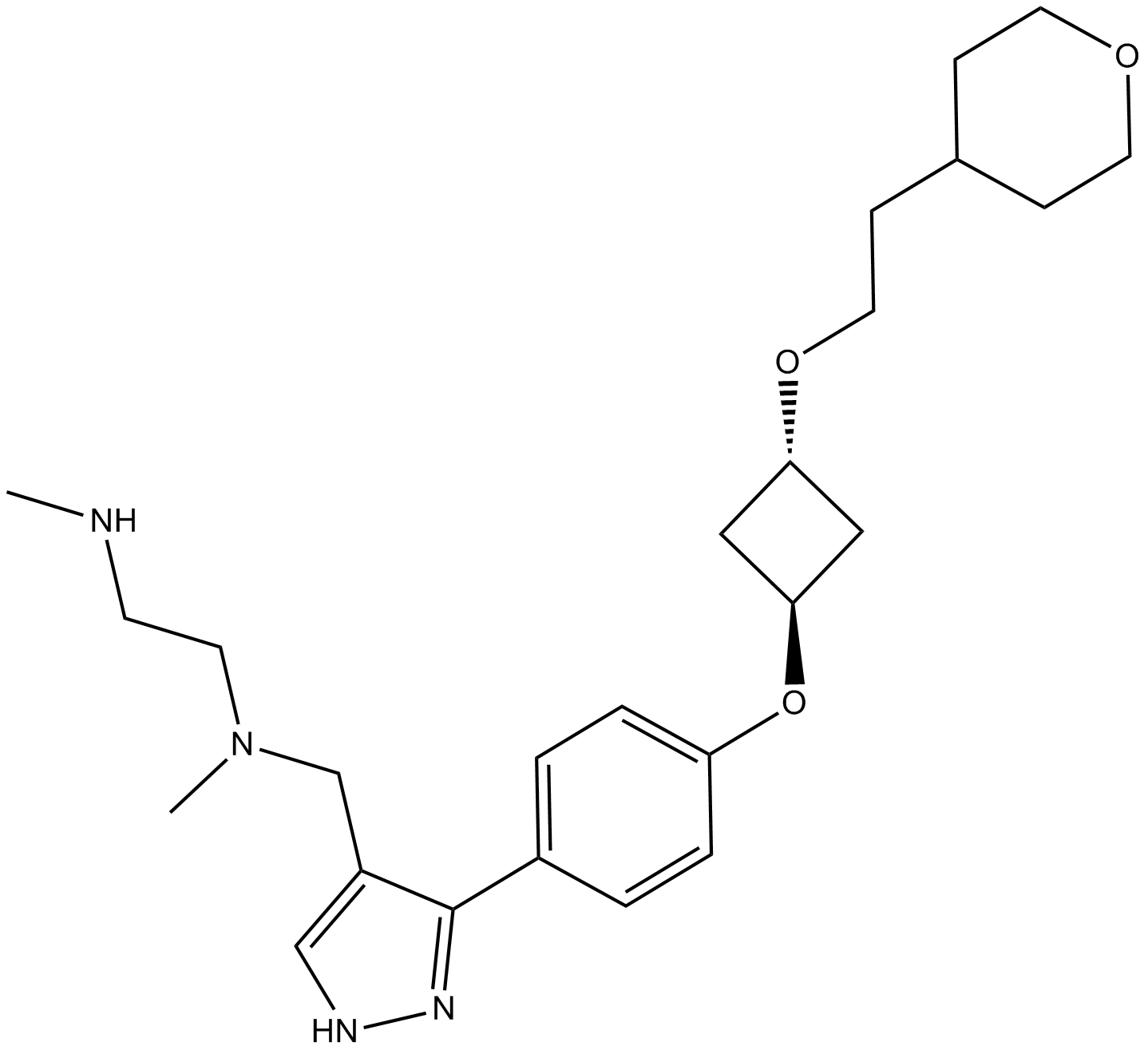
-
GC36000
EPZ020411 hydrochloride
EPZ020411 Hydrochlorid ist ein selektiver Inhibitor von PRMT6 mit einem IC50 von 10 nM, es hat eine >10-fache SelektivitÄt fÜr PRMT6 gegenÜber PRMT1 und PRMT8. EPZ020411 Hydrochlorid kann fÜr die Krebsforschung verwendet werden.
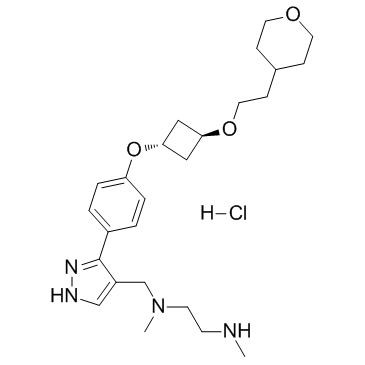
-
GC16224
EPZ031686
EPZ031686 ist ein potenter und oral aktiver SMYD3-Inhibitor mit einem IC50-Wert von 3 nM. EPZ031686 kann fÜr die Krebsforschung verwendet werden.
-
GC12932
EPZ5676
A highly potent DOT1L inhibitor
-
GC65980
EZH2-IN-13
EZH2-IN-13 ist ein potenter EZH2-Inhibitor, fÜr Details siehe Verbindung 73 im Patent WO2017139404. EZH2-IN-13 kann verwendet werden, um Krebserkrankungen oder prÄkanzerÖse LÄsionen im Zusammenhang mit EZH2-AktivitÄt zu untersuchen.
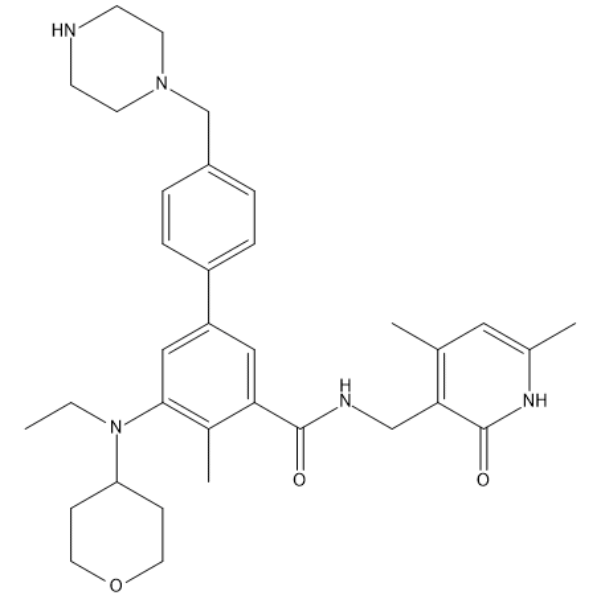
-
GC19149
EZM 2302
EZM 2302 ist ein Inhibitor der Coaktivator-assoziierten Arginin-Methyltransferase 1 (CARM1) mit einem IC50 von 6nM.
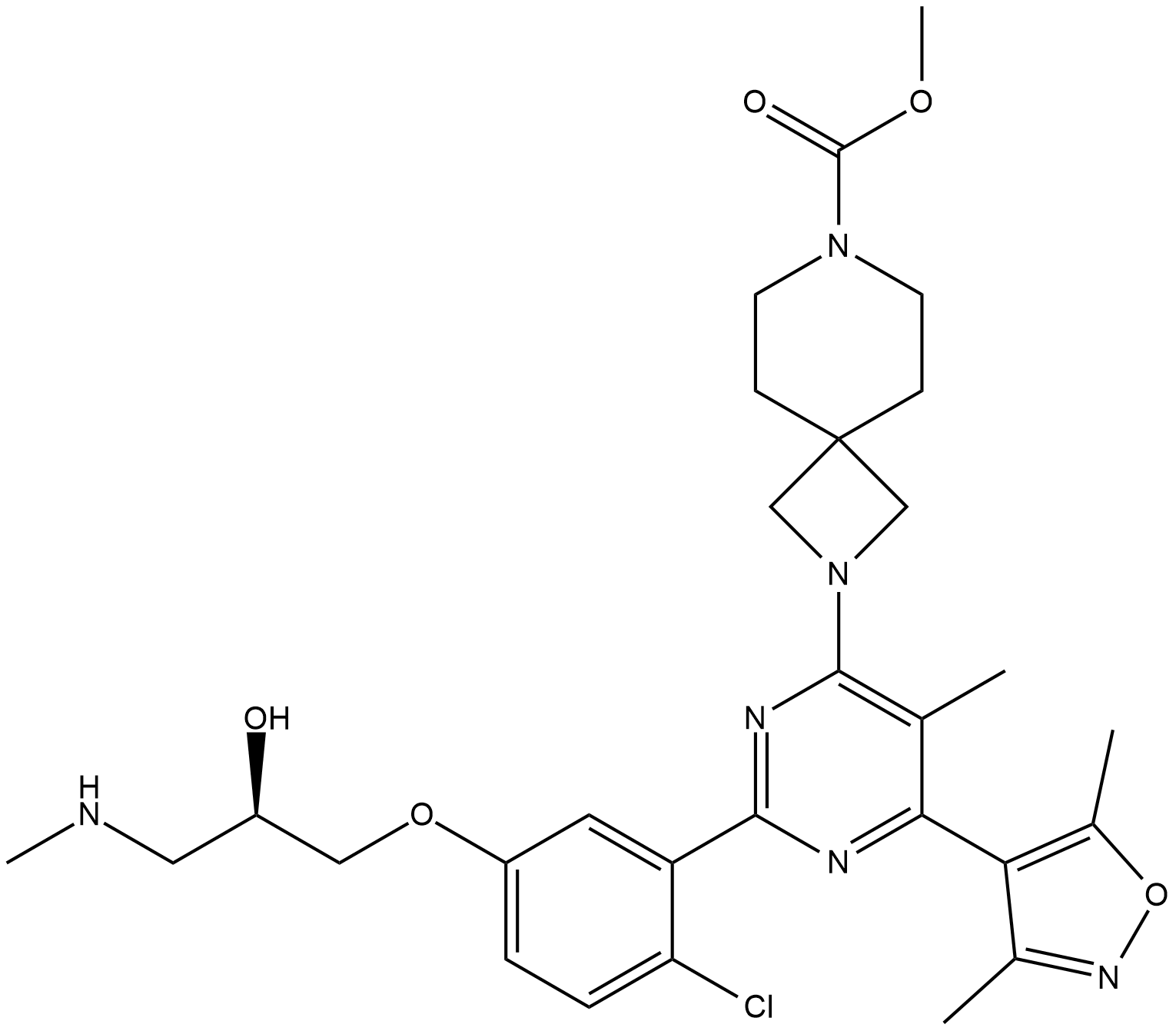
-
GC64900
EZM0414
EZM0414 ist ein potenter, selektiver, oral bioverfÜgbarer Inhibitor von SETD2 (IC50 = 18 nM im biochemischen SETD2-Assay; IC50 = 34 nM im zellulÄren Assay). EZM0414 kann fÜr die Erforschung des rezidivierten oder refraktÄren multiplen Myeloms und des diffusen großzelligen B-Zell-Lymphoms verwendet werden.
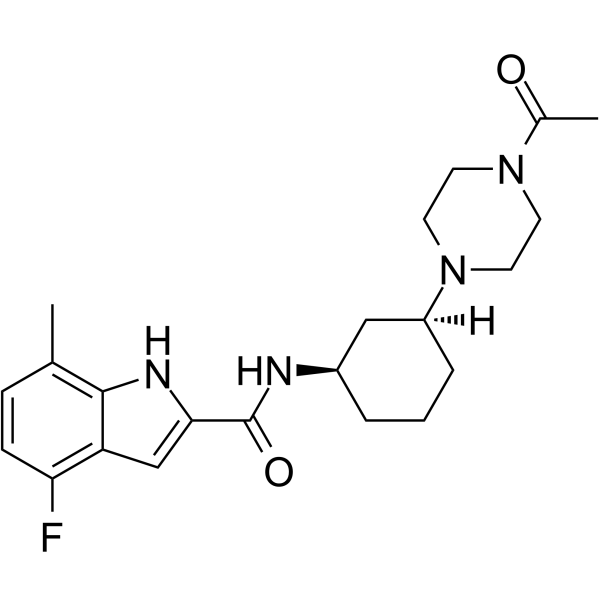
-
GC63545
FTX-6058
FTX-6058 ((S)-FTX-6058) ist ein potenter und oral aktiver Inhibitor der embryonalen Ektodermentwicklung (EED).
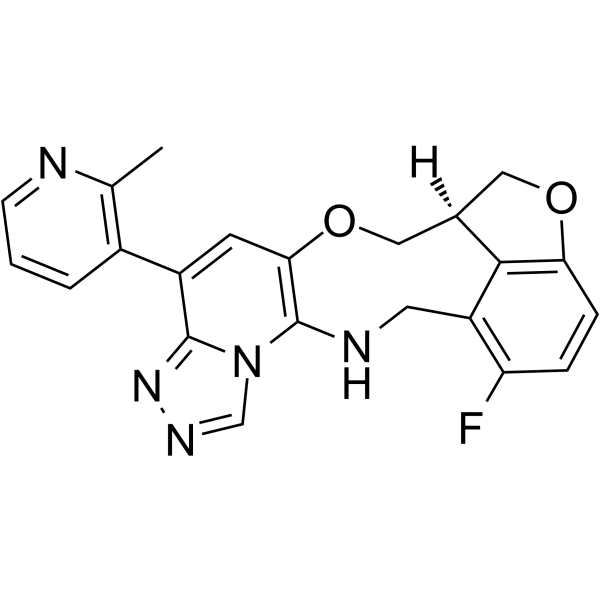
-
GC63546
FTX-6058 hydrochloride
(S)-Pociredir ((S)-FTX-6058) Hydrochlorid ist ein potenter und oral aktiver Inhibitor der embryonalen Ektodermentwicklung (EED).
-
GC43715
Furamidine (hydrochloride)
Furamidin (Hydrochlorid) (DB75-Dihydrochlorid) ist ein selektiver Inhibitor der Protein-Arginin-Methyltransferase 1 (PRMT1) mit einem IC50-Wert von 9,4 μM.
-
GC38062
Gambogenic acid
GambogensÄure ist ein Wirkstoff in Gamboge mit krebsbekÄmpfender Wirkung. GambogensÄure wirkt als wirksamer Inhibitor von EZH2, bindet spezifisch und kovalent an Cys668 innerhalb der EZH2-SET-DomÄne und induziert die EZH2-Ubiquitinierung.
-
GC36168
GNA002
GNA002 ist ein hochpotenter, spezifischer und kovalenter EZH2 (Enhancer of zeste homolog 2)-Inhibitor mit einem IC50 von 1,1 μM. GNA002 kann spezifisch und kovalent an Cys668 innerhalb der EZH2-SET-DomÄne binden und den EZH2-Abbau durch den COOH-Terminus der Hsp70-interagierenden Protein (CHIP)-vermittelten Ubiquitinierung auslÖsen. GNA002 reduziert effizient die EZH2-vermittelte H3K27-Trimethylierung und reaktiviert durch den Polycomb-Repressor-Komplex 2 (PRC2) stummgeschaltete Tumorsuppressorgene.
-
GC50697
GSK 591 dihydrochloride
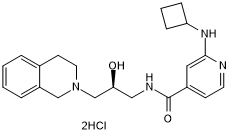
-
GC15783
GSK126
Ein selektiver EZH2-Inhibitor
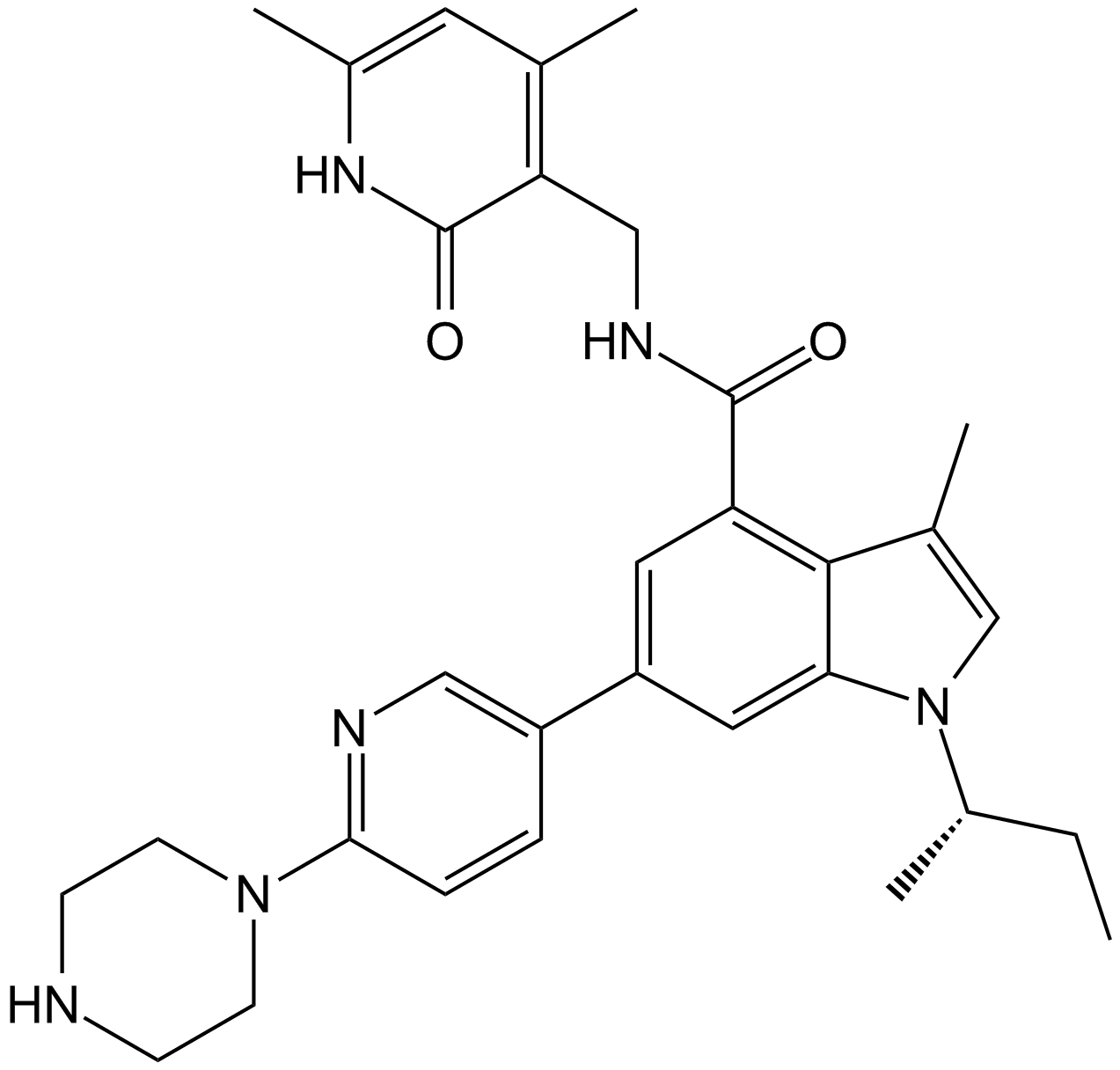
-
GC19181
GSK2807 Trifluoroacetate
GSK2807 Trifluoracetat ist ein potenter, selektiver und SAM-kompetitiver Inhibitor von SMYD3 mit einem Ki von 14 nM und einem IC50 von 130 nM.
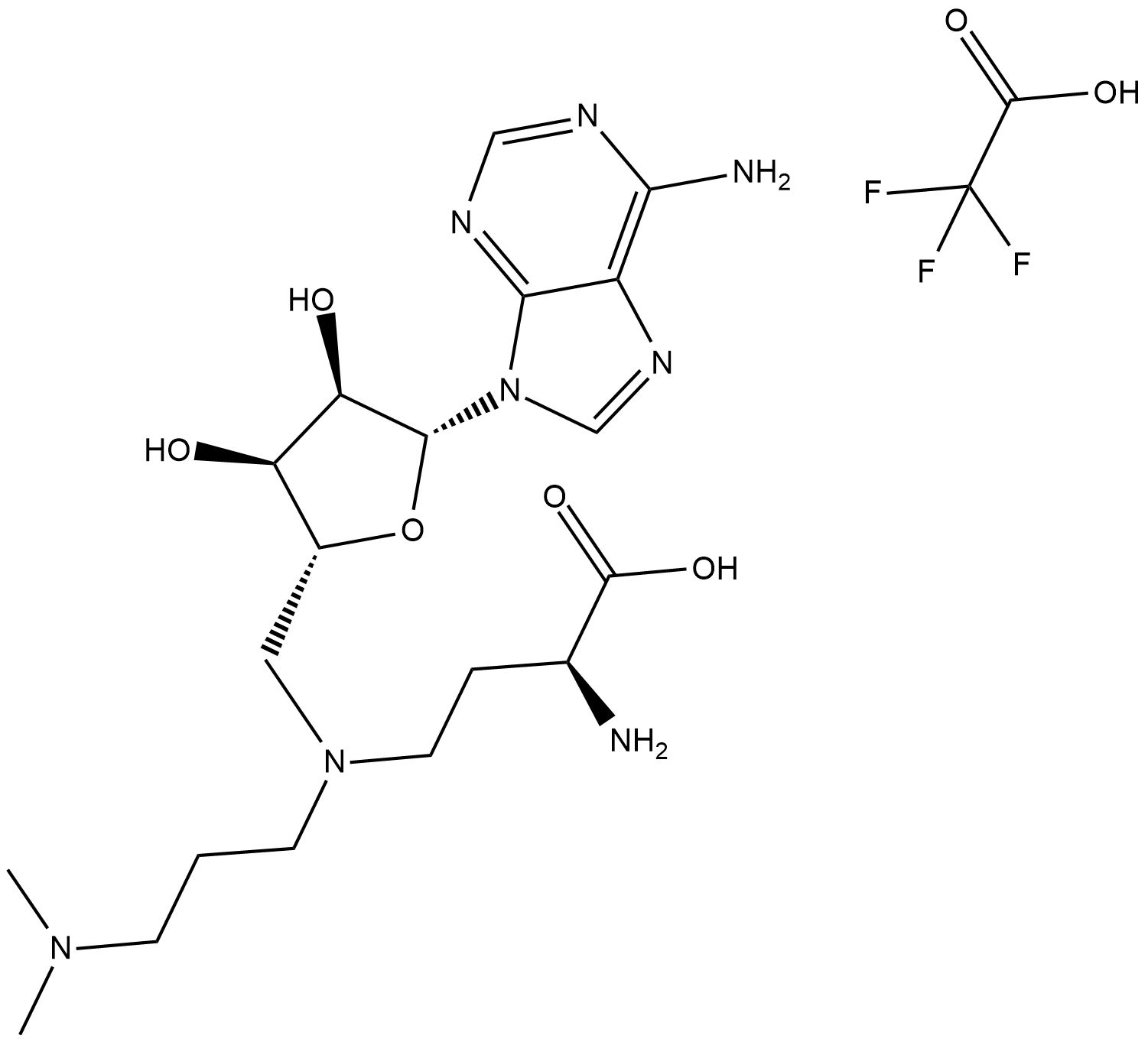
-
GC32693
GSK3326595 (EPZ015938)
GSK3326595 (EPZ015938) (EPZ015938) ist ein potenter, selektiver, reversibler Inhibitor der Protein-Arginin-Methyltransferase 5 (PRMT5) mit einem IC50 von 6,2 nM.
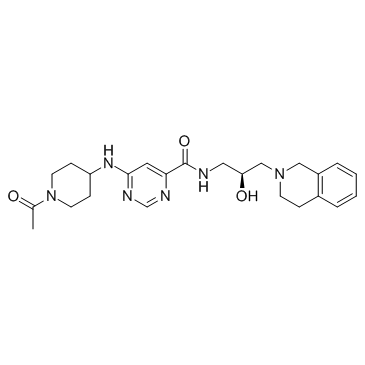
-
GC36191
GSK3368715
GSK3368715 (EPZ019997) ist ein oral aktiver, reversibler und S-Adenosyl-L-Methionin (SAM) nicht kompetitiver Typ-I-Protein-Arginin-Methyltransferase (PRMTs)-Inhibitor (IC50=3,1 nM (PRMT1), 48 nM (PRMT3), 1148 nM ( PRMT4), 5,7 nM (PRMT6), 1,7 nM (PRMT8)). GSK3368715 (EPZ019997) bewirkt eine Verschiebung der Arginin-MethylierungszustÄnde, verÄndert die Exon-Nutzung und hat eine starke Anti-Krebs-AktivitÄt.
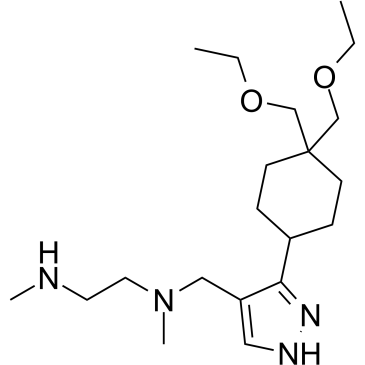
-
GC49398
GSK3368715 (hydrochloride)
An inhibitor of type I PRMTs
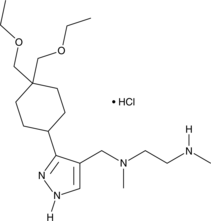
-
GC36192
GSK3368715 dihydrochloride
GSK3368715 Dihydrochlorid (EPZ019997 Dihydrochlorid) ist ein oral aktiver, reversibler und S-Adenosyl-L-Methionin (SAM) nicht kompetitiver Inhibitor der Typ-I-Protein-Arginin-Methyltransferase (PRMTs) (IC50=3,1 nM (PRMT1), 48 nM (PRMT3), 1148 nM (PRMT4), 5,7 nM (PRMT6), 1,7 nM (PRMT8)). GSK3368715-Dihydrochlorid (EPZ019997-Dihydrochlorid) erzeugt eine Verschiebung der Arginin-MethylierungszustÄnde, verÄndert die Exon-Nutzung und hat eine starke Anti-Krebs-AktivitÄt.
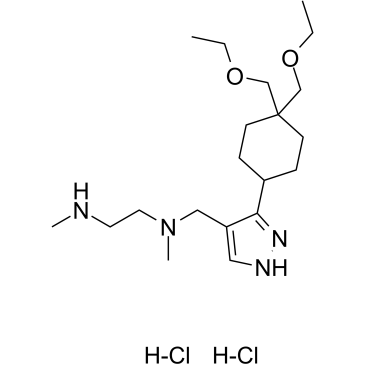
-
GC17534
GSK343
A selective, cell-permeable EZH2 inhibitor
-
GC11414
GSK503
GSK503 ist ein potenter und spezifischer Inhibitor der EZH2-Methyltransferase mit Kiapp-Werten von 3 bis 27 nM.