PROTAC
PROTACs or Proteolysis Targeting Chimeric Molecules are heterobifunctional nanomolecules that theoretically target any protein for ubiquitination and degradation. In terms of the structure, PROTACs consist of one moiety which is recognized by the E3 ligase. This moiety is then chemically and covalently linked to a small molecule or a protein that recognizes the target protein. The trimeric complex formation leads to the transfer of ubiquitins to the target protein.
By removing target proteins directly rather than merely blocking them, PROTACs can provide multiple advantages over small molecule inhibitors, which can require high systemic exposure to achieve sufficient inhibition, often resulting in toxic side effects and eventual drug resistance. PROTAC molecules possess good tissue distribution and the ability to target intracellular proteins, thus can be directly applied to cells or injected into animals without the use of vectors.
Targeted protein degradation using the PROTAC technology is emerging as a novel therapeutic method to address diseases, such as cancer, driven by the aberrant expression of a disease-causing protein. In addition to the use of PROTACs for the treatment of human disease, these molecules provide a chemical genetic approach to “knock down” proteins to study their function. Currently, there are several small molecule inhibitors that have been found to show good biological activity by specifically targeting BET, estrogen receptor (ER), androgen receptor, etc.
References:
[1] Sakamoto KM. Pediatr Res. 2010 May;67(5):505-8.
[2] Neklesa TK, et al. Pharmacol Ther. 2017 Jun;174:138-144.
Ziele für PROTAC
Produkte für PROTAC
- Bestell-Nr. Artikelname Informationen
-
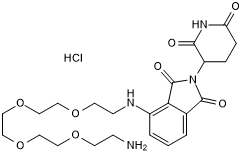
GC68352
(S,R,S)-AHPC-C2-amide-benzofuranylmethyl-pyridine
-
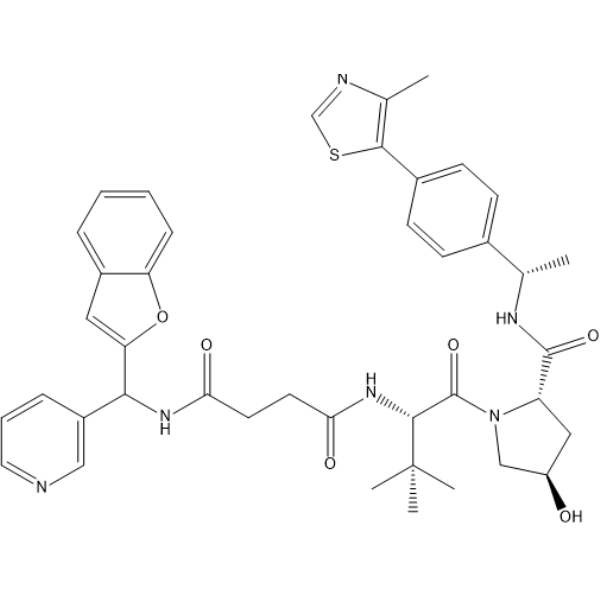
GC60009
(S,R,S)-AHPC-PEG5-COOH
(S,R,S)-AHPC-PEG5-COOH (VH032-PEG5-COOH) ist ein synthetisiertes E3-Ligase-Ligand-Linker-Konjugat, das den (S,R,S)-AHPC-basierten VHL-Liganden und einen 5-Einheiten-PEG-Linker enthÄlt in der PROTAC-Technologie verwendet.
-
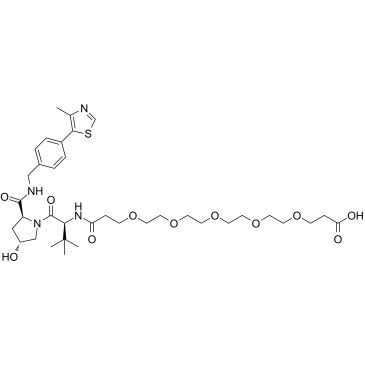
GC50469
A 410099.1 amide-alkylC4-amine
A 410099.1 amide-alkylC4-amine
-
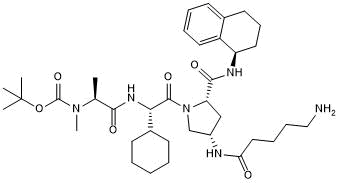
GC50468
A 410099.1 amide-PEG2-amine
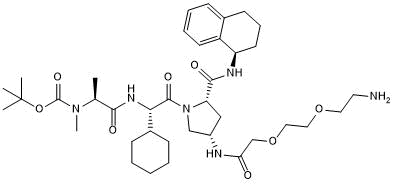
A 410099.1 amide-PEG2-amine
-
GC50467
A 410099.1 amide-PEG3-amine
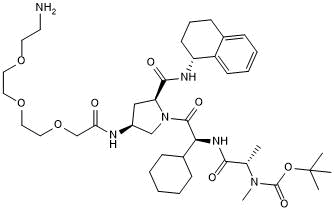
A 410099.1 amide-PEG3-amine
-
GC50737
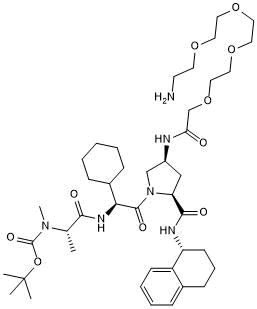
A 410099.1 amide-PEG4-amine
-
GC50739
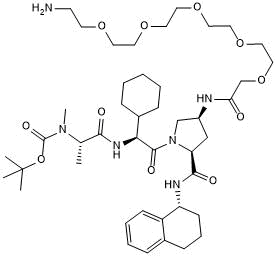
A 410099.1 amide-PEG5-amine
-
GC33280
A1874
A1874 ist ein Nutlin-basiertes (MDM2-Ligand) und BRD4-abbauendes PROTAC mit einem DC50 von 32 nM (induziert BRD4-Abbau in Zellen). Wirksam bei der Hemmung der Proliferation vieler Krebszelllinien.
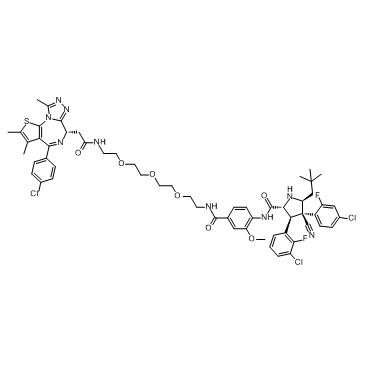
-
GC35227
ACBI1
ACBI1 ist ein potenter und kooperativer SMARCA2-, SMARCA4- und PBRM1-Abbaustoff mit DC50-Werten von 6, 11 bzw. 32 nM. ACBI1 ist ein PROTAC-Degrader. ACBI1 zeigt antiproliferative AktivitÄt. ACBI1 induziert Apoptose.
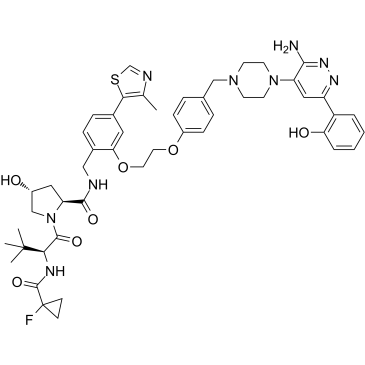
-
GC50744
ARCC 4 negative control
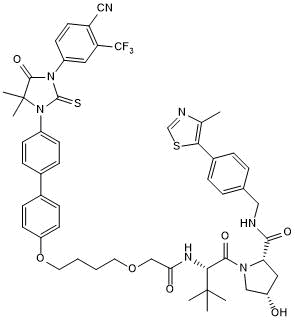
-
GC60594
ARCC-4
ARCC-4 ist ein niedrig-nanomolarer Androgenrezeptor (AR)-Abbaustoff auf Basis von PROTAC mit einem DC50 von 5nM. ARCC-4 ist ein auf Enzalutamid basierendes von Hippel-Lindau (VHL)-rekrutierendes AR PROTAC und Übertrifft Enzalutamid. ARCC-4 baut klinisch relevante AR-Mutanten, die mit einer Antiandrogentherapie assoziiert sind, effektiv ab.
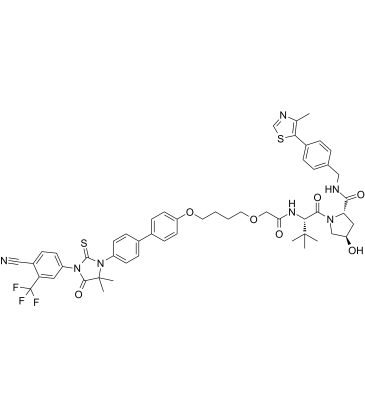
-
GC63492
ARD-2128
ARD-2128 ist ein hochwirksamer, oral bioverfÜgbarer PROTAC-Androgenrezeptor (AR)-Abbaustoff. ARD-2128 reduziert effektiv das AR-Protein, unterdrÜckt AR-regulierte Gene in Tumorgeweben und hemmt das Tumorwachstum ohne Anzeichen von ToxizitÄt. ARD-2128 hat das Potenzial fÜr die Erforschung des Prostatakrebses.
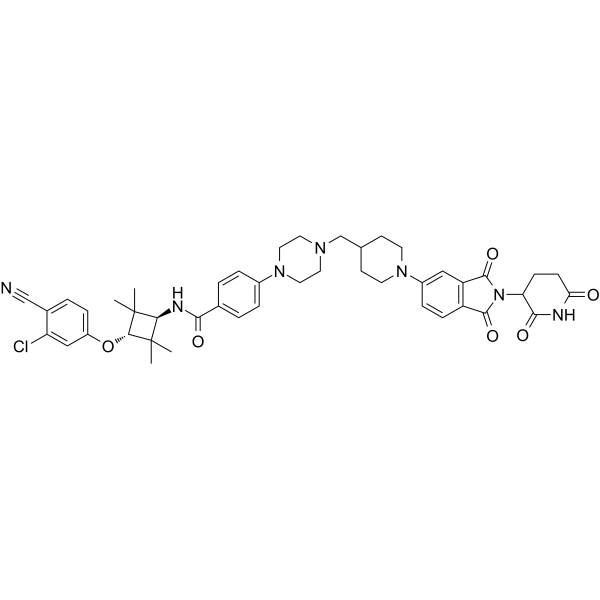
-
GC63704
ARD-2585
ARD-2585 ist ein außergewÖhnlich starker und oral aktiver PROTAC-Abbauer des Androgenrezeptors.
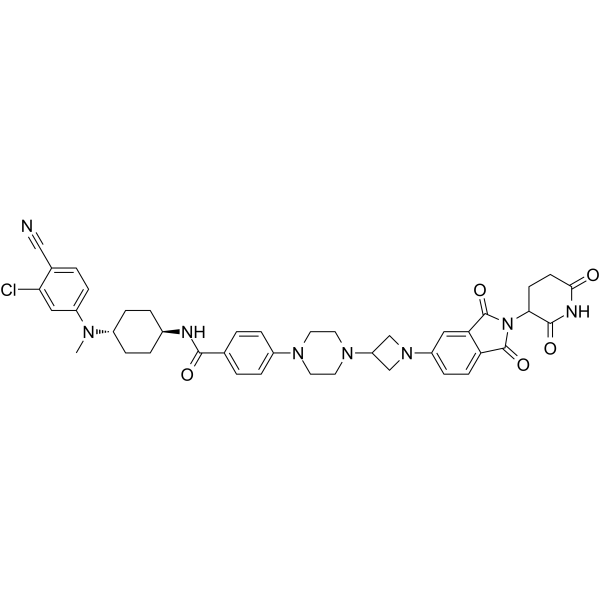
-
GC32685
ARV-771
ARV-771 ist ein potenter BET-PROTAC basierend auf E3-Ligase von Hippel-Lindau mit Kds von 34 nM, 4,7 nM, 8,3 nM, 7,6 nM, 9,6 nM und 7,6 nM fÜr BRD2(1), BRD2(2), BRD3( 1), BRD3(2), BRD4(1) bzw. BRD4(2).
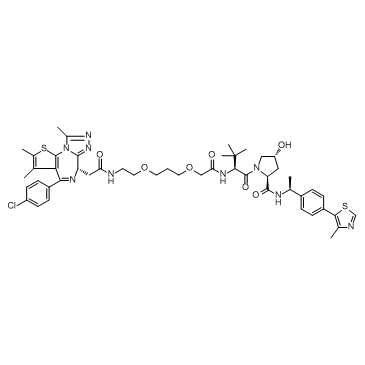
-
GC19038
ARV-825
ARV-825 ist ein PROTAC, das durch Liganden fÜr Cereblon und BRD4 verbunden ist. ARV-825 bindet an BD1 und BD2 von BRD4 mit Kds von 90 bzw. 28 nM.
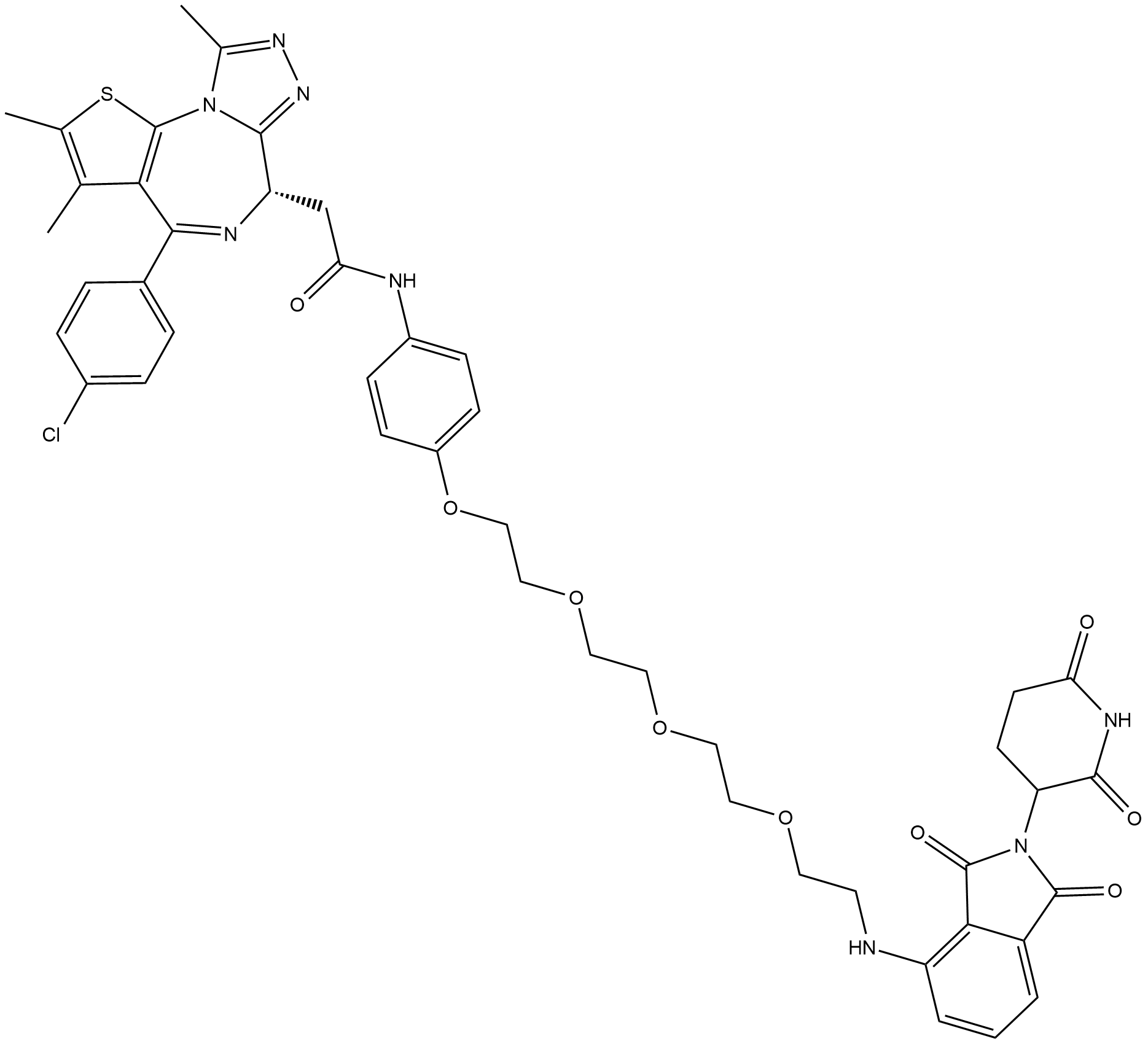
-
GC33354
AT6
AT6 ist ein PROTAC AT1-Analogon, das ein PROTAC ist, das durch Liganden fÜr von Hippel-Lindau und BRD4 mit hoher SelektivitÄt fÜr die BromodomÄne (Brd4) verbunden ist.
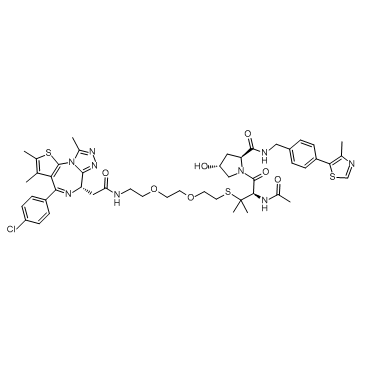
-
GC50630
aTAG 2139
Degrader of MTH1 fusion proteins for use within the aTAG system

-
GC50631
aTAG 4531
Degrader of MTH1 fusion proteins for use within the aTAG system
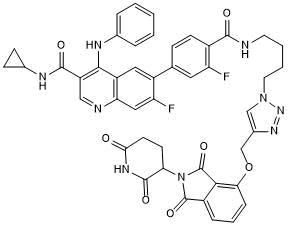
-
GC65506
BETd-246
BETd-246 ist ein PROTAC-basierter BET-BromdomÄnen(BRD)-Inhibitor der zweiten Generation, der durch Liganden fÜr Cereblon und BET verbunden ist und eine Überlegene SelektivitÄt, Wirksamkeit und AntitumoraktivitÄt aufweist.
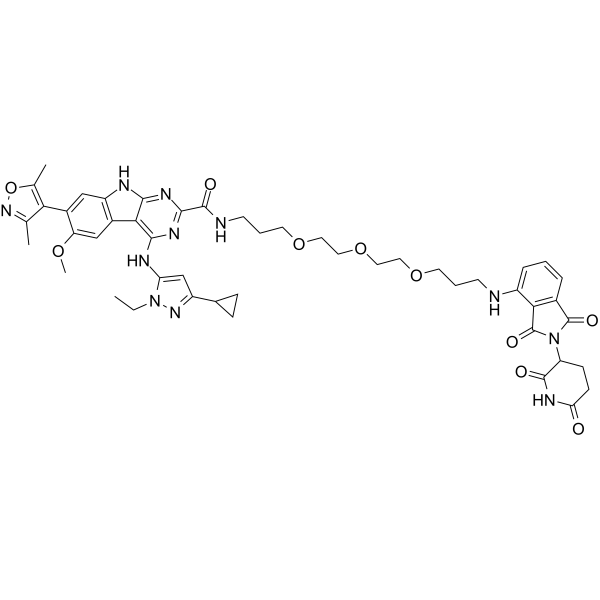
-
GC32791
BETd-260 (ZBC 260)
BETd-260 (ZBC 260) (ZBC 260) ist ein PROTAC, das durch Liganden fÜr Cereblon und BET verbunden ist, mit nur 30 pM gegen BRD4-Protein in der RS4;11-LeukÄmie-Zelllinie. BETd-260 (ZBC 260) unterdrÜckt wirksam die ZelllebensfÄhigkeit und induziert robust Apoptose in hepatozellulÄren Karzinomzellen (HCC).
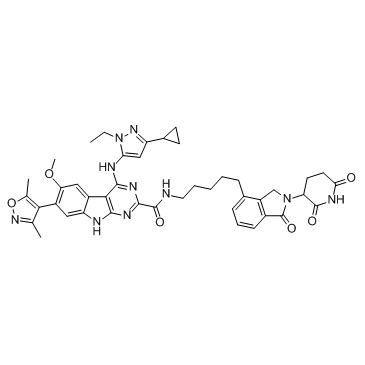
-
GC65457
BI-3663
BI-3663 ist ein hochselektives PTK2/FAK-PROTAC (DC50=30 nM) mit Cereblon-Liganden, um E3-Ligasen fÜr den PTK2-Abbau zu entfÜhren. BI-3663 hemmt PTK2 mit einem IC50 von 18 nM. BI-3663 ist ein PROTAC, das aus BI-4464 besteht, das mit einem Linker an Pomalidomid gebunden ist. Anti-Krebs-AktivitÄt.
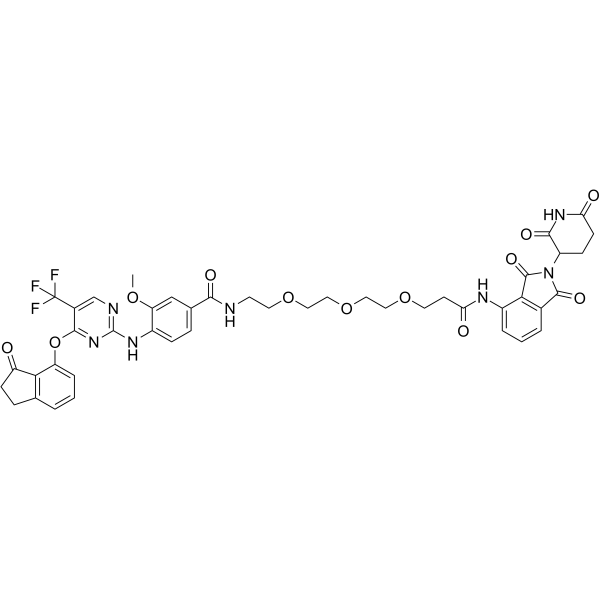
-
GC33017
BRD4 degrader AT1
BRD4-Degrader AT1 ist ein PROTAC, verbunden durch Liganden fÜr von Hippel-Lindau und BRD4 als ein hochselektiver Brd4-Degrader, mit einem Kd von 44 nM fÜr Brd4BD2 in Zellen.
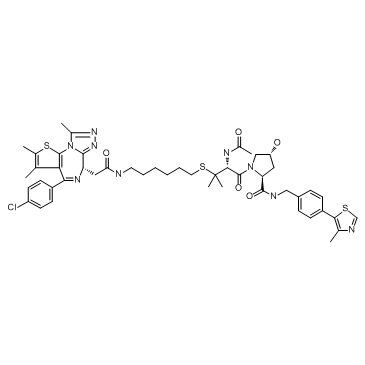
-
GC65128
BSJ-03-123
BSJ-03-123 ist ein PROTAC, das durch Liganden fÜr Cereblon und CDK als potenter und neuartiger CDK6-selektiver niedermolekularer Abbauer verbunden ist.
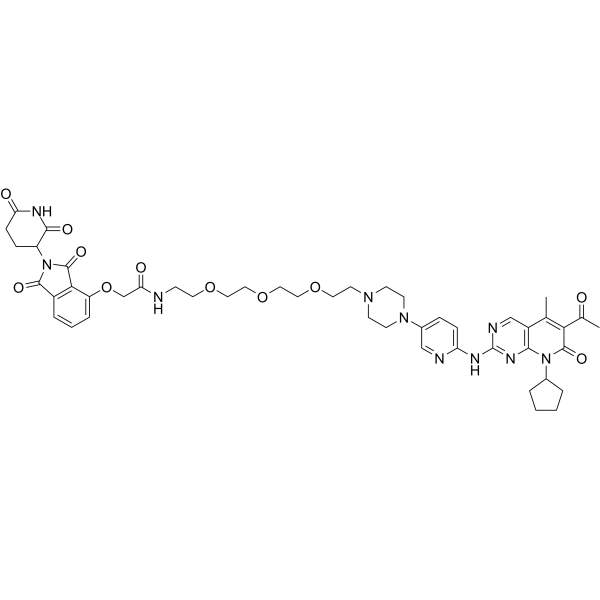
-
GC50615
BSJ-03-204
BSJ-03-204 ist ein PROTAC, das durch Liganden fÜr Cereblon und CDK verbunden ist. BSJ-03-204 ist ein potenter und selektiver CDK4/6-Doppelabbauer (PROTAC) auf Palbociclib-Basis mit IC50-Werten von 26,9 nM und 10,4 nM fÜr CDK4/D1 bzw. CDK6/D1. BSJ-03-204 induziert keinen IKZF1/3-Abbau und hat Anti-Krebs-AktivitÄt.
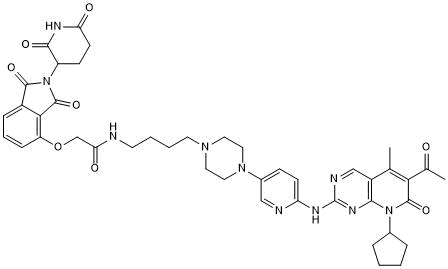
-
GC50614
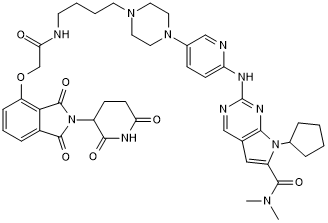
BSJ-04-132
BSJ-04-132 ist ein PROTAC, das durch Liganden fÜr Cereblon und CDK verbunden ist. BSJ-04-132 ist ein potenter und selektiver Ribociclib-basierter CDK4-Abbaustoff (PROTAC) mit IC50-Werten von 50,6 nM und 30 nM fÜr CDK4/D1 bzw. CDK6/D1. BSJ-04-132 induziert keinen Abbau von CDK6 und IKZF1/3. BSJ-04-132 hat Anti-Krebs-AktivitÄt.
-
GC50610
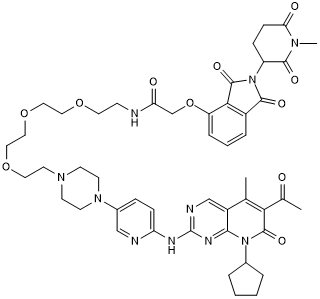
BSJ-Bump
Negative control for BSJ-03-123
-
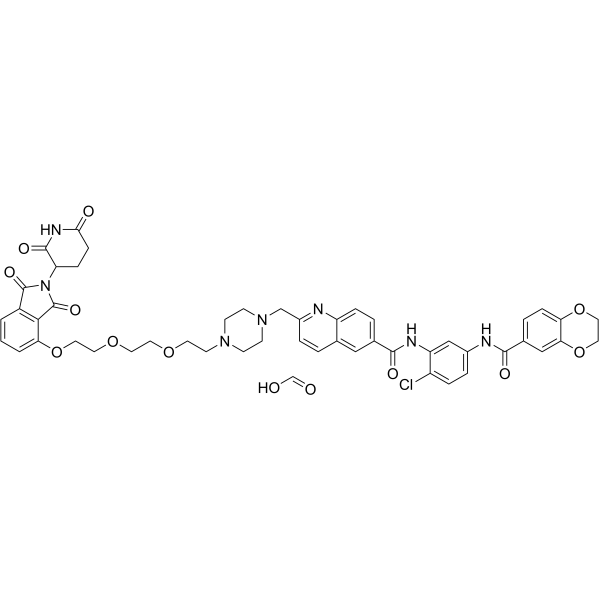
GC67861
CCT367766 formic
-
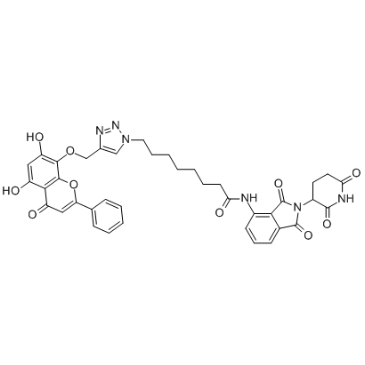
GC35635
CDK9 Antagonist-1
-
GC50363
cis MZ 1
Negative Control for MZ 1
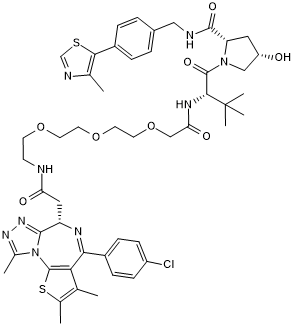
-
GC50629
cis VH 032, amine dihydrochloride
Negative control for VH 032, amine
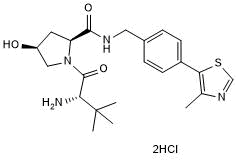
-
GC50616
cis-VZ 185
Negative control for VZ 185
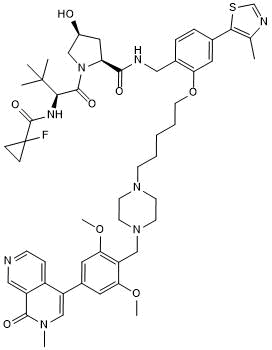
-
GC35739
CP-10
CP-10 ist ein PROTAC, verbunden durch Liganden fÜr Cereblon und CDK, mit hochselektivem, spezifischem und bemerkenswertem CDK6-Abbau (DC50=2,1 nM). Es hemmt die Proliferation mehrerer hÄmatopoetischer Krebszellen mit beeindruckender Potenz, einschließlich des multiplen Myeloms, und kann immer noch mutiertes und Überexprimiertes CDK6 abbauen.
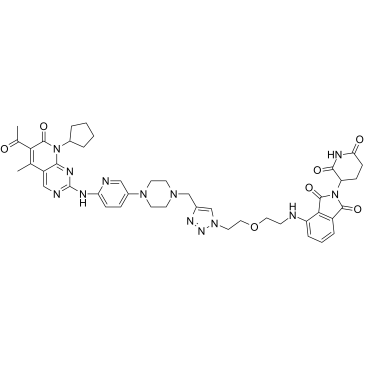
-
GC50619
CRBN-6-5-5-VHL
CRBN-6-5-5-VHL ist ein potenter und selektiver Abbauer von Cereblon (CRBN) auf der Basis von Hippel-Lindau mit einem DC50-Wert von 1,5 nM. CRBN-6-5-5-VHL hat fast keinen Einfluss auf den Abbau der Neo-Substrate IKZF1 und IKZF3.
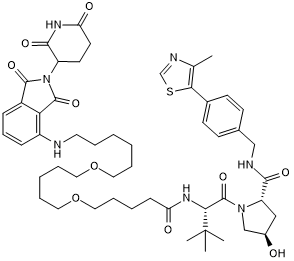
-
GC66361
DB-0646
DB-0646, ein PROTAC, ist ein Multi-Kinase-Abbauer.
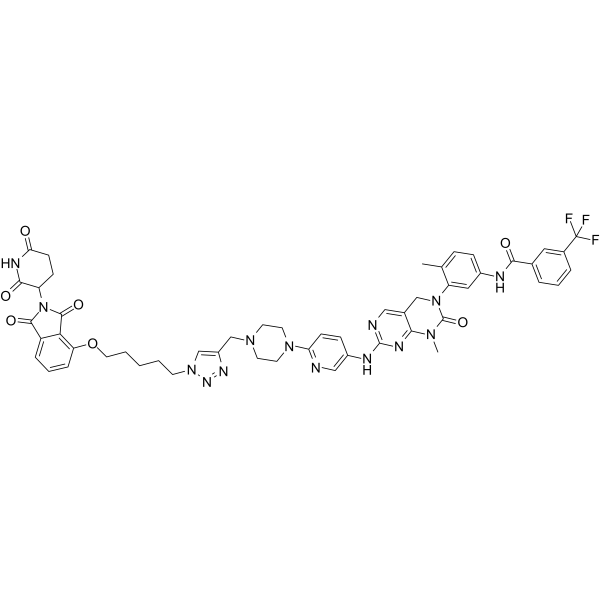
-
GC19119
dBET1
dBET1 ist ein PROTAC, verbunden durch Liganden fÜr Cereblon und BRD4 mit einem EC50 von 430 nM. dBET1 ist ein PROTAC, das aus (+)-JQ1 besteht, das mit einem Linker mit NSC 527179 verknÜpft ist.
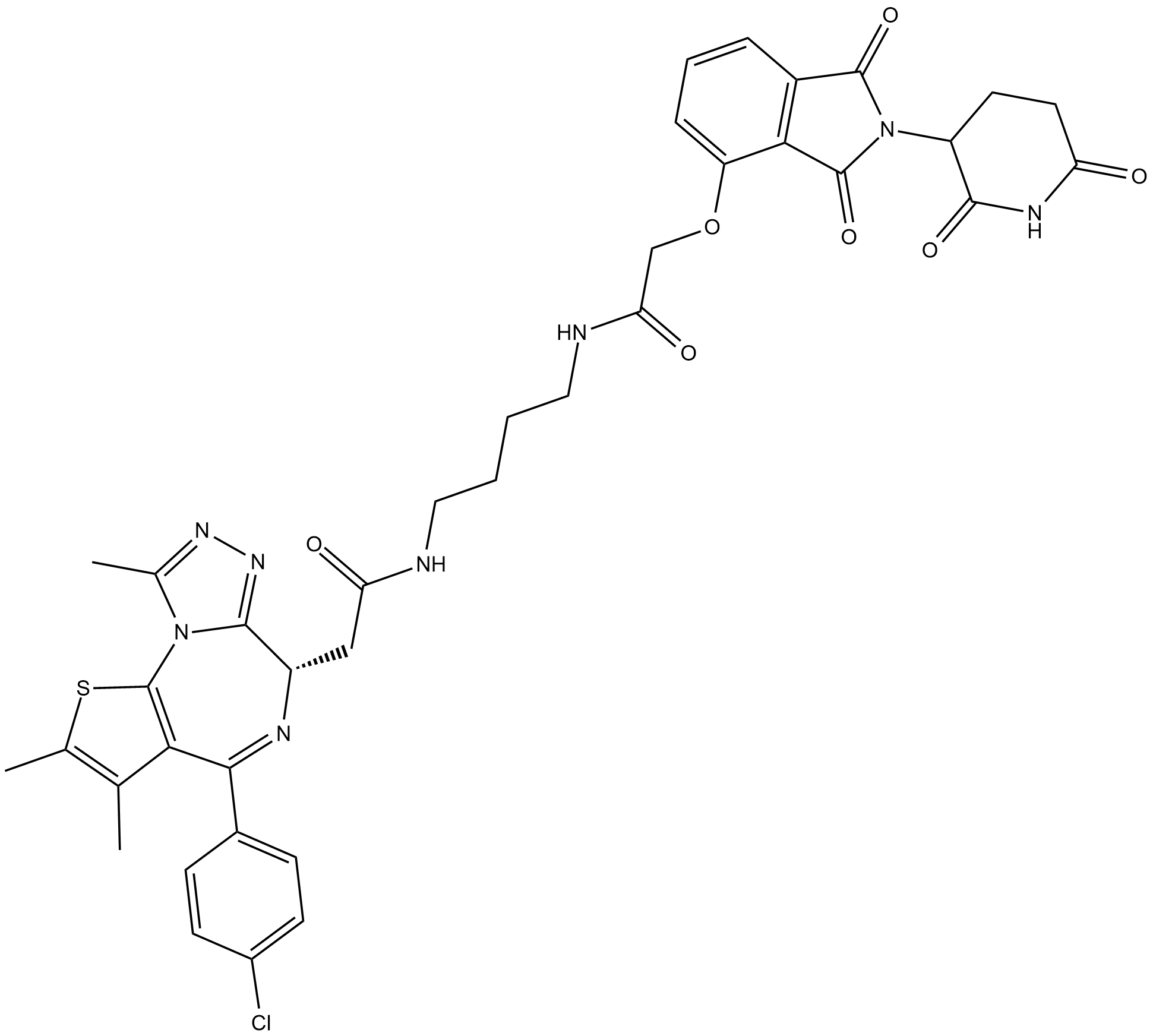
-
GC63445
dBET23
dBET23 ist ein hochwirksamer und selektiver PROTAC BRD4-Abbauer mit einem DC50/5h von ~ 50 nM fÜr BRD4BD1-Protein.
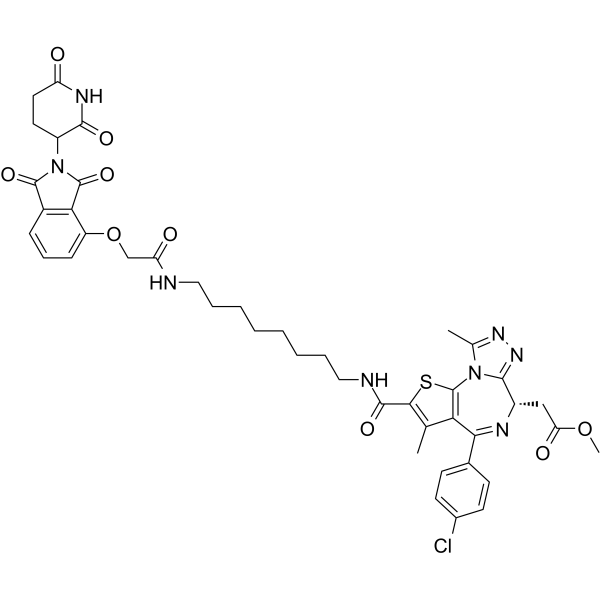
-
GC35815
dBET57
dBET57 ist ein potenter und selektiver Abbauer von BRD4BD1 auf Basis der PROTAC-Technologie. dBET57 vermittelt die Rekrutierung an die CRL4Cereblon E3-Ubiquitinligase mit einem DC50/5h von 500 nM fÜr BRD4BD1 und ist auf BRD4BD2 inaktiv.
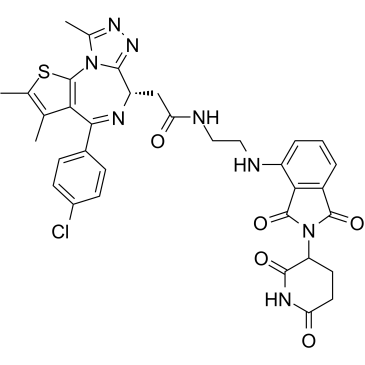
-
GC32719
dBET6
dBET6 ist ein hochpotentes, selektives und zellgÄngiges PROTAC, das durch Liganden fÜr Cereblon und BET verbunden ist, mit einem IC50 von 14 nM und AntitumoraktivitÄt hat.
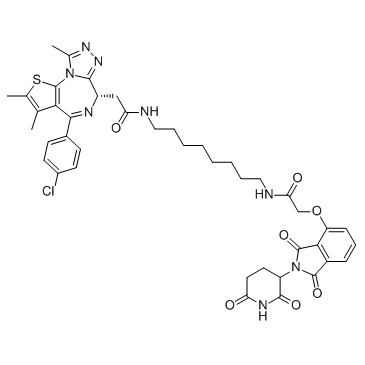
-
GC50518
dBRD9
Potent and selective BRD9 degrading PROTAC
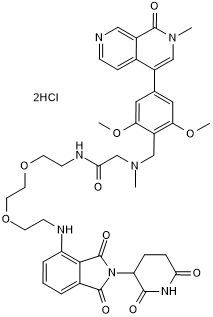
-
GC50736
dBRD9-A
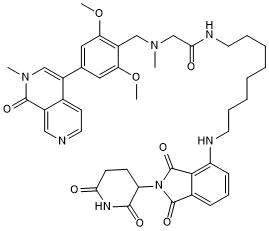
-
GC50749
DD 03-171
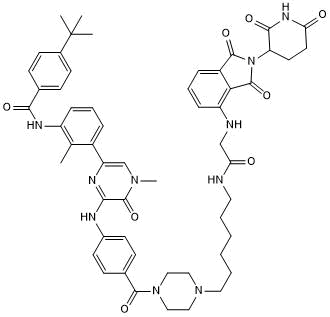
-
GC35882
dMCL1-2
dMCL1-2 ist ein potenter und selektiver PROTAC von myeloid cell leukemia 1 (MCL1) (Mitglied der Bcl-2-Familie) auf der Basis von Cereblon, das mit einer KD von 30 nM an MCL1 bindet. dMCL1-2 aktiviert die zellulÄre Apoptosemaschinerie durch Abbau von MCL1.
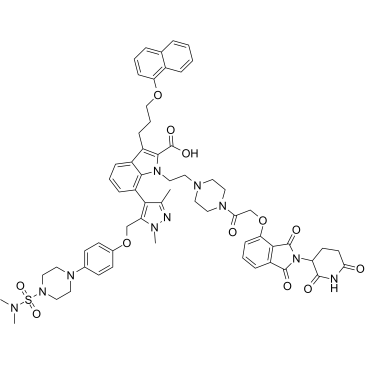
-
GC62942
DP-C-4
DP-C-4 ist ein Cereblon-basiertes duales PROTAC zum gleichzeitigen Abbau von EGFR und PARP.
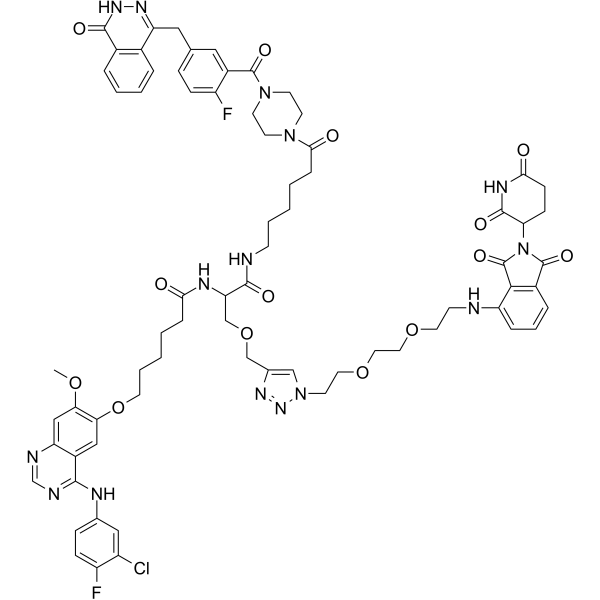
-
GC50754
dTAG-13-NEG
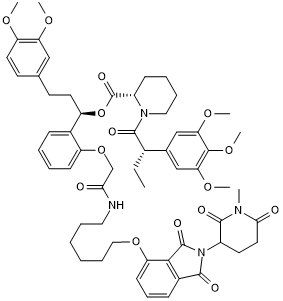
-
GC50756
dTAGV-1
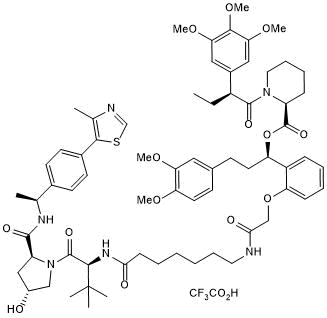
-
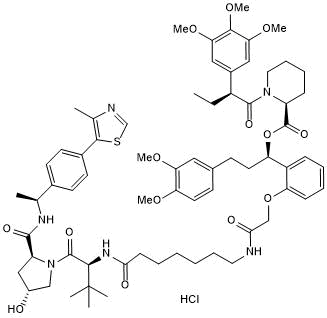
GC50757
dTAGV-1 hydrochloride
-
GC67914
dTAGV-1 TFA

-
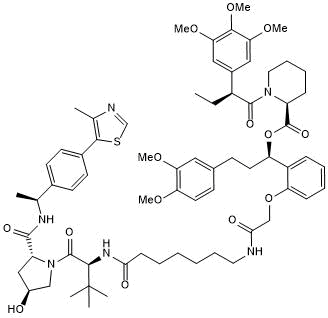
GC50755
dTAGV-1-NEG
dTAGV-1-NEG ist ein Diastereomer und als heterobifunktionelle Negativkontrolle von dTAGV-1. dTAGV-1 ist ein FKBP12F36V-selektiver Abbauer.
-
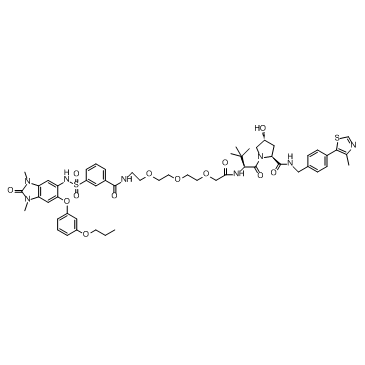
GC35904
dTRIM24
dTRIM24 ist ein selektiver bifunktionaler Abbauer von TRIM24 auf Basis von PROTAC, bestehend aus Liganden fÜr von Hippel-Lindau und TRIM24.
-
GC32902
E3 ligase Ligand-Linker Conjugates 10
E3-Ligase-Ligand-Linker-Konjugate 10 (VH032-PEG2-C4-Cl) ist ein Konjugat aus Liganden fÜr E3 und einem 13 Atom langen Linker. Der Verbinder des Linkers ist eine Halogengruppe. E3-Ligase-Ligand-Linker-Konjugate 10 enthalten den (S,R,S)-AHPC-basierten VHL-Liganden und einen Alkyl/Ether-basierten Linker. E3-Ligase-Ligand-Linker-Konjugate 10 kÖnnen den Abbau von GFP-HaloTag7 in zellbasierten Assays induzieren.
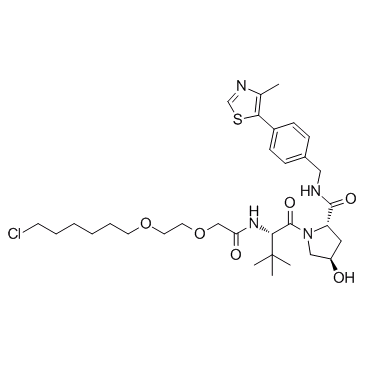
-
GC32987
E3 ligase Ligand-Linker Conjugates 8
E3-Ligase-Ligand-Linker-Konjugate 8 (VH032-C6-PEG3-C4-Cl) ist ein Konjugat aus Liganden fÜr E3 und einem Linker mit einer LÄnge von 20 Atomen. Der Verbinder des Linkers ist eine Halogengruppe. E3-Ligase-Ligand-Linker-Konjugate 8 enthalten den (S,R,S)-AHPC-basierten VHL-Liganden und einen Alkyl/Ether-basierten Linker. E3-Ligase-Ligand-Linker-Konjugate 8 kÖnnen den Abbau von GFP-HaloTag7 in zellbasierten Assays induzieren.
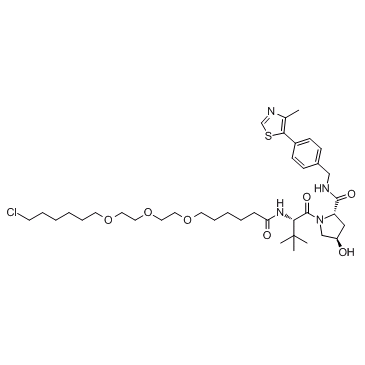
-
GC32976
E3 ligase Ligand-Linker Conjugates 9
E3-Ligase-Ligand-Linker-Konjugate 9 ist ein Konjugat aus Liganden fÜr E3 und einem Linker mit einer LÄnge von 25 Atomen. Der Verbinder des Linkers ist eine Halogengruppe. E3-Ligase-Ligand-Linker-Konjugate 9 enthalten den (S,R,S)-AHPC-basierten VHL-Liganden und den 6-Einheiten-PEG-Linker. E3-Ligase-Ligand-Linker-Konjugate 9 kÖnnen den Abbau von GFP-HaloTag7 in zellbasierten Assays induzieren.
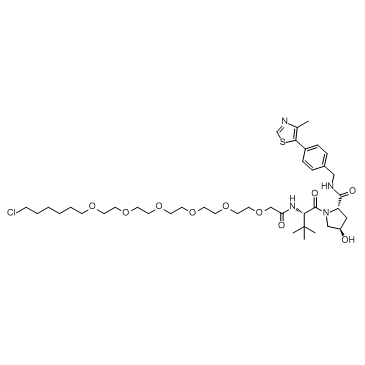
-
GC36001
ERD-308
ERD-308 ist ein hochwirksamer von Hippel-Lindau-basierter PROTAC-Degrader des Östrogenrezeptors (ER) fÜr die Behandlung von ER-positivem Brustkrebs. ERD-308 induziert >95 % des ER-Abbaus bei so niedrigen Konzentrationen wie 5 nM in beiden Zelllinien (DC50 (Konzentration, die 50 % des Proteinabbaus verursacht) von 0,17 nM und 0,43 nM in MCF-7- und T47D-ER+-Zellen , beziehungsweise).
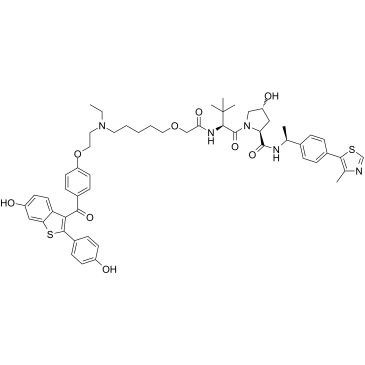
-
GC39264
FKBP12 PROTAC dTAG-13
FKBP12 PROTAC dTAG-13 (dTAG-13), ein PROTAC-basierter heterobifunktioneller Abbauer, ist ein selektiver Abbauer von FKBP12F36V mit Expression von FKBP12F36V im Leserahmen mit einem interessierenden Protein. FKBP12 PROTAC dTAG-13 greift effektiv in FKBP12F36V und CRBN ein, wodurch FKBP12F36V selektiv abgebaut wird.
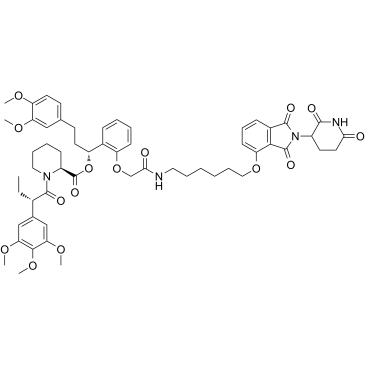
-
GC39289
FKBP12 PROTAC dTAG-7
FKBP12 PROTAC dTAG-7 (dTAG-7) ist ein heterobifunktioneller Abbauer. FKBP12 PROTAC dTAG-7 (dTAG-7) ist ein Abbauer von FKBP12F36V mit Expression von FKBP12F36V im Leserahmen mit einem interessierenden Protein. FKBP12 PROTAC dTAG-7 (dTAG-7) ist auch ein selektiver Abbauer des BET-BromodomÄnen-Transkriptions-Co-Aktivators BRD4, indem BET-BromodomÄnen mit einer E3-Ubiquitin-Ligase CRBN verbrÜckt werden.
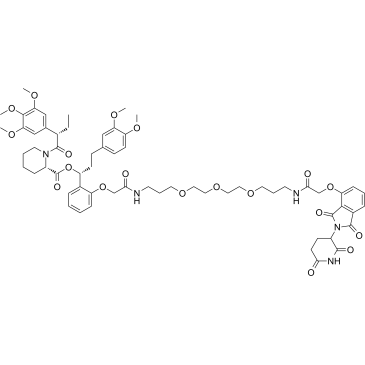
-
GC19509
Gefitinib-based PROTAC 3
Gefitinib-basiertes PROTAC 3, das ein EGFR-Bindungselement Über einen Linker an einen von Hippel-Lindau-Liganden konjugiert, induziert den EGFR-Abbau mit DC50s von 11,7 nM und 22,3 nM in HCC827 (Exon 19 del) bzw. H3255 (L858R-Mutation)-Zellen.
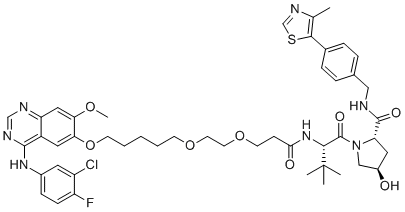
-
GC36167
GMB-475
GMB-475 ist ein auf PROTAC basierender Abbauer der BCR-ABL1-Tyrosinkinase, der die BCR-ABL1-abhÄngige Arzneimittelresistenz Überwindet. GMB-475 zielt auf das BCR-ABL1-Protein ab und rekrutiert die E3-Ligase Von Hippel Lindau (VHL), was zur Ubiquitinierung und anschließenden Degradation des onkogenen Fusionsproteins fÜhrt.
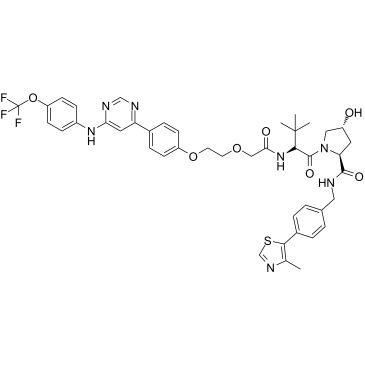
-
GC32831
HaloPROTAC 2
HaloPROTAC 2 (HaloPROTAC 2) ist ein Konjugat aus Liganden fÜr E3 und einem Linker mit einer LÄnge von 21 Atomen. Der Verbinder des Linkers ist eine Halogengruppe. HaloPROTAC 2 enthÄlt den auf VH032 basierenden VHL-Liganden und einen 5-Einheiten-PEG-Linker. HaloPROTAC 2 ist in der Lage, den Abbau von GFP-HaloTag7 in zellbasierten Assays zu induzieren.
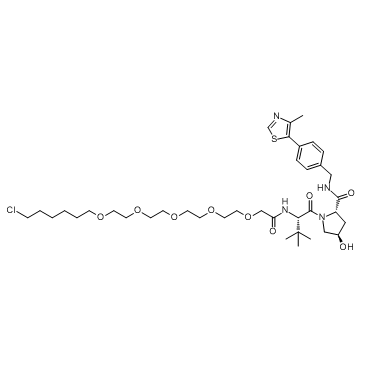
-
GC40877
Heclin
Heclin ist ein HECT E3-Ubiquitin-Ligasen-Inhibitor.
-
GC33391
Homo-PROTAC cereblon degrader 1
Homo-PROTAC-Cereblon-Degrader 1 (Verbindung 15a) ist ein hochwirksamer und effizienter Cereblon (CRBN)-Degrader mit nur minimalen Auswirkungen auf IKZF1 und IKZF3.
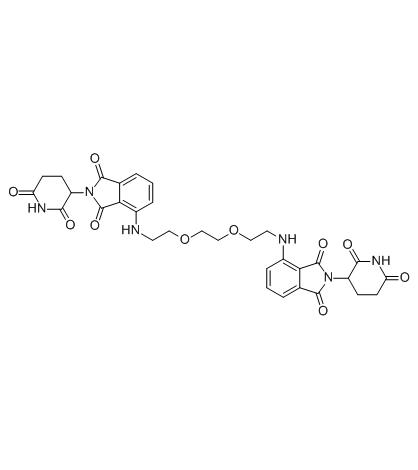
-
GC65285
Homo-PROTAC pVHL30 degrader 1
Homo-PROTAC pVHL30-Degrader 1 ist ein potenter pVHL30-Degrader auf Basis von PROTAC, bestehend aus zwei Liganden von von Hippel-Lindau.
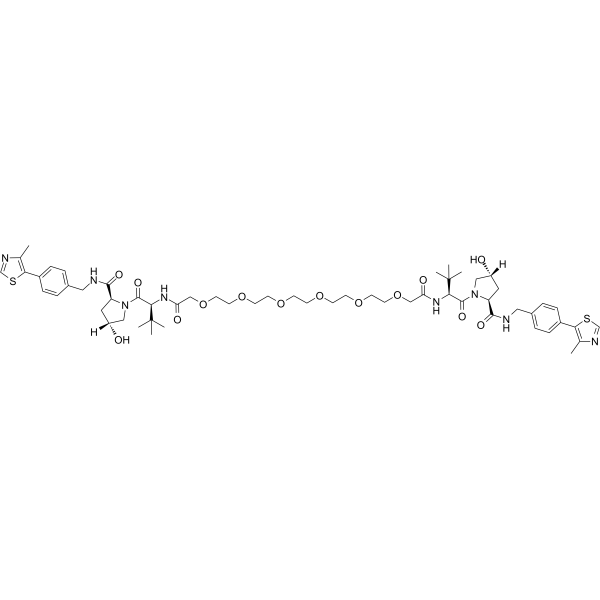
-
GC65559
INY-03-041
INY-03-041 ist ein potenter, hochselektiver und PROTAC-basierter pan-AKT-Abbaustoff, bestehend aus dem ATP-kompetitiven AKT-Inhibitor GDC-0068, konjugiert an Lenalidomid (Cereblon-Ligand). INY-03-041 hemmt AKT1, AKT2 und AKT3 mit IC50-Werten von 2,0 nM, 6,8 nM bzw. 3,5 nM.
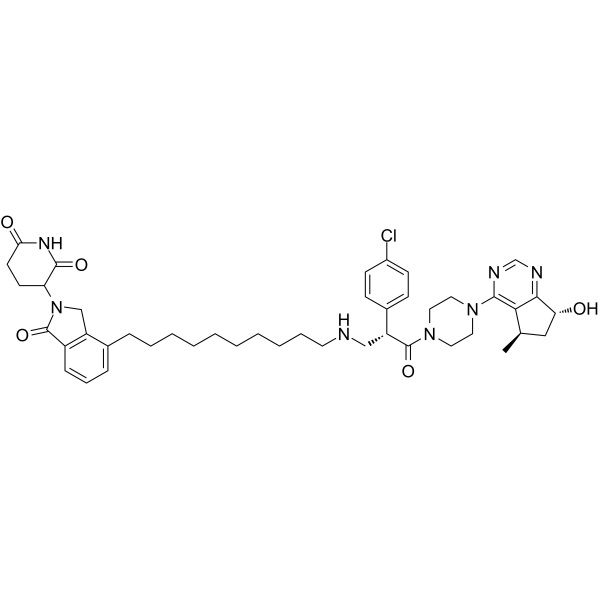
-
GC67757
INY-03-041 trihydrochloride
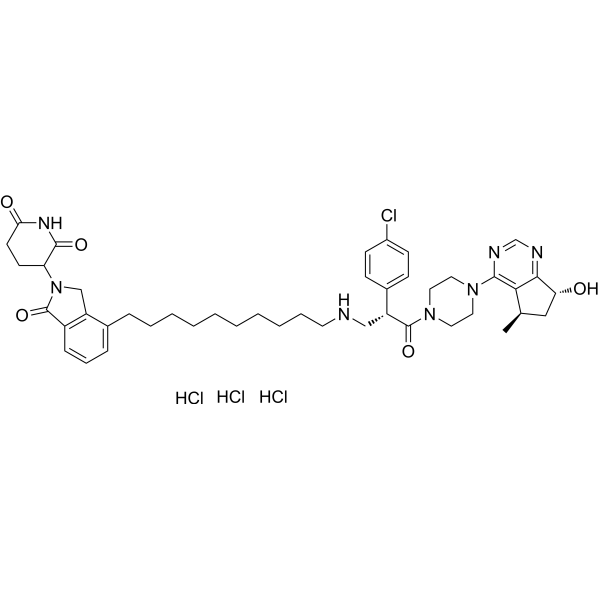
-
GC63707
JB170
JB170 ist ein potenter und hochspezifischer PROTAC-vermittelter AURORA-A (Aurora Kinase) Abbauer (DC50=28 nM) durch VerknÜpfung von Alisertib mit dem Cereblon-bindenden MolekÜl Thalidomid. JB170 bindet vorzugsweise AURORA-A (EC50 = 193 nM) gegenÜber AURORA-B (EC50 = 1,4 μM). JB170-vermittelter S-Phasen-Stillstand wird speziell durch AURORA-A-Verarmung verursacht. JB170 hat eine ausgezeichnete FÄhigkeit, die nicht-katalytische Funktion der AURORA-A-Kinase zu hemmen.
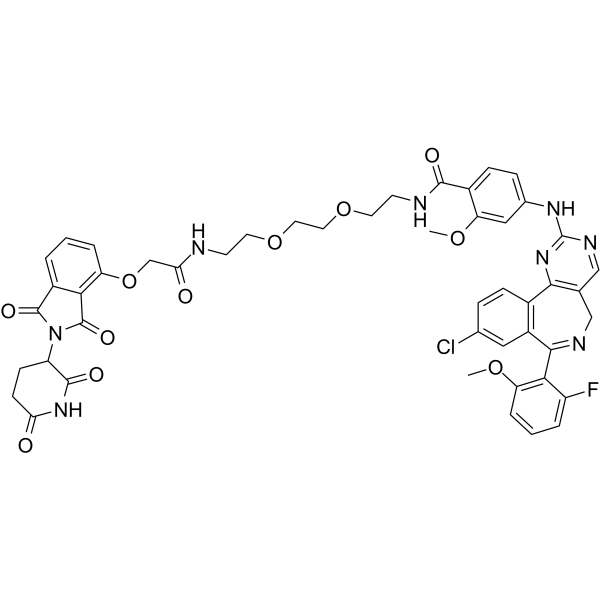
-
GC65471
JH-XI-10-02
JH-XI-10-02 ist ein PROTAC, der durch Liganden für Cereblon und CDK verbunden ist. JH-XI-10-02 ist ein hochpotenter und selektiver PROTAC CDK8-Degrader mit einer IC50 von 159 nM. JH-XI-10-02 verursacht proteasomale Degradation und beeinflusst nicht die mRNA-Level von CDK8. JH-XI-10-02 zeigt keine Wirkung auf CDK19.
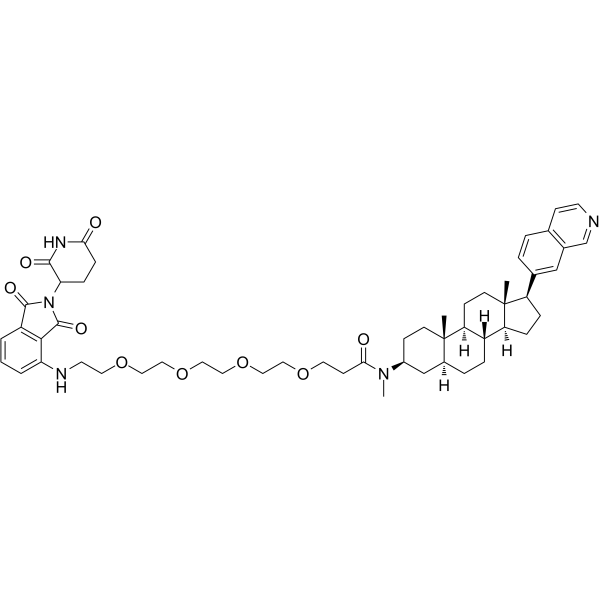
-
GC61766
LC-2
LC-2 ist ein potenter und erster seiner Klasse von Hippel-Lindau-basierter PROTAC, der in der Lage ist, endogenes KRAS G12C mit DC50-Werten zwischen 0,25 und 0,76 μM abzubauen. LC-2 bindet KRAS G12C kovalent mit einem MRTX849-Gefechtskopf und rekrutiert die E3-Ligase VHL, was einen schnellen und anhaltenden Abbau von KRAS G12C induziert, was zur UnterdrÜckung der MAPK-SignalÜbertragung sowohl in homozygoten als auch in heterozygoten KRAS G12C-Zelllinien fÜhrt.
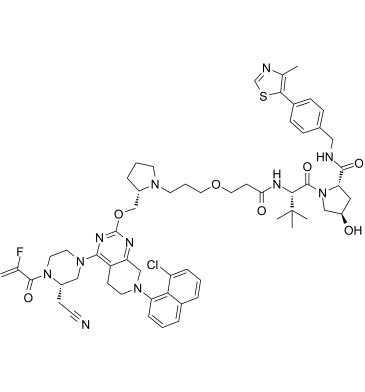
-
GC50669
Lenalidomide 4'-PEG1-amine
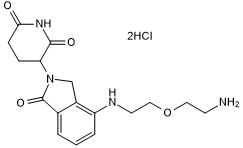
-
GC50684
Lenalidomide 4'-PEG2-amine
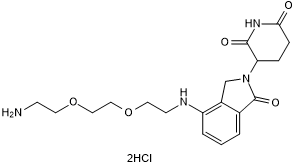
-
GC50694
Lenalidomide 4'-PEG3-amine
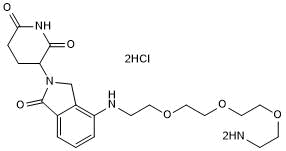
-
GC38812
MD-224
MD-224 ist ein erstklassiger und hochwirksamer niedermolekularer Human-Maus-Doppelminuten-2-Abbaustoff (MDM2), der auf dem Proteolyse-Targeting-ChimÄren-Konzept (PROTAC) basiert. MD-224 besteht aus Liganden fÜr Cereblon und MDM2. MD-224 induziert einen schnellen Abbau von MDM2 bei Konzentrationen <1 nM in menschlichen LeukÄmiezellen und erreicht einen IC50-Wert von 1,5 nM bei der Hemmung des Wachstums von RS4;11-Zellen. MD-224 hat das Potenzial, eine neue Klasse von Krebsmitteln zu werden.
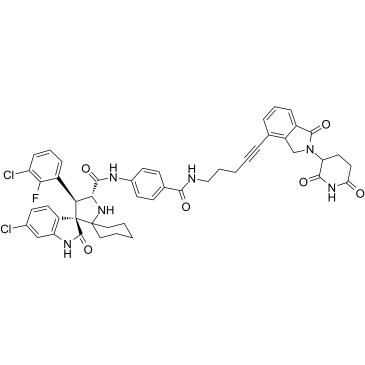
-
GC64651
MI-389
MI-389 ist ein PROTAC Translation Termination Factor GSPT1 Degrader. MI-389 unterbricht ein Ziel, das eine gemeinsame AbhÄngigkeit in verschiedenen AML- und ALL-Zelllinien darstellt, und dass die Wirkung von MI-389 von der CRL4CRBN-E3-Ligase abhÄngt.
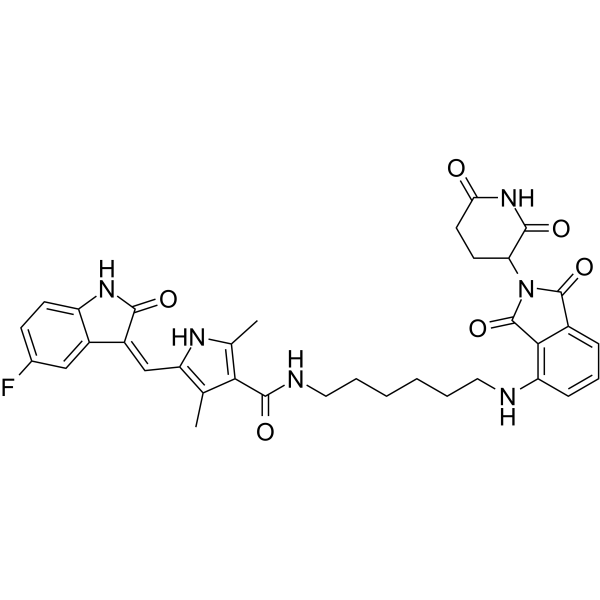
-
GC69501
MS159
MS159 is an effective nuclear receptor binding SET domain protein 2 (NSD2) PROTAC degrader. MS159 can inhibit the growth of tumor cells. MS159 is an effective chemical tool for exploring the role of NSD2 in health and disease.
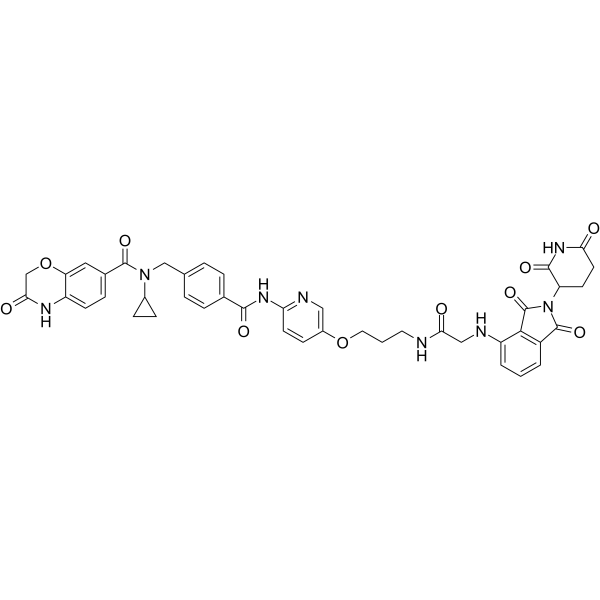
-
GC65466
MS170
MS170 ist ein potenter und selektiver Abbauer von PROTAC AKT. MS170 verringert die zellulÄre Gesamt-AKT (T-AKT) mit dem DC50-Wert von 32 nM. MS170 bindet an AKT1, AKT2 und AKT3 mit Kds von 1,3 nM, 77 nM bzw. 6,5 nM.
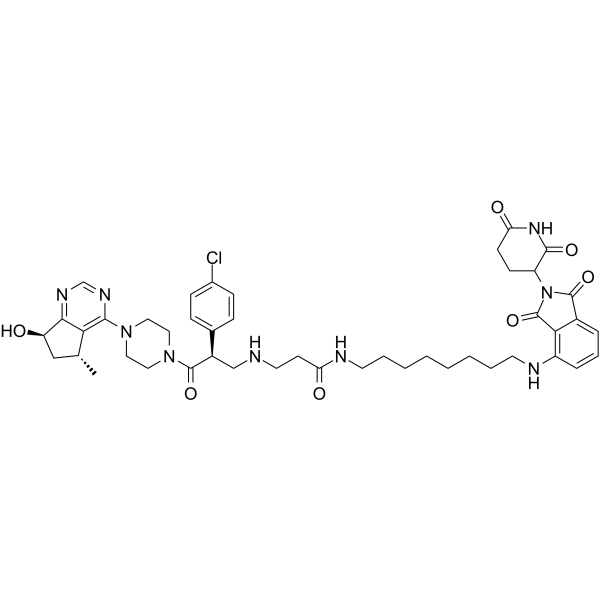
-
GC67710
MS177
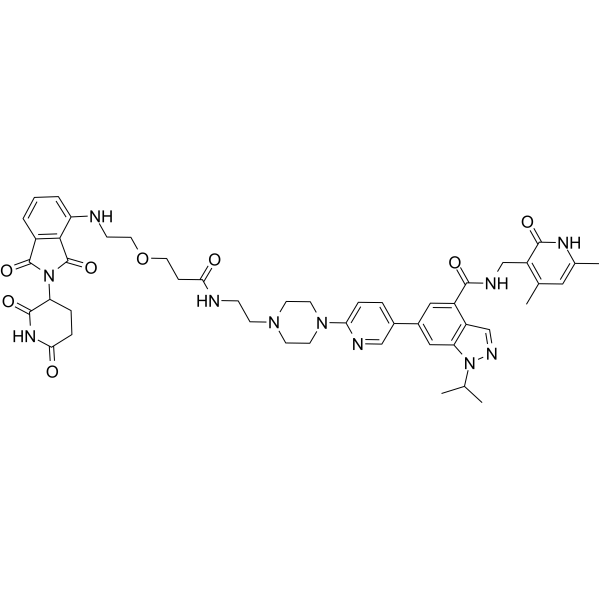
-
GC69502
MS21
MS21 ist eine neue Art von AKT-Abbauprodukt, das selektiv das Wachstum von Krebsmutationen durch die Hemmung des PI3K/PTEN-Signalwegs unterdrückt.
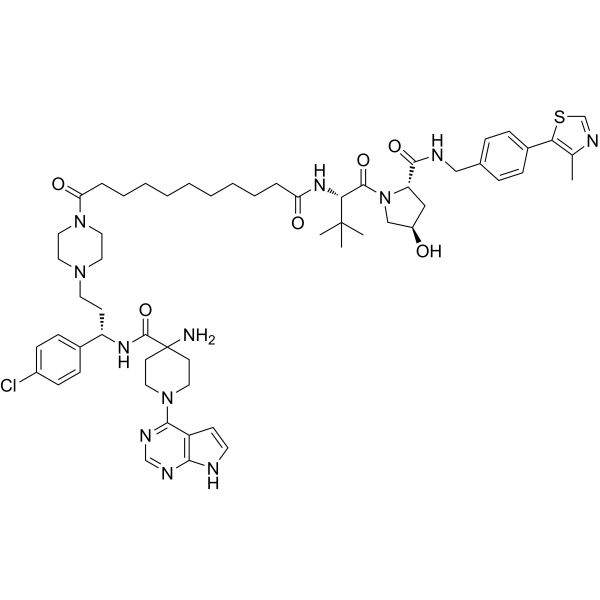
-
GC65243
MS4077
MS4077 ist ein anaplastisches Lymphomkinase (ALK) PROTAC (Abbaumittel) basierend auf einem Cereblon-Liganden mit einem Kd von 37 nM fÜr die BindungsaffinitÄt zu ALK.
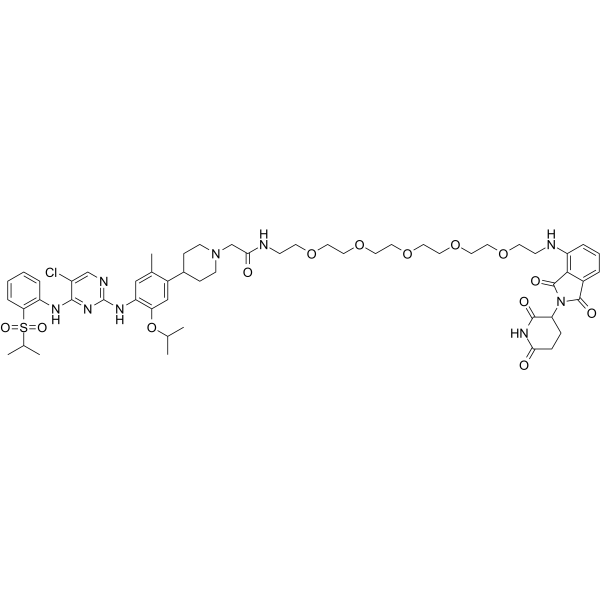
-
GC64966
MS4078
MS4078 ist ein PROTAC (Abbaumittel) einer anaplastischen Lymphomkinase (ALK) basierend auf einem Cereblon-Liganden mit einer Kd von 19 nM fÜr die BindungsaffinitÄt zu ALK.
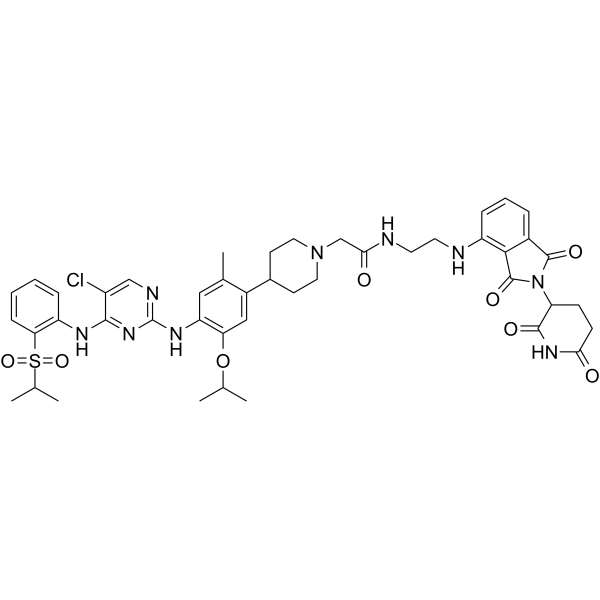
-
GC65052
MS4322
MS4322 (YS43-22) ist ein erstklassiger PRMT5-Abbaustoff und ein wertvolles chemisches Werkzeug (PROTAC) zur Erforschung der PRMT5-Funktionen bei Gesundheit und Krankheit.
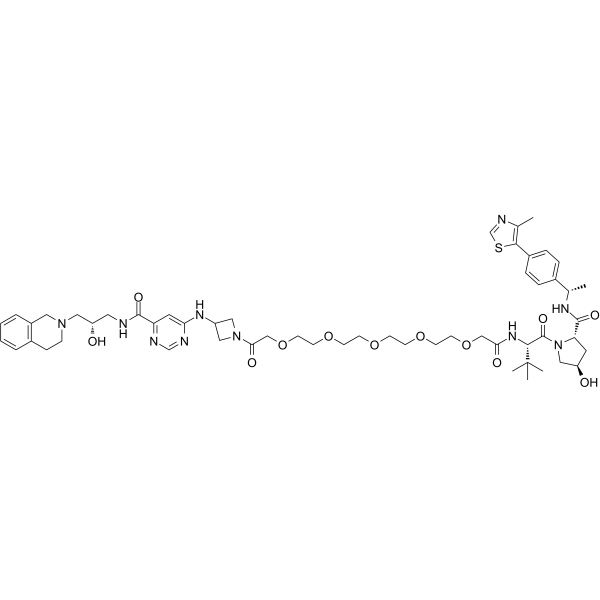
-
GC65876
MS4322 (isomer)
MS4322 (YS43-22)-Isomer ist ein Isomer von MS4322. MS4322 ist ein potenter und selektiver Abbauer von PRMT5 (Protein-Arginin-Methyltransferase 5) und hemmt das Wachstum mehrerer Krebszelllinien.
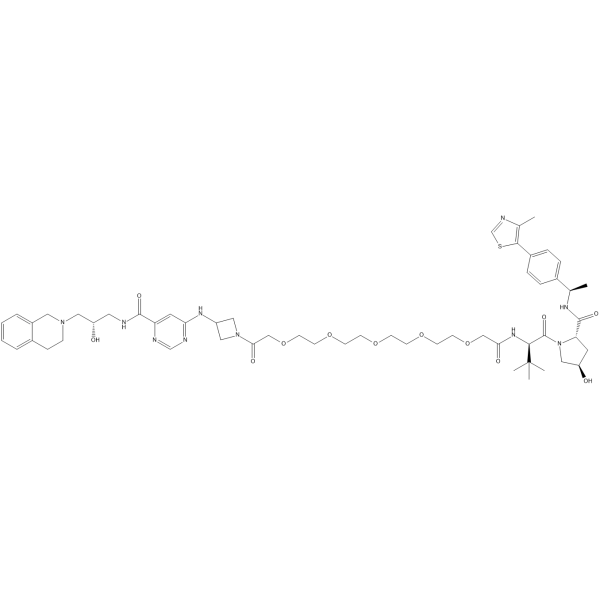
-
GC69505
MS9427
MS9427 ist ein wirksamer PROTAC-EGFR-Degradationshemmer mit Kd-Werten von 7,1 nM für Wildtyp-EGFR und 4,3 nM für die EGFR-L858R-Mutation. MS9427 baut selektiv Mutanten über das Ubiquitin/Proteasom-System (UPS) und den Autophagie/Lysosomenweg ab, aber nicht WT EGFR. MS9427 hat eine starke hemmende Wirkung auf das Zellwachstum von NSCLC-Zellen. MS9427 kann in der Krebsforschung eingesetzt werden.
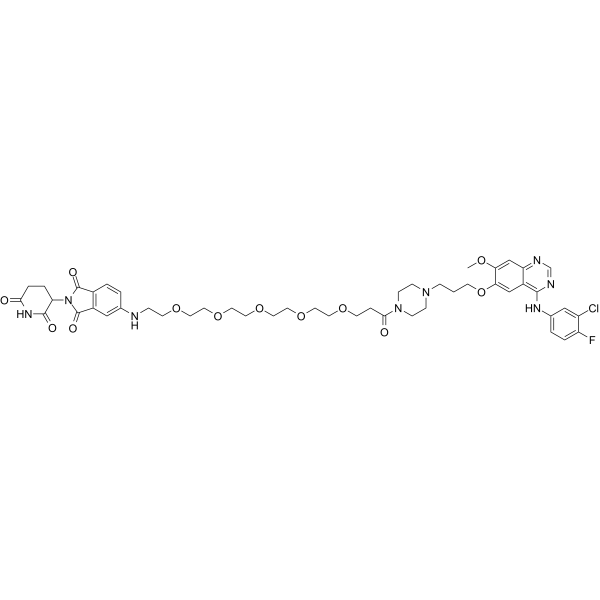
-
GC69506
MS9427 TFA
MS9427 TFA ist ein wirksamer PROTAC-EGFR-Degradationshemmer mit einer Kd von 7,1 nM für Wildtyp-EGFR und 4,3 nM für die EGFR-L858R-Mutation. MS9427 TFA baut selektiv Mutanten durch das Ubiquitin/Proteasom-System (UPS) und den Autophagie/Lysosomen-Weg ab, aber nicht WT EGFR. MS9427 TFA hat eine starke hemmende Wirkung auf das Zellwachstum von NSCLC-Zellen. MS9427 TFA kann in der Krebsforschung eingesetzt werden.
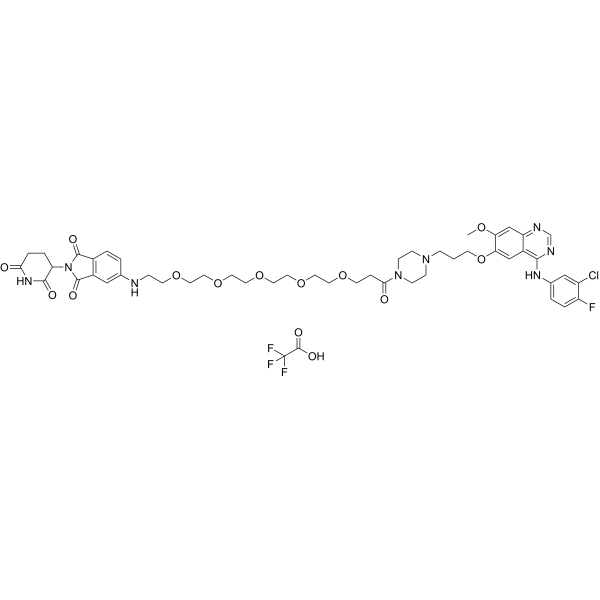
-
GC36661
MT-802
MT-802 ist ein potenter BTK-Abbauer auf Basis von Cereblon-Liganden mit einem DC50 von 1 nM.
-
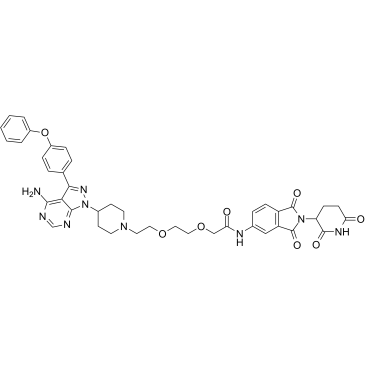
GC69744
MTX-23
MTX-23 ist ein auf AR basierendes PROTAC. MTX-23 hemmt das Wachstum von CaP-Zellen durch Abbau von AR-V7 und AR-FL. MTX-23 induziert Zellapoptose (Apoptose).
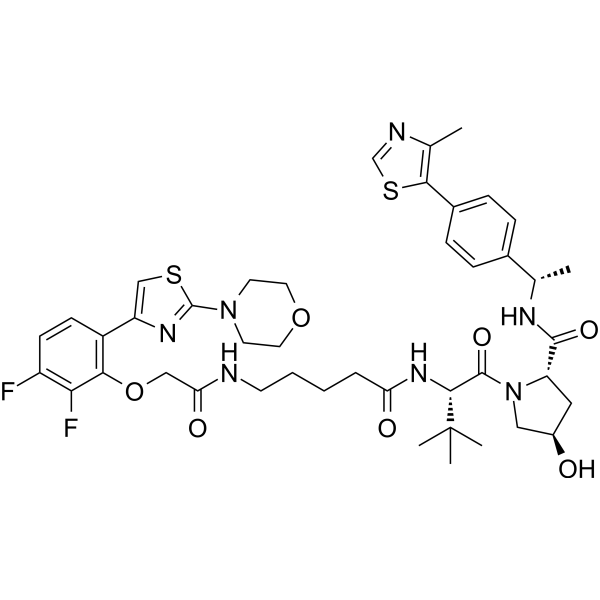
-
GC18729
MZ1
MZ1 ist ein PROTAC, verbunden durch Liganden für von Hippel-Lindau und BRD4. MZ1 induziert wirksam und schnell eine reversible, lang anhaltende und selektive Entfernung von BRD4 gegenüber BRD2 und BRD3. Kds von 382/120, 119/115 und 307/228 nM für BRD4 BD1/2, BRD3 BD1/2 bzw. BRD2 BD1/2.
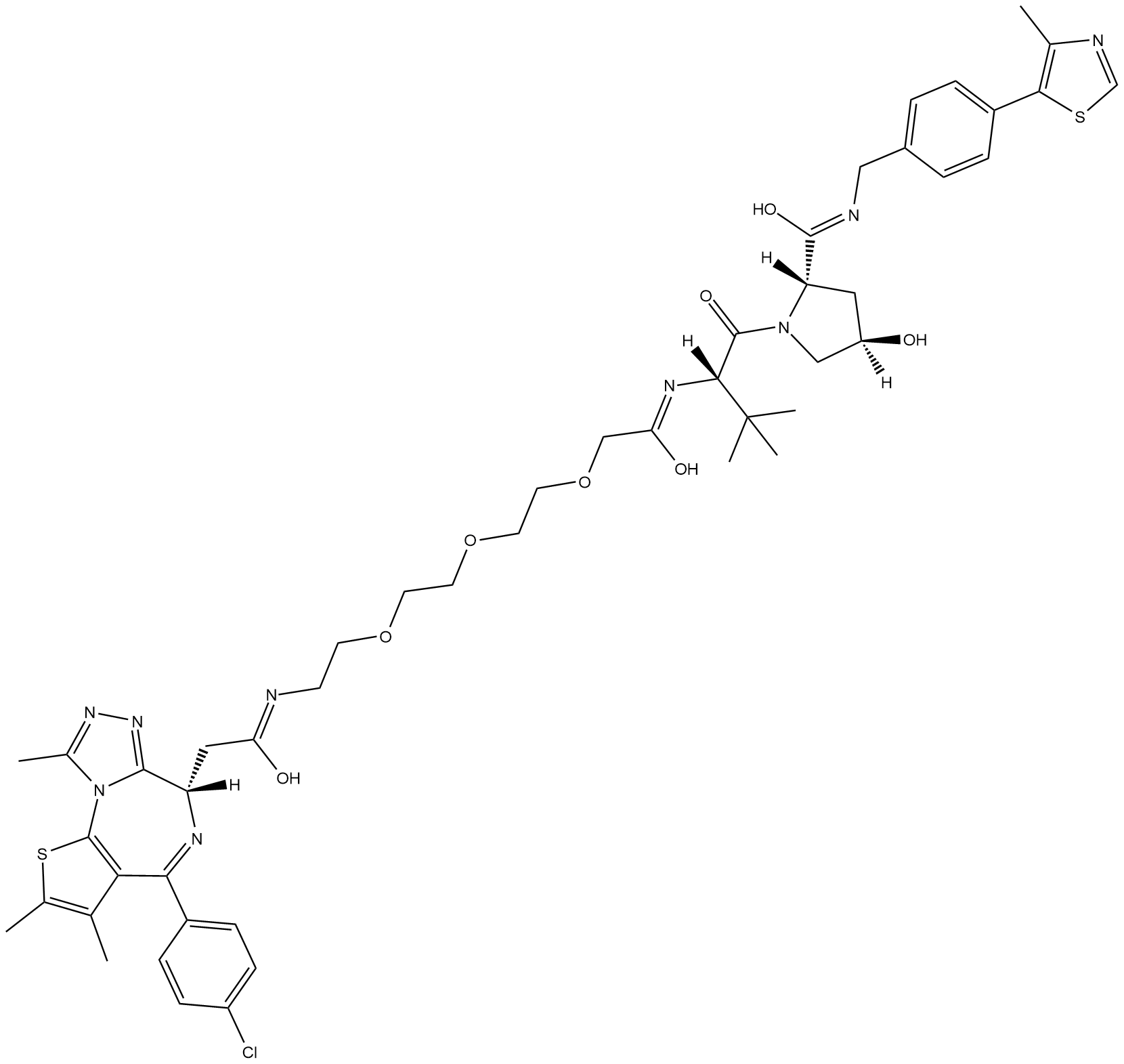
-
GC33102
MZP-54
MZP-54 ist ein PROTAC, verbunden durch Liganden fÜr von Hippel-Lindau und BRD3/4, mit einem Kd von 4 nM fÜr Brd4BD2.
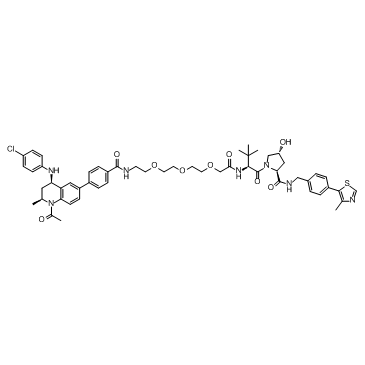
-
GC33363
MZP-55
MZP-55 ist ein PROTAC, verbunden durch Liganden fÜr von Hippel-Lindau und BRD3/4, mit einem Kd von 8 nM fÜr Brd4BD2.
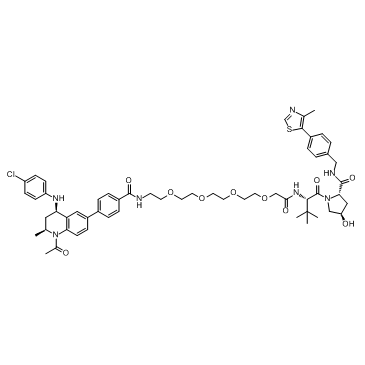
-
GC67889
NJH-2-056
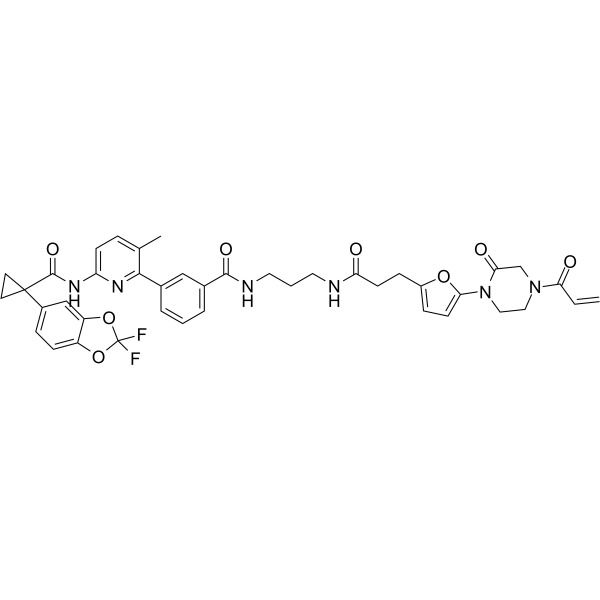
-
GC50740
NR 7h
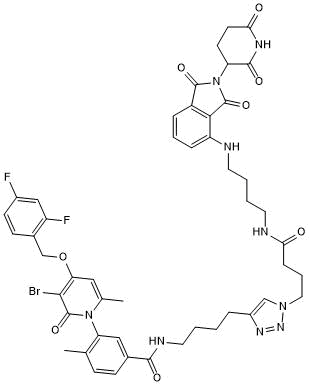
-
GC50665
Pomalidomide 4'-alkylC3-acid
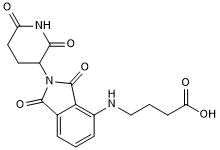
-
GC50658
Pomalidomide 4'-alkylC3-amine
Pomalidomide 4'-AlkylC3-amin ist ein synthetisiertes E3-Ligase-Ligand-Linker-Konjugat, das den auf Pomalidomid basierenden Cereblon-Liganden und einen Linker enthÄlt, der in der PROTAC-Technologie verwendet wird.
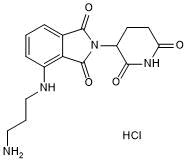
-
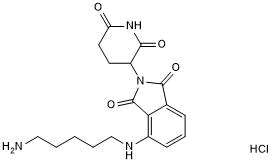
GC50674
Pomalidomide 4'-alkylC4-acid
-
GC50546
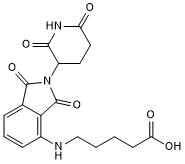
Pomalidomide 4'-alkylC5-acid
Pomalidomid-4'-Alkyl-C5-SÄure ist ein synthetisiertes E3-Ligase-Ligand-Linker-Konjugat, das den auf Pomalidomid basierenden Cereblon-Liganden und einen PEG-Linker enthÄlt, der in der PROTAC-Technologie verwendet wird.
-
GC50554
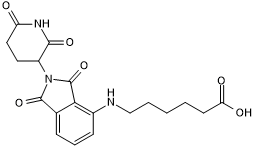
Pomalidomide 4'-alkylC5-amine
Pomalidomide 4'-AlkylC5-amin ist ein synthetisiertes E3-Ligase-Ligand-Linker-Konjugat, das den auf Thalidomid basierenden Cereblon-Liganden und einen in der PROTAC-Technologie verwendeten Linker enthÄlt.
-
GC50689
Pomalidomide 4'-alkylC6-acid
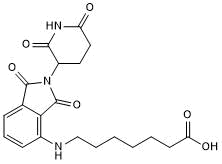
-
GC50696
Pomalidomide 4'-alkylC8-acid
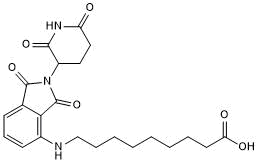
-
GC50675
Pomalidomide 4'-PEG1-acid
Pomalidomide 4'-PEG1-SÄure ist ein synthetisiertes E3-Ligase-Ligand-Linker-Konjugat, das den Pomalidomid-basierten Cereblon-Liganden und den 1-Einheit-PEG-Linker enthÄlt, die in der PROTAC-Technologie verwendet werden.
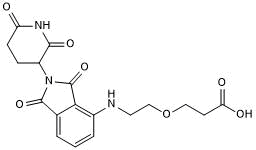
-
GC50690
Pomalidomide 4'-PEG2-acid
Pomalidomide 4'-PEG2-SÄure (Pomalidomide 4'-PEG2-SÄure) ist ein synthetisiertes E3-Ligase-Ligand-Linker-Konjugat, das den Pomalidomid-basierten Cereblon-Liganden und den 2-Einheiten-PEG-Linker enthÄlt, die in der PROTAC-Technologie verwendet werden.
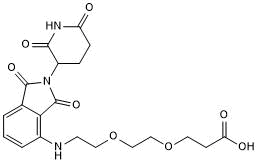
-
GC50530
Pomalidomide 4'-PEG2-amine
Pomalidomide 4'-PEG2-Amin ist ein synthetisiertes E3-Ligase-Ligand-Linker-Konjugat, das den auf Thalidomid basierenden Cereblon-Liganden und den 2-Einheiten-PEG-Linker enthÄlt, die in der PROTAC-Technologie verwendet werden.
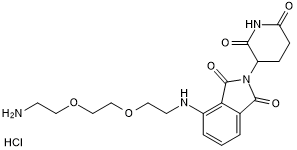
-
GC50705
Pomalidomide 4'-PEG4-acid
Pomalidomid 4'-PEG4-SÄure ist ein E3-Ligase-Ligand-Linker-Konjugat.
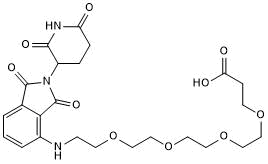
-
GC50625
Pomalidomide 4'-PEG4-amine
Pomalidomid 4'-PEG4-Amin ist ein synthetisiertes E3-Ligase-Ligand-Linker-Konjugat, das den auf Pomalidomid basierenden Cereblon-Liganden und den 4-Einheiten-PEG-Linker enthÄlt, die bei der Synthese von PROTACs verwendet werden.