Proteases
Proteases is a general term for a class of enzymes that hydrolyze protein peptide chains. According to the way they degrade polypeptides, they are divided into two categories: endopeptidases and telopeptidases. The former can cut the large molecular weight polypeptide chain from the middle to form prions and peptones with smaller molecular weights; the latter can be divided into carboxypeptidase and aminopeptidase, which respectively remove the peptide from the free carboxyl terminus or free amino terminus of the polypeptide one by one. Chain hydrolysis produces amino acids.
A general term for a class of enzymes that hydrolyze peptide bonds in proteins. According to the way they hydrolyze polypeptides, they can be divided into endopeptidases and exopeptidases. Endopeptidase cleaves the interior of the protein molecule to form smaller molecular weight peptones and peptones. Exopeptidase hydrolyzes peptide bonds one by one from the end of the free amino group or carboxyl group of protein molecules, and frees amino acids, the former is aminopeptidase and the latter is carboxypeptidase. Proteases can be classified into serine proteases, sulfhydryl proteases, metalloproteases and aspartic proteases according to their active centers and optimum pH. According to the optimum pH value of its reaction, it is divided into acidic protease, neutral protease and alkaline protease. The proteases used in industrial production are mainly endopeptidases.
Proteases are widely found in animal offal, plant stems and leaves, fruits and microorganisms. Microbial proteases are mainly produced by molds and bacteria, followed by yeast and actinomycetes.
Enzymes that catalyze the hydrolysis of proteins. There are many kinds, the important ones are pepsin, trypsin, cathepsin, papain and subtilisin. Proteases have strict selectivity for the reaction substrates they act on. A protease can only act on certain peptide bonds in protein molecules, such as the peptide bonds formed by the hydrolysis of basic amino acids catalyzed by trypsin. Proteases are widely distributed, mainly in the digestive tract of humans and animals, and are abundant in plants and microorganisms. Due to limited animal and plant resources, the industrial production of protease preparations is mainly prepared by fermentation of microorganisms such as Bacillus subtilis and Aspergillus terrestris.
Ziele für Proteases
- Caspase(114)
- Aminopeptidase(24)
- ACE(74)
- Calpains(20)
- Carboxypeptidase(10)
- Cathepsin(81)
- DPP-4(31)
- Elastase(26)
- Gamma Secretase(67)
- HCV Protease(59)
- HSP(113)
- HIV Integrase(37)
- HIV Protease(47)
- MMP(228)
- NS3/4a protease(8)
- Serine Protease(18)
- Thrombin(58)
- Urokinase(4)
- Cysteine Protease(0)
- Other Proteases(18)
- Tyrosinases(47)
- 15-PGDH(1)
- Acetyl-CoA Carboxylase(13)
- Acyltransferase(59)
- Aldehyde Dehydrogenase (ALDH)(28)
- Aminoacyl-tRNA Synthetase(9)
- ATGL(1)
- Dipeptidyl Peptidase(56)
- Drug Metabolite(457)
- E1/E2/E3 Enzyme(90)
- Endogenous Metabolite(1636)
- FABP(30)
- Farnesyl Transferase(23)
- Glutaminase(14)
- Glutathione Peroxidase(14)
- Isocitrate Dehydrogenase (IDH)(28)
- Lactate Dehydrogenase(17)
- Lipoxygenase(234)
- Mitochondrial Metabolism(207)
- NEDD8-activating Enzyme(7)
- Neprilysin(12)
- PAI-1(13)
- Ser/Thr Protease(41)
- Tryptophan Hydroxylase(13)
- Xanthine Oxidase(18)
- MALT1(10)
- PCSK9(1)
Produkte für Proteases
- Bestell-Nr. Artikelname Informationen
-
GC30649
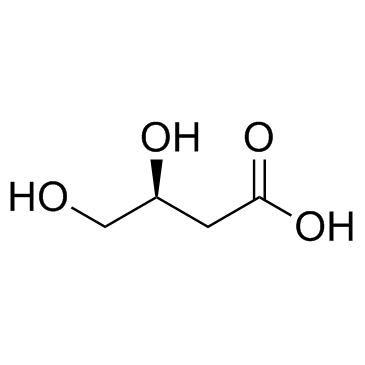
(S)-3,4-Dihydroxybutyric acid
(S)-3,4-DihydroxybuttersÄure ist ein normaler menschlicher Metabolit im Urin, der bei Patienten mit Succinat-Semialdehyd-Dehydrogenase (SSADH)-Mangel in erhÖhter Konzentration ausgeschieden wird.
-
GC64473
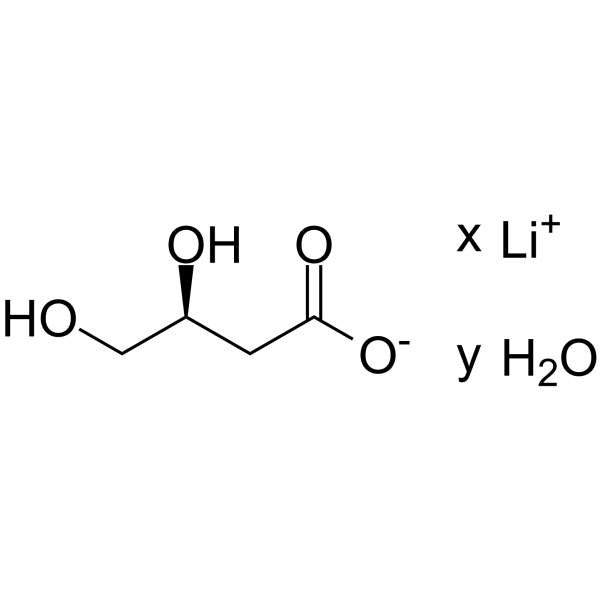
(S)-3,4-Dihydroxybutyric acid lithium hydrate
(S)-3,4-DihydroxybuttersÄure (Lithiumhydrat) ist ein normaler menschlicher Metabolit im Urin, der bei Patienten mit Succinat-Semialdehyd-Dehydrogenase (SSADH)-Mangel in erhÖhter Konzentration ausgeschieden wird.
-
GC62749
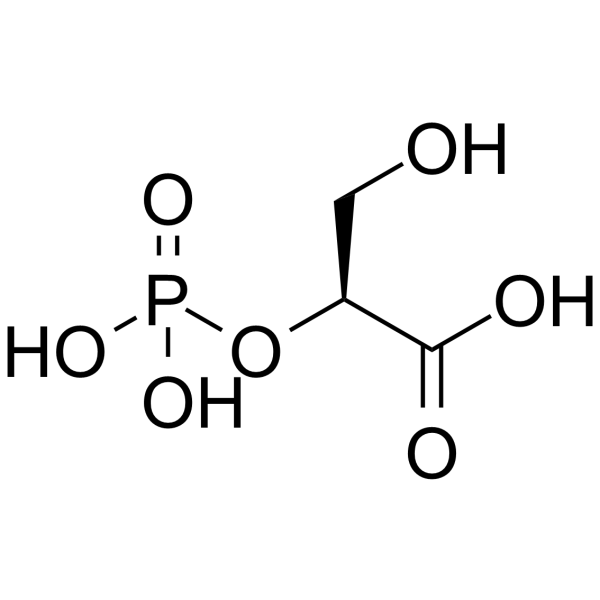
(S)-3-Hydroxy-2-(Phosphonooxy)Propanoic Acid
(S)-3-Hydroxy-2-(Phosphonooxy)propansÄure ist ein kÖrpereigener Metabolit.
-
GC30304
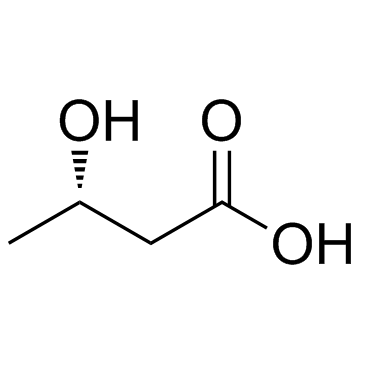
(S)-3-Hydroxybutanoic acid ((S)-β-Hydroxybutanoic acid)
(S)-3-HydroxybutansÄure ((S)-β-HydroxybutansÄure) ist ein normaler menschlicher Metabolit, der bei geriatrischen Patienten, die von einer Depression zurÜckgekehrt sind, erhÖht gefunden wurde.
-
GC30623
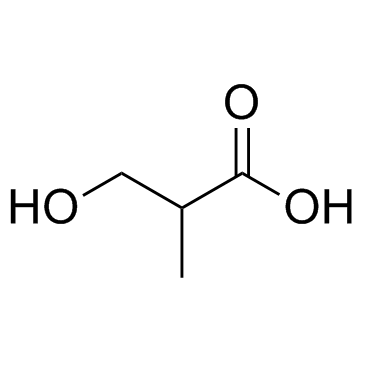
(S)-3-Hydroxyisobutyric acid
(S)-3-HydroxyisobuttersÄure ist ein wichtiger interorganischer Metabolit, ein Zwischenprodukt in den Stoffwechselwegen von L-Valin und Thymin und ein gutes gluconeogenes Substrat.
-
GC30148
(S)-b-aminoisobutyric acid
(S)-b-AminoisobuttersÄure ist eine Nicht-Protein-AminosÄure, die aus dem Katabolismus von Thymin und Valin stammt.

-
GC32993
(S)-GNE-140
(S)-GNE-140 ist das weniger aktive Enantiomer von GNE-140, das die Laktatdehydrogenase A (LDHA) hemmen kann.

-
GC62751
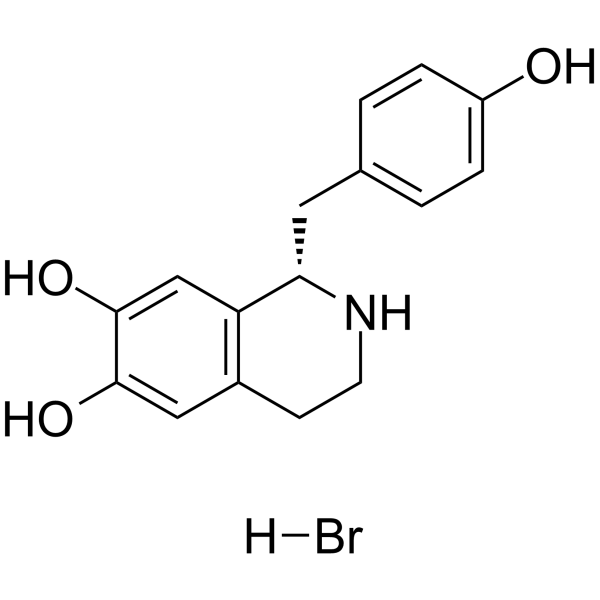
(S)-Higenamine hydrobromide
(S)-Higenamin ((S)-Norcolaurin)-Hydrobromid, ein S-Enantiomer von Higenamin, ist die Einstiegsverbindung in die Benzylisochinolin-Alkaloid-Biosynthese.
-
GC40145
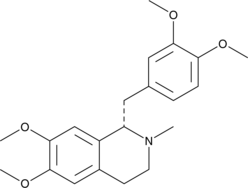
(S)-Laudanosine
(S)-Laudanosine is the (S) enantiomer of laudanosine, a metabolite of the neuromuscular blocking agents atracurium and cisatracurium.
-
GC30735
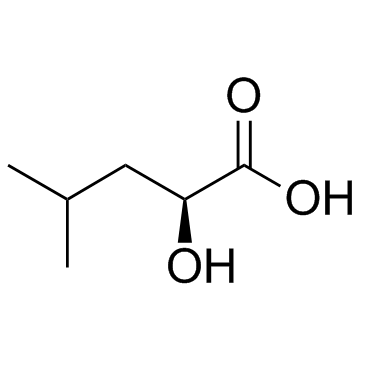
(S)-Leucic acid
(S)-LeucinsÄure ist ein AminosÄuremetabolit.
-
GC69914
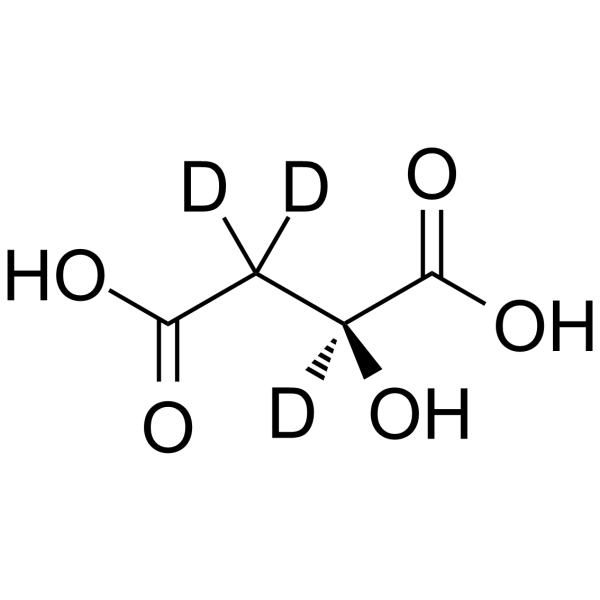
(S)-Malic acid-d3
(S)-Malic acid-d3 ist das Deuterium-Isotop von (S)-Malic acid. (S)-Malic acid ((S)-2-Hydroxysuccinic acid) ist eine natürlich vorkommende Dicarbonsäure und Quelle für den fruchtigen Geschmack, die häufig als Lebensmittelzusatzstoff verwendet wird.
-
GC35003
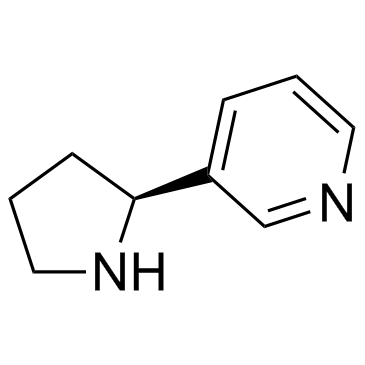
(S)-Nornicotine
-
GC49179
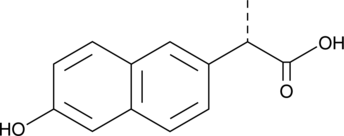
(S)-O-Desmethyl Naproxen
A metabolite of (S)-naproxen
-
GC60419
(S)-O-Desmethyl Venlafaxine N-Oxide
(S)-O-Desmethyl-Venlafaxin-N-Oxid ist ein N-Oxid von (S)-O-Desmethyl-Venlafaxin.
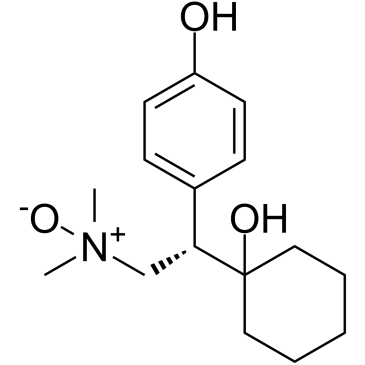
-
GC65883
(S,R)-WT IDH1 Inhibitor 2
(S,R)-WT IDH1 Inhibitor 2 (GSK321) ist ein potenter, selektiver mutierter IDH1-Inhibitor mit IC50-Werten von 2,9, 3,8, 4,6 und 46 nM fÜr R132G, R132C, R132H bzw. WT IDH1 und >100-fach SelektivitÄt gegenÜber IDH2. (S,R)-WT IDH1 Inhibitor 2 induziert eine Abnahme des intrazellulÄren 2-HG, eine Aufhebung der myeloischen Differenzierungsblockade und eine Induktion der granulozytÄren Differenzierung auf der Ebene von LeukÄmieblasten und unreiferen stammÄhnlichen Zellen. (S,R)-WT IDH1 Inhibitor 2 kann fÜr die Erforschung der akuten myeloischen LeukÄmie (AML) und anderer Krebsarten verwendet werden.
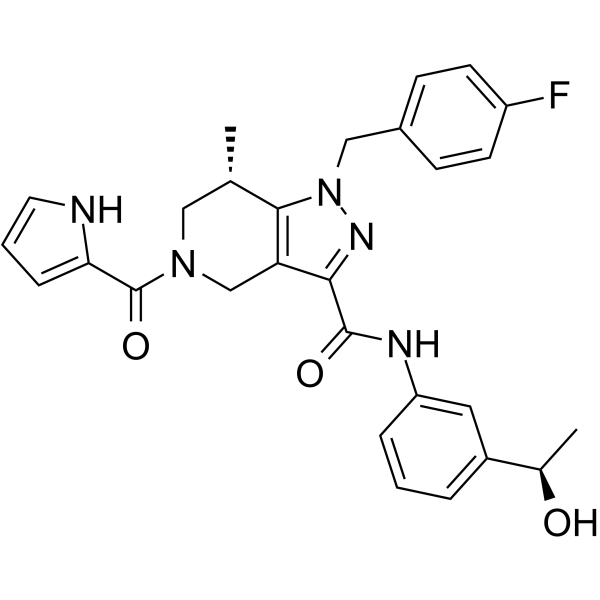
-
GC67682
(S,S)-GSK321
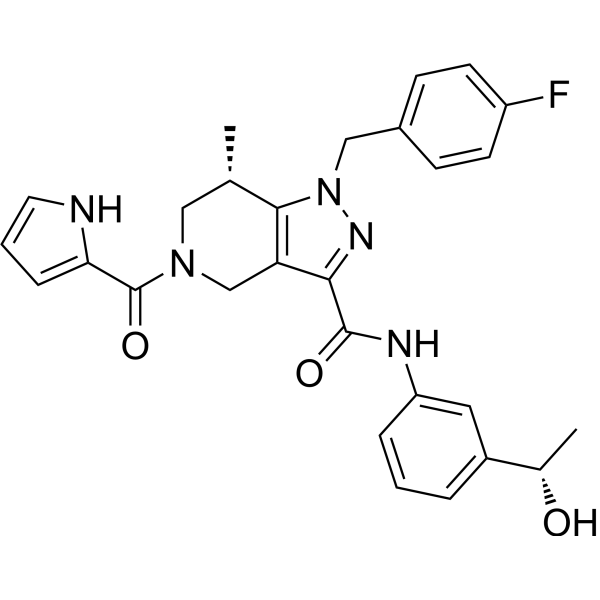
-
GC64535
(S,S)-TAPI-1
(S,S)-TAPI-1 ist ein Isomer von TAPI-1.

-
GC60427
(Z)-10-Hydroxynortriptyline
(Z)-10-Hydroxynortriptylin ist ein Metabolit von Nortriptylin.
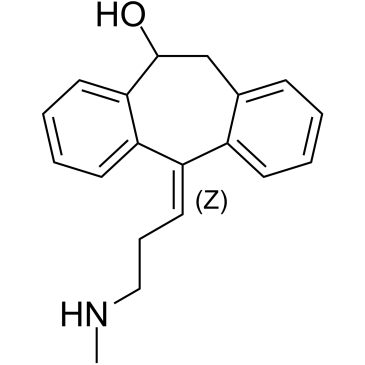
-
GC67761
(Z)-10-Hydroxynortriptyline-d3
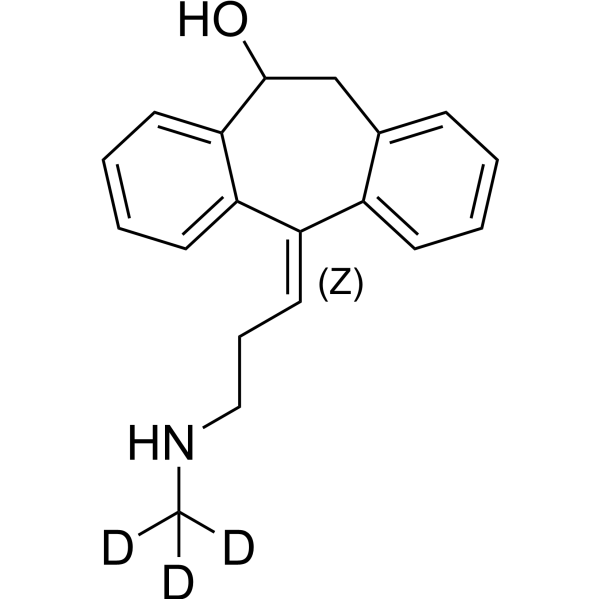
-
GC31253
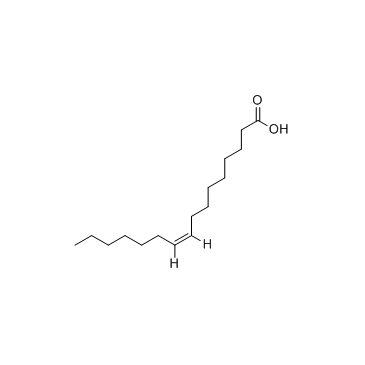
(Z)-Hexadec-9-enoic acid
(Z)-Hexadec-9-ensÄure, eine FettsÄurezusammensetzung, ist an der Verhinderung des Todes durch zerebrovaskulÄre StÖrungen bei SHRSP-Ratten beteiligt.
-
GC11847
(Z-Ala-Ala-Ala-Ala)2Rh110
fluorogenic elastase substrate

-
GC18596
(±)-2-propyl-4-Pentenoic Acid
(±)-2-Propyl-4-pentensÄure (4-en-VPA) ist ein wichtiger toxischer Metabolit von ValproinsÄure.

-
GC16375
(±)-Jasmonic Acid methyl ester
(±)-JasmonsÄuremethylester ist ein kÖrpereigener Metabolit.
-
GC41995
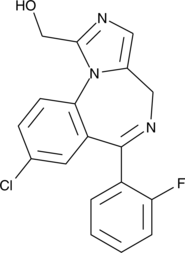
1'-hydroxy Midazolam
Hauptmetabolit des Anästhetikums Midazolam.
-
GC45978
1,10-Phenanthroline (hydrate)
o-Phenanthrolin (1,10-Phenanthrolin)-Monohydrat, ein Metallchelator, verhindert die Induktion von Chromosomenaberrationen in mit Streptozotocin behandelten Zellen.
-
GC41765
1,2,3-Tri-13(Z)-Docosenoyl-rac-glycerol
1,2,3-Tri-13(Z)-Docosenoyl-rac-glycerol ist ein TrierucasÄuretriglycerid aus dem SamenÖl.
-
GC41767
1,2,3-Triarachidonoyl-rac-glycerol
1,2,3-Triarachidonoyl-rac-glycerol ist ein kÖrpereigener Metabolit.
-
GC41768
1,2,3-Tridecanoyl-rac-glycerol
1,2,3-Tridecanoyl-rac-glycerol (Glyceryl tridecanoate) ist eine oral verfÜgbare Vorstufe von DecansÄure (DA) und kann zu DA hydrolysiert werden.
-
GC41770
1,2,3-Trieicosapentaenoyl-rac-glycerol
1,2,3-Trieicosapentaenoyl-rac-glycerol (EPA-TG) is a glycerol ester of eicosapentaenoic acid, which is an ω-3 fatty acid.
-
GC45948
1,2,3-Trilinoelaidoyl-rac-glycerol
A triacylglycerol
-
GC46041
1,2,3-Trilinoleoyl-rac-glycerol
1,2,3-Trilinoleoyl-rac-glycerol ist ein kÖrpereigener Metabolit.
-
GC41774
1,2,3-Trimyristoyl-rac-glycerol
1,2,3-Trimyristoyl-rac-glycerol, eine aktive molluskizide Komponente von Myristica fragransHoutt, hemmt signifikant die AktivitÄten von Acetylcholinesterase (AChE), saurer und alkalischer Phosphatase (ACP/ALP) im Nervengewebe von Lymnaea acuminata.
-
GC41776
1,2,3-Trioleoyl-rac-glycerol
1,2,3-Trioleoyl-rac-glycerol ist ein symmetrisches Triacylglycerol, reduziert die MMP-1-Hochregulierung, mit starken antioxidativen und entzÜndungshemmenden Eigenschaften.
-
GC41778
1,2,3-Tripalmitoyl-rac-glycerol
1,2,3-Tripalmitoyl-rac-glycerol ist ein kÖrpereigener Metabolit.

-
GC39766
1,2-Cyclohexanedione
-
GC41804
1,2-Dimyristoyl-sn-glycero-3-PE
1,2-Dimyristoyl-sn-glycero-3-PE ist ein kÖrpereigener Metabolit.

-
GC41822
1,2-Dipalmitoyl-sn-glycero-3-PE
1,2-Dipalmitoyl-sn-glycero-3-PE ist ein kÖrpereigener Metabolit.
-
GC41825
1,2-Dipalmitoyl-sn-glycero-3-phosphate (sodium salt)
1,2-Dipalmitoyl-sn-glycero-3-phosphat (Natriumsalz) (Verbindung 3-F7) ist eine PhosphatidsÄure und ein menschlicher endogener Metabolit[1]In Vitro: PhosphatidsÄuren sind biologisch aktive Lipide, die eine große Bandbreite stimulieren kÖnnen von Reaktionen in vielen verschiedenen Zelltypen, wie Thrombozytenaggregation, Kontraktion glatter Muskeln, vasoaktive Wirkungen in vivo, Chemotaxis, Expression von AdhÄsionsmolekÜlen, erhÖhte Tight-Junction-PermeabilitÄt von Endothelzellen, Induktion von Stressfasern, Modulation der HerzkontraktilitÄt und viele andere .
-
GC13877
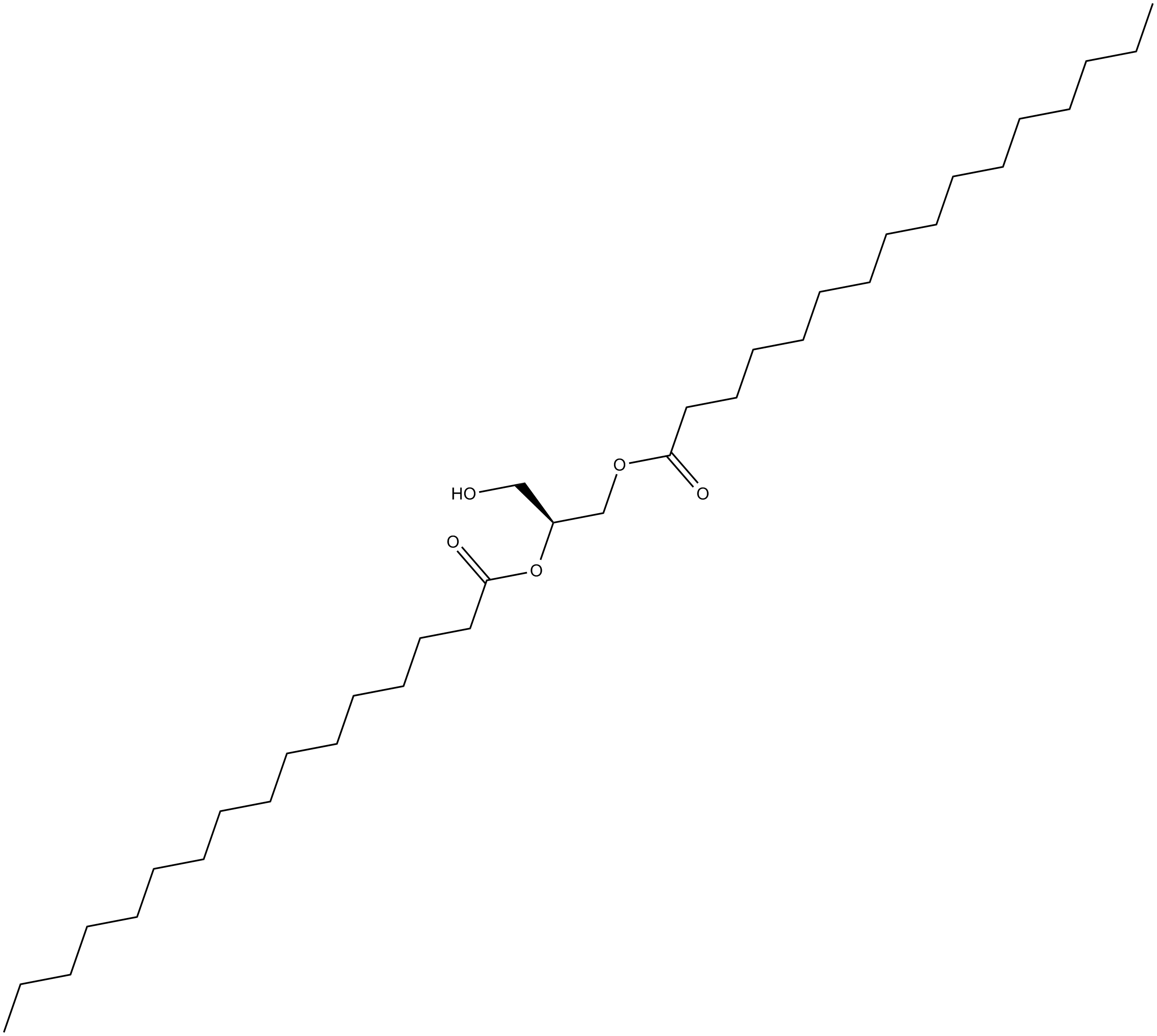
1,2-Dipalmitoyl-sn-glycerol
1,2-Dipalmitoyl-sn-glycerol ist ein kÖrpereigener Metabolit.
-
GC33621
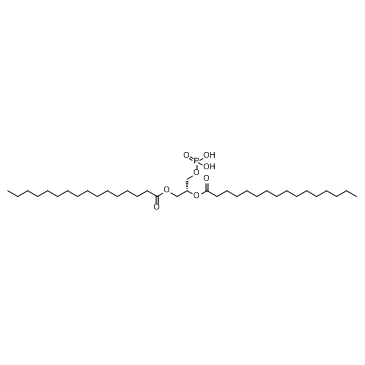
1,2-Dipalmitoyl-sn-glycerol 3-phosphate
1,2-Dipalmitoyl-sn-glycerol-3-phosphat (Verbindung 3-F7) ist eine PhosphatidsÄure und ein menschlicher endogener Metabolit.
-
GC33789
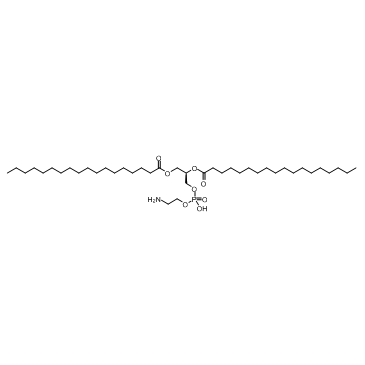
1,2-Distearoyl-sn-glycero-3-phosphorylethanolamine (1,2-DSPE)
1,2-Distearoyl-sn-glycero-3-phosphorylethanolamin (1,2-DSPE) (DSPE) ist ein Phosphoethanolamin (PE)-Lipid, das bei der Synthese von Liposomen verwendet werden kann.
-
GC41837
1,3,7-Trimethyluric Acid
1,3,7-TrimethylursÄure ist der Metabolit von Koffein. Das metabolische VerhÄltnis von 1,3,7-TrimethylursÄure zu Koffein kann als Biomarker zur Beschreibung der VariabilitÄt der CYP3A-AktivitÄt in einer Kohorte ausgewertet werden.

-
GC46387
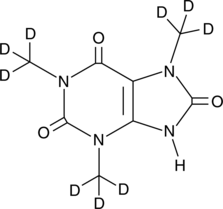
1,3,7-Trimethyluric Acid-d9
An internal standard for the quantification of 1,3,7-trimethyluric acid
-
GC62352
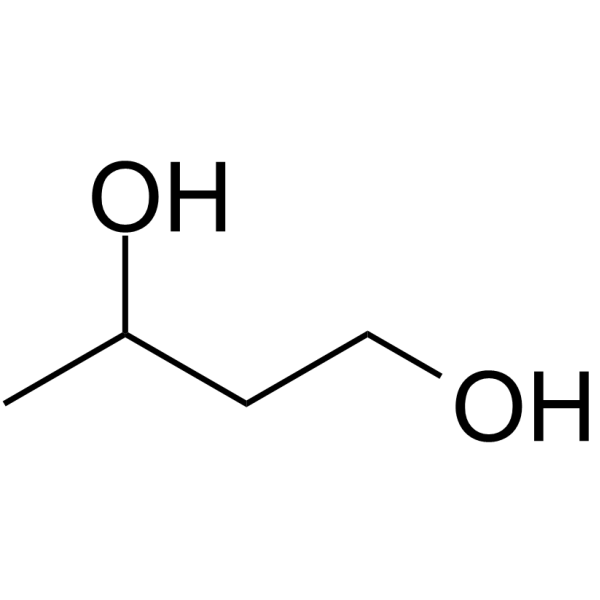
1,3-Butanediol
1,3-Butandiol, ein Ethanoldimer, das eine Kalorienquelle fÜr die menschliche ErnÄhrung darstellt.
-
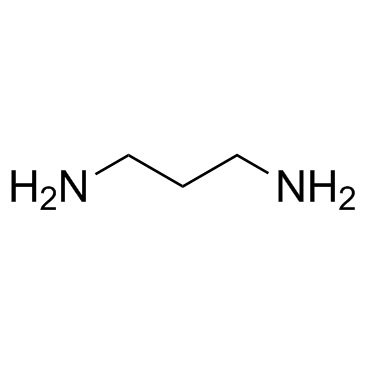
GC30742
1,3-Diaminopropane
-
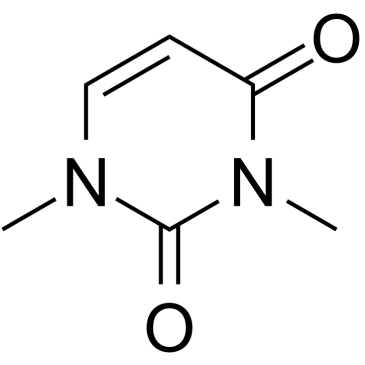
GC60433
1,3-Dimethyluracil
1,3-Dimethyluracil ist ein Pyrimidon, das sich von einem Uracil ableitet.
-
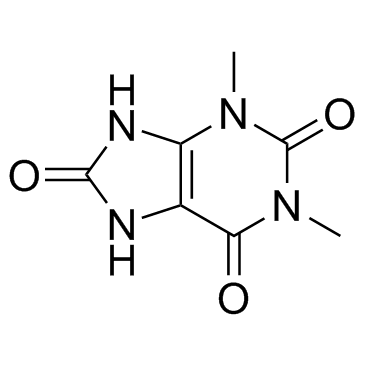
GC30749
1,3-Dimethyluric acid
1,3-DimethylurinsÄure ist ein Produkt des Theophyllin-Stoffwechsels beim Menschen.
-
GC60011
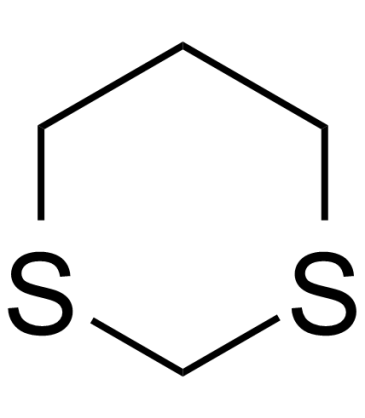
1,3-Dithiane
1,3-Dithian ist ein geschÜtztes Formaldehyd-Anion-Äquivalent, das als nÜtzliches markiertes Synthon dienen kÖnnte.
-
GC19528
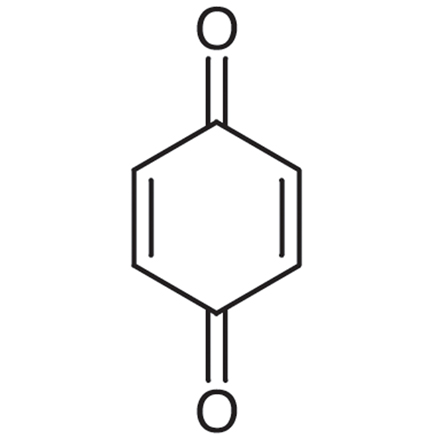
1,4-Benzoquinone
A toxic metabolite of benzene
-
GC62755
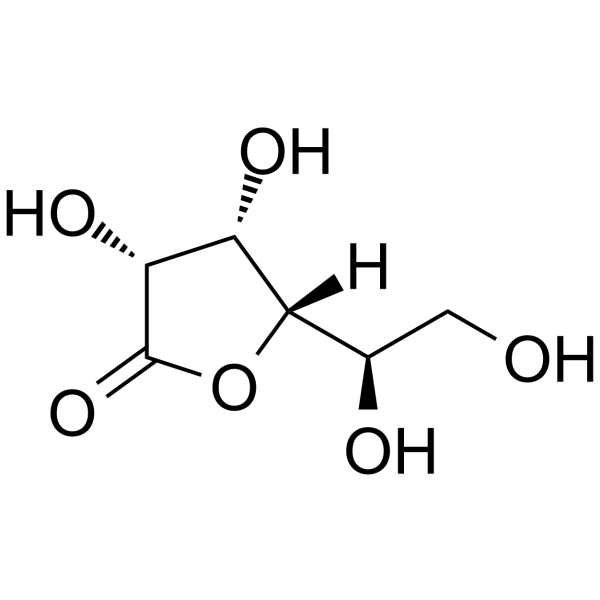
1,4-D-Gulonolactone
1,4-D-Gulonolacton ist ein kÖrpereigener Metabolit.
-
GC35039
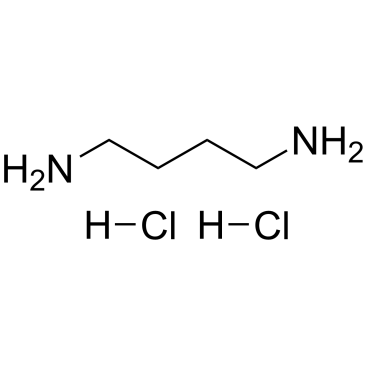
1,4-Diaminobutane dihydrochloride
1,4-Diaminobutan (Putrescin)-Dihydrochlorid ist ein endogenes Metabolit, das als Indikator fÜr umweltbedingten Stress in hÖheren Pflanzen dient: Gerste und Raps, die mit Cr(III) oder Cr(VI) gestresst sind.
-
GC68486
1,4-Diaminobutane-d8 dihydrochloride
1,4-Diaminobutan-d8 (Dihydrochlorid) ist das deuterierte Analogon von 1,4-Diaminobutan Dihydrochlorid. 1,4-Diaminobutan (Putrescin) Dihydrochlorid ist ein endogenes Stoffwechselprodukt und dient als Indikator für Verschmutzung durch Cr(III) oder Cr(VI)-Stress bei höheren Pflanzen wie Gerste und Raps.
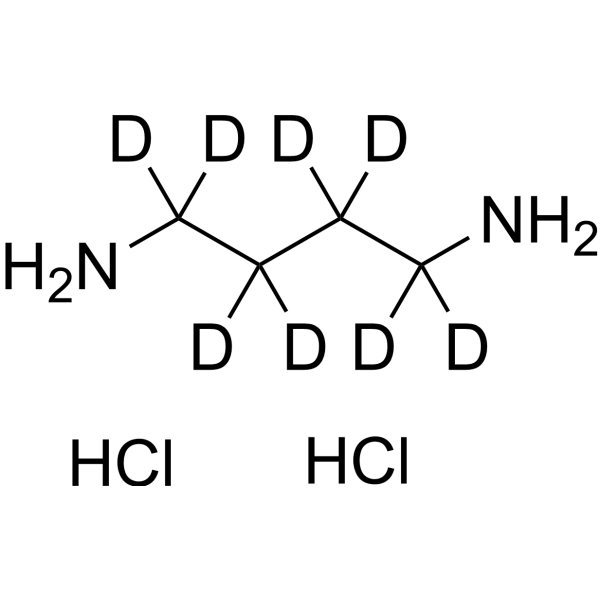
-
GC60435
1,4-Dimethoxybenzene
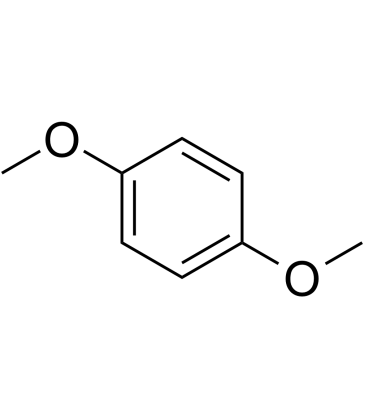
-
GC38242
1,4-Dioxane-2,5-diol
1,4-Dioxan-2,5-diol ist ein kÖrpereigener Metabolit.
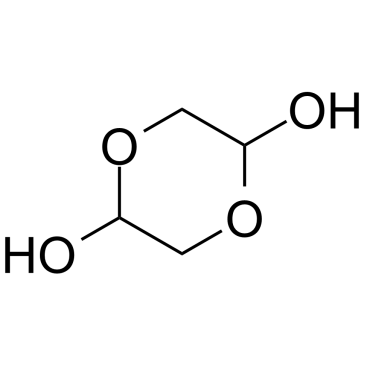
-
GC33801
1,5-Anhydrosorbitol
1,5-Anhydrosorbitol ist ein Kurzzeitmarker fÜr die glykÄmische Kontrolle.
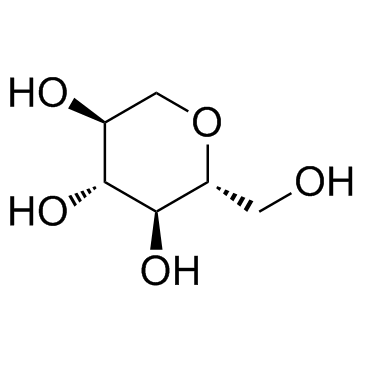
-
GC19717
1,6-anhydroglucose
1,6-Anhydroglucose (1,6-Anhydro-β-D-Glucopyranose) ist ein Anhydrozucker, der durch Glucan-Pyrolyse hergestellt wird und in der Natur weit verbreitet ist.
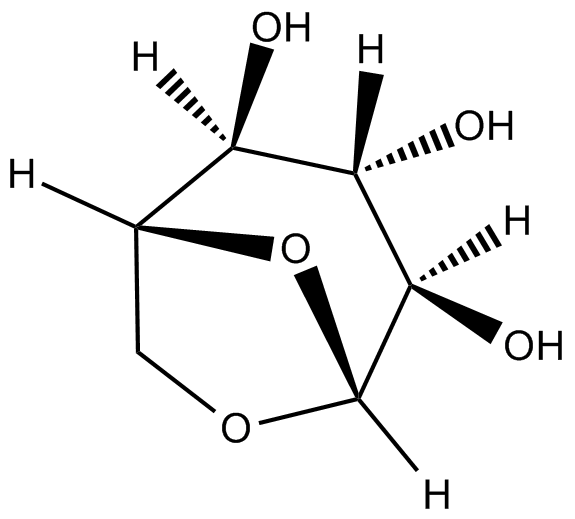
-
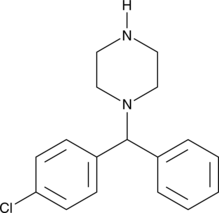
GC49294
1-(4-Chlorobenzhydryl)piperazine
An inactive metabolite of meclizine and chlorcyclizine
-
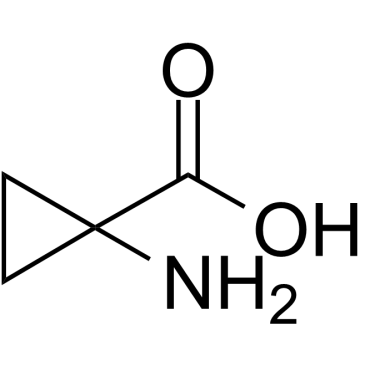
GC38270
1-Aminocyclopropane-1-carboxylic acid
1-Aminocyclopropan-1-carbonsÄure ist ein kÖrpereigener Metabolit.
-
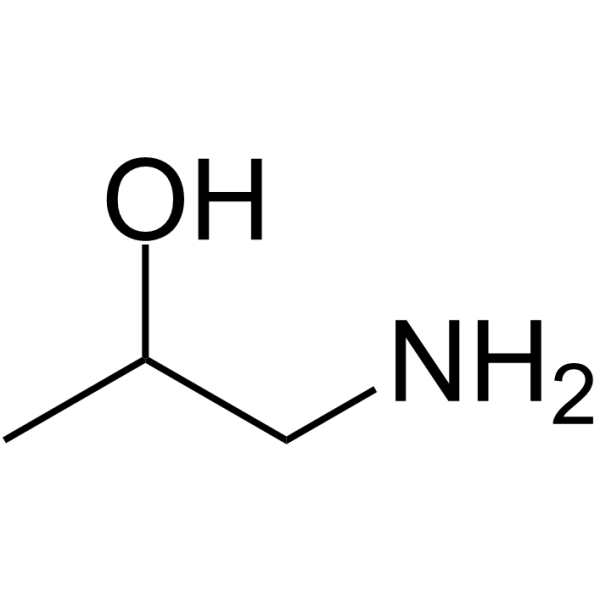
GC64404
1-Aminopropan-2-ol
1-Aminopropan-2-ol ist ein mikrobieller Metabolismus des Aminoalkoholstoffwechsels Über Propionaldehyd und Acetaldehyd in einer Pseudomonas-Art .
-
GC35029
1-Arachidoyl-sn-glycero-3-phosphocholine
1-Arachidoyl-sn-glycero-3-phosphocholin ist ein Lysophospholipid (LyP).
-
GC60445
1-Dodecanol
1-Dodecanol ist ein kÖrpereigener Metabolit.
-
GC17829
1-Hexadecanol
1-Hexadecanol ist ein Fettalkohol, ein lipophiles Substrat.

-
GC68495
1-Hexadecanol-d4
1-Hexadecanol-d4 ist das Deuterium-Isotop von 1-Hexadecanol. 1-Hexadecanol ist ein Fettalkohol und ein lipophiles Substrat.
-
GC68496
1-Hexadecanol-d5
1-Hexadecanol-d5 ist das Deuterium-Isotop von 1-Hexadecanol. 1-Hexadecanol ist ein Fettalkohol und ein lipophiles Substrat.
-
GC38271
1-Hydroxy-2-naphthoic acid
1-Hydroxy-2-naphthoesÄure ist ein kÖrpereigener Metabolit.
-
GC38670
1-Hydroxyoctadecane
1-Hydroxyoctadecan ist ein kÖrpereigener Metabolit.
-
GC60448
1-Hydroxypyrene
1-Hydroxypyren, ein Biomarker fÜr die Exposition gegenÜber polyzyklischen aromatischen Kohlenwasserstoffen (PAKs), wird in Urinproben analysiert.
-
GC32294
1-Kestose
1-Kestose, die kleinste Fructooligosaccharid-Komponente, die sowohl Faecalibacterium prausnitzii als auch Bifidobakterien effizient stimuliert.
-
GC46482
1-Linoleoyl-2-Hydroxy-sn-glycero-3-PC
1-Linoleoyl-2-Hydroxy-sn-glycero-3-PC (1-Linoleoyl-2-Hydroxy-sn-glycero-3-PC), ein Lysophospholipid, ist ein potenzieller Biomarker, der aus dem polyzystischen Ovarialsyndrom mit Insulinresistenz (IR) identifiziert wurde (PCOS).
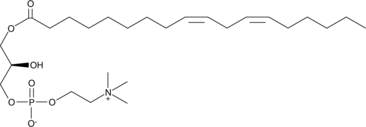
-
GC64598
1-Methyl-L-histidine-d3
1-Methyl-L-Histidin-d3 ist das mit Deuterium markierte 1-Methyl-L-Histidin.

-
GC33646
1-Methyladenine
1-Methyladenin ist ein Produkt von AlkylierungsschÄden in der DNA, das durch Schadensumkehr durch oxidative Demethylierung repariert werden kann.
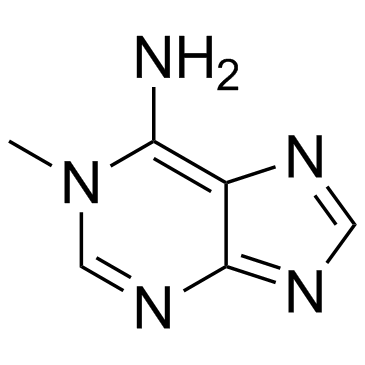
-
GC33463
1-Methyladenosine
1-Methyladenosin ist eine RNA-Modifikation, die im Wesentlichen aus zwei unterschiedlichen Reaktionstypen stammt, von denen einer durch Enzyme katalysiert wird und der andere das Ergebnis der Reaktion von RNA mit bestimmten Alkylierungsmitteln ist.
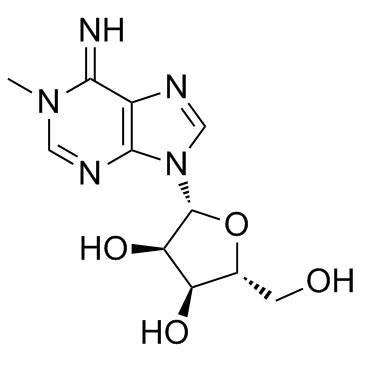
-
GC61442
1-Methylguanidine hydrochloride
1-Methylguanidinhydrochlorid ist ein kÖrpereigener Metabolit.
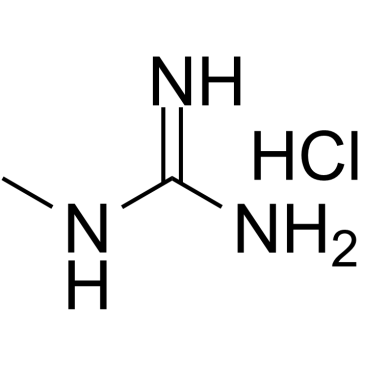
-
GC35067
1-Methylguanosine
1-Methylguanosin ist ein methyliertes Nukleosid, das aus dem RNA-Abbau stammt. 1-Methylguanosin ist ein Tumormarker.

-
GC41999
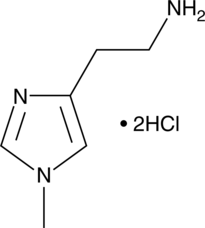
1-Methylhistamine (hydrochloride)
1-Methylhistamin (Hydrochlorid) ist ein Histamin-Metabolit.
-
GC65038
1-Methylinosine
1-Methylinosin ist ein modifiziertes Nukleotid, das an Position 37 in tRNA 3' zum Anticodon von eukaryotischer tRNA gefunden wird.

-
GC42000
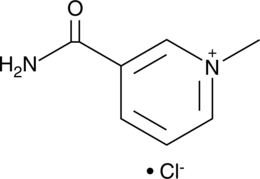
1-Methylnicotinamide (chloride)
1-Methylnicotinamid (Chlorid) (1-Methylnicotinamidchlorid) ist ein kÖrpereigener Metabolit.
-
GC31610
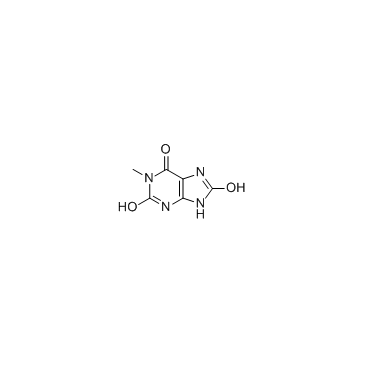
1-Methyluric acid
1-MethylursÄure wirkt auf die Schleimhaut der Harnblase und erhÖht den Blutzucker-, Insulin-, Triglycerid- und Cholesterinspiegel.
-
GC60449
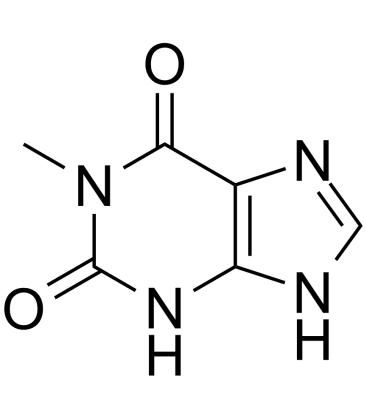
1-Methylxanthine
1-Methylxanthin, ein Koffeinderivat, ist ein essentieller Metabolit von Koffein und Theophyllin (1,3-Dimethylxanthin, TP) im menschlichen Urin. 1-Methylxanthin verstÄrkt die Strahlenempfindlichkeit von Tumorzellen.
-
GC35068
1-Monomyristin
1-Monomyristin, extrahiert aus Serenoa repens, hemmt die Hydrolyse von 2-Oleoylglycerol (IC50=32 μM) und die AktivitÄt der FettsÄureamidhydrolase (FAAH) (IC50=18 μM).

-
GC38556
1-Myristoyl-2-stearoyl-sn-glycero-3-phosphocholine
1-Myristoyl-2-stearoyl-sn-glycero-3-phosphocholin ist ein kÖrpereigener Metabolit.
-
GC30641
1-Naphthol (Fourrine ERN)
1-Naphthol ist eine fluoreszierende Molekularsonde mit Protonentransfer im angeregten Zustand (ESPT).
-
GA20382
1-O-Octadecyl-rac-glycerol

-
GC39470
1-Octanol
1-Octanol (Octanol), ein gesÄttigter Fettalkohol, ist ein Inhibitor der T-Typ-CalciumkanÄle (T-KanÄle) mit einem IC50 von 4 μM fÜr native T-StrÖme.
-
GC40183
1-Oleoyl Glycerol
1-Oleoylglycerol ist ein kÖrpereigener Metabolit.
-
GC46488
1-Palmitoyl-2-Docosahexaenoyl-sn-glycero-3-PC
A phospholipid
-
GC68501
1-Palmitoyl-2-oleoyl-sn-glycerol
1-Palmitoyl-2-oleoyl-sn-glycerol ist eine endogene Metabolit und auch der Hauptdiacylglycerin in Hypoxie-induzierbaren Faktor (HIF)-1 bei Algen.
-
GC49366
1-Salicylate Glucuronide
A metabolite of salicylic acid and aspirin
-
GC42039
1-Stearoyl-2-15(S)-HETE-sn-glycero-3-PC
1-Stearoyl-2-15(S)-HETE-sn-glycero-3-PC is a phospholipid that contains stearic acid at the sn-1 position and 15(S)-HETE at the sn-2 position.
-
GC42040
1-Stearoyl-2-15(S)-HETE-sn-glycero-3-PE
1-Stearoyl-2-15(S)-HETE-sn-glycero-3-PE is a phospholipid that contains stearic acid at the sn-1 position and 15(S)-HETE at the sn-2 position.

-
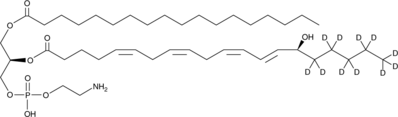
GC49730
1-Stearoyl-2-15(S)-HETE-sn-glycero-3-PE-d11
An internal standard for the quantification of 1-stearoyl-2-15(S)-HETE-sn-glycero-3-PE
-
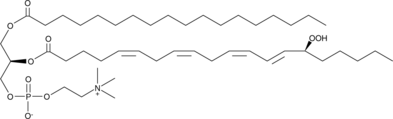
GC42041
1-Stearoyl-2-15(S)-HpETE-sn-glycero-3-PC
1-Stearoyl-2-15(S)-HpETE-sn-glycero-3-PC is a phospholipid that contains stearic acid at the sn-1 position and 15(S)-HpETE at the sn-2 position.
-
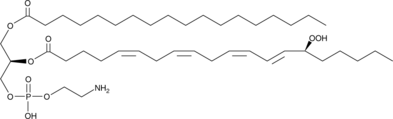
GC42042
1-Stearoyl-2-15(S)-HpETE-sn-glycero-3-PE
1-Stearoyl-2-15(S)-HpETE-sn-glycero-3-PE is a phospholipid that contains stearic acid at the sn-1 position and 15(S)-HpETE at the sn-2 position.
-
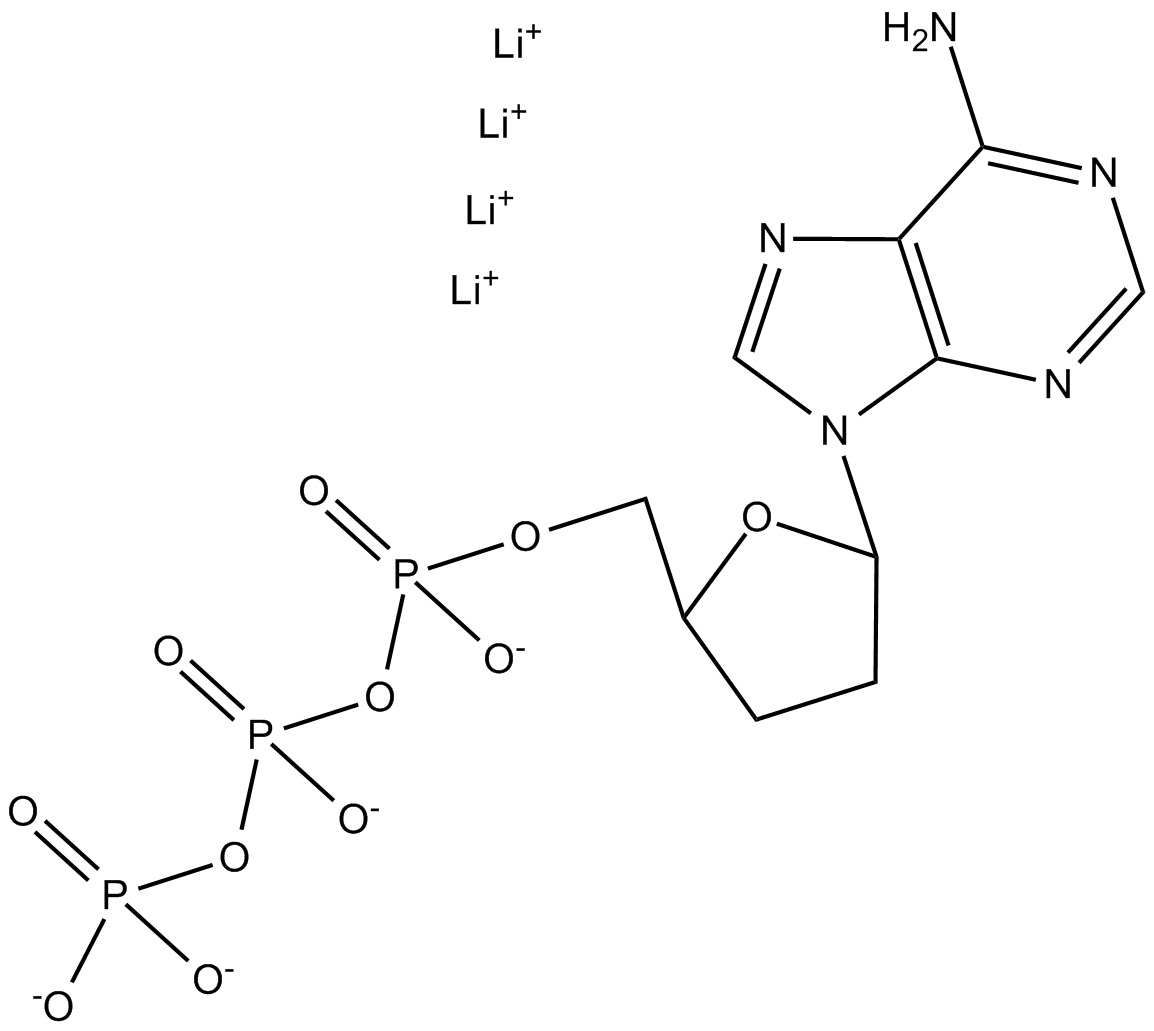
GC11139
1-Stearoyl-2-Arachidonoyl-sn-glycero-3-PC
A phospholipid
-
GC68502
1-Stearoyl-2-linoleoyl-sn-glycero-3-phosphocholine
1-Stearoyl-2-linoleoyl-sn-glycero-3-phosphocholin kann als Modell für flüchtige Verbindungen verwendet werden, die zur Untersuchung von Phosphatidylcholin-Molekülarten eingesetzt werden. Diese Methode wurde auf tatsächliche Lebensmittelproben angewendet, nämlich Sojalecithin.
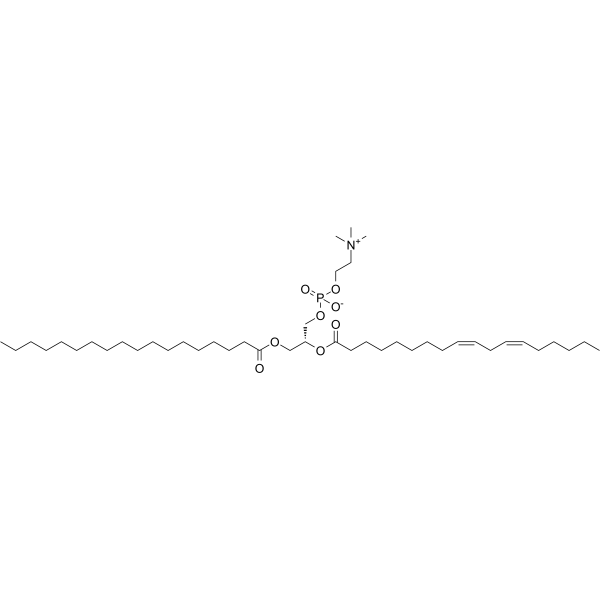
-
GC40971
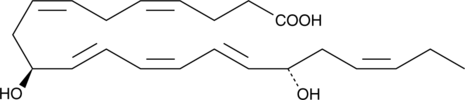
10(S),17(S)-DiHDHA
10(S),17(S)-DiHDHA (auch als Neuroprotectin D1 bekannt, wenn es in neuronalen Geweben produziert wird) ist eine von DHA abgeleitete DihydroxyfettsÄure, die starke schÜtzende und entzÜndungshemmende AktivitÄten aufweist.
-
GC49820
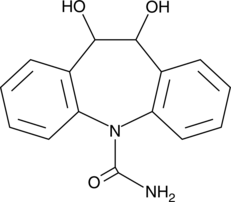
10,11-dihydro-10,11-dihydroxy Carbamazepine
A metabolite of carbamazepine and oxcarbazepine
-
GC66057
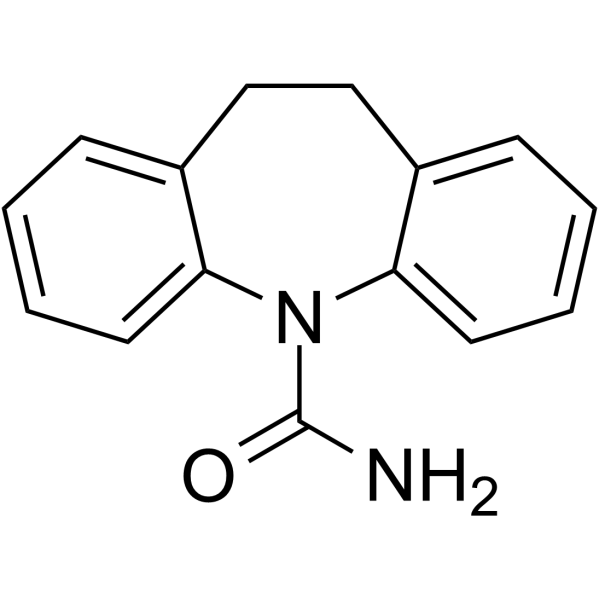
10,11-Dihydrocarbamazepine
10,11-Dihydrocarbamazepin ist der aktive Metabolit von Oxcarbazepin. 10,11-Dihydrocarbamazepin ist ebenfalls ein Zwischenprodukt. Oxcarbazepin wird schnell und fast vollstÄndig in 10,11-Dihydrocarbazepin mit wahrscheinlicher antikonvulsiver Wirksamkeit umgewandelt.
-
GC38881
10,12-Tricosadiynoic acid
10,12-TricosadiinsÄure ist ein hochspezifischer, selektiver, hochaffiner und oral aktiver Acyl-CoA-Oxidase-1 (ACOX1)-Inhibitor.
-
GC49872
10-Formyltetrahydrofolate (sodium salt) (technical grade)
10-Formyltetrahydrofolat (Natriumsalz) (technische QualitÄt) ist eine Form von TetrahydrofolsÄure, die als Spender von Formylgruppen im Anabolismus wirkt.