Proteases
Proteases is a general term for a class of enzymes that hydrolyze protein peptide chains. According to the way they degrade polypeptides, they are divided into two categories: endopeptidases and telopeptidases. The former can cut the large molecular weight polypeptide chain from the middle to form prions and peptones with smaller molecular weights; the latter can be divided into carboxypeptidase and aminopeptidase, which respectively remove the peptide from the free carboxyl terminus or free amino terminus of the polypeptide one by one. Chain hydrolysis produces amino acids.
A general term for a class of enzymes that hydrolyze peptide bonds in proteins. According to the way they hydrolyze polypeptides, they can be divided into endopeptidases and exopeptidases. Endopeptidase cleaves the interior of the protein molecule to form smaller molecular weight peptones and peptones. Exopeptidase hydrolyzes peptide bonds one by one from the end of the free amino group or carboxyl group of protein molecules, and frees amino acids, the former is aminopeptidase and the latter is carboxypeptidase. Proteases can be classified into serine proteases, sulfhydryl proteases, metalloproteases and aspartic proteases according to their active centers and optimum pH. According to the optimum pH value of its reaction, it is divided into acidic protease, neutral protease and alkaline protease. The proteases used in industrial production are mainly endopeptidases.
Proteases are widely found in animal offal, plant stems and leaves, fruits and microorganisms. Microbial proteases are mainly produced by molds and bacteria, followed by yeast and actinomycetes.
Enzymes that catalyze the hydrolysis of proteins. There are many kinds, the important ones are pepsin, trypsin, cathepsin, papain and subtilisin. Proteases have strict selectivity for the reaction substrates they act on. A protease can only act on certain peptide bonds in protein molecules, such as the peptide bonds formed by the hydrolysis of basic amino acids catalyzed by trypsin. Proteases are widely distributed, mainly in the digestive tract of humans and animals, and are abundant in plants and microorganisms. Due to limited animal and plant resources, the industrial production of protease preparations is mainly prepared by fermentation of microorganisms such as Bacillus subtilis and Aspergillus terrestris.
Ziele für Proteases
- Caspase(114)
- Aminopeptidase(24)
- ACE(74)
- Calpains(20)
- Carboxypeptidase(10)
- Cathepsin(81)
- DPP-4(31)
- Elastase(26)
- Gamma Secretase(67)
- HCV Protease(59)
- HSP(113)
- HIV Integrase(37)
- HIV Protease(47)
- MMP(228)
- NS3/4a protease(8)
- Serine Protease(18)
- Thrombin(58)
- Urokinase(4)
- Cysteine Protease(0)
- Other Proteases(18)
- Tyrosinases(47)
- 15-PGDH(1)
- Acetyl-CoA Carboxylase(13)
- Acyltransferase(59)
- Aldehyde Dehydrogenase (ALDH)(28)
- Aminoacyl-tRNA Synthetase(9)
- ATGL(1)
- Dipeptidyl Peptidase(56)
- Drug Metabolite(457)
- E1/E2/E3 Enzyme(90)
- Endogenous Metabolite(1636)
- FABP(30)
- Farnesyl Transferase(23)
- Glutaminase(14)
- Glutathione Peroxidase(14)
- Isocitrate Dehydrogenase (IDH)(28)
- Lactate Dehydrogenase(17)
- Lipoxygenase(234)
- Mitochondrial Metabolism(207)
- NEDD8-activating Enzyme(7)
- Neprilysin(12)
- PAI-1(13)
- Ser/Thr Protease(41)
- Tryptophan Hydroxylase(13)
- Xanthine Oxidase(18)
- MALT1(10)
- PCSK9(1)
Produkte für Proteases
- Bestell-Nr. Artikelname Informationen
-
GC41282
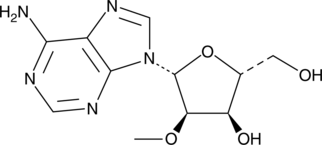
2'-O-Methyladenosine
2'-O-Methyladenosin, ein methylierter Adeninrest, wird im Urin von Normalpersonen sowie im Urin von Patienten mit Adenosindeaminase (ADA)-Mangel gefunden.
-
GC60452
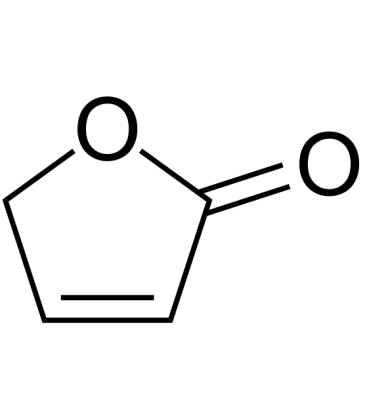
2(5H)-Furanone
-
GC39686
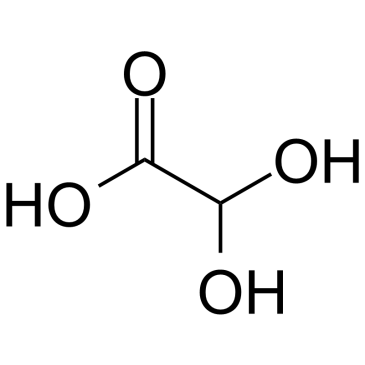
2,2-Dihydroxyacetic acid
2,2-DihydroxyessigsÄure ist ein kÖrpereigener Metabolit.
-
GC60457
2,3,5-Trimethylpyrazine
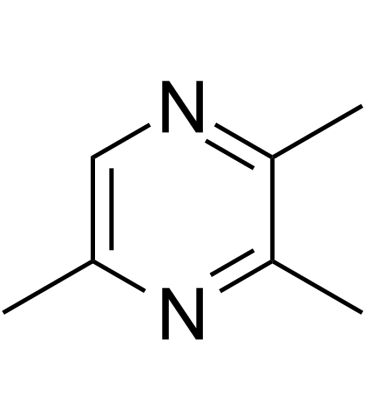
-
GC60458
2,3-Butanediol
2,3-Butandiol ist ein Butandiol, das aus der biologischen Umwandlung natÜrlicher Ressourcen gewonnen wird.
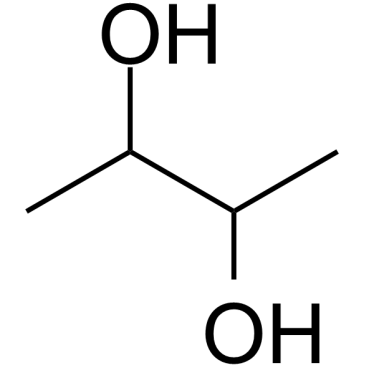
-
GC38243
2,3-Diaminopropanoic acid hydrochloride
2,3-DiaminopropansÄurehydrochlorid ist ein kÖrpereigener Metabolit.
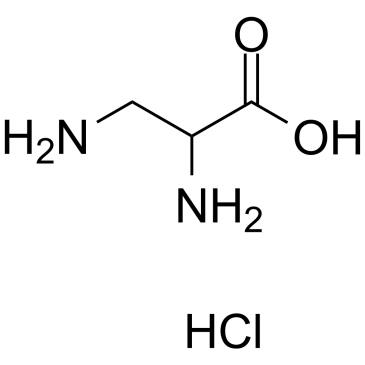
-
GC30672
2,3-Diaminopropionic acid
2, 3-DiaminopropionsÄure ist ein Metabolit von b-Oxalyl-L-a, b-DiaminopropionsÄure ist eine neurotoxische AminosÄure (ODAP).
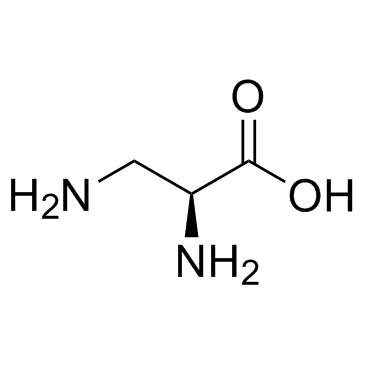
-
GC60463
2,4-Di-tert-butylphenol
-
GC39772
2,4-Dihydroxybenzaldehyde
2,4-Dihydroxybenzaldehyd ist ein kÖrpereigener Metabolit.
-
GC33626
2,4-Dihydroxybenzoic acid
2,4-DihydroxybenzoesÄure ist ein Abbauprodukt von cyanidierendem Glycosid aus SauerkÄse in Zellkultur.
-
GC38367
2,4-Dihydroxypyrimidine-5-carboxylic Acid
2,4-Dihydroxypyrimidin-5-carbonsÄure ist ein kÖrpereigener Metabolit.
-
GC33519
2,5-Dihydroxybenzoic acid
2,5-DihydroxybenzoesÄure ist ein Derivat von BenzoesÄure und ein starker Inhibitor von Fibroblasten-Wachstumsfaktoren.
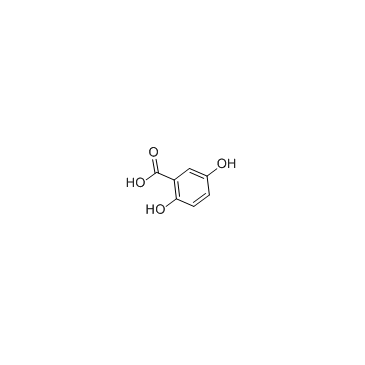
-
GC46057
2,5-Dihydroxycinnamic Acid phenethyl ester
An inhibitor of 5-LO
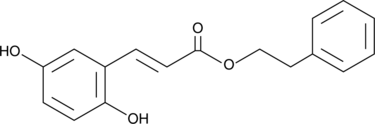
-
GC38729
2,5-Dimethylpyrazine
2,5-Dimethylpyrazin ist ein kÖrpereigener Metabolit.
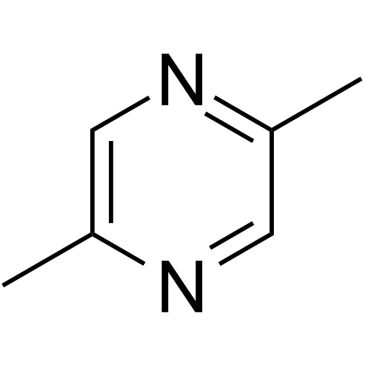
-
GC33637
2,5-Furandicarboxylic acid
Die im menschlichen Urin nachgewiesene 2,5-FurandicarbonsÄure ist ein wichtiger nachwachsender biotechnologischer Baustein, da sie als umweltfreundlicher Ersatz fÜr TerephthalsÄure bei der Herstellung von Polyestern dient.
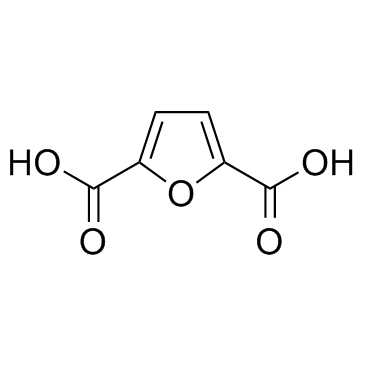
-
GA20387
2,6-Diaminopimelic acid
2,6-DiaminopimelinsÄure ist ein kÖrpereigener Metabolit.

-
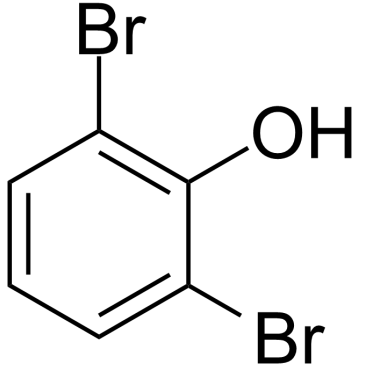
GC39773
2,6-Dibromophenol
2,6-Dibromphenol ist ein kÖrpereigener Metabolit.
-
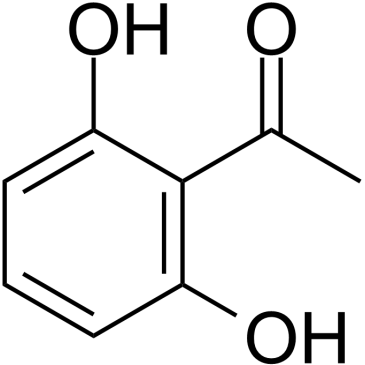
GC39767
2,6-Dihydroxyacetophenone
2,6-Dihydroxyacetophenon ist ein kÖrpereigener Metabolit.
-
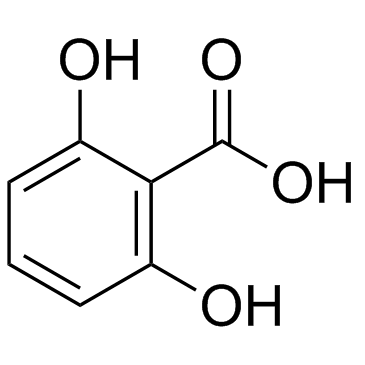
GC33642
2,6-Dihydroxybenzoic acid
2,6-DihydroxybenzoesÄure ist ein sekundÄrer Metabolit der SalicylsÄure, die wÄhrend der Metabolisierungsphase I durch Leberenzyme hydrolysiert wurde.
-
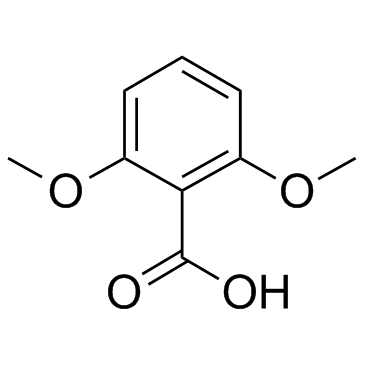
GC33643
2,6-Dimethoxybenzoic acid
2,6-DimethoxybenzoesÄure ist ein Mitglied der organischen Verbindungen, die als o-MethoxybenzoesÄuren und Derivate bekannt sind.
-
GC39768
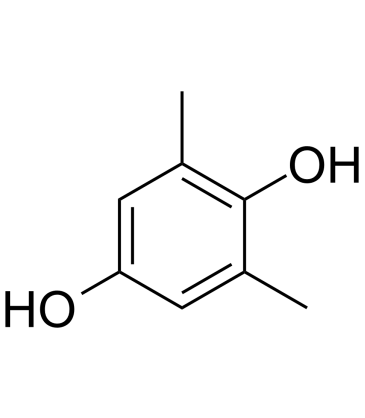
2,6-Dimethylhydroquinone
-
GC62766
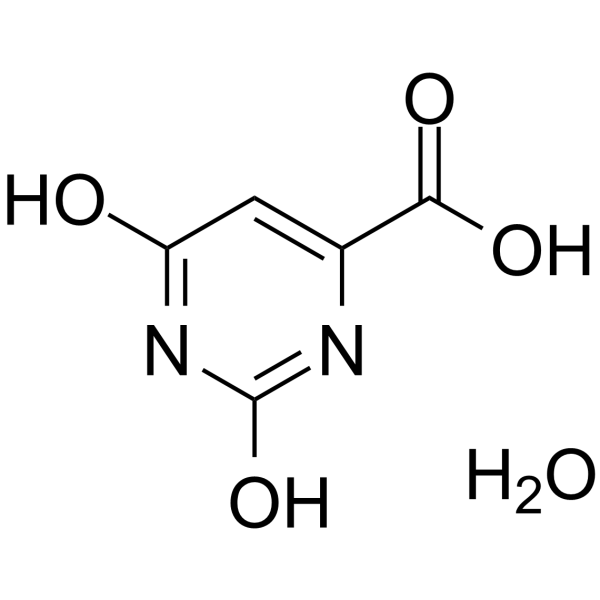
2,6-Dioxo-1,2,3,6-tetrahydropyrimidine-4-carboxylic acid hydrate
2,6-Dioxo-1,2,3,6-tetrahydropyrimidin-4-carbonsÄurehydrat ist ein kÖrpereigener Metabolit.
-
GC65461
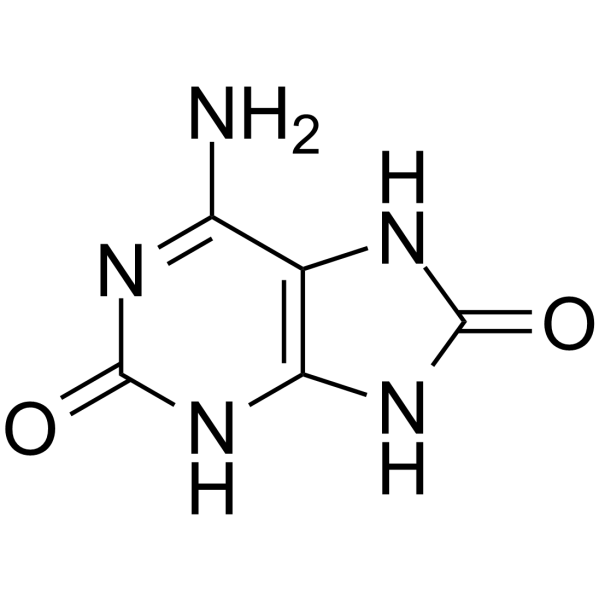
2,8-Dihydroxyadenine
2,8-Dihydroxyadenin, ein kÖrpereigener Metabolit, kann zur Bildung von Harnkristallen und Nierensteinen fÜhren.
-
GC30697
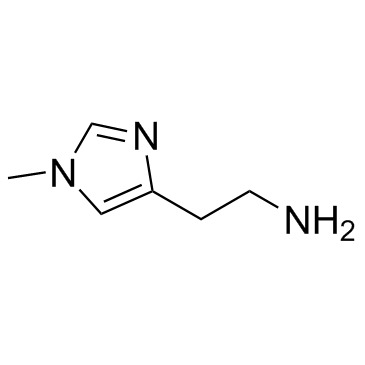
2-(1-Methyl-1H-imidazol-4-yl)ethan-1-amine
2-(1-Methyl-1H-imidazol-4-yl)ethan-1-amin ist ein Metabolit von Histamin (Him).
-
GC49159
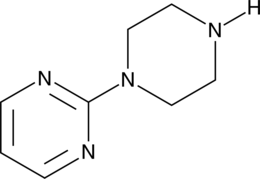
2-(1-Piperazinyl)pyrimidine
An α2-AR antagonist and active metabolite of azapirones
-
GC38301
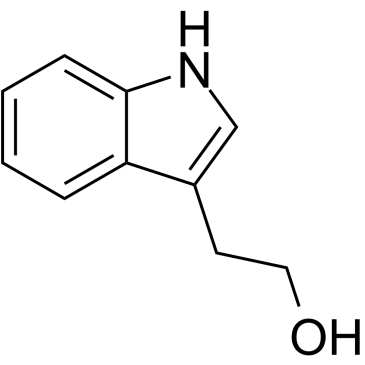
2-(1H-Indol-3-yl)ethan-1-ol
2-(1H-Indol-3-yl)ethan-1-ol ist ein kÖrpereigener Metabolit.
-
GC62763
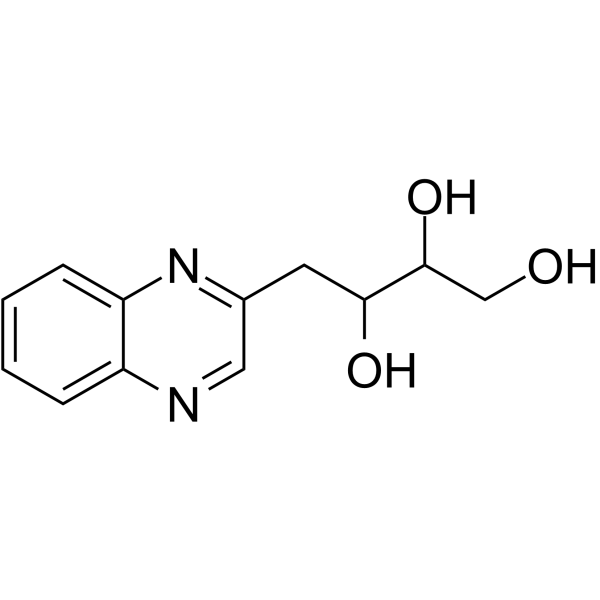
2-(2’,3’,4’-Trihydroxybutyl)quinoxaline
2-(&2rsquo;,3’,4’-Trihydroxybutyl)chinoxalin ist ein Lebensmittelmetabolit.
-
GC49330
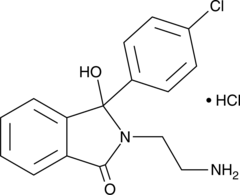
2-(2-Aminoethyl)-3-(4-chlorophenyl)-3-hydroxyisoindol-1-one (hydrochloride)
A metabolite of mazindol
-
GC33633
2-(2-Methylbenzamido)acetic acid
2-(2-Methylbenzamido)essigsÄure ist ein Metabolit, der im Urin nachgewiesen wird.
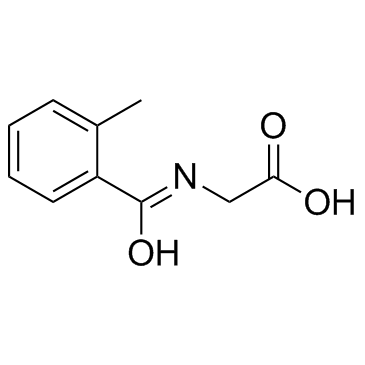
-
GC68504
2-(2-Methylbenzamido)acetic acid-d7
2-(2-Methylbenzamido)essigsäure-d7 ist das Deuterium-Isotop von 2-(2-Methylbenzamido)essigsäure. 2-(2-Methylbenzamido)essigsäure ist ein Metabolit, der im Urin nachgewiesen werden kann.
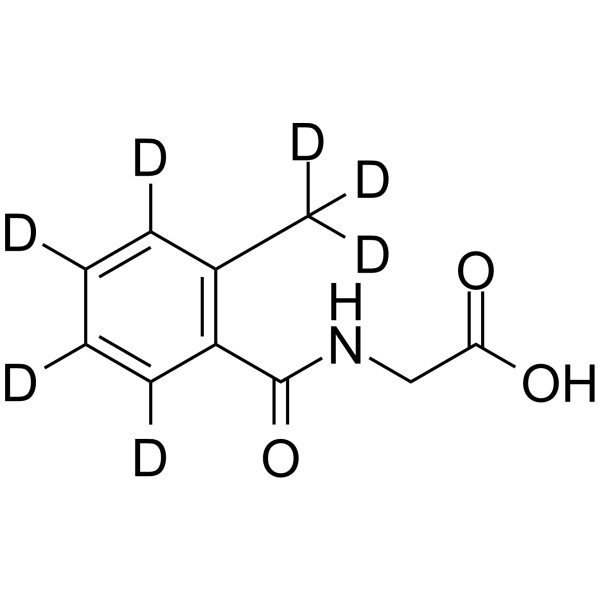
-
GC34193
2-(4-Methoxyphenyl)acetic acid (4-Methoxyphenylacetic acid)
2-(4-Methoxyphenyl)essigsÄure (4-MethoxyphenylessigsÄure) ist ein Plasmametabolit mit hohem SensitivitÄts- und SpezifitÄtswert als Biomarker zur Unterscheidung zwischen NSCLC und gesunden Kontrollen.
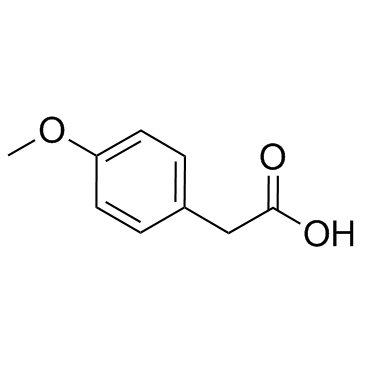
-
GC33496
2-(Methylamino)-1H-purin-6(7H)-one (N2-methylguanine)
2-(Methylamino)-1H-purin-6(7H)-on (N2-Methylguanin) (N2-Methylguanin) ist ein modifiziertes Nukleosid.
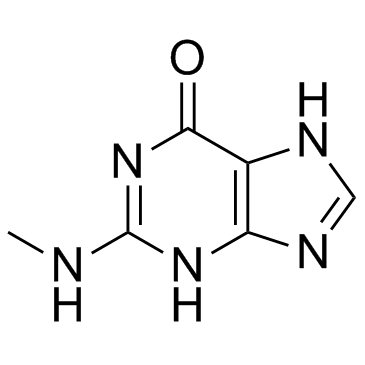
-
GC12964
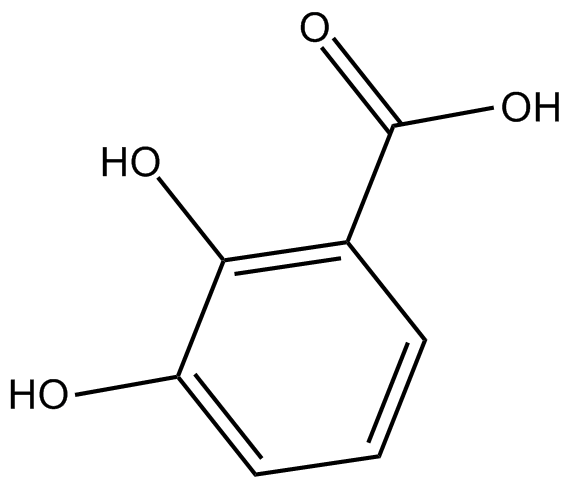
2-3-Dihydroxybenzoic acid
2-3-DihydroxybenzoesÄure ist ein normaler menschlicher BenzoesÄure-Metabolit, der im Plasma vorkommt und nach der Einnahme von Aspirin erhÖhte Konzentrationen aufweist.
-
GC39844
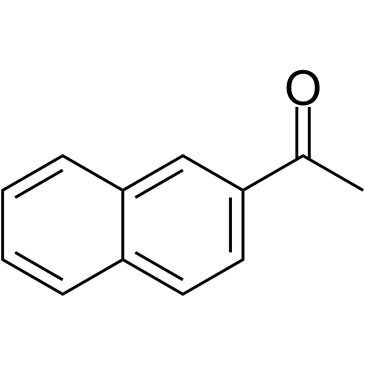
2-Acetonaphthone
2-Acetonaphthon ist ein kÖrpereigener Metabolit.
-
GC60469
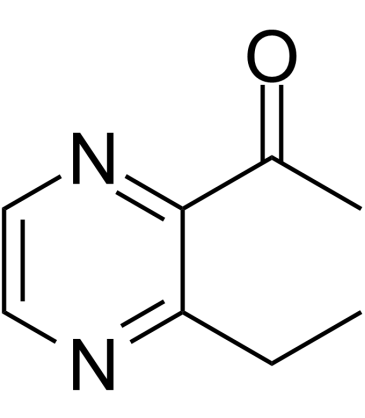
2-Acetyl-3-ethylpyrazine
2-Acetyl-3-ethylpyrazin ist ein kÖrpereigener Metabolit.
-
GC30718
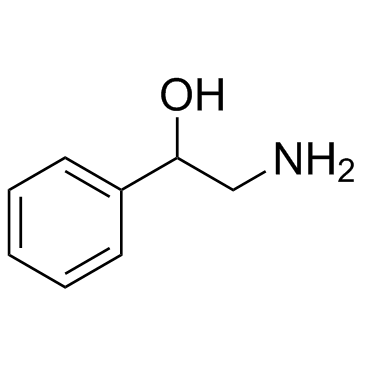
2-Amino-1-phenylethanol
2-Amino-1-phenylethanol ist ein Analogon von Noradrenalin.
-
GC66717
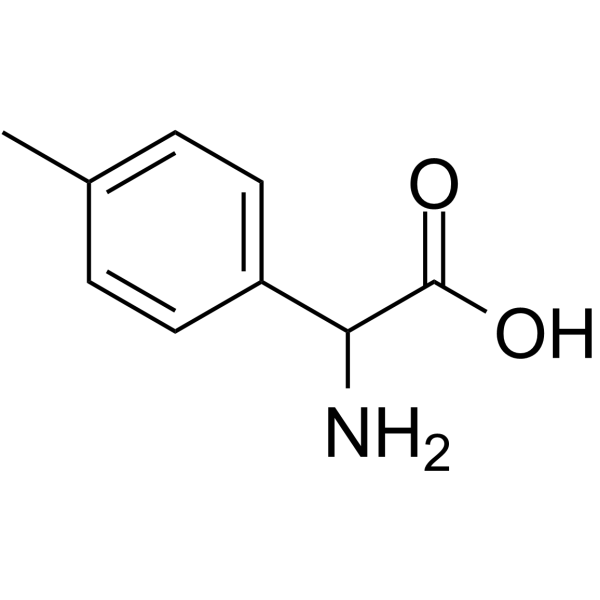
2-Amino-2-(p-tolyl)acetic acid
2-Amino-2-(p-tolyl)essigsäure wird zur Optimierung des Azidgerüsts verwendet und ist das Zwischenprodukt bei der Synthese von 1,3,4-Thiadiazolverbindungen. 1,3,4-Thiadiazol-Verbindungen zeigen potentielle Anti-Krebs-Aktivität und hemmen Glutaminase (GLSI).
-
GC62769
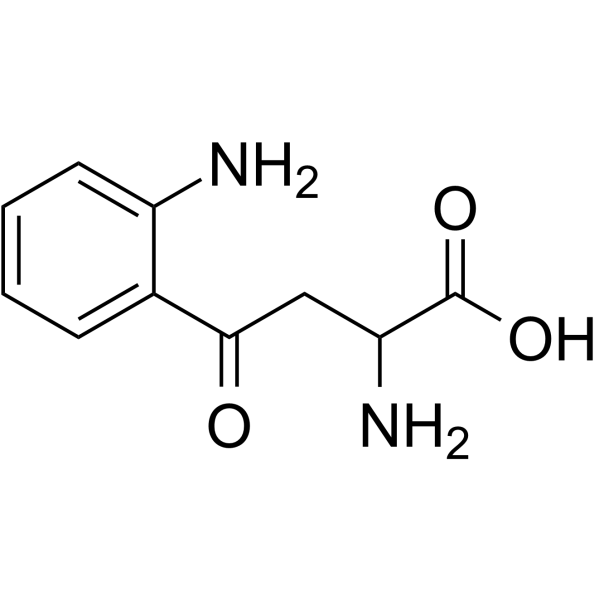
2-Amino-4-(2-aminophenyl)-4-oxobutanoic acid
2-Amino-4-(2-aminophenyl)-4-oxobutansÄure ist ein kÖrpereigener Metabolit.
-
GC60470
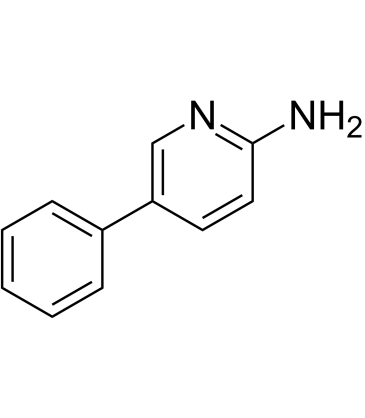
2-Amino-5-phenylpyridine
2-Amino-5-phenylpyridin ist ein mutagenes heterocyclisches aromatisches Amin, das durch Pyrolyse von Phenylalanin in Proteinen gebildet wird. 2-Amino-5-phenylpyridin ist in gegrillten Sardinen enthalten und gilt als potenziell krebserregend.
-
GC62770
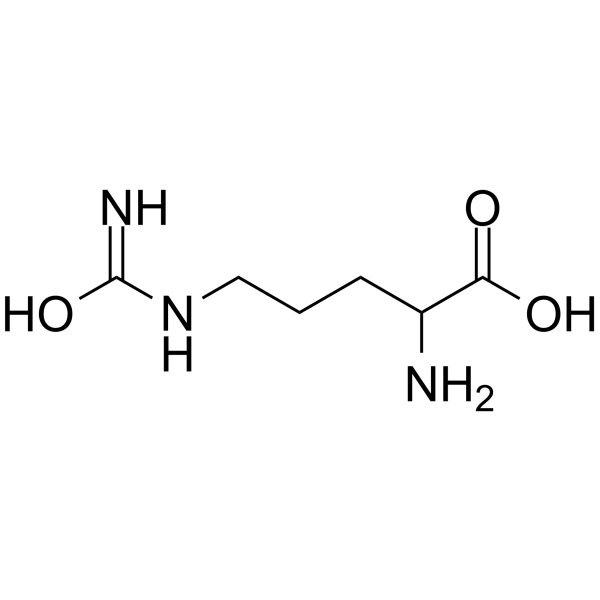
2-Amino-5-ureidopentanoic acid
2-Amino-5-ureidopentansÄure ist ein kÖrpereigener Metabolit.
-
GC38260
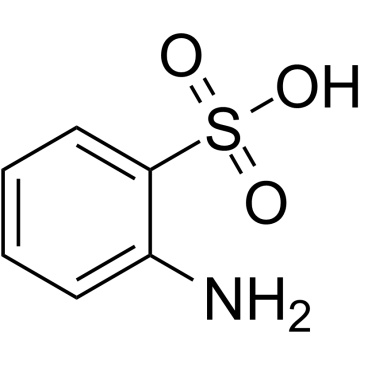
2-Aminobenzenesulfonic acid
2-AminobenzolsulfonsÄure ist ein kÖrpereigener Metabolit.
-
GC52029
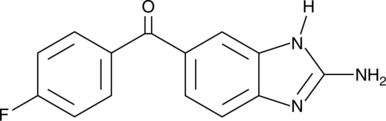
2-Aminoflubendazole
2-Aminoflubendazol ist der Metabolit von Benzimidazolen.
-
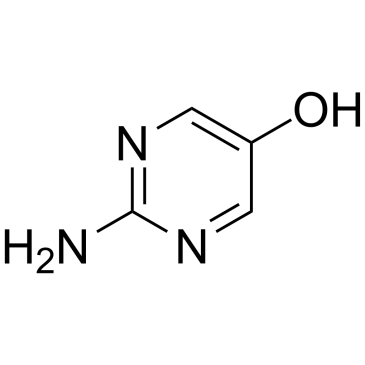
GC38321
2-Aminopyrimidin-5-ol
-
GC16403
2-Arachidonoyl Glycerol
Ein endogener Agonist der Cannabinoid-Rezeptoren CB1 und CB2.

-
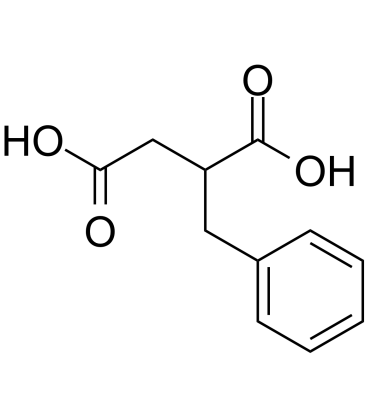
GC60471
2-Benzylsuccinic acid
2-BenzylbernsteinsÄure (DL-BenzylbernsteinsÄure) ist ein potenter Inhibitor der Carboxypeptidase A (CPA).
-
GC46539
2-Chloroadenine
A heterocyclic building block
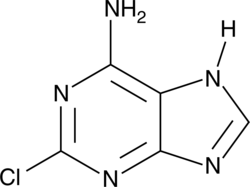
-
GC11553
2-cyano-Pyrimidine
2-Cyano-Pyrimidin ist ein potenter und nicht-selektiver Cysteinprotease-Cathepsin-K-Inhibitor mit einem IC50 von 170 nM.
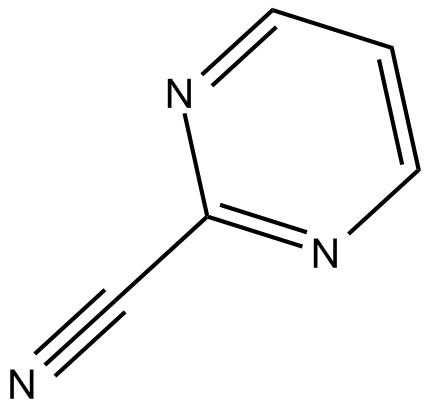
-
GC10408
2-D08
2-D08 ist ein zellgÄngiger, mechanistisch einzigartiger Inhibitor der Protein-SUMOylierung. 2-D08 hemmt auch Axl mit einem IC50 von 0,49 nM.
-
GC40675
2-deoxy-Artemisinin
2-deoxy-Artemisinin is an inactive metabolite of the antimalarial agent artemisinin.
-
GC12850
2-Deoxy-D-ribose
2-Desoxy-D-Ribose ist ein kÖrpereigener Metabolit.
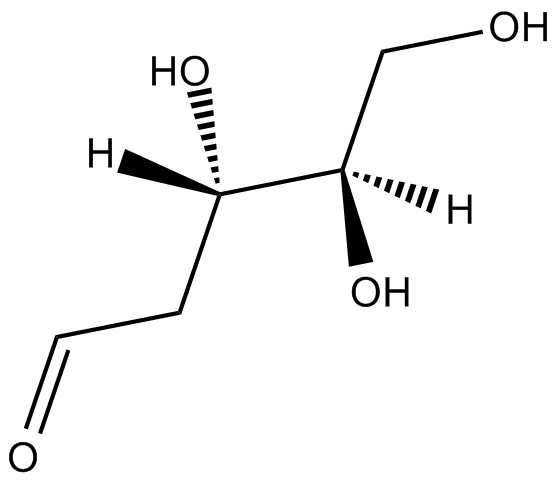
-
GC11642
2-Deoxyadenosine
2-Desoxyadenosin ist ein Nukleosid-Adenosin-Derivat, das sich in doppelsträngiger DNA mit Desoxythymidin (T) paart.
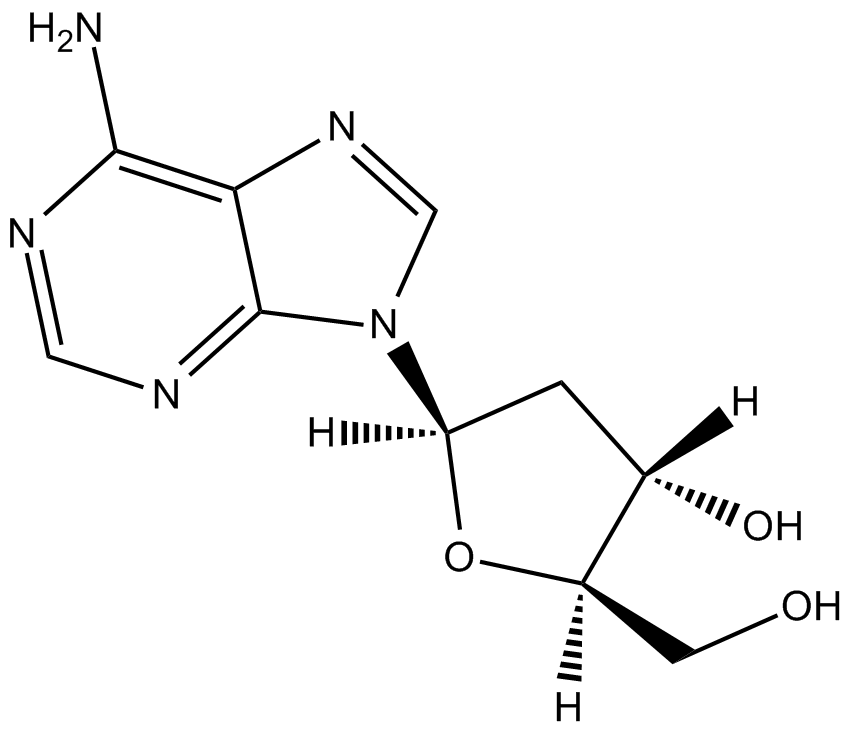
-
GC17478
2-Deoxycytidine
2-Desoxycytidin, ein Desoxyribonukleosid, könnte die biologischen Wirkungen von Bromodeoxyuridin (Brdu) hemmen.
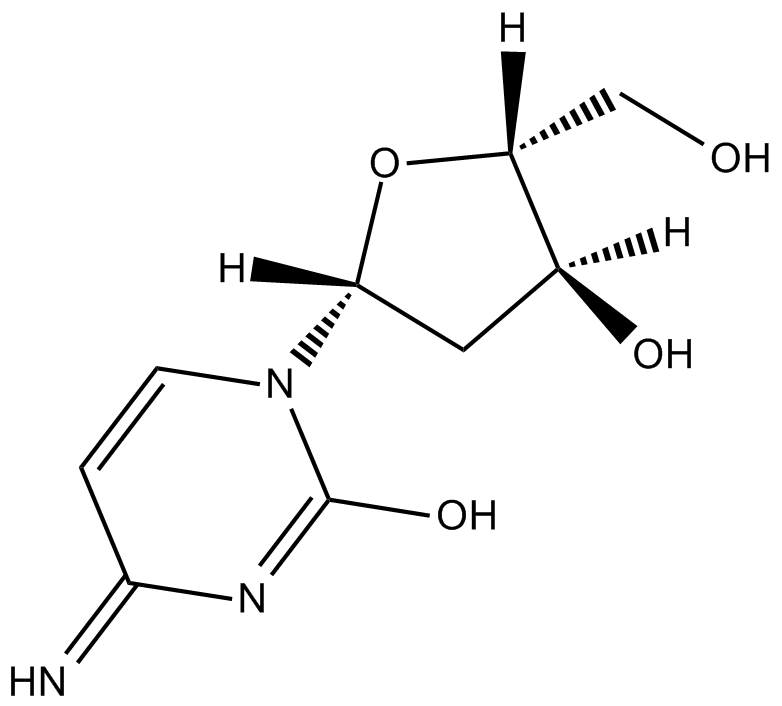
-
GC11581
2-Deoxyinosine
2’-Desoxyadenosin hemmt das Wachstum menschlicher Kolonkarzinom-Zelllinien und steht im Zusammenhang mit Purin-Nukleosid-Phosphorylase (PNP)-Mangel.
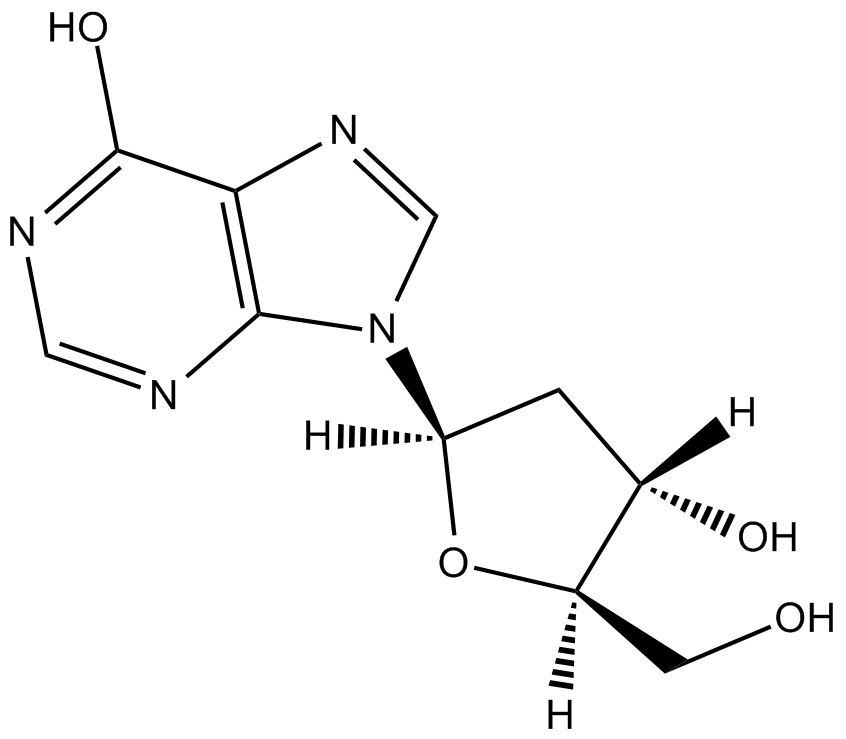
-
GC13571
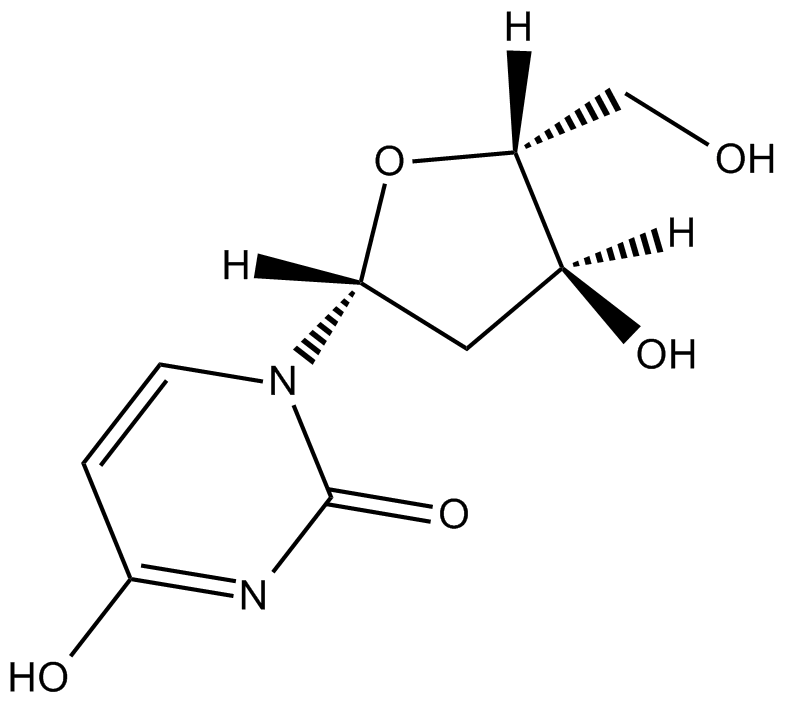
2-Deoxyuridine
2-Desoxyuridin könnte den Chromosomenbruch verstärken und führt zu einer verminderten Thymidylat-Synthetase-Aktivität.
-
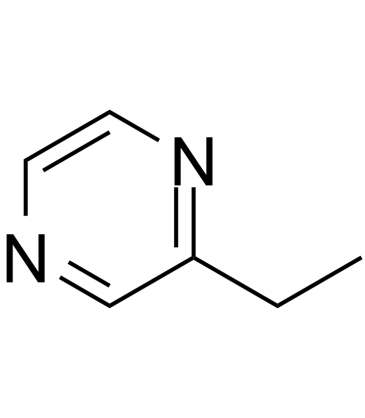
GC60472
2-Ethylpyrazine
-
GC31632
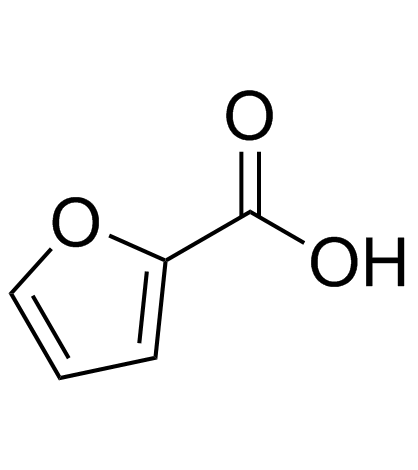
2-Furoic acid (Furan-2-carboxylic acid)
2-FurinsÄure (Furan-2-CarbonsÄure) (Furan-2-CarbonsÄure) ist eine organische Verbindung, die durch Furfuraloxidation hergestellt wird.
-
GC33645
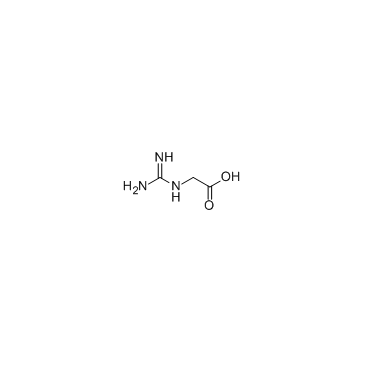
2-Guanidinoacetic acid
2-GuanidinoessigsÄure (GuanidinoessigsÄure), eine Vorstufe von Kreatin, ist ein Ersatz fÜr Arginin in der Nahrung und kÖnnte die allgemeine EnergiehomÖostase des Vogels unterstÜtzen.
-
GC11804
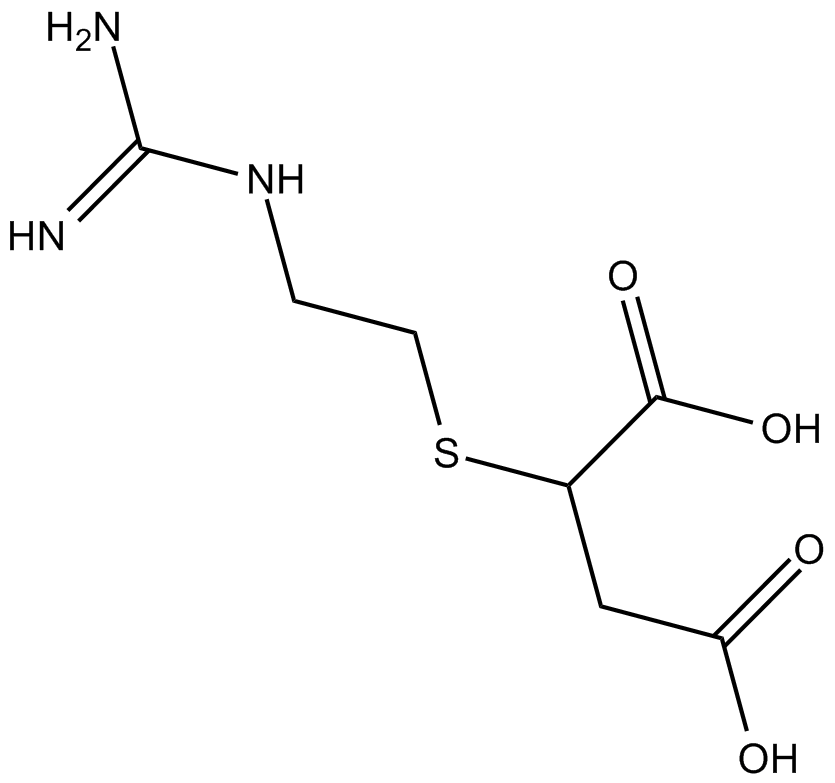
2-Guanidinoethylmercaptosuccinic Acid
2-GuanidinoethylmercaptobernsteinsÄure (2-GuanidinoethylmercaptobernsteinsÄure) ist ein potenter spezifischer Inhibitor des Carboxypeptidase B-Ähnlichen Verarbeitungsenzyms (Enzephalinkonvertase) mit einem Ki-Wert von 8,8 nM.
-
GC12545
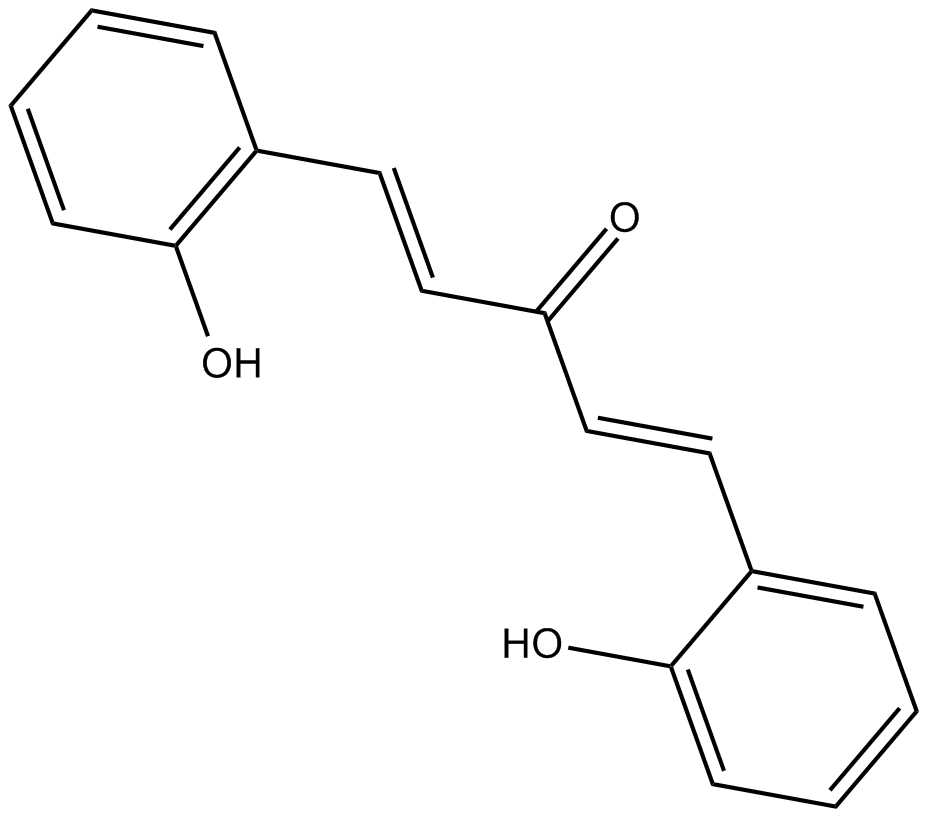
2-HBA
2-HBA ist ein starker Induktor der NAD(P)H:Chinon-Akzeptor-Oxidoreduktase 1 (NQO1), die auch Caspase-3 und Caspase-10 aktivieren kann.
-
GC35090
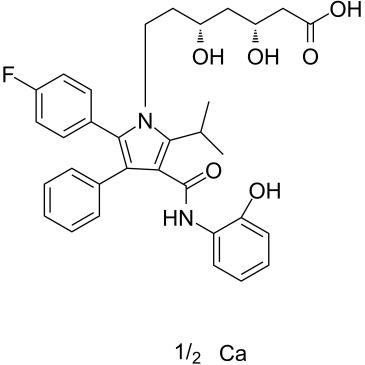
2-Hydroxy atorvastatin calcium salt
2-Hydroxy-Atorvastatin-Calciumsalz ist ein Hydroxy-Metabolit von Atorvastatin-Calciumsalz.
-
GC60474
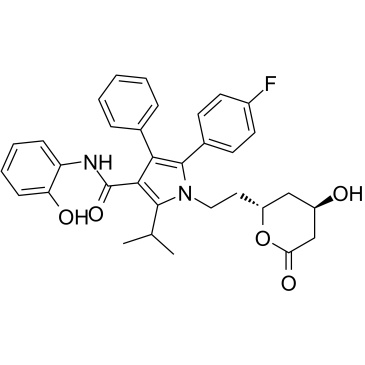
2-Hydroxy atorvastatin lactone
2-Hydroxy-Atorvastatin-Lacton ist ein Metabolit von Atorvastatin.
-
GC31544
2-Hydroxy-2-methylbutanoic acid
2-Hydroxy-2-methylbutansÄure, ein ungewÖhnlicher Metabolit, wird mit 2-HydroxyglutarsÄureurie und der Ahornsirup-Urinkrankheit in Verbindung gebracht.
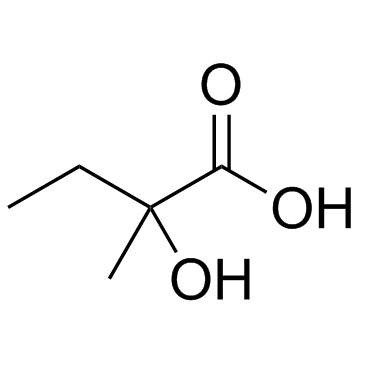
-
GC38281
2-Hydroxy-2-phenylacetic acid
2-Hydroxy-2-phenylessigsÄure ((±)-2-Hydroxy-2-phenylessigsÄure), eine alpha-HydroxycarbonsÄure, wurde weithin als Zwischenprodukt fÜr pharmazeutische und Feinchemikalien verwendet.
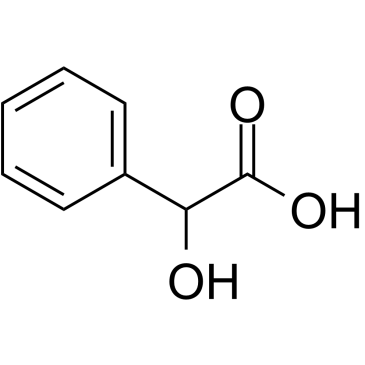
-
GC31222
2-Hydroxy-3-methylbutanoic acid
2-Hydroxy-3-methylbutansÄure ist ein Analogon von GHB mit enger Struktur, das ein natÜrlich vorkommender Neurotransmitter und ein psychoaktives Medikament ist.
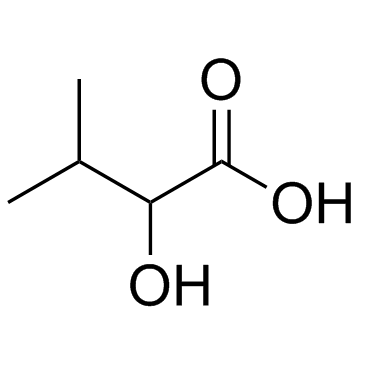
-
GC30564
2-Hydroxy-4-methoxybenzaldehyde
2-Hydroxy-4-methoxybenzaldehyd, eine chemische Verbindung und ein Isomer von Vanillin, kÖnnte zur Synthese von Urolithin M7 verwendet werden.
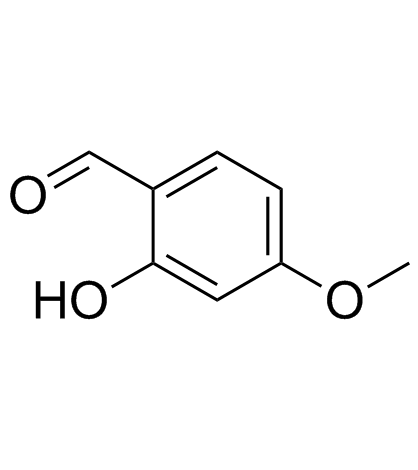
-
GC60475
2-Hydroxy-4-methylbenzaldehyde
2-Hydroxy-4-methylbenzaldehyd ist ein kÖrpereigener Metabolit.
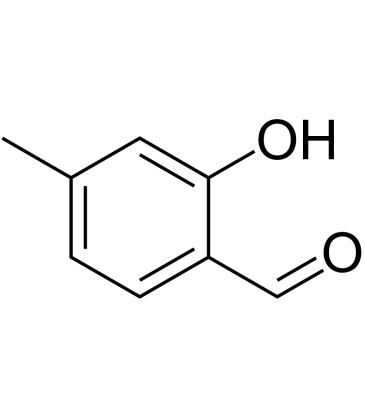
-
GC30612
2-Hydroxyadipic acid
2-HydroxyadipinsÄure ist eine organische SÄure, die durch Reduktion von 2-KetoadipinsÄure gebildet wird.
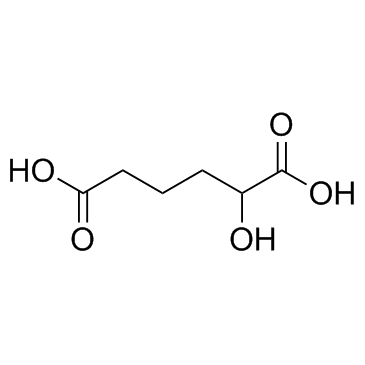
-
GC64336
2-Hydroxyemodin
2-Hydroxyemodin ist ein aktiver Metabolit von Emodin in den Lebermikrosomen.
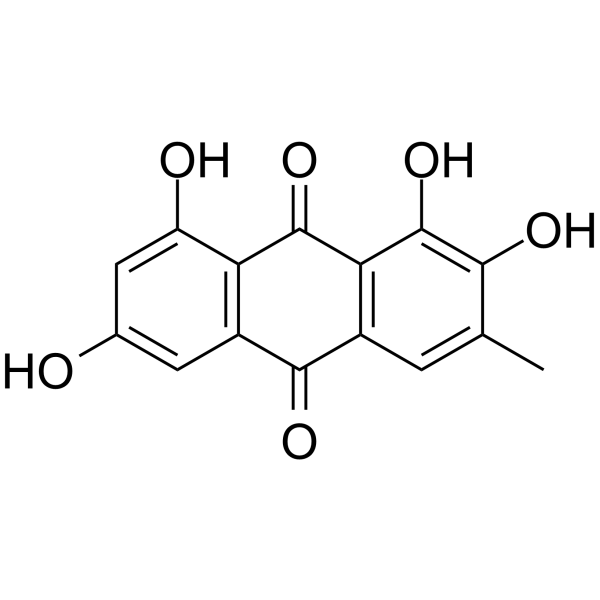
-
GC38190
2-Hydroxyhexanoic acid
2-HydroxyhexansÄure ist ein kÖrpereigener Metabolit.
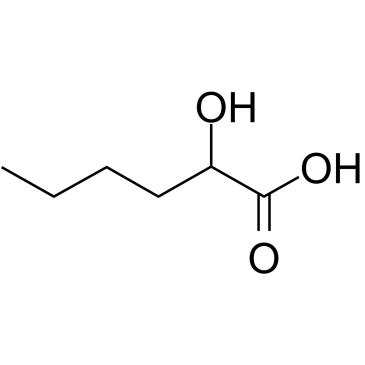
-
GC19715
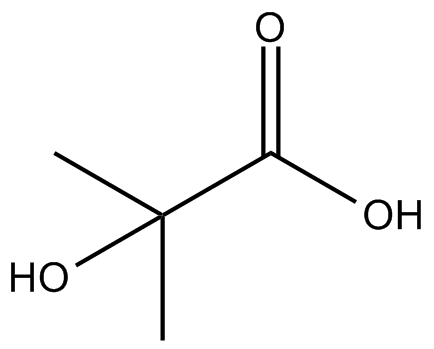
2-hydroxyisobutyrate
2-HydroxyisobuttersÄure ist ein kÖrpereigener Metabolit.
-
GC68519
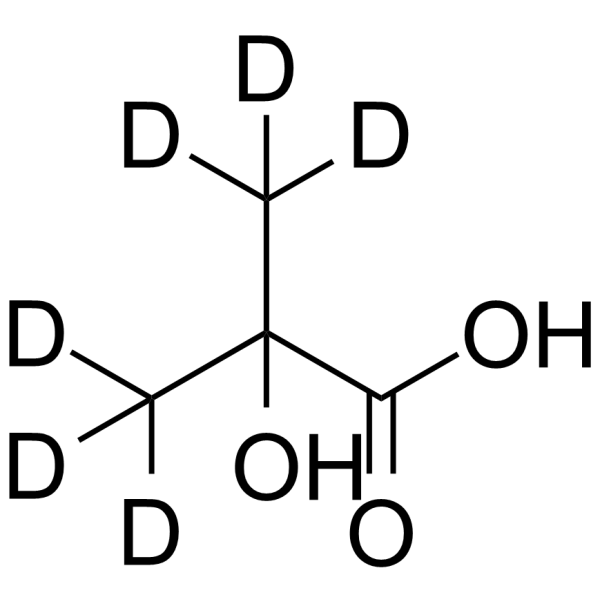
2-Hydroxyisobutyric acid-d6
2-Hydroxyisobutyric acid-d6 ist das Deuterium-Isotop von 2-Hydroxyisobuttersäure. 2-Hydroxyisobuttersäure ist ein endogenes Stoffwechselprodukt.
-
GC60476
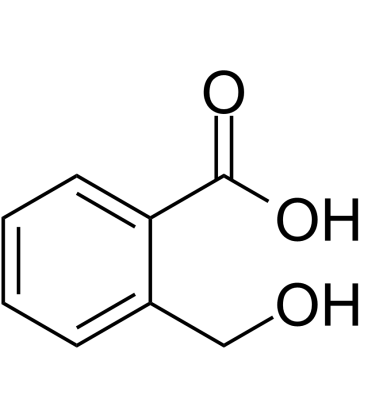
2-hydroxymethyl benzoic acid
2-HydroxymethylbenzoesÄure ist ein kÖrpereigener Metabolit.
-
GC33742
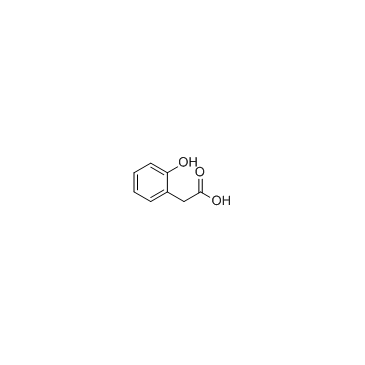
2-Hydroxyphenylacetic acid
2-HydroxyphenylessigsÄure ist ein potenzieller Biomarker fÜr Lebensmittelprodukte und steht im Zusammenhang mit Phenylketonurie (PKU).
-
GC30136
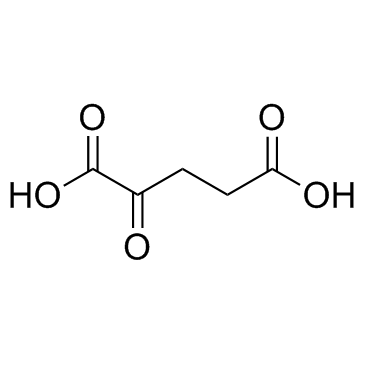
2-Ketoglutaric acid
2-KetoglutarsÄure (Alpha-KetoglutarsÄure) ist ein Zwischenprodukt bei der Produktion von ATP oder GTP im Krebszyklus.
-
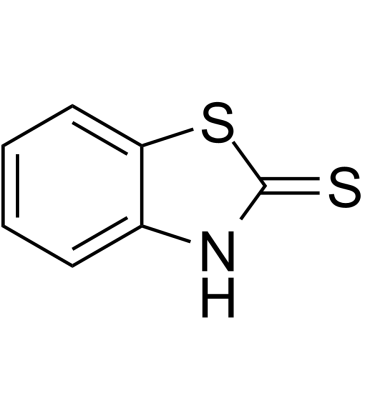
GC39761
2-Mercaptobenzothiazole
-
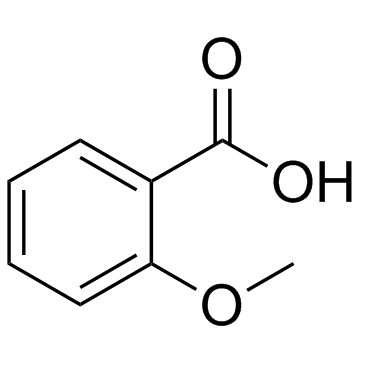
GC34179
2-Methoxybenzoic acid (NSC 3778)
2-MethoxybenzoesÄure (NSC 3778) (NSC 3778) wird als interner Standard fÜr SalicylsÄure und ihre mutmaßlichen biosynthetischen VorlÄufer in GurkenblÄttern verwendet. Eine andere bekannte Verwendung ist die Synthese von Benextramin.
-
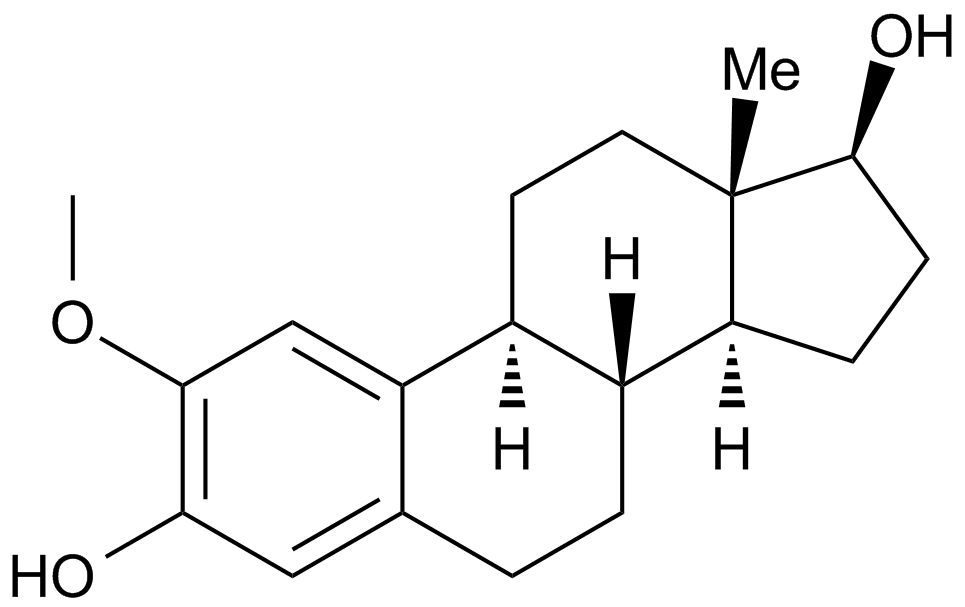
GC15084
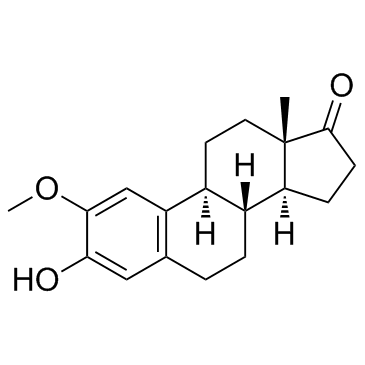
2-Methoxyestradiol (2-MeOE2)
2-Methoxyestradiol (2-MeOE2/2-Me) is a HIF-1α inhibitor that inhibits HIF-1α accumulation and HIF transcriptional activity. 2-Methoxyestradiol can trigger p53-induced apoptosis and has potential antitumor activity..
-
GC33810
2-Methoxyestrone
2-MethoxyÖstron ist ein methoxyliertes Catechin-Östrogen und Metabolit von Östron mit einem pKa-Wert von 10,81.
-
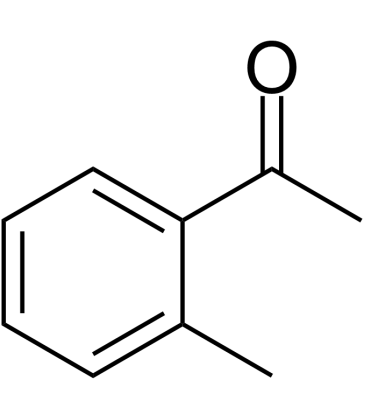
GC39776
2-Methylacetophenone
-
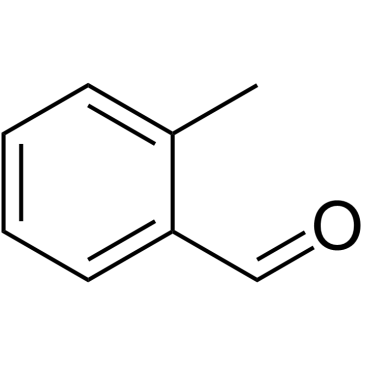
GC60477
2-Methylbenzaldehyde
2-Methylbenzaldehyd ist ein kÖrpereigener Metabolit.
-
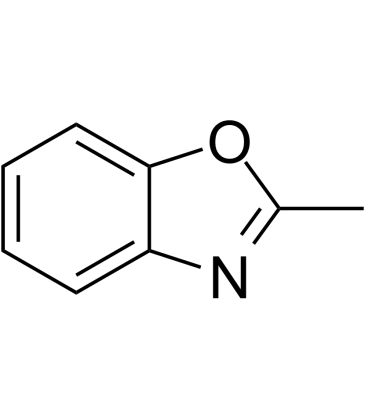
GC60478
2-Methylbenzoxazole
-
GC60479
2-Methylcitric acid
2-MethylzitronensÄure (MethylzitronensÄure) ist ein kÖrpereigener Metabolit im 2-MethylzitronensÄurezyklus.
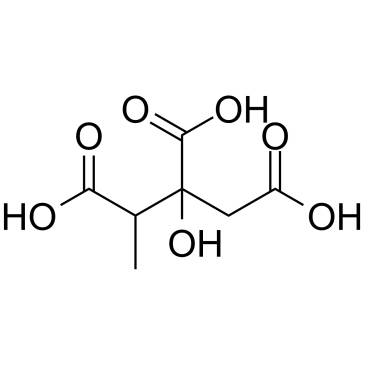
-
GC60480
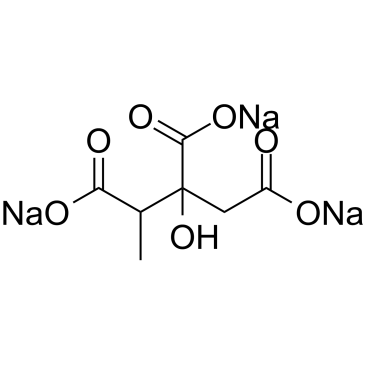
2-Methylcitric acid trisodium
2-MethylzitronensÄuretrinatrium (MethylzitronensÄuretrinatrium) ist ein kÖrpereigener Metabolit im 2-MethylzitronensÄurezyklus.
-
GC62779
2-Methylcyclohexanone
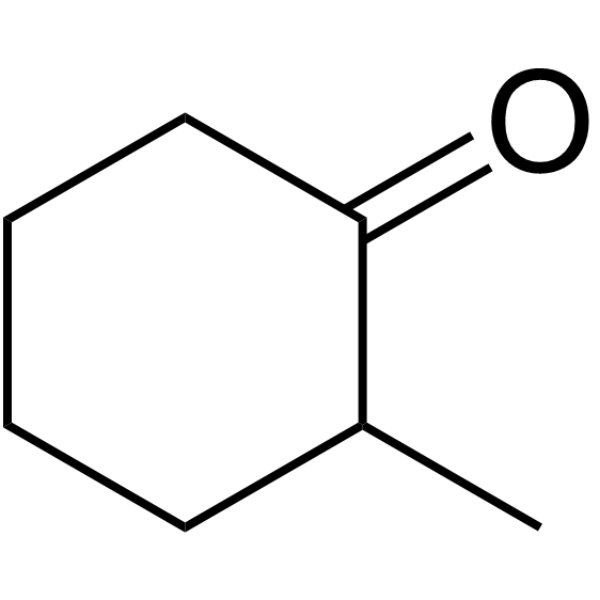
-
GC30671
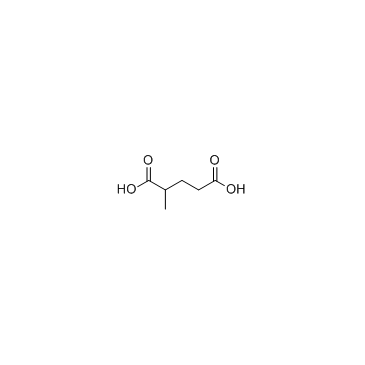
2-Methylpentanedioic acid
2-MethylpentandisÄure ist ein Metabolit von BernsteinsÄure, einem Zwischenprodukt des ZitronensÄurezyklus.
-
GC31435
2-Methylsuccinic acid
2-MethylbernsteinsÄure ist ein normaler Metabolit in menschlichen FlÜssigkeiten und das wichtigste biochemisch messbare Merkmal bei Ethylmalon-Enzephalopathie.
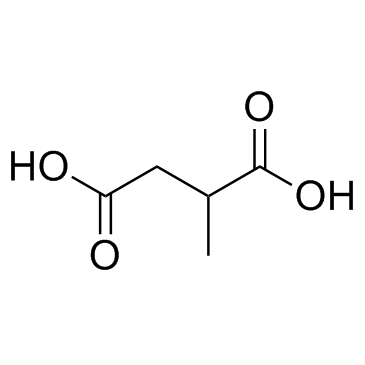
-
GC60481
2-Methylvaleric acid
2-MethylvaleriansÄure (2-MethylpentansÄure) ist eine kurzkettige FettsÄure, die aus Campomanesia adamantium und Milchprodukten isoliert wird.
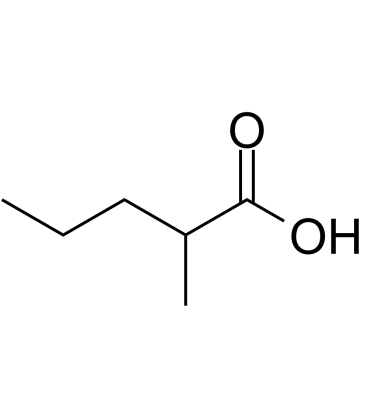
-
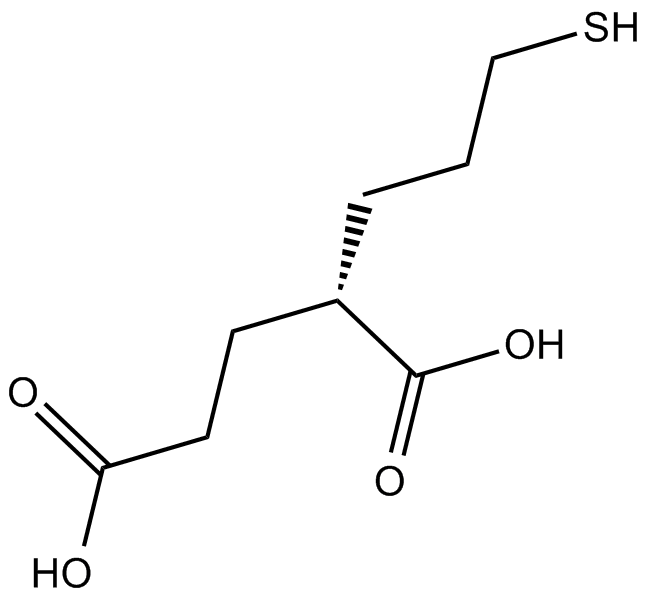
GC17937
2-MPPA
2-MPPA (GPI-5693) ist ein oral aktiver und selektiver Glutamat-Carboxypeptidase II (GCP II; PSMA)-Inhibitor mit einem IC50 von 90 nM.
-
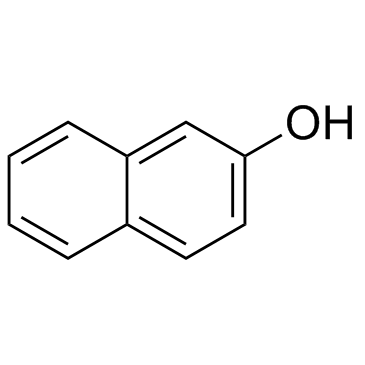
GC30720
2-Naphthol
2-Naphthol ist ein Metabolit von Naphthalin, der durch Cytochrom P450 (CYP)-Isoenzyme (CYP 1A1, CYP 1A2, CYP 2A1, CYP 2E1 und CYP 2F2) katalysiert wird.
-
GC39845
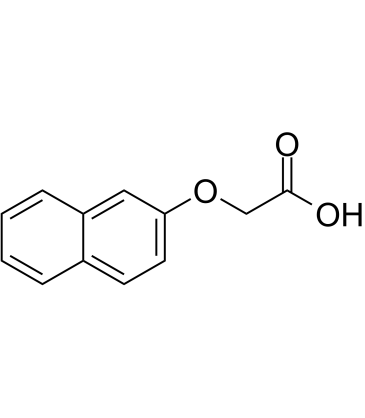
2-Naphthoxyacetic acid
2-NaphthoxyessigsÄure ist ein kÖrpereigener Metabolit.
-
GC39527
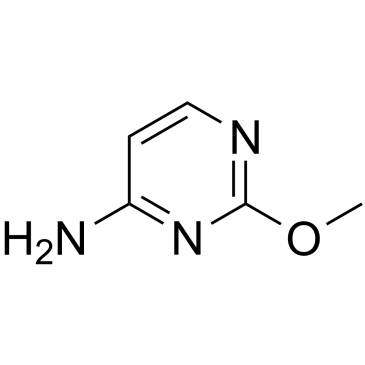
2-O-Methylcytosine
2-O-Methylcytosin, ein O-alkyliertes Analogon eines DNA-Addukts, ist die beschÄdigte Nukleobase.
-
GC42189
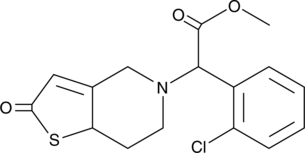
2-oxo Clopidogrel
2-oxo Clopidogrel is an intermediary metabolite of clopidogrel.
-
GC30622
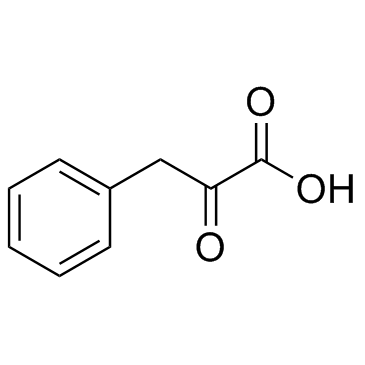
2-Oxo-3-phenylpropanoic acid
2-Oxo-3-phenylpropansÄure wird bei der Synthese von 3-PhenylmilchsÄure (PLA) durch Lactatdehydrogenase verwendet.
-
GC30629
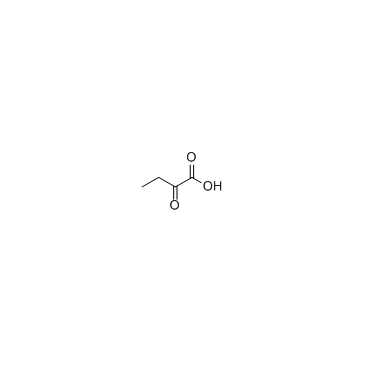
2-Oxobutanoic acid
2-OxobutansÄure ist ein Produkt der enzymatischen Spaltung von Cystathionin.
-
GC38276
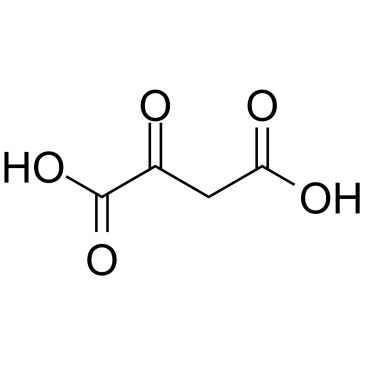
2-Oxosuccinic acid
2-OxosuccinsÄure (2-OxosuccinsÄure) ist ein metabolisches Zwischenprodukt, das auf verschiedene Weise beteiligt ist, wie z. B. am ZitronensÄurezyklus, der Gluconeogenese, dem Harnstoffzyklus, dem Glyoxylatzyklus, der AminosÄuresynthese und der FettsÄuresynthese.
-
GC30713
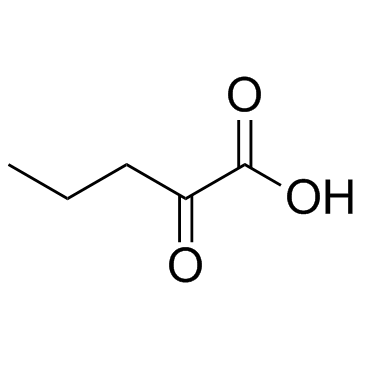
2-Oxovaleric acid
2-OxovaleriansÄure ist eine KetosÄure, die im menschlichen Blut vorkommt.
-
GC60018
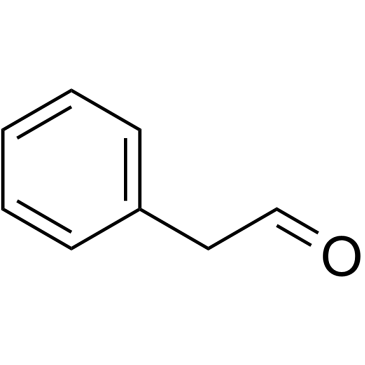
2-Phenylacetaldehyde
2-Phenylacetaldehyd ist ein kÖrpereigener Metabolit.
-
GC60483
2-Phenylacetamide
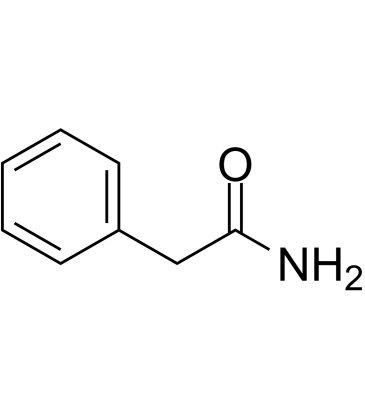
-
GC62784
2-Phenylbutanoic acid
2-PhenylbutansÄure ist ein kÖrpereigener Metabolit.
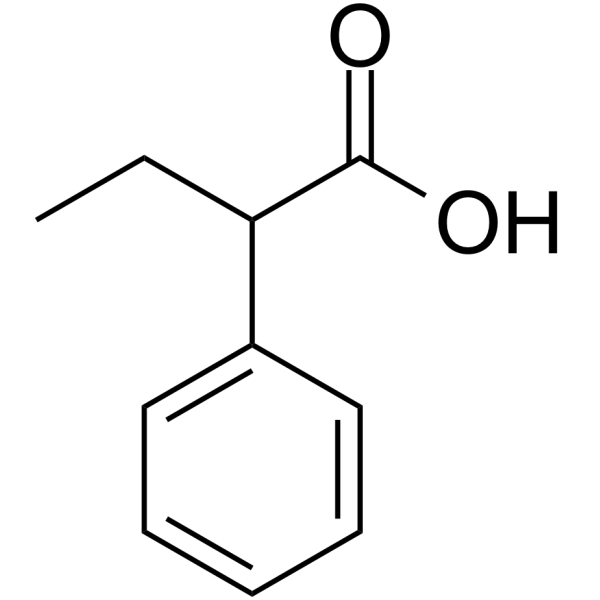
-
GC60485
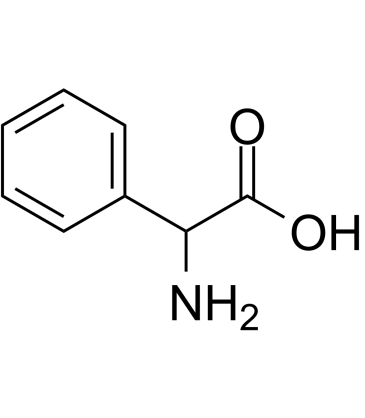
2-Phenylglycine
2-Phenylglycin (DL-α-Phenylglycin) ist ein Metabolit in der Muttermilch wÄhrend der Laktation W2 bis W4.