Proteases
Proteases is a general term for a class of enzymes that hydrolyze protein peptide chains. According to the way they degrade polypeptides, they are divided into two categories: endopeptidases and telopeptidases. The former can cut the large molecular weight polypeptide chain from the middle to form prions and peptones with smaller molecular weights; the latter can be divided into carboxypeptidase and aminopeptidase, which respectively remove the peptide from the free carboxyl terminus or free amino terminus of the polypeptide one by one. Chain hydrolysis produces amino acids.
A general term for a class of enzymes that hydrolyze peptide bonds in proteins. According to the way they hydrolyze polypeptides, they can be divided into endopeptidases and exopeptidases. Endopeptidase cleaves the interior of the protein molecule to form smaller molecular weight peptones and peptones. Exopeptidase hydrolyzes peptide bonds one by one from the end of the free amino group or carboxyl group of protein molecules, and frees amino acids, the former is aminopeptidase and the latter is carboxypeptidase. Proteases can be classified into serine proteases, sulfhydryl proteases, metalloproteases and aspartic proteases according to their active centers and optimum pH. According to the optimum pH value of its reaction, it is divided into acidic protease, neutral protease and alkaline protease. The proteases used in industrial production are mainly endopeptidases.
Proteases are widely found in animal offal, plant stems and leaves, fruits and microorganisms. Microbial proteases are mainly produced by molds and bacteria, followed by yeast and actinomycetes.
Enzymes that catalyze the hydrolysis of proteins. There are many kinds, the important ones are pepsin, trypsin, cathepsin, papain and subtilisin. Proteases have strict selectivity for the reaction substrates they act on. A protease can only act on certain peptide bonds in protein molecules, such as the peptide bonds formed by the hydrolysis of basic amino acids catalyzed by trypsin. Proteases are widely distributed, mainly in the digestive tract of humans and animals, and are abundant in plants and microorganisms. Due to limited animal and plant resources, the industrial production of protease preparations is mainly prepared by fermentation of microorganisms such as Bacillus subtilis and Aspergillus terrestris.
Ziele für Proteases
- Caspase(114)
- Aminopeptidase(24)
- ACE(74)
- Calpains(20)
- Carboxypeptidase(10)
- Cathepsin(81)
- DPP-4(31)
- Elastase(26)
- Gamma Secretase(67)
- HCV Protease(59)
- HSP(113)
- HIV Integrase(37)
- HIV Protease(47)
- MMP(228)
- NS3/4a protease(8)
- Serine Protease(18)
- Thrombin(58)
- Urokinase(4)
- Cysteine Protease(0)
- Other Proteases(18)
- Tyrosinases(47)
- 15-PGDH(1)
- Acetyl-CoA Carboxylase(13)
- Acyltransferase(59)
- Aldehyde Dehydrogenase (ALDH)(28)
- Aminoacyl-tRNA Synthetase(9)
- ATGL(1)
- Dipeptidyl Peptidase(56)
- Drug Metabolite(457)
- E1/E2/E3 Enzyme(90)
- Endogenous Metabolite(1636)
- FABP(30)
- Farnesyl Transferase(23)
- Glutaminase(14)
- Glutathione Peroxidase(14)
- Isocitrate Dehydrogenase (IDH)(28)
- Lactate Dehydrogenase(17)
- Lipoxygenase(234)
- Mitochondrial Metabolism(207)
- NEDD8-activating Enzyme(7)
- Neprilysin(12)
- PAI-1(13)
- Ser/Thr Protease(41)
- Tryptophan Hydroxylase(13)
- Xanthine Oxidase(18)
- MALT1(10)
- PCSK9(1)
Produkte für Proteases
- Bestell-Nr. Artikelname Informationen
-
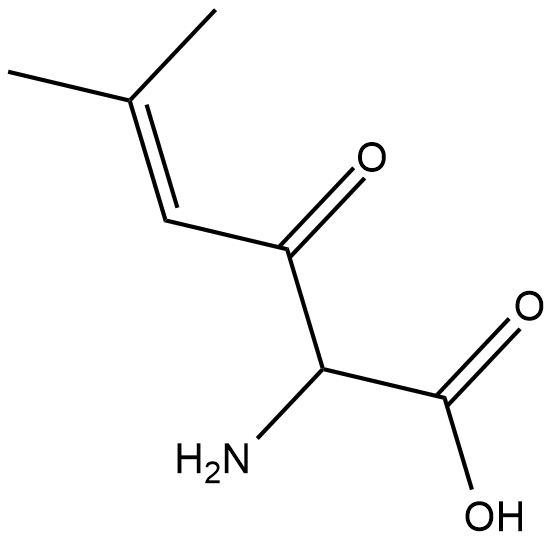
GC31611
2-Phenylpropionic acid
2-PhenylpropionsÄure ist ein Zwischenprodukt im alpha-Methylstyrol-Stoffwechsel.
-
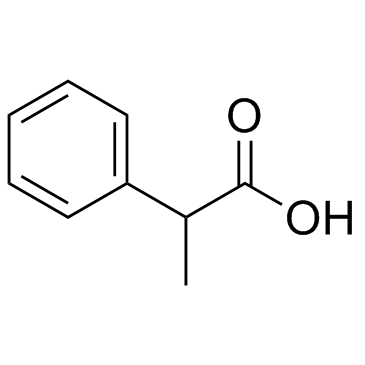
GC35095
2-Phospho-L-ascorbic acid trisodium salt
2-Phospho-L-AscorbinsÄure-Trinatriumsalz (2-Phospho-L-AscorbinsÄure-Trinatrium) ist ein lang wirkendes Vitamin-C-Derivat, das die Kollagenbildung und -expression stimulieren kann.
-
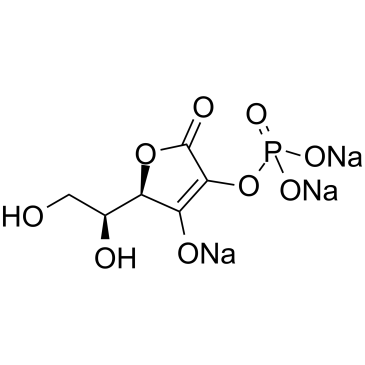
GC60486
2-Piperidone
2-Piperidon ist ein kÖrpereigener Metabolit.
-
GC19539
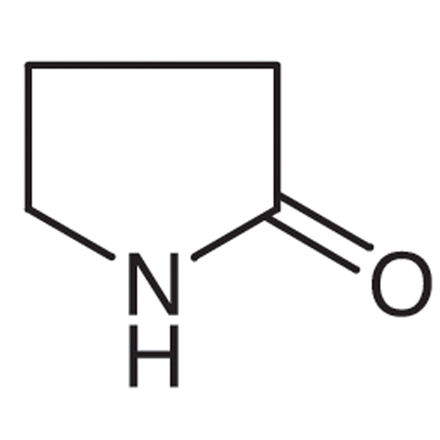
2-Pyrrolidinone
2-Pyrrolidon
-
GC42193
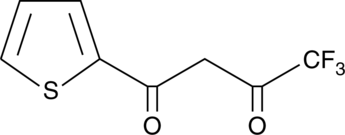
2-Thenoyltrifluoroacetone
2-Thenoyltrifluoraceton ist ein Chelatbildner.
-
GC60487
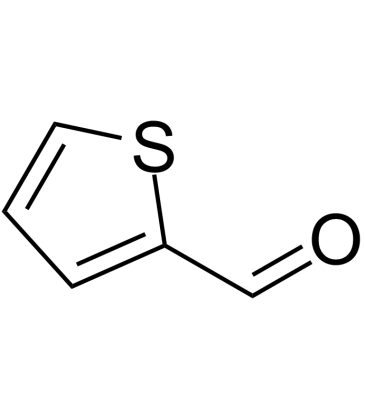
2-Thiophenecarboxaldehyde
-
GC42082
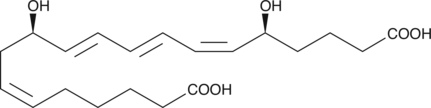
20-carboxy Leukotriene B4
20-carboxy LTB4 is a metabolite of LTB4 in human neutrophils.
-
GC35075
20-HETE
Ein quantitativer analytischer Standard, der garantiert den MaxSpec® Identitäts-, Reinheits-, Stabilitäts- und Konzentrationsspezifikationen zu entsprechen.
-
GC41421
20-hydroxy Leukotriene B4
20-hydroxy LTB4 is a metabolite of LTB4 in human neutrophils.
-
GC40968
21-hydroxy Eplerenone
21-hydroxy Eplerenone is a major metabolite of the mineralocorticoid receptor antagonist eplerenone.
-
GC33798
21-Hydroxypregnenolone
21-Hydroxypregnenolon ist ein essentielles Zwischenprodukt bei der Corticosteron-Synthese.
-
GC62767
23,25-Dihydroxy-24-oxovitamin D3
23,25-Dihydroxy-24-oxovitamin D3 ist ein Hauptmetabolit von 24(R),25-Dihydroxyvitamin D3.
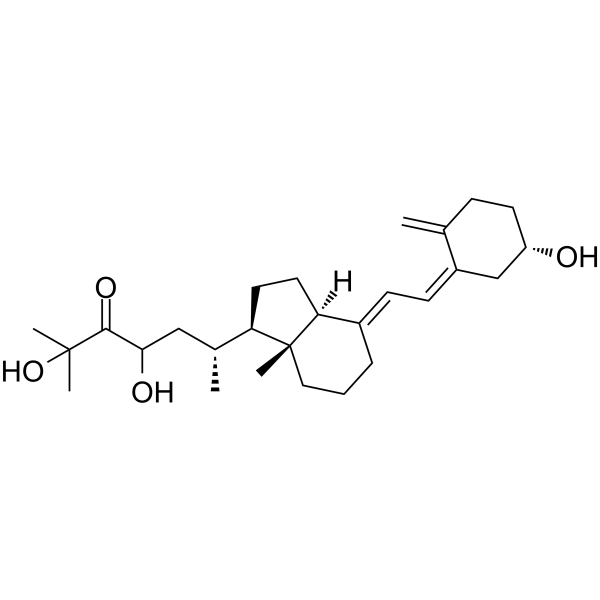
-
GC35079
24, 25-Dihydroxy VD2
24, 25-Dihydroxy VD2 ist ein hydroxylierter Metabolit von Vitamin D2; ein synthetisches Analogon von Vitamin D.
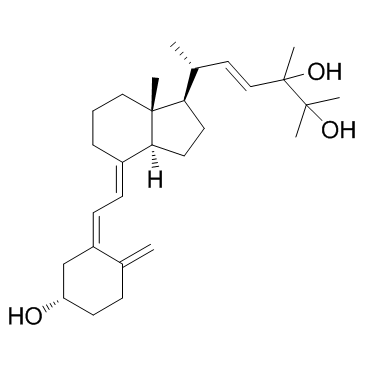
-
GC17566
24, 25-Dihydroxy VD3
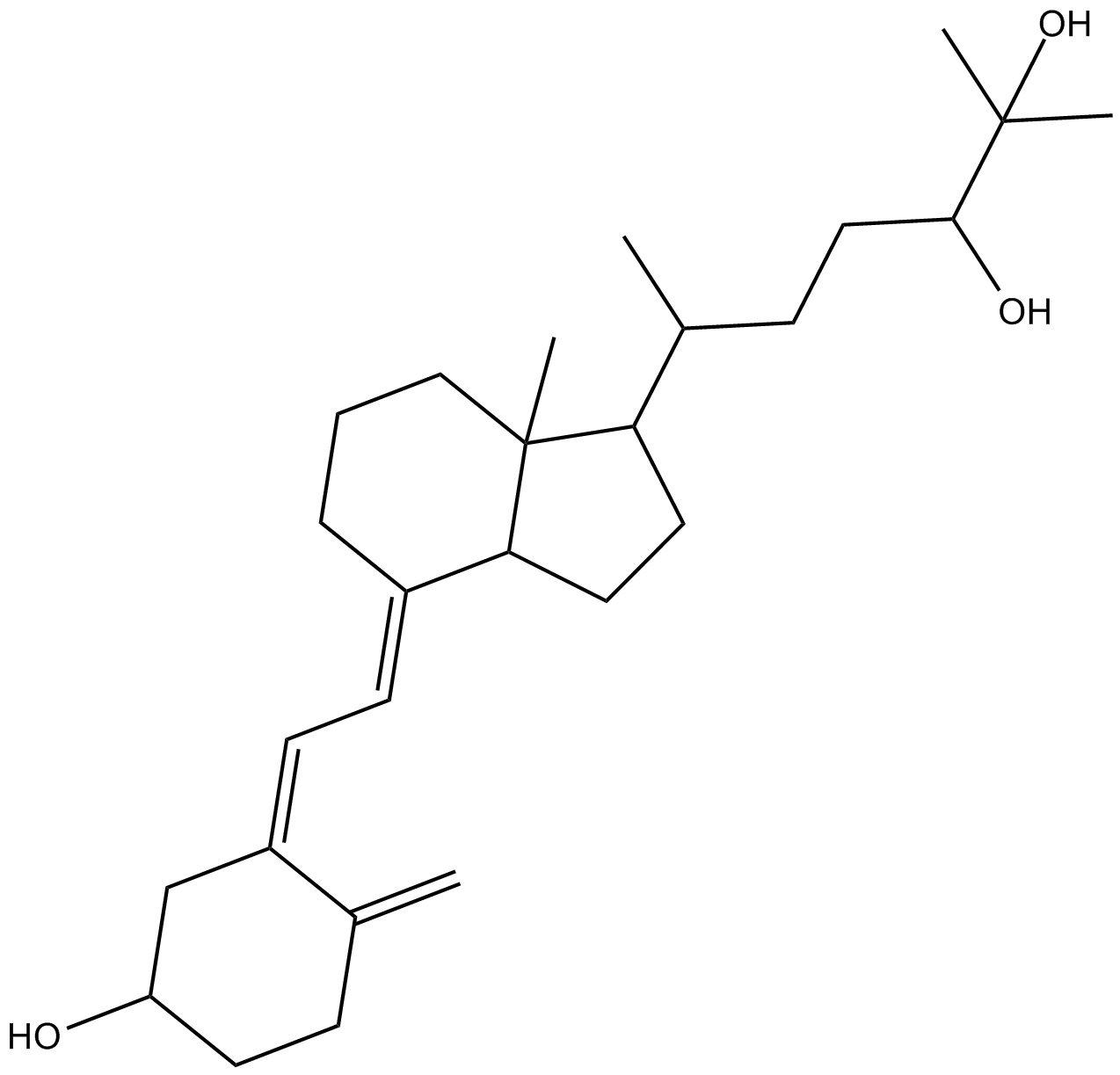
-
GC13887
25(R)-27-hydroxy Cholesterol
25(R)-27-Hydroxycholesterol ist ein selektiver Östrogenrezeptormodulator und ein Agonist des Leber-X-Rezeptors.
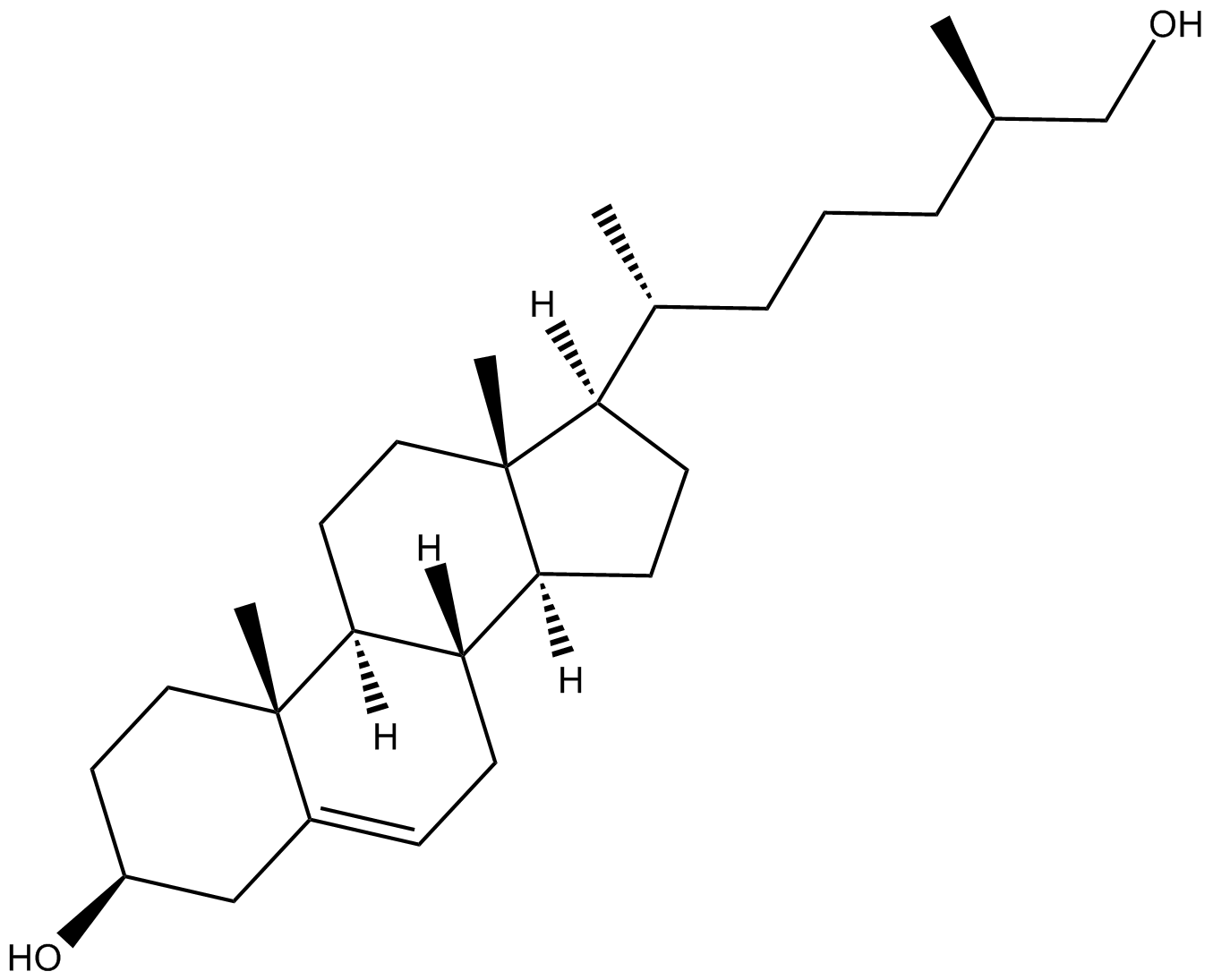
-
GC35081
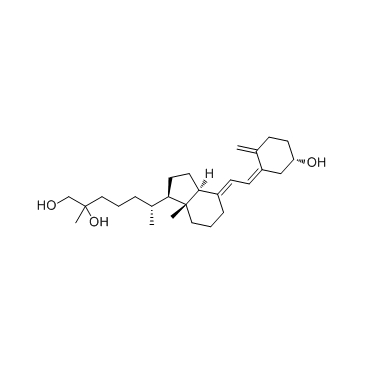
25,26-Dihydroxyvitamin D3
25,26-Dihydroxyvitamin D3 (25,26-Dihydroxycholecalciferol) ist ein Metabolit von Vitamin D3 mit intestinaler CalciumtransportaktivitÄt.
-
GC49365
25-Desacetyl Rifampicin
A major active metabolite of rifampicin

-
GC33860
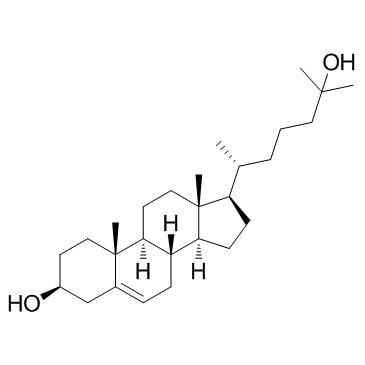
25-Hydroxycholesterol
25-Hydroxycholesterol ist ein Metabolit von Cholesterin, der von Makrophagen als Reaktion auf die Aktivierung des Toll-like-Rezeptors (TLR) produziert und ausgeschieden wird.
-
GC64985
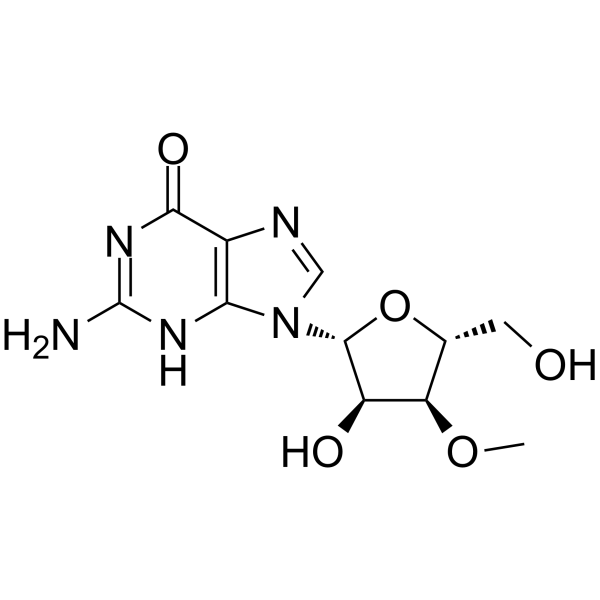
3'-O-Methylguanosine
3'-O-Methylguanosin ist ein methyliertes Nukleosidanaloga und ein RNA-Kettenterminator.
-
GC60493
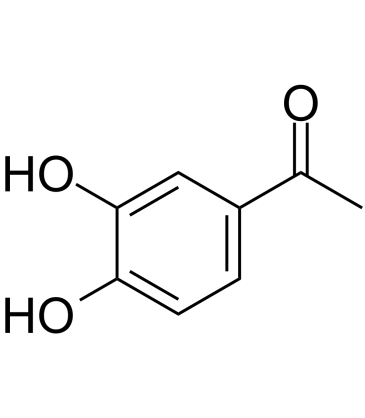
3',4'-Dihydroxyacetophenone
3',4'-Dihydroxyacetophenon (3,4-DHAP), isoliert aus Picea-Schrenkiana-Nadeln, zeigt eine stark unterdrÜckende Wirkung gegen die Tyrosinase-AktivitÄt mit einem IC50 von 10 μM. 3',4'-Dihydroxyacetophenon (3,4-DHAP) ist ein vasoaktives Mittel und Antioxidans.
-
GC60023
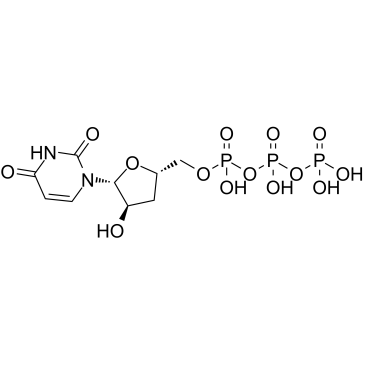
3'-Deoxyuridine-5'-triphosphate
3'-Desoxyuridin-5'-Triphosphat (3'-dUTP) ist ein Nukleotidanalogon, das die DNA-abhÄngigen RNA-Polymerasen I und II hemmt.
-
GC61862
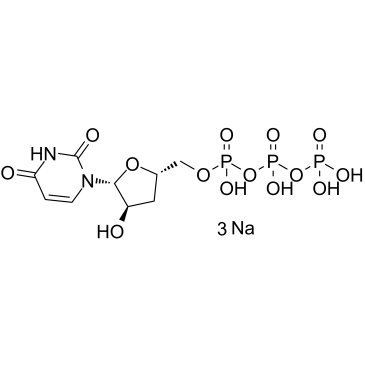
3'-Deoxyuridine-5'-triphosphate trisodium
3'-Desoxyuridin-5'-Triphosphat-Trinatrium (3'-dUTP-Trinatrium) ist ein Nukleotidanalogon, das die DNA-abhÄngigen RNA-Polymerasen I und II hemmt.
-
GC41480
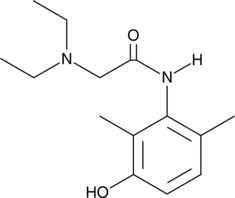
3'-hydroxy Lidocaine
3'-hydroxy Lidocaine is an active metabolite of lidocaine formed by the cytochrome P450 (CYP) isoforms CYP1A2 and CYP3A4.
-
GC39611
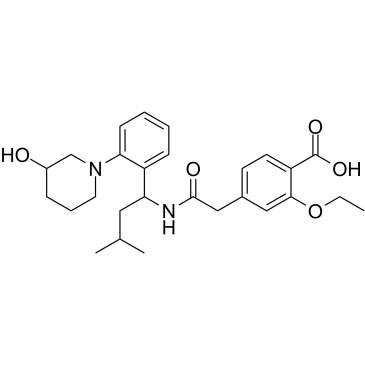
3'-Hydroxy Repaglinide
3'-Hydroxy-Repaglinid ist ein CYP2C8-Hauptmetabolit von Repaglinid.
-
GC60500
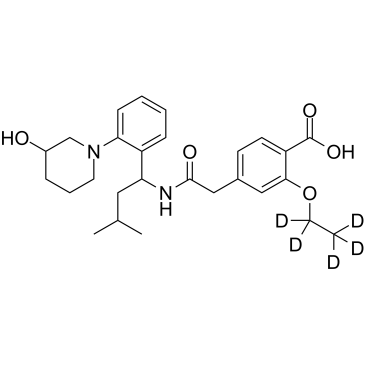
3'-Hydroxy Repaglinide D5
3'-Hydroxy-Repaglinid D5 ist das mit Deuterium markierte 3'-Hydroxy-Repaglinid.
-
GC42201
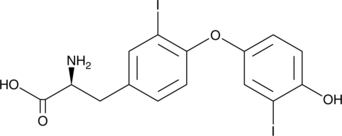
3,3'-Diiodo-L-thyronine
3,3'-Diiod-L-thyronin (3,3'-T2) ist ein kÖrpereigener Metabolit des SchilddrÜsenhormons.
-
GC60491
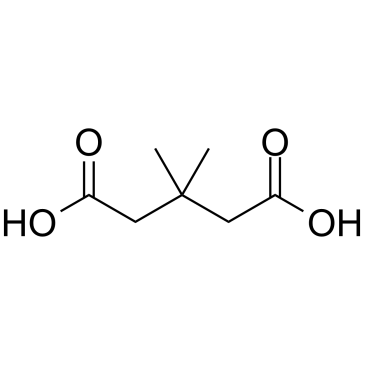
3,3-Dimethylglutaric acid
3,3-DimethylglutarsÄure, ein Mitglied der methylverzweigten FettsÄuren, ist ein endogener Metabolit, der gelegentlich im menschlichen Urin gefunden wird.
-
GC33703
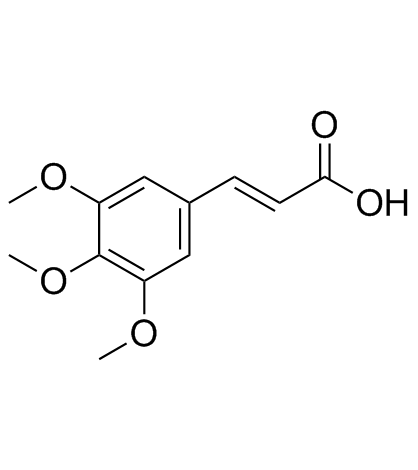
3,4,5-Trimethoxycinnamic acid
3,4,5-TrimethoxyzimtsÄure ist ein aus den Wurzeln von Polygala tenuifoliaWILLD isoliertes Phenylpropanoid mit Anti-Stress-Wirkung, das die Schlafzeit bei Tieren verlÄngert.
-
GC68082
3,4,5-Trimethoxyphenylacetic acid

-
GC39841
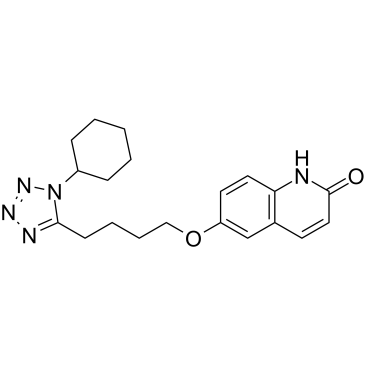
3,4-Dehydro Cilostazol
3,4-Dehydro Cilostazol (OPC-13015) ist ein aktiver Metabolit von Cilostazol.
-
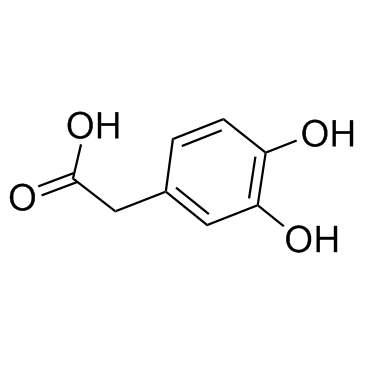
GC33649
3,4-Dihydroxybenzeneacetic acid
3,4-DihydroxybenzolessigsÄure ist der wichtigste neuronale Metabolit von Dopamin.
-
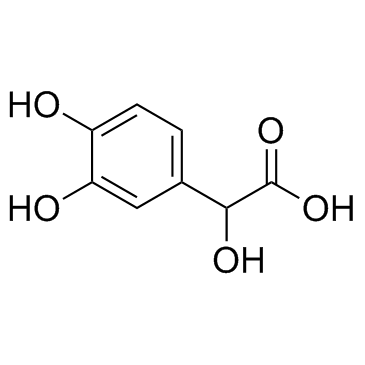
GC30146
3,4-Dihydroxymandelic acid
3,4-DihydroxymandelsÄure ist ein Metabolit von Norepinephrin.
-
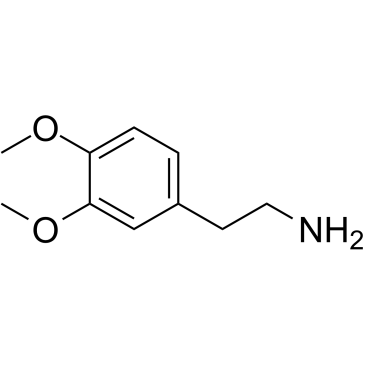
GC39777
3,4-Dimethoxyphenethylamine
-
GC39327
3,4-Dimethoxyphenol
3,4-Dimethoxyphenol ist eine aus Pflanzen gewonnene Phenylpropanoidverbindung und kann als Weißmacher in Kosmetika verwendet werden.
-
GC33615
3,4-Dimethoxyphenylacetic acid
3,4-DimethoxyphenylessigsÄure ist ein Baustein in der chemischen Synthese.
-
GC60497
3,5-Dihydroxyacetophenone
3,5-Dihydroxyacetophenon ist ein kÖrpereigener Metabolit.
-
GC33817
3,5-Dihydroxybenzoic acid
-
GC61716
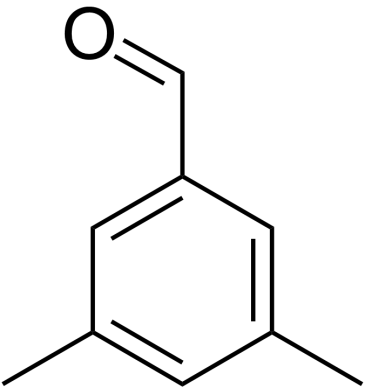
3,5-Dimethylbenzaldehyde
3,5-Dimethylbenzaldehyd ist ein Baustein in der chemischen Synthese.
-
GC62788
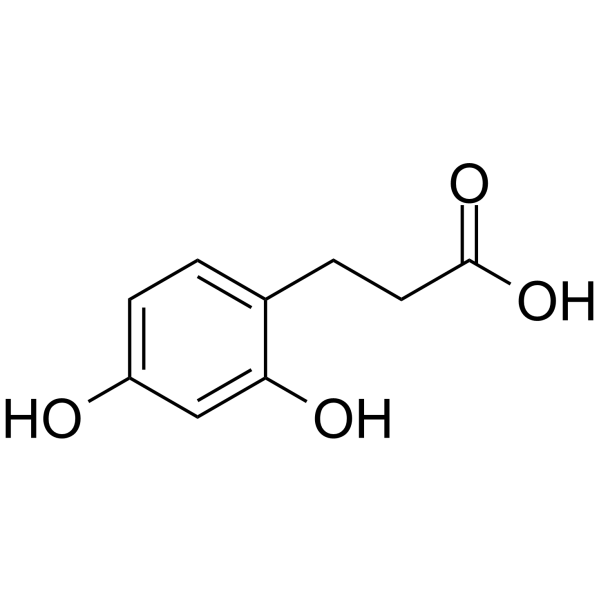
3-(2,4-Dihydroxyphenyl)propanoic acid
3-(2,4-Dihydroxyphenyl)propansÄure (DPPacid) ist ein potenter und kompetitiver Tyrosinase-Inhibitor, hemmt L-Tyrosin und DL-DOPA mit einem IC50 und einem Ki von 3,02 μM bzw. 11,5 μM.
-
GC62789
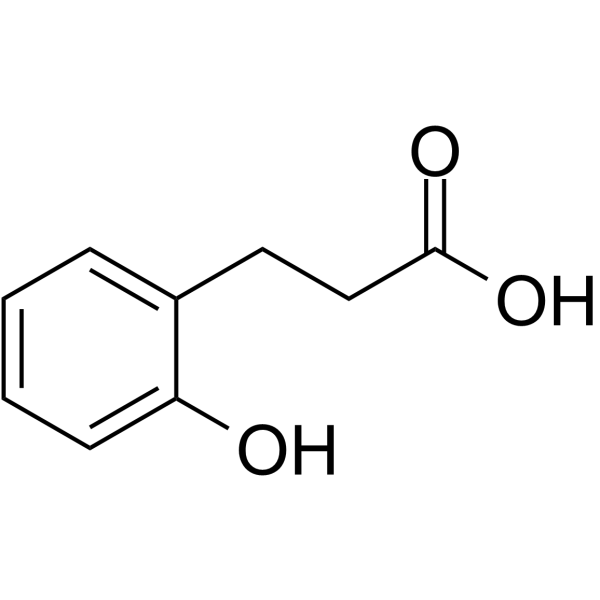
3-(2-Hydroxyphenyl)propanoic acid
3-(2-Hydroxyphenyl)propansÄure ist ein kÖrpereigener Metabolit.
-
GC33618
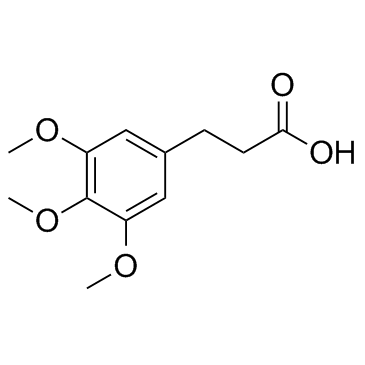
3-(3,4,5-Trimethoxyphenyl)propanoic acid
3-(3,4,5-Trimethoxyphenyl)propansÄure kommt in KrÄutern und GewÜrzen vor.
-
GC60488
3-(3-Hydroxyphenyl)propionic acid
3-(3-Hydroxyphenyl)propionsÄure ist ein Flavonoid-Metabolit, der von der menschlichen Mikroflora gebildet wird.
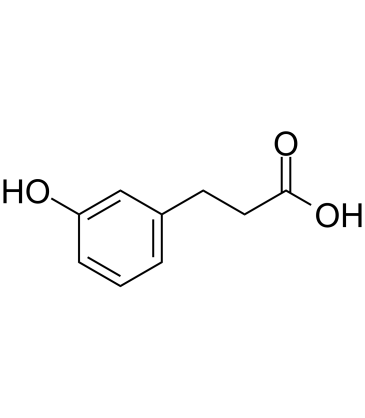
-
GC52324
3-(3-Hydroxyphenyl)propionic Acid sulfate
A metabolite of certain phenols and glycosides
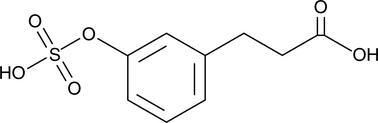
-
GC31625
3-(3-Methoxyphenyl)propionic acid
3-(3-Methoxyphenyl)propionsÄure ist eine organische SÄure, ein natÜrlich vorkommender menschlicher Metabolit, der im menschlichen Urin ausgeschieden wird.
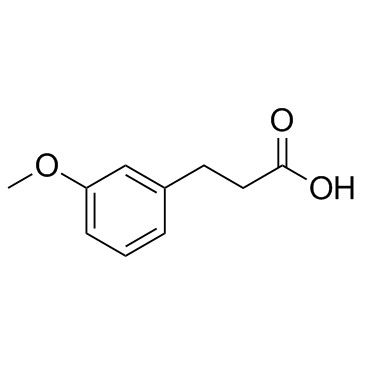
-
GC42198
3-(4-Chlorophenyl)-4-hydroxybutyric Acid (sodium salt)
An inactive metabolite of baclofen
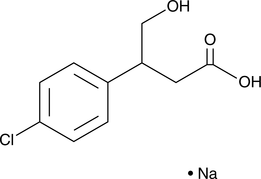
-
GC32943
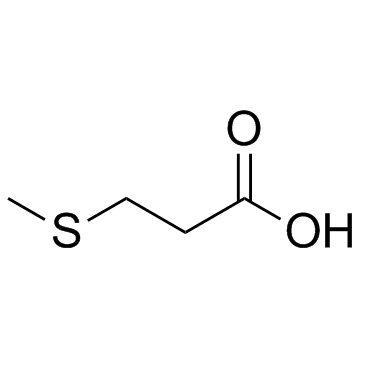
3-(Methylthio)propionic acid (3-Methylsulfanylpropionic acid)
3-(Methylthio)propionsÄure (3-MethylsulfanylpropionsÄure) ist ein Zwischenprodukt im Methioninstoffwechsel.
-
GC46583
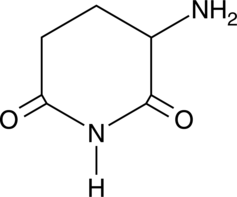
3-Amino-2,6-Piperidinedione
An active metabolite of (±)-thalidomide
-
GC31569
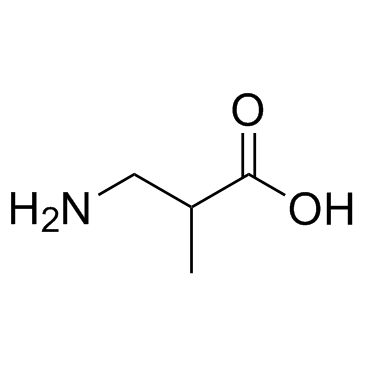
3-Amino-2-methylpropanoic acid
3-Amino-2-methylpropansÄure kann eine BrÄunung des weißen Fetts und eine hepatische β-Oxidation induzieren und korreliert umgekehrt mit kardiometabolischen Risikofaktoren.
-
GC62793
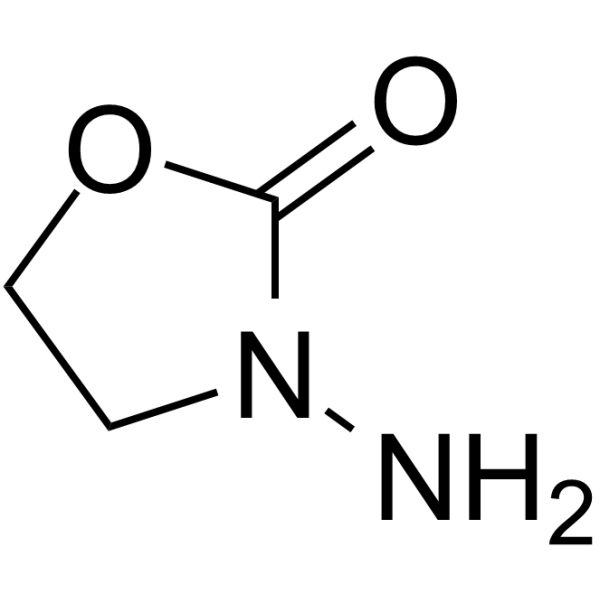
3-Amino-2-oxazolidinone
3-Amino-2-oxazolidinon (AOZ) ist der Metabolit von Furazolidon.
-
GC67902
3-Amino-2-oxazolidinone-d4

-
GC39688
3-Amino-2-piperidinone
3-Aminopiperidin-2-on ist ein Stoffwechselprodukt aller lebenden Organismen.
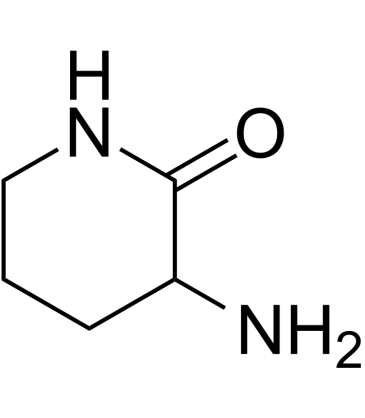
-
GC38293
3-Amino-4-hydroxybenzoic acid
3-Amino-4-hydroxybenzoesÄure ist ein kÖrpereigener Metabolit.
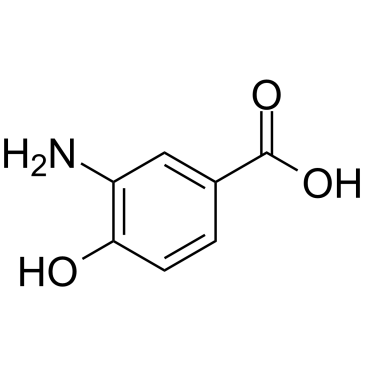
-
GC30719
3-Amino-4-methylpentanoic acid
3-Amino-4-methylpentansÄure ist eine Beta-AminosÄure und ein Positionsisomer von L-Leucin, das natÜrlicherweise im Menschen durch den Metabolismus von L-Leucin durch das Enzym Leucin-2,3-Aminomutase produziert wird.
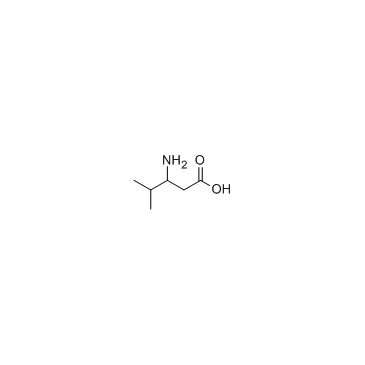
-
GC49849
3-Aminosalicylic Acid
A salicylic acid derivative
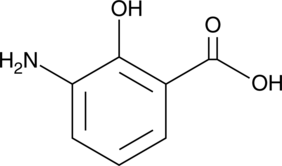
-
GC33631
3-Chloro-L-tyrosine
3-Chlor-L-Tyrosin ist ein spezifischer Marker der durch Myeloperoxidase katalysierten Oxidation und ist deutlich erhÖht in Lipoprotein niedriger Dichte, das aus humaner atherosklerotischer Intima isoliert wurde.
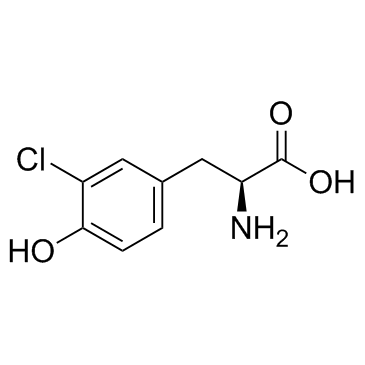
-
GC35106
3-Dehydrotrametenolic acid
3-DehydrotrametenolsÄure, isoliert aus dem Sclerotium von Poria cocos, ist ein Lactatdehydrogenase (LDH)-Hemmer. 3-DehydrotrametenolsÄure fÖrdert die Adipozytendifferenzierung in vitro und wirkt in vivo als Insulinsensibilisator. 3-DehydrotrametenolsÄure induziert Apoptose und wirkt gegen Krebs.
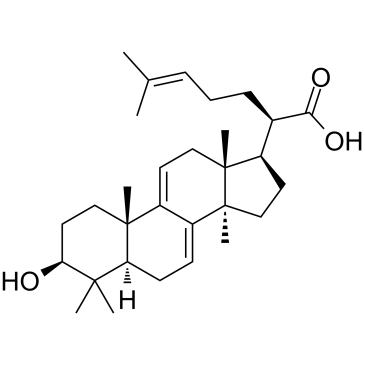
-
GC40743
3-deoxy Galactosone
3-Desoxygalactoson ist eine 1,2-Dicarbonylverbindung, die beim Abbau von Galactose entsteht.
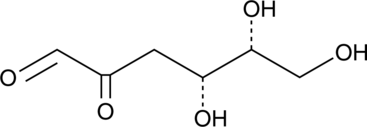
-
GC35111
3-Epiursolic Acid
3-EpiursolsÄure ist ein Triterpenoid, das aus Myrtaceae isoliert werden kann und als kompetitiver Inhibitor von Cathepsin L wirkt (IC50, 6,5 μM; Ki, 19,5 μM), ohne offensichtliche Wirkung auf Cathepsin B.
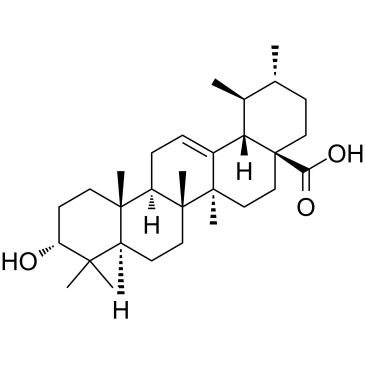
-
GC38257
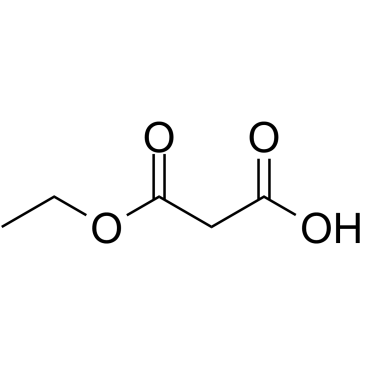
3-Ethoxy-3-oxopropanoic acid
3-Ethoxy-3-oxopropansÄure ist ein kÖrpereigener Metabolit.
-
GC38368
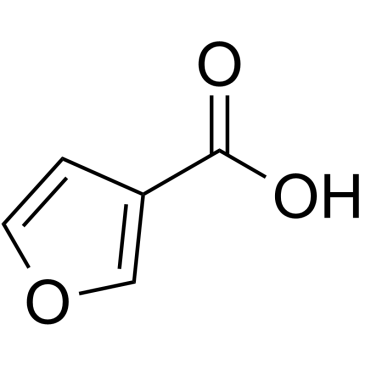
3-Furanoic acid
3-FuransÄure ist ein kÖrpereigener Metabolit.
-
GC13377
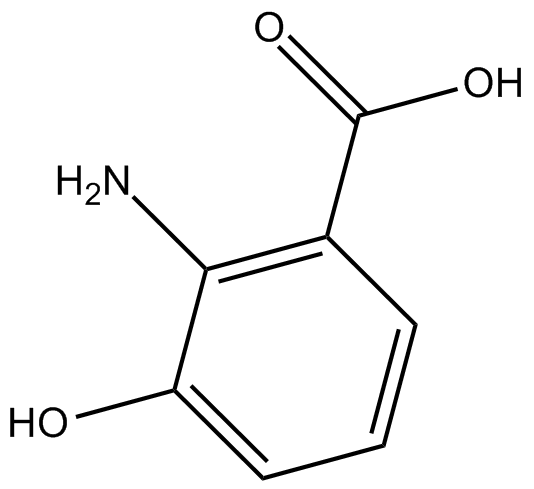
3-hydroxy Anthranilic Acid
3-Hydroxy-Anthranilsäure ist ein Tryptophan-Metabolit im Kynurenin-Weg.
-
GC42274
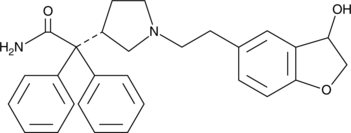
3-hydroxy Darifenacin
3-hydroxy Darifenacin is a metabolite of darifenacin.
-
GC42275
3-hydroxy Desloratidine
3-hydroxy Desloratidine is a major metabolite of desloratadine , a tricyclic antagonist of the histamine H1 receptor.
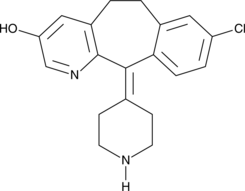
-
GC41565
3-hydroxy Medetomidine
3-hydroxy Medetomidine is a metabolite of the α2-adrenergic receptor agonist medetomidine.
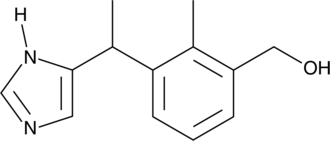
-
GC60501
3-Hydroxy-4-aminopyridine
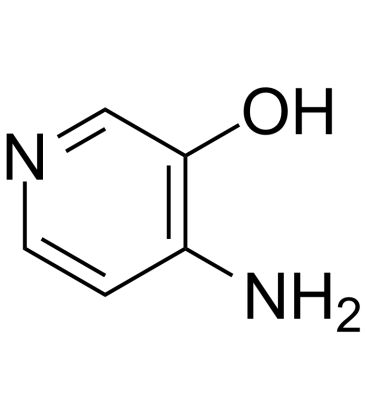
-
GC39328
3-Hydroxybenzaldehyde
3-Hydroxybenzaldehydist eine VorlÄuferverbindung fÜr phenolische Verbindungen wie Protocatechualdehyd.
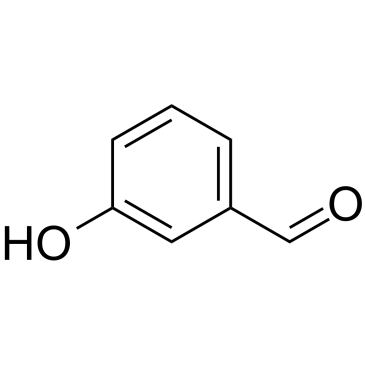
-
GC38669
3-Hydroxybenzoic acid
-
GC30616
3-Hydroxybutyric acid
3-HydroxybuttersÄure (β-HydroxybuttersÄure) ist ein Metabolit, der bei Typ-I-Diabetes erhÖht ist.
-
GC60502
3-Hydroxybutyric acid sodium
3-HydroxybuttersÄure-Natrium (β-HydroxybuttersÄure-Natrium) ist ein Metabolit, der bei Typ-I-Diabetes erhÖht ist.
-
GC33383
3-Hydroxycapric acid
3-HydroxycaprinsÄure ist ein Inhibitor der mitotischen Progression.
-
GC49364
3-Hydroxycoumarin
3-Hydroxycumarin ist ein starker Redox-Inhibitor von menschlichem 15-LOX-1.
-
GC30642
3-Hydroxydodecanoic acid
3-HydroxydodecansÄure ist eine mittelkettige FettsÄure, die mit StÖrungen des FettsÄurestoffwechsels in Verbindung gebracht wird.
-
GC41477
3-Hydroxyglutaric Acid
3-HydroxyglutarsÄure ist ein GlutarsÄurederivat.
-
GC66424
3-Hydroxyglutaric acid-d5
3-HydroxyglutarsÄure-d5 ist die mit Deuterium markierte 3-HydroxyglutarsÄure. 3-HydroxyglutarsÄure ist ein GlutarsÄurederivat.
-
GC30603
3-Hydroxyhippuric acid
3-HydroxyhippursÄure ist ein Acylglycin.
-
GC30660
3-Hydroxyisovaleric acid
3-HydroxyisovaleriansÄure ist ein normaler endogener Metabolit, der im Urin ausgeschieden wird.
-
GC30638
3-Hydroxymandelic Acid
3-HydroxymandelsÄure, ein Metabolit von Phenylephrin, Phenylephrin ist ein α-Rezeptoragonist.
-
GC38269
3-Hydroxyphenylacetic acid
-
GC14549
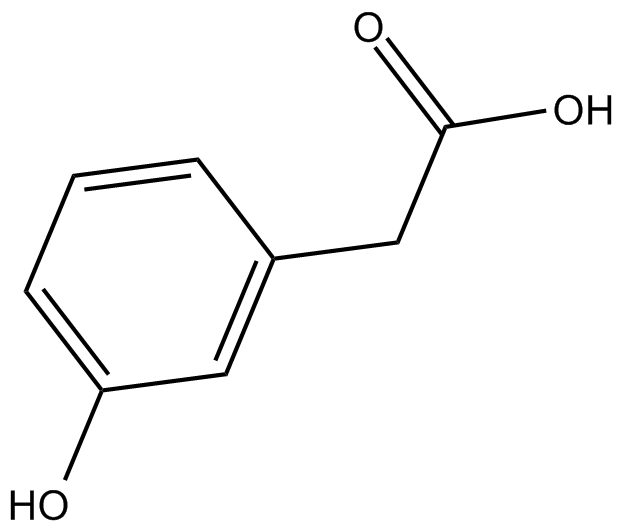
3-Hydroxyphenylacetic acid
3-HydroxyphenylessigsÄure ist ein kÖrpereigener Metabolit.
-
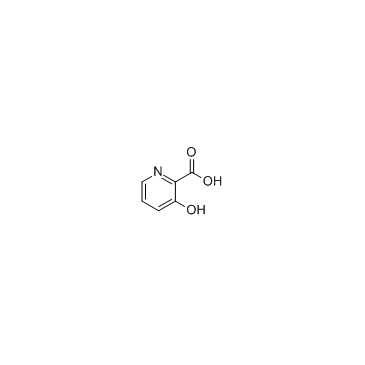
GC30636
3-Hydroxypicolinic acid (Picolinic acid, 3-hydroxy- (6CI,7CI,8CI))
3-HydroxypicolinsÄure (PicolinsÄure, 3-Hydroxy-(6CI,7CI,8CI)) ist ein PicolinsÄurederivat und gehÖrt zur Familie der Pyridine.
-
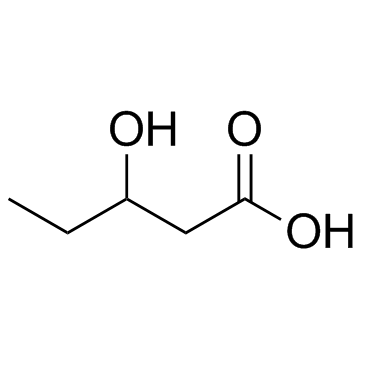
GC30729
3-Hydroxyvaleric acid
3-HydroxyvaleriansÄure ist ein 5-Kohlenstoff-KetonkÖrper.
-
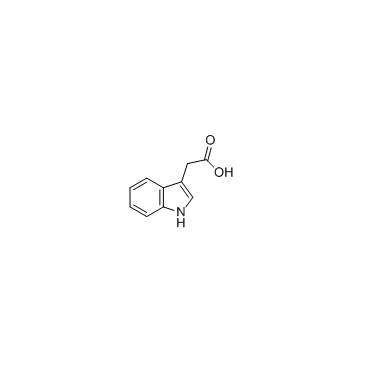
GC33436
3-Indoleacetic acid (Indole-3-acetic acid)
3-IndolessigsÄure (Indole-3-EssigsÄure) (Indole-3-EssigsÄure) ist das hÄufigste natÜrliche Pflanzenwachstumshormon aus der Klasse der Auxine.
-
GC39778
3-Indoleacetonitrile
3-Indolacetonitril ist ein kÖrpereigener Metabolit.
-
GC31290
3-Indolepropionic acid
3-IndolpropionsÄure hat sich als starkes Antioxidans erwiesen und hat Potenzial bei der Behandlung der Alzheimer-Krankheit.

-
GC48457
3-keto Fusidic Acid
An active metabolite of fusidic acid
-
GC33653
3-Methoxybenzoic acid (3-Anisic acid)
3-MethoxybenzoesÄure (3-AnissÄure) kann bei der Synthese von 3-Methoxybenzoaten von Europium(III) und Gadolinium(III) verwendet werden.
-
GC33739
3-Methoxytyramine (3-O-methyl Dopamine)
3-Methoxytyramin (3-O-Methyldopamin), ein bekannter extrazellulÄrer Metabolit von 3-Hydroxytyramin/Dopamin, ist ein Neuromodulator.
-
GC33738
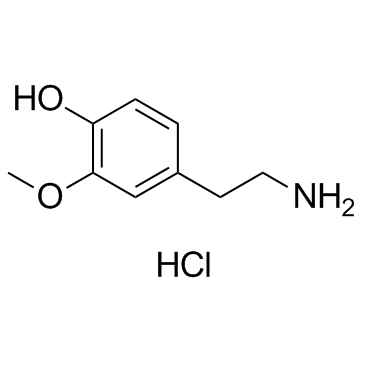
3-Methoxytyramine hydrochloride (3-O-methyl Dopamine hydrochloride)
3-Methoxytyraminhydrochlorid (3-O-Methyldopaminhydrochlorid) ist ein inaktiver Metabolit von Dopamin, der den Spurenamin-assoziierten Rezeptor 1 (TAAR1) aktivieren kann.
-
GC68324
3-Methoxytyramine-d4 hydrochloride

-
GC49869
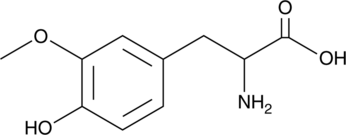
3-Methoxytyrosine
An active metabolite of L-DOPA
-
GC39690
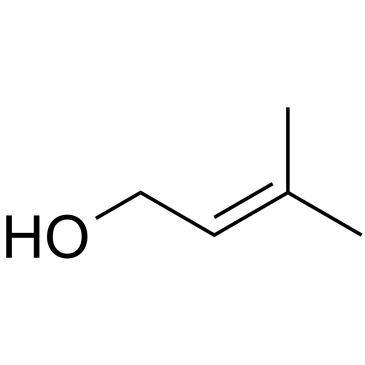
3-Methyl-2-buten-1-ol
3-Methyl-2-buten-1-ol ist ein kÖrpereigener Metabolit.
-
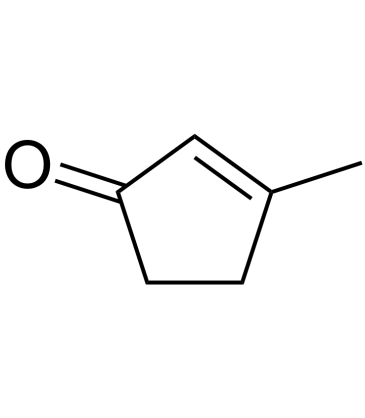
GC39846
3-Methyl-2-cyclopenten-1-one
-
GC19716
3-methyl-2-oxobutyrate
3-Methyl-2-oxobutyrat ist eine Vorstufe der PantothensÄure in Escherichia coli.
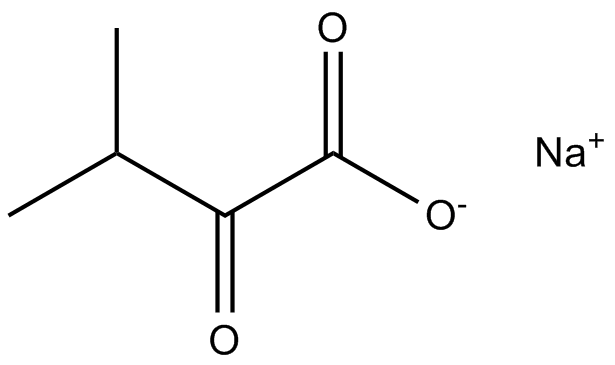
-
GC30593
3-Methyl-2-oxovaleric acid
3-Methyl-2-oxovaleriansÄure ist ein Neurotoxin, ein Acidogen und ein Metabotoxin sowie ein abnormaler Metabolit, der aus dem unvollstÄndigen Abbau von verzweigtkettigen AminosÄuren entsteht.
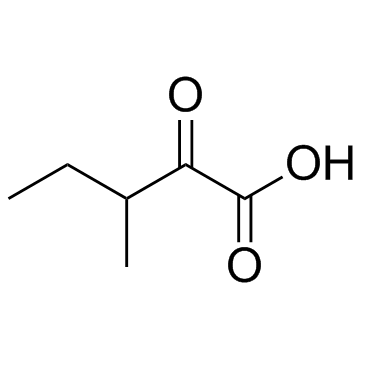
-
GC31535
3-Methyl-L-histidine
3-Methyl-L-Histidin ist ein Biomarker fÜr Fleischverzehr, insbesondere Huhn.
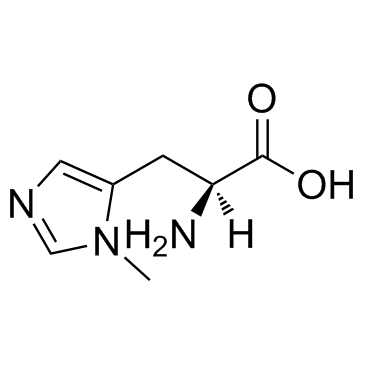
-
GC10710
3-Methyladenine
3-Methyladenin ist ein klassischer Autophagie-Inhibitor.
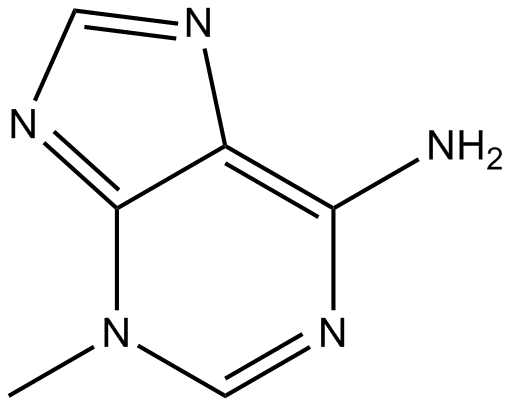
-
GC31546
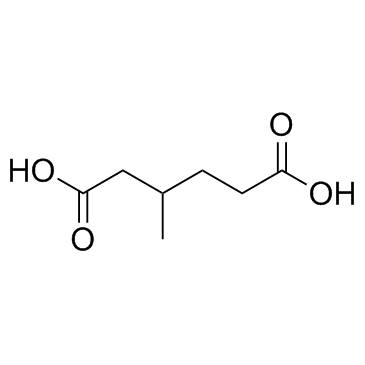
3-Methyladipic acid
3-MethyladipinsÄure ist der Endmetabolit im ω-Oxidationsweg.
-
GC38360
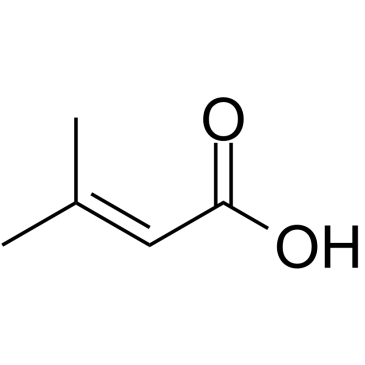
3-Methylbut-2-enoic acid
3-Methylbut-2-ensÄure ist ein kÖrpereigener Metabolit.
-
GC31595
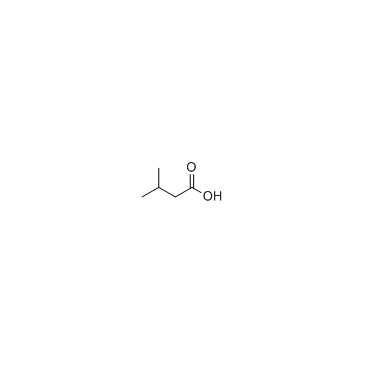
3-Methylbutanoic acid
3-MethylbutansÄure ist eine natÜrliche FettsÄure und dafÜr bekannt, den Tod von Neugeborenen und die mÖgliche jamaikanische Erbrechenskrankheit beim Menschen zu beeinflussen.
-
GC18628
3-Methylcrotonyl Glycine
3-Methylcrotonylglycin ist ein Acylglycin, ein normaler Aminosäuremetabolit, der im Urin vorkommt.