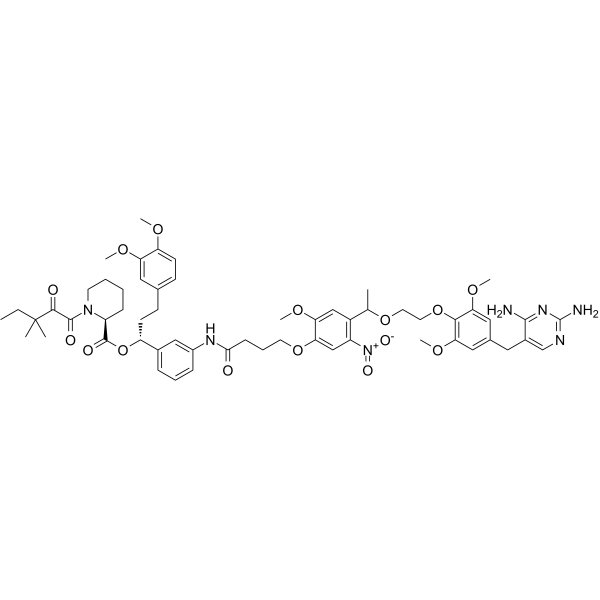FKBP
FKBP (FK506-binding protein) is one of two major immunophilins and most of FKBP family members bind FK506 and show peptidylprolyl cis/trans isomerase (PPIase) activity. Small size FKBP family members contain only FK506-binding domain, while FKBPs with large molecular weights possess extra domains such as tetratricopeptide repeat domains, calmodulin binding and transmembrane motifs. FKBPs are involved in several biochemical processes including protein folding, receptor signaling, protein trafficking and transcription. FKBP family proteins play important functional roles in the T-cell activation, when complexed with their ligands.
FK506-binding proteins 1a and 1b (FKBP1a/1b) are immunophilin proteins that bind the immunosuppressant agent FK506 and AY 22989. FKBP12 is a ubiquitous abundant protein that acts as a receptor for FK506, binds tightly to intracellular calcium release channels and to the transforming growth factor β (TGF-β) type I receptor.
Targets for FKBP
Products for FKBP
- Cat.No. Nom du produit Informations
-
GC34172
AP1867
AP1867 est un ligand synthétique dirigé par FKBP12F36V.
-
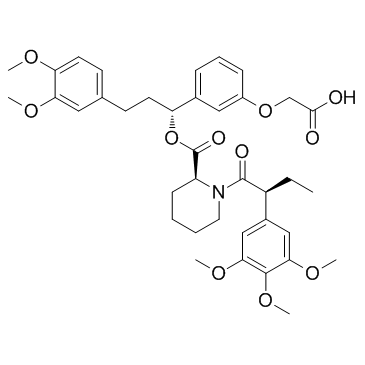
GC61745
AP1867-2-(carboxymethoxy)
AP1867-2-(carboxyméthoxy), le fragment basé sur AP1867 (un ligand synthétique dirigé par FKBP12F36V), se lie au ligand CRBN via un lieur pour former des molécules dTAG.

-
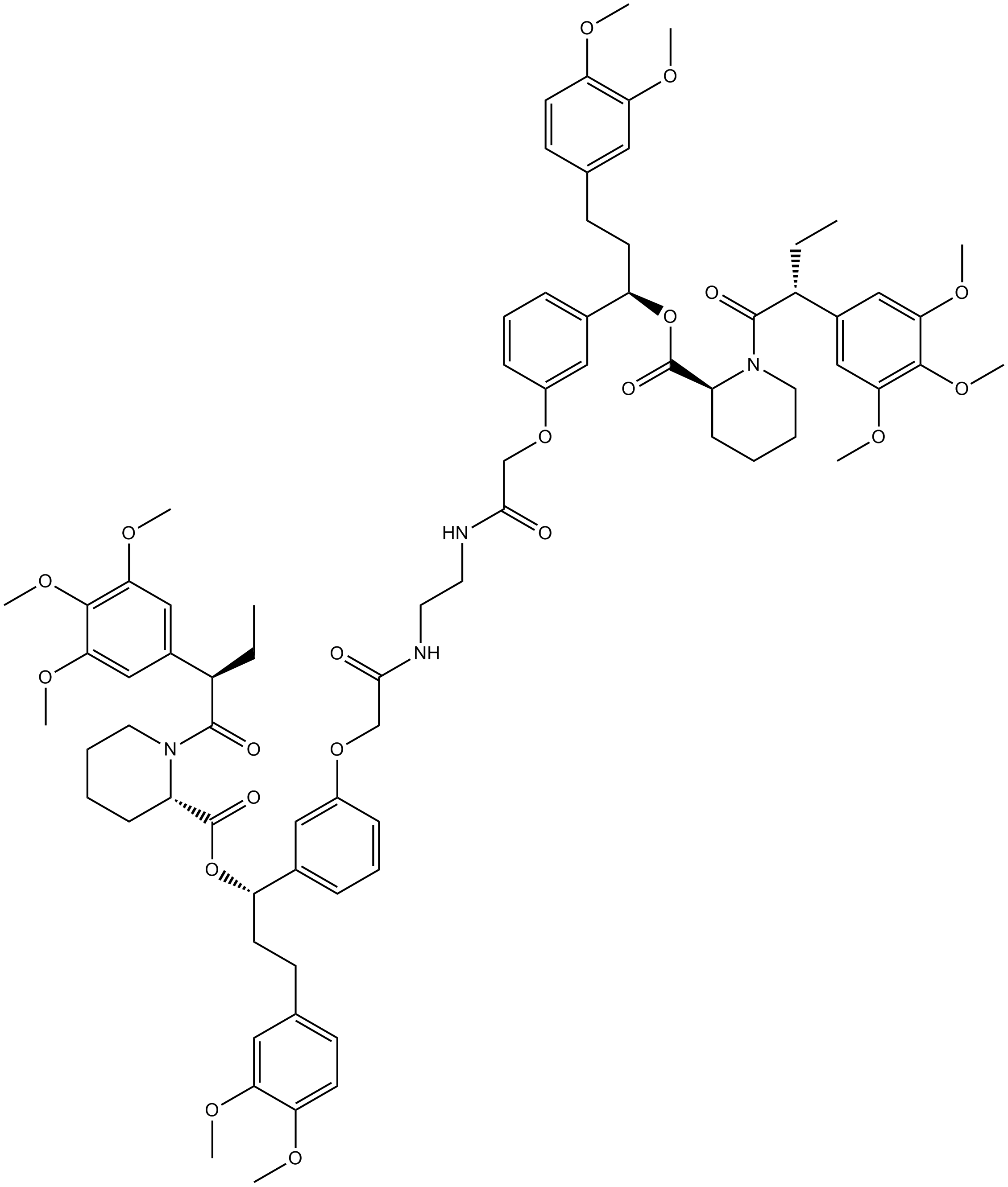
GC15586
AP1903
AP1903 (AP1903) est un agent dimérisant qui agit en réticulant les domaines FKBP. AP1903 (AP1903) dimérise l'interrupteur suicide Caspase 9 et induit rapidement l'apoptose.
-
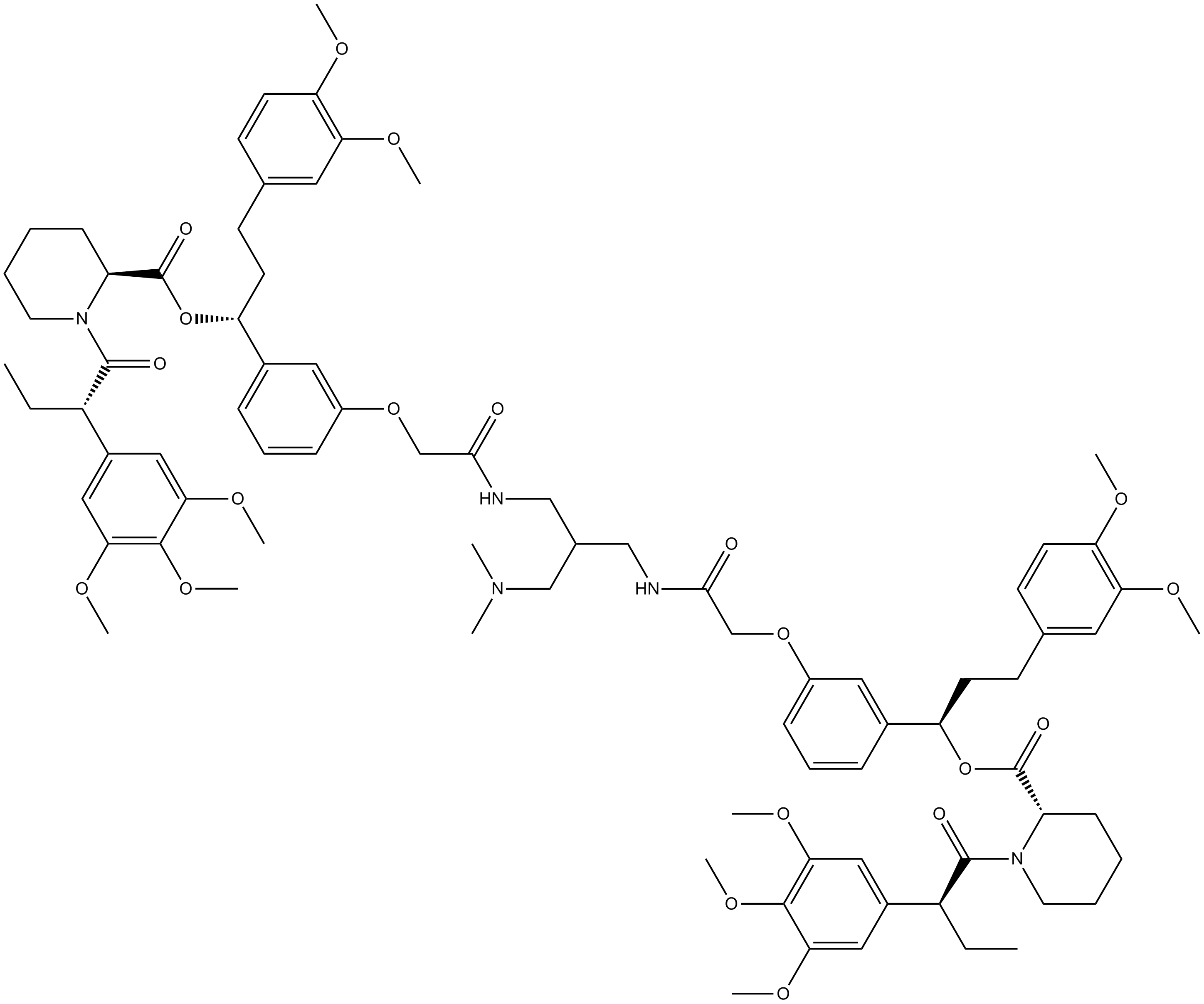
GC14498
AP20187
AP20187 (B/B Homodimerizer) est un ligand perméable aux cellules utilisé pour dimériser les protéines de fusion de la protéine de liaison FK506 (FKBP) et initier des cascades de signalisation biologique et l'expression génique ou perturber les interactions protéine-protéine.
-
GC13215
Ascomycin(FK 520)
A potent macrolide immunosuppressant
-
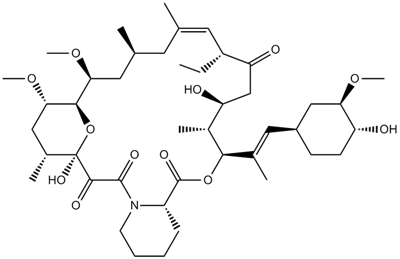
GC13601
Everolimus (RAD001)
Un dérivé de la rapamycine
-
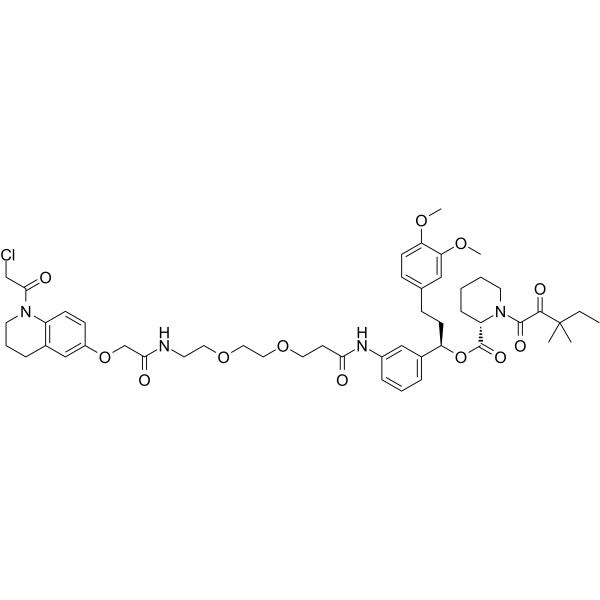
GC62186
KB02-SLF
KB02-SLF est un dégradeur nucléaire FKBP12 basé sur PROTAC (colle moléculaire). KB02-SLF favorise la dégradation nucléaire de FKBP12 en modifiant de manière covalente DCAF16 (ligase E3) et peut améliorer la durabilité de la dégradation des protéines dans les systèmes biologiques. SLF lie le ligand KB02 de l'ubiquitine E3 ligase via un lieur pour former KB02-SLF.
-
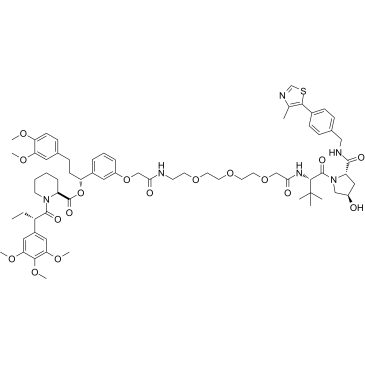
GC39503
PROTAC FKBP Degrader-3
PROTAC FKBP Degrader-3 est un PROTAC qui comprend un groupe de liaison au ligand FKBP, un lieur et un groupe de liaison von Hippel-Lindau. PROTAC FKBP Degrader-3 est un puissant dégradeur de FKBP.
-
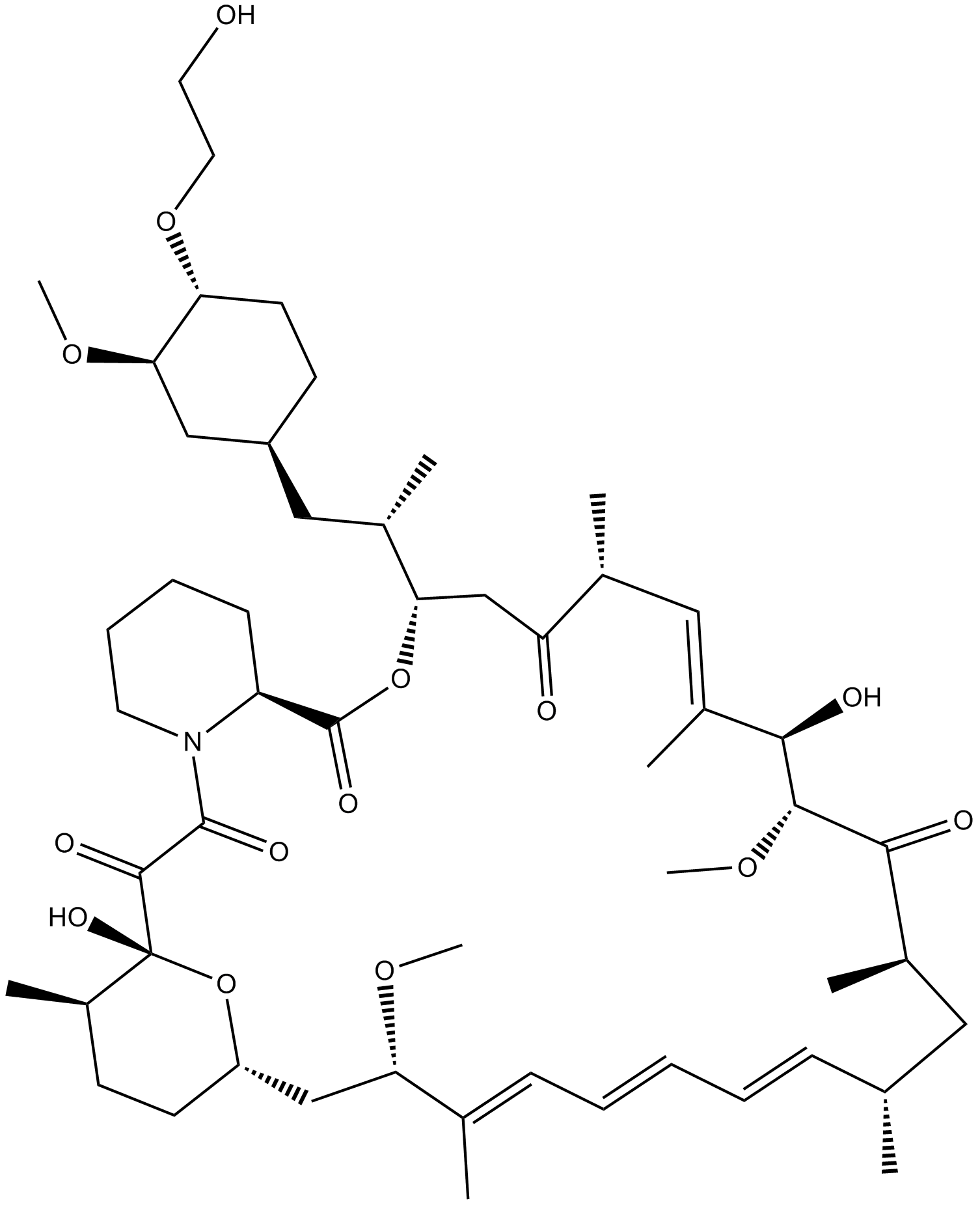
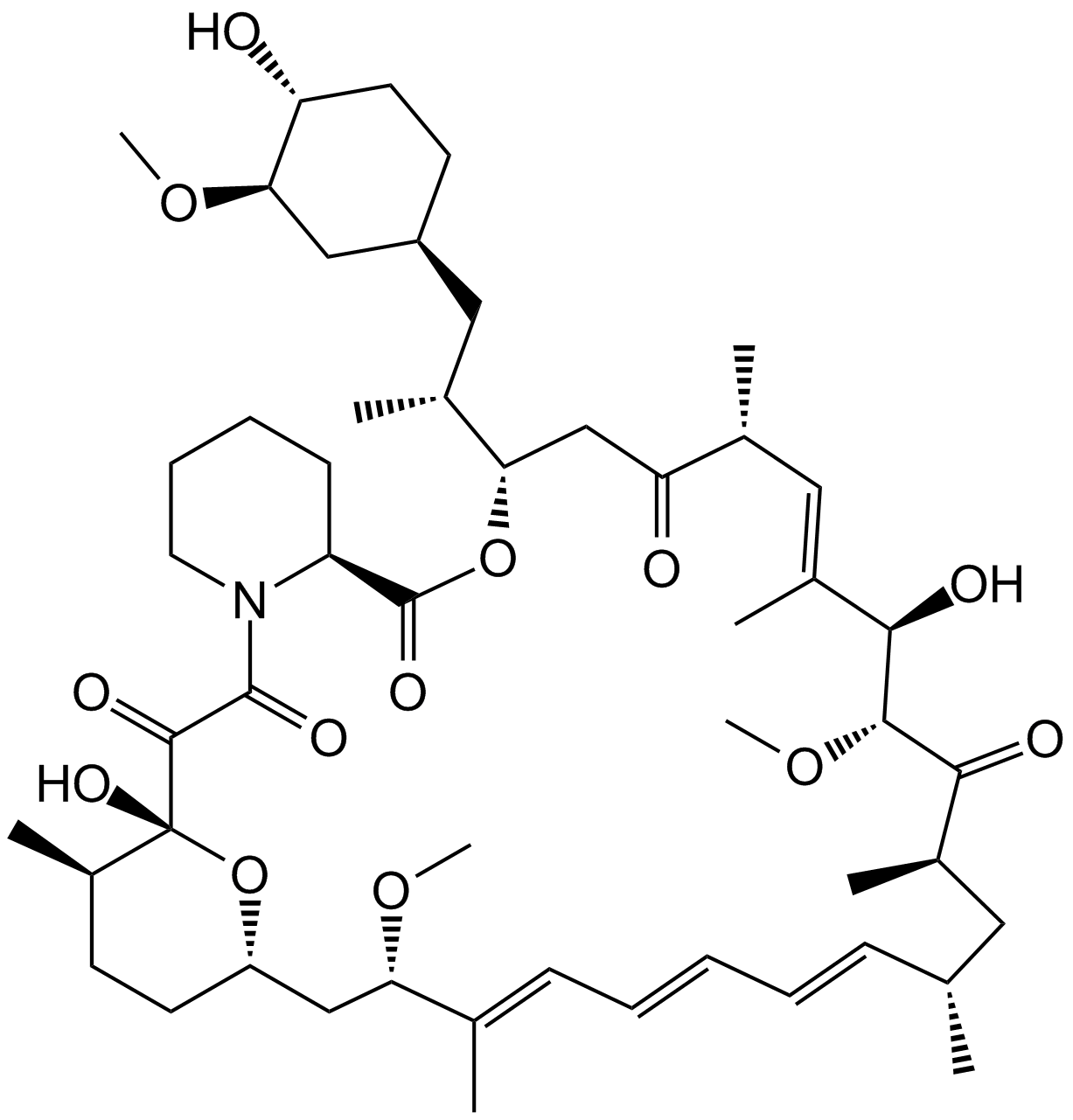
GC15031
Rapamycin (Sirolimus)
Un inhibiteur allostérique de mTORC1
-
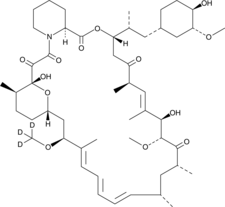
GC48027
Rapamycin-d3
La rapamycine-d3 (sirolimus-d3) est la rapamycine marquée au deutérium. La rapamycine est un inhibiteur puissant et spécifique de mTOR avec une IC50 de 0,1 nM dans les cellules HEK293. La rapamycine se lie À FKBP12 et agit spécifiquement comme un inhibiteur allostérique de mTORC1. La rapamycine est un activateur de l'autophagie, un immunosuppresseur.
-
GC33699
SAFit1
SAFit1 est un inhibiteur spécifique de la protéine de liaison FK506 51 (FKBP51) avec un Ki de 4 ± 0,3 nM .

-
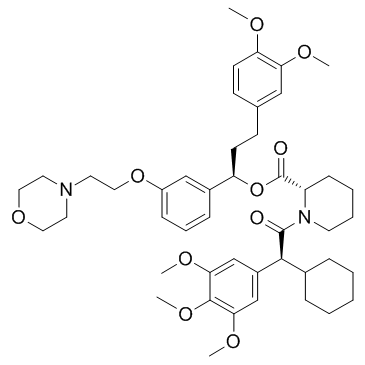
GC34093
SAFit2
SAFit2 est un inhibiteur de la protéine 51 de liaison au FK506 (FKBP51) très puissant et hautement sélectif avec un Ki de 6 nM et améliore également la liaison de l'AKT2-AS160.
-
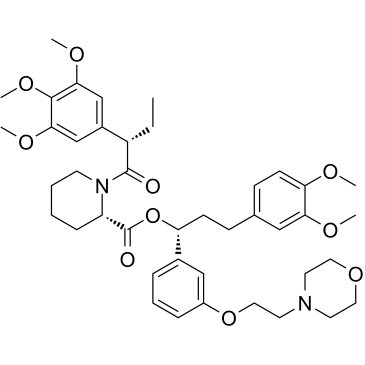
GC38060
Shield-1
Shield-1 (Shld1) est un ligand spécifique, perméable aux cellules et à haute affinité de la protéine de liaison FK506-12 (FKBP), et inverse l'instabilité en se liant à la FKBP mutée (mtFKBP), permettant une expression conditionnelle des protéines fusionnées mtFKBP.
-
GC44899
SLF
SLF est un ligand synthétique pour la protéine de liaison FK506 (FKBP) avec une affinité de 3,1 μM pour FKBP51 et une IC50 de 2,6 μM pour FKBP12. SLF peut être utilisé dans la synthèse de PROTAC.
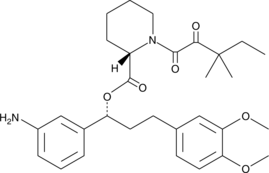
-
GC61461
SLF TFA
Le SLF TFA est un ligand synthétique pour la protéine de liaison FK506 (FKBP) avec une affinité de 3,1 μM pour FKBP51 et une IC50 de 2,6 μM pour FKBP12. Le SLF TFA peut être utilisé dans la synthèse de PROTAC.
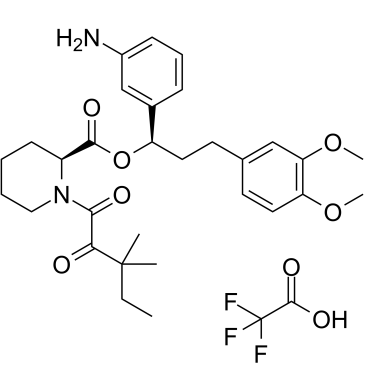
-
GC16233
Tacrolimus (FK506)
Le tacrolimus (FK506), un antibiotique macrolide aux puissants effets immunosuppresseurs, a été isolé à partir de Streptomyces tsukubaensis et a déjà été utilisé pour prévenir les greffes d'organes et traiter des troubles auto-immuns chez l'homme.
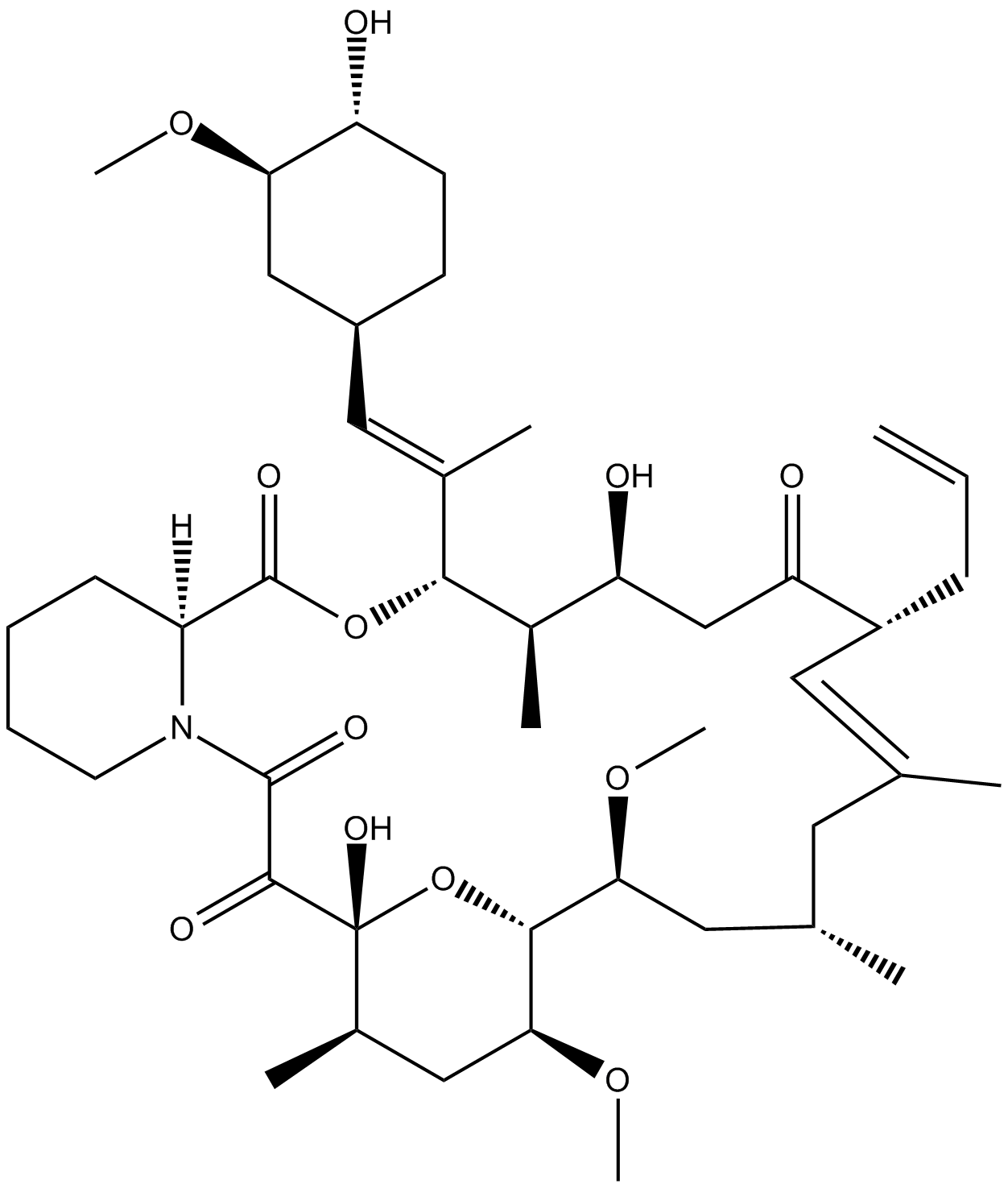
-
GC17995
Tacrolimus monohydrate
Le tacrolimus monohydraté (FK506 monohydraté), une lactone macrocyclique, se lie À la protéine de liaison FK506 (FKBP) pour former un complexe et inhibe la calcineurine phosphatase, qui inhibe la transduction du signal des lymphocytes T et la transcription de l'IL-2.
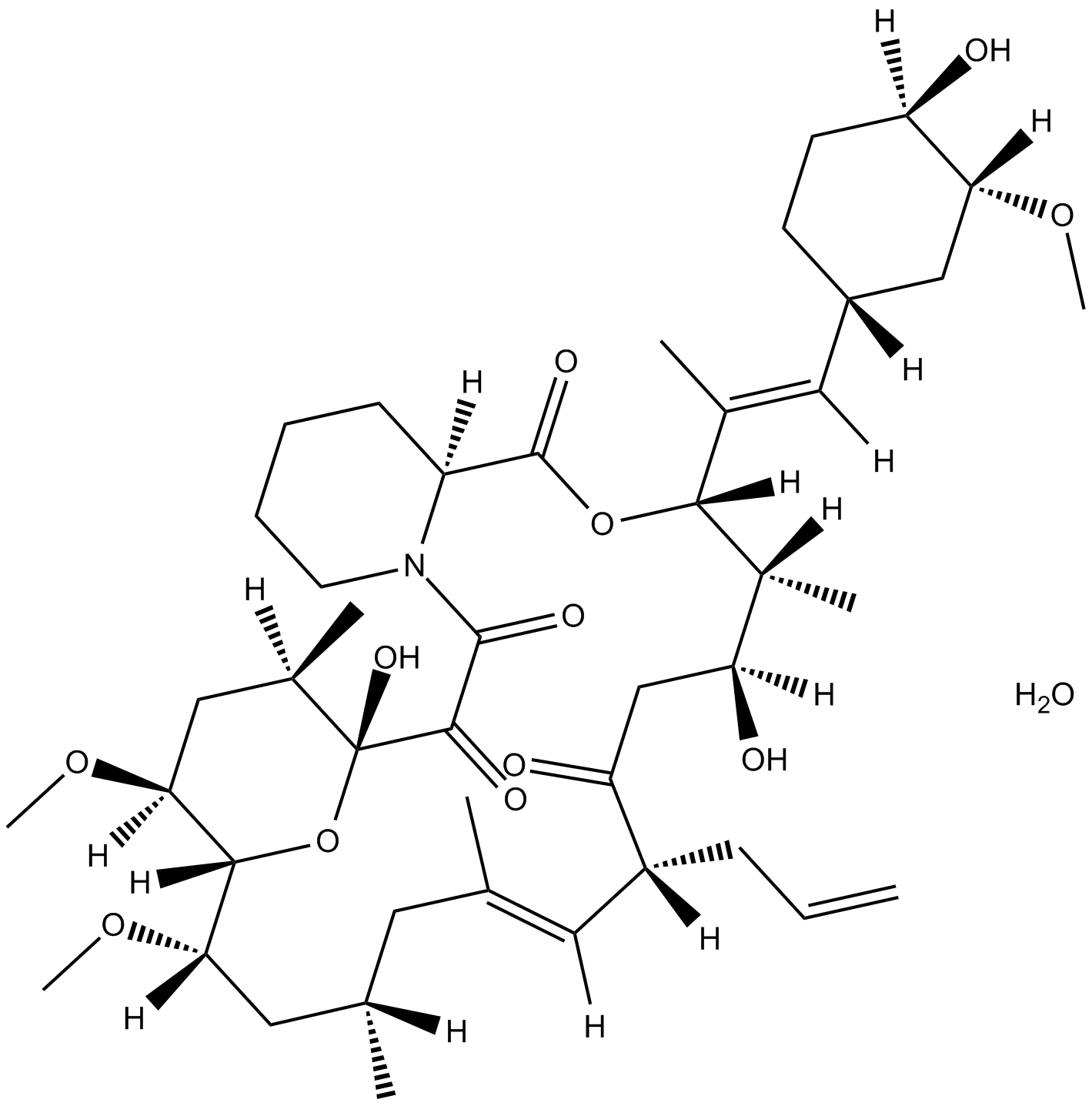
-
GC33200
Target Protein-binding moiety 13
Target Protein-binding fragment 13 (PROTAC FKBP12-binding fragment 1) est un ligand synthétique pour FKBP (SLF). Le groupement de liaison aux protéines cibles 13 (groupement de liaison PROTAC FKBP12 1) peut être utilisé dans la synthèse des PROTAC.
-
GC66370
Zapalog
Zapalog est un hétérodimériseur photoclivable À petite molécule qui peut être utilisé pour initier de manière répétée et mettre fin instantanément À une interaction physique entre deux protéines cibles. Zapalog dimérise deux protéines quelconques marquées avec les domaines FKBP et DHFR jusqu'À ce que l'exposition À la lumière provoque sa photolyse.