DNA Damage/DNA Repair
- MTH1(4)
- PARP(84)
- ATM/ATR(30)
- DNA Alkylating(20)
- DNA Ligases(3)
- DNA Methyltransferase(25)
- DNA-PK(30)
- HDAC(136)
- Nucleoside Antimetabolite/Analogue(140)
- Telomerase(17)
- Topoisomerase(151)
- tankyrase(5)
- Antifolate(38)
- CDK(250)
- Checkpoint Kinase (Chk)(31)
- CRISPR/Cas9(9)
- Deubiquitinase(72)
- DNA Alkylator/Crosslinker(71)
- DNA/RNA Synthesis(400)
- Eukaryotic Initiation Factor (eIF)(23)
- IRE1(23)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(61)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Products for DNA Damage/DNA Repair
- Cat.No. Nom du produit Informations
-
GC45353
4-oxo-27-TBDMS Withaferin A
La 4-oxo-27-TBDMS Withaferin A, un dérivé de la withaferine A, présente de puissants effets antiprolifératifs sur les cellules tumorales. La 4-oxo-27-TBDMS Withaferin A induit l'apoptose des cellules tumorales. 4-oxo-27-TBDMS Withaferin A est un agent anticancéreux.
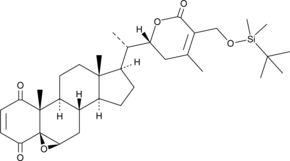
-
GC68557
4-Thio-2'-deoxyuridine
4-Thio-2'-désoxyuridine est un analogue de nucléoside purique. Les analogues de nucléosides puriques ont une activité antitumorale étendue, ciblant les tumeurs malignes du système lymphatique inerte. Le mécanisme anticancéreux dans ce processus dépend de l'inhibition de la synthèse d'ADN, induisant l'apoptose cellulaire.
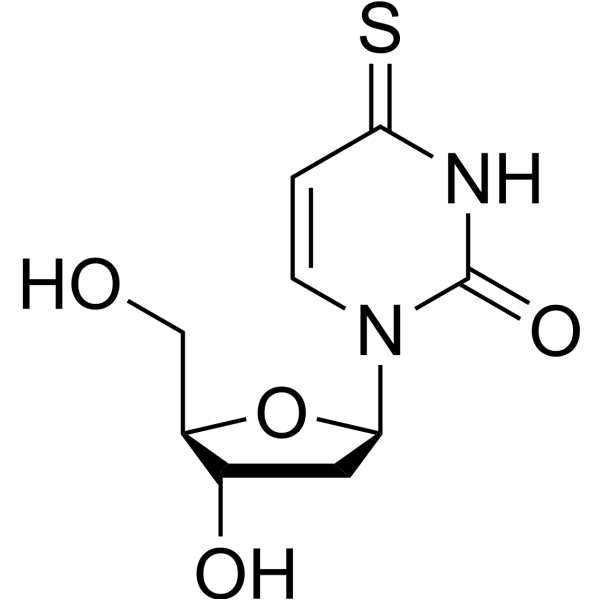
-
GC40477
4-Thiouracil
4-Thiouracil is a site-specific, photoactivatable probe used to detect RNA structures and nucleic acid-nucleic acid contacts.
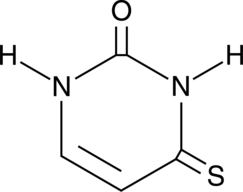
-
GC42474
4-Thiouridine
La 4-thiouridine est un analogue de ribonucléoside, elle est largement utilisée dans l'analyse de l'ARN et le marquage (m)ARN.
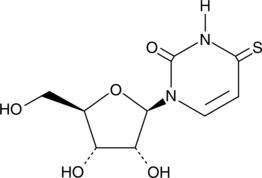
-
GC13104
4E1RCat
4E1RCat est un inhibiteur de la traduction cap-dépendante et inhibe l'interaction eIF4E:eIF4GI, avec une IC50 an de ~4 μM.
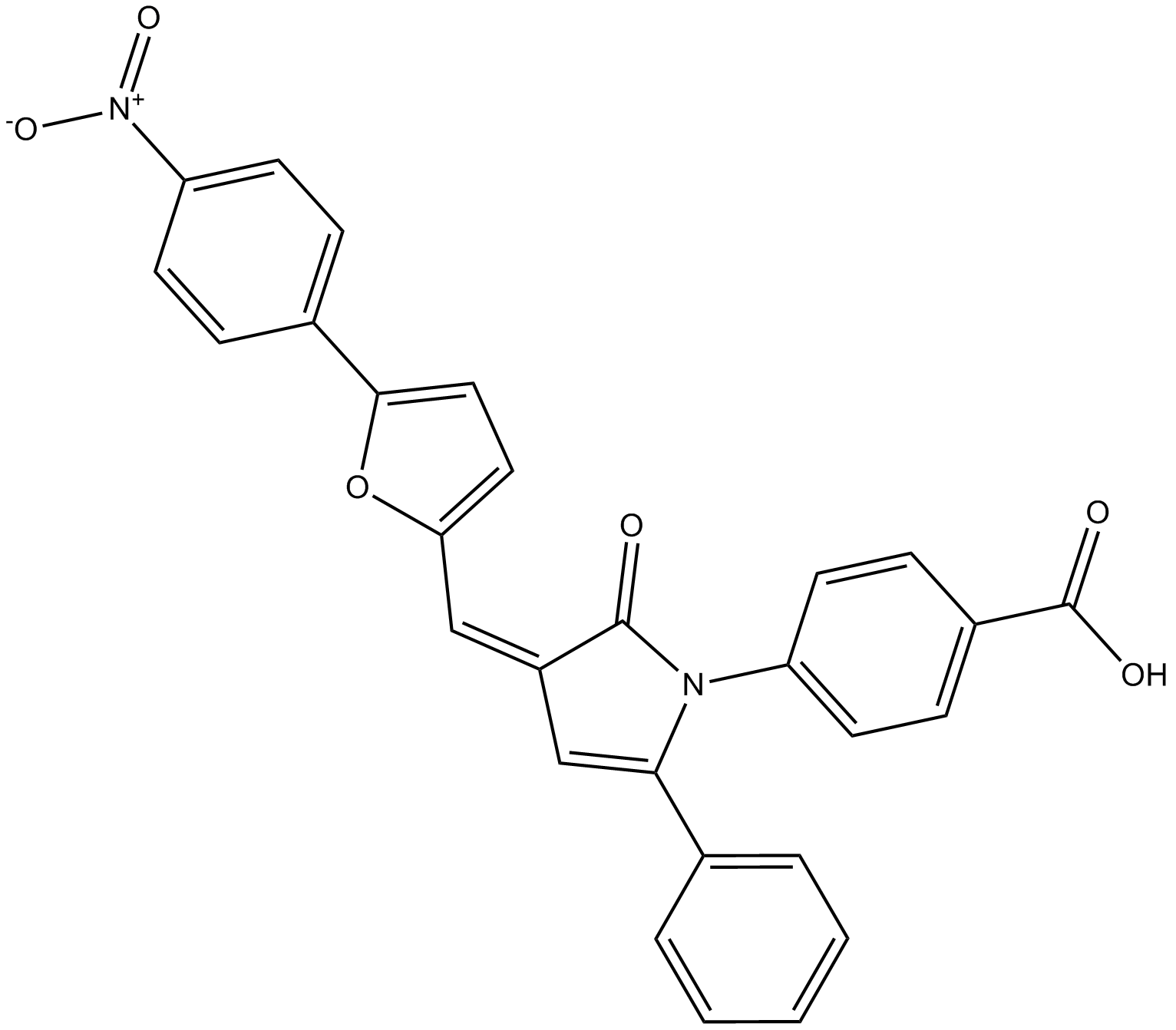
-
GC30785
4E2RCat
4E2RCat est un inhibiteur de l'interaction eIF4E-eIF4G avec une IC50 de 13,5 μM.
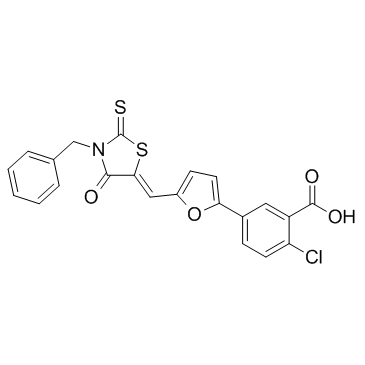
-
GC10468
4EGI-1
4EGI-1 est un inhibiteur de l'interaction eIF4E/eIF4G, avec un Kd de 25 μM contre la liaison eIF4E.
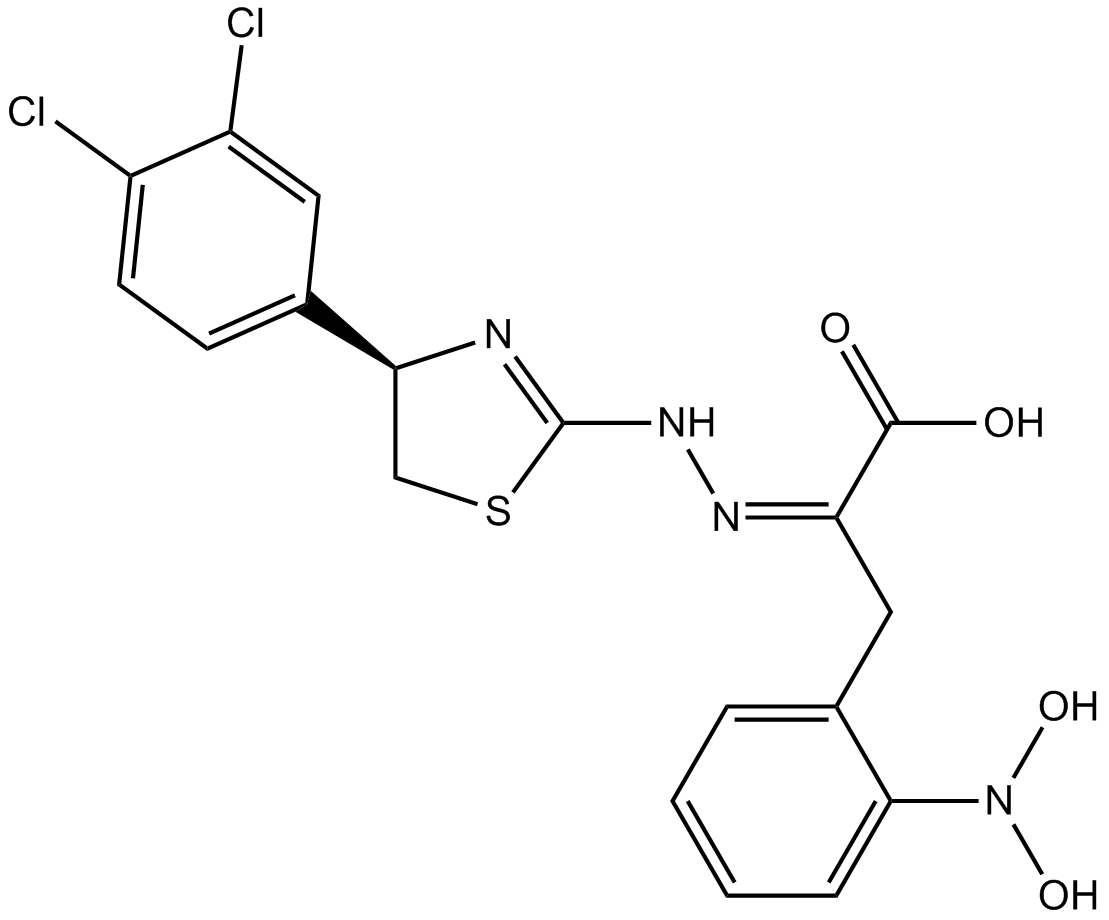
-
GC12667
4SC-202
4SC-202 (4SC-202) est un inhibiteur sélectif d'HDAC de classe I avec une IC50 de 1,20 μM, 1,12 μM et 0,57 μM pour HDAC1, HDAC2 et HDAC3, respectivement. Il affiche également une activité inhibitrice contre la déméthylase spécifique de la lysine 1 (LSD1).
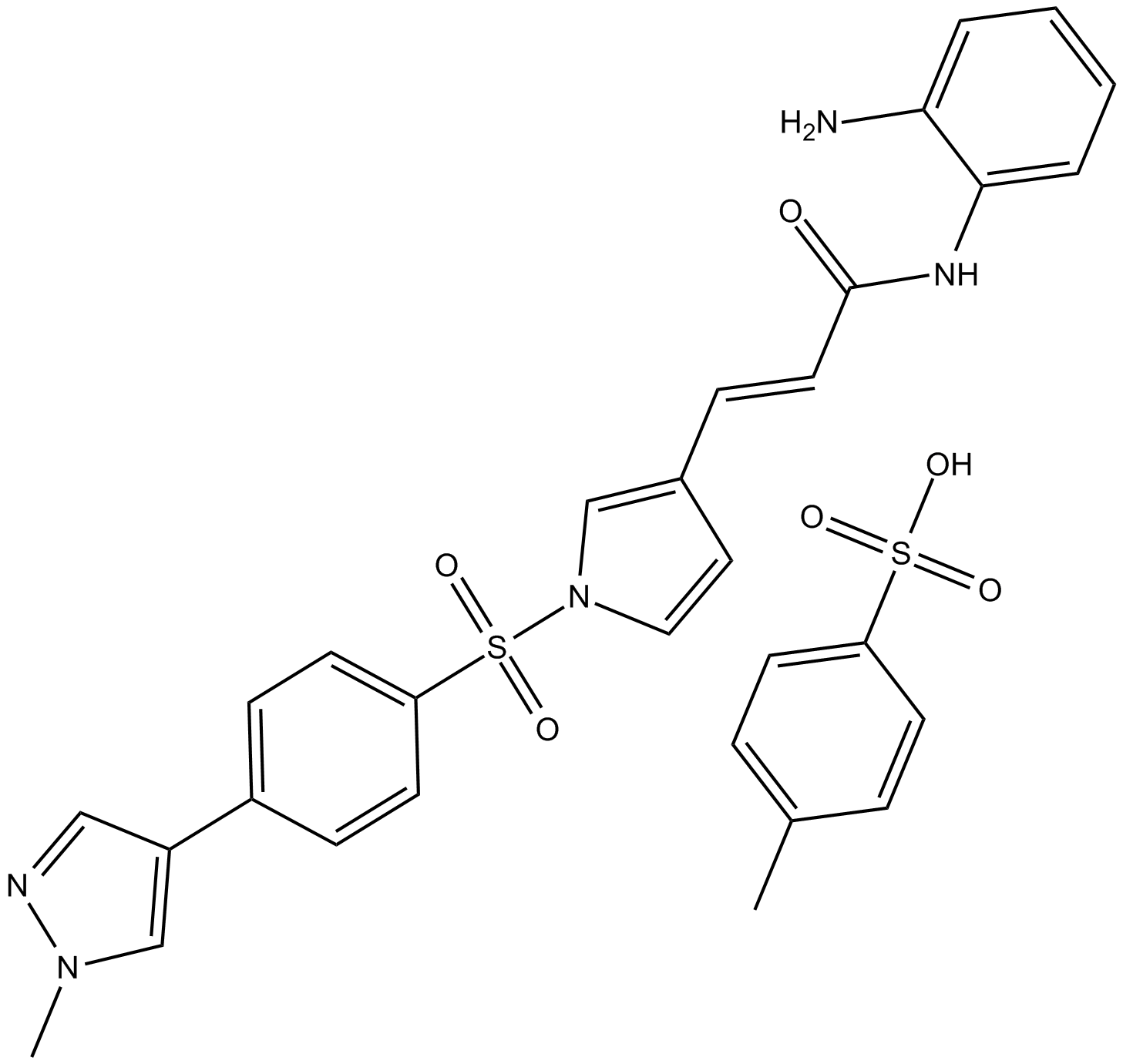
-
GC65520
5'-DMT-3'-TBDMS-ibu-rG
5'-DMT-3'-TBDMS-ibu-rG est un nucléoside modifié.

-
GC65402
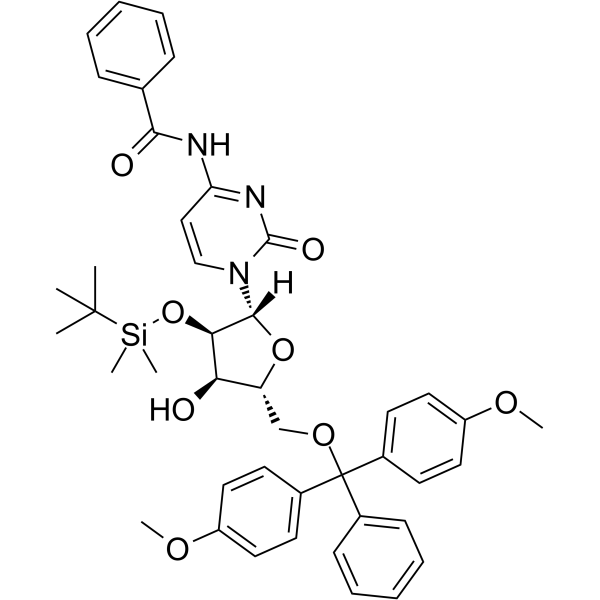
5'-O-DMT-2'-O-TBDMS-Bz-rC
5'-O-DMT-&2#39;-O-TBDMS-Bz-rC est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
-
GC65164
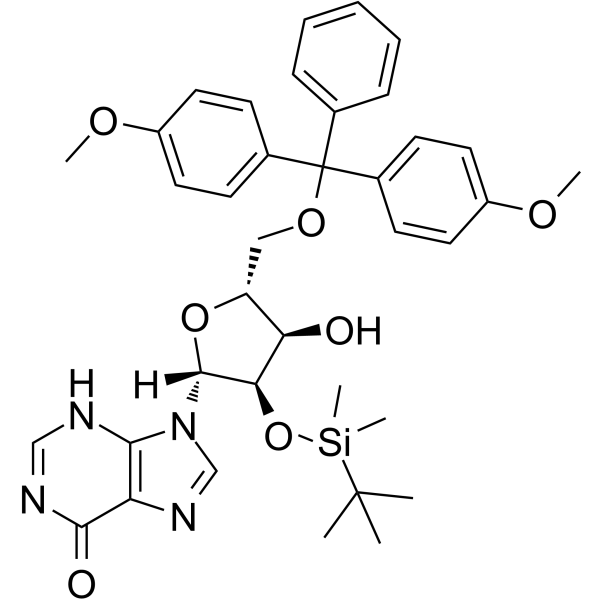
5'-O-DMT-2'-O-TBDMS-rI
5'-O-DMT-&2#39;-O-TBDMS-rI est un nucléoside modifié.
-
GC65396
5'-O-DMT-ibu-dC
5'-O-DMT-ibu-dC peut être utilisé dans la synthèse d'oligodésoxyribonucléotides.

-
GC64982
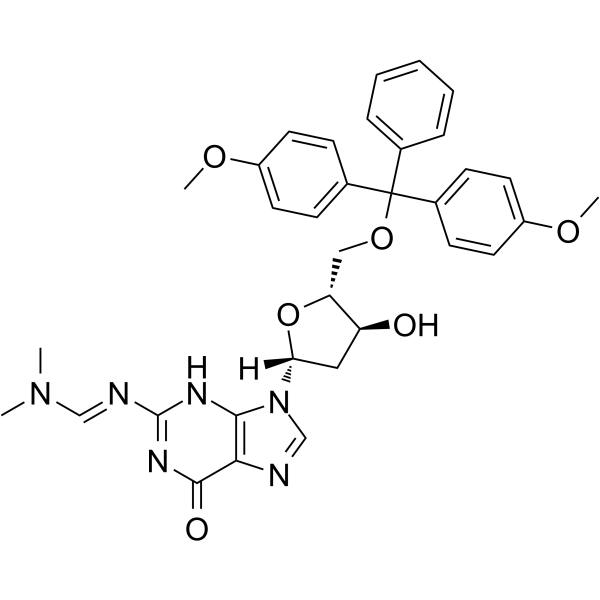
5'-O-DMT-N2-DMF-dG
Le 5'-O-DMT-2'-O-TBDMS-rI est un nucléoside modifié.
-
GC65482
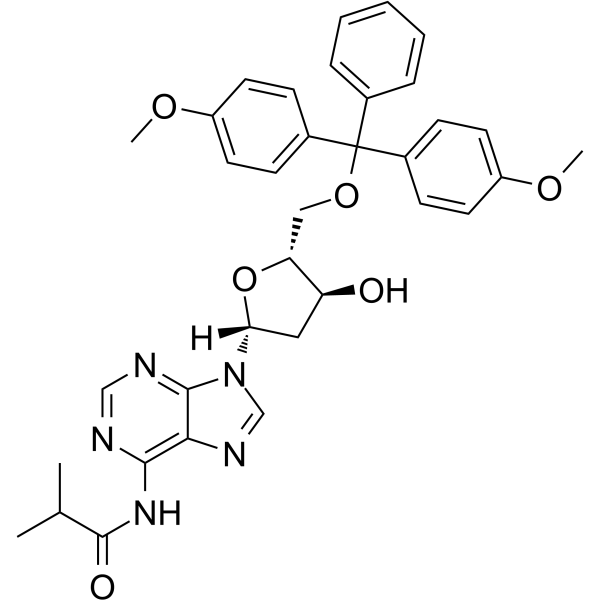
5'-O-DMT-N6-ibu-dA
5'-O-DMT-N6-ibu-dA peut être utilisé dans la synthèse d'oligodésoxyribonucléotides.
-
GC65562
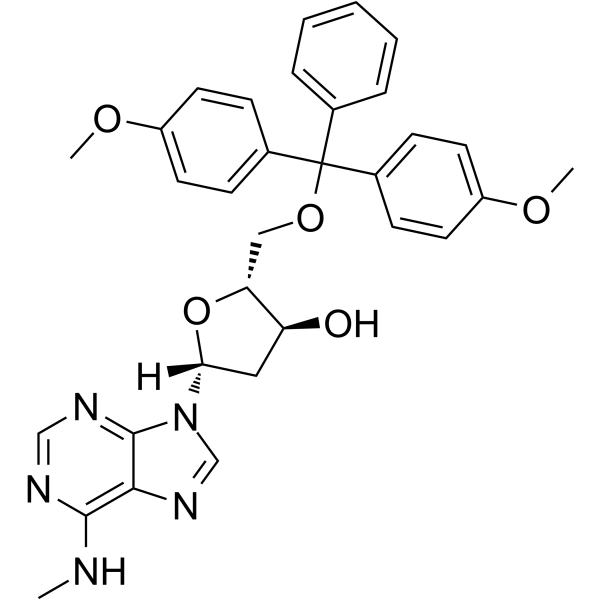
5'-O-DMT-N6-Me-2'-dA
5'-O-DMT-N6-Me-&2#39;-dA est un nucléoside ayant des effets protecteurs et modificateurs.
-
GC65401
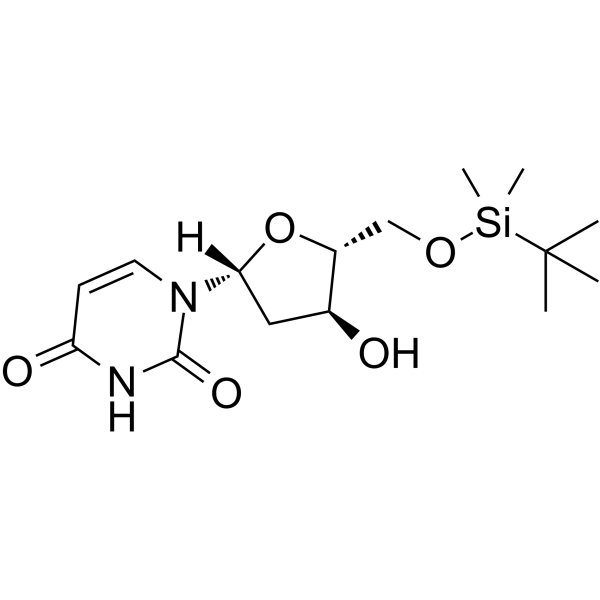
5'-O-TBDMS-dU
5'-O-TBDMS-dU peut être utilisé dans la synthèse d'oligoribonucléotides.
-
GC62157
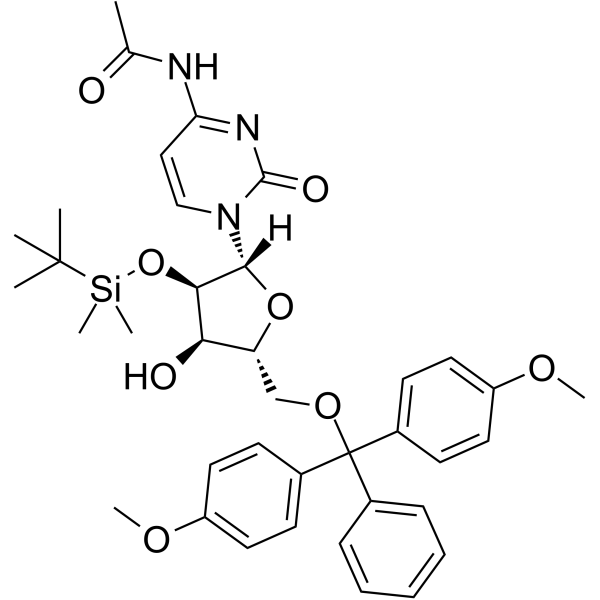
5’-O-DMT-2’-O-TBDMS-Ac-rC
5’-O-DMT-&2rsquo;-O-TBDMS-Ac-rC est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
-
GC62149
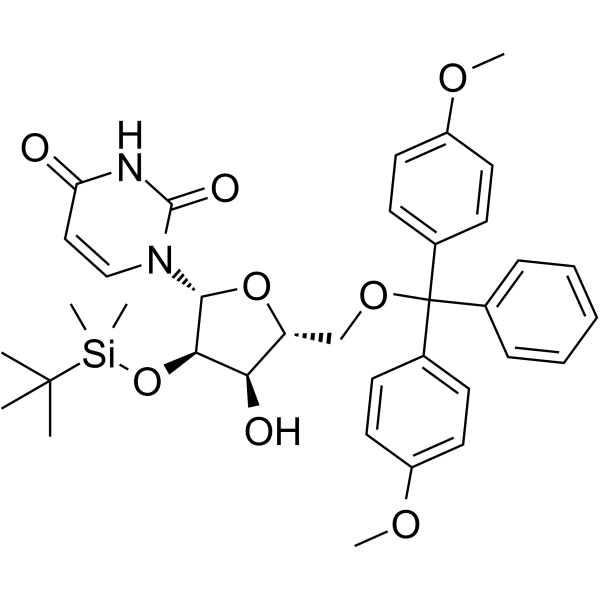
5’-O-DMT-2’-TBDMS-Uridine
5’-O-DMT-&2rsquo;-TBDMS-Uridine est un désoxyribonucléoside utilisé pour la synthèse d'oligonucléotides.
-
GC62532
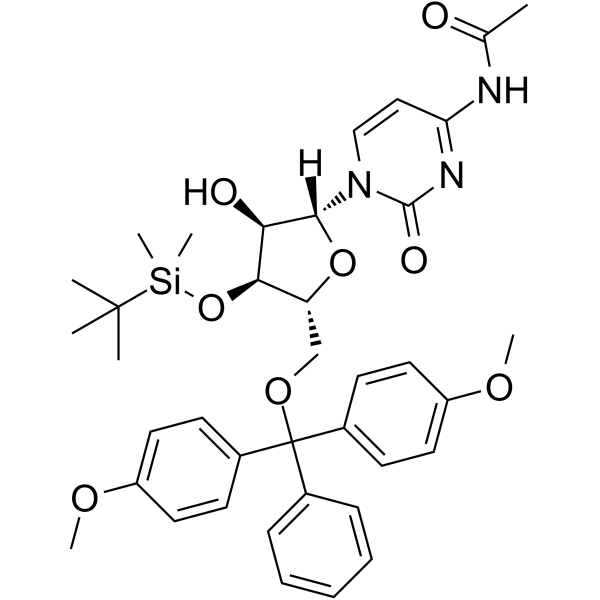
5’-O-DMT-3’-O-TBDMS-Ac-rC
5’-O-DMT-3’-O-TBDMS-Ac-rC est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
-
GC62573
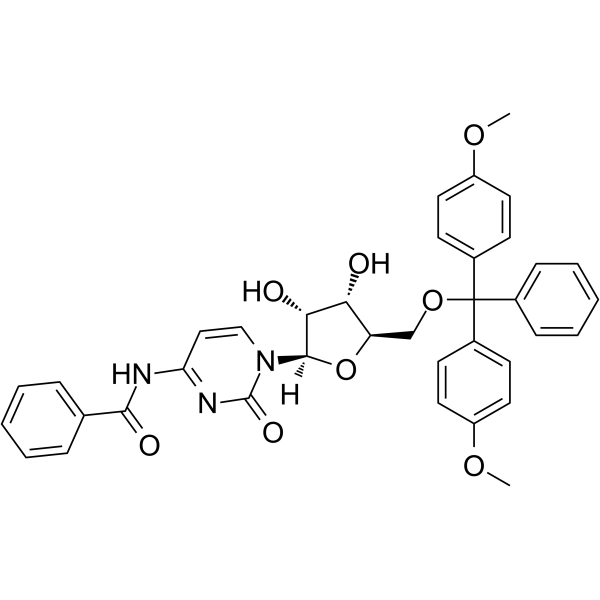
5’-O-DMT-Bz-rC
Le 5'-O-DMT-Bz-Rc est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
-
GC62574
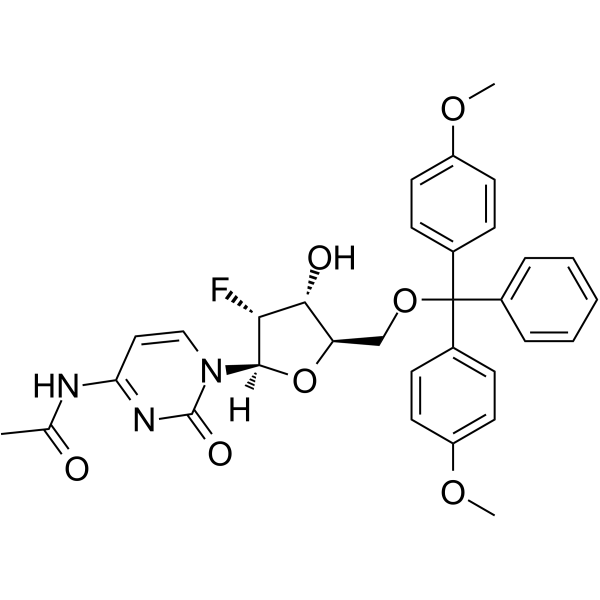
5’-O-DMT-N4-Ac-2’-F-dC
5’-O-DMT-N4-Ac-&2rsquo;-F-dC est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
-
GC62575
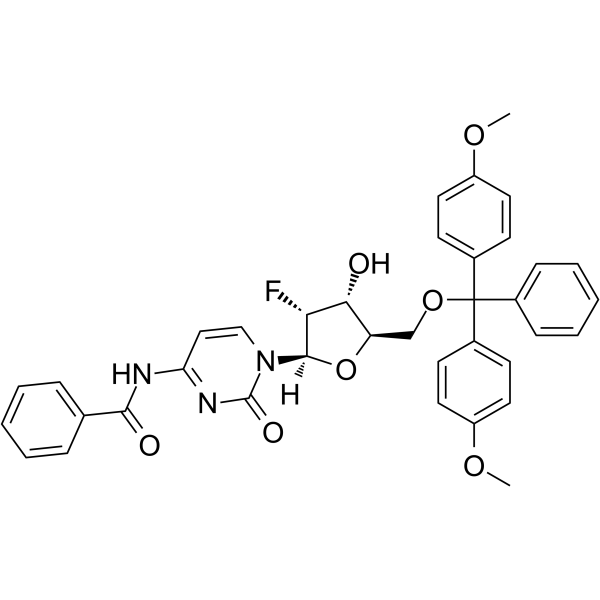
5’-O-DMT-N4-Bz-2’-F-dC
5’-O-DMT-N4-Bz-&2rsquo;-F-dC est un nucléoside ayant des effets protecteurs et modificateurs.
-
GC62576
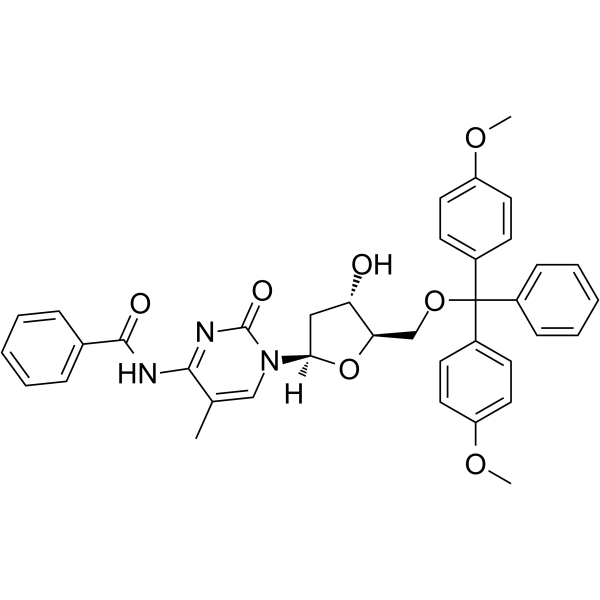
5’-O-DMT-N4-Bz-5-Me-dC
5’-O-DMT-N4-Bz-5-Me-dC est un nucléoside modifié.
-
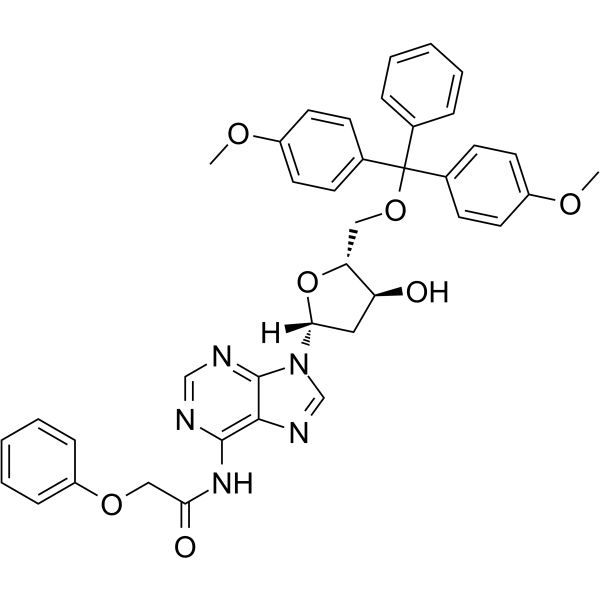
GC62812
5’-O-DMT-PAC-dA
5’-O-DMT-PAC-dA peut être utilisé dans la synthèse d'oligoribonucléotides.
-
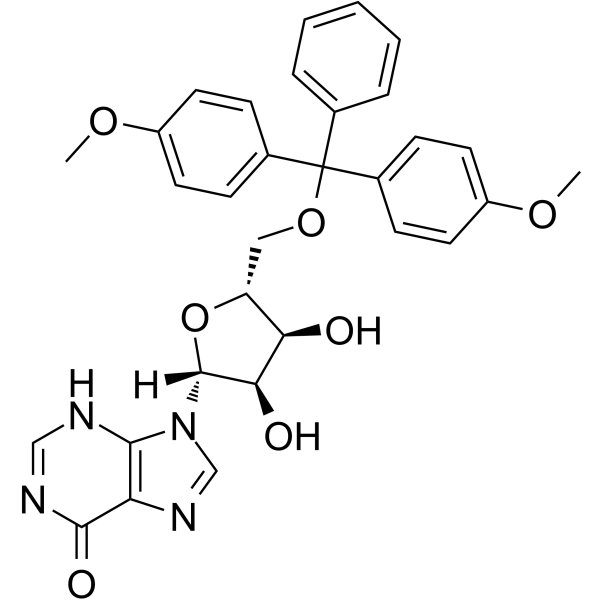
GC62813
5’-O-DMT-rI
Le 5'-O-DMT-Ri peut être utilisé dans la synthèse d'oligoribonucléotides.
-
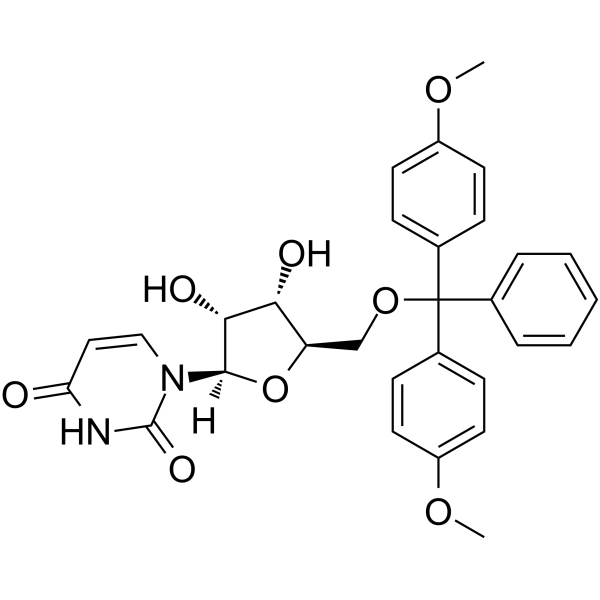
GC62577
5’-O-DMT-rU
5’-O-DMT-rU est un nucléoside modifié et peut être utilisé pour synthétiser l'ARN.
-
GC62578
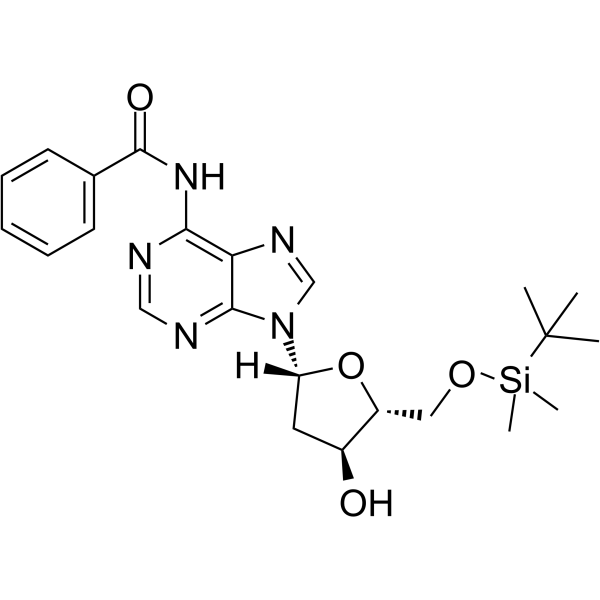
5’-O-TBDMS-Bz-dA
5’-O-TBDMS-Bz-dA est un nucléoside ayant des effets protecteurs et modificateurs.
-
GC62579
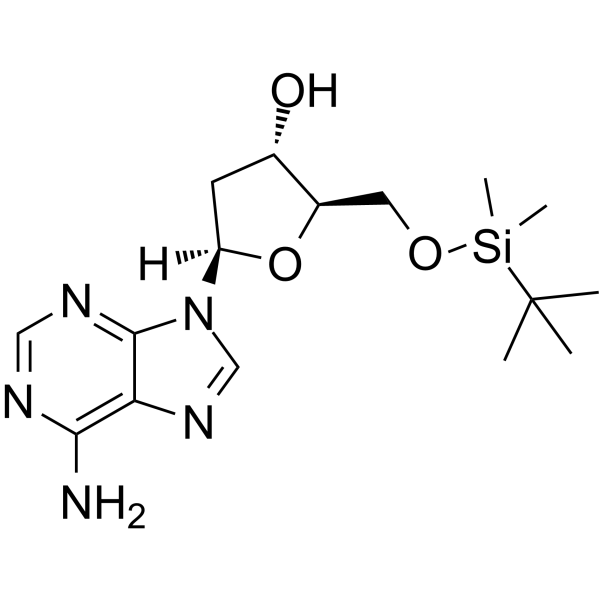
5’-O-TBDMS-dA
5’-O-TBDMS-dA est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
-
GC62580
5’-O-TBDMS-dG
5’-O-TBDMS-dG est un nucléoside modifié.

-
GC62581
5’-O-TBDMS-dT
5’-O-TBDMS-dT est un nucléoside ayant des effets protecteurs et modificateurs.

-
GC40641
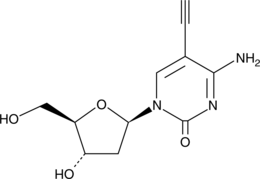
5'-Ethynyl-2'-deoxycytidine
5'-Ethynyl-2'-deoxycytidine (EdC) is a nucleoside analog that inhibits replication of the herpes simplex virus-1 (HSV-1) KOS strain (ID50 = 0.2 μg/mL).
-
GC61432
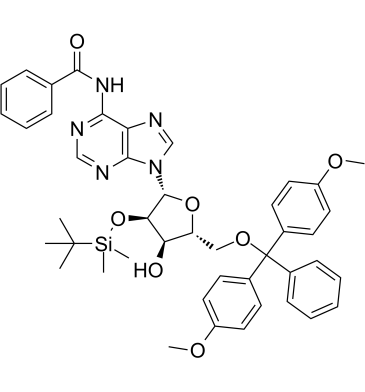
5'-O-DMT-2'-O-TBDMS-N-Bz-Adenosine
5'-O-DMT-2'-O-TBDMS-N-Bz-Adenosineest un dérivé de l'adénosine et peut être utilisé comme intermédiaire pour la synthèse d'oligonucléotides.
-
GC61726
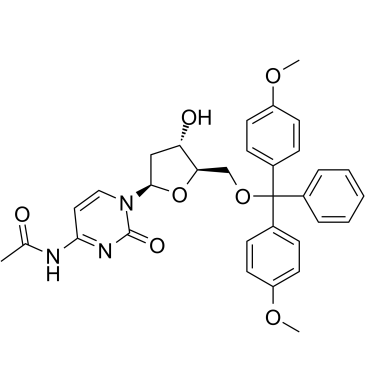
5'-O-DMT-N4-Ac-dC
Le 5'-O-DMT-N4-Ac-dC (N4-Acétyl-2'-désoxy-5'-O-DMT-cytidine, composé 7), un désoxynucléoside, peut être utilisé pour synthétiser le dodécyl phosphoramidite qui est le brut matériel pour la synthèse de dod-DNA (ADN amphiphile contenant une région hydrophobe interne constituée de liaisons dodécyl phosphotriester) .
-
GC48662
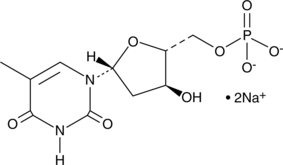
5'-Thymidylic Acid (sodium salt)
5'-L'acide thymidylique (sel de sodium) est un métabolite endogène.
-
GC35150
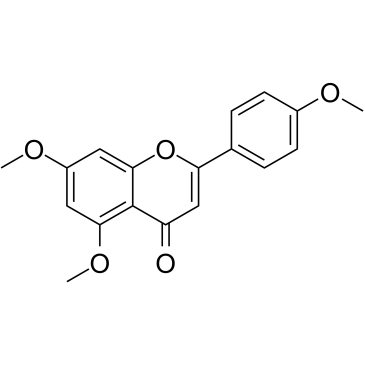
5,7,4'-Trimethoxyflavone
La 5,7,4'-triméthoxyflavone est isolée de Kaempferia parviflora (KP) qui est une célèbre plante médicinale de Thaïlande. La 5,7,4'-triméthoxyflavone induit l'apoptose, comme en témoignent les augmentations de la phase sous-G1, la fragmentation de l'ADN, la coloration annexine-V/PI, le rapport Bax/Bcl-xL, l'activation protéolytique de la caspase-3 et la dégradation de la poly (ADP-ribose) polymérase (PARP). La 5,7,4'-triméthoxyflavone est significativement efficace pour inhiber la prolifération des cellules cancéreuses gastriques humaines SNU-16 d'une manière dépendante de la concentration.
-
GC68161
5-AIQ
-
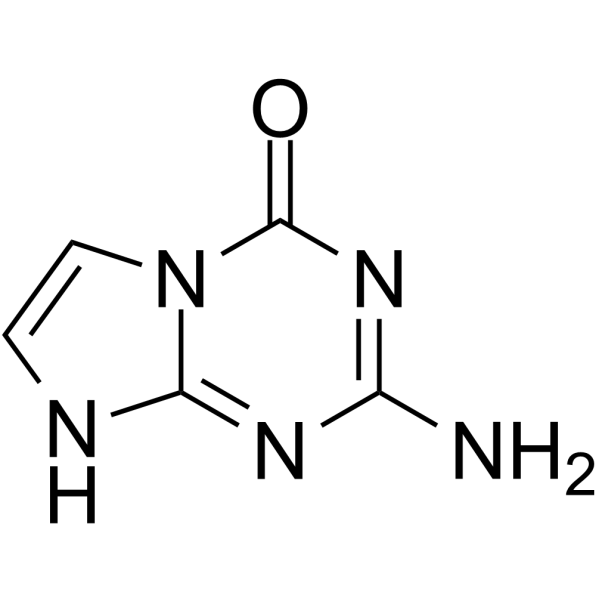
GC64952
5-Aza-7-deazaguanine
La 5-aza-7-déazaguanine est un substrat de la purine nucléoside phosphorylase d'E. coli de type sauvage (WT) et de son mutant Ser90Ala dans la synthèse de nucléosides À base modifiée.
-
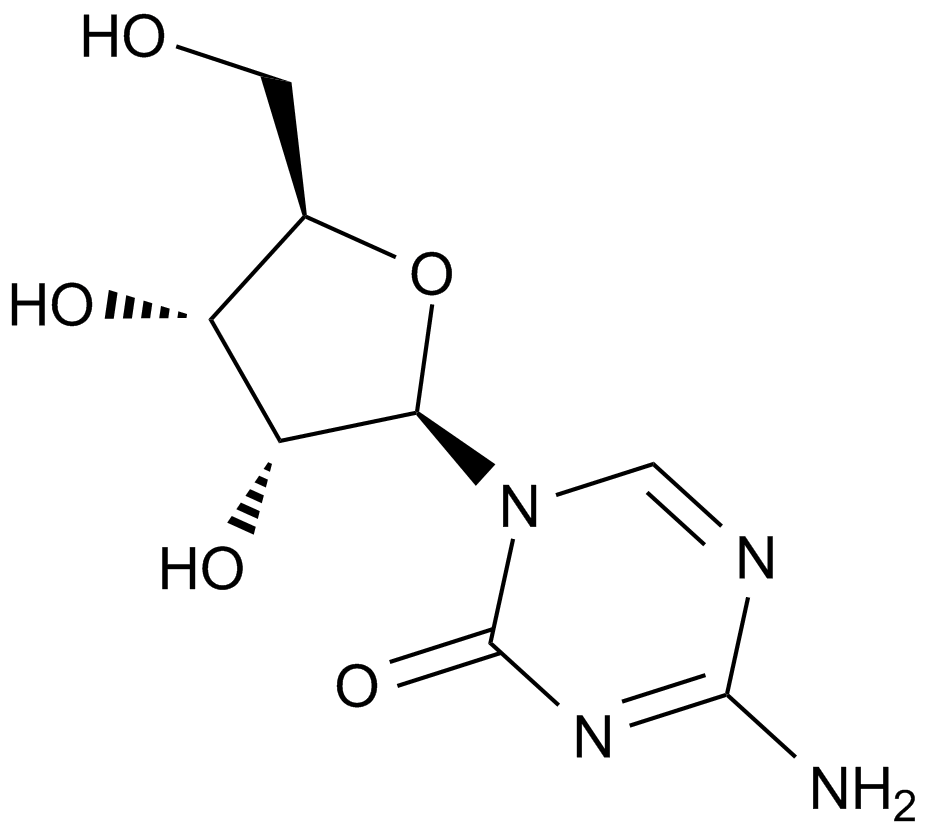
GC10946
5-Azacytidine
A DNA methyltransferase inhibitor
-
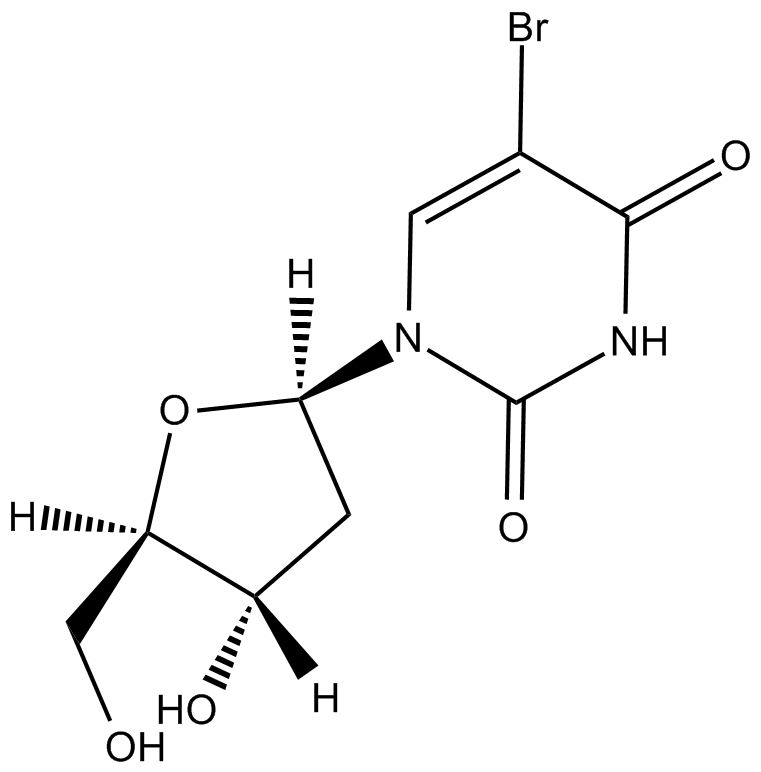
GC11940
5-BrdU
Analogie synthétique de la thymidine
-
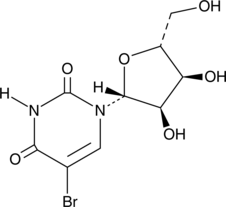
GC46681
5-Bromouridine
A brominated uridine analog
-
GC49245
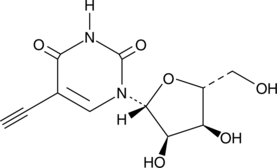
5-Ethynyluridine
La 5-éthynyluridine (5-EU) est un puissant nucléoside perméable aux cellules qui peut être utilisé pour marquer l'ARN nouvellement synthétisé.
-
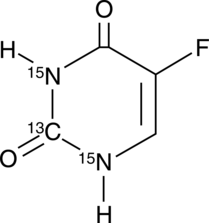
GC42545
5-Fluorouracil-13C,15N2
5-Fluorouracil-13C,15N2 is intended for use as an internal standard for the quantification of 5-flurouracil by GC- or LC-MS.
-
GC46033
5-Heneicosylresorcinol
An alkylresorcinol

-
GC62389
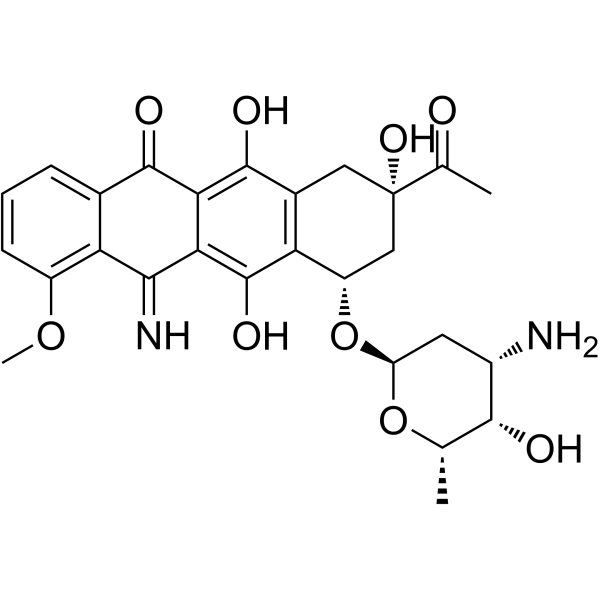
5-Iminodaunorubicin
La 5-iminodaunorubicine est une anthracycline modifiée par une quinone qui conserve une activité antitumorale. La 5-iminodaunorubicine produit des ruptures de brins d'ADN dissimulées par des protéines dans les cellules cancéreuses.
-
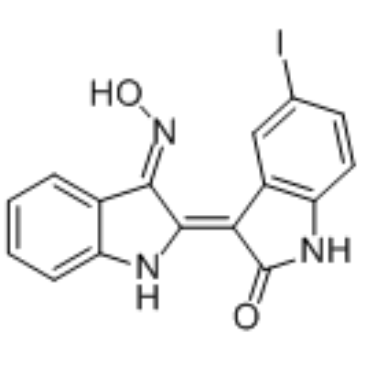
GC35162
5-Iodo-indirubin-3'-monoxime
La 5-iodo-indirubine-3'-monoxime est un puissant inhibiteur de GSK-3β, CDK5/P25 et CDK1/cycline B, en compétition avec l'ATP pour la liaison au site catalytique de la kinase, avec des IC50 de 9, 20 et 25 nM, respectivement.
-
GC66658
5-Me-dC(Ac) amidite
L'amidite 5-Me-dC(Ac) est utilisé pour synthétiser l'ADN ou l'ARN.
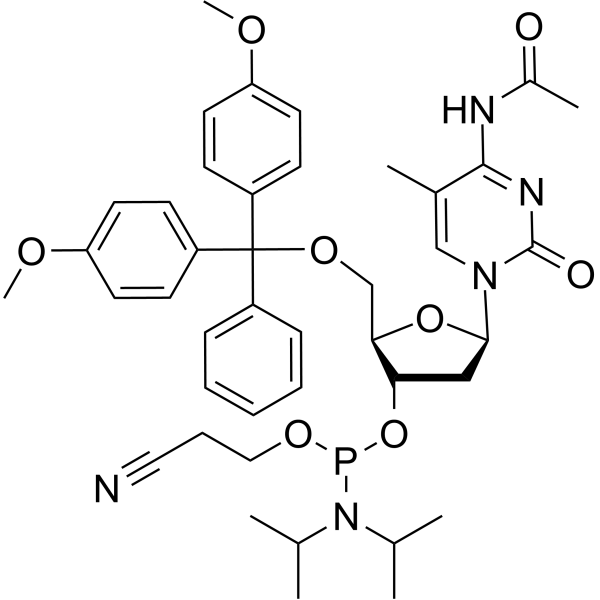
-
GC31187
5-Methoxyflavone
La 5-méthoxyflavone, appartenant À la famille des flavonoÏdes, est un inhibiteur de l'ADN polymérase-bêta et un agent neuroprotecteur contre la toxicité bêta-amyloÏde.
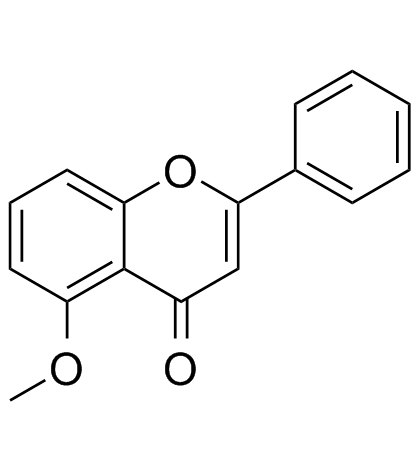
-
GC42562
5-Methyl-2'-deoxycytidine
La 5-méthyl-2'-désoxycytidine dans l'ADN simple brin peut agir en cis pour signaler la méthylation de novo de l'ADN .
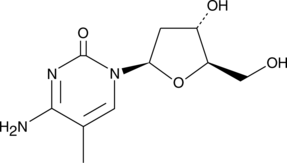
-
GC67671
5-Methylcytidine 5′-triphosphate trisodium
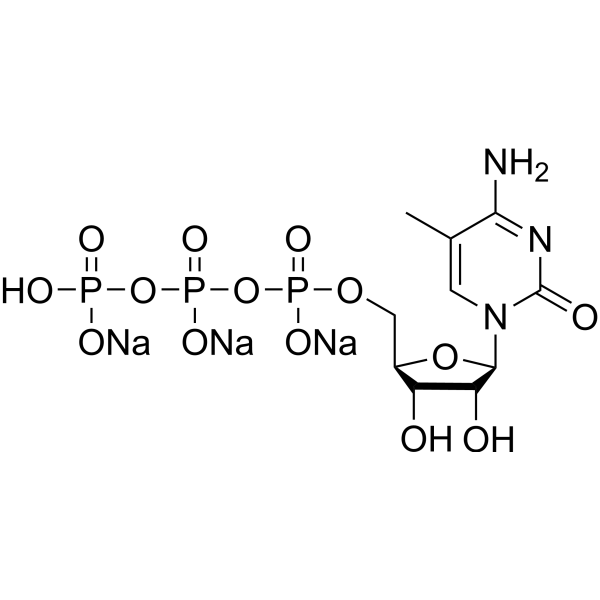
-
GC35166
5-Methylcytosine
La 5-méthylcytosine est une modification de l'ADN bien caractérisée et se trouve également principalement dans les ARN non codants abondants chez les procaryotes et les eucaryotes.
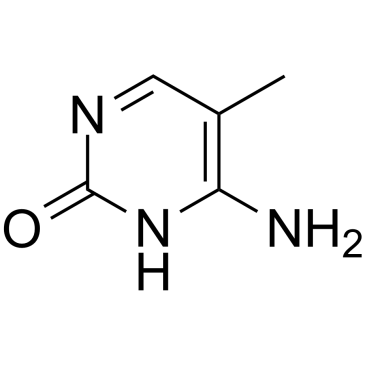
-
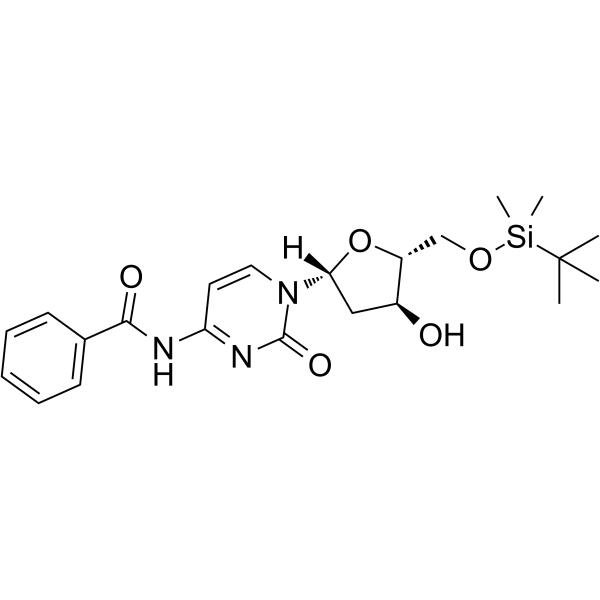
GC62547
5-O-TBDMS-N4-Benzoyl-2-deoxycytidine
La 5-O-TBDMS-N4-Benzoyl-2-désoxycytidine est un nucléoside modifié.
-
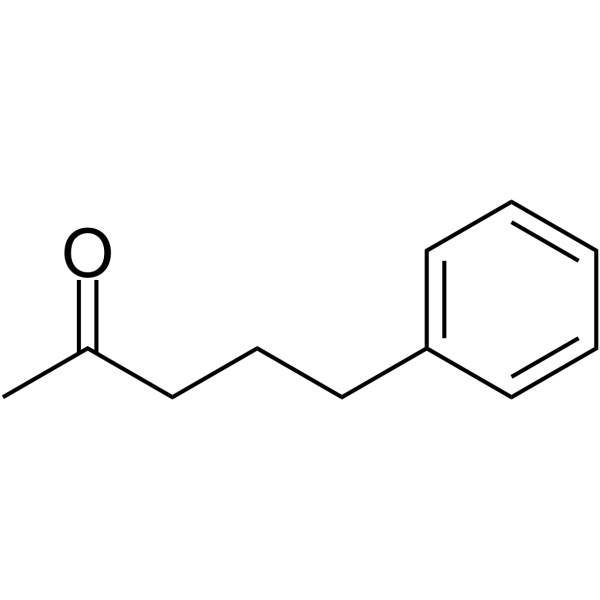
GC66055
5-Phenylpentan-2-one
La 5-phénylpentane-2-one est un puissant inhibiteur des histones désacétylases (HDAC). Le 5-phénylpentan-2-one peut être utilisé pour la recherche sur les troubles du cycle de l'urée.
-
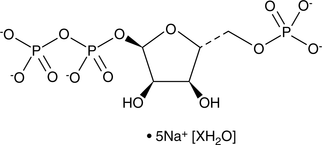
GC46713
5-Phospho-D-ribose 1-diphosphate (sodium salt hydrate)
An intermediate in several biochemical pathways
-
GC46079
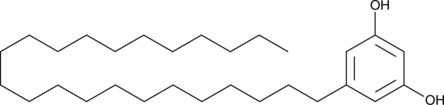
5-Tricosylresorcinol
Le 5-tricosylresorcinolthe est le premier lipide du kyste.
-
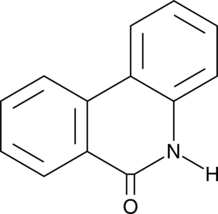
GC45772
6(5H)-Phenanthridinone
An inhibitor of PARP1 and 2
-
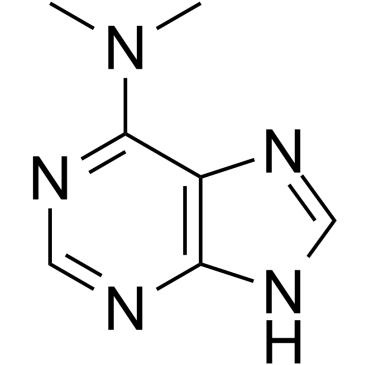
GC60532
6-(Dimethylamino)purine
La 6-(diméthylamino)purine est un double inhibiteur de la protéine kinase et de la CDK.
-
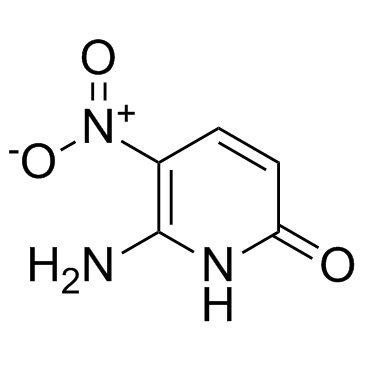
GC30548
6-Amino-5-nitropyridin-2-one
La 6-amino-5-nitropyridin-2-one est une base pyridinique et utilisée comme nucléobase de l'ADN hachimoji, dans laquelle elle s'apparie avec la 5-aza-7-déazaguanine.
-
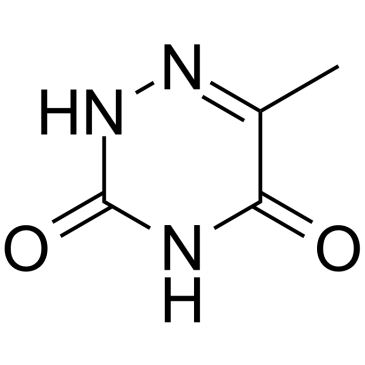
GC60031
6-Azathymine
La 6-azathymine, un analogue 6-azoté de la thymine, est un puissant inhibiteur de la D-3-aminoisobutyrate-pyruvate aminotransférase.
-
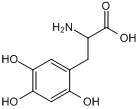
GC50249
6-Hydroxy-DL-DOPA
La 6-hydroxy-DL-DOPA est un inhibiteur allostérique sélectif et efficace du domaine de liaison À l'ADNsb de RAD52. La 6-hydroxy-DL-DOPA peut être utilisée pour la recherche sur le cancer.
-
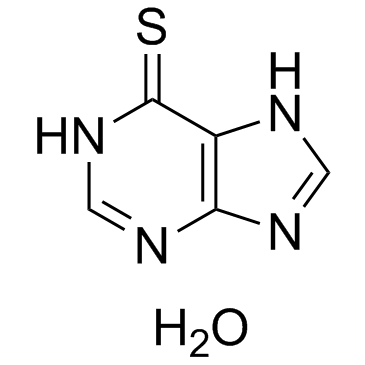
GC32975
6-Mercaptopurine hydrate
L'hydrate de 6-mercaptopurine (hydrate de mercaptopurine; hydrate de 6-MP) est un analogue de la purine qui agit comme un antagoniste des purines endogènes et a été largement utilisé comme agent antileucémique et médicament immunosuppresseur.
-
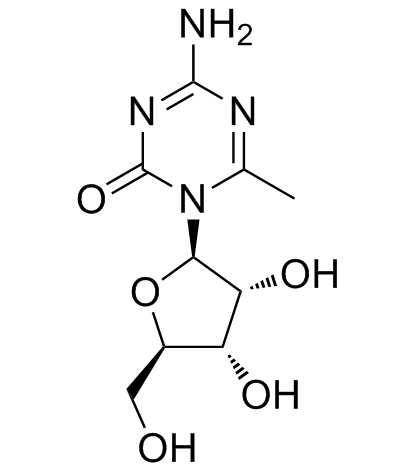
GC35180
6-Methyl-5-azacytidine
La 6-méthyl-5-azacytidine est un puissant inhibiteur de la DNMT.
-
GC49235
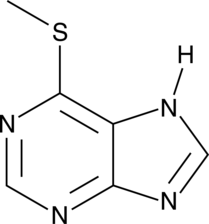
6-Methylmercaptopurine
A metabolite of 6-mercaptopurine
-
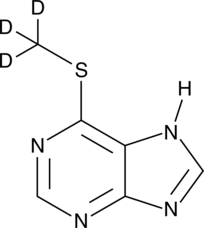
GC49488
6-Methylmercaptopurine-d3
An internal standard for the quantification of 6-MMP
-
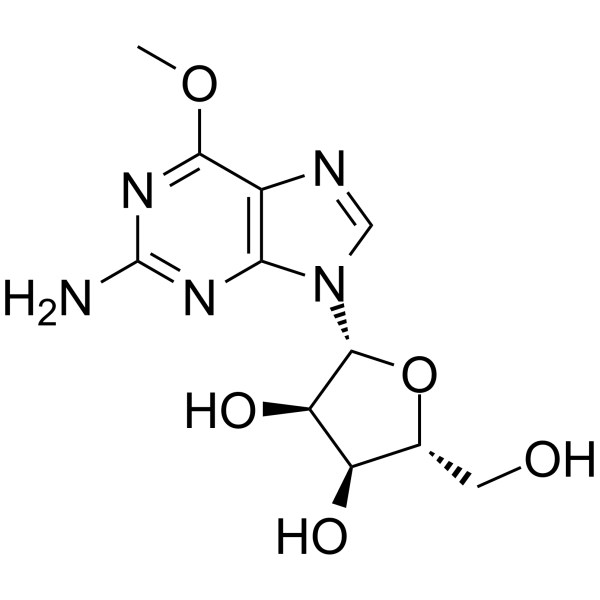
GC65082
6-O-Methyl Guanosine
La 6-O-méthyl guanosine est un nucléoside modifié.
-
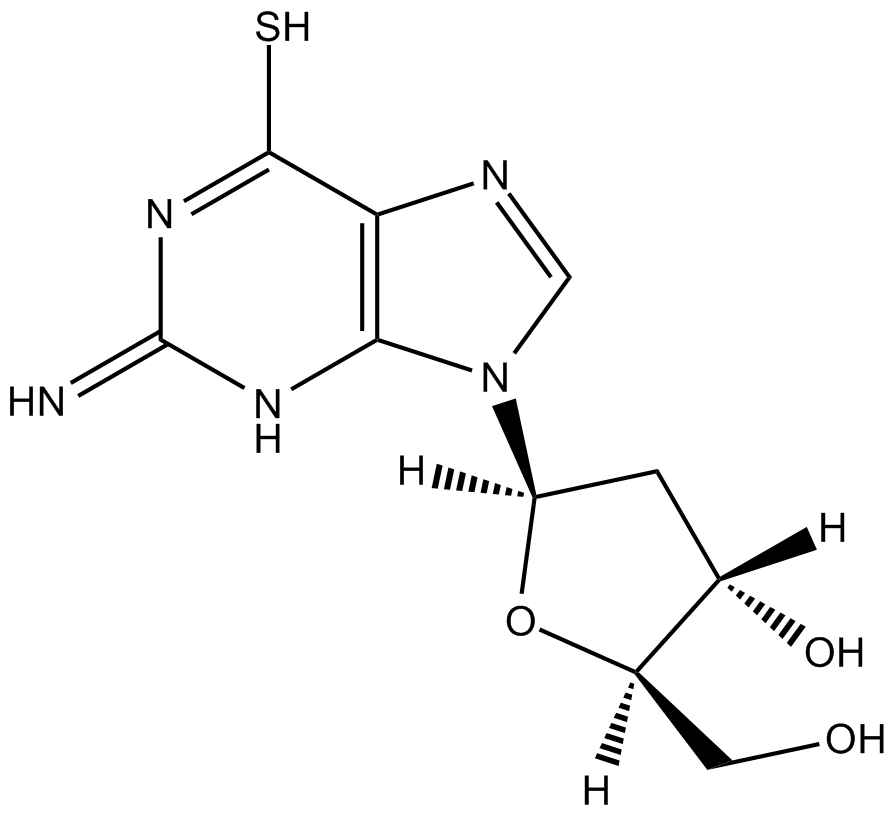
GC12193
6-Thio-dG
Le 6-Thio-dG est un analogue nucléosidique qui peut être incorporé dans des télomères synthétisés de novo par la télomérase.
-
GC35171
6-Thioinosine
La 6-thioinosine (6TI) est un antimétabolite purique, agit comme un agent anti-adipogenèse, régule à la baisse les niveaux d'ARNm de PPAR γ et C/EBPα, ainsi que la protéine cible PPAR γ telle que LPL, CD36, aP2 et LXRα.
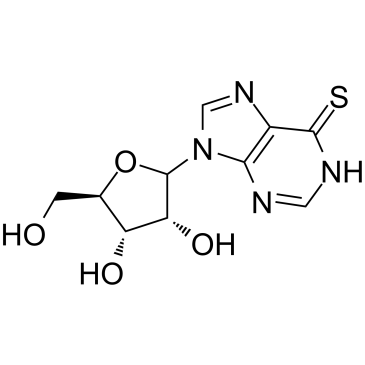
-
GC39625
6RK73
6RK73 est un inhibiteur covalent irréversible et spécifique de l'UCHL1 avec une IC50 de 0,23 μM. 6RK73 ne montre presque aucune inhibition de UCHL3 (IC50 = 236 μM). 6RK73 inhibe spécifiquement l'activité UCHL1 dans le cancer du sein.
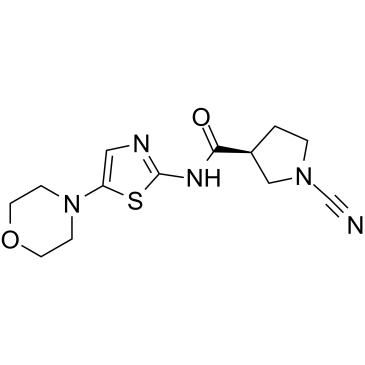
-
GC46733
7,12-Dimethylbenz[a]anthracene
Le 7,12-diméthylbenz[a]anthracène a une activité cancérigène en tant qu'hydrocarbure aromatique polycyclique (HAP). Le 7,12-diméthylbenz[a]anthracène est utilisé pour induire la formation de tumeurs chez divers modèles de rongeurs.
![7,12-Dimethylbenz[a]anthracene Chemical Structure 7,12-Dimethylbenz[a]anthracene Chemical Structure](/media/struct/GC4/GC46733.png)
-
GC33577
7-Aminoactinomycin D (7-AAD)
La 7-aminoactinomycine D (7-AAD) (7-AAD), une coloration fluorescente de l'ADN, est un puissant inhibiteur de l'ARN polymérase.
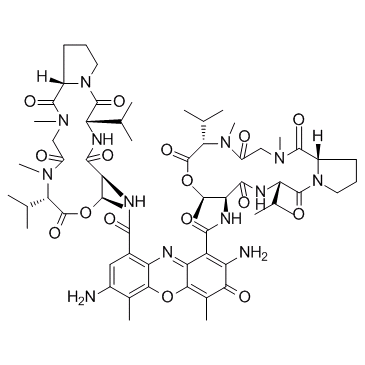
-
GC65408
7-Deaza-2'-deoxy-7-iodoadenosine
7-Deaza-'-désoxy-7-iodoadénosine est un oligonucléotide modifié contenant de la 7-Déazaadénine.
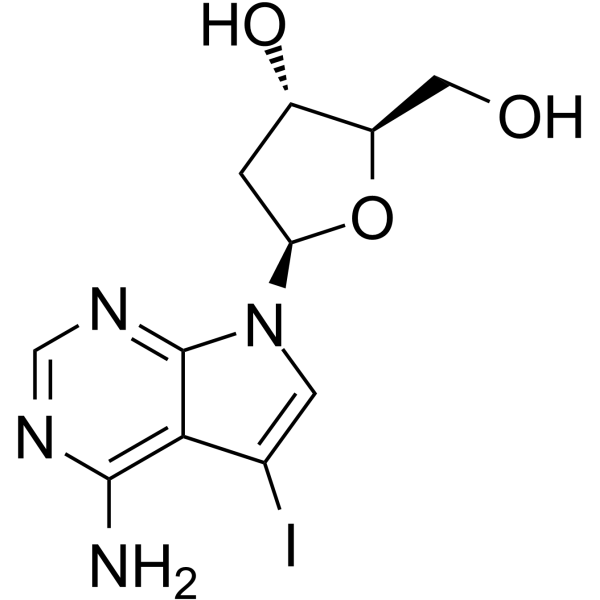
-
GC66060
7-Iodo-2',3'-dideoxy-7-deaza-guanosine
7-Iodo-2',3'-didésoxy-7-déaza-guanosine est un didésoxynucléoside qui peut être utilisé dans la synthèse d'ADN et les réactions de séquenÇage.
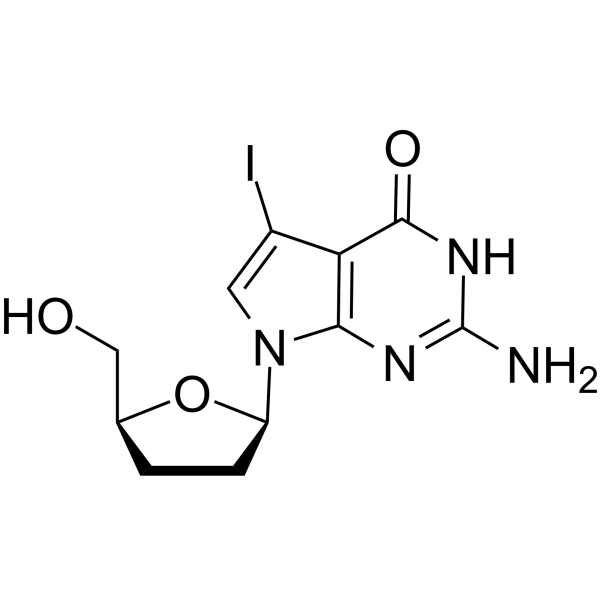
-
GC65675
7-Iodo-7-deaza-2'-deoxyguanosine
7-Iodo-7-deaza-2'-désoxyguanosine (7-Deaza-7-Iodo-2'-deoxyguanosine) est un dérivé de désoxyguanosine qui peut être utilisé dans la synthèse d'ADN et les réactions de séquenÇage.
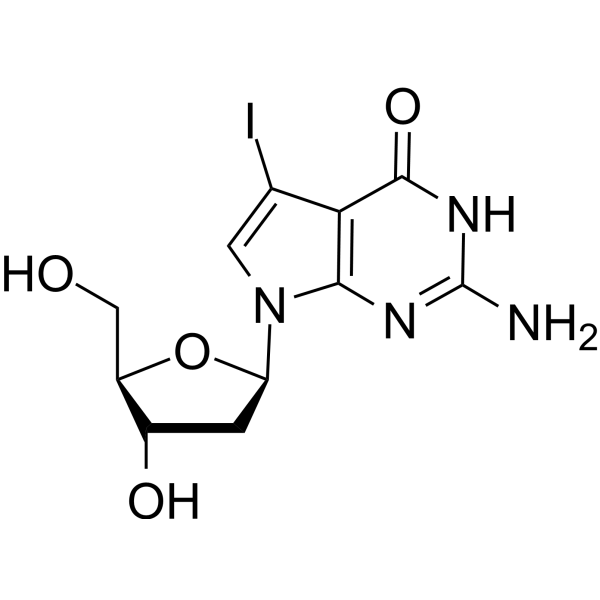
-
GC30531
7-Methylguanosine
La 7-méthylguanosine est un nouvel inhibiteur de la nucléotidase cNIIIB avec une valeur IC50 de 87,8 ± 7,5 μM.
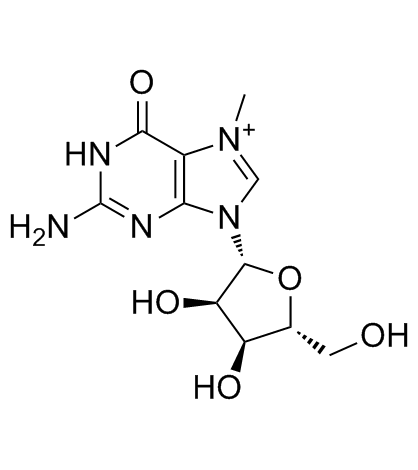
-
GC49561
7-Methylguanosine-d3
An internal standard for the quantification of 7-methylguanosine
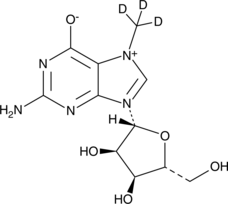
-
GC66058
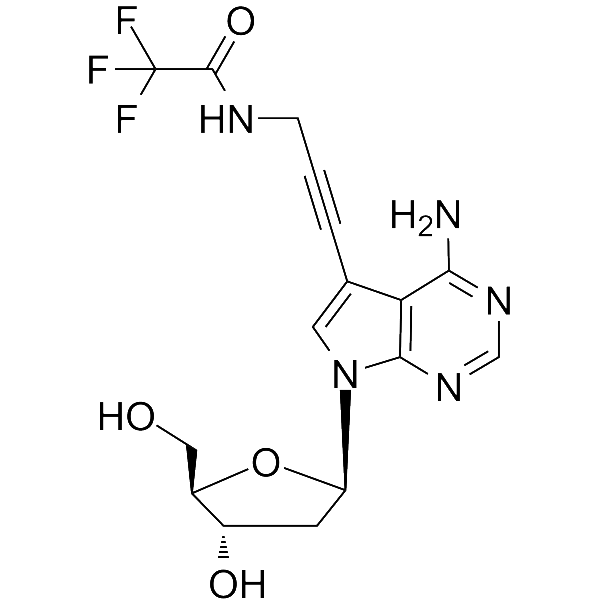
7-TFA-ap-7-Deaza-dA
Le 7-TFA-ap-7-Deaza-dA est un nucléoside modifié. Le 7-TFA-ap-7-Deaza-dA peut être utilisé dans la synthèse d'acide désoxyribonucléique ou d'acide nucléique.
-
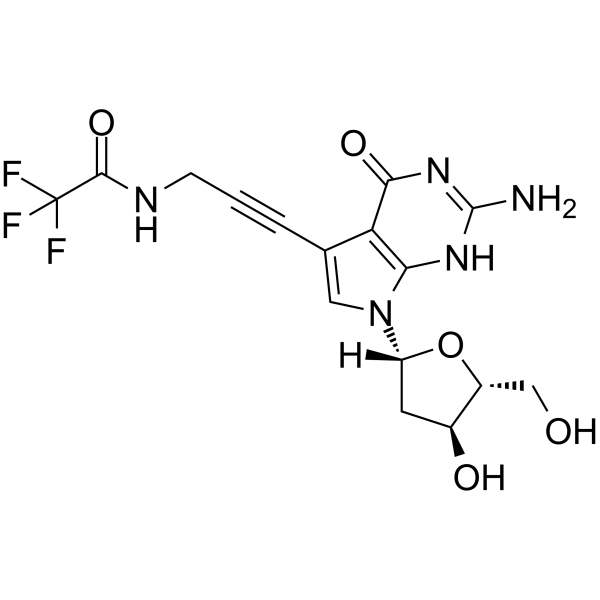
GC65525
7-TFA-ap-7-Deaza-dG
Le 5'-O-TBDMS-dG est un nucléoside modifié.
-
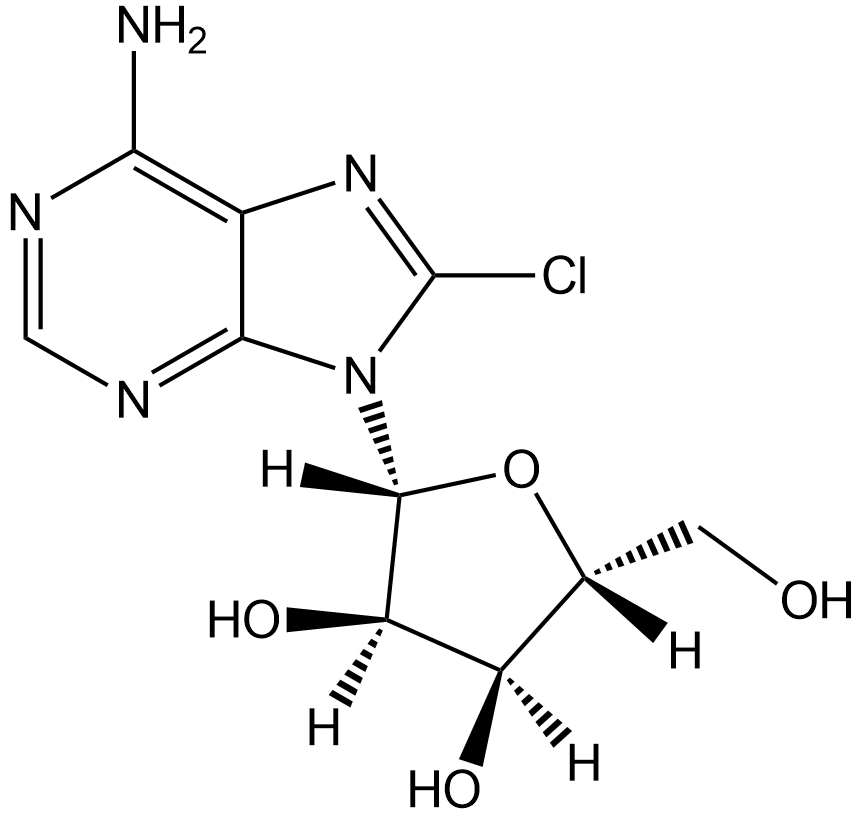
GC12777
8-Chloroadenosine
Nucleoside analog, inhibits RNA synthesis
-
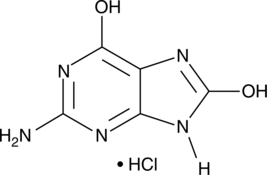
GC42627
8-Hydroxyguanine (hydrochloride)
8-Hydroxyguanine is produced by oxidative degradation of DNA by hydroxyl radical.
-
GC18426
8-Nitroguanine
8-Nitroguanine is a nitrative guanine derivative formed by oxidative damage to the guanine base in DNA by reactive nitrogen species (RNS) during inflammation and in vitro by reaction of DNA with peroxynitrite and other RNS reagents.
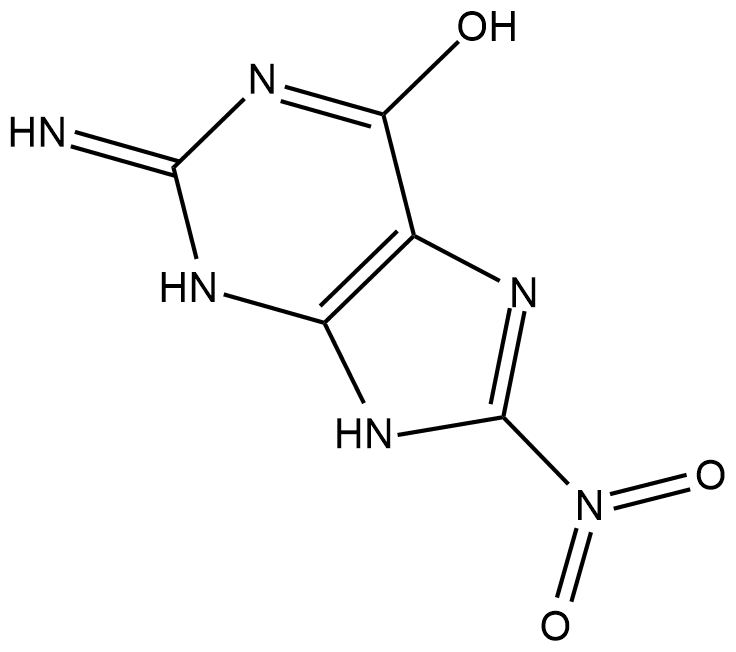
-
GC41643
9(Z),11(E),13(E)-Octadecatrienoic Acid
9(Z),11(E),13(E)-Octadecatrienoic Acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.

-
GC11903
9-amino Camptothecin
Le 9-amino Camptothecin (9-amino-20(S)-camptothecin) est un inhibiteur de la topoisomérase I avec une puissante activité anticancéreuse.
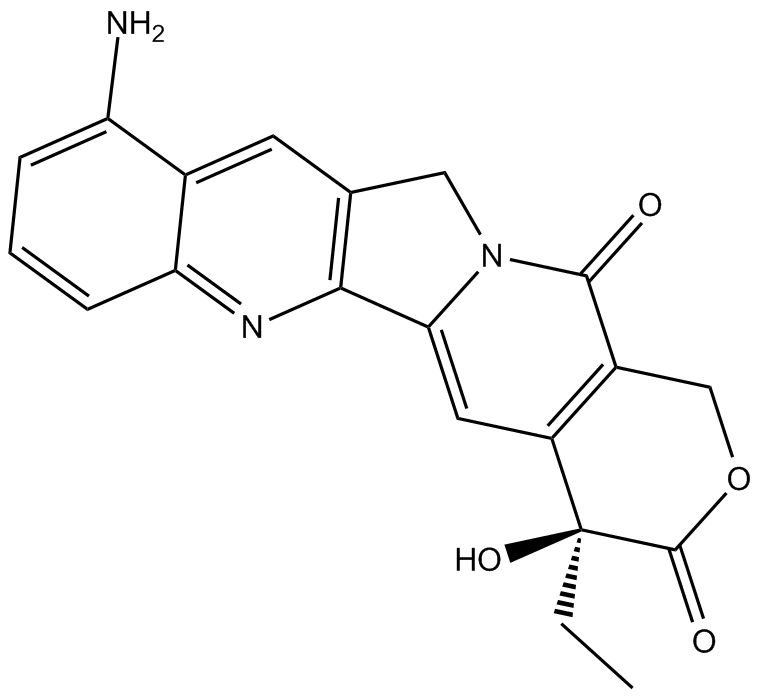
-
GC45367
9-Hydroxyellipticine (hydrochloride)
La 9-hydroxyellipticine (chlorhydrate) est un inhibiteur de Topo II et RyR.
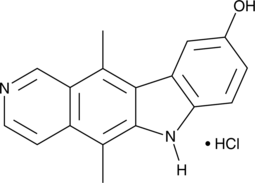
-
GN10035
9-Methoxycamptothecin
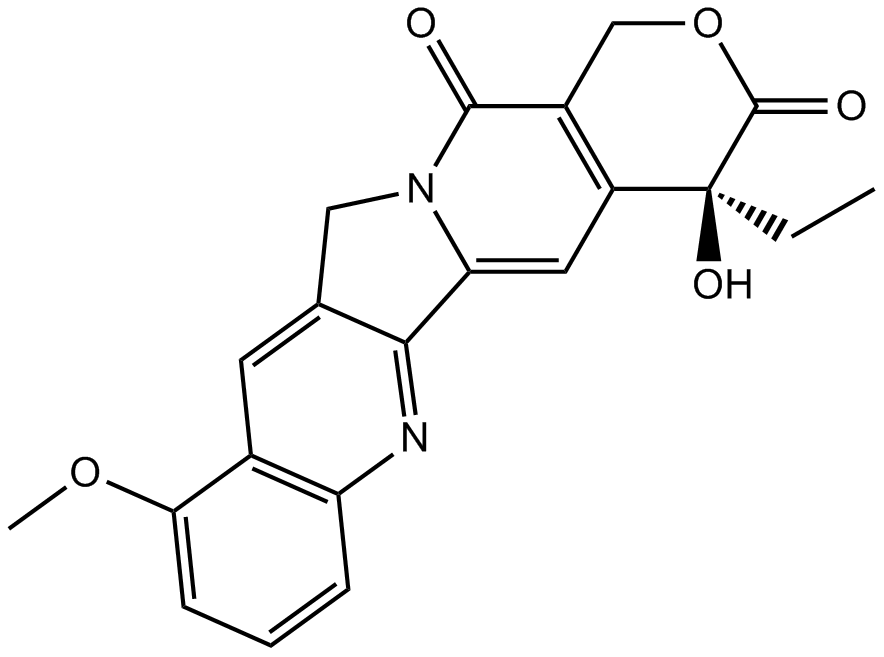
-
GC48840
9-Methoxyellipticine
An alkaloid with anticancer activity
-
GC60037
A-3 hydrochloride
Le chlorhydrate d'A-3 est un antagoniste non sélectif puissant, perméable aux cellules, réversible et compétitif pour l'ATP de diverses kinases.
-
GC13805
Abacavir
-
GC63728
Abemaciclib metabolite M18 hydrochloride
Le chlorhydrate du métabolite M18 (LSN3106729) d'Abemaciclib, le métabolite de l'Abemaciclib, est un inhibiteur de CDK avec une activité antitumorale. Le chlorhydrate du métabolite M18 de l'abémaciclib et un ligand CRBN ont été utilisés pour concevoir le dégradeur PROTAC CDK4/6.
-
GC38749
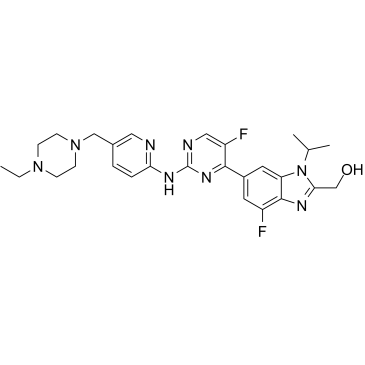
Abemaciclib metabolite M20
Le métabolite M20 de l'abémaciclib (LSN3106726), le métabolite actif de l'abémaciclib, est un inhibiteur sélectif de CDK4/6 pour le traitement du cancer.
-
GC35218
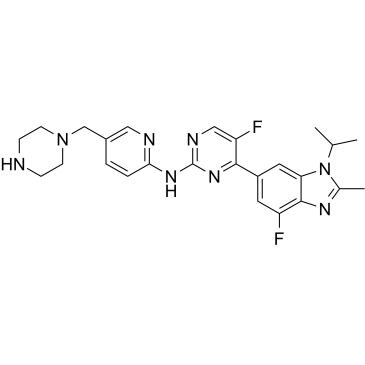
Abemaciclib Metabolites M2
Abemaciclib Metabolites M2 (LSN2839567) est un métabolite de l'abemaciclib, agit comme un puissant inhibiteur de CDK4 et CDK6, avec des IC50 de 1,2 et 1,3 nM, respectivement. Activité anticancéreuse.
-
GC64280
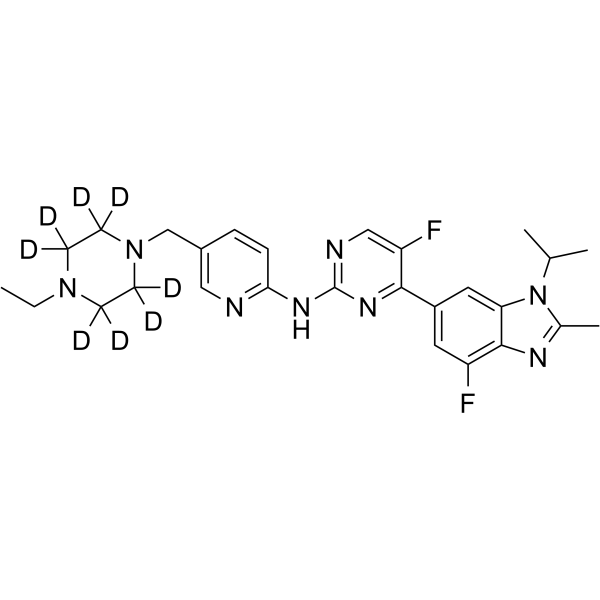
Abemaciclib-d8
Abemaciclib-d8 (LY2835219-d8) est le deutérium marqué Abemaciclib. Abemaciclib (LY2835219) est un inhibiteur sélectif de CDK4/6 avec des valeurs IC50 de 2 nM et 10 nM pour CDK4 et CDK6, respectivement.
-
GA20605
Ac-Lys-AMC
L'Ac-Lys-AMC (Hexanamide), également appelé MAL, est un substrat fluorescent pour les HDAC d'histone désacétylase.

-
GC61763
Ac-rC Phosphoramidite
Le phosphoramidite Ac-rC est utilisé pour la modification du phosphorodithioate d'oligoribonucléotide (ARN PS2) .

-
GC34090
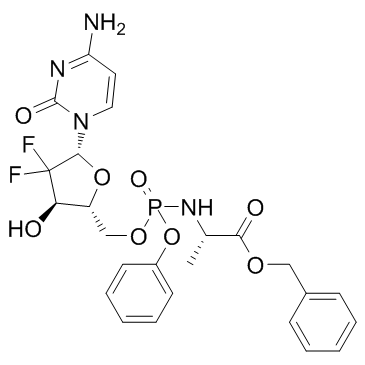
Acelarin (NUC-1031)
Acelarin (NUC-1031) (NUC-1031) est une transformation ProTide et une amélioration de l'analogue nucléoside largement utilisé, la gemcitabine.
-
GC35238
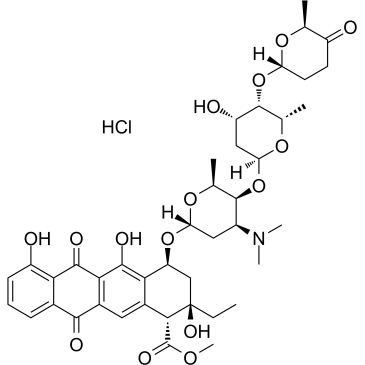
Aclacinomycin A hydrochloride
Chlorhydrate d'aclacinomycine A (chlorhydrate d'aclarubicine), une molécule fluorescente et le premier inhibiteur non peptidique décrit présentant une spécificité discrète pour l'activité CTRL (chymotrypsine-like) du protéasome 20S. Le chlorhydrate d'aclacinomycine A est également un double inhibiteur de la topoisomérase I et II. Un agent chimiothérapeutique anthracycline efficace pour les cancers hématologiques et les tumeurs solides.
-
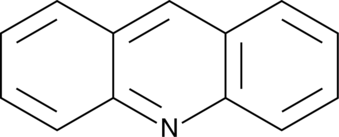
GC49804
Acridine
An azaarene
-
GC16866
Actinomycin D
Un bloqueur de transcription interagissant avec l'ADN ayant une activité anti-cancer.

-
GC35247
ACX-362E
ACX-362E (ACX-362E) est un premier inhibiteur de l'ADN polymérase IIIC (pol IIIC) actif par voie orale, avec un Ki de 0,325 μM pour l'ADN pol IIIC de C.
-
GC34458
ACY-1083
ACY-1083 est un inhibiteur sélectif de HDAC6 pénétrant dans le cerveau avec une IC50 de 3 nM et est 260 fois plus sélectif pour HDAC6 que toutes les autres classes d'isoformes HDAC. ACY-1083 inverse efficacement la neuropathie périphérique induite par la chimiothérapie.
-
GC10417
ACY-241
ACY-241 (ACY241) est un inhibiteur HDAC6 puissant, actif par voie orale et hautement sélectif de deuxième génération avec une IC50 de 2,6 nM (IC50 de 35 nM, 45 nM, 46 nM et 137 nM pour HDAC1, HDAC2, HDAC3 et HDAC8, respectivement ). ACY-241 a des effets anticancéreux.
-
GC19020
ACY-738
ACY-738 est un inhibiteur HDAC6 puissant, sélectif et biodisponible par voie orale, avec une IC50 de 1,7 nM ; ACY-738 inhibe également HDAC1, HDAC2 et HDAC3, avec des IC50 de 94, 128 et 218 nM.



