DNA/RNA Synthesis
RNA synthesis, which is also called DNA transcription, is a highly selective process. Transcription by RNA polymerase II extends beyond RNA synthesis, towards a more active role in mRNA maturation, surveillance and export to the cytoplasm.
Single-strand breaks are repaired by DNA ligase using the complementary strand of the double helix as a template, with DNA ligase creating the final phosphodiester bond to fully repair the DNA.DNA ligases discriminate against substrates containing RNA strands or mismatched base pairs at positions near the ends of the nickedDNA. Bleomycin (BLM) exerts its genotoxicity by generating free radicals, whichattack C-4′ in the deoxyribose backbone of DNA, leading to opening of the ribose ring and strand breakage; it is an S-independentradiomimetic agent that causes double-strand breaks in DNA.
First strand cDNA is synthesized using random hexamer primers and M-MuLV Reverse Transcriptase (RNase H). Second strand cDNA synthesis is subsequently performed using DNA Polymerase I and RNase H. The remaining overhangs are converted into blunt ends using exonuclease/polymerase activity. After adenylation of the 3′ ends of DNA fragments, NEBNext Adaptor with hairpin loop structure is ligated to prepare the samples for hybridization. Cell cycle and DNA replication are the top two pathways regulated by BET bromodomain inhibition. Cycloheximide blocks the translation of mRNA to protein.
Targets for DNA/RNA Synthesis
Products for DNA/RNA Synthesis
- Cat.No. Nom du produit Informations
-
GC38000
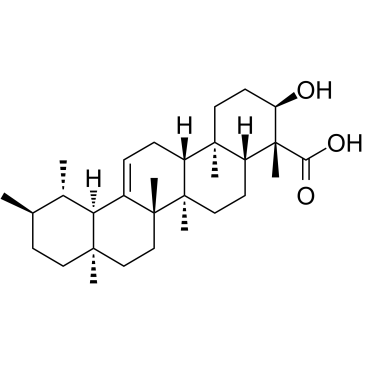
β-Boswellic acid
β ; - L'acide boswellique est isolé de la résine de gomme de Boswellia serrate.β ; - L'acide boswellique est un inhibiteur de type non réducteur de la formation de produit de 5-lipoxygénase (5-LO) qui interagit directement avec le 5-LO ou bloque sa translocation . β-L'acide boswellique inhibe la synthèse d'ADN, d'ARN et de protéines dans les cellules de leucémie humaine HL-60.
-
GC10867
(+)-Aphidicolin
L'aphidicolin ((+)-Aphidicolin), un inhibiteur réversible de la réplication de l'ADN nucléaire eucaryote, peut bloquer le cycle cellulaire à la phase pré-S[1].
-
GC48635
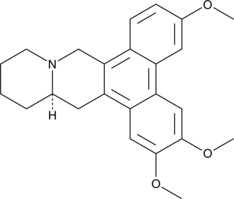
(-)-Cryptopleurine
An alkaloid with diverse biological activities
-
GC41695
(6R,S)-5,6,7,8-Tetrahydrofolic Acid (hydrochloride)
(6R,S)-5,6,7,8-Tetrahydrofolic acid (THFA), the reduced form of folic acid, serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
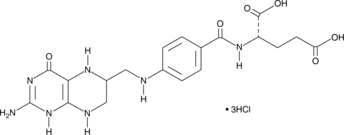
-
GC41088
(6S)-Tetrahydrofolic Acid
(6S)-Tetrahydrofolic acid is a diastereomer of tetrahydrofolic acid, a reduced form of folic acid that serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
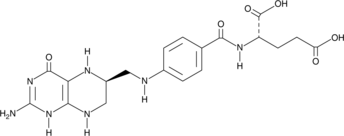
-
GC41633
(R)-Prunasin
(R)-Prunasin est un inhibiteur de l'ADN polymérase β.
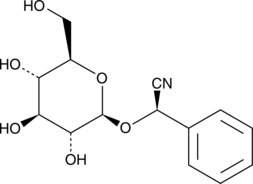
-
GC41859
1,5,7-Triazabicyclo[4.4.0]dec-5-ene
1,5,7-Triazabicyclo[4.4.0]dec-5-ene is a bicyclic guanidine base that can be used as a catalyst for a variety of base-mediated organic transformations, including Michael additions, Wittig reactions, Henry reactions, and transesterification reactions.
![1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure 1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure](/media/struct/GC4/GC41859.png)
-
GC48909
1-Hydroxyanthraquinone
La 1-hydroxyanthraquinone, un composé naturel À activité orale provenant de certaines plantes comme Tabebuia avellanedae, présente un effet cancérigène.

-
GC49872
10-Formyltetrahydrofolate (sodium salt) (technical grade)
Le 10-formyltétrahydrofolate (sel de sodium) (qualité technique) est une forme d'acide tétrahydrofolique qui agit comme donneur de groupes formyle dans l'anabolisme.
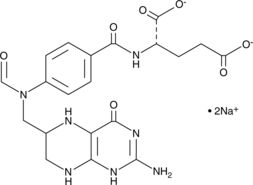
-
GC63796
116-9e
116-9e (MAL2-11B) est un inhibiteur de l'ADNJA1 co-chaperon Hsp70.
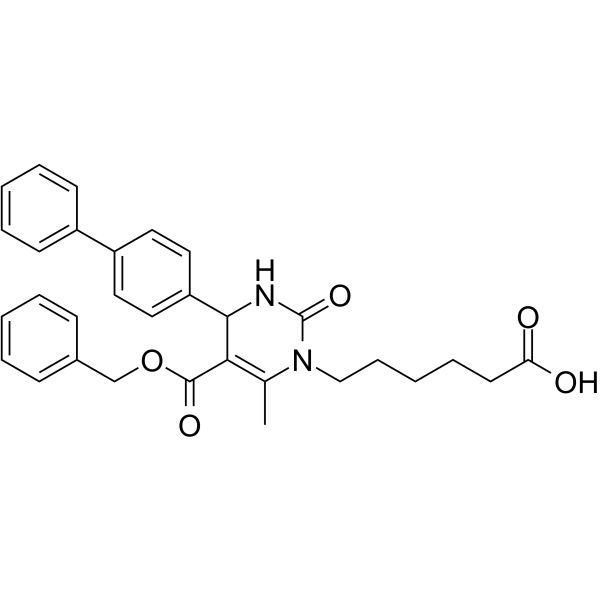
-
GC49759
13C17-Mycophenolic Acid
An internal standard for the quantification of mycophenolic acid
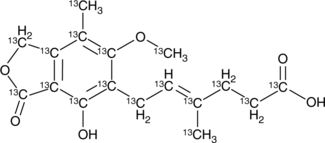
-
GC46474
18-Deoxyherboxidiene
Le 18-désoxyherboxidiène (RQN-18690A) est un puissant inhibiteur de l'angiogenèse.

-
GC65489
2'-F-Bz-dC Phosphoramidite
&2#39;-F-Bz-dC Le phosphoramidite peut être utilisé dans la synthèse d'oligoribonucléotides.
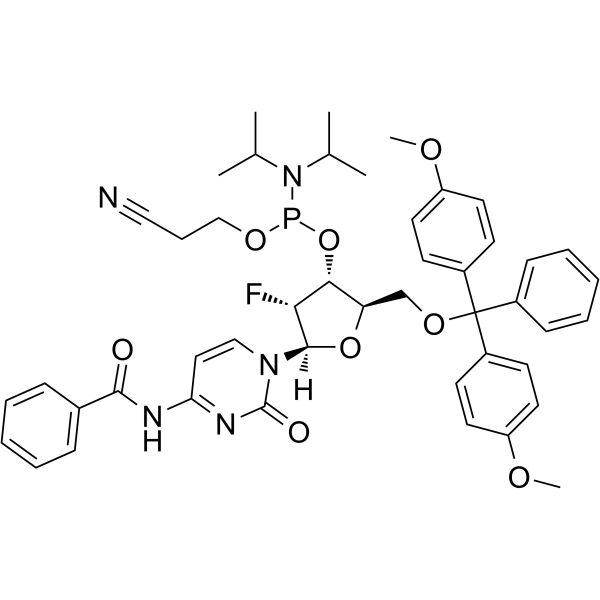
-
GC52183
2'-O-Methyl-5-methyluridine
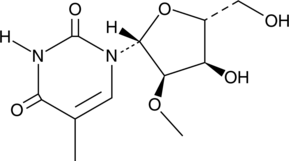
-
GC66713
2'-O-MOE-5-Me-C(Bz)
2'-O-MOE-5-Me-C (Bz) est un nucléotide pour la synthèse stéréosélective d'alkylphosphonates de nucléoside.
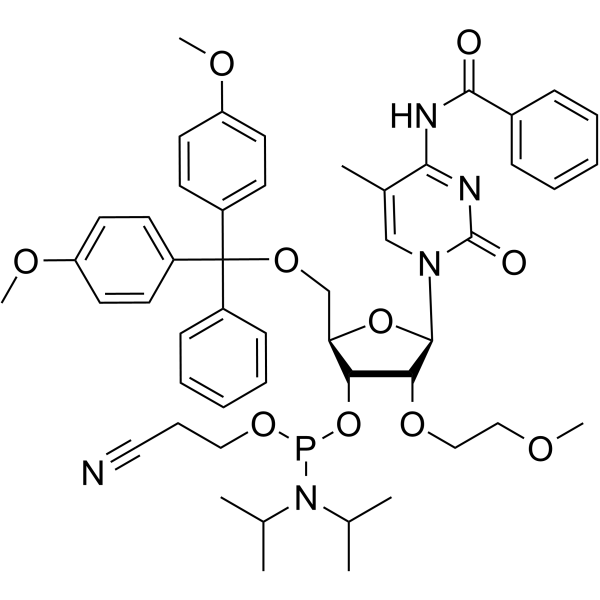
-
GC66657
2'-O-MOE-5-Me-rC
2'-O-MOE-5-Me-rC est un composé actif. 2'-O-MOE-5-Me-rC peut être utilisé pour la synthèse d'oligonucléotides.
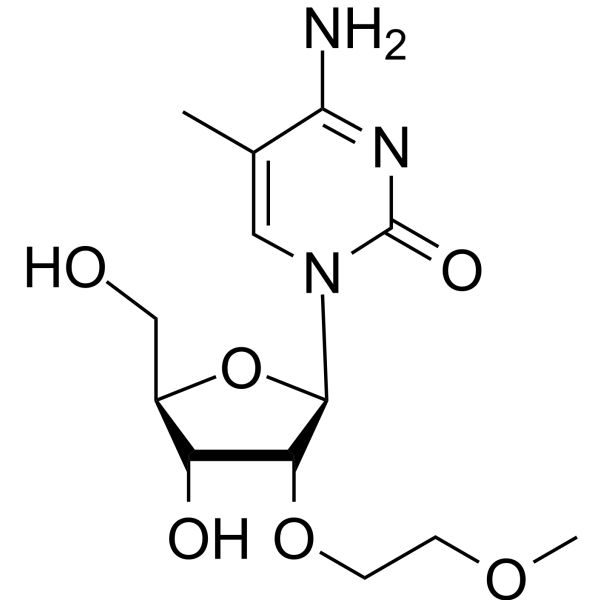
-
GC66091
2'-O-MOE-5-Me-rU
2'-O-MOE-5-Me-rU est un composé actif. 2'-O-MOE-5-Me-rU peut être utilisé pour la synthèse d'oligonucléotides.
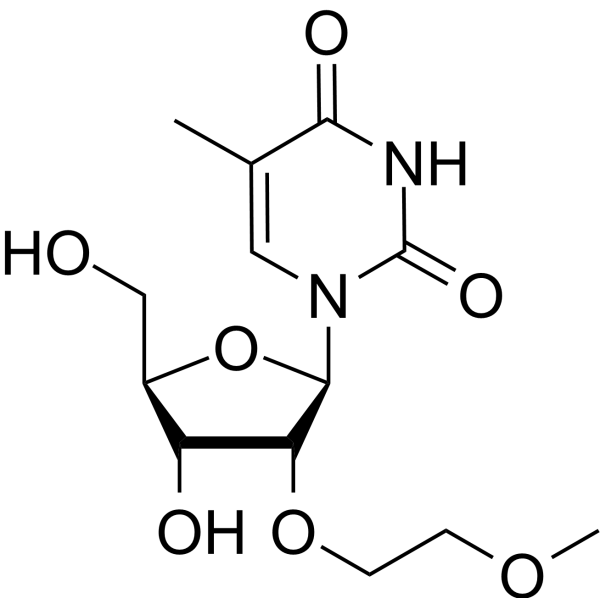
-
GC66656
2'-O-MOE-rC
2'-O-MOE-rC est un nucléoside modifié par 2'-O-MOE. 2'-O-MOE-rC peut être utilisé pour la synthèse d'ADN.
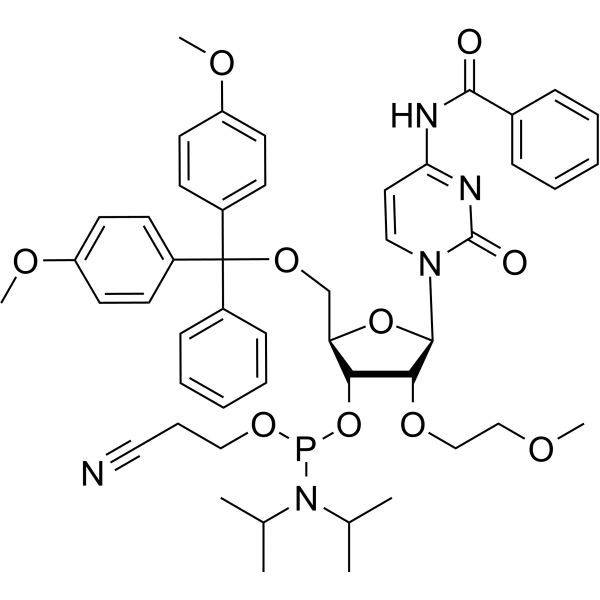
-
GC66652
2'-O-MOE-U
2'-O-MOE-U est un phosphoramidite, peut être utilisé pour la synthèse d'oligonucléotides.

-
GC62530
2’-O-Me-C(Bz) Phosphoramidite
&2rsquo;-O-Me-C(Bz) Phosphoramidite est un monomère phosphoramidite modifié, qui peut être utilisé pour la synthèse d'oligonucléotides.
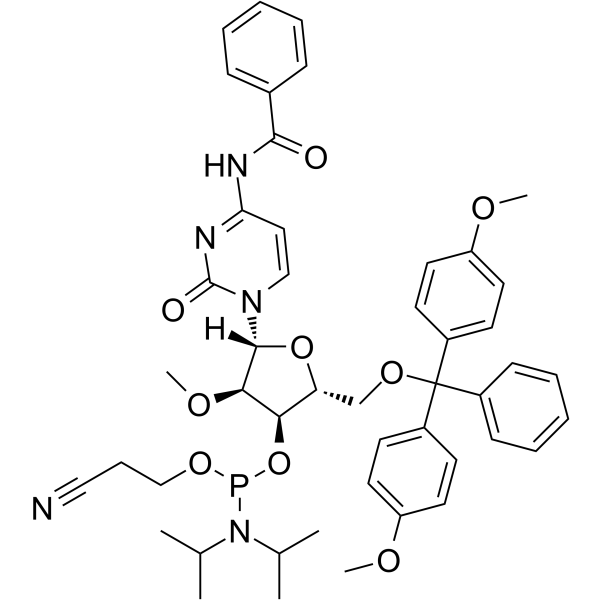
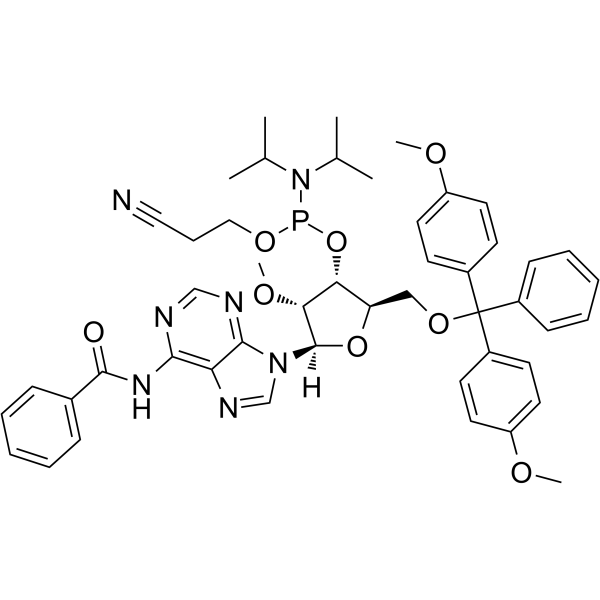
-
GC62529
2’-OMe-A(Bz) Phosphoramidite
&2rsquo;-OMe-A(Bz) Phosphoramidite est un monomère phosphoramidite modifié, qui peut être utilisé pour la synthèse d'oligonucléotides.
-
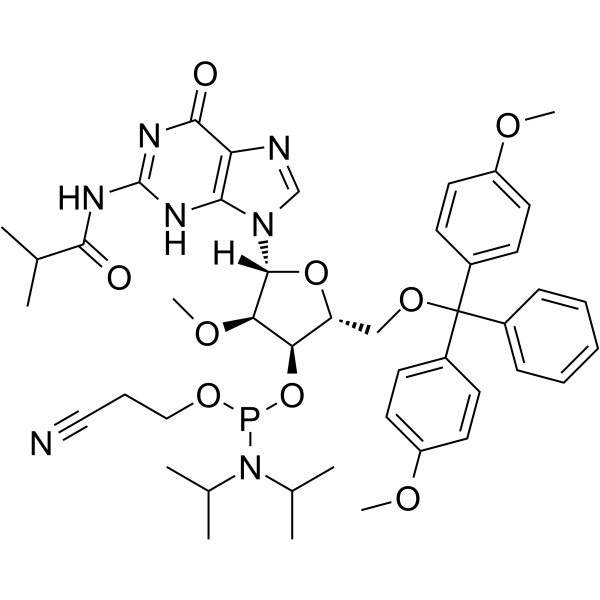
GC62531
2’-OMe-G(ibu) Phosphoramidite
&2rsquo;-OMe-G(ibu) Phosphoramidite est un monomère phosphoramidite modifié, qui peut être utilisé pour la synthèse d'oligonucléotides.
-
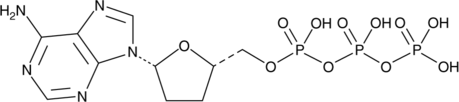
GC42061
2',3'-Dideoxyadenosine 5'-triphosphate
2',3'-Dideoxyadenosine 5'-triphosphate (2',3'-Dideoxyadenosine 5'-triphosphate), an active metabolite of 2',3'-dideoxyinosine, is a chain-elongating inhibitor of DNA polymerase.
-
GC46519
2',3'-O-Isopropylideneguanosine
A building block
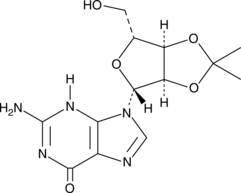
-
GC46540
2'-Deoxyadenosine-5'-triphosphate (sodium salt hydrate)
A purine nucleotide
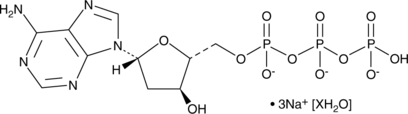
-
GC48440
2'-Deoxycytidine-5'-triphosphate (sodium salt)
2'-Deoxycytidine-5'-triphosphate (sel de sodium) (dCTP sel trisodique) est un nucléoside triphosphate qui peut être utilisé pour la synthèse d'ADN.
-
GC42151
2'-Deoxyguanosine 5'-monophosphate (sodium salt hydrate)
2'-Deoxyguanosine 5'-monophosphate (dGMP) is used as a substrate of guanylate kinases to generate dGDP, which in turn is phosphorylated to dGTP, a nucleotide precursor used in DNA synthesis.
-
GC42080
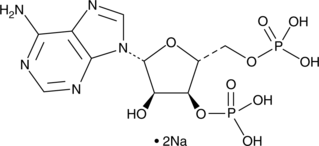
2'2'-cGAMP (sodium salt)
2'2'-cGAMP is a synthetic dinucleotide (CDN) that contains non-canonical 2'5'-phosphodiester bonds.
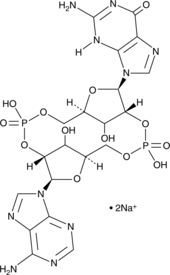
-
GC60459
2,4-D (2,4-Dichlorophenoxyacetic acid)
Le 2,4-D (acide 2,4-dichlorophénoxyacétique) (acide 2,4-D (acide 2,4-dichlorophénoxyacétique) ichlorophénoxyacétique) est un herbicide systémique sélectif pour le contrÔle des mauvaises herbes À feuilles larges.
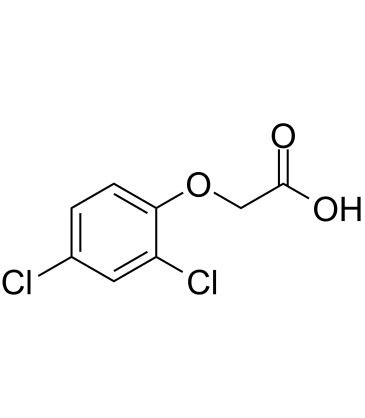
-
GC46524
2,6-Dichloropurine-9-β-D-riboside
A building block
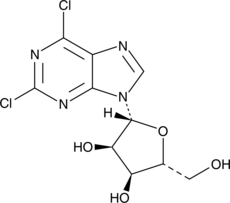
-
GC33496
2-(Methylamino)-1H-purin-6(7H)-one (N2-methylguanine)
La 2-(méthylamino)-1H-purine-6(7H)-one (N2-méthylguanine) (N2-méthylguanine) est un nucléoside modifié.
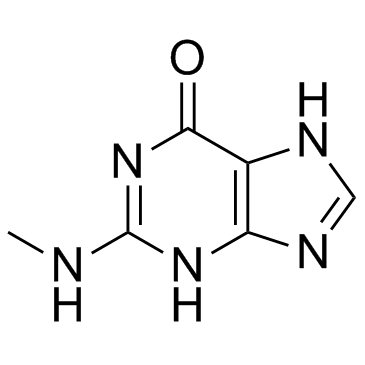
-
GC64983
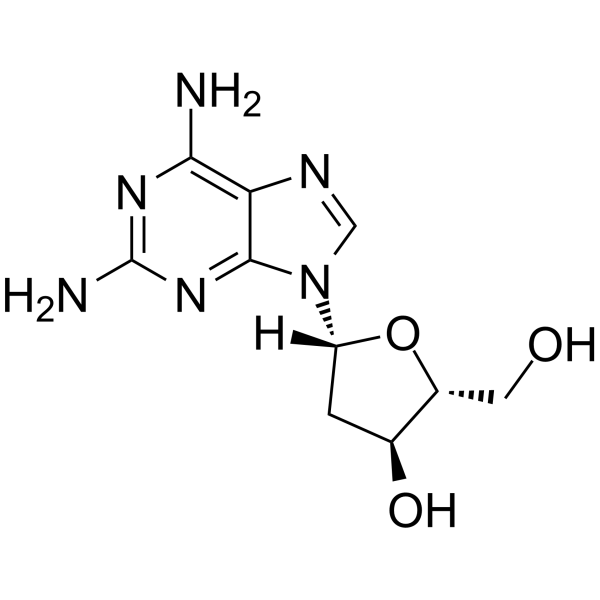
2-Amino-2'-deoxyadenosine
La 2-amino-&2#39;-désoxyadénosine est un désoxyribonucléoside utilisé pour la synthèse d'oligonucléotides.
-
GC39527
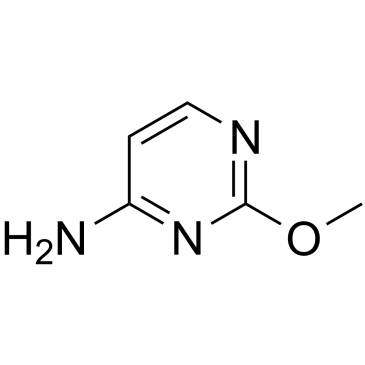
2-O-Methylcytosine
La 2-O-méthylcytosine, un analogue O-alkylé d'un adduit d'ADN, est la nucléobase endommagée.
-
GC49348
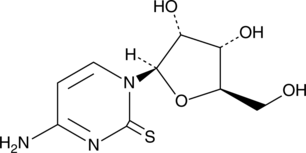
2-Thiocytidine
A modified nucleobase
-
GC42197
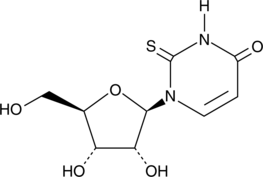
2-Thiouridine
2-Thiouridine (s2U) is a modified nucleobase found in tRNAs that is known to stabilize U:A pairs and modestly destabilize U:G wobble pairs.
-
GC40618
3',4',7-Trihydroxyisoflavone
3',4',7-Trihydroxyisoflavone, un métabolite majeur de Daidzein, est un inhibiteur compétitif de l'ATP de Cot (Tpl2/MAP3K8) et MKK4. 3',4',7-Trihydroxyisoflavone a des activités anticancéreuses, anti-angiogéniques, chimioprotectrices et antiradicalaires.
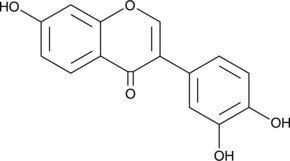
-
GC60023
3'-Deoxyuridine-5'-triphosphate
Le 3'-désoxyuridine-5'-triphosphate (3'-dUTP) est un analogue nucléotidique qui inhibe les ARN polymérases I et II dépendantes de l'ADN.
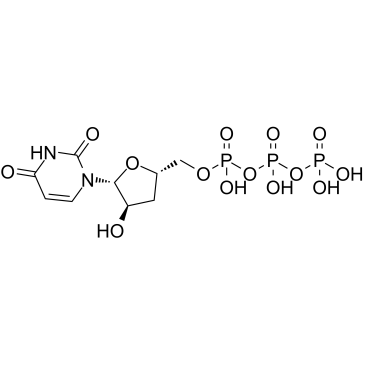
-
GC61862
3'-Deoxyuridine-5'-triphosphate trisodium
Le 3'-désoxyuridine-5'-triphosphate trisodique (3'-dUTP trisodique) est un analogue nucléotidique qui inhibe les ARN polymérases I et II dépendantes de l'ADN.
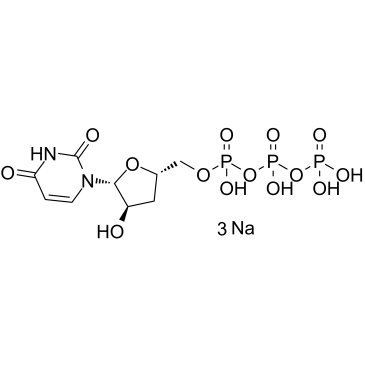
-
GC45332
3'-Dephosphocoenzyme A
An intermediate in the biosynthesis of CoA
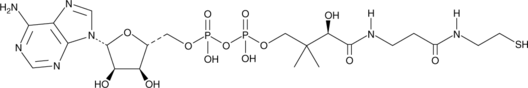
-
GC34452
3,7,4'-Trihydroxyflavone
3,7,4'-Trihydroxyflavone, isolée de Rhus javanica var.
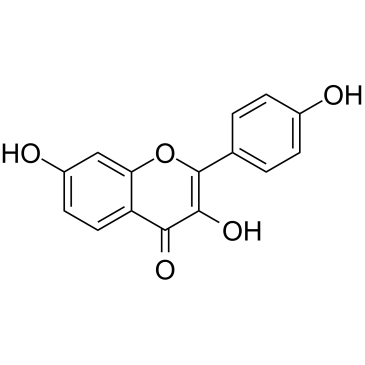
-
GC13510
3-AP
Le 3-AP (PAN-811) est un puissant inhibiteur de la sous-unité M2 de la ribonucléotide réductase (RR) et un puissant radiosensibilisateur.
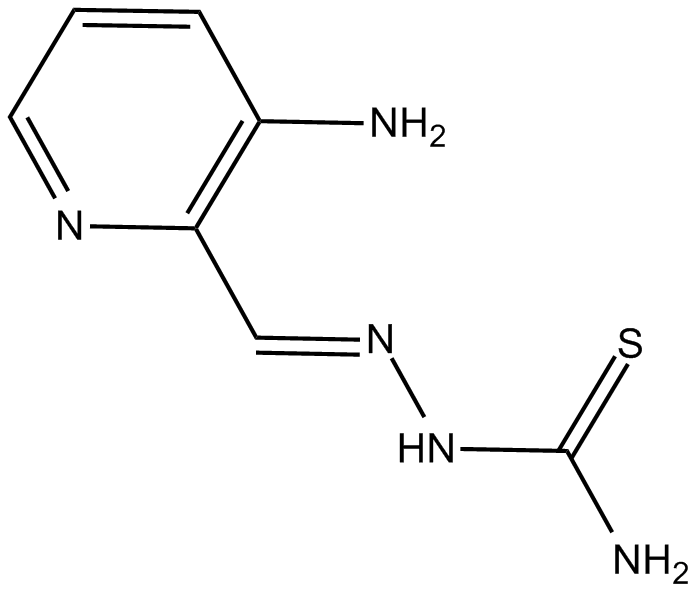
-
GC48457
3-keto Fusidic Acid
An active metabolite of fusidic acid
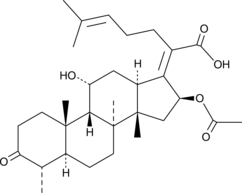
-
GC52391
306-O12B-3
An ionizable cationic lipidoid
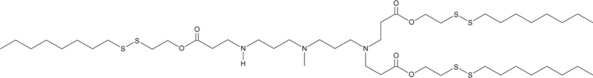
-
GC18853
4-isocyanato TEMPO
4-isocyanato TEMPO is a spin labeling reagent used to label the 2'-position in RNA.
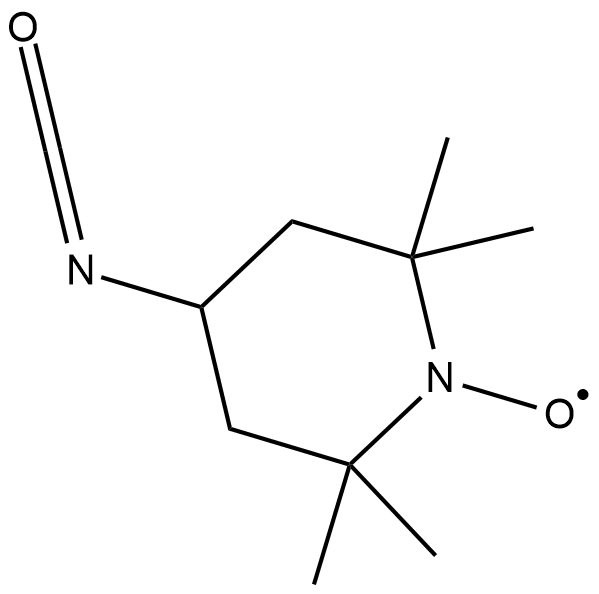
-
GC61683
4-Methoxyphenethyl alcohol
L'alcool 4-méthoxyphénéthylique, un alcool aromatique, est le principal composant de l'odeur anisée produite par A.
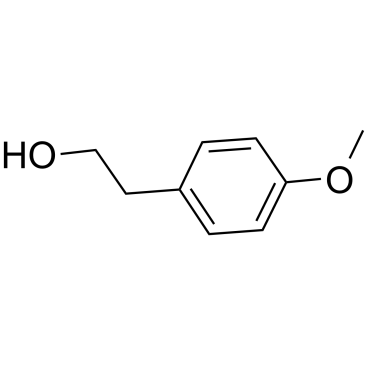
-
GC65520
5'-DMT-3'-TBDMS-ibu-rG
5'-DMT-3'-TBDMS-ibu-rG est un nucléoside modifié.

-
GC65402
5'-O-DMT-2'-O-TBDMS-Bz-rC
5'-O-DMT-&2#39;-O-TBDMS-Bz-rC est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
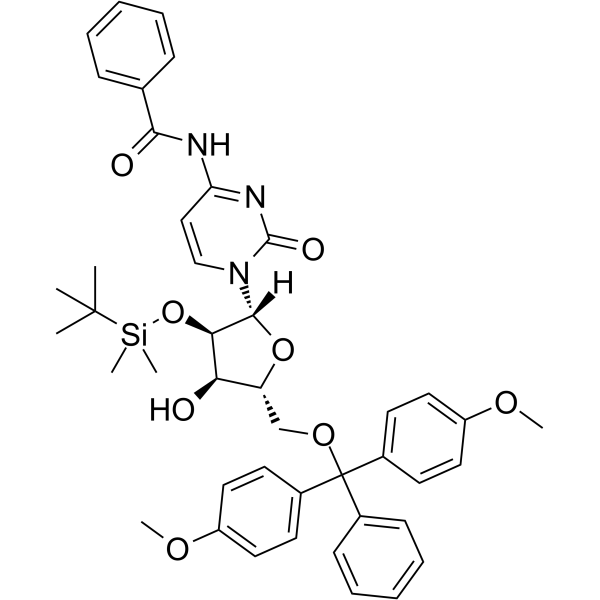
-
GC65164
5'-O-DMT-2'-O-TBDMS-rI
5'-O-DMT-&2#39;-O-TBDMS-rI est un nucléoside modifié.
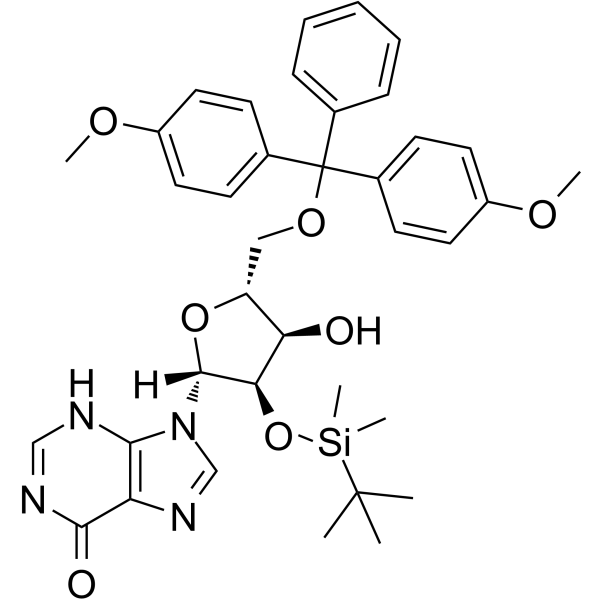
-
GC65396
5'-O-DMT-ibu-dC
5'-O-DMT-ibu-dC peut être utilisé dans la synthèse d'oligodésoxyribonucléotides.

-
GC64982
5'-O-DMT-N2-DMF-dG
Le 5'-O-DMT-2'-O-TBDMS-rI est un nucléoside modifié.
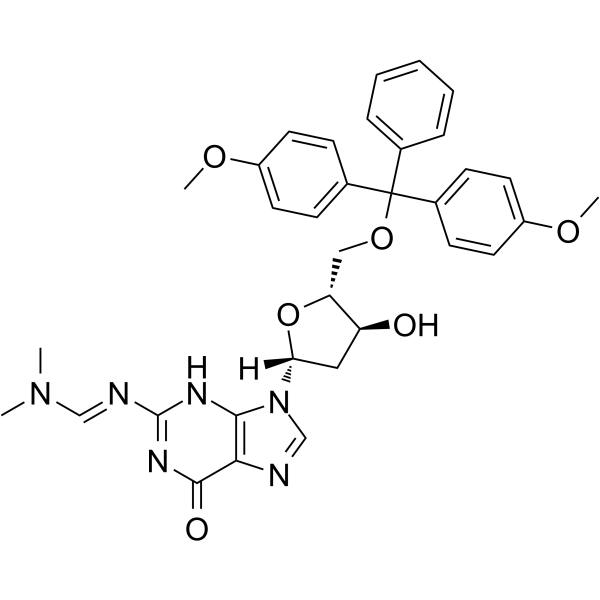
-
GC65482
5'-O-DMT-N6-ibu-dA
5'-O-DMT-N6-ibu-dA peut être utilisé dans la synthèse d'oligodésoxyribonucléotides.
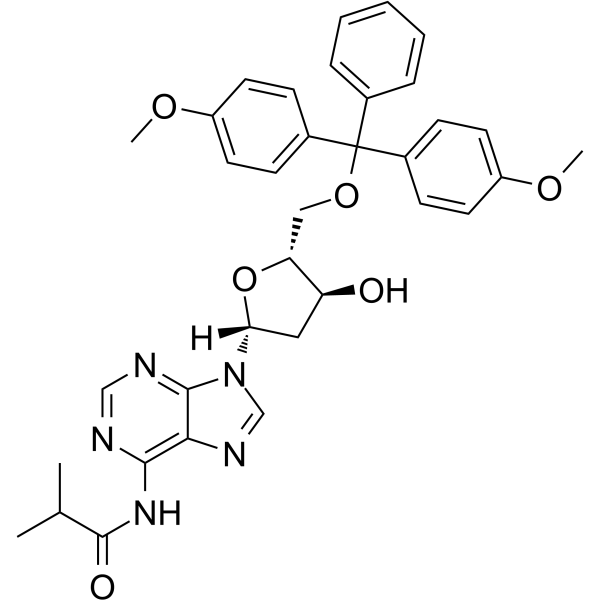
-
GC65562
5'-O-DMT-N6-Me-2'-dA
5'-O-DMT-N6-Me-&2#39;-dA est un nucléoside ayant des effets protecteurs et modificateurs.
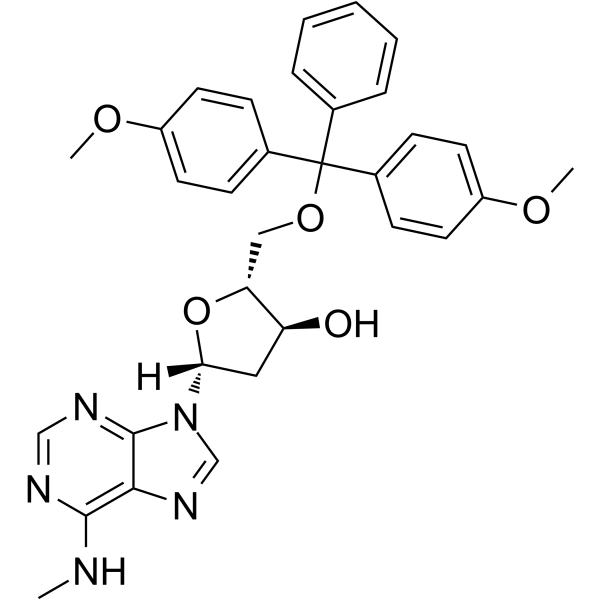
-
GC65401
5'-O-TBDMS-dU
5'-O-TBDMS-dU peut être utilisé dans la synthèse d'oligoribonucléotides.
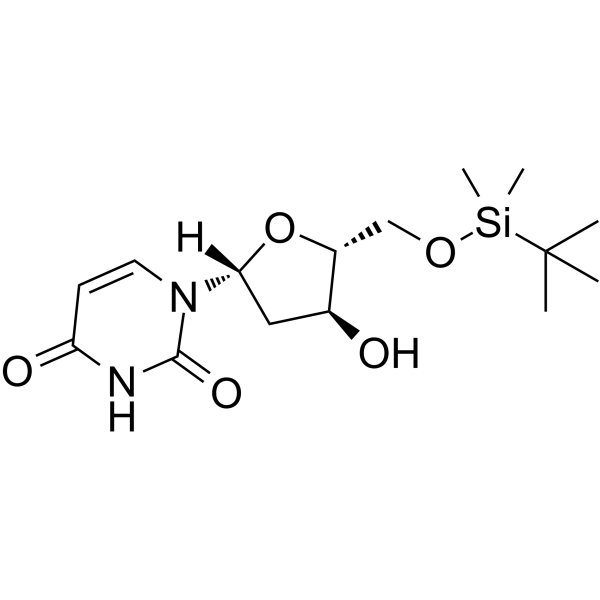
-
GC62157
5’-O-DMT-2’-O-TBDMS-Ac-rC
5’-O-DMT-&2rsquo;-O-TBDMS-Ac-rC est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
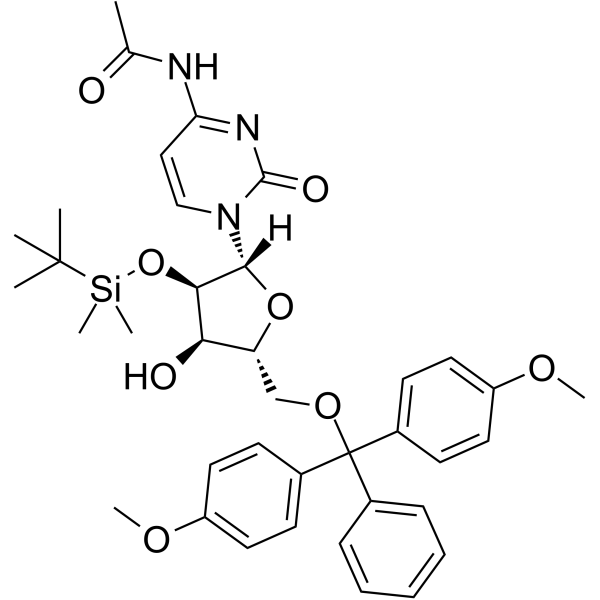
-
GC62149
5’-O-DMT-2’-TBDMS-Uridine
5’-O-DMT-&2rsquo;-TBDMS-Uridine est un désoxyribonucléoside utilisé pour la synthèse d'oligonucléotides.
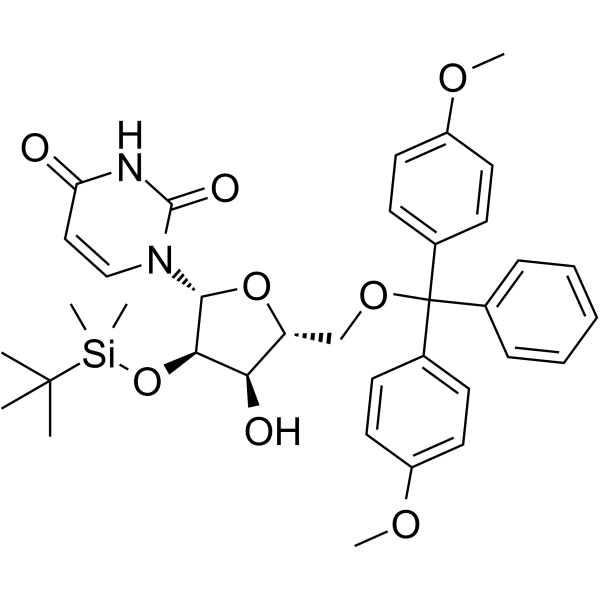
-
GC62532
5’-O-DMT-3’-O-TBDMS-Ac-rC
5’-O-DMT-3’-O-TBDMS-Ac-rC est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
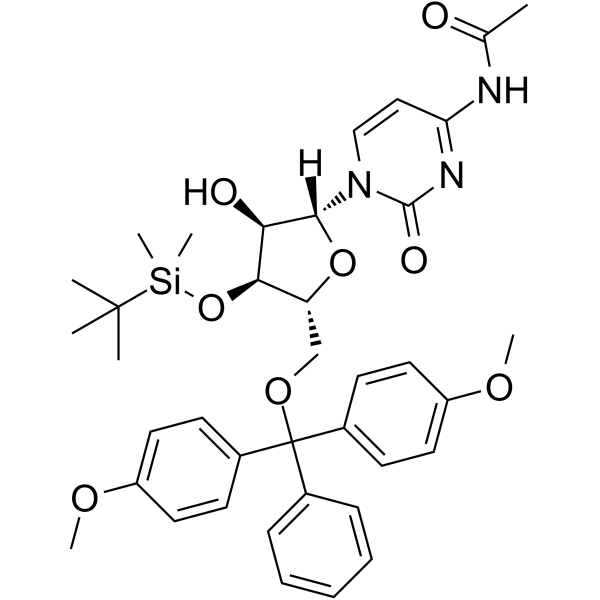
-
GC62573
5’-O-DMT-Bz-rC
Le 5'-O-DMT-Bz-Rc est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
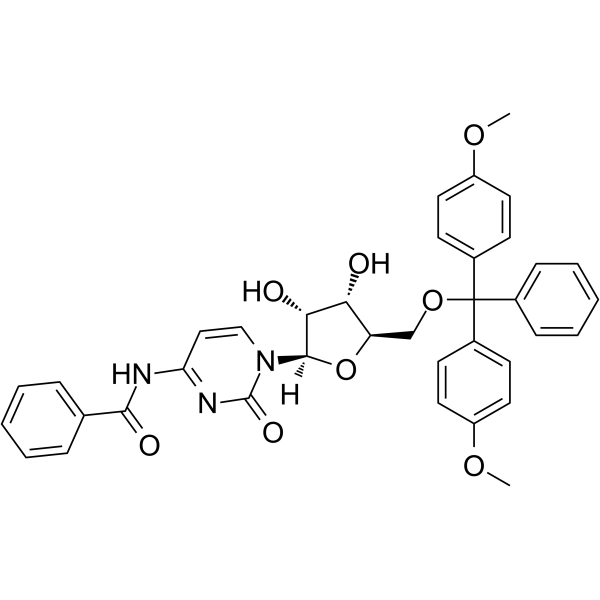
-
GC62574
5’-O-DMT-N4-Ac-2’-F-dC
5’-O-DMT-N4-Ac-&2rsquo;-F-dC est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
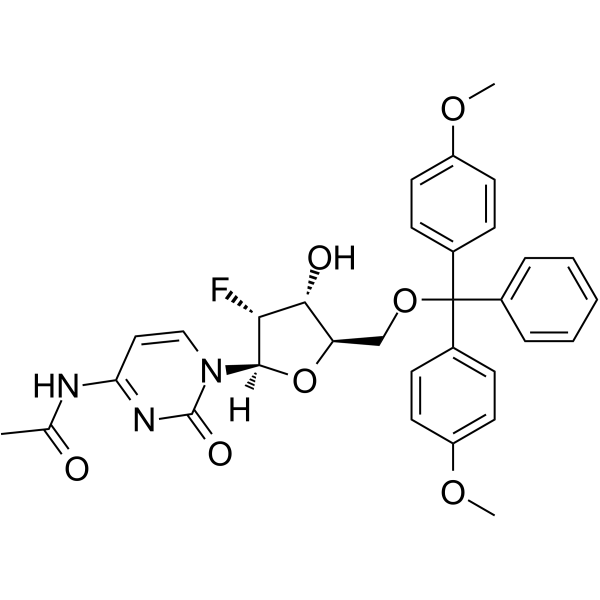
-
GC62575
5’-O-DMT-N4-Bz-2’-F-dC
5’-O-DMT-N4-Bz-&2rsquo;-F-dC est un nucléoside ayant des effets protecteurs et modificateurs.
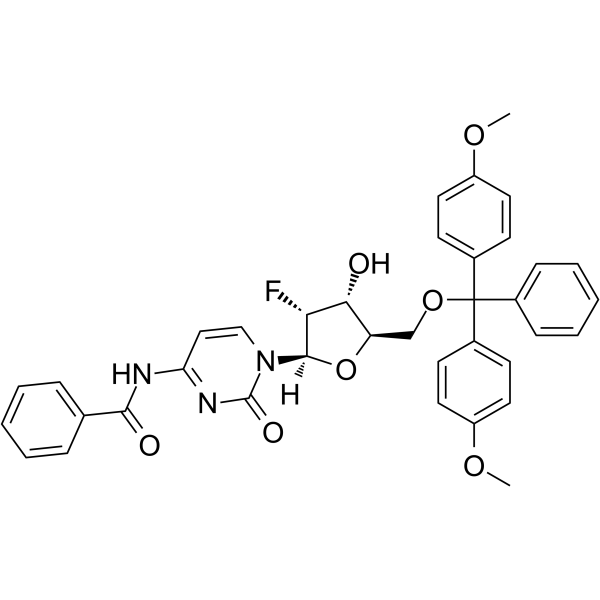
-
GC62576
5’-O-DMT-N4-Bz-5-Me-dC
5’-O-DMT-N4-Bz-5-Me-dC est un nucléoside modifié.
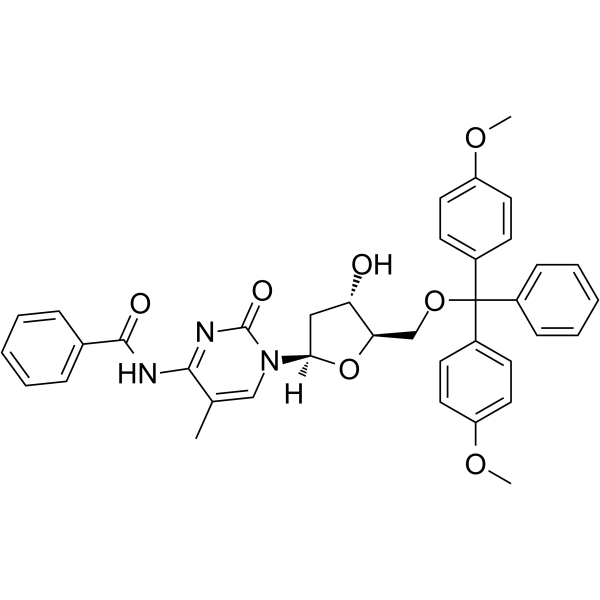
-
GC62812
5’-O-DMT-PAC-dA
5’-O-DMT-PAC-dA peut être utilisé dans la synthèse d'oligoribonucléotides.
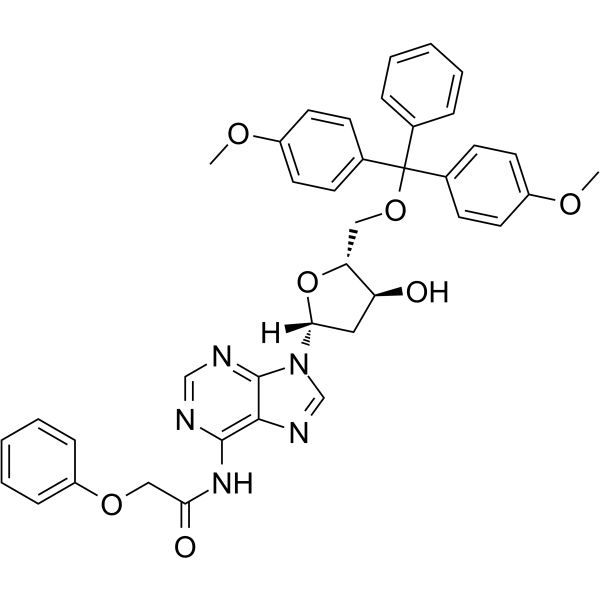
-
GC62813
5’-O-DMT-rI
Le 5'-O-DMT-Ri peut être utilisé dans la synthèse d'oligoribonucléotides.
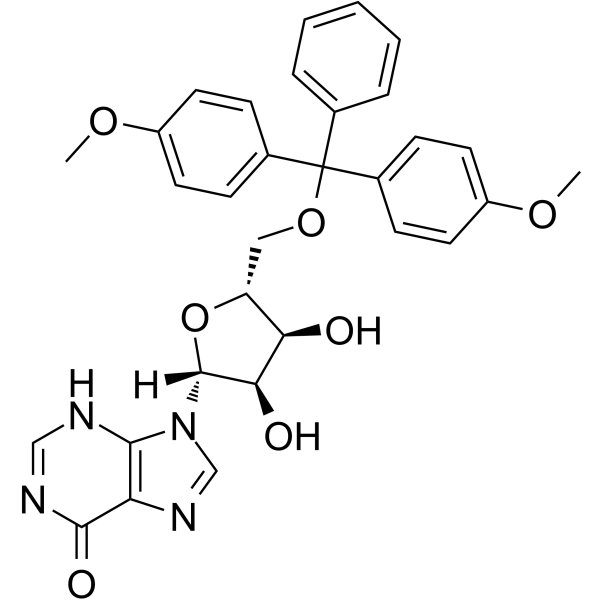
-
GC62577
5’-O-DMT-rU
5’-O-DMT-rU est un nucléoside modifié et peut être utilisé pour synthétiser l'ARN.
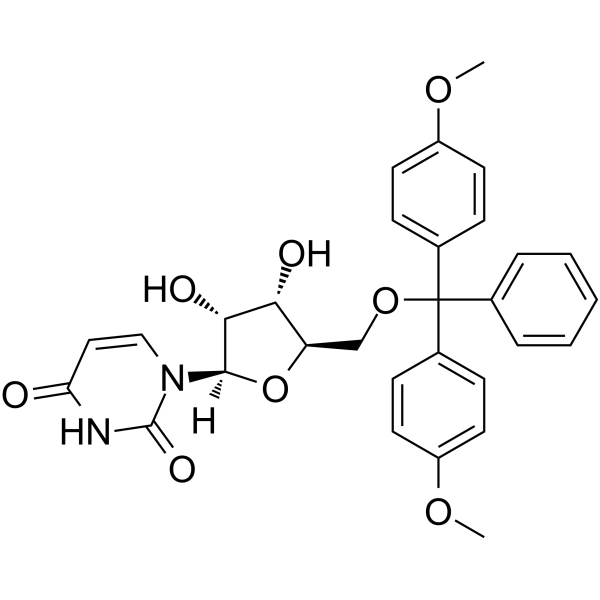
-
GC62578
5’-O-TBDMS-Bz-dA
5’-O-TBDMS-Bz-dA est un nucléoside ayant des effets protecteurs et modificateurs.
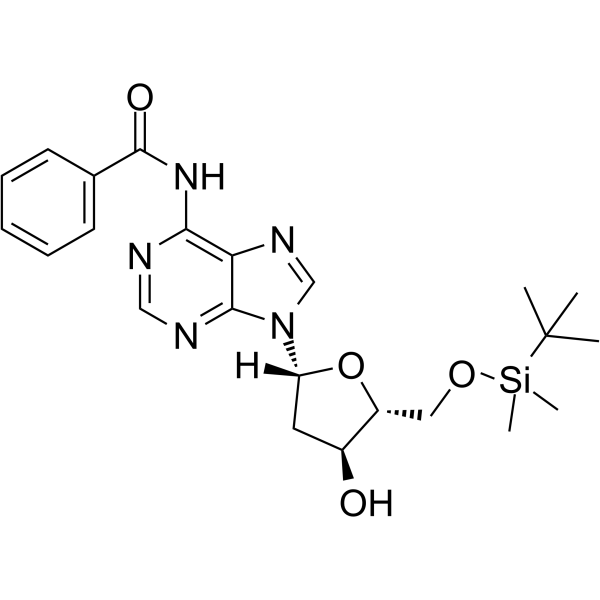
-
GC62579
5’-O-TBDMS-dA
5’-O-TBDMS-dA est un nucléoside modifié et peut être utilisé pour synthétiser de l'ADN ou de l'ARN.
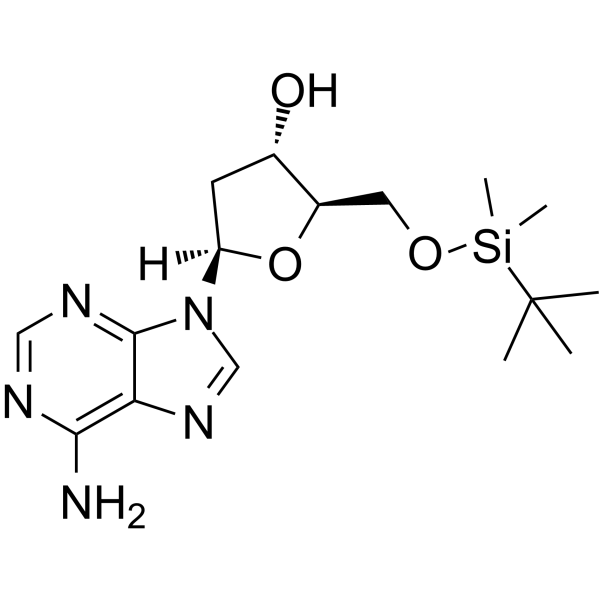
-
GC62580
5’-O-TBDMS-dG
5’-O-TBDMS-dG est un nucléoside modifié.

-
GC62581
5’-O-TBDMS-dT
5’-O-TBDMS-dT est un nucléoside ayant des effets protecteurs et modificateurs.

-
GC40641
5'-Ethynyl-2'-deoxycytidine
5'-Ethynyl-2'-deoxycytidine (EdC) is a nucleoside analog that inhibits replication of the herpes simplex virus-1 (HSV-1) KOS strain (ID50 = 0.2 μg/mL).
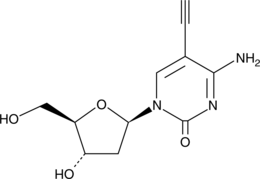
-
GC61726
5'-O-DMT-N4-Ac-dC
Le 5'-O-DMT-N4-Ac-dC (N4-Acétyl-2'-désoxy-5'-O-DMT-cytidine, composé 7), un désoxynucléoside, peut être utilisé pour synthétiser le dodécyl phosphoramidite qui est le brut matériel pour la synthèse de dod-DNA (ADN amphiphile contenant une région hydrophobe interne constituée de liaisons dodécyl phosphotriester) .
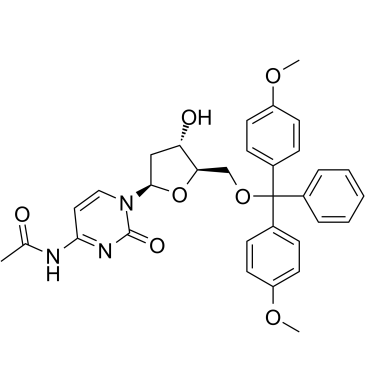
-
GC48662
5'-Thymidylic Acid (sodium salt)
5'-L'acide thymidylique (sel de sodium) est un métabolite endogène.
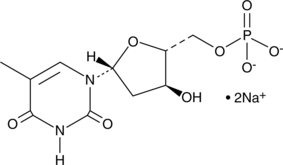
-
GC46681
5-Bromouridine
A brominated uridine analog
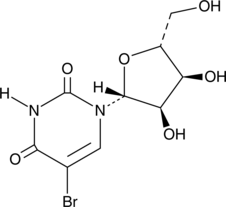
-
GC49245
5-Ethynyluridine
La 5-éthynyluridine (5-EU) est un puissant nucléoside perméable aux cellules qui peut être utilisé pour marquer l'ARN nouvellement synthétisé.
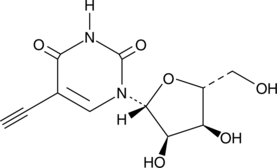
-
GC62389
5-Iminodaunorubicin
La 5-iminodaunorubicine est une anthracycline modifiée par une quinone qui conserve une activité antitumorale. La 5-iminodaunorubicine produit des ruptures de brins d'ADN dissimulées par des protéines dans les cellules cancéreuses.
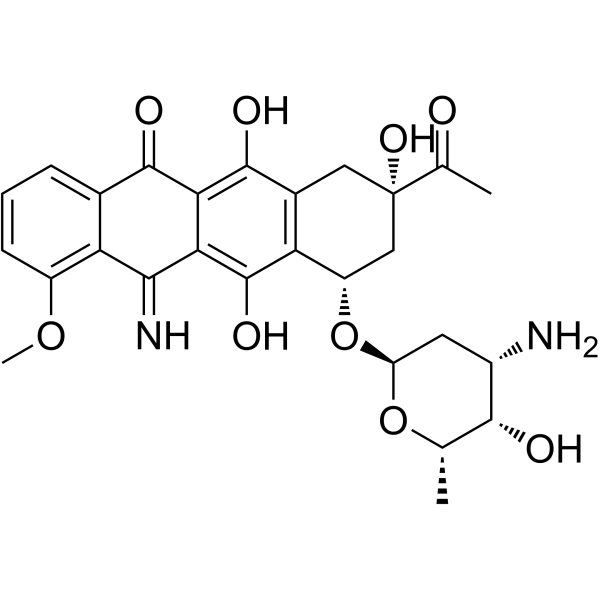
-
GC66658
5-Me-dC(Ac) amidite
L'amidite 5-Me-dC(Ac) est utilisé pour synthétiser l'ADN ou l'ARN.
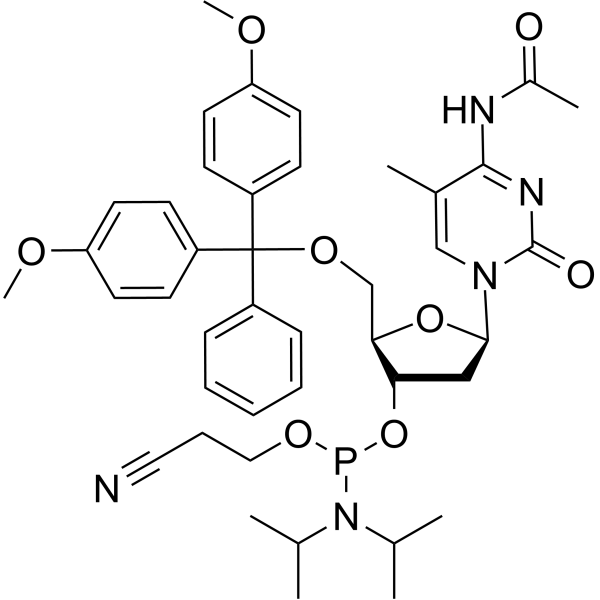
-
GC31187
5-Methoxyflavone
La 5-méthoxyflavone, appartenant À la famille des flavonoÏdes, est un inhibiteur de l'ADN polymérase-bêta et un agent neuroprotecteur contre la toxicité bêta-amyloÏde.
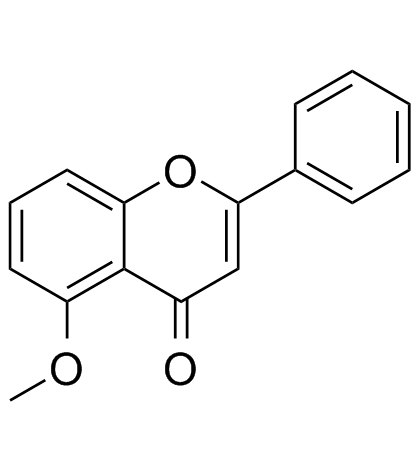
-
GC67671
5-Methylcytidine 5′-triphosphate trisodium
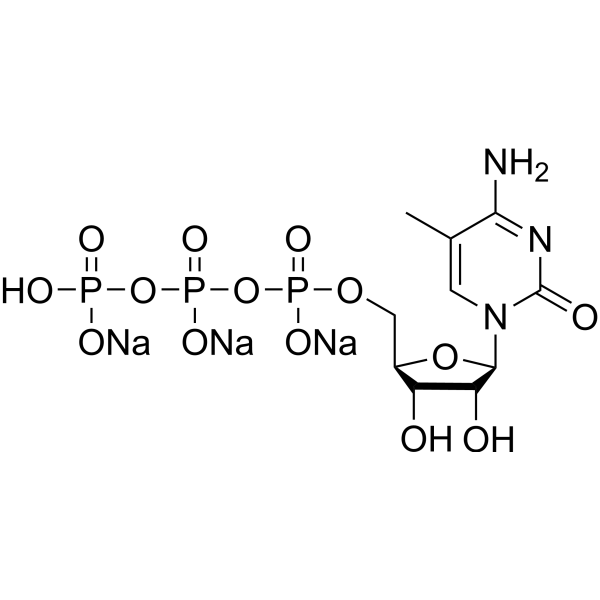
-
GC35166
5-Methylcytosine
La 5-méthylcytosine est une modification de l'ADN bien caractérisée et se trouve également principalement dans les ARN non codants abondants chez les procaryotes et les eucaryotes.
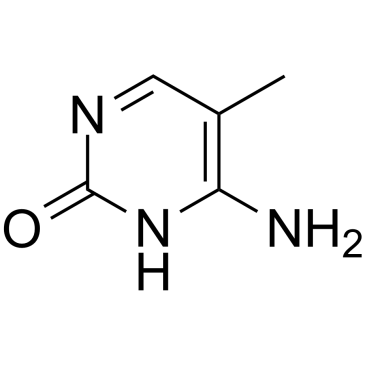
-
GC62547
5-O-TBDMS-N4-Benzoyl-2-deoxycytidine
La 5-O-TBDMS-N4-Benzoyl-2-désoxycytidine est un nucléoside modifié.
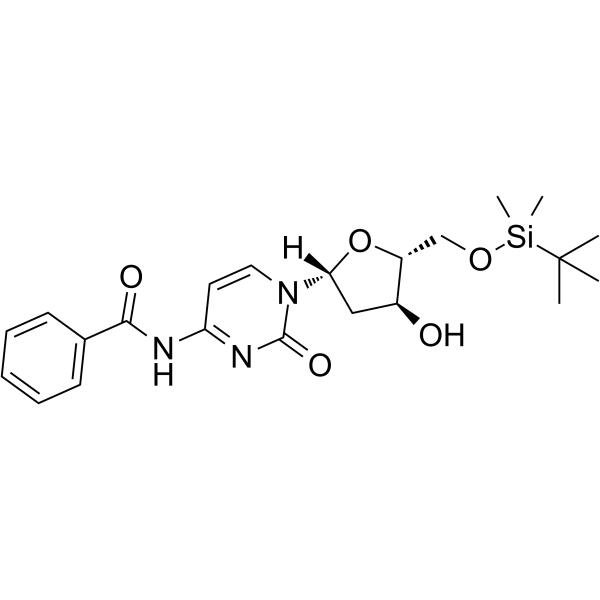
-
GC46713
5-Phospho-D-ribose 1-diphosphate (sodium salt hydrate)
An intermediate in several biochemical pathways
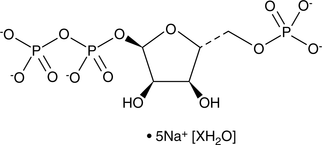
-
GC60031
6-Azathymine
La 6-azathymine, un analogue 6-azoté de la thymine, est un puissant inhibiteur de la D-3-aminoisobutyrate-pyruvate aminotransférase.
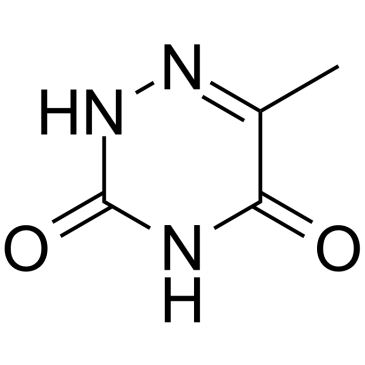
-
GC50249
6-Hydroxy-DL-DOPA
La 6-hydroxy-DL-DOPA est un inhibiteur allostérique sélectif et efficace du domaine de liaison À l'ADNsb de RAD52. La 6-hydroxy-DL-DOPA peut être utilisée pour la recherche sur le cancer.
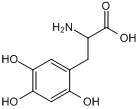
-
GC12193
6-Thio-dG
Le 6-Thio-dG est un analogue nucléosidique qui peut être incorporé dans des télomères synthétisés de novo par la télomérase.
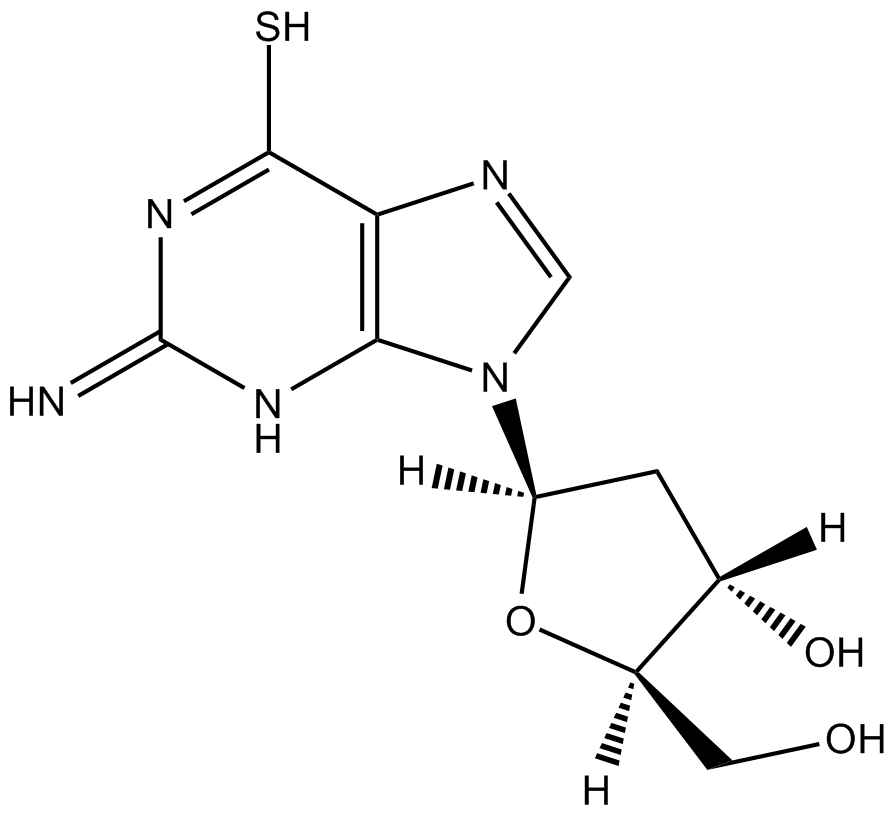
-
GC33577
7-Aminoactinomycin D (7-AAD)
La 7-aminoactinomycine D (7-AAD) (7-AAD), une coloration fluorescente de l'ADN, est un puissant inhibiteur de l'ARN polymérase.
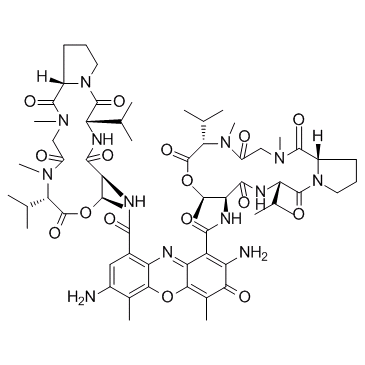
-
GC65408
7-Deaza-2'-deoxy-7-iodoadenosine
7-Deaza-'-désoxy-7-iodoadénosine est un oligonucléotide modifié contenant de la 7-Déazaadénine.
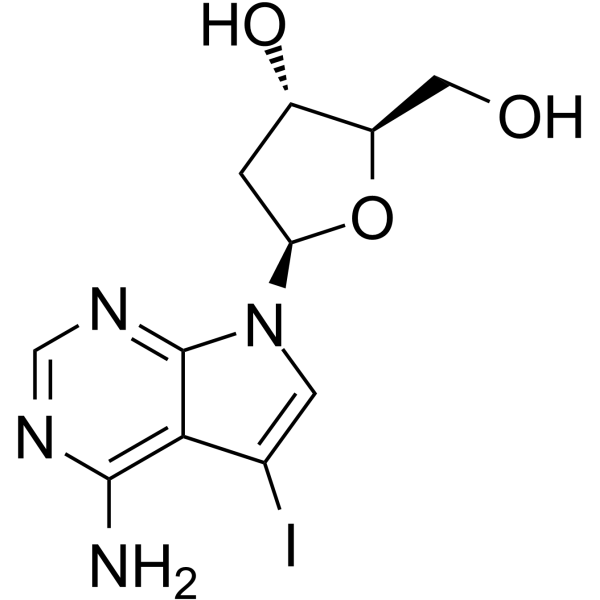
-
GC65675
7-Iodo-7-deaza-2'-deoxyguanosine
7-Iodo-7-deaza-2'-désoxyguanosine (7-Deaza-7-Iodo-2'-deoxyguanosine) est un dérivé de désoxyguanosine qui peut être utilisé dans la synthèse d'ADN et les réactions de séquenÇage.
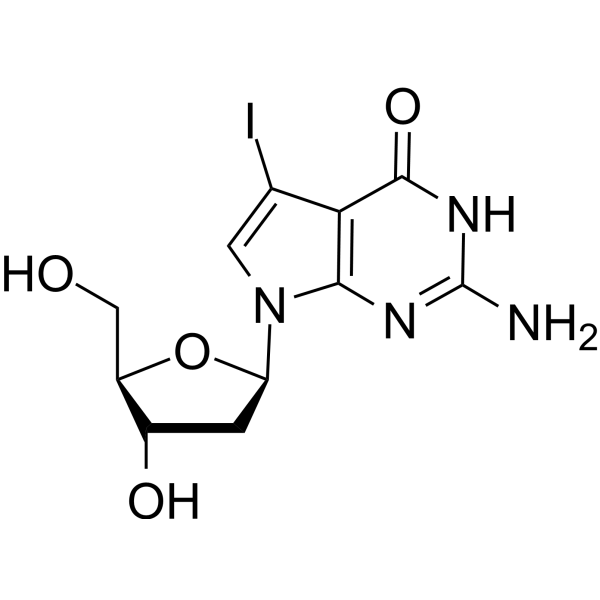
-
GC49561
7-Methylguanosine-d3
An internal standard for the quantification of 7-methylguanosine
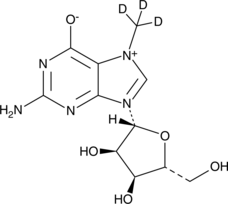
-
GC66058
7-TFA-ap-7-Deaza-dA
Le 7-TFA-ap-7-Deaza-dA est un nucléoside modifié. Le 7-TFA-ap-7-Deaza-dA peut être utilisé dans la synthèse d'acide désoxyribonucléique ou d'acide nucléique.
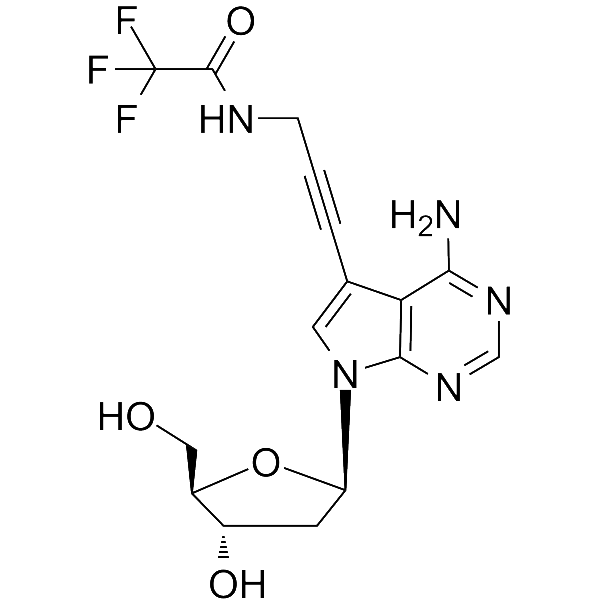
-
GC65525
7-TFA-ap-7-Deaza-dG
Le 5'-O-TBDMS-dG est un nucléoside modifié.
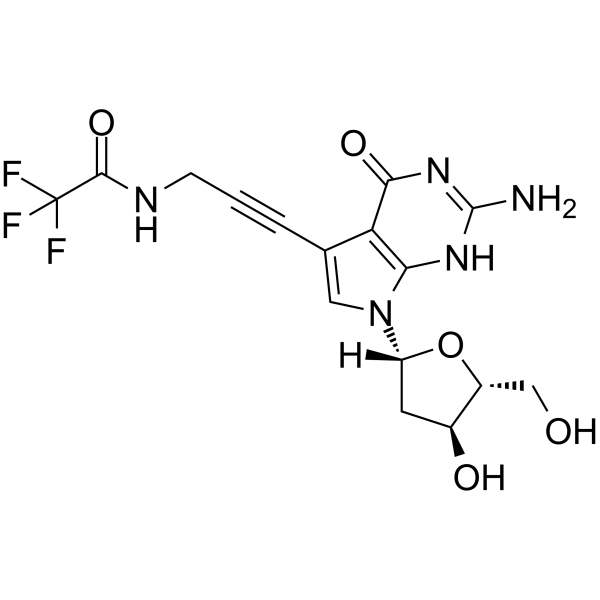
-
GC12777
8-Chloroadenosine
Nucleoside analog, inhibits RNA synthesis
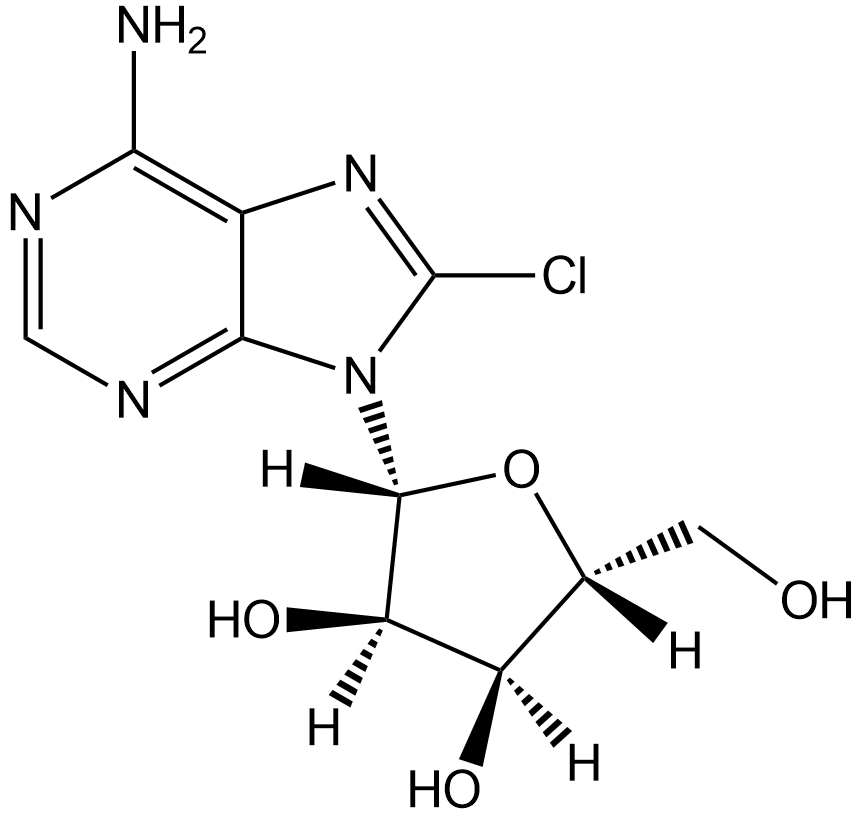
-
GC61763
Ac-rC Phosphoramidite
Le phosphoramidite Ac-rC est utilisé pour la modification du phosphorodithioate d'oligoribonucléotide (ARN PS2) .

-
GC34090
Acelarin (NUC-1031)
Acelarin (NUC-1031) (NUC-1031) est une transformation ProTide et une amélioration de l'analogue nucléoside largement utilisé, la gemcitabine.
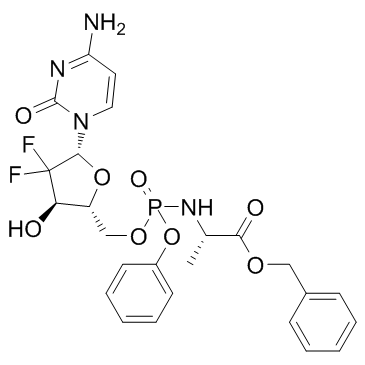
-
GC16866
Actinomycin D
Un bloqueur de transcription interagissant avec l'ADN ayant une activité anti-cancer.

-
GC35247
ACX-362E
ACX-362E (ACX-362E) est un premier inhibiteur de l'ADN polymérase IIIC (pol IIIC) actif par voie orale, avec un Ki de 0,325 μM pour l'ADN pol IIIC de C.
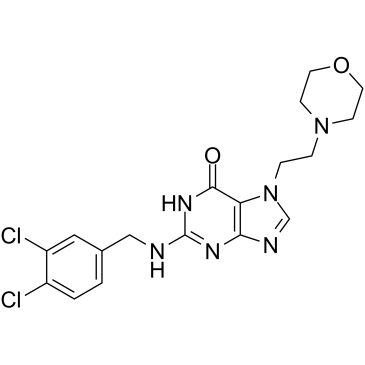
-
GC17186
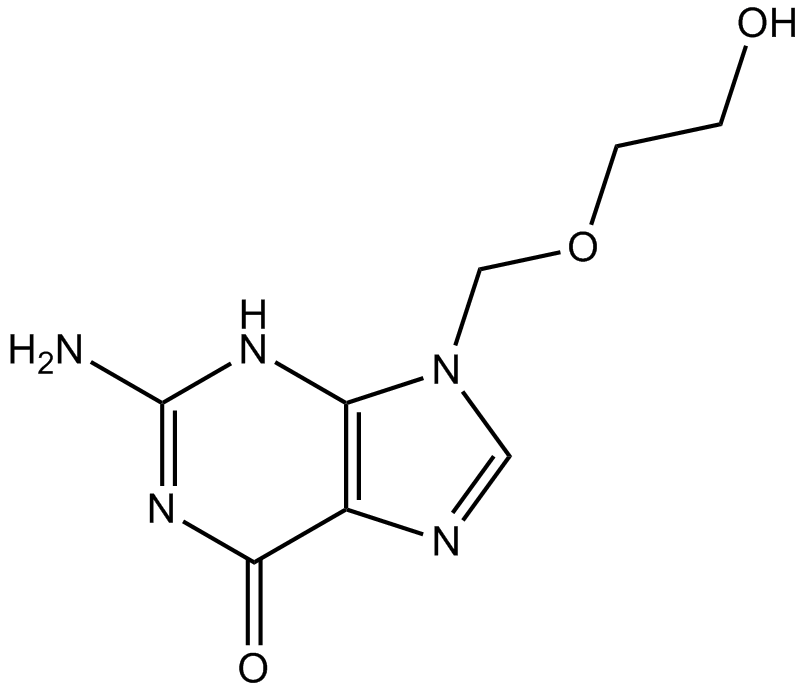
Acyclovir
L'acyclovir (aciclovir) est un puissant agent antiviral actif par voie orale.
-
GC13432
Adenine
L'adénine (6-aminopurine), une purine, est l'une des quatre nucléobases de l'acide nucléique de l'ADN. L'adénine agit comme un composant chimique de l'ADN et de l'ARN. L'adénine joue également un rÔle important dans la biochimie impliquée dans la respiration cellulaire, la forme À la fois de l'ATP et des cofacteurs (NAD et FAD), et la synthèse des protéines.

-
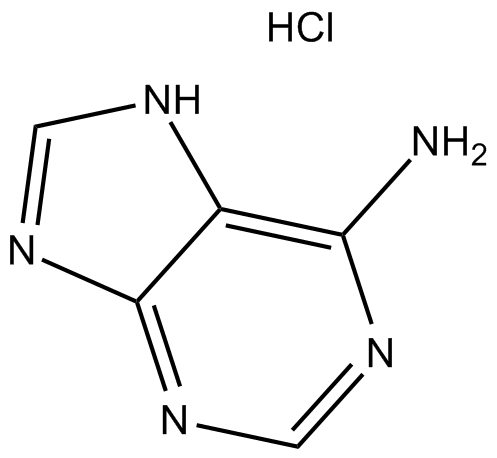
GC11825
Adenine HCl
L'adénine HCl (chlorhydrate de 6-aminopurine), une purine, est l'une des quatre nucléobases de l'acide nucléique de l'ADN. L'adénine HCl agit comme un composant chimique de l'ADN et de l'ARN. L'adénine HCl joue également un rÔle important dans la biochimie impliquée dans la respiration cellulaire, la forme À la fois de l'ATP et des cofacteurs (NAD et FAD), et la synthèse des protéines.
-
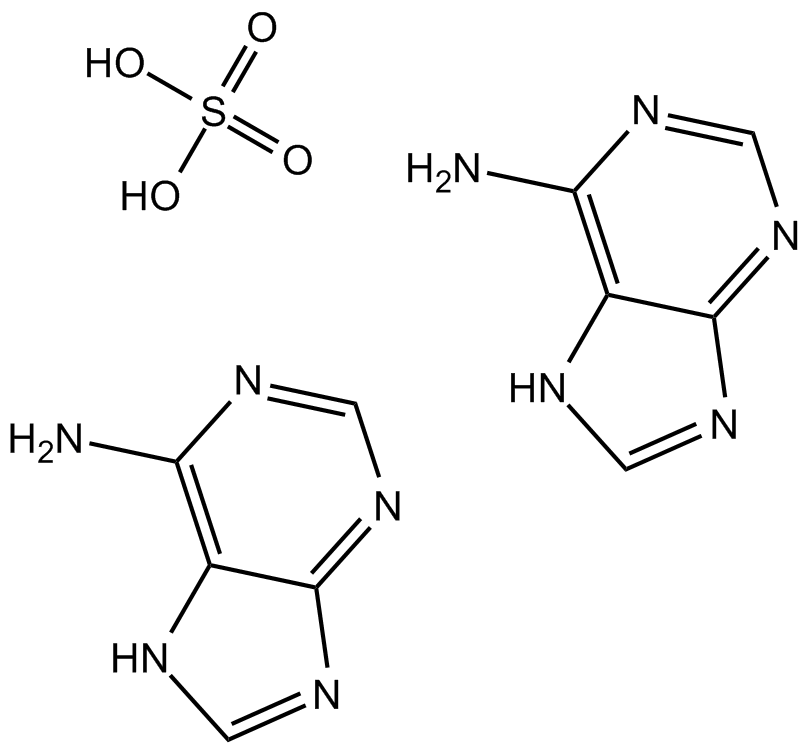
GC17278
Adenine sulfate
Le sulfate d'adénine (hémisulfate de 6-aminopurine), une purine, est l'une des quatre nucléobases de l'acide nucléique de l'ADN. Le sulfate d'adénine agit comme un composant chimique de l'ADN et de l'ARN. Le sulfate d'adénine joue également un rÔle important dans la biochimie impliquée dans la respiration cellulaire, la forme À la fois de l'ATP et des cofacteurs (NAD et FAD), et la synthèse des protéines.
-
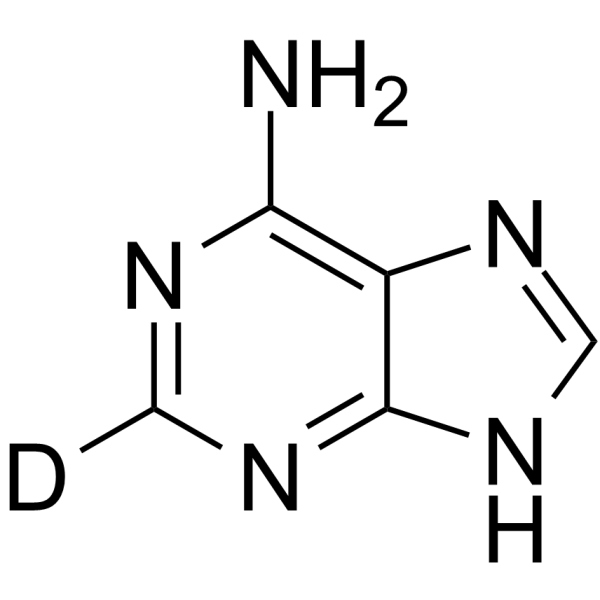
GC67946
Adenine-d1
-
GC49004
Adenosine 3’-monophosphate (sodium salt hydrate)
A nucleotide
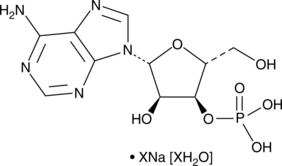
-
GC42732
Adenosine 3',5'-diphosphate (sodium salt)
L'adénosine 3',5'-diphosphate (sel de sodium) est un inhibiteur des hydroxystéroÏdes sulfotransférases.