Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nom du produit Informations
-
GC16509
Adox
Adox, un analogue nucléosidique purique, est un puissant inhibiteur de la S-adénosylhomocystéine hydrolase (SAHH) (Ki = 3,3 nM). L'adénosine dialdéhyde présente une puissante activité anti-tumorale in vivo et peut être utilisée pour la recherche sur le cancer.
-
GC64527
ADTL-SA1215
L'ADTL-SA1215 est un activateur de petite molécule spécifique de première classe de SIRT3 qui module l'autophagie dans le cancer du sein triple négatif.
-
GC65330
AES-135
L'AES-135, un inhibiteur pan-HDAC À base d'acide hydroxamique, prolonge la survie dans un modèle murin orthotopique de cancer du pancréas. AES-135 inhibe HDAC3, HDAC6, HDAC8 et HDAC11 avec des IC50 allant de 190 À 1100 nM.
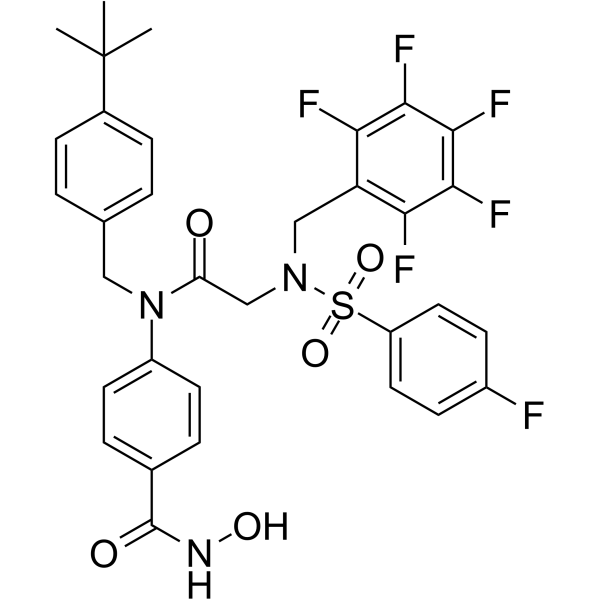
-
GC48775
AES-350
AES-350 est un inhibiteur HDAC6 puissant et actif par voie orale avec une CI50 et un Ki de 0,0244 μM et 0,035 μM, respectivement. AES-350 est également contre HDAC3, HDAC8 dans un test d'activité enzymatique avec des valeurs IC50 de 0,187 μM et 0,245 μM, respectivement. L'AES-350 déclenche l'apoptose dans les cellules AML par inhibition des HDAC et peut être utilisé pour la recherche sur la leucémie myéloÏde aiguë (LAM).
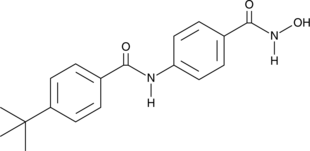
-
GC66379
AFM-30a hydrochloride
Le chlorhydrate d'AFM-30a est un puissant inhibiteur de la protéine arginine désiminase 2 (PAD2) et possède une excellente sélectivité pour PAD2. Le chlorhydrate d'AFM-30a se lie À PAD2 avec une valeur EC50 de 9,5 μM. Le chlorhydrate d'AFM-30a inhibe également la citrullination H3 avec une valeur EC50 de 0,4 μM. Le chlorhydrate d'AFM-30a peut être utilisé pour la recherche de certains cancers et de diverses maladies auto-immunes, notamment la polyarthrite rhumatoÏde (PR), la sclérose en plaques, le lupus et la colite ulcéreuse.
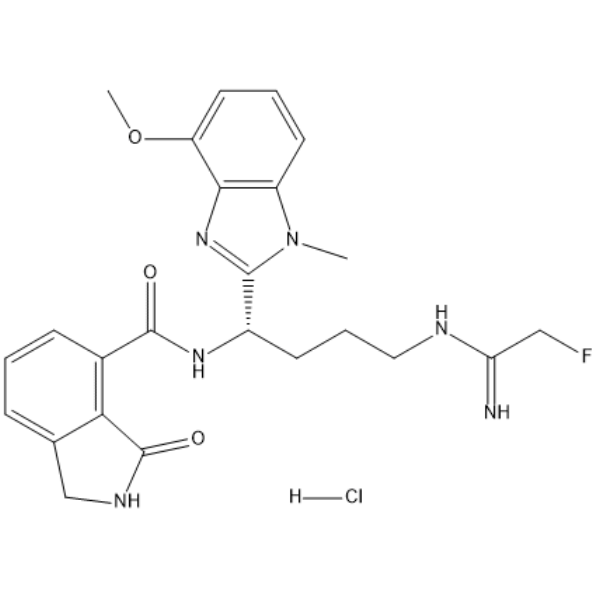
-
GC33416
AFP464
AFP464 (base libre NSC710464), est un HIF-1α actif ; inhibiteur avec une IC50 de 0,25 μM, est également un puissant activateur du récepteur d'aryle hydrocarbure (AhR).

-
GC16318
AG-14361
A PARP1 inhibitor
-
GC17881
AGK 2
AGK 2 est un inhibiteur sélectif de SIRT2 avec une IC50 de 3,5 μM. AGK 2 inhibe SIRT1 et SIRT3 avec des IC50 de 30 et 91 μM, respectivement.
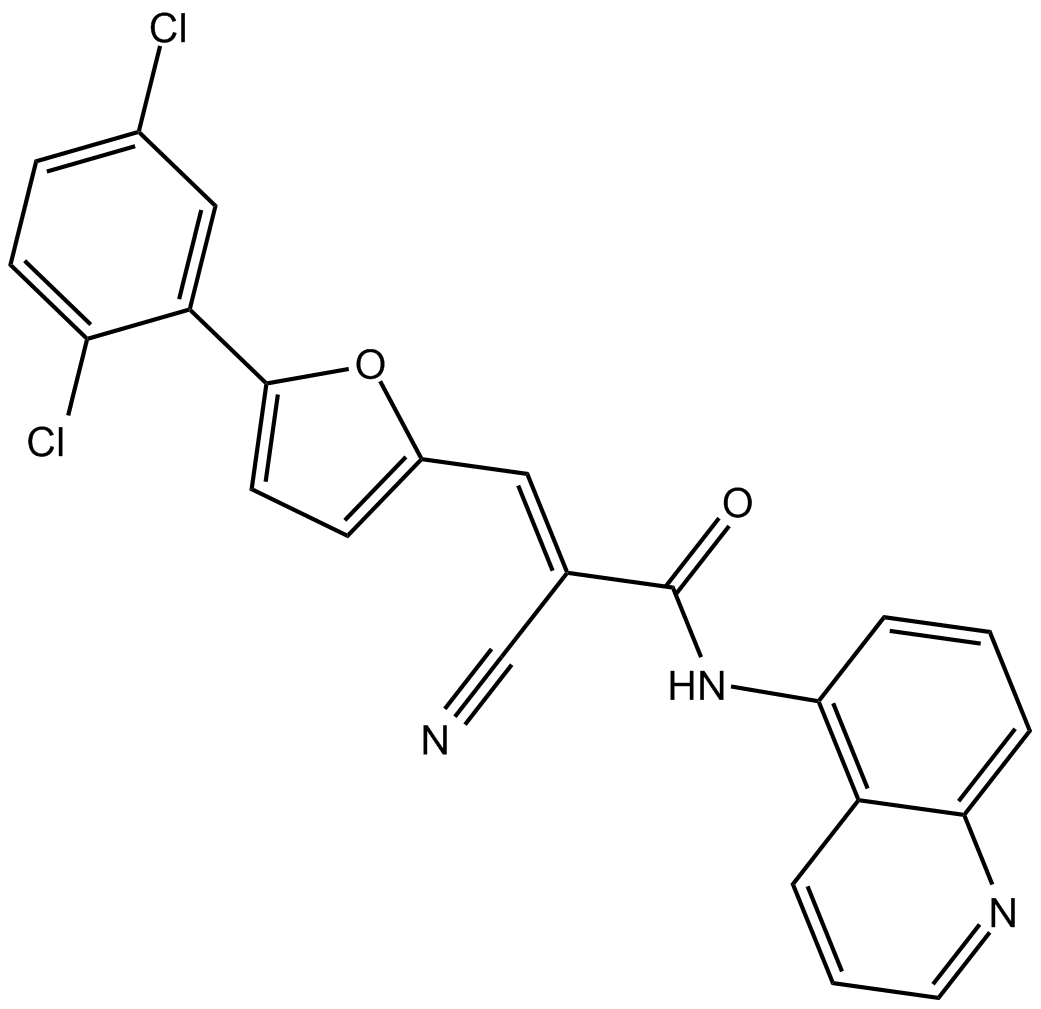
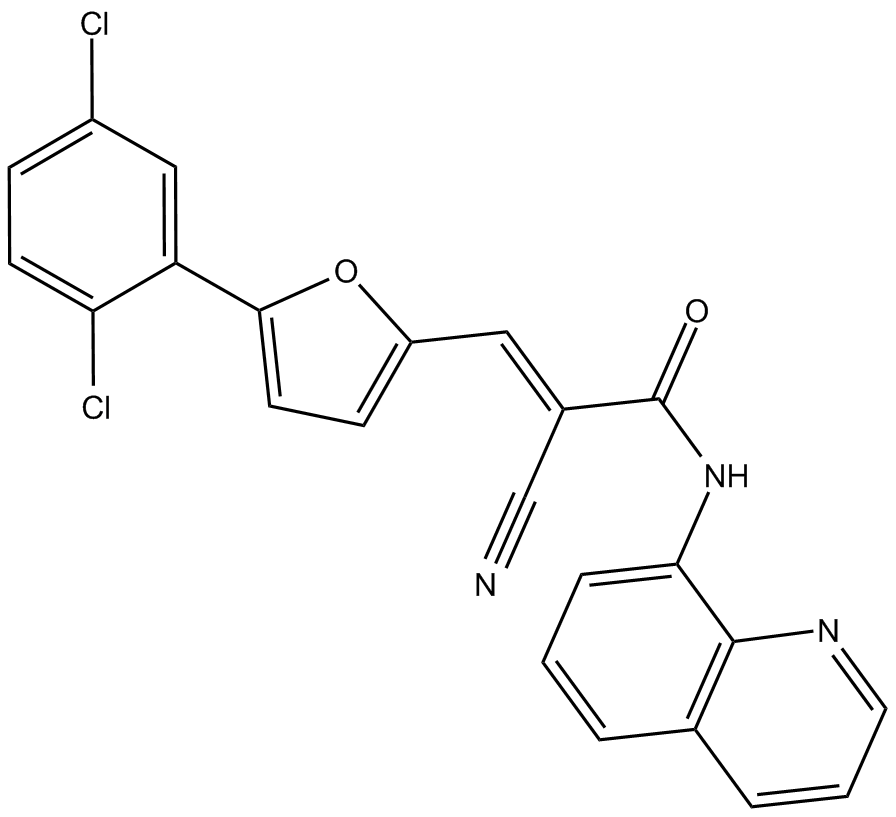
-
GC15931
AGK7
AGK7 est un puissant inhibiteur de la sirtuine 2 (SIRT2).
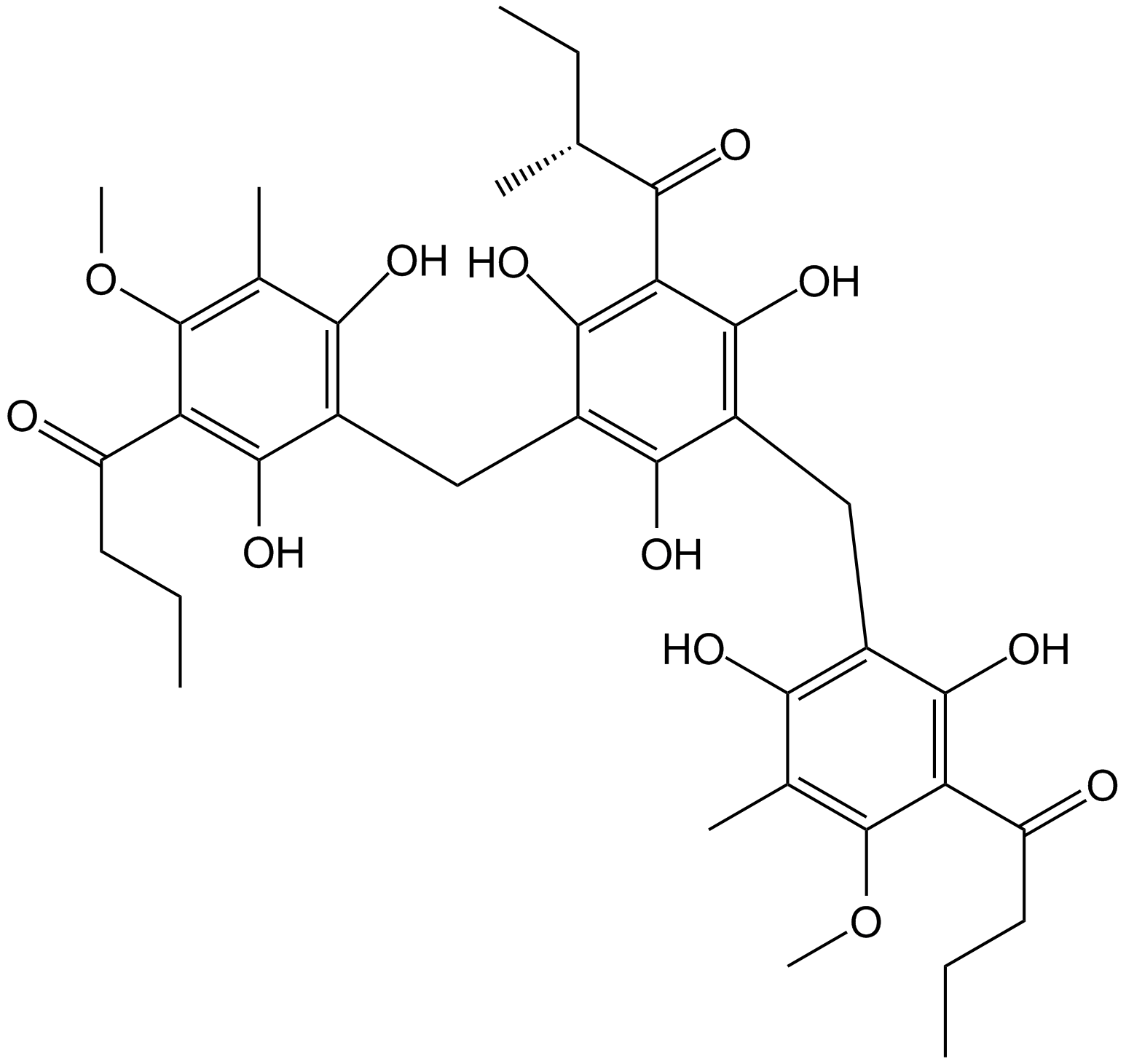
-
GN10413
Agrimol B
-
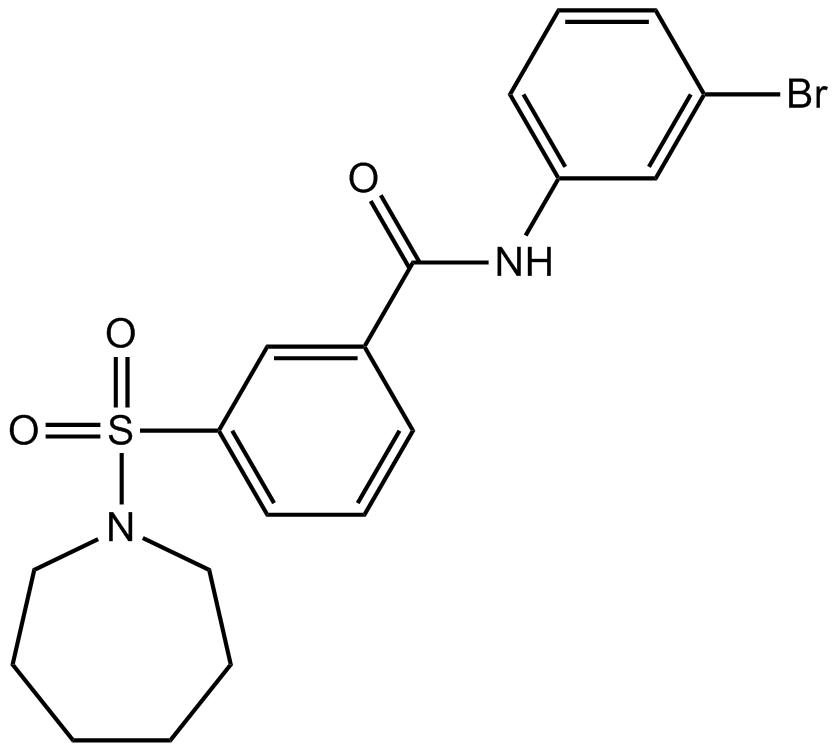
GC10676
AK-7
AK-7 est un inhibiteur sélectif de SIRT2 perméable aux cellules et au cerveau, avec une IC50 de 15,5 μM.
-
GC62277
AKB-6899
Le Ro24-7429 est un antagoniste de la protéine Tat transactivatrice du VIH-1 puissant et actif par voie orale. Le Ro24-7429 est également un inhibiteur du facteur de transcription 1 lié aux avortons (RUNX1). Le Ro24-7429 a des effets anti-VIH, antifibrotiques et anti-inflammatoires.
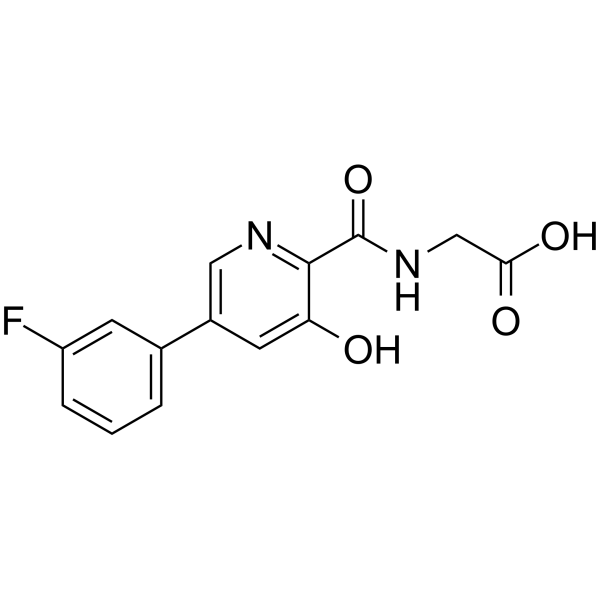
-
GC62433
AKI603
AKI603 est un inhibiteur de l'Aurora kinase A (AurA), avec une IC50 de 12,3 nM. AKI603 est développé pour surmonter la résistance médiée par la mutation BCR-ABL-T315I. AKI603 présente une forte activité anti-proliférative dans les cellules leucémiques.
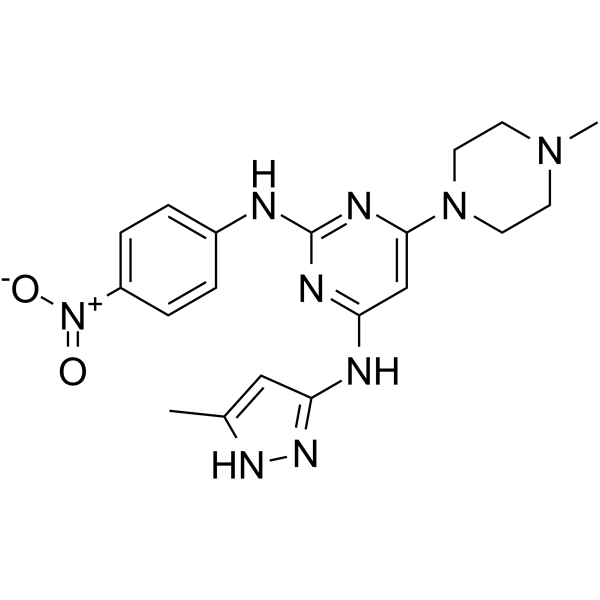
-
GC17344
Alexidine dihydrochloride
An alkyl bis(biguanide) antiseptic
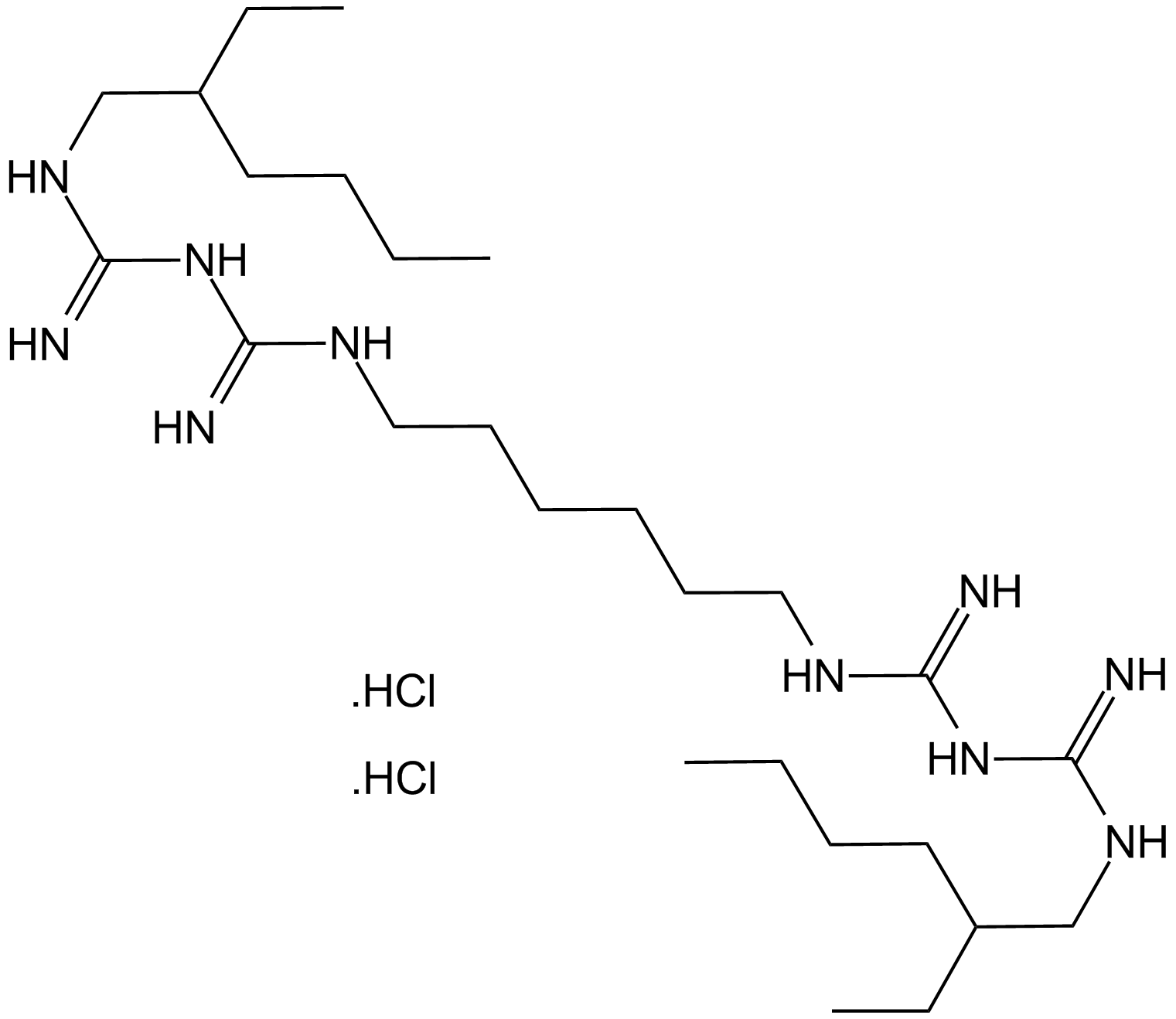
-
GC49393
all-trans-13,14-Dihydroretinol
A metabolite of all-trans retinoic acid

-
GC35297
Alobresib
L'alobrésib (GS-5829) est un inhibiteur du bromodomaine BET, qui représente un agent thérapeutique hautement efficace contre le carcinome séreux utérin (USC) récurrent/résistant À la chimiothérapie surexprimant c-Myc.
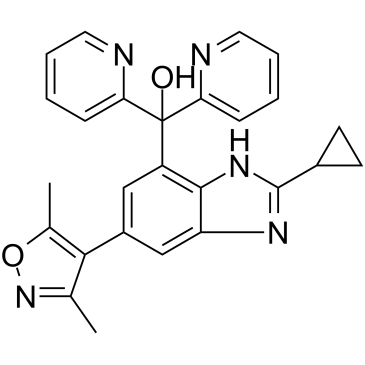
-
GC67984
Alteminostat
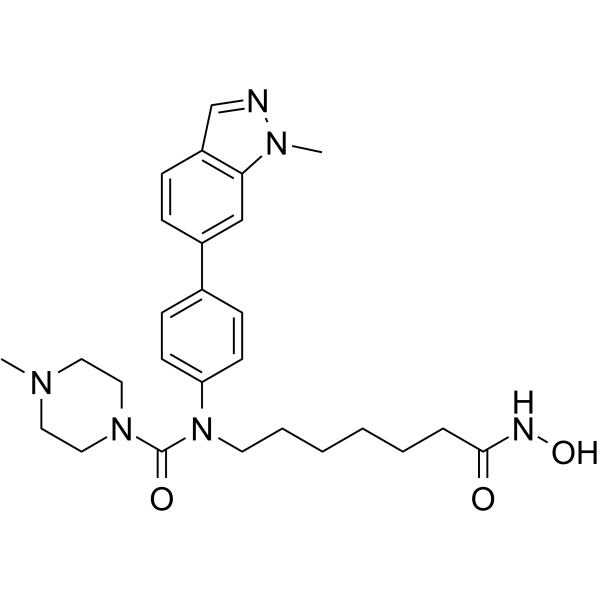
-
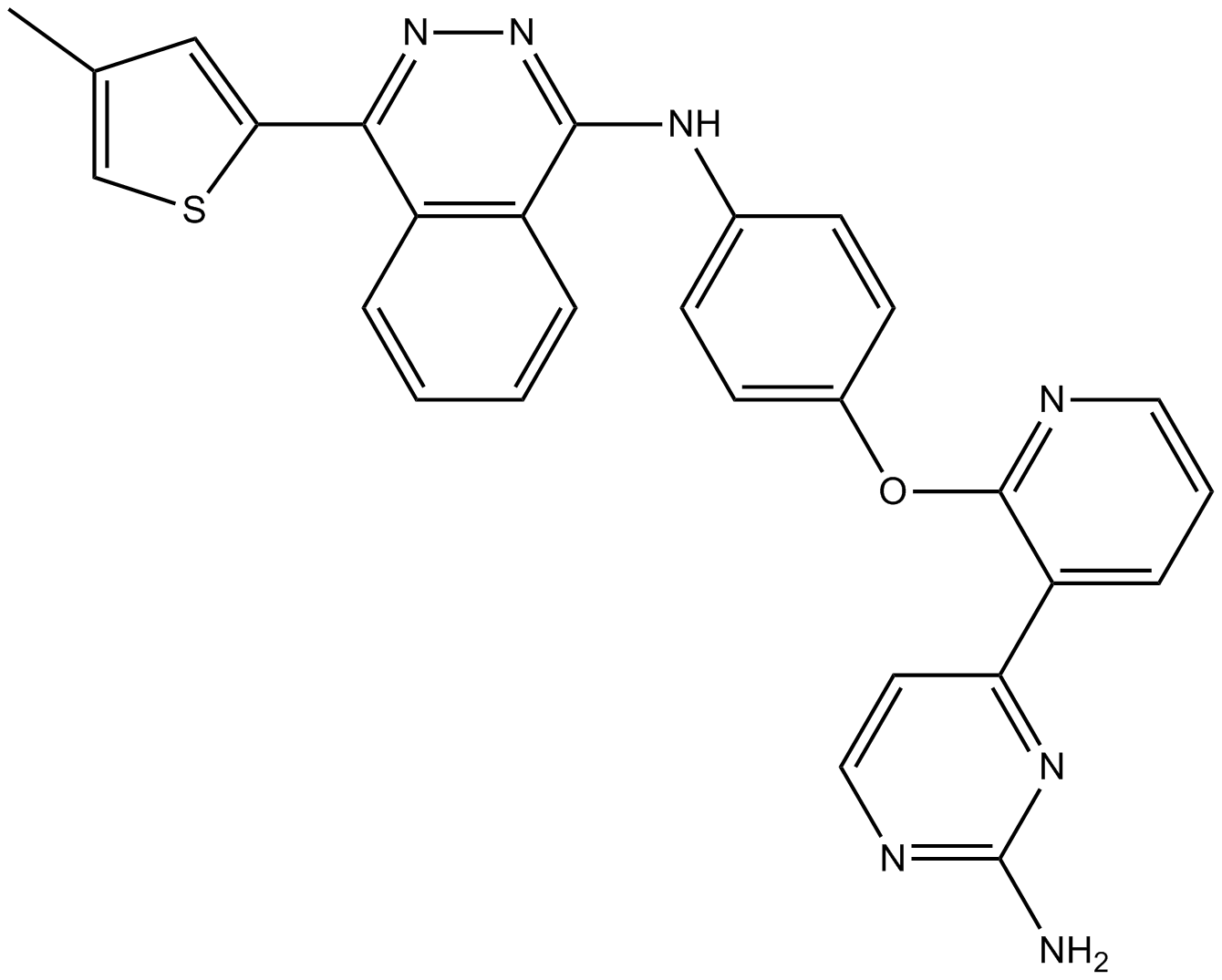
GC13198
AMG-900
A selective pan-Aurora kinase inhibitor
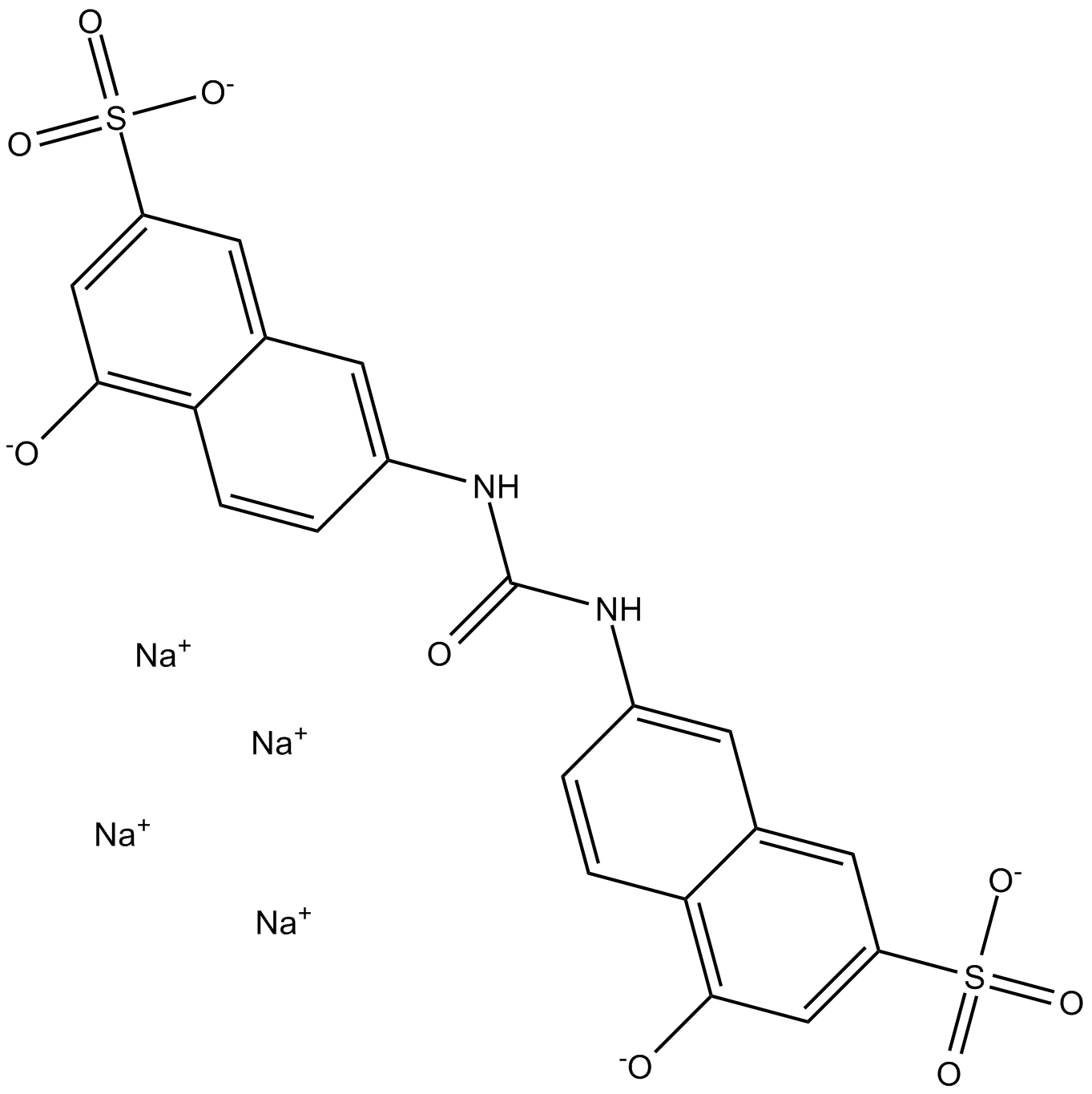
-
GC17275
AMI-1
A cell permeable inhibitor of PRMTs
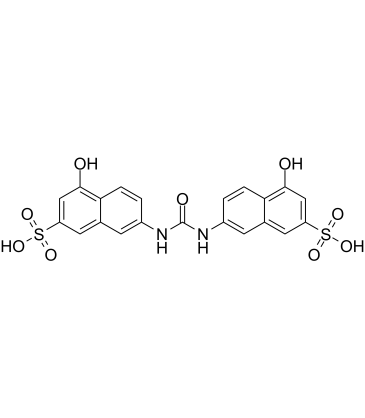
-
GC39840
AMI-1 free acid
L'acide libre AMI-1 est un inhibiteur puissant, perméable aux cellules et réversible des protéines arginine N-méthyltransférases (PRMT), avec des IC50 de 8,8 μM et 3,0 μM pour le PRMT1 humain et la levure-Hmt1p, respectivement. L'acide libre AMI-1 exerce des effets inhibiteurs des PRMT en bloquant la liaison peptide-substrat.
-
GC17546
AMI5
L'éosine Y (disodique) est une molécule de colorant rouge acide soluble.
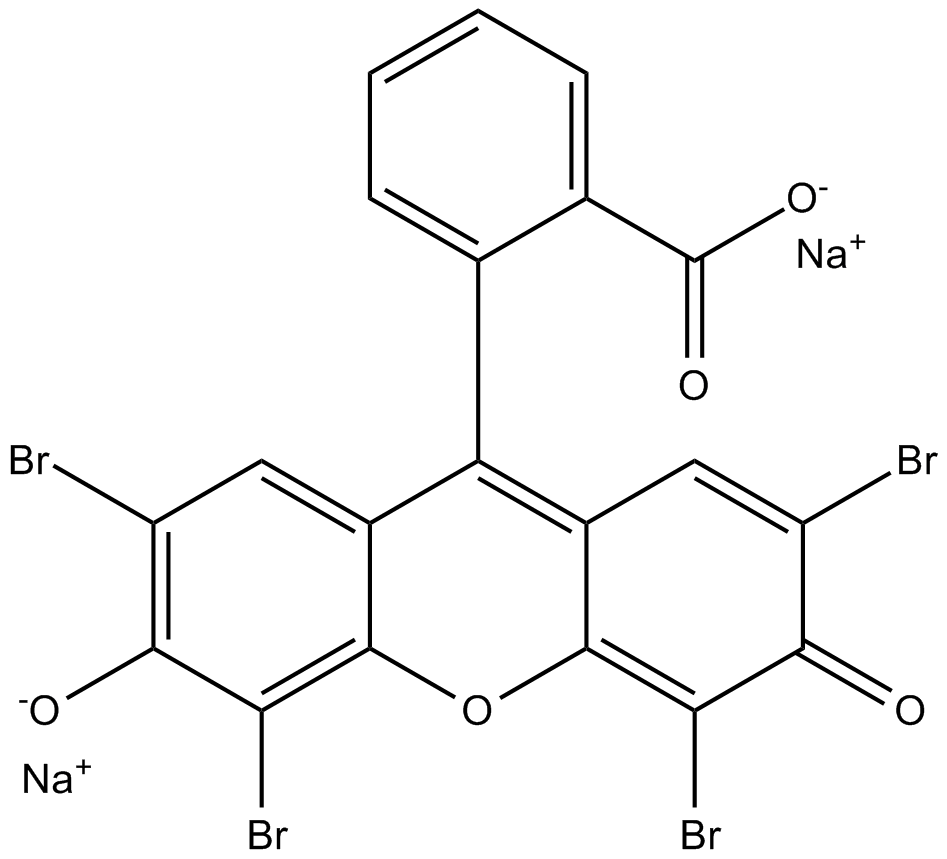
-
GC42785
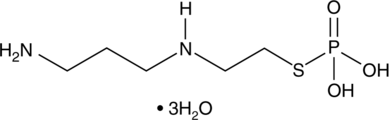
Amifostine (hydrate)
L'amifostine (hydrate) (WR2721 trihydraté) est un agent cytoprotecteur À large spectre et un radioprotecteur. L'amifostine (hydrate) protège sélectivement les tissus normaux des dommages causés par la radiothérapie et la chimiothérapie. L'amifostine (hydrate) est un puissant facteur inductible par l'hypoxie-α1 (HIF-α1) et un inducteur de p53. L'amifostine (hydrate) protège les cellules contre les dommages en éliminant les radicaux libres dérivés de l'oxygène. L'amifostine (hydrate) réduit la toxicité rénale et a une action anti-angiogénique.
-
GC42790
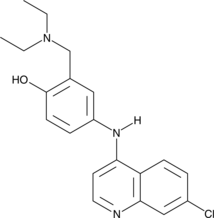
Amodiaquine
Amodiaquine est un composé antipaludique aminoquinoléine.
-
GC60579
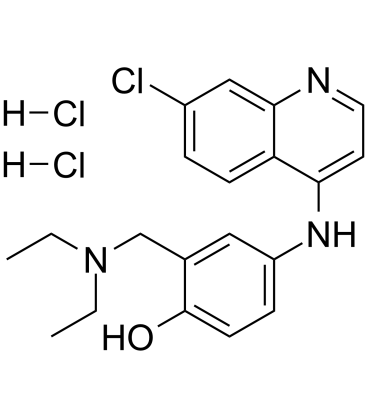
Amodiaquine dihydrochloride
Le dichlorhydrate d'amodiaquine (dichlorhydrate d'amodiaquine), une classe d'agents antipaludéens de la 4-aminoquinoléine, est un inhibiteur puissant et actif par voie orale de l'histamine N-méthyltransférase avec un Ki de 18,6 nM.
-
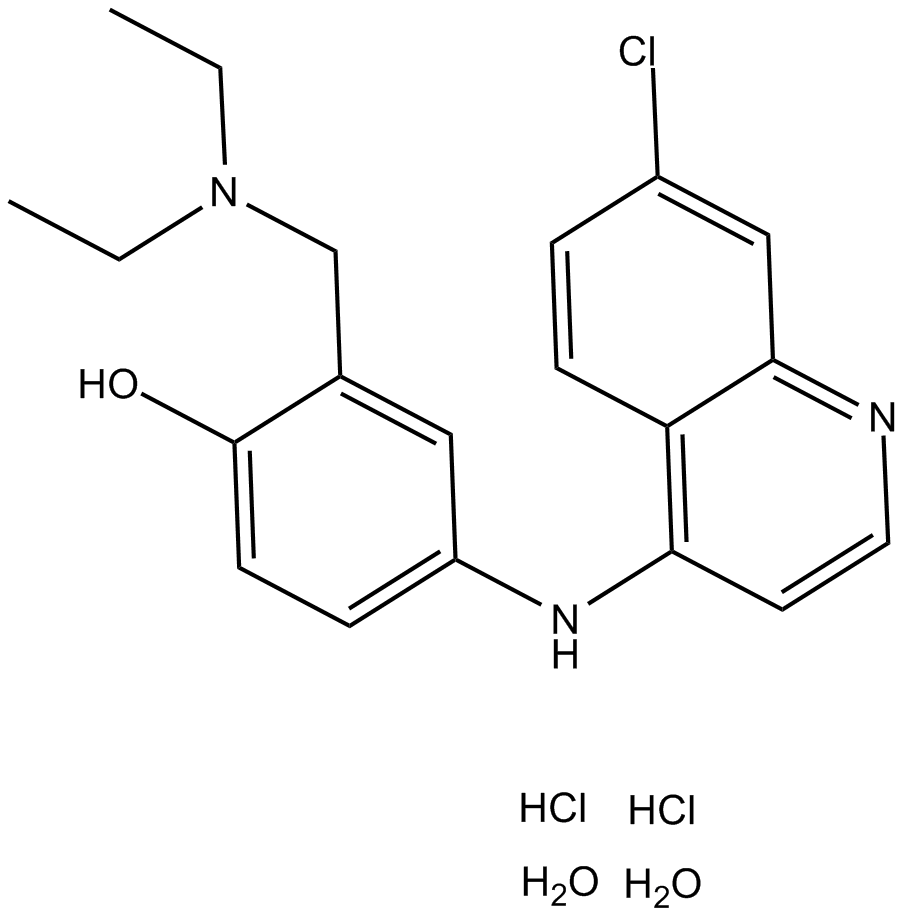
GC10905
Amodiaquine dihydrochloride dihydrate
Le dichlorhydrate d'amodiaquine dihydraté (dichlorhydrate d'amodiaquine dihydraté), une classe d'agents antipaludiques de la 4-aminoquinoléine, est un puissant inhibiteur de l'histamine N-méthyltransférase actif par voie orale.
-
GC67912
Amredobresib

-
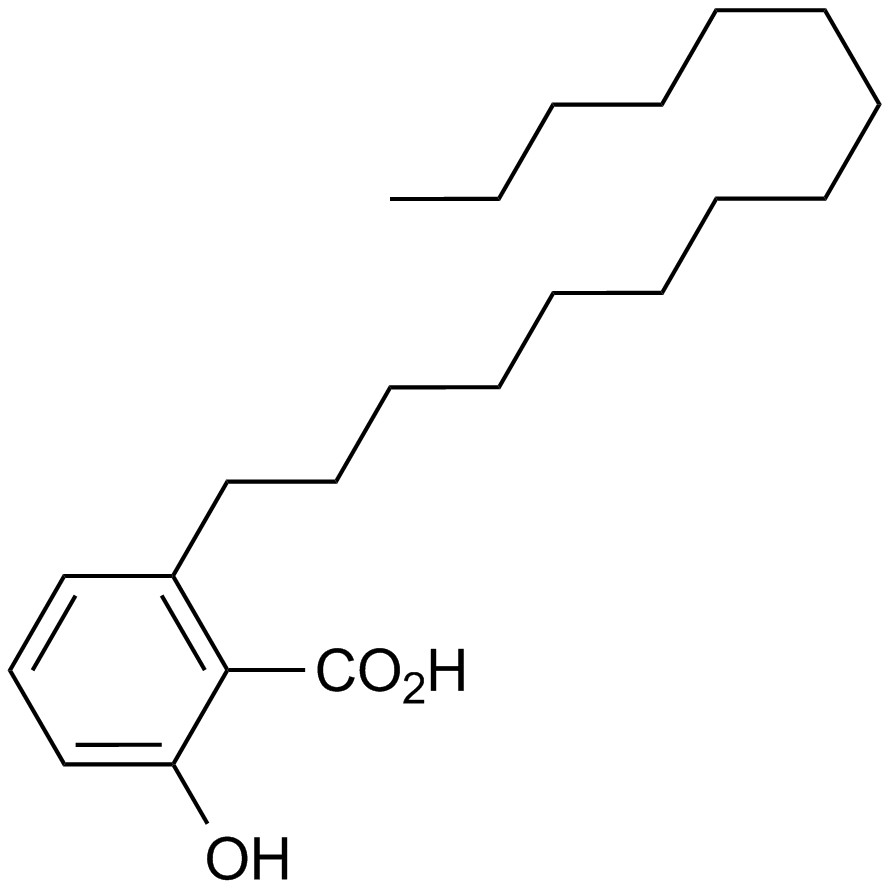
GC17284
Anacardic acid
A histone acetyltransferase inhibitor
-
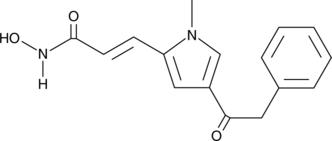
GC40674
APHA Compound 8
A class I and II HDAC inhibitor
-
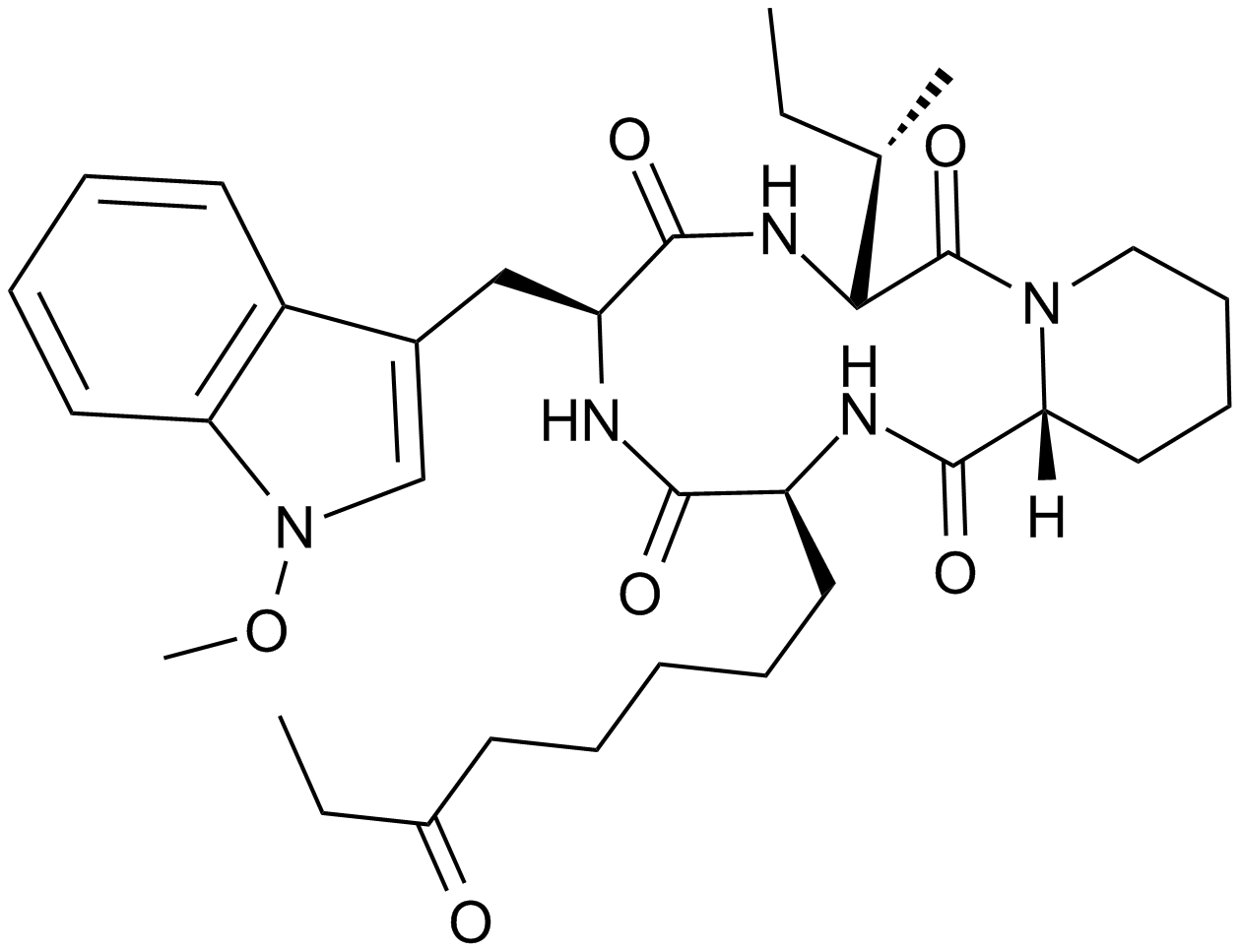
GC12961
Apicidin
Un inhibiteur d'HDAC perméable à la cellule.
-
GC14590
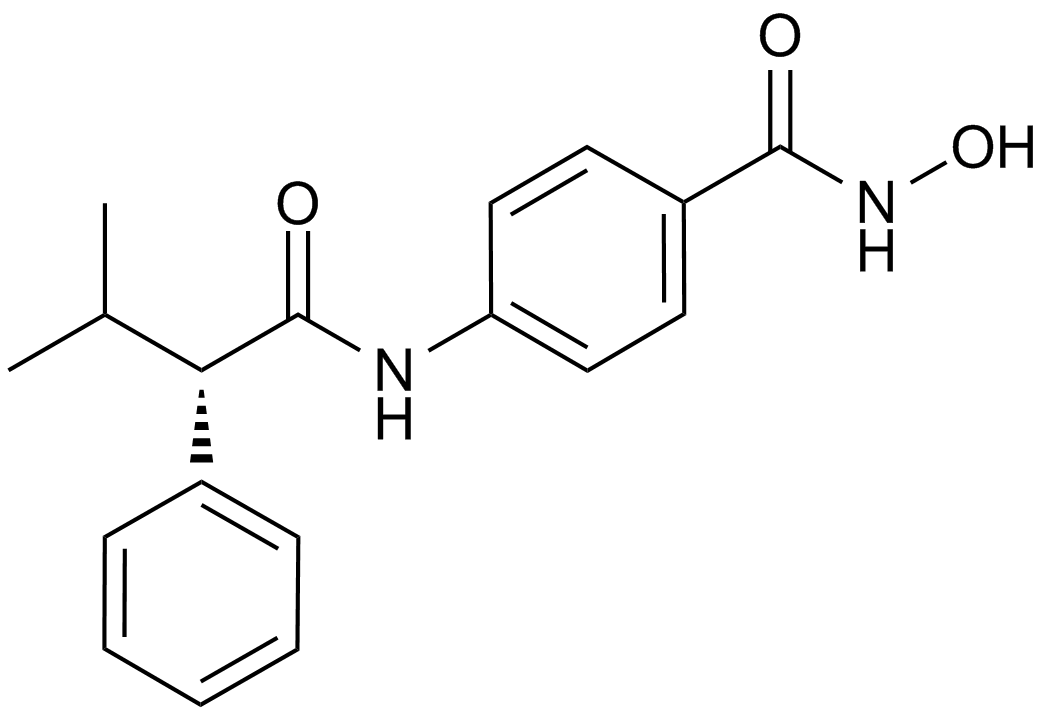
AR-42 (OSU-HDAC42)
HDAC inhibitor,novel and potent
-
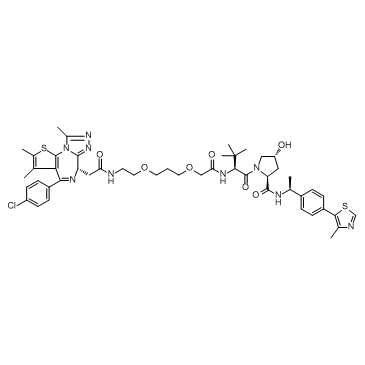
GC32685
ARV-771
ARV-771 est un puissant BET PROTAC basé sur la ligase E3 de Hippel-Lindau avec des Kd de 34 nM, 4,7 nM, 8,3 nM, 7,6 nM, 9,6 nM et 7,6 nM pour BRD2(1), BRD2(2), BRD3( 1), BRD3(2), BRD4(1) et BRD4(2), respectivement.
-
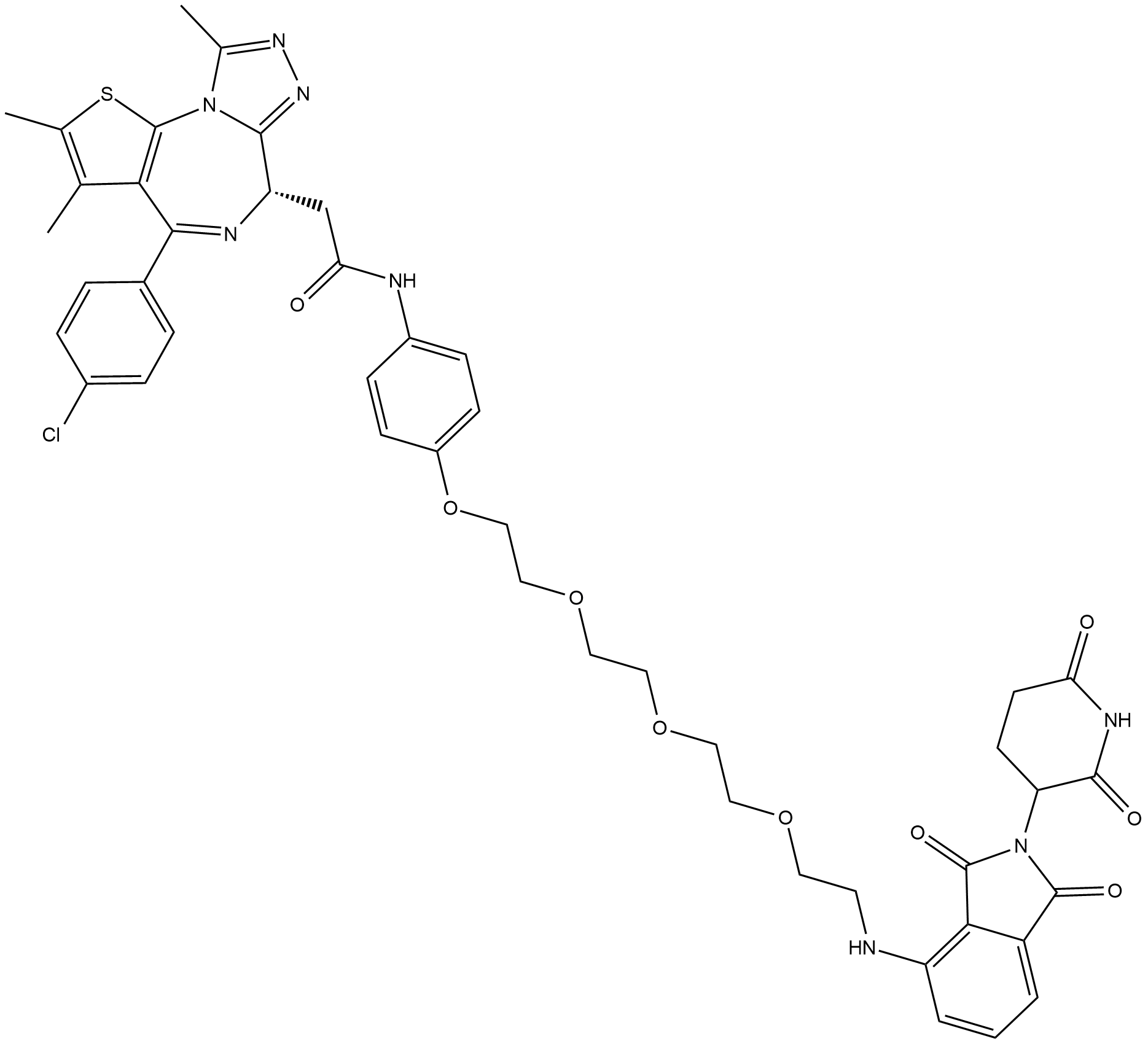
GC19038
ARV-825
ARV-825 est un PROTAC connecté par des ligands pour Cereblon et BRD4. ARV-825 se lie À BD1 et BD2 de BRD4 avec des Kd de 90 et 28 nM, respectivement.
-
GC65918
AS-85
L'AS-85 est un puissant inhibiteur de l'histone méthyltransférase ASH1L (IC50=0,6 μM) avec une activité anti-leucémique. AS-85 se lie fortement au domaine ASH1L SET, avec la valeur Kd de 0,78μ ;M.
-
GC62615
AS-99
L'AS-99 est un inhibiteur puissant et sélectif de l'histone méthyltransférase ASH1L (IC50 = 0,79μ ; M, Kd = 0,89⋼ ; M) avec une activité anti-leucémique. L'AS-99 bloque la prolifération cellulaire, induit l'apoptose et la différenciation, régule À la baisse les gènes cibles de fusion MLL et réduit le fardeau de la leucémie in vivo.
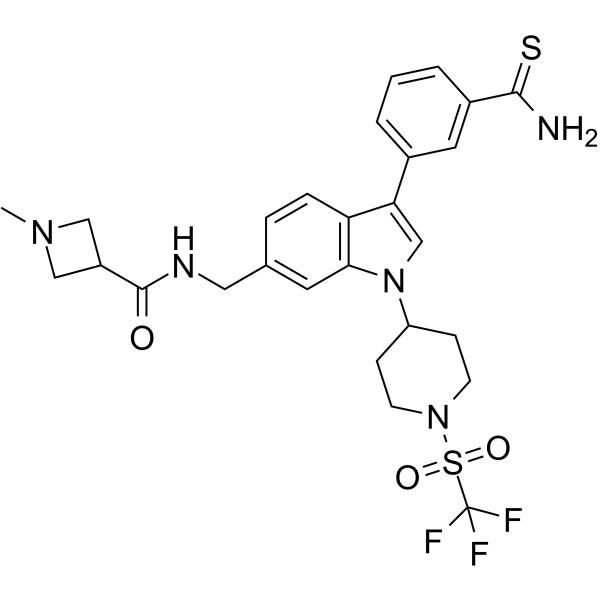
-
GC62849
AS-99 TFA
L'AS-99 TFA est un inhibiteur puissant et sélectif de l'histone méthyltransférase ASH1L (IC50=0,79μM, Kd=0,89μM) avec une activité anti-leucémique. L'AS-99 TFA bloque la prolifération cellulaire, induit l'apoptose et la différenciation, régule À la baisse les gènes cibles de fusion MLL et réduit le fardeau de la leucémie in vivo.
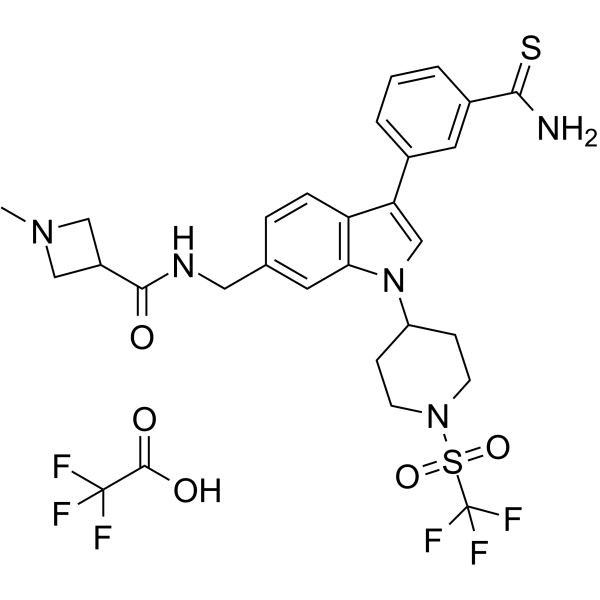
-
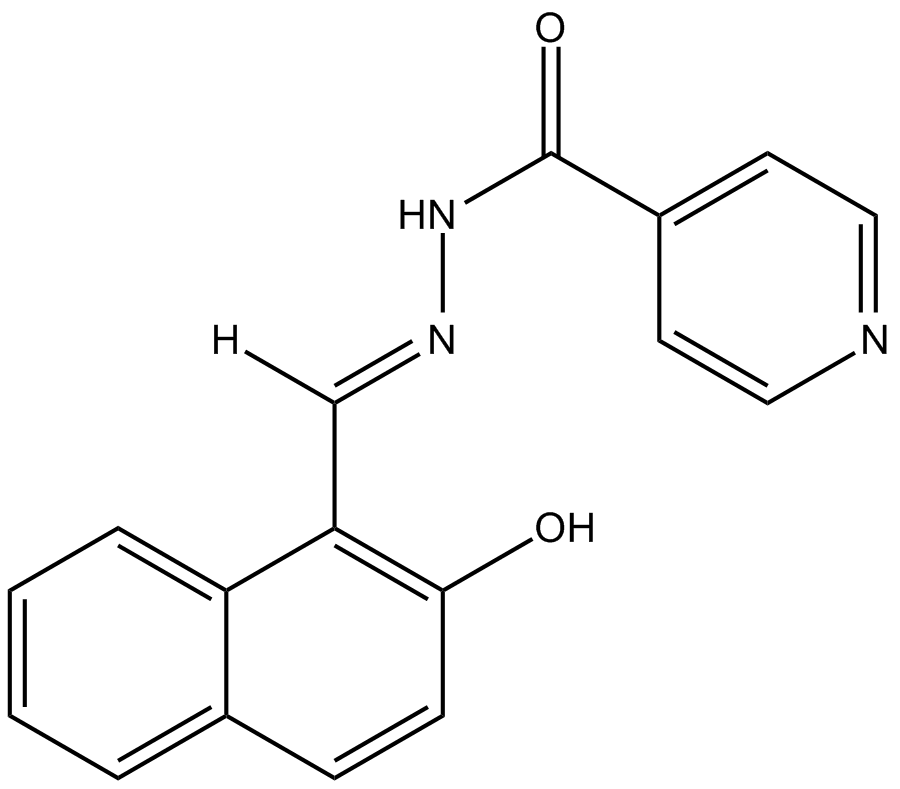
GC17820
AS8351
AS8351 (NSC51355) est un inhibiteur de KDM5B, qui peut induire et maintenir des marques de chromatine actives pour faciliter l'induction de cellules de type cardiomyocyte .
-
GC10638
AT9283
A broad spectrum kinase inhibitor
-
GC50066
Atiprimod dihydrochloride
JAK2 inhibitor
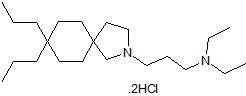
-
GC46892
ATRA-BA Hybrid
A prodrug form of all-trans retinoic acid and butyric acid
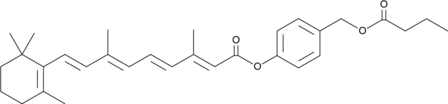
-
GN10627
Atractylenolide I
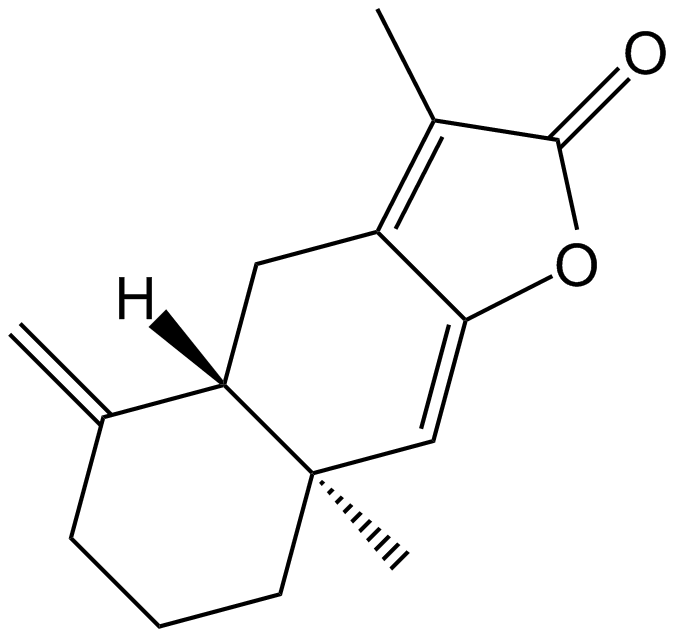
-
GC64271
AU-15330
AU-15330 est un dégradeur chimère ciblant la protéolyse (PROTAC) des sous-unités SWI/SNF ATPase, SMARCA2 et SMARCA4. L'AU-15330 induit une puissante inhibition de la croissance tumorale dans les modèles de xénogreffe du cancer de la prostate et agit en synergie avec l'enzalutamide, un antagoniste de l'AR. L'AU-15330 induit une rémission de la maladie dans des modèles de cancer de la prostate résistant À la castration (CRPC) sans toxicité.
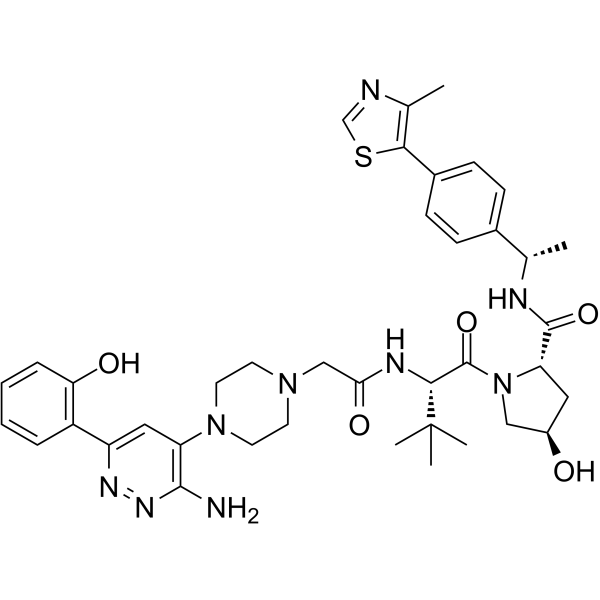
-
GC39699
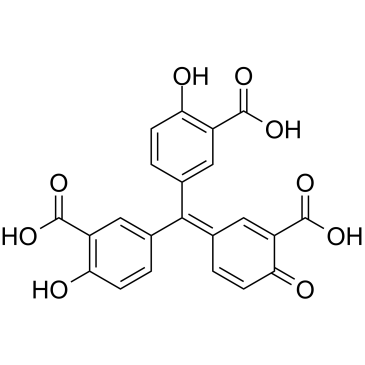
Aurintricarboxylic acid
L'acide aurintricarboxylique est un antagoniste allostérique de puissance nanomolaire avec une sélectivité envers les P2X1R et P2X3R sensibles À l'αβ-méthylène-ATP, avec des IC50 de 8,6 nM et 72,9 nM pour rP2X1R et rP2X3R, respectivement .
-
GC13332
Aurora A Inhibitor I
A potent and selective inhibitor of Aurora A kinase

-
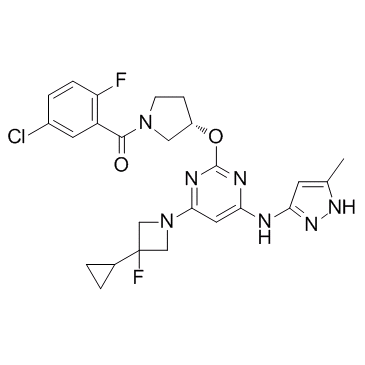
GC33379
Aurora B inhibitor 1
L'inhibiteur Aurora B 1 est un inhibiteur Aurora B (Aurora-1) extrait du brevet WO2007059299A1, composé 1-3, a une valeur Ki <0,010 uM.
-
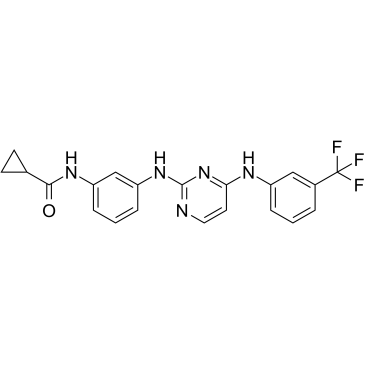
GC38441
Aurora Kinase Inhibitor 3
A potent inhibitor of Aurora A kinase
-
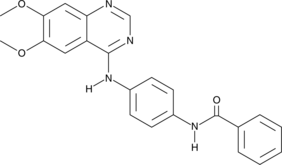
GC40667
Aurora Kinase Inhibitor II
Aurora Kinase Inhibitor II est un inhibiteur de kinase Aurora sélectif et compétitif pour l'ATP avec des IC50 de 310 nM et 240 nM pour Aurora A et Aurora B, respectivement.
-
GC15711
Aurora Kinase Inhibitor III
Aurora Kinase Inhibitor III est un inhibiteur puissant et sélectif de la kinase Aurora A avec une IC50 de 42 nM et inhibe faiblement l'EGFR avec une IC50 >10 μM.
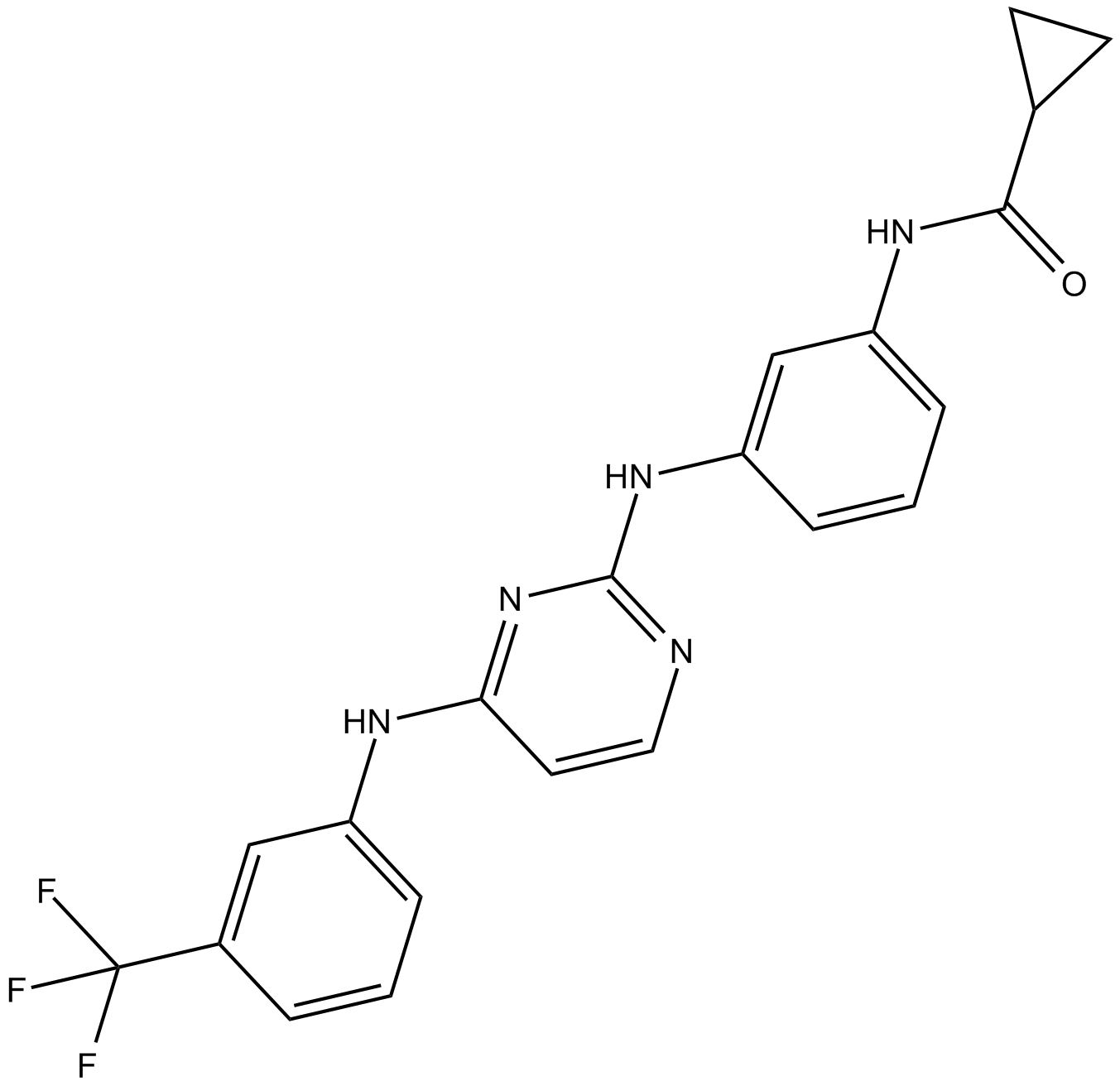
-
GC67899
Aurora kinase inhibitor-8

-
GC13433
AZ 960
A JAK2 inhibitor
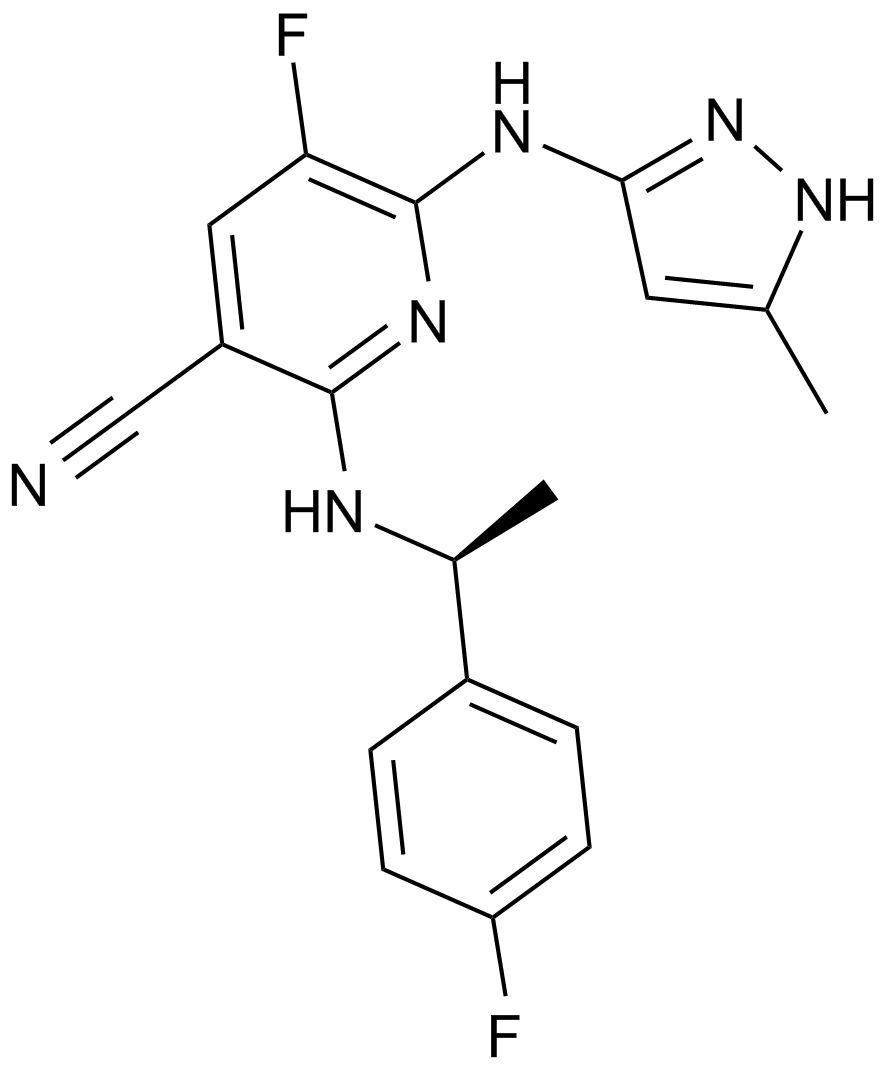
-
GC65899
AZ3391
AZ3391 est un puissant inhibiteur de PARP. AZ3391 est un dérivé de la quinoxaline. La famille d'enzymes PARP joue un rÔle important dans un certain nombre de processus cellulaires, tels que la réplication, la recombinaison, le remodelage de la chromatine et la réparation des dommages À l'ADN. AZ3391 a le potentiel pour la recherche de maladies et d'affections survenant dans les tissus du système nerveux central, tels que le cerveau et la moelle épinière (extrait du brevet WO2021260092A1, composé 23).
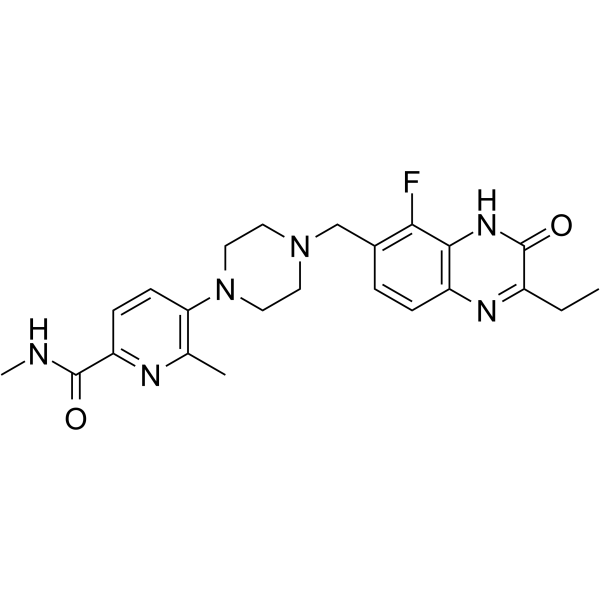
-
GC13744
AZ505
AZ505 est un inhibiteur puissant et sélectif de SMYD2 avec une IC50 de 0,12 μM.
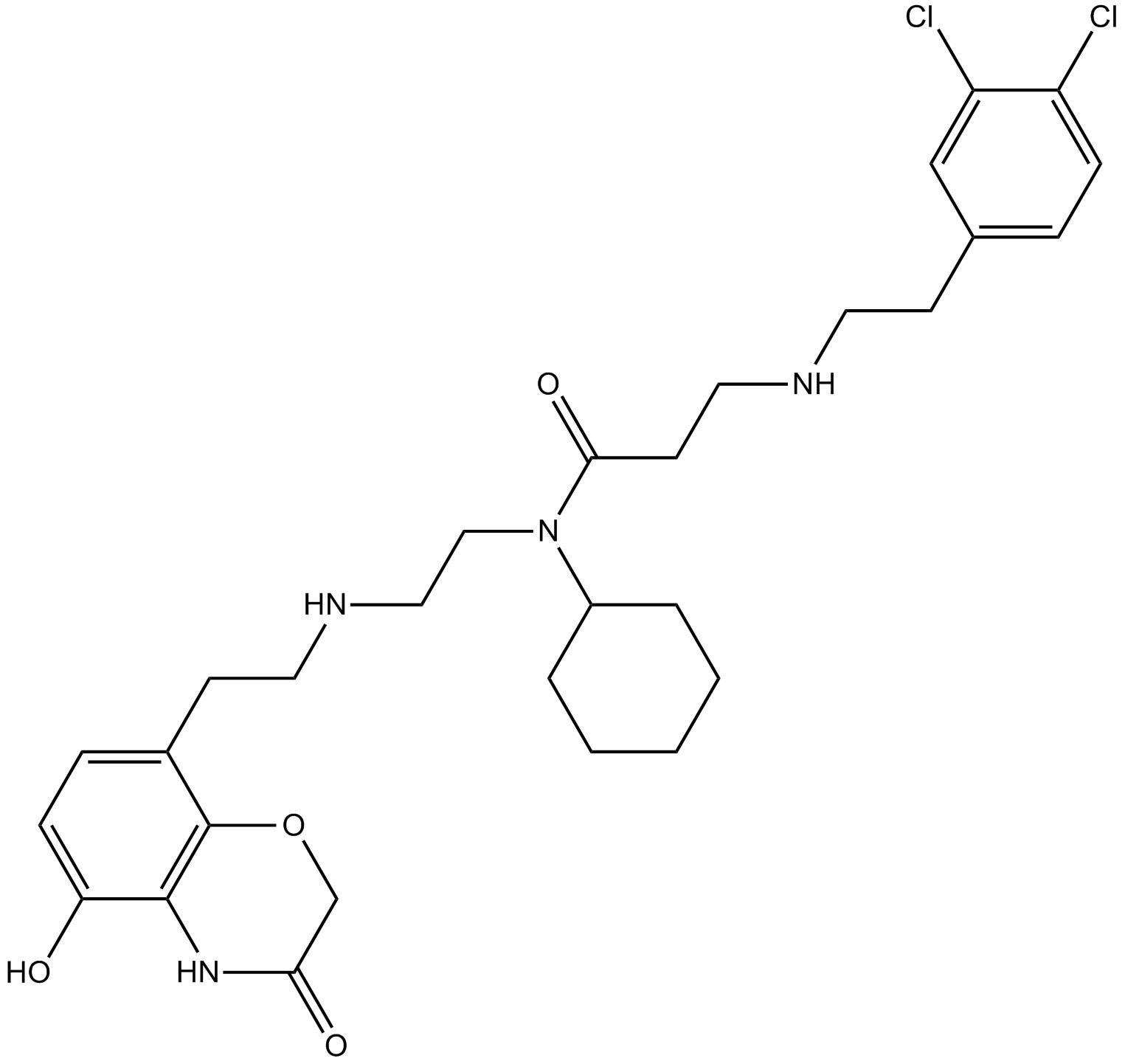
-
GC13103
AZ505 ditrifluoroacetate
Le ditrifluoroacétate d'AZ505 est un inhibiteur puissant et sélectif de SMYD2 avec une IC50 de 0,12 μM.
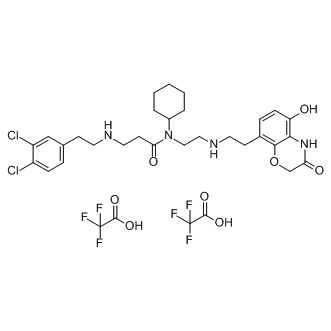
-
GC64124
AZ506
AZ506 est un puissant inhibiteur de SMYD2 avec une IC50 de 17 nM. AZ506 inhibe l'activité de la méthyltransférase SMYD2 dans les cellules, entraÎnant une diminution du signal de méthylation médié par SMYD2.
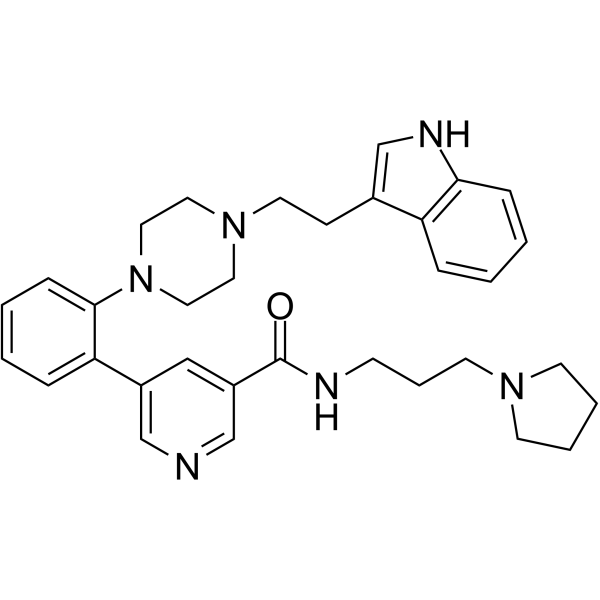
-
GC16725
AZ6102
AZ6102 est un puissant inhibiteur double de TNKS1 et TNKS2, avec des IC50 de 3 nM et 1 nM, respectivement, et a également une sélectivité 100 fois supérieure contre d'autres enzymes de la famille PARP, avec des IC50 de 2,0 μM, 0,5 μM et> 3 μM, pour PARP1 , PARP2 et PARP6, respectivement.
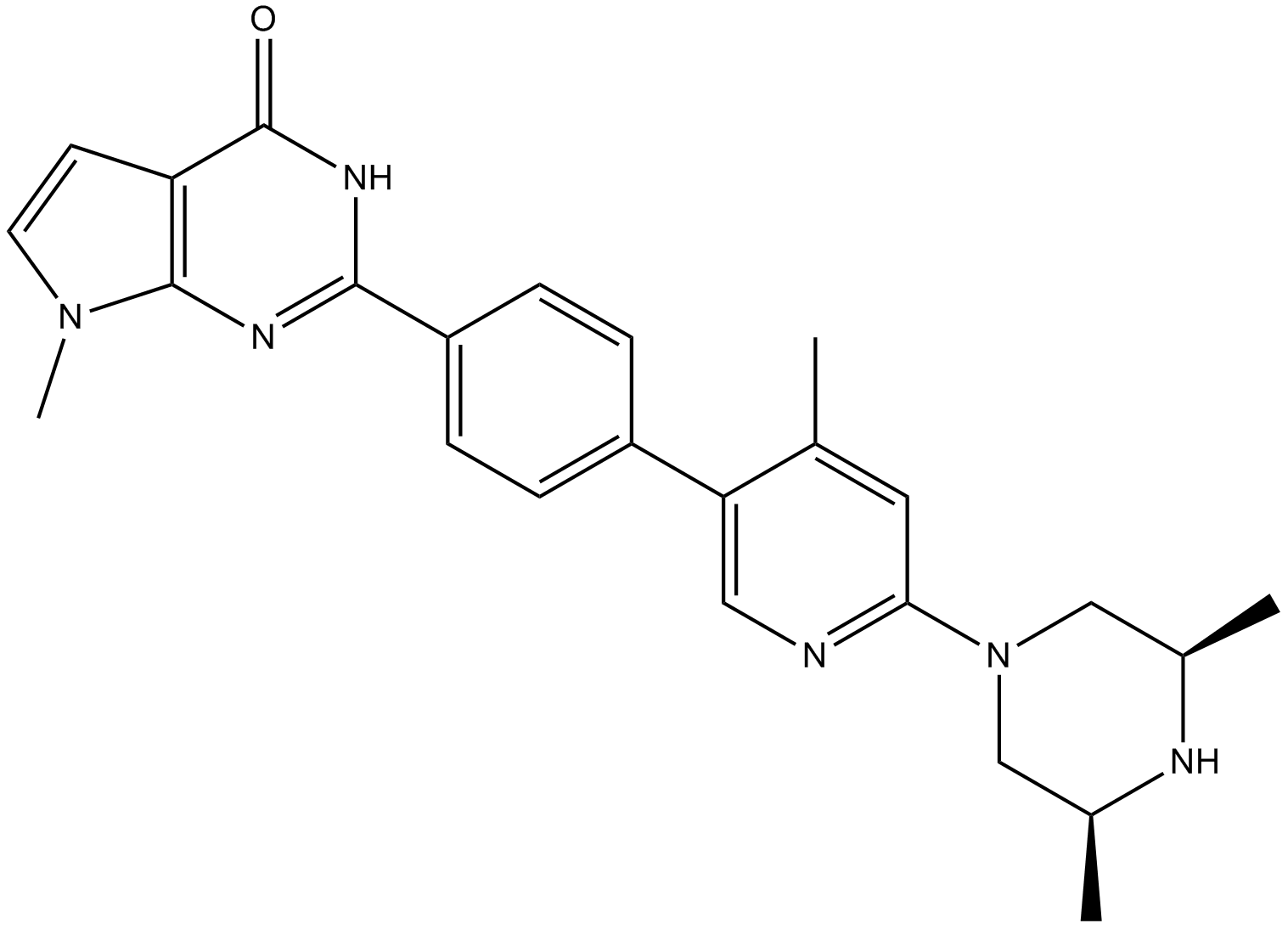
-
GC46900
AZ9482
AZ9482 est un triple inhibiteur de PARP1/2/6, avec des valeurs IC50 de 1 nM, 1 nM et 640 nM pour PARP1, PARP2 et PARP6, respectivement.
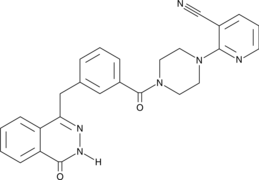
-
GC48971
AZD 1152 (hydrochloride)
A prodrug for a potent Aurora B inhibitor
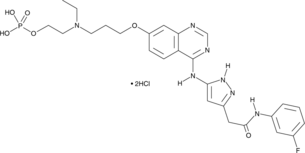
-
GC13014
AZD 5153
orally available, bivalent inhibitor of the bromodomain and extraterminal (BET) protein BRD4.
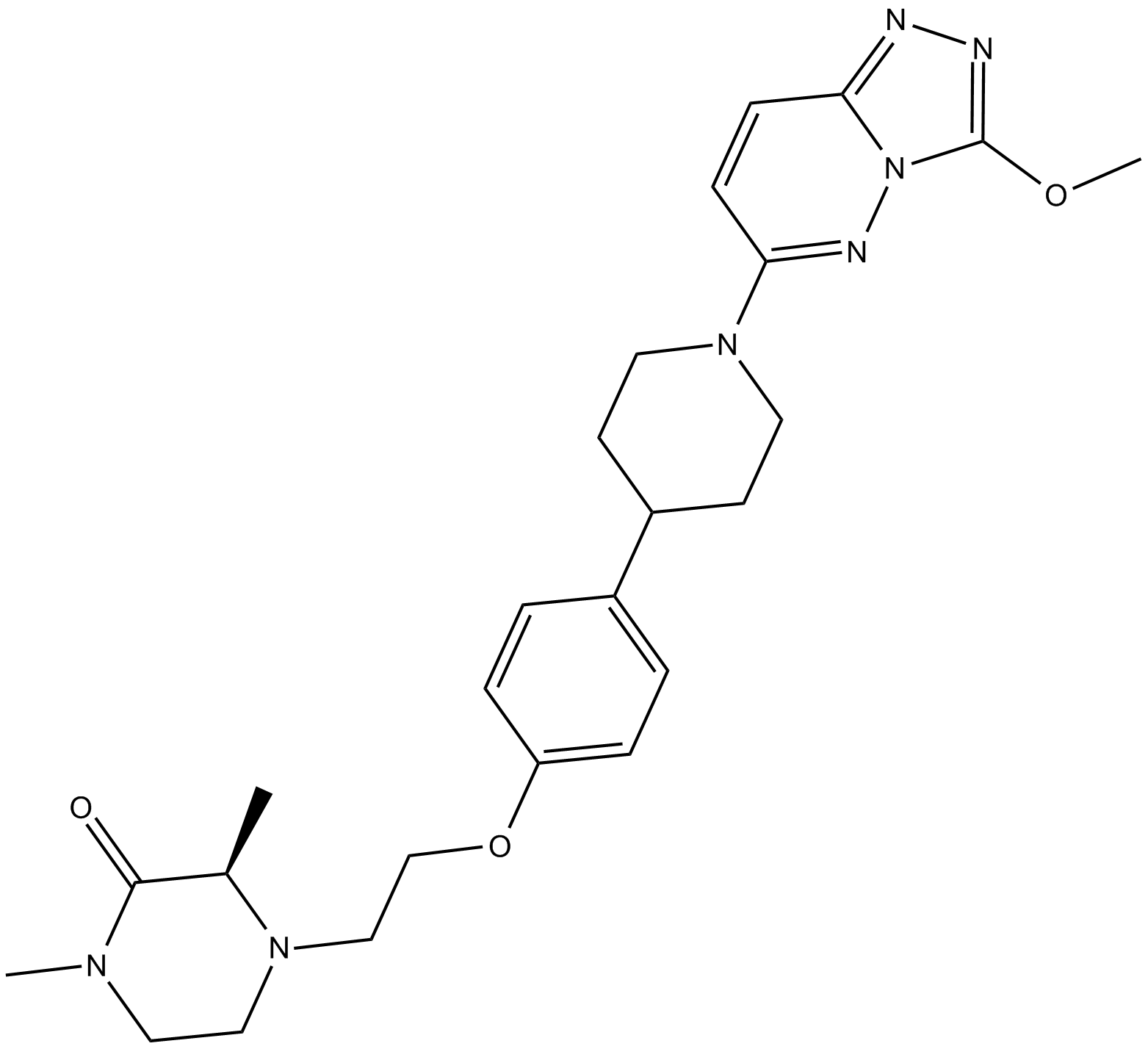
-
GC14709
AZD1152
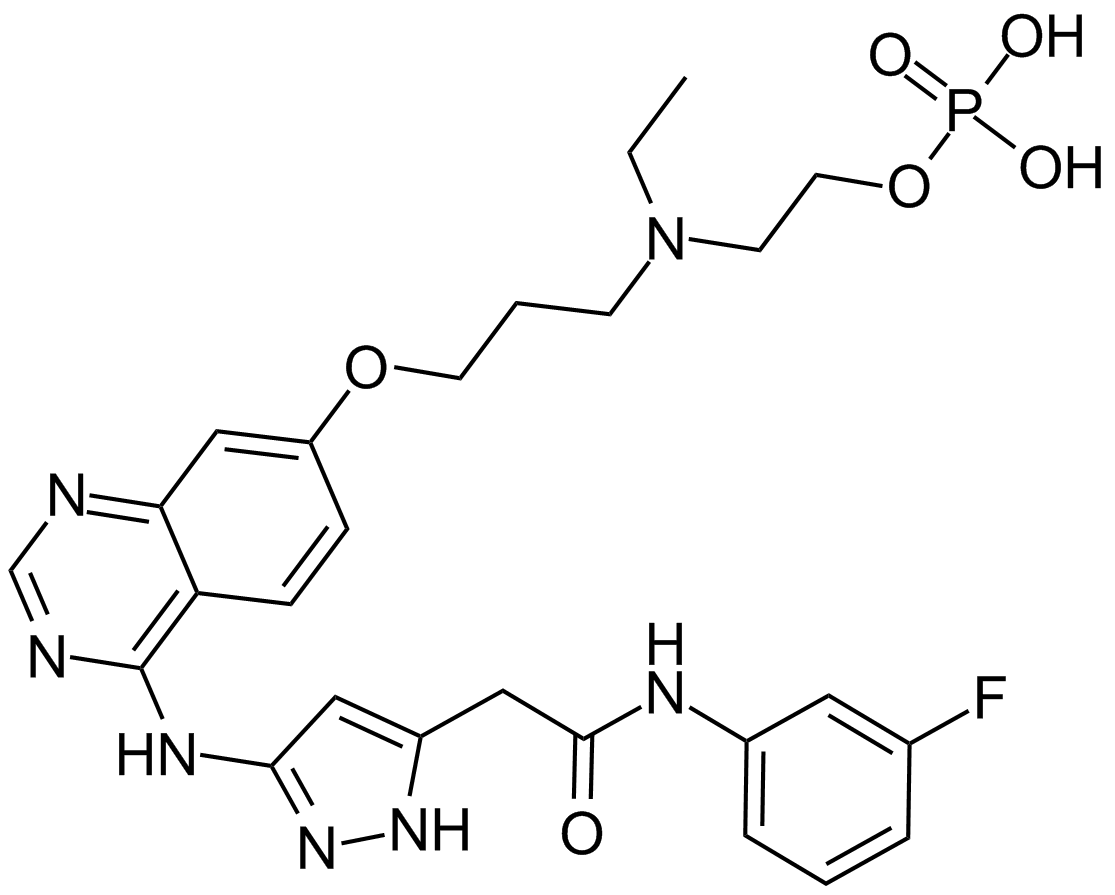
-
GC12660
AZD1208
A pan-Pim kinase inhibitor
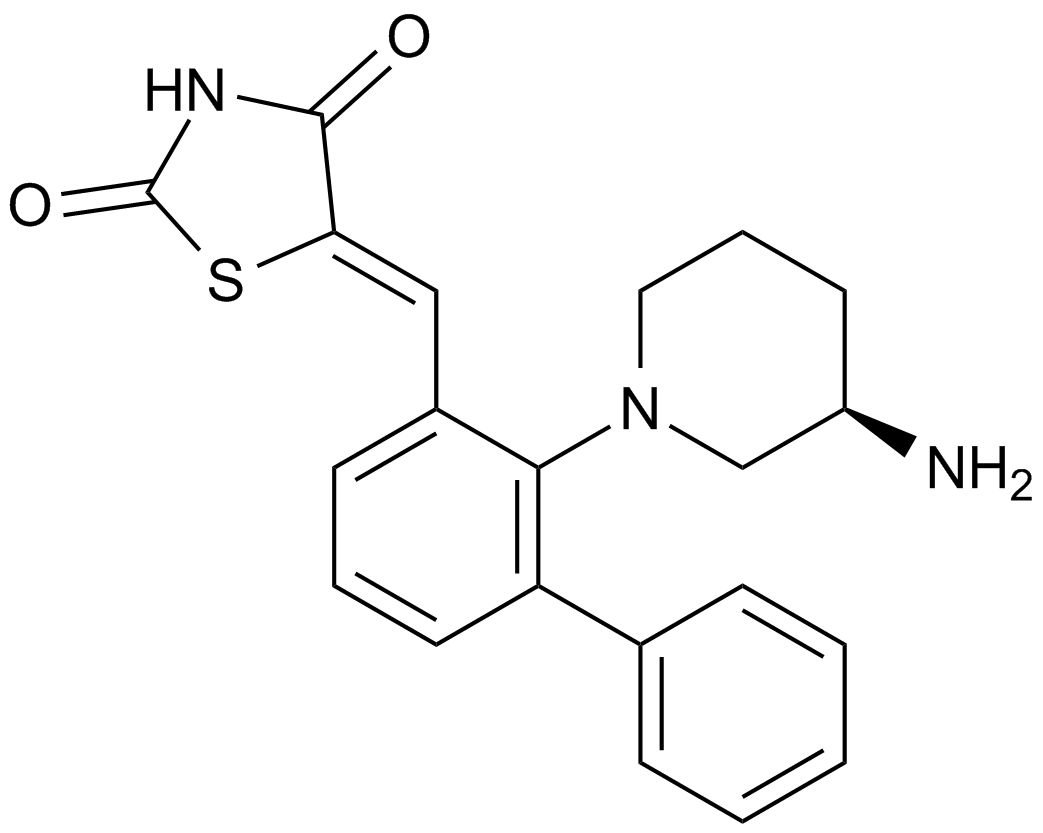
-
GC35447
AZD1208 hydrochloride
Le chlorhydrate d'AZD1208 est un inhibiteur des kinases PIM hautement sélectif et biodisponible par voie orale.
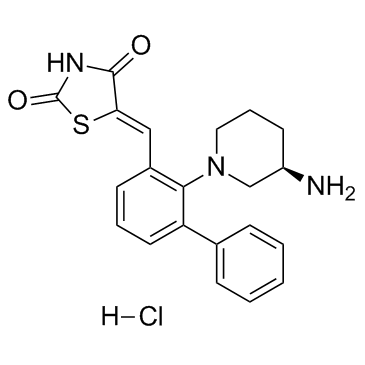
-
GC12504
AZD1480
A potent JAK2 inhibitor
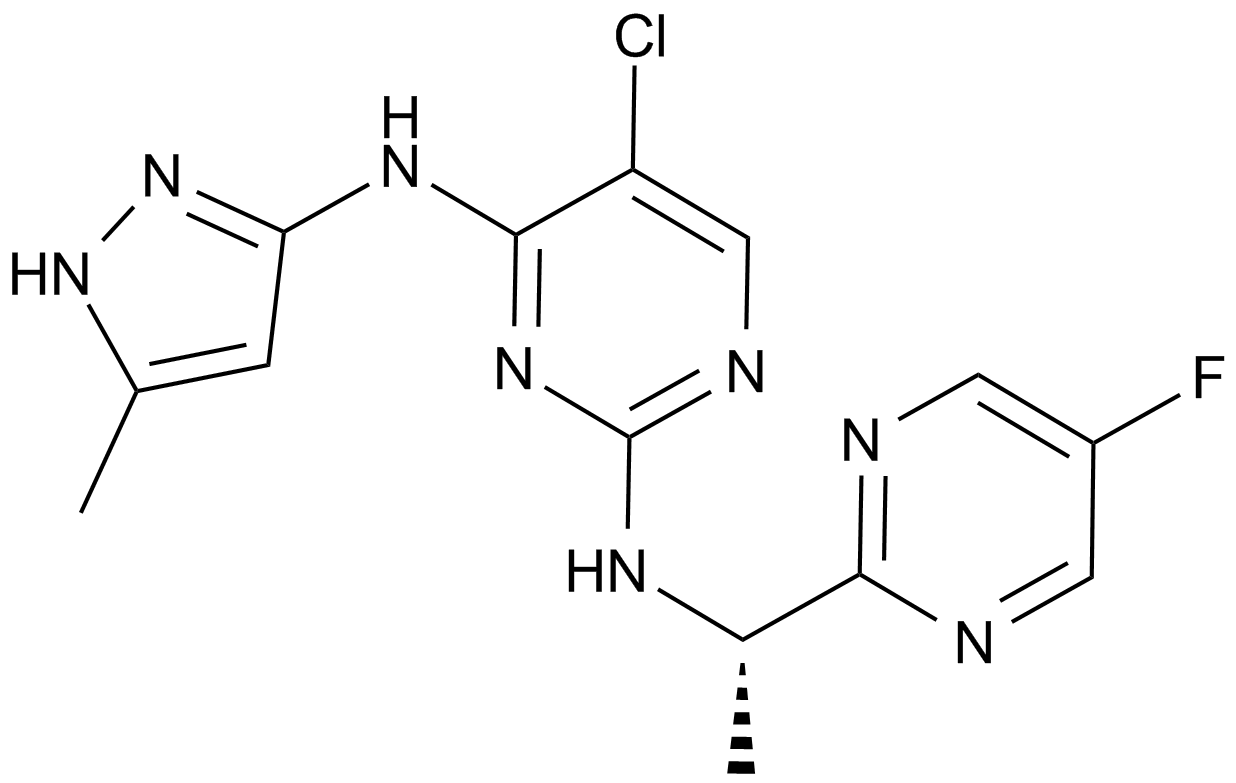
-
GC17965
AZD2461
A PARP inhibitor
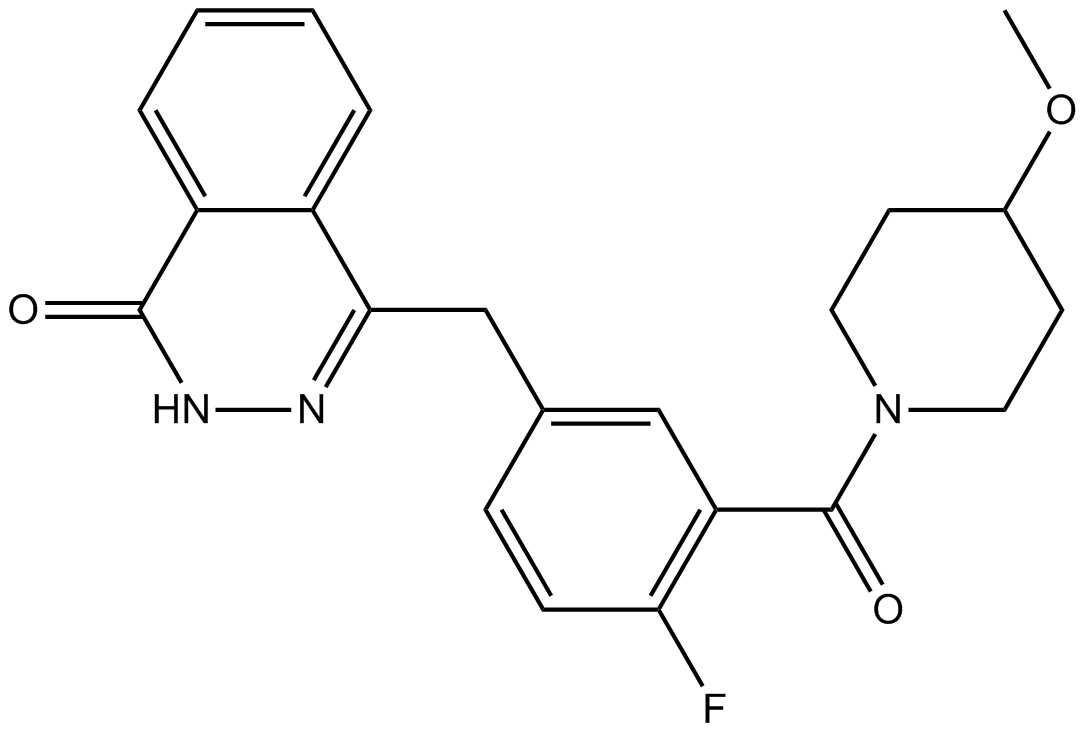
-
GC35448
AZD5153 6-Hydroxy-2-naphthoic acid
AZD5153 L'acide 6-hydroxy-2-naphtoÏque est l'acide 6-hydroxy-2-naphtoÏque de l'AZD5153. AZD5153 est un inhibiteur de bromodomaine BET/BRD4 puissant, sélectif et disponible par voie orale; perturbe BRD4 avec une IC50 de 1,7 nM.
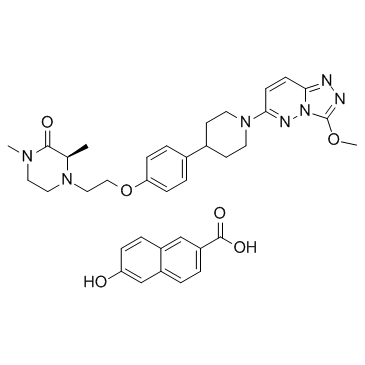
-
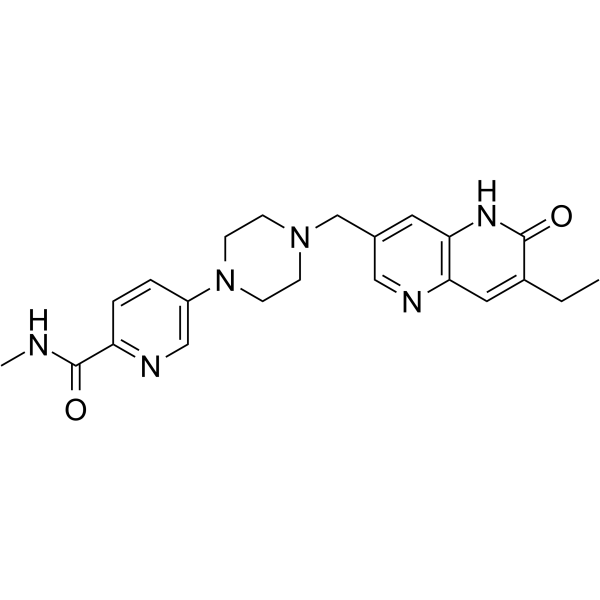
GC62310
AZD5305
AZD5305 est un inhibiteur PARP actif puissant, sélectif et oral. AZD5305 est puissant et efficace dans les xénogreffes animales et les modèles PDX.
-
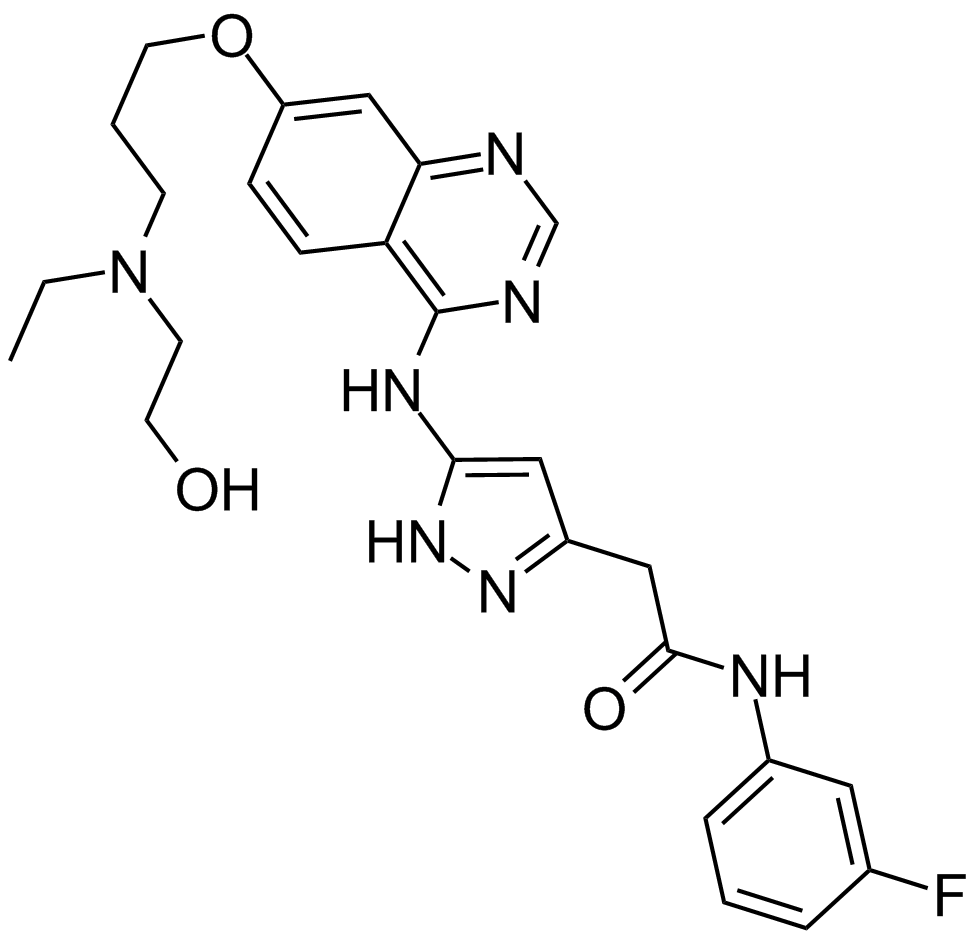
GC14955
Barasertib (AZD1152-HQPA)
A selective Aurora kinase B inhibitor
-
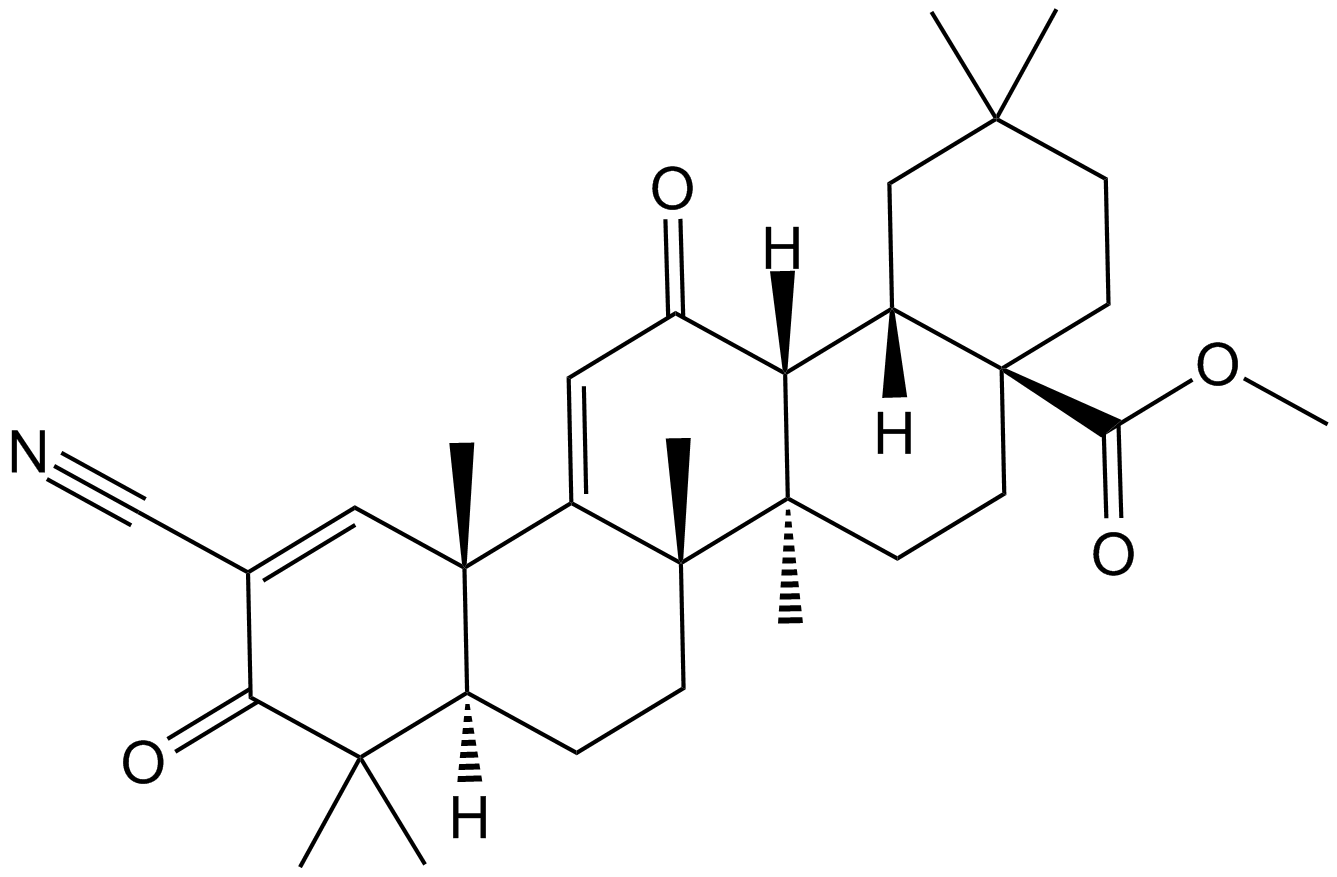
GC11572
Bardoxolone methyl
A synthetic triterpenoid with potent anticancer and antidiabetic activity
-
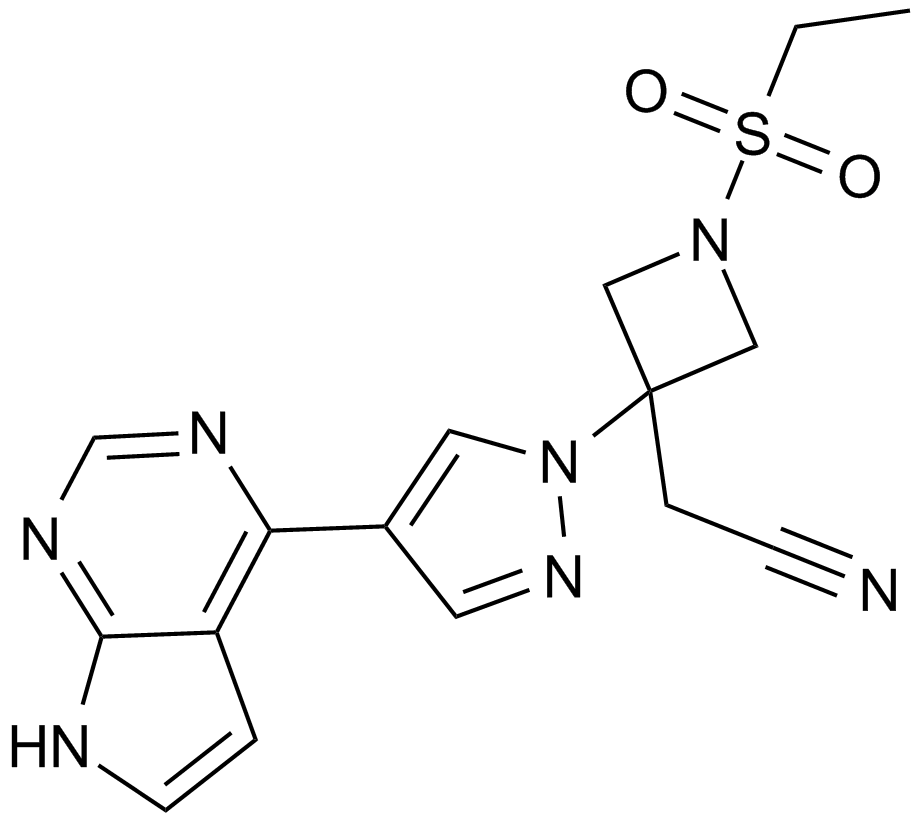
GC14844
Baricitinib (LY3009104, INCB028050)
A JAK1 and JAK2 inhibitor
-
GC13830
Baricitinib phosphate
A JAK1 and JAK2 inhibitor

-
GC12698
BAY 87-2243
BAY 87-2243 est un inhibiteur du facteur 1 induit par l'hypoxie (HIF-1) très puissant et sélectif.
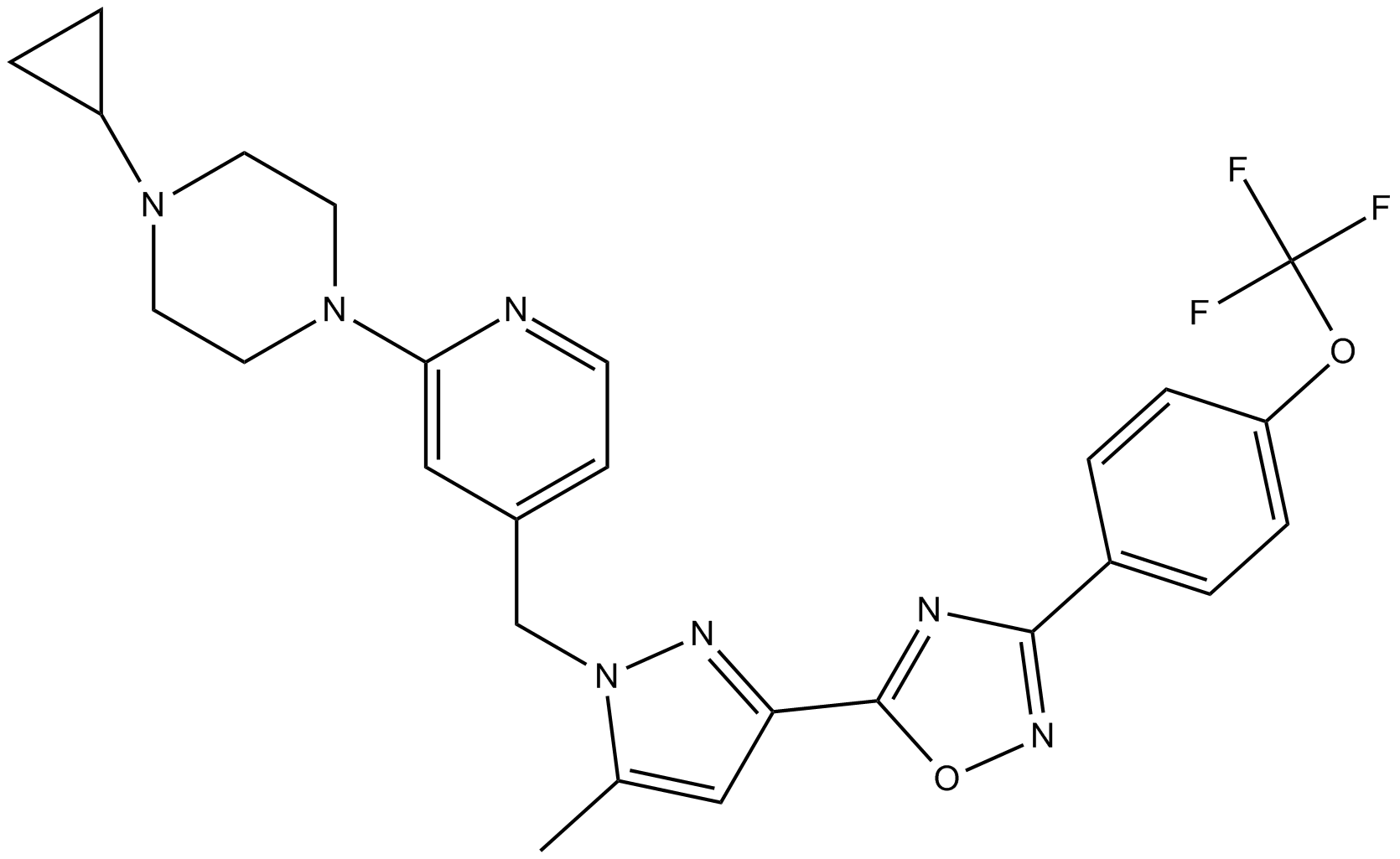
-
GC18508
BAY-299
BAY-299 est un double inhibiteur très puissant avec des IC50 de 67 nM pour les bromodomaines BRPF2 (BD), 8 nM pour TAF1 BD2 et 106 nM pour TAF1L BD2.
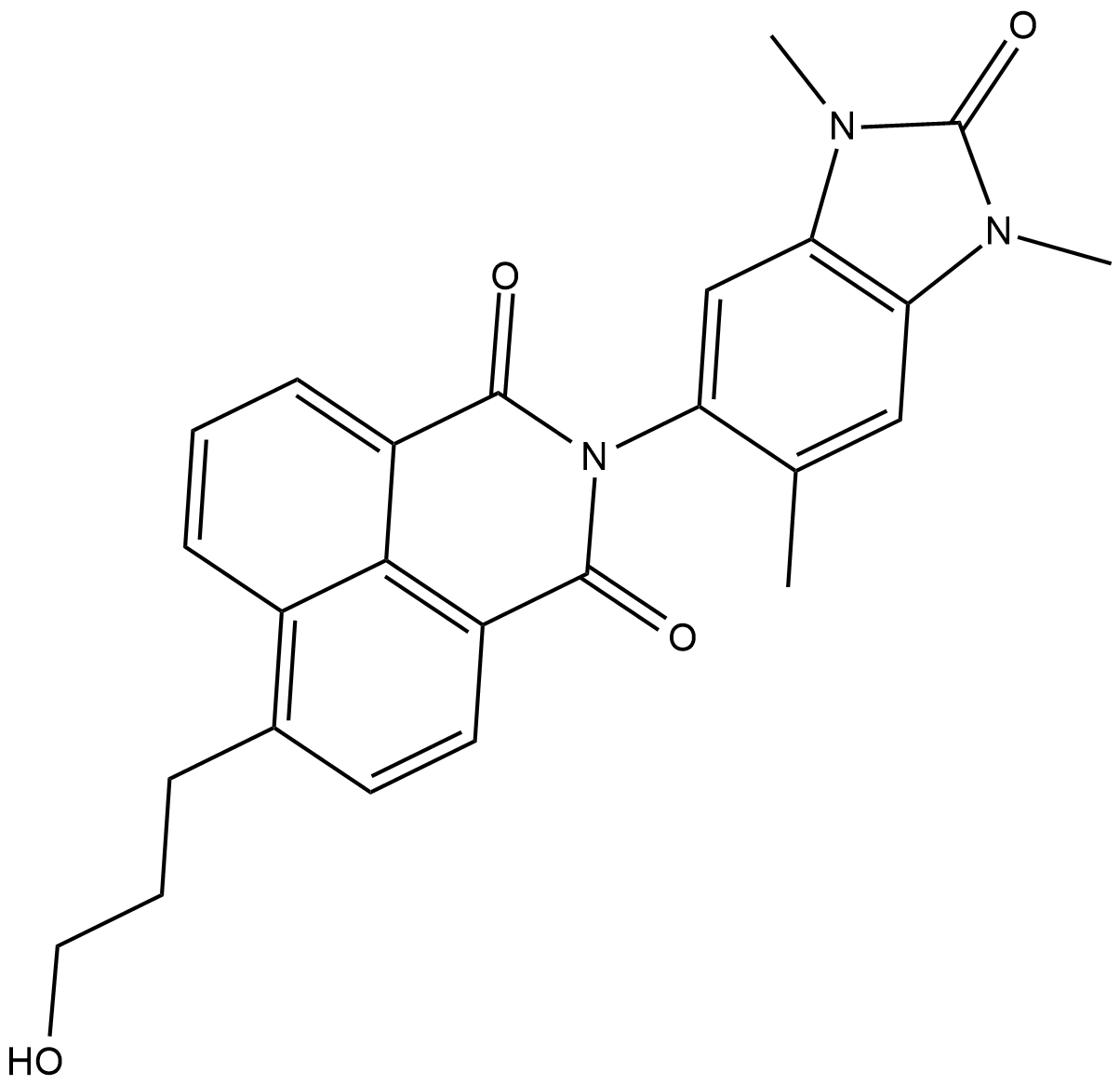
-
GC18159
BAY-598
BAY-598 est un inhibiteur sélectif À petite molécule de SMYD2 avec une IC50 de 27 nM.
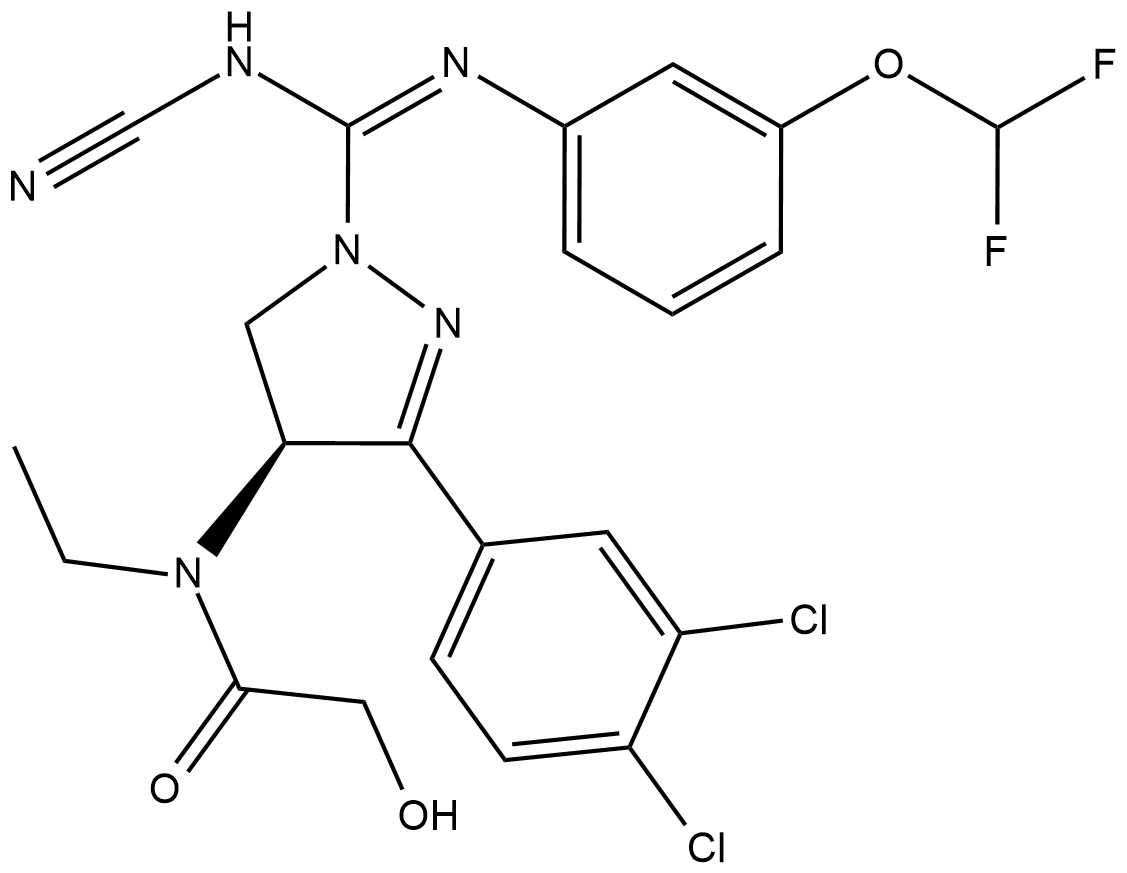
-
GC45389
BAY-6035
BAY-6035 est un inhibiteur puissant, sélectif et compétitif du substrat de SMYD3. BAY-6035 inhibe la méthylation du peptide MEKK2 avec une IC50 de 88 nM.
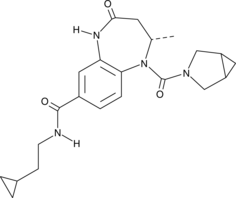
-
GC33104
BAY1238097
BAY1238097 est un inhibiteur puissant et sélectif de la liaison de BET aux histones et possède une forte activité antiproliférative dans différents modèles de LMA (leucémie myéloÏde aiguë) et de MM (myélome multiple) grÂce À la régulation À la baisse des niveaux de c-Myc et de son transcriptome en aval (IC50 < 100nM).
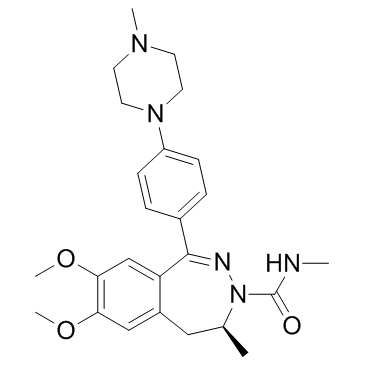
-
GC63694
BAZ1A-IN-1
BAZ1A-IN-1 est un puissant inhibiteur de BAZ1A (protéine contenant du bromodomaine). BAZ1A-IN-1 montre une valeur KD de 0,52 μM contre le bromodomaine BAZ1A. BAZ1A-IN-1 montre une bonne activité anti-viabilité contre les lignées cellulaires cancéreuses exprimant un niveau élevé de BAZ1A, mais une activité faible ou nulle contre les cellules cancéreuses avec un faible niveau d'expression de BAZ1A.
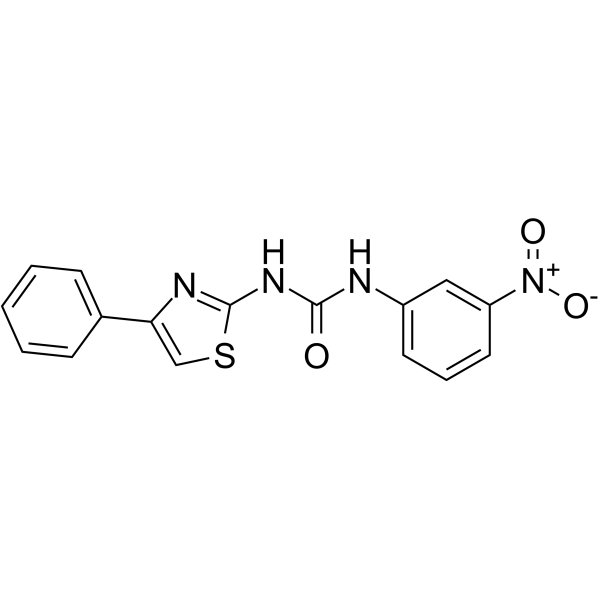
-
GC15175
BAZ2-ICR
BAZ2-ICR est un inhibiteur des bromodomaines BAZ2A/B puissant, sélectif, actif sur les cellules et actif par voie orale avec des IC50 de 130 nM et 180 nM, et des Kd de 109 nM et 170 nM, respectivement. BAZ2-ICR montre une sélectivité de 10 À 15 fois pour la liaison de BAZ2A/B sur CECR2 et une sélectivité > 100 fois sur tous les autres bromodomaines. BAZ2-ICR est une sonde chimique épigénétique.
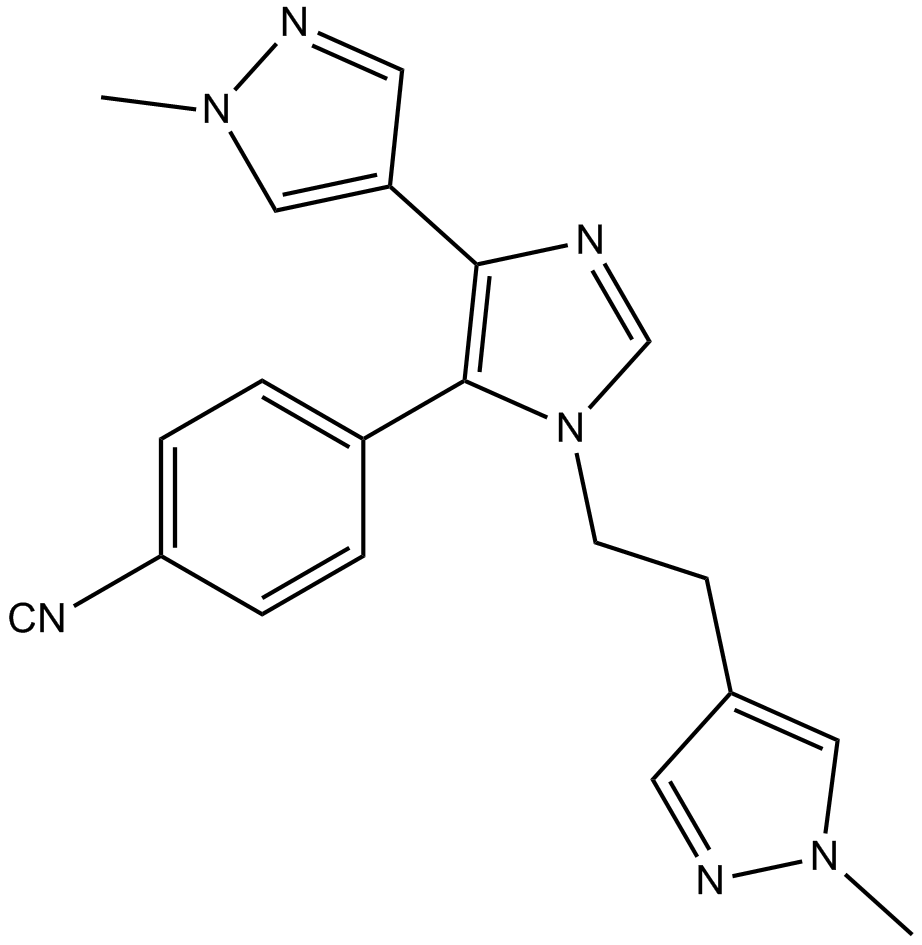
-
GC35480
BB-Cl-Amidine hydrochloride
Le chlorhydrate de BB-Cl-Amidine est un inhibiteur de peptidylarginine déminase (PAD).
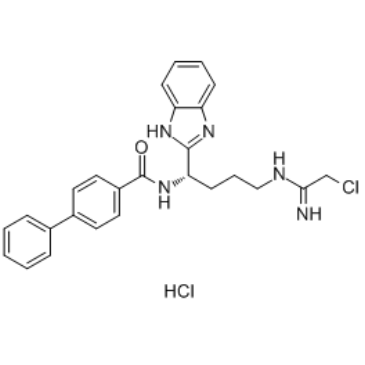
-
GC42910
BB-F-Yne
BB-F-Yne is a cell-permeable derivative of the protein arginine diminase (PAD) inhibitor BB-Cl-amidine that contains an alkyne moiety for use in click chemistry reactions.
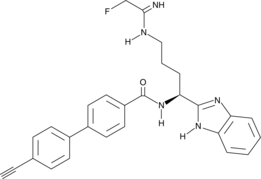
-
GC18161
BCI-121
Le BCI-121 est un inhibiteur de SMYD3 qui entrave la prolifération des cellules cancéreuses.
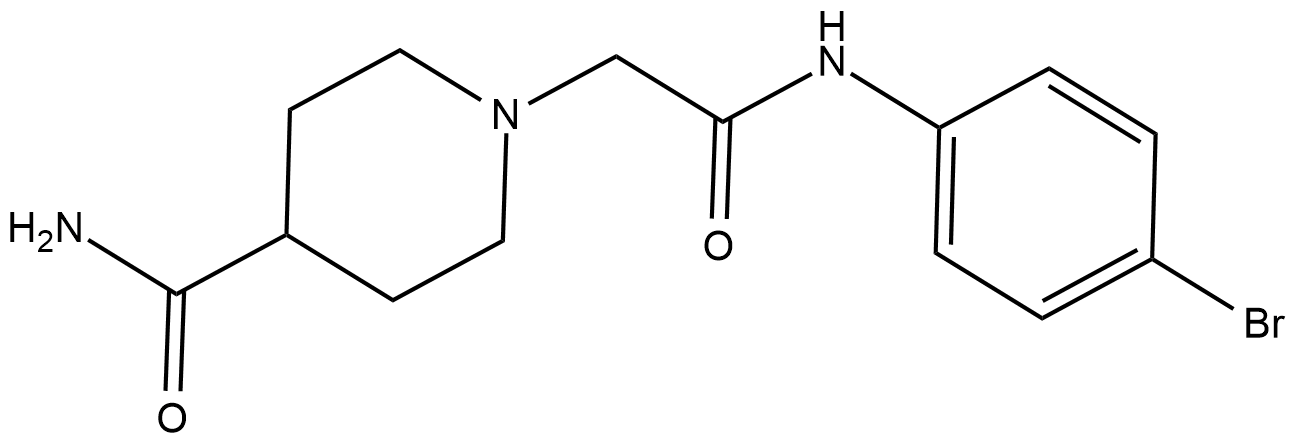
-
GC15962
Belinostat (PXD101)
Belinostat is an effective HDAC inhibitor with an IC50 of 27 nM in HeLa cell extracts.
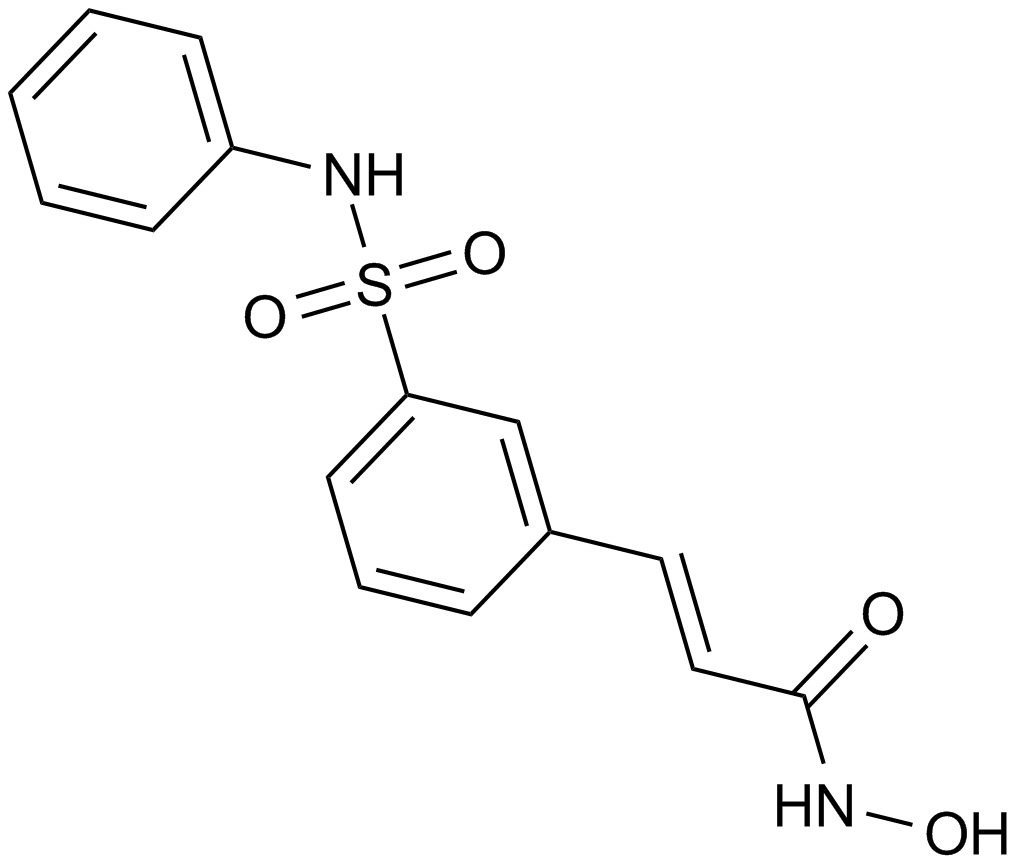
-
GC12844
Benzamide
Le benzamide (benzènecarboxamide) est un puissant inhibiteur de la poly(ADP-ribose) polymérase (PARP).
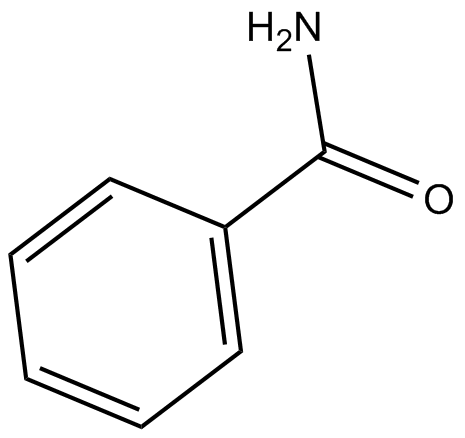
-
GC30763
Benzenebutyric acid (4-Phenylbutyric acid)
L'acide benzènebutyrique (acide 4-phénylbutyrique) (4-PBA) est un inhibiteur de HDAC et du stress endoplasmique (ER), utilisé dans la recherche sur le cancer et les infections.
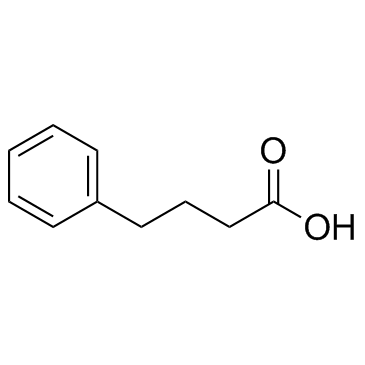
-
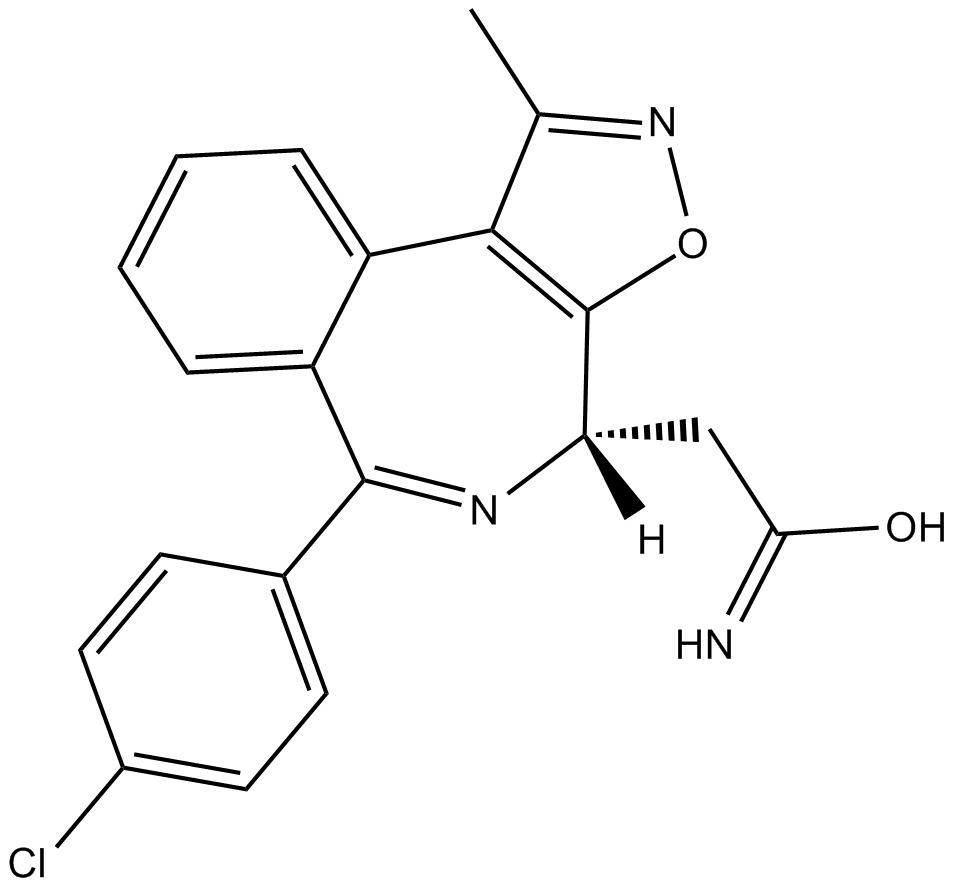
GC16299
BET bromodomain inhibitor
L'inhibiteur de bromodomaine BET est un puissant inhibiteur de BET extrait du brevet WO/2015/153871A2, exemple de composé 11.
-
GC63528
BET bromodomain inhibitor 1
L'inhibiteur 1 du bromodomaine BET est un inhibiteur sélectif du bromodomaine et du bromodomaine extra-terminal (BET) actif par voie orale avec une IC50 de 2,6 nM pour BRD4. L'inhibiteur de bromodomaine BET 1 se lie À BRD2(2), BRD3(2), BRD4(1), BRD4(2) et BRDT(2) avec des affinités élevées (valeurs Kd de 1,3 nM, 1,0 nM, 3,0 nM, 1,6 nM, 2,1 nM, respectivement). l'inhibiteur de bromodomaine 1 a une activité anticancéreuse.
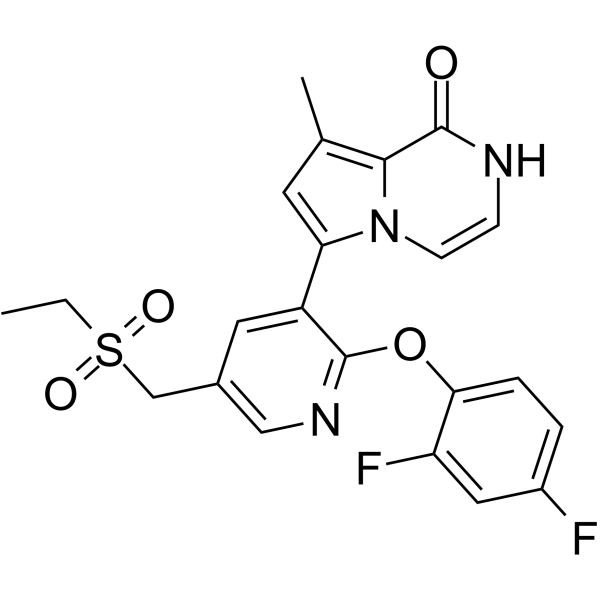
-
GC35505
BET-BAY 002
BET-BAY 002 est un puissant inhibiteur de BET; montre son efficacité dans un modèle de myélome multiple.
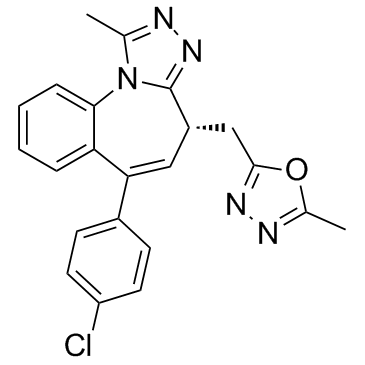
-
GC35506
BET-BAY 002 S enantiomer
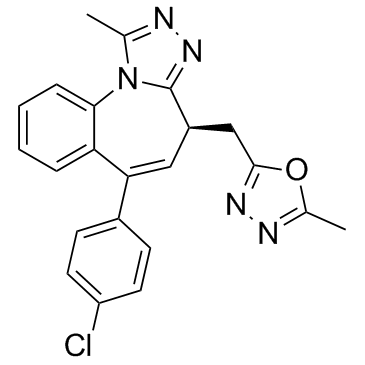
-
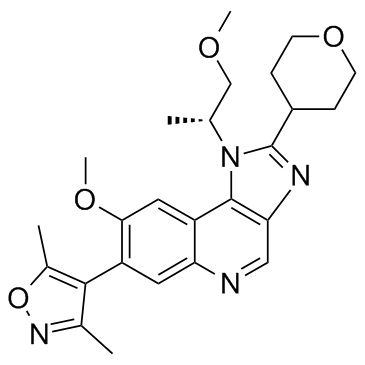
GC31831
BET-IN-1
BET-IN-1 est un pan-inhibiteur des huit bromodomaines BET et une sélectivité par rapport À d'autres protéines représentatives contenant des bromodomaines.
-
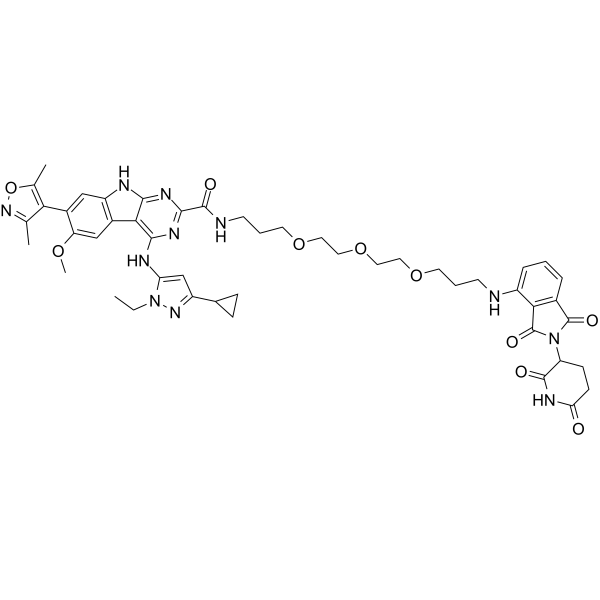
GC65506
BETd-246
BETd-246 est un inhibiteur du bromodomaine BET (BRD) de deuxième génération et basé sur PROTAC, connecté par des ligands pour Cereblon et BET, présentant une sélectivité, une puissance et une activité antitumorale supérieures.
-
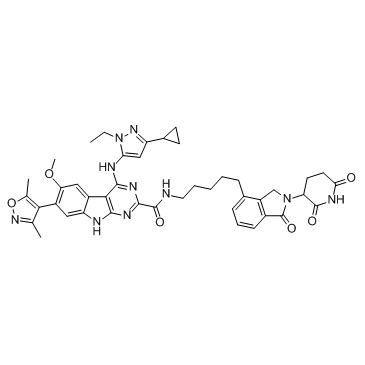
GC32791
BETd-260 (ZBC 260)
BETd-260 (ZBC 260) (ZBC 260) est un PROTAC connecté par des ligands pour Cereblon et BET, avec aussi peu que 30 pM contre la protéine BRD4 dans la lignée cellulaire de leucémie RS4;11. BETd-260 (ZBC 260) supprime puissamment la viabilité cellulaire et induit de manière robuste l'apoptose dans les cellules de carcinome hépatocellulaire (HCC).
-
GC12074
BG45
BG45 est un inhibiteur de HDAC de classe I avec une sélectivité pour HDAC3 (IC50 = 289 nM).
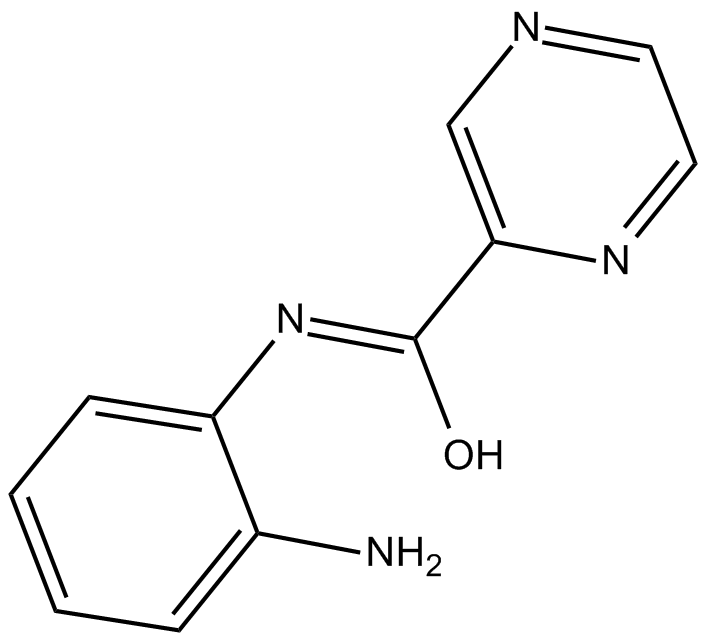
-
GC14380
BGP-15
BGP-15 est un inhibiteur de PARP, avec un IC50 et un Ki de 120 et 57 μM, respectivement.
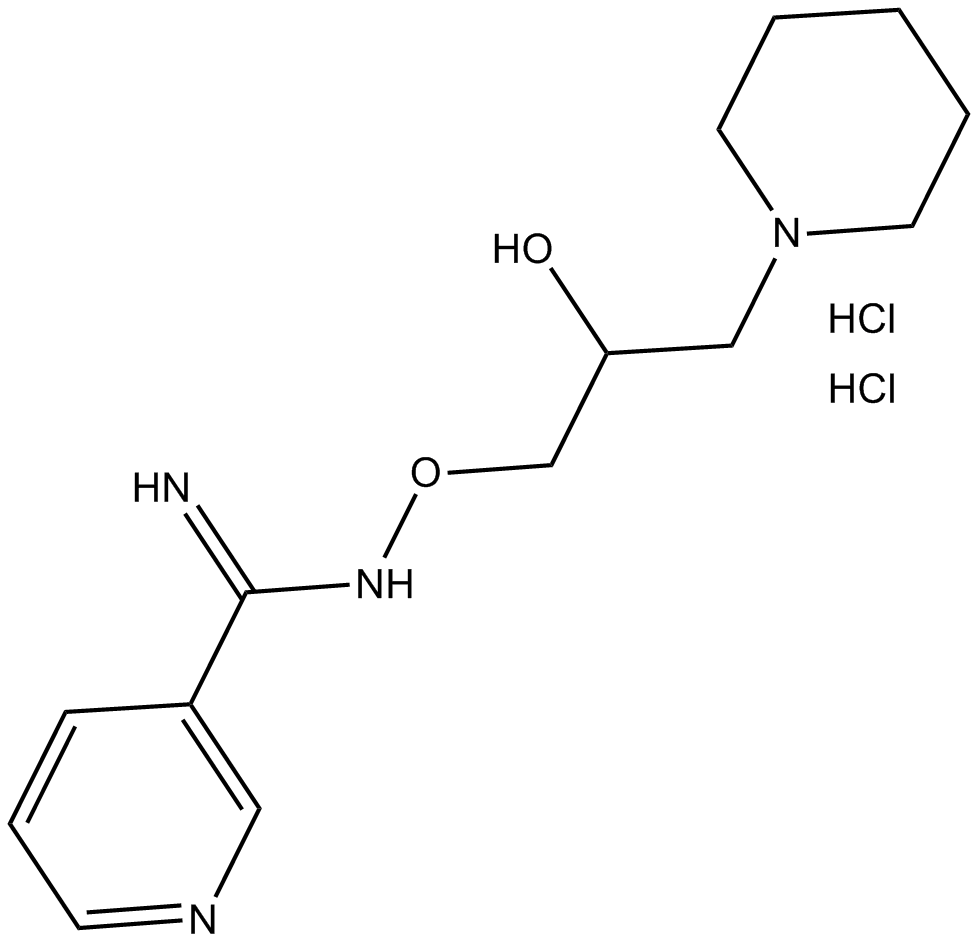
-
GC12450
BI 2536
Un inhibiteur puissant de Plk1
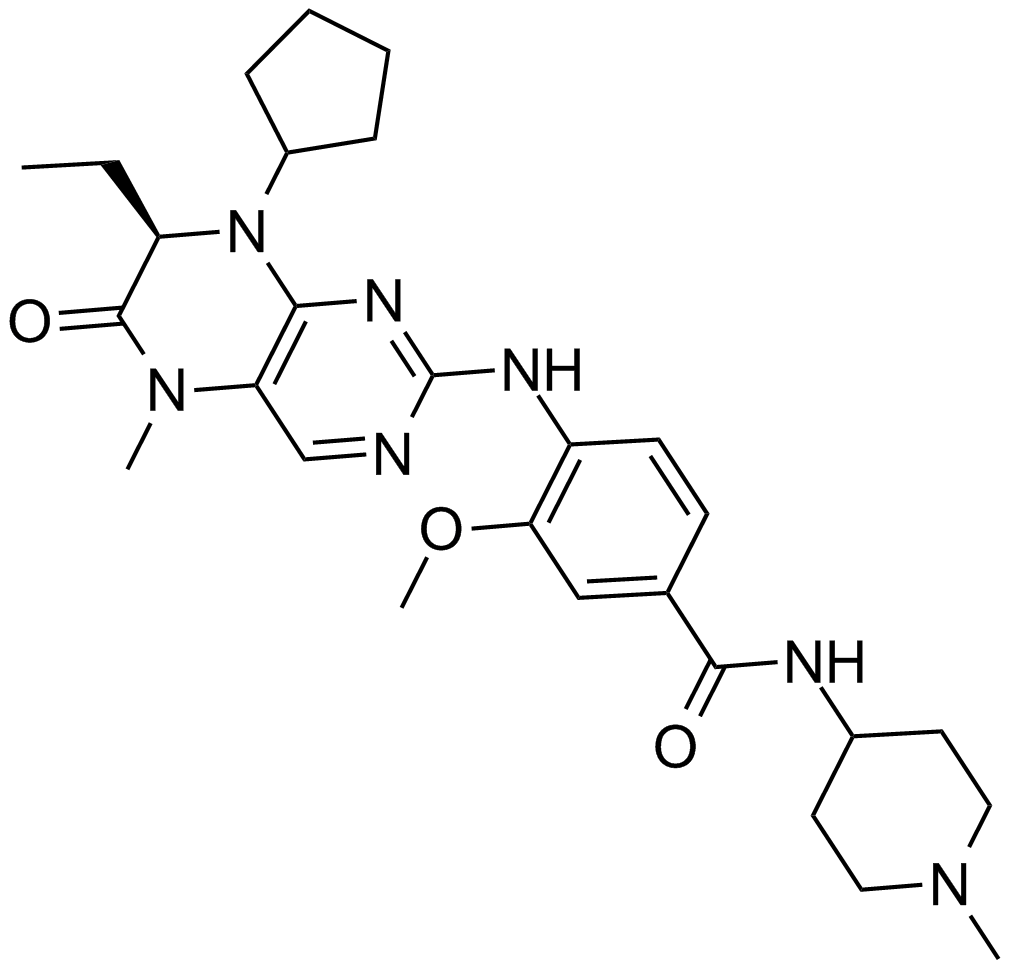
-
GC50540
BI 9321
Nuclear receptor-binding SET domain (NSD) 3 antagonist; selectively binds PWWP1 domain
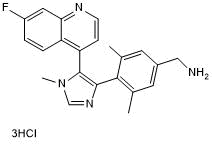
-
GC16726
BI-7273
BI-7273 est un inhibiteur de BRD9 sélectif et perméable aux cellules, avec une IC50 et un Kd de 19 et 0,75 nM; montre également un effet élevé sur BRD7, avec une IC50 et un Kd de 117 nM et 0,3 nM.
-
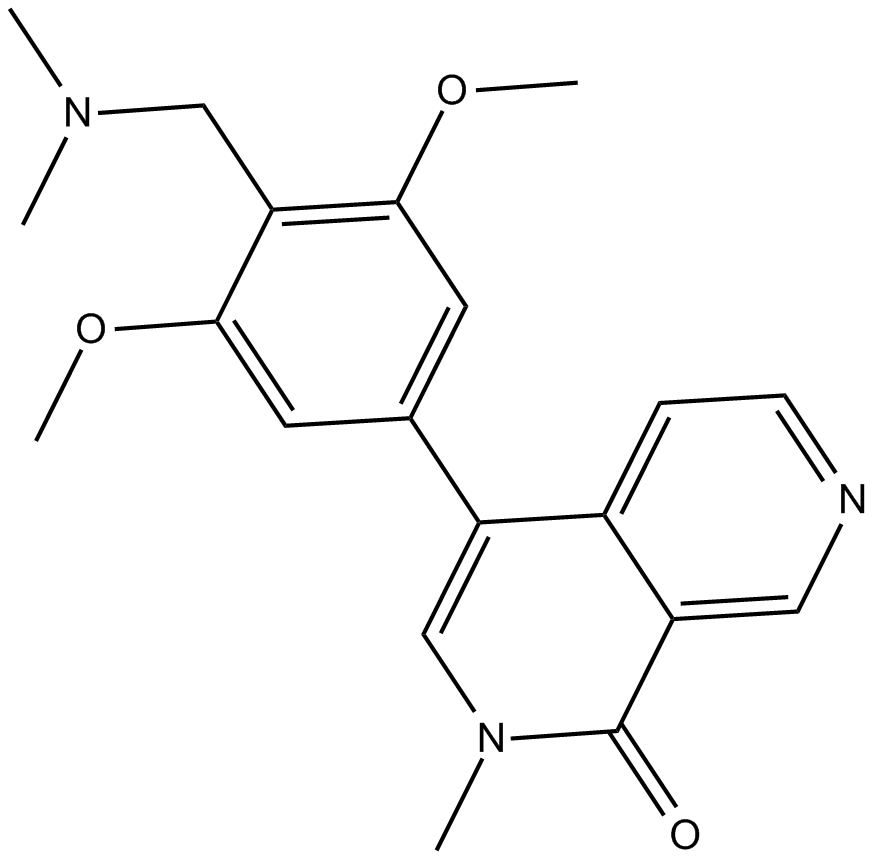
GC17828
BI-847325
Le BI-847325 est un double inhibiteur compétitif de l'ATP des kinases MEK et Aurora (AK) avec des valeurs IC50 de 4 et 15 nM pour la MEK2 humaine et l'AK-C, respectivement.

-
GC39663
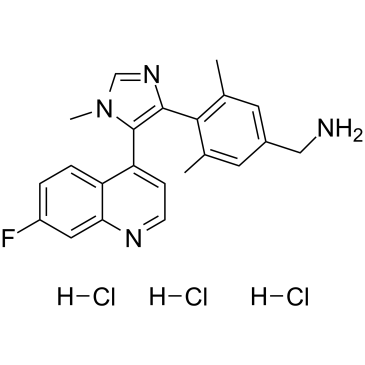
BI-9321 trihydrochloride
Le trichlorhydrate de BI-9321 est un antagoniste du domaine SET 3 (NSD3)-PWWP1 puissant, sélectif et actif cellulaire avec une valeur Kd de 166 nM. Le trichlorhydrate de BI-9321 est inactif contre NSD2-PWWP1 et NSD3-PWWP2. Le trichlorhydrate de BI-9321 perturbe spécifiquement les interactions des histones du domaine NSD3-PWWP1 avec une IC50 de 1,2 μM dans les cellules U2OS.
-
GC16817
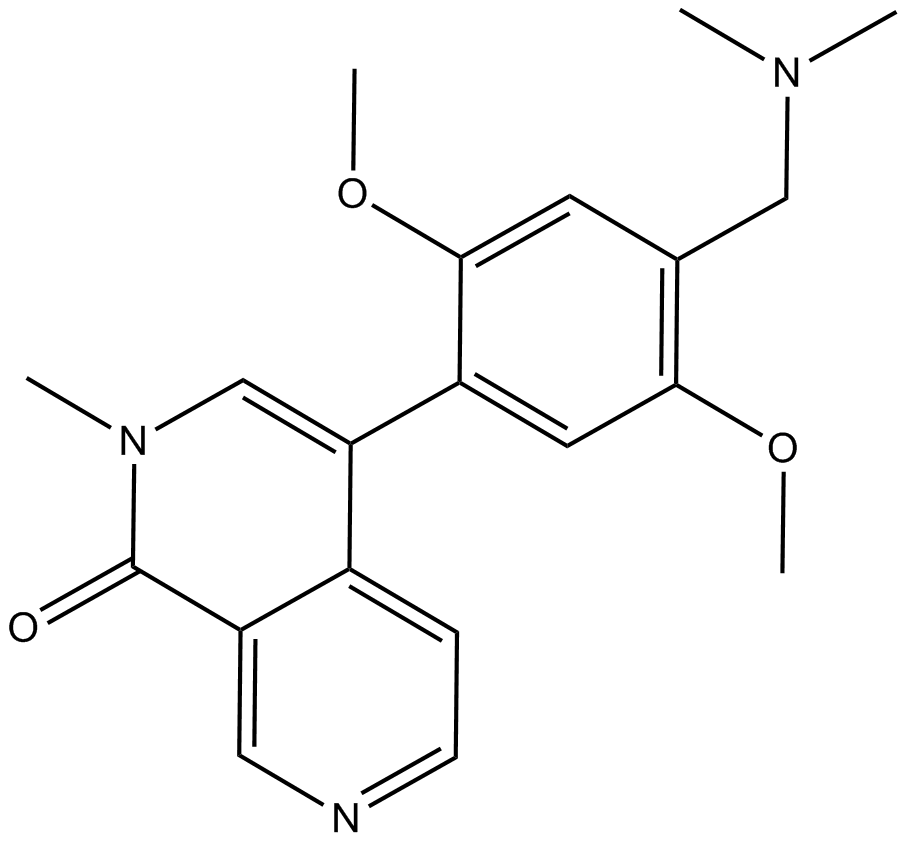
BI-9564
Le BI-9564 est un inhibiteur des bromodomaines BRD9/BRD7 puissant, sélectif et perméable aux cellules, avec des IC50 de 75 nM et 3,4 μM et des Kd de 14 nM et 239 nM, respectivement. Le BI-9564 a une IC50 > 100 μM pour la famille BET.
-
GC42933
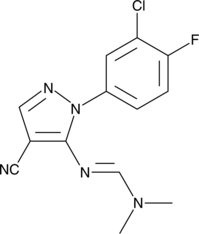
Binucleine 2
Binucleine 2 is an isoform-specific and ATP-competitive inhibitor of Drosophila Aurora B kinase (Ki = 0.36 μM), a kinase involved in cell division.
-
GC64789
Biotinylated-JQ1
Biotinylé-JQ1 (biotine-JQ1) est un dérivé biotinylé de JQ1 avec une haute affinité pour le bromodomaine de BRD4. Le JQ1 biotinylé inhibe la prolifération des cellules de myélome multiple MM1.S avec une EC50 de 0,4μM.
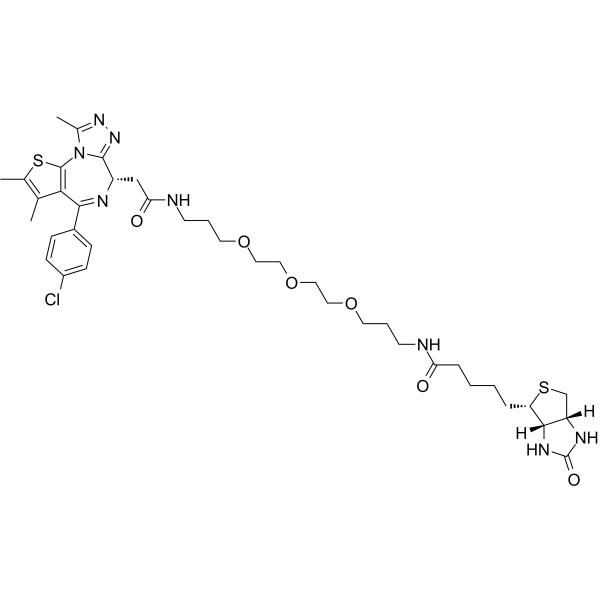
-
GC25139
Biphenyl-4-sulfonyl chloride
Biphenyl-4-sulfonyl chloride (p-Phenylbenzenesulfonyl, 4-Phenylbenzenesulfonyl, p-Biphenylsulfonyl) is a HDAC inhibitor with synthetic applications in palladium-catalyzed desulfitative C-arylation.
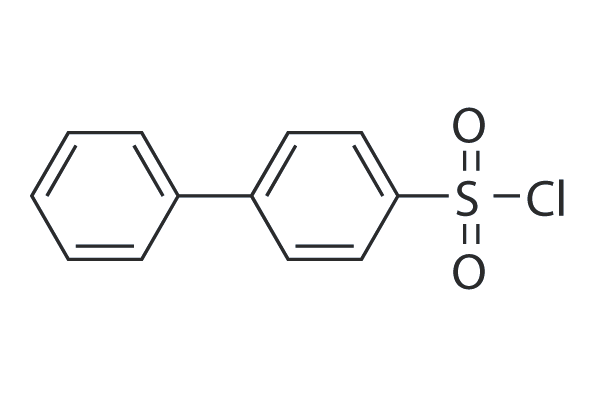
-
GC48463
Bisubstrate Inhibitor 78
An inhibitor of NNMT