Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nom du produit Informations
-
GC30530
Dot1L-IN-2
Dot1L-IN-2 est un inhibiteur puissant, sélectif et biodisponible par voie orale de Dot1L (une histone méthyltransférase), avec une IC50 et un Ki de 0,4 nM et 0,08 nM, respectivement.
-
GC62613
Dot1L-IN-4
Dot1L-IN-4 est un puissant perturbateur de l'inhibiteur de la protéine de type 1 de silenÇage télomérique (DOT1L) avec une IC50 SPA DOT1L de 0,11 nM.
-
GC65962
Dot1L-IN-5
Dot1L-IN-5 est un puissant perturbateur de l'inhibiteur de la protéine de type 1 de silenÇage télomérique (DOT1L) avec une IC50 SPA DOT1L de 0,17 nM.
-
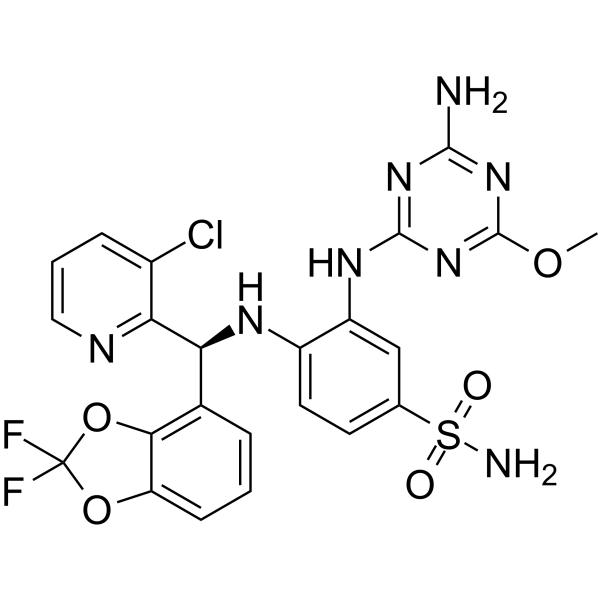
GC12680
DR 2313
A PARP inhibitor
-
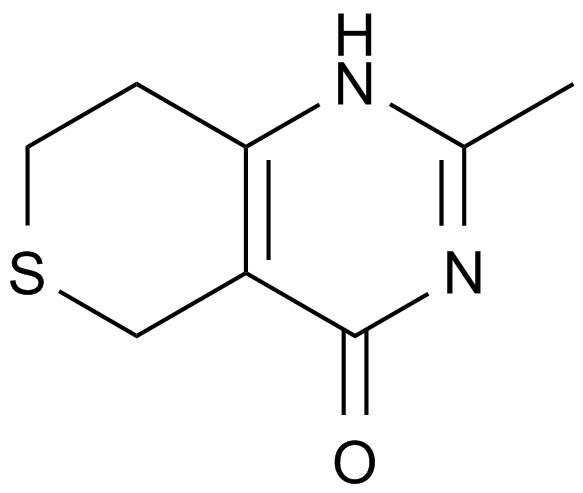
GC13706
Droxinostat
An inhibitor of HDAC3, HDAC6, and HDAC8
-
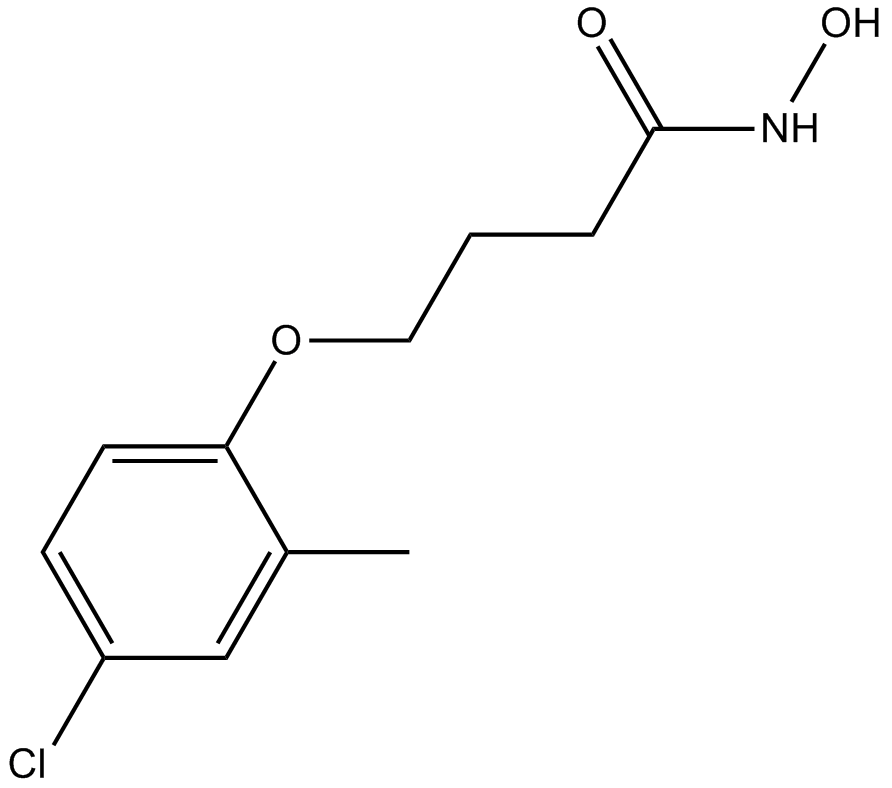
GC45927
DS-437
Le DS-437 est un double inhibiteur de PRMT5/7 (CI50 de PRMT5/7 = 6 μM).
-
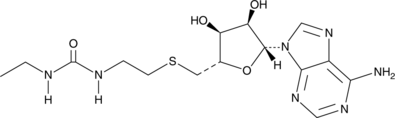
GC35904
dTRIM24
dTRIM24 est un dégradeur bifonctionnel sélectif de TRIM24 basé sur PROTAC, composé de ligands pour von Hippel-Lindau et TRIM24.
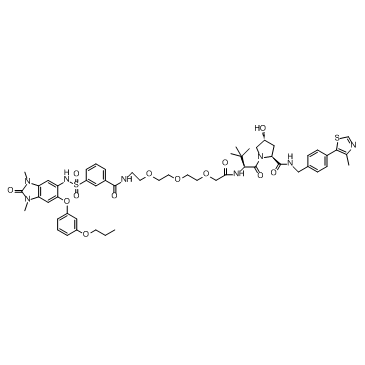
-
GC35914
DW14800
DW14800 est un inhibiteur de la protéine arginine méthyltransférase 5 (PRMT5), avec une IC50 de 17 nM. DW14800 réduit les niveaux de H4R3me2s et améliore la transcription de HNF4α, mais ne modifie pas l'expression de PRMT5. Activité anticancéreuse.
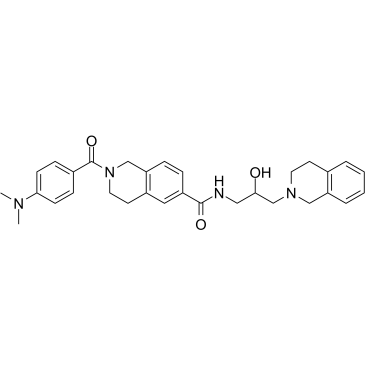
-
GC33403
E-7386
E-7386 est un modulateur CBP/bêta-caténine actif par voie orale.
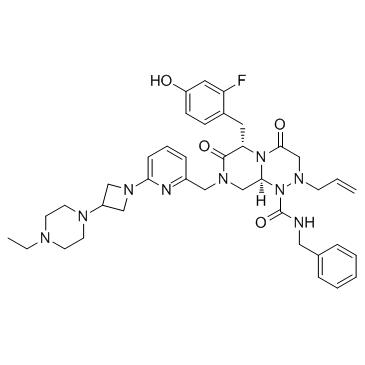
-
GC64209
E7016
E7016 (GPI 21016) est un inhibiteur de PARP disponible par voie orale. E7016 peut améliorer la radiosensibilité des cellules tumorales in vitro et in vivo grÂce À l'inhibition de la réparation de l'ADN. E7016 agit comme un agent anticancéreux potentiel.
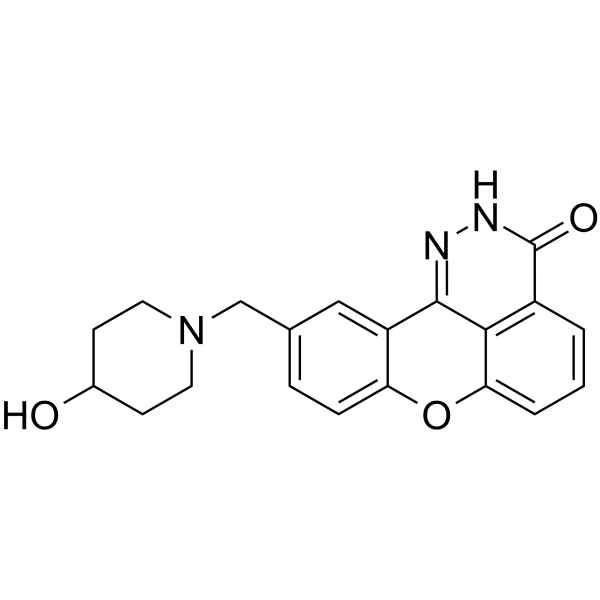
-
GC18172
E7449
E7449 est un puissant inhibiteur de PARP1 et PARP2 et inhibe également TNKS1 et TNKS2, avec des IC50 de 2,0, 1,0, ~50 et ~50 nM pour PARP1, PARP2, TNKS1 et TNKS2, respectivement, en utilisant 32P-NAD+ comme substrat.
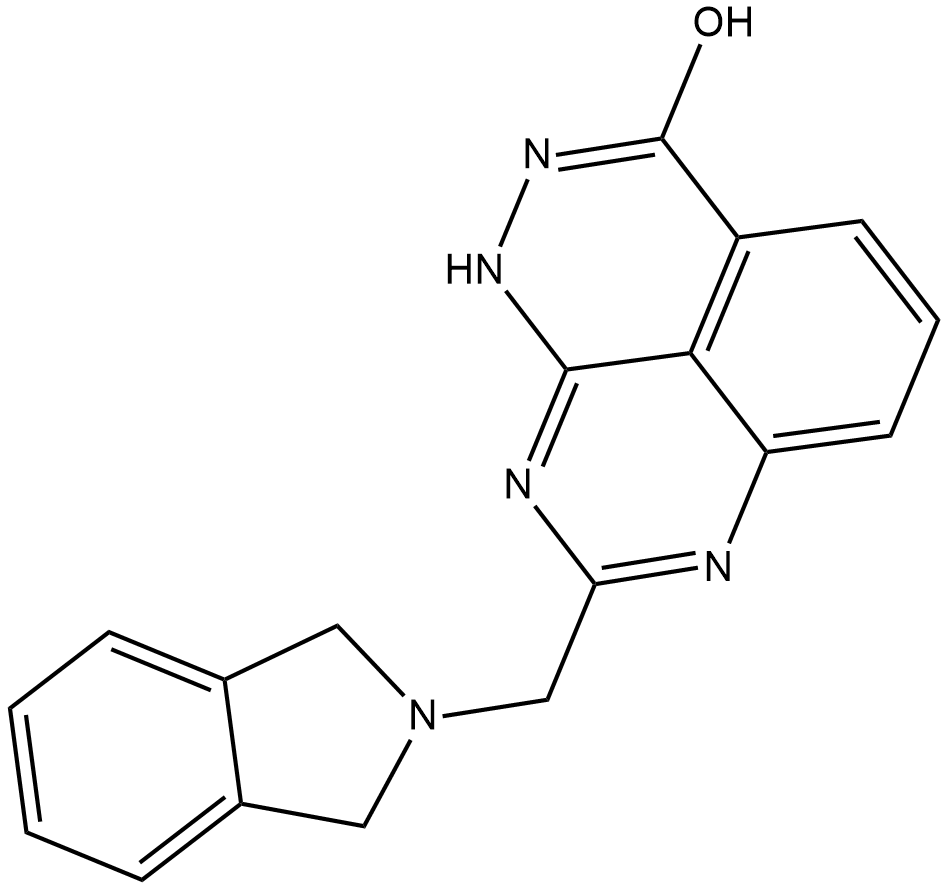
-
GC12991
EB 47
A PARP1 and TNKS2 inhibitor
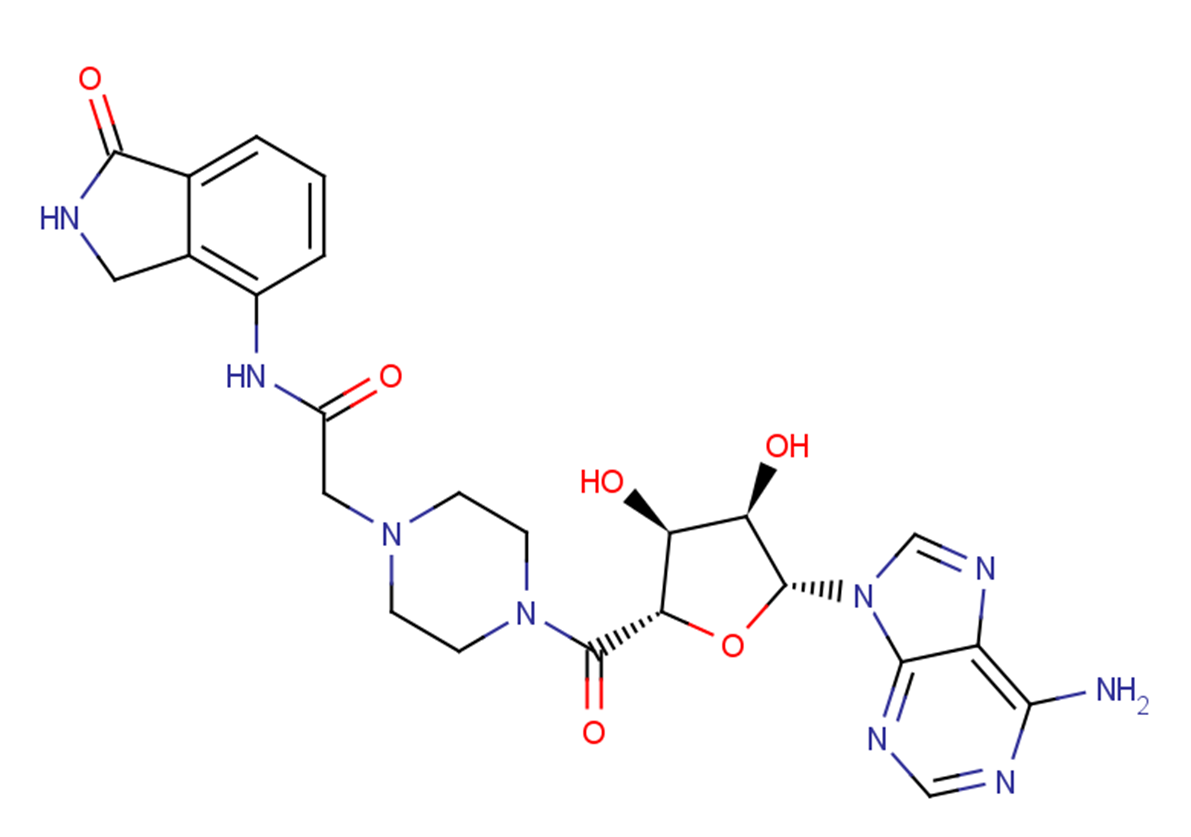
-
GC33220
EBI-2511
EBI-2511 est un inhibiteur EZH2 très puissant et actif par voie orale, avec une IC50 de 6 nM dans les lignées cellulaires Pfeffiera, respectivement.
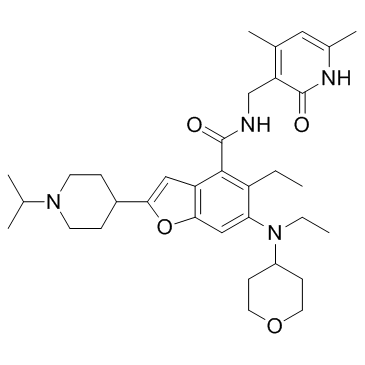
-
GC18236
Echinomycin
Un inhibiteur de la transcription génique médiée par HIF1.

-
GC19129
EDO-S101
EDO-S101 (Tinostamustine) est un inhibiteur pan HDAC; inhibe HDAC6, HDAC1, HDAC2 et HDAC3 avec des valeurs IC50 de 6 nM, 9 nM, 9 nM et 25 nM, respectivement.
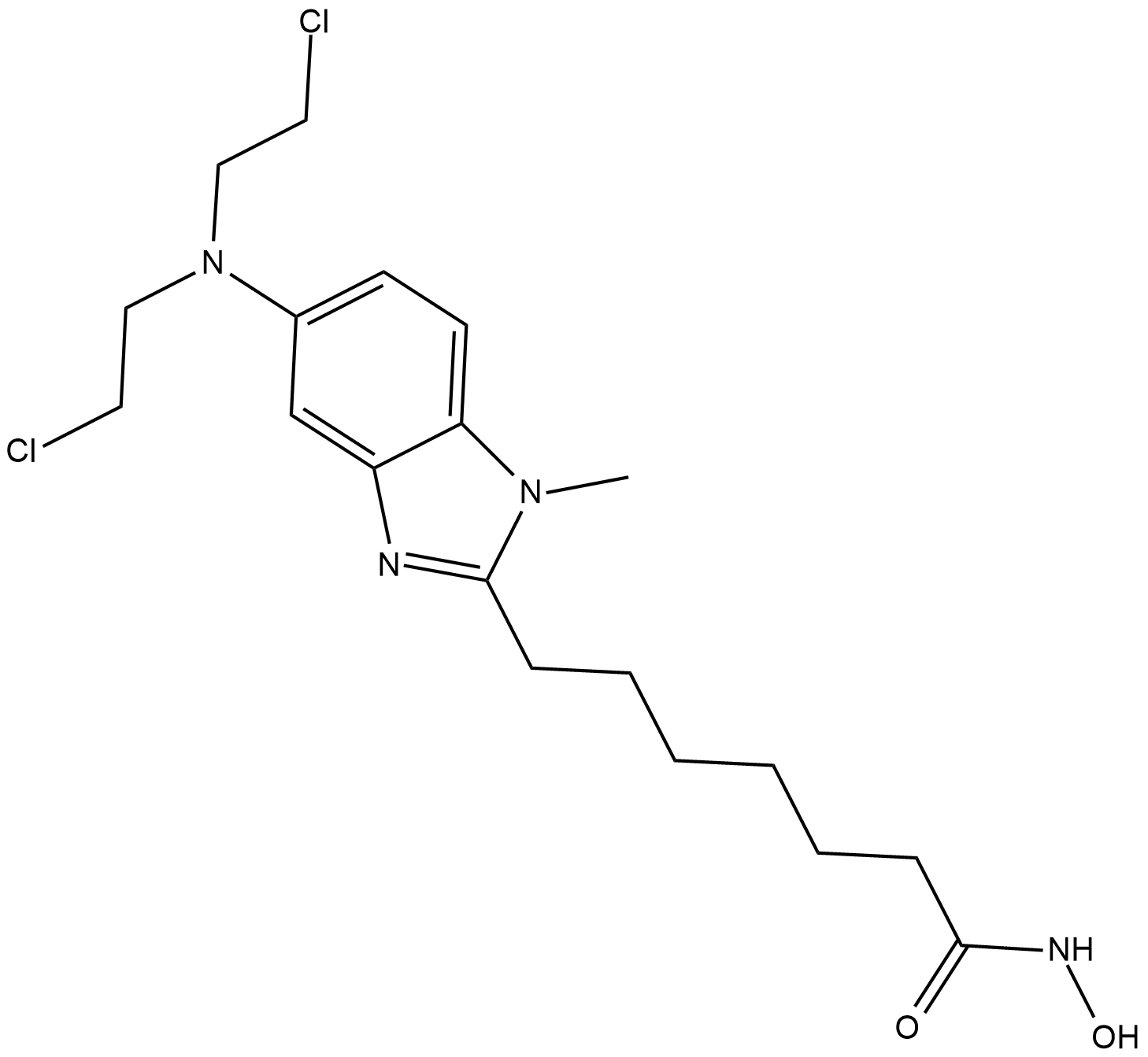
-
GC19130
EED226
EED226 est un inhibiteur du complexe répressif polycomb 2 (PRC2), qui se lie À la poche K27me3 lors du développement de l'ectoderme embryonnaire (EED) et présente une forte activité antitumorale dans le modèle de souris xénogreffe. L'EED226 est un inhibiteur de l'EED puissant, sélectif et biodisponible par voie orale. EED226 inhibe PRC2 avec une IC50 de 23,4 nM lorsque le peptide H3K27me0 est utilisé comme substrat dans les essais enzymatiques in vitro.
-
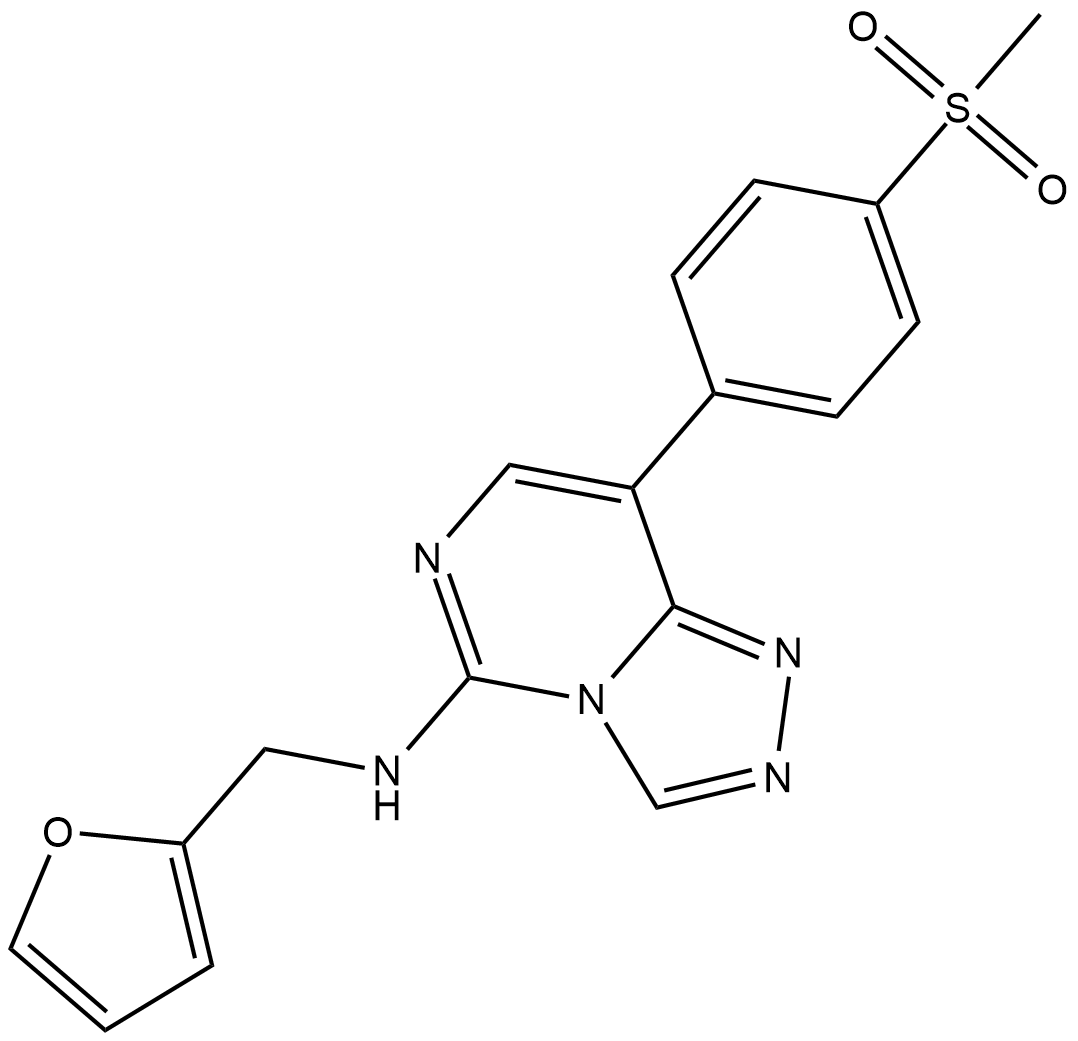
GC67934
EEDi-5285
-
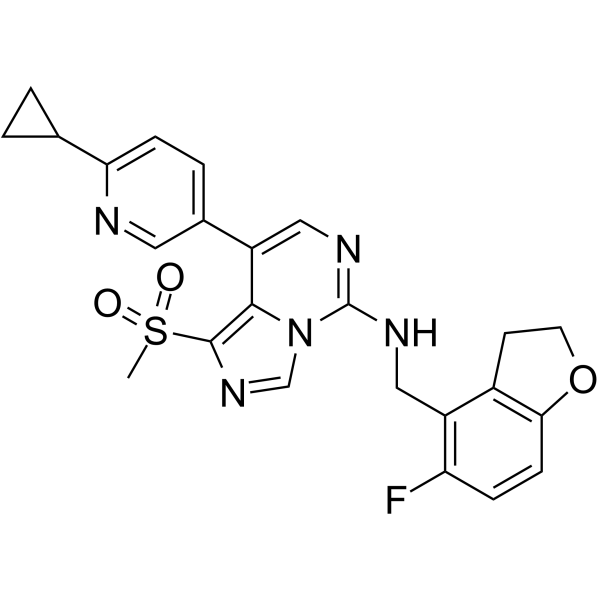
GC34567
EHMT2-IN-1
EHMT2-IN-1 est un puissant inhibiteur de l'EHMT, avec des IC50 de tous <100 nM pour le peptide EHMT1, le peptide EHMT2 et l'EHMT2 cellulaire. Utilisé dans la recherche de troubles sanguins ou de cancer.
-
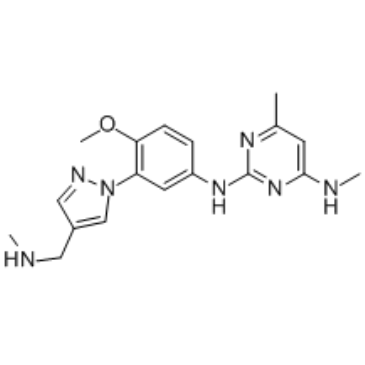
GC34568
EHMT2-IN-2
EHMT2-IN-2 est un puissant inhibiteur de l'EHMT, avec des IC50 de tous <100 nM pour le peptide EHMT1, le peptide EHMT2 et l'EHMT2 cellulaire. Utilisé dans la recherche d'une maladie du sang ou d'un cancer.
-
GC14756
EI1
EI1 (KB-145943) est un inhibiteur EZH2 puissant et sélectif avecIC50de 15nM et 13nM pour EZH2 (WT) et EZH2 (Y641F), respectivement.
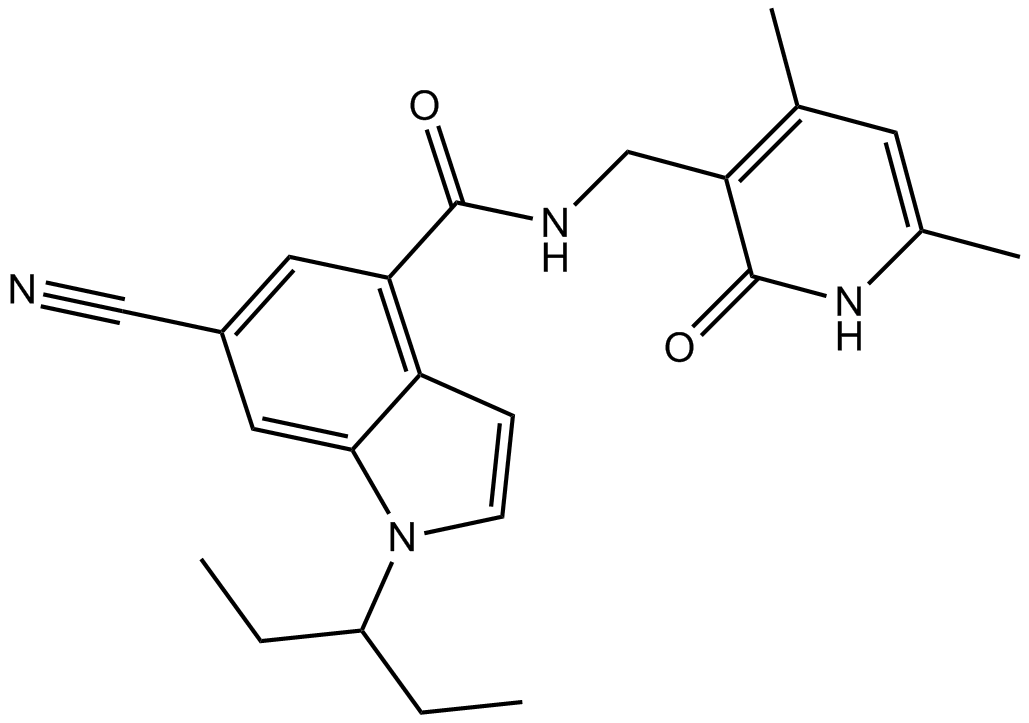
-
GC33043
EL-102
EL-102 est un inhibiteur du facteur 1 induit par l'hypoxie (Hif1α). EL-102 induit l'apoptose, inhibe la polymérisation de la tubuline et présente des activités contre le cancer de la prostate. EL-102 peut être utilisé pour la recherche sur le cancer.
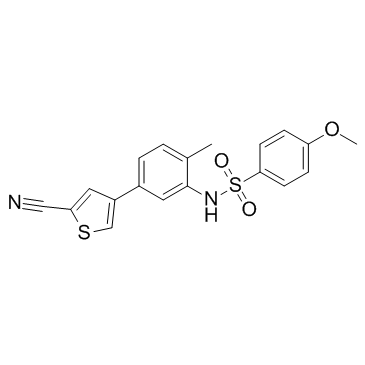
-
GC64191
Elevenostat
Elevenostat (JB3-22) est un inhibiteur sélectif de HDAC11 (IC50=0,235μM). Activité anti-myélome multiple (MM).
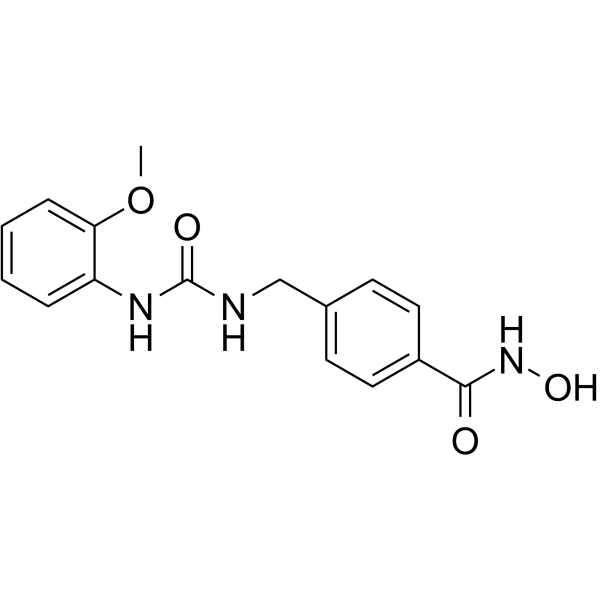
-
GC31245
EML 425
EML425 est un inhibiteur puissant et sélectif de la protéine de liaison CREB (CBP)/p300 avec des IC50 de 2,9 et 1,1 μM, respectivement.
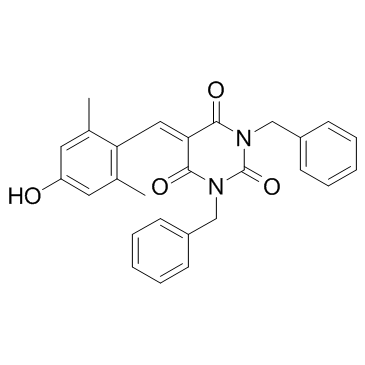
-
GC49124
EN219
EN219 est un ligand covalent synthétique modérément sélectif contre une cystéine N-terminale (C8) de RNF114 avec une IC50 de 470 nM. EN219 inhibe l'auto-ubiquitination médiée par RNF114 et l'ubiquitination p21.
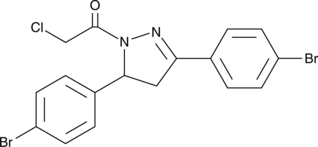
-
GC33634
Enarodustat (JTZ-951)
L'énarodustat (JTZ-951) est un inhibiteur de la prolyl hydroxylase du facteur inductible par l'hypoxie puissant et actif par voie orale, avec une CE50 de 0,22 μM.
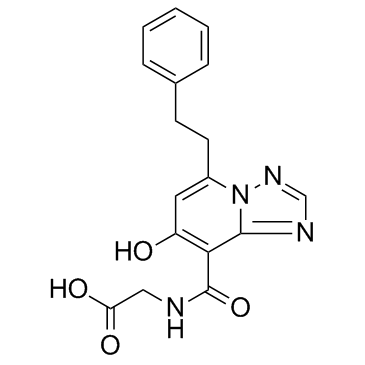
-
GC33170
ENMD-119 (ENMD 1198)
ENMD-119 (ENMD 1198) (IRC-110160), un agent déstabilisateur des microtubules actif par voie orale, est un analogue du 2-méthoxyestradiol avec une activité antiproliférative et antiangiogénique. ENMD-119 (ENMD 1198) convient pour inhiber HIF-1alpha et STAT3 dans les cellules HCC humaines et conduit À une réduction de la croissance tumorale et de la vascularisation.
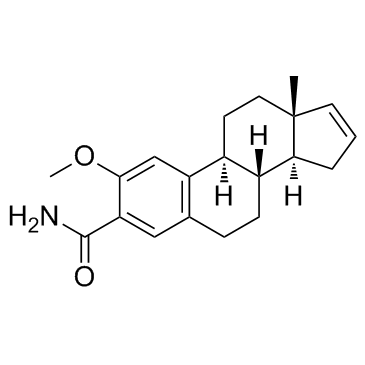
-
GC16519
ENMD-2076
A multi-kinase inhibitor
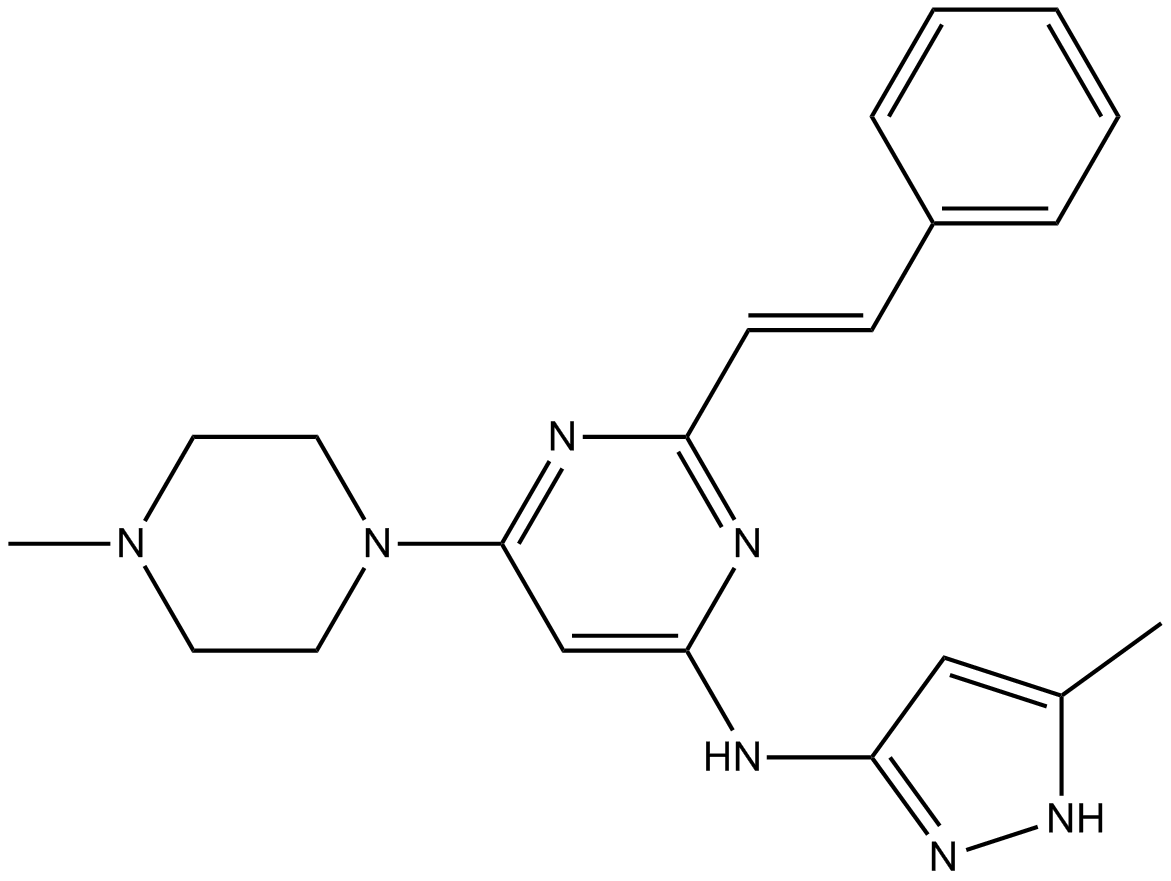
-
GC12145
ENMD-2076 L-(+)-Tartaric acid
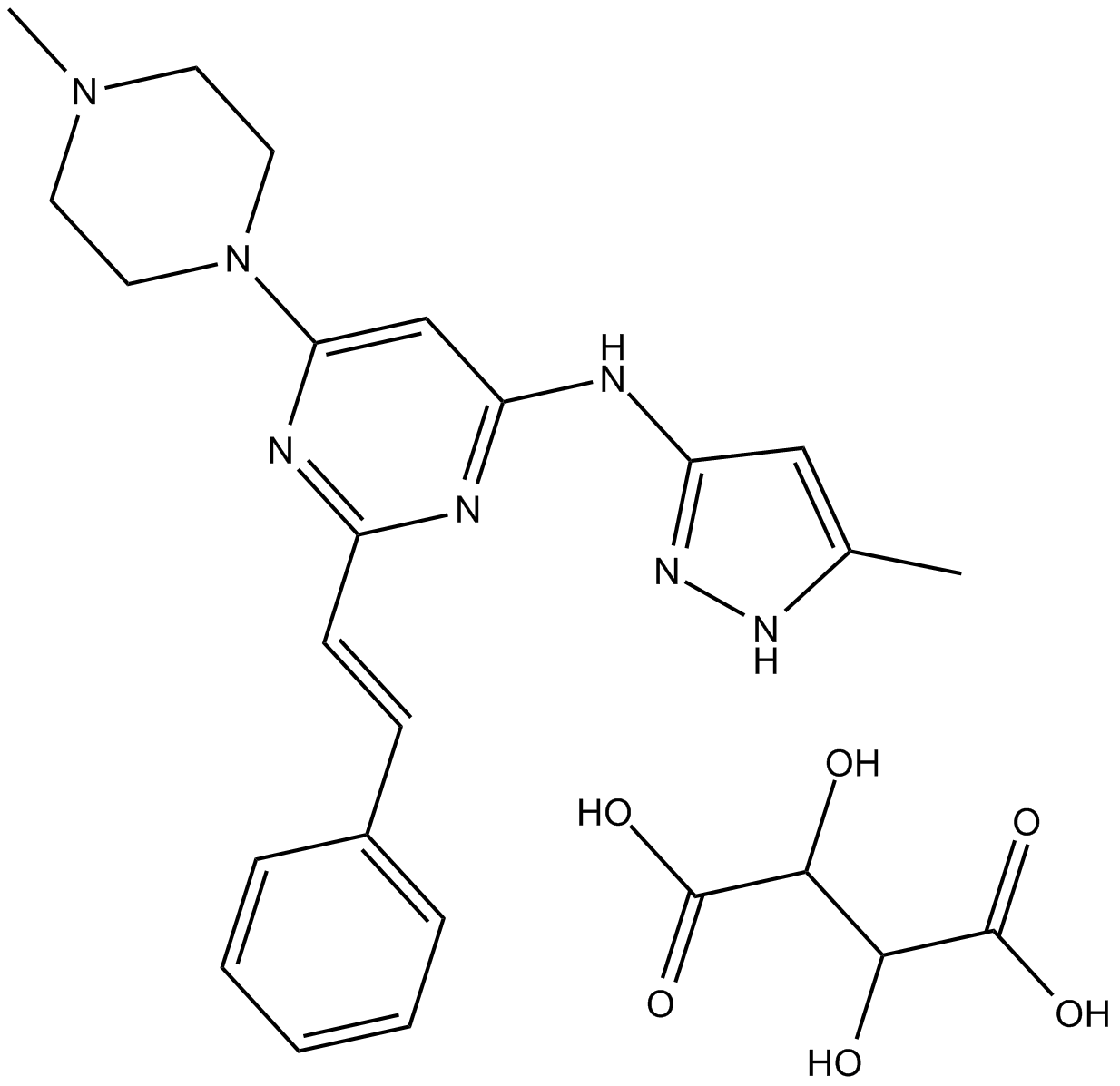
-
GC50182
ent-LP 99
ent-LP 99 est un inhibiteur puissant et sélectif de BRD7 et BRD9 avec un KD de 99 nM pour BRD9.
-
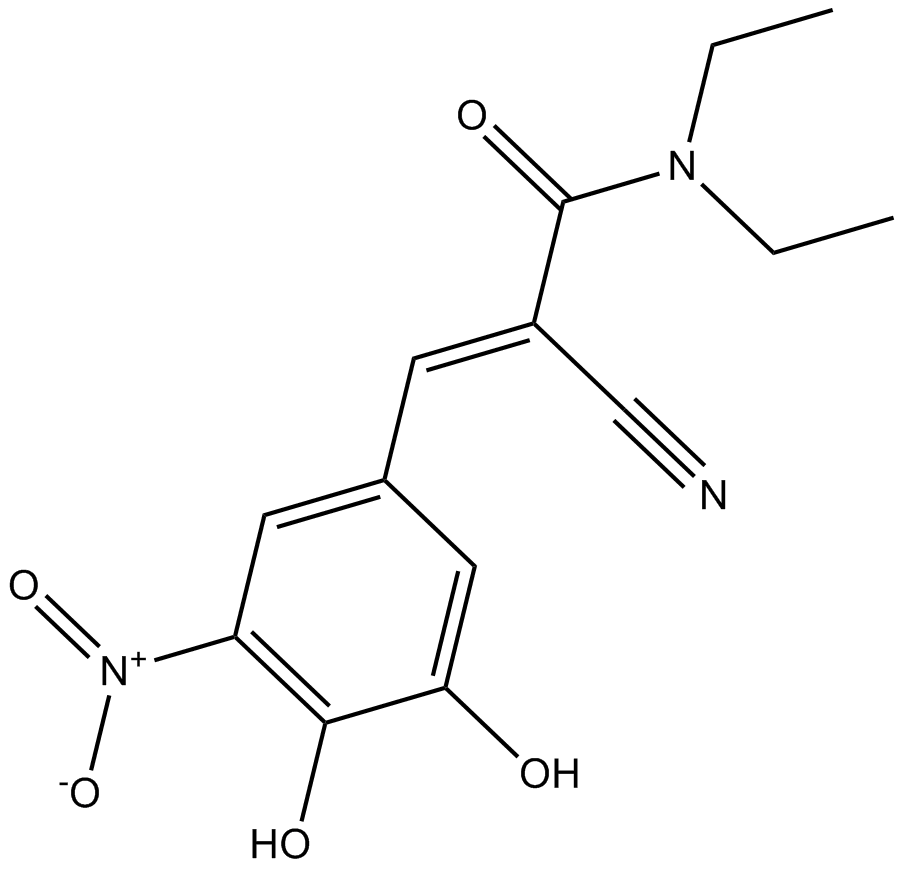
GC17334
Entacapone
A reversible COMT inhibitor
-
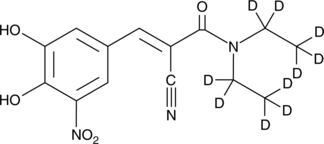
GC47294
Entacapone-d10
Entacapone-d10 est le deutérium marqué Entacapone.
-
GC11625
Entinostat (MS-275,SNDX-275)
A histone deacetylase inhibitor
-
GC14062
EPZ-6438
A EZH2 inhibitor,potent and selective
-
GC64343
EPZ-719
EPZ-719 est un nouvel inhibiteur puissant de SETD2 (IC50 = 0,005 μM) avec une sélectivité élevée par rapport aux autres histones méthyltransférases.
-
GC13383
EPZ004777
A potent inhibitor of DOT1L
-
GC48980
EPZ004777 (formate)
A potent inhibitor of DOT1L

-
GC15259
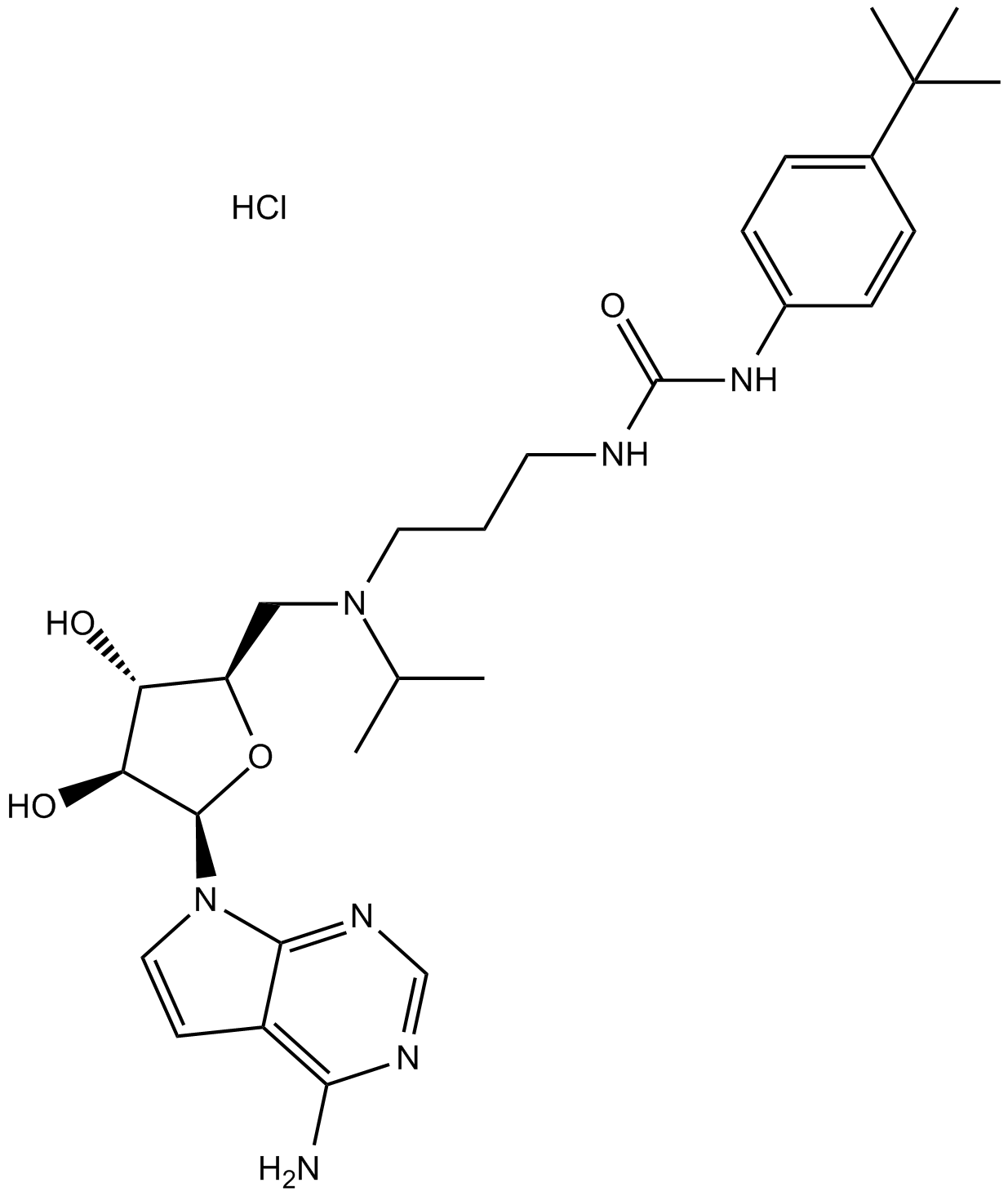
EPZ004777 HCl
EPZ004777 HCl est un puissant inhibiteur sélectif de DOT1L avec une IC50 de 0,4 nM.
-
GC13878
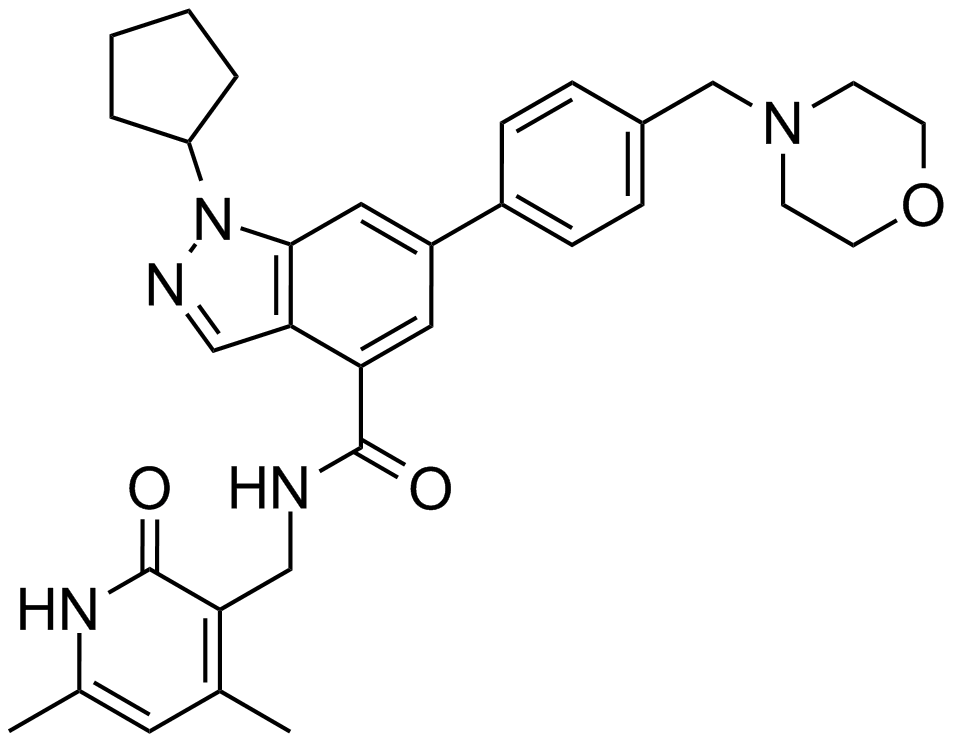
EPZ005687
A potent, selective inhibitor of EZH2
-
GC19141
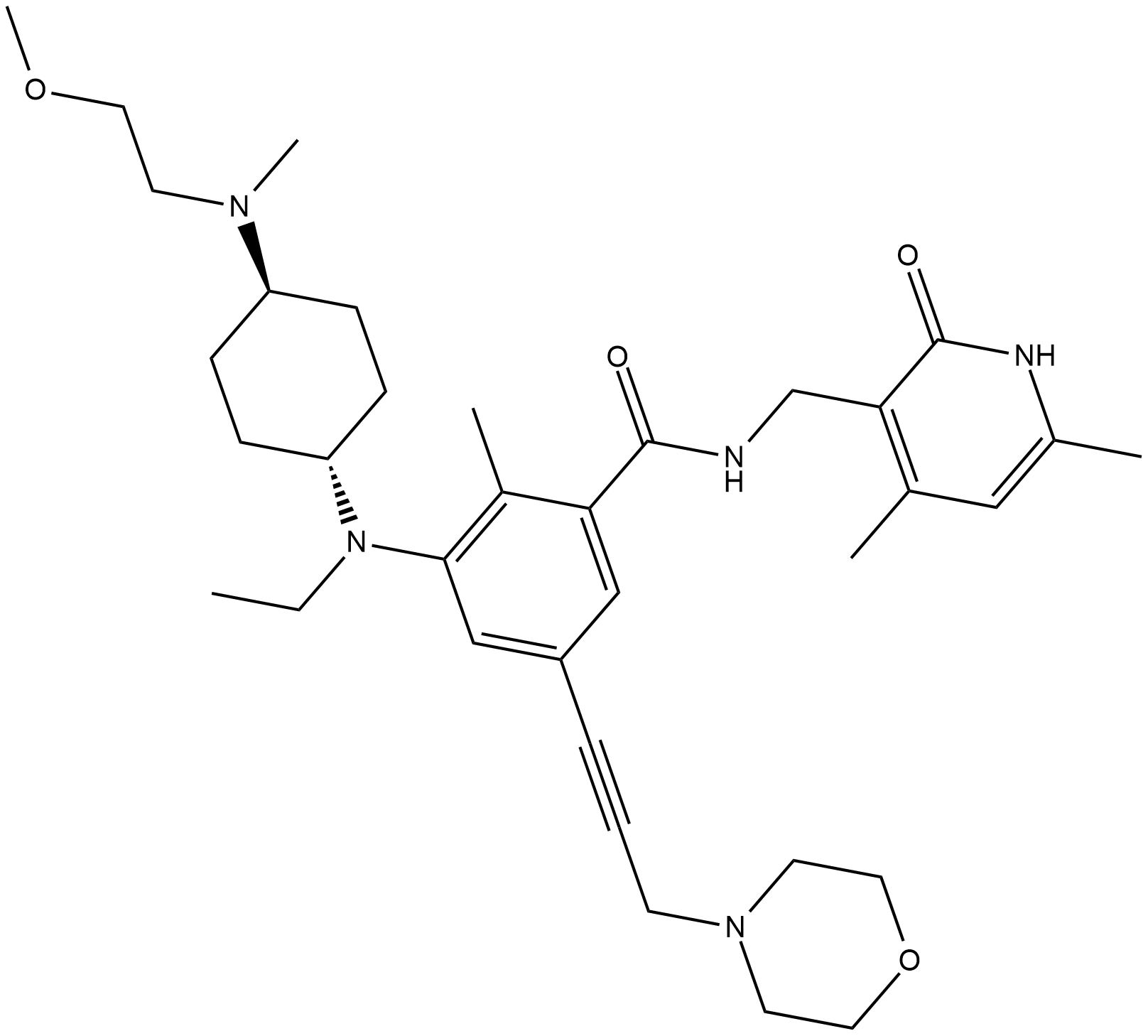
EPZ011989
EPZ011989 est un inhibiteur Zeste Homolog 2 (EZH2) puissant et actif par voie orale avec une stabilité métabolique. EPZ011989 a une inhibition inhibitrice pour EZH2 avec une valeur Ki <3 nM. EPZ011989 montre une inhibition robuste de la marque méthyle et une activité anti-tumorale. EPZ011989 peut être utilisé pour la recherche de divers cancers.
-
GC34136
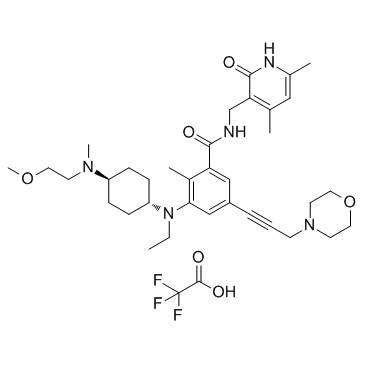
EPZ011989 trifluoroacetate (EPZ-011989 trifluoroacetate)
Le trifluoroacétate EPZ-011989 est un inhibiteur de Zeste Homolog 2 (EZH2) puissant et actif par voie orale avec une stabilité métabolique. Le trifluoroacétate EPZ-011989 a une inhibition inhibitrice pour EZH2 avec une valeur Ki <3 nM. Le trifluoroacétate EPZ-011989 présente une inhibition robuste de la marque méthyle et une activité anti-tumorale. Le trifluoroacétate EPZ-011989 peut être utilisé pour la recherche de divers cancers.
-
GC15302
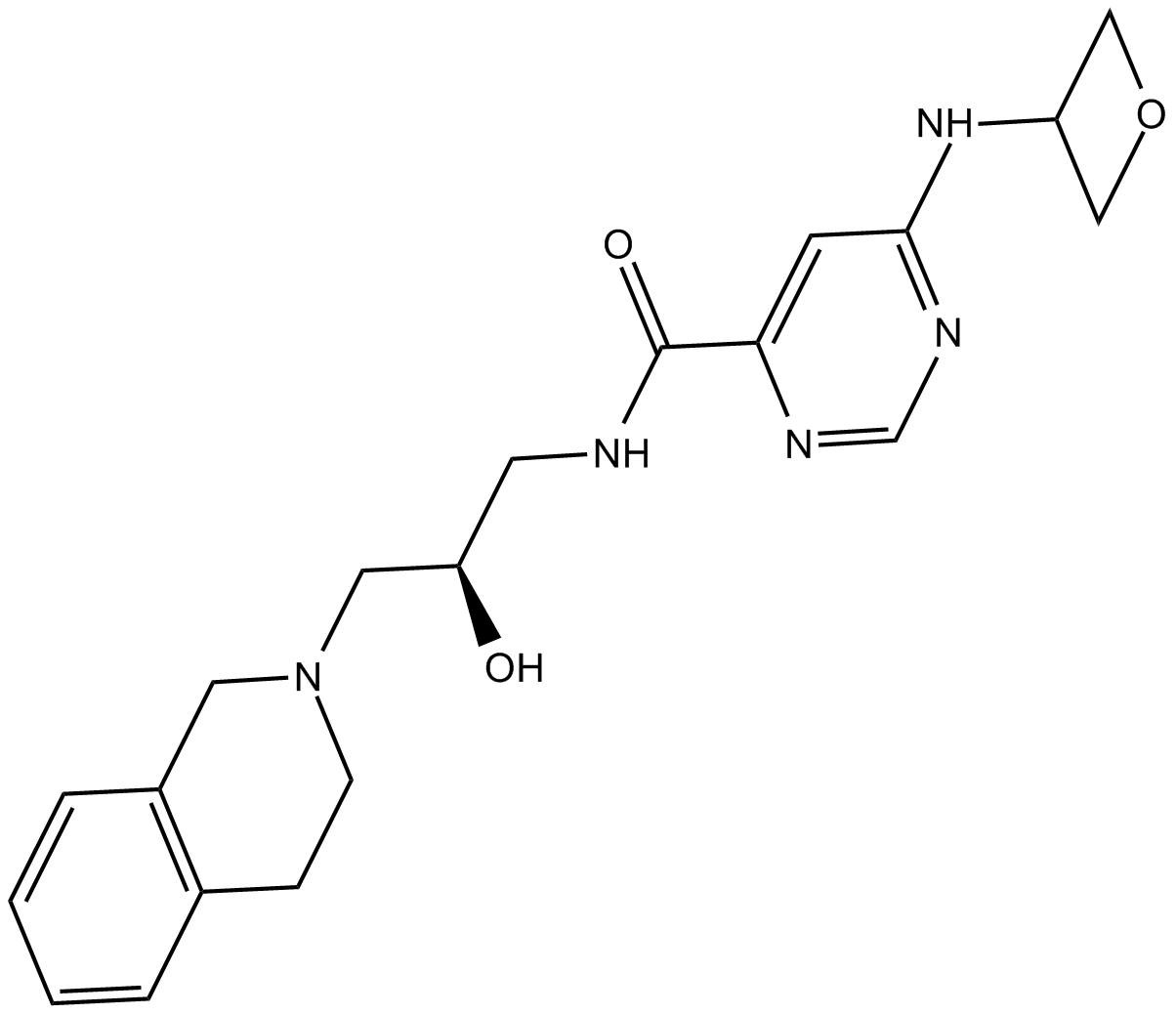
EPZ015666
EPZ015666 (GSK3235025) est un inhibiteur de PRMT5 disponible par voie orale avec une IC50 de 22 nM.
-
GC15102
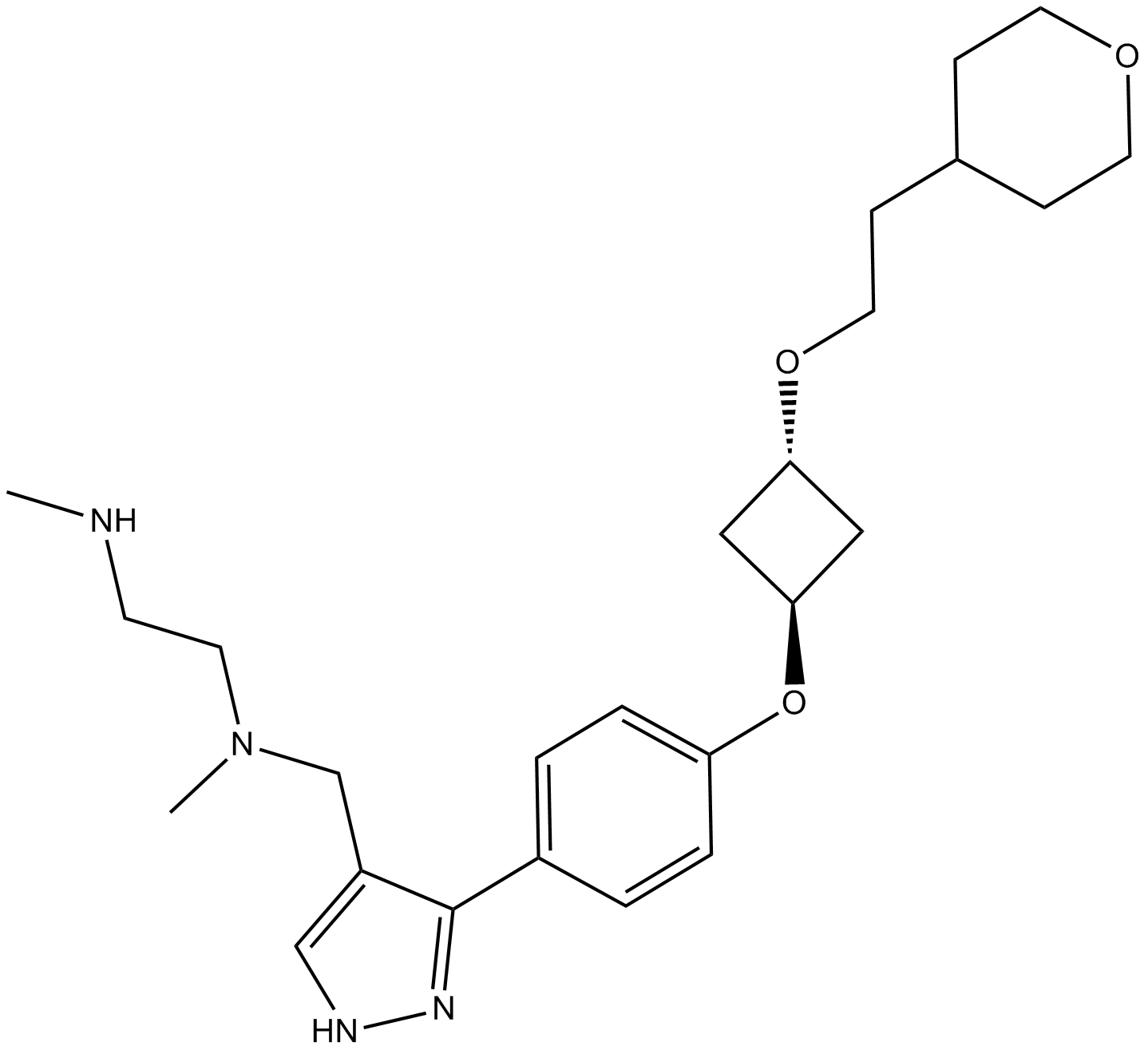
EPZ020411
EPZ020411 est un inhibiteur sélectif de PRMT6 avec une IC50 de 10 nM, a une sélectivité >10 fois pour PRMT6 par rapport À PRMT1 et PRMT8. EPZ020411 peut être utilisé pour la recherche sur le cancer.
-
GC36000
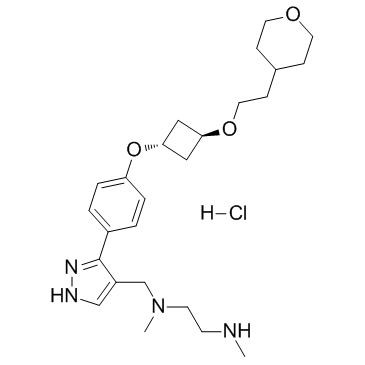
EPZ020411 hydrochloride
Le chlorhydrate d'EPZ020411 est un inhibiteur sélectif de PRMT6 avec une IC50 de 10 nM, il a une sélectivité >10 fois pour PRMT6 par rapport À PRMT1 et PRMT8. Le chlorhydrate d'EPZ020411 peut être utilisé pour la recherche sur le cancer.
-
GC16224
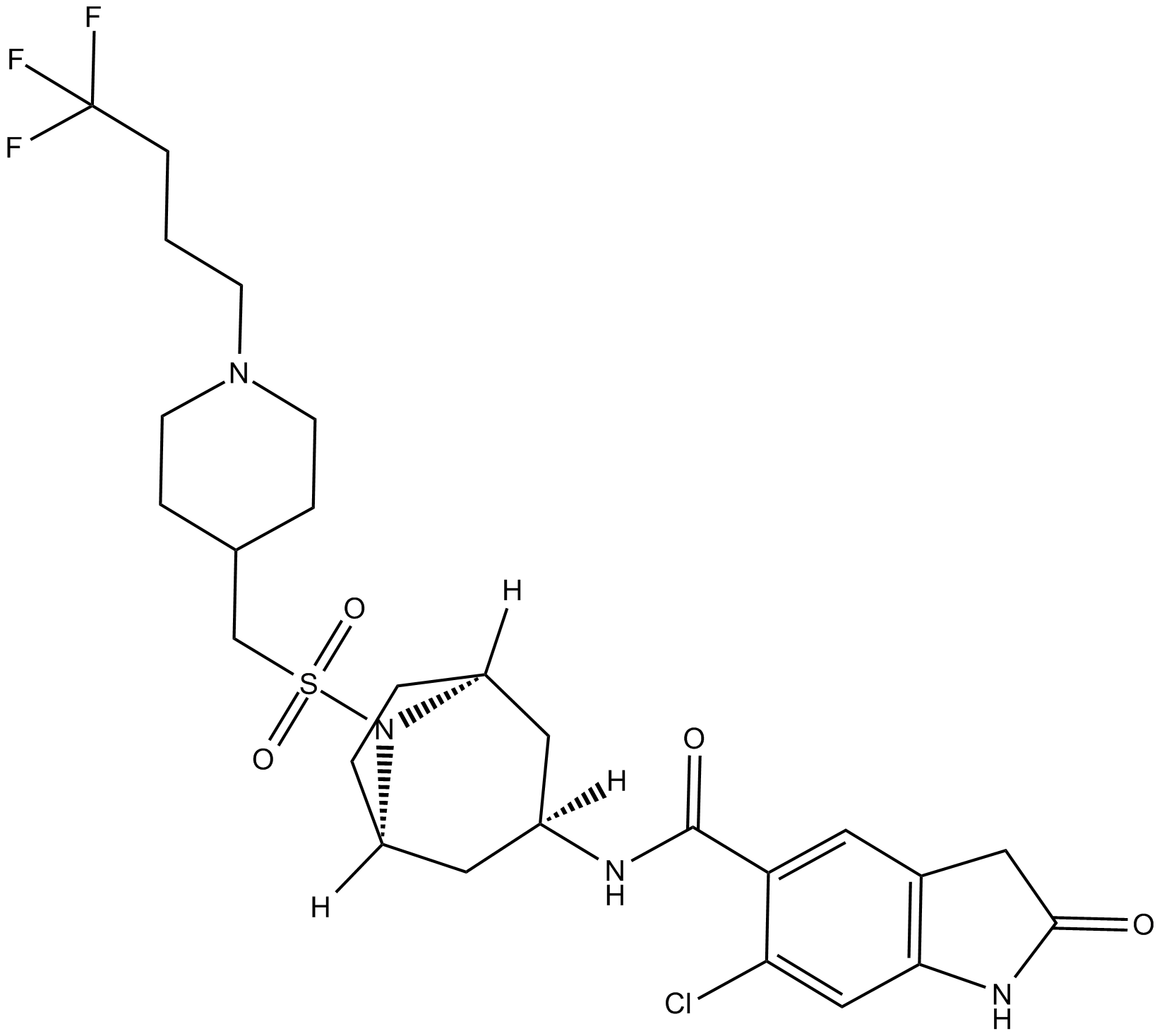
EPZ031686
EPZ031686 est un inhibiteur SMYD3 puissant et actif par voie orale et avec une valeur IC50 de 3 nM. EPZ031686 peut être utilisé pour la recherche sur le cancer.
-
GC12932
EPZ5676
A highly potent DOT1L inhibitor
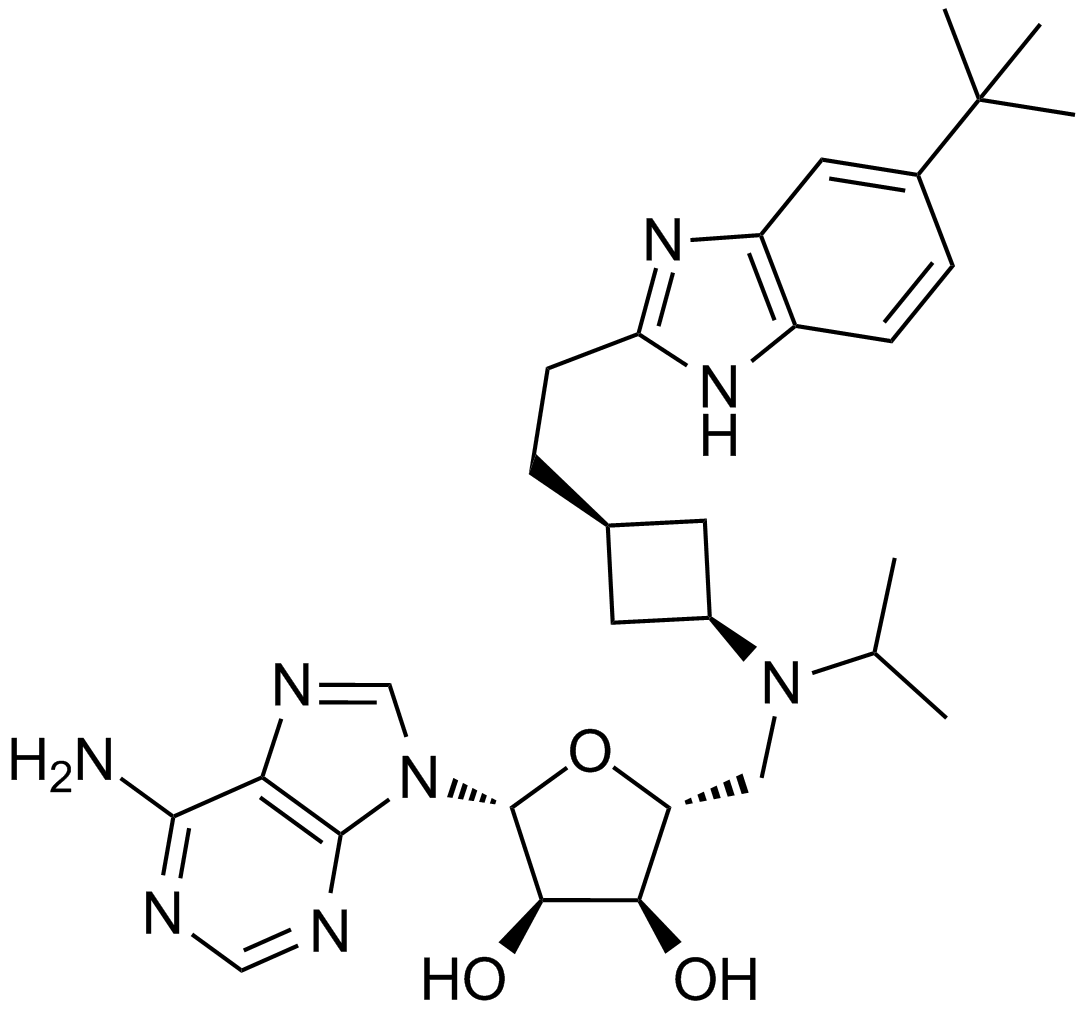
-
GC64575
Et-29
Et-29 est un inhibiteur puissant et sélectif de SIRT5 (Ki = 40 nM).
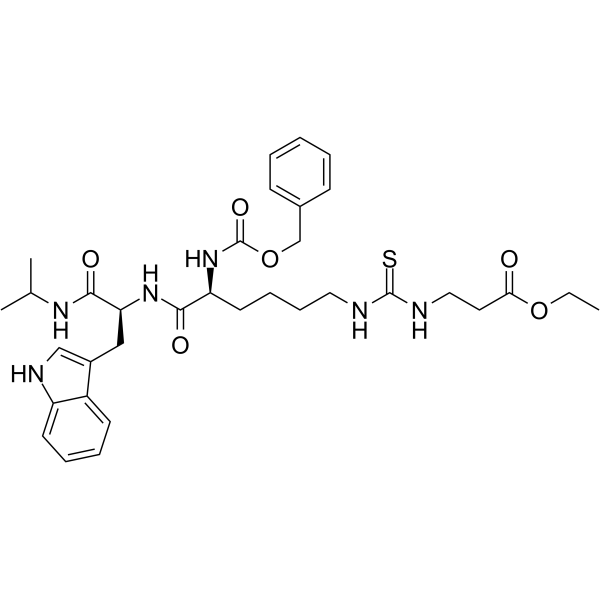
-
GC61669
Ethyl 3,4-dihydroxybenzoate
Le 3,4-dihydroxybenzoate d'éthyle (protocatéchuate d'éthyle), un antioxydant, est un inhibiteur de la prolyl-hydroxylase présent dans le testa des graines d'arachide.
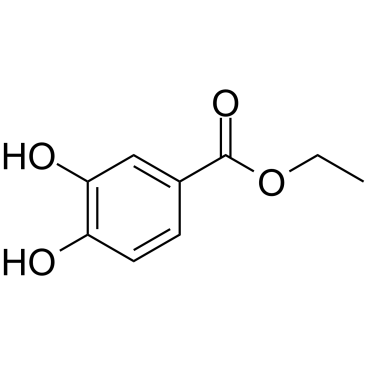
-
GC10635
EX 527 (SEN0014196)
A SIRT1 inhibitor
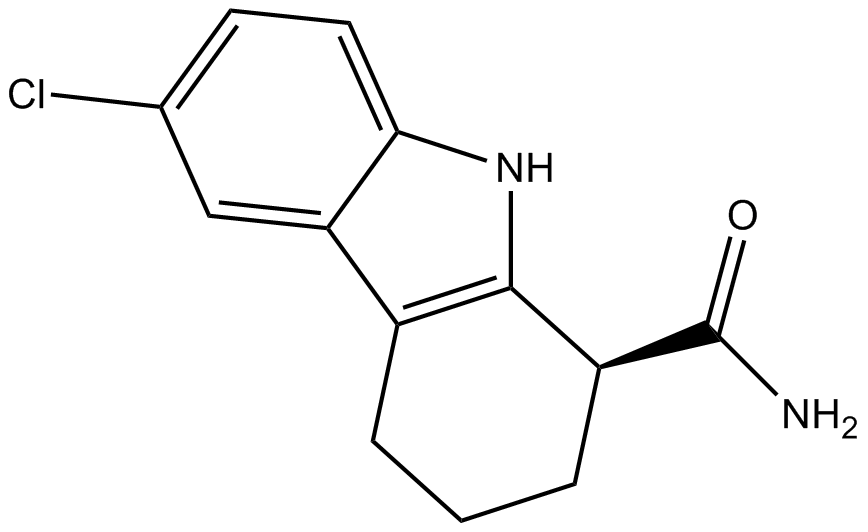
-
GC17126
EX-527 R-enantiomer
L'énantiomère R EX-527 ((R)-EX-527) est un énantiomère R de Selisistat. Selisistat (EX-527) est un inhibiteur puissant et sélectif de SIRT1 avec IC50 de 98 nM.
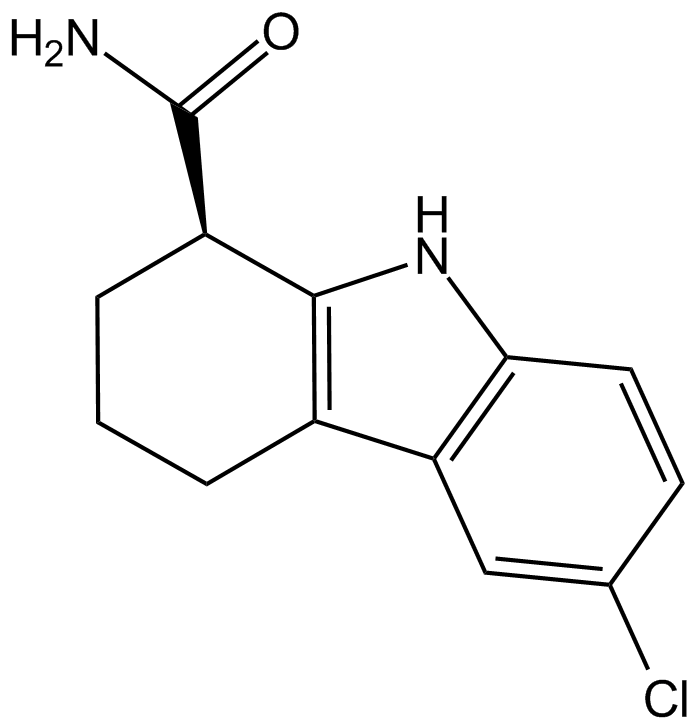
-
GC13417
EX-527 S-enantiomer
L'énantiomère S EX-527 ((S)-EX-527) est un inhibiteur puissant et sélectif de SIRT1, avec une IC50 de 98 nM.
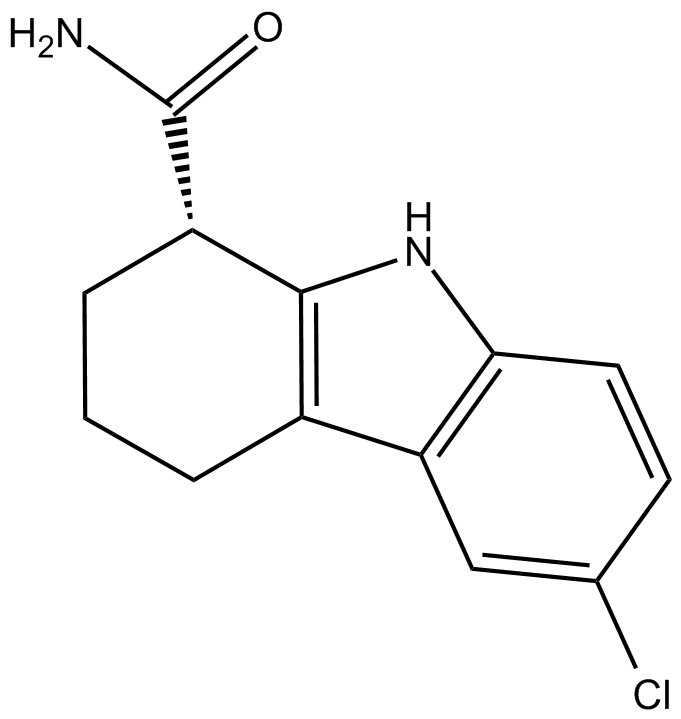
-
GC65980
EZH2-IN-13
EZH2-IN-13 est un puissant inhibiteur d'EZH2, pour plus de détails, veuillez vous référer au composé 73 dans le brevet WO2017139404. EZH2-IN-13 peut être utilisé pour étudier les cancers ou les lésions précancéreuses associés À l'activité EZH2.
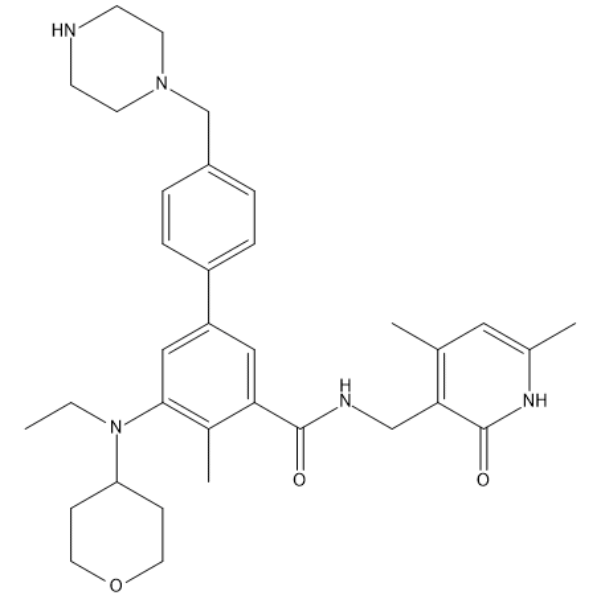
-
GC19149
EZM 2302
EZM 2302 est un inhibiteur de l'arginine méthyltransférase 1 associée au coactivateur (CARM1) avec une IC50 de 6nM.
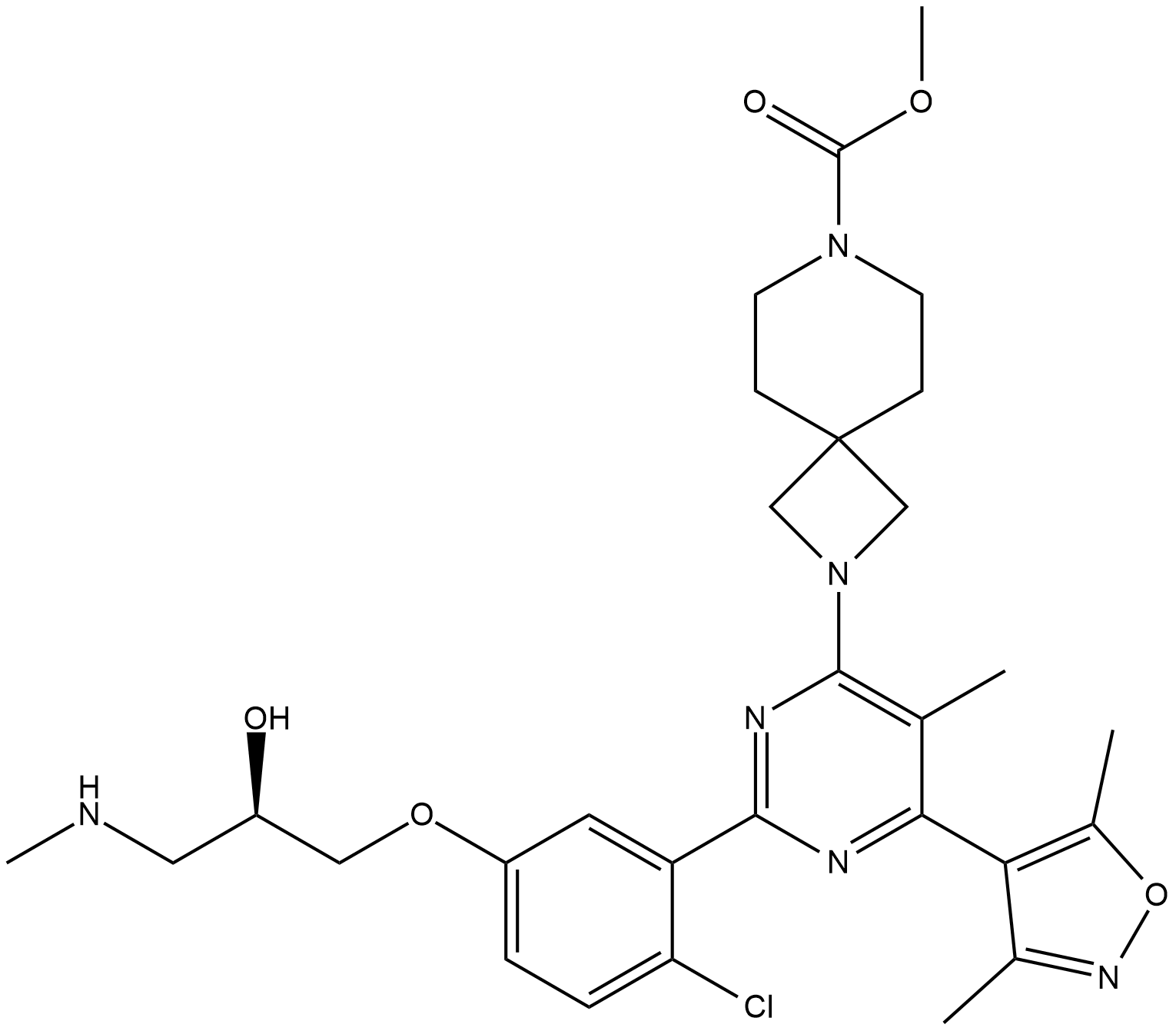
-
GC64900
EZM0414
EZM0414 est un inhibiteur puissant, sélectif et biodisponible par voie orale de SETD2 (IC50 = 18 nM dans le test biochimique SETD2; IC50 = 34 nM dans le test cellulaire). EZM0414 peut être utilisé pour la recherche du myélome multiple récidivant ou réfractaire et du lymphome diffus À grandes cellules B.
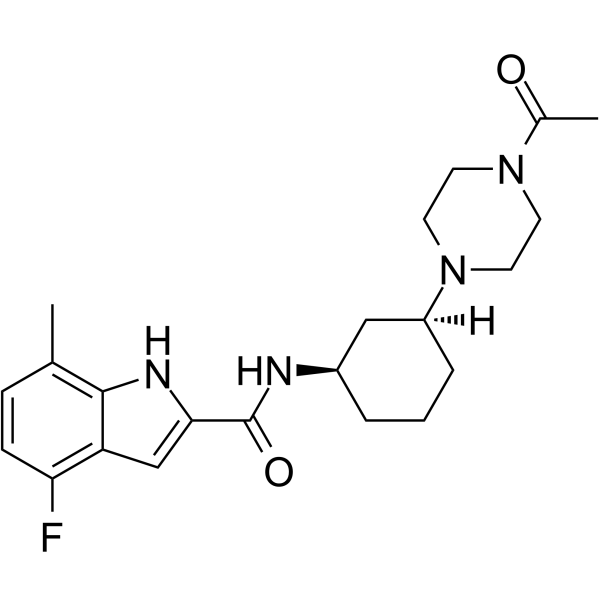
-
GC43651
F-Amidine (trifluoroacetate salt)
F-amidine is an inhibitor of protein arginine deiminases (PADs) that is selective for PAD1 and PAD4 (IC50s = 29.5, 350, and 21.6 μM for PAD1, PAD3, and PAD4 in vitro, respectively).
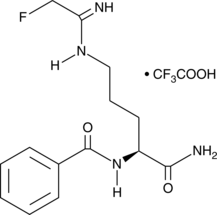
-
GC13139
FG-4592 (ASP1517)
Un inhibiteur des enzymes HIF-PH

-
GC16638
FG2216
Le FG2216 (IOX3) est un inhibiteur puissant et oralement actif de la HIF prolyl hydroxylase-2 (PHD2), avec une IC50 de 3,9 μM.
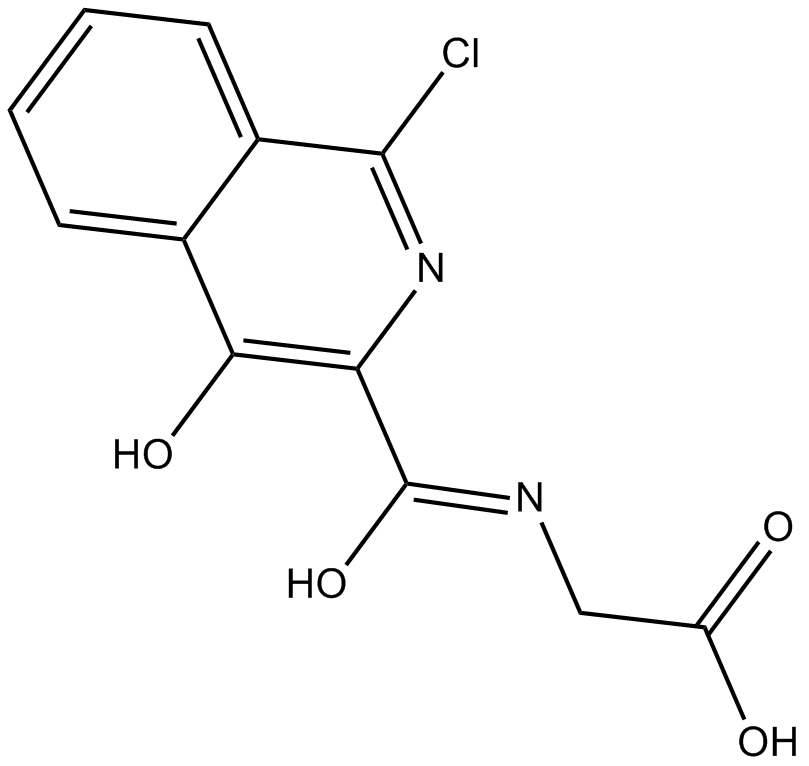
-
GC64821
FHD-286
Le FHD-286 est un inhibiteur de BRG1/BRM ATPase pour le traitement des troubles liés au BAF tels que la leucémie myéloÏde aiguë.
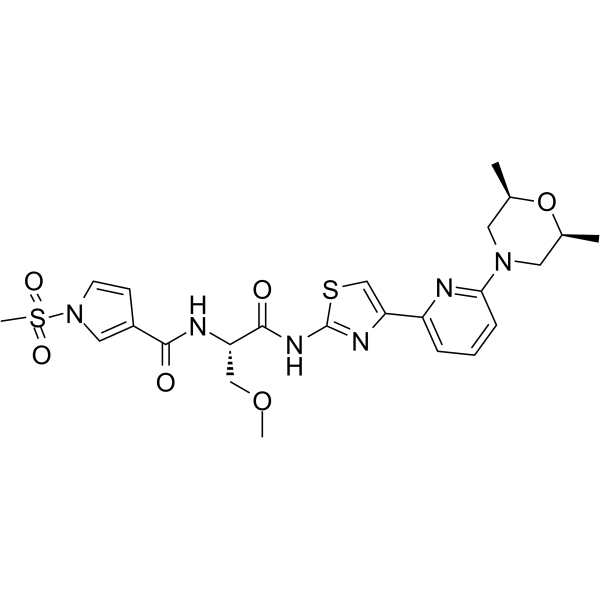
-
GC65160
FHT-1015
FHT-1205 est un puissant inhibiteur de SMARCA4/SMARCA2 ATPase (BRG1 et BRM) avec des CI50 ≤ 10 nM (WO2020160180A1 ; composé 67).
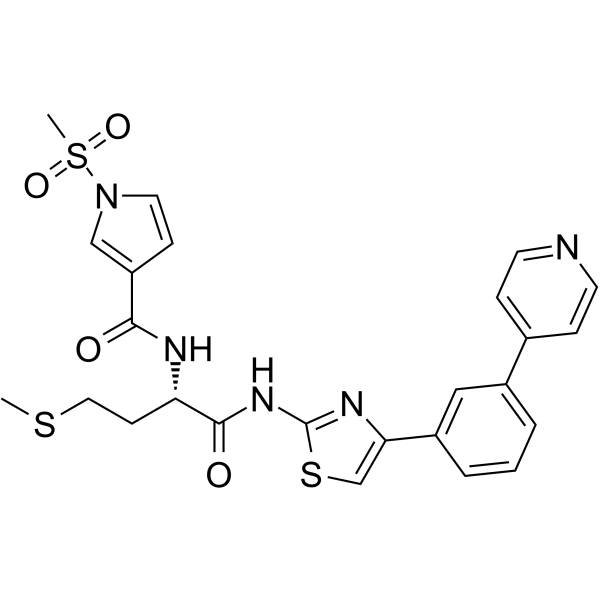
-
GC64777
FHT-1204
Le FHT-1204 est un puissant inhibiteur de SMARCA4/SMARCA2 ATPase (BRG1 et BRM) avec des CI50 ≤ 10 nM (WO2020160180A1 ; composé 70).
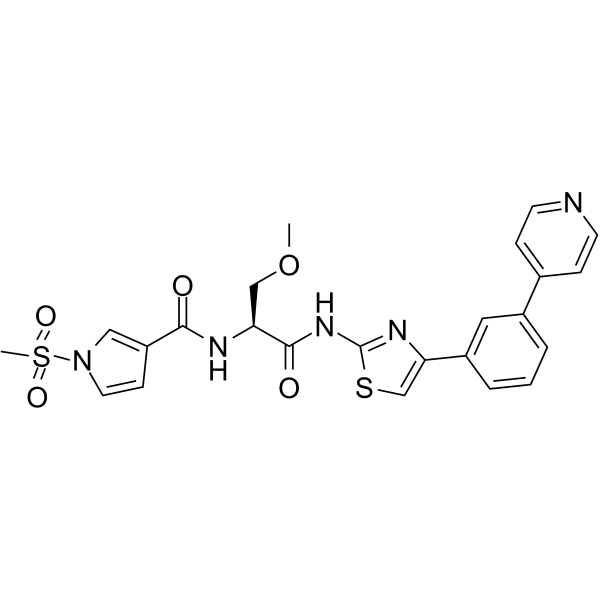
-
GC46148
Filgotinib-d4
Le filgotinib-d4 (GLPG0634-d4) est le filgotinib marqué au deutérium. Le filgotinib (GLPG0634) est un inhibiteur sélectif de JAK1 avec une IC50 de 10 nM, 28 nM, 810 nM et 116 nM pour JAK1, JAK2, JAK3 et TYK2, respectivement.
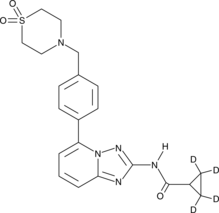
-
GN10030
Fisetin
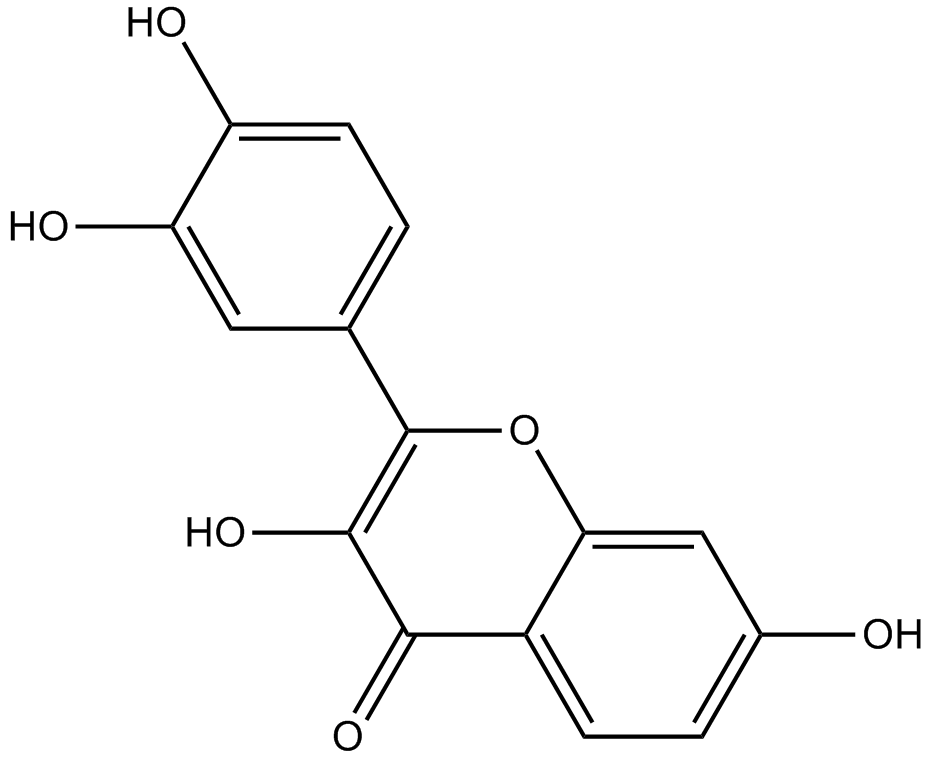
-
GC33015
FL-411 (BRD4-IN-1)
FL-411 (BRD4-IN-1) est un inhibiteur puissant et sélectif de BRD4 avec une IC50 de 0,43± ; 0,09 μ ; M pour BRD4(1).
-
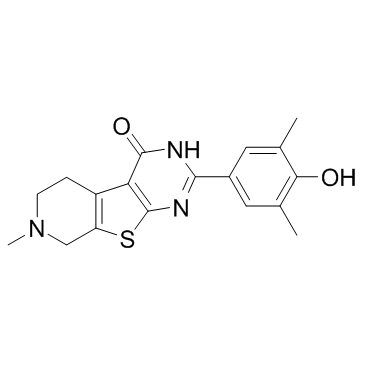
GC16875
FLLL32
FLLL32, un analogue synthétique de la curcumine, est un double inhibiteur JAK2/STAT3 À activité anti-tumorale. FLLL32 peut inhiber l'induction de la phosphorylation de STAT3 par l'IFNα et l'IL-6 dans les cellules cancéreuses du sein.
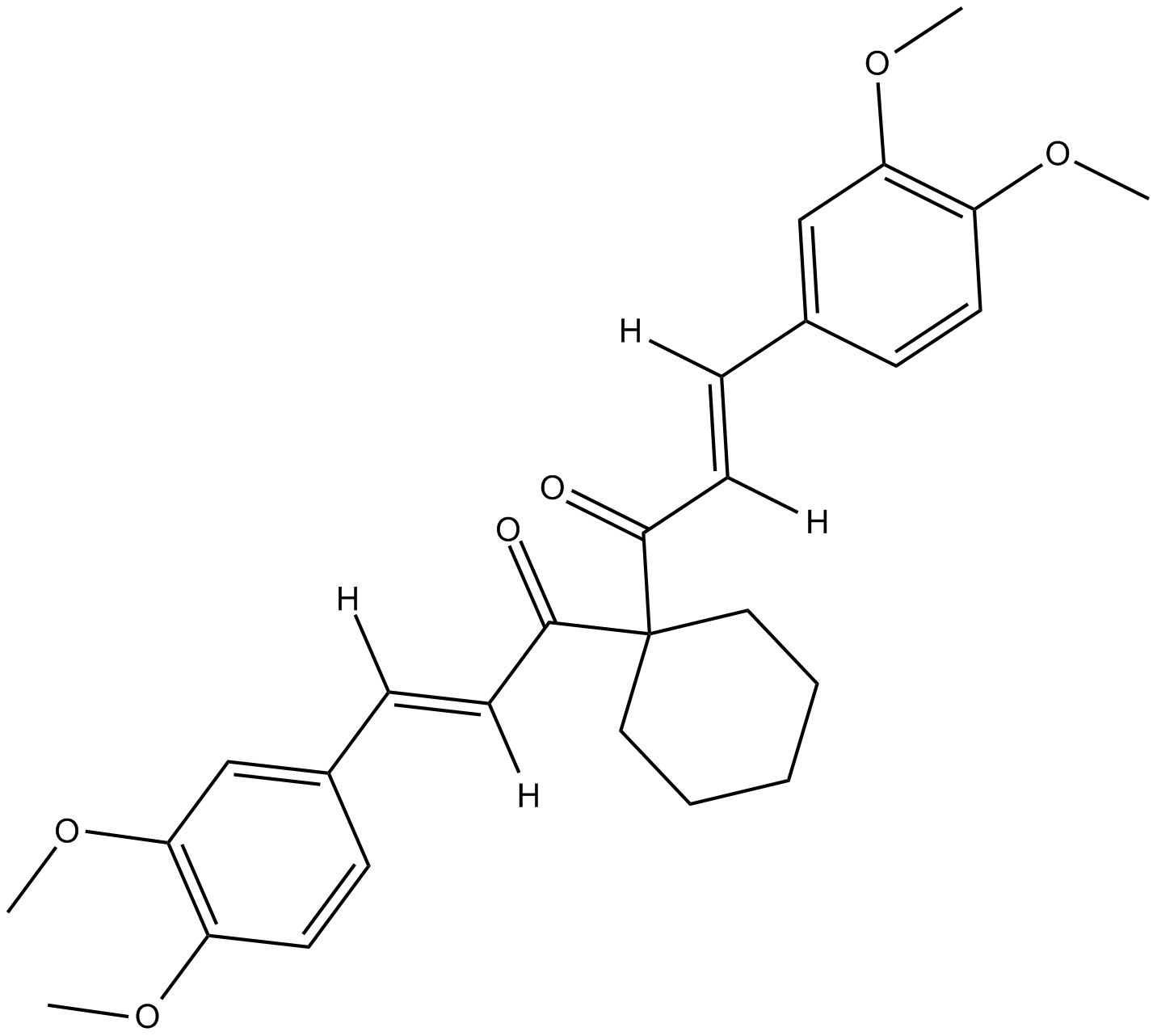
-
GC50506
Fluorescein-NAD+
La fluorescéine-NAD+ est une alternative au NAD radiomarqué et un substrat pour l'ADP-ribosylation.
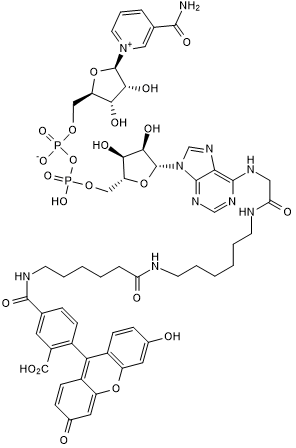
-
GC62121
Fluzoparib
Le fluzoparib (SHR3162) est un inhibiteur de PARP1 puissant et actif par voie orale (IC50 = 1,46 ± 0,72 nM, un test enzymatique acellulaire) avec une activité antitumorale supérieure. Le fluzoparib inhibe sélectivement la prolifération des cellules déficientes en réparation par recombinaison homologue (RH) et sensibilise les cellules déficientes en RH et les cellules compétentes en RH aux agents cytotoxiques. Le fluzoparib présente de bonnes propriétés pharmacocinétiques in vivo et peut être utilisé pour la recherche sur le cancer de l'ovaire récidivant avec mutation BRCA1/2.
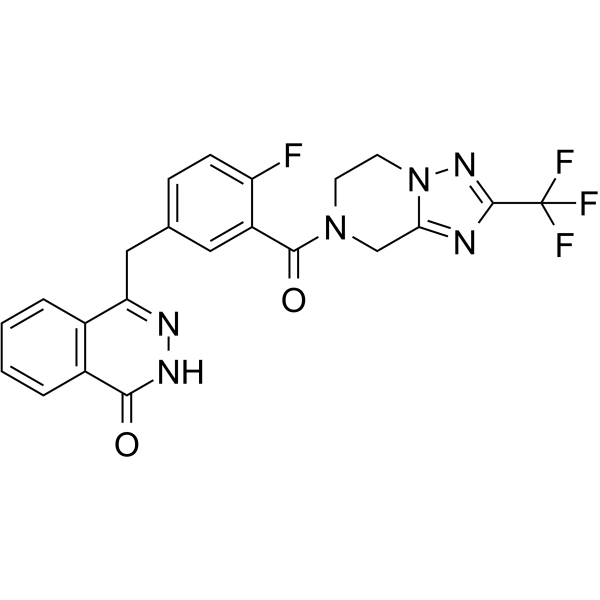
-
GC50537
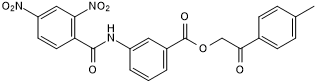
FM19G11
FM19G11 est un inhibiteur du facteur 1-alpha (HIF-1α) inductible par l'hypoxie, et il inhibe l'activité de la luciférase induite par l'hypoxie avec une IC50 de 80 nM dans les cellules HeLa.
-
GC19408
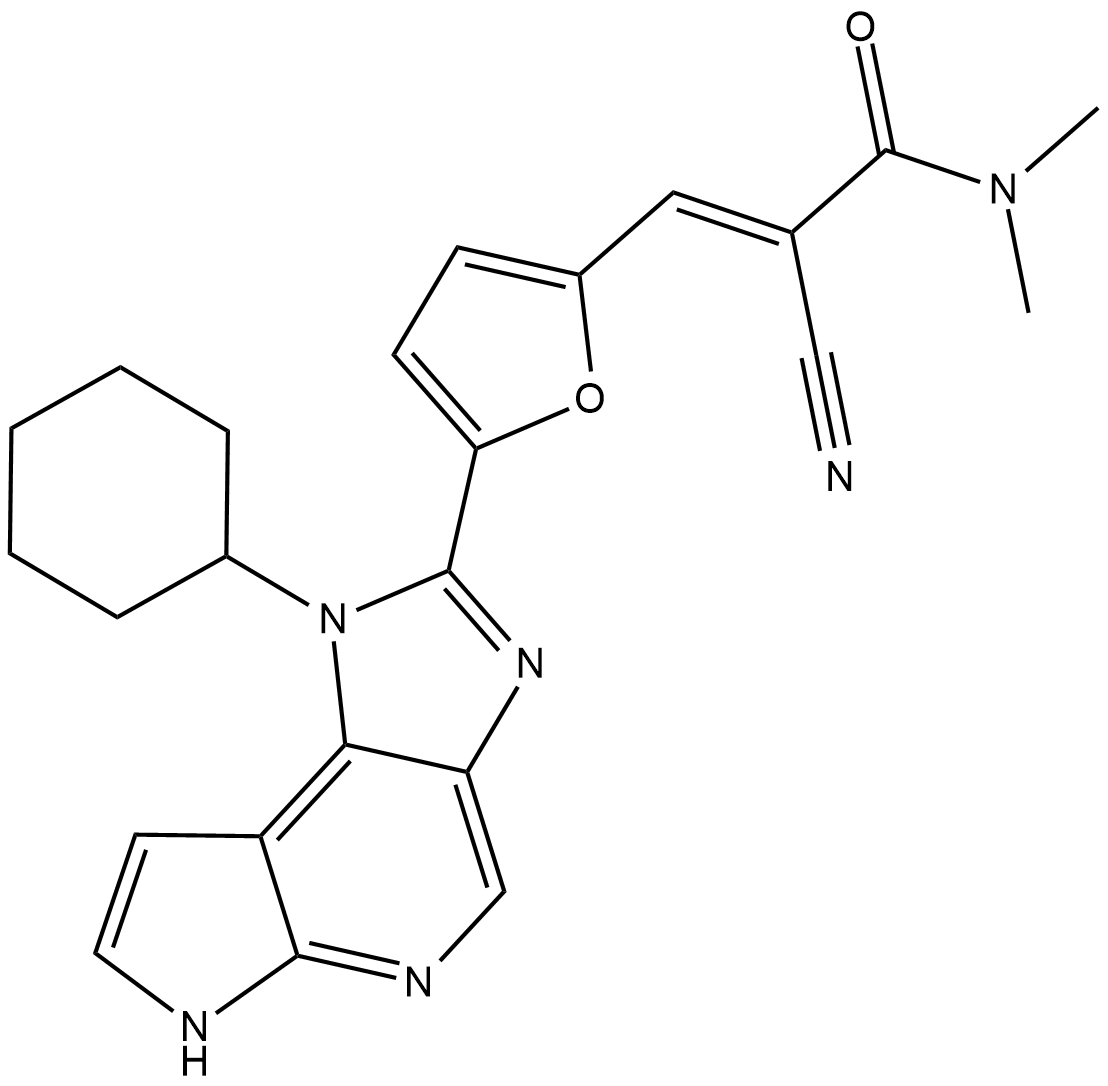
FM381
FM-381 is a highly potent and JAK3-selective janus kinase (JAK) inhibitor
-
GC62461
FNDR-20123
Le FNDR-20123 est un inhibiteur d'HDAC antipaludéen sÛr, premier de sa catégorie et actif par voie orale avec des IC50 de 31 nM et 3 nM pour Plasmodium et HDAC humain, respectivement.

-
GC14395
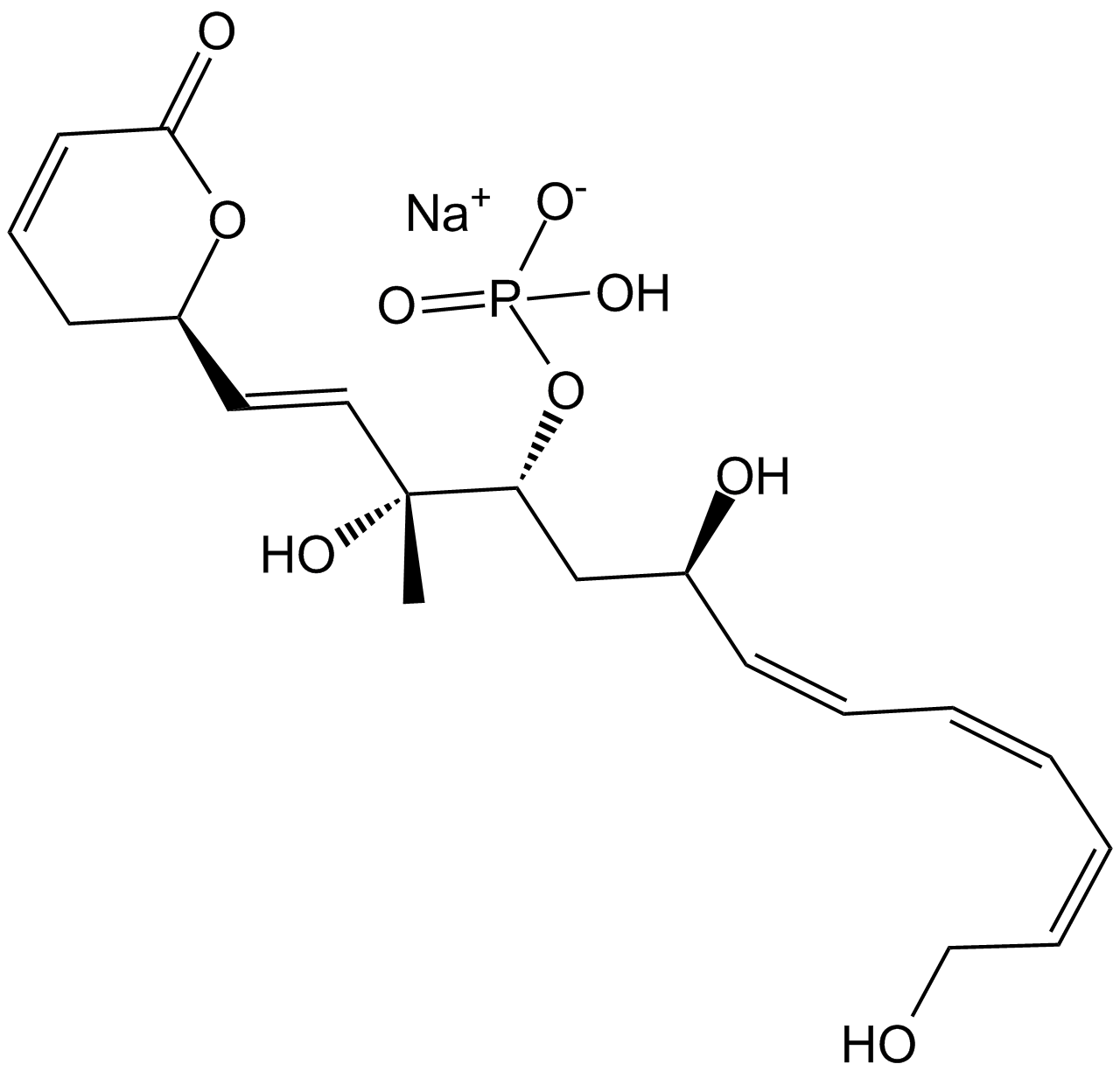
Fostriecin sodium salt
A potent inhibitor of protein phosphatases 2A and 4
-
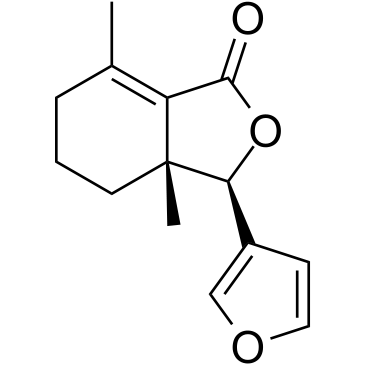
GC38044
Fraxinellone
-
GC65206
FT895
Le FT895 est un inhibiteur de HDAC11 puissant et sélectif avec une IC50 de 3 nM.
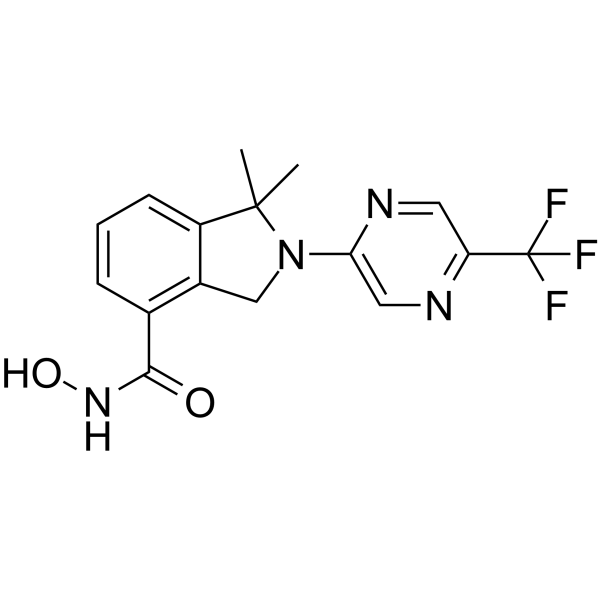
-
GC63545
FTX-6058
Le FTX-6058 ((S)-FTX-6058) est un inhibiteur puissant et oralement actif du développement de l'ectoderme embryonnaire (EED).
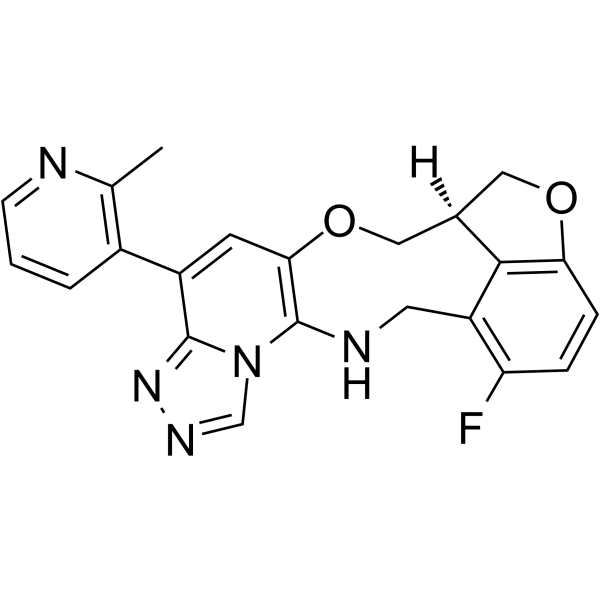
-
GC63546
FTX-6058 hydrochloride
Le chlorhydrate de (S)-Pociredir ((S)-FTX-6058) est un inhibiteur puissant et oralement actif du développement de l'ectoderme embryonnaire (EED).
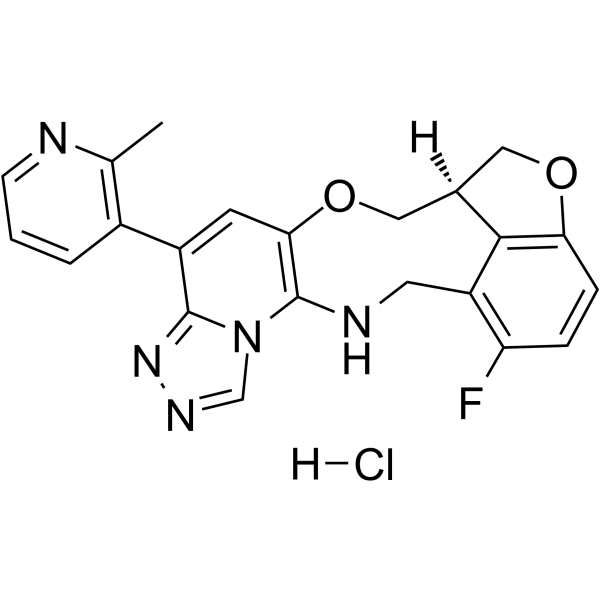
-
GC10456
Fucosterol
Le fucostérol est un stérol isolé d'algues, d'algues ou de diatomées.
-
GC43715
Furamidine (hydrochloride)
La furamidine (chlorhydrate) (dichlorhydrate DB75) est un inhibiteur sélectif de la protéine arginine méthyltransférase 1 (PRMT1) avec une IC50 de 9,4 μM.
-
GC13541
G007-LK
G007-LK est un inhibiteur puissant et sélectif de TNKS1 et TNKS2, avec des IC50 de 46 nM et 25 nM, respectivement.
-
GC62246
G5-7
G5-7, un inhibiteur de JAK2 actif par voie orale et allostérique, inhibe sélectivement la phosphorylation médiée par JAK2 et l'activation d'EGFR (Tyr1068) et de STAT3 en se liant À JAK2. G5-7 induit l'arrêt du cycle cellulaire, l'apoptose et possède un effet anti-angiogénique. G5-7 a le potentiel pour l'étude des gliomes.
-
GC38062
Gambogenic acid
L'acide gambogénique est un ingrédient actif du gamboge, avec une activité anticancéreuse. L'acide gambogénique agit comme un inhibiteur efficace d'EZH2, se lie spécifiquement et de manière covalente À Cys668 dans le domaine EZH2-SET et induit l'ubiquitination d'EZH2.
-
GC40900
Ganoderic Acid D
L'acide ganodérique D, un triterpénoÏde tétracyclique hautement oxygéné, est le principal composant actif du Ganoderma lucidum. L'acide ganodérique D régule À la hausse l'expression protéique de SIRT3 et induit la cyclophiline D désacétylée (CypD) par SIRT3. L'acide ganodérique D inhibe la reprogrammation énergétique des cellules cancéreuses du cÔlon, y compris l'absorption de glucose, la production de lactate, la production de pyruvate et d'acétyl-coenzyme dans les cellules cancéreuses du cÔlon. L'acide ganodérique D induit l'apoptose du carcinome cervical humain HeLa.
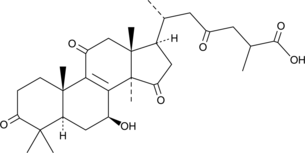
-
GC13474
Garcinol
Le garcinol, une benzophénone polyisoprénylée récoltée À partir de Garcinia indica, exerce des propriétés anticholinestérases vis-À-vis de l'acétylcholinestérase (AChE) et de la butyrylcholinestérase (BChE) avec des IC50 de 0,66 μM et 7,39 μM, respectivement.
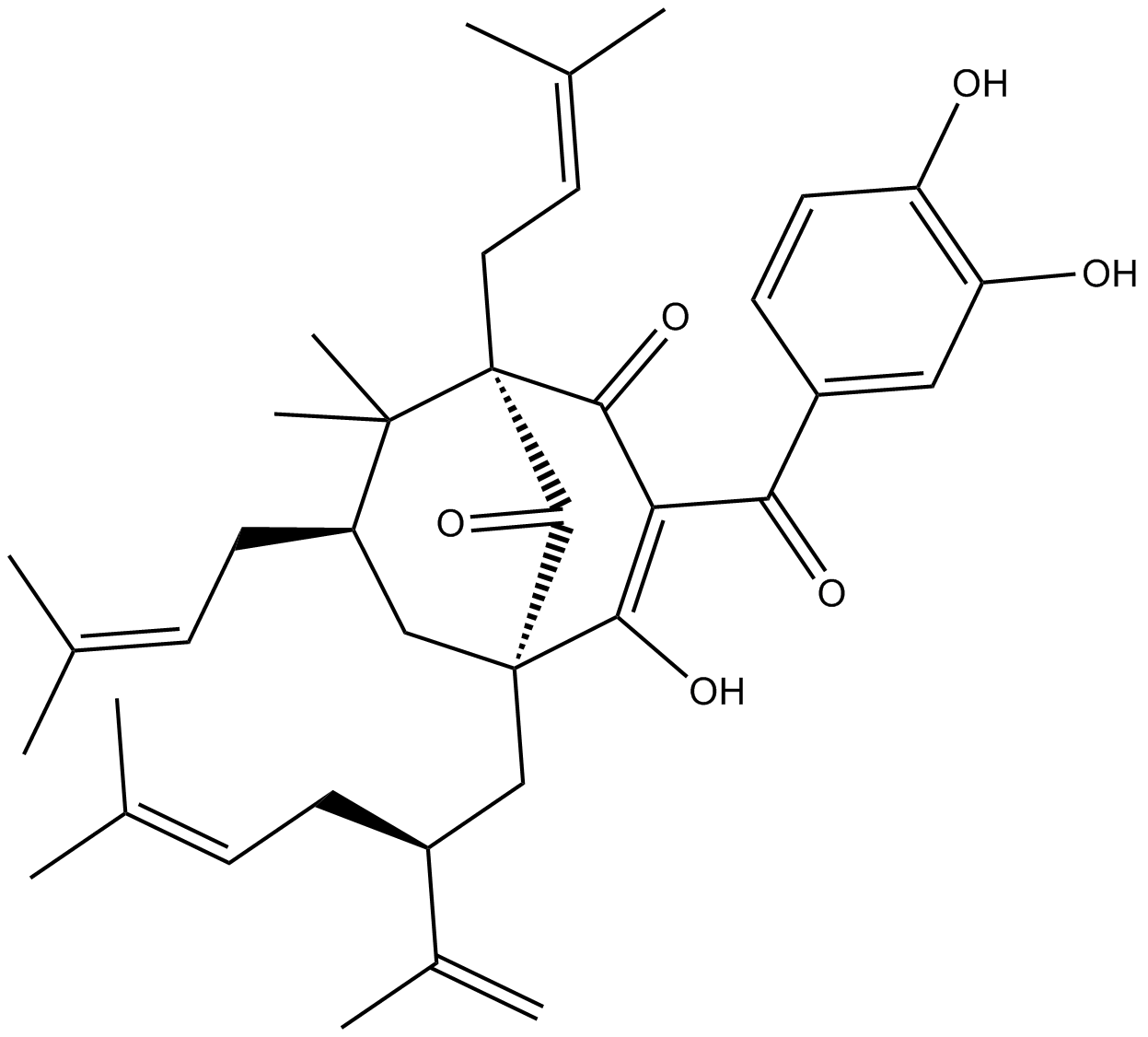
-
GC60172
Gardenia yellow
Le jaune de gardénia est un membre actif de la crocine, augmente l'expression de l'ARNm de SIRT3 et agit comme un agent antidépresseur actif par voie orale .

-
GC32958
GDC-0339
Le GDC-0339 est un inhibiteur de pan-Pim kinase puissant, biodisponible par voie orale et bien toléré, avec un Kis de 0,03 nM, 0,1 nM et 0,02 nM pour Pim1, Pim2 et Pim3, respectivement. Le GDC-0339 est découvert comme traitement potentiel du myélome multiple.
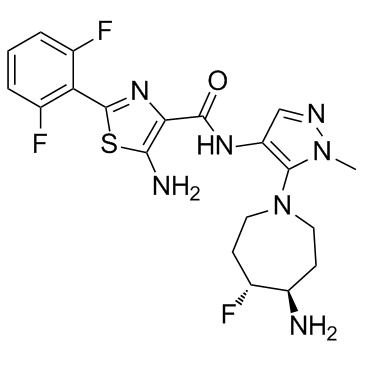
-
GC68378
GDC-4379

-
GC19542
GeA-69
GeA-69 est un inhibiteur allostérique sélectif de la poly-adénosine-diphosphate-ribose polymérase 14 (PARP14) ciblant le macrodomaine 2 (MD2), avec une valeur Kd de 2,1 µM. GeA-69 implique des mécanismes de réparation des dommages à l'ADN et empêche le recrutement de PARP14 MD2 sur les sites de dommages à l'ADN induits par laser.

-
GN10473
Ginkgolide C
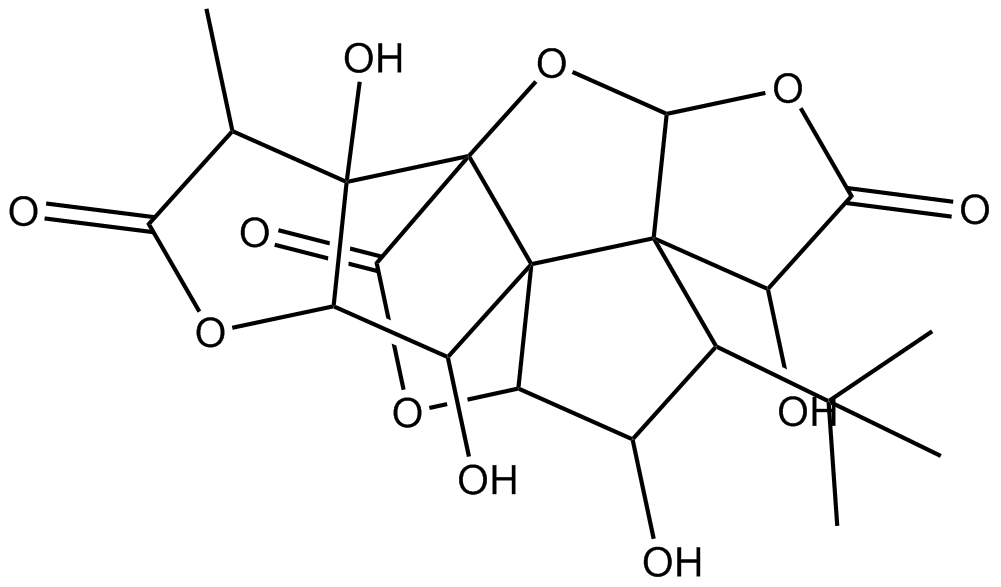
-
GN10584
Ginsenoside Rk1
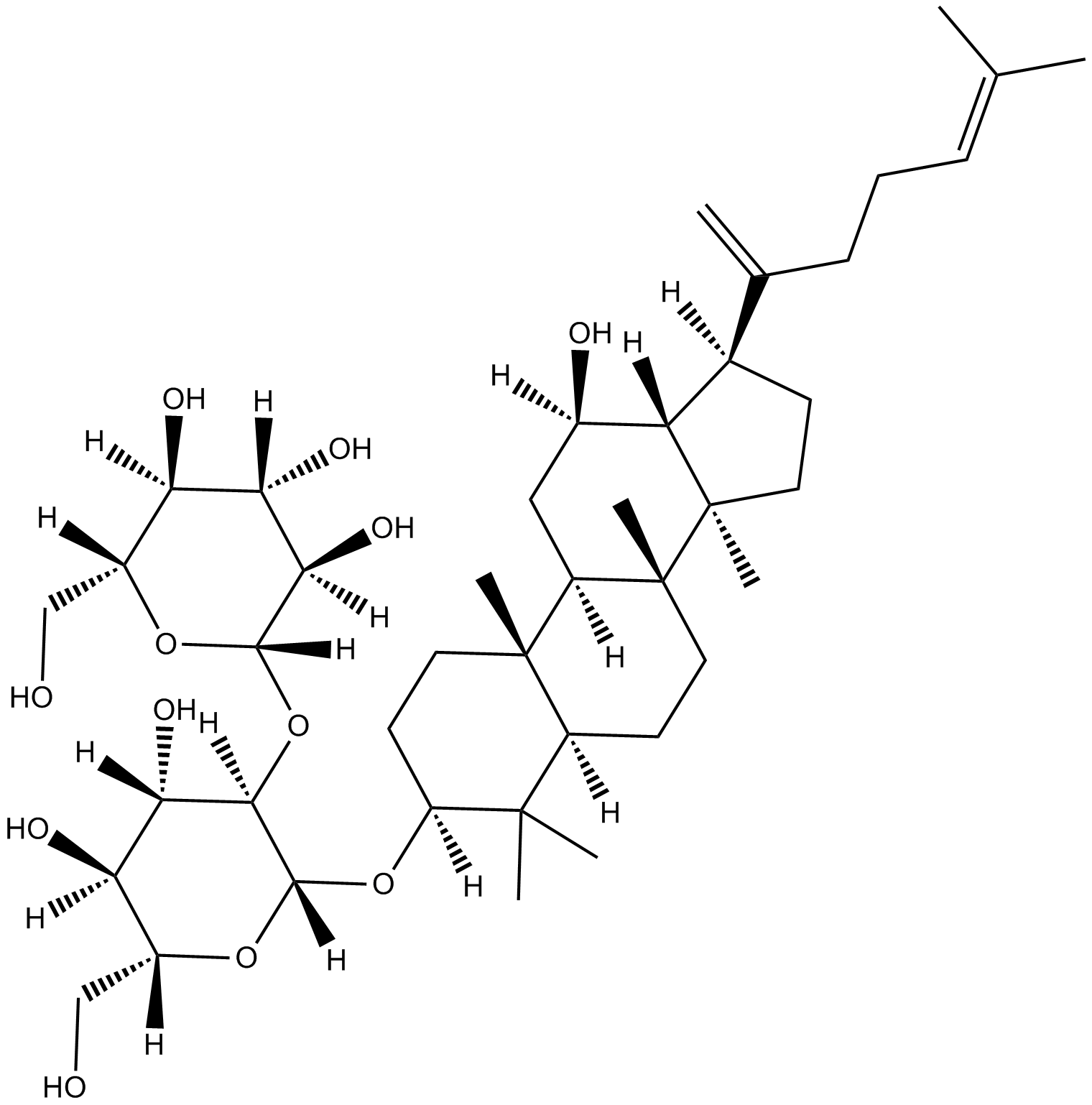
-
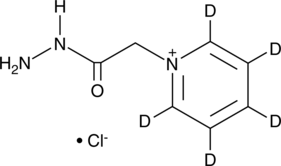
GC48994
Girard’s Reagent P-d5
An internal standard for the quantification of Girard’s reagent P
-
GC33050
Givinostat (ITF-2357)
Givinostat (ITF-2357) (ITF-2357) est un inhibiteur d'HDAC avec une IC50 de 198 et 157 nM pour HDAC1 et HDAC3, respectivement.

-
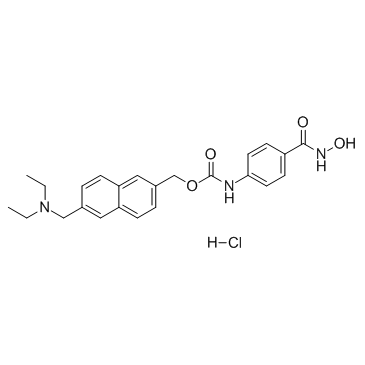
GC33159
Givinostat hydrochloride (ITF-2357 hydrochloride)
Le chlorhydrate de givinostat (ITF-2357) est un inhibiteur d'HDAC avec une CI50 de 198 et 157 nM pour HDAC1 et HDAC3, respectivement.
-
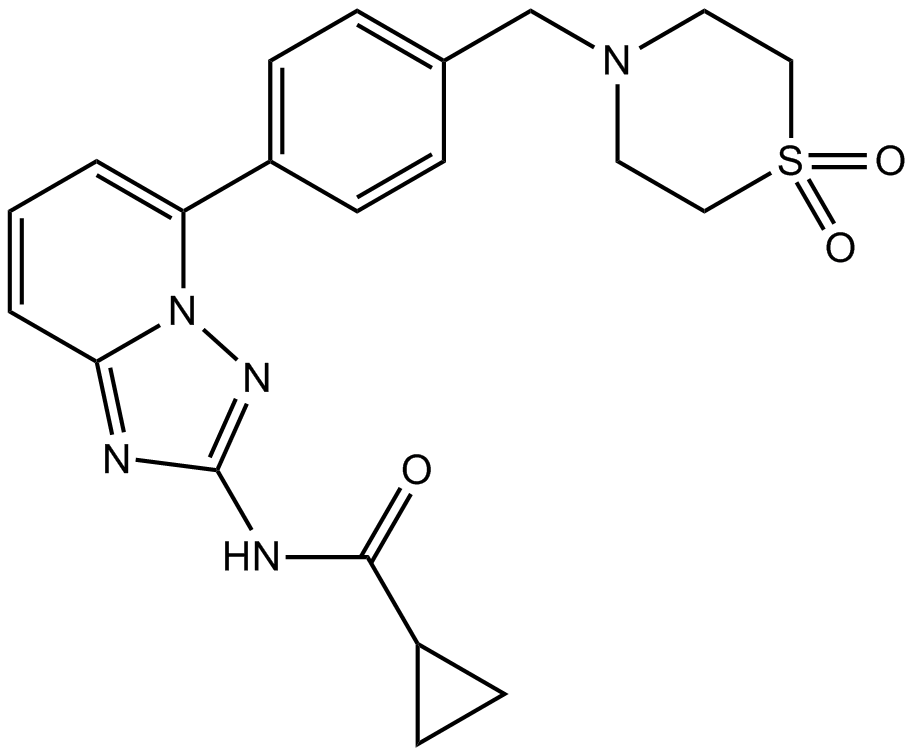
GC12579
GLPG0634
GLPG0634 (GLPG0634) est un inhibiteur de JAK1 sélectif et actif par voie orale avec une IC50 de 10 nM, 28 nM, 810 nM et 116 nM pour JAK1, JAK2, JAK3 et TYK2, respectivement.
-
GC17222
GLPG0634 analogue
L'analogue GLPG0634 (Compoun 176) est un inhibiteur de JAK À large spectre avec des valeurs IC50 <100 nM contre JAK1, JAK2 et JAK3.
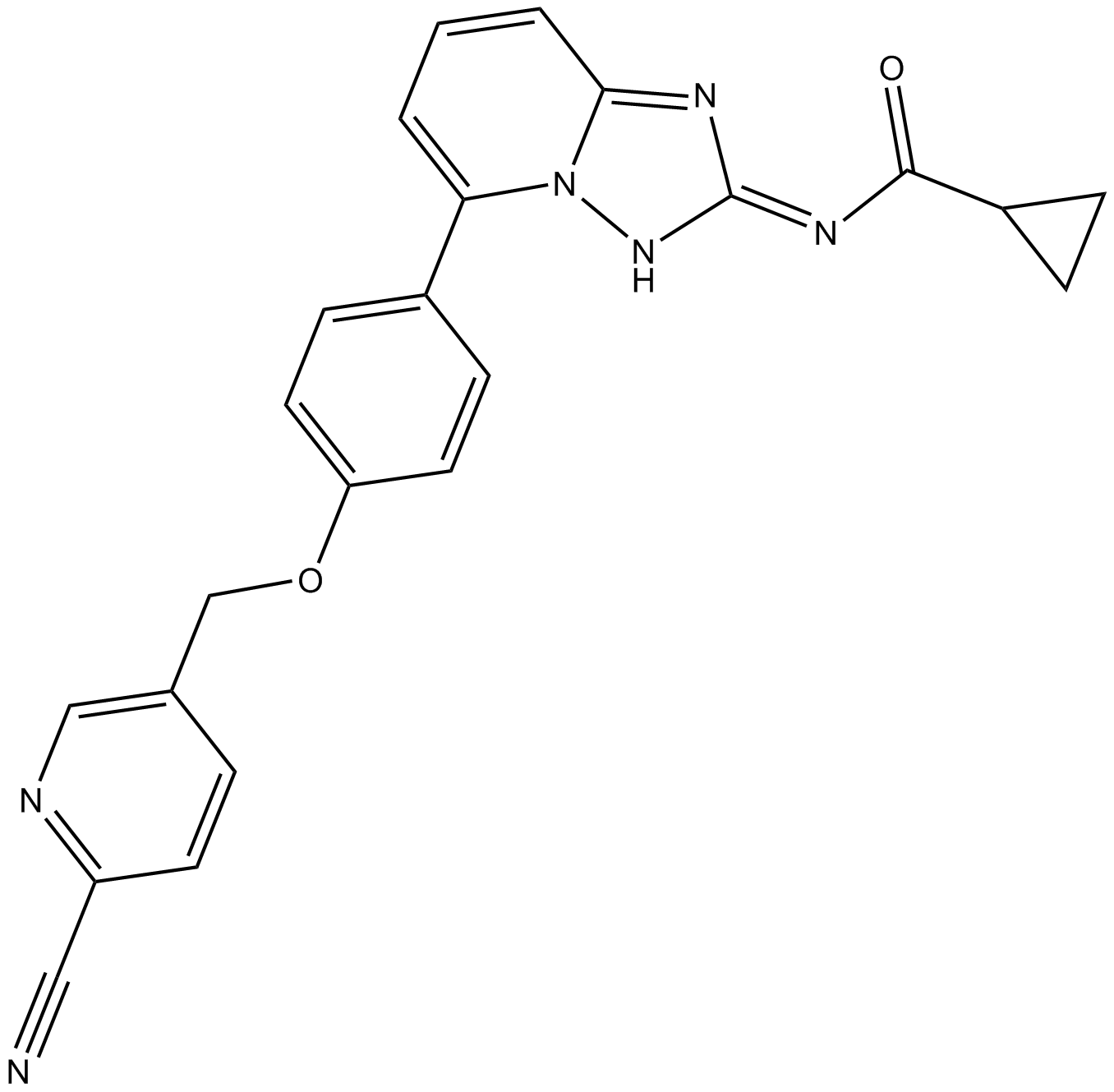
-
GC30263
Glucosamine (D-Glucosamine)
La glucosamine (D-Glucosamine) (D-Glucosamine (D-Glucosamine)) est un sucre aminé et un précurseur important dans la synthèse biochimique des protéines et des lipides glycosylés, est utilisée comme complément alimentaire.
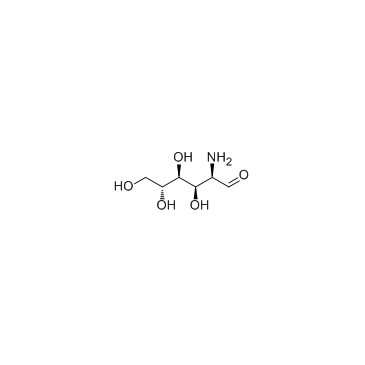
-
GN10669
Glucosamine sulfate
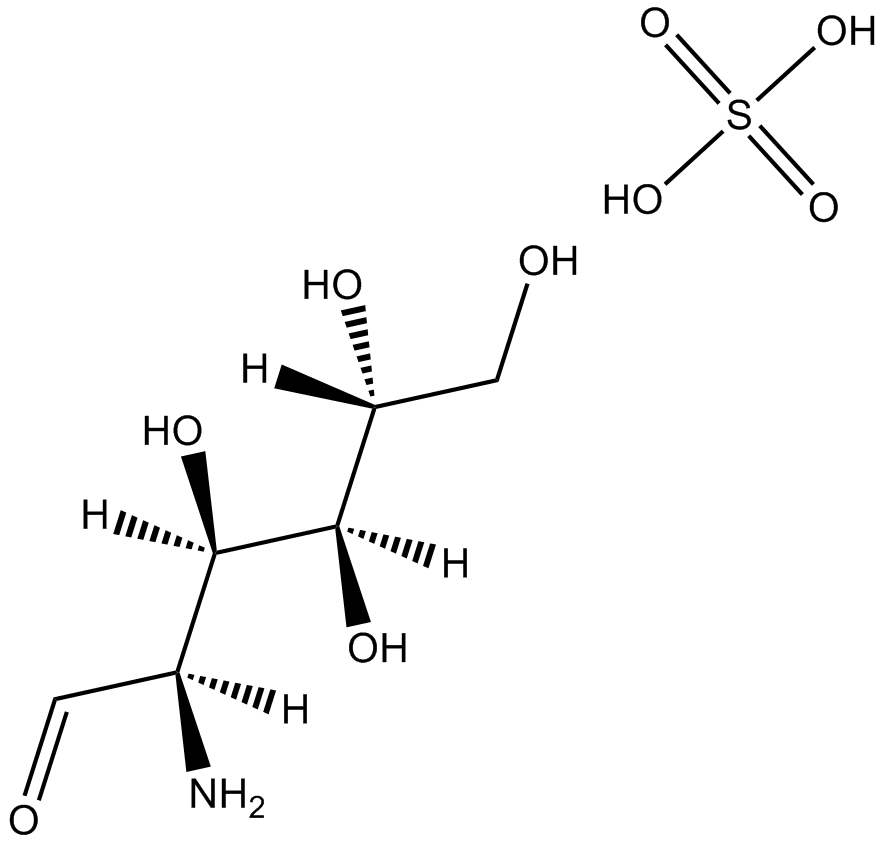
-
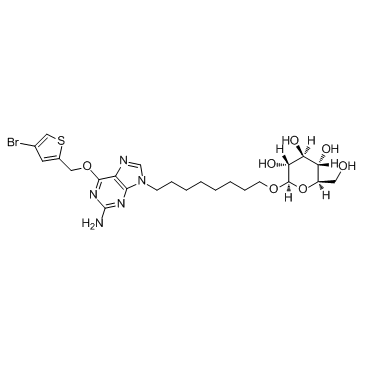
GC36153
Glucose-conjugated MGMT inhibitor
L'inhibiteur de MGMT conjugué au glucose est un puissant inhibiteur de la O6-méthylguanine-ADNméthyl-transférase (MGMT), avec des CI50 de 32 nM in vitro (extraits cellulaires) et de 10 nM dans les cellules HeLa S3.
-
GC34595
GN44028
GN44028 est un inhibiteur puissant et actif par voie orale du facteur inductible par l'hypoxie (HIF)-1α, avec une IC50 de 14 nM. GN44028 inhibe l'activité transcriptionnelle de HIF-1α induite par l'hypoxie sans supprimer l'expression de l'ARNm de HIF-1α, l'accumulation de protéines HIF-1α ou l'hétérodimérisation HIF-1α/HIF-1β. Le GN44028 peut être utilisé dans la recherche sur les cancers.
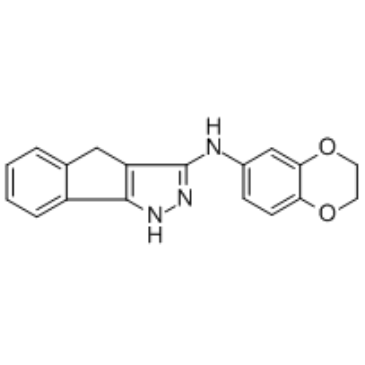
-
GC36168
GNA002
GNA002 est un inhibiteur EZH2 (Enhancer of zeste homolog 2) très puissant, spécifique et covalent avec une IC50 de 1,1 μM. GNA002 peut se lier spécifiquement et de manière covalente À Cys668 dans le domaine EZH2-SET, déclenchant la dégradation de EZH2 par l'extrémité COOH de l'ubiquitination médiée par la protéine interagissant avec Hsp70 (CHIP). GNA002 réduit efficacement la triméthylation H3K27 médiée par EZH2, réactive les gènes suppresseurs de tumeurs silencieux du complexe répresseur polycomb 2 (PRC2).
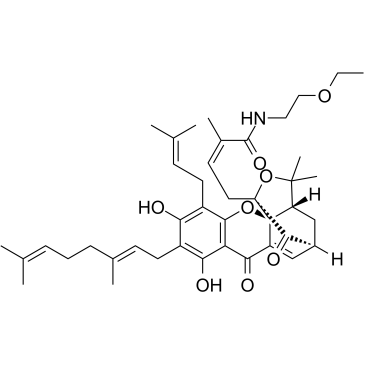
-
GC32960
GNE-049
GNE-049 est un inhibiteur de CBP très puissant et sélectif avec une IC50 de 1,1 nM dans le test TR-FRET. GNE-049 inhibe également BRET et BRD4(1) avec des IC50 de 12 nM et 4200 nM, respectivement.
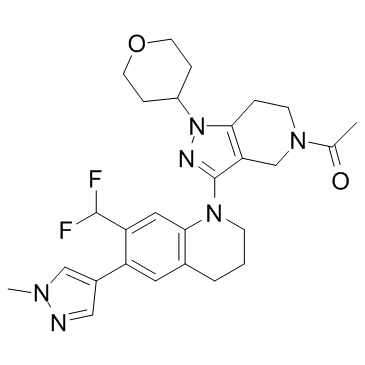
-
GC68439
GNE-064

-
GC33212
GNE-207
GNE-207 est un inhibiteur puissant, sélectif et biodisponible par voie orale du bromodomaine de CBP, avec une IC50 de 1 nM, présente un indice sélectif de> 2500 fois contre BRD4 (1). GNE-207 montre une excellente puissance CBP, avec une CE50 de 18 nM pour l'expression de MYC dans les cellules MV-4-11.
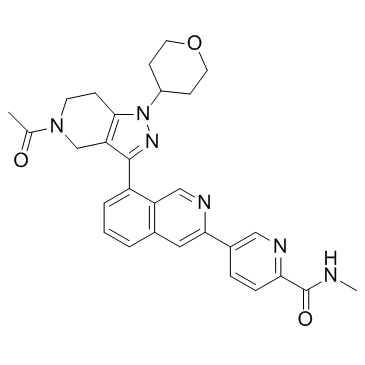
-
GC32747
GNE-272
GNE-272 est un inhibiteur puissant et sélectif de CBP/EP300 avec des valeurs IC50 de 0,02, 0,03 et 13 μM pour CBP, EP300 et BRD4, respectivement. GNE-272 est également une sonde sélective in vivo pour CBP/EP300.