DNA Damage/DNA Repair
- MTH1(4)
- PARP(84)
- ATM/ATR(30)
- DNA Alkylating(20)
- DNA Ligases(3)
- DNA Methyltransferase(25)
- DNA-PK(30)
- HDAC(136)
- Nucleoside Antimetabolite/Analogue(140)
- Telomerase(17)
- Topoisomerase(151)
- tankyrase(5)
- Antifolate(38)
- CDK(250)
- Checkpoint Kinase (Chk)(31)
- CRISPR/Cas9(9)
- Deubiquitinase(72)
- DNA Alkylator/Crosslinker(71)
- DNA/RNA Synthesis(400)
- Eukaryotic Initiation Factor (eIF)(23)
- IRE1(23)
- LIM Kinase (LIMK)(9)
- TOPK(5)
- Casein Kinase(61)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Products for DNA Damage/DNA Repair
- Cat.No. Nombre del producto Información
-
GC38000
β-Boswellic acid
β-El ácido boswélico se aísla de la resina de goma del serrado de Boswellia. β-El ácido boswélico es un inhibidor de tipo no reductor de la formación del producto 5-lipoxigenasa (5-LO) que interactúa directamente con la 5-LO o bloquea su translocación . β-El ácido boswélico inhibe la síntesis de ADN, ARN y proteína en células de leucemia humana HL-60.
-
GC31365
γ-Oryzanol
γ-Oryzanol es un potente inhibidor de las ADN metiltransferasas (DNMT) en el cuerpo estriado de ratones.
-
GC38194
(±)-10-Hydroxycamptothecin
(±)-10-hidroxicamptotecina es un alcaloide de indol que inhibe la actividad de la topoisomerasa I y tiene un amplio espectro de actividad anticancerígena.
-
GC33107
(±)-BAY-1251152
(±)-BAY-1251152 ((±)-BAY-1251152) es una mezcla racémica de BAY-1251152. BAY-1251152 es un inhibidor potente y altamente selectivo de PTEF/CDK9.

-
GC10867
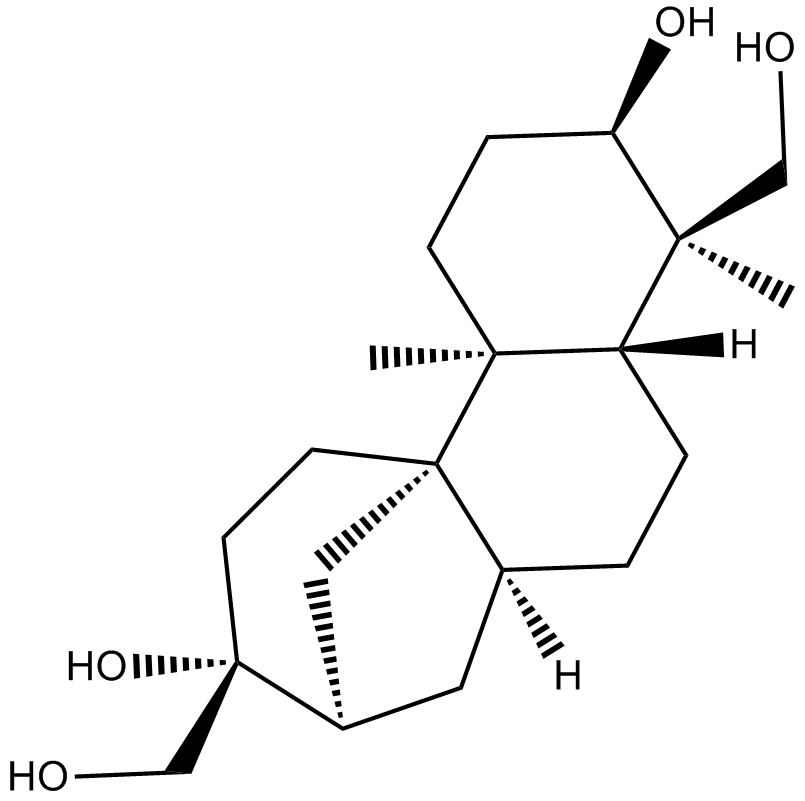
(+)-Aphidicolin
Aphidicolina ((+)-Aphidicolina), un inhibidor reversible de la replicación del ADN nuclear eucariota, puede bloquear el ciclo celular en la fase pre-S[1].
-
GC34955
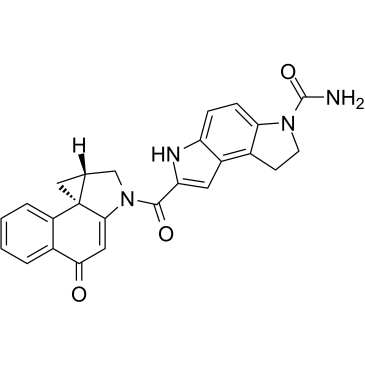
(+)-CBI-CDPI1
(+)-CBI-CDPI1 es un anÁlogo funcional mejorado de CC-1065. (+)-CBI-CDPI1 es un agente alquilante de ADN. (+)-CBI-CDPI1 es una toxina de conjugados de fÁrmacos de anticuerpos (ADC).
-
GC34956
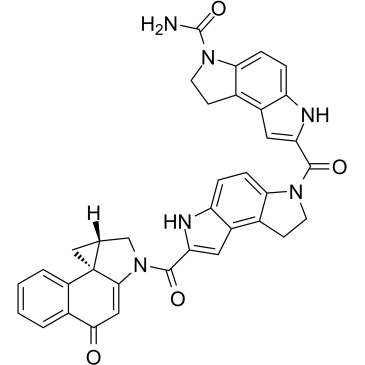
(+)-CBI-CDPI2
(+)-CBI-CDPI2 es un anÁlogo funcional mejorado de CC-1065. (+)-CBI-CDPI1 es un agente alquilante de ADN. (+)-CBI-CDPI2 es una toxina de conjugados de fÁrmacos de anticuerpos (ADC).
-
GC32429
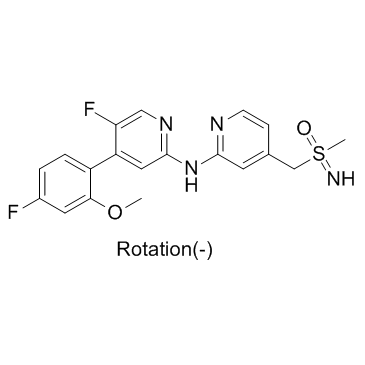
(-)-BAY-1251152
(-)-BAY-1251152 ((-)-BAY-1251152) es un enantiÓmero de BAY-1251152 con rotaciÓn (-). BAY-1251152 es un inhibidor potente y altamente selectivo de PTEF/CDK9.
-
GC48635
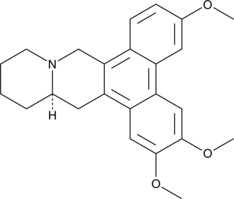
(-)-Cryptopleurine
An alkaloid with diverse biological activities
-
GC40076
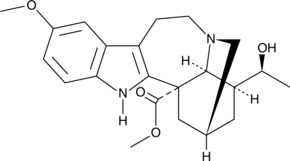
(-)-Voacangarine
(-)-Voacangarine is an indole alkaloid originally isolated from V.
-
GC38377
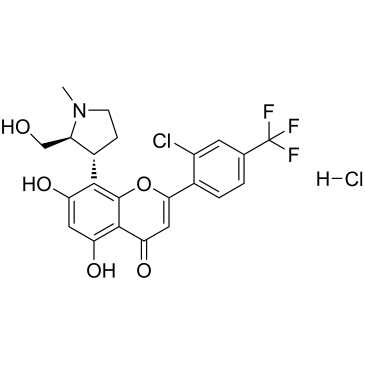
(2S,3R)-Voruciclib hydrochloride
El clorhidrato de (2S,3R)-voruciclib es el enantiÓmero del clorhidrato de voruciclib. (2S,3R)-Voruciclib es un inhibidor de CDK activo por vÍa oral.
-
GC41695
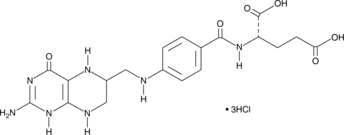
(6R,S)-5,6,7,8-Tetrahydrofolic Acid (hydrochloride)
(6R,S)-5,6,7,8-Tetrahydrofolic acid (THFA), the reduced form of folic acid, serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
-
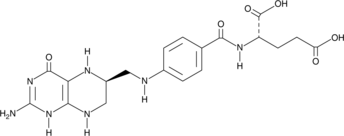
GC41088
(6S)-Tetrahydrofolic Acid
(6S)-Tetrahydrofolic acid is a diastereomer of tetrahydrofolic acid, a reduced form of folic acid that serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
-
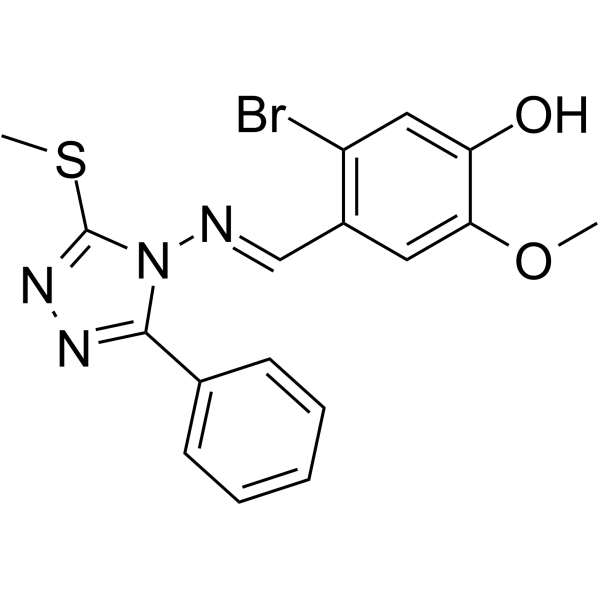
GC62735
(E/Z)-GO289
(E/Z)-GO289 es un inhibidor potente y selectivo de la caseÍna quinasa 2 (CK2) (IC50=7 nM). (E/Z)-GO289 alarga fuertemente el perÍodo circadiano. (E/Z)-GO289 exhibe una inhibiciÓn dependiente del tipo de célula del crecimiento de células cancerosas que se correlaciona con la funciÓn del reloj celular.
-
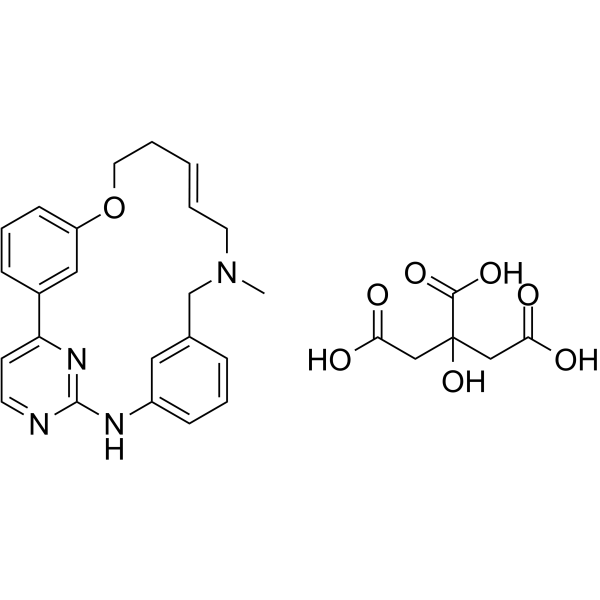
GC64429
(E/Z)-Zotiraciclib citrate
El citrato de (E/Z)-zotiraciclib es un potente inhibidor de CDK2, JAK2 y FLT3.
-
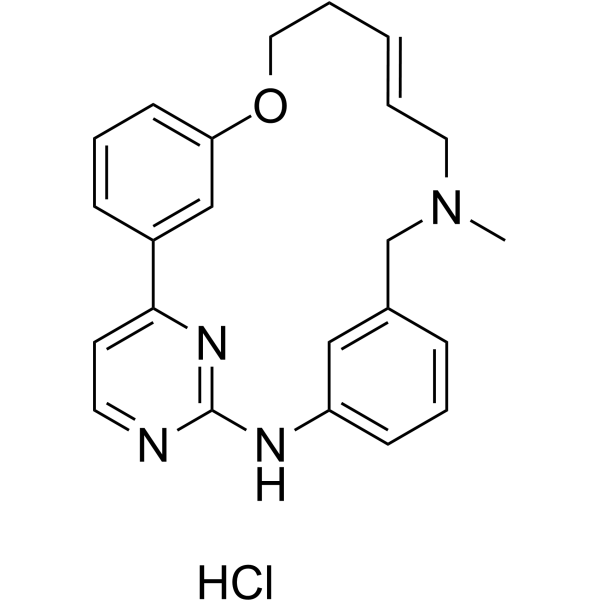
GC63864
(E/Z)-Zotiraciclib hydrochloride
El clorhidrato de (E/Z)-zotiraciclib ((E/Z)-TG02) es un potente inhibidor de CDK2, JAK2 y FLT3.
-
GC69793
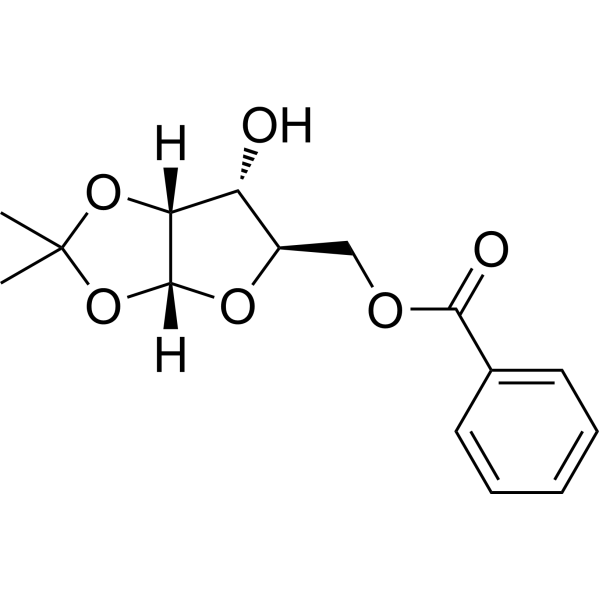
(R)-5-O-Benzoyl-1,2-di-O-isopropylidene-alpha-D-xylofuranose
(R)-5-O-Benzoyl-1,2-di-O-isopropilideno-alpha-D-xilofuranosa es un análogo de nucleósido de purina. Los análogos de nucleósidos de purina tienen una amplia actividad antitumoral y se dirigen a los tumores malignos del sistema linfático inactivo. El mecanismo anticancerígeno en este proceso depende de la inhibición de la síntesis del ADN, inducción de apoptosis y otros factores.
-
GC41716
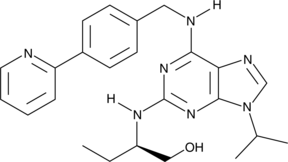
(R)-CR8
(R)-CR8 (CR8), un análogo de segunda generación de roscovitina, es un potente inhibidor de CDK1/2/5/7/9.
-
GC39281
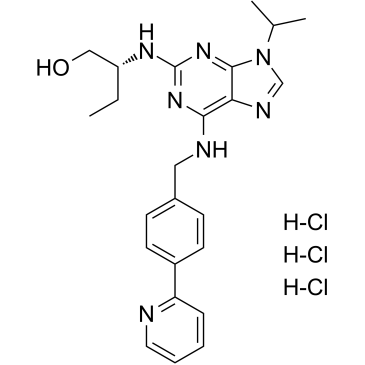
(R)-CR8 trihydrochloride
El triclorhidrato de (R)-CR8 (CR8), un anÁlogo de segunda generaciÓn de la roscovitina, es un potente inhibidor de CDK1/2/5/7/9.
-
GC64210
(R)-GSK-3685032
(R)-GSK-3685032 es el enantiÓmero R de GSK-3685032. GSK-3685032 es un inhibidor selectivo de DNMT1 reversible no dependiente del tiempo, no covalente, primero en su clase, con una IC50 de 0,036 μM. GSK-3685032 induce una fuerte pérdida de metilaciÓn del ADN, activaciÓn transcripcional e inhibiciÓn del crecimiento de células cancerosas.
-
GC41633
(R)-Prunasin
(R)-prunasina es un inhibidor de la ADN polimerasa β.
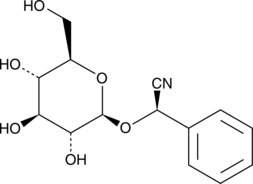
-
GC34124
(rel)-MC180295
(rel)-MC180295 ((rel)-(rel)-MC180295) es un inhibidor potente y selectivo de CDK9-Cyclin T1, con una IC50 de 5 nM, al menos 22 veces mÁs selectivo para CDK9 que para otras CDK. (rel)-MC180295 también inhibe GSK-3α y GSK-3β. (rel)-MC180295 ((rel)-(rel)-MC180295) tiene un potente efecto antitumoral.
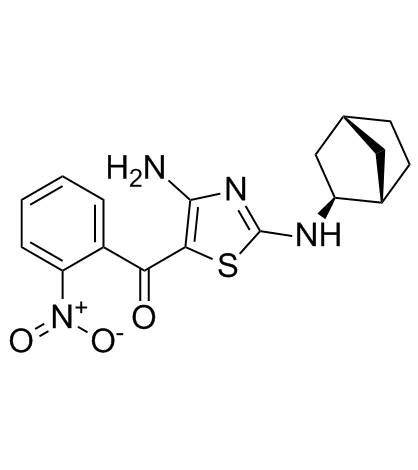
-
GC10098
(S)-10-Hydroxycamptothecin
La (S)-10-hidroxicamptotecina (10-HCPT;10-hidroxicamptotecina) es un inhibidor de la topoisomerasa I del ADN aislado de la planta china Camptotheca accuminata. La (S)-10-hidroxicamptotecina muestra un notable efecto inductor de la apoptosis. La (S)-10-hidroxicamptotecina tiene potencial para el tratamiento de hepatoma, carcinoma gÁstrico, cÁncer de colon y leucemia.
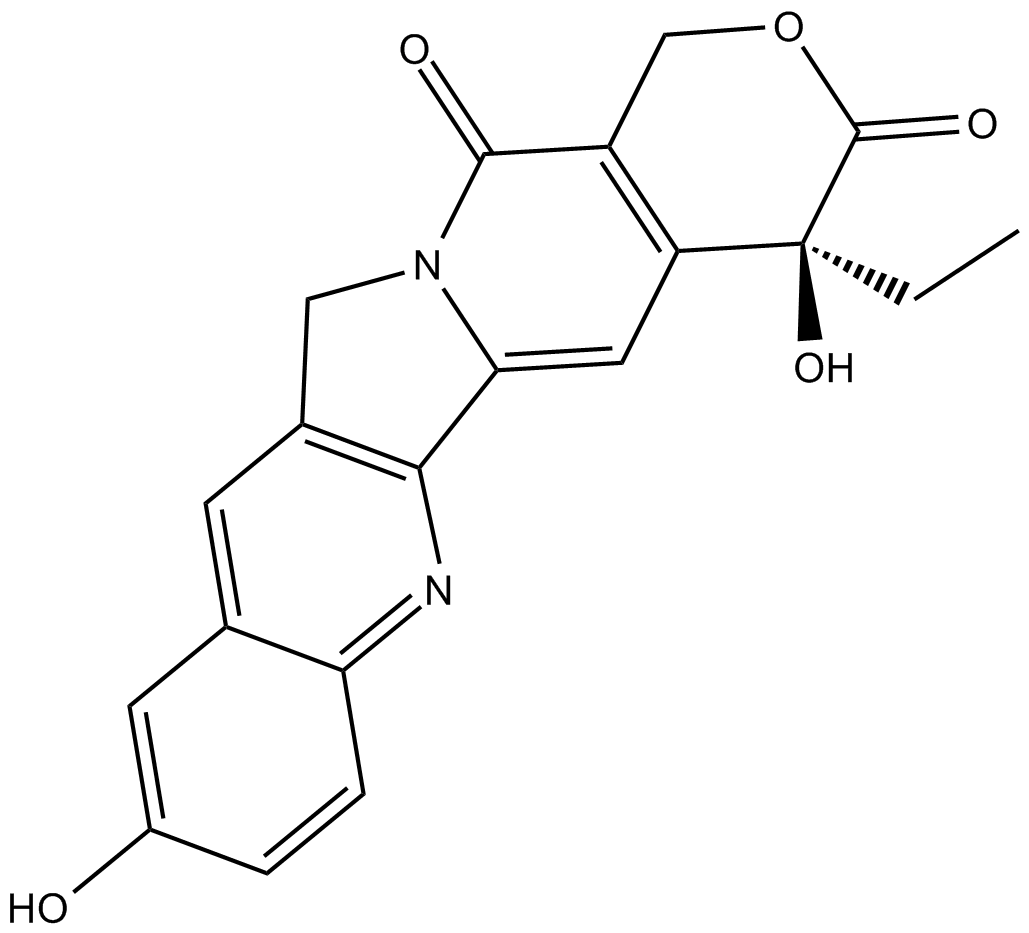
-
GC34999
(S)-Ceralasertib
(S)-Ceralasertib ((S)-AZD6738) se extrae de la patente WO2011154737A1, Compuesto II, presenta una IC50 de 2,578 nM.(S)-Ceralasertib es un potente y selectivo inhibidor de ATR de sulfoximina morfolinopirimidina con excelente farmacocinética y fisicoquÍmica preclÍnica (PK ) caracterÍsticas.(S)-Ceralasertib se desarrolla mejorando la solubilidad acuosa y elimina la inhibiciÓn dependiente del tiempo de CYP3A4.
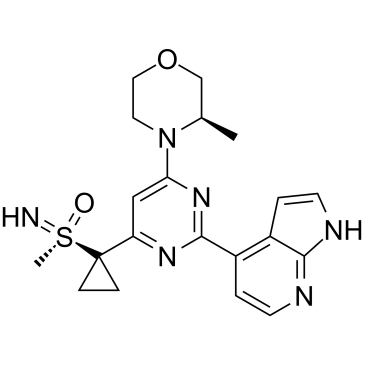
-
GC46351
(S)-CR8
(S)-CR8 es el isÓmero S de CR8. (S)-CR8 es un inhibidor de CDK potente y selectivo con IC50 de 0,060, 0,080, 0,11, 0,12 y 0,15 μM para CDK2/ciclina E, CDK2/ciclina A, CDK9/ciclina T, CDK5/p25 y CDK1/ciclina B, respectivamente. (S)-CR8 reduce la supervivencia de las células SH-SY5Y (IC50 0,40μM).
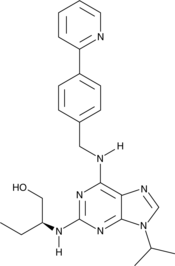
-
GC13136
(S)-Crizotinib
(S)-Crizotinib es un inhibidor potente y selectivo de MTH1 (homÓlogo de mutT) con una IC50 de 330 nM. (S)-Crizotinib interrumpe la homeostasis del grupo de nucleÓtidos a través de la inhibiciÓn de MTH1, induce un aumento en las roturas de cadena simple de ADN, activa la reparaciÓn de ADN en células de carcinoma de colon humano y suprime de manera efectiva el crecimiento tumoral en modelos animales.
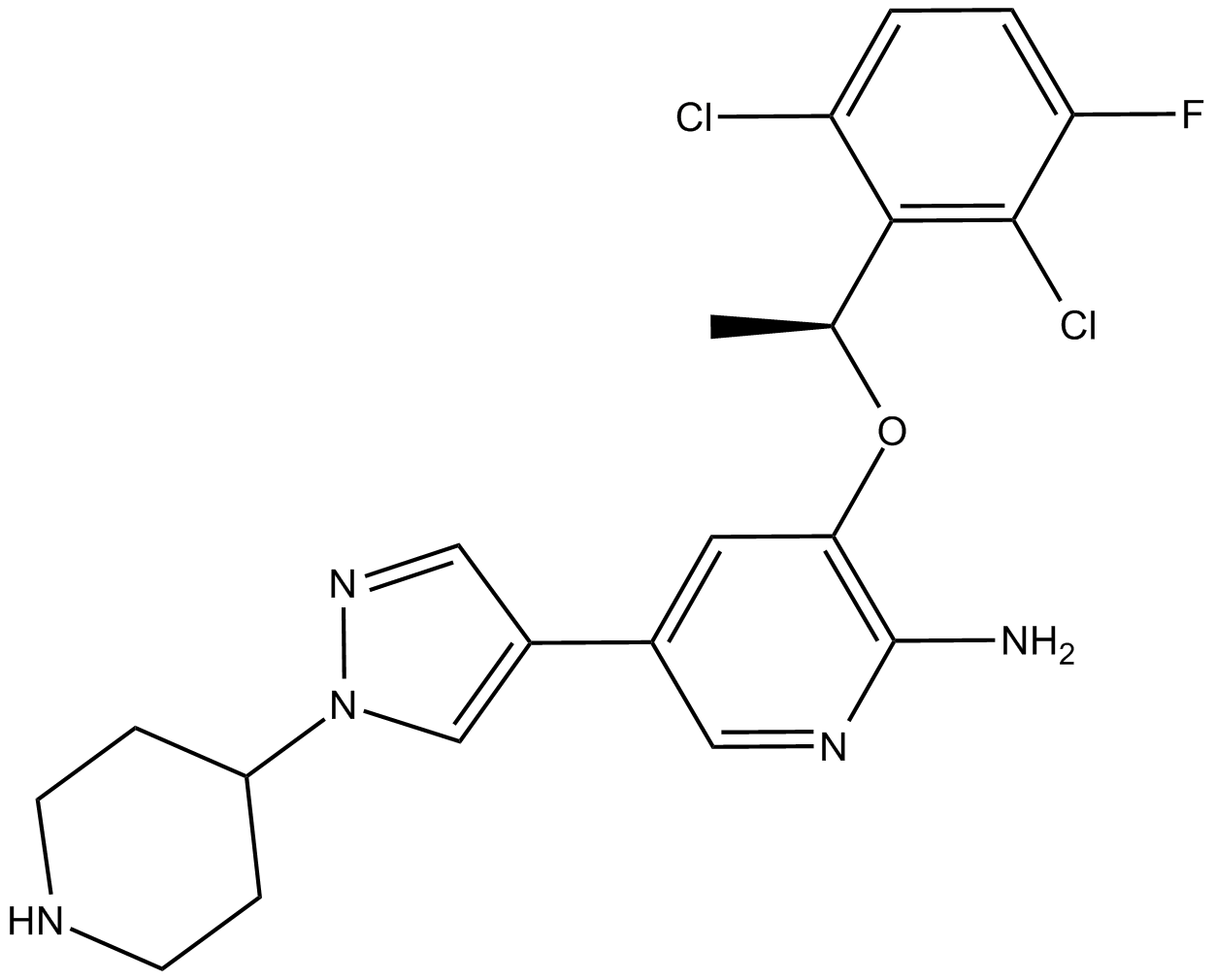
-
GC65877
(S)-GFB-12811
(S)-GFB-12811 (compuesto 596) es un inhibidor potente y selectivo de CDK5, con un valor IC50 inferior a 10 nM. (S)-GFB-12811 se puede utilizar en la investigaciÓn de la progresiÓn del ciclo celular, el desarrollo neuronal y la tumorigénesis.
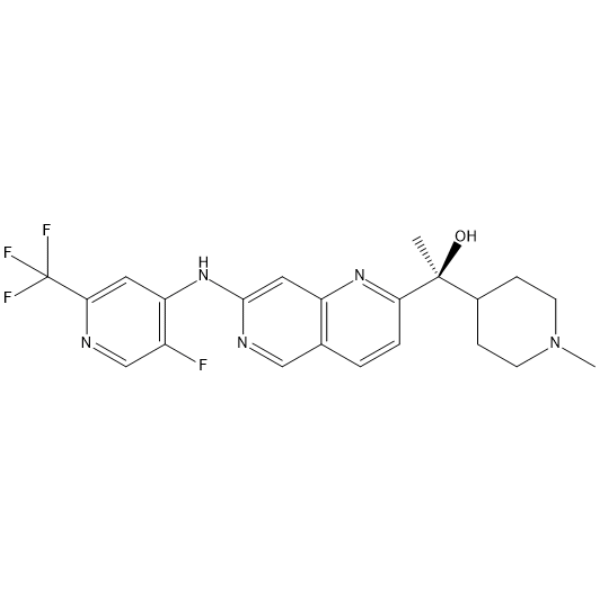
-
GC65997
(S)-LY3177833 hydrate
El hidrato de (S)-LY3177833 ((S)-Ejemplo 2) es un inhibidor de la cinasa CDC7 activo por vÍa oral. El hidrato de (S)-LY3177833 muestra una amplia actividad anticancerÍgena in vitro.
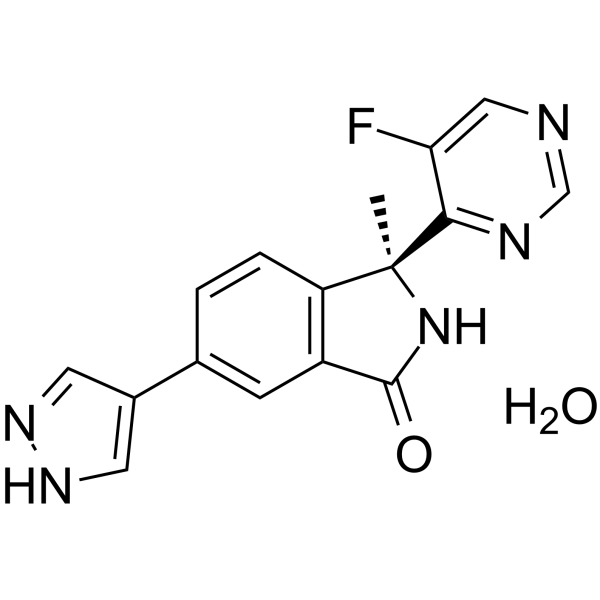
-
GC60421
(S)-Seco-Duocarmycin SA
(S)-Seco-Duocarmycin SA es un alquilador de ADN, citotÓxico para las células cancerosas y actÚa como una citotoxina ADC para conjugados de anticuerpo-fÁrmaco.
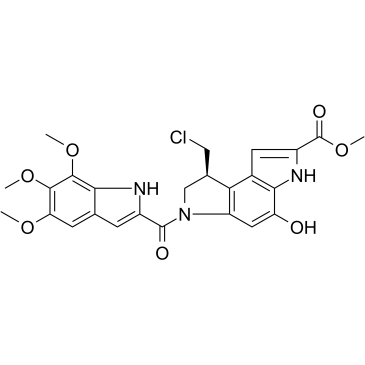
-
GC39842
(Z)-4EGI-1
(Z)-4EGI-1 es el isÓmero Z de 4EGI-1 y es un inhibidor de la interacciÓn eIF4E/eIF4G y del inicio de la traducciÓn. (Z)-4EGI-1 se une eficazmente a eIF4E con una IC50 de 43,5 μM y un valor de Kd de 8,74 μM. (Z)-4EGI-1 tiene actividad anticancerÍgena.
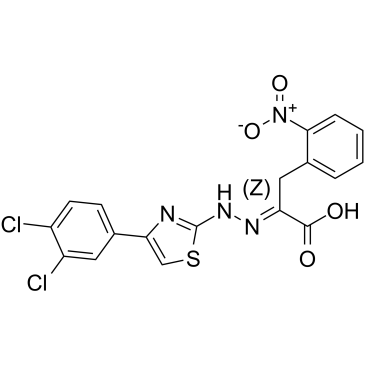
-
GC68484
1,2-O-Isopropylidene-5-O-p-toluoyl-a-D-xylofuranose
1,2-O-Isopropilideno-5-O-p-toluoil-a-D-xilofuranosa es un análogo de nucleósido de purina. Los análogos de nucleósidos de purina tienen una amplia actividad antitumoral y se dirigen a los tumores malignos del sistema linfático inactivo. El mecanismo anticancerígeno en este proceso depende de la inhibición de la síntesis del ADN, inducción de apoptosis y otros factores.
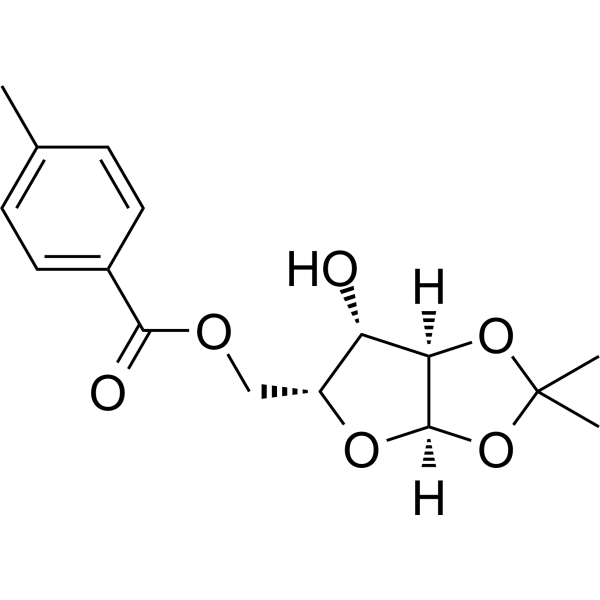
-
GC19528
1,4-Benzoquinone
A toxic metabolite of benzene
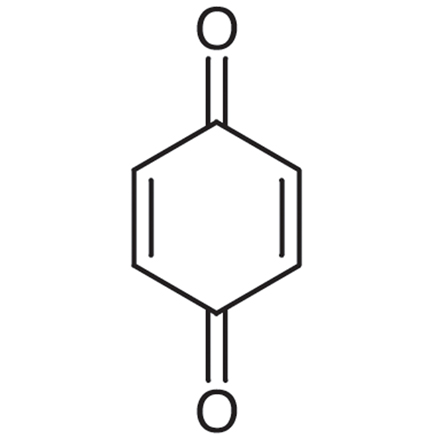
-
GC41859
1,5,7-Triazabicyclo[4.4.0]dec-5-ene
1,5,7-Triazabicyclo[4.4.0]dec-5-ene is a bicyclic guanidine base that can be used as a catalyst for a variety of base-mediated organic transformations, including Michael additions, Wittig reactions, Henry reactions, and transesterification reactions.
![1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure 1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure](/media/struct/GC4/GC41859.png)
-
GC65551
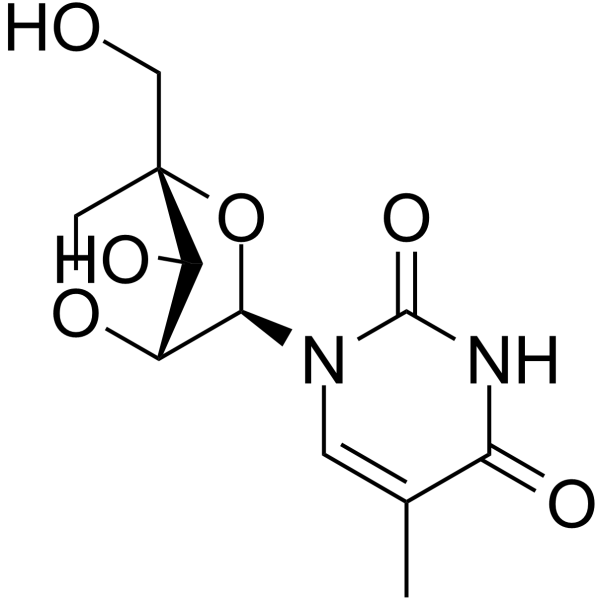
1-(2'-O-4-C-Methylene-beta-D-ribofuranosyl)thymine
1-(&2#39;-O-4-C-Metileno-beta-D-ribofuranosil)timina es un nucleósido bicíclico.
-
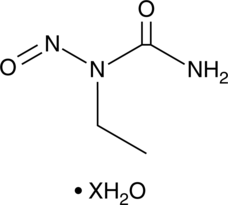
GC49470
1-Ethyl-1-nitrosourea (hydrate)
A DNA alkylating agent
-
GC48909
1-Hydroxyanthraquinone
La 1-hidroxiantraquinona, un compuesto natural con actividad oral de algunas plantas como Tabebuia avellanedae, exhibe un efecto cancerÍgeno.

-
GC65038
1-Methylinosine
La 1-metilinosina es un nucleÓtido modificado que se encuentra en la posiciÓn 37 del ARNt 3' del anticodÓn del ARNt eucariÓtico.

-
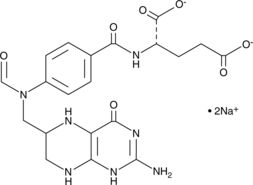
GC49872
10-Formyltetrahydrofolate (sodium salt) (technical grade)
El 10-formiltetrahidrofolato (sal de sodio) (grado técnico) es una forma de Ácido tetrahidrofÓlico que actÚa como donante de grupos formilo en el anabolismo.
-
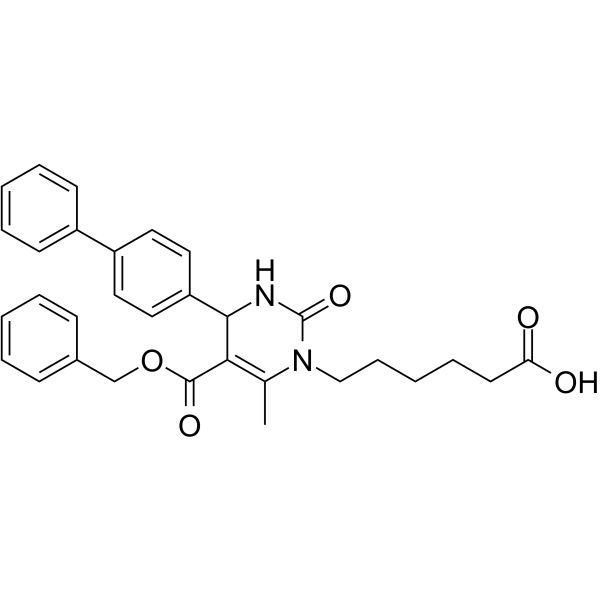
GC63796
116-9e
116-9e (MAL2-11B) es un inhibidor DNAJA1 de la cochaperona Hsp70.
-
GC49759
13C17-Mycophenolic Acid
An internal standard for the quantification of mycophenolic acid
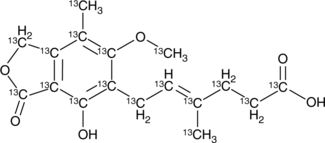
-
GC46474
18-Deoxyherboxidiene
El 18-desoxiherboxidieno (RQN-18690A) es un potente inhibidor de la angiogénesis.

-
GC65489
2'-F-Bz-dC Phosphoramidite
&2#39;-F-Bz-dC La fosforamidita se puede utilizar en la síntesis de oligorribonucleótidos.
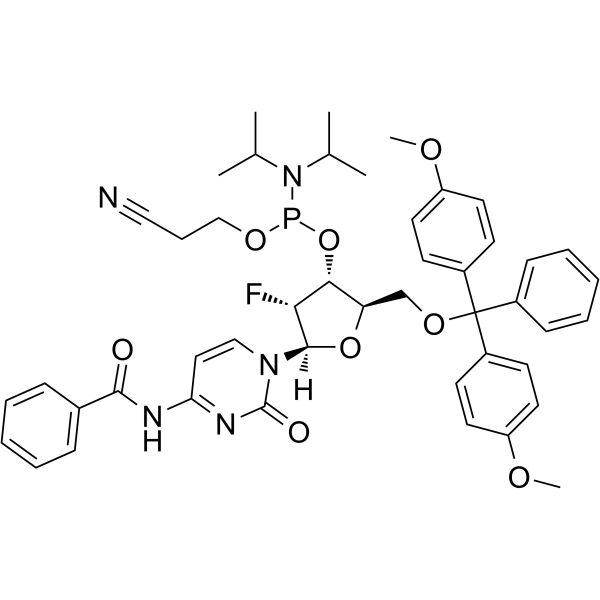
-
GC66651
2'-O,4'-C-Methyleneadenosine
2'-O,4'-C-Metilenadenosina (LNA-A) es un ácido nucleico bloqueado (LNA) y también es un análogo de adenosina.
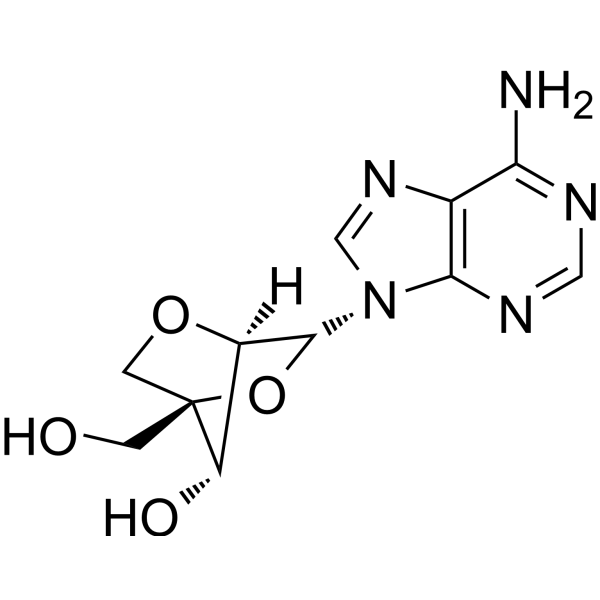
-
GC66654
2'-O,4'-C-Methylenecytidine
2'-O,4'-C-metilencitidina (LNA-C(Bz)) es un análogo de nucleósido bicíclico con conformación de tipo N fija. 2'-O,4'-C-metilencitidina se puede utilizar para sintetizar oligonucleótidos. 2'-O,4'-C-Metilencitidina forma dúplex con hebras complementarias de ADN y ARN.
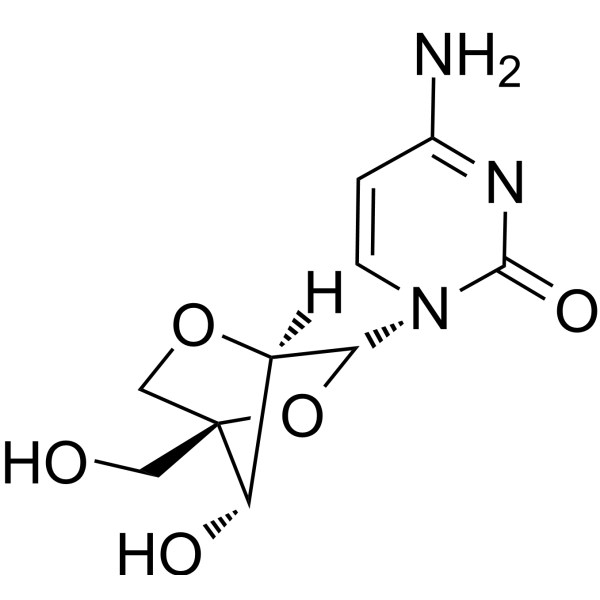
-
GC66655
2'-O,4'-C-Methyleneguanosine
2′-O,4′-C-metilenguanosina (LNA-G) es un análogo de guanina inverso, donde LNA (ácido nucleico bloqueado) es un análogo de ácido nucleico. La modificación de LNA se puede usar en una variedad de aplicaciones, como la afinidad de unión efectiva a secuencias complementarias y una mayor resistencia a la nucleasa que los nucleótidos naturales, lo que ofrece un gran potencial para aplicaciones en el diagnóstico y la investigación de enfermedades. LNA-G también está disponible a través de la ADN polimerasa KOD, que permite la integración de los nucleótidos de LNA-G en la cadena de ADN.
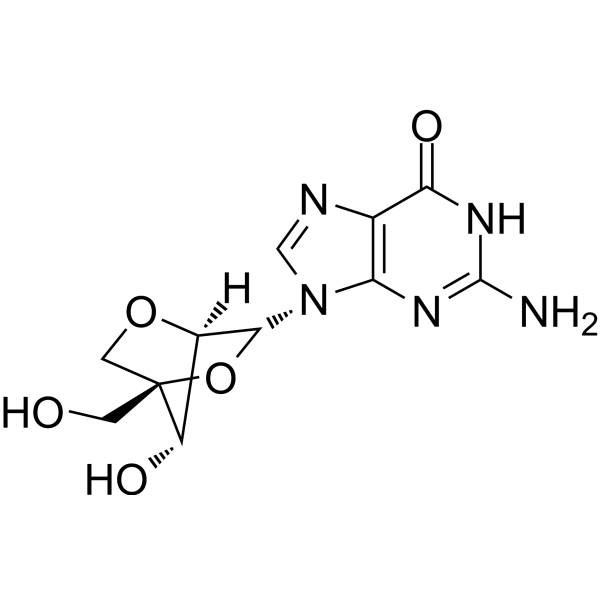
-
GC52183
2'-O-Methyl-5-methyluridine
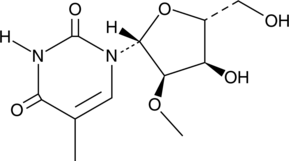
-
GC66713
2'-O-MOE-5-Me-C(Bz)
2'-O-MOE-5-Me-C (Bz) es un nucleótido para la síntesis estereoselectiva de alquilfosfonatos de nucleósidos.
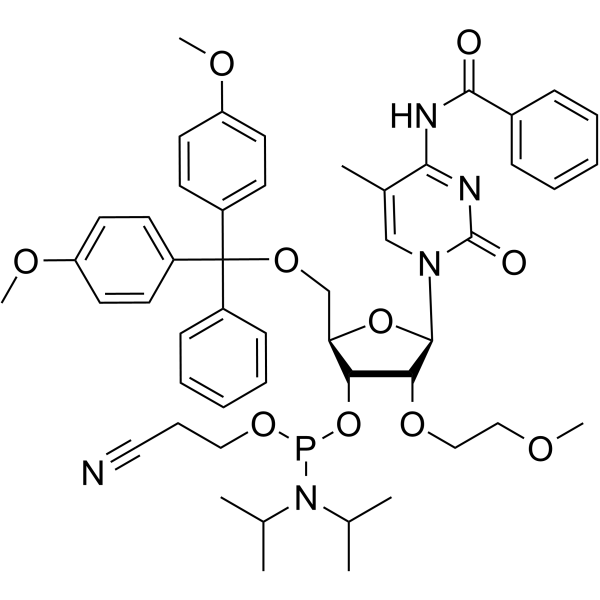
-
GC66657
2'-O-MOE-5-Me-rC
2'-O-MOE-5-Me-rC es un compuesto activo. 2'-O-MOE-5-Me-rC se puede utilizar para la síntesis de oligonucleótidos.
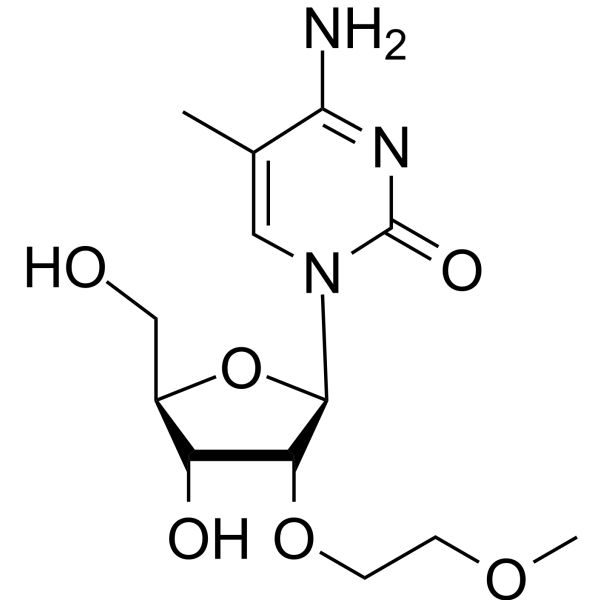
-
GC66091
2'-O-MOE-5-Me-rU
2'-O-MOE-5-Me-rU es un compuesto activo. 2'-O-MOE-5-Me-rU se puede utilizar para la sÍntesis de oligonucleÓtidos.
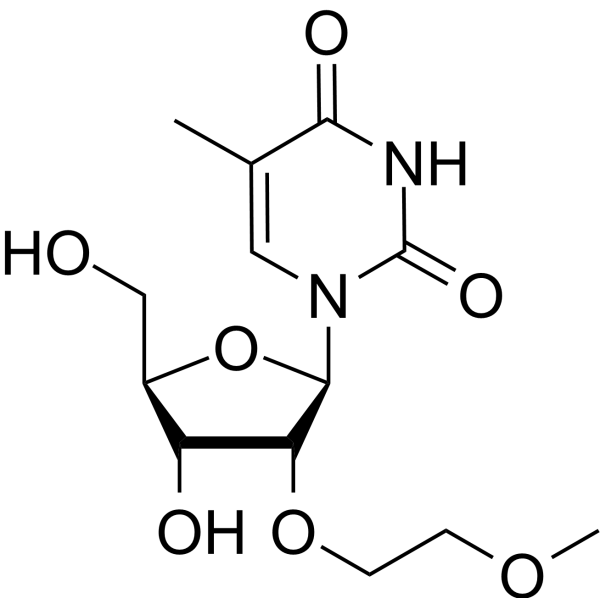
-
GC66656
2'-O-MOE-rC
2'-O-MOE-rC es un nucleósido modificado 2'-O-MOE. 2'-O-MOE-rC se puede utilizar para la síntesis de ADN.
-
GC66652
2'-O-MOE-U
2'-O-MOE-U es una fosforamidita, se puede utilizar para la síntesis de oligonucleótidos.

-
GC65170
2′,3′-Di-O-acetylguanosine
&2#8242;,3′-Di-O-acetilguanosina es un análogo de nucleósido.
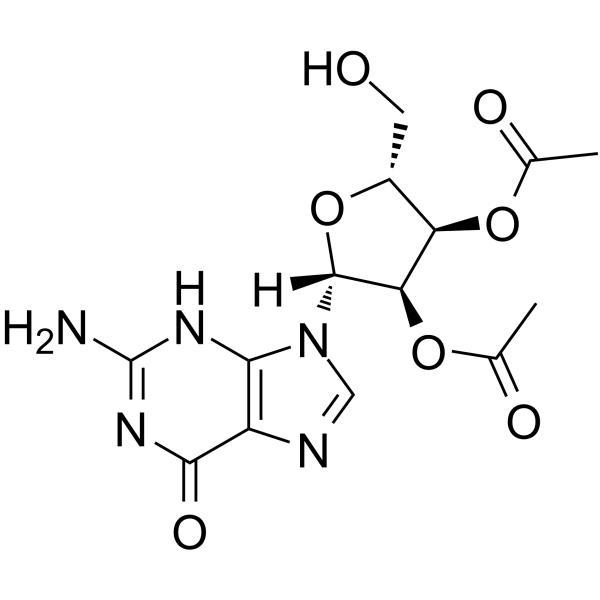
-
GC67384
2′-Deoxy-β-L-uridine
2'-Deoxy-β-L-uridina es un análogo de nucleido y un sustrato específico para la enzima viral, no muestra estereoespecificidad contra el herpes simplex 1 (HSV1) timidina quinasa (TK). 2′-Desoxi-β-L-uridina ejerce actividad antiviral a través de la interacción de 5'-trifosfatos con la ADN polimerasa viral.
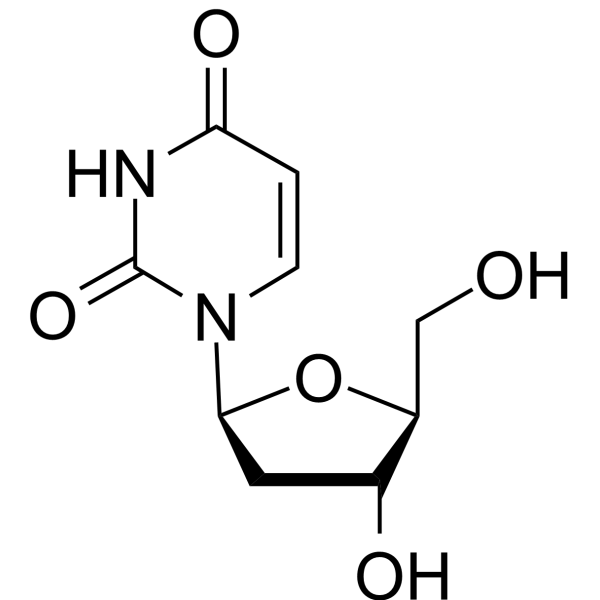
-
GC61667
2′-Deoxy-2′-fluoroadenosine
&2#8242;-Desoxi-&22#8242;-fluoroadenosina se puede utilizar para la síntesis de oligonucleótidos modificados con 2′-desoxi-2′-fluoro hibridados con ARN. &2#8242;-Desoxi-&22#8242;-fluoroadenosina se puede escindir de manera eficiente por E. coli nucleósido de purina fosforilasa (PNP) al agente tóxico 2-fluoroadenina (FAde). &2#8242;-Desoxi-&22#8242;-fluoroadenosina muestra una excelente actividad in vivo contra tumores que expresan E. coli PNP.
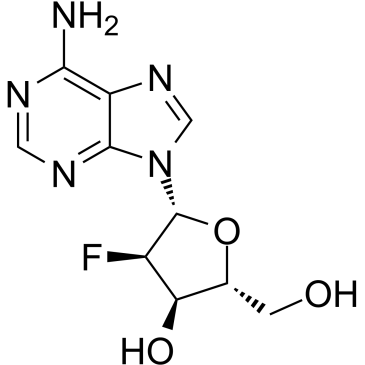
-
GC62530
2’-O-Me-C(Bz) Phosphoramidite
&2rsquo;-O-Me-C(Bz) La fosforamidita es un monómero de fosforamidita modificado, que puede usarse para la síntesis de oligonucleótidos.
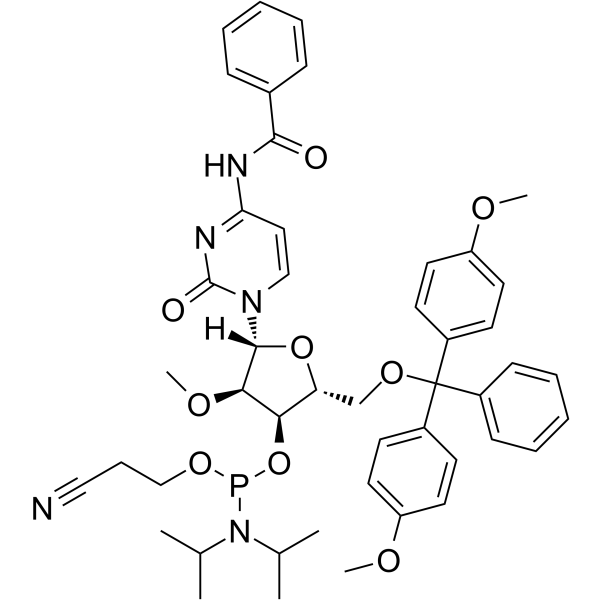
-
GC62529
2’-OMe-A(Bz) Phosphoramidite
&2rsquo;-OMe-A(Bz) Phosphoramidita es un monómero de fosforamidita modificada, que se puede utilizar para la síntesis de oligonucleótidos.
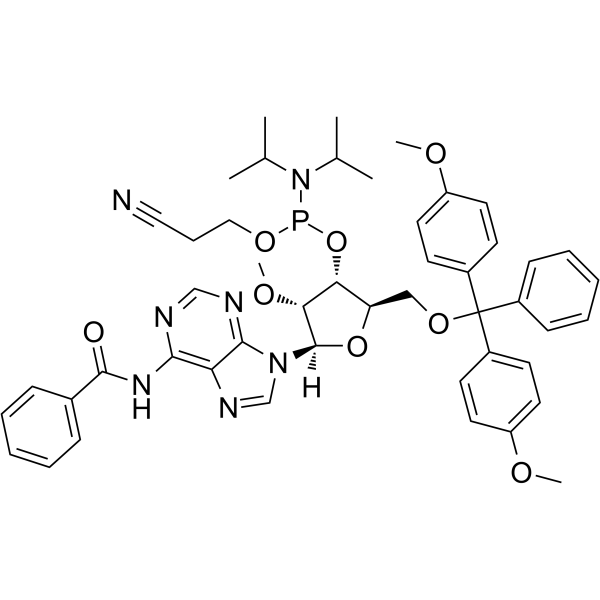
-
GC62531
2’-OMe-G(ibu) Phosphoramidite
&2rsquo;-OMe-G(ibu) Phosphoramidita es un monómero de fosforamidita modificada, que se puede utilizar para la síntesis de oligonucleótidos.
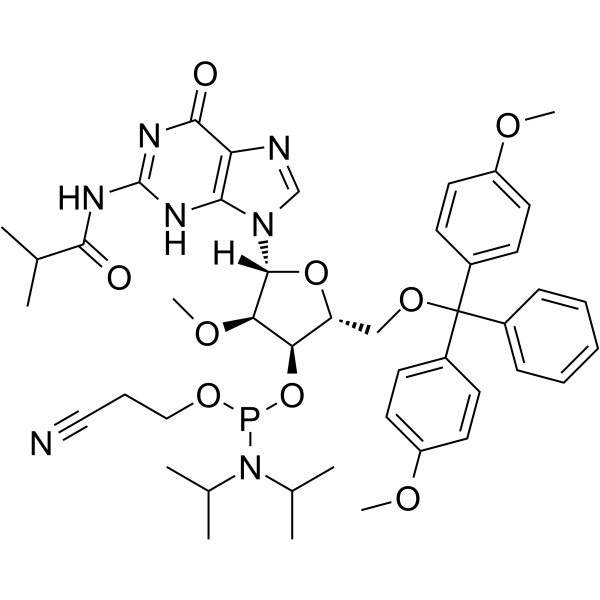
-
GC46508
2',2'-Difluoro-2'-deoxyuridine
An active metabolite of gemcitabine
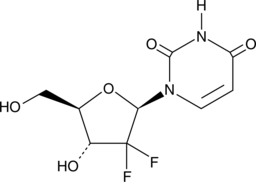
-
GC42061
2',3'-Dideoxyadenosine 5'-triphosphate
2',3'-Dideoxyadenosine 5'-triphosphate (2',3'-Dideoxyadenosine 5'-triphosphate), an active metabolite of 2',3'-dideoxyinosine, is a chain-elongating inhibitor of DNA polymerase.
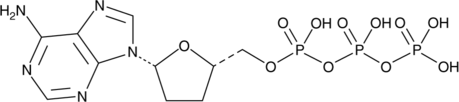
-
GC46519
2',3'-O-Isopropylideneguanosine
A building block
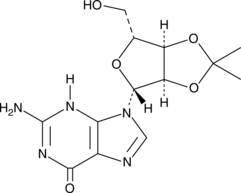
-
GC46540
2'-Deoxyadenosine-5'-triphosphate (sodium salt hydrate)
A purine nucleotide
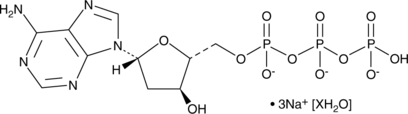
-
GC48440
2'-Deoxycytidine-5'-triphosphate (sodium salt)
2'-Desoxicitidina-5'-trifosfato (sal de sodio) (sal trisÓdica de dCTP) es un nucleÓsido trifosfato que se puede utilizar para la sÍntesis de ADN.
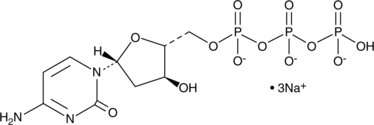
-
GC42151
2'-Deoxyguanosine 5'-monophosphate (sodium salt hydrate)
2'-Deoxyguanosine 5'-monophosphate (dGMP) is used as a substrate of guanylate kinases to generate dGDP, which in turn is phosphorylated to dGTP, a nucleotide precursor used in DNA synthesis.
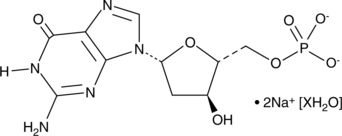
-
GC33430
2'-Deoxypseudoisocytidine
La 2'-desoxipseudoisocitidina es un anÁlogo de nucleÓsido.
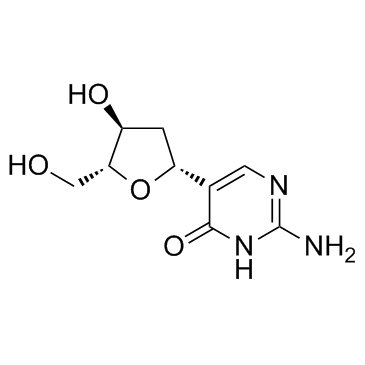
-
GC35072
2'-O,4'-C-Methyleneuridine
2'-O,4'-C-Metilenouridina (Compuesto 15a) es un nucleÓsido bicÍclico.

-
GC42080
2'2'-cGAMP (sodium salt)
2'2'-cGAMP is a synthetic dinucleotide (CDN) that contains non-canonical 2'5'-phosphodiester bonds.
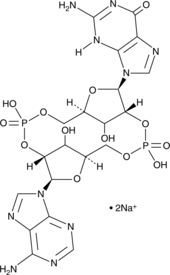
-
GC64399
2,4,6-Trihydroxybenzoic acid
El Ácido 2,4,6-trihidroxibenzoico, el metabolito flavonoide, es un inhibidor de CDK. El Ácido 2,4,6-trihidroxibenzoico se puede utilizar para la investigaciÓn del cÁncer.
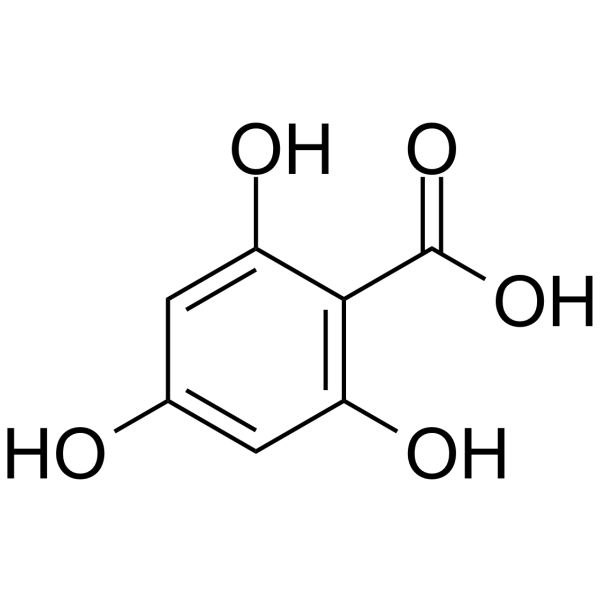
-
GC60459
2,4-D (2,4-Dichlorophenoxyacetic acid)
El 2,4-D (Ácido 2,4-diclorofenoxiacético) (2,4-D (Ácido 2,4-diclorofenoxiacético) Ácido iclorofenoxiacético) es un herbicida sistémico selectivo para el control de malezas de hoja ancha.
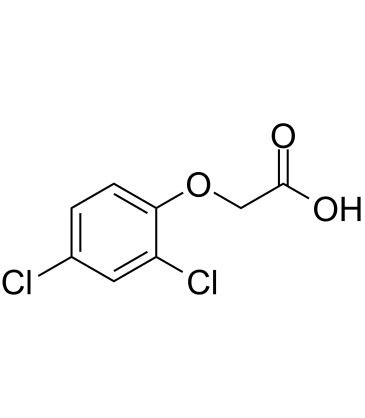
-
GC46524
2,6-Dichloropurine-9-β-D-riboside
A building block
-
GC33496
2-(Methylamino)-1H-purin-6(7H)-one (N2-methylguanine)
La 2-(metilamino)-1H-purina-6(7H)-ona (N2-metilguanina) (N2-metilguanina) es un nucleÓsido modificado.
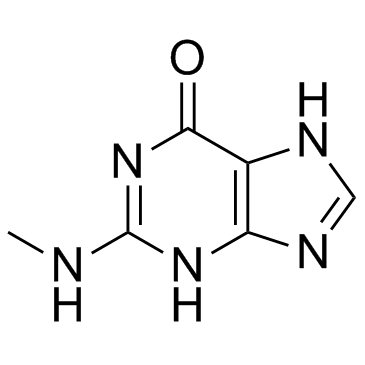
-
GC64983
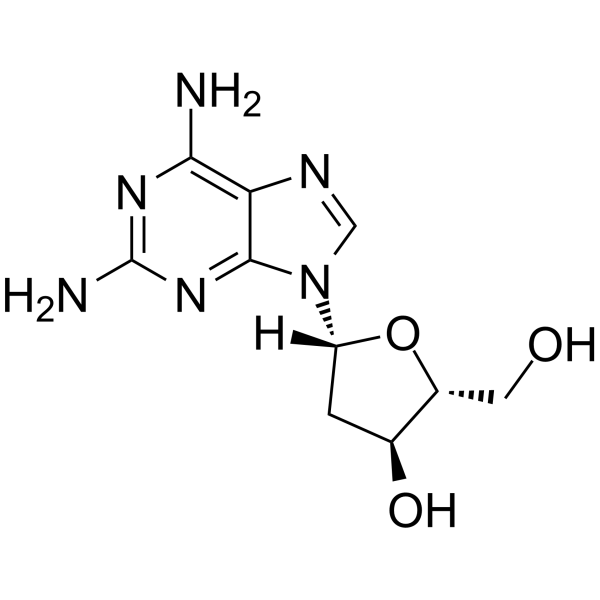
2-Amino-2'-deoxyadenosine
2-Amino-&2#39;-desoxiadenosina es un desoxirribonucleósido utilizado para la síntesis de oligonucleótidos.
-
GC39527
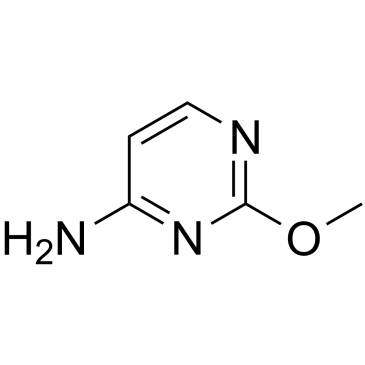
2-O-Methylcytosine
La 2-O-metilcitosina, un anÁlogo O-alquilado de un aducto de ADN, es la nucleobase daÑada.
-
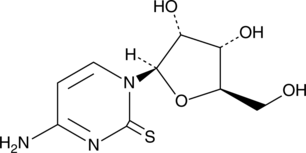
GC49348
2-Thiocytidine
A modified nucleobase
-
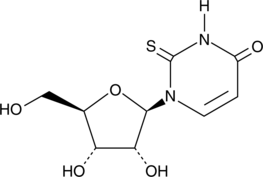
GC42197
2-Thiouridine
2-Thiouridine (s2U) is a modified nucleobase found in tRNAs that is known to stabilize U:A pairs and modestly destabilize U:G wobble pairs.
-
GC65083
3'-Azido-3'-deoxy-5-fluorocytidine
3'-Azido-3&2#39;-desoxi-5-fluorocitidina (Compuesto 12) es un derivado de citidina.
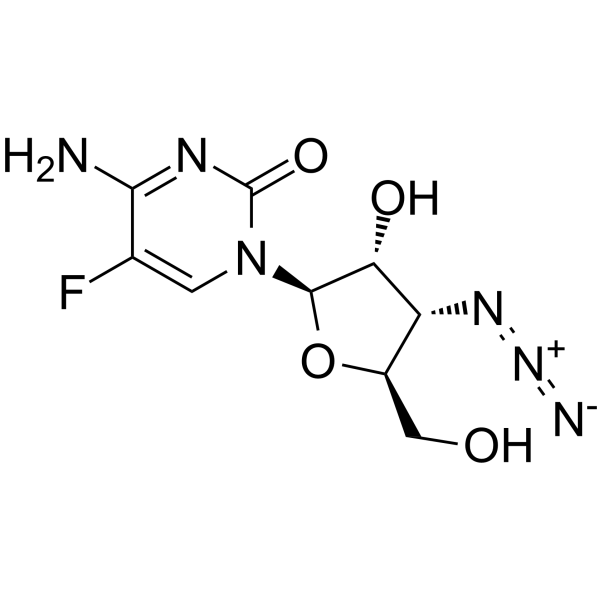
-
GC64985
3'-O-Methylguanosine
3'-O-metilguanosina es un análogo de nucleósido metilado y un terminador de cadena de ARN.
-
GC40618
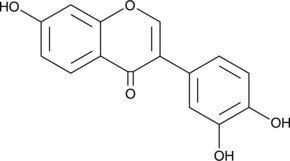
3',4',7-Trihydroxyisoflavone
3',4',7-trihidroxiisoflavona, un metabolito principal de la daidzeÍna, es un inhibidor competitivo de ATP de Cot (Tpl2/MAP3K8) y MKK4. 3',4',7-trihidroxiisoflavona tiene actividades anticancerÍgenas, antiangiogénicas, quimioprotectoras y de eliminaciÓn de radicales libres.
-
GC35105
3'-Azido-3'-deoxy-beta-L-uridine
La 3'-azido-3'-desoxi-beta-L-uridina (Compuesto 25) es un derivado de nucleÓsido.
-
GC60023
3'-Deoxyuridine-5'-triphosphate
El 3'-desoxiuridina-5'-trifosfato (3'-dUTP) es un anÁlogo de nucleÓtido que inhibe las ARN polimerasas I y II dependientes de ADN.
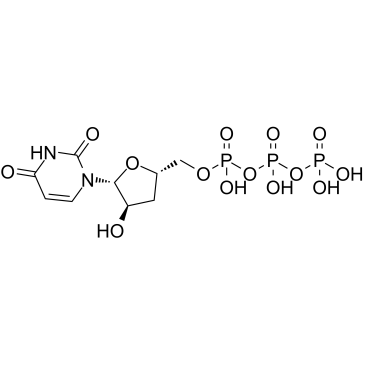
-
GC61862
3'-Deoxyuridine-5'-triphosphate trisodium
El 3'-desoxiuridina-5'-trifosfato trisÓdico (3'-dUTP trisÓdico) es un anÁlogo de nucleÓtido que inhibe las ARN polimerasas I y II dependientes de ADN.
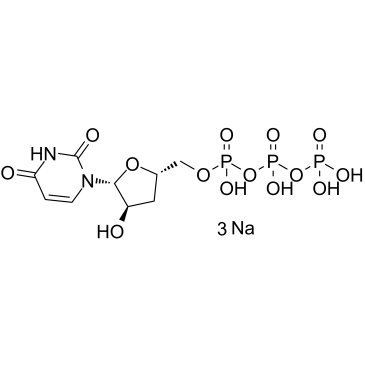
-
GC45332
3'-Dephosphocoenzyme A
An intermediate in the biosynthesis of CoA
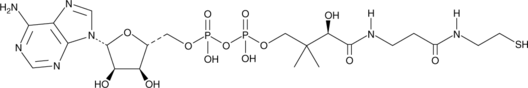
-
GC34384
3,6-DMAD hydrochloride
El clorhidrato de 3,6-DMAD, un derivado de la acridina, es un potente inhibidor de la vÍa IRE1α-XBP1s. El clorhidrato de 3,6-DMAD promueve la secreciÓn de IL-6 a través de la vÍa IRE1α-XBP1s. El clorhidrato de 3,6-DMAD inhibe la oligomerizaciÓn de IRE1α y la actividad de la endorribonucleasa (RNasa). El clorhidrato de 3,6-DMAD se puede utilizar para la investigaciÓn del cÁncer.
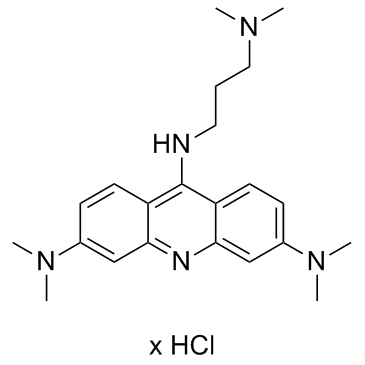
-
GC34452
3,7,4'-Trihydroxyflavone
3,7,4'-trihidroxiflavona, aislada de Rhus javanica var.
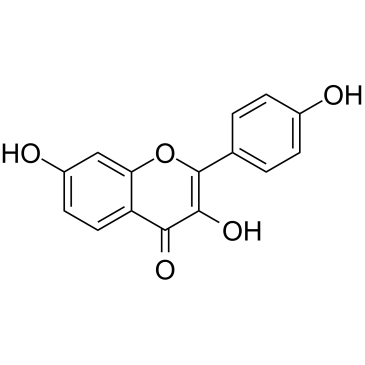
-
GC52129
3-Amino-5-hydroxybenzoic Acid
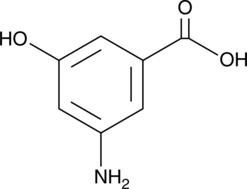
-
GC13510
3-AP
3-AP (PAN-811) es un potente inhibidor de la subunidad M2 de la ribonucleÓtido reductasa (RR) y es un potente radiosensibilizador.
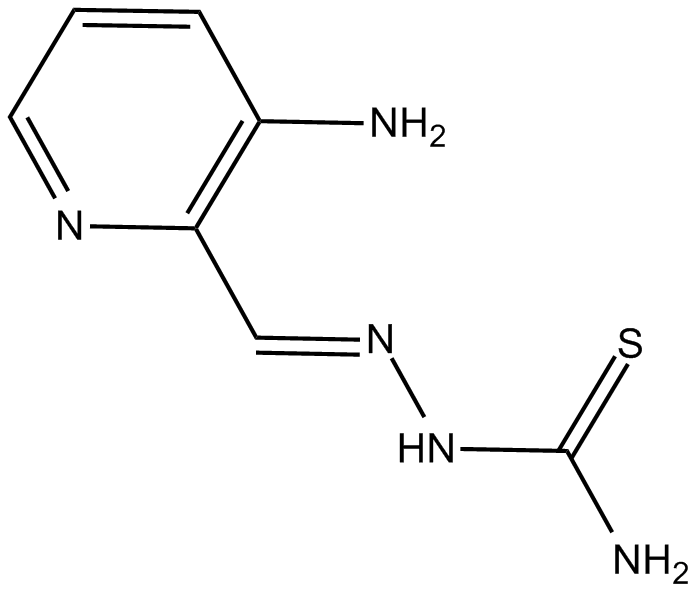
-
GC48457
3-keto Fusidic Acid
An active metabolite of fusidic acid
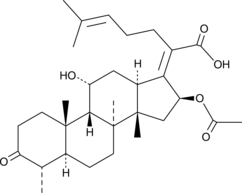
-
GC65084
3-Methylcytidine
La 3-metilcitidina, un nucleÓsido urinario, se puede utilizar como biomarcador de cuatro tipos diferentes de cÁncer: cÁncer de pulmÓn, cÁncer gÁstrico, cÁncer de colon y cÁncer de mama.
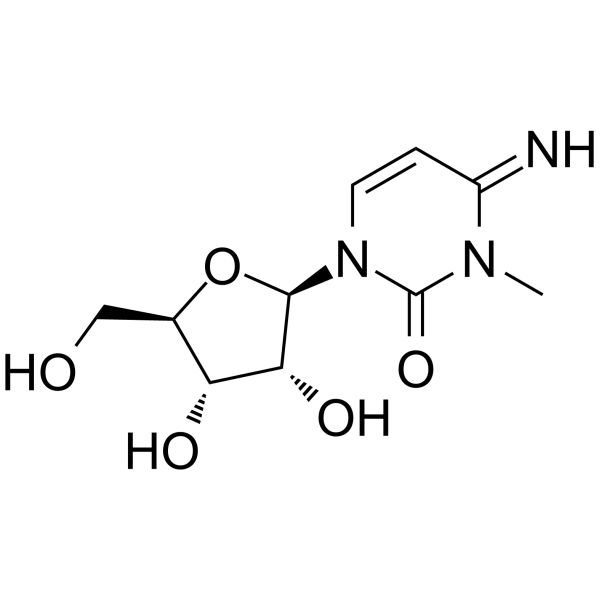
-
GC52391
306-O12B-3
An ionizable cationic lipidoid
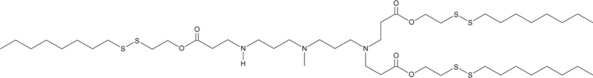
-
GC15389
360A
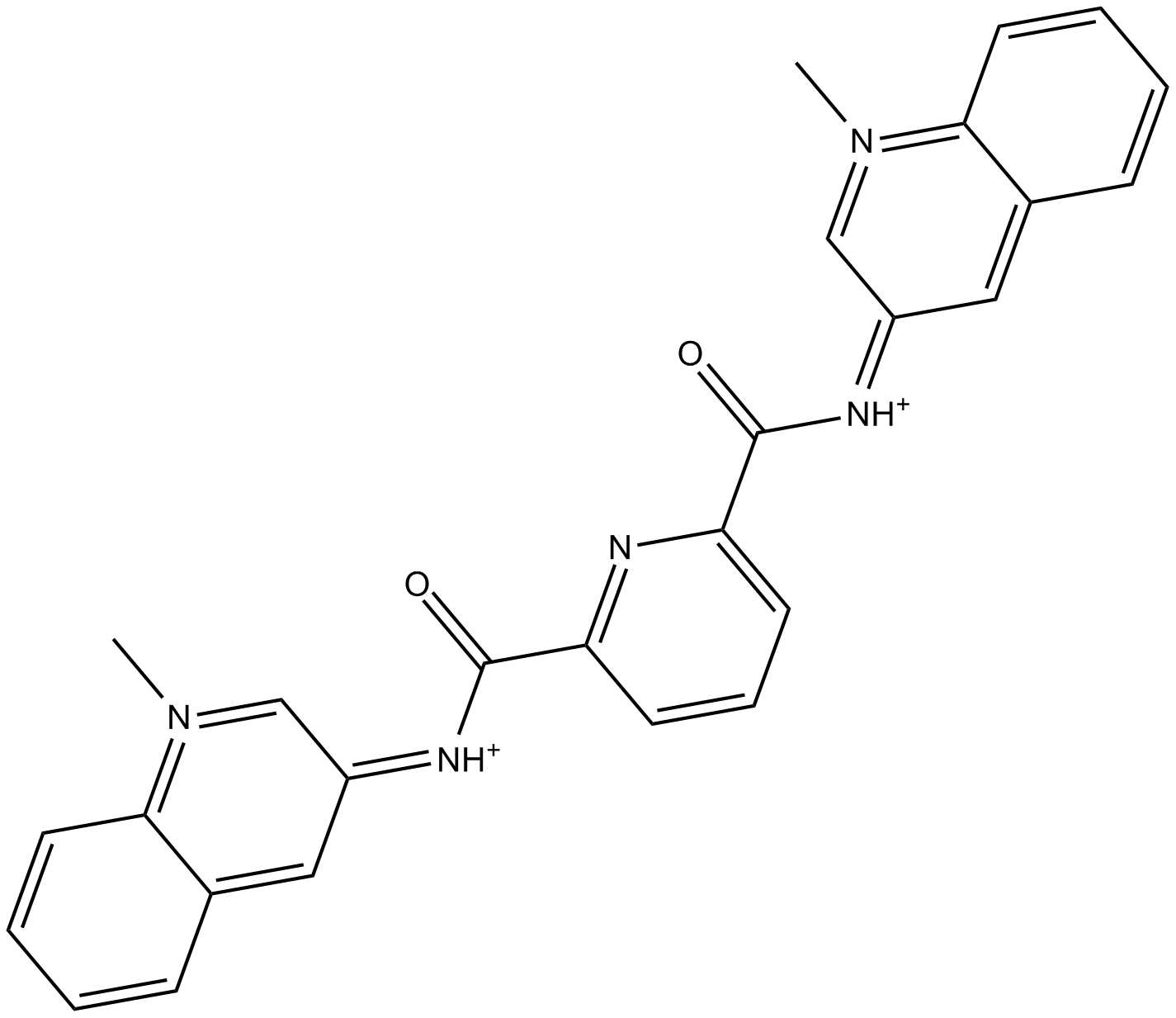
-
GC10115
360A iodide
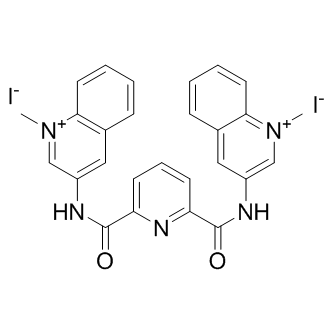
-
GC14493
4μ8C
Inhibidor de la Rnasa IRE1, potente y no tóxico.
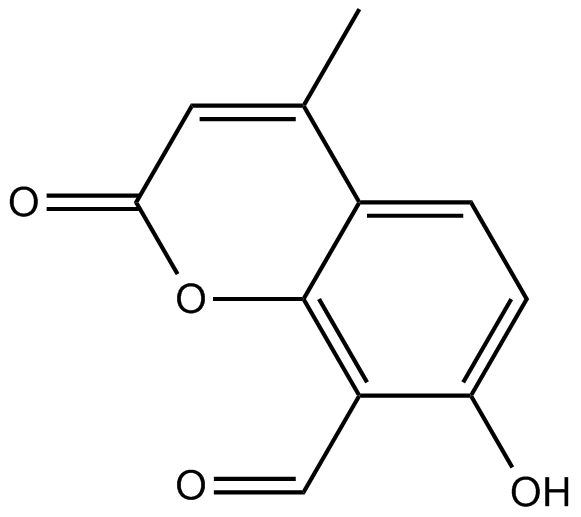
-
GC17271
4'-Demethylepipodophyllotoxin
An inhibitor of tubulin polymerization
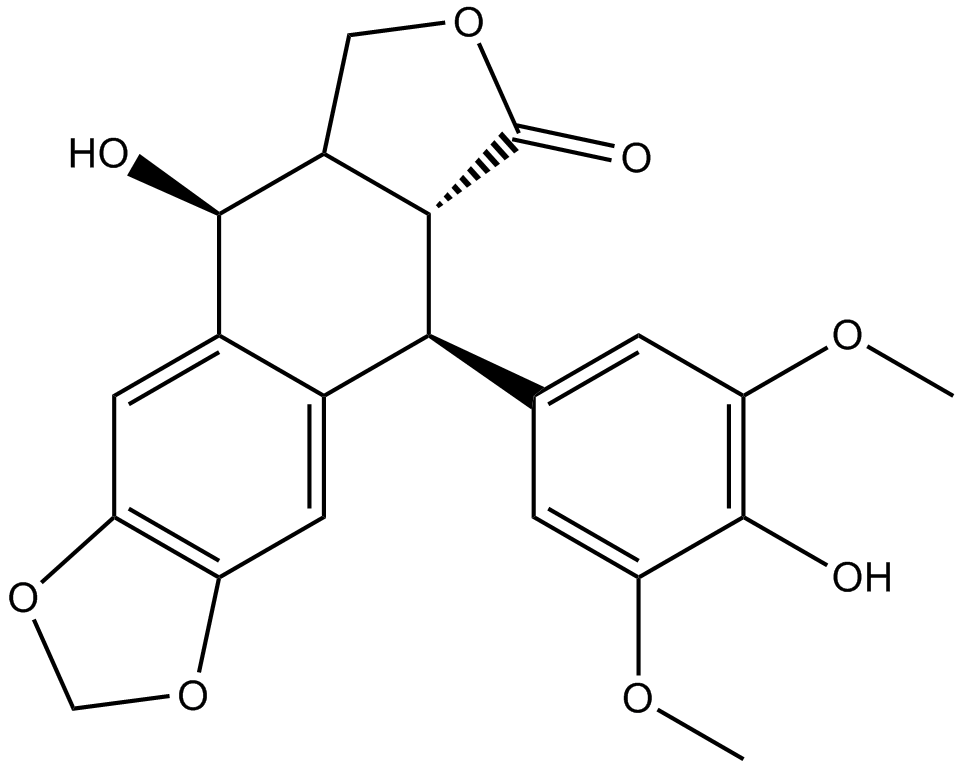
-
GC46607
4-(Methylnitrosamino)-1-(3-pyridyl)-1-butanone
A tobacco-specific nitrosamine carcinogen
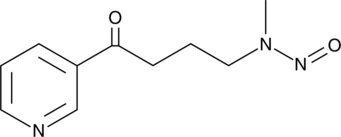
-
GC11761
4-amino-1,8-Naphthalimide
La 4-amino-1,8-naftalimida es un potente inhibidor de PARP y potencia la citotoxicidad de la γ-radiaciÓn en células cancerosas.
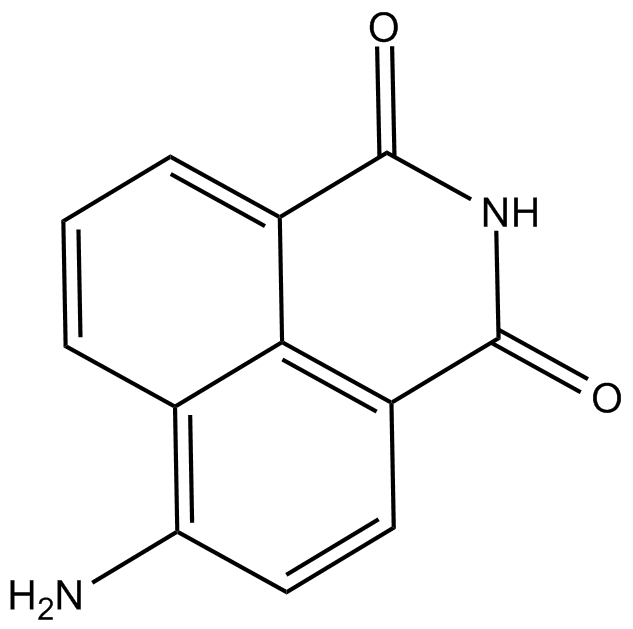
-
GC42401
4-hydroperoxy Cyclophosphamide
Un análogo activado de ciclofosfamida.
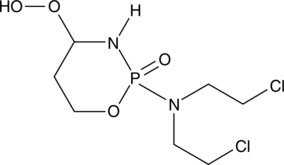
-
GC68332
4-Hydroperoxy Cyclophosphamide-d4

-
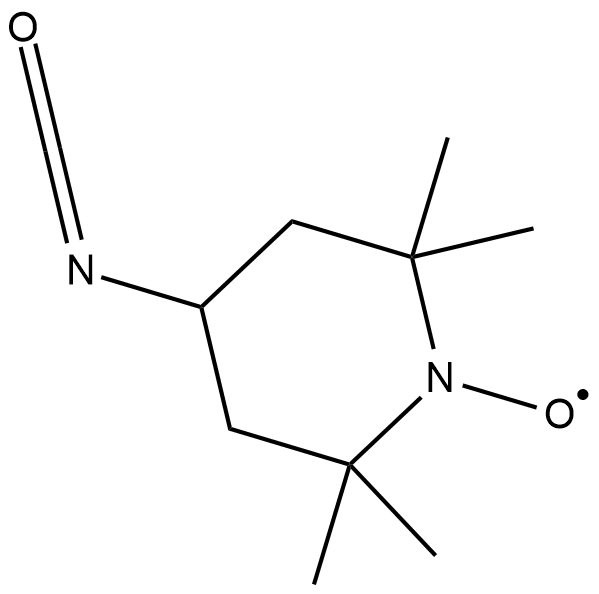
GC18853
4-isocyanato TEMPO
4-isocyanato TEMPO is a spin labeling reagent used to label the 2'-position in RNA.
-
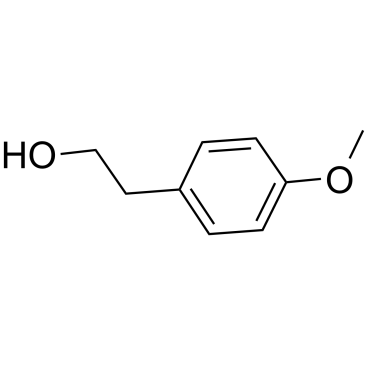
GC61683
4-Methoxyphenethyl alcohol
El alcohol 4-metoxifenetÍlico, un alcohol aromÁtico, es el principal componente del olor a anÍs producido por A.
-
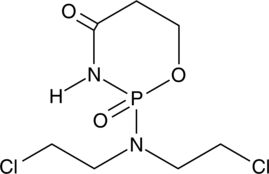
GC49127
4-oxo Cyclophosphamide
An inactive metabolite of cyclophosphamide
-
GC42464
4-oxo-2-Nonenal
4-hydroxy Nonenal is a lipid peroxidation product derived from oxidized ω-6 polyunsaturated fatty acids such as arachidonic acid and linoleic acid.