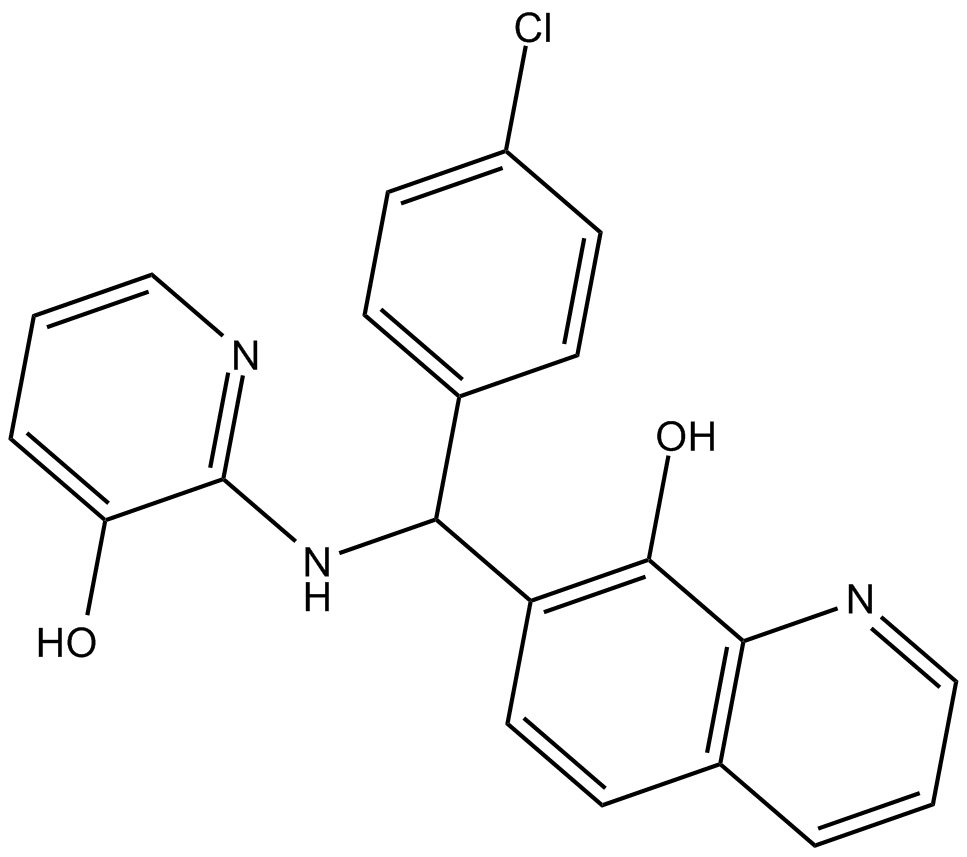
Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nombre del producto Información
-
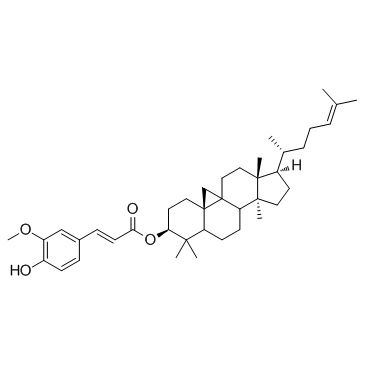
GC31365
γ-Oryzanol
γ-Oryzanol es un potente inhibidor de las ADN metiltransferasas (DNMT) en el cuerpo estriado de ratones.
-
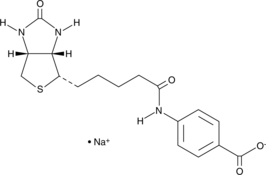
GC45258
(+)-Biotin 4-Amidobenzoic Acid (sodium salt)
(+)-Biotin 4-amidobenzoic acid is a substrate of biotinidase, which cleaves biotin amide to give biotin in vivo.
-
GC61595
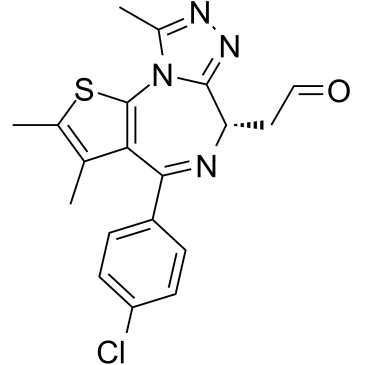
(+)-JQ-1-aldehyde
(+)-JQ-1-aldehÍdo es la forma de aldehÍdo de (+)-JQ1. (+)-JQ-1-aldehÍdo se puede usar como precursor para sintetizar PROTAC, que se dirige a los bromodominios BET.
-
GC34958
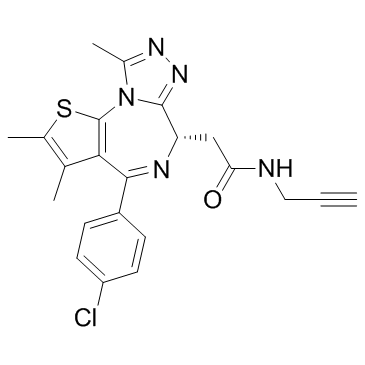
(+)-JQ1 PA
(+)-JQ1 PA es un derivado del bromodominio y del inhibidor extraterminal (BET) JQ1, con una IC50 de 10,4 nM.
-
GC13822
(-)-JQ1
A selective inhibitor of BET bromodomains
-
GC34964
(1S,3R,5R)-PIM447 dihydrochloride
(1S,3R,5R)-PIM447 (diclorhidrato) un inhibidor de PIM extraÍdo de la patente US 20100056576 A1, compuesto ejemplo 72, tiene valores IC50 de 0,095 μM para Pim1, 0,522 μM para Pim2 y 0,369 μM para Pim3.
-
GC62130
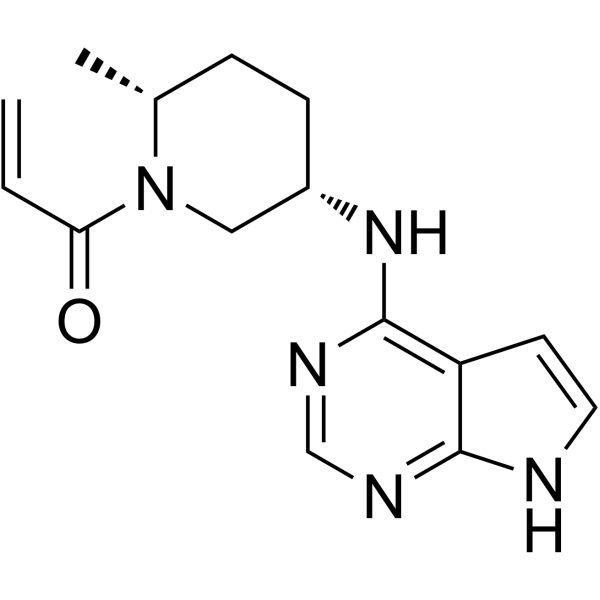
(2R,5S)-Ritlecitinib
(2R,5S)-Ritlecitinib ((2R,5S)-PF-06651600) es un inhibidor potente y selectivo de JAK3 (IC50=144,8 nM) extraÍdo de la patente US20150158864A1, ejemplo 68.
-
GC34971
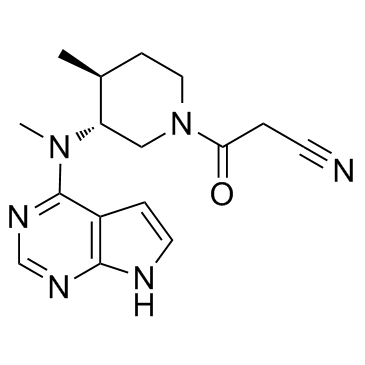
(3R,4S)-Tofacitinib
(3R,4S)-Tofacitinib es un enantiÓmero menos activo de Tofacitinib.
-
GC34972
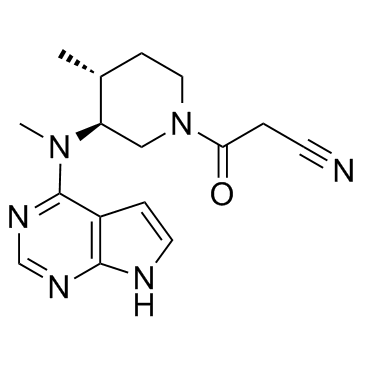
(3S,4R)-Tofacitinib
(3S,4R)-Tofacitinib es un enantiÓmero menos activo de Tofacitinib.
-
GC34973
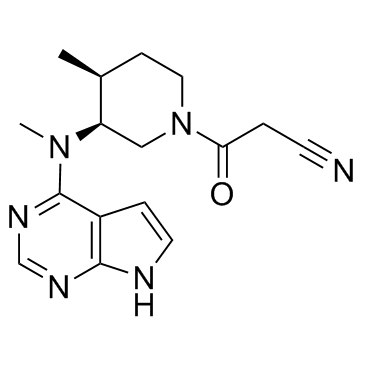
(3S,4S)-Tofacitinib
(3S,4S)-Tofacitinib es el enantiÓmero S menos activo de Tofacitinib.
-
GC41695
(6R,S)-5,6,7,8-Tetrahydrofolic Acid (hydrochloride)
(6R,S)-5,6,7,8-Tetrahydrofolic acid (THFA), the reduced form of folic acid, serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
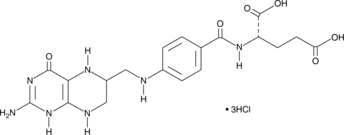
-
GC41088
(6S)-Tetrahydrofolic Acid
(6S)-Tetrahydrofolic acid is a diastereomer of tetrahydrofolic acid, a reduced form of folic acid that serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
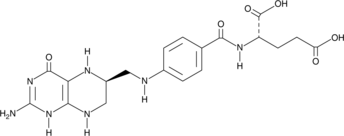
-
GC61807
(E/Z)-AG490
(E/Z)-AG490 ((E/Z)-Tyrphostin AG490) es un compuesto racémico de los isÓmeros (E)-AG490 y (Z)-AG490. (E)-AG490 es un inhibidor de tirosina quinasa que inhibe EGFR, Stat-3 y JAK2/3.

-
GC63864
(E/Z)-Zotiraciclib hydrochloride
El clorhidrato de (E/Z)-zotiraciclib ((E/Z)-TG02) es un potente inhibidor de CDK2, JAK2 y FLT3.
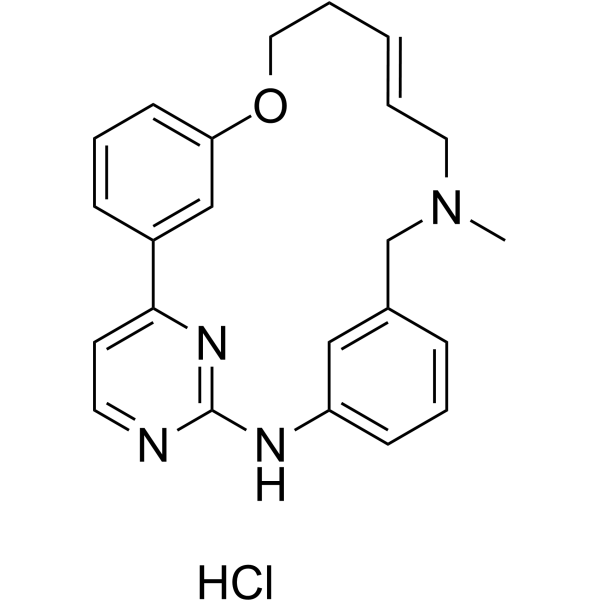
-
GC15104
(R)-(+)-Etomoxir sodium salt
La sal sÓdica de etomoxir((R)-(+)-Etomoxir) es un inhibidor irreversible de la carnitina palmitoiltransferasa 1a (CPT-1a), inhibe la oxidaciÓn de Ácidos grasos (FAO) a través de CPT-1a e inhibe la oxidaciÓn del palmitato en humanos, ratas y conejillo de indias.
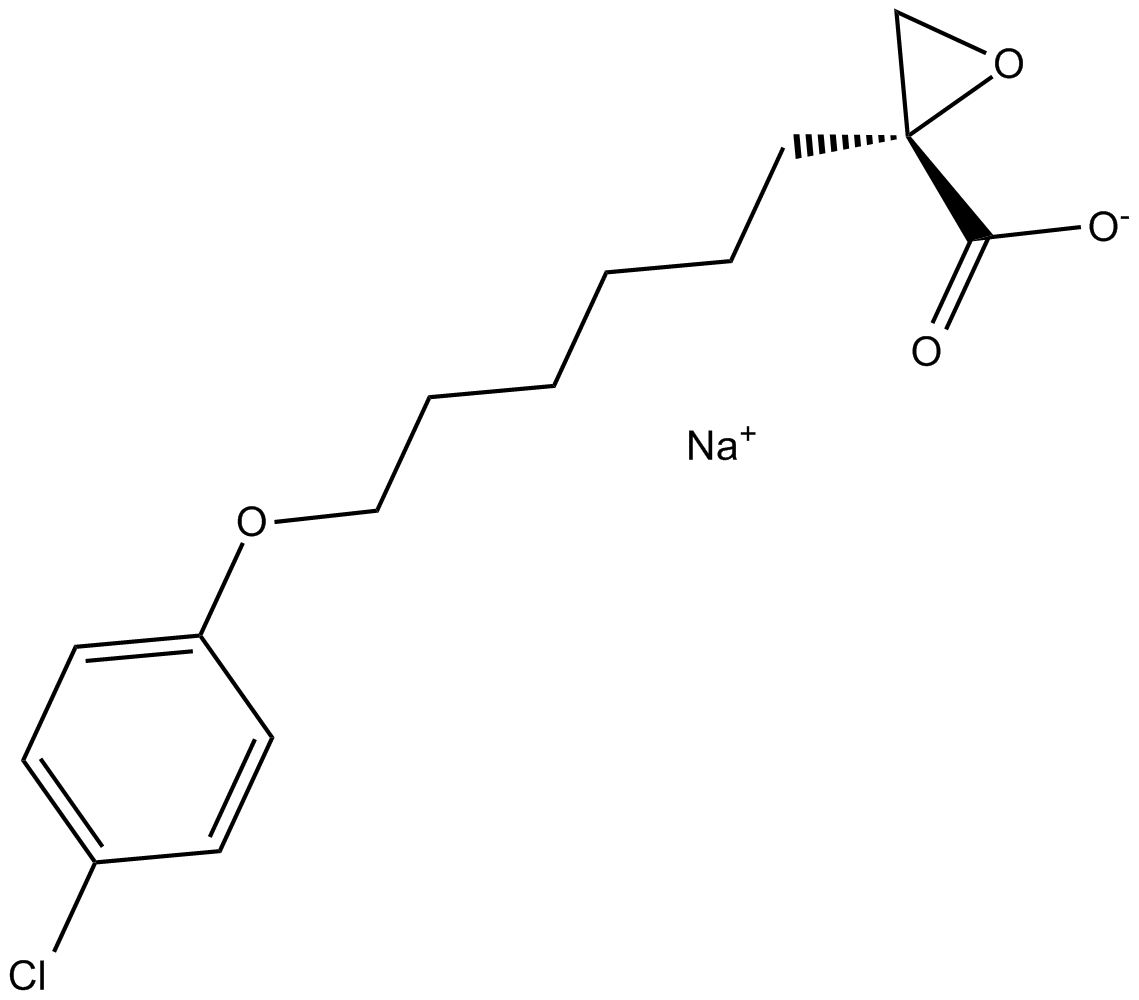
-
GC63791
(R)-(-)-JQ1 Enantiomer
El enantiÓmero (R)-(-)-JQ1 es el estereoisÓmero de (+)-JQ1. (+)-JQ1 reduce potentemente la expresiÓn de ambos genes diana BRD4, mientras que el enantiÓmero (R)-(-)-JQ1 no tiene ningÚn efecto.
-
GC45908
(R)-BAY-598
An inhibitor of SMYD2
-
GC34443
(R)-BAY1238097
(R)-BAY1238097 es el isÓmero R con menor actividad de BAY1238097. BAY1238097 es un inhibidor potente y selectivo de la uniÓn de BET a las histonas y tiene una fuerte actividad antiproliferativa en diferentes modelos de AML (leucemia mieloide aguda) y MM (mieloma mÚltiple) a través de la regulaciÓn negativa de los niveles de c-Myc y su transcriptoma posterior.
-
GC64210
(R)-GSK-3685032
(R)-GSK-3685032 es el enantiÓmero R de GSK-3685032. GSK-3685032 es un inhibidor selectivo de DNMT1 reversible no dependiente del tiempo, no covalente, primero en su clase, con una IC50 de 0,036 μM. GSK-3685032 induce una fuerte pérdida de metilaciÓn del ADN, activaciÓn transcripcional e inhibiciÓn del crecimiento de células cancerosas.
-
GC11340
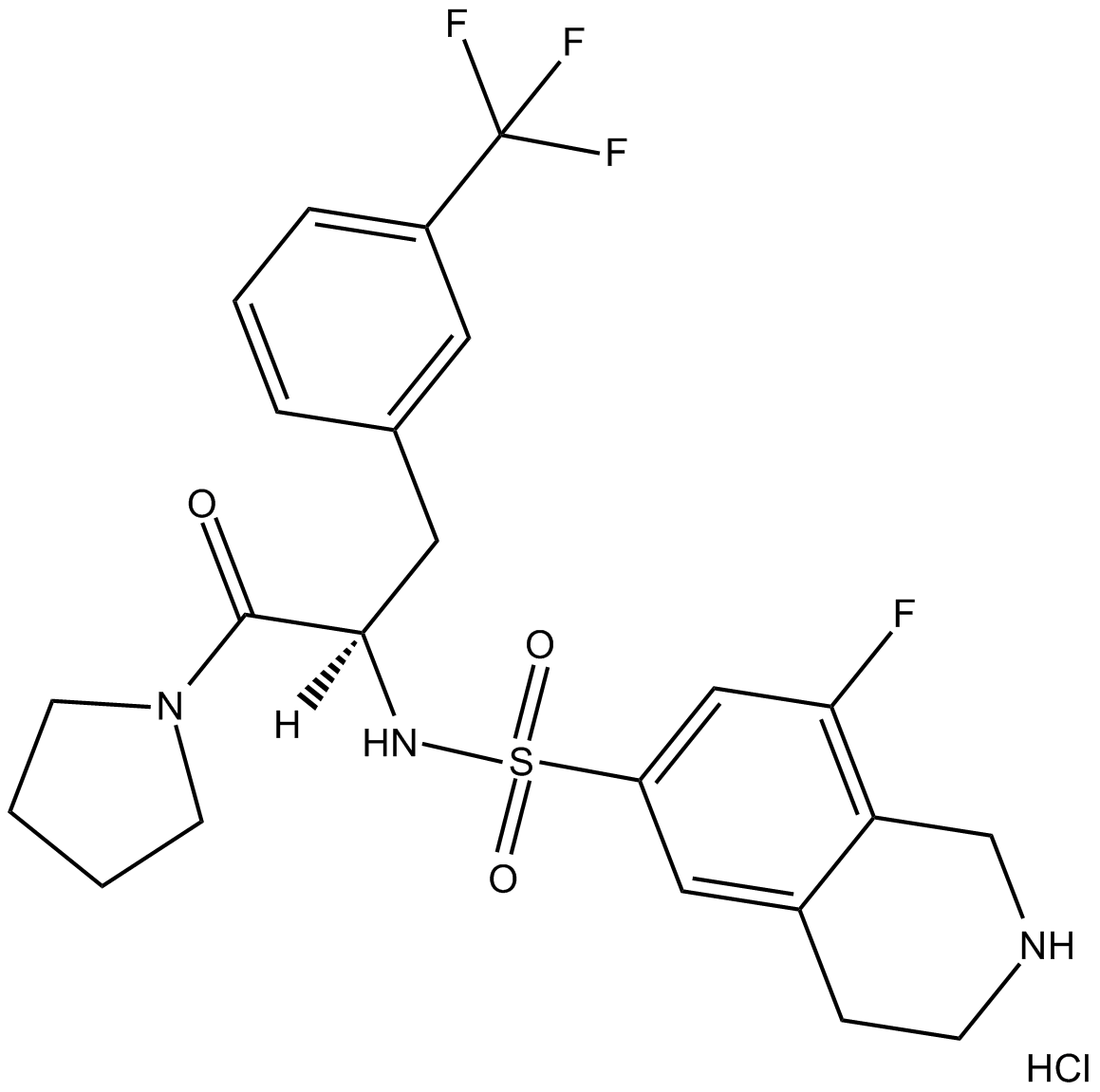
(R)-PFI 2 hydrochloride
El clorhidrato de PFI-2 ((R)-(R)-PFI 2 clorhidrato) es un dominio SET potente y selectivo que contiene un inhibidor de la lisina metiltransferasa 7 (SETD7).
-
GC69794
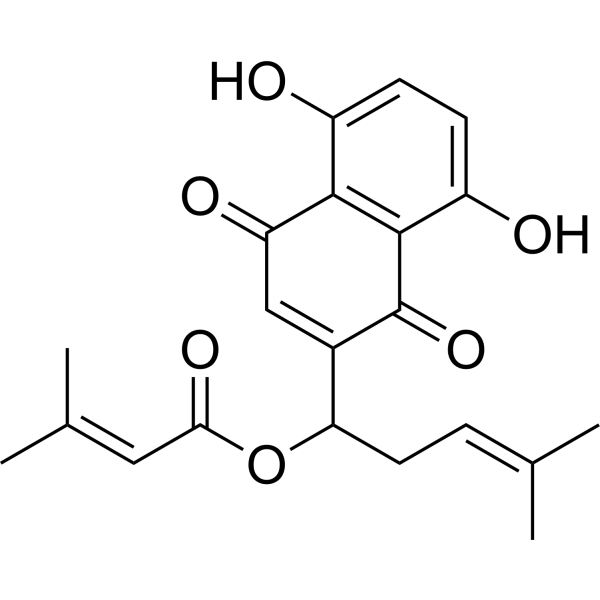
(Rac)-Arnebin 1
(Rac)-Arnebin 1 ((Rac)-β,β-Dimetilacrilalcanina) es el enantiómero de β,β-Dimetilacrilalcanina y/o β,β-Dimetilacrilshikonina. La β,β-Dimetilacrilalcanina y la β,β-Dimetilacrilshikonina son compuestos quinona aislados de plantas del género Lithospermum. La β,β-Dimetilacrilshikonina tiene actividad antitumoral.
-
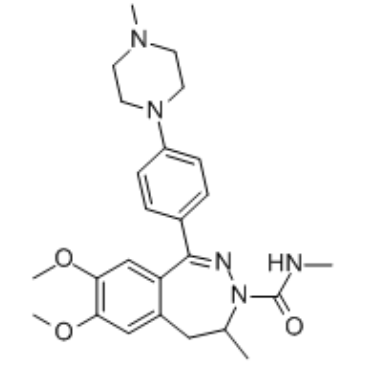
GC34446
(rac)-BAY1238097
(Rac)-BAY1238097 es un inhibidor de BET, con una IC50 de 1,02 μM para BRD4. Utilizado en la investigaciÓn del cÁncer.
-
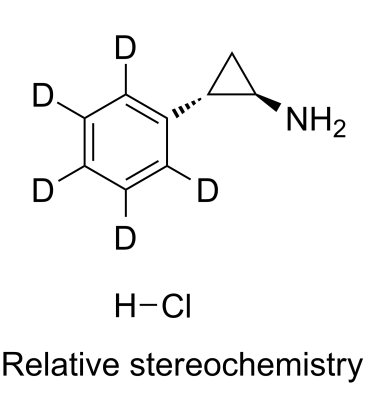
GC60004
(rel)-Tranylcypromine D5 hydrochloride
El clorhidrato de tranilcipromina-d5 (SKF 385-d5) es un clorhidrato de (rel)-tranilcipromina marcado con deuterio.
-
GC33198
(S)-JQ-35 (TEN-010)
(S)-JQ-35 (TEN-010) (TEN-010) es un inhibidor de las proteÍnas que contienen bromodominio de la familia Bromodomain y Extra-Terminal (BET) con actividad antineoplÁsica potencial.
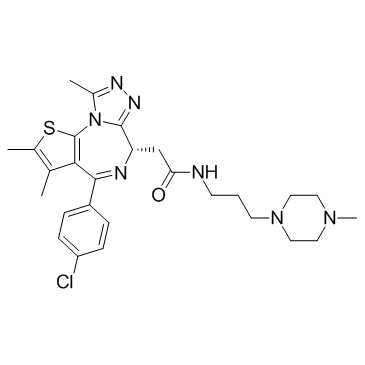
-
GC65045
(S)-MRTX-1719
(S)-MRTX-1719 (ejemplo 16-7) es el enantiÓmero S de MRTX-1719. (S)-MRTX-1719 es un inhibidor del complejo PRMT5/MTA, con una IC50 de 7070 nM.
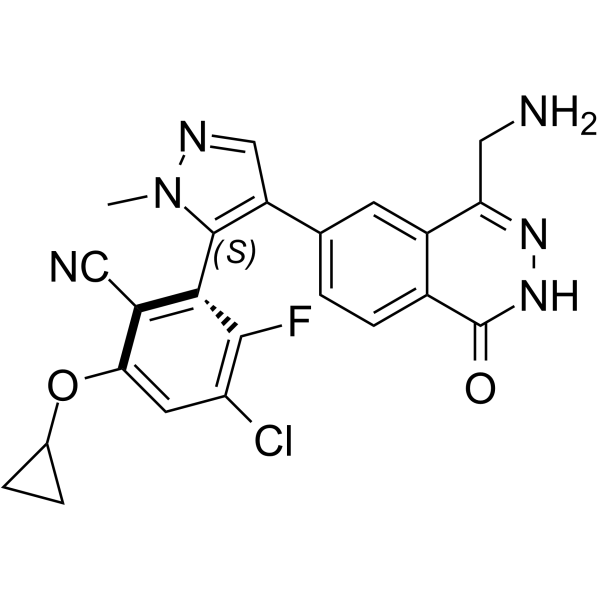
-
GC13634
(S)-PFI-2 (hydrochloride)
Negative control of (R)-PFI 2 hydrochloride
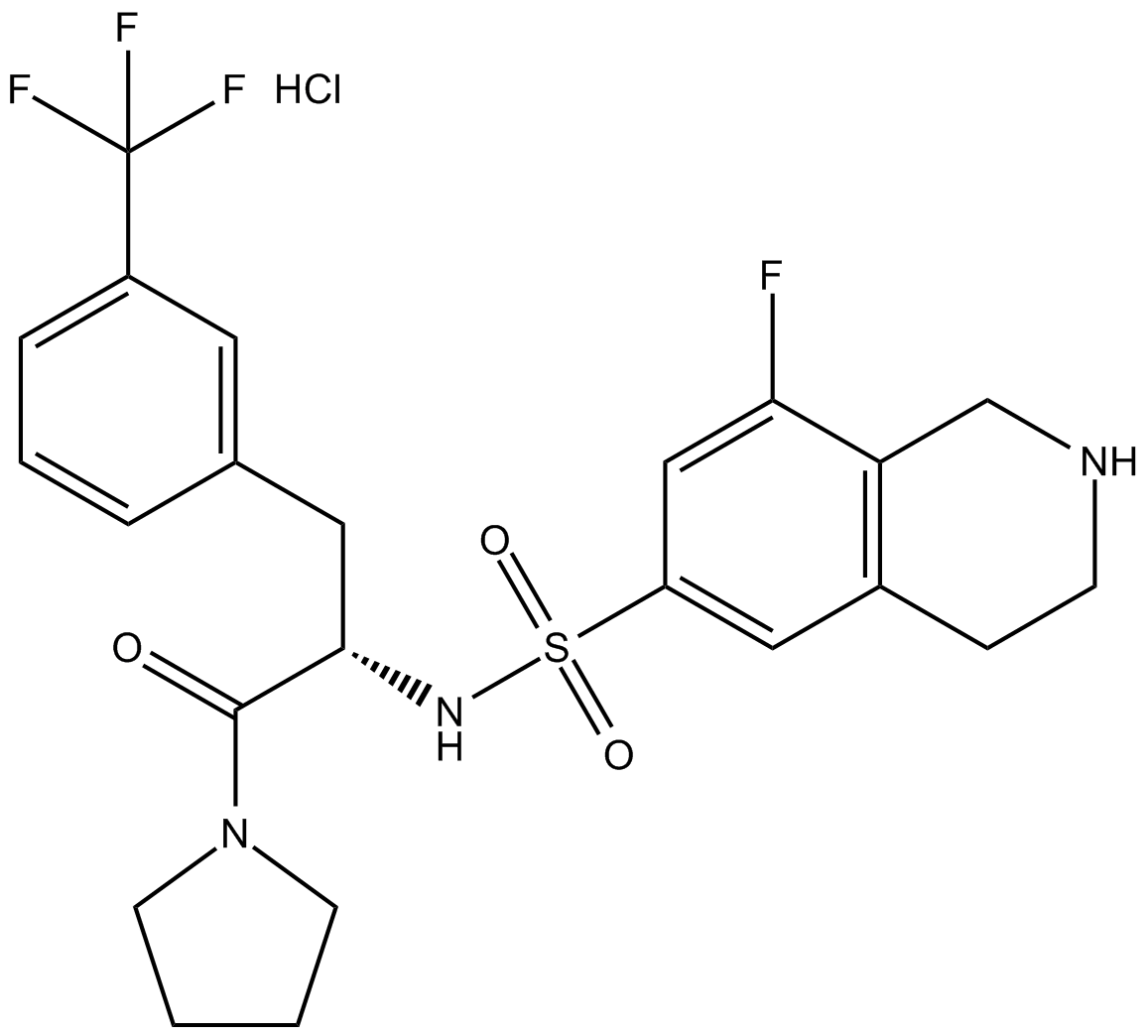
-
GC65133
(S,R,S)-AHPC-C5-COOH
(S,R,S)-AHPC-C5-COOH (VH032-C5-COOH) es un conjugado enlazador-ligando de ligasa E3 sintetizado que contiene el ligando basado en VHL VH032 y un enlazador para formar PROTAC. VH-032 es un inhibidor selectivo y potente de la interacciÓn VHL/HIF-1α con una Kd de 185 nM, tiene potencial para el estudio de anemia y enfermedades isquémicas.
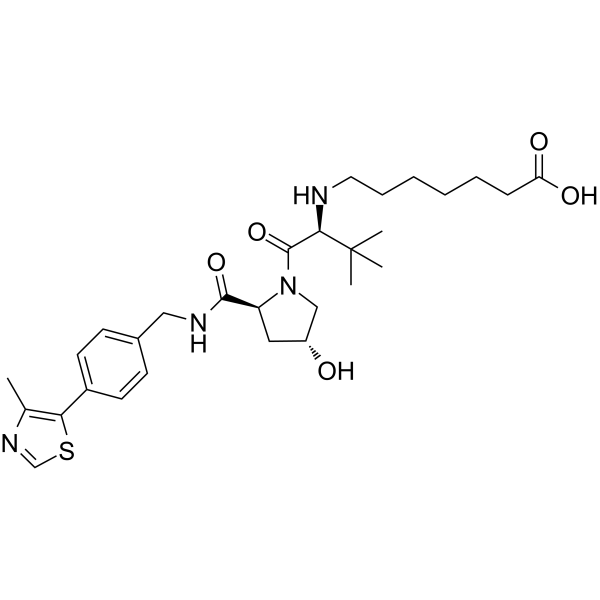
-
GC17055
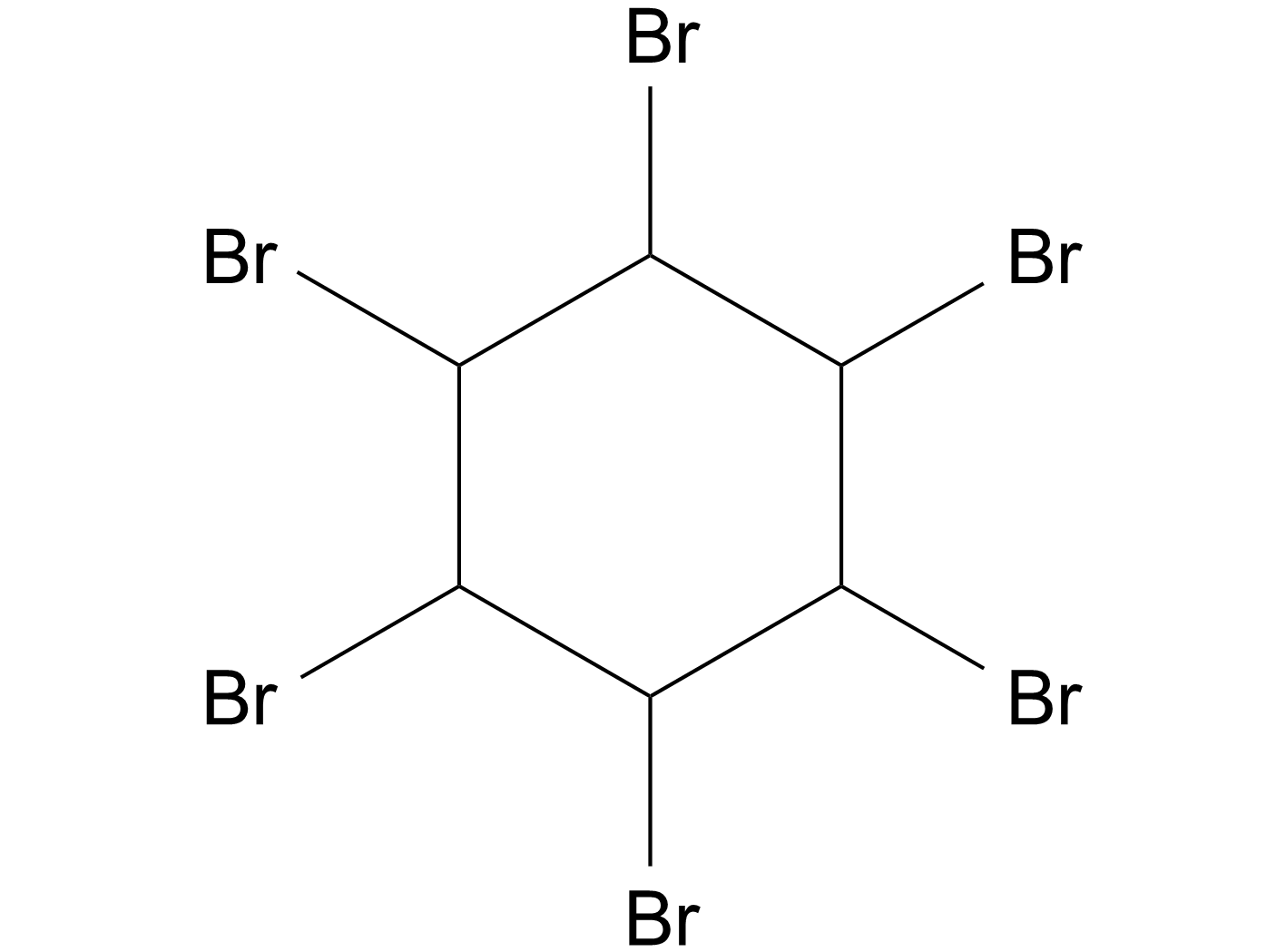
1,2,3,4,5,6-Hexabromocyclohexane
1,2,3,4,5,6-六溴环己烷(1,2,3,4,5,6-Hexabromocyclohexane)直接抑制JAK2激酶结构域内的口袋,抑制自身磷酸化。
-
GC61949
1,4-DPCA ethyl ester
El éster etÍlico de 1,4-DPCA es el éster etÍlico de 1,4-DPCA y puede inhibir el factor inhibidor HIF (FIH).

-
GC40468
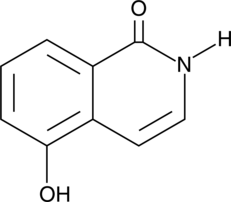
1,5-Isoquinolinediol
El 1,5-isoquinolinediol es un potente inhibidor de PARP, con una IC50 de 0,18-0,37 μM.
-
GC41984
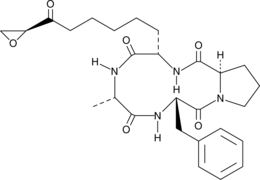
1-Alaninechlamydocin
La 1-alanina clamidocina, un tetrapéptido cÍclico, es un potente inhibidor de HDAC (IC50 = 6,4 nM). La 1-alaninaclamidocina induce la detenciÓn del ciclo celular G2/M y la apoptosis en células MIA PaCa-2.
-
GC39214
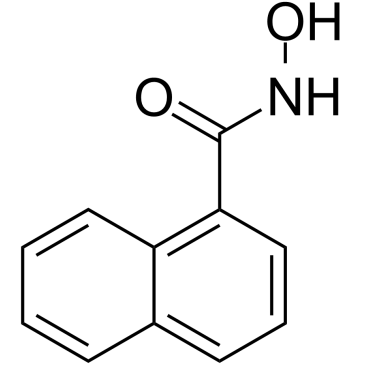
1-Naphthohydroxamic acid
El Ácido 1-naftohidroxÁmico (Compuesto 2) es un inhibidor HDAC8 potente y selectivo con una IC50 de 14 μM. El Ácido 1-naftohidroxÁmico es mÁs selectivo para HDAC8 que HDAC1 de clase I y HDAC6 de clase II (IC50 >100 μM). El Ácido 1-naftohidroxÁmico no aumenta la acetilaciÓn global de la histona H4 y tampoco reduce la actividad HDAC intracelular total. El Ácido 1-naftohidroxÁmico puede inducir la acetilaciÓn de la tubulina.
-
GC46508
2',2'-Difluoro-2'-deoxyuridine
An active metabolite of gemcitabine
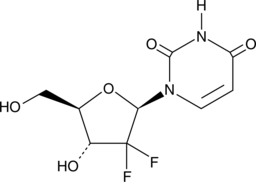
-
GC12775
2',3',5'-triacetyl-5-Azacytidine
2',3',5'-triacetil-5-azacitidina es un profÁrmaco oralmente activo de 5-azacitidina. La 5-azacitidina es un inhibidor de la ADN metiltransferasa.
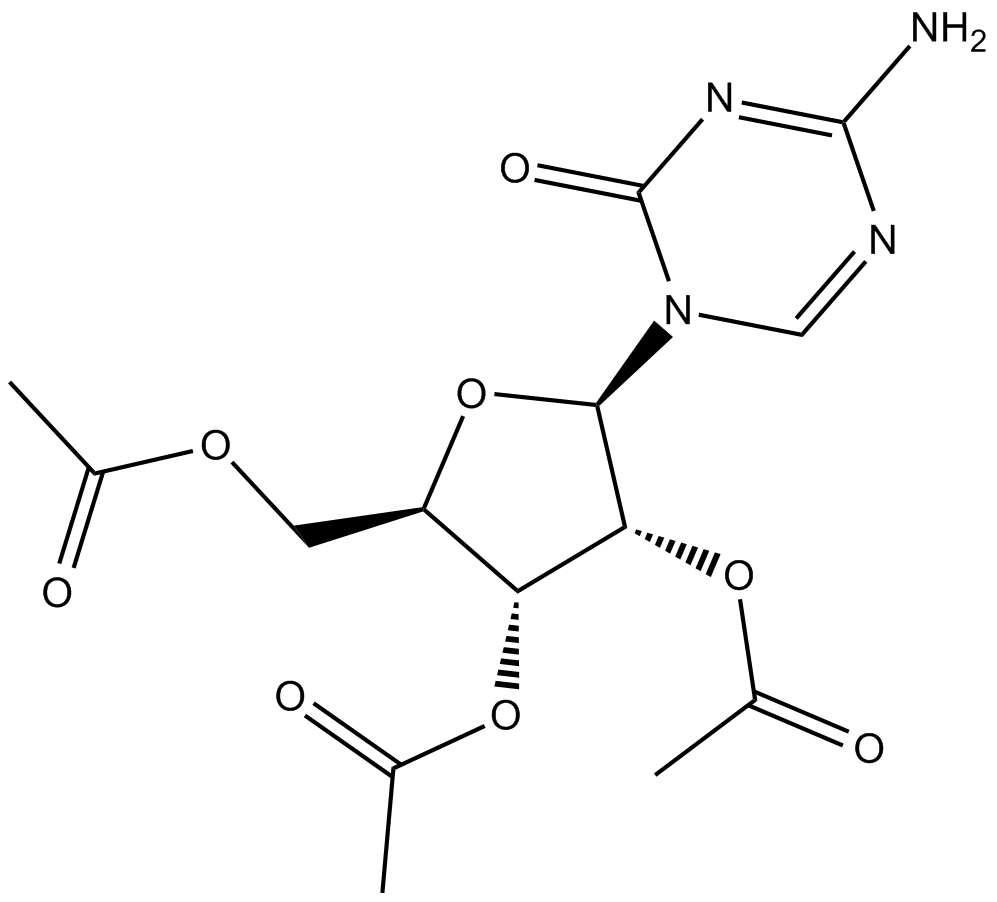
-
GC16195
2,4-DPD
El dietilpiridin-2,4-dicarb es un proinhibidor potente dirigido por la prolil 4-hidroxilasa.
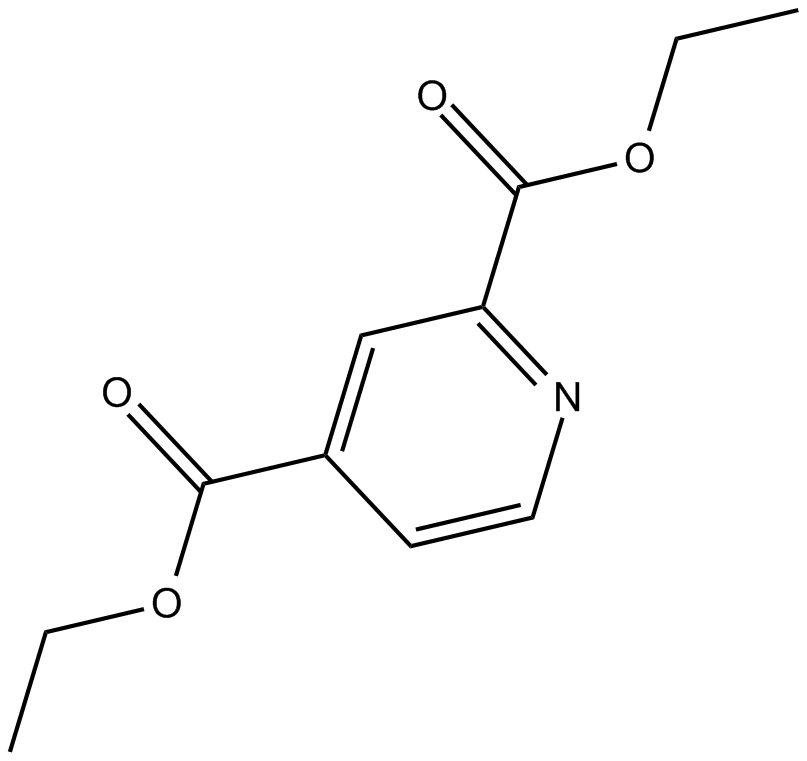
-
GC11873
2,4-Pyridinedicarboxylic Acid
El Ácido 2,4-piridindicarboxÍlico (Ácido 2,4 piridindicarboxÍlico) es un inhibidor de amplio espectro de la oxigenasa 2OG, incluida la familia de histona desmetilasas (JHDM) que contiene el dominio JmjC. El Ácido 2,4-piridindicarboxÍlico es un quÍmico objetivo en el campo de los plÁsticos de base biolÓgica.
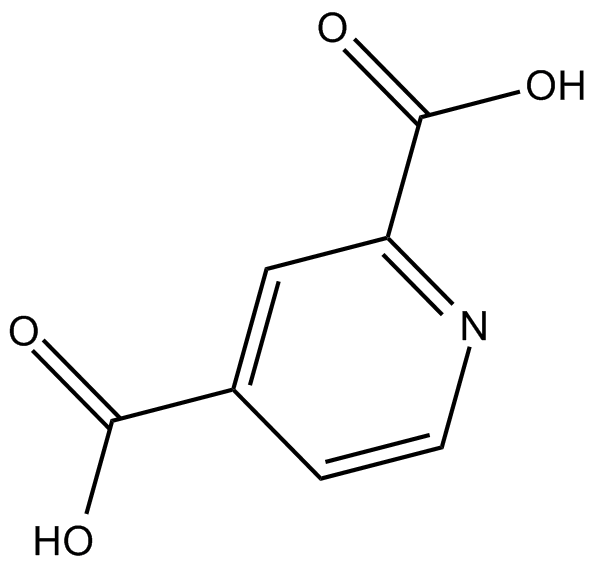
-
GC42075
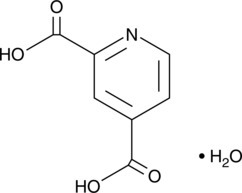
2,4-Pyridinedicarboxylic Acid (hydrate)
2,4-Pyridinedicarboxylic Acid (2,4-PDCA) is a compound that structurally mimics 2-oxoglutarate (2-OG, also known as α-ketoglutarate) and chelates zinc, thus affecting a range of enzymes.
-
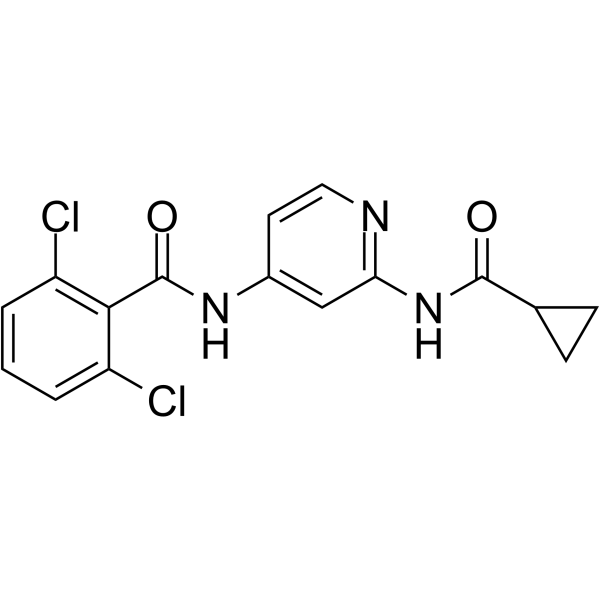
GC62233
2,6-Dichloro-N-(2-(cyclopropanecarboxamido)pyridin-4-yl)benzamide
GDC-046 es un inhibidor de TYK2 potente, selectivo y biodisponible por vÍa oral con Kis de 4,8, 0,7, 0,7 y 0,4 nM para TYK2, JAK1, JAK2 y JAK3, respectivamente.
-
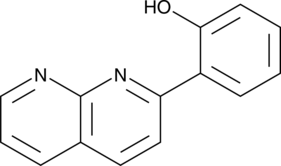
GC46503
2-(1,8-Naphthyridin-2-yl)phenol
El 2-(1,8-naftiridin-2-il)fenol es un potenciador selectivo de la transcripciÓn de STAT1. 2-(1,8-naftiridin-2-il)fenol puede mejorar la capacidad de IFN-γ para inhibir la proliferaciÓn de células de cÁncer de mama humano y fibrosarcoma.
-
GC10408
2-D08
2-D08 es un inhibidor mecÁnicamente Único, permeable a las células, de la SUMOilaciÓn de proteÍnas. 2-D08 también inhibe Axl con un IC50 de 0,49 nM.
-
GC11249
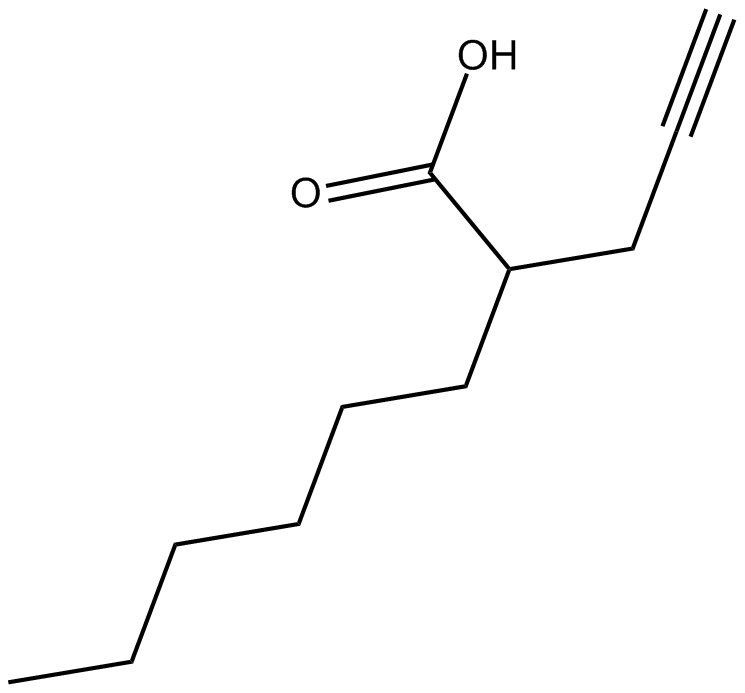
2-hexyl-4-Pentynoic Acid
Potent and robust HDACs inhibitor
-
GC15084
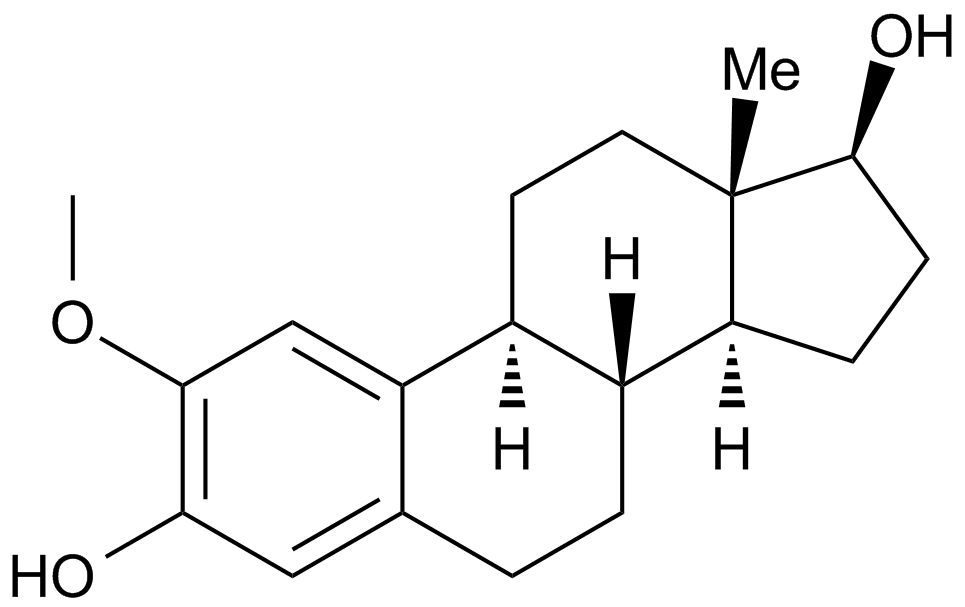
2-Methoxyestradiol (2-MeOE2)
2-Methoxyestradiol (2-MeOE2/2-Me) is a HIF-1α inhibitor that inhibits HIF-1α accumulation and HIF transcriptional activity. 2-Methoxyestradiol can trigger p53-induced apoptosis and has potential antitumor activity..
-
GC62781
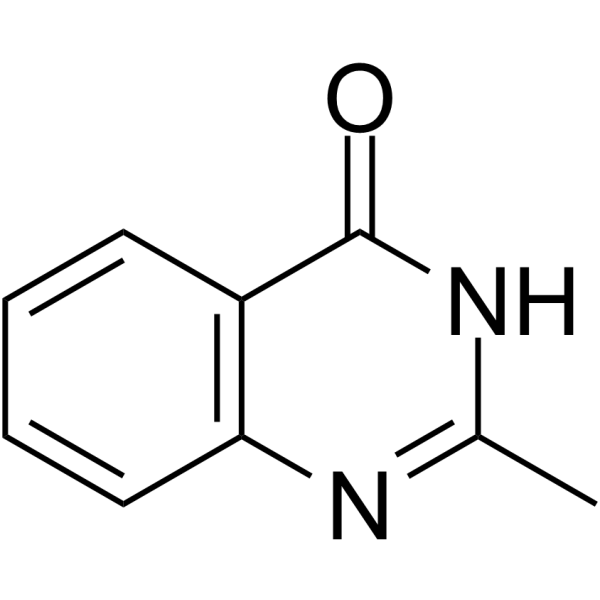
2-Methylquinazolin-4-ol
El 2-metilquinazolin-4-ol es un potente inhibidor competitivo de la poli(ADP-ribosa) sintetasa, con una Ki de 1,1 μM.
-
GC14282
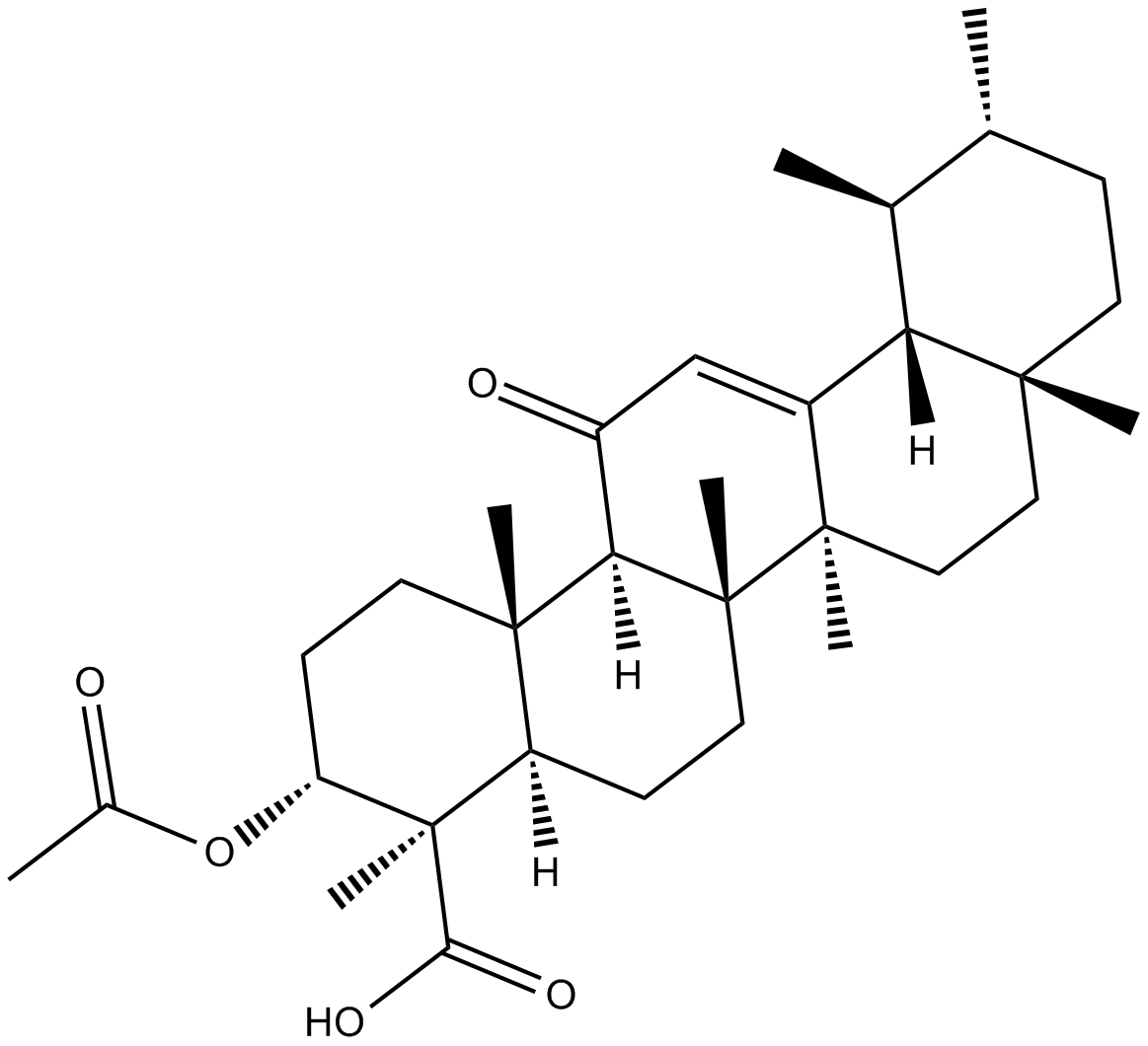
3-acetyl-11-keto-β-Boswellic Acid
El Ácido 3-acetil-11-ceto-β-boswélico (acetil-11-ceto-β-Ácido boswélico) es un compuesto triterpenoide activo del extracto de serrato de Boswellia y un nuevo activador Nrf2.
-
GC17907
3-Deazaneplanocin A (DZNep) hydrochloride
Un inhibidor de la lisina metiltransferasa EZH2.
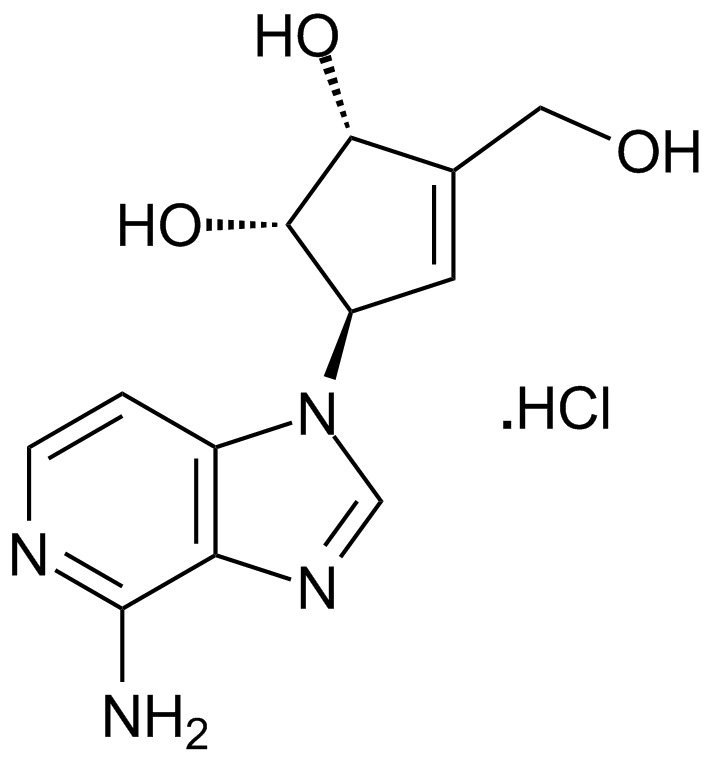
-
GC13145
3-Deazaneplanocin,DZNep
An inhibitor of lysine methyltransferase EZH2
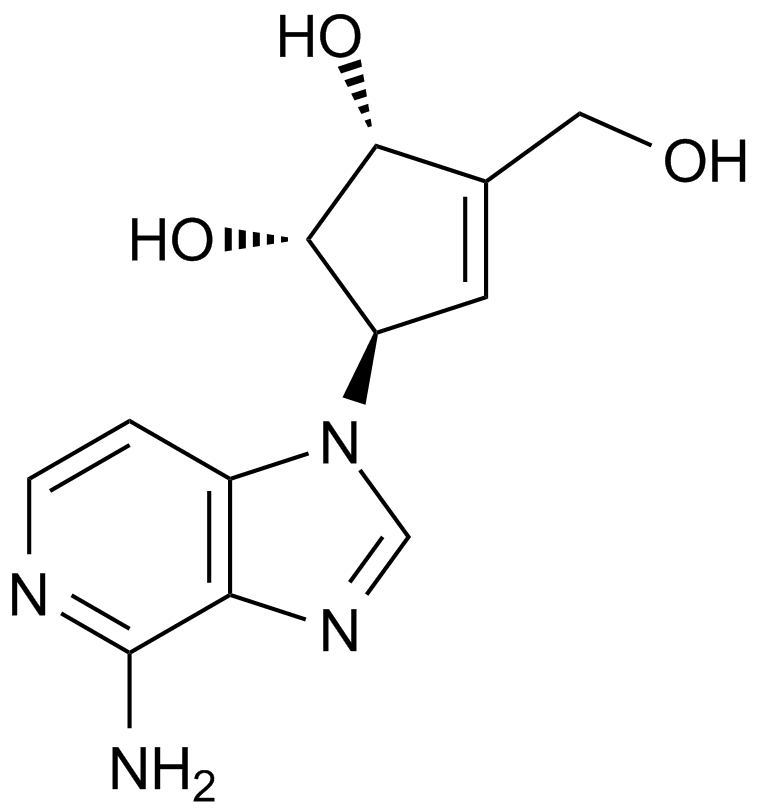
-
GC18509
3-Methylcytidine (methosulfate)
3-Methylcytidine (methosulfate) is a cytidine derivative used as an internal standard for HPLC.
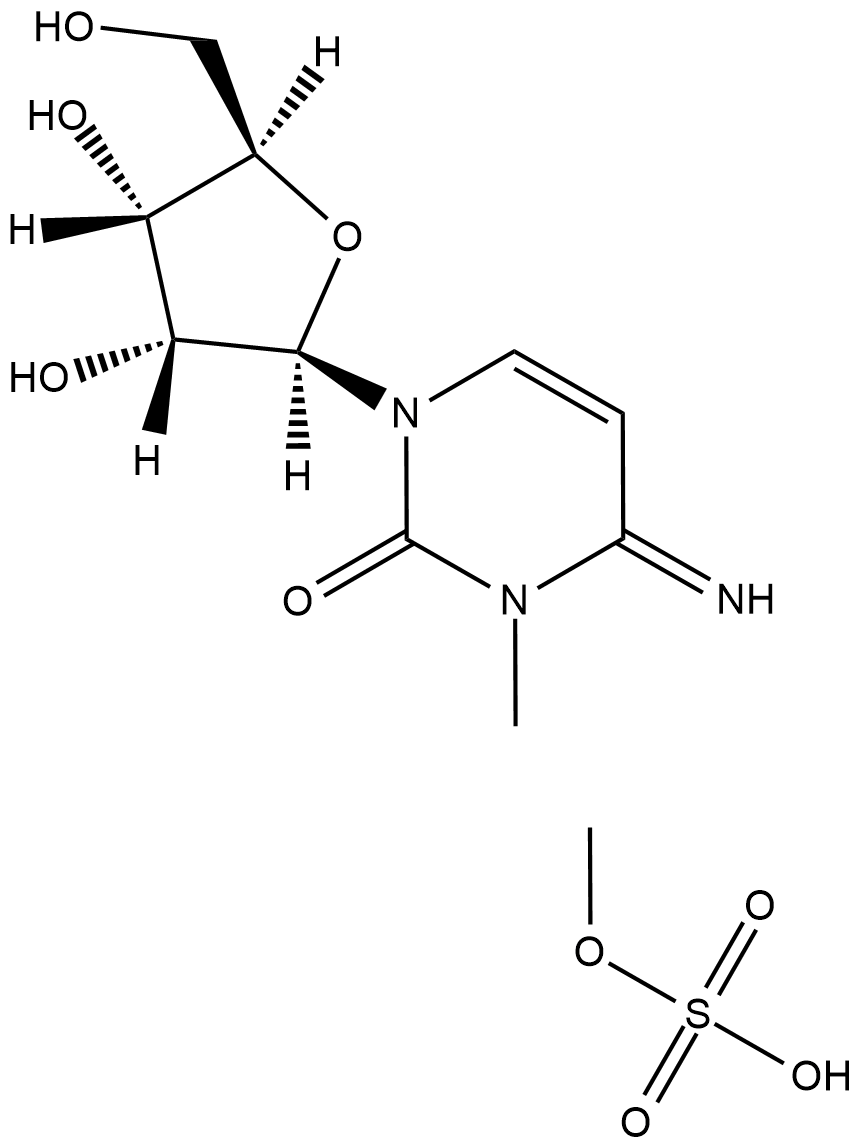
-
GC19013
3-TYP
3-TYP inhibe SIRT3 con un IC50 de 16 nM, y es más potente sobre SIRT1 y SIRT2 con IC50 de 88 nM y 92 nM, respectivamente.
-
GC62805
4’-Methoxychalcone
4’-Methoxychalcone regula la diferenciación de adipocitos a través de la activación de PPARγ.

-
GC17922
4'-bromo-Resveratrol
El 4'-Bromo-resveratrol es un inhibidor potente y dual de la Sirtuina-1 y la Sirtuina-3. El 4'-bromo-resveratrol inhibe el crecimiento de células de melanoma a través de la reprogramaciÓn metabÓlica mitocondrial. El 4'-bromo-resveratrol imparte efectos antiproliferativos en las células de melanoma a través de una reprogramaciÓn metabÓlica y afecta el ciclo celular y la seÑalizaciÓn de la apoptosis.
-
GC46607
4-(Methylnitrosamino)-1-(3-pyridyl)-1-butanone
A tobacco-specific nitrosamine carcinogen
-
GC11761
4-amino-1,8-Naphthalimide
La 4-amino-1,8-naftalimida es un potente inhibidor de PARP y potencia la citotoxicidad de la γ-radiaciÓn en células cancerosas.
-
GC46628
4-Chloro-6,7-bis(2-methoxyethoxy)quinazoline
A building block and synthetic intermediate
-
GC17005
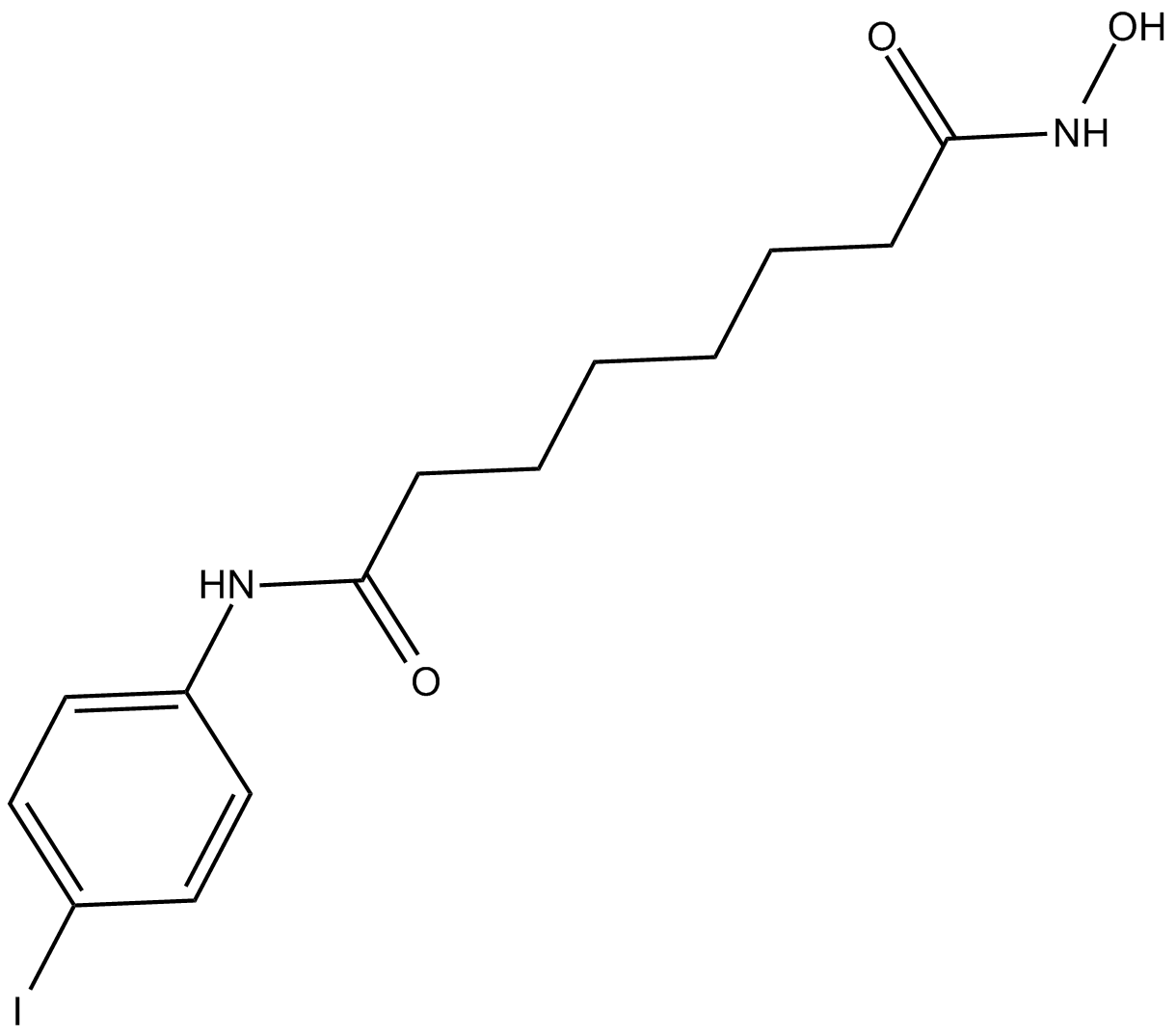
4-iodo-SAHA
4-Iodo-SAHA (1k) es un inhibidor de histona desacetilasa (HDAC) de clase I y clase II activo por vÍa oral con CE50 de 1,1, 0,95, 0,12, 0,24, 0,85 y 1,3 μM para las lÍneas celulares Skbr3, HT29, U937, JA16 y HL60 , respectivamente. 4-Iodo-SAHA (1k) se puede utilizar para la investigaciÓn del cÁncer.
-
GC46675
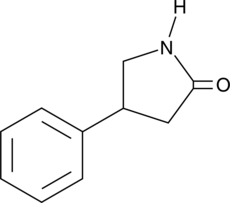
4-Phenyl-2-pyrrolidinone
A precursor and synthetic intermediate
-
GC12667
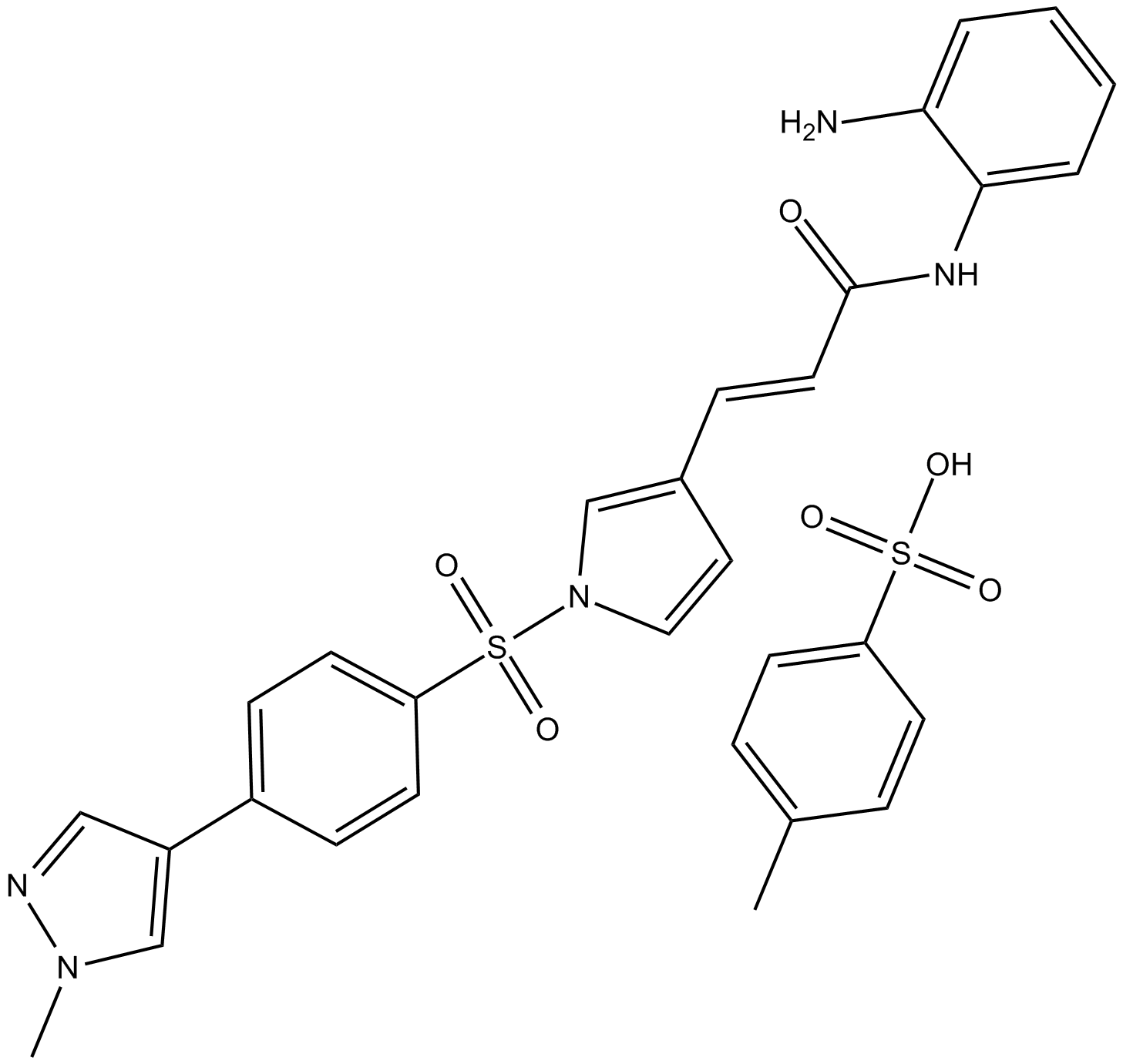
4SC-202
4SC-202 (4SC-202) es un inhibidor selectivo de HDAC de clase I con IC50 de 1,20 μM, 1,12 μM y 0,57 μM para HDAC1, HDAC2 y HDAC3, respectivamente. También muestra actividad inhibitoria contra la desmetilasa 1 especÍfica de lisina (LSD1).
-
GC35150
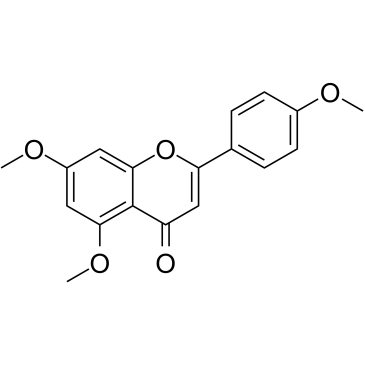
5,7,4'-Trimethoxyflavone
La 5,7,4'-trimetoxiflavona se aísla de Kaempferia parviflora (KP), una famosa planta medicinal de Tailandia. La 5,7,4'-trimetoxiflavona induce la apoptosis, como lo demuestran los incrementos de la fase sub-G1, la fragmentación del ADN, la tinción de anexina-V/PI, la relación Bax/Bcl-xL, la activación proteolítica de la caspasa-3 y la degradación de poli (ADP-ribosa) polimerasa (PARP) proteína. La 5,7,4'-trimetoxiflavona es significativamente eficaz para inhibir la proliferación de células de cáncer gástrico humano SNU-16 de una manera dependiente de la concentración.
-
GN10629
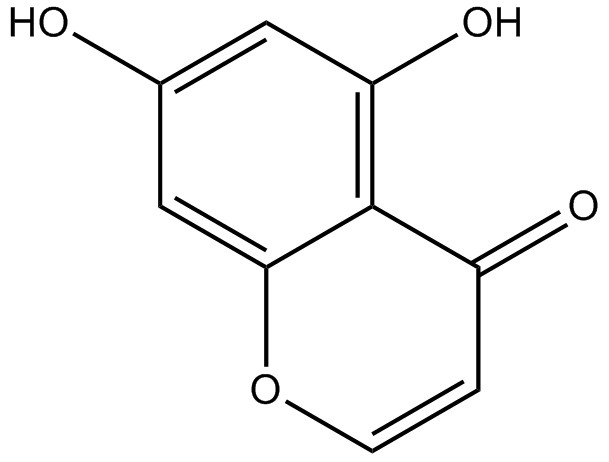
5,7-dihydroxychromone
-
GC10898
5-(N,N-dimethyl)-Amiloride (hydrochloride)
NHE1, NHE2, and NHE3 inhibitor
-
GC68161
5-AIQ
-
GC10946
5-Azacytidine
A DNA methyltransferase inhibitor
-
GC61662
5-Fluoro-2'-deoxycytidine
La 5-fluoro-2'-desoxicitidina, un anÁlogo de nucleÓsido de fluoropirimidina, es un inhibidor de la ADN metiltransferasa (DNMT). La 5-fluoro-2'-desoxicitidina es un profÁrmaco selectivo de tumores del potente inhibidor de la timidilato sintasa 5-fluoro-2'-dUMP.
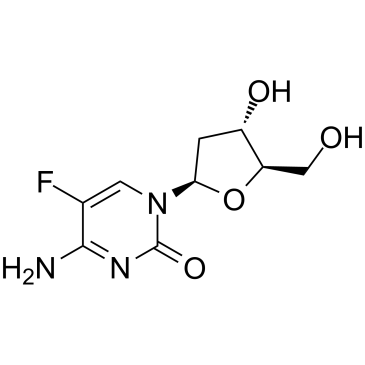
-
GC42562
5-Methyl-2'-deoxycytidine
La 5-metil-2'-desoxicitidina en el ADN monocatenario puede actuar en cis para seÑalar la metilaciÓn del ADN de novo.
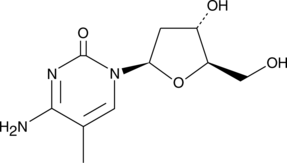
-
GC66055
5-Phenylpentan-2-one
La 5-fenilpentan-2-ona es un potente inhibidor de las histonas desacetilasas (HDAC). 5-Phenylpentan-2-one se puede utilizar para la investigaciÓn del trastorno del ciclo de la urea.
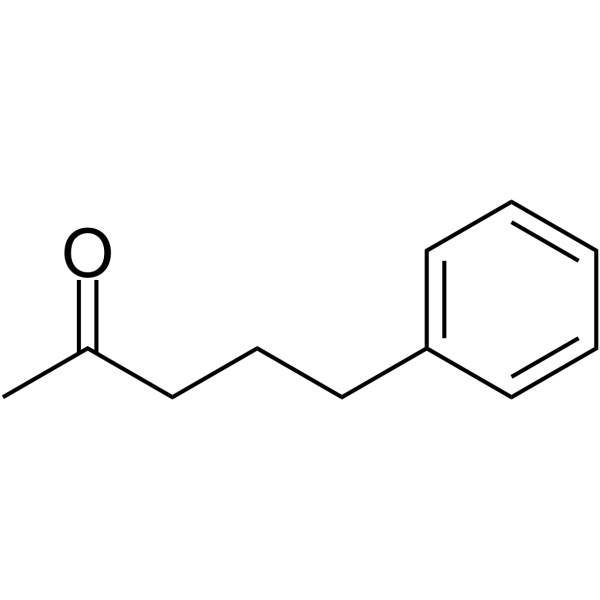
-
GC45772
6(5H)-Phenanthridinone
An inhibitor of PARP1 and 2
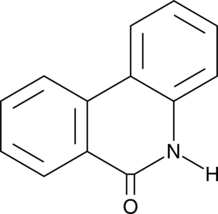
-
GC35175
6-Demethoxytangeretin
La 6-demetoxitangeretina es un flavonoide cÍtrico aislado de Citrus depressa.
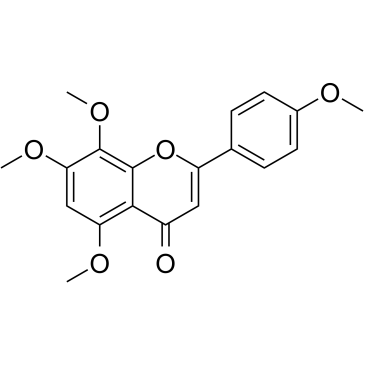
-
GC35180
6-Methyl-5-azacytidine
La 6-metil-5-azacitidina es un potente inhibidor de la DNMT.
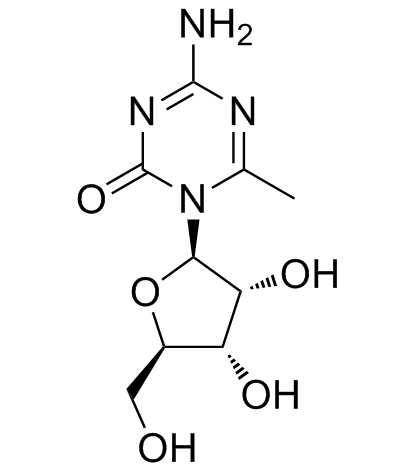
-
GC62279
653-47
653-47, un potenciador, potencia significativamente la actividad inhibidora de la proteÍna de uniÓn al elemento de respuesta cAMP (CREB) de 666-15. 653-47 también es un inhibidor de CREB muy débil con IC50 de 26,3 μM.
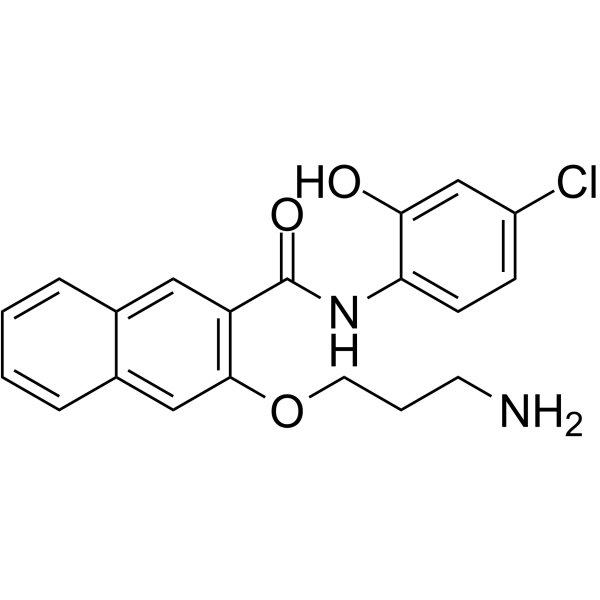
-
GC62144
653-47 hydrochloride
El clorhidrato de 653-47, un potenciador, potencia significativamente la actividad inhibidora de la proteÍna de uniÓn al elemento de respuesta cAMP (CREB) de 666-15. El clorhidrato de 653-47 también es un inhibidor de CREB muy débil con IC50 de 26,3 μM.
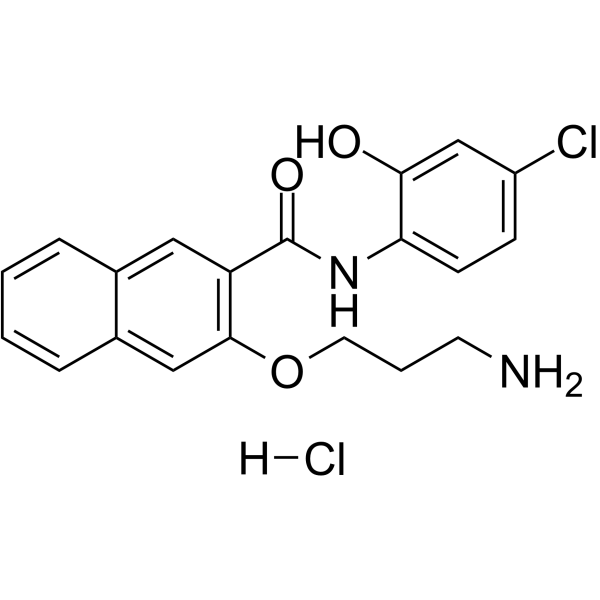
-
GC32689
666-15
666-15 es un inhibidor de CREB potente y selectivo con una IC50 de 81 nM. 666-15 suprime el crecimiento tumoral en un modelo de xenoinjerto de cÁncer de mama.
-
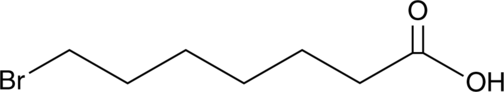
GC46736
7-Bromoheptanoic Acid
A building block
-
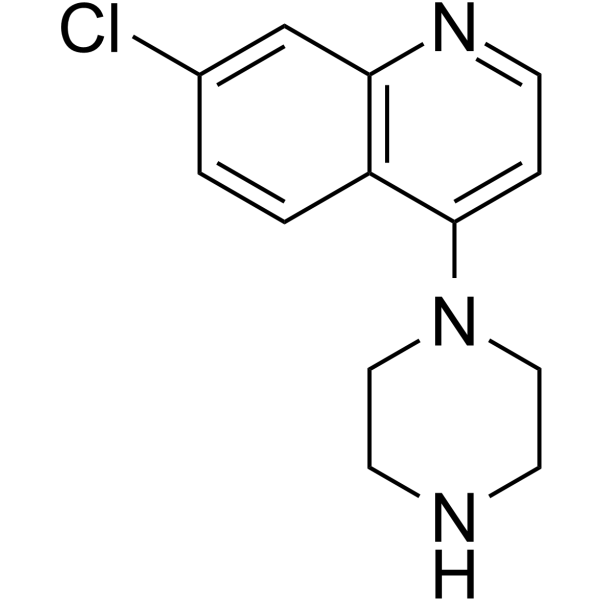
GC62651
7-Chloro-4-(piperazin-1-yl)quinoline
La 7-cloro-4-(piperazin-1-il)quinolona es un andamio importante en la quÍmica médica.
-
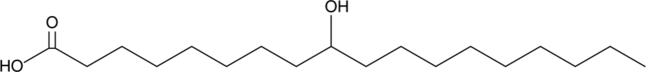
GC48986
9-hydroxy Stearic Acid
A hydroxy fatty acid
-
GC16015
A 366
A 366 es un inhibidor de histona metiltransferasa G9a potente, altamente selectivo y competitivo con péptidos con IC50 de 3,3 y 38 nM para G9a y GLP (EHMT1), respectivamente. A 366 muestra una selectividad de \u003e1000 veces sobre otras 21 metiltransferasas. A 366 también es un potente inhibidor nanomolar de la interacción Spindlin1-H3K4me3 (IC50 \u003d 182,6 nM). A 366 muestra alta afinidad por el receptor H3 de histamina humano (Ki \u003d 17 nM) y muestra selectividad de subtipo entre los subconjuntos de las familias de receptores histaminérgicos y dopaminérgicos.
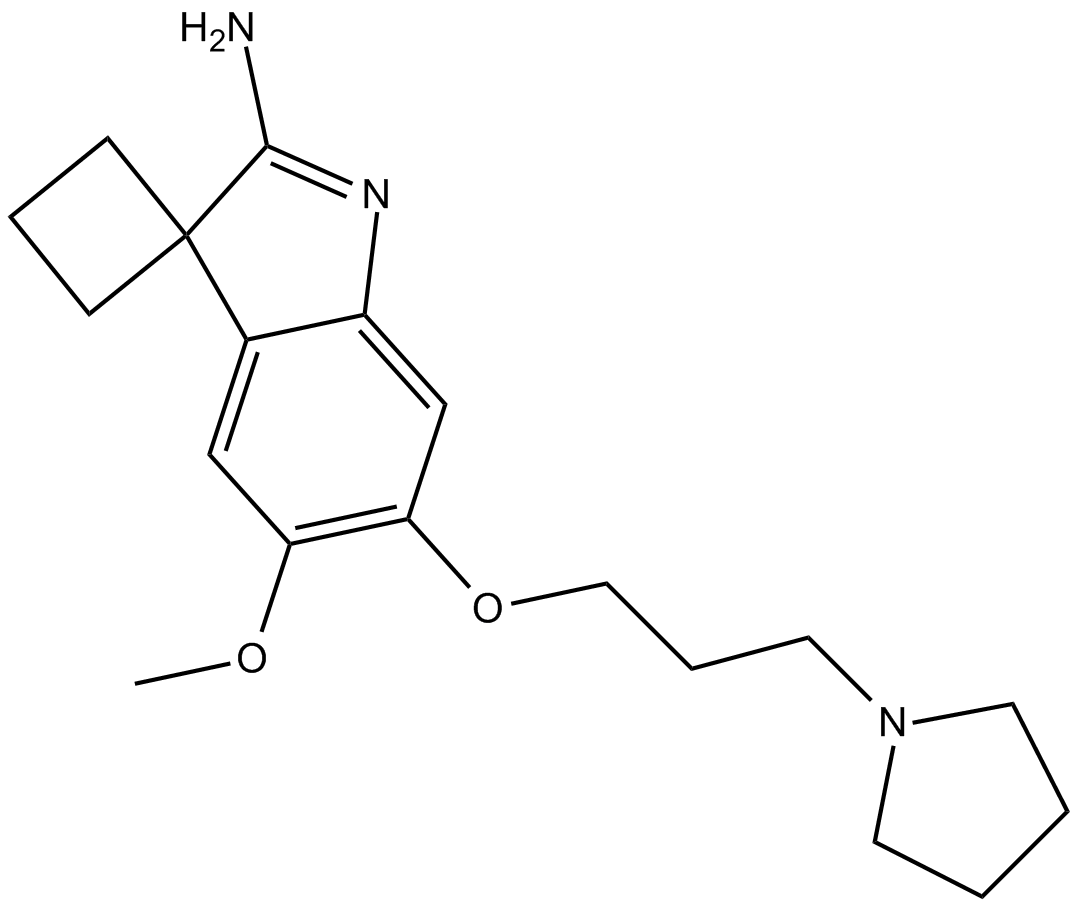
-
GC32861
A-196
A-196 es un inhibidor potente y selectivo de SUV420H1 y SUV420H2 con valores IC50 de 25 nM y 144 nM, respectivamente. A-196 inhibe SUV4-20 bioquÍmicamente de manera competitiva con el sustrato. A-196 representa una sonda quÍmica de primera clase de SUV4-20 para investigar el papel de las histonas metiltransferasas en la integridad genÓmica.
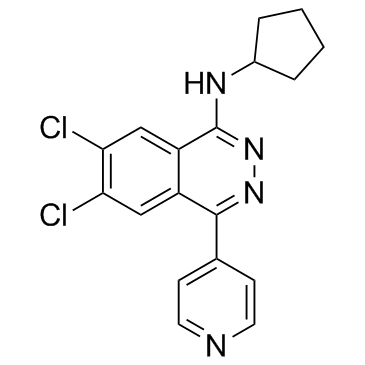
-
GC33187
A-395 (A395)
A-395 (A395) es un antagonista de las interacciones proteÍna-proteÍna del complejo represivo 2 de Polycomb (PRC2) que inhibe potentemente el complejo trimérico PRC2 (EZH2-EED-SUZ12) con una IC50 de 18 nM.
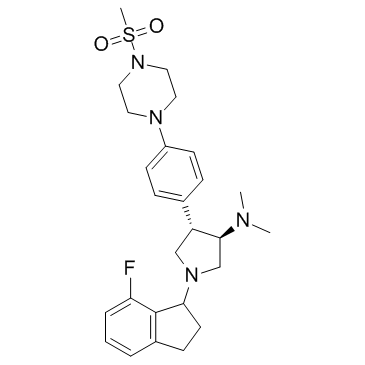
-
GC32677
A-485
A-485 es un inhibidor catalÍtico potente y selectivo de p300/CBP con IC50 de 9,8nM y 2,6nM para p300 y CBP histona acetiltransferasa (HAT), respectivamente.
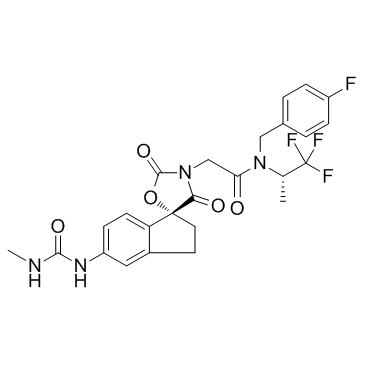
-
GC30503
A-893
A-893 es un inhibidor celular activo de la metiltransferasa SMYD2, con una IC50 de 2,8 nM.
-
GC12390
A-966492
A PARP1 and PARP2 inhibitor
-
GC33280
A1874
A1874 es un PROTAC basado en nutlina (ligando MDM2) y degradante de BRD4 con una DC50 de 32 nM (induce la degradaciÓn de BRD4 en las células). Eficaz para inhibir la proliferaciÓn de muchas lÍneas celulares de cÁncer.
-
GC35216
AAPK-25
AAPK-25 es un inhibidor dual Aurora/PLK potente y selectivo con actividad antitumoral, que puede provocar un retraso mitÓtico y detener las células en una prometafase, reflejada por la fosforilaciÓn de la histona H3Ser10 del biomarcador y seguida de un aumento de la apoptosis. AAPK-25 se dirige a Aurora-A, -B y -C con valores de Kd que oscilan entre 23 y 289 nM, asÍ como a PLK-1, -2 y -3 con valores de Kd que oscilan entre 55 y 456 nM.
-
GC19409
ABBV-744
ABBV-744 es un inhibidor selectivo, activo por vÍa oral, primero en su clase, del dominio BDII de las proteÍnas de la familia BET con valores de IC50 que van de 4 a 18 nM para BRD2, BRD3, BRD4 y BRDT.
-
GC10154
ABC294640
ABC294640 (ABC294640) es un inhibidor selectivo y competitivo de la esfingosina quinasa 2 (SK2) con Ki de 9,8 μM.
-
GC32023
Abrocitinib (PF-04965842)
Abrocitinib (PF-04965842) (PF-04965842) es un inhibidor de JAK1 potente, activo por vÍa oral y selectivo, con IC50 de 29 y 803 nM para JAK1 y JAK2, respectivamente.
-
GC12422
ABT-888 (Veliparib)
-
GA20481
Ac-Arg-Gly-Lys(Ac)-AMC
Ac-Arg-Gly-Lys(Ac)-AMC es un sustrato para HDAC.

-
GA20605
Ac-Lys-AMC
Ac-Lys-AMC (hexanamida), también denominada MAL, es un sustrato fluorescente para las histonas desacetilasas HDAC.

-
GC48382
Ac-QPKK(Ac)-AMC
A fluorogenic substrate for SIRT1, SIRT2, and SIRT3

-
GC35227
ACBI1
ACBI1 es un degradador potente y cooperativo de SMARCA2, SMARCA4 y PBRM1 con DC50 de 6, 11 y 32 nM, respectivamente. ACBI1 es un degradador PROTAC. ACBI1 muestra actividad antiproliferativa. ACBI1 induce la apoptosis.
-
GC12917
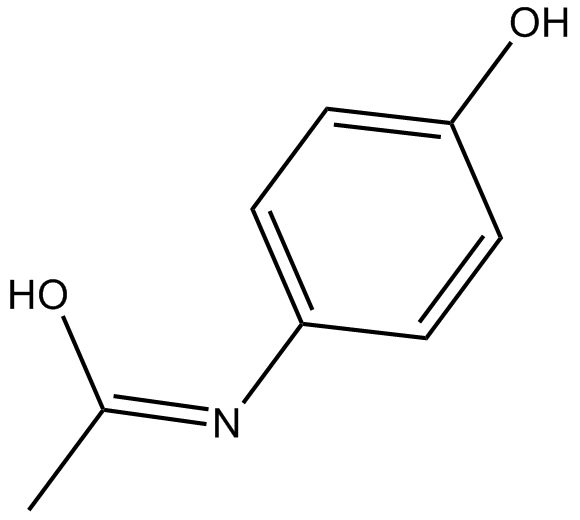
Acetaminophen
Un inhibidor de COX
-
GC64137
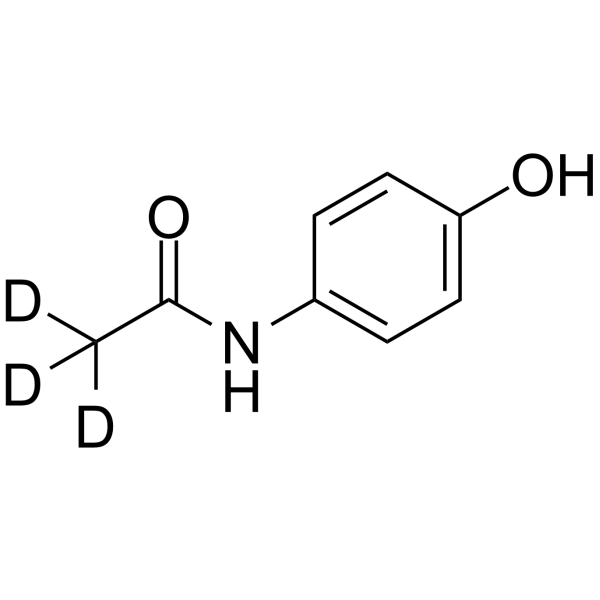
Acetaminophen-d3
-
GC35230
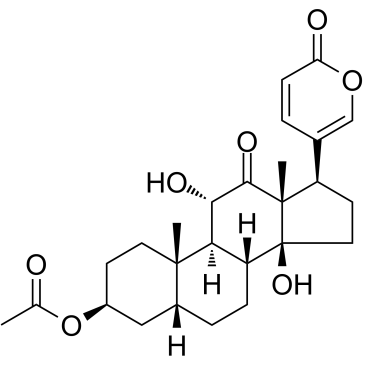
Acetylarenobufagin
Acetylarenobufagin es un modulador del factor 1 inducible por hipoxia esteroide (HIF-I).
-
GC32083
Acriflavine
La acriflavina es un tinte fluorescente para marcar ARN de alto peso molecular.

-
GC62354
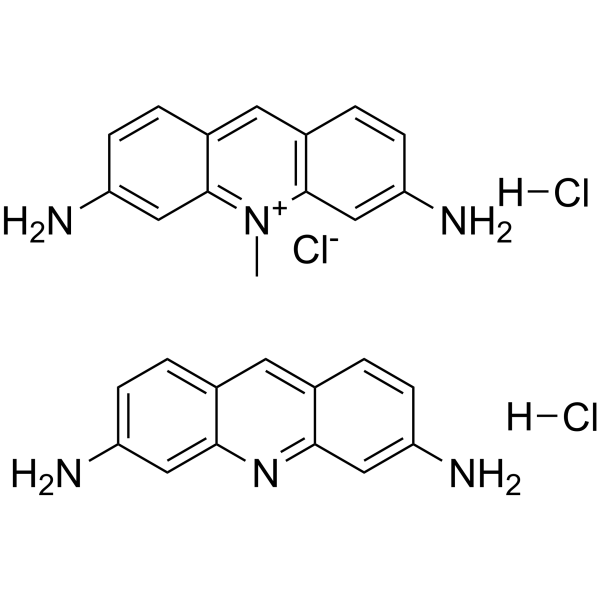
Acriflavine hydrochloride
El clorhidrato de acriflavina (clorhidrato de cloruro de acriflavinio) es un tinte fluorescente de acridina que se puede usar para marcar el Ácido nucleico.
-
GC34458
ACY-1083
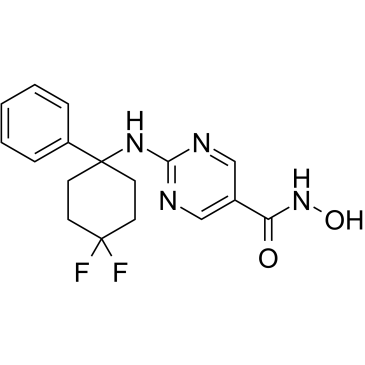
ACY-1083 es un inhibidor selectivo de HDAC6 que penetra en el cerebro con una IC50 de 3 nM y es 260 veces más selectivo para HDAC6 que todas las demás clases de isoformas de HDAC. ACY-1083 revierte eficazmente la neuropatía periférica inducida por quimioterapia.
-
GC10417
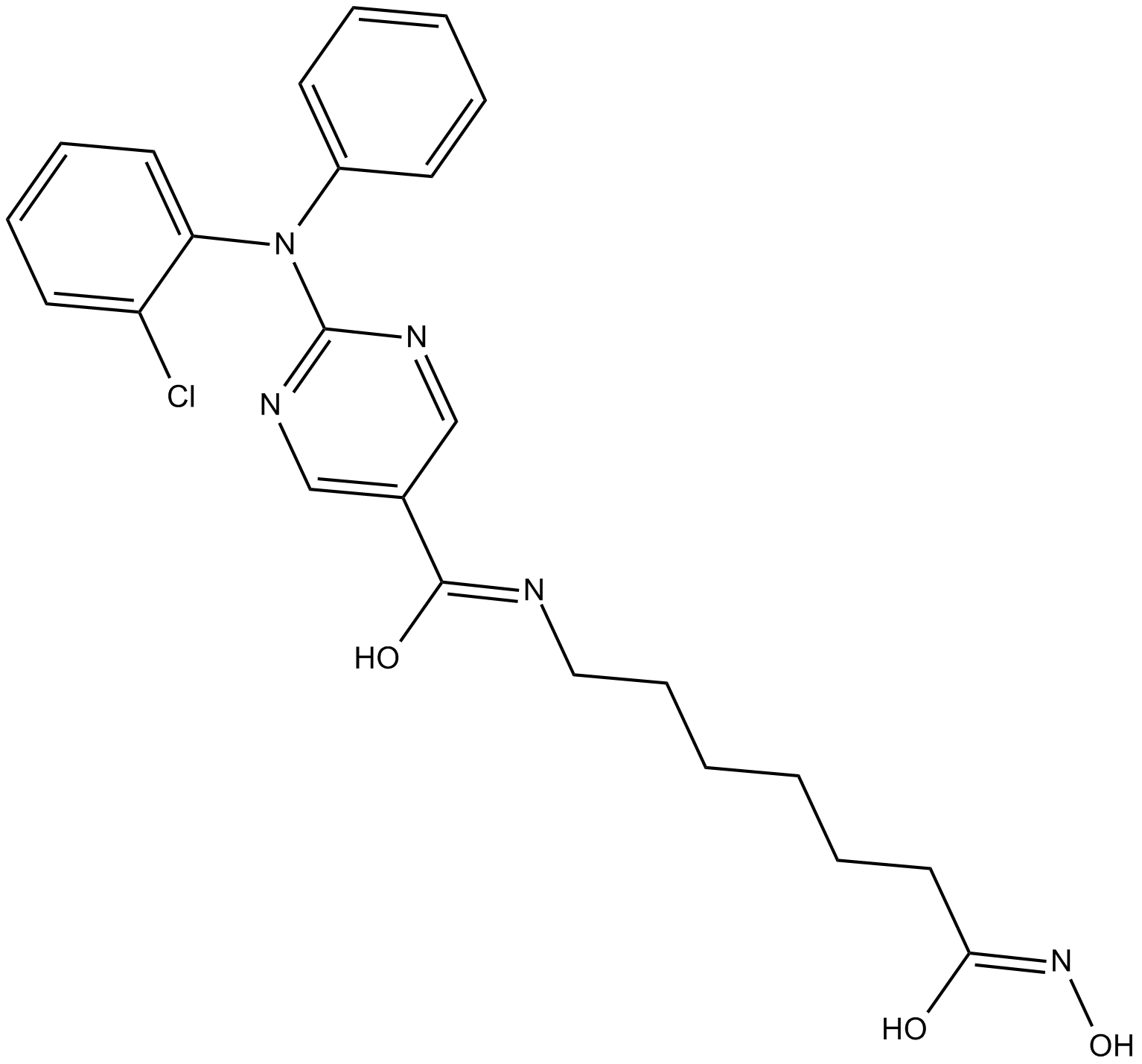
ACY-241
ACY-241 (ACY241) es un inhibidor de HDAC6 potente, activo por vÍa oral y altamente selectivo de segunda generaciÓn con una IC50 de 2,6 nM (IC50 de 35 nM, 45 nM, 46 nM y 137 nM para HDAC1, HDAC2, HDAC3 y HDAC8, respectivamente) ). ACY-241 tiene efectos anticancerÍgenos.
-
GC19020
ACY-738
ACY-738 es un inhibidor de HDAC6 potente, selectivo y biodisponible por vÍa oral, con una IC50 de 1,7 nM; ACY-738 también inhibe HDAC1, HDAC2 y HDAC3, con IC50 de 94, 128 y 218 nM.

-
GC30782
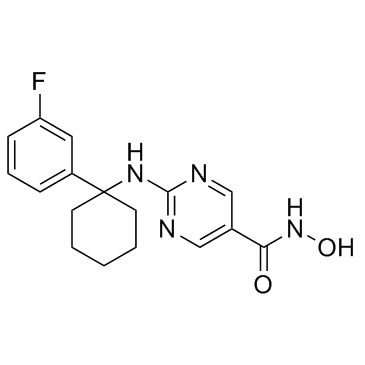
ACY-775
ACY-775 es un inhibidor potente y selectivo de la histona desacetilasa 6 (HDAC6) con una IC50 de 7,5nM.
-
GC30526
ACY-957
ACY-957 es un inhibidor selectivo y activo por vÍa oral de HDAC1 y HDAC2, con IC50 de 7 nM, 18 nM y 1300 nM contra HDAC1/2/3, respectivamente, y no muestra inhibiciÓn sobre HDAC4/5/6/7/8 /9.
-
GC12487
Adaptaquin
Adaptaquin es un inhibidor del factor prolil hidroxilasa 2 inducible por hipoxia (HIF-PHD2), con una IC50 de 2 μM.