- Cat.No. Nombre del producto Información
-
GC26270
Bovine Serum Albumin(Fatty Acid & lgG Free, BioPremium)
BSA is a highly purified, lyophilized albumin, suitable for Westerns, enzyme systems, protein standards, hybridizations and as a protease-sensitive immunoassays.

-
GC26269
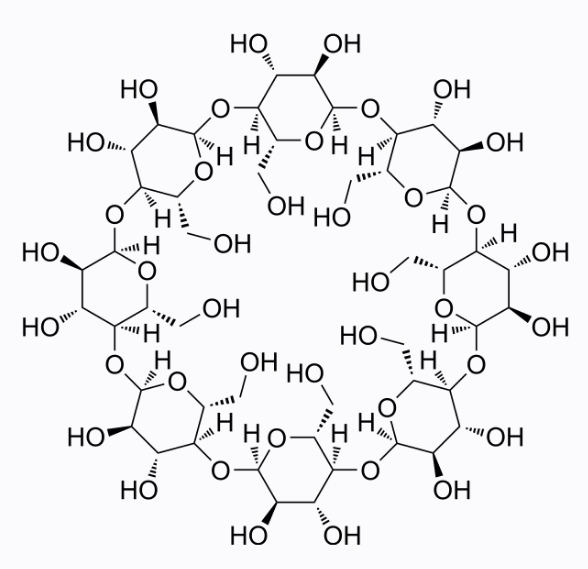
γ-Cyclodextrin
γ-Cyclodextrin is an endogenous metabolite.
-
GC26268
Galectin-3-IN-1
Galectin-3-IN-1 (Compound 1) is a potent multivalent inhibitor of galectin-3 (Gal-3).

-
GC26267
JG-2016
JG-2016 is a potent inhibitor of histone acetyltransferase 1 (HAT1), with an IC50 of 14.8 μM in the HAT1 acetylation assays.

-
GC26266
Lactyl-CoA
Lactyl-CoA is an acyl-CoA formally condensed from the sulfhydryl group of CoA and the carboxyl group of lactic acid,

-
GC26265
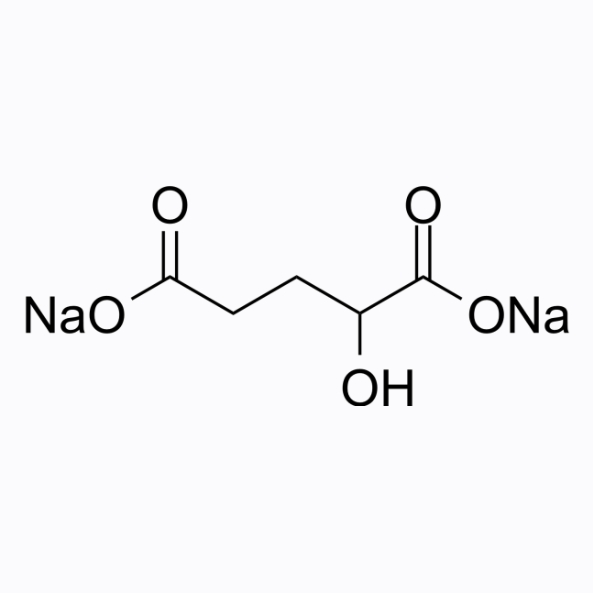
α-Hydroxyglutaric acid disodium
α-Hydroxyglutaric acid (2-Hydroxyglutarate) disodium is an α-hydroxy acid form of glutaric acid.
-
GC26264
AR antagonist 3
AR antagonist 3 is an effective selective androgen receptor (AR) antagonist with an IC50 of 0.47 µ M. AR antagonist 3 shows a dose-dependent decrease in FRET signal (IC50=18.05) μ M) . When administered intratumorally, AR antagonist 3 can effectively inhibit tumor growth.

-
GD91233
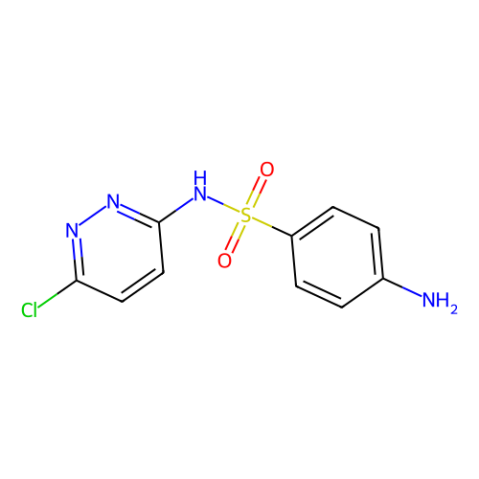
Sulfachloropyridazine
Sulfachloropyridazine is a sulfonamide antibiotic that blocks the synthesis of dihydrofolic acid by inhibiting the enzyme dihydropteroate synthase.
-
GD85600
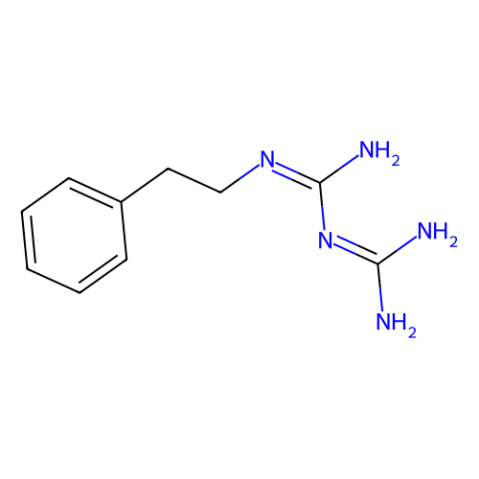
N-Phenethylbiguanide
-
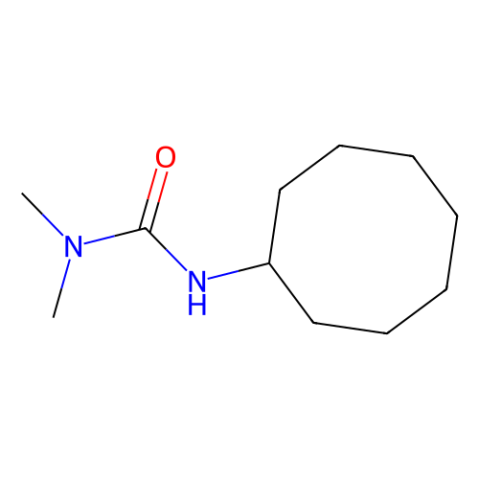
GB10412
Cycluron
Analytical standard, 97%
-
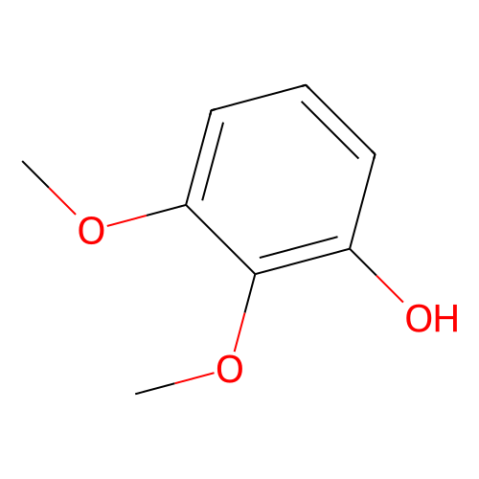
GD36838
2,3-Dimethoxyphenol
2,3-Dimethoxyphenol is a plant-derived phenylpropanoid compound that is glycosidated to form glycoside compound.
-
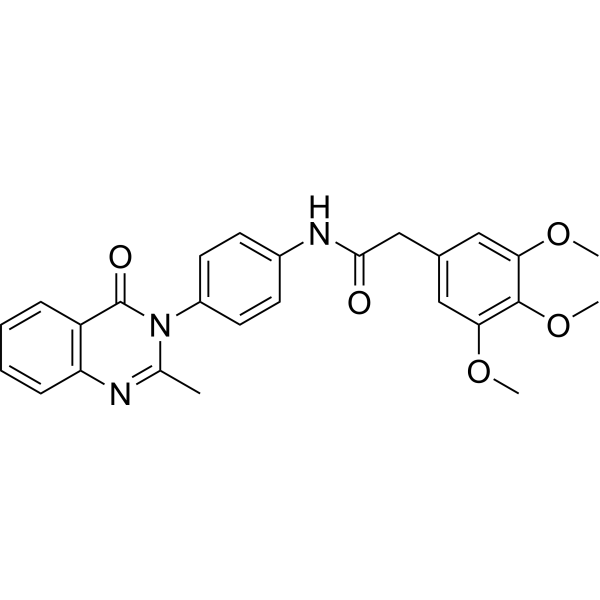
GC26263
icFSP1
icFSP1 is a potent ferroptosis suppressor protein-1 (FSP1) inhibitor.
-
GC26262
PEG800
PEG800 is a strongly hydrophilic polyethylene glycol used as an excellent solvent for a large number of substances.

-
GC26261
ODN TTAGGG
ODN TTAGGG (A151), an inhibitory oligonucleotide (ODN), is an antagonist of TLR9, AIM2, and cGAS. ODN TTAGGG has immunosuppressive effects and can inhibit AIM2 inflammasome activation and cGAS activation through competition with DNA. ODN TTAGGG can be used for research on autoimmune diseases such as lupus erythematosus. ODN TTAGGG sequence: 5 '- T-T-A-G-G-T-T-A-G-G-G-T-A-G-G-T-A-G-G-G-G-3'.

-
GC26260
Cal-590 Potassium Salt
Calcium ion measurement plays an important role in various physiological activities. Fluorescent probes that bind with calcium ions and exhibit spectral reactions allow researchers to observe changes in intracellular free calcium ion levels, which can be detected through fluorescence microscopy, flow cytometry, fluorescence spectrophotometer, and fluorescence enzyme-linked immunosorbent assay (ELISA) plates.

-
GC26259
Cal-590 AM,Cell Permeant
Calcium ion measurement plays an important role in various physiological activities. Fluorescent probes that bind with calcium ions and exhibit a light channel reaction allow researchers to observe changes in intracellular free calcium ion levels, which can be detected through English light microscopy, flow cytometry, fluorescence spectrophotometer, and fluorescence enzyme assay plates.

-
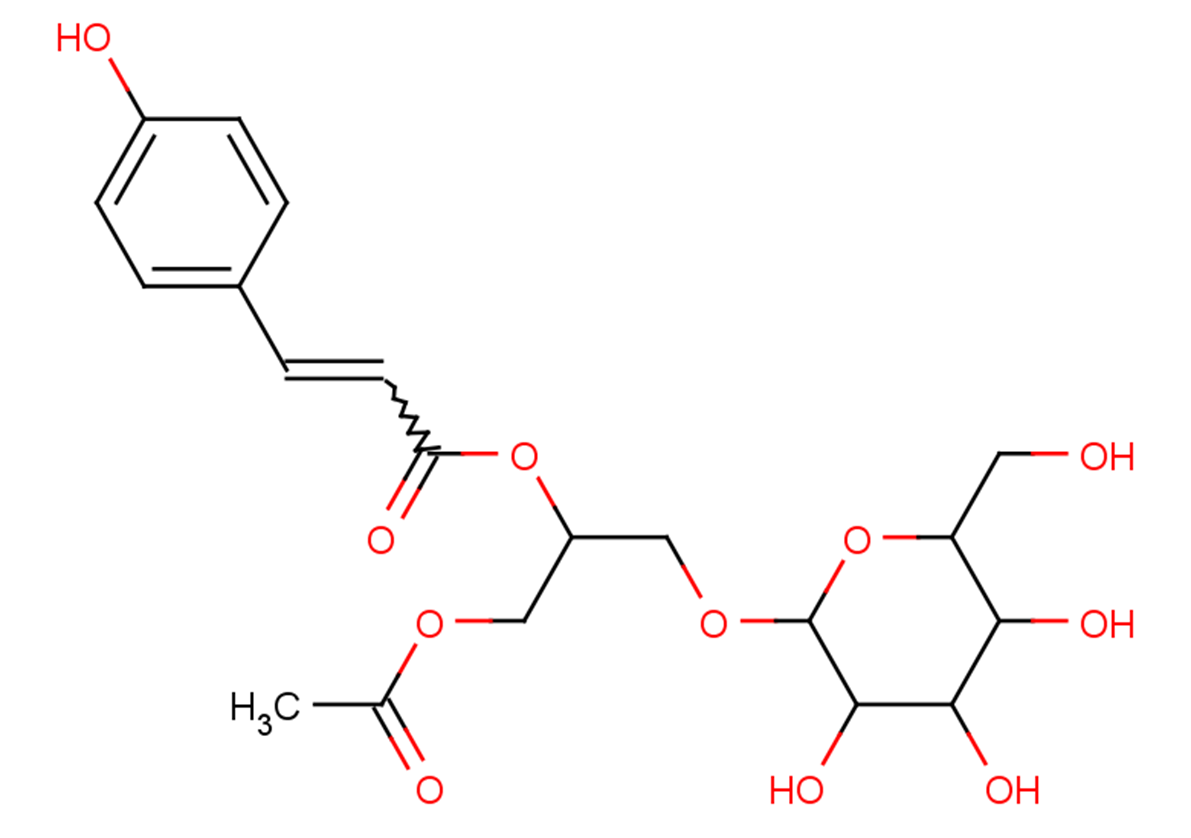
GF60005
regaloside I
Regaloside I is the monomeric component in lily.
-
GF55002
DNA Ladder(1-10kb,8bands)

-
GF55001
DNA Ladder(0.1-10kb, 21bands)

-
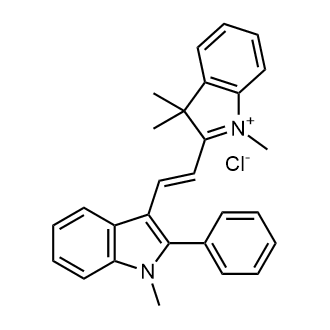
GC26258
Basic Orange 22
Cationic Orange 2GL is mainly used for dyeing acrylic fibers and direct printing of acrylic fabrics. It is a bright orange with a slight red light and stable color light.
-
GC26257
Ammonium bisulfite
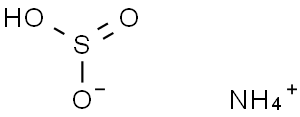
-
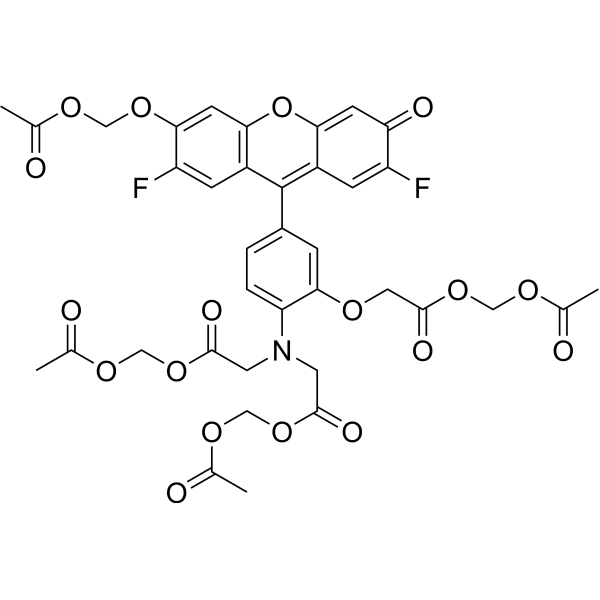
GC26256
Mag-Fluo-4 AM
Mag-Fluo-4 AM is a fluorecent Ca2+ chelator, with high affinity for calcium.
-
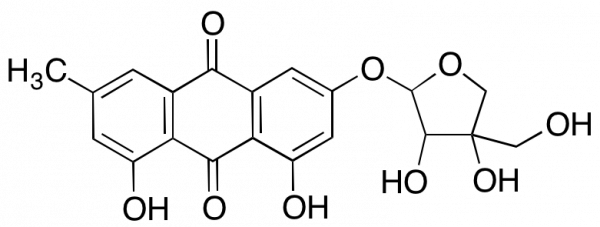
GD74269
Frangulin B
-
GC26255
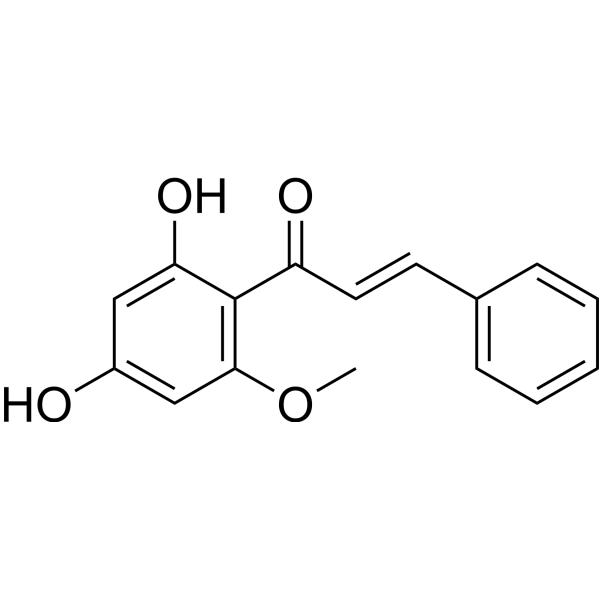
(E)-Cardamonin
Cardamonin can be found from cardamom, and target various signaling molecules, transcriptional factors, cytokines and enzymes.
-
GC26253
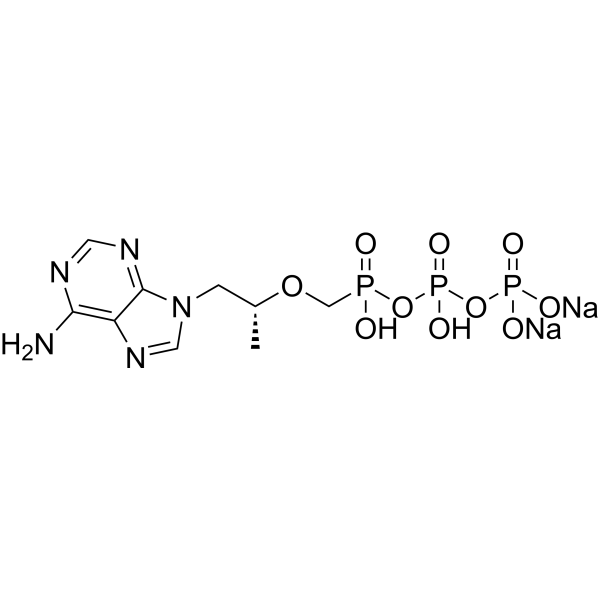
Tenofovir diphosphate disodium
Tenofovir diphosphate disodium is an antiretroviral agent and an inhibitor of DNA polymerases.
-
GC26252
Chitosan quaternary ammonium salt (degree of substitution: 92%)

-
GC26251
Chitosan quaternary ammonium salt (degree of substitution: 95%)

-
GC26250
Chitosan quaternary ammonium salt (degree of substitution: 98%)

-
GC26249
(Z)-Hexadec-11-en-1-yl acetate

-
GC26248
FITC-Dextran (MW 2000000)
FITC Extran (MW 2000000) is a compound belonging to the fluorescent dye class.

-
GC26247
Helenien
-
GC26246
SYTOX Green Nucleic Acid Stain (5 mM in DMSO)
SYTOX Green Nucleic Acid Stain is a high affinity nucleic acid dye that easily penetrates into damaged cell membranes, but cannot penetrate live cell membranes.

-
GK10026
Coomassie Blue Fast Staining Solution
Coomassie Blue Fast Staining Solution can be used for polyacrylamide protein gel without pollution, fast, and high-sensitive staining. The minimum protein detection is 10 ng.

-
GC26245
Sodium Citrate Buffer
0.1M,pH4.0

-
GC26244
(S)-2-Amino-3-(4-((fluorosulfonyl)oxy)phenyl)propanoic acid hydrochloride

-
GC26243
ITS Cell Culture Additives (100 ×)
ITS (Insulin, Transferrin, Selenium) cell culture additives (100 ×), Including the three most commonly used additives in cell culture, Insulin Insulin, Human Transferrin Human Transferrin, and Selenous Acid Selenite

-
GK10006
Phos-tag Biotin BTL-104
Phos-tag Biotin BTL-104 is a monobiotinylated Phos-tag derivative containing ZnCl2 that can be used to detect phosphopeptides and purified phosphoproteins. Phos-tag is a functional molecule that can specifically bind to phosphate ions.

-
GC26242
Alexa Fluor 594 labeled Concanavalin A
Concanavalin ACon A, derived from Canavalia ensiformis beans, is a lectin protein (Mw 104 kDa).

-
GC26241
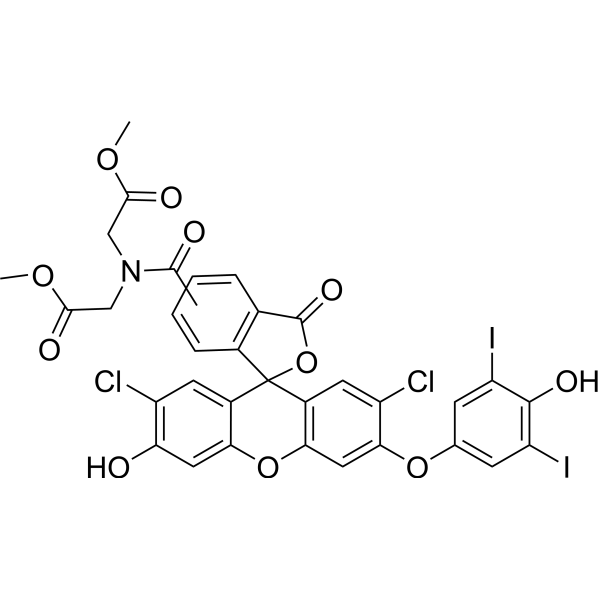
HKOH-1r
HKOH-1r is a living cell reactive oxygen species fluorescent probe with a maximum absorption/emission wavelength of 500/520nm.
-
GC26240
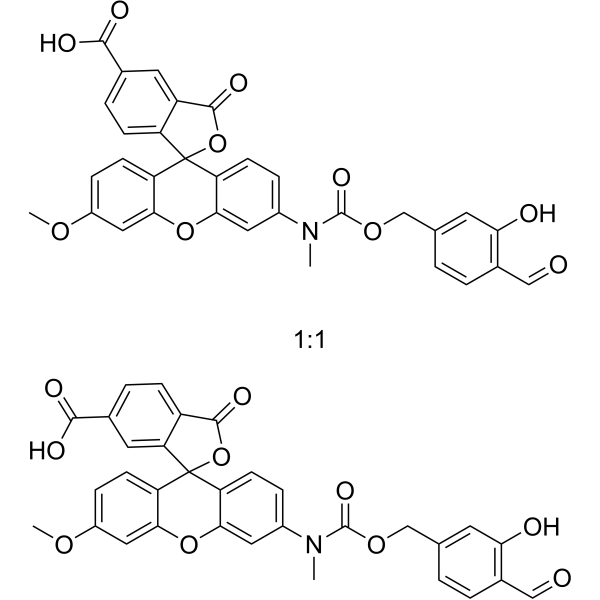
HKPerox-2
HKPerox-2 is a live cell green fluorescent probe that specifically binds to intracellular hydroxyl radicals with a maximum absorption/emission wavelength of 520/543nm.
-
GC26239
Sodium carboxymethyl cellulose (Viscosity:800-1200 mPa.s)
Sodium carboxymethyl cellulose (Viscosity: 800-1200 mPa. s) is a sodium salt of carboxymethyl cellulose, commonly used as a thickener, paste, and barrier.

-
GC26238
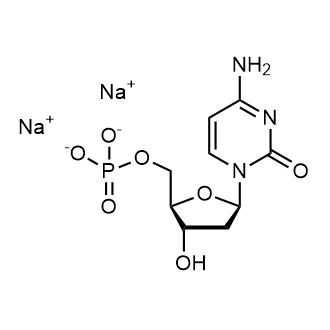
2′-Deoxycytidine 5′-monophosphate sodium salt
-
GC91477
740 Y-P (trifluoroacetate salt)
740 Y-P es un activador sintético de fosfopeptido de PI3K.

-
GC91473
A11 (trifluoroacetate salt)
A11 es un péptido que penetra en la célula compuesto por el dominio de transducción de proteína Tat del VIH-1 unido a un péptido de 11 aminoácidos correspondiente a los residuos 20-30 del extremo N-terminal de la anexina A1.
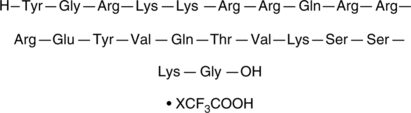
-
GC91468
A20FMDV2 (trifluoroacetate salt)
A20FMDV2 es un péptido que comprende el dominio celular interactuante RGD α-helicoidal en la proteína de cápside VP1 del virus de la fiebre aftosa (FMDV) serotipo O y se une al integrina αVβ6.
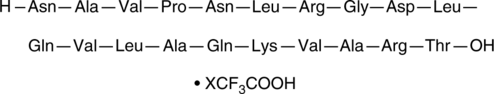
-
GC91474
Azurin (50-77) (P. aeruginosa) (trifluoroacetate salt)
Azurina (50-77) es un fragmento de péptido de la proteína bacteriana que contiene cobre, azurina, que se encuentra en P. aeruginosa y tiene actividades de detención del ciclo celular, proliferación del cáncer y regulación de la angiogénesis.

-
GC91480
Exendin-4 (3-39) amide (trifluoroacetate salt)
Exendin-4 (3-39) amida es un agonista del receptor de péptido similar al glucagón 1 (GLP-1R) y un metabolito activo de exendin-4 (48-86) amida.

-
GC91466
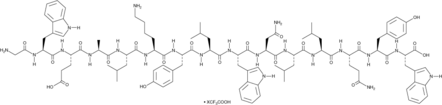
F9170 (trifluoroacetate salt)
F9170 es un péptido antiviral correspondiente a los aminoácidos 789-803 de la glicoproteína de envoltura del VIH-1.
-
GC91470
Fyn Peptide (410-430) (human, mouse, rat, porcine, bovine) (trifluoroacetate salt)
El péptido Fyn (410-430) es un fragmento de péptido de la quinasa tirosina no receptora de la familia Src, llamada Fyn, que contiene Tyr420, el cual se somete a autofosforilación al activarse la forma completa de Fyn.

-
GC91469
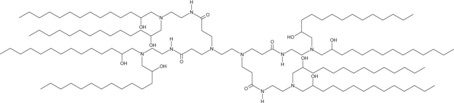
G0-C14 analog
El análogo G0-C14 es un derivado del lípido catiónico ionizable G0-C14.
-
GC91471
Interleukin-8 (54-72) (human) (trifluoroacetate salt)
La interleucina-8 (IL-8) (54-72) es un péptido que corresponde a los aminoácidos 54-72 de la secuencia madura humana de IL-8 y 81-99 de la secuencia preprocesada.

-
GC91481
Myr5A Peptide
El péptido Myr5A es un péptido acilado compuesto por el péptido mimético de la apolipoproteína A1 (ApoA1) 5A conjugado al ácido graso saturado ácido mirístico.

-
GC91475
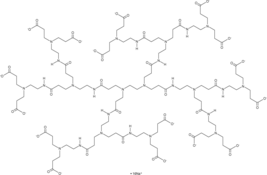
PAMAM Dendrimer G1.5 Carboxylate (sodium salt) (water solution)
El dendrímero PAMAM G1.5 carboxilato es un dendrímero de poliamidoamina (PAMAM) con terminaciones carboxilato.
-
GC91478
PAMAM Dendrimer G2.0 Amidoethanol (water solution)
El dendrímero PAMAM G2.0 amidoetanol (PAMAM G2.0 amidoetanol) es un dendrímero de poliamidoamina (PAMAM) con terminaciones hidroxilo que se ha utilizado como sistema de administración de fármacos in vitro.

-
GC91476
PAMAM Dendrimer G2.0 Amine (water solution)
El dendrímero PAMAM G2.0 amina es un dendrímero de poliamidoamina (PAMAM) con terminaciones de amina.
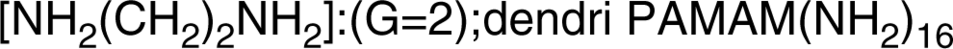
-
GC91467
Ser159]-PKCε (149-164) (trifluoroacetate salt)
El péptido sustrato Ser159]-PKCε (149-164) es un sustrato de péptido para PKCγ, PKCδ, PKCε, PKCΗ y PKCΞ, quinasas serina/treonina involucradas en varios procesos celulares como la proliferación, diferenciación y supervivencia.
![Ser159]-PKCε (149-164) (trifluoroacetate salt) Chemical Structure Ser159]-PKCε (149-164) (trifluoroacetate salt) Chemical Structure](/media/struct/GC9/GC91467.png)
-
GC91479
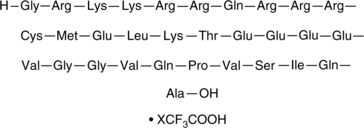
Tat-ASIC1a (1-20) (mouse, rat) (trifluoroacetate salt)
Tat-ASIC1a (1-20) es un péptido sintético compuesto por la secuencia de péptido que penetra en la célula del dominio de transducción de proteína Tat del VIH-1, unida a un péptido de 20 aminoácidos correspondiente a los aminoácidos 1-20 del canal iónico sensor ácido 1a (ASIC1a).
-
GC91472
Tat-Gap 19 (trifluoroacetate salt)
Tat-Gap 19 es un inhibidor de péptido de los hemicanales de conexina43 (Cx43).
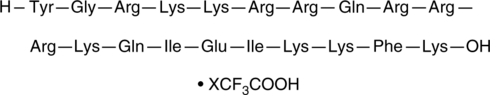
-
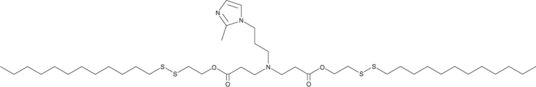
GC91454
9322-O16B
9322-O16B es un lípido catiónico ionizable.
-
GC91462
Abl Substrate Peptide (trifluoroacetate salt)
El péptido sustrato Abl es un sustrato de péptido para la tirosina quinasa Abl.
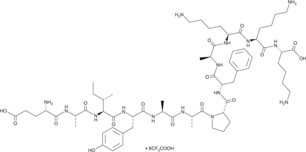
-
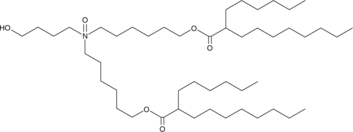
GC91453
ALC-0315 N-oxide
El N-óxido de ALC-0315 es una posible impureza en las preparaciones comerciales del lípido amino catiónico ionizable ALC-0315.
-
GC91442
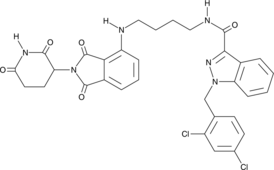
C-02
C-02 es una quimera dirigida a la proteólisis (PROTAC) compuesta por el inhibidor de hexoquinasa lonidamina unido al ligando cereblón talidomida.
-
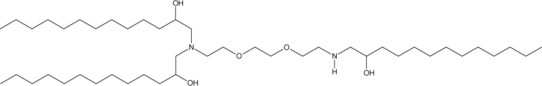
GC91452
C13-112-tri-tail
C13-112-tri-trail es un lípido catiónico ionizable.
-
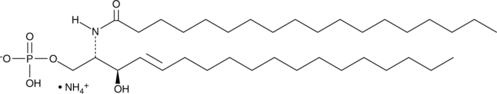
GC91445
C18 Ceramide-1-phosphate (d18:1/18:0) (ammonium salt)
C18 Ceramida-1-fosfato (d18:1/18:0) es una ceramida-1-fosfato de cadena larga que se ha encontrado en la piel de ratones.
-
GC91460
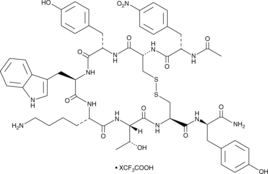
CYN 154806 (trifluoroacetate salt)
CYN 154806 es un antagonista cíclico de péptido del receptor de somatostatina 2 (SST2) (IC50 = 2.62 nM).
-
GC91450
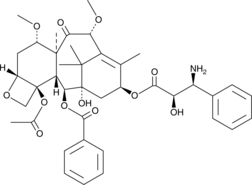
De-Boc-Cabazitaxel
De-Boc-Cabazitaxel es una posible impureza en las preparaciones comerciales del taxano semisintético cabazitaxel.
-
GC91444
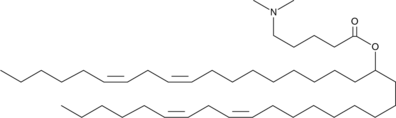
DLin-MC4-DMA
DLin-MC4-DMA es un lípido catiónico ionizable (pKa = 6.93).
-
GC91465
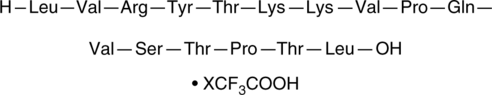
EPI-X4 (trifluoroacetate salt)
EPI-X4 es un fragmento de péptido endógeno que corresponde a los aminoácidos 408-423 de la albúmina sérica humana y es un antagonista del receptor 4 de quimiocina (motivo C-X-C) (CXCR4).
-
GC91448
Fumonisin B4
La fumonisina B4 (FB4) es una micotoxina que se ha encontrado en F. verticillioides.
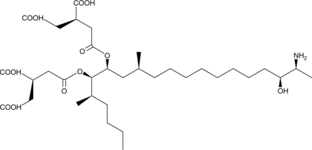
-
GC91464
Fz7-21 (trifluoroacetate salt)
Fz7-21 es un antagonista peptídico del receptor acoplado a proteína G de la familia frizzled 7 (FZD7; IC50 = 100 nM).
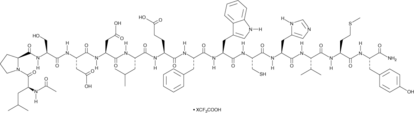
-
GC91447
IAJD249
IAJD249 es un sistema de entrega de ARNm con dendrímeros Janus anfifílicos ionizables (IAJD) (pKa = 6,35).
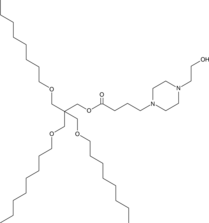
-
GC91446
IAJD93
IAJD93 es un sistema de entrega de ARNm con dendrímeros Janus anfifílicos ionizables (IAJD) (pKa = 6,25).
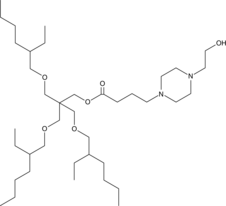
-
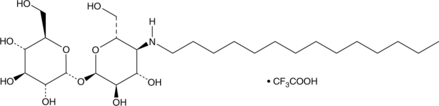
GC91443
IMCTA-C14 (trifluoroacetate salt)
IMCTA-C14 es un detergente y derivado del metabolito microbiano 4-trehalosamina.
-
GC91449
Ipamorelin (acetate)
Ipamorelin es un secretagogo de la hormona del crecimiento pentapéptido (GHS) y agonista del receptor GHS 1a (GHS-R1a).
-
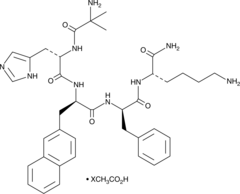
GC91459
LIH383 (trifluoroacetate salt)
LIH383 es un agonista peptídico del receptor 7 de quimiocina (motivo C-X-C) (CXCR7), un receptor scavenger de quimiocinas y péptidos opioides.
-
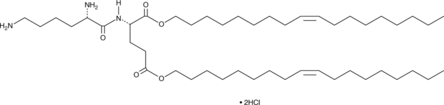
GC91455
Lipid OA2 (hydrochloride)
El lípido OA2 es un lípido catiónico ionizable que se ha utilizado en la generación de nanopartículas lipídicas (LNPs) de un solo componente para la entrega de siRNA.
-
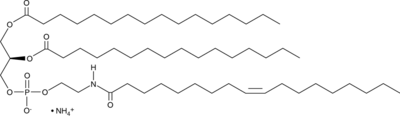
GC91457
N-Oleoyl-DPPE (ammonium salt)
N-Oleoyl-DPPE es una N-acil-fosfatidiletanolamina (NAPE).
-
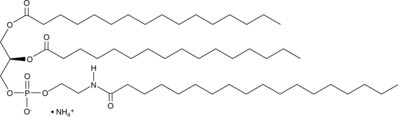
GC91458
N-Stearoyl-DPPE (ammonium salt)
N-Stearoyl-DPPE es un N-acil-fosfatidiletanolamina (NAPE).
-
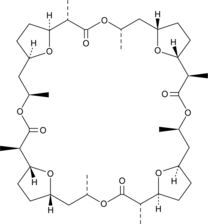
GC91451
Nonactin, Monactin, and Dinactin Mixture
La mezcla de nonactina, monactina y dinactina es un antibiótico macrotetralídico.
-
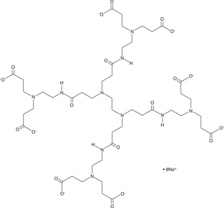
GC91461
PAMAM Dendrimer G0.5 Carboxylate (sodium salt) (water solution)
El dendrímero PAMAM G0.5 carboxilato es un dendrímero de poliamidoamina (PAMAM) con terminaciones de carboxilato.
-
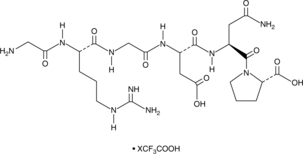
GC91441
RGD Peptide (trifluoroacetate salt)
El péptido RGD es un compuesto sintético compuesto por el motivo arginina-glicina-aspartato que se ha utilizado ampliamente para inhibir las interacciones integrina-ligando en estudios relacionados con la adhesión celular, migración, crecimiento y diferenciación.
-
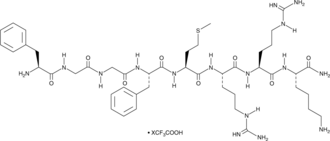
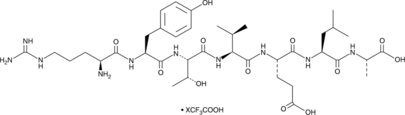
GC91456
RYTVELA (trifluoroacetate salt)
RYTVELA es un derivado del antagonista peptídico del receptor 1 de interleucina-1 (IL-1R1) rytvela que contiene todos los aminoácidos L.
-
GC91463
SAP15 (trifluoroacetate salt)
SAP15 es un péptido que penetra en las células y está compuesto por los aminoácidos C-terminales 52-67 de la β-defensina-3 humana.
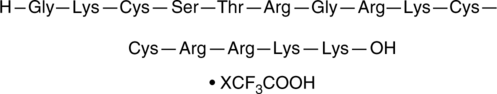
-
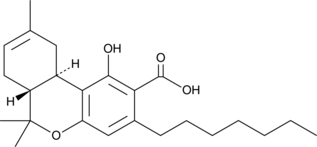
GC91400
δ8-THCPA-A
δ8-THCPA-A es un estándar de referencia analítico que es estructuralmente similar a los fitocannabinoides conocidos.
-
GC91386
(±)-Cannabichromebutolic Acid
El ácido cannabichromebutólico (±) es un estándar de referencia analítico que es estructuralmente similar a los fitocannabinoides conocidos.
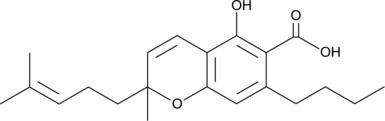
-
GC91399
(±)-Cannabichromephorolic Acid
El ácido cannabichromephorolic (±) es un estándar de referencia analítico que es estructuralmente similar a los fitocannabinoides conocidos.
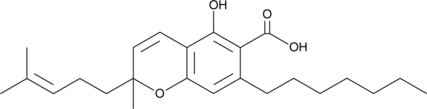
-
GC91346
(+)-2-Fluorodeoxyarbutin
(+)-2-Fluorodeoxyarbutin es un derivado fluorado del inhibidor de la tirosinasa deoxiarbutina.
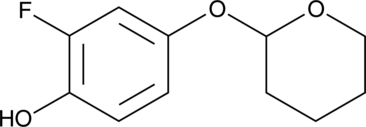
-
GC91418
(E)-KPT-330
(E)-KPT-330 es un metabolito del inhibidor de exportina 1 (XPO1/CRM1) selinexor.
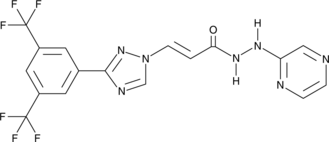
-
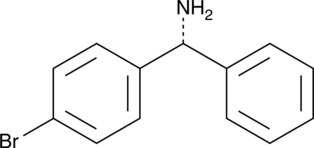
GC91359
(R)-(4-Bromophenyl)(phenyl)methanamine
(R)-(4-Bromofenil)(fenil)metanamina es un intermediario sintético.
-
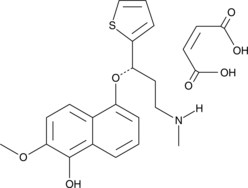
GC91425
(S)-5-hydroxy-6-methoxy Duloxetine (maleate)
El (S)-5-hidroxi-6-metoxi duloxetina es un metabolito activo del inhibidor de la recaptación de serotonina (5-HT) y norepinefrina (S)-duloxetina.
-
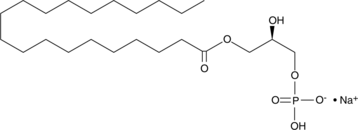
GC91429
1-Arachidoyl Lysophosphatidic Acid (sodium salt)
1-El ácido lisofosfatídico araquidilo (1-arachidoyl LPA) es un agonista del receptor de ácido lisofosfatídico 1 (LPA1) y un glicerofosfolípido que contiene ácido araquídico en la posición sn-1.
-
GC91394
1-Myristyl-2-hydroxy-sn-glycero-3-PA
1-Miristil-2-hidroxi-sn-glicero-3-PA (1-miristil LPA) es un derivado de LPA que contiene ácido mirístico en la posición sn-1 y es agonista del receptor 1 de ácido lisofosfatídico (LPA1) y LPA2.

-
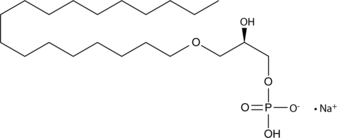
GC91421
1-Oleyl-2-hydroxy-sn-glycero-3-PA (sodium salt)
1-Oleil-2-hidroxi-sn-glicero-3-PA (1-Octadecyl LPA) es un análogo de LPA que contiene alcohol estearílico en la posición sn-1.
-
GC91413
1-Palmityl-2-hydroxy-sn-glycero-3-PA (sodium salt)
1-Palmityl-2-hidroxi-sn-glicero-3-PA (sal de sodio) es un análogo éter de ácido lisofosfatídico (LPA) que contiene un grupo hexadecilo en la posición sn-1.

-
GC91341
1-Phenylethylamine (hydrochloride)
La feniletilamina es un estándar de referencia analítico categorizado como una fenetilamina.
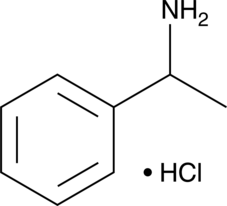
-
GC91374
12-oxo Stearic Acid
El ácido 12-oxo esteárico es un ácido graso oxo saturado (SOFA) y metabolito del ácido ricinoleico que se ha encontrado en la leche de vaca y cabra.
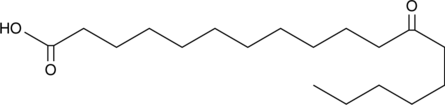
-
GC91402
15-deoxy-δ12,14-Prostaglandin J2-2-glyceryl ester
El éster de glicerilo 15-deoxi-δ12,14-prostaglandina J2-2 (éster de glicerilo 15-deoxi-δ12,14-PGJ2-2) se forma a partir de PGD2 mediante la eliminación de dos moléculas de agua.
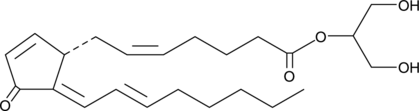
-
GC91367
3-(4-Chloro-2-methylphenyl)-2-methyl-4(3H)-quinazolinone
La 3-(4-cloro-2-metilfenil)-2-metil-4(3H)-quinazolinona es un estándar de referencia analítico categorizado como una quinazolinona.
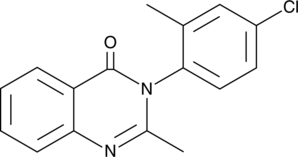
-
GC91351
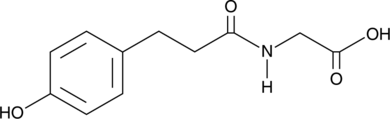
4-Hydroxyphenylpropionylglycine
4-Hidroxifenilpropionilglicina es un metabolito del aminoácido condicionalmente esencial tirosina.
-
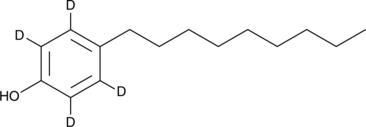
GC91352
4-Nonylphenol-2,3,5,6-d4
El 4-Nonilfenol-2,3,5,6-d4 está destinado a ser utilizado como estándar interno para la cuantificación del 4-nonylphenol por GC o LC-MS.
- GlpBio - Master of Small Molecules | Compounds - Peptides - Kits.
- Entrar | Register
- sales@glpbio.com
- (909) 407-4943
-
LenguajeES - Español
-
Research Areas
-
Signaling Pathways
- Proteases
- Apoptosis
- Chromatin/Epigenetics
- Metabolism
- MAPK Signaling
- Tyrosine Kinase
- DNA Damage/DNA Repair
- PI3K/Akt/mTOR Signaling
- Microbiology & Virology
- Cell Cycle/Checkpoint
- Ubiquitination/ Proteasome
- JAK/STAT Signaling
- TGF-β / Smad Signaling
- Angiogenesis
- GPCR/G protein
- Stem Cell
- Cancer Biology
- Endocrinology and Hormones
- Immunology/Inflammation
- Neuroscience
- Membrane Transporter/Ion Channel
- Other Signal Transduction
- Product Type
-
Signaling Pathways
- Screening Library
- Kits
- Herramientas
- Ordering
- About us
- Contact us
- Blog


