MAPK Signaling

Targets for MAPK Signaling
- ERK(85)
- MEK1/2(74)
- NKCC(5)
- MNK(6)
- PKA(56)
- p38(103)
- Rac(2)
- Raf(76)
- RasGAP (Ras- P21)(1)
- JNK(66)
- cAMP(33)
- Protein Kinase G(2)
- RSK(33)
- Other(759)
- MAPKAPK(1)
- MKK(2)
- KLF(3)
- MAP3K(22)
- MAP4K(21)
- MAPKAPK2 (MK2)(9)
- Mixed Lineage Kinase(9)
Products for MAPK Signaling
- Cat.No. Nombre del producto Información
-
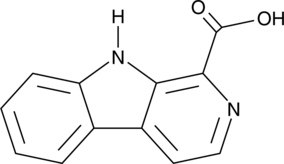
GC48920
β-Carboline-1-carboxylic Acid
An alkaloid with diverse biological activities
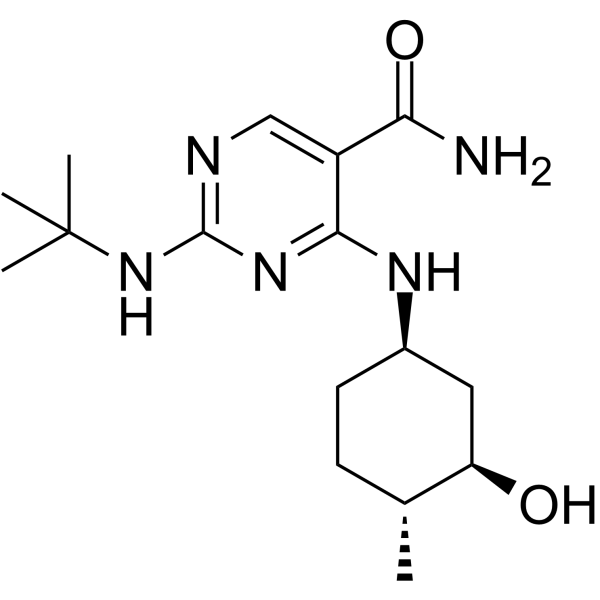
-
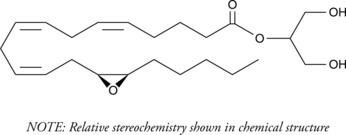
GC41656
(±)2-(14,15-Epoxyeicosatrienoyl) Glycerol
2-Arachidonoyl glycerol (2-AG) is an endogenous central cannabinoid (CB1) receptor agonist that is present at relatively high levels in the central nervous system.
-
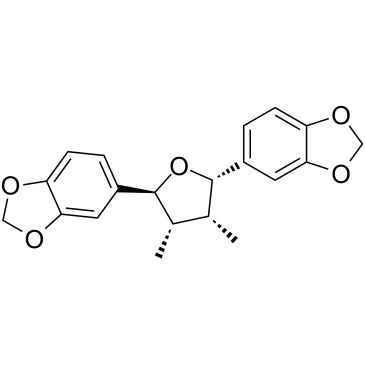
GC60393
(-)-Zuonin A
La (-)-zuonina A (D-epigalbacina), una lignina natural, es un potente inhibidor selectivo de JNK, con IC50 de 1,7 μM, 2,9 μM y 1,74 μM para JNK1, JNK2 y JNK3, respectivamente.
-
GC13944
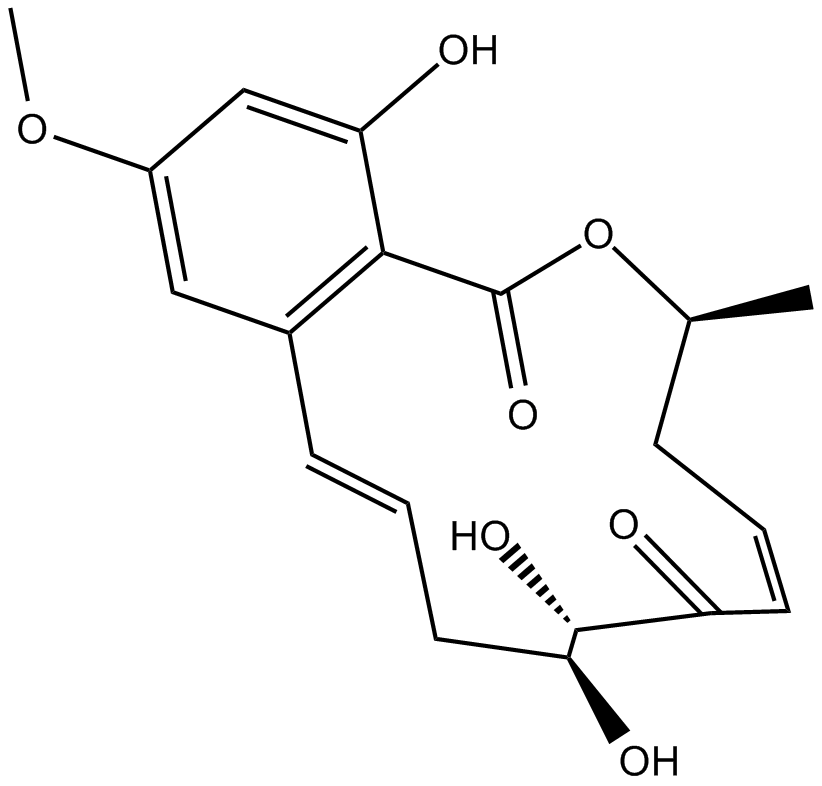
(5Z)-7-Oxozeaenol
TAK1 mitogen-activated protein kinase kinase kinase (MAPKKK) inhibitor
-
GC63903
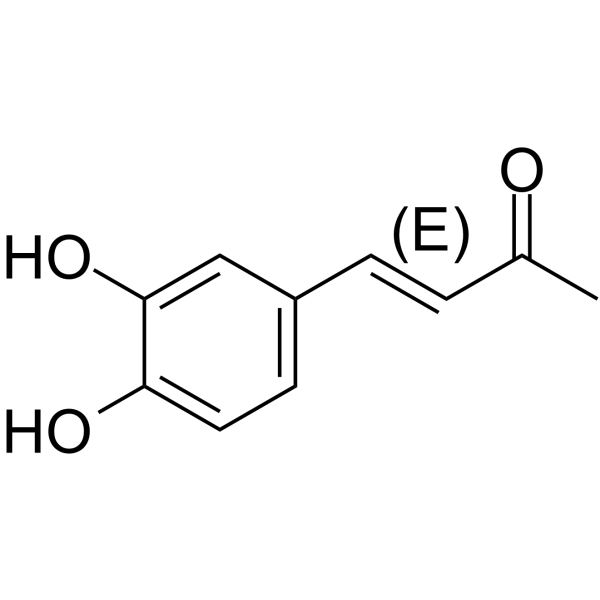
(E)-Osmundacetone
(E)-Osmundacetone es el isÓmero de Osmundacetone.
-
GC62528
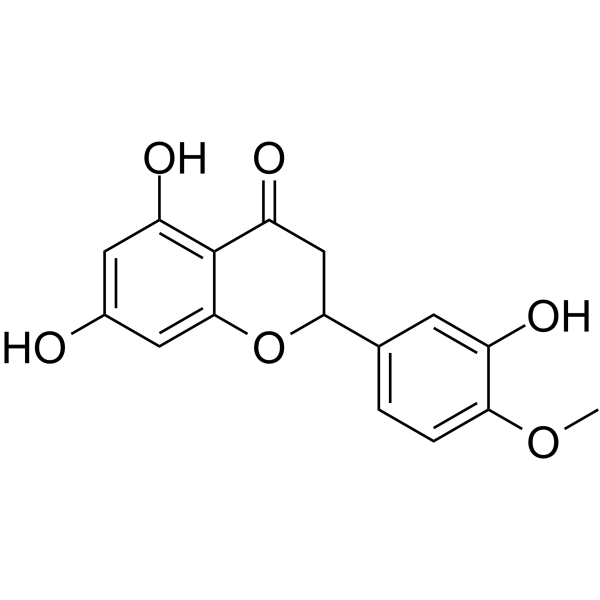
(Rac)-Hesperetin
(Rac)-Hesperetin es el racemato de Hesperetin.
-
GC41740
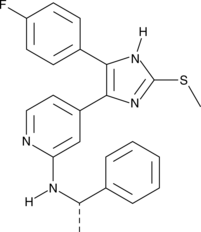
(S)-p38 MAPK Inhibitor III
(S)-p38 MAPK inhibitor III is a methylsulfanylimidazole that inhibits p38 MAP kinase (IC50 = 0.90 μM in vitro).
-
GC12851
10Z-Hymenialdisine
10Z-Hymenialdisine ((Z)-Hymenialdisine) es un alcaloide de pirrol bioactivo natural. La 10Z-himenialdisina es un inhibidor de la pancinasa y tiene actividades anticancerÍgenas.
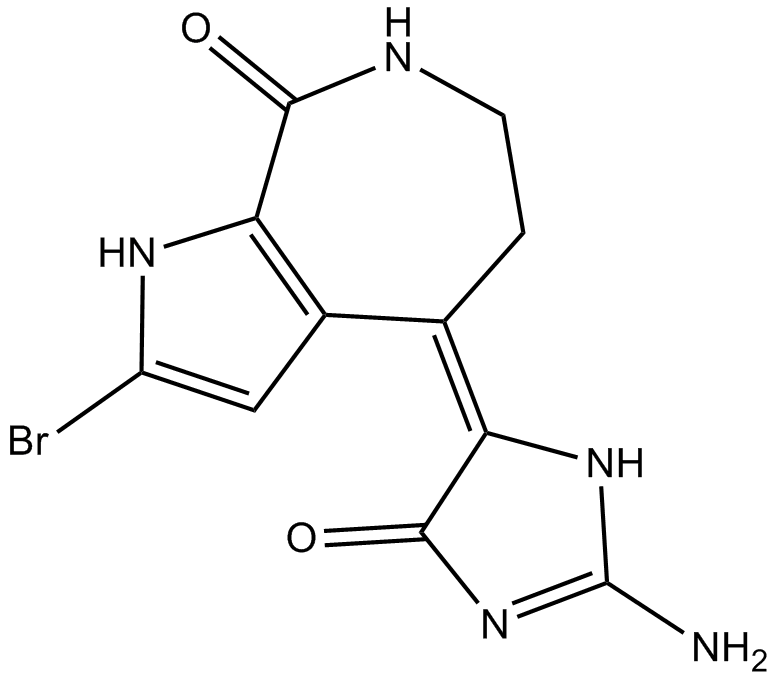
-
GC46554
2'-O-Monosuccinyladenosine-3',5'-cyclic monophosphate
2'-O-Monosuccinyladenosine-3',5'-el monofosfato cÍclico es un anÁlogo de AMPc que puede acoplarse covalentemente a la acetilcolinesterasa.
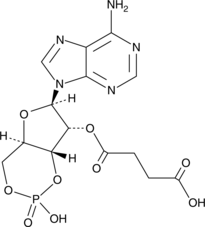
-
GC39325
2,5-Dihydroxyacetophenone
La 2,5-dihidroxiacetofenona, aislada de Rehmanniae Radix Preparata, inhibe la producciÓn de mediadores inflamatorios en los macrÓfagos activados mediante el bloqueo de las vÍas de seÑalizaciÓn ERK1/2 y NF-κB.
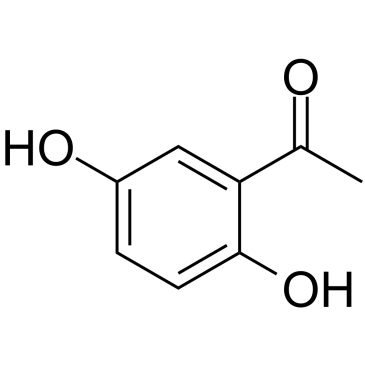
-
GC35132
4-Hydroxylonchocarpin
4-Hydroxylonchocarpin es un compuesto de chalcona de un extracto de Psoralea corylifolia.
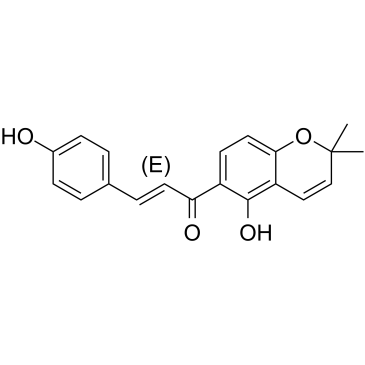
-
GC48381
5'-pApA (sodium salt)
A linearized form of cyclic di-AMP
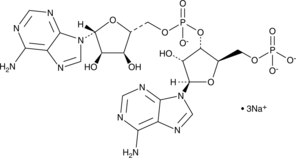
-
GC63700
5,6,7-Trimethoxyflavone
La 5,6,7-trimetoxiflavona es un nuevo inhibidor de p38-α MAPK con efecto antiinflamatorio.
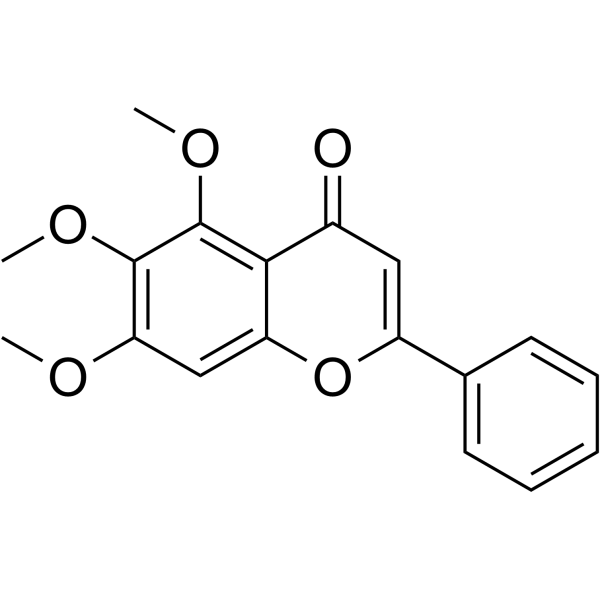
-
GC41423
5-trans Prostaglandin E2
5-trans PGE2 occurs naturally in some gorgonian corals and is a common impurity in commercial lots of PGE1.
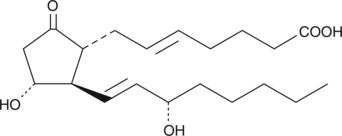
-
GC12834
6-Bnz-cAMP sodium salt
cAMP analog,PKA activator
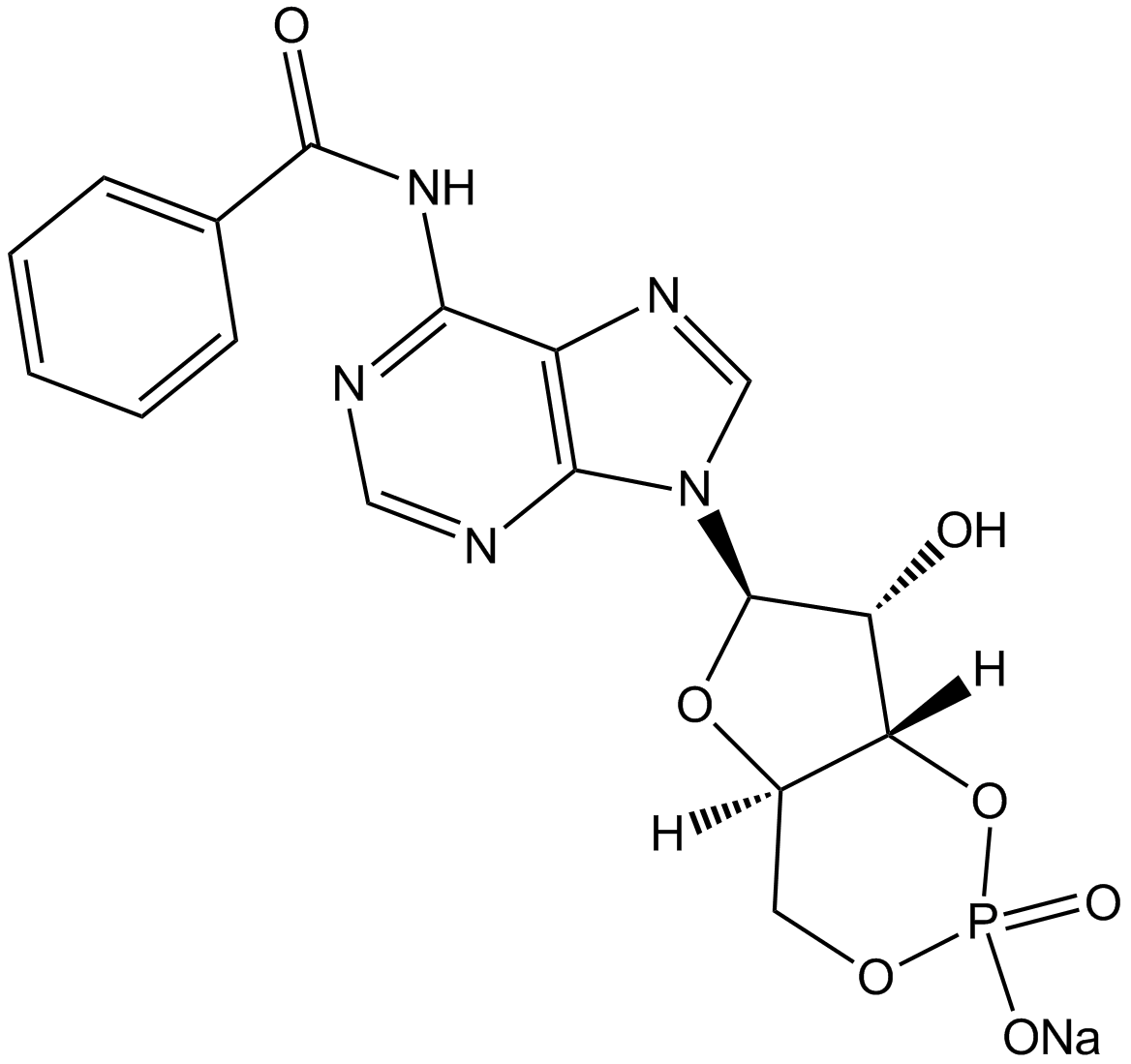
-
GC42616
7-oxo Staurosporine
7-oxo Staurosporine is an antibiotic originally isolated from S.
-
GC16929
8-Bromo-cAMP, sodium salt
Análogo de cAMP permeable a la célula que activa PKA.
-
GC42622
8-bromo-Cyclic AMP
8-bromo-Cyclic AMP is a brominated derivative of cAMP that remains long-acting due to its resistance to degradation by cAMP phosphodiesterase.
-
GC64460
8-Chloro-cAMP
El 8-cloro-cAMP es un anÁlogo de cAMP que induce la detenciÓn del crecimiento y modula la actividad de PKA dependiente de cAMP. El 8-cloro-cAMP tiene actividad anticancerÍgena.
-
GC15352
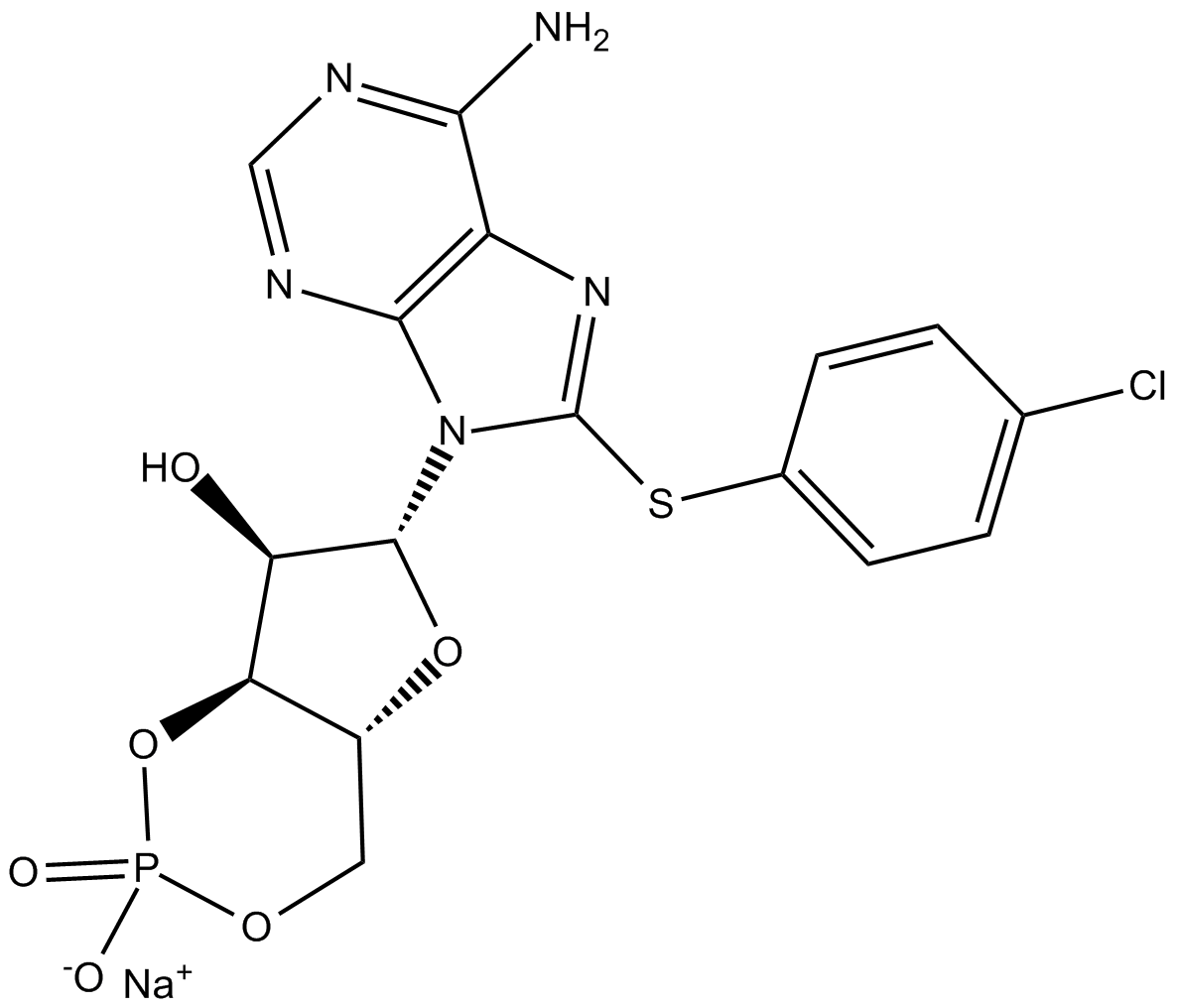
8-CPT-Cyclic AMP (sodium salt)
El 8-CPT-AMP cÍclico (8-CPT-cAMP) de sodio es un activador selectivo de la proteÍna quinasa dependiente de AMP cÍclico (PKA).
-
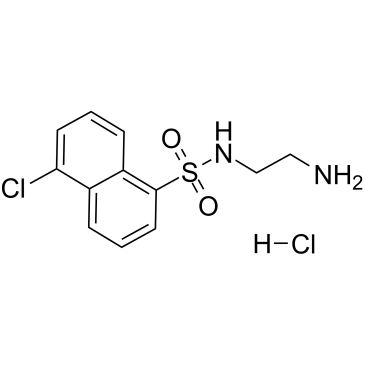
GC60037
A-3 hydrochloride
El clorhidrato de A-3 es un potente antagonista no selectivo de varias quinasas, permeable a las células, reversible y competitivo con ATP.
-
GA20623
Ac-muramyl-Ala-D-Glu-NH₂
Ac-muramyl-Ala-D-Glu-NH₂ (MDP) es un péptido inmunorreactivo sintético que consta de Ácido N-acetilmurÁmico unido a una cadena de aminoÁcidos corta de L-Ala-D-isoGln.

-
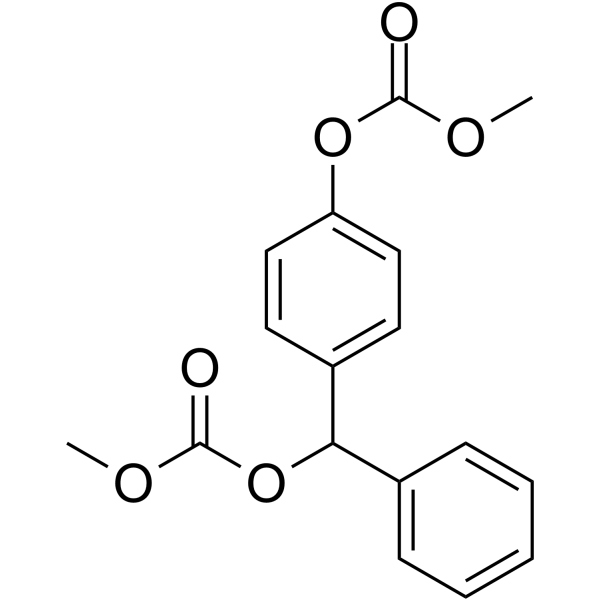
GC68597
ACA-28
ACA-28 (compuesto 2a) is an effective ERK MAPK signal regulator. ACA-28 induces apoptosis through excessive activation of ERK and selectively inhibits cancer cell growth. ACA-28 inhibits the growth of melanoma cells (SK-MEL-28) and normal melanocytes (NHEM), with IC50 values of 5.3 and 10.1 μM, respectively.
-
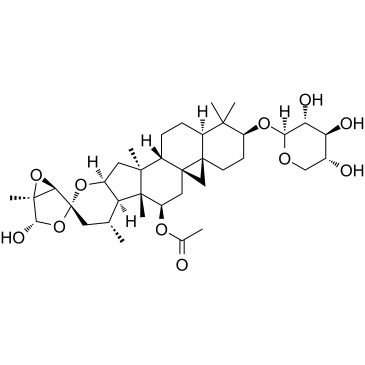
GC35242
Actein
La acteÍna es un glucÓsido triterpénico aislado de los rizomas de Cimicifuga foetida. La acteÍna suprime la proliferaciÓn celular, induce la autofagia y la apoptosis mediante la promociÓn de la activaciÓn de ROS/JNK y el bloqueo de la vÍa AKT en el cÁncer de vejiga humano. La acteÍna tiene poca toxicidad in vivo.
-
GC19019
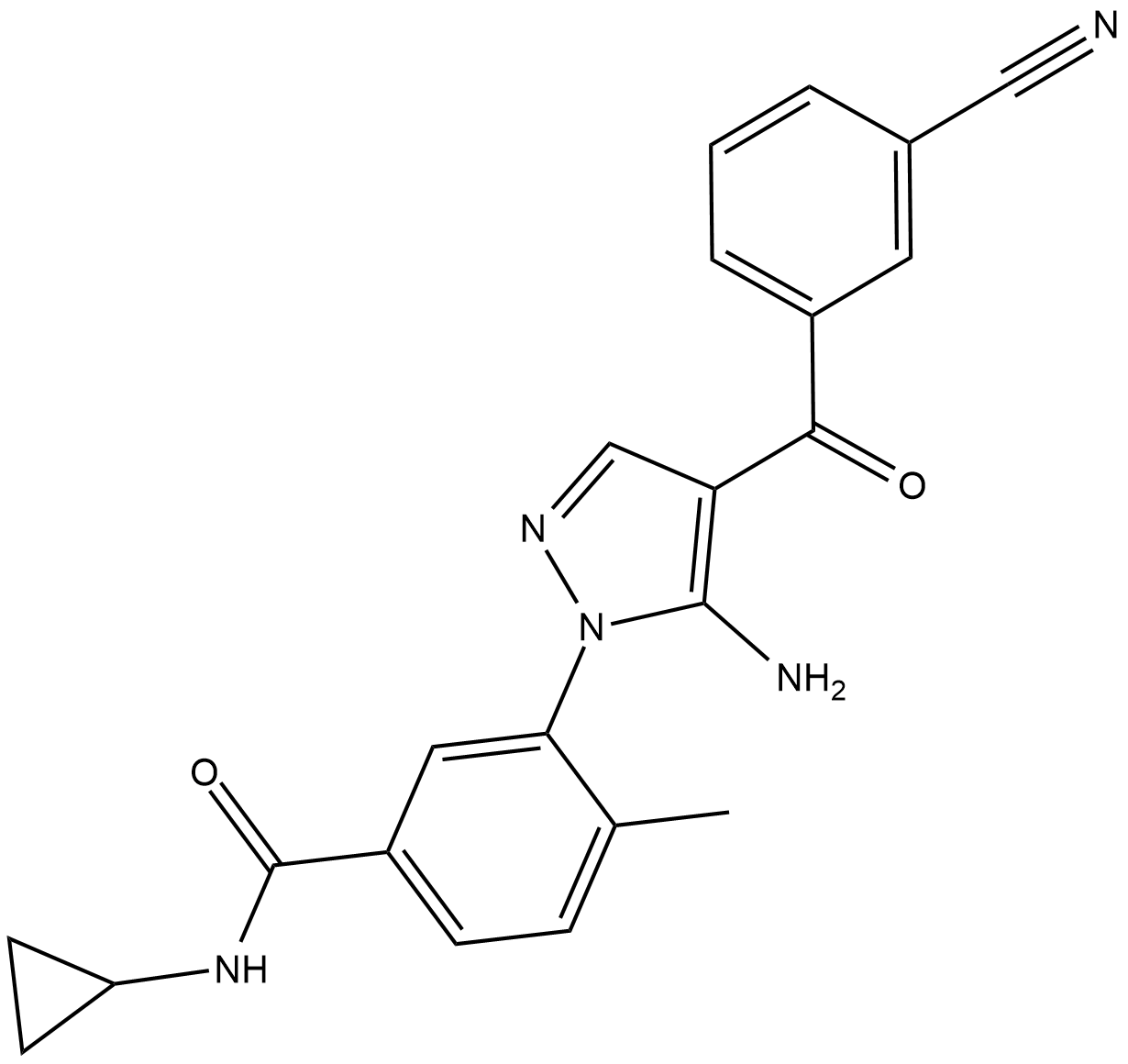
Acumapimod
Acumapimod (BCT197) es un inhibidor de la MAP quinasa p38 activo por vÍa oral, con una IC50 de menos de 1 μM para p38α.
-
GC32797
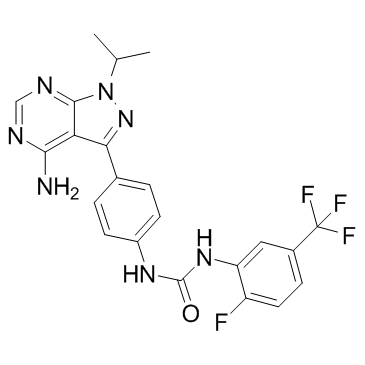
AD80
AD80, un inhibidor multiquinasa, inhibe RET, RAF, SRC y S6K, con una actividad mTOR muy reducida.
-
GC49285
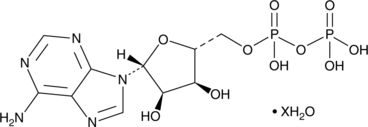
Adenosine 5’-methylenediphosphate (hydrate)
An inhibitor of ecto-5’-nucleotidase
-
GC46808
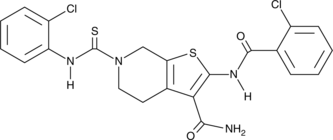
ADTL-EI1712
A dual ERK1 and ERK5 inhibitor
-
GC10204
AEG 3482
AEG 3482 es un potente compuesto antiapoptótico que inhibe la actividad de la quinasa Jun (JNK) a través de la expresión inducida de la proteína de choque térmico 70 (HSP70).
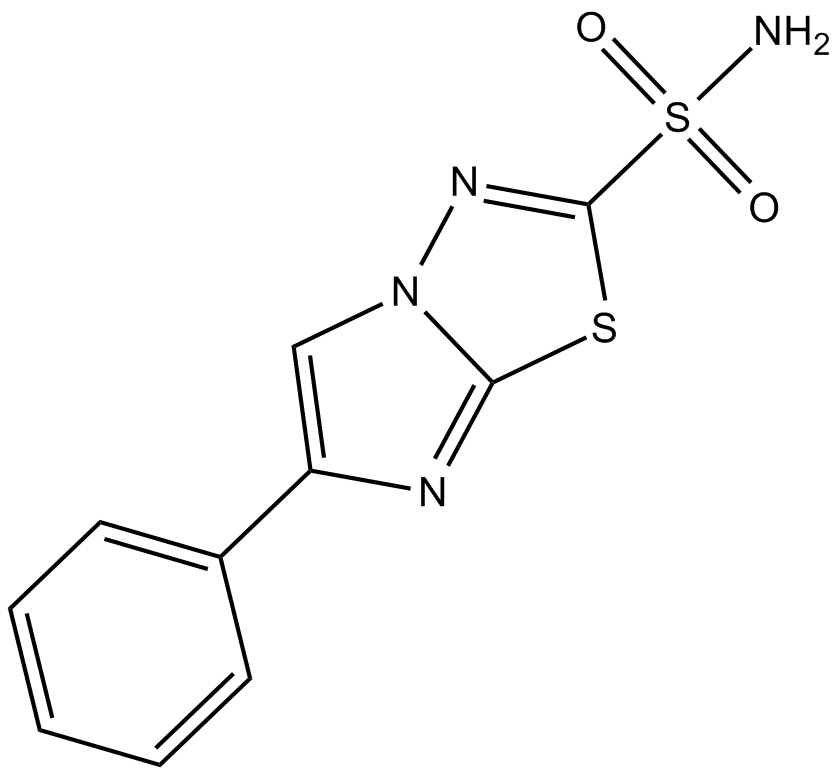
-
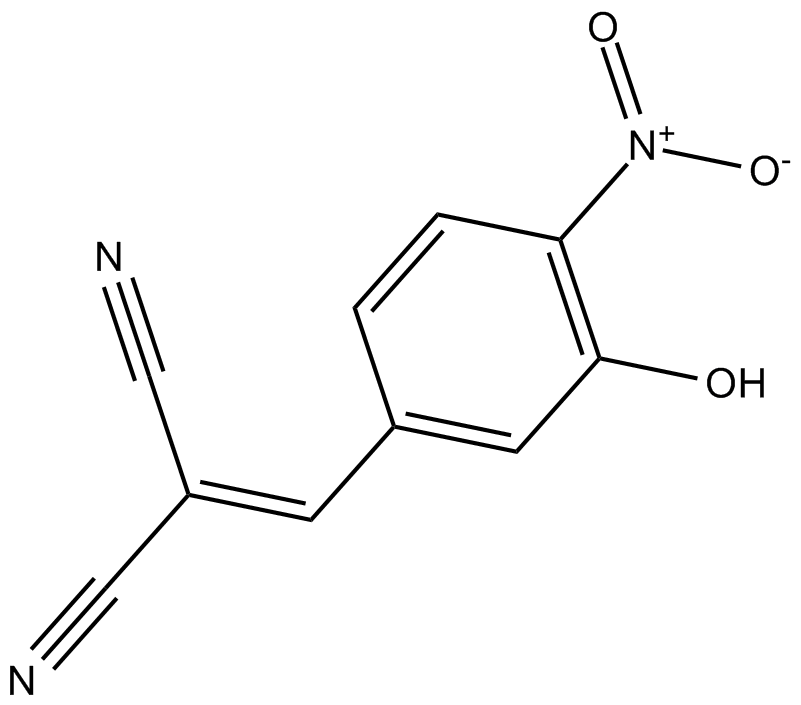
GC17265
AG-126
AG-126 es un inhibidor de la tirosina quinasa, puede inhibir la fosforilación de ERK1 y ERK2 a 25-50 μM.
-
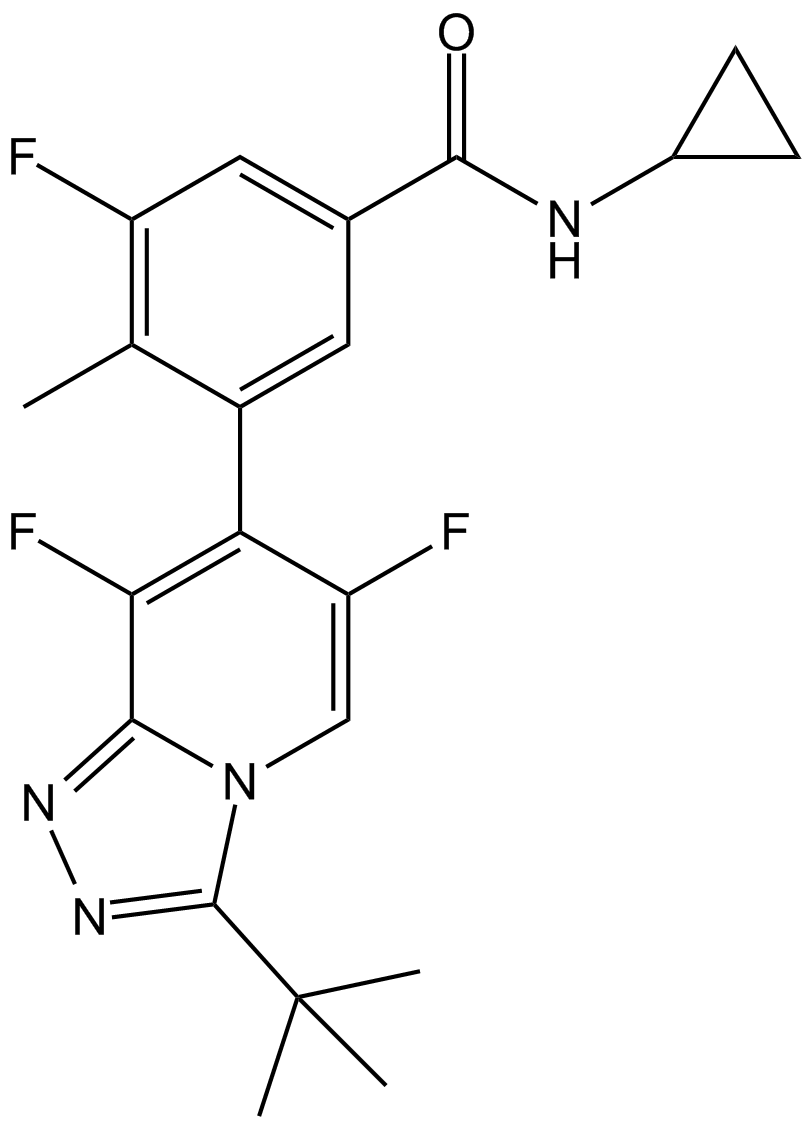
GC12646
AL 8697
AL 8697 es un inhibidor de p38α MAPK especÍfico y activo por vÍa oral con una IC50 de 6 nM.
-
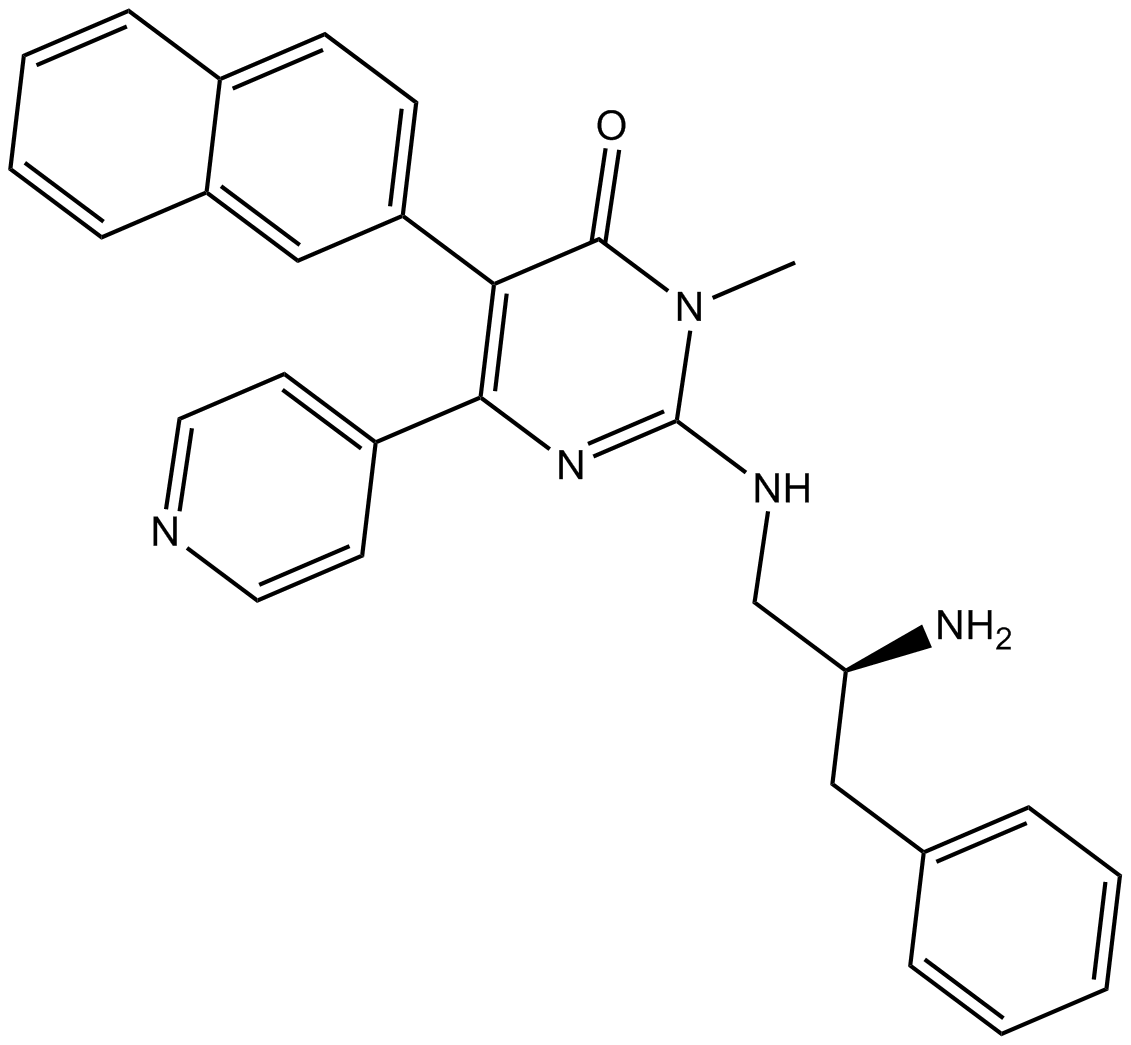
GC14899
AMG 548
AMG 548, un inhibidor de p38α selectivo y activo por vía oral (Ki \u003d 0,5 nM), se muestra ligeramente selectivo sobre p38β (Ki \u003d 36 nM) y \u003e1000 veces selectivo contra p38γ y p38δ.
-
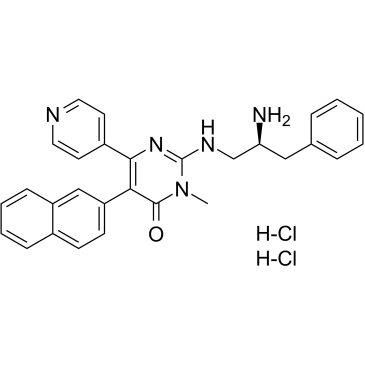
GC38518
AMG-548 dihydrochloride
El diclorhidrato de AMG-548, un inhibidor de p38α selectivo y activo por vÍa oral (Ki = 0,5 nM), se muestra ligeramente selectivo sobre p38β (Ki = 36 nM) y >1000 veces selectivo contra p38γ y p38δ.
-
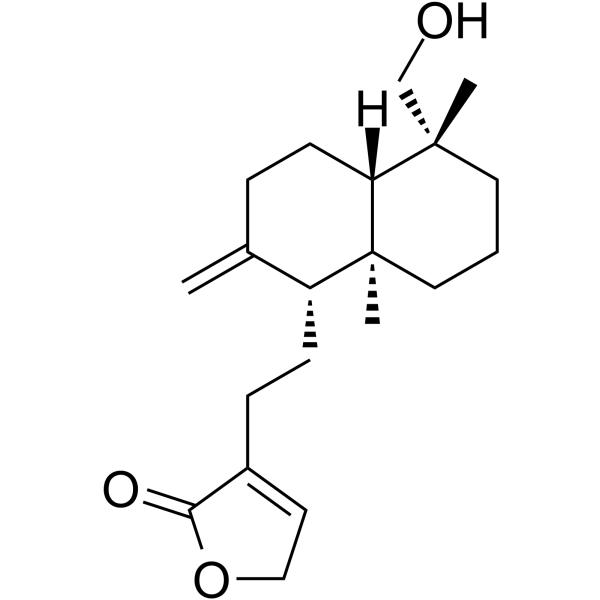
GC65917
Andrograpanin
Andrograpanin, un compuesto bioactivo de Andrographis paniculata, exhibe propiedades antiinflamatorias y antiinfecciosas.
-
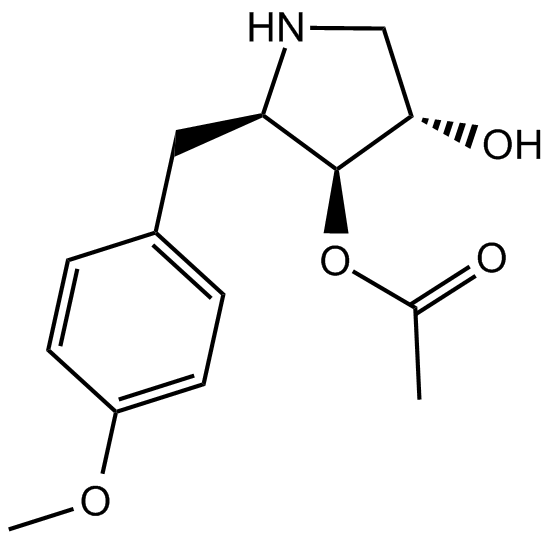
GC11559
Anisomycin
Agonista de JNK, potente y específico.
-
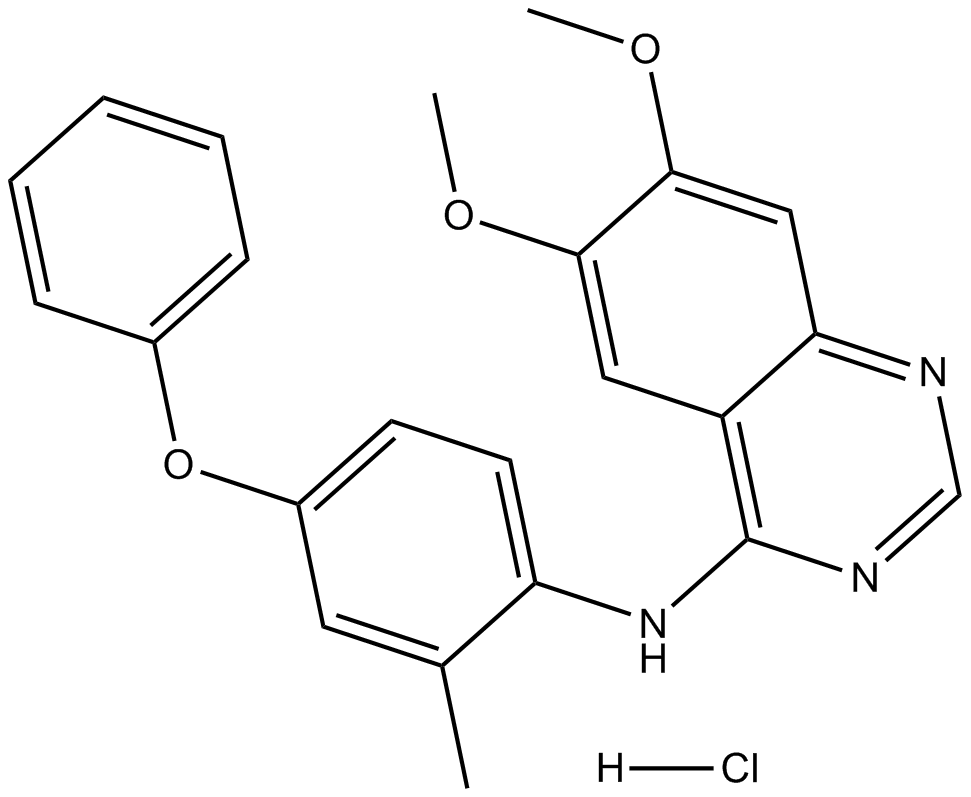
GC15240
APS-2-79
APS-2-79 es un antagonista de MEK dependiente de KSR. APS-2-79 inhibe la uniÓn de ATPbiotina a KSR2 dentro del complejo KSR2-MEK1 con una IC50 de 120 nM. APS-2-79 hace que la estabilizaciÓn del estado inactivo de KSR antagonice la seÑalizaciÓn oncogénica de Ras-MAPK.
-
GC32692
APTO-253 (LOR-253)
APTO-253 es una molécula pequeña novedosa que ejerce una potente actividad antitumoral induciendo la expresión del gen del factor de transcripción maestro Kruppel-like factor 4 (KLF4), inhibiendo así el ciclo celular y conduciendo a la muerte celular programada.
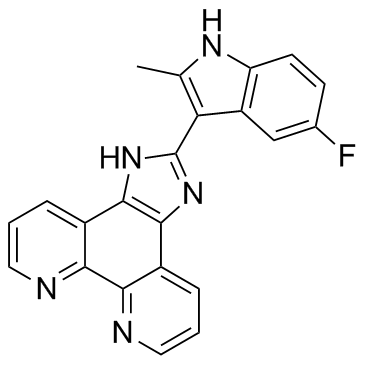
-
GN10063
Arctigenin
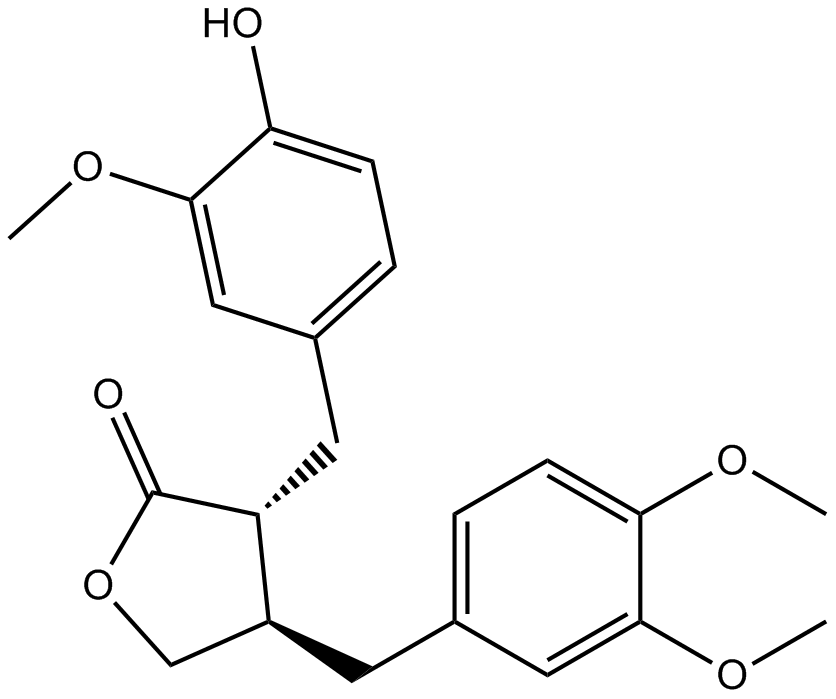
-
GC12882
AS 602801
A selective, orally bioavailable JNK inhibitor
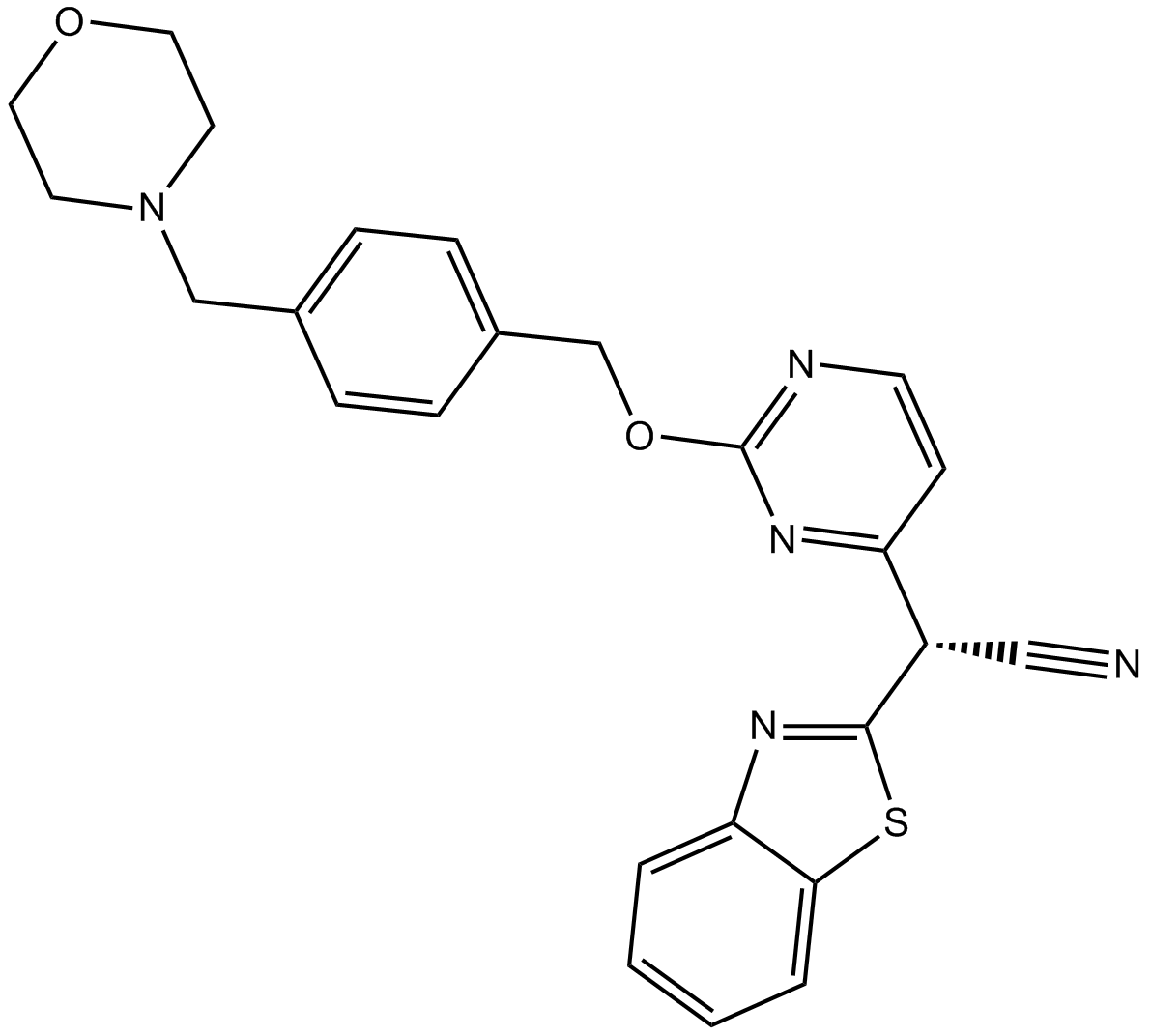
-
GC10010
AS601245
AS601245 es un inhibidor de JNK (proteÍna quinasa c-Jun NH2-terminal) competitivo con ATP, selectivo y activo por vÍa oral con IC50 de 150, 220 y 70 nM para tres isoformas humanas de JNK (hJNK1, hJNK2 y hJNK3), respectivamente.
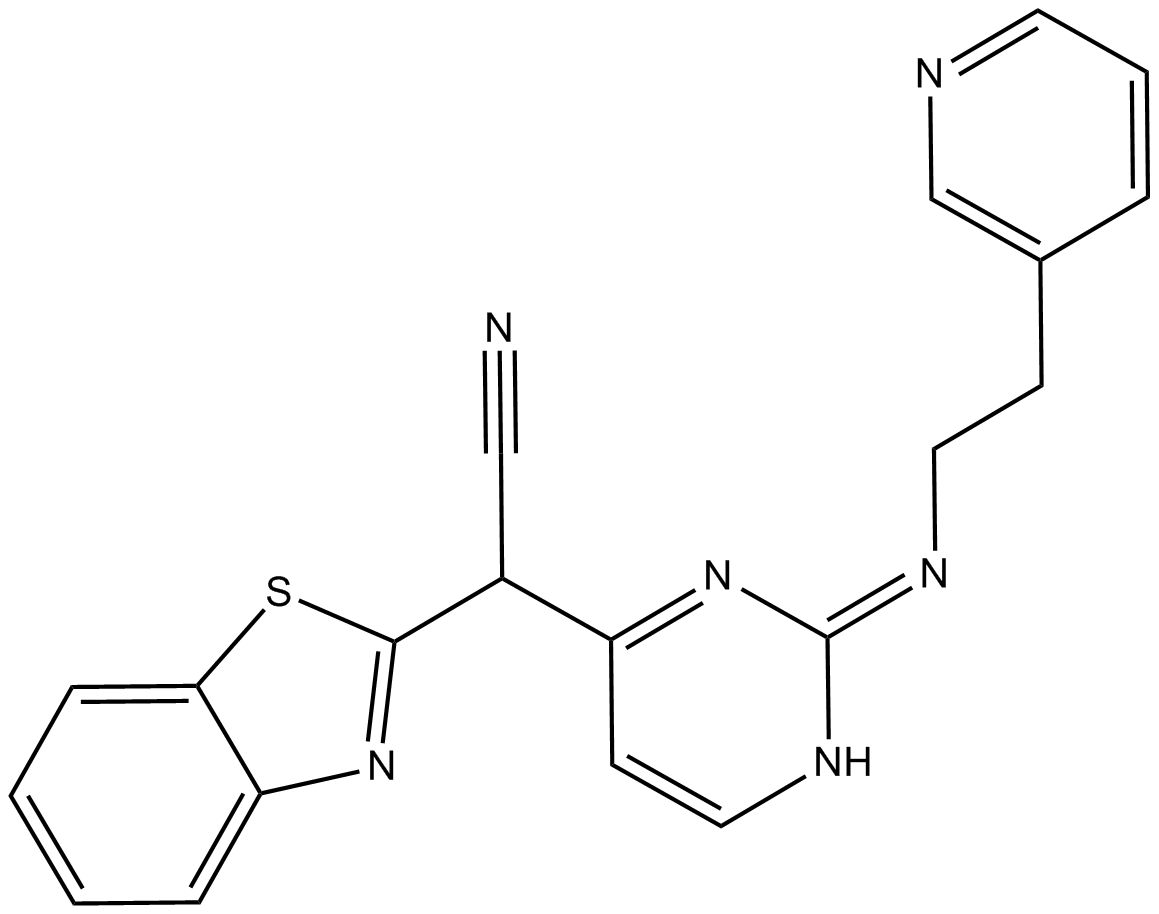
-
GC19041
ASK1-IN-1
ASK1-IN-1 es un potente inhibidor selectivo de ATP competitivo disponible por vÍa oral de la quinasa 1 reguladora de la seÑal de apoptosis (ASK1) con una IC50 de 2,87 nM.
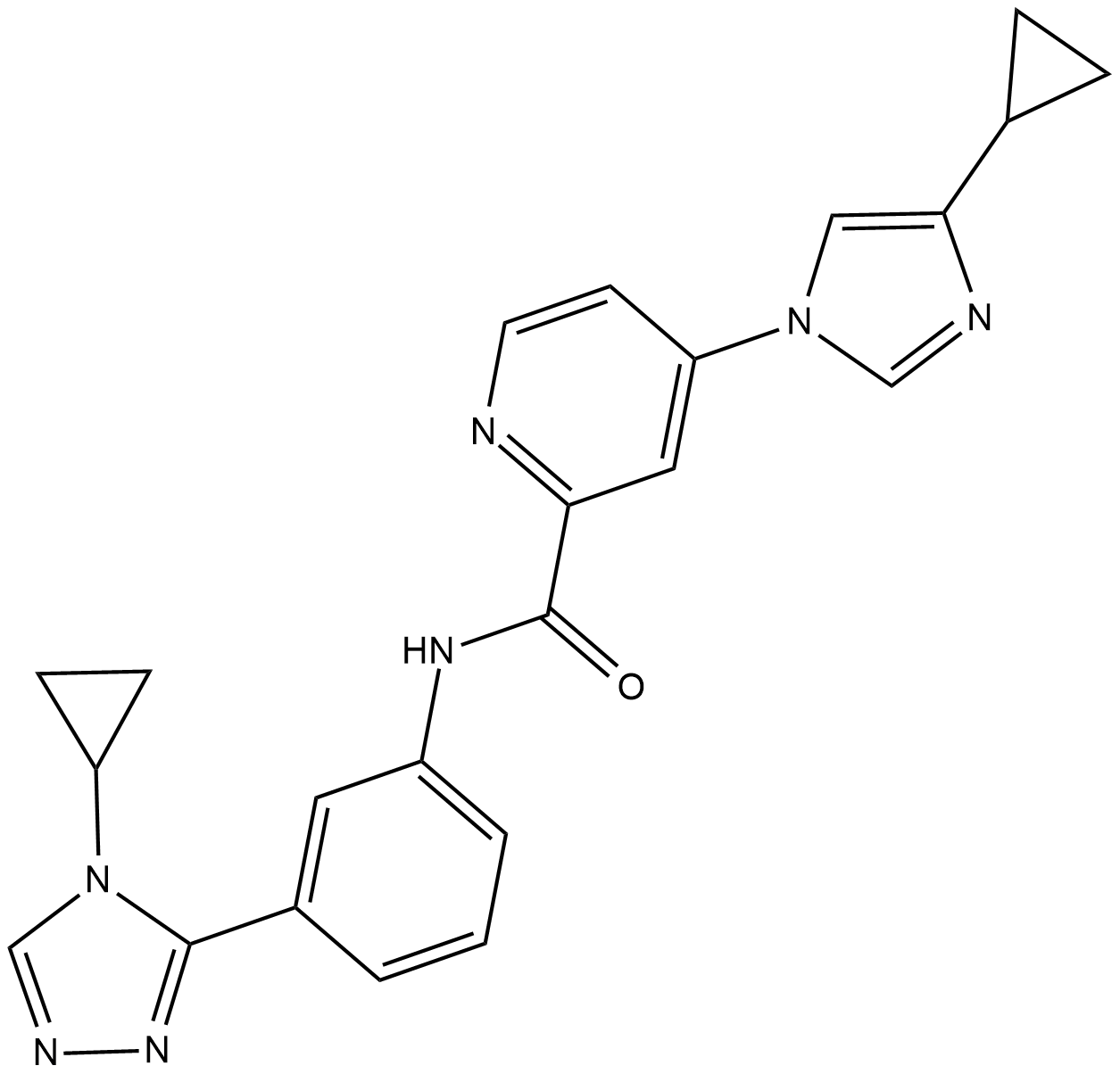
-
GC62426
ASK1-IN-2
ASK1-IN-2 es un inhibidor potente y activo por vÍa oral de la quinasa 1 reguladora de la seÑal de apoptosis (ASK1), con una IC50 de 32,8 nM.
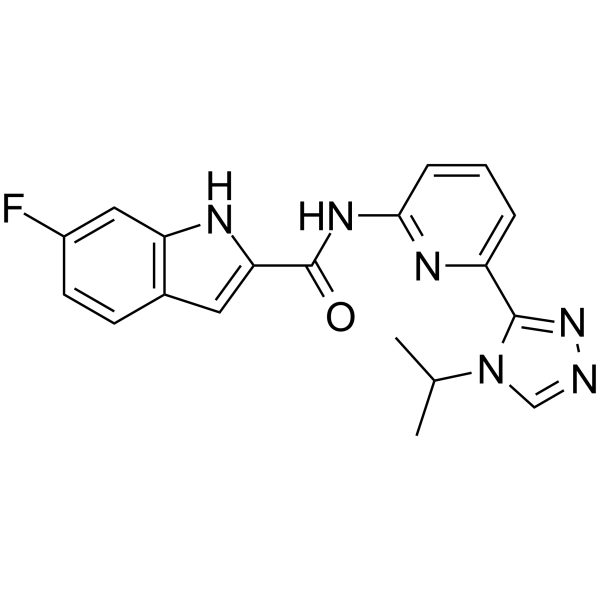
-
GC35412
Asperulosidic Acid
El Ácido asperulosÍdico (ASPA), un glucÓsido iridoide bioactivo, se extrae de las hierbas de Hedyotis diffusa Willd.
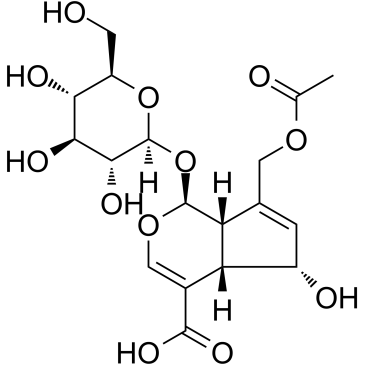
-
GN10494
Astragaloside IV
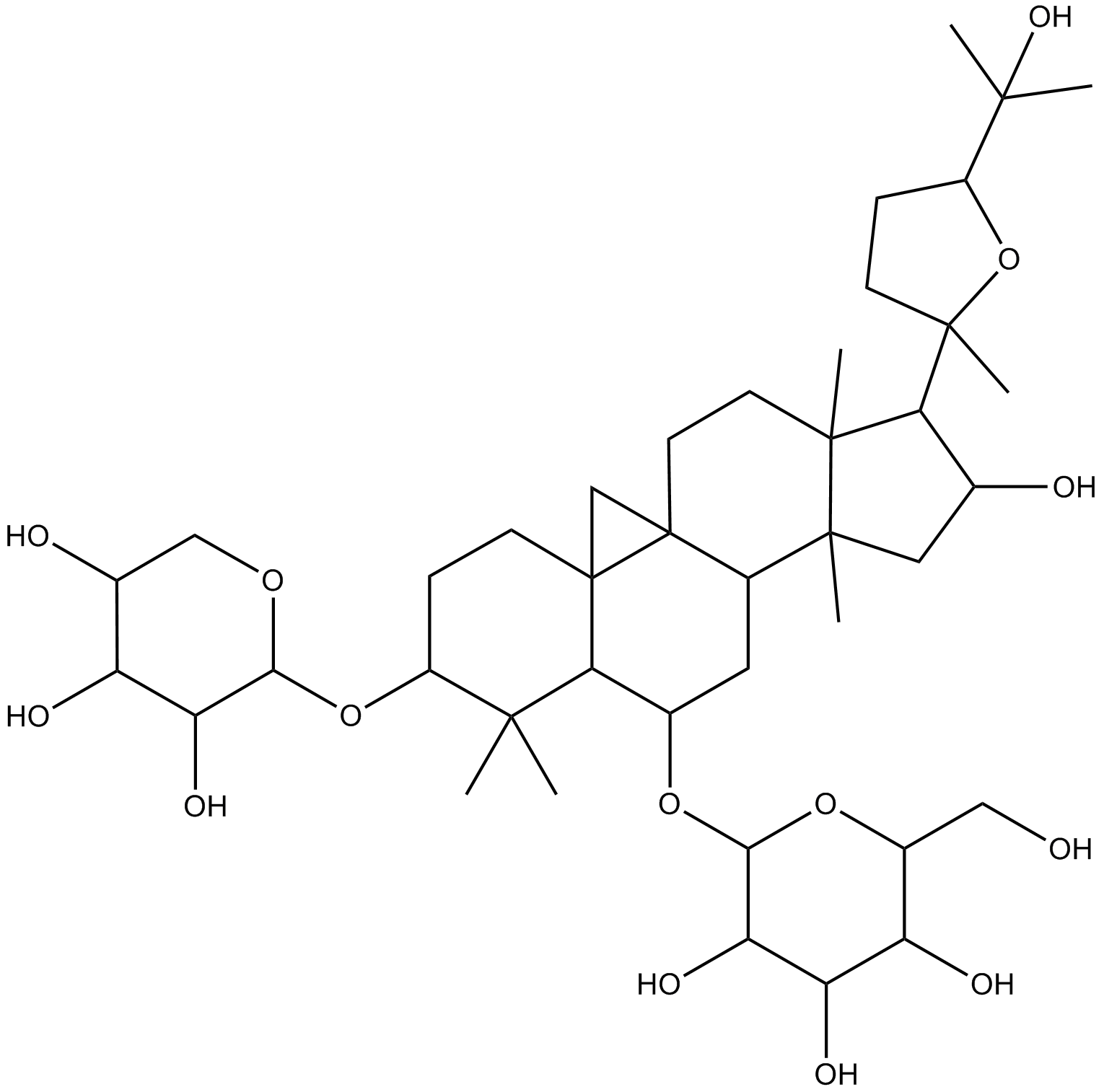
-
GC17712
AT13148
AT13148 es un inhibidor multi-AGC cinasa activo por vÍa oral y competitivo con ATP con IC50 de 38 nM/402 nM/50 nM, 8 nM, 3 nM y 6 nM/4 nM para Akt1/2/3, p70S6K, PKA, y ROCKI/II, respectivamente.
-
GC12297
AT7867
AT7867 es un potente inhibidor competitivo de ATP de Akt1/Akt2/Akt3 y p70S6K/PKA con IC50 de 32 nM/17 nM/47 nM y 85 nM/20 nM, respectivamente.
-
GC10918
AT7867 dihydrochloride
A potent and orally bioavailable pan-Akt inhibitor
-
GC31667
AX-15836
AX-15836 es un inhibidor de ERK5 potente y selectivo con una IC50 de 8 nM.
-
GC15055
AZ 628
AZ 628 es un inhibidor de la cinasa pan-Raf con IC50 de 105, 34 y 29 nM para B-Raf, B-RafV600E y c-Raf-1, respectivamente.

-
GC19479
AZ304
AZ304 es un inhibidor de la quinasa BRAF dual competitivo con ATP, inhibe potentemente BRAF de tipo salvaje, BRAF mutante V600E y CRAF de tipo salvaje, con IC50 de 79 nM, 38 nM y 68 nM, respectivamente. AZ304 también tiene un efecto significativo sobre otras quinasas, como p38 (IC50, 6 nM), CSF1R (IC50, 35 nM). Actividad antitumoral.

-
GC19048
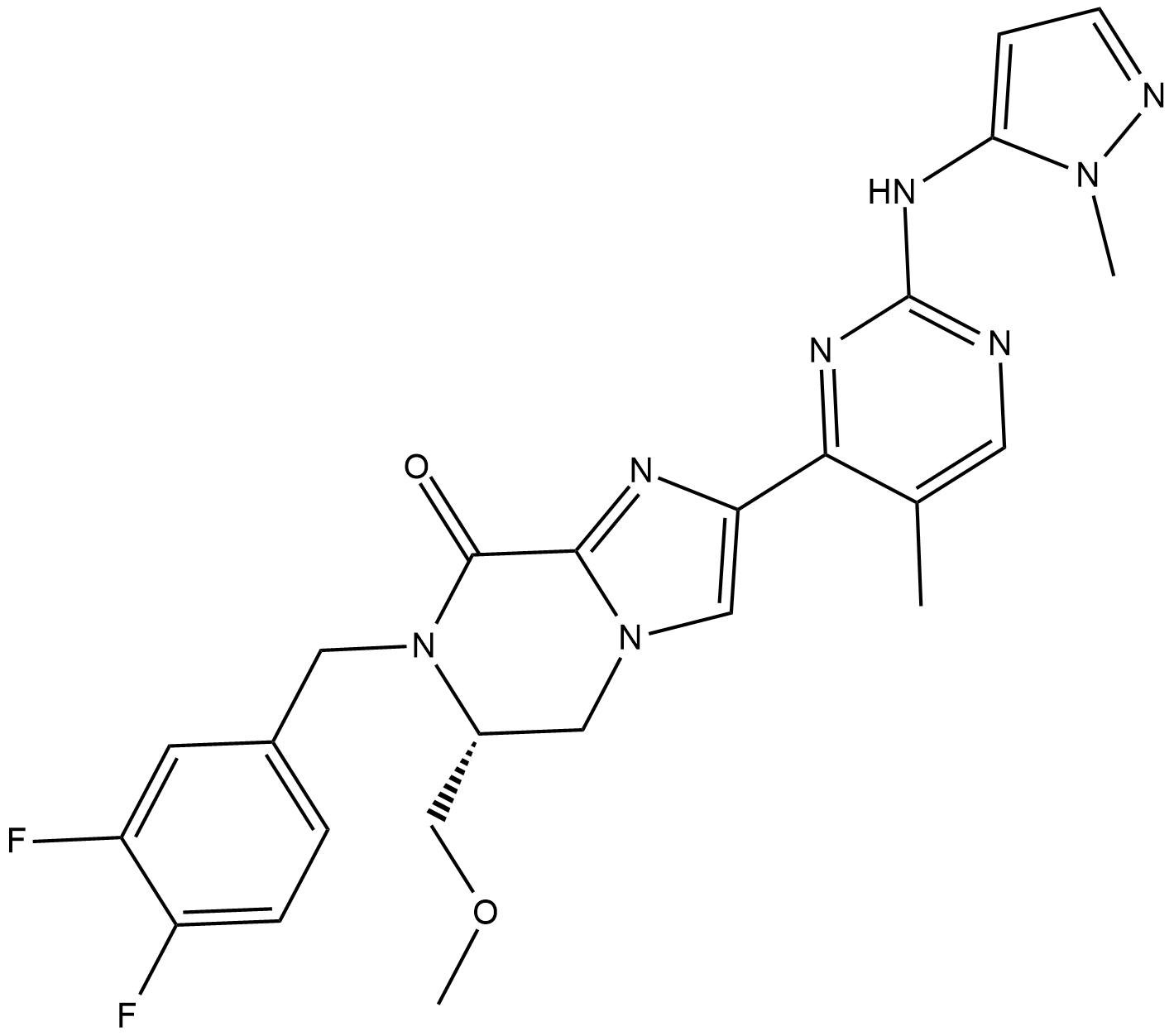
AZD-0364
AZD-0364 (AZD0364) es un potente y selectivo inhibidor de ERK2 extraÍdo de la patente WO2017080979A1, compuesto ejemplo 18, tiene una IC50 de 0,6 nM.
-
GC17030
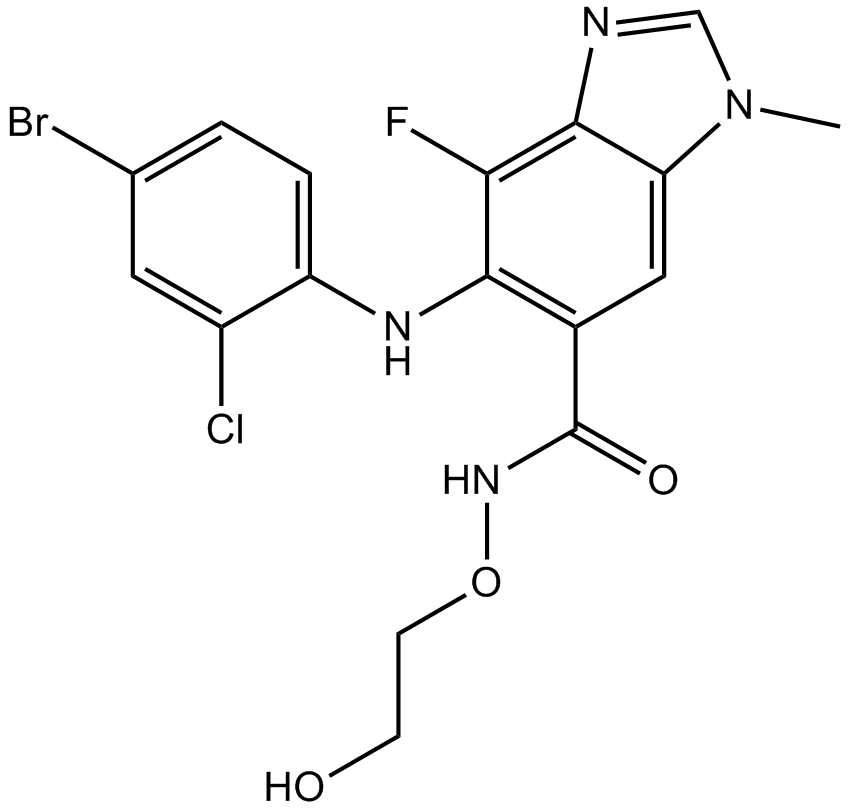
AZD6244(Selumetinib)
A highly selective inhibitor of MEK1/2
-
GC31924
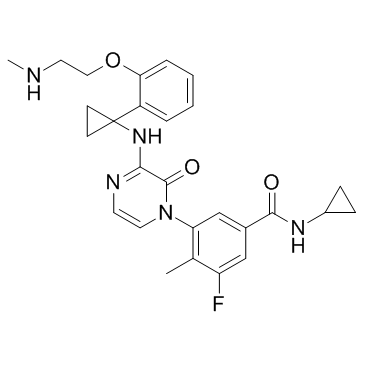
AZD7624
AZD7624 es un inhibidor de p38 inhalado, con una potente actividad antiinflamatoria.
-
GC14643
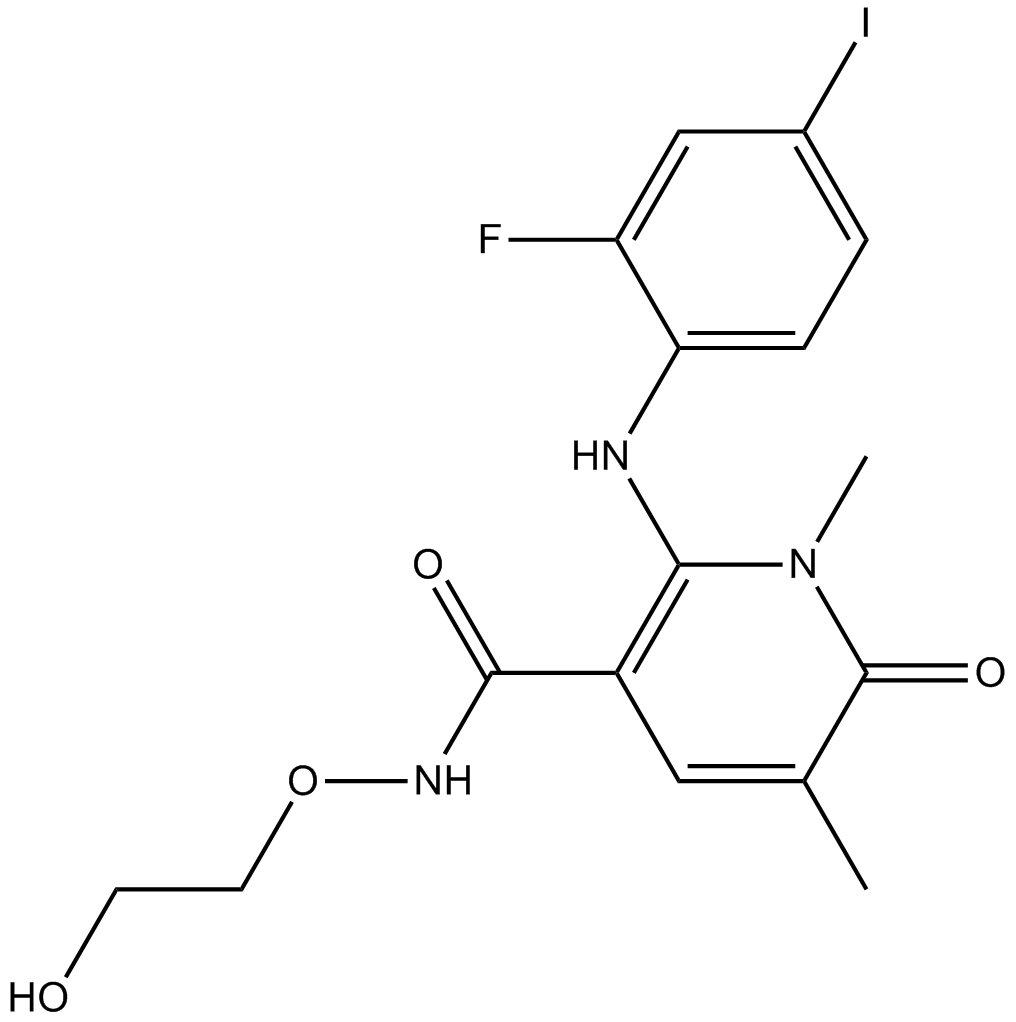
AZD8330
AZD8330 (ARRY-424704) es un inhibidor de MEK1/MEK2 potente y no competitivo, con una IC50 de 7 nM.
-
GC38738
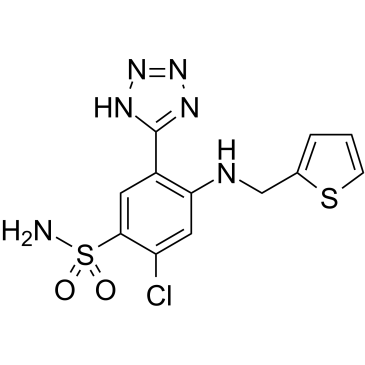
Azosemide
La azosemida, un diurético de asa de sulfonamida, es un potente inhibidor de NKCC1 con IC50 de 0,246μM y 0,197μM para hNKCC1A y NKCC1B, respectivamente.
-
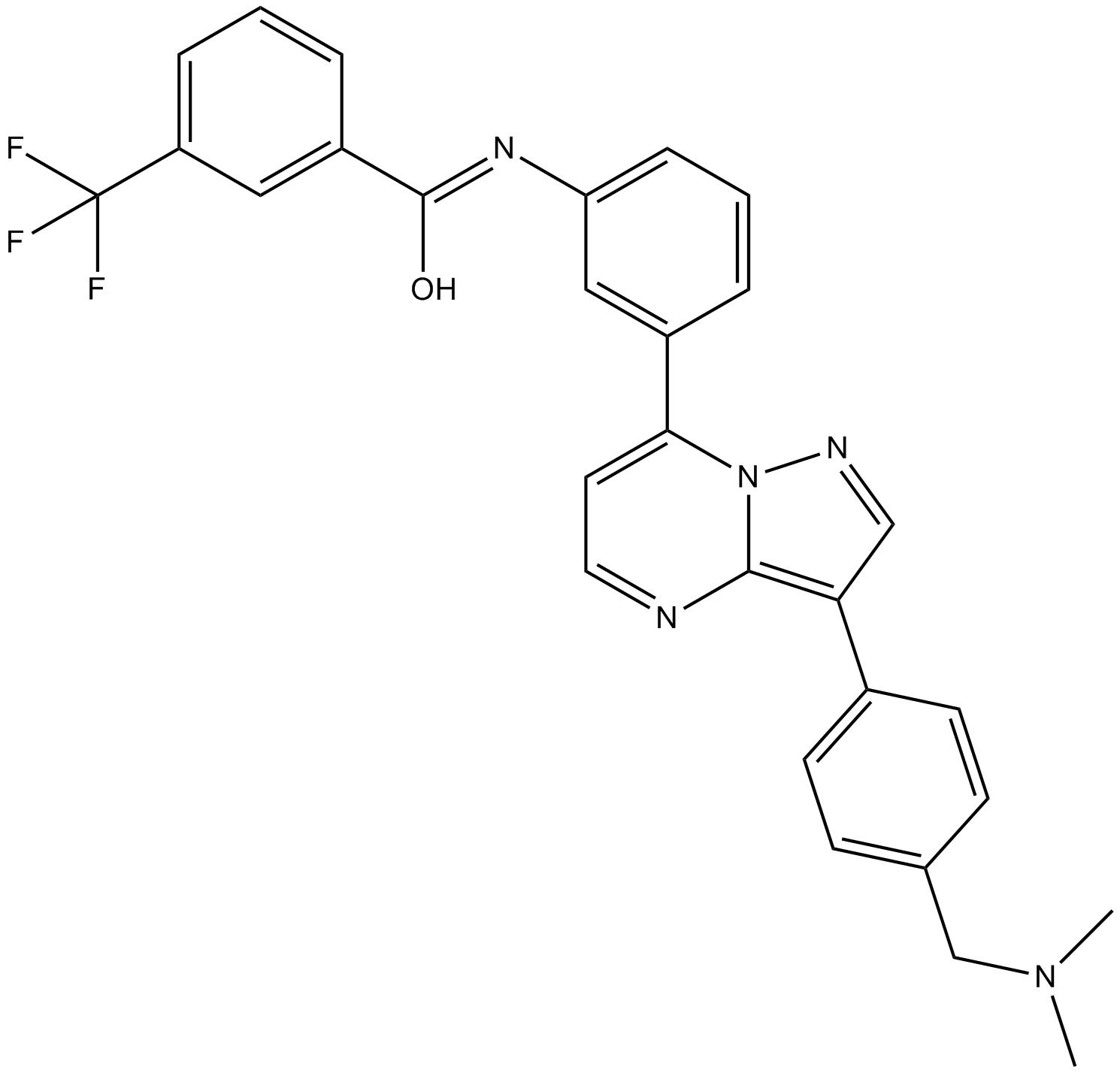
GC10534
B-Raf IN 1
B-Raf IN 1 es un inhibidor potente y selectivo de la quinasa B-Raf con una IC50 de 24 nM.
-
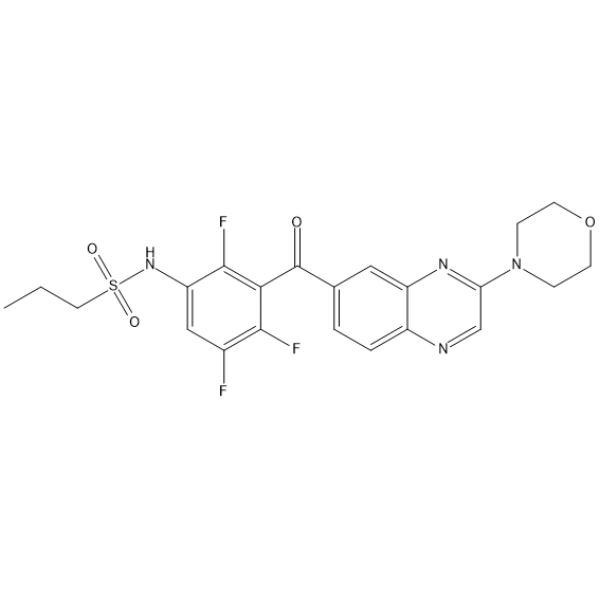
GC65978
B-Raf IN 10
B-Raf IN 10 (Compuesto C09) es un inhibidor de BRAF con un IC50 entre 50 y 100 nM. B-Raf IN 10 muestra actividad antitumoral.
-
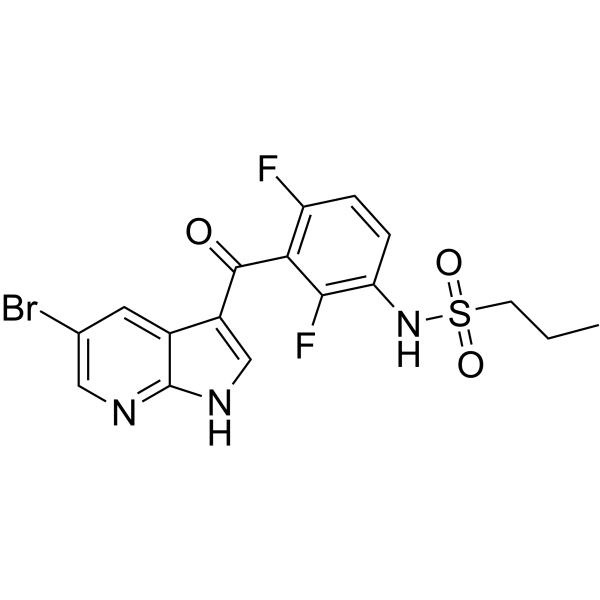
GC67800
B-Raf IN 11
-
GC64550
B-Raf IN 2
B-Raf IN 2 es un potente y selectivo inhibidor de BRAF extraÍdo de la patente WO2021116055A1, compuesto Ia. B-Raf IN 2 se puede utilizar para la investigaciÓn del cÁncer.
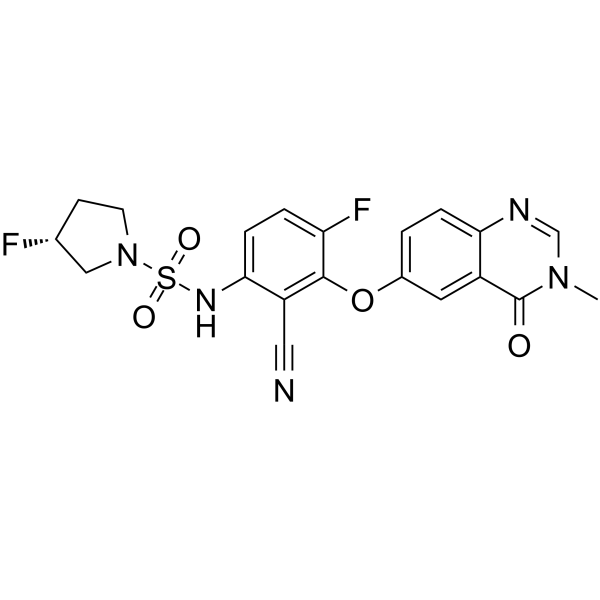
-
GC12151
B-Raf inhibitor
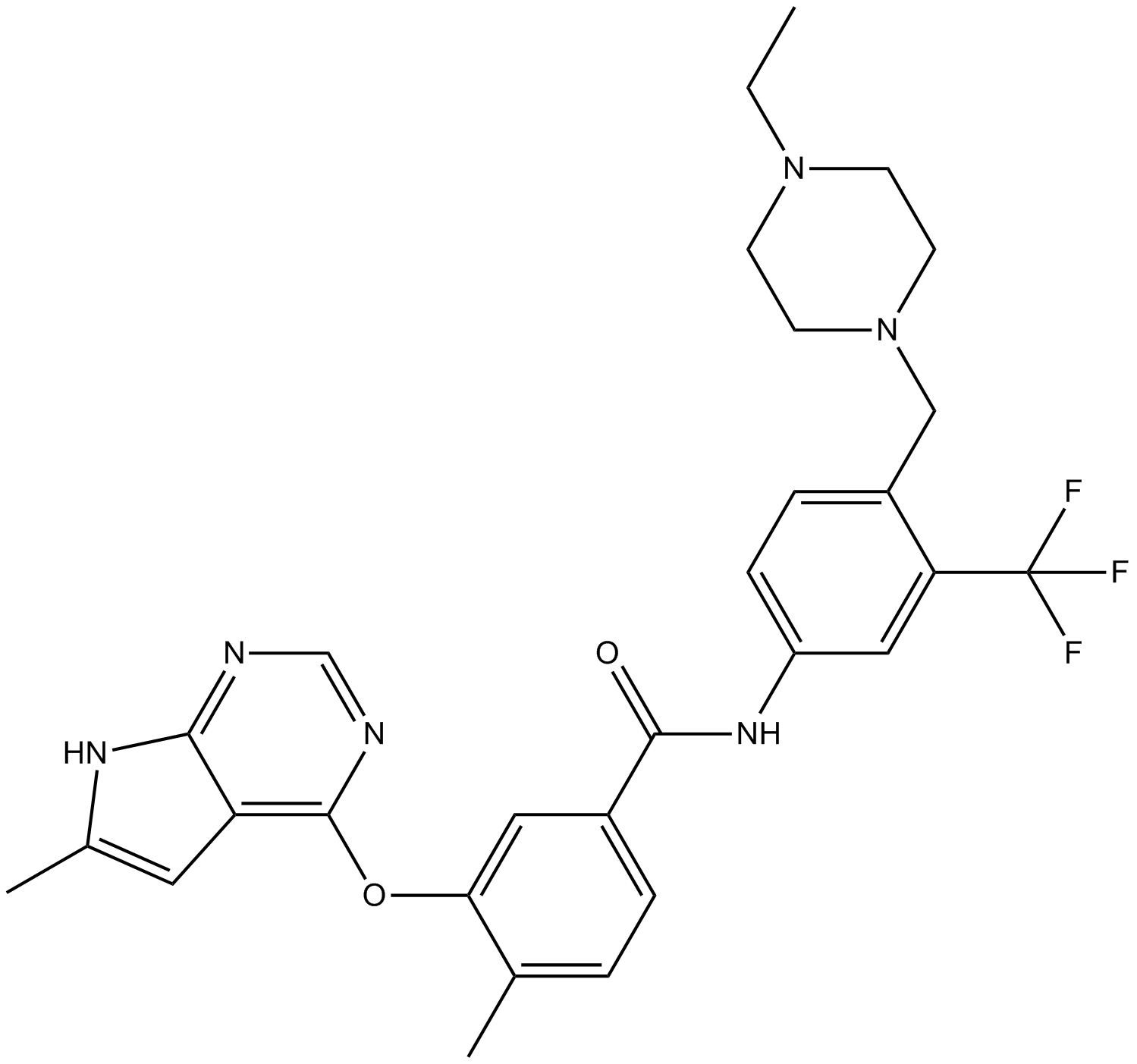
-
GC11599
B-Raf inhibitor 1
El inhibidor 1 de B-Raf es un potente inhibidor de la quinasa Raf con Kis de 1 nM, 1 nM y 0,3 nM para B-RafWT, B-RafV600E y C-Raf, respectivamente.
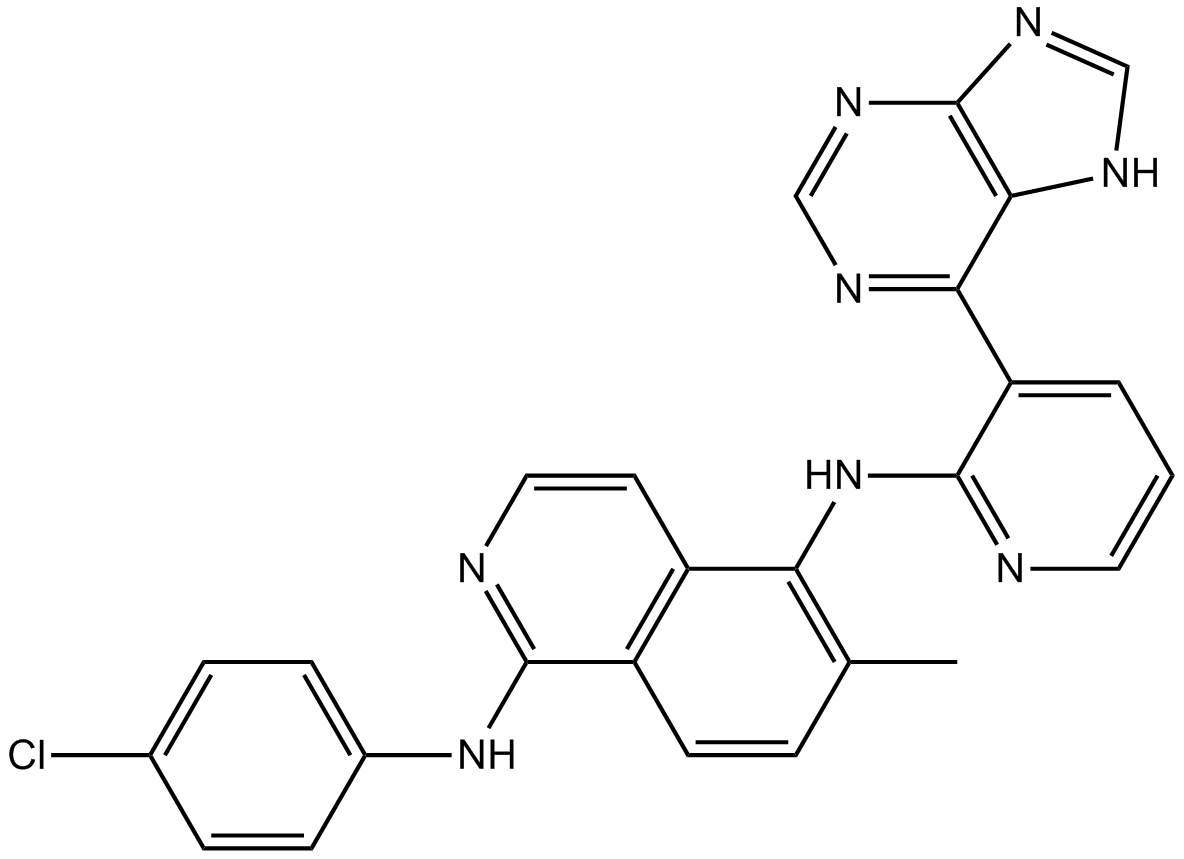
-
GC14907
B-Raf inhibitor 1 dihydrochloride
El diclorhidrato del inhibidor 1 de B-B-Raf es un potente inhibidor de la quinasa Raf con Kis de 1 nM, 1 nM y 0,3 nM para B-RafWT, B-RafV600E y C-Raf, respectivamente.
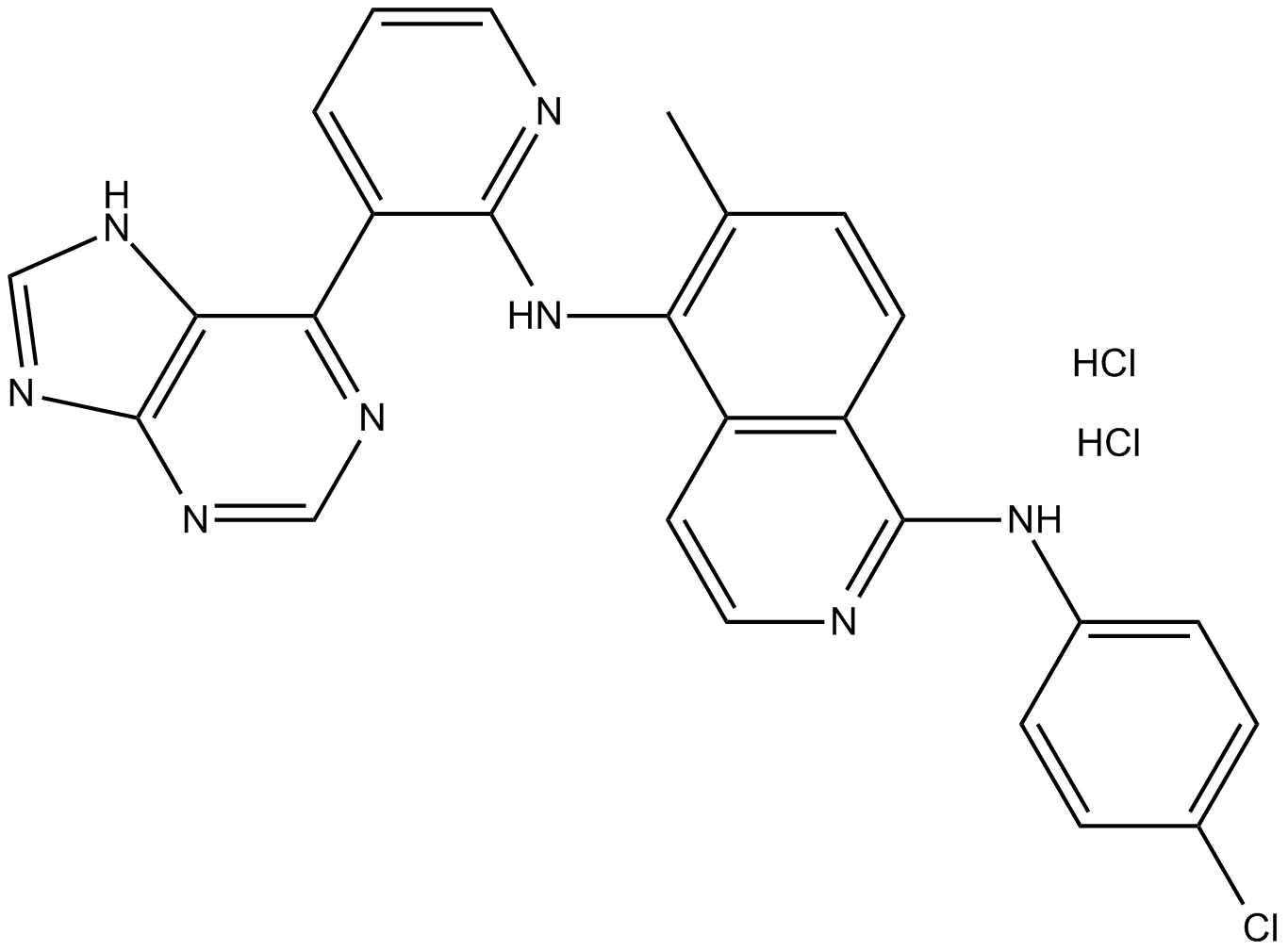
-
GN10590
Bakuchiol
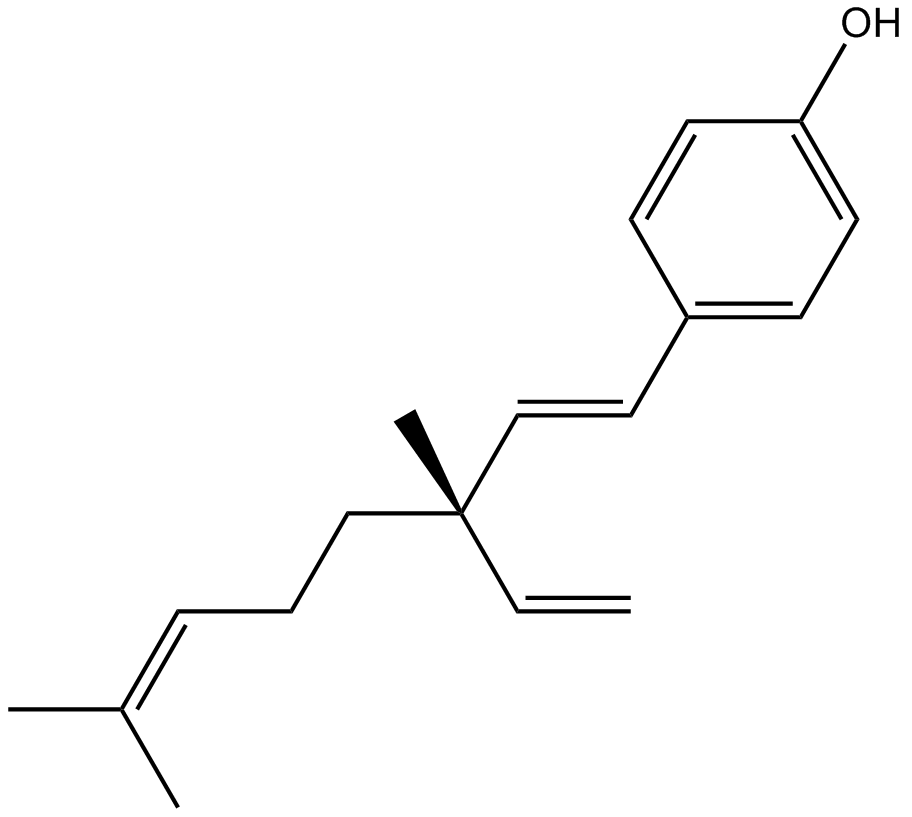
-
GC33305
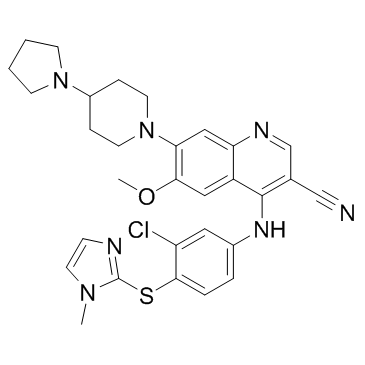
Balamapimod (MKI 833)
Balamapimod (MKI 833) (MKI 833) es un inhibidor reversible de Ras/Raf/MEK con potencial actividad antitumoral.
-
GC34342
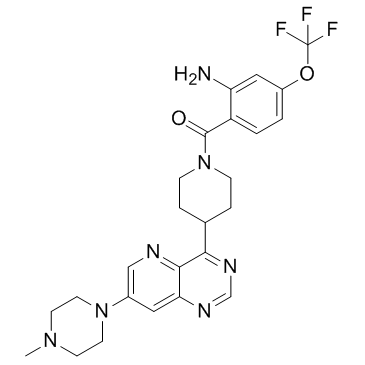
BAY885
BAY885 es un inhibidor de ERK5 muy potente y selectivo con una IC50 de 35 nM. BAY885 muestra una débil inhibiciÓn de otras quinasas.
-
GC32950
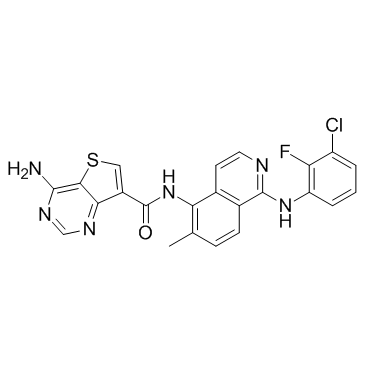
Belvarafenib
Belvarafenib (HM95573) es un inhibidor potente y pan RAF (fibrosarcoma acelerado rÁpidamente), con IC50 de 56 nM, 7 nM y 5 nM para B-RAF, B-RAFv600E y C-RAF respectivamente.
-
GC35488
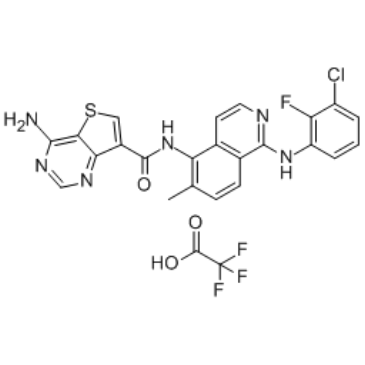
Belvarafenib TFA
Belvarafenib TFA (HM95573 TFA) es un inhibidor potente y pan RAF (fibrosarcoma acelerado rÁpidamente), con IC50 de 56 nM, 7 nM y 5 nM para B-RAF, B-RAFv600E y C-RAF respectivamente.
-
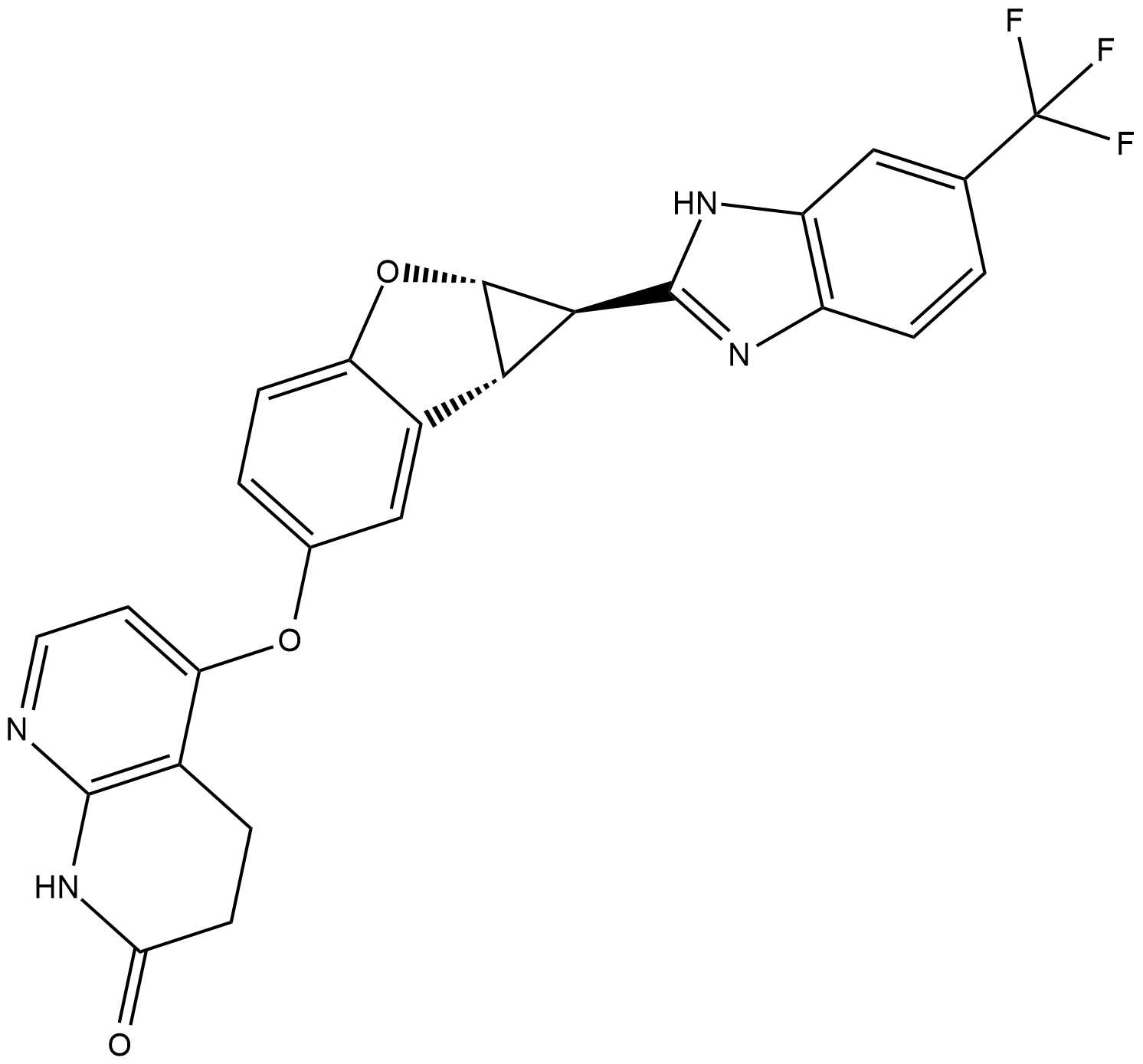
GC19066
BGB-283
BGB-283 is a novel and potent Raf Kinase and EGFR inhibitor with IC50 values of 23 and 29 nM for recombinant BRafV600E and EGFR, respectively.
-
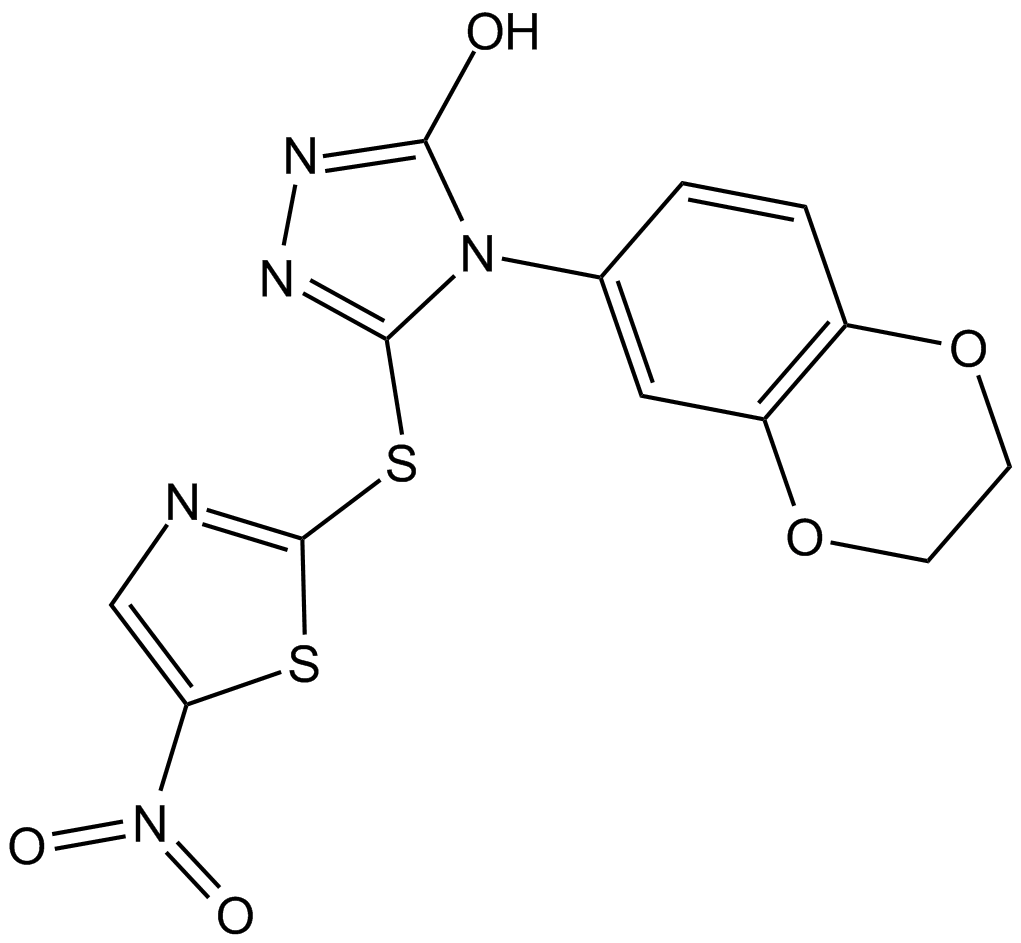
GC12904
BI 78D3
BI 78D3 funciona como un inhibidor competitivo de sustrato de JNK, inhibe la actividad quinasa de JNK (IC50 \u003d 280 nM).
-
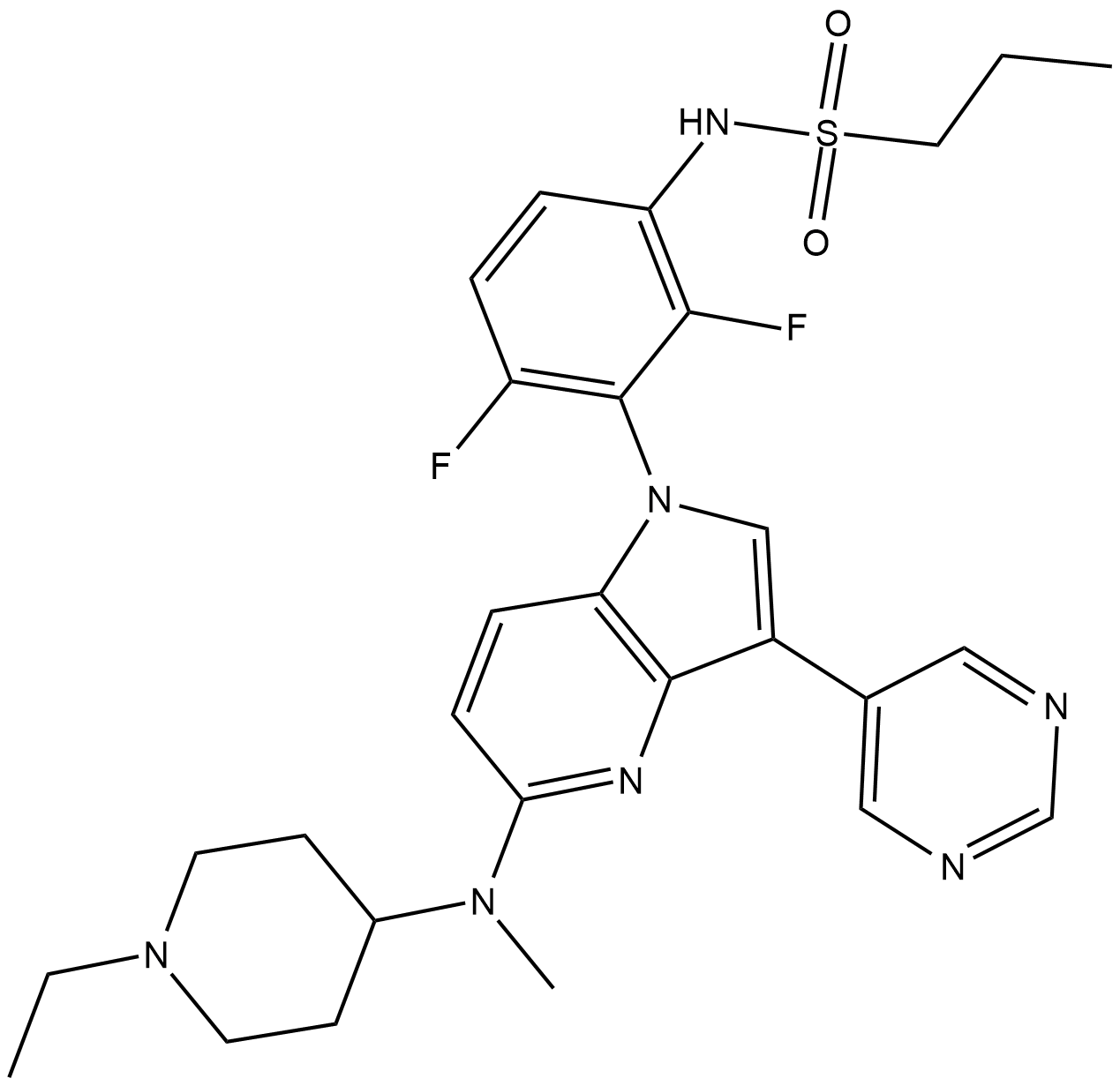
GC17828
BI-847325
BI-847325 es un inhibidor dual competitivo de ATP de MEK y aurora quinasas (AK) con valores IC50 de 4 y 15 nM para MEK2 y AK-C humanos, respectivamente.

-
GC18178
BI-882370
BI-882370 es un inhibidor potente y selectivo de la quinasa RAF que se une al sitio de uniÓn de ATP de la quinasa posicionada en la conformaciÓn DFG-out (inactiva) de la quinasa BRAF. BI-882370 (BI 882370) inhibe el mutante oncogénico BRAFV600E, las quinasas WT BRAF y CRAF con IC50 de 0,4, 0,8 y 0,6 nM, respectivamente. BI-882370 también inhibe las quinasas de la familia SRC.
-
GC13468
BI-D1870
BI-D1870 es un inhibidor de las isoformas RSK competitivo con ATP, permeable a las células y penetrado en el cerebro, con IC50 de 31 nM/24 nM/18 nM/15 nM para RSK1/RSK2/RSK3/RSK4, respectivamente.

-
GC35518
Bilobetin
Bilobetin, un componente activo de Ginkgo biloba, puede reducir los lÍpidos en sangre y mejorar los efectos de la insulina.
-
GC11497
BIRB 796 (Doramapimod)
BIRB 796 (Doramapimod) (BIRB 796) es un inhibidor de p38 MAPK muy potente, activo por vÍa oral, que tiene una CI50 para p38α = 38 nM, para p38β = 65 nM, para p38γ = 7 para p748; 520 nm.
-
GC35525
Bisabolangelone
La bisabolangelona, un derivado del sesquiterpeno, se aÍsla de las raÍces de Osterici Radix.
-
GC15693
BIX 02188
BIX 02188 es un potente inhibidor selectivo de MEK5 con una IC50 de 4,3 nM. BIX 02188 inhibe la actividad catalítica de ERK5, con un IC50 de 810 nM.
-
GC12220
BIX 02189
BIX 02189 es un inhibidor de MEK5 potente y selectivo con una IC50 de 1,5 nM. BIX 02189 también inhibe la actividad catalítica de ERK5 con un IC50 de 59 nM.
-
GC12982
BIX 02565
BIX 02565 es un potente inhibidor de la quinasa 2 ribosomal S6 (RSK2) con IC50 de 1,1 nM.
-
GC16997
BMS-582949
p38 MAPK inhibitor
-
GC10593
BMS-582949 hydrochloride
El clorhidrato de BMS-582949 es un inhibidor de p38α MAPK activo por vÍa oral y altamente selectivo, con una IC50 de 13 nM.

-
GC42968
BPIQ-II (hydrochloride)
BPIQ-II is a linear imidazoloquinazoline that potently inhibits the tyrosine kinase activity of the epidermal growth factor receptor (EGFR; IC50 = 8 pM).
-
GC12482
BRAF inhibitor
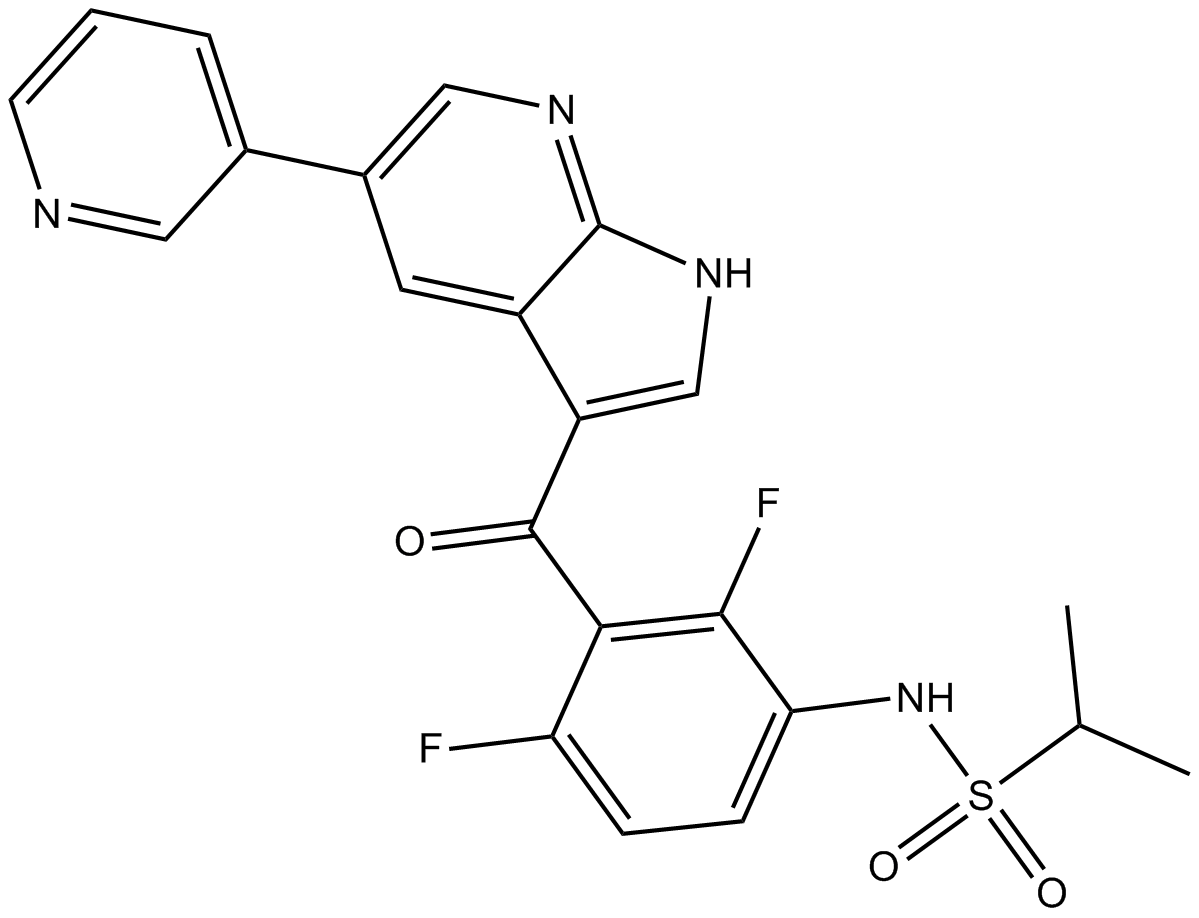
-
GC16779
BRD 7389
BRD 7389 es un inhibidor específico de la quinasa de la familia RSK con IC50 de 1,5 μM, 2,4 μM y 1,2 μM para RSK1, RSK2 y RSK3, respectivamente.
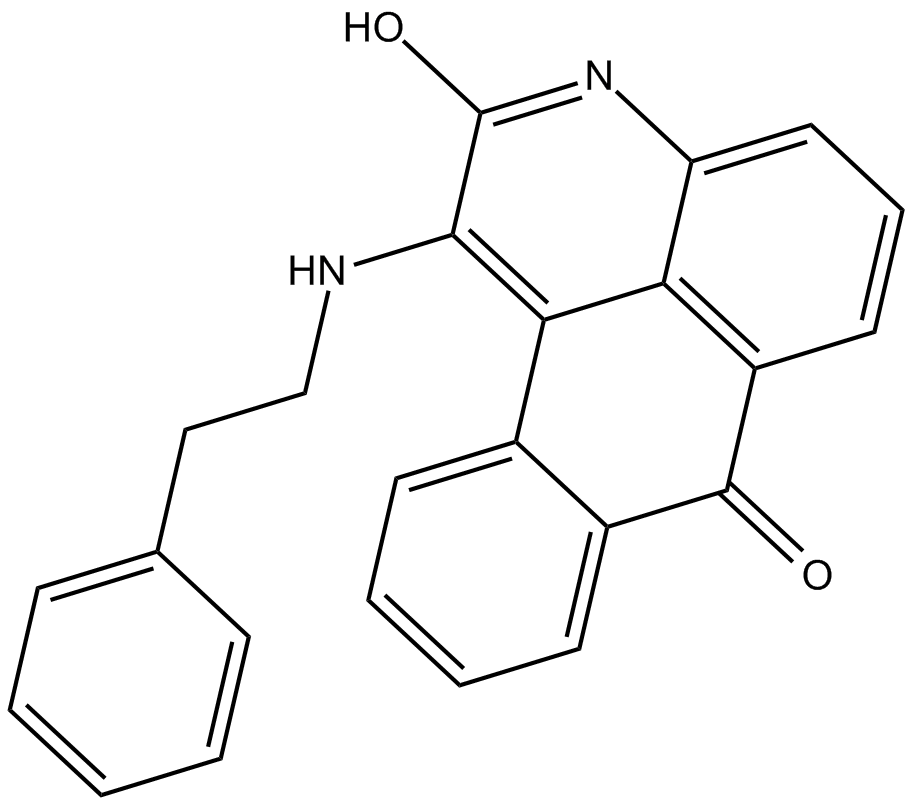
-
GC35565
Bucladesine calcium salt
La sal de sal de calcio de bucladesina (sal de calcio de dibutiril-cAMP; sal de calcio DC2797) es un anÁlogo de AMP cÍclico permeable a las células (cAMP) y activa selectivamente la proteÍna quinasa dependiente de cAMP (PKA) al aumentar el nivel intracelular de cAMP.
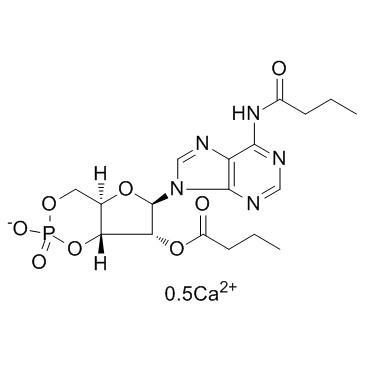
-
GC15524
Bumetanide
La bumetanida (Ro 10-6338; PF 1593), un diurético de asa muy potente, es un bloqueador del cotransportador Na+-K+-Cl+ (NKCC).
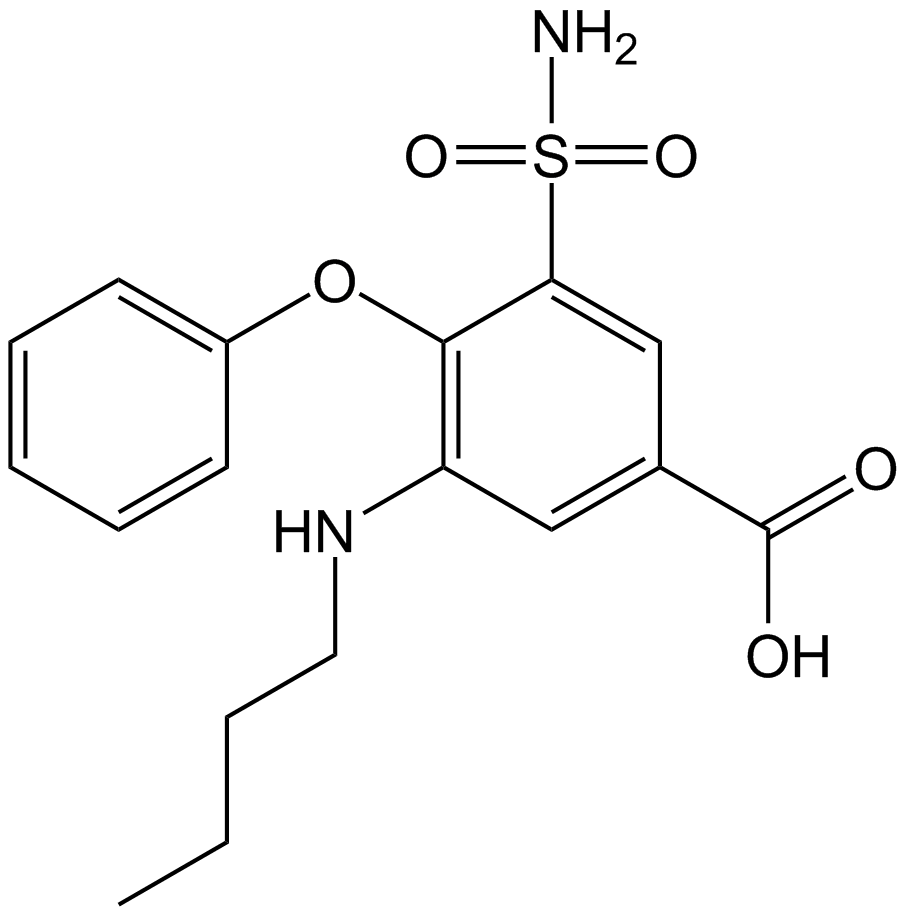
-
GC11605
C-1
C-1 es un potente inhibidor de la proteÍna quinasa, con IC50 de 4 μM, 8 μM, 12 μM y 240 μM para proteÍna quinasa dependiente de cGMP (PKG), proteÍna quinasa dependiente de cAMPPKA , proteÍna quinasa C (PKC) y MLC-quinasa, respectivamente. C-1 también se usa como inhibidor de ROCK.

-
GC10693
c-JUN peptide
JNK/c-Jun interaction inhibitor

-
GC43065
C2 Phytoceramide (t18:0/2:0)
C2 Phytoceramide is a bioactive semisynthetic sphingolipid that inhibits formyl peptide-induced oxidant release (IC50 = 0.38 μM) in suspended polymorphonuclear cells.
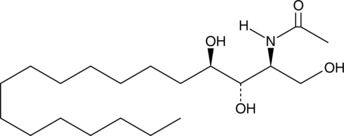
-
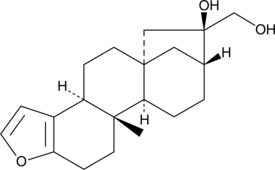
GC40352
Cafestol
El cafestol, uno de los principales componentes del café, es un diterpeno especÍfico del café.
-
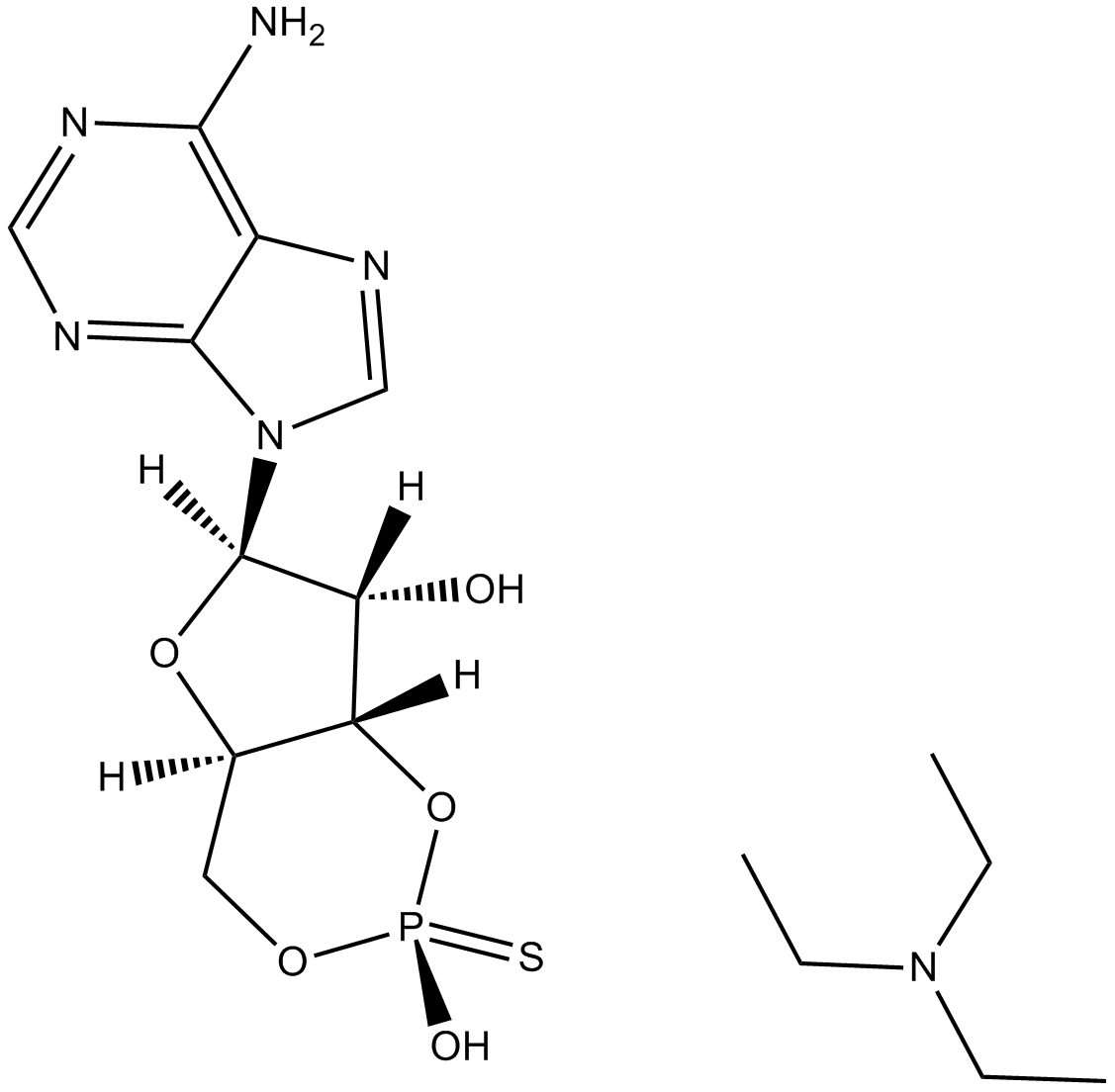
GC10941
cAMPS-Rp, triethylammonium salt
cAMPS-Rp, sal de trietilamonio, un anÁlogo de cAMP, es un antagonista potente y competitivo de la activaciÓn inducida por cAMP de PKA I y II dependiente de cAMP (Kis de 12,5 μM y 4,5 μM, respectivamente).
-
GC12706
cAMPS-Sp, triethylammonium salt
cAMPS-Sp, sal de trietilamonio, un anÁlogo de cAMP, es un potente antagonista competitivo inducido por la activaciÓn de cAMP de cAMP dependiente de PKA I y II (Kis de 12,5 μM y 4,5 μM, respectivamente).

-
GN10016
Carnosol
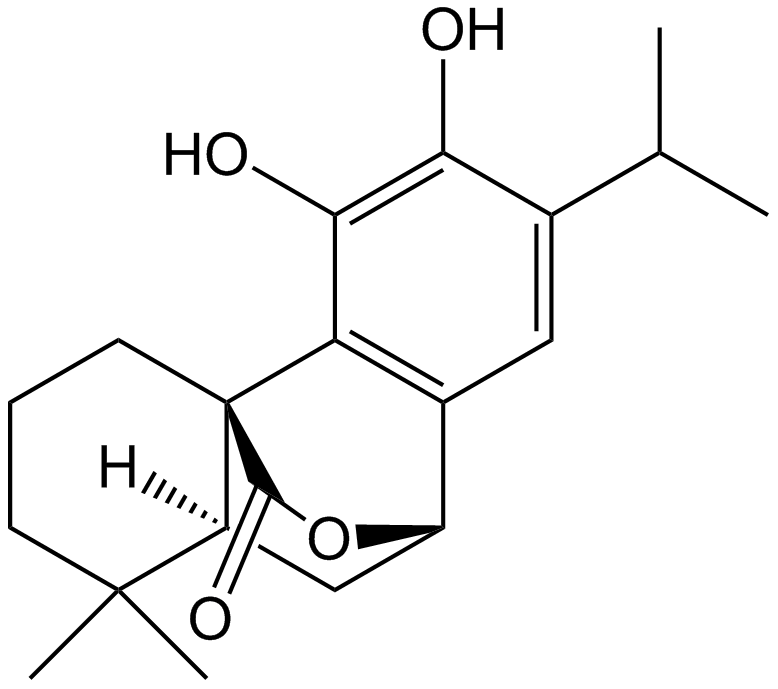
-
GC47045
Carvedilol-d5
An internal standard for the quantification of carvedilol
-
GC43167
CAY10561
The extracellular signal-regulated kinase (ERK) signal transduction pathway regulates a diverse array of cellular processes.
-
GC40650
CAY10706
CAY10706 is a ligustrazine-curcumin hybrid that promotes intracellular reactive oxygen species accumulation preferentially in lung cancer cells.
-
GC43198
CAY10717
CAY10717 is a multi-targeted kinase inhibitor that exhibits greater than 40% inhibition of 34 of 104 kinases in an enzymatic assay at a concentration of 100 nM.
-
GC50400
CC 401 dihydrochloride
High affinity JNK inhibitor; also inhibits HCMV replication
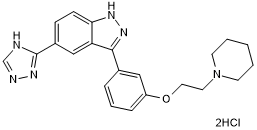
-
GC13529
CC-401
CC-401 es un potente inhibidor de las tres formas de JNK con Ki de 25 a 50 nM.
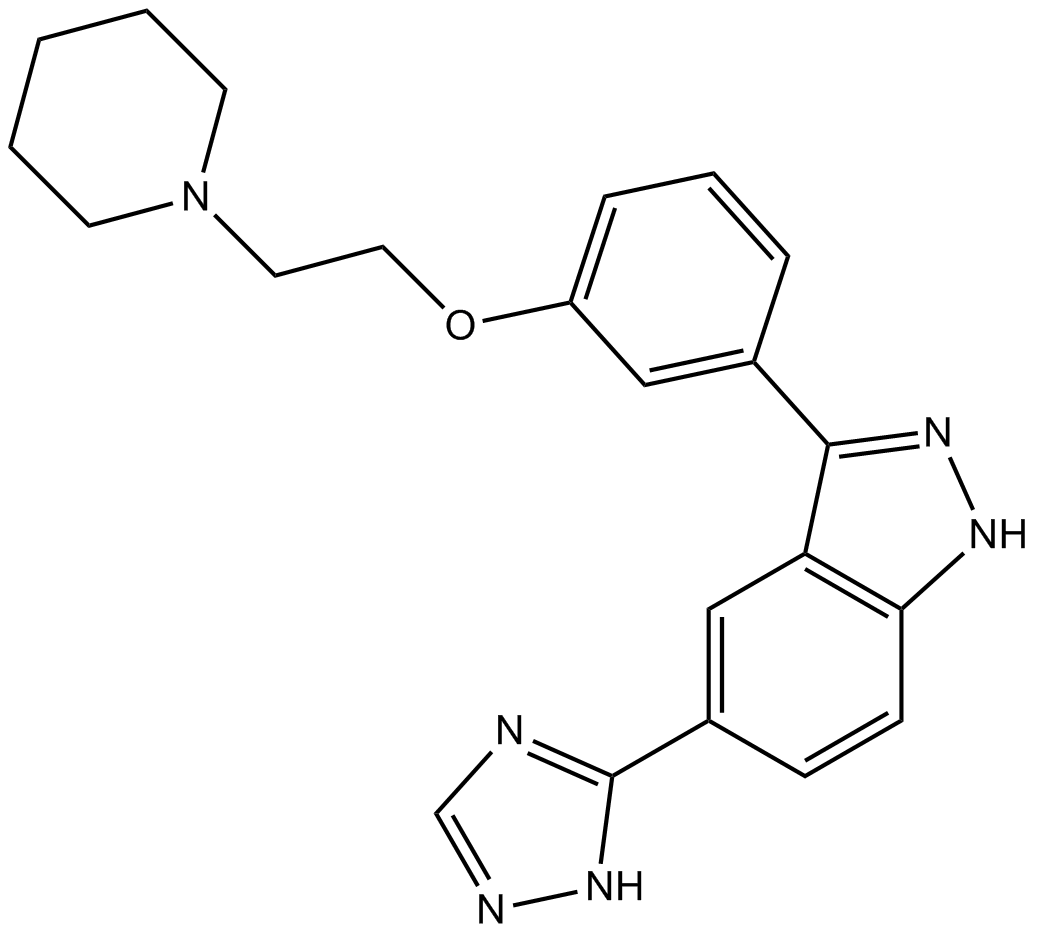
-
GC14197
CC-401 hydrochloride
A potent, specific pan-JNK inhibitor
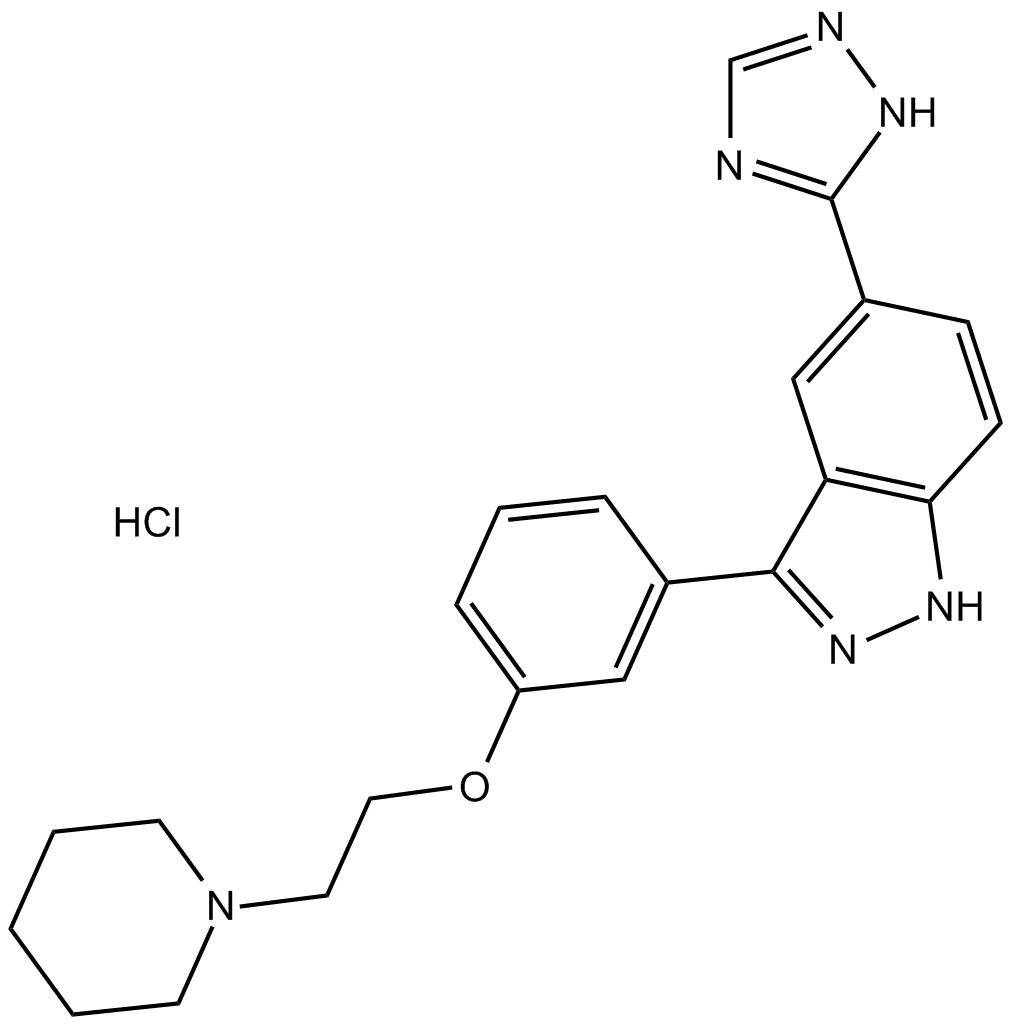
-
GC62492
CC-90001
CC-90001 es un inhibidor potente, selectivo y oralmente activo de la quinasa N-terminal c-Jun (JNK).