E3 Ligase
E3 ligases perform the last step in the ubiquitination cascade. They transfer the ubiquitin from the active site cysteine of E2 enzyme to the substrate lysine or N terminus. The loss of function or mis-targeting of the E3 ligases are associated with various cancer types.
Products for E3 Ligase
- Cat.No. Nom du produit Informations
-
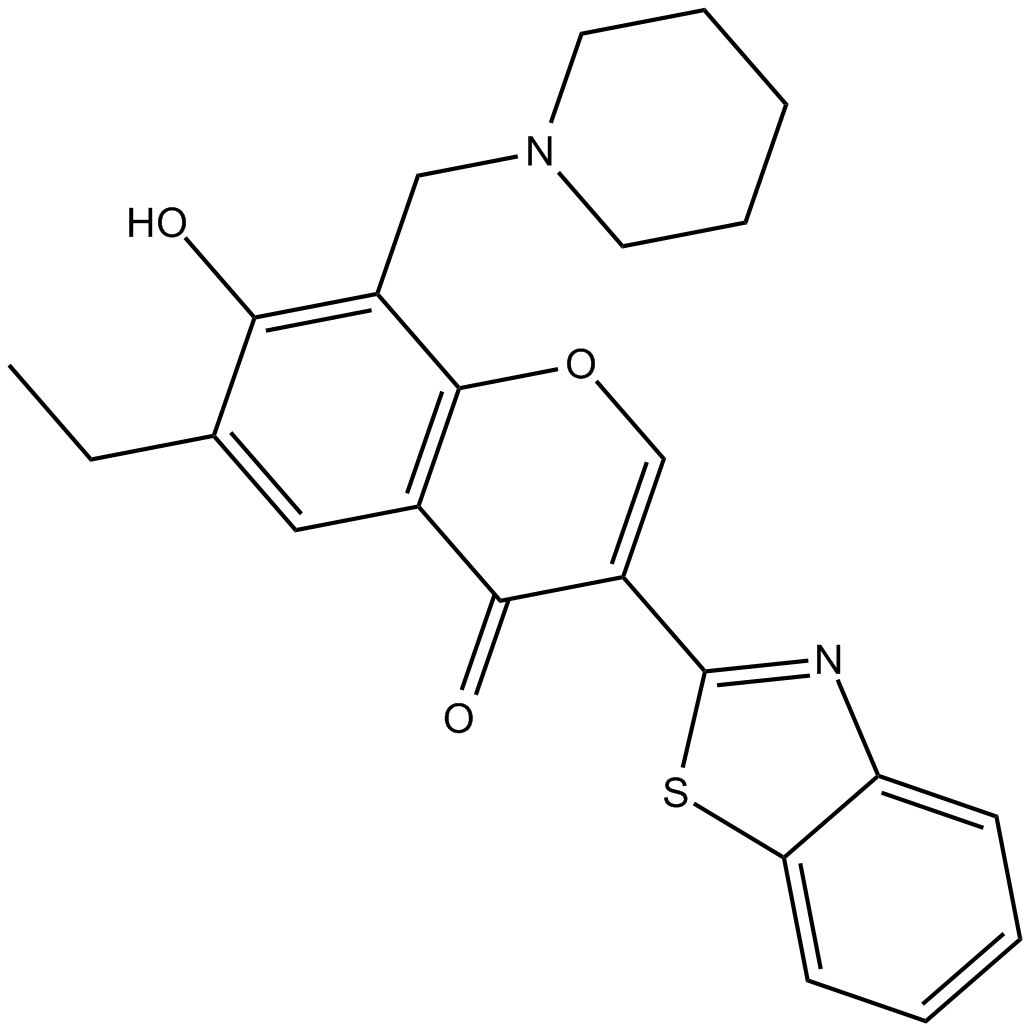
GC25019
5-amino-2,4-dimethylpyridine (5A-DMP)
-
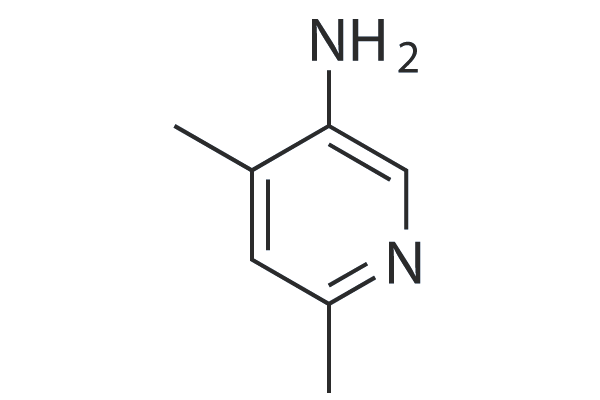
GC50234
N106
N106 est un activateur de SUMOylation de l'ATPase calcique du réticulum sarcoplasmique (SERCA2a) de première classe.
-
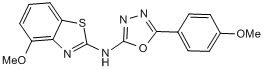
GC44291
NAB 2
NAB 2 est un agent protecteur des neurones.
-
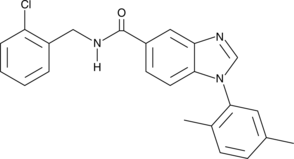
GC15017
NSC 624206
NSC 624206 est un inhibiteur de l'ubiquitine E1 (UBA1), avec une IC50 d'environ 9 μM. NSC 624206 bloque spécifiquement la formation d'ubiquitine-thioester (IC50 \u003d 13 μM) mais n'a aucun effet sur l'adénylation de l'ubiquitine.
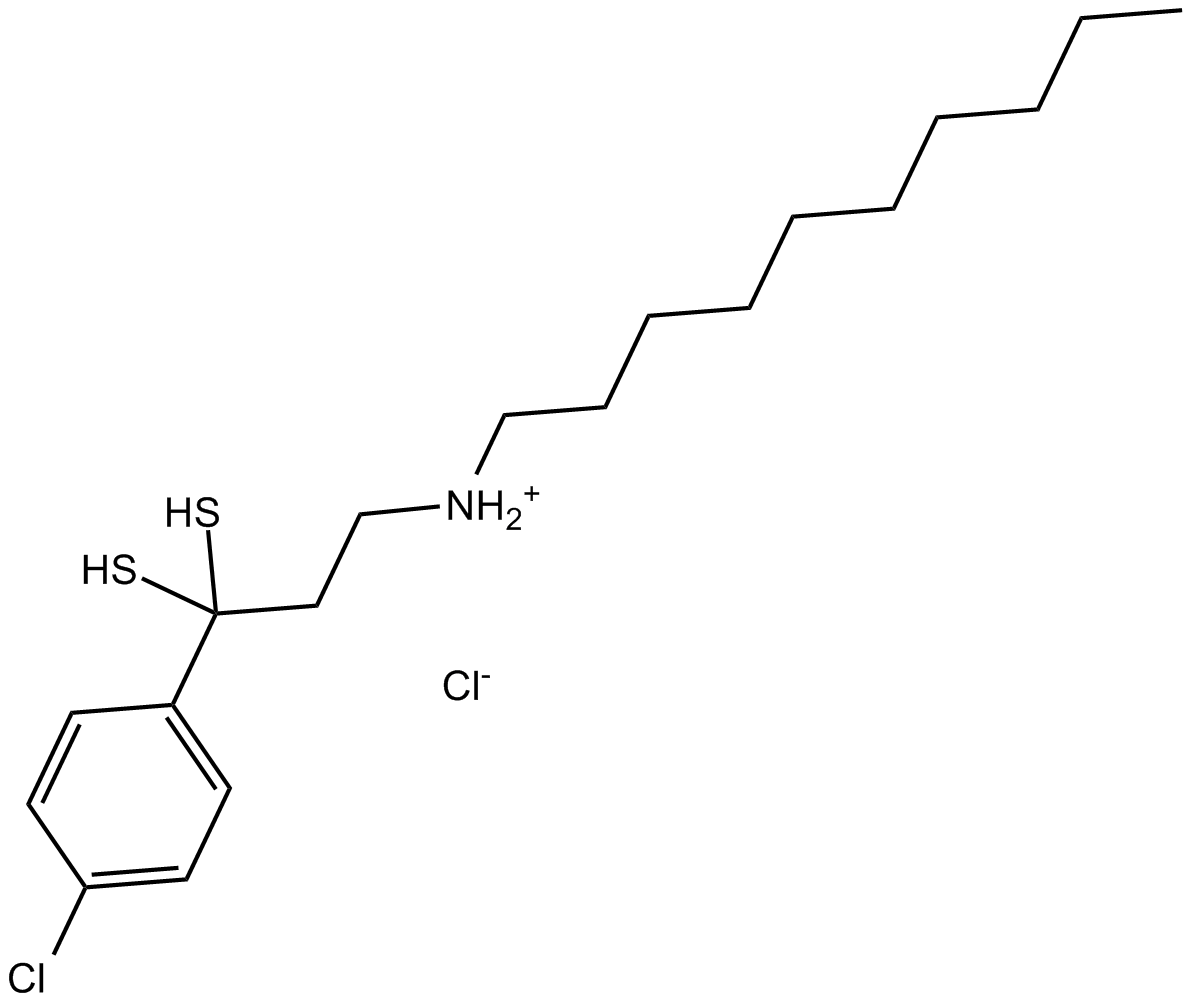
-
GC10940
PRT 4165
PRT 4165 est un puissant inhibiteur de l'ubiquitylation de H2A médiée par PRC1.
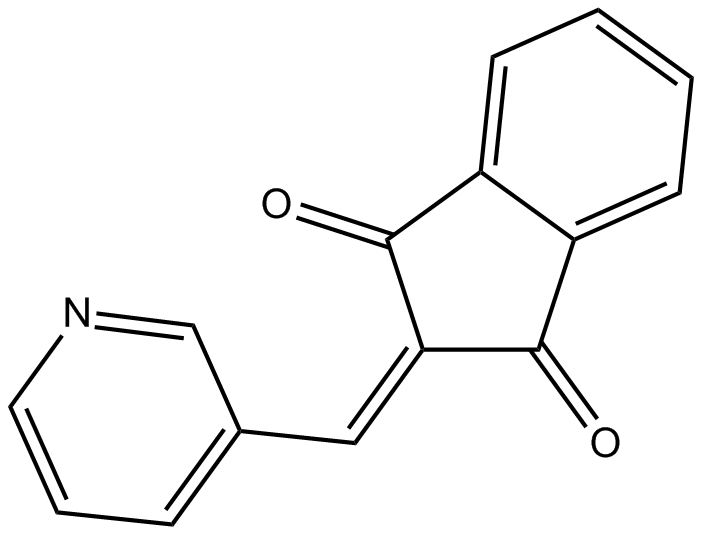
-
GC15492
SMER 3
SMER 3, un activateur de la rapamycine, est un inhibiteur sélectif de l'ubiquitine ligase Skp1-Cullin-F-box (SCF) Met30. SMER 3 améliore l'effet inhibiteur de la croissance de la rapamycine en inhibant SCFmet30.
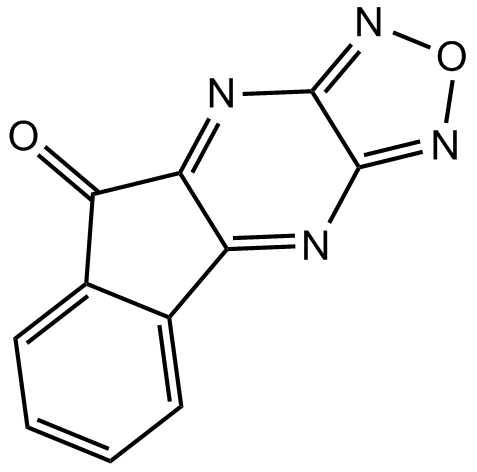
-
GC11869
SZL P1-41
SZL P1-41 est un inhibiteur spécifique de Skp2, se lie au domaine F-box de Skp2 pour empêcher l'association Skp1 et la formation du complexe Skp2 SCF. SZL P1-41, comme le déficit en Skp2, augmente l'apoptose/sénescence médiée par p27, alors qu'il altère la glycolyse induite par Akt. Activités anti-tumorales.