Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Targets for Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1145)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Products for Apoptosis
- Cat.No. Nom du produit Informations
-
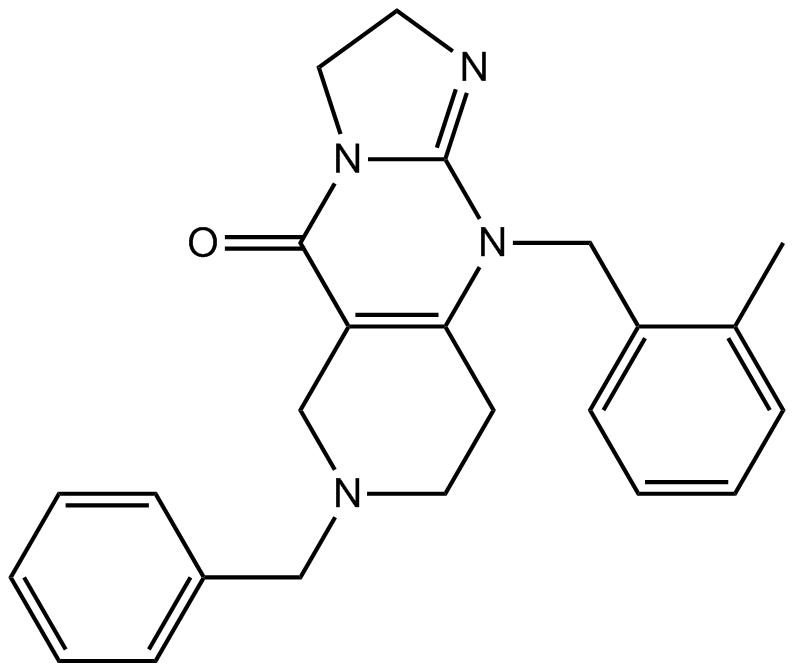
GC10350
TIC10 isomer
L'isomère TIC10 est l'isomère de TIC10.
-
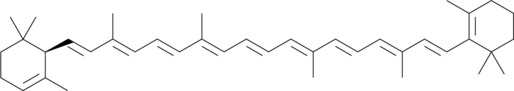
GC41183
α-Carotene
α-Le carotène, précurseur de la vitamine A, est utilisé comme agent anti-métastatique ou comme adjuvant pour les médicaments anticancéreux. α-Le carotène est isolé À partir de légumes jaune-orange et vert foncé.
-
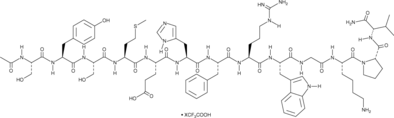
GC48292
α-MSH (human, mouse, rat, porcine, bovine, ovine) (trifluoroacetate salt)
α-MSH (α-Melanocyte-Stimulating Hormone) TFA, un neuropeptide endogène, est un agoniste endogène du récepteur 4 de la mélanocortine (MC4R) avec des activités anti-inflammatoires et antipyrétiques.
-
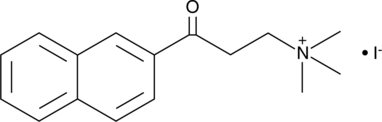
GC45213
α-NETA
α-NETA est un inhibiteur puissant et non compétitif de la choline acétyltransférase (ChA; IC50 \u003d 76 μM) et de la cholinestérase (ChE; IC50 \u003d 40 μM). α-NETA inhibe faiblement l'acétylcholinestérase (AChE ; IC50 \u003d 1 mM).
-
GC41499
α-Phellandrene
α-Phellandrene is a cyclic monoterpene that has been found in various plants, including Cannabis, and has diverse biological activities.
-
GC63941
α-Solanine
Il a été observé que la α-solanine, un composant bioactif et l'un des principaux glycoalcaloÏdes stéroÏdiens de la pomme de terre, inhibe la croissance et induit l'apoptose des cellules cancéreuses.
-
GC67618
α-Tocopherol phosphate disodium
α-Le phosphate de tocophérol (phosphate d'alpha-tocophérol) disodique, un antioxydant prometteur, peut protéger contre la mort cellulaire induite par les UVA1 à ondes longues et piéger les ROS induites par les UVA1 dans un modèle de cellule cutanée. α ; - Le phosphate de tocophérol disodique possède un potentiel thérapeutique dans l'inhibition de l'apoptose et augmente la capacité migratoire des cellules progénitrices endothéliales dans des conditions de glucose élevé/hypoxique et favorise l'angiogenèse.
-
GC48920
β-Carboline-1-carboxylic Acid
An alkaloid with diverse biological activities
-
GC41623
β-Elemonic Acid
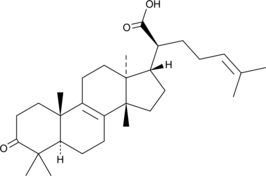
β ; - L'acide élémonique est un triterpène isolé de Boswellia papyrifera.
-
GC64619
β-Ionone
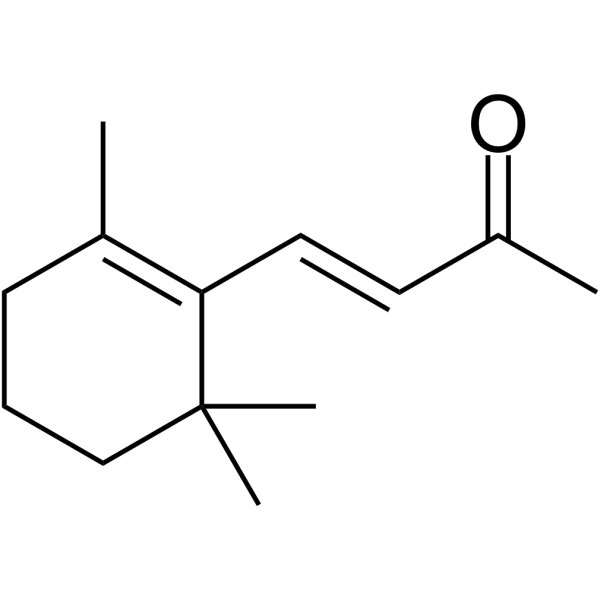
β-Ionone est efficace dans l'induction de l'apoptose dans les cellules d'adénocarcinome gastrique SGC7901. Activité anticancéreuse.
-
GC66048
δ-Secretase inhibitor 11
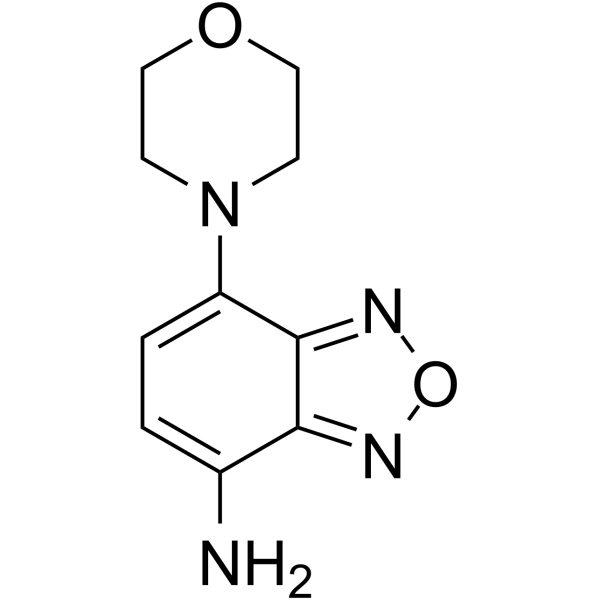
δ-L'inhibiteur de sécrétase 11 (composé 11) est un &7#948;-inhibiteur de sécrétase actif par voie orale, puissant, pénétré dans la BHE, non toxique, sélectif et spécifique, avec une IC50 de 0,7 μM. δ-l'inhibiteur de sécrétase 11 interagit À la fois avec le site actif et le site allostérique de la δ-sécrétase. δ ; - L'inhibiteur de sécrétase 11 atténue le clivage de tau et de l'APP (protéine précurseur de l'amyloÏde). δ-L'inhibiteur de sécrétase 11 améliore le dysfonctionnement synaptique et les troubles cognitifs dans les modèles de souris transgéniques tau P301S et 5XFAD. δ ; - L'inhibiteur de sécrétase 11 peut être utilisé pour la recherche sur la maladie d'Alzheimer.
-
GC46008
(±)-Thalidomide-d4
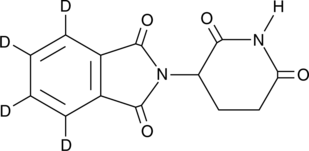
(±)-Thalidomide-d4 est un deutérium marqué Thalidomide.
-
GC45618
(±)-trans-GK563
A GVIA iPLA2 inhibitor
-
GC45270
(±)10(11)-EDP Ethanolamide
(±)10(11)-EDP ethanolamide is an ω-3 endocannabinoid epoxide and cannabinoid (CB) receptor agonist (EC50s = 0.43 and 22.5 nM for CB1 and CB2 receptors, respectively).
-
GC49268
(+)-δ-Cadinene
A sesquiterpene with antimicrobial and anticancer activities
-
GC18516
(+)-Aeroplysinin-1
(+)-Aeroplysinin-1 ((+)-Aeroplysinin-1), un métabolite secondaire isolé des éponges marines, montre de puissants effets antibiotiques sur les bactéries Gram-positives et exerce une activité antivirale contre le VIH-1 (IC50 \u003d 14,6 μM).
-
GC17008
(+)-Apogossypol
(+)-Apogossypol est un antagoniste pan-BCL-2. (+)-Apogossypol se lie À Mcl-1, Bcl-2 et Bcl-xL avec des EC50 de 2,6, 2,8 et 3,69 μM, respectivement.
-
GC45256
(+)-ar-Turmerone
(+)-ar-Turmerone ((+)-(+)-ar-Turmerone) est un composé bioactif majeur de l'herbe Curcuma longa avec des activités anti-tumorigenèse et anti-inflammatoires.
-
GN10654
(+)-Corynoline
-
GC31691
(+)-DHMEQ
(+)-DHMEQ est un activateur du facteur de transcription antioxydant Nrf2.
-
GC45265
(+)-Goniothalesdiol
(+)-Goniothalesdiol, isolated from the bark of the Malaysian tree G.

-
GC45274
(+)-Pinoresinol
(+) - Le pinorésinol est un lignol d'origine végétale servant de défense chez une chenille. (+) - Le pinorésinol sensibilise considérablement les cellules cancéreuses contre l'apoptose induite par le ligand induisant l'apoptose liée au TNF (TRAIL).
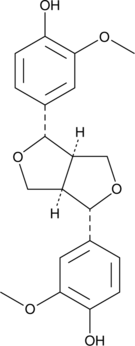
-
GC18749
(+)-Rugulosin
(+)-La rugulosine est une matière colorante cristalline de Penicillium rugulosum Thom.
-
GC41345
(-)-α-Bisabolol
(-)-α-Bisabolol ((-)-α-Bisabolol), un alcool sesquiterpénique monocyclique, exerce des activités antioxydantes, anti-inflammatoires et anti-apoptotique.
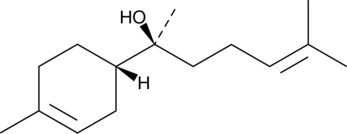
-
GC49502
(-)-β-Sesquiphellandrene
A sesquiterpene with antiviral and anticancer activities
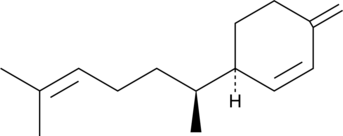
-
GC45244
(-)-(α)-Kainic Acid (hydrate)
Un puissant stimulant du système nerveux central pour l'induction de crises convulsives.

-
GC45246
(-)-Chaetominine
La (-)-chétominine est un métabolite alcaloïde. La (-)-chaetomine a une cytotoxicité contre la leucémie humaine K562 et les lignées cellulaires SW1116 du cancer du côlon. La (-)-chaetomine réduit la résistance aux médicaments médiée par MRP1 en inhibant la voie de signalisation PI3K/Akt/Nrf2 dans les cellules leucémiques humaines K562/Adr.
-
GC40698
(-)-Perillyl Alcohol
L'alcool (-)-périllylique est un monoterpène présent dans la lavande, inhibe la farnésylation de Ras, régule À la hausse le récepteur du mannose-6-phosphate et induit l'apoptose. Activité anticancéreuse.
-
GC40076
(-)-Voacangarine
(-)-Voacangarine is an indole alkaloid originally isolated from V.
-
GC62193
(1S,2S)-Bortezomib
Le (1S,2S)-bortézomib est un énantiomère du bortézomib. Le bortézomib est un inhibiteur du protéasome perméable aux cellules, réversible et sélectif, et inhibe puissamment le protéasome 20S (Ki de 0,6 nM) en ciblant un résidu thréonine. Le bortézomib perturbe le cycle cellulaire, induit l'apoptose et inhibe le NF-κB. Le bortézomib est un agent anticancéreux et le premier inhibiteur thérapeutique du protéasome À être utilisé chez l'homme.
-
GC34965
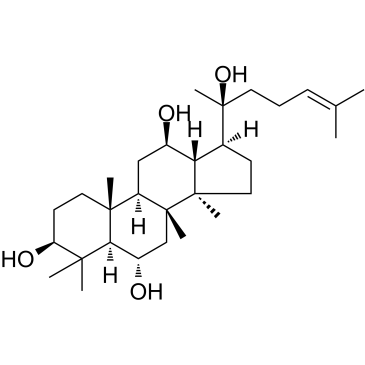
(20S)-Protopanaxatriol
Un métabolite actif de ginsénoside
-
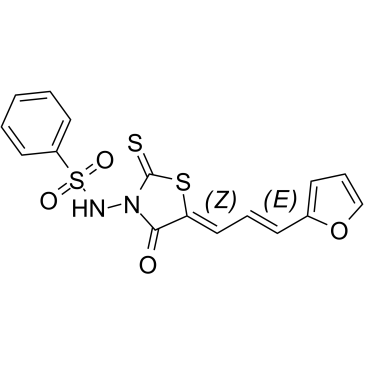
GC60397
(5Z,2E)-CU-3
(5Z,2E)-CU-3 est un inhibiteur puissant et sélectif contre l'isozyme α de DGK avec une valeur IC50 de 0,6 μM, inhibe de manière compétitive l'affinité de DGKα pour l'ATP avec une valeur Km de 0,48 mM. (5Z,2E)-CU-3 cible la région catalytique, mais pas la région régulatrice de DGKα. (5Z,2E)-CU-3 a des effets antitumoraux et proimmunogènes, améliore l'apoptose des cellules cancéreuses et l'activation des cellules T.
-
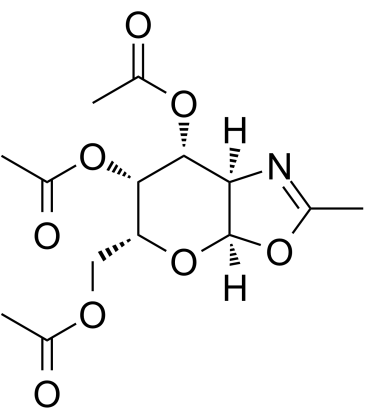
GC60398
(6R)-FR054
(6R)-FR054 est un isomère moins actif de FR054.
-
GC50482
(D)-PPA 1
PD-1/PD-L1 interaction inhibitor

-
GA20156
(D-Ser(tBu)⁶,Azagly¹⁰)-LHRH (free base)

-
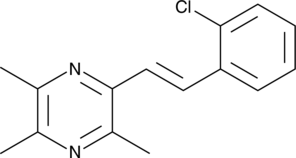
GC41700
(E)-2-(2-Chlorostyryl)-3,5,6-trimethylpyrazine
(E)-2-(2-Chlorostyryl)-3,5,6-trimethylpyrazine (CSTMP) is a stilbene derivative with antioxidant and anticancer activities.
-
GC41268
(E)-2-Hexadecenal
Sphingosine-1-phosphate (S1P), a bioactive lipid involved in many signaling processes, is irreversibly degraded by the membrane-bound S1P lyase.
-
GC41701
(E)-2-Hexadecenal Alkyne
(E)-2-Hexadecenal alkyne is an alkyne version of the sphingolipid degradation product (E)-2-hexadecenal that can be used as a click chemistry probe.

-
GC34980
(E)-Ferulic acid
L'acide (E)-férulique est un isomère de l'acide férulique qui est un composé aromatique, abondant dans les parois cellulaires des plantes. L'acide (E)-férulique provoque la phosphorylation de la β-caténine, entraÎnant une dégradation protéasomique de la β-caténine et augmente l'expression du facteur pro-apoptotique Bax et diminue l'expression du facteur pro-survie survivine. L'acide (E)-férulique montre une puissante capacité À éliminer les espèces réactives de l'oxygène (ROS) et inhibe la peroxydation des lipides. L'acide (E)-férulique exerce À la fois des effets anti-prolifération et anti-migration dans la lignée cellulaire H1299 du cancer du poumon humain.
-
GC34981
(E)-Flavokawain A
La (E)-FlavokawaÏne A, une chalcone extraite du kava, a un effet anticancérogène. La (E)-flavokawaÏne A induit l'apoptose dans les cellules cancéreuses de la vessie par l'implication de la voie apoptotique dépendante de la protéine bax et dépendante des mitochondries et supprime la croissance tumorale chez la souris.
-
GC61437
(E)-Methyl 4-coumarate
(E)-Methyl 4-coumarate (Methyl 4-hydroxycinnamate), trouvé dans plusieurs plantes, comme l'oignon vert (Allium cepa) ou le noni (Morinda citrifolia L.
-
GC34125
(E)-[6]-Dehydroparadol
Le (E)-[6]-déhydroparadol, un métabolite oxydatif du [6]-shogaol, est un puissant activateur de Nrf2. Le (E)-[6]-déhydroparadol peut inhiber la croissance et induire l'apoptose des cellules cancéreuses humaines.
![(E)-[6]-Dehydroparadol Chemical Structure (E)-[6]-Dehydroparadol Chemical Structure](/media/struct/GC3/GC34125.png)
-
GC49189
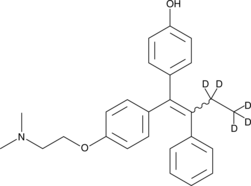
(E/Z)-4-hydroxy Tamoxifen-d5
An internal standard for the quantification of (E/Z)-4-hydroxy tamoxifen
-
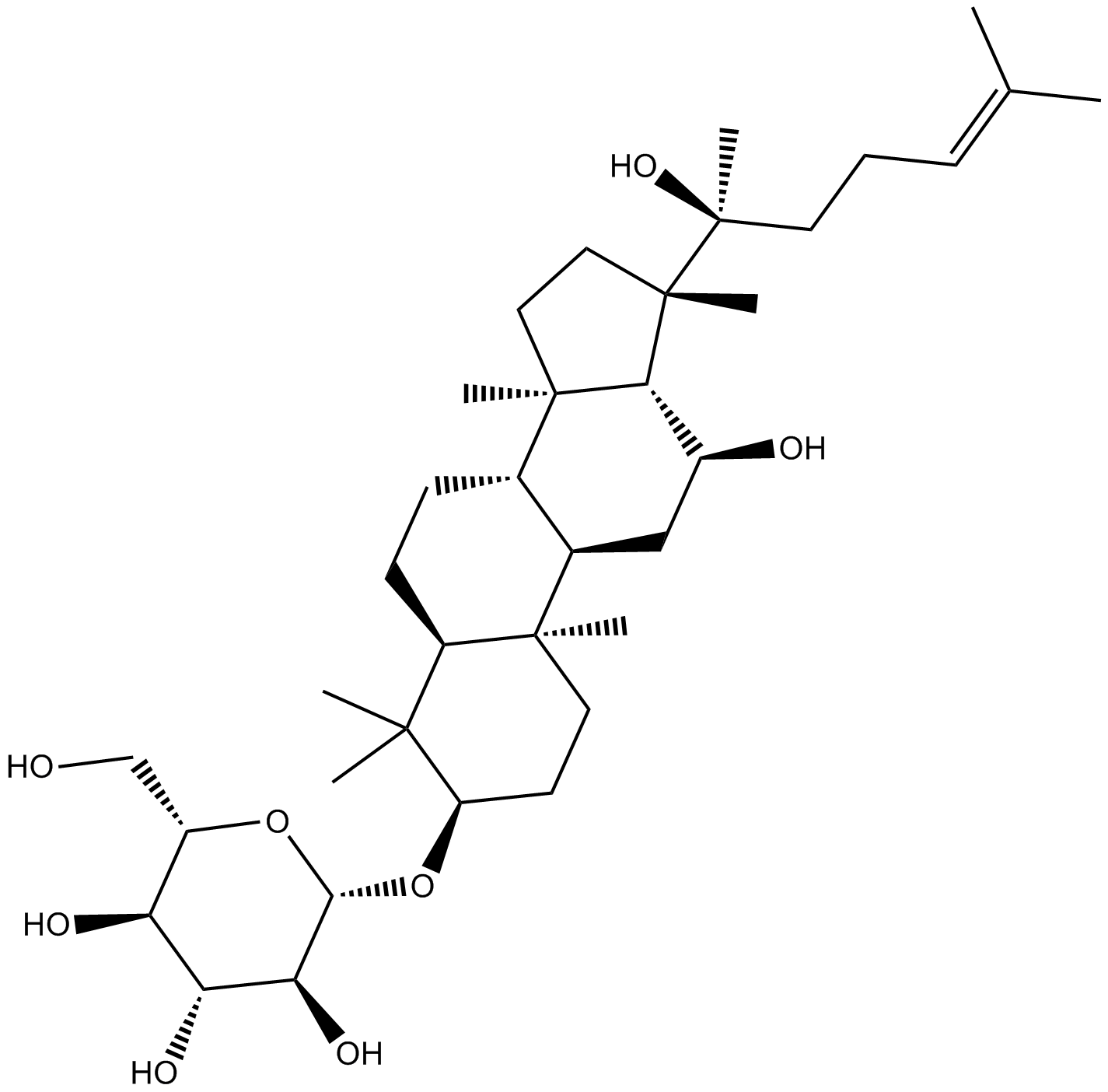
GN10783
(R) Ginsenoside Rh2
-
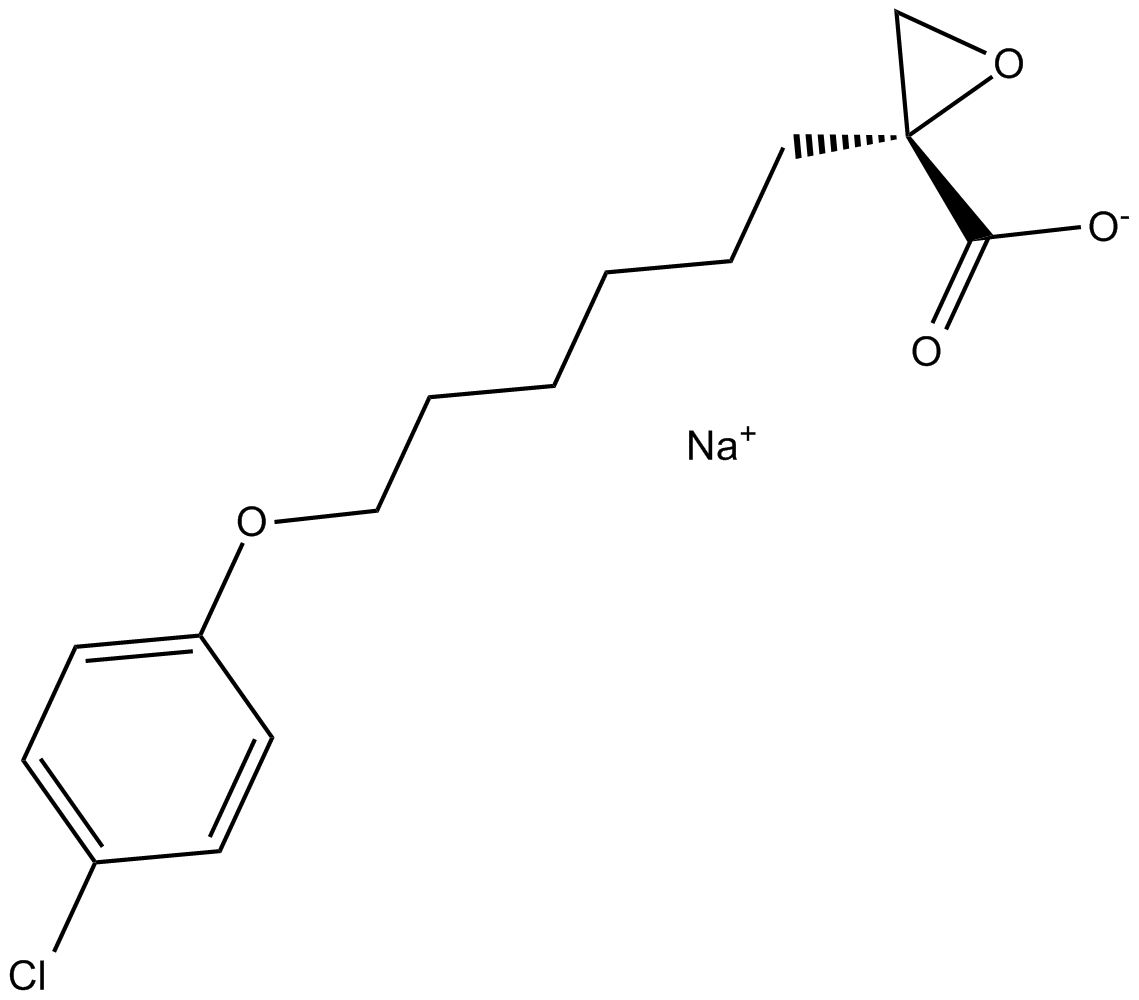
GC15104
(R)-(+)-Etomoxir sodium salt
Le sel de sodium d'Etomoxir((R)-(+)-Etomoxir) est un inhibiteur irréversible de la carnitine palmitoyltransférase 1a (CPT-1a), inhibe l'oxydation des acides gras (FAO) par CPT-1a et inhibe l'oxydation du palmitate β ; chez l'homme, le rat et cochon d'inde.
-
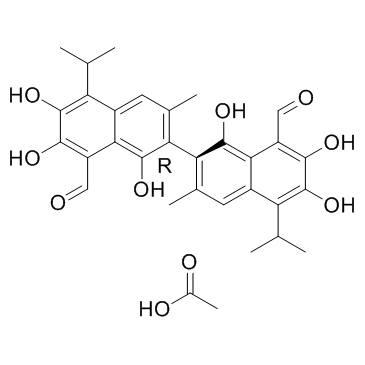
GC34096
(R)-(-)-Gossypol acetic acid (AT-101 (acetic acid))
L'acide (R)-(-)-Gossypol acétique (AT-101 (acide acétique)) (AT-101 (acide acétique)) est l'isomère lévogyre d'un produit naturel Gossypol. AT-101 est déterminé À se lier aux protéines Bcl-2, Mcl-1 et Bcl-xL avec Kis de 260± 30 nM, 170± 10 nM et 480± 40 nM, respectivement.
-
GC65610
(R)-5-Hydroxy-1,7-diphenyl-3-heptanone
La (R)-5-hydroxy-1,7-diphényl-3-heptanone est un diarylheptanoÏde présent dans Alpinia officinarum.

-
GC41716
(R)-CR8
(R)-CR8 (CR8), un analogue de deuxième génération de la Roscovitine, est un puissant inhibiteur de CDK1/2/5/7/9.
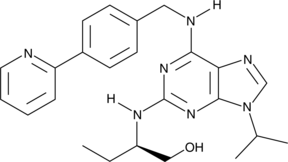
-
GC39281
(R)-CR8 trihydrochloride
Le trichlorhydrate de (R)-CR8 (CR8), un analogue de deuxième génération de la roscovitine, est un puissant inhibiteur de CDK1/2/5/7/9.
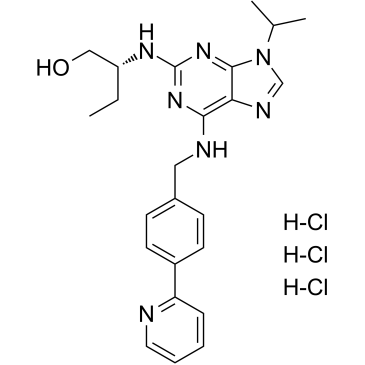
-
GC41719
(R)-nitro-Blebbistatin
(R)-nitro-Blebbistatin is a more stable form of (+)-blebbistatin, which is the inactive form of (-)-blebbistatin.
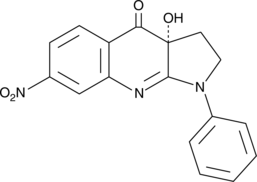
-
GC60407
(R)-Verapamil D7 hydrochloride
Le chlorhydrate de (R)-vérapamil D7 ((R)-(+)-chlorhydrate de vérapamil D7) est un chlorhydrate de (R)-vérapamil marqué au deutérium. Le chlorhydrate de (R)-vérapamil ((R)-(+)-chlorhydrate de vérapamil) est un inhibiteur de la glycoprotéine P. Le chlorhydrate de (R)-vérapamil bloque le transport médié par MRP1, entraÎnant une chimiosensibilisation des cellules surexprimant MRP1 aux médicaments anticancéreux.
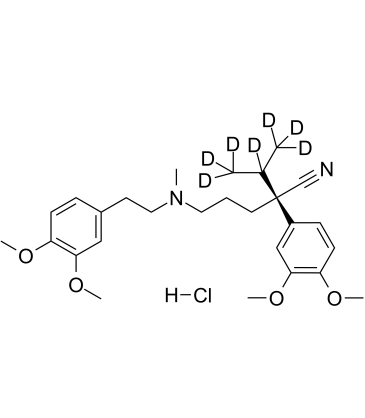
-
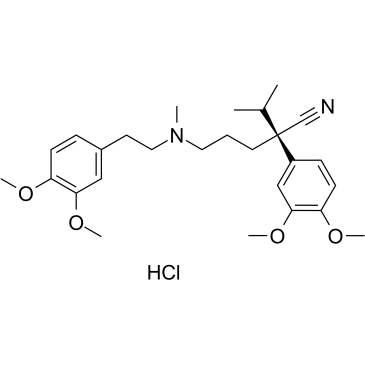
GC60408
(R)-Verapamil hydrochloride
Le chlorhydrate de (R)-vérapamil ((R)-(+)-chlorhydrate de vérapamil) est un inhibiteur de la glycoprotéine P. Le chlorhydrate de (R)-vérapamil bloque le transport médié par MRP1, entraÎnant une chimiosensibilisation des cellules surexprimant MRP1 aux médicaments anticancéreux.
-
GC19541
(rac)-Antineoplaston A10
(rac)-Antinéoplaston A10 est le racémate de l'Antinéoplaston A10. L'antinéoplaston A10 est un inhibiteur de Ras potentiellement pour le traitement du gliome, du lymphome, de l'astrocytome et du cancer du sein.

-
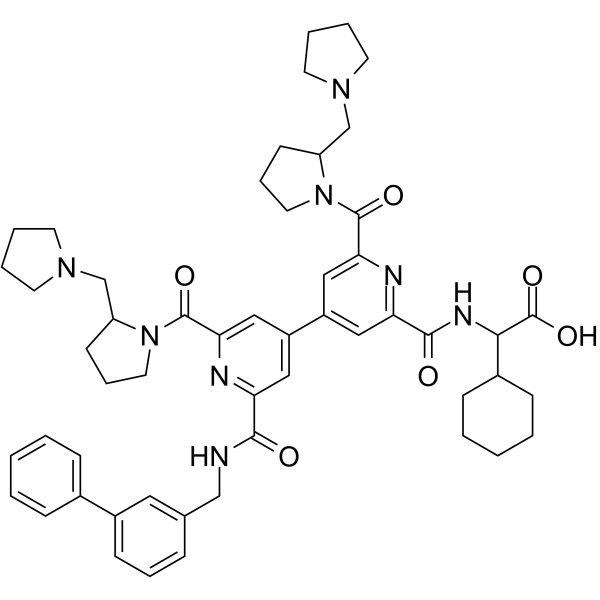
GC69795
(Rac)-BIO8898
(Rac)-BIO8898 est un inhibiteur de l'interaction co-stimulatrice CD40-CD154. (Rac)-BIO8898 inhibe la liaison de CD154 à CD40-Ig, avec une IC50 de 25 μM.
-
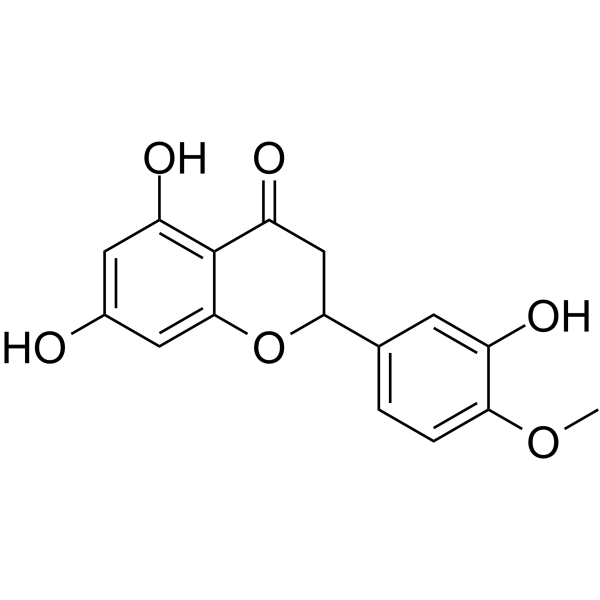
GC62528
(Rac)-Hesperetin
(Rac)-Hesperetin est le racémate de l'Hesperetin.
-
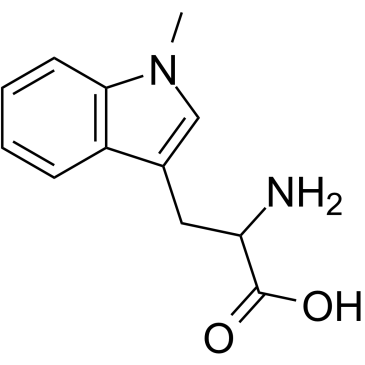
GC61750
(Rac)-Indoximod
Le (Rac)-indoximod (1-méthyl-DL-tryptophane) est un inhibiteur de l'indoleamine 2,3-dioxygénase (IDO).
-
GC10098
(S)-10-Hydroxycamptothecin
La (S)-10-hydroxycamptothécine (10-HCPT;10-hydroxycamptothécine) est un inhibiteur de l'ADN topoisomérase I isolé de la plante chinoise Camptotheca accuminata. La (S)-10-hydroxycamptothécine présente un effet inducteur d'apoptose remarquable. La (S)-10-hydroxycamptothécine a le potentiel de traiter l'hépatome, le carcinome gastrique, le cancer du cÔlon et la leucémie.
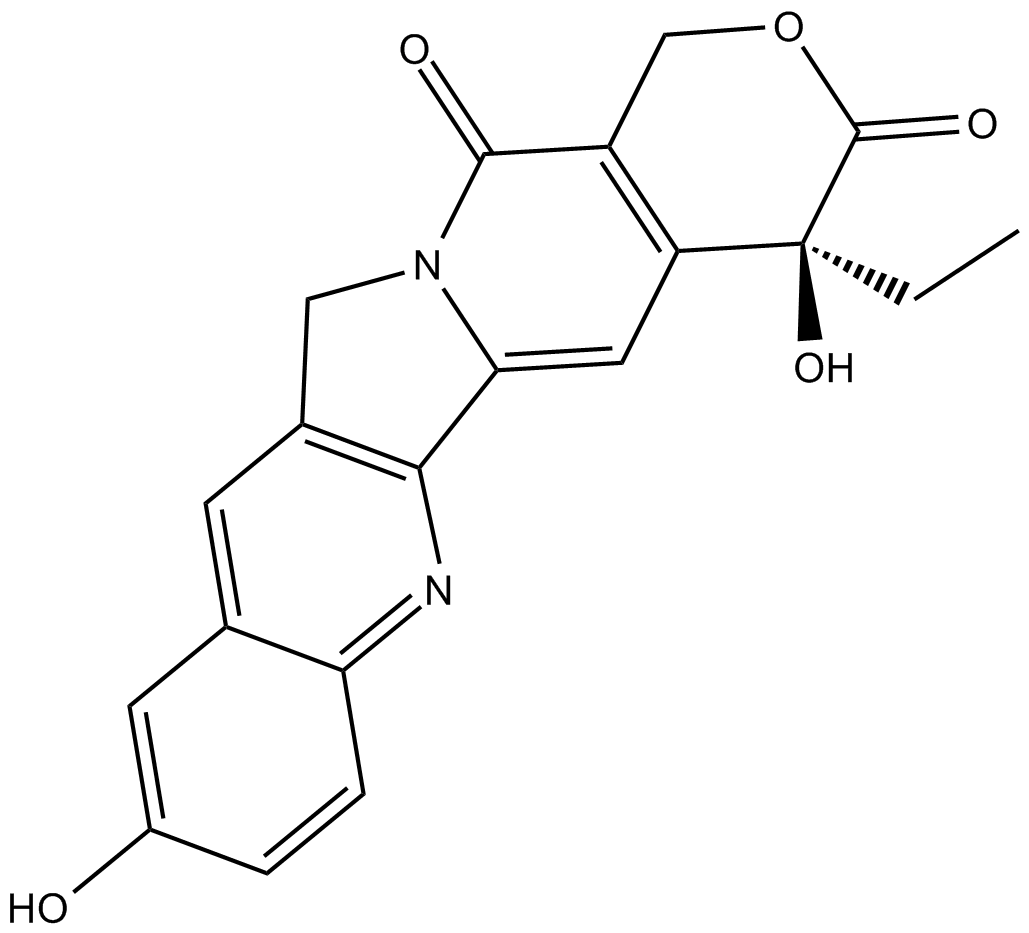
-
GC41557
(S)-3'-amino Blebbistatin
(S)-3'-amino Blebbistatin is a more stable and less phototoxic form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
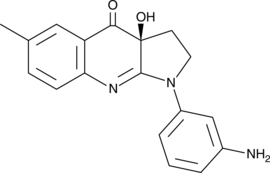
-
GC41484
(S)-3'-hydroxy Blebbistatin
(S)-3'-hydroxy Blebbistatin is a more stable and less phototoxic form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
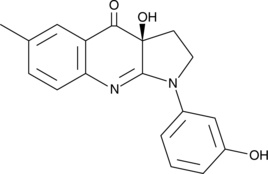
-
GC52192
(S)-4'-nitro-Blebbistatin
(S)-4'-nitro-Blebbistatin est un inhibiteur non cytotoxique, photostable, fluorescent et spécifique de la myosine II, utilisé dans l'étude du rÔle spécifique de la myosine II dans les études biologiques physiologiques, développementales et cellulaires.
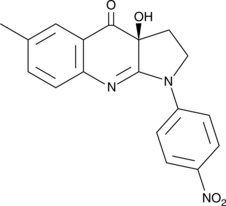
-
GC35001
(S)-Gossypol acetic acid
Le (S)-Gossypol est l'isomère d'un produit naturel Gossypol. Le (S)-gossypol se lie au sillon de liaison BH3 des protéines Bcl-xL et Bcl-2 avec une haute affinité.
-
GC41739
(S)-nitro-Blebbistatin
(S)-nitro-Blebbistatin is a more stable form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
-
GC60425
(S)-Verapamil D7 hydrochloride
Le chlorhydrate de (S)-vérapamil D7 (chlorhydrate de (S)-(-)-vérapamil D7) est un chlorhydrate de (S)-vérapamil marqué au deutérium. Le chlorhydrate de (S)-vérapamil (chlorhydrate de S(-)-vérapamil) inhibe le transport des leucotriènes C4 (LTC4) et de la calcéine par MRP1. Le chlorhydrate de (S)-vérapamil entraÎne la mort des cellules tumorales potentiellement résistantes.
-
GC60008
(S)-Verapamil hydrochloride
Le chlorhydrate de (S)-vérapamil (chlorhydrate de S(-)-vérapamil) inhibe le transport des leucotriènes C4 (LTC4) et de la calcéine par MRP1. Le chlorhydrate de (S)-vérapamil entraÎne la mort des cellules tumorales potentiellement résistantes.
-
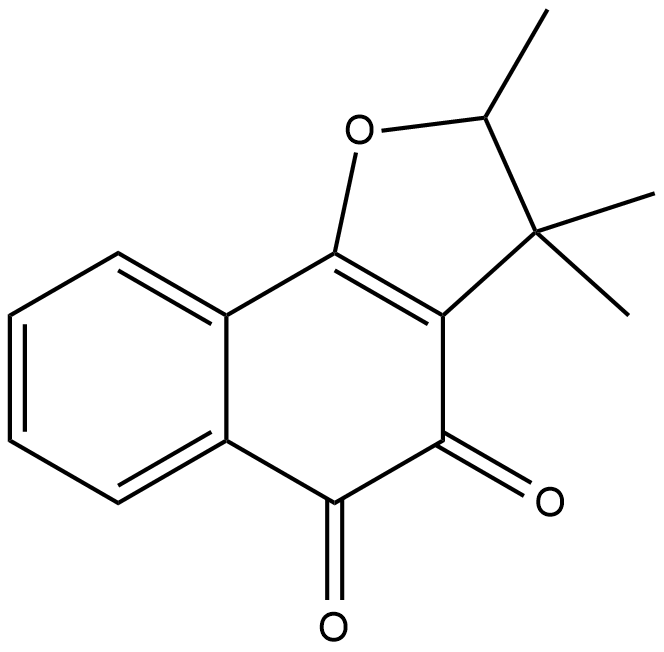
GC18787
(±)-Dunnione
(±)-Dunnione is a naturally occurring naphthoquinone with diverse biological activities.
-
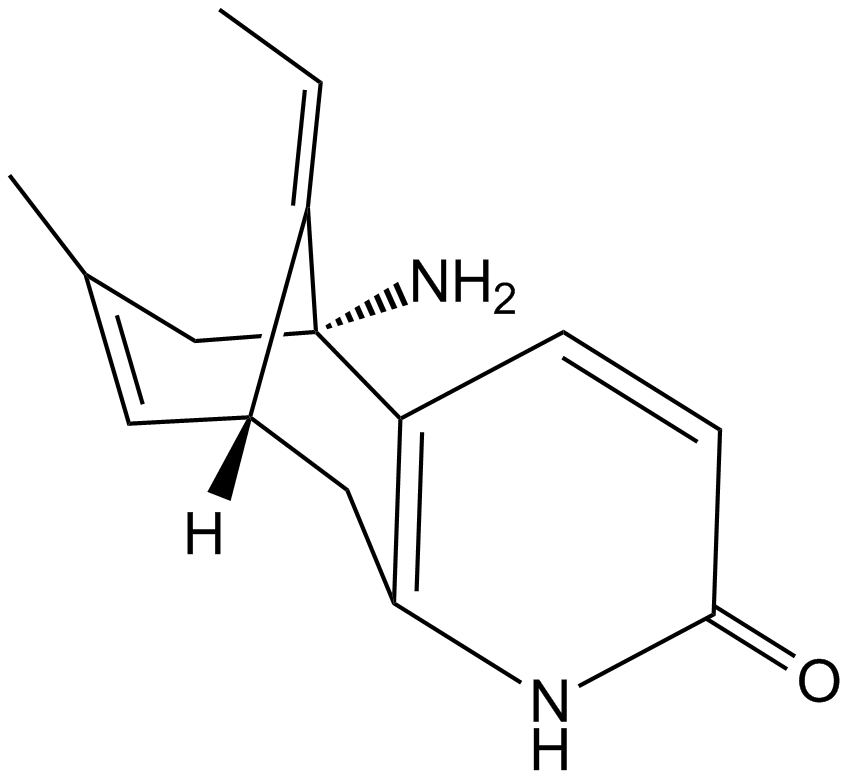
GC11965
(±)-Huperzine A
A neuroprotective AChE inhibitor
-
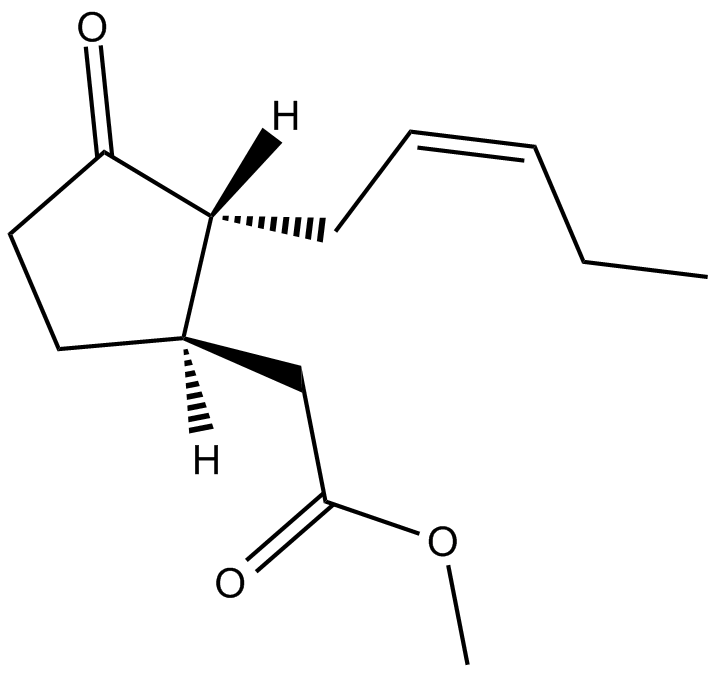
GC16375
(±)-Jasmonic Acid methyl ester
(±) - L'ester méthylique de l'acide jasmonique est un métabolite endogène.
-
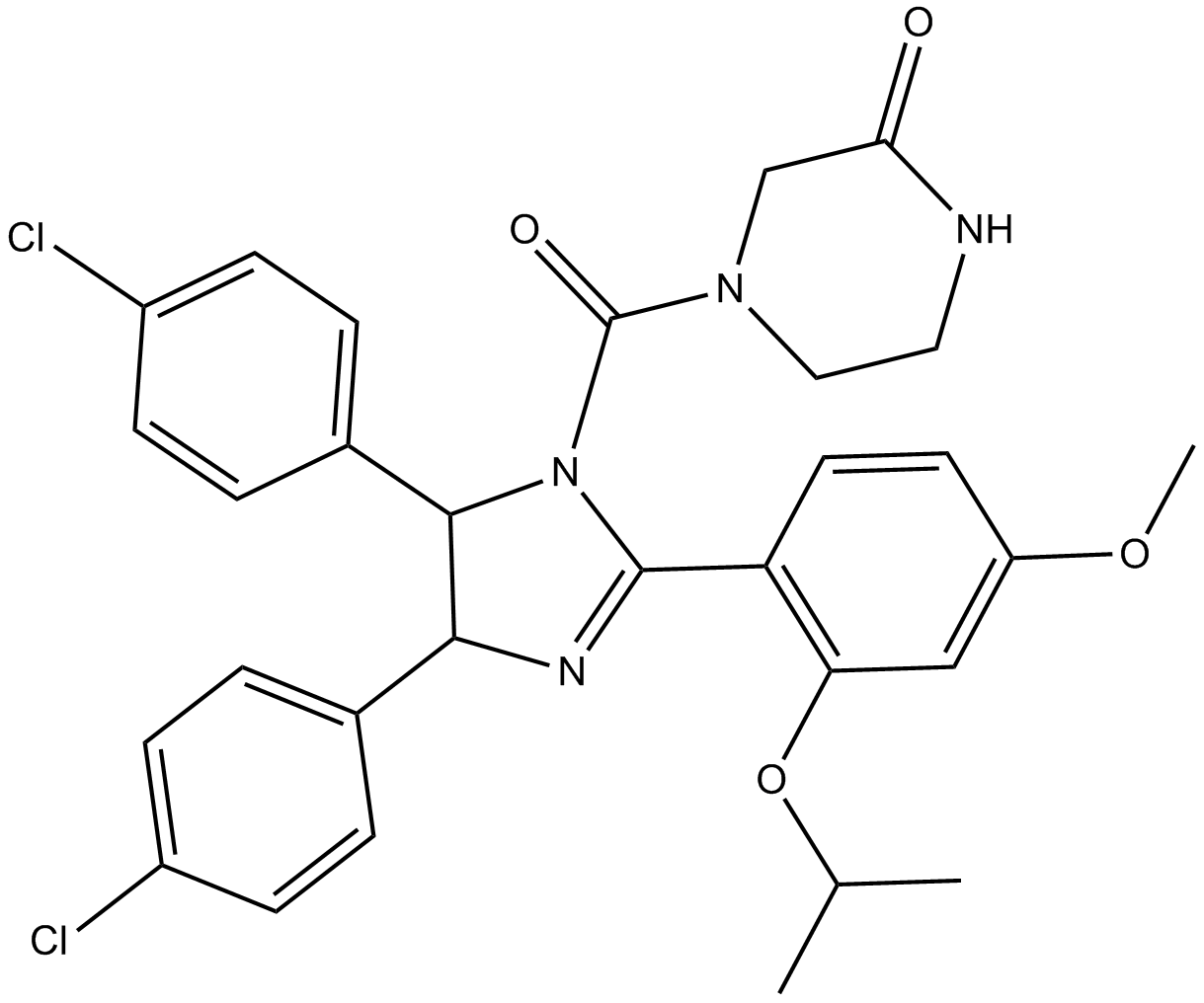
GC14154
(±)-Nutlin-3
MDM2 antagonist, potent and selective
-
GC46379
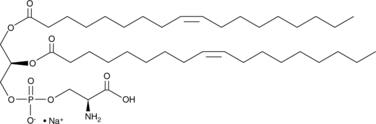
1,2-Dioleoyl-sn-glycero-3-PS (sodium salt)
Le 1,2-dioléoyl-sn-glycéro-3-PS (sel de sodium) est un substitut de la phosphosérine/phosphatidylsérine.
-
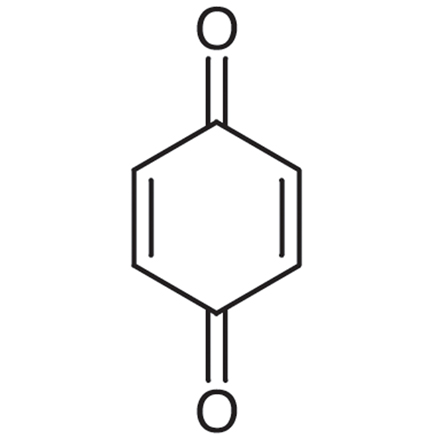
GC19528
1,4-Benzoquinone
A toxic metabolite of benzene
-
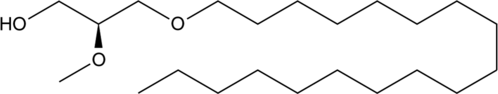
GC42018
1-O-Octadecyl-2-O-methyl-sn-glycerol
1-O-Octadecyl-2-O-methyl-sn-glycerol is a metabolite of a phosphotidylinositol ether lipid analog (PIA).
-
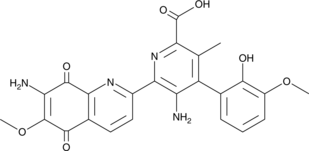
GC41865
10'-Desmethoxystreptonigrin
10'-Desmethoxystreptonigrin is an antibiotic originally isolated from Streptomyces and a derivative of the antibiotic streptonigrin.
-
GC49736
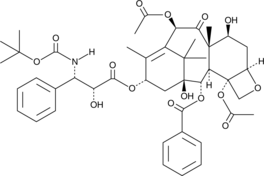
10-acetyl Docetaxel
Le 10-acétyl docétaxel (10-acétyl docétaxel) est un analogue du docétaxel, avec une activité anticancéreuse. Le docétaxel est un inhibiteur du désassemblage des microtubules, avec une activité antimitotique.
-
GC64726
10-Formyl-5,8-dideazafolic acid
L'acide 10-formyl-5,8-didéazafolique est un inhibiteur de la thymidylate synthase.

-
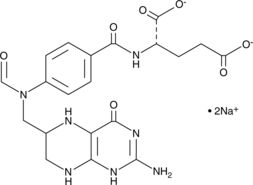
GC49872
10-Formyltetrahydrofolate (sodium salt) (technical grade)
Le 10-formyltétrahydrofolate (sel de sodium) (qualité technique) est une forme d'acide tétrahydrofolique qui agit comme donneur de groupes formyle dans l'anabolisme.
-
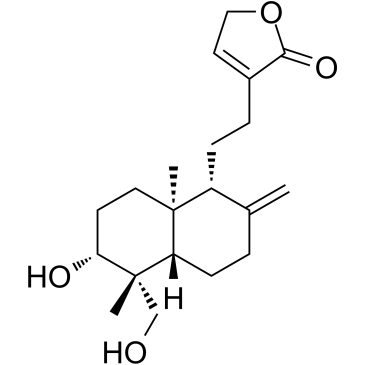
GC35057
14-Deoxyandrographolide
Le 14-désoxyandrographolide est un diterpène labdane ayant une activité de blocage des canaux calciques.
-
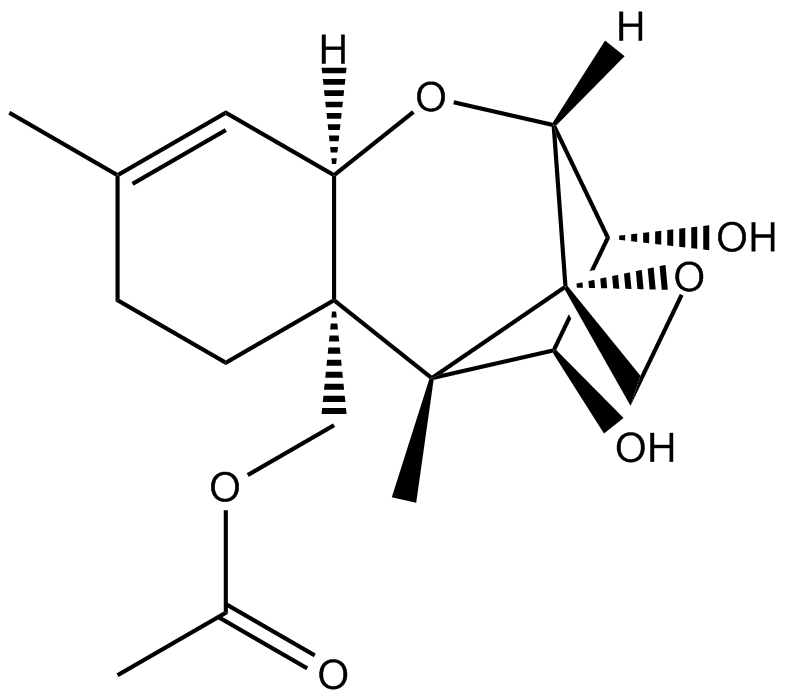
GC11988
15-acetoxy Scirpenol
Le 15-acétoxyscirpénol, l'une des mycotoxines du groupement acétoxyscirpénol (ASM), induit fortement l'apoptose et inhibe la croissance des lymphocytes T Jurkat de manière dose-dépendante en activant d'autres caspases indépendantes de la caspase-3.
-
GC41938
15-Lipoxygenase Inhibitor 1
L'inhibiteur de 15-lipoxygénase 1 est un inhibiteur sélectif de la 15-lipoxygénase, avec une CI50 de 18 μM. L'inhibiteur de la 15-lipoxygénase 1 a des CI50 de 19,5 μM et 19,1 μM pour la 15-lipoxygénase de soja (SLO) et la 15-lipoxygénase-1 humaine (15-LOX-1), respectivement. L'inhibiteur de la 15-lipoxygénase 1 a un potentiel pour la recherche sur le cancer de la prostate.
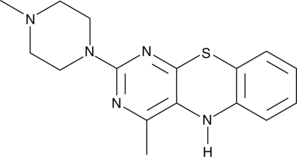
-
GC46451
16F16
A PDI inhibitor

-
GC11720
17-AAG (KOS953)
An inhibitor of Hsp90
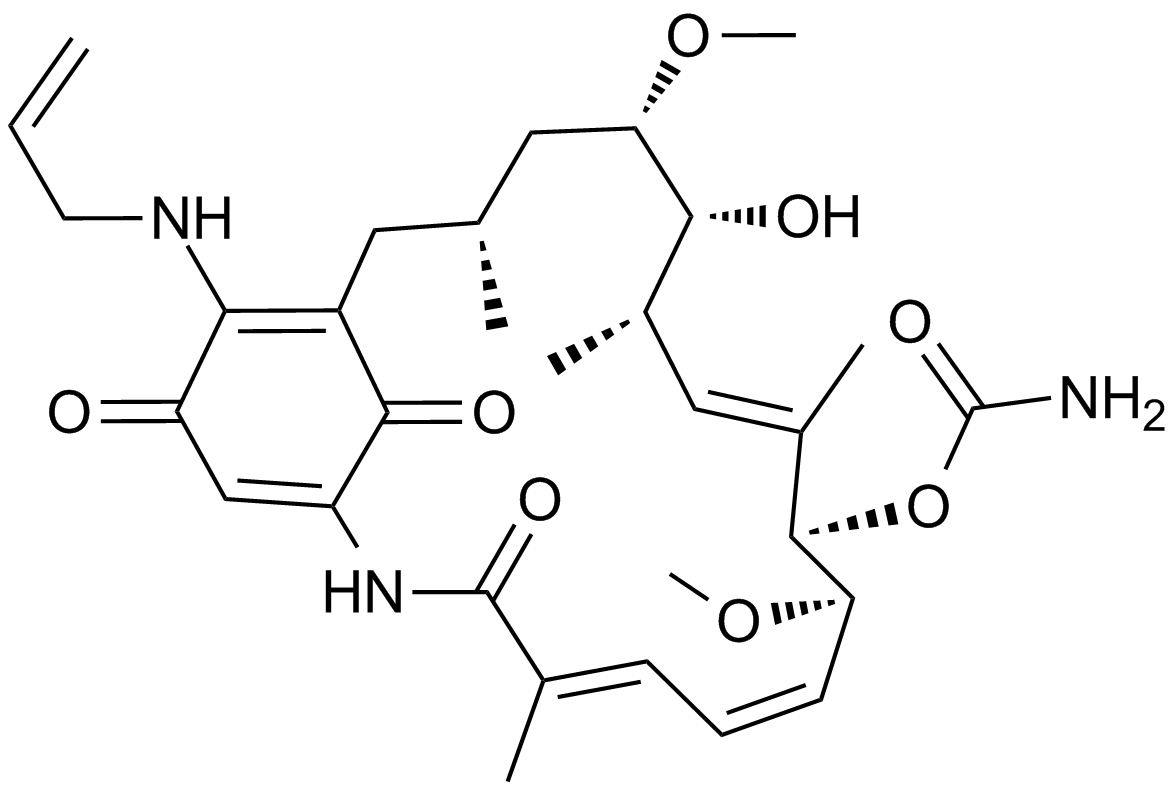
-
GC13044
17-DMAG (Alvespimycin) HCl
Le 17-DMAG (alvespimycine) HCl (chlorhydrate de 17-DMAG ; KOS-1022 ; BMS 826476) est un puissant inhibiteur de Hsp90, se liant À Hsp90 avec une EC50 de 62± ; 29 nM.

-
GC41983
19,20-Epoxycytochalasin D
La 19,20-époxycytochalasine D, une cytochalasine, est un métabolite fongique de Nemania sp.
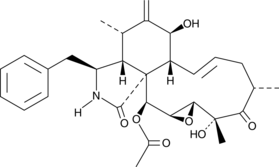
-
GC48423
19-O-Acetylchaetoglobosin A
La 19-O-acétylchaetoglobosine A, un alcaloÏde du cytochalasane, est un métabolite fongique isolé À l'origine de C. globosum qui a des activités inhibitrices et cytotoxiques de la polymérisation de l'actine. La 19-O-acétylchaetoglobosine A est cytotoxique pour les cellules cancéreuses cervicales HeLa.
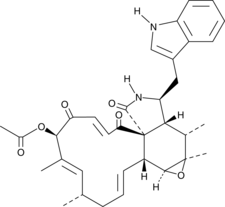
-
GC39296
1G244
1G244 est un puissant inhibiteur de DPP8/9 avec des IC50 de 12 nM et 84 nM, respectivement. 1G244 n'inhibe pas DPPIV et DPPII. 1G244 induit l'apoptose dans les cellules de myélome multiple et a des effets anti-myélome.
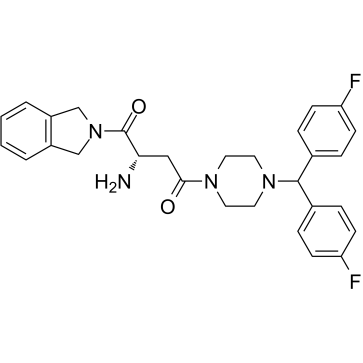
-
GC46508
2',2'-Difluoro-2'-deoxyuridine
An active metabolite of gemcitabine
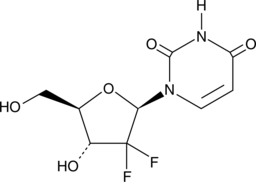
-
GC41612
2'-O-Methylguanosine
La 2'-O-méthylguanosine est un nucléoside modifié produit dans les ARNt par l'action de l'ARNt guanosine-2'-O-méthyltransférase.
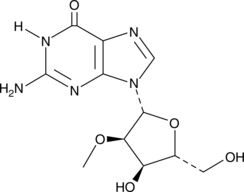
-
GC12258
2,3-DCPE hydrochloride
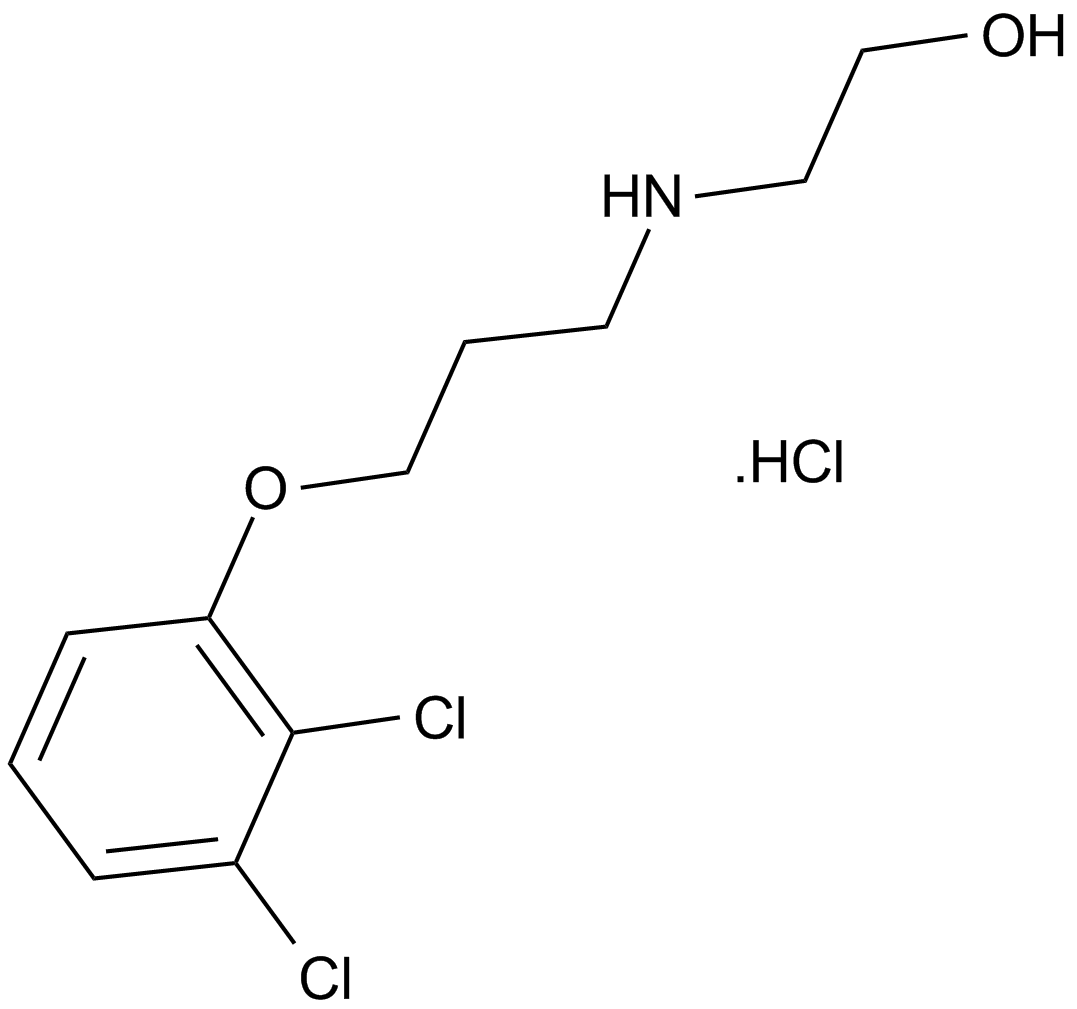
-
GC40947
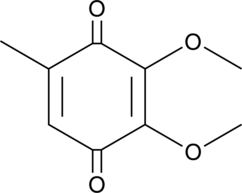
2,3-Dimethoxy-5-methyl-p-benzoquinone
La 2,3-diméthoxy-5-méthyl-p-benzoquinone (CoQ0) est un puissant composé d'ubiquinone actif oral qui peut être dérivé d'Antrodia cinnamomea.
-
GC68452
2,4,6-Triiodophenol

-
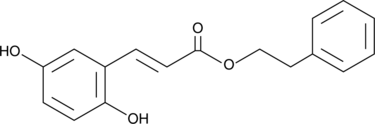
GC46057
2,5-Dihydroxycinnamic Acid phenethyl ester
An inhibitor of 5-LO
-
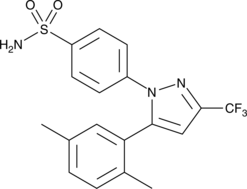
GC45324
2,5-dimethyl Celecoxib
-
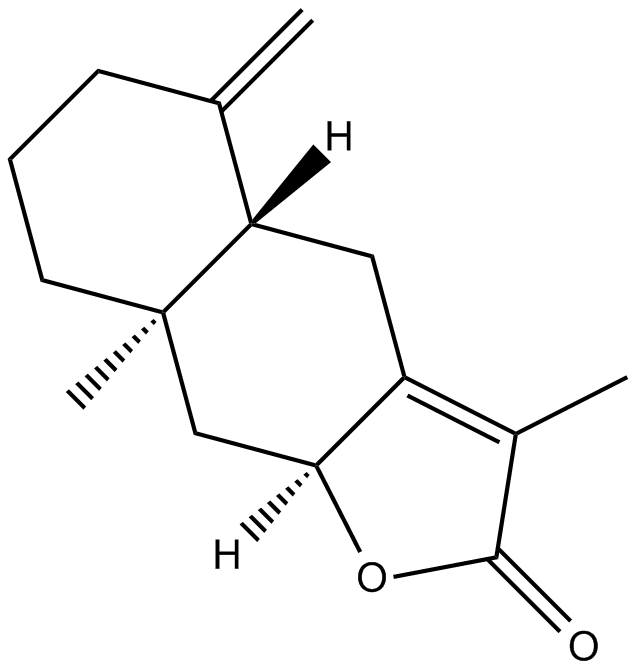
GN10065
2-Atractylenolide
-
GC40675
2-deoxy-Artemisinin
2-deoxy-Artemisinin is an inactive metabolite of the antimalarial agent artemisinin.
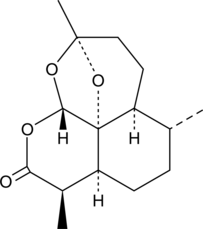
-
GC17430
2-Deoxy-D-glucose
2-Désoxy-D-glucose (2DG) est un analogue du glucose qui agit comme inhibiteur glycolytique compétitif.
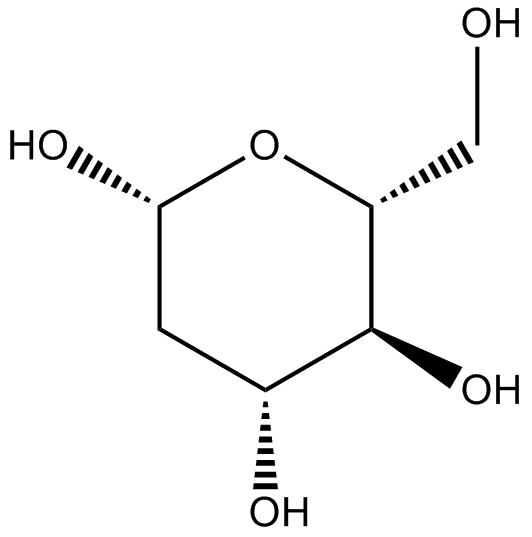
-
GC49223
2-deoxy-D-Glucose-13C6
An internal standard for the quantification of 2-deoxy-D-glucose
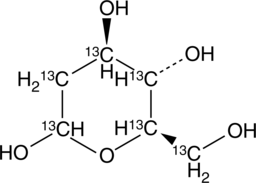
-
GC46545
2-Fluoroadenine
La 2-fluoroadénine est une base purique toxique. La 2-fluoroadénine a une toxicité dans les cellules tumorales non proliférantes et proliférantes. La 2-fluoroadénine peut être utilisée pour la recherche anticancéreuse.
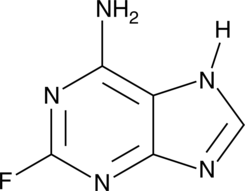
-
GC12545
2-HBA
Le 2-HBA est un puissant inducteur de la NAD(P)H:accepteur de quinone oxydoréductase 1 (NQO1) qui peut également activer la caspase-3 et la caspase-10.
-
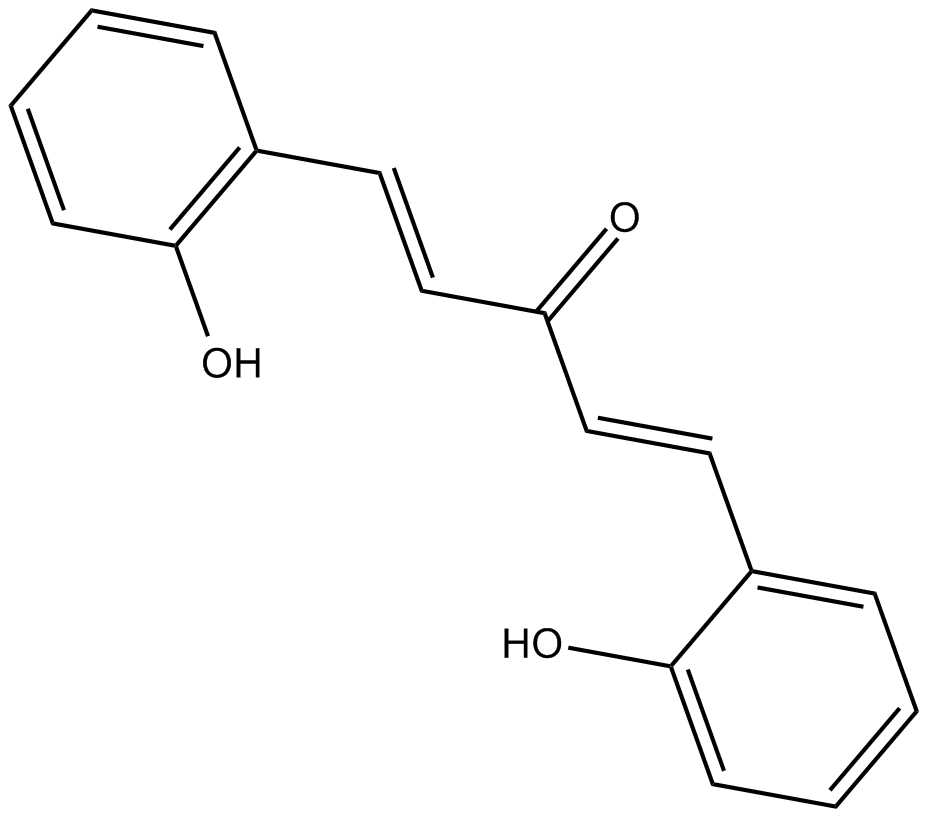
GC38318
2-Methoxycinnamaldehyde
Le 2-méthoxycinnamaldéhyde (o-méthoxycinnamaldéhyde) est un composé naturel de Cinnamomum cassia, avec une activité antitumorale. Le 2-méthoxycinnamaldéhyde inhibe la prolifération et induit l'apoptose par la perte du potentiel de membrane mitochondriale (δψm), l'activation de la caspase-3 et de la caspase-9. Le 2-méthoxycinnamaldéhyde inhibe efficacement la migration des HASMC induite par le facteur de croissance dérivé des plaquettes (PDGF).
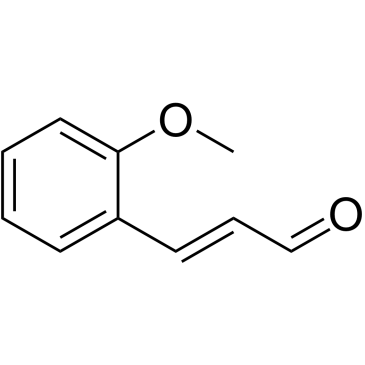
-
GC15084
2-Methoxyestradiol (2-MeOE2)
2-Methoxyestradiol (2-MeOE2/2-Me) is a HIF-1α inhibitor that inhibits HIF-1α accumulation and HIF transcriptional activity. 2-Methoxyestradiol can trigger p53-induced apoptosis and has potential antitumor activity..
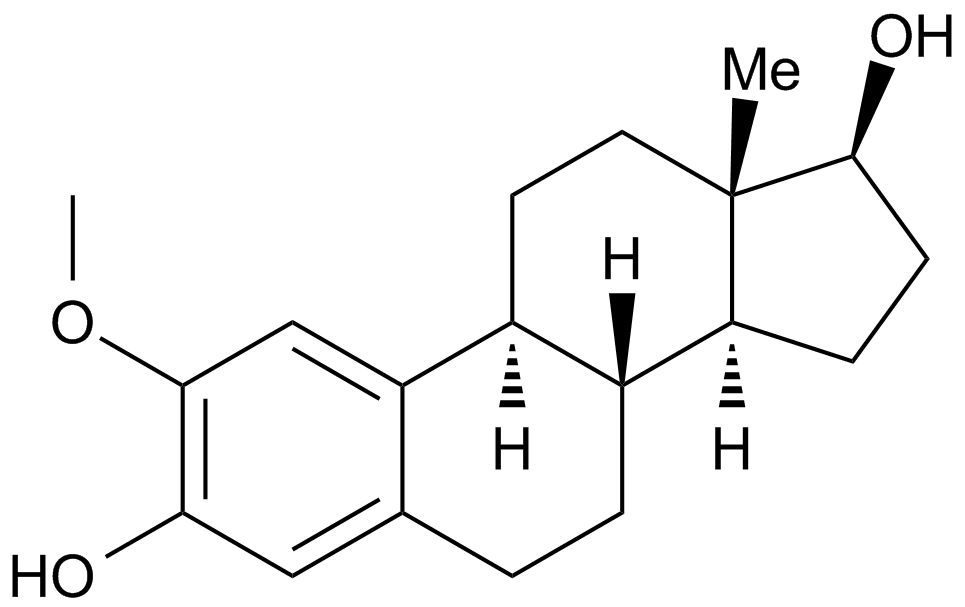
-
GC68043
2-tert-Butyl-1,4-benzoquinone



