DNA Damage/DNA Repair
- MTH1(4)
- PARP(61)
- ATM/ATR(27)
- DNA Alkylating(19)
- DNA Ligases(3)
- DNA Methyltransferase(18)
- DNA-PK(27)
- HDAC(99)
- Nucleoside Antimetabolite/Analogue(146)
- Telomerase(14)
- Topoisomerase(134)
- tankyrase(5)
- Antifolate(32)
- CDK(234)
- Checkpoint Kinase (Chk)(29)
- CRISPR/Cas9(9)
- Deubiquitinase(64)
- DNA Alkylator/Crosslinker(65)
- DNA/RNA Synthesis(410)
- Eukaryotic Initiation Factor (eIF)(23)
- IRE1(22)
- LIM Kinase (LIMK)(10)
- TOPK(6)
- Casein Kinase(55)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Products for DNA Damage/DNA Repair
- Cat.No. Nom du produit Informations
-
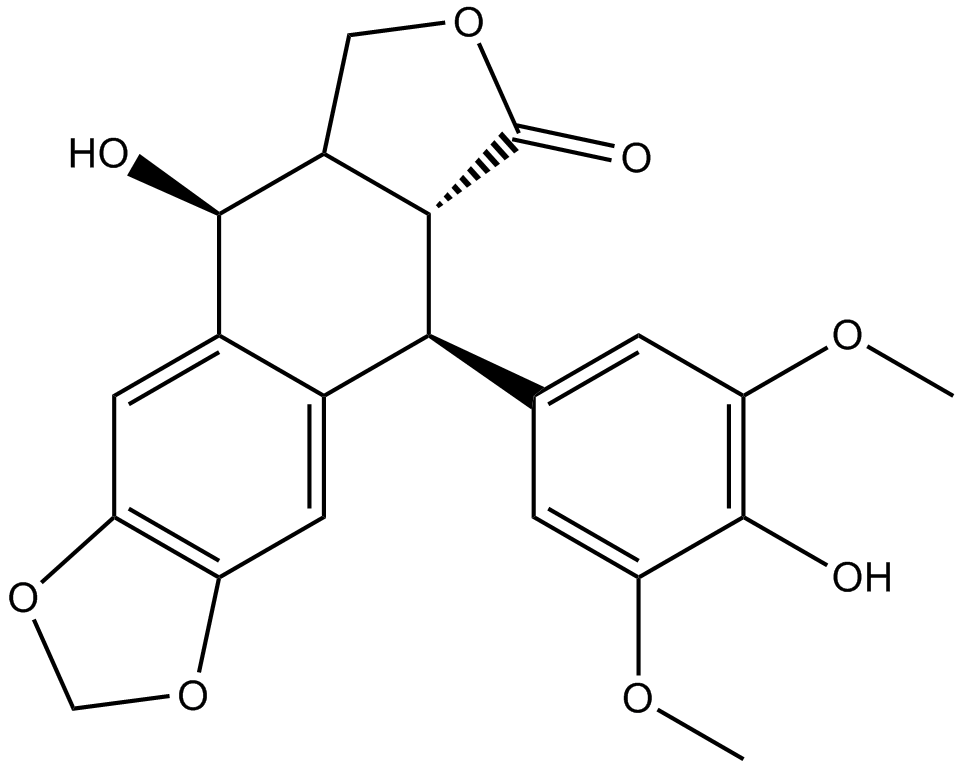
GC38000
β-Boswellic acid
β ; - L'acide boswellique est isolé de la résine de gomme de Boswellia serrate.β ; - L'acide boswellique est un inhibiteur de type non réducteur de la formation de produit de 5-lipoxygénase (5-LO) qui interagit directement avec le 5-LO ou bloque sa translocation . β-L'acide boswellique inhibe la synthèse d'ADN, d'ARN et de protéines dans les cellules de leucémie humaine HL-60.
-
GC31365
γ-Oryzanol
γ-Oryzanol est un puissant inhibiteur de l'ADN méthyltransférase (DNMT) dans le striatum des souris.
-
GC38194
(±)-10-Hydroxycamptothecin
(M)-10-Hydroxycamptothécine est un alcaloïde indole qui inhibe l'activité de la topoisomérase I et possède un large spectre d'activité anticancéreuse.
-
GC33107
(±)-BAY-1251152
(±)-BAY-1251152; (±)-VIP152
(±)-BAY-1251152 ((±)-BAY-1251152) est un mélange racémique de BAY-1251152. BAY-1251152 est un inhibiteur puissant et hautement sélectif de PTEF/CDK9.
-
GC10867
(+)-Aphidicolin
ICI 69653, NSC 234714
L'aphidicolin ((+)-Aphidicolin), un inhibiteur réversible de la réplication de l'ADN nucléaire eucaryote, peut bloquer le cycle cellulaire à la phase pré-S[1].
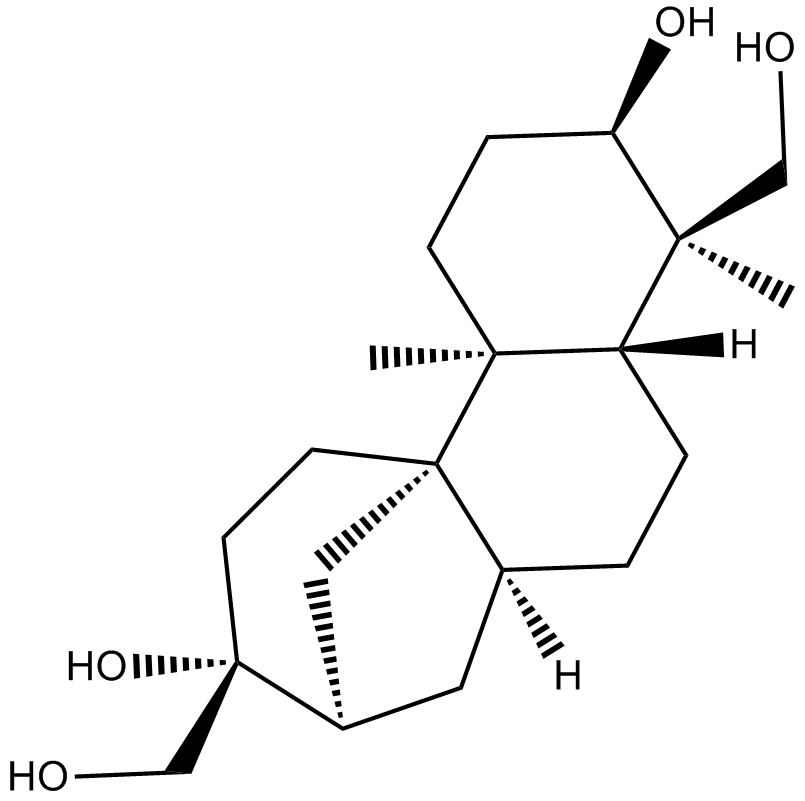
-
GC34955
(+)-CBI-CDPI1
(+)-CBI-CDPI1 est un analogue fonctionnel amélioré du CC-1065. (+)-CBI-CDPI1 est un agent alkylant de l'ADN. (+)-CBI-CDPI1 est une toxine de conjugués anticorps-médicaments (ADC).
-
GC34956
(+)-CBI-CDPI2
(+)-CBI-CDPI2 est un analogue fonctionnel amélioré du CC-1065. (+)-CBI-CDPI1 est un agent alkylant de l'ADN. (+)-CBI-CDPI2 est une toxine de conjugués anticorps-médicaments (ADC).
-
GC32429
(-)-BAY-1251152
(-)-BAY-1251152; (-)-VIP152
(-)-BAY-1251152 ((-)-BAY-1251152) est un énanthimère de BAY-1251152 avec rotation (-). BAY-1251152 est un inhibiteur puissant et hautement sélectif de PTEF/CDK9.
-
GC48635
(-)-Cryptopleurine
(R)-Cryptopleurine, NSC 19912
An alkaloid with diverse biological activities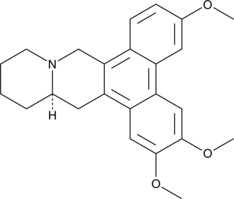
-
GC40076
(-)-Voacangarine
NSC 306219, (-)-Voacristine
(-)-Voacangarine is an indole alkaloid originally isolated from V.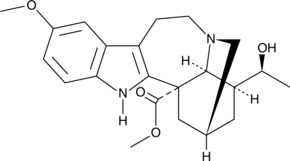
-
GC38377
(2S,3R)-Voruciclib hydrochloride
Le chlorhydrate de (2S,3R)-Voruciclib est l'énantiomère du chlorhydrate de Voruciclib. Le (2S,3R)-Voruciclib est un inhibiteur de CDK actif par voie orale.
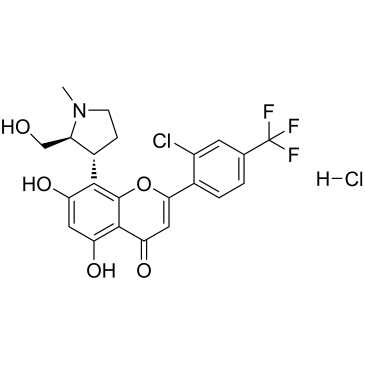
-
GC41695
(6R,S)-5,6,7,8-Tetrahydrofolic Acid (hydrochloride)
Tetrahydrofolate, THFA
(6R,S)-5,6,7,8-Tetrahydrofolic acid (THFA), the reduced form of folic acid, serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.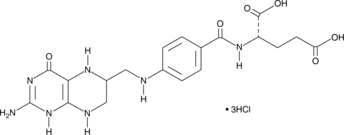
-
GC41088
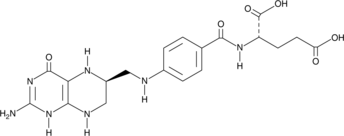
(6S)-Tetrahydrofolic Acid
(6S)-Tetrahydrofolic acid is a diastereomer of tetrahydrofolic acid, a reduced form of folic acid that serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
-
GC72591
(E,E)-RGFP966
(E,E)-RGFP966 is a selective and CNS permeable HDAC3 inhibitor that can be used for the research of Huntington’s disease.

-
GC62735
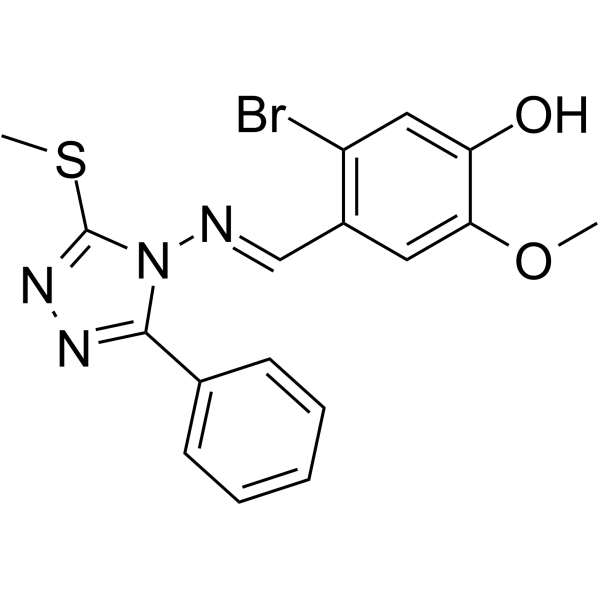
(E/Z)-GO289
(E/Z)-GO289 est un inhibiteur puissant et sélectif de la caséine kinase 2 (CK2) (IC50 = 7 nM). (E/Z)-GO289 allonge fortement la période circadienne. (E/Z)-GO289 présente une inhibition dépendante du type de cellule de la croissance des cellules cancéreuses qui est corrélée avec la fonction de l'horloge cellulaire.
-
GC64429
(E/Z)-Zotiraciclib citrate
(E/Z)-TG02 citrate; (E/Z)-SB1317 citrate
Le citrate de (E/Z)-zotiraciclib est un puissant inhibiteur de CDK2, JAK2 et FLT3.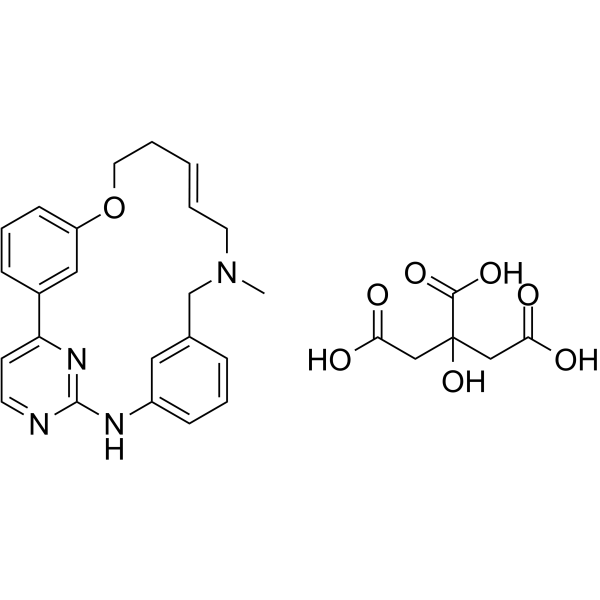
-
GC63864
(E/Z)-Zotiraciclib hydrochloride
(E/Z)-TG02 hydrochloride; (E/Z)-SB1317 hydrochloride
Le chlorhydrate de (E/Z)-Zotiracilib ((E/Z)-TG02) est un puissant inhibiteur de CDK2, JAK2 et FLT3.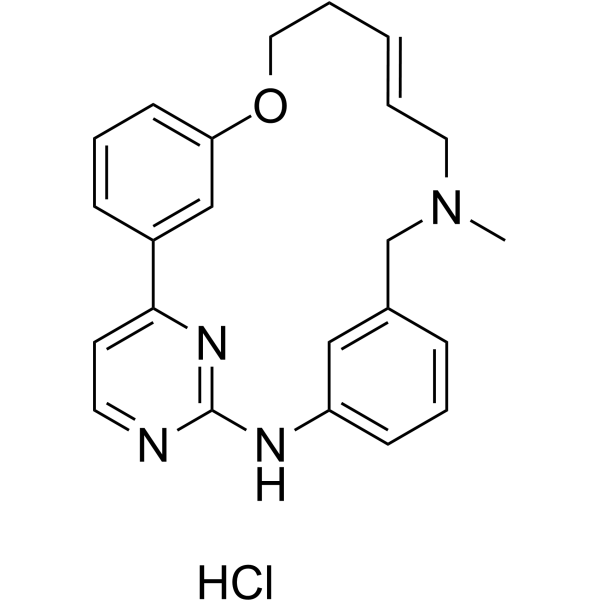
-
GC69793
(R)-5-O-Benzoyl-1,2-di-O-isopropylidene-alpha-D-xylofuranose
(R)-5-O-Benzoyl-1,2-di-O-isopropylidene-alpha-D-xylofuranose est un analogue de nucléoside purique. Les analogues de nucléosides puriques ont une activité anticancéreuse étendue et ciblent les tumeurs malignes du système lymphatique inerte. Le mécanisme anticancéreux dans ce processus dépend de l'inhibition de la synthèse d'ADN, l'induction de l'apoptose cellulaire, etc.
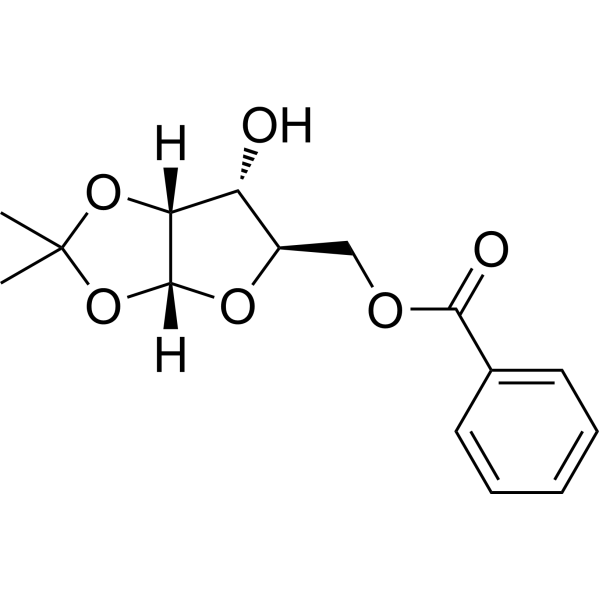
-
GC41716
(R)-CR8
(R)-CR8 (CR8), un analogue de deuxième génération de la Roscovitine, est un puissant inhibiteur de CDK1/2/5/7/9.
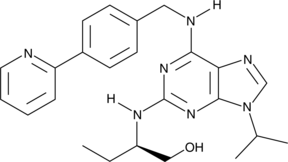
-
GC39281
(R)-CR8 trihydrochloride
CR8, (R)-Isomer trihydrochloride
Le trichlorhydrate de (R)-CR8 (CR8), un analogue de deuxième génération de la roscovitine, est un puissant inhibiteur de CDK1/2/5/7/9.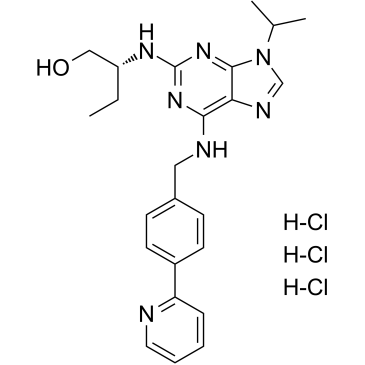
-
GC64210
(R)-GSK-3685032
(R)-GSK-3685032 est l'énantiomère R de GSK-3685032. GSK-3685032 est un inhibiteur sélectif de la DNMT1 réversible, non dépendant du temps et non covalent, avec une IC50 de 0,036 μM. GSK-3685032 induit une perte robuste de méthylation de l'ADN, une activation de la transcription et une inhibition de la croissance des cellules cancéreuses.
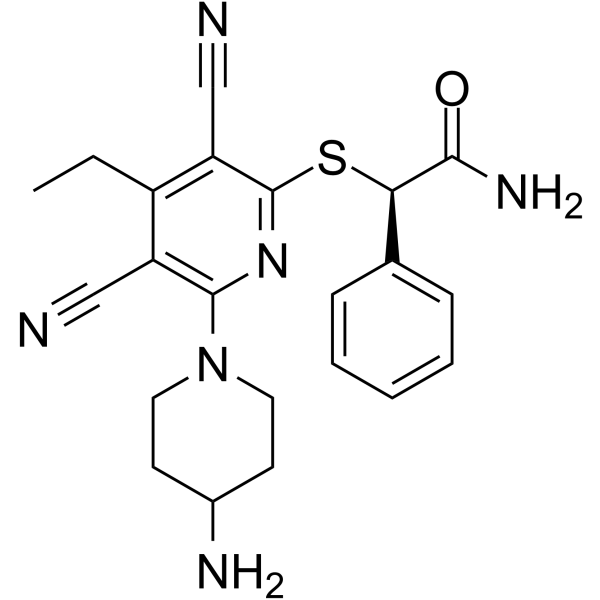
-
GC41633
(R)-Prunasin
(R)-Prunasin est un inhibiteur de l'ADN polymérase β.
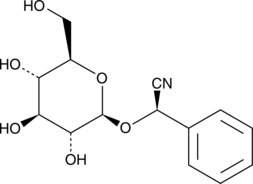
-
GC34124
(rel)-MC180295
(rel)-MC180295
(rel)-MC180295 ((rel)-(rel)-MC180295) est un inhibiteur puissant et sélectif de CDK9-Cycline T1, avec une IC50 de 5 nM, au moins 22 fois plus sélectif pour CDK9 que pour les autres CDK. (rel)-MC180295 inhibe également GSK-3α ; et GSK-3β ;. (rel)-MC180295 ((rel)-(rel)-MC180295) a un puissant effet antitumoral.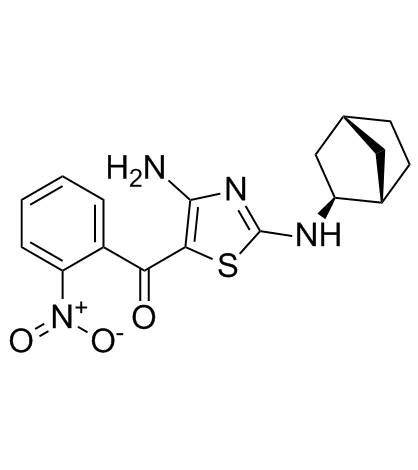
-
GC10098
(S)-10-Hydroxycamptothecin
ChEMBL 273862, NSC 107124
La (S)-10-hydroxycamptothécine (10-HCPT;10-hydroxycamptothécine) est un inhibiteur de l'ADN topoisomérase I isolé de la plante chinoise Camptotheca accuminata. La (S)-10-hydroxycamptothécine présente un effet inducteur d'apoptose remarquable. La (S)-10-hydroxycamptothécine a le potentiel de traiter l'hépatome, le carcinome gastrique, le cancer du cÔlon et la leucémie.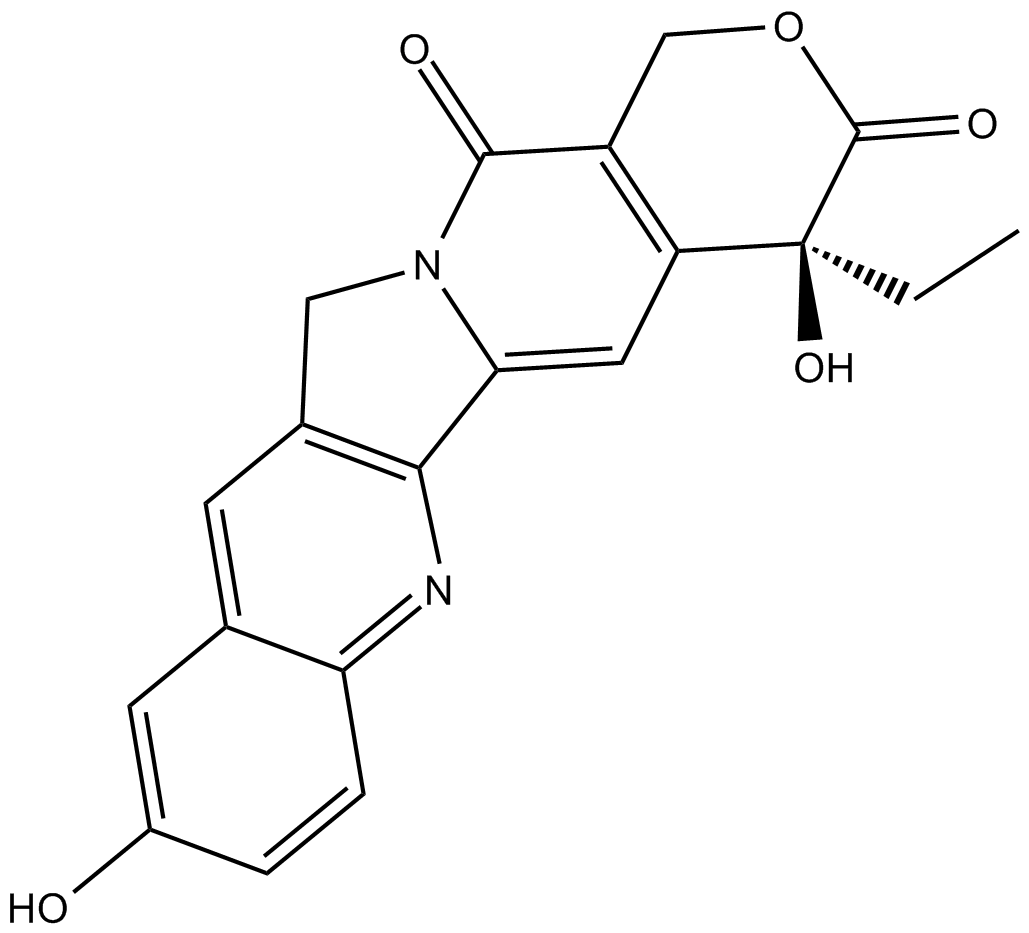
-
GC34999
(S)-Ceralasertib
(S)-AZD6738
Le (S)-ceralasertib ((S)-AZD6738) est extrait du brevet WO2011154737A1, composé II, présente une IC50 de 2,578 nM.(S)-ceralasertib est un inhibiteur puissant et sélectif de la sulfoximine morpholinopyrimidine ATR avec d'excellentes propriétés physicochimiques et pharmacocinétiques précliniques (PK ) caractéristiques.(S)-Ceralasertib est développé pour améliorer la solubilité aqueuse et élimine l'inhibition dépendante du temps du CYP3A4.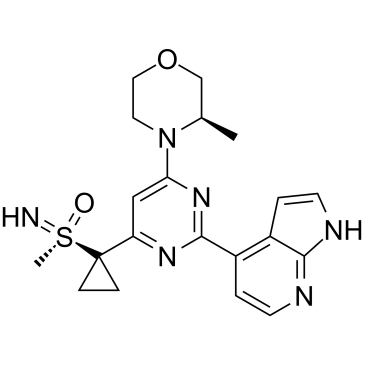
-
GC46351
(S)-CR8
(S)-CR8 est l'isomère S de CR8. (S)-CR8 est un inhibiteur de CDK puissant et sélectif avec des IC50 de 0,060, 0,080, 0,11, 0,12 et 0,15 μM pour CDK2/cycline E, CDK2/cycline A, CDK9/cycline T, CDK5/p25 et CDK1/cycline B, respectivement. (S)-CR8 réduit la survie des cellules SH-SY5Y (IC50 0,40μM).
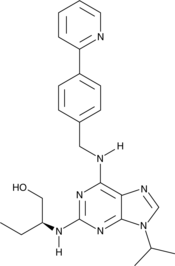
-
GC13136
(S)-Crizotinib
Le (S)-crizotinib est un inhibiteur puissant et sélectif de MTH1 (homologue mutT) avec une IC50 de 330 nM. Le (S)-crizotinib perturbe l'homéostasie du pool de nucléotides via l'inhibition de MTH1, induit une augmentation des cassures simple brin de l'ADN, active la réparation de l'ADN dans les cellules de carcinome du cÔlon humain et supprime efficacement la croissance tumorale dans les modèles animaux.
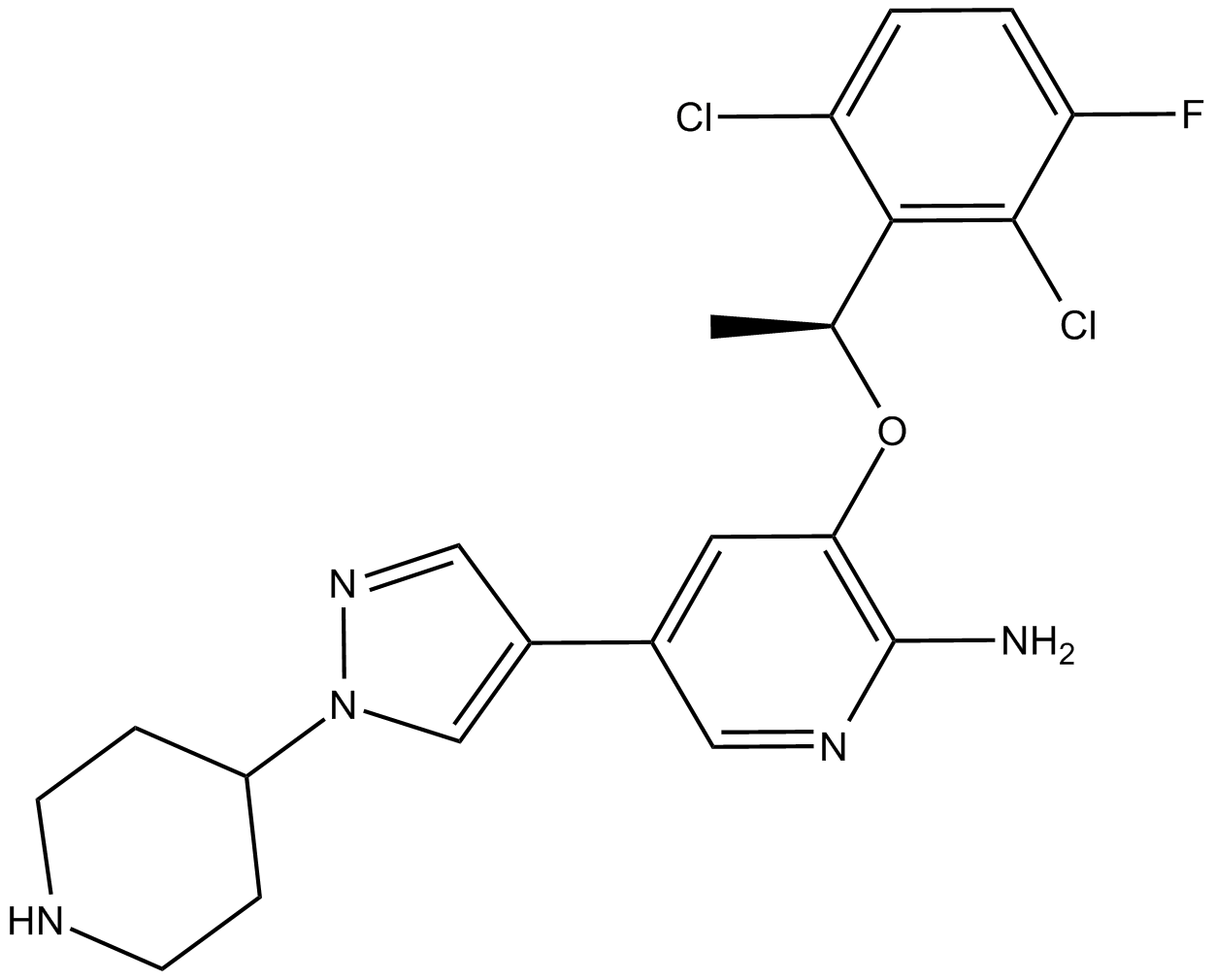
-
GC65877
(S)-GFB-12811
Le (S)-GFB-12811 (composé 596) est un inhibiteur puissant et sélectif de CDK5, avec une valeur IC50 inférieure À 10 nM. Le (S)-GFB-12811 peut être utilisé dans la recherche sur la progression du cycle cellulaire, le développement neuronal, la tumorigenèse.
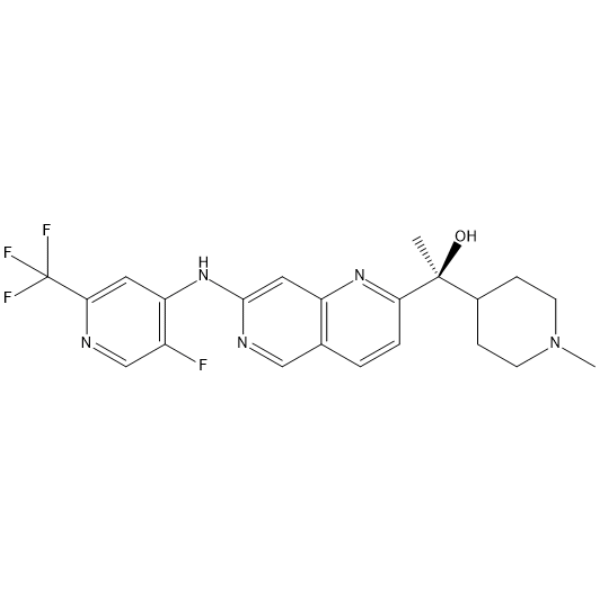
-
GC65997
(S)-LY3177833 hydrate
L'hydrate de (S)-LY3177833 ((S)-Exemple 2) est un inhibiteur de kinase CDC7 actif par voie orale. L'hydrate de (S)-LY3177833 présente une large activité anticancéreuse in vitro.
-
GC60421
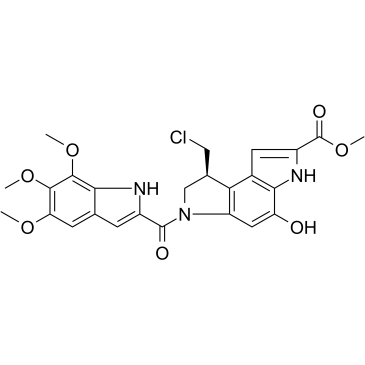
(S)-Seco-Duocarmycin SA
(S)-Seco-Duocarmycin SA est un alkylateur d'ADN, cytotoxique pour les cellules cancéreuses et agit comme une cytotoxine ADC pour les conjugués anticorps-médicament.
-
GC39842
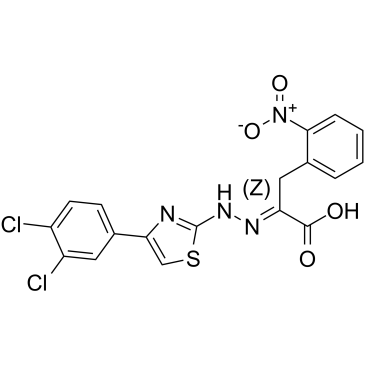
(Z)-4EGI-1
(Z)-4EGI-1 est l'isomère Z de 4EGI-1 et est un inhibiteur de l'interaction eIF4E/eIF4G et de l'initiation de la traduction. (Z)-4EGI-1 se lie efficacement À eIF4E avec une IC50 de 43,5 μM et une valeur Kd de 8,74 μM. (Z)-4EGI-1 a une activité anticancéreuse.
-
GC68484
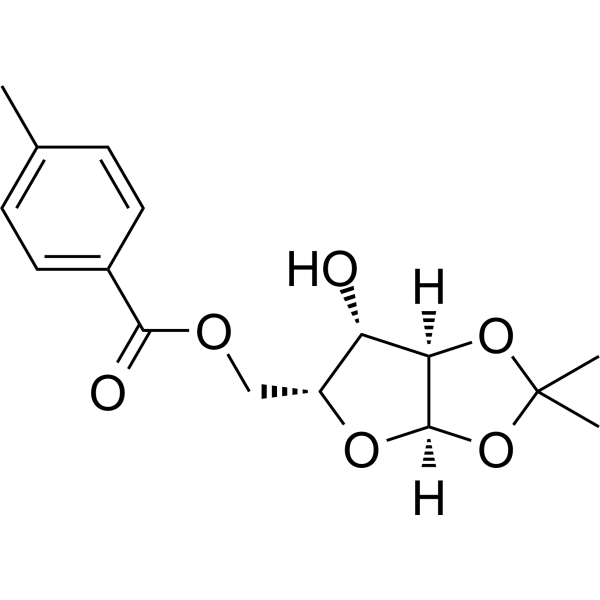
1,2-O-Isopropylidene-5-O-p-toluoyl-a-D-xylofuranose
1,2-O-Isopropylidene-5-O-p-toluoyl-a-D-xylofuranose est un analogue de nucléoside purique. Les analogues de nucléosides puriques ont une activité antitumorale étendue et ciblent les tumeurs malignes du système lymphatique inerte. Le mécanisme anticancéreux dans ce processus dépend de l'inhibition de la synthèse d'ADN, l'induction de l'apoptose cellulaire, etc.
-
GC19528
1,4-Benzoquinone
p-Benzoquinone, NSC 36324, p-Quinone
A toxic metabolite of benzene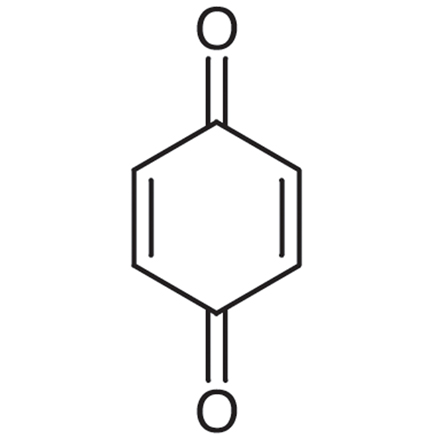
-
GC41859
1,5,7-Triazabicyclo[4.4.0]dec-5-ene
TBD, Triazabicyclodecene
1,5,7-Triazabicyclo[4.4.0]dec-5-ene is a bicyclic guanidine base that can be used as a catalyst for a variety of base-mediated organic transformations, including Michael additions, Wittig reactions, Henry reactions, and transesterification reactions.![1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure 1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure](/media/struct/GC4/GC41859.png)
-
GC65551
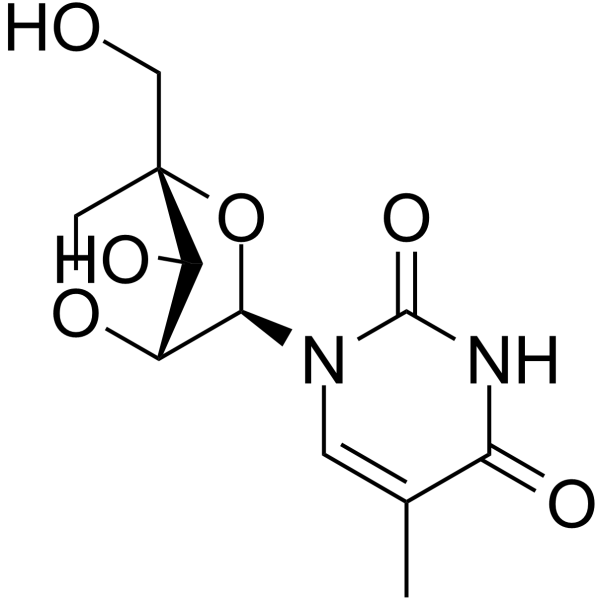
1-(2'-O-4-C-Methylene-beta-D-ribofuranosyl)thymine
La 1-('-O-4-C-méthylène-bêta-D-ribofuranosyl)thymine est un nucléoside bicyclique.
-
GC71294
1-(2-Deoxy-β-D-threo-pentofuranosyl)thymine
1-(2-Deoxy-β-D-threo-pentofuranosyl)tmine is a tmidine analog.

-
GC49470
1-Ethyl-1-nitrosourea (hydrate)
ENU, Ethylnitrosourea, N-Ethyl-N-nitrosourea, N-Nitroso-N-ethylurea
A DNA alkylating agent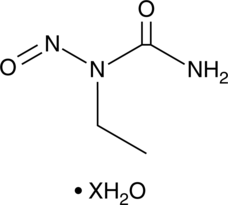
-
GC48909
1-Hydroxyanthraquinone
NSC 8640
La 1-hydroxyanthraquinone, un composé naturel À activité orale provenant de certaines plantes comme Tabebuia avellanedae, présente un effet cancérigène.
-
GC65038
1-Methylinosine
1-methyl Inosine, N1-Methylinosine
La 1-méthylinosine est un nucléotide modifié trouvé en position 37 dans l'ARNt en 3' de l'anticodon de l'ARNt eucaryote.
-
GC49872
10-Formyltetrahydrofolate (sodium salt) (technical grade)
10-CHO-FH4, 10-CHO-THF, N10-Formyltetrahydrofolate, 10-formyl H4PteGlu, 10-fTHF
Le 10-formyltétrahydrofolate (sel de sodium) (qualité technique) est une forme d'acide tétrahydrofolique qui agit comme donneur de groupes formyle dans l'anabolisme.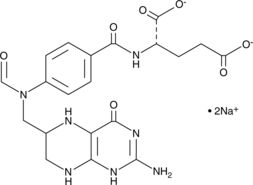
-
GC63796
116-9e
MAL2-11B
116-9e (MAL2-11B) est un inhibiteur de l'ADNJA1 co-chaperon Hsp70.
-
GC71326
12R-LOX-IN-2
12R-LOX-IN-2 (compound 7b) is an inhibitor of 12R-lipoxygenase (12R-LOX).

-
GC49759
13C17-Mycophenolic Acid
13C17-MPA
An internal standard for the quantification of mycophenolic acid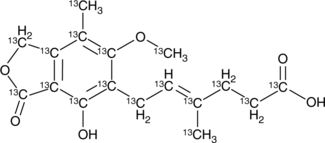
-
GC46474
18-Deoxyherboxidiene
RQN-18690A
Le 18-désoxyherboxidiène (RQN-18690A) est un puissant inhibiteur de l'angiogenèse.
-
GC65489
2'-F-Bz-dC Phosphoramidite
&2#39;-F-Bz-dC Le phosphoramidite peut être utilisé dans la synthèse d'oligoribonucléotides.
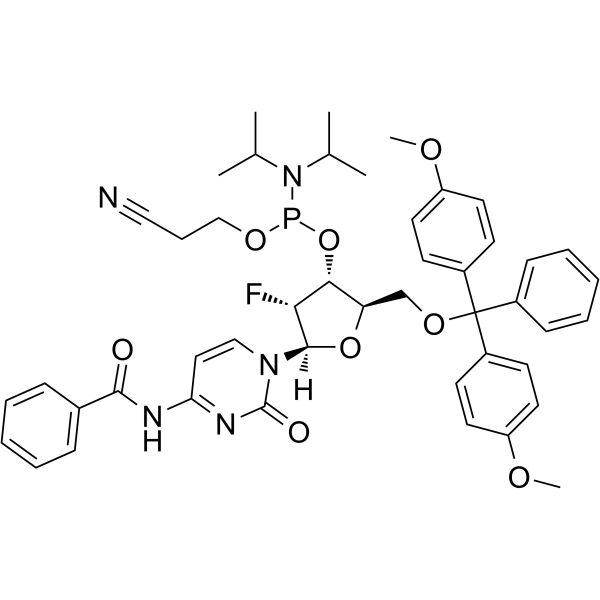
-
GC66651
2'-O,4'-C-Methyleneadenosine
LNA-A
2'-O,4'-C-méthylèneadénosine (LNA-A) est un acide nucléique verrouillé (LNA) et est également un analogue de l'adénosine.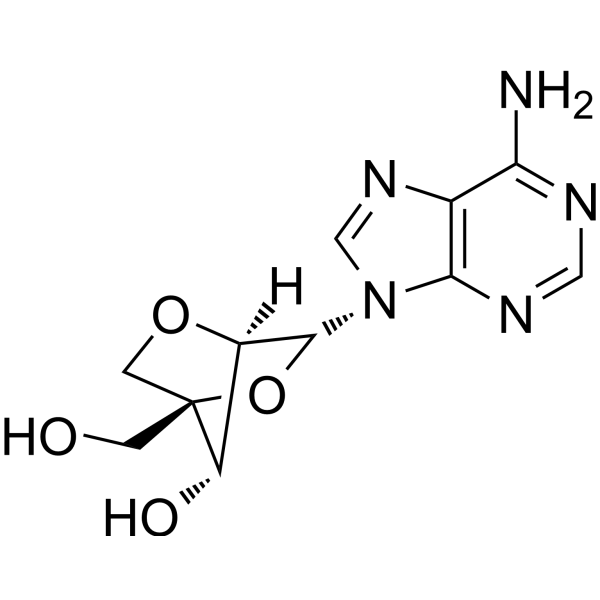
-
GC66654
2'-O,4'-C-Methylenecytidine
LNA-C(Bz)
2'-O,4'-C-Méthylènecytidine (LNA-C(Bz)) est un analogue de nucléoside bicyclique avec une conformation de type N fixe. 2'-O,4'-C-Méthylènecytidine peut être utilisé pour synthétiser des oligonucléotides. 2'-O,4'-C-Méthylènecytidine forme des duplex avec des brins d'ADN et d'ARN complémentaires.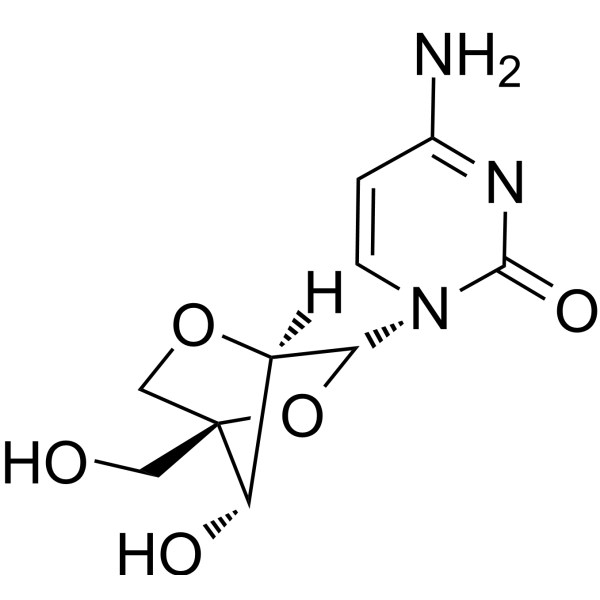
-
GC66655
2'-O,4'-C-Methyleneguanosine
LNA-G
2ò-O,4ò-C-Méthylèneguanosine (LNA-G) est un analogue de guanine inverse, où LNA (acide nucléique verrouillé) est un analogue d'acide nucléique. La modification de LNA peut être utilisée dans une variété d'applications telles qu'une affinité de liaison efficace avec des séquences complémentaires et une plus grande résistance aux nucléases que les nucléotides naturels, offrant un grand potentiel pour des applications dans le diagnostic et la recherche de maladies. LNA-G est également disponible via l'ADN polymérase KOD, qui permet l'intégration des nucléotides LNA-G dans le brin d'ADN.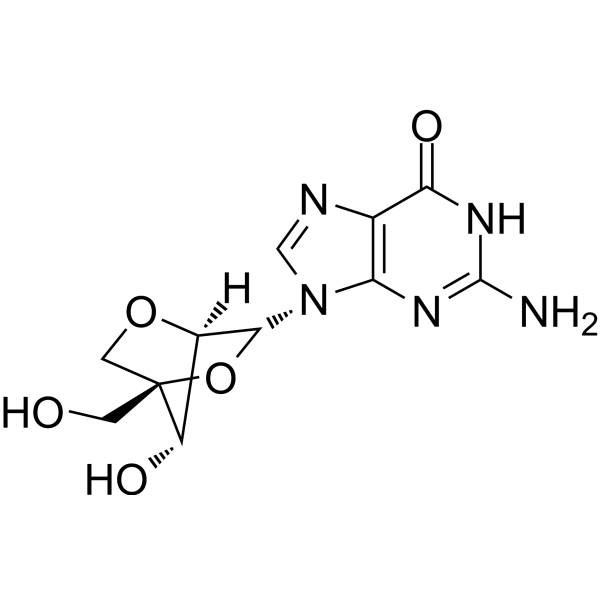
-
GC52183
2'-O-Methyl-5-methyluridine
2'-O-Methylribothymidine, 2'-O-Methylthymidine
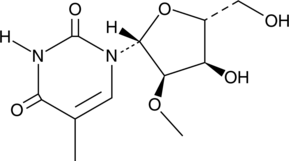
-
GC66713
2'-O-MOE-5-Me-C(Bz)
2'-O-MOE-5-Me-C (Bz) est un nucléotide pour la synthèse stéréosélective d'alkylphosphonates de nucléoside.
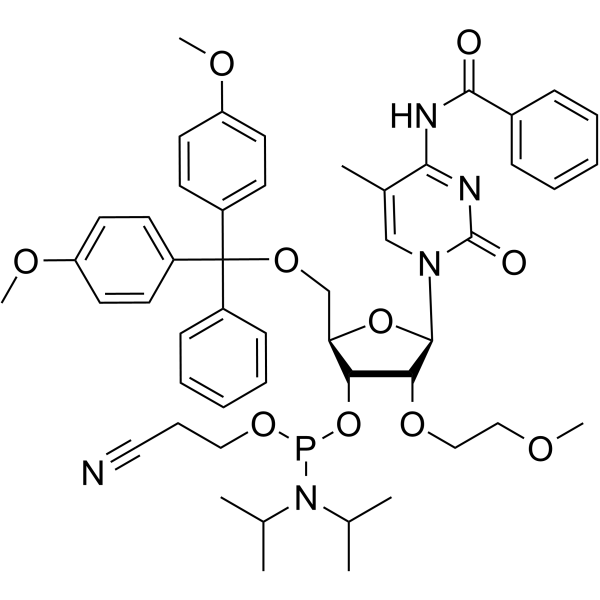
-
GC66657
2'-O-MOE-5-Me-rC
2'-O-MOE-5-Me-rC est un composé actif. 2'-O-MOE-5-Me-rC peut être utilisé pour la synthèse d'oligonucléotides.
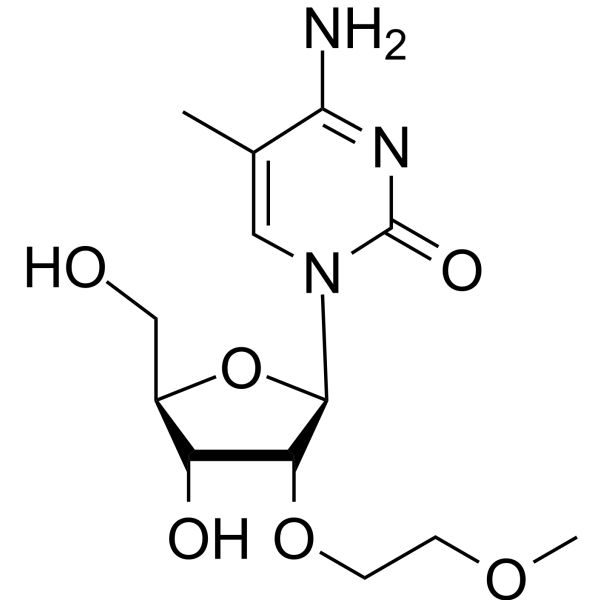
-
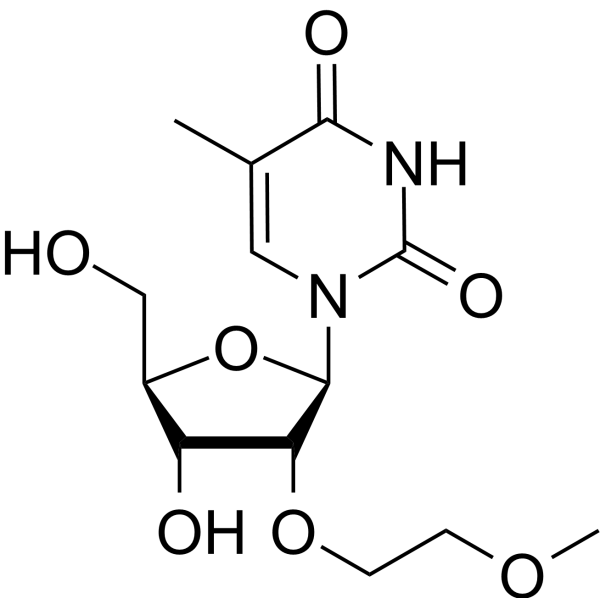
GC66091
2'-O-MOE-5-Me-rU
2'-O-MOE-5-Me-rU est un composé actif. 2'-O-MOE-5-Me-rU peut être utilisé pour la synthèse d'oligonucléotides.
-
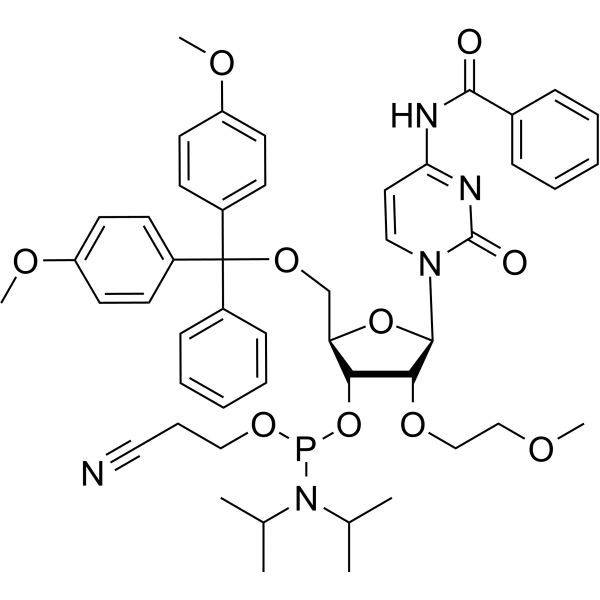
GC66656
2'-O-MOE-rC
2'-O-MOE-rC est un nucléoside modifié par 2'-O-MOE. 2'-O-MOE-rC peut être utilisé pour la synthèse d'ADN.
-
GC66652
2'-O-MOE-U
2'-O-MOE-U est un phosphoramidite, peut être utilisé pour la synthèse d'oligonucléotides.

-
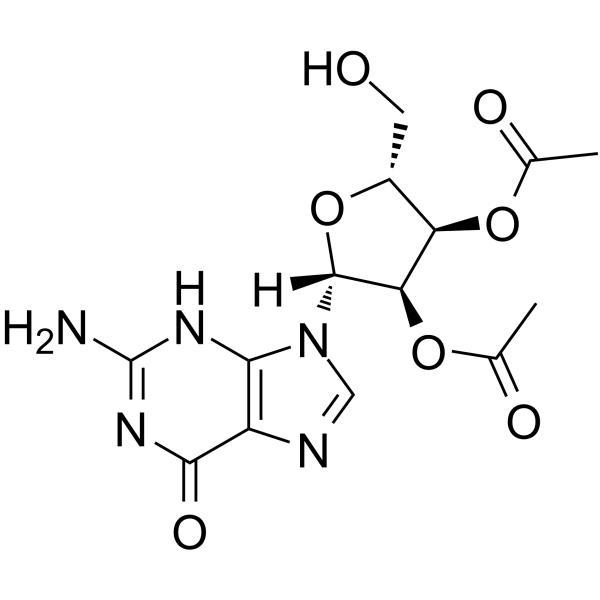
GC65170
2′,3′-Di-O-acetylguanosine
La di-O-acétylguanosine est un analogue nucléosidique.
-
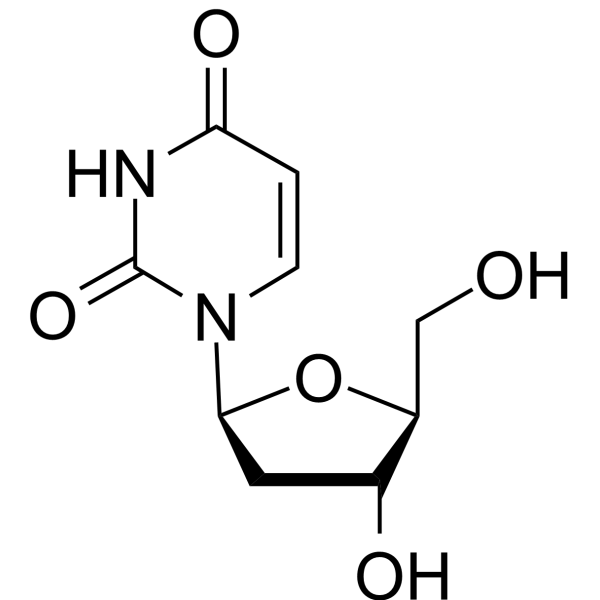
GC67384
2′-Deoxy-β-L-uridine
2'-Deoxy-β-L-uridine est un analogue de nucléotide et un substrat spécifique pour l'enzyme virale, ne montre aucune stéréospécificité contre l'herpès simplex 1 (HSV1) thymidine kinase (TK). 2ò-Deoxy-β-L-uridine exerce une activité antivirale via l'interaction de 5'-triphosphates avec l'ADN polymérase virale.
-
GC61667
2′-Deoxy-2′-fluoroadenosine
&2#8242;-Deoxy-&2#8242;-fluoroadenosine peut être utilisé pour la synthèse d'oligonucléotides 2′-Deoxy-2′-fluoro-modifiés hybridés avec de l'ARN. &2#8242;-Déoxy-&2#8242;-fluoroadénosine peut être clivée efficacement par E. coli purine nucléoside phosphorylase (PNP) en agent toxique 2-fluoroadénine (FAde). &2#8242;-Déoxy-&2#8242;-fluoroadénosine montre une excellente activité in vivo contre les tumeurs exprimant E. coli PNP.
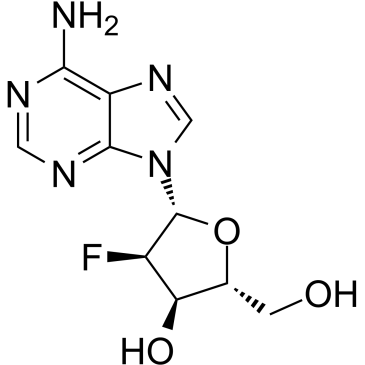
-
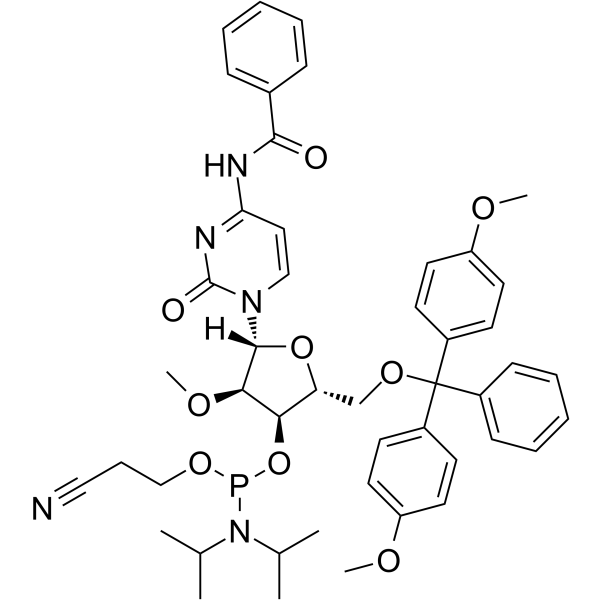
GC62530
2’-O-Me-C(Bz) Phosphoramidite
&2rsquo;-O-Me-C(Bz) Phosphoramidite est un monomère phosphoramidite modifié, qui peut être utilisé pour la synthèse d'oligonucléotides.
-
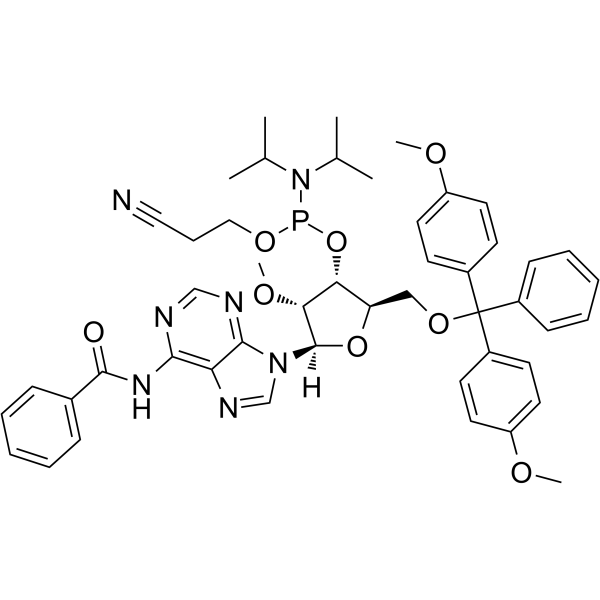
GC62529
2’-OMe-A(Bz) Phosphoramidite
&2rsquo;-OMe-A(Bz) Phosphoramidite est un monomère phosphoramidite modifié, qui peut être utilisé pour la synthèse d'oligonucléotides.
-
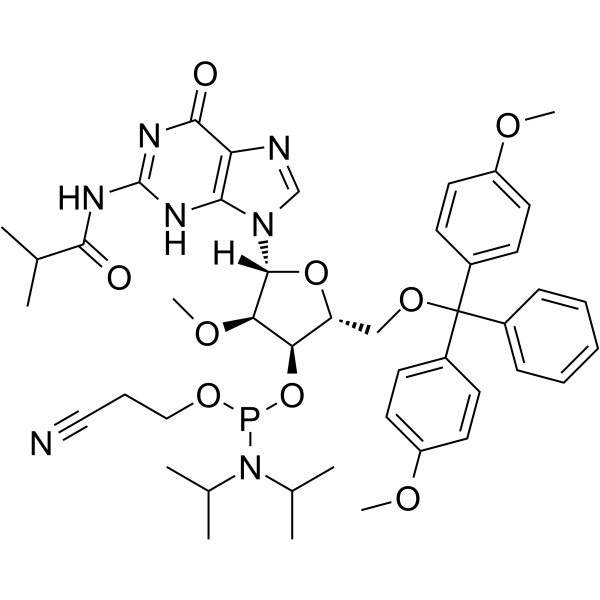
GC62531
2’-OMe-G(ibu) Phosphoramidite
&2rsquo;-OMe-G(ibu) Phosphoramidite est un monomère phosphoramidite modifié, qui peut être utilisé pour la synthèse d'oligonucléotides.
-
GC46508
2',2'-Difluoro-2'-deoxyuridine
dFdU
An active metabolite of gemcitabine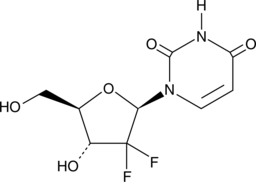
-
GC42061
2',3'-Dideoxyadenosine 5'-triphosphate
2',3'-Dideoxyadenosine 5'-triphosphate
2',3'-Dideoxyadenosine 5'-triphosphate (2',3'-Dideoxyadenosine 5'-triphosphate), an active metabolite of 2',3'-dideoxyinosine, is a chain-elongating inhibitor of DNA polymerase.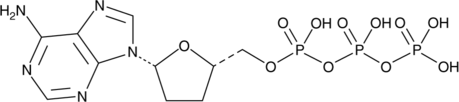
-
GC46519
2',3'-O-Isopropylideneguanosine
A building block
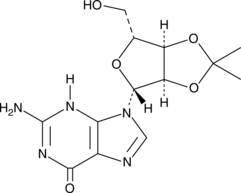
-
GC46540
2'-Deoxyadenosine-5'-triphosphate (sodium salt hydrate)
A purine nucleotide
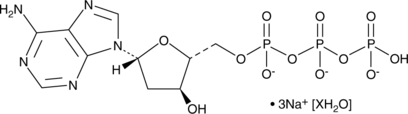
-
GC48440
2'-Deoxycytidine-5'-triphosphate (sodium salt)
dCTP
2'-Deoxycytidine-5'-triphosphate (sel de sodium) (dCTP sel trisodique) est un nucléoside triphosphate qui peut être utilisé pour la synthèse d'ADN.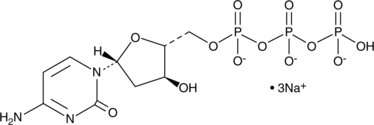
-
GC42151
2'-Deoxyguanosine 5'-monophosphate (sodium salt hydrate)
dGMP
2'-Deoxyguanosine 5'-monophosphate (dGMP) is used as a substrate of guanylate kinases to generate dGDP, which in turn is phosphorylated to dGTP, a nucleotide precursor used in DNA synthesis.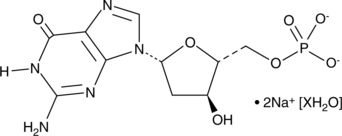
-
GC33430
2'-Deoxypseudoisocytidine
La 2'-désoxypseudoisocytidine est un analogue nucléosidique.
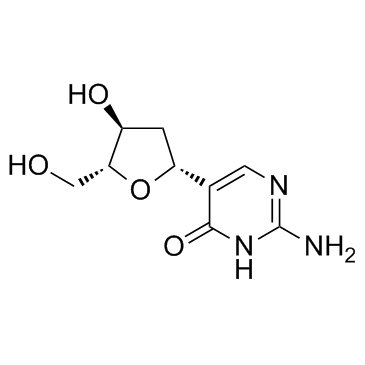
-
GC35072
2'-O,4'-C-Methyleneuridine
2'-O,4'-C-Méthylèneuridine (Composé 15a) est un nucléoside bicyclique.

-
GC42080
2'2'-cGAMP (sodium salt)
Adenosine-Guanosine 2’,2’-cyclic monophosphate, cGAMP(2’-5’), 2’,2’-Cyclic GMP-AMP
2'2'-cGAMP is a synthetic dinucleotide (CDN) that contains non-canonical 2'5'-phosphodiester bonds.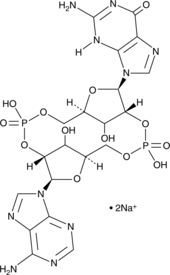
-
GC64399
2,4,6-Trihydroxybenzoic acid
L'acide 2,4,6-trihydroxybenzoÏque, le métabolite flavonoÏde, est un inhibiteur de CDK. L'acide 2,4,6-trihydroxybenzoÏque peut être utilisé pour la recherche sur le cancer.
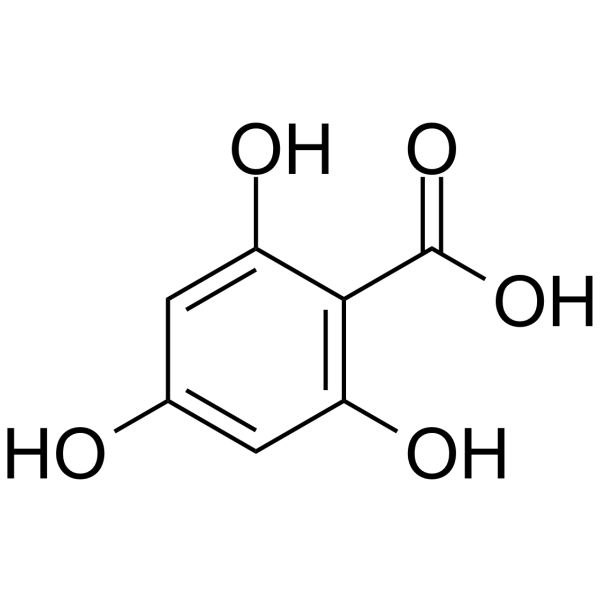
-
GC60459
2,4-D (2,4-Dichlorophenoxyacetic acid)
2,4D, Diclordon, Vidon 638
Le 2,4-D (acide 2,4-dichlorophénoxyacétique) (acide 2,4-D (acide 2,4-dichlorophénoxyacétique) ichlorophénoxyacétique) est un herbicide systémique sélectif pour le contrÔle des mauvaises herbes À feuilles larges.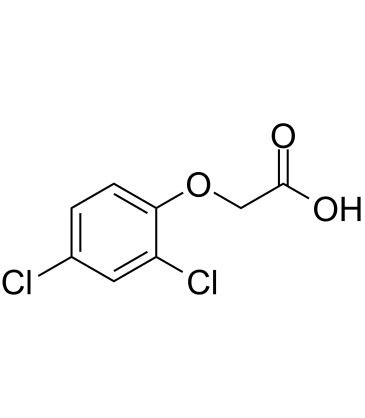
-
GC71530
2,4-D-13C6
2,4-D-13C6 is the 13C-labeled 2,4-D.

-
GC46524
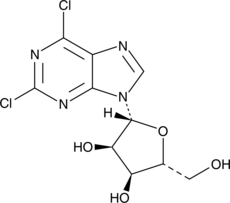
2,6-Dichloropurine-9-β-D-riboside
2,6-Dichloropurine riboside
A building block
-
GC33496
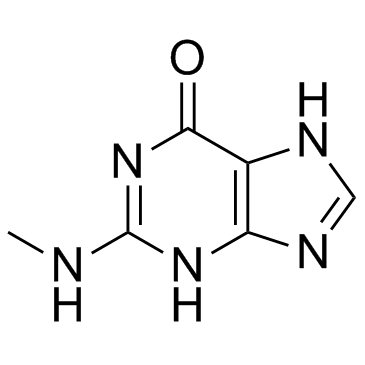
2-(Methylamino)-1H-purin-6(7H)-one (N2-methylguanine)
La 2-(méthylamino)-1H-purine-6(7H)-one (N2-méthylguanine) (N2-méthylguanine) est un nucléoside modifié.
-
GC64983
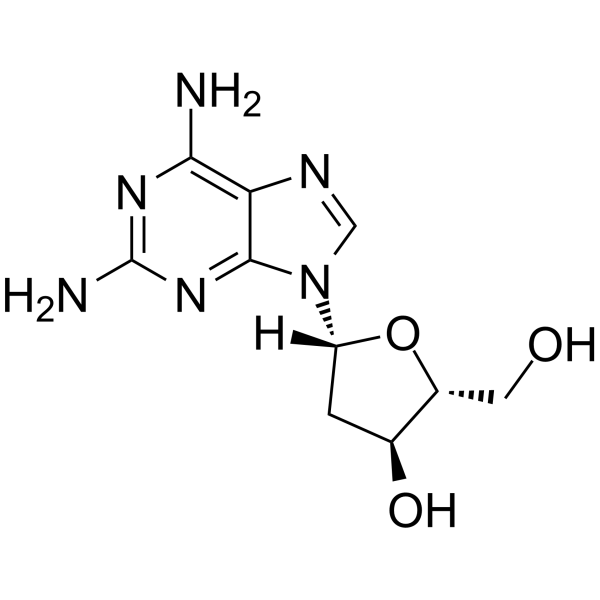
2-Amino-2'-deoxyadenosine
La 2-amino-&2#39;-désoxyadénosine est un désoxyribonucléoside utilisé pour la synthèse d'oligonucléotides.
-
GC71560
2-Iodoacetamide-d4
2-Iodoacetamide-d4 is the deuterium labeled 2-Iodoacetamide.

-
GC71208
2-Methyladenosine
2-Metladenosine is an adenosine analogue.

-
GC39527
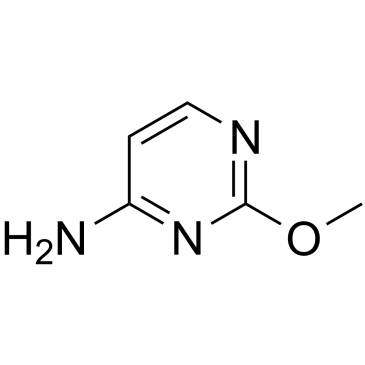
2-O-Methylcytosine
La 2-O-méthylcytosine, un analogue O-alkylé d'un adduit d'ADN, est la nucléobase endommagée.
-
GC49348
2-Thiocytidine
α-2-Thioribocytidine
A modified nucleobase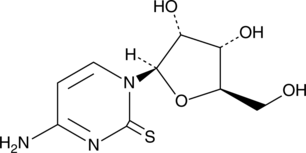
-
GC42197
2-Thiouridine
1-β-D-ribofuranosyl-2-thiouracil, s2U
2-Thiouridine (s2U) is a modified nucleobase found in tRNAs that is known to stabilize U:A pairs and modestly destabilize U:G wobble pairs.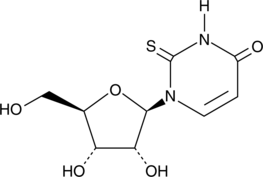
-
GC65083
3'-Azido-3'-deoxy-5-fluorocytidine
3'-Azido-3'-désoxy-5-fluorocytidine (Composé 12) est un dérivé de la cytidine.
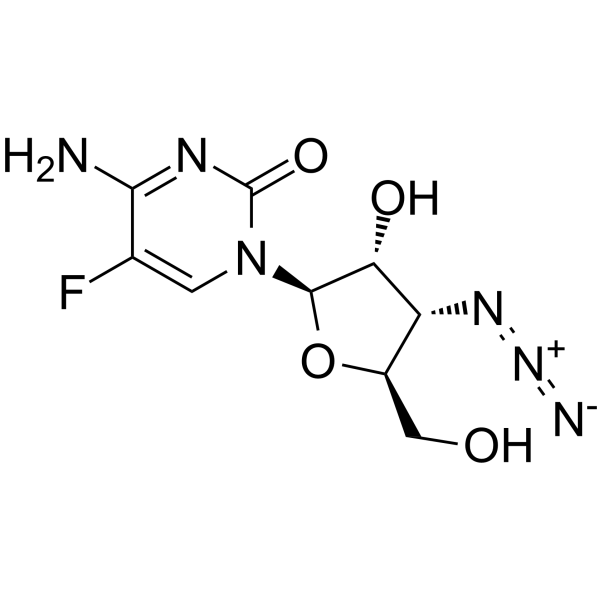
-
GC71204
3'-O-(2-Methoxyethyl)guanosine
3’-O-(2-Methoxyetl)guanosine is a guanosine analogue.

-
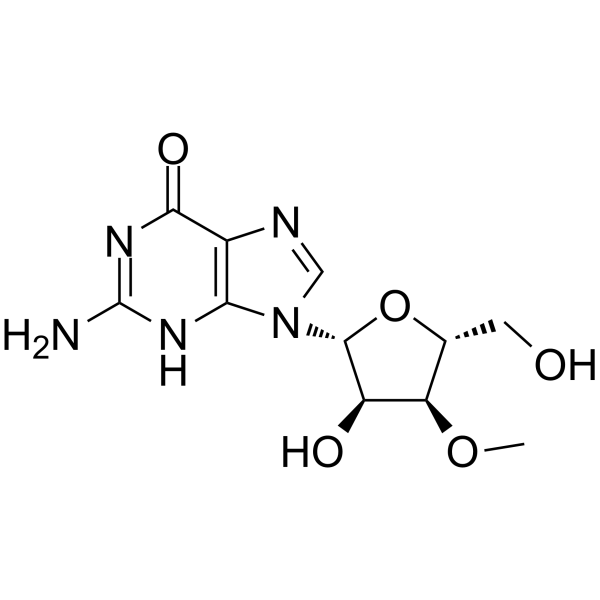
GC64985
3'-O-Methylguanosine
3'-O-Méthylguanosine est un analogue de nucléoside méthylé et un terminateur de chaîne d'ARN.
-
GC68542
3'Ome-m7GpppAmpG ammonium
3'Ome-m7GpppAmpG ammonium est un analogue de coiffe à trois nucléotides contenant une molécule d'acide ribonucléique verrouillée (LNA). 3'Ome-m7GpppAmpG ammonium présente une efficacité de traduction significative. Il peut être utilisé comme outil potentiel en biologie moléculaire pour les vaccins à ARNm et la transfection d'ARNm, tels que la production de protéines, la thérapie génique et l'immunothérapie contre le cancer.

-
GC40618
3',4',7-Trihydroxyisoflavone
3'-hydroxy Daidzein, 3’,4’,7-THIF
3',4',7-Trihydroxyisoflavone, un métabolite majeur de Daidzein, est un inhibiteur compétitif de l'ATP de Cot (Tpl2/MAP3K8) et MKK4. 3',4',7-Trihydroxyisoflavone a des activités anticancéreuses, anti-angiogéniques, chimioprotectrices et antiradicalaires.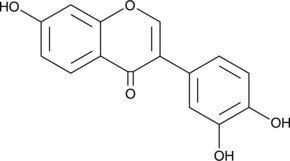
-
GC35105
3'-Azido-3'-deoxy-beta-L-uridine
La 3'-azido-3'-désoxy-bêta-L-uridine (composé 25) est un dérivé de nucléoside.
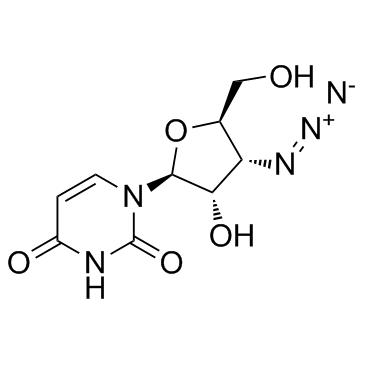
-
GC60023
3'-Deoxyuridine-5'-triphosphate
3'-dUTP
Le 3'-désoxyuridine-5'-triphosphate (3'-dUTP) est un analogue nucléotidique qui inhibe les ARN polymérases I et II dépendantes de l'ADN.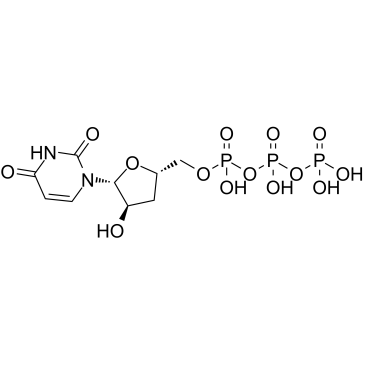
-
GC61862
3'-Deoxyuridine-5'-triphosphate trisodium
3'-dUTP trisodium
Le 3'-désoxyuridine-5'-triphosphate trisodique (3'-dUTP trisodique) est un analogue nucléotidique qui inhibe les ARN polymérases I et II dépendantes de l'ADN.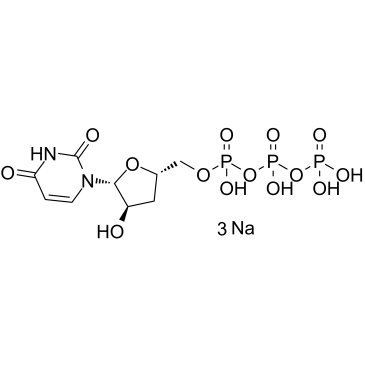
-
GC45332
3'-Dephosphocoenzyme A
depCoA, Dephospho-CoA
An intermediate in the biosynthesis of CoA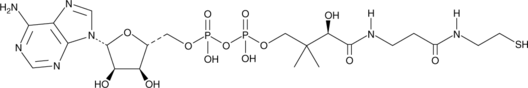
-
GC34384
3,6-DMAD hydrochloride
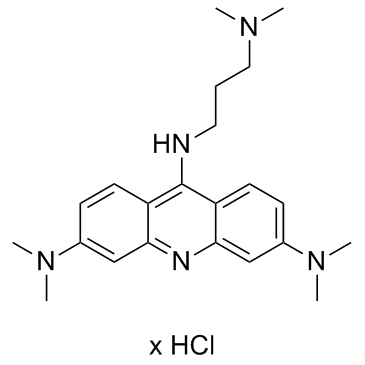
Le chlorhydrate de 3,6-DMAD, un dérivé de l'acridine, est un puissant inhibiteur de la voie IRE1α-XBP1s. Le chlorhydrate de 3,6-DMAD favorise la sécrétion d'IL-6 via la voie IRE1α-XBP1s. Le chlorhydrate de 3,6-DMAD inhibe l'oligomérisation de l'IRE1α et l'activité de l'endoribonucléase (RNase). Le chlorhydrate de 3,6-DMAD peut être utilisé pour la recherche sur le cancer.
-
GC34452
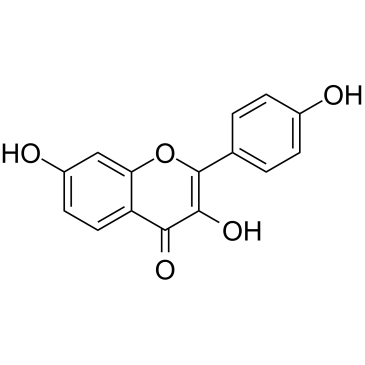
3,7,4'-Trihydroxyflavone
3,7,4'-Trihydroxyflavone, isolée de Rhus javanica var.
-
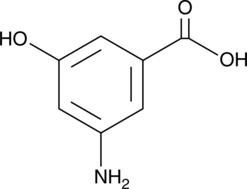
GC52129
3-Amino-5-hydroxybenzoic Acid
AHBA
-
GC13510
3-AP
3-Aminopyridine-2-Carboxyaldehyde Thiosemicarbazone,NSC 663249,Triapine™
Le 3-AP (PAN-811) est un puissant inhibiteur de la sous-unité M2 de la ribonucléotide réductase (RR) et un puissant radiosensibilisateur.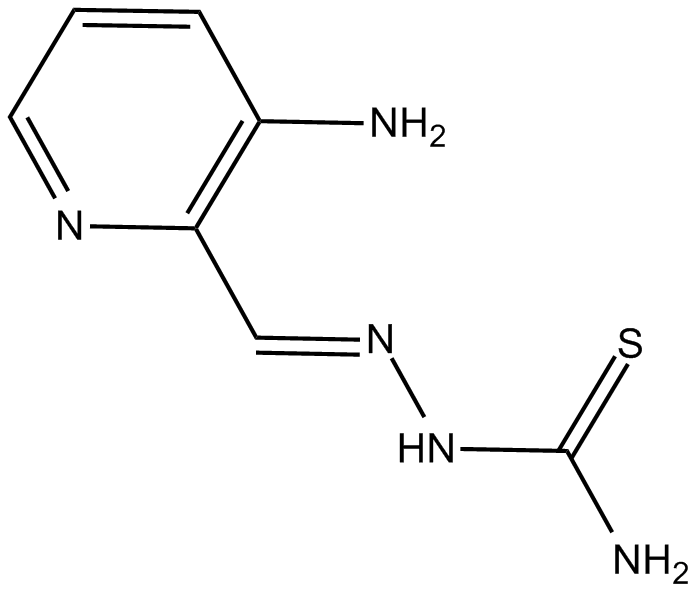
-
GC48457
3-keto Fusidic Acid
3-keto FA, 3-Oxofusidic Acid
An active metabolite of fusidic acid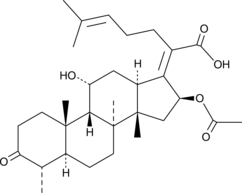
-
GC65084
3-Methylcytidine
La 3-méthylcytidine, un nucléoside urinaire, peut être utilisée comme biomarqueur de quatre types différents de cancer : cancer du poumon, cancer gastrique, cancer du cÔlon et cancer du sein.
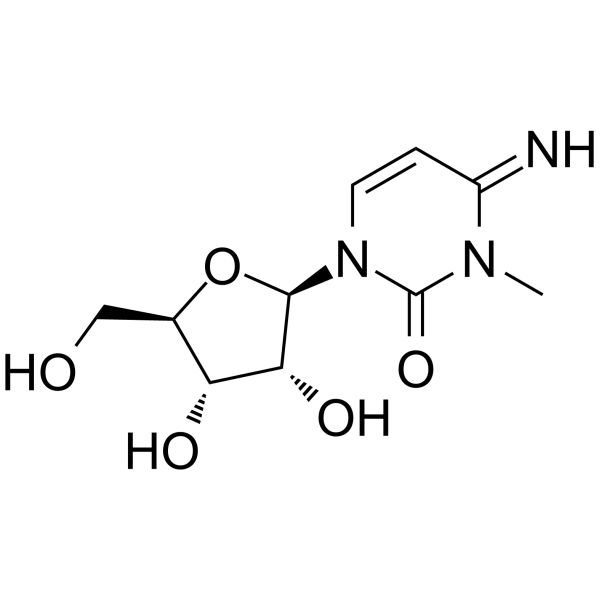
-
GC52391
306-O12B-3
An ionizable cationic lipidoid
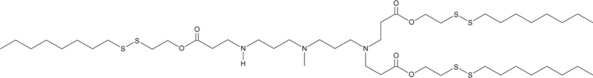
-
GC15389
360A
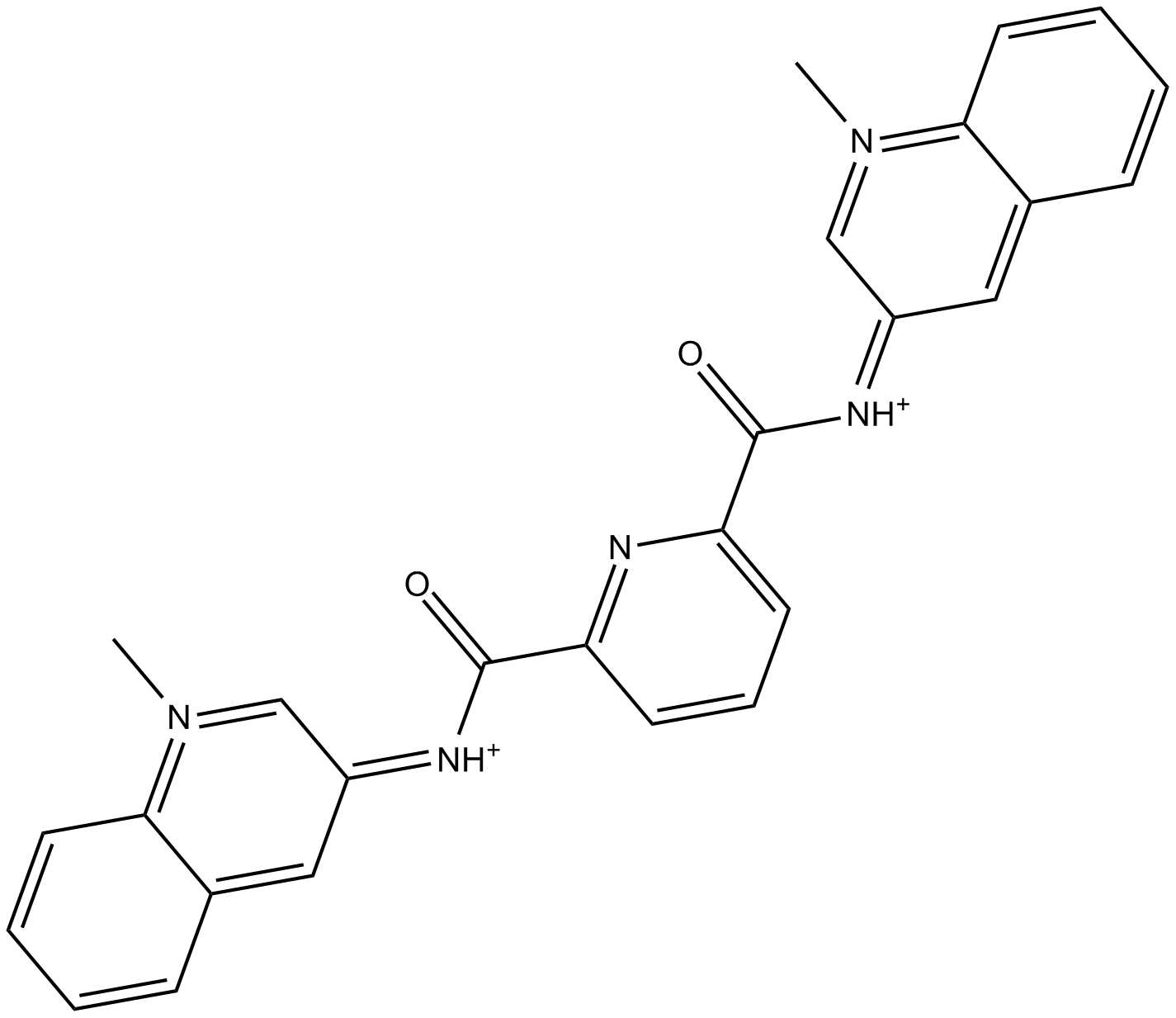
-
GC10115
360A iodide
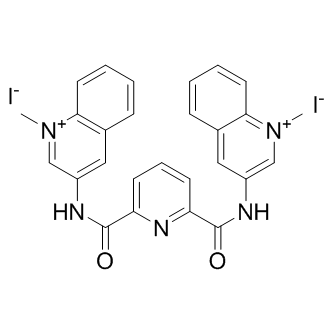
-
GC14493
4μ8C
4u8C
Inhibiteur de l'ARNase IRE1, puissant et non toxique.
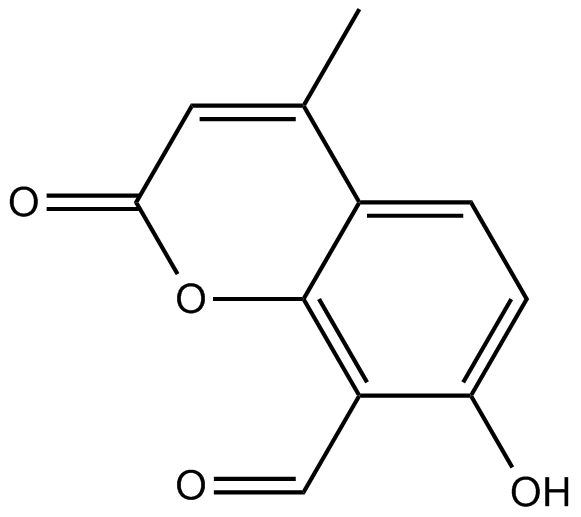
-
GC17271
4'-Demethylepipodophyllotoxin
(-)-4′-Demethylepipodophyllotoxin
An inhibitor of tubulin polymerization