Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Targets for Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Products for Chromatin/Epigenetics
- Cat.No. Nom du produit Informations
-
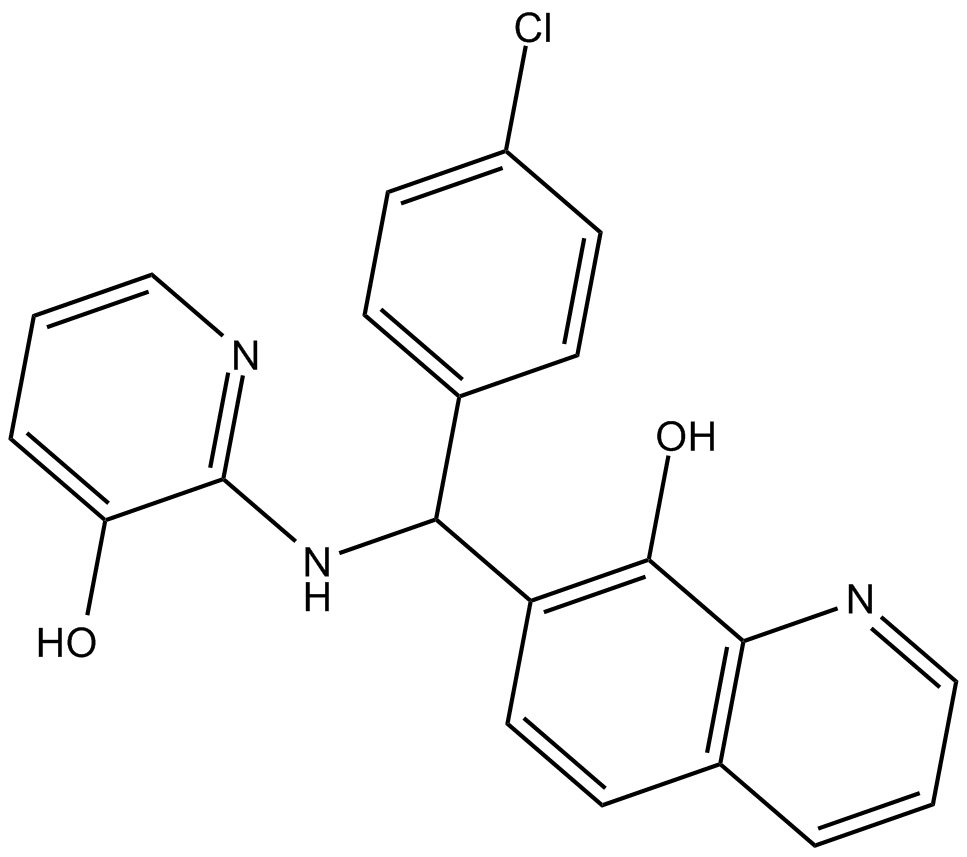
GC31365
γ-Oryzanol
γ-Oryzanol est un puissant inhibiteur de l'ADN méthyltransférase (DNMT) dans le striatum des souris.
-
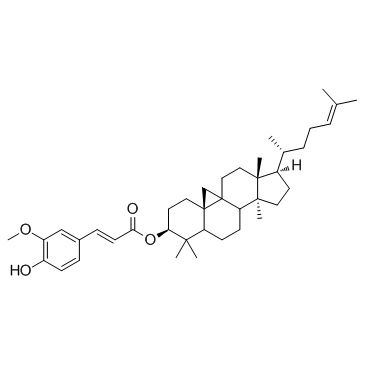
GC45258
(+)-Biotin 4-Amidobenzoic Acid (sodium salt)
(+)-Biotin 4-amidobenzoic acid is a substrate of biotinidase, which cleaves biotin amide to give biotin in vivo.
-
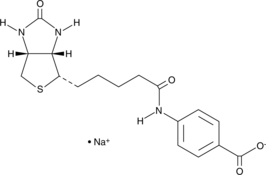
GC61595
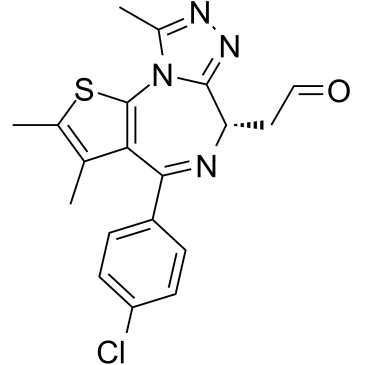
(+)-JQ-1-aldehyde
(+)-JQ-1-aldéhyde est la forme aldéhyde de (+)-JQ1. (+)-JQ-1-aldéhyde peut être utilisé comme précurseur pour synthétiser les PROTAC, qui ciblent les bromodomaines BET.
-
GC34958
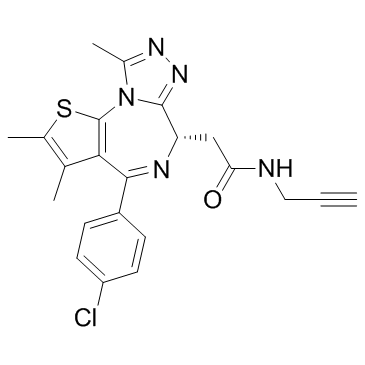
(+)-JQ1 PA
(+)-JQ1 PA est un dérivé du bromodomaine et de l'inhibiteur extra-terminal (BET) JQ1, avec une IC50 de 10,4 nM.
-
GC13822
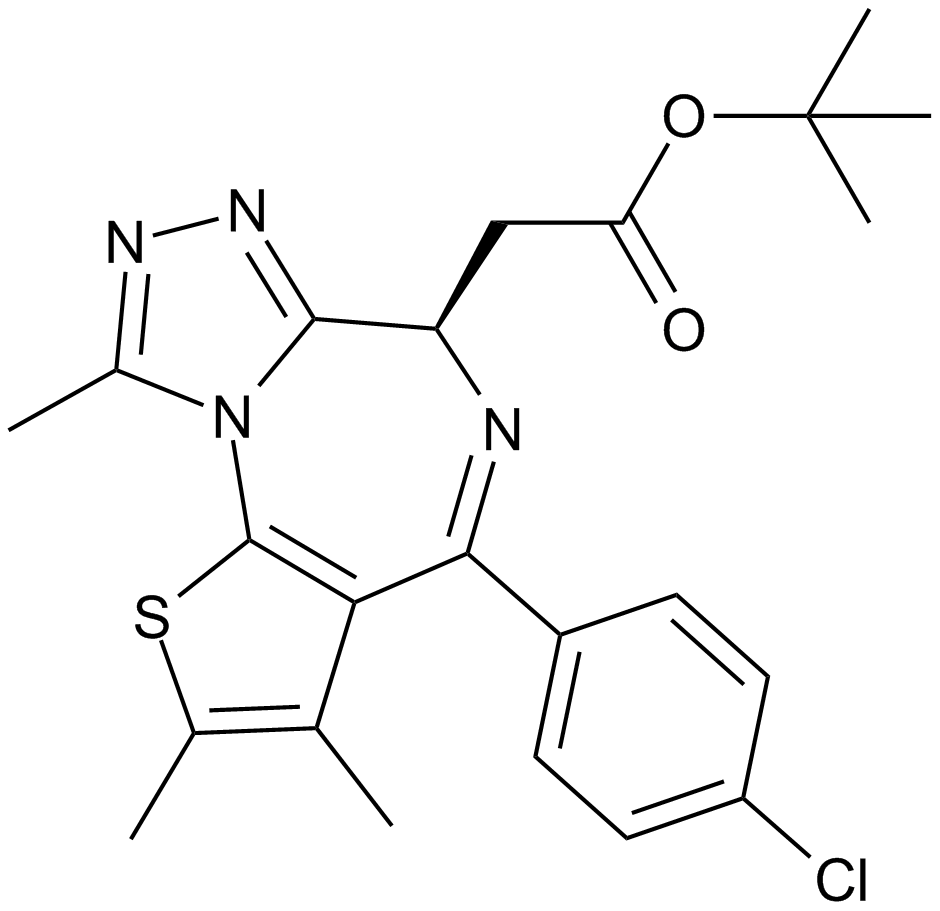
(-)-JQ1
A selective inhibitor of BET bromodomains
-
GC34964
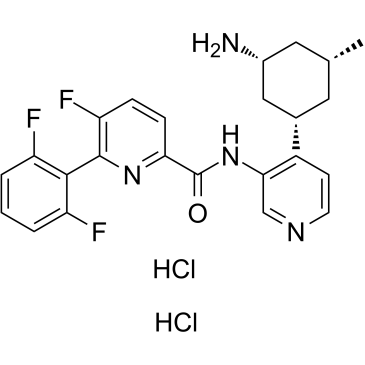
(1S,3R,5R)-PIM447 dihydrochloride
Le (1S,3R,5R)-PIM447 (dichlorhydrate) un inhibiteur de PIM extrait du brevet US 20100056576 A1, exemple composé 72, a des valeurs d'IC50 de 0,095 μM pour Pim1, 0,522 μM pour Pim2 et 0,369 μM pour Pim3.
-
GC62130
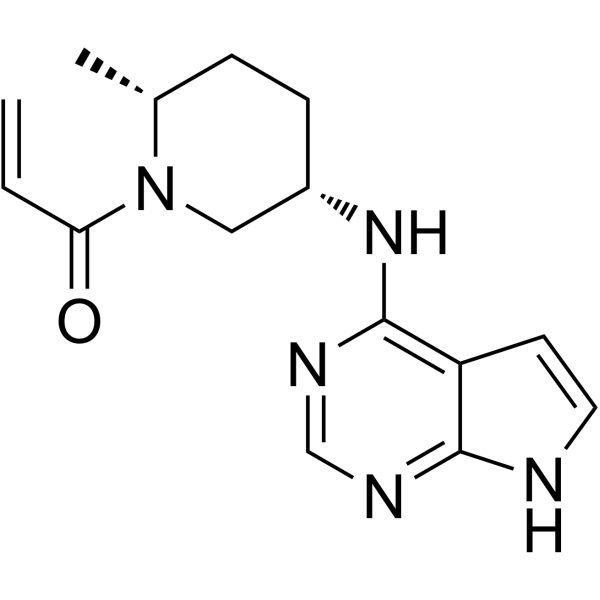
(2R,5S)-Ritlecitinib
Le (2R,5S)-Ritlecitinib ((2R,5S)-PF-06651600) est un inhibiteur puissant et sélectif de JAK3 (IC50=144,8 nM) extrait du brevet US20150158864A1, exemple 68.
-
GC34971
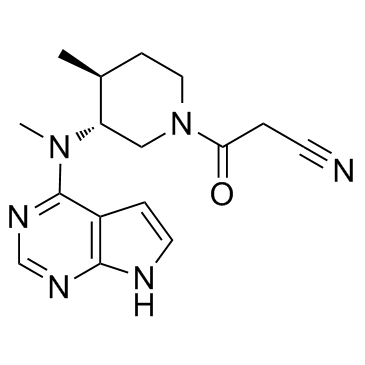
(3R,4S)-Tofacitinib
Le (3R,4S)-tofacitinib est un énantiomère moins actif du tofacitinib.
-
GC34972
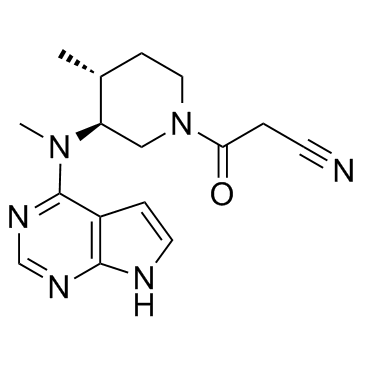
(3S,4R)-Tofacitinib
Le (3S,4R)-tofacitinib est un énantiomère moins actif du tofacitinib.
-
GC34973
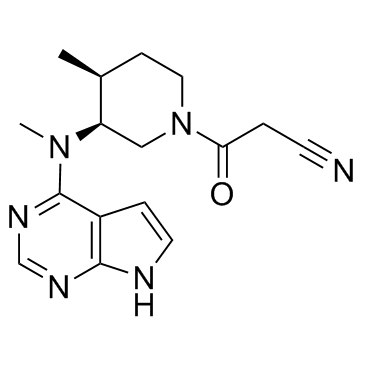
(3S,4S)-Tofacitinib
Le (3S,4S)-tofacitinib est l'énantiomère S le moins actif du tofacitinib.
-
GC41695
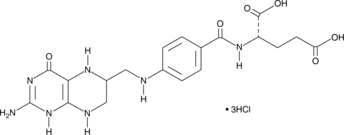
(6R,S)-5,6,7,8-Tetrahydrofolic Acid (hydrochloride)
(6R,S)-5,6,7,8-Tetrahydrofolic acid (THFA), the reduced form of folic acid, serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
-
GC41088
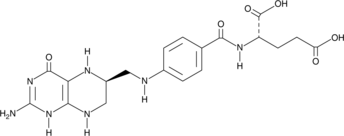
(6S)-Tetrahydrofolic Acid
(6S)-Tetrahydrofolic acid is a diastereomer of tetrahydrofolic acid, a reduced form of folic acid that serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
-
GC61807
(E/Z)-AG490
(E/Z)-AG490 ((E/Z)-Tyrphostine AG490) est un composé racémique des isomères (E)-AG490 et (Z)-AG490. (E)-AG490 est un inhibiteur de la tyrosine kinase qui inhibe l'EGFR, Stat-3 et JAK2/3.

-
GC63864
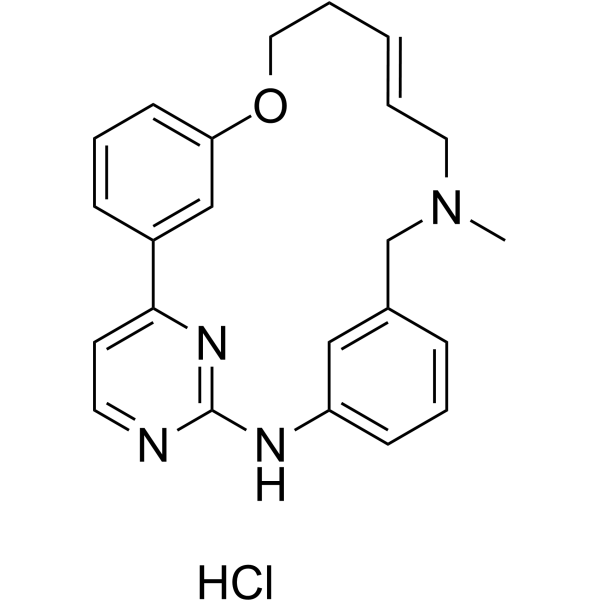
(E/Z)-Zotiraciclib hydrochloride
Le chlorhydrate de (E/Z)-Zotiracilib ((E/Z)-TG02) est un puissant inhibiteur de CDK2, JAK2 et FLT3.
-
GC15104
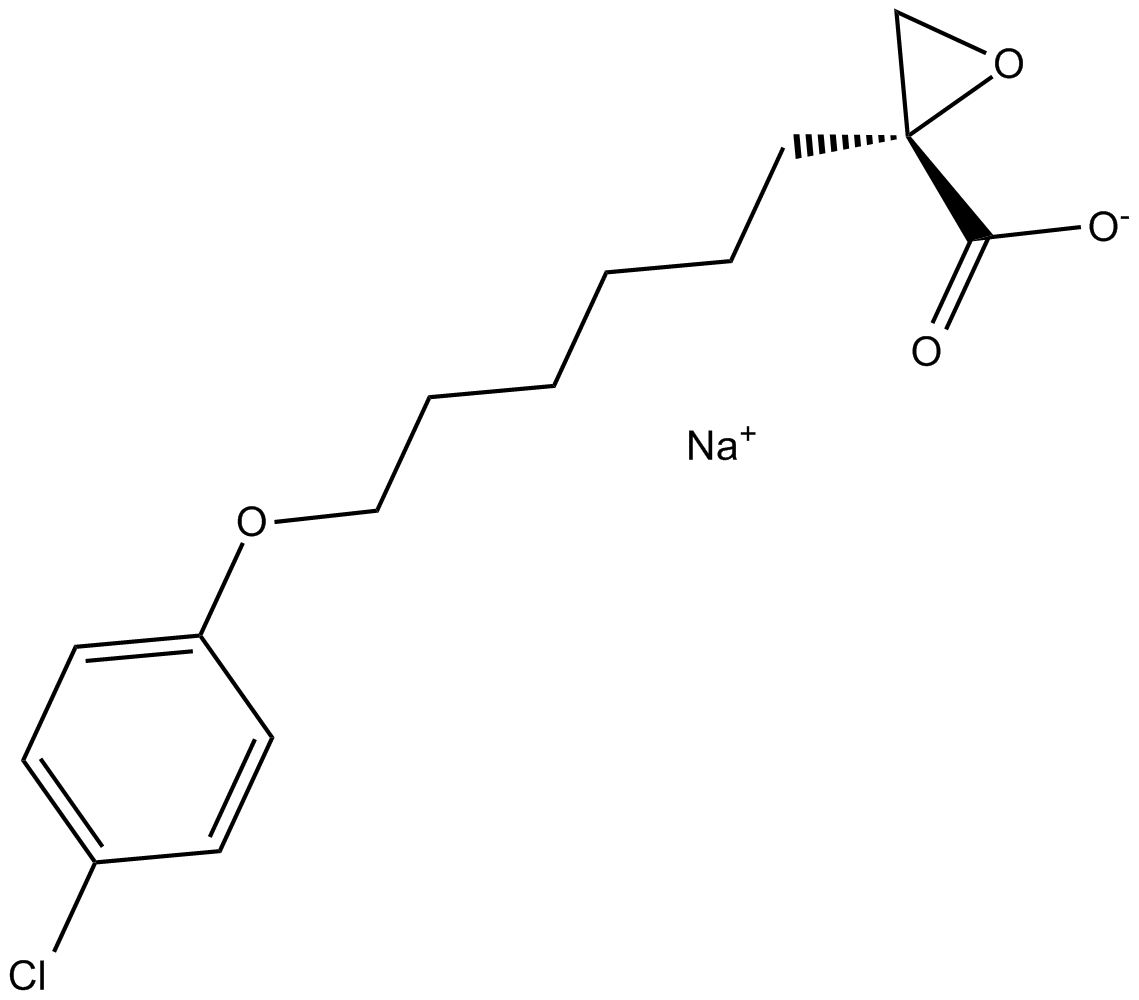
(R)-(+)-Etomoxir sodium salt
Le sel de sodium d'Etomoxir((R)-(+)-Etomoxir) est un inhibiteur irréversible de la carnitine palmitoyltransférase 1a (CPT-1a), inhibe l'oxydation des acides gras (FAO) par CPT-1a et inhibe l'oxydation du palmitate β ; chez l'homme, le rat et cochon d'inde.
-
GC63791
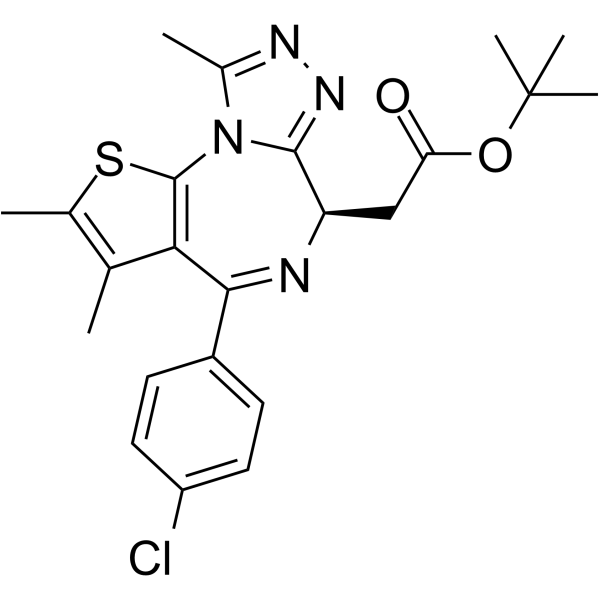
(R)-(-)-JQ1 Enantiomer
L'énantiomère (R)-(-)-JQ1 est le stéréoisomère de (+)-JQ1. (+)-JQ1 diminue puissamment l'expression des deux gènes cibles de BRD4, alors que l'énantiomère (R)-(-)-JQ1 n'a aucun effet.
-
GC45908
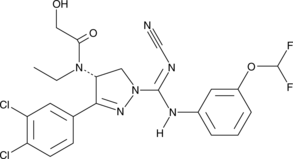
(R)-BAY-598
An inhibitor of SMYD2
-
GC34443
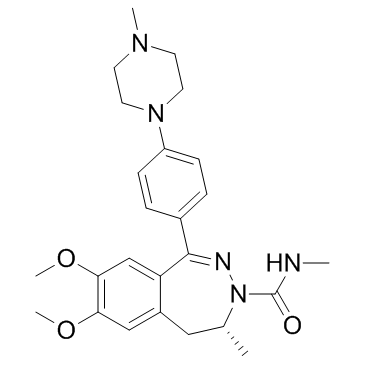
(R)-BAY1238097
(R)-BAY1238097 est l'isomère R avec une activité plus faible de BAY1238097. BAY1238097 est un inhibiteur puissant et sélectif de la liaison BET aux histones et possède une forte activité anti-proliférative dans différents modèles AML (leucémie myéloÏde aiguë) et MM (myélome multiple) grÂce À la régulation À la baisse des niveaux de c-Myc et de son transcriptome en aval.
-
GC64210
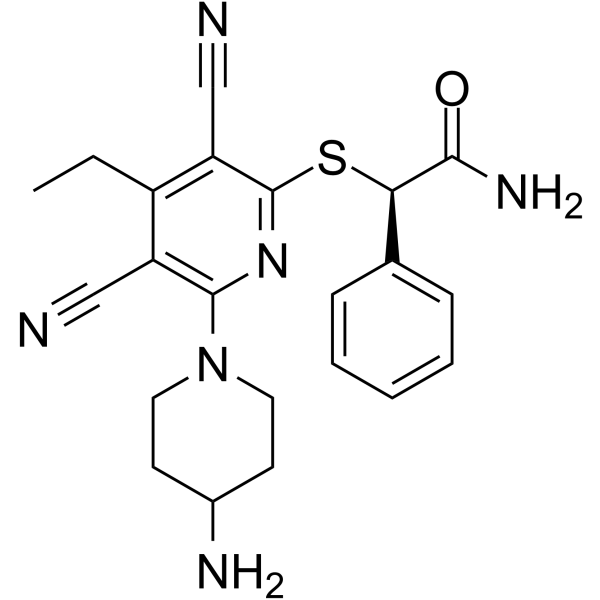
(R)-GSK-3685032
(R)-GSK-3685032 est l'énantiomère R de GSK-3685032. GSK-3685032 est un inhibiteur sélectif de la DNMT1 réversible, non dépendant du temps et non covalent, avec une IC50 de 0,036 μM. GSK-3685032 induit une perte robuste de méthylation de l'ADN, une activation de la transcription et une inhibition de la croissance des cellules cancéreuses.
-
GC11340
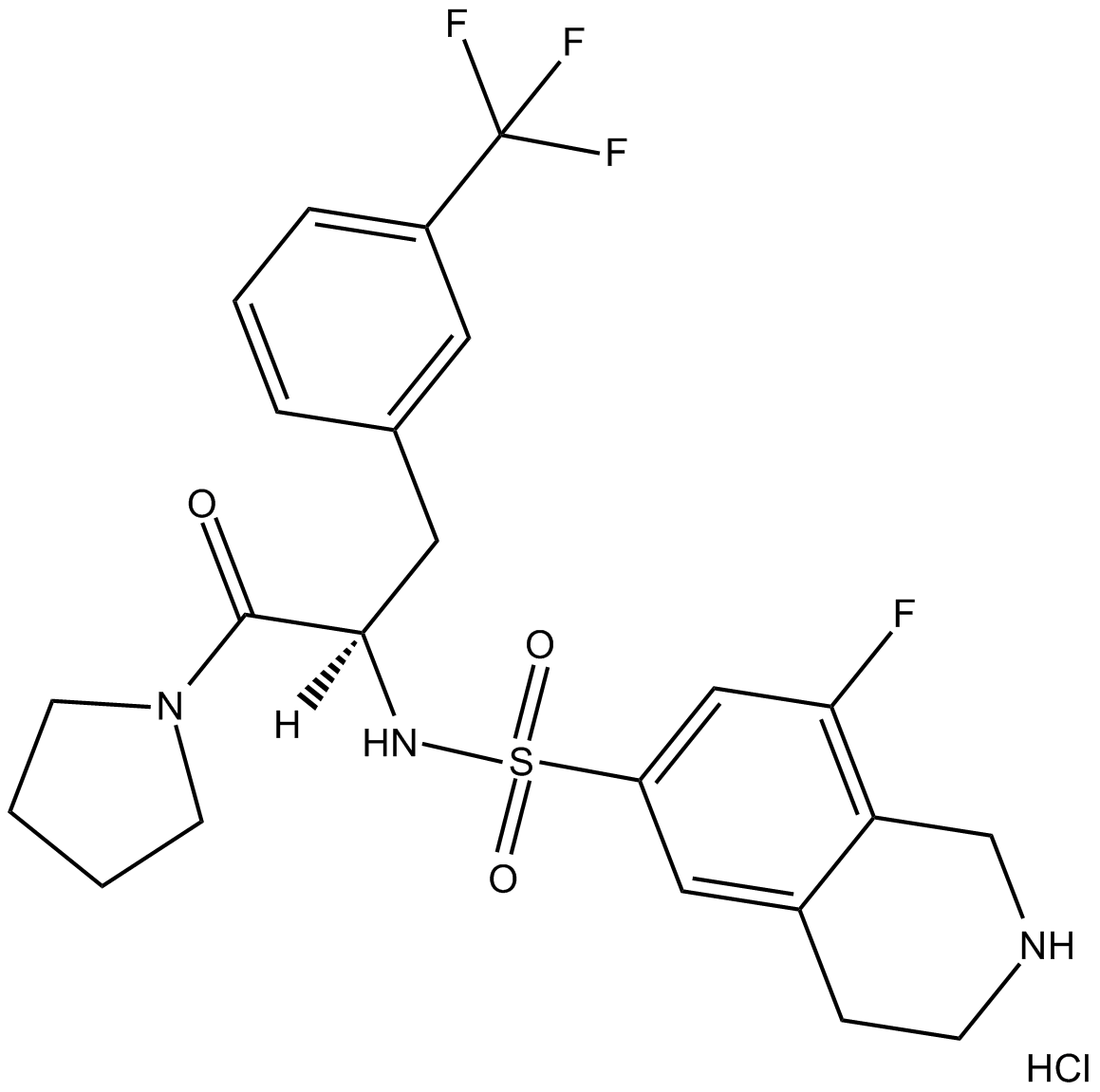
(R)-PFI 2 hydrochloride
Le chlorhydrate de PFI-2 ((R)-(R)-PFI 2 hydrochloride) est un domaine SET puissant et sélectif contenant un inhibiteur de la lysine méthyltransférase 7 (SETD7).
-
GC69794
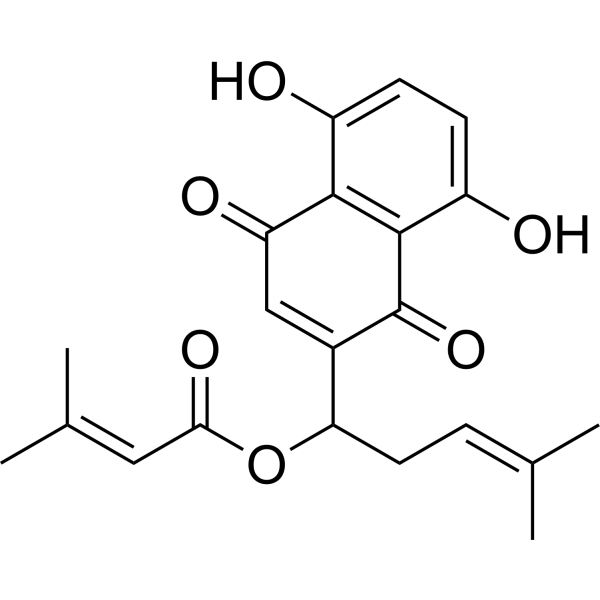
(Rac)-Arnebin 1
(Rac)-Arnebin 1 ((Rac)-β,β-Diméthylacrylalkannin) est un énantiomère de β,β-Diméthylacrylalkannin et/ou de β,β-Diméthylacrylshikonin. Ces derniers sont des composés quinoniques isolés à partir de plantes du genre Lithospermum. Le β,β-Diméthylacrylshikonin possède une activité anti-tumorale.
-
GC34446
(rac)-BAY1238097
(Rac)-BAY1238097 est un inhibiteur de BET, avec une IC50 de 1,02 μM pour BRD4. Utilisé dans la recherche sur le cancer.
-
GC60004
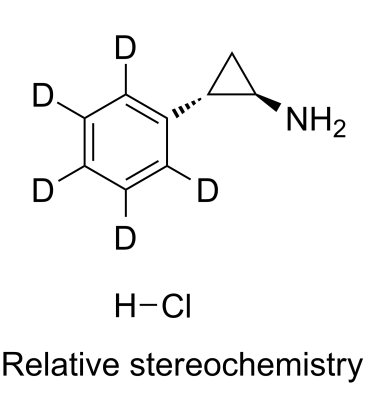
(rel)-Tranylcypromine D5 hydrochloride
Le chlorhydrate de tranylcypromine-d5 (SKF 385-d5) est un chlorhydrate de (rel)-tranylcypromine marqué au deutérium.
-
GC33198
(S)-JQ-35 (TEN-010)
(S)-JQ-35 (TEN-010) (TEN-010) est un inhibiteur des protéines contenant des bromodomaines de la famille Bromodomain et Extra-Terminal (BET) avec une activité antinéoplasique potentielle.
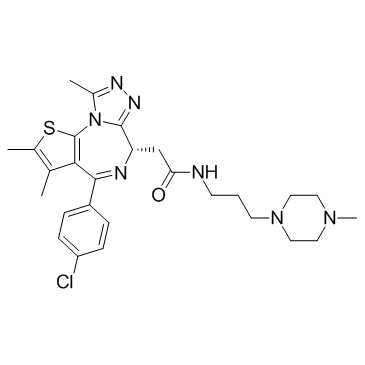
-
GC65045
(S)-MRTX-1719
(S)-MRTX-1719 (exemple 16-7) est l'énantiomère S du MRTX-1719. (S)-MRTX-1719 est un inhibiteur du complexe PRMT5/MTA, avec une IC50 de 7070 nM.
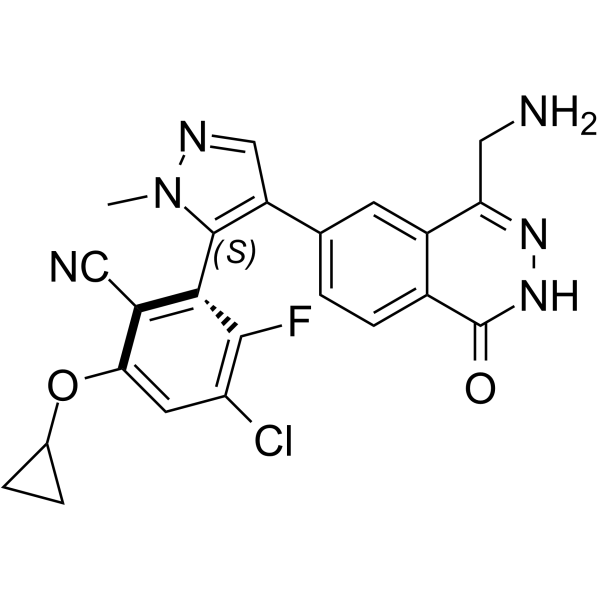
-
GC13634
(S)-PFI-2 (hydrochloride)
Negative control of (R)-PFI 2 hydrochloride
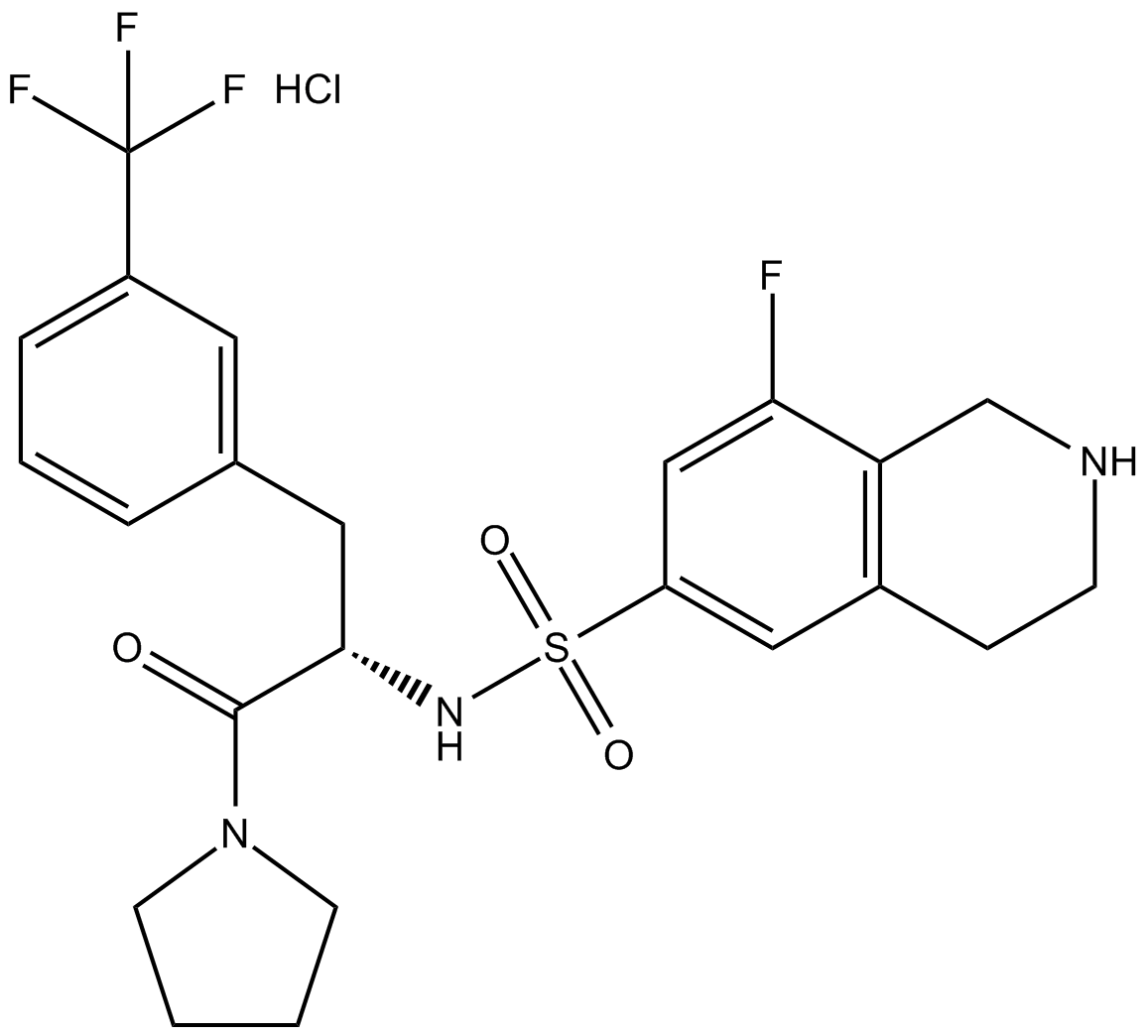
-
GC65133
(S,R,S)-AHPC-C5-COOH
(S,R,S)-AHPC-C5-COOH (VH032-C5-COOH) est un conjugué ligand-lieur de ligase E3 synthétisé, contient le ligand À base de VH032 VHL et un lieur pour former des PROTAC. VH-032 est un inhibiteur sélectif et puissant de l'interaction VHL/HIF-1α avec un Kd de 185 nM, a le potentiel pour l'étude de l'anémie et des maladies ischémiques.
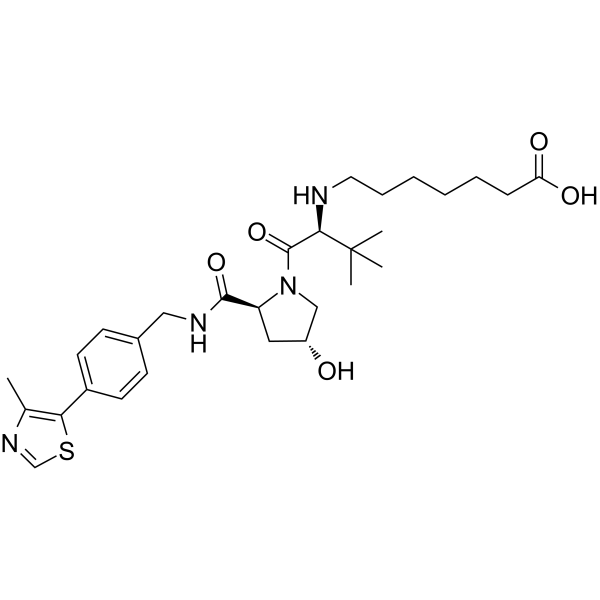
-
GC17055
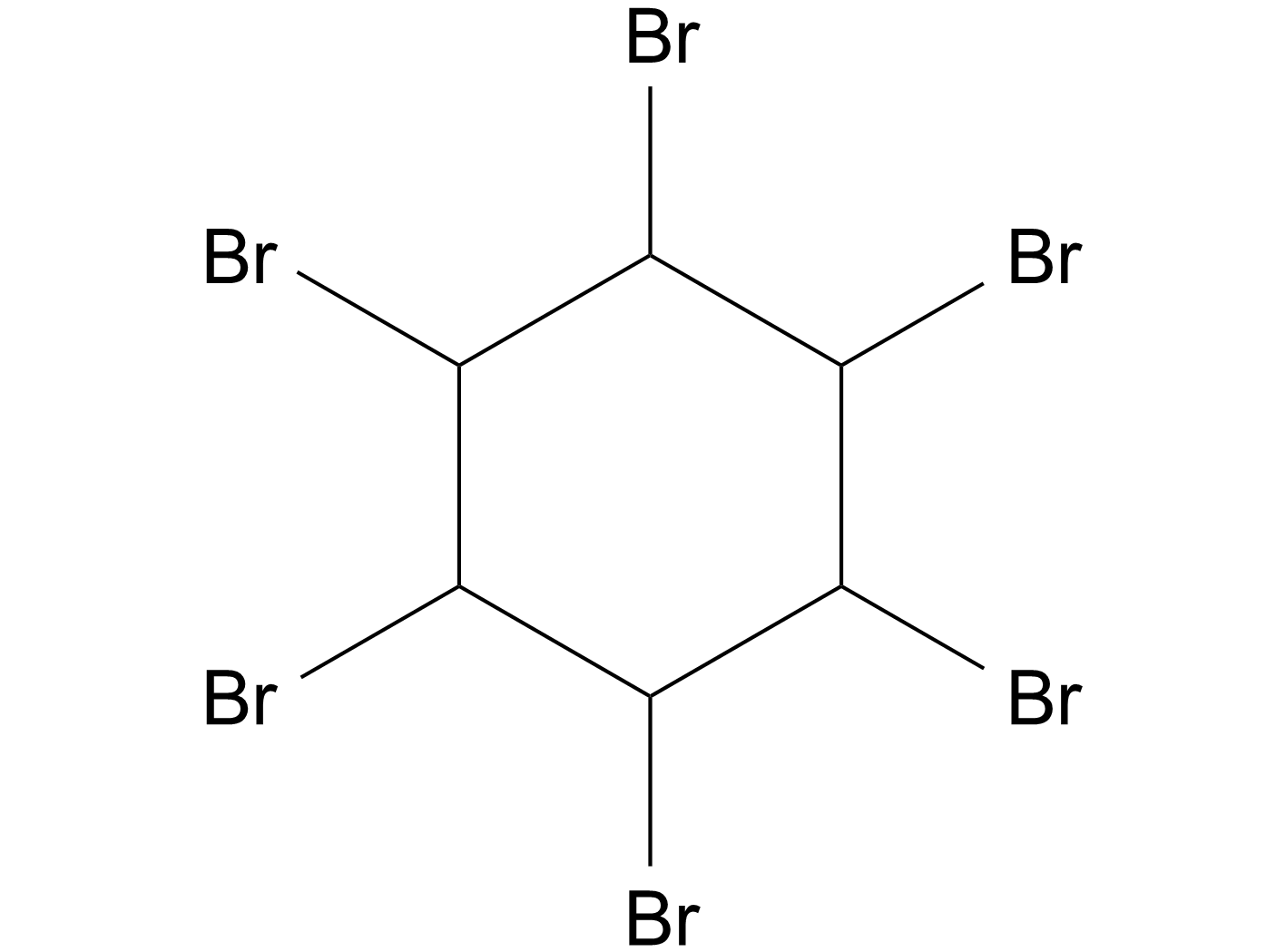
1,2,3,4,5,6-Hexabromocyclohexane
1,2,3,4,5,6-六溴环己烷(1,2,3,4,5,6-Hexabromocyclohexane)直接抑制JAK2激酶结构域内的口袋,抑制自身磷酸化。
-
GC61949
1,4-DPCA ethyl ester
L'ester éthylique de 1,4-DPCA est l'ester éthylique du 1,4-DPCA et peut inhiber le facteur inhibiteur du HIF (FIH).

-
GC40468
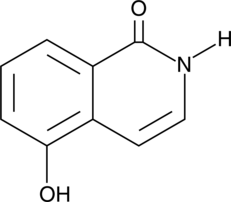
1,5-Isoquinolinediol
Le 1,5-isoquinolinediol est un puissant inhibiteur de PARP, avec une IC50 de 0,18-0,37 μM.
-
GC41984
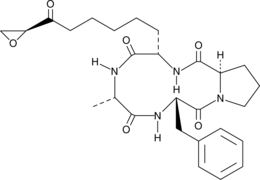
1-Alaninechlamydocin
La 1-alaninechlamydocine, un tétrapeptide cyclique, est un puissant inhibiteur d'HDAC (IC50 = 6,4 nM). La 1-alaninechlamydocine induit l'arrêt du cycle cellulaire G2/M et l'apoptose dans les cellules MIA PaCa-2.
-
GC39214
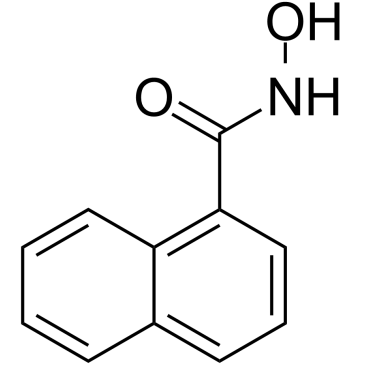
1-Naphthohydroxamic acid
L'acide 1-naphtohydroxamique (composé 2) est un inhibiteur HDAC8 puissant et sélectif avec une IC50 de 14 μM. L'acide 1-naphtohydroxamique est plus sélectif pour HDAC8 que la classe I HDAC1 et la classe II HDAC6 (IC50> 100 μM). L'acide 1-naphtohydroxamique n'augmente pas l'acétylation globale de l'histone H4 et ne réduit pas non plus l'activité intracellulaire totale des HDAC. L'acide 1-naphtohydroxamique peut induire l'acétylation de la tubuline.
-
GC46508
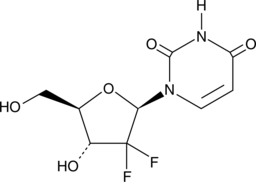
2',2'-Difluoro-2'-deoxyuridine
An active metabolite of gemcitabine
-
GC12775
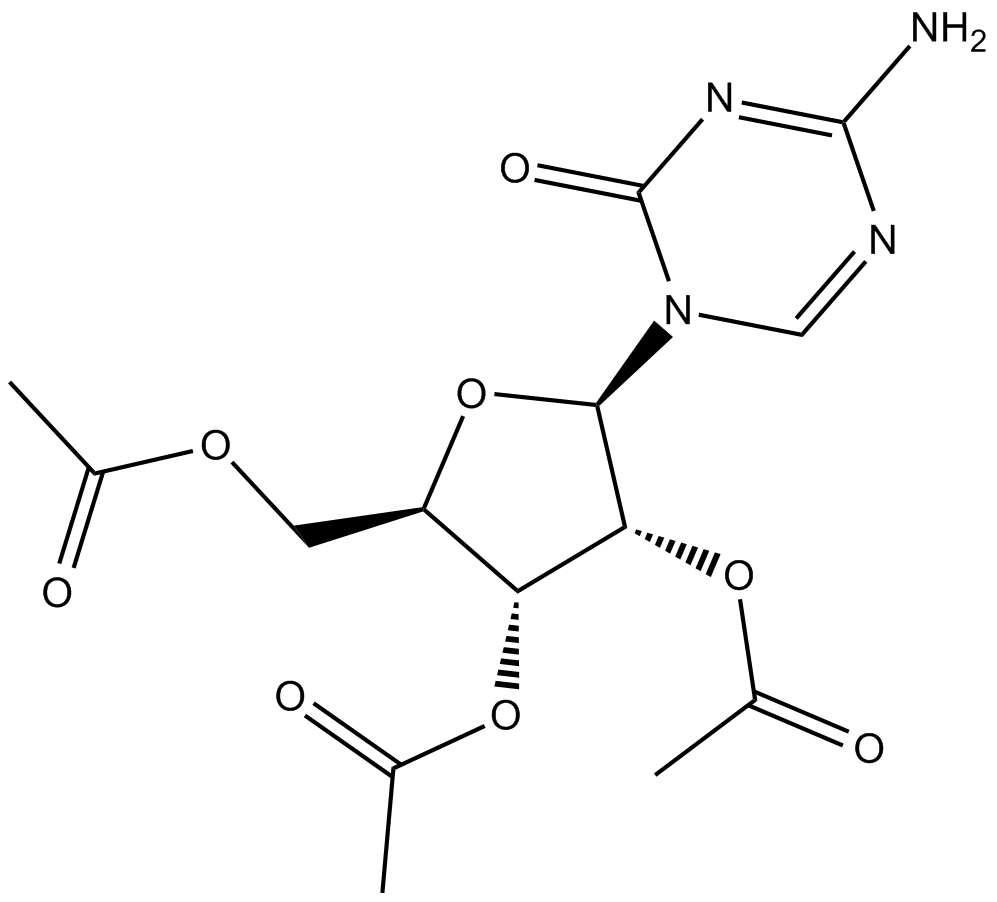
2',3',5'-triacetyl-5-Azacytidine
2',3',5'-triacétyl-5-azacytidine est un promédicament actif par voie orale de la 5-azacytidine. La 5-azacytidine est un inhibiteur de l'ADN méthyltransférase.
-
GC16195
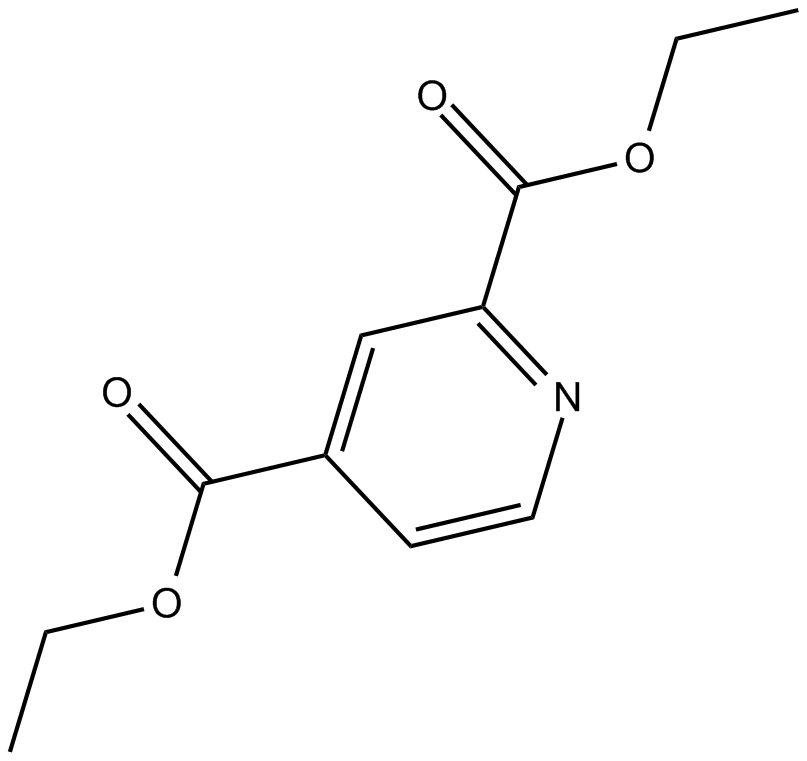
2,4-DPD
Le diéthyl pyridine-2,4-dicarb est un puissant pro-inhibiteur dirigé par la prolyl 4-hydroxylase.
-
GC11873
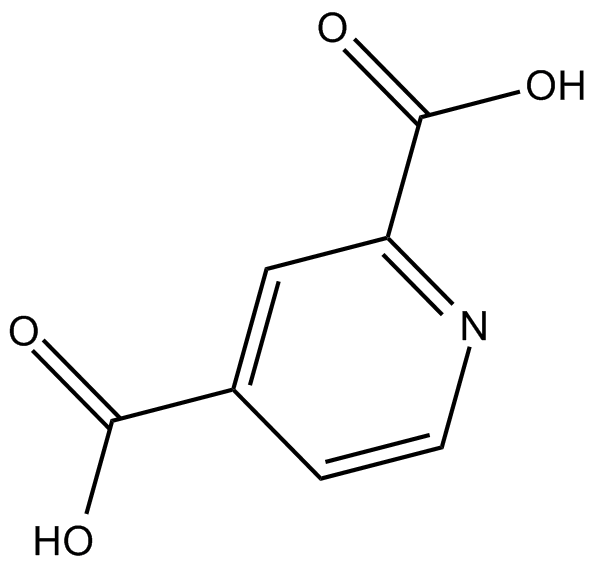
2,4-Pyridinedicarboxylic Acid
L'acide 2,4-pyridinedicarboxylique (acide 2,4 pyridine dicarboxylique) est un inhibiteur À large spectre de la 2OG oxygénase, y compris la famille des histones déméthylases (JHDM) contenant le domaine JmjC. L'acide 2,4-pyridinedicarboxylique est un produit chimique cible dans le domaine des bioplastiques.
-
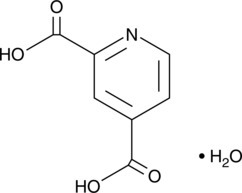
GC42075
2,4-Pyridinedicarboxylic Acid (hydrate)
2,4-Pyridinedicarboxylic Acid (2,4-PDCA) is a compound that structurally mimics 2-oxoglutarate (2-OG, also known as α-ketoglutarate) and chelates zinc, thus affecting a range of enzymes.
-
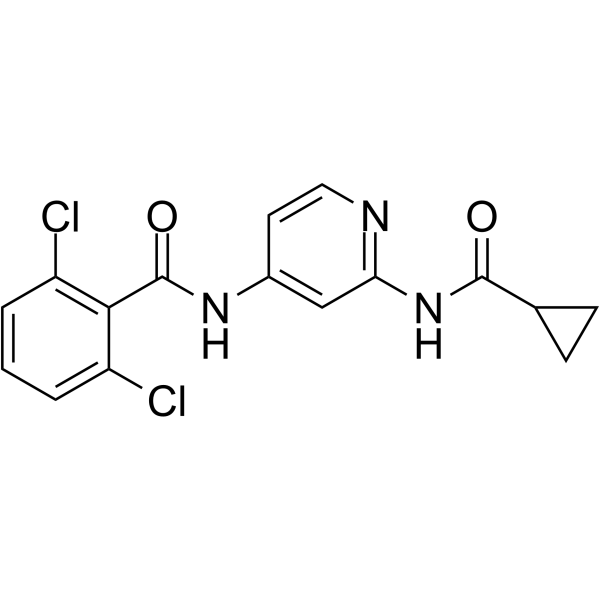
GC62233
2,6-Dichloro-N-(2-(cyclopropanecarboxamido)pyridin-4-yl)benzamide
Le GDC-046 est un inhibiteur de TYK2 puissant, sélectif et biodisponible par voie orale avec un Kis de 4,8, 0,7, 0,7 et 0,4 nM pour TYK2, JAK1, JAK2 et JAK3, respectivement .
-
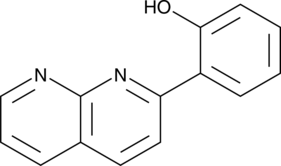
GC46503
2-(1,8-Naphthyridin-2-yl)phenol
Le 2-(1,8-naphtyridin-2-yl)phénol est un activateur sélectif de la transcription de STAT1. Le 2-(1,8-naphtyridin-2-yl)phénol peut améliorer la capacité de l'IFN-γ ; pour inhiber la prolifération des cellules humaines du cancer du sein et du fibrosarcome.
-
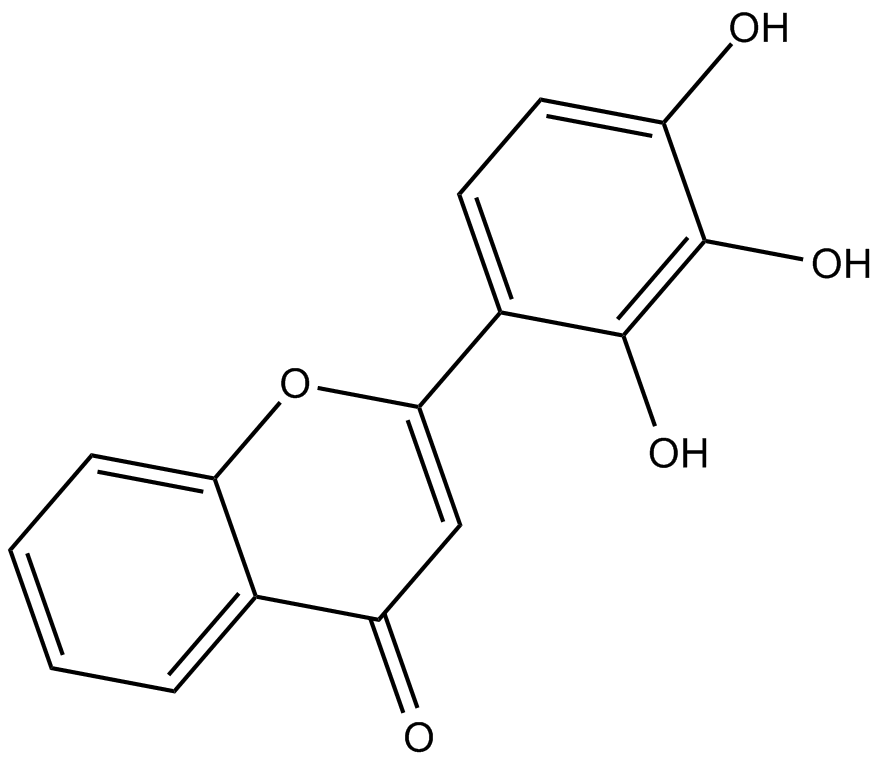
GC10408
2-D08
Le 2-D08 est un inhibiteur perméable aux cellules et mécaniquement unique de la SUMOylation des protéines. Le 2-D08 inhibe également Axl avec une IC50 de 0,49 nM.
-
GC11249
2-hexyl-4-Pentynoic Acid
Potent and robust HDACs inhibitor
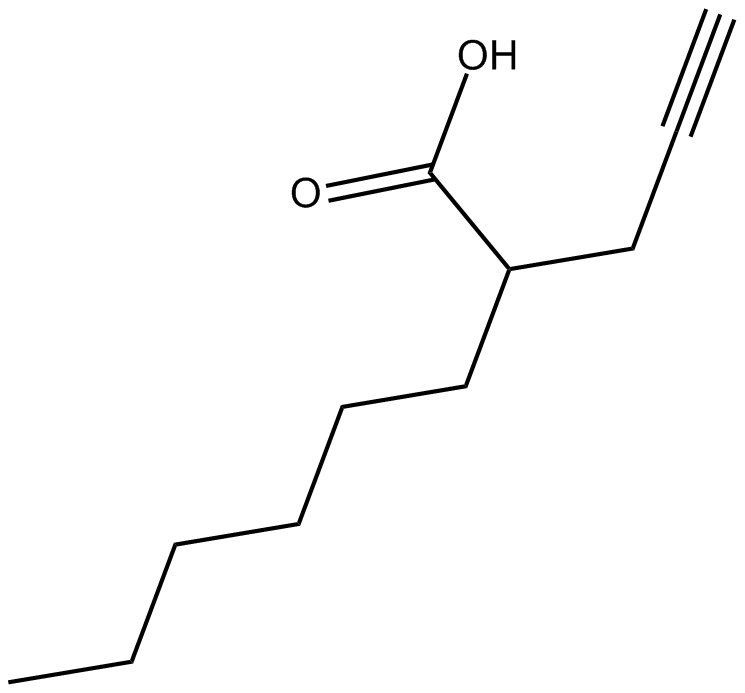
-
GC15084
2-Methoxyestradiol (2-MeOE2)
2-Methoxyestradiol (2-MeOE2/2-Me) is a HIF-1α inhibitor that inhibits HIF-1α accumulation and HIF transcriptional activity. 2-Methoxyestradiol can trigger p53-induced apoptosis and has potential antitumor activity..
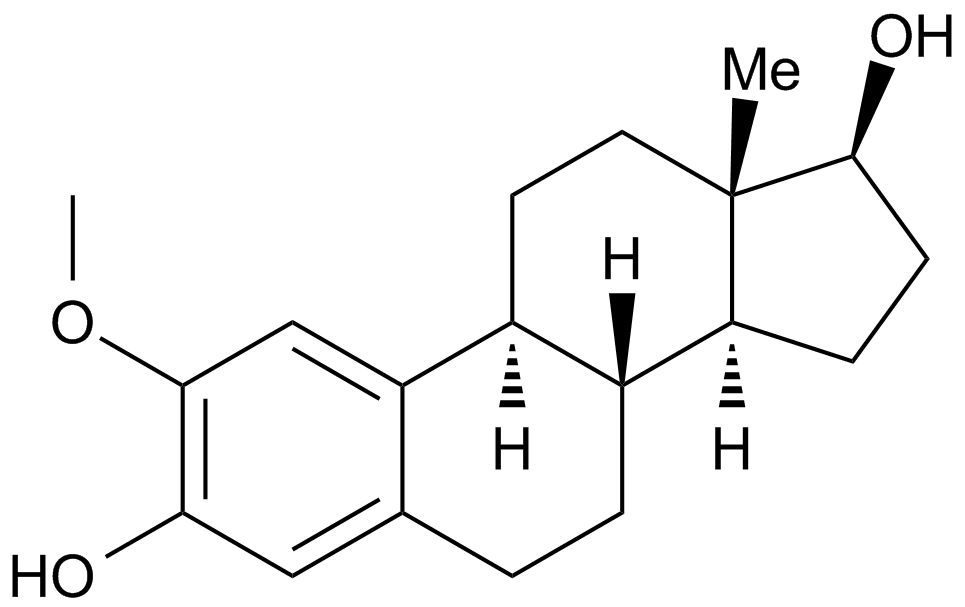
-
GC62781
2-Methylquinazolin-4-ol
Le 2-méthylquinazolin-4-ol est un puissant inhibiteur compétitif de la poly(ADP-ribose) synthétase, avec un Ki de 1,1 μM.
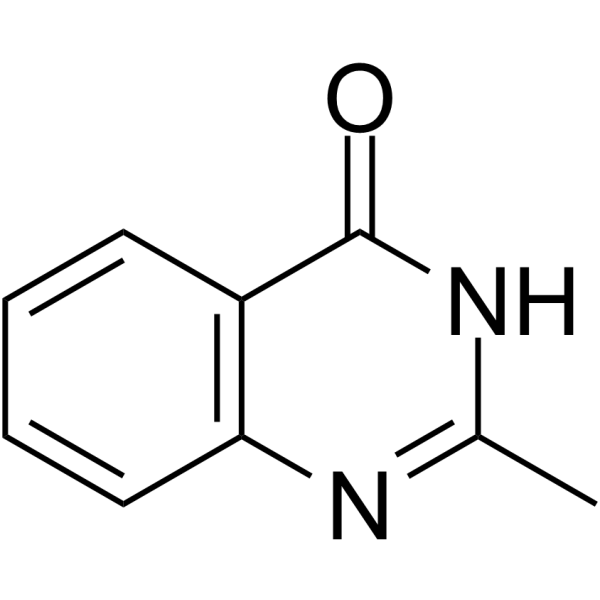
-
GC14282
3-acetyl-11-keto-β-Boswellic Acid
L'acide 3-acétyl-11-céto-β-boswellique (acétyl-11-céto-β-acide boswellique) est un composé triterpénoÏde actif issu de l'extrait de boswellia serrate et un nouvel activateur Nrf2.
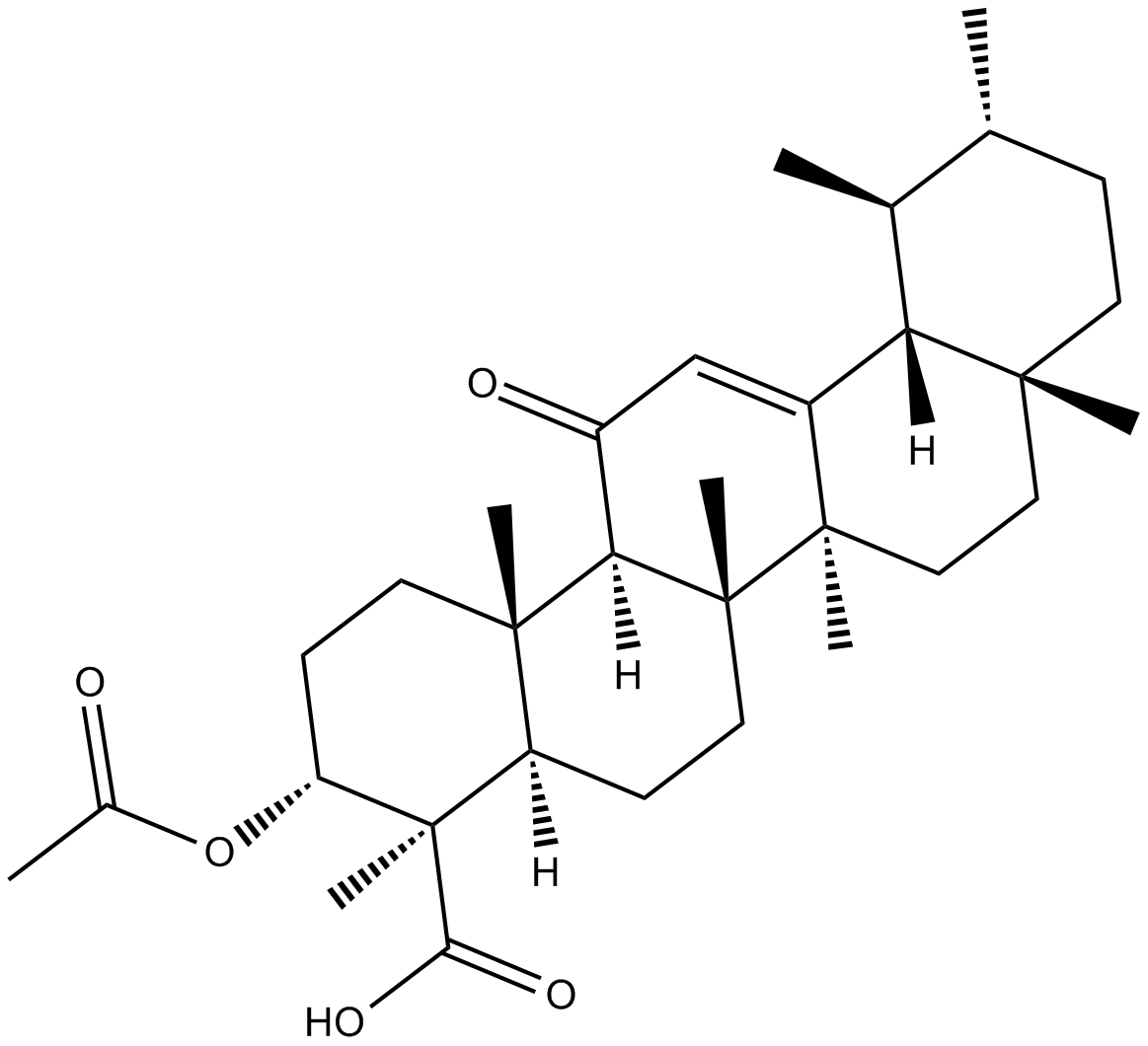
-
GC17907
3-Deazaneplanocin A (DZNep) hydrochloride
Un inhibiteur de la lysine méthyltransférase EZH2.
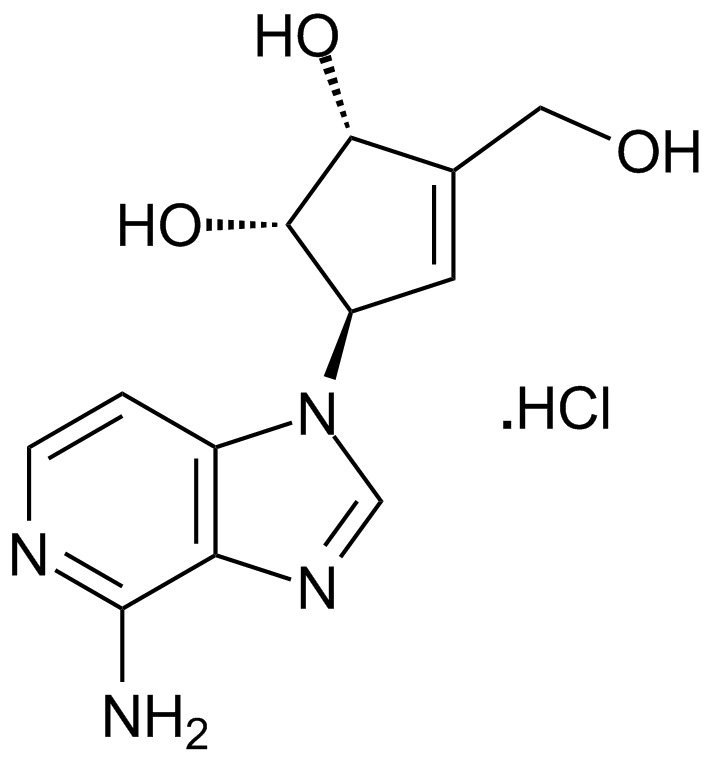
-
GC13145
3-Deazaneplanocin,DZNep
An inhibitor of lysine methyltransferase EZH2
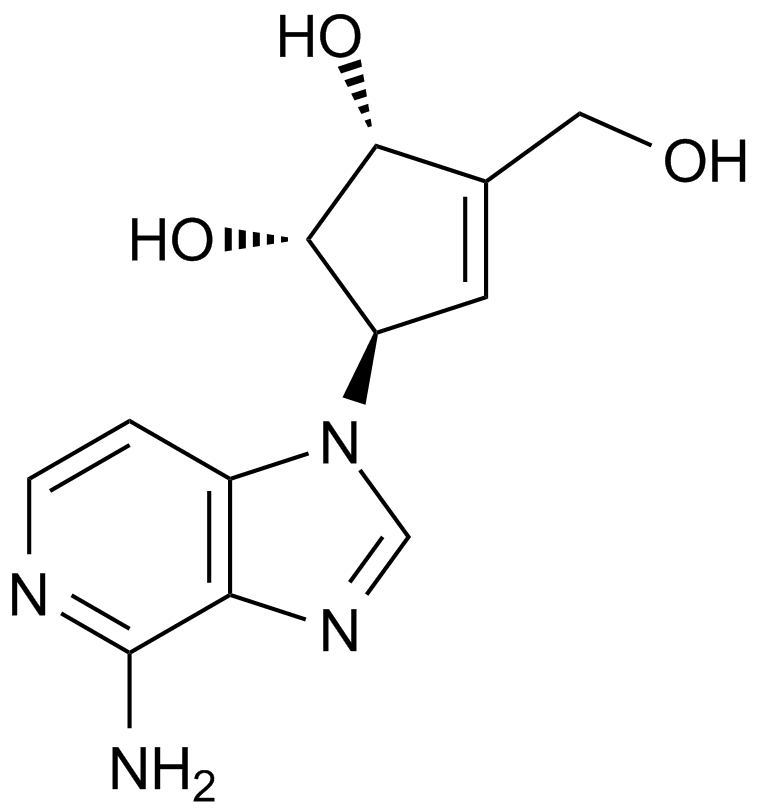
-
GC18509
3-Methylcytidine (methosulfate)
3-Methylcytidine (methosulfate) is a cytidine derivative used as an internal standard for HPLC.
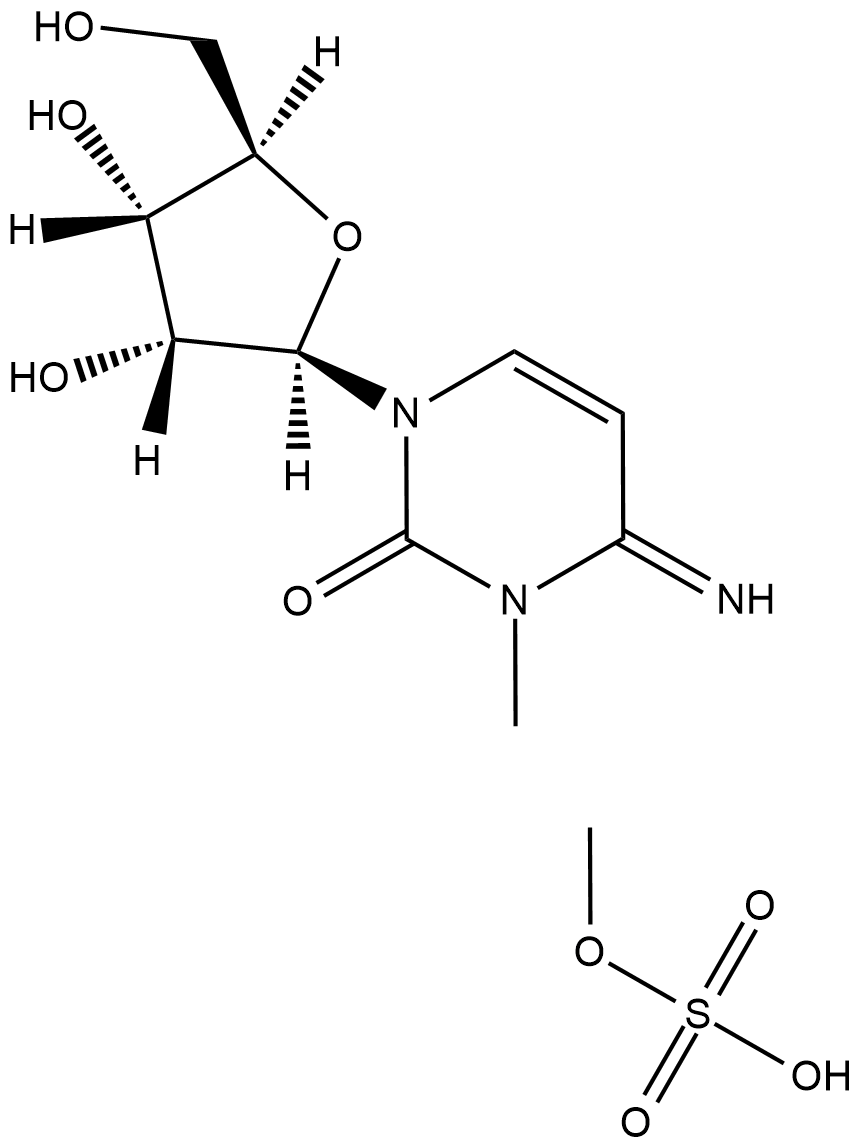
-
GC19013
3-TYP
3-TYP inhibe SIRT3 avec un IC50 de 16 nM, et est plus puissant sur SIRT1 et SIRT2 avec des IC50 de 88 nM et 92 nM, respectivement.
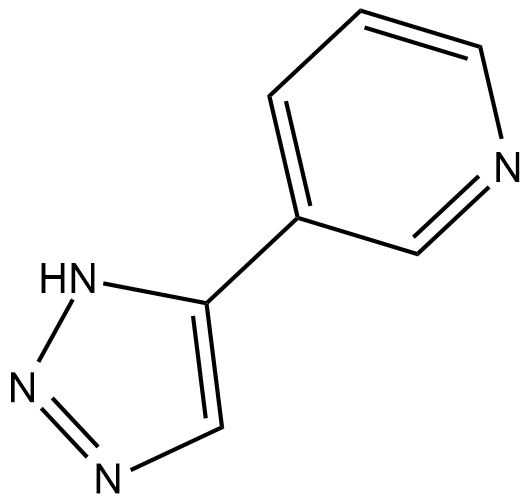
-
GC62805
4’-Methoxychalcone
4’-Methoxychalcone régule la différenciation des adipocytes par l'activation de PPARγ.

-
GC17922
4'-bromo-Resveratrol
Le 4'-bromo-resvératrol est un inhibiteur puissant et double de la sirtuine-1 et de la sirtuine-3. Le 4'-bromo-resvératrol inhibe la croissance des cellules de mélanome par la reprogrammation métabolique mitochondriale. Le 4'-bromo-resvératrol confère des effets antiprolifératifs dans les cellules de mélanome par une reprogrammation métabolique et affecte le cycle cellulaire et la signalisation de l'apoptose.
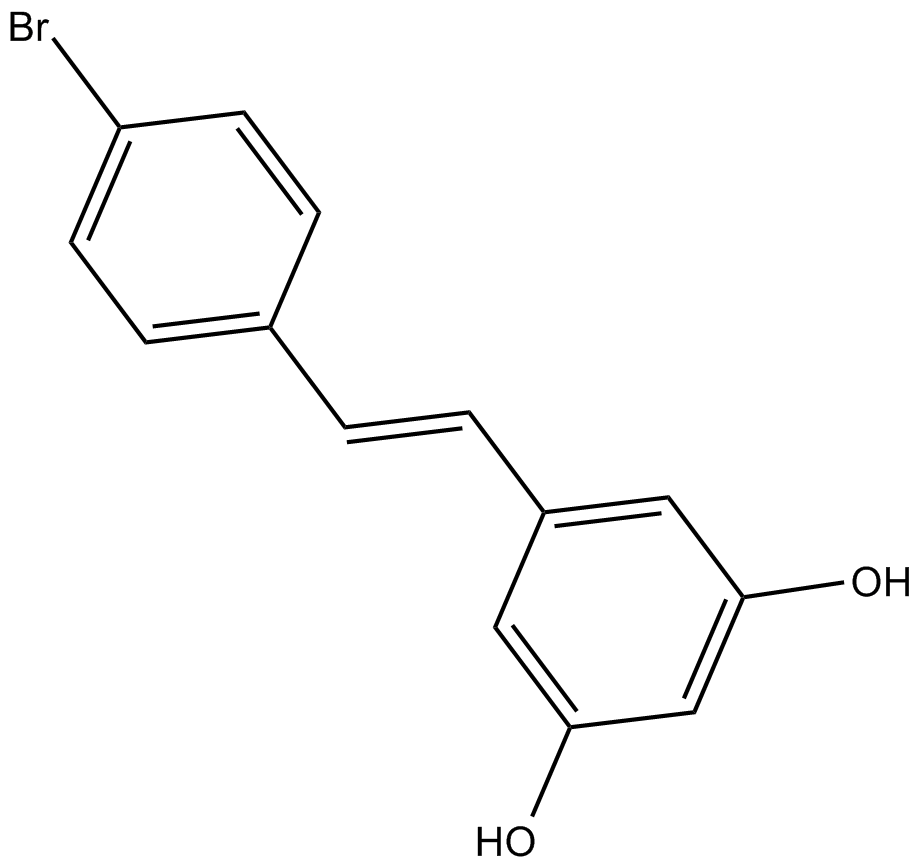
-
GC46607
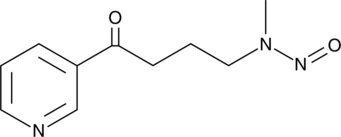
4-(Methylnitrosamino)-1-(3-pyridyl)-1-butanone
A tobacco-specific nitrosamine carcinogen
-
GC11761
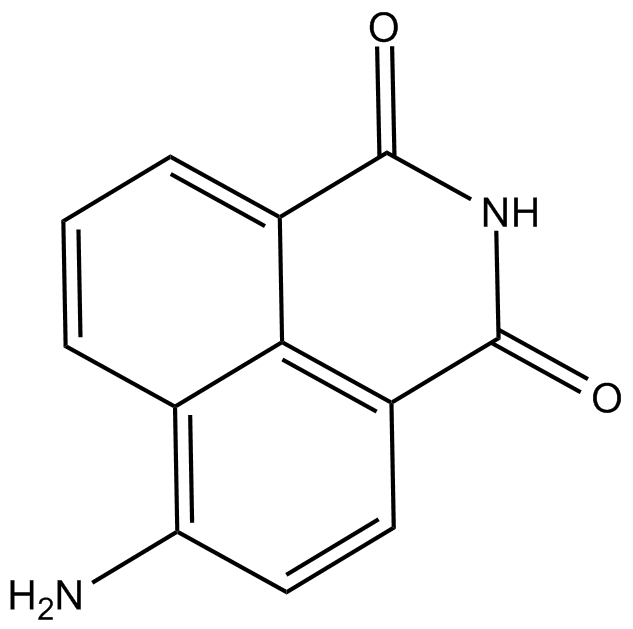
4-amino-1,8-Naphthalimide
Le 4-amino-1,8-naphtalimide est un puissant inhibiteur de PARP et potentialise la cytotoxicité des rayonnements γ-dans les cellules cancéreuses.
-
GC46628
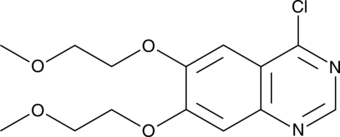
4-Chloro-6,7-bis(2-methoxyethoxy)quinazoline
A building block and synthetic intermediate
-
GC17005
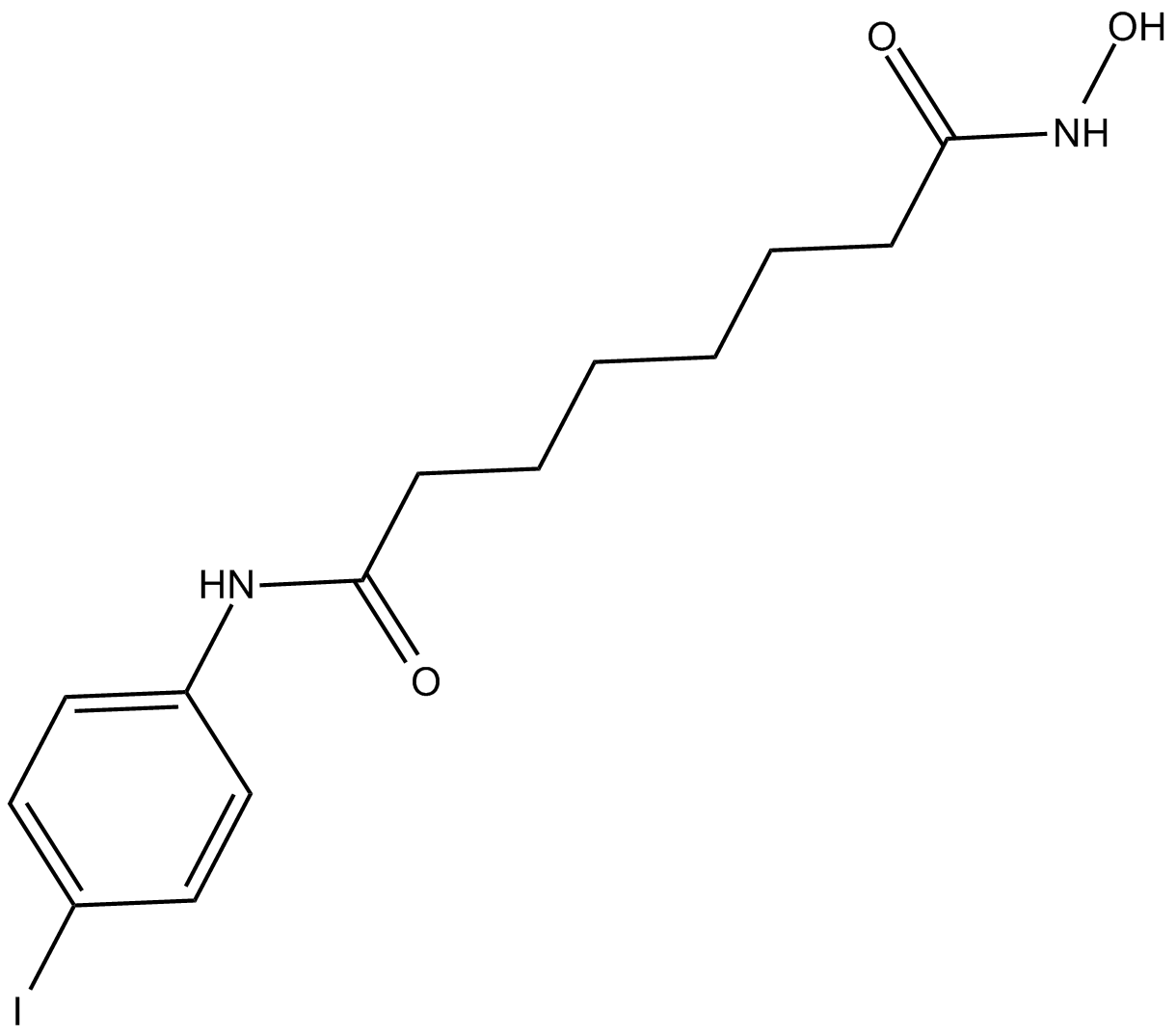
4-iodo-SAHA
Le 4-Iodo-SAHA (1k) est un inhibiteur d'histone désacétylase (HDAC) de classe I et de classe II actif par voie orale avec des CE50 de 1,1, 0,95, 0,12, 0,24, 0,85 et 1,3 μM pour les lignées cellulaires Skbr3, HT29, U937, JA16 et HL60 , respectivement. Le 4-Iodo-SAHA (1k) peut être utilisé pour la recherche sur le cancer.
-
GC46675
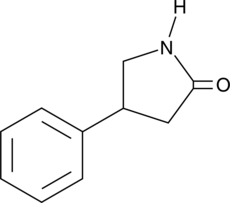
4-Phenyl-2-pyrrolidinone
A precursor and synthetic intermediate
-
GC12667
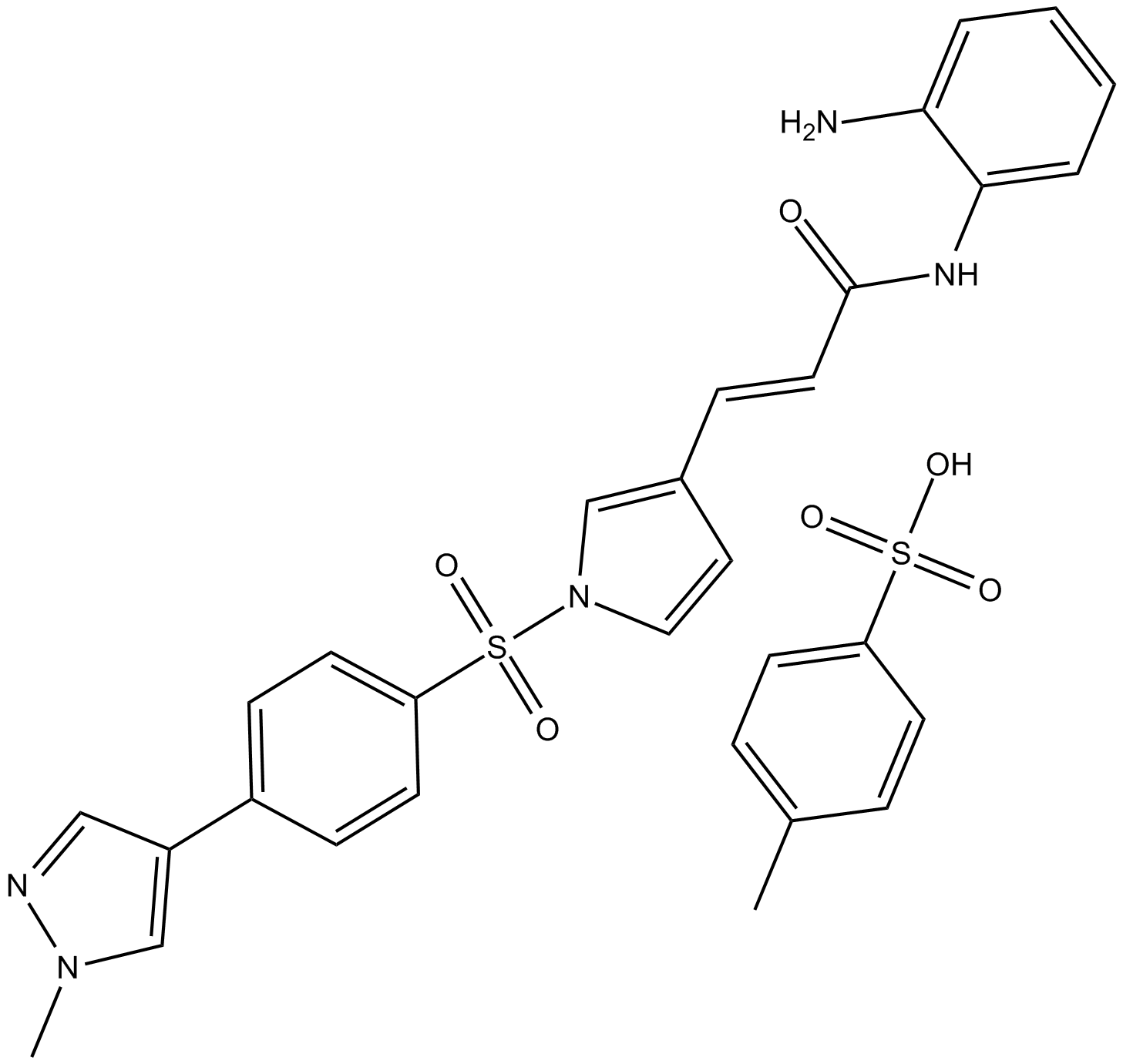
4SC-202
4SC-202 (4SC-202) est un inhibiteur sélectif d'HDAC de classe I avec une IC50 de 1,20 μM, 1,12 μM et 0,57 μM pour HDAC1, HDAC2 et HDAC3, respectivement. Il affiche également une activité inhibitrice contre la déméthylase spécifique de la lysine 1 (LSD1).
-
GC35150
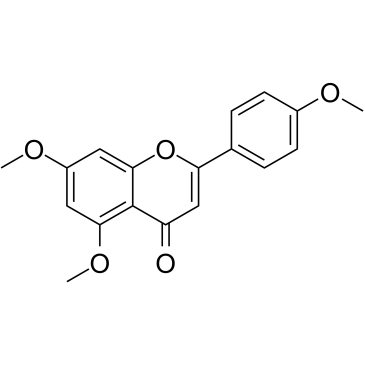
5,7,4'-Trimethoxyflavone
La 5,7,4'-triméthoxyflavone est isolée de Kaempferia parviflora (KP) qui est une célèbre plante médicinale de Thaïlande. La 5,7,4'-triméthoxyflavone induit l'apoptose, comme en témoignent les augmentations de la phase sous-G1, la fragmentation de l'ADN, la coloration annexine-V/PI, le rapport Bax/Bcl-xL, l'activation protéolytique de la caspase-3 et la dégradation de la poly (ADP-ribose) polymérase (PARP). La 5,7,4'-triméthoxyflavone est significativement efficace pour inhiber la prolifération des cellules cancéreuses gastriques humaines SNU-16 d'une manière dépendante de la concentration.
-
GN10629
5,7-dihydroxychromone
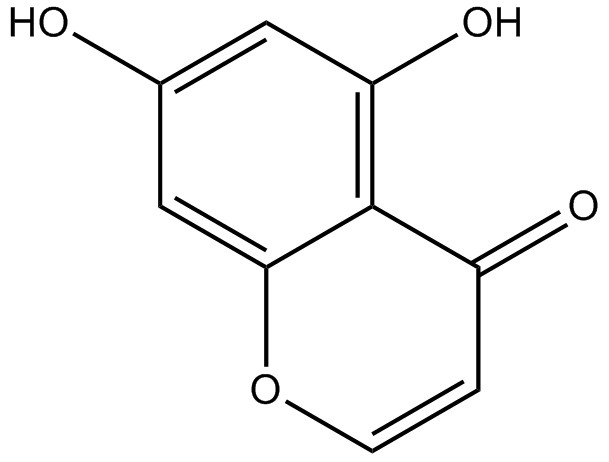
-
GC10898
5-(N,N-dimethyl)-Amiloride (hydrochloride)
NHE1, NHE2, and NHE3 inhibitor
-
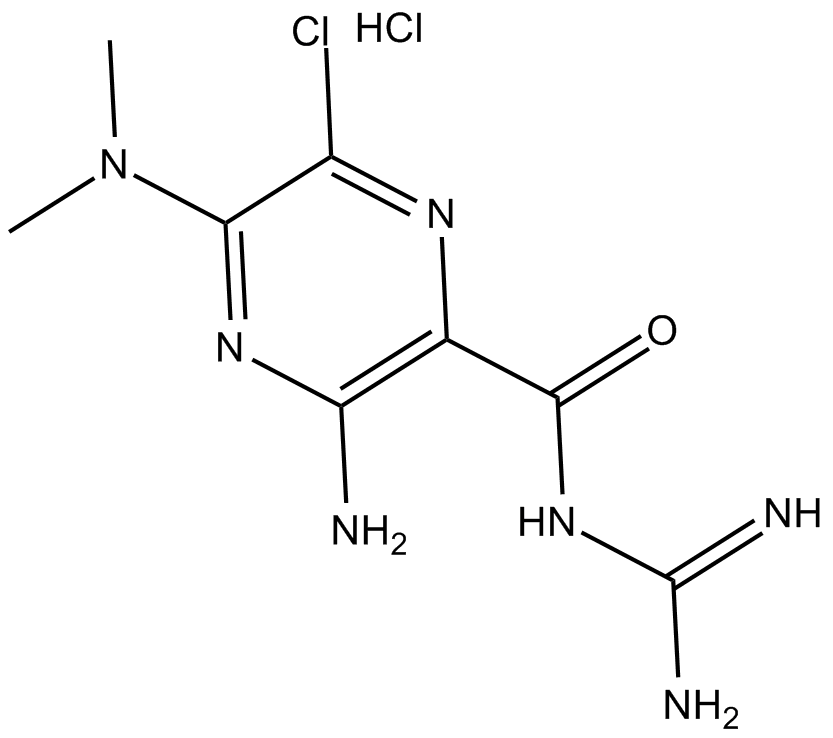
GC68161
5-AIQ
-
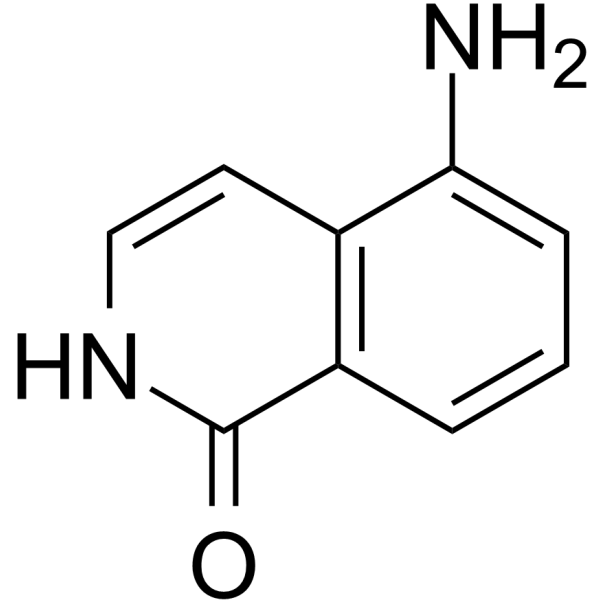
GC10946
5-Azacytidine
A DNA methyltransferase inhibitor
-
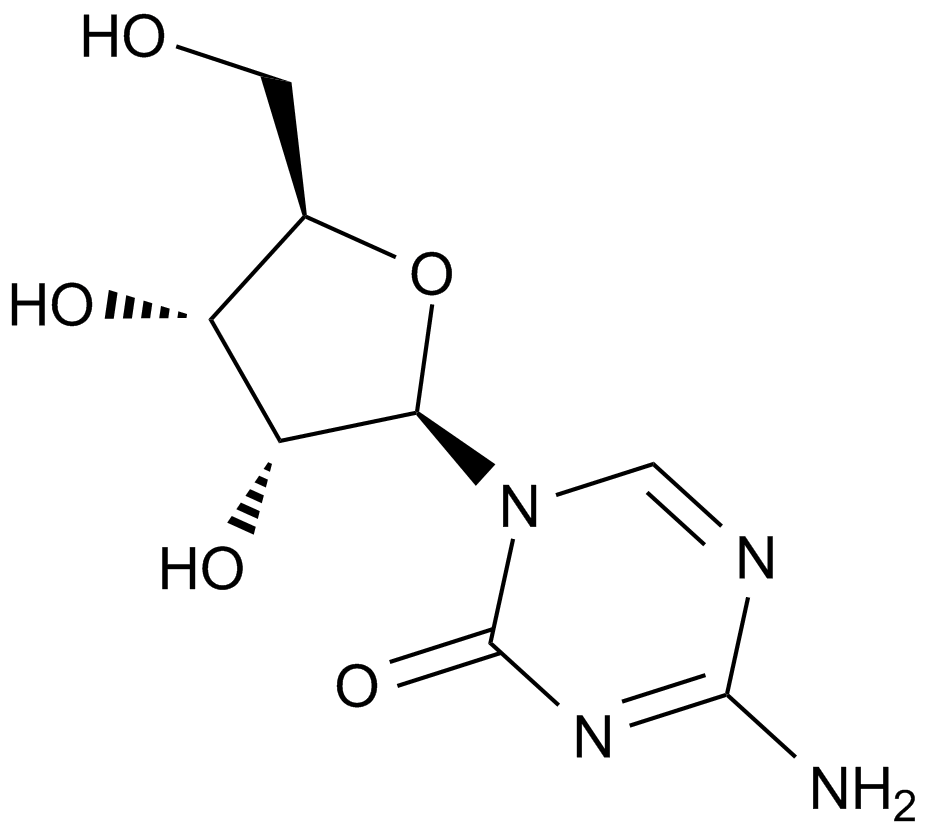
GC61662
5-Fluoro-2'-deoxycytidine
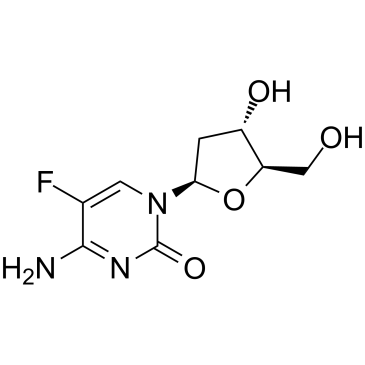
La 5-fluoro-2'-désoxycytidine, un analogue nucléosidique de la fluoropyrimidine, est un inhibiteur de l'ADN méthyltransférase (DNMT). La 5-fluoro-2'-désoxycytidine est une prodrogue sélective pour les tumeurs du puissant inhibiteur de la thymidylate synthase, le 5-fluoro-2'-dUMP.
-
GC42562
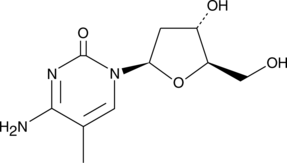
5-Methyl-2'-deoxycytidine
La 5-méthyl-2'-désoxycytidine dans l'ADN simple brin peut agir en cis pour signaler la méthylation de novo de l'ADN .
-
GC66055
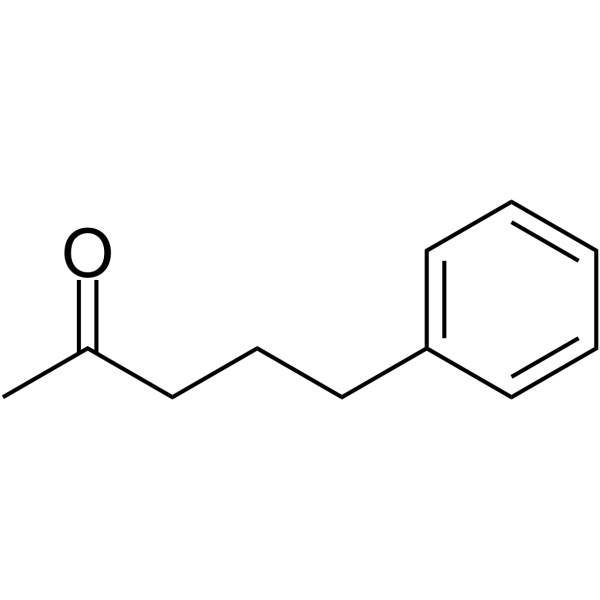
5-Phenylpentan-2-one
La 5-phénylpentane-2-one est un puissant inhibiteur des histones désacétylases (HDAC). Le 5-phénylpentan-2-one peut être utilisé pour la recherche sur les troubles du cycle de l'urée.
-
GC45772
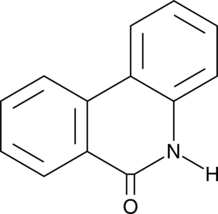
6(5H)-Phenanthridinone
An inhibitor of PARP1 and 2
-
GC35175
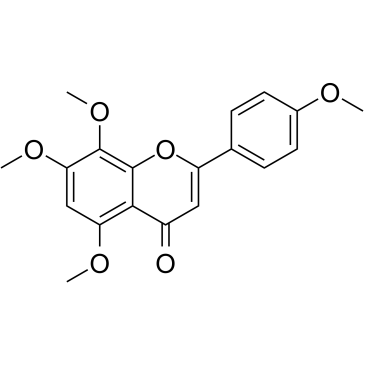
6-Demethoxytangeretin
La 6-déméthoxytangérétine est un flavonoÏde d'agrumes isolé de Citrus depressa.
-
GC35180
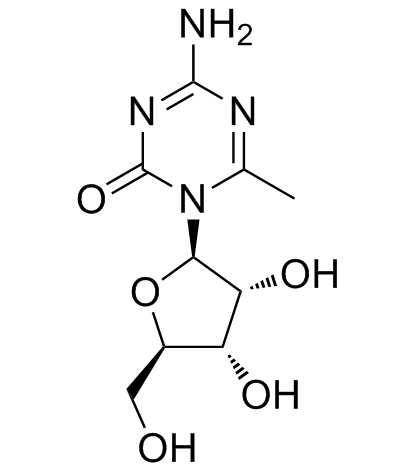
6-Methyl-5-azacytidine
La 6-méthyl-5-azacytidine est un puissant inhibiteur de la DNMT.
-
GC62279
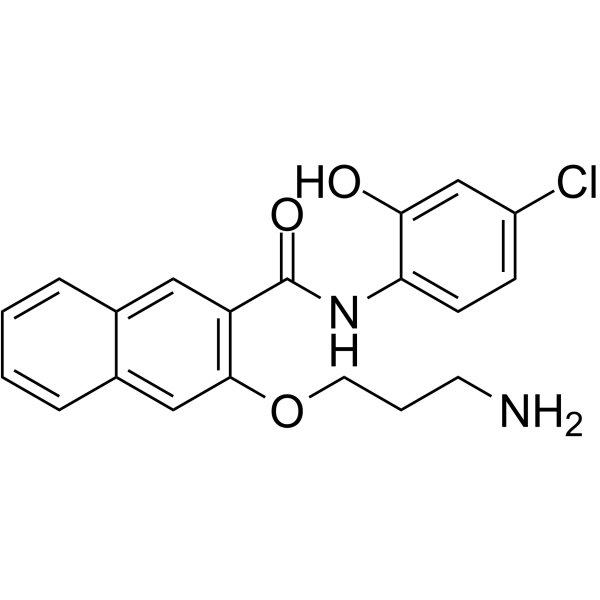
653-47
653-47, un potentialisateur, potentialise significativement l'activité inhibitrice de la protéine de liaison À l'élément de réponse À l'AMPc (CREB) de 666-15. Le 653-47 est également un inhibiteur CREB très faible avec une IC50 de 26,3 μM.
-
GC62144
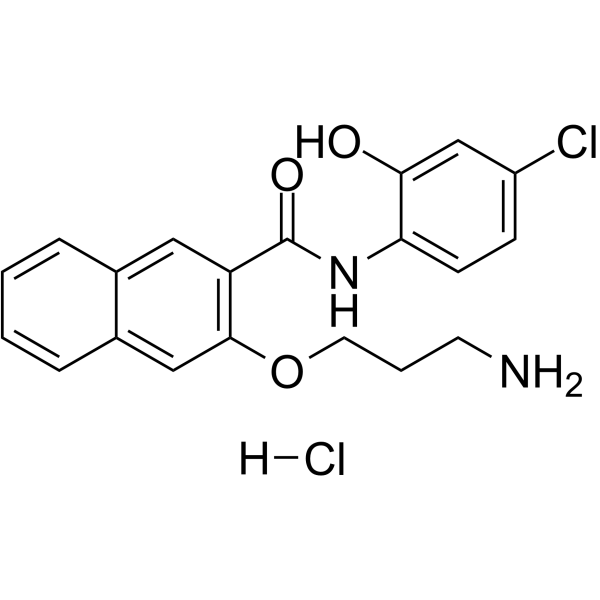
653-47 hydrochloride
Le chlorhydrate de 653-47, un potentialisateur, potentialise de manière significative l'activité inhibitrice de la protéine de liaison À l'élément de réponse À l'AMPc (CREB) de 666-15. Le chlorhydrate de 653-47 est également un très faible inhibiteur de CREB avec une IC50 de 26,3 μM.
-
GC32689
666-15
666-15 est un inhibiteur CREB puissant et sélectif avec une IC50 de 81 nM. 666-15 supprime la croissance tumorale dans un modèle de xénogreffe de cancer du sein.
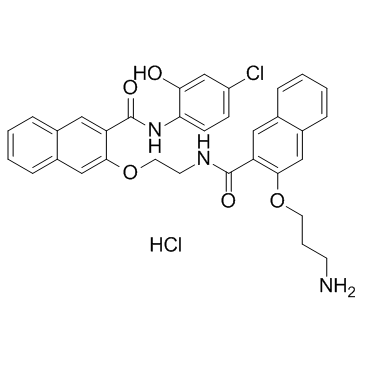
-
GC46736
7-Bromoheptanoic Acid
A building block
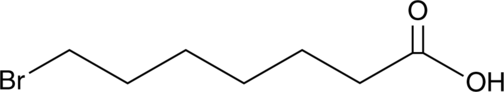
-
GC62651
7-Chloro-4-(piperazin-1-yl)quinoline
La 7-chloro-4-(pipérazin-1-yl)quinolone est un échafaudage important en chimie médicinale.
-
GC48986
9-hydroxy Stearic Acid
A hydroxy fatty acid
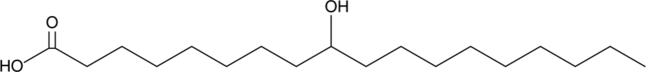
-
GC16015
A 366
A 366 est un puissant inhibiteur de l'histone méthyltransférase G9a, hautement sélectif et compétitif avec les peptides, avec des CI50 de 3,3 et 38 nM pour G9a et GLP (EHMT1), respectivement. A 366 montre une sélectivité \u003e 1000 fois supérieure à 21 autres méthyltransférases. A 366 est également un puissant inhibiteur nanomolaire de l'interaction Spindlin1-H3K4me3 (IC50 \u003d 182,6 nM). A 366 affiche une affinité élevée pour le récepteur de l'histamine H3 humain (Ki \u003d 17 nM) et présente une sélectivité de sous-type parmi les sous-ensembles des familles de récepteurs histaminergiques et dopaminergiques.
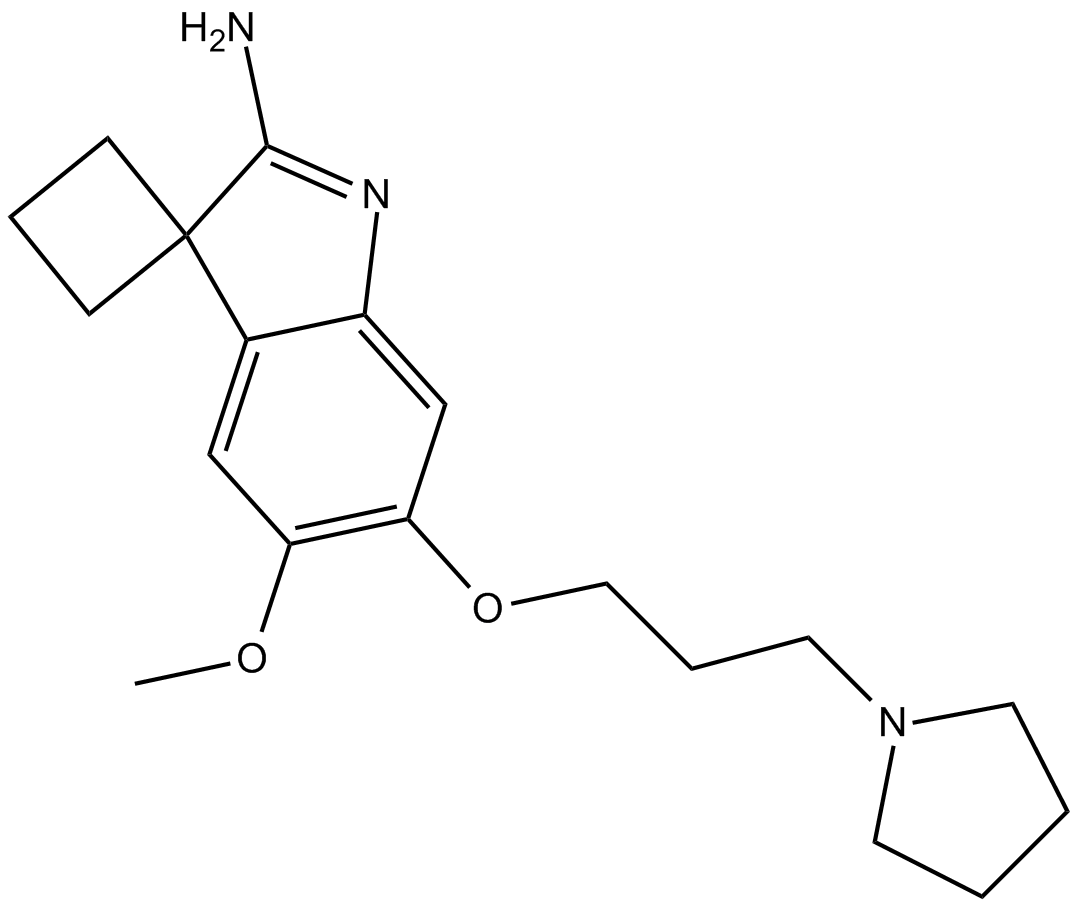
-
GC32861
A-196
A-196 est un inhibiteur puissant et sélectif de SUV420H1 et SUV420H2 avec des valeurs IC50 de 25 nM et 144 nM, respectivement. A-196 inhibe biochimiquement SUV4-20 d'une manière compétitive avec le substrat. A-196 représente une sonde chimique de premier ordre de SUV4-20 pour étudier le rÔle des histones méthyltransférases dans l'intégrité génomique.
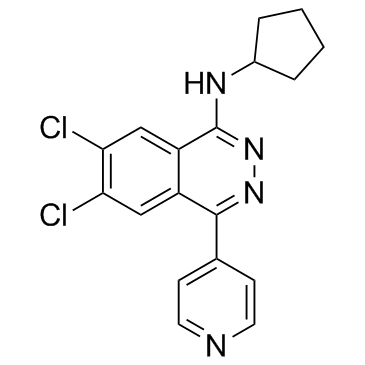
-
GC33187
A-395 (A395)
A-395 (A395) est un antagoniste des interactions protéine-protéine du complexe répressif polycomb 2 (PRC2) qui inhibe puissamment le complexe trimérique PRC2 (EZH2-EED-SUZ12) avec une IC50 de 18 nM.
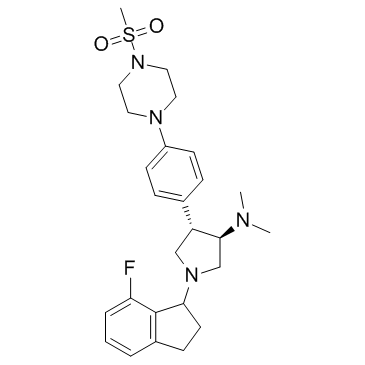
-
GC32677
A-485
A-485 est un inhibiteur catalytique puissant et sélectif de p300/CBP avec des IC50 de 9,8nM et 2,6nM pour p300 et CBP histone acétyltransférase (HAT), respectivement.
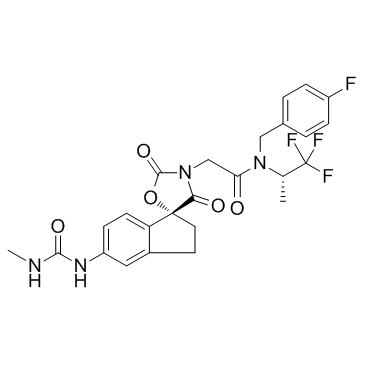
-
GC30503
A-893
A-893 est un inhibiteur cellulaire actif de la méthyltransférase SMYD2, avec une IC50 de 2,8 nM.
-
GC12390
A-966492
A PARP1 and PARP2 inhibitor
-
GC33280
A1874
A1874 est un PROTAC À base de nutline (ligand MDM2) et dégradant BRD4 avec un DC50 de 32 nM (induit la dégradation de BRD4 dans les cellules). Efficace pour inhiber la prolifération de nombreuses lignées cellulaires cancéreuses.
-
GC35216
AAPK-25
AAPK-25 est un double inhibiteur Aurora/PLK puissant et sélectif avec une activité anti-tumorale, qui peut provoquer un retard mitotique et arrêter les cellules dans une prométaphase, reflétée par la phosphorylation de l'histone H3Ser10 du biomarqueur et suivie d'une augmentation de l'apoptose. AAPK-25 cible Aurora-A, -B et -C avec des valeurs de Kd allant de 23 À 289 nM, ainsi que PLK-1, -2 et -3 avec des valeurs de Kd allant de 55 À 456 nM.
-
GC19409
ABBV-744
ABBV-744 est un inhibiteur sélectif et actif par voie orale, premier de sa catégorie, du domaine BDII des protéines de la famille BET avec des valeurs IC50 allant de 4 À 18 nM pour BRD2, BRD3, BRD4 et BRDT.
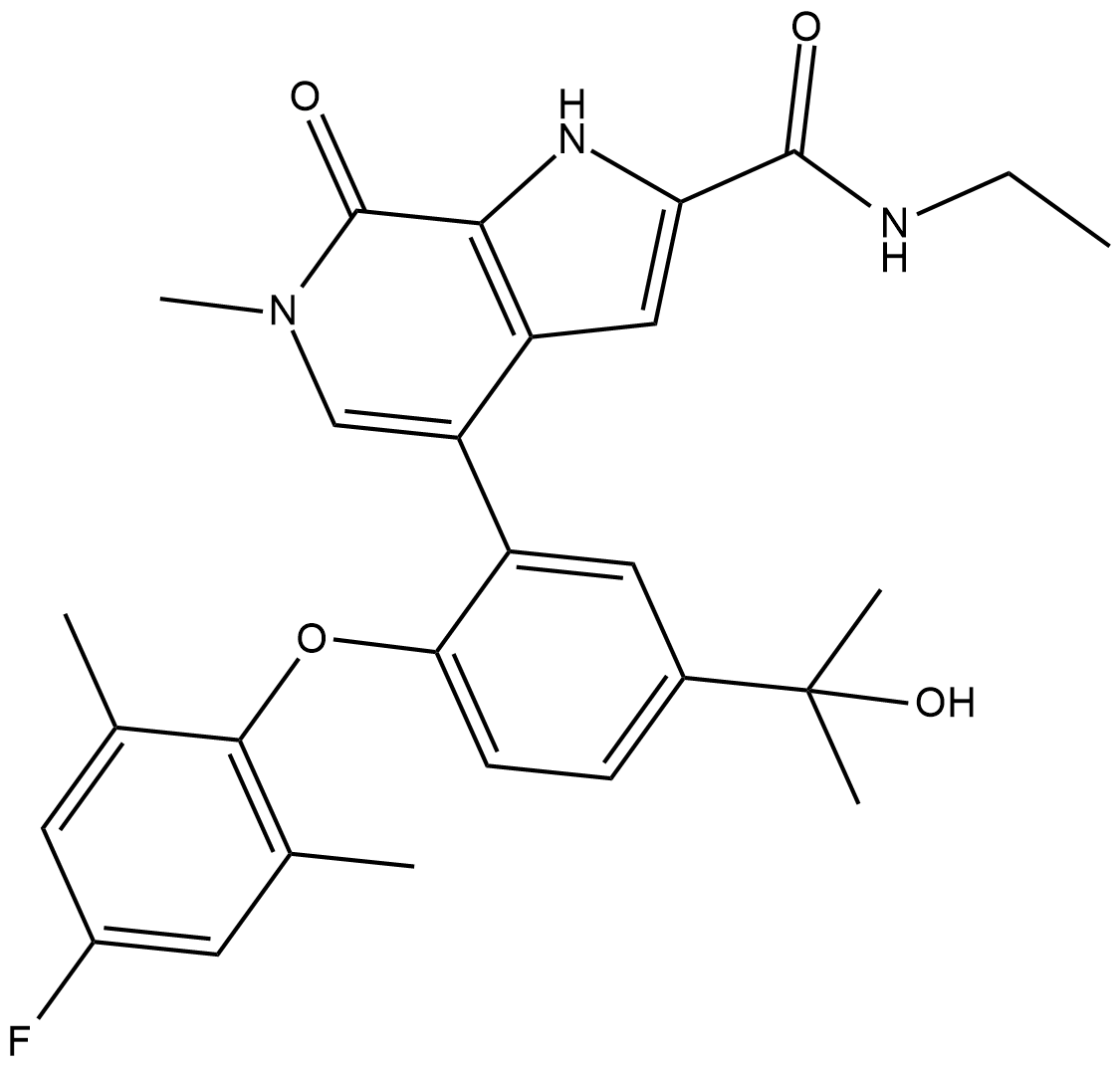
-
GC10154
ABC294640
ABC294640 (ABC294640) est un inhibiteur sélectif et compétitif de la sphingosine kinase 2 (SK2) avec un Ki de 9,8 μM.
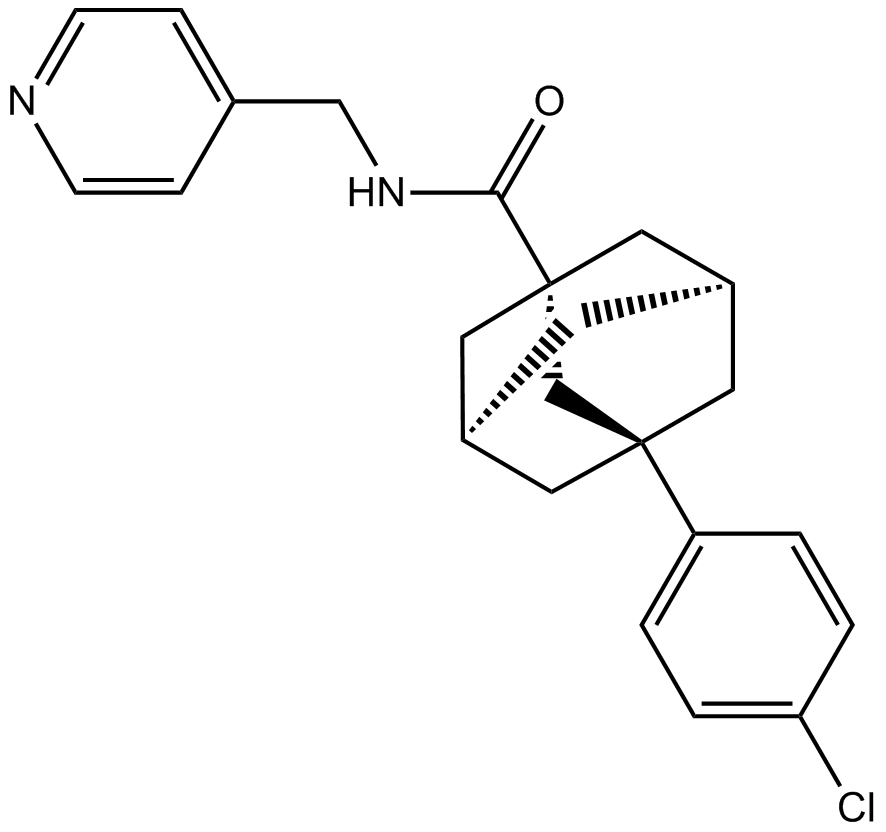
-
GC32023
Abrocitinib (PF-04965842)
L'abrocitinib (PF-04965842) (PF-04965842) est un inhibiteur de JAK1 puissant, actif par voie orale et sélectif, avec des IC50 de 29 et 803 nM pour JAK1 et JAK2, respectivement.
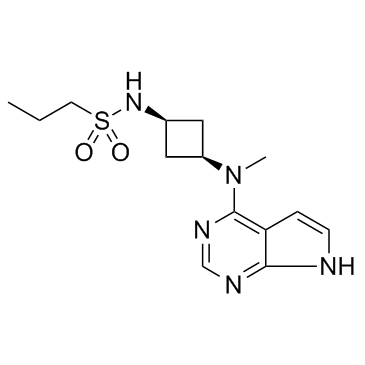
-
GC12422
ABT-888 (Veliparib)
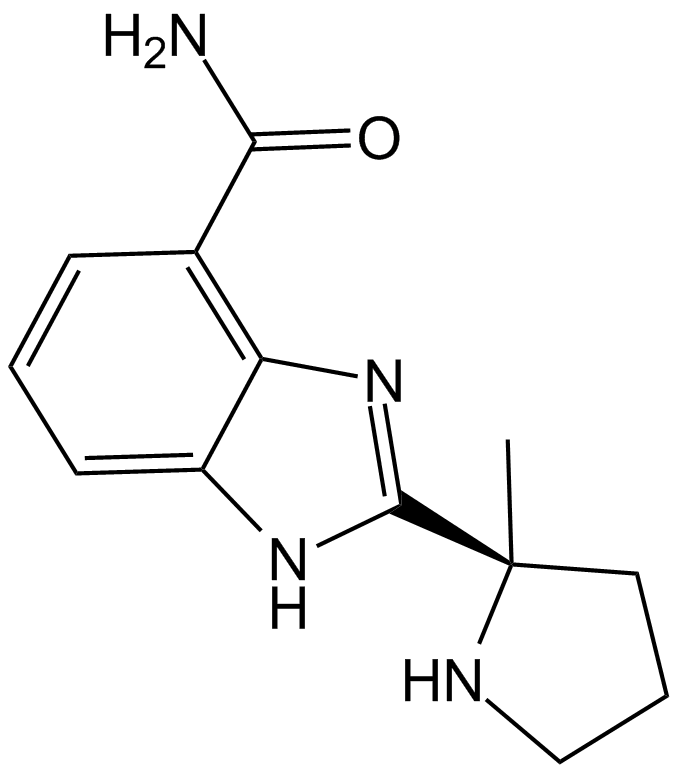
-
GA20481
Ac-Arg-Gly-Lys(Ac)-AMC
Ac-Arg-Gly-Lys(Ac)-AMC est un substrat pour HDAC.

-
GA20605
Ac-Lys-AMC
L'Ac-Lys-AMC (Hexanamide), également appelé MAL, est un substrat fluorescent pour les HDAC d'histone désacétylase.

-
GC48382
Ac-QPKK(Ac)-AMC
A fluorogenic substrate for SIRT1, SIRT2, and SIRT3

-
GC35227
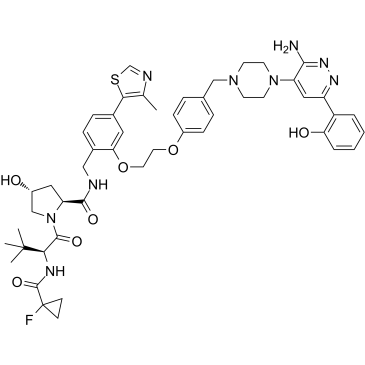
ACBI1
ACBI1 est un dégradeur puissant et coopératif de SMARCA2, SMARCA4 et PBRM1 avec des DC50 de 6, 11 et 32 nM, respectivement. ACBI1 est un dégradeur de PROTAC. ACBI1 montre une activité anti-proliférative. ACBI1 induit l'apoptose.
-
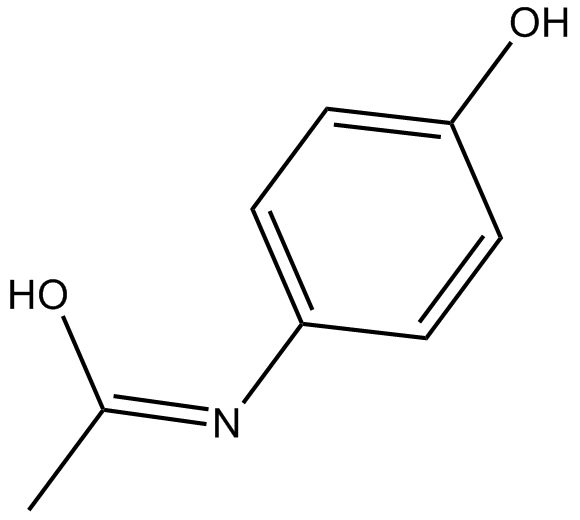
GC12917
Acetaminophen
Un inhibiteur de COX
-
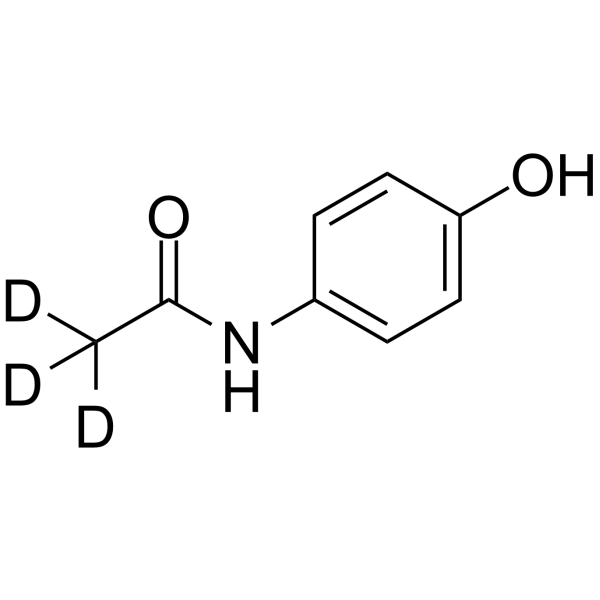
GC64137
Acetaminophen-d3
-
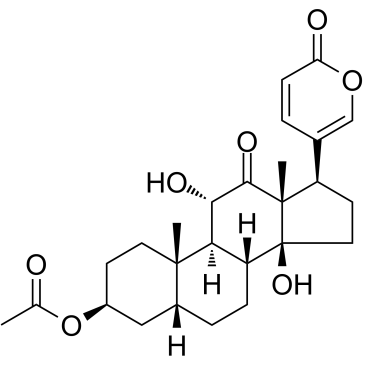
GC35230
Acetylarenobufagin
L'acétylarénobufagine est un modulateur du facteur 1 inductible par l'hypoxie stéroÏdienne (HIF-I).
-
GC32083
Acriflavine
L'acriflavine est un colorant fluorescent pour marquer l'ARN de haut poids moléculaire.

-
GC62354
Acriflavine hydrochloride
Le chlorhydrate d'acriflavine (chlorhydrate de chlorure d'acriflavinium) est un colorant d'acridine fluorescent qui peut être utilisé pour marquer l'acide nucléique.
-
GC34458
ACY-1083
ACY-1083 est un inhibiteur sélectif de HDAC6 pénétrant dans le cerveau avec une IC50 de 3 nM et est 260 fois plus sélectif pour HDAC6 que toutes les autres classes d'isoformes HDAC. ACY-1083 inverse efficacement la neuropathie périphérique induite par la chimiothérapie.
-
GC10417
ACY-241
ACY-241 (ACY241) est un inhibiteur HDAC6 puissant, actif par voie orale et hautement sélectif de deuxième génération avec une IC50 de 2,6 nM (IC50 de 35 nM, 45 nM, 46 nM et 137 nM pour HDAC1, HDAC2, HDAC3 et HDAC8, respectivement ). ACY-241 a des effets anticancéreux.
-
GC19020
ACY-738
ACY-738 est un inhibiteur HDAC6 puissant, sélectif et biodisponible par voie orale, avec une IC50 de 1,7 nM ; ACY-738 inhibe également HDAC1, HDAC2 et HDAC3, avec des IC50 de 94, 128 et 218 nM.

-
GC30782
ACY-775
ACY-775 est un inhibiteur puissant et sélectif de l'histone désacétylase 6 (HDAC6) avec une IC50 de 7,5nM.
-
GC30526
ACY-957
ACY-957 est un inhibiteur oralement actif et sélectif de HDAC1 et HDAC2, avec des IC50 de 7 nM, 18 nM et 1300 nM contre HDAC1/2/3, respectivement, et ne montre aucune inhibition sur HDAC4/5/6/7/8 /9.
-
GC12487
Adaptaquin
Adaptaquin est un inhibiteur du facteur prolyl hydroxylase 2 inductible par l'hypoxie (HIF-PHD2), avec une IC50 de 2 μM.