Ubiquitination/ Proteasome
- Autophagy(1089)
- DUB(20)
- E1 Activating(1)
- E2 conjugating(1)
- E2 Ligases(0)
- E3 Ligase(8)
- Proteasome(99)
- p97(12)
- Mitophagy(82)
- ULK(9)
Products for Ubiquitination/ Proteasome
- Cat.No. Nom du produit Informations
-
GC30761
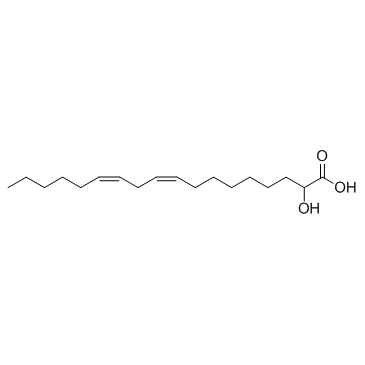
α-Hydroxylinoleic acid
L'acide α-hydroxylinoléique (α-acide hydroxylinoléique) induit l'autophagie médiée par le stress du réticulum endoplasmique (RE).
-
GC46008
(±)-Thalidomide-d4
N-Phthaloylglutamimide-d4
(±)-Thalidomide-d4 est un deutérium marqué Thalidomide.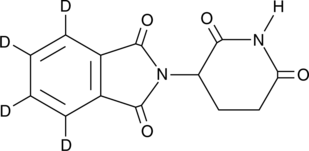
-
GC34960
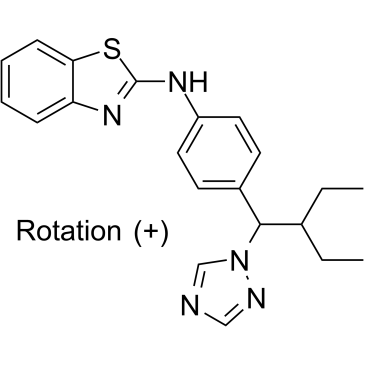
(+)-Talarozole
(+)-Talarozole est un puissant inhibiteur du métabolisme de l'acide rétinoÏque extrait du brevet WO 1997049704 A1.
-
GC10603
(-)-epicatechin gallate
ECG
Le gallate de (-)-épicatéchine (Epicatechin gallate) inhibe la cyclooxygénase-1 (COX-1) avec une IC50 de 7,5 μM.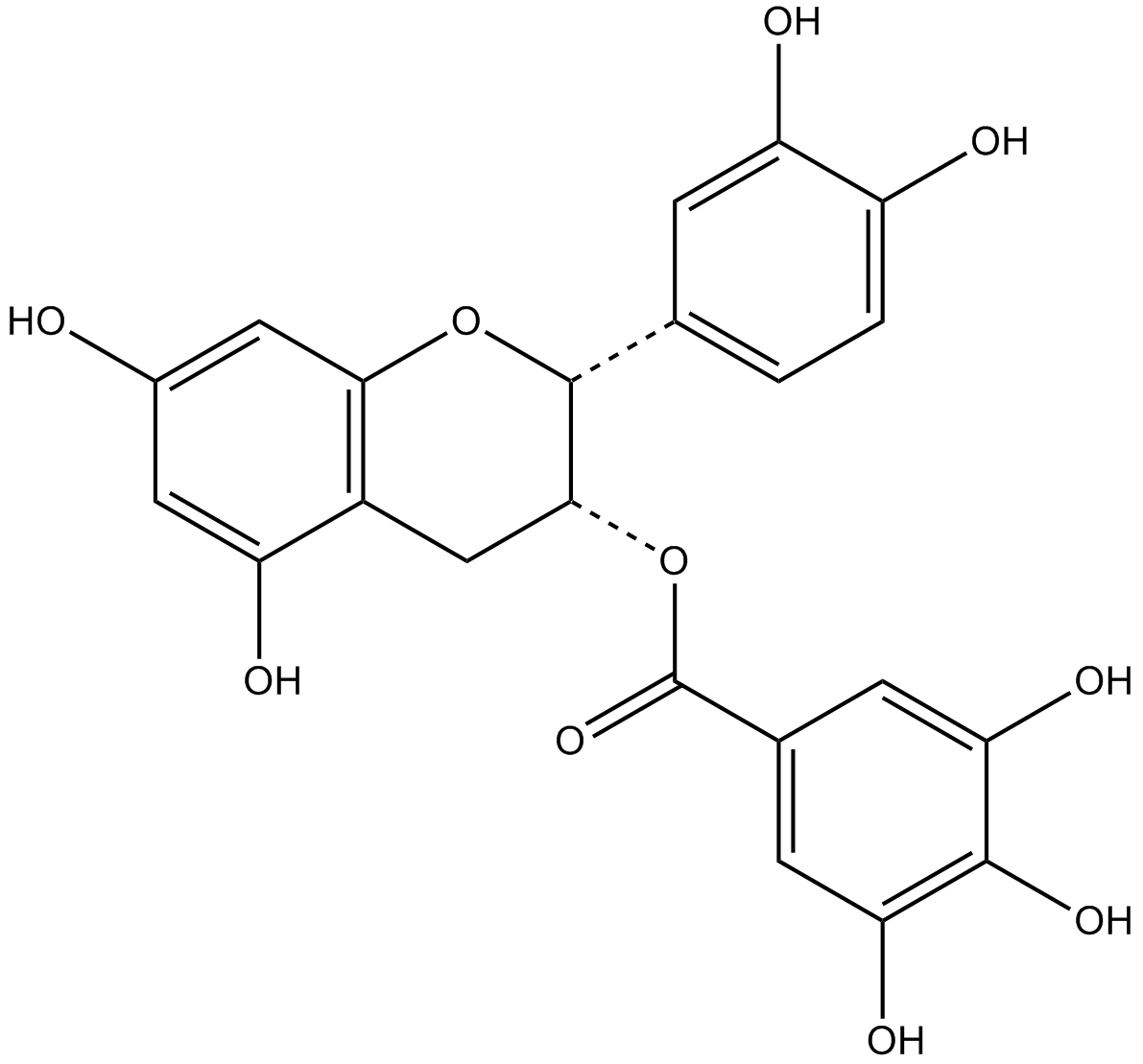
-
GC17242
(-)-epigallocatechin
(-)EGC, epi-Gallocatechin, NSC 674039
La (-)-épigallocatéchine (épigallocatéchine) est le flavonoÏde le plus abondant dans le thé vert, peut se lier aux polypeptides natifs dépliés et empêcher la conversion en fibrilles amyloÏdes.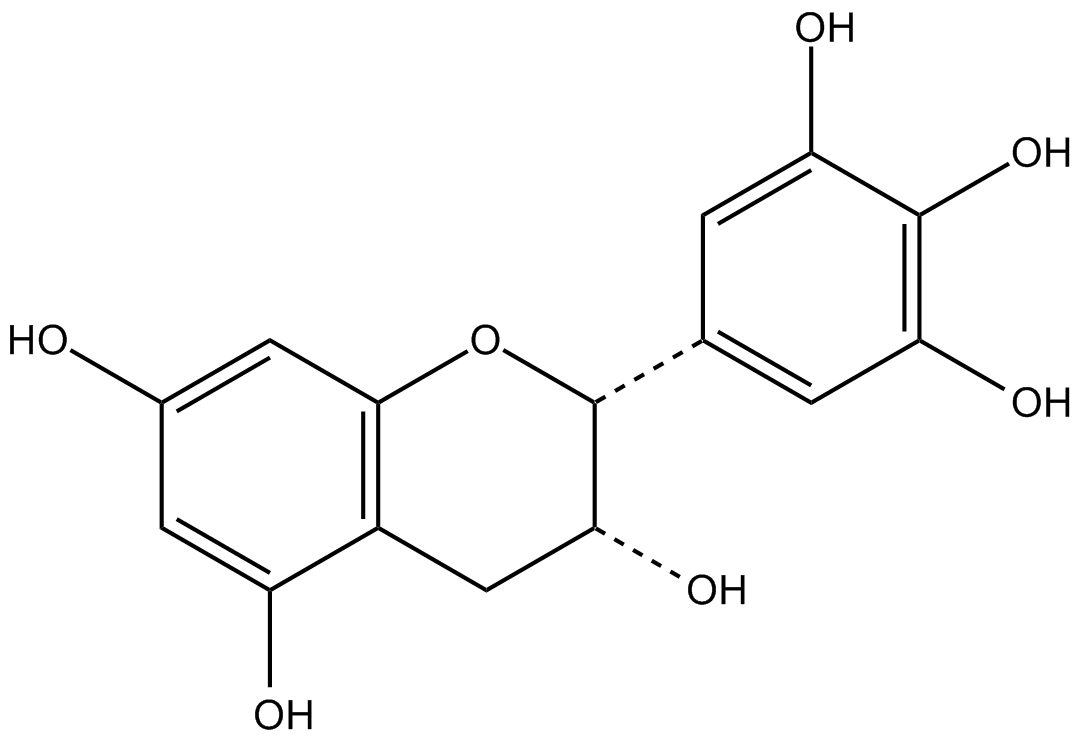
-
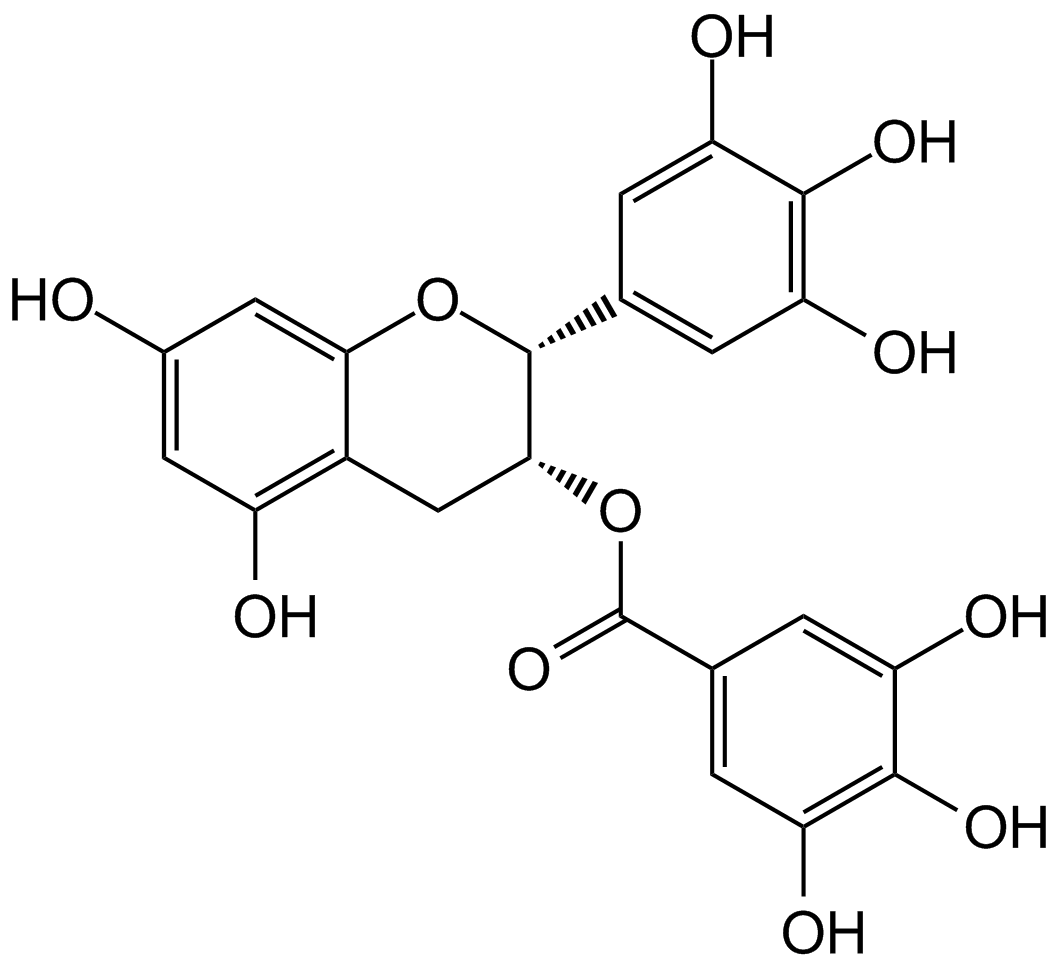
GC14049
(-)-Epigallocatechin gallate (EGCG)
EGCG
Un phénol avec des activités biologiques diverses.
-
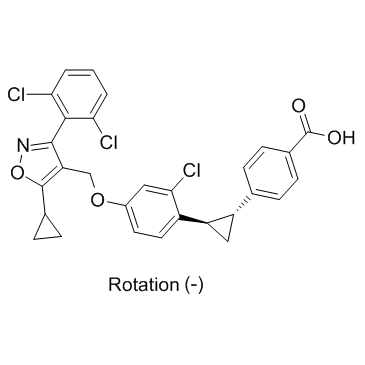
GC31386
(-)-PX20606 trans isomer ((-)-PX-102 trans isomer)
L'isomère trans (-)-PX20606 (isomère trans ((-)-PX-102) est un agoniste de FXR avec des EC50 de 18 et 29 nM pour FXR dans les tests FRET et M1H, respectivement.
-
GC65547
(1S,2R)-Alicapistat
(1S,2R)-ABT-957
(1S,2R)-Alicapistat ((1S,2R)-ABT-957) est un inhibiteur sélectif actif par voie orale des calpaÏnes humaines 1 et 2 pour l'application potentielle de la maladie d'Alzheimer (MA).
-
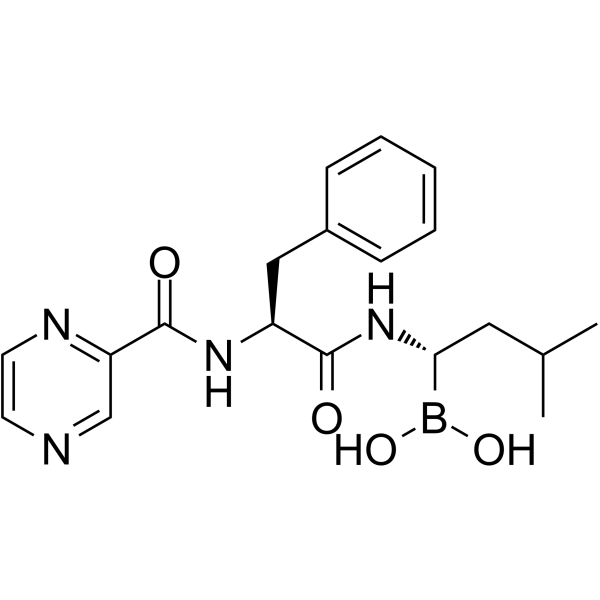
GC62193
(1S,2S)-Bortezomib
Le (1S,2S)-bortézomib est un énantiomère du bortézomib. Le bortézomib est un inhibiteur du protéasome perméable aux cellules, réversible et sélectif, et inhibe puissamment le protéasome 20S (Ki de 0,6 nM) en ciblant un résidu thréonine. Le bortézomib perturbe le cycle cellulaire, induit l'apoptose et inhibe le NF-κB. Le bortézomib est un agent anticancéreux et le premier inhibiteur thérapeutique du protéasome À être utilisé chez l'homme.
-
GC60395
(3S,5R)-Fluvastatin D6
(3S,5R)-XU 62-320 free acid D6
La (3S,5R)-fluvastatine D6 est la (3S,5R)-fluvastatine sodique marquée au deutérium.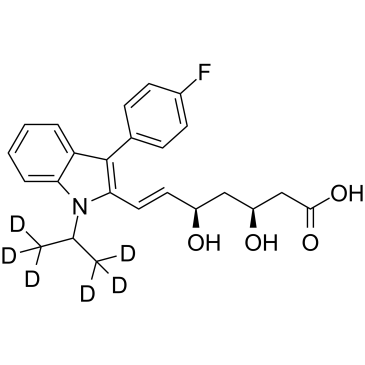
-
GC49189
(E/Z)-4-hydroxy Tamoxifen-d5
Afimoxifene-d5, 4-OHT-d5
An internal standard for the quantification of (E/Z)-4-hydroxy tamoxifen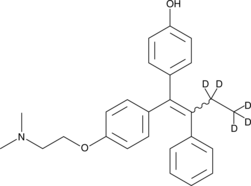
-
GC34096
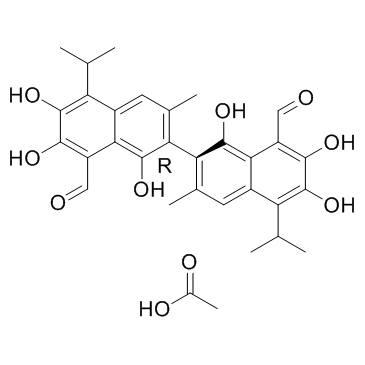
(R)-(-)-Gossypol acetic acid (AT-101 (acetic acid))
L'acide (R)-(-)-Gossypol acétique (AT-101 (acide acétique)) (AT-101 (acide acétique)) est l'isomère lévogyre d'un produit naturel Gossypol. AT-101 est déterminé À se lier aux protéines Bcl-2, Mcl-1 et Bcl-xL avec Kis de 260± 30 nM, 170± 10 nM et 480± 40 nM, respectivement.
-
GC31665
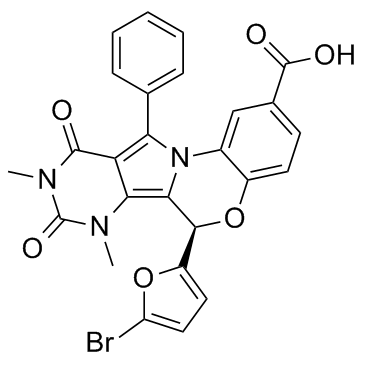
(R)-BPO-27
Le (R)-BPO-27, l'énantiomère R du BPO-27, est un inhibiteur CFTR puissant, actif par voie orale et compétitif pour l'ATP, avec une IC50 de 4nM.
-
GC41233
(R)-MG132
Un inhibiteur puissant de protéasome
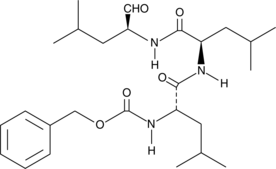
-
GC62528
(Rac)-Hesperetin
(Rac)-Hesperetin est le racémate de l'Hesperetin.
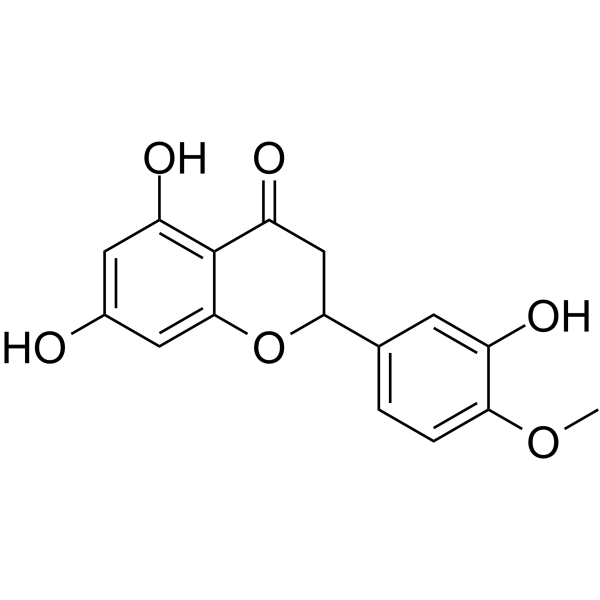
-
GC13136
(S)-Crizotinib
Le (S)-crizotinib est un inhibiteur puissant et sélectif de MTH1 (homologue mutT) avec une IC50 de 330 nM. Le (S)-crizotinib perturbe l'homéostasie du pool de nucléotides via l'inhibition de MTH1, induit une augmentation des cassures simple brin de l'ADN, active la réparation de l'ADN dans les cellules de carcinome du cÔlon humain et supprime efficacement la croissance tumorale dans les modèles animaux.
-
GC14820
(S)-Naproxen
CG 3117, (S)-Naproxen
Le (S)-naproxène est un inhibiteur de la COX-1 et de la COX-2 avec des CI50 de 8,72 et 5,15 μM, respectivement dans le test cellulaire.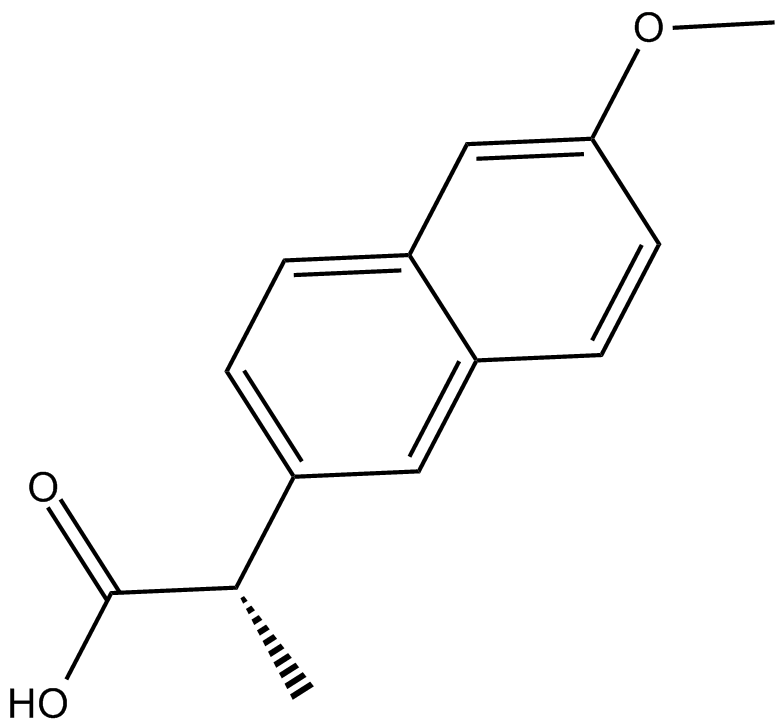
-
GC15015
(±)-Bay K 8644
SQ 28,873
(±)-Bay K 8644 ((±)-(±)-Bay K 8644) est un racémate constitué de deux isomères (R)-(+)-Bay-K-8644 et (S)-(-)-Bay -K-8644.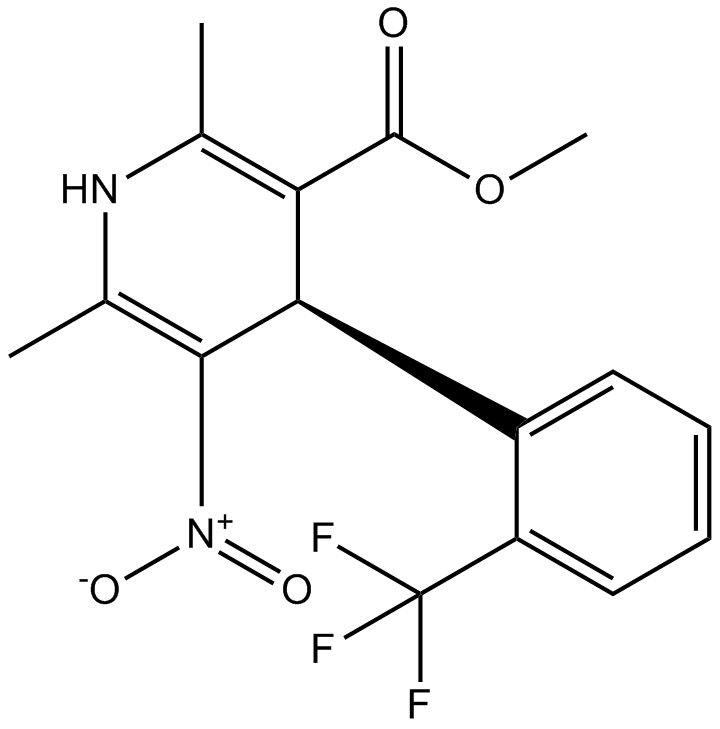
-
GC35068
1-Monomyristin
MG(14:0/0:0/0:0), 1-Monomyristin
La 1-monomyristine, extraite de Serenoa repens, inhibe l'hydrolyse du 2-oléoylglycérol (IC50=32 μM) et l'activité de l'amide hydrolase d'acide gras (FAAH) (IC50=18 μM).
-
GC17295
10058-F4
10058-F4 est un inhibiteur de c-Myc qui empêche la dimérisation de c-Myc-Max et la transactivation de l'expression du gène cible de c-Myc.

-
GC14918
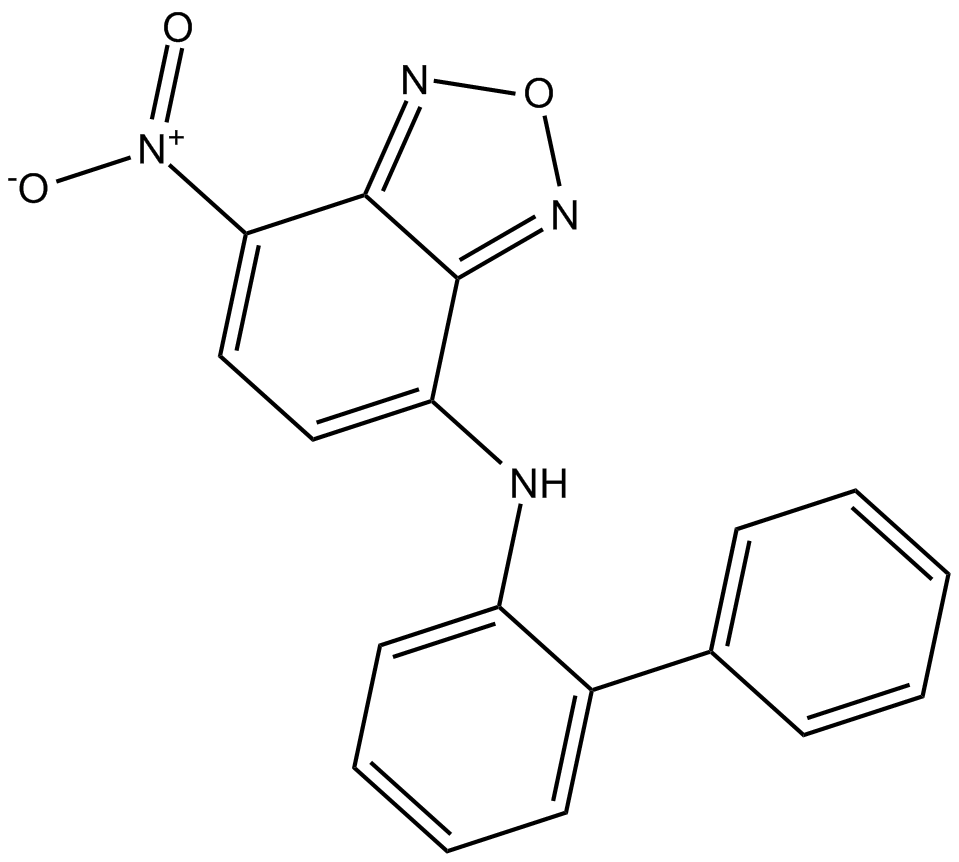
10074-G5
10074-G5 est un inhibiteur de la dimérisation c-Myc-Max avec une IC50 de 146 μM.
-
GC11720
17-AAG (KOS953)
BMS 722782, CP 127374, KOS 953, NSC 330507, Tanespimycin
17-AAG(Geldanamycin), a natural benzoquinone ansamycin antibiotic, is the first established inhibitor of Hsp90.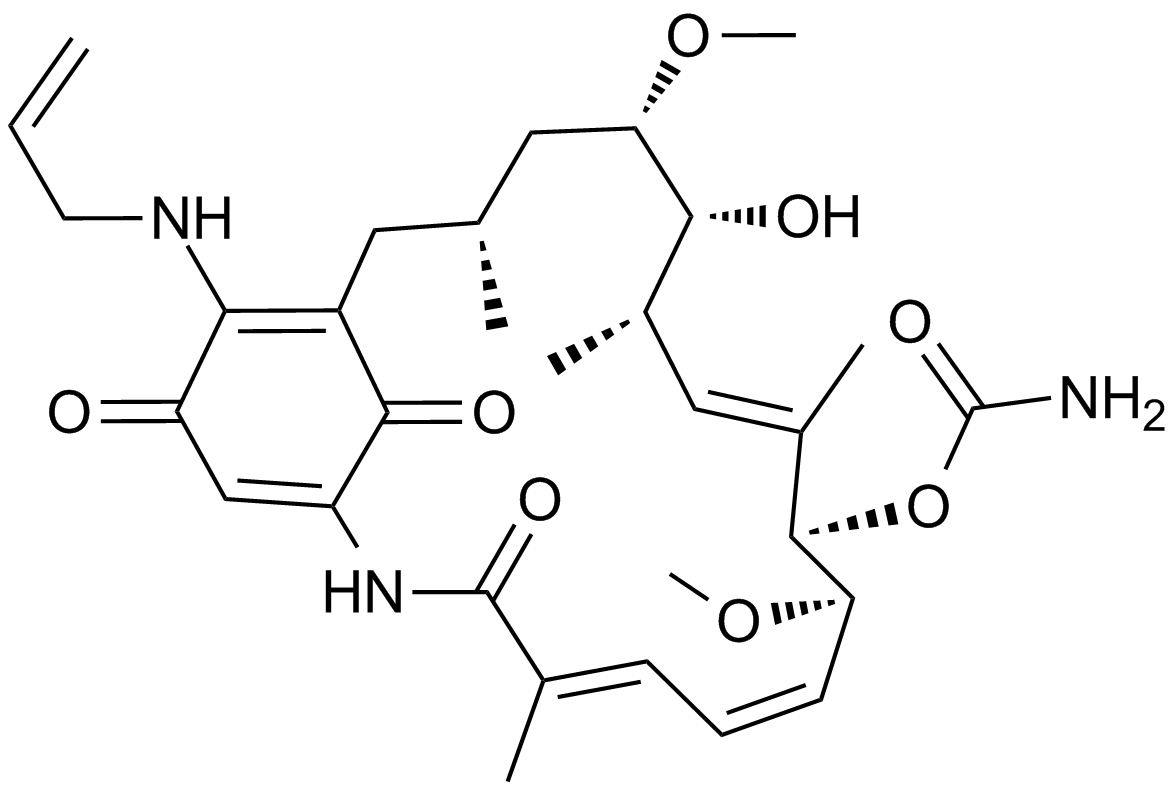
-
GC40947
2,3-Dimethoxy-5-methyl-p-benzoquinone
Coenzyme Q0, CoQ0
La 2,3-diméthoxy-5-méthyl-p-benzoquinone (CoQ0) est un puissant composé d'ubiquinone actif oral qui peut être dérivé d'Antrodia cinnamomea.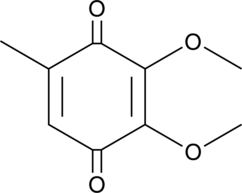
-
GC46057
2,5-Dihydroxycinnamic Acid phenethyl ester
An inhibitor of 5-LO
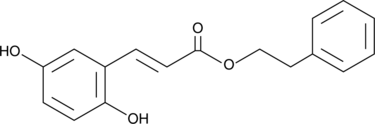
-
GC15084
2-Methoxyestradiol (2-MeOE2)
2Hydroxyestradiol 2methyl ether, 2ME2, NSC 659853, Panzem
2-Methoxyestradiol (2-MeOE2/2-Me) is an HIF-1α inhibitor.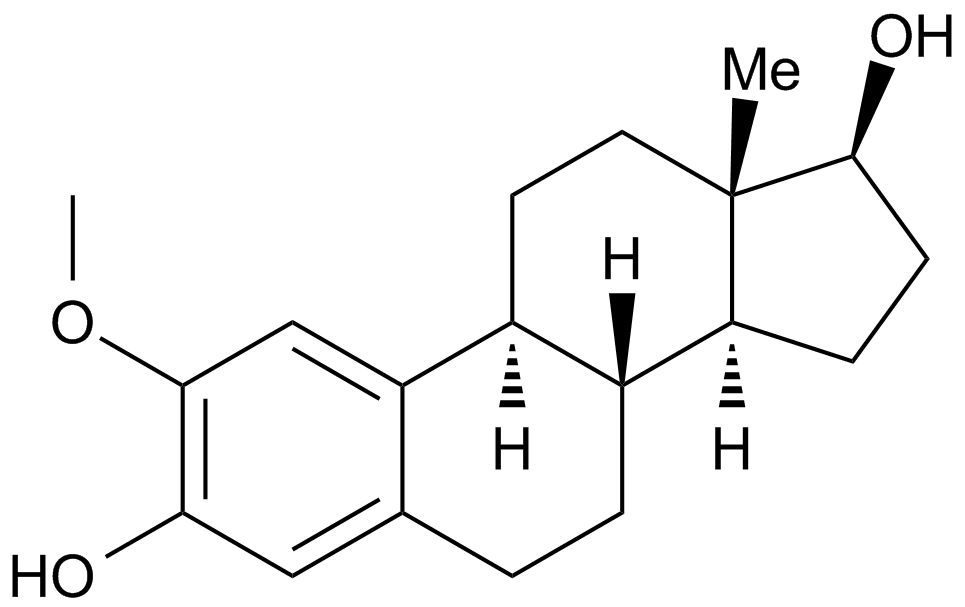
-
GC30011
20-Deoxyingenol
Le 20-désoxyingénol, un diterpène, est isolé des racines d'Euphorbia kansui.
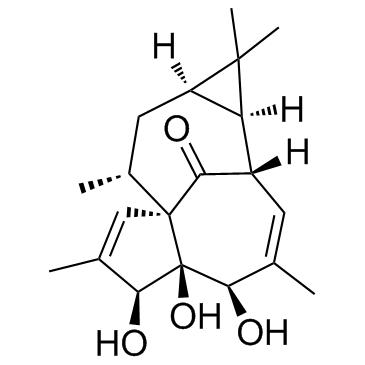
-
GC68503
20S Proteasome activator 1
Le activateur de protéasome 1 à 20S est un activateur efficace de la protéase à 20S, avec des IC50 respectifs de 0,3 μM, 0,7 μM et 1,8 μM pour les sites similaires à ceux de la trypsine pancréatique, chymotrypsine pancréatique et cathepsine B. Le activateur de protéasome 1 à 20S peut être traduit dans le système cellulaire pour empêcher l'accumulation du variant pathogène A53T d'α-synucléine. Le activateur de protéasome 1 à 20S peut être utilisé dans la recherche sur les maladies neurodégénératives.
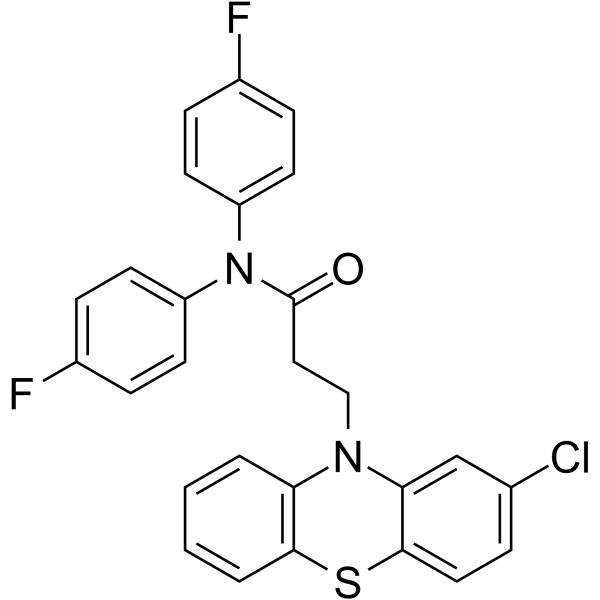
-
GC64472
20S Proteasome-IN-1
20S Proteasome-IN-1 est un inhibiteur de protéasome 26S extrait du brevet WO2006128196A2 composé 2.
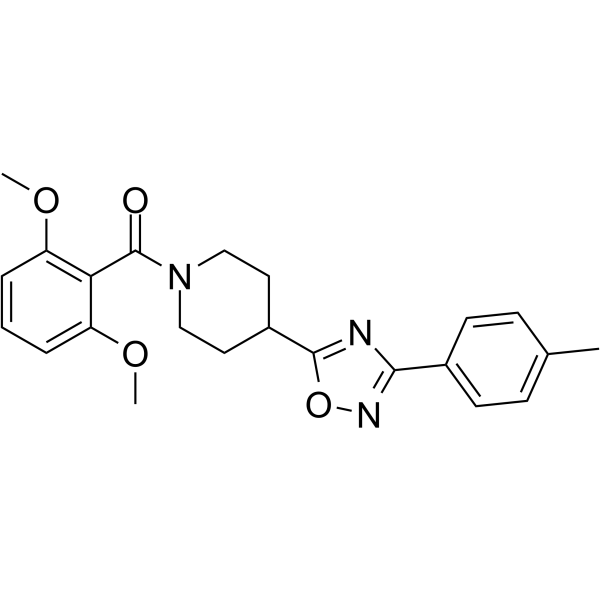
-
GC35112
3'-Hydroxypterostilbene
Le 3'-hydroxyptérostilbène est un analogue du ptérostilbène. Le 3'-hydroxyptérostilbène inhibe la croissance des cellules COLO 205, HCT-116 et HT-29 avec des CI50 de 9,0, 40,2 et 70,9 μM, respectivement. Le 3'-hydroxyptérostilbène régule À la baisse de manière significative les voies de signalisation PI3K/Akt et MAPK et inhibe efficacement la croissance des cellules cancéreuses du cÔlon humain en induisant l'apoptose et l'autophagie. Le 3'-hydroxyptérostilbène peut être utilisé pour la recherche sur le cancer.
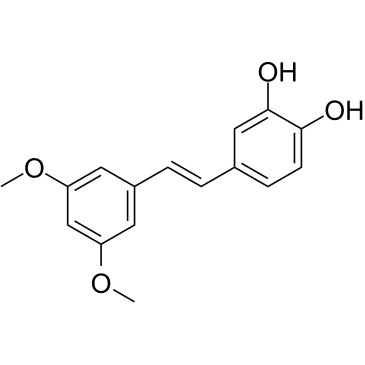
-
GC12791
3,3'-Diindolylmethane
DIM
A phytochemical with antiradiation and chemopreventative effects
-
GC10710
3-Methyladenine
3-MA
La 3-méthyladénine est un inhibiteur classique de l'autophagie.
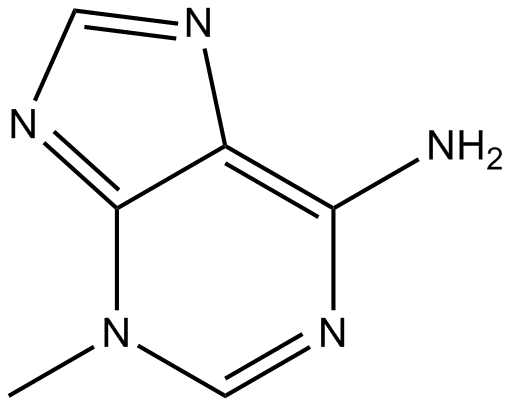
-
GC32767
3BDO
3-Benzyl-5-((2-nitrophenoxy)methyl)dihydrofuran-2(3H)-one
3BDO est un nouvel activateur mTOR qui peut également inhiber l'autophagie.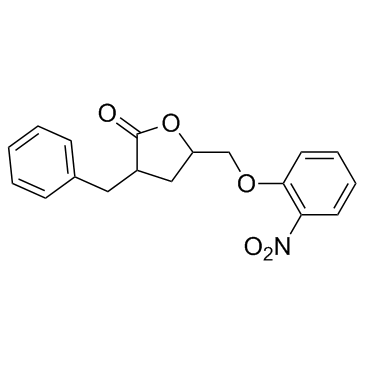
-
GC39658
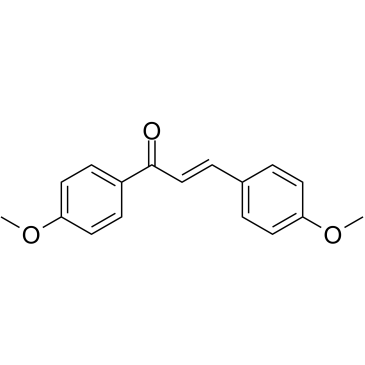
4,4'-Dimethoxychalcone
La 4,4'-diméthoxychalcone agit comme un inducteur naturel de l'autophagie aux propriétés anti-Âge.
-
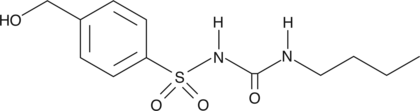
GC42414
4-hydroxy Tolbutamide
Le 4-hydroxy tolbutamide (hydroxytolbutamide) est un métabolite du tolbutamide.
-
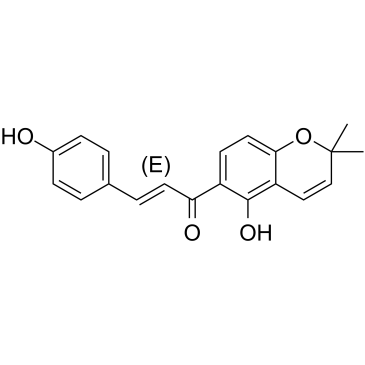
GC35132
4-Hydroxylonchocarpin
La 4-hydroxylonchocarpine est un composé de chalcone issu d'un extrait de Psoralea corylifolia.
-
GC70208
4-Hydroxytolbutamide-d9
4-droxytolbutamide-d9 is the deuterium labeled 4-droxytolbutamide.

-
GC13104
4E1RCat
eIF4E/eIF4G Interaction Inhibitor II
4E1RCat est un inhibiteur de la traduction cap-dépendante et inhibe l'interaction eIF4E:eIF4GI, avec une IC50 an de ~4 μM.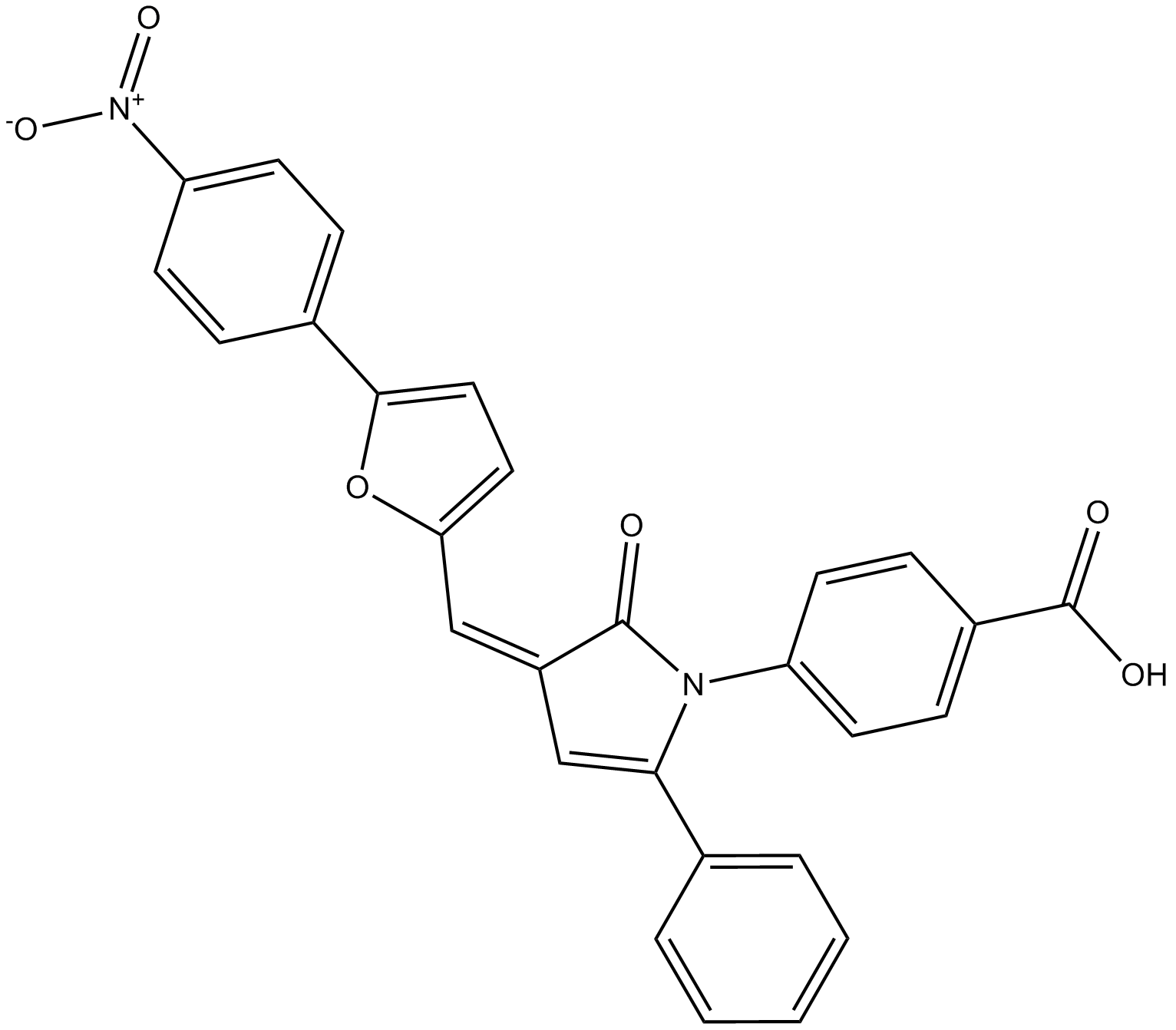
-
GC30785
4E2RCat
4E2RCat est un inhibiteur de l'interaction eIF4E-eIF4G avec une IC50 de 13,5 μM.
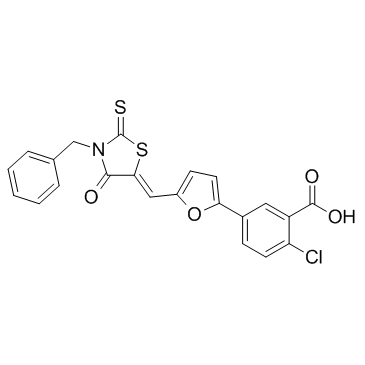
-
GC25019
5-amino-2,4-dimethylpyridine (5A-DMP)
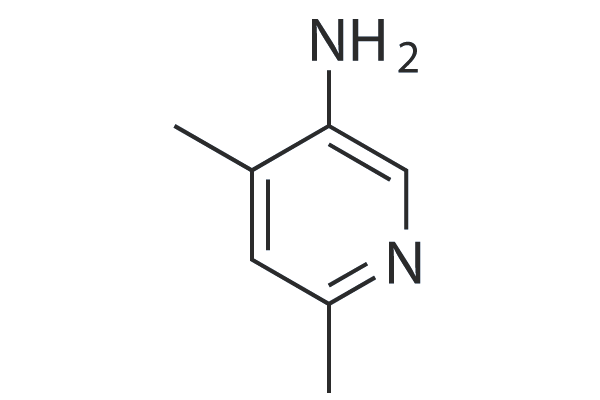
-
GC65668
5-Amino-8-hydroxyquinoline
5A8HQ
La 5-amino-8-hydroxyquinoléine (5A8HQ), un candidat anticancéreux potentiel, a une activité inhibitrice prometteuse du protéasome.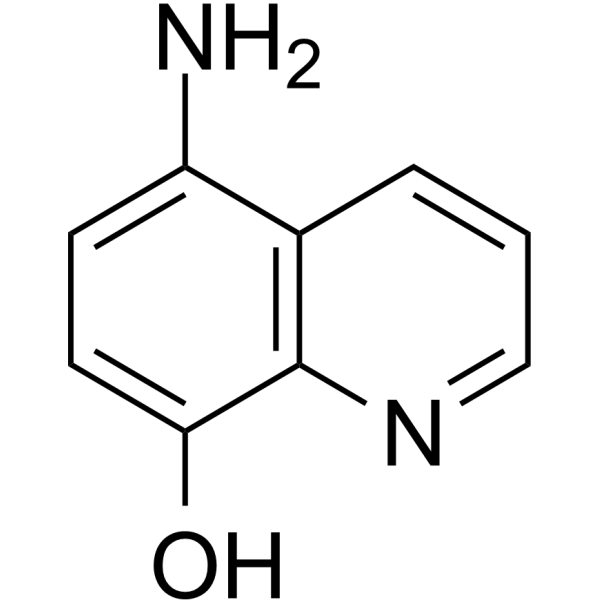
-
GC45356
5-Aminolevulinic Acid (hydrochloride)
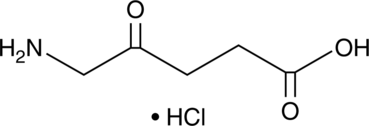
-
GC10946
5-Azacytidine
Antibiotic U 18496, 5AzaC, Ladakamycin, Mylosar, NSC 102816, NSC 103627, U 18496, WR 183027
5-Azacytidine (also known as 5-AzaC), a compound belonging to a class of cytosine analogues, is a DNA methyl transferase (DNMT) inhibitor that exerts potent cytotoxicity against multiple myeloma (MM) cells, including MM.1S, MM.1R, RPMI-8266, RPMI-LR5, RPMI-Dox40 and Patient-derived MM, with the half maximal inhibition concentration IC50 values of 1.5 μmol/L, 0.7 μmol/L, 1.1 μmol/L, 2.5 μmol/L, 3.2 μmol/L and 1.5 μmol/L respectively.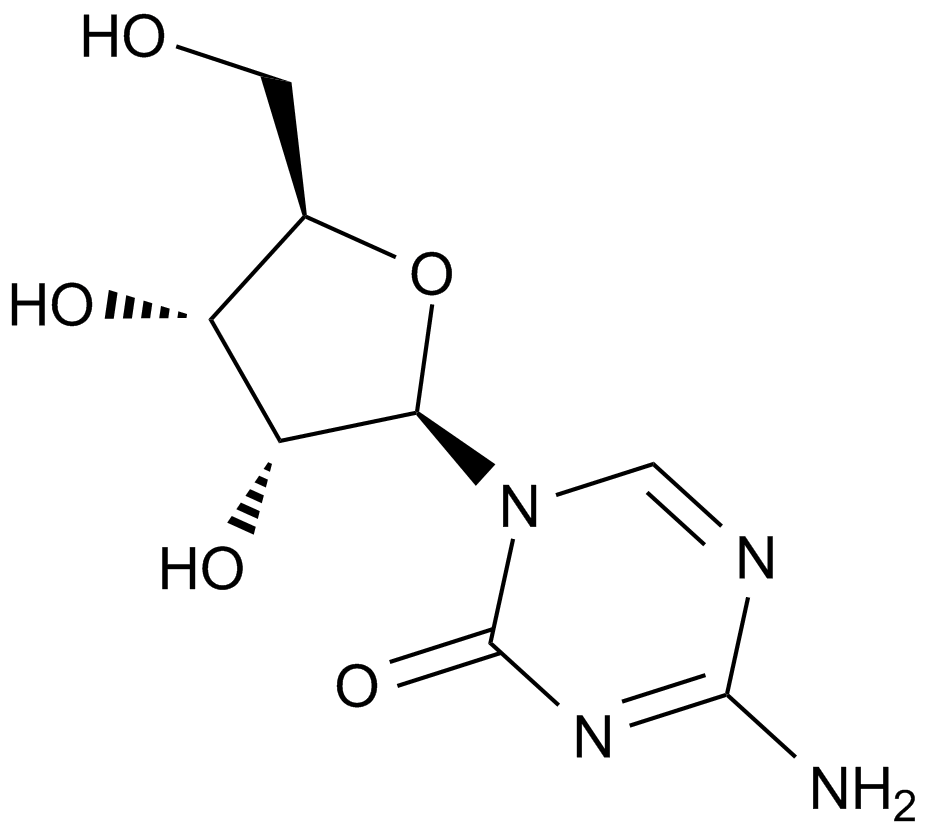
-
GN10241
5-Methoxypsoralen
5-Methoxypsoralen, 5-MOP, Heraclin, NSC 95437
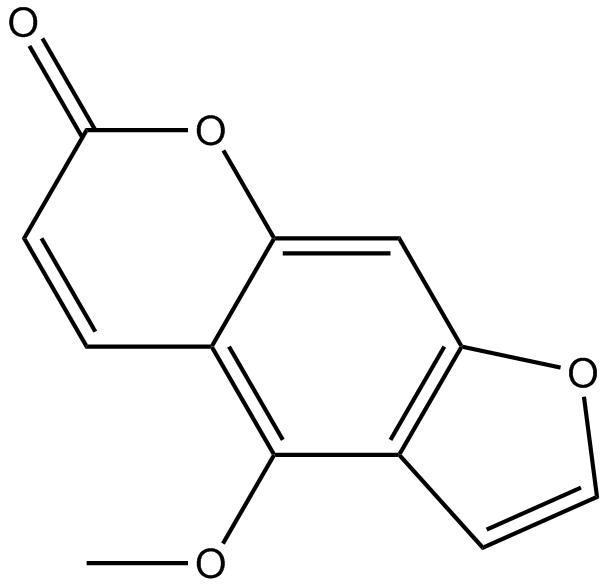
-
GC16267
6-Hydroxydopamine hydrobromide
6-hydroxy Dopamine, Oxidopamine, 2,4,5-Trihydroxyphenethylamine
6-Hydroxydopamine hydrobromide (6-OHDA) est un analogue structurel des catécholamines, de la dopamine et de la noradrénaline, et exerce ses effets toxiques sur les neurones catécholaminergiques.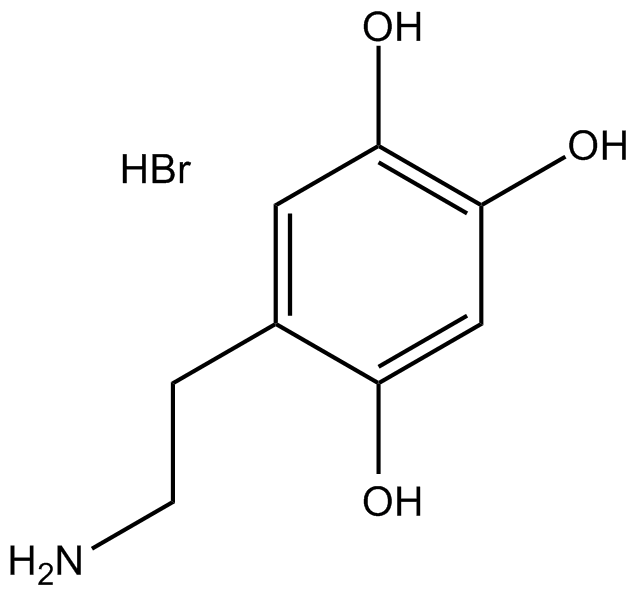
-
GN10228
6-Shogaol
Un composant bioactif du gingembre
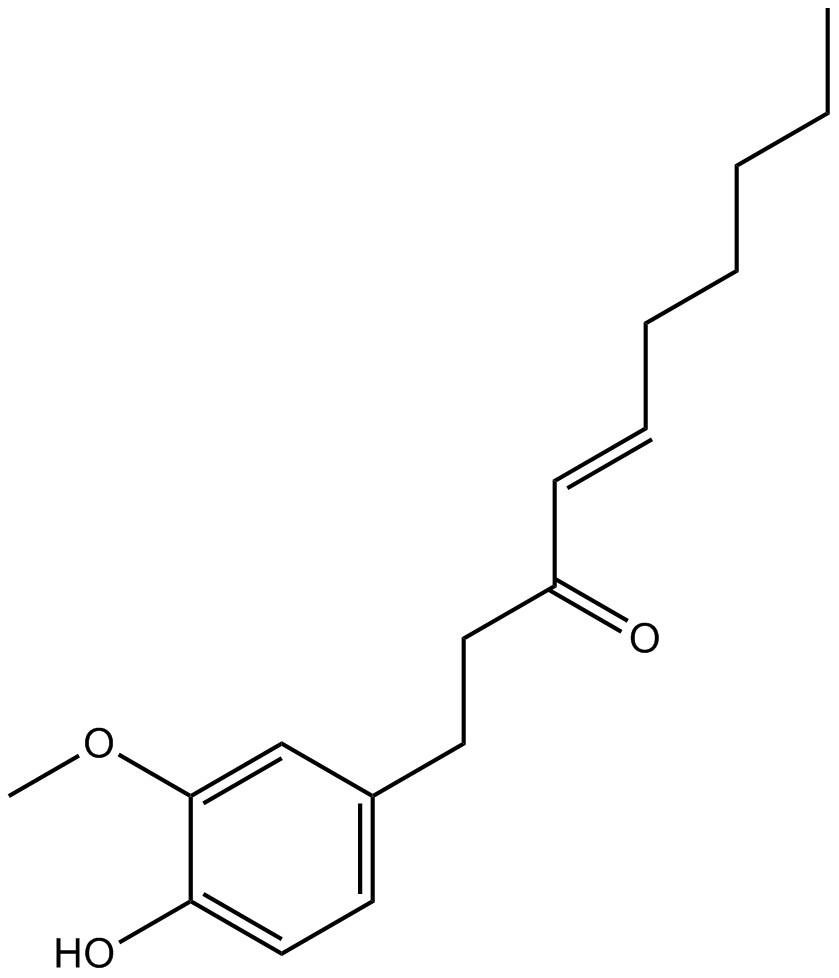
-
GC12739
A 484954
A 484954 est un inhibiteur hautement sélectif du facteur d'élongation eucaryote 2 (eEF2), avec une IC50 de 280 nM.
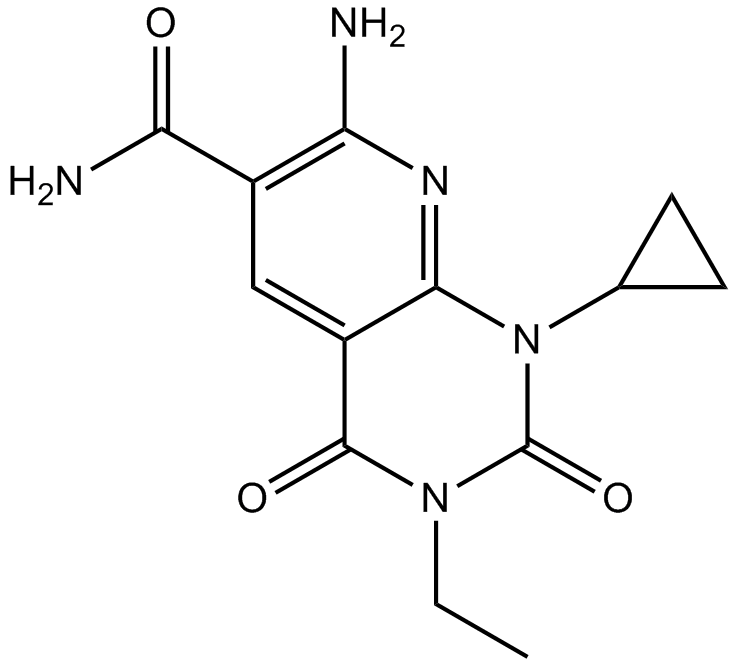
-
GC17710
A-317491
A 317491;A317491
A P2X3 and P2X2/3 receptor antagonist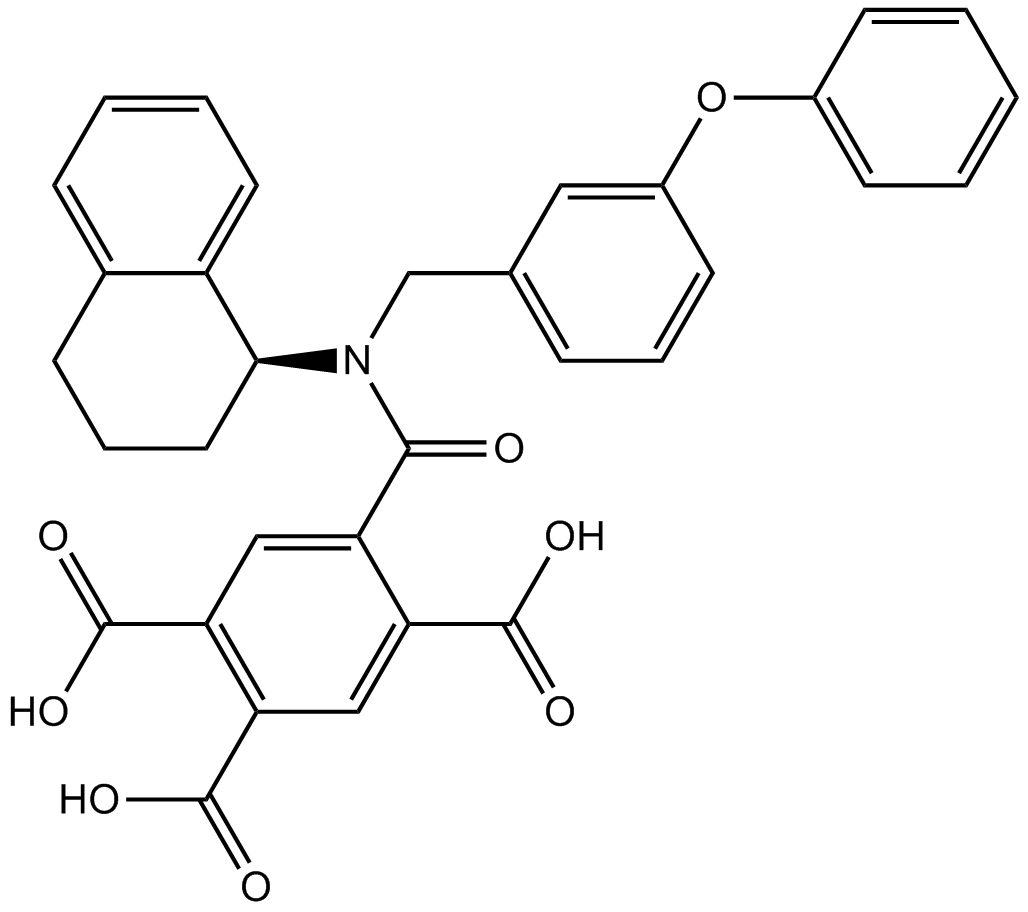
-
GC35211
A-317491 sodium salt hydrate
L'hydrate de sel de sodium A-317491 est un antagoniste puissant, sélectif et non nucléotidique des récepteurs P2X3 et P2X2/3, avec Kis de 22, 22, 9 et 92 nM pour hP2X3, rP2X3, hP2X2/3 et rP2X2/3, respectivement.
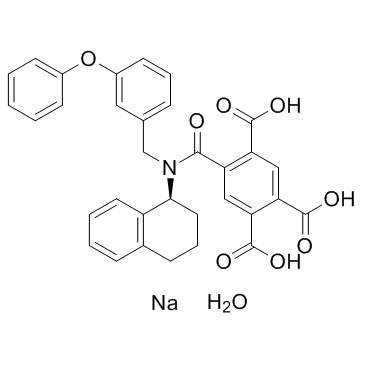
-
GC15014
A-867744
A positive allosteric modulator of α7 nAChRs
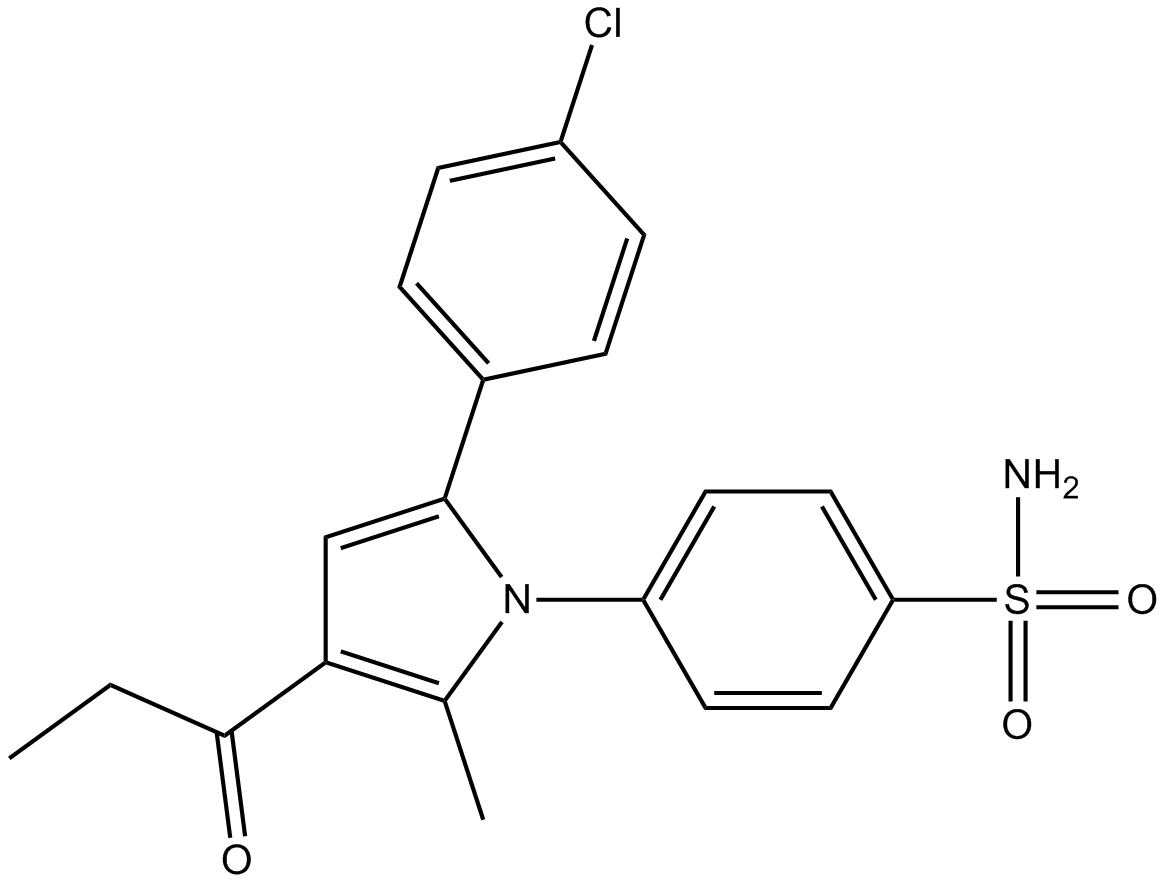
-
GC11200
A23187
Calcimycin
A23187 (A-23187) est un antibiotique et un ionophore de cations divalents unique (comme le calcium et le magnésium).
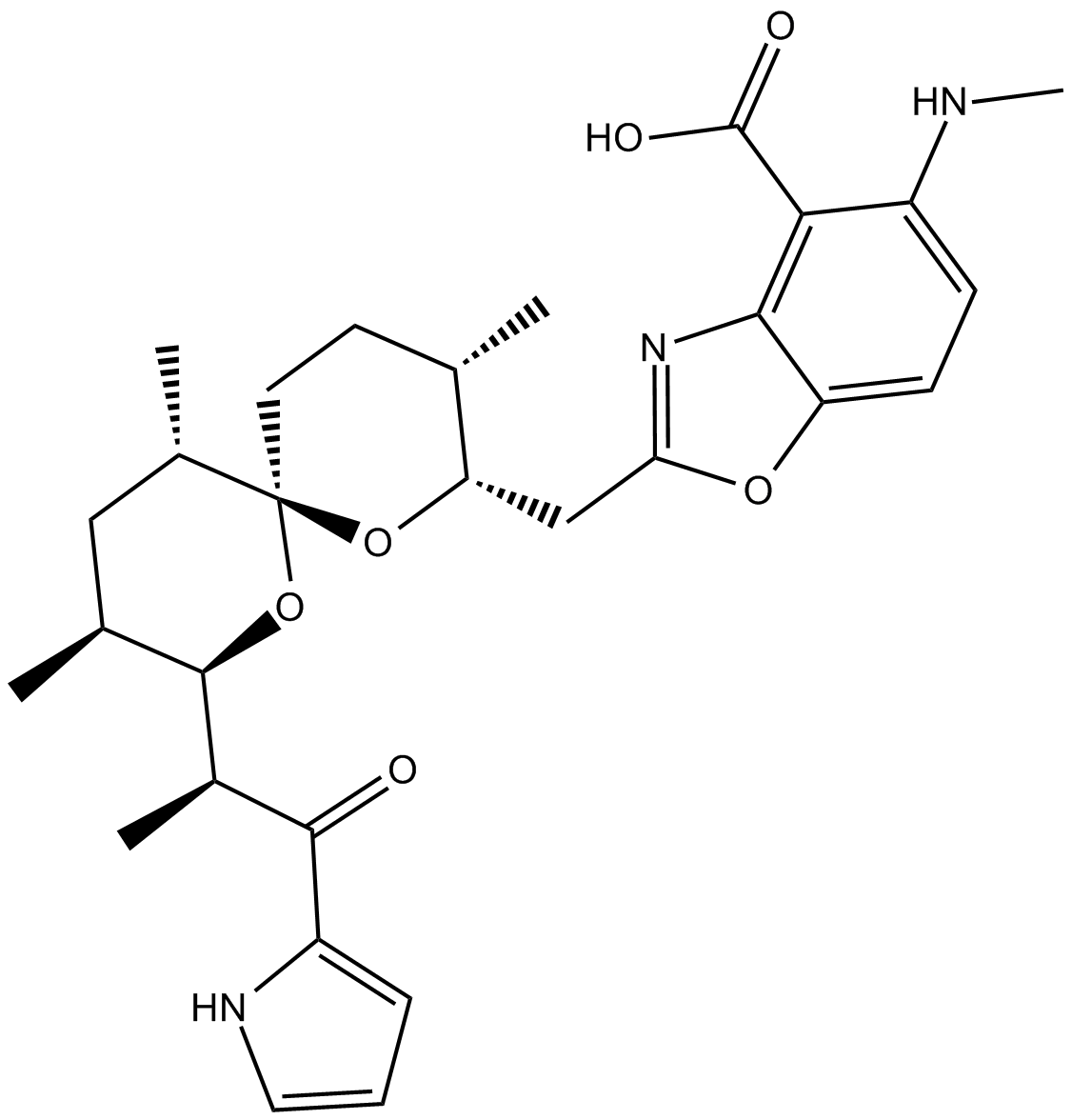
-
GC14069
ABT-199
GDC 0199, Venetoclax
Un inhibiteur de Bcl-2
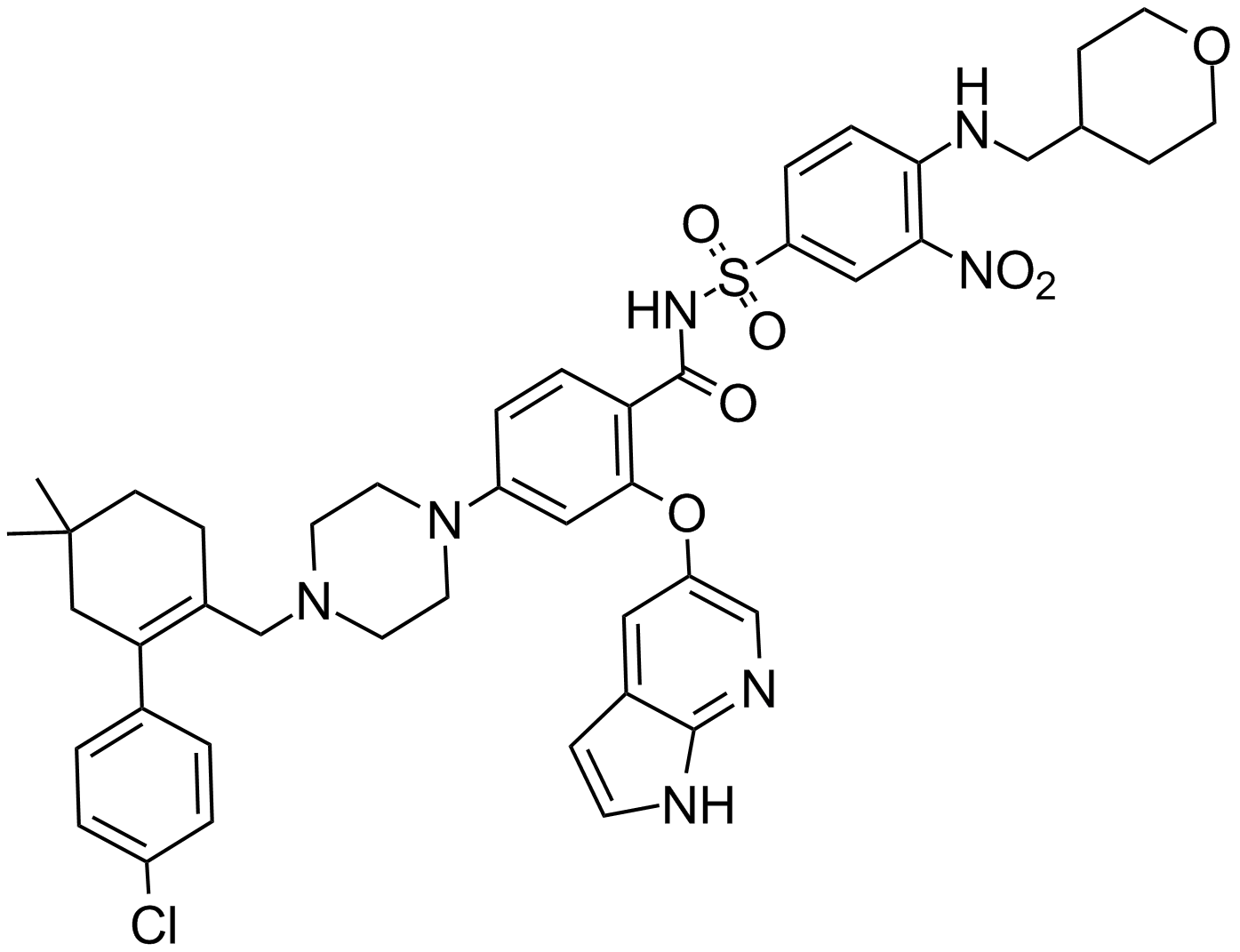
-
GC17234
ABT-737
ABT 737, ABT737
An inhibitor of anti-apoptotic Bcl-2 proteins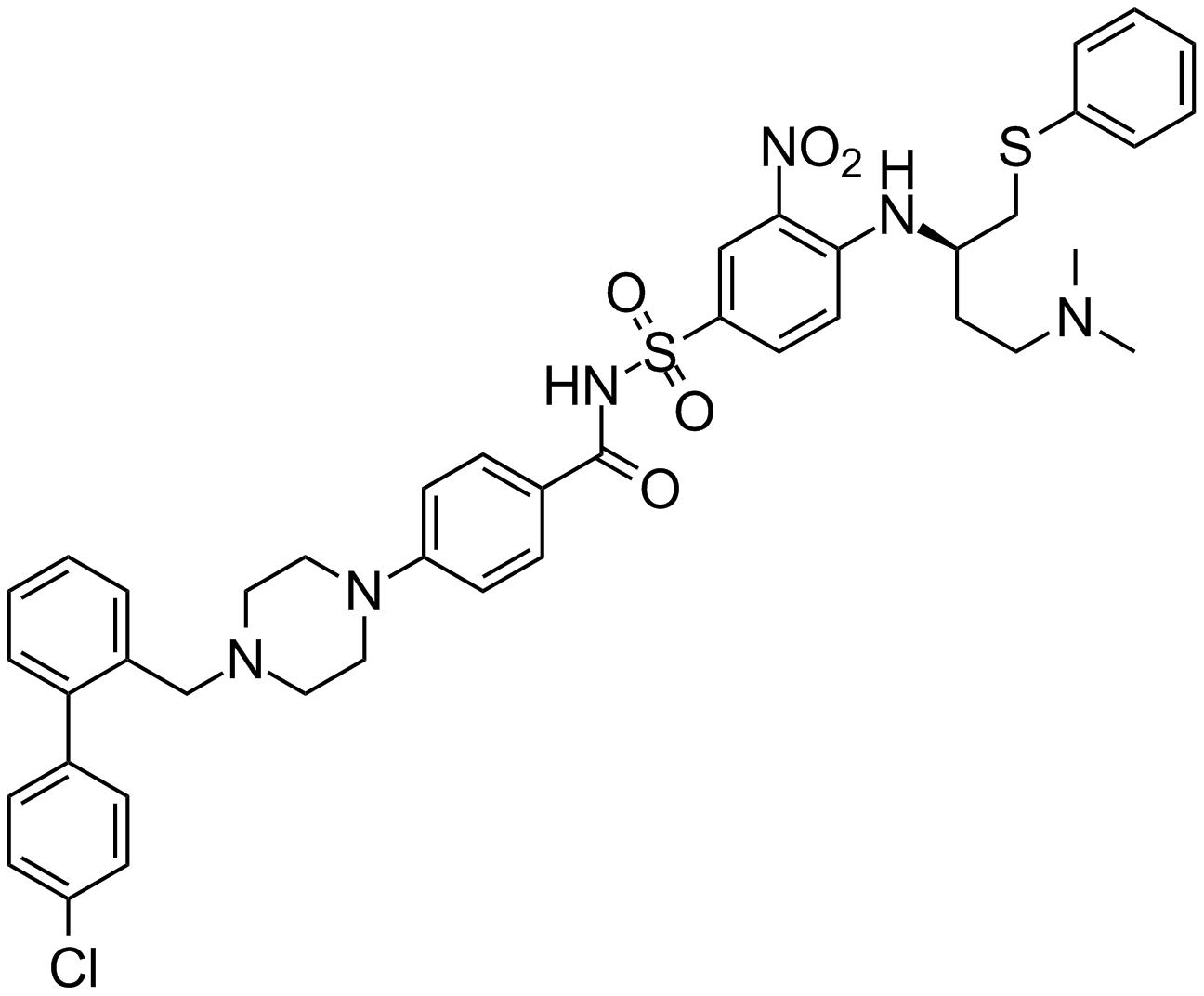
-
GC15875
ABT-751 (E7010)
ABT-751 (E7010)(E 7010) est un nouvel agent biodisponible de liaison À la tubuline et de sulfamide antimitotique avec une IC50 d'environ 1,5 et 3,4 μM dans les lignées cellulaires de neuroblastome et non neuroblastome, respectivement.
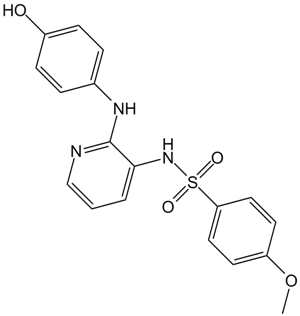
-
GC12422
ABT-888 (Veliparib)
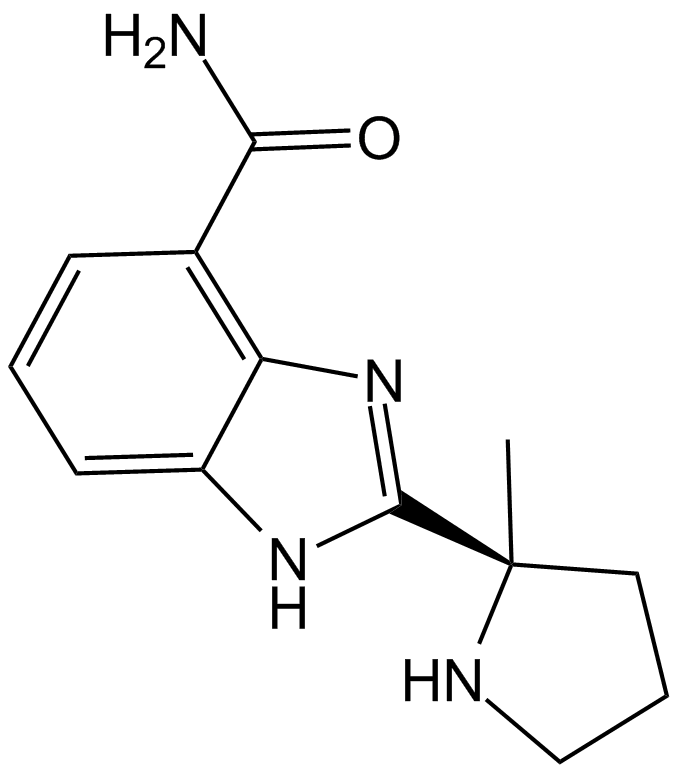
-
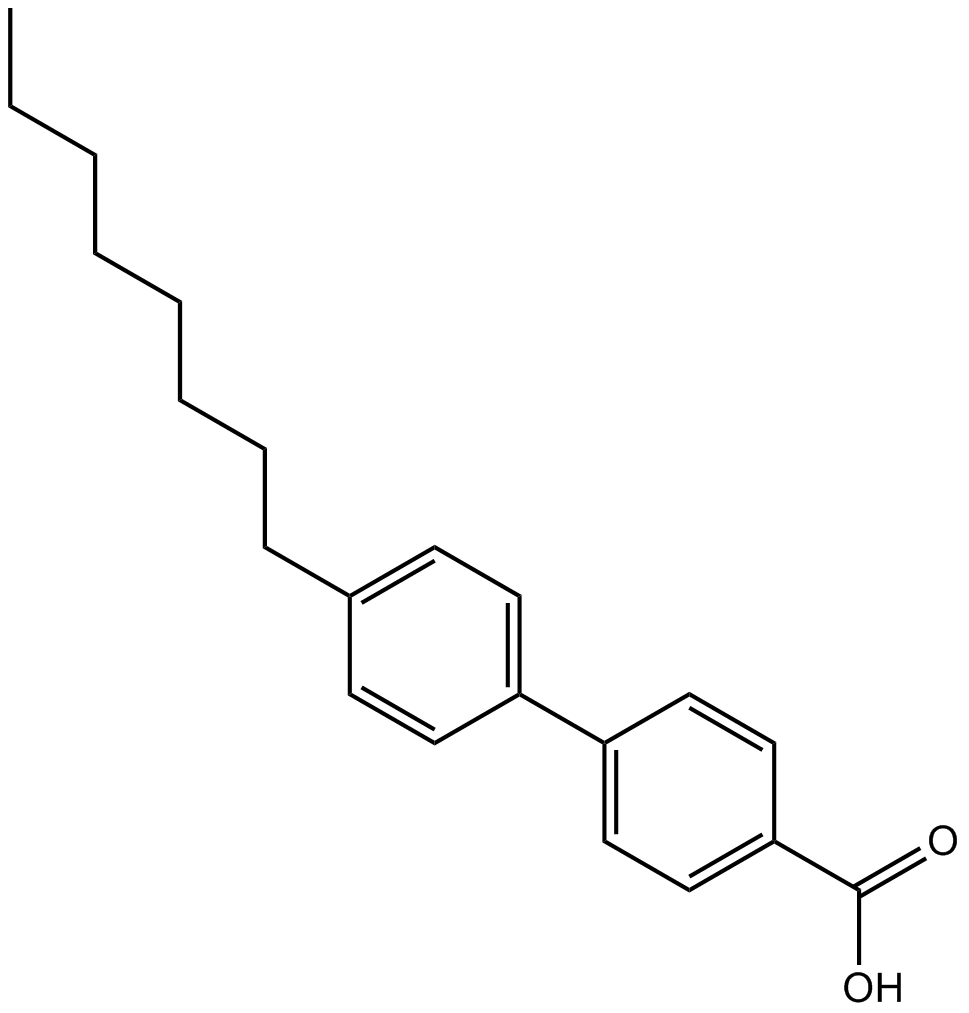
GC10871
AC 55649
L'AC 55649 est un puissant agoniste hautement sélectif des isoformes du récepteur RARβ2 humain, avec une pEC50 de 6,9.
-
GC42685
Ac-ANW-AMC
Ac-Ala-Asn-Trp-AMC
Ac-ANW-AMC est un substrat fluorogène pour l'immunoprotéasome.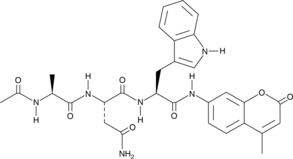
-
GC42710
Ac-PAL-AMC
Acetyl-Pro-Ala-Leu-7-amino-4-Methylcoumarin, Ac-Pro-Ala-Leu-AMC
Ac-PAL-AMC est un substrat fluorogène spécifique de l'activité LMP2/β1i du protéasome 20S.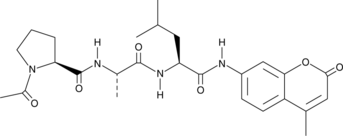
-
GC42720
Ac-WLA-AMC
Acetyl-Trp-Leu-Ala-7-amino-4-Methylcoumarin, Ac-Trp-Leu-Ala-AMC
Ac-WLA-AMC est un substrat fluorogène spécifique du protéasome constitutif 20S β5.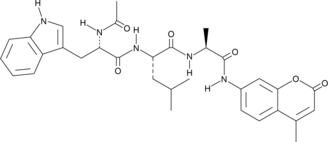
-
GC35228
Aceglutamide
Aceglutamide, Acetylglutamine, N-acetyl Gln, NSC 186896
L'acéglutamide (α-N-acétyl-L-glutamine) est un psychostimulant et nootropique, utilisé pour améliorer la mémoire et la concentration.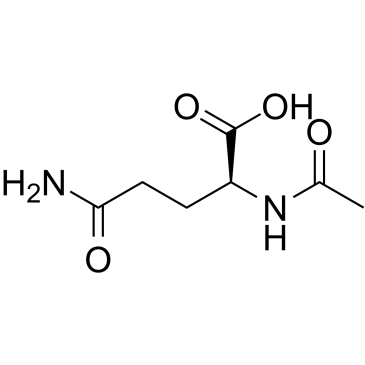
-
GC11567
Acetazolamide
L-579,486, NSC 145177
L'acétazolamide est un inhibiteur de l'anhydrase carbonique (CA) IX avec une IC50 de 30 nM pour hCA IX.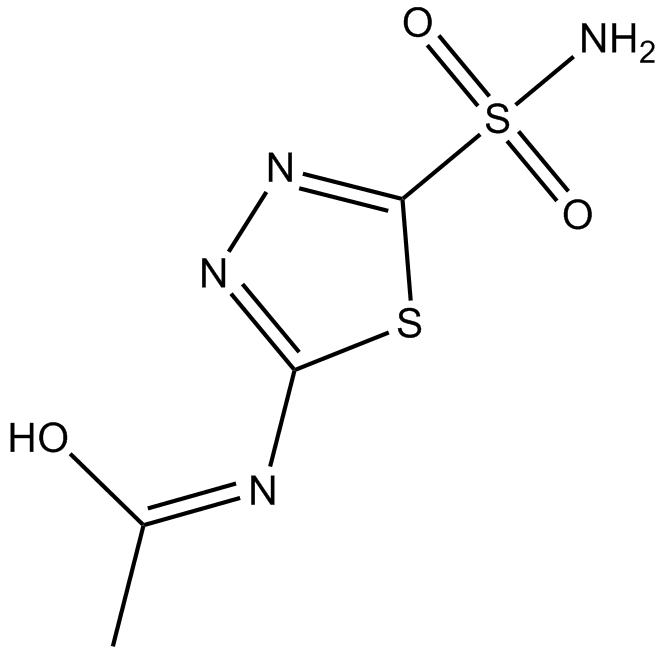
-
GC14142
Acetylcholine Chloride
ACh
Le chlorure d'acétylcholine (chlorure d'ACh), un neurotransmetteur, est un puissant agoniste cholinergique.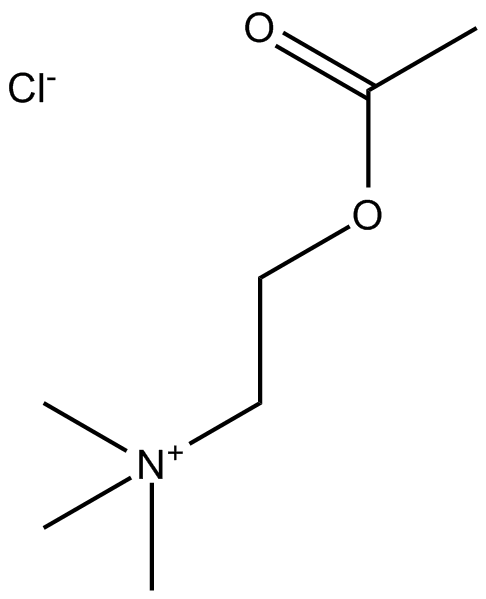
-
GC17094
Acitretin
all-trans Acitretin, Ro 10-1670, Ro 10-1670/000
L'acitrétine (Ro 10-1670) est un rétinoÏde systémique de deuxième génération qui a été utilisé dans le traitement du psoriasis.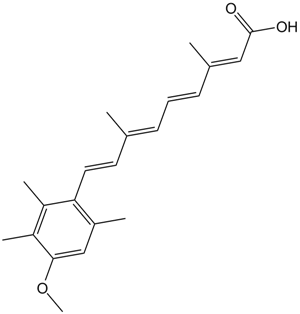
-
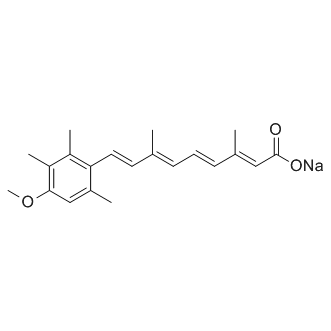
GC17113
Acitretin sodium
L'acitrétine (Ro 10-1670) sodique est un rétinoÏde systémique de deuxième génération qui a été utilisé dans le traitement du psoriasis.
-
GC10102
Aclacinomycin A
NSC 208734
An anthracycline with antibiotic and anticancer activities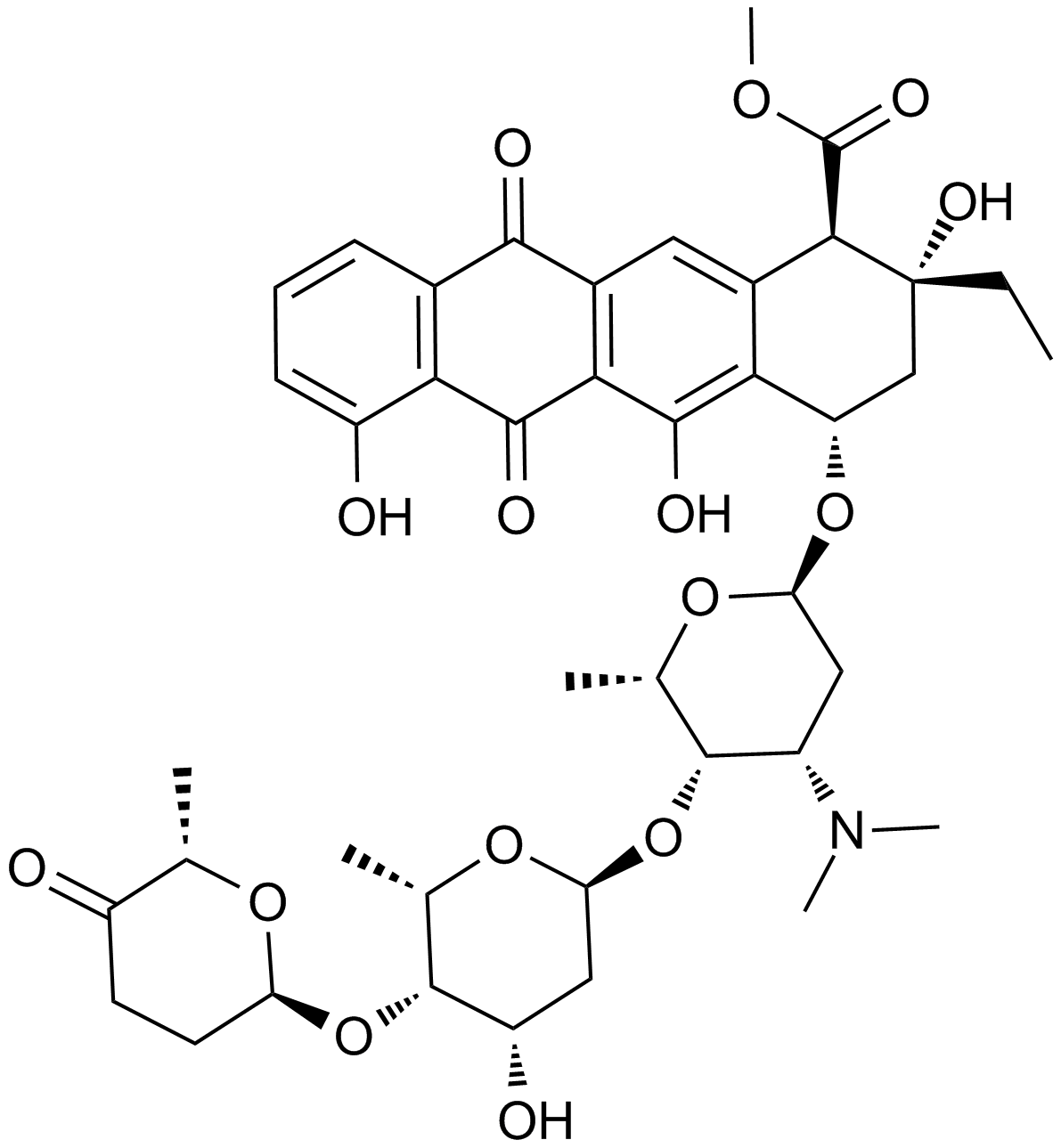
-
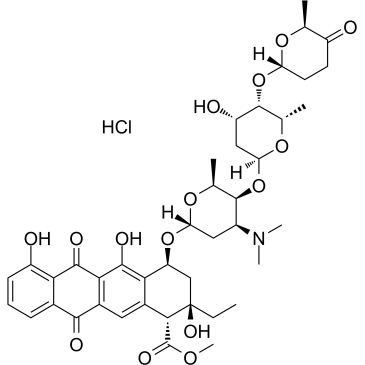
GC35238
Aclacinomycin A hydrochloride
Chlorhydrate d'aclacinomycine A (chlorhydrate d'aclarubicine), une molécule fluorescente et le premier inhibiteur non peptidique décrit présentant une spécificité discrète pour l'activité CTRL (chymotrypsine-like) du protéasome 20S. Le chlorhydrate d'aclacinomycine A est également un double inhibiteur de la topoisomérase I et II. Un agent chimiothérapeutique anthracycline efficace pour les cancers hématologiques et les tumeurs solides.
-
GC41242
Acridine Orange
NSC 194350
L'acridine orange est un colorant fluorescent perméable aux cellules qui colore les organismes (bactéries, parasites, virus, etc.
-
GC35242
Actein
L'actéine est un glycoside triterpénique isolé des rhizomes de Cimicifuga foetida. L'actéine supprime la prolifération cellulaire, induit l'autophagie et l'apoptose en favorisant l'activation des ROS/JNK et en atténuant la voie AKT dans le cancer de la vessie humaine. L'actéine a peu de toxicité in vivo.
-
GC16866
Actinomycin D
Cosmegen, Dactinomycin, Meractinomycin, NCI C04682, NSC 3053, Oncostatin K
Un bloqueur de transcription interagissant avec l'ADN ayant une activité anti-cancer.

-
GC19019
Acumapimod
BCT-197
L'acumapimod (BCT197) est un inhibiteur de la p38 MAP kinase actif par voie orale, avec une IC50 inférieure À 1 μM pour p38α.
-
GC10610
Adapalene
CD 271
L'adapalène (CD271), un rétinoÏde synthétique de troisième génération, est largement utilisé pour la recherche sur l'acné.
-
GC14379
Adapalene sodium salt
Le sel de sodium de l'adapalène (CD271), un rétinoÏde synthétique de troisième génération, est largement utilisé pour la recherche sur l'acné.
-
GC14106
Adenosine
NSC 7652
nucléoside
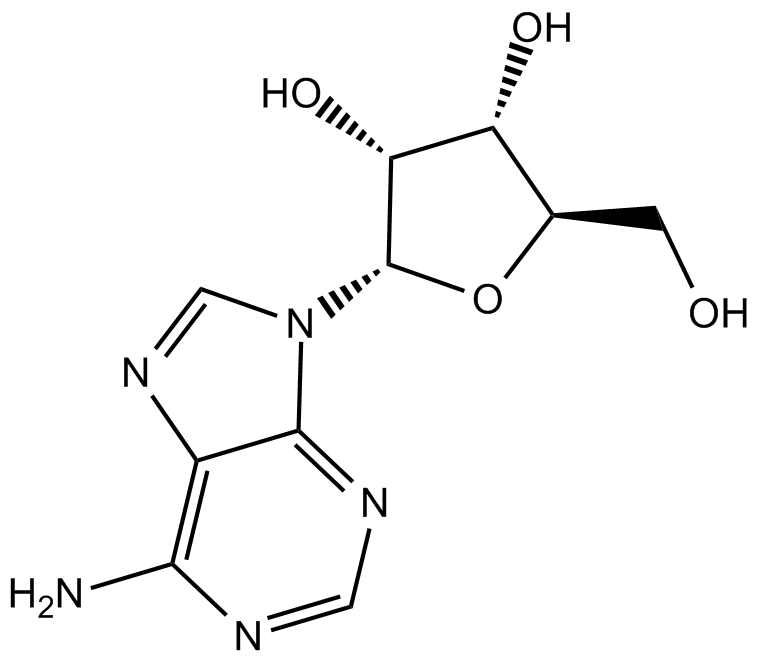
-
GC19729
Adenosine 5′-diphosphoribose sodium
ADP ribose sodium
L'adénosine 5′-diphosphoribose sodique (ADP ribose sodique) est un métabolite nicotinamide adénine nucléotide (NAD+).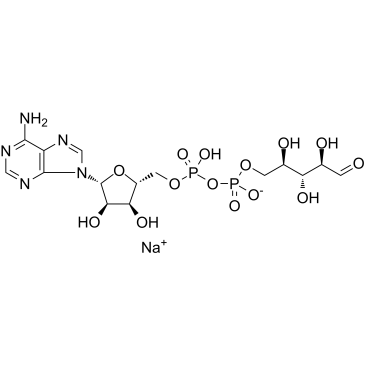
-
GC10707
Afatinib dimaleate
Afatinib dimaleate, BIBW 2992, BIBW 2992MA2
An inhibitor of EGFR and ErbB2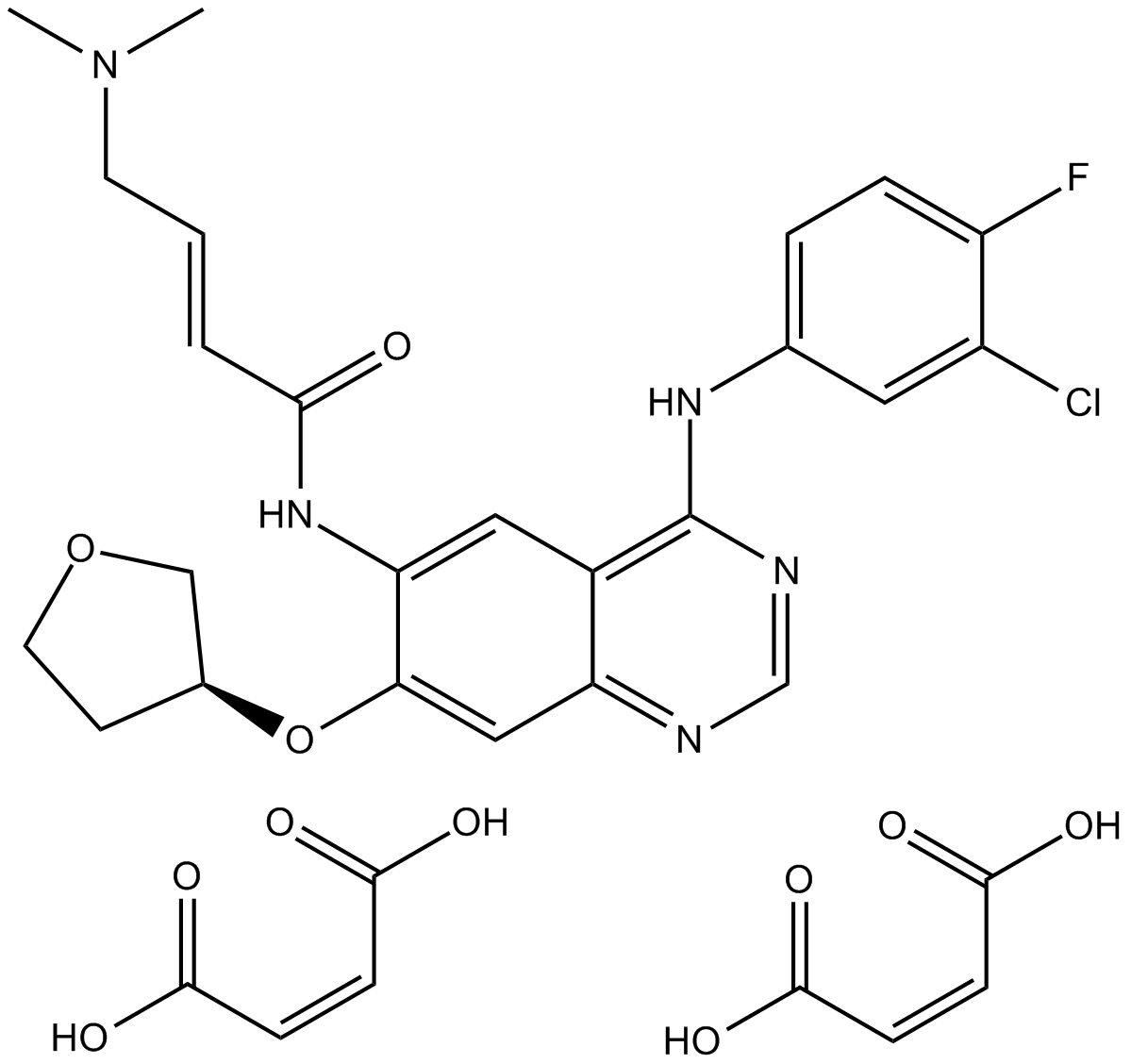
-
GC60567
Afatinib impurity 11
L'impureté 11 de l'afatinib est une impureté de l'afatinib. L'afatinib est un inhibiteur irréversible de la famille de l'EGFR avec des IC50 de 0,5 nM, 0,4 nM, 10 nM et 14 nM pour EGFRwt, EGFRL858R, EGFRL858R/T790M et HER2, respectivement.
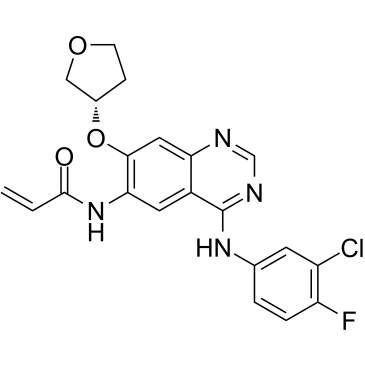
-
GC35262
Afzelin
Kaempferin, Kaempferol 3-O-rhamnoside, Kaempferol 3-O-α-L-rhamnopyranoside
Afzelin (Kaempferol-3-O-rhamnoside) est un glycoside de flavonol trouvé dans Houttuynia cordata Thunberg et est largement utilisé dans la préparation d'agents antibactériens et antipyrétiques, de détoxifiants et pour le traitement de l'inflammation.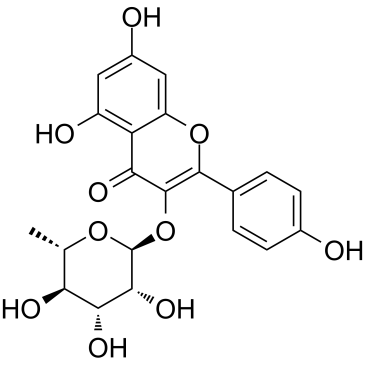
-
GC13697
AG-1024
AGS 200, Tyrphostin AG1024
AG-1024 (Tyrphostin AG 1024) est un inhibiteur réversible, compétitif et sélectif de l'IGF-1R avec une IC50 de 7 μM.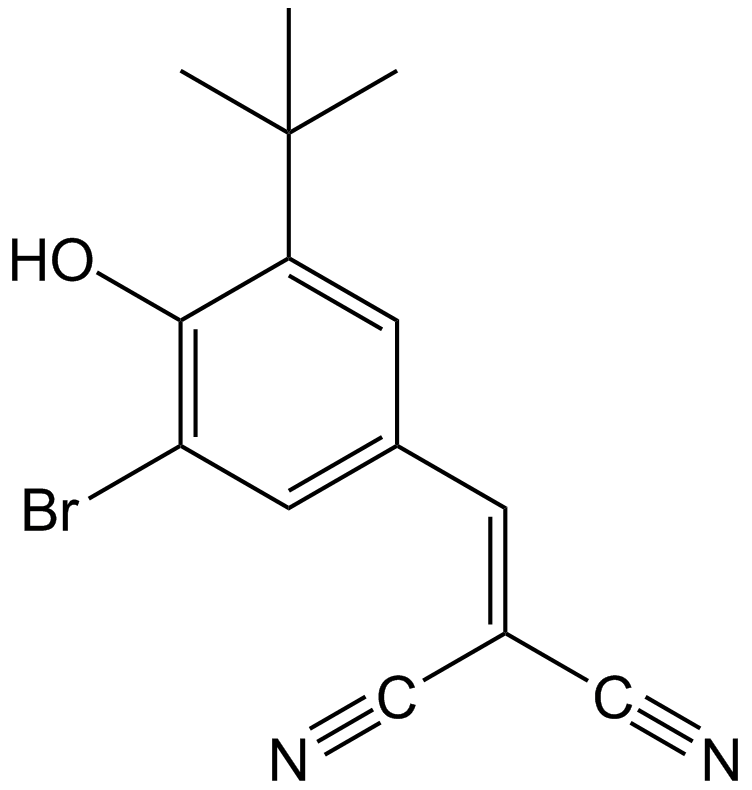
-
GC13854
AG-490 (Tyrphostin B42)
Tyrphostin AG-490
AG-490 (Tyrphostin B42) (Tyrphostin AG-490 (Tyrphostin B42)) est un inhibiteur de la tyrosine kinase qui inhibe EGFR, Stat-3 et JAK2/3.
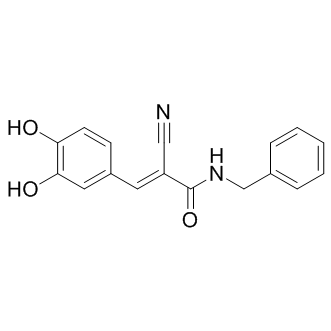
-
GC32817
AGN 193109
AGN 193109 est un analogue de rétinoÏde et agit comme un antagoniste spécifique et très efficace des récepteurs de l'acide rétinoÏque (RAR), avec des Kd de 2 nM, 2 nM et 3 nM pour RARα, RARβ et RARγ, respectivement.
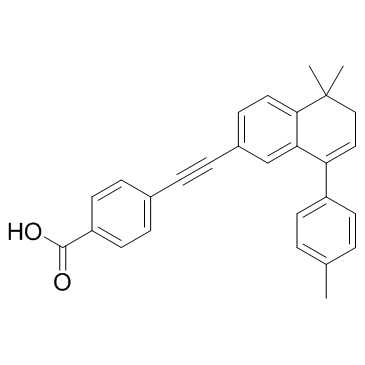
-
GC33315
AGN 194078
AGN 194078 est un RARα sélectif ; agoniste avec un Kd et une CE50 de 3 et 112 nM, respectivement.
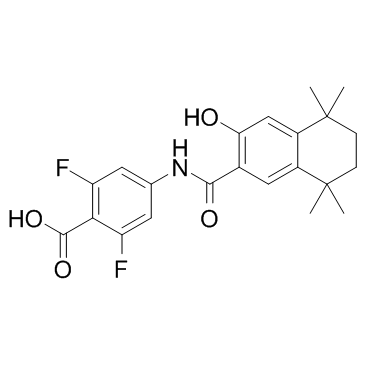
-
GC12598
AGN 194310
VTP-194310
AGN 194310 (VTP-194310) est un pan-antagoniste des récepteurs de l'acide rétinoÏque (RAR) À haute affinité, puissant et sélectif avec des valeurs de Kd de 3 nM, 2 nM, 5 nM pour RARα, RARβ, RARγ, respectivement.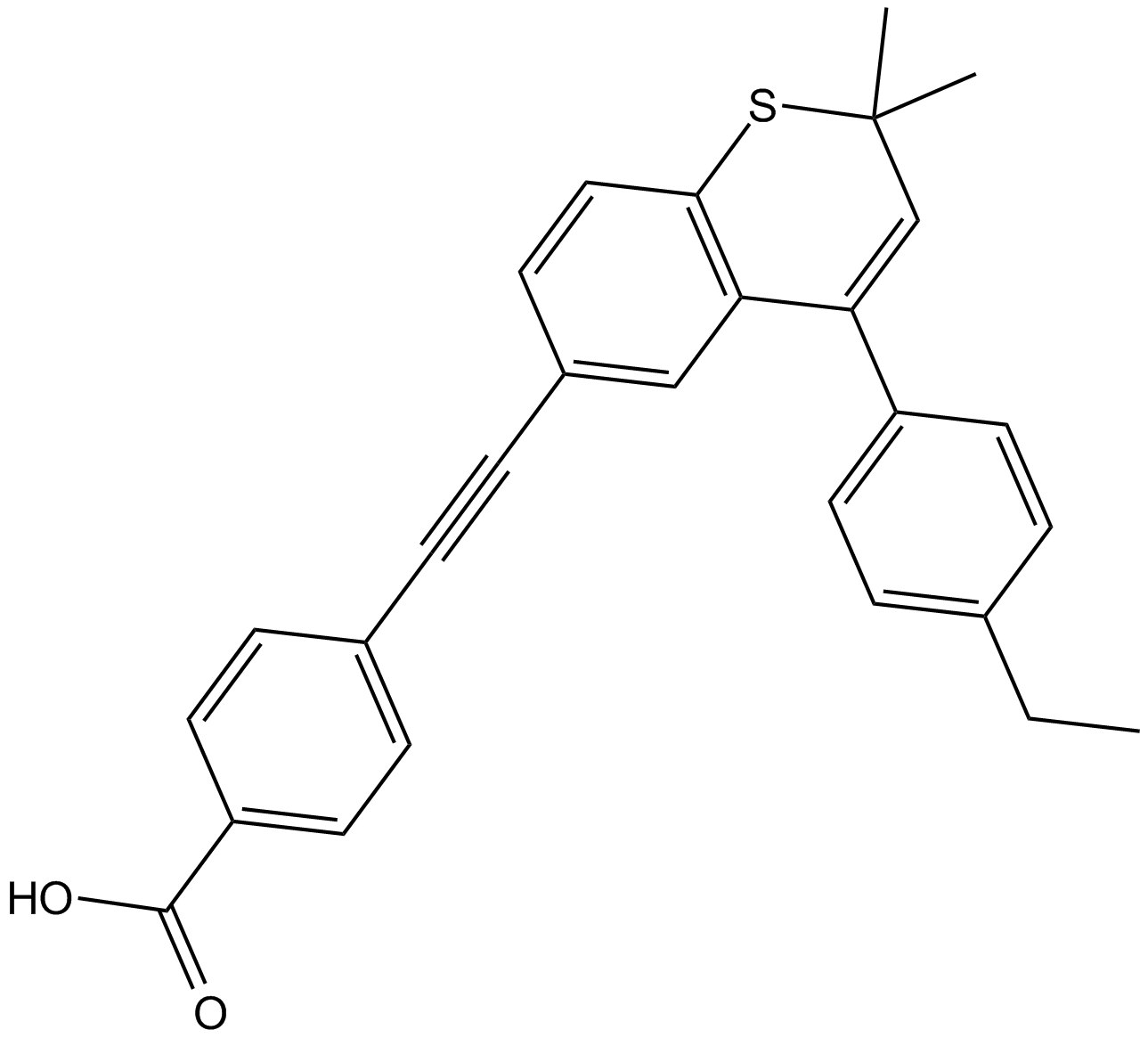
-
GC35265
AGN 195183
IRX-5183; VTP-195183; NRX-195183
AGN 195183 (IRX-5183) est un agoniste puissant et sélectif de RARα (Kd \u003d 3 nM) avec une sélectivité de liaison améliorée par rapport à AGN 193836. AGN 195183 n'a aucune activité sur RARβ/γ.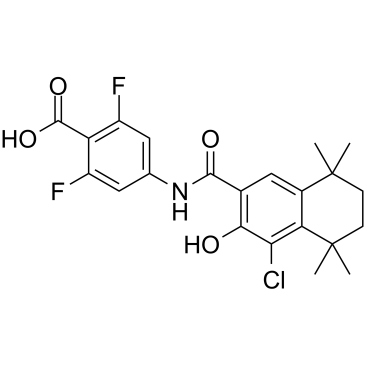
-
GC35266
AGN 196996
AGN 196996 est un antagoniste RARα puissant et sélectif avec une valeur Ki de 2 nM ; peu d'affinité de liaison pour RARβ(Ki=1087 nM) et RARγ(Ki=8523 nM).
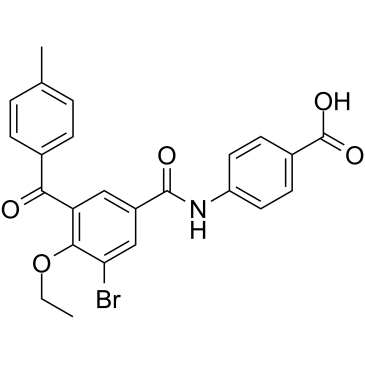
-
GC35267
AGN 205327
AGN 205327 est un puissant agoniste synthétique des RAR avec une CE50 de 3766/734/32 nM pour RARα/β/γ respectivement; aucune inhibition sur RXR.
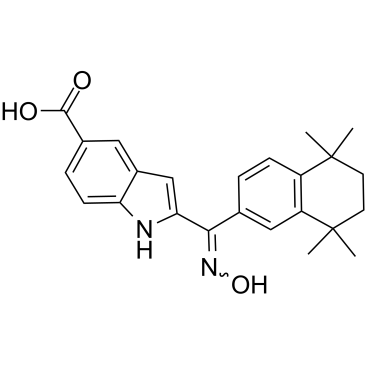
-
GC35268
AGN 205728
AGN 205728 est un antagoniste RARγ puissant et sélectif avec des valeurs Ki/IC95 de 3 nM/ 0,6 nM ; pas d'inhibition sur RARα et RARβ.
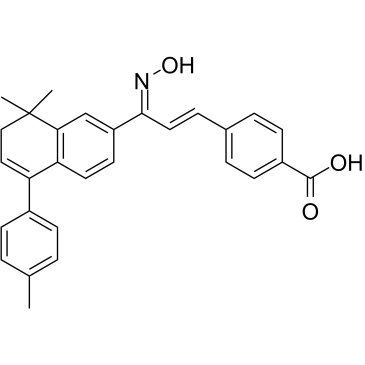
-
GC10518
AICAR
Acadesine, AICA-Riboside, NSC 105823
Un activateur de l'AMPK
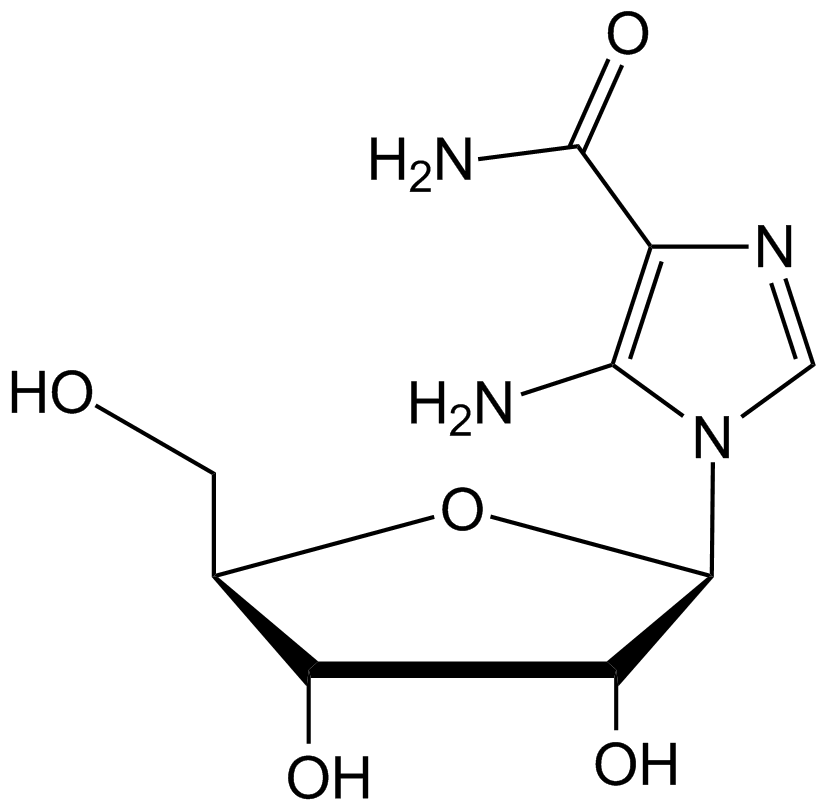
-
GC10580
AICAR phosphate
AICAR phosphate (Acadesine phosphate) est un analogue de l'adénosine et un activateur de l'AMPK. Le phosphate AICAR régule le métabolisme du glucose et des lipides et inhibe la production de cytokines pro-inflammatoires et d'iNOS. Le phosphate AICAR est également un inhibiteur de l'autophagie, du YAP et de la mitophagie.
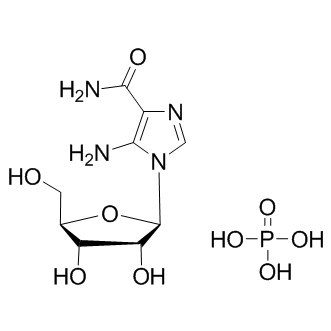
-
GC12646
AL 8697
AL 8697 est un inhibiteur de p38α MAPK spécifique et actif par voie orale avec une IC50 de 6 nM.
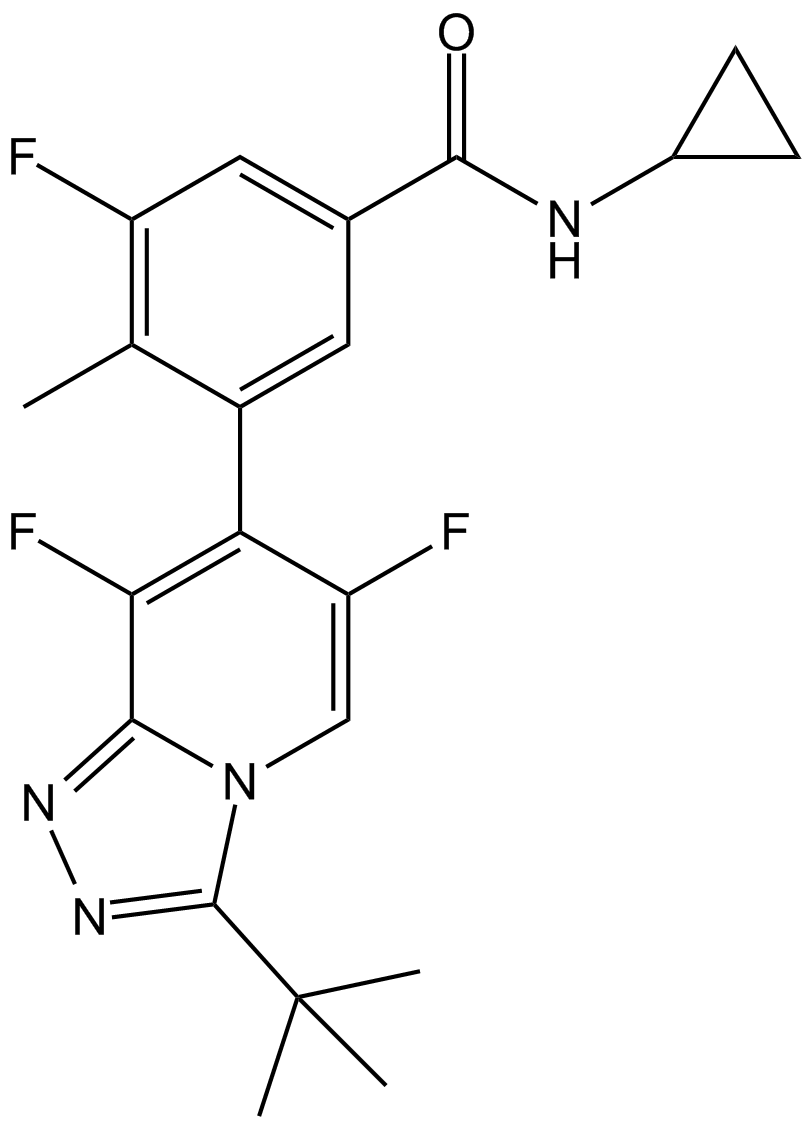
-
GC33743
Alicapistat (ABT-957)
ABT-957
Alicapistat (ABT-957) (ABT-957) est un inhibiteur sélectif actif par voie orale des calpaÏnes humaines 1 et 2 pour l'application potentielle de la maladie d'Alzheimer (MA).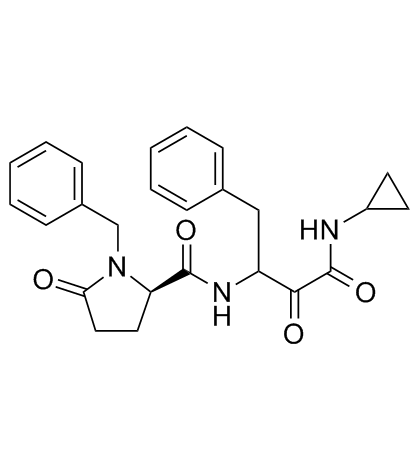
-
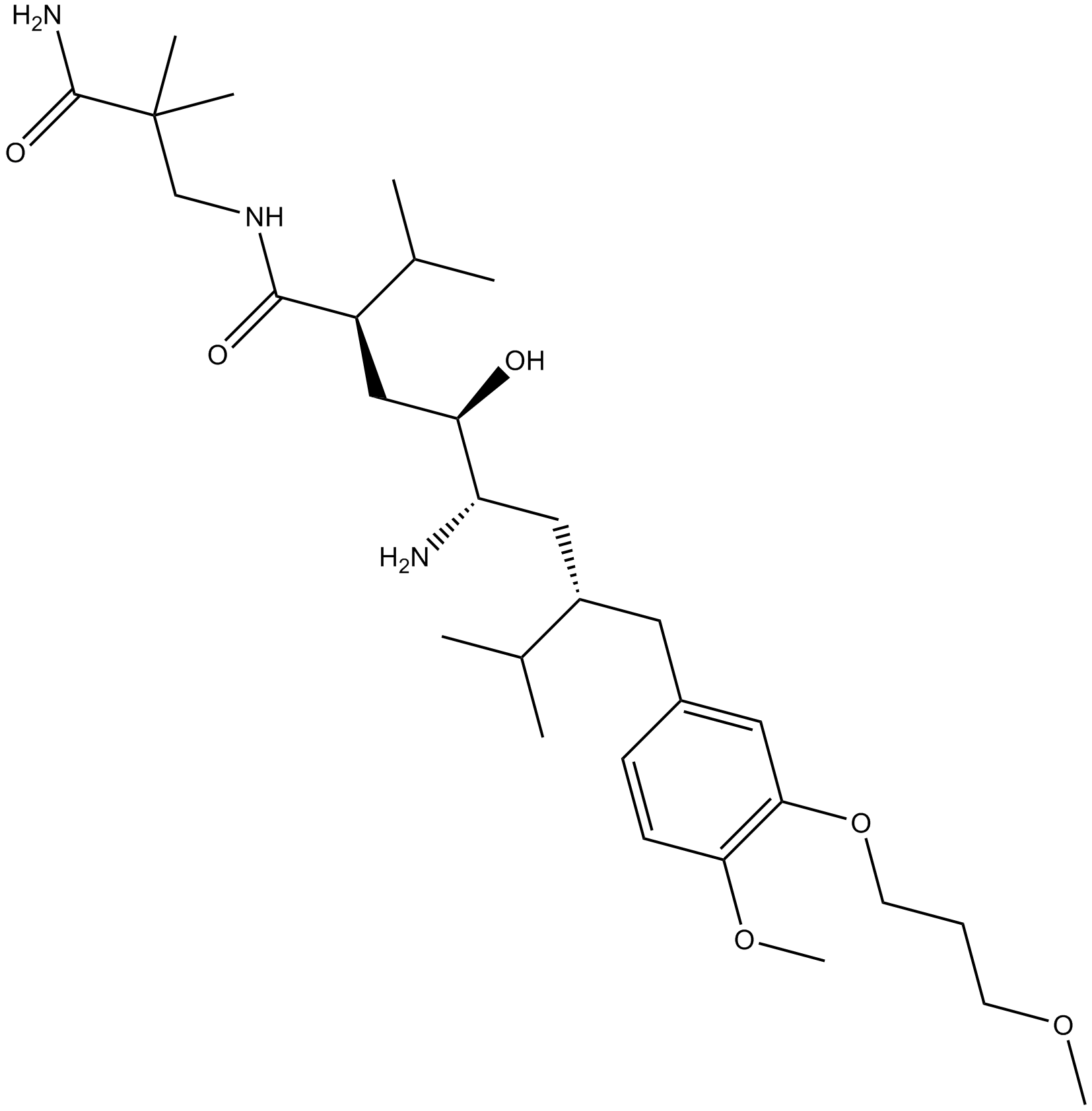
GC15451
Aliskiren
-
GC13223
Aliskiren Hemifumarate
CGP 60536, SPP 100
L'hémifumarate d'aliskirène (CGP 60536; CGP60536B; SPP 100) est un inhibiteur de la rénine actif et sélectif par voie orale, avec une IC50 de 1,5 nM.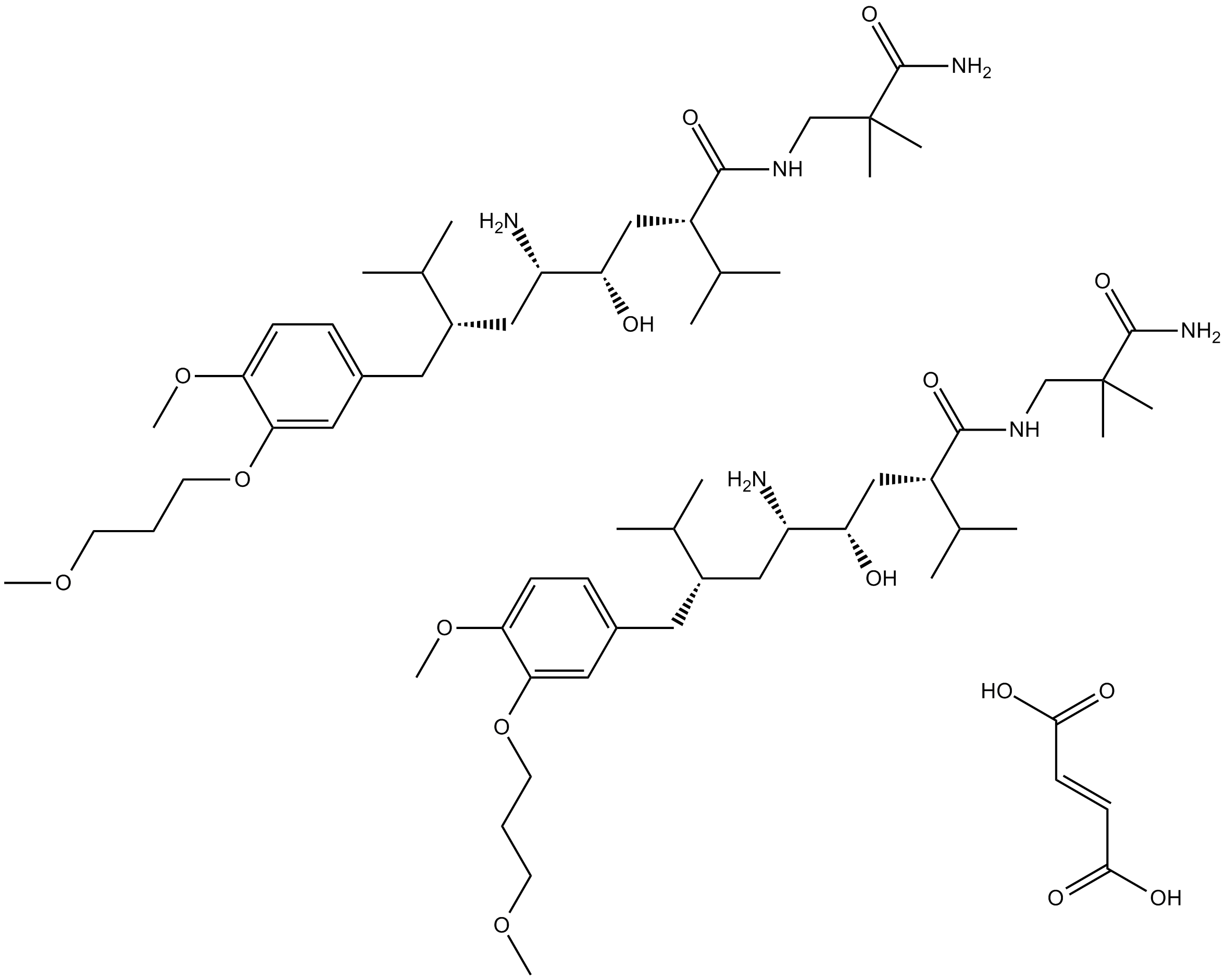
-
GN10407
Alisol A
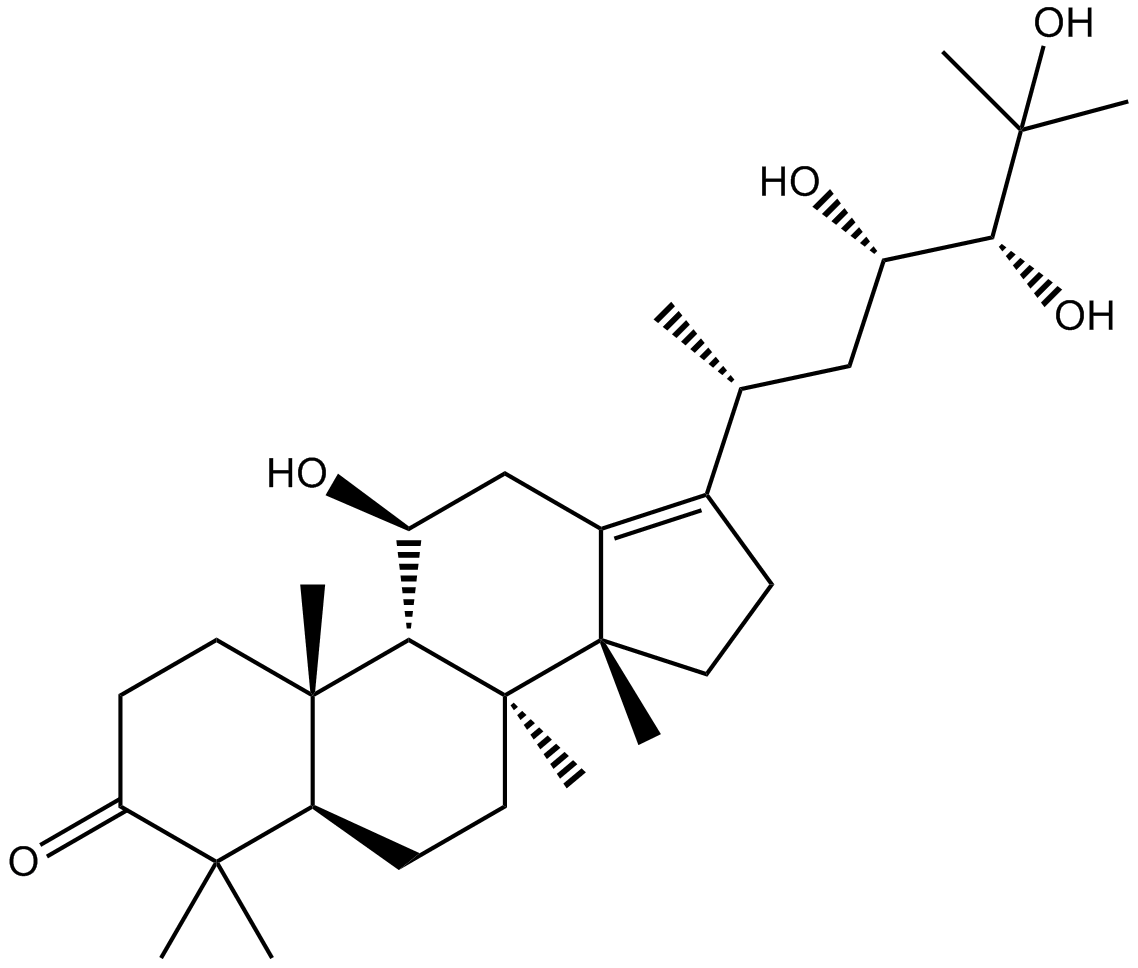
-
GC14749
ALLO-1
ALLO-1, un récepteur de l'autophagie, est essentiel À la formation d'autophagosomes autour des organites paternels et se lie directement À l'homologue du ver LC3 LGG-1 via son motif de région d'interaction avec LC3 (LIR).
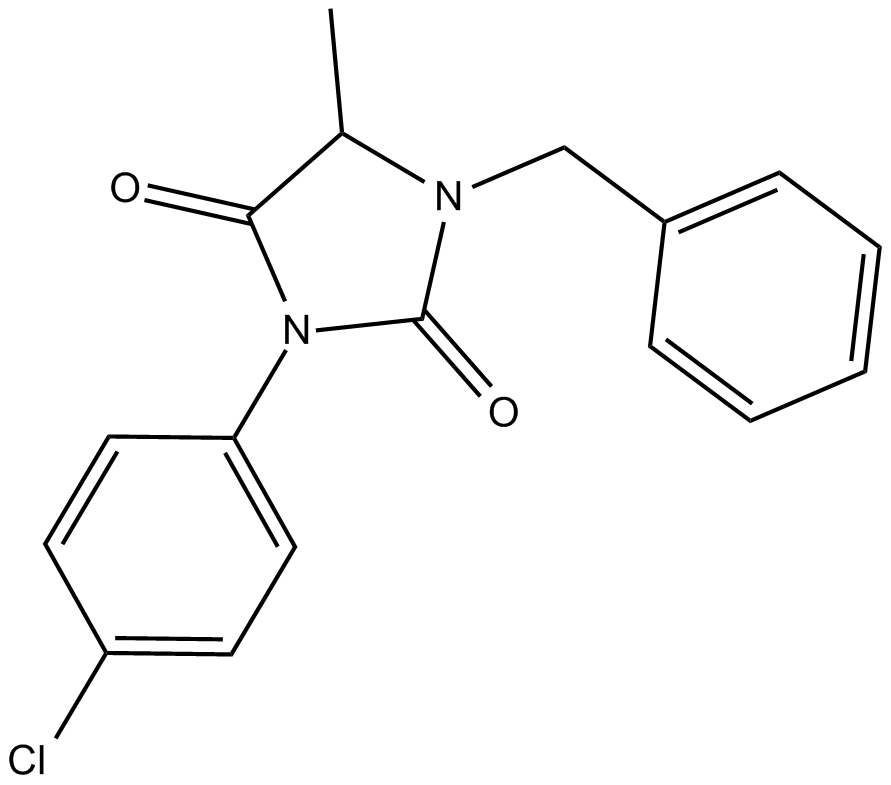
-
GN10516
Aloeemodin
3-Hydroxymethylchrysazine, NSC 38628, Rhabarberone
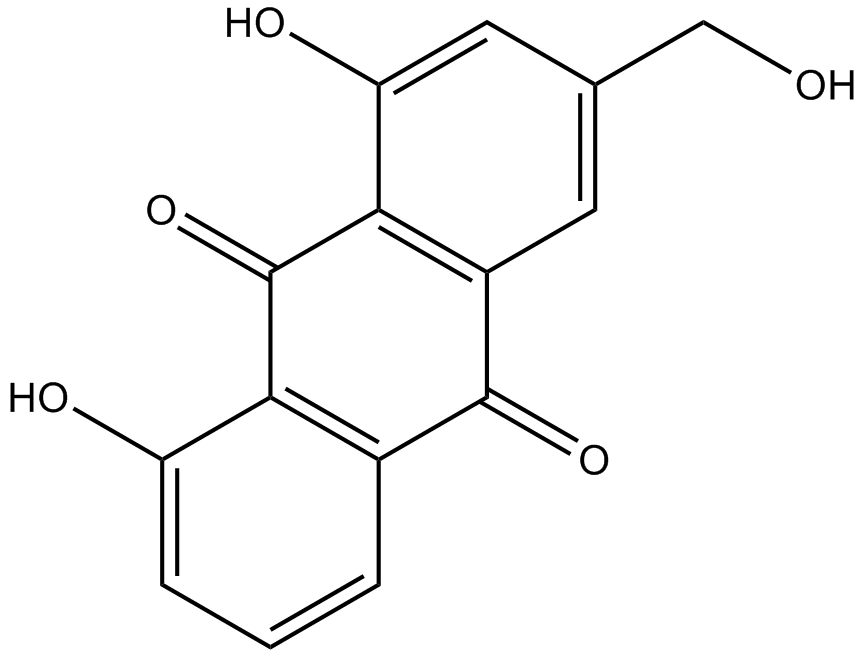
-
GC14314
Aloperine
L'alopérine est un alcaloÏde des plantes sophora telles que Sophora alopecuroides L, qui a montré des propriétés anticancéreuses, anti-inflammatoires et anti-virales.
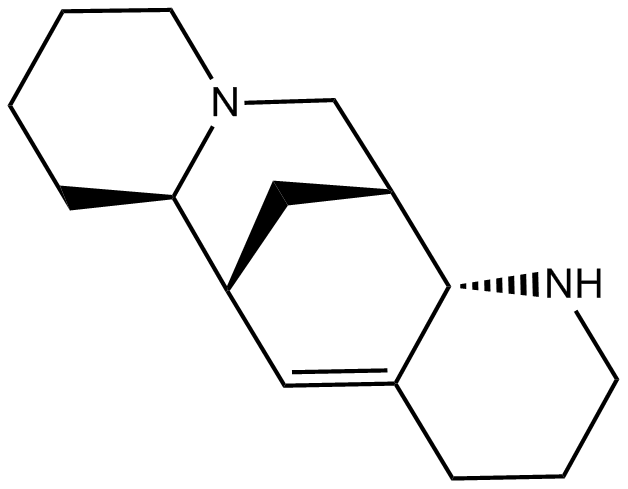
-
GC13789
AM 114
PS-IX; AM114
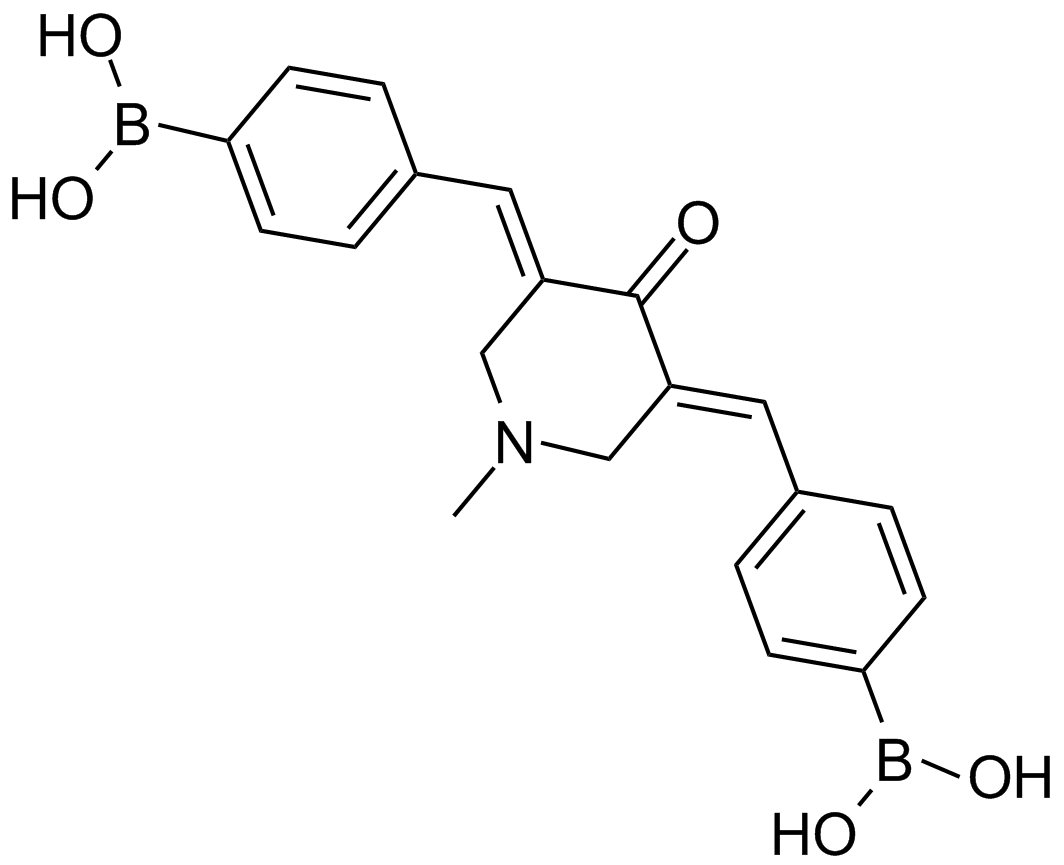
-
GC13664
AM580
CD336, NSC 608001, Ro 406055
AM580 est un agoniste sélectif de RARα avec IC50 et EC50 de 8 nM et 0,36 nM, respectivement.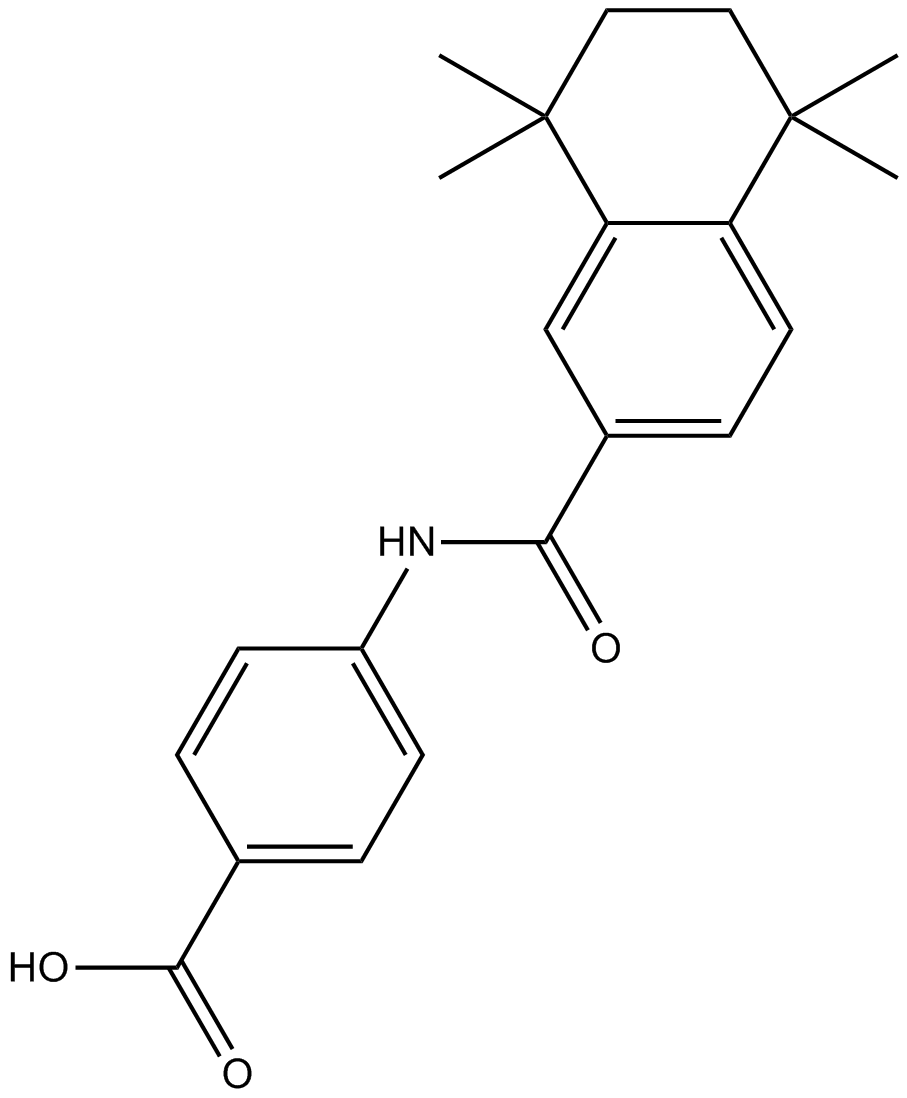
-
GC35322
Amiodarone
L'amiodarone est un médicament anti-arythmique pour l'inhibition du canal potassique sensible À l'ATP avec une IC50 de 19,1 μM.
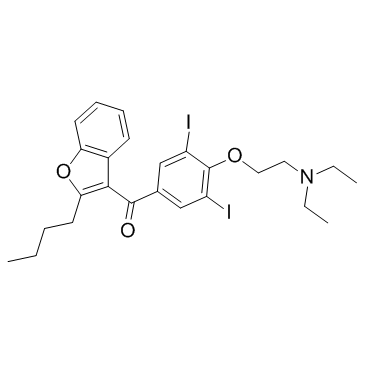
-
GC11432
Amiodarone HCl
Amiodarone HCl, un agent antiarythmique de classe III À base de benzofurane, inhibe les queues WT outwardIhERG avec une IC50 de ~ 45 nM.
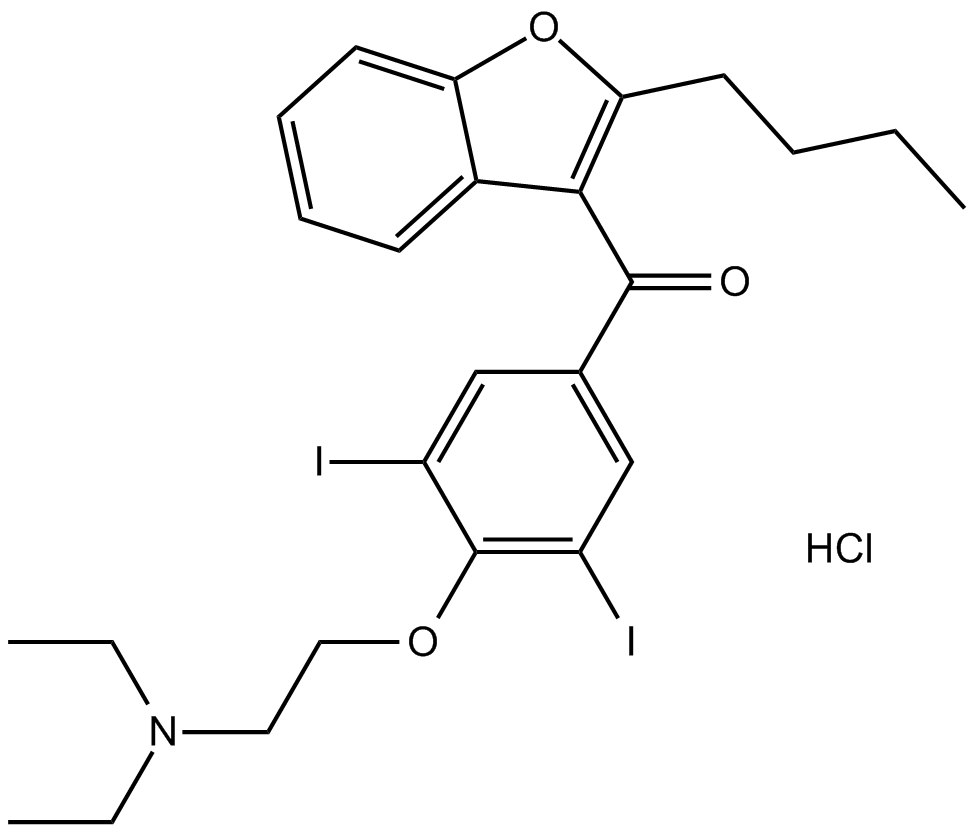
-
GC63316
Ammonium chloride
Le chlorure d'ammonium, en tant que composé hétéropolaire avec régulation de la valeur du pH, peut provoquer une alcalinisation intracellulaire et une acidose métabolique, affectant ainsi l'activité enzymatique et influenÇant le processus du système biologique. Le chlorure d'ammonium est un inhibiteur de l'autophagie.



