CDK
<div class="colum_1 clearer"><p>CDKs (Cyclin-dependent kinases) are serine-threonine kinases first discovered for their role in regulating the cell cycle. They are also involved in regulating transcription, mRNA processing, and the differentiation of nerve cells. CDKs are relatively small proteins, with molecular weights ranging from 34 to 40 kDa, and contain little more than the kinase domain. In fact, yeast cells can proliferate normally when their CDK gene has been replaced with the homologous human gene. By definition, a CDK binds a regulatory protein called a cyclin. Without cyclin, CDK has little kinase activity; only the cyclin-CDK complex is an active kinase.</p><p>There are around 20 Cyclin-dependent kinases (CDK1-20) known till date. CDK1, 4 and 5 are involved in cell cycle, and CDK 7, 8, 9 and 11 are associated with transcription.</p><p>CDK levels remain relatively constant throughout the cell cycle and most regulation is post-translational. Most knowledge of CDK structure and function is based on CDKs of <i>S. pombe</i> (Cdc2), <i>S. cerevisia</i> (CDC28), and vertebrates (CDC2 and CDK2). The four major mechanisms of CDK regulation are cyclin binding, CAK phosphorylation, regulatory inhibitory phosphorylation, and binding of CDK inhibitory subunits (CKIs).</p></div>
Targets for CDK
Products for CDK
- Cat.No. Nombre del producto Información
-
GC65346
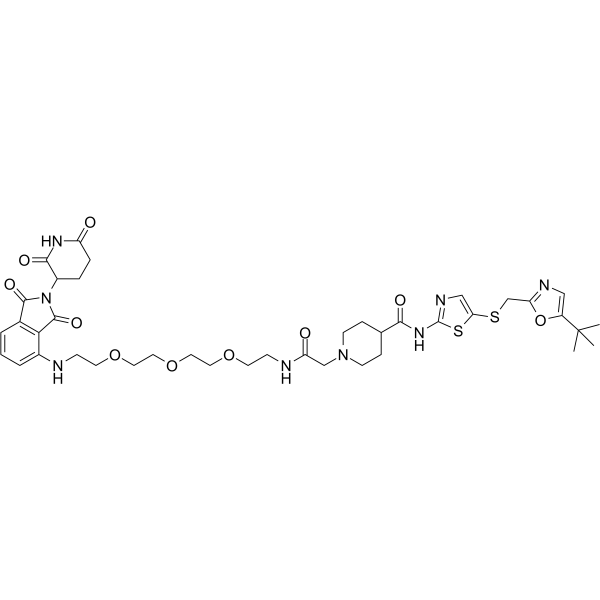
THAL-SNS-032
THAL-SNS-032 es un degradador selectivo de CDK9 PROTAC que consta de un ligando SNS-032 de uniÓn a CDK unido a un derivado de talidomida que se une a la ubiquitina ligasa E3 Cereblon (CRBN).
-
GC10840
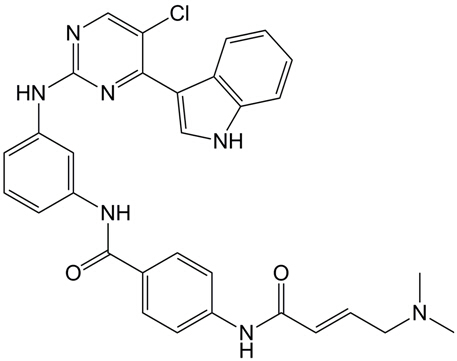
THZ1
THZ1 es un inhibidor covalente selectivo y potente de CDK7 con una IC50 de 3,2 nM. THZ1 también inhibe las quinasas estrechamente relacionadas CDK12 y CDK13 y regula a la baja la expresiÓn de MYC.
-
GC25996
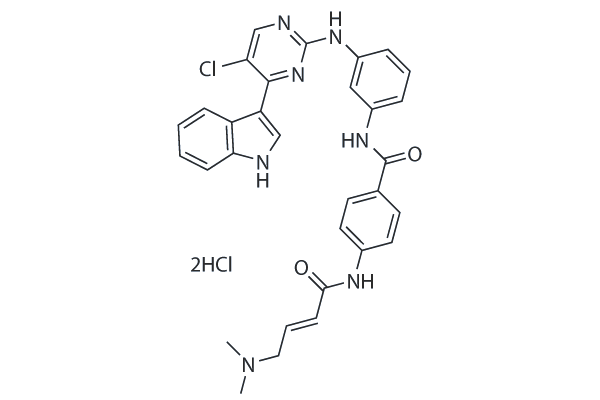
THZ1 2HCl
THZ1 is a covalent CDK7 inhibitor which has the unprecedented ability to target a remote cysteine residue located outside of the canonical kinase domain, providing an unanticipated means of achieving selectivity for CDK7.
-
GC12642
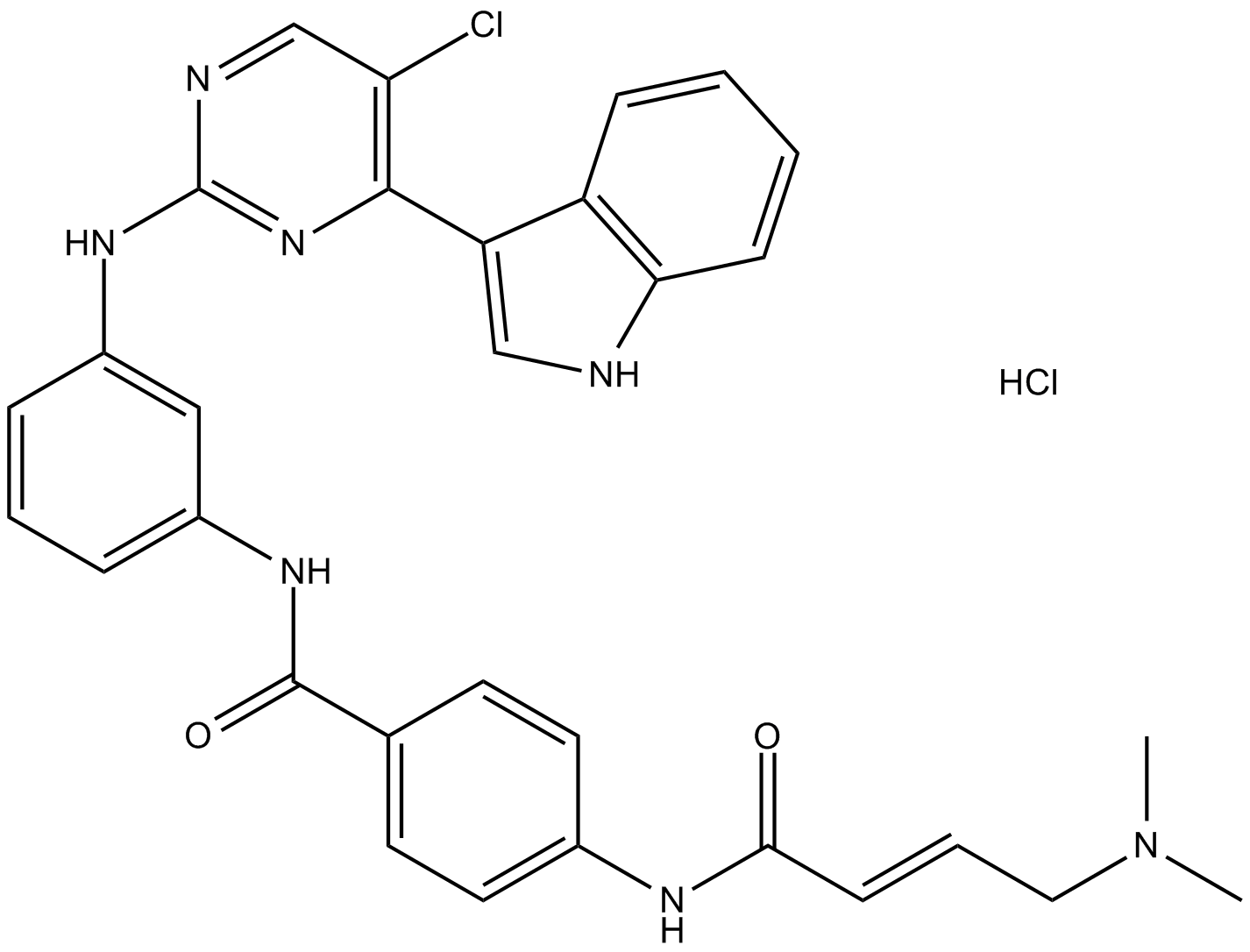
THZ1 Hydrochloride
El clorhidrato de THZ1 es un inhibidor covalente selectivo y potente de CDK7 con una IC50 de 3,2 nM. El clorhidrato de THZ1 también inhibe las quinasas estrechamente relacionadas CDK12 y CDK13 y regula a la baja la expresiÓn de MYC.
-
GC37786
THZ1-R
THZ1-R es un anÁlogo de THZ1 no reactivo con cisteÍna que muestra una actividad disminuida para la inhibiciÓn de CDK7. THZ1-R se une a CDK7 con una Kd de 142 nM.
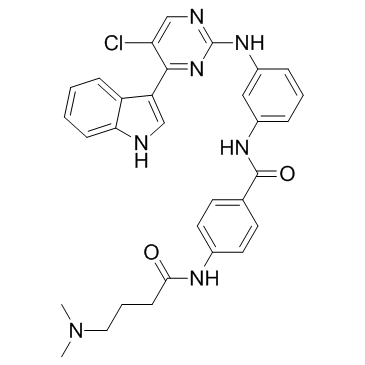
-
GC13511
THZ2
THZ2 es un inhibidor potente y selectivo de CDK7 con una IC50 de 13,9 nM.
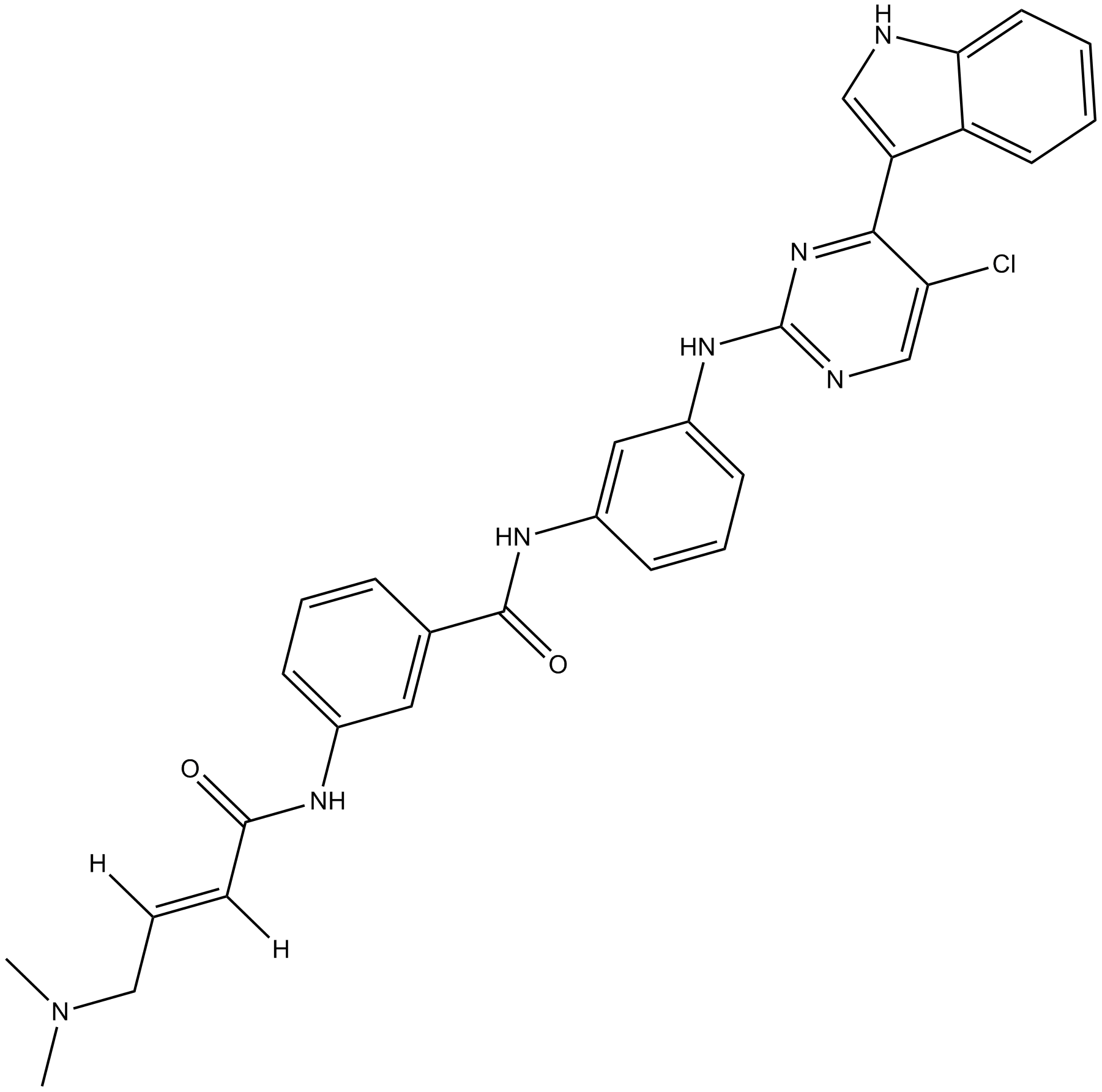
-
GC16420
THZ531
THZ531 es un inhibidor selectivo y covalente de CDK12 y CDK13 con IC50 de 158 nM y 69 nM, respectivamente.
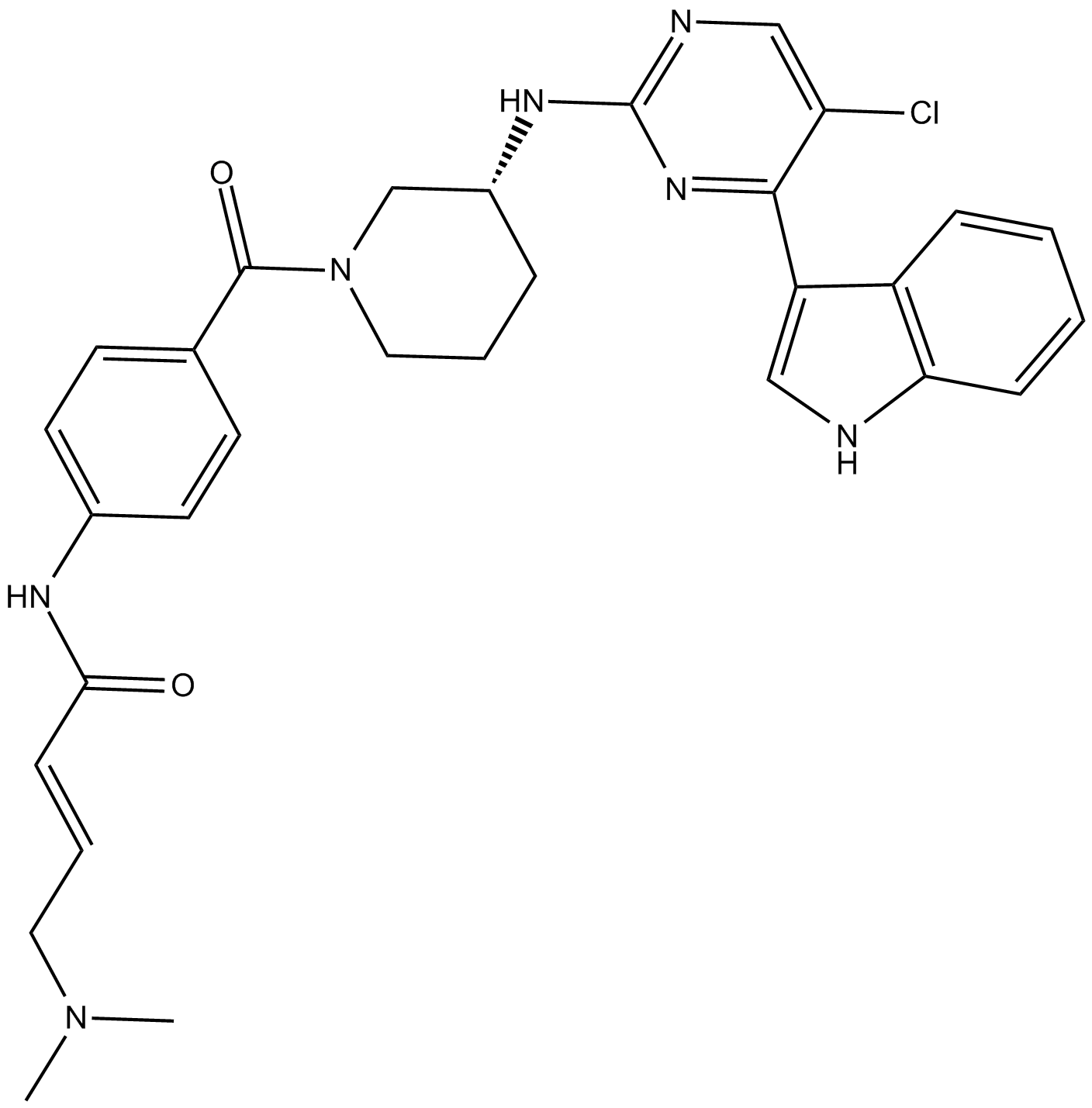
-
GC50484
TL 12-186
TL 12-186 es un degradador de PROTAC multiquinasa dependiente de Cereblon. Las multiquinasas incluyen CDK, BTK, FLT3, Aurora quinasas, TEC, ULK, ITK, et al. TL 12-186 inhibe CDK2/ciclina A (IC50\u003d73 nM) y CDK9/ciclina T1 (IC50\u003d55 nM).
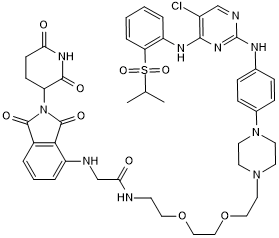
-
GC34118
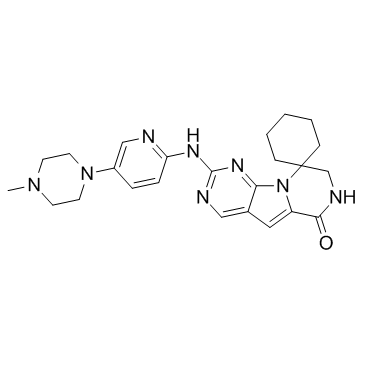
Trilaciclib (G1T28)
Trilaciclib (G1T28) es un inhibidor de CDK4/6 con IC50 de 1 nM y 4 nM para CDK4 y CDK6, respectivamente.
-
GC34087
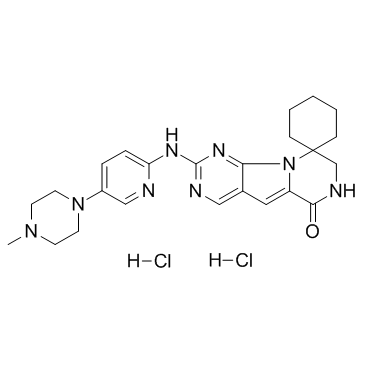
Trilaciclib hydrochloride (G1T28 hydrochloride)
El clorhidrato de trilaciclib (clorhidrato de G1T28) (clorhidrato de G1T28) es un inhibidor de CDK4/6 con IC50 de 1 nM y 4 nM para CDK4 y CDK6, respectivamente.
-
GC45983
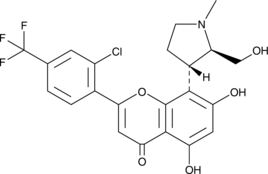
Voruciclib
Voruciclib es un inhibidor de CDK selectivo y activo por vÍa oral con valores de Ki de 0,626 nM-9,1 nM. Voruciclib bloquea potentemente CDK9, el regulador transcripcional de MCL-1. Voruciclib reprime la expresiÓn de MCL-1 en mÚltiples modelos de linfoma difuso de células B grandes (DLBCL).
-
GC65311
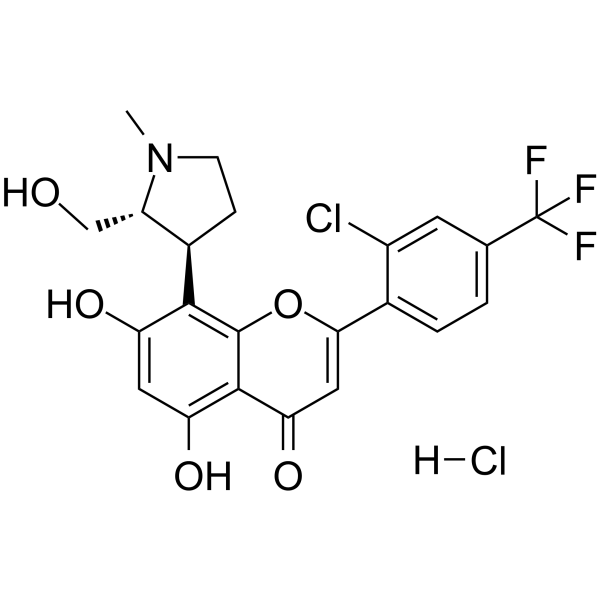
Voruciclib hydrochloride
El clorhidrato de voruciclib es un inhibidor de CDK selectivo y activo por vÍa oral con valores de Ki de 0,626 nM-9,1 nM. El clorhidrato de voruciclib bloquea de manera potente a CDK9, el regulador transcripcional de MCL-1. El clorhidrato de voruciclib reprime la expresiÓn de MCL-1 en mÚltiples modelos de linfoma difuso de células B grandes (DLBCL).
-
GN10017
Wogonin
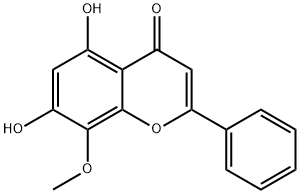
-
GC18117
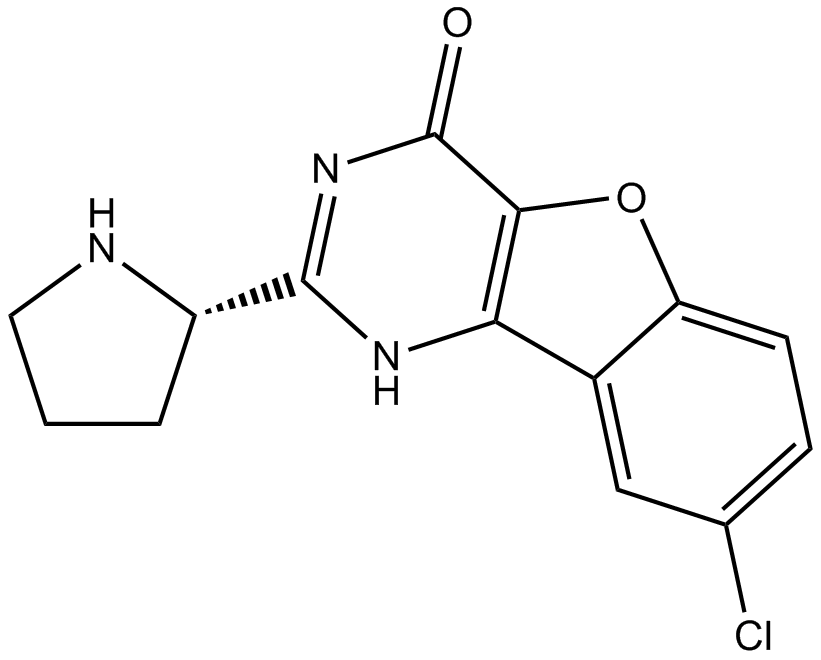
XL413
XL413 es un inhibidor potente, selectivo y competitivo de ATP de Cdc7, con una IC50 de 3,4 nM, y también muestra un efecto potente con IC50 de 215, 42 nM en CK2, PIM1, respectivamente, y una EC50 de 118 nM en pMCM.
-
GC26084
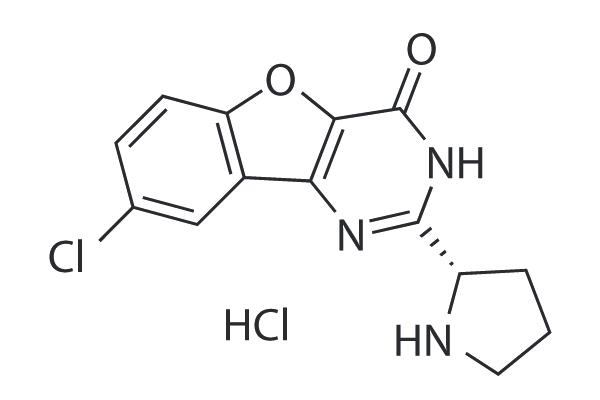
XL413 (BMS-863233)
XL413 (BMS-863233) is a potent and selective cell division cycle 7 homolog (CDC7) kinase inhibitor with IC50 of 3.4 nM, showing 63-, 12- and 35-fold selectivity over CK2, Pim-1 and pMCM2, respectively. Phase 1/2.
-
GC13328
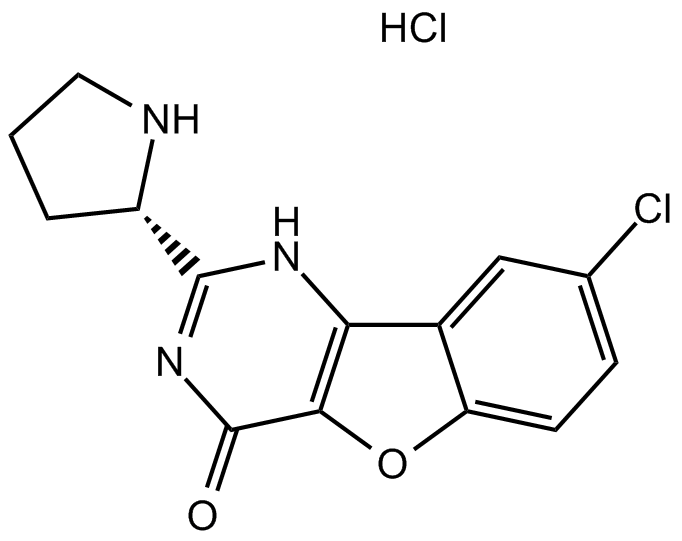
XL413 hydrochloride
El clorhidrato de XL413 (BMS-863233) es un inhibidor potente, selectivo y competitivo de ATP de Cdc7, con una IC50 de 3,4 nM, y también muestra un efecto potente con IC50 de 215, 42 nM en CK2, PIM1, respectivamente, y una EC50 de 118 nM en pMCM.
-
GC62701
XY028-133
XY028-133 (ejemplo 14) es un degradador de CDK4/6 basado en PROTAC con actividad antitumoral, que consta de ligandos para von Hippel-Lindau y CDK.
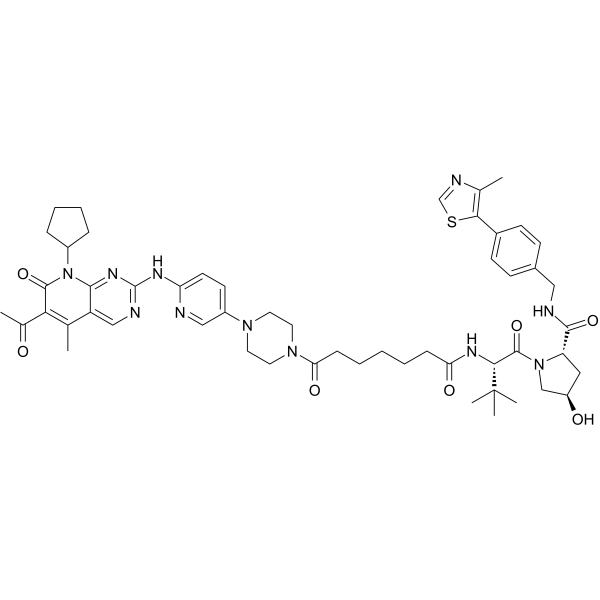
-
GC62139
XY028-140
XY028-140 es un PROTAC conectado por ligandos para Cereblon y CDK. XY028-140 inhibe tanto la expresiÓn de CDK4/6 como la actividad de CDK4/6 en las células cancerosas.
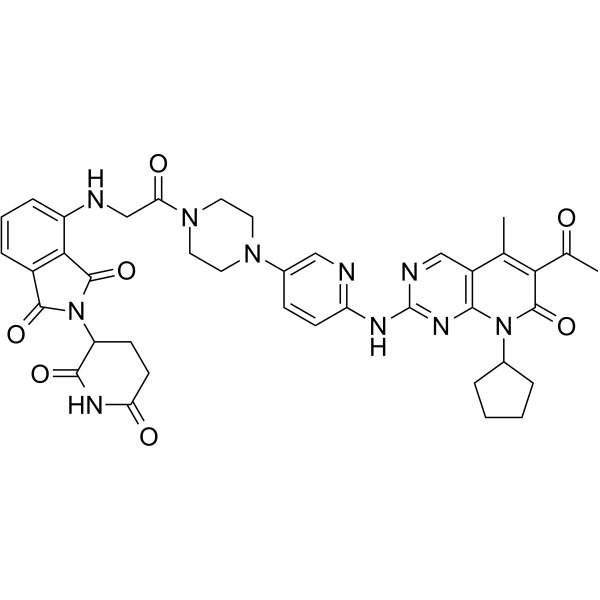
-
GC62294
YKL-5-124
YKL-5-124 es un inhibidor de CDK7 potente, selectivo, irreversible y covalente con IC50 de 53,5 nM y 9,7 nM para CDK7 y CDK7/Mat1/CycH, respectivamente. YKL-5-124 es >100 veces mÁs selectivo para CDK7 que para CDK9 y CDK2, e inactivo contra CDK12 y CDK13. YKL-5-124 induce una fuerte detenciÓn del ciclo celular, inhibe la expresiÓn génica impulsada por E2F y muestra poco efecto sobre el estado de fosforilaciÓn de la ARN polimerasa II.
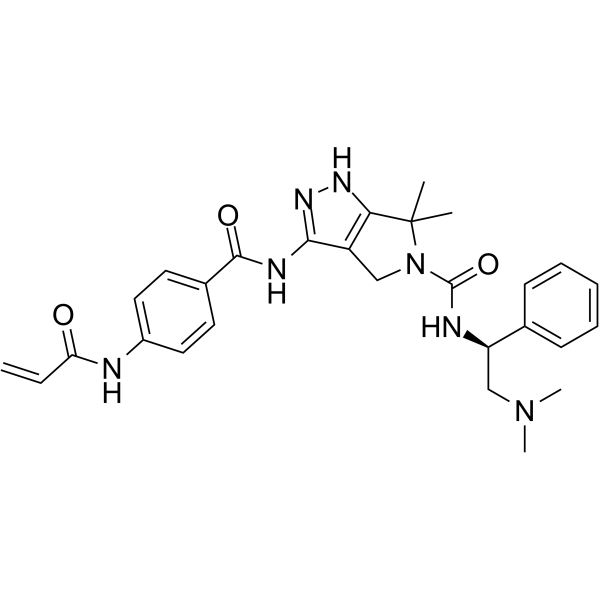
-
GC62112
Zotiraciclib
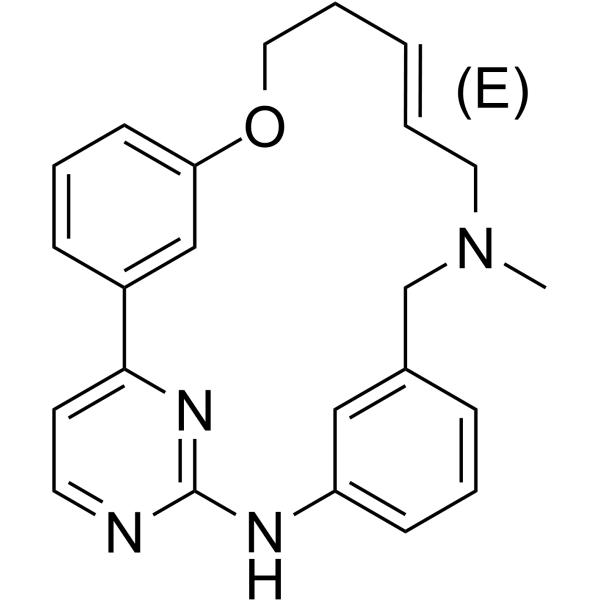
-
GC35021
[pSer2, pSer5, pSer7]-CTD TFA
[pSer2, pSer5, pSer7]-CTD (TFA), un sustrato para CDK7 (proteÍna quinasa dependiente de ciclina), es un polipéptido fosforilado en los sitios ser2, ser5 y ser7 del dominio carboxi-terminal (CTD) de la ARN polimerasa II.
![[pSer2, pSer5, pSer7]-CTD TFA Chemical Structure [pSer2, pSer5, pSer7]-CTD TFA Chemical Structure](/media/struct/GC3/GC35021.png)
-
GC35022
[pThr3]-CDK5 Substrate
El sustrato [pThr3]-CDK5 es un sustrato Phospho-Thr3CDK5 eficaz.
![[pThr3]-CDK5 Substrate Chemical Structure [pThr3]-CDK5 Substrate Chemical Structure](/media/struct/GC3/GC35022.png)


