iGluR
iGluR (ionotropic glutamate receptor) is a ligand-gated ion channel that is activated by the neurotransmitter glutamate. iGluR are integral membrane proteins compose of four large subunits that form a central ion channel pore. Sequence similarity among all known glutamate receptor subunits, including the AMPA, kainate, NMDA, and δ receptors.
AMPA receptors are the main charge carriers during basal transmission, permitting influx of sodium ions to depolarise the postsynaptic membrane. NMDA receptors are blocked by magnesium ions and therefore only permit ion flux following prior depolarisation. This enables them to act as coincidence detectors for synaptic plasticity. Calcium influx through NMDA receptors leads to persistent modifications in the strength of synaptic transmission.
Targets for iGluR
Products for iGluR
- Cat.No. Nom du produit Informations
-
GC16986
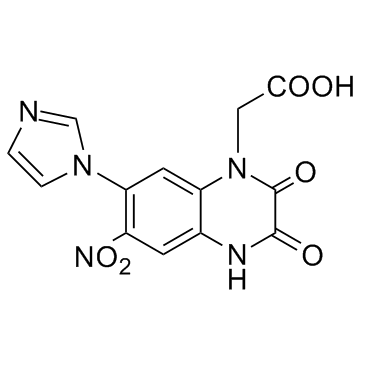
SDZ 220-581 hydrochloride
Le chlorhydrate de SDZ 220-581 est un antagoniste des récepteurs NMDA actif, puissant et compétitif par voie orale avec une valeur de pKi de 7,7 .
-
GC37703
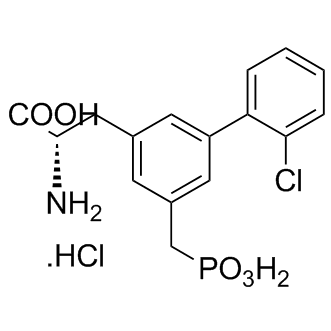
Sunifiram
Le sunifiram (DM-235) est un médicament de type ampakine dérivé de la pipérazine qui a des effets nootropiques dans les études animales avec une puissance significativement plus élevée que le piracétam.
-
GC16101
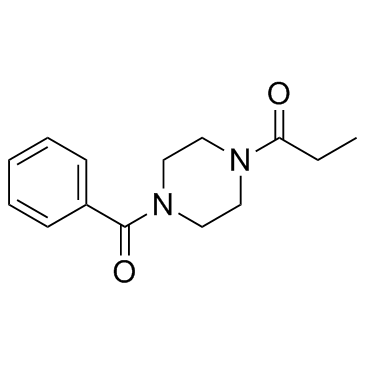
SYM 2081
SYM 2081 est un ligand de haute affinité et un puissant agoniste sélectif des récepteurs kaÏnate, inhibe la liaison [3H]-kaÏnate avec une IC50 de 35 nM, une sélectivité de près de 3000 et 200 fois pour les récepteurs kaÏnate par rapport aux récepteurs AMPA et NMDA respectivement [1 ].
-
GC13728
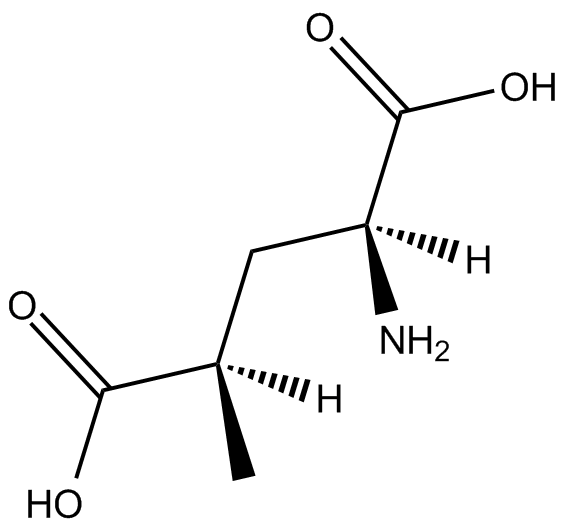
SYM 2206
Le SYM 2206 est un antagoniste puissant et non compétitif des récepteurs AMPA, avec une IC50 de 1,6 μM.
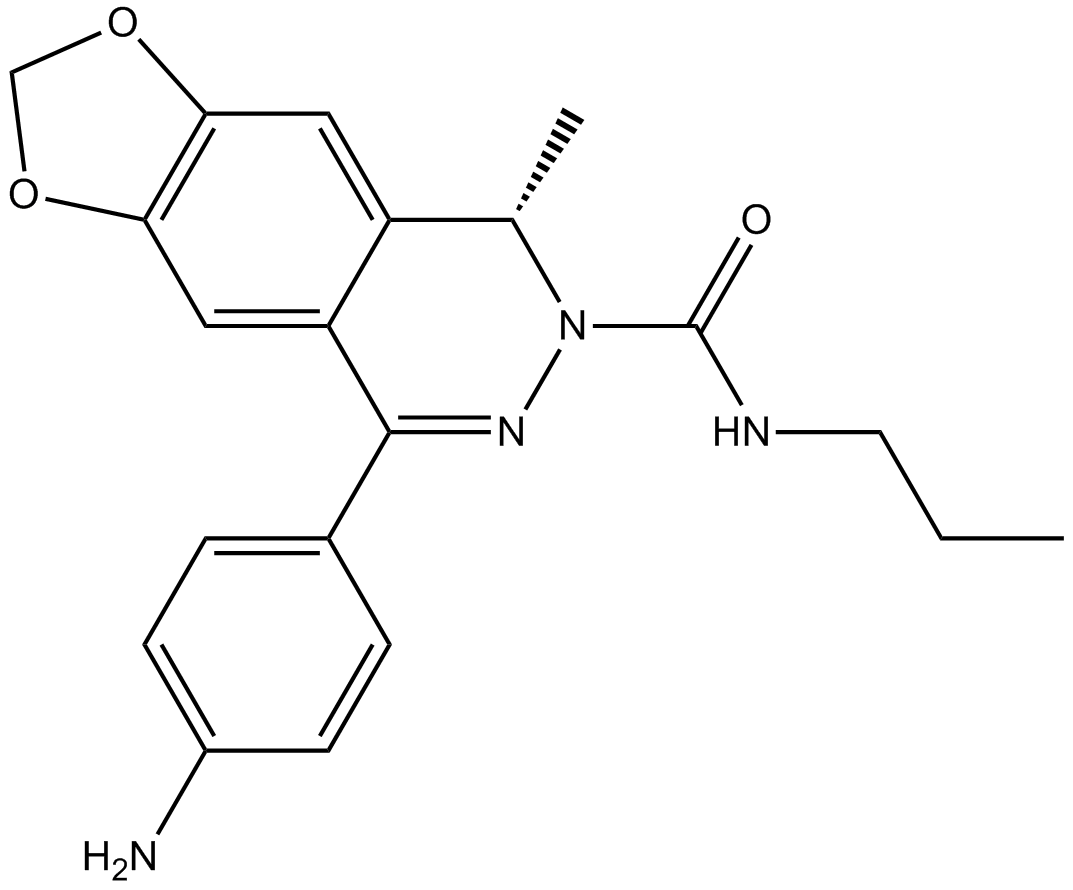
-
GC64650
TAK-653
Le TAK-653, un potentialisateur des récepteurs AMPA avec une activité agoniste minimale, produit un effet antidépresseur avec un profil de sécurité favorable chez le rat.
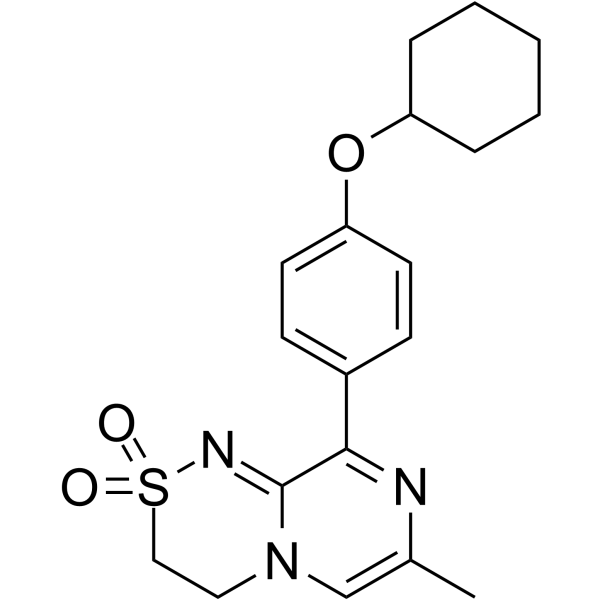
-
GC19346
Talampanel
Talampanel (LY300164) est un antagoniste des récepteurs α-amino-3-hydroxy-5-méthyl-4-isoxazolepropionate (AMPA) oral et sélectif avec une activité anti-épileptique.
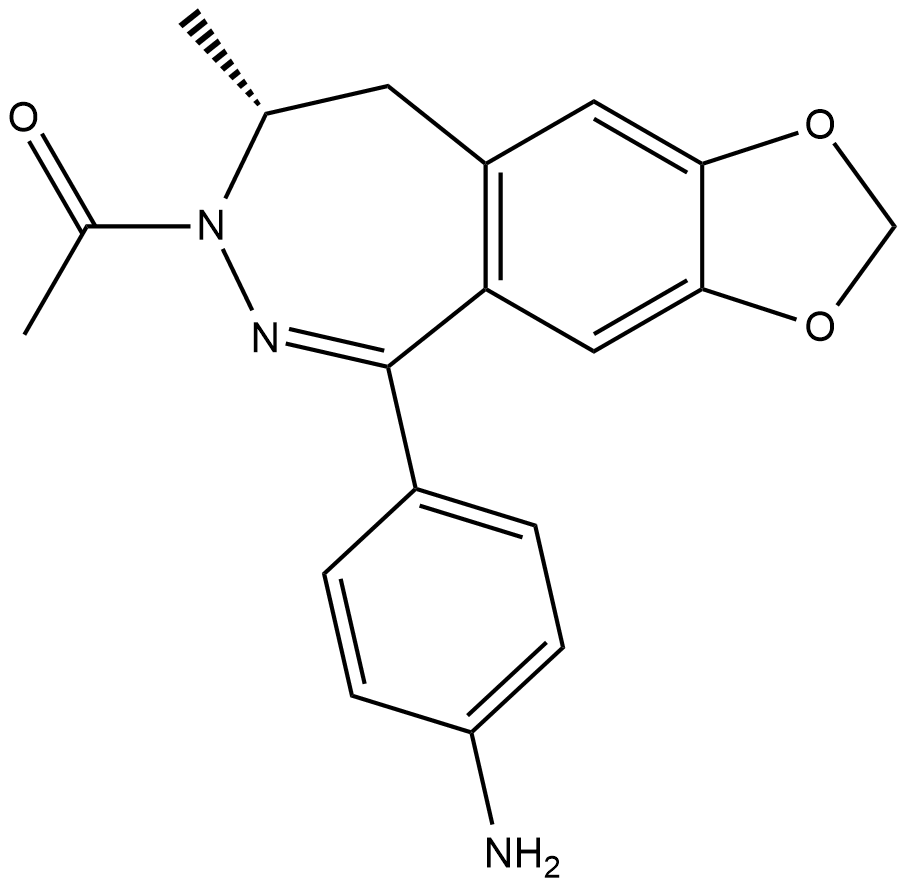
-
GC30774
Tat-NR2B9c
Tat-NR2B9c est conçu pour empêcher la production d'oxyde nitrique (NO) en empêchant la liaison de la protéine 95 de densité postsynaptique (PSD-95) aux récepteurs N-méthyl-D-aspartate (NMDA) et à la synthase d'oxyde nitrique neuronale.

-
GC34828
Tat-NR2B9c TFA
Tat-NR2B9c TFA (Tat-NR2Bct TFA) est un inhibiteur de densité postsynaptique-95 (PSD-95), avec des valeurs EC50 de 6,7 nM et 670 nM pour PSD-95d2 (PSD-95 PDZ domaine 2) et PSD-95d1, respectivement .
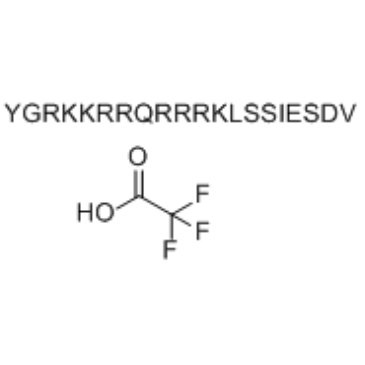
-
GC11585
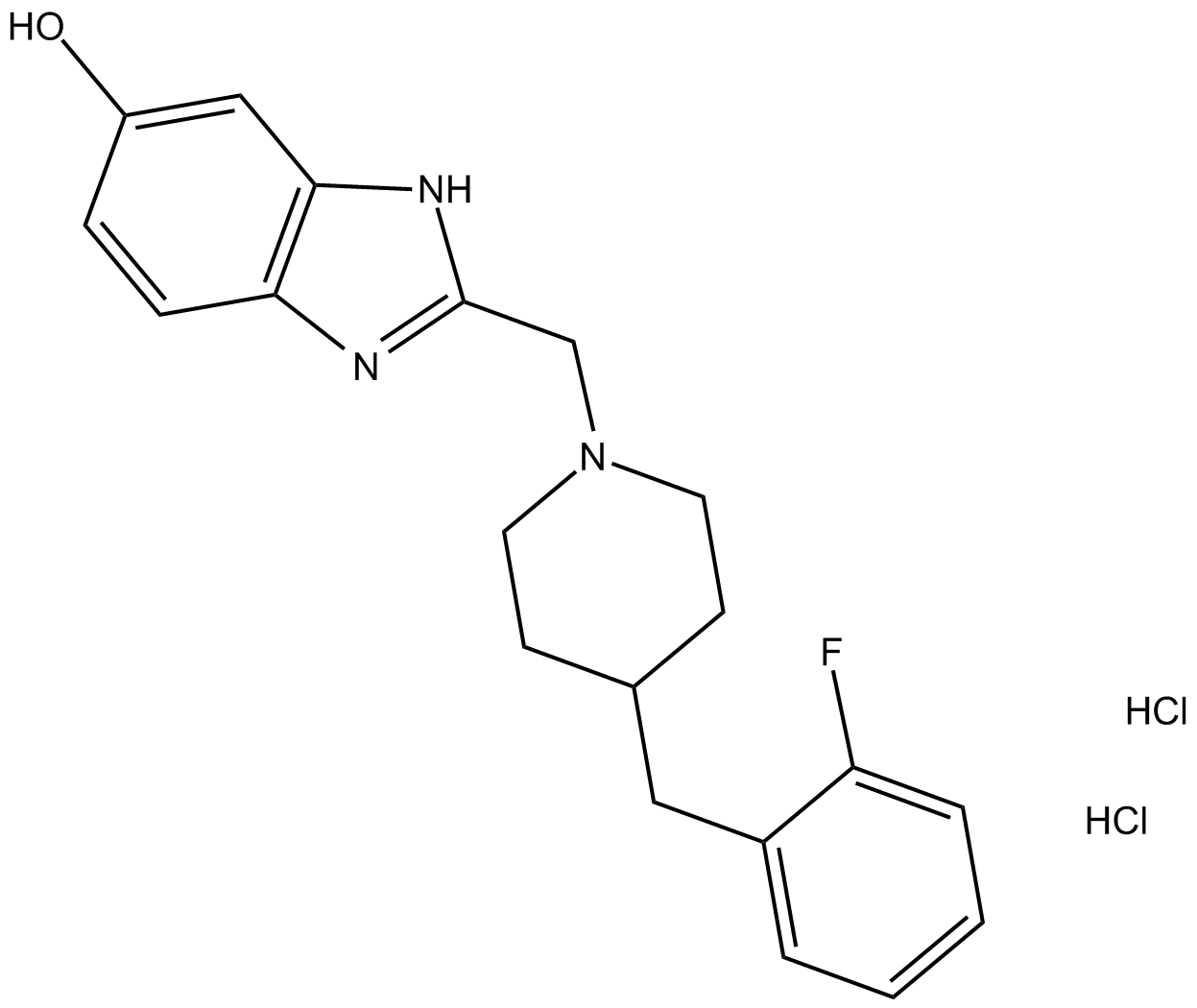
TCN 237 dihydrochloride
Le dichlorhydrate de TCN 237 est un antagoniste NMDA puissant et sélectif pour NR2B avec un Ki de 0,85 nM ; La CI50 de l'influx NR2B Ca2+ est de 9,7 nM ; aucune activité sur NR2A, NR2C, NR2D, canal hERG et récepteur α1-adrénergique.
-
GC33341
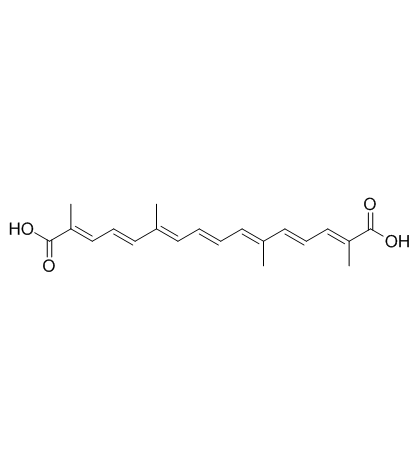
Transcrocetin (trans-Crocetin)
La transcrocétine (trans-Crocetin) (trans-Crocetin), extraite du safran (Crocus sativus L.), agit comme un antagoniste des récepteurs NMDA avec une haute affinité. La transcrocétine (trans-crocétine) (trans-crocétine) est capable de traverser la barrière hémato-encéphalique et d'atteindre le système nerveux central (SNC).
-
GC37821
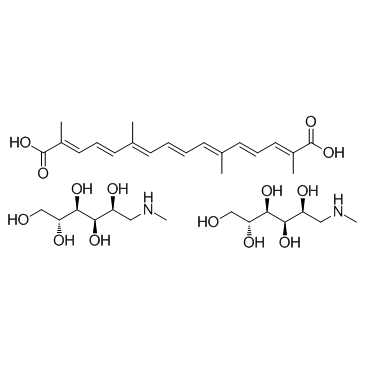
Transcrocetin meglumine salt
Le sel de méglumine de transcrocétine, extrait du safran (Crocus sativus L.), agit comme un antagoniste des récepteurs NMDA avec une haute affinité.
-
GC32447
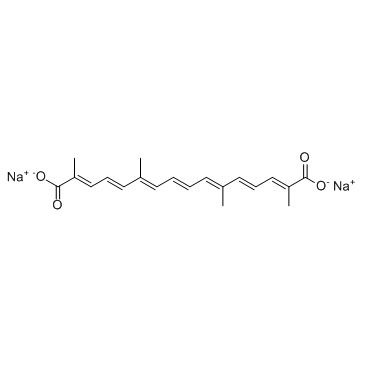
Transcrocetinate disodium (Disodium trans-crocetinate)
Le transcrocétinate disodique (Disodium trans-crocetinate), extrait du safran (Crocus sativus L.), agit comme un antagoniste des récepteurs NMDA avec une haute affinité.
-
GC30839
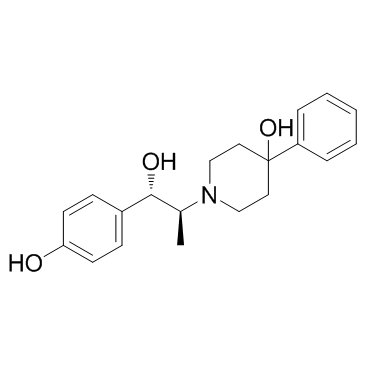
Traxoprodil
Le traxoprodil (CP101,606) est un antagoniste puissant et sélectif du NMDA et protège les neurones de l'hippocampe avec une IC50 de 10 nM.
-
GC33685
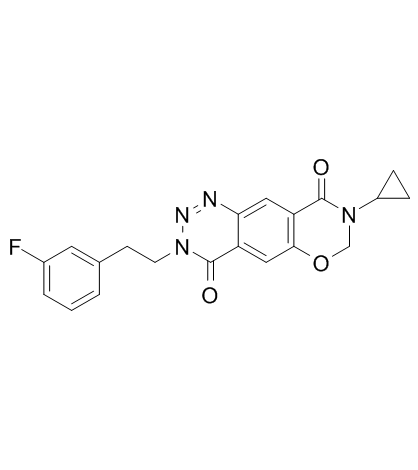
Tulrampator (CX-1632)
Tulrampator (CX-1632) (CX-1632) est un AMPAR positif biodisponible par voie orale (modulateur allostérique du récepteur AMPA).
-
GC38446
UBP 302
An antagonist of GluR5 subunit-containing kainate receptors
-
GC70084
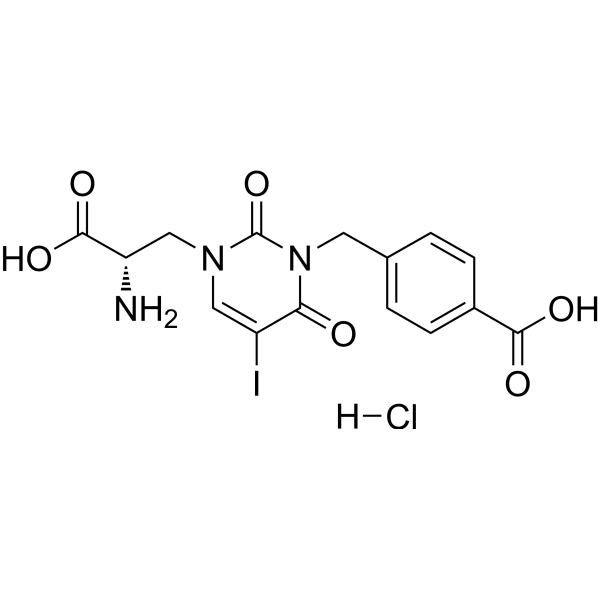
UBP301 hydrochloride
UBP301 hydrochloride est un antagoniste sélectif efficace des récepteurs de la cétone (kainate receptor), avec des valeurs d'IC50 et de KD respectives de 164 μM et 5,94 μM. UBP301 hydrochloride présente une sélectivité pour les récepteurs de la cétone rouge supérieure à celle des récepteurs AMPA, jusqu'à 30 fois plus élevée. UBP301 hydrochloride est un dérivé du Willardiine.
-
GC31153
UK-240455
UK-240455 est un antagoniste puissant et sélectif du site N-méthyl D-aspartate (NMDA) glycine.
-
GC67922
Zelquistinel

-
GC16300
ZK 200775
ZK 200775 (ZK200775) est un antagoniste AMPA/kainate hautement sélectif avec peu d'activité contre le NMDA ; ont des valeurs Ki de 3,2 nM, 100 nM et 8,5 μM contre le quisqualate, le kainate et le NMDA, respectivement.
-
GC30818
ZL006
ZL006 est un puissant inhibiteur de l'interaction nNOS/PSD-95 et inhibe la synthèse de NO médiée par les récepteurs NMDA.
-
GC30457
Zonampanel (YM 872)
Zonampanel (YM 872) (YM 872) est un antagoniste sélectif du sous-type de récepteur du glutamate, récepteur de l'acide α-amino-3-hydroxy-5-méthylisoxazole-4-propionique (AMPA).