Histone Deacetylation
Products for Histone Deacetylation
- Cat.No. Nombre del producto Información
-
GC46628
4-Chloro-6,7-bis(2-methoxyethoxy)quinazoline
A building block and synthetic intermediate
-
GC46736
7-Bromoheptanoic Acid
A building block
-
GC48986
9-hydroxy Stearic Acid
A hydroxy fatty acid
-
GC48382
Ac-QPKK(Ac)-AMC
A fluorogenic substrate for SIRT1, SIRT2, and SIRT3

-
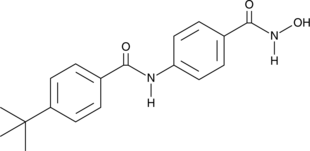
GC48775
AES-350
AES-350 es un inhibidor de HDAC6 potente y activo por vÍa oral con una IC50 y una Ki de 0,0244 μM y 0,035 μM, respectivamente. AES-350 también es contra HDAC3, HDAC8 en un ensayo de actividad enzimÁtica con valores IC50 de 0,187 μM y 0,245 μM, respectivamente. AES-350 desencadena la apoptosis en las células de AML a través de la inhibiciÓn de HDAC y se puede utilizar para la investigaciÓn de la leucemia mieloide aguda (AML).
-
GC49393
all-trans-13,14-Dihydroretinol
A metabolite of all-trans retinoic acid

-
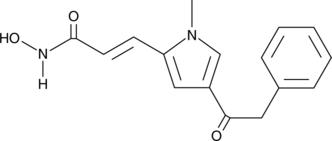
GC40674
APHA Compound 8
A class I and II HDAC inhibitor
-
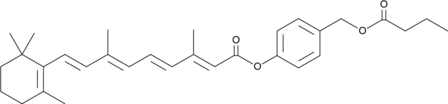
GC46892
ATRA-BA Hybrid
A prodrug form of all-trans retinoic acid and butyric acid
-
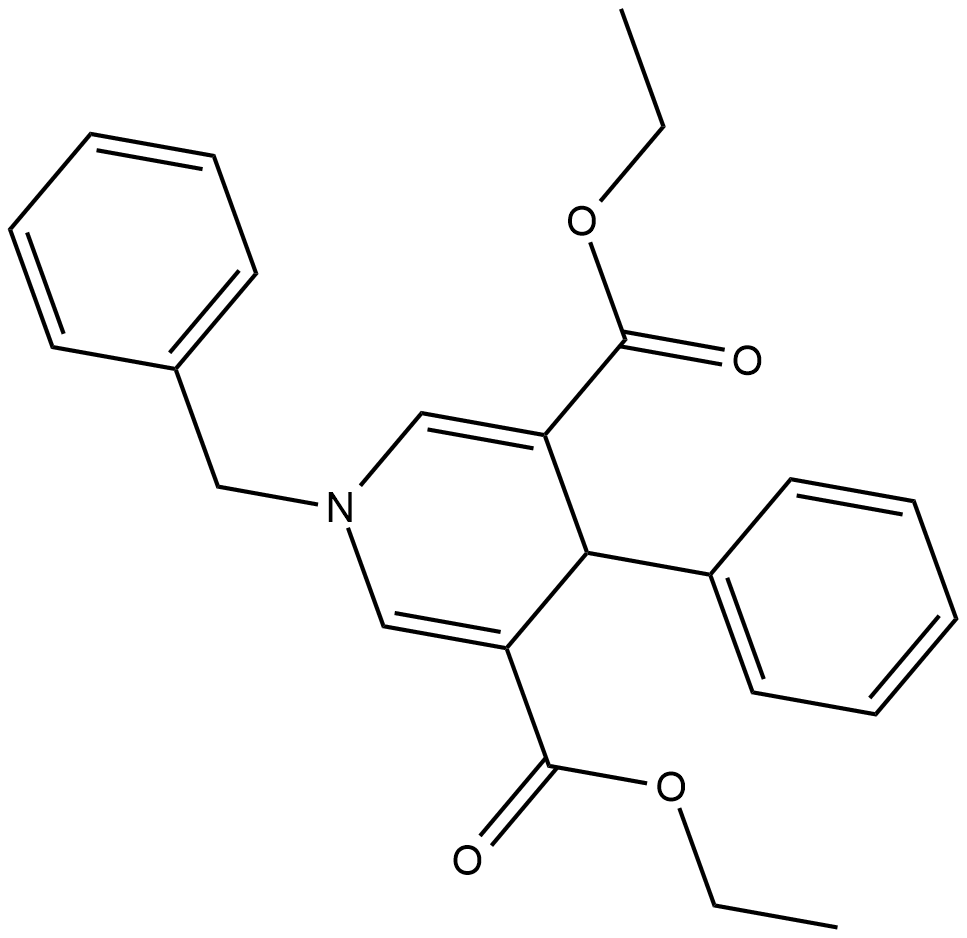
GC18408
BML-278
BML-278 is an activator of sirtuin 1 (SIRT1) that has an EC150 value (effective concentration able to increase the enzyme by 150%) of 1 uM.
-
GC11641
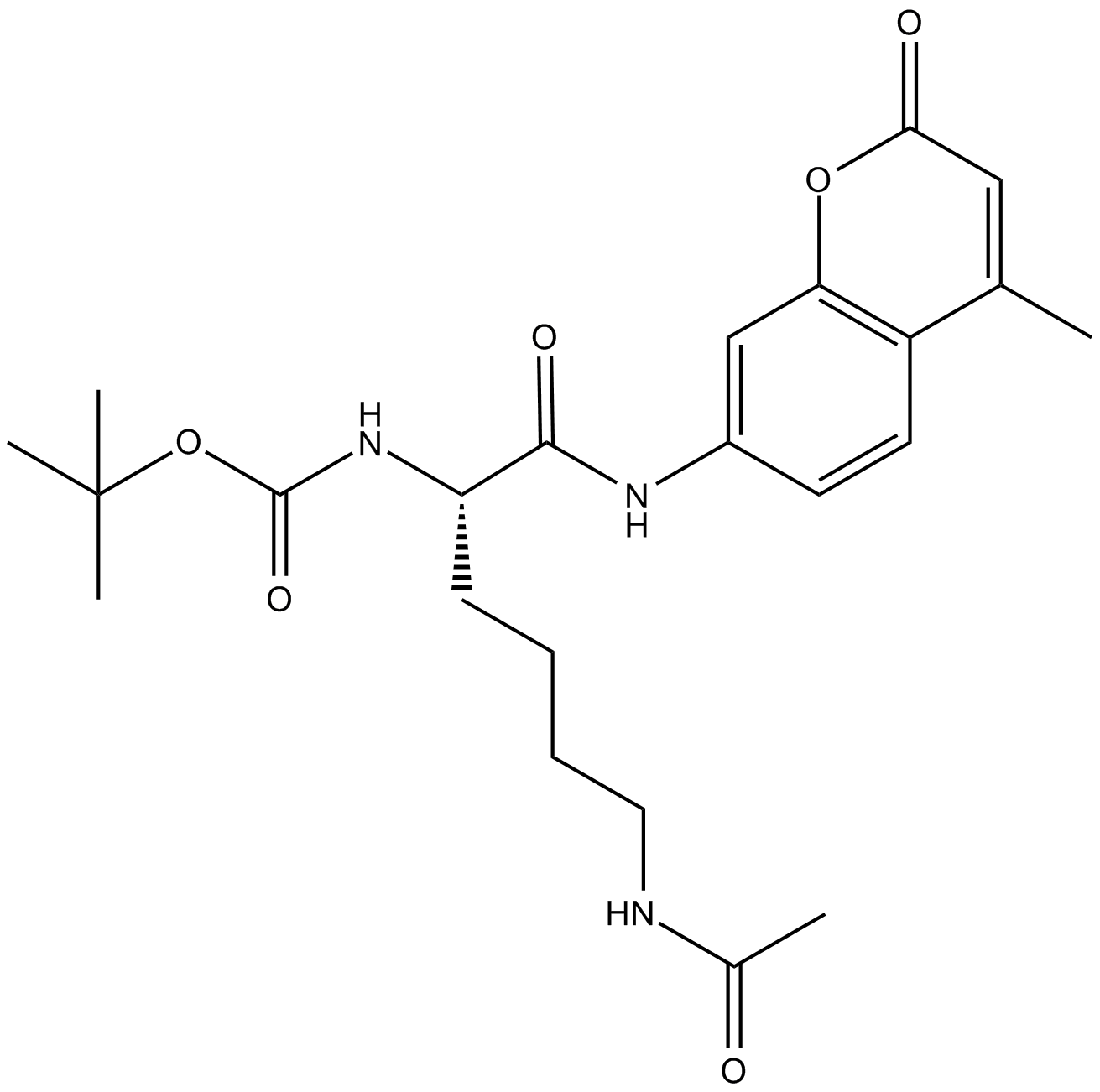
Boc-Lys(Ac)-AMC
GPCR G2A/GPR132 agonist
-
GC48620
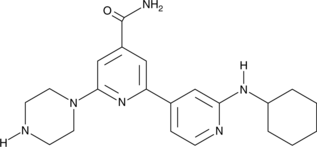
BPKDi
BPKDi es un potente inhibidor de bipiridil PKD con IC50 de 1 nM, 9 nM y 1 nM para PKD1, PKD2 y PKD3, respectivamente.
-
GC40923
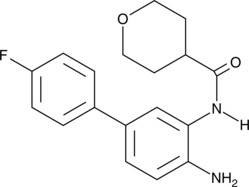
BRD4884
BRD4884 es un potente inhibidor de HDAC con valores IC50 de 29 nM, 62 nM y 1,09 μM para HDAC1, 2 y 3, respectivamente.
-
GC46104
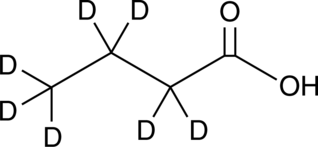
Butyric Acid-d7
An internal standard for the quantification of sodium butyrate
-
GC43147
CAY10398
CAY10398 is an inhibitor of histone deacetylase (HDAC1) with an IC50 value of 10 μM.
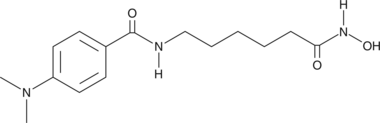
-
GC41601
CAY10591
CAY10591 es un potente activador de Sirt1 y suprime TNF-α de manera dependiente de la dosis.
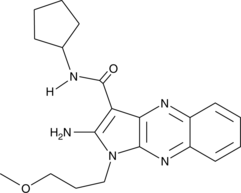
-
GC43201
CAY10722
CAY10722 is an inhibitor of sirtuin 3 (SIRT3), a class III HDAC (71% inhibition at 200 μM).
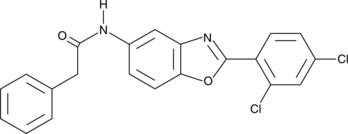
-
GC49029
CAY17c
An inhibitor of BRD4 and class I and class IIb HDACs
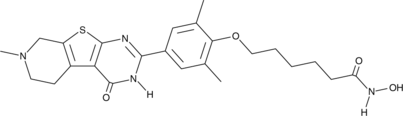
-
GC43318
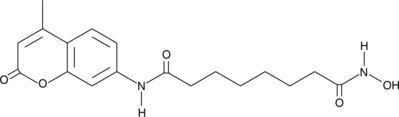
coumarin-SAHA
Coumarin-SAHA es una sonda fluorescente para determinar las afinidades de uniÓn (kd) y las tasas de disociaciÓn (koff) de los complejos inhibidores de HDAC8.
-
GC52368
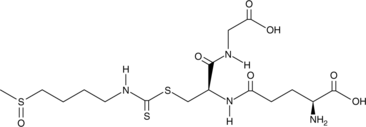
DL-Sulforaphane Glutathione
A metabolite of sulforaphane
-
GC43806
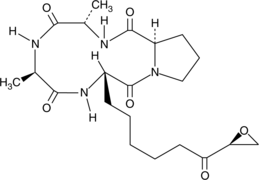
HC Toxin
La toxina HC, un tetrapéptido cíclico, es un potente inhibidor de HDAC con una IC50 de 30 nM. La Toxina HC induce la apoptosis de las células tumorales y tiene efectos anticancerígenos.
-
GC41495
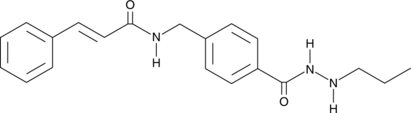
HDAC3 Inhibitor
El inhibidor de HDAC3 (compuesto 5) es un inhibidor de HDAC3 potente y selectivo, con una IC50 de 5,96 nM.
-
GC49693
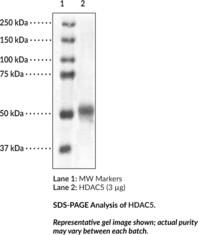
HDAC5 (human, recombinant)
Active, pure human recombinant enzyme
-
GC47579
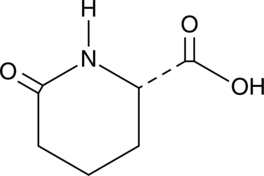
L-Pyrohomoglutamic Acid
An amino acid building block
-
GC52188
MDL 800

-
GC48805
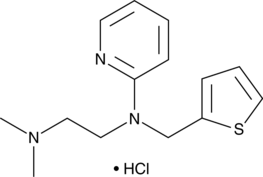
Methapyrilene (hydrochloride)
El clorhidrato de metapirileno (tenilpiramina) es un antihistamínico receptor H1 activo por vía oral y un agente anticolinérgico de la clase química de la piridina.
-
GC49270
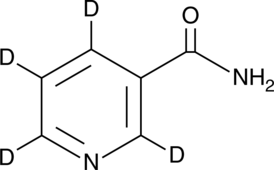
Nicotinamide-d4
La nicotinamida-d4 (niacinamida-d4) es la nicotinamida marcada con deuterio.
-
GC52166
NN-390
NN-390 es un inhibidor potente y selectivo de HDAC6, con una IC50 de 9,8 nM. NN-390 penetra la barrera hematoencefÁlica (BBB). NN-390 muestra potencial de estudio en metÁstasis del Grupo 3 MB (meduloblastoma).

-
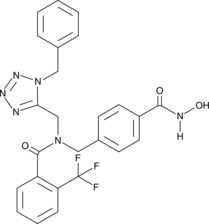
GC48460
NR-160
An inhibitor of HDAC6
-
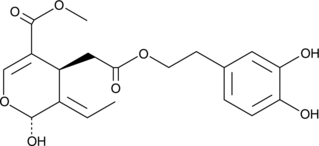
GC49815
Oleuropein aglycone
A polyphenol with diverse biological activities
-
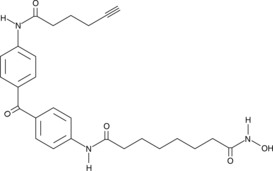
GC44865
SAHA-BPyne
Suberoylanilide hydroxamic acid (SAHA) is a class I and class II histone deacetylase (HDAC) inhibitor that binds directly to the catalytic site of the enzyme thereby blocking substrate access.
-
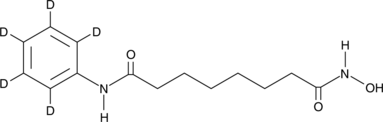
GC44866
SAHA-d5
SAHA-d5 (SAHA-d5) es el Vorinostat marcado con deuterio.
-
GC48084
Sodium 4-Phenylbutyrate-d11
El fenilbutirato-d11 (sodio) es 4-fenilbutirato de sodio marcado con deuterio. El 4-fenilbutirato de sodio (4-PBA de sodio) es un inhibidor de la HDAC y el estrés del retÍculo endoplÁsmico (ER), que se utiliza en la investigaciÓn del cÁncer y las infecciones.
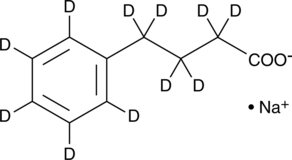
-
GC46226
SS-208
SS-208 es un inhibidor selectivo de HDAC6, con una IC50 de 12 nM. SS-208 posee actividad antitumoral en melanoma.
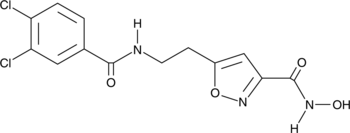
-
GC18915
Trapoxin A
Trapoxin A is a cyclotetrapeptide histone deacetylase (HDAC) inhibitor.
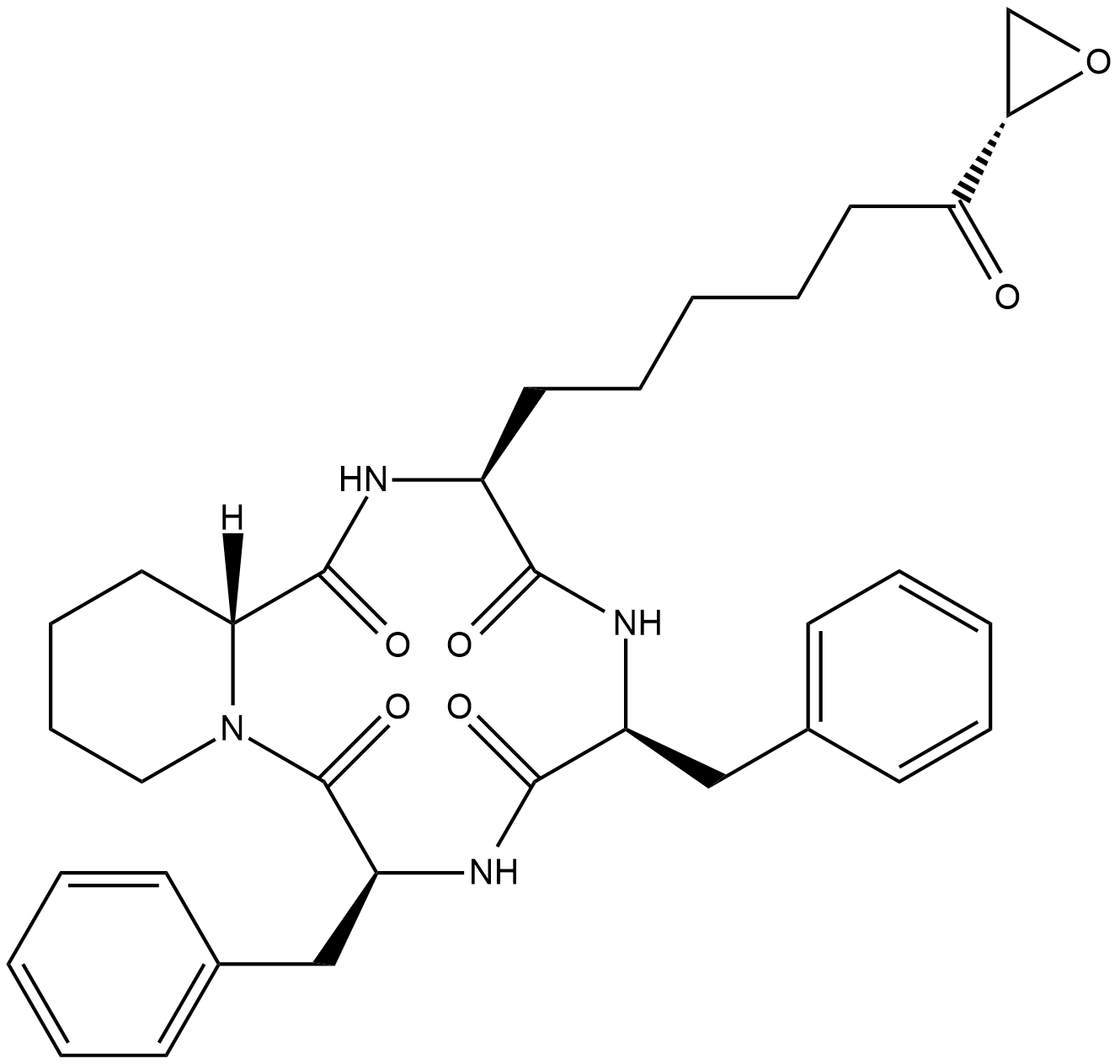
-
GC45080
Trichostatin C
Trichostatin C is a glycosylated derivative of trichostatin A, the antifungal antibiotic that reversibly inhibits histone deacetylase.
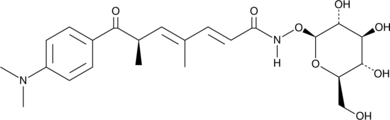
-
GC45095
Tubastatin A (trifluoroacetate salt)
Tubastatin A is a potent HDAC6 inhibitor with an IC50 value of 15 nM.
-
GC48408
TW9
A dual inhibitor of BRD4 and HDAC1
-
GC45137
Valproic Acid Acyl-D-Glucuronide
Valproic acid acyl-D-glucuronide is the major urinary metabolite of valproic acid.