PKA
PKA (protein kinase A), also known as cAMP-dependent protein kinase, has several functions in the cell, including regulation of glycogen, sugar, and lipid metabolism.
Products for PKA
- Cat.No. Nombre del producto Información
-
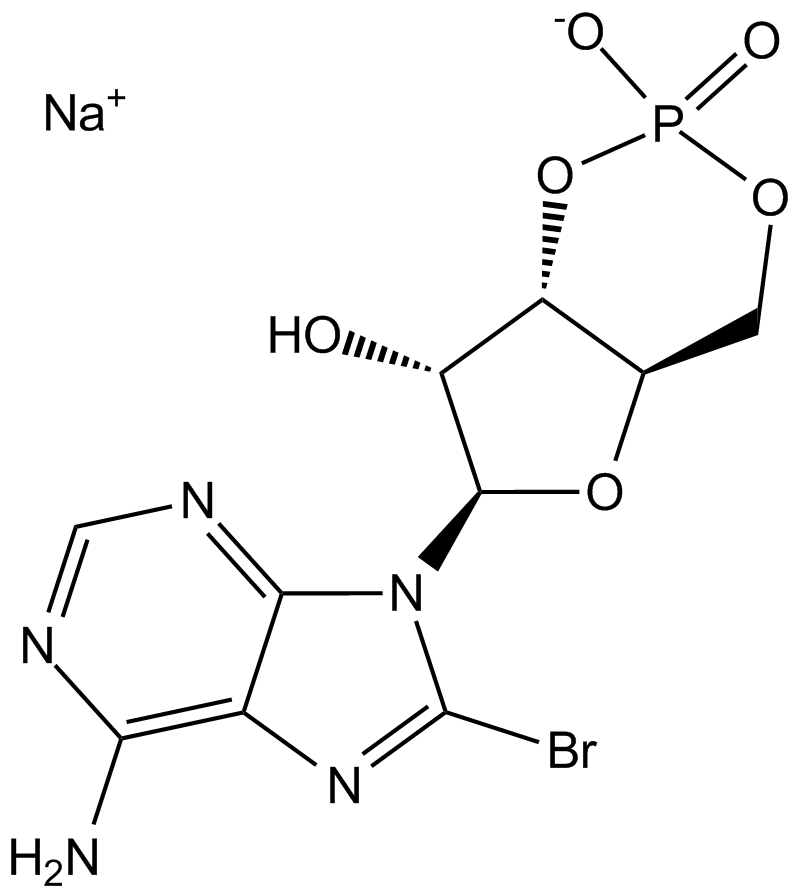
GC16929
8-Bromo-cAMP, sodium salt
Análogo de cAMP permeable a la célula que activa PKA.
-
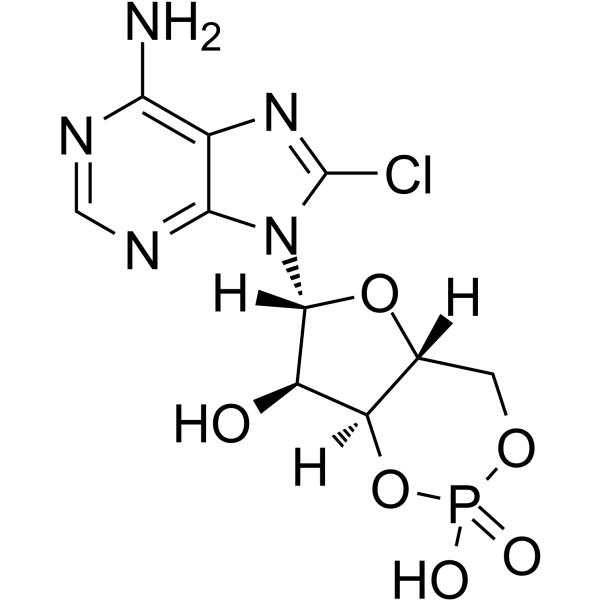
GC64460
8-Chloro-cAMP
El 8-cloro-cAMP es un anÁlogo de cAMP que induce la detenciÓn del crecimiento y modula la actividad de PKA dependiente de cAMP. El 8-cloro-cAMP tiene actividad anticancerÍgena.
-
GC15352
8-CPT-Cyclic AMP (sodium salt)
El 8-CPT-AMP cÍclico (8-CPT-cAMP) de sodio es un activador selectivo de la proteÍna quinasa dependiente de AMP cÍclico (PKA).
-
GC60037
A-3 hydrochloride
El clorhidrato de A-3 es un potente antagonista no selectivo de varias quinasas, permeable a las células, reversible y competitivo con ATP.
-
GC17712
AT13148
AT13148 es un inhibidor multi-AGC cinasa activo por vÍa oral y competitivo con ATP con IC50 de 38 nM/402 nM/50 nM, 8 nM, 3 nM y 6 nM/4 nM para Akt1/2/3, p70S6K, PKA, y ROCKI/II, respectivamente.
-
GC12297
AT7867
AT7867 es un potente inhibidor competitivo de ATP de Akt1/Akt2/Akt3 y p70S6K/PKA con IC50 de 32 nM/17 nM/47 nM y 85 nM/20 nM, respectivamente.
-
GC10918
AT7867 dihydrochloride
A potent and orally bioavailable pan-Akt inhibitor
-
GC35518
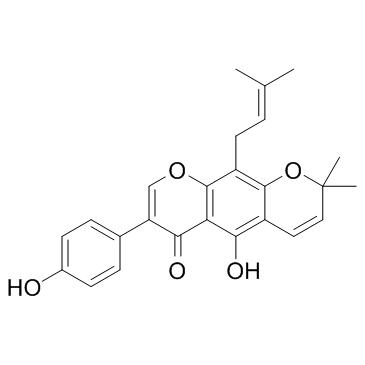
Bilobetin
Bilobetin, un componente activo de Ginkgo biloba, puede reducir los lÍpidos en sangre y mejorar los efectos de la insulina.
-
GC35565
Bucladesine calcium salt
La sal de sal de calcio de bucladesina (sal de calcio de dibutiril-cAMP; sal de calcio DC2797) es un anÁlogo de AMP cÍclico permeable a las células (cAMP) y activa selectivamente la proteÍna quinasa dependiente de cAMP (PKA) al aumentar el nivel intracelular de cAMP.
-
GC11605
C-1
C-1 es un potente inhibidor de la proteÍna quinasa, con IC50 de 4 μM, 8 μM, 12 μM y 240 μM para proteÍna quinasa dependiente de cGMP (PKG), proteÍna quinasa dependiente de cAMPPKA , proteÍna quinasa C (PKC) y MLC-quinasa, respectivamente. C-1 también se usa como inhibidor de ROCK.

-
GC10941
cAMPS-Rp, triethylammonium salt
cAMPS-Rp, sal de trietilamonio, un anÁlogo de cAMP, es un antagonista potente y competitivo de la activaciÓn inducida por cAMP de PKA I y II dependiente de cAMP (Kis de 12,5 μM y 4,5 μM, respectivamente).
-
GC19090
CCG215022
CCG215022 es un inhibidor de las quinasas del receptor acoplado a proteÍna G (GRK) con IC50 de 0,15 ± 0,07 μM, 0,38 ± 0,06 μM y 3,9 ± 1 μM para GRK2, GRK5 y GRK1, respectivamente.
-
GC15525
cGMP Dependent Kinase Inhibitor Peptide
El péptido inhibidor de la quinasa dependiente de cGMP es un inhibidor competitivo de ATP de la proteÍna quinasa dependiente de cGMP (PKG), con una Ki de 86 μM.
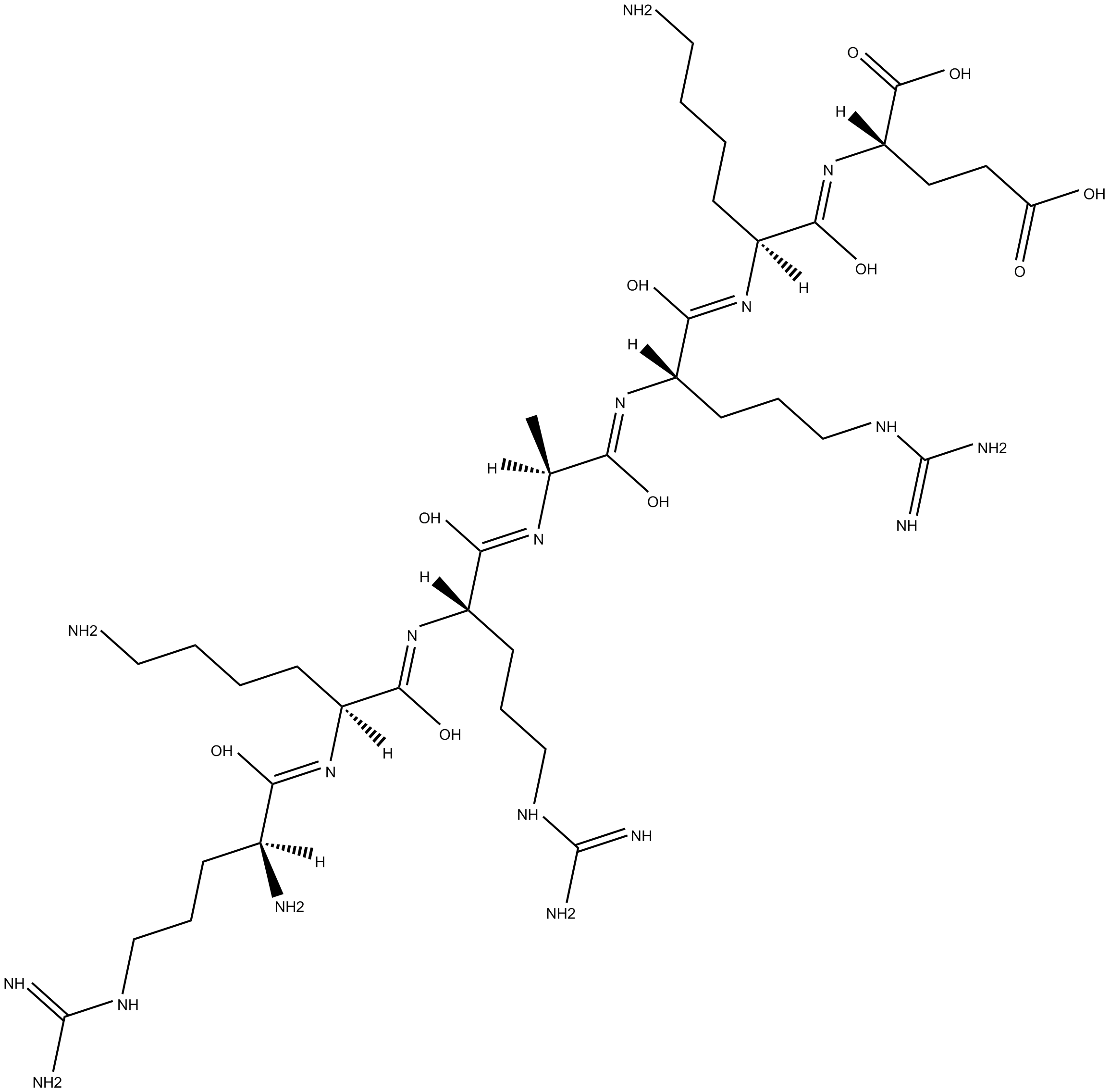
-
GC31525
CREBtide
CREBtide, un péptido sintético de 13 aminoÁcidos, se ha informado como sustrato de PKA.
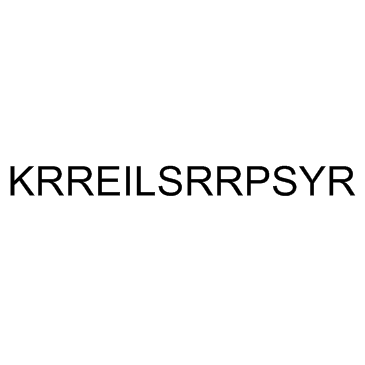
-
GC50181
CW 008
PKA signaling activator; promotes osteogenesis from hMSCs

-
GN10336
Daphnetin
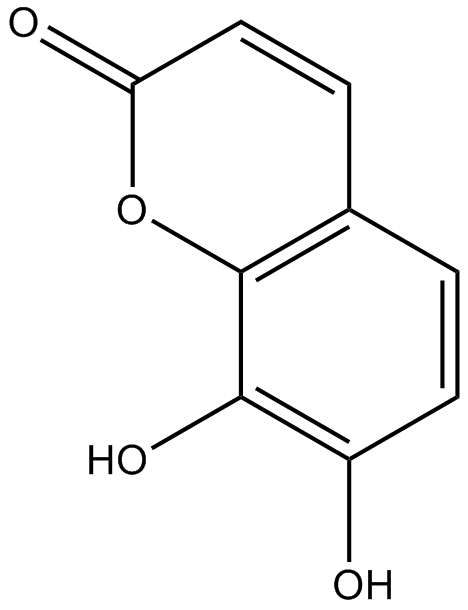
-
GC12824
Dibutyryl-cAMP, sodium salt
La sal sÓdica de dibutiril-cAMP (sal sÓdica de dibutiril-cAMP) es un anÁlogo del AMP cÍclico estabilizado (cAMP) y un activador selectivo de la PKA.
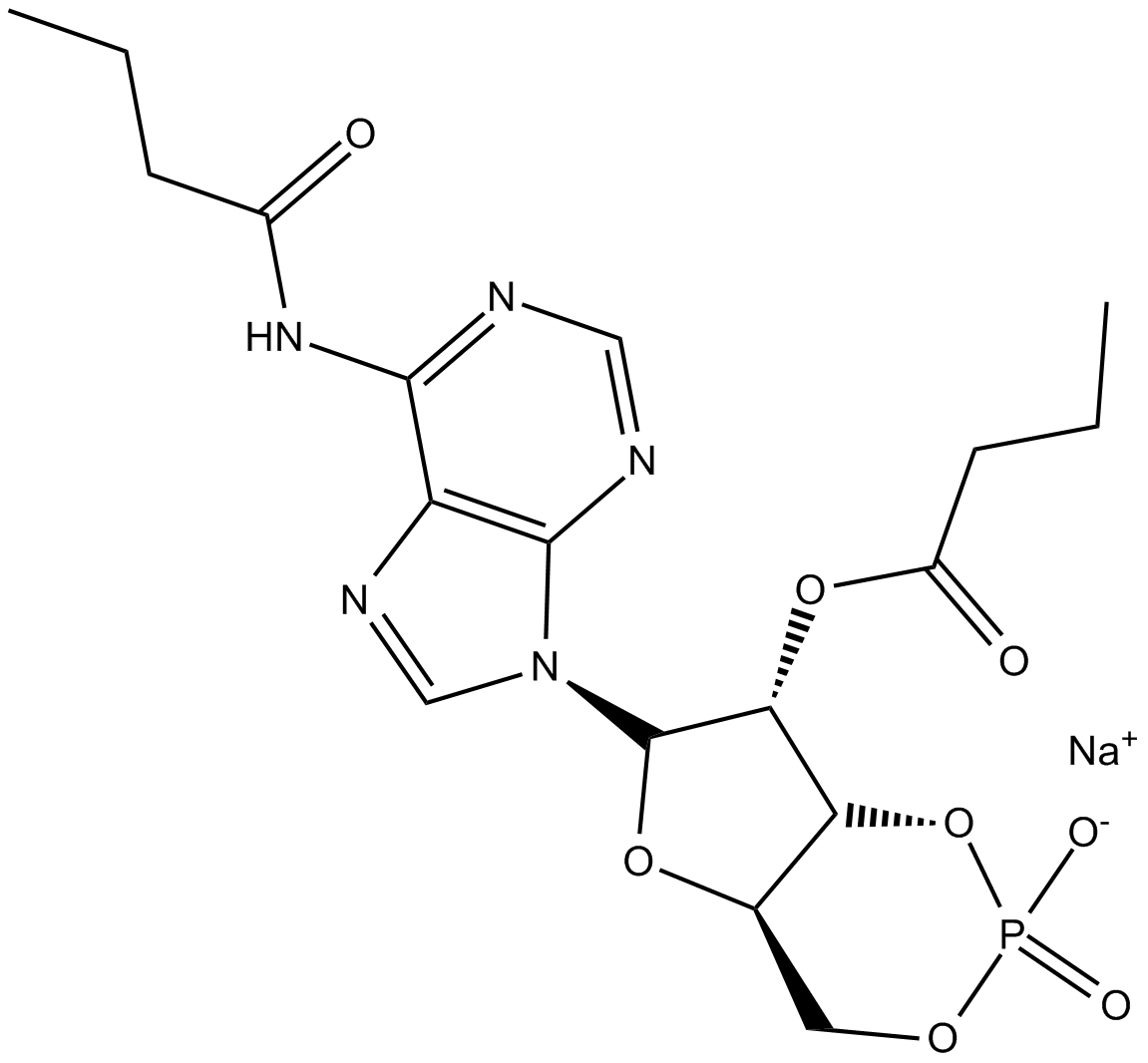
-
GC13869
Fasudil
Fasudil (HA-1077; AT877) es un inhibidor inespecÍfico de RhoA/ROCK y también tiene un efecto inhibitorio sobre las proteÍnas quinasas, con una Ki de 0,33 μM para ROCK1, IC50 de 0,158 μM y 4,58 μM, 12,30 μM, 1,650 μM para ROCK2 y PKA , PKC, PKG, respectivamente. Fasudil es también un potente antagonista y vasodilatador de los canales de Ca2+.

-
GC14289
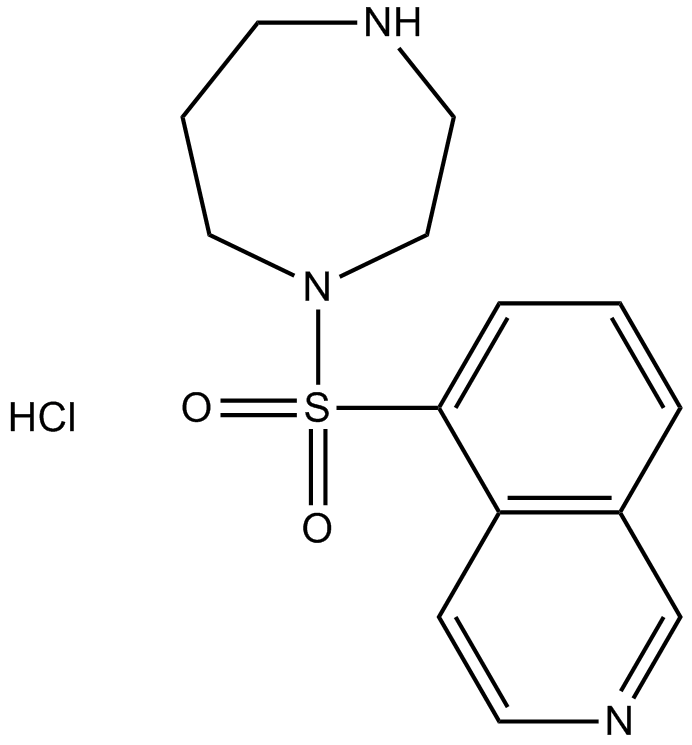
Fasudil (HA-1077) HCl
Fasudil (HA-1077; AT877) El clorhidrato es un inhibidor rhoa/roca inespecÍfico y también tiene un efecto inhibitorio en las proteÍnas quinasas, con un KI de 0.33 μ m para rock115&&&&7777 que offlineefficient_models_2022q2.md;M, 1.650 μM for ROCK2 and PKA, PKC, PKG, respectively.en_es_2021q4.mdFasudil (HA-1077) HCl is also a potent Ca2+ channel antagonist and vasodilator.en_es_2021q4.md
-
GC17255
Gliotoxin
An immunosuppressive mycotoxin with diverse biological effects
-
GC19396
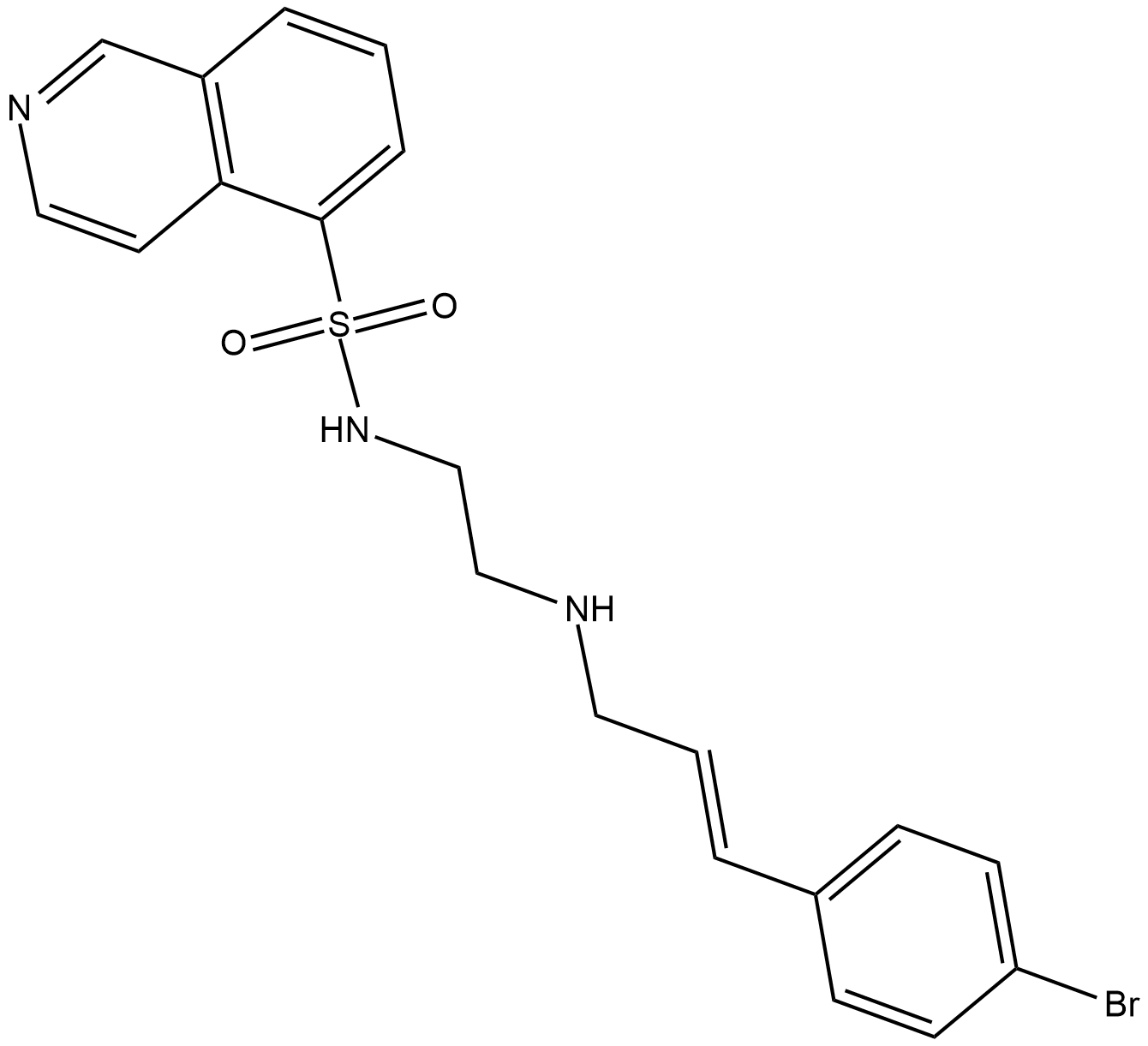
H 89
Un potente inhibidor de la proteína quinasa dependiente del AMP cíclico.
-
GC10074
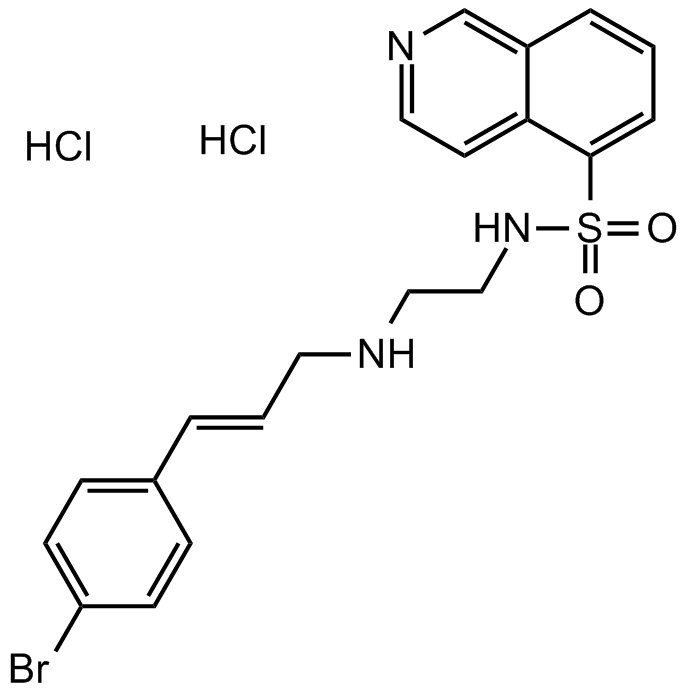
H 89 2HCl
H-89 2HCl es un inhibidor potente y selectivo de la proteína quinasa A dependiente de cAMP con un valor IC50 de 48 nM, mostrando débil inhibición de PKG, PKC, quinasa de caseína y otras quinasas.
-
GC12799
H-8 (hydrochloride)
H-8 (diclorhidrato) es un inhibidor de PKA permeable a las células, reversible y competitivo con ATP.
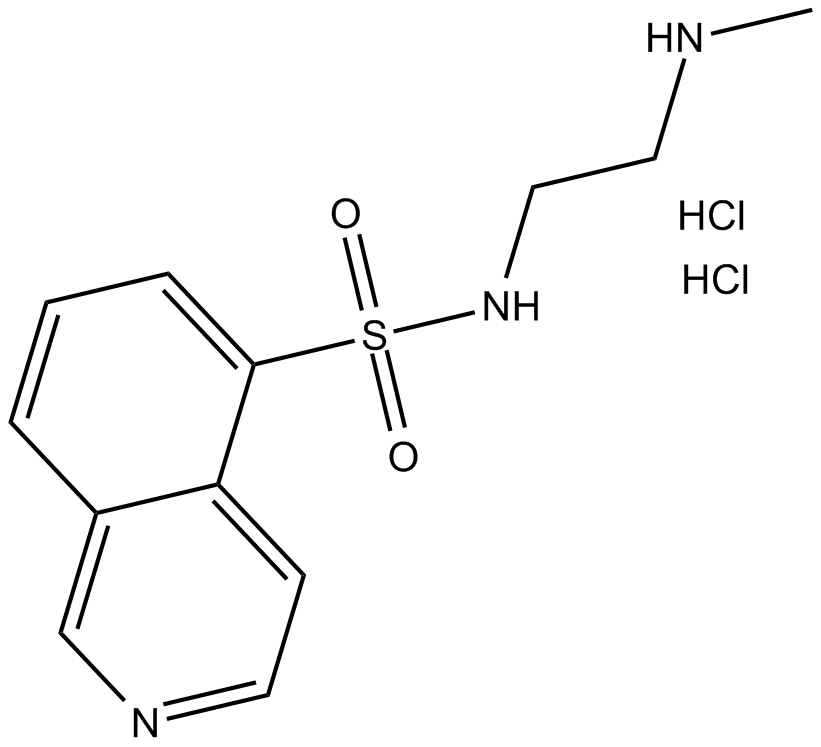
-
GC65303
Jaspamycin
La jaspamicina (7-CN-7-C-Ino) es un potente activador de la PKA, de uniÓn al sitio R (PKAR), con una EC50 de 6,5 nM y una Kd de 8 nM en Trypanosoma brucei.
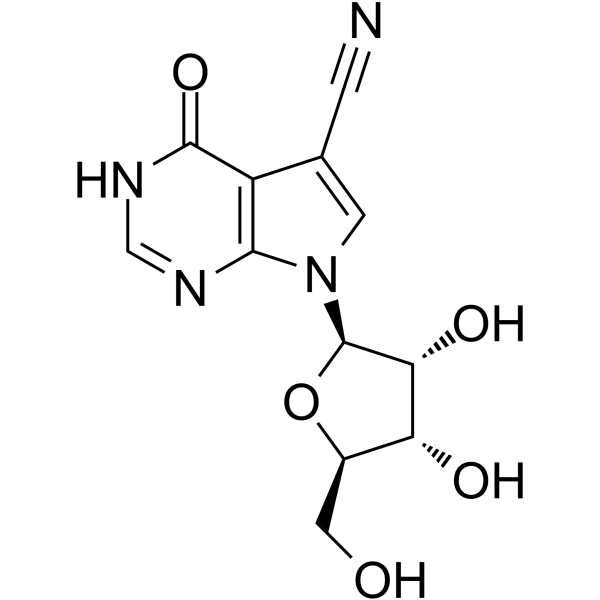
-
GC11362
K 252a
Un inhibidor de la proteína quinasa.
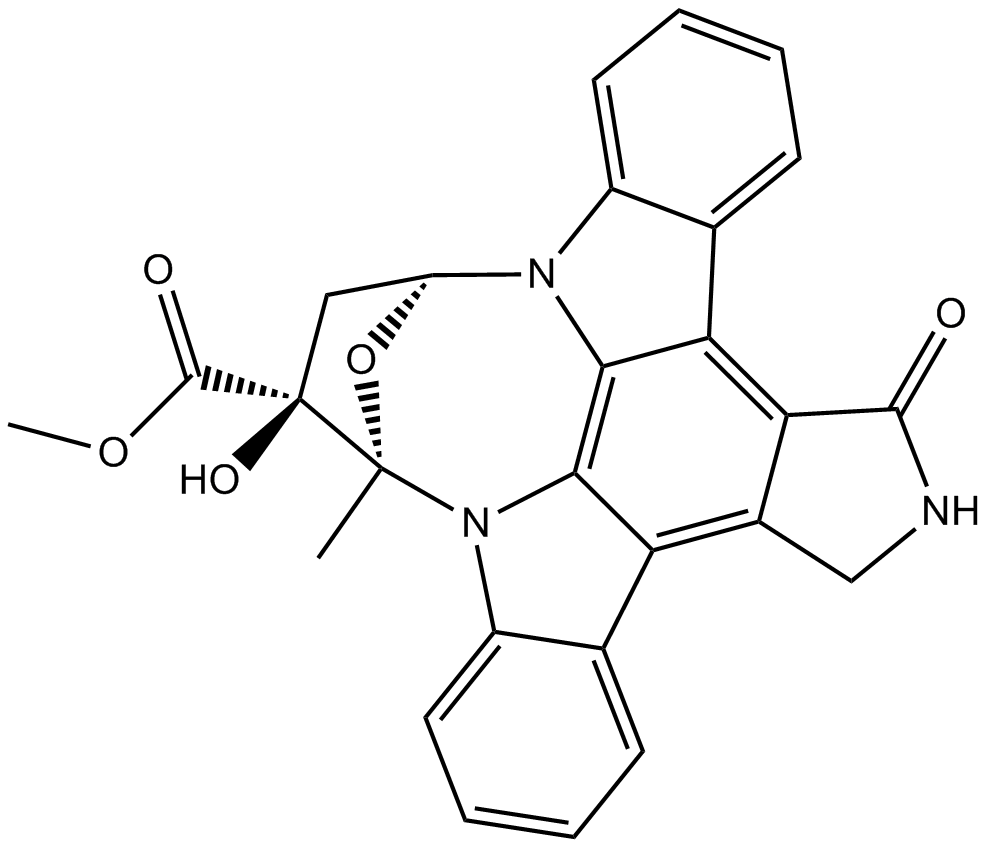
-
GC13640
Kemptide
Kemptide es un heptapéptido sintético que actÚa como sustrato especÍfico para la proteÍna quinasa dependiente de cAMP (PKA).
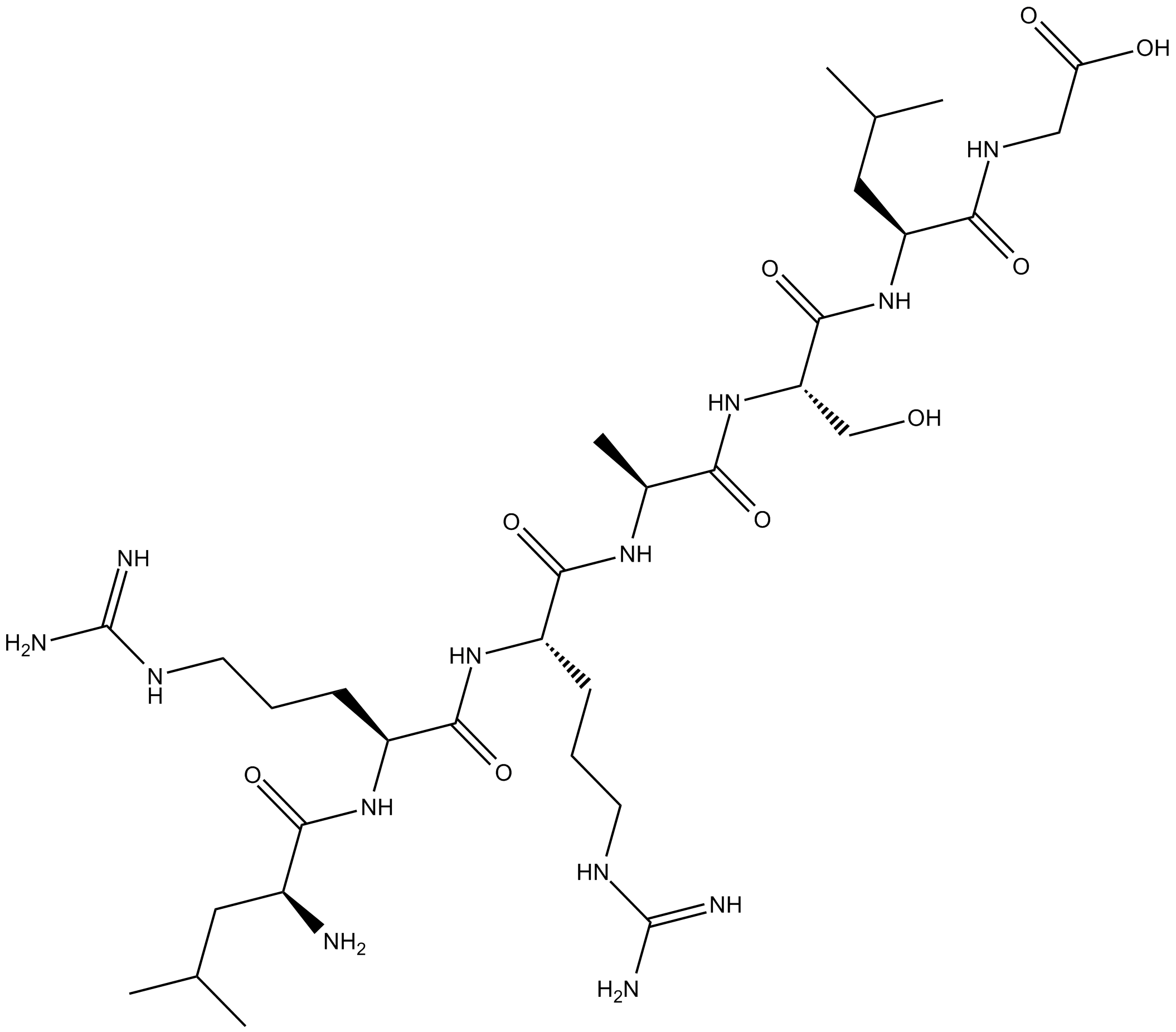
-
GC36391
Kemptide Phospho-Ser5
Kemptide (Phospho-Ser5) es un péptido aceptor de fosfato que sirve como sustrato especÍfico para la proteÍna quinasa dependiente de cAMP (PKA).

-
GC14648
KT 5720
KT 5720 es un inhibidor competitivo de ATP, permeable a las células, potente, específico, reversible de la proteína quinasa A (PKA), con una Ki de 60 nM.
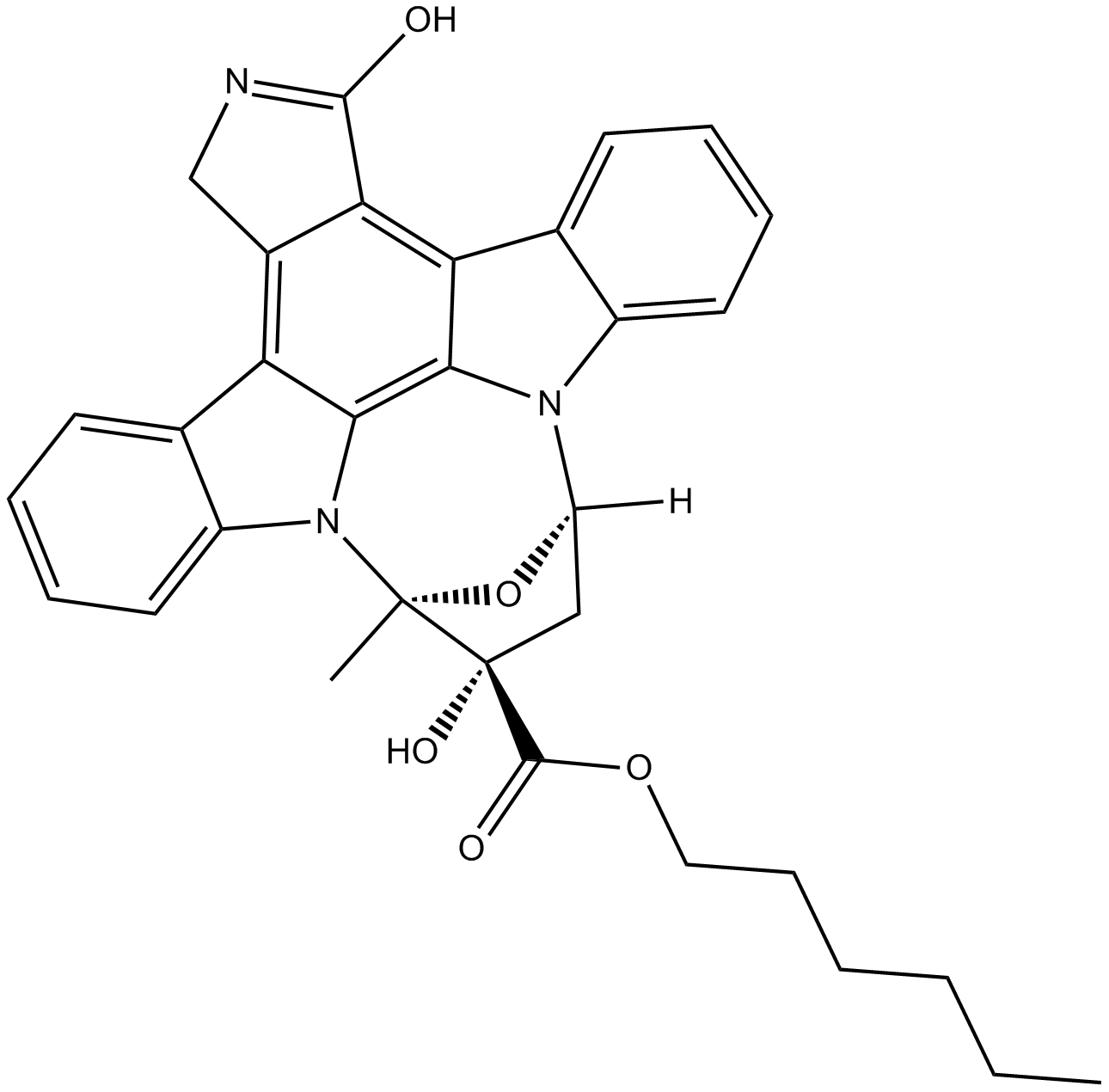
-
GC17346
KT 5823
KT 5823, un inhibidor selectivo de la proteína quinasa dependiente de cGMP (PKG) con un valor Ki de 0,23 μM, también inhibe PKA y PKC con valores Ki de 10 μM y 4 μM, respectivamente.
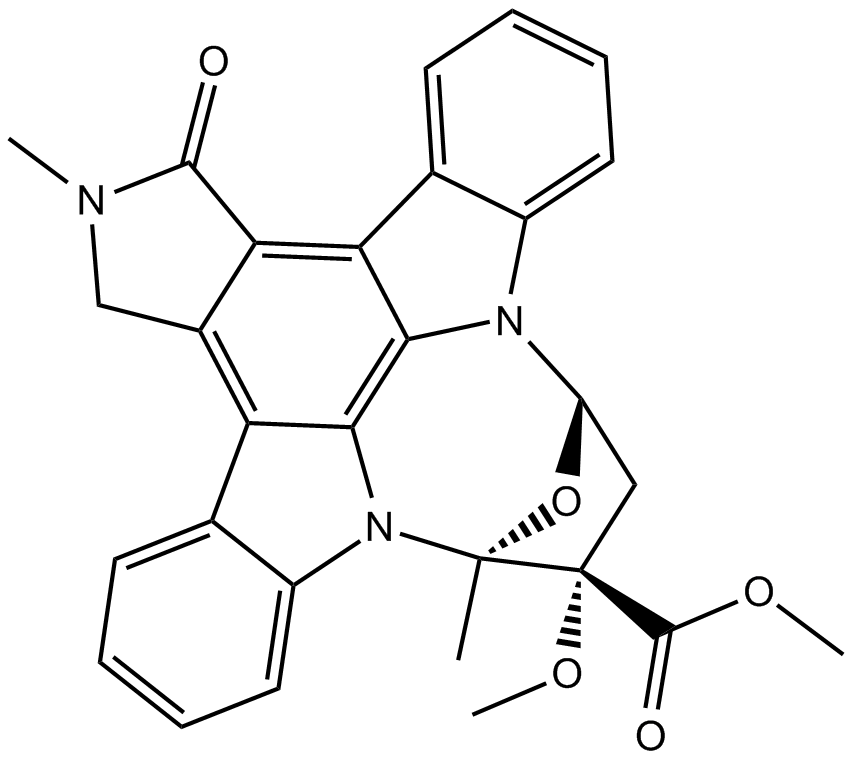
-
GC30545
Malantide
Malantide es un dodecapéptido sintético derivado del sitio fosforilado por la proteÍna quinasa dependiente de cAMP (PKA) en la subunidad β de la fosforilasa quinasa. Malantide es un sustrato altamente especÍfico para PKA con una Km de 15 μM y muestra una inhibiciÓn del inhibidor de proteÍnas (PKI) >90 % de la fosforilaciÓn del sustrato en varios extractos de tejido de rata. Malantide también es un sustrato eficiente para PKC con una Km de 16 μM.
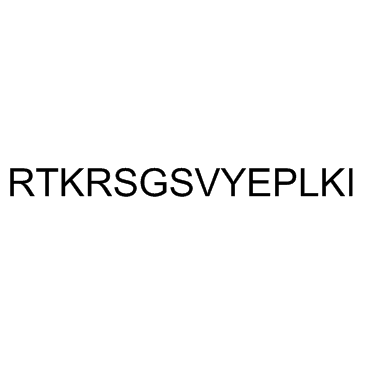
-
GC33771
Metadoxine
La metadoxina bloquea la diferenciaciÓn de los adipocitos en asociaciÓn con la inhibiciÓn de la vÍa de la proteÍna de uniÓn al elemento de respuesta de la proteÍna quinasa A-cAMP (PKA-CREB).
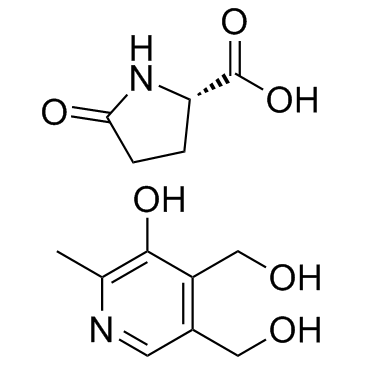
-
GC49440
ML-9
ML-9 es un inhibidor selectivo y potente de la quinasa Akt, inhibe la quinasa de cadena ligera de miosina (MLCK) y la actividad de la molécula de interacciÓn estromal 1 (STIM1). ML-9 inhibe la actividad de MLCK, PKA y PKC con valores de Ki de 4, 32 y 54 μM, respectivamente. ML-9 induce la autofagia al estimular la formaciÓn de autofagosomas e inhibir su degradaciÓn.
-
GC44653
PKA Inhibitor (5-24)
El inhibidor de PKA (5-24) es un inhibidor peptÍdico sintético, competitivo y potente de la PKA (proteÍna quinasa dependiente de AMPc), con una Ki de 2,3 nM.

-
GP10075
PKA inhibitor fragment (6-22) amide
A synthetic peptide inhibitor of PKA
-
GC61512
PKA Inhibitor Fragment (6-22) amide TFA
Fragmento inhibidor de PKA (6-22) amida TFA es un inhibidor de la proteÍna quinasa A (PKA) dependiente de cAMP, con una Ki de 2,8 nM.

-
GC49265
PKI (14-22) amide (myristoylated) (trifluoroacetate salt)
A PKA inhibitor
-
GC69715
PKI (14-24)amide TFA
PKI (14-24)amide TFA es un inhibidor efectivo de PKA. En el citosol celular, PKI (14-24)amide inhibe fuertemente la actividad de la proteína quinasa dependiente del AMP cíclico.
-
GC12321
PKI 14-22 amide, myristoylated
PKI 14-22 amida, miristoilada es un potente inhibidor de PKA dependiente de cAMP. PKI 14-22 amida, miristoilada reduce la respuesta fagocítica mediada por IgG y también inhibe la adhesión de neutrófilos.
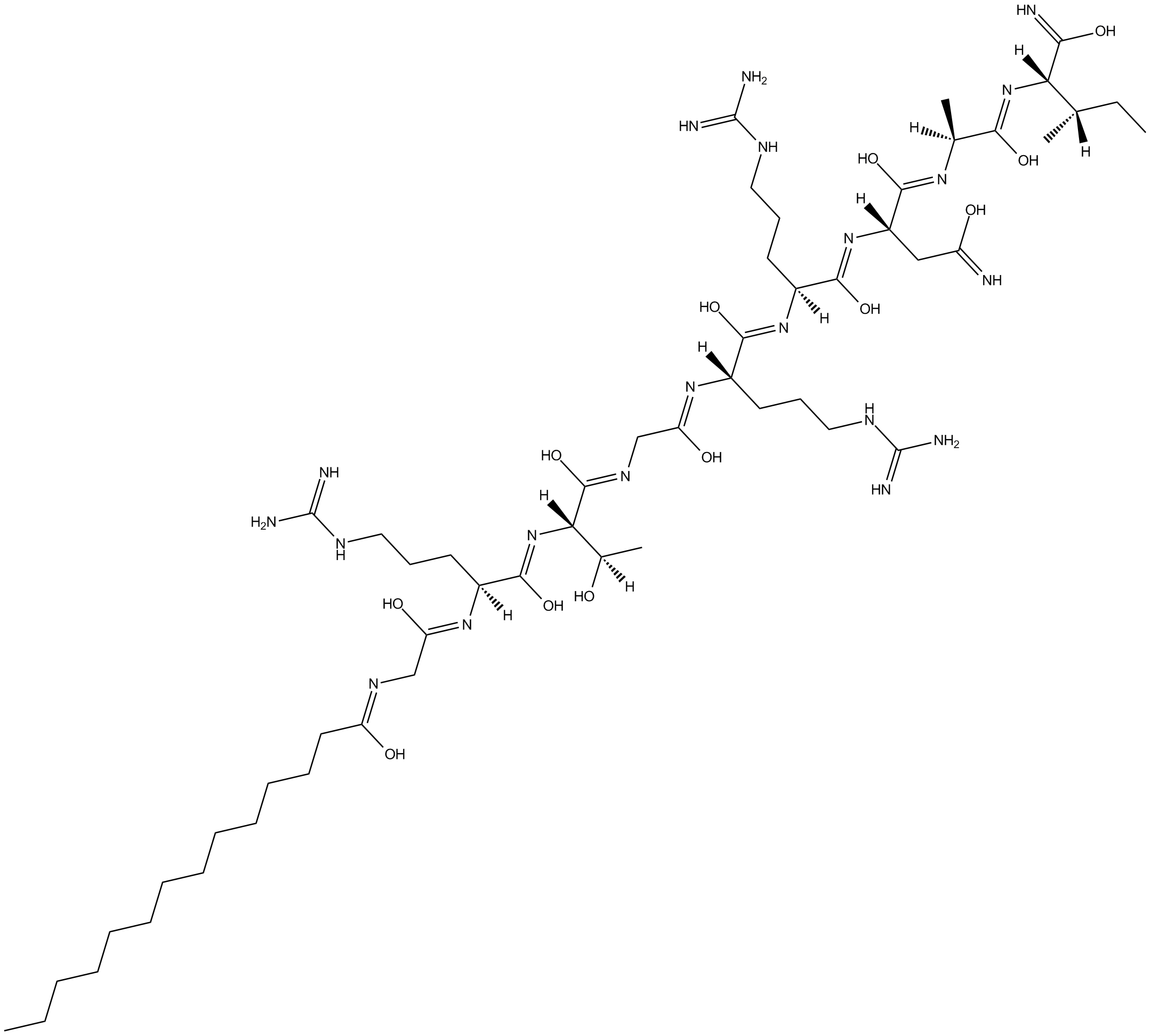
-
GC61849
PKI 14-22 amide,myristoylated TFA
PKI 14-22 amida, miristoilado TFA es un potente inhibidor de PKA dependiente de cAMP. PKI 14-22 amida, miristoilado TFA reduce la respuesta fagocÍtica mediada por IgG y también inhibe la adhesiÓn de neutrÓfilos.

-
GC50502
RI-STAD 2
AKAP disruptor; selectively binds PKA-RI with high affinity and blocks its interaction with AKAP; cell permeable
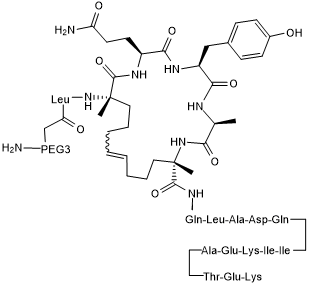
-
GC44853
Rp-8-CPT-Cyclic AMP (sodium salt)
Rp-8-CPT-Cyclic AMP (sal de sodio), un anÁlogo de cAMP, es un antagonista potente y competitivo de la activaciÓn inducida por cAMP de PKA I y II dependiente de cAMP.
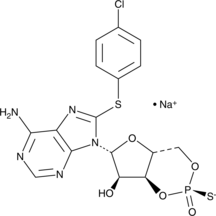
-
GC63176
Rp-cAMPS sodium salt
La sal sÓdica de Rp-cAMPS, un anÁlogo de cAMP, es una activaciÓn potente y competitiva inducida por cAMP de antagonistas de PKA I y II dependientes de cAMP (Kis de 12,5 μM y 4,5 μM, respectivamente).
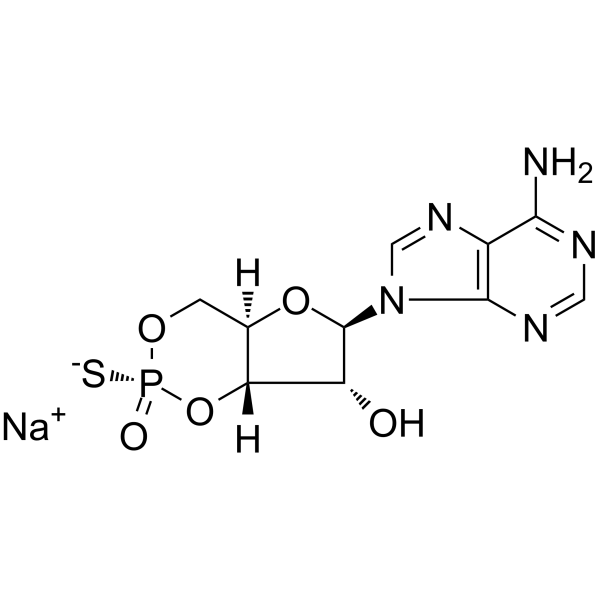
-
GC10428
Sp-5,6-dichloro-cBIMPS (sodium salt)
PKA activator
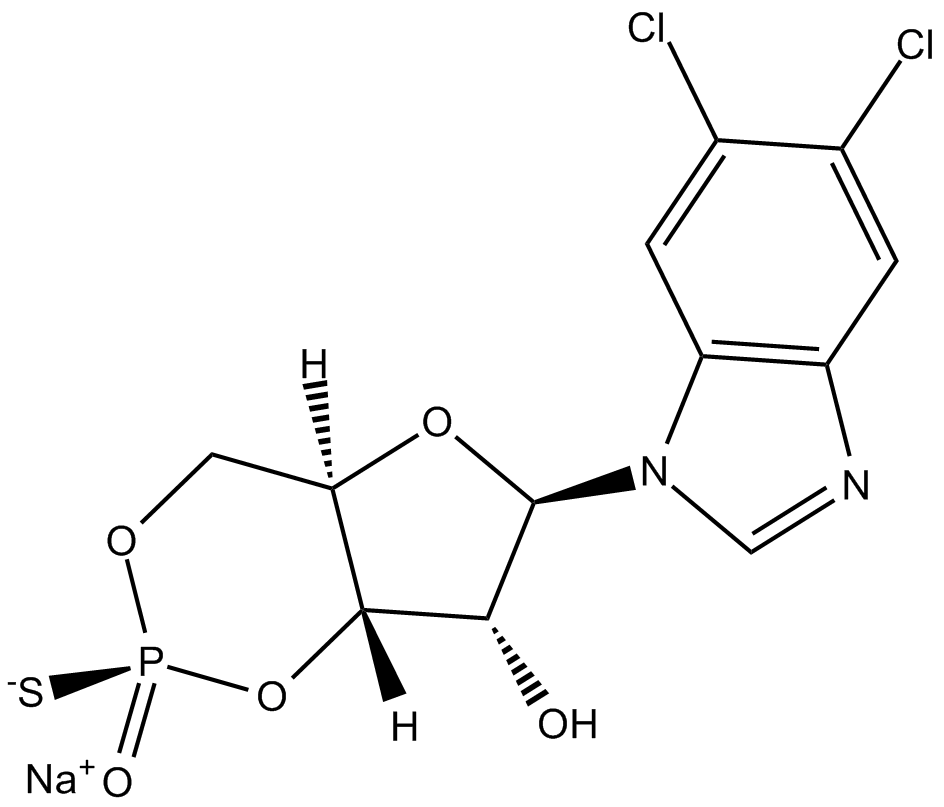
-
GC61747
Sp-cAMPS
Sp-cAMPS, un anÁlogo de cAMP, es un potente activador de PKA I y PKA II dependientes de cAMP.
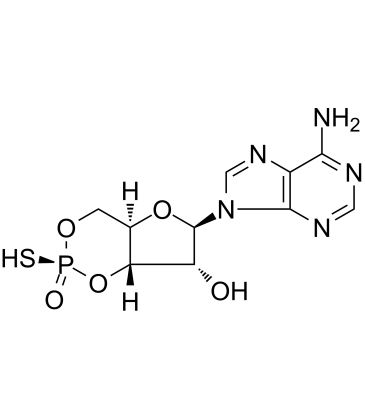
-
GC50406
st-Ht31
Inhibits PKA/AKAP interactions; cell permeable
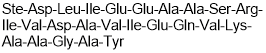
-
GC50407
st-Ht31 P
Negative control for st-Ht31
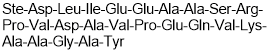
-
GC50501
STAD 2
STAD 2 es un disruptor potente y selectivo de PKA-RII, con una Kd de 6,2 nM.
-
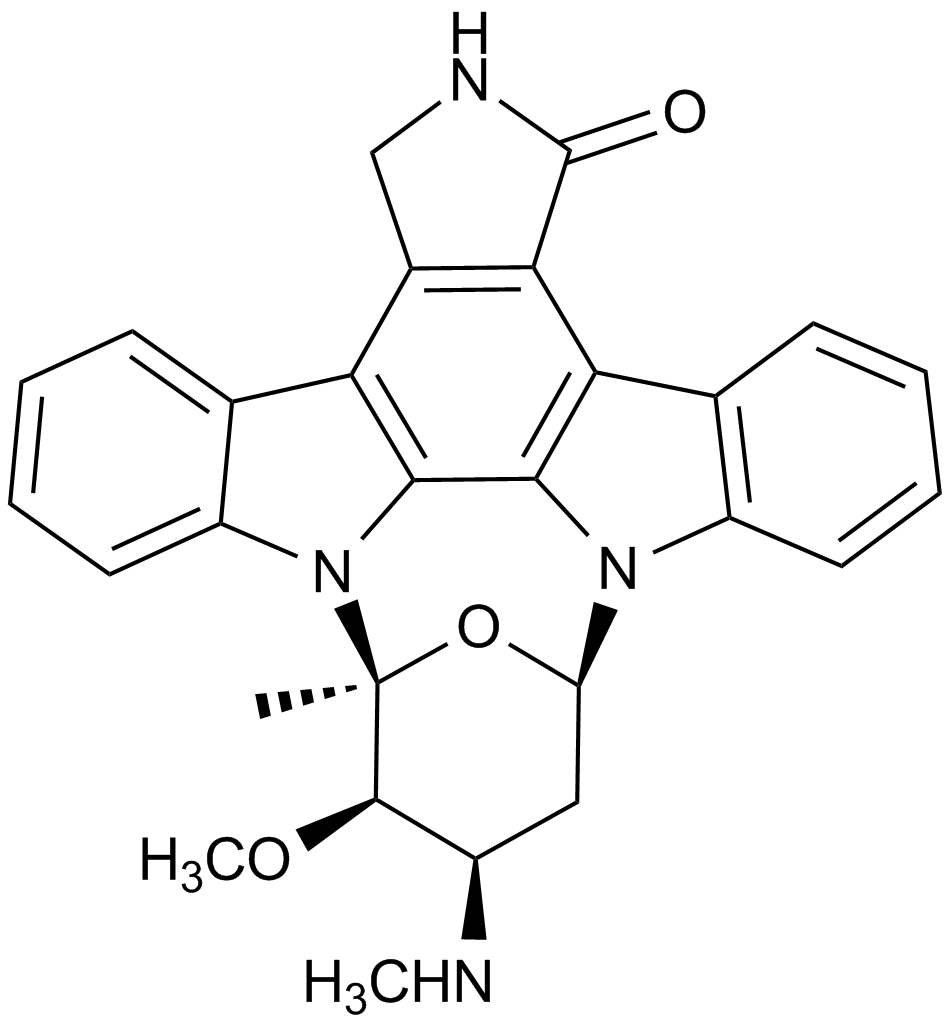
GC15299
Staurosporine(CGP 41251)
A potent inhibitor of protein kinase C
-
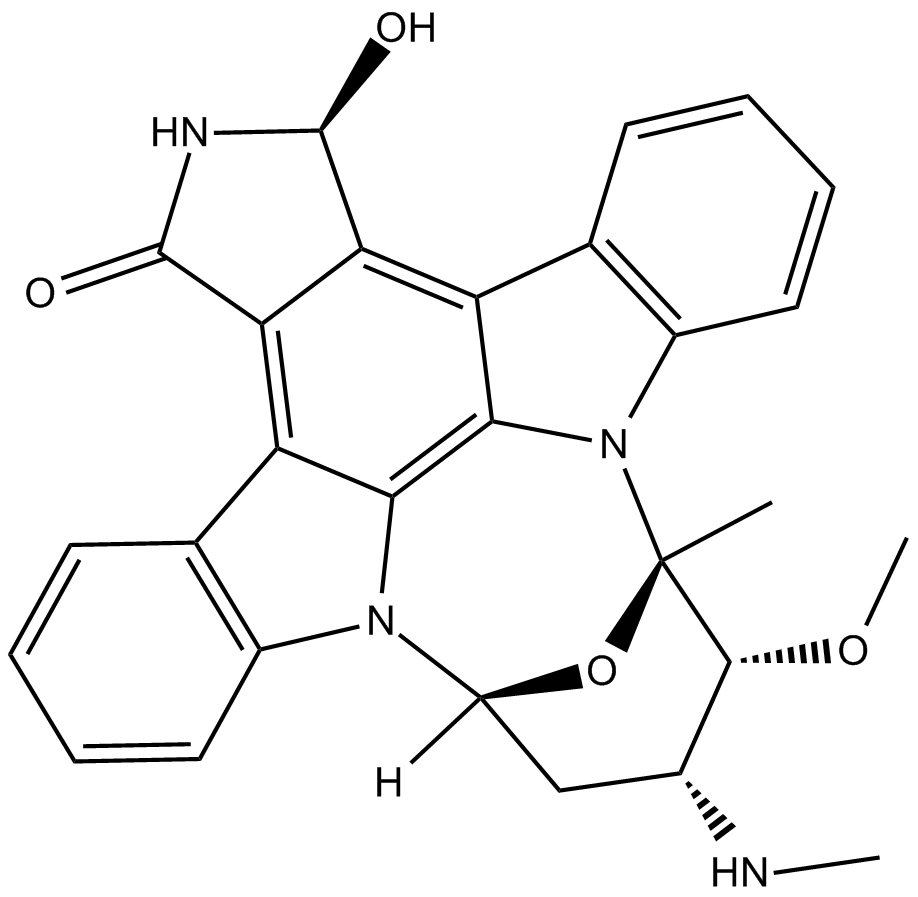
GC16678
UCN-02
UCN-02 (7-epi-Hydroxystaurosporine) es un inhibidor selectivo de la proteÍna quinasa C (PKC) producido por la cepa N-12 de Streptomyces, con IC50 de 62 nM y 250 nM para PKC y proteÍna quinasa A (PKA), respectivamente. UCN-02 (7-epi-Hydroxystaurosporine) muestra un efecto citotÓxico en el crecimiento de las células HeLa S3.
-
GC30857
Warangalone (Scandenolone)
Warangalone (Scandenolone) es un compuesto contra la malaria que puede inhibir el crecimiento de ambas cepas del parÁsito 3D7 (sensible a la cloroquina) y K1 (resistente a la cloroquina) con IC50 de 4.8 μg/mL y 3.7 μg/mL, respectivamente.