p53
The p53 tumor suppressor is a 53 kDa nuclear phosphoprotein of 393 amino acids that is encoded by the TP53 gene (20 kb with 11 exons and 10 introns) and characterized by the presence of several structural and functional domains, including a N-terminus, a central core domain, a C-terminal region, a strongly basic carboxyl-terminal regulatory domain, a nuclear localization signal sequence and three nuclear export signal sequence. The p53 is considered as a major “guardian of genome” for its activities in a wide range of cellular events, including cell-cycle regulation, induction of apoptosis, gene amplification, DNA recombination, chromosomal segregation and cellular senescence.
Products for p53
- Cat.No. Nombre del producto Información
-
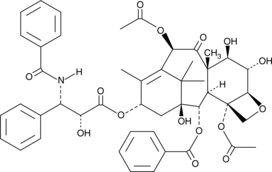
GC42586
6α-hydroxy Paclitaxel
6α-hidroxi Paclitaxel es un metabolito primario de Paclitaxel. 6α-hidroxi Paclitaxel conserva un efecto dependiente del tiempo sobre los polipéptidos transportadores de aniones orgÁnicos 1B1/SLCO1B1 (OATP1B1) con una potencia de inhibiciÓn similar a Paclitaxel, mientras que ya no mostrÓ inhibiciÓn dependiente del tiempo de OATP1B3. 6α-hidroxi Paclitaxel se puede utilizar para la investigaciÓn del cÁncer.
-
GC41643
9(Z),11(E),13(E)-Octadecatrienoic Acid
9(Z),11(E),13(E)-Octadecatrienoic Acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.

-
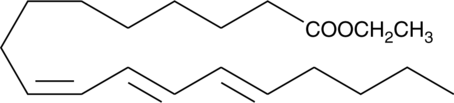
GC40785
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
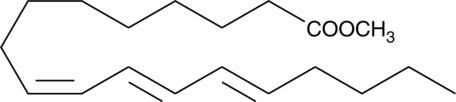
GC40710
9(Z),11(E),13(E)-Octadecatrienoic Acid methyl ester
9Z,11E,13E-octadecatrienoic acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
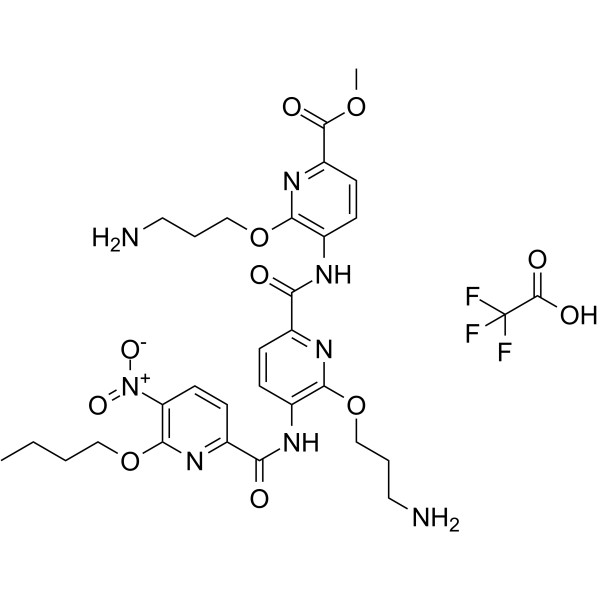
GC65880
ADH-6 TFA
ADH-6 TFA es un compuesto de tripiridilamida. ADH-6 anula el autoensamblaje del subdominio de nucleaciÓn de agregaciÓn del mutante p53 DBD. ADH-6 TFA se dirige y disocia agregados de p53 mutantes en células cancerosas humanas, lo que restaura la actividad transcripcional de p53', lo que lleva a la detenciÓn del ciclo celular y la apoptosis. ADH-6 TFA tiene potencial para la investigaciÓn de enfermedades cancerosas.
-
GC33356
AM-8735
AM-8735 es un inhibidor de MDM2 potente y selectivo con una IC50 de 25 nM.
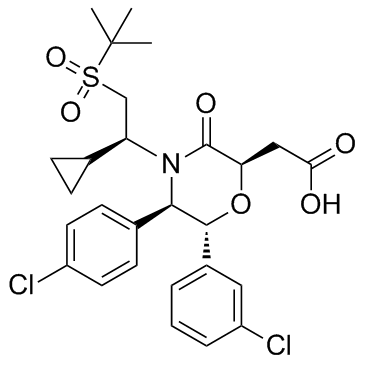
-
GC15828
AMG232
AMG232 (AMG 232) es un inhibidor potente, selectivo y disponible por vÍa oral de la interacciÓn p53-MDM2, con una IC50 de 0,6 nM. AMG232 se une a MDM2 con una Kd de 0,045 nM.
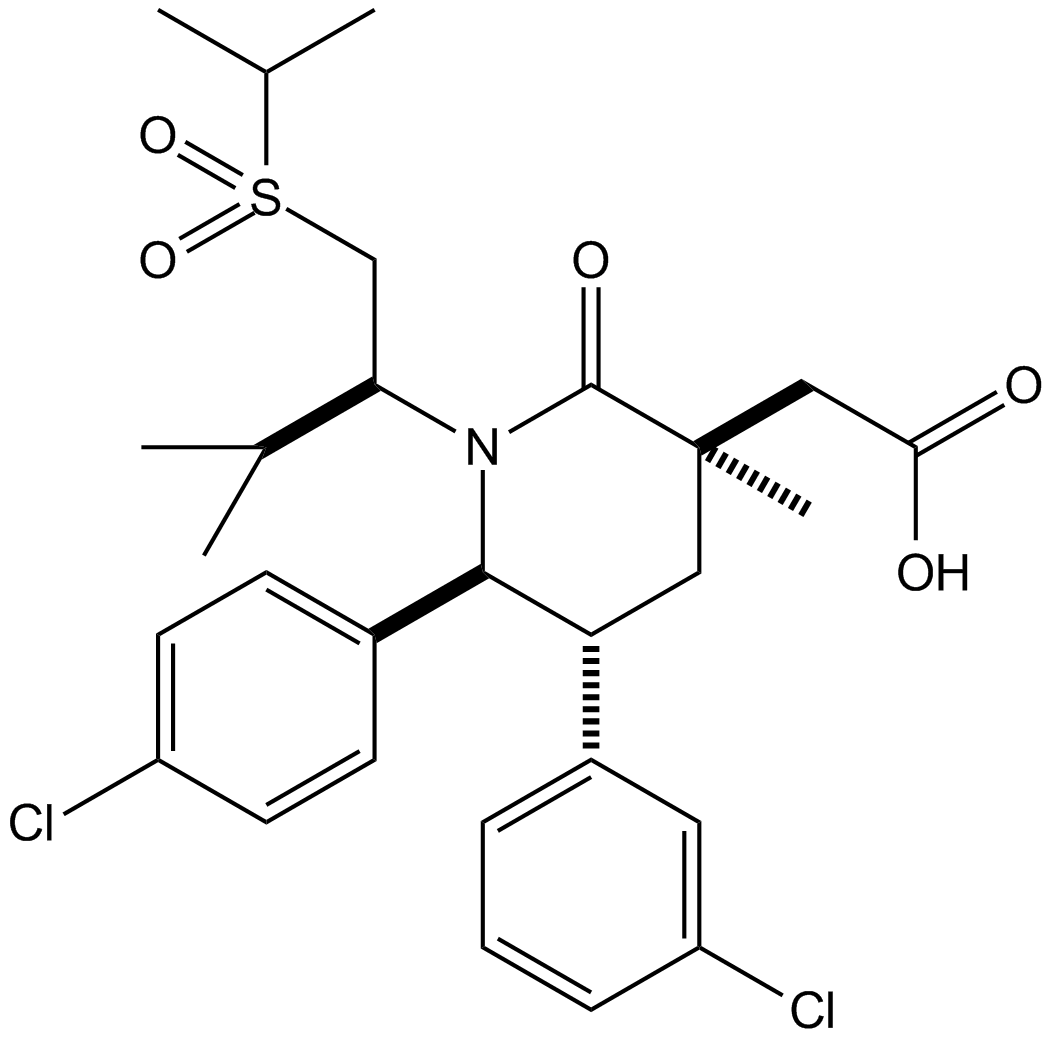
-
GC42785
Amifostine (hydrate)
La amifostina (hidrato) (WR2721 trihidrato) es un agente citoprotector de amplio espectro y un radioprotector. La amifostina (hidrato) protege selectivamente los tejidos normales del daÑo causado por la radiaciÓn y la quimioterapia. La amifostina (hidrato) es un potente factor inducible por hipoxia-α1 (HIF-α1) e inductor de p53. La amifostina (hidrato) protege las células del daÑo al eliminar los radicales libres derivados del oxÍgeno. La amifostina (hidrato) reduce la toxicidad renal y tiene acciÓn antiangiogénica.
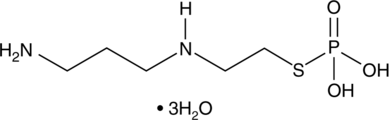
-
GC61804
Amifostine thiol
El tiol de amifostina (WR-1065) es un metabolito activo del citoprotector Amifostina. El tiol de amifostina es un agente citoprotector con capacidades radioprotectoras. El tiol de amifostina activa p53 a través de una vÍa de seÑalizaciÓn dependiente de JNK.
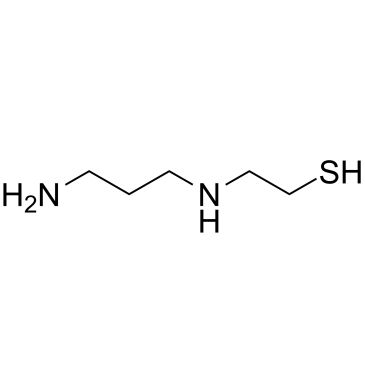
-
GC35367
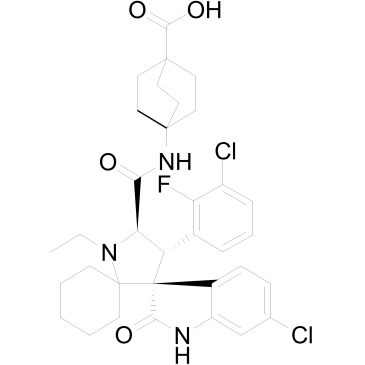
APG-115
APG-115 (APG-115) es un inhibidor de la proteÍna MDM2 activo por vÍa oral que se une a la proteÍna MDM2 con valores IC50 y Ki de 3,8 nM y 1 nM, respectivamente. APG-115 bloquea la interacciÓn de MDM2 y p53 e induce la detenciÓn del ciclo celular y la apoptosis de manera dependiente de p53.
-
GC18136
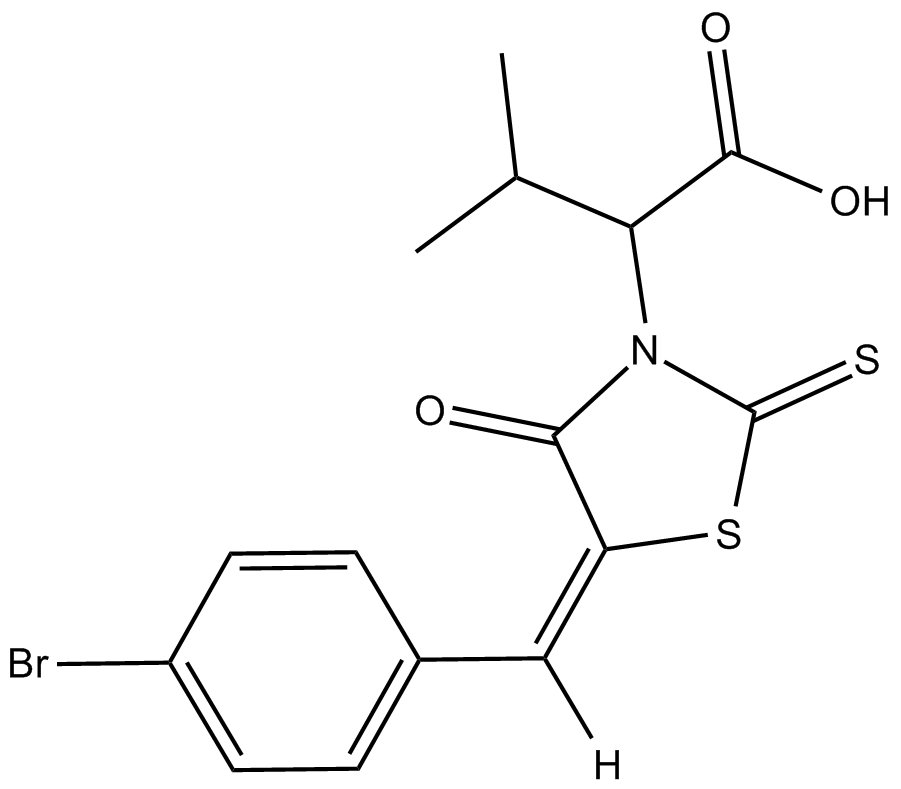
BH3I-1
BH3I-1 es un antagonista de la familia Bcl-2, que inhibe la uniÓn del péptido Bak BH3 a Bcl-xL con una Ki de 2,4±0,2 μM en el ensayo FP. BH3I-1 tiene una Kd de 5,3 μM frente al par p53/MDM2.
-
GC35511
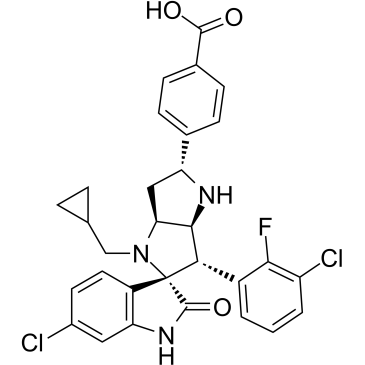
BI-0252
BI-0252 es un inhibidor selectivo de MDM2-p53 activo por vÍa oral con una IC50 de 4 nM. BI-0252 puede inducir regresiones tumorales en todos los animales de un xenoinjerto SJSA-1 de ratÓn, con la inducciÓn concomitante de los genes diana de la proteÍna tumoral p53 (TP53) y marcadores de apoptosis.
-
GC43189
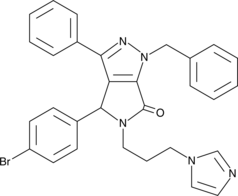
CAY10681
Inactivation of the tumor suppressor p53 commonly coincides with increased signaling through NF-κB in cancer.
-
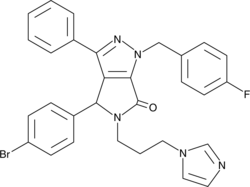
GC43190
CAY10682
(±)-Nutlin-3 blocks the interaction of p53 with its negative regulator Mdm2 (IC50 = 90 nM), inducing the expression of p53-regulated genes and blocking the growth of tumor xenografts in vivo.
-
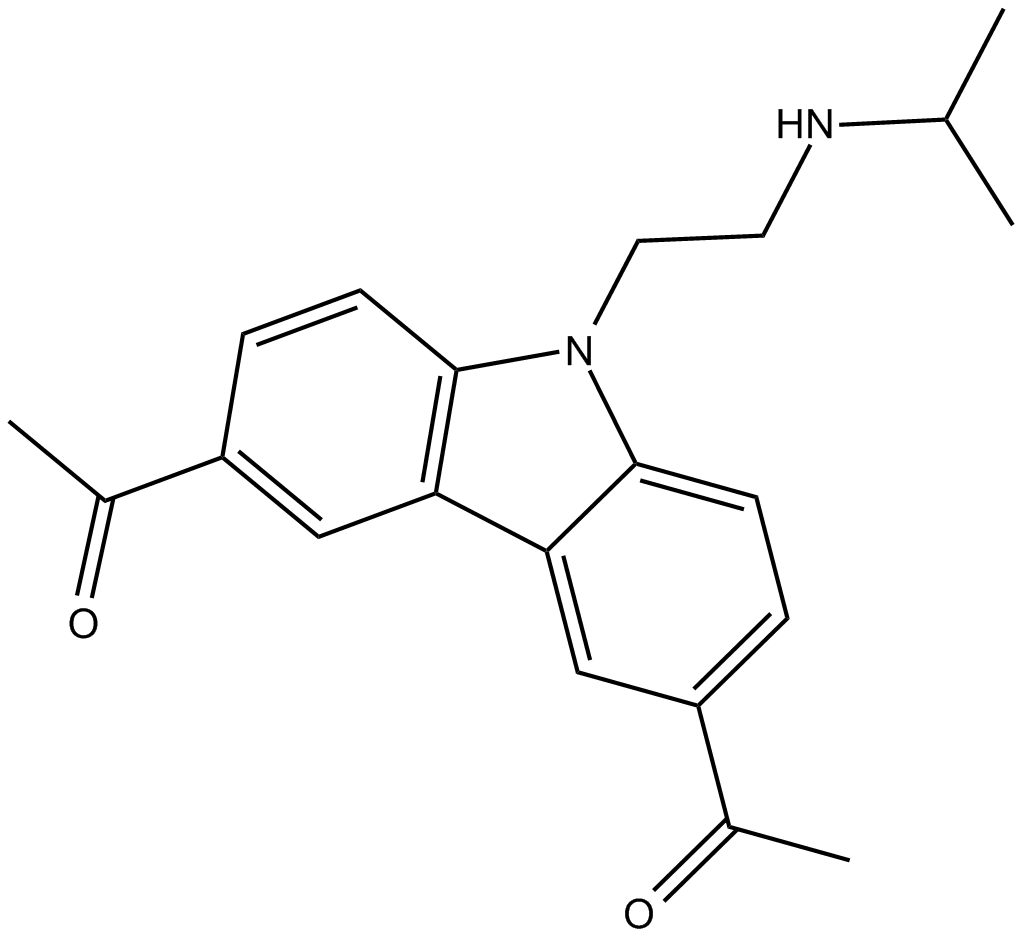
GC14634
CBL0137
curaxin that activates p53 and inhibits NF-κB
-
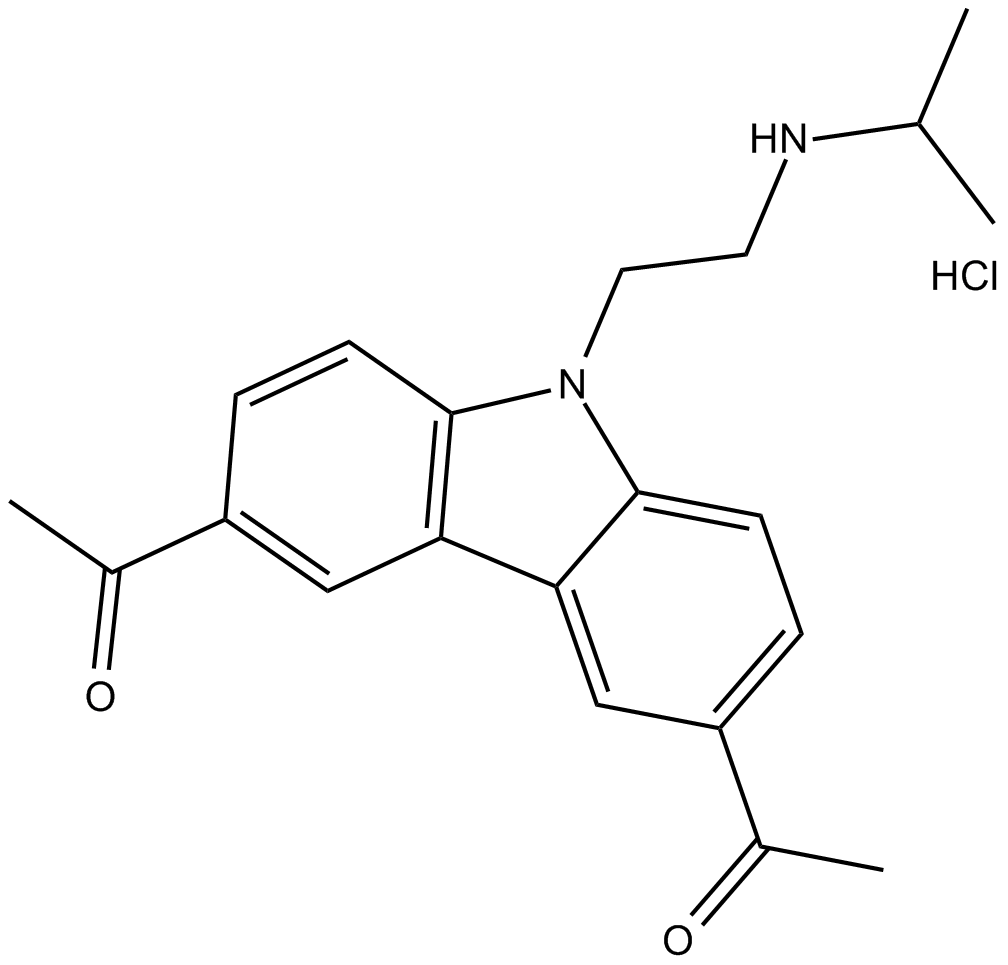
GC15394
CBL0137 (hydrochloride)
CBL0137 (clorhidrato) es un inhibidor de la histona chaperona, FACT. CBL0137 (clorhidrato) también puede activar p53 e inhibe NF-κB con EC50 de 0,37 y 0,47 µM, respectivamente.
-
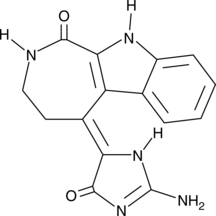
GC43239
Chk2 Inhibitor
El inhibidor de Chk2 (compuesto 1) es un inhibidor potente y selectivo de la quinasa de punto de control 2 (Chk2), con IC50 de 13,5 nM y 220,4 nM para Chk2 y Chk1, respectivamente. El inhibidor de Chk2 puede provocar un fuerte efecto de radioprotecciÓn mediado por Chk2 dependiente de ataxia telangiectasia mutada (ATM).
-
GC64649
Cjoc42
Cjoc42 es un compuesto capaz de unirse a gankyrin. Cjoc42 inhibe la actividad de la ganquirina de forma dependiente de la dosis. Cjoc42 previene la disminuciÓn en los niveles de proteÍna p53 normalmente asociados con altas cantidades de gankyrin. Cjoc42 restaura la transcripciÓn dependiente de p53 y la sensibilidad al daÑo del ADN.
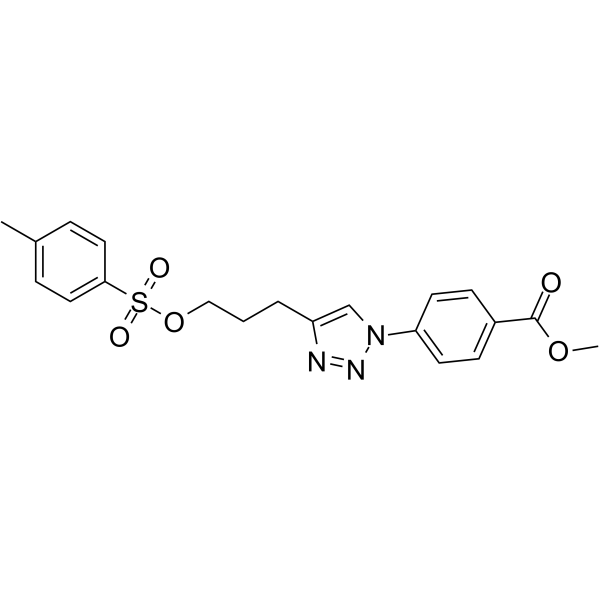
-
GC43297
Coenzyme Q2
Coenzyme Q10 is a component of the electron transport chain and participates in aerobic cellular respiration, generating energy in the form of ATP.
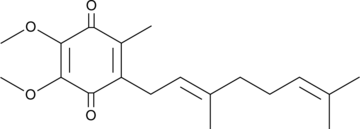
-
GC15225
COTI-2
COTI-2, un fÁrmaco contra el cÁncer con baja toxicidad, es un activador de tercera generaciÓn disponible por vÍa oral de formas mutantes de p53. COTI-2 actÚa reactivando p53 mutante e inhibiendo la vÍa PI3K/AKT/mTOR. COTI-2 induce la apoptosis en mÚltiples lÍneas de células tumorales humanas. COTI-2 exhibe actividad antitumoral en HNSCC a través de mecanismos dependientes e independientes de p53. COTI-2 convierte la p53 mutante en una conformaciÓn de tipo salvaje.
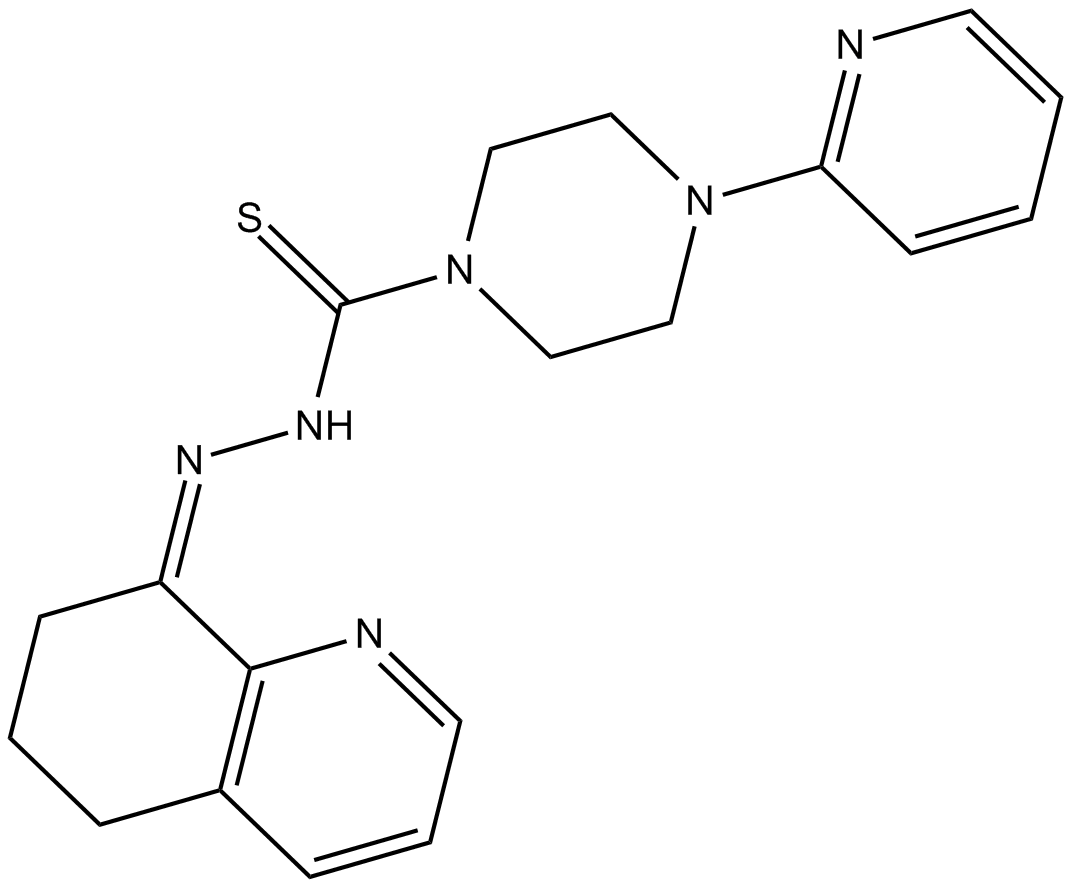
-
GC15840
CP 31398 dihydrochloride
A p53 stabilizing agent
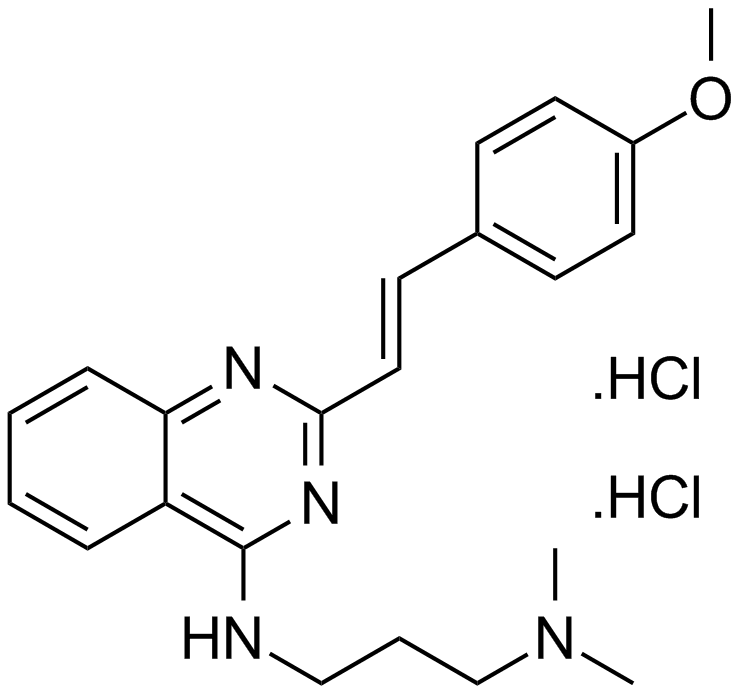
-
GC32911
CTX1
CTX1 es un activador de p53 que supera la represiÓn de p53 mediada por HdmX. CTX1 exhibe una potente actividad anticancerÍgena en un sistema modelo de leucemia mieloide aguda (LMA) de ratÓn.
-
GC43408
Deoxycholic Acid (sodium salt hydrate)
El Ácido desoxicÓlico (Ácido colanoico) hidrato de sodio, un Ácido biliar, es un subproducto del metabolismo intestinal, que activa el receptor de Ácidos biliares acoplado a proteÍna G TGR5.
-
GC47187
Deoxycholic Acid-d4
El Ácido desoxicÓlico-d4 es el Ácido desoxicÓlico marcado con deuterio.
-
GC33384
DPBQ
DPBQ activa p53 y desencadena la apoptosis de una manera poliploide especÍfica, pero no inhibe la topoisomerasa ni se une al ADN. DPBQ provoca la expresiÓn y fosforilaciÓn de p53 y este efecto es especÍfico de las células tetraploides.
-
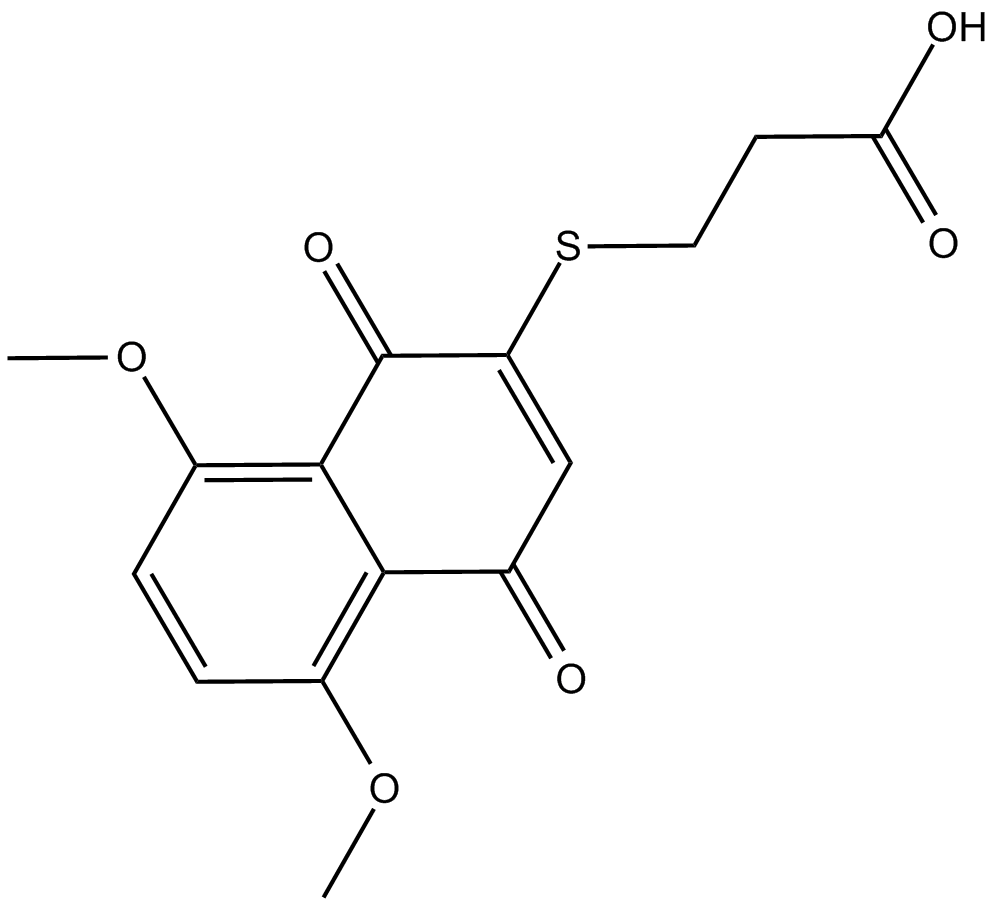
GC15258
GN25
p53-Snail binding Inhibitor
-
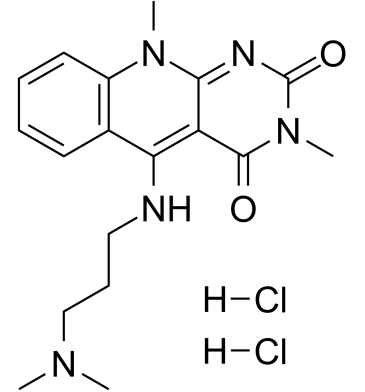
GC61608
HLI373 dihydrochloride
El diclorhidrato de HLI373 es un inhibidor eficaz de Hdm2.
-
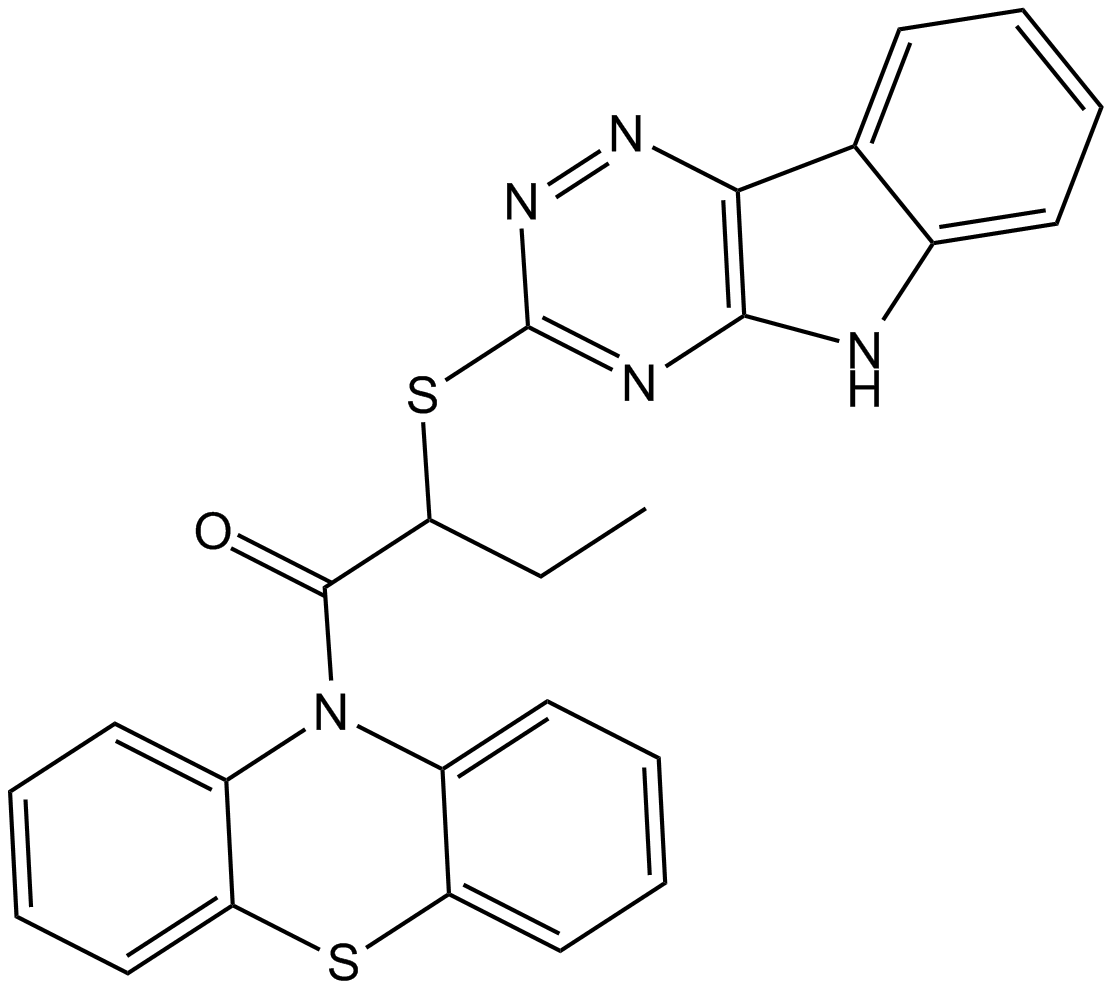
GC14755
Inauhzin
Inauhzin es un inhibidor dual SirT1/IMPDH2 y actÚa como un activador p53, utilizado en la investigaciÓn del cÁncer.
-
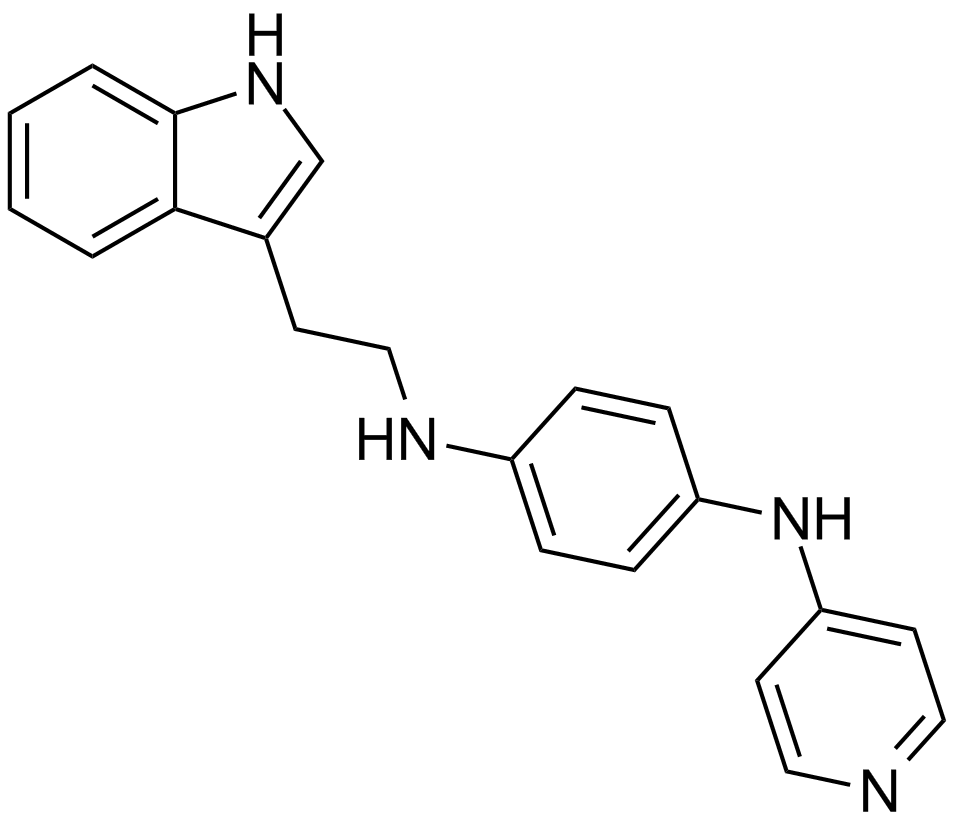
GC12117
JNJ-26854165 (Serdemetan)
An antagonist of MDM2 action
-
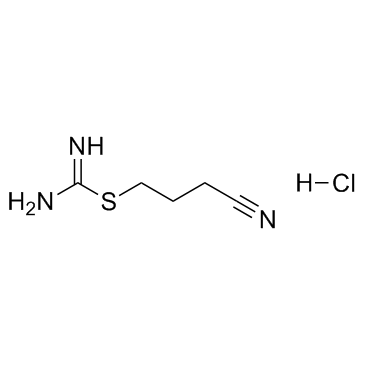
GC32919
Kevetrin hydrochloride (4-Isothioureidobutyronitrile hydrochloride)
El clorhidrato de kevetrin (clorhidrato de 4-isotioureidobutironitrilo) es un potente activador de p53, induce la apoptosis en células de leucemia mieloide aguda mutante y de tipo salvaje TP53. Kevetrin una actividad citotÓxica preferencial contra las células blÁsticas.
-
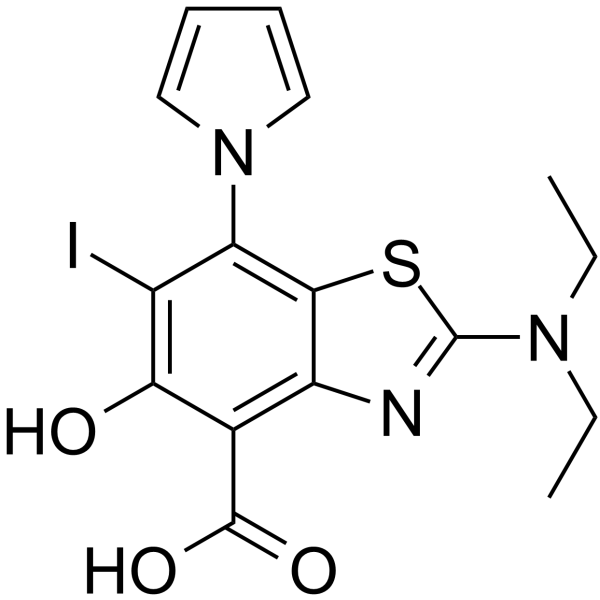
GC62629
MB710
MB710, un derivado de aminobenzotiazol, es un estabilizador de la mutaciÓn oncogénica p53 Y220C. MB710 se une firmemente al bolsillo Y220C y estabiliza p53-Y220C, con una Kd de 4,1 μM. MB710 muestra actividad anticancerÍgena en lÍneas celulares p53-Y220C.
-
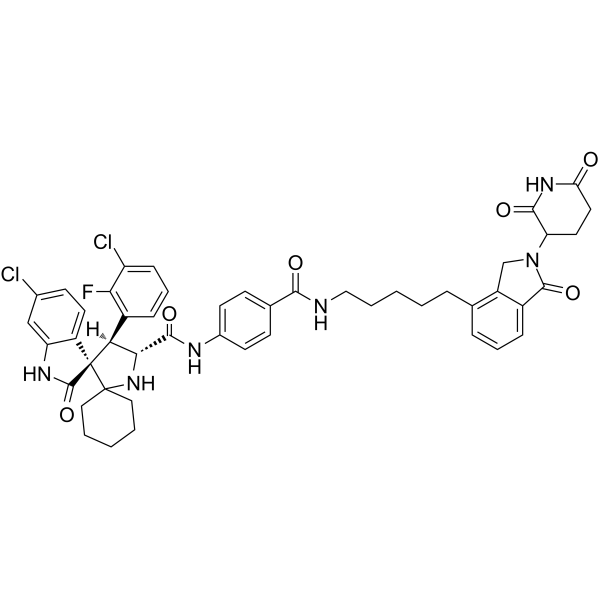
GC62716
MD-222
MD-222 es el primer degradador PROTAC altamente potente de MDM2 de su clase. MD-222 consta de ligandos para Cereblon y MDM2. MD-222 induce una rÁpida degradaciÓn de la proteÍna MDM2 y la activaciÓn de p53 de tipo salvaje en las células. MD-222 tiene efectos anticancerÍgenos.
-
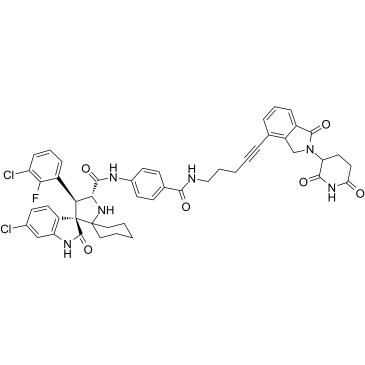
GC38812
MD-224
MD-224 es un degradador de doble minuto 2 (MDM2) murino humano de molécula pequeÑa altamente potente y primero en su clase basado en el concepto de quimera dirigida a la proteÓlisis (PROTAC). MD-224 consta de ligandos para Cereblon y MDM2. MD-224 induce una rÁpida degradaciÓn de MDM2 a concentraciones <1 nM en células de leucemia humana y logra un valor IC50 de 1,5 nM en la inhibiciÓn del crecimiento de células RS4;11. MD-224 tiene el potencial de ser una nueva clase de agente anticancerÍgeno.
-
GC62557
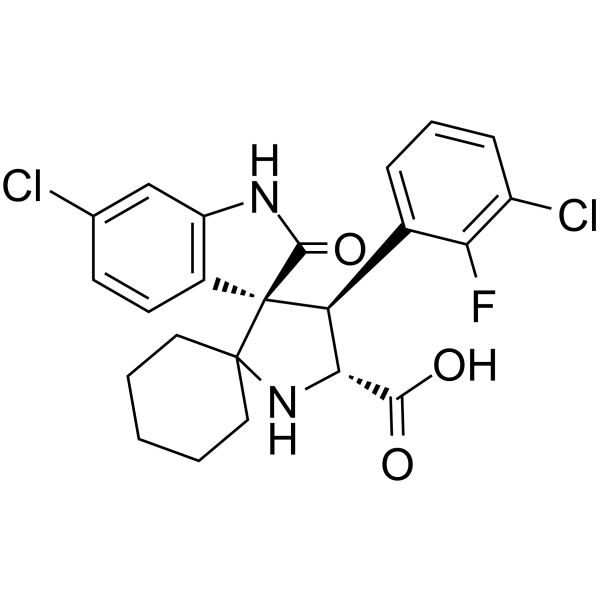
MDM2-IN-1
MDM2-IN-1 (Compuesto 30) es un inhibidor sintético de la interacciÓn MDM2-p53 (MDM2) y contiene la configuraciÓn trans (D-).
-
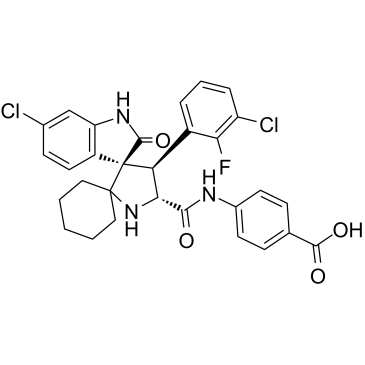
GC36605
MI-1061
MI-1061 es un inhibidor de MDM2 (interacciÓn MDM2-p53) potente, biodisponible por vÍa oral y quÍmicamente estable (IC50 = 4,4 nM; Ki = 0,16 nM). MI-1061 activa p53 de forma potente e induce la apoptosis en el tejido tumoral de xenoinjerto SJSA-1 en ratones. Actividad antitumoral.
-
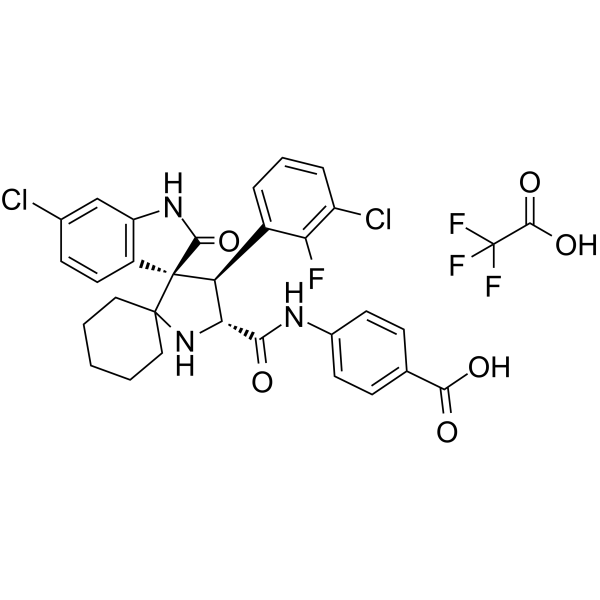
GC62598
MI-1061 TFA
MI-1061 TFA es un inhibidor de MDM2 (interacciÓn MDM2-p53) potente, biodisponible por vÍa oral y quÍmicamente estable (IC50 = 4,4 nM; Ki = 0,16 nM). MI-1061 TFA activa p53 de forma potente e induce la apoptosis en el tejido tumoral de xenoinjerto SJSA-1 en ratones. Actividad antitumoral.
-
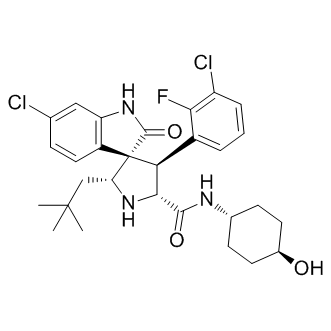
GC16296
MI-773
MI-773 es un potente inhibidor de la interacciÓn proteÍna-proteÍna (PPI) de MDM2-p53 con alta afinidad de uniÓn contra MDM2 (Kd=8,2 nM). MI-773 tiene actividad antitumoral.
-
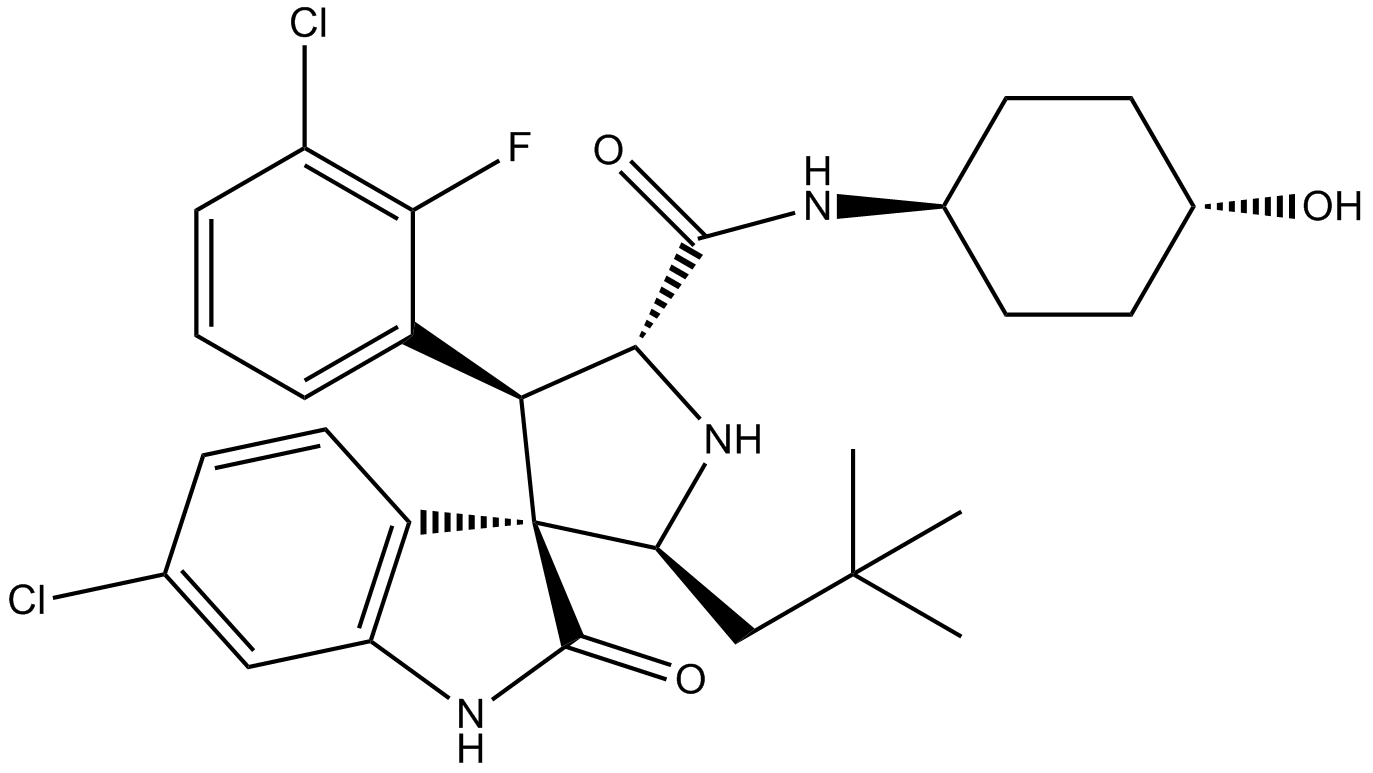
GC11547
MI-773 (SAR405838)
MI-773 (SAR405838) (MI-77301), un anÁlogo de MI-773, es un inhibidor de interacciÓn MDM2-p53 muy potente y selectivo. MI-773 (SAR405838) se une a MDM2 con una Ki de 0,88 nM. MI-773 (SAR405838) induce la apoptosis y tiene una potente actividad antitumoral.
-
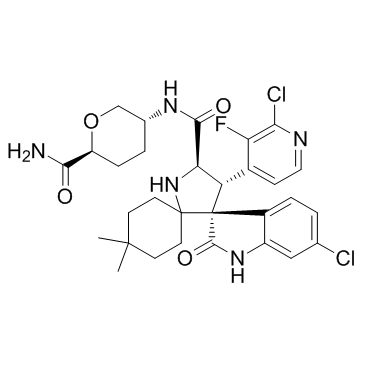
GC32881
Milademetan (DS-3032)
Milademetan (DS-3032) (DS-3032) es un inhibidor de MDM2 especÍfico y activo por vÍa oral para la investigaciÓn de leucemia mieloide aguda (LMA) o tumores sÓlidos. Milademetan (DS-3032) (DS-3032) induce la detenciÓn del ciclo celular G1, la senescencia y la apoptosis.
-
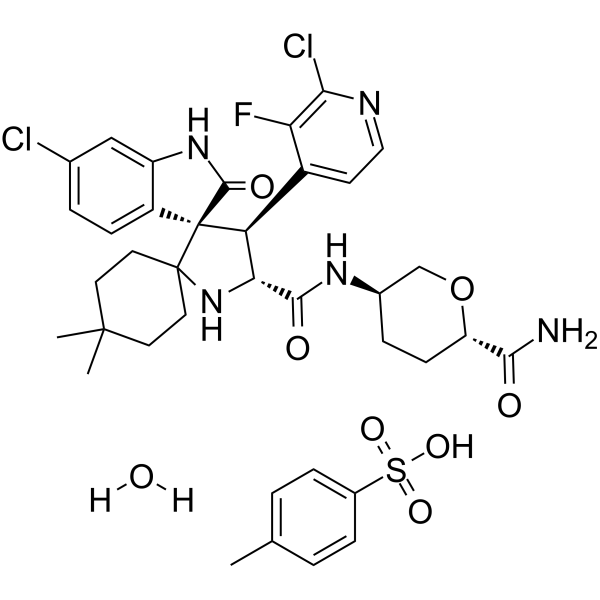
GC62621
Milademetan tosylate hydrate
El hidrato de tosilato de milademetÁn (DS-3032) es un inhibidor de MDM2 especÍfico y activo por vÍa oral para la investigaciÓn de la leucemia mieloide aguda (LMA) o tumores sÓlidos. El hidrato de tosilato de milademetÁn (DS-3032) induce la detenciÓn del ciclo celular G1, la senescencia y la apoptosis.
-
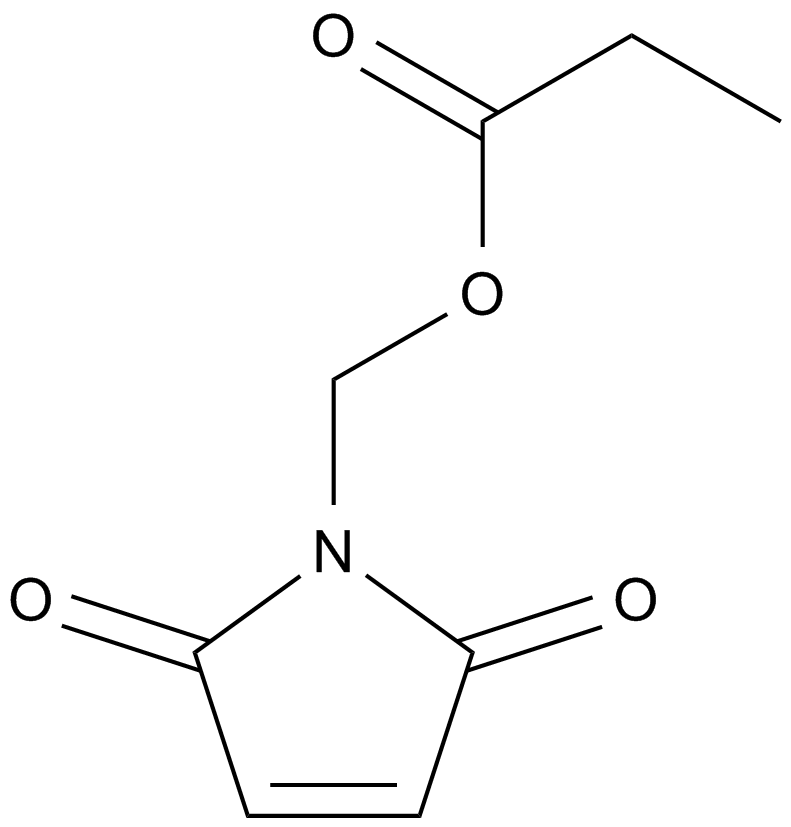
GC12893
MIRA-1
A mutant p53 reactivator
-
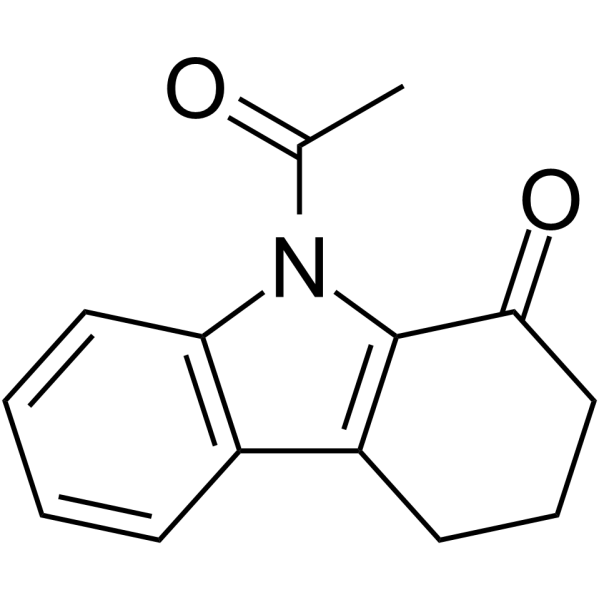
GC62322
MS7972
MS7972 es una molécula pequeÑa que bloquea la asociaciÓn de la proteÍna de uniÓn p53 humana y CREB. MS7972 puede bloquear casi por completo esta interacciÓn BRD en 50 μM.
-
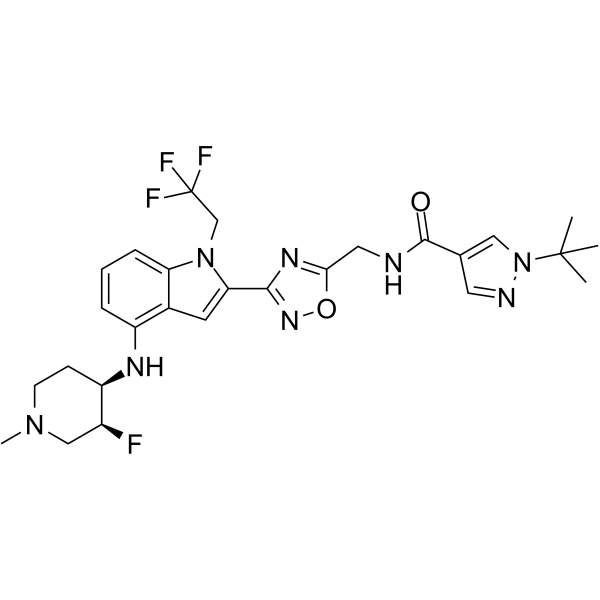
GC68371
Mutant p53 modulator-1
-
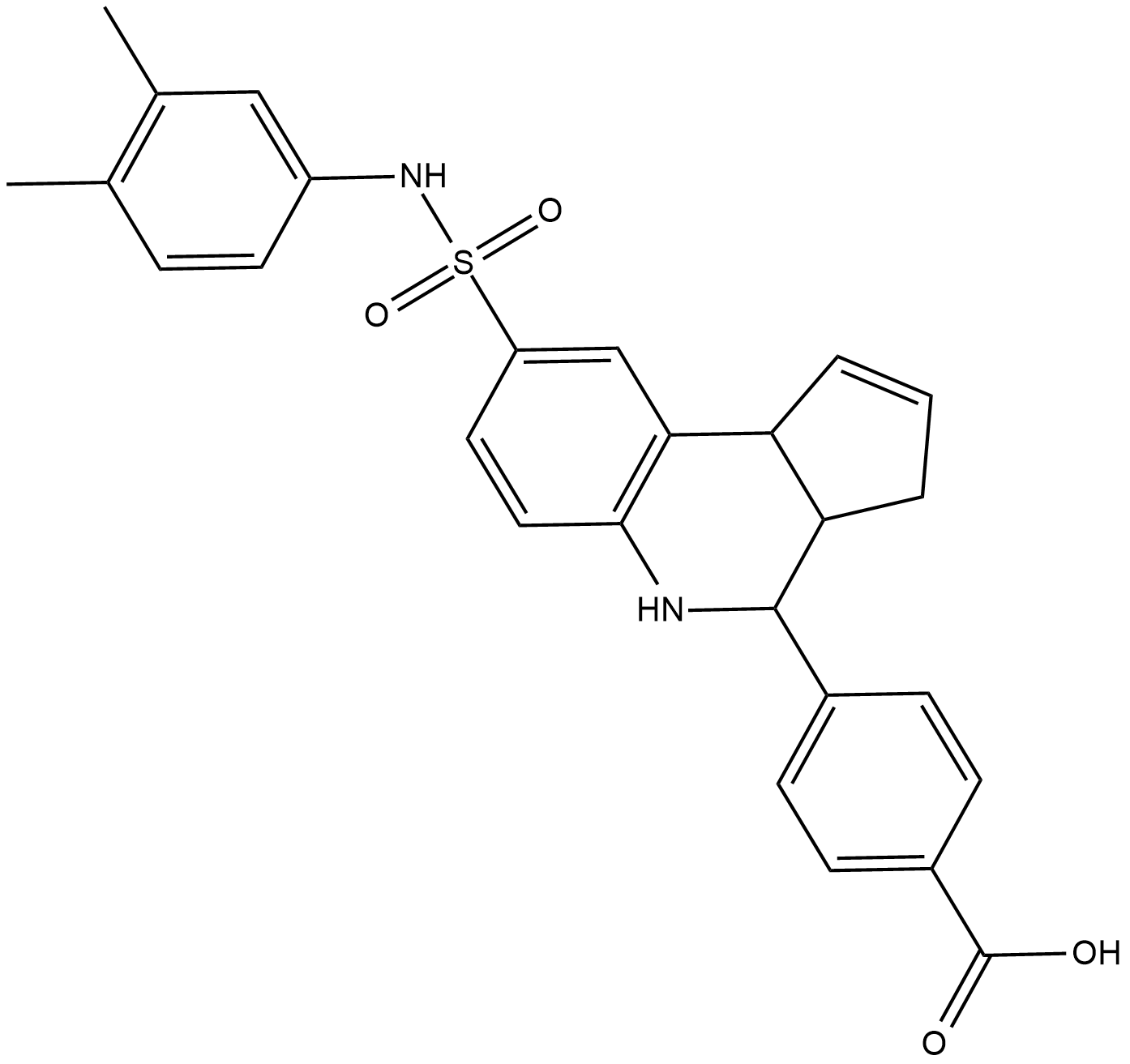
GC19257
MX69
MX69 es un inhibidor de MDM2/XIAP, utilizado para el tratamiento del cÁncer.
-
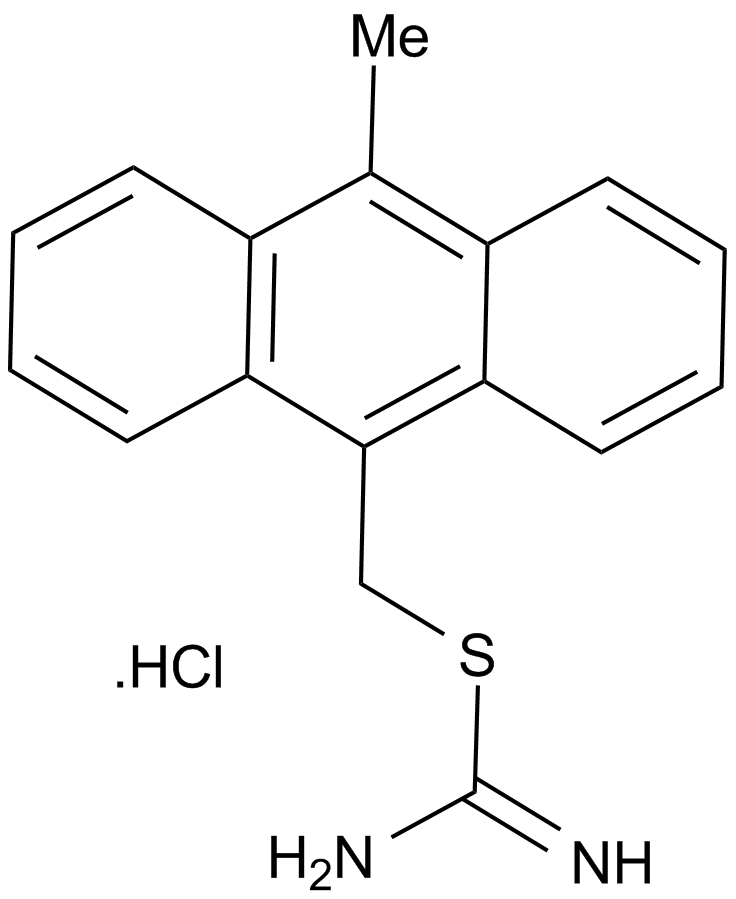
GC15621
NSC 146109 hydrochloride
An activator of p53
-
GC15404
NSC 319726
A p53 reactivator
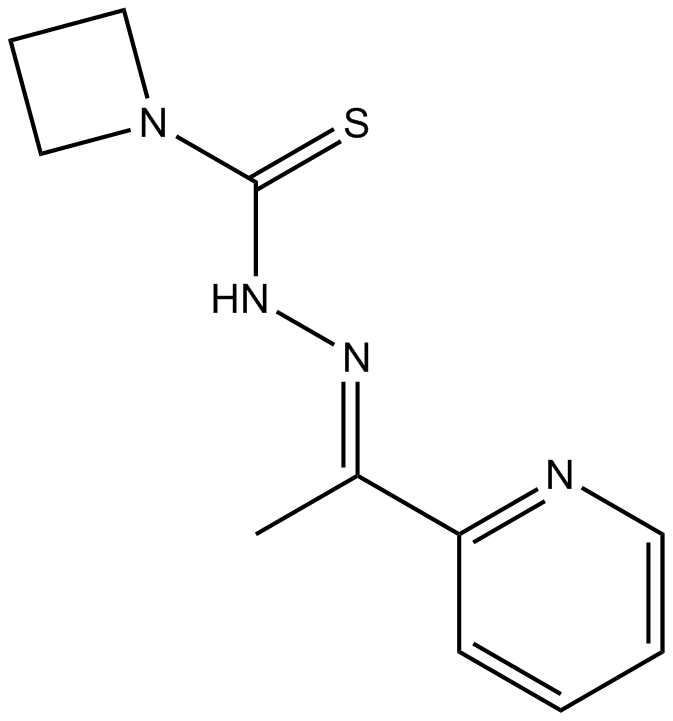
-
GC16151
NSC348884
NSC348884 es un inhibidor de la nucleofosmina (NPM), interrumpe la formaciÓn de oligÓmeros e induce la apoptosis, inhibe la proliferaciÓn celular con IC50 de 1,7-4,0 μM en distintas lÍneas celulares de cÁncer. NSC348884 se puede utilizar para la investigaciÓn del cÁncer.
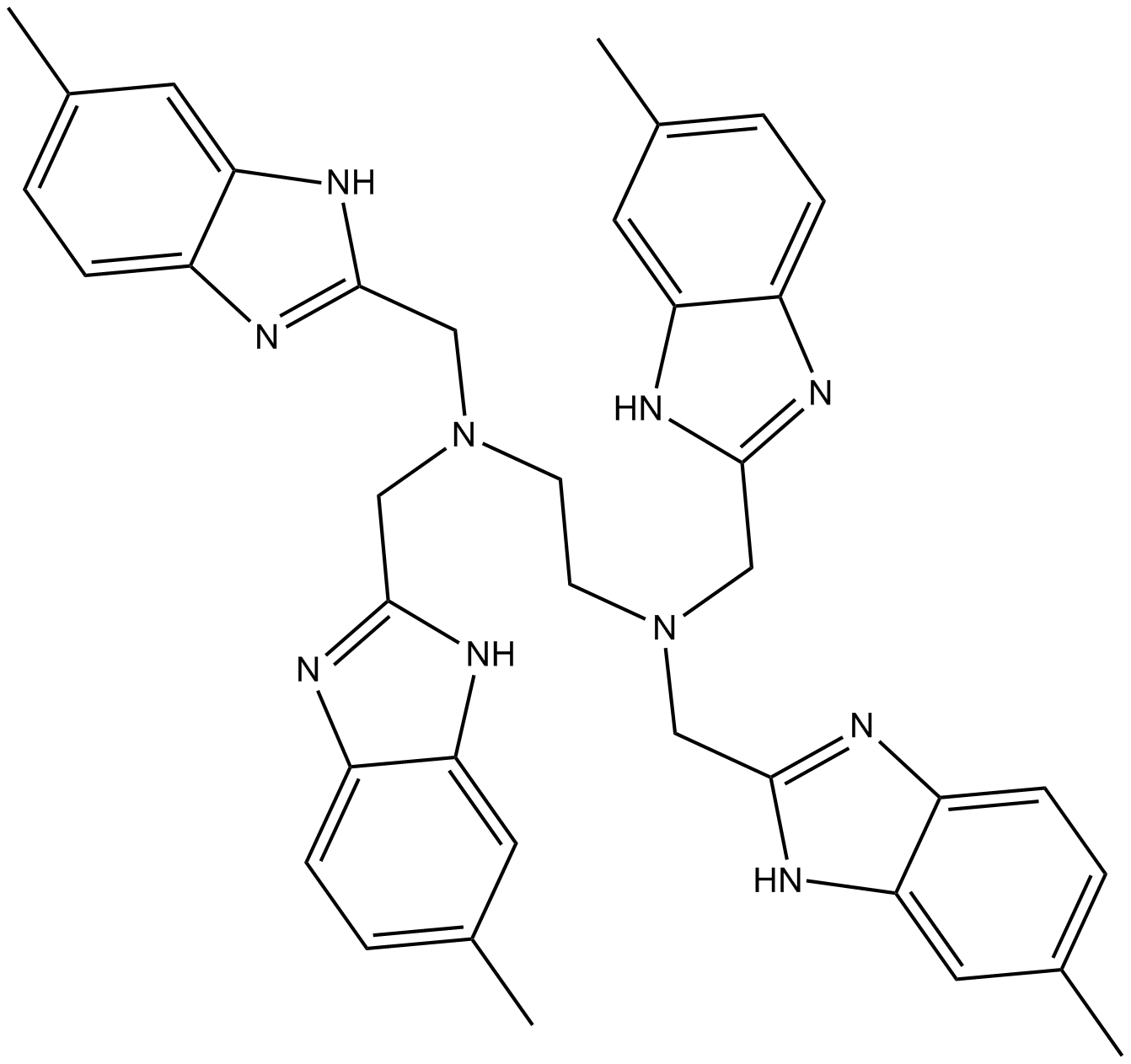
-
GC16179
NSC59984
NSC59984 induce la degradaciÓn de la proteÍna p53 mutante a través de MDM2 y la vÍa ubiquitina-proteasoma. NSC59984 actÚa apuntando a p53 mutante GOF y estimula a p73 para restaurar la seÑalizaciÓn de la vÍa p53.
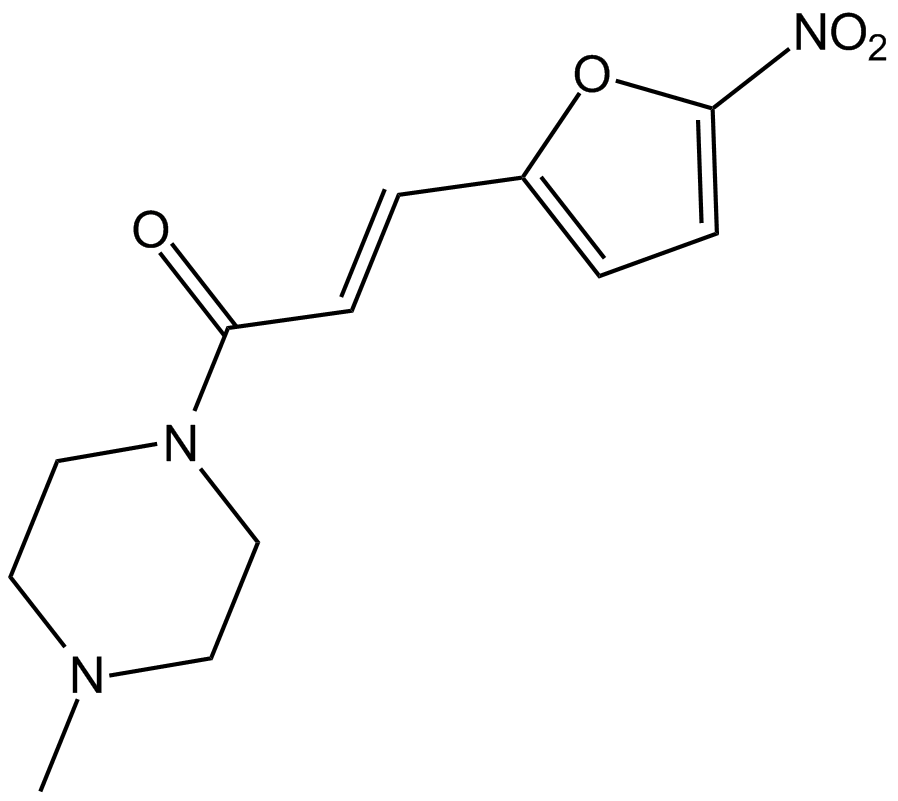
-
GC16051
Nutlin-3
A racemic mixture of (?)-nutlin-3 and (+)-nutlin-3
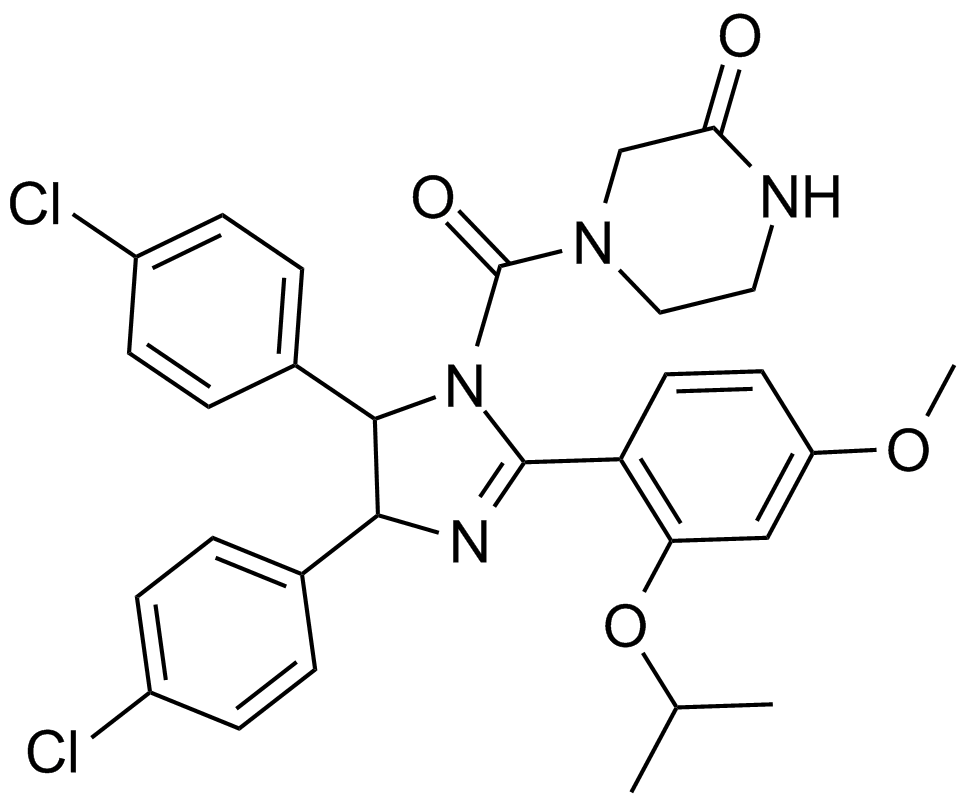
-
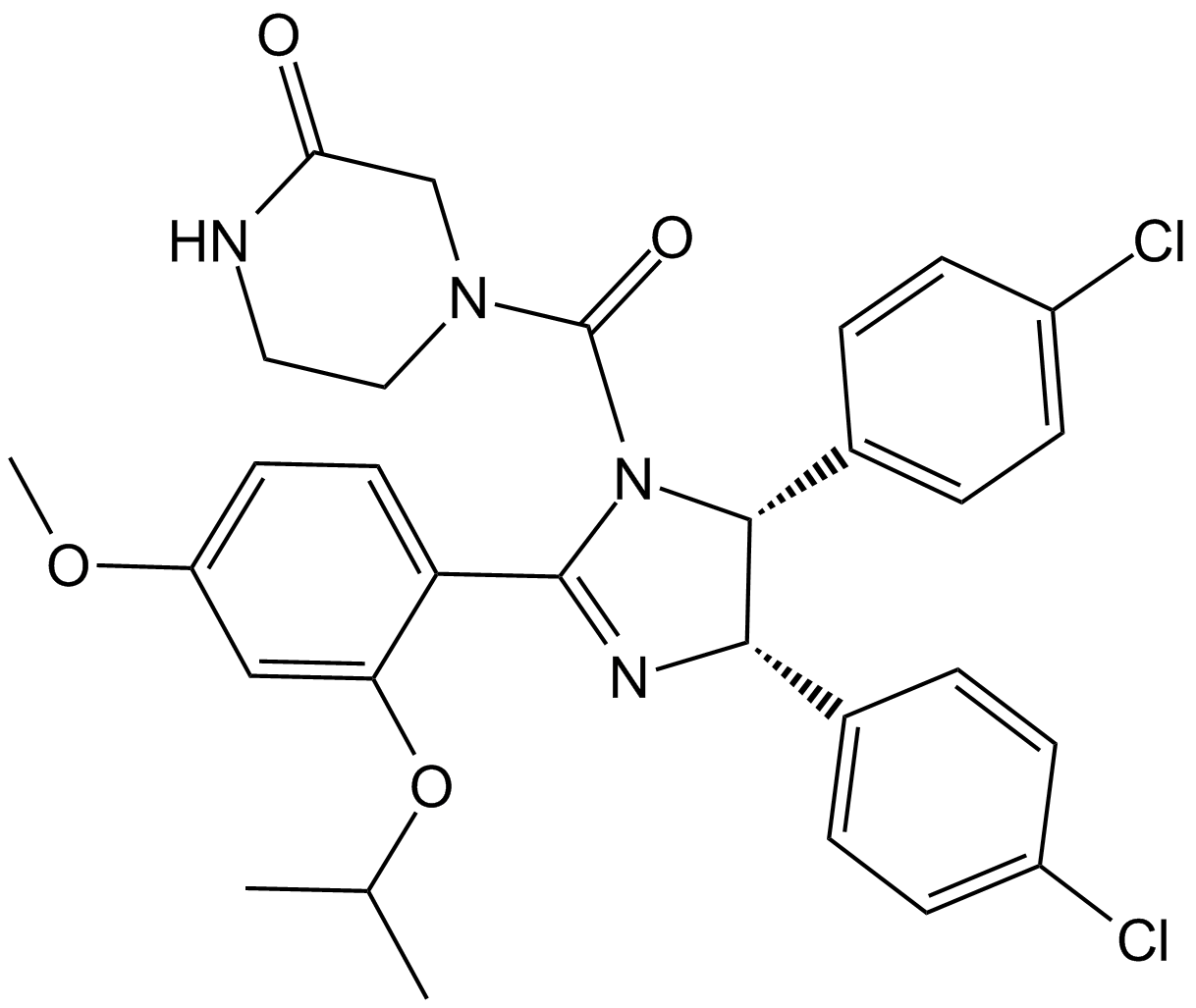
GC10470
Nutlin-3a chiral
An inhibitor of the p53-Mdm2 interaction
-
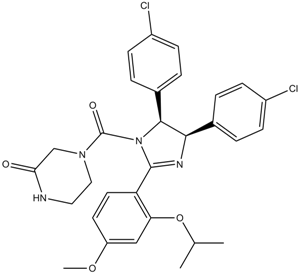
GC12508
Nutlin-3b
Nutlin-3b es un inhibidor de p53/MDM2 con un IC50 de 13,6 μM. Nutlin-3b es 150 veces menos potente para unirse a MDM2 que Nutlin-3a.
-
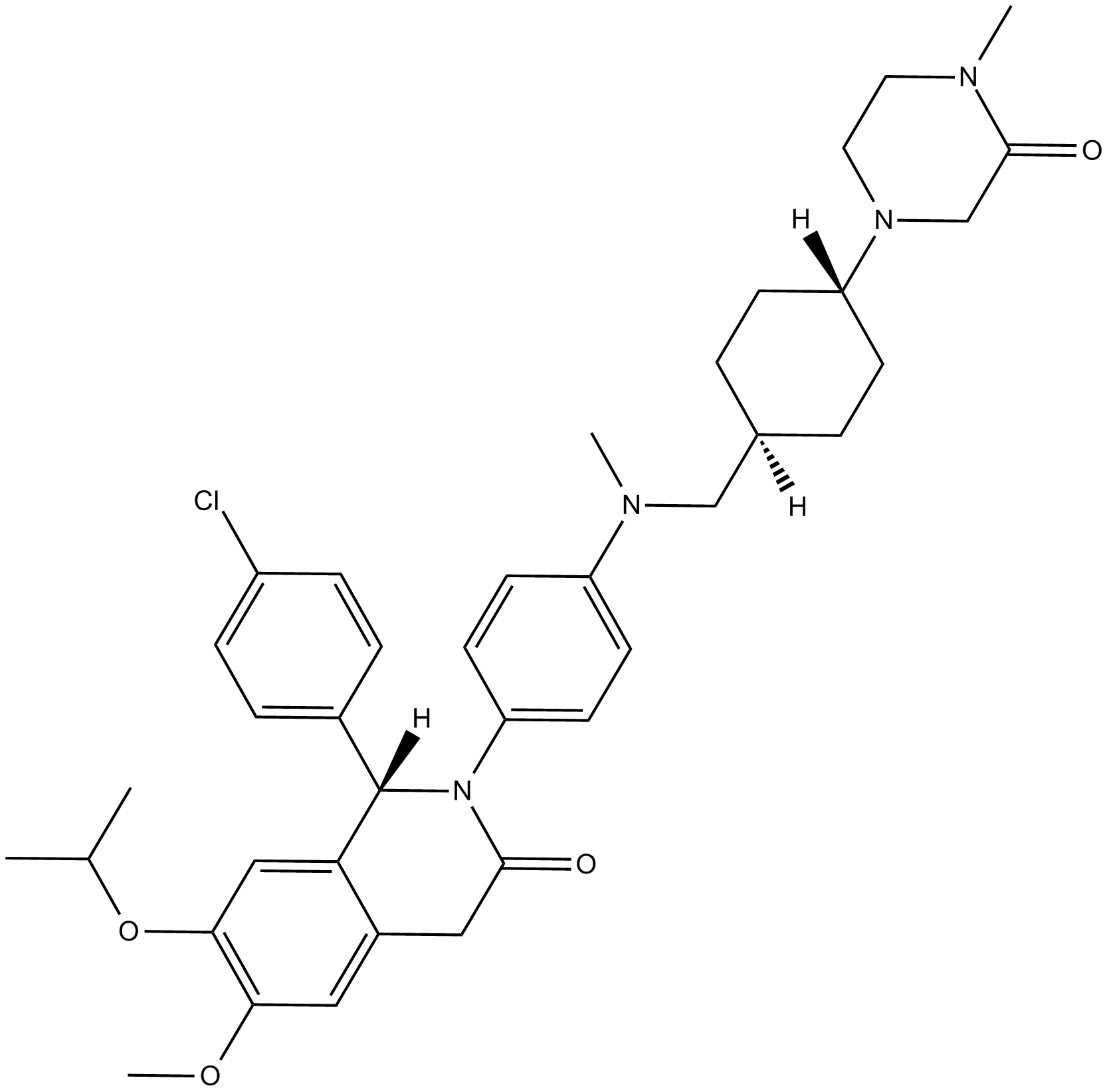
GC12647
NVP-CGM097
NVP-CGM097 es un inhibidor de MDM2 potente y selectivo con IC50 de 1,7±0,1 nM para hMDM2.
-
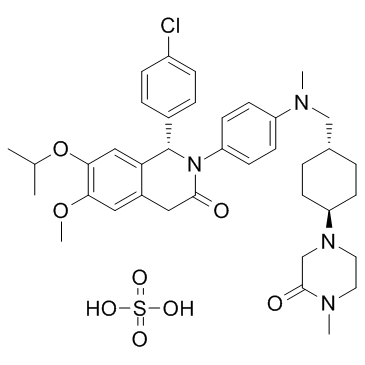
GC36785
NVP-CGM097 sulfate
El sulfato de NVP-CGM097 es un inhibidor de MDM2 potente y selectivo con IC50 de 1,7 ± 0,1 nM para hMDM2.
-
GC19268
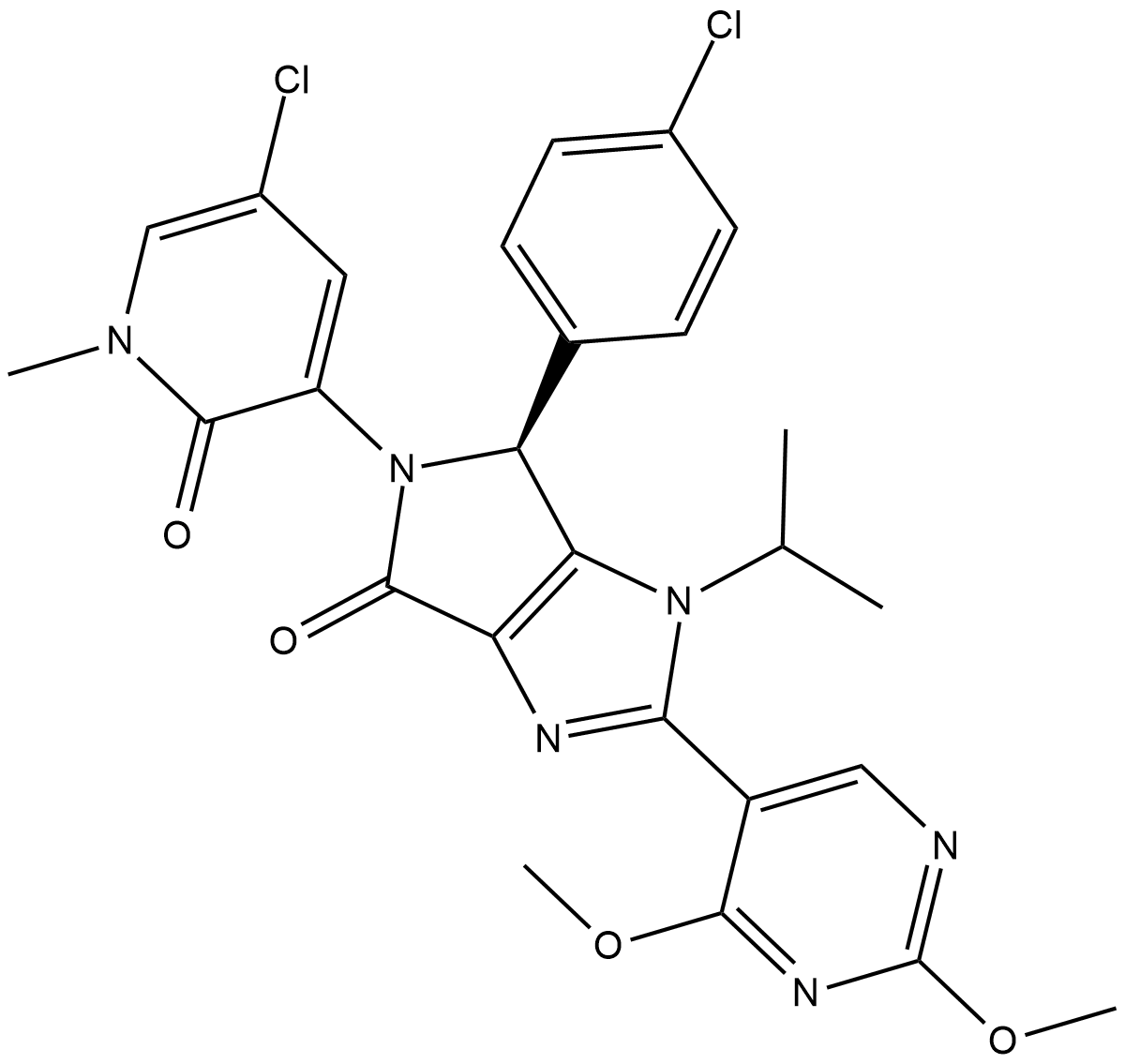
NVP-HDM201
NVP-HDM201 (NVP-HDM201) es un inhibidor de la interacciÓn p53-MDM2 potente, biodisponible por vÍa oral y altamente especÍfico.
-
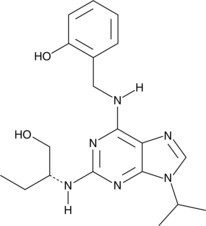
GC48652
Olomoucine II
A CDK inhibitor
-
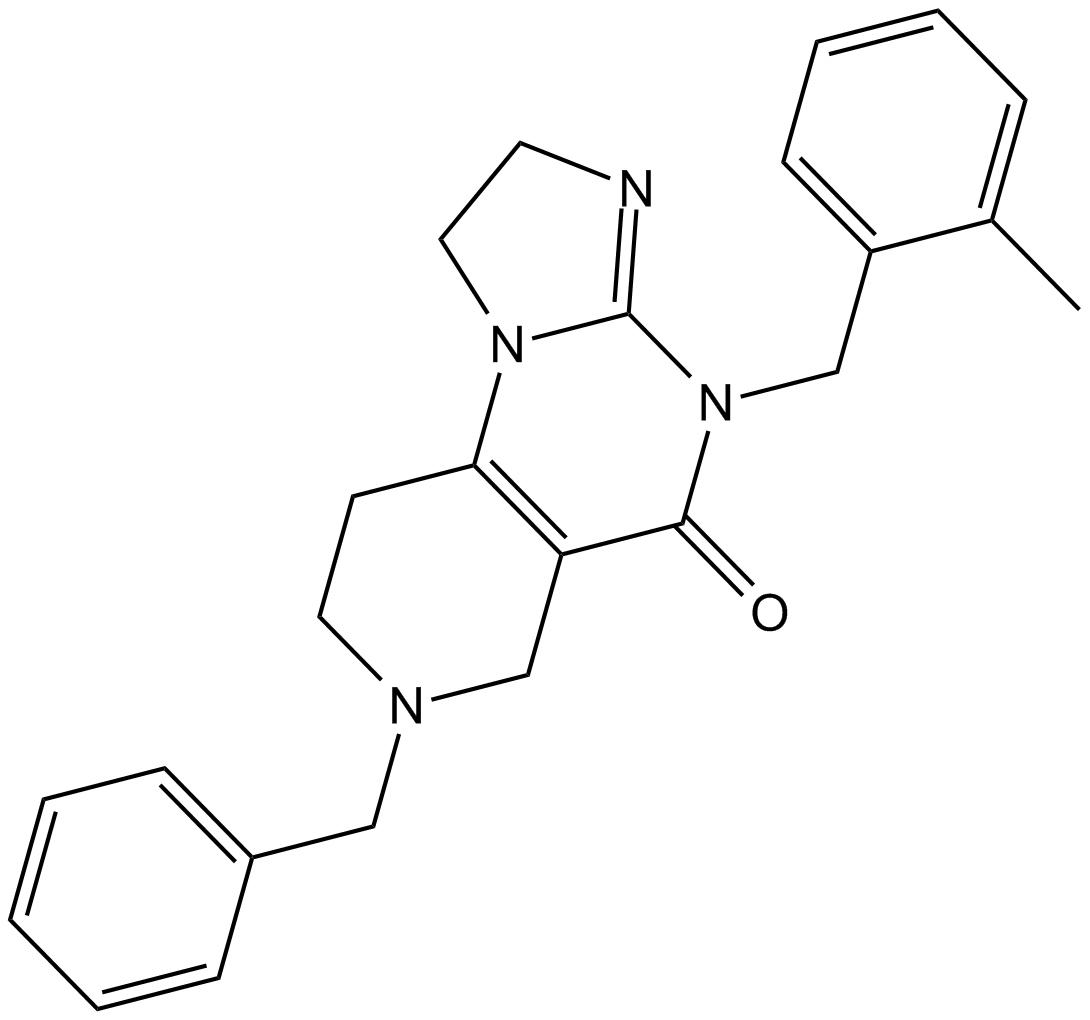
GC11711
ONC201
ONC201 (ONC-201) es un inductor de ligando inductor de apoptosis relacionado con el factor de necrosis tumoral (TRAIL) potente, activo por vÍa oral y estable que actÚa inhibiendo Akt y ERK, activando en consecuencia Foxo3a e induciendo significativamente TRAIL en la superficie celular. ONC201 puede cruzar la barrera hematoencefÁlica.
-
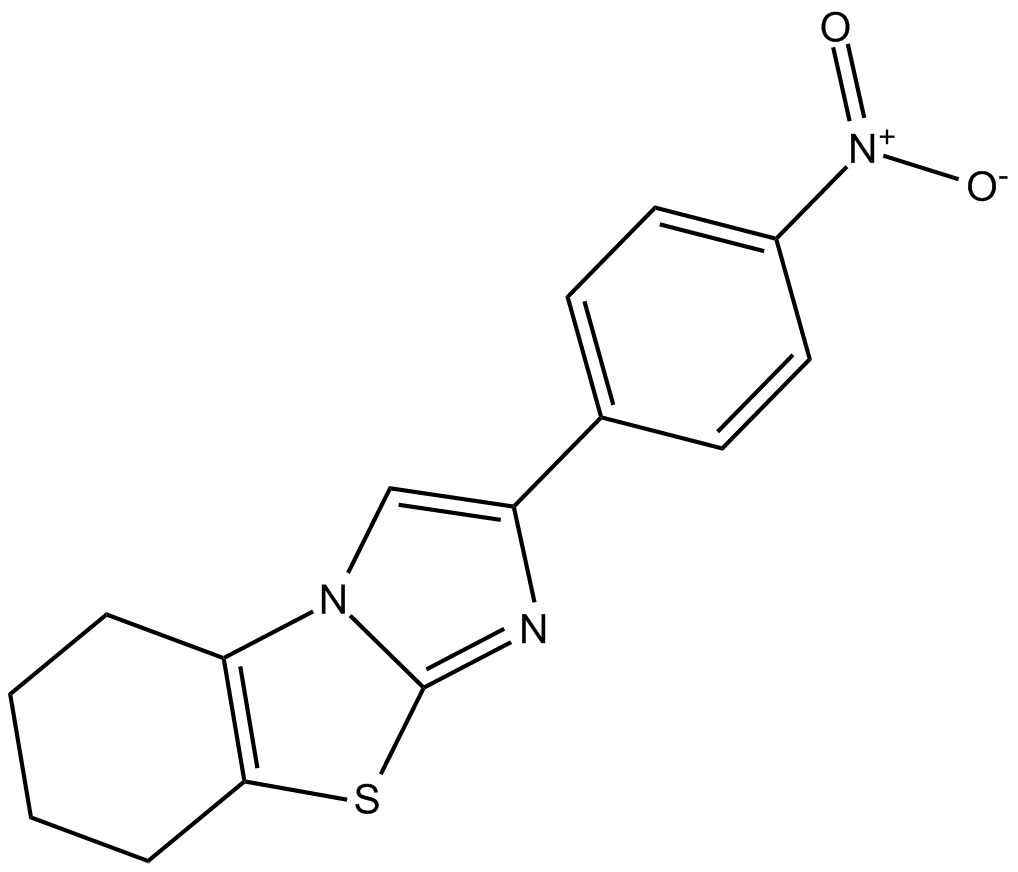
GC15613
p-nitro-Cyclic Pifithrin-α
pifitrina p-nitro-cÍclica-α (PFN-α) es un inhibidor de p53 permeable a las células y de forma activa.
-
GC15812
p-nitro-Pifithrin-α
p-nitro-Pifithrin-α, un anÁlogo permeable a las células de pifitrin-α, es un potente inhibidor de p53.
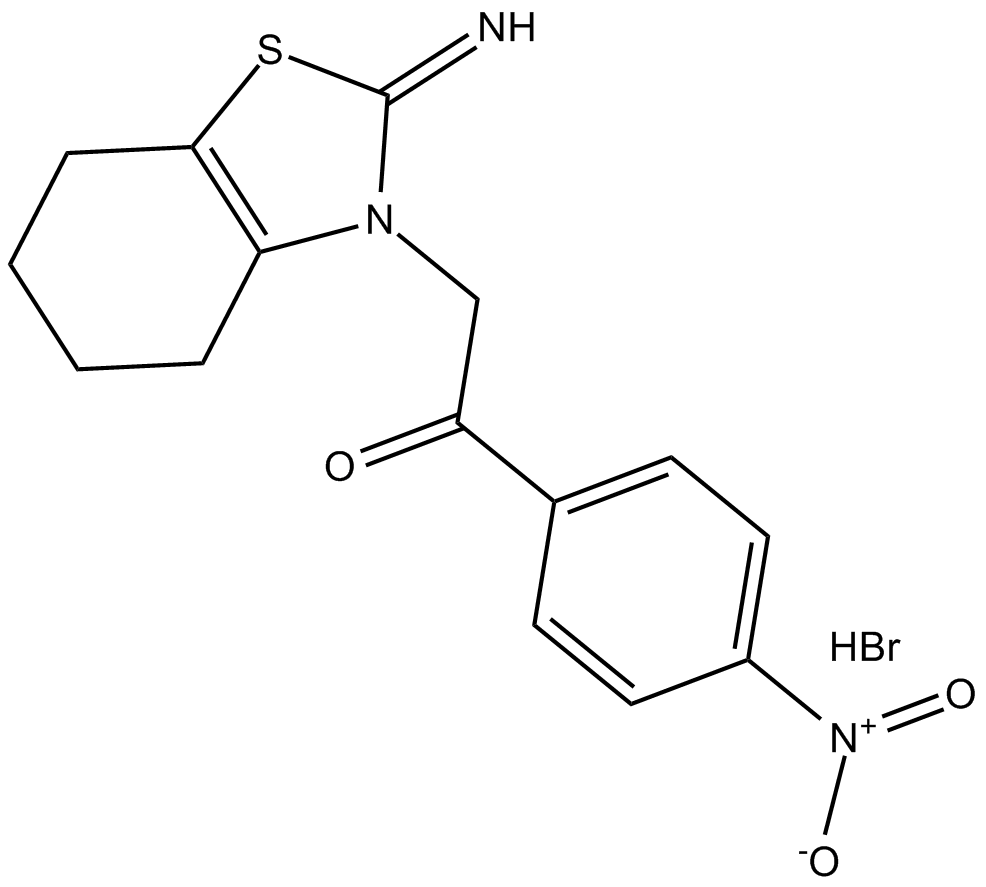
-
GC67765
p53 Activator 5
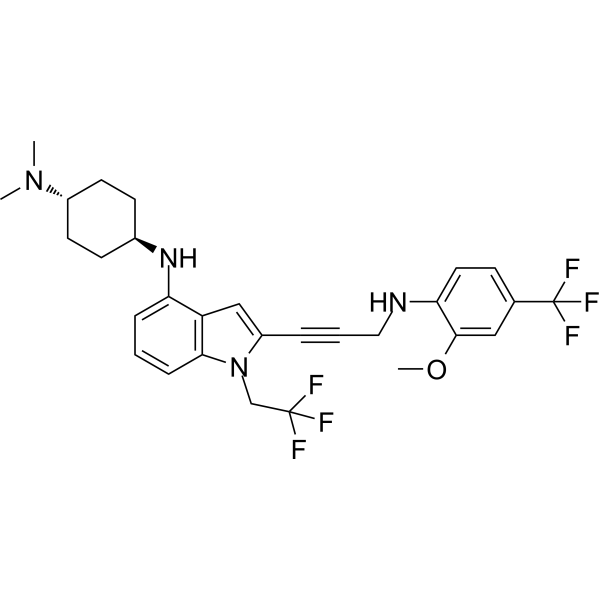
-
GC69648
p53 Activator 7
P53 Activator 7 es un activador de p53 mutado Y220C (MDM-2/p53), con una EC50 de 104 nM. Puede unirse a la variante mutada de p53 y restaurar su capacidad para unirse al ADN (WO2022213975A1; Ejemplo B-1).
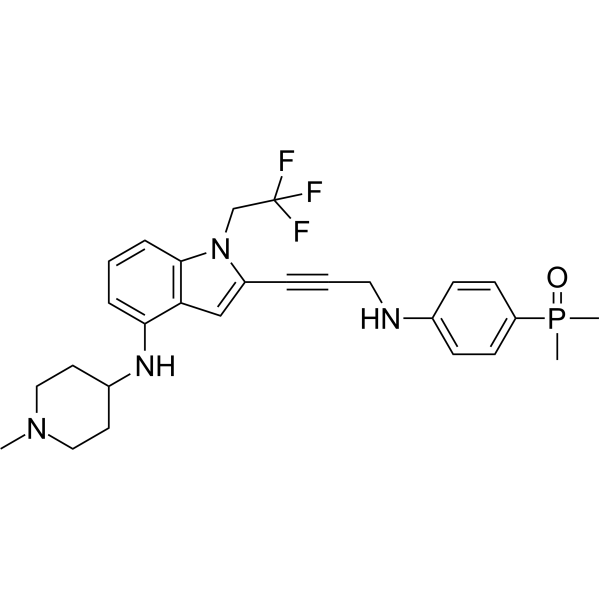
-
GC36835
p53 and MDM2 proteins-interaction-inhibitor chiral
El inhibidor de la interacción de las proteínas p53 y MDM2 quiral (Compuesto 32) es un inhibidor de la interacción entre las proteínas p53 y MDM2.
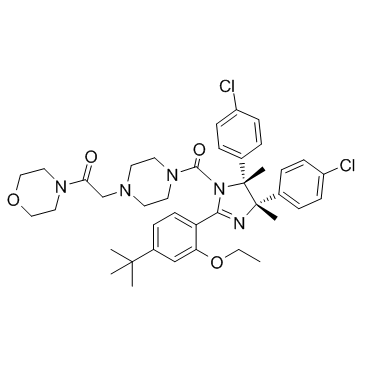
-
GC36836
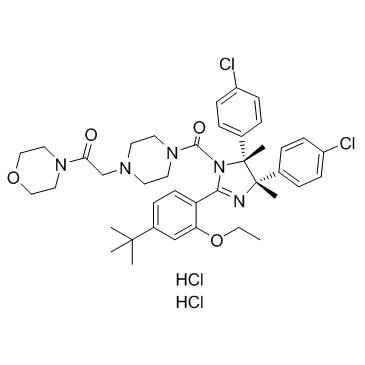
p53 and MDM2 proteins-interaction-inhibitor dihydrochloride
El diclorhidrato del inhibidor de la interacciÓn de las proteÍnas p53 y MDM2 es un inhibidor de la interacciÓn entre las proteÍnas p53 y MDM2.
-
GC36837
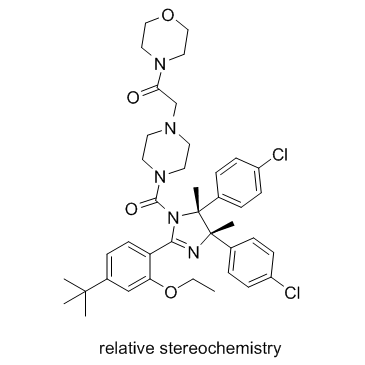
p53 and MDM2 proteins-interaction-inhibitor racemic
El inhibidor de la interacción de las proteínas p53 y MDM2 racémico (Compuesto 2j) es un inhibidor de la interacción entre las proteínas p53 y MDM2.
-
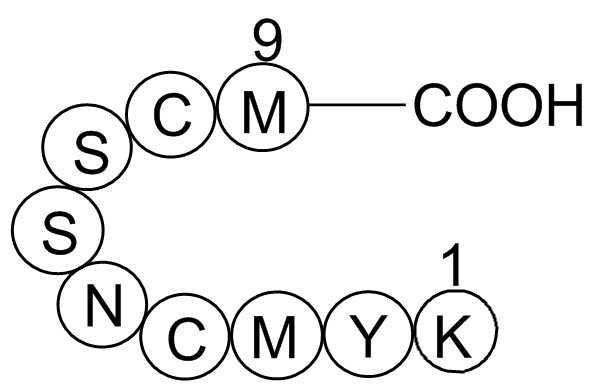
GP10036
p53 tumor suppressor fragment
Regulates cell cycle
-
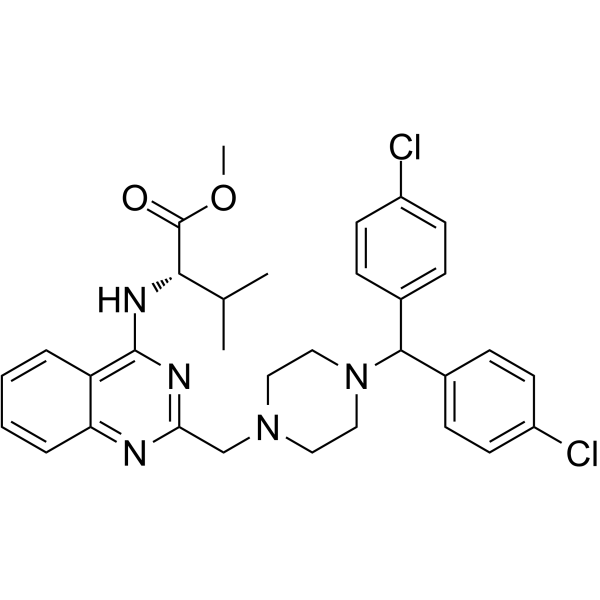
GC65961
P53R3
P53R3 es un potente reactivador de p53 y restaura la uniÓn de ADN especÍfica de secuencia de mutantes de puntos calientes de p53, incluidos p53R175H, p53R248W y p53R273H. P53R3 induce efectos antiproliferativos dependientes de p53 con una especificidad mucho mayor que PRIMA-1. P53R3 mejora el reclutamiento de p53 y p53M237I de tipo salvaje para varios promotores de genes diana. P53R3 aumenta fuertemente el ARNm, la proteÍna total y la expresiÓn en la superficie celular del receptor de muerte 5 (DR5). P53R3 se utiliza para la investigaciÓn del cÁncer.
-
GC45758
Paclitaxel octadecanedioate
A prodrug form of paclitaxel
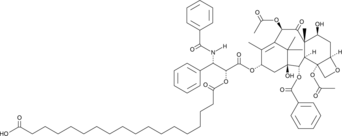
-
GC47853
Paclitaxel-d5
Paclitaxel-d5 es un Paclitaxel marcado con deuterio. Paclitaxel es un agente antineoplÁsico natural y estabiliza la polimerizaciÓn de tubulina.
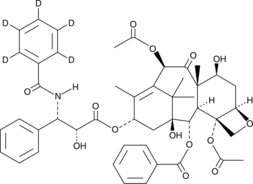
-
GC12658
PhiKan 083
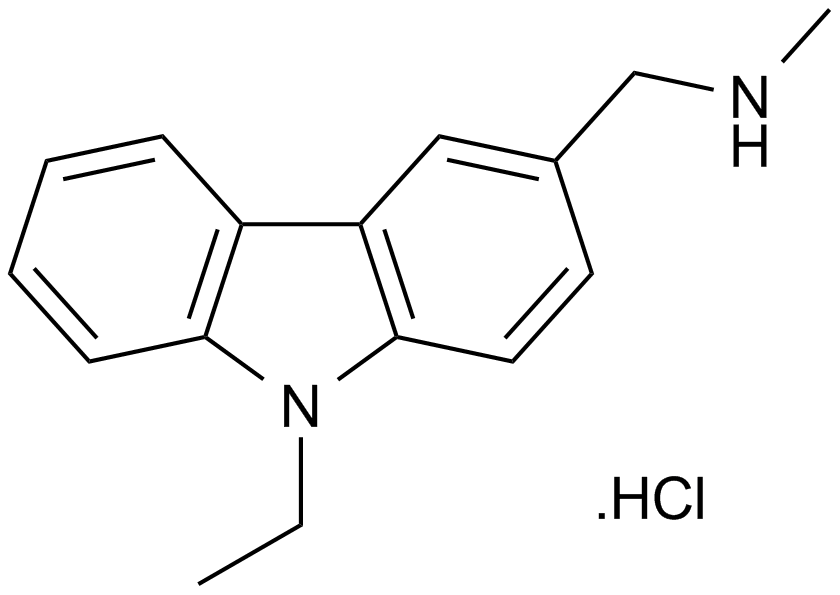
-
GC36896
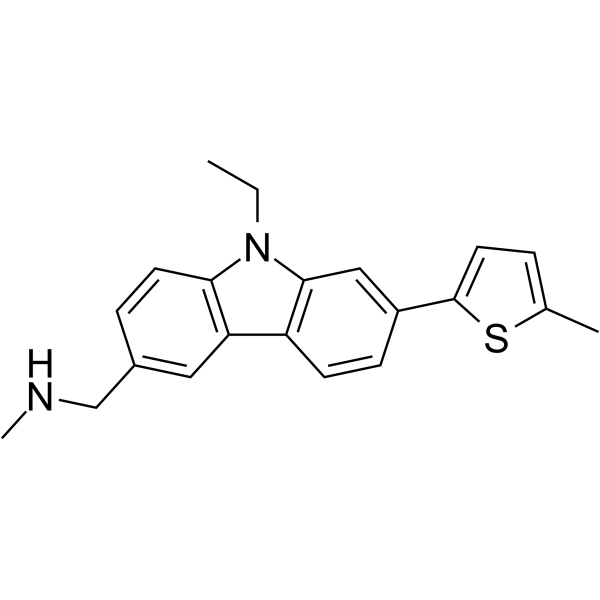
PhiKan 083 hydrochloride
El clorhidrato de PhiKan 083 es un derivado de carbazol, que se une a la cavidad superficial y estabiliza Y220C (un mutante de p53), con una Kd de 167 μM y una afinidad de uniÓn relativa (Kd) de 150 μM en células Ln229.
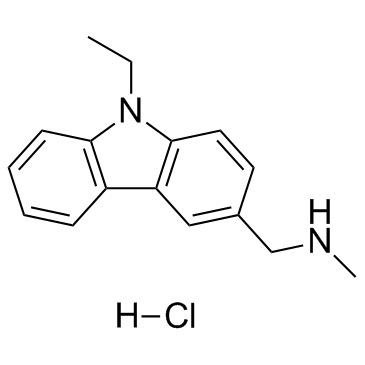
-
GC10538
Pifithrin-α (PFTα)
Un inactivador de p53
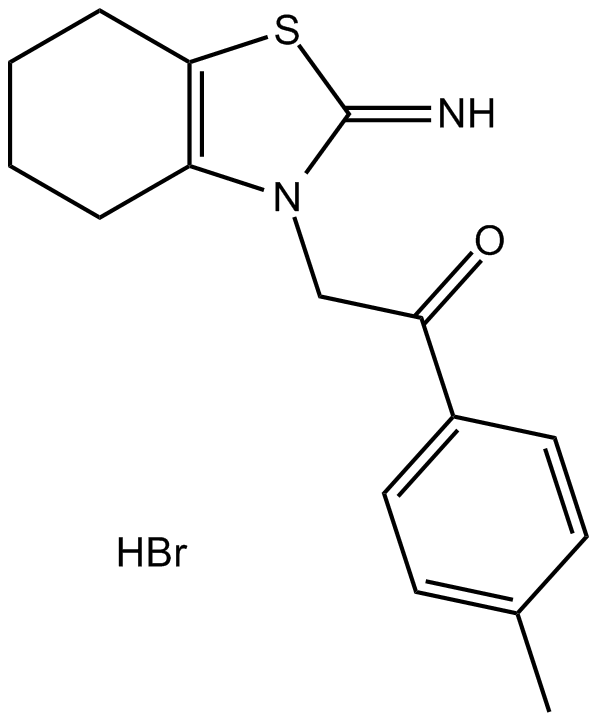
-
GC17262
Pifithrin-β
La pifitrina-β (PFT β) es un potente inhibidor de p53 con una IC50 de 23 μM.
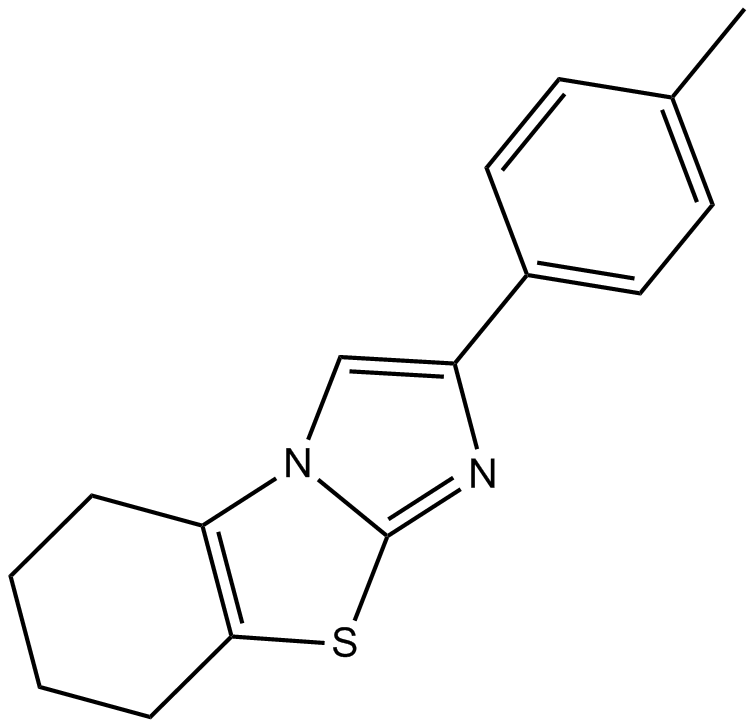
-
GC10282
Piperlongumine
An alkaloid with anticancer and antioxidant activities
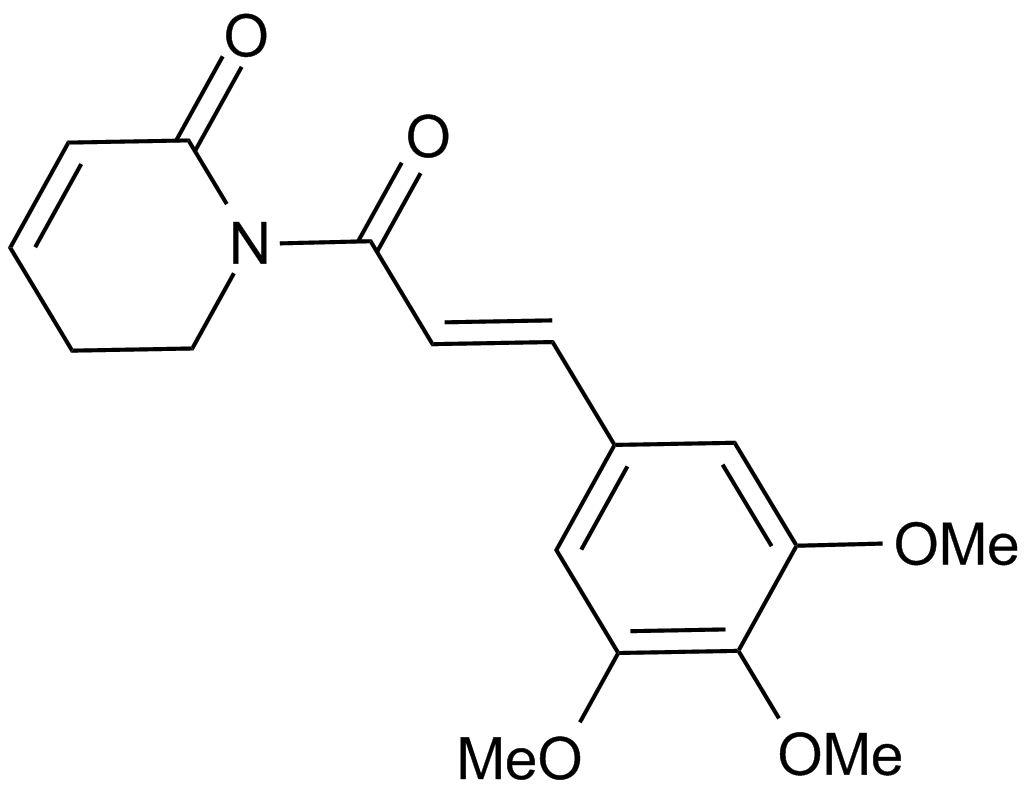
-
GC32946
PK11007
PK11007 es un alquilante de tiol suave con actividad anticancerÍgena. PK11007 estabiliza p53 a través de la alquilaciÓn selectiva de dos cisteÍnas expuestas en la superficie sin comprometer su actividad de uniÓn al ADN. PK11007 induce la muerte de células cancerosas mutantes p53 al aumentar los niveles de especies reactivas de oxÍgeno (ROS).
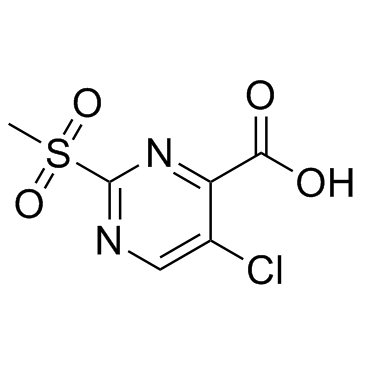
-
GC44652
PK7242 (maleate)
The protein p53, often called the 'guardian of the genome,' is a transcription factor that is activated in response to cellular stress (low oxygen levels, heat shock, DNA damage, etc.) and acts to prevent further proliferation of the stressed cell by promoting cell cycle arrest or apoptosis.
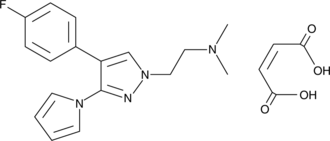
-
GC64768
PK9327
PK9327 es un estabilizador de molécula pequeÑa que se dirige a las mutaciones cancerosas p53 que crean cavidades.
-
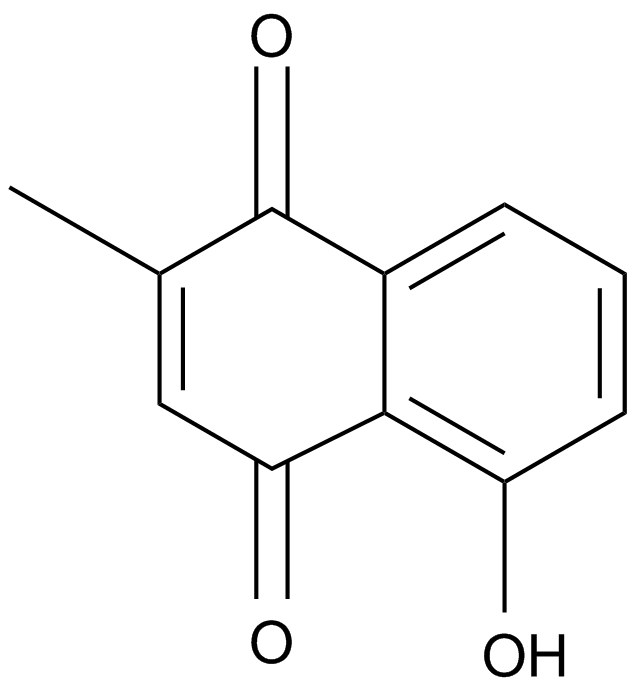
GC10315
Plumbagin
A natural naphthoquinone
-
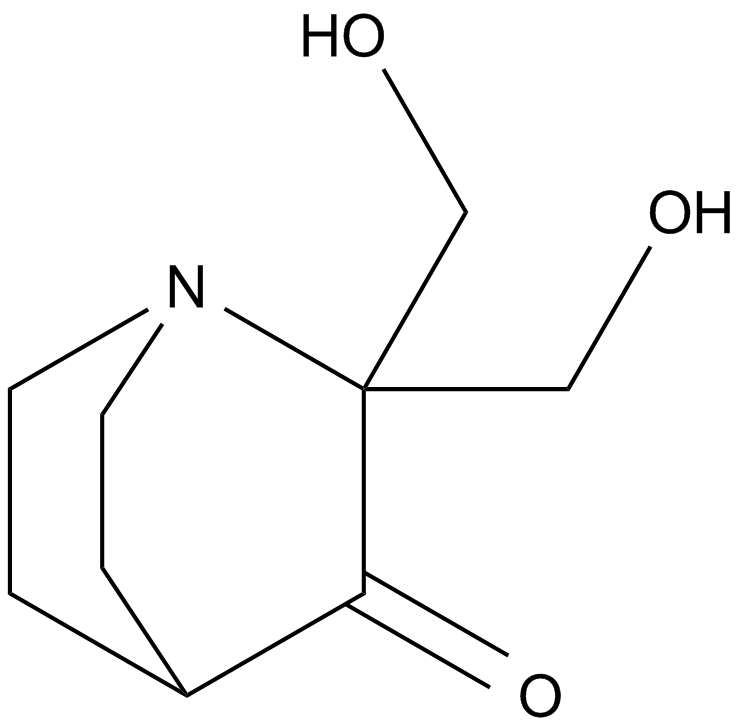
GC12086
PRIMA-1
A re-activator of the apoptotic function of mutant p53 proteins
-
GC37010
PROTAC MDM2 Degrader-1
PROTAC MDM2 Degrader-1 es un degradador MDM2 basado en la tecnologÍa PROTAC. PROTAC MDM2 Degrader-1 se compone de un potente inhibidor de MDM2, enlazador y el ligando de MDM2 para la ubiquitina ligasa E3.
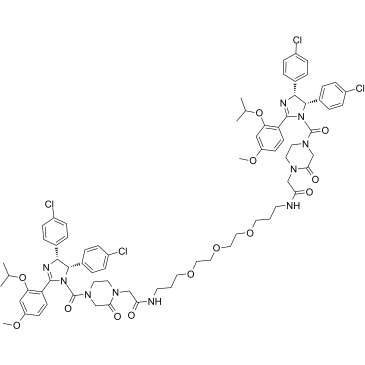
-
GC37011
PROTAC MDM2 Degrader-2
PROTAC MDM2 Degrader-2 es un degradador MDM2 basado en la tecnologÍa PROTAC. PROTAC MDM2 Degrader-2 se compone de un potente inhibidor de MDM2, enlazador y el ligando de MDM2 para la ubiquitina ligasa E3.
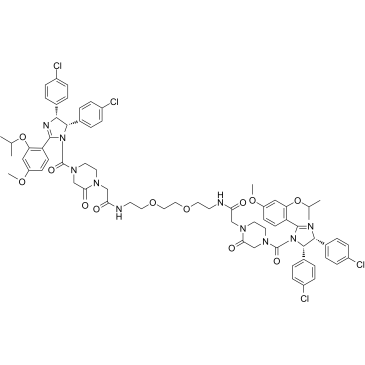
-
GC37012
PROTAC MDM2 Degrader-3
PROTAC MDM2 Degrader-3 es un degradador MDM2 basado en la tecnologÍa PROTAC. PROTAC MDM2 Degrader-3 se compone de un potente inhibidor de MDM2, enlazador y el ligando de MDM2 para la ubiquitina ligasa E3.
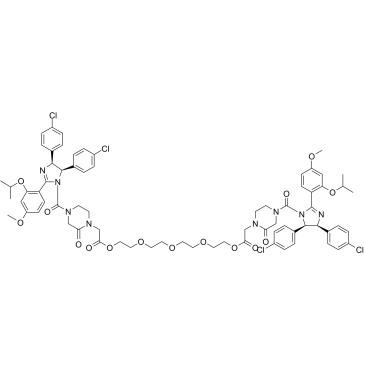
-
GC37013
PROTAC MDM2 Degrader-4
PROTAC MDM2 Degrader-4 es un degradador MDM2 basado en la tecnologÍa PROTAC. PROTAC MDM2 Degrader-4 se compone de un potente inhibidor de MDM2, enlazador y el ligando de MDM2 para la ubiquitina ligasa E3.
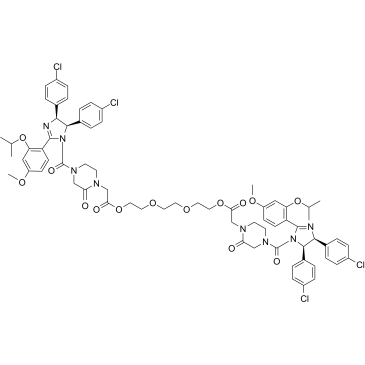
-
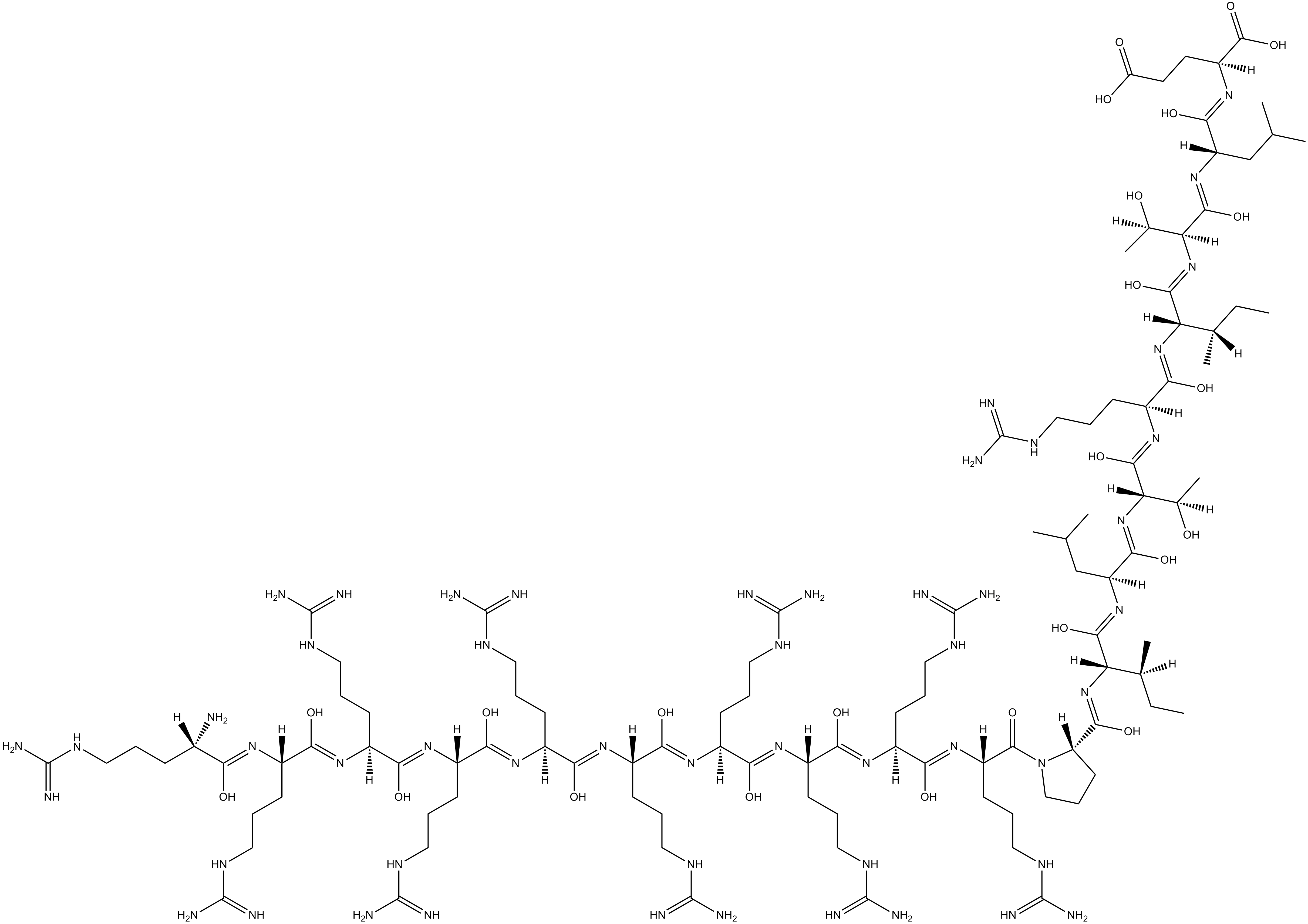
GC15946
ReACp53
ReACp53 podrÍa inhibir la formaciÓn de amiloide p53 y rescatar la funciÓn de p53 en lÍneas celulares de cÁncer.
-
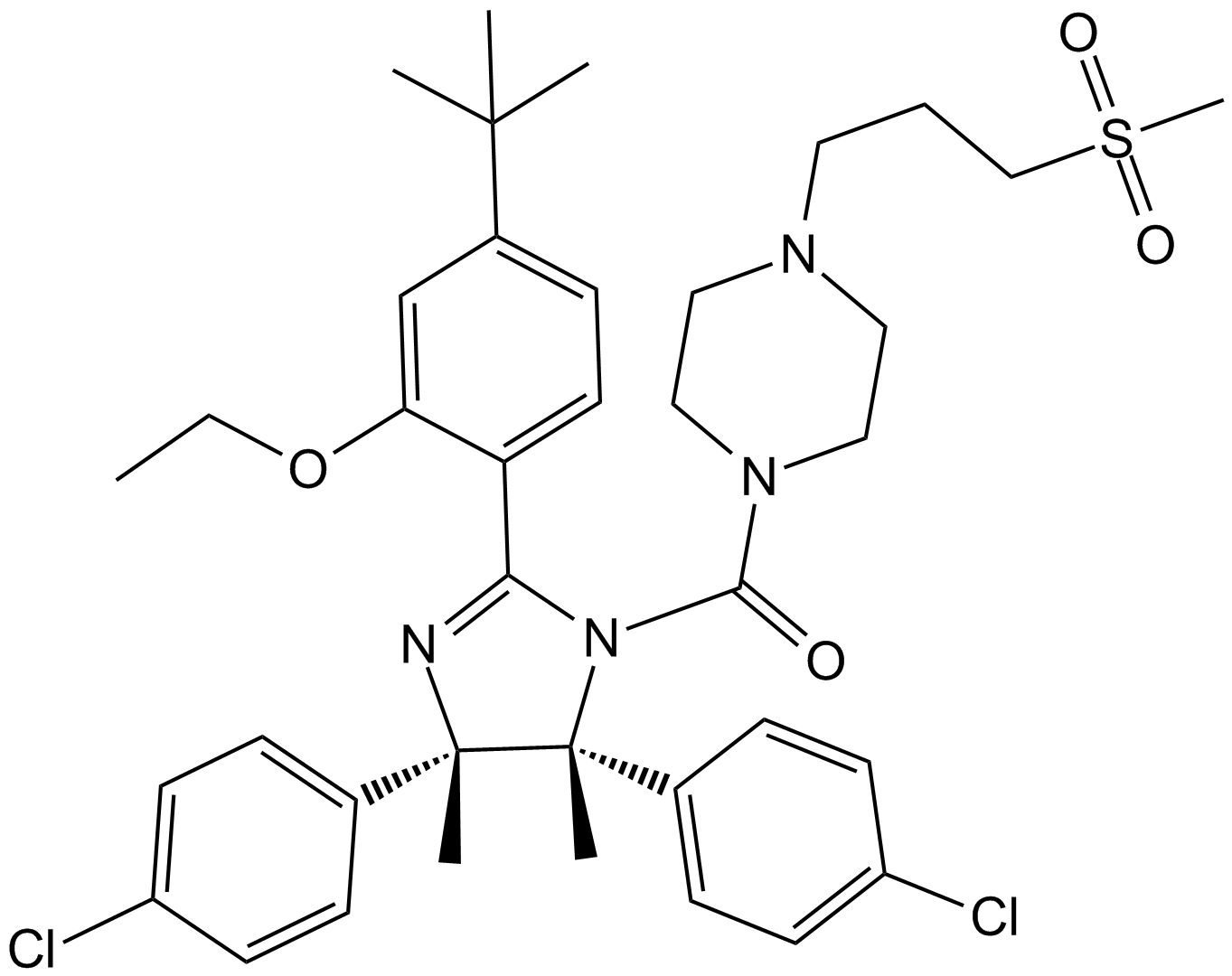
GC13019
RG7112
An inhibitor of the MDM2-p53 interaction
-
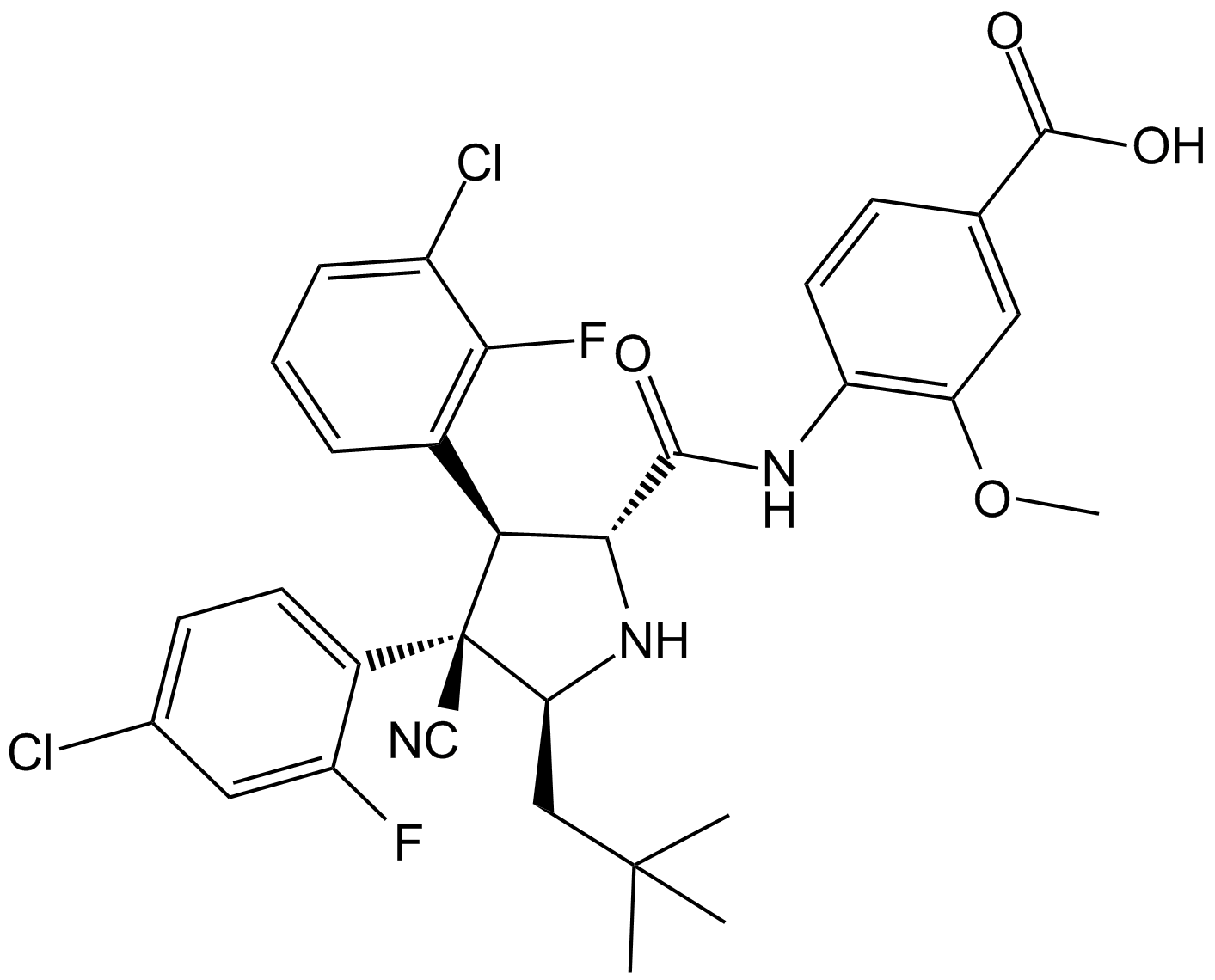
GC11594
RG7388
An inhibitor of the MDM2-p53 interaction
-
GC12793
RITA (NSC 652287)
An inhibitor of the p53-HDM-2 interaction
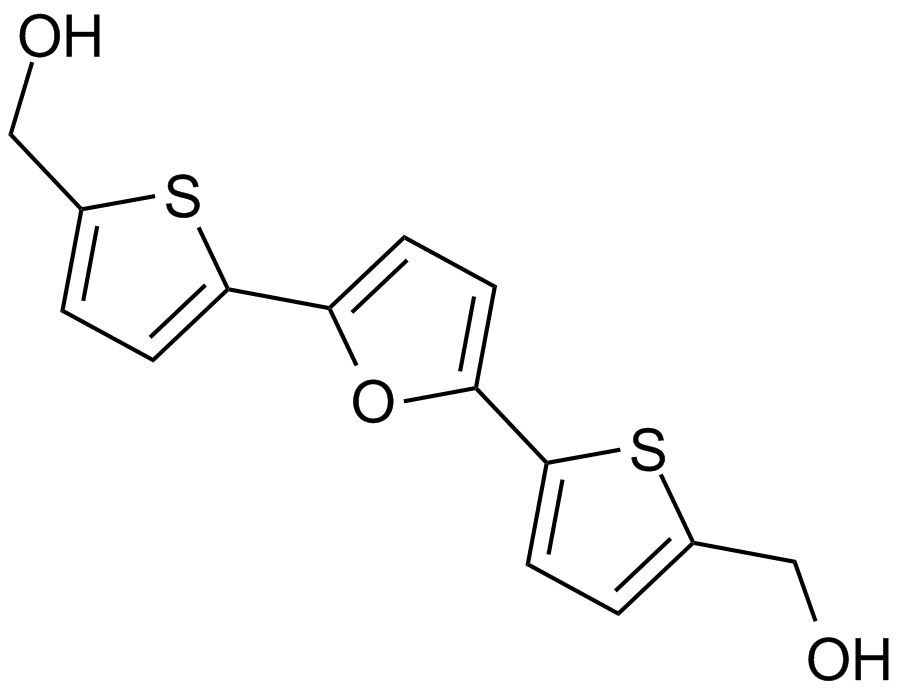
-
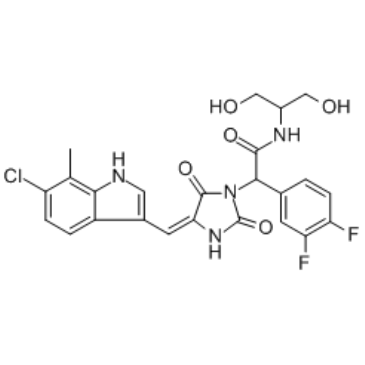
GC37549
RO-5963
RO-5963 es un inhibidor dual de p53-MDM2 y p53-MDMX con IC50 de ~17 nM y ~24 nM, respectivamente.
-
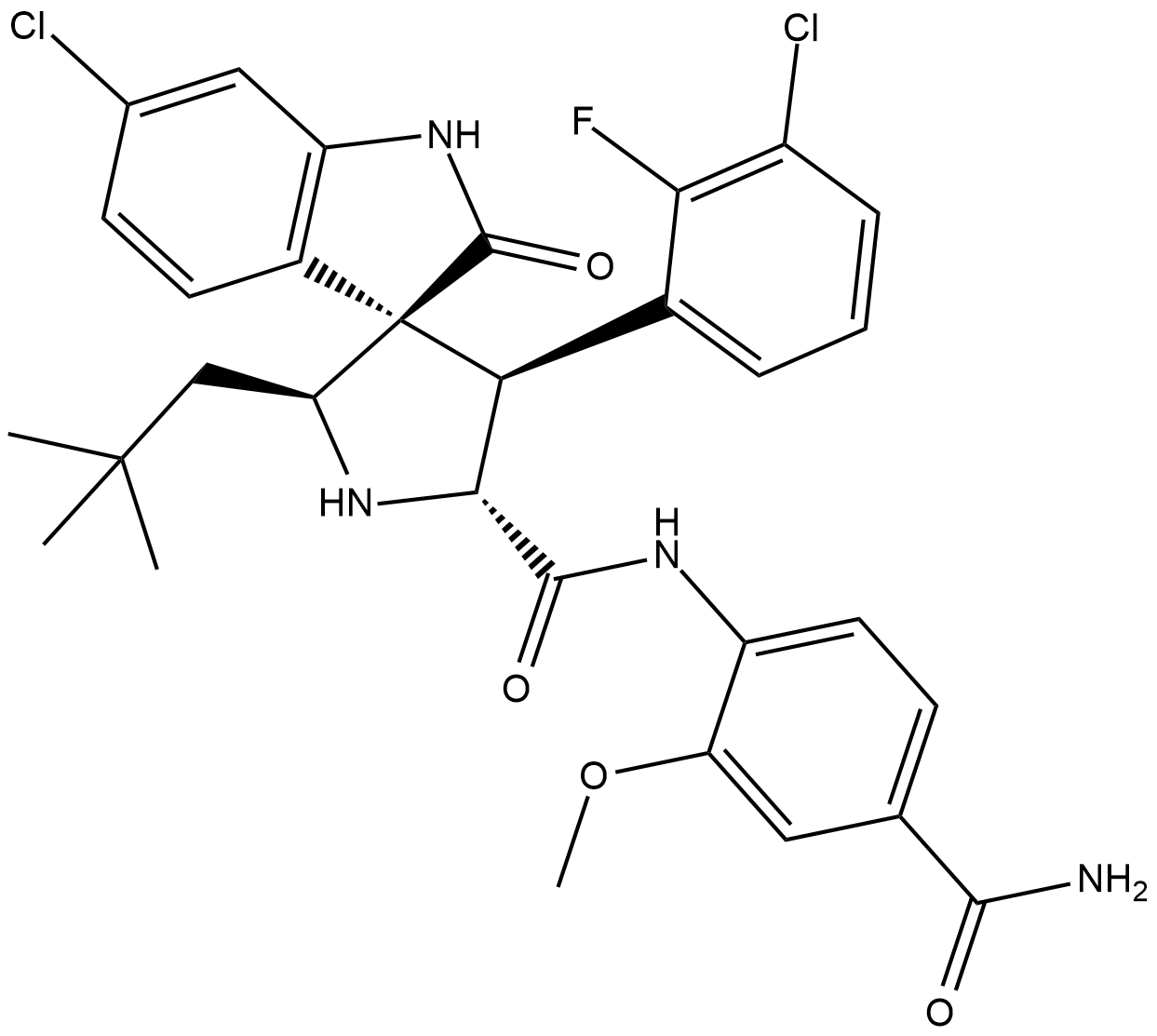
GC19312
RO8994
RO8994 es una serie altamente potente y selectiva de inhibidores de MDM2 de molécula pequeÑa de espiroindolinona, con IC50 de 5 nM (ensayos de uniÓn a HTRF) y 20 nM (ensayos de proliferaciÓn de MTT).
-
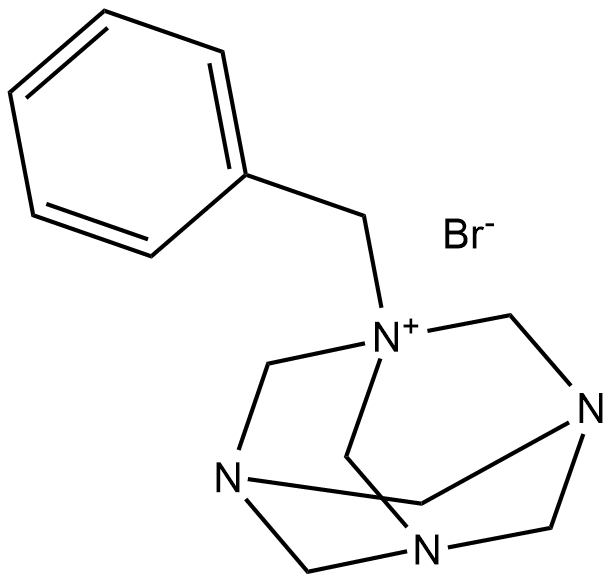
GC18624
Roslin-2
Roslin-2 (bromuro de bencilhexametilenotetramina) es un reactivador de p53 con efectos anticancerÍgenos. Roslin-2 se une a FAK, interrumpe la uniÓn de FAK y p53.
-
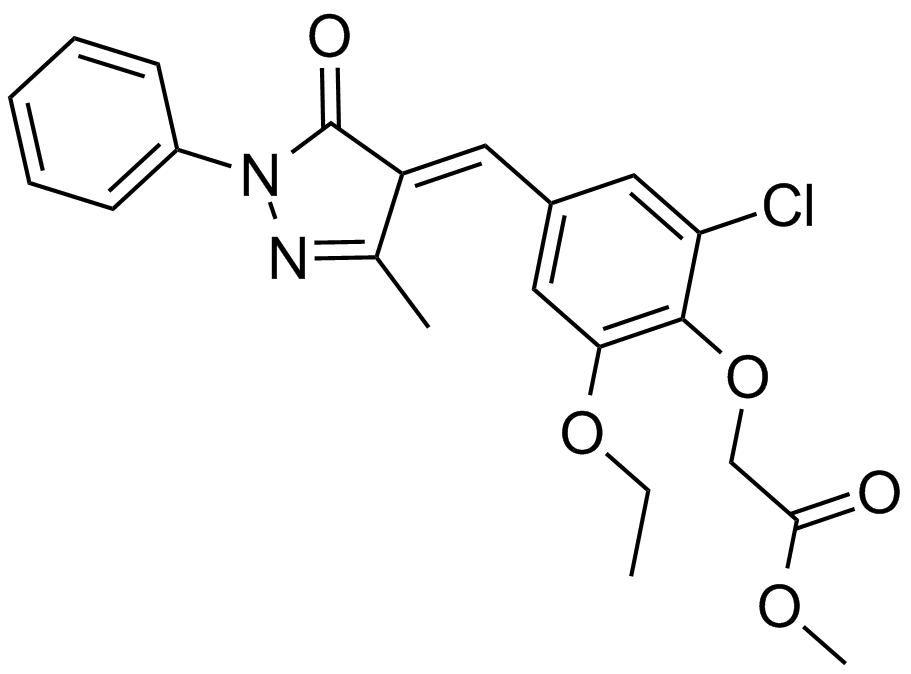
GC13590
SJ 172550
A small molecule inhibitor of MDMX
-
GC65284
SLMP53-1
SLMP53-1 es un reactivador de p53 de tipo salvaje y mutante con actividad antitumoral prometedora. SLMP53-1 media la reprogramaciÓn del metabolismo de la glucosa en las células cancerosas. SLMP53-1 agota la angiogénesis, disminuyendo la formaciÓn de tubos de células endoteliales y los niveles de expresiÓn del factor de crecimiento endotelial vascular (VEGF).
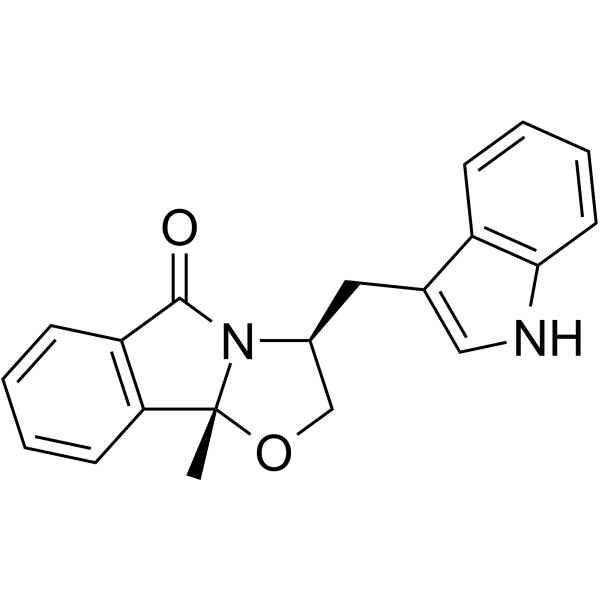
-
GC16371
Solasodine
An alkaloid with diverse biological activities
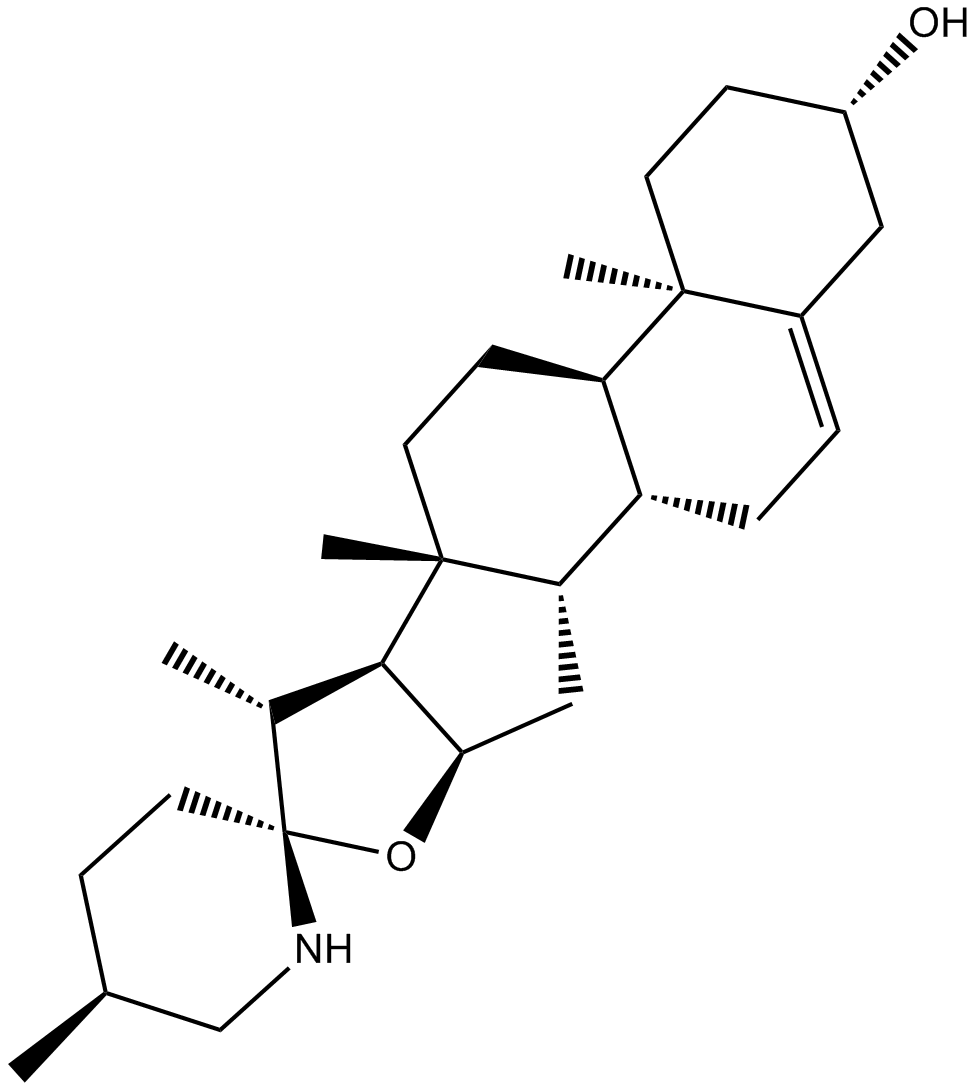
-
GC69968
Sulanemadlin
Sulanemadlin (ALRN-6924) es un potente péptido cíclico basado en p53. Sulanemadlin es un inhibidor de la interacción proteína-proteína entre p53-MDM2, p53-MDMX o p53 y MDM2 y MDMX. Sulanemadlin se puede utilizar en investigaciones sobre el cáncer.
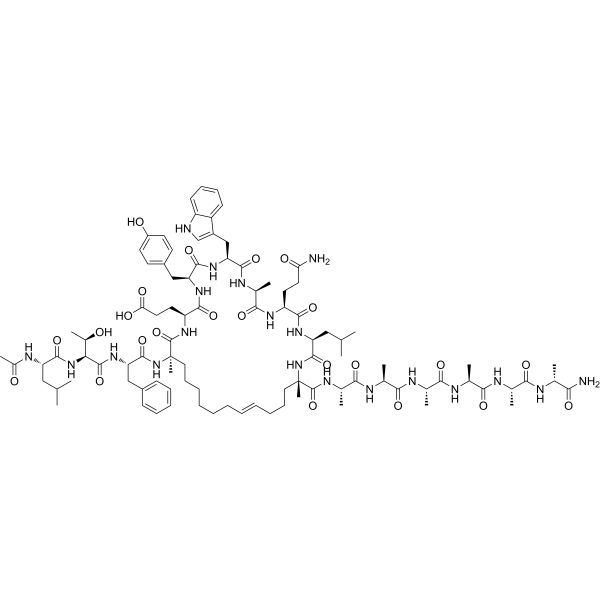
-
GC14165
Tenovin-1
A small molecule activator of p53
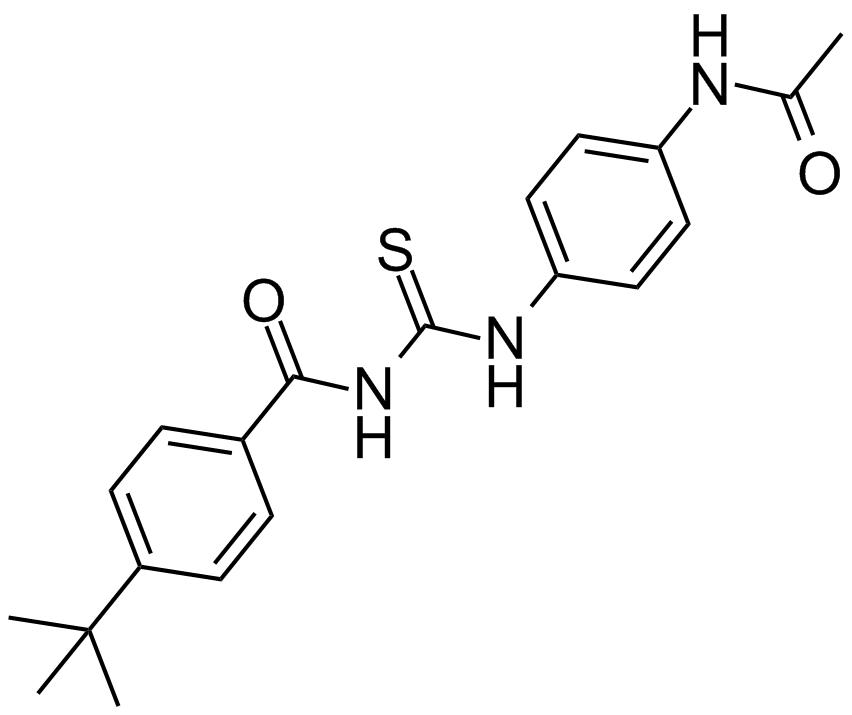
-
GC12337
Tenovin-3
Tenovin-3 es un activador de p53.
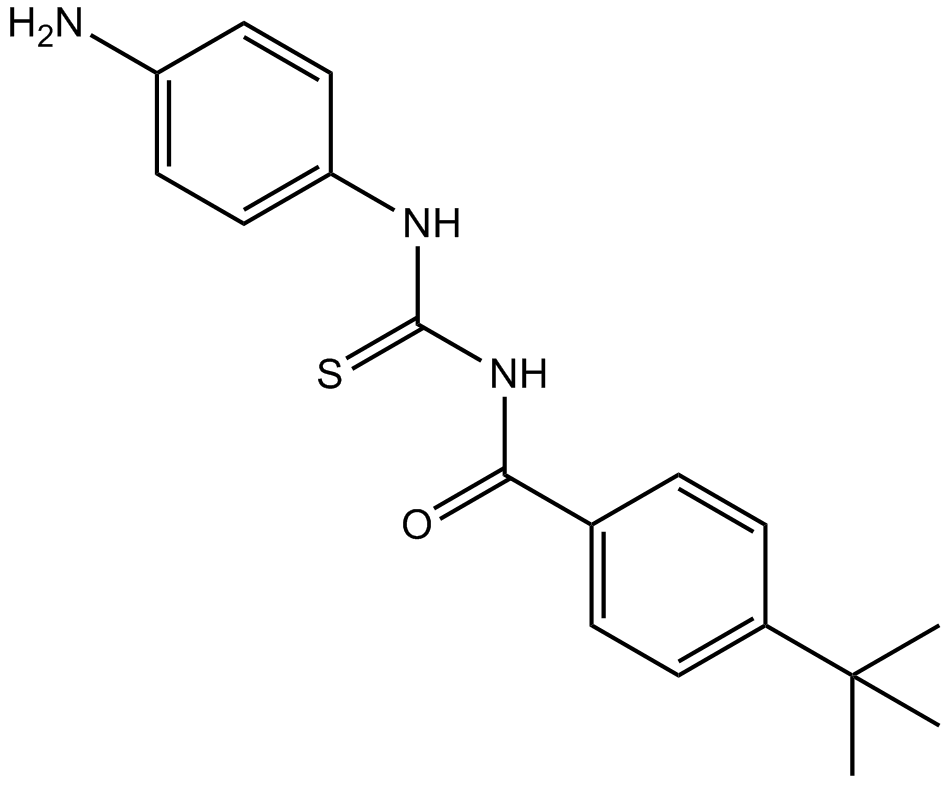
-
GC16436
Tenovin-6
Tenovin-6, un anÁlogo de Tenovin-1, es un activador de la actividad transcripcional de p53. Tenovin-6 inhibe las actividades de la proteÍna desacetilasa de SIRT1, SIRT2 y SIRT3 humanas purificadas con IC50 de 21 μM, 10 μM y 67 μM, respectivamente. Tenovin-6 también inhibe la dihidroorotato deshidrogenasa (DHODH).
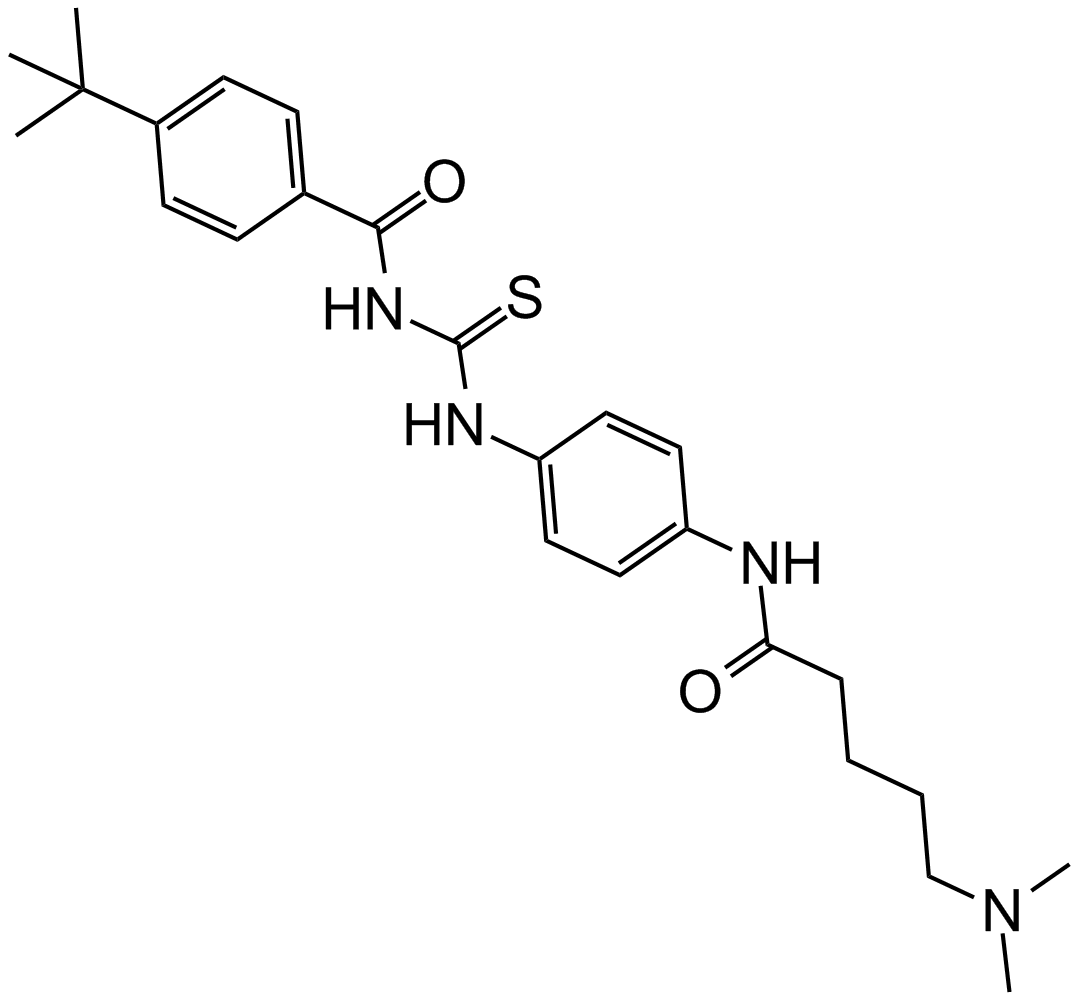
-
GC37761
Tenovin-6 Hydrochloride
El clorhidrato de Tenovin-6, un anÁlogo de Tenovin-1, es un activador de la actividad transcripcional de p53. El clorhidrato de tenovin-6 inhibe las actividades de la proteÍna desacetilasa de SIRT1, SIRT2 y SIRT3 humanos purificados con IC50 de 21 μM, 10 μM y 67 μM, respectivamente. El clorhidrato de Tenovin-6 también inhibe la dihidroorotato deshidrogenasa (DHODH).
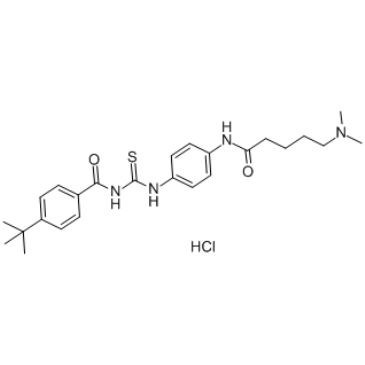
-
GC61352
Triglycidyl isocyanurate
El isocianurato de triglicidilo (TGIC; Teroxirona) es un triepÓxido de triazeno con actividades antiangiogénicas y antineoplÁsicas.
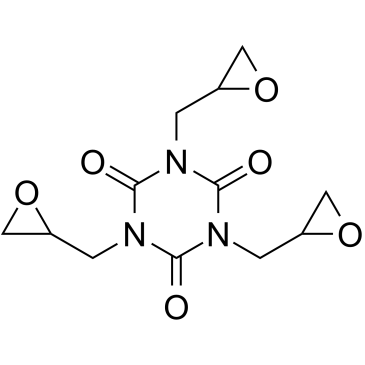
-
GP10028
tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus]
P53 binding protein fragment
![tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus] Chemical Structure tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus] Chemical Structure](/media/struct/GP1/GP10028.png)
-
GC11557
WR 1065
A radioprotective agent
-
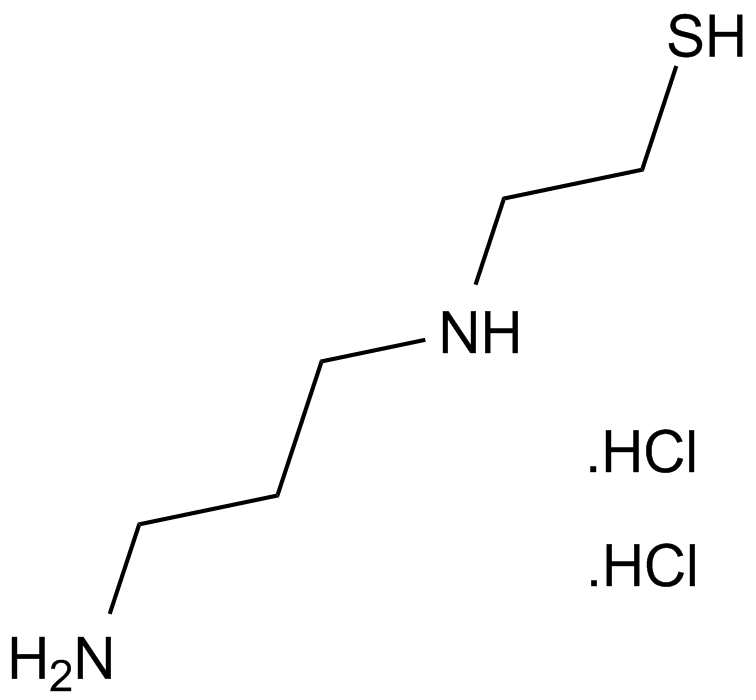
GC19545
WR-1065 dihydrochloride
WR-1065 dihydrochloride can protect normal tissues from the toxic effects of certain cancer drugs and activate p53 through a JNK-dependent signaling pathway.



