Nucleoside Antimetabolite/Analogue
Nucleoside analouges belong to the fmalily of antimetablits that resemble the neleosieds for uptake and metaolism that inibitr dna sytnse and couase cain termination. It is a durg target for cacner and viral infcations.
Products for Nucleoside Antimetabolite/Analogue
- Cat.No. Nombre del producto Información
-
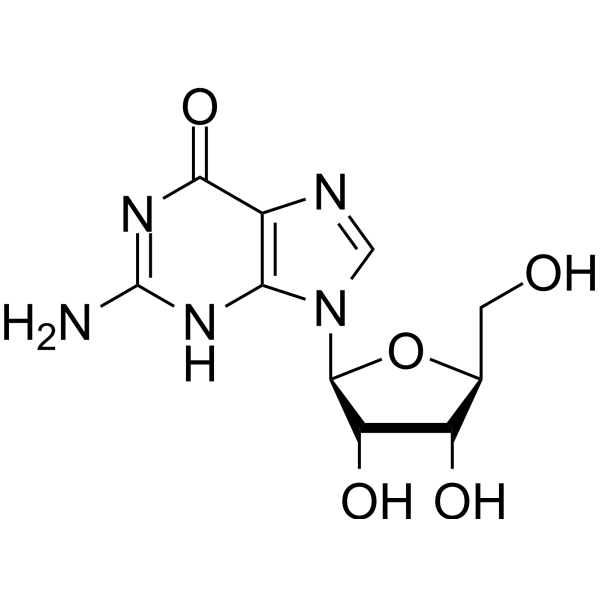
GC69793
(R)-5-O-Benzoyl-1,2-di-O-isopropylidene-alpha-D-xylofuranose
(R)-5-O-Benzoyl-1,2-di-O-isopropilideno-alpha-D-xilofuranosa es un análogo de nucleósido de purina. Los análogos de nucleósidos de purina tienen una amplia actividad antitumoral y se dirigen a los tumores malignos del sistema linfático inactivo. El mecanismo anticancerígeno en este proceso depende de la inhibición de la síntesis del ADN, inducción de apoptosis y otros factores.
-
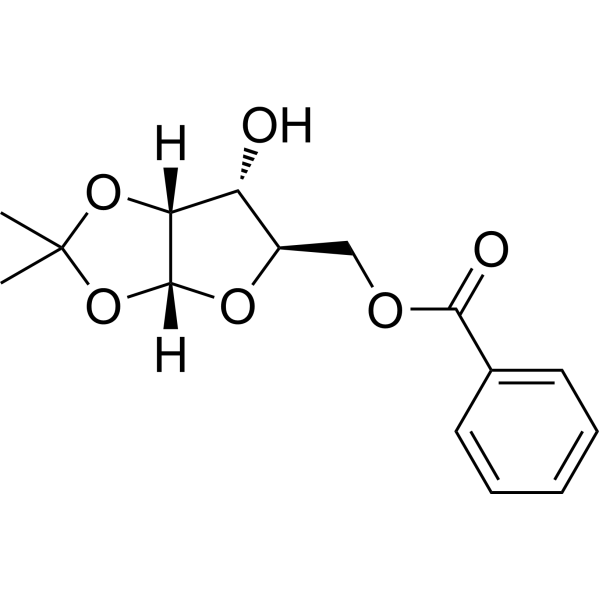
GC68484
1,2-O-Isopropylidene-5-O-p-toluoyl-a-D-xylofuranose
1,2-O-Isopropilideno-5-O-p-toluoil-a-D-xilofuranosa es un análogo de nucleósido de purina. Los análogos de nucleósidos de purina tienen una amplia actividad antitumoral y se dirigen a los tumores malignos del sistema linfático inactivo. El mecanismo anticancerígeno en este proceso depende de la inhibición de la síntesis del ADN, inducción de apoptosis y otros factores.
-
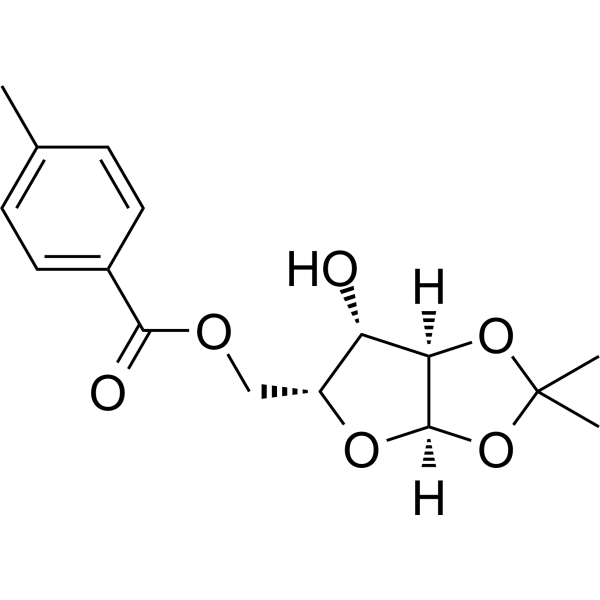
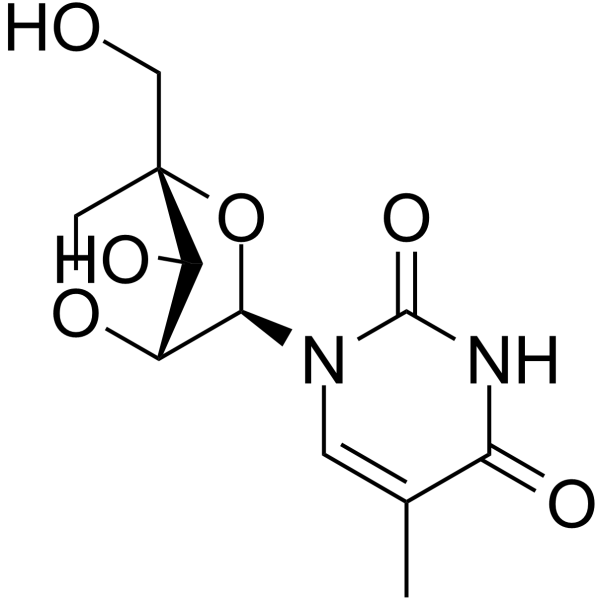
GC65551
1-(2'-O-4-C-Methylene-beta-D-ribofuranosyl)thymine
1-(&2#39;-O-4-C-Metileno-beta-D-ribofuranosil)timina es un nucleósido bicíclico.
-
GC65038
1-Methylinosine
La 1-metilinosina es un nucleÓtido modificado que se encuentra en la posiciÓn 37 del ARNt 3' del anticodÓn del ARNt eucariÓtico.

-
GC65489
2'-F-Bz-dC Phosphoramidite
&2#39;-F-Bz-dC La fosforamidita se puede utilizar en la síntesis de oligorribonucleótidos.
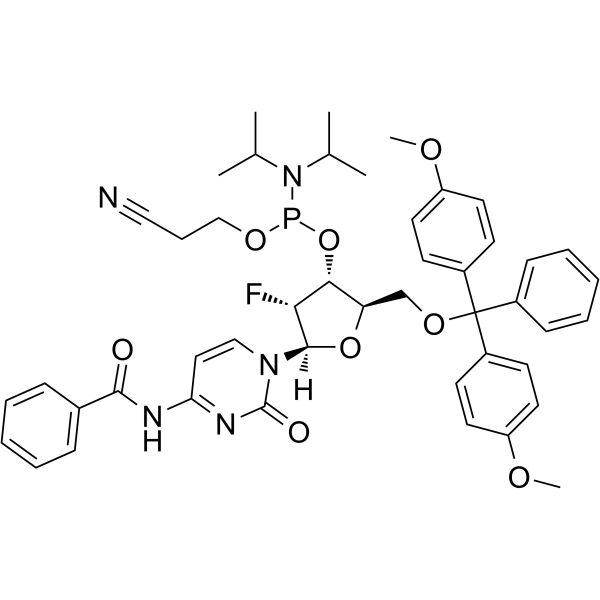
-
GC66651
2'-O,4'-C-Methyleneadenosine
2'-O,4'-C-Metilenadenosina (LNA-A) es un ácido nucleico bloqueado (LNA) y también es un análogo de adenosina.
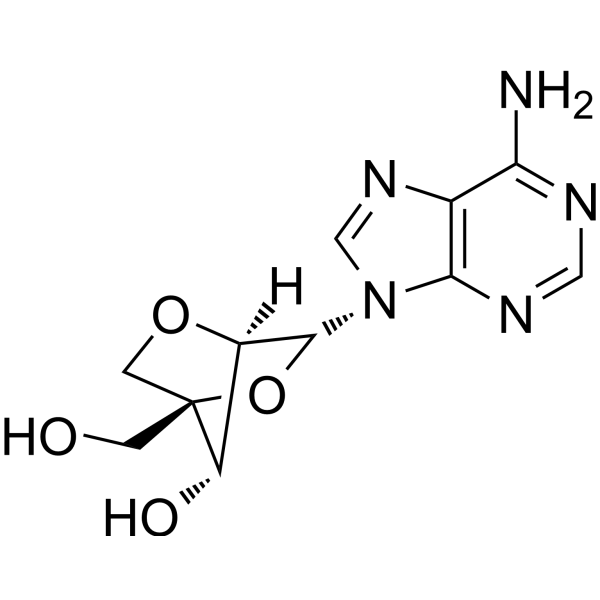
-
GC66654
2'-O,4'-C-Methylenecytidine
2'-O,4'-C-metilencitidina (LNA-C(Bz)) es un análogo de nucleósido bicíclico con conformación de tipo N fija. 2'-O,4'-C-metilencitidina se puede utilizar para sintetizar oligonucleótidos. 2'-O,4'-C-Metilencitidina forma dúplex con hebras complementarias de ADN y ARN.
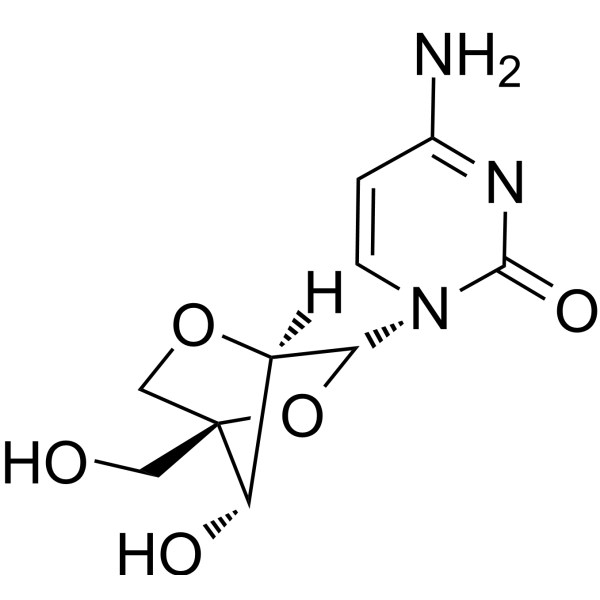
-
GC66655
2'-O,4'-C-Methyleneguanosine
2′-O,4′-C-metilenguanosina (LNA-G) es un análogo de guanina inverso, donde LNA (ácido nucleico bloqueado) es un análogo de ácido nucleico. La modificación de LNA se puede usar en una variedad de aplicaciones, como la afinidad de unión efectiva a secuencias complementarias y una mayor resistencia a la nucleasa que los nucleótidos naturales, lo que ofrece un gran potencial para aplicaciones en el diagnóstico y la investigación de enfermedades. LNA-G también está disponible a través de la ADN polimerasa KOD, que permite la integración de los nucleótidos de LNA-G en la cadena de ADN.
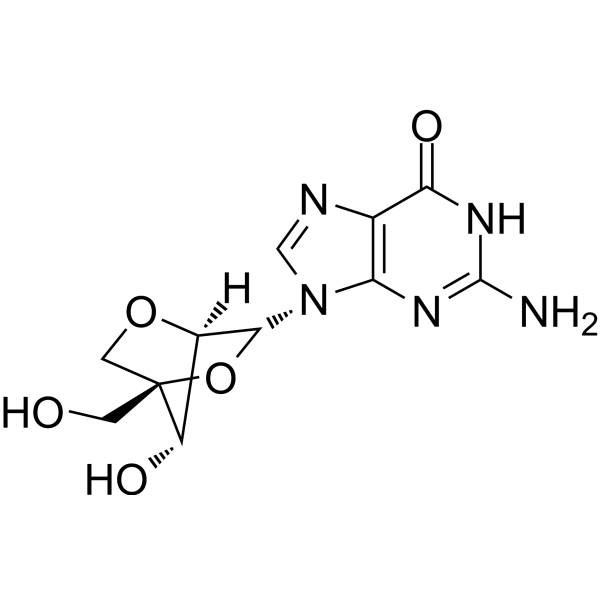
-
GC65170
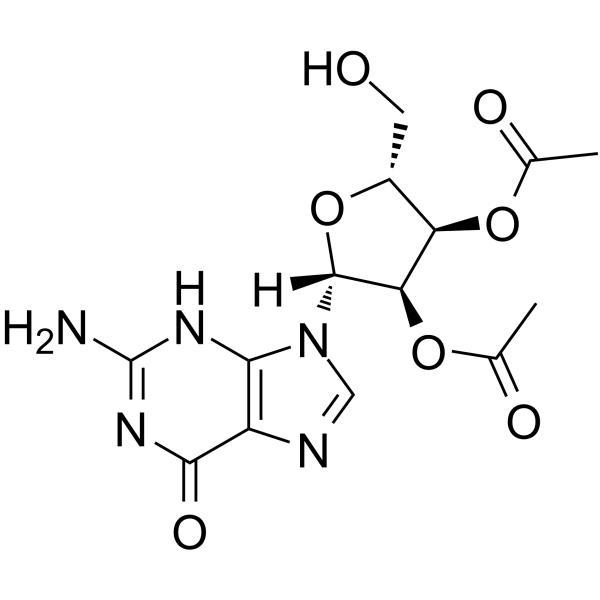
2′,3′-Di-O-acetylguanosine
&2#8242;,3′-Di-O-acetilguanosina es un análogo de nucleósido.
-
GC67384
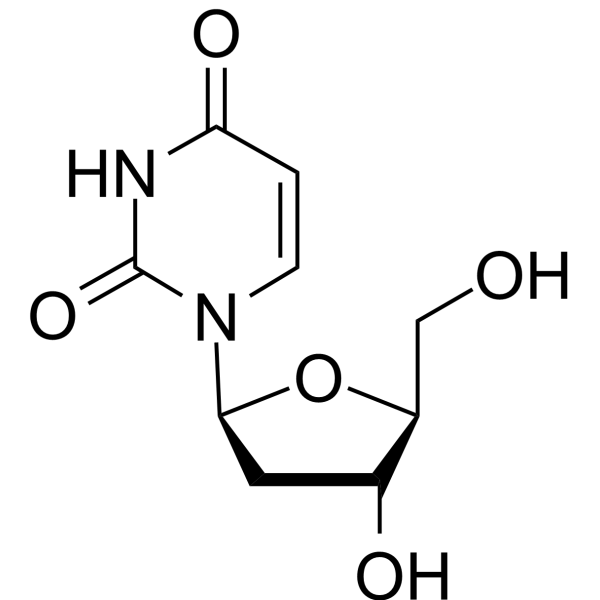
2′-Deoxy-β-L-uridine
2'-Deoxy-β-L-uridina es un análogo de nucleido y un sustrato específico para la enzima viral, no muestra estereoespecificidad contra el herpes simplex 1 (HSV1) timidina quinasa (TK). 2′-Desoxi-β-L-uridina ejerce actividad antiviral a través de la interacción de 5'-trifosfatos con la ADN polimerasa viral.
-
GC61667
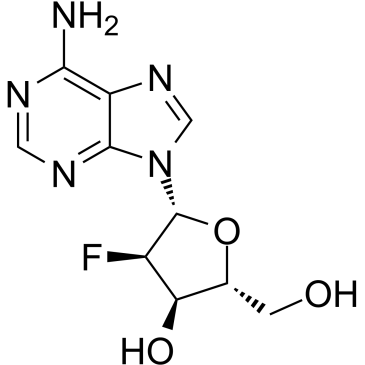
2′-Deoxy-2′-fluoroadenosine
&2#8242;-Desoxi-&22#8242;-fluoroadenosina se puede utilizar para la síntesis de oligonucleótidos modificados con 2′-desoxi-2′-fluoro hibridados con ARN. &2#8242;-Desoxi-&22#8242;-fluoroadenosina se puede escindir de manera eficiente por E. coli nucleósido de purina fosforilasa (PNP) al agente tóxico 2-fluoroadenina (FAde). &2#8242;-Desoxi-&22#8242;-fluoroadenosina muestra una excelente actividad in vivo contra tumores que expresan E. coli PNP.
-
GC62530
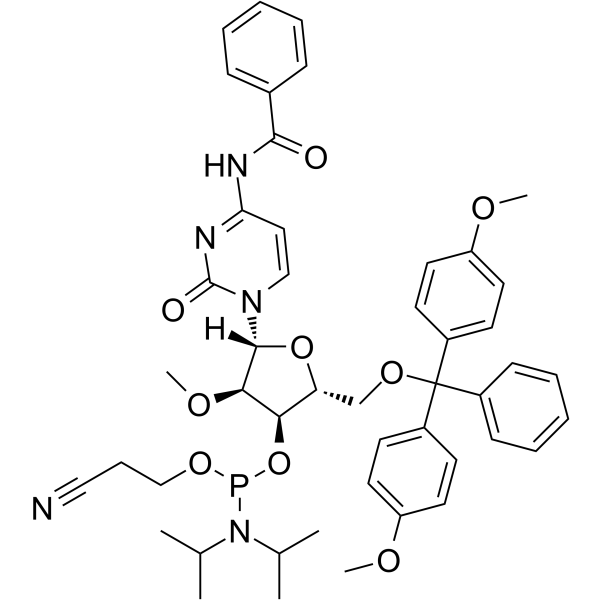
2’-O-Me-C(Bz) Phosphoramidite
&2rsquo;-O-Me-C(Bz) La fosforamidita es un monómero de fosforamidita modificado, que puede usarse para la síntesis de oligonucleótidos.
-
GC62529
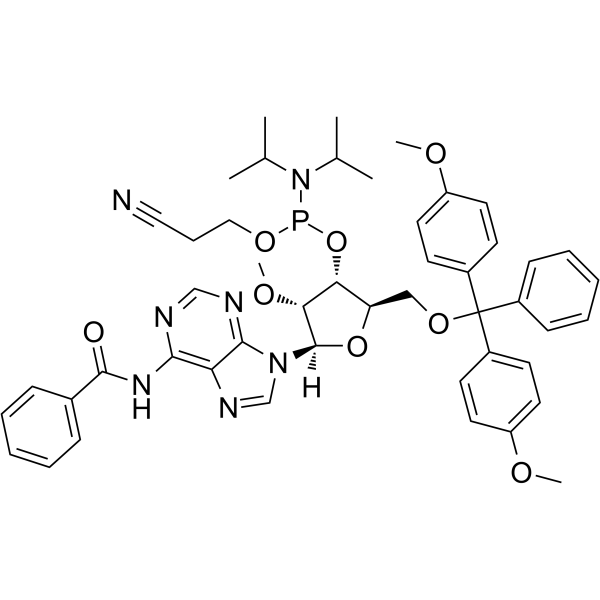
2’-OMe-A(Bz) Phosphoramidite
&2rsquo;-OMe-A(Bz) Phosphoramidita es un monómero de fosforamidita modificada, que se puede utilizar para la síntesis de oligonucleótidos.
-
GC62531
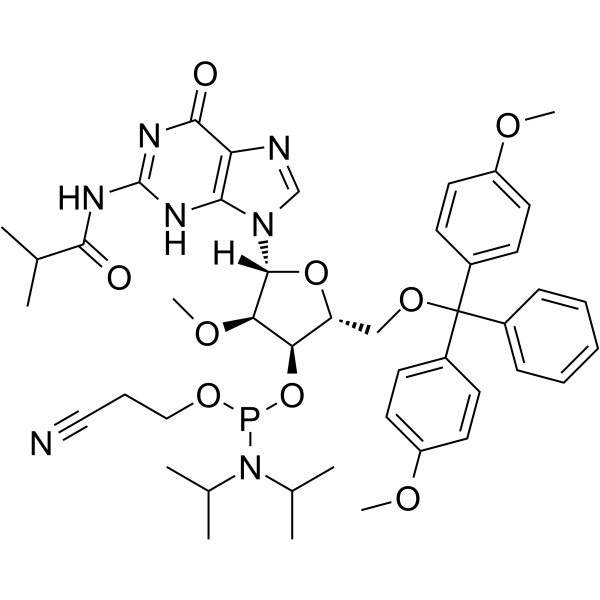
2’-OMe-G(ibu) Phosphoramidite
&2rsquo;-OMe-G(ibu) Phosphoramidita es un monómero de fosforamidita modificada, que se puede utilizar para la síntesis de oligonucleótidos.
-
GC48440
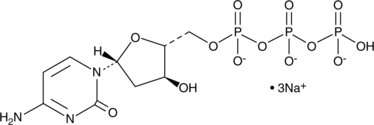
2'-Deoxycytidine-5'-triphosphate (sodium salt)
2'-Desoxicitidina-5'-trifosfato (sal de sodio) (sal trisÓdica de dCTP) es un nucleÓsido trifosfato que se puede utilizar para la sÍntesis de ADN.
-
GC33430
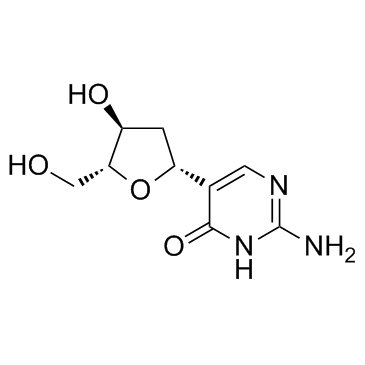
2'-Deoxypseudoisocytidine
La 2'-desoxipseudoisocitidina es un anÁlogo de nucleÓsido.
-
GC35072
2'-O,4'-C-Methyleneuridine
2'-O,4'-C-Metilenouridina (Compuesto 15a) es un nucleÓsido bicÍclico.

-
GC64983
2-Amino-2'-deoxyadenosine
2-Amino-&2#39;-desoxiadenosina es un desoxirribonucleósido utilizado para la síntesis de oligonucleótidos.
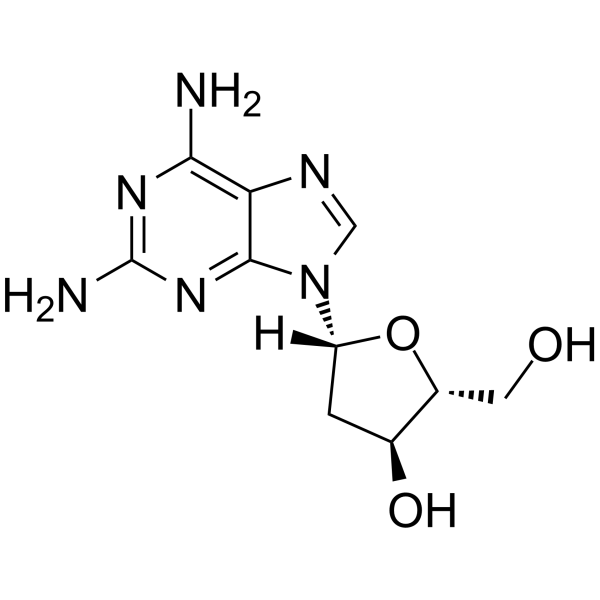
-
GC65083
3'-Azido-3'-deoxy-5-fluorocytidine
3'-Azido-3&2#39;-desoxi-5-fluorocitidina (Compuesto 12) es un derivado de citidina.
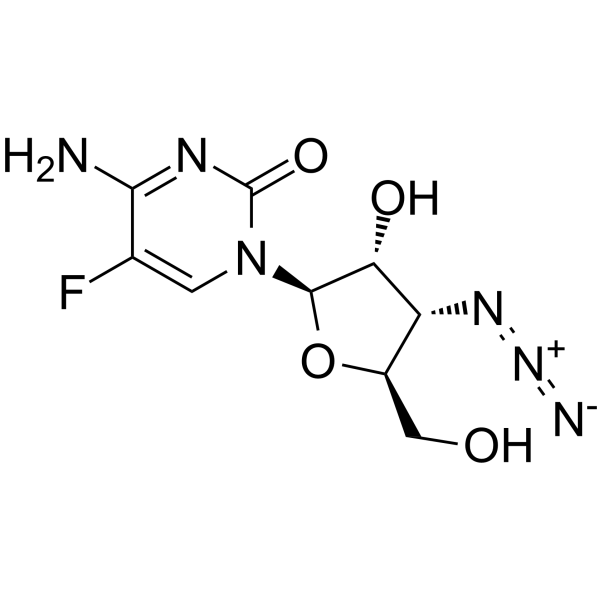
-
GC64985
3'-O-Methylguanosine
3'-O-metilguanosina es un análogo de nucleósido metilado y un terminador de cadena de ARN.
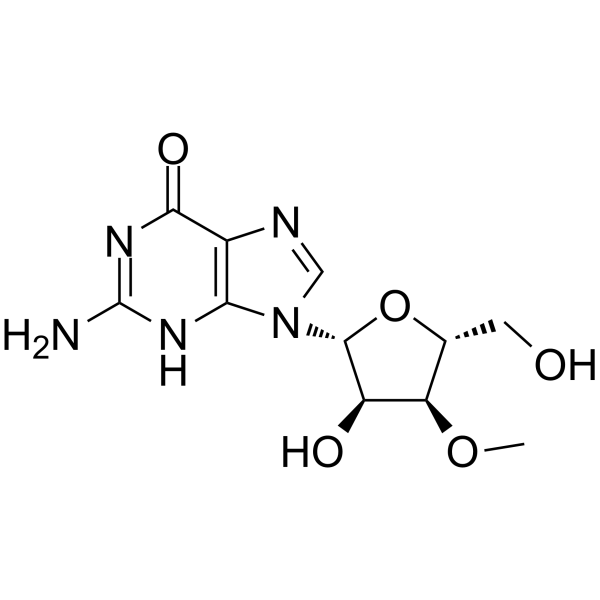
-
GC35105
3'-Azido-3'-deoxy-beta-L-uridine
La 3'-azido-3'-desoxi-beta-L-uridina (Compuesto 25) es un derivado de nucleÓsido.
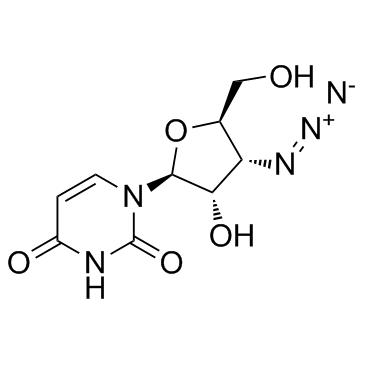
-
GC60023
3'-Deoxyuridine-5'-triphosphate
El 3'-desoxiuridina-5'-trifosfato (3'-dUTP) es un anÁlogo de nucleÓtido que inhibe las ARN polimerasas I y II dependientes de ADN.
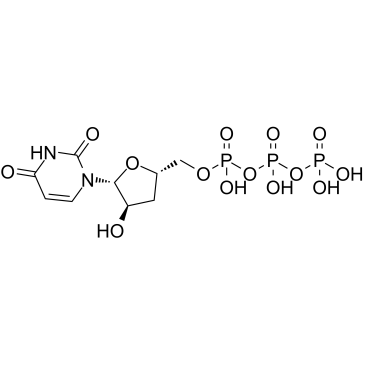
-
GC61862
3'-Deoxyuridine-5'-triphosphate trisodium
El 3'-desoxiuridina-5'-trifosfato trisÓdico (3'-dUTP trisÓdico) es un anÁlogo de nucleÓtido que inhibe las ARN polimerasas I y II dependientes de ADN.
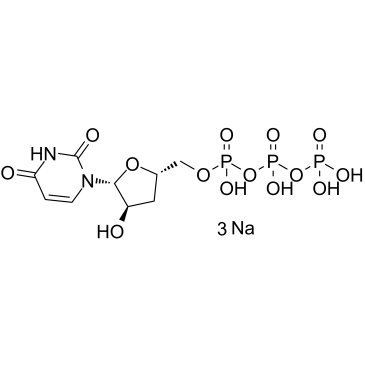
-
GC65084
3-Methylcytidine
La 3-metilcitidina, un nucleÓsido urinario, se puede utilizar como biomarcador de cuatro tipos diferentes de cÁncer: cÁncer de pulmÓn, cÁncer gÁstrico, cÁncer de colon y cÁncer de mama.
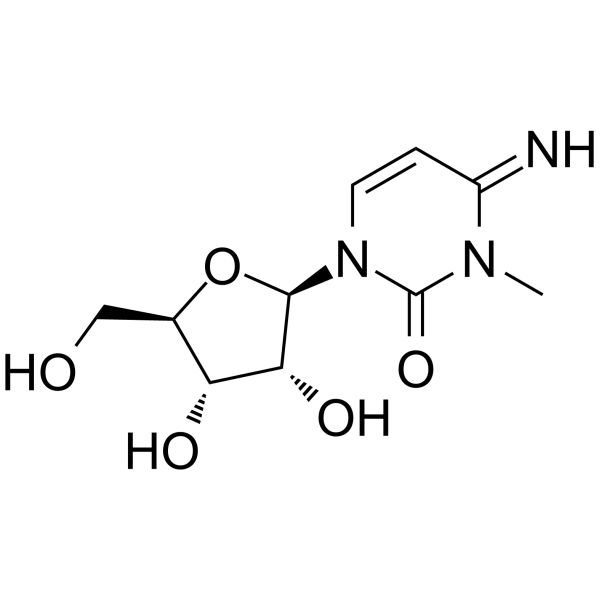
-
GC68557
4-Thio-2'-deoxyuridine
4-Tio-2'-deoxiuridina es un análogo de nucleósido de purina. Los análogos de nucleósidos de purina tienen una amplia actividad antitumoral, dirigida a los tumores malignos del sistema linfático inactivo. El mecanismo anticancerígeno en este proceso depende de la inhibición de la síntesis del ADN, inducción de apoptosis y otros factores.
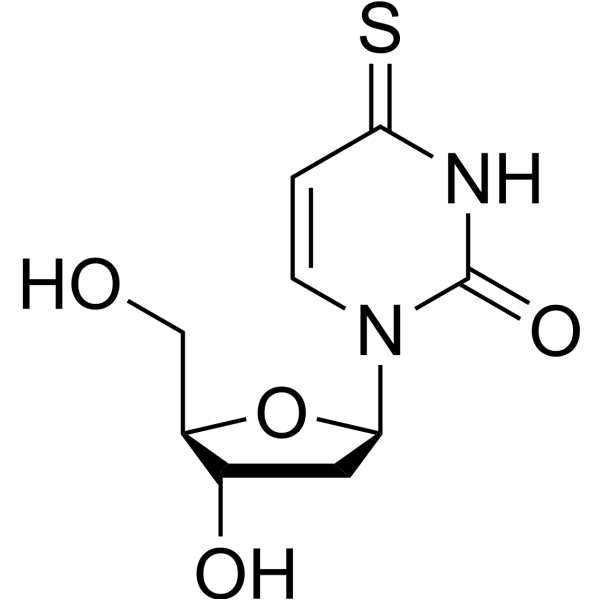
-
GC42474
4-Thiouridine
La 4-tiouridina es un anÁlogo de ribonucleÓsido, se usa ampliamente en anÁlisis de ARN y marcaje de (m)ARN.
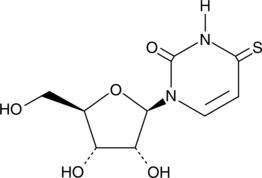
-
GC65520
5'-DMT-3'-TBDMS-ibu-rG
5'-DMT-3&2#39;-TBDMS-ibu-rG es un nucleósido modificado.

-
GC65402
5'-O-DMT-2'-O-TBDMS-Bz-rC
5'-O-DMT-&2#39;-O-TBDMS-Bz-rC es un nucleósido modificado y se puede utilizar para sintetizar ADN o ARN.
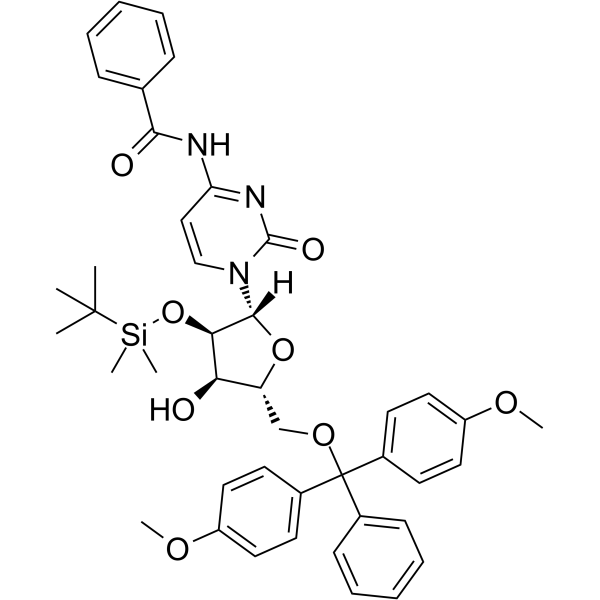
-
GC65164
5'-O-DMT-2'-O-TBDMS-rI
5'-O-DMT-&22#39;-O-TBDMS-rI es un nucleósido modificado.
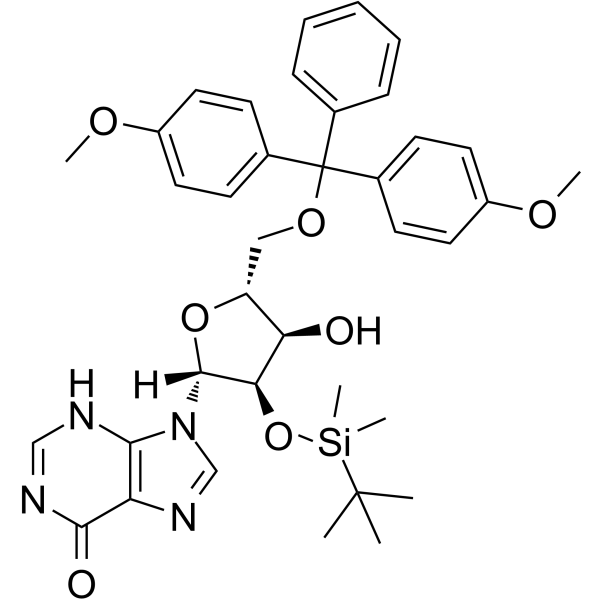
-
GC65396
5'-O-DMT-ibu-dC
5'-O-DMT-ibu-dC se puede utilizar en la síntesis de oligodesoxirribonucleótidos.

-
GC64982
5'-O-DMT-N2-DMF-dG
5'-O-DMT-2'-O-TBDMS-rI es un nucleósido modificado.
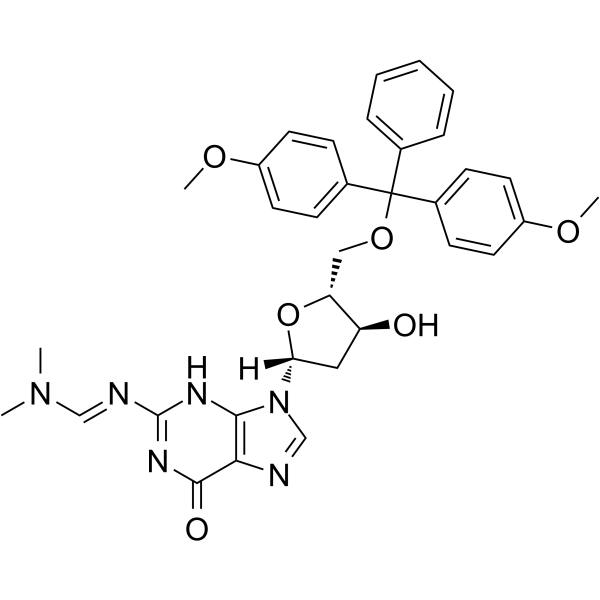
-
GC65482
5'-O-DMT-N6-ibu-dA
5'-O-DMT-N6-ibu-dA se puede utilizar en la síntesis de oligodesoxirribonucleótidos.
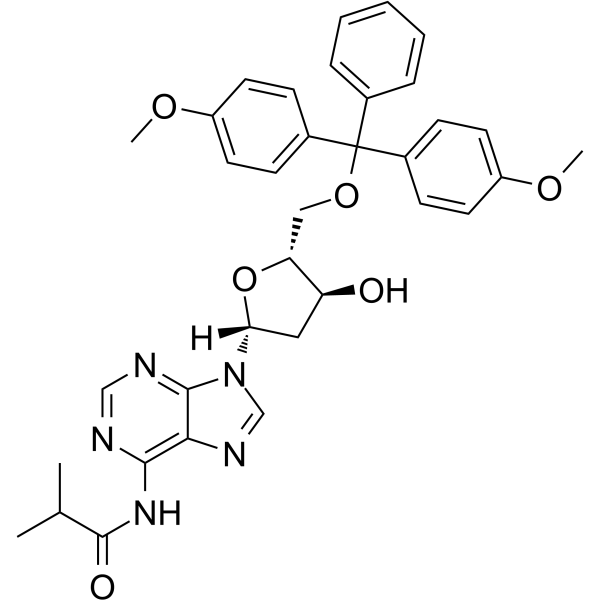
-
GC65562
5'-O-DMT-N6-Me-2'-dA
5'-O-DMT-N6-Me-&2#39;-dA es un nucleósido con efectos protectores y de modificación.
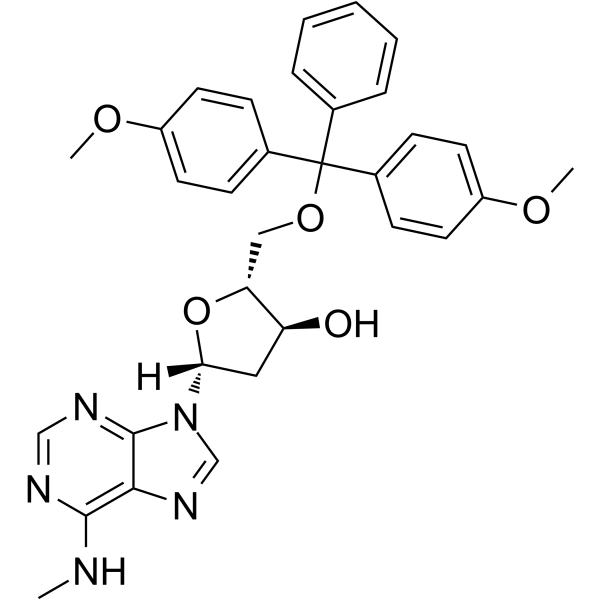
-
GC65401
5'-O-TBDMS-dU
5'-O-TBDMS-dU se puede utilizar en la síntesis de oligorribonucleótidos.
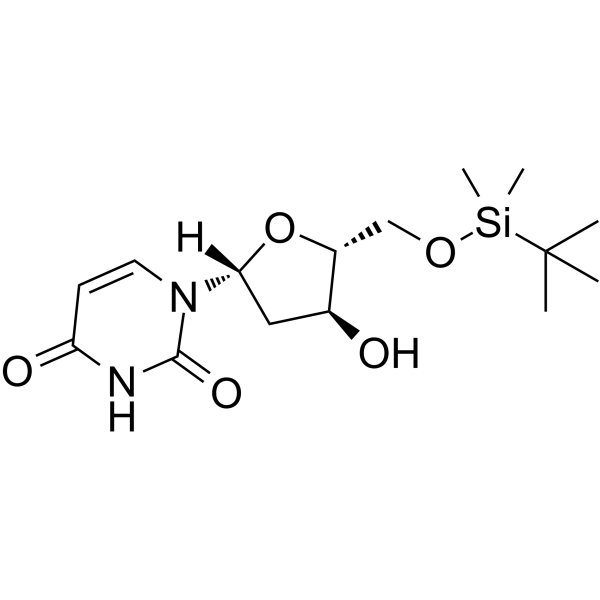
-
GC62157
5’-O-DMT-2’-O-TBDMS-Ac-rC
5’-O-DMT-&2rsquo;-O-TBDMS-Ac-rC es un nucleósido modificado y puede usarse para sintetizar ADN o ARN.
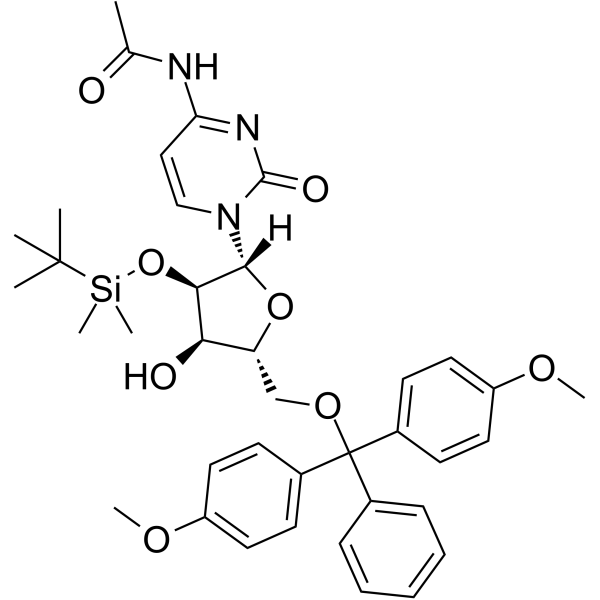
-
GC62149
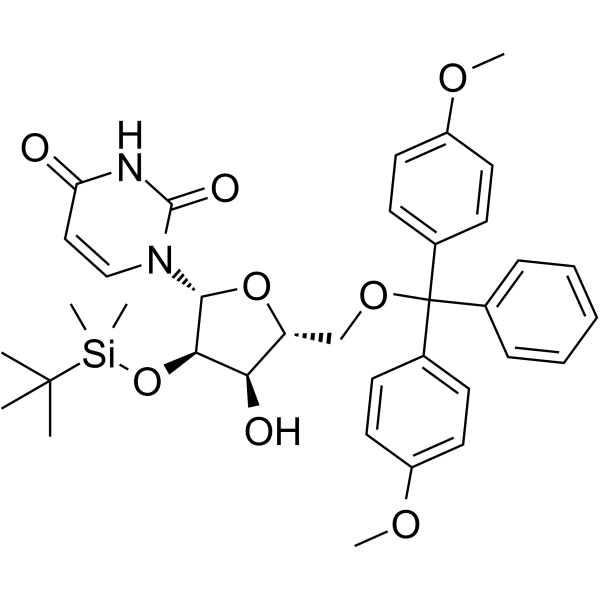
5’-O-DMT-2’-TBDMS-Uridine
5’-O-DMT-&2rsquo;-TBDMS-uridina es un desoxirribonucleósido utilizado para la síntesis de oligonucleótidos.
-
GC62532
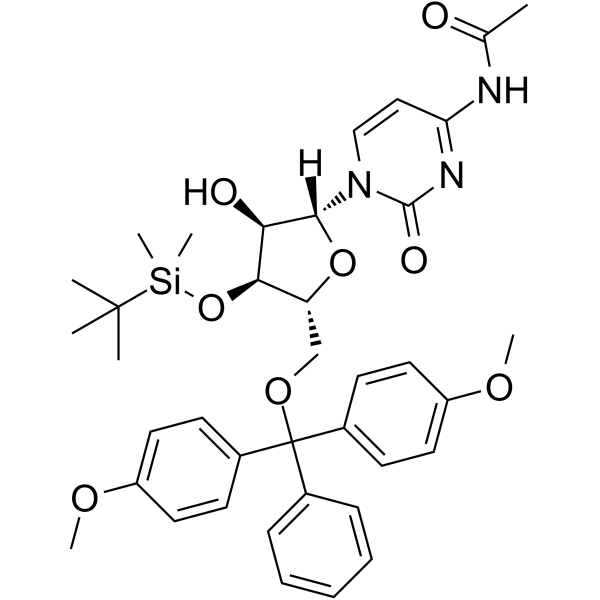
5’-O-DMT-3’-O-TBDMS-Ac-rC
5’-O-DMT-3’-O-TBDMS-Ac-rC es un nucleósido modificado y puede usarse para sintetizar ADN o ARN.
-
GC62573
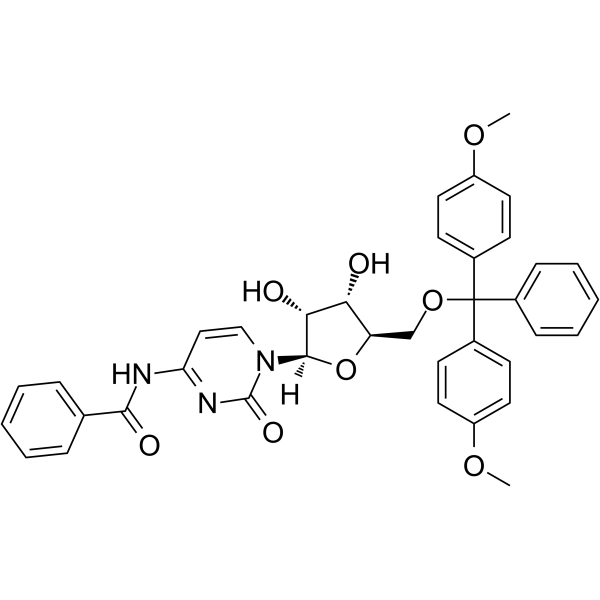
5’-O-DMT-Bz-rC
5'-O-DMT-Bz-Rc es un nucleósido modificado y se puede utilizar para sintetizar ADN o ARN.
-
GC62574
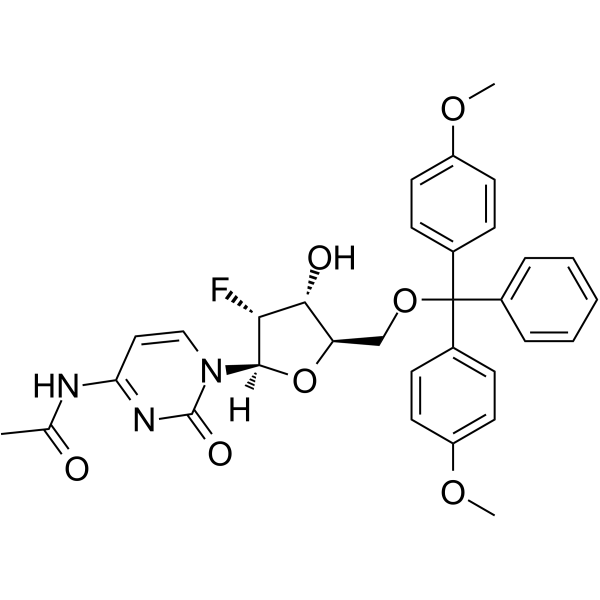
5’-O-DMT-N4-Ac-2’-F-dC
5’-O-DMT-N4-Ac-&2rsquo;-F-dC es un nucleósido modificado y se puede utilizar para sintetizar ADN o ARN.
-
GC62575
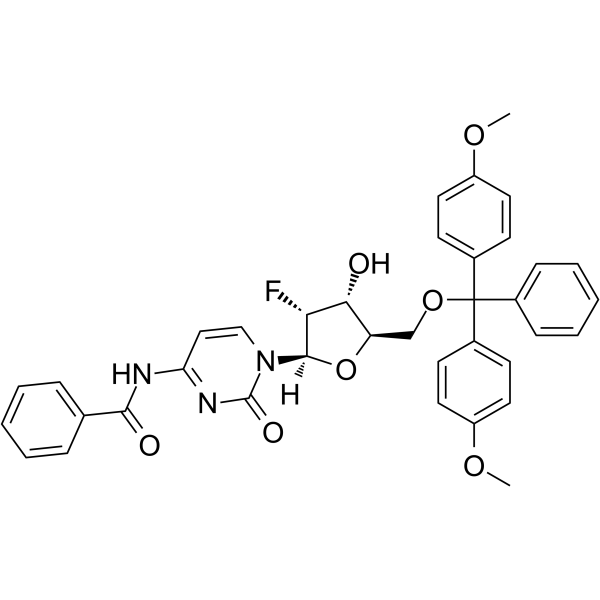
5’-O-DMT-N4-Bz-2’-F-dC
5’-O-DMT-N4-Bz-&2rsquo;-F-dC es un nucleósido con efectos protectores y de modificación.
-
GC62576
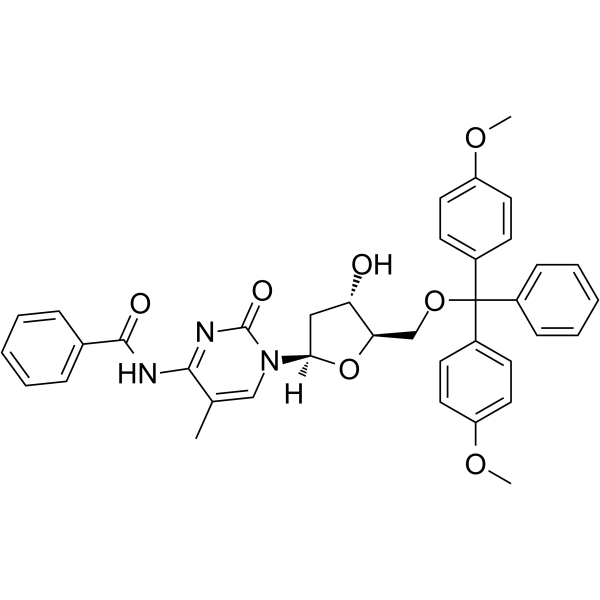
5’-O-DMT-N4-Bz-5-Me-dC
5’-O-DMT-N4-Bz-5-Me-dC es un nucleósido modificado.
-
GC62577
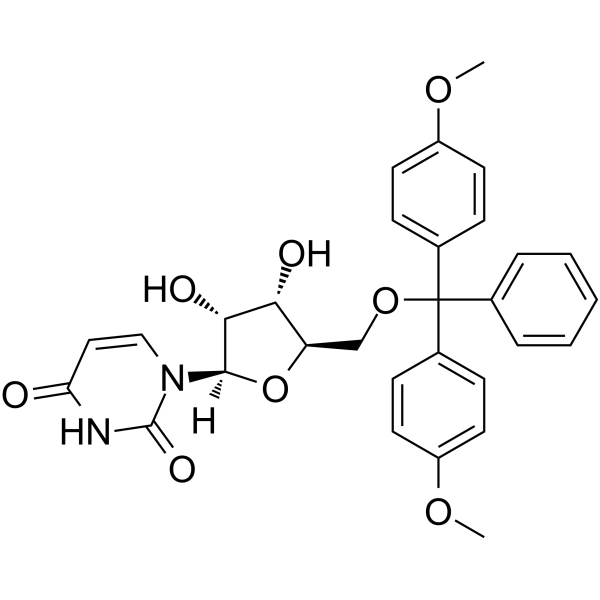
5’-O-DMT-rU
5’-O-DMT-rU es un nucleósido modificado y se puede utilizar para sintetizar ARN.
-
GC62578
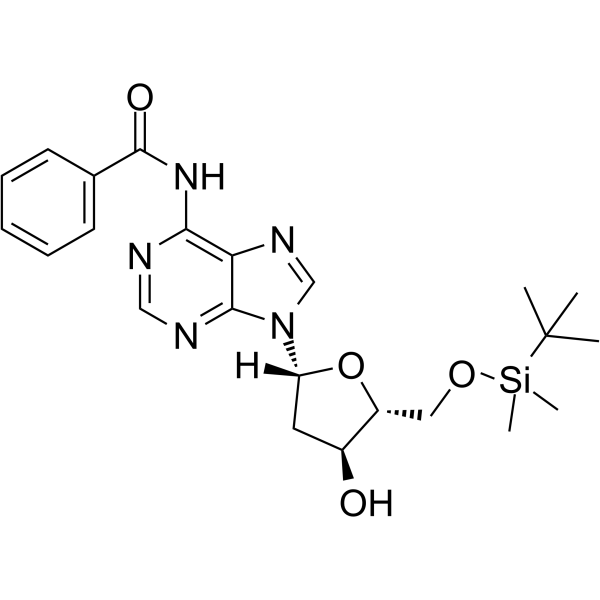
5’-O-TBDMS-Bz-dA
5’-O-TBDMS-Bz-dA es un nucleósido con efectos protectores y de modificación.
-
GC62579
5’-O-TBDMS-dA
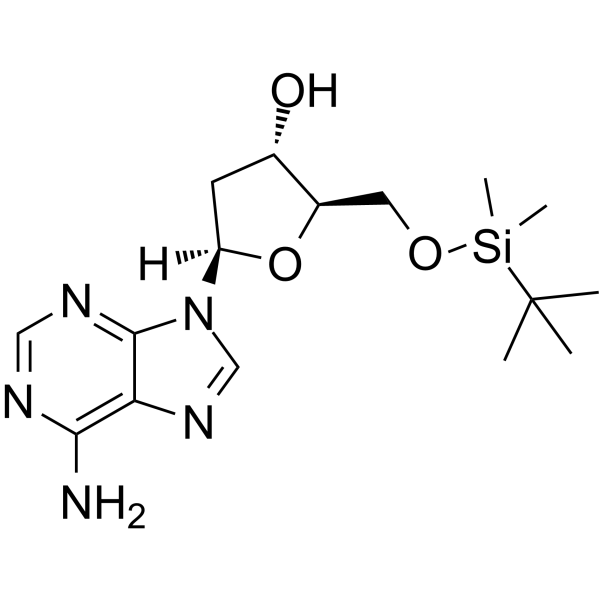
5’-O-TBDMS-dA es un nucleósido modificado y se puede utilizar para sintetizar ADN o ARN.
-
GC62580
5’-O-TBDMS-dG
5’-O-TBDMS-dG es un nucleósido modificado.

-
GC62581
5’-O-TBDMS-dT
5’-O-TBDMS-dT es un nucleósido con efectos protectores y de modificación.

-
GC61432
5'-O-DMT-2'-O-TBDMS-N-Bz-Adenosine
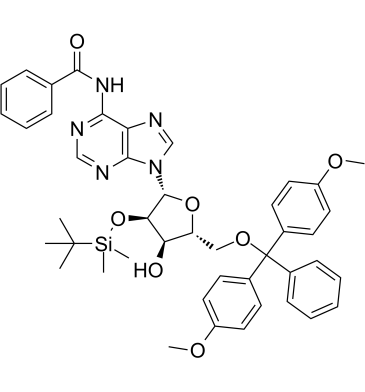
5'-O-DMT-2'-O-TBDMS-N-Bz-Adenosinees un derivado de adenosina y se puede usar como intermediario para la sÍntesis de oligonucleÓtidos.
-
GC64952
5-Aza-7-deazaguanine
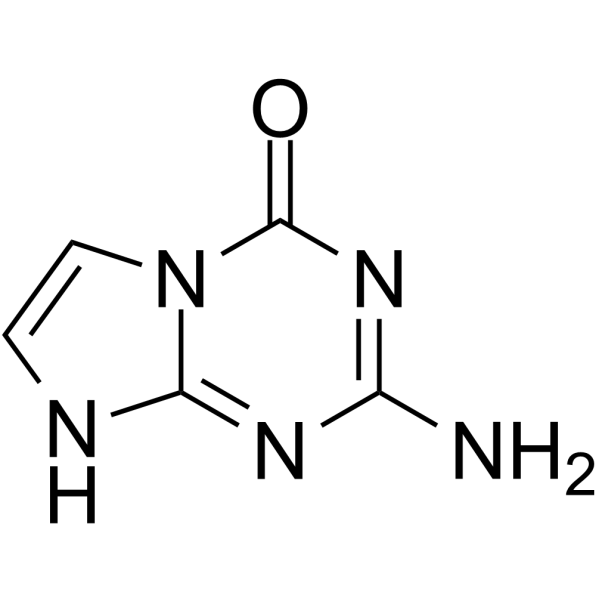
La 5-aza-7-desazaguanina es un sustrato para la fosforilasa de nucleÓsido de purina de E. coli de tipo salvaje (WT) y su mutante Ser90Ala en la sÍntesis de nucleÓsidos modificados con base.
-
GC10946
5-Azacytidine
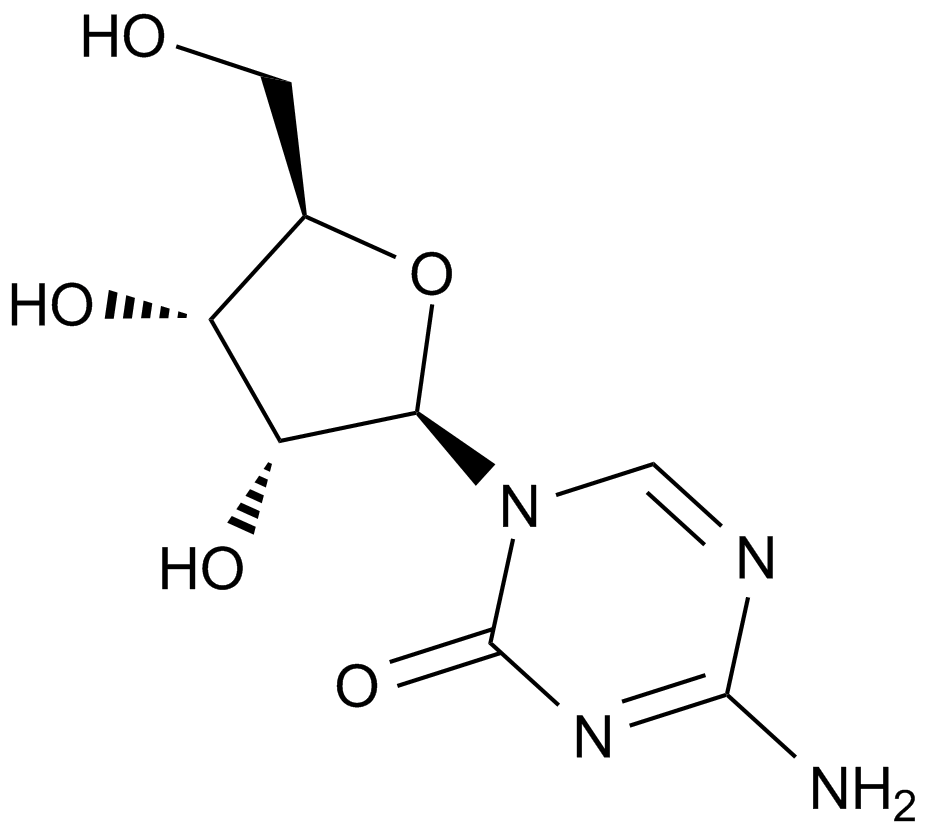
A DNA methyltransferase inhibitor
-
GC11940
5-BrdU
Análogo sintético de timidina.
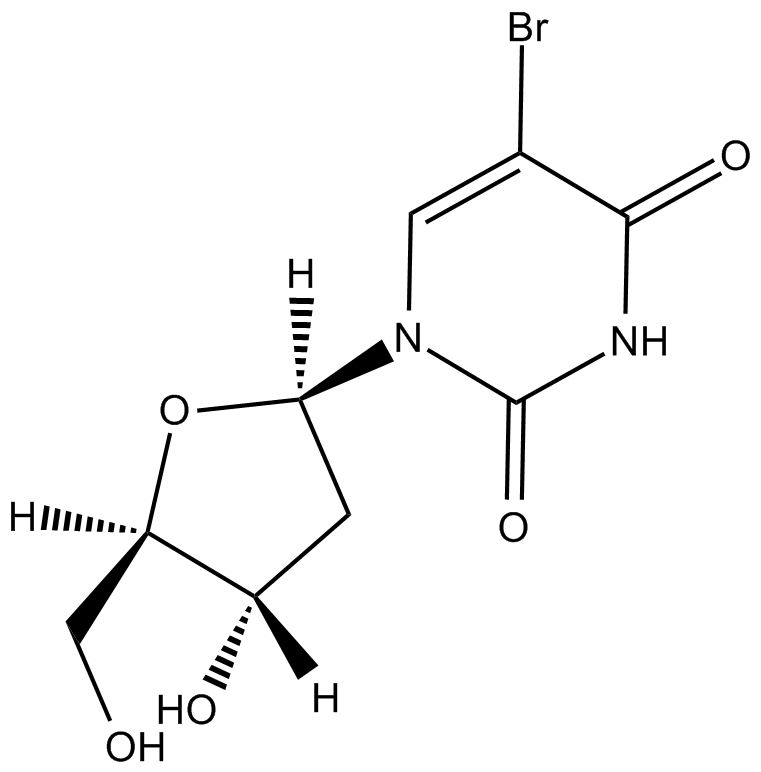
-
GC62547
5-O-TBDMS-N4-Benzoyl-2-deoxycytidine
La 5-O-TBDMS-N4-Benzoil-2-desoxicitidina es un nucleÓsido modificado.
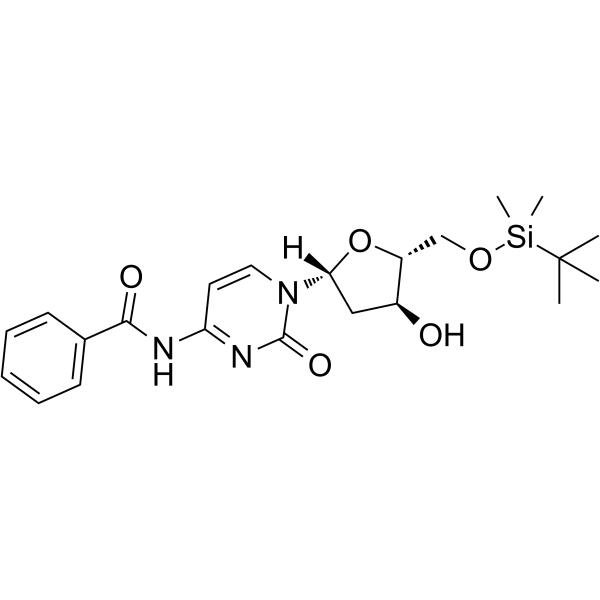
-
GC30548
6-Amino-5-nitropyridin-2-one
La 6-amino-5-nitropiridin-2-ona es una base de piridina y se utiliza como nucleobase del ADN de hachimoji, en el que se empareja con la 5-aza-7-deazaguanina.
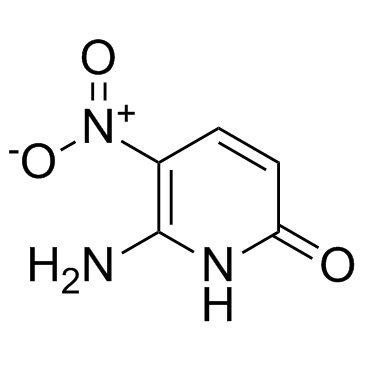
-
GC60031
6-Azathymine
La 6-azatimina, un anÁlogo de 6-nitrÓgeno de la timina, es un potente inhibidor de la D-3-aminoisobutirato-piruvato aminotransferasa.
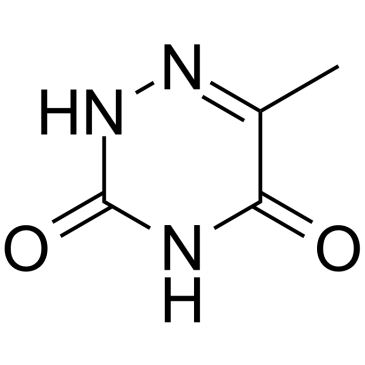
-
GC32975
6-Mercaptopurine hydrate
El hidrato de 6-mercaptopurina (hidrato de mercaptopurina; hidrato de 6-MP) es un anÁlogo de purina que actÚa como antagonista de las purinas endÓgenas y se ha utilizado ampliamente como agente antileucémico y fÁrmaco inmunosupresor.
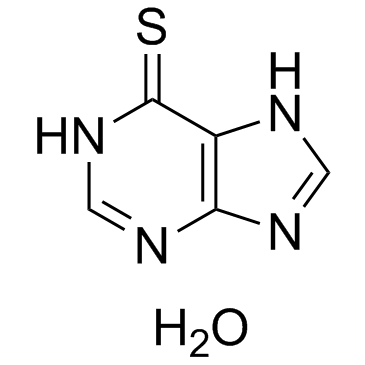
-
GC65082
6-O-Methyl Guanosine
La 6-O-metil guanosina es un nucleÓsido modificado.
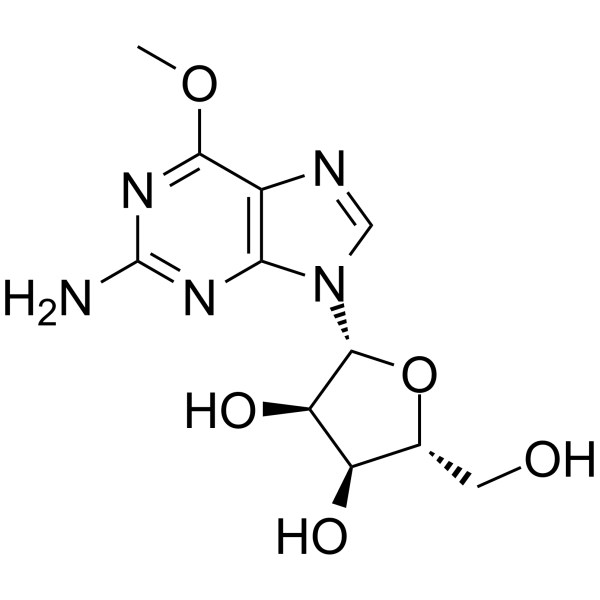
-
GC35171
6-Thioinosine
La 6-tioinosina (6TI) es un antimetabolito de purina, actúa como un agente antiadipogénesis, regula a la baja los niveles de ARNm de PPAR γ y C/EBPα, así como la proteína diana de PPAR γ como LPL, CD36, aP2 y LXRα.
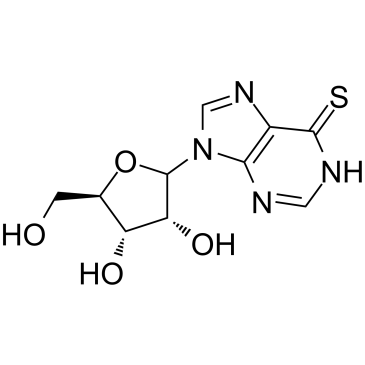
-
GC66060
7-Iodo-2',3'-dideoxy-7-deaza-guanosine
7-Iodo-2',3'-dideoxy-7-deaza-guanosine es un didesoxinucleÓsido que se puede utilizar en la sÍntesis de ADN y reacciones de secuenciaciÓn.
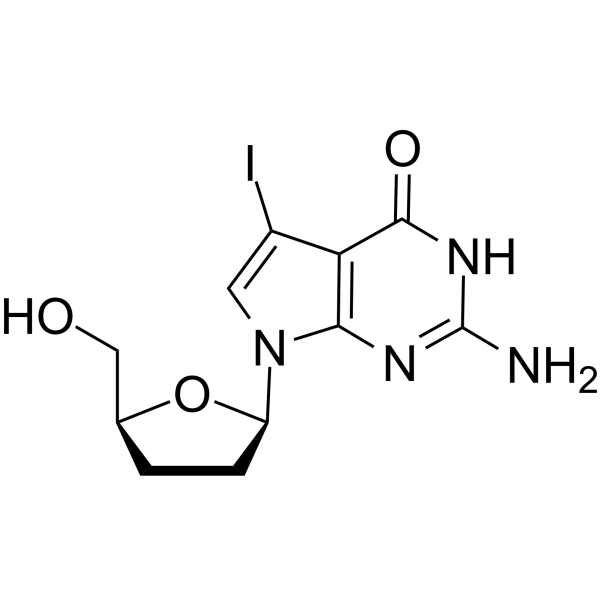
-
GC30531
7-Methylguanosine
La 7-metilguanosina es un nuevo inhibidor de la nucleotidasa cNIIIB con un valor IC50 de 87,8±7,5 μM.
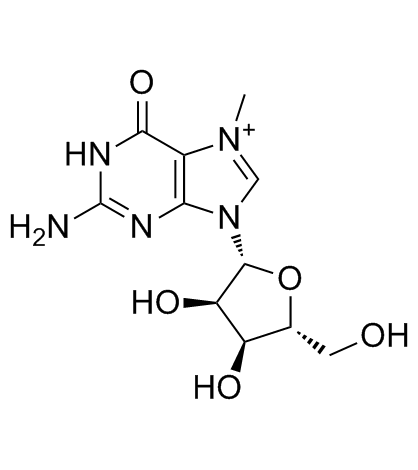
-
GC65525
7-TFA-ap-7-Deaza-dG
5'-O-TBDMS-dG es un nucleÓsido modificado.
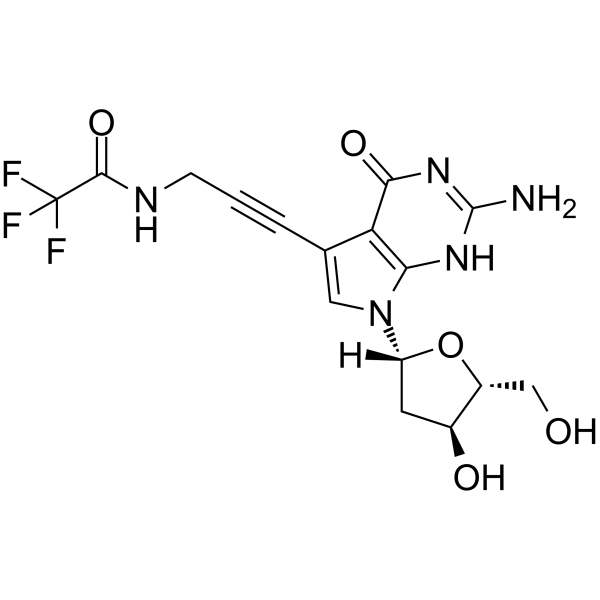
-
GC13805
Abacavir
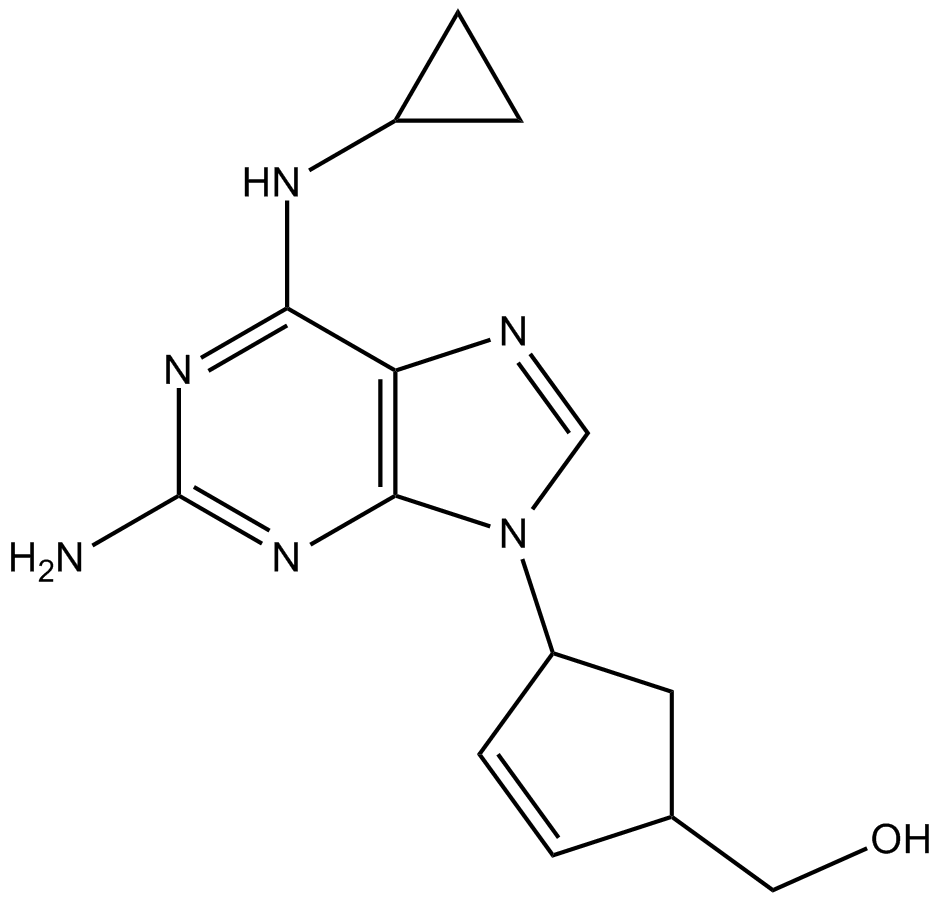
-
GC13432
Adenine
La adenina (6-aminopurina), una purina, es una de las cuatro bases nitrogenadas del Ácido nucleico del ADN. La adenina actÚa como un componente quÍmico del ADN y el ARN. La adenina también juega un papel importante en la bioquÍmica involucrada en la respiraciÓn celular, la forma tanto de ATP como de los cofactores (NAD y FAD) y la sÍntesis de proteÍnas.

-
GC14106
Adenosine
nucleósido
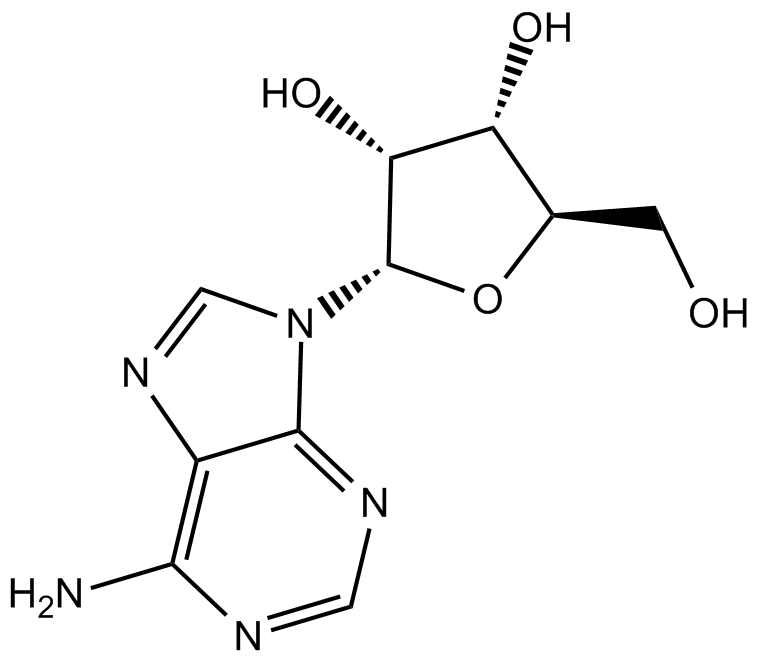
-
GC65462
Adenosine 5′-monophosphoramidate sodium
La adenosina 5′-monofosforamidato de sodio es un derivado de la adenosina y se puede utilizar como intermediario para la síntesis de nucleótidos.
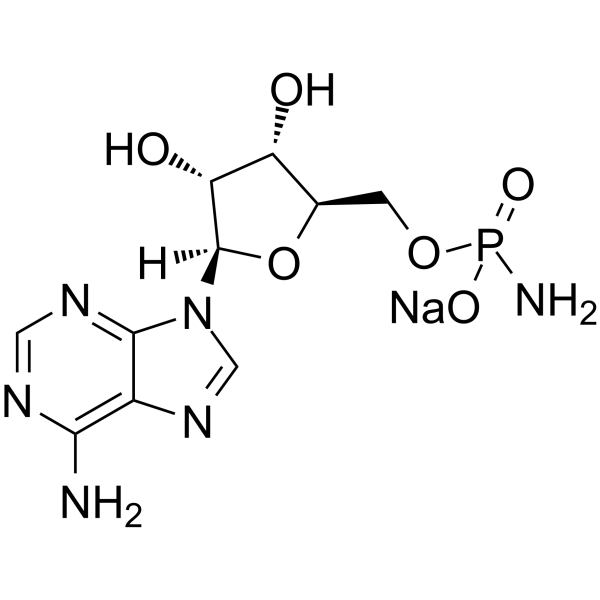
-
GC11843
Azaguanine-8
La azaguanina-8 es un anÁlogo de purina que muestra actividad antineoplÁsica. La azaguanina-8 funciona como un antimetabolito y se incorpora fÁcilmente a los Ácidos ribonucleicos, lo que interfiere con las vÍas biosintéticas normales y, por lo tanto, inhibe el crecimiento celular.
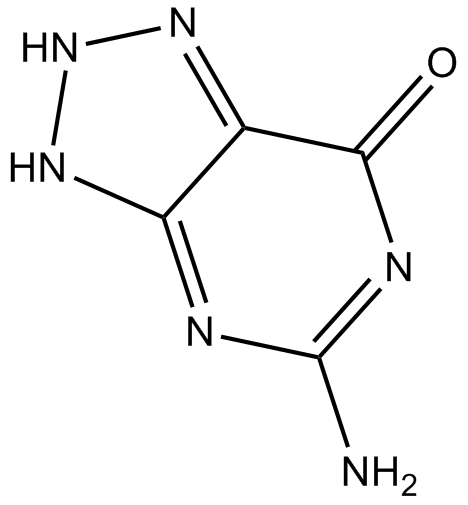
-
GC15866
Capecitabine
La capecitabina es un profÁrmaco oral que se convierte en su metabolito activo, 5-FU, mediante la timidina fosforilasa.
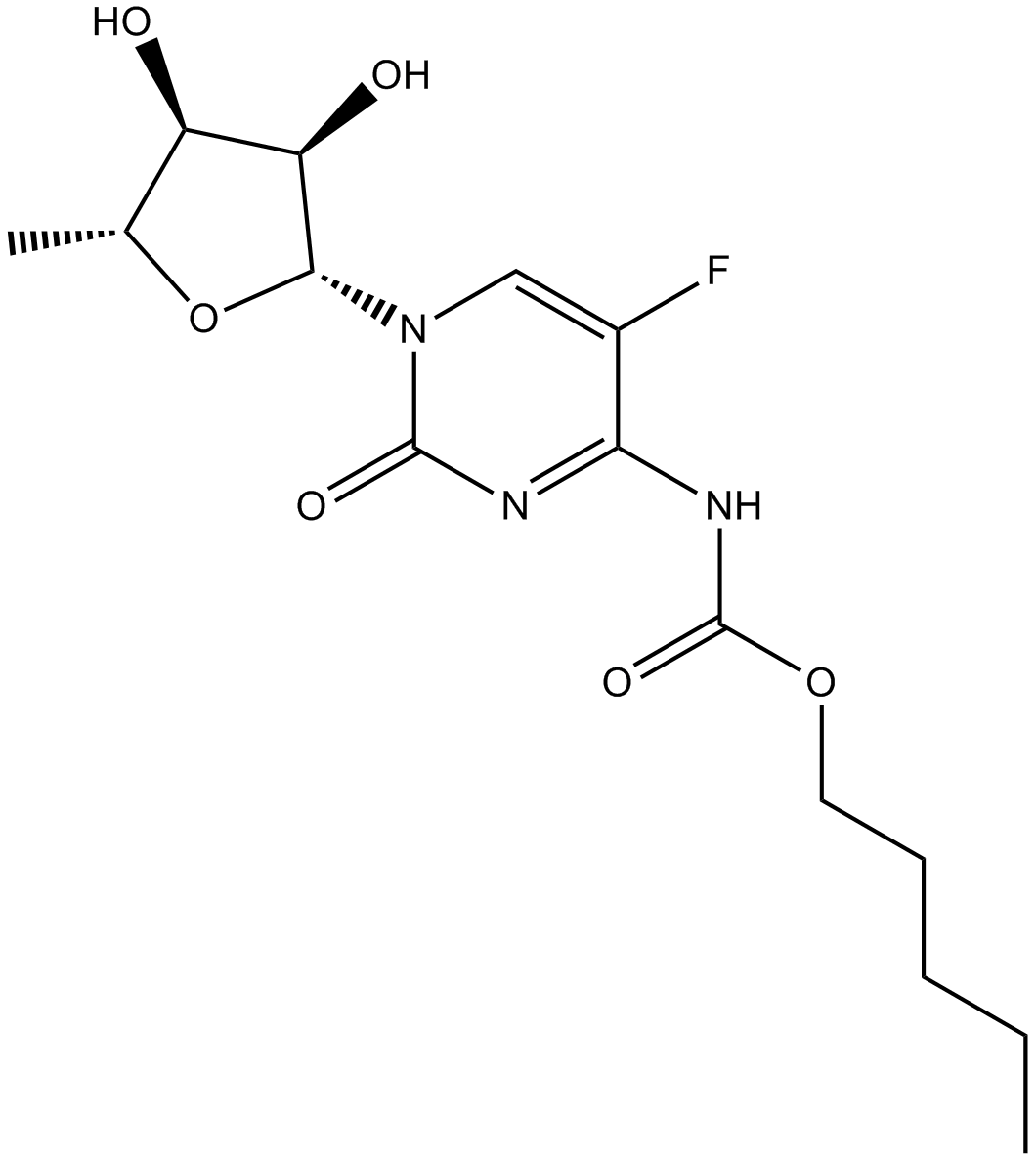
-
GC12748
Carmofur
El carmofur (HCFU), un derivado del 5-fluorouracilo, es un agente antineoplÁsico. Carmofur es un inhibidor de la ceramidasa Ácida con una IC50 de 79 nM para la enzima de rata. Carmofur inhibe la proteasa principal del SARS-CoV-2 (Mpro). Carmofur inhibe el SARS-CoV-2 en células Vero E6 con un EC50 de 24,3 μM.
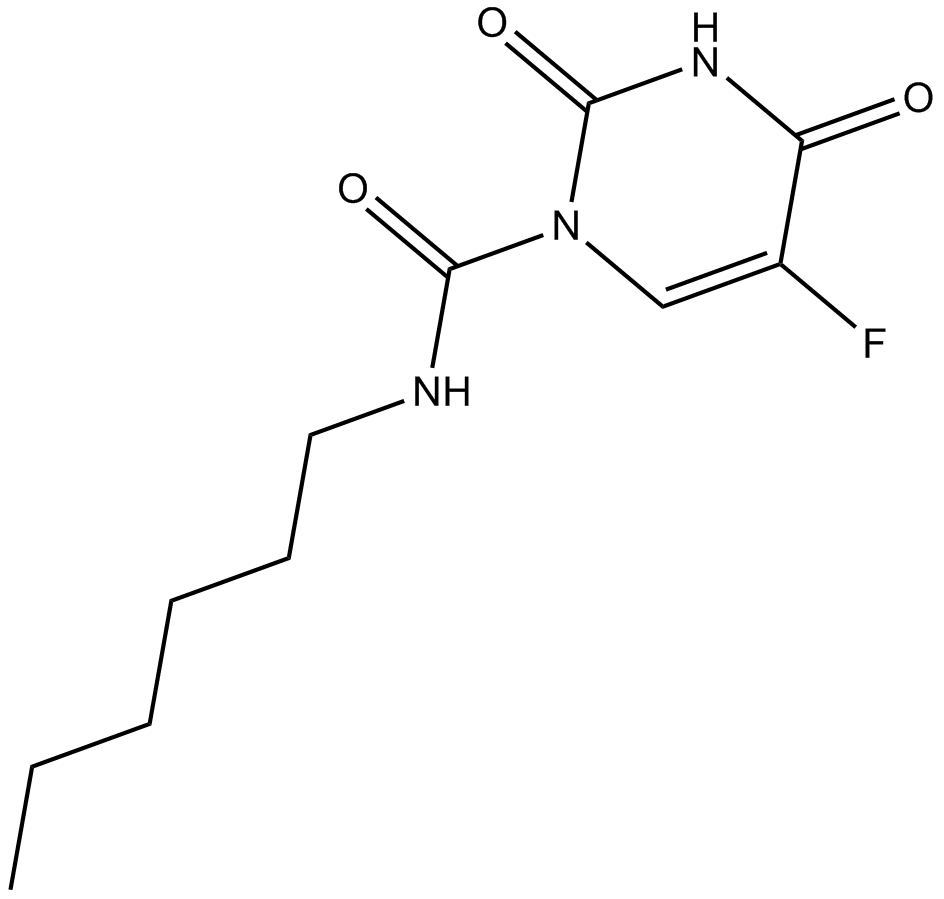
-
GC64261
Censavudine
La censavudina (OBP-601; BMS-986001), un anÁlogo de nucleÓsido, es un inhibidor de la transcriptasa inversa de nucleÓsido.
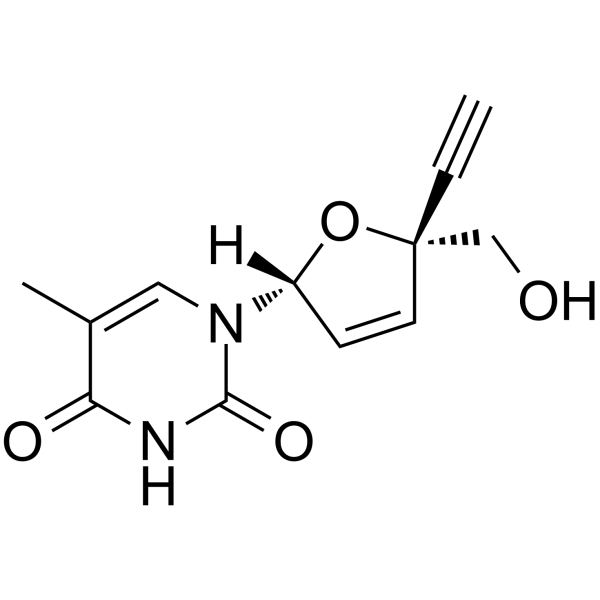
-
GC35693
CI 972 (anhydrous)
CI 972 (anhidro) es un inhibidor potente, activo por vía oral y competitivo de la purina nucleósido fosforilasa (PNP) (Ki \u003d 0,83 μM) en desarrollo como agente inmunosupresor selectivo de células T.
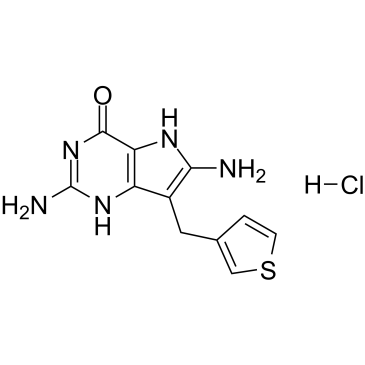
-
GC15219
Clofarabine
La clofarabina, un anÁlogo de nucleÓsido para la investigaciÓn del cÁncer, es un potente inhibidor de la ribonucleÓtido reductasa (IC50=65 nM) al unirse al sitio alostérico de la subunidad reguladora.
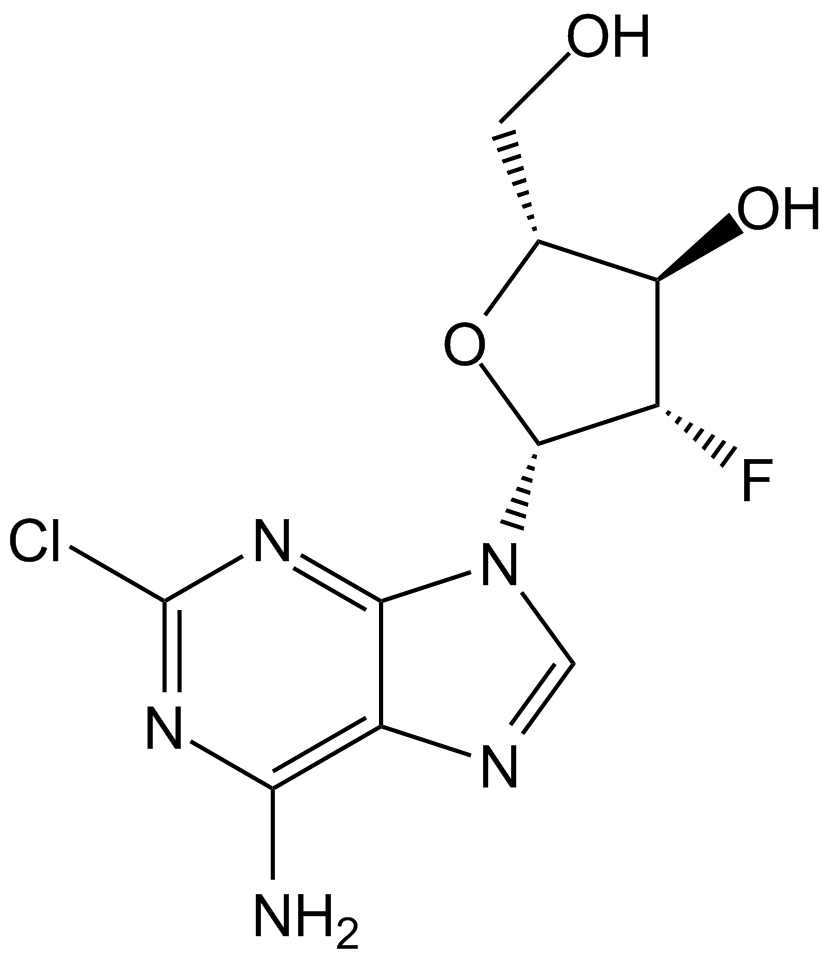
-
GC33177
CNDAC
CNDAC es un metabolito principal del fÁrmaco oral sapacitabina y un anÁlogo de nucleÓsido.
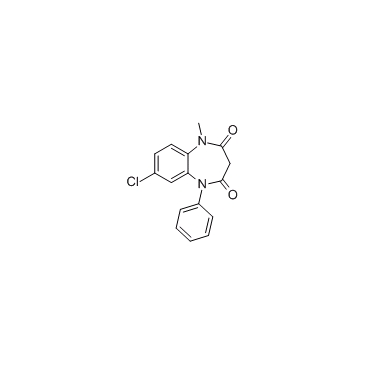
-
GC38382
CNDAC hydrochloride
El clorhidrato de CNDAC es un metabolito del agente activo por vÍa oral sapacitabina y un anÁlogo de nucleÓsido.
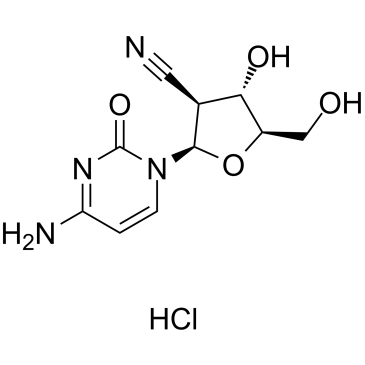
-
GC13070
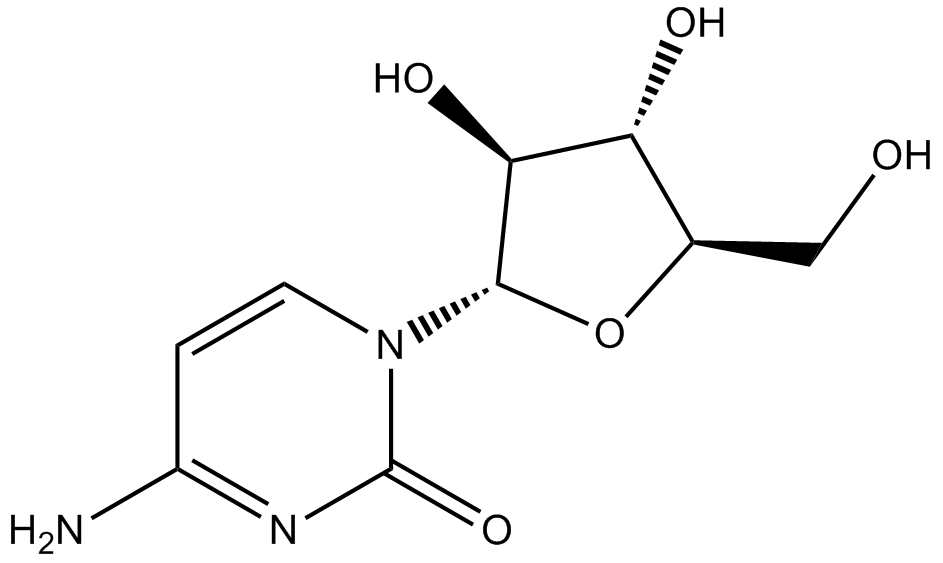
Cytarabine
Agente citotóxico, bloquea la síntesis de ADN.
-
GC14961
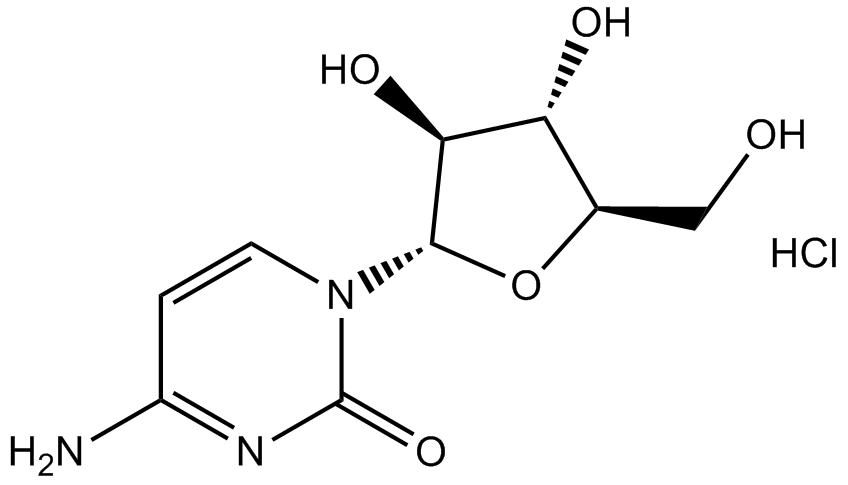
Cytarabine hydrochloride
El clorhidrato de citarabina, un anÁlogo de nucleÓsido, provoca la detenciÓn del ciclo celular en fase S e inhibe la ADN polimerasa.
-
GC13729
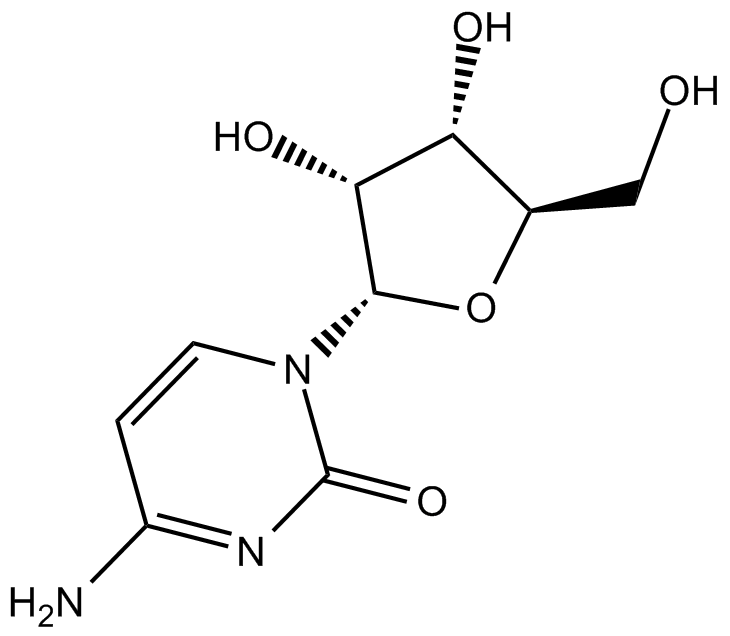
Cytidine
La citidina es un nucleÓsido de pirimidina y actÚa como componente del ARN.
-
GC14485
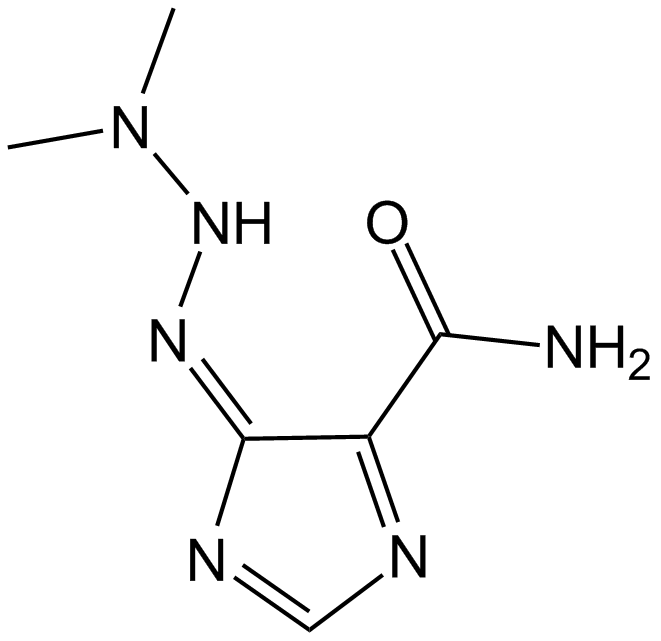
Dacarbazine
La dacarbazina es un agente alquilante antineoplÁsico no especÍfico del ciclo celular. La dacarbazina inhibe la respuesta linfoblÁstica T y B, con valores de IC50 de 50 y 10 μg/ml, respectivamente. La dacarbazina se puede utilizar para la investigaciÓn del melanoma maligno metastÁsico.
-
GC34123
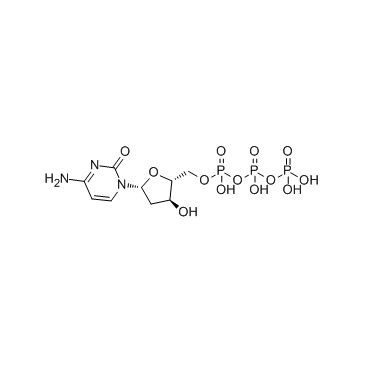
Deoxycytidine triphosphate (dCTP)
El trifosfato de desoxicitidina (dCTP) (dCTP) es un trifosfato de nucleÓsido que se puede utilizar para la sÍntesis de ADN.
-
GC33494
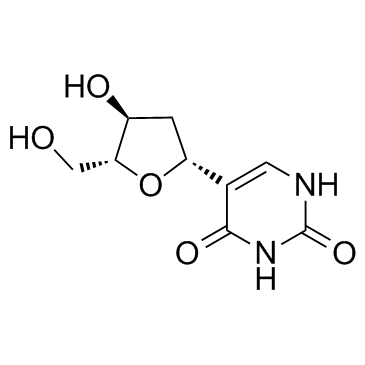
Deoxypseudouridine
La desoxipseudouridina es un anÁlogo de nucleÓsido.
-
GC62150
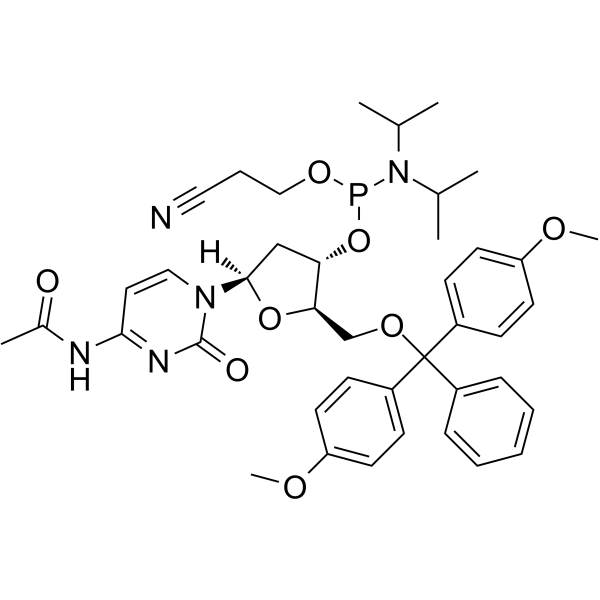
DMT-dC(ac) Phosphoramidite
DMT-dC(ac) Fosforamidita es un monÓmero de fosforamidita modificada, que se puede utilizar para la sÍntesis de oligonucleÓtidos.
-
GC62538
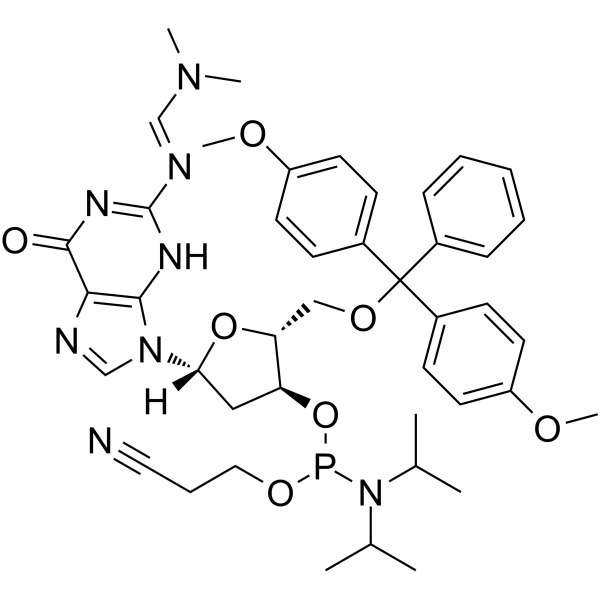
DMT-dG(dmf) Phosphoramidite
DMT-dG(dmf) La fosforamidita es un monÓmero de fosfinamida que se puede utilizar en la preparaciÓn de oligonucleÓtidos
-
GC66711
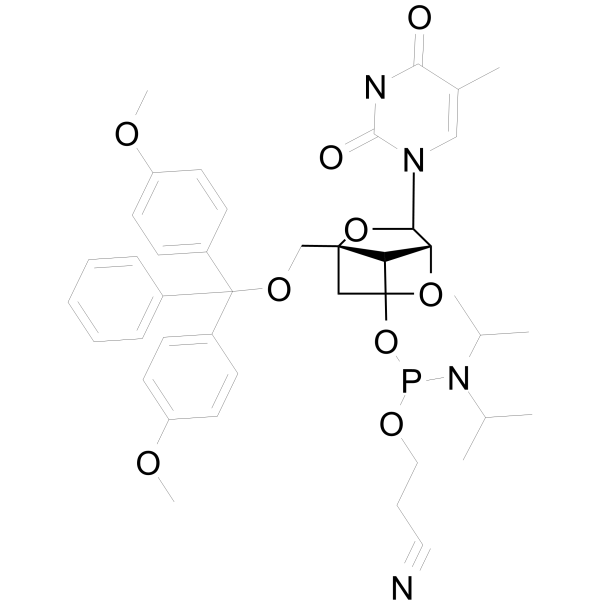
DMTr-LNA-5MeU-3-CED-phosphoramidite
DMTr-LNA-5MeU-3-CED-fosforamidita es un derivado de nucleósido.
-
GC11099
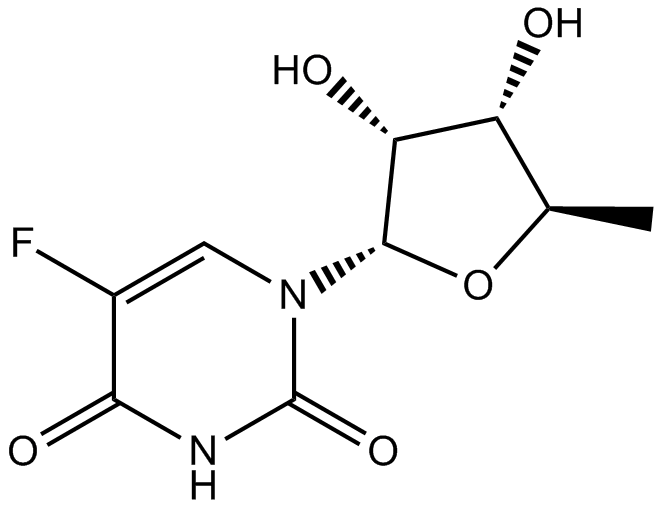
Doxifluridine
La doxifluridina (Ro 21-9738; 5'-DFUR) es un activador de la timidina fosforilasa para las células PC9-DPE2 con IC50 de 0,62 μM.
-
GC11654
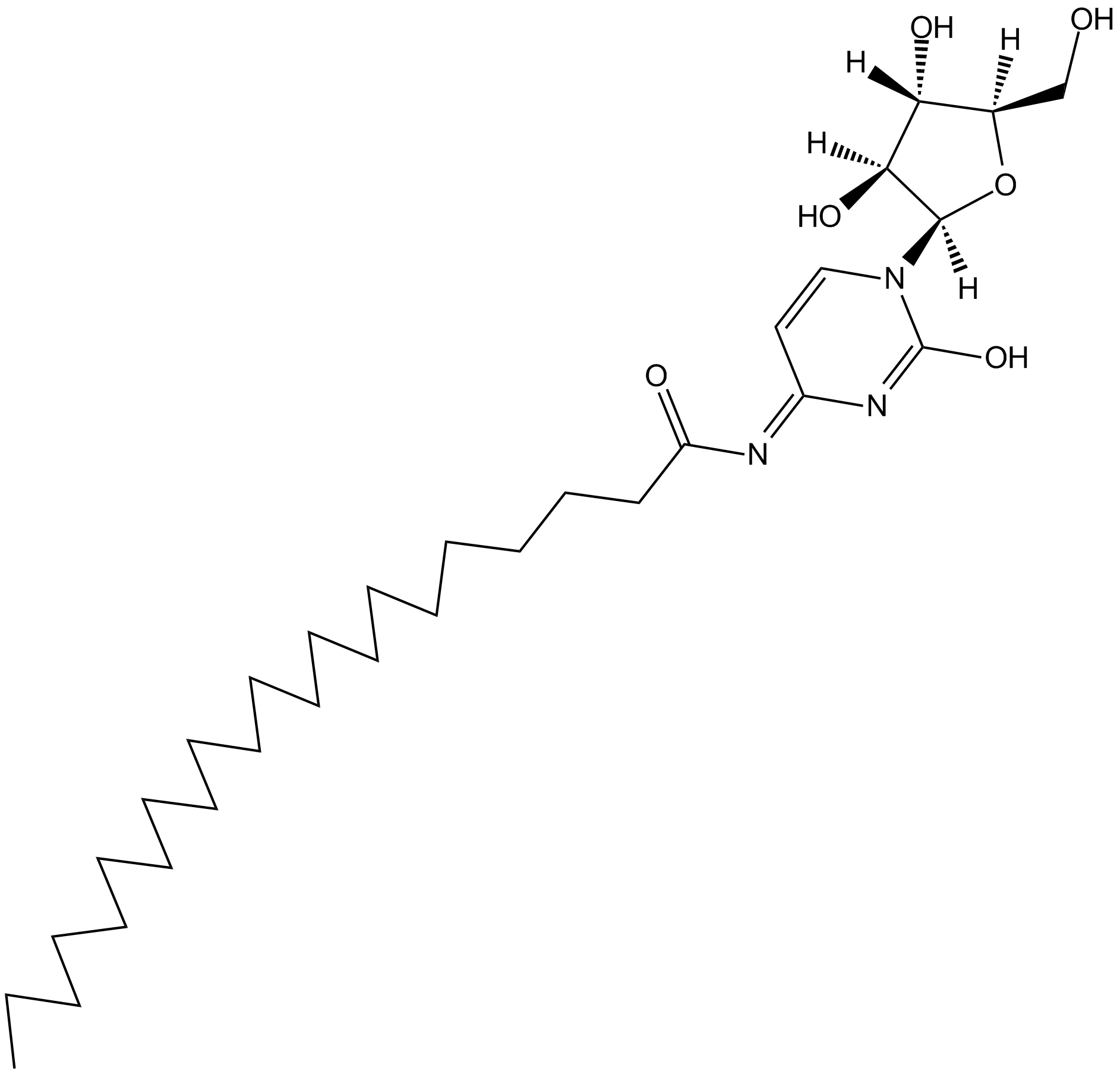
Enocitabine
La enocitabina es un anÁlogo de nucleÓsido y es un potente inhibidor de la replicaciÓn del ADN y un terminador de la cadena del ADN.
-
GC34112
Ethynylcytidine (ECyD)
La etinilescitidina (ECyD) (ECyD), un anÁlogo de nucleÓsido y un potente inhibidor de la sÍntesis de ARN, inhibe las ARN polimerasas I, II y II. La etinilescitidina (ECyD) tiene una sÓlida actividad antitumoral en una amplia gama de modelos de cÁncer.
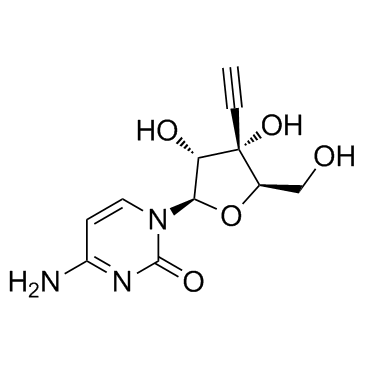
-
GC18014
Floxuridine
La floxuridina (5-fluorouracilo 2'-desoxirribosido) es un anÁlogo de la pirimidina y se conoce como antimetabolito oncolÓgico.
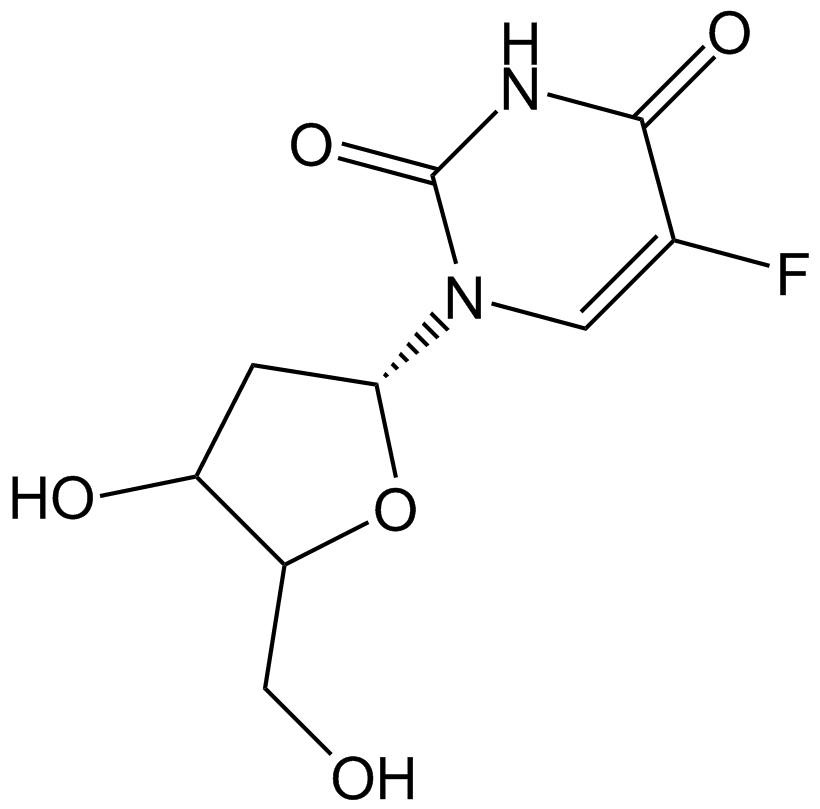
-
GC14144
Fludarabine
La fludarabina (NSC 118218) es un inhibidor de la sÍntesis de ADN y un anÁlogo de purina fluorada con actividad antineoplÁsica en tumores malignos linfoproliferativos. La fludarabina inhibe la activaciÓn inducida por citoquinas de STAT1 y la transcripciÓn de genes dependientes de STAT1 en linfocitos normales en reposo o activados.
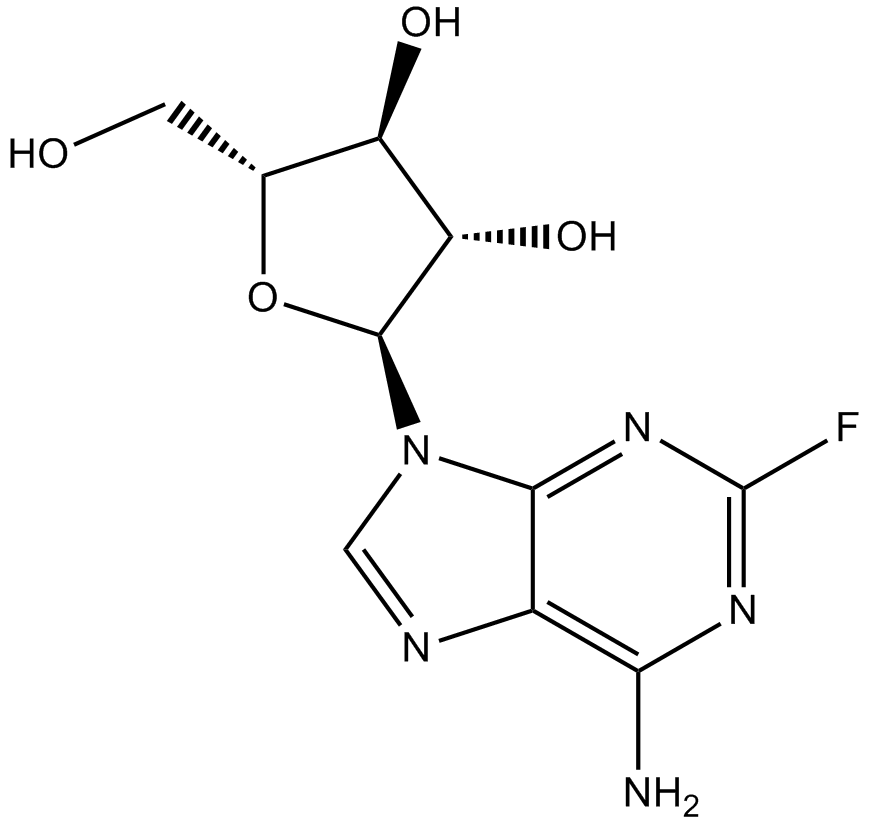
-
GC15134
Fludarabine Phosphate (Fludara)
La fludarabina (fosfato) es un anÁlogo de la adenosina y la desoxiadenosina, que puede competir con el dATP para incorporarse al ADN e inhibir la sÍntesis de ADN.
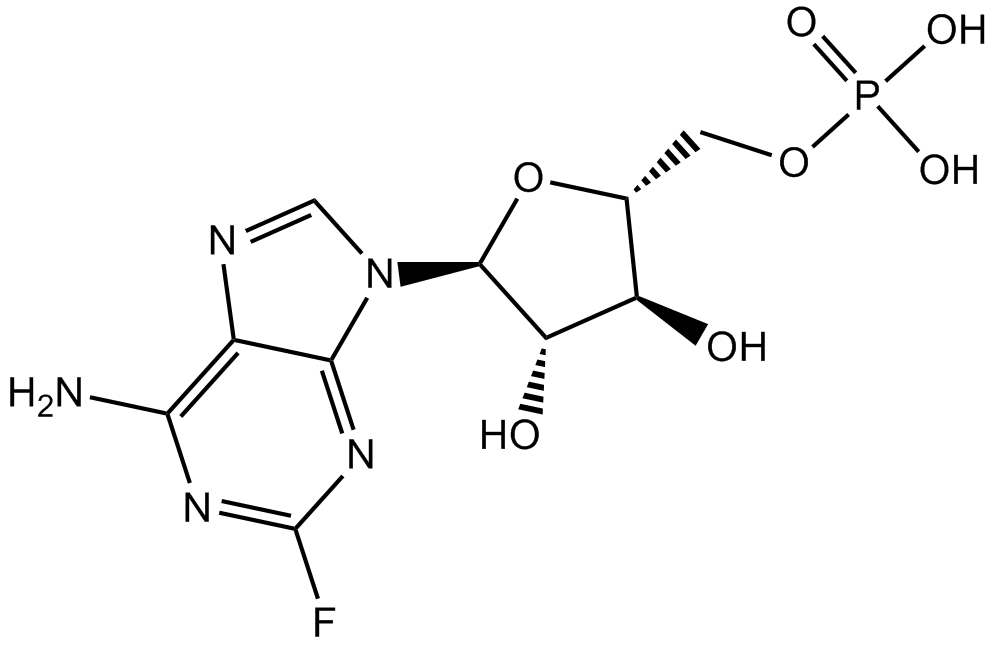
-
GC66430
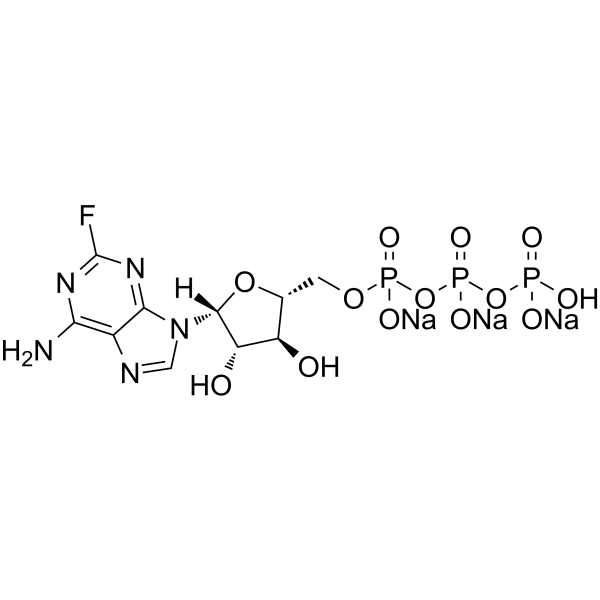
Fludarabine triphosphate trisodium
El trifosfato de fludarabina (F-ara-ATP) trisÓdico, el metabolito activo de la fludarabina (HY-B0069), es un inhibidor potente, no competitivo y especÍfico de la ADN primasa, con una IC50 de 2,3 μM y una Ki de 6,1 μM . La fludarabina trifosfato trisÓdica inhibe la sÍntesis de ADN bloqueando la formaciÓn de ADN primasa y ARN cebador. La fludarabina trifosfato trisÓdica inhibe la ribonucleÓtido reductasa y la ADN polimerasa y, en Última instancia, conduce a la apoptosis celular.
-
GC14466
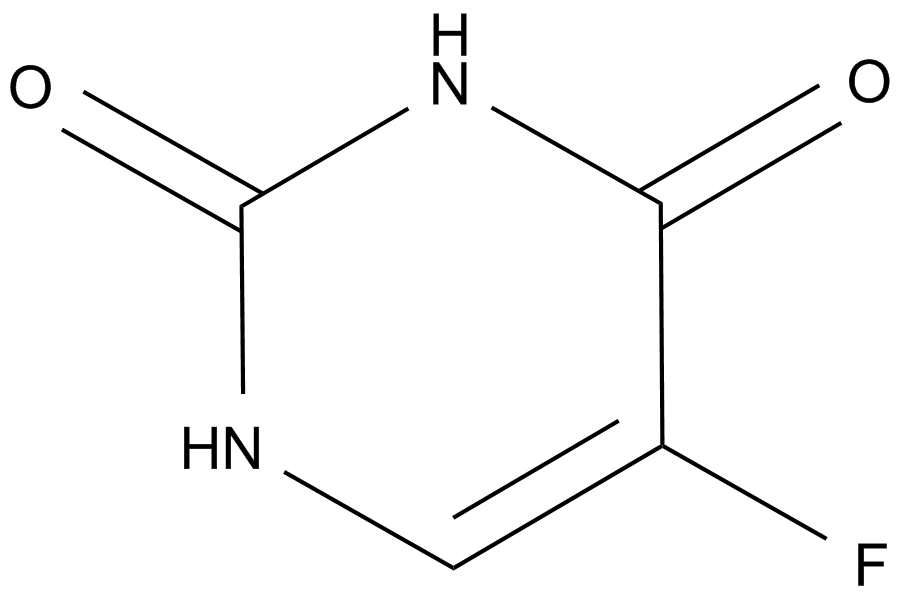
Fluorouracil (Adrucil)
Una forma de prodroga de FdUMP.
-
GC33029
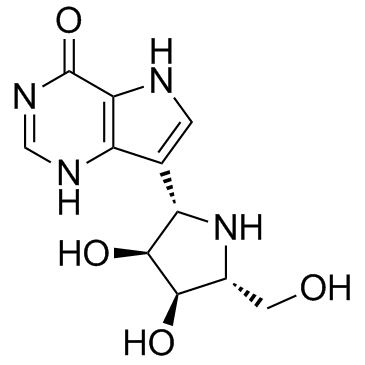
Forodesine (BCX-1777 freebase)
Forodesina (BCX-1777 base libre) (BCX-1777) es un inhibidor de la fosforilasa (PNP) de nucleÓsido de purina muy potente y activo por vÍa oral con valores de IC50 que oscilan entre 0,48 y 1,57 nM para PNP de humanos, ratones, ratas, monos y perros. Forodesine (BCX-1777 freebase) es un potente inhibidor de la proliferaciÓn de linfocitos humanos. Forodesine (BCX-1777 freebase) podrÍa inducir la apoptosis en células leucémicas al aumentar los niveles de dGTP.
-
GC32708
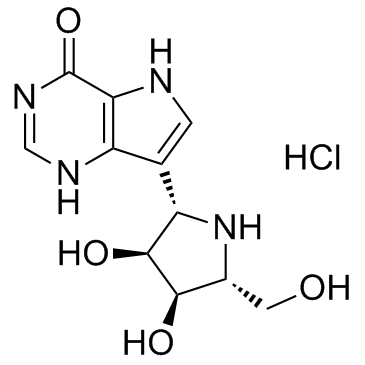
Forodesine hydrochloride (BCX-1777)
El clorhidrato de forodesina (BCX-1777) (BCX-1777 clorhidrato) es un inhibidor de la fosforilasa (PNP) de nucleÓsido de purina muy potente y activo por vÍa oral con valores de IC50 que oscilan entre 0,48 y 1,57 nM para PNP de humanos, ratones, ratas, monos y perros. El clorhidrato de forodesina (BCX-1777) es un potente inhibidor de la proliferaciÓn de linfocitos humanos. El clorhidrato de forodesina (BCX-1777) podrÍa inducir la apoptosis en células leucémicas al aumentar los niveles de dGTP.
-
GC36071
Fosteabine
La fosteabina es un anÁlogo oral y profÁrmaco de la citarabina que es resistente a la desoxicitidina desaminasa.
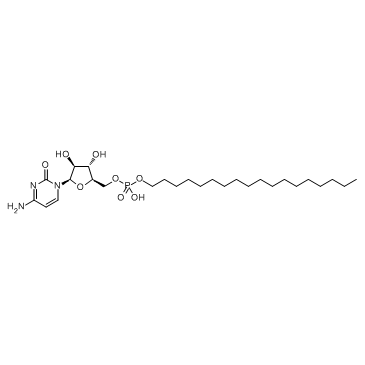
-
GC13831
FT-207 (NSC 148958)
FT-207 (NSC 148958) (FT 207; NSC 148958) es un profÁrmaco quimioterapéutico de 5-FU utilizado en el tratamiento de cÁnceres; es un componente de tegafur-uracilo.
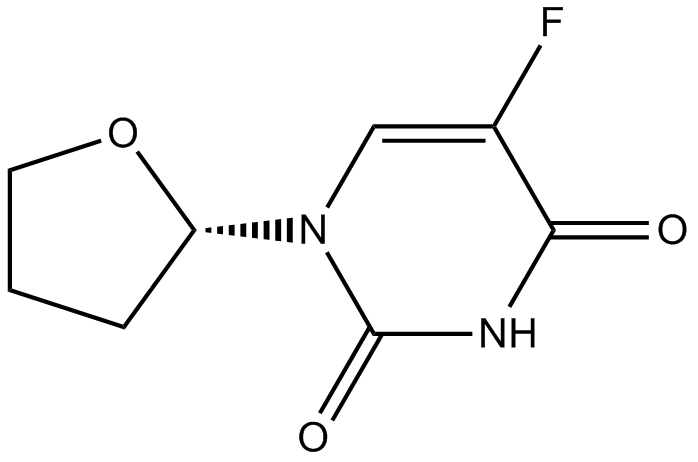
-
GC60865
Ganciclovir sodium
El ganciclovir (BW 759) sÓdico, un anÁlogo de nucleÓsido, es un agente antiviral activo por vÍa oral con actividad contra el CMV.
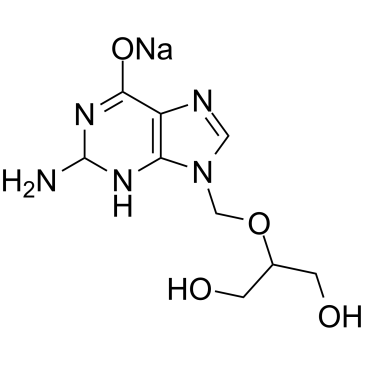
-
GC16805
Gemcitabine
La gemcitabina (LY 188011) es un antimetabolito anÁlogo del nucleÓsido de pirimidina y un agente antineoplÁsico. La gemcitabina inhibe la sÍntesis y reparaciÓn del ADN, lo que produce autofagia y apoptosis.
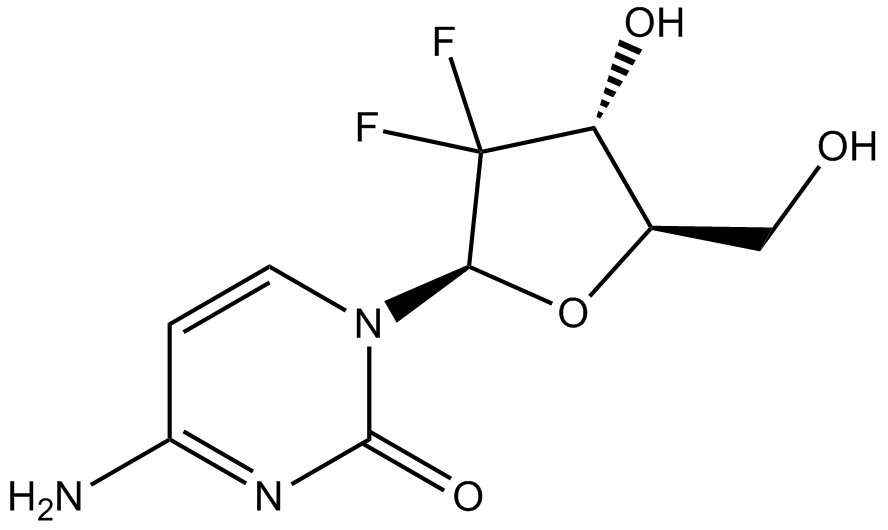
-
GC36130
Gemcitabine elaidate
Elaidato de gemcitabina (CP-4126) es un profÁrmaco lipofÍlico de la gemcitabina. El elaidato de gemcitabina se convierte en gemcitabina por acciÓn de las esterasas para fosforilarse. El elaidato de gemcitabina muestra actividad antitumoral.
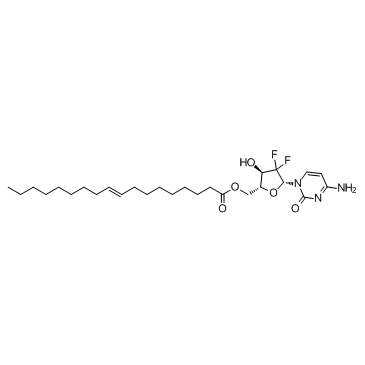
-
GC63777
Gemcitabine elaidate hydrochloride
El clorhidrato de elaidato de gemcitabina (CP-4126) es un profÁrmaco lipofÍlico de la gemcitabina. El hidrocloruro de elaidato de gemcitabina se convierte en gemcitabina por acciÓn de las esterasas para ser fosforilado. El hidrocloruro de elaidato de gemcitabina exhibe actividad antitumoral.
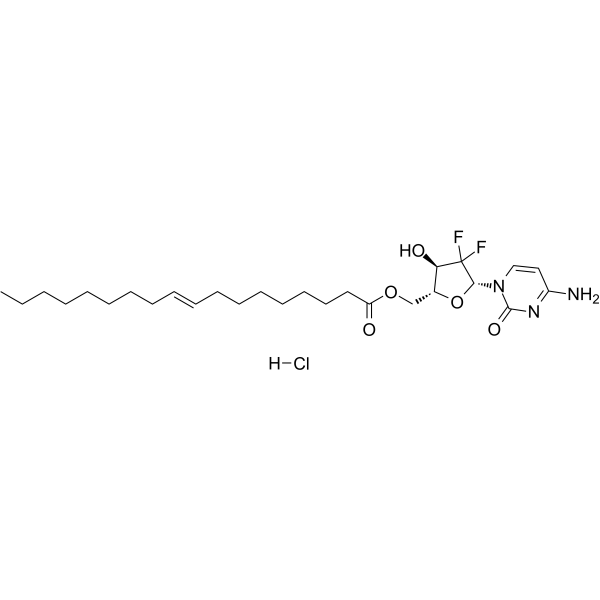
-
GC14447
Gemcitabine HCl
El clorhidrato de gemcitabina (clorhidrato LY 188011) es un antimetabolito anÁlogo del nucleÓsido de pirimidina y un agente antineoplÁsico. El clorhidrato de gemcitabina inhibe la sÍntesis y reparaciÓn del ADN, lo que produce autofagia y apoptosis.
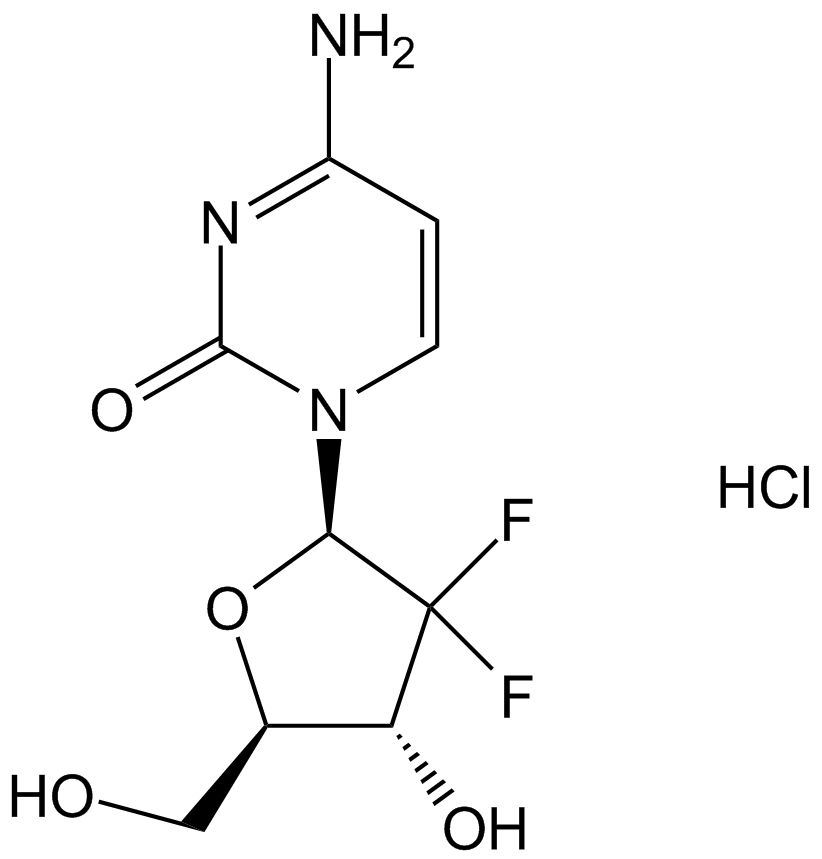
-
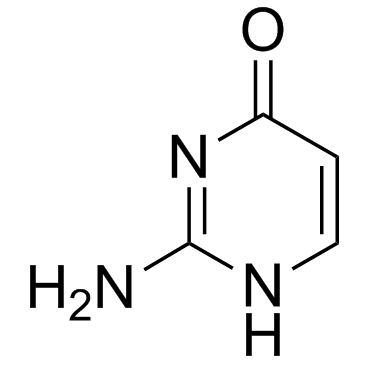
GC30554
Isocytosine
La isocitosina es una nucleobase no natural y un isÓmero de la citosina.
-
GC33662
Isoguanine
La isoguanina es una base de purina que es un isÓmero de la guanina.
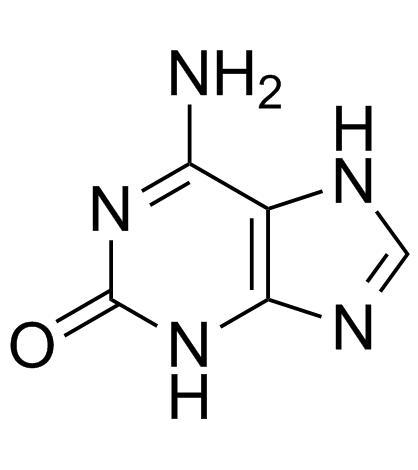
-
GC66622
L-Guanosine
L-guanosina es la configuración L de la guanosina. La guanosina es un nucleósido de purina con actividad anti-herpesvirus.