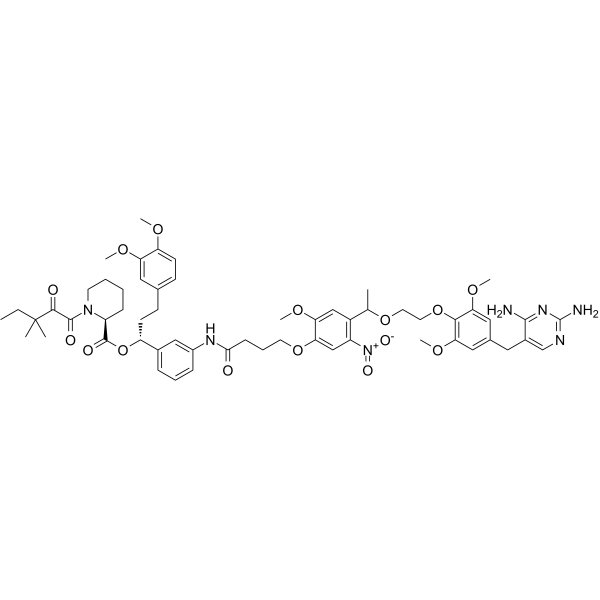FKBP
FKBP (FK506-binding protein) is one of two major immunophilins and most of FKBP family members bind FK506 and show peptidylprolyl cis/trans isomerase (PPIase) activity. Small size FKBP family members contain only FK506-binding domain, while FKBPs with large molecular weights possess extra domains such as tetratricopeptide repeat domains, calmodulin binding and transmembrane motifs. FKBPs are involved in several biochemical processes including protein folding, receptor signaling, protein trafficking and transcription. FKBP family proteins play important functional roles in the T-cell activation, when complexed with their ligands.
FK506-binding proteins 1a and 1b (FKBP1a/1b) are immunophilin proteins that bind the immunosuppressant agent FK506 and AY 22989. FKBP12 is a ubiquitous abundant protein that acts as a receptor for FK506, binds tightly to intracellular calcium release channels and to the transforming growth factor β (TGF-β) type I receptor.
Targets for FKBP
Products for FKBP
- Cat.No. Nombre del producto Información
-
GC34172
AP1867
AP1867 es un ligando dirigido por FKBP12F36V sintético.
-
GC61745
AP1867-2-(carboxymethoxy)
AP1867-2-(carboximetoxi), el resto basado en AP1867 (un ligando sintético dirigido por FKBP12F36V), se une al ligando CRBN a través de un conector para formar moléculas dTAG.

-
GC15586
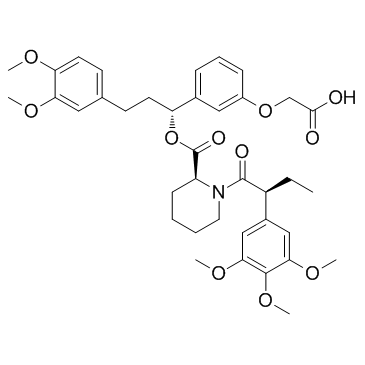
AP1903
AP1903 (AP1903) es un agente dimerizador que actÚa entrecruzando los dominios FKBP. AP1903 (AP1903) dimeriza el interruptor suicida Caspase 9 e induce rÁpidamente la apoptosis.
-
GC14498
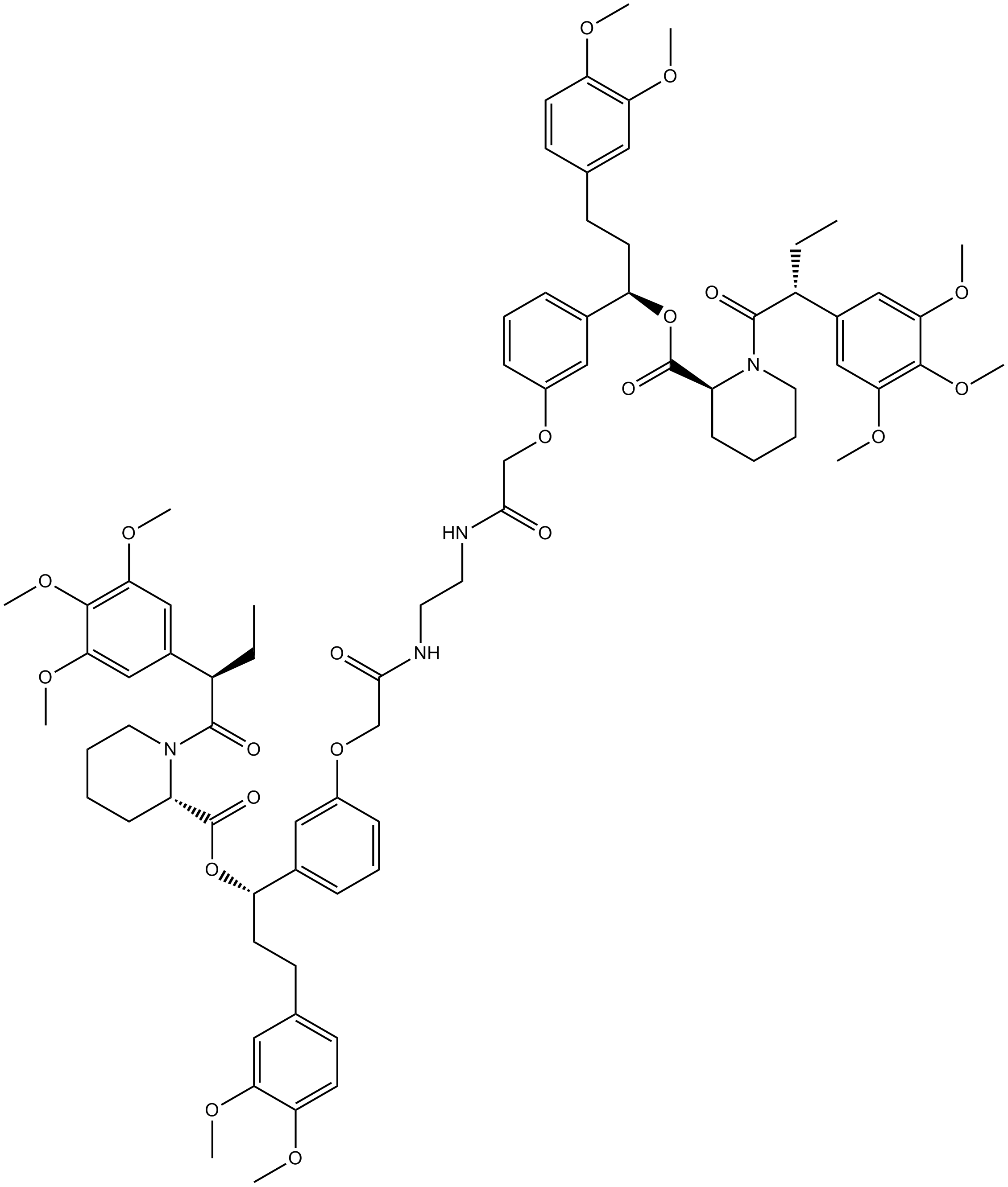
AP20187
AP20187 (B/B Homodimerizer) es un ligando permeable a las células que se utiliza para dimerizar las proteÍnas de fusiÓn de la proteÍna de uniÓn a FK506 (FKBP) e iniciar cascadas de seÑalizaciÓn biolÓgica y expresiÓn génica o interrumpir las interacciones proteÍna-proteÍna.
-
GC13215
Ascomycin(FK 520)
A potent macrolide immunosuppressant
-
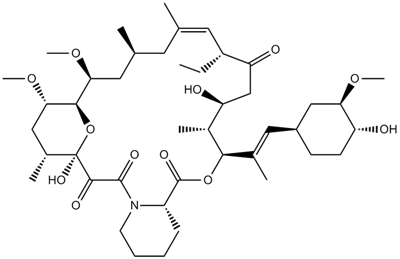
GC13601
Everolimus (RAD001)
Un derivado de rapamicina.
-
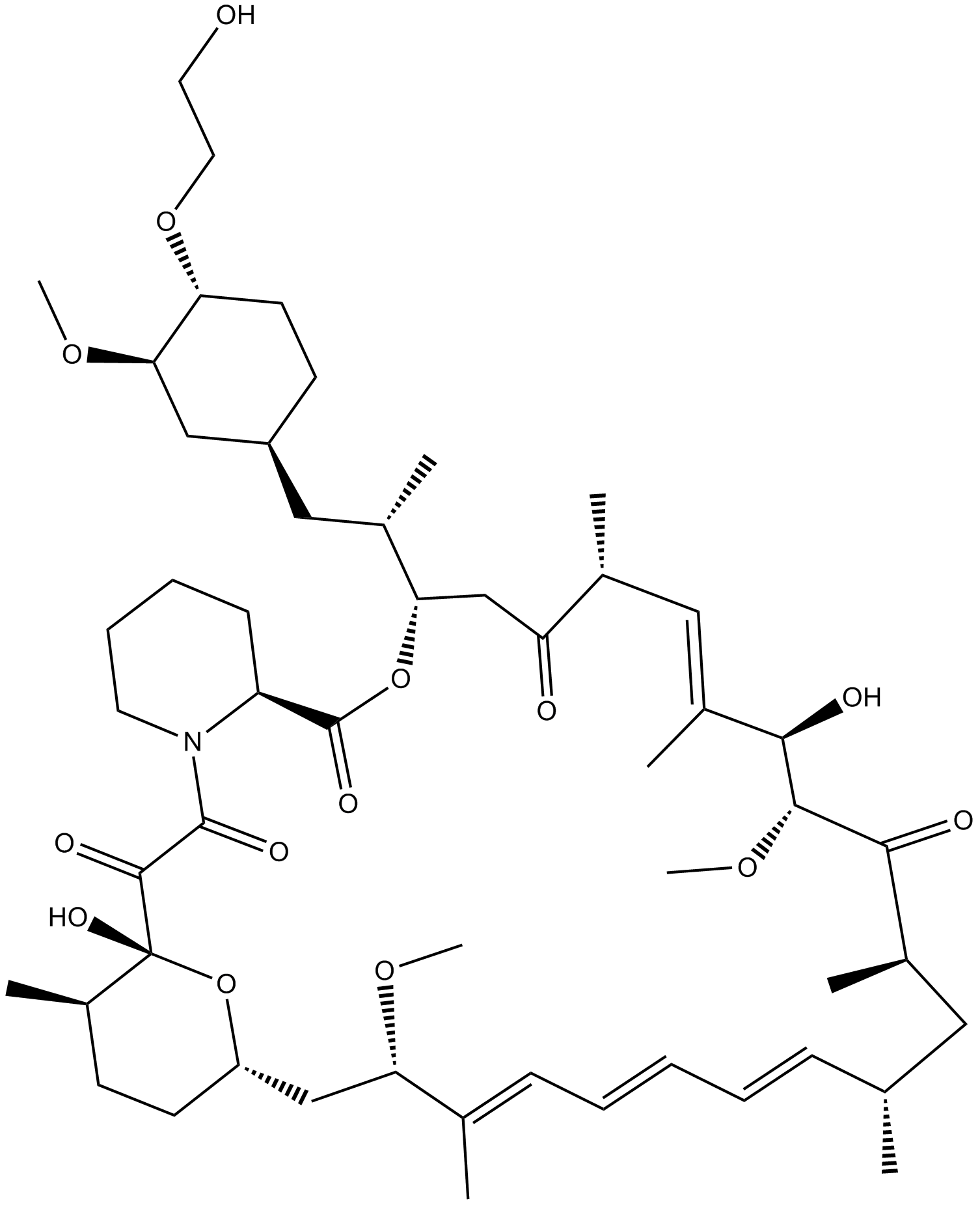
GC62186
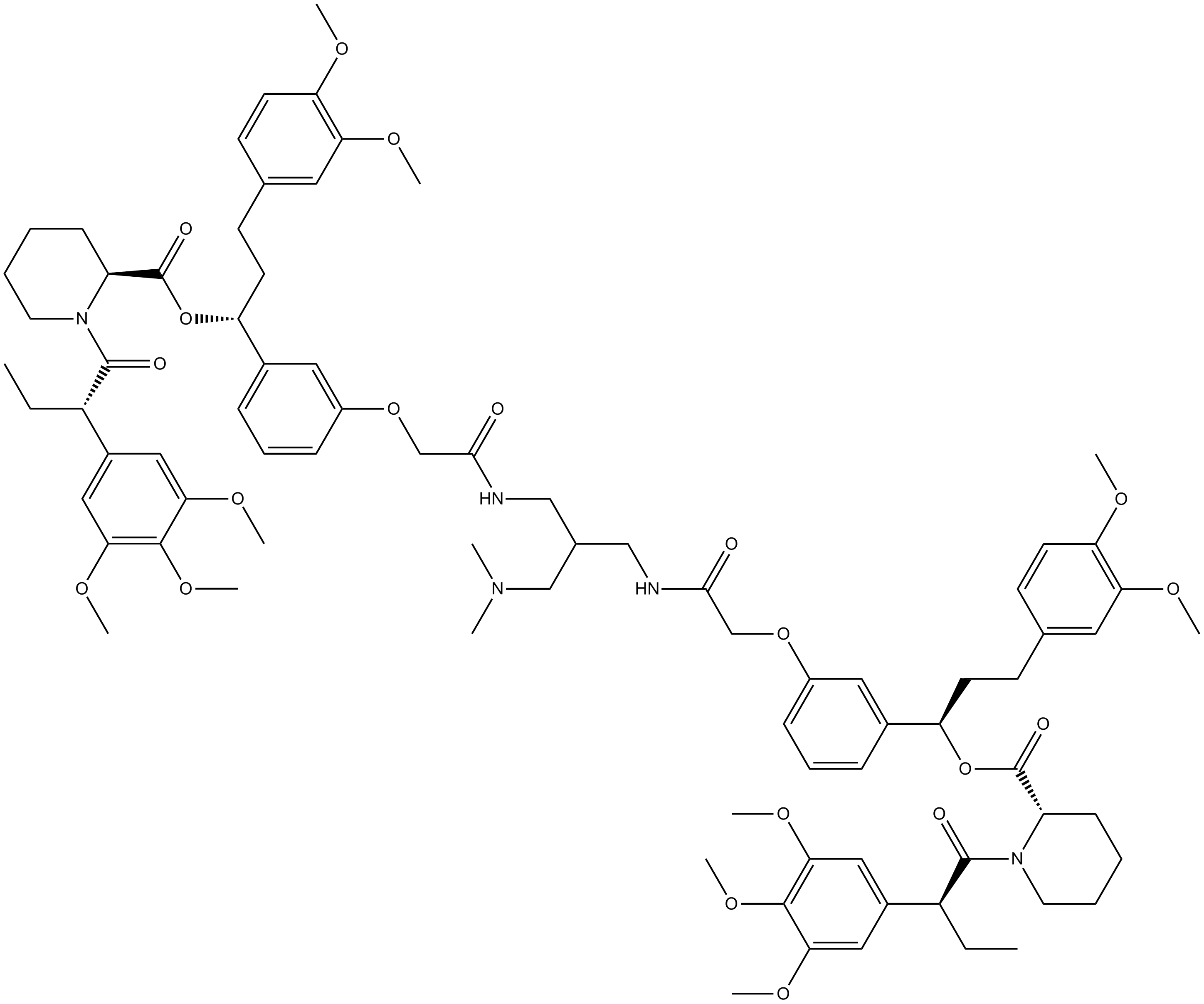
KB02-SLF
KB02-SLF es un degradador nuclear FKBP12 basado en PROTAC (pegamento molecular). KB02-SLF promueve la degradaciÓn nuclear de FKBP12 mediante la modificaciÓn covalente de DCAF16 (ligasa E3) y puede mejorar la durabilidad de la degradaciÓn de proteÍnas en sistemas biolÓgicos. SLF se une al ligando KB02 de ubiquitina E3 ligasa a través de un conector para formar KB02-SLF.
-
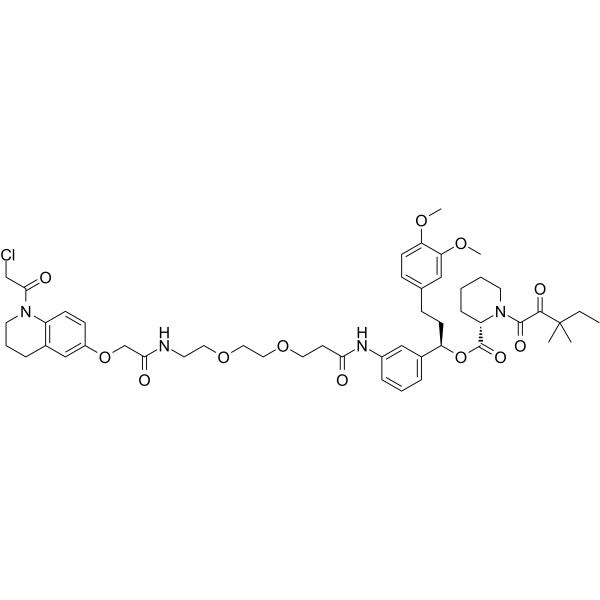
GC39503
PROTAC FKBP Degrader-3
PROTAC FKBP Degrader-3 es un PROTAC que comprende un grupo de uniÓn a ligando FKBP, un enlazador y un grupo de uniÓn de von Hippel-Lindau. PROTAC FKBP Degrader-3 es un potente degradador de FKBP.
-
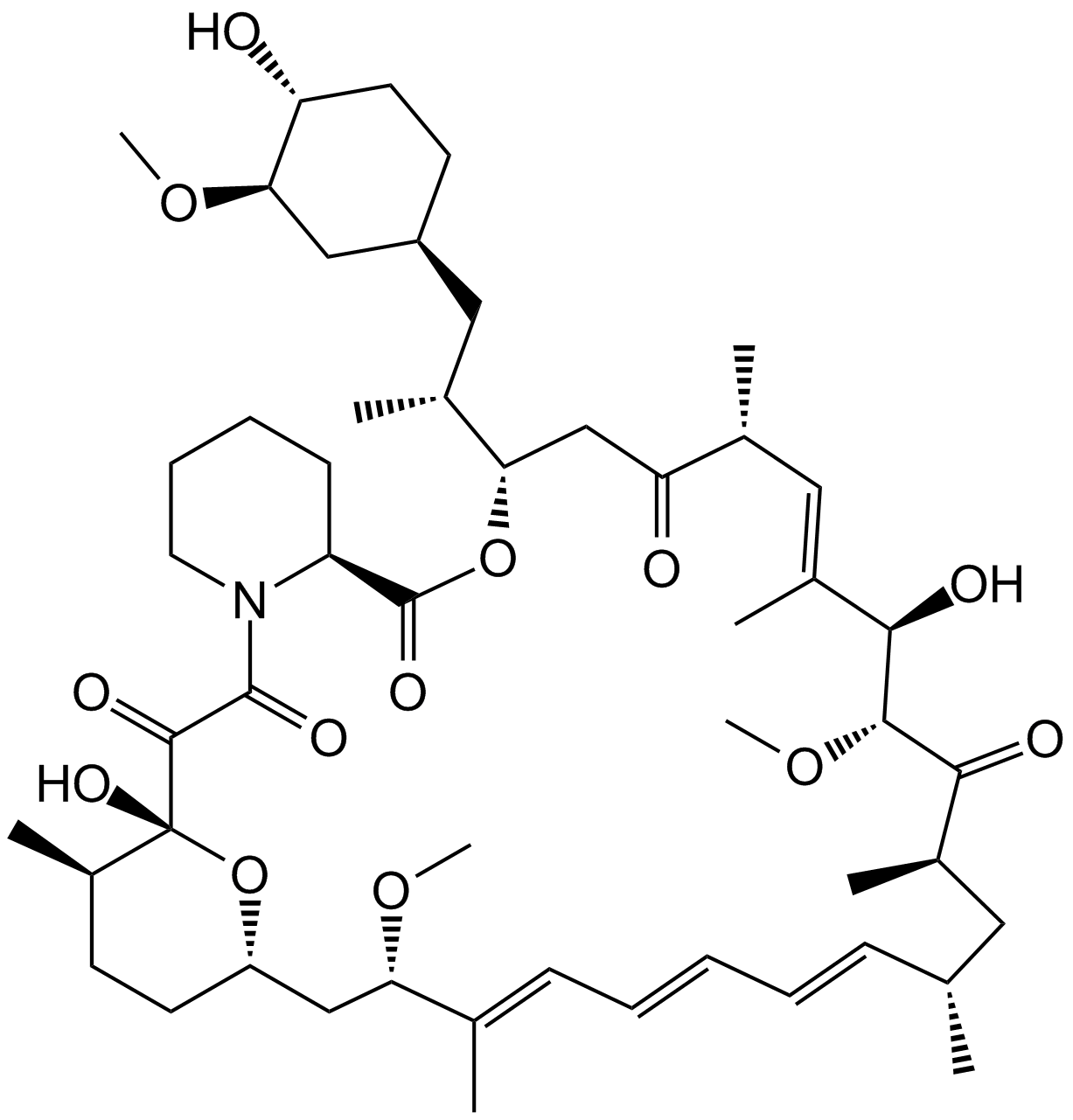
GC15031
Rapamycin (Sirolimus)
Un inhibidor alostérico de mTORC1
-
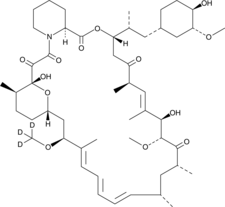
GC48027
Rapamycin-d3
Rapamicina-d3 (Sirolimus-d3) es la rapamicina marcada con deuterio. La rapamicina es un inhibidor de mTOR potente y especÍfico con una IC50 de 0,1 nM en células HEK293. La rapamicina se une a FKBP12 y actÚa especÍficamente como un inhibidor alostérico de mTORC1. La rapamicina es un activador de la autofagia, un inmunosupresor.
-
GC33699
SAFit1
SAFit1 es un inhibidor especÍfico de la proteÍna de uniÓn a FK506 51 (FKBP51) con una Ki de 4±0,3 nM.

-
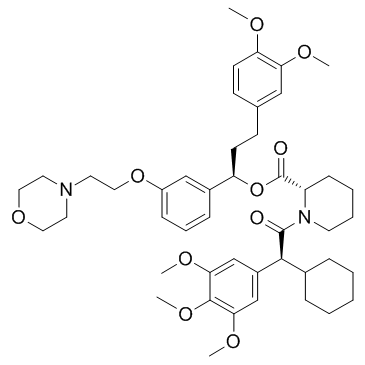
GC34093
SAFit2
SAFit2 es un inhibidor de la proteÍna 51 (FKBP51) de uniÓn a FK506 altamente potente y altamente selectivo con una Ki de 6 nM y también mejora la uniÓn de AKT2-AS160.
-
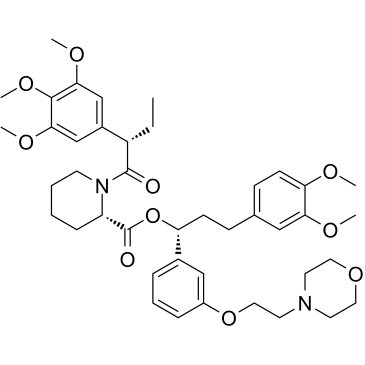
GC38060
Shield-1
Shield-1 (Shld1) es un ligando específico, permeable a la célula y de alta afinidad para la proteína de unión a FK506-12 (FKBP), y revierte la inestabilidad al unirse a FKBP mutado (mtFKBP), permitiendo así la expresión condicional de proteínas fusionadas con mtFKBP.
-
GC44899
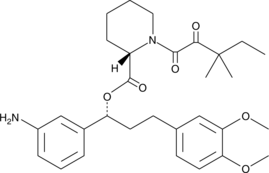
SLF
SLF es un ligando sintético para la proteÍna de uniÓn a FK506 (FKBP) con una afinidad de 3,1 μM por FKBP51 y una IC50 de 2,6 μM por FKBP12. SLF se puede utilizar en la sÍntesis de PROTAC.
-
GC61461
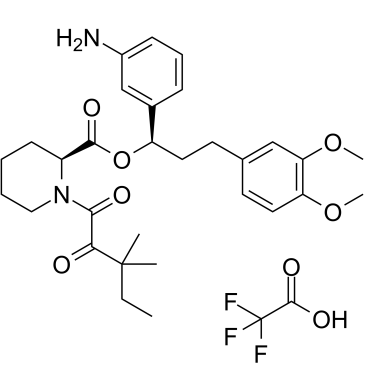
SLF TFA
SLF TFA es un ligando sintético para la proteÍna de uniÓn a FK506 (FKBP) con una afinidad de 3,1 μM por FKBP51 y una IC50 de 2,6 μM por FKBP12. SLF TFA se puede utilizar en la sÍntesis de PROTAC.
-
GC16233
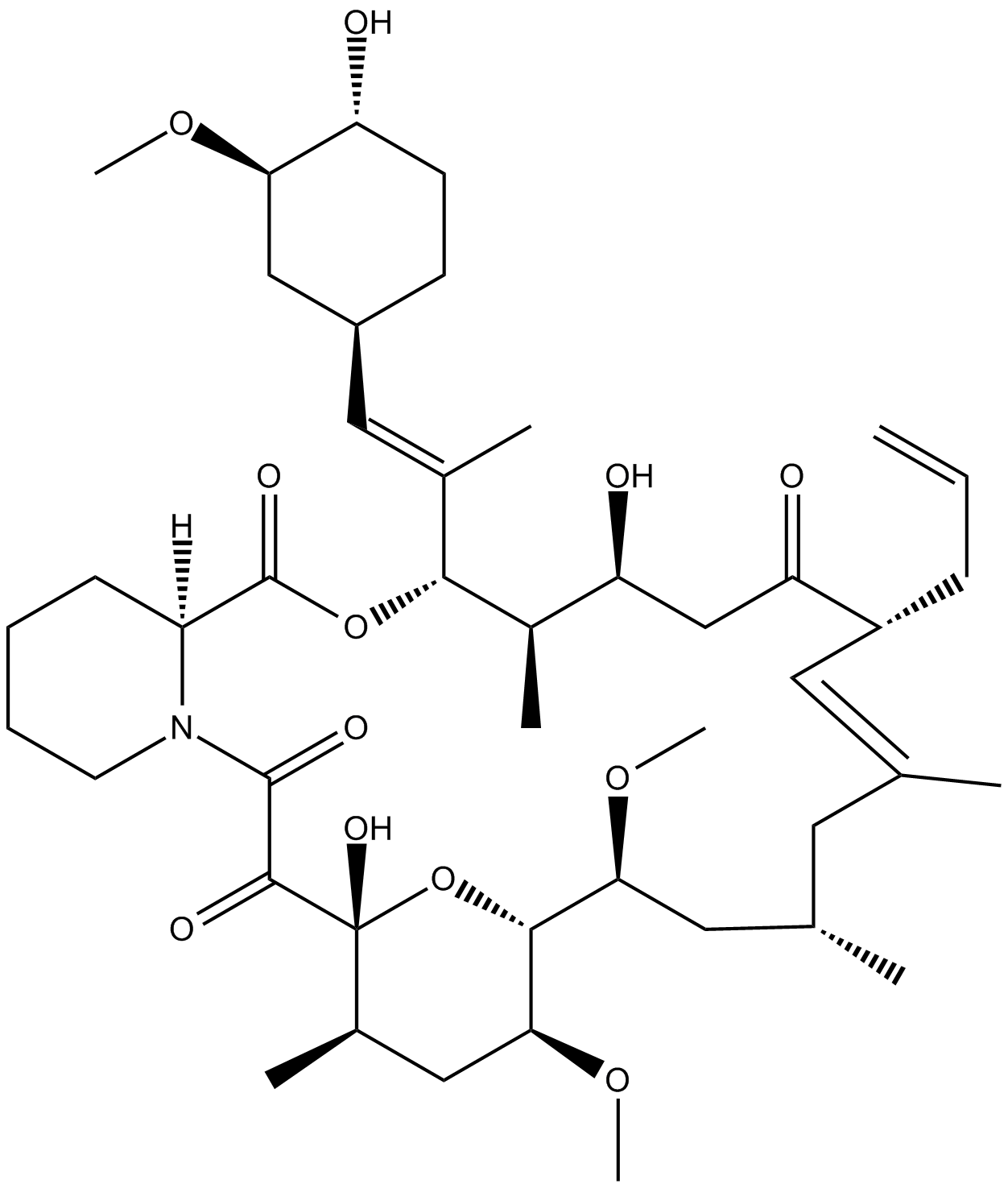
Tacrolimus (FK506)
Tacrolimus (FK506), un antibiótico macrólido con potentes efectos inmunosupresores, se aisló de Streptomyces tsukubaensis y se ha utilizado anteriormente para prevenir aloinjertos y para el tratamiento de trastornos autoinmunes en humanos.
-
GC17995
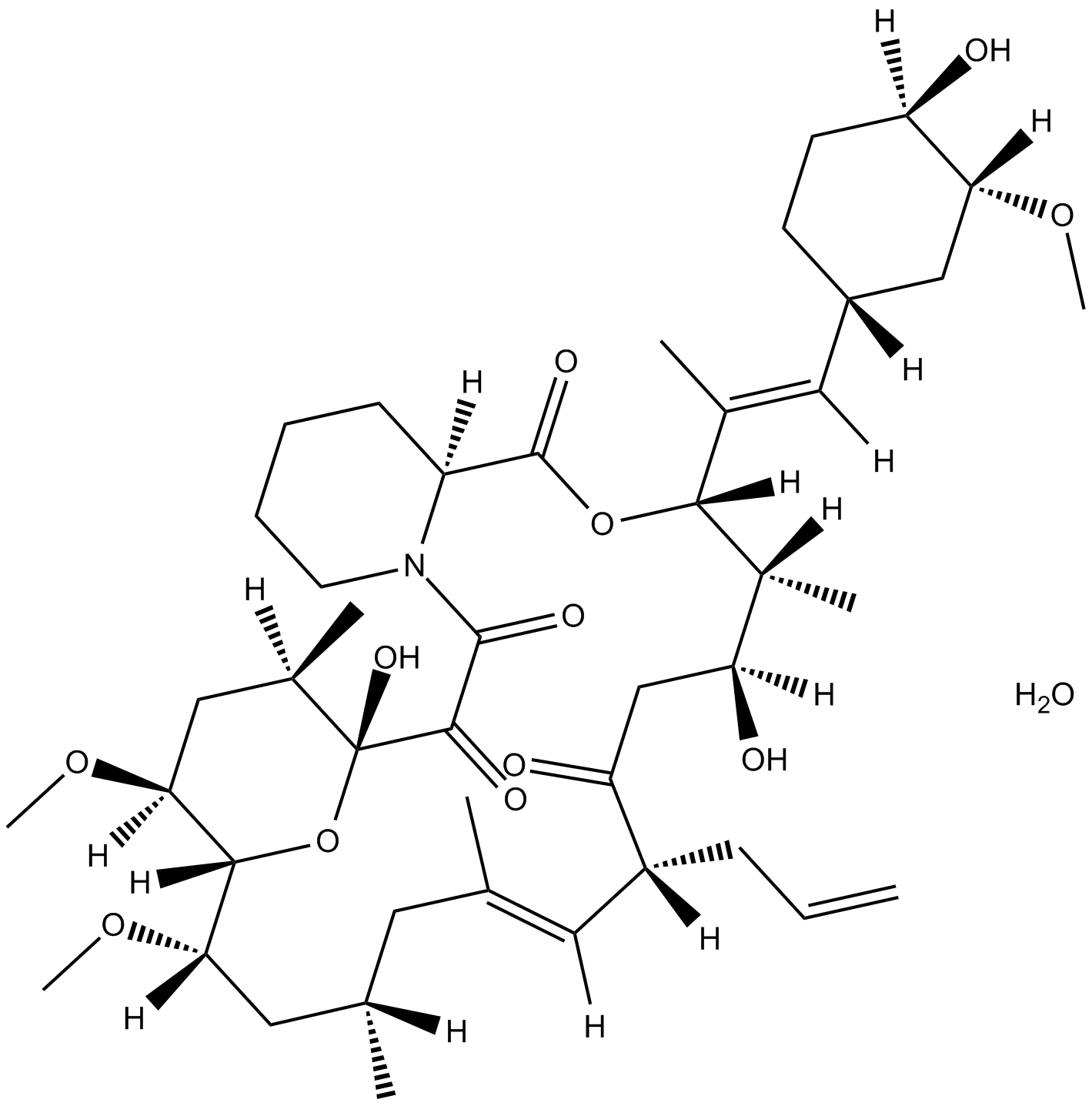
Tacrolimus monohydrate
El monohidrato de tacrolimus (FK506 monohidrato), una lactona macrocÍclica, se une a la proteÍna de uniÓn a FK506 (FKBP) para formar un complejo e inhibe la calcineurina fosfatasa, que inhibe la transducciÓn de seÑales de linfocitos T y la transcripciÓn de IL-2.
-
GC33200
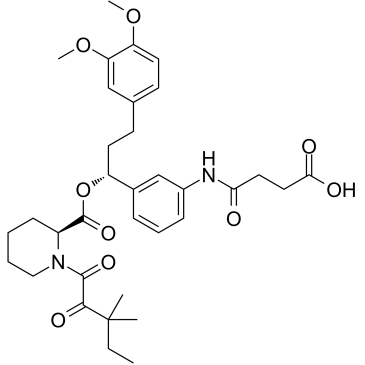
Target Protein-binding moiety 13
El resto 13 de uniÓn a proteÍna diana (resto 1 de uniÓn a PROTAC FKBP12) es un ligando sintético para FKBP (SLF). El resto de uniÓn a proteÍna diana 13 (resto de uniÓn a PROTAC FKBP12 1) se puede utilizar en la sÍntesis de PROTAC.
-
GC66370
Zapalog
Zapalog es un heterodimerizador de molécula pequeÑa fotoescindible que se puede usar para iniciar repetidamente y terminar instantÁneamente una interacciÓn fÍsica entre dos proteÍnas objetivo. Zapalog dimeriza dos proteÍnas marcadas con los dominios FKBP y DHFR hasta que la exposiciÓn a la luz provoca su fotÓlisis.