JAK/STAT Signaling

Targets for JAK/STAT Signaling
Products for JAK/STAT Signaling
- Cat.No. Nombre del producto Información
-
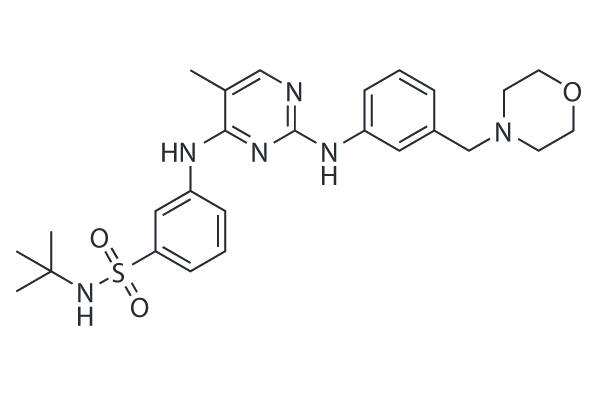
GC14170
Nifuroxazide
La nifuroxazida es un inhibidor eficaz de STAT3, ademÁs ejerce una potente actividad antitumoral y antimetastasis.
-
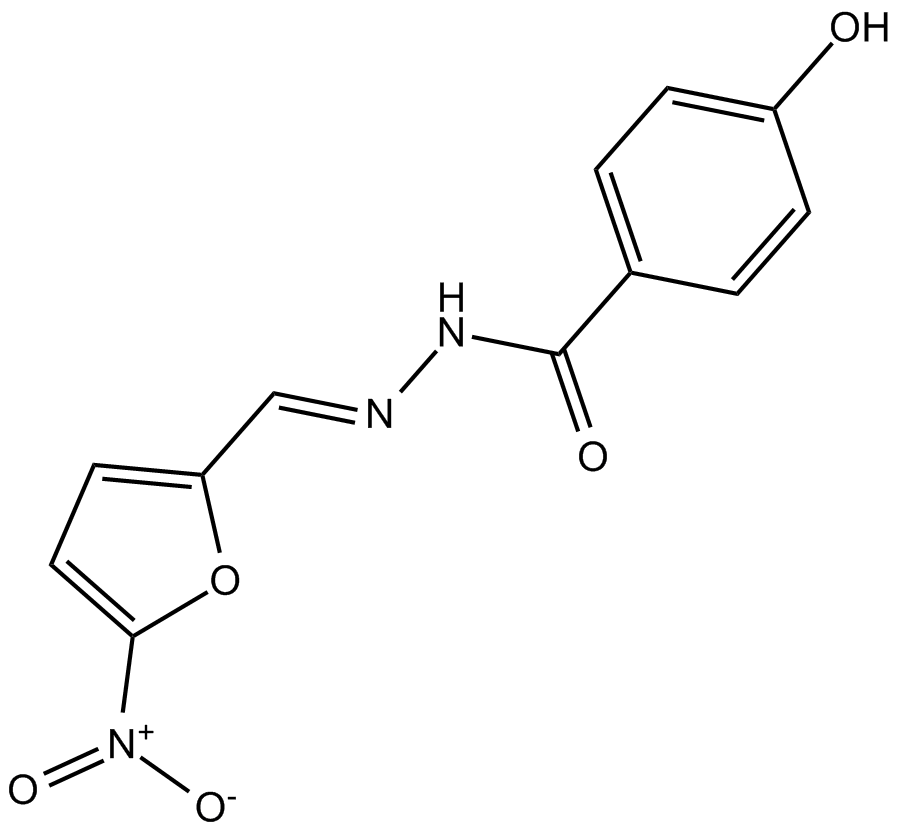
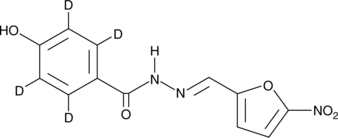
GC48839
Nifuroxazide-d4
An internal standard for the quantification of nifuroxazide
-
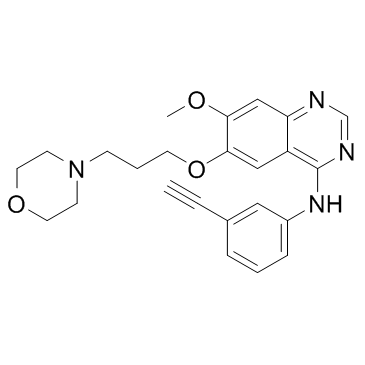
GC33131
NRC-2694
NRC-2694 es un antagonista del receptor del factor de crecimiento epidérmico (EGFR) con propiedades anticancerÍgenas y antiproliferativas.
-
GC14653
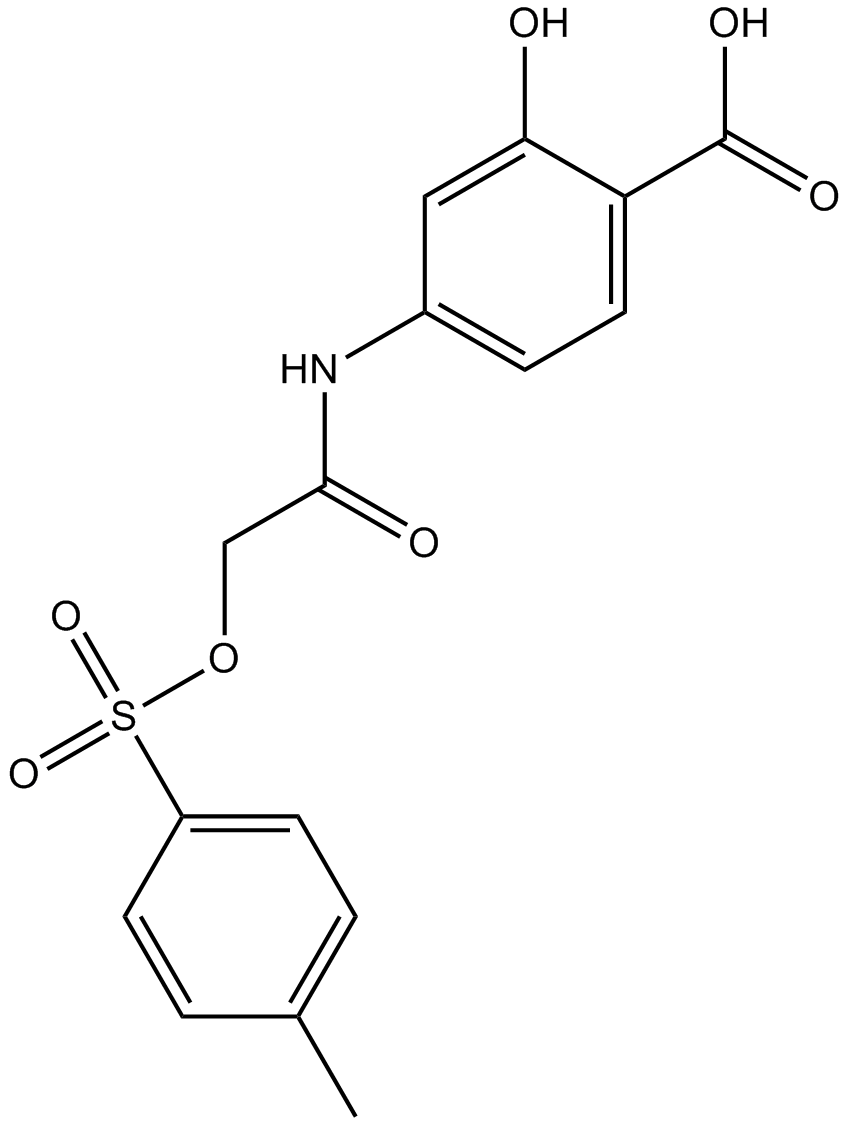
NSC 74859
NSC 74859 (S3I-201) es un inhibidor selectivo de Stat3 con un IC50 de 86 μM.
-
GC69594
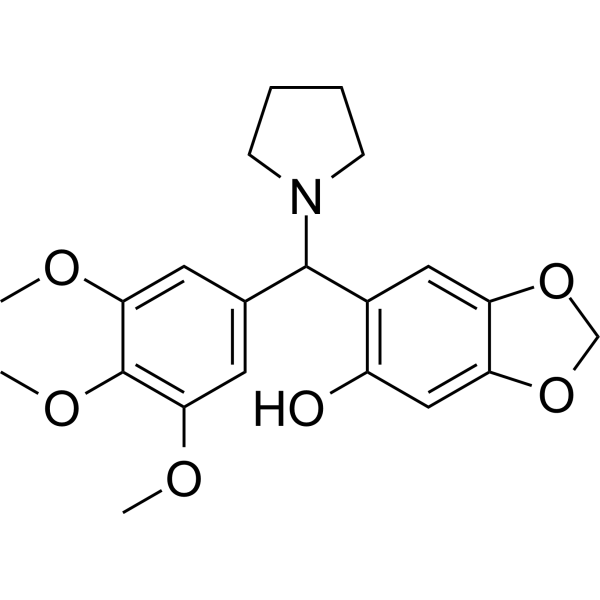
NSC-370284
NSC-370284 es un inhibidor selectivo de la 10-11 translocación 1 (TET1) y la 5-hidroximetilcitosina (5hmC). NSC-370284 inhibe significativamente los niveles de expresión de TET1 al dirigirse a STAT3/5.
-
GC14103
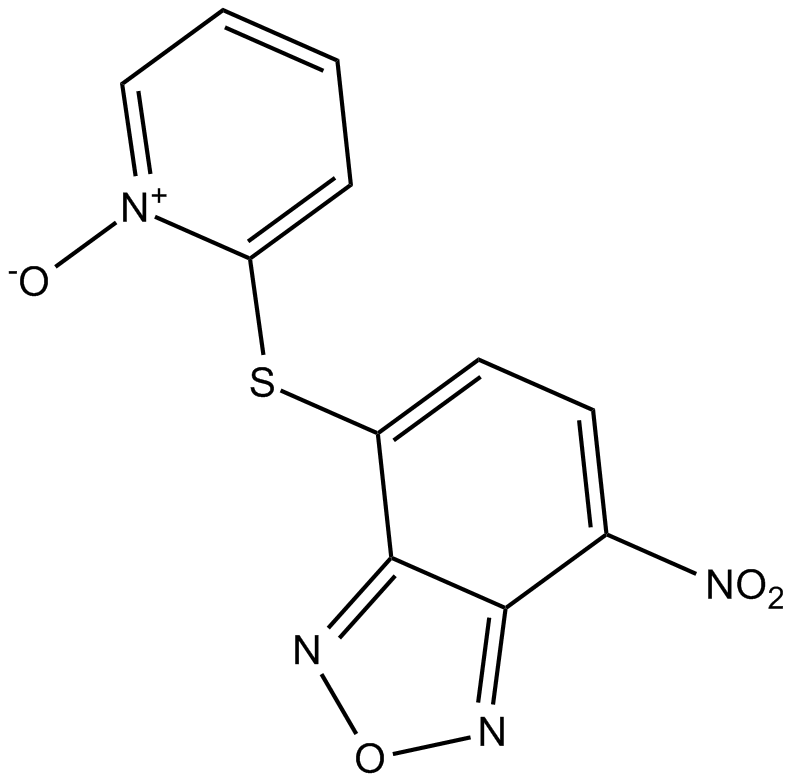
NSC228155
NSC228155 es un activador de EGFR, se une a la región extracelular de EGFR y mejora la fosforilación de tirosina de EGFR. NSC228155 también es un potente inhibidor de la interacción KIX-KID, inhibe el dominio inducible por quinasa (KID) de CREB y el dominio que interactúa con KID (KIX) de CBP, con una IC50 de 0,36 μM.
-
GC69596
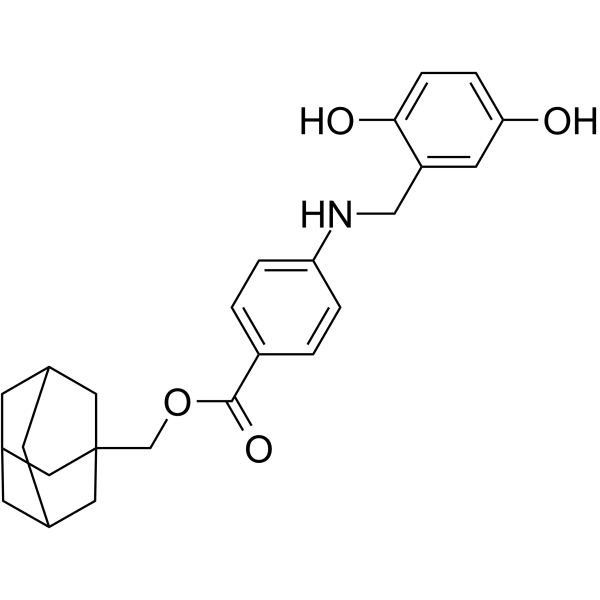
NSC689857
NSC689857 es un inhibidor efectivo de EGFR y SCFSKP2, con un IC50 de 36 μM para Skp2-Cks1. NSC689857 puede inhibir la fosforilación de p27 (IC50=30 μM). NSC689857 tiene diferentes niveles de actividad en distintos tipos de cáncer, siendo más resistente a las células leucémicas que a otros tipos de células cancerosas.
-
GC12712
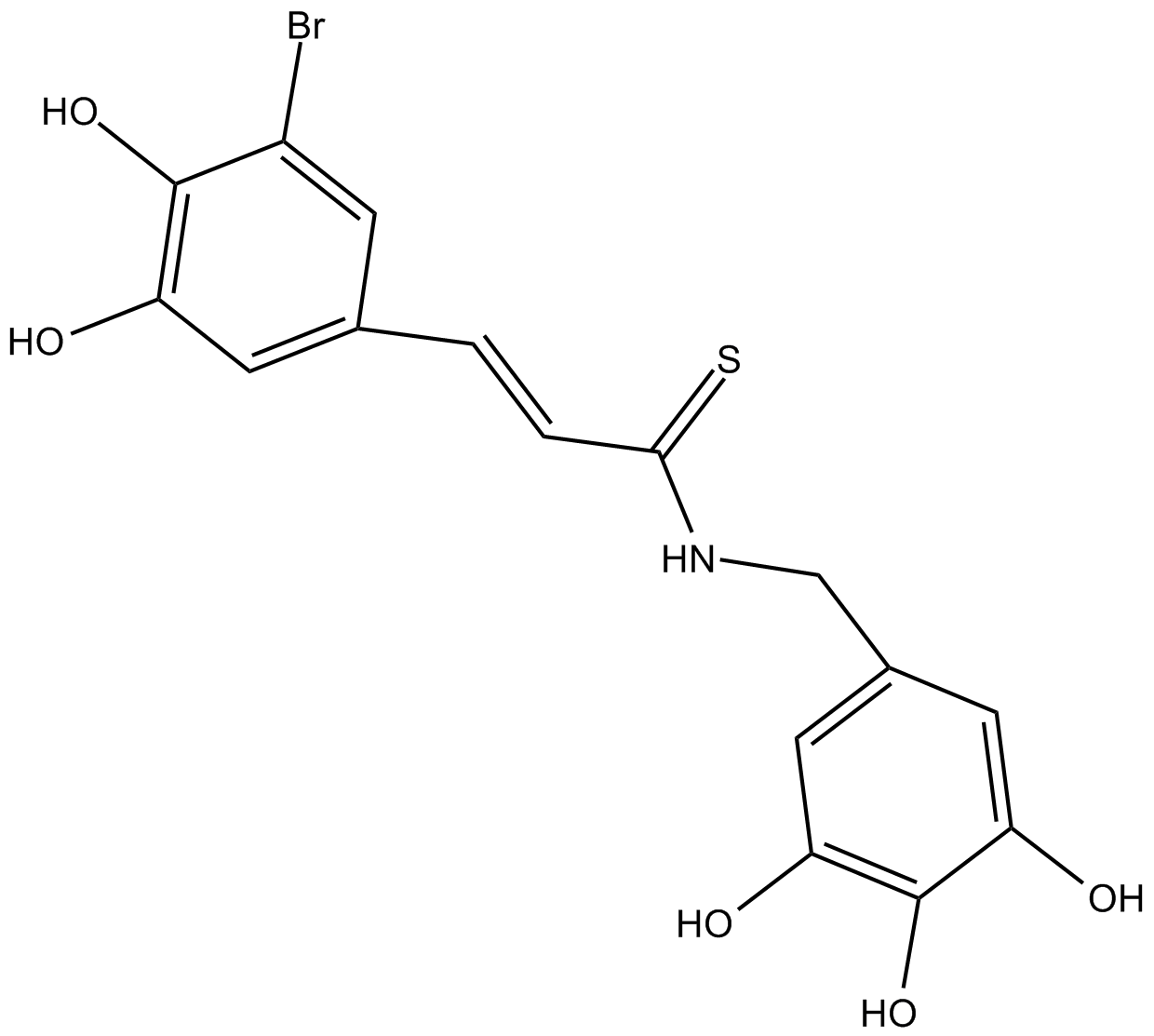
NT157
IRS-1/2 inhibitor, inhibits IGF-1R and STAT3 signaling pathway
-
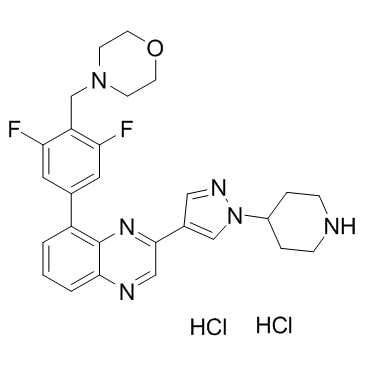
GC36783
NVP-BSK805 dihydrochloride
El diclorhidrato de NVP-BSK805 es un inhibidor de JAK2 competitivo con ATP, con IC50 de 0,48 nM, 31,63 nM, 18,68 nM y 10,76 nM para JAK2 JH1 (homologÍa 1 de JAK), JAK1 JH1, JAK3 JH1 y TYK2 JH1, respectivamente.
-
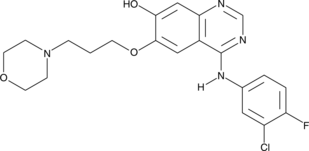
GC44491
O-Desmethyl Gefitinib
O-desmetil gefitinib es un metabolito activo de gefitinib en plasma humano. La formaciÓn de O-desmetil gefitinib depende de la actividad de CYP2D6. O-desmetil gefitinib inhibe EGFR con una IC50 de 36 nM en ensayos subcelulares.
-
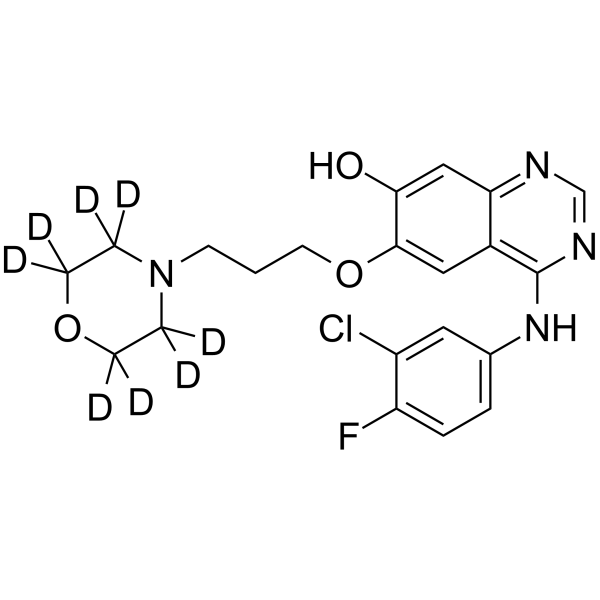
GC68321
O-Desmethyl gefitinib-d8
-
GC61627
Ochromycinone
La ocromicinona ((Rac)-STA-21) es un antibiÓtico natural y un inhibidor de STAT3.

-
GC31677
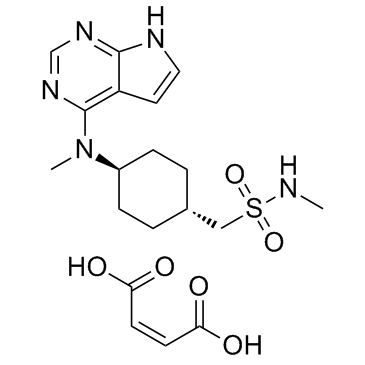
Oclacitinib maleate (PF-03394197 maleate)
El maleato de oclacitinib (PF-03394197 maleato) (PF-03394197 maleato) es un nuevo inhibidor de JAK.
-
GC15370
Olmutinib (HM61713, BI 1482694)
Olmutinib (HM61713, BI 1482694) (HM61713; BI-1482694) es un tercer inhibidor irreversible de la tirosina quinasa del EGFR activo por vÍa oral que se une a un residuo de cisteÍna cerca del dominio quinasa. Olmutinib (HM61713, BI 1482694) se usa para NSCLC.
-
GC64770
Oritinib
Oritinib (SH-1028), un TKI EGFR irreversible de tercera generaciÓn, supera la resistencia mediada por T790M en el cÁncer de pulmÓn de células no pequeÑas. Oritinib (SH-1028), un inhibidor selectivo mutante de la actividad de la cinasa de EGFR, inhibe las cinasas EGFRWT, EGFRL858R, EGFRL861Q, EGFRL858R/T790M, EGFRd746-750 y EGFRd746-750/T790M, con IC50 de 18, 0,7, 4, 0,1, 1,4 y 0,89 nM, respectivamente.

-
GC17530
OSI-420
OSI-420 (OSI-420) es un metabolito activo de erlotinib. Erlotinib es un potente inhibidor de la tirosina cinasa del EGFR.
-
GC36819
Osimertinib dimesylate
El dimesilato de osimertinib (dimesilato de AZD-9291) es un inhibidor de EGFR selectivo irreversible y mutante con IC50 de 12 y 1 nM contra EGFRL858R y EGFRL858R/T790M, respectivamente.
-
GC69635
OSM-SMI-10B
OSM-SMI-10B es un derivado de OSM-SMI-10. La co-incubación de OSM-SMI-10B y OSM (Oncostatina M) reduce significativamente la fosforilación de STAT3 inducida por OSM en células cancerosas.

-
GC41329
Pacritinib
FMS-like tyrosine kinase 3 (FLT3) and Janus kinase 2 (JAK2) are tyrosine kinases that mediate cytokine signaling and are frequently mutated in cancers, particularly acute myeloid leukemia.
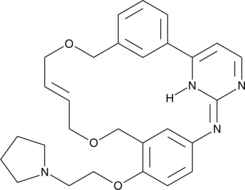
-
GC66405
Panitumumab
Panitumumab (ABX-EGF) es un anticuerpo monoclonal IgG2 anti-EGFR totalmente humano con actividad antitumoral. Panitumumab inhibe la proliferaciÓn, supervivencia y angiogénesis de las células tumorales. Panitumumab se puede utilizar en la investigaciÓn de cÁnceres, como el cÁncer de colon.

-
GC66333
Panitumumab (anti-EGFR)
Panitumumab (anti-EGFR) es un anticuerpo monoclonal IgG2 anti-EGFR completamente humano con actividad antitumoral. Panitumumab (anti-EGFR) inhibe la proliferaciÓn, supervivencia y angiogénesis de las células tumorales. Panitumumab (anti-EGFR) se puede utilizar en la investigaciÓn de cÁnceres, como el cÁncer de colon.

-
GC69665
Patritumab
Patritumab (anticuerpo recombinante humano anti-ERBB3) es un anticuerpo monoclonal neutralizante dirigido contra ERBB3. Patritumab tiene un efecto sinérgico con Cetuximab y puede inhibir eficazmente la fosforilación de EGFR, HER2, HER3, ERK y AKT. Patritumab también puede inducir apoptosis celular y suprimir el crecimiento de tumores xenotransplantados de cáncer de páncreas, cáncer de pulmón no microcítico y cáncer colorrectal.

-
GC15925
PD 158780
PD 158780 es un potente inhibidor de la familia EGFR con IC50 de 8 pM, 49, 52, 52 nM para EGFR, ErbB2, ErbB3 y ErbB4, respectivamente.
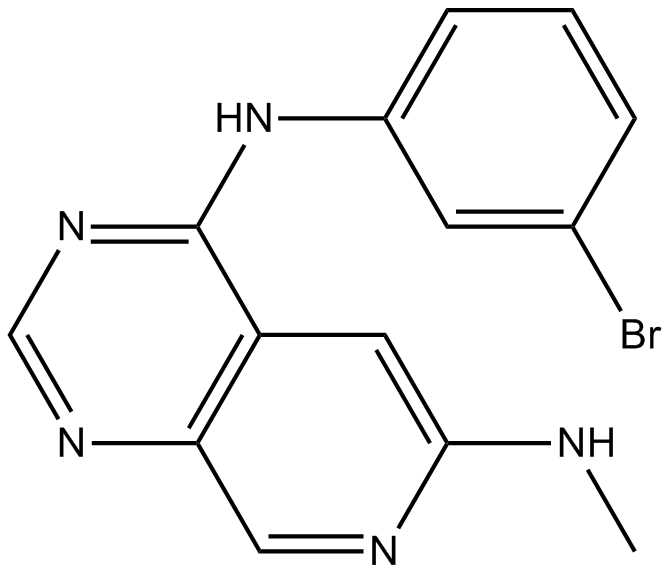
-
GC34105
PD153035 Hydrochloride (ZM 252868)
A highly potent EGFR inhibitor
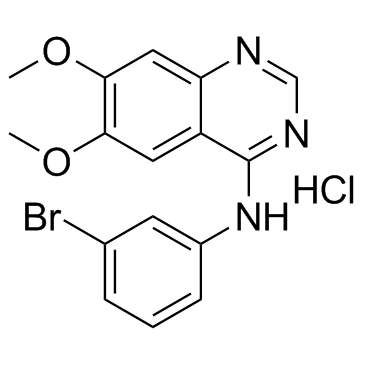
-
GC11015
PD168393
PD168393es un inhibidor potente, selectivo y permeable a las células de la tirosina quinasa EGFR y ErbB2. PD168393 inactiva irreversiblemente el receptor EGF (IC50 = 0,7 nM) y es inactivo frente al receptor de insulina, PDGFR, FGFR y PKC.
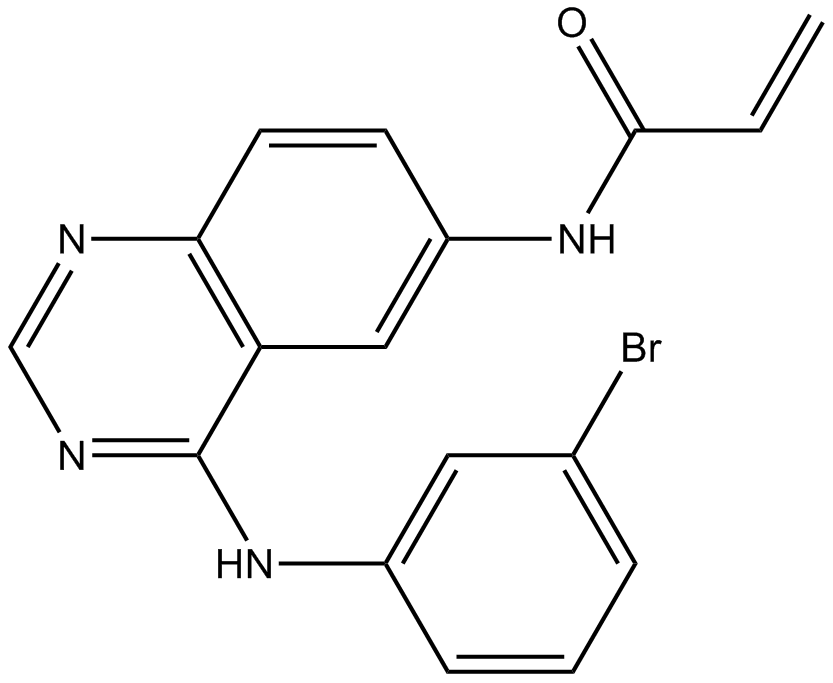
-
GC10341
Peficitinb (ASP015K, JNJ-54781532)
Peficitinb (ASP015K, JNJ-54781532) (ASP015K) es un inhibidor de JAK activo por vÍa oral, con IC50 de 3,9, 5,0, 0,7 y 4,8 nM para JAK1, JAK2, JAK3 y Tyk2, respectivamente.
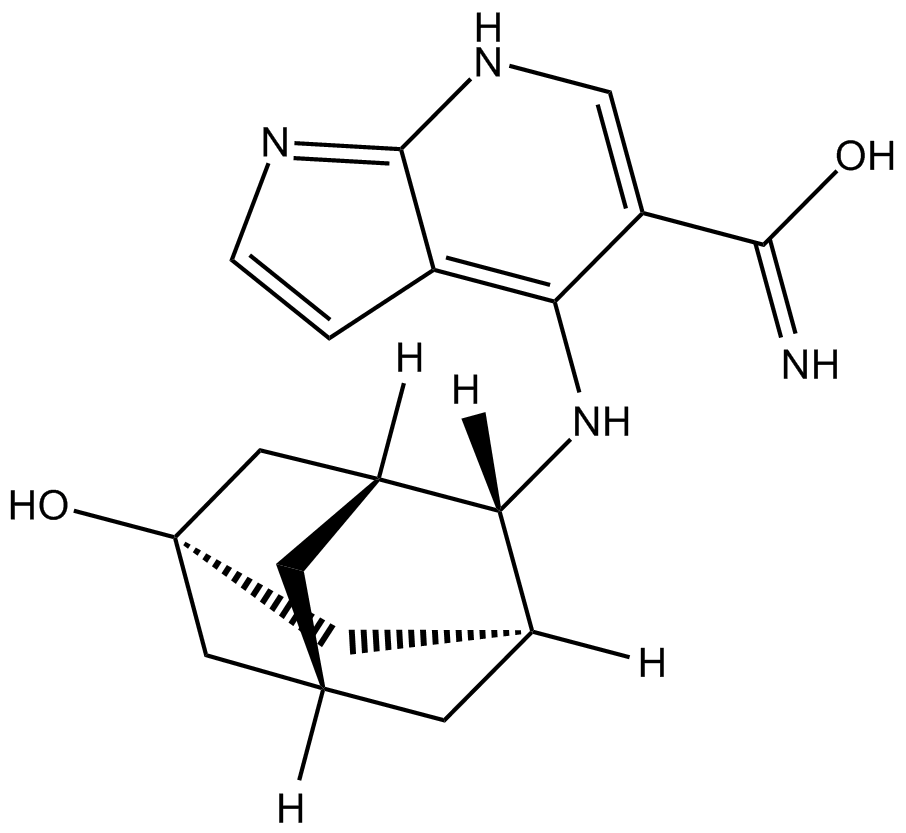
-
GC17473
Pelitinib (EKB-569)
Pelitinib (EKB-569) (EKB-569; WAY-EKB 569) es un inhibidor irreversible de EGFR con una IC50 de 38,5 nM; también inhibe ligeramente Src, MEK/ERK y ErbB2 con IC50 de 282, 800 y 1255 nM, respectivamente.
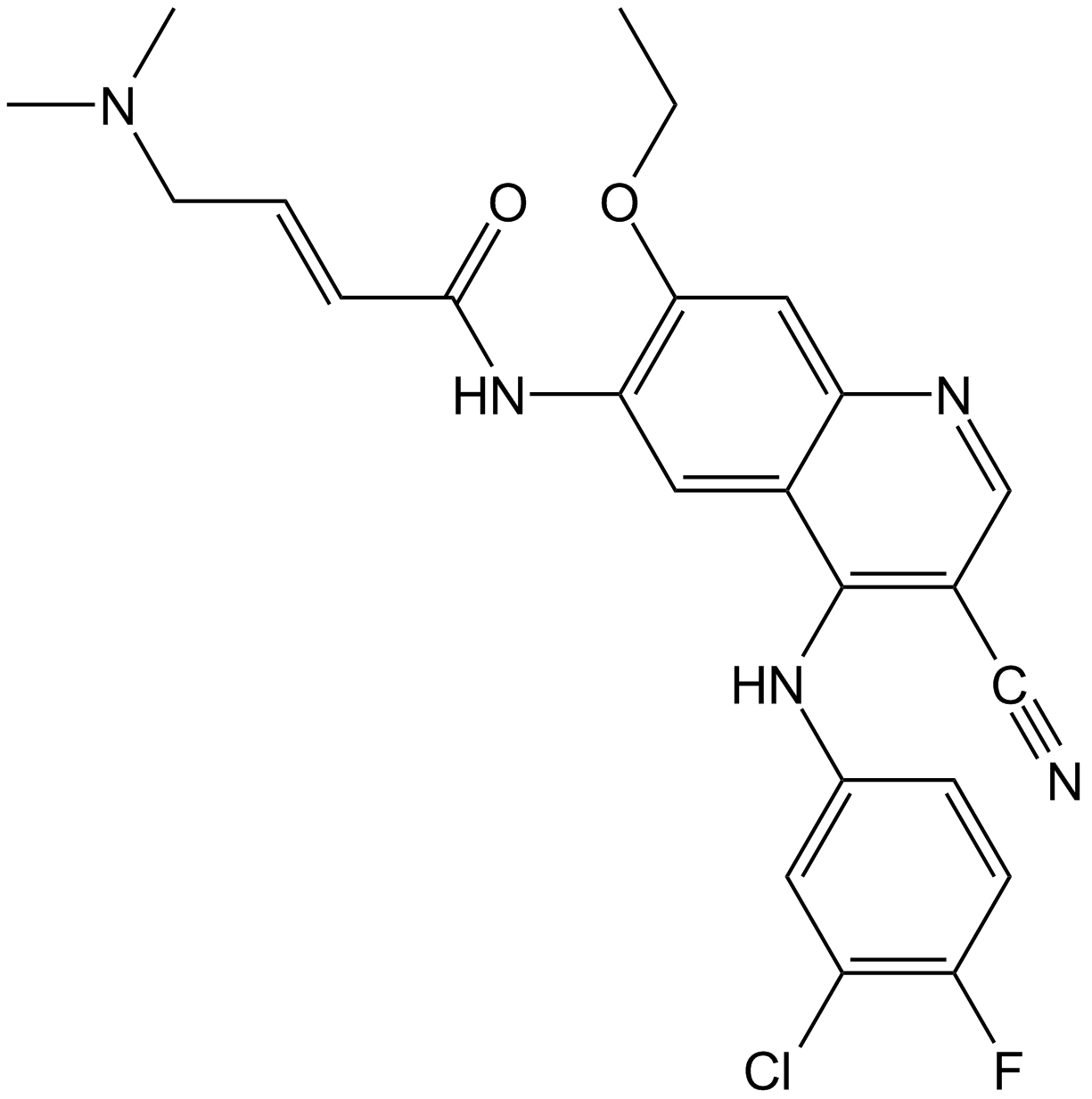
-
GC34210
Pertuzumab (Anti-Human HER2, Humanized Antibody)
Pertuzumab (Anticuerpo humanizado Anti-Humano HER2), el primero de una nueva clase de agentes designados como inhibidores de la dimerización de HER, es un anticuerpo monoclonal IgG1 humanizado que se une estéricamente al dominio II del receptor erbB2.

-
GC69690
Petosemtamab
Petosemtamab (MCLA 158) is a monoclonal antibody (mAb) that targets both EGFR (Kd: 0.22 nM) and LGR5 (Kd: 0.86 nM). Petosemtamab blocks EGFR signaling and receptor degradation in LGR5+ cancer cells. It can be used for research on solid tumors such as head and neck squamous cell carcinoma (HNSCC), metastatic colorectal cancer (CRC), among others.

-
GC14938
PF-03394197(Oclacitinib)
Novel Janus kinase inhibitor
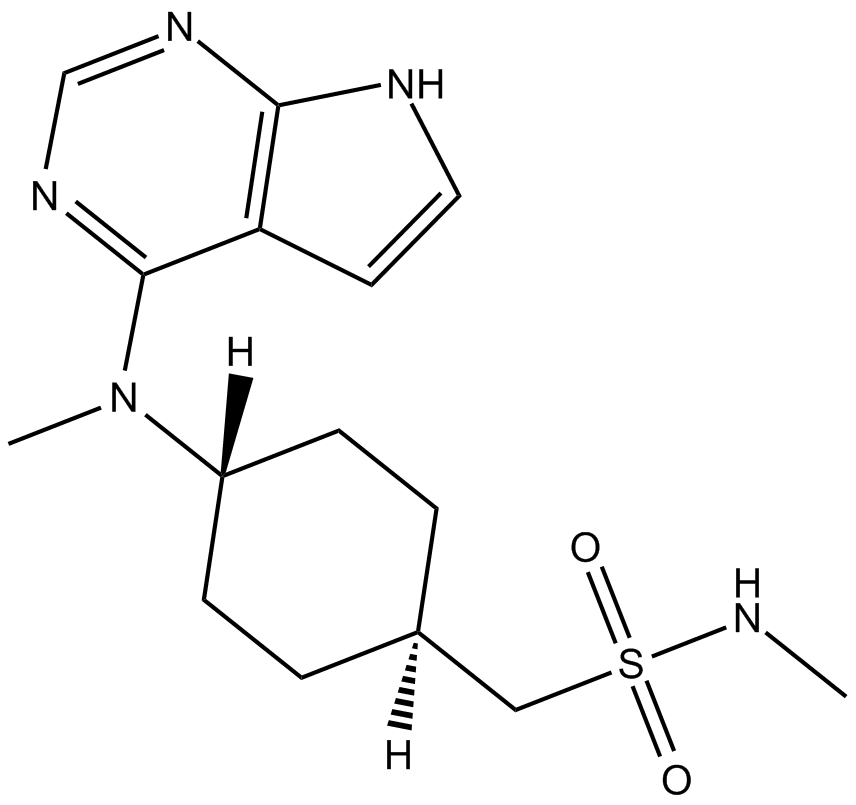
-
GC36882
PF-06263276
PF-06263276 (PF 6263276) es un inhibidor pan-JAK potente y selectivo, con IC50 de 2,2 nM, 23,1 nM, 59,9 nM y 29,7 nM para JAK1, JAK2, JAK3 y TYK2, respectivamente.
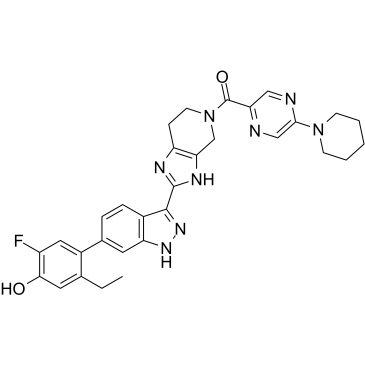
-
GC32927
PF-06459988
PF-06459988 es un inhibidor selectivo de mutantes, irreversible y de actividad oral de las formas mutantes de EGFR. PF-06459988 demuestra una alta potencia y especificidad para los EGFR mutantes dobles que contienen T790M. PF-06459988 se puede utilizar para la investigaciÓn del cÁncer.
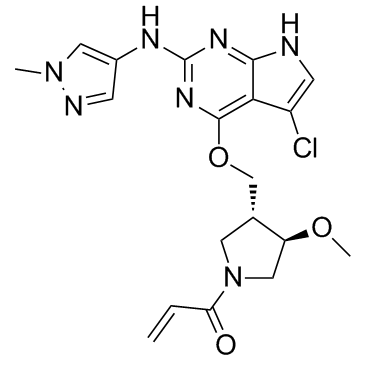
-
GC19288
PF-06651600
PF-06651600 (PF-06651600) es un inhibidor de JAK3 selectivo y activo por vÍa oral con una IC50 de 33,1 nM.
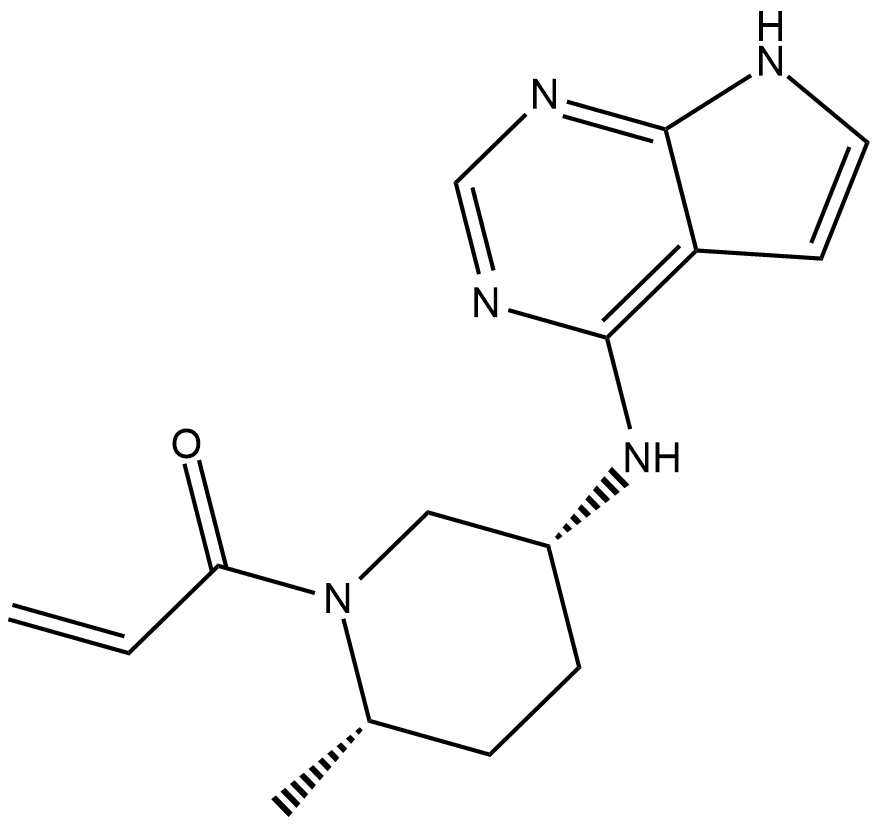
-
GC19405
PF-06700841 P-Tosylate
Brepocitinib (PF-06700841) P-tosilato es un potente inhibidor dual de Janus quinasa 1 (JAK1) y TYK2 con IC50 de 17 nM y 23 nM, respectivamente.
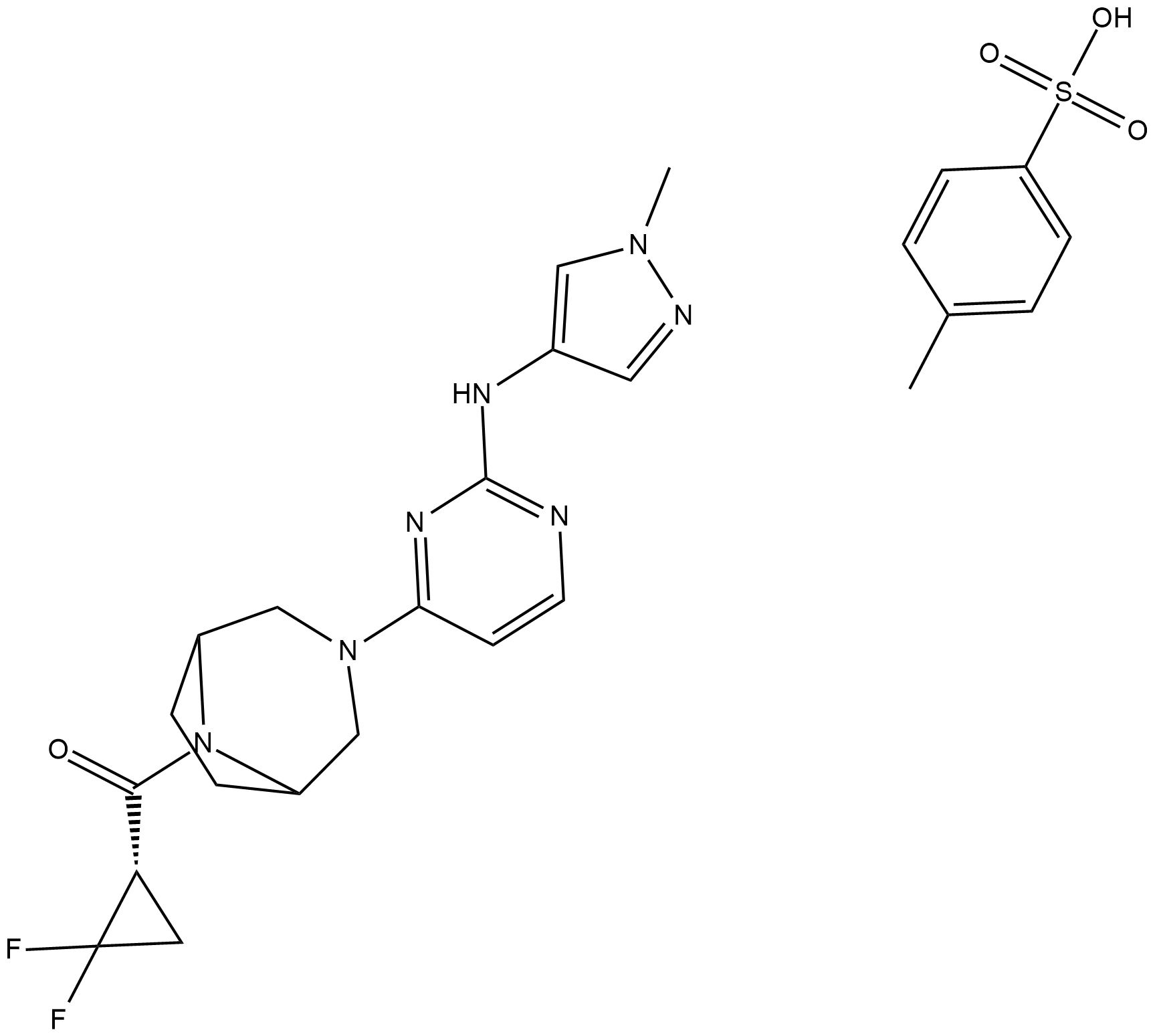
-
GN10314
Picroside I
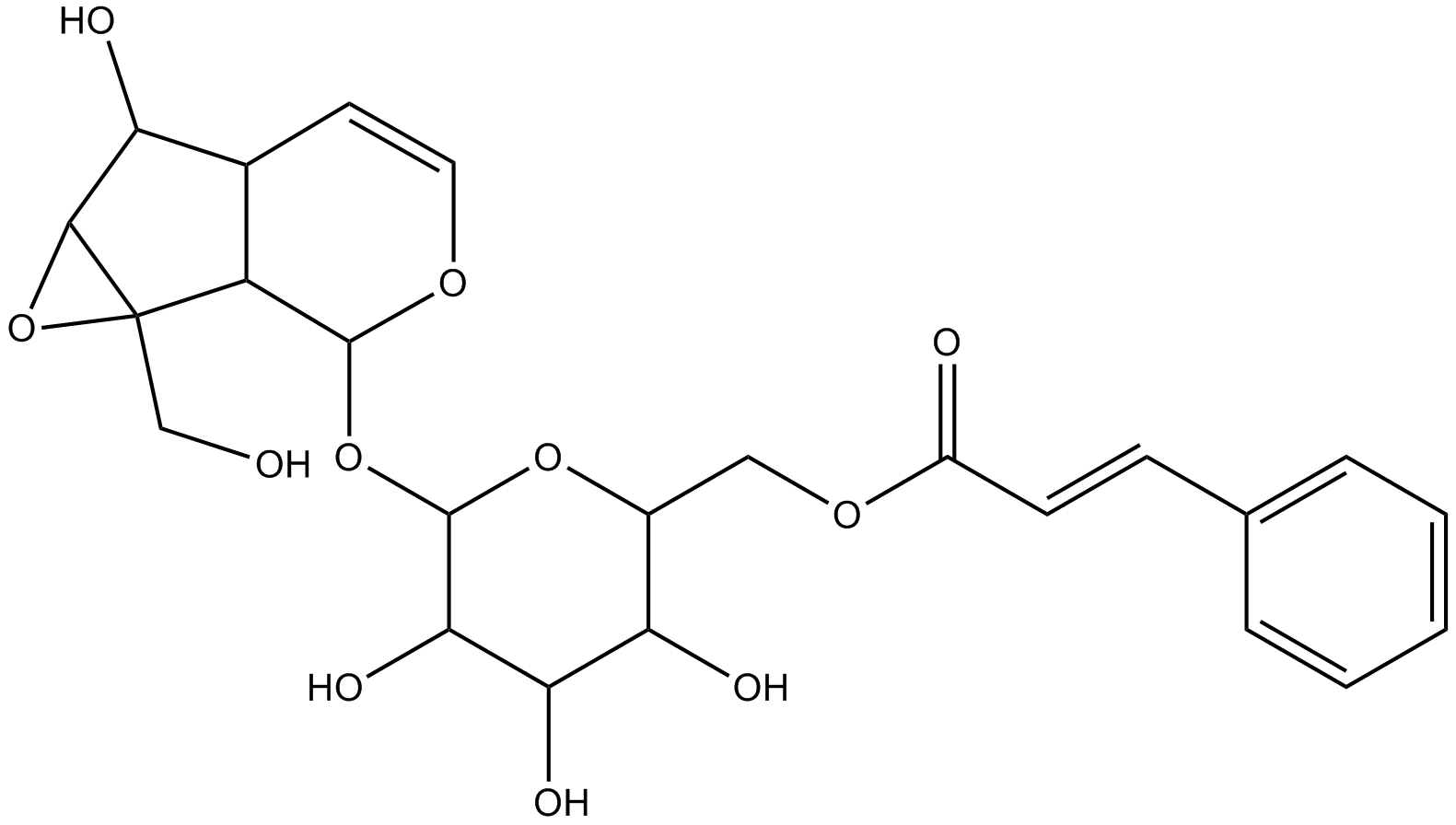
-
GC19419
PIM inhibitor 1 phosphate
El fosfato de uzansertib (INCB053914) es un inhibidor de la cinasa pan-PIM competitivo con ATP, activo por vÍa oral, con IC50 de 0,24 nM, 30 nM, 0,12 nM para PIM1, PIM2, PIM3, respectivamente. El inhibidor de PIM 1 fosfato tiene una amplia actividad antiproliferativa contra una variedad de lÍneas de células tumorales hematolÓgicas.

-
GC36918
PIM-447 dihydrochloride
El diclorhidrato de PIM447 (diclorhidrato de LGH447) es un inhibidor pan-PIM quinasa selectivo, potente y disponible por vÍa oral, con valores de Ki de 6, 18 y 9 pM para PIM1, PIM2 y PIM3, respectivamente. El diclorhidrato de PIM447 muestra efectos duales antimieloma y protectores Óseos. El diclorhidrato de PIM447 induce la apoptosis.

-
GC65278
PIM1-IN-1
PIM1-IN-1 es un inhibidor potente y altamente selectivo de PIM1/3, con IC50 de 7, 5530 y 70 nM para PIM1, PIM2 y PIM3, respectivamente, inhibe la fosforilaciÓn de BAD, un objetivo aguas abajo de PIM, con un EC50 de 262 nm. PIM1-IN-1 no muestra ningÚn efecto evidente sobre la uniÓn de FLT3 o hERG. Actividad antiproliferativa y anticancerÍgena.
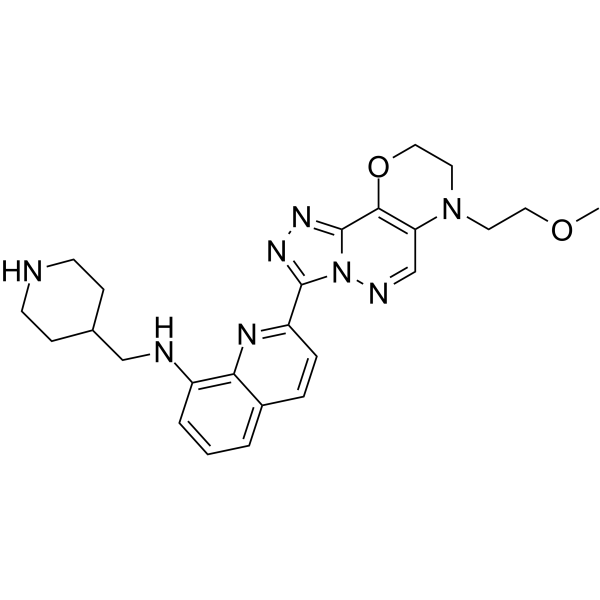
-
GC19398
PIM447
PIM447 (LGH447) es un potente inhibidor pan-PIM cinasa selectivo, disponible por vÍa oral, con valores de Ki de 6, 18 y 9 pM para PIM1, PIM2 y PIM3, respectivamente. PIM447 muestra efectos duales antimieloma y protectores Óseos. PIM447 induce la apoptosis.
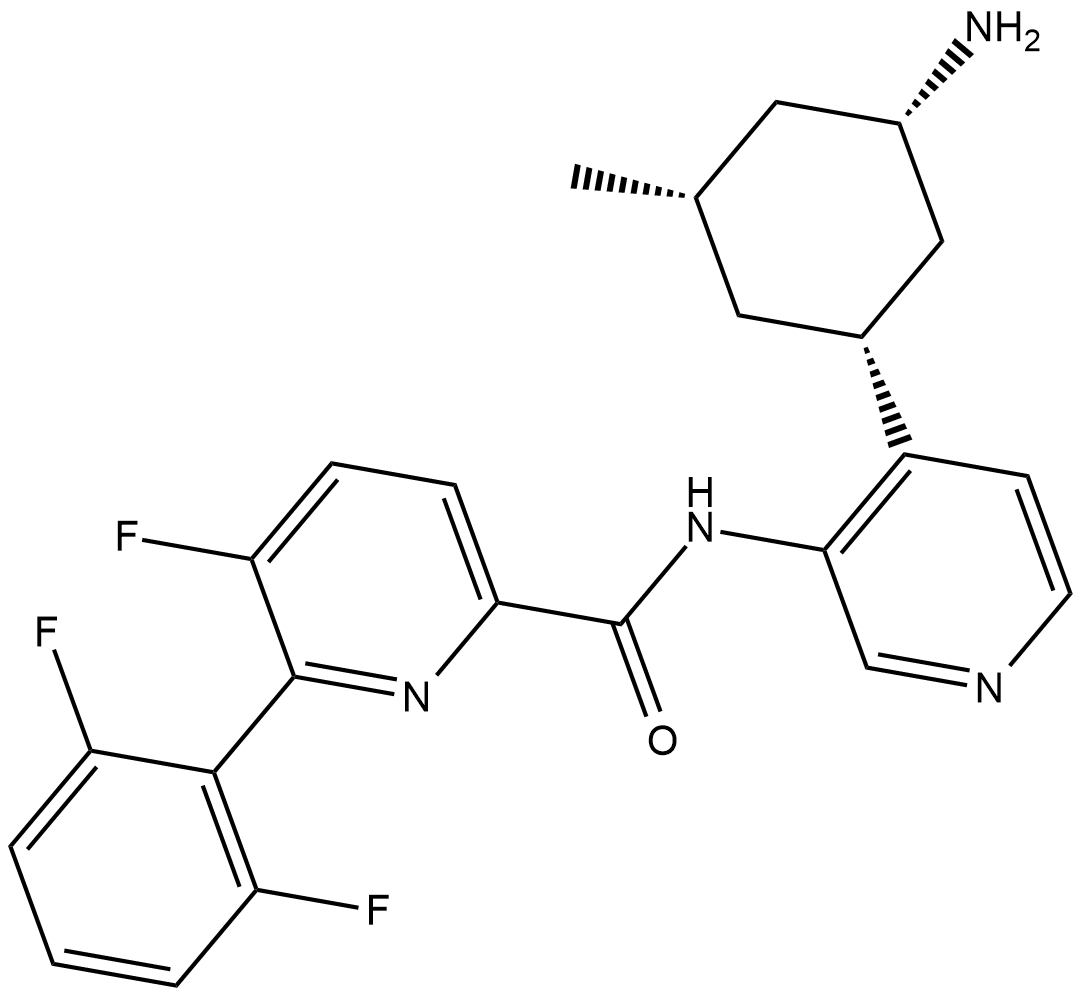
-
GC10643
Pimozide
La pimozida es un antagonista de los receptores de dopamina, con Kis de 1,4 nM, 2,5 nM y 588 nM para los receptores de dopamina D2, D3 y D1, respectivamente, y también tiene afinidad por el adrenoceptor α1, con una Ki de 39 nM; La pimozida también inhibe STAT3 y STAT5.

-
GC45756
Pimozide-d4
La pimozida D4 (R6238 D4) es una pimozida marcada con deuterio.
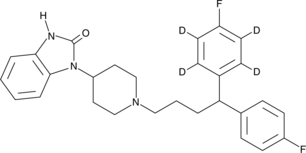
-
GC40915
PKI-166
PKI-166 es un inhibidor de la tirosina quinasa EGFR potente, selectivo y biodisponible por vÍa oral, con una IC50 de 0,7 nM.
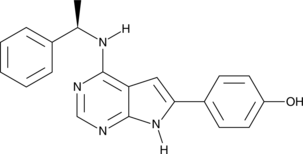
-
GC69728
Ponezumab
Ponezumab (PF-04360365) es un anticuerpo monoclonal humanizado contra la proteína amiloide tipo beta IgG2. Ponezumab puede reducir los niveles de Aβ en el sistema nervioso central y mejorar el rendimiento de ratones en diversos modelos de aprendizaje y memoria. Ponezumab se puede utilizar para investigar la enfermedad de Alzheimer.

-
GC64701
Povorcitinib
Povorcitinib es un inhibidor potente y selectivo de JAK1.
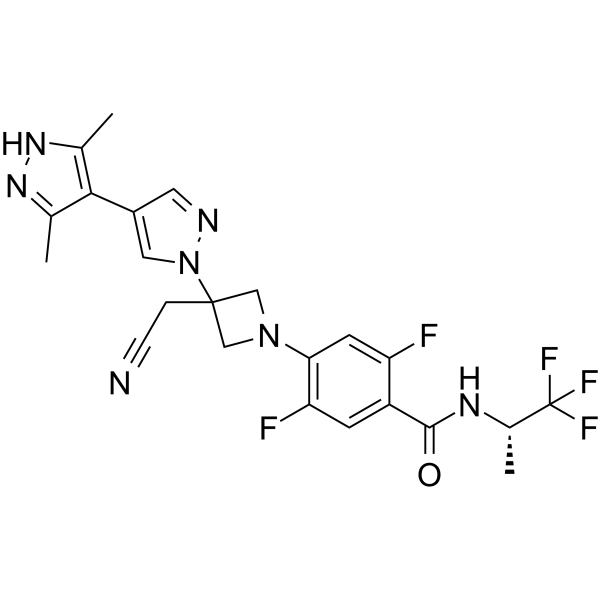
-
GC64700
Povorcitinib phosphate
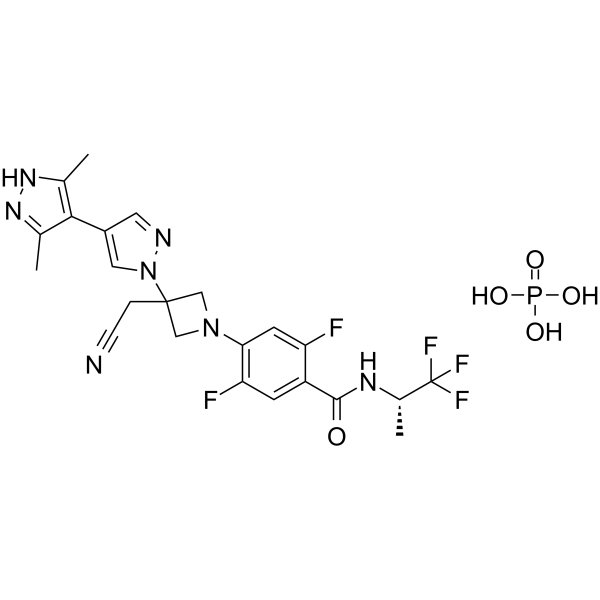
-
GC17916
Poziotinib
A irreversible pan-HER inhibitor
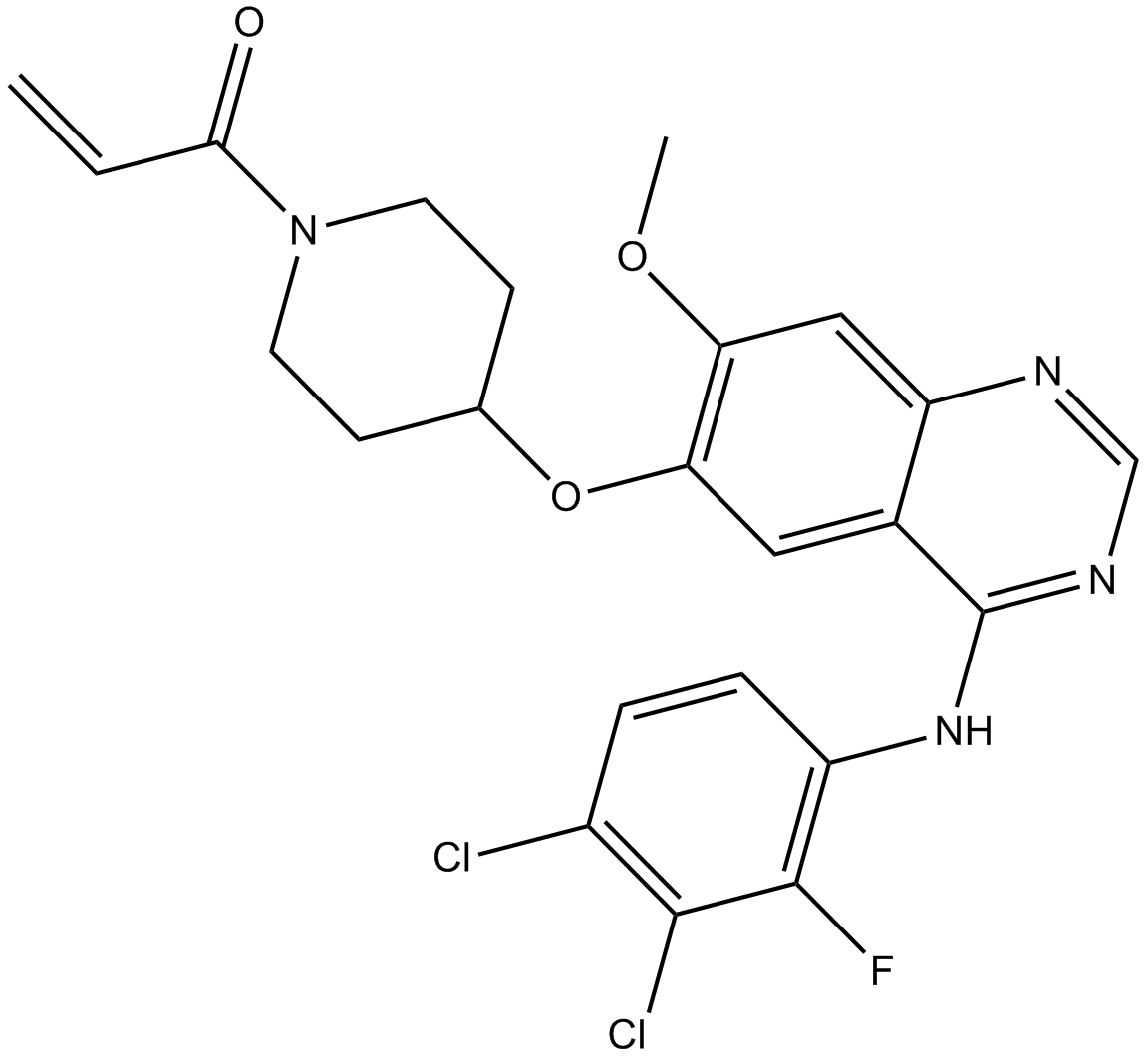
-
GC30601
Propanamide
La propanamida es un inhibidor de JAK potente y selectivo extraÍdo de la patente WO2012122452A1, Compuesto II, tiene el potencial para el tratamiento de trastornos de la piel (como el lupus cutÁneo).
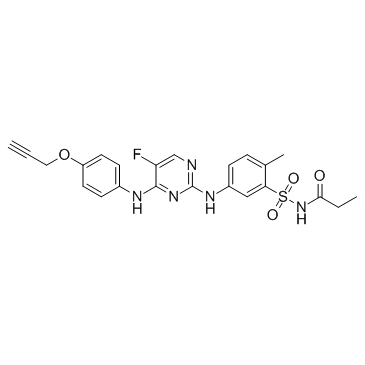
-
GC34742
Protosappanin A
La protosappanina A (PTA), un ingrediente inmunosupresor y el principal compuesto de bifenilo aislado de Caesalpinia sappan L, suprime la vÍa de inflamaciÓn dependiente de JAK2/STAT3 mediante la regulaciÓn negativa de la fosforilaciÓn de JAK2 y STAT3.
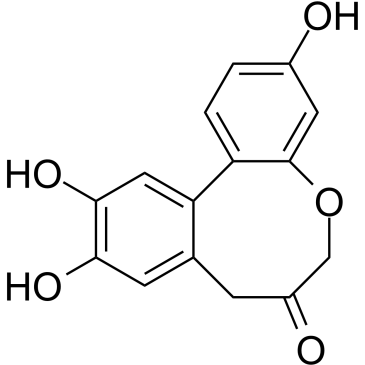
-
GC69780
Pumecitinib
Pumecitinib es un inhibidor de JAK con actividad antiinflamatoria.
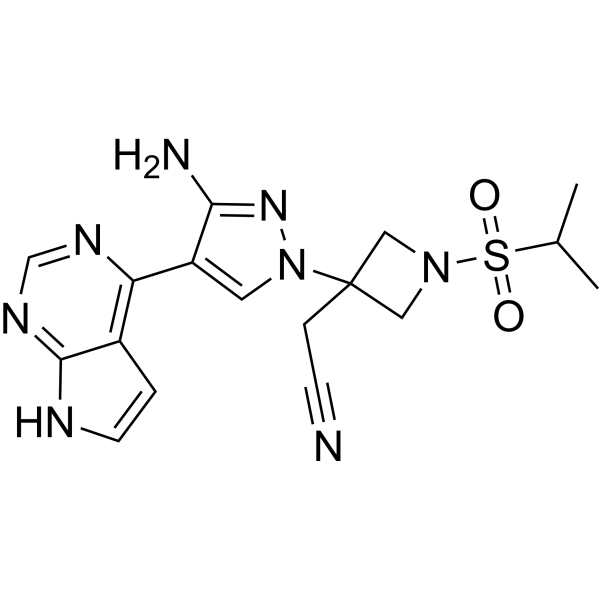
-
GC32733
Pyrotinib (SHR-1258)
Pyrotinib (SHR-1258) (SHR-1258) es un inhibidor dual potente y selectivo de EGFR/HER2 con IC50 de 13 y 38 nM, respectivamente.
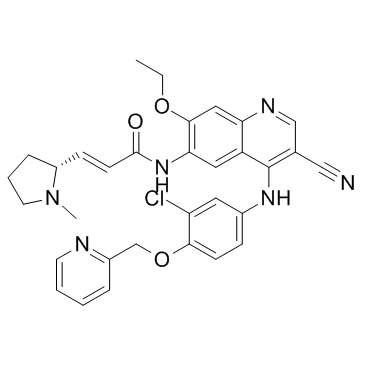
-
GC32989
Pyrotinib dimaleate (SHR-1258 dimaleate)
El dimaleato de pirotinib (dimaleato de SHR-1258) (dimaleato de SHR-1258) es un inhibidor dual potente y selectivo de EGFR/HER2 con IC50 de 13 y 38 nM, respectivamente.
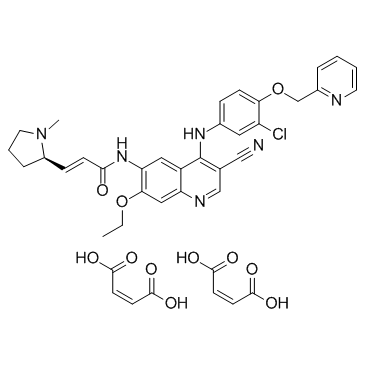
-
GC65201
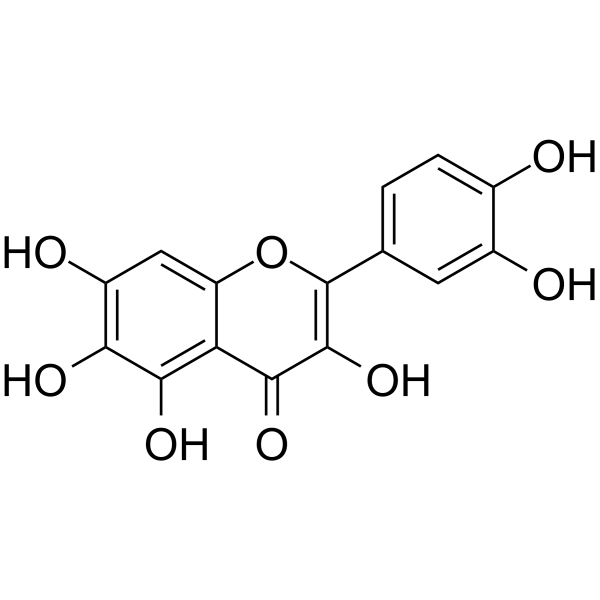
Quercetagetin
La quercetagetina (6-hidroxiquercetina) es un flavonoide.
-
GC39081
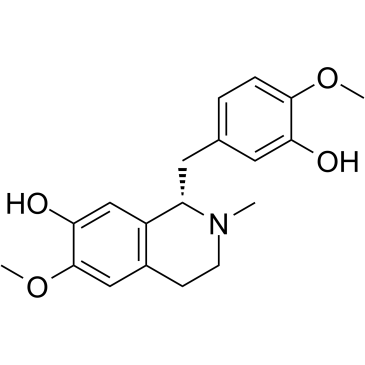
Reticuline
La reticulina muestra efectos antiinflamatorios a través de las vÍas de seÑalizaciÓn JAK2/STAT3 y NF-κB.
-
GC12038
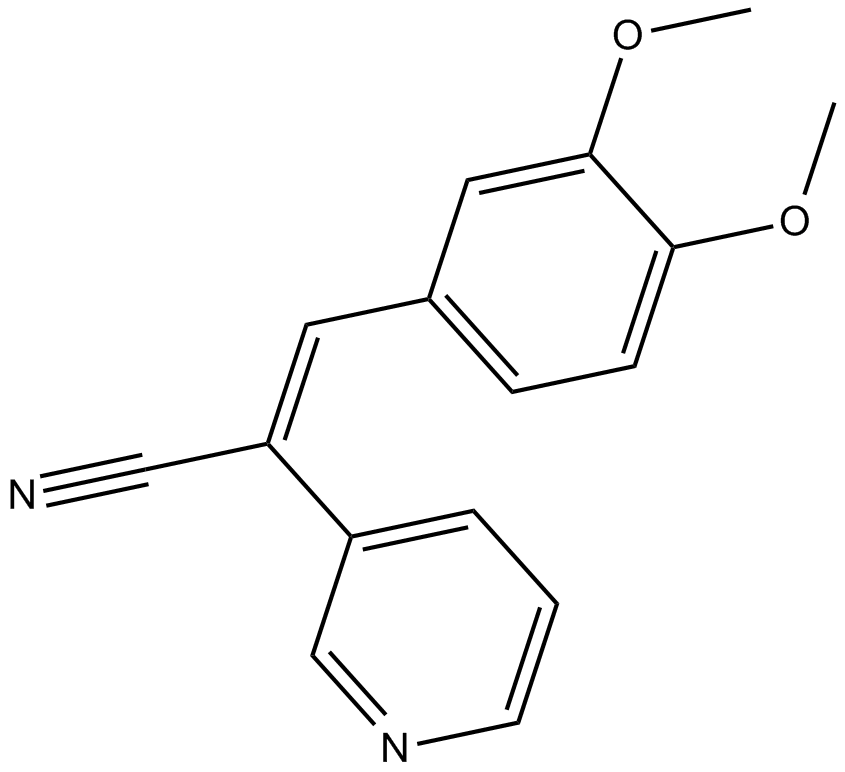
RG 13022
RG 13022 es un inhibidor de tirosina quinasa; inhibe la reacción de autofosforilación del receptor EGF con una IC50 de 4 μM.
-
GC10217
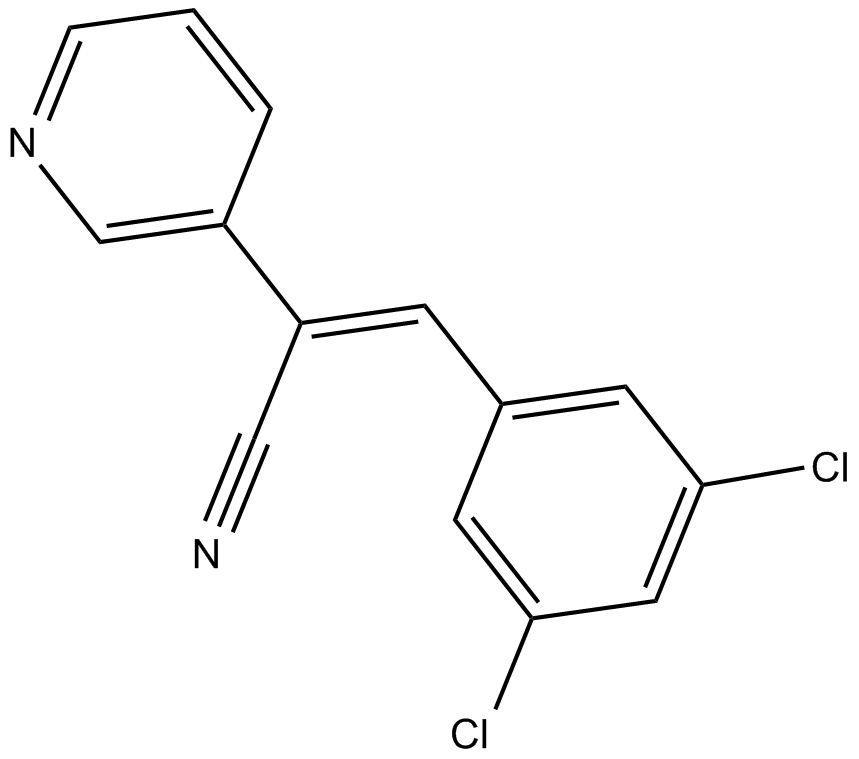
RG-14620
RG-14620 es un inhibidor de EGFR con un IC50 de 3 μM.
-
GC37522
RGB-286638
RGB-286638 es un inhibidor de CDK que inhibe la actividad quinasa de ciclina T1-CDK9, ciclina B1-CDK1, ciclina E-CDK2, ciclina D1-CDK4, ciclina E-CDK3 y p35-CDK5 con IC50 de 1, 2, 3 , 4, 5 y 5 nM, respectivamente; también inhibe GSK-3β, TAK1, Jak2 y MEK1, con IC50 de 3, 5, 50 y 54 nM.
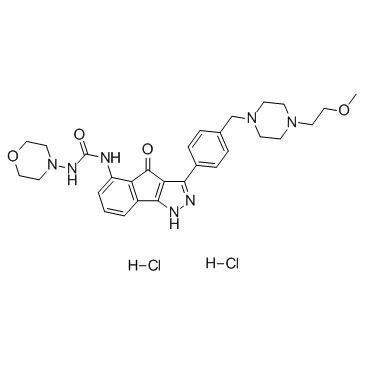
-
GC37523
RGB-286638 free base
RGB-286638 es un inhibidor de CDK que inhibe la actividad quinasa de ciclina T1-CDK9, ciclina B1-CDK1, ciclina E-CDK2, ciclina D1-CDK4, ciclina E-CDK3 y p35-CDK5 con IC50 de 1, 2, 3 , 4, 5 y 5 nM, respectivamente; también inhibe GSK-3β, TAK1, Jak2 y MEK1, con IC50 de 3, 5, 50 y 54 nM.
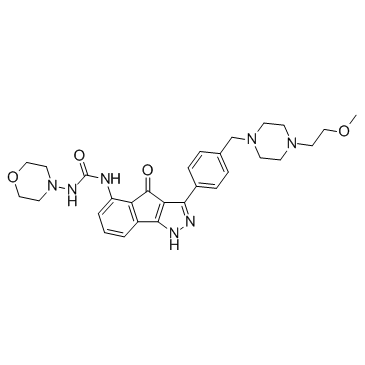
-
GC25869
RO495
RO495 (CS-2667) is a potent inhibitor of Non-receptor tyrosine-protein kinase 2 (TYK2).
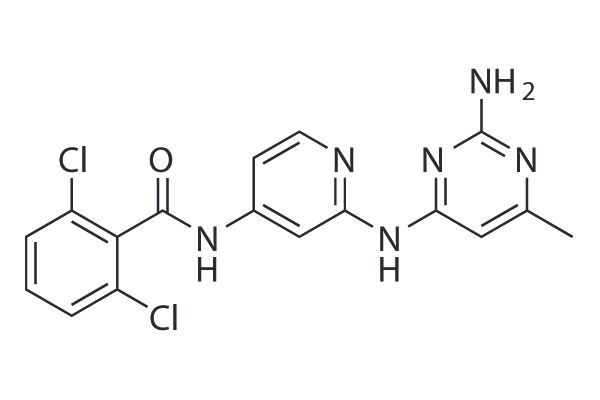
-
GC16956
RO8191
RO8191 (CDM-3008), un compuesto de imidazonaftiridina, es un agonista del receptor de interferÓn (IFN) potente y activo por vÍa oral.
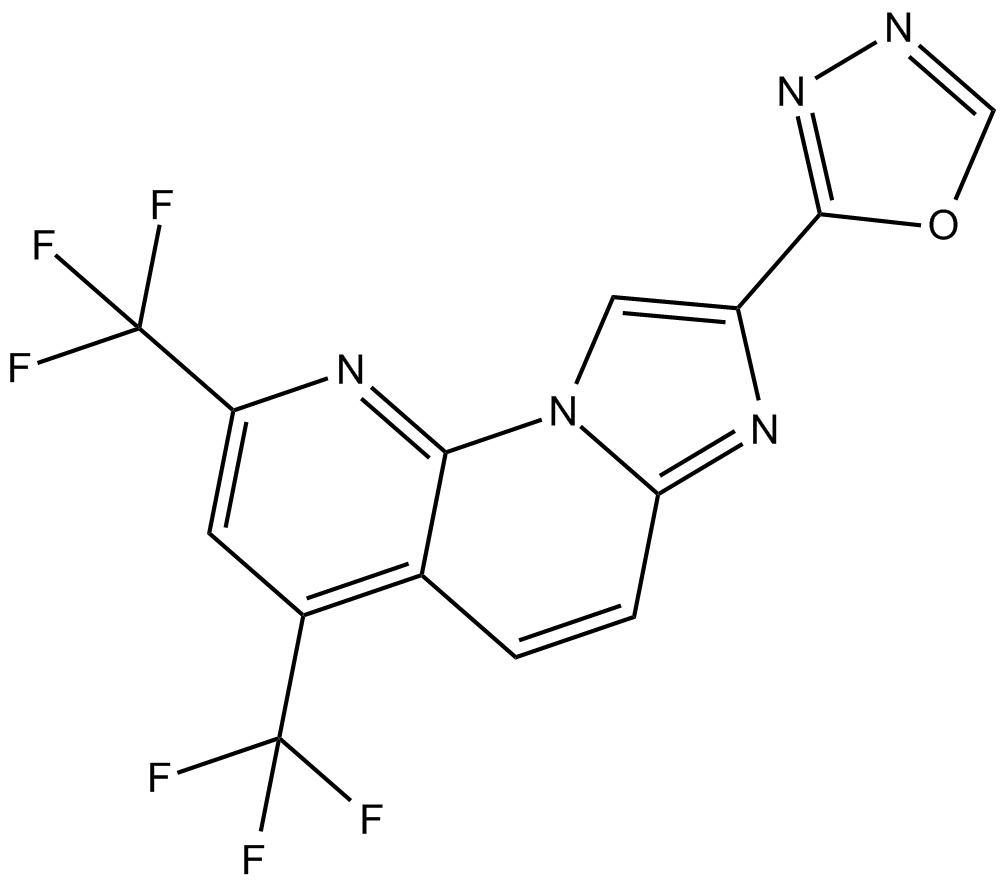
-
GC33061
Rociletinib hydrobromide (CO-1686 (hydrobromide))
El bromhidrato de rociletinib (CO-1686 (bromhidrato)) (bromhidrato de CO-1686) es un inhibidor de la cinasa administrado por vÍa oral que se dirige especÍficamente a las formas mutantes de EGFR, incluido T790M, y los valores de Ki para EGFRL858R/T790M y EGFRWT son 21,5 nM y 303,3 nM, respectivamente.
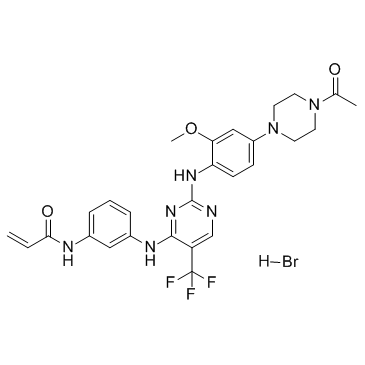
-
GC37568
RTC-5
RTC-5 (TRC-382) es una fenotiazina optimizada con potencia anticancerÍgena. RTC-5 demuestra eficacia contra un modelo de xenoinjerto de un cÁncer impulsado por EGFR, sus efectos se atribuyen a la regulaciÓn negativa concomitante de la seÑalizaciÓn de PI3K-AKT y RAS-ERK.

-
GC69843
Ruserontinib
Ruserontinib (SKLB1028) es un inhibidor de múltiples quinasas orales con actividad contra EGFR, FLT3 y Abl. Su IC50 en humanos FLT3 es de 55 nM y tiene actividad antitumoral.
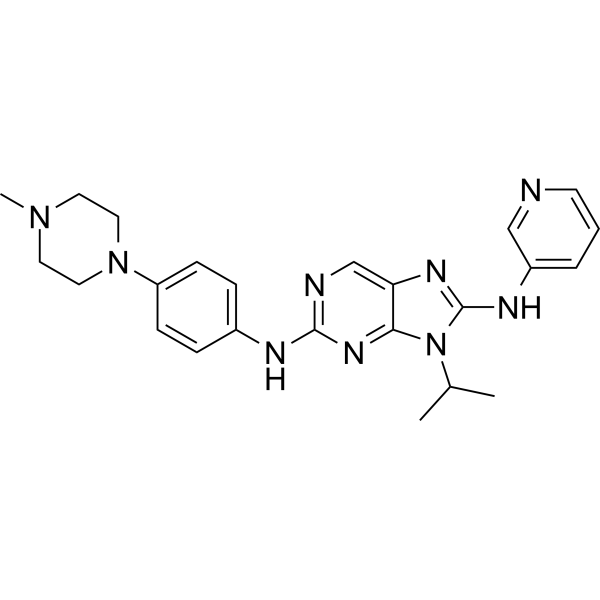
-
GC37575
Ruxolitinib sulfate
El sulfato de ruxolitinib (sulfato INCB018424) es el primer inhibidor potente y selectivo de JAK1/2 que ingresa a la clÍnica con IC50 de 3,3 nM/2,8 nM, y tiene una selectividad > 130 veces mayor para JAK1/2 que para JAK3.
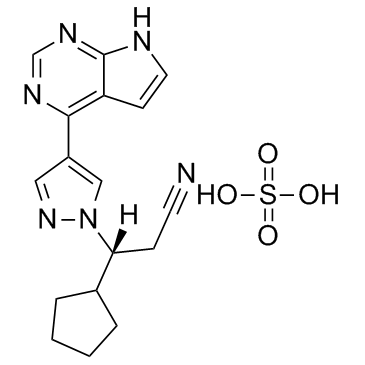
-
GN10164
Saikosaponin D
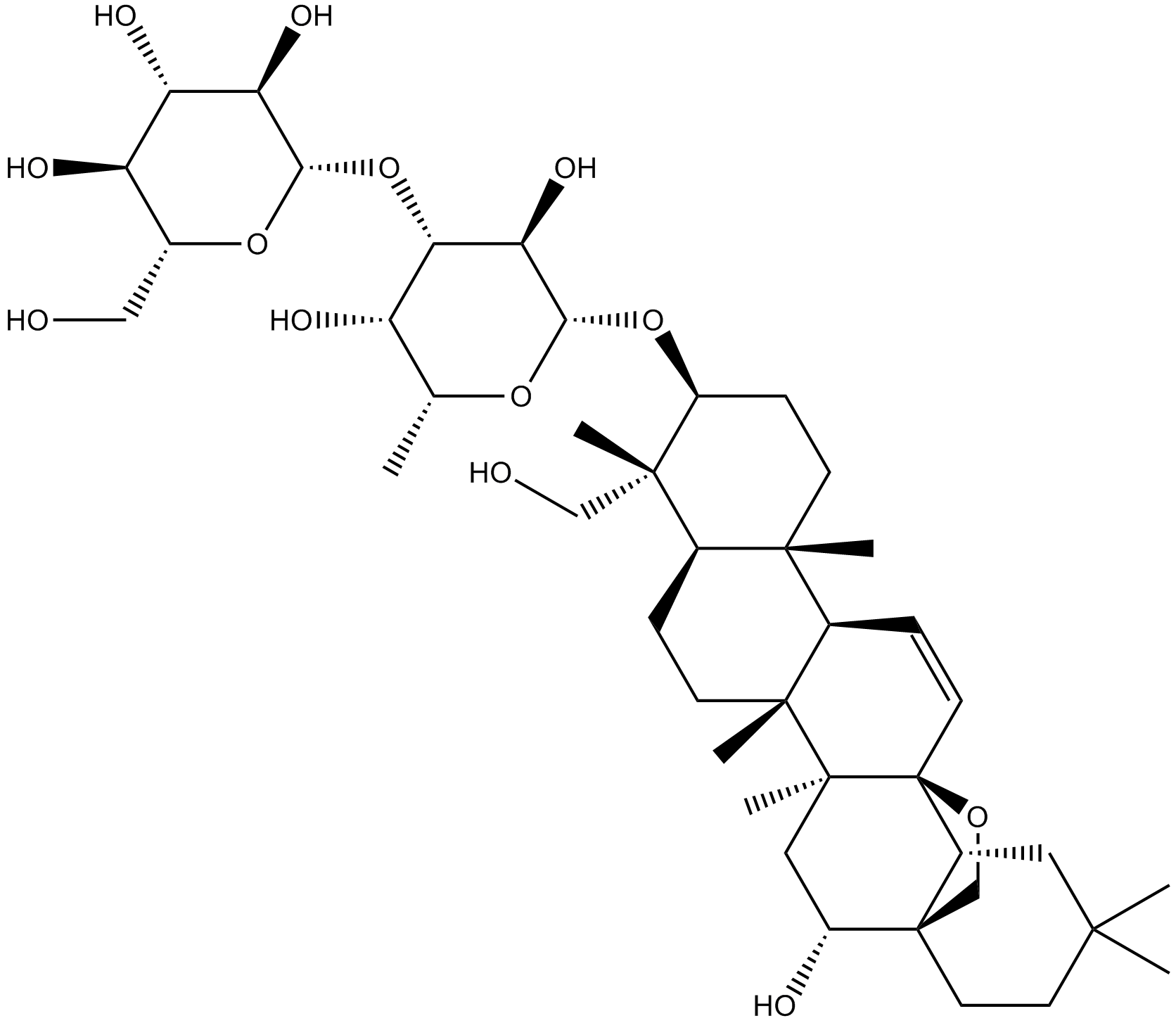
-
GC69854
Salviolone
Salviolone es un derivado natural de diterpeno que puede combatir las células del melanoma. Salviolone tiene una acción multifacética en el melanoma al bloquear el proceso del ciclo celular, la señalización STAT3 y el fenotipo maligno de las células del melanoma A375.
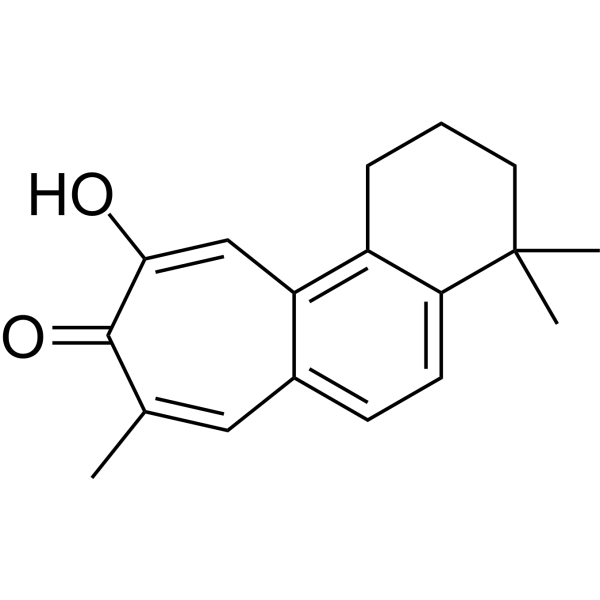
-
GC19320
SAR-20347
SAR-20347 es un inhibidor de TYK2, JAK1, JAK2 y JAK3 con IC50 de 0,6, 23, 26 y 41 nM, respectivamente.
-
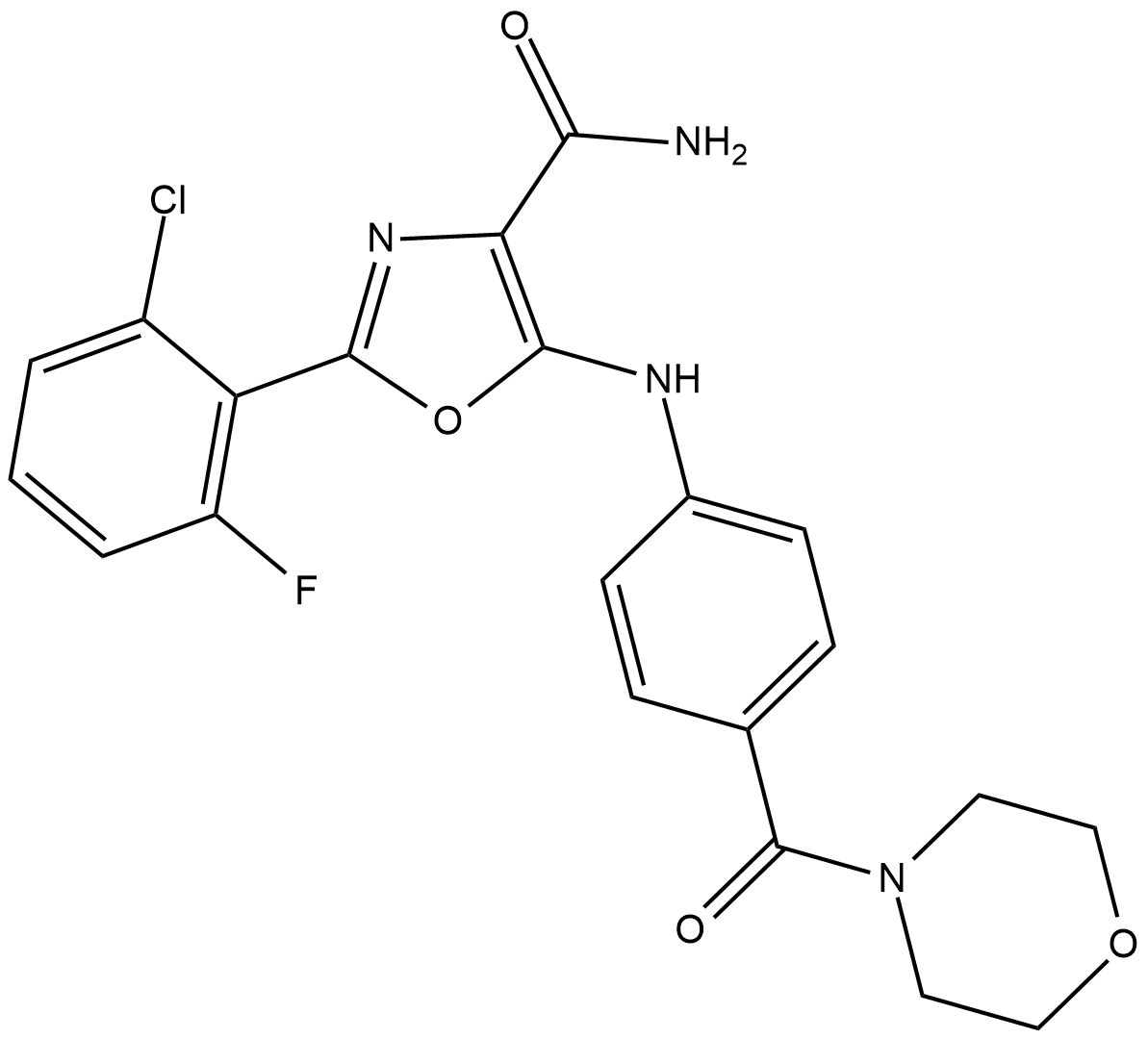
GC25899
SC-1
SC-1 (1-(4-Chloro-3-(trifluoromethyl)phenyl)-3-(4-(4-cyanophenoxy)phenyl)urea, STAT3-IN-7), a Sorafenib analogue and potently inhibits the phosphorylation of STAT3, induces cell apoptosis through SHP-1 dependent STAT3 inactivation.
-
GC61524
SC-43
SC-43, un derivado de sorafenib, es un agonista de SHP-1 (PTPN6) potente y activo por vÍa oral. SC-43 inhibe la fosforilaciÓn de STAT3 e induce la apoptosis celular. SC-43 tiene efectos antifibrÓticos y anticancerÍgenos.

-
GN10624
Scutellarin
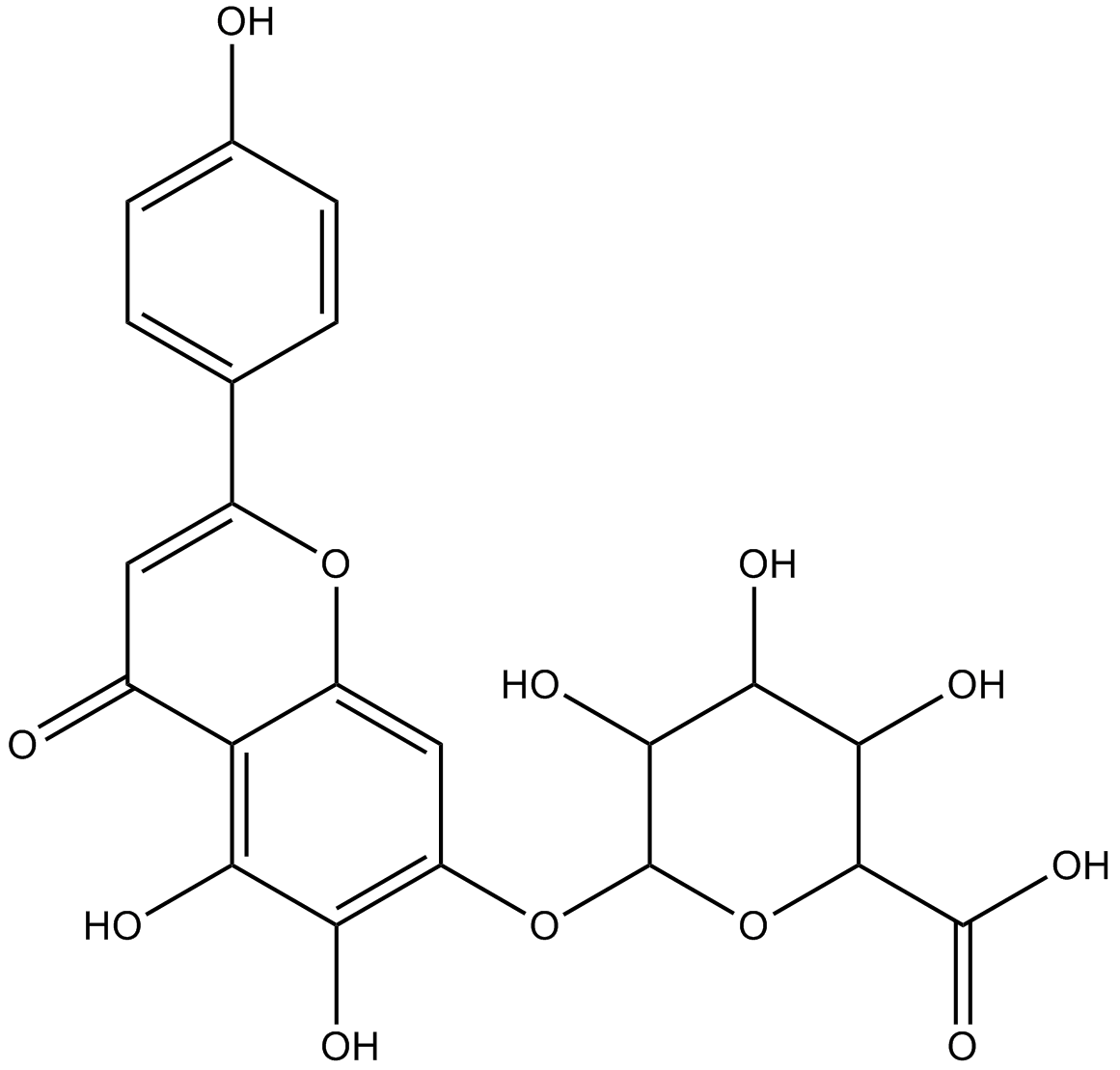
-
GC44880
SD 1029
SD 1029 es un inhibidor de JAK2/STAT3. SD 1029 inhibe la translocación nuclear STAT3. SD 1029 es un inhibidor de la activación de STAT3 debido a la inhibición de la fosforilación de JAK2.
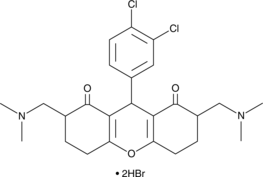
-
GC64774
SEL24-B489
SEL24-B489 es un potente inhibidor dual de PIM y FLT3-ITD de tipo I, activo por vÍa oral, con valores de Kd de 2 nM para PIM1, 2 nM para PIM2 y 3 nM para PIM3, respectivamente.
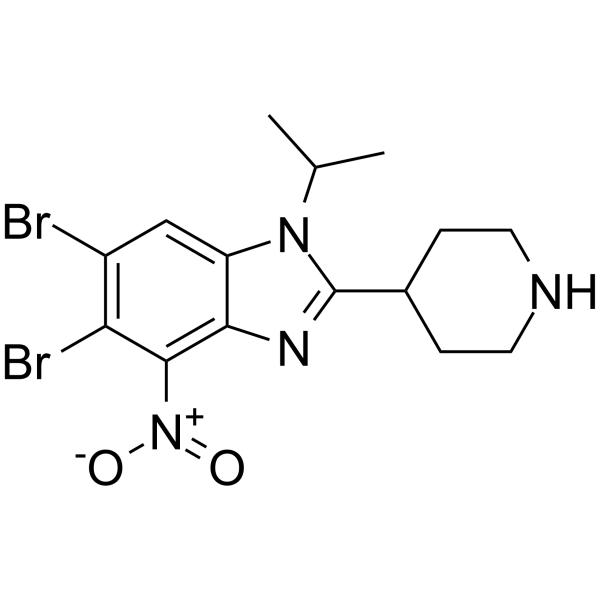
-
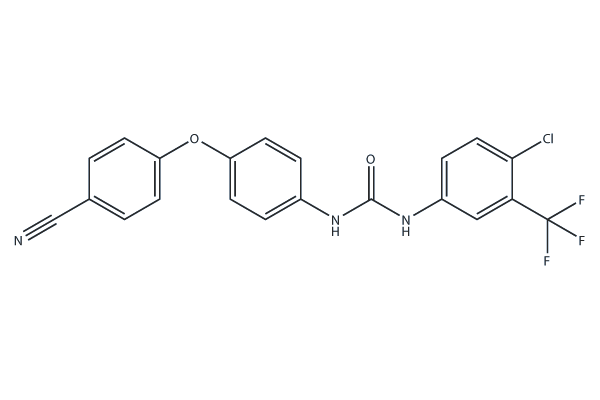
GC68430
Selatinib
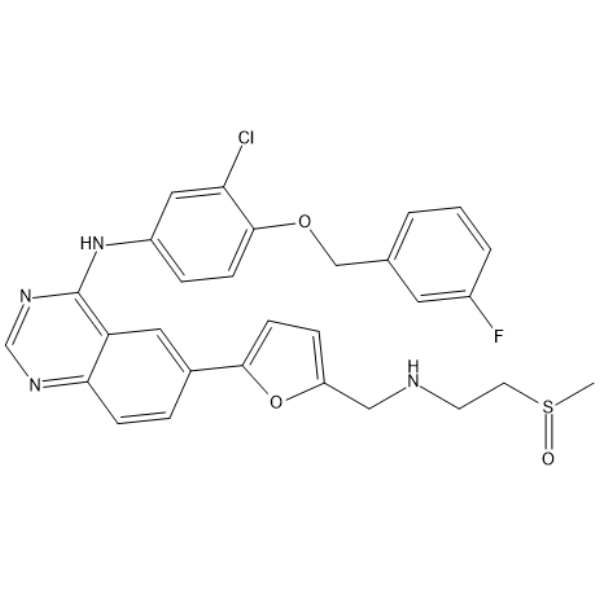
-
GC14658
SH-4-54
SH-4-54 es un inhibidor de STAT que se une a STAT3 y STAT5 con KD de 300, 464 nM, respectivamente.
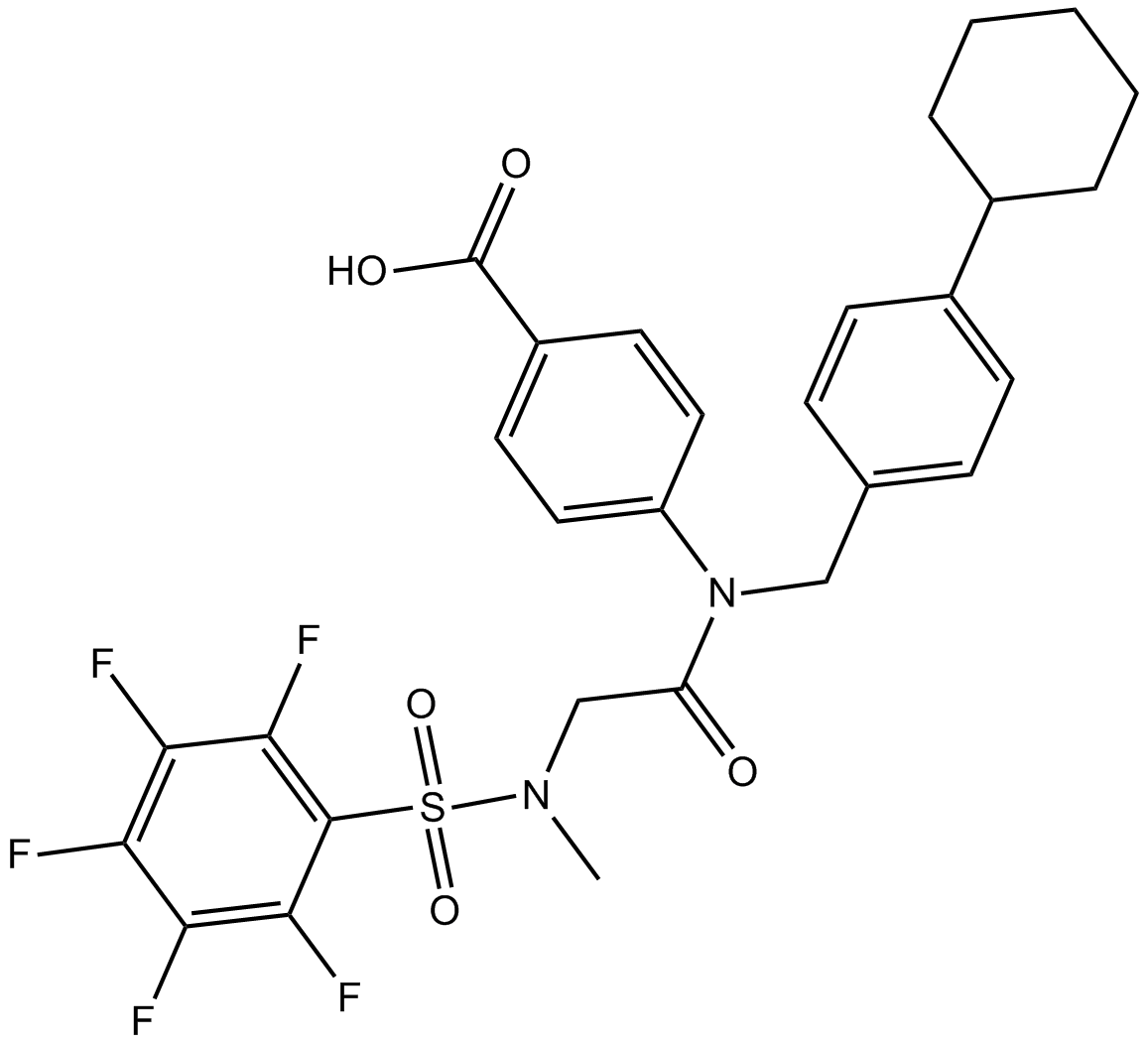
-
GC32928
SH5-07
SH5-07 es un inhibidor de Stat3 basado en Ácido hidroxÁmico con una IC50 de 3,9 μM en ensayo in vitro.
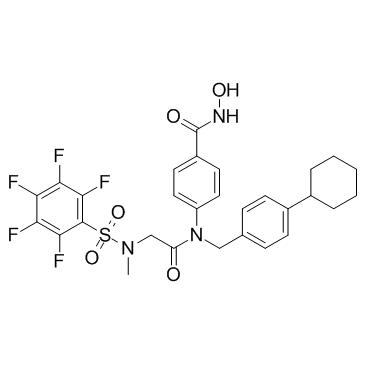
-
GC37651
SMI-16a
SMI-16a es un inhibidor selectivo de la quinasa Pim con valores IC50 de 0,15, 0,02 y 48 μM para células Pim1, Pim2 y PC3, respectivamente.

-
GC19334
Solcitinib
Solcitinib es un inhibidor de JAK1 selectivo, potente, competitivo y activo por vÍa oral, con una IC50 de 9,8 nM y una selectividad de 11, 55 y 23 veces sobre JAK2, JAK3 y TYK2, respectivamente; Solcitinib se utiliza en la investigaciÓn de la psoriasis en placas de moderada a grave.
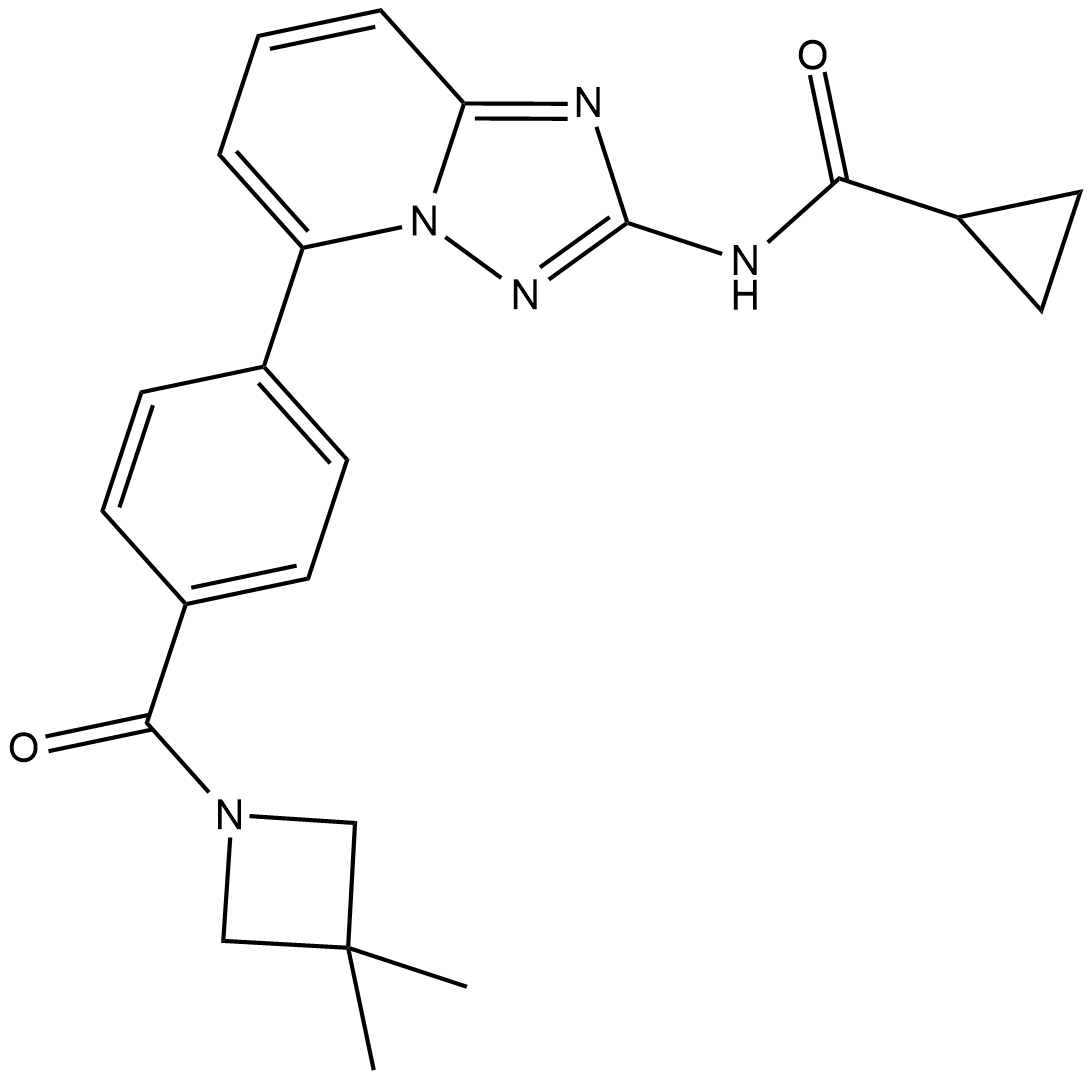
-
GC17761
STA-21
A STAT3 inhibitor
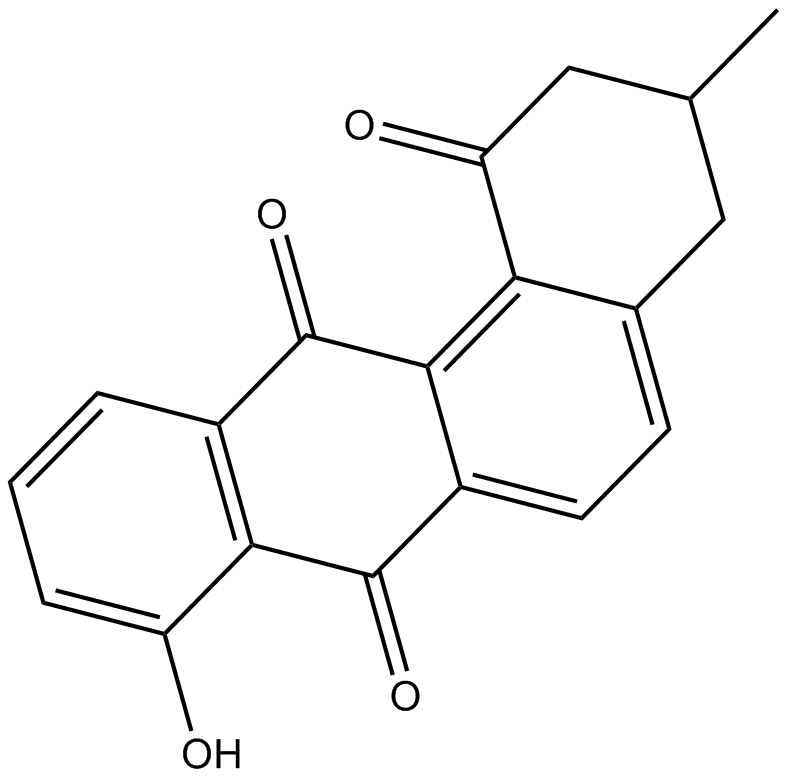
-
GC62559
Stafia-1
Stafia-1 es un potente inhibidor de STAT5a (K i = 10,9 μM, IC50 = 22,2 μM). Stafia-1 muestra una alta selectividad sobre STAT5b y otros miembros de la familia STAT.
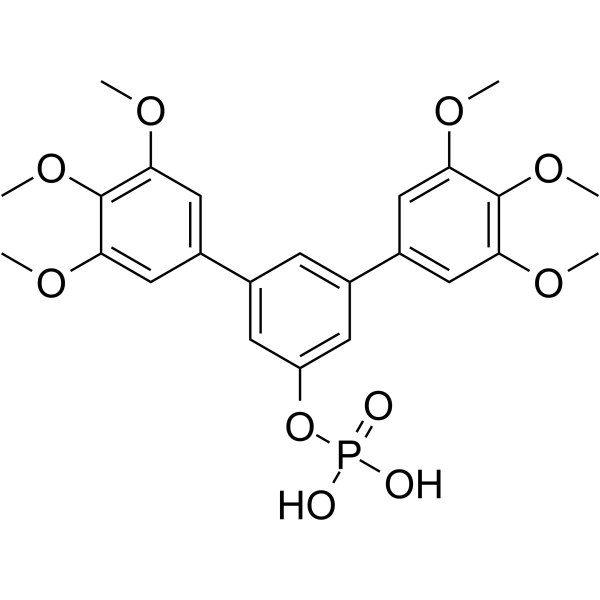
-
GC63203
Stafia-1-dipivaloyloxymethyl ester
El éster de estafia-1-dipivaloiloximetilo (compuesto 27, 0-200 μM) reduce significativamente la expresiÓn de pSTAT5a y no tiene una inhibiciÓn obvia sobre pSTAT5b.
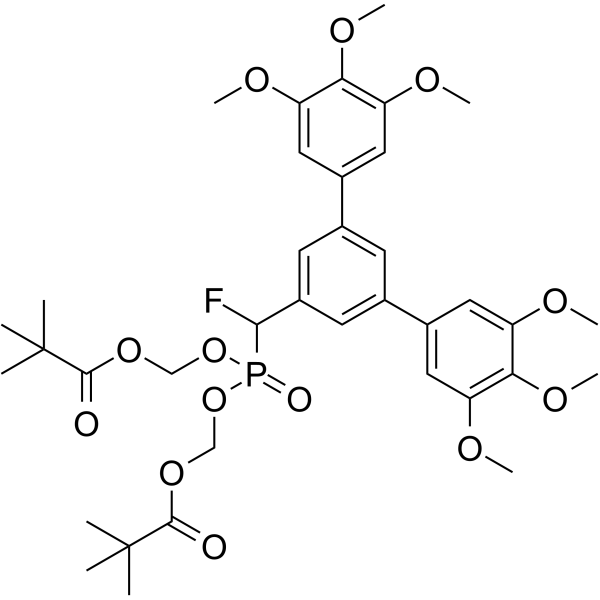
-
GC62254
Stafib-1
Stafib-1 es el primer inhibidor selectivo del dominio STAT5b SH2, con una Ki de 44 nM y una IC50 de 154 nM.
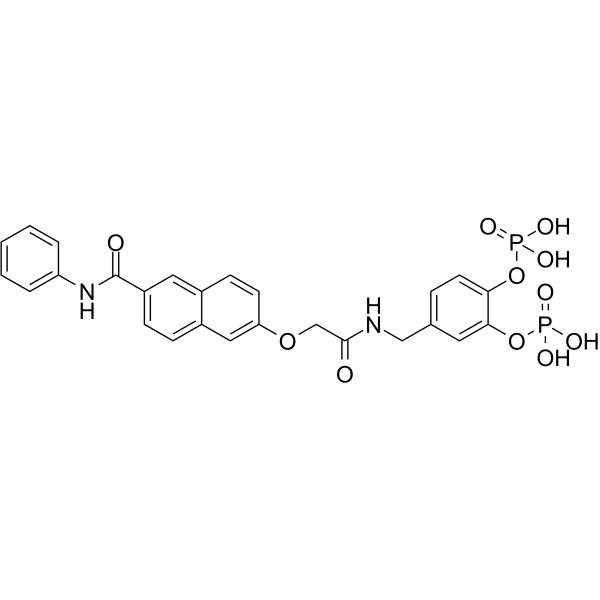
-
GC63204
Stafib-2
Stafib-2 es un inhibidor potente y selectivo del factor de transcripciÓn STAT5b, con una IC50 de 82 nM y 1,7 μM para STAT5b y STAT5a, respectivamente.
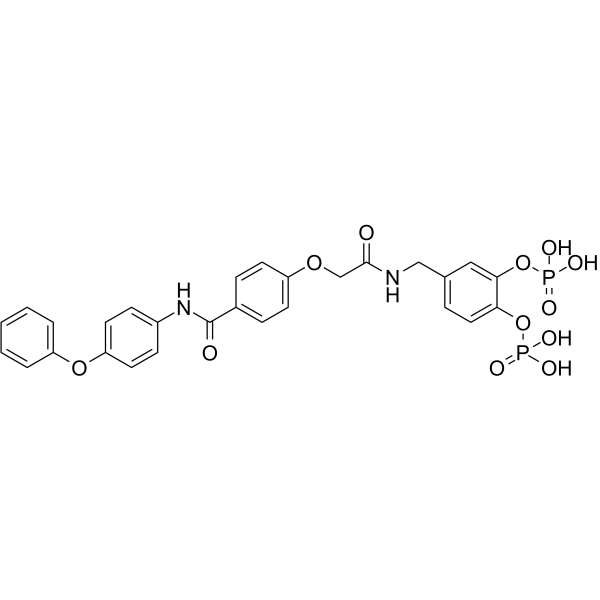
-
GC52293
STAT3 Inhibitor 4m
A STAT3 inhibitor
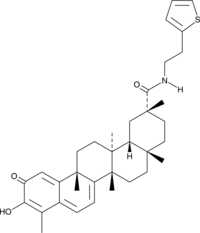
-
GC37688
STAT3-IN-1
STAT3-IN-1 (compuesto 7d) es un inhibidor de STAT3 excelente, selectivo y activo por vÍa oral, con valores de IC50 de 1,82 μM y 2,14 μM en células HT29 y MDA-MB 231, respectivamente. STAT3-IN-1 (compuesto 7d) induce la apoptosis tumoral.
-
GC69952
STAT3-IN-11
STAT3-IN-11 (7a) es un inhibidor selectivo de STAT3 que puede inhibir la fosforilación del sitio Tyr705 de STAT3. STAT3-IN-11 puede inhibir la fosforilación de genes aguas abajo (Survivin y Mcl-1), sin afectar la expresión de quinasas tirosina upstream (Src y JAK2) y p-STAT1. STAT3-IN-11 puede inducir apoptosis en células cancerosas, lo que podría ser útil para el descubrimiento de inhibidores de STAT3 y agentes antitumorales.
-
GC69953
STAT3-IN-12
STAT3-IN-12 is an effective STAT3 signal inhibitor that can suppress the activation of IL-6-induced JAK/STAT3 signaling pathway. STAT3-IN-12 can inhibit cancer cell growth, migration, and induce apoptosis and cycle arrest. STAT3-IN-12 can be used in cancer-related research such as hepatocellular carcinoma (HCC) and esophageal cancer.
-
GC37689
STAT3-IN-3
STAT3-IN-3 es un potente y selectivo inhibidor del transductor de seÑales y activador de la transcripciÓn 3 (STAT3), con actividad antiproliferativa. STAT3-IN-3 induce la apoptosis en células de cÁncer de mama. STAT3-IN-3 actÚa como un prometedor inhibidor de STAT3 dirigido a las mitocondrias para la investigaciÓn del cÁncer.
-
GC13647
STAT5 Inhibitor
El inhibidor de STAT5 es un inhibidor de STAT5 con un IC50 de 47 μ M para STAT5β isoforma.
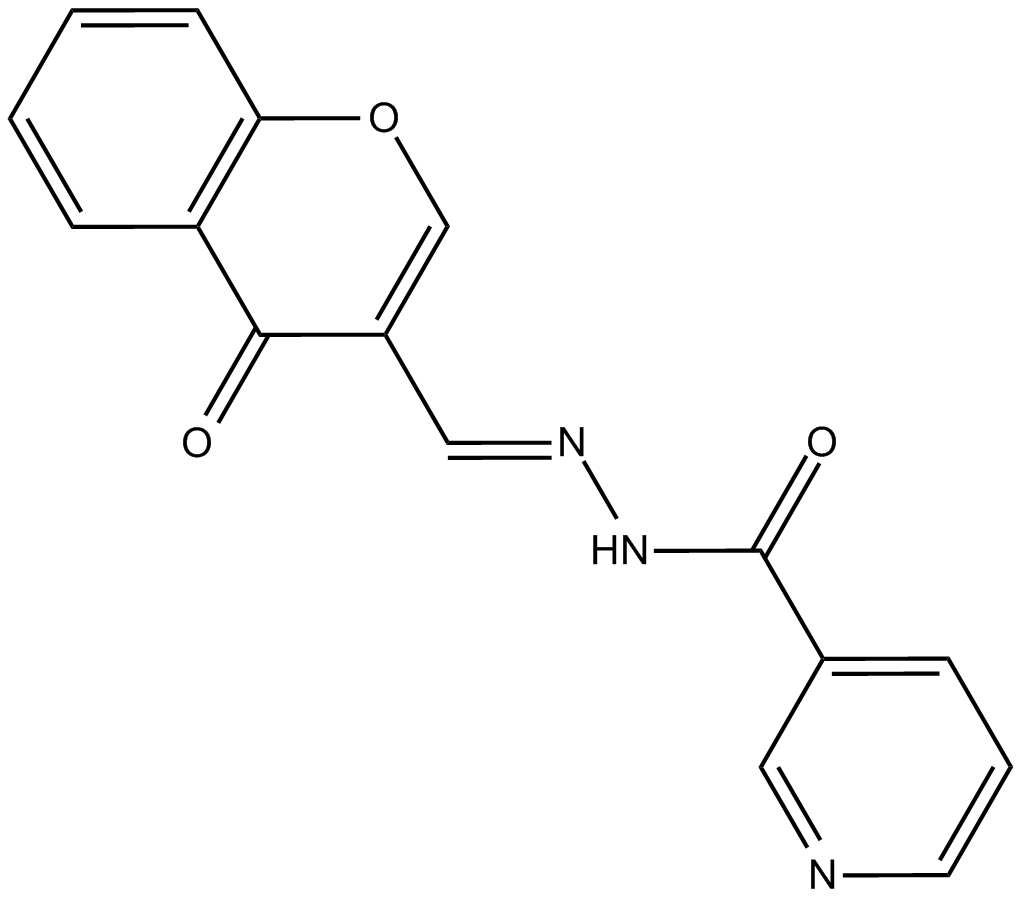
-
GC65344
STAT5-IN-2
STAT5-IN-2 es un inhibidor de STAT5, extraÍdo de la referencia 1, ejemplo 17f. STAT5-IN-2 tiene un potente efecto antileucémico.
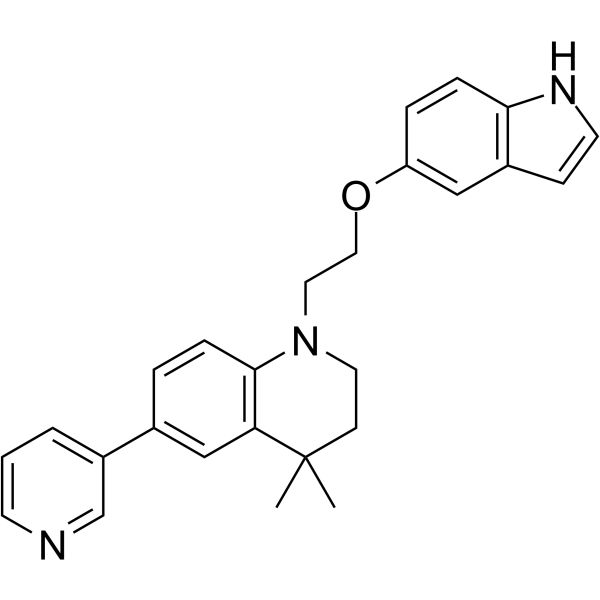
-
GC17886
Stattic
Stattic es el primer inhibidor no peptídico de molécula pequeña de STAT3, que inhibe efectivamente la activación de STAT3 y su translocación nuclear.
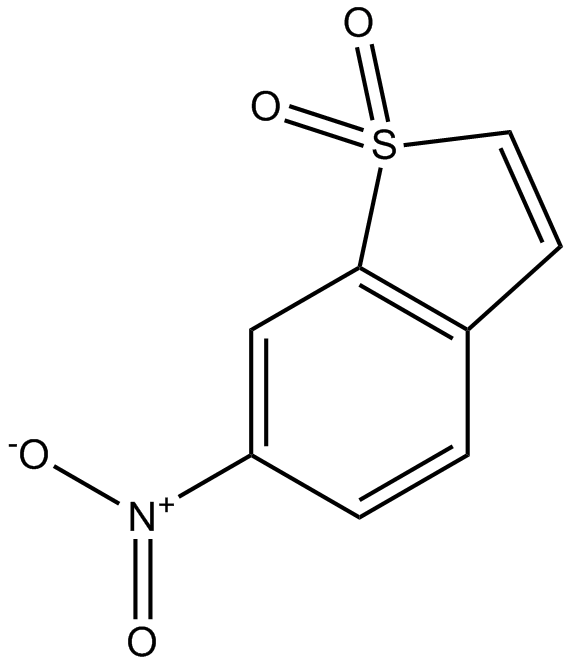
-
GC67779
STX-0119
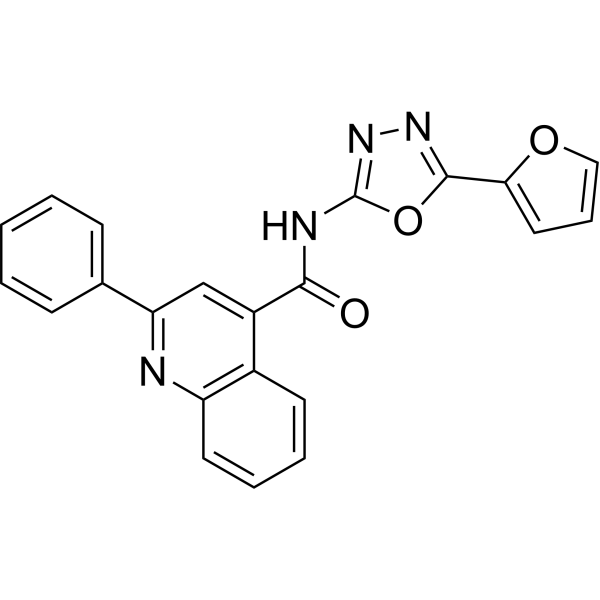
-
GC11172
TAK-285
TAK-285 es un inhibidor de HER2 y EGFR (HER1) potente, selectivo, competitivo con ATP y activo por vÍa oral con IC50 de 17 nM y 23 nM, respectivamente. TAK-285 tiene una selectividad >10 veces mayor para HER1/2 que para HER4, y menos potente para MEK1/5, c-Met, Aurora B, Lck, CSK, etc. TAK-285 tiene actividad antitumoral eficaz. TAK-285 puede cruzar la barrera hematoencefÁlica (BBB).
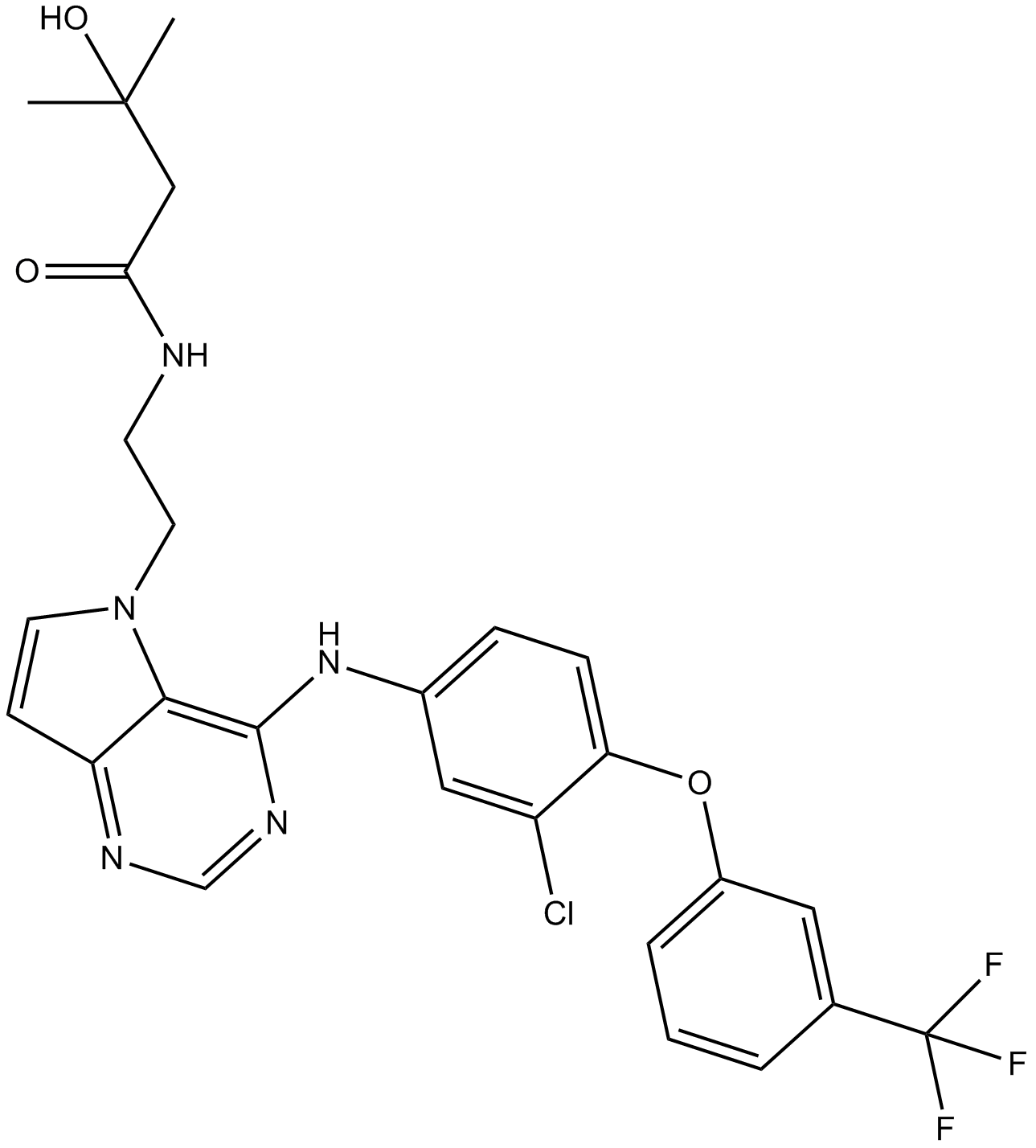
-
GC32100
Tarloxotinib bromide (TH-4000)
El bromuro de tarloxotinib (TH-4000) (TH-4000) es un inhibidor irreversible de EGFR/HER2.
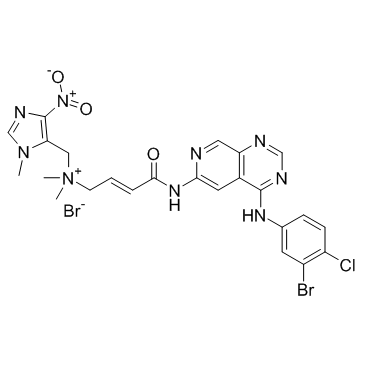
-
GC65310
TAS0728
TAS0728 es un inhibidor de HER2 potente, selectivo, activo por vÍa oral, irreversible y de uniÓn covalente, con una IC50 de 13 nM. TAS0728 también muestra IC50 de 4.9, 8.5, 31, 65, 33, 25 y 86 nM para BMX, HER4, BLK, EGFR, JAK3, SLK y LOK respectivamente. AdemÁs, TAS0728 exhibe una inhibiciÓn sÓlida y sostenida de la fosforilaciÓn de HER2
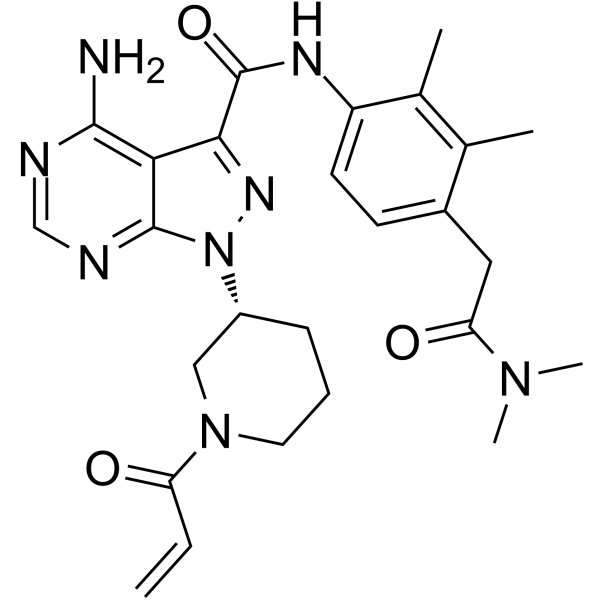
-
GC32752
TAS6417
TAS6417 (CLN-081) es un inhibidor de la tirosina cinasa del EGFR altamente eficaz, activo por vÍa oral y pan-mutaciÓn selectivo con un andamio Único que se ajusta al sitio de uniÓn al ATP de la regiÓn bisagra del EGFR, con valores de CI50 que oscilan entre 1,1 y 8,0 nM.
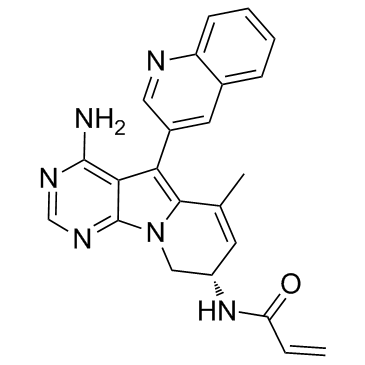
-
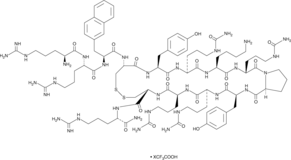
GC45004
TC 14012 (trifluoroacetate salt)
C-X-C chemokine receptor type 4 (CXCR4) is a receptor for the stromal cell-derived factor-1 which is designated as chemokine ligand 12 (CXCL12).
-
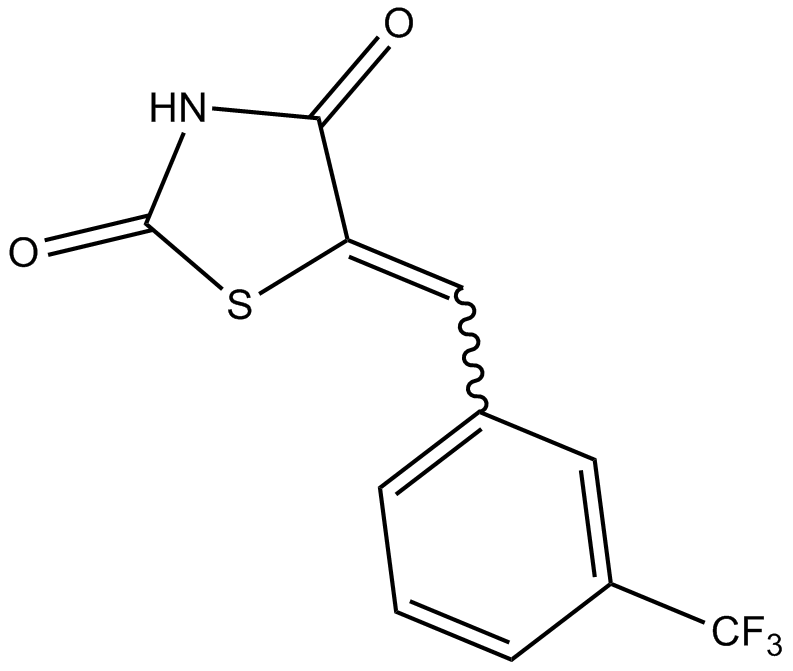
GC11990
TCS-PIM-1-4a
TCS-PIM-1-4a (SMI-4a) es un inhibidor de las cinasas pan-Pim que bloquea la actividad de mTORC1 mediante la activaciÓn de AMPK. TCS-PIM-1-4a mata una amplia gama de lÍneas celulares mieloides y linfoides (valores IC50 que van desde 0,8 μM a 40 μM).
-
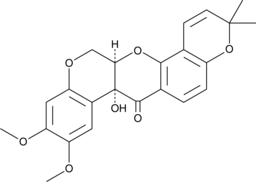
GC41577
Tephrosin (synthetic)
La tefrosina (sintética) es un rotenoide natural que tiene potentes actividades antitumorales. La tefrosina (sintética) induce la degradaciÓn de EGFR y ErbB2 al inducir la internalizaciÓn de los receptores.
-
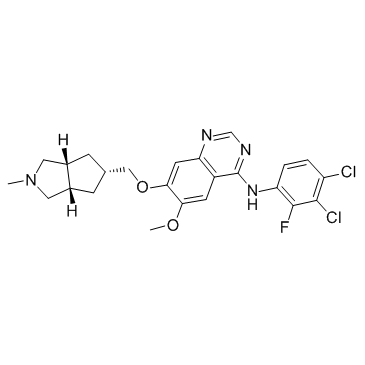
GC31752
Tesevatinib (XL-647)
Tesevatinib (XL-647) (XL-647; EXEL-7647; KD-019) es un inhibidor de la tirosina cinasa multidiana disponible por vÍa oral; inhibe la cinasa EGFR, ErbB2, KDR, Flt4 y EphB4 con IC50 de 0,3, 16, 1,5, 8,7 y 1,4 nM.
-
GC39022
Tetramethylcurcumin
La tetrametilcurcumina (FLLL31), derivada de la curcumina, suprime especÍficamente la fosforilaciÓn de STAT3 al unirse selectivamente a Janus quinasa 2 y el dominio de homologÍa-2 de STAT3 Src.
-
GC25992
TG-89
TG-89 is an inhibitor of JAK2 with IC50 of 11.2 μM.