Chromatin/Epigenetics
Epigenetics
Epigenetics means above genetics. It determines how much and whether a gene is expressed without changing DNA sequences. Epigenetic regulations include, 1. DNA methylation: the addition of methyl group to DNA, converting cytosine to 5-methylcytosine, mostly at CpG sites; 2. Histone modifications: posttranslational modificationEpigeneticss of histone proteins including acetylation, methylation, ubiquitylation, phosphorylation and sumoylation; 3. miRNAs: non-coding microRNA downregulating gene expression; 4. Prions: infectious proteins viewed as epigenetic agents capable of inducing a phenotype without changing the genome.
Ziele für Chromatin/Epigenetics
- Bromodomain(52)
- Aurora Kinase(89)
- DNA Methyltransferase(40)
- HDAC(218)
- Histone Acetyltransferases(67)
- Histone Demethylases(98)
- Histone Methyltransferase(212)
- HIF(101)
- JAK(178)
- MBT Domains(1)
- PARP(128)
- Pim(35)
- Protein Ser/Thr Phosphatases(41)
- RNA Polymerase(8)
- Sirtuin(84)
- Sphingosine Kinase-2(1)
- Polycomb repressive complex(2)
- SUMOylation(3)
- PAD(18)
- Epigenetic Reader Domain(207)
- MicroRNA(13)
- Protein Arginine Deiminase(12)
- Chromodomain(1)
- Citrullination(15)
- DNA/RNA Demethylation(1)
- DNA/RNA Methylation(6)
- Histone Deacetylation(38)
- Histones/Histone Peptides(7)
- PHD Domains(0)
- Tandem Tudor & Tudor-like Domains(1)
- PRMT(2)
Produkte für Chromatin/Epigenetics
- Bestell-Nr. Artikelname Informationen
-
GC33028
CF53
CF53 ist ein hochpotenter, selektiver und oral aktiver Inhibitor des BET-Proteins mit einem Ki von <1 nM, Kd von 2,2 nM und einem IC50 von 2 nM fÜr BRD4 BD1. CF53 bindet sowohl an die BD1- als auch an die BD2-DomÄne von BRD2-, BRD3-, BRD4- und BRDT-BET-Proteinen mit hoher AffinitÄt, sehr selektiv gegenÜber Nicht-BET-BromdomÄnen-enthaltenden Proteinen. CF53 zeigt sowohl in vitro als auch in vivo eine starke AntitumoraktivitÄt.
-
GC65602
CFT8634
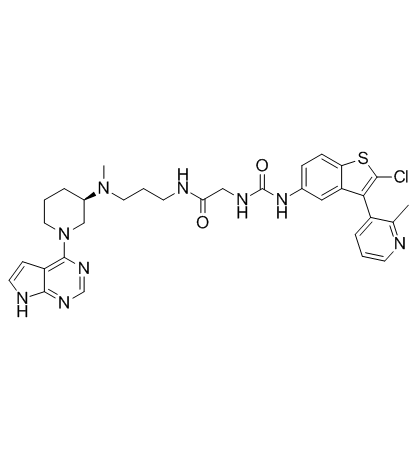
CFT8634 ist ein Abbauer, der auf BRD9 abzielt, das aus dem Patent WO2021178920A1, Verbindung 174, extrahiert wurde. CFT8634 kann fÜr die Erforschung von Synovialsarkom und SMARCB1-deletierten soliden Tumoren verwendet werden.
-
GC35668
CG-200745
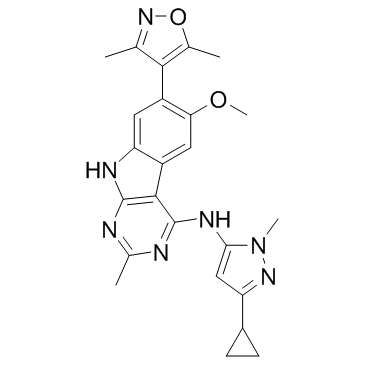
CG-200745 (CG-200745) ist ein oral aktiver, potenter pan-HDAC-Inhibitor, der die HydroxamsÄureeinheit hat, um Zink am Boden der katalytischen Tasche zu binden. CG-200745 hemmt die Deacetylierung von Histon H3 und Tubulin. CG-200745 induziert die Akkumulation von p53, fÖrdert die p53-abhÄngige Transaktivierung und verstÄrkt die Expression der Proteine MDM2 und p21 (Waf1/Cip1). CG-200745 erhÖht die Empfindlichkeit von Gemcitabin-resistenten Zellen gegenÜber Gemcitabin und 5-Fluorouracil (5-FU; ). CG-200745 induziert Apoptose und hat Antitumorwirkungen.
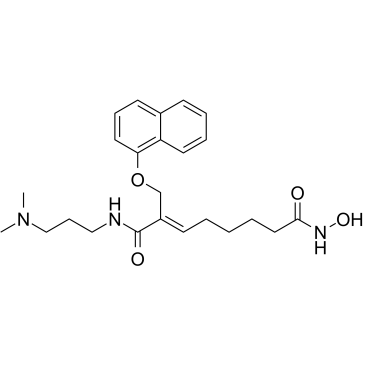
-
GC39679
CG347B
CG347B ist ein selektiver HDAC6-Inhibitor, der auch an der Synthese anderer Metalloenzym-Inhibitoren beteiligt ist.
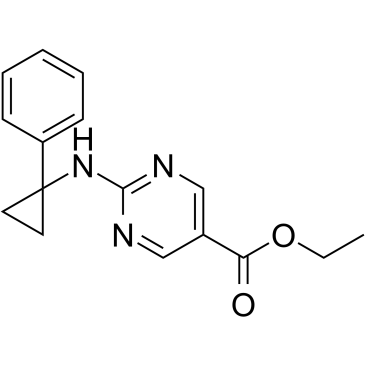
-
GC14936
Chaetocin
Inhibitor of lys9-specific HMTs
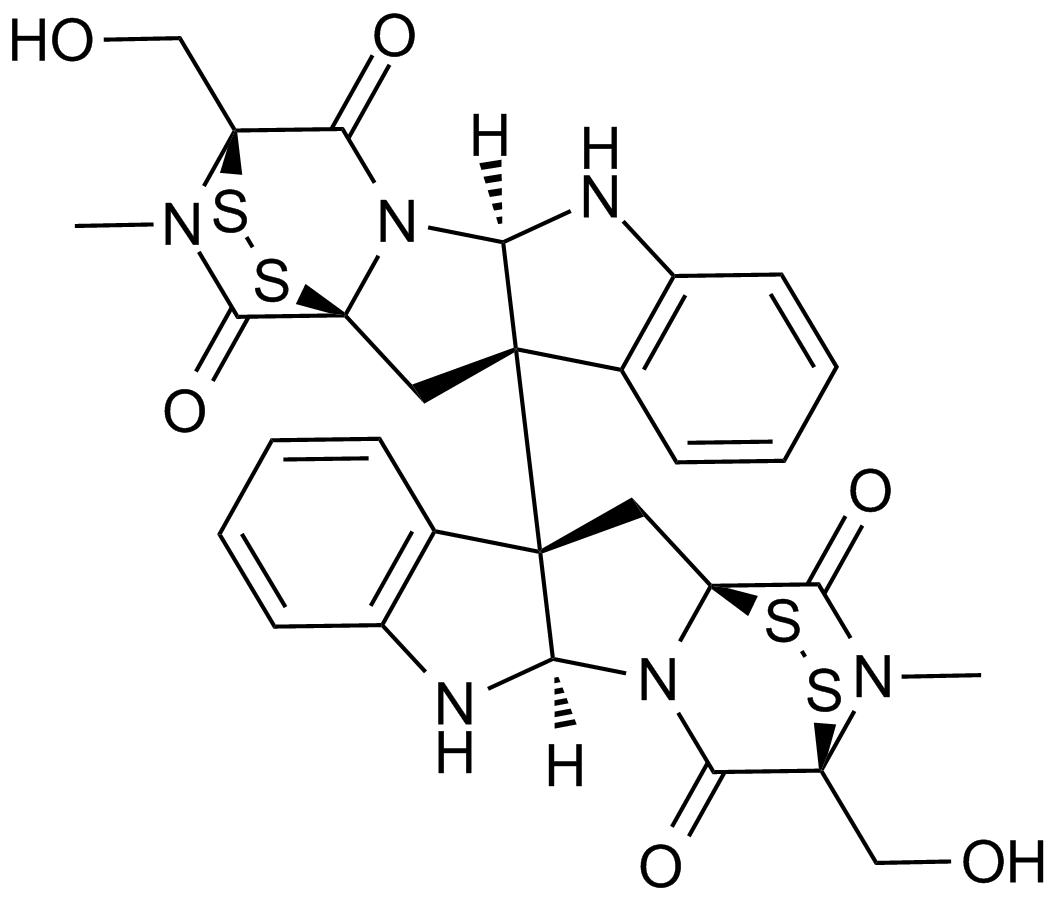
-
GC11975
CHAPS
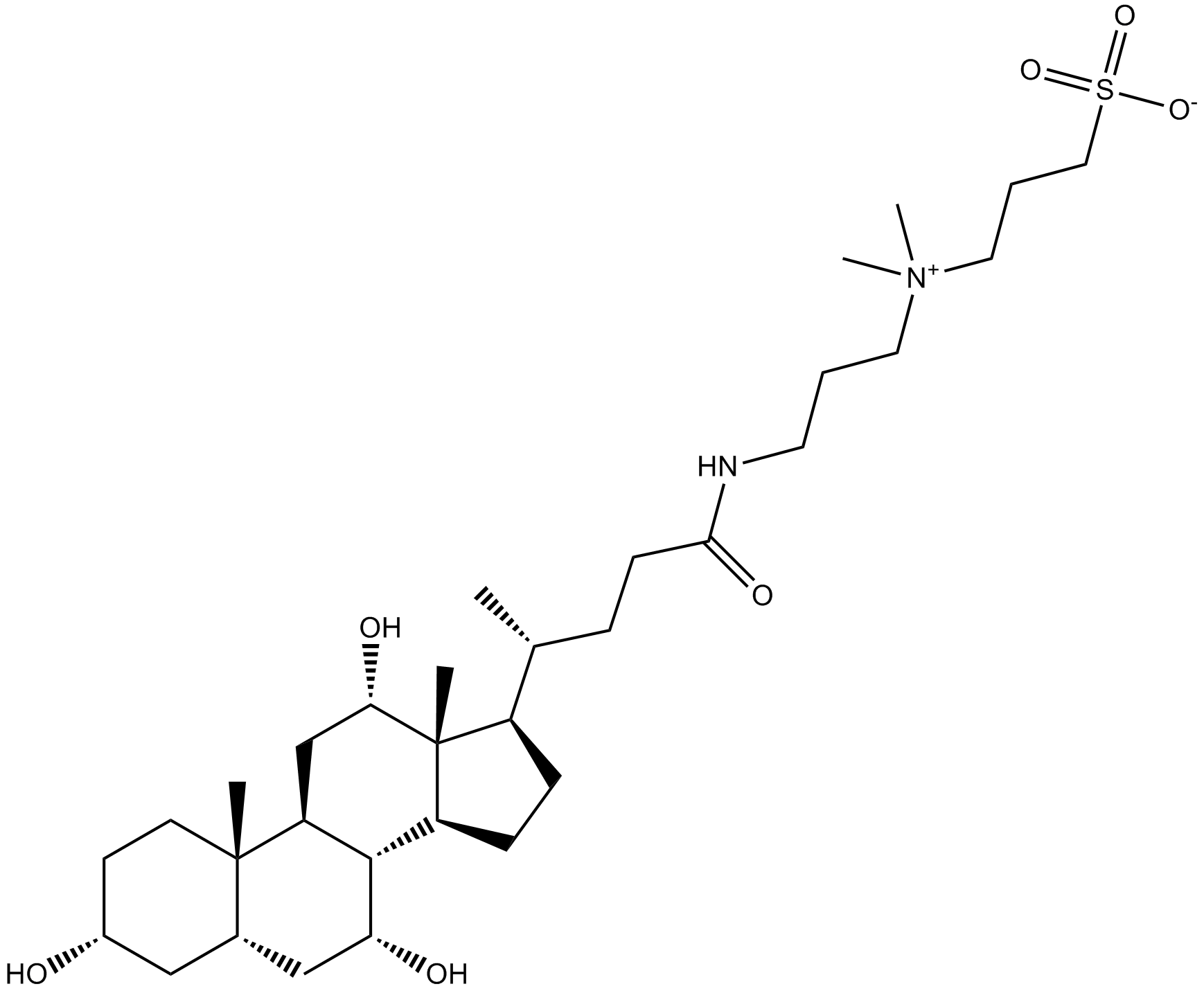
-
GC64942
CHDI-390576
CHDI-390576, ein potenter, zelldurchlÄssiger und ZNS-penetranter Histon-Deacetylase (HDAC)-Hemmer der Klasse IIa mit IC50-Werten von 54 nM, 60 nM, 31 nM, 50 nM fÜr HDAC4, HDAC5, HDAC7, HDAC9 der Klasse IIa, zeigt >500 -fache SelektivitÄt gegenÜber Klasse-I-HDACs (1, 2, 3) und ~150-fache SelektivitÄt gegenÜber HDAC8 und der Klasse-IIb-HDAC6-Isoform.
-
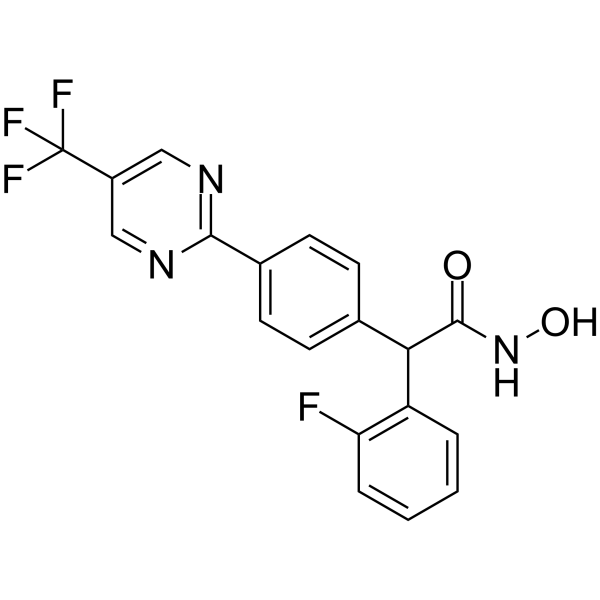
GC17405
Chetomin
An inhibitor of HIF signaling
-
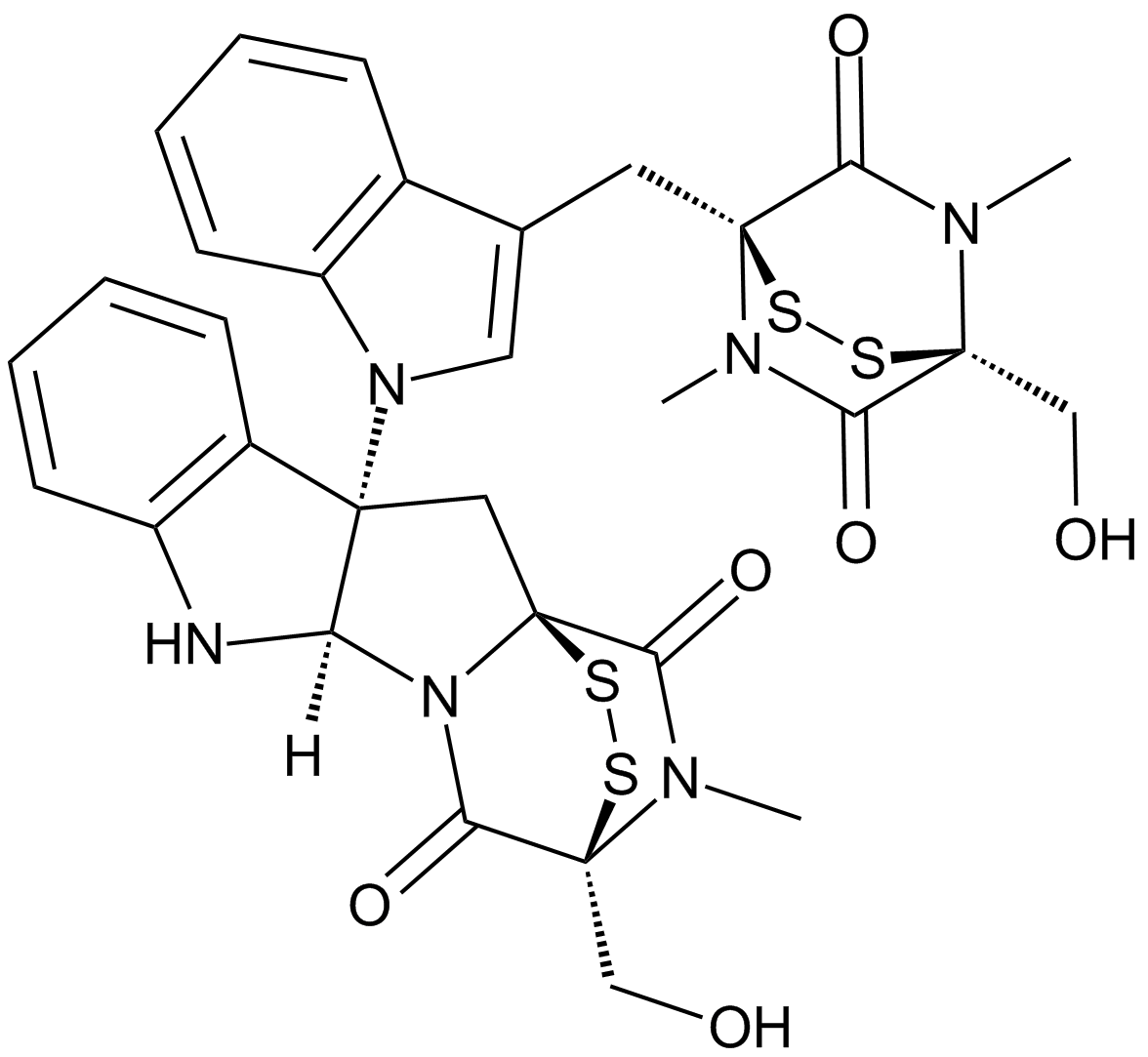
GC62145
Chiauranib
Chiauranib (CS2164) ist ein oral aktiver Multi-Target-Inhibitor gegen Tumorangiogenese. Chiauranib hemmt wirksam die Angiogenese-bezogenen Kinasen (VEGFR1, VEGFR2, VEGFR3, PDGFRα und c-Kit), die mitosebezogene Kinase Aurora B und die chronisch entzÜndungsbezogene Kinase CSF-1R mit IC50-Werten im Bereich von 1-9 nM. Chiauranib hat stark krebshemmende Wirkungen.

-
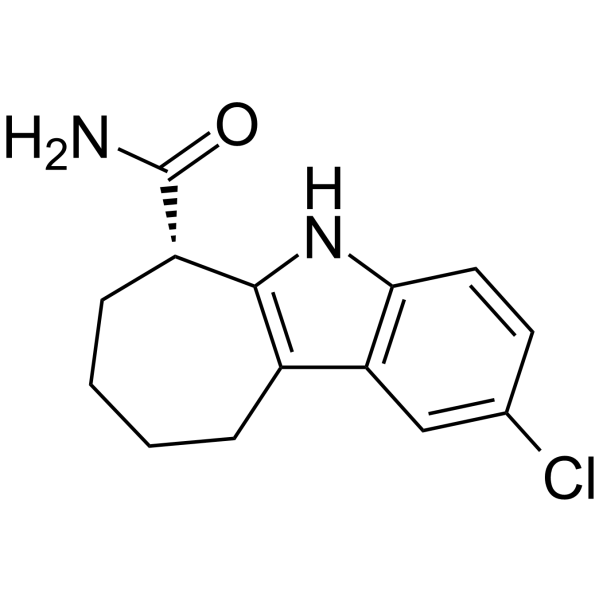
GC64255
CHIC35
CHIC35, ein Analogon von EX-527, ist ein potenter und selektiver Inhibitor von SIRT1 (IC50=0,124 μM).
-
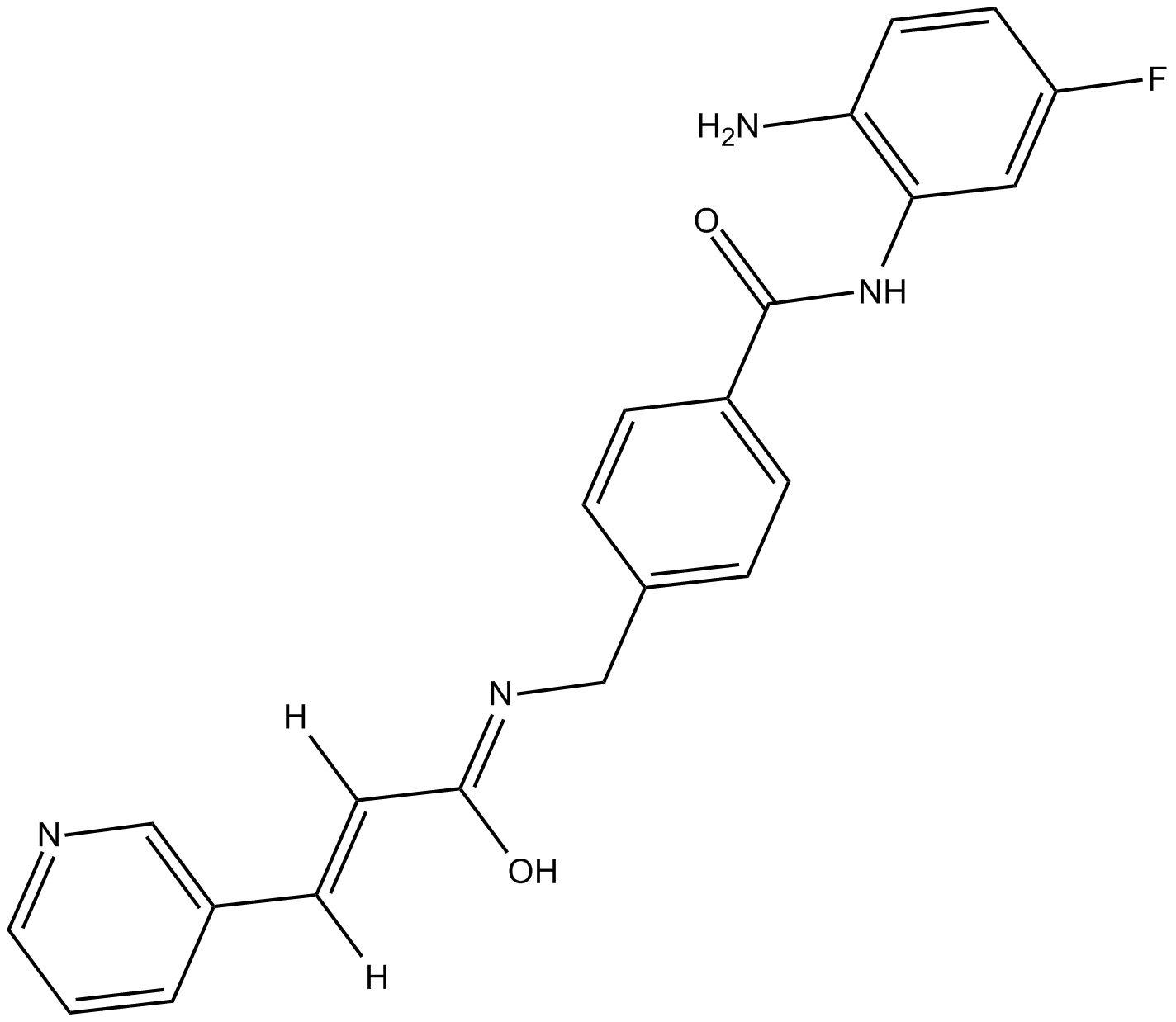
GC16042
Chidamide
Chidamid (Chidamid-Verunreinigung) ist eine Verunreinigung von Chidamid. Chidamid ist ein potenter und oral bioverfÜgbarer Hemmer der HDAC-Enzyme Klasse I (HDAC1/2/3) und Klasse IIb (HDAC10).
-
GN10518
Chitosamine hydrochloride
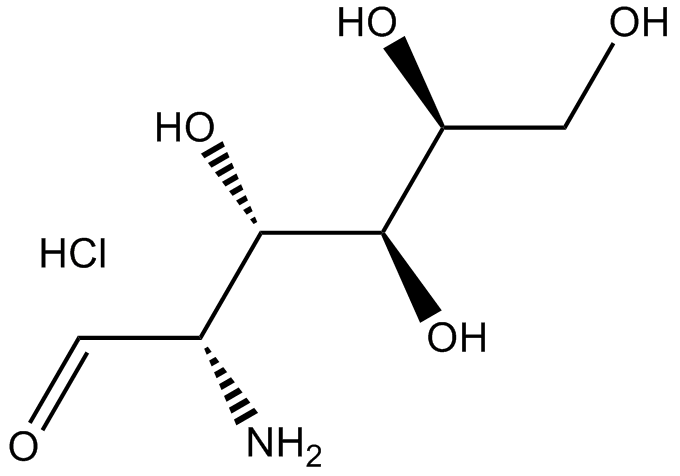
-
GC45717
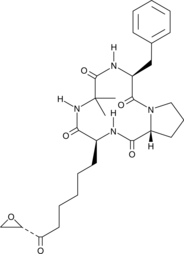
Chlamydocin
Chlamydocin, ein Pilzmetabolit, ist ein hochpotenter HDAC-Inhibitor mit einem IC50 von 1,3 nM. Chlamydocin zeigt starke antiproliferative und AntikrebsaktivitÄten. Chlamydocin induziert Apoptose durch Aktivierung von Caspase-3.
-
GN10308
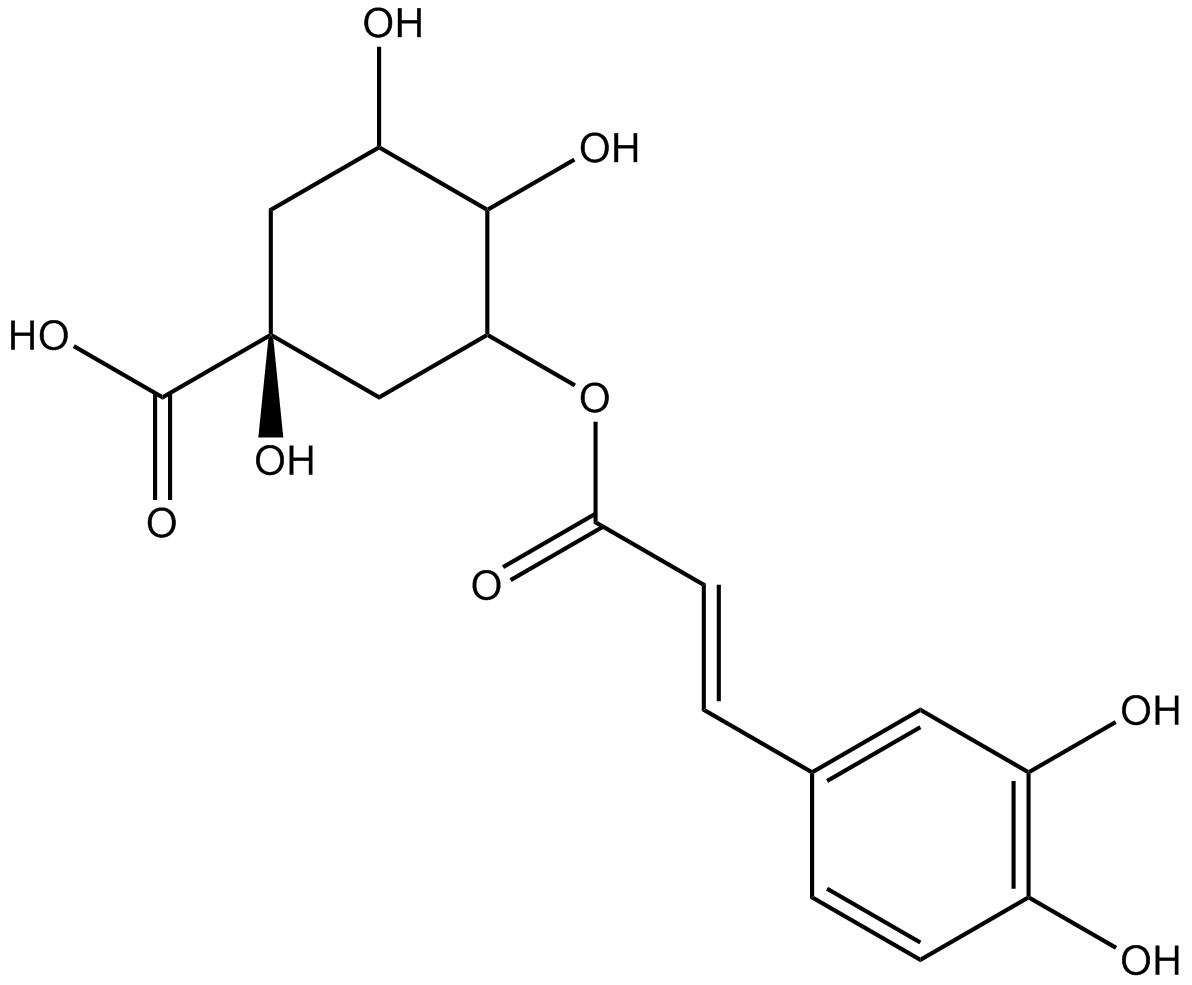
Chlorogenic acid
Chlorogensäure ist eine wichtige phenolische Verbindung in Kaffee und Tee.
-
GC11652
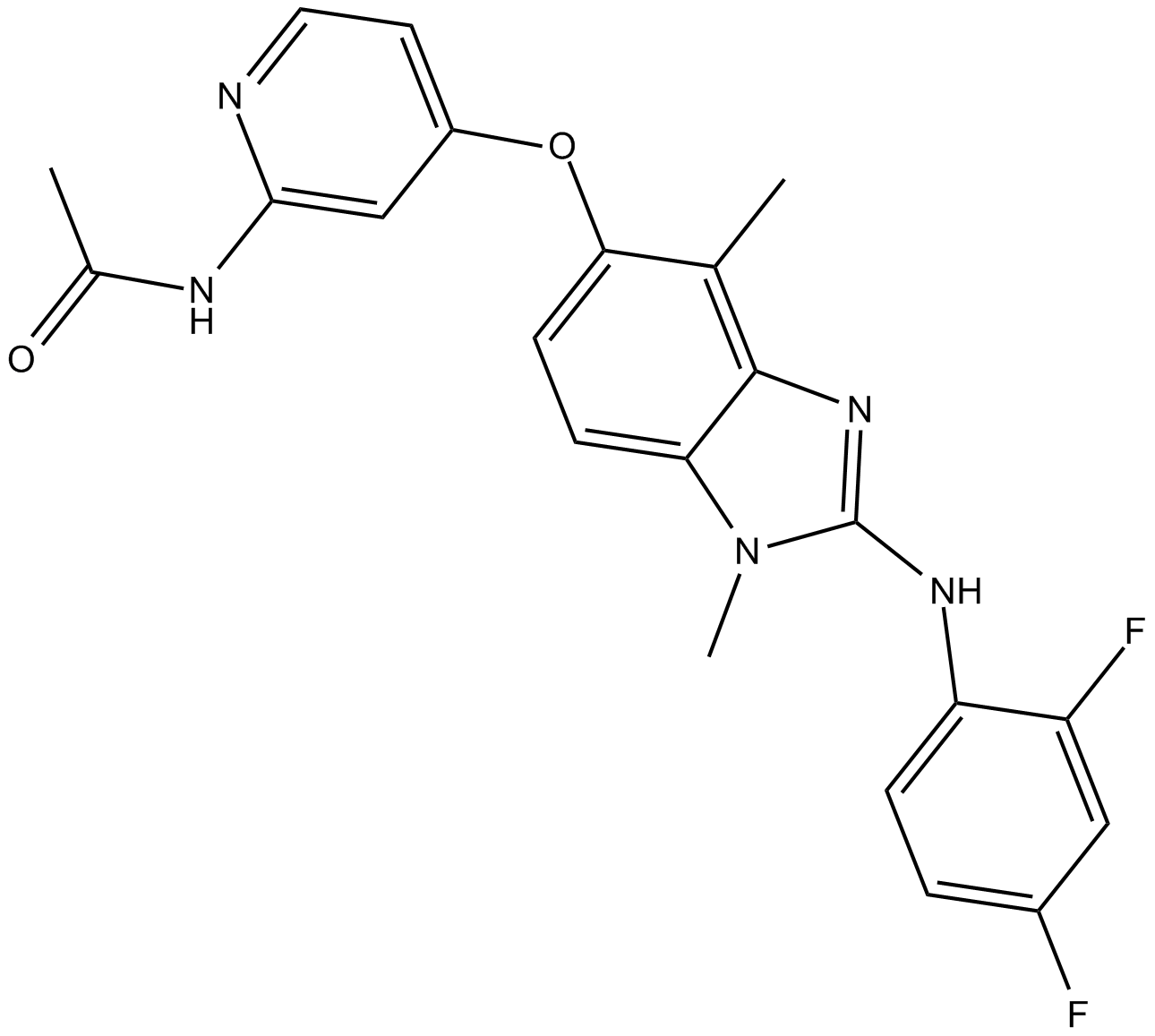
CHZ868
CHZ868 ist ein Typ II JAK2-Inhibitor mit einem IC50 von 0,17 μM in EPOR JAK2 WT Ba/F3-Zelle.
-
GC11539
CI 976
CI 769 (CI 769) ist ein potenter, oral aktiver und selektiver Inhibitor von ACAT (Acyl-Coenzym A:Cholesterin-Acyltransferase) mit einem IC50-Wert von 73 nM.
-
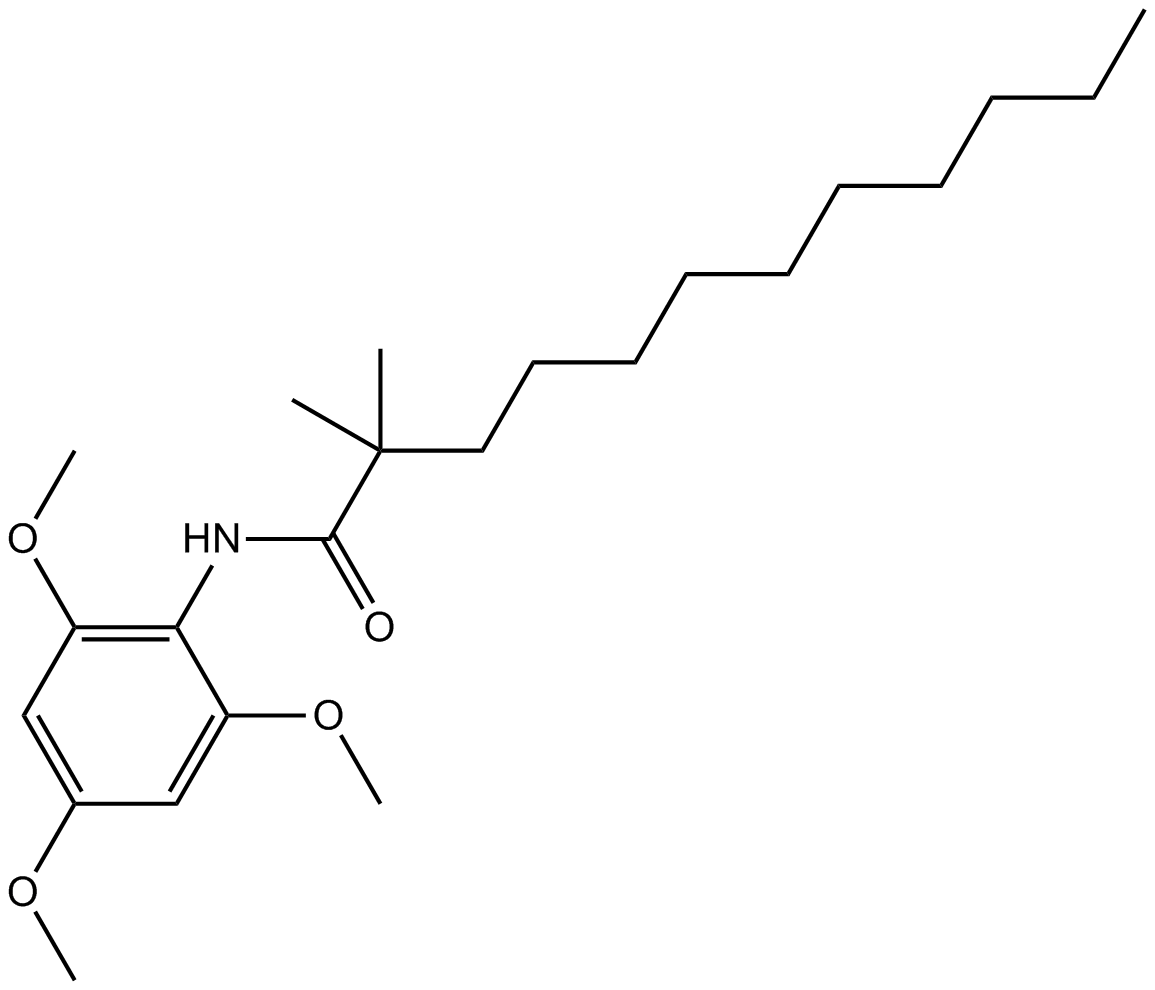
GC13408
CI994 (Tacedinaline)
An inhibitor of HDAC1, -2, and -3
-
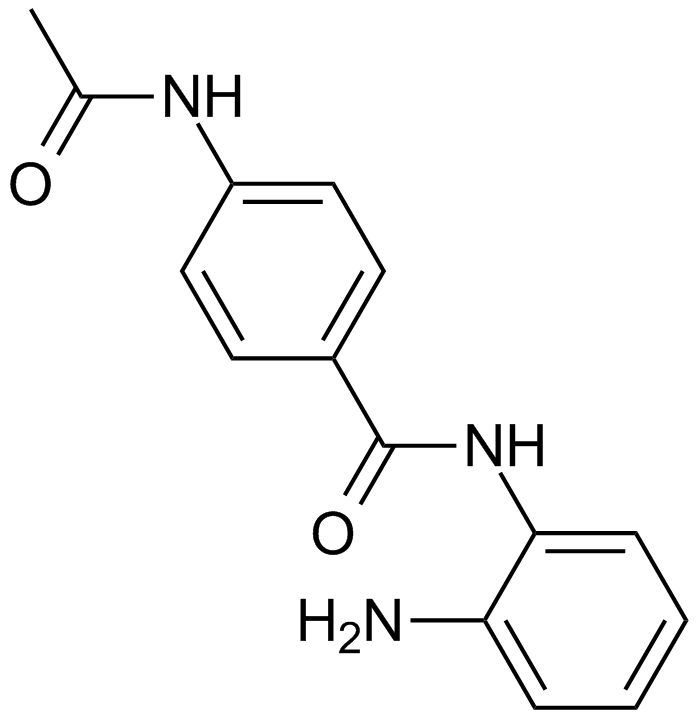
GC15718
CID 2011756
An inhibitor of protein kinase D
-
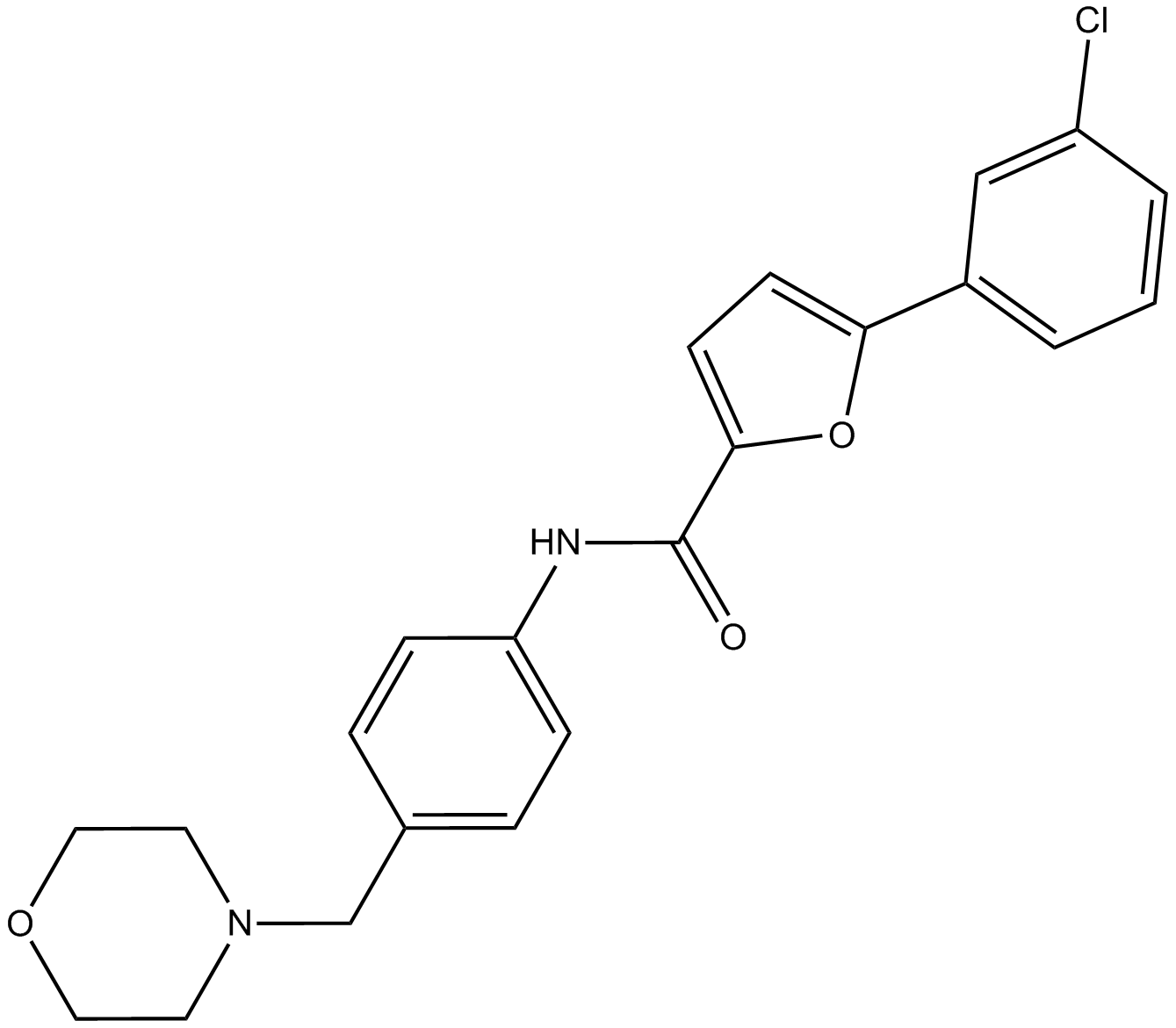
GC18577
CID-2818500
An inhibitor of PRMT1
-
GC52351
Citrullinated α-Enolase (R8 + R14) (1-19)-biotin Peptide
A biotinylated and citrullinated α-enolase peptide

-
GC52363
Citrullinated Histone H3 (R2 + R8 + R17) (2-22)-biotin Peptide
A biotinylated and citrullinated histone H3 peptide

-
GC52367
Citrullinated Vimentin (G146R) (R144 + R146) (139-159)-biotin Peptide
A biotinylated and citrullinated mutant vimentin peptide

-
GC52370
Citrullinated Vimentin (R144) (139-159)-biotin Peptide
A biotinylated and citrullinated vimentin peptide

-
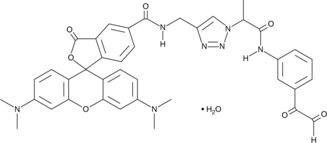
GC43275
Citrulline-specific Probe-Rhodamine
Protein arginine deiminases (PADs) catalyze the posttranslational modification of arginine residues on proteins to form citrulline, which plays a large role in regulating gene expression.
-
GC39485
CK2/ERK8-IN-1
CK2/ERK8-IN-1 ist ein dualer Caseinkinase 2 (CK2) (Ki von 0,25 μM) und ERK8 (MAPK15, ERK7) Inhibitor mit IC50s von 0,50 μM. CK2/ERK8-IN-1 bindet auch an PIM1, HIPK2 (homÖodomÄnen-interagierende Proteinkinase 2) und DYRK1A mit Kis von 8,65 μM, 15,25 μM bzw. 11,9 μM. CK2/ERK8-IN-1 hat pro-apoptotische Wirksamkeit.
-
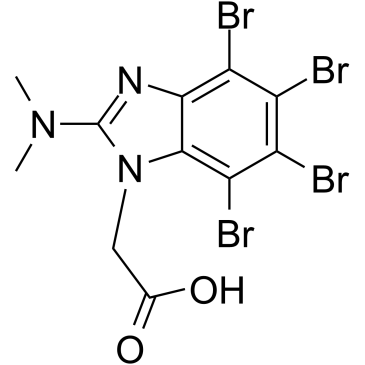
GC35706
Cl-amidine
Cl-Amidin ist ein oral aktiver Peptidylarginin-Deminase (PAD)-Inhibitor mit IC50-Werten von 0,8 μM, 6,2 μM und 5,9 μM fÜr PAD1, PAD3 bzw. PAD4.
-
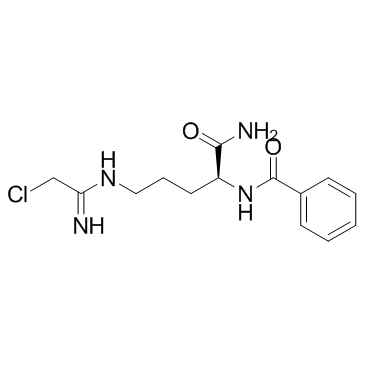
GC18925
Cl-Amidine (hydrochloride)
Cl-Amidin (Hydrochlorid) ist ein oral aktiver Peptidylarginin-Deminase (PAD)-Inhibitor mit IC50-Werten von 0,8 μM, 6,2 μM und 5,9 μM für PAD1, PAD3 bzw. PAD4.
-
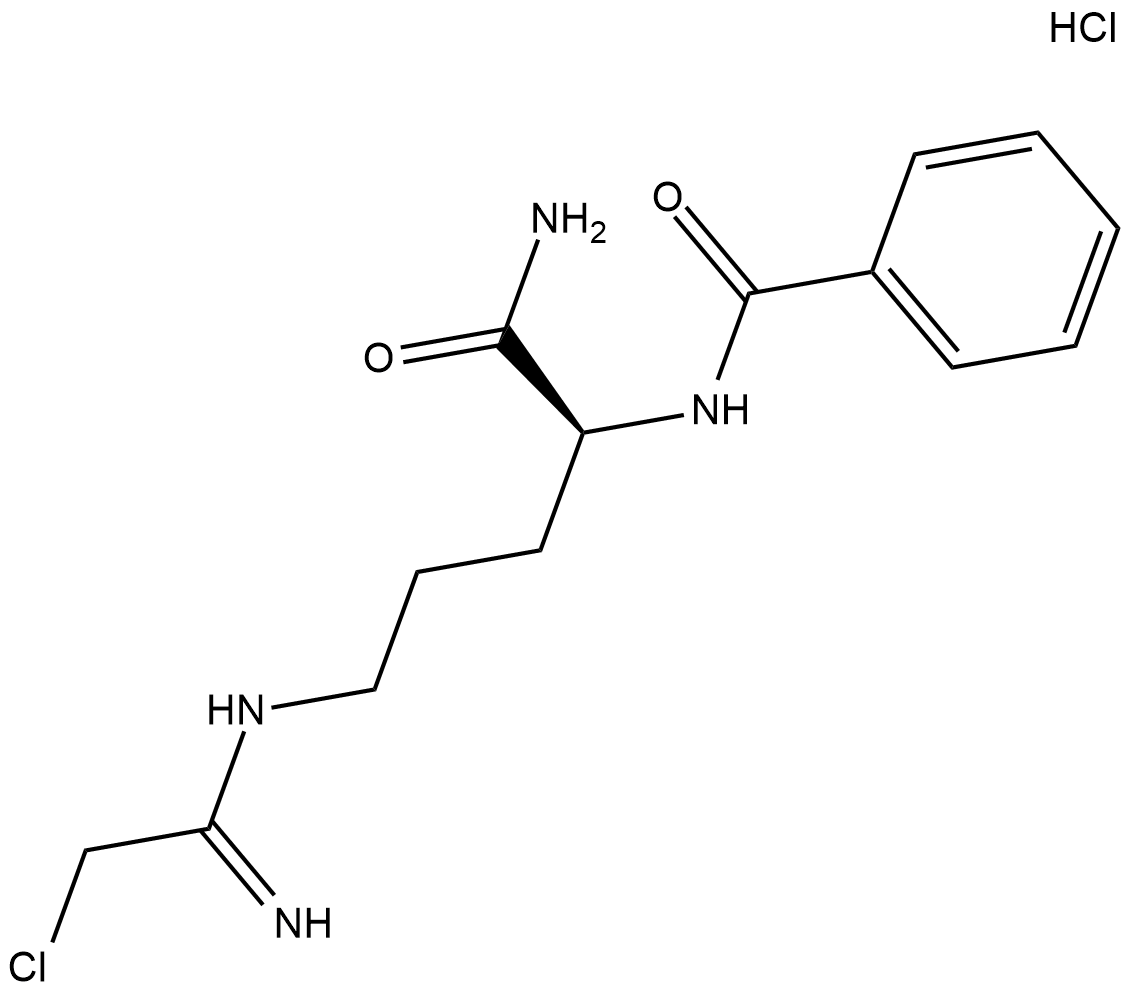
GC11032
Cl-Amidine (trifluoroacetate salt)
Cl-Amidin (Trifluoracetat-Salz) ist ein oral aktiver Peptidylarginindeminase (PAD)-Inhibitor mit IC50-Werten von 0,8 μM, 6,2 μM und 5,9 μM für PAD1, PAD3 und PAD4.
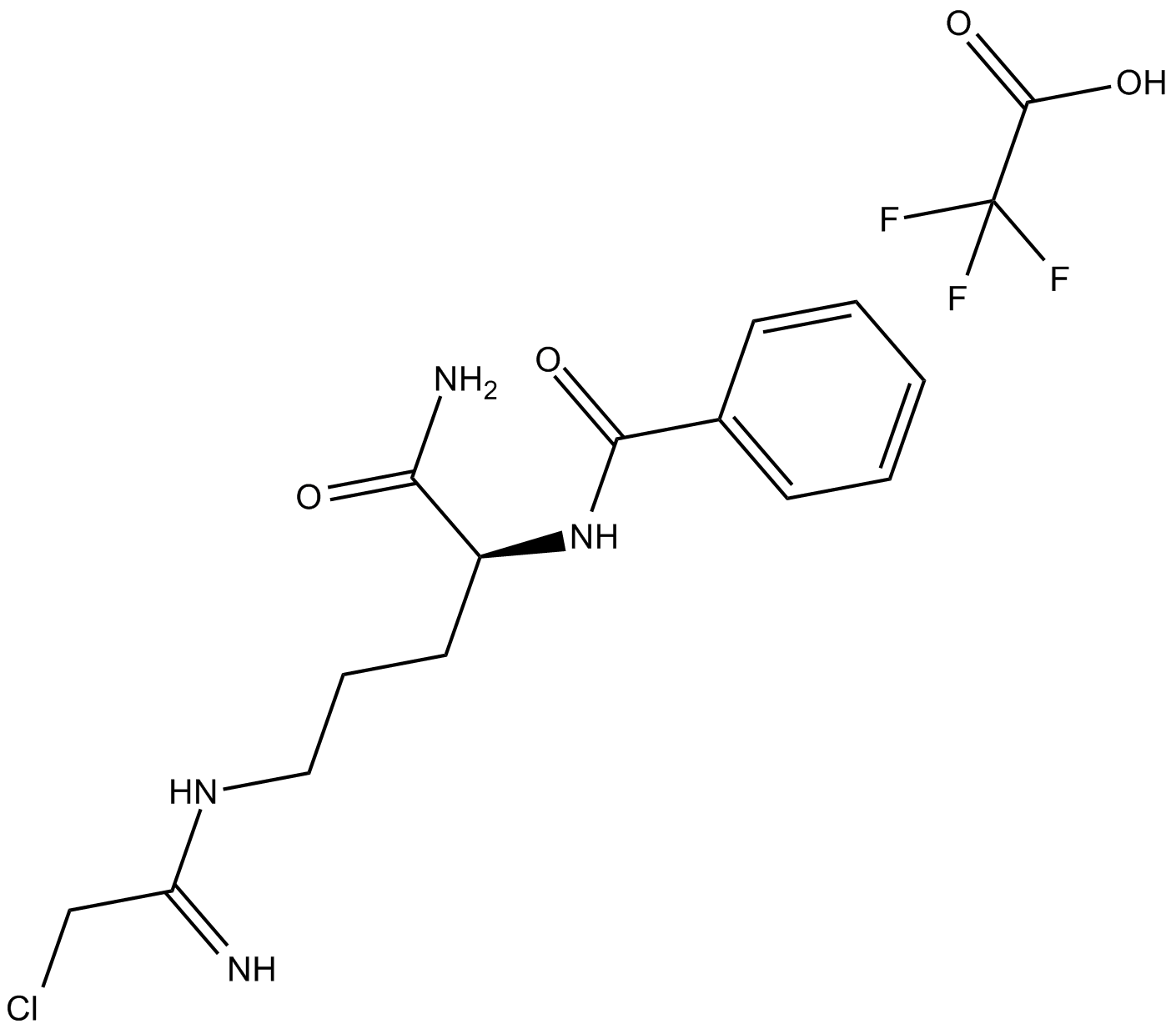
-
GC12367
CM-272
CM-272 ist ein erstklassiger, potenter, selektiver, substratkompetitiver und reversibler dualer G9a/DNA-Methyltransferase (DNMTs)-Inhibitor mit AntitumoraktivitÄten. CM-272 hemmt G9a, DNMT1, DNMT3A, DNMT3B und GLP mit IC50-Werten von 8 nM, 382 nM, 85 nM, 1200 nM bzw. 2 nM. CM-272 hemmt die Zellproliferation und fÖrdert die Apoptose, wodurch IFN-stimulierte Gene und immunogener Zelltod induziert werden.
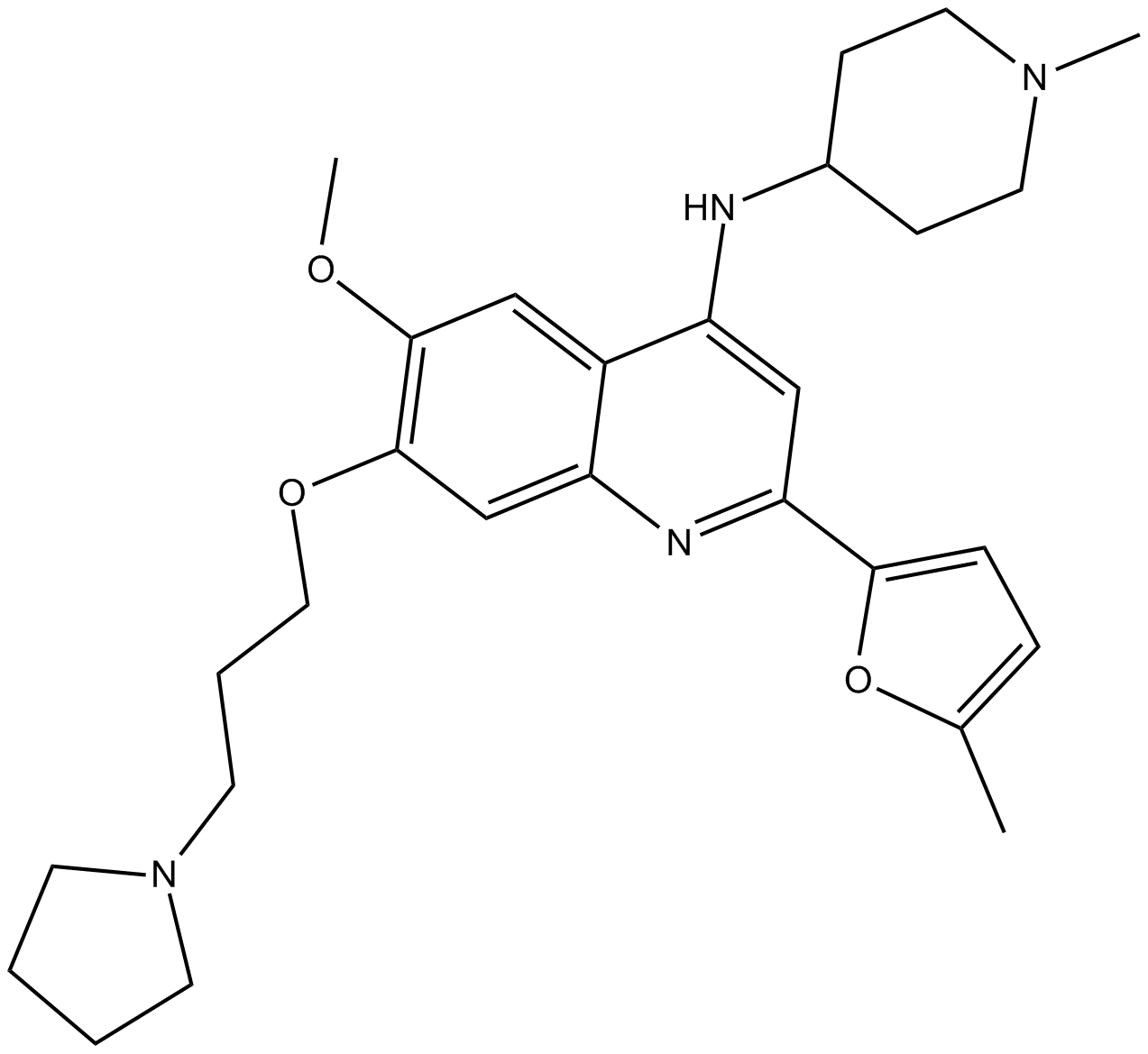
-
GC33320
CM-579
CM-579 ist ein erstklassiger reversibler dualer Inhibitor von G9a und DNMT mit IC50-Werten von 16 nM, 32 nM fÜr G9a bzw. DNMT. Hat eine starke zellulÄre In-vitro-AktivitÄt in einer Vielzahl von Krebszellen.
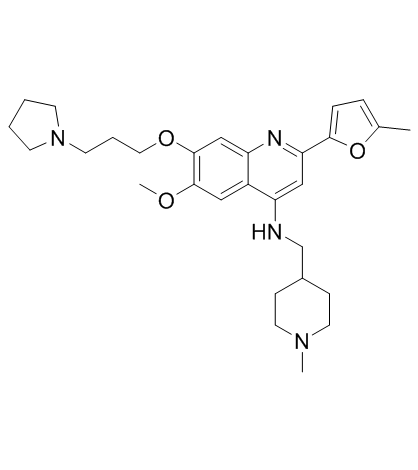
-
GC35714
CM-579 trihydrochloride
CM-579-Trihydrochlorid ist ein erstklassiger reversibler dualer Inhibitor von G9a und DNMT mit IC50-Werten von 16 nM bzw. 32 nM fÜr G9a und DNMT. Hat eine starke zellulÄre In-vitro-AktivitÄt in einer Vielzahl von Krebszellen.
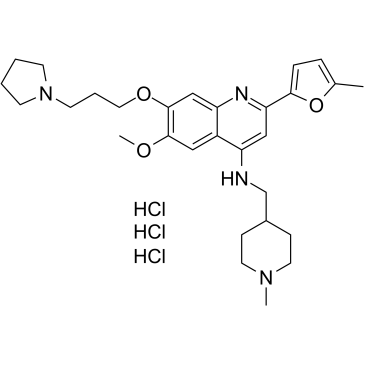
-
GC65426
CM-675
CM-675 ist ein dualer, fÜr Phosphodiesterase 5 (PDE5) und Histon-Deacetylasen der Klasse I selektiver Inhibitor mit IC50-Werten von 114 nM und 673 nM fÜr PDE5 bzw. HDAC1.
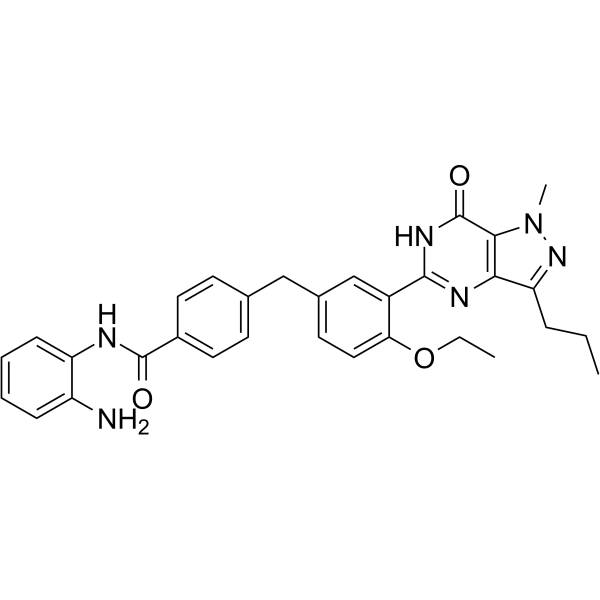
-
GC39665
CMP-5
CMP-5 ist ein potenter, spezifischer und selektiver PRMT5-Inhibitor, wÄhrend es keine AktivitÄt gegen PRMT1-, PRMT4- und PRMT7-Enzyme zeigt. CMP-5 blockiert selektiv S2Me-H4R3, indem es die PRMT5-Methyltransferase-AktivitÄt auf HistonprÄparaten hemmt. CMP-5 verhindert die vom Epstein-Barr-Virus (EBV) getriebene B-Lymphozyten-Transformation, lÄsst aber normale B-Zellen unbeeinflusst.
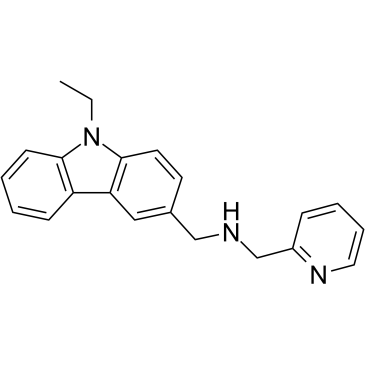
-
GC34165
Corin
Corin ist ein dualer Inhibitor der Histon-Lysin-spezifischen Demethylase (LSD1) und Histon-Deacetylase (HDAC) mit einem Ki(inact) von 110 nM fÜr LSD1 und einem IC50 von 147 nM fÜr HDAC1.
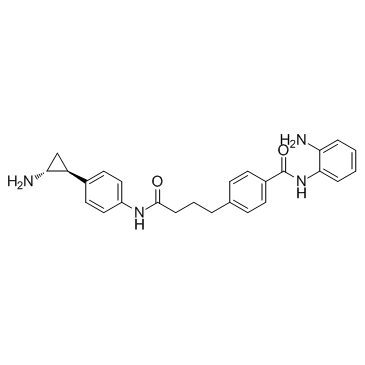
-
GC43318
coumarin-SAHA
Coumarin-SAHA ist eine Fluoreszenzsonde zur Bestimmung der BindungsaffinitÄten (kd) und der Dissoziationsraten (koff) der HDAC8-Inhibitor-Komplexe.
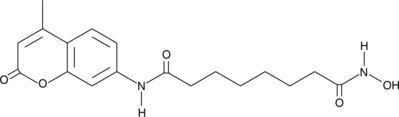
-
GC62908
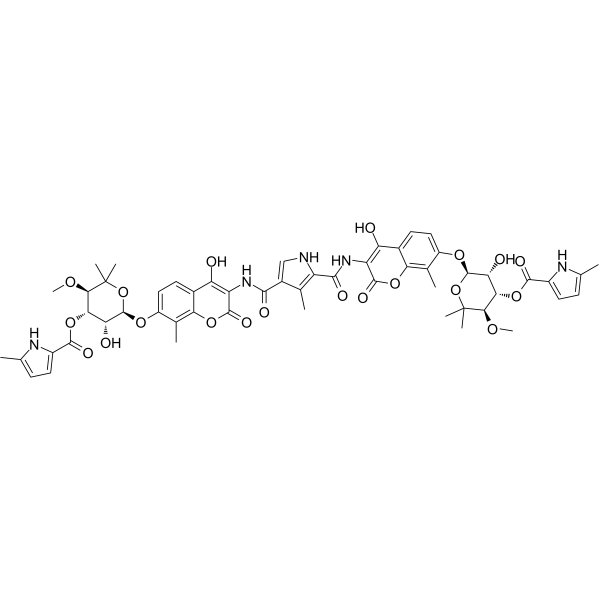
Coumermycin A1
Coumermycin A1 ist ein JAK2-Signalaktivator.
-
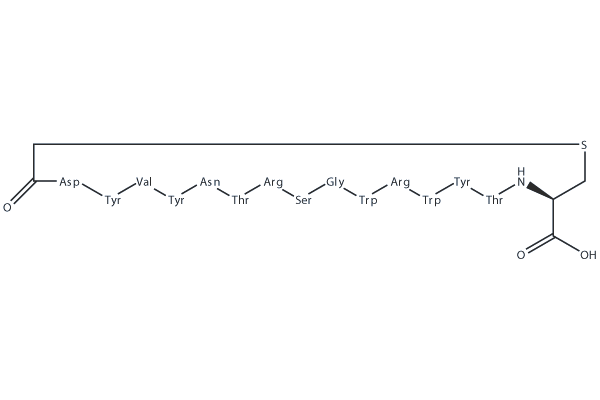
GC25304
CP2
CP2 is a cyclic peptide that inhibits the JmjC histone demethylases KDM4 with IC50 values of 42 nM and 29 nM for KDM4A and KDM4C, respectively.
-
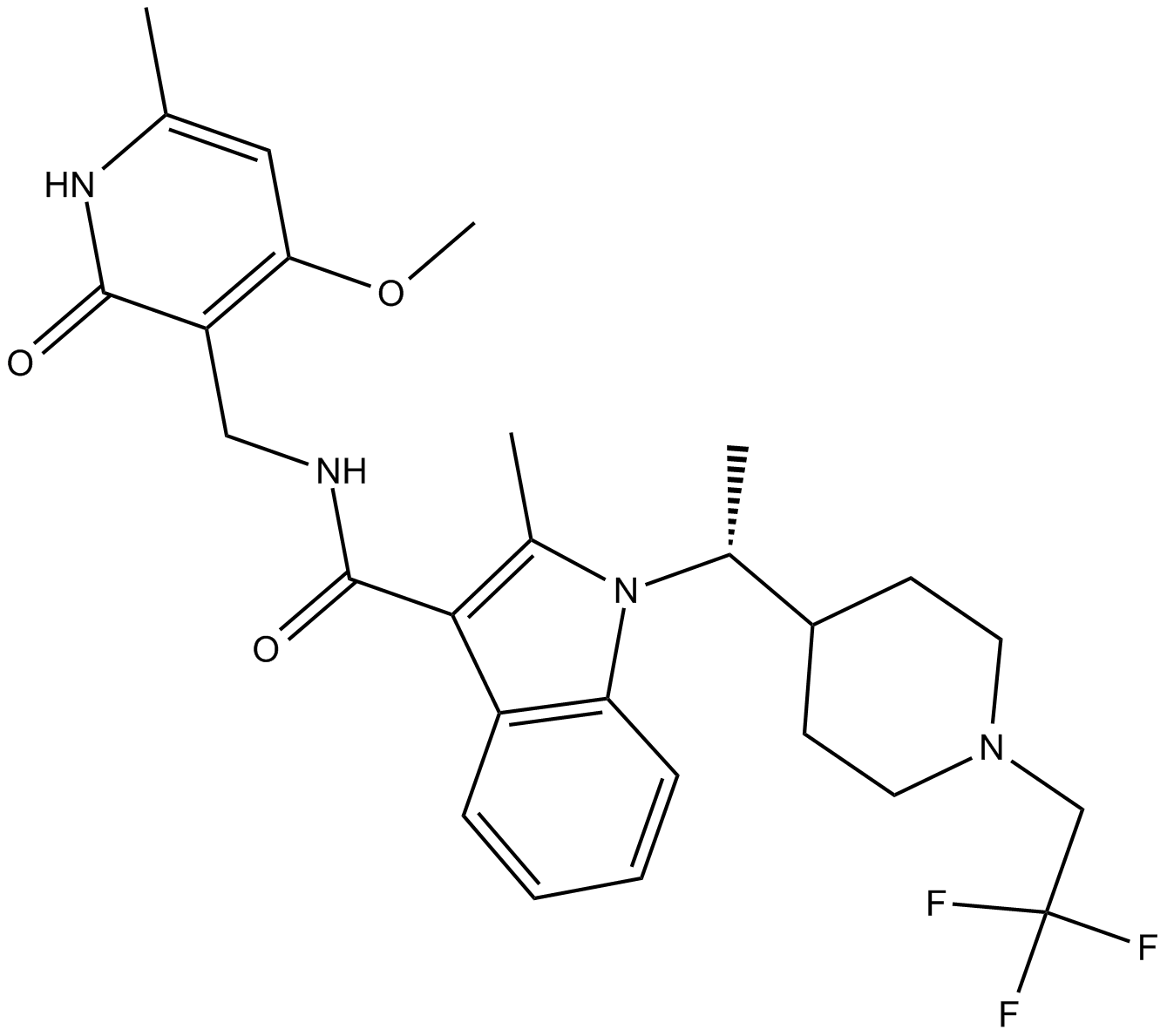
GC16298
CPI-1205
EZH2 inhibitor
-
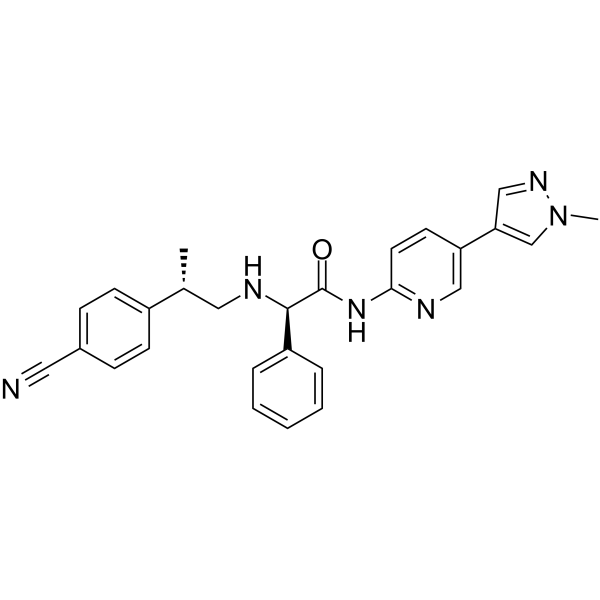
GC62669
CPI-1612
CPI-1612 ist ein hochpotenter, oral aktiver EP300/CBP-Histonacetyltransferase (HAT)-Inhibitor mit einem IC50 von 8,1 nM fÜr EP300 HAT. CPI-1612 hat eine AntikrebsaktivitÄt.
-
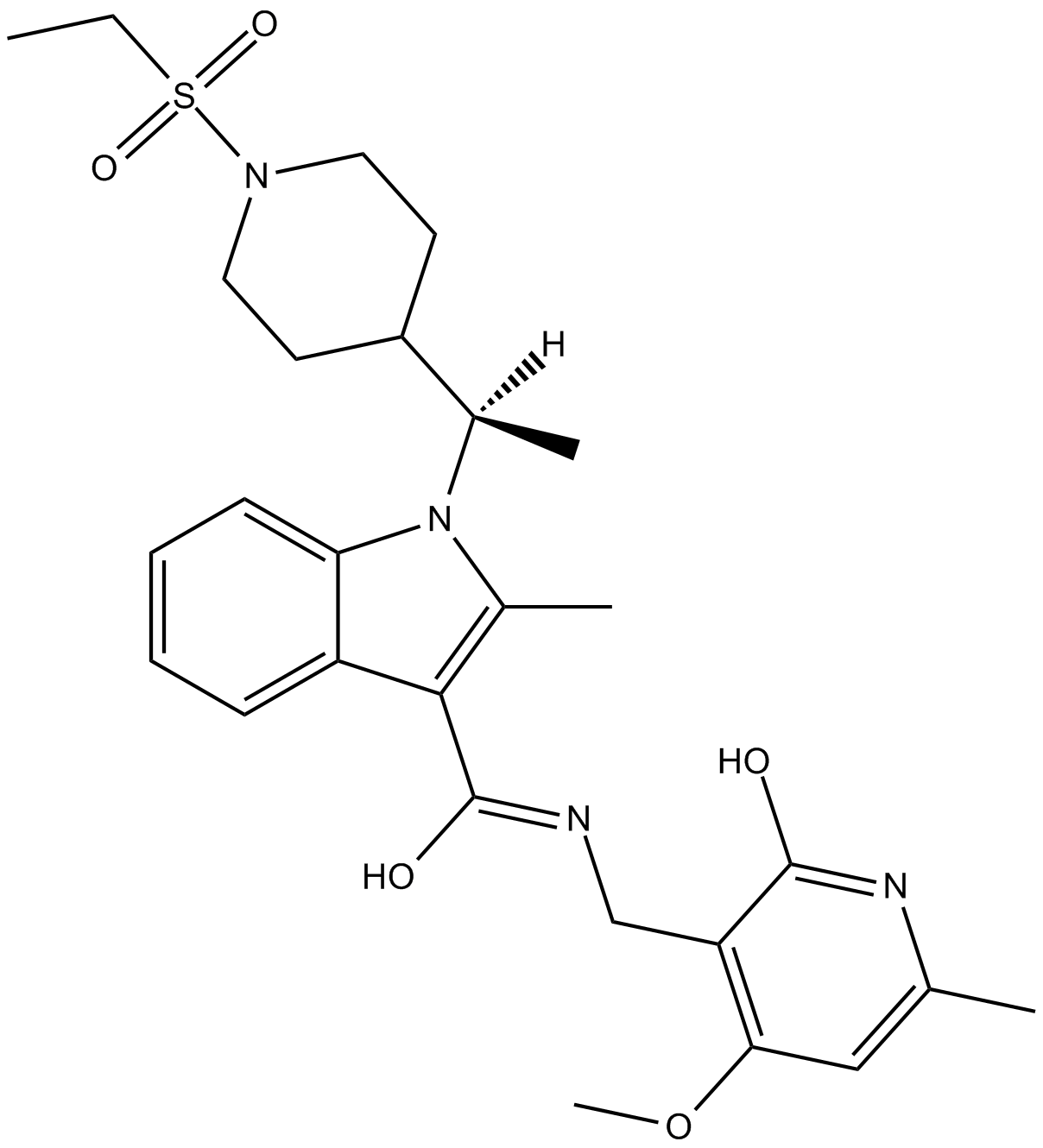
GC16599
CPI-169
EZH2 inhibitor
-
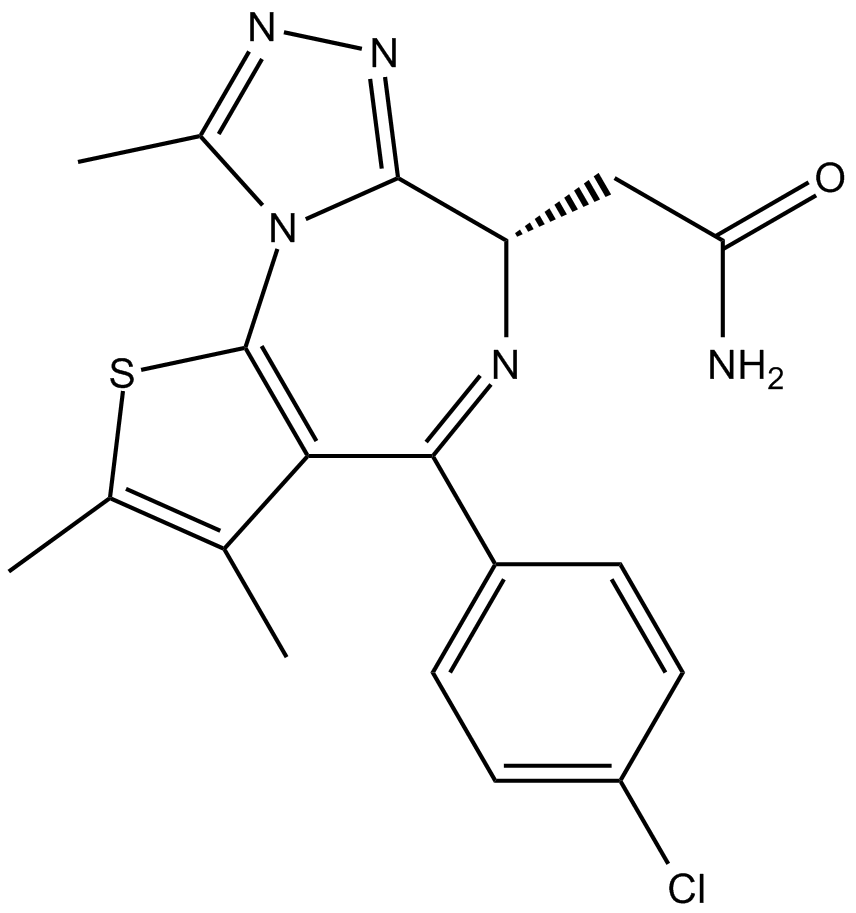
GC14699
CPI-203
CPI-203 ist ein neuartiger potenter, selektiver und zellgÄngiger Inhibitor der BET-BromodomÄne mit einem IC50-Wert von ca. 37 nM (BRD4 α-Screen-Assay).
-
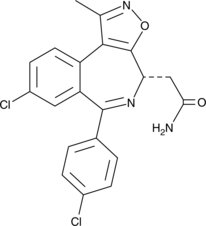
GC43320
CPI-268456
CPI-268456 (Verbindung 141) ist ein potenter BRD4-Inhibitor mit einem IC50 von < 0,5 μM.
-
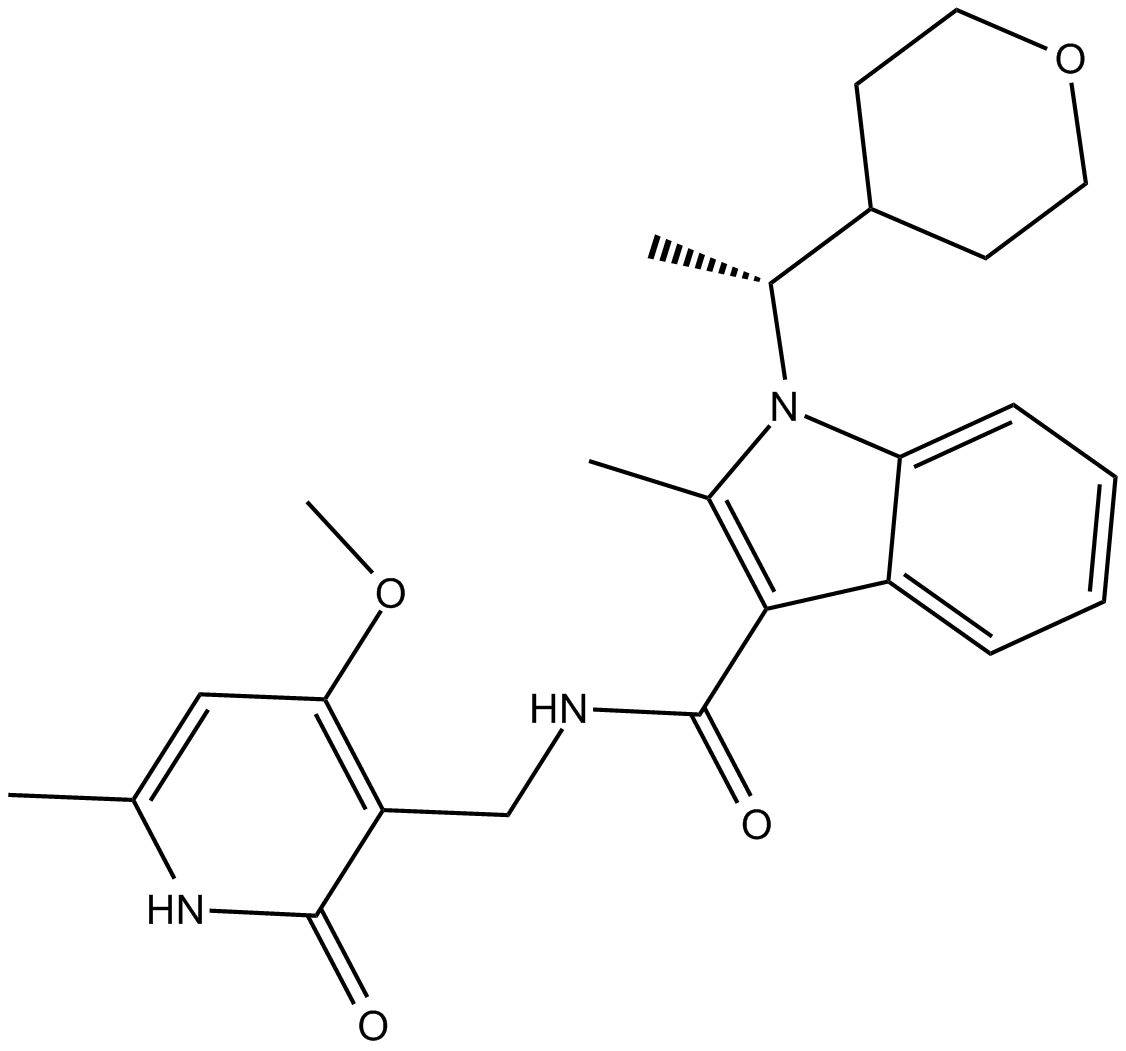
GC10021
CPI-360
EZH2 inhibitor
-
GC10774
CPI-455
KDM5-Inhibitor
-
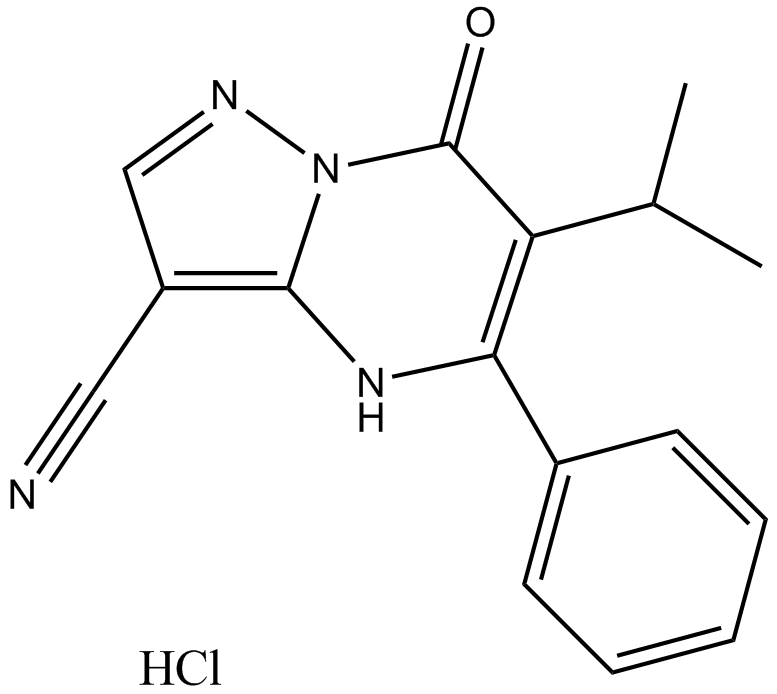
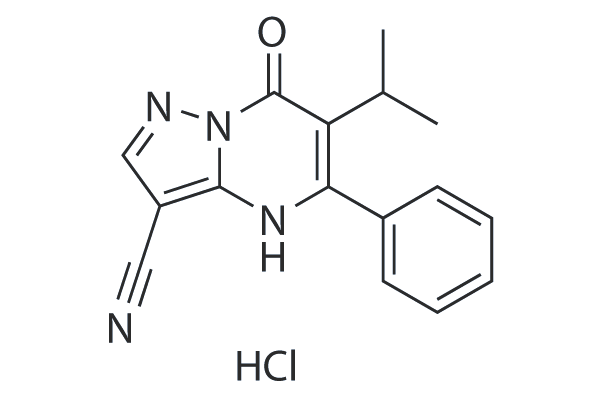
GC25305
CPI-455 HCl
CPI-455 HCl is a specific KDM5 inhibitor with a half-maximal inhibitory concentration (IC50) of 10 ± 1 nM for full-length KDM5A in enzymatic assays, elevating global levels of H3K4 trimethylation (H3K4me3) and decreased the number of DTPs in multiple cancer cell line models treated with standard chemotherapy or targeted agents.This product has poor solubility, animal experiments are available, cell experiments please choose carefully!
-
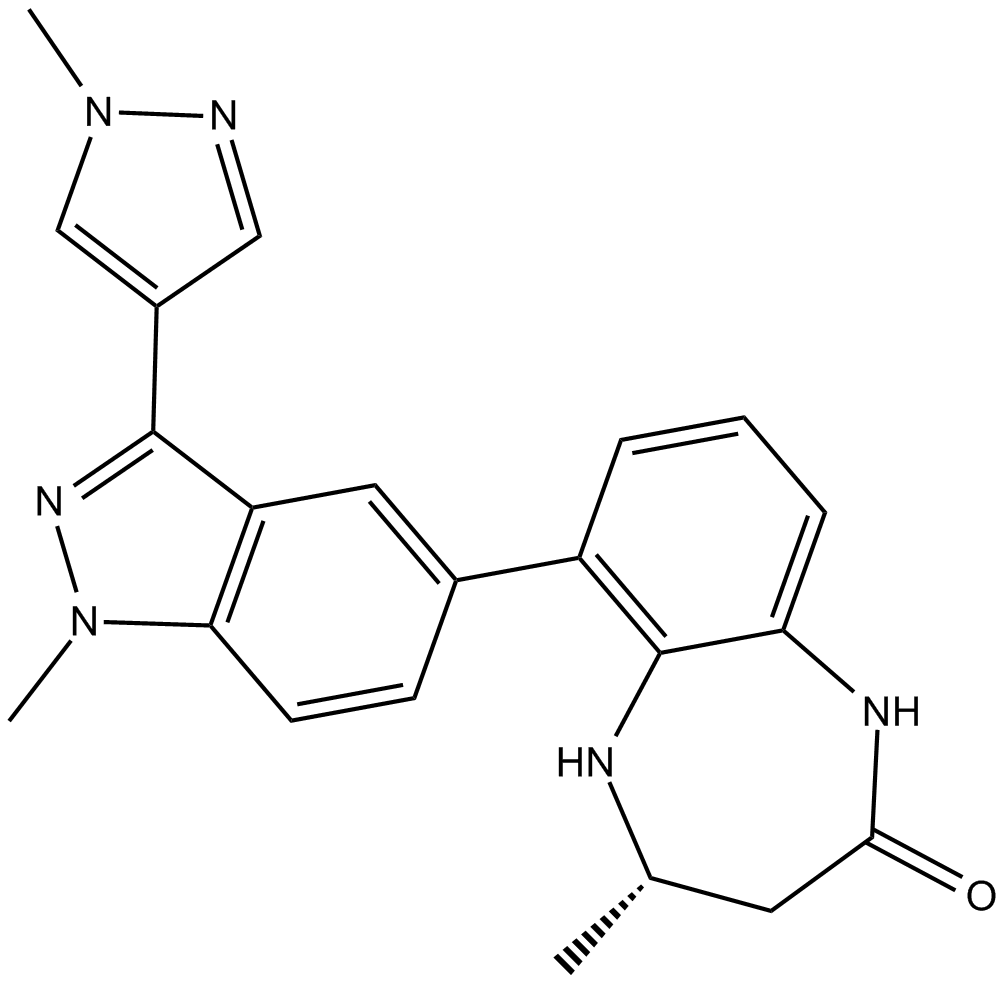
GC10382
CPI-637
CPI-637 ist ein selektiver und potenter CBP/EP300-BromodomÄnen-Inhibitor mit IC50-Werten von 0,03 μM, 0,051 μM und 11,0 μM fÜr CBP, EP300 bzw. BRD4 BD-1 und einem EC50 von 0,3 μM fÜr CBP.
-
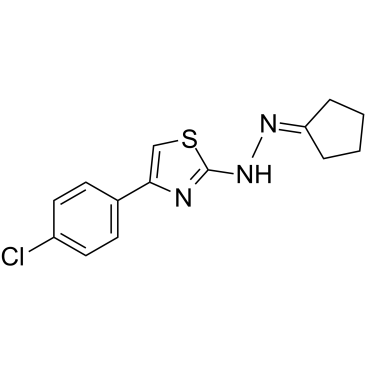
GC39365
CPTH2
CPTH2 ist ein potenter Hemmer der Histonacetyltransferase (HAT). CPTH2 hemmt selektiv die Acetylierung von Histon H3 durch Gcn5. CPTH2 induziert Apoptose und verringert die InvasivitÄt einer Zelllinie des klarzelligen Nierenkarzinoms (ccRCC) durch die Hemmung der Acetyltransferase p300 (KAT3B).
-
GC14428
CPTH2 (hydrochloride)
CPTH2 (Hydrochlorid) ist ein potenter Hemmer der Histonacetyltransferase (HAT). CPTH2 (Hydrochlorid) hemmt selektiv die Acetylierung von Histon H3 durch Gcn5. CPTH2 (Hydrochlorid) induziert Apoptose und verringert die Invasivität einer Zelllinie des klarzelligen Nierenkarzinoms (ccRCC) durch die Hemmung der Acetyltransferase p300 (KAT3B).
-
GC35742
CPUY074020
CPUY074020 ist ein potenter und oraler bioverfÜgbarer Inhibitor der Histon-Methyltransferase G9a mit einem IC50 von 2,18 μM. CPUY074020 besitzt antiproliferative AktivitÄt.
-
GC32565
CRA-026440
CRA-026440 ist ein potenter Breitspektrum-HDAC-Hemmer.
-
GC67674
CRA-026440 hydrochloride
-
GC38412
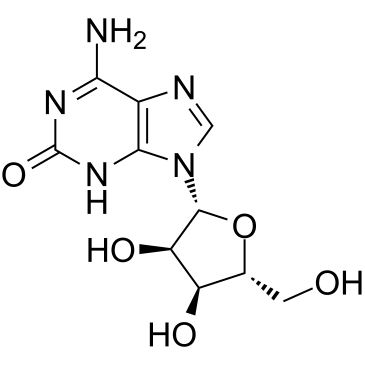
Crotonoside
A guanosine analog with diverse biological activities
-
GC14524
CTPB
CTPB ist ein guter Aktivator des p300-Enzyms Histonacetyltransferase (HAT).
-
GC16008
CUDC-101
A multi-target inhibitor of HDACs, EGFR, and HER2
-
GC12115
CUDC-907
A dual inhibitor of HDACs and PI3Ks
-
GN10442
Curculigoside
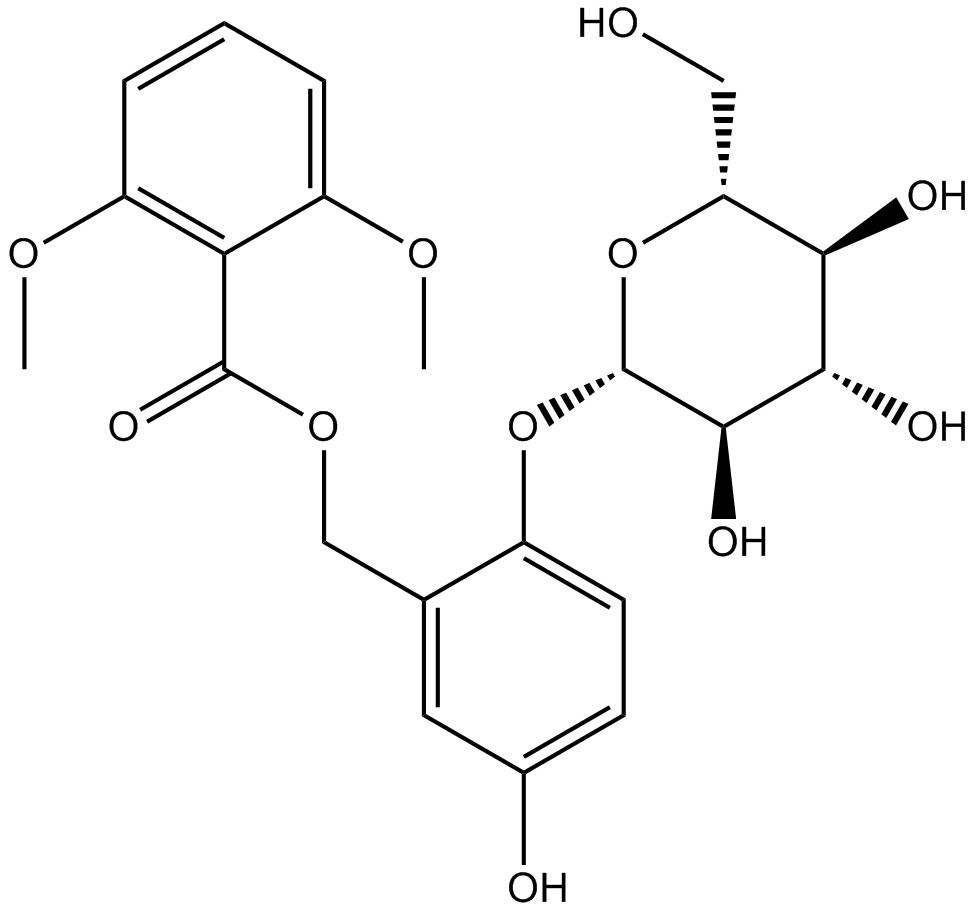
-
GC14787
Curcumin
Ein gelbes Pigment mit vielfältigen biologischen Aktivitäten.
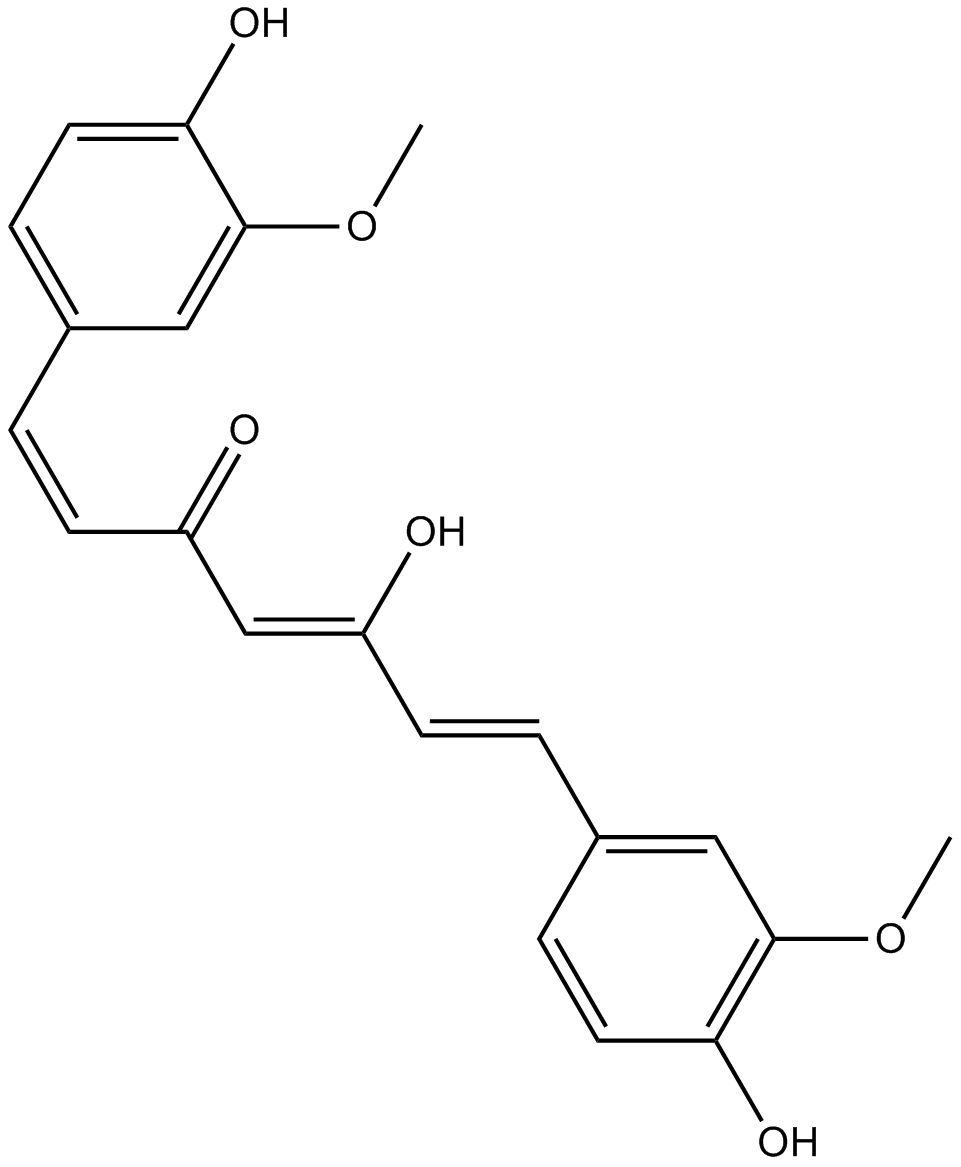
-
GC40226
Curcumin-d6
Curcumin D6 (Diferuloylmethan D6) ist ein Deuterium mit der Bezeichnung Curcumin (gelbe Kurkuma).
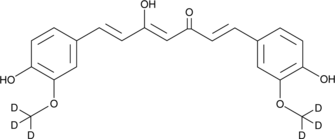
-
GC13056
CX-6258
A pan-Pim kinase inhibitor
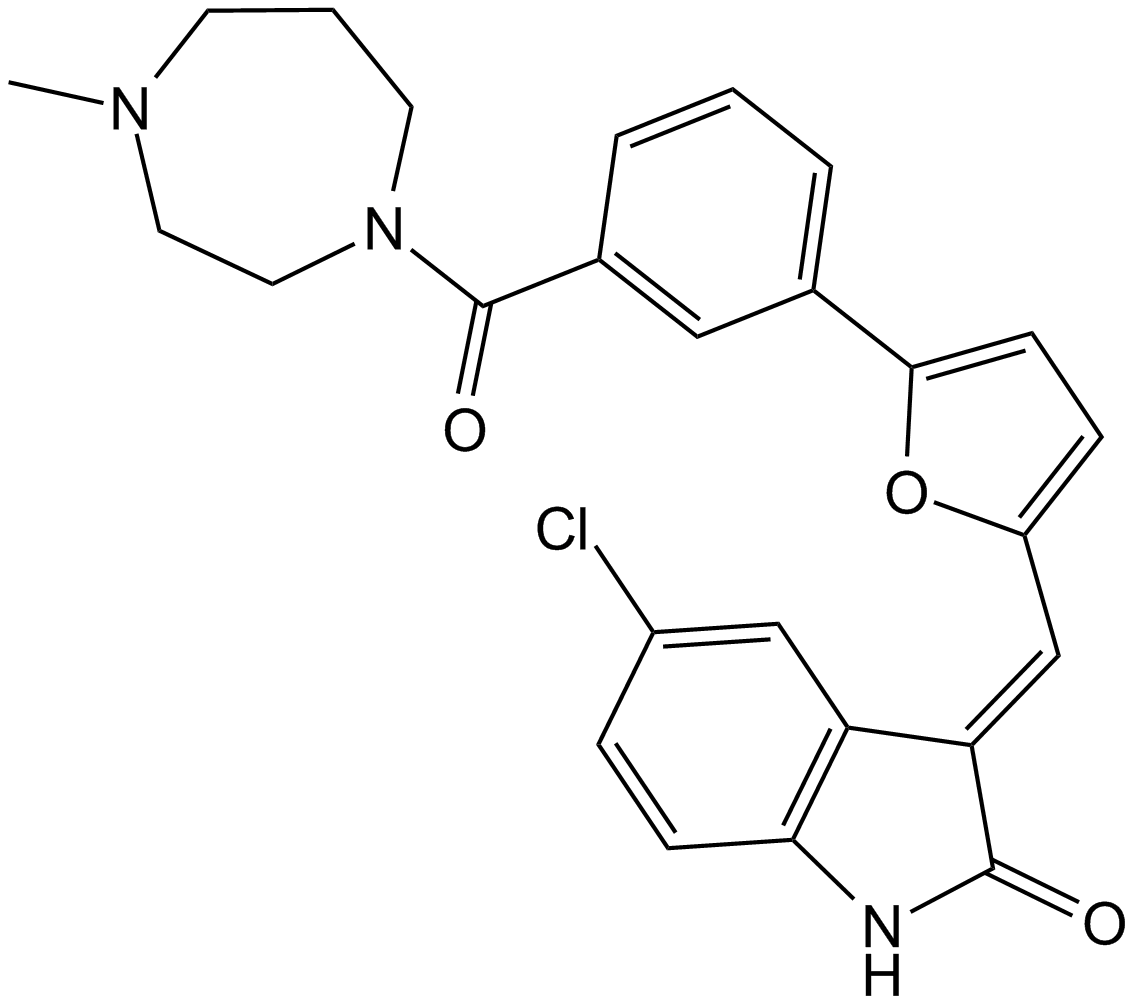
-
GC19747
CX-6258 HCl
CX-6258 HCl ist ein potenter und Kinase-selektiver Pan-Pim-Kinasen-Inhibitor mit IC50-Werten von 5 nM, 25 nM und 16 nM fÜr Pim-1, Pim-2 bzw. Pim-3.
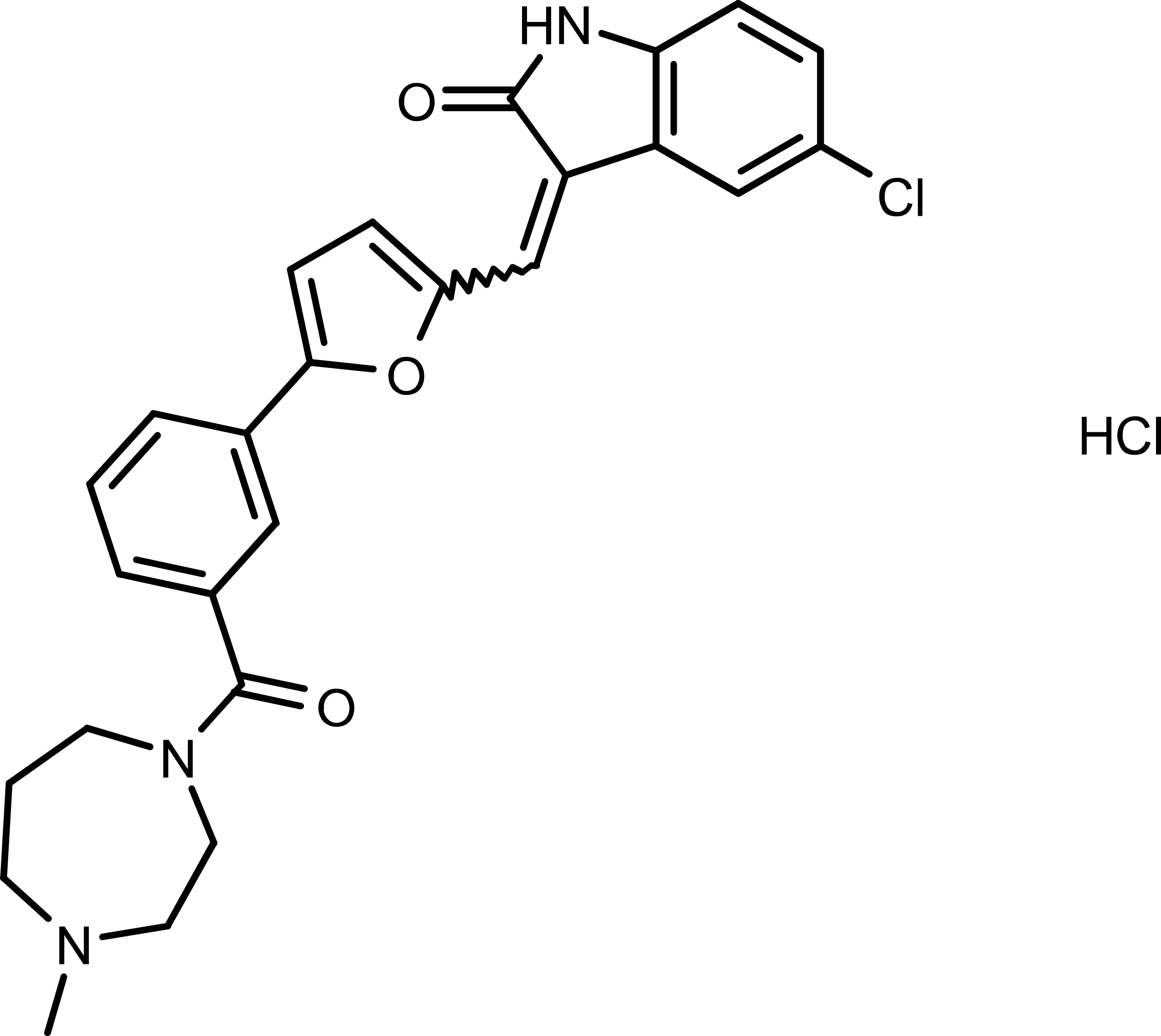
-
GC35762
CX-6258 hydrochloride hydrate
CX-6258-Hydrochloridhydrat ist ein potenter und Kinase-selektiver Pan-Pim-Kinasen-Inhibitor mit IC50-Werten von 5 nM, 25 nM und 16 nM fÜr Pim-1, Pim-2 bzw. Pim-3.
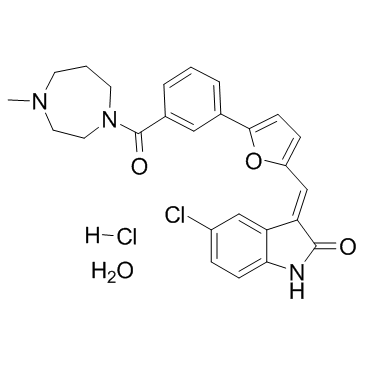
-
GC15106
CYC116
A potent Aurora kinase inhibitor
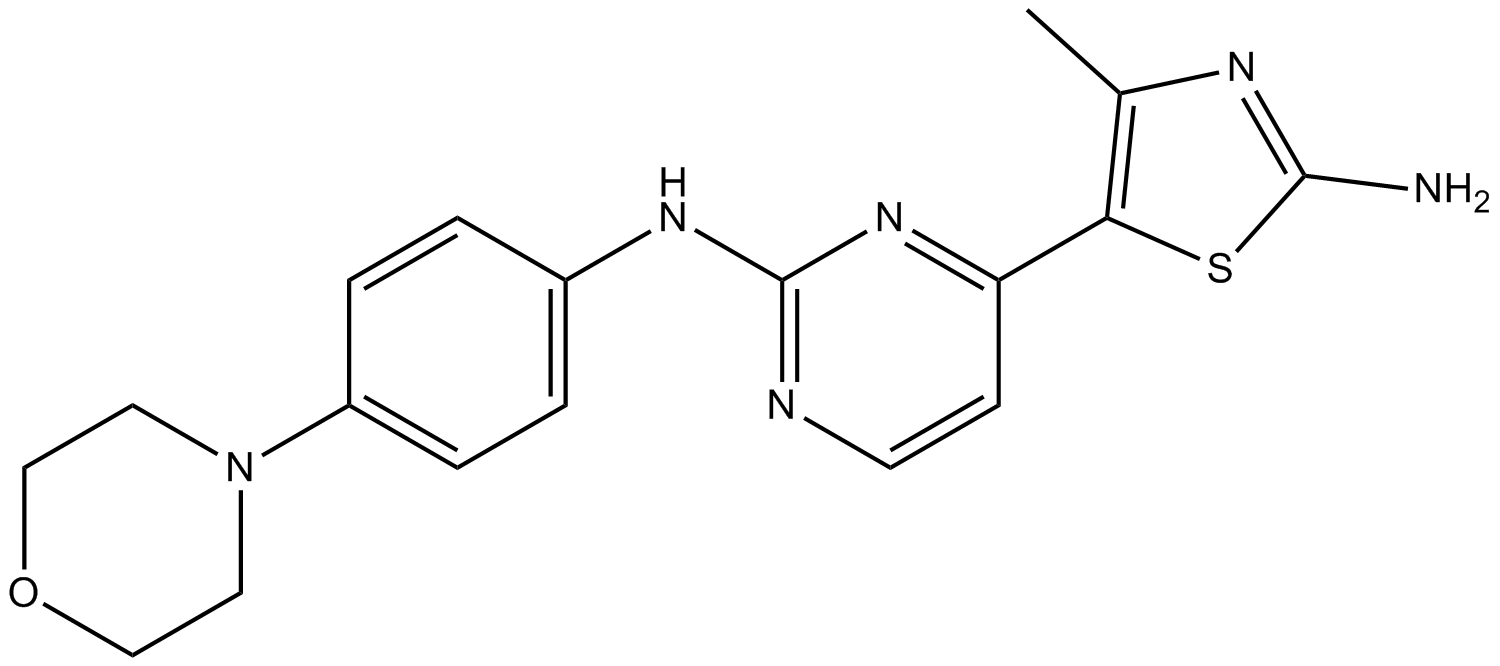
-
GC43354
Cysmethynil
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.

-
GC17050
CYT387
A potent inhibitor of JAK1 and JAK2
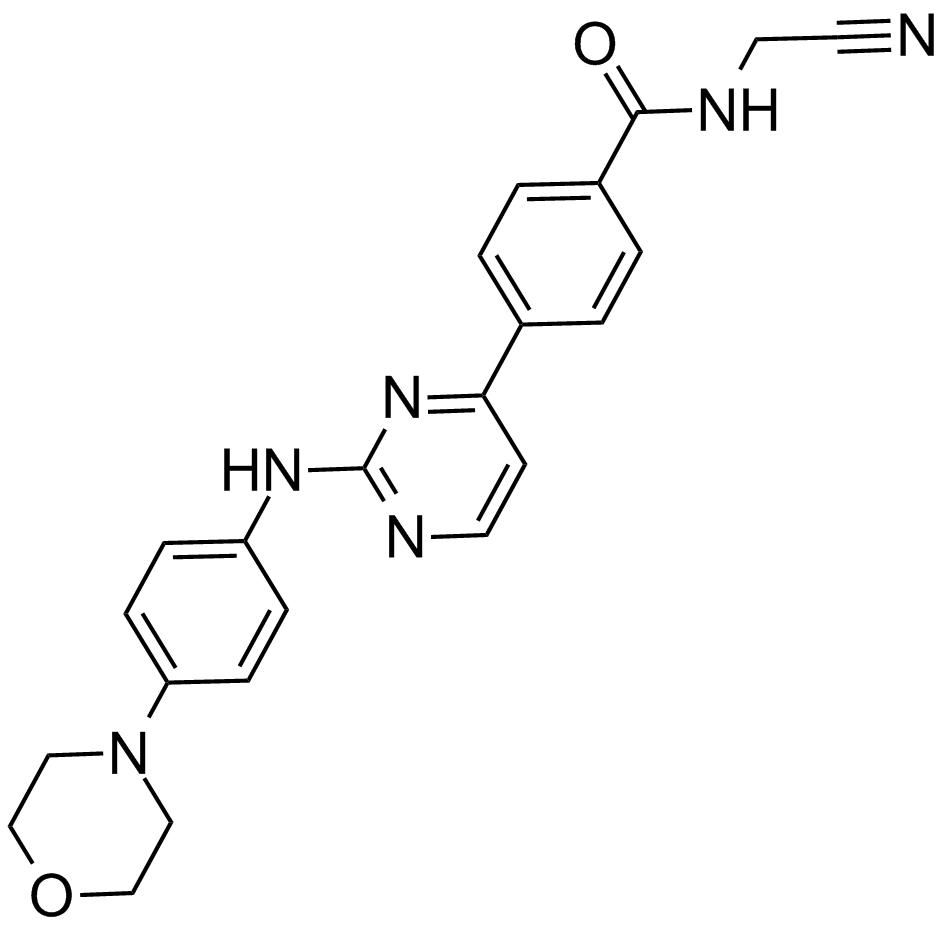
-
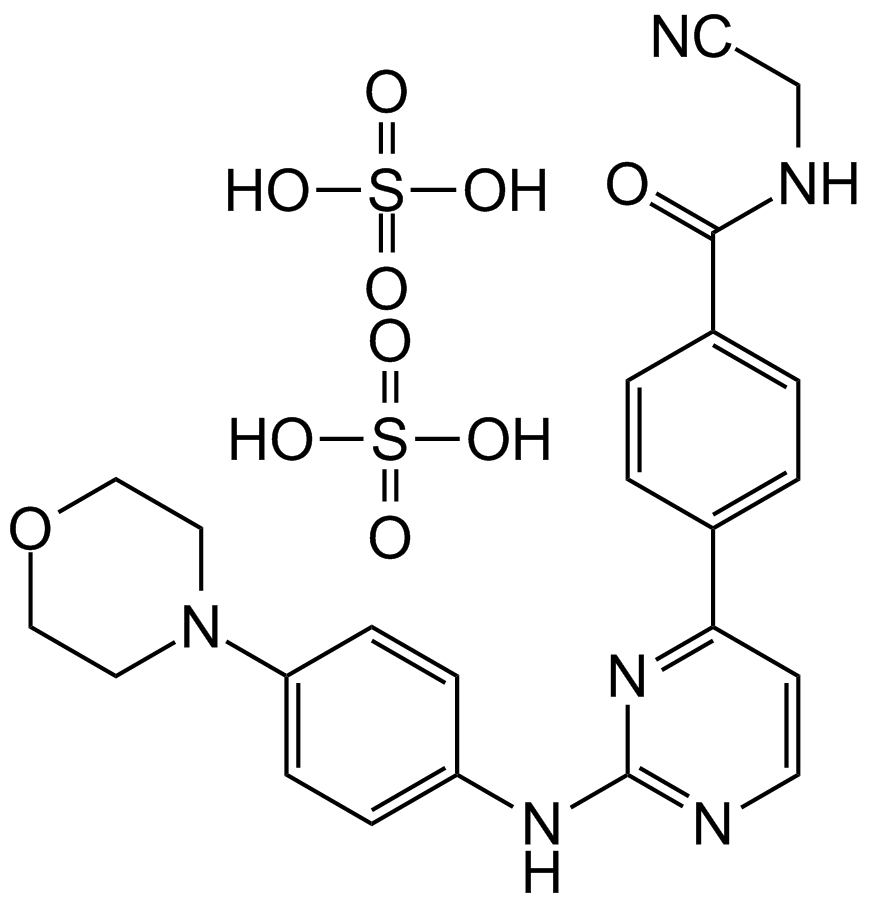
GC16468
CYT387 sulfate salt
A potent inhibitor of JAK1 and JAK2
-
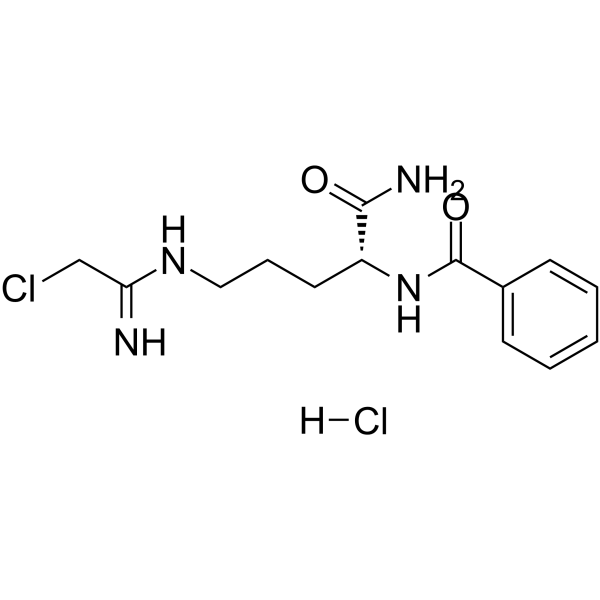
GC63780
D-Cl-amidine hydrochloride
D-Cl-Amidinhydrochlorid ist ein potenter und hochselektiver PAD1-Inhibitor. D-Cl-Amidin ist gut verträglich und weist keine signifikante Toxizität auf.
-
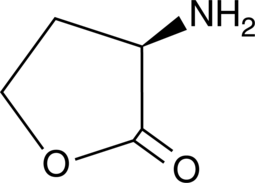
GC48967
D-Homoserine lactone
An enantiomer of L-homoserine lactone
-
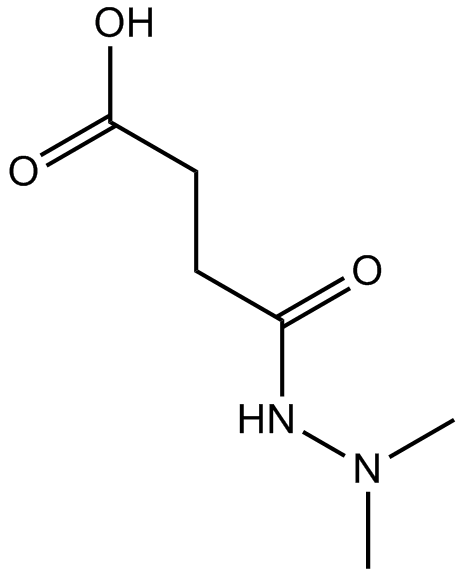
GC15830
Daminozide
Selective inhibitor of KDM2/7 histone demethylases
-
GC15217
Danusertib (PHA-739358)
A pan-Aurora kinase and Abl inhibitor
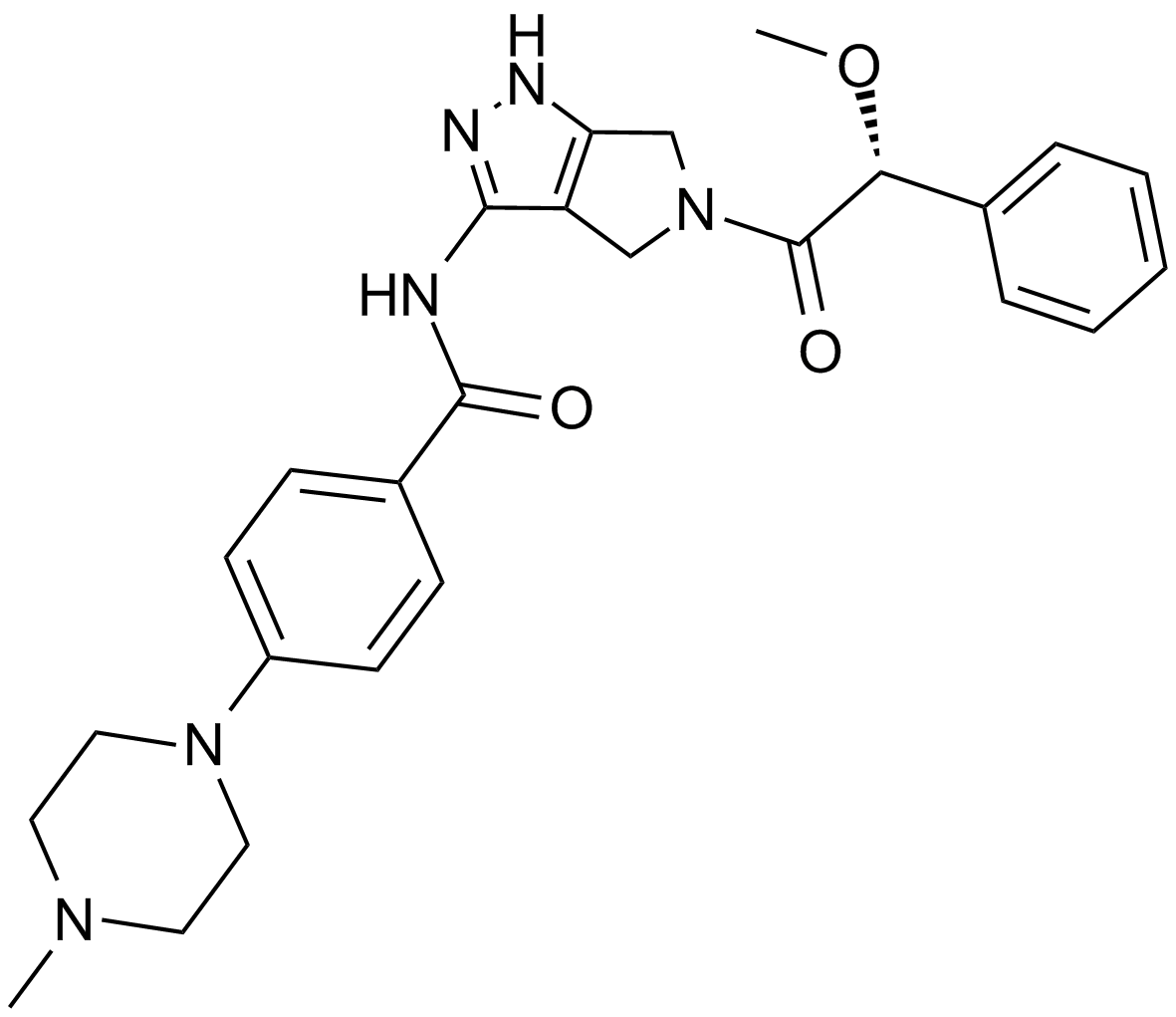
-
GC16647
Daprodustat(GSK1278863)
Daprodustat(GSK1278863) (GSK1278863) ist ein oral aktiver Prolylhydroxylase (HIF-PH)-Inhibitor des Hypoxie-induzierbaren Faktors, der zur Behandlung von AnÄmie im Zusammenhang mit chronischen Nierenerkrankungen entwickelt wird.

-
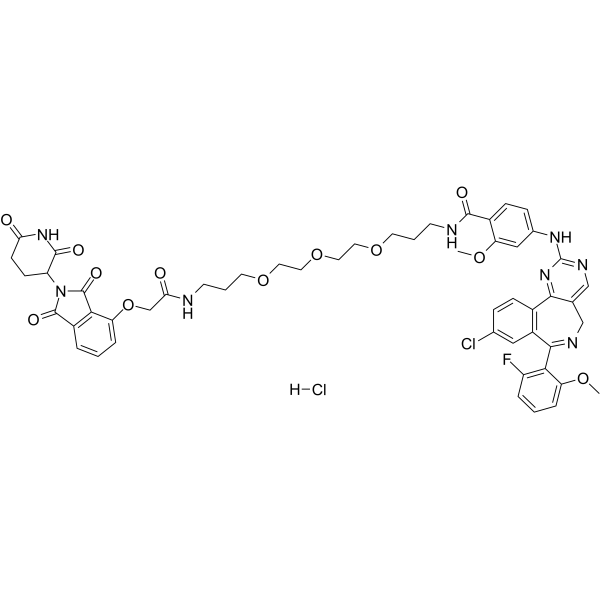
GC68147
dAURK-4 hydrochloride
-
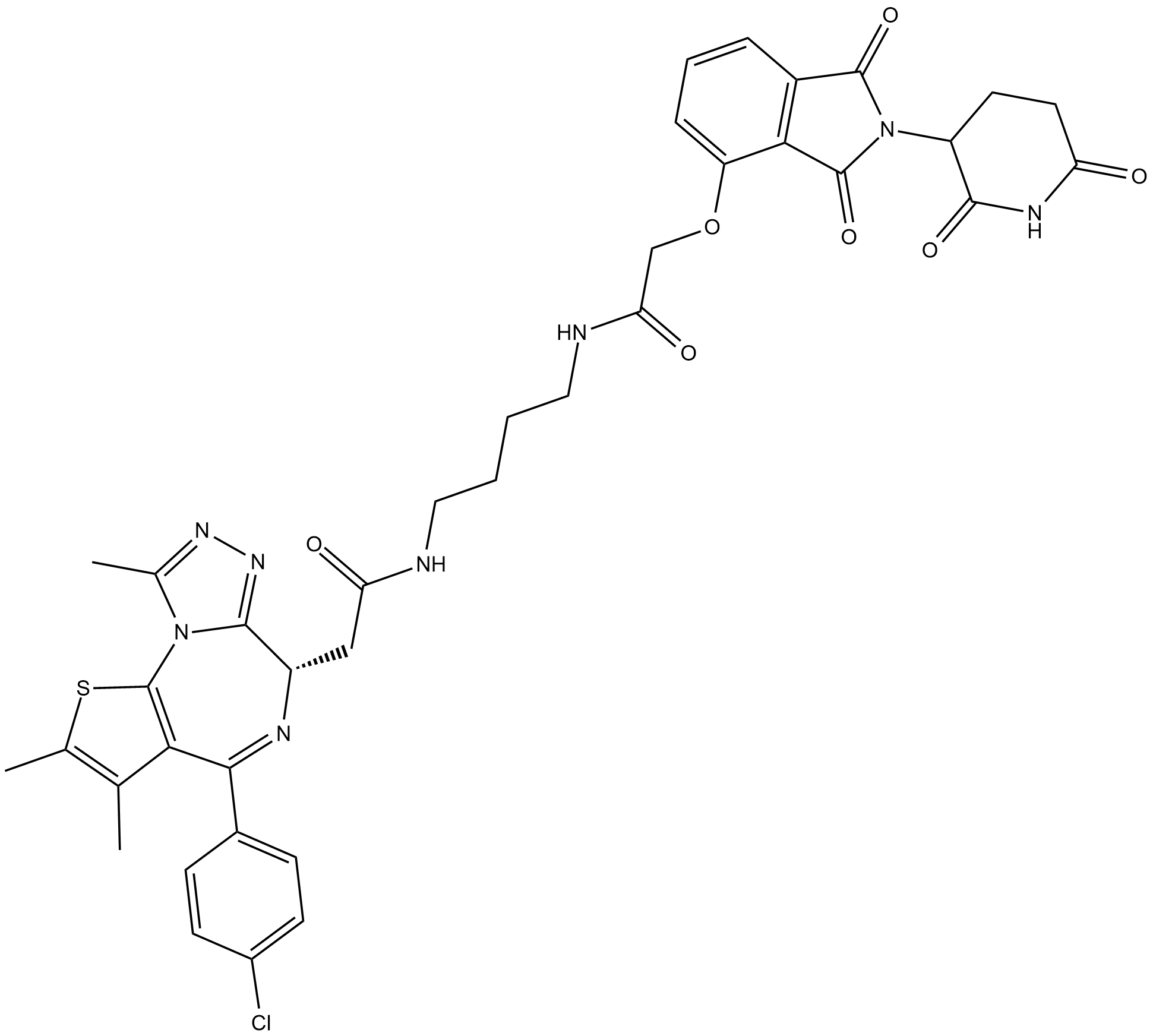
GC19119
dBET1
dBET1 ist ein PROTAC, verbunden durch Liganden fÜr Cereblon und BRD4 mit einem EC50 von 430 nM. dBET1 ist ein PROTAC, das aus (+)-JQ1 besteht, das mit einem Linker mit NSC 527179 verknÜpft ist.
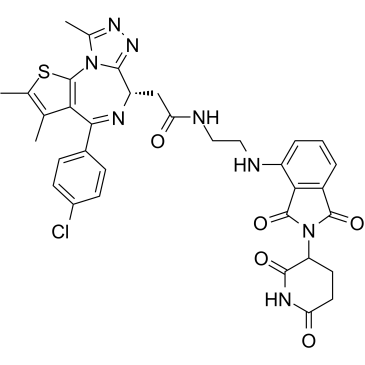
-
GC35815
dBET57
dBET57 ist ein potenter und selektiver Abbauer von BRD4BD1 auf Basis der PROTAC-Technologie. dBET57 vermittelt die Rekrutierung an die CRL4Cereblon E3-Ubiquitinligase mit einem DC50/5h von 500 nM fÜr BRD4BD1 und ist auf BRD4BD2 inaktiv.
-
GC32719
dBET6
dBET6 ist ein hochpotentes, selektives und zellgÄngiges PROTAC, das durch Liganden fÜr Cereblon und BET verbunden ist, mit einem IC50 von 14 nM und AntitumoraktivitÄt hat.
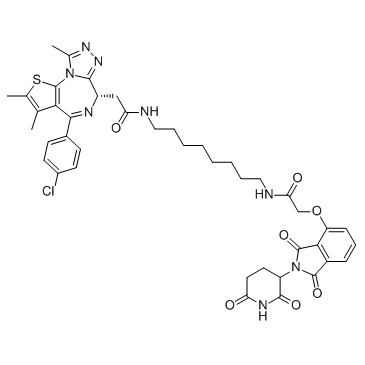
-
GC33148
DC-05
DC-05 ist ein DNA-Methyltransferase 1 (DNMT1)-Inhibitor mit einem IC50- und einem Kd-Wert von 10,3 μM bzw. 1,09 μM.
-
GC65186
DC-S239
DC-S239 ist ein selektiver Histon-Methyltransferase-SET7-Inhibitor mit einem IC50-Wert von 4,59 μM. DC-S239 zeigt auch SelektivitÄt fÜr DNMT1, DOT1L, EZH2, NSD1, SETD8 und G9a. DC-S239 hat AntikrebsaktivitÄt.
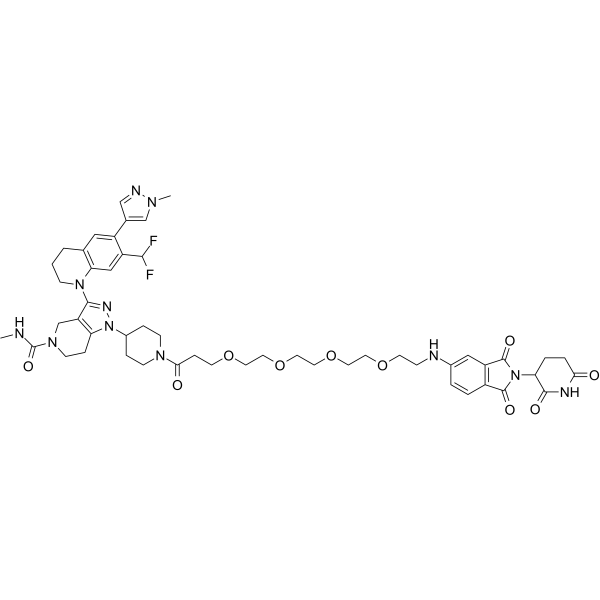
-
GC62211
dCBP-1
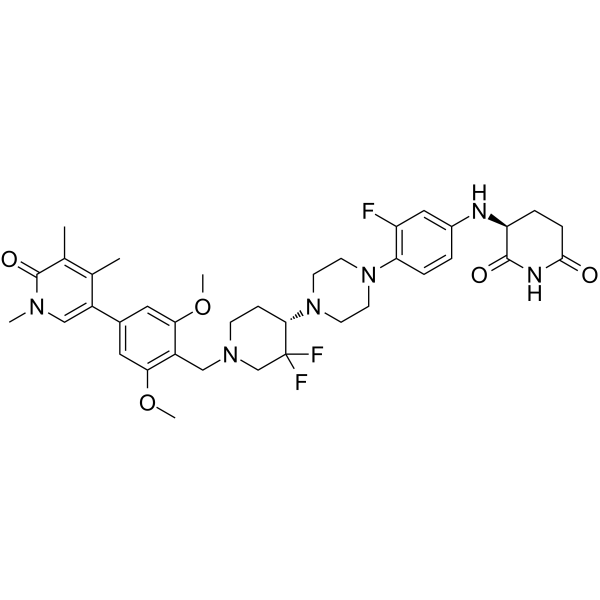
dCBP-1 ist ein potenter und selektiver heterobifunktioneller Abbauer von p300/CBP auf der Basis von Cereblon-Liganden. dCBP-1 ist außergewÖhnlich wirksam bei der AbtÖtung multipler Myelomzellen und schwÄcht die onkogene Enhancer-AktivitÄt ab, die die MYC-Expression antreibt.
-
GC63627
DCH36_06
DCH36_06 ist ein potenter und selektiver p300/CBP-Inhibitor mit IC50-Werten von 0,6 μM und 3,2 μM fÜr p300 bzw. CBP. DCH36_06-vermittelte p300/CBP-Hemmung, die zu einer Hypoacetylierung von H3K18 in LeukÄmiezellen fÜhrt. Anti-Tumor-AktivitÄt.
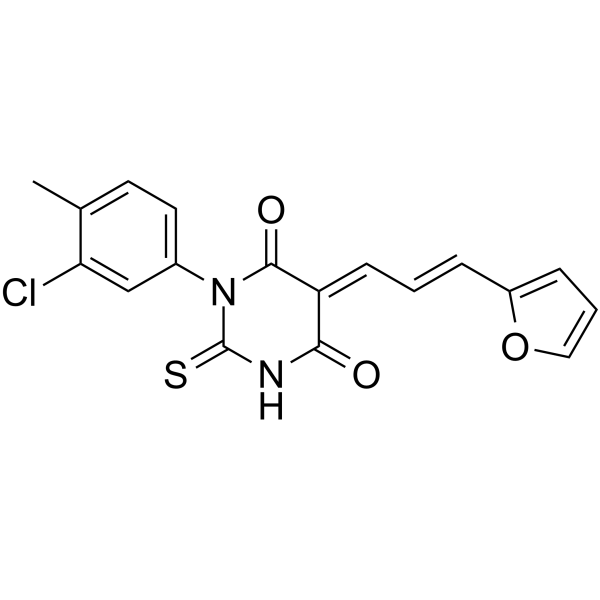
-
GC63662
DCLX069
DCLX069 ist ein selektiver Protein-Arginin-Methyltransferase-1 (PRMT1)-Inhibitor mit einem IC50-Wert von 17,9 μM. DCLX069 zeigt weniger AktivitÄt gegenÜber PRMT4 und PRMT6. DCLX069 hat Antikrebswirkungen.
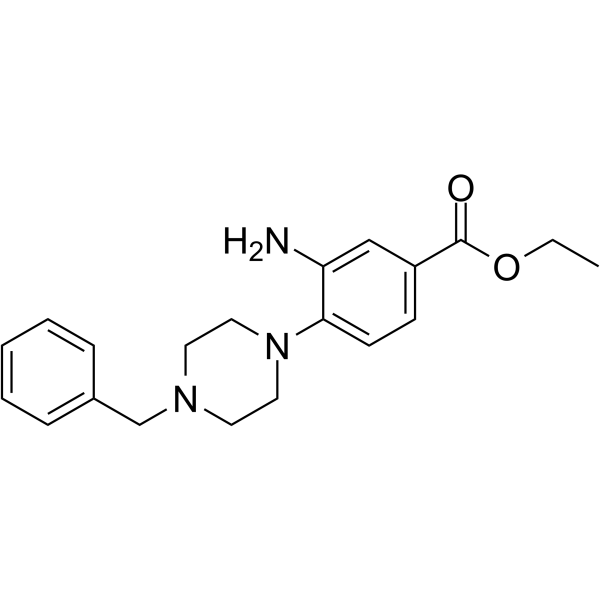
-
GC32872
DC_517
DC_517 ist ein Inhibitor der DNA-Methyltransferase 1 (DNMT1) mit einem IC50- und einem Kd-Wert von 1,7 μM bzw. 0,91 μM.
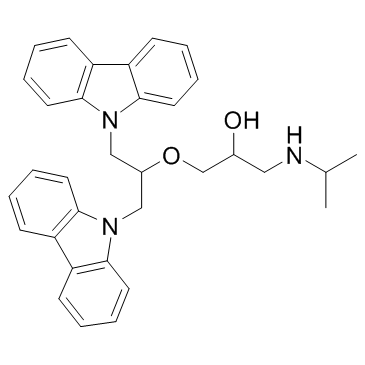
-
GC35816
DC_C66
DC_C66 ist ein zelldurchlÄssiger, selektiver Coaktivator-assoziierter Arginin-Methyltransferase-1 (CARM1)-Inhibitor mit einem IC50 von 1,8 μM. DC_C66 hat eine gute SelektivitÄt fÜr CARM1 gegenÜber PRMT1 (IC50 = 21 μM), PRMT6 (IC50 = 47 μM) und PRMT5.
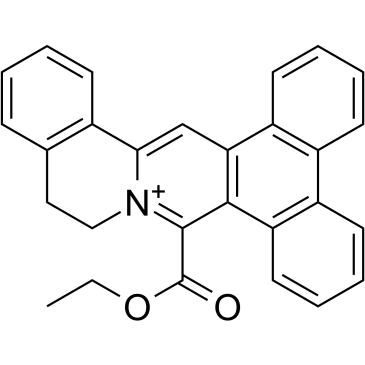
-
GC67863
DDO-2093 dihydrochloride

-
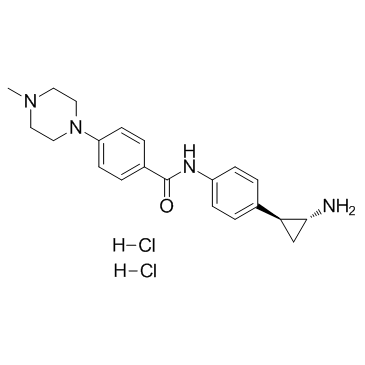
GC33013
DDP-38003 dihydrochloride
DDP-38003 Dihydrochlorid ist ein neuartiger, oral verfÜgbarer Inhibitor der Histon-Lysin-spezifischen Demethylase 1A (KDM1A/LSD1) mit einem IC50 von 84 nM.
-
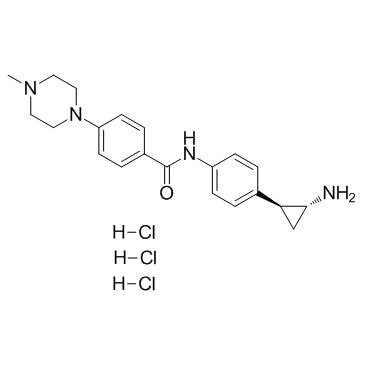
GC34296
DDP-38003 trihydrochloride
DDP-38003 Trihydrochlorid ist ein neuartiger, oral verfÜgbarer Inhibitor der Histon-Lysin-spezifischen Demethylase 1A (KDM1A/LSD1) mit einem IC50 von 84 nM.
-
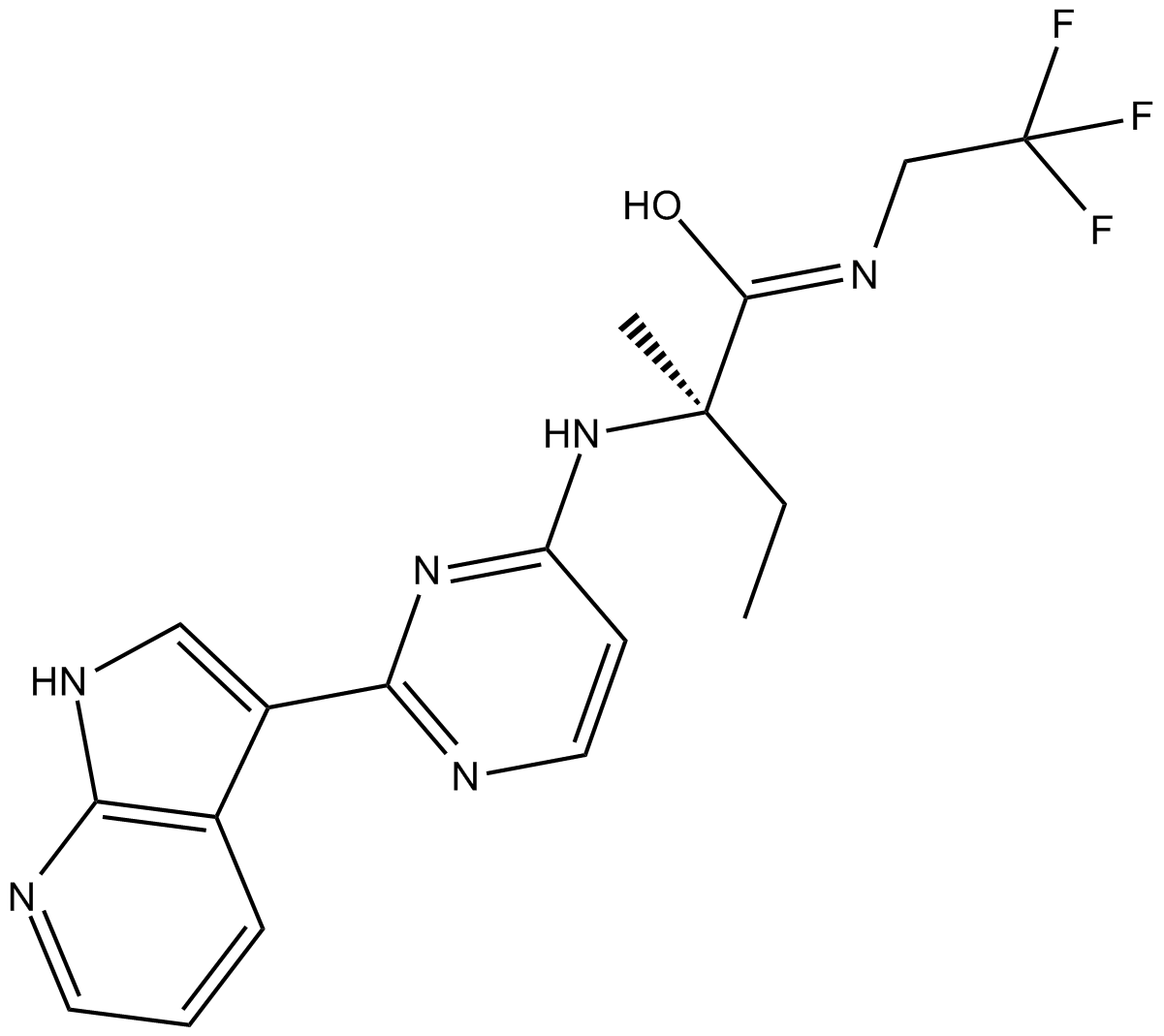
GC10770
Decernotinib(VX-509)
Decernotinib (VX-509) ist ein potenter, oral aktiver JAK3-Inhibitor mit Kis-Werten von 2,5, 11, 13 und 11 nM fÜr JAK3, JAK1, JAK2 bzw. TYK2.
-
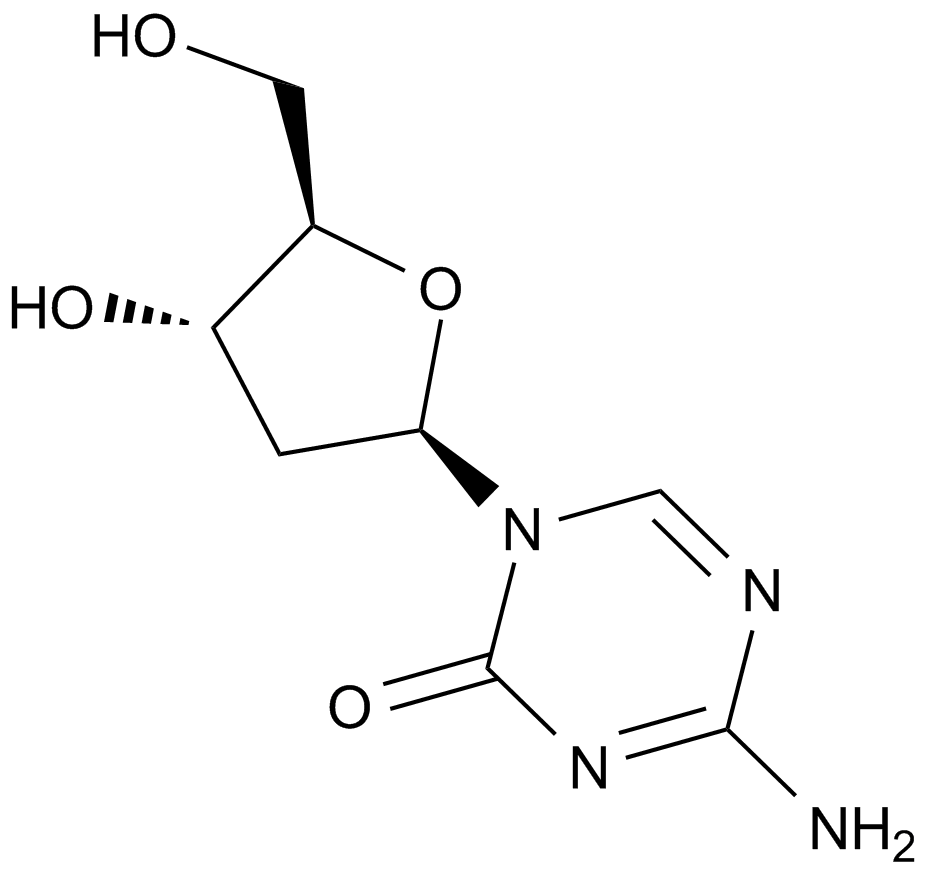
GC15255
Decitabine(NSC127716, 5AZA-CdR)
Ein 2'-Deoxy-Analogon von 5-Azacytidin.
-
GC68955
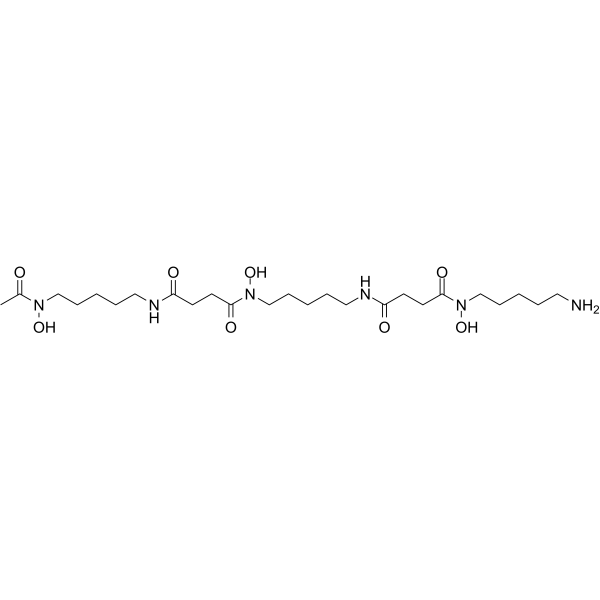
Deferoxamine
Deferoxamin (Deferoxamin B) ist ein Eisenchelator (bindet Fe(III) und viele andere Metallkationen), der weit verbreitet zur Reduzierung von Eisenablagerungen in Geweben eingesetzt wird. Deferoxamin hat eine gute antioxidative Aktivität und kann den HIF-1α -Spiegel erhöhen. Deferoxamin hat auch antiproliferative Aktivität, indem es Krebszellen zum Absterben und Autophagie induziert. Deferoxamin kann für die Erforschung von Diabetes, neurodegenerativen Erkrankungen sowie Anti-Krebs- und Anti-COVID-19 verwendet werden.
-
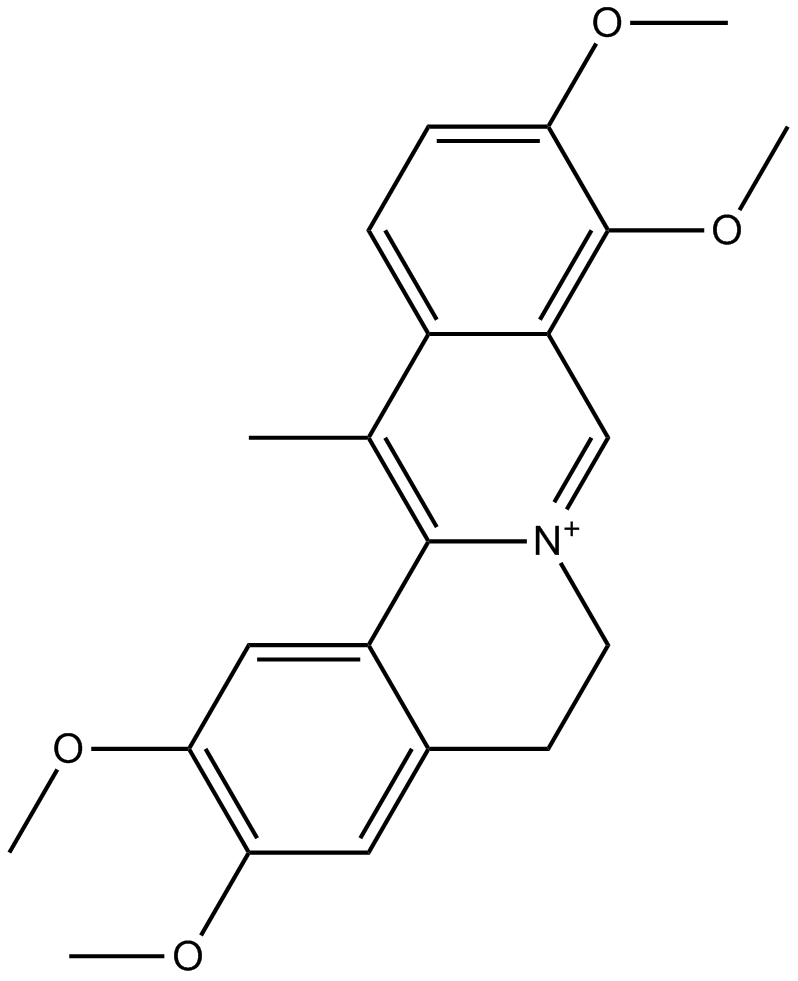
GN10040
Dehydrocorydaline
-
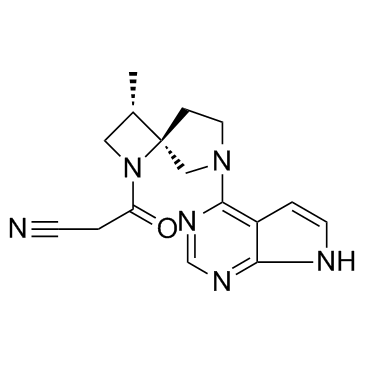
GC31660
Delgocitinib (JTE-052)
Delgocitinib (JTE-052) (JTE-052) ist ein spezifischer JAK-Inhibitor mit IC50-Werten von 2,8, 2,6, 13 und 58 nM fÜr JAK1, JAK2, JAK3 bzw. Tyk2.
-
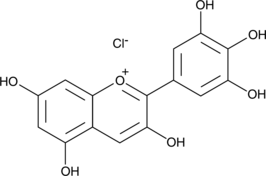
GC43406
Delphinidin (chloride)
Delphinidin (Chlorid), ein Anthocyanidin, wird aus Beeren und Rotwein isoliert.
-
GC31230
Dencichin (Dencichine)
Dencichin ist eine Nicht-Protein-AminosÄure, die ursprÜnglich aus Panax Notoginseng extrahiert wurde und die AktivitÄt der HIF-Prolylhydroxylase-2 (PHD-2) hemmen kann.
-
GC38767
Deoxyshikonin
Deoxyshikonin wird aus Lithospermum erythrorhizon Sieb mit AntitumoraktivitÄt isoliert.
-
GC32449
Desidustat
Desidustat ist ein oral wirksamer HIF-Hydroxylase-Hemmer.
-
GC34187
Dihydrocoumarin (Hydrocoumarin)
Dihydrocumarin (Hydrocumarin) ist eine Verbindung, die in Melilotus officinalis vorkommt. Dihydrocumarin (Hydrocumarin) ist ein Hefe-Sir2p-Inhibitor. Dihydrocumarin (Hydrocumarin) hemmt auch menschliches SIRT1 und SIRT2 mit IC50-Werten von 208 μM bzw. 295 μM.
-
GC14024
DL-AP3
A metabotropic glutamate receptor antagonist
-
GC52368
DL-Sulforaphane Glutathione
A metabolite of sulforaphane
-
GC65546
DNMT3A-IN-1
DNMT3A-IN-1 ist ein potenter und selektiver DNMT3A-Inhibitor.
-
GC35893
Domatinostat
Domatinostat (freie Base von 4SC-202) ist ein selektiver HDAC-Inhibitor der Klasse I mit IC50-Werten von 1,20 μM, 1,12 μM und 0,57 μM fÜr HDAC1, HDAC2 bzw. HDAC3. Es zeigt auch eine hemmende AktivitÄt gegen Lysin-spezifische Demethylase 1 (LSD1).
-
GC14490
Donepezil HCl
Donepezil HCl (E2020) ist ein reversibler, selektiver AChE-Hemmer mit einer IC50 von 6,7 nM fÜr die AChE-AktivitÄt.
-
GC33208
Dot1L-IN-1
Dot1L-IN-1 ist ein hochwirksamer, selektiver und strukturell neuer Dot1L-Inhibitor mit einem Ki von 2 pM.