Epigenetic Reader Domain
Epigenetic regulators of gene expression and chromatin state include so-called writers, erasers, and readers of chromatin modifications.Well-characterized examples of reader domains include bromodomains typically binding acetyllysine and chromatin organization modifier (chromo), malignant brain tumor (MBT), plant homeodomain (PHD), and Tudor domains generally associating with methyllysine. Research on epigenetic readers has been tremendously influenced by the discovery of selective inhibitors targeting the bromodomain and extraterminal motif (BET) family of acetyl-lysine readers. The human genome encodes 46 proteins containing 61 bromodomains clustered into eight families. Distinct experimental approaches are used to identify the first BET inhibitors, GSK 525762A and (+)-JQ-1.
The Polycomb group (PcG) protein, enhancer of zeste homologue 2 (EZH2), has an essential role in promoting histone H3 lysine 27 trimethylation (H3K27me3) and epigenetic gene silencing. This function of EZH2 is important for cell proliferation and inhibition of cell differentiation, and is implicated in cancer progression. Cyclin-dependent kinases regulate epigenetic gene silencing through phosphorylation of EZH2. In many types of cancers including lymphomas and leukemia, EZH2 is postulated to exert its oncogenic effects via aberrant histone and DNA methylation, causing silencing of tumor suppressor genes.
p300/CBP is not only a transcriptional adaptor but also a histone acetyltransferase.
Ziele für Epigenetic Reader Domain
Produkte für Epigenetic Reader Domain
- Bestell-Nr. Artikelname Informationen
-
GC61595
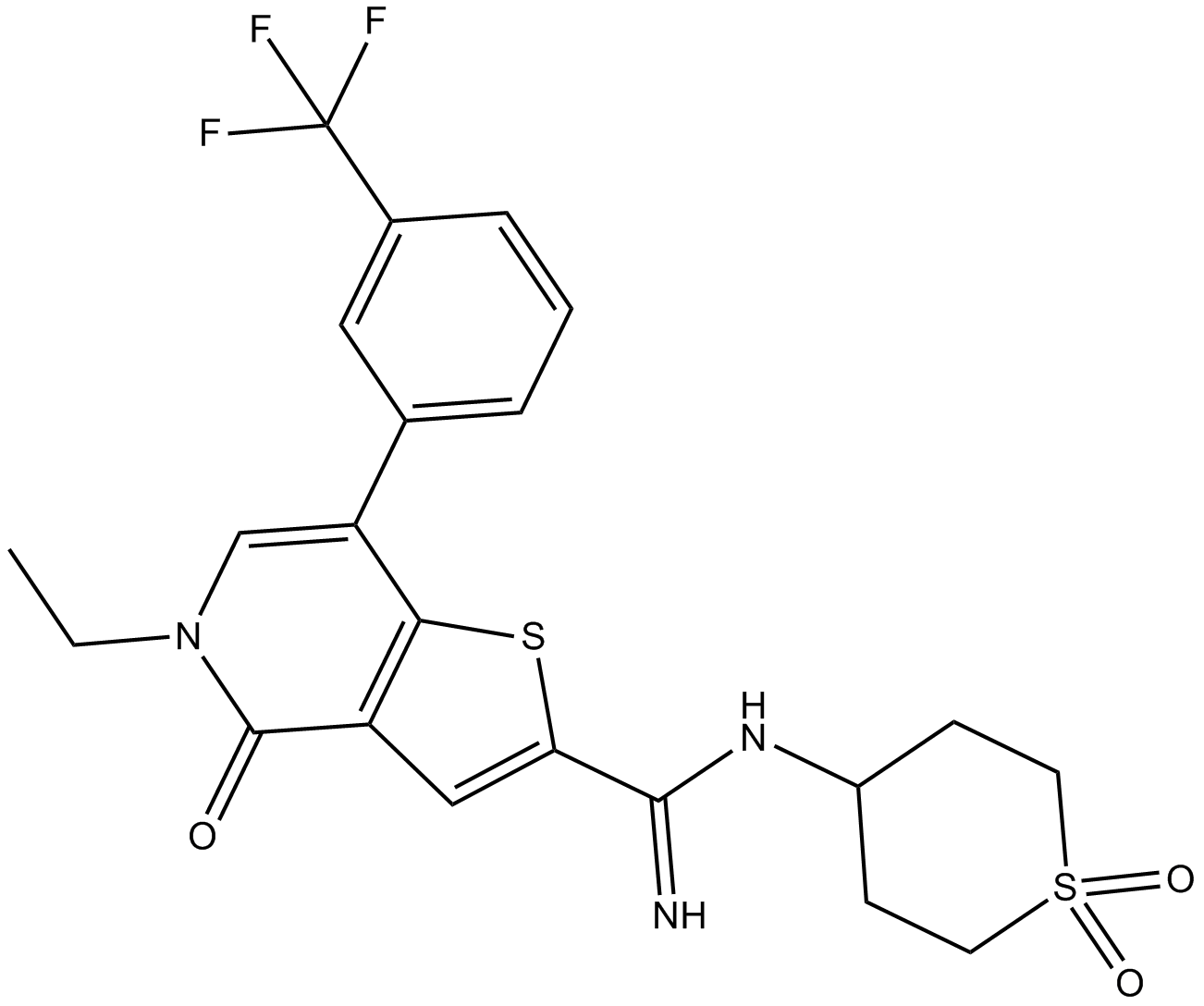
(+)-JQ-1-aldehyde
(+)-JQ-1-Aldehyd ist die Aldehydform von (+)-JQ1. (+)-JQ-1-Aldehyd kann als Vorstufe zur Synthese von PROTACs verwendet werden, die auf BET-BromodomÄnen abzielen.
-
GC34958
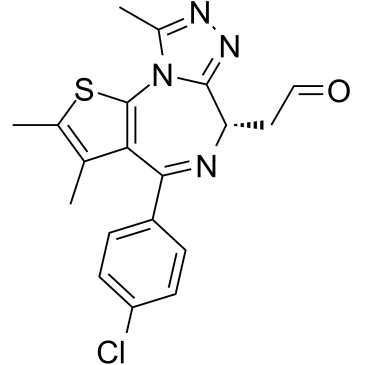
(+)-JQ1 PA
(+)-JQ1 PA ist ein Derivat der BromodomÄne und des extraterminalen (BET) Inhibitors JQ1 mit einem IC50 von 10,4 nM.
-
GC13822
(-)-JQ1
A selective inhibitor of BET bromodomains
-
GC63791
(R)-(-)-JQ1 Enantiomer
(R)-(-)-JQ1 Enantiomer ist das Stereoisomer von (+)-JQ1. (+)-JQ1 verringert stark die Expression beider BRD4-Zielgene, wÄhrend (R)-(-)-JQ1-Enantiomer keine Wirkung hat.
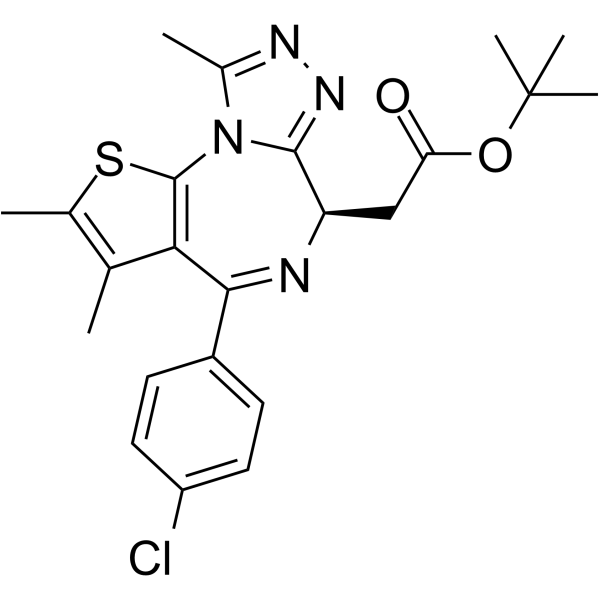
-
GC34443
(R)-BAY1238097
(R)-BAY1238097 ist das R-Isomer mit geringerer AktivitÄt von BAY1238097. BAY1238097 ist ein potenter und selektiver Inhibitor der BET-Bindung an Histone und hat eine starke antiproliferative AktivitÄt in verschiedenen AML- (akute myeloische LeukÄmie) und MM- (multiples Myelom) Modellen durch Herunterregulierung der c-Myc-Spiegel und seines nachgeschalteten Transkriptoms.
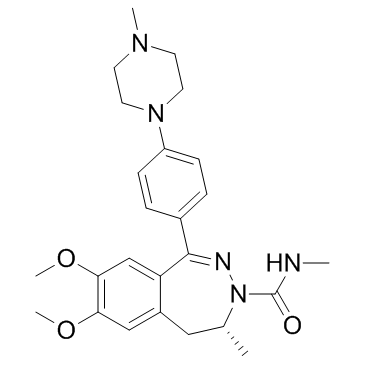
-
GC34446
(rac)-BAY1238097
(Rac)-BAY1238097 ist ein BET-Inhibitor mit einem IC50-Wert von 1,02 μM fÜr BRD4. Wird in der Krebsforschung verwendet.
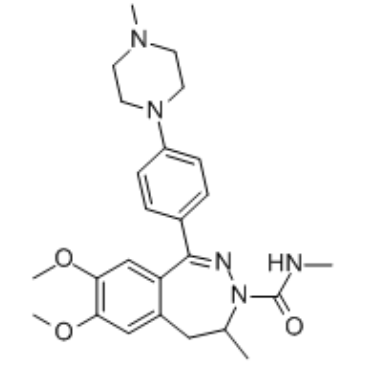
-
GC33198
(S)-JQ-35 (TEN-010)
(S)-JQ-35 (TEN-010) (TEN-010) ist ein Inhibitor der BromodomÄne und der Familie der extraterminalen (BET) BromodomÄne enthaltenden Proteine mit potenzieller antineoplastischer AktivitÄt.
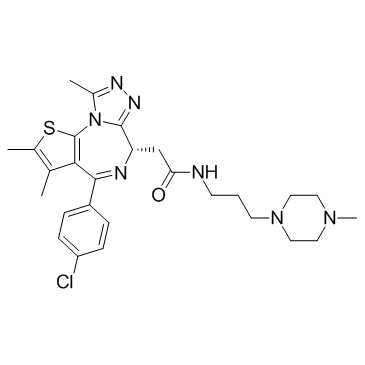
-
GC35175
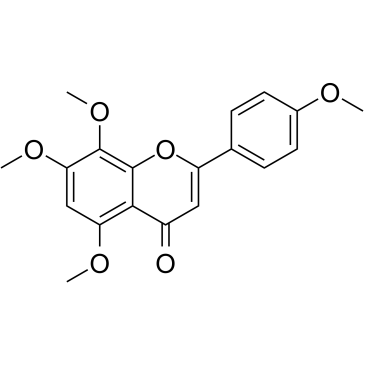
6-Demethoxytangeretin
6-Demethoxytangeretin ist ein aus Citrus depressa isoliertes Zitrusflavonoid.
-
GC62279
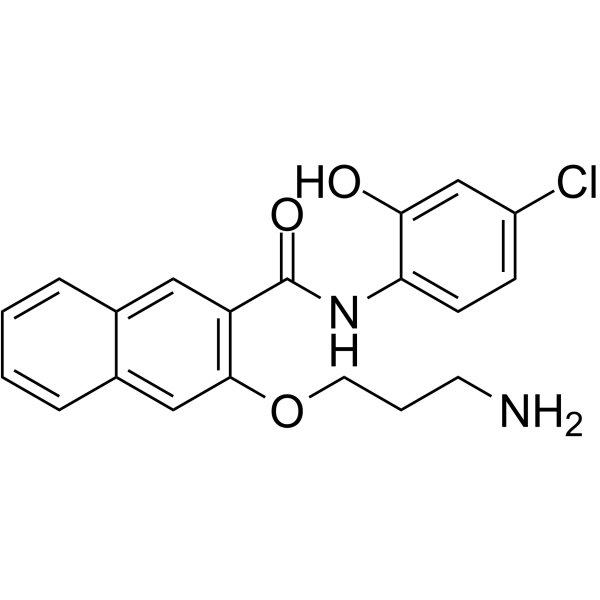
653-47
653-47, ein Potenzierer, potenziert signifikant die cAMP-Response-Element-Bindungsprotein (CREB)-InhibitoraktivitÄt von 666-15. 653-47 ist ebenfalls ein sehr schwacher CREB-Inhibitor mit IC50 von 26,3 μM.
-
GC62144
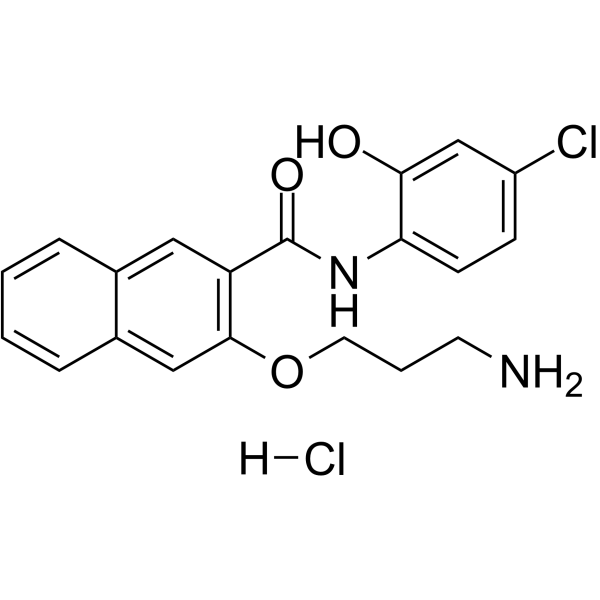
653-47 hydrochloride
653-47-Hydrochlorid, ein Potentiator, potenziert die HemmaktivitÄt des cAMP-Response-Element-Bindungsproteins (CREB) von 666-15 signifikant. 653-47 Hydrochlorid ist auch ein sehr schwacher CREB-Inhibitor mit IC50 von 26,3 μM.
-
GC32689
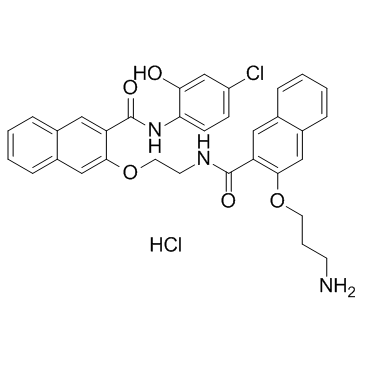
666-15
666-15 ist ein potenter und selektiver CREB-Inhibitor mit einem IC50 von 81 nM. 666-15 unterdrÜckt das Tumorwachstum in einem Brustkrebs-Xenotransplantatmodell.
-
GC32677
A-485
A-485 ist ein potenter und selektiver katalytischer Inhibitor von p300/CBP mit IC50-Werten von 9,8nM und 2,6nM fÜr p300 bzw. CBP-Histonacetyltransferase (HAT).
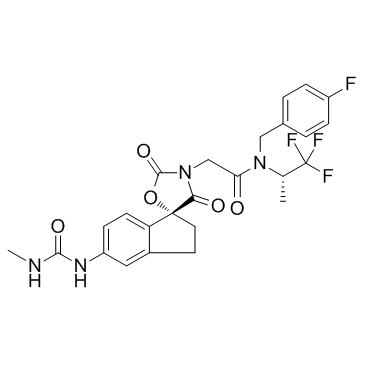
-
GC33280
A1874
A1874 ist ein Nutlin-basiertes (MDM2-Ligand) und BRD4-abbauendes PROTAC mit einem DC50 von 32 nM (induziert BRD4-Abbau in Zellen). Wirksam bei der Hemmung der Proliferation vieler Krebszelllinien.
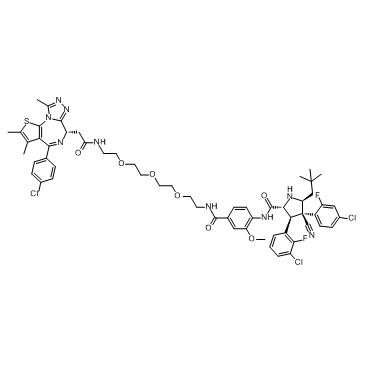
-
GC19409
ABBV-744
ABBV-744 ist ein erstklassiger, oral aktiver und selektiver Inhibitor der BDII-DomÄne von Proteinen der BET-Familie mit IC50-Werten im Bereich von 4 bis 18 nM fÜr BRD2, BRD3, BRD4 und BRDT.
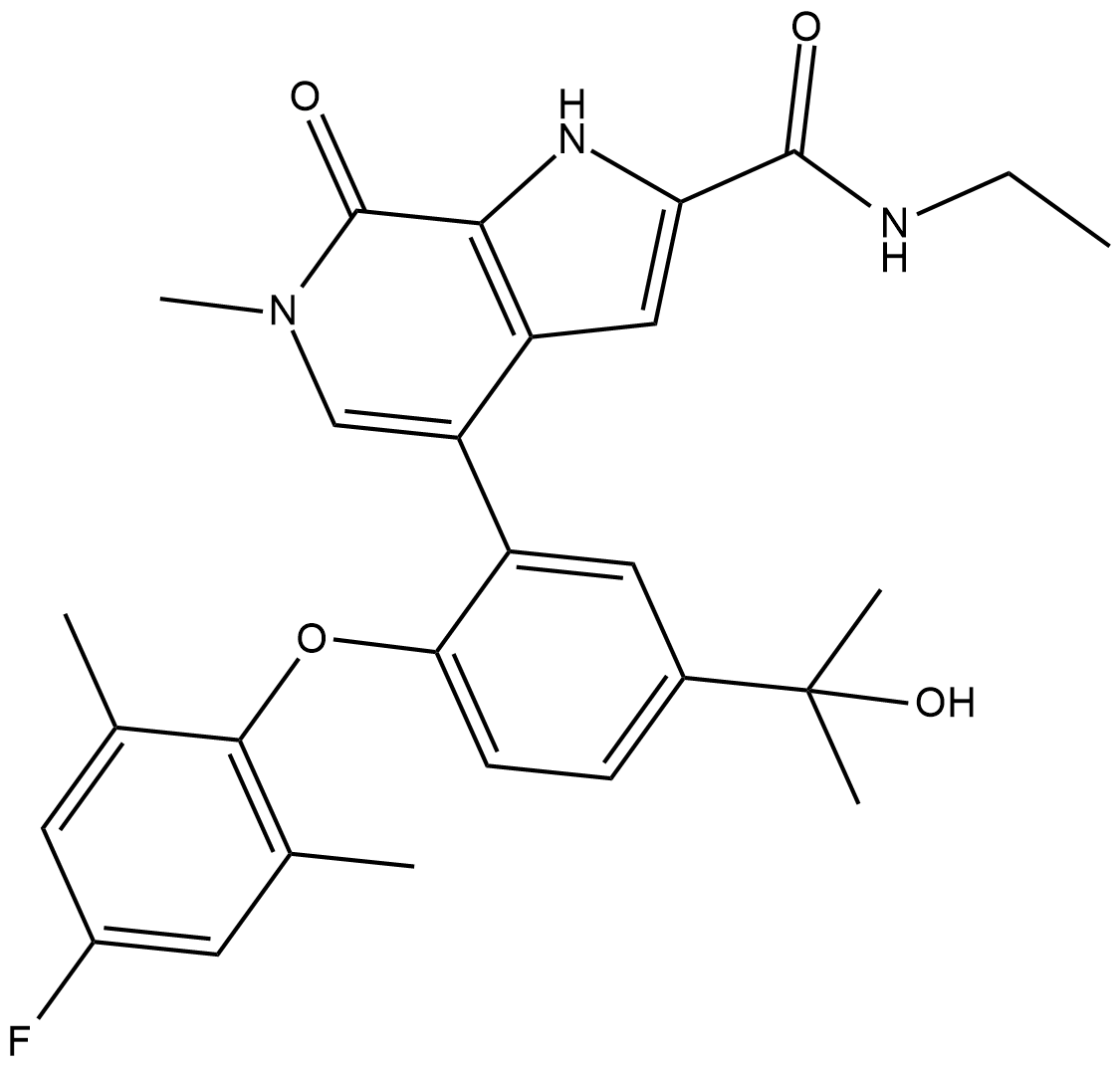
-
GC35227
ACBI1
ACBI1 ist ein potenter und kooperativer SMARCA2-, SMARCA4- und PBRM1-Abbaustoff mit DC50-Werten von 6, 11 bzw. 32 nM. ACBI1 ist ein PROTAC-Degrader. ACBI1 zeigt antiproliferative AktivitÄt. ACBI1 induziert Apoptose.
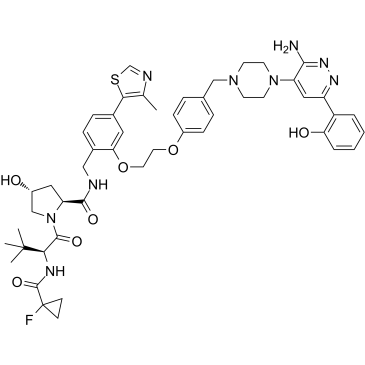
-
GC35297
Alobresib
Alobresib (GS-5829) ist ein BET-BromodomÄnen-Inhibitor, der ein hochwirksames Therapeutikum gegen rezidivierendes/chemotherapieresistentes serÖses Uteruskarzinom (USC) darstellt, das c-Myc Überexprimiert.
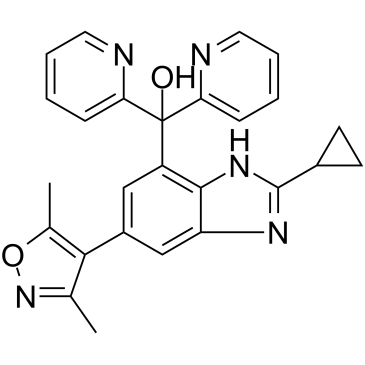
-
GC67912
Amredobresib

-
GC17284
Anacardic acid
A histone acetyltransferase inhibitor
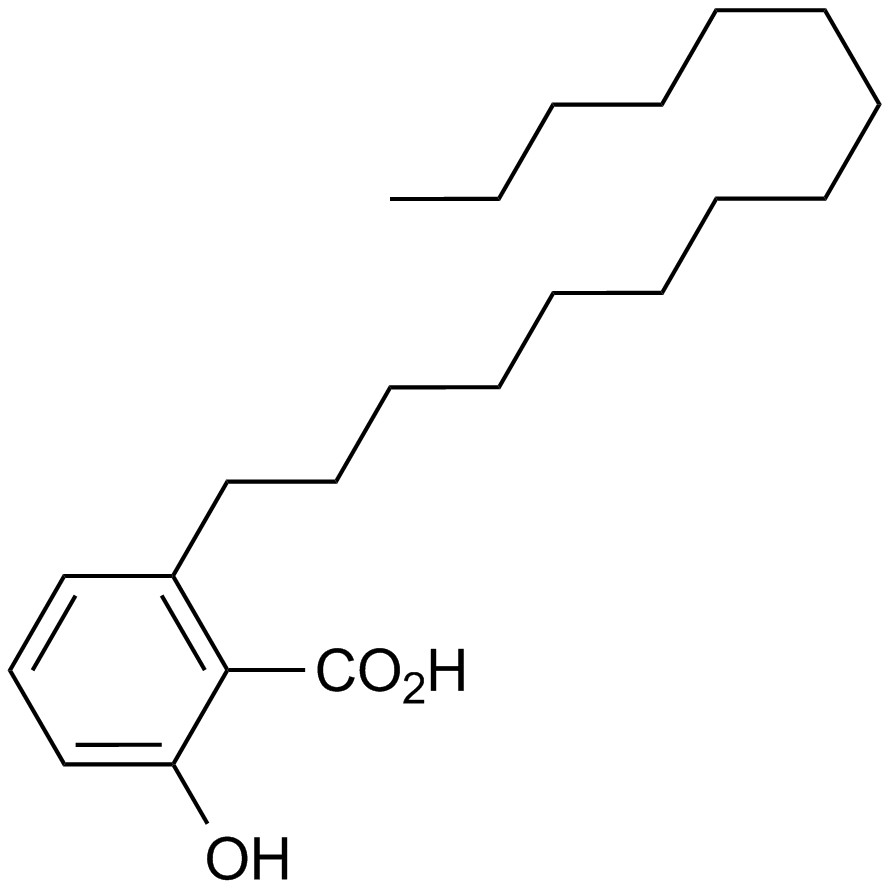
-
GC32685
ARV-771
ARV-771 ist ein potenter BET-PROTAC basierend auf E3-Ligase von Hippel-Lindau mit Kds von 34 nM, 4,7 nM, 8,3 nM, 7,6 nM, 9,6 nM und 7,6 nM fÜr BRD2(1), BRD2(2), BRD3( 1), BRD3(2), BRD4(1) bzw. BRD4(2).
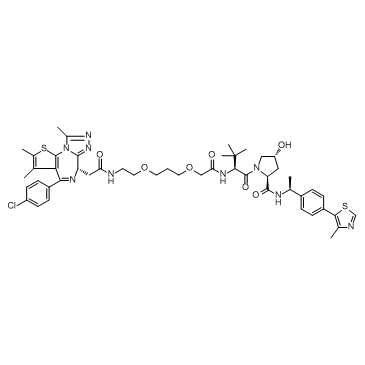
-
GC19038
ARV-825
ARV-825 ist ein PROTAC, das durch Liganden fÜr Cereblon und BRD4 verbunden ist. ARV-825 bindet an BD1 und BD2 von BRD4 mit Kds von 90 bzw. 28 nM.
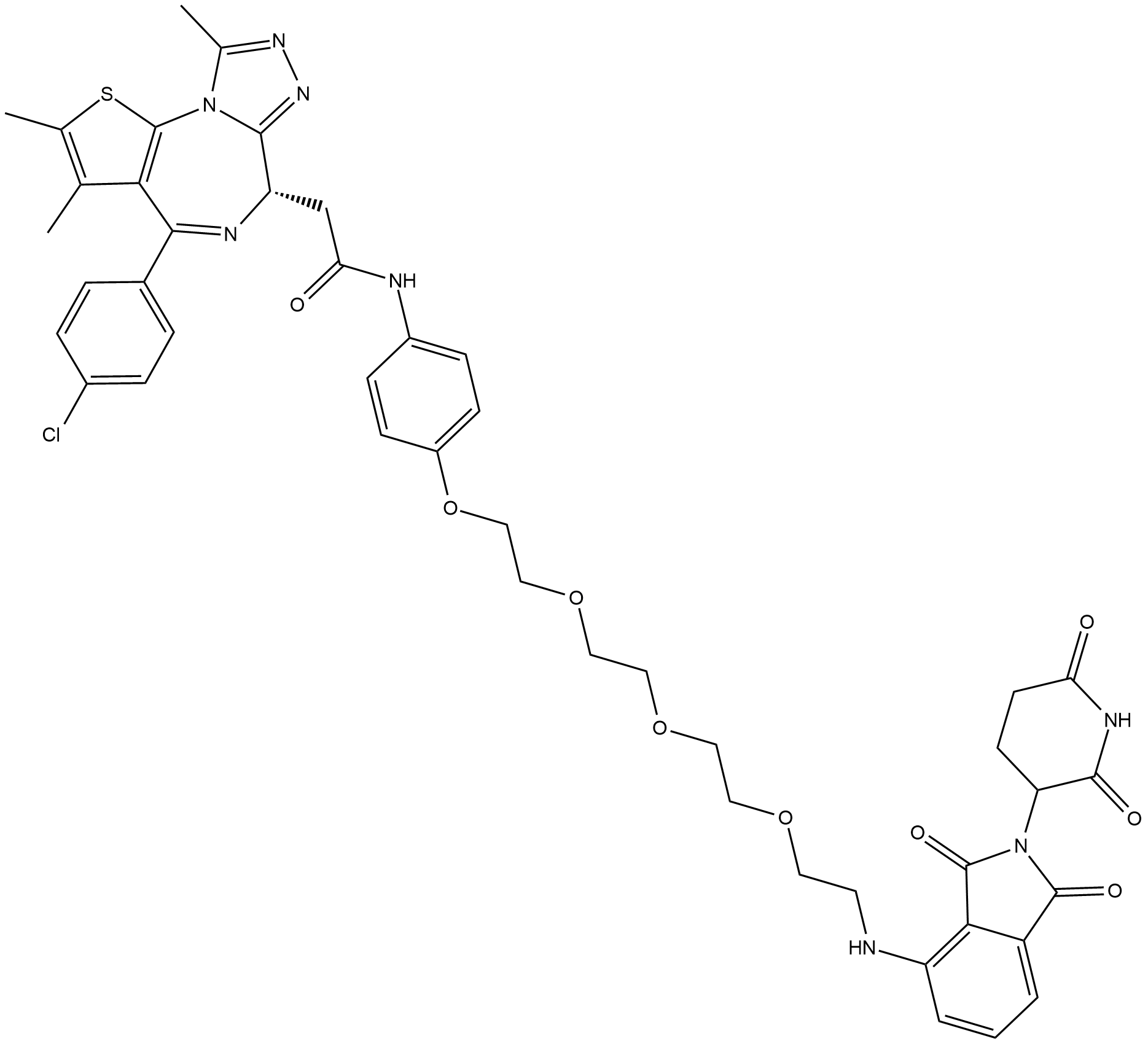
-
GC64271
AU-15330
AU-15330 ist ein Proteolyse-Targeting-ChimÄre (PROTAC)-Degrader der SWI/SNF-ATPase-Untereinheiten, SMARCA2 und SMARCA4. AU-15330 induziert eine starke Hemmung des Tumorwachstums in Xenograft-Modellen von Prostatakrebs und wirkt synergetisch mit dem AR-Antagonisten Enzalutamid. AU-15330 induziert eine Krankheitsremission in Modellen mit kastrationsresistentem Prostatakrebs (CRPC) ohne ToxizitÄt.
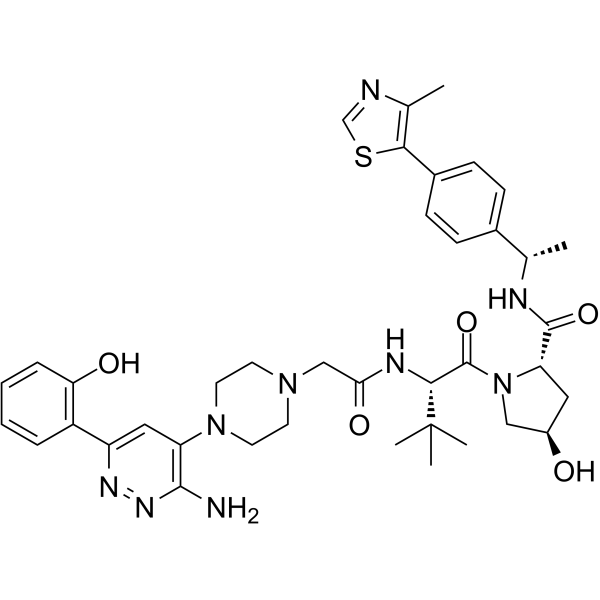
-
GC35448
AZD5153 6-Hydroxy-2-naphthoic acid
AZD5153 6-Hydroxy-2-naphthoesÄure ist die 6-Hydroxy-2-naphthoesÄure von AZD5153. AZD5153 ist ein potenter, selektiver und oral verfÜgbarer BET/BRD4-BromodomÄnen-Inhibitor; stÖrt BRD4 mit einem IC50 von 1,7 nM.
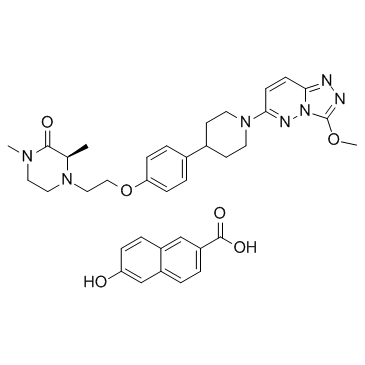
-
GC18508
BAY-299
BAY-299 ist ein sehr potenter dualer Inhibitor mit IC50-Werten von 67 nM fÜr BRPF2-BromodomÄnen (BD), 8 nM fÜr TAF1 BD2 und 106 nM fÜr TAF1L BD2.
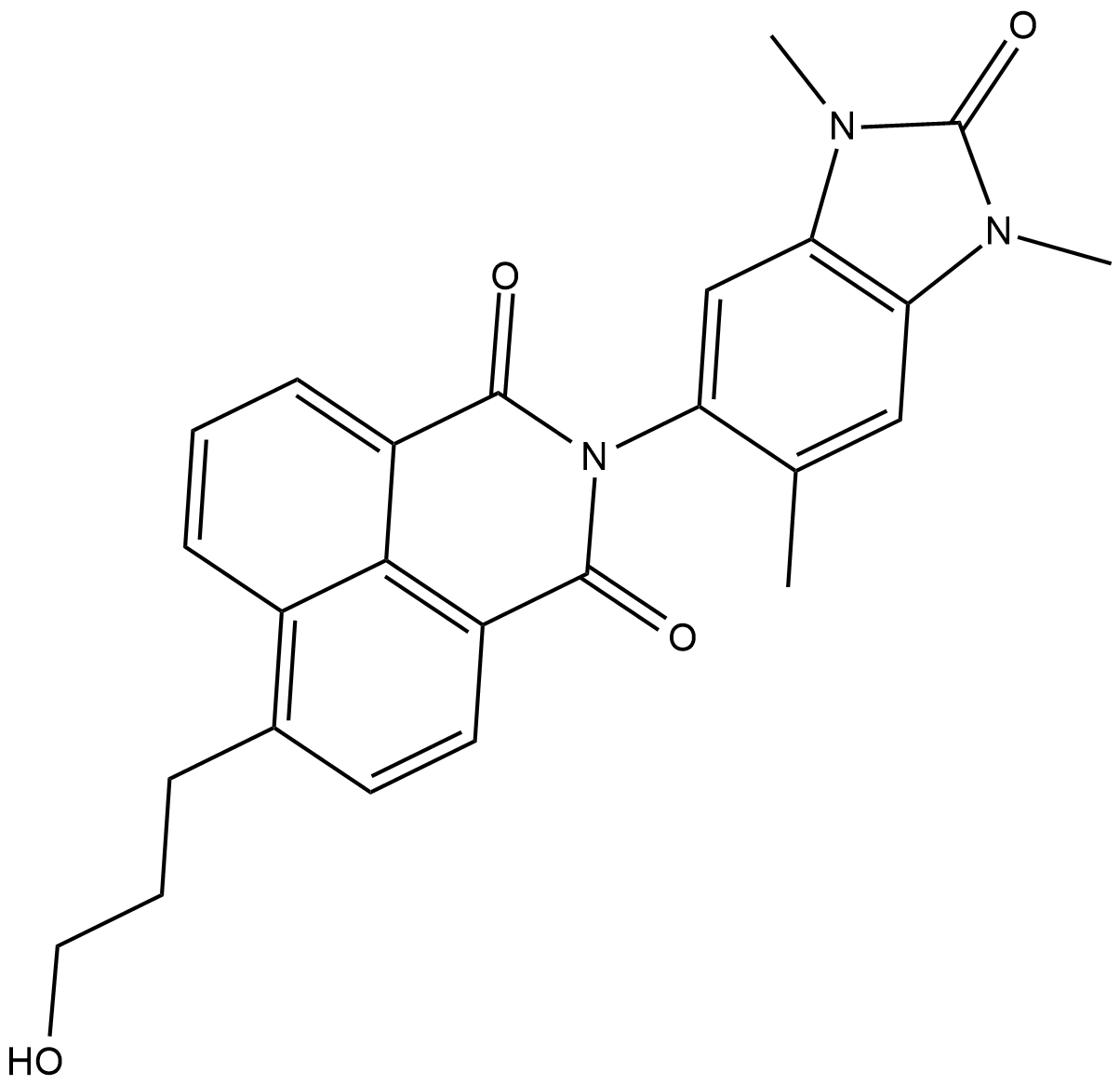
-
GC33104
BAY1238097
BAY1238097 ist ein potenter und selektiver Inhibitor der BET-Bindung an Histone und hat eine starke antiproliferative AktivitÄt in verschiedenen AML- (akute myeloische LeukÄmie) und MM- (multiples Myelom) Modellen durch Herunterregulierung der c-Myc-Spiegel und seines nachgeschalteten Transkriptoms (IC50 < 100 nM).
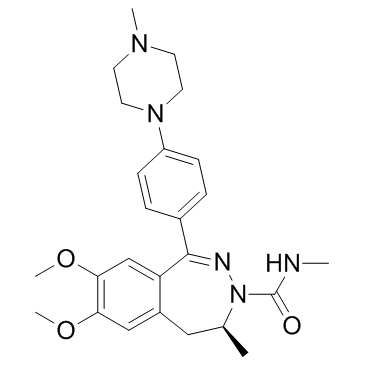
-
GC63694
BAZ1A-IN-1
BAZ1A-IN-1 ist ein potenter Inhibitor von BAZ1A (bromodomÄnenhaltiges Protein). BAZ1A-IN-1 zeigt einen KD-Wert von 0,52 μM gegen die BAZ1A-BromodomÄne. BAZ1A-IN-1 zeigt eine gute Anti-LebensfÄhigkeitsaktivitÄt gegen Krebszelllinien, die einen hohen BAZ1A-Spiegel exprimieren, aber eine schwache oder keine AktivitÄt gegen Krebszellen mit einem niedrigen BAZ1A-Expressionsspiegel.
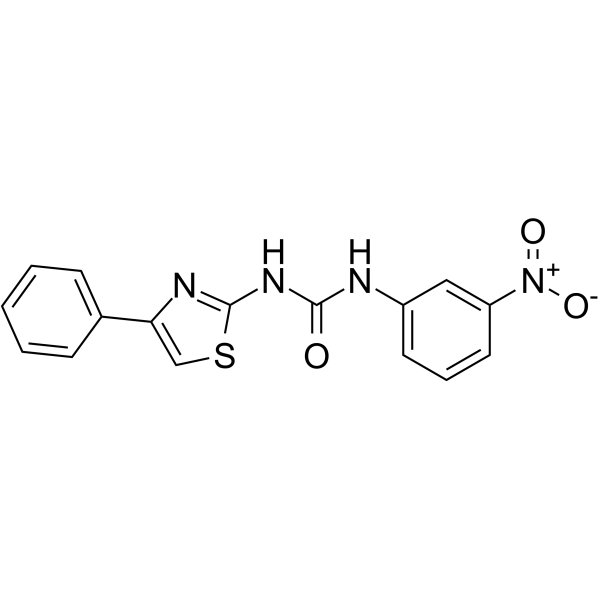
-
GC15175
BAZ2-ICR
BAZ2-ICR ist ein potenter, selektiver, zellaktiver und oral aktiver BAZ2A/B-BromodomÄnen-Inhibitor mit IC50s von 130 nM und 180 nM und Kds von 109 nM bzw. 170 nM. BAZ2-ICR zeigt eine 10-15-fache SelektivitÄt fÜr die Bindung von BAZ2A/B gegenÜber CECR2 und eine >100-fache SelektivitÄt gegenÜber allen anderen BromodomÄnen. BAZ2-ICR ist eine epigenetische chemische Sonde.
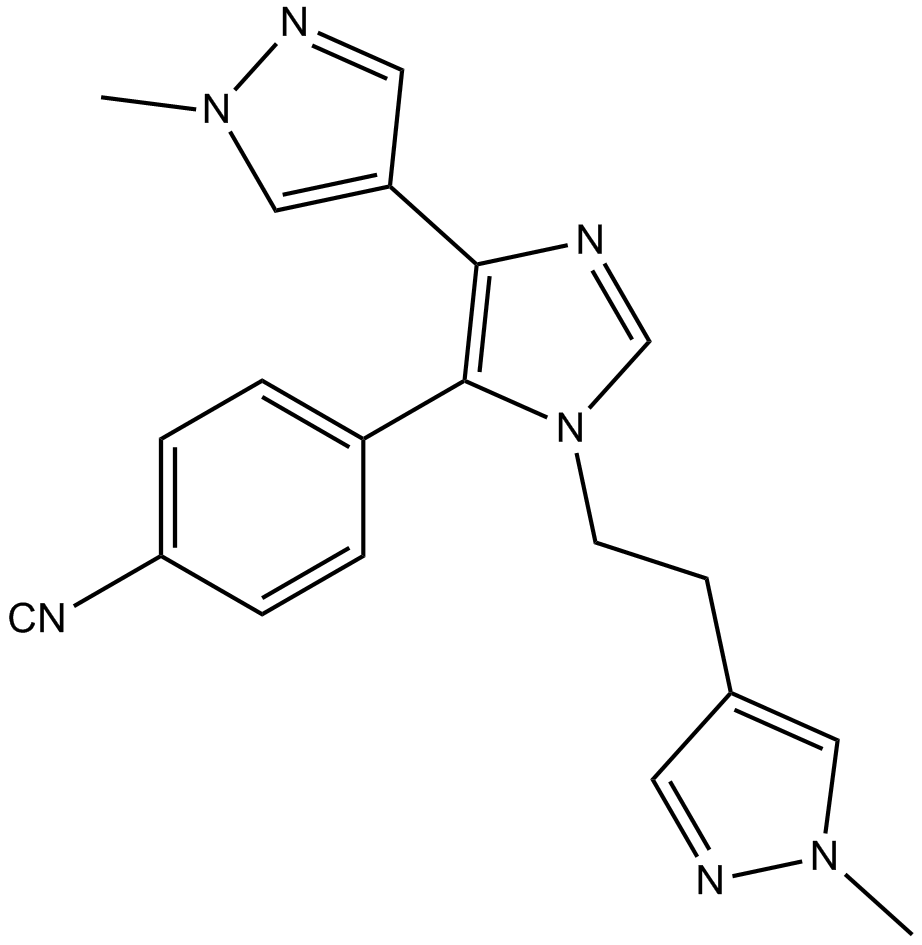
-
GC16299
BET bromodomain inhibitor
BET-BromdomÄnen-Inhibitor ist ein potenter BET-Inhibitor, extrahiert aus Patent WO/2015/153871A2, Verbindungsbeispiel 11.
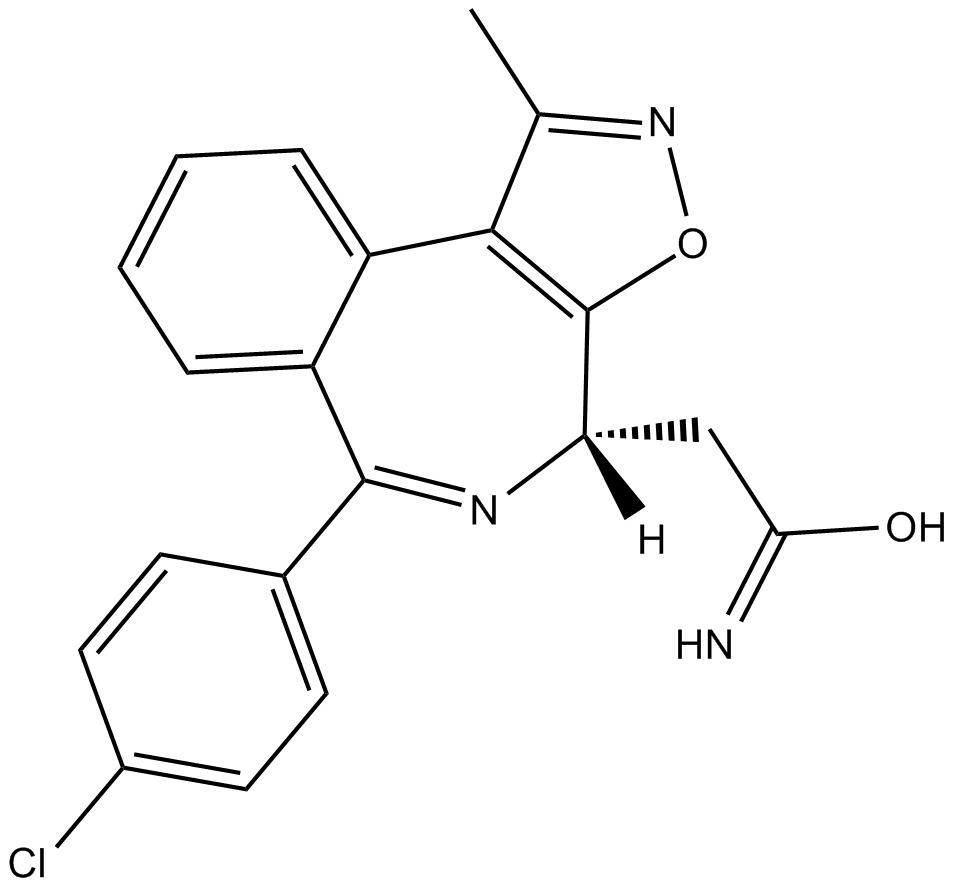
-
GC63528
BET bromodomain inhibitor 1
BET-BromodomÄnen-Inhibitor 1 ist ein oral aktiver, selektiver BromodomÄnen- und extraterminaler (BET) BromodomÄnen-Inhibitor mit einem IC50-Wert von 2,6 nM fÜr BRD4. BET-BromodomÄnen-Inhibitor 1 bindet an BRD2(2), BRD3(2), BRD4(1), BRD4(2) und BRDT(2) mit hohen AffinitÄten (Kd-Werte von 1,3 nM, 1,0 nM, 3,0 nM, 1,6 nM, 2,1 nM). BromodomÄnen-Inhibitor 1 hat Anti-Krebs-AktivitÄt.
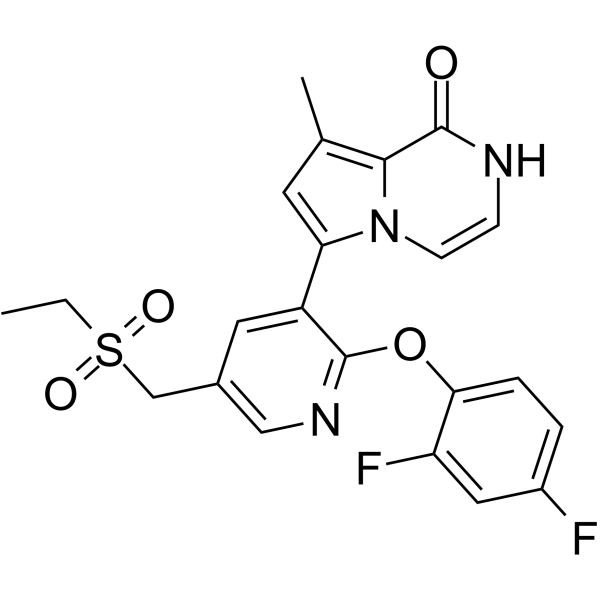
-
GC35505
BET-BAY 002
BET-BAY 002 ist ein starker BET-Hemmer; zeigt die Wirksamkeit in einem multiplen Myelommodell.
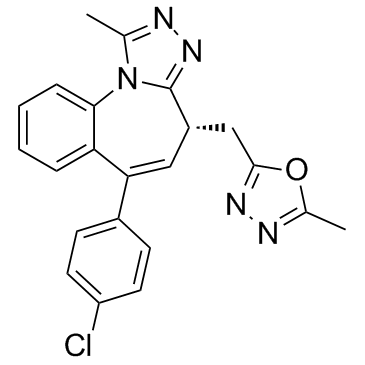
-
GC35506
BET-BAY 002 S enantiomer
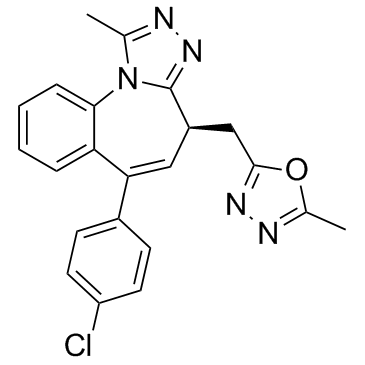
-
GC31831
BET-IN-1
BET-IN-1 ist ein Pan-Inhibitor aller acht BET-BromodomÄnen und selektiv gegenÜber anderen reprÄsentativen BromodomÄnen-enthaltenden Proteinen.
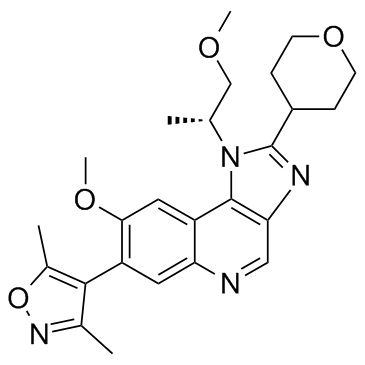
-
GC65506
BETd-246
BETd-246 ist ein PROTAC-basierter BET-BromdomÄnen(BRD)-Inhibitor der zweiten Generation, der durch Liganden fÜr Cereblon und BET verbunden ist und eine Überlegene SelektivitÄt, Wirksamkeit und AntitumoraktivitÄt aufweist.
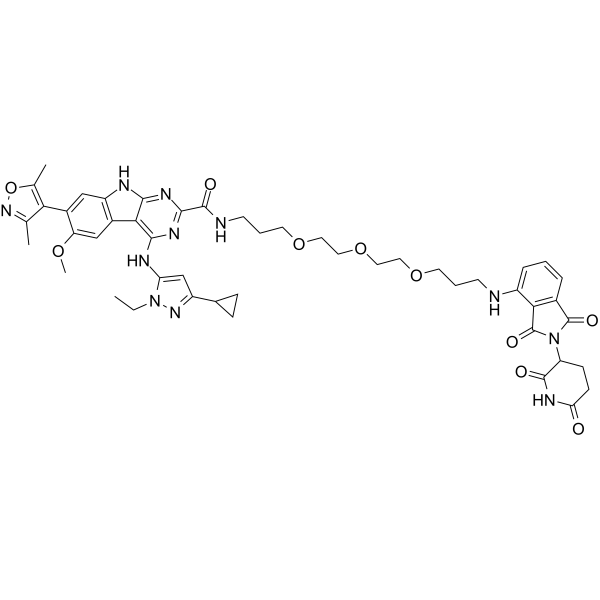
-
GC32791
BETd-260 (ZBC 260)
BETd-260 (ZBC 260) (ZBC 260) ist ein PROTAC, das durch Liganden fÜr Cereblon und BET verbunden ist, mit nur 30 pM gegen BRD4-Protein in der RS4;11-LeukÄmie-Zelllinie. BETd-260 (ZBC 260) unterdrÜckt wirksam die ZelllebensfÄhigkeit und induziert robust Apoptose in hepatozellulÄren Karzinomzellen (HCC).
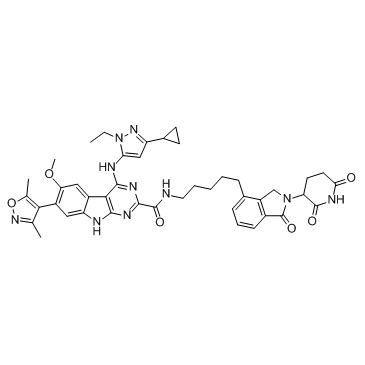
-
GC12450
BI 2536
Ein potenter Hemmstoff von Plk1.
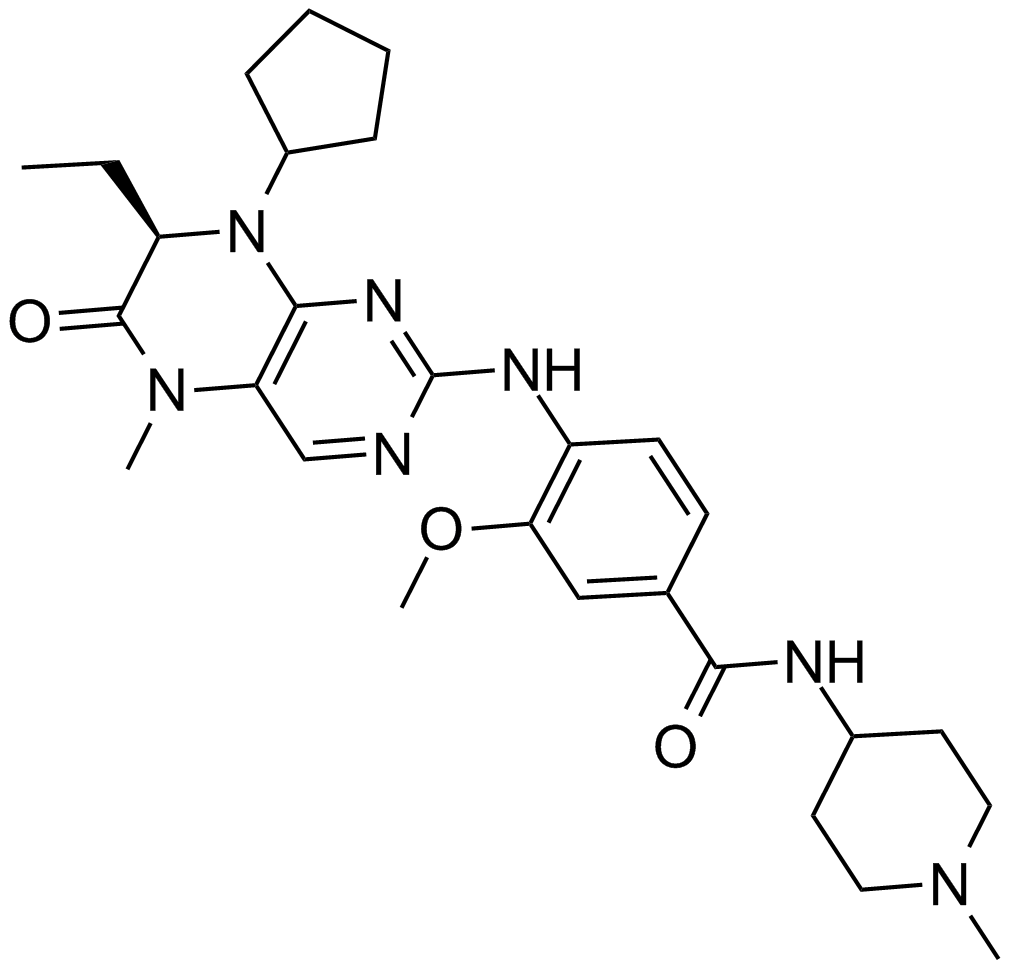
-
GC16726
BI-7273
BI-7273 ist ein selektiver und zellgÄngiger BRD9-Inhibitor mit einem IC50 und einem Kd von 19 und 0,75 nM; zeigt auch eine hohe Wirkung auf BRD7 mit einem IC50 und einem Kd von 117 nM und 0,3 nM.
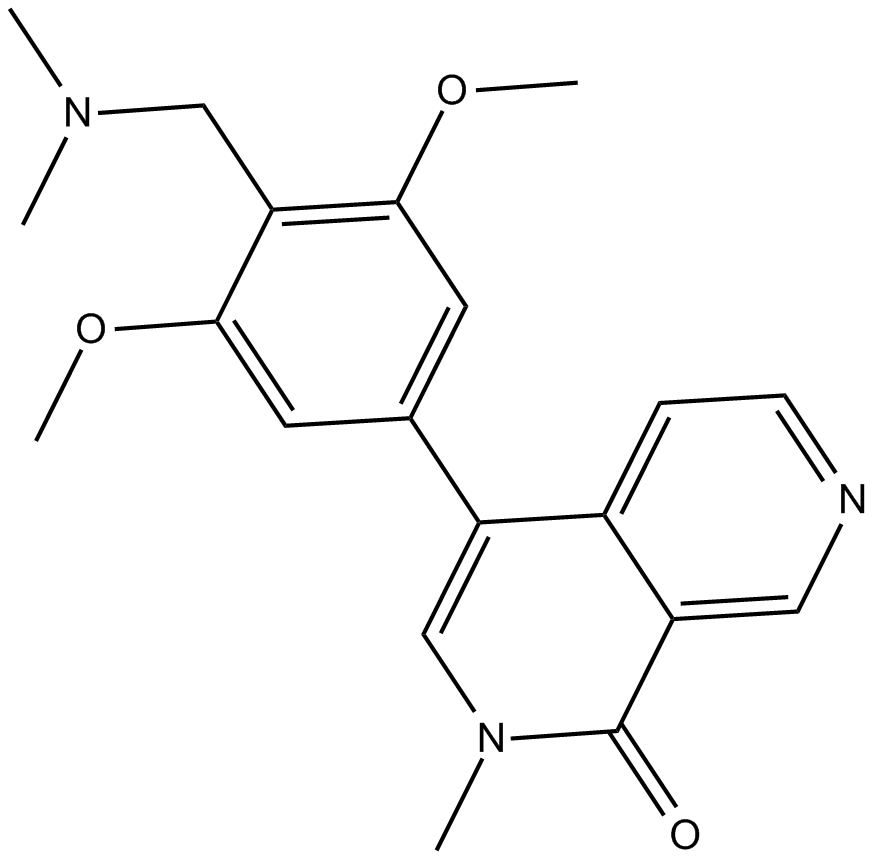
-
GC16817
BI-9564
BI-9564 ist ein potenter, selektiver und zellgÄngiger BRD9/BRD7-BromodomÄnen-Inhibitor mit IC50s von 75 nM und 3,4 μM und Kds von 14 nM bzw. 239 nM. BI-9564 hat einen IC50 von > 100 μM fÜr die BET-Familie.
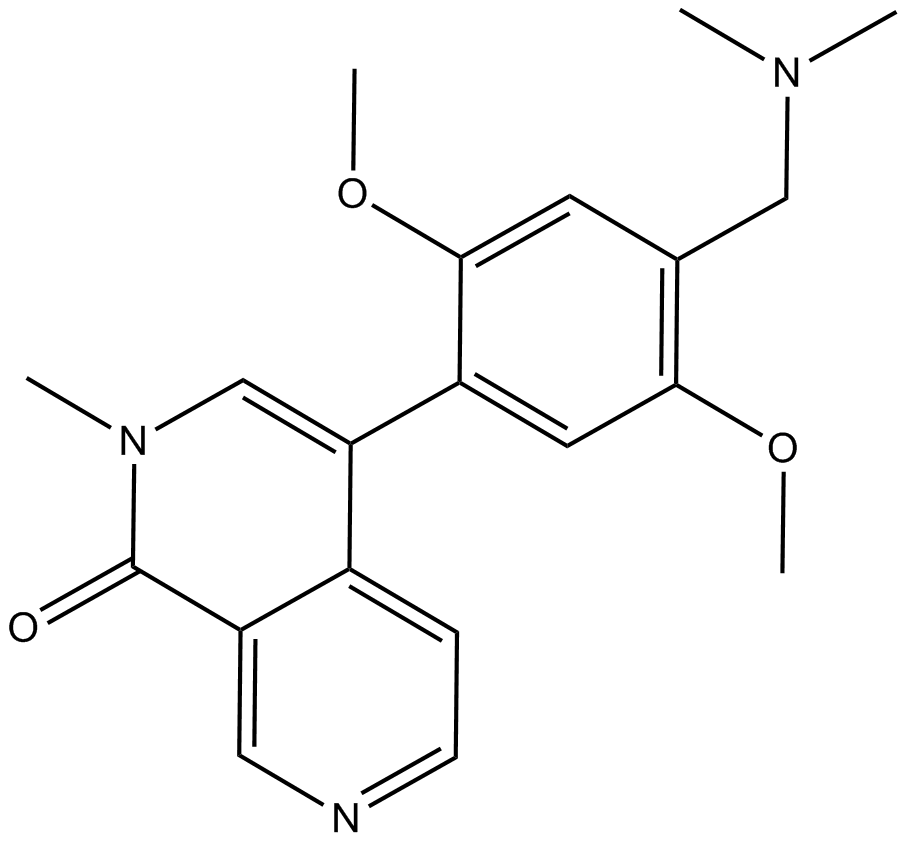
-
GC64789
Biotinylated-JQ1
Biotinyliertes JQ1 (Biotin-JQ1) ist ein biotinyliertes Derivat von JQ1 mit hoher AffinitÄt fÜr die BromodomÄne von BRD4. Biotinyliertes JQ1 hemmt die Proliferation von multiplen MM1.S-Myelomzellen mit einem EC50-Wert von 0,4μM.
-
GC32812
BMS-986158
BMS-986158 ist ein potenter BET-Inhibitor mit IC50-Werten von 6,6 und 5nM in NCI-H211-Zellen von kleinzelligem Lungenkrebs (SCLC) bzw. MDA-MB231-Zellen von dreifach negativem Brustkrebs (TNBC).
-
GC64538
BPTF-IN-BZ1
BPTF-IN-BZ1, ein BPTF-Inhibitor, besitzt eine hohe Potenz (Kd = 6,3 nM).
-
GC35552
BRD-IN-3
BRD-IN-3 ((R,R)-36n) ist ein hochpotenter PCAF-BromdomÄnen(BRD)-Inhibitor mit einem IC50 von 7 nM. BRD-IN-3 zeigt auch AktivitÄt gegen GCN5 und FALZ.
-
GC33017
BRD4 degrader AT1
BRD4-Degrader AT1 ist ein PROTAC, verbunden durch Liganden fÜr von Hippel-Lindau und BRD4 als ein hochselektiver Brd4-Degrader, mit einem Kd von 44 nM fÜr Brd4BD2 in Zellen.
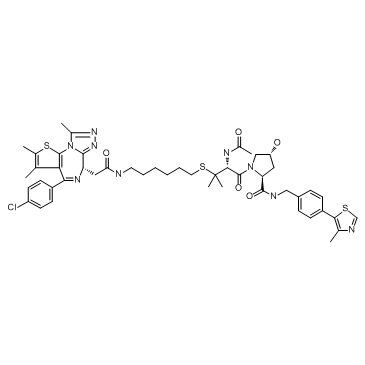
-
GC64960
BRD4 Inhibitor-10
BRD4-Inhibitor-10 ist ein potenter BRD4-BD1-Inhibitor, extrahiert aus Patent WO2015022332A1, Verbindung II-25, hat einen IC50 von 8 nM.
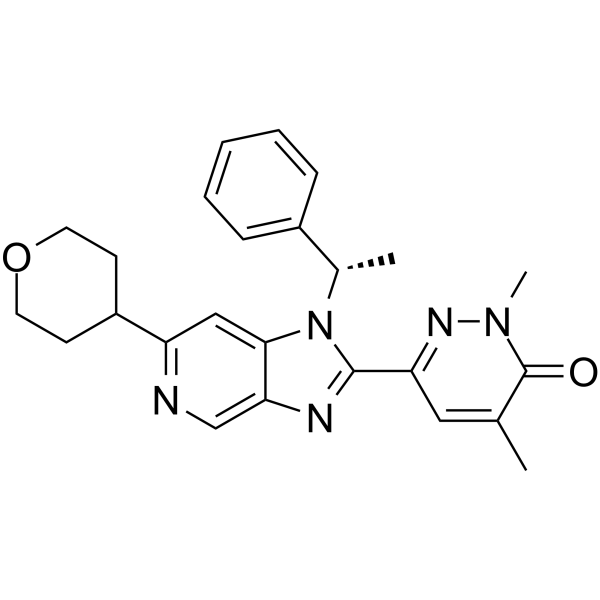
-
GC65569
BRD4 Inhibitor-24
BRD4-Inhibitor-24 (Verbindung 3U) ist ein potenter BRD4-Inhibitor, BRD4-Inhibitor-24 zeigt AntitumoraktivitÄt gegen MCF7- und K652-Zellen mit IC50-Werten von 33,7 bzw. 45,9 μM (aus Patent CN107721975A entnommen).
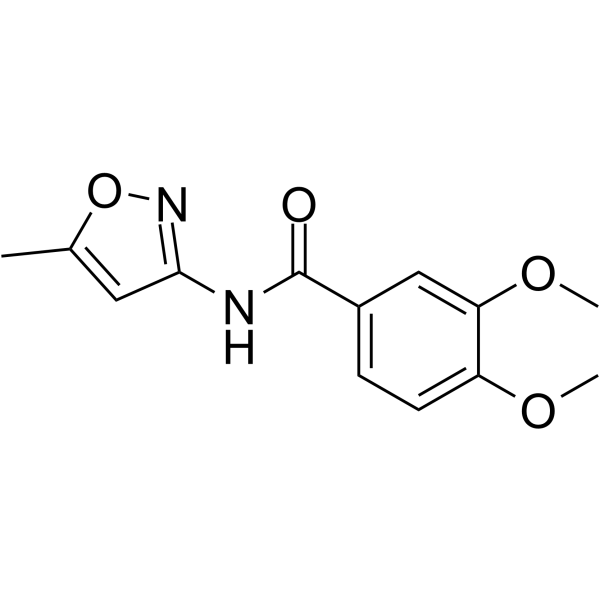
-
GC66441
BRD4/CK2-IN-1
BRD4/CK2-IN-1 ist der erste hochwirksame und oral aktive Dual-Target-Inhibitor von BRD4/CK2 (bromdomÄnenhaltiges Protein 4/Caseinkinase 2) mit IC50-Werten von 180 nM bzw. 230 nM fÜr BRD4 und CK2. BRD4/CK2-IN-1 hat eine starke AntikrebsaktivitÄt ohne offensichtliche ToxizitÄt. BRD4/CK2-IN-1 induziert Apoptose und Autophagie-assoziierten Zelltod bei dreifach negativem Brustkrebs (TNBC)
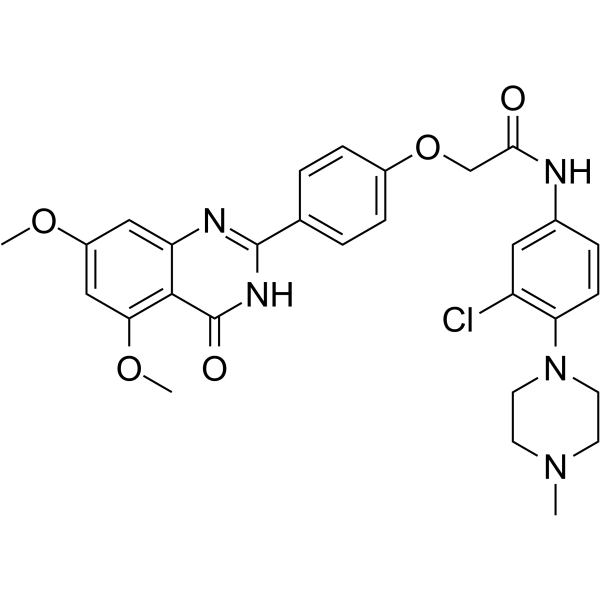
-
GC34505
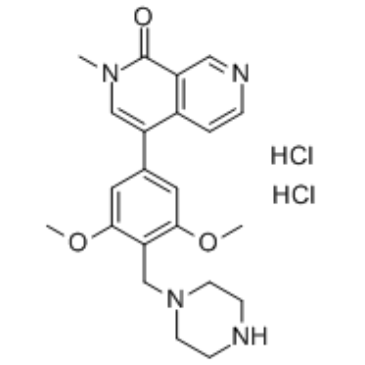
BRD7-IN-1
BRD7-IN-1, ein modifiziertes Derivat von BI7273 (BRD7/9-Inhibitor), bindet Über einen Linker an einen VHL-Liganden, um ein PROTAC VZ185 (VZ185 gegen BRD7/9 mit DC50s von 4,5 bzw. 1,8 nM) zu bilden.
-
GC64541
BRM/BRG1 ATP Inhibitor-2
BRM/BRG1 ATP Inhibitor-2 ist ein BRG1/BRM ATPase-Inhibitor zur Behandlung von BAF-bedingten Erkrankungen.
-
GC35557
Bromodomain IN-1
BromodomÄne IN-1 ist ein BromodomÄnen-Inhibitor, extrahiert aus dem Patent WO2016069578A1, Verbindung 4.
-
GC16531
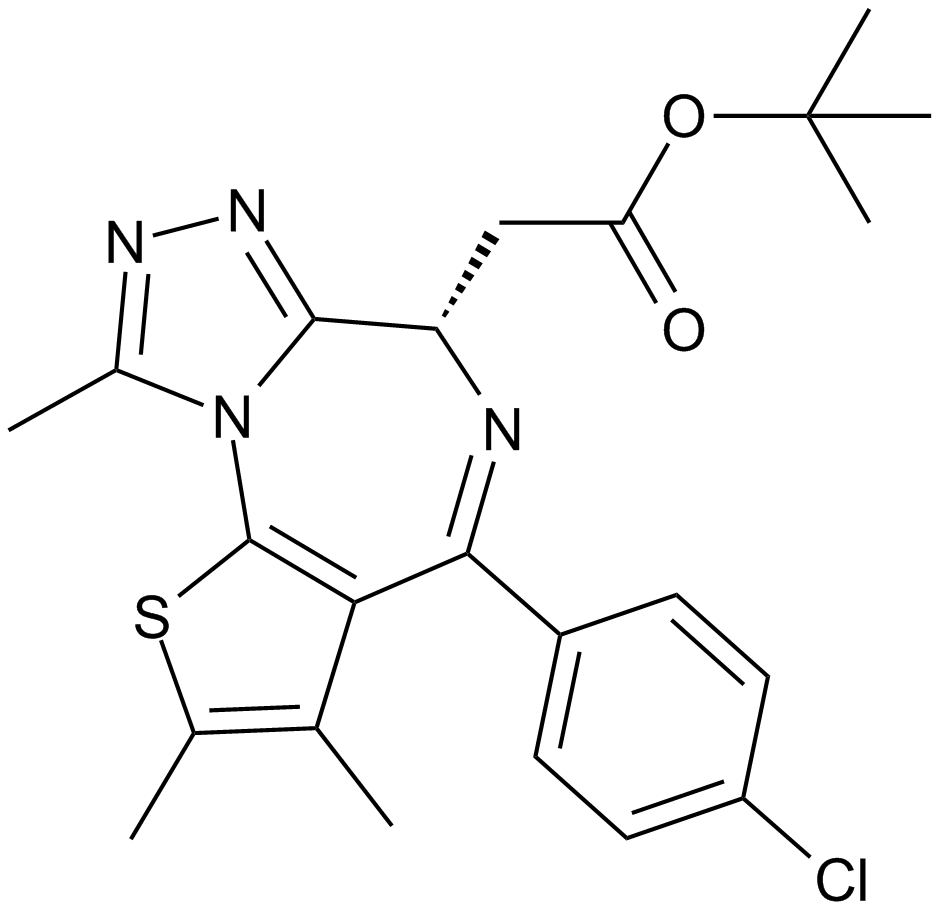
Bromodomain Inhibitor, (+)-JQ1
A selective inhibitor of BET bromodomains
-
GC35558
Bromodomain inhibitor-8
BromodomÄnen-Inhibitor-8 (Intermediate 21) ist ein BET-BromodomÄnen-Inhibitor zur Behandlung von Autoimmun- und EntzÜndungskrankheiten.
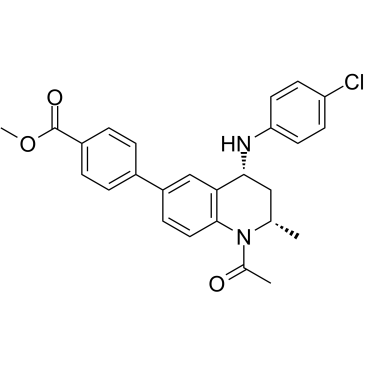
-
GC10402
Bromosporine
A non-specific bromodomain inhibitor
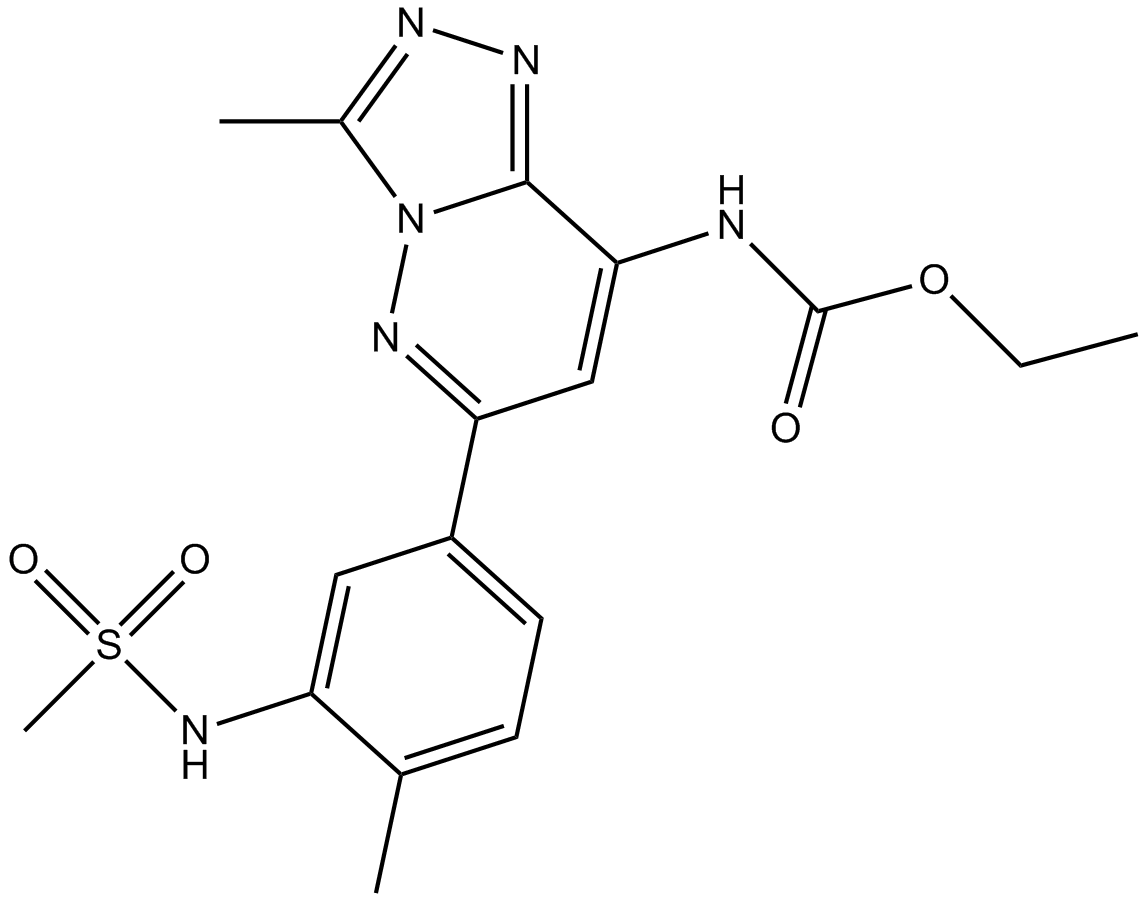
-
GC12733
C646
C646 ist ein selektiver und kompetitiver Histon-Acetyltransferase-p300-Inhibitor mit einem Ki von 400 nM und ist fÜr andere Acetyltransferasen weniger wirksam.
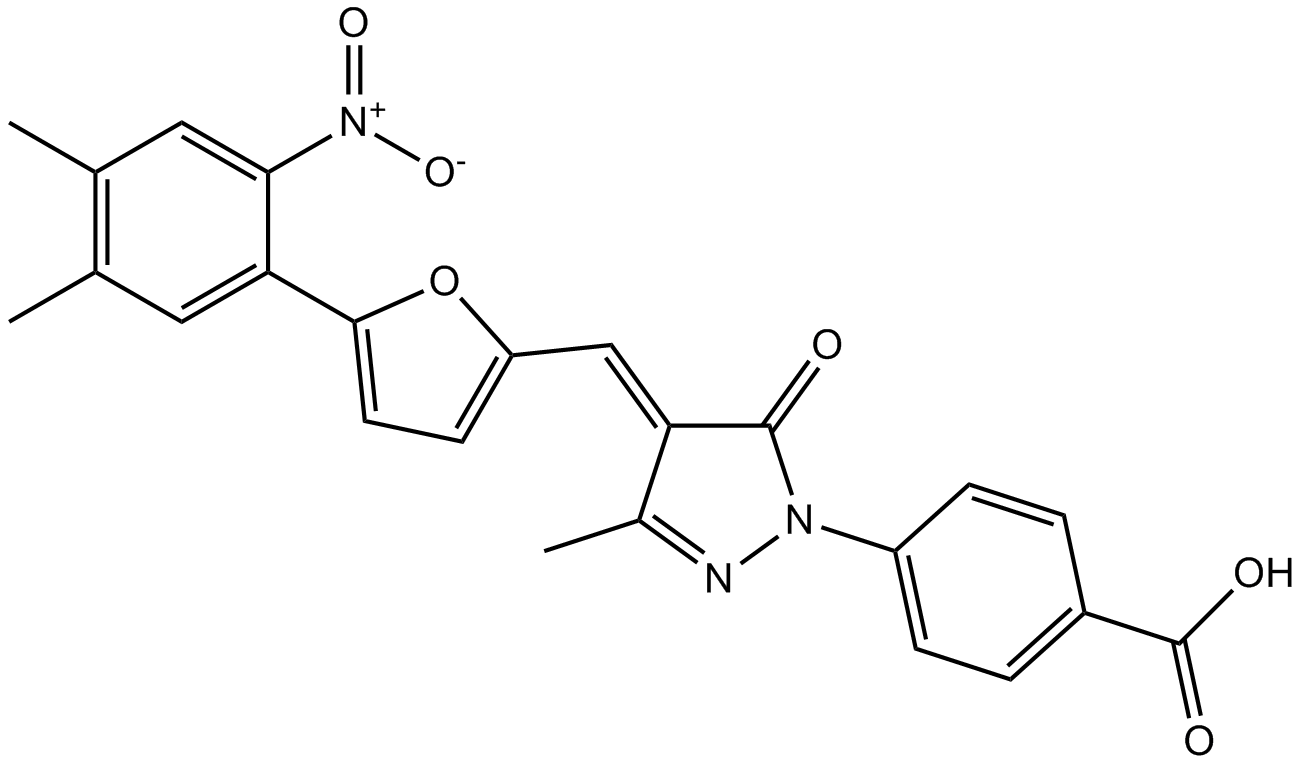
-
GC34517
CBP-IN-1
CBP-IN-1 (CCS1477) ist ein oral aktiver, potenter und selektiver Inhibitor des p300/CBP-Bromodomäne.
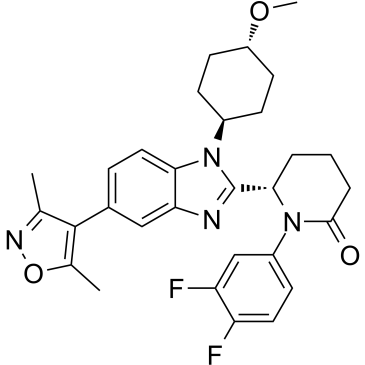
-
GC61577
CBP/p300-IN-1
CBP/p300-IN-1 ist ein CBP/EP300-BromodomÄnen-Inhibitor.
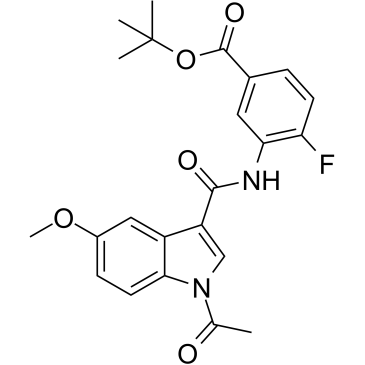
-
GC64696
CBP/p300-IN-12
CBP/p300-IN-12 ist ein potenter und selektiver kovalenter Histon-Acetyltransferase-p300- (IC50 von 166 nM) und CBP-Inhibitor. CBP/p300-IN-12 senkt die Spiegel von H3K27Ac von PC-3-Zellen (EC50 von 37 nM). CBP/p300-IN-12 bildet mit C1450 ein kovalentes Addukt.
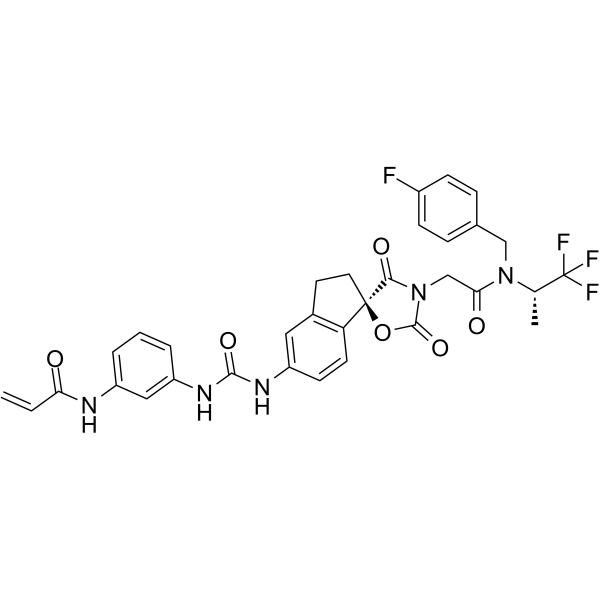
-
GC62330
CC-90010
CC-90010 (Verbindung 1) ist ein reversibler und oral aktiver BET-Inhibitor. CC-90010 wird in der Studie fÜr fortgeschrittene solide Tumore angewendet.
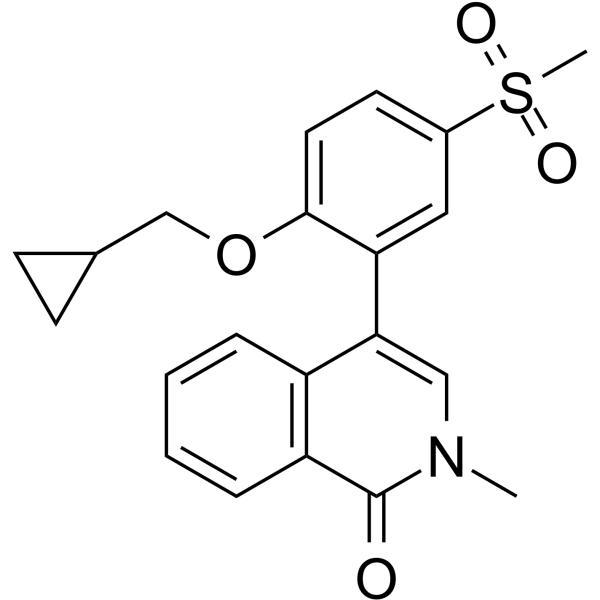
-
GC19098
CeMMEC1
CeMMEC1 ist ein Inhibitor von BRD4 und hat auch eine hohe AffinitÄt zu TAF1 mit einem IC50 von 0,9 μM fÜr TAF1 und einem Kd von 1,8 μM fÜr TAF1 (2).
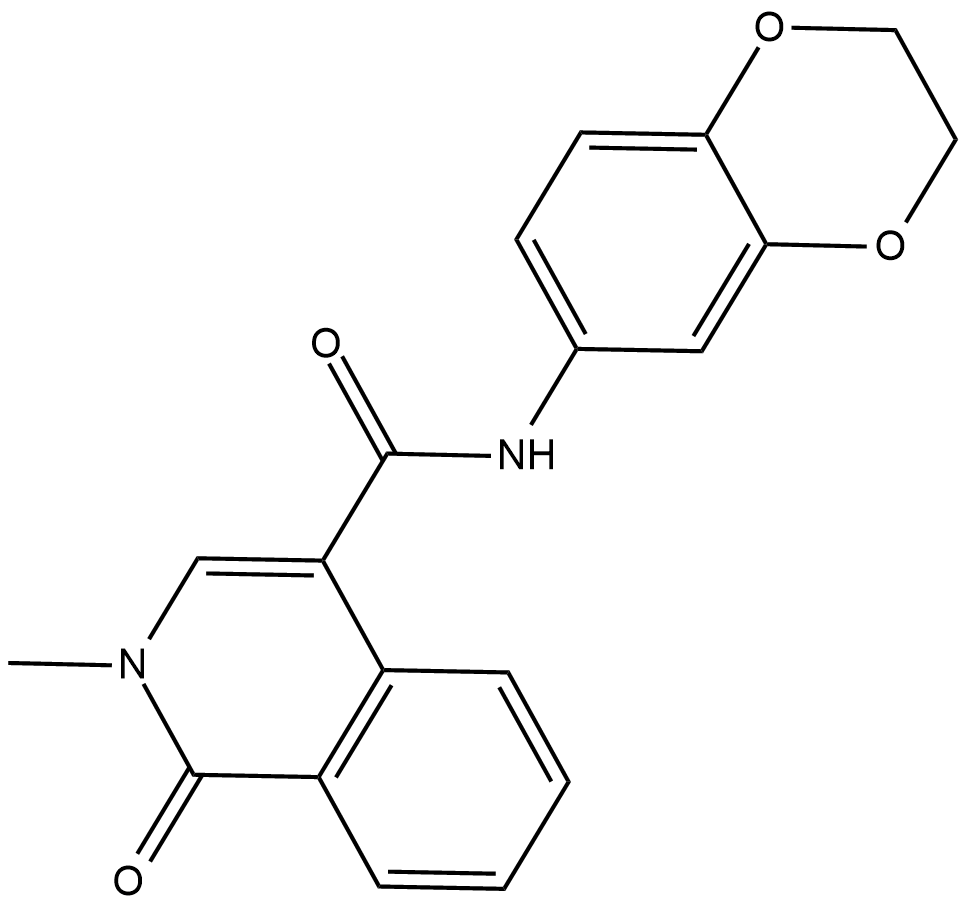
-
GC33028
CF53
CF53 ist ein hochpotenter, selektiver und oral aktiver Inhibitor des BET-Proteins mit einem Ki von <1 nM, Kd von 2,2 nM und einem IC50 von 2 nM fÜr BRD4 BD1. CF53 bindet sowohl an die BD1- als auch an die BD2-DomÄne von BRD2-, BRD3-, BRD4- und BRDT-BET-Proteinen mit hoher AffinitÄt, sehr selektiv gegenÜber Nicht-BET-BromdomÄnen-enthaltenden Proteinen. CF53 zeigt sowohl in vitro als auch in vivo eine starke AntitumoraktivitÄt.
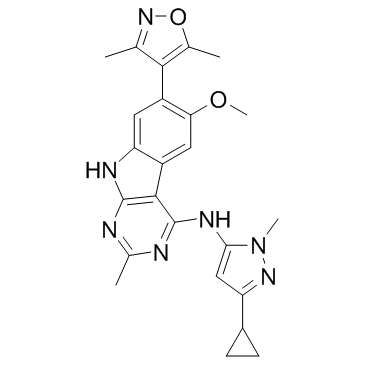
-
GC65602
CFT8634
CFT8634 ist ein Abbauer, der auf BRD9 abzielt, das aus dem Patent WO2021178920A1, Verbindung 174, extrahiert wurde. CFT8634 kann fÜr die Erforschung von Synovialsarkom und SMARCB1-deletierten soliden Tumoren verwendet werden.
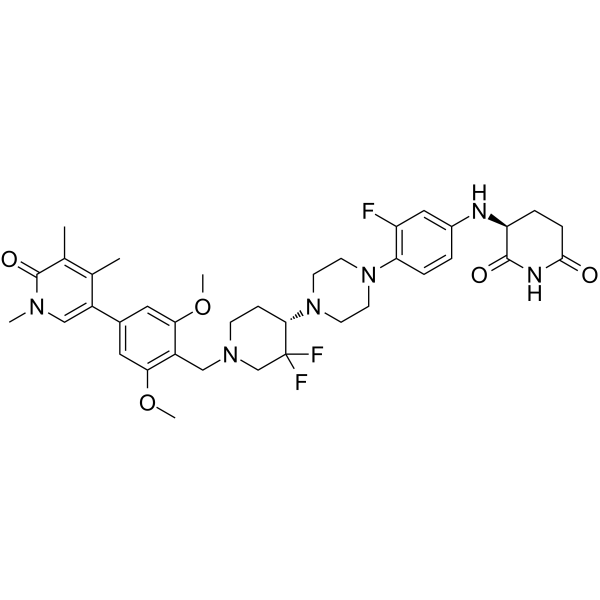
-
GC16599
CPI-169
EZH2 inhibitor
-
GC14699
CPI-203
CPI-203 ist ein neuartiger potenter, selektiver und zellgÄngiger Inhibitor der BET-BromodomÄne mit einem IC50-Wert von ca. 37 nM (BRD4 α-Screen-Assay).
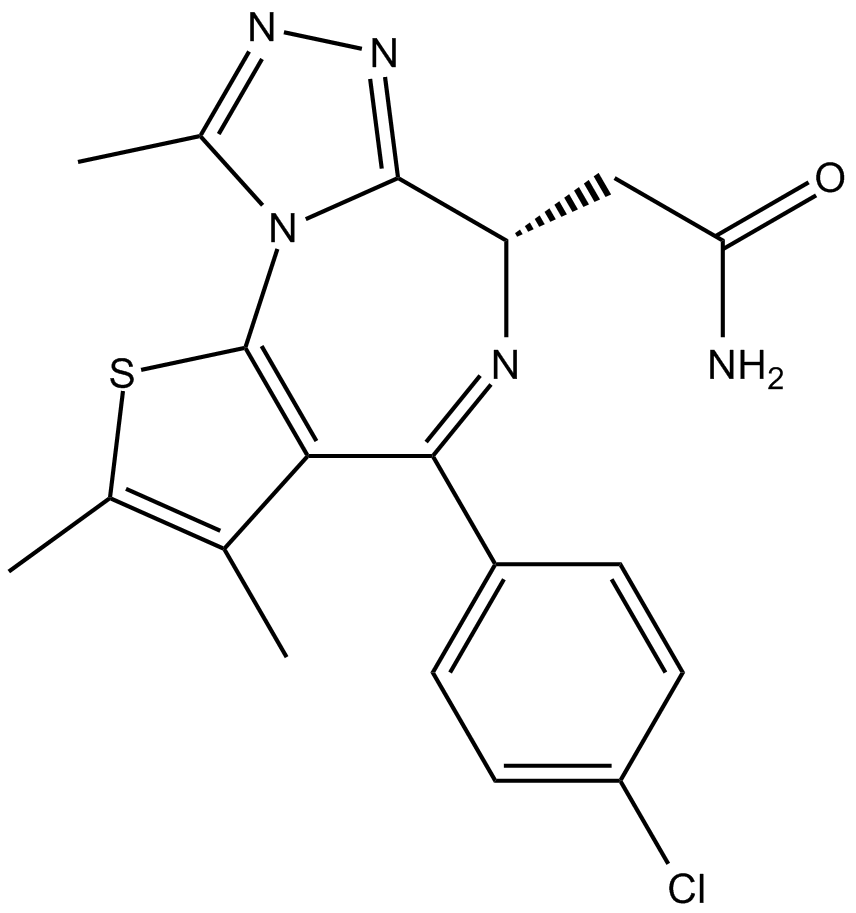
-
GC10382
CPI-637
CPI-637 ist ein selektiver und potenter CBP/EP300-BromodomÄnen-Inhibitor mit IC50-Werten von 0,03 μM, 0,051 μM und 11,0 μM fÜr CBP, EP300 bzw. BRD4 BD-1 und einem EC50 von 0,3 μM fÜr CBP.
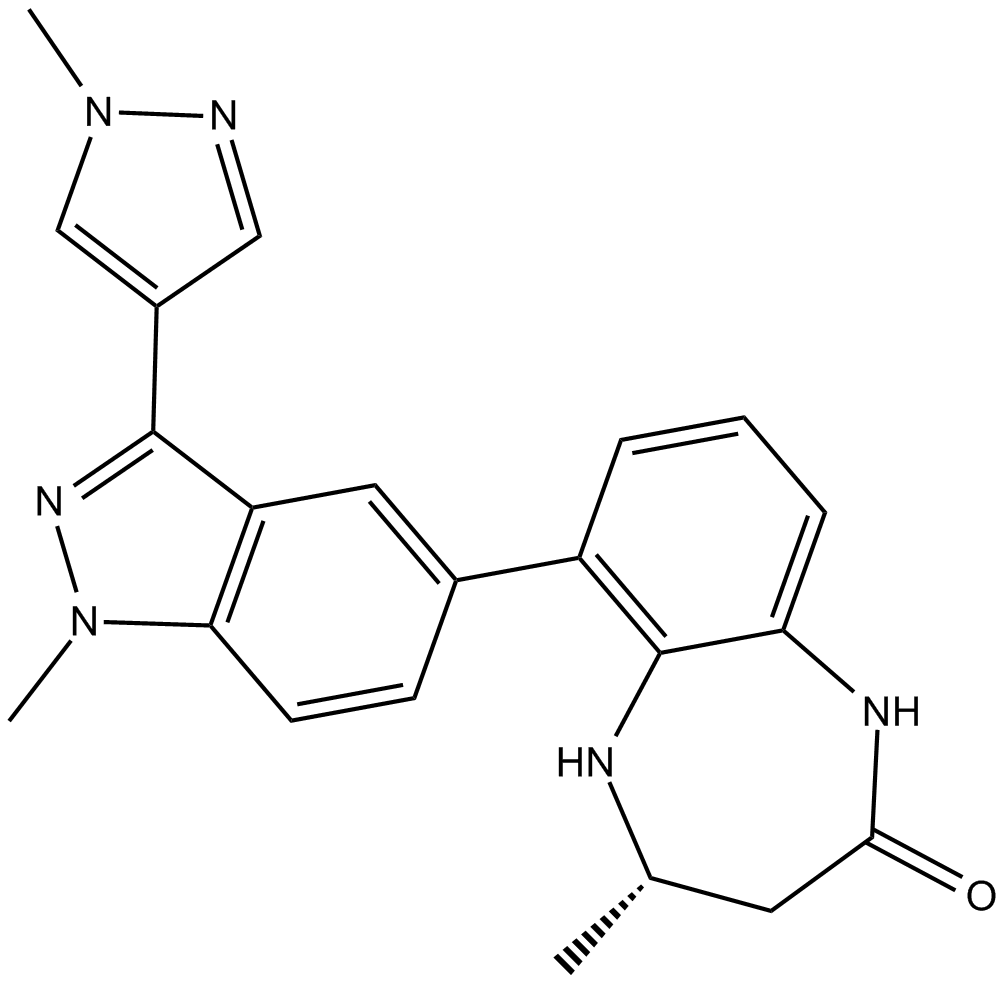
-
GC14787
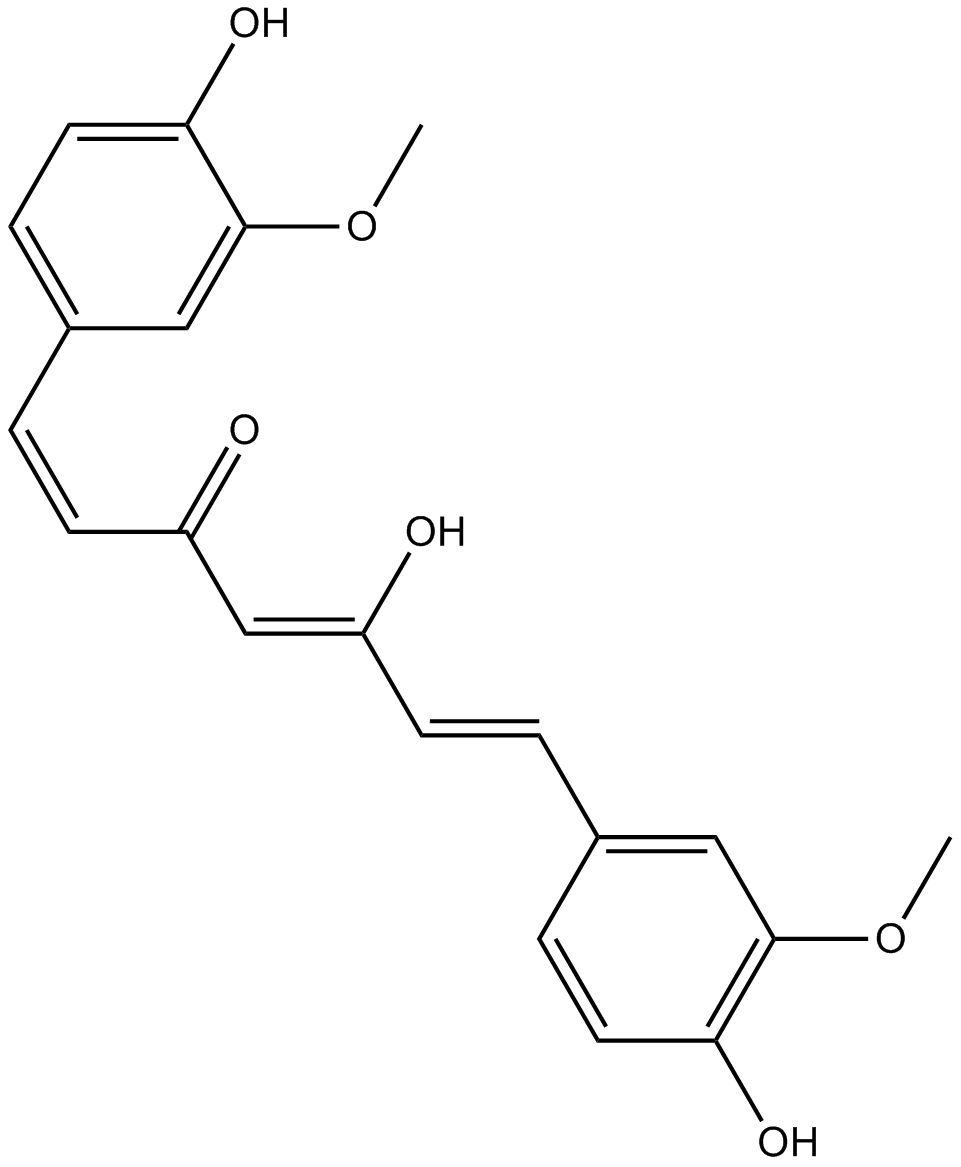
Curcumin
Ein gelbes Pigment mit vielfältigen biologischen Aktivitäten.
-
GC40226
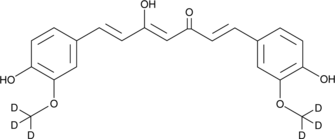
Curcumin-d6
Curcumin D6 (Diferuloylmethan D6) ist ein Deuterium mit der Bezeichnung Curcumin (gelbe Kurkuma).
-
GC19119
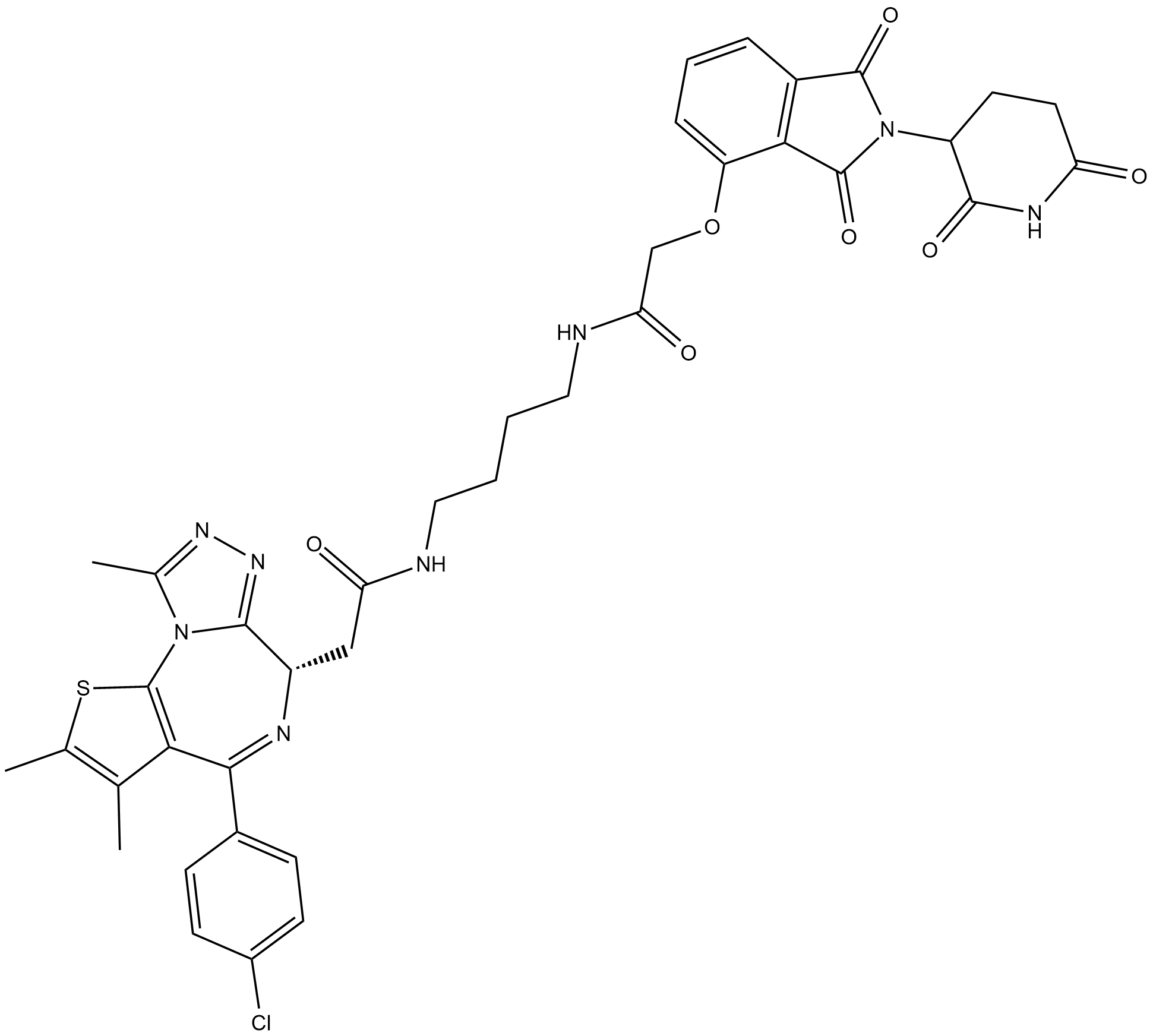
dBET1
dBET1 ist ein PROTAC, verbunden durch Liganden fÜr Cereblon und BRD4 mit einem EC50 von 430 nM. dBET1 ist ein PROTAC, das aus (+)-JQ1 besteht, das mit einem Linker mit NSC 527179 verknÜpft ist.
-
GC35815
dBET57
dBET57 ist ein potenter und selektiver Abbauer von BRD4BD1 auf Basis der PROTAC-Technologie. dBET57 vermittelt die Rekrutierung an die CRL4Cereblon E3-Ubiquitinligase mit einem DC50/5h von 500 nM fÜr BRD4BD1 und ist auf BRD4BD2 inaktiv.
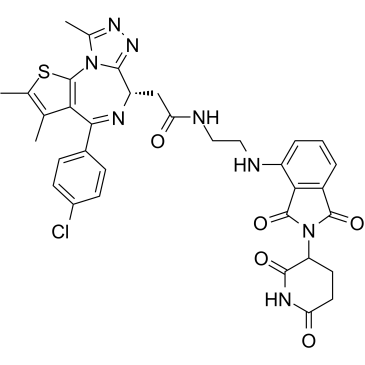
-
GC32719
dBET6
dBET6 ist ein hochpotentes, selektives und zellgÄngiges PROTAC, das durch Liganden fÜr Cereblon und BET verbunden ist, mit einem IC50 von 14 nM und AntitumoraktivitÄt hat.
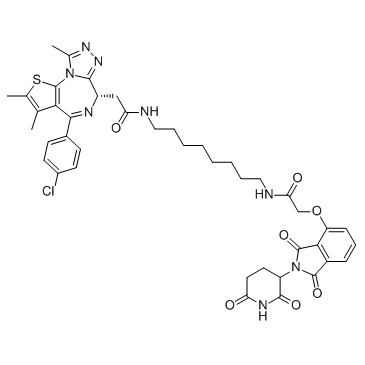
-
GC62211
dCBP-1
dCBP-1 ist ein potenter und selektiver heterobifunktioneller Abbauer von p300/CBP auf der Basis von Cereblon-Liganden. dCBP-1 ist außergewÖhnlich wirksam bei der AbtÖtung multipler Myelomzellen und schwÄcht die onkogene Enhancer-AktivitÄt ab, die die MYC-Expression antreibt.
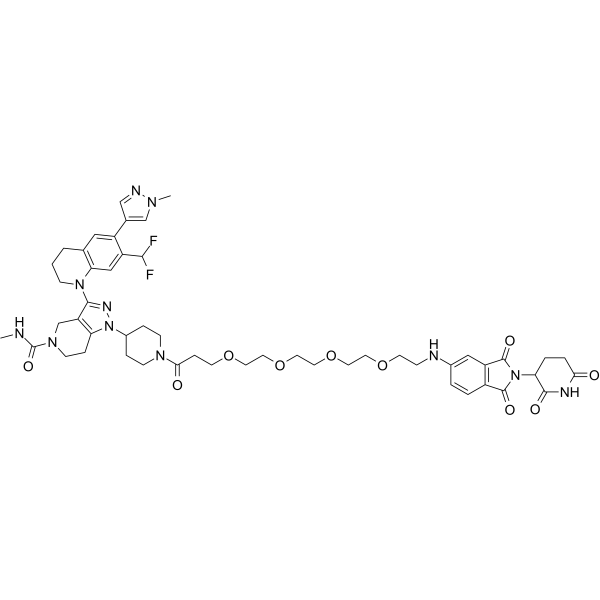
-
GC35904
dTRIM24
dTRIM24 ist ein selektiver bifunktionaler Abbauer von TRIM24 auf Basis von PROTAC, bestehend aus Liganden fÜr von Hippel-Lindau und TRIM24.
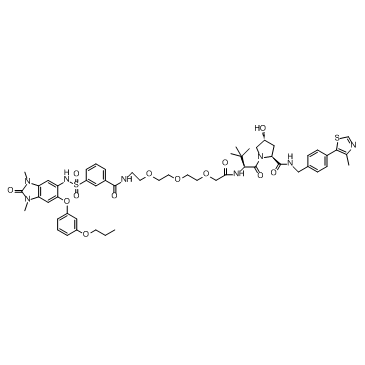
-
GC33403
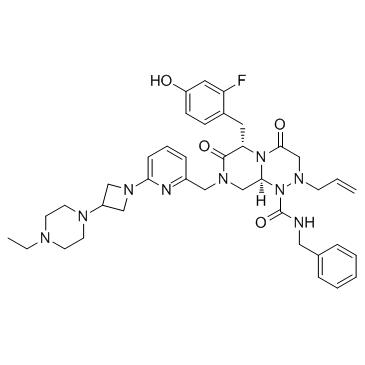
E-7386
E-7386 ist ein oral aktiver CBP/Beta-Catenin-Modulator.
-
GC31245
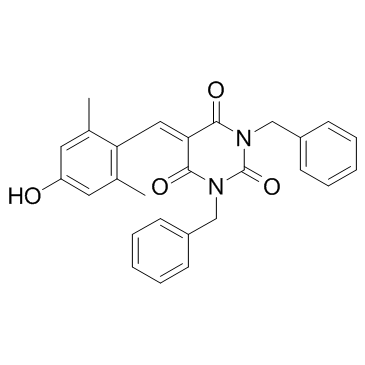
EML 425
EML425 ist ein potenter und selektiver CREB-Bindungsprotein (CBP)/p300-Inhibitor mit IC50-Werten von 2,9 bzw. 1,1 μM.
-
GC50182
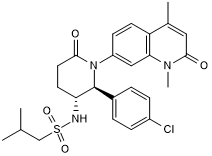
ent-LP 99
ent-LP 99 ist ein potenter und selektiver BRD7- und BRD9-Inhibitor mit einer KD von 99 nM fÜr BRD9.
-
GC64821
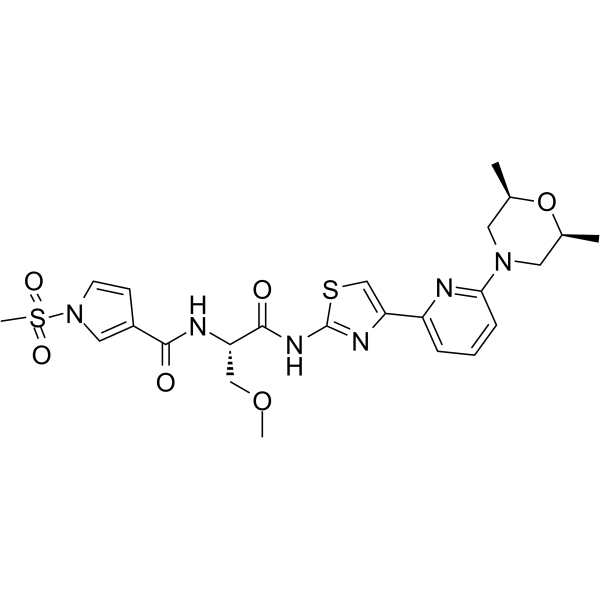
FHD-286
FHD-286 ist ein BRG1/BRM-ATPase-Inhibitor zur Behandlung von BAF-bedingten Erkrankungen wie akuter myeloischer LeukÄmie.
-
GC65160
FHT-1015
FHT-1205 ist ein potenter SMARCA4/SMARCA2 ATPase (BRG1 und BRM) Inhibitor mit IC50s von ≤10 nM (WO2020160180A1; Verbindung 67).
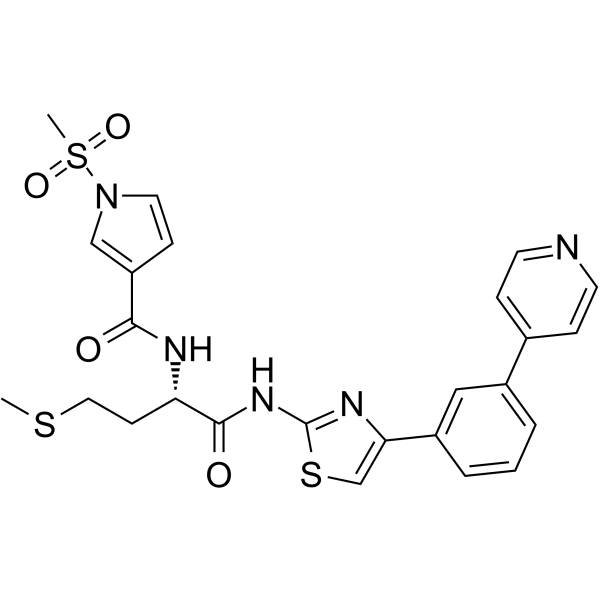
-
GC64777
FHT-1204
FHT-1204 ist ein potenter SMARCA4/SMARCA2 ATPase (BRG1 und BRM) Inhibitor mit IC50s von ≤10 nM (WO2020160180A1; Verbindung 70).
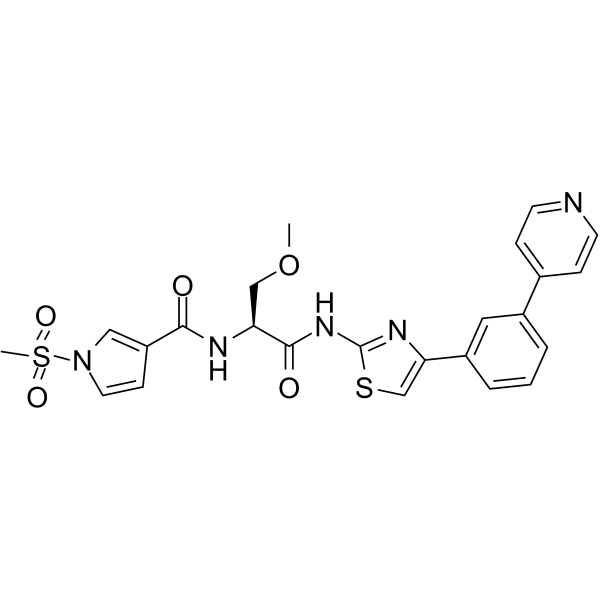
-
GC33015
FL-411 (BRD4-IN-1)
FL-411 (BRD4-IN-1) ist ein potenter und selektiver BRD4-Inhibitor mit einem IC50 von 0,43&7#177;0,09 μM fÜr BRD4(1).
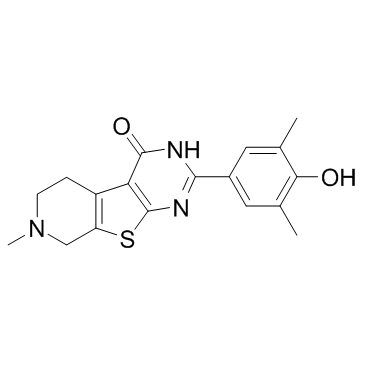
-
GC32960
GNE-049
GNE-049 ist ein hochpotenter und selektiver CBP-Inhibitor mit einem IC50-Wert von 1,1 nM im TR-FRET-Assay. GNE-049 hemmt auch BRET und BRD4(1) mit IC50-Werten von 12 nM bzw. 4200 nM.
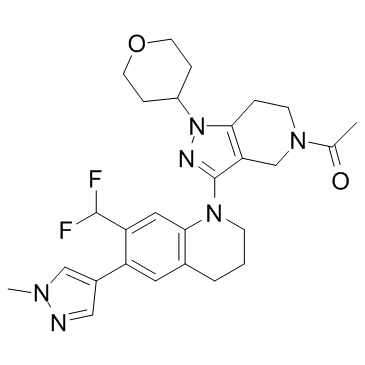
-
GC68439
GNE-064

-
GC33212
GNE-207
GNE-207 ist ein potenter, selektiver und oral bioverfÜgbarer Inhibitor der BromodomÄne von CBP mit einem IC50 von 1 nM, der einen >2500-fachen selektiven Index gegenÜber BRD4 aufweist (1). GNE-207 zeigt eine hervorragende CBP-Potenz mit einem EC50-Wert von 18 nM fÜr die MYC-Expression in MV-4-11-Zellen.
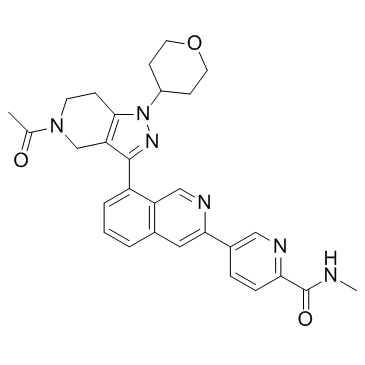
-
GC32747
GNE-272
GNE-272 ist ein potenter und selektiver CBP/EP300-Inhibitor mit IC50-Werten von 0,02, 0,03 und 13 μM fÜr CBP, EP300 bzw. BRD4. GNE-272 ist auch eine selektive In-vivo-Sonde fÜr CBP/EP300.
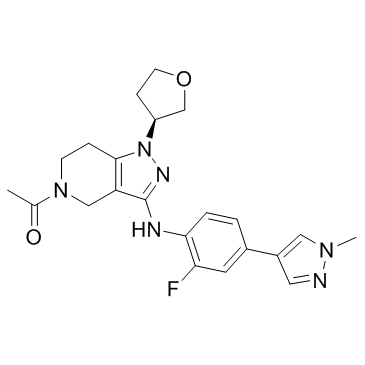
-
GC65326
GNE-375
GNE-375 ist ein potenter und hochselektiver BRD9-Inhibitor mit einem IC50 von 5 nM. GNE-375 zeigt >100-fach selektiv fÜr BRD9 gegenÜber BRD4, TAF1 und CECR2. GNE-375 verringert die Bindung von BRD9 an Chromatin.
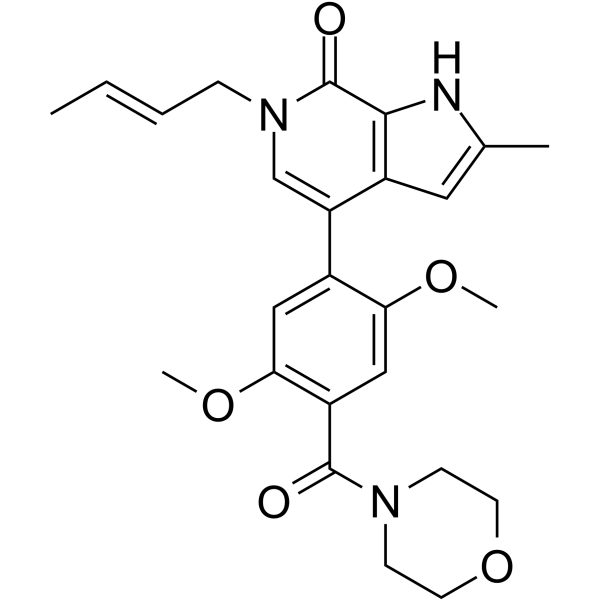
-
GC32081
GNE-781
GNE-781 ist ein oral aktiver, hochpotenter und selektiver CBP-Inhibitor mit einem IC50-Wert von 0,94 nM im TR-FRET-Assay. GNE-781 hemmt auch BRET und BRD4(1) mit IC50-Werten von 6,2 nM bzw. 5100 nM. GNE-781 zeigt AntitumoraktivitÄt in einem MOLM-16-AML-Xenotransplantatmodell.
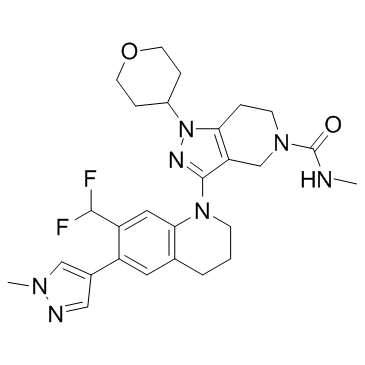
-
GC33204
GS-626510
GS-626510 ist ein potenter und oral aktiver BromodomÄnen-Inhibitor der BET-Familie mit Kd-Werten von 0,59-3,2 nM fÜr BRD2/3/4, mit IC50-Werten von 83 nM und 78 nM fÜr BD1 bzw. BD2.
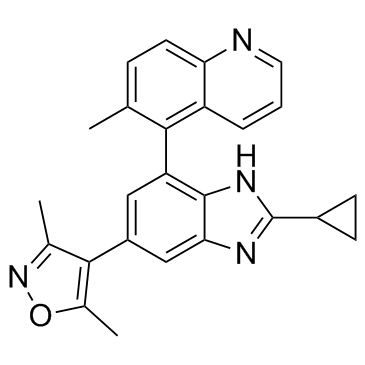
-
GC31731
GSK 4027
GSK 4027 ist eine chemische Sonde fÜr die PCAF/GCN5-BromodomÄne mit einem pIC50 von 7,4 ± 0,11 fÜr PCAF in einem zeitaufgelÖsten Fluoreszenz-Resonanz-Energietransfer (TR-FRET)-Assay.
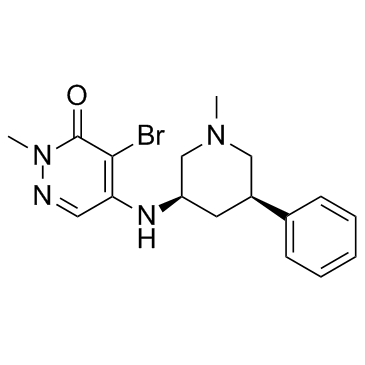
-
GC12440
GSK 5959
GSK 5959 ist ein potenter, selektiver und zellgängiger BRPF1-Bromodomänen-Inhibitor mit einem IC50 von ~ 80 nM.
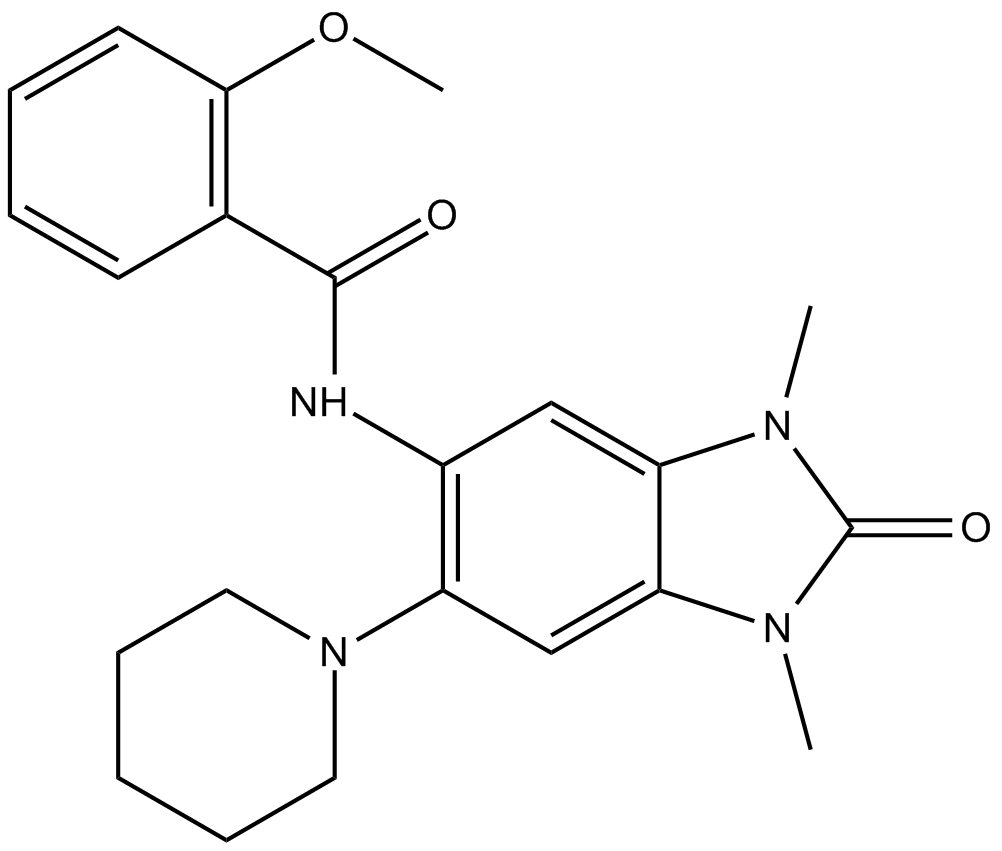
-
GC50378
GSK 9311 hydrochloride
GSK 9311-Hydrochlorid, ein weniger aktives Analogon von GSK6853, kann als Negativkontrolle verwendet werden. GSK 9311-Hydrochlorid hemmt die BRPF-Bromodomäne mit pIC50-Werten von 6,0 und 4,3 für BRPF1 bzw. BRPF2.
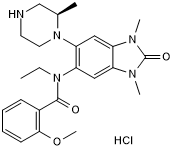
-
GC14063
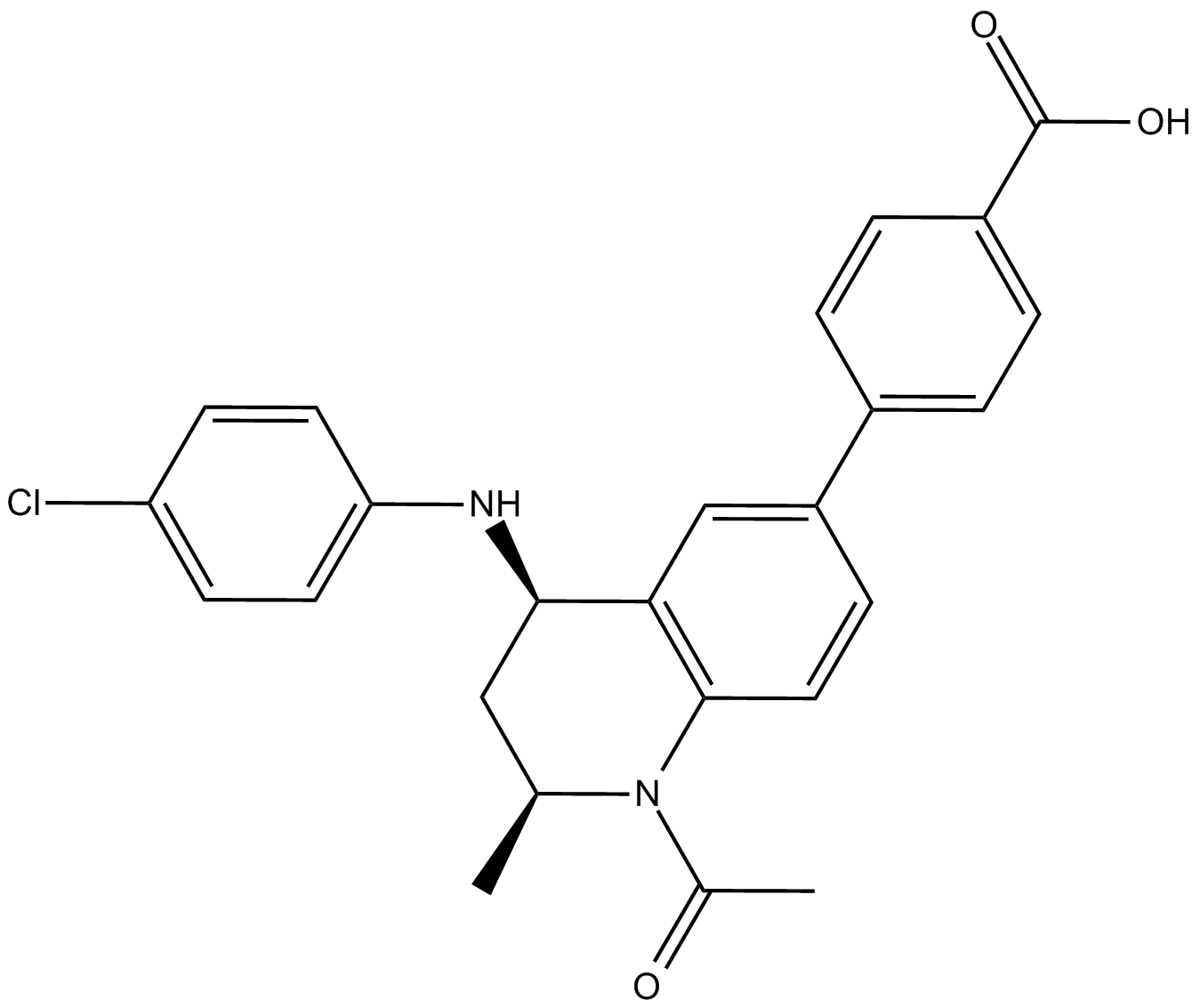
GSK1324726A
GSK1324726A ist ein neuer, potenter und selektiver Inhibitor von BET-Proteinen mit hoher AffinitÄt zu BRD2 (IC50=41 nM), BRD3 (IC50=31 nM) und BRD4 (IC50=22 nM).
-
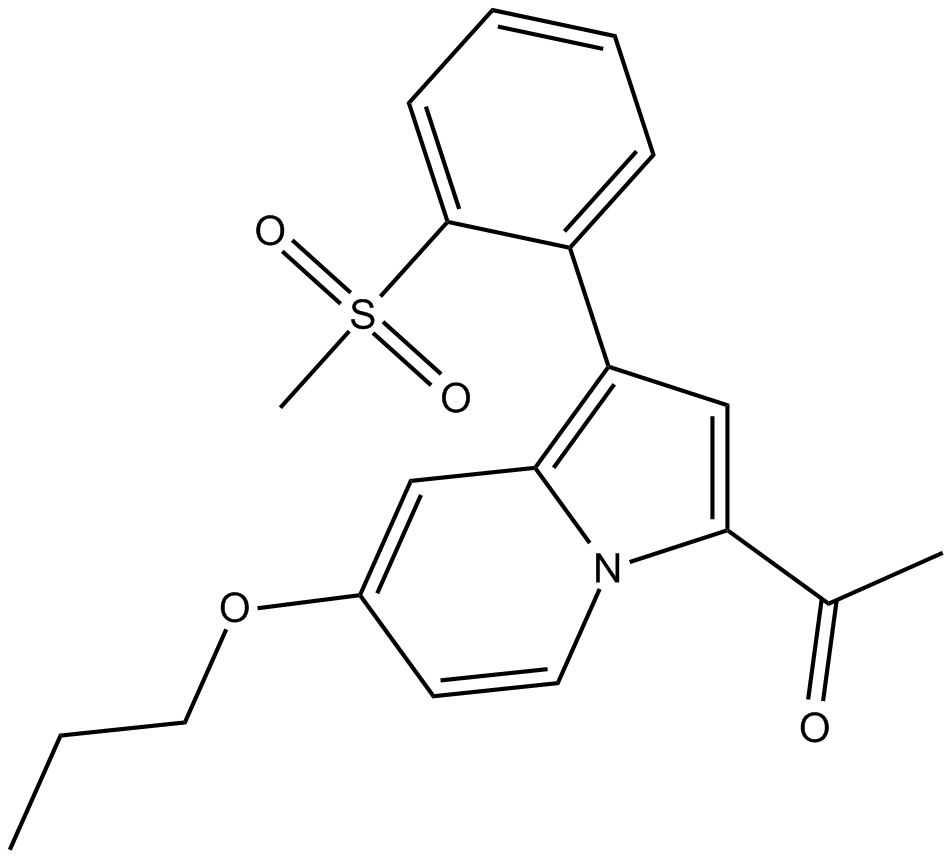
GC15789
GSK2801
GSK2801 ist ein potenter, selektiver, oral aktiver und zellaktiver Acetyl-Lysin-kompetitiver BAZ2A- und BAZ2B-BromodomÄnen-Inhibitor mit Kd-Werten von 136 nM bzw. 257 nM. GSK2801 zeigt eine >50-fache SelektivitÄt fÜr BAZ2A/B gegenÜber BRD4.
-
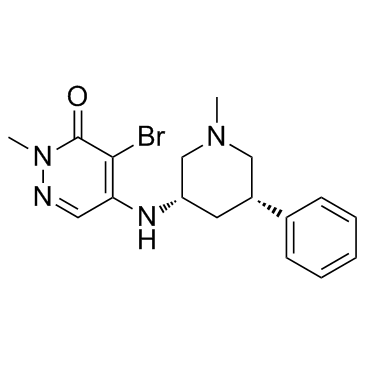
GC30714
GSK4028
GSK4028 ist die enantiomere Negativkontrolle von GSK4027, einer chemischen PCAF/GCN5-BromodomÄnensonde, der pIC50 von GSK4028 betrÄgt 4,9 in einem zeitaufgelÖsten Fluoreszenzresonanzenergietransfer (TR-FRET)-Assay.
-
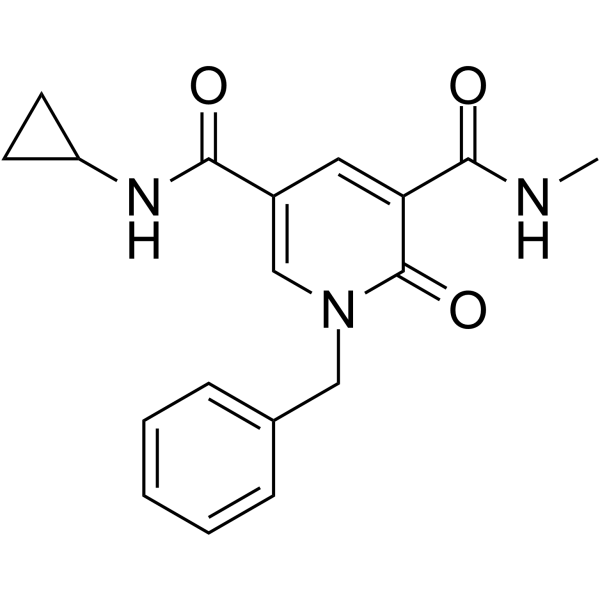
GC62312
GSK620
GSK620 ist ein potenter und oral wirksamer Pan-BD2-Inhibitor mit exzellenter breiter Selektivität, Entwickelbarkeit und in vivo oralen Pharmakokinetik.
-
GC13025
GSK6853
GSK6853 ist ein potenter und selektiver Inhibitor der BRPF1-BromodomÄne.
-
GC62654
GSK778
GSK778 (iBET-BD1) ist ein potenter und selektiver BD1-BromodomÄnen-Inhibitor der BET-Proteine mit IC50-Werten von 75 nM (BRD2 BD1), 41 nM (BRD3 BD1), 41 nM (BRD4 BD1) und 143 nM (BRDT BD1) , beziehungsweise.
-
GC38049
GSK8573
GSK8573 ist eine inaktive Kontrollverbindung fÜr GSK2801 (kompetitiver Acetyl-Lysin-Inhibitor der BAZ2A- und BAZ2B-BromdomÄnen). GSK8573 hat BindungsaktivitÄt an BRD9 mit einem Kd-Wert von 1,04 μM und ist inaktiv gegen BAZ2A/B und andere BromodomÄnenfamilien. GSK8573 kann als strukturell verwandte negative Kontrollverbindung in biologischen Experimenten verwendet werden.
-
GC60184
GSK8814
GSK8814 ist eine potente, selektive und chemische ATAD2/2B-BromdomÄnensonde und ein Inhibitor mit einer Bindungskonstante von pKd=8,1 und einem pKi=8,9 in BROMOscan. GSK8814 bindet an ATAD2 und BRD4 BD1 mit pIC50s von 7,3 bzw. 4,6. GSK8814 zeigt eine 500-fache SelektivitÄt fÜr ATAD2 gegenÜber BRD4 BD1.
-
GC33324
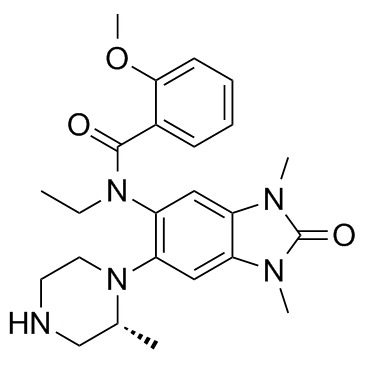
GSK9311
GSK9311, ein weniger aktives Analogon von GSK6853, kann als negative Kontrolle verwendet werden. GSK9311 hemmt die BRPF-BromodomÄne mit pIC50-Werten von 6,0 und 4,3 fÜr BRPF1 bzw. BRPF2.
-
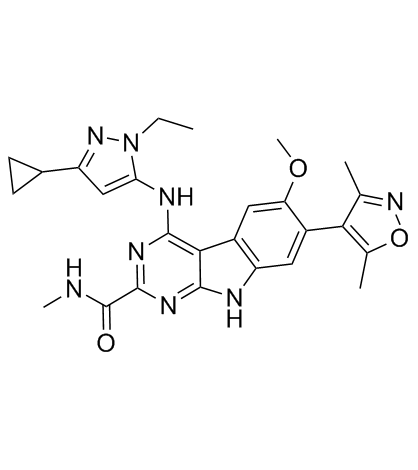
GC33211
HJB97
HJB97 ist ein hochaffiner BET-Inhibitor mit Kis von 0,9 nM (BRD2 BD1), 0,27 nM (BRD2 BD2), 0,18 nM (BRD3 BD1), 0,21 nM (BRD3 BD2), 0,5 nM (BRD4 BD1), 1,0 nM (BRD4 BD2), bzw. HJB97 wird fÜr das Design eines potenziellen PROTAC BET-Abbaumittels verwendet und hat AntitumoraktivitÄt.
-
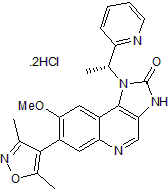
GC50070
I-BET 151 dihydrochloride
I-BET 151-Dihydrochlorid (GSK1210151A-Dihydrochlorid) ist ein BET-Bromodomänen-Inhibitor, der BRD4, BRD2 und BRD3 mit pIC50 von 6,1, 6,3 bzw. 6,6 hemmt.
-
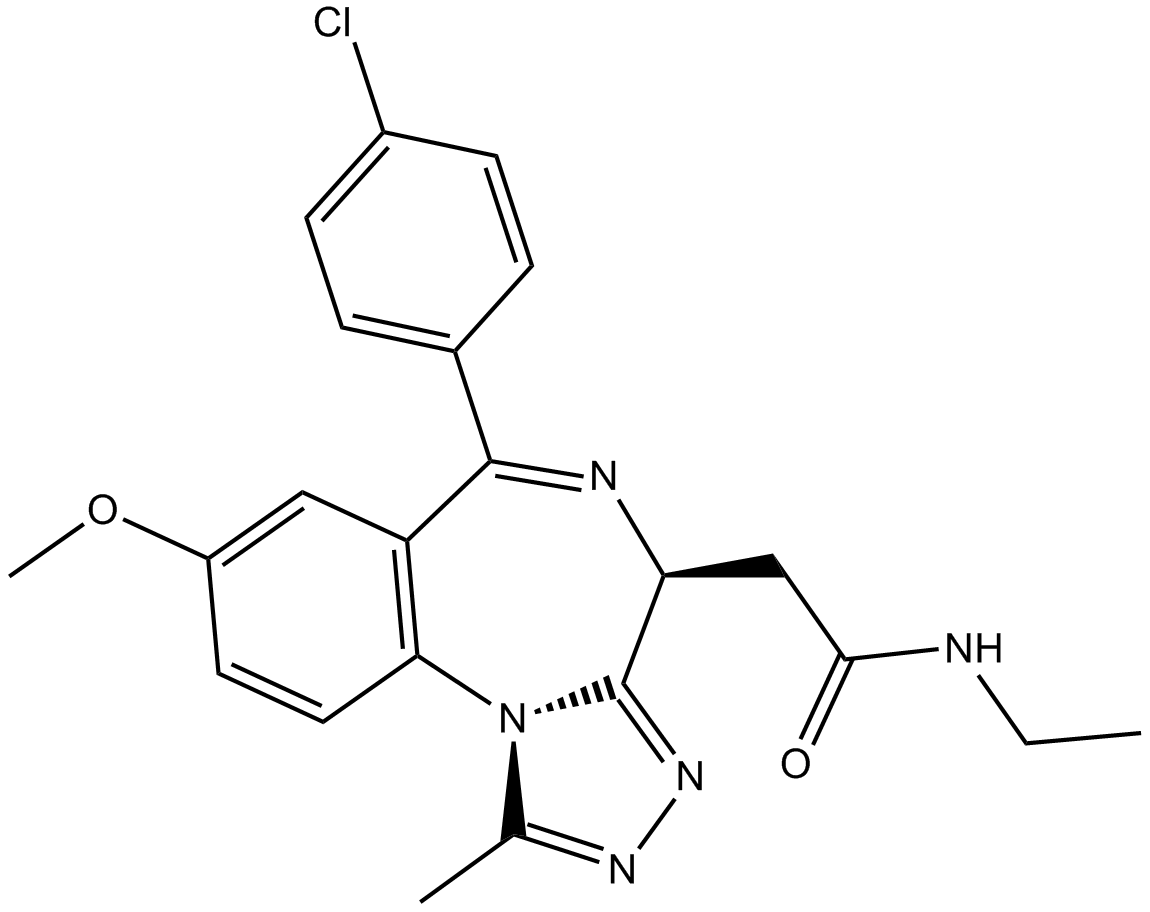
GC17073
I-BET-762
I-BET-762 (I-BET762; GSK525762) ist ein BET-BromodomÄnen-Inhibitor mit IC50 von 32,5-42,5 nM.
-
GC15747
I-BET151 (GSK1210151A)
I-BET151 (GSK1210151A) (GSK1210151A) ist ein BET-BromodomÄnen-Inhibitor, der BRD4, BRD2 und BRD3 mit pIC50 von 6,1, 6,3 bzw. 6,6 hemmt.
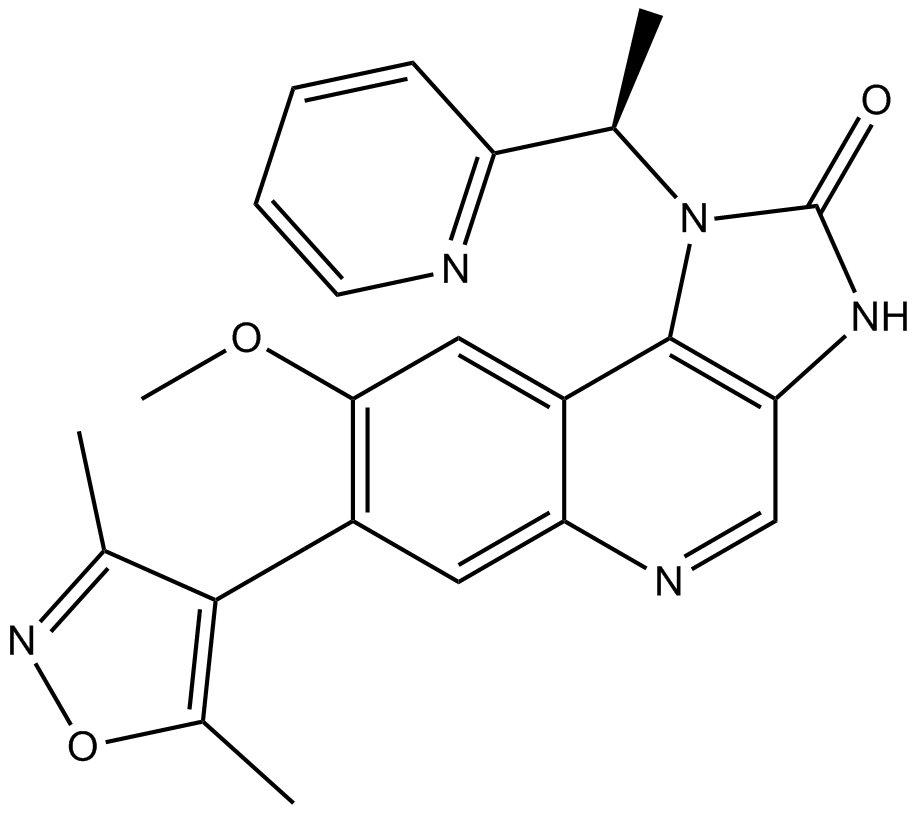
-
GC64297
I-BET567
I-BET567 ist ein potenter und oral aktiver Inhibitor des Pan-BET-Kandidaten mit pIC50-Werten von 6,9 und 7,2 fÜr BRD4 BD1 bzw. BD2.
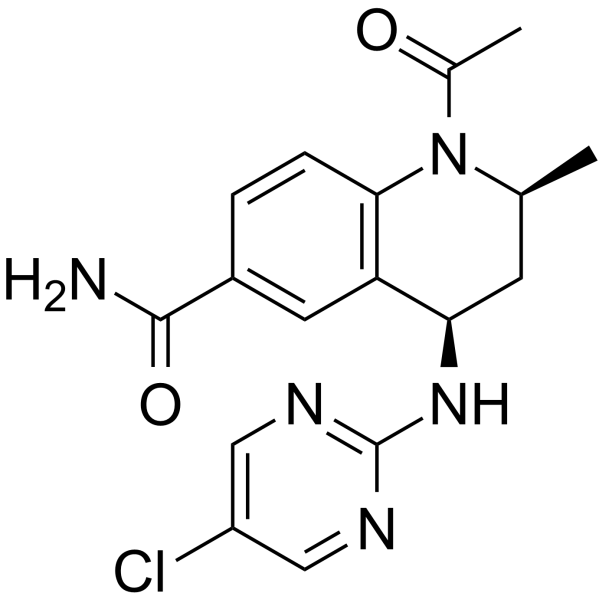
-
GC12588
I-BRD9
I-BRD9 ist eine selektive zellulÄre chemische Sonde des BromodomÄnen-enthaltenden Proteins 9 (BRD9) mit einem pIC50-Wert von 7,3 μM.