Histone Demethylases
Histone demethylases are a diverse group of enzymes catalyzing a process to reverse histone methylation, in which methyl (CH3-) groups are removed from instead of being transferred to histone. So far, two evolutionarily conserved histone demethylase families have been identified, including LSD demethylases and JMJC demethylases. LSD1 and LSD2 are two well-established members of LSD demethylase family, which catalyze demethylation through a flavin adenine dinucleotide (FAD)-dependent amine oxidation reaction. LSD1 has been found to demethylate H3K4mel, H3K4me2, H3K9me1, H3K9me2 and a few non-histone targets; while LSD2 has been found to demethylate only H3K4me1 and H3K4me2. Members of JMJC demethylase family is characterized by containing the catalytic JMJC domain, which demethylate substrates, including H3K4, H3K9, H3K27, H3K36 and H4K20, through a Fe(II)- and α-ketogutarate-dependent dioxygenase reaction.
Produkte für Histone Demethylases
- Bestell-Nr. Artikelname Informationen
-
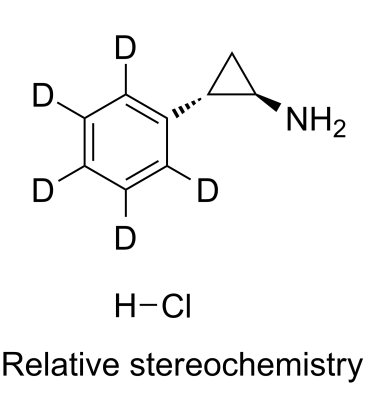
GC60004
(rel)-Tranylcypromine D5 hydrochloride
Tranylcypromin-d5 (SKF 385-d5)-Hydrochlorid ist ein mit Deuterium markiertes (rel)-Tranylcypromin-Hydrochlorid.
-
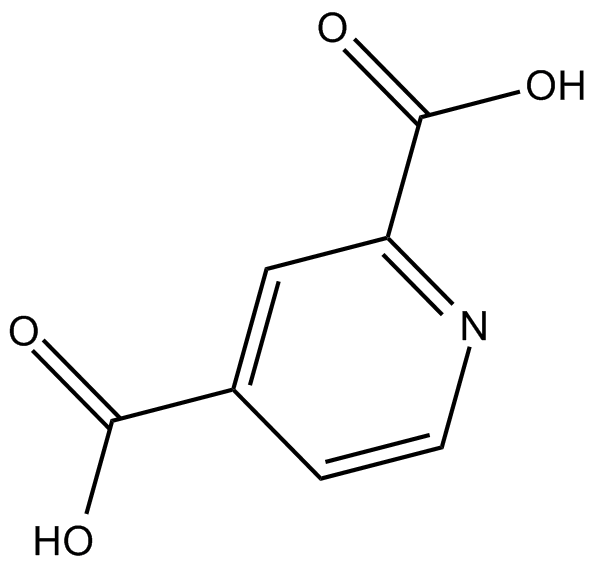
GC11873
2,4-Pyridinedicarboxylic Acid
2,4-PyridindicarbonsÄure (2,4-PyridindicarbonsÄure) ist ein Breitband-Inhibitor der 2OG-Oxygenase, einschließlich der JmjC-DomÄnen-enthaltenden Familie der Histon-Demethylasen (JHDMs). 2,4-PyridindicarbonsÄure ist eine Zielchemikalie im Bereich der biobasierten Kunststoffe.
-
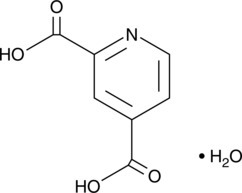
GC42075
2,4-Pyridinedicarboxylic Acid (hydrate)
2,4-Pyridinedicarboxylic Acid (2,4-PDCA) is a compound that structurally mimics 2-oxoglutarate (2-OG, also known as α-ketoglutarate) and chelates zinc, thus affecting a range of enzymes.
-
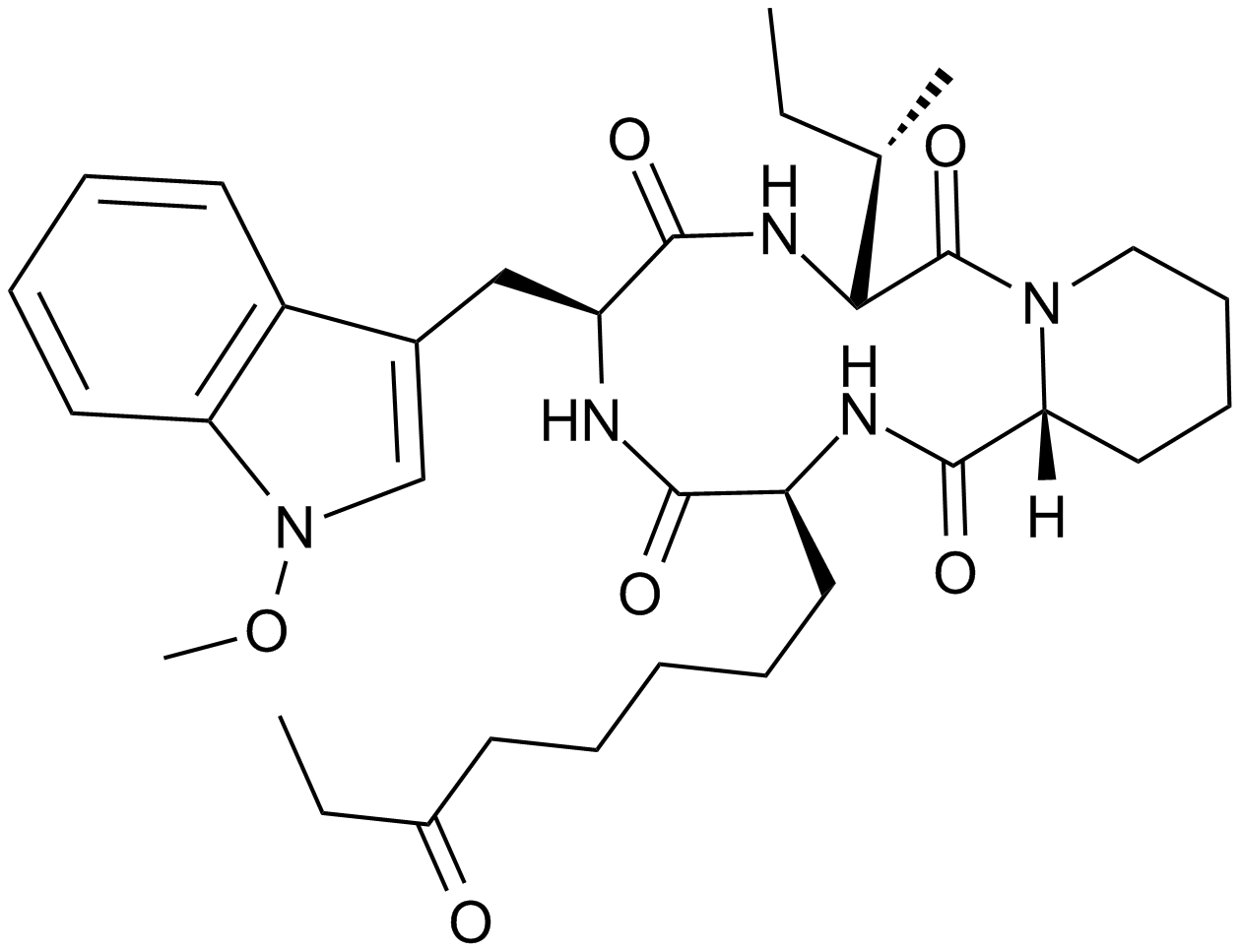
GC12961
Apicidin
Ein HDAC-Inhibitor, der die Zellmembran durchdringen kann.
-
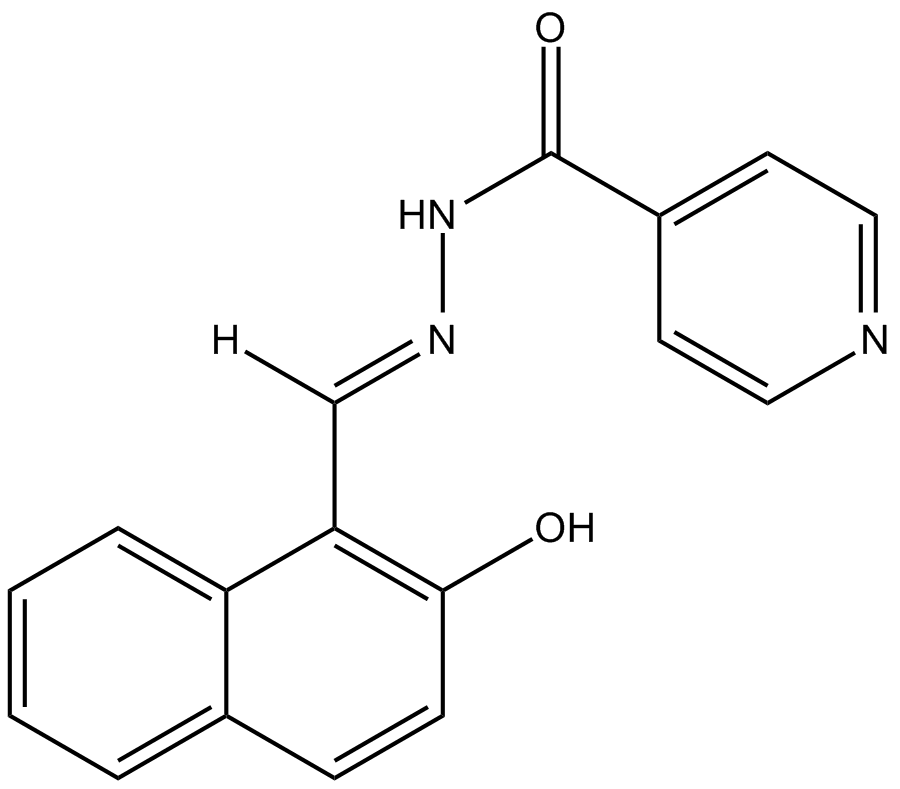
GC17820
AS8351
AS8351 (NSC51355) ist ein KDM5B-Inhibitor, der aktive Chromatinmarkierungen induzieren und aufrechterhalten kann, um die Induktion von Kardiomyozyten-Ähnlichen Zellen zu erleichtern.
-
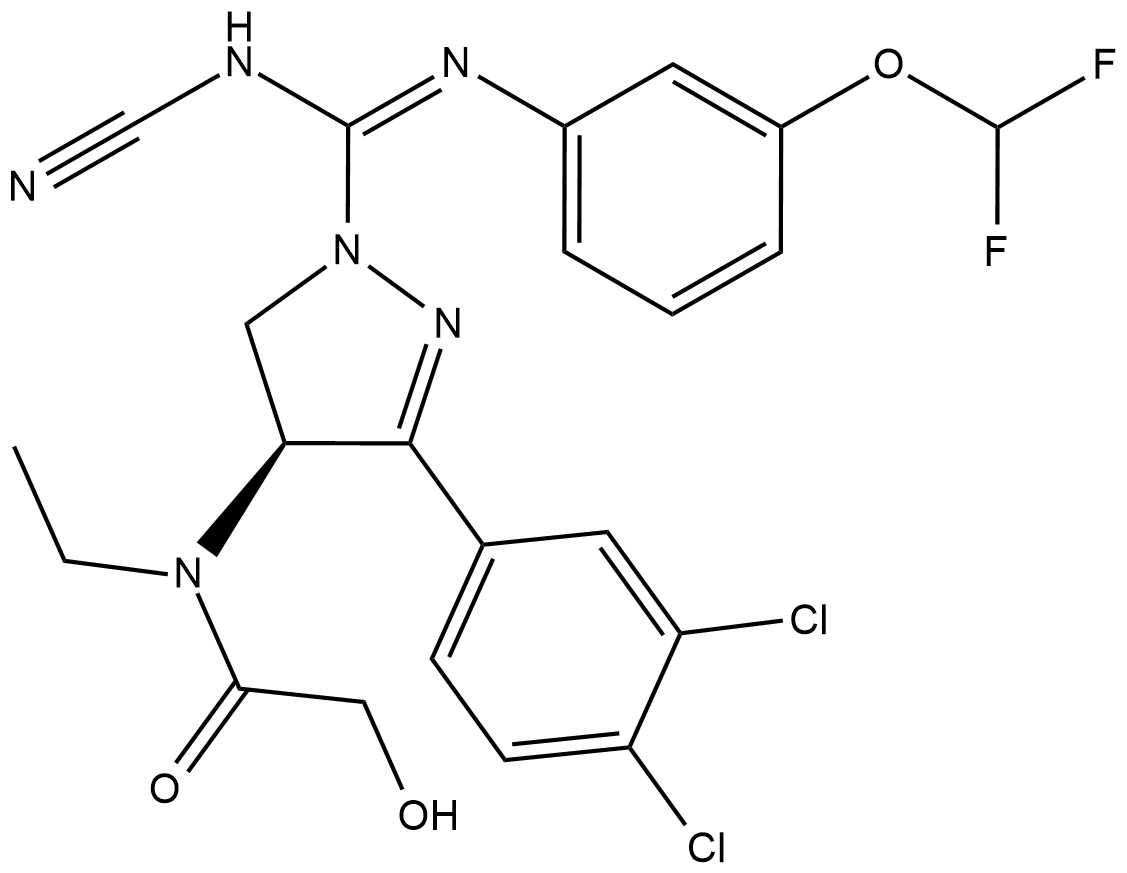
GC18159
BAY-598
BAY-598 ist ein selektiver niedermolekularer Inhibitor von SMYD2 mit einem IC50 von 27 nM.
-
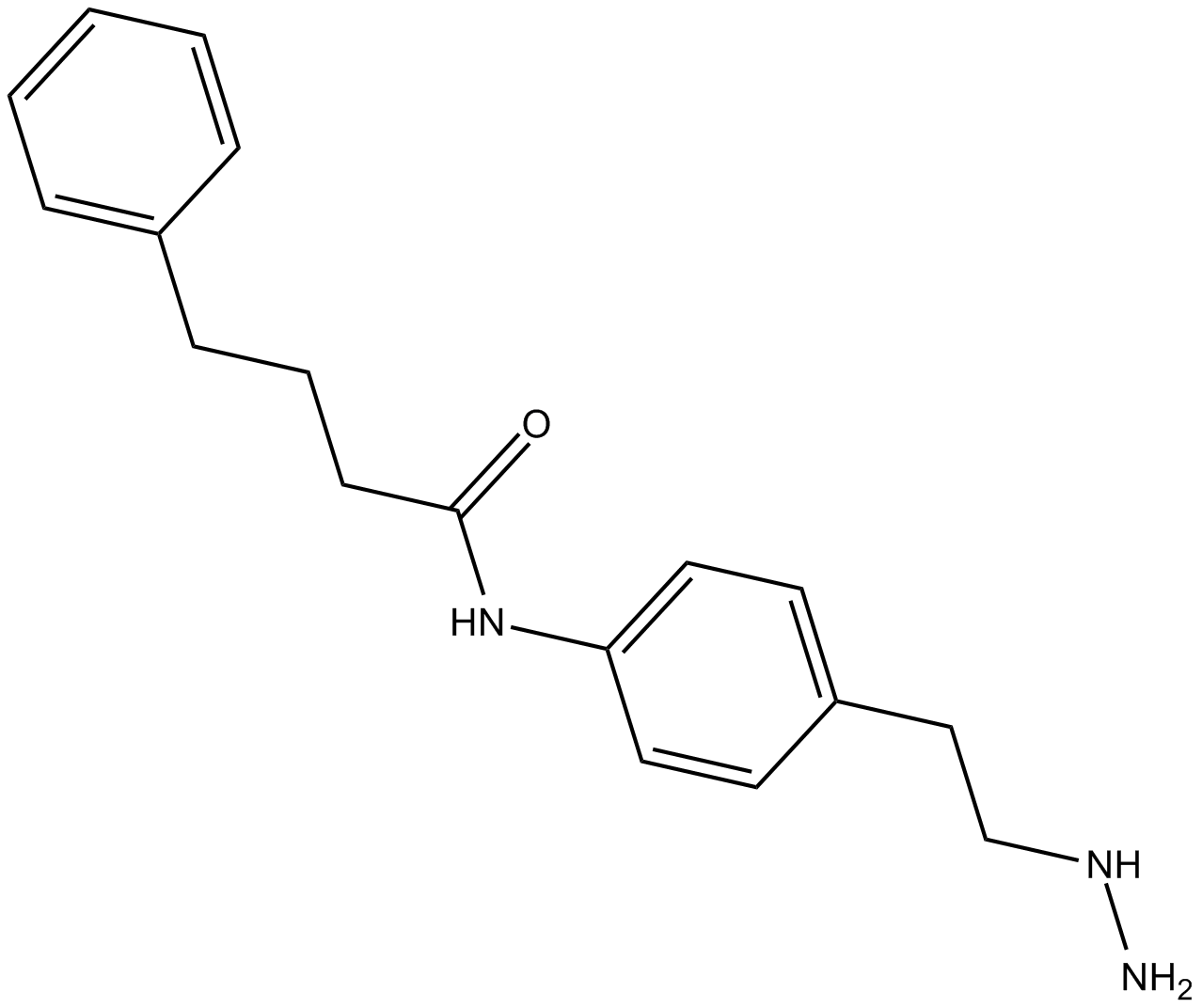
GC16114
Bizine
Bizin, ein Phenelzin-Analogon, ist ein potenter und selektiver LSD1-Hemmer mit einem b>Ki von 59 nM.
-
GC64865
Bomedemstat
Bomedemstat (IMG-7289) ist ein oral aktiver und irreversibler Hemmer der Lysin-spezifischen Demethylase 1 (LSD1). Bomedemstat kann die H3K4- und H3K9-Methylierung erhÖhen und dann die Genexpression verÄndern. Bomedemstat zeigt krebshemmende AktivitÄten, hemmt die Proliferation von Krebszellen und induziert Apoptose.
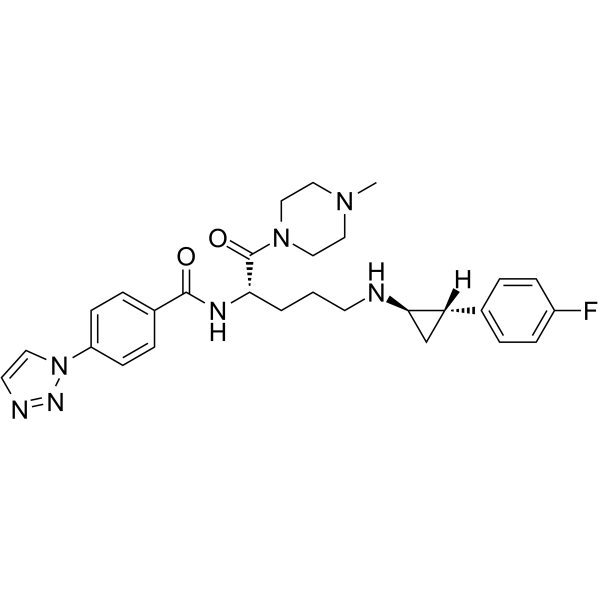
-
GC67921
Bomedemstat ditosylate

-
GC65879
Bomedemstat hydrochloride
Bomedemstat (IMG-7289) Hydrochlorid ist ein oral aktiver und irreversibler Lysin-spezifischer Demethylase-1 (LSD1)-Hemmer. Bomedemstathydrochlorid kann die H3K4- und H3K9-Methylierung erhÖhen und dann die Genexpression verÄndern. Bomedemstat-Hydrochlorid zeigt krebshemmende AktivitÄten, hemmt die Proliferation von Krebszellen und induziert Apoptose.
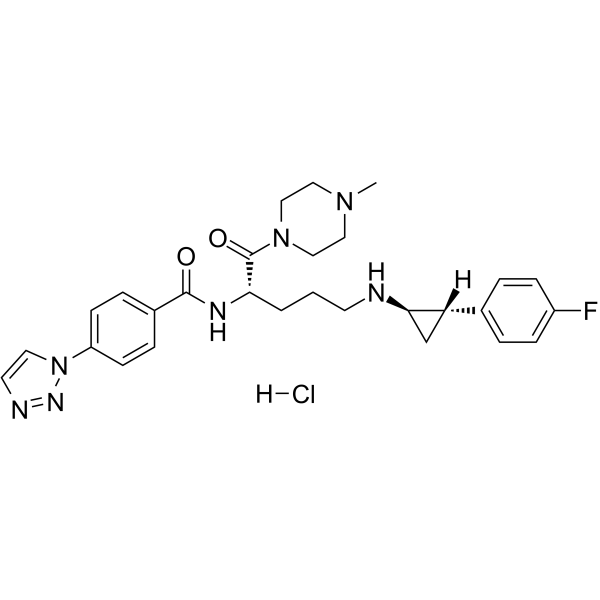
-
GC35621
CBB1003
CBB1003 ist ein neuartiger Histon-Demethylase-LSD1-Inhibitor mit einem IC50-Wert von 10,54 uM.
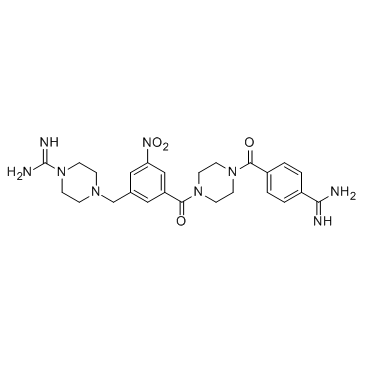
-
GC35622
CBB1003 hydrochloride
CBB1003 Hcl ist ein neuartiger Histon-Demethylase-LSD1-Inhibitor mit einem IC50-Wert von 10,54 uM.
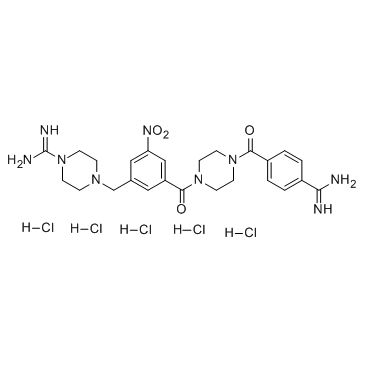
-
GC14151
CBB1007
CBB1007 ist eine zellgÄngige Amidino-Guanidinium-Verbindung, die als potenter, reversibler und substratkompetitiver LSD1-selektiver Inhibitor wirkt (IC50 = 5,27 μM fÜr hLSD1).
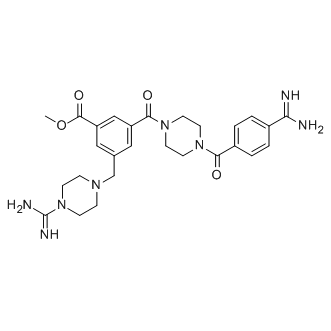
-
GC35623
CBB1007 hydrochloride
CBB1007 Hcl ist eine zellgÄngige Amidino-Guanidinium-Verbindung, die als potenter, reversibler und substratkompetitiver LSD1-selektiver Inhibitor wirkt (IC50 = 5,27 μM fÜr hLSD1).
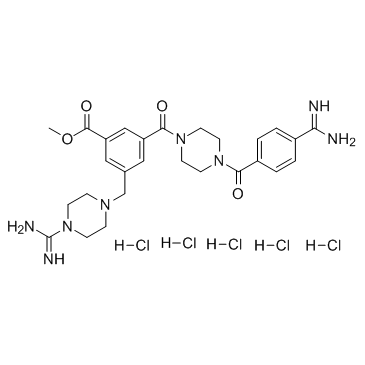
-
GC35624
CBB1007 trihydrochloride
CBB1007-Trihydrochlorid ist eine zellgÄngige Amidino-Guanidinium-Verbindung, die als potenter, reversibler und substratkompetitiver LSD1-selektiver Inhibitor wirkt (IC50 = 5,27 μM fÜr hLSD1).
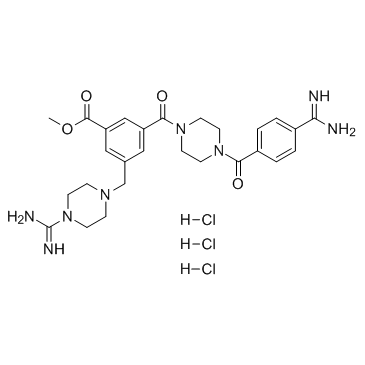
-
GC34165
Corin
Corin ist ein dualer Inhibitor der Histon-Lysin-spezifischen Demethylase (LSD1) und Histon-Deacetylase (HDAC) mit einem Ki(inact) von 110 nM fÜr LSD1 und einem IC50 von 147 nM fÜr HDAC1.
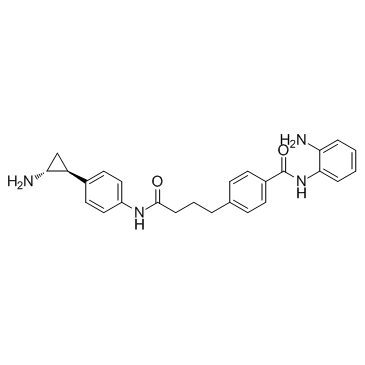
-
GC25304
CP2
CP2 is a cyclic peptide that inhibits the JmjC histone demethylases KDM4 with IC50 values of 42 nM and 29 nM for KDM4A and KDM4C, respectively.
-
GC10774
CPI-455
KDM5-Inhibitor
-
GC25305
CPI-455 HCl
CPI-455 HCl is a specific KDM5 inhibitor with a half-maximal inhibitory concentration (IC50) of 10 ± 1 nM for full-length KDM5A in enzymatic assays, elevating global levels of H3K4 trimethylation (H3K4me3) and decreased the number of DTPs in multiple cancer cell line models treated with standard chemotherapy or targeted agents.This product has poor solubility, animal experiments are available, cell experiments please choose carefully!
-
GC15830
Daminozide
Selective inhibitor of KDM2/7 histone demethylases
-
GC33013
DDP-38003 dihydrochloride
DDP-38003 Dihydrochlorid ist ein neuartiger, oral verfÜgbarer Inhibitor der Histon-Lysin-spezifischen Demethylase 1A (KDM1A/LSD1) mit einem IC50 von 84 nM.
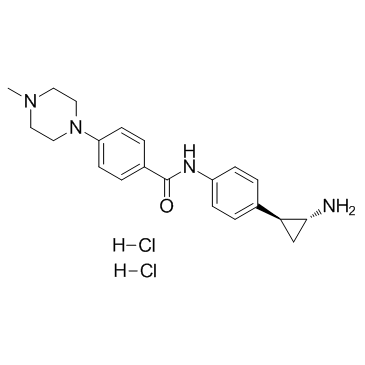
-
GC34296
DDP-38003 trihydrochloride
DDP-38003 Trihydrochlorid ist ein neuartiger, oral verfÜgbarer Inhibitor der Histon-Lysin-spezifischen Demethylase 1A (KDM1A/LSD1) mit einem IC50 von 84 nM.
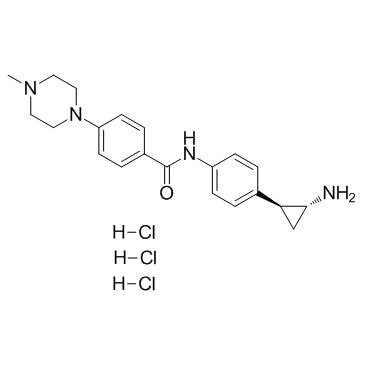
-
GC34314
GSK 690 Hydrochloride
GSK 690 (Hydrochlorid) ist ein reversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1) mit einem Kd-Wert von 9 nM und einem biochemischen IC50-Wert von 37 nM.
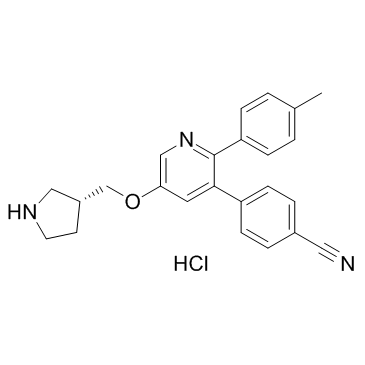
-
GC10617
GSK J1
A dual inhibitor of JMJD3 and UTX
-
GC13086
GSK J2
GSK J2 ist ein Isomer von GSK-J1, das keine spezifische Aktivität aufweist. GSK-J1 ist ein potenter Inhibitor der H3K27me3/me2-Demethylasen JMJD3/KDM6B und UTX/KDM6A.
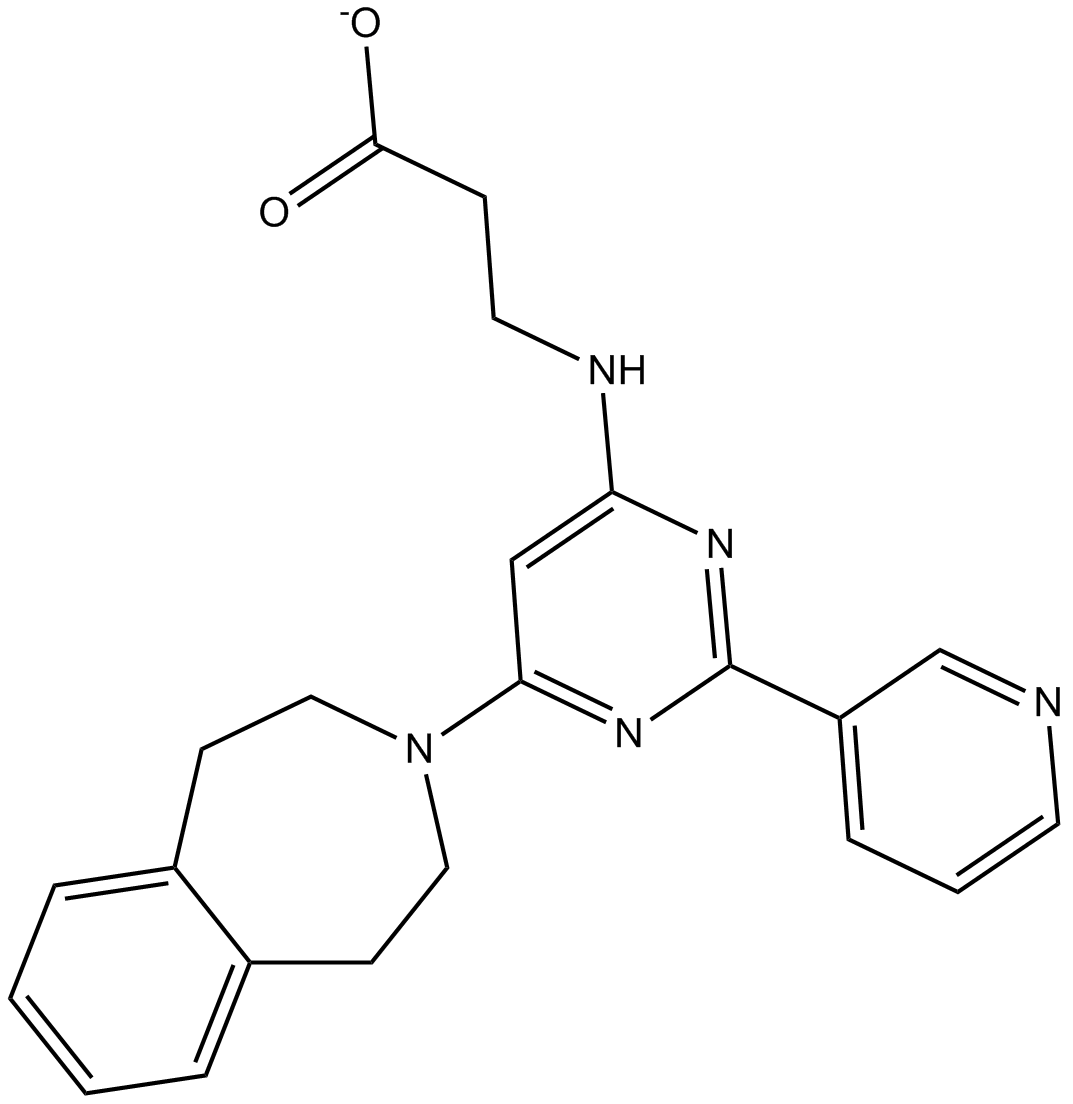
-
GC12997
GSK J4 free base
Die freie Base GSK J4 ist ein potenter dualer Inhibitor der H3K27me3/me2-Demethylasen JMJD3/KDM6B und UTX/KDM6A mit IC50-Werten von 8,6 bzw. 6,6 μM. Die freie Base von GSK J4 inhibiert LPS-induziertes TNF-⋱ Produktion in humanen primÄren Makrophagen mit einem IC50 von 9 μM. GSK J4 ist ein zelldurchlÄssiges Prodrug von GSK-J1. Die freie Base von GSK J4 induziert eine stressbedingte Apoptose des endoplasmatischen Retikulums.
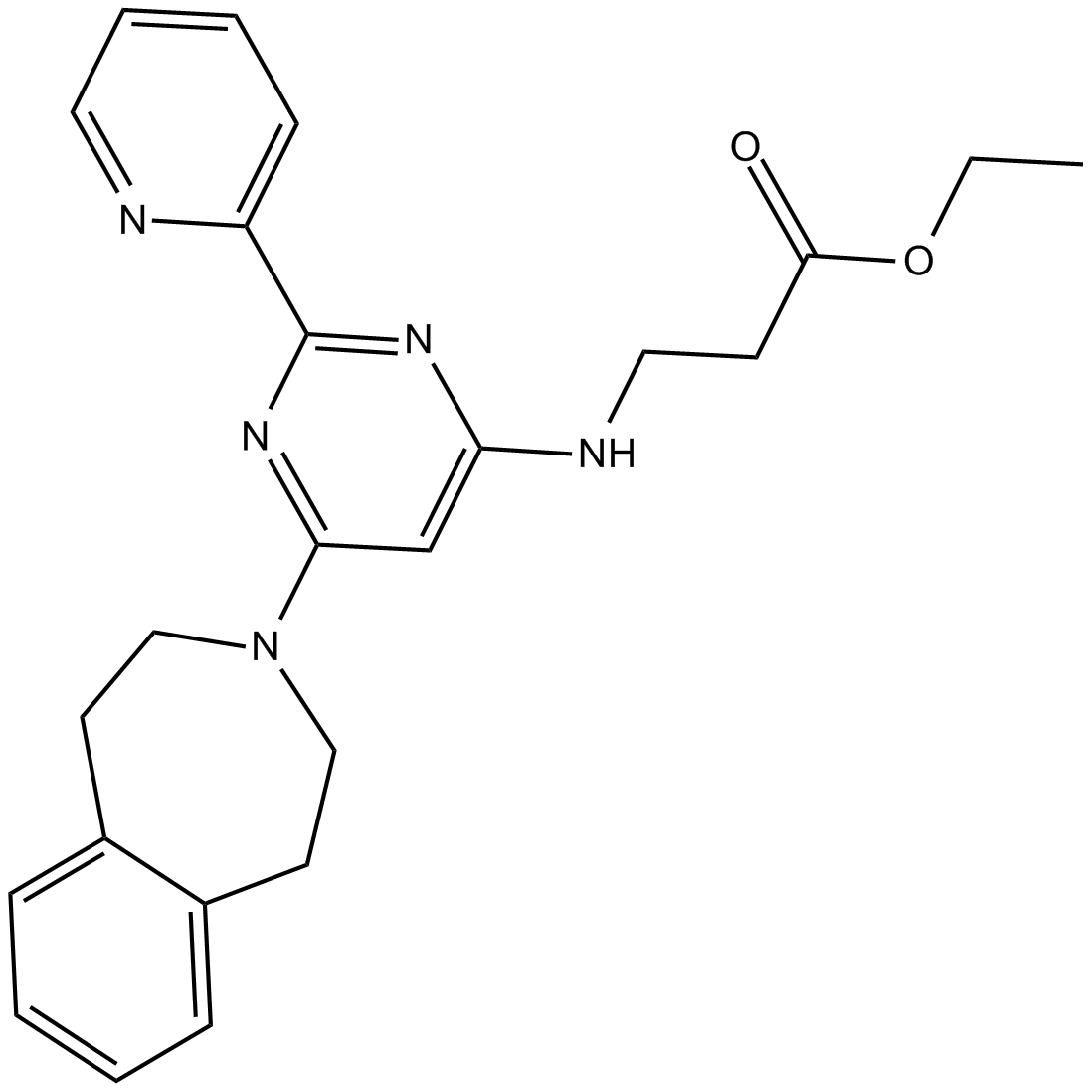
-
GC15497
GSK J4 HCl
Prodrug eines selektiven H3K27-Histon-Demethylase-Inhibitors.
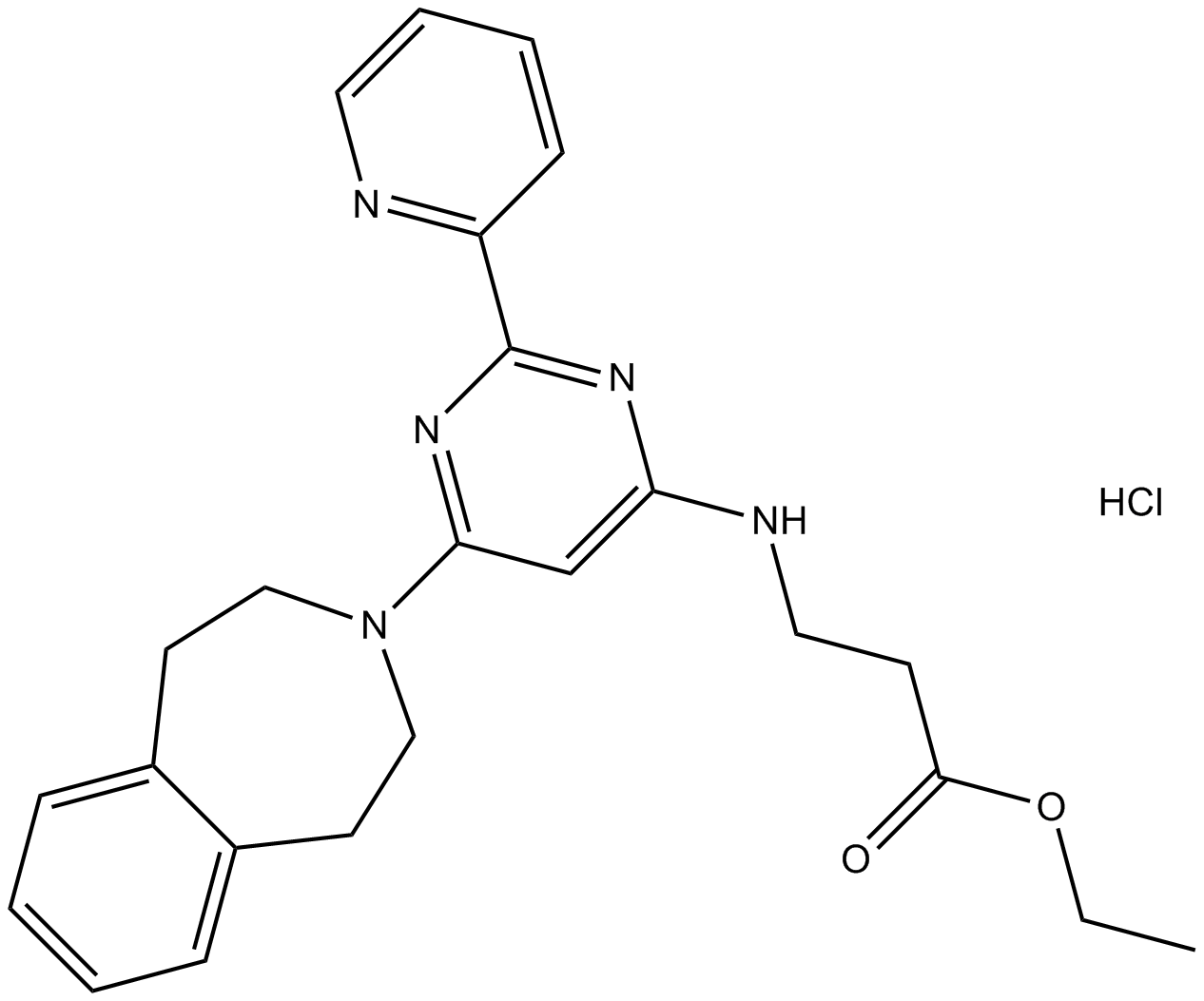
-
GC17118
GSK J5
inactive isomer of GSK J4, KDM inhibitor
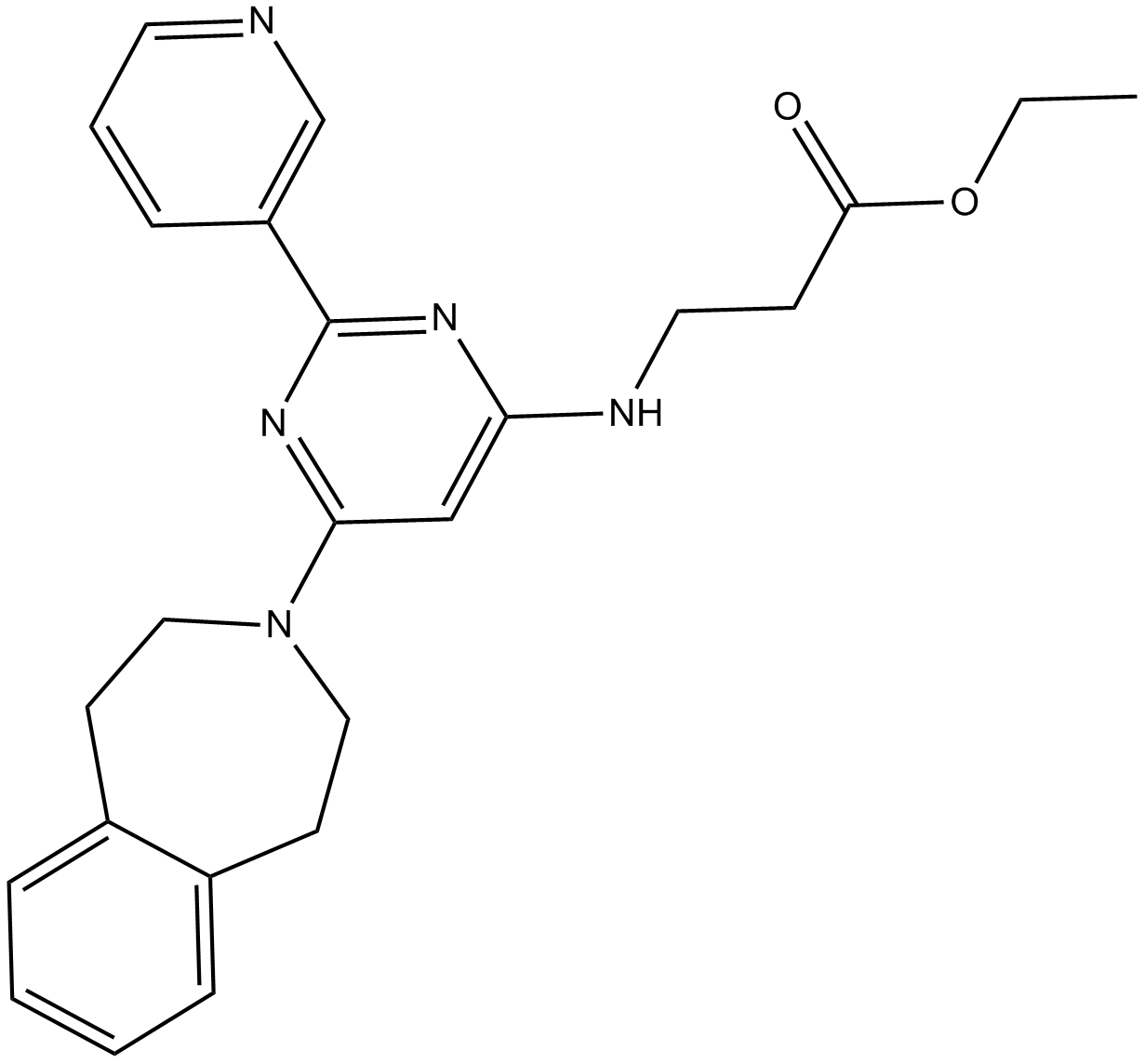
-
GC36195
GSK-J1 lithium salt
GSK-J1-Lithiumsalz ist ein potenter Inhibitor der H3K27me3/me2-Demethylasen JMJD3/KDM6B und UTX/KDM6A mit einem IC50-Wert von 60 nM gegenÜber KDM6B.
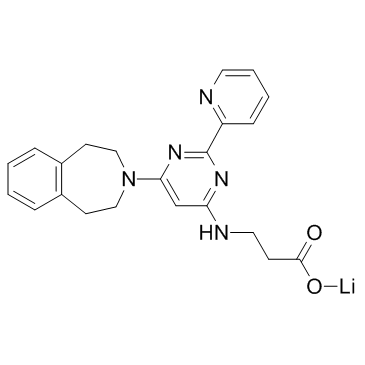
-
GC43792
GSK-J2 (sodium salt)
The histone H3 lysine 27 (H3K27) demethylase JMJD3 plays important roles in the transcriptional regulation of cell differentiation, development, the inflammatory response, and cancer.
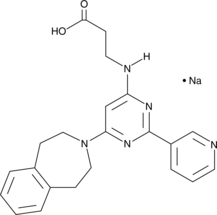
-
GC43794
GSK-J5 (hydrochloride)
GSK-J5 (Hydrochlorid) ist ein zellgängiges Esterderivat von GSK J2, inaktiv. GSK-J5 (Hydrochlorid) ist ebenfalls ein Isomer von GSK-J4 und wird häufig als negative Gruppe verwendet.
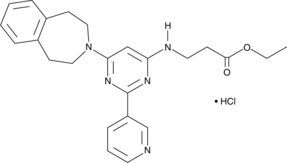
-
GC18012
GSK-LSD1 (hydrochloride)
LSD1 inhibitor
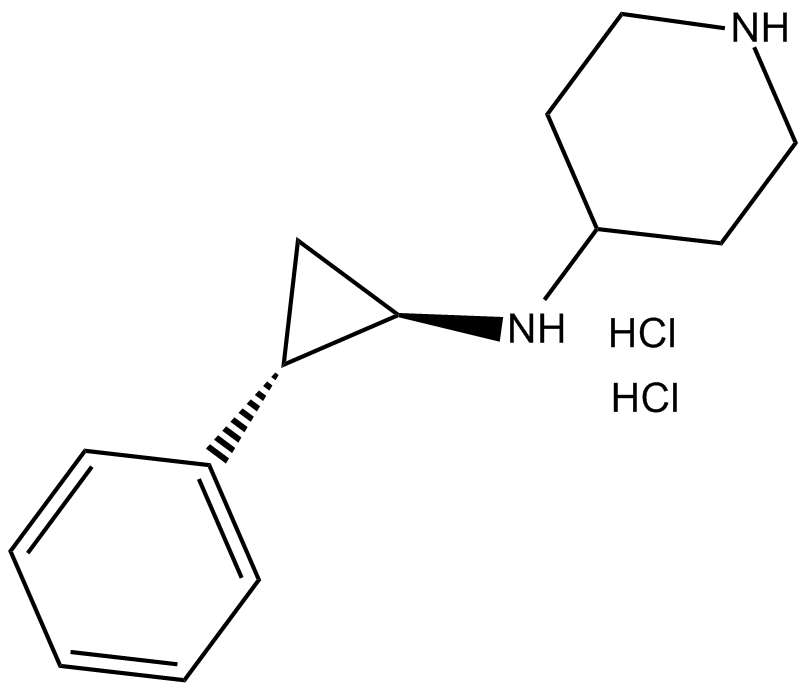
-
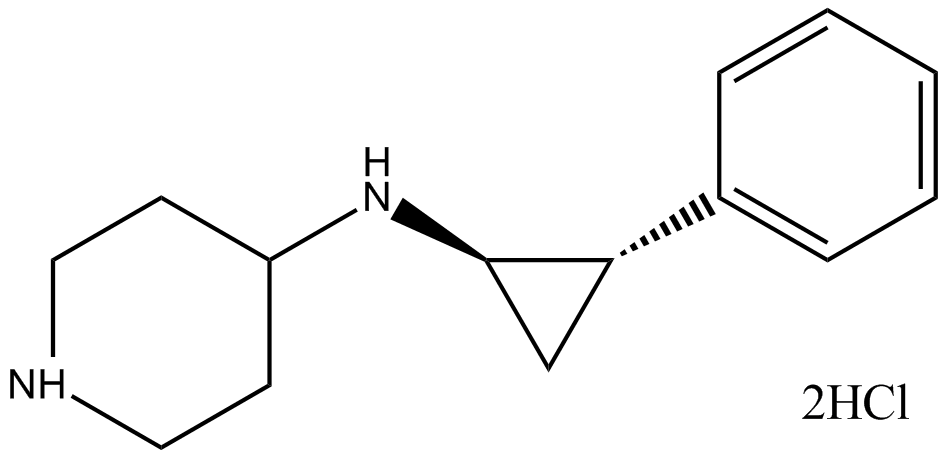
GC15368
GSK-LSD1 2HCl
irreversible, and selective LSD1 inhibitor
-
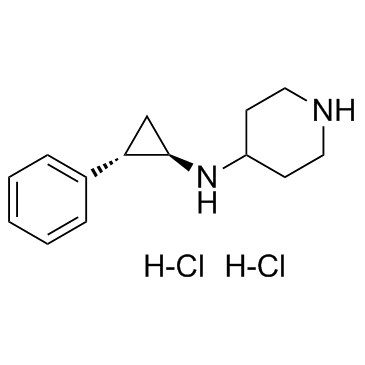
GC32764
GSK-LSD1 Dihydrochloride
GSK-LSD1-Dihydrochlorid ist ein potenter, selektiver und irreversibler Hemmer der Lysin-spezifischen Demethylase 1 (LSD1) mit einem IC50-Wert von 16 nM.
-
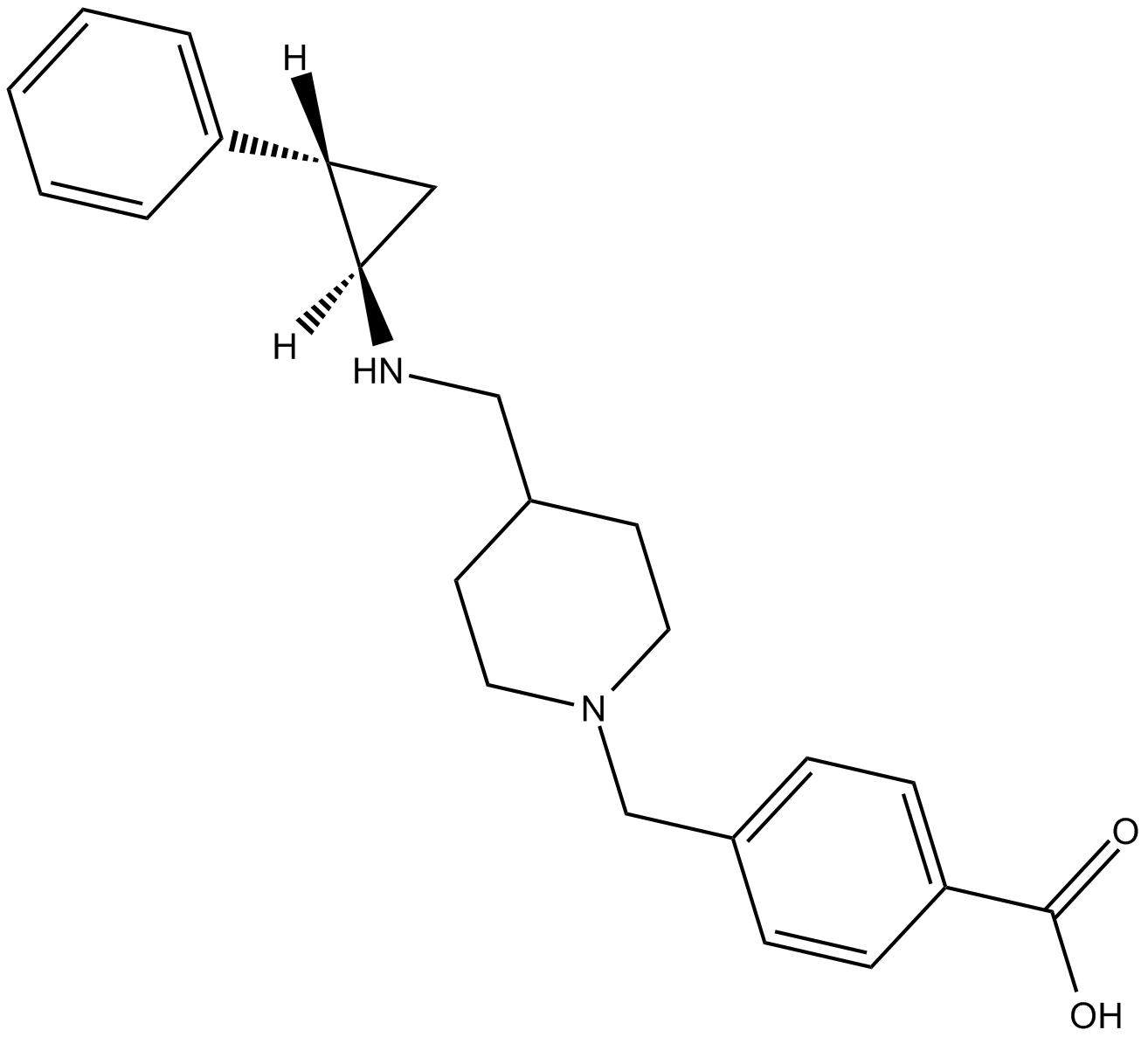
GC11578
GSK2879552
GSK2879552 ein oral aktiver, selektiver und irreversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1/KDM1A) mit potenzieller antineoplastischer AktivitÄt.
-
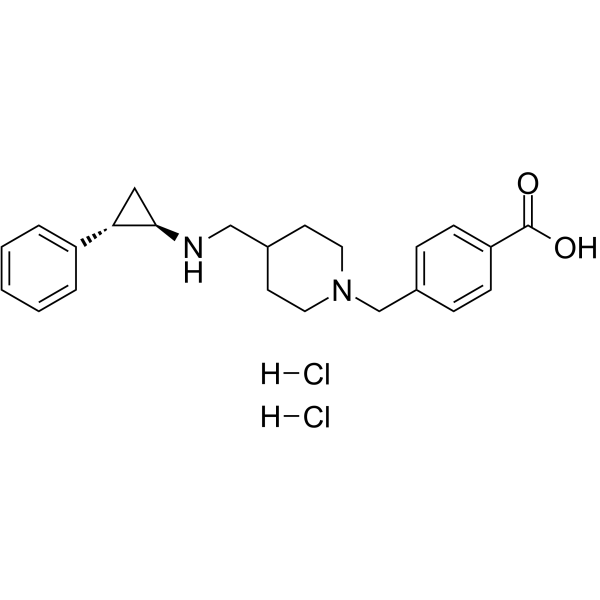
GC62719
GSK2879552 dihydrochloride
GSK2879552 Dihydrochlorid, ein oral aktiver, selektiver und irreversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1/KDM1A) mit potenzieller antineoplastischer AktivitÄt.
-
GC34604
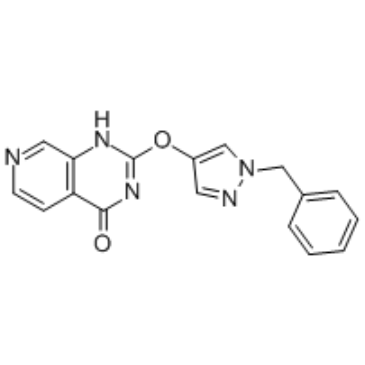
GSK467
GSK467 ist ein zellgÄngiger und selektiver KDM5B (JARID1B oder PLU1)-Inhibitor mit einem Ki von 10 nM und einem IC50 von 26 nM. GSK467 zeigt eine 180-fache SelektivitÄt fÜr KDM4C und keine messbaren inhibitorischen Wirkungen gegenÜber KDM6 oder anderen Mitgliedern der Jumonji-Familie.
-
GC17754
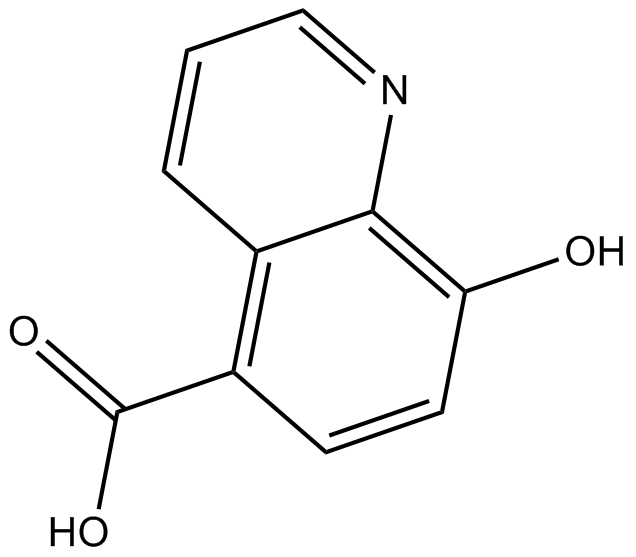
IOX 1
IOX 1, 5-Carboxy-8-hydroxychinolin, ist ein potenter Breitbandinhibitor von 2OG-Oxygenasen, einschließlich der JmjC-Demethylasen. IOX 1 hemmt KDM4C, KDM4E, KDM2A, KDM3A und KDM6B mit IC50-Werten von 0,6 μM, 2,3 μM, 1,8 μM, 0,1 μM bzw. 1,4 μM. IOX 1 hemmt auch ALKBH5.
-
GC15603
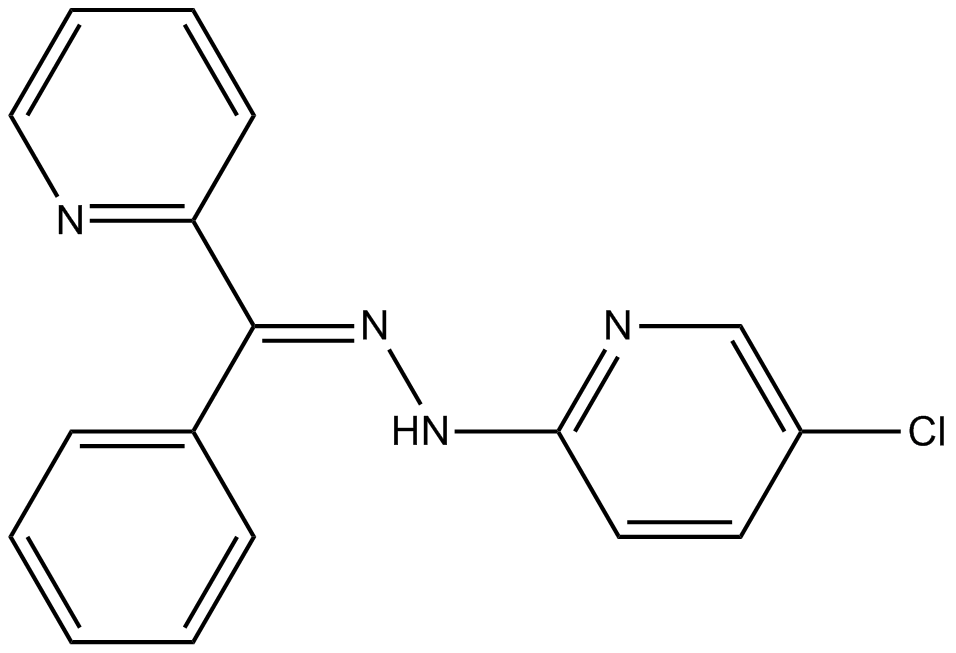
JIB-04
JIB-04 ist ein panselektiver Jumonji-Histondemethylase-Inhibitor mit IC50-Werten von 230, 340, 855, 445, 435, 1100 und 290 nM fÜr JARID1A, JMJD2E, JMJD3, JMJD2A, JMJD2B, JMJD2C bzw. JMJD2D.
-
GC64841
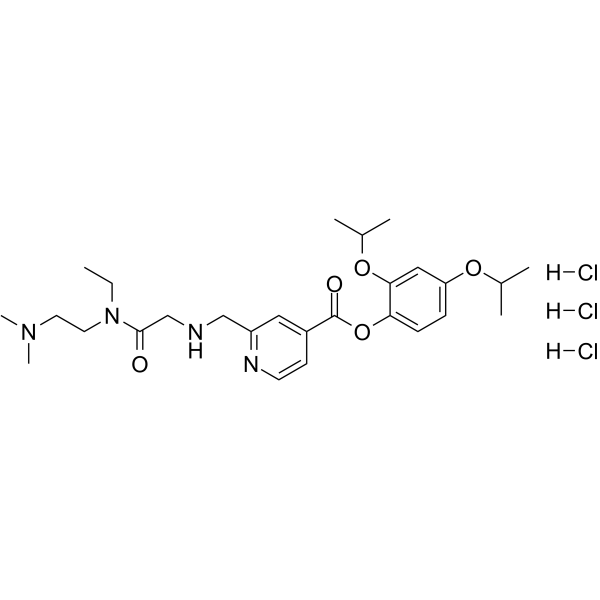
JQKD82 trihydrochloride
JQKD82 (JADA82) Trihydrochlorid ist ein zellgÄngiger und selektiver KDM5-Inhibitor. JQKD82-Trihydrochlorid erhÖht H3K4me3 und kann fÜr die Erforschung des multiplen Myeloms verwendet werden.
-
GC36388
KDM2A/7A-IN-1
KDM2A/7A-IN-1 ist ein erstklassiger, selektiver und zellgÄngiger Inhibitor von Histon-Lysin-Demethylasen. KDM2A/7A mit einem IC50-Wert von 0,16μM fÜr KDM2A weist eine 75-fache SelektivitÄt gegenÜber anderen JmjC-Lysin-Demethylasen auf und ist es inaktiv gegenÜber Methyltransferasen und Histonacetyltransferasen.
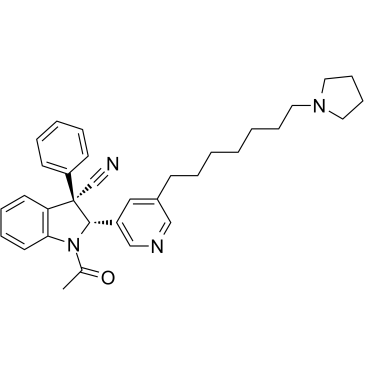
-
GC69327
KDM2B-IN-4
KDM2B-IN-4 ist ein Inhibitor der Histonde-Methyltransferase KDM2B. KDM2B-IN-4 kann für Krebsforschung verwendet werden. Weitere Informationen finden Sie im Patentdokument WO2016112284A1 unter Verwendung von Verbindung 182b.
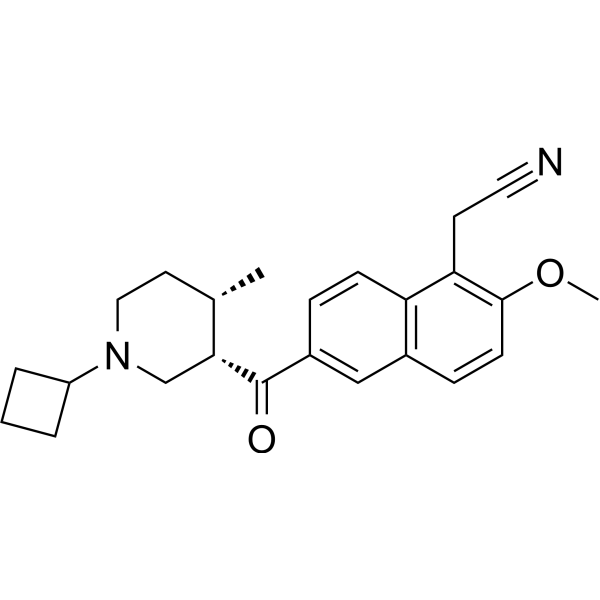
-
GC36389
KDM4-IN-2
KDM4-IN-2 (Verbindung 19a) ist ein potenter und selektiver dualer KDM4/KDM5-Inhibitor mit Kis von 4 und 7 nM fÜr KDM4A bzw. KDM5B.
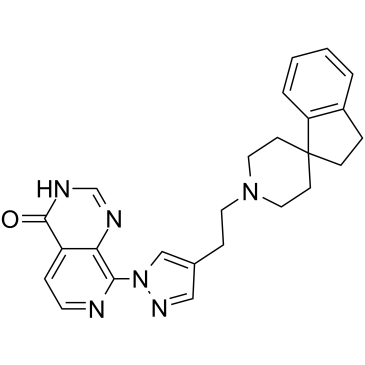
-
GC31887
KDM4D-IN-1
KDM4D-IN-1 ist ein neuer Inhibitor der Histon-Lysin-Demethylase 4D (KDM4D) mit einem IC50-Wert von 0,41 ± 0,03 μM.
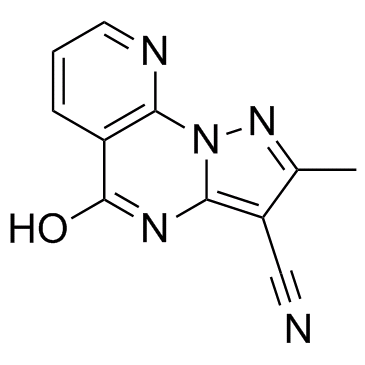
-
GC32790
KDM5-IN-1
KDM5-IN-1 ist ein potenter, selektiver und oral bioverfÜgbarer KDM5-Inhibitor mit einem IC50 von 15,1 nM.
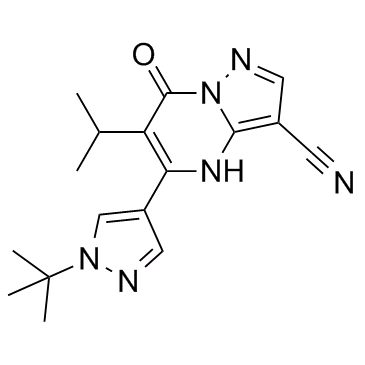
-
GC32842
KDM5A-IN-1
KDM5A-IN-1 ist ein potenter, oral bioverfÜgbarer Pan-Histon-Lysin-Demethylase-5 (KDM5)-Inhibitor mit IC50-Werten von 45 nM, 56 nM und 55 nM fÜr KDM5A, KDM5B bzw. KDM5C und mit einem EC50-Wert von 960 nM fÜr PC9 H3K4Me3. KDM5A-IN-1 ist deutlich weniger wirksam gegenÜber anderen KDM5B-Enzymen (1A, 2B, 3B, 4C, 6A, 7B).
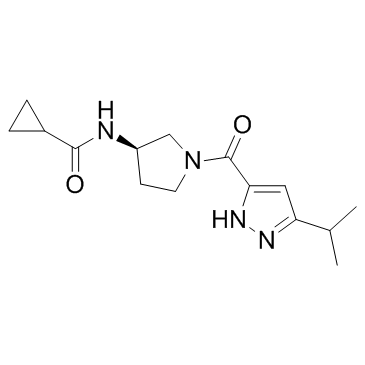
-
GC38709
KDOAM-25 citrate
KDOAM-25-Citrat ist ein potenter und hochselektiver Inhibitor der Histon-Lysin-Demethylase 5 (KDM5) mit IC50-Werten von 71 nM, 19 nM, 69 nM, 69 nM fÜr KDM5A, KDM5B, KDM5C bzw. KDM5D. KDOAM-25-Citrat erhÖht die globale H3K4-Methylierung an Transkriptionsstartstellen und beeintrÄchtigt die Proliferation in MM1S-Zellen des multiplen Myeloms.
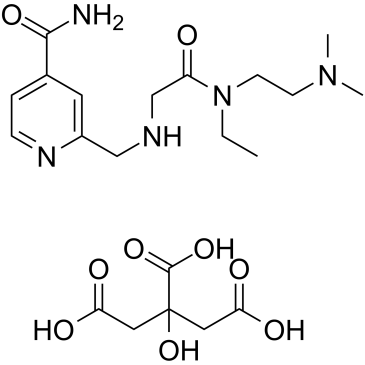
-
GC36484
LSD1-IN-5
LSD1-IN-5 (Verbindung 4e) ist ein potenter und reversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1) mit einem IC50 von 121 nM. LSD1-IN-5 erhÖht dimethyliertes Lys4 von Histon H3, zeigt keine Wirkung auf die Expression von LSD1.
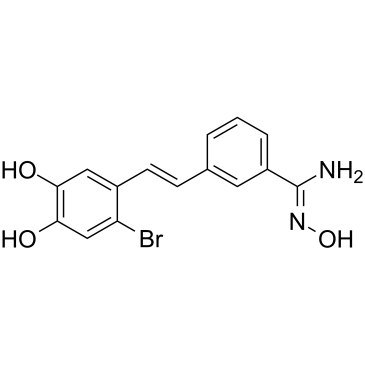
-
GC36485
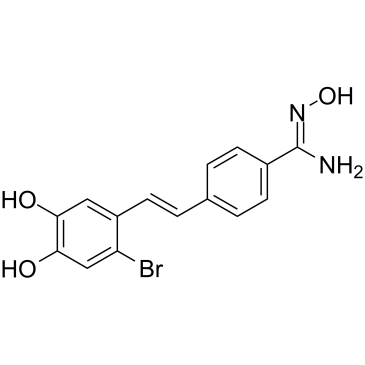
LSD1-IN-6
LSD1-IN-6 (Verbindung 4m) ist ein potenter und reversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1) mit einem IC50 von 123 nM. LSD1-IN-6 erhÖht dimethyliertes Lys4 von Histon H3, zeigt keine Wirkung auf die Expression von LSD1.
-
GC62434
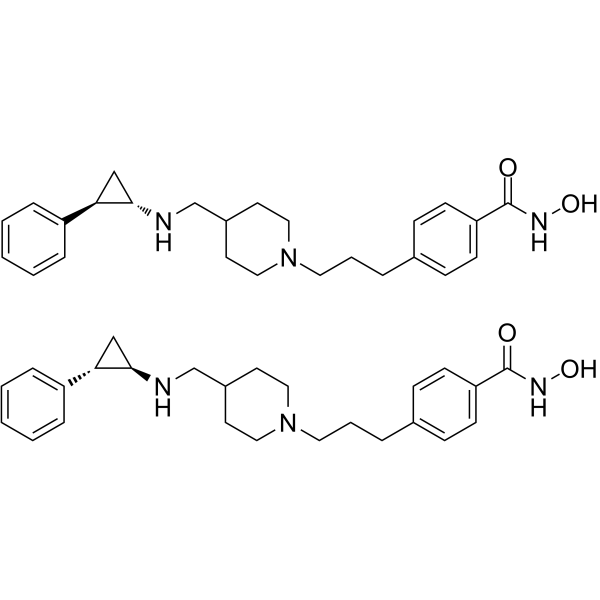
LSD1/HDAC6-IN-1
LSD1/HDAC6-IN-1 ist ein oral aktiver dualer Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1)/Histondeacetylase 6 (HDAC6) mit AntitumoraktivitÄt. LSD1/HDAC6-IN-1 kann fÜr die Erforschung des multiplen Myeloms (MM) verwendet werden.
-
GC13534
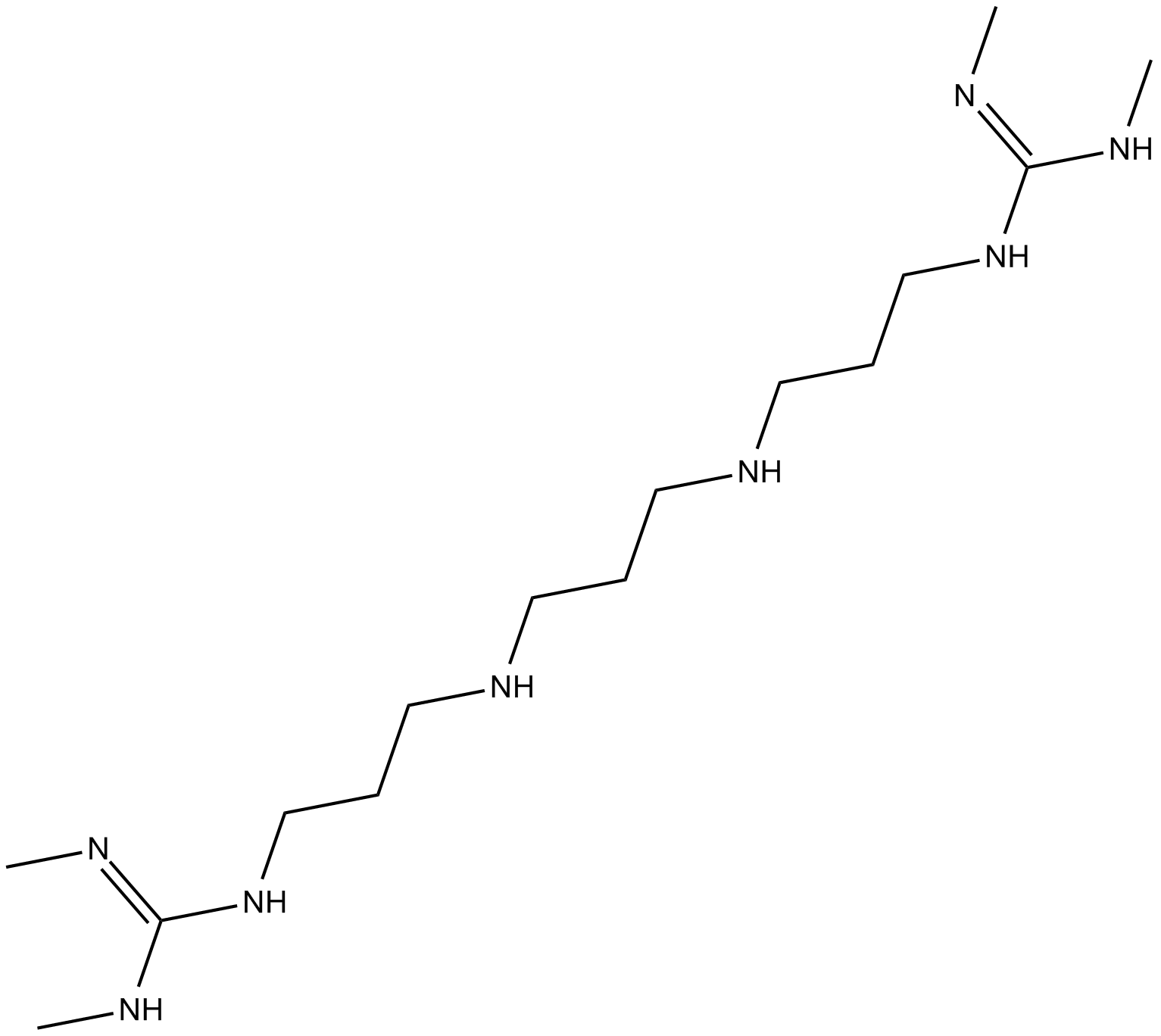
Lysine-specific Demethylase Inhibitor (1C)
lysine-specific demethylase Inhibitor
-
GC47586
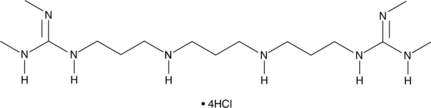
Lysine-specific Demethylase Inhibitor (1C) (hydrochloride)
An LSD1 inhibitor
-
GC65254
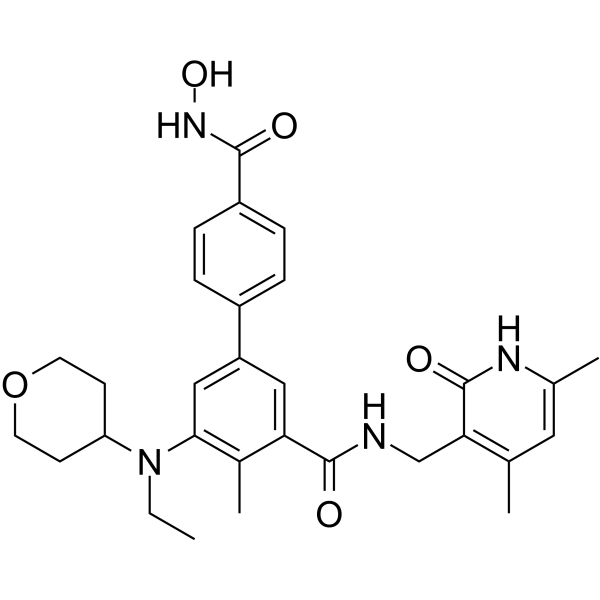
MC4355
MC4355 ist ein dualer Inhibitor von EZH2 und Histon-Deacetylase (HDAC).
-
GC44184
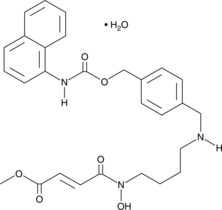
Methylstat (hydrate)
Methylstat is a methyl ester prodrug of a Jumonji C domain-containing histone demethylase (JMJD) inhibitor that has favorable cell permeability.
-
GC12557
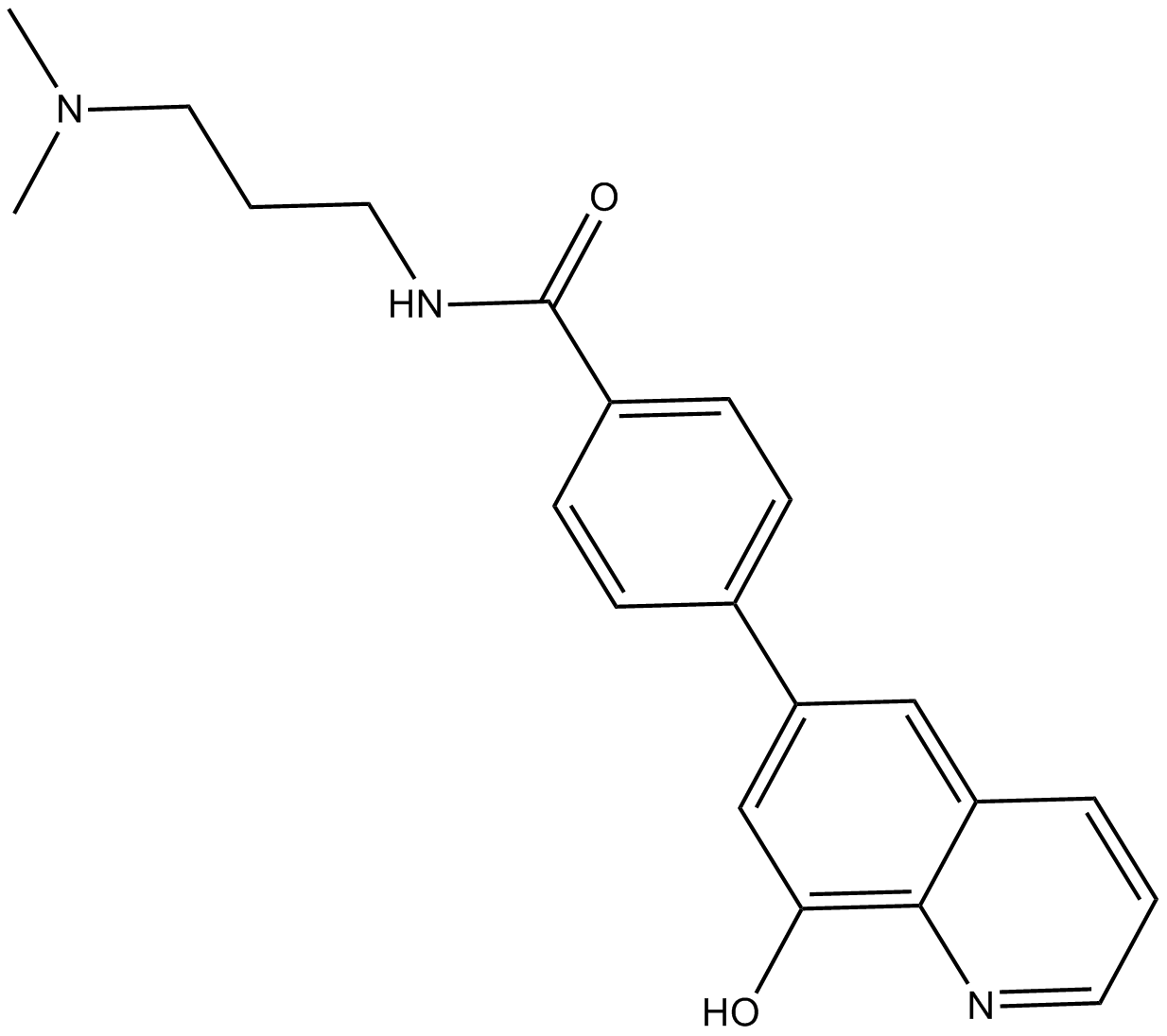
ML324
ML324 ist ein potenter JMJD2-Demethylase-Inhibitor mit antiviraler AktivitÄt. ML324 zeigt auch eine Hemmung der Histon-Demethylase KDM4B mit einer IC50 von 4,9 μM. ML324 hat eine starke antivirale AktivitÄt gegen Infektionen mit dem Herpes-simplex-Virus (HSV) und dem humanen Cytomegalovirus (hCMV) durch Hemmung der viralen IE-Genexpression.
-
GC12458
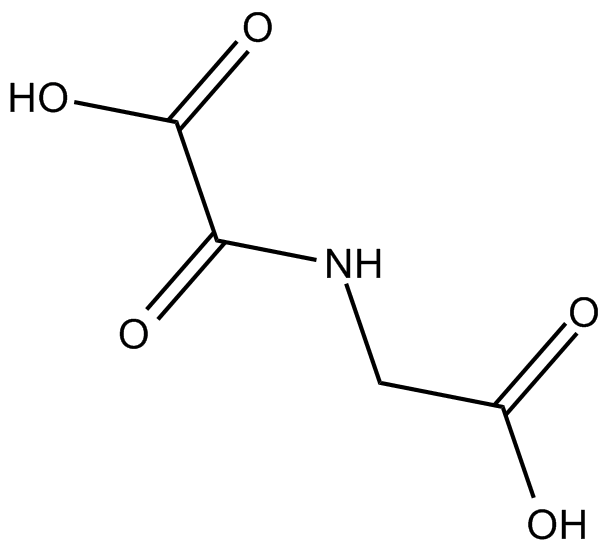
N-Oxalylglycine
cell permeable inhibitor of α-ketoglutarate-dependent enzymes, including JMJD2A, JMJD2C, and JMJD2E.
-
GC65138
NCDM-32B
NCDM-32B ist ein potenter und selektiver KDM4-Inhibitor, der die LebensfÄhigkeit und transformierende PhÄnotypen von Brustkrebs beeintrÄchtigt.
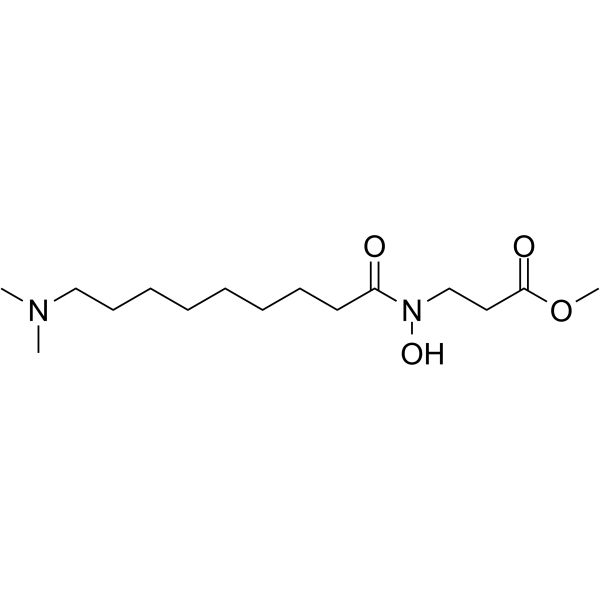
-
GC19410
NCGC00244536
NCGC00244536 ist ein potenter KDM4B-Inhibitor mit einem IC50 von 10 nM.
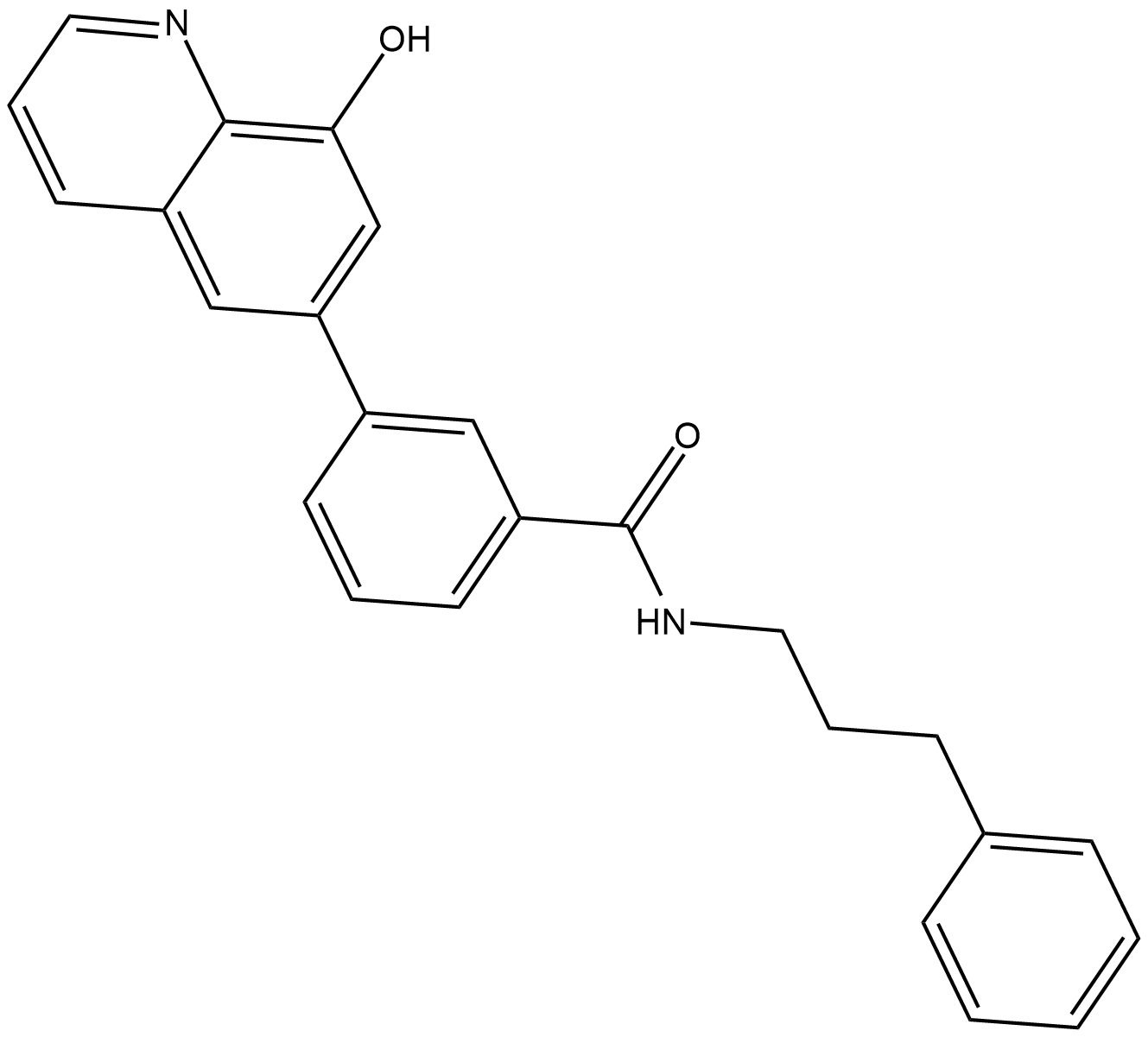
-
GC32896
NCGC00247743
NCGC00247743 ist ein Histon-Lysin-Demethylase-KDM4-Inhibitor.
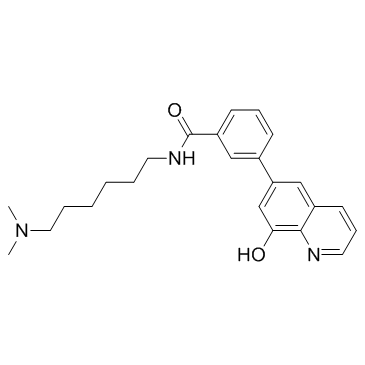
-
GC50136
NSC 636819
NSC 636819 ist ein kompetitiver und selektiver Inhibitor von KDM4A/KDM4B. KDM4A/KDM4B sind potenzielle Progressionsfaktoren für Prostatakrebs. NSC 636819 hat das Potenzial für die Erforschung von Krebserkrankungen, insbesondere Prostatakrebs.
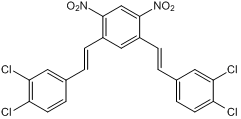
-
GC17314
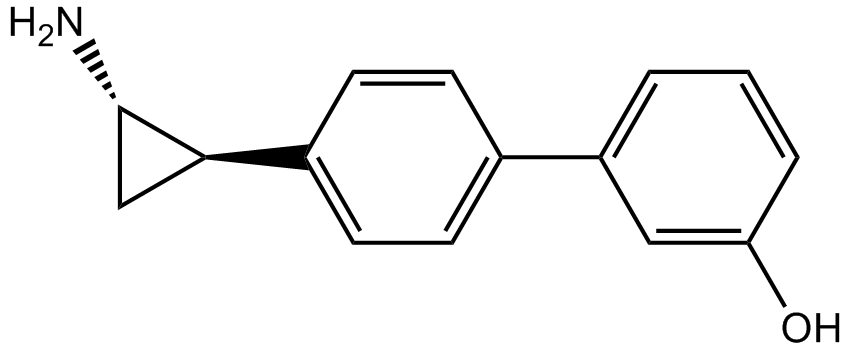
OG-L002
OG-L002 ist ein potenter und hochselektiver LSD1-Hemmer mit einem IC50 von 0,02 μM.
-
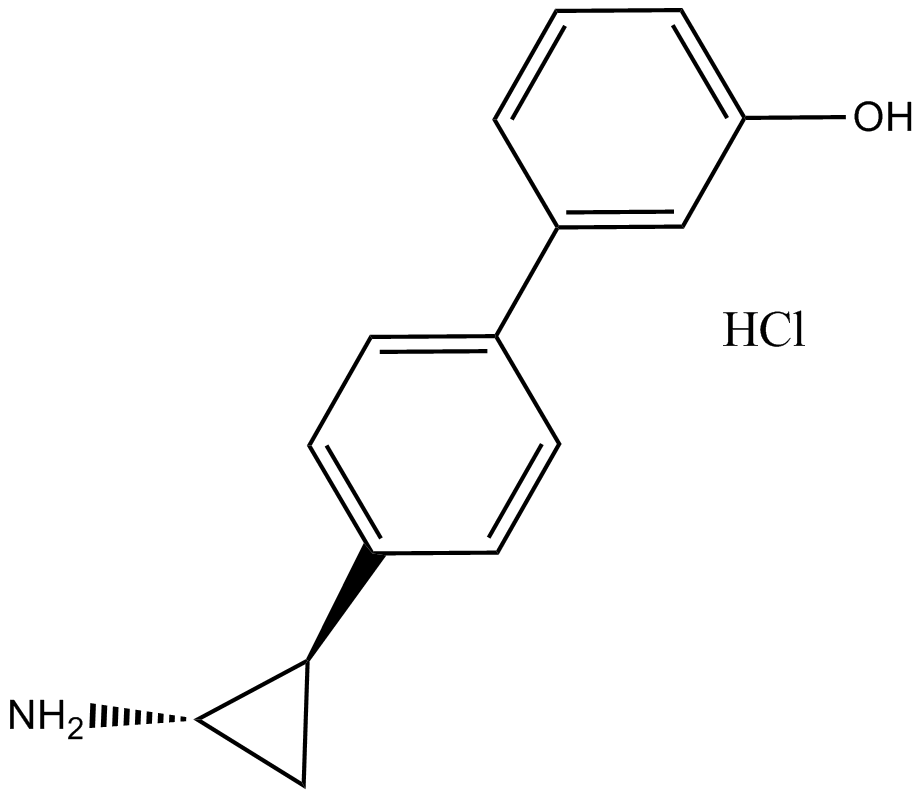
GC11792
OG-L002 HCl
LSD1 inhibitor,potent and specific
-
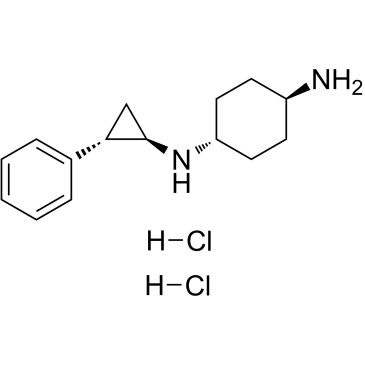
GC36818
ORY-1001(trans)
Iadademstat (ORY-1001) Dihydrochlorid ist ein selektiver irreversibler Lysin (K)-spezifischer Demethylase 1A (KDM1A/LSD1)-Hemmer.
-
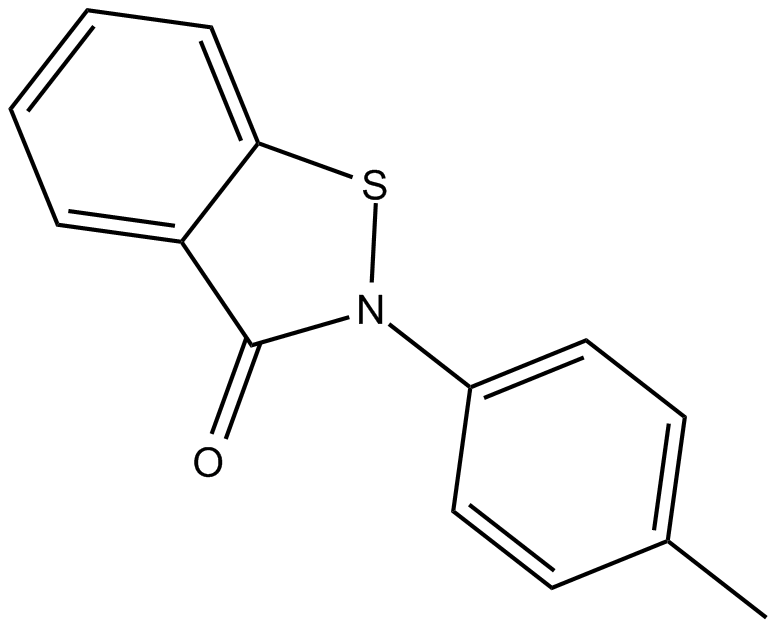
GC15301
PBIT
PBIT ist ein spezifischer Inhibitor der Enzyme Jumonji AT-rich Interactive Domain 1 (JARID1). PBIT hemmt die Histon-Demethylase JARID1B (KDM5B oder PLU1) mit einem IC50 von etwa 3 μM . PBIT hemmt auch JARID1A und JARID1C mit IC50-Werten von 6 μM bzw. 4,9 μM.
-
GC62678
PFI-90
PFI-90 ist ein selektiver Inhibitor der Histon-Demethylase (KDM3B), der die Wirkung von PAX3-FOXO1 hemmt. PFI-90 induziert Apoptose und myogene Differenzierung, was zu einem erhÖhten Zelltod fÜhrt. PFI-90 hat das Potenzial fÜr die AntitumoraktivitÄt. (Patent WO2021101929A1).
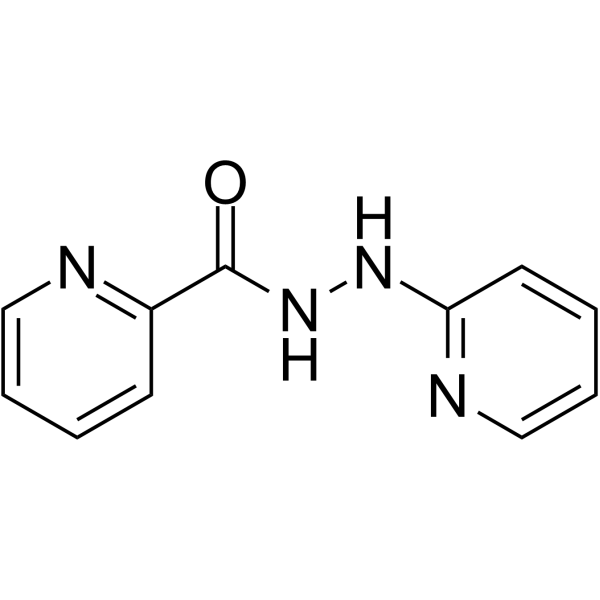
-
GC14066
Procaine
Procain ist ein DNA-demethylierendes Mittel.
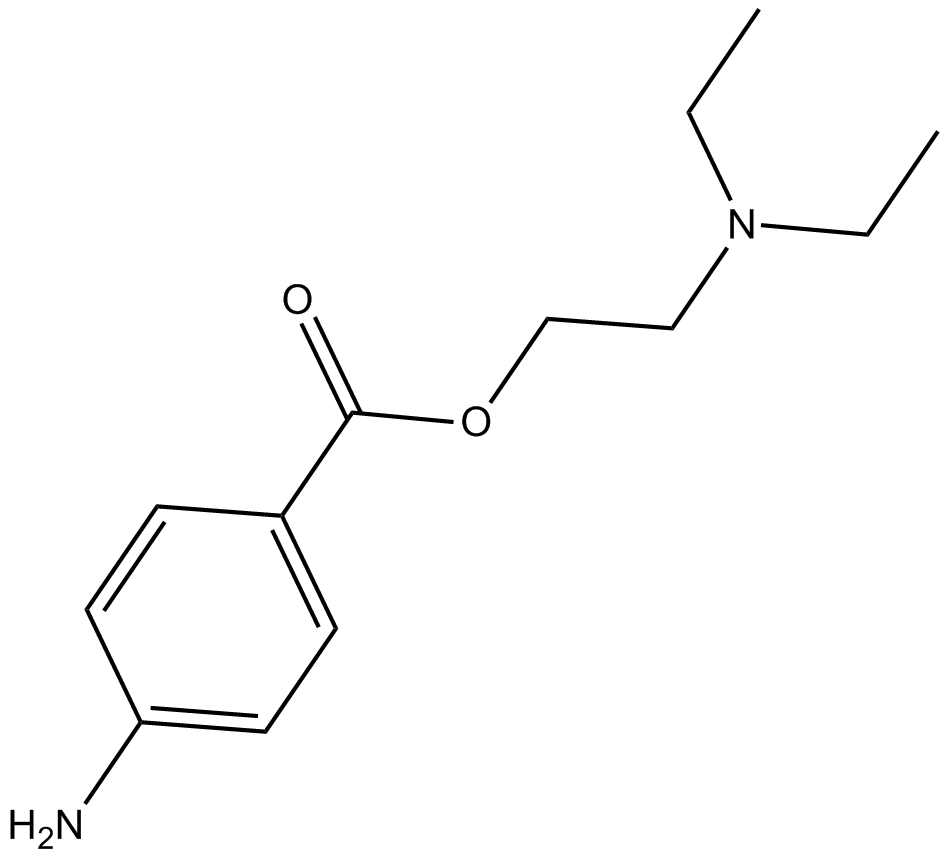
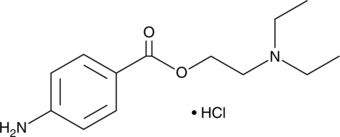
-
GC44685
Procaine (hydrochloride)
Procaine (hydrochloride) is an analytical reference standard categorized as a local anesthetic that is used as an adulterant.
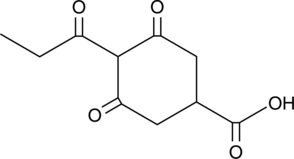
-
GC49116
Prohexadione
A plant growth regulator
-
GC32699
QC6352
QC6352 ist ein oral verfÜgbarer, selektiver und potenter KDM4C-Inhibitor mit einem IC50 von 35 nM.
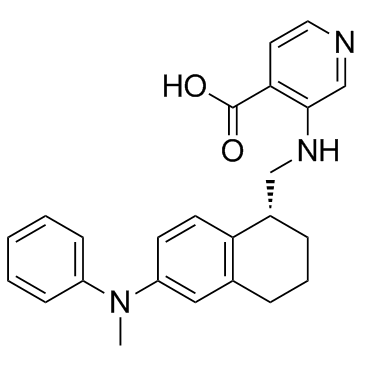
-
GC10148
RN 1 dihydrochloride
RN 1 Dihydrochlorid ist ein potenter, ins Gehirn eindringender, irreversibler und selektiver Lysin-spezifischer Demethylase 1 (LSD1)-Hemmer mit einem IC50 von 70 nM.
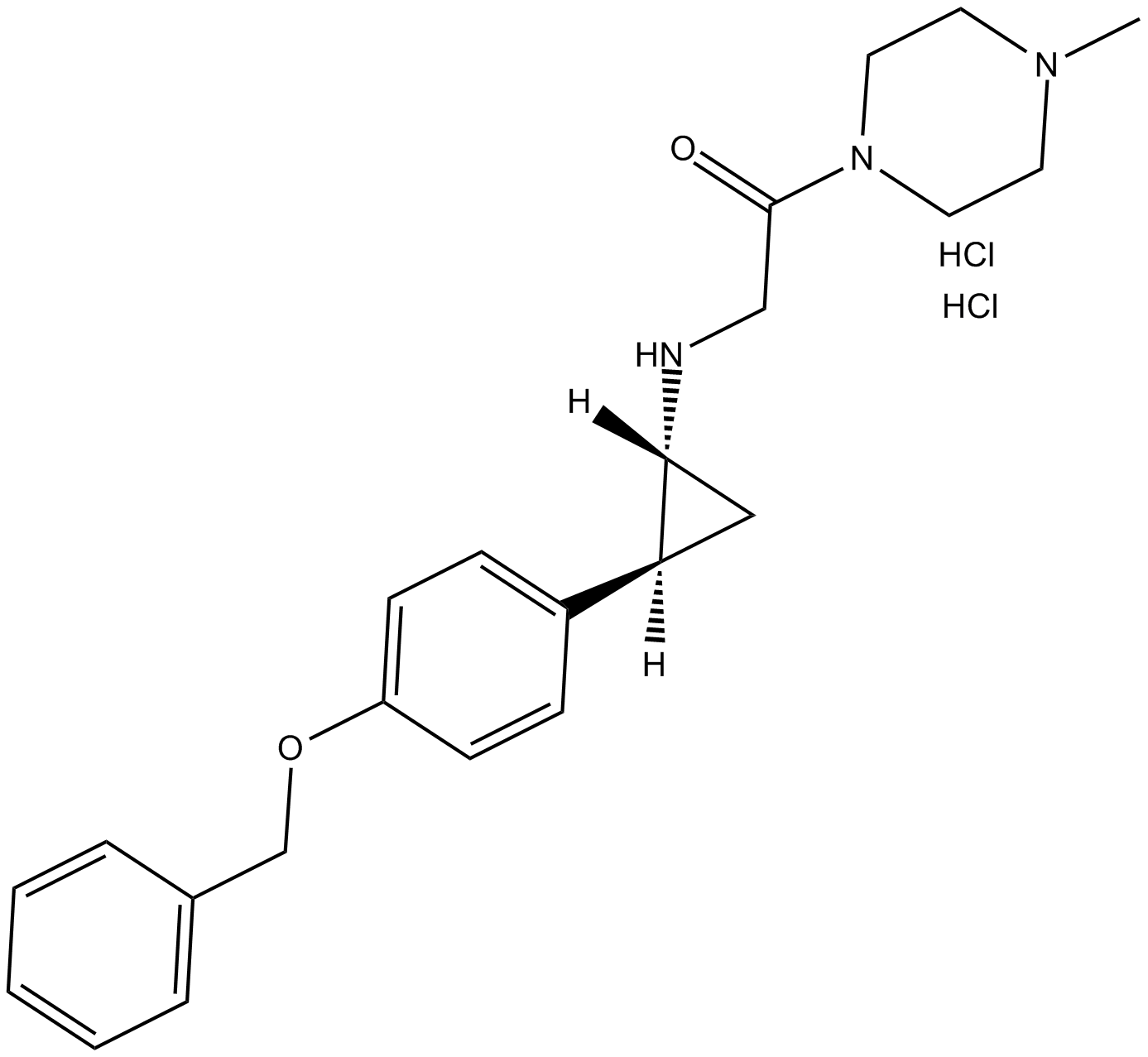
-
GC16743
RN-1 (hydrochloride)
LSD1 inhibitor
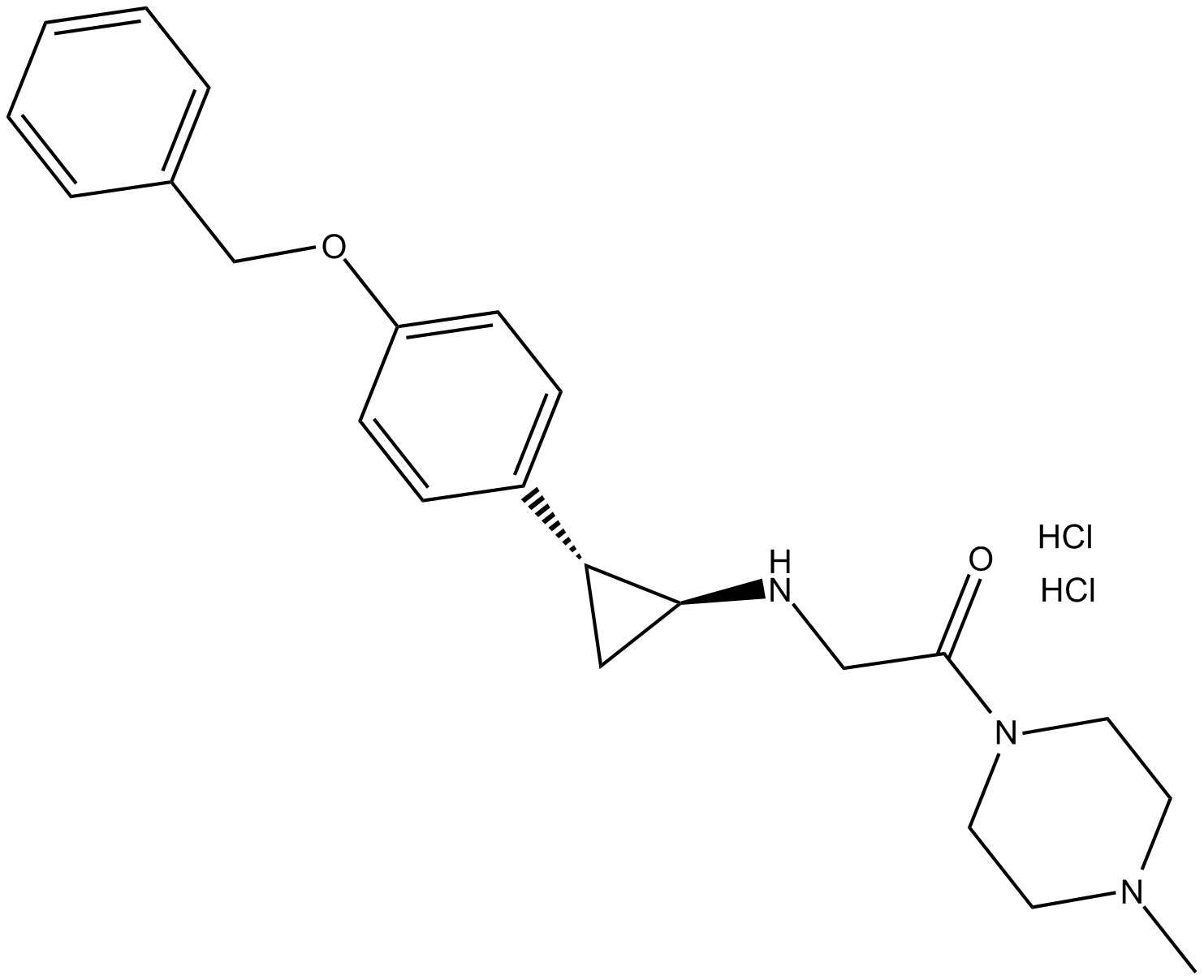
-
GC33339
S 2101
S 2101 ist ein Lysin-spezifischer Demethylase 1 (LSD1)-Hemmer mit einem IC50 von 0,99 μM, Ki von 0,61 μM und Kinact/Ki von 4560 M/s.
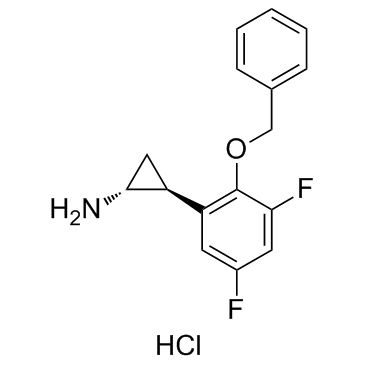
-
GC64303
S2116
S2116, ein N-alkyliertes Tranylcypromin (TCP)-Derivat, ist ein potenter Hemmer der Lysin-spezifischen Demethylase 1 (LSD1). S2116 erhÖht die H3K9-Methylierung und die reziproke H3K27-Deacetylierung in Super-Enhancer-Regionen. S2116 induziert Apoptose in TCP-resistenten T-Zell-Zellen der akuten lymphoblastischen LeukÄmie (T-ALL) durch Repression der Transkription der NOTCH3- und TAL1-Gene. S2116 verzÖgert signifikant das Wachstum von T-ALL-Zellen in xenotransplantierten MÄusen.
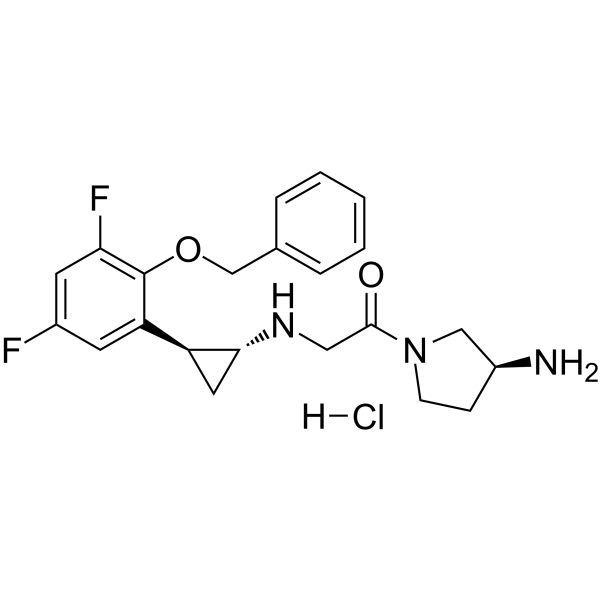
-
GC64902
S2157
S2157, ein N-alkyliertes Tranylcypromin (TCP)-Derivat, ist ein potenter Hemmer der Lysin-spezifischen Demethylase 1 (LSD1). S2157 erhÖht die H3K9-Methylierung und die reziproke H3K27-Deacetylierung in Super-Enhancer-Regionen. S2157 induziert Apoptose in TCP-resistenten T-Zell-Zellen der akuten lymphoblastischen LeukÄmie (T-ALL) durch Repression der Transkription der NOTCH3- und TAL1-Gene. S2157 passiert effizient die Blut-Hirn-Schranke und kann ZNS-LeukÄmie bei MÄusen, denen T-ALL-Zellen transplantiert wurden, fast vollstÄndig beseitigen.
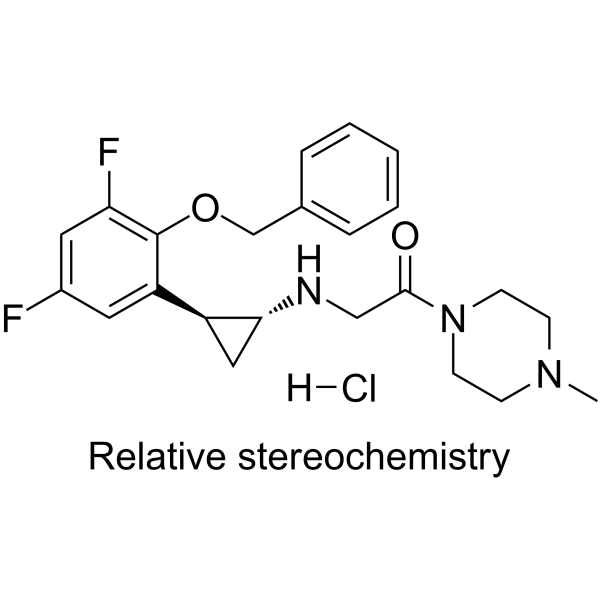
-
GC32820
Seclidemstat (SP-2577)
Seclidemstat (SP-2577) ist ein potenter, nicht kompetitiver und reversibler KDM1A (LSD1)-Hemmer (Ki=31 nM, IC50=13 nM). Seclidemstat (SP-2577) fÖrdert die Antitumor-ImmunitÄt bei Eierstockkrebs mit Switch/Sucrose Nonfermentable (SWI/SNF)-Mutation und hemmt die Virusproduktion, die virale DNA-Replikation und die spÄte Genexpression. Secldemstat (SP-2577) kann fÜr die Erforschung des Ewing-Sarkoms verwendet werden.
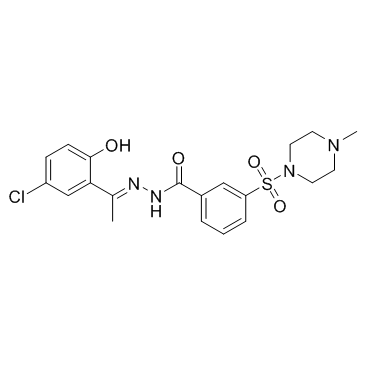
-
GC63456
Seclidemstat mesylate
Seclidemstat (SP-2577)-Mesylat ist ein potenter, nicht kompetitiver und reversibler KDM1A (LSD1)-Inhibitor (Ki=31 nM, IC50=13 nM). Seclidemstat-Mesylat fÖrdert die Antitumor-ImmunitÄt bei Eierstockkrebs mit Switch/Sucrose Nonfermentable (SWI/SNF)-Mutation und hemmt die Virusproduktion, die virale DNA-Replikation und die spÄte Genexpression. Secldemstat-Mesylat kann fÜr die Erforschung des Ewing-Sarkoms verwendet werden.
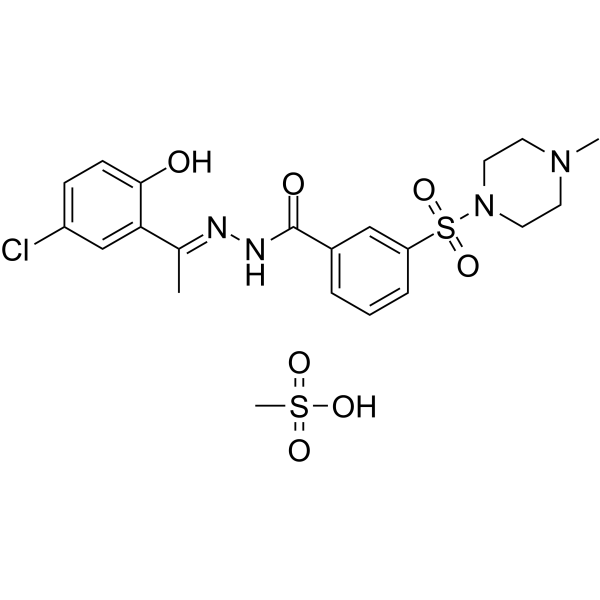
-
GC14631
SP2509
SP2509 ist ein potenter und selektiver Antagonist der Lysin-spezifischen Demethylase 1 (LSD1) mit einem IC50 von 13 nM.
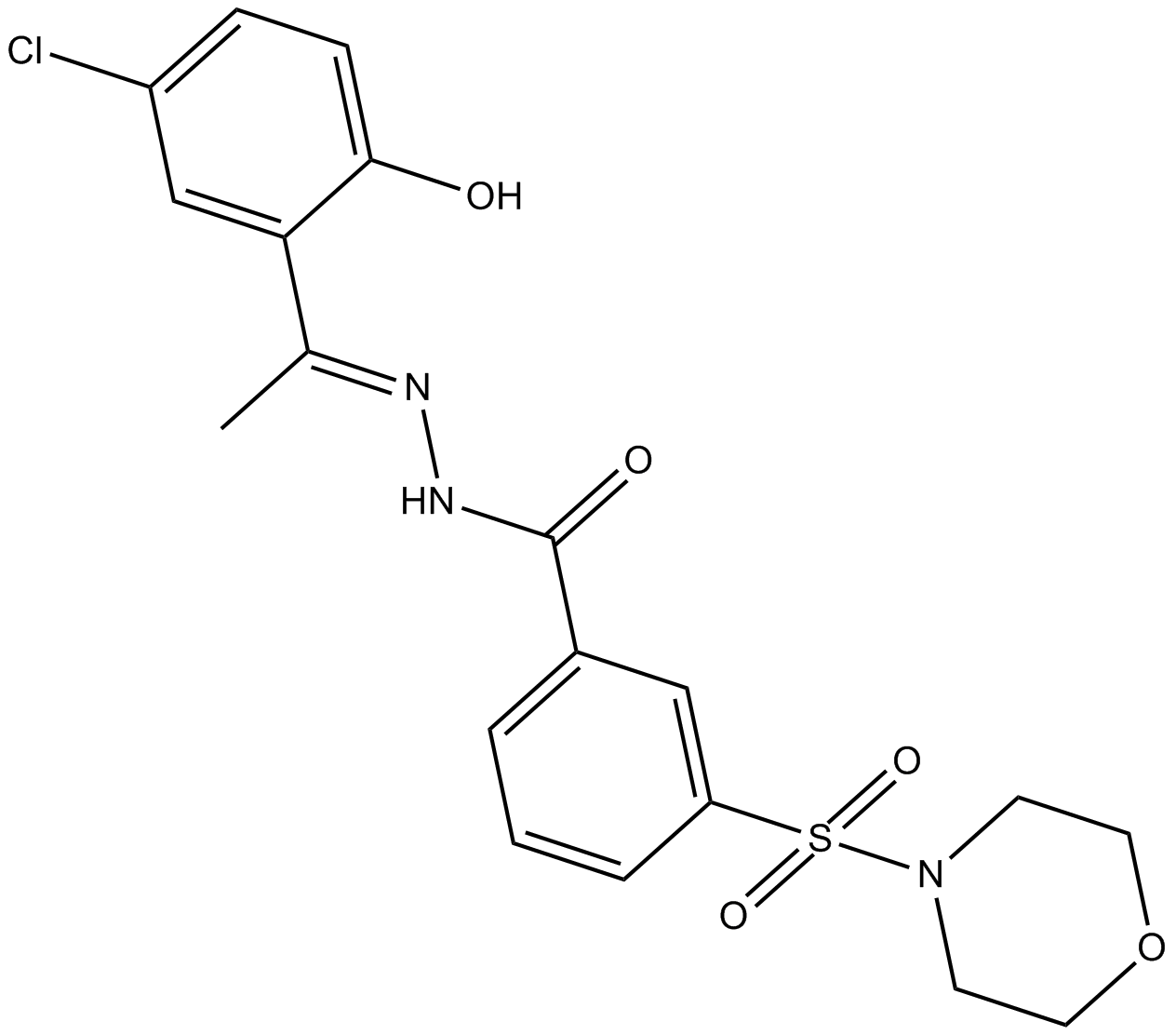
-
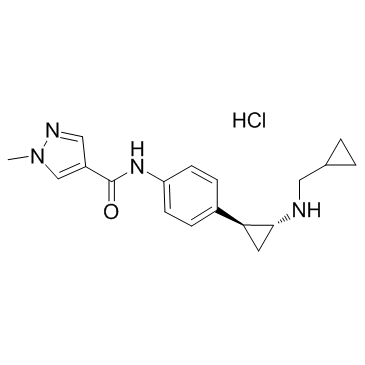
GC33038
T-3775440 hydrochloride
T-3775440 (Hydrochlorid) ist ein irreversibler Lysin-spezifischer Histon-Demethylase (LSD1)-Inhibitor mit einem IC50-Wert von 2,1 nM.
-
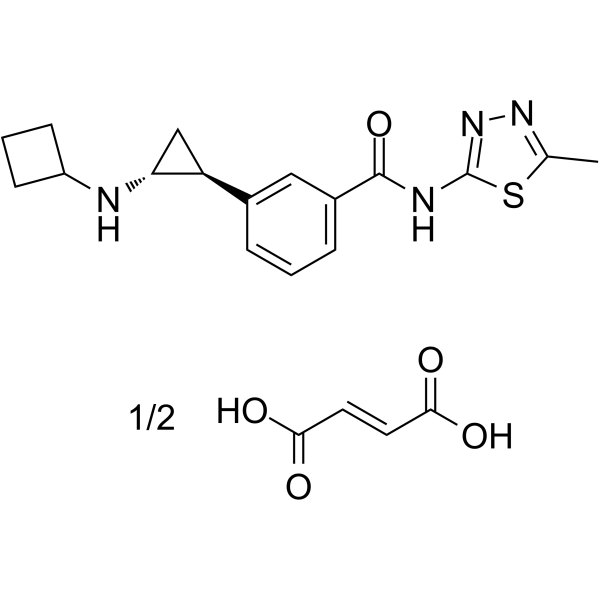
GC65456
T-448
T-448 ist ein spezifischer, oral aktiver und irreversibler Inhibitor der Lysin-spezifischen Demethylase 1 (LSD1, eine H3K4-Demethylase) mit einem IC50 von 22 nM.
-
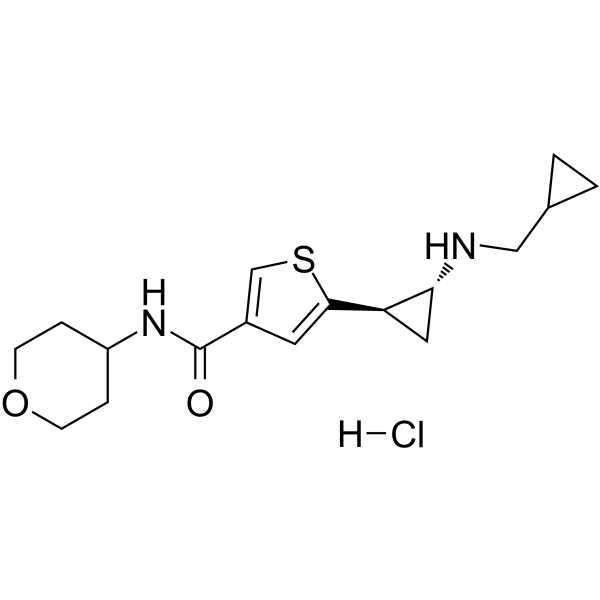
GC63437
TAK-418
TAK-418 ist ein selektiver, oral aktiver LSD1 (KDM1A)-Enzyminhibitor mit einem IC50 von 2,9 nM.
-
GC10319
TC-E 5002
TC-E 5002 (TC-E 5002) ist ein selektiver Inhibitor der Histon-Demethylase der KDM2/7-Unterfamilie (IC50-Werte sind 0,2, 1,2, 6,8, 55, 83, >100 und >120 μ M fÜr KDM7A, KDM7B, KDM2A, KDM5A , KDM4C, KDM6A bzw. KDM4A). TC-E 5002 hemmt das Wachstum von HeLa- und KYSE-150-Krebszellen in vitro.
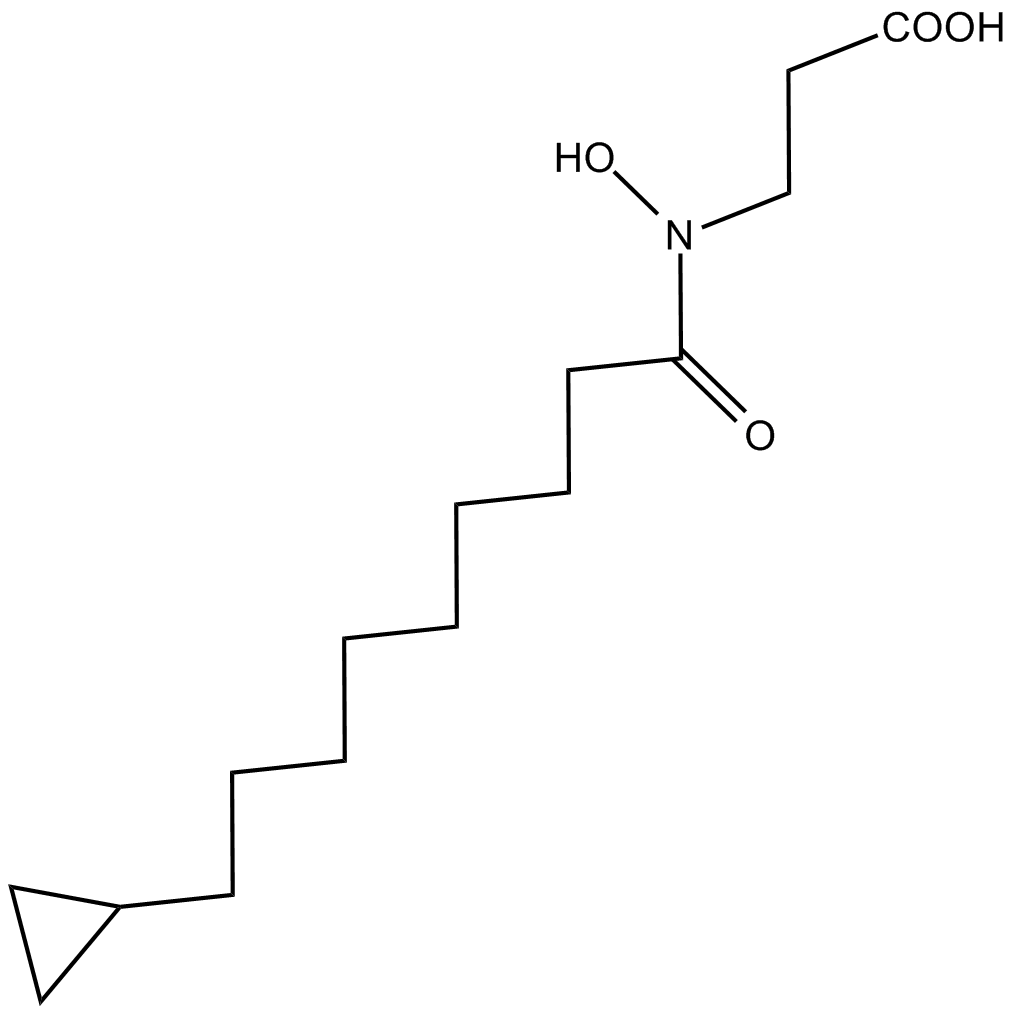
-
GC70033
TK-129
TK-129 ist ein wirksamer und niedrigtoxischer KDM5B-Inhibitor (mit hoher Affinität; IC50=44 nM) zur oralen Einnahme. TK-129 schützt das Herz, indem es KDM5B hemmt und den mit KDM5B verbundenen Wnt-Signalweg blockiert. In vitro reduziert TK-129 die Aktivierung von Herzmuskelzellen durch Ang II und in vivo verringert es die Myokardumgestaltung und Fibrose durch Isoprenalin. TK-129 kann für die Erforschung kardiovaskulärer Erkrankungen eingesetzt werden.
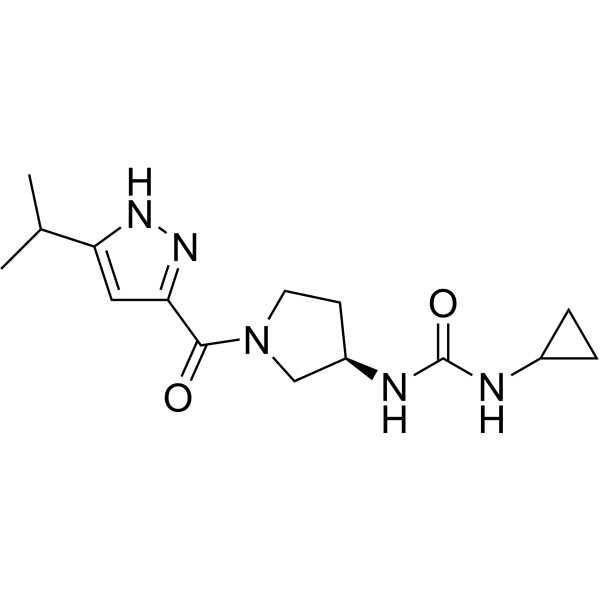
-
GC11576
Tranylcypromine (2-PCPA) HCl
(1S,2R)-Tranylcypromin ((1S,2R)-SKF 385) Hydrochlorid ist ein starkes Antidepressivum.
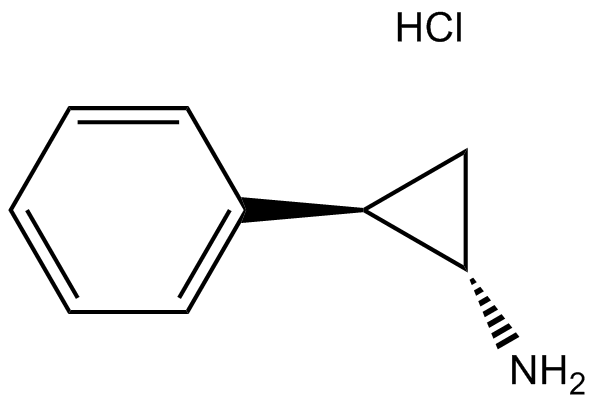
-
GC30766
Tranylcypromine hemisulfate (dl-Tranylcypromine hemisulfate)
Tranylcypromin (SKF 385) Hemisulfat ist ein irreversibler, nicht selektiver Monoaminooxidase (MAO)-Hemmer, der zur Behandlung von Depressionen eingesetzt wird.
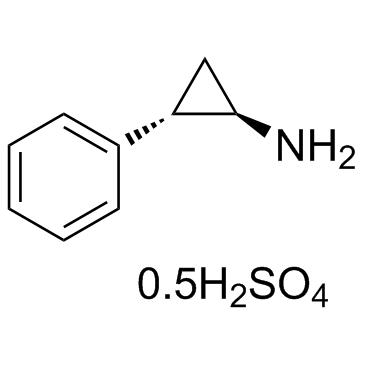
-
GC13919
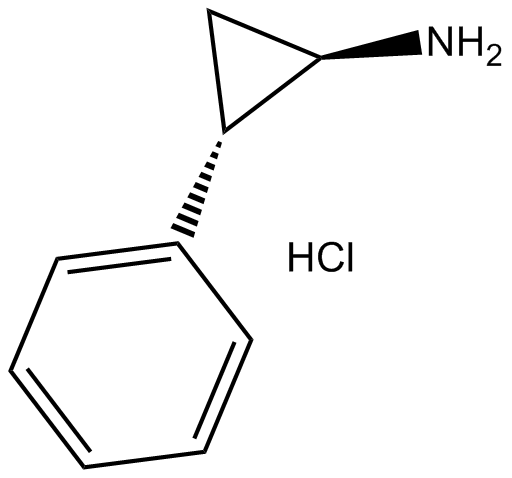
Tranylcypromine hydrochloride
LSD1/BHC110 and monoamine oxidase (MAO) inhibitor
-
GC37956
YUKA1
YUKA1 ist ein potenter und zellgÄngiger Lysin-Demethylase-5A (KDM5A)-Inhibitor mit einem IC50 von 2,66 μM, weniger aktiv auf KDM5C (IC50, 7,12 μM) und inaktiv auf KDM5B, KDM6A oder KDM6B. YUKA1 erhÖht die H3K4me3-Spiegel in menschlichen Zellen mit Anti-Krebs-AktivitÄt.
-
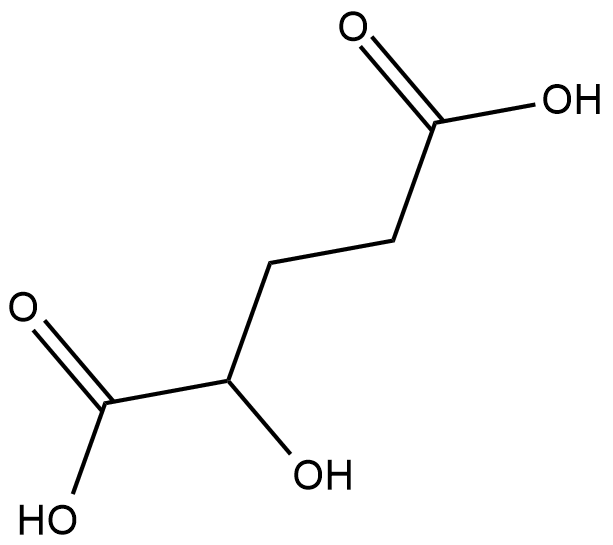
GC18623
α-Hydroxyglutaric Acid
α-HydroxyglutarsÄure (2-Hydroxyglutarat) ist eine α-HydroxysÄureform der GlutarsÄure. α-HydroxyglutarsÄure ist ein kompetitiver Inhibitor mehrerer α-Ketoglutarat-abhÄngiger Dioxygenasen, einschließlich Histon-Demethylasen und der TET-Familie von 5-Methlycytosin (5mC)-Hydroxylasen.