p53
The p53 tumor suppressor is a 53 kDa nuclear phosphoprotein of 393 amino acids that is encoded by the TP53 gene (20 kb with 11 exons and 10 introns) and characterized by the presence of several structural and functional domains, including a N-terminus, a central core domain, a C-terminal region, a strongly basic carboxyl-terminal regulatory domain, a nuclear localization signal sequence and three nuclear export signal sequence. The p53 is considered as a major “guardian of genome” for its activities in a wide range of cellular events, including cell-cycle regulation, induction of apoptosis, gene amplification, DNA recombination, chromosomal segregation and cellular senescence.
Products for p53
- Cat.No. Nom du produit Informations
-
GC42586
6α-hydroxy Paclitaxel
6α-hydroxy Paclitaxel est un métabolite primaire du Paclitaxel. 6α-hydroxy Le paclitaxel conserve un effet dépendant du temps sur les polypeptides transporteurs d'anions organiques 1B1/SLCO1B1 (OATP1B1) avec une puissance d'inhibition similaire À celle du paclitaxel, alors qu'il n'a plus montré d'inhibition dépendante du temps de l'OATP1B3. 6α-hydroxy Paclitaxel peut être utilisé pour la recherche sur le cancer.
-
GC41643
9(Z),11(E),13(E)-Octadecatrienoic Acid
9(Z),11(E),13(E)-Octadecatrienoic Acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.

-
GC40785
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester
9(Z),11(E),13(E)-Octadecatrienoic Acid ethyl ester (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
GC40710
9(Z),11(E),13(E)-Octadecatrienoic Acid methyl ester
9Z,11E,13E-octadecatrienoic acid (α-ESA) is a conjugated polyunsaturated fatty acid commonly found in plant seed oil.
-
GC65880
ADH-6 TFA
ADH-6 TFA est un composé tripyridylamide. ADH-6 abroge l'auto-assemblage du sous-domaine d'agrégation-nucléation du mutant p53 DBD. L'ADH-6 TFA cible et dissocie les agrégats de p53 mutants dans les cellules cancéreuses humaines, ce qui restaure l'activité transcriptionnelle de p53', entraÎnant l'arrêt du cycle cellulaire et l'apoptose. L'ADH-6 TFA a le potentiel pour la recherche sur les maladies cancéreuses.
-
GC33356
AM-8735
L'AM-8735 est un inhibiteur puissant et sélectif de MDM2 avec une IC50 de 25 nM.
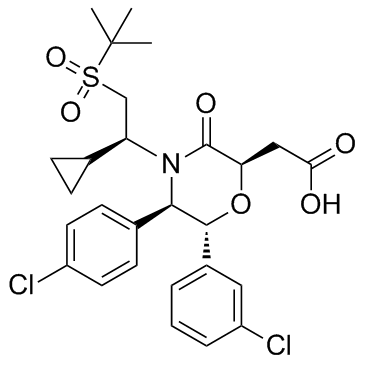
-
GC15828
AMG232
AMG232 (AMG 232) est un inhibiteur puissant, sélectif et disponible par voie orale de l'interaction p53-MDM2, avec une IC50 de 0,6 nM. AMG232 se lie À MDM2 avec un Kd de 0,045 nM.
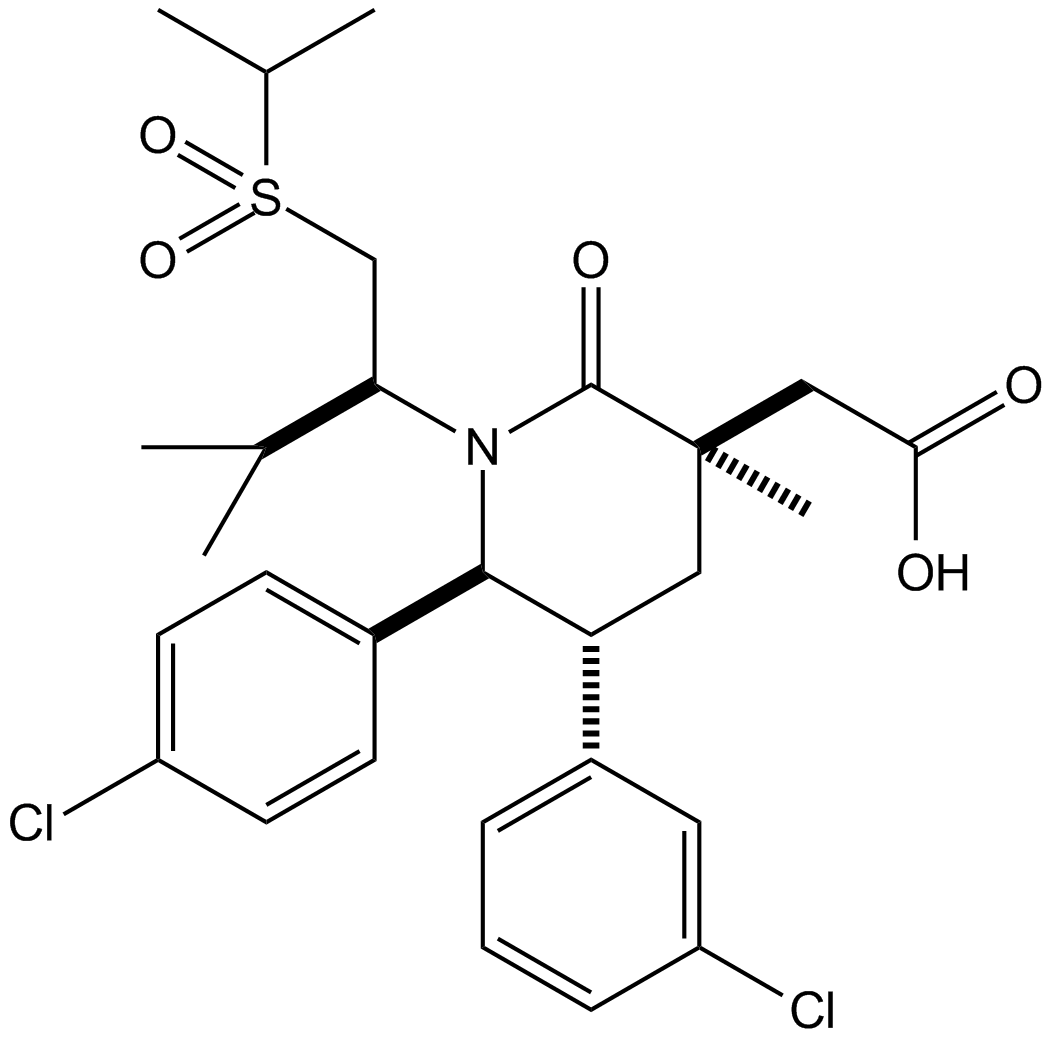
-
GC42785
Amifostine (hydrate)
L'amifostine (hydrate) (WR2721 trihydraté) est un agent cytoprotecteur À large spectre et un radioprotecteur. L'amifostine (hydrate) protège sélectivement les tissus normaux des dommages causés par la radiothérapie et la chimiothérapie. L'amifostine (hydrate) est un puissant facteur inductible par l'hypoxie-α1 (HIF-α1) et un inducteur de p53. L'amifostine (hydrate) protège les cellules contre les dommages en éliminant les radicaux libres dérivés de l'oxygène. L'amifostine (hydrate) réduit la toxicité rénale et a une action anti-angiogénique.
-
GC61804
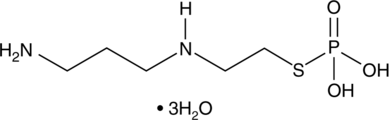
Amifostine thiol
Amifostine thiol (WR-1065) est un métabolite actif du cytoprotecteur Amifostine. Le thiol d'amifostine est un agent cytoprotecteur doté de capacités radioprotectrices. Le thiol d'amifostine active p53 via une voie de signalisation dépendante de JNK.
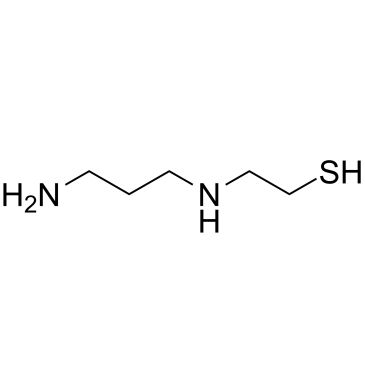
-
GC35367
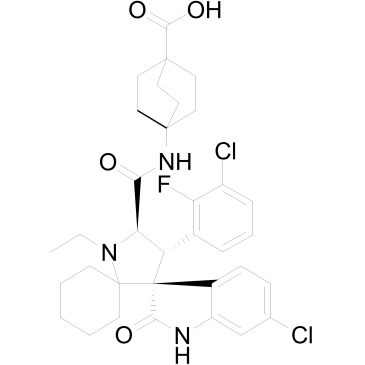
APG-115
APG-115 (APG-115) est un inhibiteur de la protéine MDM2 actif par voie orale se liant À la protéine MDM2 avec des valeurs IC50 et Ki de 3,8 nM et 1 nM, respectivement. L'APG-115 bloque l'interaction de MDM2 et de p53 et induit l'arrêt du cycle cellulaire et l'apoptose d'une manière dépendante de p53.
-
GC18136
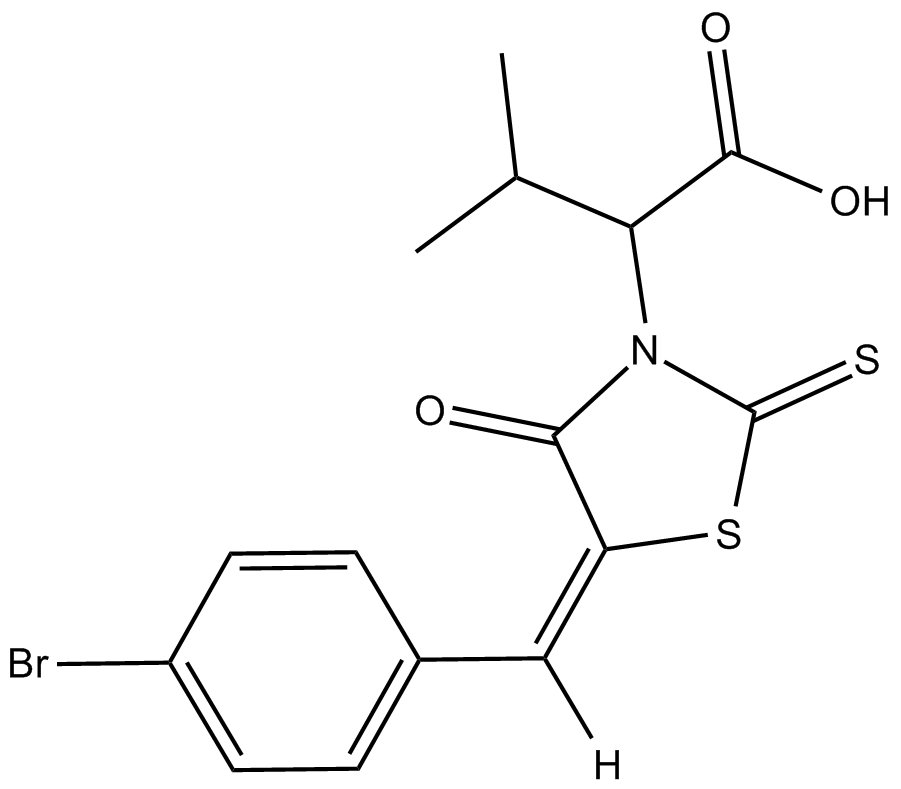
BH3I-1
BH3I-1 est un antagoniste de la famille Bcl-2, qui inhibe la liaison du peptide Bak BH3 À Bcl-xL avec un Ki de 2,4 ± 0,2 μM dans le test FP. BH3I-1 a un Kd de 5,3 μM contre le couple p53/MDM2.
-
GC35511
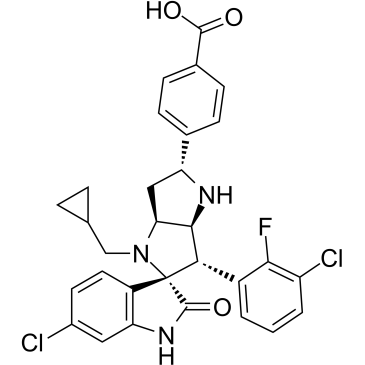
BI-0252
Le BI-0252 est un inhibiteur sélectif de MDM2-p53 actif par voie orale avec une IC50 de 4 nM. Le BI-0252 peut induire des régressions tumorales chez tous les animaux d'une xénogreffe SJSA-1 de souris, avec induction concomitante des gènes cibles de la protéine tumorale p53 (TP53) et des marqueurs d'apoptose.
-
GC43189
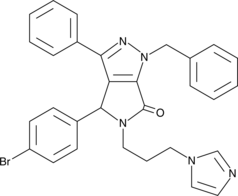
CAY10681
Inactivation of the tumor suppressor p53 commonly coincides with increased signaling through NF-κB in cancer.
-
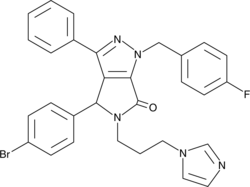
GC43190
CAY10682
(±)-Nutlin-3 blocks the interaction of p53 with its negative regulator Mdm2 (IC50 = 90 nM), inducing the expression of p53-regulated genes and blocking the growth of tumor xenografts in vivo.
-
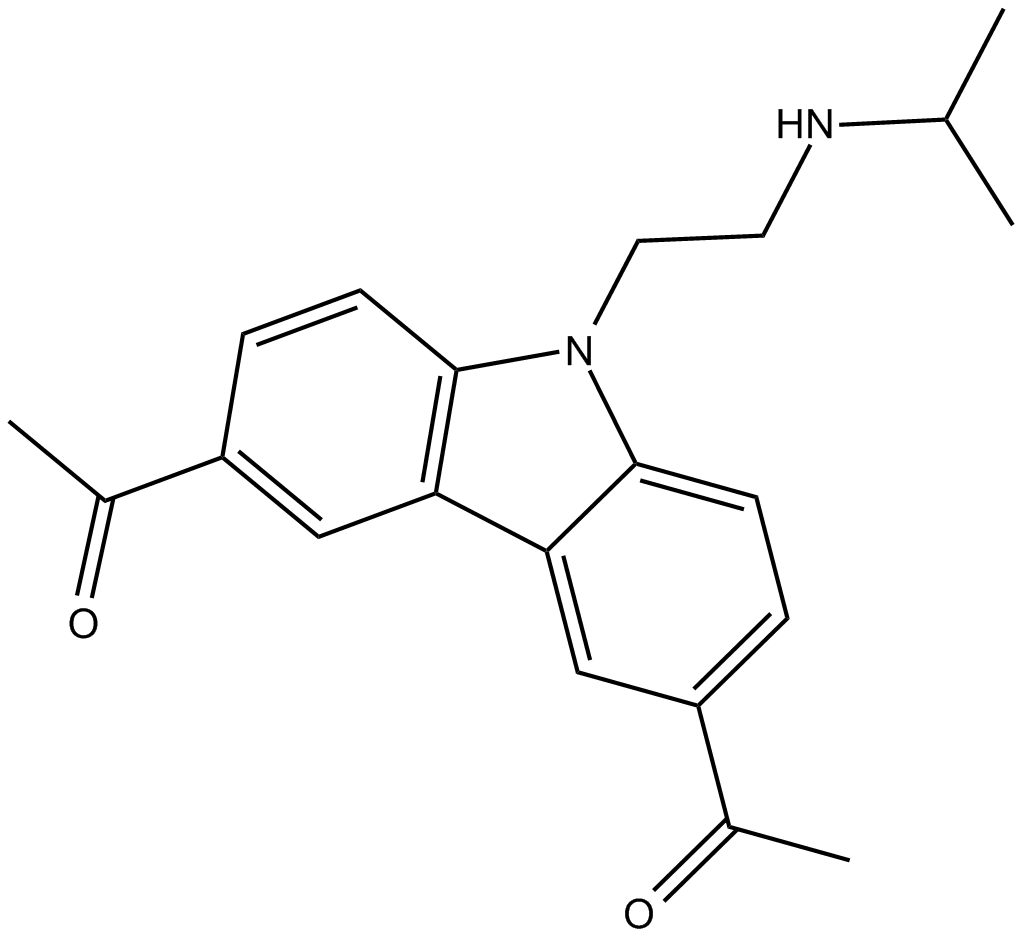
GC14634
CBL0137
curaxin that activates p53 and inhibits NF-κB
-
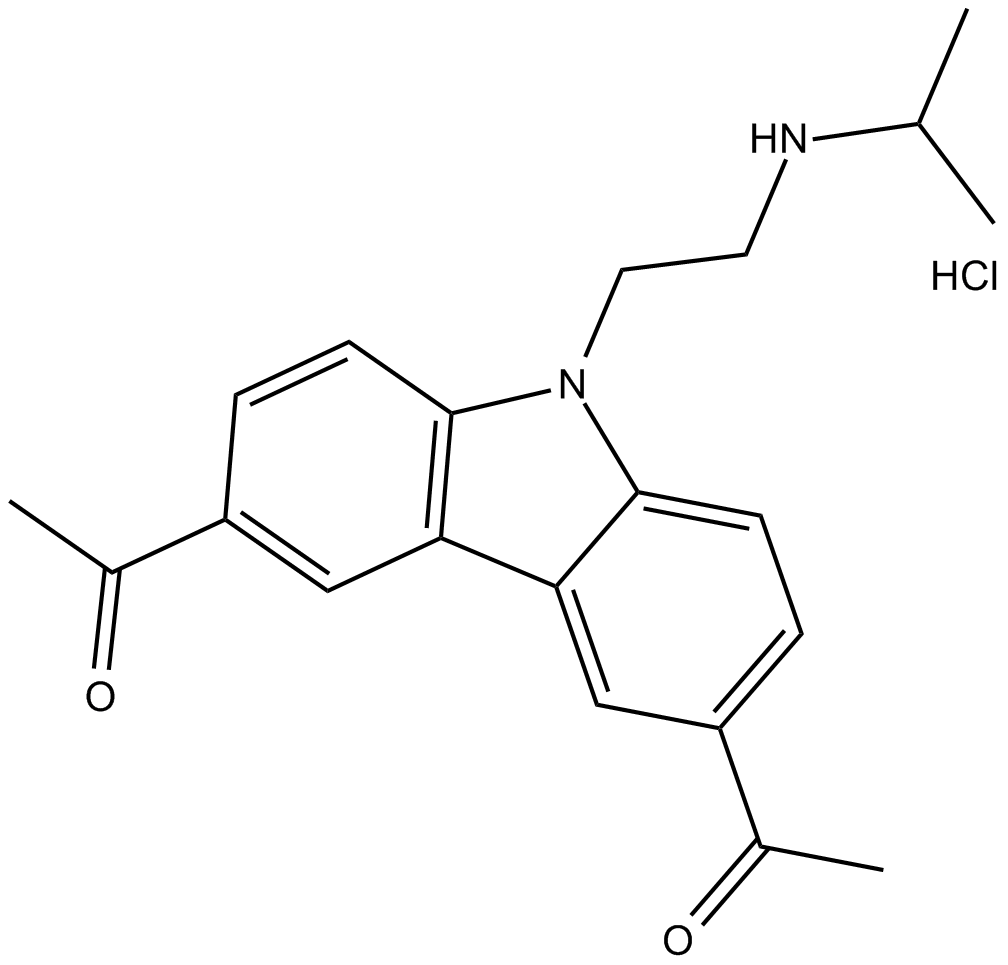
GC15394
CBL0137 (hydrochloride)
CBL0137 (chlorhydrate) est un inhibiteur de l'histone chaperon, FACT. CBL0137 (chlorhydrate) peut également activer p53 et inhiber NF-κB avec des CE50 de 0,37 et 0,47 µM, respectivement.
-
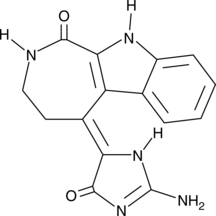
GC43239
Chk2 Inhibitor
L'inhibiteur de Chk2 (composé 1) est un inhibiteur puissant et sélectif de la kinase de point de contrÔle 2 (Chk2), avec des IC50 de 13,5 nM et 220,4 nM pour Chk2 et Chk1, respectivement. L'inhibiteur de Chk2 peut provoquer un fort effet de radioprotection médié par Chk2 dépendant de l'ataxie télangiectasie (ATM).
-
GC64649
Cjoc42
Cjoc42 est un composé capable de se lier À la gankyrine. Cjoc42 inhibe l'activité de la gankyrine de manière dose-dépendante. Cjoc42 empêche la diminution des niveaux de protéine p53 normalement associée À des quantités élevées de gankyrine. Cjoc42 restaure la transcription dépendante de p53 et la sensibilité aux dommages À l'ADN.
-
GC43297
Coenzyme Q2
Coenzyme Q10 is a component of the electron transport chain and participates in aerobic cellular respiration, generating energy in the form of ATP.
-
GC15225
COTI-2
COTI-2, un médicament anticancéreux À faible toxicité, est un activateur de troisième génération disponible par voie orale des formes mutantes p53. COTI-2 agit À la fois en réactivant p53 mutant et en inhibant la voie PI3K/AKT/mTOR. COTI-2 induit l'apoptose dans plusieurs lignées cellulaires tumorales humaines. COTI-2 présente une activité antitumorale dans le HNSCC par le biais de mécanismes dépendants et indépendants de p53. COTI-2 convertit p53 mutant en conformation de type sauvage.
-
GC15840
CP 31398 dihydrochloride
A p53 stabilizing agent
-
GC32911
CTX1
CTX1 est un activateur de p53 qui surmonte la répression de p53 médiée par HdmX. CTX1 présente une puissante activité anticancéreuse dans un système modèle de leucémie myéloÏde aiguë (LMA) chez la souris.
-
GC43408
Deoxycholic Acid (sodium salt hydrate)
L'acide désoxycholique (acide cholanoÏque) hydrate de sodium, un acide biliaire, est un sous-produit du métabolisme intestinal, qui active le récepteur d'acide biliaire couplé À la protéine G TGR5.
-
GC47187
Deoxycholic Acid-d4
L'acide désoxycholique-d4 est l'acide désoxycholique marqué au deutérium.
-
GC33384
DPBQ
DPBQ active p53 et déclenche l'apoptose d'une manière spécifique aux polyploÏdes, mais n'inhibe pas la topoisomérase ni ne se lie À l'ADN. DPBQ provoque l'expression et la phosphorylation de p53 et cet effet est spécifique aux cellules tétraploÏdes.
-
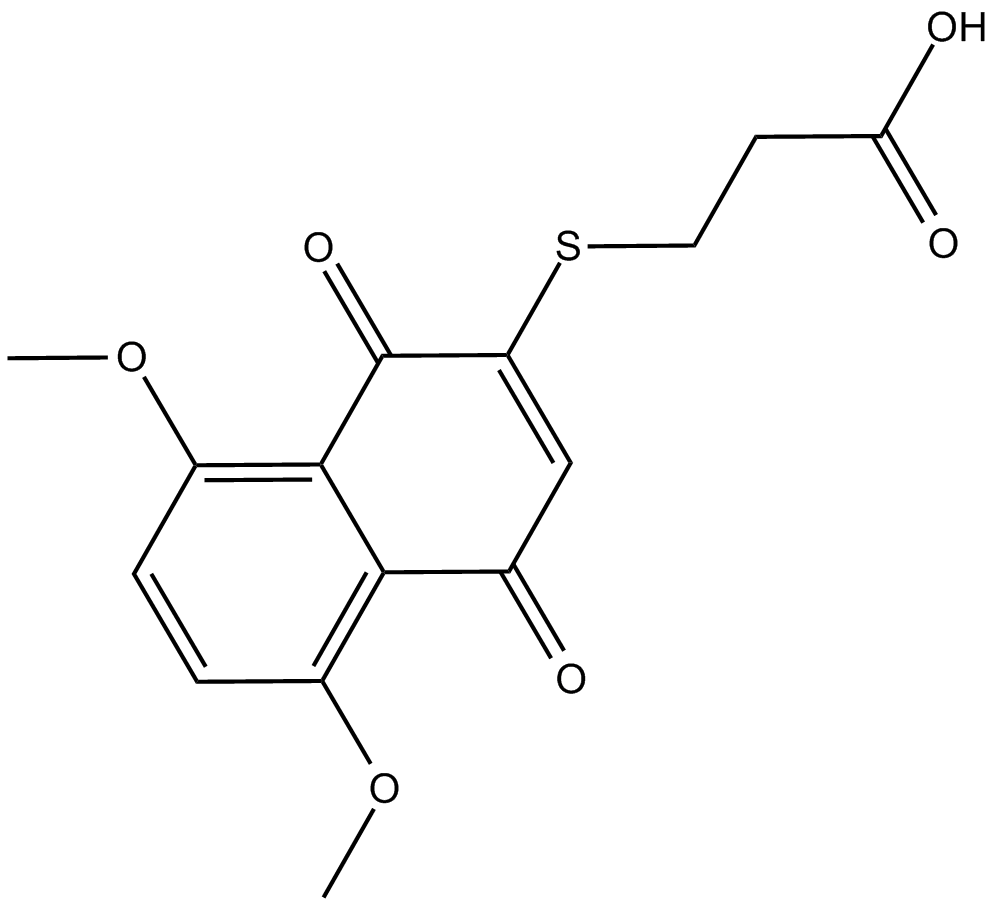
GC15258
GN25
p53-Snail binding Inhibitor
-
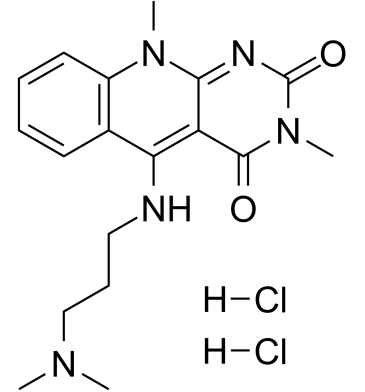
GC61608
HLI373 dihydrochloride
Le dichlorhydrate HLI373 est un inhibiteur efficace de Hdm2.
-
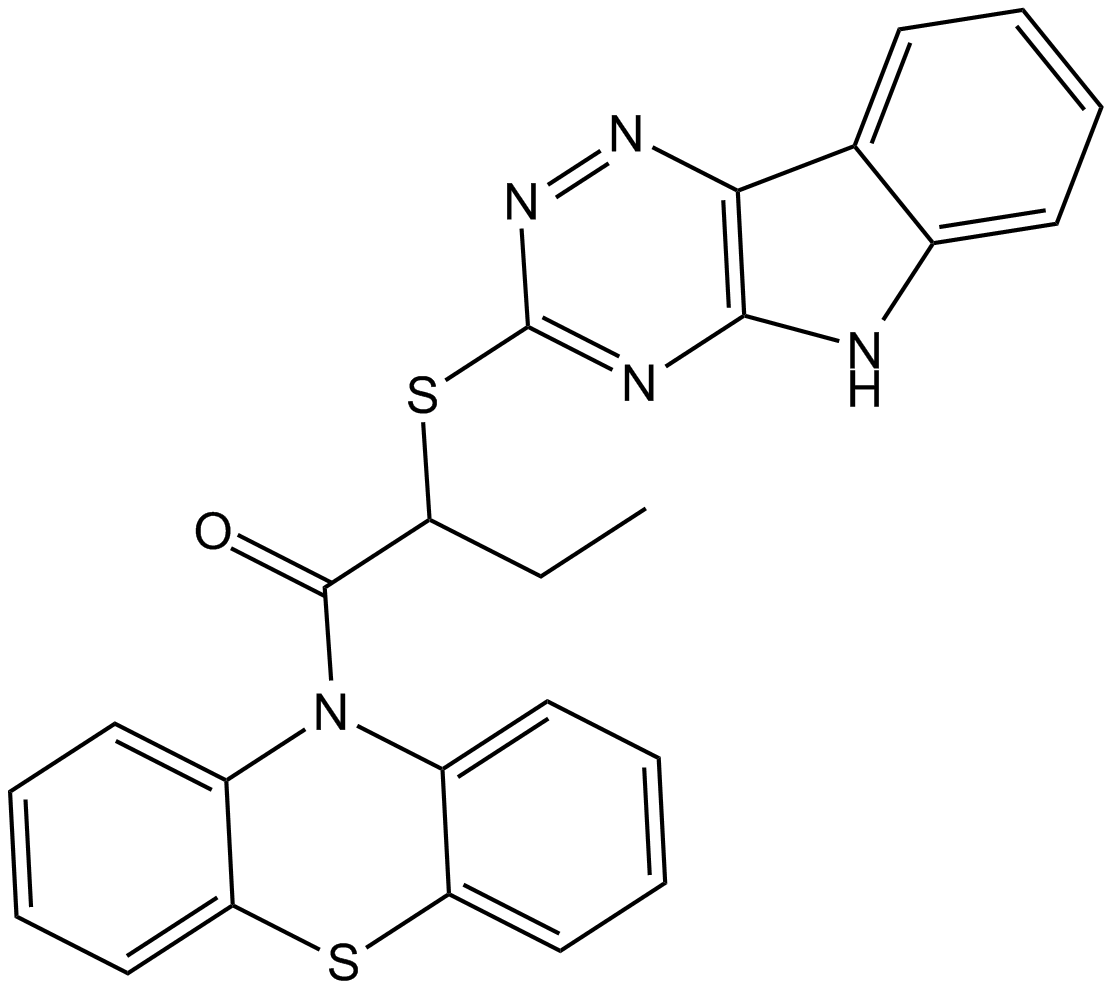
GC14755
Inauhzin
L'inauhzine est un double inhibiteur SirT1/IMPDH2, et agit comme un activateur p53, utilisé dans la recherche sur le cancer.
-
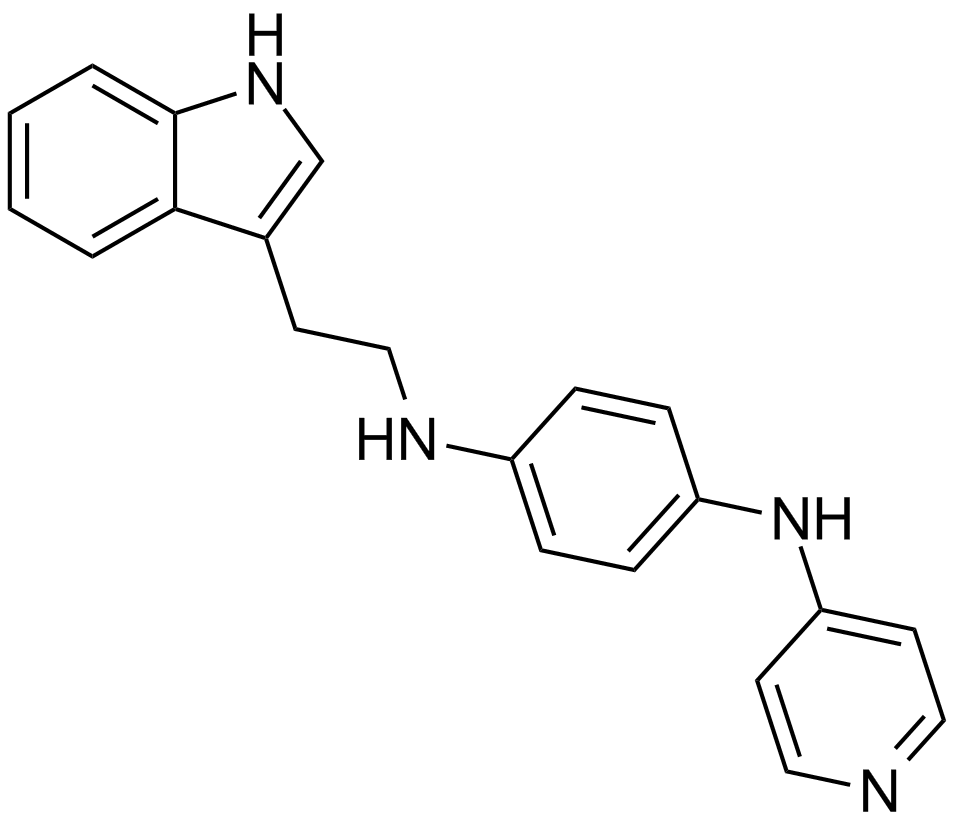
GC12117
JNJ-26854165 (Serdemetan)
An antagonist of MDM2 action
-
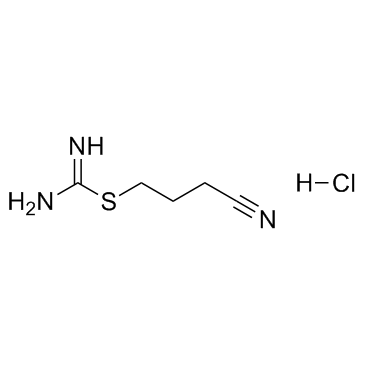
GC32919
Kevetrin hydrochloride (4-Isothioureidobutyronitrile hydrochloride)
Le chlorhydrate de kévétrine (chlorhydrate de 4-isothiouréidobutyronitrile) est un puissant activateur de p53, induit l'apoptose dans les cellules de leucémie myéloÏde aiguë TP53 de type sauvage et mutantes. Kevetrin une activité cytotoxique préférentielle contre les cellules blastiques.
-
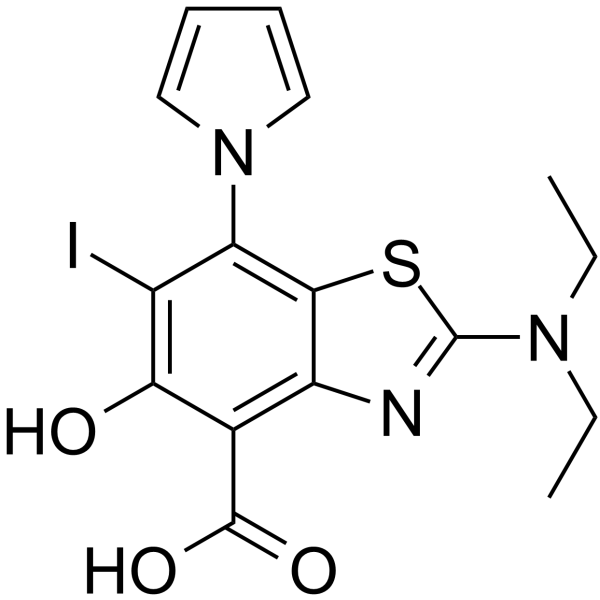
GC62629
MB710
Le MB710, un dérivé de l'aminobenzothiazole, est un stabilisateur de la mutation p53 oncogène Y220C. MB710 se lie étroitement À la poche Y220C et stabilise p53-Y220C, avec un Kd de 4,1 μM. MB710 montre une activité anticancéreuse dans les lignées cellulaires p53-Y220C.
-
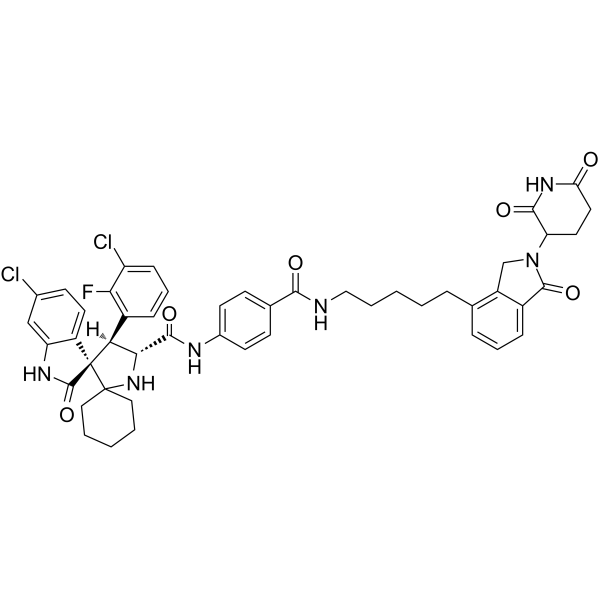
GC62716
MD-222
MD-222 est le premier dégradeur PROTAC hautement puissant de MDM2. MD-222 se compose de ligands pour Cereblon et MDM2. MD-222 induit une dégradation rapide de la protéine MDM2 et l'activation de p53 de type sauvage dans les cellules. Le MD-222 a des effets anticancéreux.
-
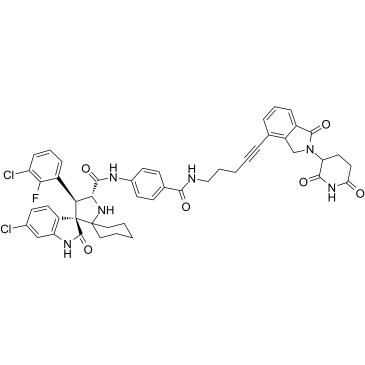
GC38812
MD-224
Le MD-224 est un dégradeur double minute 2 (MDM2) murin humain À petite molécule, premier de sa catégorie et très puissant, basé sur le concept de la chimère de ciblage de la protéolyse (PROTAC). MD-224 se compose de ligands pour Cereblon et MDM2. Le MD-224 induit une dégradation rapide de MDM2 À des concentrations <1 nM dans les cellules leucémiques humaines et atteint une valeur IC50 de 1,5 nM dans l'inhibition de la croissance des cellules RS4;11. Le MD-224 a le potentiel d'être une nouvelle classe d'agents anticancéreux.
-
GC62557
MDM2-IN-1
MDM2-IN-1 (composé 30) est un inhibiteur synthétique de l'interaction MDM2-p53 (MDM2) et contient la configuration trans (D-).
-
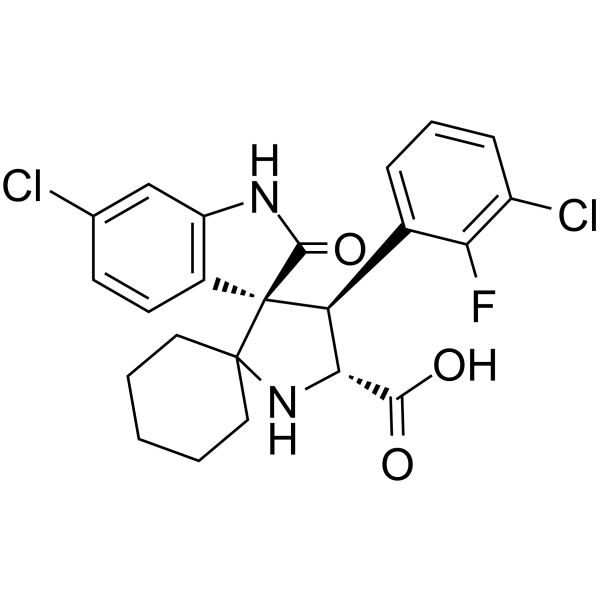
GC36605
MI-1061
Le MI-1061 est un puissant inhibiteur de MDM2 (interaction MDM2-p53) biodisponible par voie orale et chimiquement stable (IC50 = 4,4 nM ; Ki = 0,16 nM). MI-1061 active puissamment p53 et induit l'apoptose dans le tissu tumoral de xénogreffe SJSA-1 chez la souris. Activité anti-tumorale.
-
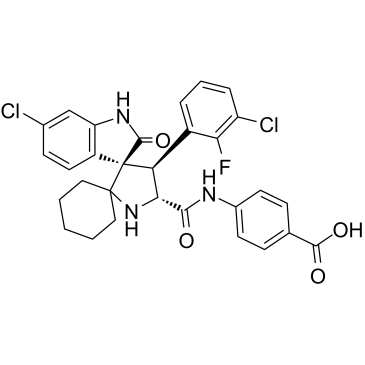
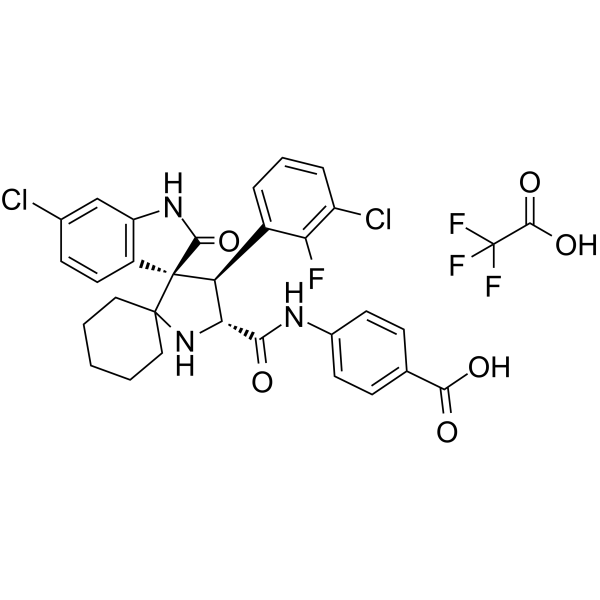
GC62598
MI-1061 TFA
Le MI-1061 TFA est un puissant inhibiteur de MDM2 (interaction MDM2-p53) biodisponible par voie orale et chimiquement stable (IC50 = 4,4 nM ; Ki = 0,16 nM). MI-1061 TFA active puissamment p53 et induit l'apoptose dans le tissu tumoral de xénogreffe SJSA-1 chez la souris. Activité anti-tumorale.
-
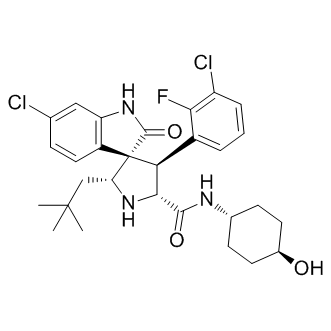
GC16296
MI-773
MI-773 est un puissant inhibiteur de l'interaction protéine-protéine (PPI) MDM2-p53 avec une affinité de liaison élevée contre MDM2 (Kd = 8,2 nM). Le MI-773 a une activité antitumorale.
-
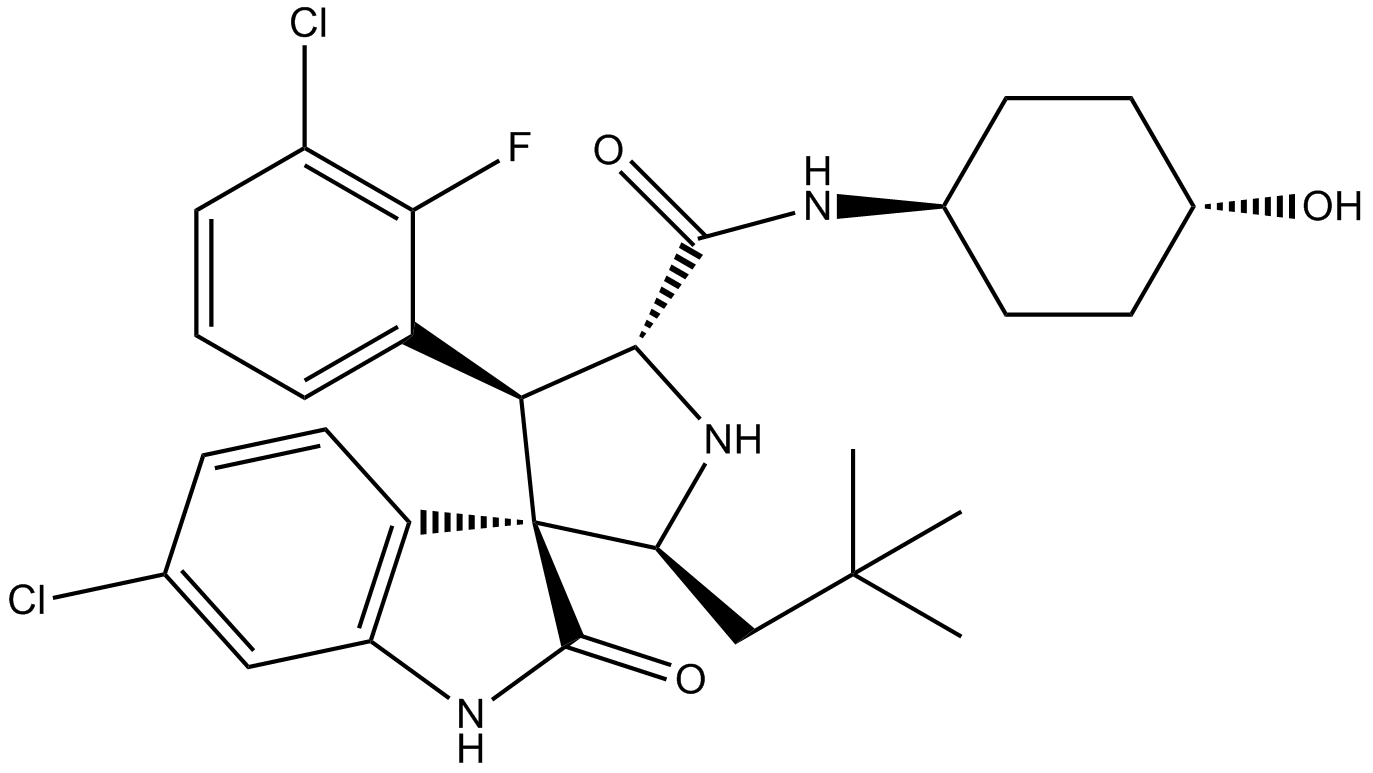
GC11547
MI-773 (SAR405838)
Le MI-773 (SAR405838) (MI-77301), un analogue du MI-773, est un inhibiteur de l'interaction MDM2-p53 très puissant et sélectif. MI-773 (SAR405838) se lie À MDM2 avec un Ki de 0,88 nM. Le MI-773 (SAR405838) induit l'apoptose et possède une puissante activité antitumorale.
-
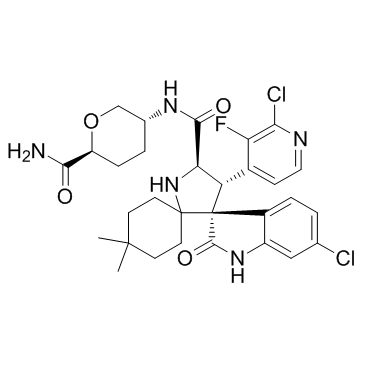
GC32881
Milademetan (DS-3032)
Milademetan (DS-3032) (DS-3032) est un inhibiteur MDM2 spécifique et actif par voie orale pour la recherche de la leucémie myéloÏde aiguë (LMA) ou des tumeurs solides. Le milademétan (DS-3032) (DS-3032) induit l'arrêt du cycle cellulaire G1, la sénescence et l'apoptose.
-
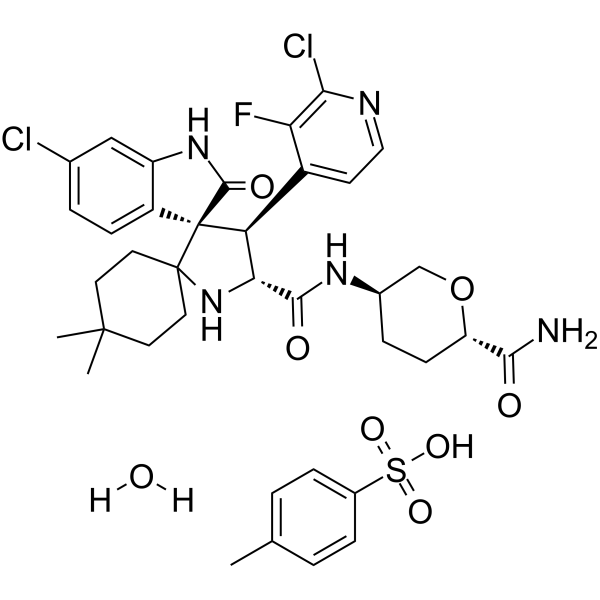
GC62621
Milademetan tosylate hydrate
L'hydrate de tosylate de milademétan (DS-3032) est un inhibiteur MDM2 spécifique et actif par voie orale pour la recherche de la leucémie myéloÏde aiguë (LMA) ou des tumeurs solides. L'hydrate de tosylate de milademétan (DS-3032) induit l'arrêt du cycle cellulaire G1, la sénescence et l'apoptose.
-
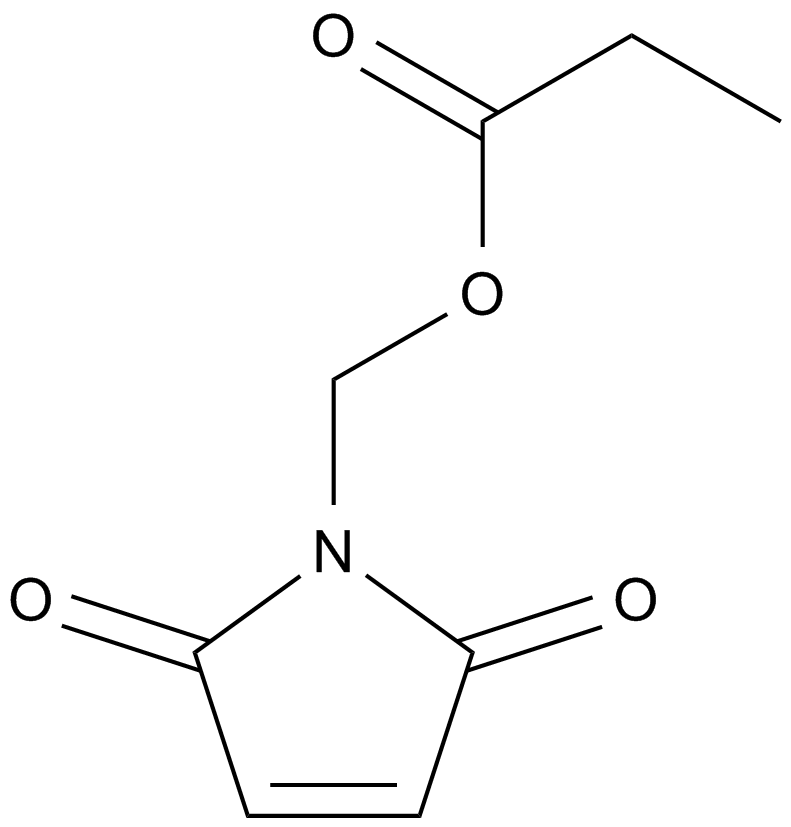
GC12893
MIRA-1
A mutant p53 reactivator
-
GC62322
MS7972
MS7972 est une petite molécule qui bloque l'association de la protéine de liaison p53 humaine et CREB. MS7972 peut presque complètement bloquer cette interaction BRD À 50 μM.
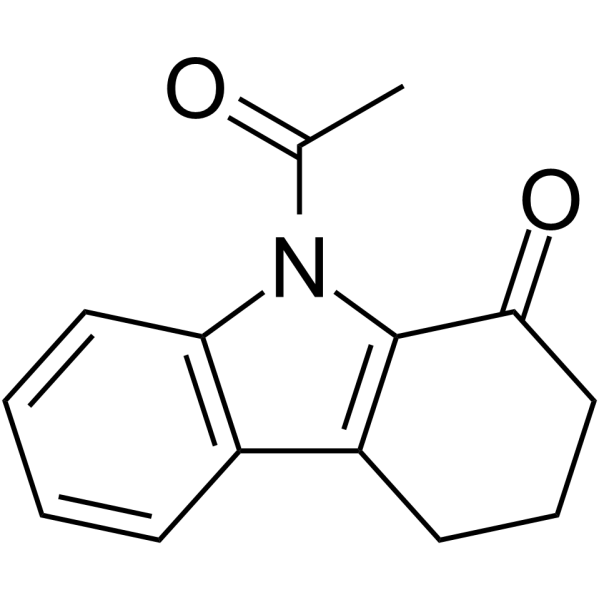
-
GC68371
Mutant p53 modulator-1
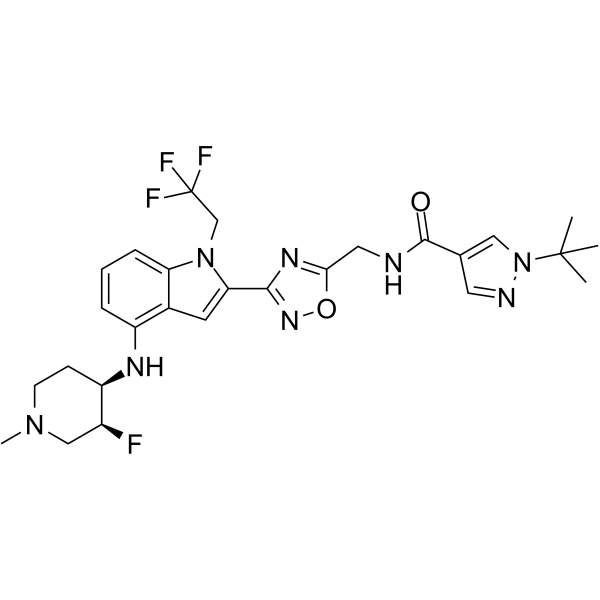
-
GC19257
MX69
MX69 est un inhibiteur de MDM2/XIAP, utilisé pour le traitement du cancer.
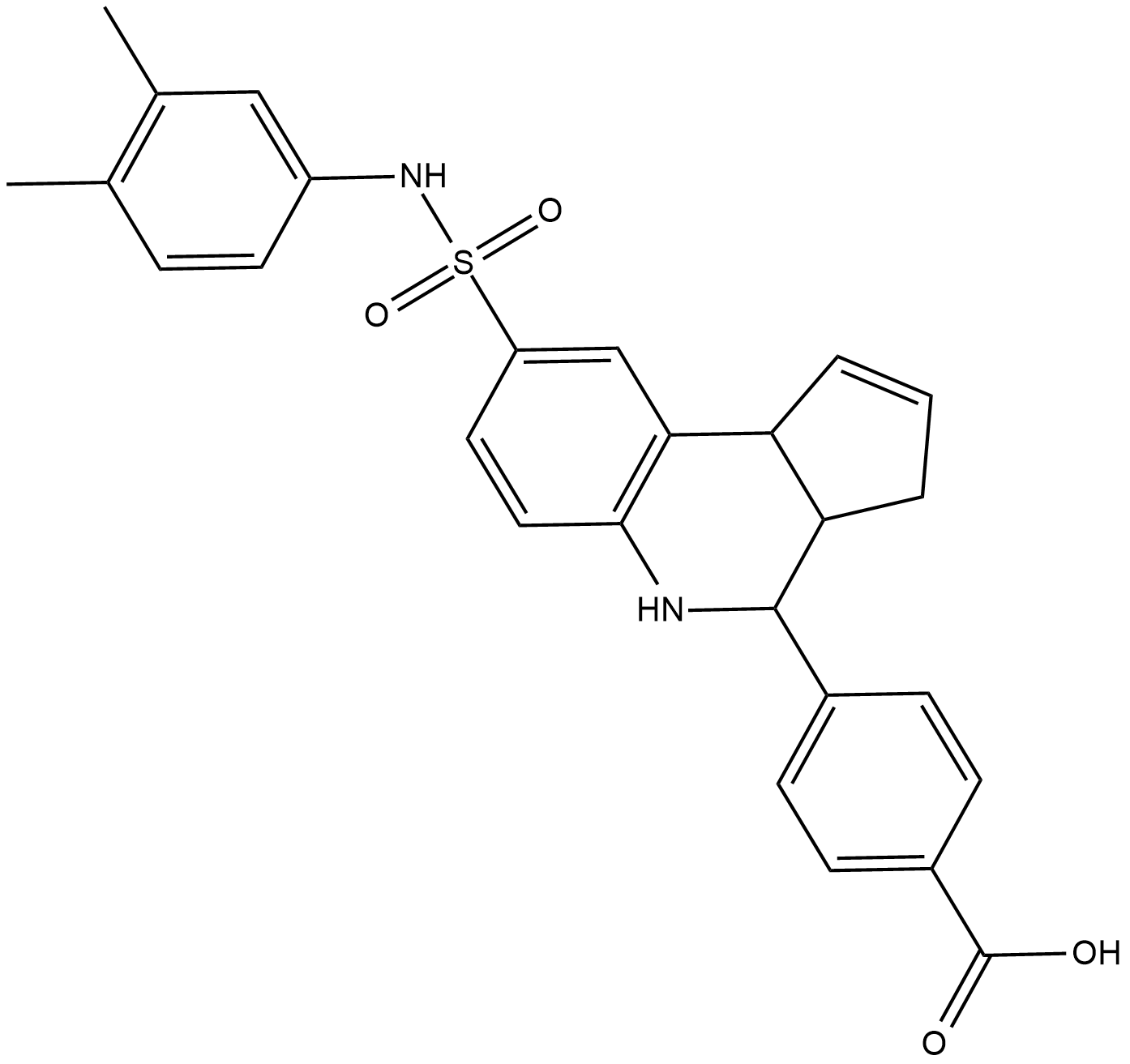
-
GC15621
NSC 146109 hydrochloride
An activator of p53
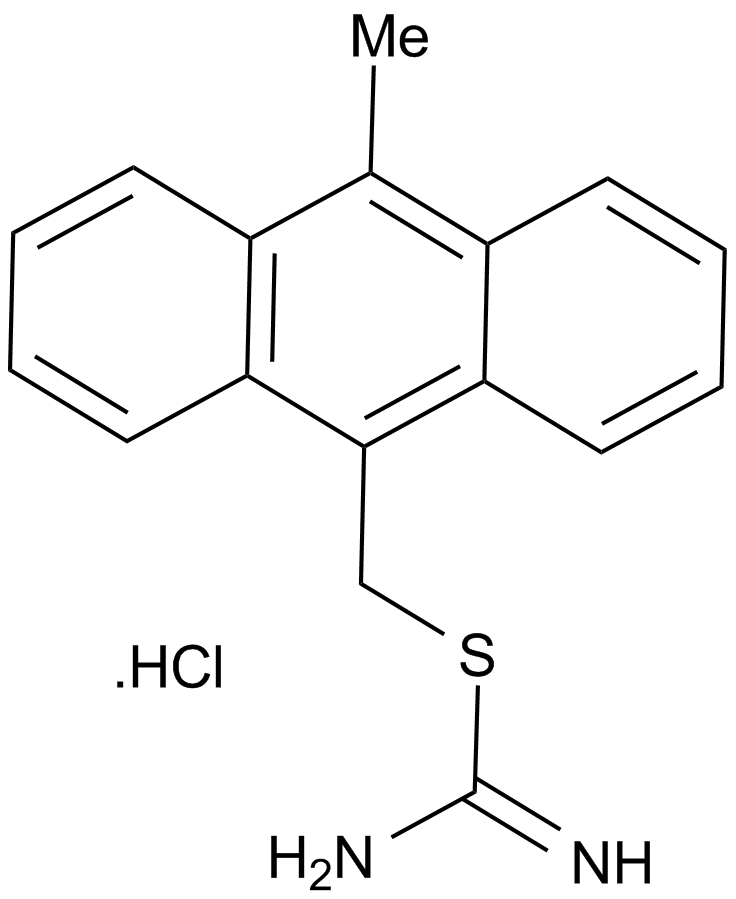
-
GC15404
NSC 319726
A p53 reactivator
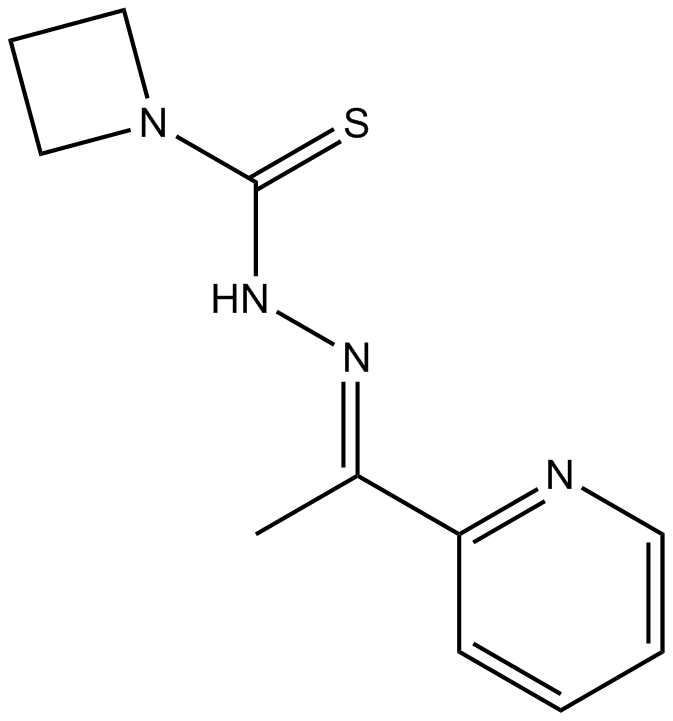
-
GC16151
NSC348884
NSC348884 est un inhibiteur de la nucléophosmine (NPM), il perturbe la formation d'oligomères et induit l'apoptose, inhibe la prolifération cellulaire avec des IC50 de 1,7 À 4,0 μM dans des lignées cellulaires cancéreuses distinctes. NSC348884 peut être utilisé pour la recherche sur le cancer.
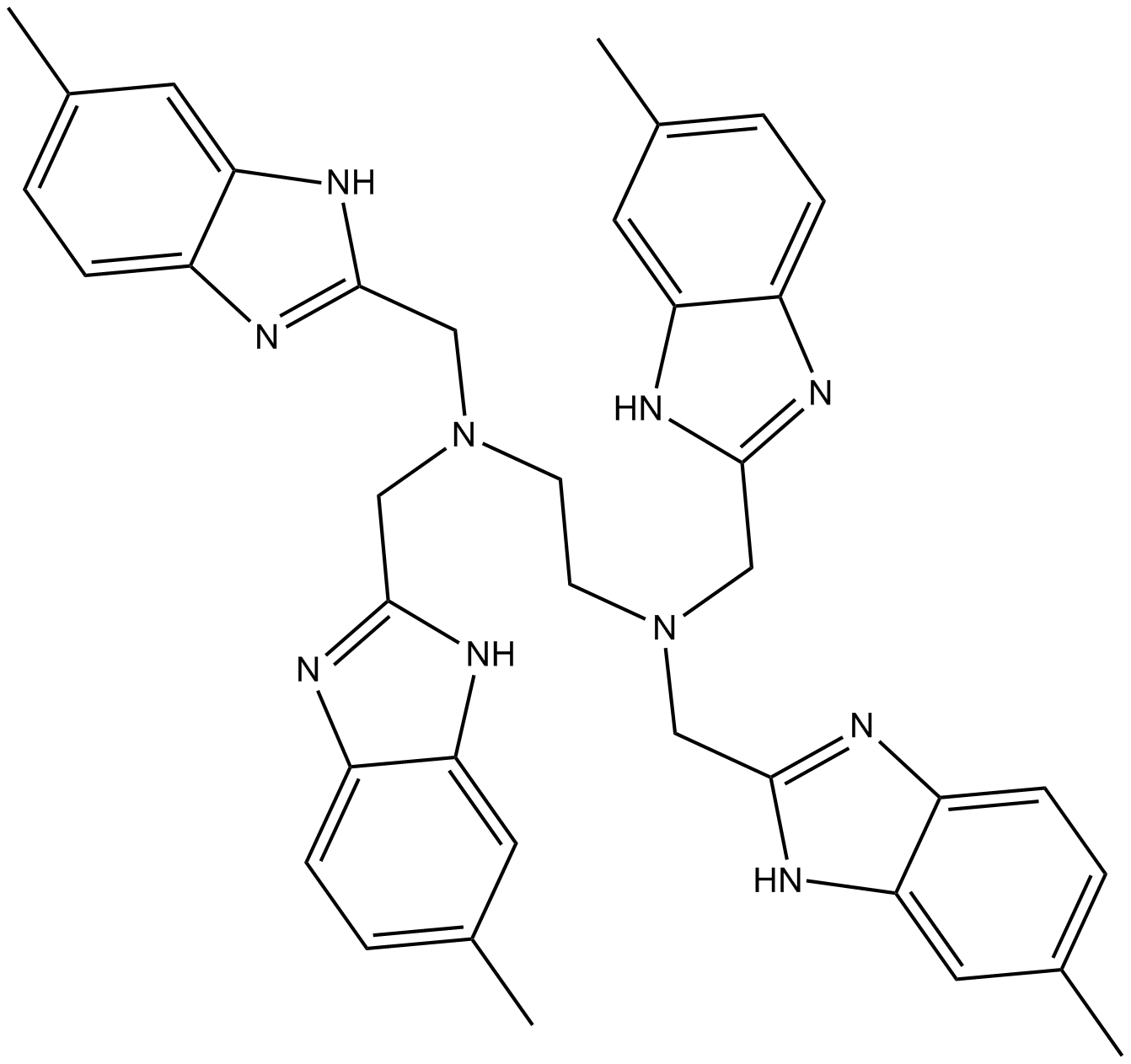
-
GC16179
NSC59984
NSC59984 induit la dégradation de la protéine p53 mutante via MDM2 et la voie ubiquitine-protéasome. NSC59984 agit en ciblant p53 mutant GOF et stimule p73 pour restaurer la voie de signalisation p53.
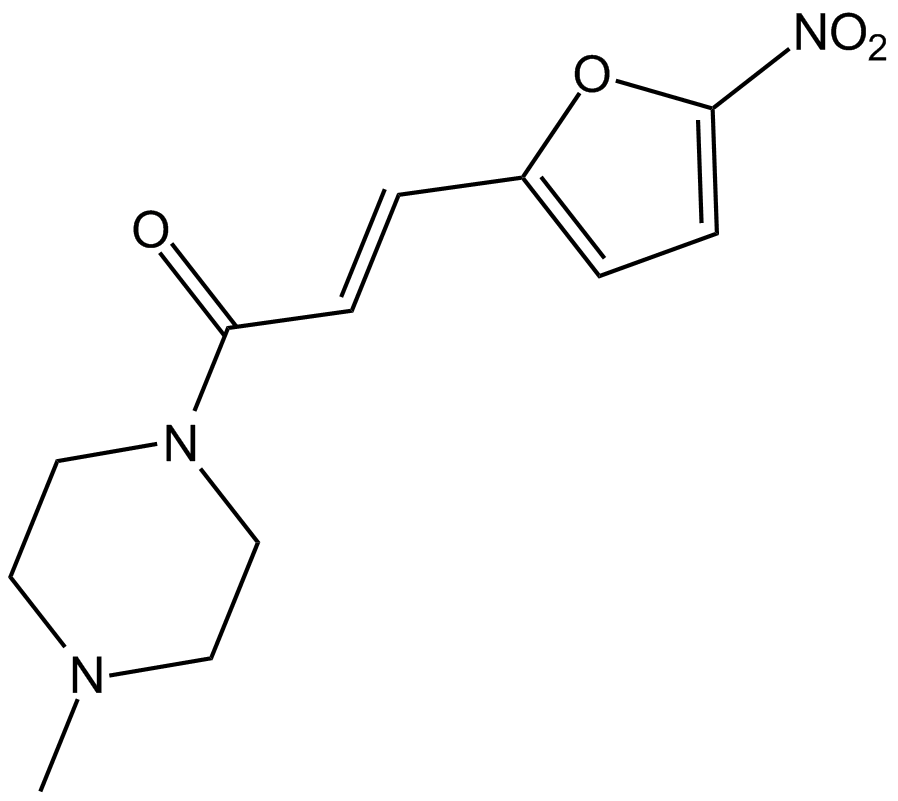
-
GC16051
Nutlin-3
A racemic mixture of (?)-nutlin-3 and (+)-nutlin-3
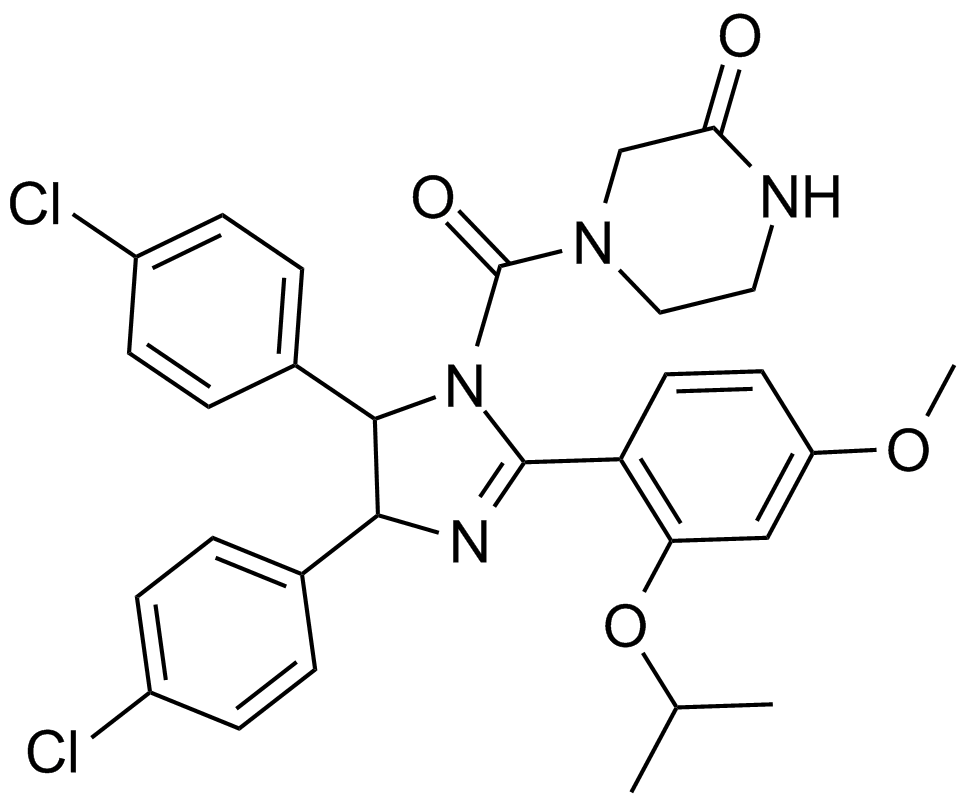
-
GC10470
Nutlin-3a chiral
An inhibitor of the p53-Mdm2 interaction
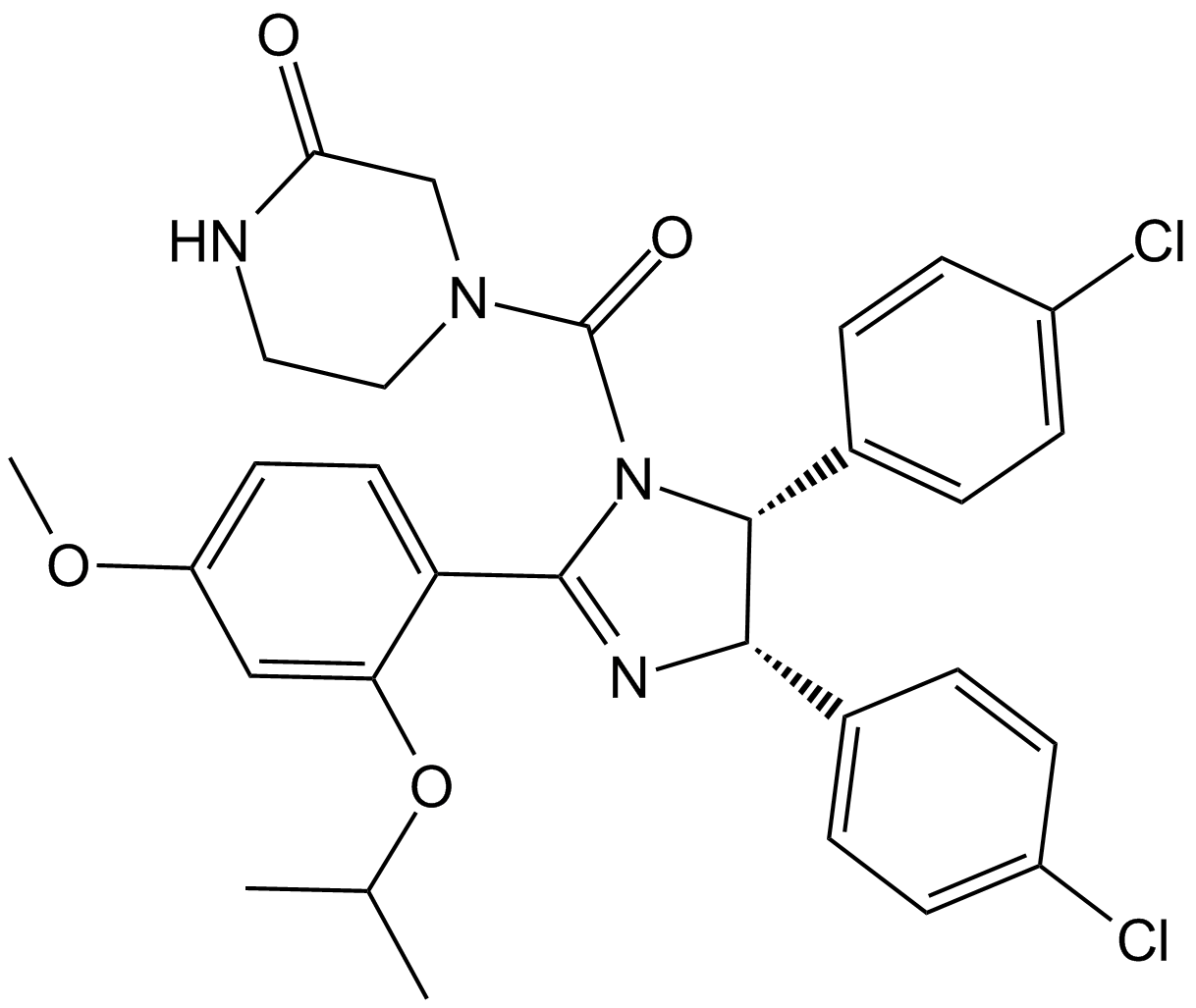
-
GC12508
Nutlin-3b
Nutlin-3b est un inhibiteur de p53/MDM2 avec une IC50 de 13,6 μM. Nutlin-3b est 150 fois moins puissant pour se lier au MDM2 que Nutlin-3a.
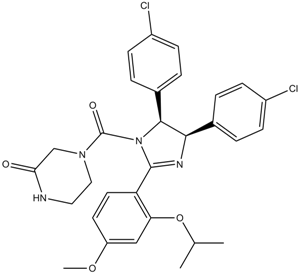
-
GC12647
NVP-CGM097
NVP-CGM097 est un inhibiteur puissant et sélectif de MDM2 avec une IC50 de 1,7 ± 0,1 nM pour hMDM2.
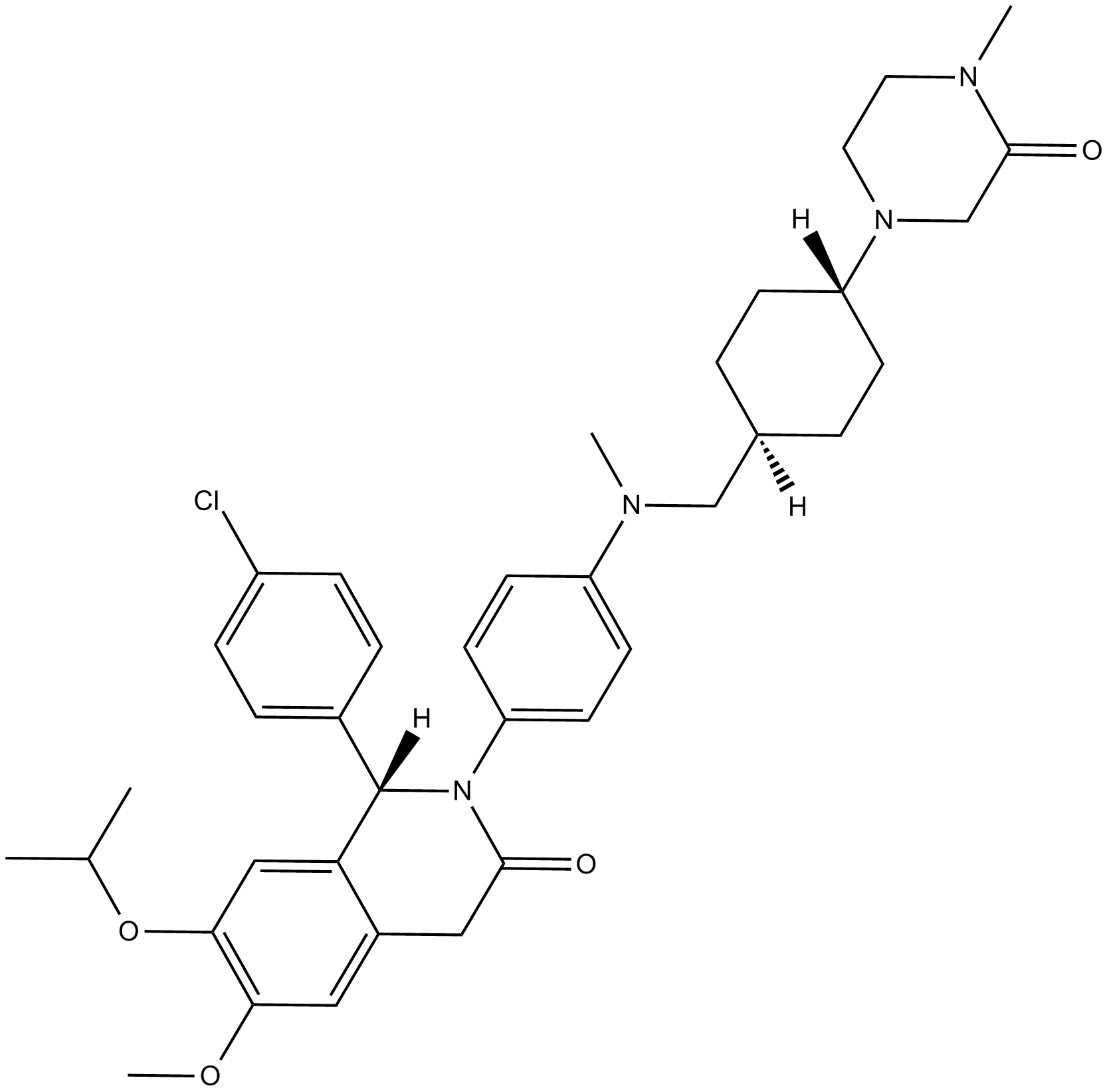
-
GC36785
NVP-CGM097 sulfate
Le sulfate de NVP-CGM097 est un inhibiteur puissant et sélectif de MDM2 avec une IC50 de 1,7 ± 0,1 nM pour hMDM2.
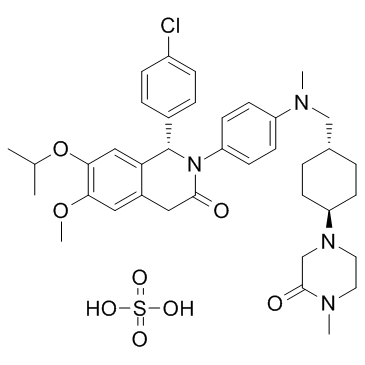
-
GC19268
NVP-HDM201
NVP-HDM201 (NVP-HDM201) est un puissant inhibiteur de l'interaction p53-MDM2 biodisponible par voie orale et hautement spécifique.
-
GC48652
Olomoucine II
A CDK inhibitor
-
GC11711
ONC201
ONC201 (ONC-201) est un inducteur puissant, actif par voie orale et stable du ligand induisant l'apoptose lié au facteur de nécrose tumorale (TRAIL) qui agit en inhibant Akt et ERK, activant par conséquent Foxo3a et induisant de manière significative le TRAIL de surface cellulaire. ONC201 peut traverser la barrière hémato-encéphalique.
-
GC15613
p-nitro-Cyclic Pifithrin-α
p-nitro-Cyclic Pifithrin-α (PFN-α) est un inhibiteur de p53 perméable aux cellules et sous forme active.
-
GC15812
p-nitro-Pifithrin-α
p-nitro-Pifithrin-α, un analogue perméable aux cellules de pifithrin-α, est un puissant inhibiteur de p53.
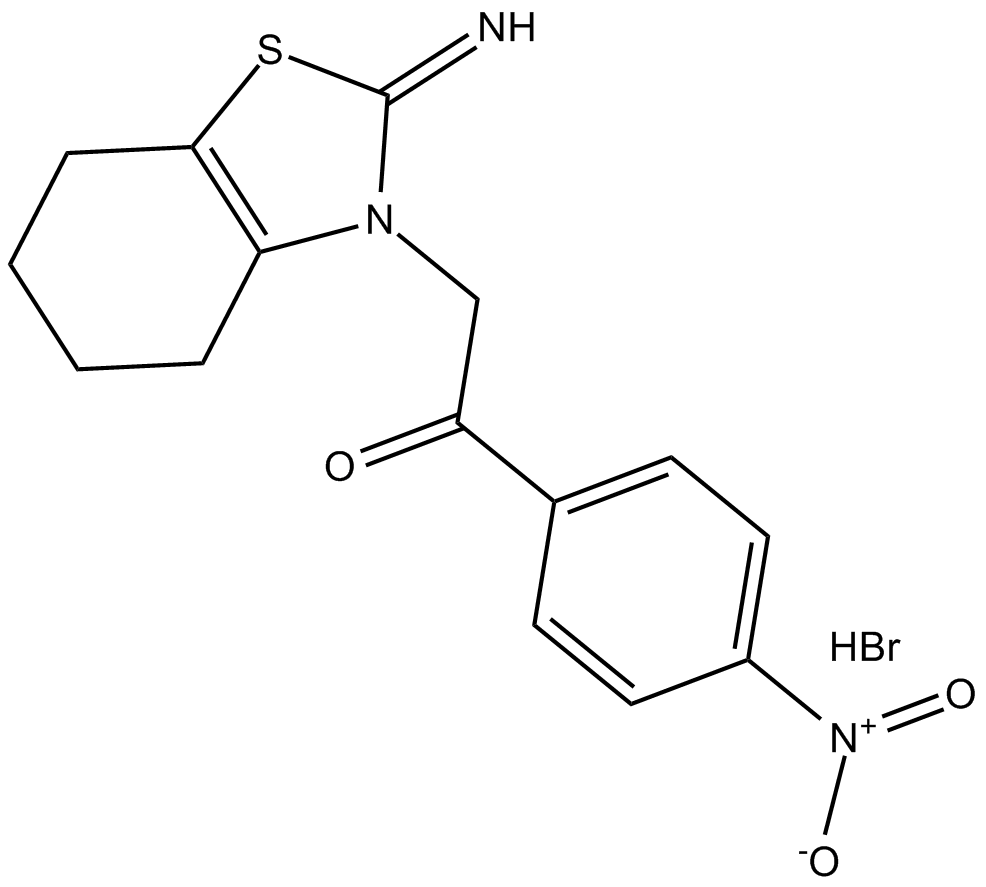
-
GC67765
p53 Activator 5
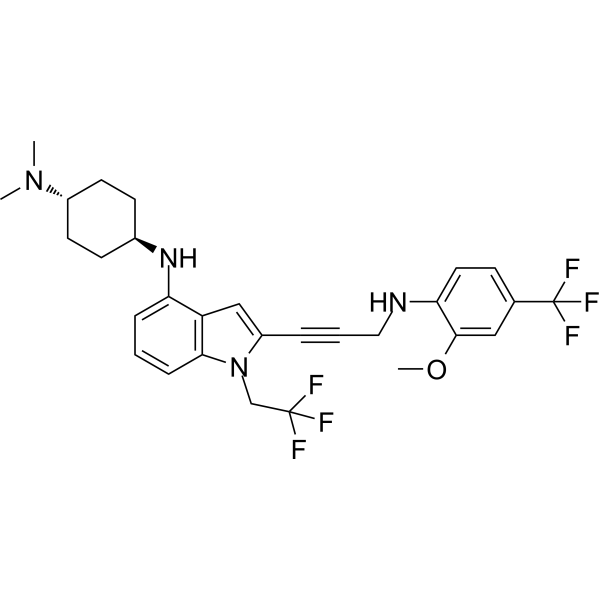
-
GC69648
p53 Activator 7
Le p53 Activator 7 est un activateur de la mutation Y220C (MDM-2/p53) de p53, avec une EC50 de 104 nM. Le p53 Activator 7 peut se lier aux variants mutés de p53 et restaurer leur capacité à se lier à l'ADN (WO2022213975A1 ; Exemple B-1).
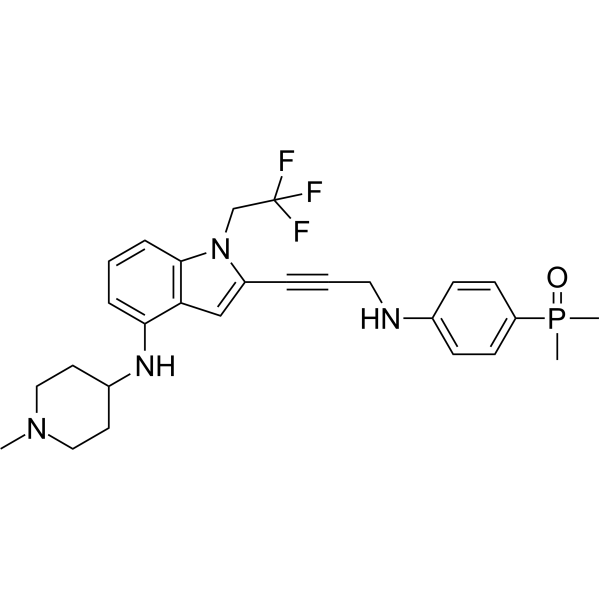
-
GC36835
p53 and MDM2 proteins-interaction-inhibitor chiral
L'inhibiteur chiral de l'interaction des protéines p53 et MDM2 (composé 32) est un inhibiteur de l'interaction entre les protéines p53 et MDM2.
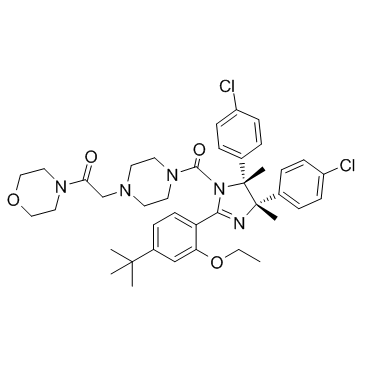
-
GC36836
p53 and MDM2 proteins-interaction-inhibitor dihydrochloride
Le dichlorhydrate inhibiteur de l'interaction des protéines p53 et MDM2 est un inhibiteur de l'interaction entre les protéines p53 et MDM2.
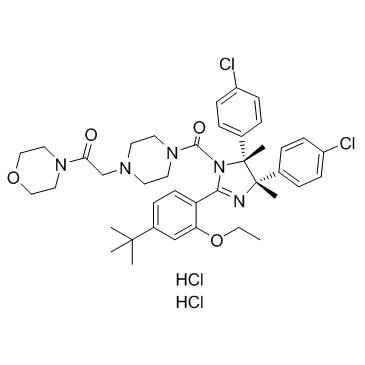
-
GC36837
p53 and MDM2 proteins-interaction-inhibitor racemic
Le racémique inhibiteur de l'interaction des protéines p53 et MDM2 (composé 2j) est un inhibiteur de l'interaction entre les protéines p53 et MDM2.
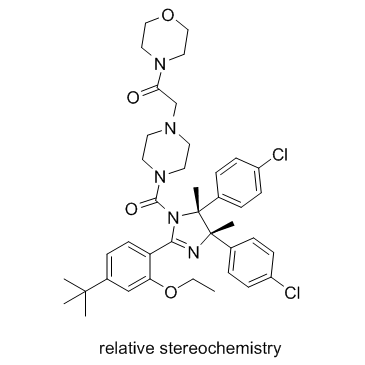
-
GP10036
p53 tumor suppressor fragment
Regulates cell cycle
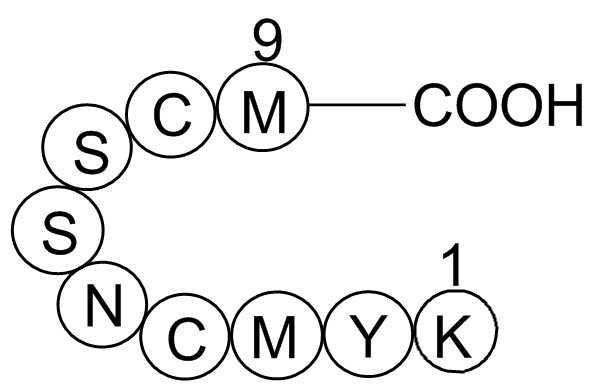
-
GC65961
P53R3
P53R3 est un puissant réactivateur de p53 et restaure la liaison À l'ADN spécifique À la séquence des mutants de points chauds de p53, y compris p53R175H, p53R248W et p53R273H. P53R3 induit des effets antiprolifératifs dépendants de p53 avec une spécificité beaucoup plus élevée que PRIMA-1. P53R3 améliore le recrutement de p53 et p53M237I de type sauvage sur plusieurs promoteurs de gènes cibles. P53R3 améliore fortement l'expression de l'ARNm, des protéines totales et de la surface cellulaire du récepteur de mort récepteur de mort 5 (DR5). Le P53R3 est utilisé pour la recherche sur le cancer.
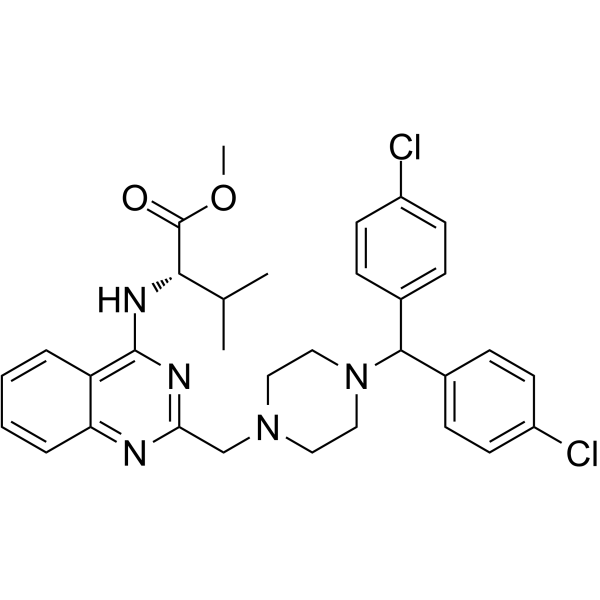
-
GC45758
Paclitaxel octadecanedioate
A prodrug form of paclitaxel
-
GC47853
Paclitaxel-d5
Le paclitaxel-d5 est un paclitaxel marqué au deutérium. Le paclitaxel est un agent antinéoplasique naturel qui stabilise la polymérisation de la tubuline.
-
GC12658
PhiKan 083
-
GC36896
PhiKan 083 hydrochloride
Le chlorhydrate de PhiKan 083 est un dérivé de carbazole, qui se lie À la cavité de surface et stabilise Y220C (un mutant p53), avec un Kd de 167 μM et une affinité de liaison relative (Kd) de 150 μM dans les cellules Ln229.
-
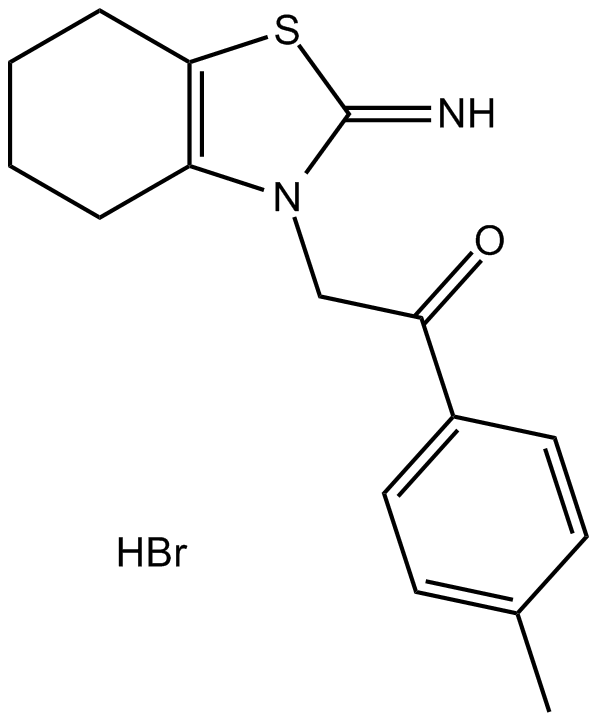
GC10538
Pifithrin-α (PFTα)
Un inactivateur de p53
-
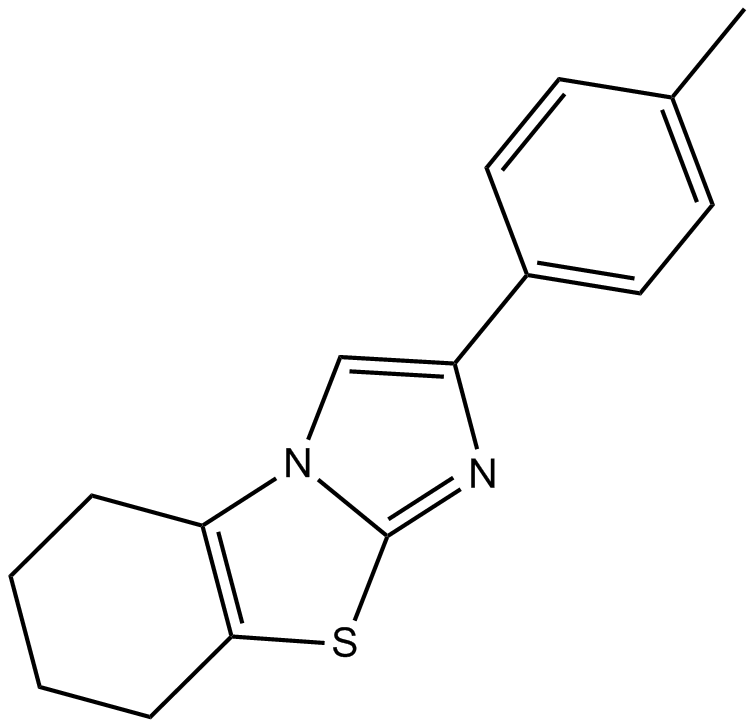
GC17262
Pifithrin-β
La pifithrine-β (PFT β) est un puissant inhibiteur de p53 avec une IC50 de 23 μM.
-
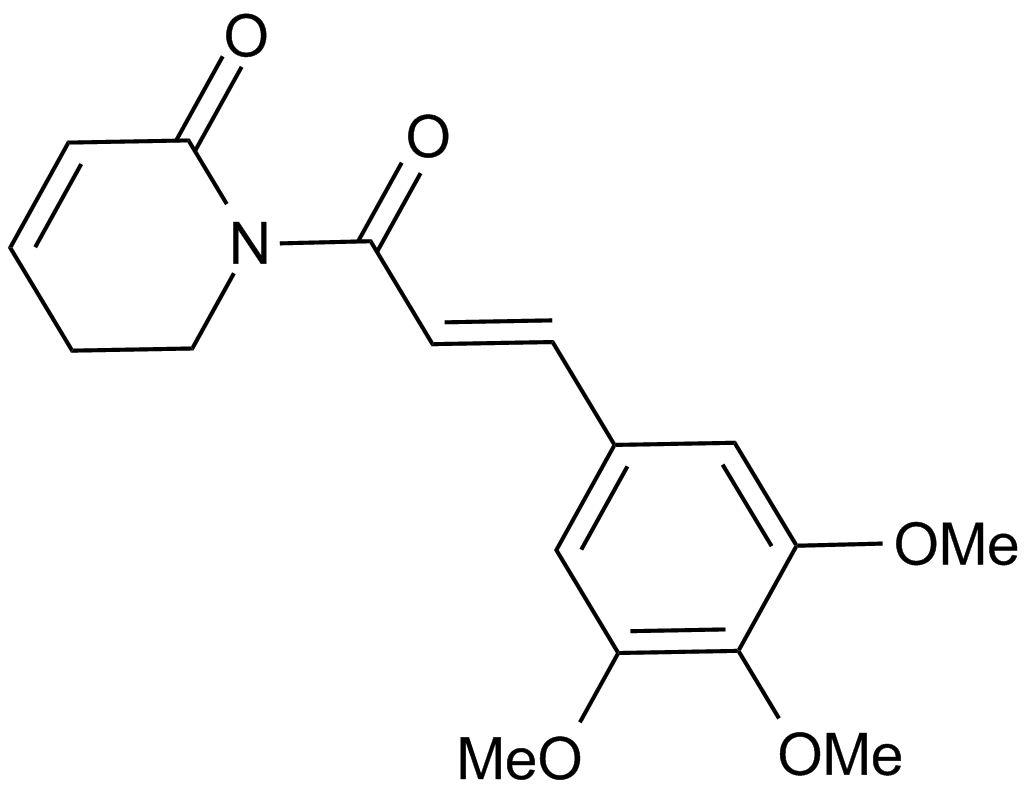
GC10282
Piperlongumine
An alkaloid with anticancer and antioxidant activities
-
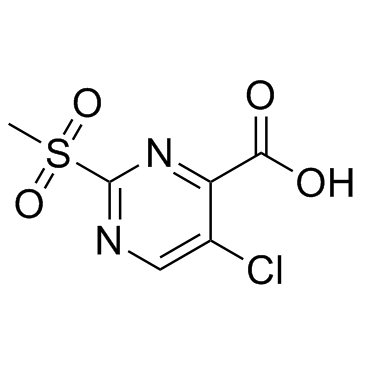
GC32946
PK11007
PK11007 est un alkylateur de thiol doux avec une activité anticancéreuse. PK11007 stabilise p53 par alkylation sélective de deux cystéines exposées en surface sans compromettre son activité de liaison À l'ADN. PK11007 induit la mort des cellules cancéreuses mutantes p53 en augmentant les niveaux d'espèces réactives de l'oxygène (ROS).
-
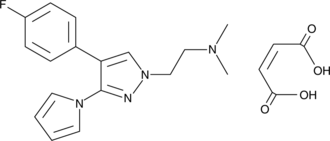
GC44652
PK7242 (maleate)
The protein p53, often called the 'guardian of the genome,' is a transcription factor that is activated in response to cellular stress (low oxygen levels, heat shock, DNA damage, etc.) and acts to prevent further proliferation of the stressed cell by promoting cell cycle arrest or apoptosis.
-
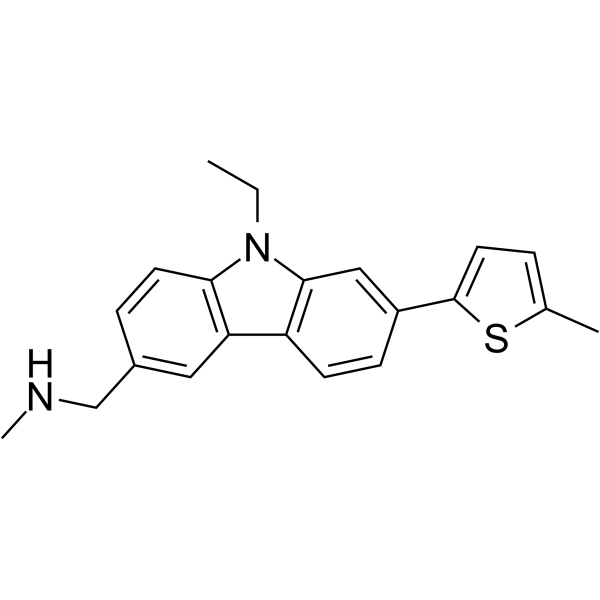
GC64768
PK9327
Le PK9327 est un stabilisateur À petite molécule ciblant les mutations cancéreuses p53 créant des cavités.
-
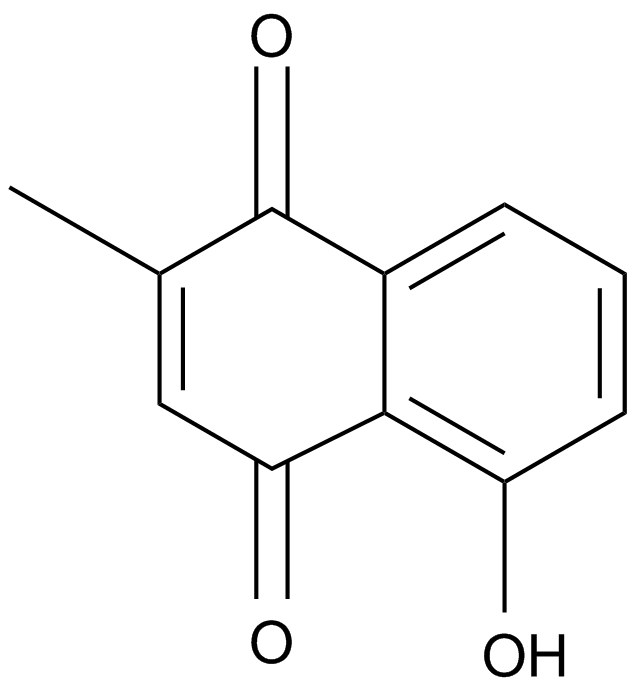
GC10315
Plumbagin
A natural naphthoquinone
-
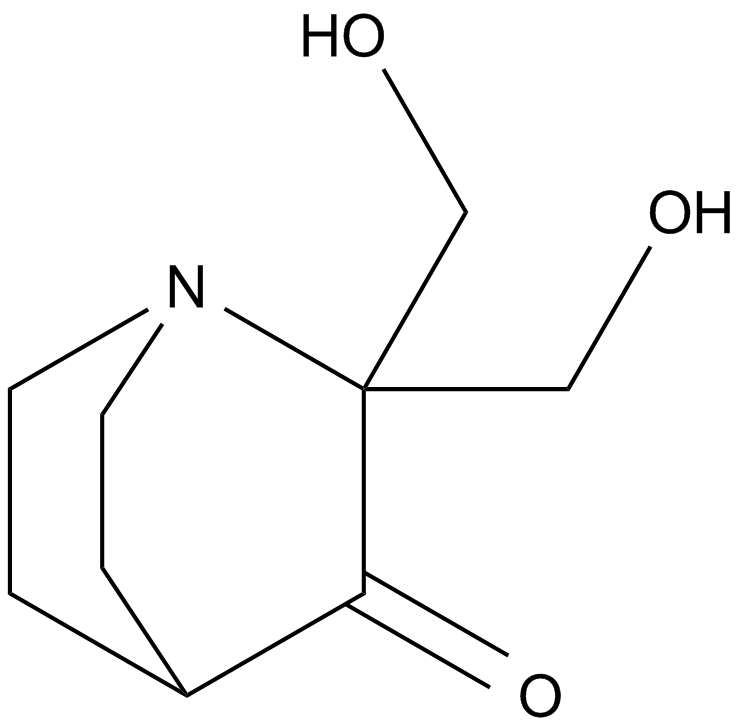
GC12086
PRIMA-1
A re-activator of the apoptotic function of mutant p53 proteins
-
GC37010
PROTAC MDM2 Degrader-1
PROTAC MDM2 Degrader-1 est un dégradeur MDM2 basé sur la technologie PROTAC. PROTAC MDM2 Degrader-1 se compose d'un puissant inhibiteur de MDM2, d'un lieur et du ligand MDM2 pour l'ubiquitine ligase E3.
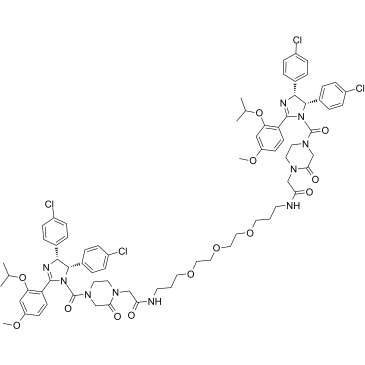
-
GC37011
PROTAC MDM2 Degrader-2
PROTAC MDM2 Degrader-2 est un dégradeur MDM2 basé sur la technologie PROTAC. PROTAC MDM2 Degrader-2 se compose d'un puissant inhibiteur de MDM2, d'un lieur et du ligand MDM2 pour l'ubiquitine ligase E3.
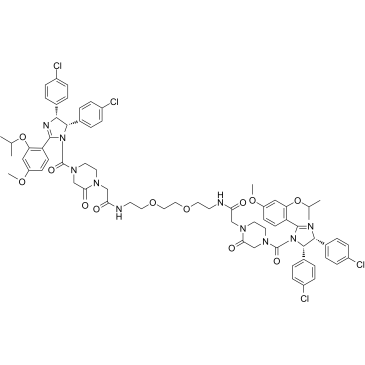
-
GC37012
PROTAC MDM2 Degrader-3
PROTAC MDM2 Degrader-3 est un dégradeur MDM2 basé sur la technologie PROTAC. PROTAC MDM2 Degrader-3 se compose d'un puissant inhibiteur de MDM2, d'un lieur et du ligand MDM2 pour l'ubiquitine ligase E3.
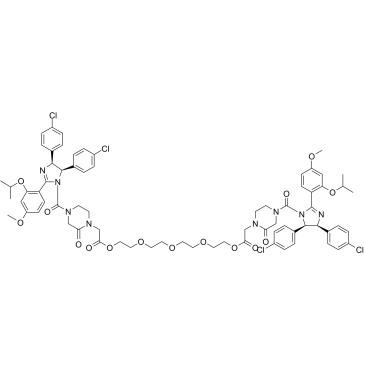
-
GC37013
PROTAC MDM2 Degrader-4
PROTAC MDM2 Degrader-4 est un dégradeur MDM2 basé sur la technologie PROTAC. PROTAC MDM2 Degrader-4 se compose d'un puissant inhibiteur de MDM2, d'un lieur et du ligand MDM2 pour l'ubiquitine ligase E3.
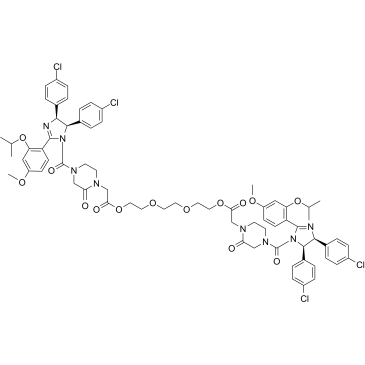
-
GC15946
ReACp53
ReACp53 pourrait inhiber la formation d'amyloÏde p53 et sauver la fonction p53 dans les lignées cellulaires cancéreuses.
-
GC13019
RG7112
An inhibitor of the MDM2-p53 interaction
-
GC11594
RG7388
An inhibitor of the MDM2-p53 interaction
-
GC12793
RITA (NSC 652287)
An inhibitor of the p53-HDM-2 interaction
-
GC37549
RO-5963
Le RO-5963 est un double inhibiteur p53-MDM2 et p53-MDMX avec des IC50 d'environ 17 nM et d'environ 24 nM, respectivement.
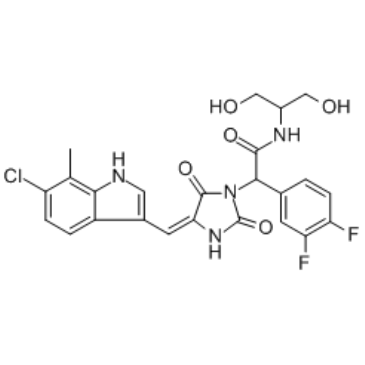
-
GC19312
RO8994
RO8994 est une série très puissante et sélective d'inhibiteurs MDM2 À petite molécule de spiroindolinone, avec une IC50 de 5 nM (tests de liaison HTRF) et 20 nM (tests de prolifération MTT).
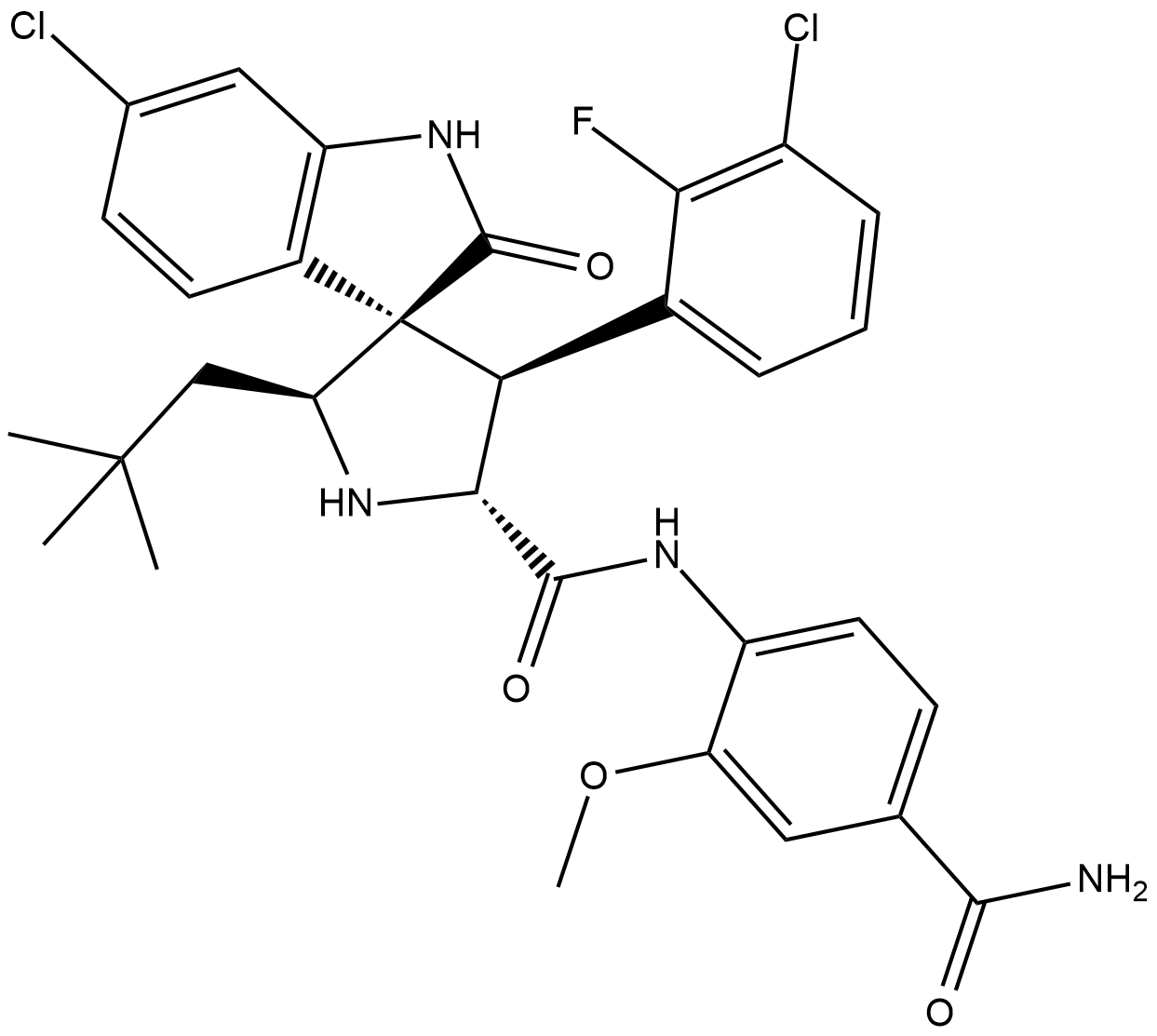
-
GC18624
Roslin-2
Roslin-2 (bromure de benzylhexaméthylènetétramine) est un réactivateur de p53 aux effets anticancéreux. Roslin-2 lie FAK, perturbe la liaison de FAK et p53.
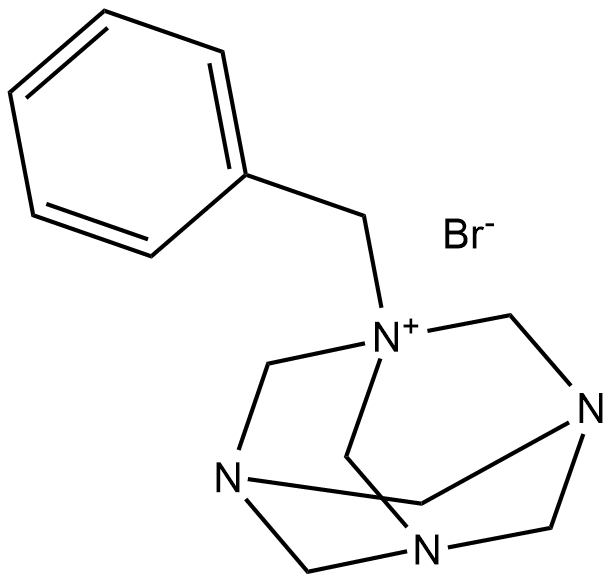
-
GC13590
SJ 172550
A small molecule inhibitor of MDMX
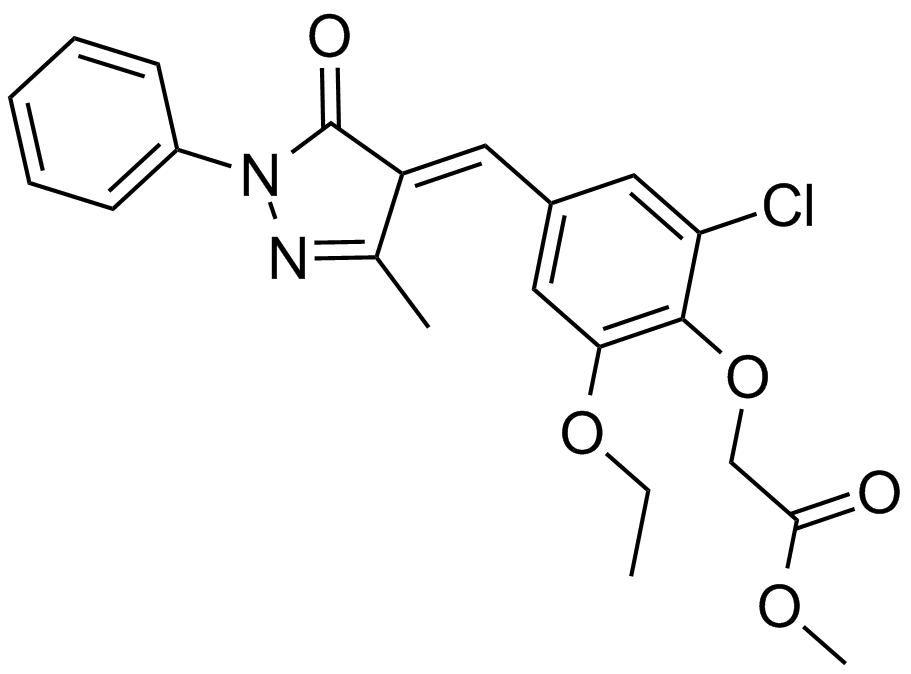
-
GC65284
SLMP53-1
SLMP53-1 est un réactivateur de p53 de type sauvage et mutant avec une activité antitumorale prometteuse. SLMP53-1 intervient dans la reprogrammation du métabolisme du glucose dans les cellules cancéreuses. SLMP53-1 épuise l'angiogenèse, diminuant la formation de tubes de cellules endothéliales et les niveaux d'expression du facteur de croissance endothélial vasculaire (VEGF).
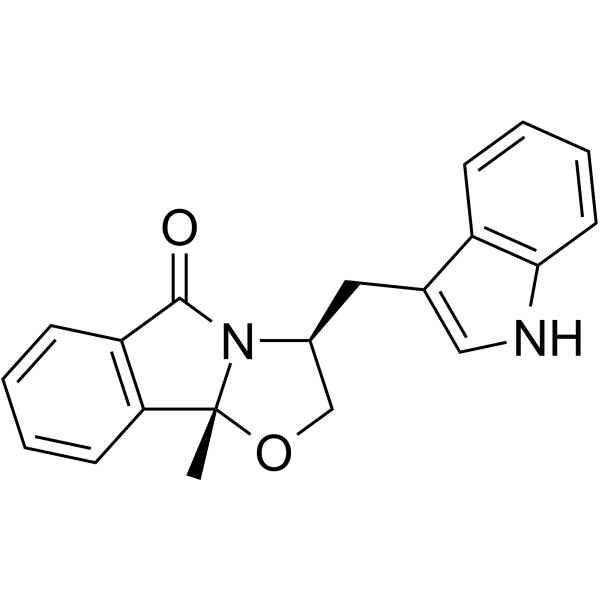
-
GC16371
Solasodine
An alkaloid with diverse biological activities
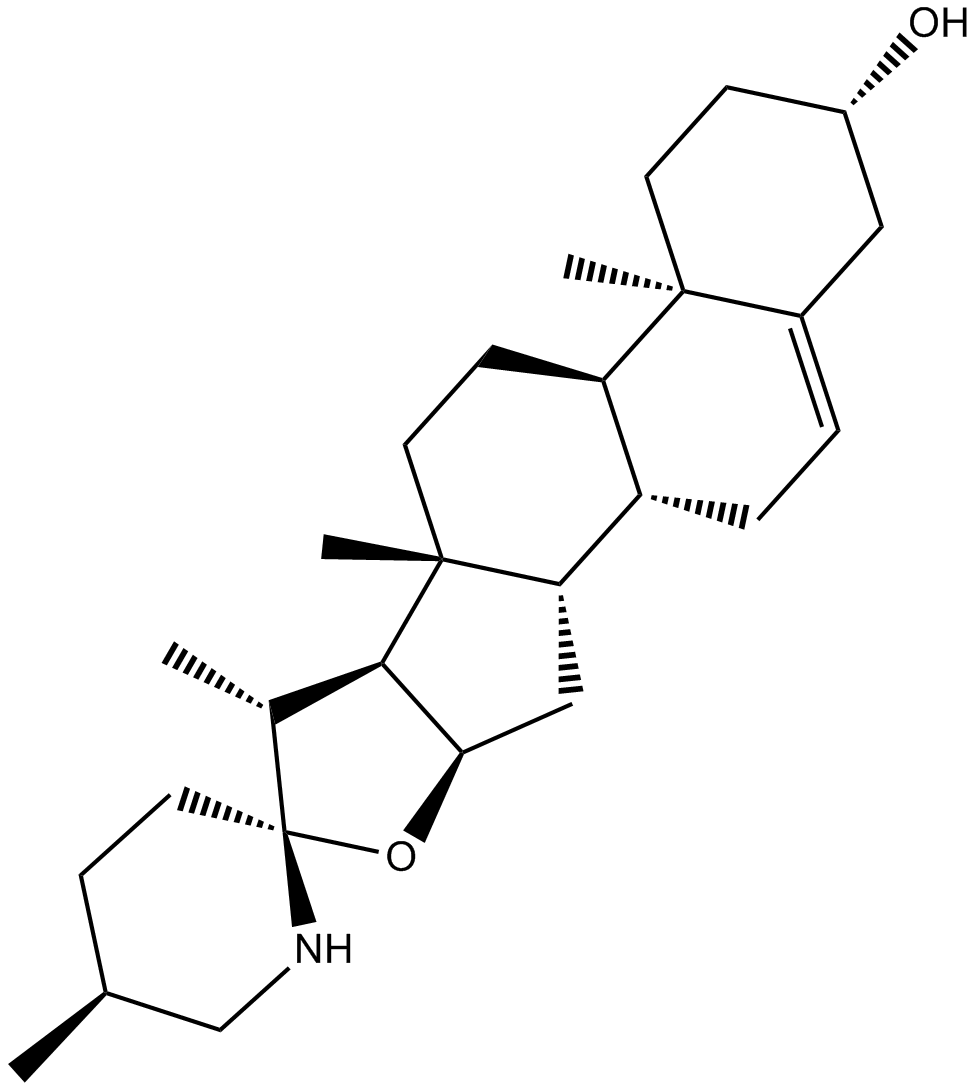
-
GC69968
Sulanemadlin
Sulanemadlin (ALRN-6924) is a potent p53-based peptidomimetic macrocycle. Sulanemadlin is an inhibitor of protein-protein interactions between p53-MDM2, p53-MDMX, or p53 and MDM2 and MDMX. Sulanemadlin can be used for cancer research.
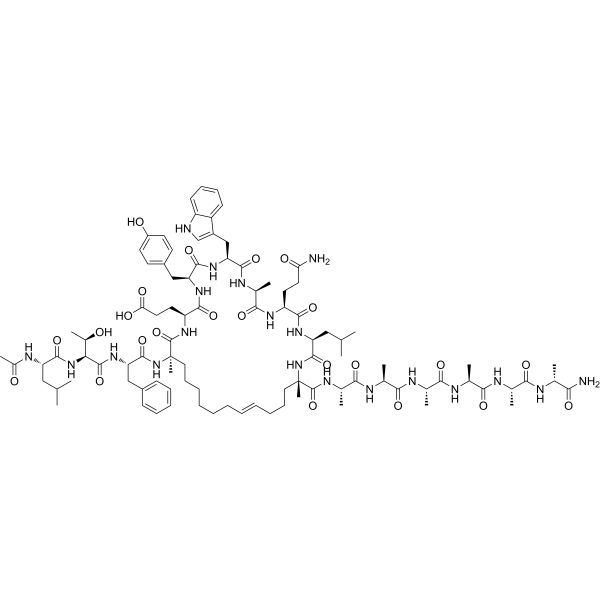
-
GC14165
Tenovin-1
A small molecule activator of p53
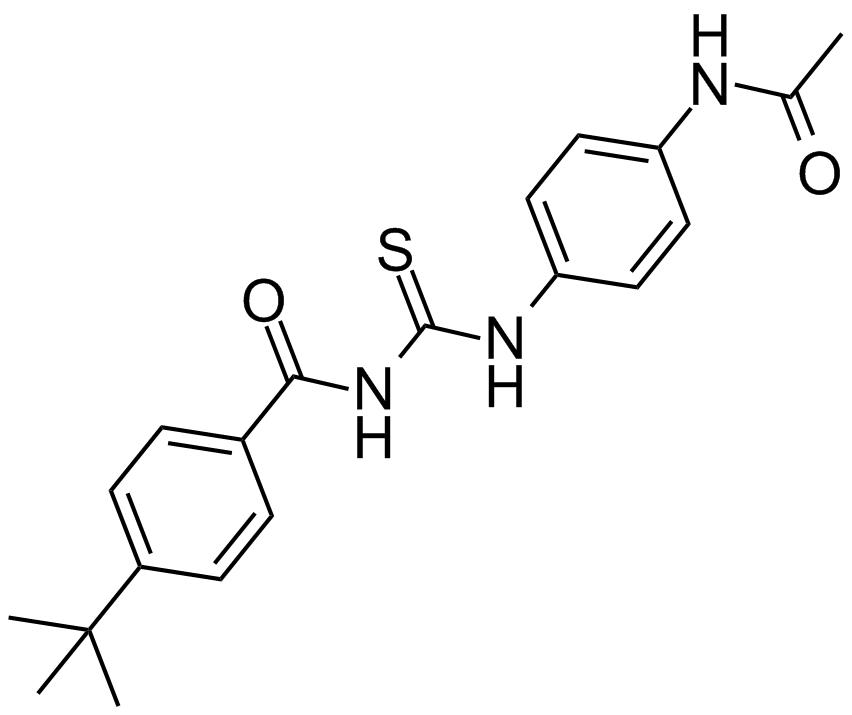
-
GC12337
Tenovin-3
Tenovin-3 est un activateur de p53.
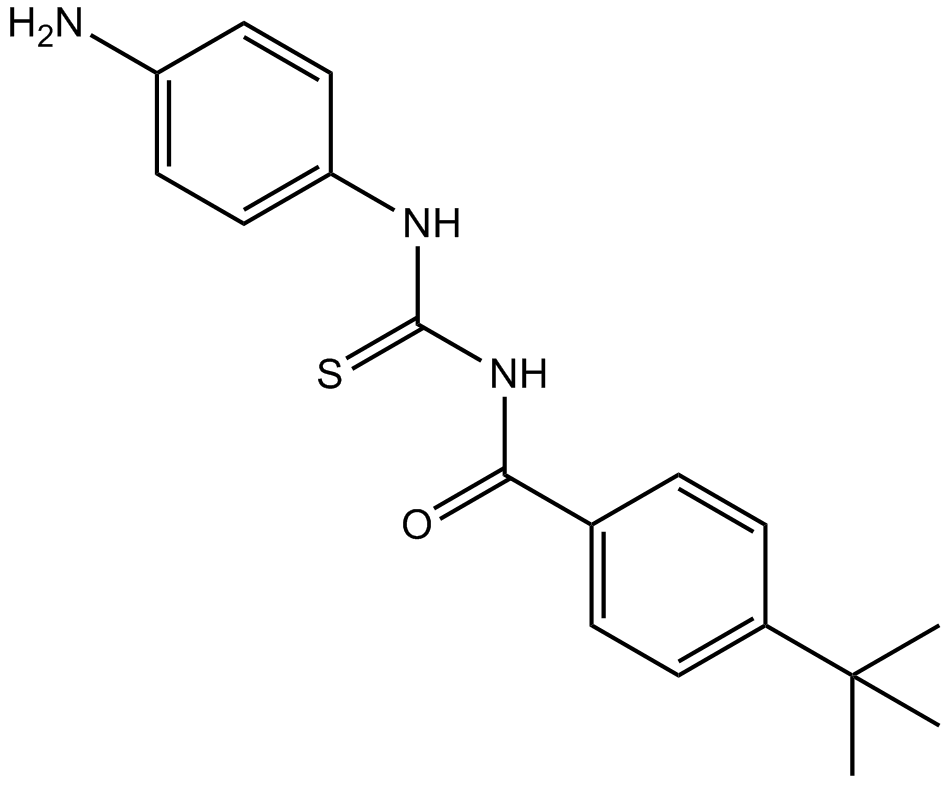
-
GC16436
Tenovin-6
La ténovine-6, un analogue de la ténovine-1, est un activateur de l'activité transcriptionnelle de p53. La ténovine-6 inhibe les activités de la protéine désacétylase des SIRT1, SIRT2 et SIRT3 humains purifiés avec des IC50 de 21 μM, 10 μM et 67 μM, respectivement. La ténovine-6 inhibe également la dihydroorotate déshydrogénase (DHODH).
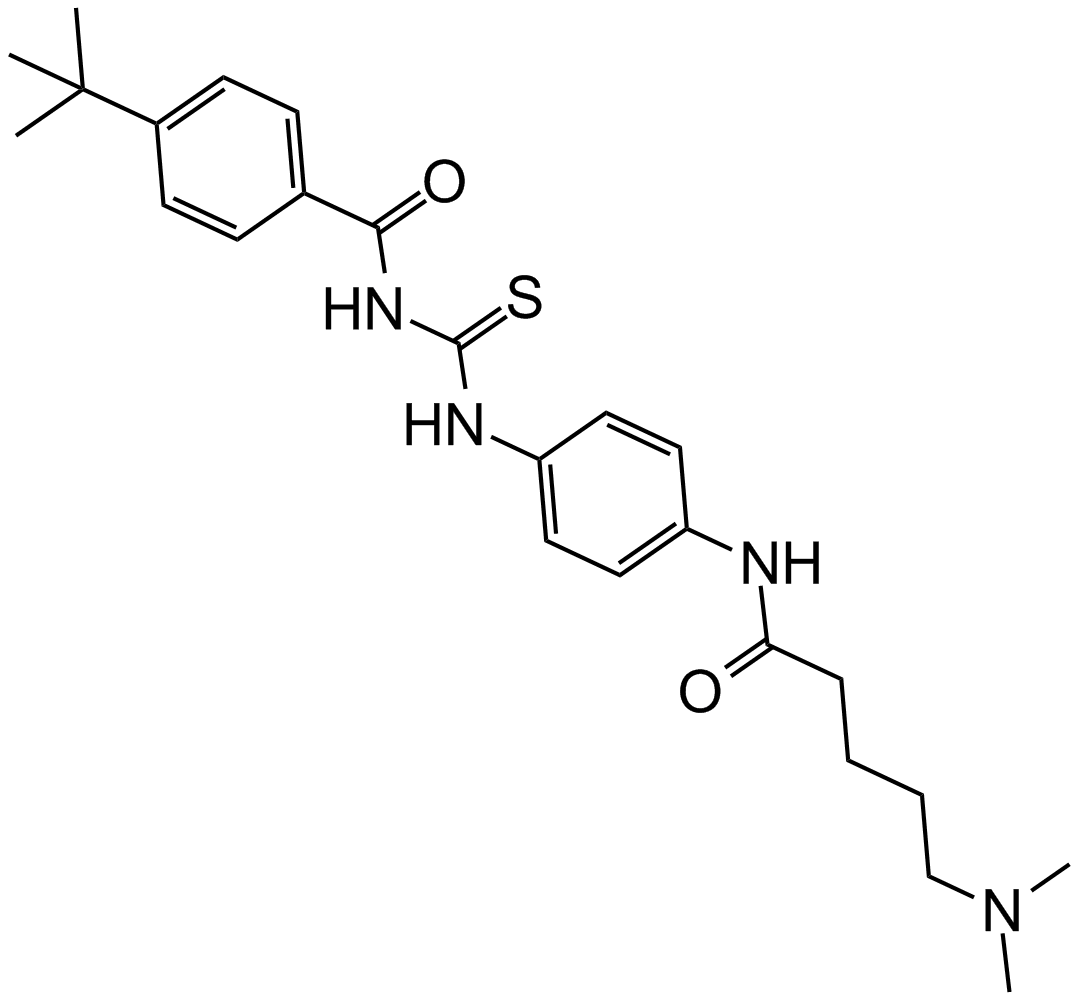
-
GC37761
Tenovin-6 Hydrochloride
Le chlorhydrate de Tenovin-6, un analogue de Tenovin-1, est un activateur de l'activité transcriptionnelle de p53. Le chlorhydrate de ténovine-6 inhibe les activités de la protéine désacétylase des SIRT1, SIRT2 et SIRT3 humains purifiés avec des CI50 de 21 μM, 10 μM et 67 μM, respectivement. Le chlorhydrate de ténovine-6 inhibe également la dihydroorotate déshydrogénase (DHODH).
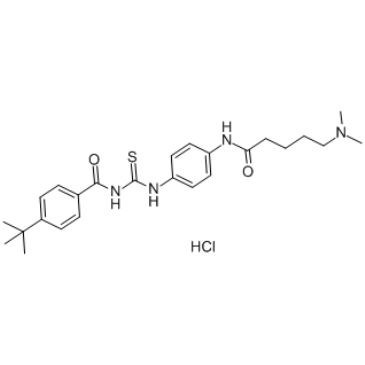
-
GC61352
Triglycidyl isocyanurate
L'isocyanurate de triglycidyle (TGIC; Teroxirone) est un triépoxyde de triazène ayant des activités antiangiogéniques et antinéoplasiques.
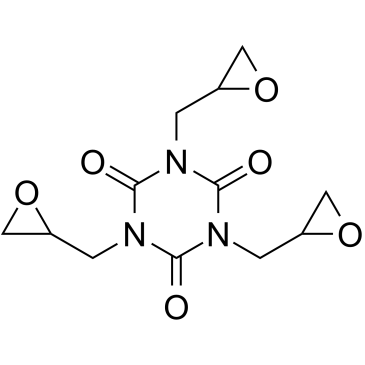
-
GP10028
tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus]
P53 binding protein fragment
![tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus] Chemical Structure tumor protein p53 binding protein fragment [Homo sapiens]/[Mus musculus] Chemical Structure](/media/struct/GP1/GP10028.png)
-
GC11557
WR 1065
A radioprotective agent
-
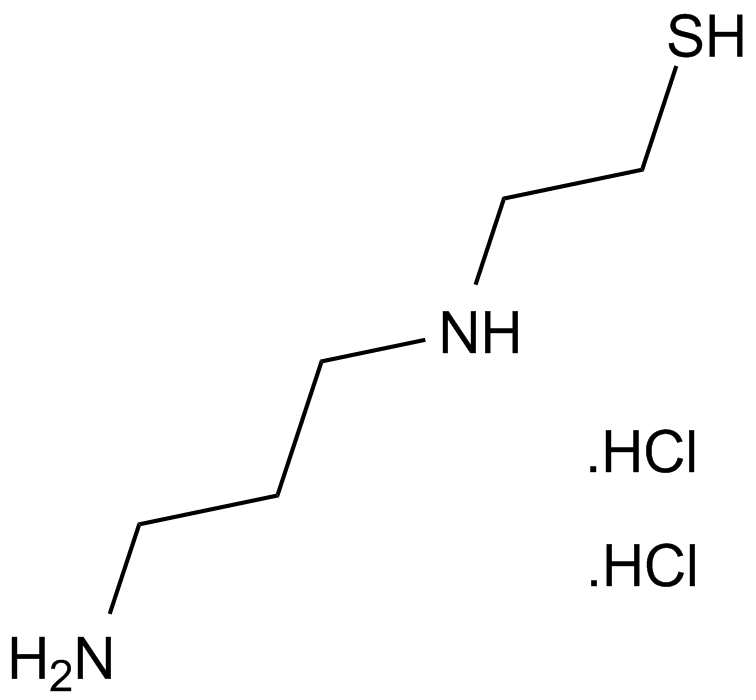
GC19545
WR-1065 dihydrochloride
WR-1065 dihydrochloride can protect normal tissues from the toxic effects of certain cancer drugs and activate p53 through a JNK-dependent signaling pathway.



