Deubiquitinase
Deubiquitinases (DUBs) are a large group of proteases that cleave ubiquitin from proteins and other molecules. Ubiquitin is attached to proteins in order to regulate the degradation of proteins via the proteasome and lysosome; coordinate thecellular localisation of proteins; activate and inactivate proteins; and modulate protein-protein interactions. DUBs can reverse these effects by cleaving the peptide or isopeptide bond between ubiquitin and its substrate protein. In humans there are nearly 100 DUB genes, which can be classified into two main classes: cysteine proteases and metalloproteases. The cysteine proteases comprise ubiquitin-specific proteases (USPs), ubiquitin C-terminal hydrolases (UCHs), Machado-Josephin domain proteases (MJDs) and ovarian tumour proteases (OTU). The metalloprotease group contains only the Jab1/Mov34/Mpr1 Pad1 N-terminal+ (MPN+) (JAMM) domain proteases. DUBs play several roles in the ubiquitin pathway. One of the best characterised functions of DUBs is the removal of monoubiqutin and polyubiquitin chainsfrom proteins.
Targets for Deubiquitinase
Products for Deubiquitinase
- Cat.No. Nombre del producto Información
-
GC39625
6RK73
6RK73 es un inhibidor covalente irreversible y especÍfico de UCHL1 con una IC50 de 0,23 μM. 6RK73 casi no muestra inhibiciÓn de UCHL3 (IC50=236 μM). 6RK73 inhibe especÍficamente la actividad de UCHL1 en el cÁncer de mama.
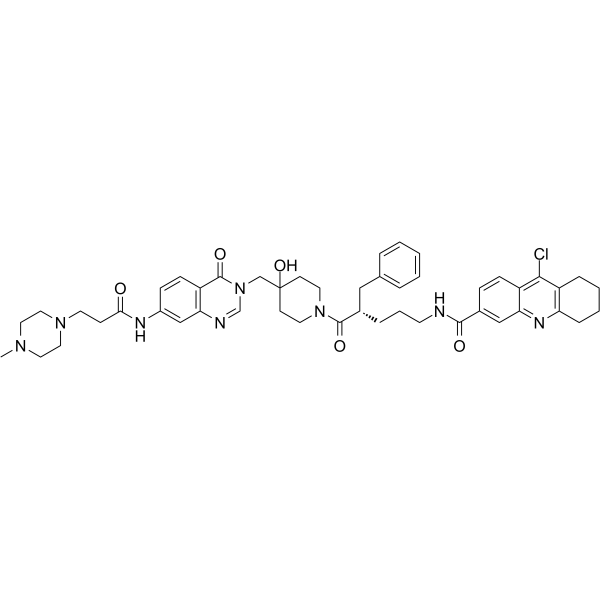
-
GC13035
Bay 11-7821
Un inhibidor selectivo e irreversible de NF-κB.
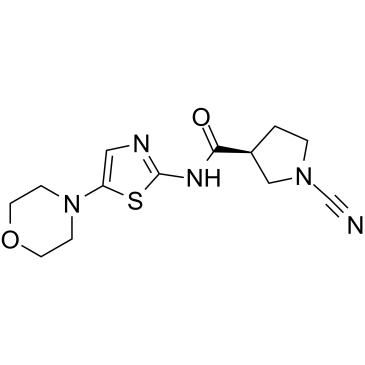
-
GC65259
BC-1471
BC-1471 es un inhibidor de la deubiquitinasa de la proteÍna de uniÓn a STAM (STAMBP).
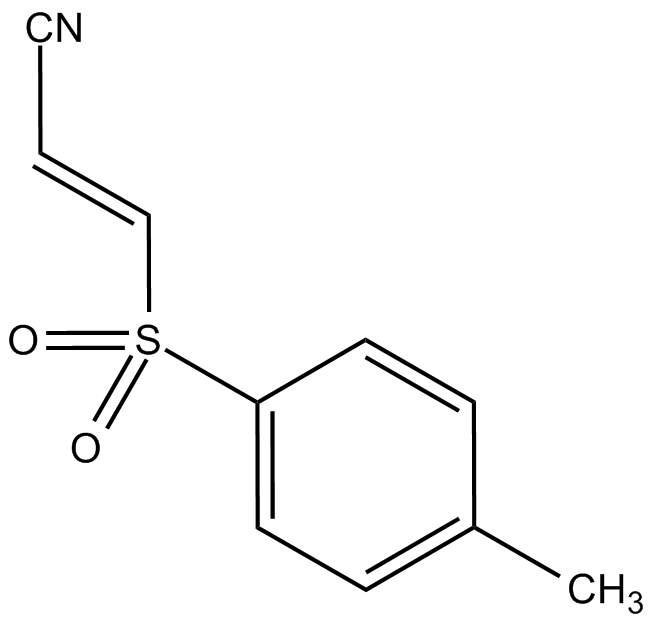
-
GC13892
C527
C527 es un inhibidor de la enzima pan DUB, con una alta potencia para el complejo USP1/UAF1 (IC50=0,88 μM).
-
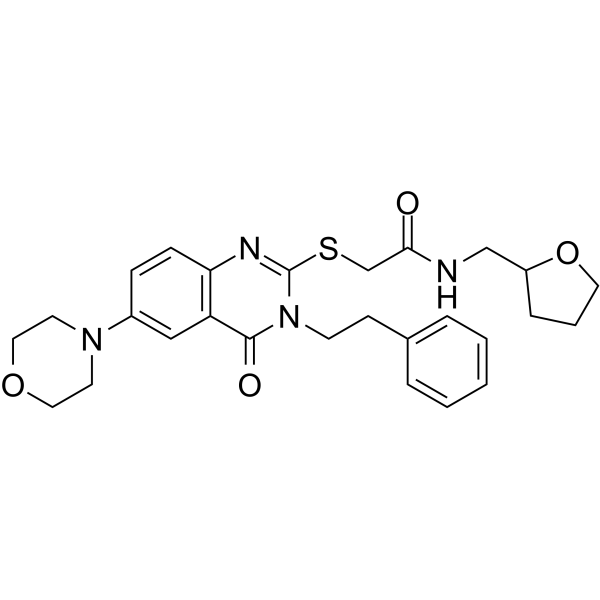
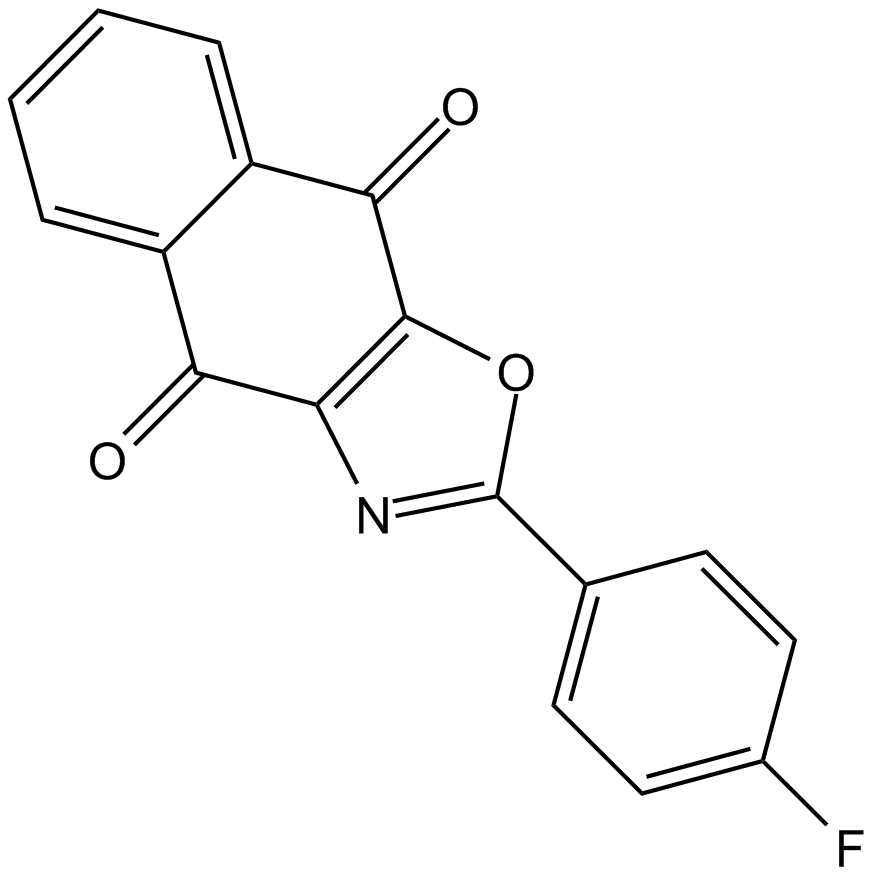
GC67886
DC-U4106

-
GC35906
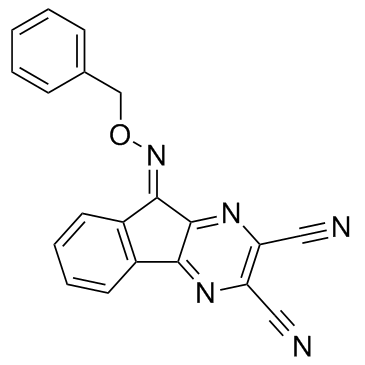
DUBs-IN-1
DUBs-IN-1 es un inhibidor activo de las proteasas especÍficas de ubiquitina (USP), con una IC50 de 0,85 μM para USP8.
-
GC17125
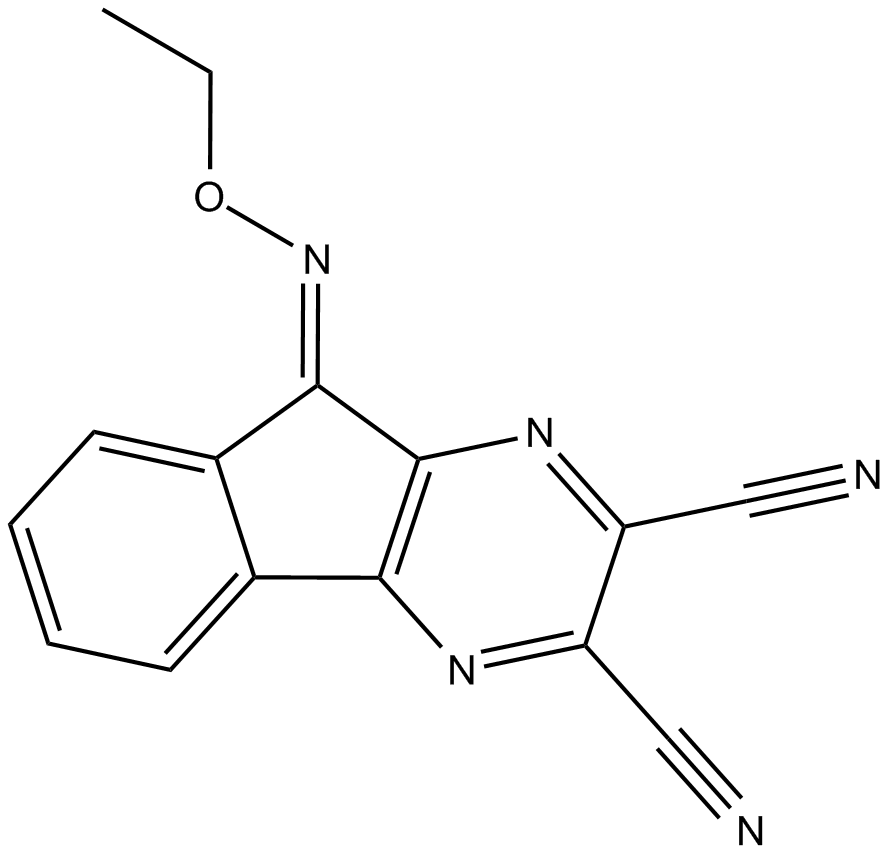
DUBs-IN-2
DUBs-IN-2 es un potente inhibidor de la deubiquitinasa con una IC50 de 0,28 μM para USP8.
-
GC10356
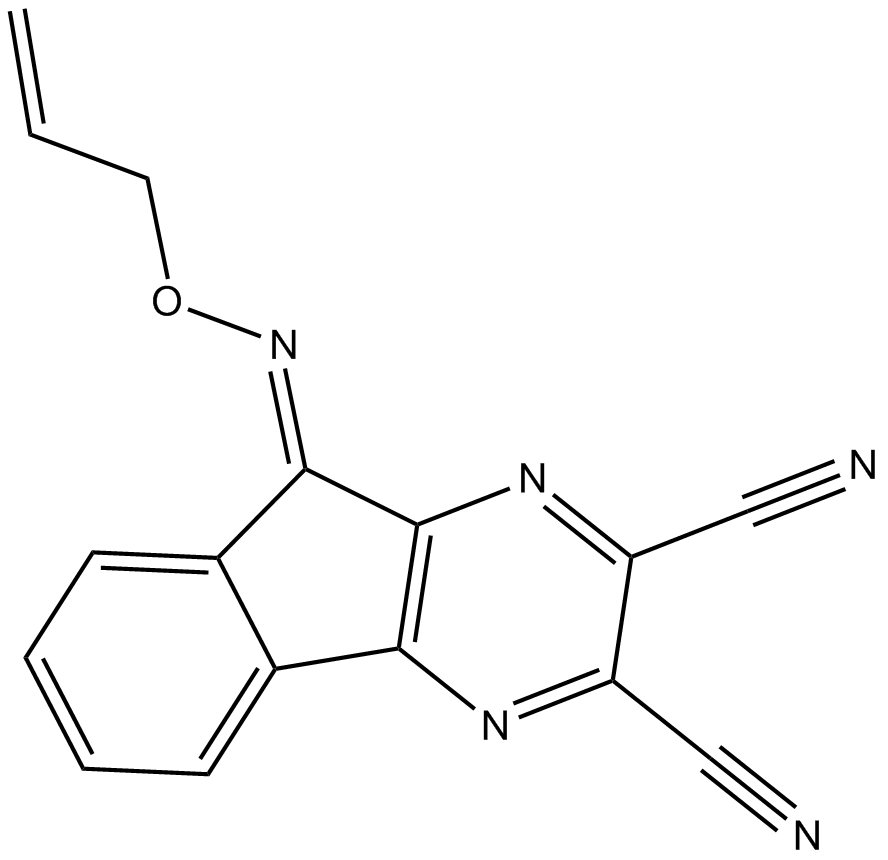
DUBs-IN-3
DUBs-IN-3 es un potente inhibidor de la enzima deubiquitinasa (USP) extraÍdo del compuesto de referencia 22c con una IC50 de 0,56 μM para USP8.
-
GC34083
EOAI3402143
EOAI3402143 es un inhibidor de la deubiquitinasa (DUB), que inhibe de forma dependiente de la dosis Usp9x/Usp24 y Usp5.
-
GC62979
FT206
FT206 es un inhibidor de carboxamidas como protrasa especÍfica de ubiquitina extraÍdo de la patente WO 2020033707 A1, ejemplo 11-1.
-
GC67915
FT3967385

-
GC32786
FT671
FT671 es un inhibidor potente, no covalente y selectivo de USP7 con una IC50 de 52 nM y se une al dominio catalítico de USP7 con un Kd de 65 nM.
-
GC69134
FT709
FT709 es un inhibidor selectivo efectivo de USP9X con un valor IC50 de 82 nM. USP9X está relacionado con la función del centrosoma, el arreglo cromosómico durante la mitosis, la degradación del receptor EGF, la sensibilización química y el ritmo circadiano.
-
GC32917
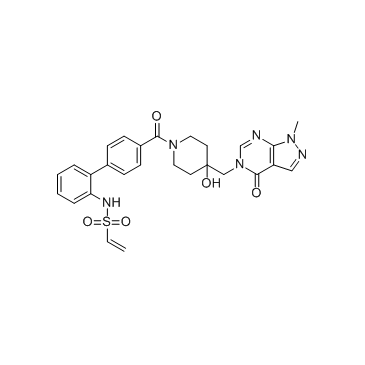
FT827
FT827 es un inhibidor selectivo y covalente de la proteasa 7 específica de ubiquitina (USP7) (Ki=4.2 ?M). FT827 se une al dominio catalítico USP7 (USP7CD; residuos 208-560) con un valor aparente de Kd de 7.8 ?M.
-
GC32856
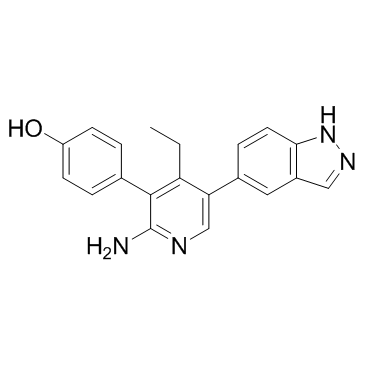
GNE-6640
GNE-6640 es un inhibidor selectivo y no covalente de la peptidasa 7 especÍfica de ubiquitina (USP7), con valores IC50 de 0,75 μM, 0,43 μM, 20,3 μM y 0,23 μM para USP7 de longitud completa, dominio catalÍtico USP7, USP43 de longitud completa y Ub- MDM2, respectivamente.
-
GC32727
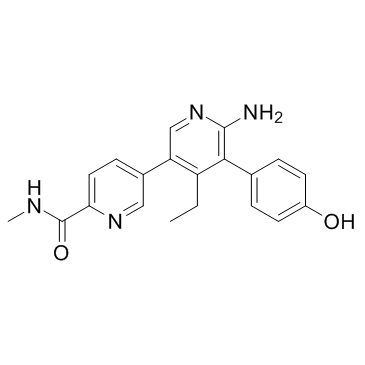
GNE-6776
GNE-6776 es un inhibidor de USP7 selectivo y biodisponible por vÍa oral.
-
GC39172
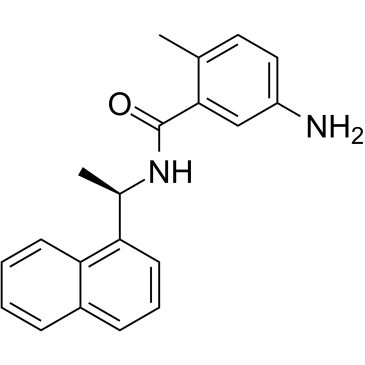
GRL0617
Un inhibidor de PLpro de SARS-CoV y SARS-CoV-2.
-
GC34354
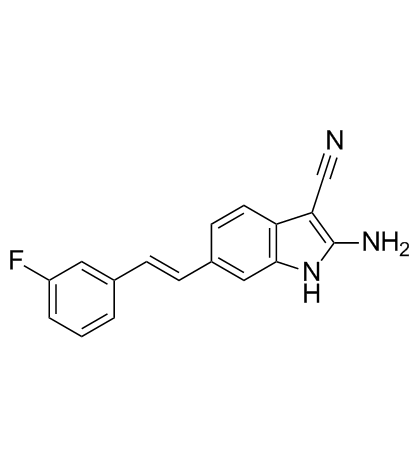
GSK2643943A
GSK2643943A es un inhibidor de la enzima desubiquitinante (DUB) dirigido a USP20. GSK2643943A tiene afinidad con un IC50 de 160 nM para USP20/Ub-Rho. GSK2643943A tiene eficacia antitumoral y se puede utilizar para la investigaciÓn del carcinoma oral de células escamosas (OSCC).
-
GC36211
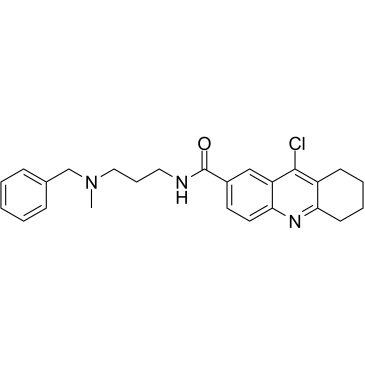
HBX 19818
HBX 19818 es un inhibidor especÍfico de la proteasa 7 especÍfica de ubiquitina (USP7), con una IC50 de 28,1 μM.
-
GC14797
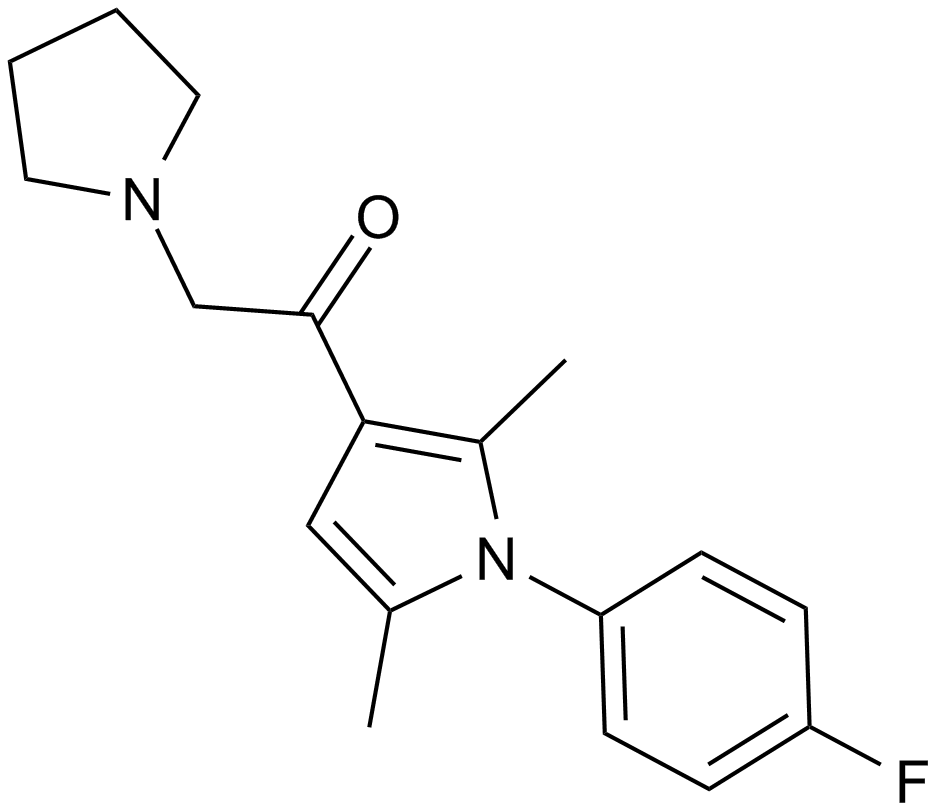
IU1
A deubiquitinating enzyme inhibitor
-
GC63027
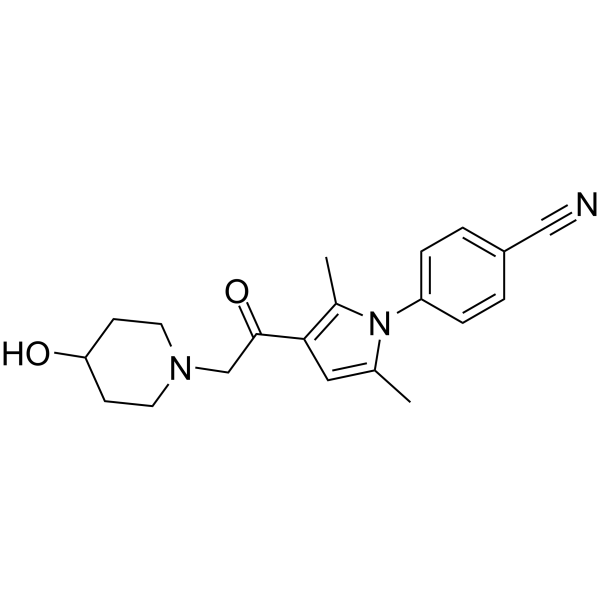
IU1-248
IU1-248, un derivado de IU1, es un inhibidor potente y selectivo de USP14 con una IC50 de 0,83μM.
-
GC64956
IU1-47
IU1-47 es un inhibidor potente y especÍfico de USP14 con una IC50 de 0,6 μM.
-
GC62460
LCAHA
LCAHA (LCA hidroxiamida) es un inhibidor de la deubiquitinasa USP2a con IC50 de 9,7 μM y 3,7 μM en el ensayo Ub-AMC y el ensayo Di-Ub, respectivamente. LCAHA desestabiliza la ciclina D1 e induce la detenciÓn de G0/G1 al inhibir la deubiquitinasa USP2a.
-
GC10510
LDN 57444
An inhibitor of UCH-L1
-
GC60981
LDN-91946
LDN-91946 es un inhibidor potente, selectivo y no competitivo de la ubiquitina C-terminal hidrolasa-L1 (UCH-L1) con una Ki app de 2,8 μM.
-
GC34670
MF-094
MF-094 es un inhibidor de USP30 potente y selectivo con una IC50 de 120 nM. MF-094 aumenta la ubiquitinaciÓn de proteÍnas y acelera la mitofagia.
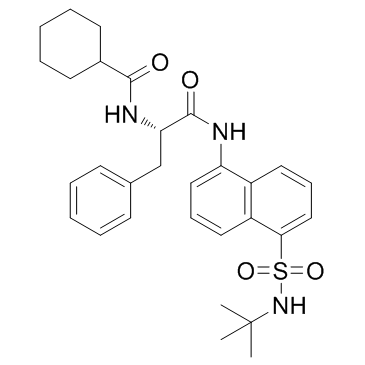
-
GC17635
ML-323
ML-323 es un inhibidor potente y reversible de USP1-UAF1 con IC50 de 76 nM en un ensayo Ub-Rho. Las constantes de inhibiciÓn medidas de ML-323 para la enzima libre (Ki) son 68 nM.
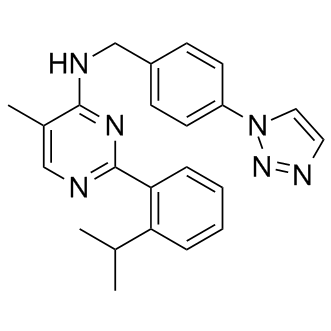
-
GC32720
ML364
ML364 es un inhibidor selectivo de la peptidasa 2 especÍfica de ubiquitina (USP2) (IC50 = 1,1 μM) con actividad antiproliferativa, que se une directamente a USP2 (Kd = 5,2 μM), induce un aumento en la degradaciÓn de la ciclina D1 celular y provoca la detenciÓn del ciclo celular. ML364 aumenta los niveles de ROS mitocondriales y disminuye el contenido intracelular de ATP.
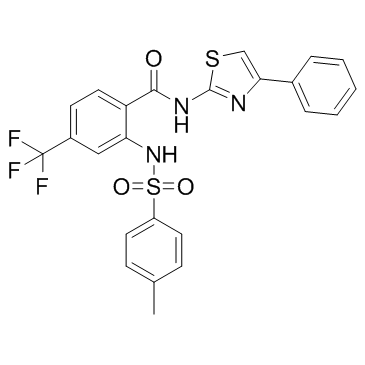
-
GC12475
NSC 632839 hydrochloride
A deubiquitylase and deSUMOylase inhibitor
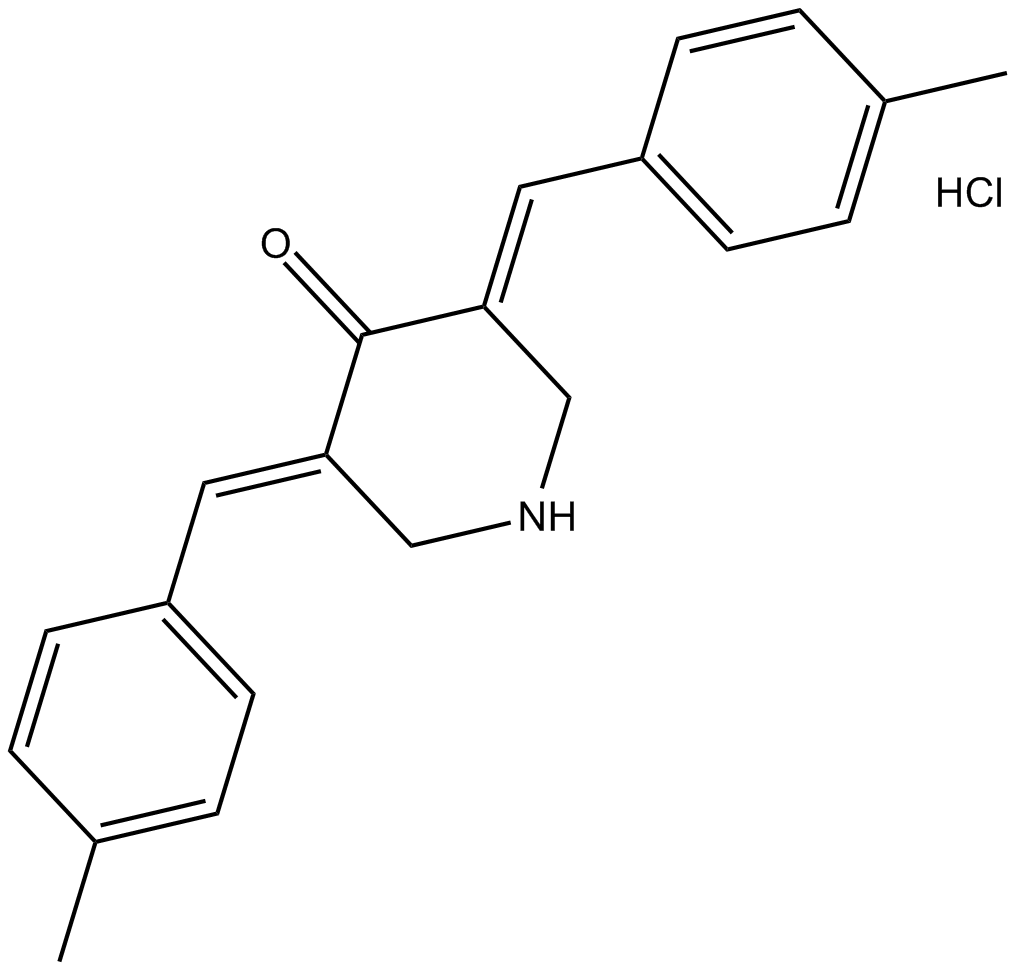
-
GC15503
NSC 687852 (b-AP15)
An inhibitor of the deubiquitinases USP14 and UCHL5
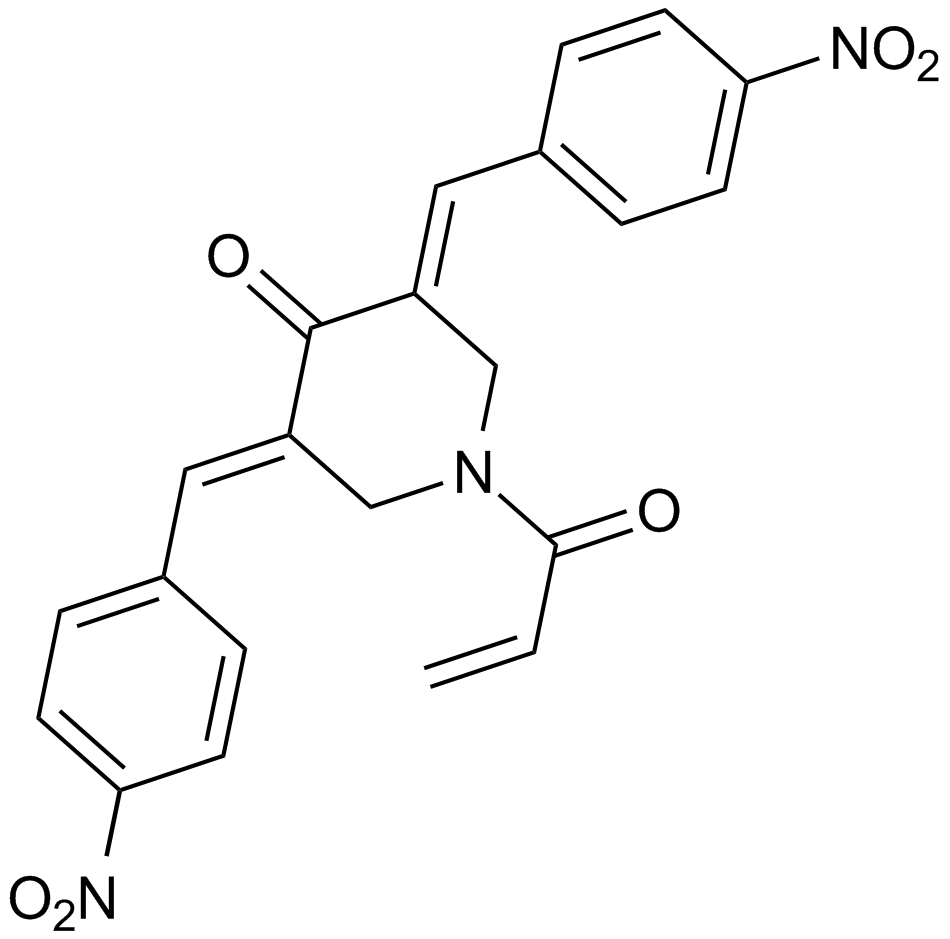
-
GC69637
OTUB1/USP8-IN-1
OTUB1/USP8-IN-1 es un inhibidor doble efectivo de OTUB1/USP8, con valores de IC50 de 0.17 y 0.28 nM para la inhibición de OTUB1 y USP8, respectivamente. OTUB1/USP8-IN-1 se puede utilizar en investigaciones sobre el cáncer.
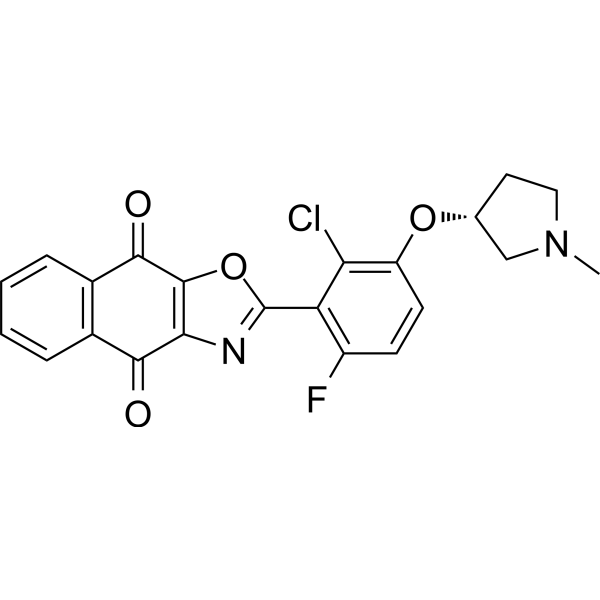
-
GC10379
P 22077
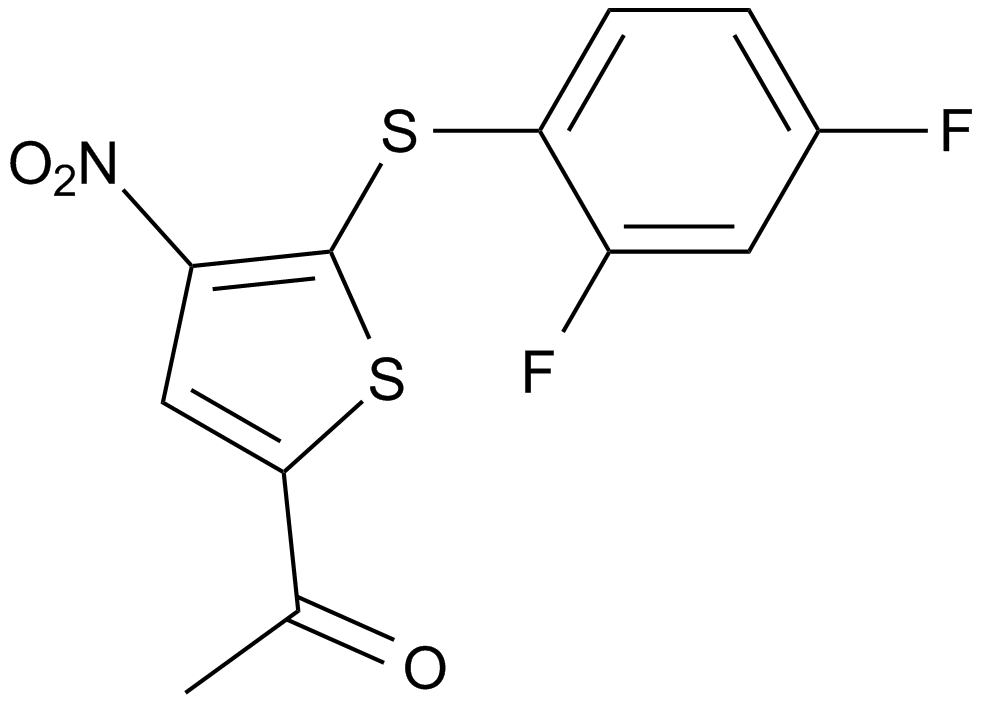
-
GC12067
P005091
A selective inhibitor of ubiquitin-specific protease 7
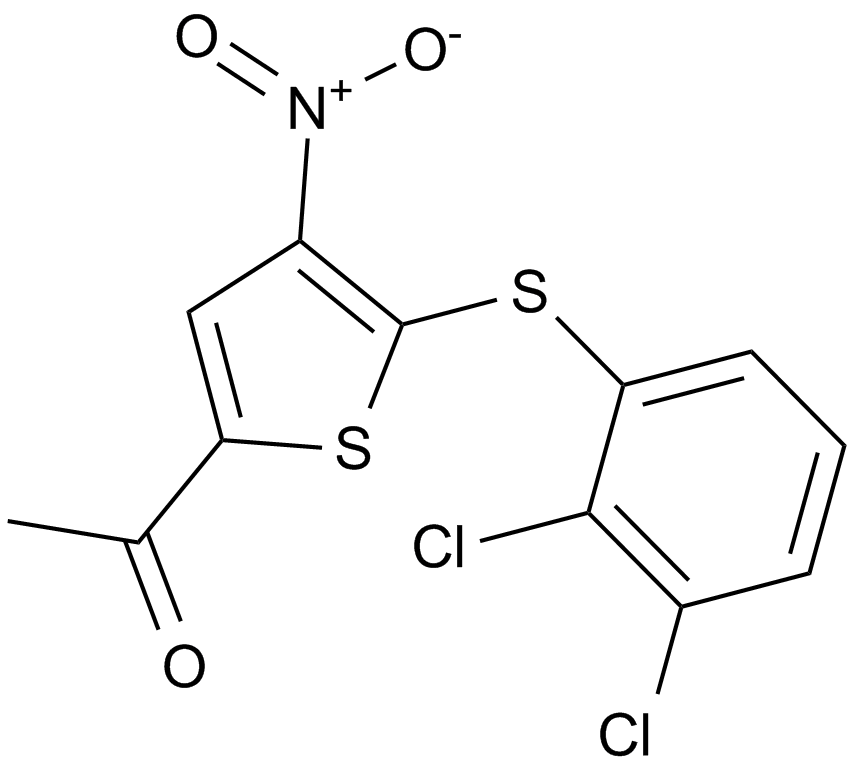
-
GC13208
PR-619
PR-619 is a broad-spectrum DUB (deubiquitinating enzyme) inhibitor. PR-619 has demonstrated robust DUB inhibitory activity (5-20 µM) and growth inhibitory activity with IC50 of 2 µM in HEK 293T cells..
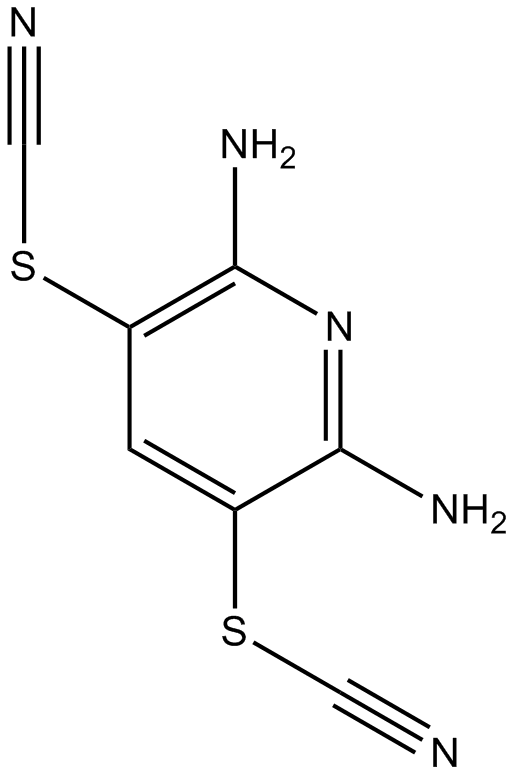
-
GC60317
RA-9
RA-9 es un inhibidor potente y selectivo de las enzimas desubiquitinantes asociadas al proteasoma (DUB) con un perfil de toxicidad favorable y actividad anticancerÍgena. RA-9 bloquea la degradaciÓn de proteÍnas dependiente de ubiquitina sin afectar la actividad proteolÍtica del proteasoma 20S. RA-9 induce selectivamente el inicio de la apoptosis en lÍneas celulares de cÁncer de ovario y cultivos primarios derivados de donantes. RA-9 induce respuestas de estrés del retÍculo endoplÁsmico (ER) en células de cÁncer de ovario.
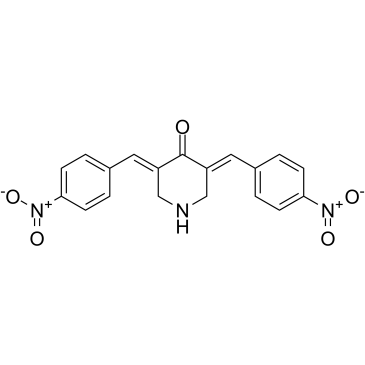
-
GC17698
SJB2-043
An inhibitor of the USP1-UAF1 complex and SARS-CoV-2 PLpro
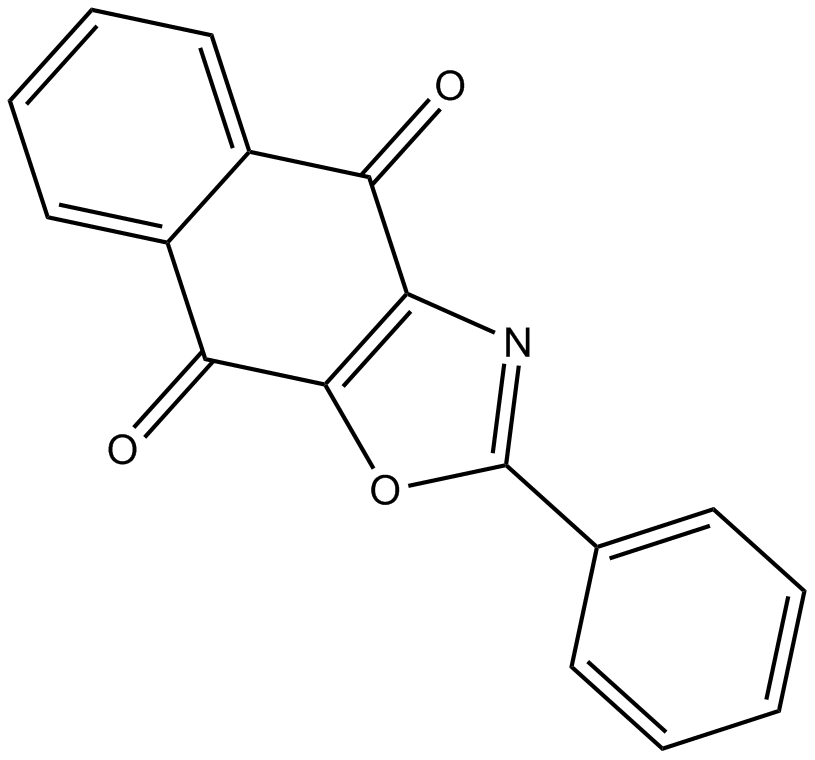
-
GC13401
SJB3-019A
SJB3-019A es un inhibidor de USP1 potente y novedoso, 5 veces mÁs potente que SJB2-043 para promover la degradaciÓn de ID1 y la citotoxicidad en células K562 con IC50 de 0,0781 μM.
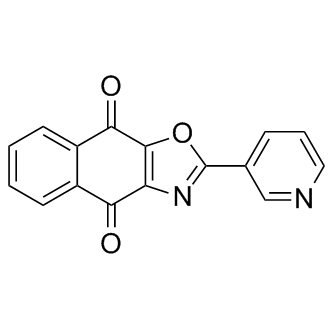
-
GC67712
STAMBP-IN-1
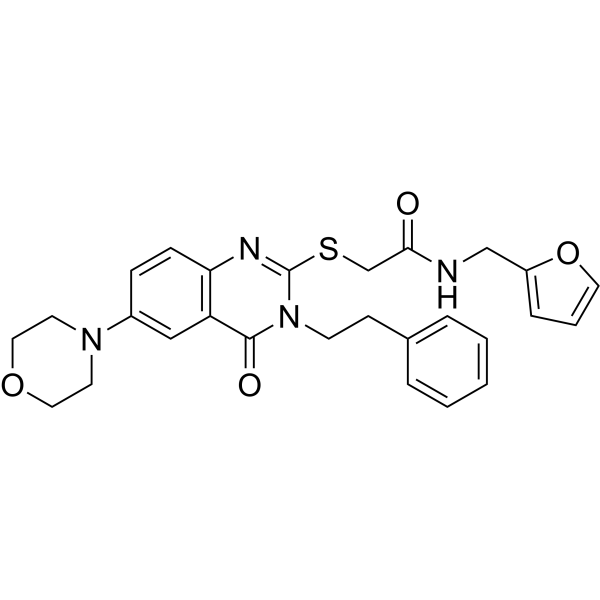
-
GC62509
STD1T
STD1T es un inhibidor de la deubiquitinasa USP2a con una IC50 de 3,3 μM en el ensayo Ub-AMC.
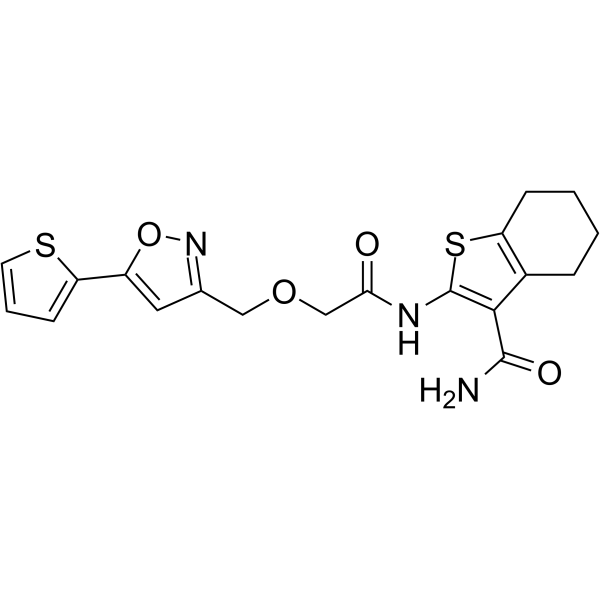
-
GC13013
TCID
TCID (4,5,6,7-tetracloroindano-1,3-diona) es un inhibidor potente y selectivo de la hidrolasa C-terminal de ubiquitina neuronal (UCH-L3) con una IC50 de 0,6 μM.
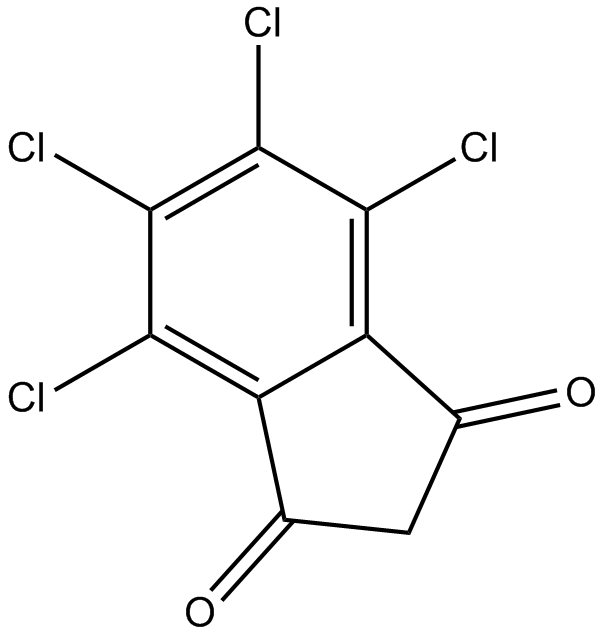
-
GC14366
Thioguanine
A thiopurine analog
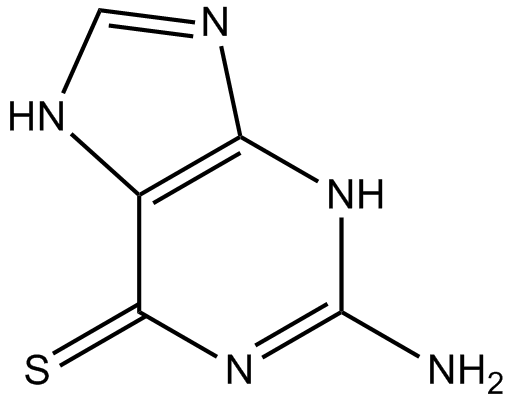
-
GC70098
USP1-IN-2
USP1-IN-2 (Compuesto I-193) is an effective inhibitor of USP1, with an IC50 less than 50 nM.
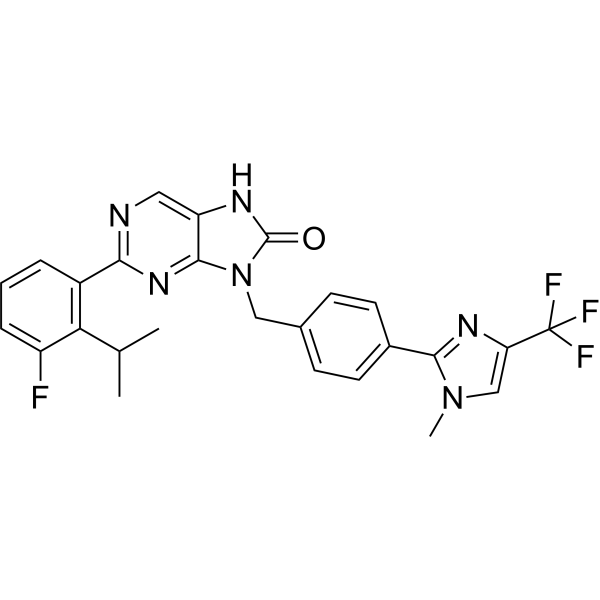
-
GC67963
USP15-IN-1
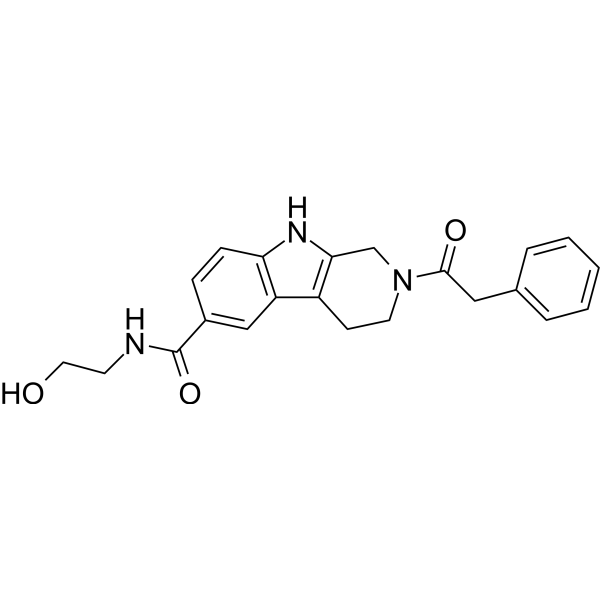
-
GC38043
USP25/28 inhibitor AZ1
El inhibidor AZ1 de USP25/28 (AZ1) es un inhibidor dual, selectivo, no competitivo y oralmente activo de las proteasas específicas de ubiquitina (USP) 25/28, con IC50s de 0.7 μM y 0.6 μM, respectivamente.
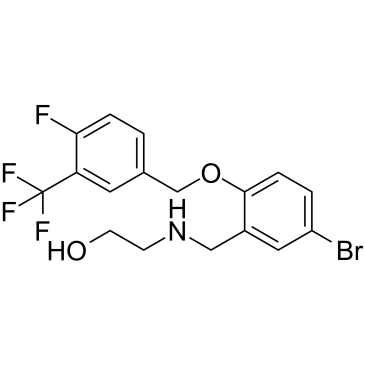
-
GC70099
USP30 inhibitor 11
El inhibidor USP30 11 es un inhibidor selectivo y efectivo de USP30, con un valor IC50 de 0.01 µμ. El inhibidor USP30 11 es el ejemplo 83, derivado del documento de patente WO2017009650A1, utilizado para investigar el cáncer y enfermedades relacionadas con disfunciones mitocondriales.
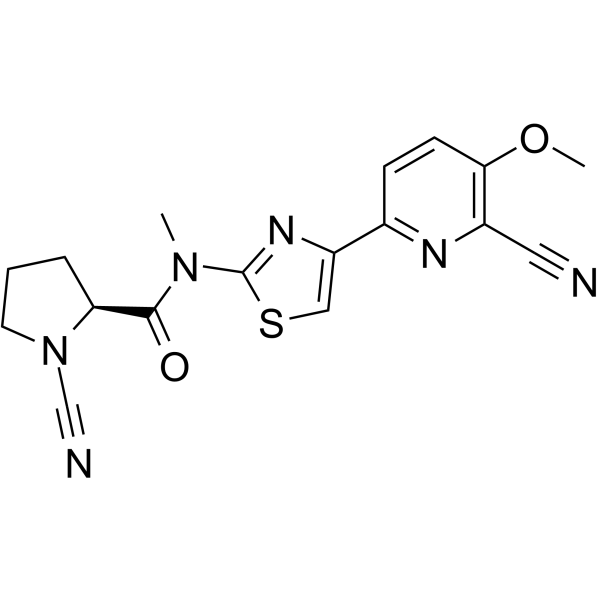
-
GC62410
USP30 inhibitor 18
El inhibidor 18 de USP30 es un inhibidor selectivo de USP30 con una IC50 de 0,02 μM. El inhibidor 18 de USP30 aumenta la ubiquitinaciÓn de proteÍnas y acelera la mitofagia.
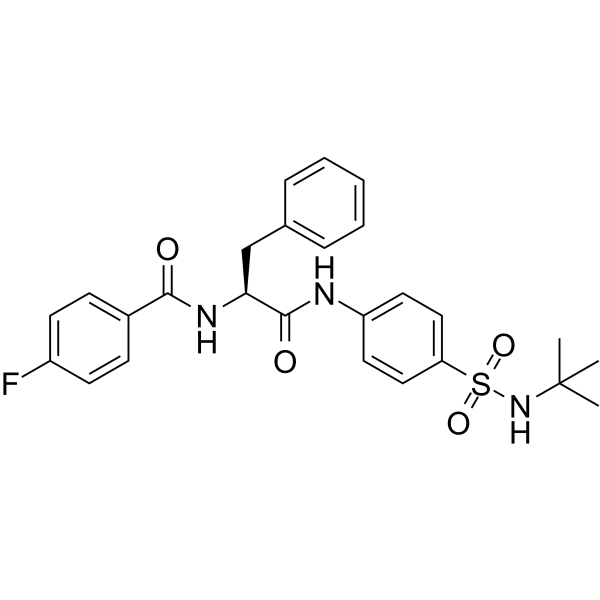
-
GC66438
USP5-IN-1
USP5-IN-1 (compuesto 64), un potente inhibidor de la desubiquitinasa USP5, se une al dominio ZnF-UBD de USP5 con una KD de 2.8 μM. USP5-IN-1 es selectivo sobre nueve proteínas que contienen dominios ZnF-UBD estructuralmente similares. USP5-IN-1 inhibe la escisión catalítica de USP5 en un sustrato di-ubiquitina.
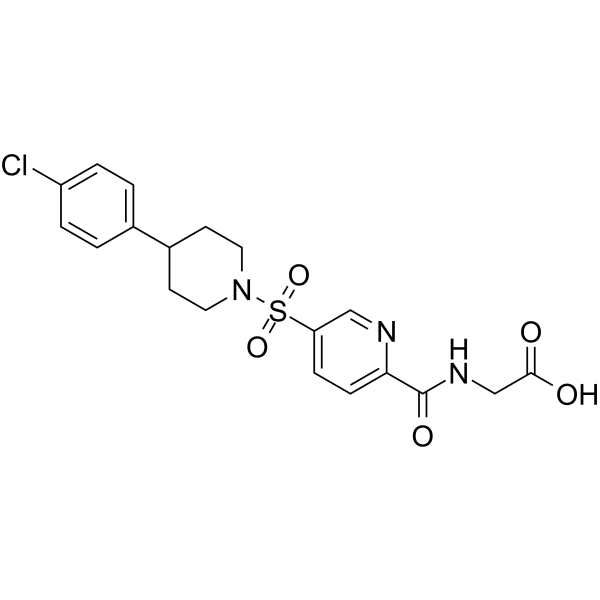
-
GC37875
USP7-IN-1
USP7-IN-1 es un inhibidor selectivo y reversible de la proteasa 7 especÍfica de ubiquitina (USP7), con una IC50 de 77 μM, y se puede utilizar para la investigaciÓn del cÁncer.
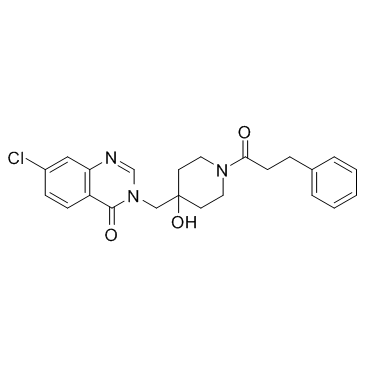
-
GC70100
USP7-IN-11
USP7-IN-11 es un inhibidor efectivo de la deubiquitinasa proteína específica de ubiquitina 7 (USP7), con una IC50 de 0.37 nM. USP7-IN-11 tiene propiedades anticancerígenas (WO2022048498A1; ejemplo 187).
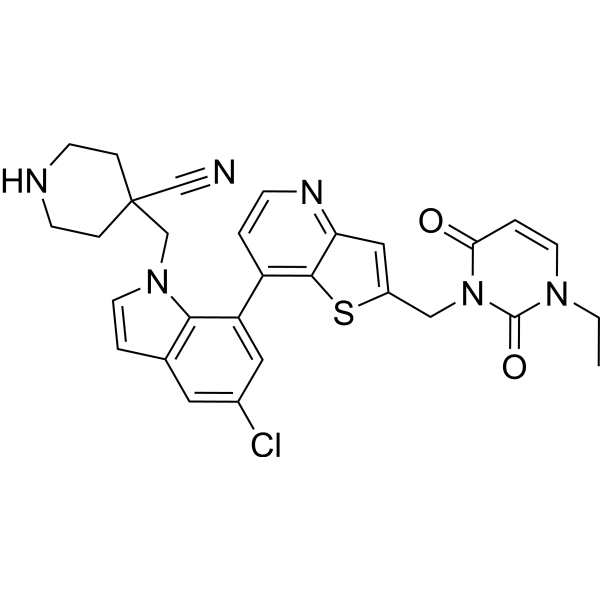
-
GC65275
USP7-IN-3
USP7-IN-3 (Compuesto 5) es un potente y selectivo inhibidor de la proteasa 7 especÍfica de ubiquitina alostérica (USP7).
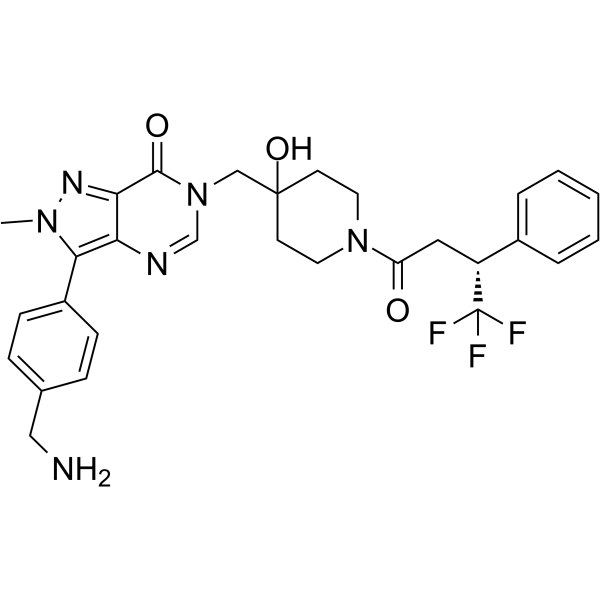
-
GC70101
USP7-IN-7
USP7-IN-7 (compuesto 124) es un inhibidor de USP7 con un valor IC50 <10 nM. USP7-IN-7 tiene una citotoxicidad en células de cáncer mutante p53, tumores sanguíneos con p53 tipo salvaje y células de neuroblastoma a niveles nanomolares bajos. USP7-IN-7 se puede utilizar para la investigación del cáncer.
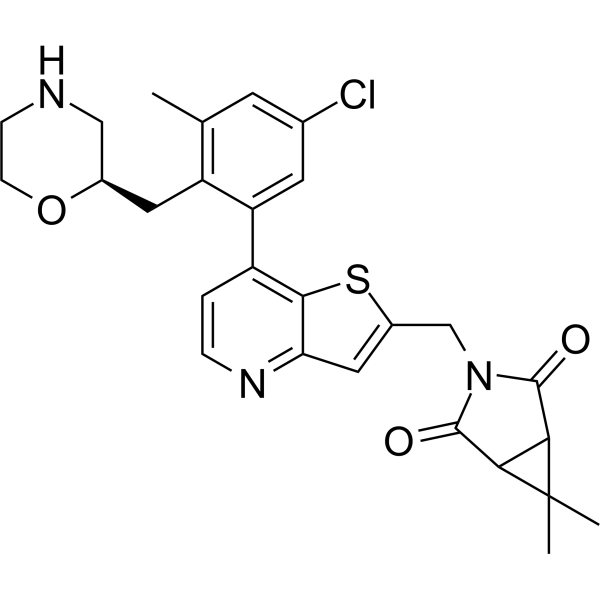
-
GC62377
USP7-IN-8
USP7-IN-8 (ejemplo 81) es un inhibidor selectivo de la proteasa 7 especÍfica de ubiquitina (USP7) con una CI50 de 1,4 μM en un ensayo Ub-Rho110. USP7-IN-8 no muestra actividad contra USP47 y USP5. USP7-IN-8 tiene efectos anticancerÍgenos.
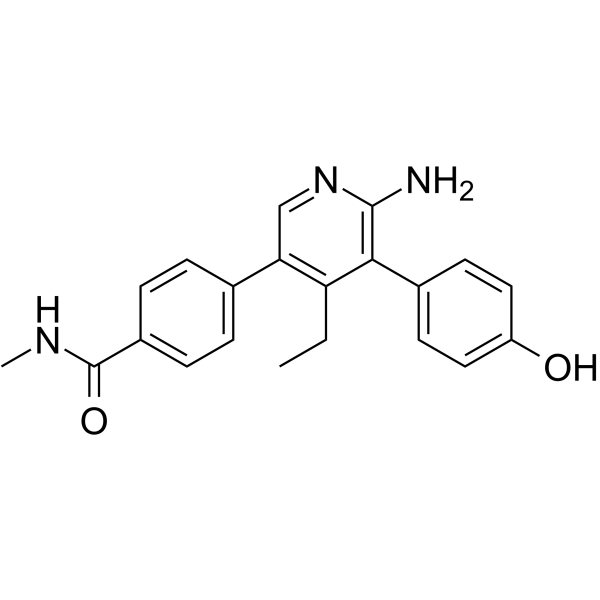
-
GC70102
USP7-IN-9
USP7-IN-9 is an efficient inhibitor of USP7 with an IC50 of 40.8 nM. USP7-IN-9 can induce apoptosis in RS4;11 cells and block the cell cycle at G0/G1 and S phase. USP7-IN-9 reduces the levels of cancer proteins MDM2 and DNMT1, while increasing the levels of tumor suppressor proteins p53 and p21.
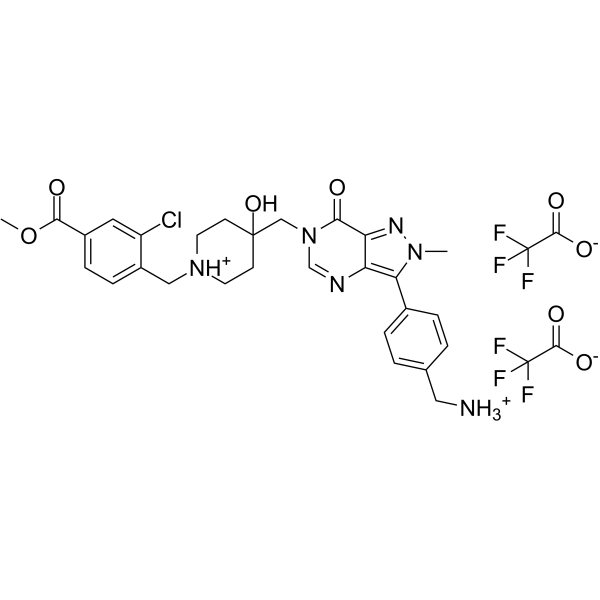
-
GC18160
USP7/USP47 inhibitor
El inhibidor de USP7/USP47 es un inhibidor selectivo de la proteasa 7/47 (USP7/USP47) especÍfico de ubiquitina, con EC50 de 0,42 μM y 1,0 μM, respectivamente.
-
GC64537
USP8-IN-1
USP8-IN-1 es un inhibidor de USP8 con una IC50 de 1,9 μM. USP8-IN-1 inhibe el crecimiento de células H1975 con un GI50 de 82,04 μM (CN111138358A; U10).
-
GC70103
USP8-IN-3
USP8-IN-3 (Compd U51) es un inhibidor de la enzima desubiquitinante USP8, con un valor IC50 de 4.0 μM. USP8-IN-3 también inhibe la proliferación celular de las células GH3 y H1957, con valores GI50 de 37.03 μM y 6.01 μM respectivamente.
-
GC19378
VLX1570
VLX1570 es un inhibidor competitivo de las proteasomas deubiquitinasas (DUB) con una IC50 de aproximadamente 10 μM.
-
GC10970
WP1130
WP1130 (WP1130) es un inhibidor de la deubiquitinasa permeable a las células (DUB), que inhibe directamente la actividad DUB de USP9x, USP5, USP14 y UCH37. Se ha demostrado que WP1130 regula a la baja las proteÍnas antiapoptÓticas Bcr-Abl y JAK2.
-
GC62517
XL177A
XL177A es un inhibidor irreversible de USP7 muy potente y selectivo con una IC50 de 0,34nM. XL177A provoca la muerte de células cancerosas a través de un mecanismo dependiente de p53.