Epigenetic Reader Domain
Epigenetic regulators of gene expression and chromatin state include so-called writers, erasers, and readers of chromatin modifications.Well-characterized examples of reader domains include bromodomains typically binding acetyllysine and chromatin organization modifier (chromo), malignant brain tumor (MBT), plant homeodomain (PHD), and Tudor domains generally associating with methyllysine. Research on epigenetic readers has been tremendously influenced by the discovery of selective inhibitors targeting the bromodomain and extraterminal motif (BET) family of acetyl-lysine readers. The human genome encodes 46 proteins containing 61 bromodomains clustered into eight families. Distinct experimental approaches are used to identify the first BET inhibitors, GSK 525762A and (+)-JQ-1.
The Polycomb group (PcG) protein, enhancer of zeste homologue 2 (EZH2), has an essential role in promoting histone H3 lysine 27 trimethylation (H3K27me3) and epigenetic gene silencing. This function of EZH2 is important for cell proliferation and inhibition of cell differentiation, and is implicated in cancer progression. Cyclin-dependent kinases regulate epigenetic gene silencing through phosphorylation of EZH2. In many types of cancers including lymphomas and leukemia, EZH2 is postulated to exert its oncogenic effects via aberrant histone and DNA methylation, causing silencing of tumor suppressor genes.
p300/CBP is not only a transcriptional adaptor but also a histone acetyltransferase.
Targets for Epigenetic Reader Domain
Products for Epigenetic Reader Domain
- Cat.No. Nombre del producto Información
-
GC34078
I-CBP112
I-CBP112 es un inhibidor de la interacciÓn proteÍna-proteÍna competitivo de acetil-lisina especÍfico y potente, que inhibe los bromodominios CBP/p300, mejora la acetilaciÓn por p300.
-
GC43889
I-CBP112 (hydrochloride)
I-CBP112 (clorhidrato) es un inhibidor selectivo de CBP/P300 que se une directamente a sus bromodominios (Kds \u003d 142 y 625 nM, respectivamente). I-CBP112 reduce significativamente el potencial iniciador de leucemia de las células de leucemia mieloide aguda MLL-AF9(+) de manera dependiente de la dosis in vitro e in vivo. I-CBP112 aumenta la actividad citotóxica del inhibidor del bromodominio BET JQ1, así como de la doxorrubicina.
-
GC33042
IACS-9571 (ASIS-P040)
IACS-9571 (ASIS-P040) es un inhibidor potente y selectivo de TRIM24 y BRPF1, con una IC50 de 8 nM para TRIM24, y Kds de 31 nM y 14 nM para TRIM24 y BRPF1, respectivamente.
-
GC60925
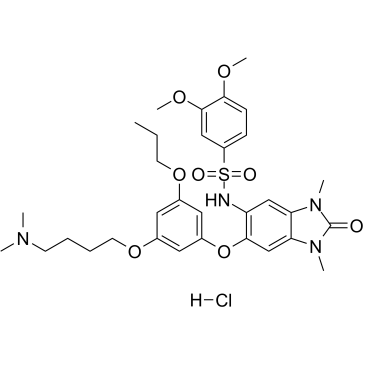
IACS-9571 hydrochloride
El clorhidrato de IACS-9571 (ASIS-P040) es un inhibidor potente y selectivo de TRIM24 y BRPF1, con una IC50 de 8 nM para TRIM24 y Kds de 31 nM y 14 nM para TRIM24 y BRPF1, respectivamente.
-
GC34437
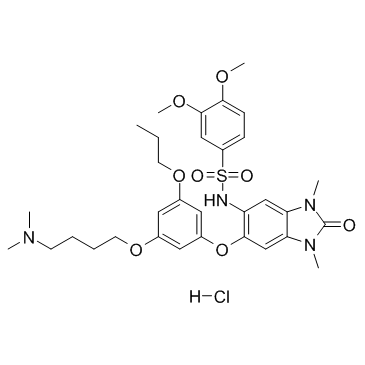
IACS-9571 Hydrochloride (ASIS-P040 Hydrochloride)
-
GC48548
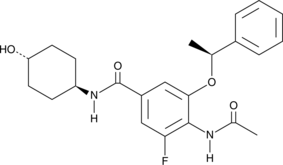
iBET-BD2
A BD2 bromodomain inhibitor
-
GC19200
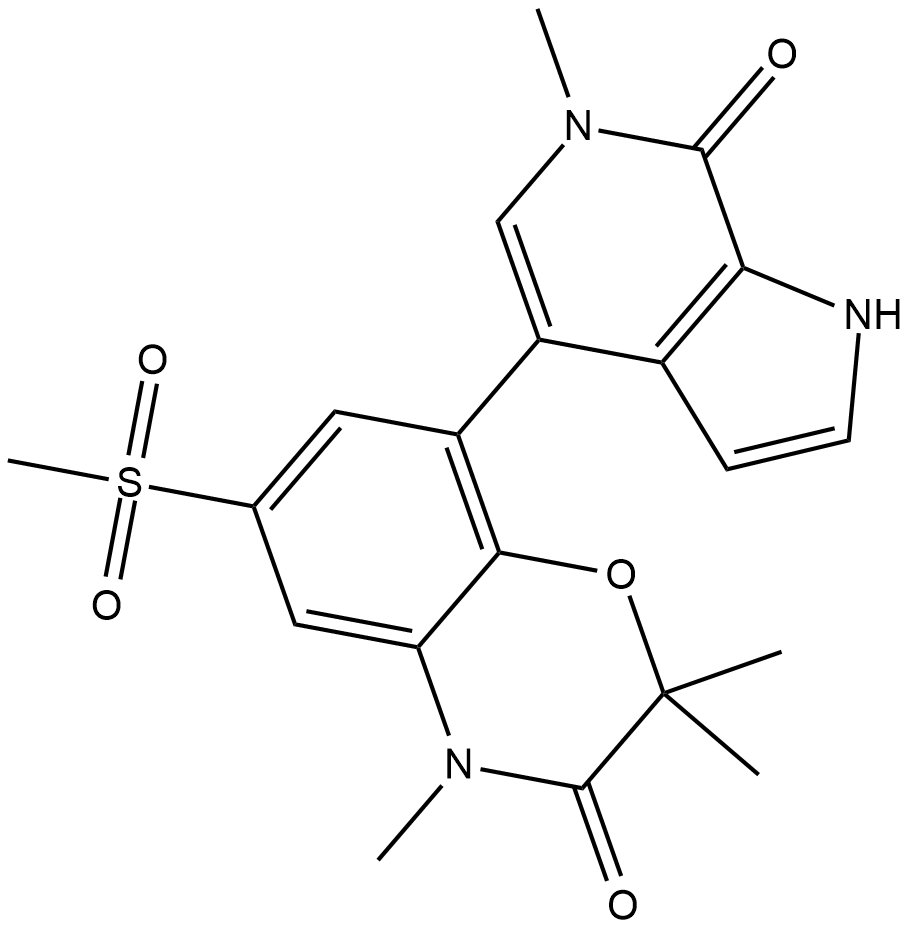
INCB-057643
INCB-057643 es un nuevo inhibidor BET biodisponible por vÍa oral.
-
GC33026
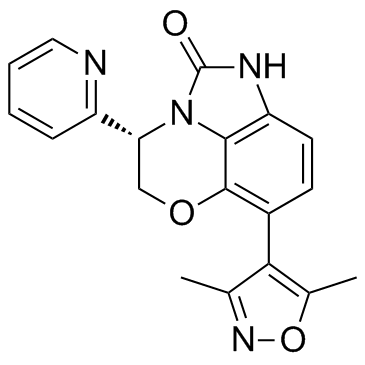
INCB054329
INCB054329 es un potente inhibidor de BET.
-
GC30644
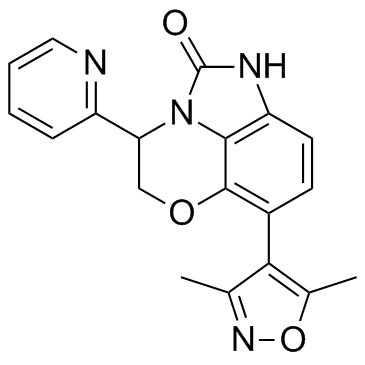
INCB054329 Racemate
INCB054329 El racemato es un inhibidor de la proteÍna BET.
-
GC19210
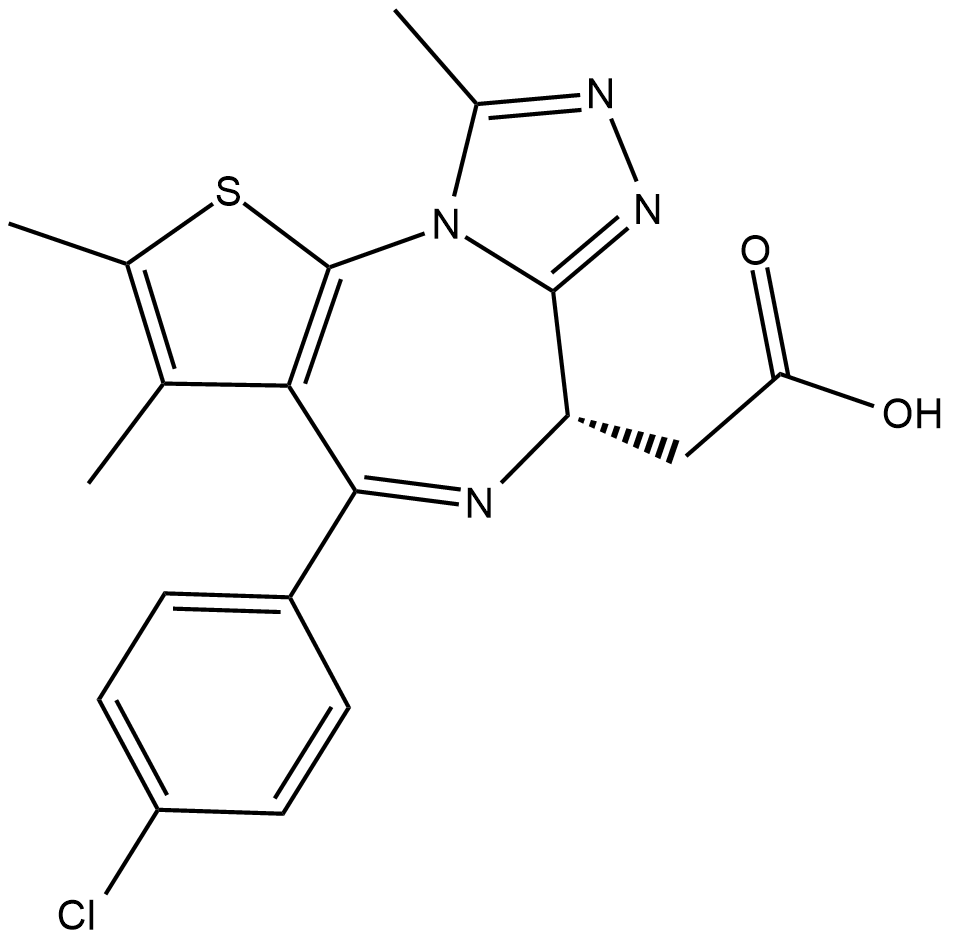
JQ-1 carboxylic acid
El ácido carboxílico JQ-1, un tipo de derivado de (+)-JQ-1, es un potente inhibidor del bromodominio BET para regular a la baja la expresión de PD-L1 en la superficie de las células tumorales. El ácido carboxílico JQ-1 se puede utilizar como precursor para sintetizar PROTAC, que se dirige a los bromodominios BET.
-
GC31654
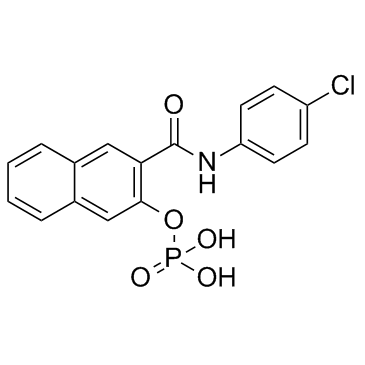
KG-501 (Naphthol AS-E phosphate)
KG-501 (fosfato de naftol AS-E) es un inhibidor de CREB, con un IC50 de 6,89 μM.
-
GC50395
L Moses dihydrochloride
High affinity and selective PCAF bromodomain inhibitor
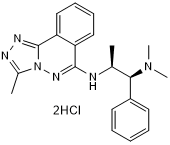
-
GC33183
L-45 (L-Moses)
L-45 (L-Moses) (L-45) es el primer inhibidor de bromodominio (Brd) del factor asociado a p300/CBP (PCAF) potente, selectivo y activo celular con una Kd de 126 nM.
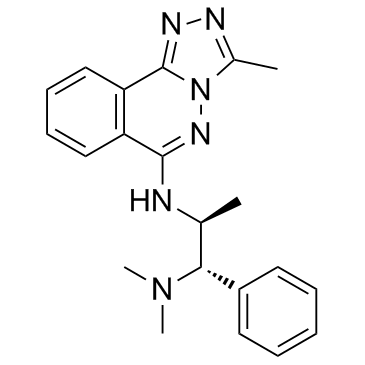
-
GC34377
L-45 dihydrochloride (L-Moses dihydrochloride)
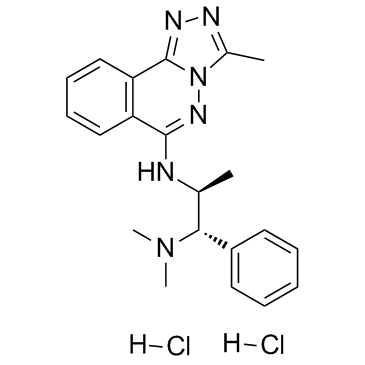
-
GC18731
LP99
LP99, una sonda epigenética, es un inhibidor potente y selectivo de los bromodominios BRD7 y BRD9 con una Kd de 99 nM contra BRD9.
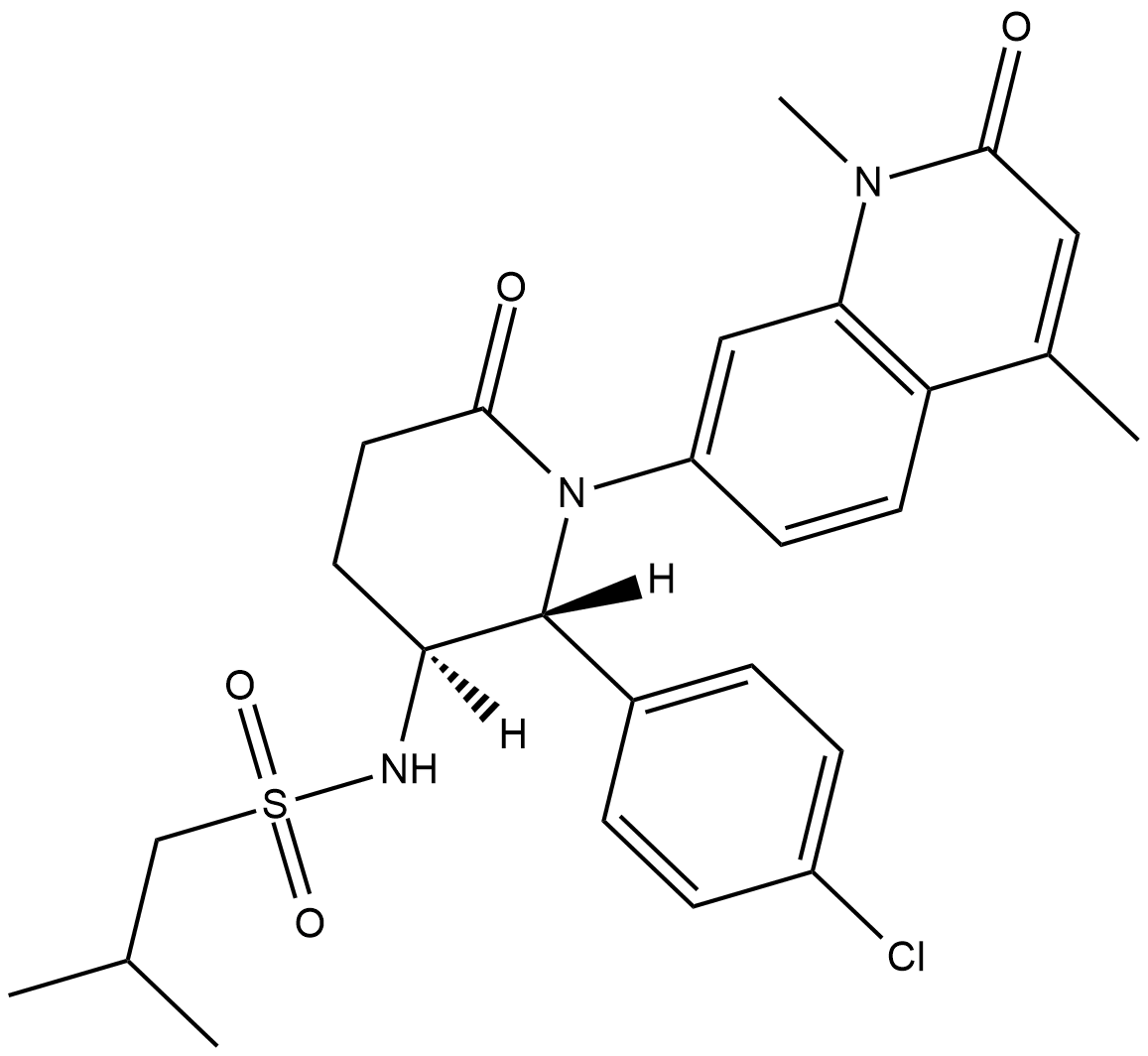
-
GC62591
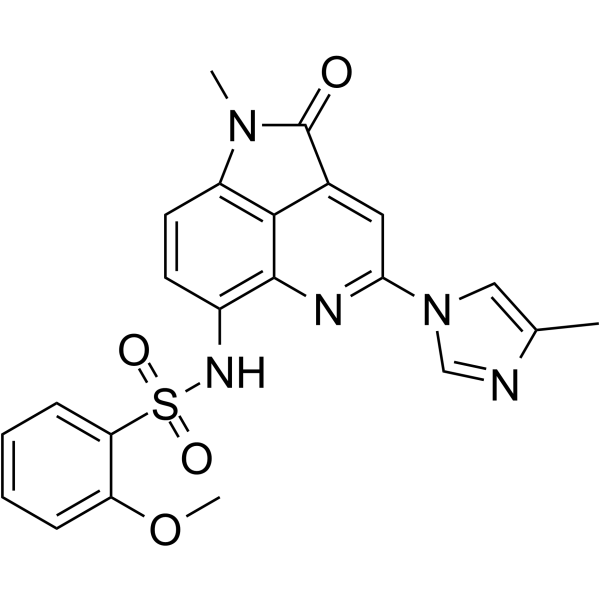
LT052
LT052 es un inhibidor BET BD1 altamente selectivo con un IC50 de 87,7 nM.
-
GC36521
M89
M-89 es un inhibidor de menina muy potente y específico, con una Kd de 1,4 nM para unirse a la menina.
-
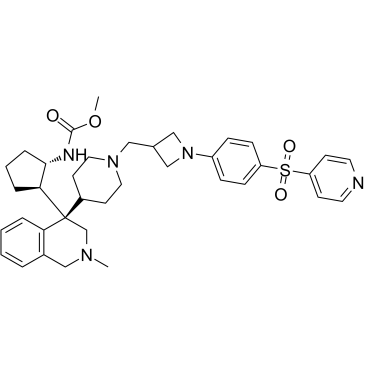
GC63064
Menin-MLL inhibitor 19
Menina-MLL inhibidor 19, un potente exo-aza espiroinhibidor de la interacciÓn menina-mll, ejemplo A17, extraÍdo de la patente WO2019120209A1.
-
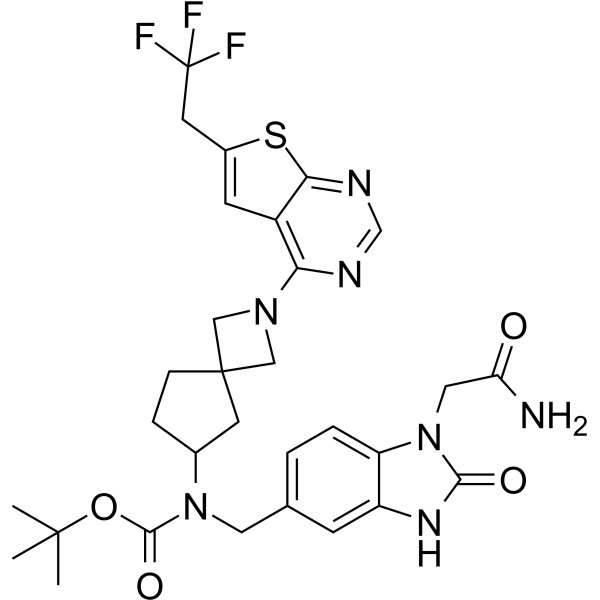
GC63538
Menin-MLL inhibitor 20
El inhibidor 20 de menina-MLL es un inhibidor irreversible de la interacciÓn menina-MLL con actividades antitumorales (documento WO2020142557A1, intermedio 6).

-
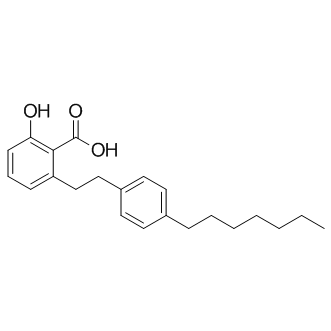
GC11439
MG 149
MG149 (inhibidor Tip60 HAT) es un inhibidor Tip60 selectivo y potente con IC50 de 74 uM, potencia similar para MOF (IC50= 47 uM); poco potente para PCAF y p300 (IC50 >200 uM).
-
GC11418
MI-2
An inhibitor of menin-MLL fusion protein interactions
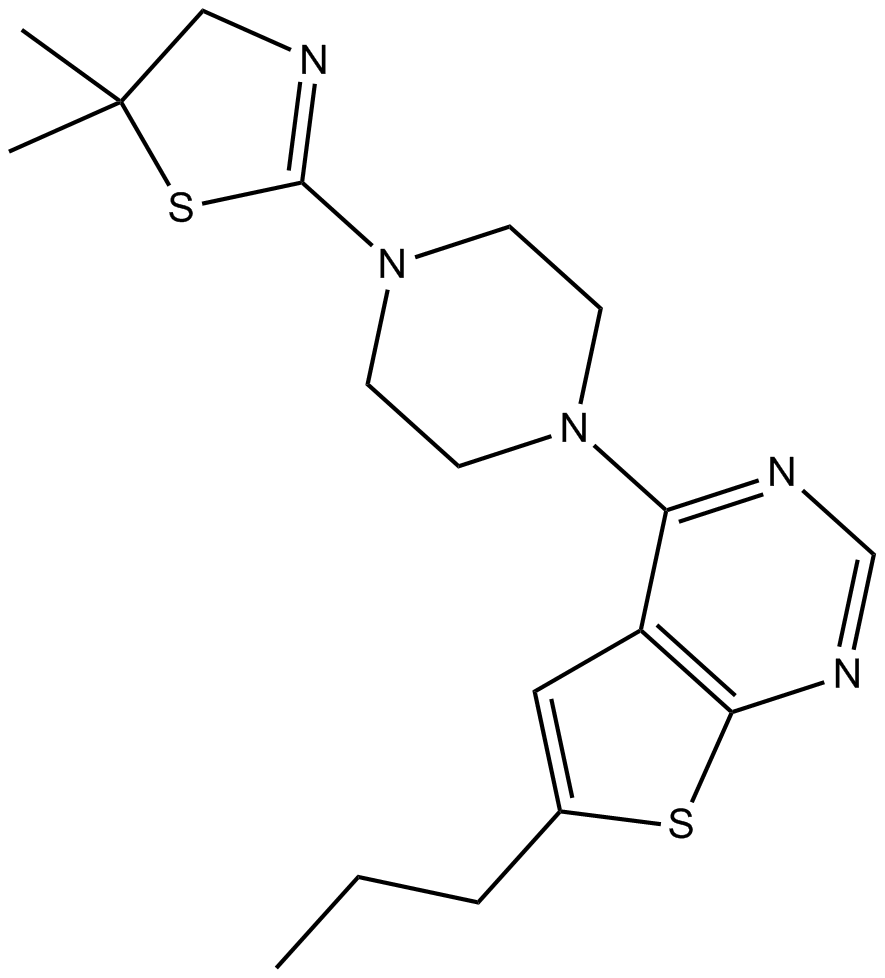
-
GC12199
MI-3
An inhibitor of menin-MLL fusion protein interactions
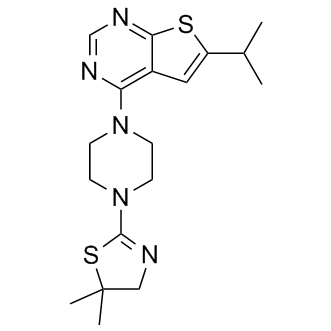
-
GC19246
MI-463
MI-463 es un inhibidor de molécula pequeÑa altamente potente y biodisponible por vÍa oral de la interacciÓn menina-mLL.
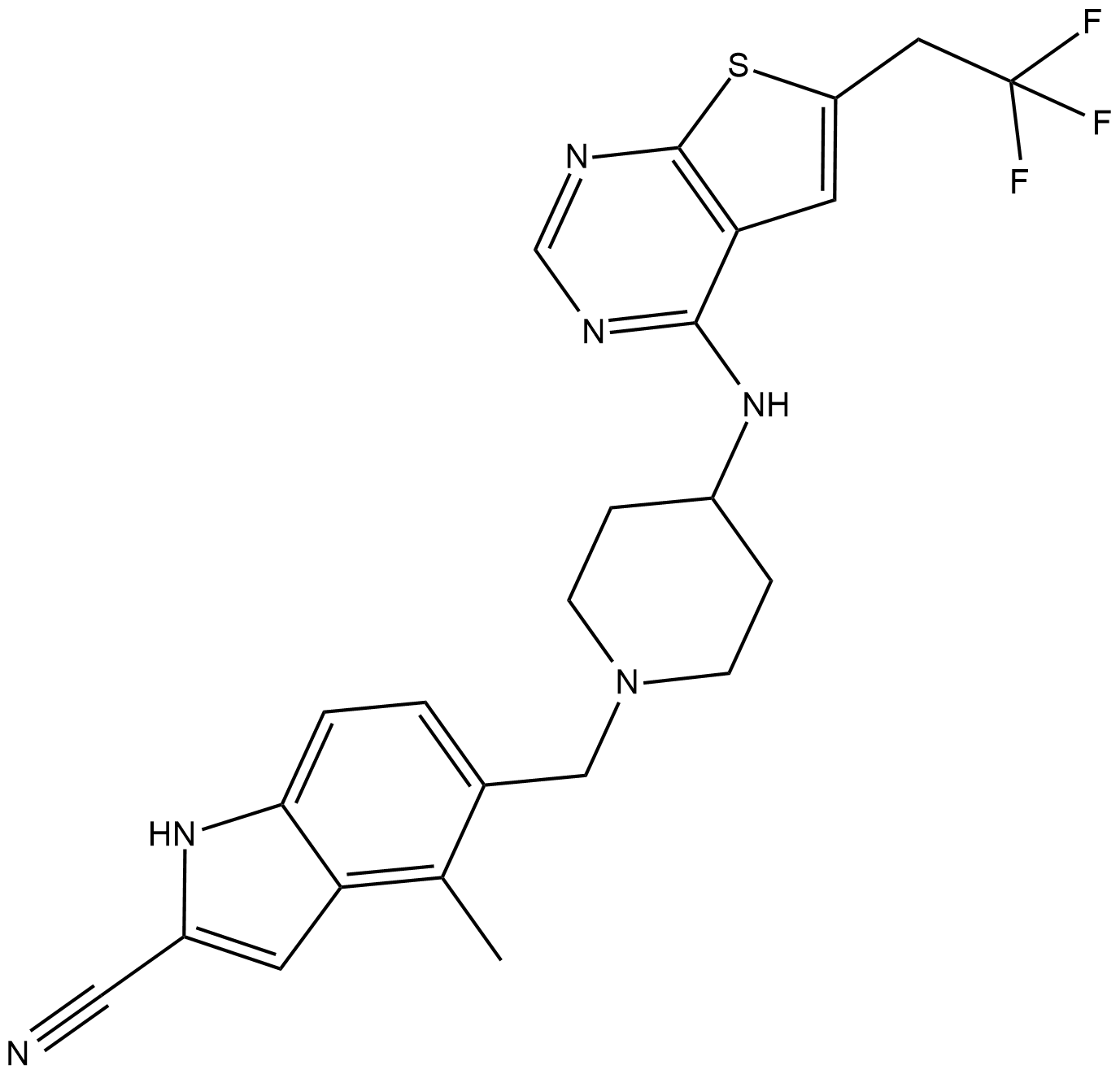
-
GC19247
MI-503
MI-503 es un inhibidor de molécula pequeÑa altamente potente y biodisponible por vÍa oral de la interacciÓn menina-mLL.
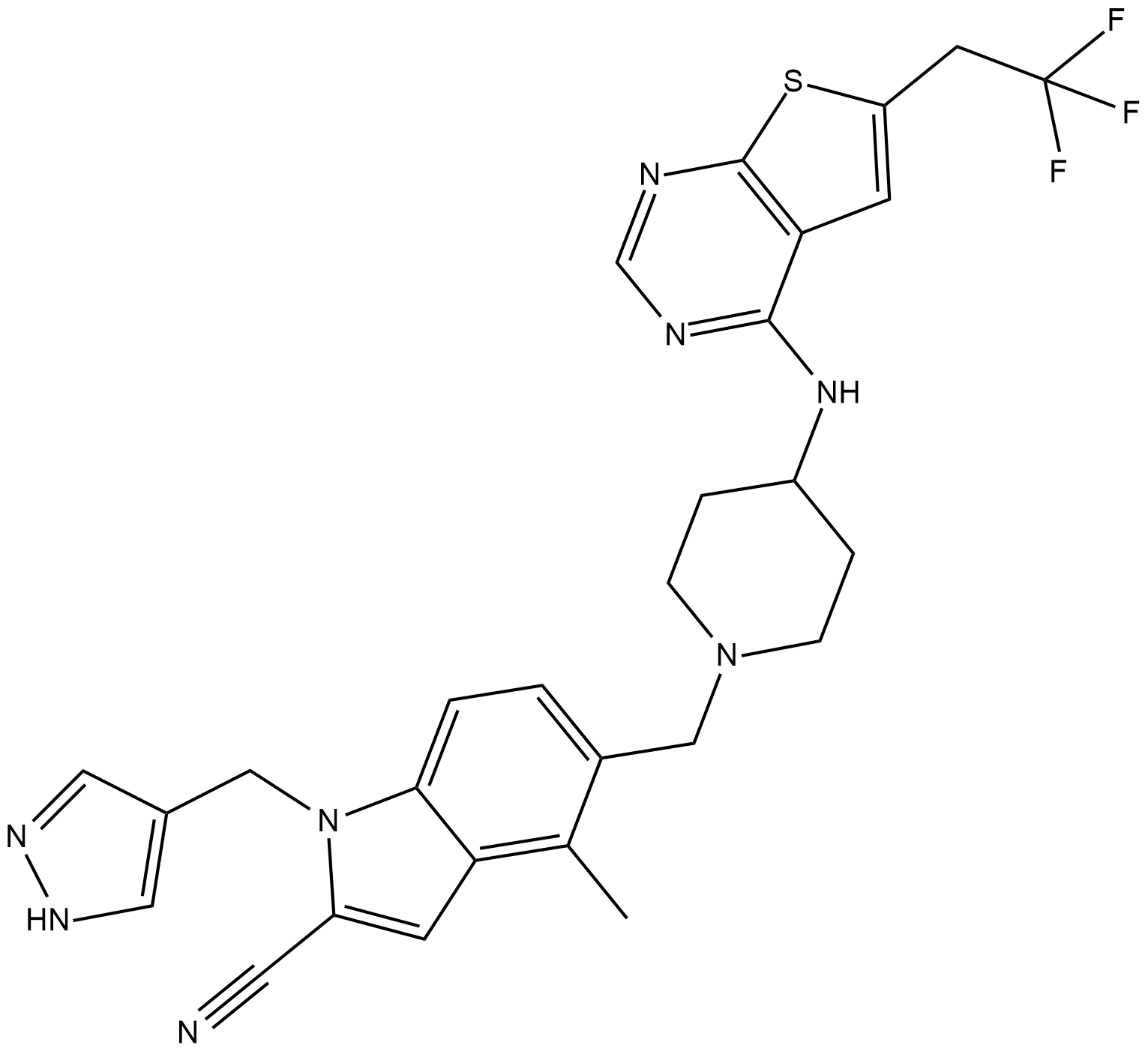
-
GC32798
MI-538
MI-538 es un inhibidor de la interacciÓn entre la menina y las proteÍnas de fusiÓn MLL con una IC50 de 21 nM.
-
GC19248
Mivebresib
Mivebresib (ABBV-075) es un inhibidor de bromodominio y de dominio extraterminal (BET) potente y activo por vÍa oral. Mivebresib se une a BRD4 con una Ki de 1,5 nM.
-
GC34675
Molibresib besylate
El besilato de molibresib (GSK 525762C; besilato de I-BET 762) es un inhibidor del bromodominio BET con IC50 de 32,5-42,5 nM.
-
GC36657
MS31
MS31 es un inhibidor de la proteÍna spindlin 1 (SPIN1) lector de metillisina similar a un fragmento potente, de alta afinidad y selectivo. MS31 inhibe potentemente las interacciones entre SPIN1 y H3K4me3 (IC50=77 nM, AlphaLISA; 243 nM, FP). MS31 se une selectivamente al dominio Tudor II de SPIN1 (Kd = 91 nM). MS31 inhibe potentemente la uniÓn de péptidos que contienen trimetillisina a SPIN1. MS31 no es tÓxico para las células no tumorigénicas.
-
GC39252
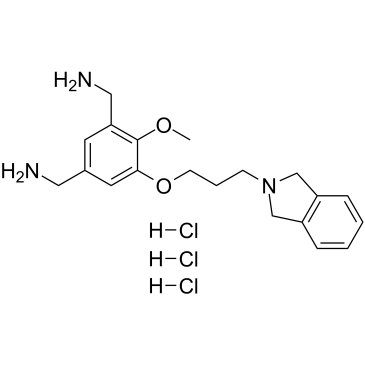
MS31 trihydrochloride
El triclorhidrato de MS31 es un inhibidor de la proteÍna spindlin 1 (SPIN1) lector de metillisina, potente, de alta afinidad y selectivo. El triclorhidrato de MS31 inhibe de forma potente las interacciones entre SPIN1 y H3K4me3 (IC50=77 nM, AlphaLISA; 243 nM, FP). El trihidrocloruro de MS31 se une selectivamente al dominio II de Tudor de SPIN1 (Kd = 91 nM). El triclorhidrato de MS31 inhibe potentemente la uniÓn de los péptidos que contienen trimetillisina a SPIN1 y no es tÓxico para las células no tumorigénicas.
-
GC44250
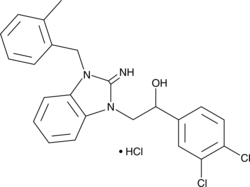
MS351
MS351 is an antagonist of chromobox 7 (CBX7) that acts by binding the CBX7 chromodomain.
-
GC64999
MS402
MS402 es un inhibidor BET BrD selectivo de BD1 con Kis de 77 nM, 718 nM, 110 nM, 200 nM, 83 nM y 240 nM para BRD4(BD1), BRD4(BD2), BRD3(BD1), BRD3(BD2) , BRD2(BD1) y BRD2(BD2), respectivamente.

-
GC31881
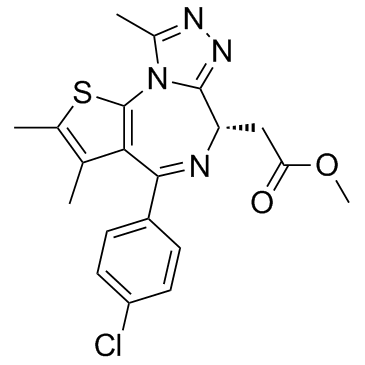
MS417 (GTPL7512)
MS417 (GTPL7512) es un inhibidor selectivo de BRD4 especÍfico de BET, se une a BRD4-BD1 y BRD4-BD2 con IC50 de 30, 46 nM y Kds de 36,1, 25,4 nM, respectivamente, con una selectividad débil en CBP BRD (IC50, 32,7 μ ;METRO).
-
GC13148
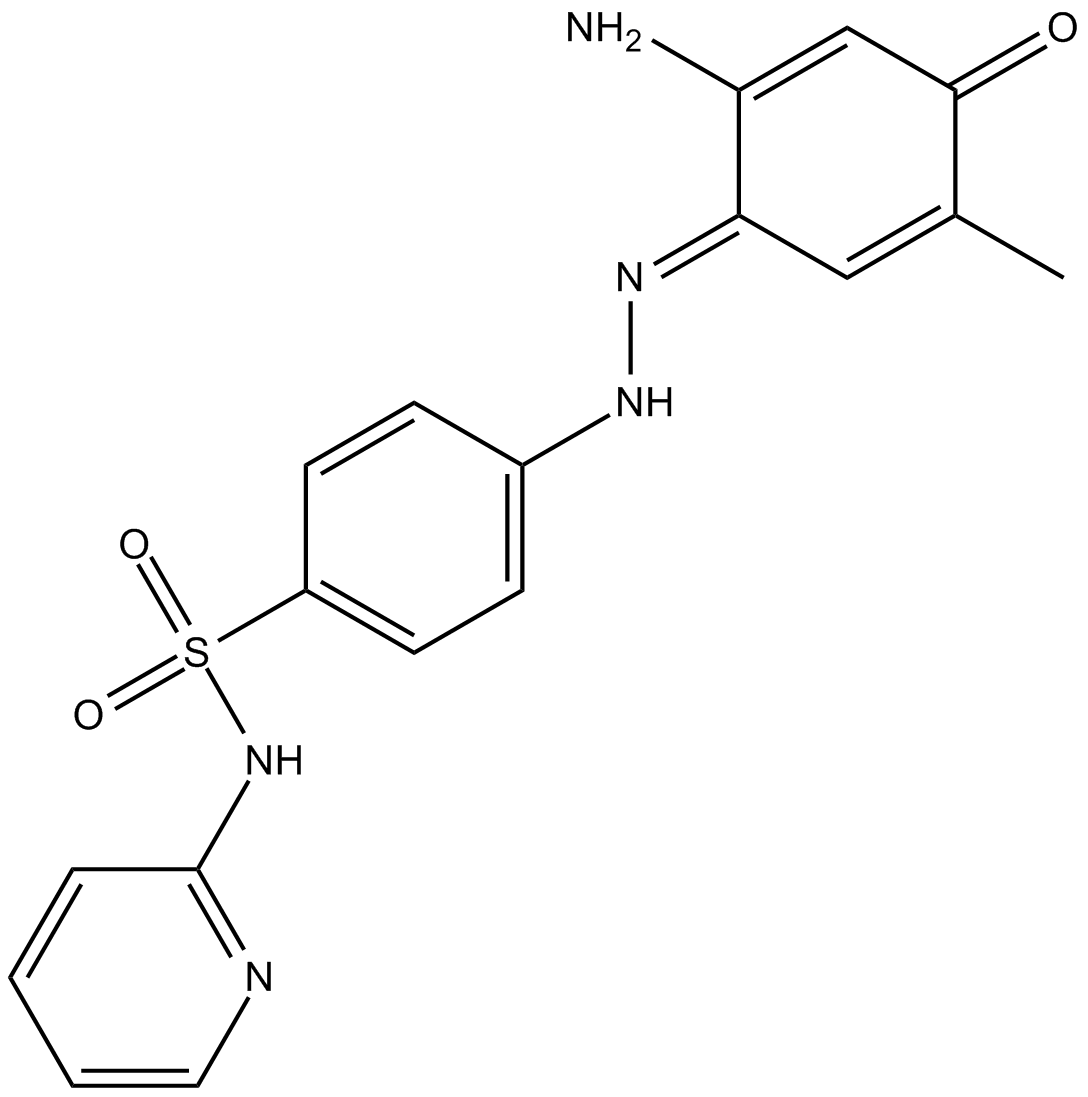
MS436
MS436 es una nueva clase de inhibidor de bromodominio, exhibe una potente afinidad de un Ki estimado = 30-50 nM para el BRD4 BrD1 y una selectividad de 10 veces sobre el BrD2.
-
GC38474
MS645
MS645 es un inhibidor bivalente de bromodominios BET (BrD) con una Ki de 18,4 nM para BRD4-BD1/BD2. MS645 restringe espacialmente la inhibiciÓn bivalente de BRD4 BrD, lo que da como resultado una represiÓn sostenida de la actividad transcripcional de BRD4 en células tumorales sÓlidas.
-
GC18729
MZ1
MZ1 es un PROTAC conectado por ligandos para von Hippel-Lindau y BRD4. MZ1 induce potente y rápidamente la eliminación reversible, duradera y selectiva de BRD4 sobre BRD2 y BRD3. Kds de 382/120, 119/115 y 307/228 nM para BRD4 BD1/2, BRD3 BD1/2 y BRD2 BD1/2, respectivamente.
-
GC33102
MZP-54
MZP-54 es un PROTAC conectado por ligandos para von Hippel-Lindau y BRD3/4, con una Kd de 4 nM para Brd4BD2.
-
GC33363
MZP-55
MZP-55 es un PROTAC conectado por ligandos para von Hippel-Lindau y BRD3/4, con una Kd de 8 nM para Brd4BD2.
-
GC61841
Naphthol AS-E
Naphthol AS-E es un inhibidor potente y permeable a las células de la interacciÓn KIX-KID. Naphthol AS-E se une directamente al dominio KIX de CBP (Kd: 8,6 μM), bloquea la interacciÓn entre el dominio KIX y el dominio KID de CREB con IC50 de 2,26 μM. Naphthol AS-E se puede utilizar para la investigaciÓn del cÁncer.
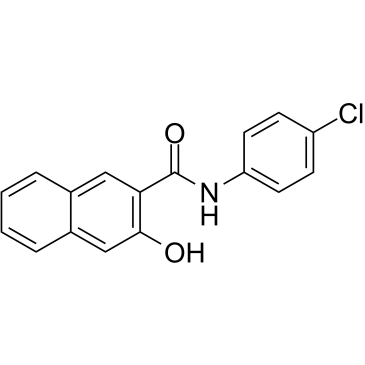
-
GC62620
NHWD-870
NHWD-870 es un inhibidor de bromodominio de la familia BET potente, activo por vÍa oral y selectivo y solo se une a bromodominios de BRD2, BRD3, BRD4 (IC50 = 2,7 nM) y BRDT. NHWD-870 tiene potentes eficacias supresoras de tumores y suprime la interacciÓn entre células cancerosas y macrÓfagos. NHWD-870 aumenta la apoptosis tumoral e inhibe la proliferaciÓn tumoral.
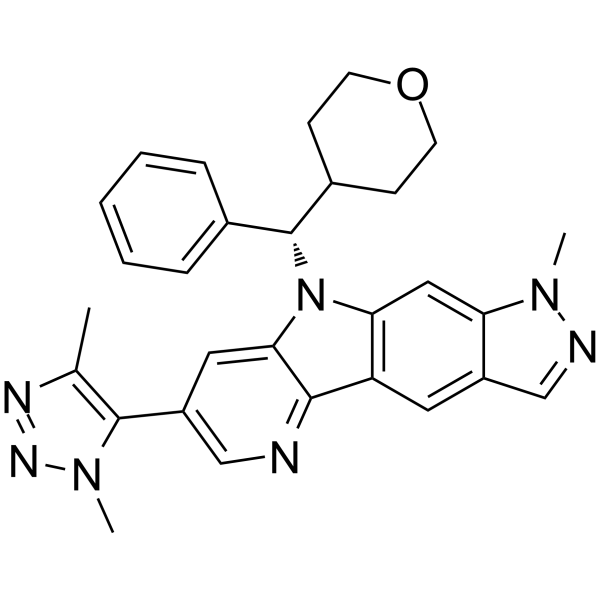
-
GC36734
NI-42
NI-42 (compuesto 13-d), una sonda quÍmica estructuralmente ortogonal para los BRPF, es un potente inhibidor sesgado del BRD de los BRPF (IC50 de BRPF1/2/3=7,9/48/260 nM; Kds de BRPF1 /2/3=40/210/940 nM) con excelente selectividad sobre proteÍnas BRD que no son de clase IV.
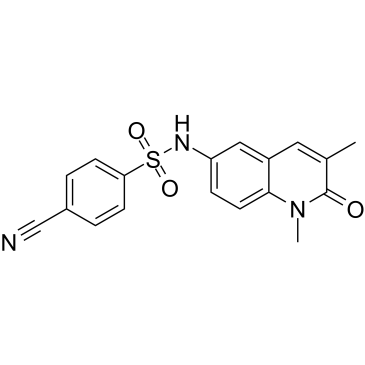
-
GC16763
NI-57
NI-57 es un inhibidor de la familia de proteÍnas bromodomain y plant homeodomain finger-containing (BRPF), con IC50 de 3,1, 46 y 140 nM para BRPF1, BRPF2 (BRD1) y BRPF3, respectivamente.
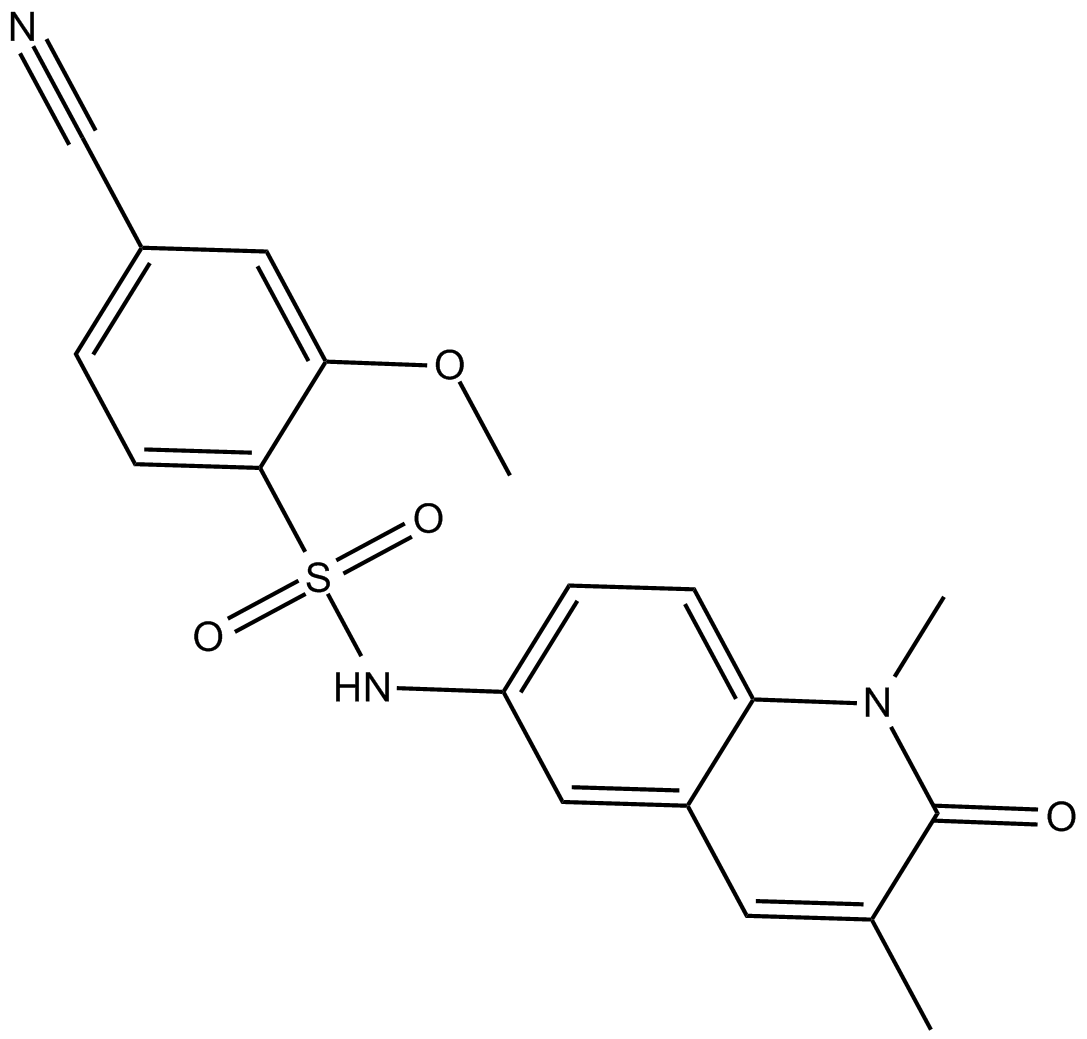
-
GC14103
NSC228155
NSC228155 es un activador de EGFR, se une a la región extracelular de EGFR y mejora la fosforilación de tirosina de EGFR. NSC228155 también es un potente inhibidor de la interacción KIX-KID, inhibe el dominio inducible por quinasa (KID) de CREB y el dominio que interactúa con KID (KIX) de CBP, con una IC50 de 0,36 μM.
-
GC44477
NVS-CECR2-1
NVS-CECR2-1, un inhibidor de bromodominio (BRD) de la familia no BET, es un inhibidor potente y selectivo de la regiÓn cromosÓmica del sÍndrome del ojo de gato, candidato 2 (CECR2). NVS-CECR2-1 se une a CECR2 BRD con alta afinidad (IC50=47 nM; KD=80 nM). NVS-CECR2-1 exhibe actividad citotÓxica e induce la apoptosis contra varias células cancerosas al dirigirse a CECR2, asÍ como a través de un mecanismo independiente de CECR2.
-
GC69601
OARV-771
OARV-771 es un tipo de agente degradador BET (PROTAC) basado en VHL, con una mejorada permeabilidad celular. OARV-771 muestra DC50 para Brd4, Brd2 y Brd3 de 6, 1 y 4 nM respectivamente.
-
GC66067
ODM-207
ODM-207 (BET-IN-4) es un potente inhibidor de la proteÍna de bromodominio BET (BRD4), con un IC50 de ≤ 1 μM.
-
GC17482
OF-1
OF-1 es un potente inhibidor de bromodominio pan-BRPF (BRD), con valores IC50 de 270 nM, 1,2 μM para TRIM24 y BRPF1B, respectivamente.
-
GC17973
OTX-015
A BRD2, BRD3, and BRD4 inhibitor
-
GC39417
OXFBD04
OXFBD04 es un inhibidor de BRD4 potente y selectivo con una IC50 de 166 nM. OXFBD04 es un potente ligando de bromodominio BET con una modesta afinidad adicional por el bromodominio CREBBP. OXFBD04 tiene actividad anticancerÍgena.
-
GC36887
PF-CBP1 hydrochloride
El clorhidrato de PF-CBP1 es un inhibidor altamente selectivo del bromodominio de la proteÍna de uniÓn a CREB (CBP BRD).
-
GC14361
PFI 3
PFI 3 es un inhibidor de bromodominio SMARCA2/4 selectivo, potente y permeable a las células con una Kd de 89 nM.
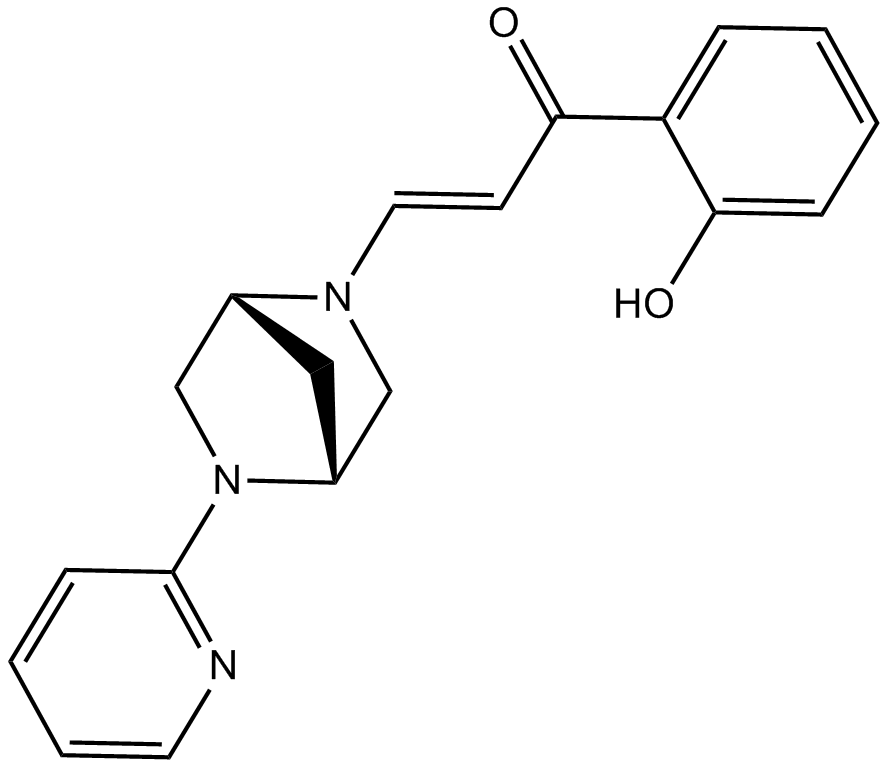
-
GC10979
PFI 4
PFI 4 (compuesto 11) es un inhibidor potente y altamente selectivo del bromodominio BRPF1 (BRPF1B), con una IC50 de 172 nM. PFI 4 se puede utilizar para explorar los mecanismos funcionales del complejo HBO1/BRPF1 y para estudiar la pérdida ósea y las lesiones óseas malignas osteolíticas.
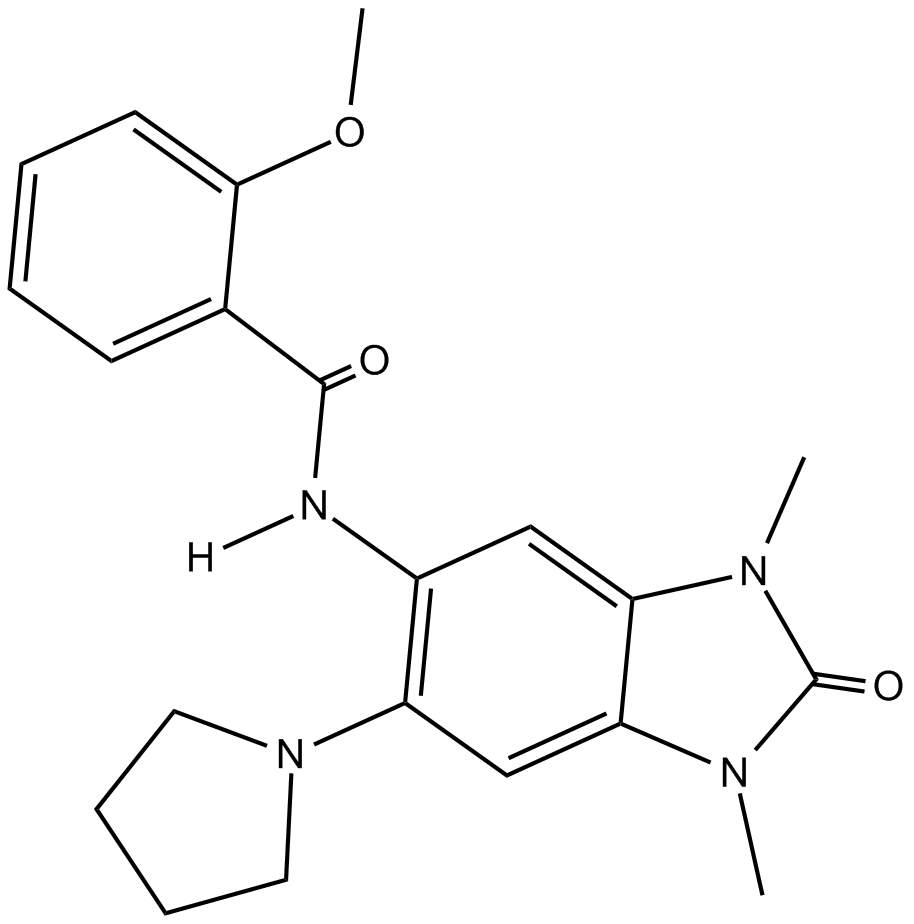
-
GC15375
PFI-1 (PF-6405761)
A BET bromodomain inhibitor
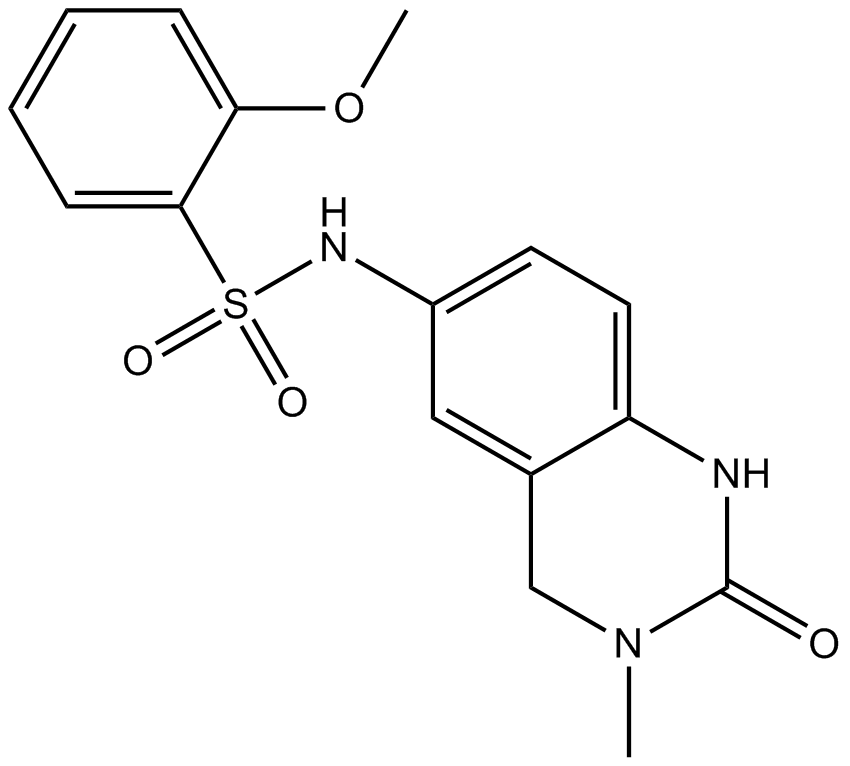
-
GC19298
PLX51107
PLX51107 es un inhibidor BET potente y selectivo, con Kds de 1,6, 2,1, 1,7 y 5 nM para BD1 y 5,9, 6,2, 6,1 y 120 nM para BD2 de BRD2, BRD3, BRD4 y BRDT, respectivamente; PLX51107 también interactÚa con los bromodominios de CBP y EP300 (Kd, en el rango de 100 nM).
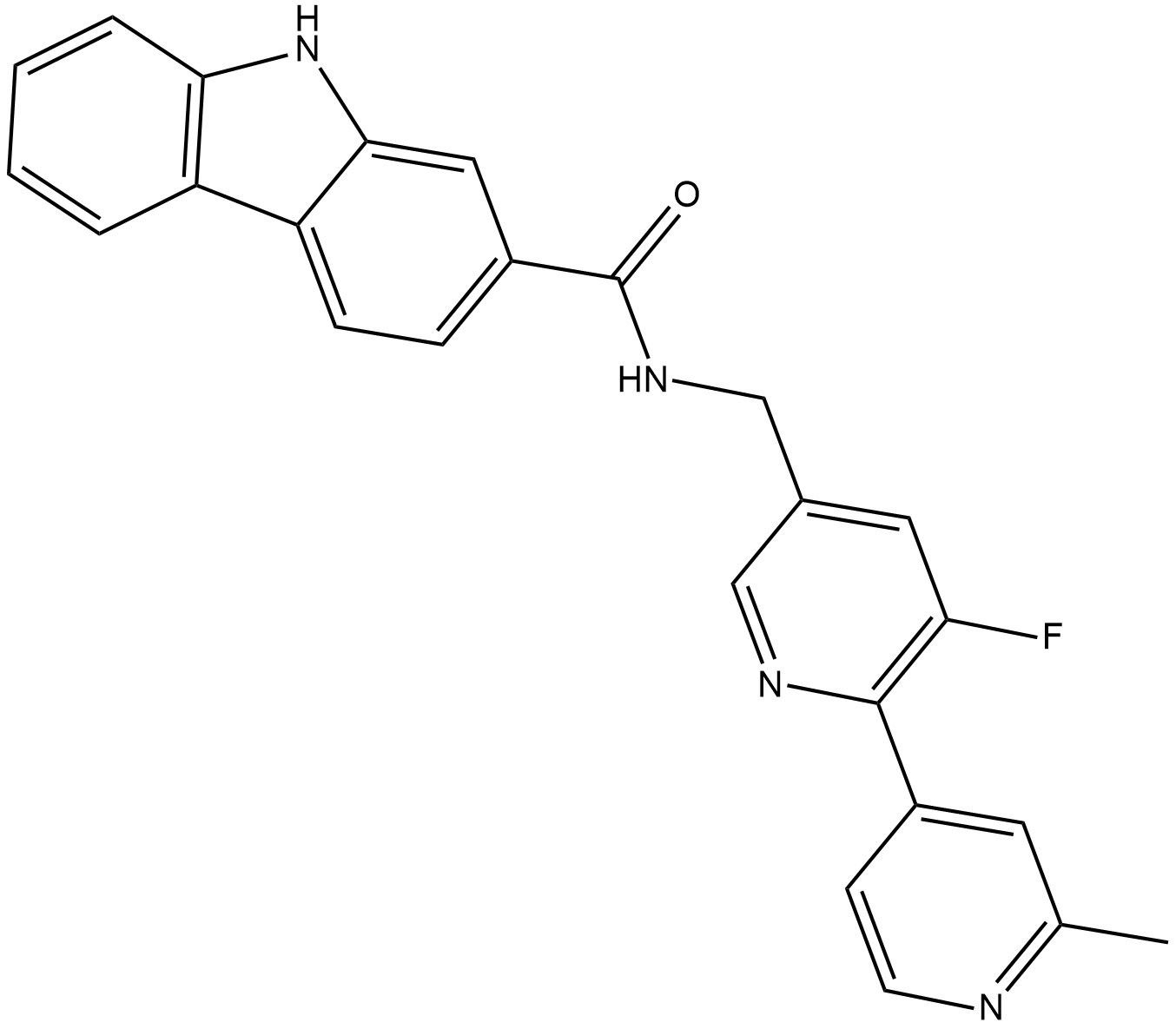
-
GC36943
PNZ5
PNZ5 es un inhibidor pan-BET potente y basado en isoxazol con alta selectividad y potencia similar al bien establecido (+)-JQ1, con una KD de 5,43 nM para BRD4(1).

-
GC33109
PROTAC BET Degrader-1
PROTAC BET Degrader-1 es un PROTAC conectado por ligandos para Cereblon y BET, que disminuye los niveles de proteÍna BRD2, BRD3 y BRD4 a baja concentraciÓn.
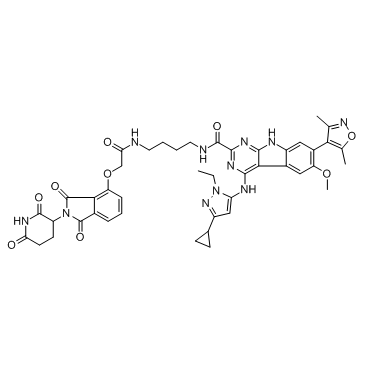
-
GC32980
PROTAC BET degrader-2
PROTAC BET degradador-2 es un PROTAC conectado por ligandos para Cereblon y BET con un valor IC50 de 9,6 nM en la inhibiciÓn del crecimiento celular en las células RS4;11 y capaz de lograr la regresiÓn tumoral.
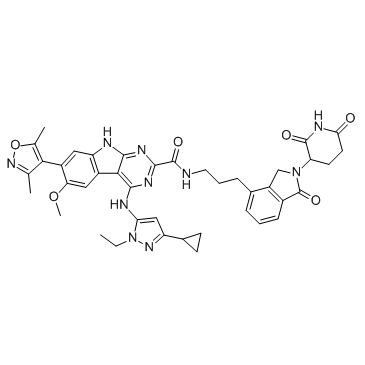
-
GC34325
PROTAC BET degrader-3
PROTAC BET Degrader-3 es un PROTAC conectado por ligandos para von Hippel-Lindau y BET.
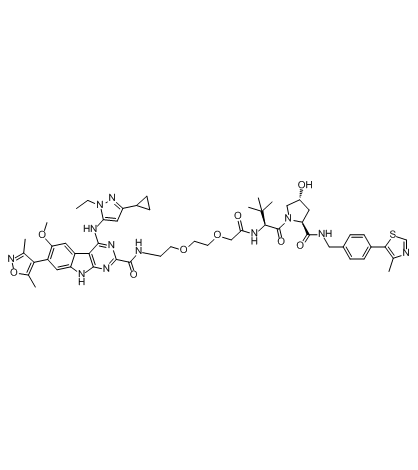
-
GC61212
PROTAC BRD4 Degrader-5
PROTAC BRD4 Degrader-5 es un PROTAC conectado por ligandos para von Hippel-Lindau y BRD4. PROTAC BRD4 Degrader-5 puede degradar potentemente BRD4 en lÍneas celulares de cÁncer de mama positivas y negativas para HER2.
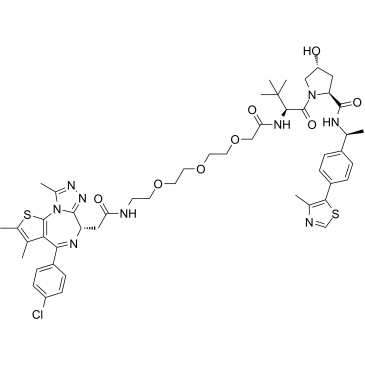
-
GC62606
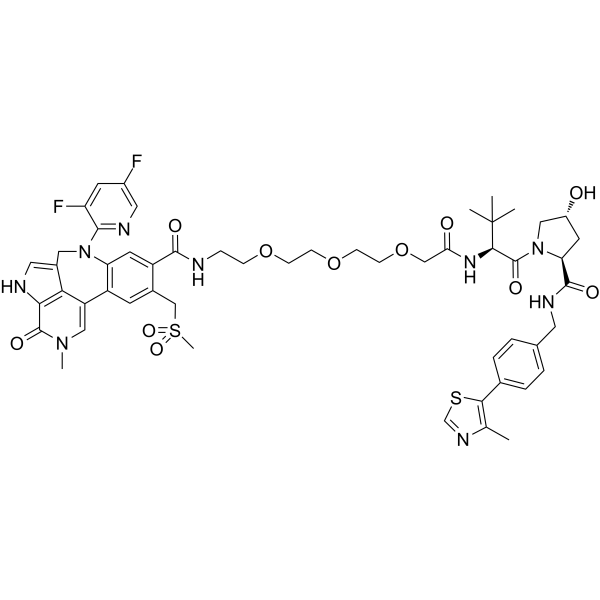
PROTAC BRD4 Degrader-8
PROTAC BRD4 Degrader-8 es un PROTAC conectado por ligandos para von Hippel-Lindau y BRD4, con IC50 de 1,1 nM y 1,4 nM para BRD4 BD1 y BD2, respectivamente. PROTAC BRD4 Degrader-8 es capaz de degradar potentemente la proteÍna BRD4 en las células de cÁncer de prÓstata PC3.
-
GC39180
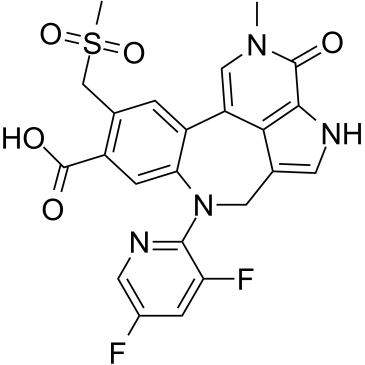
PROTAC BRD4 ligand-1
PROTAC BRD4 ligand-1 es un potente inhibidor de BET y un ligando para la proteÍna BRD4 diana para PROTACT GNE-987.
-
GC33372
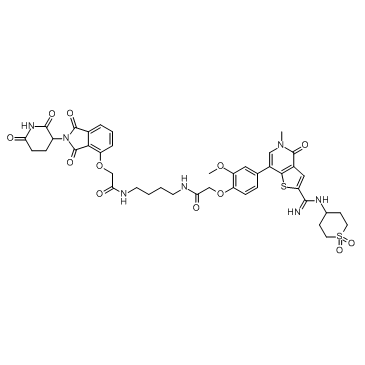
PROTAC BRD9 Degrader-1
PROTAC BRD9 Degrader-1 es un PROTAC conectado por ligandos para Cereblon y BRD9 (IC50=13,5 nM), que se puede utilizar como sonda selectiva Útil para el estudio de la biologÍa del complejo BAF.
-
GC33217
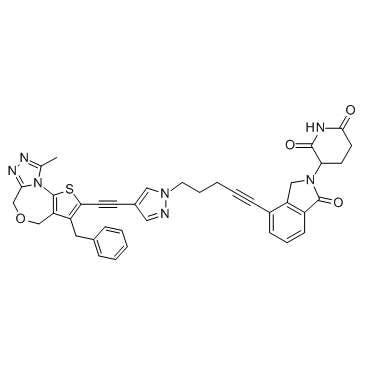
QCA570
QCA570 es un PROTAC conectado por ligandos para Cereblon y BET, con una IC50 de 10 nM para BRD4 BD1 Protein.
-
GC14199
RVX-208
RVX-208 (RVX-208) es un inhibidor de los reguladores transcripcionales BET con selectividad por el segundo bromodominio. Los IC50 son 87 μM y 0,51 μM para BD1 y BD2, respectivamente.

-
GC63178
RVX-297
RVX-297 es un potente inhibidor del bromodominio BET activo por vÍa oral con selectividad por BD2.
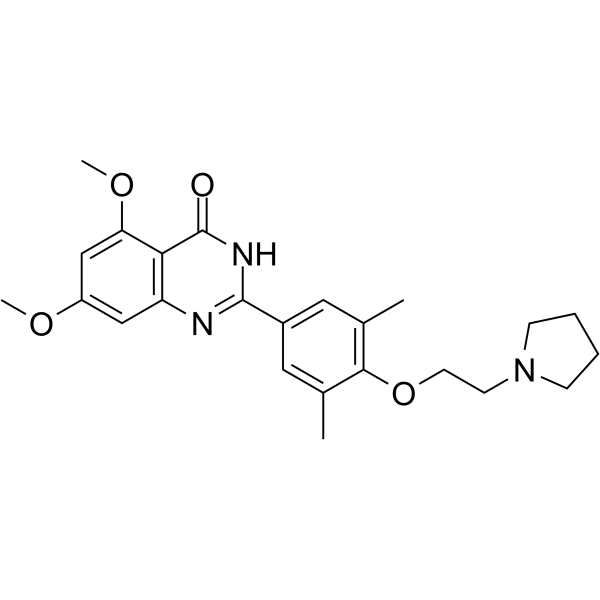
-
GC19328
SF2523
SF2523 es un inhibidor altamente selectivo y potente de PI3K con IC50 de 34 nM, 158 nM, 9 nM, 241 nM y 280 nM para PI3Kα, PI3Kγ, DNA-PK, BRD4 y mTOR, respectivamente.
-
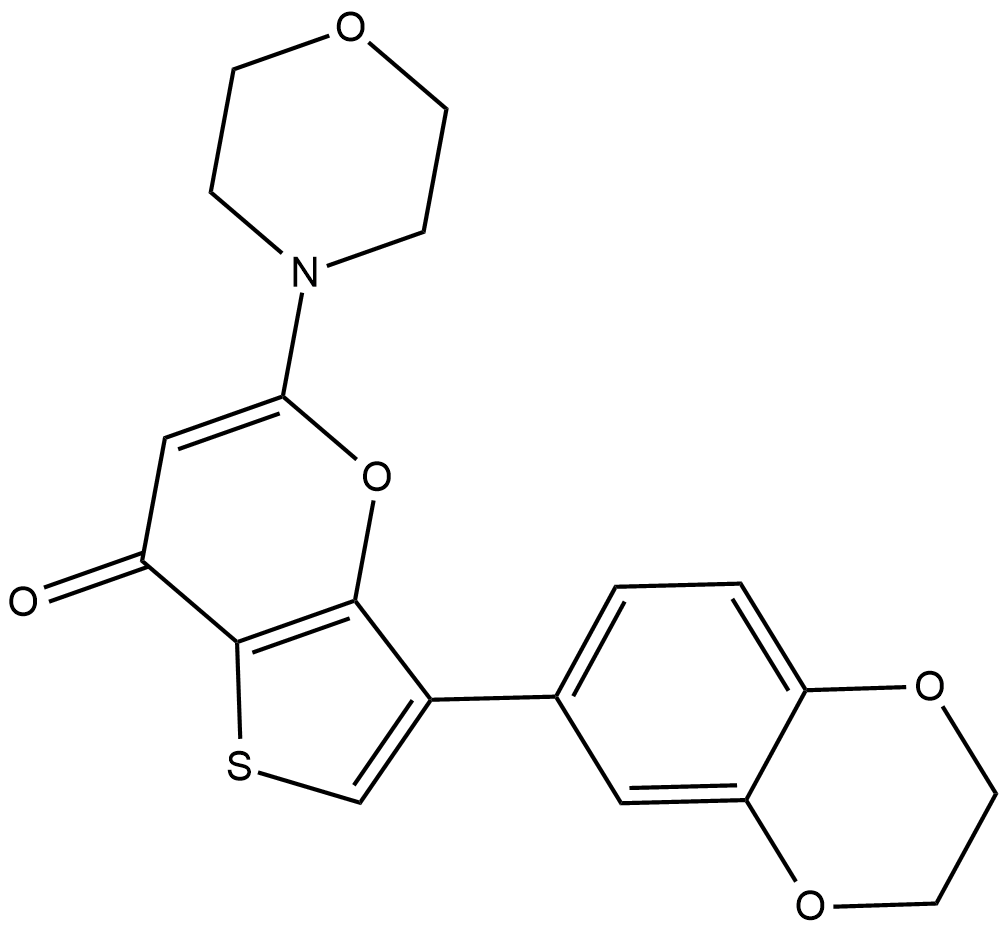
GC10174
SGC-CBP30
A potent inhibitor of CREBBP/EP300 bromodomains
-
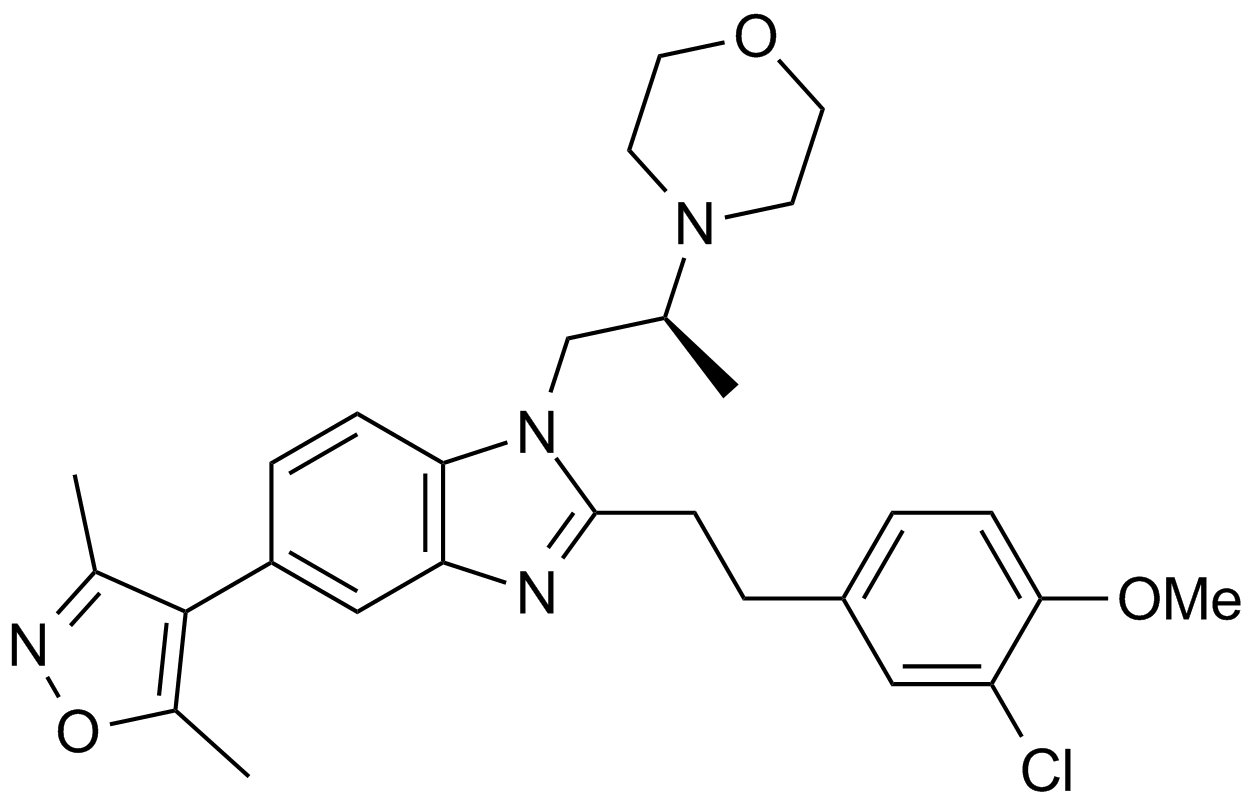
GC34778
SGC-iMLLT
SGC-iMLLT es la primera sonda quÍmica de su clase y un potente inhibidor selectivo de las interacciones MLLT1/3-histonas con una IC50 de 0,26 μM. SGC-iMLLT muestra una alta actividad de uniÓn hacia el dominio MLLT1 YEATS (YD) y MLLT3 YD (AF9/YEATS3) con Kds de 0,129 y 0,077 μM, respectivamente.
-
GC63887
SGC-SMARCA-BRDVIII
SGC-SMARCA-BRDVIII es un inhibidor potente y selectivo de SMARCA2/4 y PB1(5), con Kds de 35 nM, 36 nM y 13 nM, respectivamente.
-
GC65574
SNIPER(BRD)-1
SNIPER(BRD)-1, consta de un antagonista de IAP LCL-161 derivado y un inhibidor de BET, (+)-JQ-1, conectados por un conector. SNIPER(BRD)-1 induce la degradaciÓn de BRD4 a través de la vÍa ubiquitina-proteasoma. SNIPER(BRD)-1 también degrada cIAP1, cIAP2 y XIAP con IC50 de 6,8 nM, 17 nM y 49 nM, respectivamente.
-
GC32863
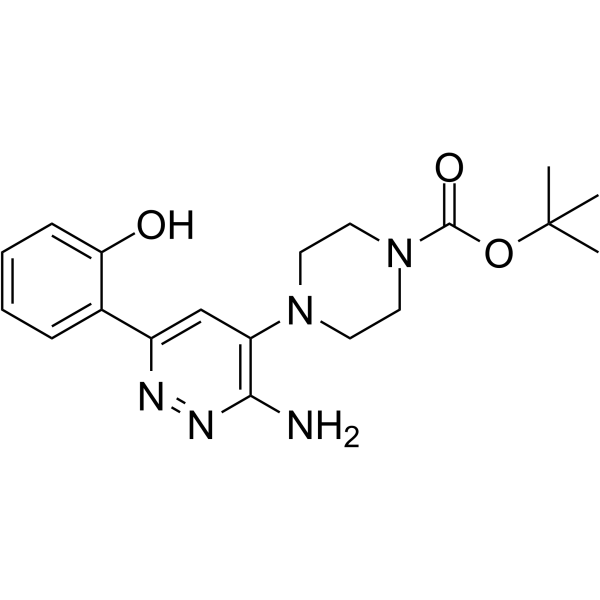
Target Protein-binding moiety 4
El resto de uniÓn a proteÍnas diana 4 (Ácido carboxÍlico de molibresib) es un ligando de ojiva basado en I-BET762 para reacciones de conjugaciÓn de PROTAC dirigido a BET. El resto de uniÓn a proteÍnas diana 4 (Ácido carboxÍlico de molibresib) es un inhibidor de BRD4 con una pIC50 de 5,1.
-
GC50340
TC AC 28
High affinity BET bromodomain ligand
-
GC34319
TD-428
TD-428 es un PROTAC conectado por ligandos para Cereblon y BRD4. TD-428 es un degradador BRD4 altamente especÍfico con un DC50 de 0,32 nM. TD-428 es un BET PROTAC, que comprende TD-106 (un ligando de CRBN) unido a JQ1 (un inhibidor de BET). TD-428 induce eficazmente la degradaciÓn de la proteÍna BET.
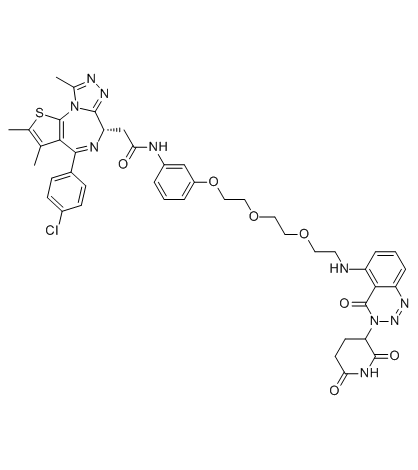
-
GC70037
TMX1
TMX1 es un inhibidor de la degradación covalente de moléculas BRD4. TMX1 recluta selectivamente DCAF16 a BRD4BD2, lo que resulta en la degradación de BRD4.
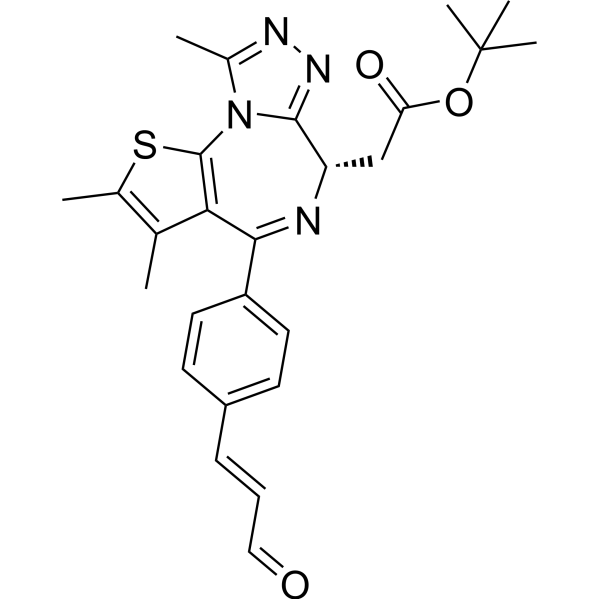
-
GC50542
TP 238
CECR2 and BPTF/FALZ inhibitor
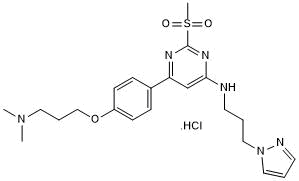
-
GC40628
TP-472
TP-472 es un inhibidor selectivo de BRD7/9, con KD de 0,34 μM y 33 nM para BRD7 y BRD9, respectivamente.
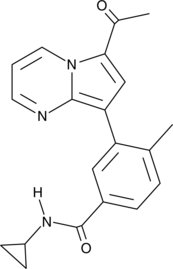
-
GC32880
TPOP146
TPOP146 es un inhibidor selectivo del bromodominio de benzoxazepina CBP/P300 con valores de Kd de 134 nM y 5,02 μM para CBP y BRD4.
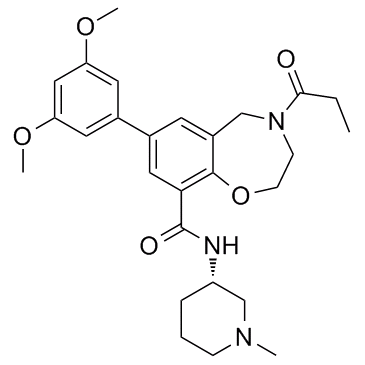
-
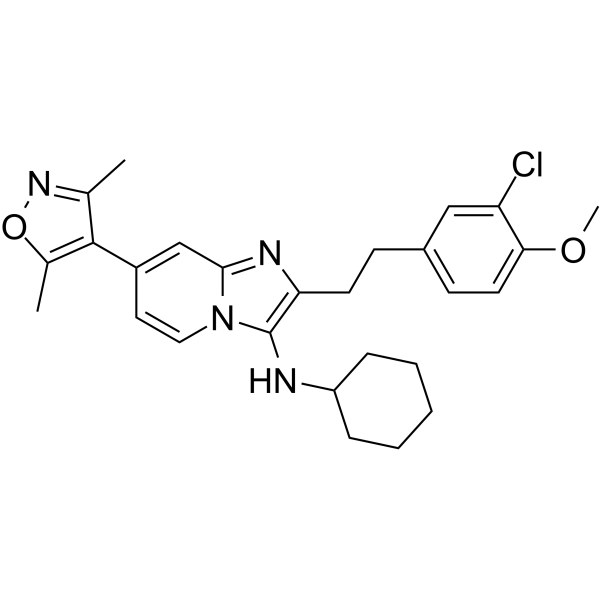
GC62405
UMB298
UMB298 es un inhibidor de bromodominio CBP/P300 potente y selectivo.
-
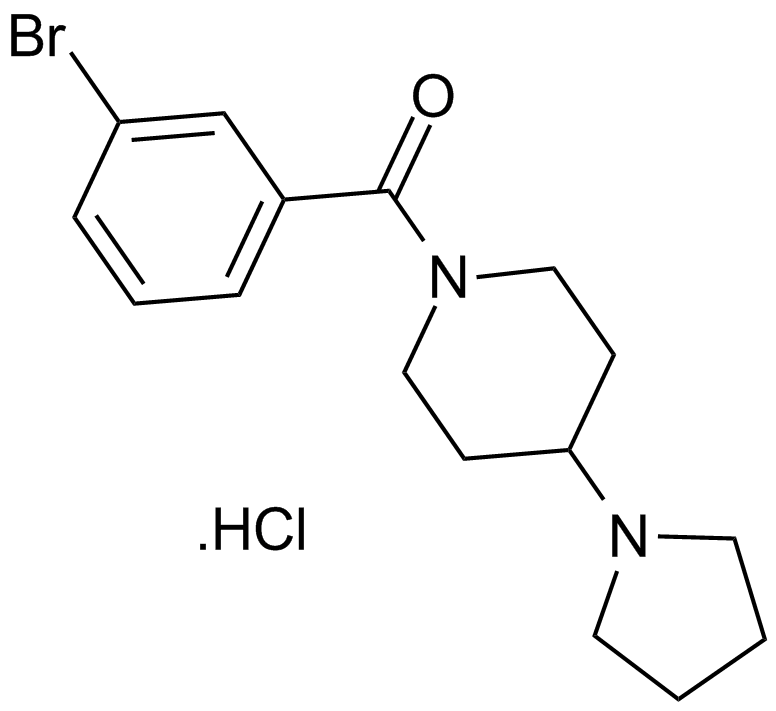
GC12419
UNC 926 hydrochloride
An antagonist of L3MBTL1, L3MBTL3, and L3MBTL4
-
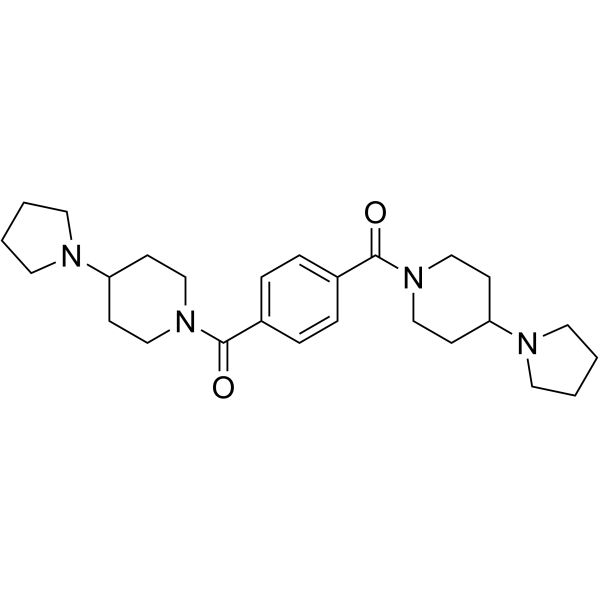
GC68025
UNC1021
-
GC64040
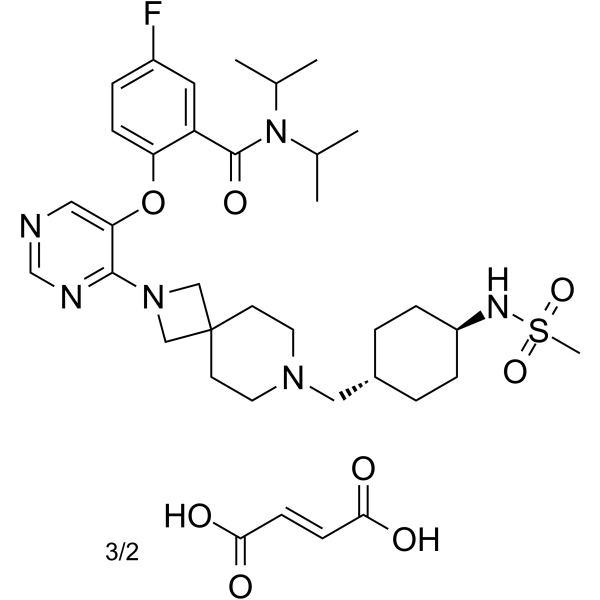
VTP50469 fumarate
El fumarato VTP50469 es un inhibidor de la interacciÓn Menina-MLL potente, altamente selectivo y activo por vÍa oral con una Ki de 104 pM. El fumarato VTP50469 tiene una actividad potente contra la leucemia.
-
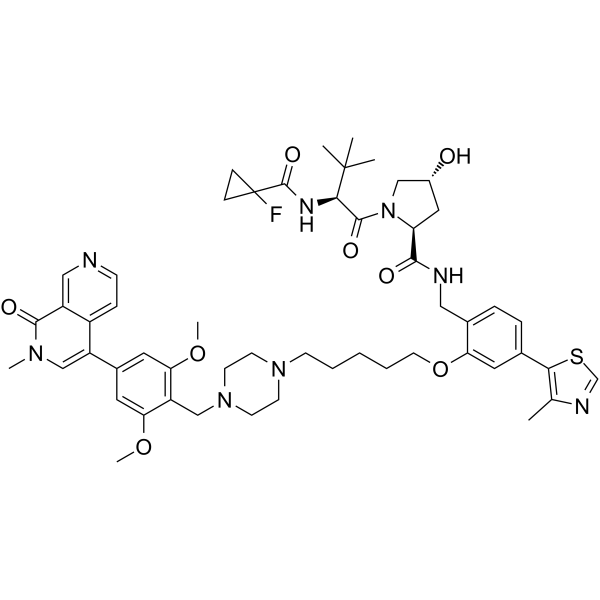
GC65135
VZ185
VZ185 es una sonda de degradaciÓn doble basada en Von Hippel-Lindau potente, rÁpida y selectiva de BRD9 y BRD7 con DC50 de 4,5 y 1,8 nM, respectivamente. VZ185 es citotÓxico en células EOL-1 y A-402, con CE50 de 3 nM y 40 nM, respectivamente.
-
GC33285
Y06036
Y06036 es un inhibidor BET potente y selectivo, que se une al bromodominio BRD4(1) con un valor Kd de 82 nM. Actividad antitumoral.

-
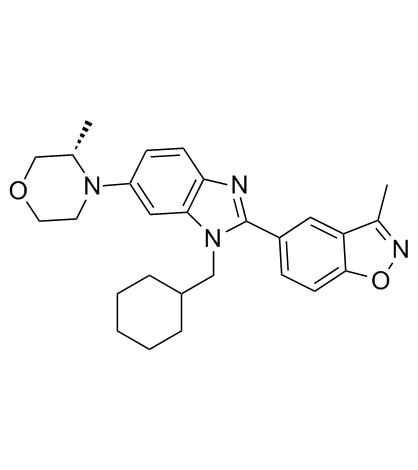
GC33264
Y06137
Y06137 es un inhibidor BET potente y selectivo para el tratamiento del cÁncer de prÓstata resistente a la castraciÓn (CPRC). Y06137 se une al bromodominio BRD4(1) con una Kd de 81 nM.
-
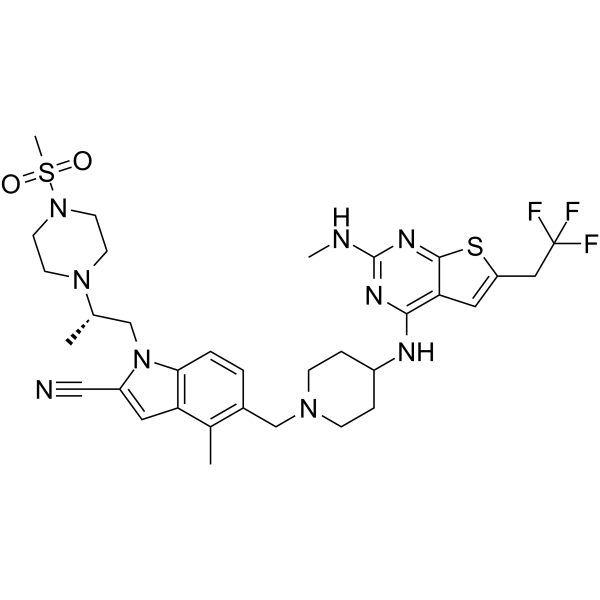
GC64518
Ziftomenib
Ziftomenib (KO-539) es un inhibidor de la interacciÓn menina-MLL con actividades antitumorales (WO2017161028A1, compuesto 151).
-
GC19455
ZL0420
ZL0420 es un inhibidor potente y selectivo de la proteÍna 4 que contiene bromodominio (BRD4) con valores IC50 de 27 nM contra BRD4 BD1 y 32 nM contra BRD4 BD2.

-
GC65204
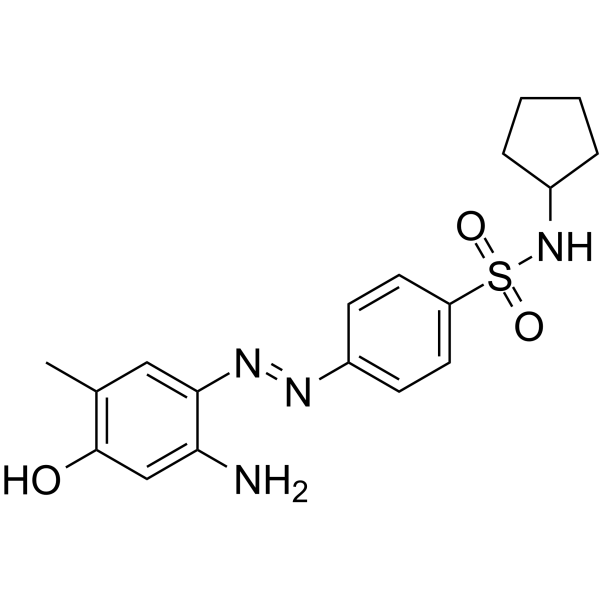
ZL0454
ZL0454 es un inhibidor potente y selectivo de la proteÍna 4 que contiene bromodominio (BRD4) con una IC50 de 49 y 32 nM para BD1 y BD2.
-
GC64529
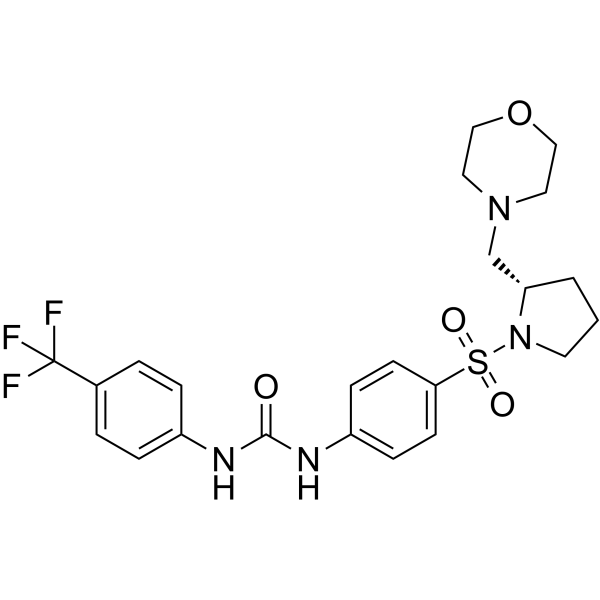
ZL0590
ZL0590 es un potente inhibidor selectivo de BRD4 BD1 activo por vÍa oral con una IC50 de 90 nM para BRD4 BD1 humano.
-
GC65269
ZXH-3-26
ZXH-3-26 es un PROTAC conectado por ligandos para Cereblon y BRD4 con un DC50/5h de 5 nM. El DC50/5h se refiere a la mitad de la degradaciÓn mÁxima después de 5 horas de tratamiento de ~ 5 nM.