Epigenetic Reader Domain
Epigenetic regulators of gene expression and chromatin state include so-called writers, erasers, and readers of chromatin modifications.Well-characterized examples of reader domains include bromodomains typically binding acetyllysine and chromatin organization modifier (chromo), malignant brain tumor (MBT), plant homeodomain (PHD), and Tudor domains generally associating with methyllysine. Research on epigenetic readers has been tremendously influenced by the discovery of selective inhibitors targeting the bromodomain and extraterminal motif (BET) family of acetyl-lysine readers. The human genome encodes 46 proteins containing 61 bromodomains clustered into eight families. Distinct experimental approaches are used to identify the first BET inhibitors, GSK 525762A and (+)-JQ-1.
The Polycomb group (PcG) protein, enhancer of zeste homologue 2 (EZH2), has an essential role in promoting histone H3 lysine 27 trimethylation (H3K27me3) and epigenetic gene silencing. This function of EZH2 is important for cell proliferation and inhibition of cell differentiation, and is implicated in cancer progression. Cyclin-dependent kinases regulate epigenetic gene silencing through phosphorylation of EZH2. In many types of cancers including lymphomas and leukemia, EZH2 is postulated to exert its oncogenic effects via aberrant histone and DNA methylation, causing silencing of tumor suppressor genes.
p300/CBP is not only a transcriptional adaptor but also a histone acetyltransferase.
Ziele für Epigenetic Reader Domain
Produkte für Epigenetic Reader Domain
- Bestell-Nr. Artikelname Informationen
-
GC34078
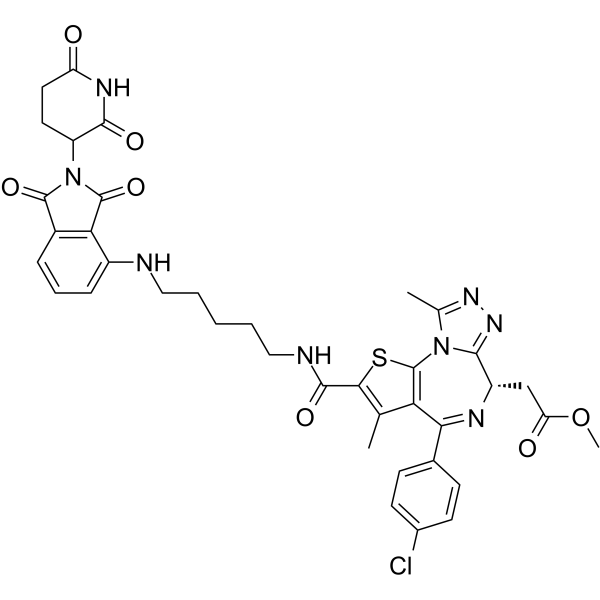
I-CBP112
I-CBP112 ist ein spezifischer und potenter Acetyl-Lysin-kompetitiver Protein-Protein-Interaktionsinhibitor, der die CBP/p300-BromodomÄnen hemmt und die Acetylierung durch p300 verstÄrkt.
-
GC43889
I-CBP112 (hydrochloride)
I-CBP112 (Hydrochlorid) ist ein selektiver Inhibitor von CBP/P300, der ihre Bromodomänen (Kds \u003d 142 bzw. 625 nM) direkt bindet. I-CBP112 reduziert signifikant das Leukämie-initiierende Potenzial von MLL-AF9(+) akuten myeloischen Leukämiezellen in einer dosisabhängigen Weise in vitro und in vivo. I-CBP112 erhöht die zytotoxische Aktivität des BET-Bromodomänen-Inhibitors JQ1 sowie von Doxorubicin.
-
GC33042
IACS-9571 (ASIS-P040)
IACS-9571 (ASIS-P040) ist ein potenter und selektiver Inhibitor von TRIM24 und BRPF1, mit einer IC50 von 8 nM für TRIM24 sowie Kds von 31 nM und 14 nM für TRIM24 bzw. BRPF1.
-
GC60925
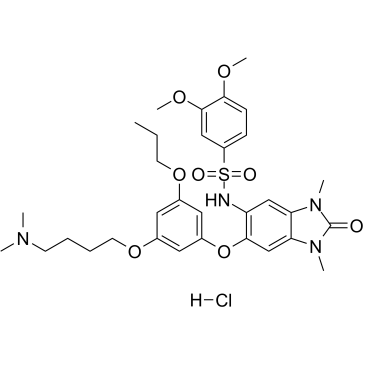
IACS-9571 hydrochloride
IACS-9571 (ASIS-P040) Hydrochlorid ist ein potenter und selektiver Inhibitor von TRIM24 und BRPF1 mit einem IC50 von 8 nM fÜr TRIM24 und Kds von 31 nM und 14 nM fÜr TRIM24 bzw. BRPF1.
-
GC34437
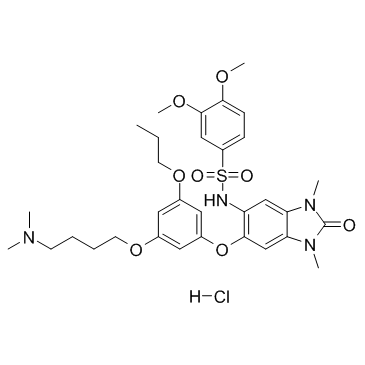
IACS-9571 Hydrochloride (ASIS-P040 Hydrochloride)
-
GC48548
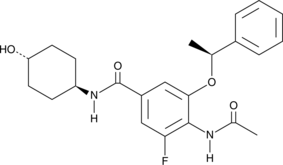
iBET-BD2
A BD2 bromodomain inhibitor
-
GC19200
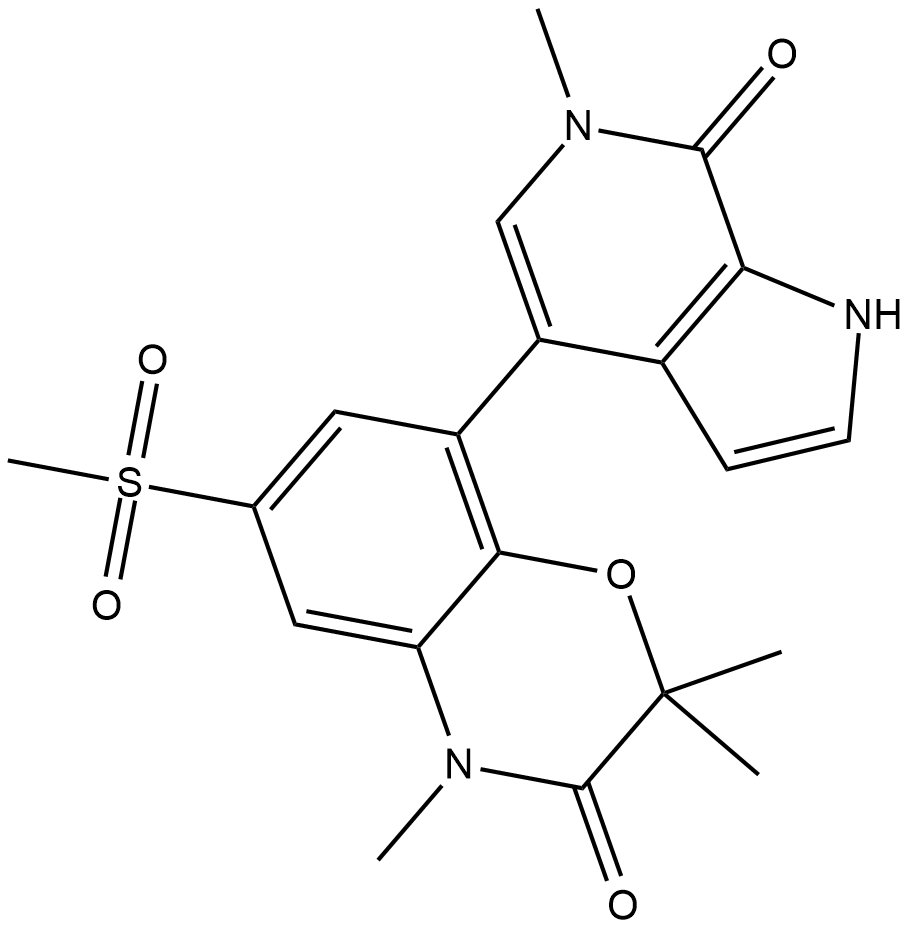
INCB-057643
INCB-057643 ist ein neuartiger, oral bioverfÜgbarer BET-Inhibitor.
-
GC33026
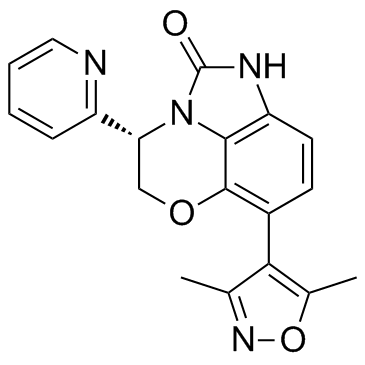
INCB054329
INCB054329 ist ein potenter BET-Hemmer.
-
GC30644
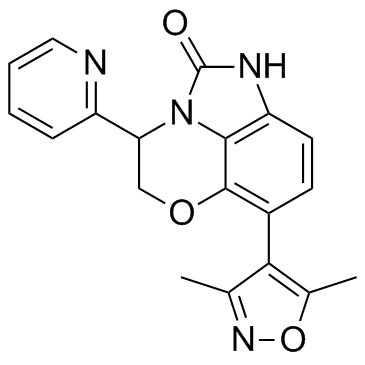
INCB054329 Racemate
INCB054329 Racemate ist ein BET-Protein-Inhibitor.
-
GC19210
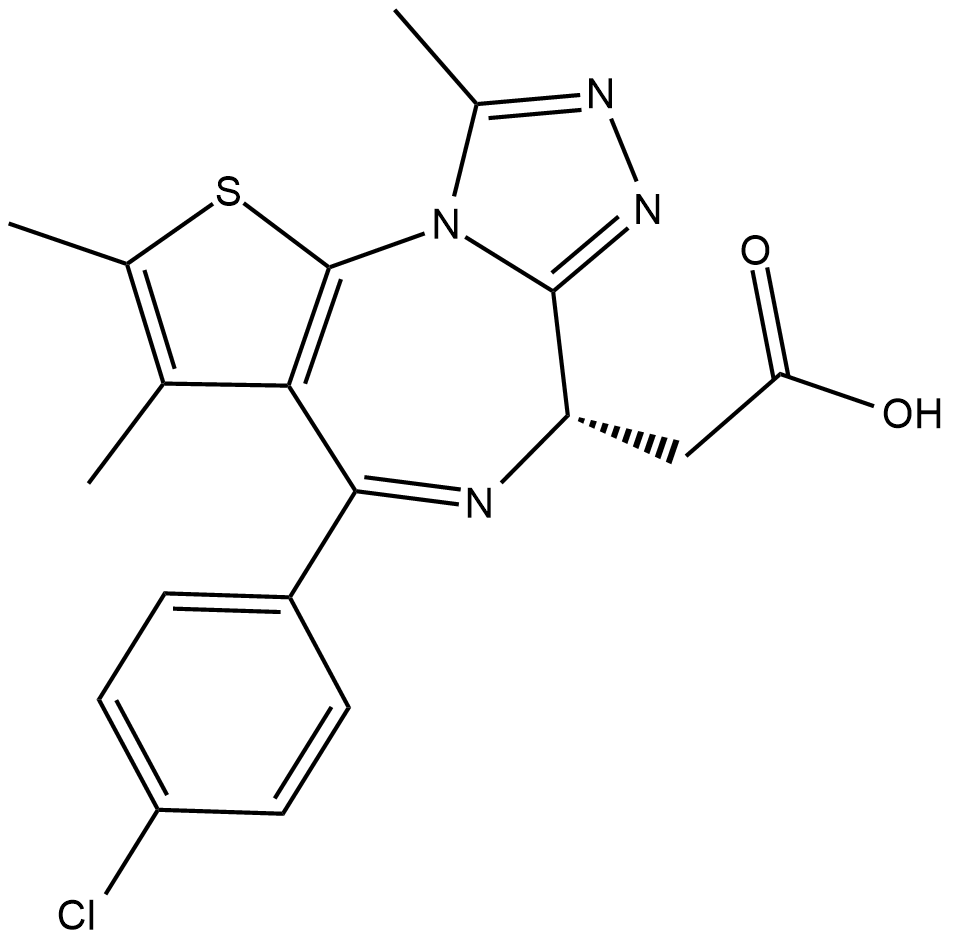
JQ-1 carboxylic acid
JQ-1-Carbonsäure, eine Art von (+)-JQ-1-Derivat, ist ein potenter BET-Bromodomänen-Inhibitor zur Herunterregulierung der PD-L1-Expression auf der Oberfläche von Tumorzellen. JQ-1-Carbonsäure kann als Vorstufe zur Synthese von PROTACs verwendet werden, die auf BET-Bromodomänen abzielen.
-
GC31654
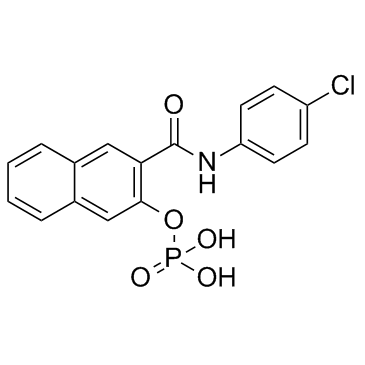
KG-501 (Naphthol AS-E phosphate)
KG-501 (Naphthol AS-E Phosphat) ist ein CREB-Inhibitor mit einem IC50 von 6,89 μM.
-
GC50395
L Moses dihydrochloride
High affinity and selective PCAF bromodomain inhibitor
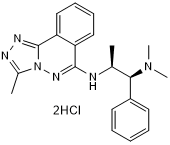
-
GC33183
L-45 (L-Moses)
L-45 (L-Moses) (L-45) ist der erste potente, selektive und zellaktive Inhibitor der p300/CBP-assoziierten Faktor (PCAF)-BromdomÄne (Brd) mit einem Kd von 126 nM.
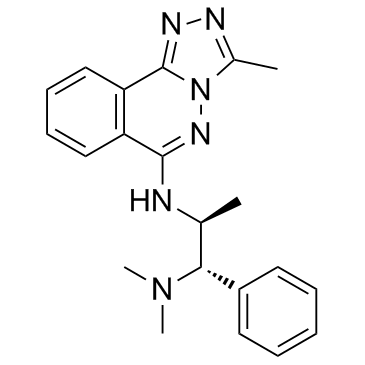
-
GC34377
L-45 dihydrochloride (L-Moses dihydrochloride)
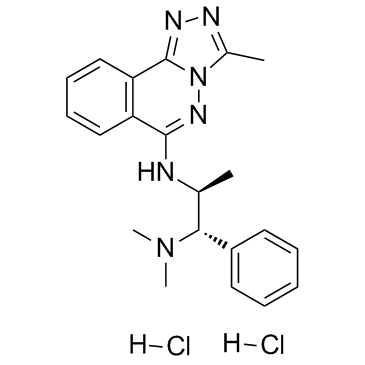
-
GC18731
LP99
LP99, eine epigenetische Sonde, ist ein potenter und selektiver Inhibitor der BRD7- und BRD9-BromodomÄnen mit einer Kd von 99 nM gegen BRD9.
-
GC62591
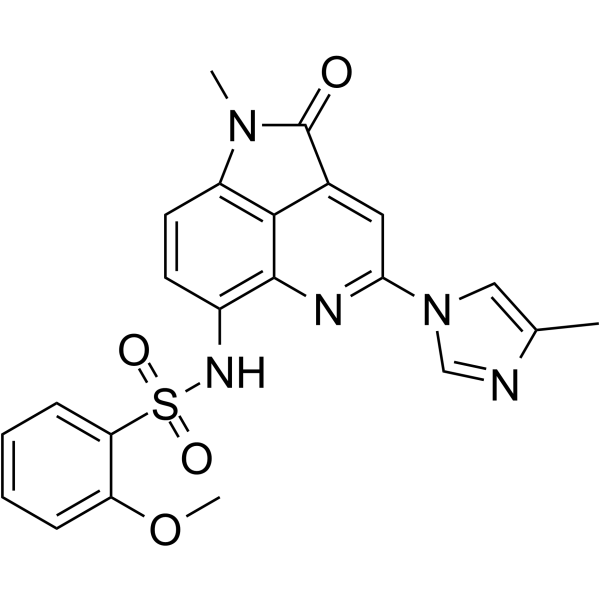
LT052
LT052 ist ein hochselektiver BET-BD1-Inhibitor mit einem IC50 von 87,7 nM.
-
GC36521
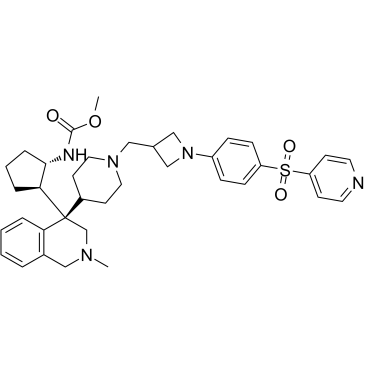
M89
M-89 ist ein hochwirksamer und spezifischer Menin-Inhibitor mit einer Kd von 1,4 nM für die Bindung an Menin.
-
GC63064
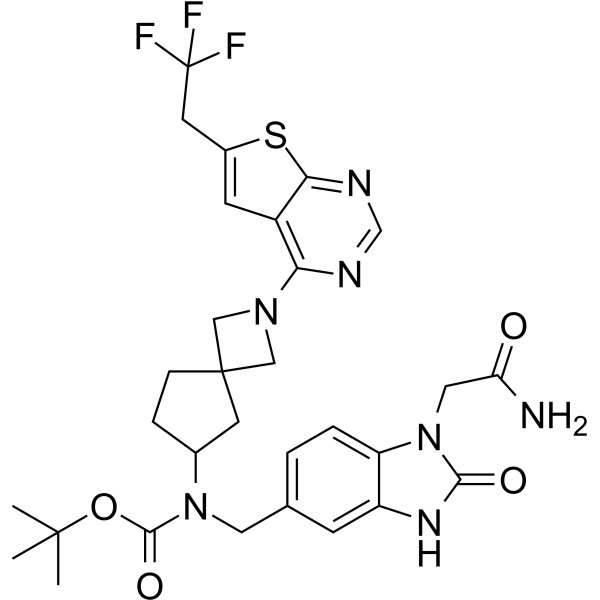
Menin-MLL inhibitor 19
Menin-MLL-Inhibitor 19, ein potenter Exo-Aza-Spiro-Inhibitor der Menin-Mll-Wechselwirkung, Beispiel A17, extrahiert aus dem Patent WO2019120209A1.
-
GC63538
Menin-MLL inhibitor 20
Menin-MLL-Inhibitor 20 ist ein irreversibler Menin-MLL-Interaktionsinhibitor mit AntitumoraktivitÄten (WO2020142557A1, Intermediat 6).

-
GC11439
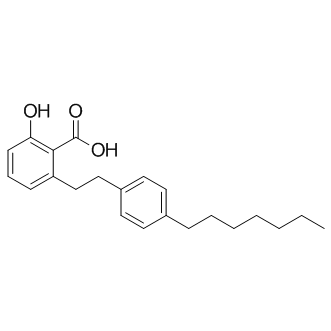
MG 149
MG149 (Tip60-HAT-Inhibitor) ist ein selektiver und potenter Tip60-Inhibitor mit IC50 von 74 uM, Ähnlicher Potenz fÜr MOF (IC50 = 47 uM); wenig potent fÜr PCAF und p300 (IC50 >200 uM).
-
GC11418
MI-2
An inhibitor of menin-MLL fusion protein interactions
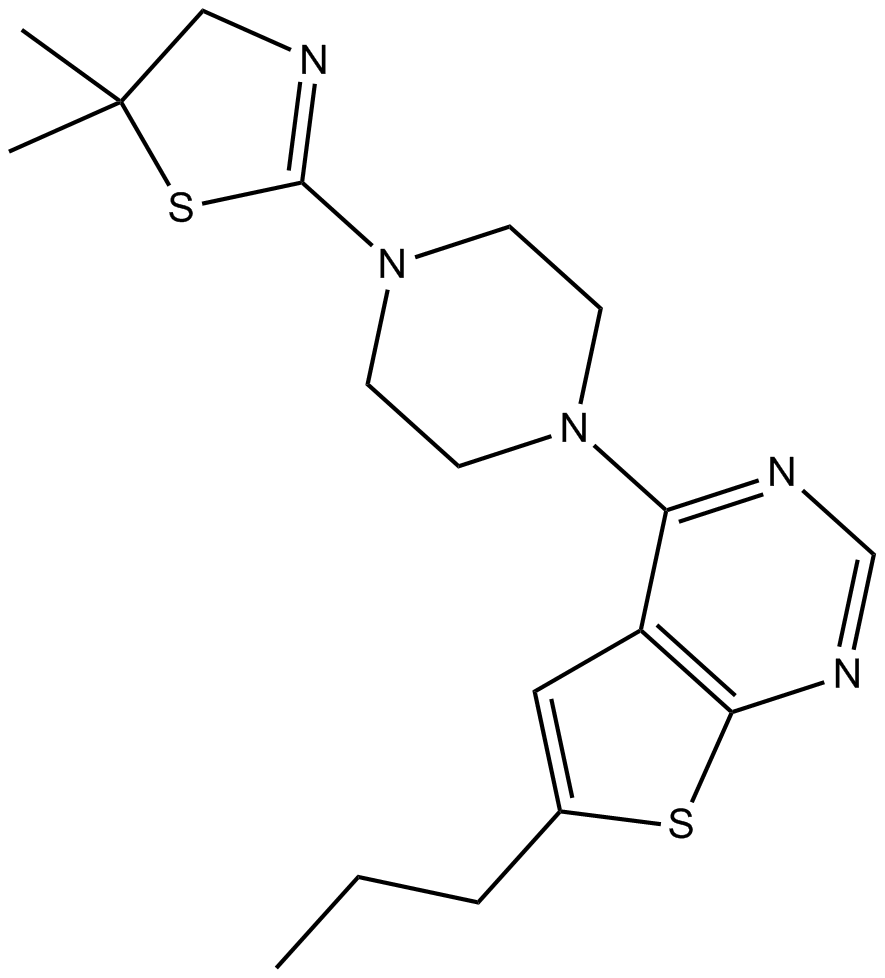
-
GC12199
MI-3
An inhibitor of menin-MLL fusion protein interactions
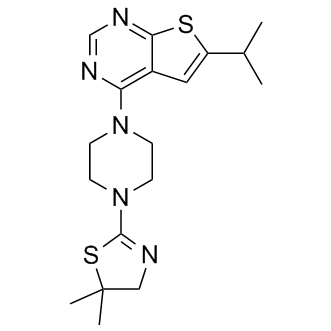
-
GC19246
MI-463
MI-463 ist ein hochwirksamer und oral bioverfÜgbarer niedermolekularer Inhibitor der Menin-mLL-Interaktion.
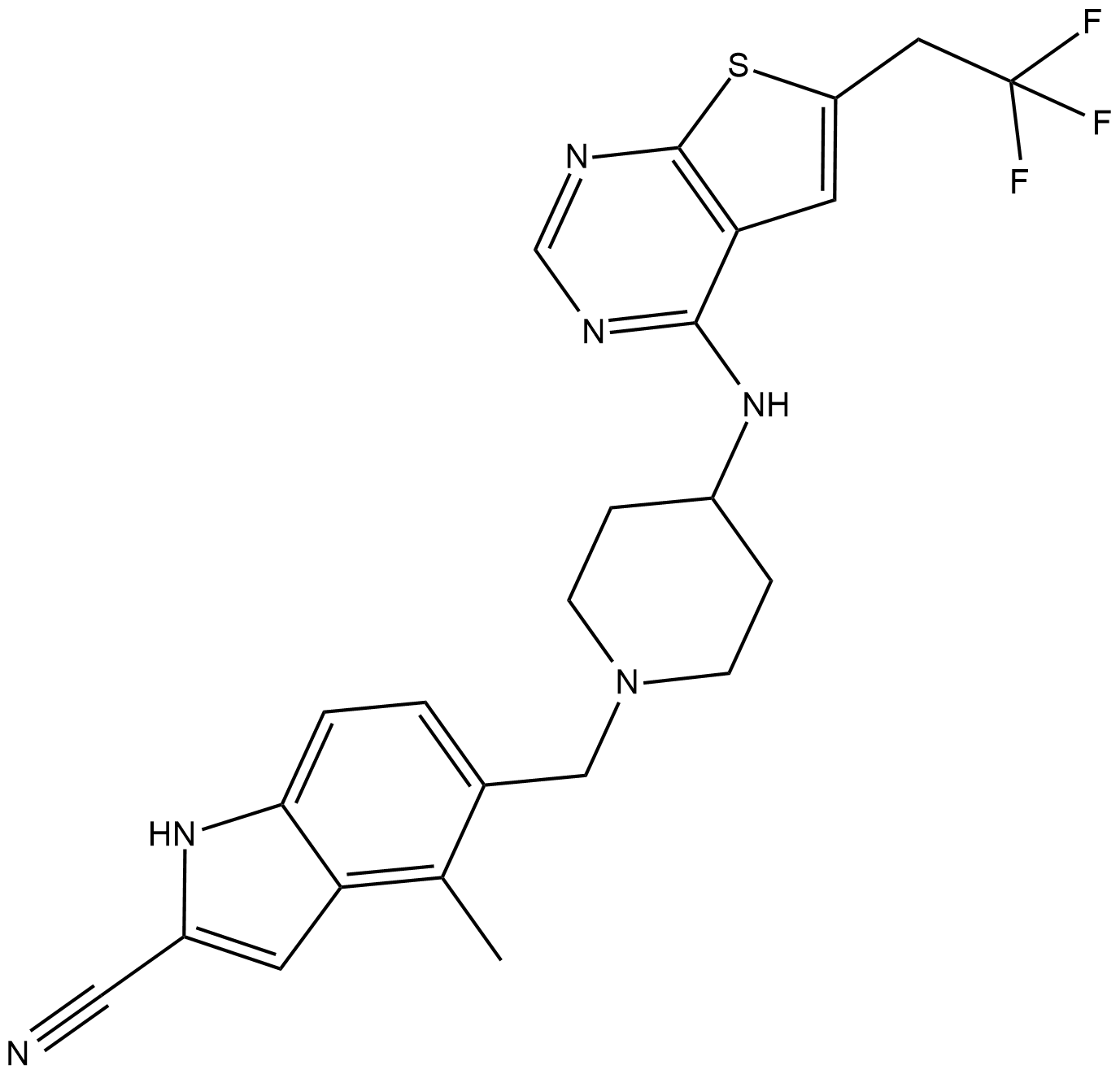
-
GC19247
MI-503
MI-503 ist ein hochwirksamer und oral bioverfÜgbarer niedermolekularer Inhibitor der Menin-mLL-Interaktion.
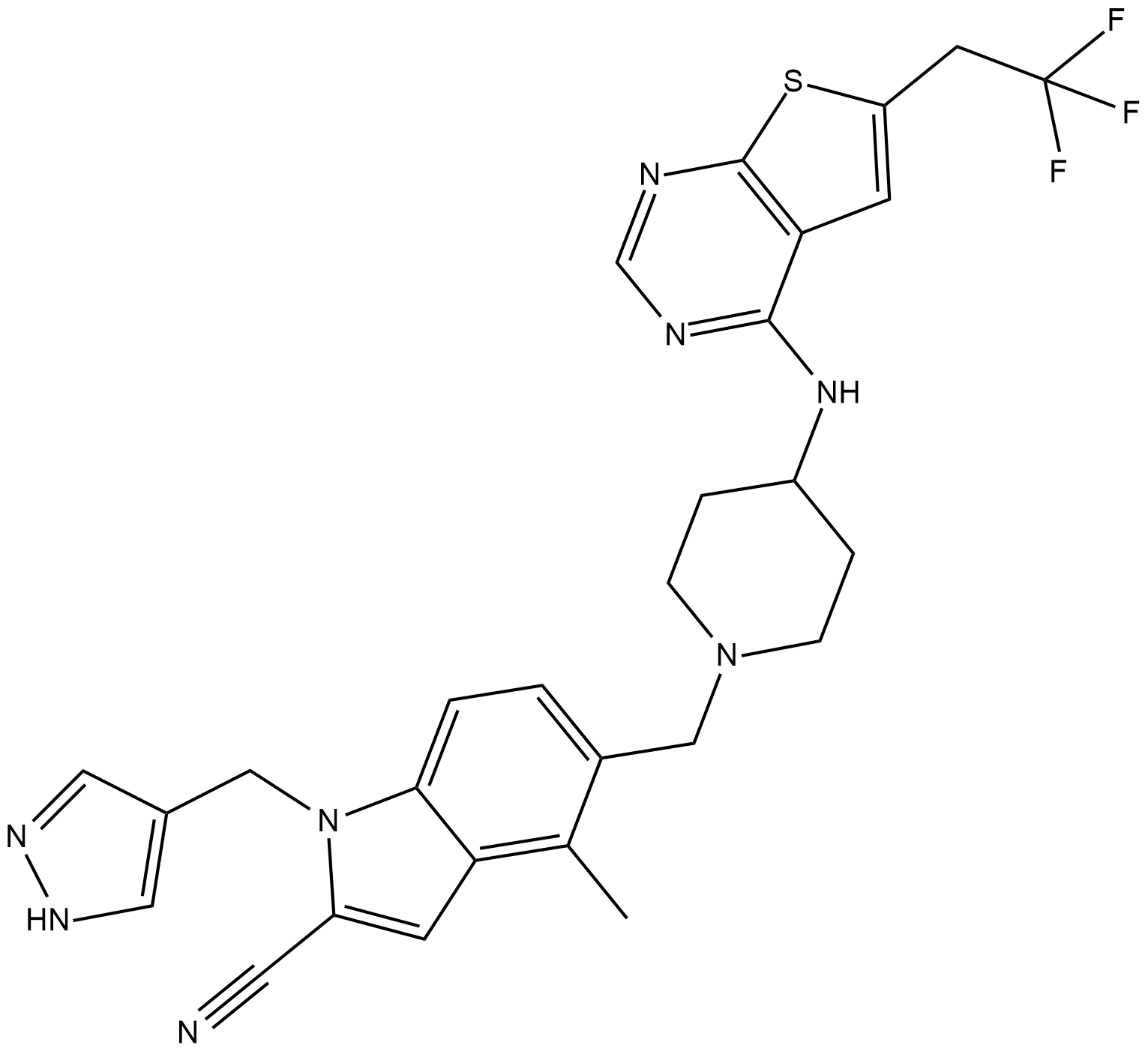
-
GC32798
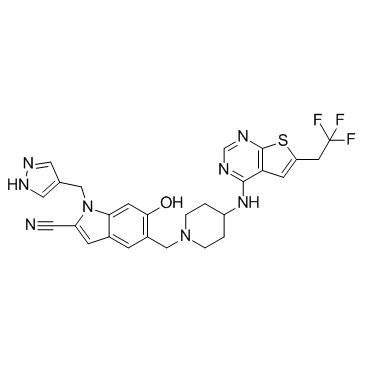
MI-538
MI-538 ist ein Inhibitor der Interaktion zwischen Menin und MLL-Fusionsproteinen mit einem IC50 von 21 nM.
-
GC19248
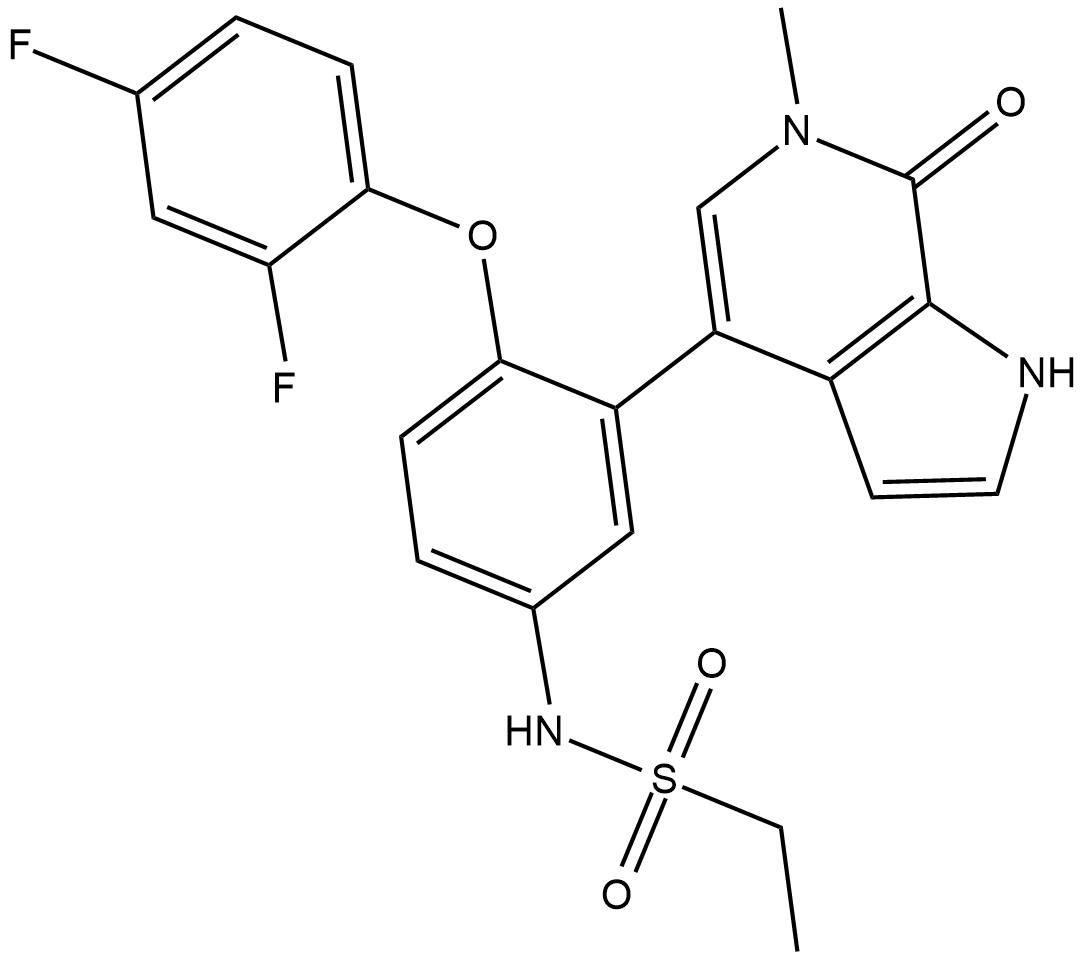
Mivebresib
Mivebresib (ABBV-075) ist ein potenter und oral wirksamer BromodomÄnen- und BET-BromdomÄnen-Inhibitor. Mivebresib bindet an BRD4 mit einem Ki von 1,5 nM.
-
GC34675
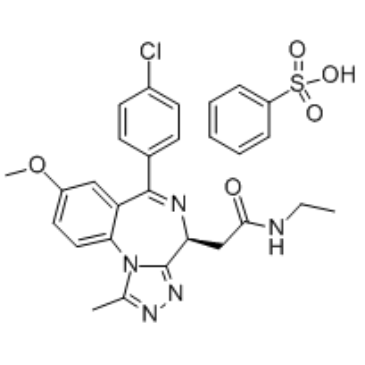
Molibresib besylate
Molibresib-Besylat (GSK 525762C; I-BET 762-Besylat) ist ein BET-BromdomÄnen-Inhibitor mit einem IC50-Wert von 32,5-42,5 nM.
-
GC36657
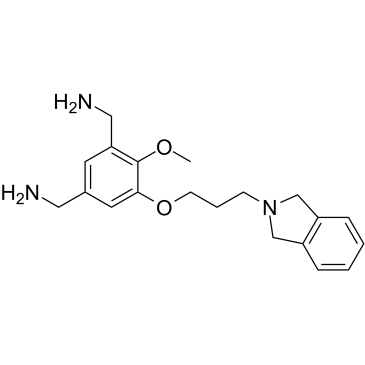
MS31
MS31 ist ein potenter, hochaffiner und selektiver Fragment-Ähnlicher Inhibitor des Methyllysin-Reader-Proteins Spinlin 1 (SPIN1). MS31 hemmt stark die Wechselwirkungen zwischen SPIN1 und H3K4me3 (IC50=77 nM, AlphaLISA; 243 nM, FP). MS31 bindet selektiv die Tudor-DomÄne II von SPIN1 (Kd = 91 nM). MS31 hemmt wirksam die Bindung von Trimethyllysin-enthaltenden Peptiden an SPIN1. MS31 ist fÜr nicht-tumorigene Zellen nicht toxisch.
-
GC39252
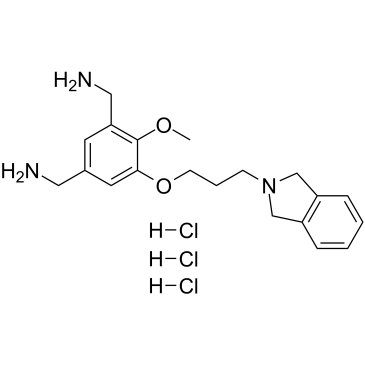
MS31 trihydrochloride
MS31-Trihydrochlorid ist ein potenter, hochaffiner und selektiver Fragment-Ähnlicher Inhibitor des Methyllysin-Reader-Proteins Spinlin 1 (SPIN1). MS31-Trihydrochlorid hemmt wirksam die Wechselwirkungen zwischen SPIN1 und H3K4me3 (IC50 = 77 nM, AlphaLISA; 243 nM, FP). MS31-Trihydrochlorid bindet selektiv die Tudor-DomÄne II von SPIN1 (Kd = 91 nM). MS31-Trihydrochlorid hemmt wirksam die Bindung von Trimethyllysin-enthaltenden Peptiden an SPIN1 und ist fÜr nicht-tumorigene Zellen nicht toxisch.
-
GC44250
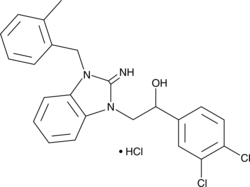
MS351
MS351 is an antagonist of chromobox 7 (CBX7) that acts by binding the CBX7 chromodomain.
-
GC64999
MS402
MS402 ist ein BD1-selektiver BET-BrD-Inhibitor mit Kis von 77 nM, 718 nM, 110 nM, 200 nM, 83 nM und 240 nM fÜr BRD4(BD1), BRD4(BD2), BRD3(BD1), BRD3(BD2) , BRD2(BD1) bzw. BRD2(BD2).

-
GC31881
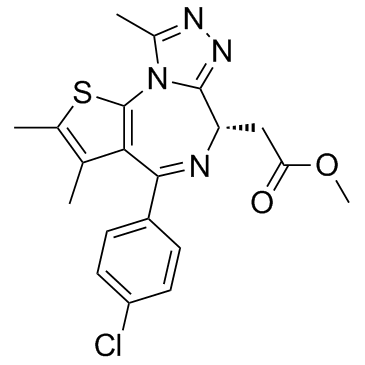
MS417 (GTPL7512)
MS417 (GTPL7512) ist ein selektiver BET-spezifischer BRD4-Inhibitor, bindet an BRD4-BD1 und BRD4-BD2 mit IC50s von 30, 46 nM bzw. Kds von 36,1, 25,4 nM, mit schwacher SelektivitÄt bei CBP BRD (IC50, 32,7 μ ;M).
-
GC13148
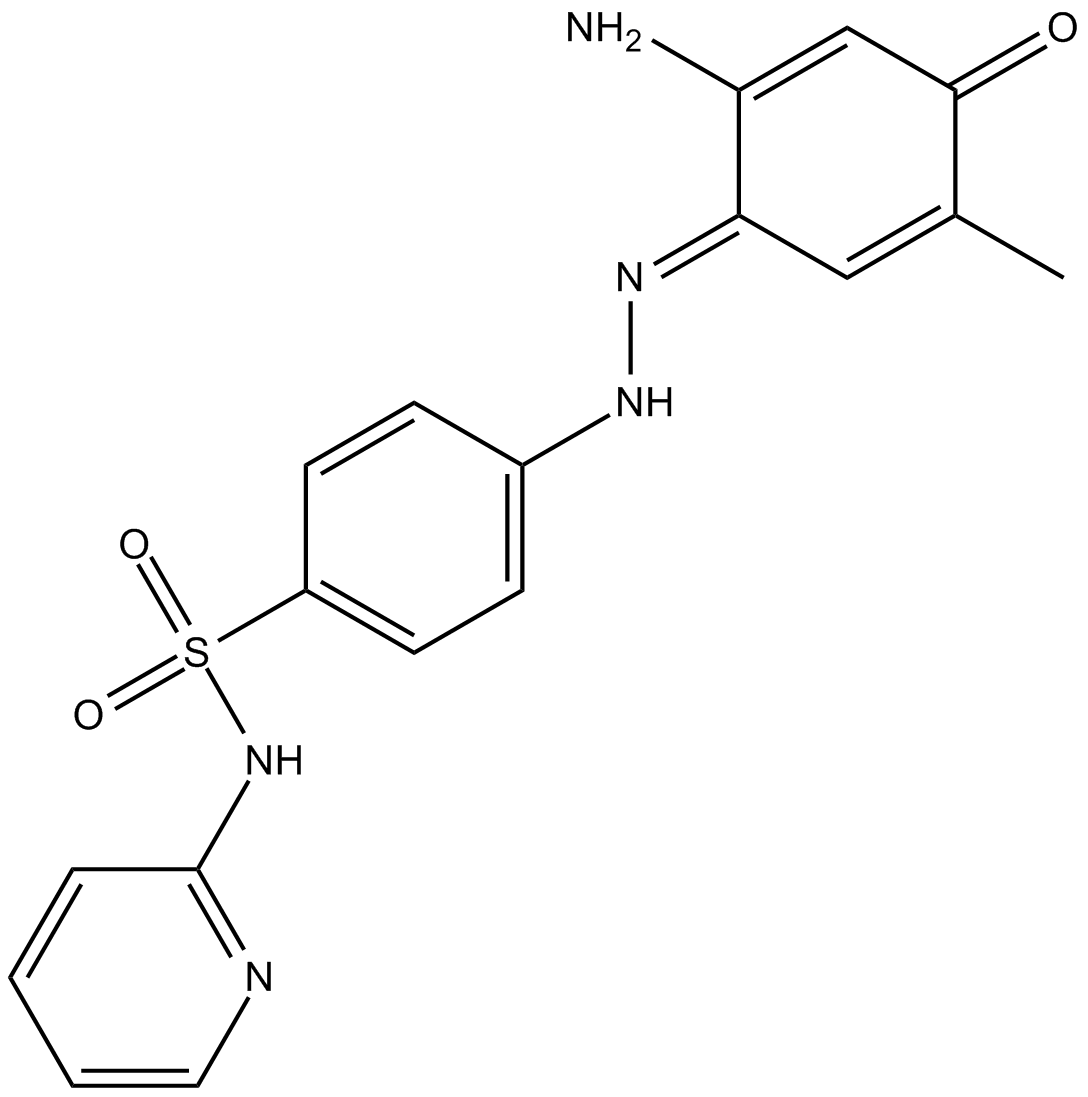
MS436
MS436 ist eine neue Klasse von BromodomÄnen-Inhibitoren, zeigt eine starke AffinitÄt von geschÄtzten Ki = 30-50 nM fÜr das BRD4-BrD1 und eine 10-fache SelektivitÄt gegenÜber dem BrD2.
-
GC38474
MS645
MS645 ist ein bivalenter BET-BromodomÄnen (BrD)-Inhibitor mit einem Ki von 18,4 nM fÜr BRD4-BD1/BD2. MS645 schrÄnkt die bivalente Hemmung von BRD4-BrDs rÄumlich ein, was zu einer anhaltenden Repression der BRD4-TranskriptionsaktivitÄt in soliden Tumorzellen fÜhrt.
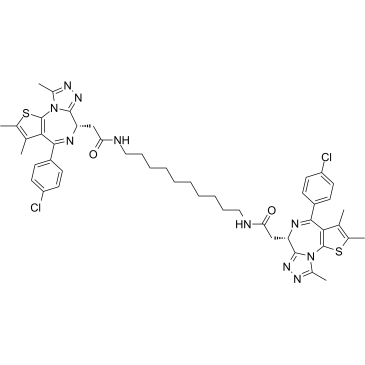
-
GC18729
MZ1
MZ1 ist ein PROTAC, verbunden durch Liganden für von Hippel-Lindau und BRD4. MZ1 induziert wirksam und schnell eine reversible, lang anhaltende und selektive Entfernung von BRD4 gegenüber BRD2 und BRD3. Kds von 382/120, 119/115 und 307/228 nM für BRD4 BD1/2, BRD3 BD1/2 bzw. BRD2 BD1/2.
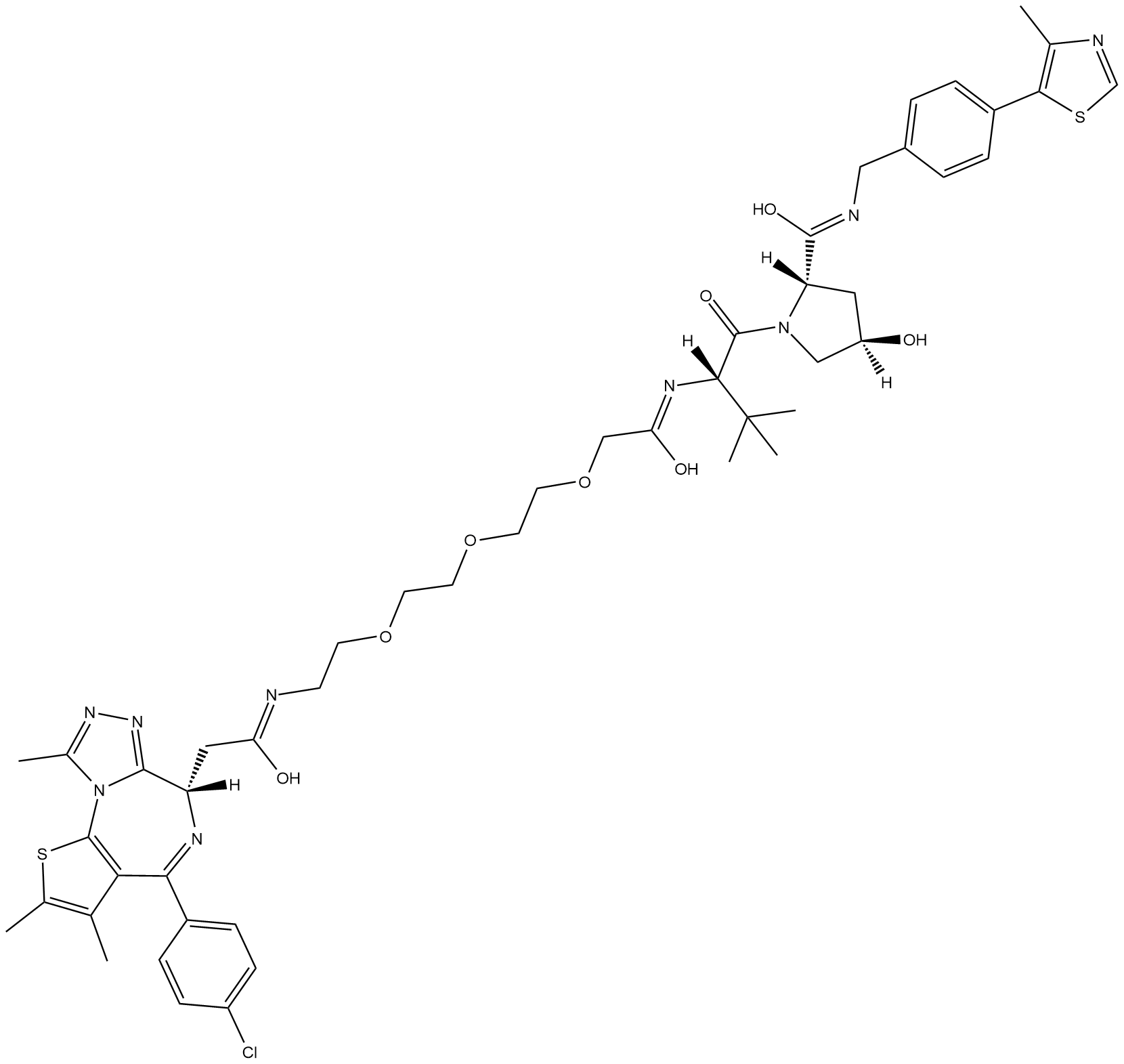
-
GC33102
MZP-54
MZP-54 ist ein PROTAC, verbunden durch Liganden fÜr von Hippel-Lindau und BRD3/4, mit einem Kd von 4 nM fÜr Brd4BD2.
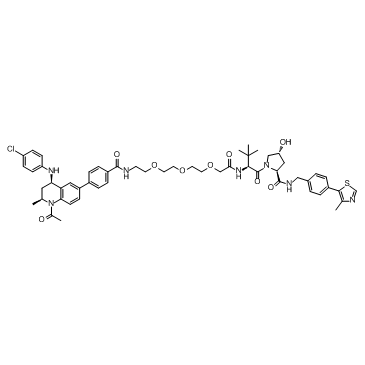
-
GC33363
MZP-55
MZP-55 ist ein PROTAC, verbunden durch Liganden fÜr von Hippel-Lindau und BRD3/4, mit einem Kd von 8 nM fÜr Brd4BD2.
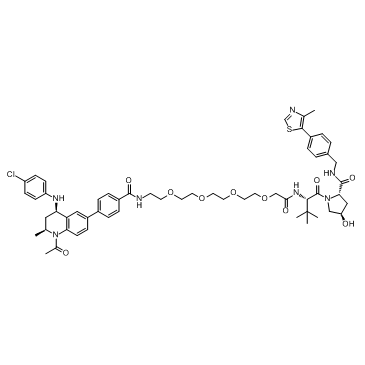
-
GC61841
Naphthol AS-E
Naphthol AS-E ist ein potenter und zellgÄngiger Inhibitor der KIX-KID-Interaktion. Naphthol AS-E bindet direkt an die KIX-DomÄne von CBP (Kd: 8,6 μM), blockiert die Wechselwirkung zwischen der KIX-DomÄne und der KID-DomÄne von CREB mit einem IC50 von 2,26 μM. Naphthol AS-E kann fÜr die Krebsforschung verwendet werden.
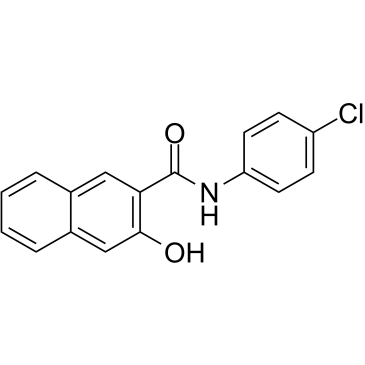
-
GC62620
NHWD-870
NHWD-870 ist ein potenter, oral aktiver und selektiver BromodomÄnen-Inhibitor der BET-Familie und bindet nur BromodomÄnen von BRD2, BRD3, BRD4 (IC50 = 2,7 nM) und BRDT. NHWD-870 hat starke tumorunterdrÜckende Wirkungen und unterdrÜckt die Wechselwirkung zwischen Krebszellen und Makrophagen. NHWD-870 erhÖht die Tumorapoptose und hemmt die Tumorproliferation.
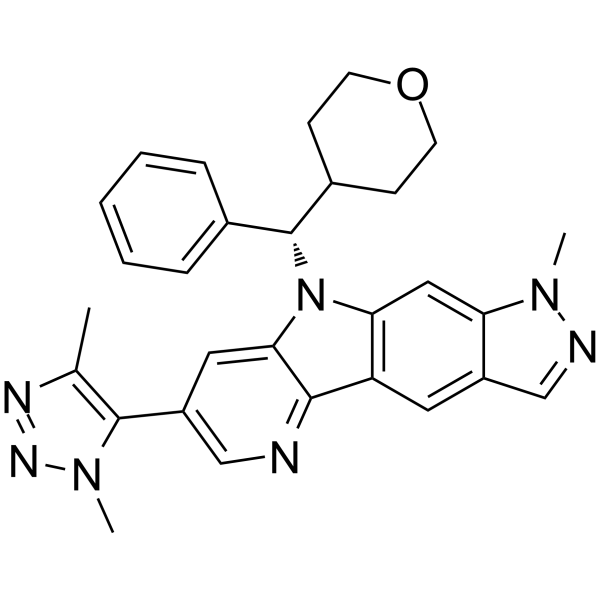
-
GC36734
NI-42
NI-42 (Verbindung 13-d), eine strukturell orthogonale chemische Sonde fÜr die BRPFs, ist ein voreingenommener, potenter Inhibitor der BRD der BRPFs (IC50s von BRPF1/2/3 = 7,9/48/260 nM; Kds von BRPF1 /2/3=40/210/940 nM) mit ausgezeichneter SelektivitÄt gegenÜber Nicht-Klasse-IV-BRD-Proteinen.
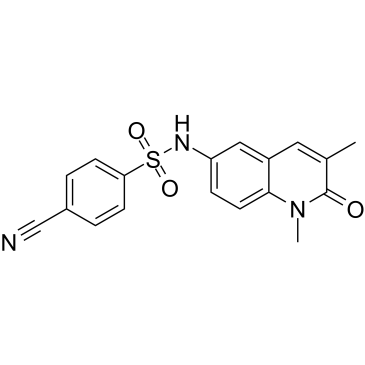
-
GC16763
NI-57
NI-57 ist ein Inhibitor der BromodomÄnen- und PflanzenhomÖodomÄnen-Finger enthaltenden (BRPF) Familie von Proteinen mit IC50-Werten von 3,1, 46 und 140 nM fÜr BRPF1, BRPF2 (BRD1) bzw. BRPF3.
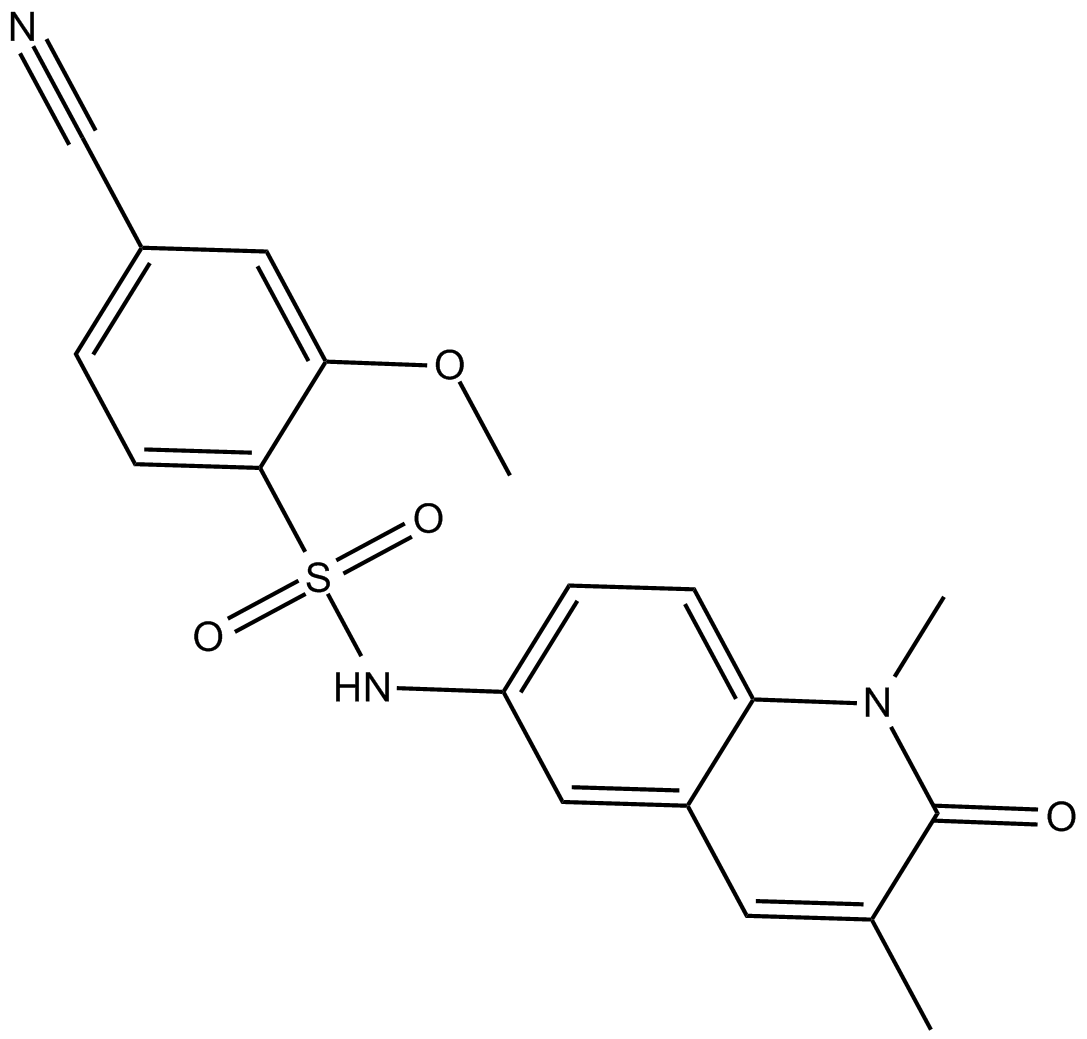
-
GC14103
NSC228155
NSC228155 ist ein Aktivator von EGFR, bindet an die extrazelluläre Region von EGFR und verstärkt die Tyrosinphosphorylierung von EGFR. NSC228155 ist auch ein potenter Inhibitor der KIX-KID-Interaktion, hemmt die Kinase-induzierbare Domäne (KID) von CREB und die KID-interagierende Domäne (KIX) von CBP mit einem IC50 von 0,36 μM.
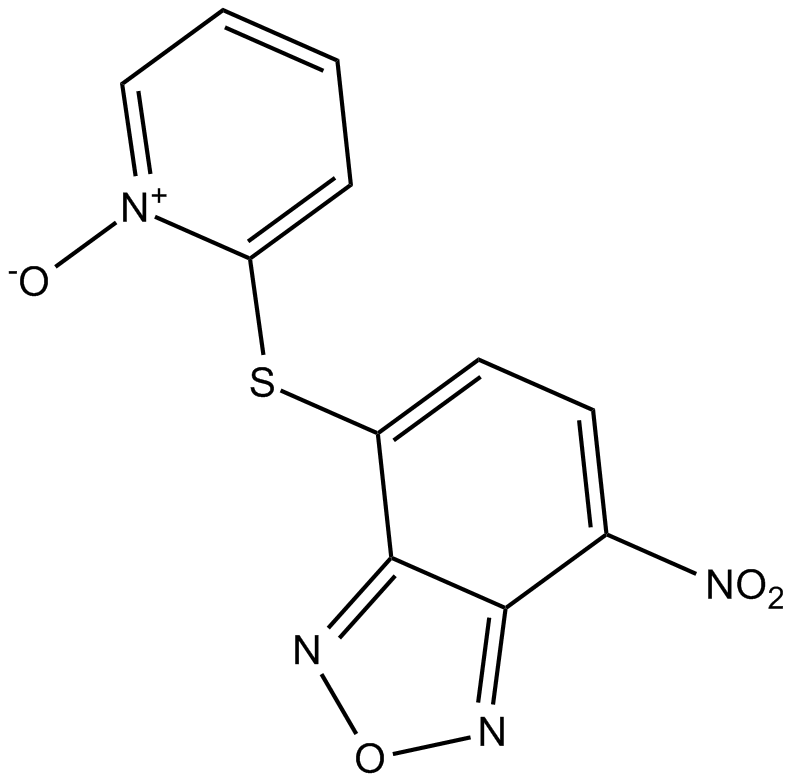
-
GC44477
NVS-CECR2-1
NVS-CECR2-1, ein nicht zur BET-Familie gehÖrender BromodomÄnen(BRD)-Inhibitor, ist ein potenter und selektiver Inhibitor der Chromosomenregion des Cat-Eye-Syndroms, Kandidat 2 (CECR2). NVS-CECR2-1 bindet an CECR2 BRD mit hoher AffinitÄt (IC50 = 47 nM; KD = 80 nM). NVS-CECR2-1 zeigt zytotoxische AktivitÄt und induziert Apoptose gegen verschiedene Krebszellen, indem es auf CECR2 als auch Über einen CECR2-unabhÄngigen Mechanismus abzielt.
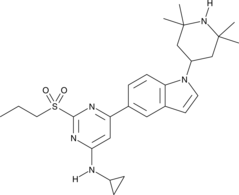
-
GC69601
OARV-771
OARV-771 ist ein VHL-basierter BET-Degradationshemmer (PROTAC) mit verbesserter Zellpermeabilität. OARV-771 zeigt DC50-Werte von 6, 1 und 4 nM für Brd4, Brd2 und Brd3.
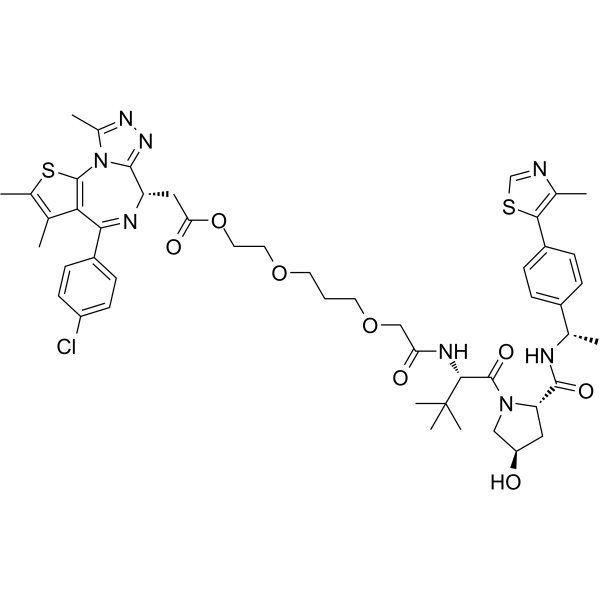
-
GC66067
ODM-207
ODM-207 (BET-IN-4) ist ein potenter Inhibitor des BET-BromdomÄnenproteins (BRD4) mit einem IC50 von ≤ 1 μM.
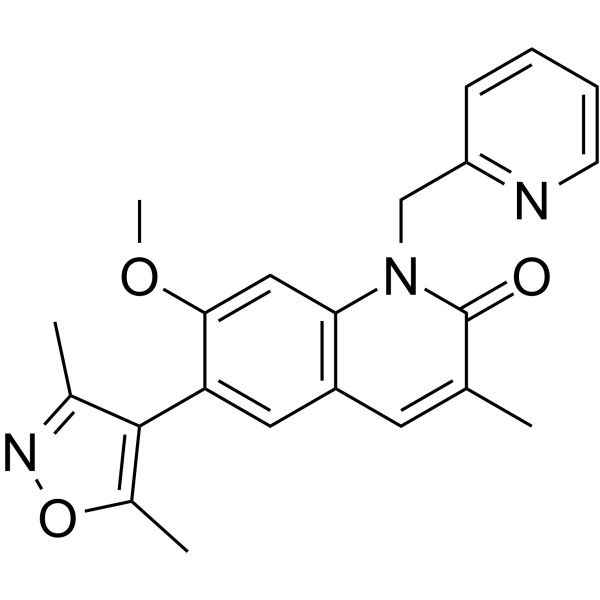
-
GC17482
OF-1
OF-1 ist ein potenter pan-BRPF-BromdomÄnen(BRD)-Inhibitor mit IC50-Werten von 270 nM, 1,2 μM fÜr TRIM24 bzw. BRPF1B.
-
GC17973
OTX-015
A BRD2, BRD3, and BRD4 inhibitor
-
GC39417
OXFBD04
OXFBD04 ist ein potenter und selektiver BRD4-Inhibitor mit einem IC50 von 166 nM. OXFBD04 ist ein potenter BET-BromodomÄnen-Ligand mit einer zusÄtzlichen bescheidenen AffinitÄt fÜr die CREBBP-BromodomÄne. OXFBD04 hat Anti-Krebs-AktivitÄt.
-
GC36887
PF-CBP1 hydrochloride
PF-CBP1-Hydrochlorid ist ein hochselektiver Inhibitor der BromodomÄne des CREB-bindenden Proteins (CBP BRD).
-
GC14361
PFI 3
PFI 3 ist ein selektiver, potenter und zellgängiger SMARCA2/4-Bromodomänen-Inhibitor mit einem Kd von 89 nM.
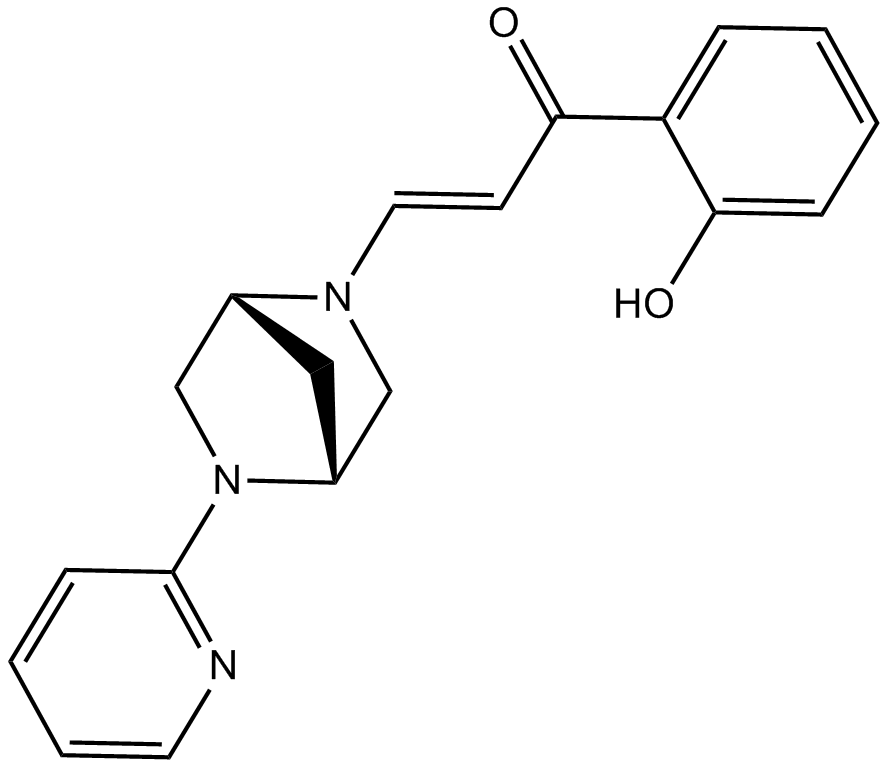
-
GC10979
PFI 4
PFI 4 (Verbindung 11) ist ein potenter und hochselektiver Inhibitor der BRPF1-Bromodomäne (BRPF1B) mit einem IC50 von 172 nM. PFI 4 kann verwendet werden, um die funktionellen Mechanismen des HBO1/BRPF1-Komplexes zu erforschen und um Knochenschwund und osteolytische maligne Knochenläsionen zu untersuchen.
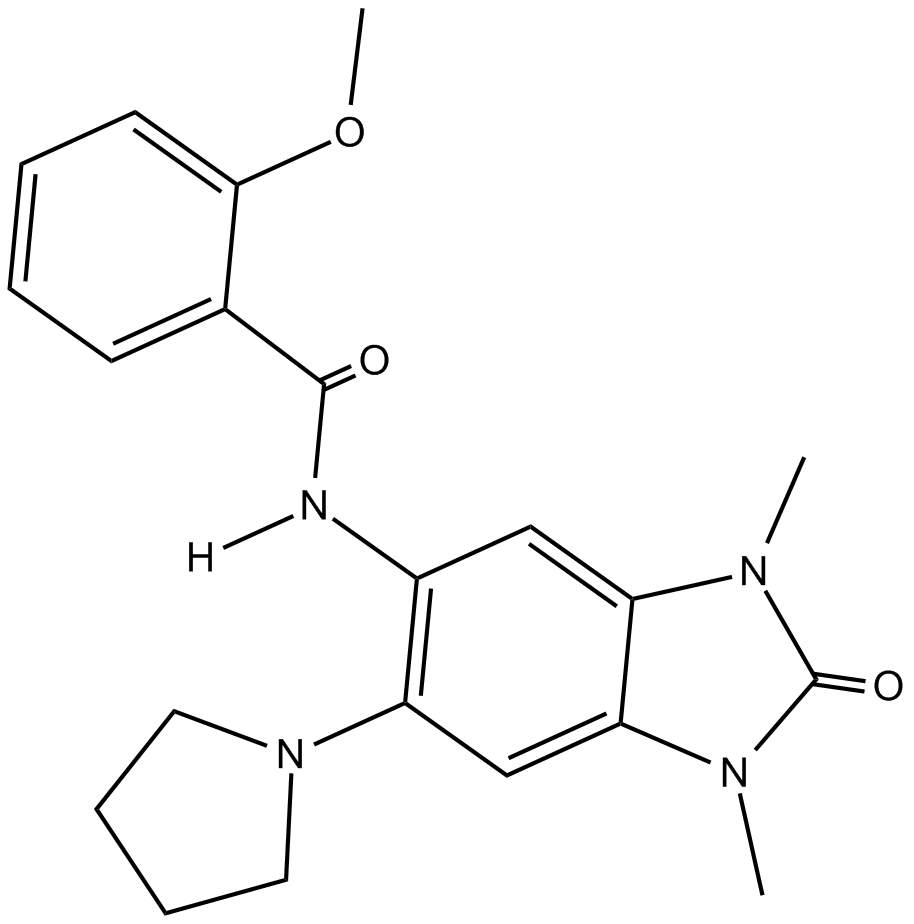
-
GC15375
PFI-1 (PF-6405761)
A BET bromodomain inhibitor
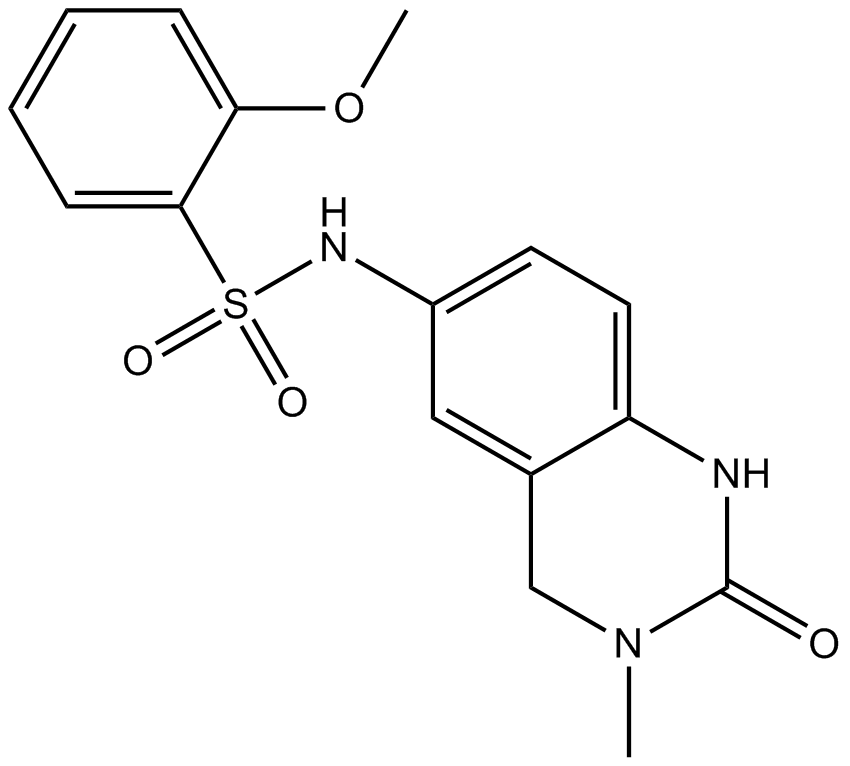
-
GC19298
PLX51107
PLX51107 ist ein potenter und selektiver BET-Inhibitor mit Kds von 1,6, 2,1, 1,7 und 5 nM fÜr BD1 und 5,9, 6,2, 6,1 und 120 nM fÜr BD2 von BRD2, BRD3, BRD4 bzw. BRDT; PLX51107 interagiert auch mit den BromodomÄnen von CBP und EP300 (Kd, im Bereich von 100 nM).
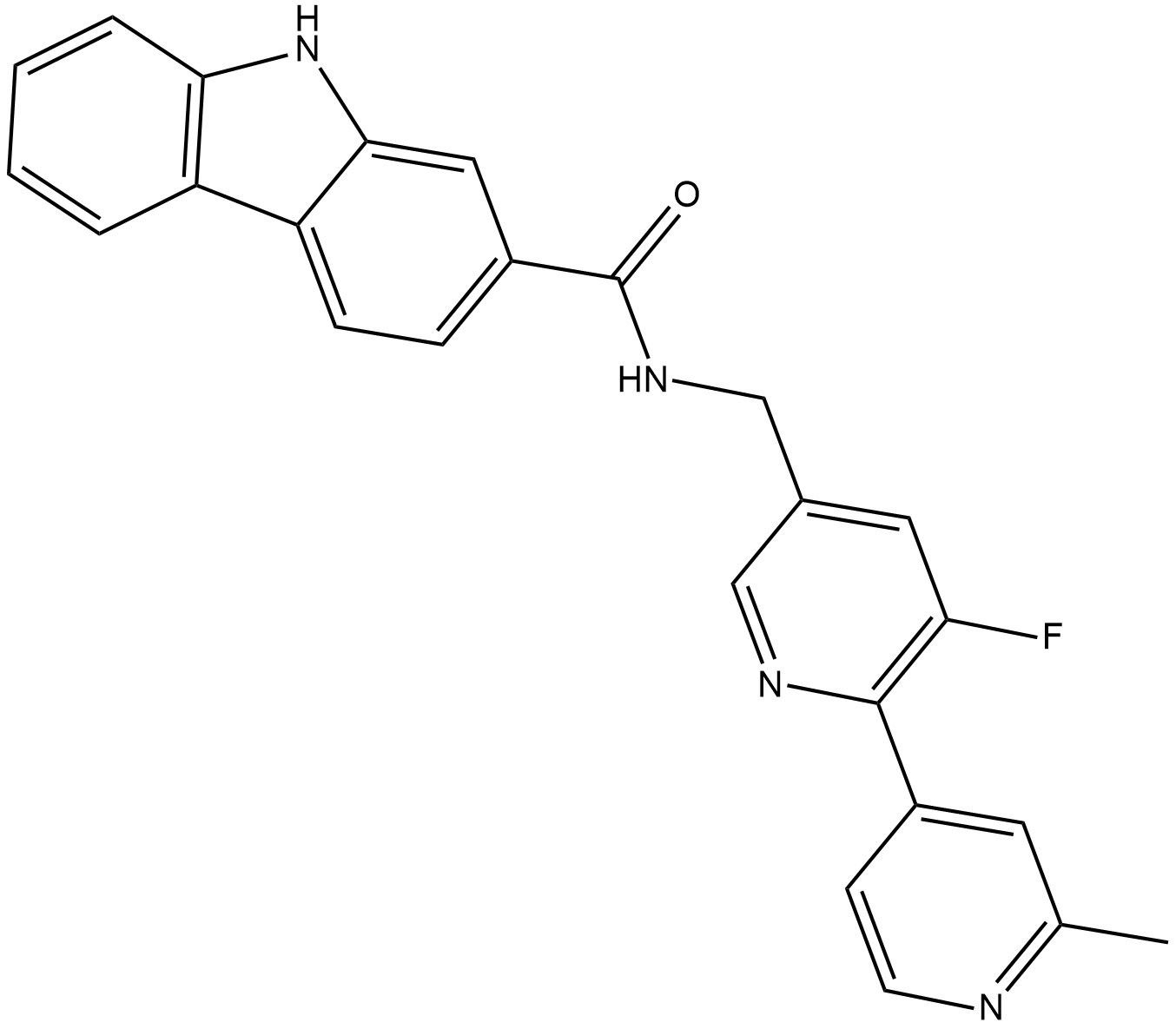
-
GC36943
PNZ5
PNZ5 ist ein potenter und auf Isoxazol basierender pan-BET-Inhibitor mit hoher SelektivitÄt und Wirksamkeit Ähnlich dem etablierten (+)-JQ1, mit einer KD von 5,43 nM fÜr BRD4(1).

-
GC33109
PROTAC BET Degrader-1
PROTAC BET Degrader-1 ist ein PROTAC, das durch Liganden fÜr Cereblon und BET verbunden ist und die BRD2-, BRD3- und BRD4-Proteinspiegel bei niedriger Konzentration senkt.
-
GC32980
PROTAC BET degrader-2
PROTAC BET degrader-2 ist ein durch Liganden fÜr Cereblon und BET verbundenes PROTAC mit einem IC50-Wert von 9,6 nM in der Zellwachstumshemmung in den RS4;11-Zellen und in der Lage, eine Tumorregression zu erreichen.
-
GC34325
PROTAC BET degrader-3
PROTAC BET Degrader-3 ist ein PROTAC, das durch Liganden fÜr von Hippel-Lindau und BET verbunden ist.
-
GC61212
PROTAC BRD4 Degrader-5
PROTAC BRD4 Degrader-5 ist ein PROTAC, das durch Liganden fÜr von Hippel-Lindau und BRD4 verbunden ist. PROTAC BRD4 Degrader-5 kann BRD4 in HER2-positiven und -negativen Brustkrebszelllinien wirksam abbauen.
-
GC62606
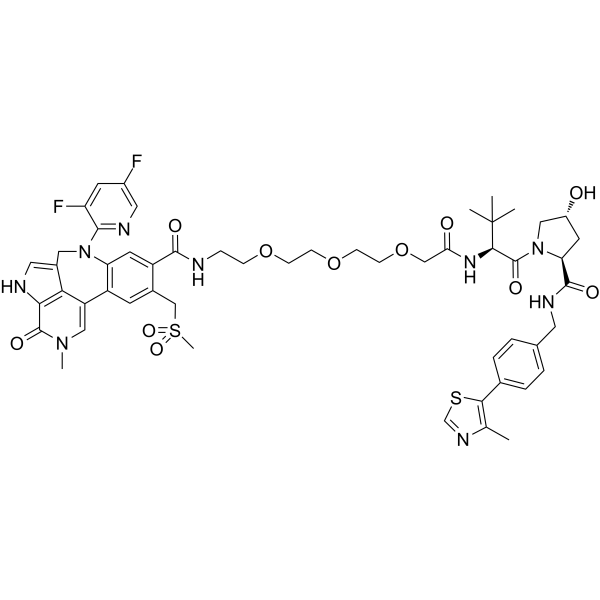
PROTAC BRD4 Degrader-8
PROTAC BRD4 Degrader-8 ist ein PROTAC, verbunden durch Liganden fÜr von Hippel-Lindau und BRD4, mit IC50-Werten von 1,1 nM und 1,4 nM fÜr BRD4 BD1 bzw. BD2. PROTAC BRD4 Degrader-8 ist in der Lage, das BRD4-Protein in PC3-Prostatakrebszellen wirksam abzubauen.
-
GC39180
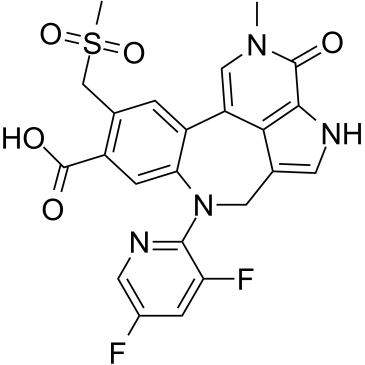
PROTAC BRD4 ligand-1
PROTAC BRD4 Ligand-1 ist ein potenter BET-Inhibitor und ein Ligand fÜr das BRD4-Zielprotein von PROTACT GNE-987.
-
GC33372
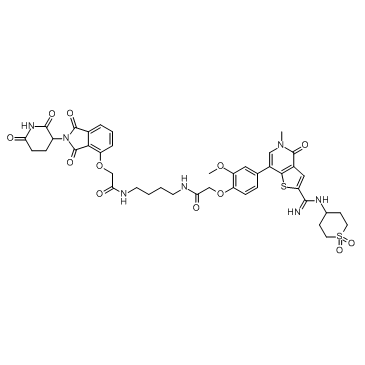
PROTAC BRD9 Degrader-1
PROTAC BRD9 Degrader-1 ist ein PROTAC, das durch Liganden fÜr Cereblon und BRD9 (IC50 = 13,5 nM) verbunden ist und als selektive Sonde verwendet werden kann, die fÜr die Untersuchung der BAF-Komplexbiologie nÜtzlich ist.
-
GC33217
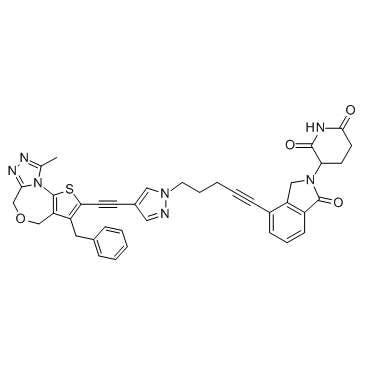
QCA570
QCA570 ist ein PROTAC, verbunden durch Liganden fÜr Cereblon und BET, mit einem IC50 von 10 nM fÜr BRD4 BD1 Protein.
-
GC14199
RVX-208
RVX-208 (RVX-208) ist ein Inhibitor von BET-Transkriptionsregulatoren mit SelektivitÄt fÜr die zweite BromodomÄne. Die IC50-Werte sind 87 μM und 0,51 μM fÜr BD1 bzw. BD2.

-
GC63178
RVX-297
RVX-297 ist ein potenter, oral aktiver BET-BromodomÄnen-Inhibitor mit SelektivitÄt fÜr BD2.
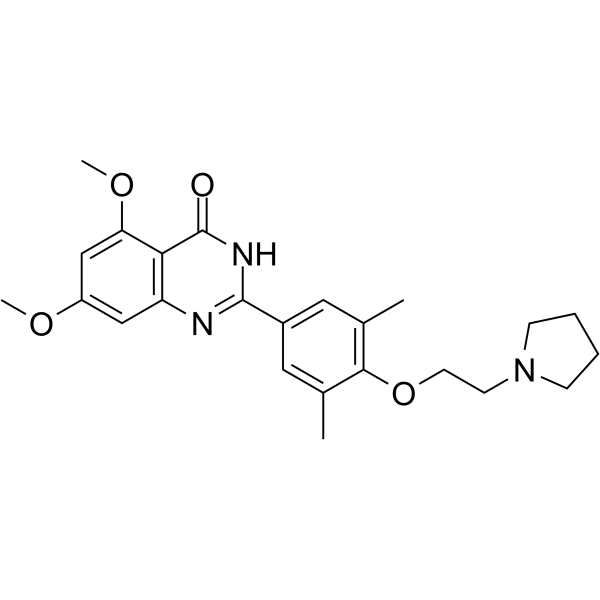
-
GC19328
SF2523
SF2523 ist ein hochselektiver und wirksamer Inhibitor von PI3K mit IC50-Werten von 34 nM, 158 nM, 9 nM, 241 nM und 280 nM fÜr PI3Kα, PI3Kγ, DNA-PK, BRD4 bzw. mTOR.
-
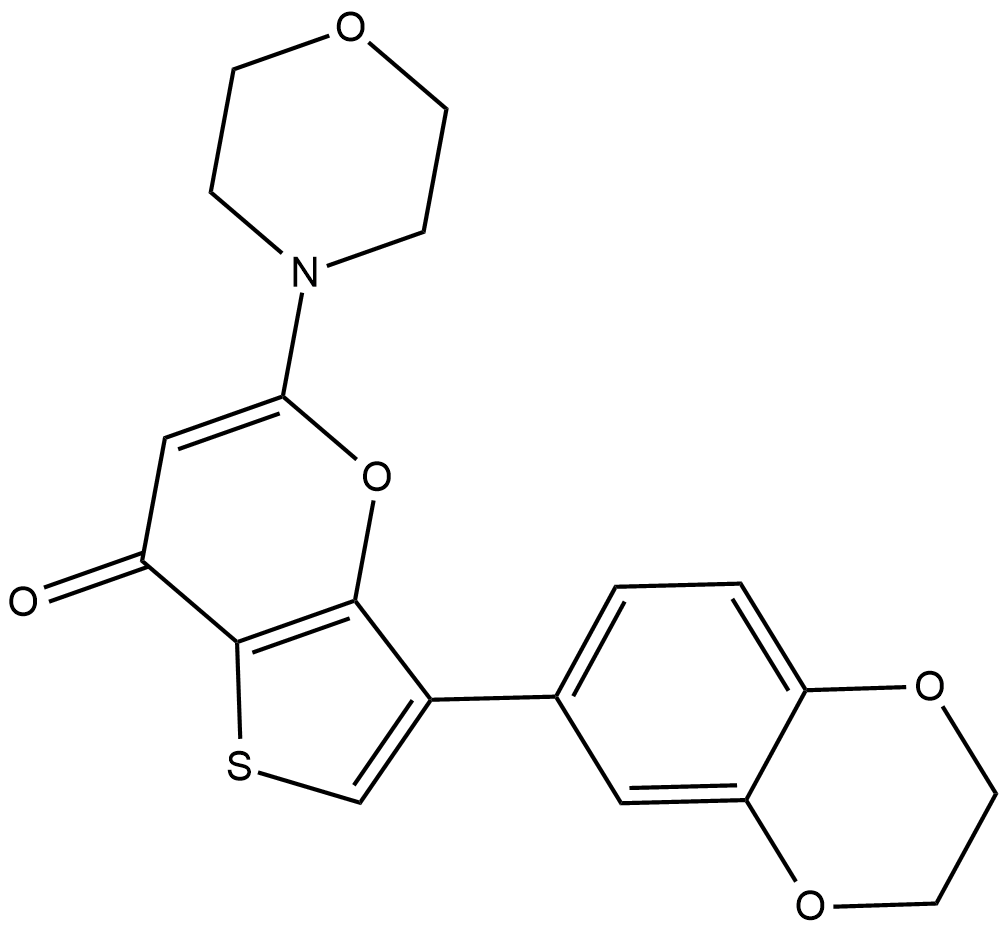
GC10174
SGC-CBP30
A potent inhibitor of CREBBP/EP300 bromodomains
-
GC34778
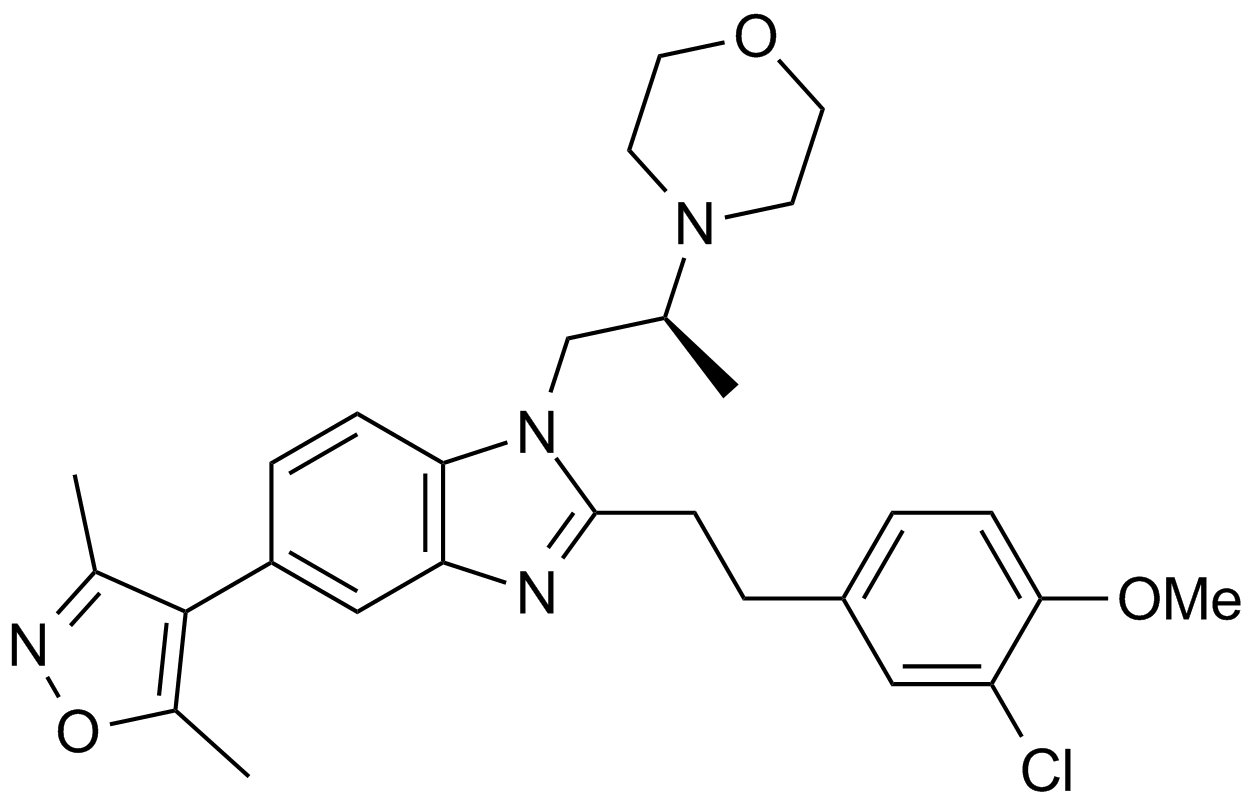
SGC-iMLLT
SGC-iMLLT ist eine erstklassige chemische Sonde und ein potenter, selektiver Inhibitor von MLLT1/3-Histon-Wechselwirkungen mit einem IC50 von 0,26 μM. SGC-iMLLT zeigt eine hohe BindungsaktivitÄt gegenÜber der MLLT1-YEATS-DomÄne (YD) und MLLT3-YD (AF9/YEATS3) mit Kds von 0,129 bzw. 0,077 μM.
-
GC63887
SGC-SMARCA-BRDVIII
SGC-SMARCA-BRDVIII ist ein potenter und selektiver Inhibitor von SMARCA2/4 und PB1(5) mit Kds von 35 nM, 36 nM bzw. 13 nM.
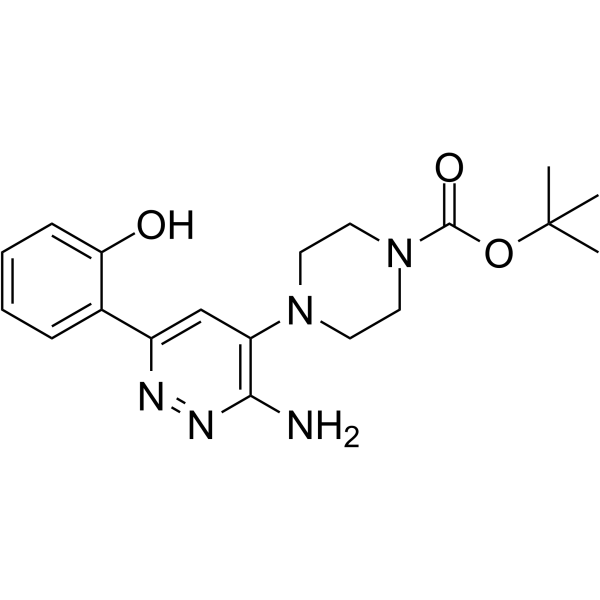
-
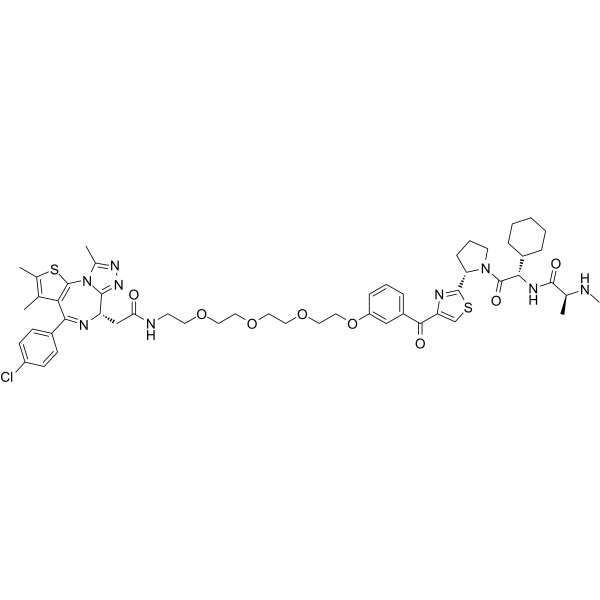
GC65574
SNIPER(BRD)-1
SNIPER(BRD)-1 besteht aus einem LCL-161-Derivat des IAP-Antagonisten und einem BET-Inhibitor (+)-JQ-1, die durch einen Linker verbunden sind. SNIPER(BRD)-1 induziert den Abbau von BRD4 Über den Ubiquitin-Proteasom-Weg. SNIPER(BRD)-1 baut auch cIAP1, cIAP2 und XIAP mit IC50-Werten von 6,8 nM, 17 nM bzw. 49 nM ab.
-
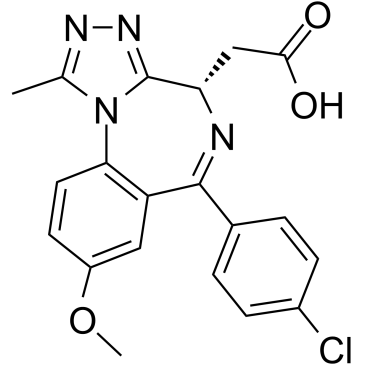
GC32863
Target Protein-binding moiety 4
Die Zielprotein-bindende Einheit 4 (Molibresib-CarbonsÄure) ist ein I-BET762-basierter Gefechtskopf-Ligand fÜr Konjugationsreaktionen von PROTAC-Targeting auf BET. Zielprotein-bindende Einheit 4 (Molibresib-CarbonsÄure) ist ein BRD4-Inhibitor mit einem pIC50 von 5,1.
-
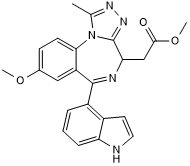
GC50340
TC AC 28
High affinity BET bromodomain ligand
-
GC34319
TD-428
TD-428 ist ein PROTAC, das durch Liganden fÜr Cereblon und BRD4 verbunden ist. TD-428 ist ein hochspezifischer BRD4-Abbaustoff mit einem DC50 von 0,32 nM. TD-428 ist ein BET-PROTAC, das TD-106 (einen CRBN-Liganden) umfasst, der an JQ1 (einen BET-Inhibitor) gebunden ist. TD-428 induziert effizient den BET-Proteinabbau.
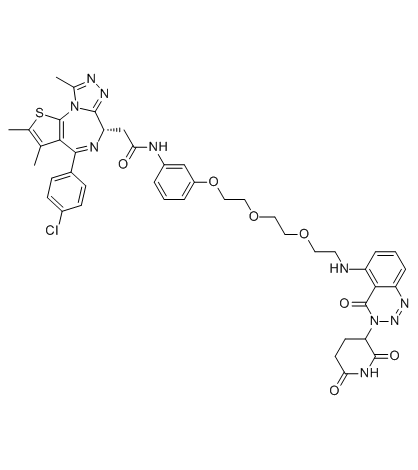
-
GC70037
TMX1
TMX1 ist ein kovalenter Molekülkleber, der BRD4 abbaut. TMX1 rekrutiert selektiv DCAF16 auf BRD4BD2 und führt zum Abbau von BRD4.
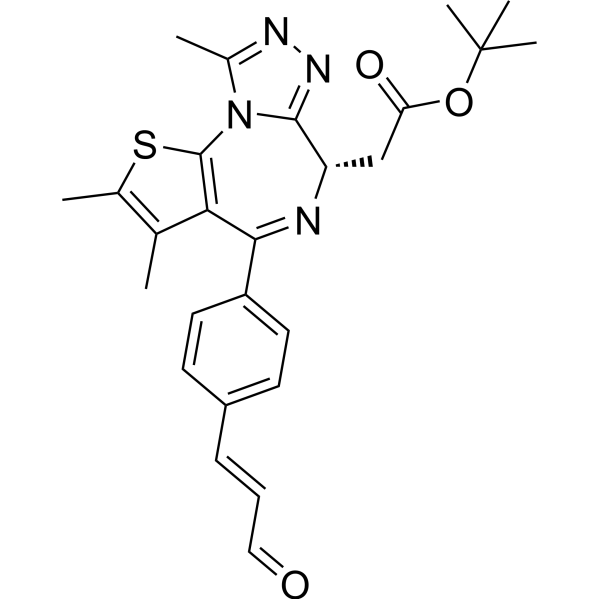
-
GC50542
TP 238
CECR2 and BPTF/FALZ inhibitor
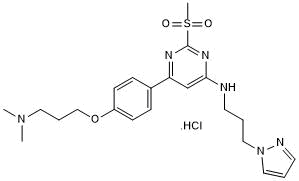
-
GC40628
TP-472
TP-472 ist ein selektiver BRD7/9-Inhibitor mit KDs von 0,34 μM und 33 nM fÜr BRD7 bzw. BRD9.
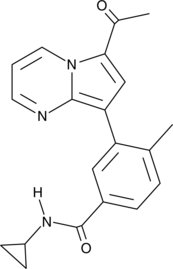
-
GC32880
TPOP146
TPOP146 ist ein selektiver CBP/P300-Benzoxazepin-BromodomÄnen-Inhibitor mit Kd-Werten von 134 nM und 5,02 μM fÜr CBP und BRD4.
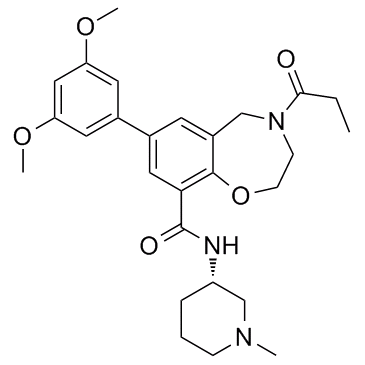
-
GC62405
UMB298
UMB298 ist ein potenter und selektiver CBP/P300-BromodomÄnen-Inhibitor.
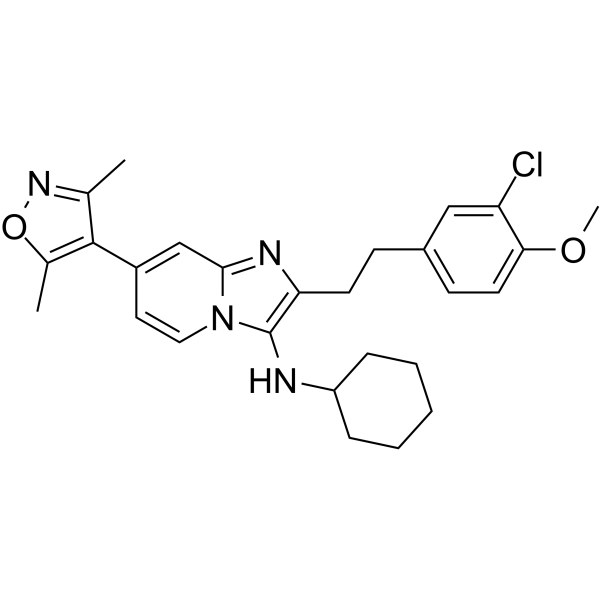
-
GC12419
UNC 926 hydrochloride
An antagonist of L3MBTL1, L3MBTL3, and L3MBTL4
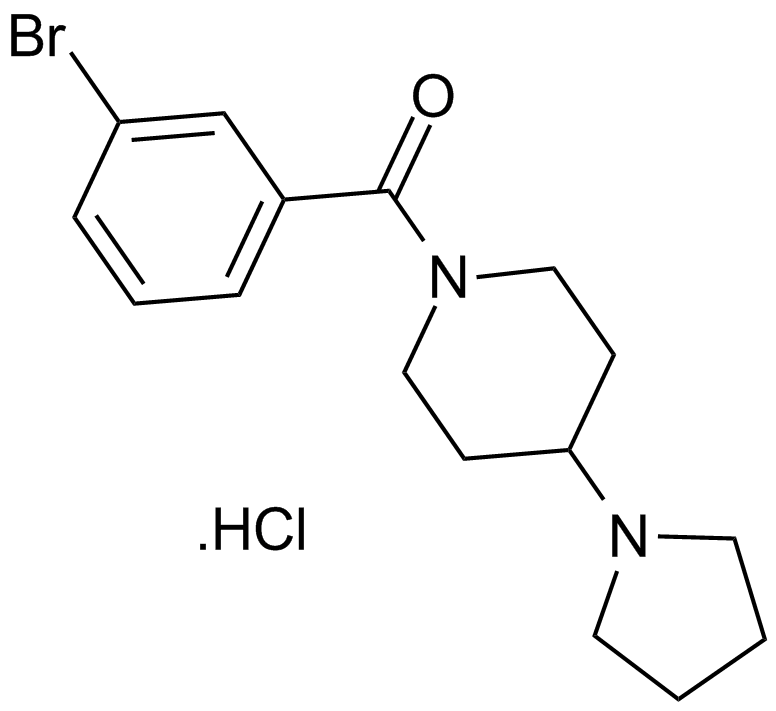
-
GC68025
UNC1021
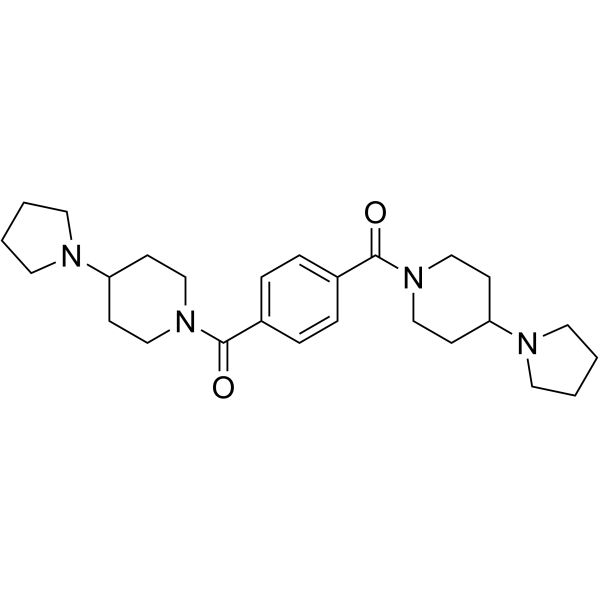
-
GC64040
VTP50469 fumarate
VTP50469-Fumarat ist ein potenter, hochselektiver und oral aktiver Inhibitor der Menin-MLL-Interaktion mit einem Ki von 104 pM. VTP50469-Fumarat hat eine starke Anti-LeukÄmie-AktivitÄt.
-
GC65135
VZ185
VZ185 ist eine potente, schnelle und selektive von Hippel-Lindau-basierte Dual-Degrader-Sonde von BRD9 und BRD7 mit DC50-Werten von 4,5 bzw. 1,8 nM. VZ185 ist in EOL-1- und A-402-Zellen mit EC50-Werten von 3 nM bzw. 40 nM zytotoxisch.
-
GC33285
Y06036
Y06036 ist ein potenter und selektiver BET-Inhibitor, der mit einem Kd-Wert von 82 nM an die BRD4(1)-BromodomÄne bindet. Antitumor-AktivitÄt.

-
GC33264
Y06137
Y06137 ist ein potenter und selektiver BET-Inhibitor zur Behandlung von kastrationsresistentem Prostatakrebs (CRPC). Y06137 bindet an die BRD4(1)-BromodomÄne mit einem Kd von 81 nM.
-
GC64518
Ziftomenib
Ziftomenib (KO-539) ist ein Inhibitor der Menin-MLL-Interaktion mit AntitumoraktivitÄten (WO2017161028A1, Verbindung 151).
-
GC19455
ZL0420
ZL0420 ist ein potenter und selektiver BromodomÄnen-haltiger Protein 4 (BRD4)-Inhibitor mit IC50-Werten von 27 nM gegen BRD4 BD1 und 32 nM gegen BRD4 BD2.

-
GC65204
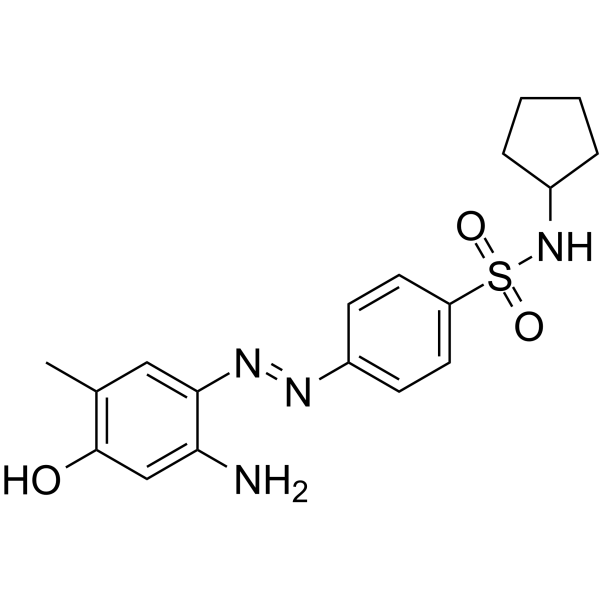
ZL0454
ZL0454 ist ein potenter und selektiver Inhibitor des BromodomÄnen-enthaltenden Proteins 4 (BRD4) mit einem IC50-Wert von 49 und 32 nM fÜr BD1 und BD2.
-
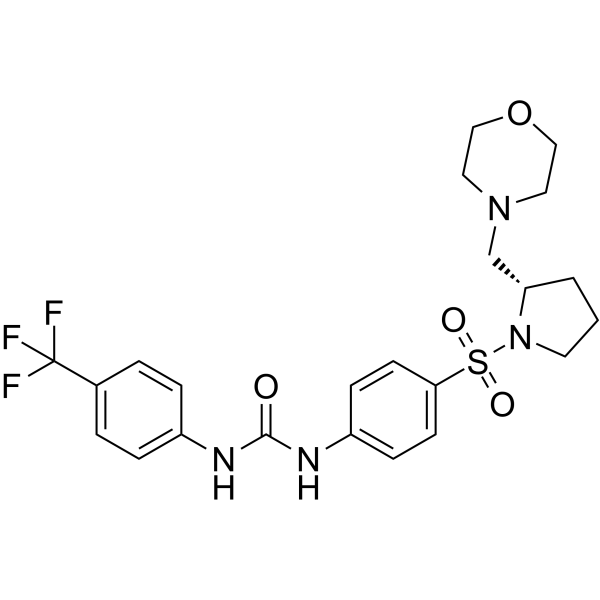
GC64529
ZL0590
ZL0590 ist ein potenter, oral aktiver BRD4 BD1-selektiver Inhibitor mit einem IC50-Wert von 90 nM fÜr humanes BRD4 BD1.
-
GC65269
ZXH-3-26
ZXH-3-26 ist ein PROTAC, verbunden durch Liganden fÜr Cereblon und BRD4 mit einem DC50/5h von 5 nM. Der DC50/5h bezieht sich auf den halbmaximalen Abbau nach 5 Stunden Behandlung von ~ 5 nM.