PARP
Poly (ADP-ribose) polymerases (PARPs) is a large family of proteins with a conserved catalytic domain that catalyze an immediate DNA-damage-dependent post-translational modification of histones and other nuclear proteins leading to the survival of injured proliferating cells. So far, a total number of 18 human PARP proteins encoded by different genes have been identified, including PARP-1 to PARP-4, PARP-5a, PARP-5b, PARP-5c and PARP-6 to PARP-16. The general structural of PARP proteins has been revealed through the extensive study of the founding family member PARP-1, which is characterized by the presence of four functional domains, including a DNA-binding domain, a caspase-cleaved domain, an automodification domain and a catalytic domain.
Products for PARP
- Cat.No. Nombre del producto Información
-
GC40468
1,5-Isoquinolinediol
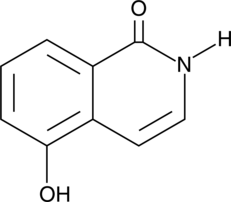
El 1,5-isoquinolinediol es un potente inhibidor de PARP, con una IC50 de 0,18-0,37 μM.
-
GC62781
2-Methylquinazolin-4-ol
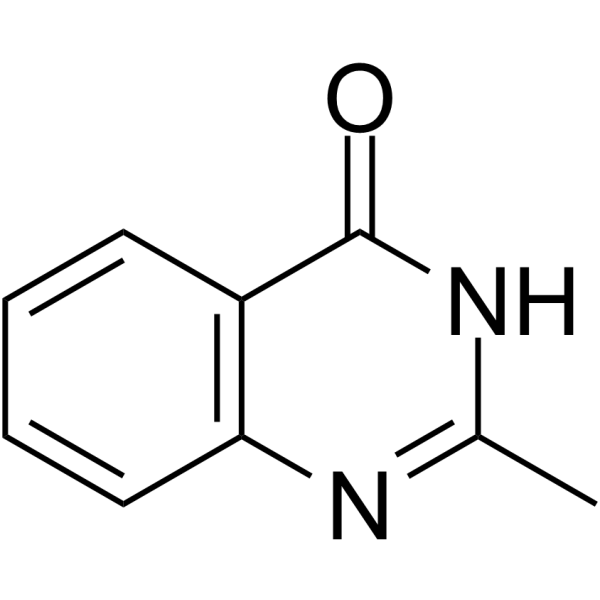
El 2-metilquinazolin-4-ol es un potente inhibidor competitivo de la poli(ADP-ribosa) sintetasa, con una Ki de 1,1 μM.
-
GC62805
4’-Methoxychalcone
4’-Methoxychalcone regula la diferenciación de adipocitos a través de la activación de PPARγ.

-
GC11761
4-amino-1,8-Naphthalimide
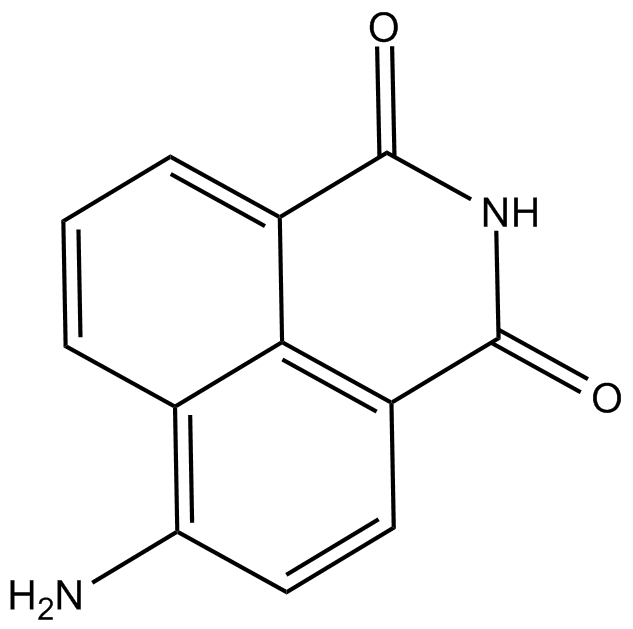
La 4-amino-1,8-naftalimida es un potente inhibidor de PARP y potencia la citotoxicidad de la γ-radiaciÓn en células cancerosas.
-
GC35150
5,7,4'-Trimethoxyflavone
La 5,7,4'-trimetoxiflavona se aísla de Kaempferia parviflora (KP), una famosa planta medicinal de Tailandia. La 5,7,4'-trimetoxiflavona induce la apoptosis, como lo demuestran los incrementos de la fase sub-G1, la fragmentación del ADN, la tinción de anexina-V/PI, la relación Bax/Bcl-xL, la activación proteolítica de la caspasa-3 y la degradación de poli (ADP-ribosa) polimerasa (PARP) proteína. La 5,7,4'-trimetoxiflavona es significativamente eficaz para inhibir la proliferación de células de cáncer gástrico humano SNU-16 de una manera dependiente de la concentración.
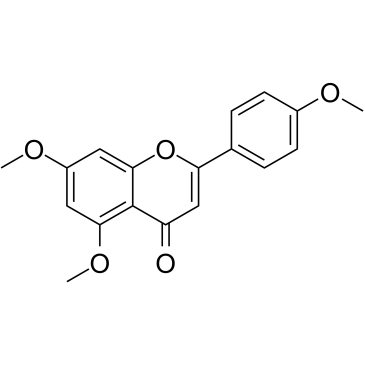
-
GN10629
5,7-dihydroxychromone
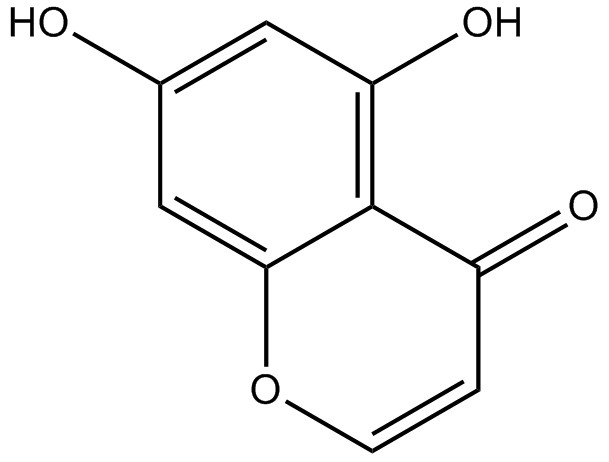
-
GC68161
5-AIQ
-
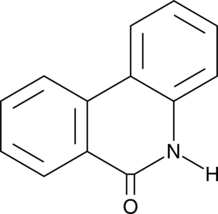
GC45772
6(5H)-Phenanthridinone
An inhibitor of PARP1 and 2
-
GC12390
A-966492
A PARP1 and PARP2 inhibitor
-
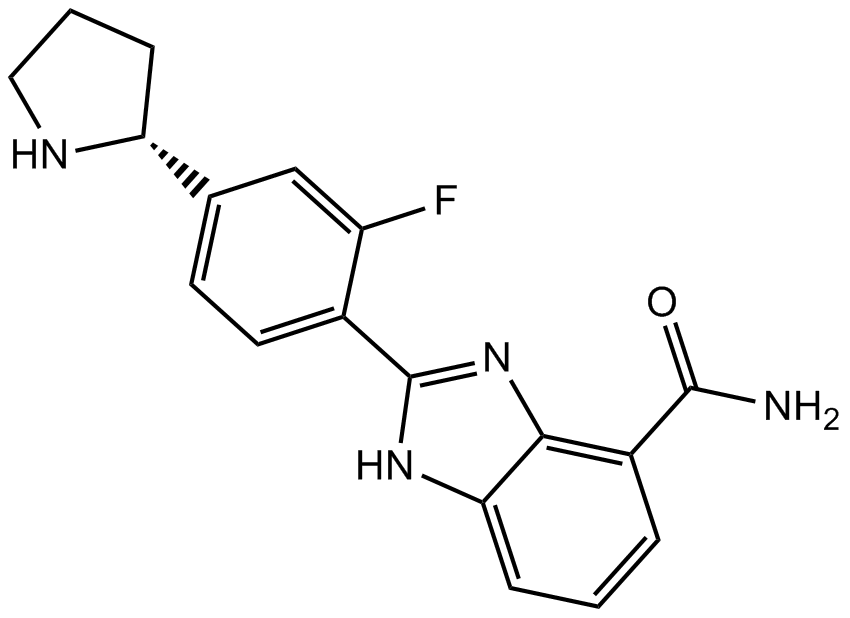
GC12422
ABT-888 (Veliparib)
-
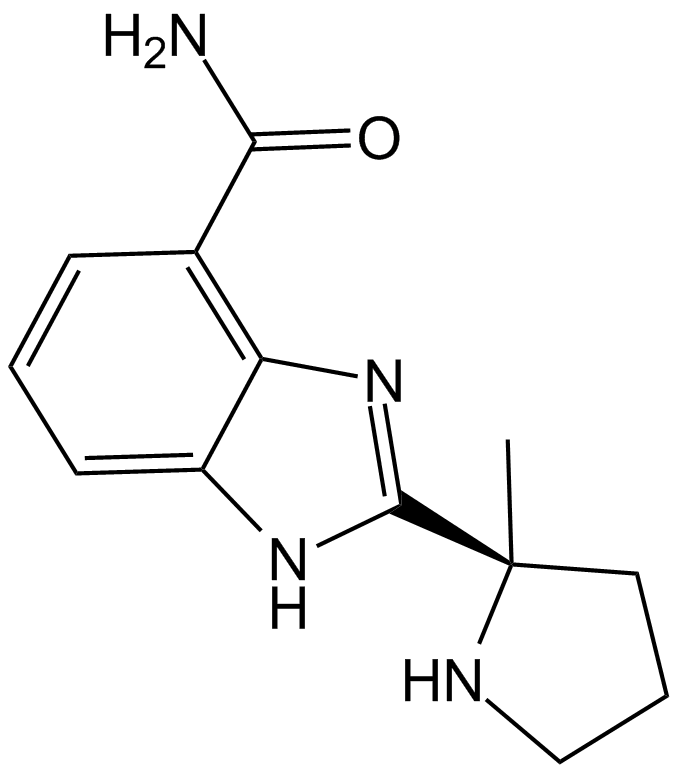
GC16318
AG-14361
A PARP1 inhibitor
-
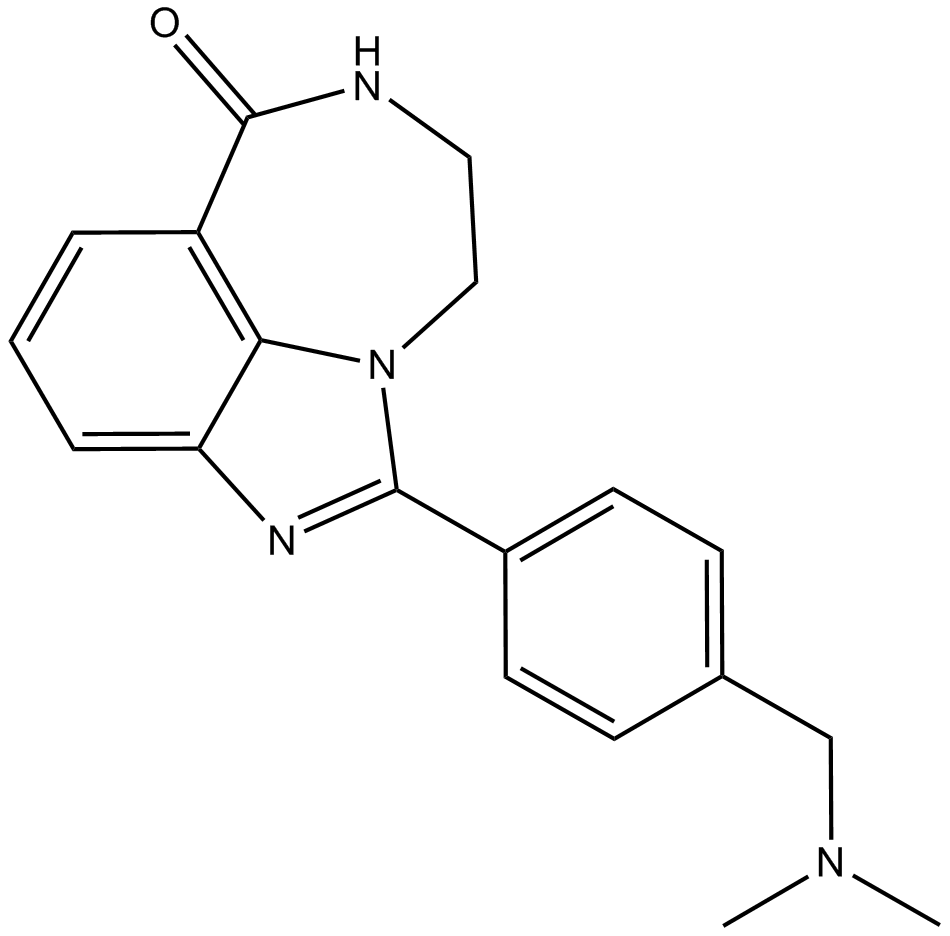
GC65899
AZ3391
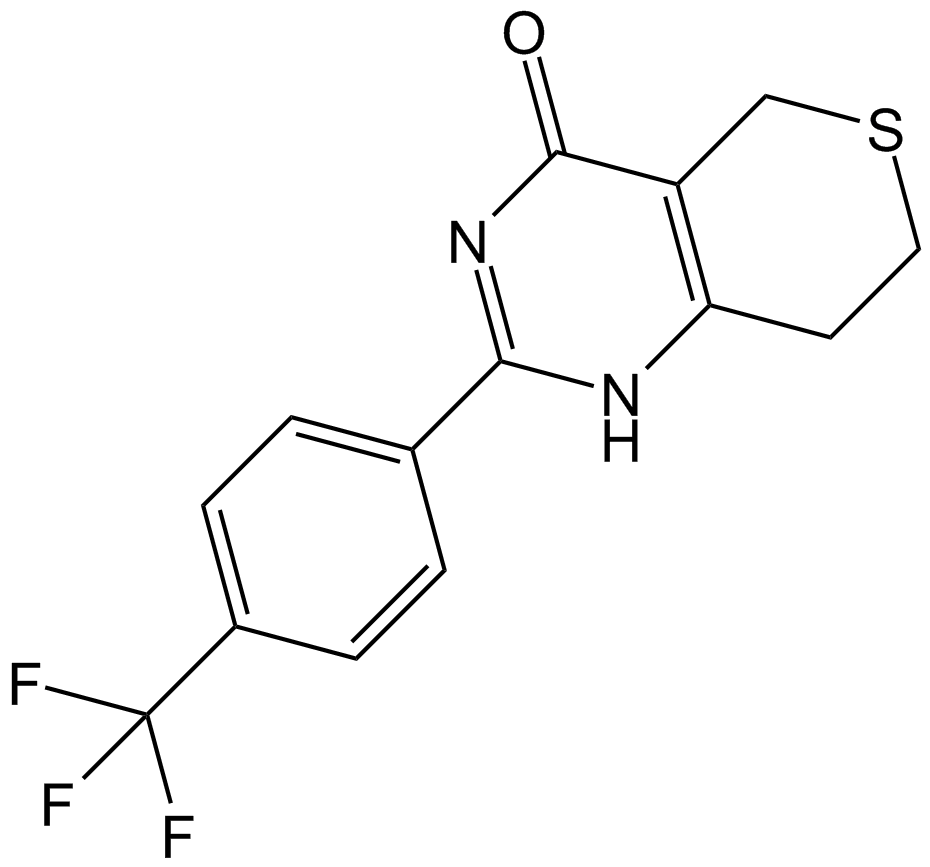
AZ3391 es un potente inhibidor de PARP. AZ3391 es un derivado de la quinoxalina. La familia de enzimas PARP desempeÑa un papel importante en una serie de procesos celulares, como la replicaciÓn, la recombinaciÓn, la remodelaciÓn de la cromatina y la reparaciÓn del daÑo del ADN. AZ3391 tiene el potencial para la investigaciÓn de enfermedades y condiciones que ocurren en los tejidos del sistema nervioso central, como el cerebro y la médula espinal (extraÍdo de la patente WO2021260092A1, compuesto 23).
-
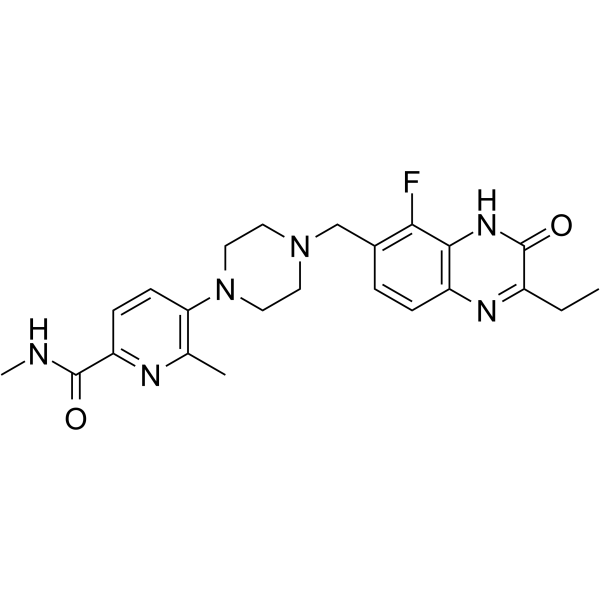
GC16725
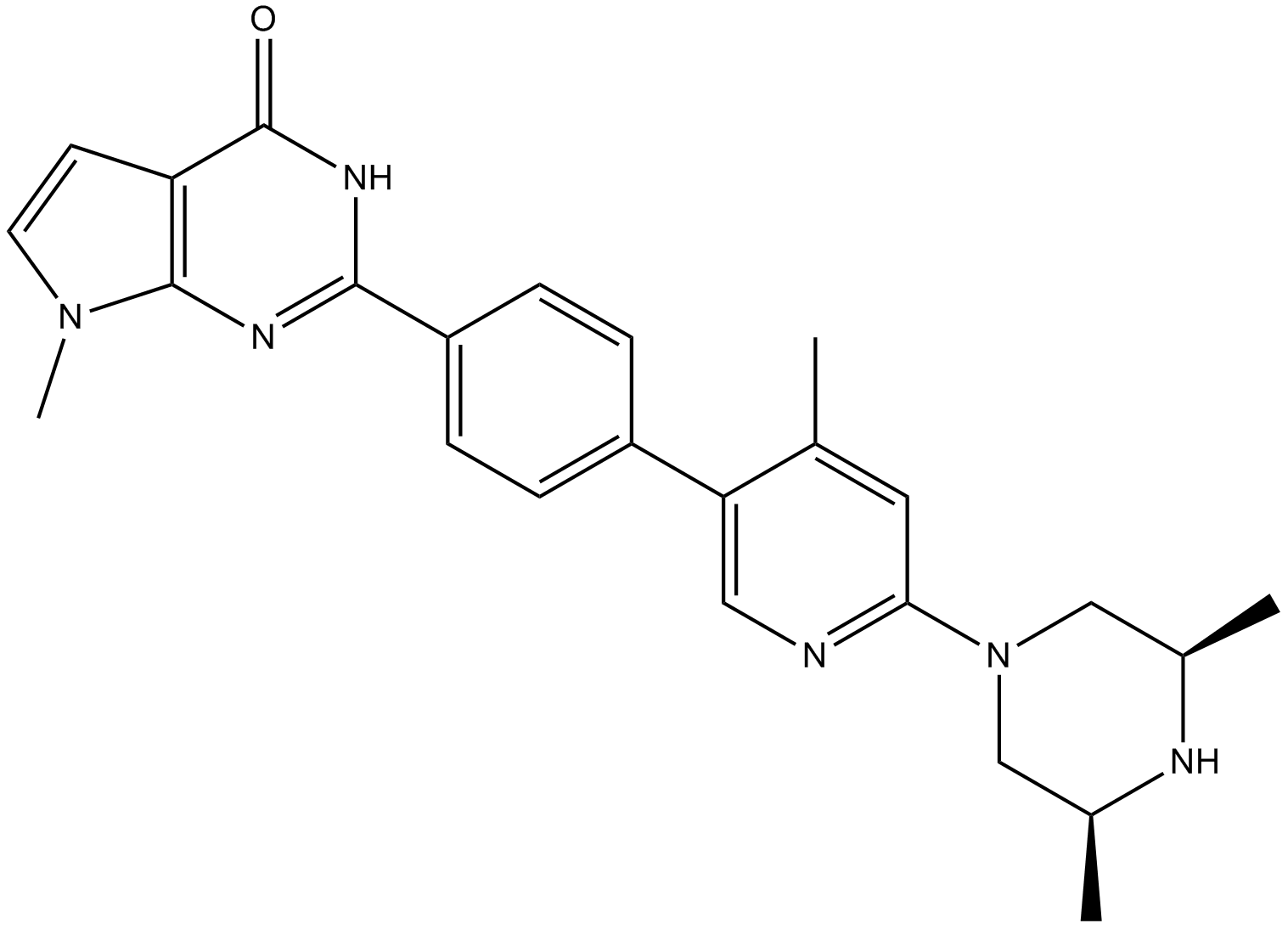
AZ6102
AZ6102 es un potente inhibidor doble de TNKS1 y TNKS2, con IC50 de 3 nM y 1 nM, respectivamente, y tiene una selectividad 100 veces mayor frente a otras enzimas de la familia PARP, con IC50 de 2,0 μM, 0,5 μM y >3 μM, para PARP1 , PARP2 y PARP6, respectivamente.
-
GC46900
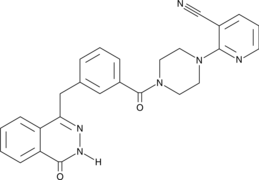
AZ9482
AZ9482 es un triple inhibidor de PARP1/2/6, con valores IC50 de 1 nM, 1 nM y 640 nM para PARP1, PARP2 y PARP6, respectivamente.
-
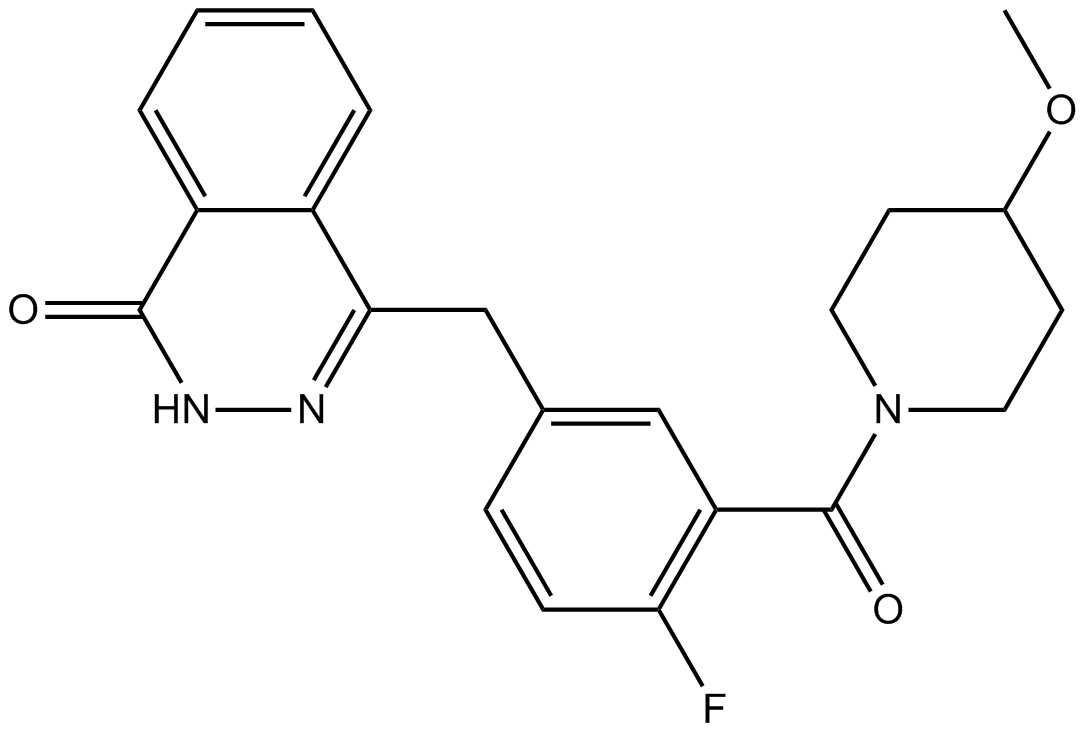
GC17965
AZD2461
A PARP inhibitor
-
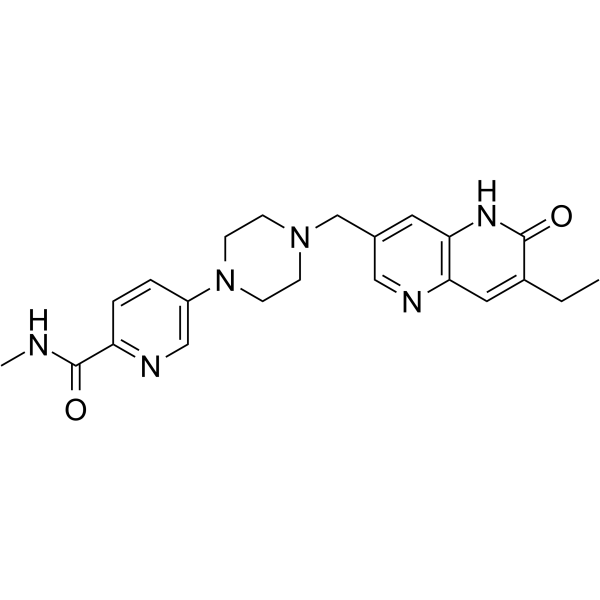
GC62310
AZD5305
AZD5305 es un inhibidor de PARP potente, selectivo y activo por vÍa oral. AZD5305 es potente y eficaz en xenoinjertos animales y modelos PDX.
-
GC12844
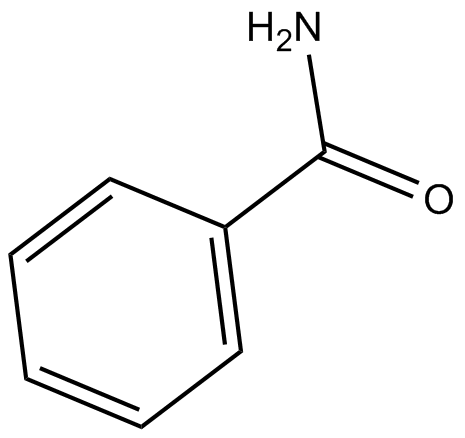
Benzamide
La benzamida (bencenocarboxamida) es un potente inhibidor de la poli(ADP-ribosa) polimerasa (PARP).
-
GC14380
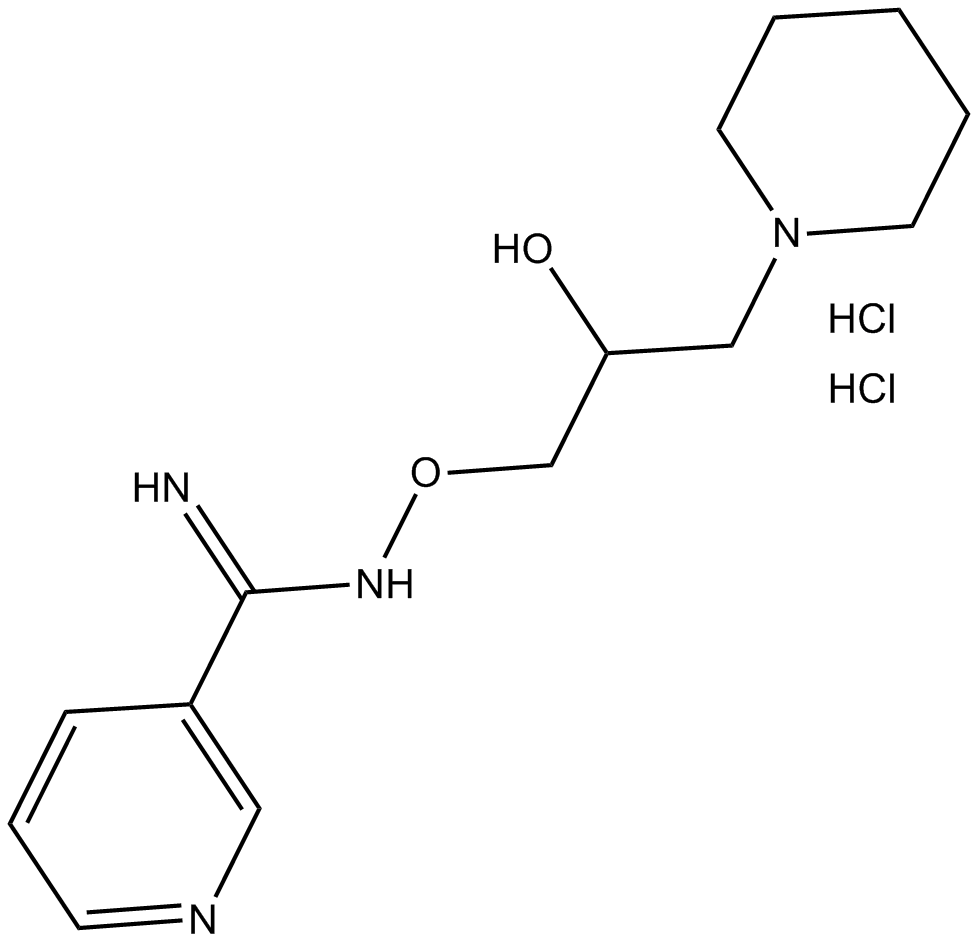
BGP-15
BGP-15 es un inhibidor de PARP, con un IC50 y un Ki de 120 y 57 μM, respectivamente.
-
GC15932
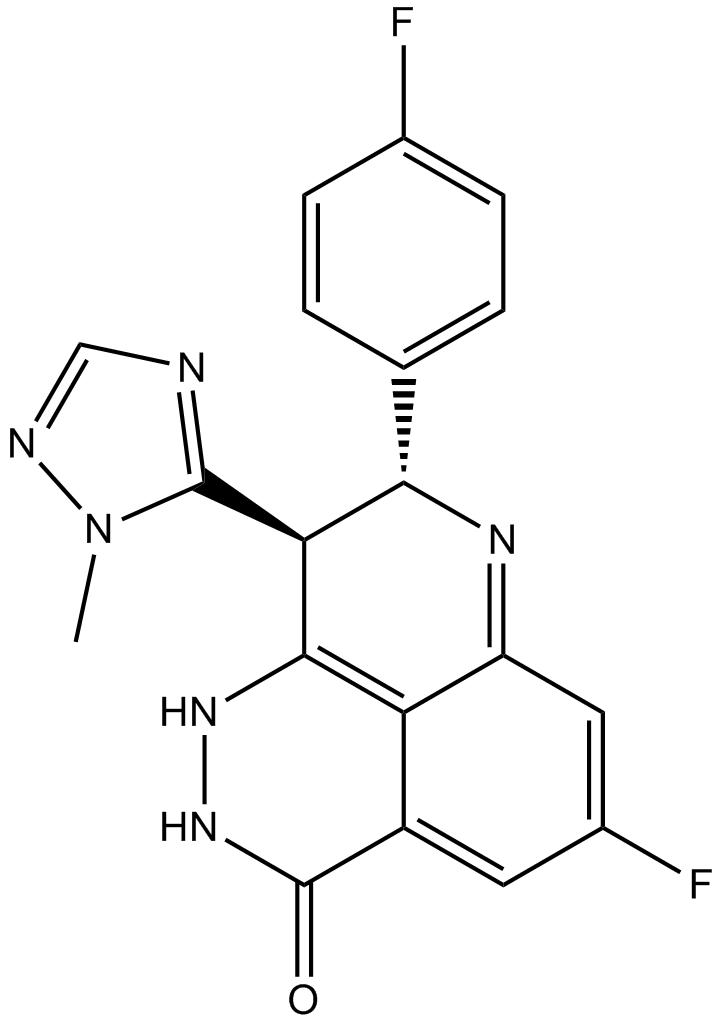
BMN 673
A PARP inhibitor
-
GC10920
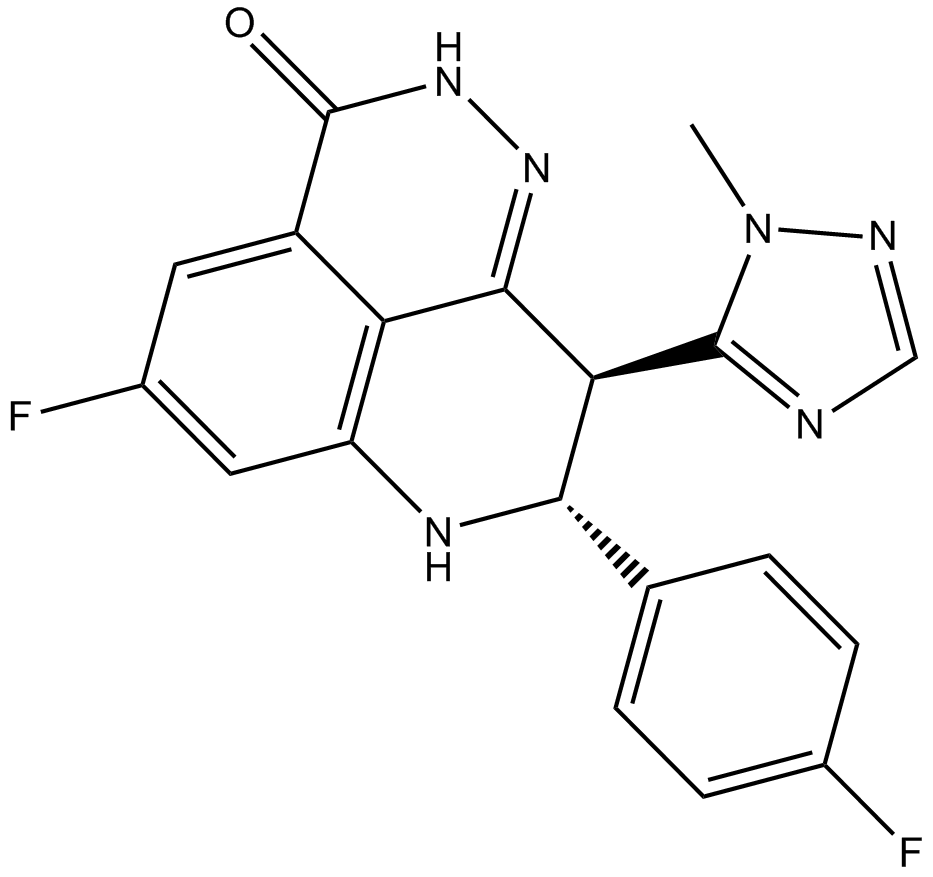
BMN-673 8R,9S
-
GC35547
BR102375
BR102375 es un agonista completo del receptor γ (PPAR γ) activado por el proliferador de peroxisomas sin TZD para el tratamiento de la diabetes tipo 2, revela un valor EC50 de 0,28 μM y una relaciÓn Amax del 98 %.
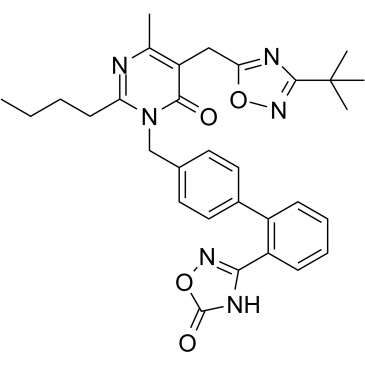
-
GC33223
BRCA1-IN-1
BRCA1-IN-1 es un nuevo inhibidor de BRCA1 similar a una molécula pequeÑa con IC50 y Ki de 0,53 μM y 0,71 μM, respectivamente.
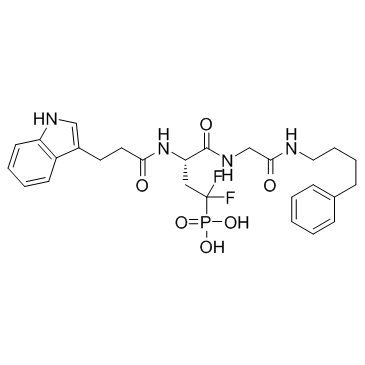
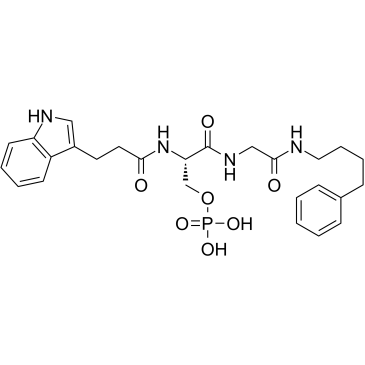
-
GC35550
BRCA1-IN-2
BRCA1-IN-2 (compuesto 15) es un inhibidor de la interacciÓn proteÍna-proteÍna (PPI) permeable a las células para BRCA1 con un IC50 de 0,31 μM y una Kd de 0,3 μM, que muestra actividades antitumorales a través de la interrupciÓn de BRCA1 (BRCT)2 /interacciones proteÍna.
-
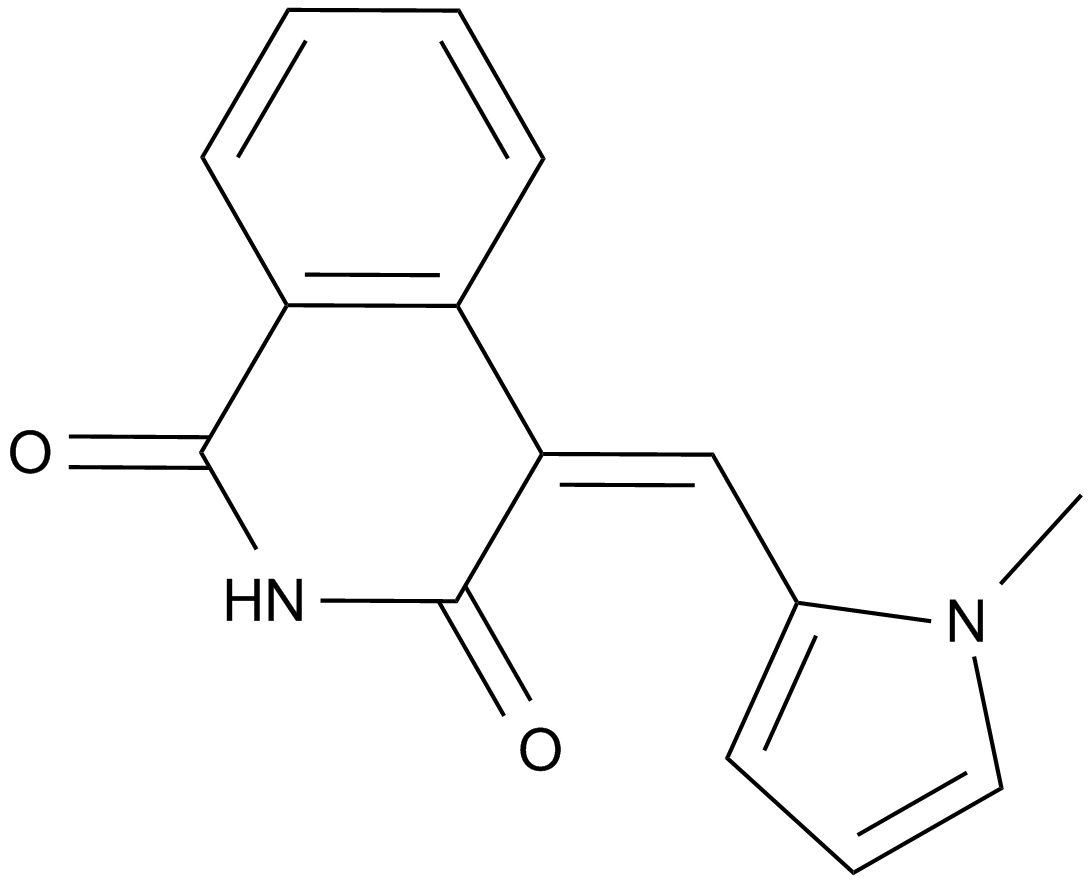
GC10690
BYK 204165
A selective inhibitor of PARP1
-
GC14434
BYK 49187
Potent PARP-1/PARP-2 inhibitor
-
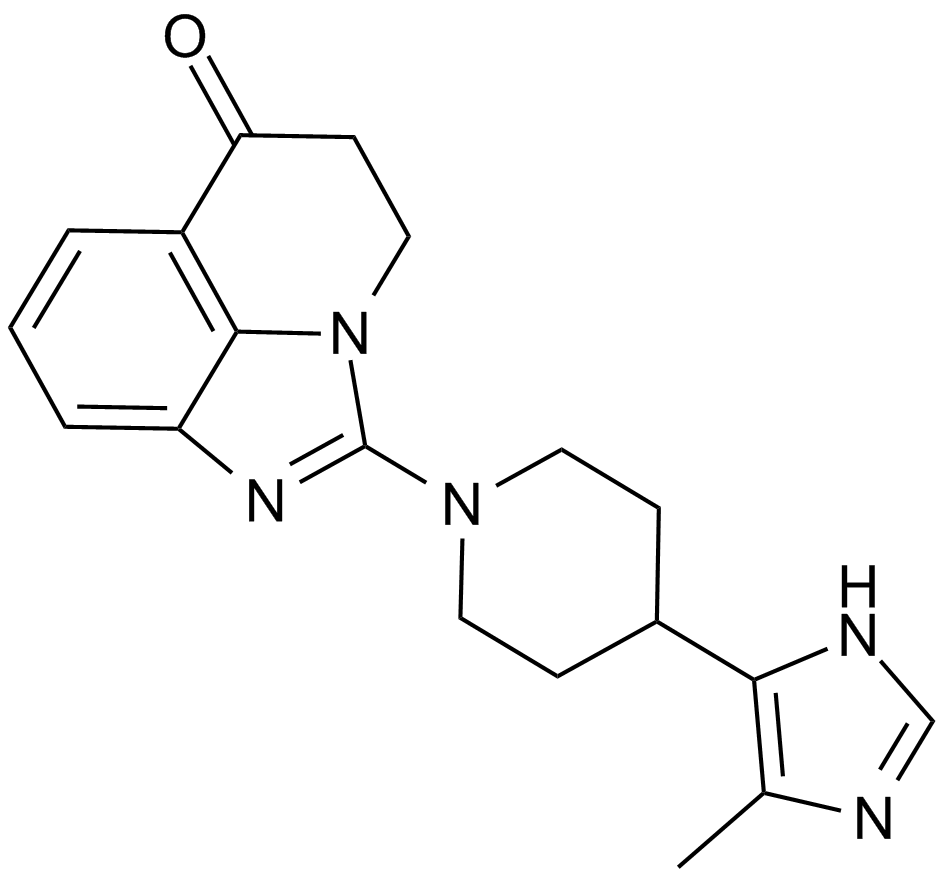
GC47055
CAY10749
CAY10749 (Compuesto 15) es un potente inhibidor PARP/PI3K con valores PIC50 de 8.22, 8.44, 8.25, 6.54, 8.13, 6.08 para PARP-1, PARP-2, PI3K⊵ ;, PI3K⋲ offlineefficient_models_2022q2.md.en_es_2021q4.mdCAY10749 is a highly effective anticancer compound targeted against a wide range of oncologic diseases.en_es_2021q4.md
-
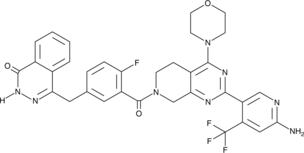
GC47056
CAY10753
A TNKS2 inhibitor
-
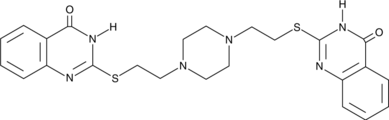
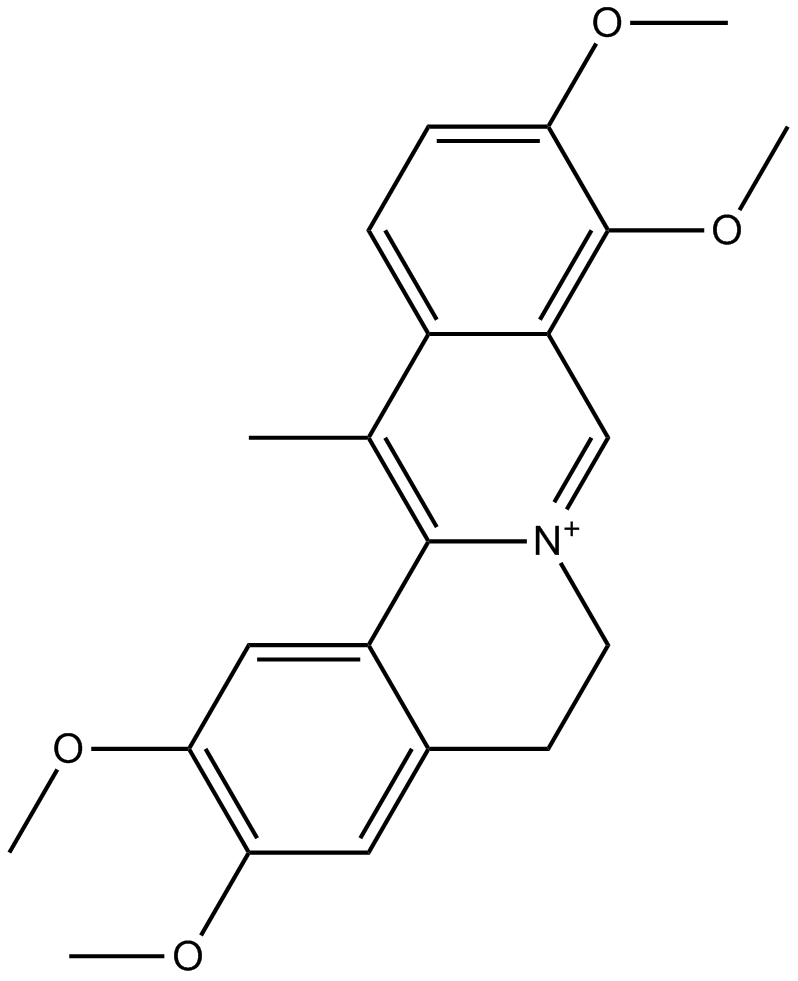
GN10040
Dehydrocorydaline
-
GC12680
DR 2313
A PARP inhibitor
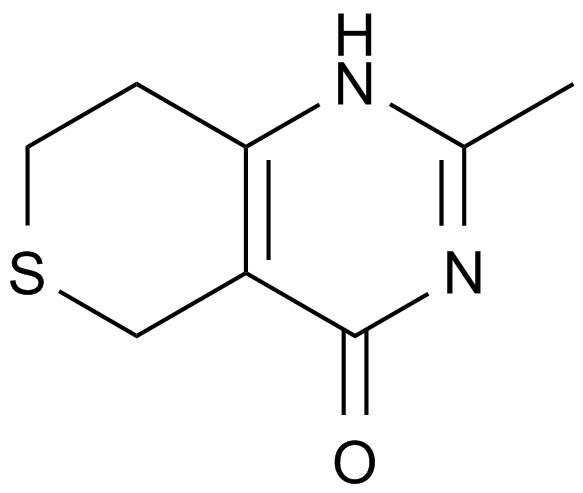
-
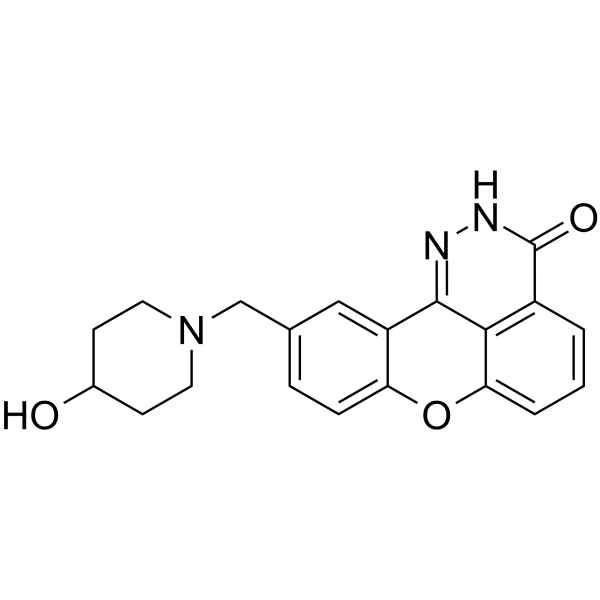
GC64209
E7016
E7016 (GPI 21016) es un inhibidor de PARP disponible por vÍa oral. E7016 puede mejorar la radiosensibilidad de las células tumorales in vitro e in vivo a través de la inhibiciÓn de la reparaciÓn del ADN. E7016 actÚa como un potencial agente anticancerÍgeno.
-
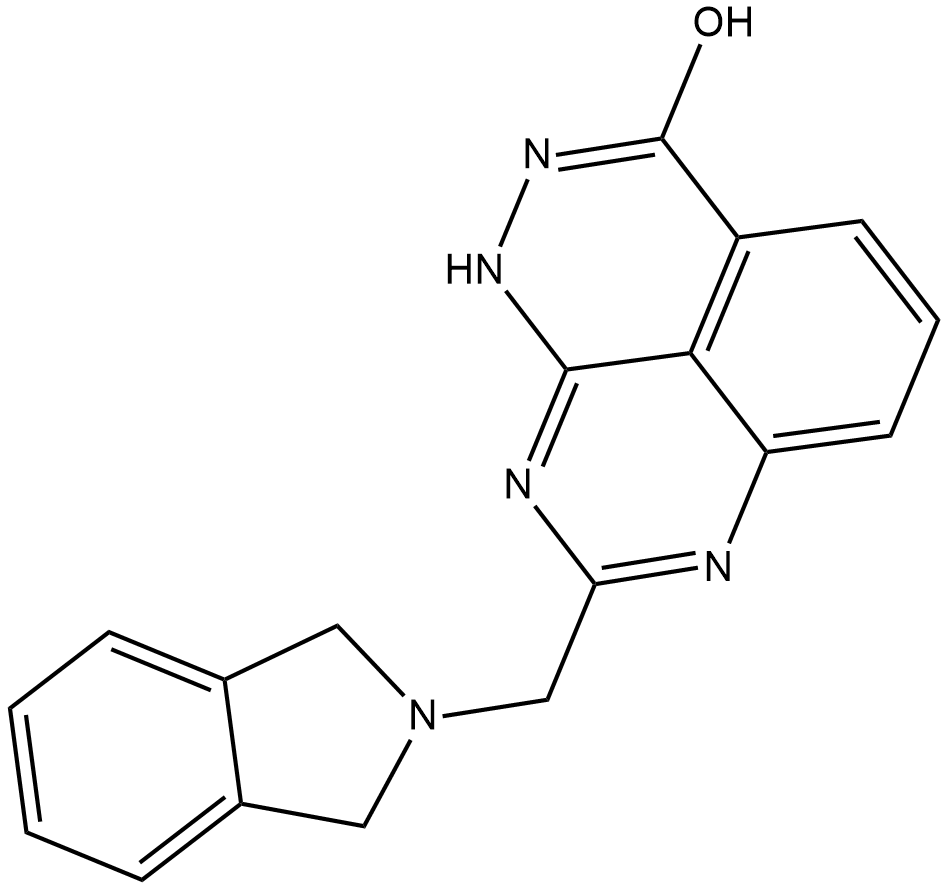
GC18172
E7449
E7449 es un potente inhibidor de PARP1 y PARP2 y también inhibe TNKS1 y TNKS2, con IC50 de 2,0, 1,0, ~50 y ~50 nM para PARP1, PARP2, TNKS1 y TNKS2, respectivamente, usando 32P-NAD+ como sustrato.
-
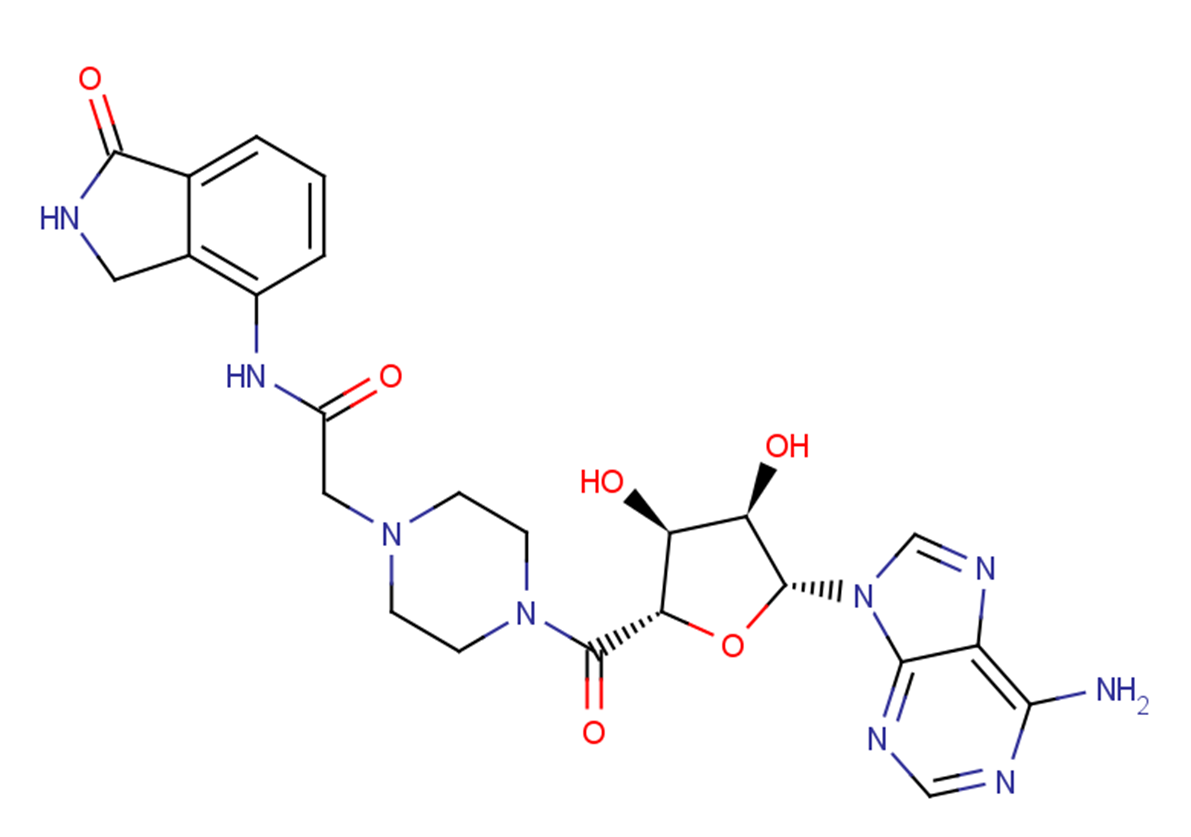
GC12991
EB 47
A PARP1 and TNKS2 inhibitor
-
GC50506
Fluorescein-NAD+
La fluoresceÍna-NAD+ es una alternativa a la NAD radiomarcada y un sustrato para la ribosilaciÓn de ADP.
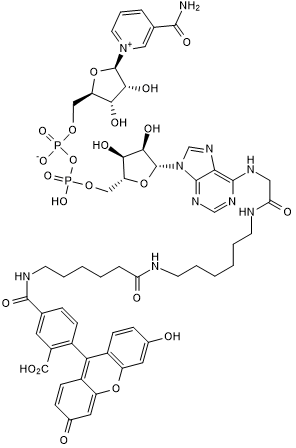
-
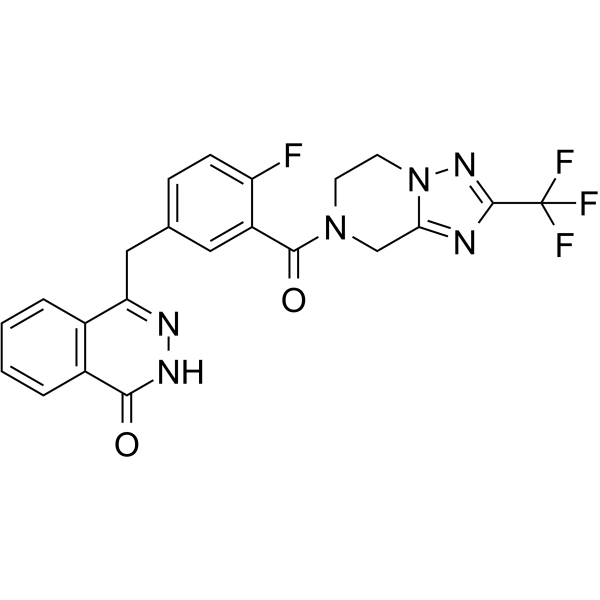
GC62121
Fluzoparib
Fluzoparib (SHR3162) es un inhibidor de PARP1 potente y activo por vÍa oral (IC50 = 1,46 ± 0,72 nM, un ensayo enzimÁtico libre de células) con una actividad antitumoral superior. Fluzoparib inhibe selectivamente la proliferaciÓn de células deficientes en reparaciÓn de recombinaciÓn homÓloga (HR) y sensibiliza tanto a las células con deficiencia de HR como a las células competentes con HR a los agentes citotÓxicos. Fluzoparib exhibe buenas propiedades farmacocinéticas in vivo y puede usarse para la investigaciÓn del cÁncer de ovario recidivante con mutaciÓn BRCA1/2.
-
GC10456
Fucosterol
El fucosterol es un esterol aislado de algas, algas o diatomeas.
-
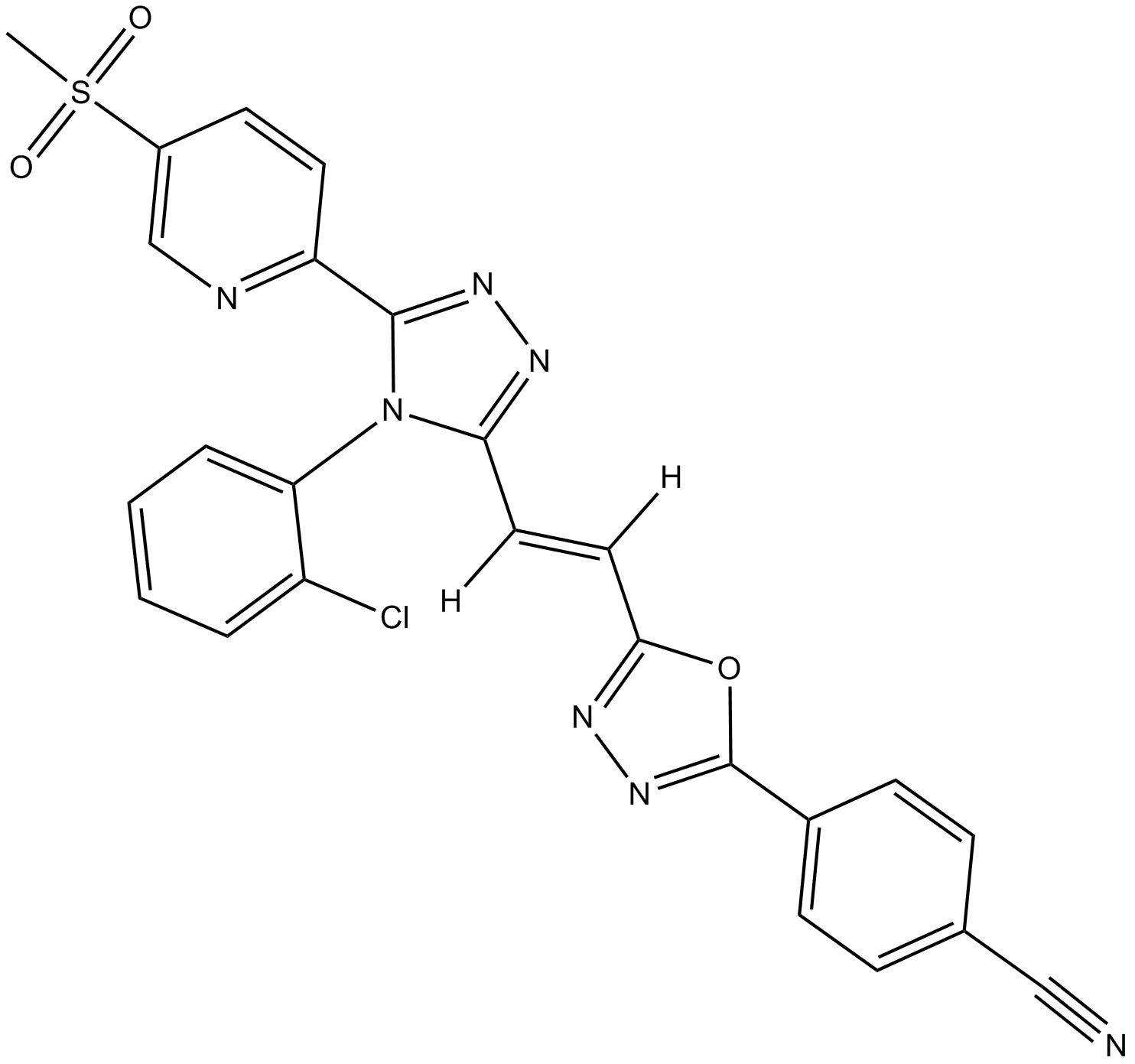
GC13541
G007-LK
G007-LK es un inhibidor potente y selectivo de TNKS1 y TNKS2, con IC50 de 46 nM y 25 nM, respectivamente.
-
GC19542
GeA-69
GeA-69 es un inhibidor alostérico selectivo de la poliadenosina-difosfato-ribosa polimerasa 14 (PARP14) que se dirige al macrodominio 2 (MD2), con un valor de Kd de 2,1 µM. GeA-69 participa en los mecanismos de reparación de daños en el ADN y evita el reclutamiento de PARP14 MD2 en los sitios de daños en el ADN inducidos por láser.

-
GC15353
Iniparib (BSI-201)
A PARP1 inhibitor
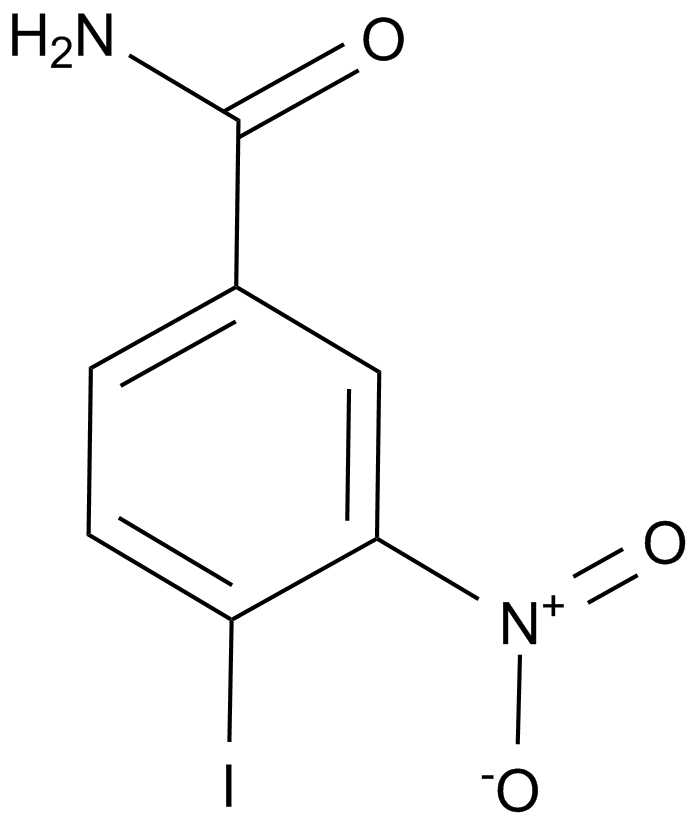
-
GC12496
INO-1001
A PARP inhibitor
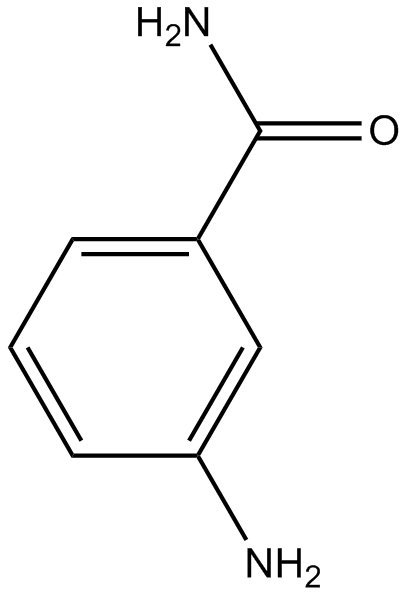
-
GC38385
INO-1001
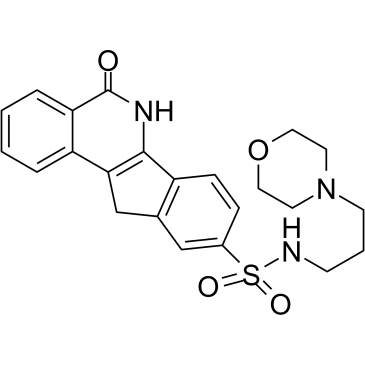
-
GC34195
K-756
K-756 es un inhibidor de tankirasa directo y selectivo (TNKS), que inhibe la actividad de ribosilaciÓn de ADP de TNKS1 y TNKS2 con IC50 de 31 y 36 nM, respectivamente.
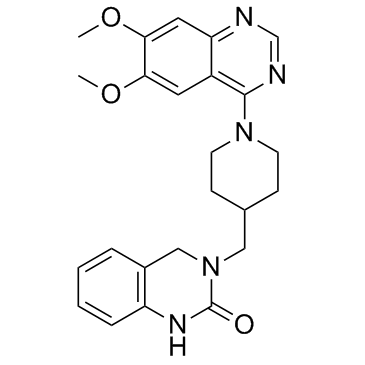
-
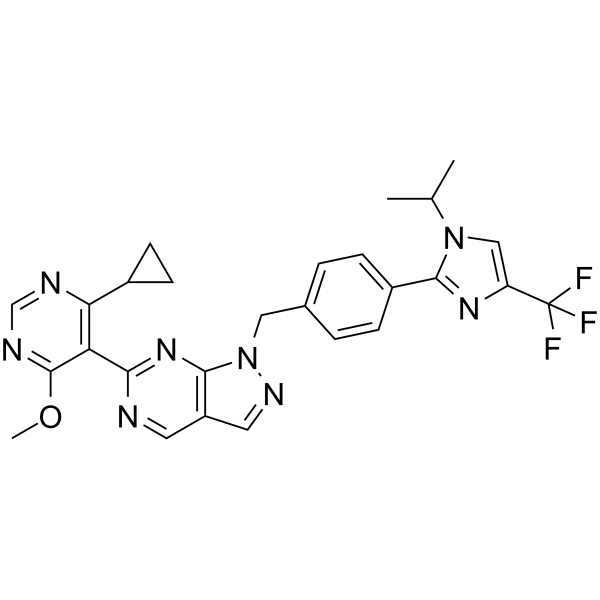
GC65907
KSQ-4279
KSQ-4279 (USP1-IN-1, FÓrmula I) es un inhibidor de USP1 y PARP (extraÍdo de la patente WO2021163530).
-
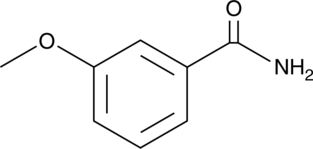
GC47693
m-Methoxybenzamide
La m-metoxibenzamida (3-MBA), un inhibidor de la ADP-ribosiltransferasa (ADPRT) y PARP, inhibe la divisiÓn celular en Bacillus subtilis, lo que lleva a la filamentaciÓn y, finalmente, a la lisis de las células. La m-metoxibenzamida (3-MBA) mejora el crecimiento de plantas in vitro, la microtuberizaciÓn y la eficiencia de transformaciÓn de la papa azul (Solanum tuberosum L. subsp. andigenum).
-
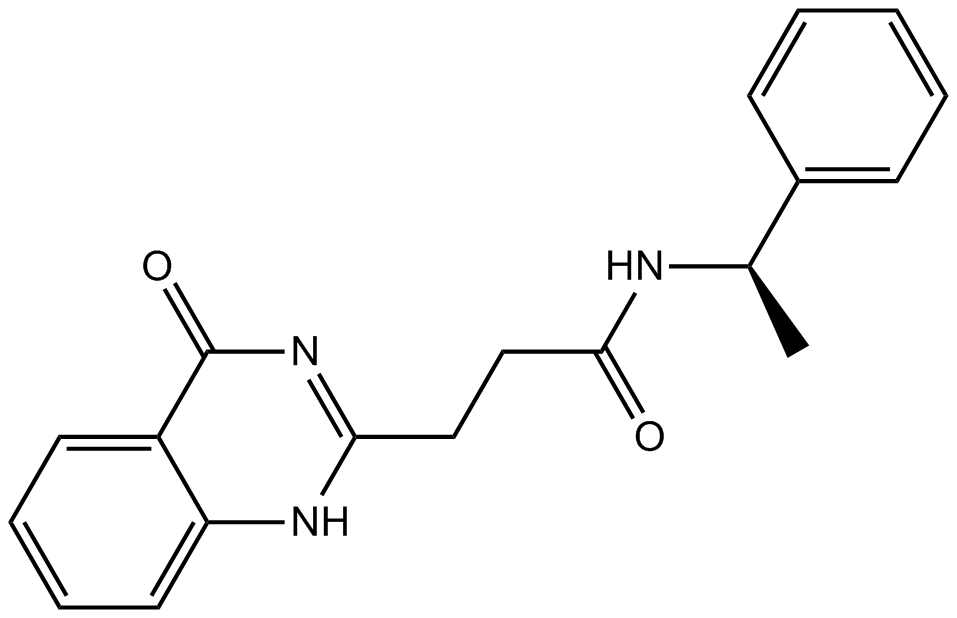
GC13419
ME0328
ME0328 es un inhibidor potente y selectivo de ARTD3/PARP3 con una IC50 de 0,89±0,28 μM.
-
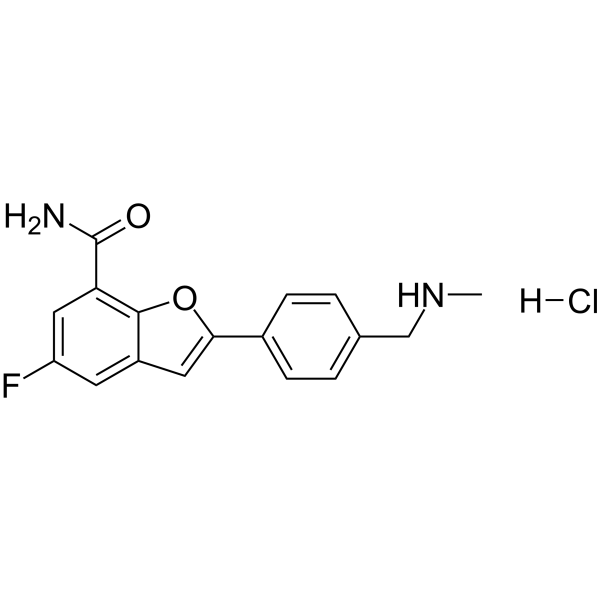
GC62252
Mefuparib hydrochloride
El clorhidrato de mefuparib (MPH) es un inhibidor PARP1/2 selectivo, competitivo con sustrato y activo por vÍa oral con IC50 de 3,2 nM y 1,9 nM, respectivamente. El clorhidrato de mefuparib induce la apoptosis y posee una importante actividad anticancerÍgena in vitro e in vivo.
-
GC17802
MK-4827
An orally bioavailable PARP1/2 inhibitor
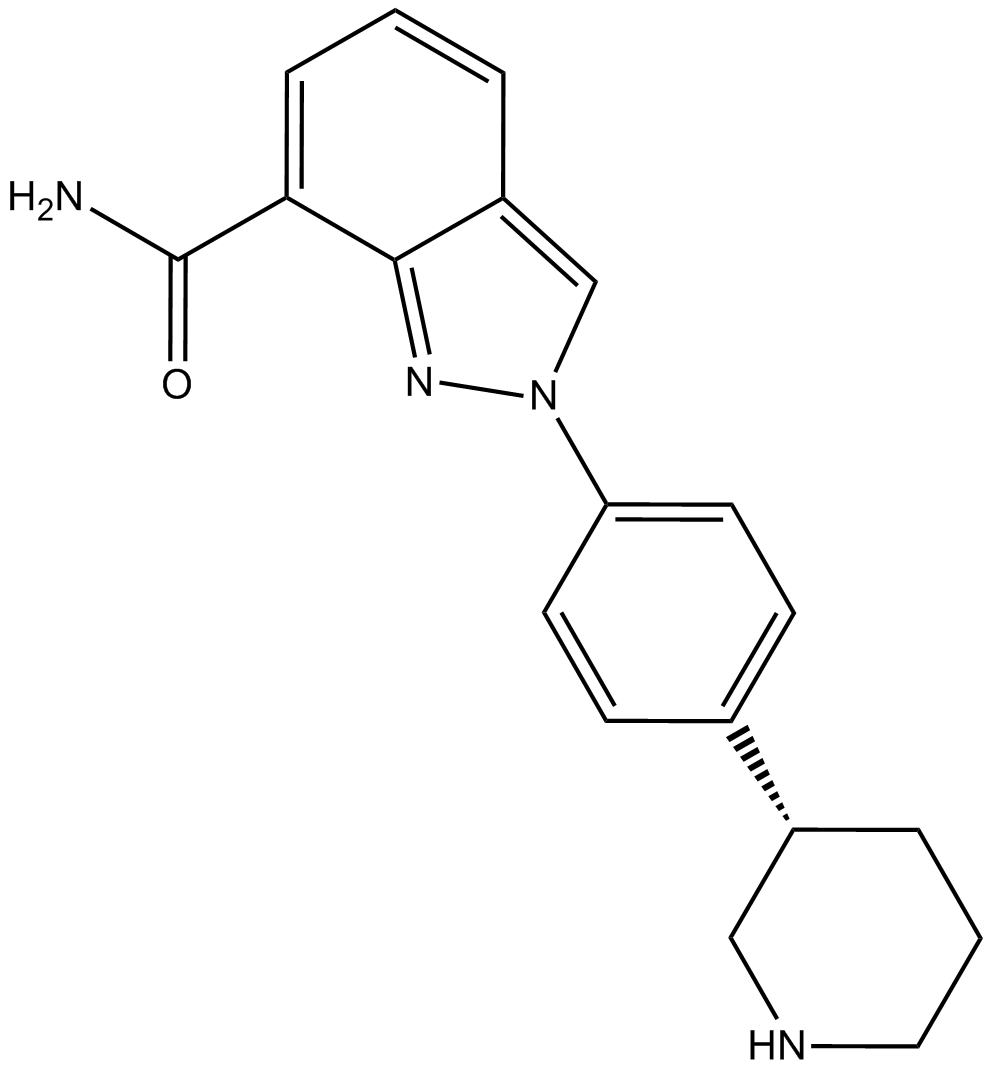
-
GC12756
MK-4827 hydrochloride
El clorhidrato de MK-4827 (clorhidrato de MK-4827) es un inhibidor de PARP1 y PARP2 muy potente y biodisponible por vÍa oral con IC50 de 3,8 y 2,1 nM, respectivamente. El clorhidrato de MK-4827 conduce a la inhibiciÓn de la reparaciÓn del daÑo del ADN, activa la apoptosis y muestra actividad antitumoral.
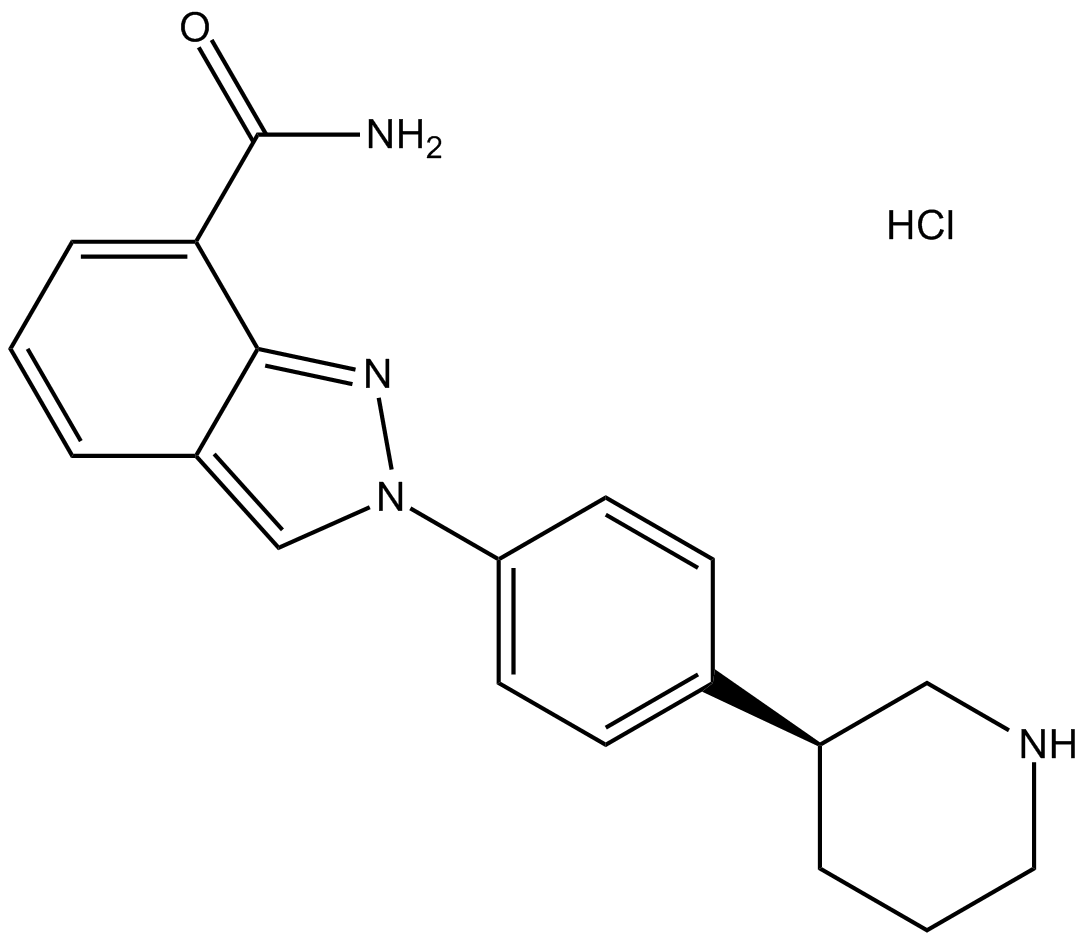
-
GC17052
MK-4827 Racemate
selective inhibitor of PARP1/PARP2
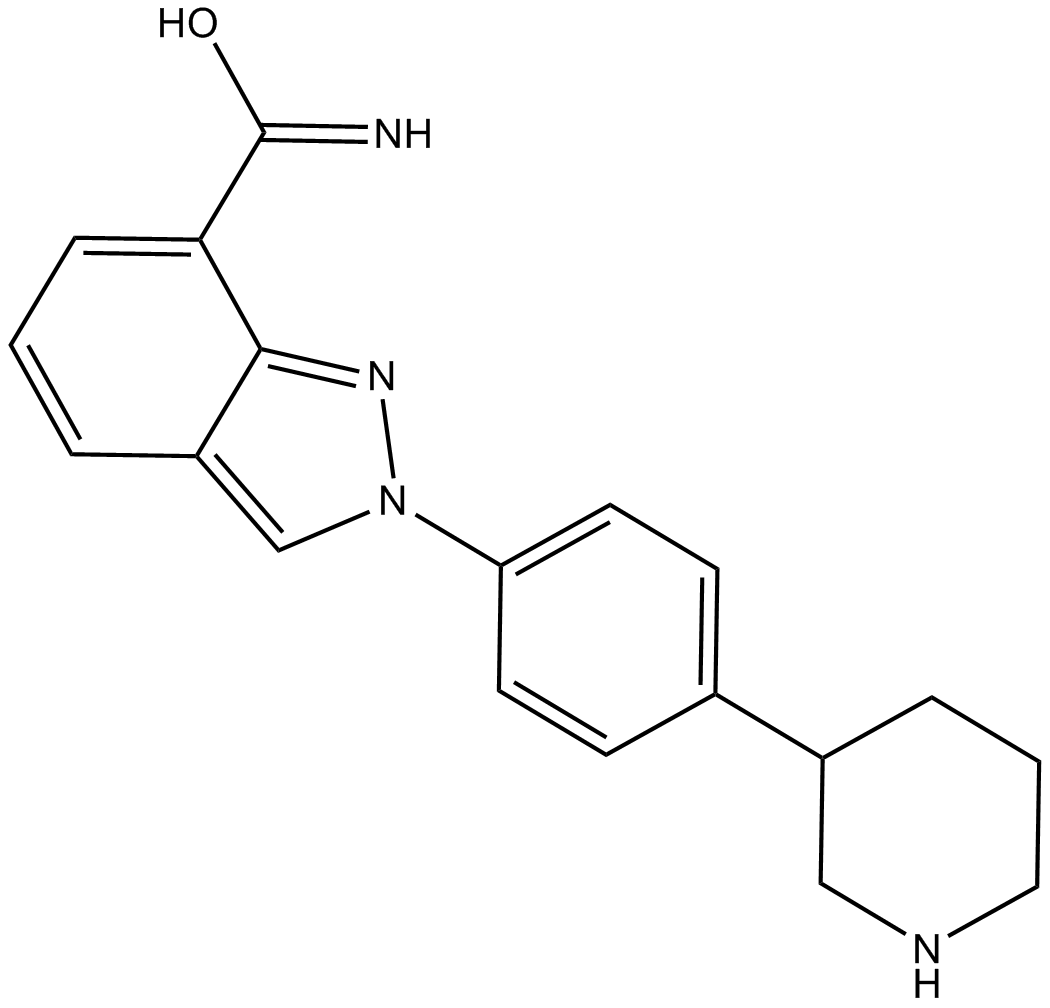
-
GC11537
MK-4827 tosylate
El tosilato de MK-4827 (tosilato de MK-4827) es un inhibidor de PARP1 y PARP2 muy potente y biodisponible por vÍa oral con una IC50 de 3,8 y 2,1 nM, respectivamente. El tosilato de MK-4827 conduce a la inhibiciÓn de la reparaciÓn del daÑo del ADN, activa la apoptosis y muestra actividad antitumoral.
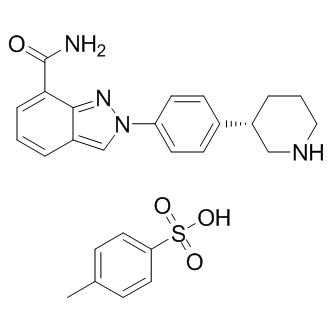
-
GC16914
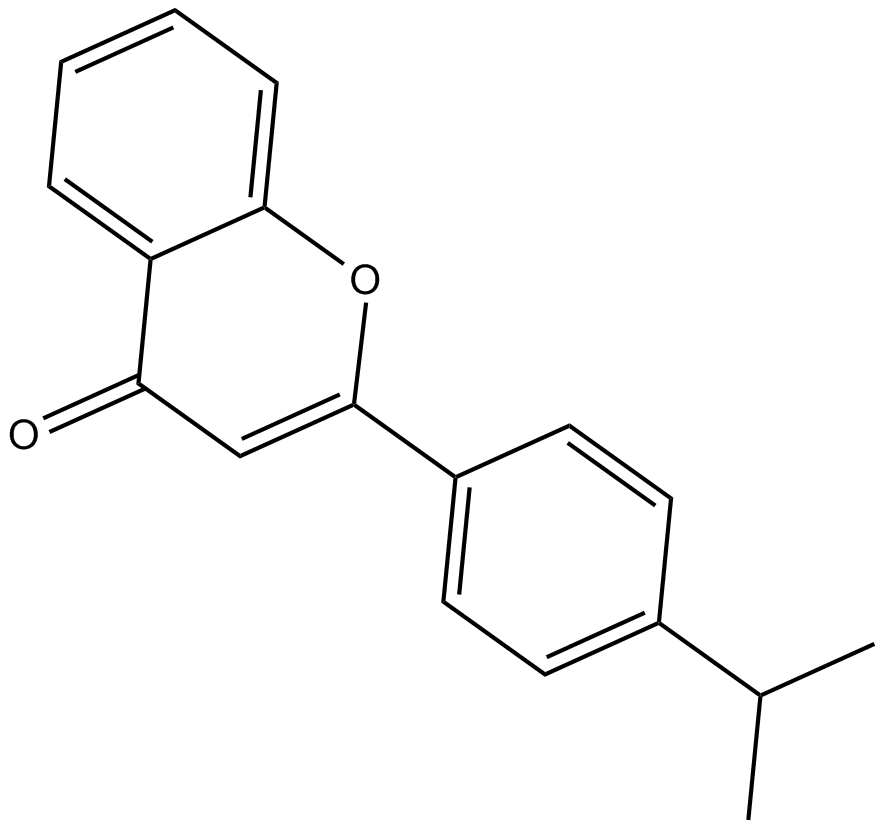
MN 64
MN 64 es un potente inhibidor de tankirasa 1, con IC50 de 6 nM, 72 nM, 19,1 μM y 39,4 μM para TNKS1, TNKS2, ARTD1 y ARTD2, respectivamente.
-
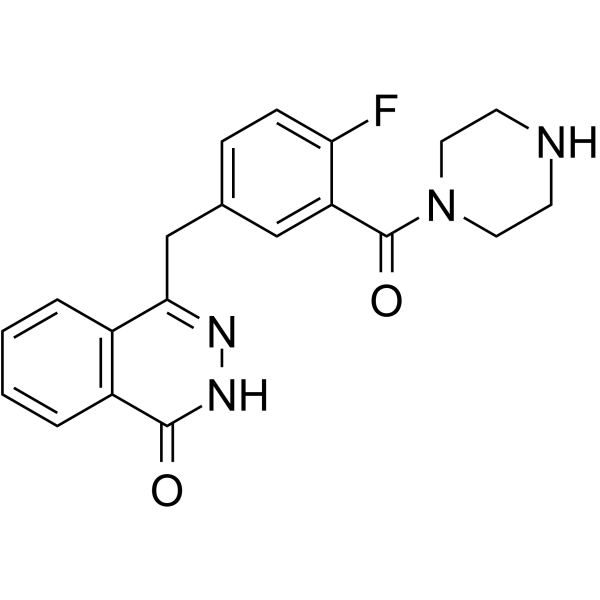
GC62154
N-Descyclopropanecarbaldehyde Olaparib
N-desciclopropanocarbaldehÍdo Olaparib es un anÁlogo de Olaparib que contiene un resto DOTA. N-DesciclopropanocarbaldehÍdo Olaparib es un ligando basado en CRBN para sintetizar nuevos EGFR duales y PARP PROTAC, DP-C-4. N-desciclopropanocarbaldehÍdo Olaparib puede ser radiomarcado F-18 o fluorÓforo para tomografÍa por emisiÓn de positrones (PET) o imÁgenes Ópticas en varios tipos de tumores.
-
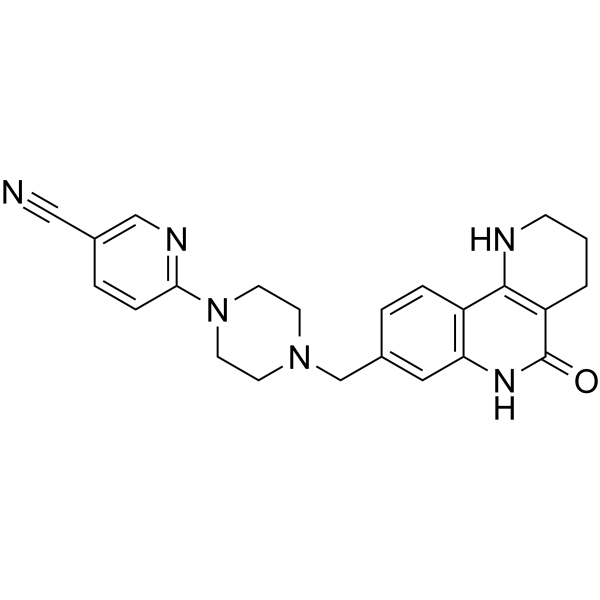
GC65202
Nesuparib
Nesuparib es un potente inhibidor de PARP.
-
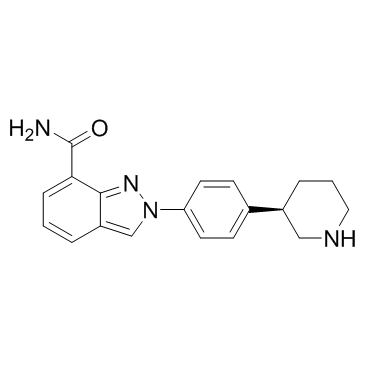
GC34120
Niraparib R-enantiomer (MK 4827 (R-enantiomer))
El enantiÓmero R de niraparib (enantiÓmero MK-4827 R) es un excelente inhibidor de PARP1 con IC50 de 2,4 nM.
-
GC19264
NMS-P118
NMS-P118 es un inhibidor de PARP-1 potente, disponible por vÍa oral y altamente selectivo para la terapia del cÁncer.
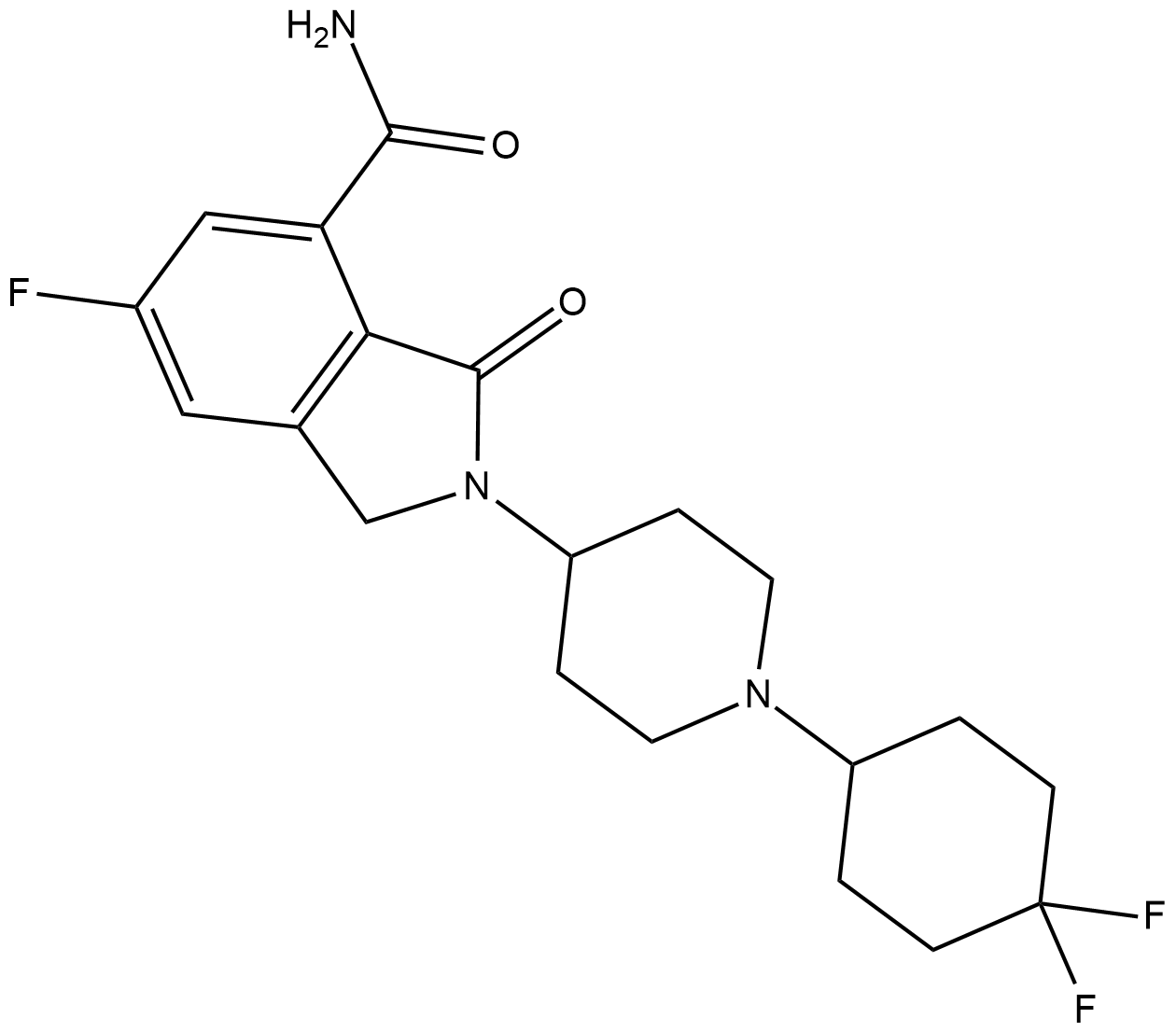
-
GC36751
NMS-P515
NMS-P515 es un potente inhibidor de PARP-1 estereoespecÍfico, activo por vÍa oral, con una Kd de 16 nM y una IC50 de 27 nM (en células Hela). Actividad antitumoral.
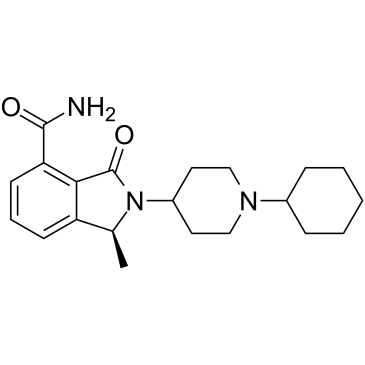
-
GC17775
NU 1025
An inhibitor of PARP
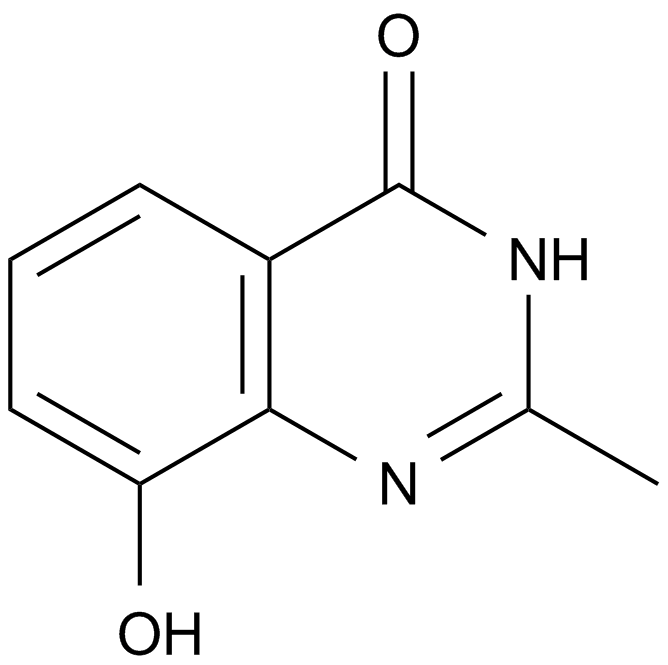
-
GC17555
NVP-TNKS656
NVP-TNKS656 es un inhibidor de TNKS2 altamente potente, selectivo y activo por vÍa oral con IC50 de 6 nM, y tiene una selectividad > 300 veces frente a PARP1 y PARP2.
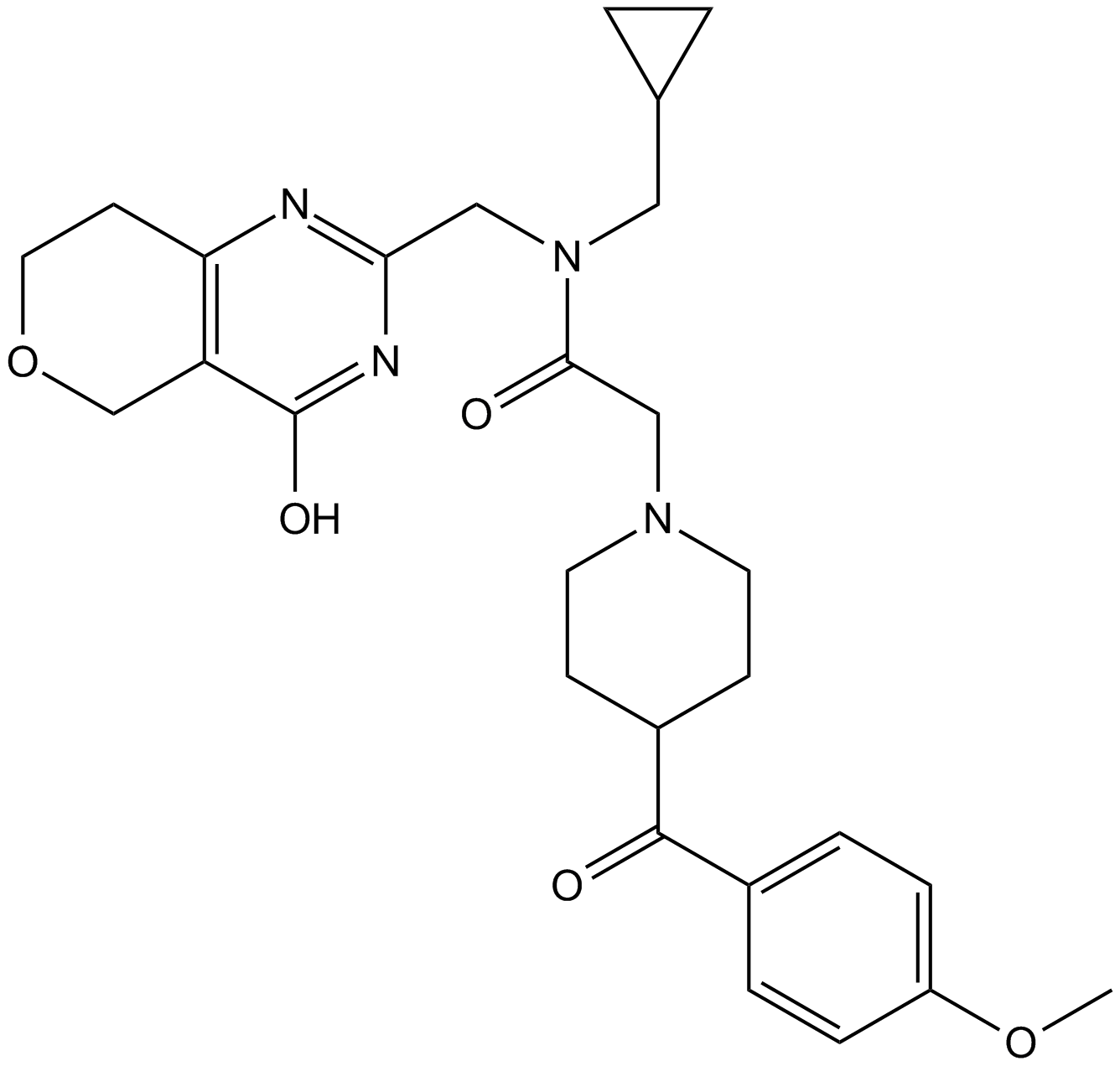
-
GC17580
Olaparib (AZD2281, Ku-0059436)
Un inhibidor de PARP
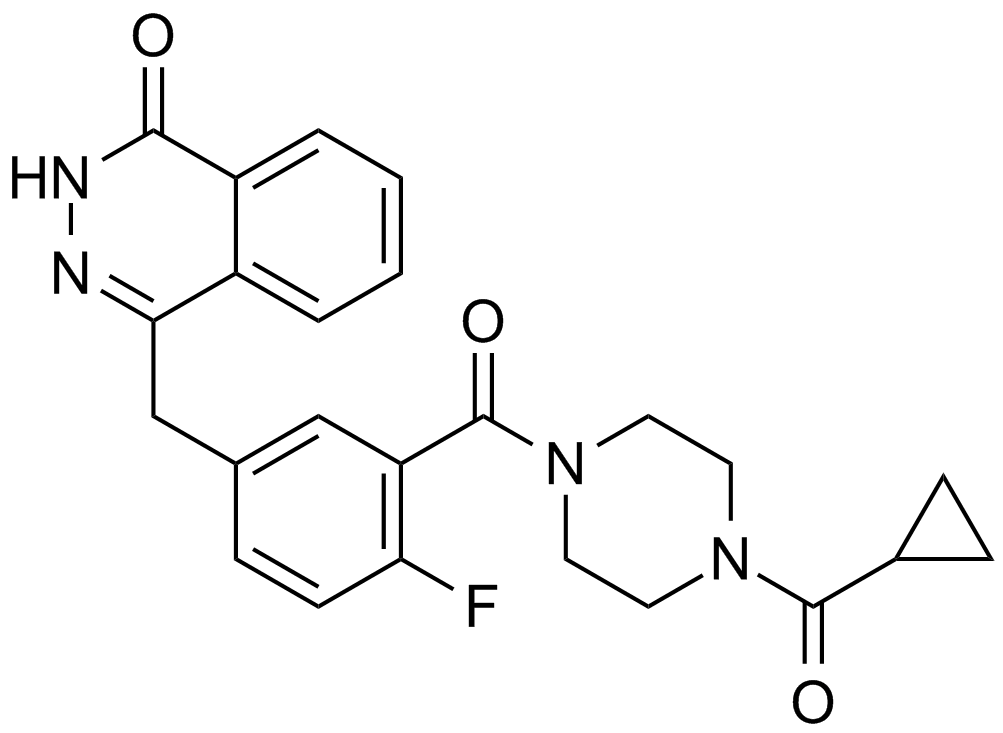
-
GC69618
Olaparib-d8
Olaparib-d8 es el deuterio sustituido de Olaparib (AZD2281). Olaparib es un inhibidor oral efectivo de PARP que inhibe PARP-1 y PARP-2 con IC50 de 5 y 1 nM, respectivamente. Olaparib es un activador de la autofagia y mitofagia.
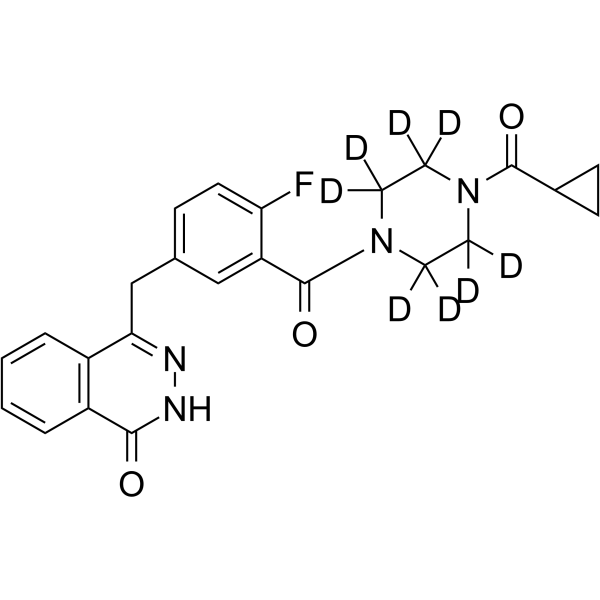
-
GC67906
OM-153

-
GN10114
Oroxin A

-
GC45808
OUL35
OUL35 (NSC39047) es un inhibidor potente y selectivo de ARTD10 (PARP-10), con una IC50 de 329 nM.
-
GC34071
Pamiparib (BGB-290)
Pamiparib (BGB-290) (BGB-290) es un inhibidor de PARP altamente selectivo, potente y activo por vÍa oral, con valores de CI50 de 0,9 nM y 0,5 nM para PARP1 y PARP2, respectivamente. Pamiparib (BGB-290) tiene una potente captura de PARP y la capacidad de penetrar en el cerebro, y puede usarse para la investigaciÓn de varios tipos de cÁncer, incluido el tumor sÓlido.
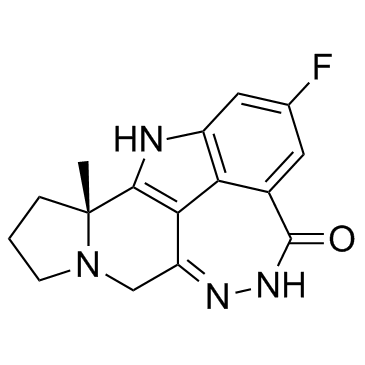
-
GC36855
Paris saponin VII
Paris saponin VII (Chonglou Saponin VII) es una saponina esteroide aislada de las raÍces y rizomas de Trillium tschonoskii Maxim. La apoptosis inducida por saponina VII de Paris en células K562/ADR estÁ asociada con Akt/MAPK y la inhibiciÓn de P-gp. La saponina VII de Paris atenÚa el potencial de la membrana mitocondrial, aumenta la expresiÓn de proteÍnas relacionadas con la apoptosis, como Bax y el citocromo c, y disminuye los niveles de expresiÓn de proteÍnas de Bcl-2, caspasa-9, caspasa-3, PARP-1 y p- Act. Paris saponin VII induce una autofagia robusta en células K562/ADR y proporciona una base bioquÍmica en el tratamiento de la leucemia.
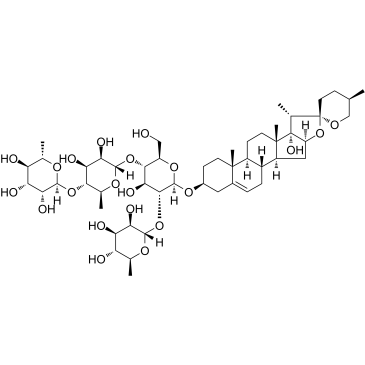
-
GC64579
PARP-1-IN-2
PARP-1-IN-2 (compuesto 11 g) es un potente inhibidor de PARP1 penetrado por BBB, con una IC50 de 149 nM. PARP1-IN-2 muestra una actividad antiproliferativa significativamente potente contra la lÍnea de células epiteliales de adenocarcinoma de pulmÓn humano A549. PARP1-IN-2 puede inducir la apoptosis de las células A549.
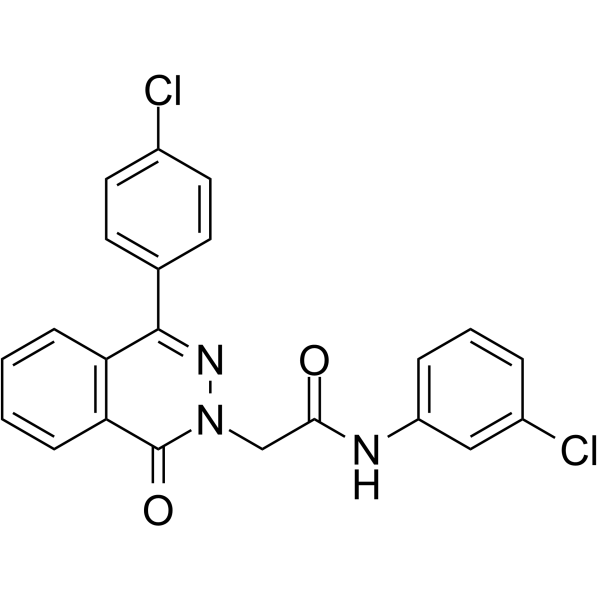
-
GC65927
PARP-2-IN-1
PARP-2-IN-1 es un inhibidor potente y selectivo de PARP-2 con una IC50 de 11,5 nM.
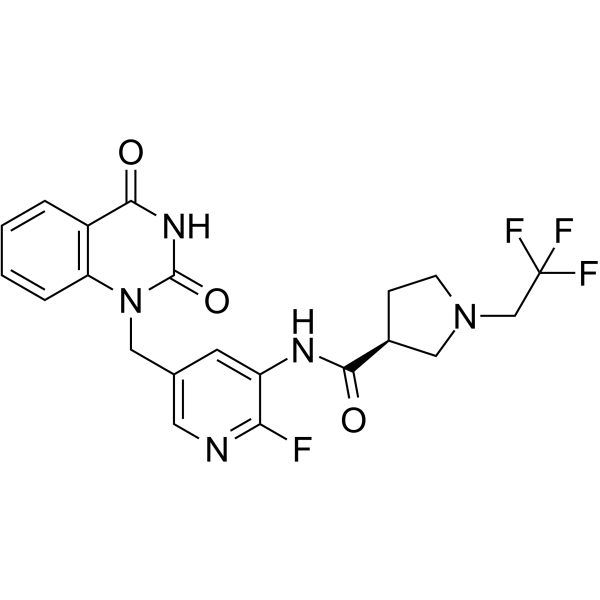
-
GC68006
PARP1-IN-11
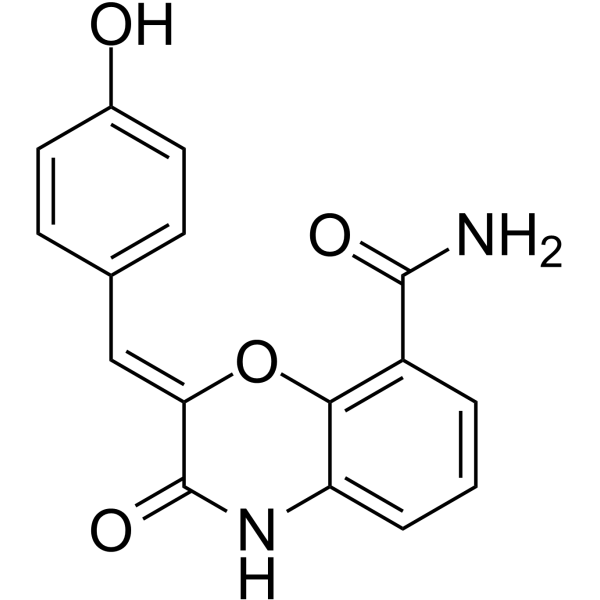
-
GC62275
PARP1-IN-5 dihydrochloride
El diclorhidrato de PARP1-IN-5 es un inhibidor de PARP-1 de baja toxicidad, activo por vÍa oral, potente y selectivo (IC50 = 14,7 nM). El diclorhidrato de PARP1-IN-5 se puede utilizar para la investigaciÓn del cÁncer.
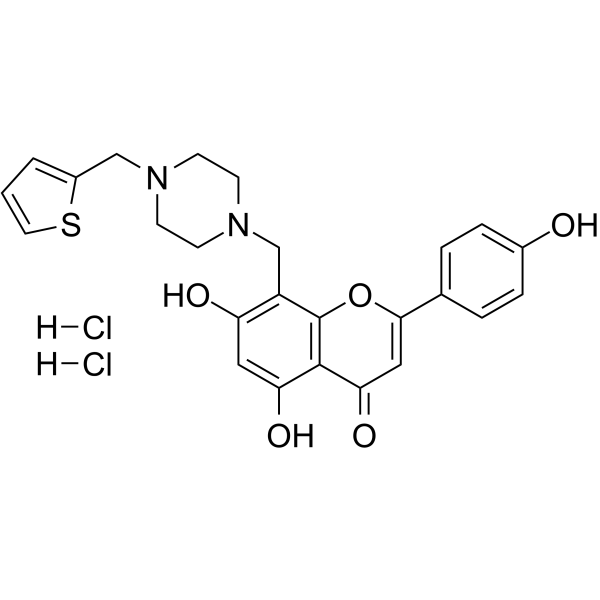
-
GC69660
PARP1-IN-7
PARP1-IN-7 es un inhibidor de la poli ADP ribosa polimerasa-1 (PARP1) utilizado como agente anticancerígeno.
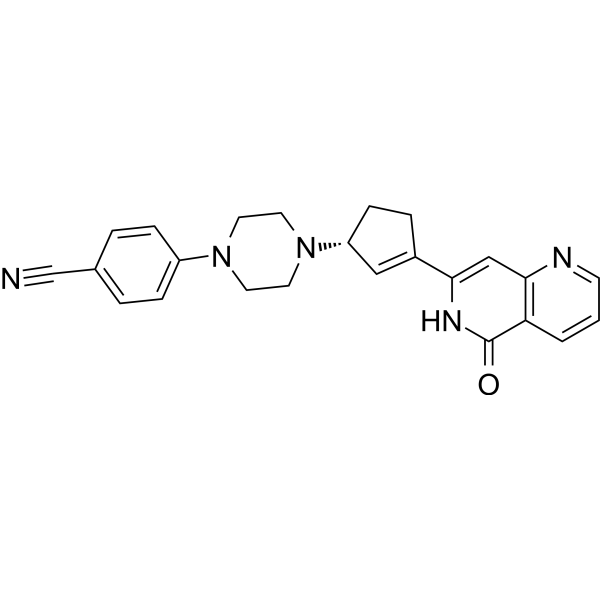
-
GC64578
PARP1-IN-8
PARP1-IN-8 (compuesto 11c) es un potente inhibidor de PARP1 penetrado por BBB, con una IC50 de 97 nM. PARP1-IN-8 muestra una actividad antiproliferativa significativamente potente contra la lÍnea de células epiteliales de adenocarcinoma de pulmÓn humano A549.
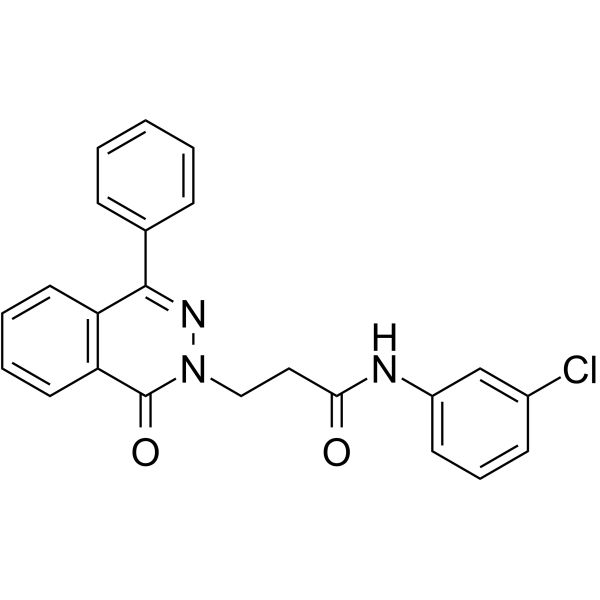
-
GC69657
PARP10-IN-2
PARP10-IN-2 es un inhibidor efectivo de la PARP10, una enzima de transferencia de mono-ADP-ribosa. Su IC50 para la PARP10 humana es de 3.64 μM. Además, también inhibe la PARP2 y la PARP15, con IC50 respectivos para las formas humanas de 27 μM y 11 μM.
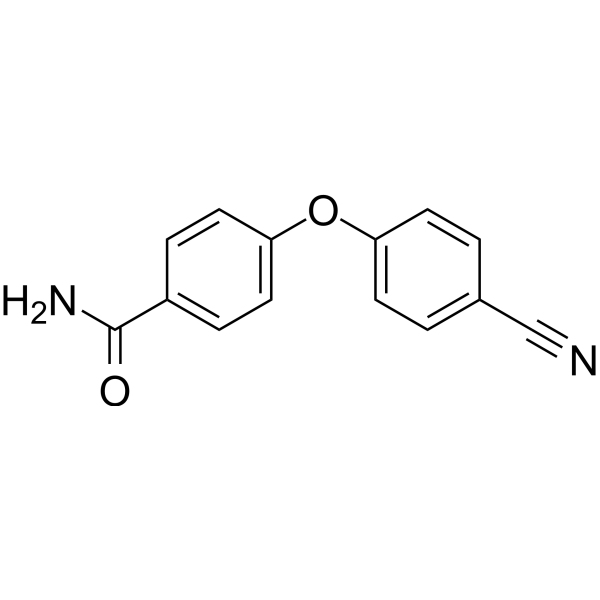
-
GC69658
PARP10-IN-3
PARP10-IN-3 es un inhibidor selectivo de la poli (ADP-ribosa) polimerasa PARP10, con una IC50 de 480 nM en humanos. También inhibe PARP2 y PARP15, con una IC50 de 1.7 μM para ambos en humanos.
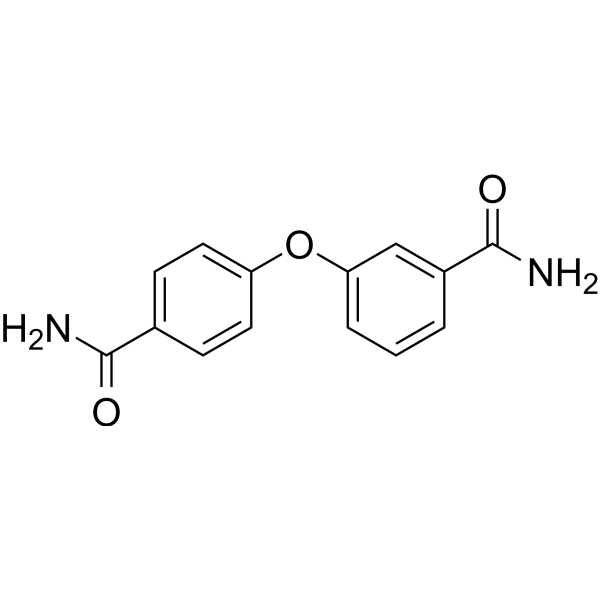
-
GC68035
PARP10/15-IN-1
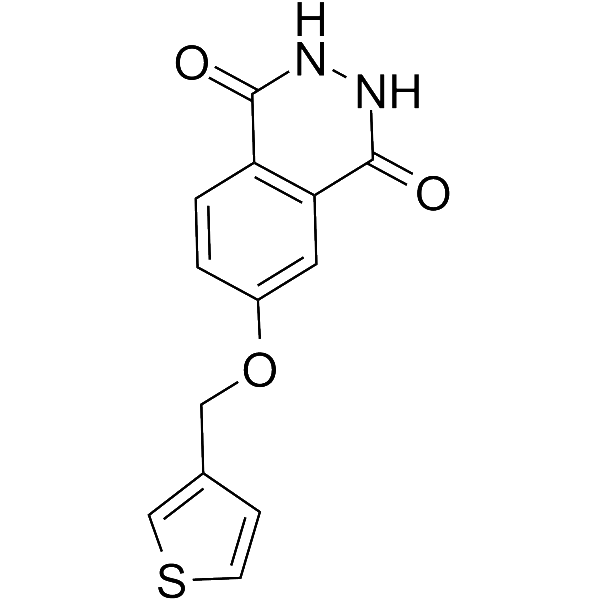
-
GC69655
PARP10/15-IN-2
PARP10/15-IN-2 (Compuesto 8h) es un inhibidor doble efectivo de PARP10 y PARP15, con valores de IC50 para PARP10 y PARP15 de 0.15 µM y 0.37 µM, respectivamente. El PARP10/15-IN-2 puede penetrar en las células y prevenir la apoptosis.
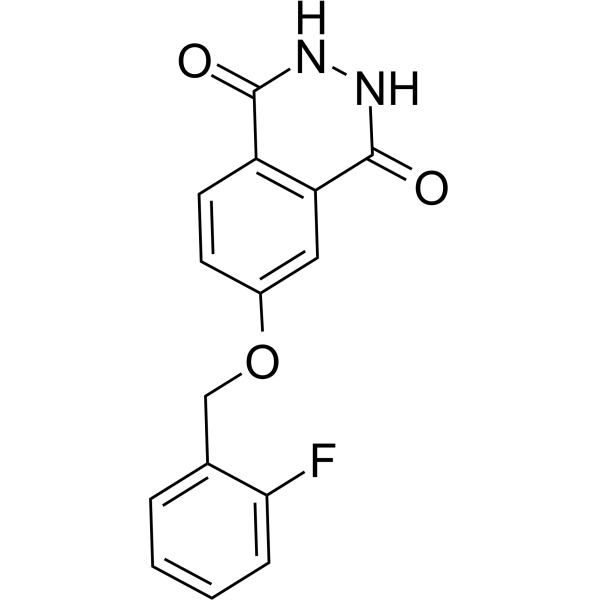
-
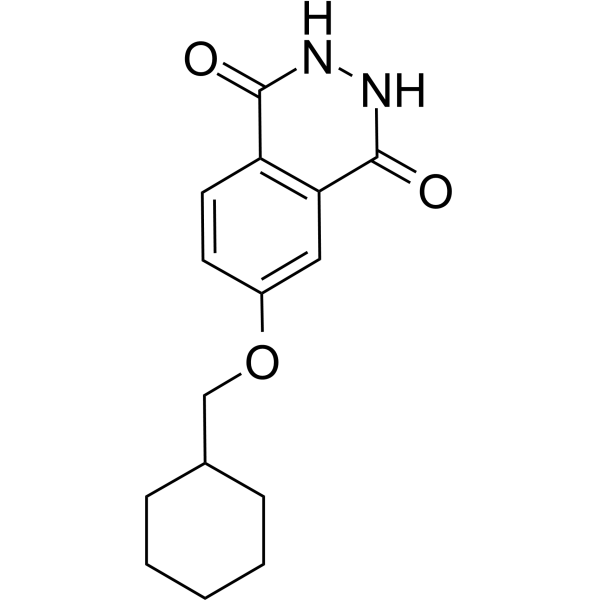
GC69656
PARP10/15-IN-3
PARP10/15-IN-3 (Compuesto 8a) es un inhibidor doble efectivo de PARP10 y PARP15, con valores de IC50 para PARP10 y PARP15 de 0.14 µM y 0.40 µM, respectivamente. El PARP10/15-IN-3 puede penetrar en las células y prevenir la apoptosis celular.
-
GC69659
PARP11 inhibitor ITK7
El inhibidor de PARP11 ITK7 (ITK7) es un inhibidor selectivo y efectivo de PARP11. El valor IC50 de ITK7 para la inhibición de PARP11 es de 14 nM. ITK7 se puede utilizar en estudios de localización celular.
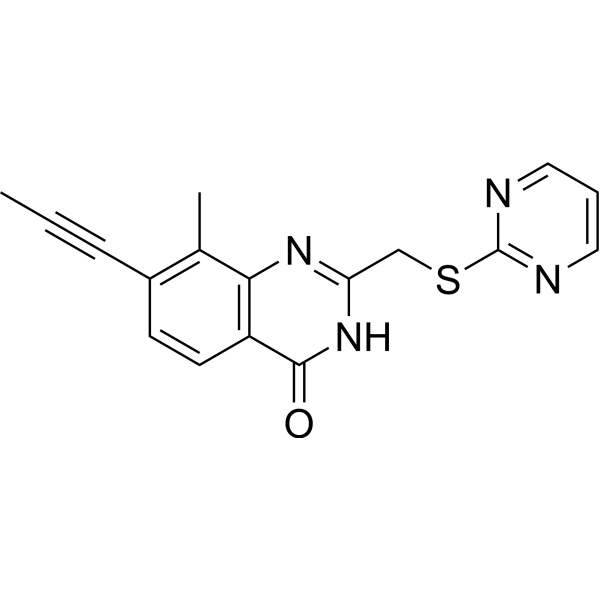
-
GC39302
PARP14 inhibitor H10
El inhibidor H10 de PARP14, compuesto H 10, es un inhibidor selectivo contra PARP14 (IC50=490 nM), sobre otras PARP (≈24 veces sobre PARP1). El inhibidor H10 de PARP14 induce la apoptosis celular mediada por caspasa-3/7.
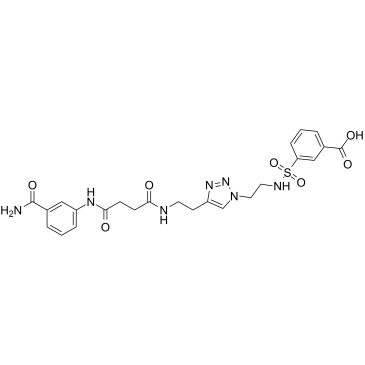
-
GC69661
PARP7-IN-14
PARP7-IN-14 (I-1) es un inhibidor selectivo efectivo de PARP7 con un valor IC50 de 7.6 nM. PARP7-IN-14 tiene actividad anticancerígena.
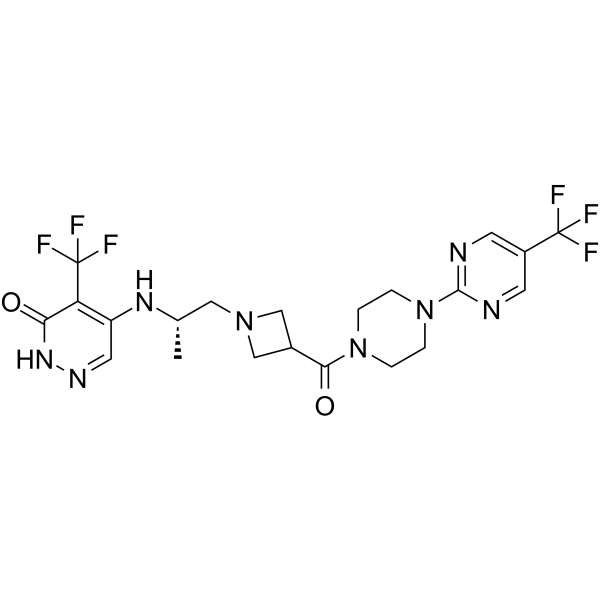
-
GC14251
Picolinamide
poly (ADP-ribose) synthetase inhibitor
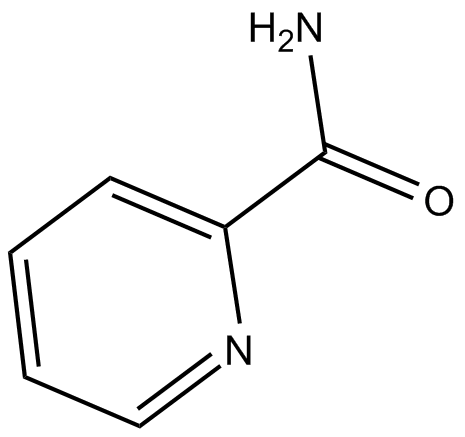
-
GC10995
PJ34
An inhibitor of poly (ADP-ribose) polymerases

-
GC10145
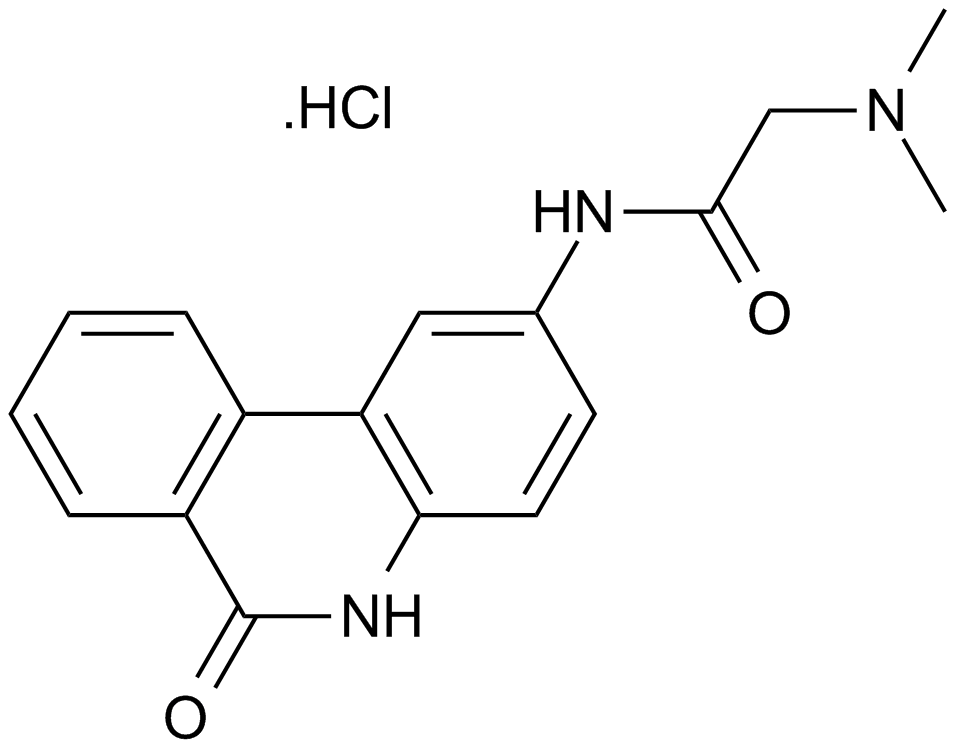
PJ34 hydrochloride
An inhibitor of poly (ADP-ribose) polymerases
-
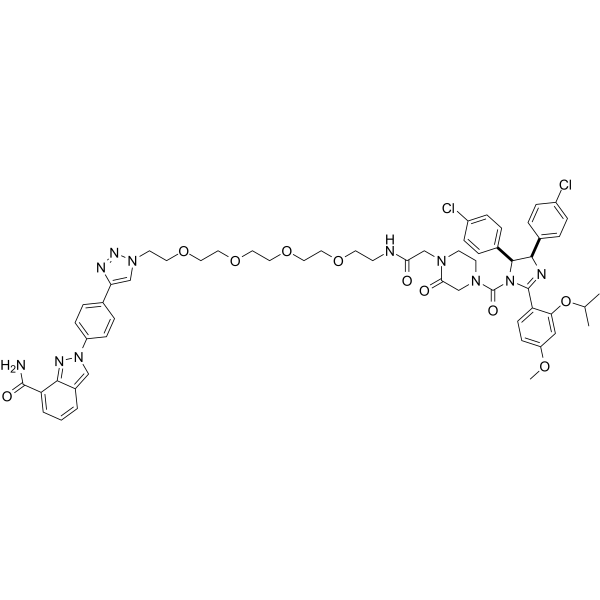
GC65147
PROTAC PARP1 degrader
El degradador PROTAC PARP1 es un degradador PARP1 basado en el ligando MDM2 E3. Induce una escisiÓn significativa de PARP1 y muerte celular programada. El degradador PROTAC PARP1 a 10 μM a las 24 h inhibe la lÍnea celular MDA-MB-231 con una IC50 de 6,12 μM.
-
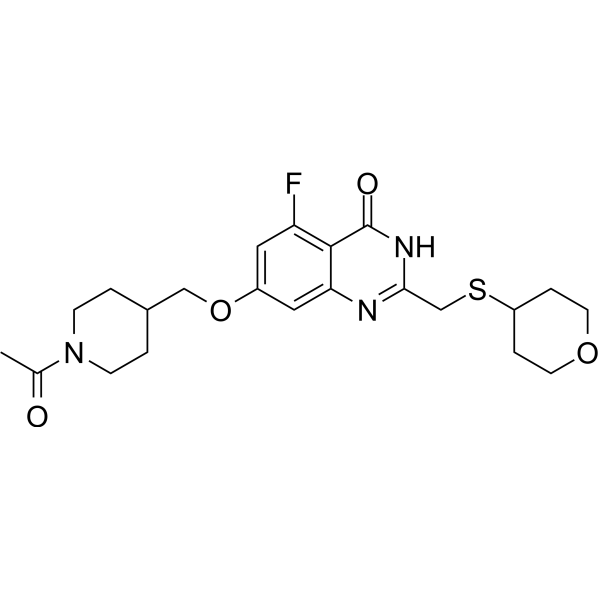
GC69804
RBN-3143
RBN-3143 es un inhibidor efectivo y competitivo de PARP14 que cataliza la competencia de NAD+, con un valor IC50 de 4 nM. RBN-3143 inhibe la ADP-ribosilación mediada por PARP14 y estabiliza a PARP14 en líneas celulares. RBN-3143 se utiliza en investigaciones sobre inflamación pulmonar.
-
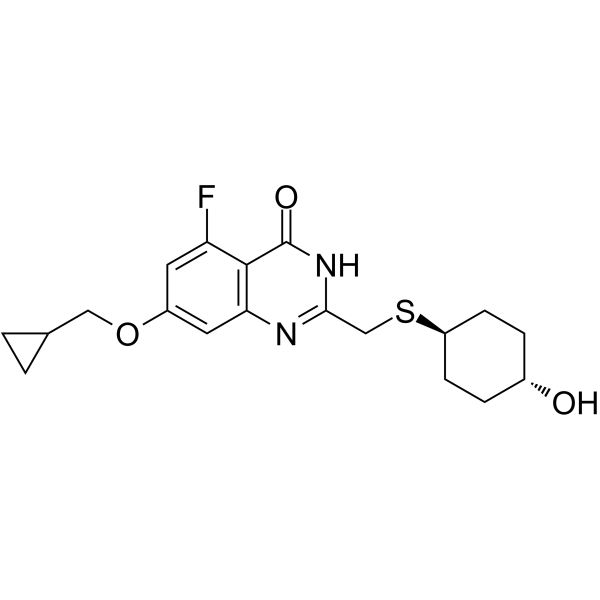
GC62473
RBN012759
RBN012759 es un inhibidor potente, selectivo y activo por vÍa oral de PARP14, con una IC50 de <3 nM.
-
GC19505
RK-287107
RK-287107 es un inhibidor de tankirasa potente y específico con IC50 de 14,3 y 10,6 nM para tankirasa-1 y tankirasa-2, respectivamente. RK-287107 bloquea el crecimiento de células de cáncer colorrectal.
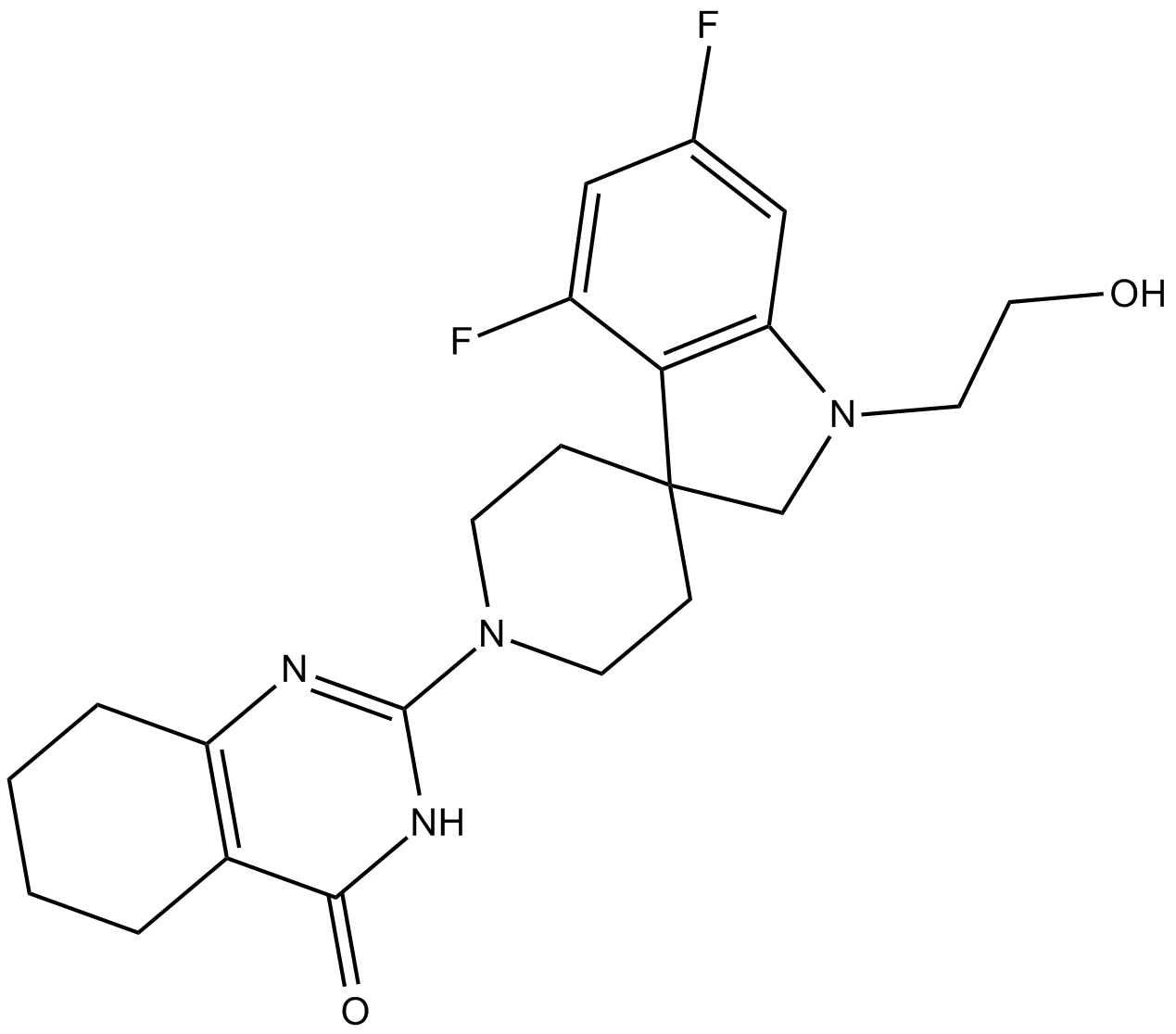
-
GC13249
Rucaparib (free base)
Rucaparib (base libre) (AG014699) es un potente inhibidor activo por vÍa oral de las proteÍnas PARP (PARP-1, PARP-2 y PARP-3) con una Ki de 1,4 nM para PARP1. Rucaparib (base libre) es un inhibidor modesto de la hexosa-6-fosfato deshidrogenasa (H6PD). Rucaparib (base libre) tiene potencial para la investigaciÓn del cÁncer de prÓstata resistente a la castraciÓn (CPRC).
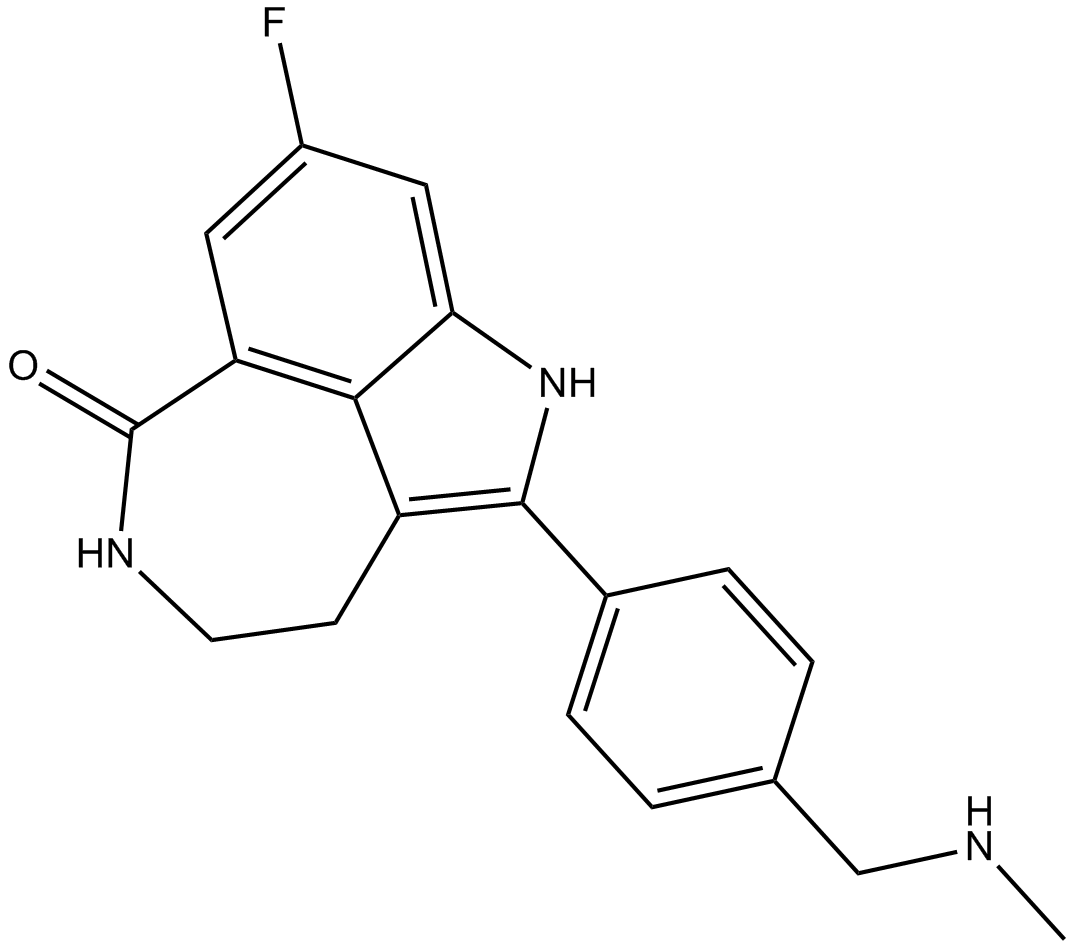
-
GC32793
Rucaparib Camsylate
El monocamsilato de rucaparib (AG014699) es un inhibidor potente y activo por vÍa oral de las proteÍnas PARP (PARP-1, PARP-2 y PARP-3) con una Ki de 1,4 nM para PARP1. El camsilato de rucaparib es un inhibidor modesto de la hexosa-6-fosfato deshidrogenasa (H6PD). El camsilato de rucaparib tiene potencial para la investigaciÓn del cÁncer de prÓstata resistente a la castraciÓn (CPRC).
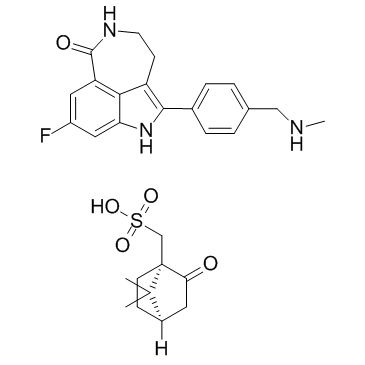
-
GC15955
Rucaparib?(AG–014699,PF–01367338) phosphate
A PARP1 inhibitor
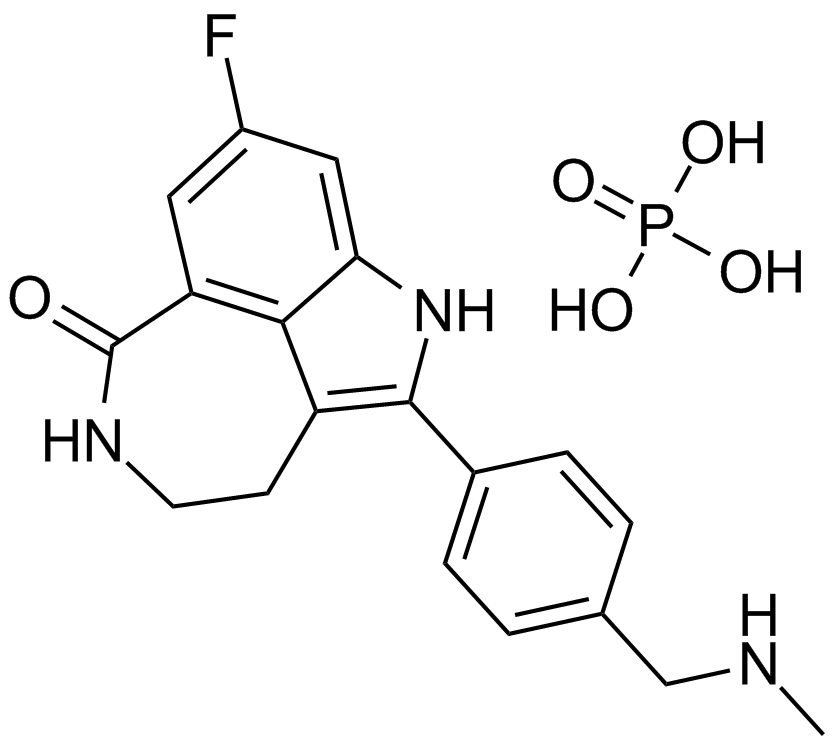
-
GC62105
Senaparib
Senaparib (IMP4297) es un inhibidor de PARP1/2 muy potente, selectivo y activo por vÍa oral. Senaparib (IMP4297) exhibe una fuerte actividad antitumoral en modelos animales.
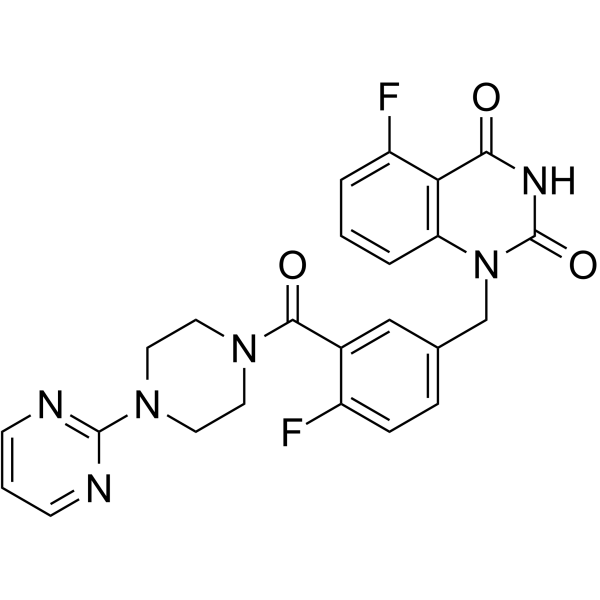
-
GC67962
Simmiparib
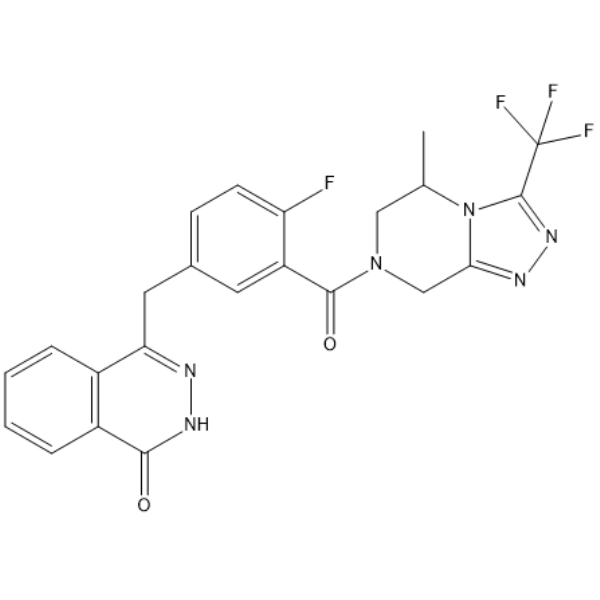
-
GC69907
SK-575
SK-575 es un inhibidor de la degradación de PARP1 altamente eficiente y específico para el complejo proteico hidrolizado dirigido (PROTAC), con un IC50 de 2.30 nM. SK-575 puede inhibir efectivamente el crecimiento celular en cánceres que portan mutaciones BRCA1/2.
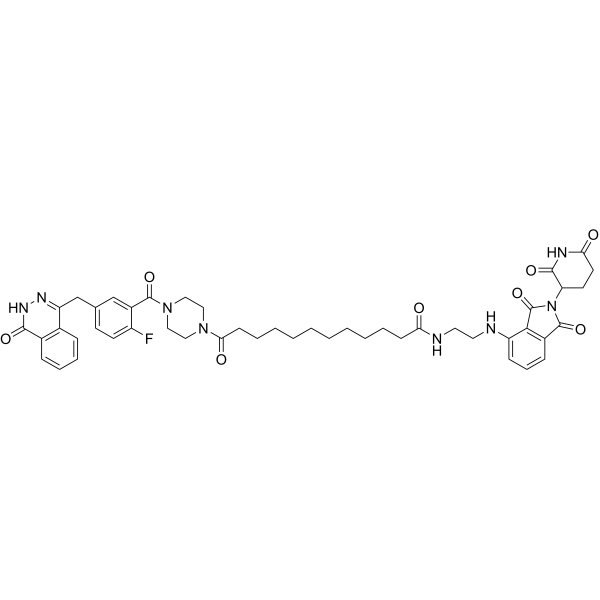
-
GC37728
Talazoparib tosylate
El tosilato de talazoparib (BMN 673ts) es un inhibidor de PARP1/2 novedoso, potente y disponible por vÍa oral con una IC50 de 0,57 nM para PARP1.
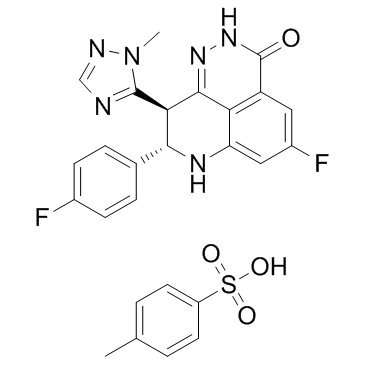
-
GC15041
Tankyrase Inhibitors (TNKS) 22
Tankyrase inhibitor
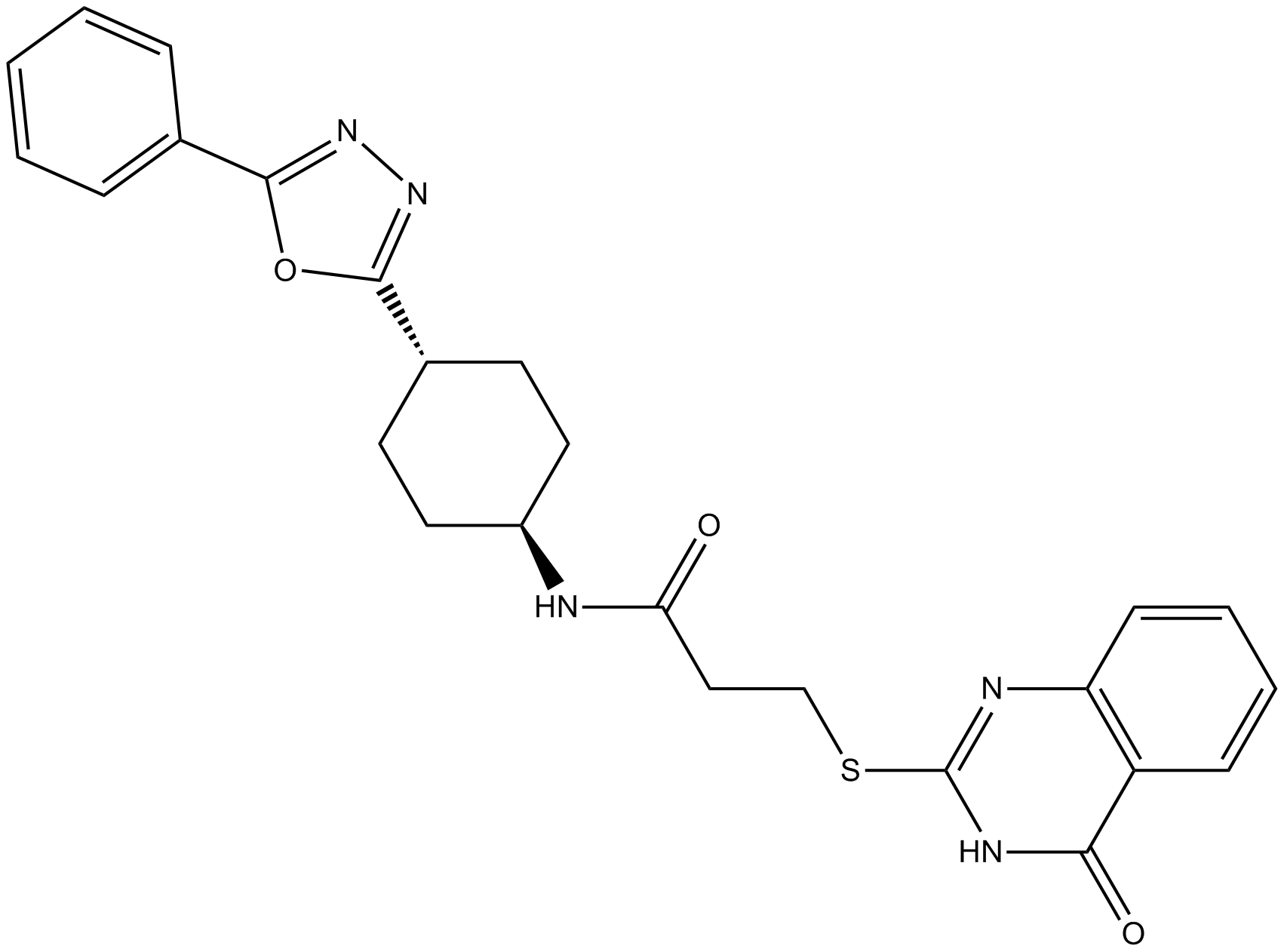
-
GC11548
Tankyrase Inhibitors (TNKS) 49
Tankyrase inhibitor
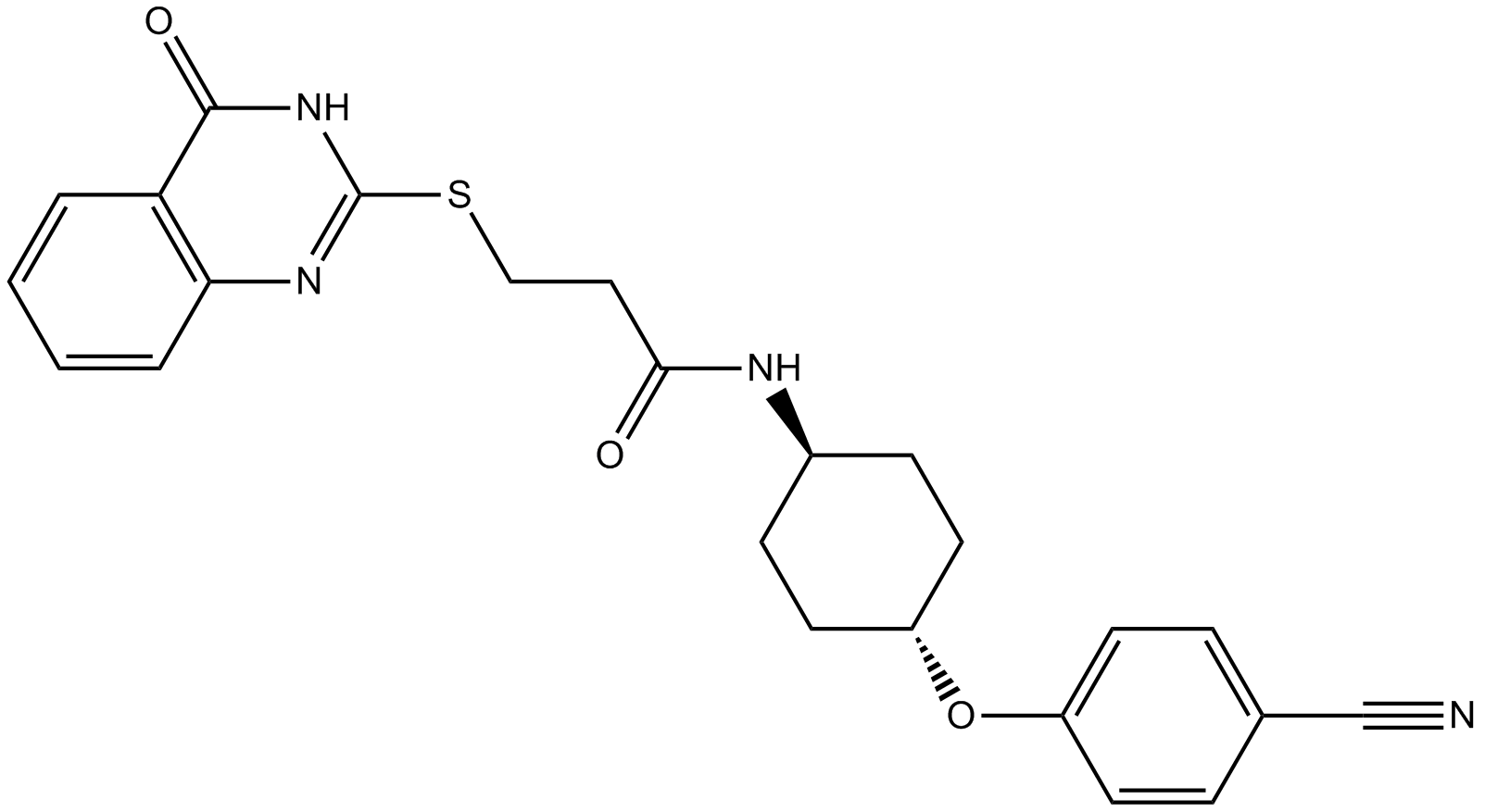
-
GC37735
Tankyrase-IN-2
Tankyrase-IN-2 (compuesto 5k) es un inhibidor de tankirasa potente, selectivo y oralmente activo (IC50 de 10, 7 y 710 nM para TNKS1, TNKS2 y PARP1, respectivamente). Tankyrase-IN-2 tiene un perfil fisicoquÍmico favorable y propiedades farmacocinéticas que modulan la actividad de la vÍa Wnt en un modelo de xenoinjerto colorrectal.
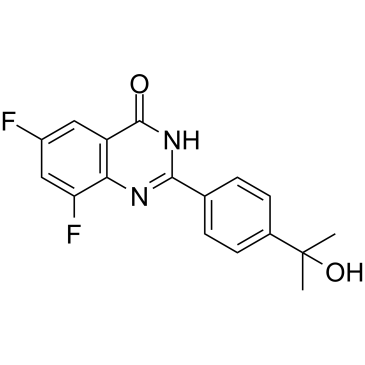
-
GC14509
UPF 1069
A selective PARP2 inhibitors
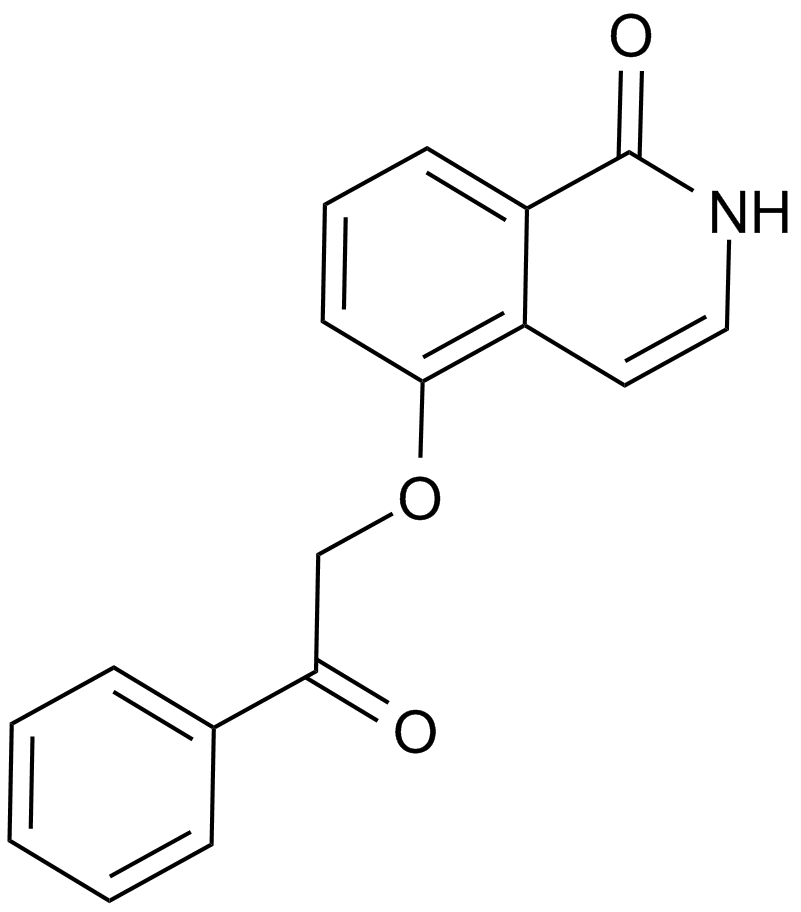
-
GC17783
Veliparib dihydrochloride
An orally bioavailable inhibitor of PARP1 and PARP2
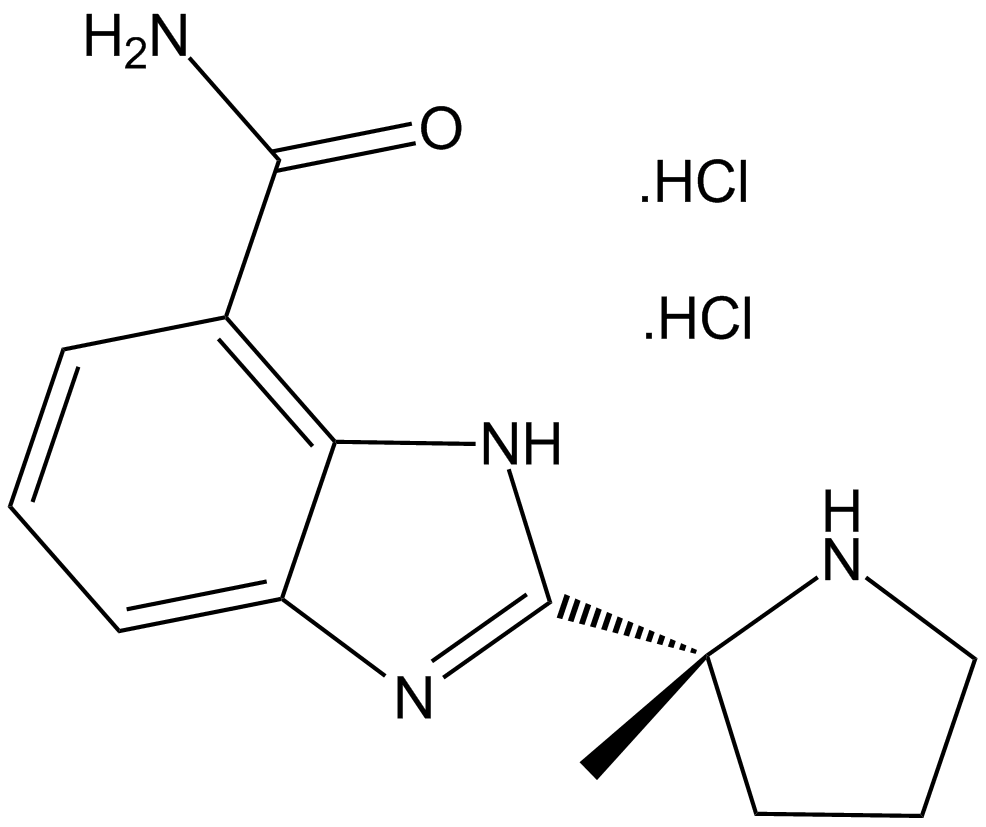
-
GC62129
Venadaparib
Venadaparib (IDX-1197) es un inhibidor de PARP potente, selectivo y activo por vÍa oral con IC50 de 1,4 nM y 1,0 nM para PARP1 y PARP2, respectivamente. Venadaparib no es sensible a PARP-5. Venadaparib previene la reparaciÓn de roturas monocatenarias (SSB) del ADN y puede utilizarse para la investigaciÓn de tumores sÓlidos.
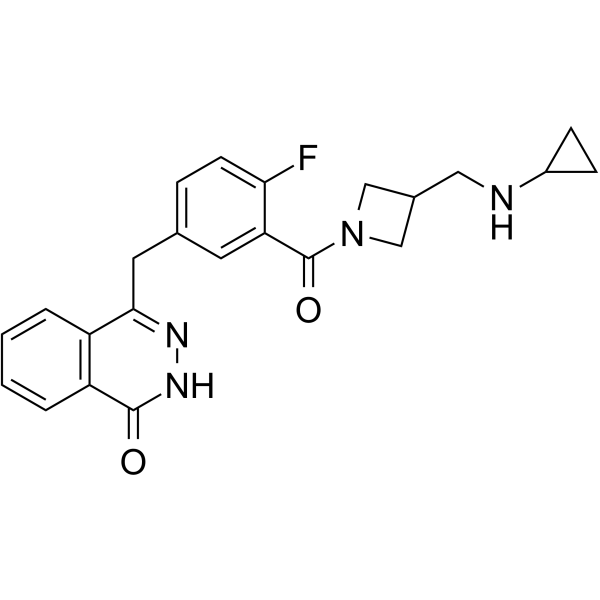
-
GC33358
WD2000-012547
WD2000-012547 es un inhibidor selectivo de la poli(ADP-ribosa)-polimerasa (PARP-1) con un pKi de 8,221.
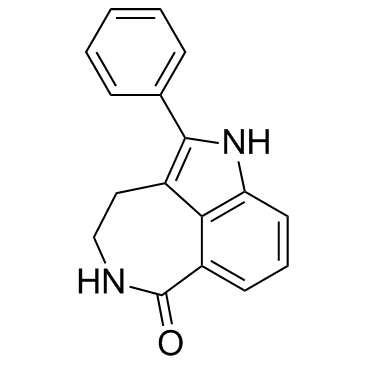
-
GC12781
XAV-939
XAV-939 inhibe de forma selectiva la transcripción mediada por β-catenina.