PKC
PKC (protein kinase C) is a serine/threonine protein kinase that mediates insulin signaling, actin cytoskeleton, cell mobility, apoptosis and tumorigenesis etc.
Products for PKC
- Cat.No. Nombre del producto Información
-
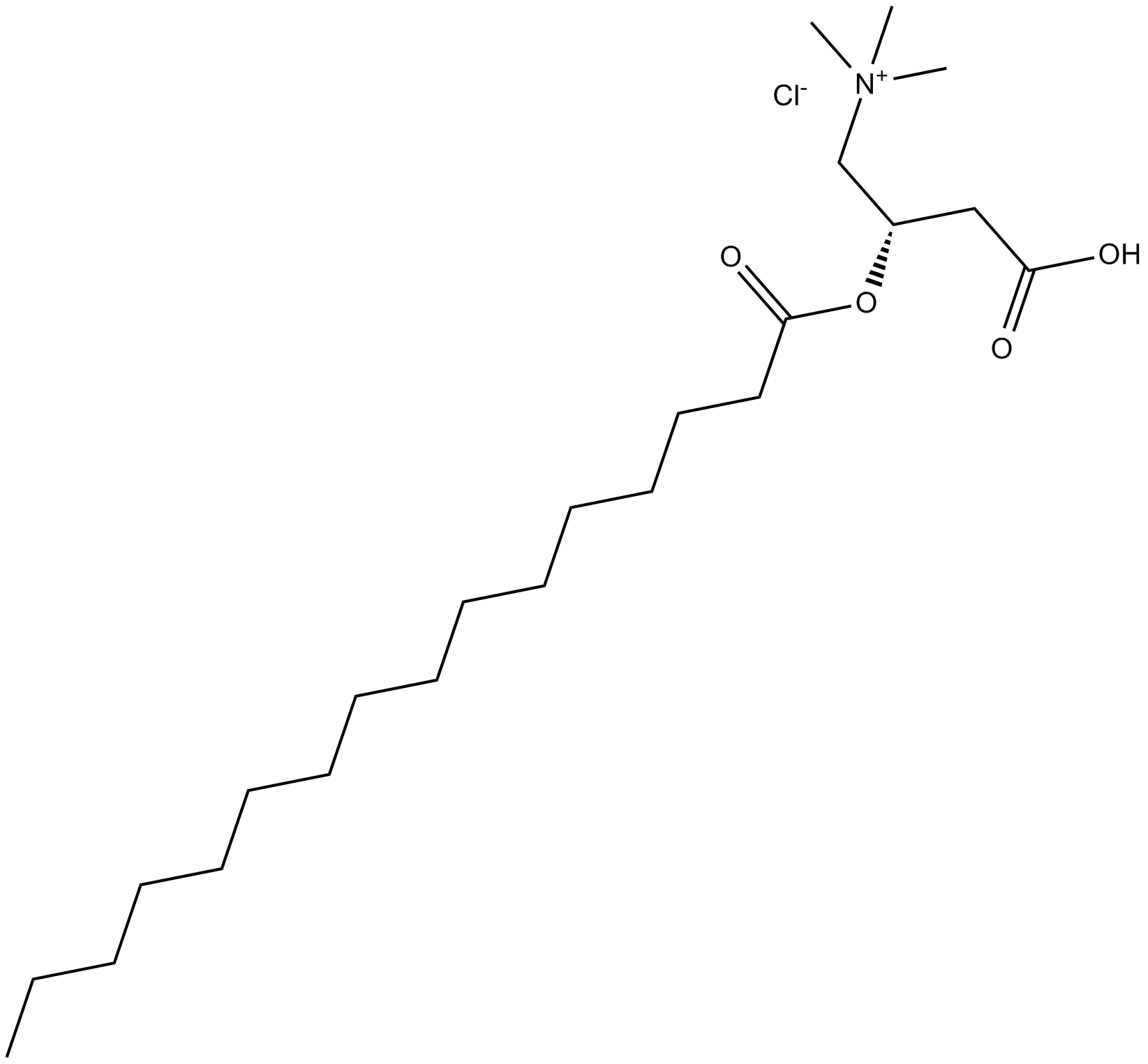
GC13890
(±)-Palmitoylcarnitine chloride
(±)-El cloruro de palmitoilcarnitina es un sustrato mitocondrial derivado de un ácido graso y reduce selectivamente la supervivencia celular en las células de cáncer colorrectal y de próstata al afectar las vías proinflamatorias, la entrada de Ca2+ y los efectos similares a los de la DHT.
-
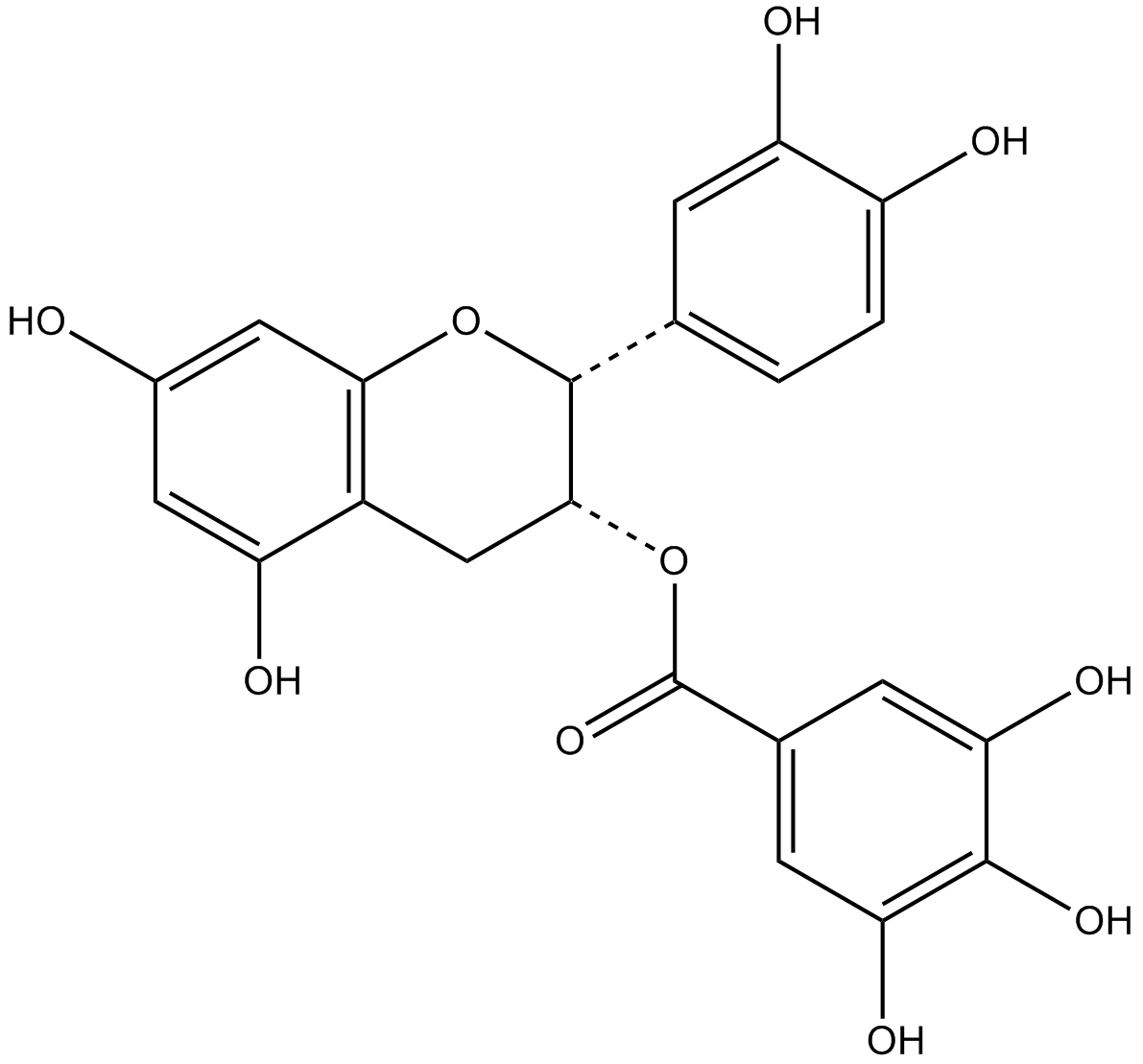
GC10603
(-)-epicatechin gallate
El galato de (-)-epicatequina (galato de epicatequina) inhibe la ciclooxigenasa-1 (COX-1) con una IC50 de 7,5 μM.
-
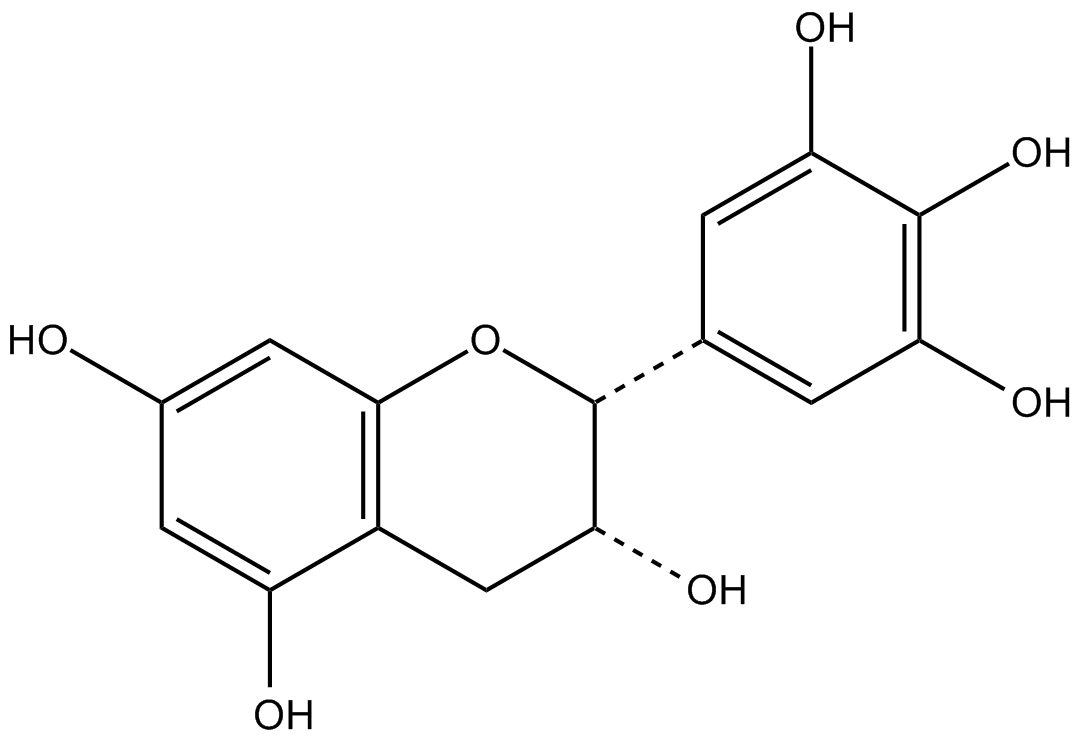
GC17242
(-)-epigallocatechin
(-)-Epigalocatequina (Epigalocatequina) es el flavonoide mÁs abundante en el té verde, puede unirse a polipéptidos nativos desplegados y prevenir la conversiÓn a fibrillas de amiloide.
-
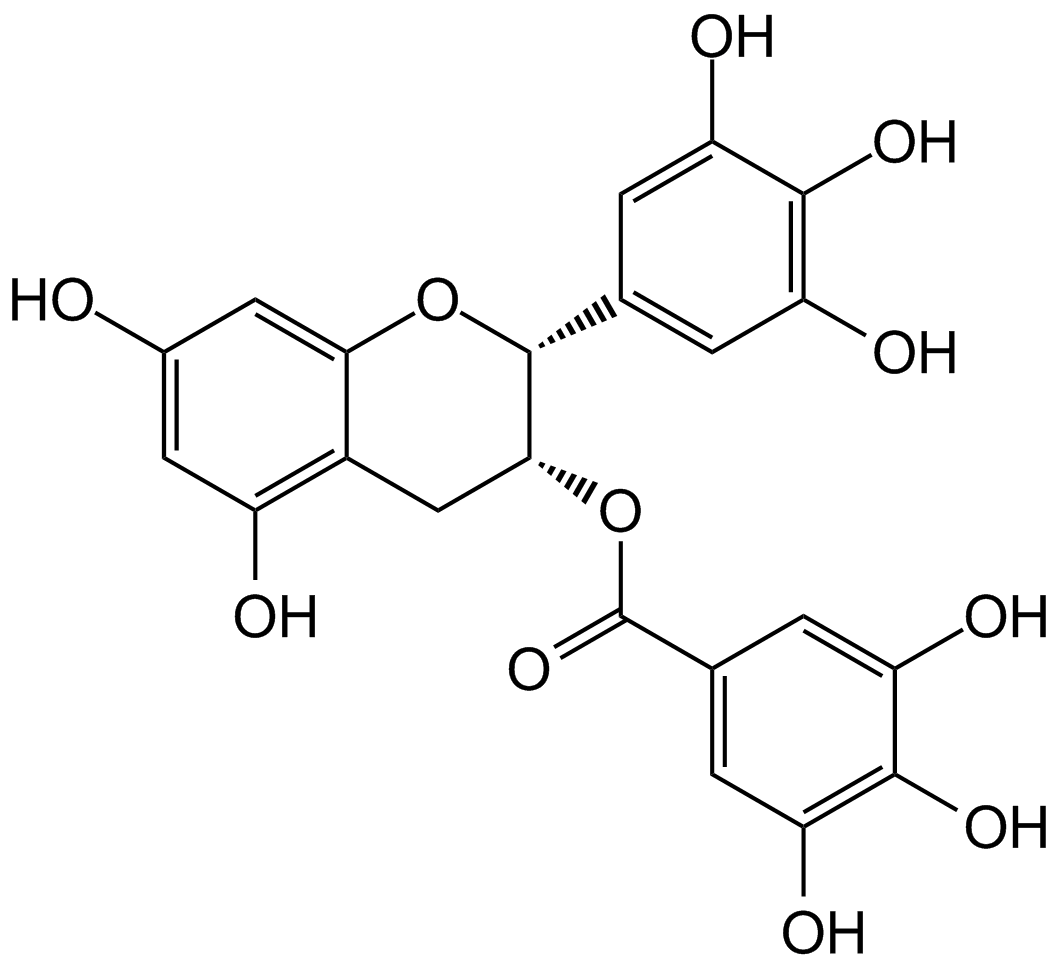
GC14049
(-)-Epigallocatechin gallate (EGCG)
Un fenol con diversas actividades biológicas.
-
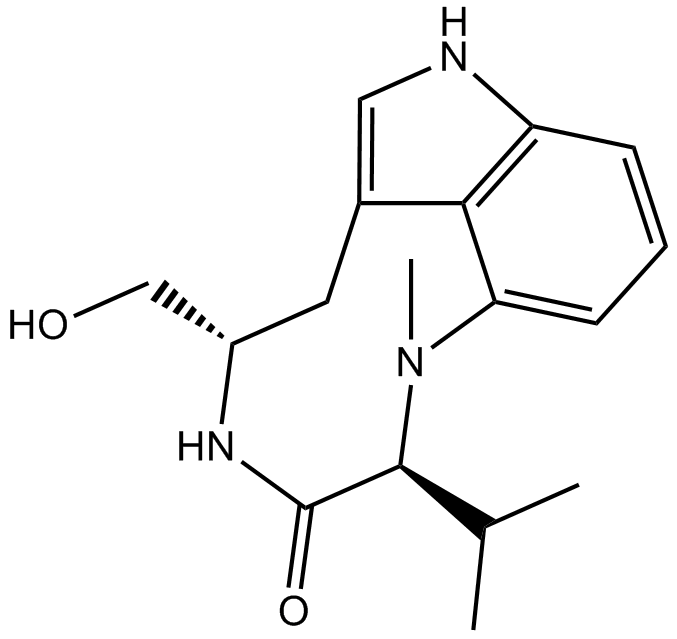
GC14012
(-)-Indolactam V
(-)-Indolactama V es un activador de PKC, con Kis de 3,36 nM, 1,03 μM para η-CRD2 (péptido sustituto de PKCη), γ-CRD2 (péptido sustituto de PKCγ) y Kds de 5,5 nM (η-C1B), 7,7 nM (ε-C1B), 8,3 nM (δ-C1B), 18,9 nM (β-C1A-largo), 20,8 nM (α-C1A-largo), 137 nM (β-C1B), 138 nM (γ-C1A) , 213 nM (γ-C1B), y tiene actividad antitumoral.
-
GC18062
1,2-Dilauroyl-sn-glycerol
El 1,2-dilauroil-sn-glicerol es un diacilglicerol saturado y puede desempeÑar un papel en la transducciÓn de seÑales de segundos mensajeros.

-
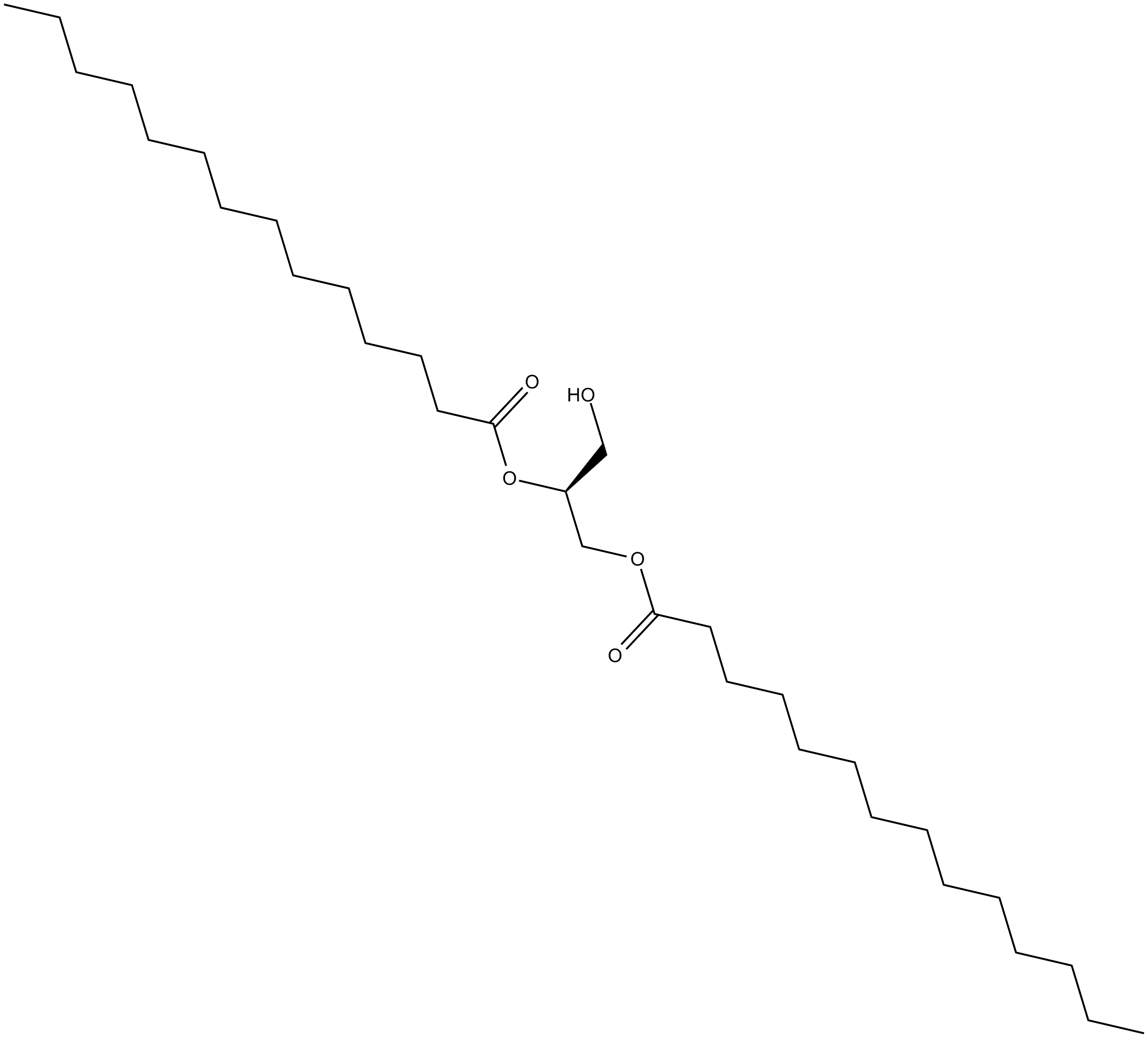
GC14134
1,2-Dimyristoyl-sn-glycerol
El 1,2-dimiristoil-sn-glicerol es un diacilglicerol saturado y un segundo mensajero débil para la activaciÓn de la PKC.
-
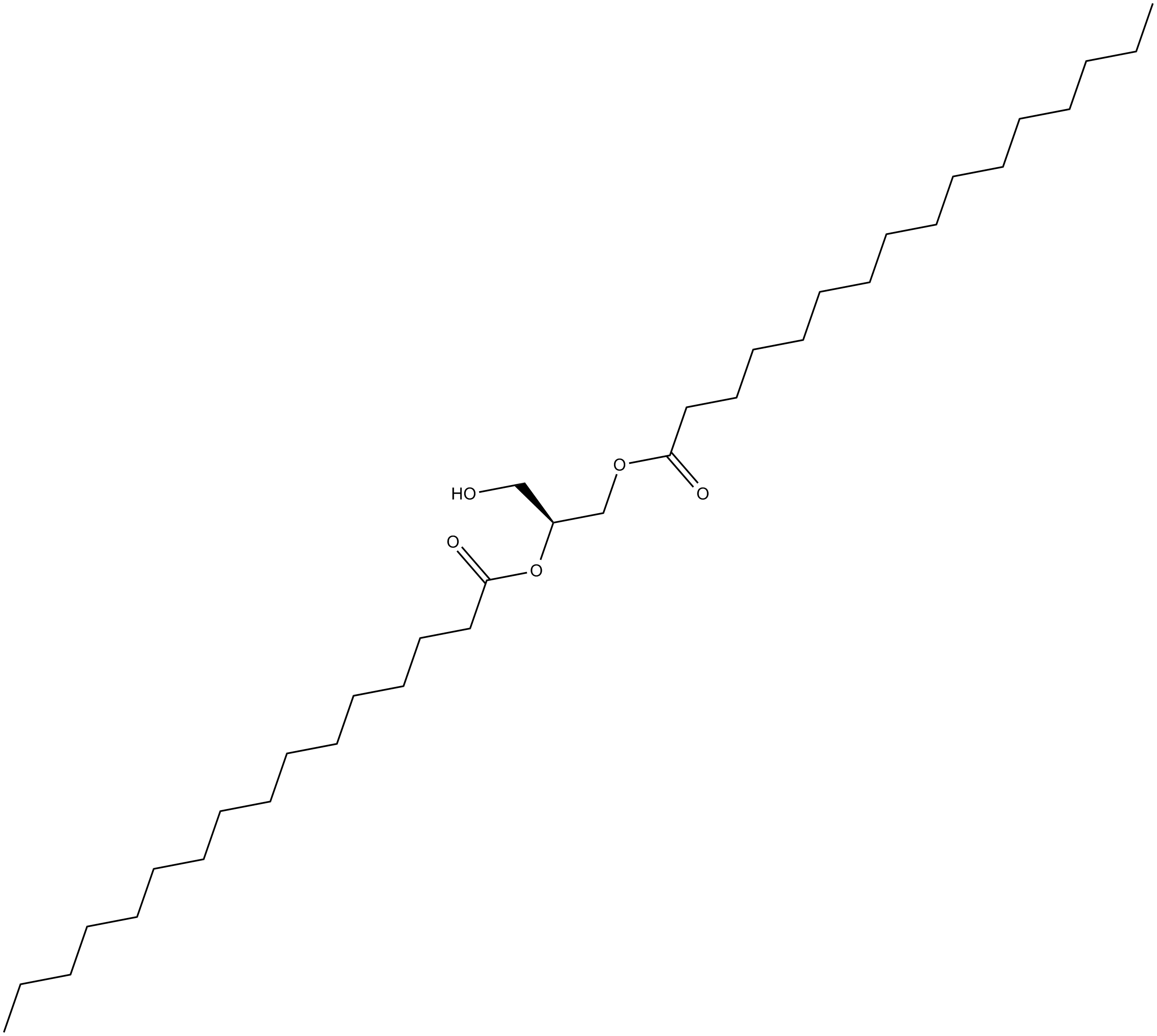
GC13877
1,2-Dipalmitoyl-sn-glycerol
El 1,2-dipalmitoil-sn-glicerol es un metabolito endÓgeno.
-
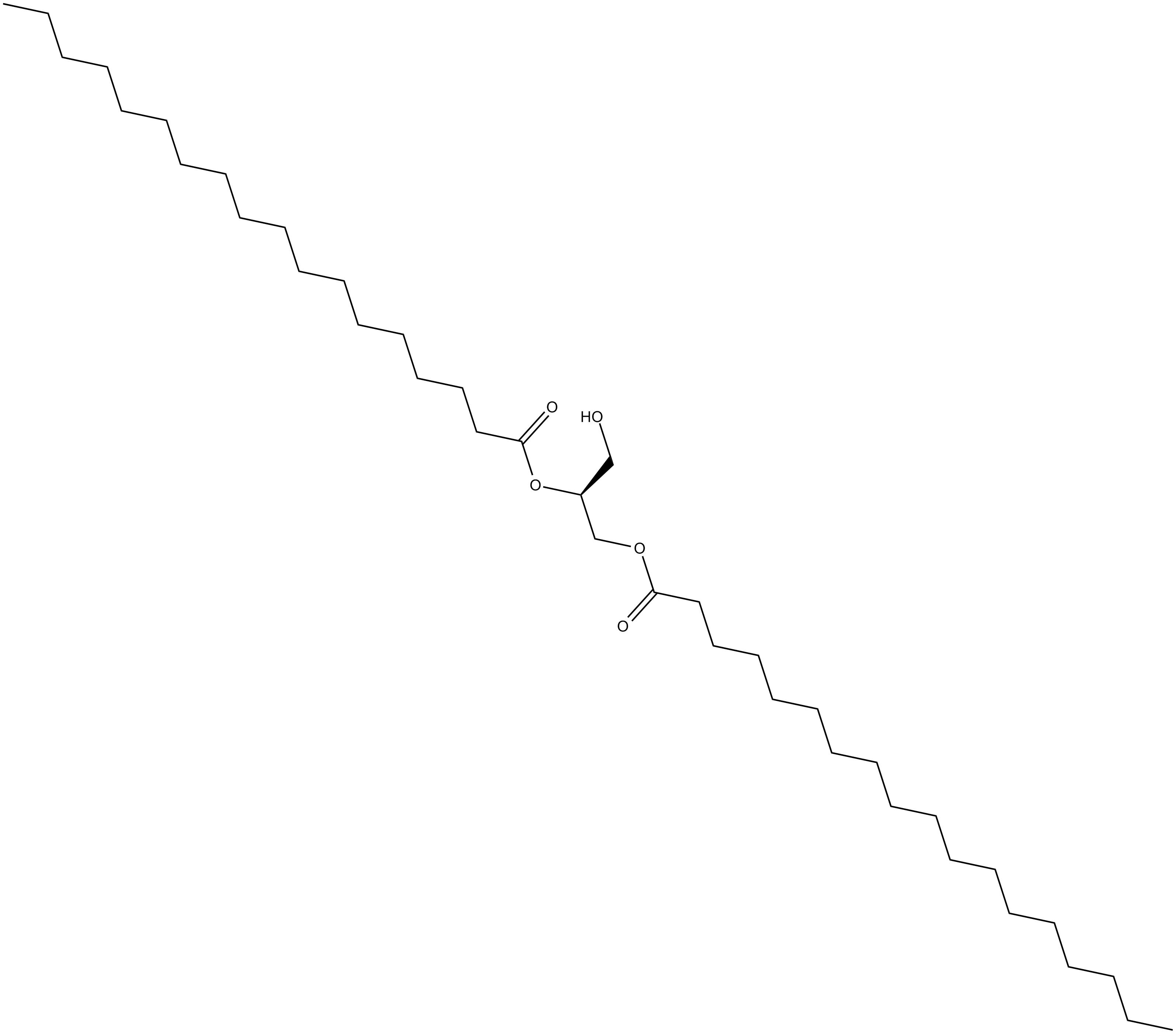
GC12662
1,2-Distearoyl-sn-glycerol
El 1,2-diestearoil-sn-glicerol (DSG; 1,2-dioctadecanoil-sn-glicerol) actÚa como estÁndar interno para la separaciÓn e identificaciÓn de especies moleculares de 1,2-diacil-sn-glicerol (DAG) .
-
GN10444
12-O-tetradecanoyl phorbol-13-acetate
Un activador de PKC
-
GC60037
A-3 hydrochloride
El clorhidrato de A-3 es un potente antagonista no selectivo de varias quinasas, permeable a las células, reversible y competitivo con ATP.
-
GC16475
Afuresertib
Afuresertib (GSK2110183) es un inhibidor pan-Akt quinasa biodisponible por vÍa oral, selectivo, ATP-competitivo y potente con Kis de 0,08/2/2,6 nM para Akt1/Akt2/Akt3, respectivamente.
-
GC42747
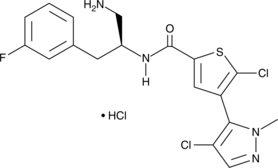
Afuresertib (hydrochloride)
Afuresertib (clorhidrato) (GSK 2110183 clorhidrato) es un inhibidor pan-Akt quinasa biodisponible por vía oral, selectivo, ATP-competitivo y potente con Kis de 0,08/2/2,6 nM para Akt1/Akt2/Akt3 respectivamente.
-
GC31959
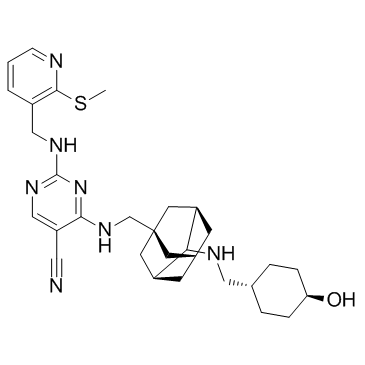
AS2521780
AS2521780 es un nuevo inhibidor selectivo de PKCθ con una IC50 de 0,48 nM.
-
GC64815
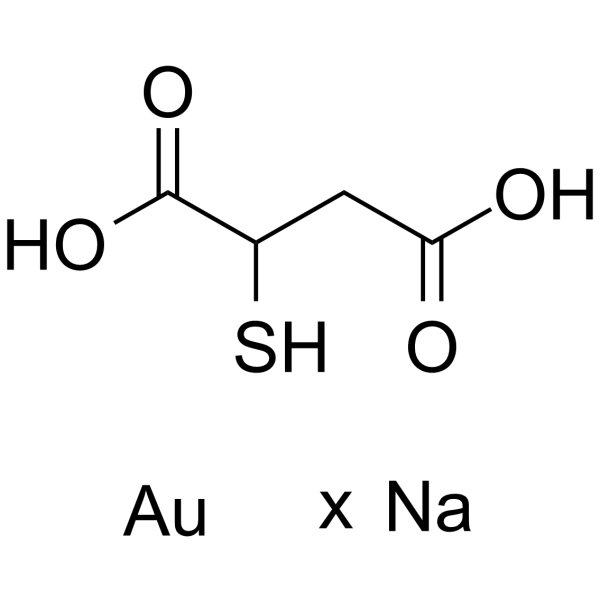
Aurothiomalate sodium
El aurotiomalato de sodio es un potente y selectivo inhibidor oncogénico de la seÑalizaciÓn de PKCΙ.
-
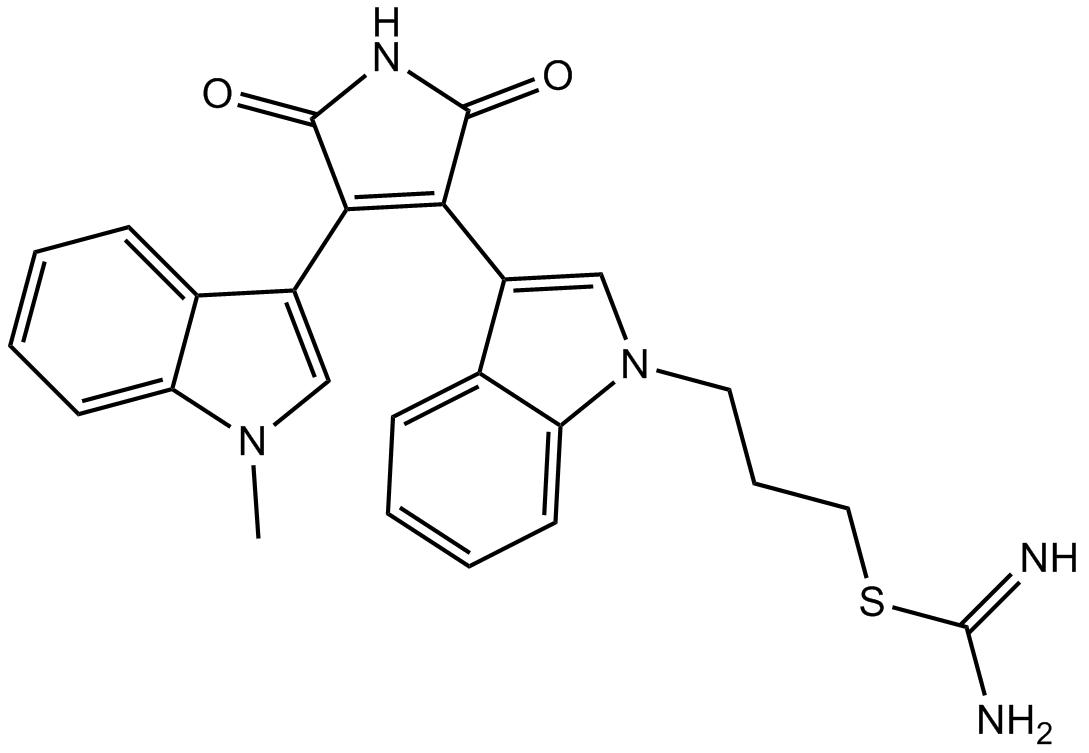
GC12135
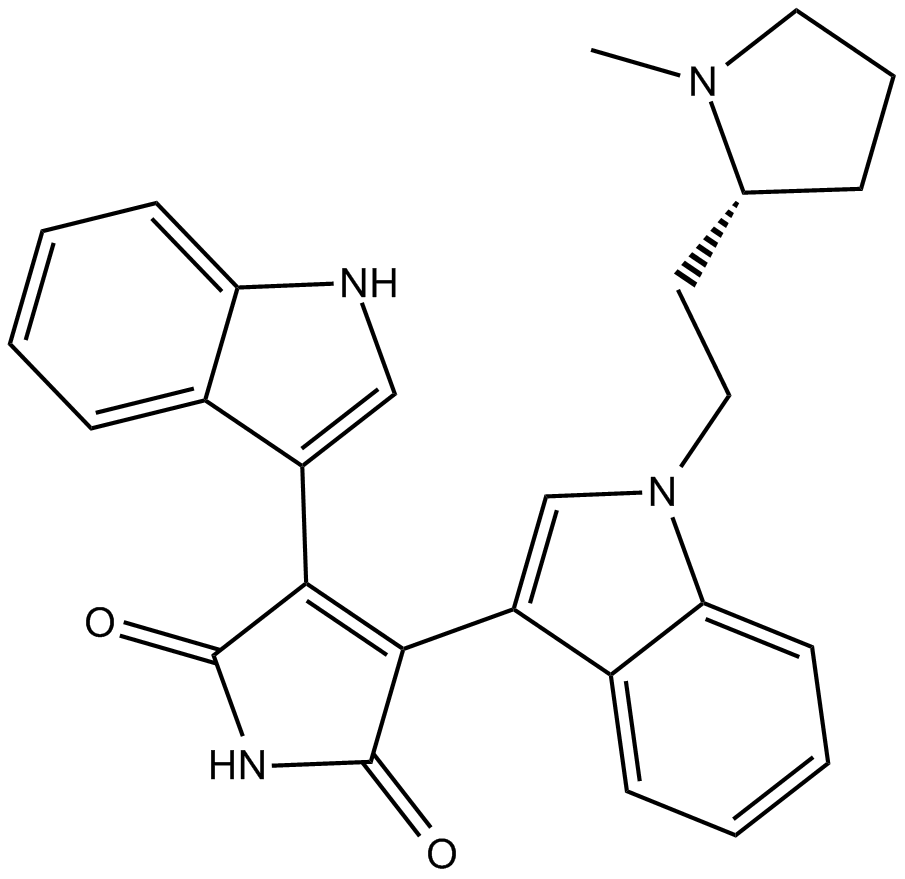
Bisindolylmaleimide II
Bisindolylmaleimide II es un inhibidor general de todos los subtipos de PKC.
-
GC14716
Bisindolylmaleimide IV
La bisindolylmaleimide IV (Arcyriarubin A) es un potente inhibidor de la proteÍna cinasa C (PKC), con IC50 que oscila entre 0,1 y 0,55 μM.
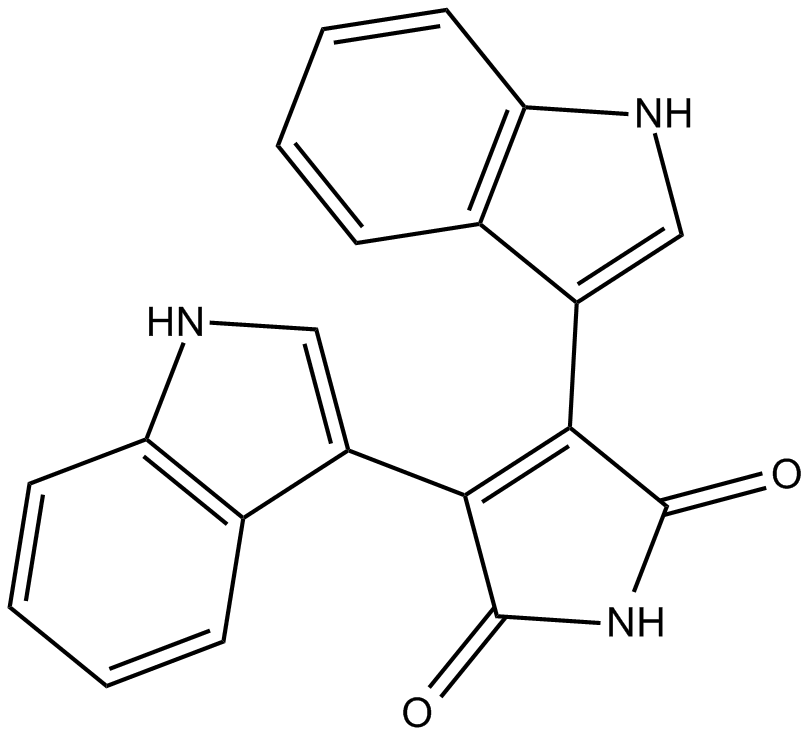
-
GC17239
Bisindolylmaleimide V
Bisindolylmaleimide V es un control negativo permeable a las células para estudios de inhibiciÓn de la proteÍna quinasa C con un valor IC50 superior a 100 μM. Bisindolylmaleimide V bloquea la activaciÓn de la proteÍna quinasa p70s6k/p85s6k (S6K) estimulada por mitÓgenos in vivo con una IC50 de 8 μM.
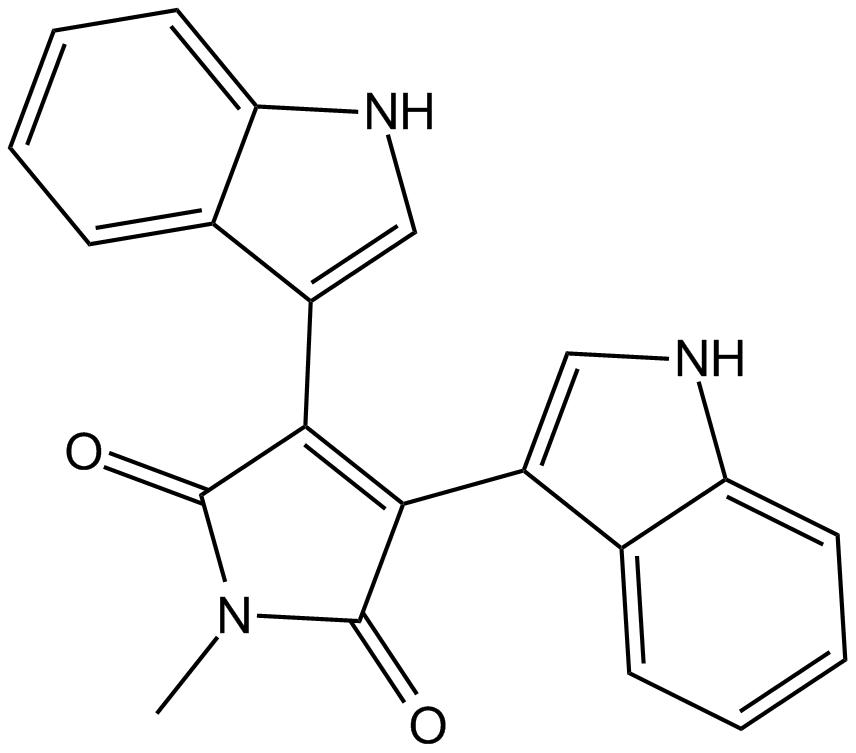
-
GC13226
Bisindolylmaleimide VIII (acetate)
La bisindolilmaleimida VIII (acetato) (acetato de Ro 31-7549) es un inhibidor potente y selectivo de la proteína quinasa C (PKC) con una CI50 de 158 nM para la PKC de cerebro de rata.
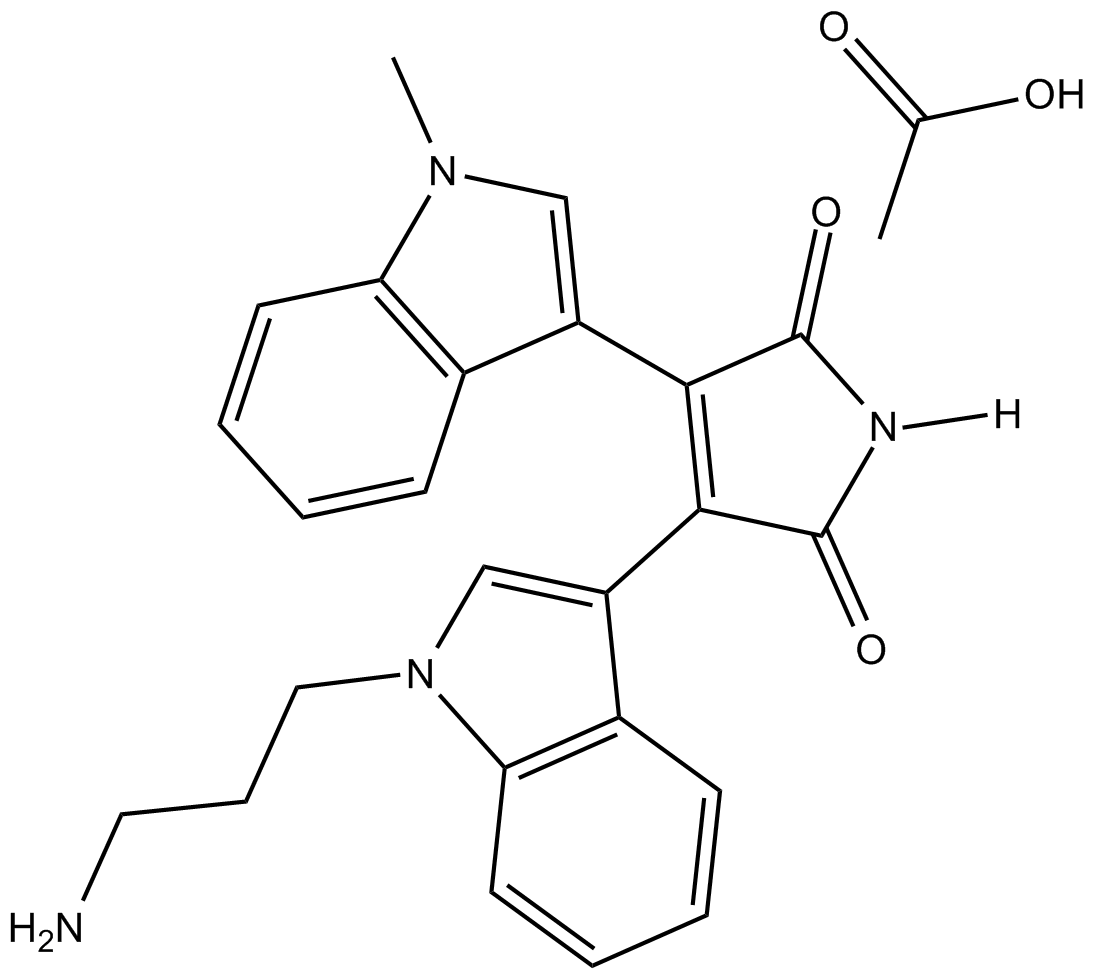
-
GC18354
Bisindolylmaleimide X (hydrochloride)
La bisindolilmaleimida X (clorhidrato) (BIM-X clorhidrato) es un inhibidor potente y selectivo de la proteína quinasa C (PKC).
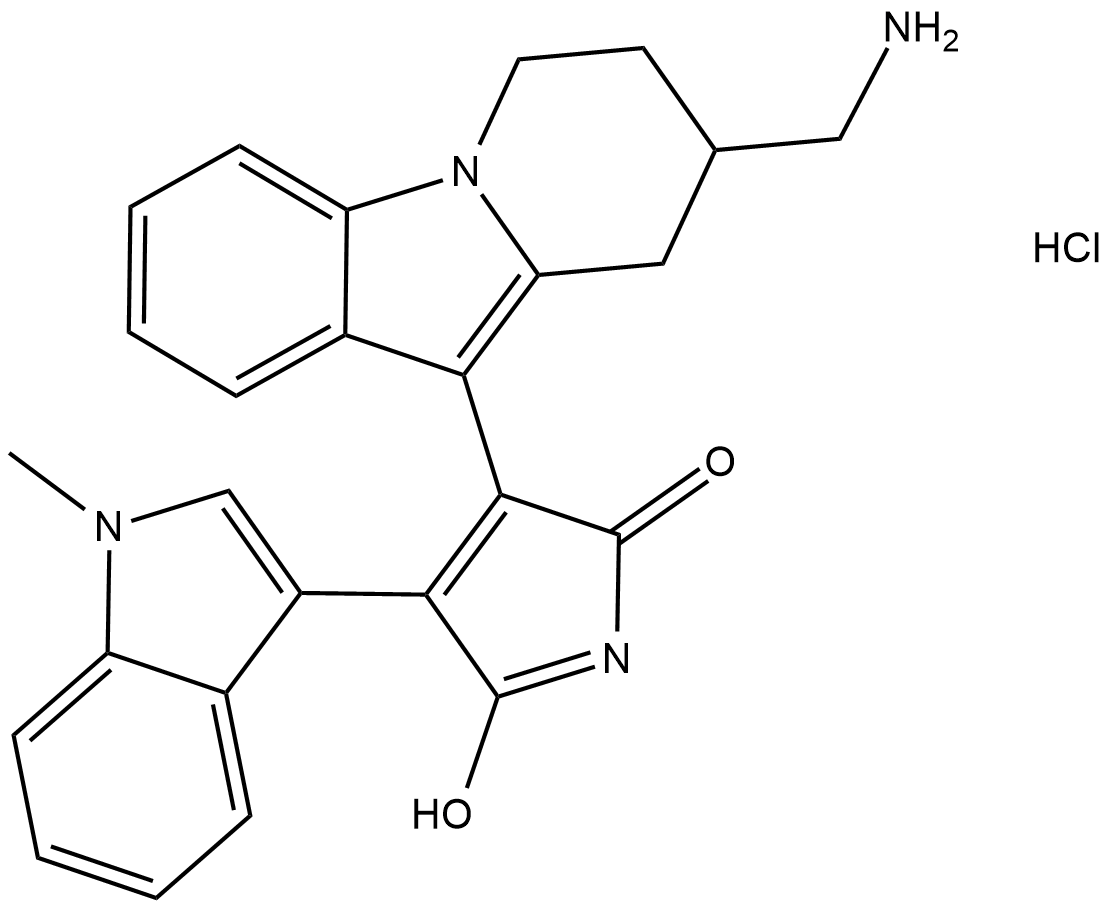
-
GC35530
BJE6-106
BJE6-106 (B106) es un potente inhibidor selectivo de PKCδ de tercera generaciÓn con una IC50 de 0,05 μM y una selectividad de objetivos sobre la isoenzima PKCα clÁsica de la PKC (IC50 = 50 μM).

-
GC17670
Bryostatin 1
La briostatina 1 es un macrÓlido natural aislado del briozoo Bugula neritina y es un modulador PKC potente y permeable al sistema nervioso central (SNC).
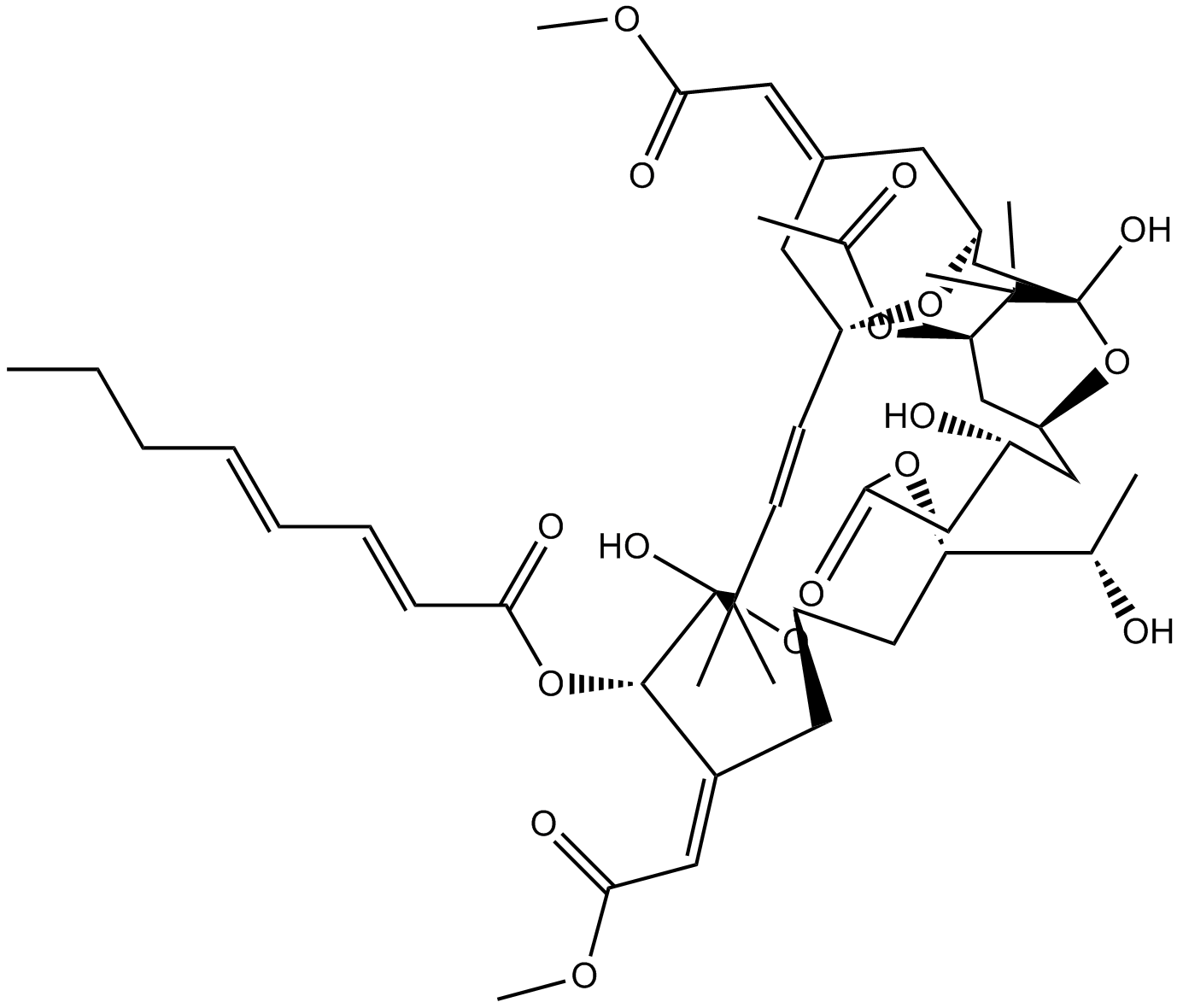
-
GC12842
Bryostatin 2
Protein kinase C (PKC) activator
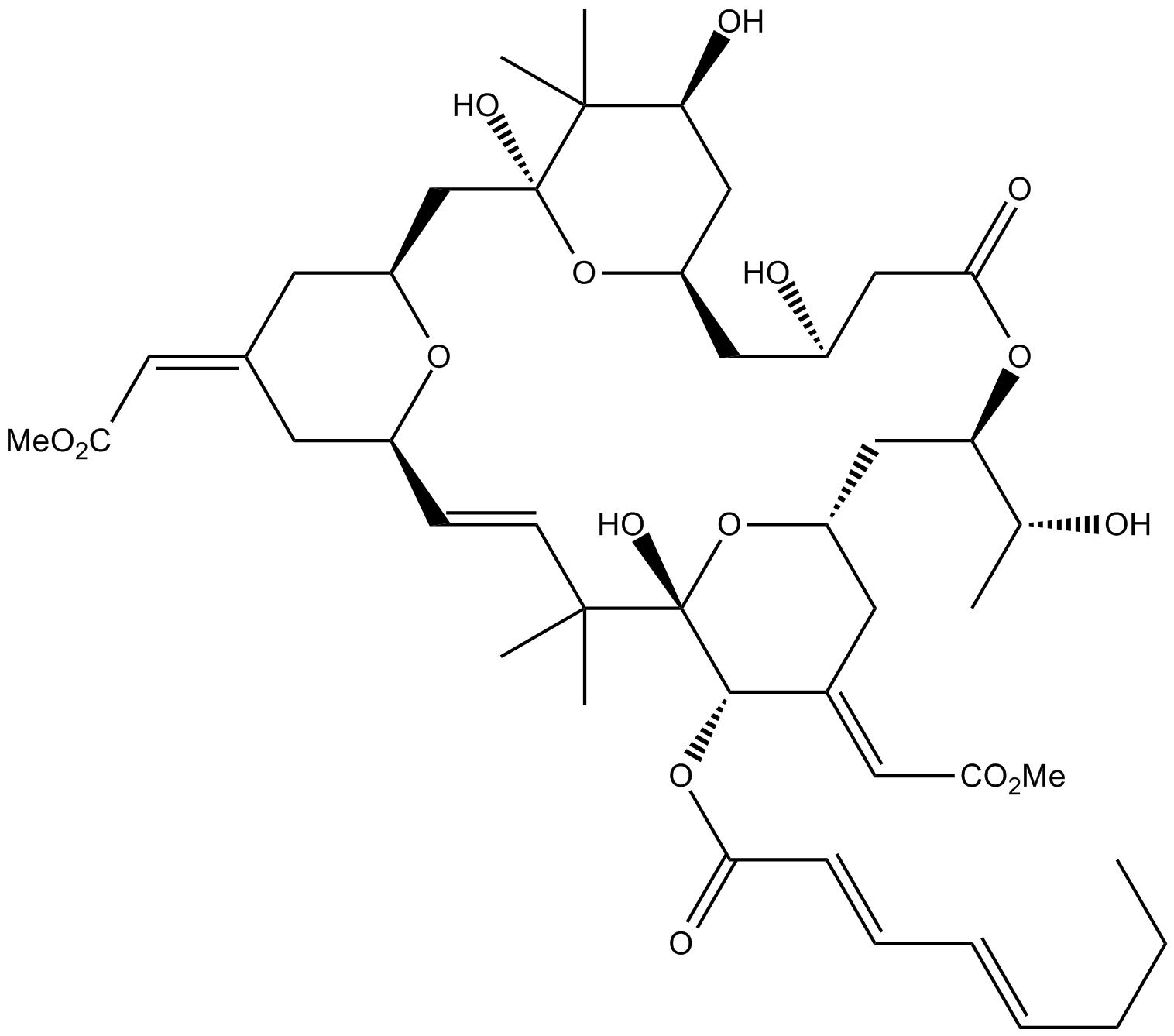
-
GC12695
Bryostatin 3
La briostatina 3, una lactona macrocÍclica, es un activador de la proteÍna quinasa C, con una Ki de 2,75 nM. La briostatina 3 puede bloquear la inhibiciÓn de la proliferaciÓn celular por 12-O-tetradecanoilforbol-13-acetato (TPA), pero no bloqueÓ la adhesiÓn del sustrato celular potenciada por TPA.
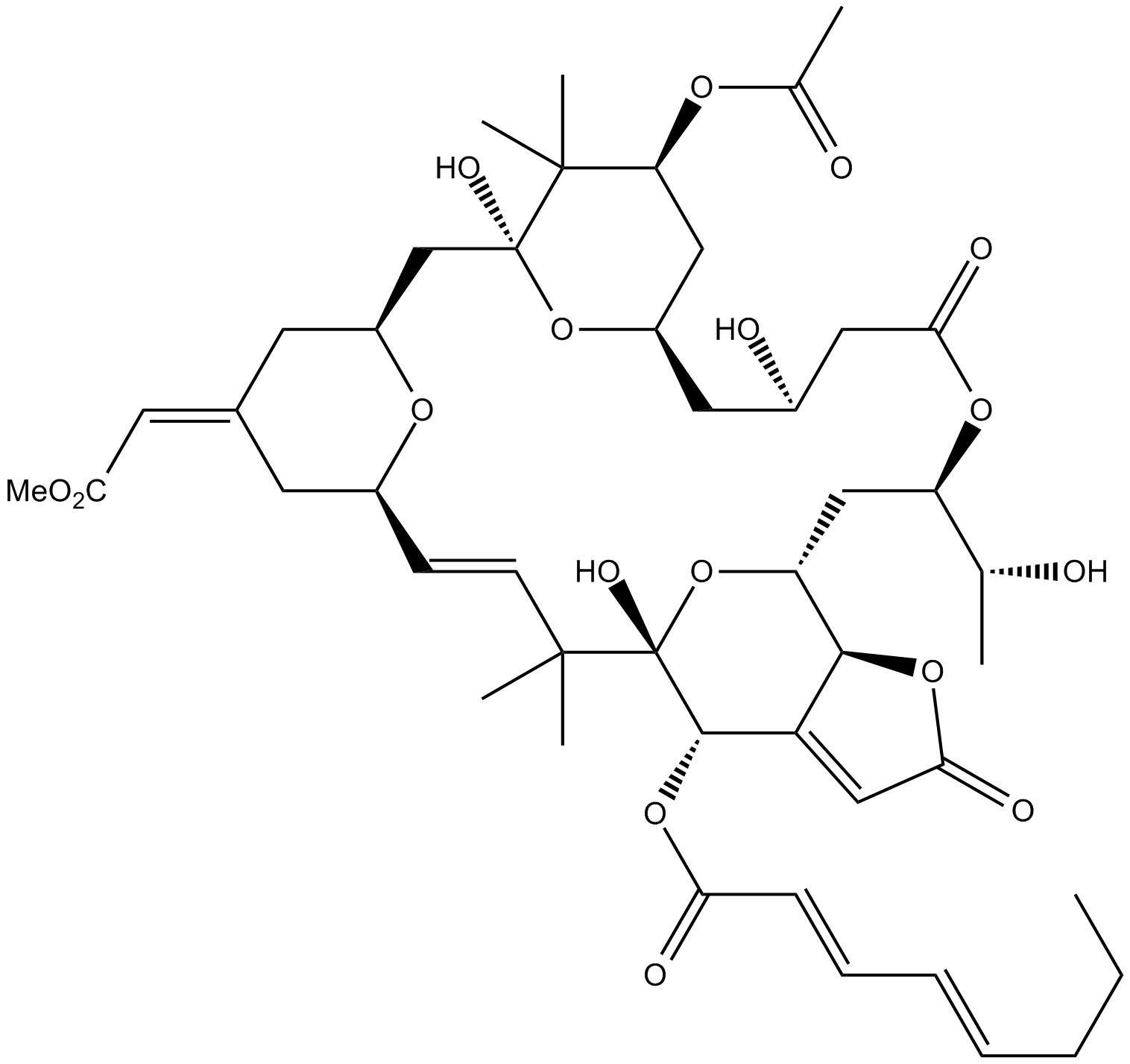
-
GC11605
C-1
C-1 es un potente inhibidor de la proteÍna quinasa, con IC50 de 4 μM, 8 μM, 12 μM y 240 μM para proteÍna quinasa dependiente de cGMP (PKG), proteÍna quinasa dependiente de cAMPPKA , proteÍna quinasa C (PKC) y MLC-quinasa, respectivamente. C-1 también se usa como inhibidor de ROCK.

-
GC43105
C8 Ceramide (d18:1.8:0)
C8 Ceramida (d18:1.8:0) (N-Octanoyl-D-eritro-esfingosina) es un análogo permeable a la célula de ceramidas que ocurren naturalmente.
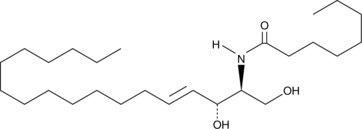
-
GC11159
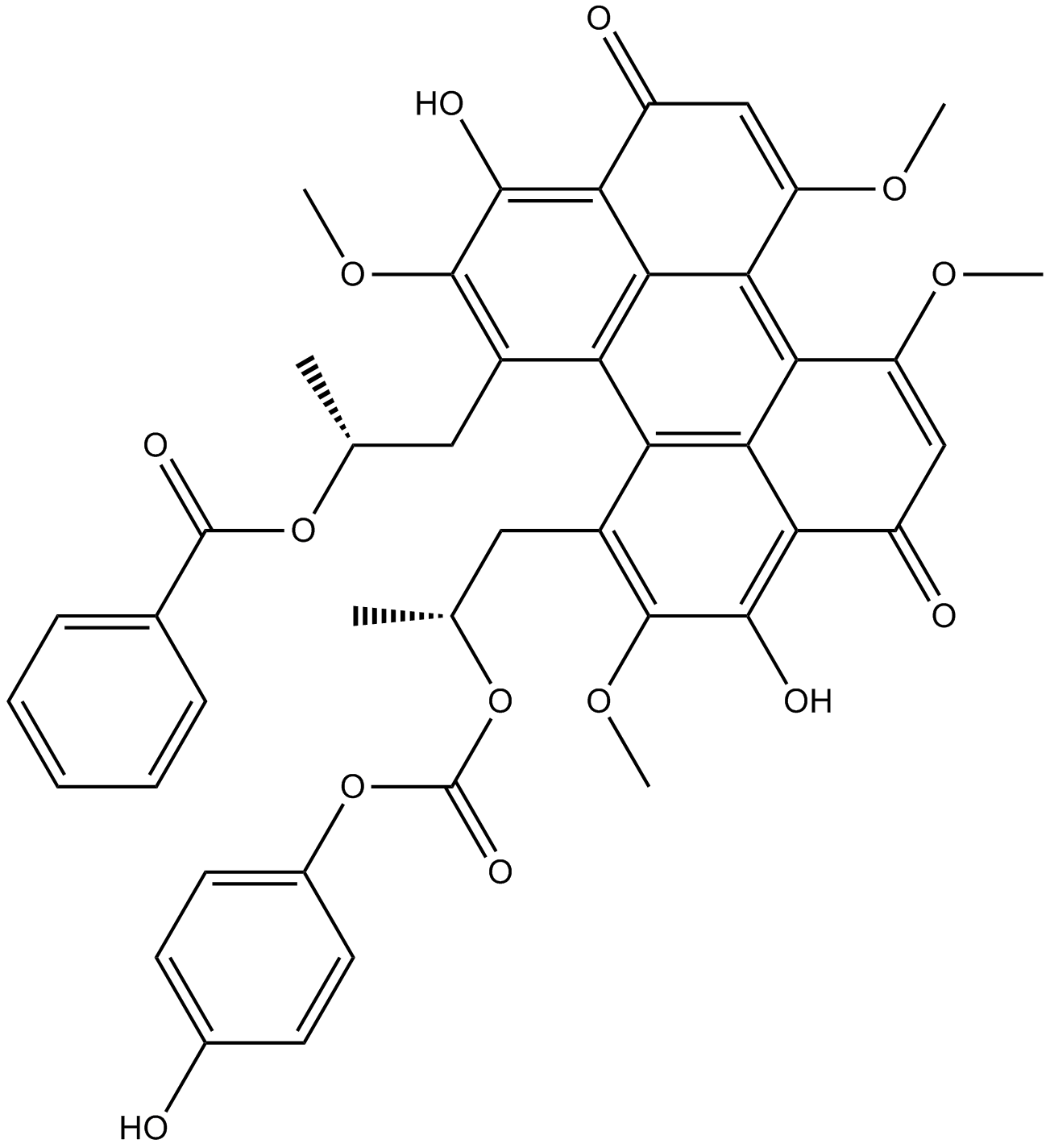
Calphostin C
La calfostina C es un inhibidor potente y especÍfico de la proteÍna quinasa C.
-
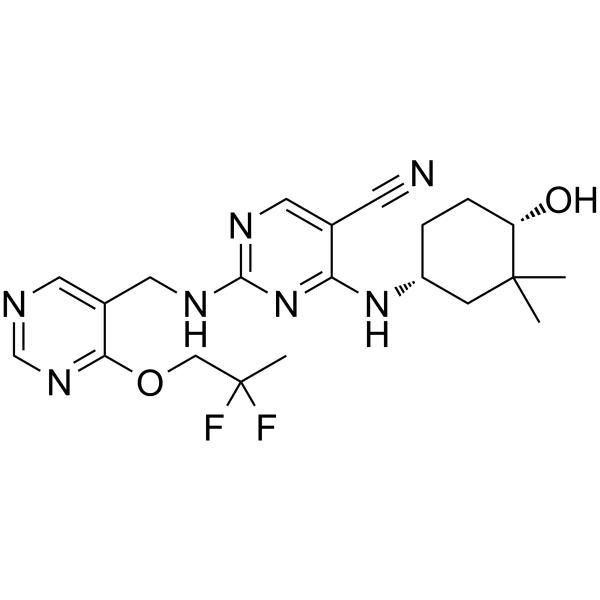
GC62612
CC-90005
CC-90005 es un inhibidor potente, selectivo y activo por vÍa oral de la proteÍna quinasa C-θ (PKC-θ), con una IC50 de 8 nM.
-
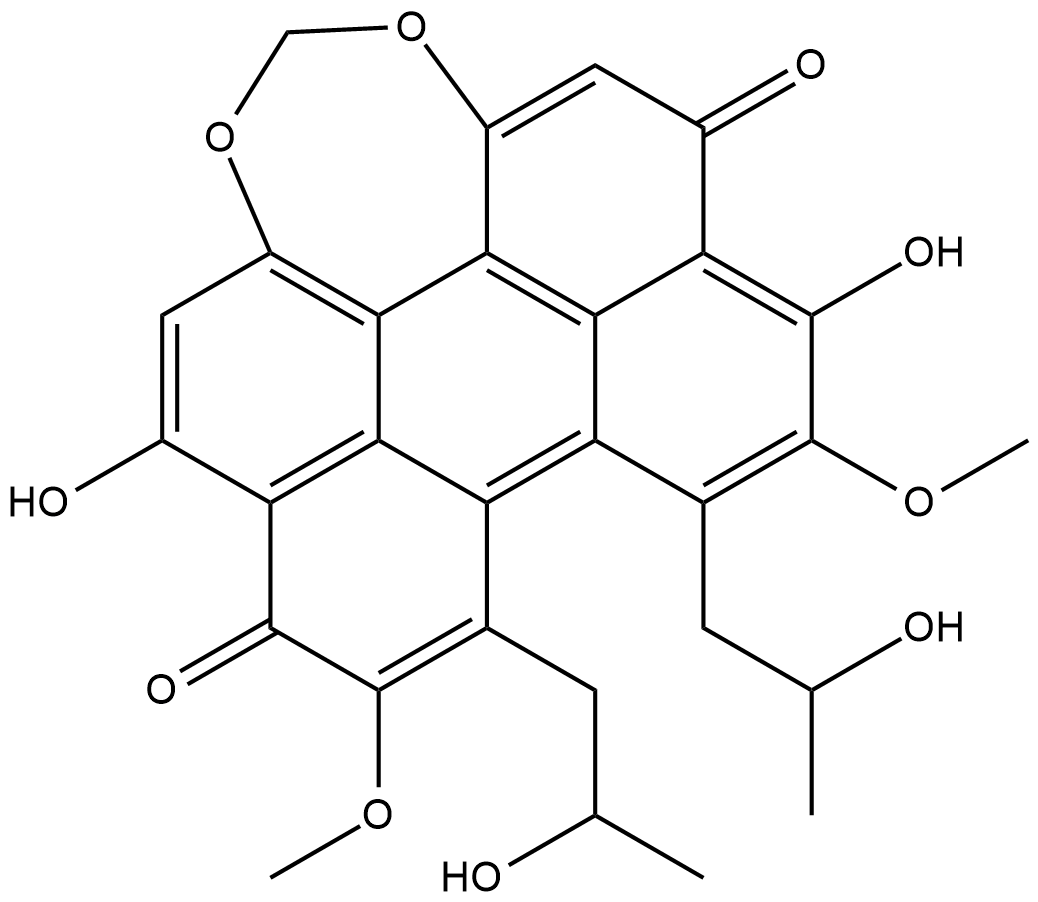
GC18521
Cercosporin
La cercosporina es producida por un patÓgeno vegetal, Cercosporakikuchii, y los elsinocromos, pigmentos de la familia de hongos elsinoe. La cercosporina es un fotosensibilizador potente con una longitud de onda de activaciÓn corta, principalmente adecuado para tratamientos de terapia fotodinÁmica superficial (TFD), especialmente cuando es necesario evitar perforaciones. La cercosporina contiene las caracterÍsticas estructurales de la perilenoquinona necesarias para la actividad de la PKC con una IC50 de 0,6-1,3 μM.
-
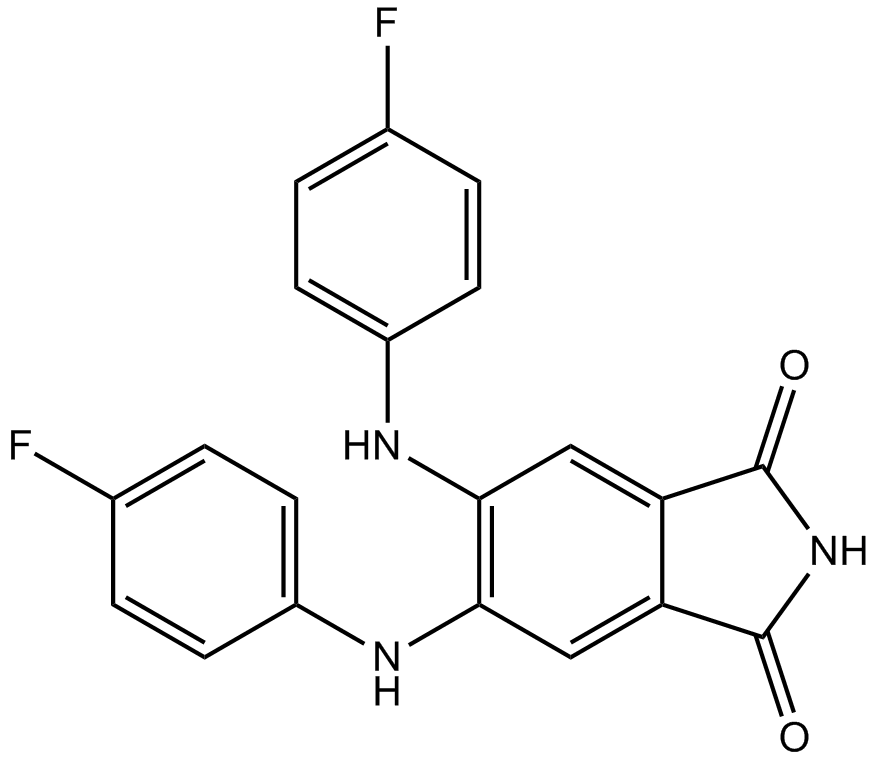
GC10687
CGP 53353
CGP 53353 (DAPH-7) es un potente inhibidor de PKC con IC50 de 0,41 mM y 3,8 mM para PKCβII y PKCβI, respectivamente.
-
GC14650
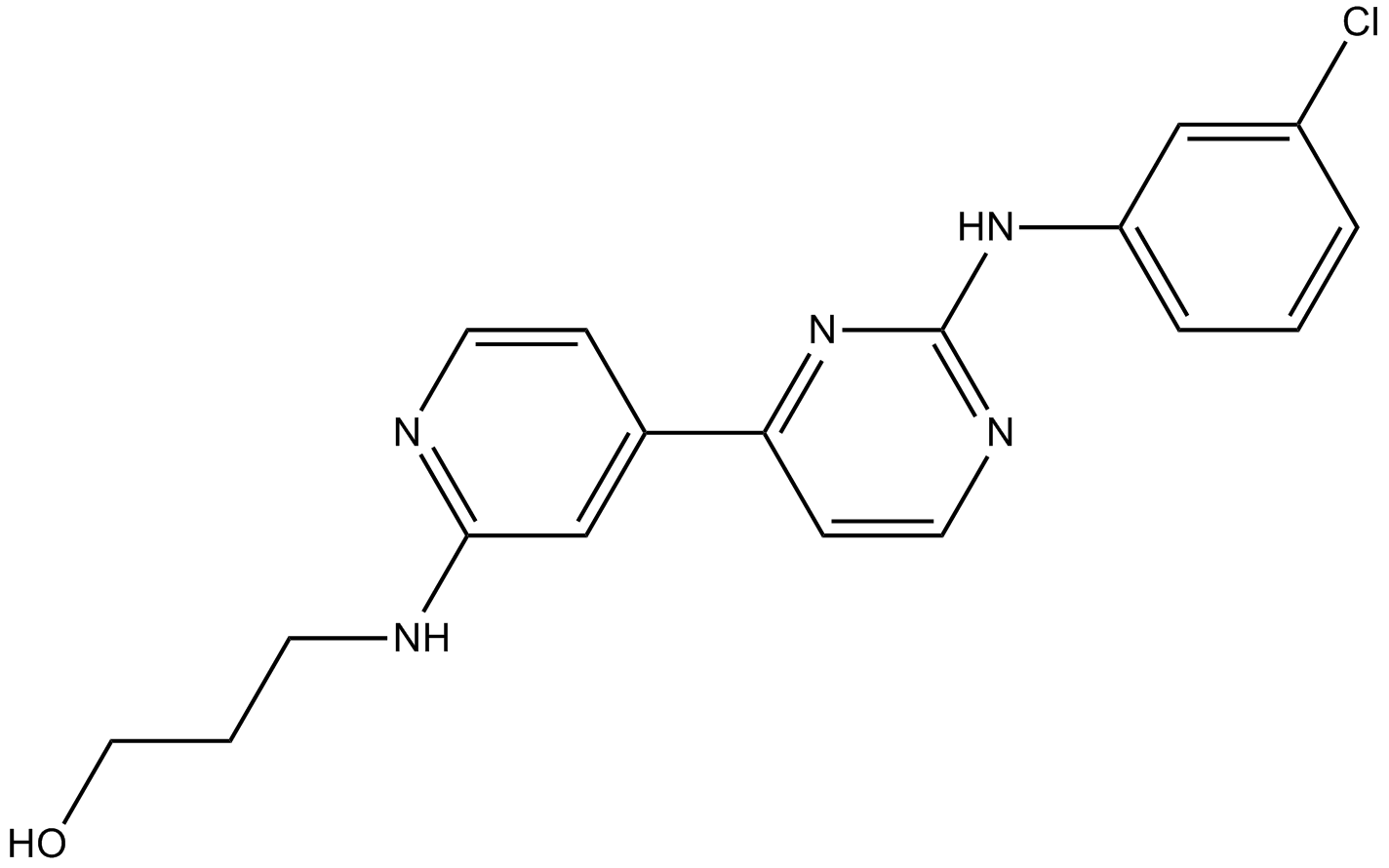
CGP60474
A CDK inhibitor
-
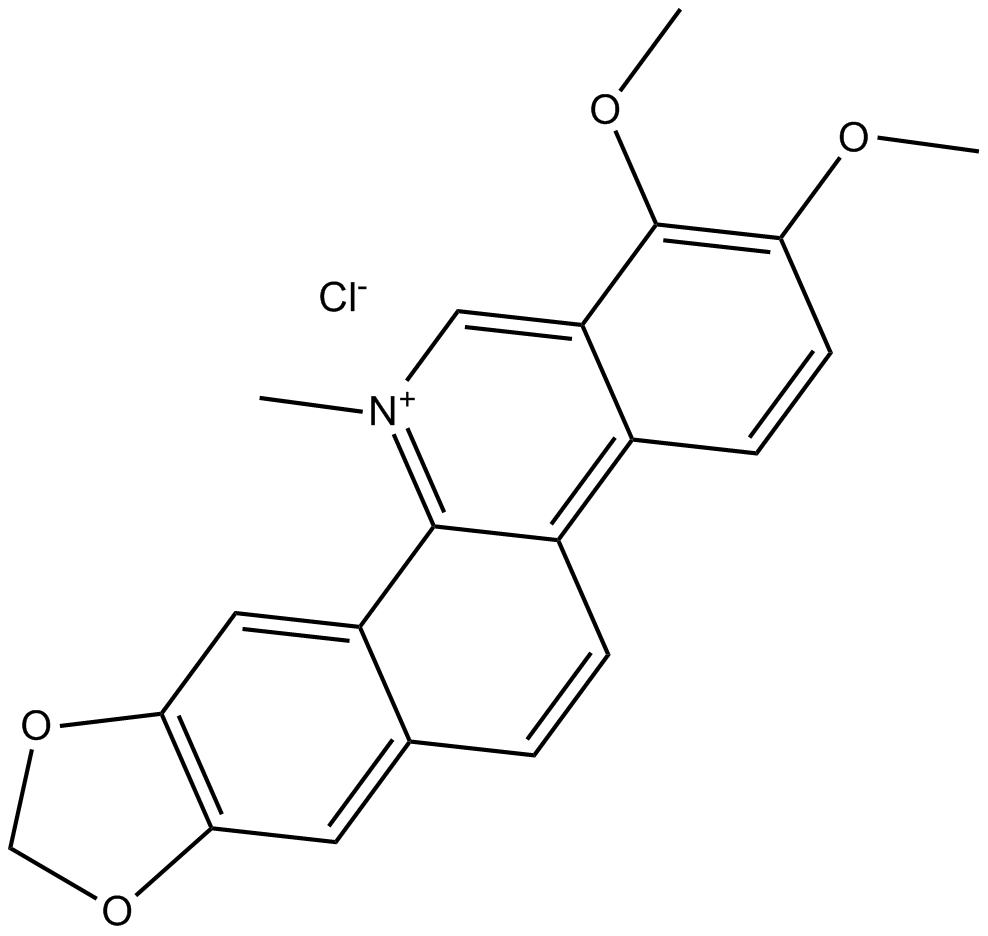
GN10463
Chelerythrine
-
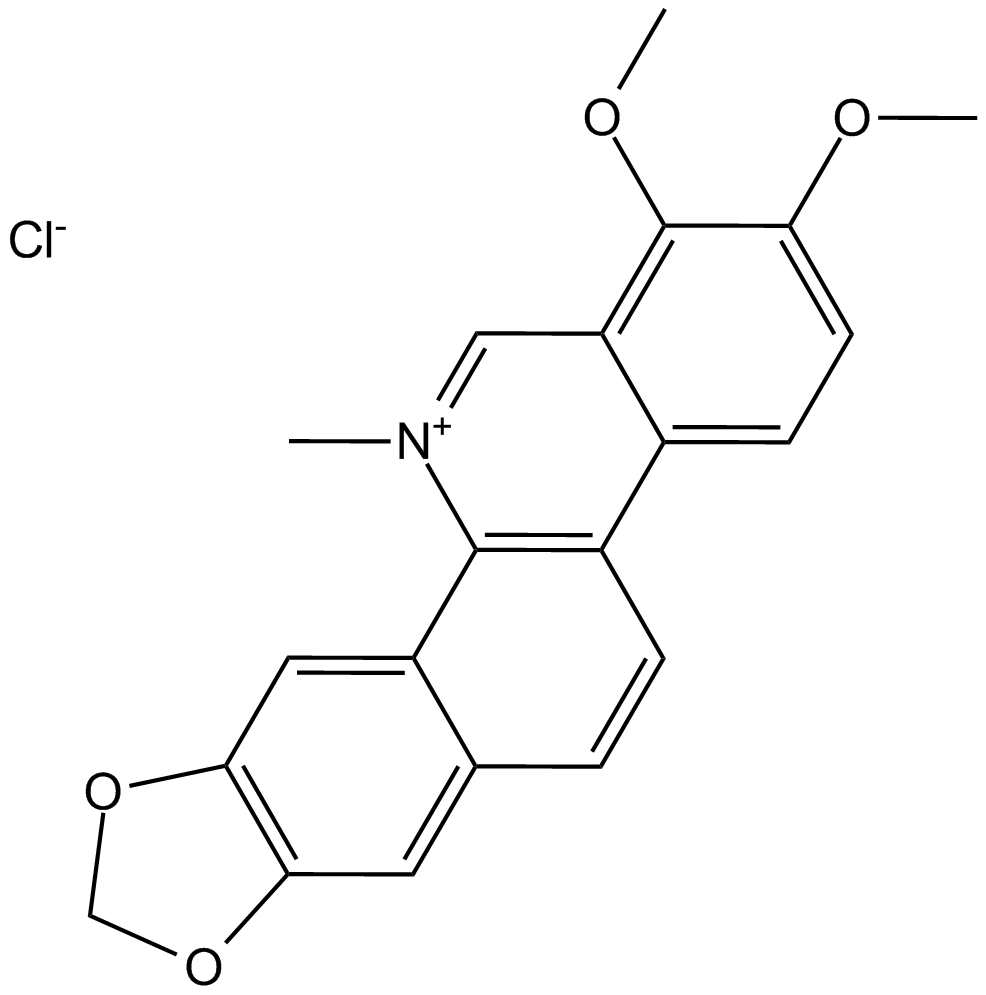
GC13065
Chelerythrine Chloride
Potent inhibitor of PKC and Bcl-xL
-
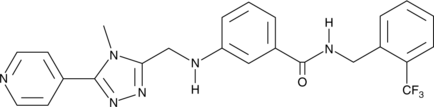
GC43286
CMPD101
CMPD101 es un inhibidor de molécula pequeÑa potente, altamente selectivo y permeable a la membrana de GRK2/3 con IC50 de 18 nM y 5,4 nM, respectivamente.
-
GC50704
CRT 0066854 hydrochloride
El clorhidrato de CRT 0066854 es un inhibidor potente y selectivo de las PKC atípicas.
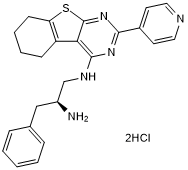
-
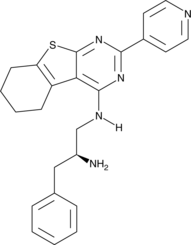
GC45414
CRT0066854
CRT0066854 es un potente y selectivo inhibidor de las isoenzimas PKC atÍpicas.
-
GC17591
D-erythro-Sphingosine (synthetic)
D-erythro-sphingosine (Erythrosphingosine) es un activador de quinasa eficaz de p32.

-
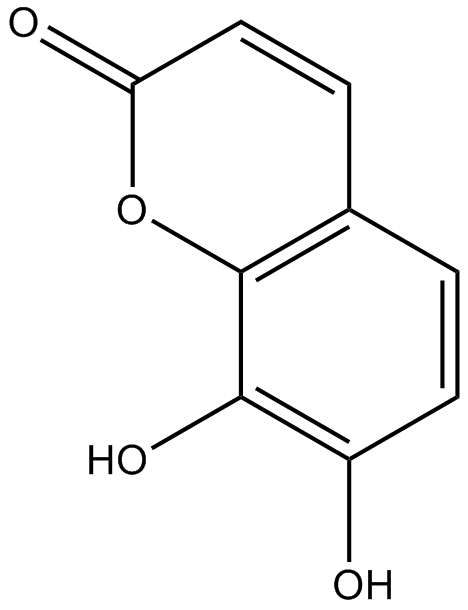
GN10336
Daphnetin
-
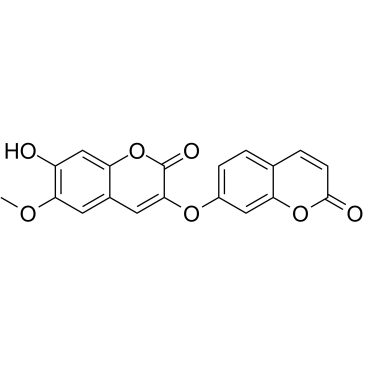
GC38186
Daphnoretin
La dafnoretina (Dephnoretin), aislada de Wikstroemia indica, posee actividad antiviral.
-
GC38388
DCPLA-ME
DCPLA-ME, la forma de éster metÍlico de DCPLA, es un potente activador de PKCε para su uso en el tratamiento de enfermedades neurodegenerativas.

-
GC31892
Decursin ((+)-Decursin)
Decursin ((+)-Decursin) ((+)-Decursin ((+)-Decursin)) es un potente agente antitumoral.
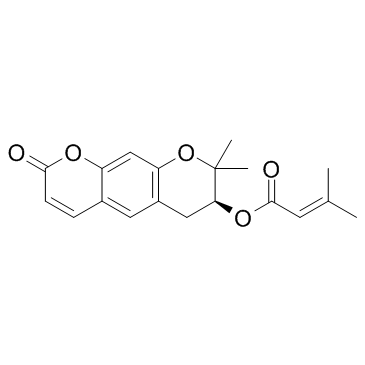
-
GC38085
Decursinol angelate
El angelato de decursinol, un agente citotÓxico y activador de la proteÍna quinasa C (PKC) de la raÍz de Angelica gigas, posee actividades antitumorales y antiinflamatorias.
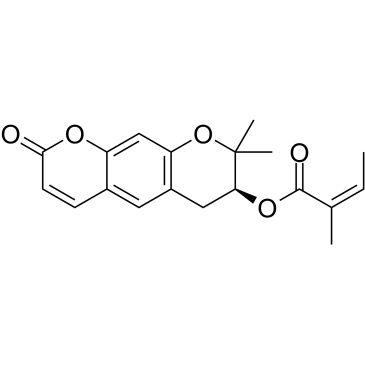
-
GC16354
Dequalinium Chloride
El cloruro de decualinio es un bloqueador selectivo de los canales K+ sensibles a la apamina.

-
GC38482
Desmethylglycitein
DesmetilgliciteÍna (4',6,7-trihidroxiisoflavona), un metabolito de la daidzeÍna, procedente de Glycine max con actividades antioxidantes y anticancerÍgenas. La desmetilgliciteÍna se une directamente a CDK1 y CDK2 in vivo, lo que suprime la actividad de CDK1 y CDK2. La desmetilgliciteÍna es un inhibidor directo de la proteÍna quinasa C (PKC) α, contra la metaloproteinasa de matriz 1 (MMP1) inducida por UV solar (sUV). La desmetilgliciteÍna se une a PI3K de manera competitiva con ATP en el citosol, donde inhibe la actividad de PI3K y las cascadas de seÑalizaciÓn aguas abajo, lo que lleva a la supresiÓn de la adipogénesis en los preadipocitos 3T3-L1.

-
GC17767
Dihydrosphingosine
La dihidroesfingosina es un potente inhibidor de la PKC y la fosfolipasa A2 (PLA2).
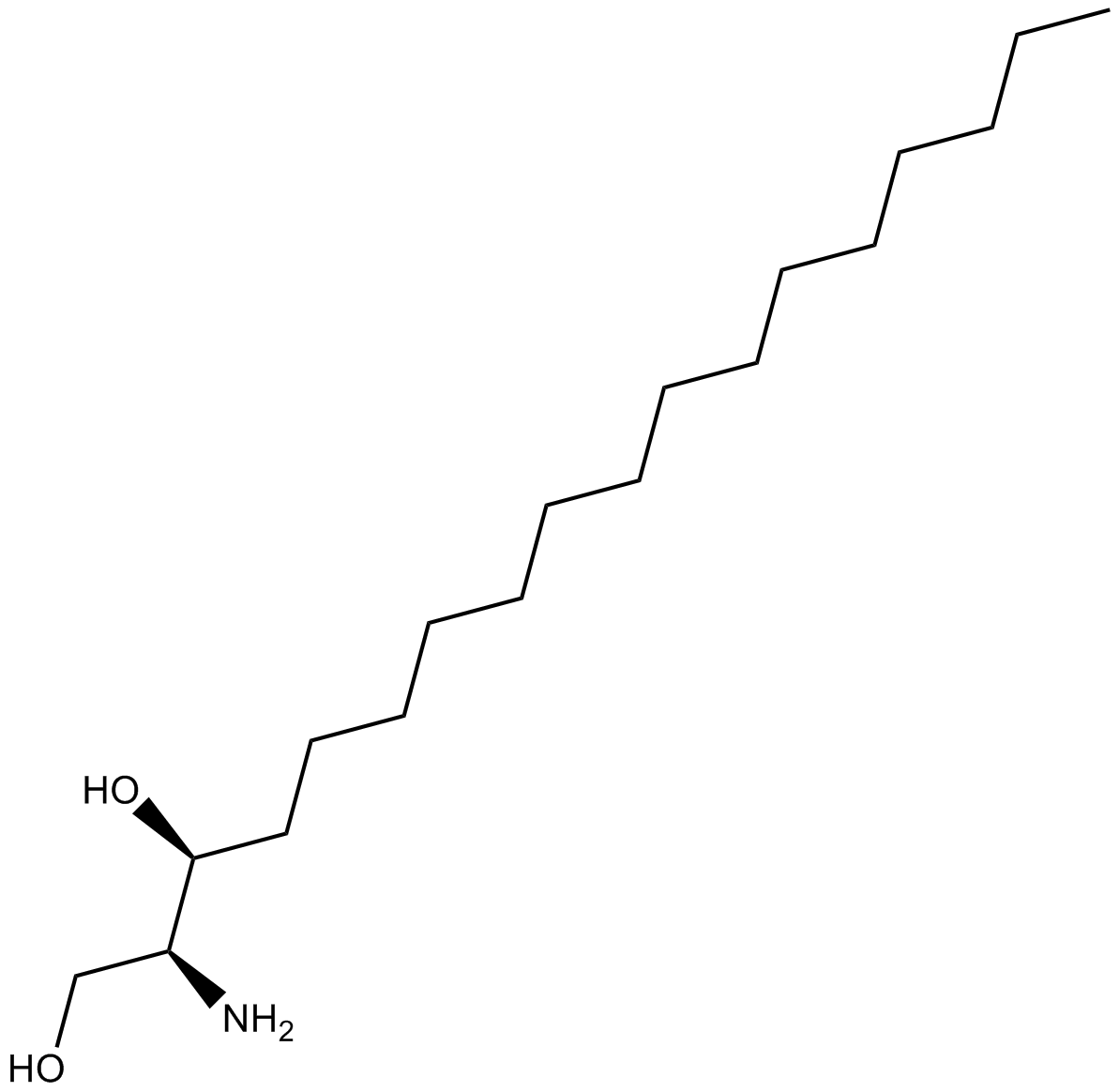
-
GC11499
Enzastaurin (LY317615)
Enzastaurin (LY317615) (LY317615) es un PKCβ potente y selectivo; inhibidor con una IC50 de 6 nM, que muestra una selectividad de 6 a 20 veces sobre PKCα PKCγ y PKCε.
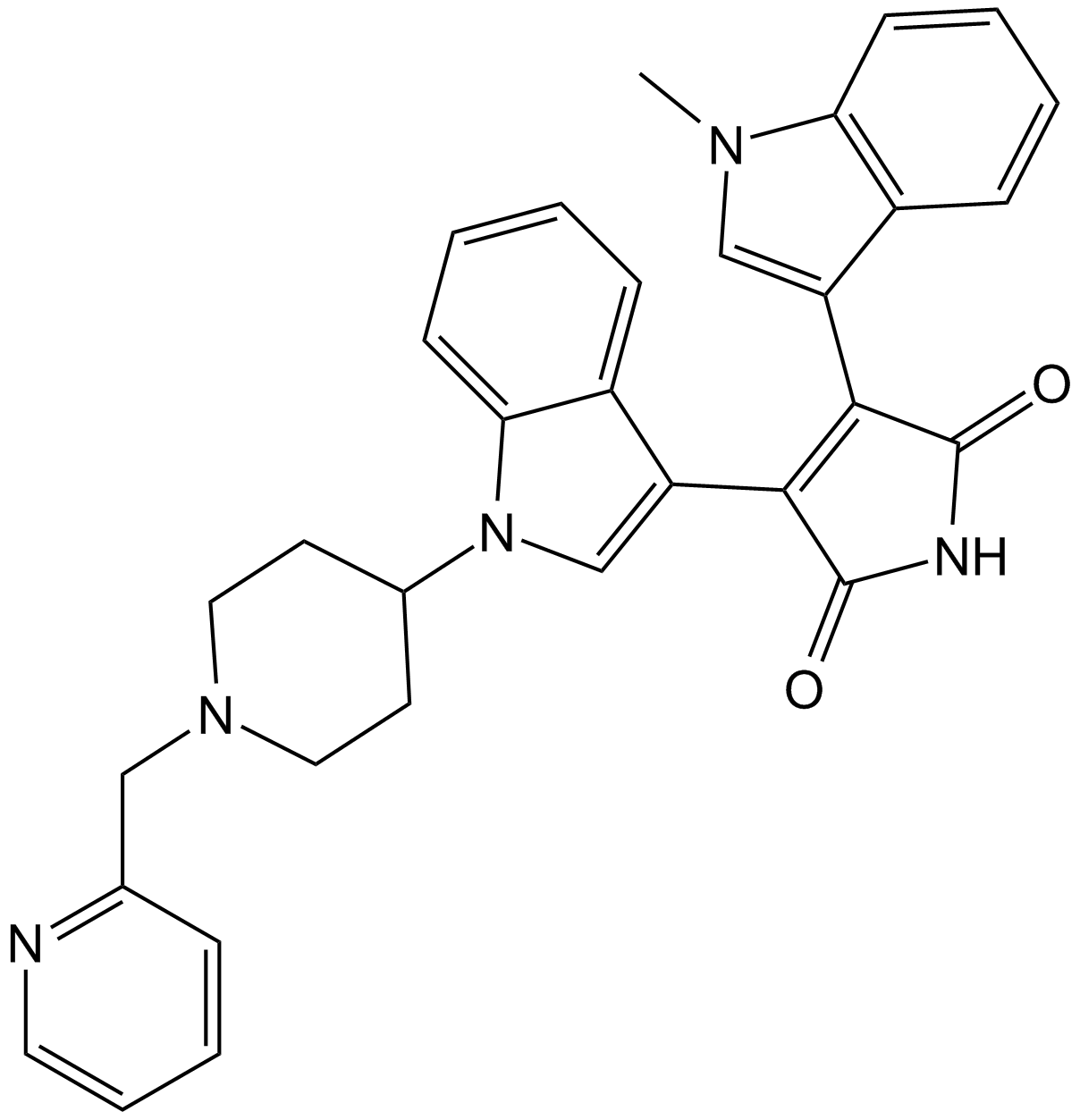
-
GC13869
Fasudil
Fasudil (HA-1077; AT877) es un inhibidor inespecÍfico de RhoA/ROCK y también tiene un efecto inhibitorio sobre las proteÍnas quinasas, con una Ki de 0,33 μM para ROCK1, IC50 de 0,158 μM y 4,58 μM, 12,30 μM, 1,650 μM para ROCK2 y PKA , PKC, PKG, respectivamente. Fasudil es también un potente antagonista y vasodilatador de los canales de Ca2+.

-
GC14289
Fasudil (HA-1077) HCl
Fasudil (HA-1077; AT877) El clorhidrato es un inhibidor rhoa/roca inespecÍfico y también tiene un efecto inhibitorio en las proteÍnas quinasas, con un KI de 0.33 μ m para rock115&&&&7777 que offlineefficient_models_2022q2.md;M, 1.650 μM for ROCK2 and PKA, PKC, PKG, respectively.en_es_2021q4.mdFasudil (HA-1077) HCl is also a potent Ca2+ channel antagonist and vasodilator.en_es_2021q4.md
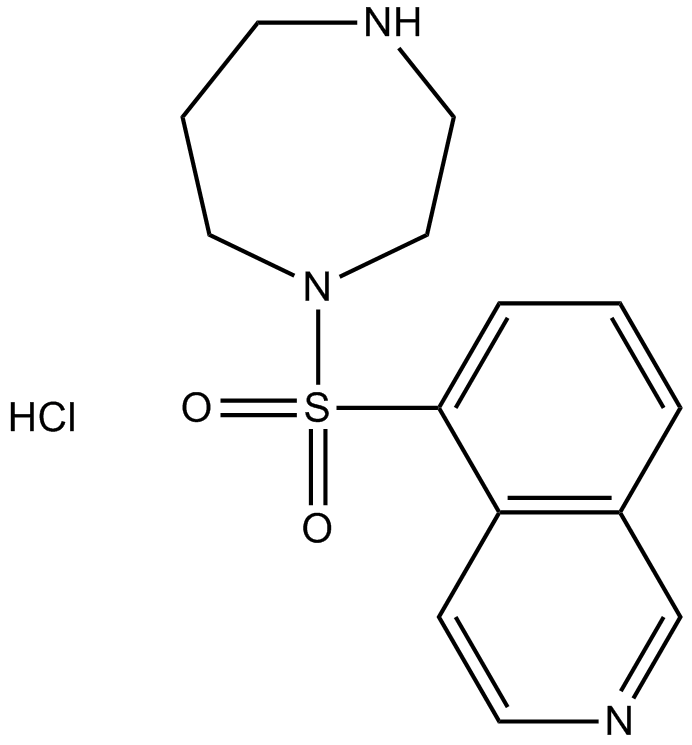
-
GC12027
FR 236924
FR 236924 (FR236924), un derivado del Ácido linoleico, activa selectiva y directamente PKCε.
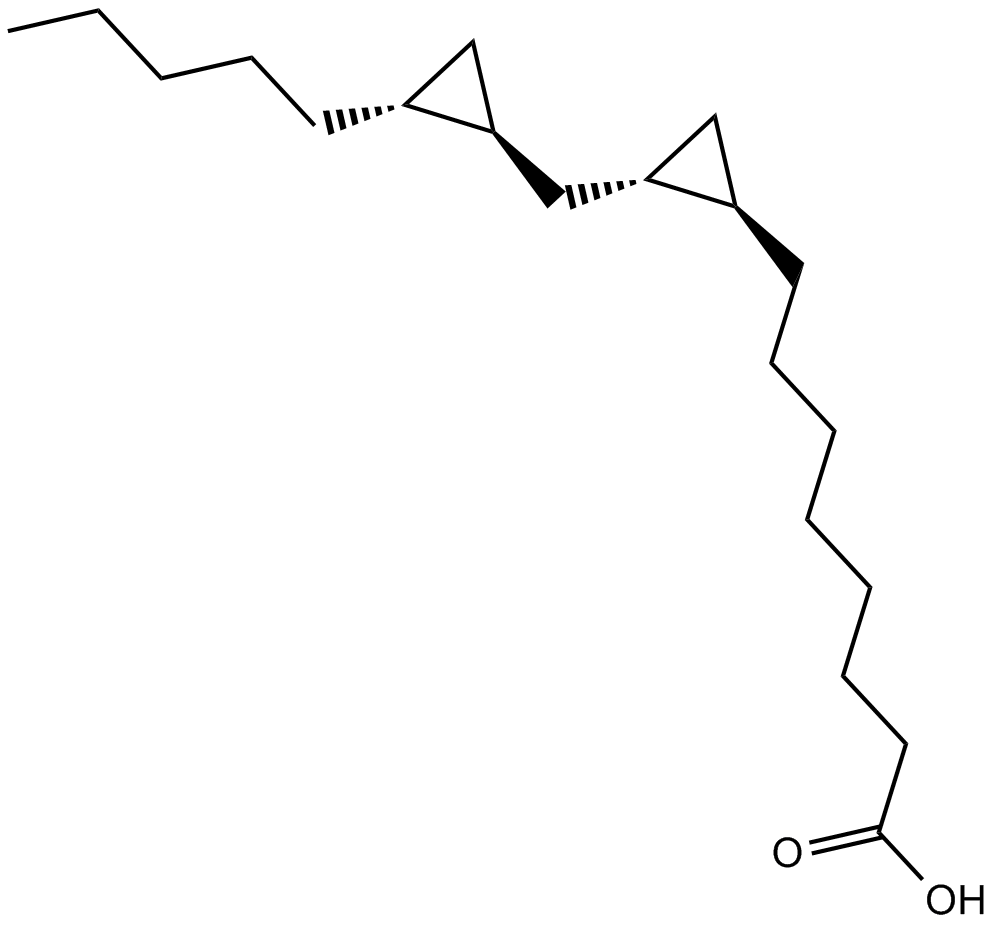
-
GC15431
GF 109203X (Bisindolylmaleimide I)
GF 109203X (GF109203X) es un inhibidor de la proteÍna quinasa C (PKC) altamente selectivo, permeable a las células y reversible con una Ki de 14 nM.

-
GC15564
Go 6976
Go 6769 es un inhibidor de la Proteína Quinasa C (PKC), con una IC50 de 20 nM.
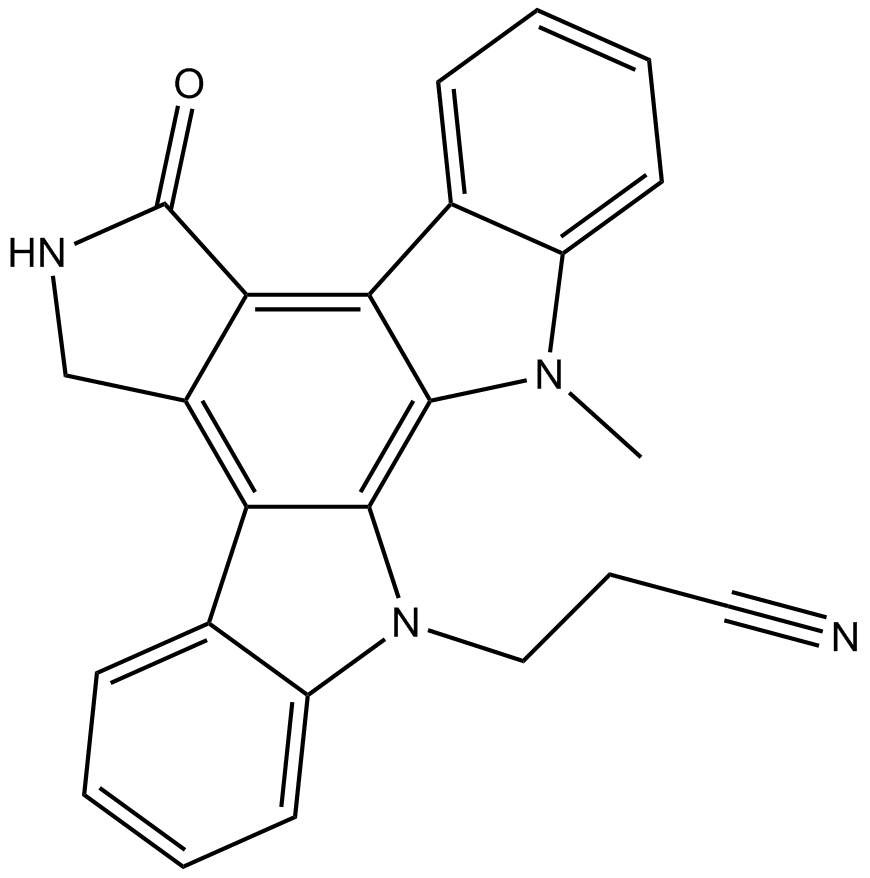
-
GC16907
Go 6983
Go 6983 (GÖ 6983) es uno del grupo bisindolilmaleimida de compuestos inhibidores de PKC, Go 6983 (GÖ 6983) pudo diferenciar entre PKC mu y otras isoenzimas de PKC.
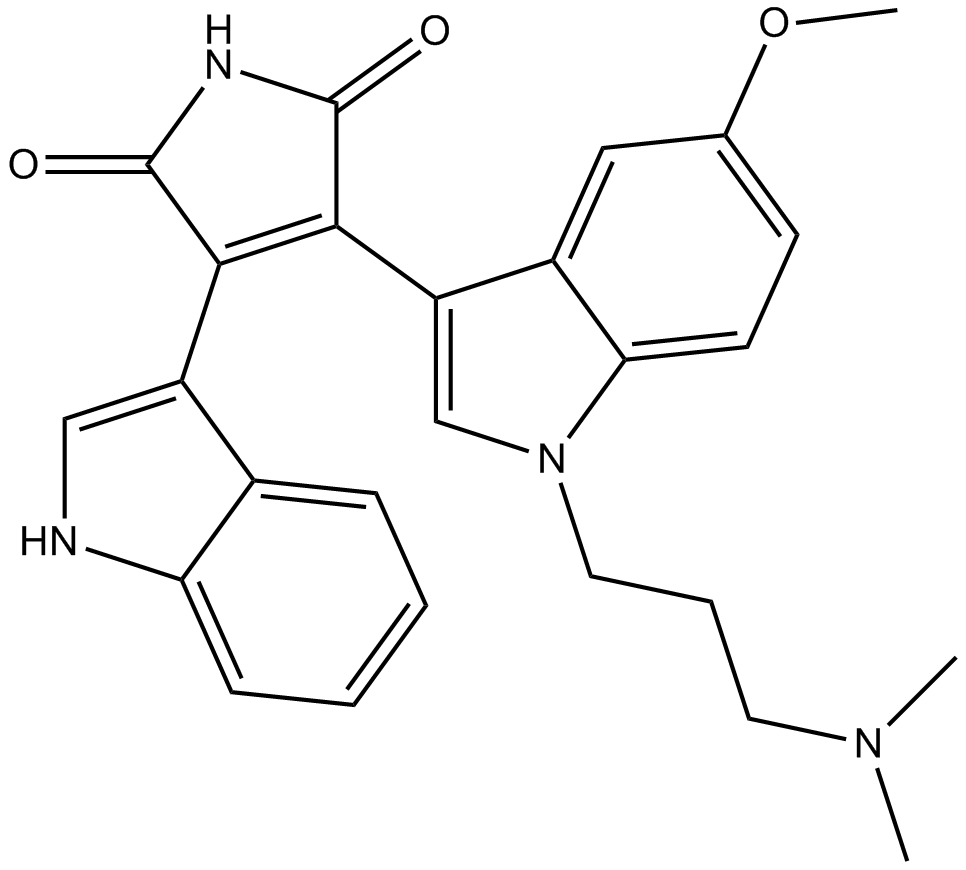
-
GC10299
Hexadecyl Methyl Glycerol
protein kinase C activity inhibitor
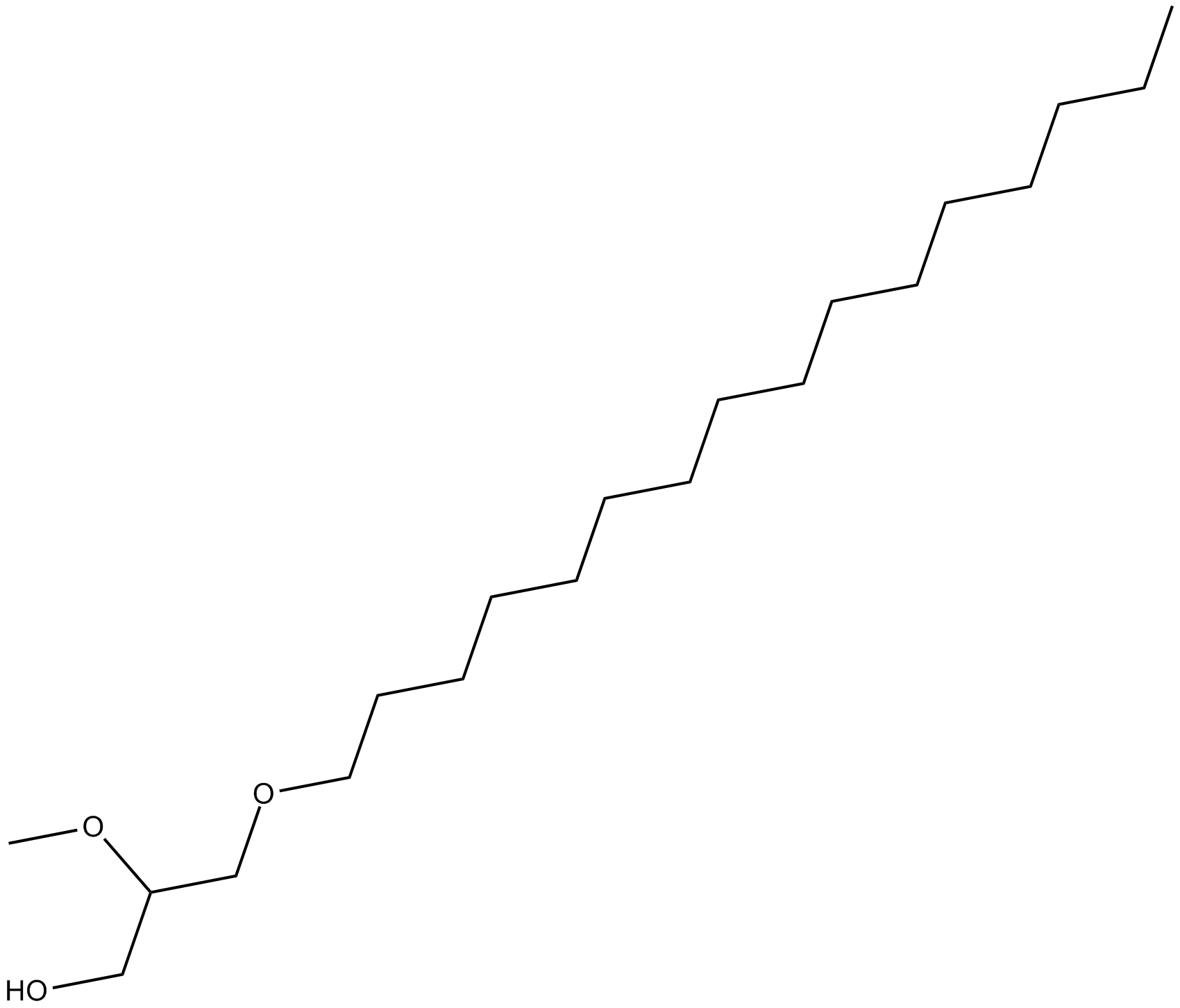
-
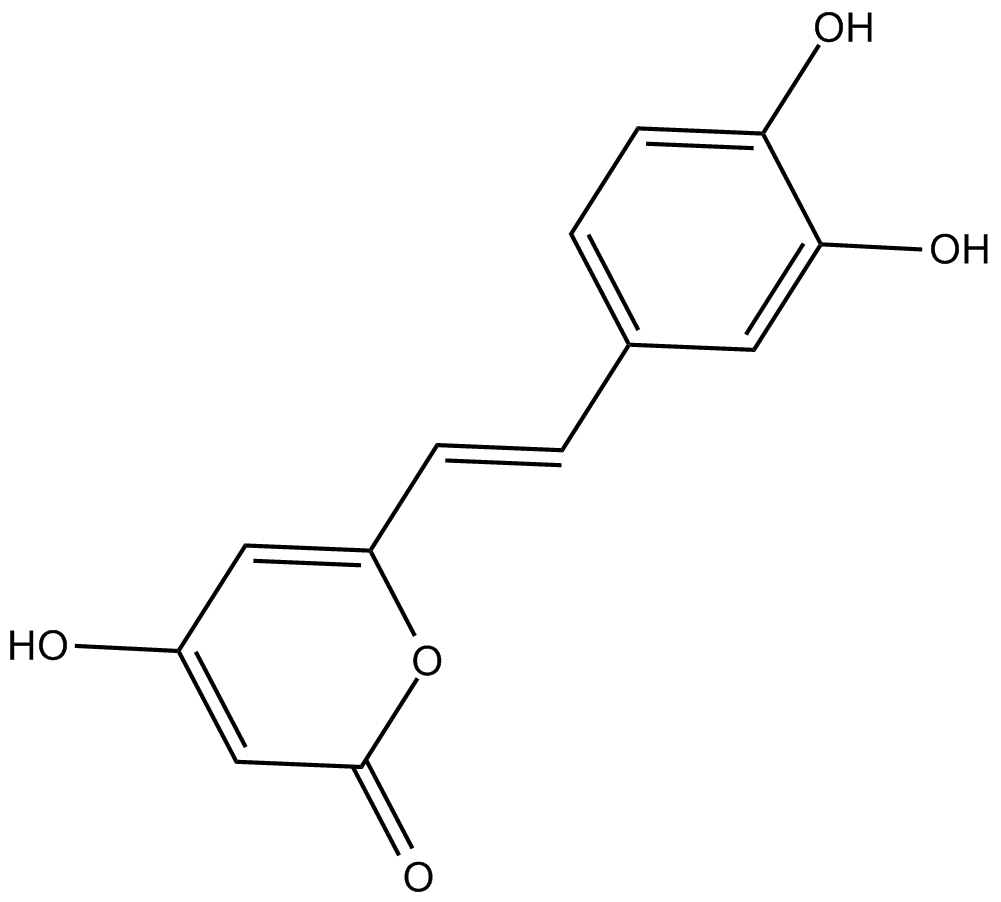
GC15018
Hispidin
Se ha demostrado que la hispidina, un inhibidor de la PKC y un compuesto fenÓlico de Phellinus linteus, posee fuertes propiedades antioxidantes, anticancerÍgenas, antidiabéticas y antidemencia.
-
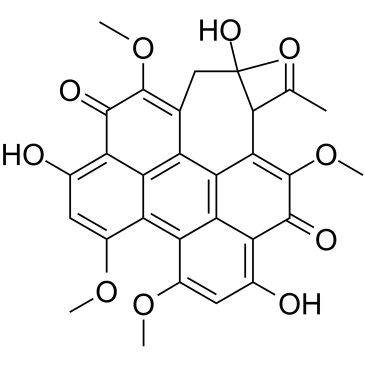
GC36282
Hypocrellin A
La hipocrelina A, un inhibidor natural de la PKC, tiene muchas propiedades biolÓgicas y farmacolÓgicas, como actividades antitumorales, antivirales, antibacterianas y contra la leishmania.
-
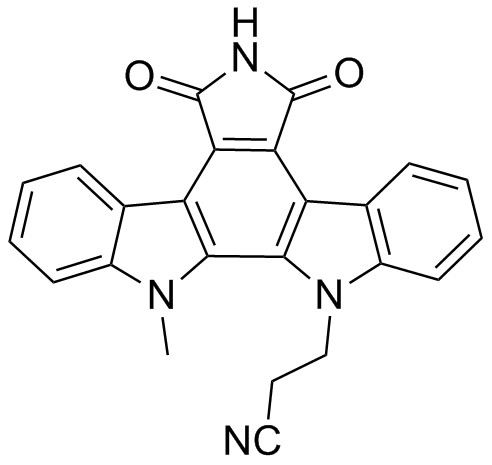
GC15420
ICP 103
Protein kinase inhibitor
-
GC32870
Ingenol ((-)-Ingenol)
Ingenol ((-)-Ingenol) es un activador de PKC, con una Ki de 30 μM, con actividad antitumoral.
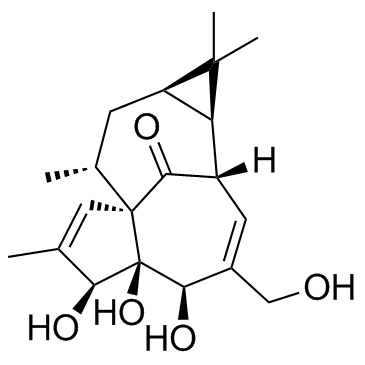
-
GC61776
Ingenol 3,20-dibenzoate
El 3,20-dibenzoato de ingenol es un potente agonista selectivo de la isoforma de la proteÍna quinasa C (PKC).

-
GC31656
Ingenol Mebutate (Ingenol 3-angelate)
El mebutato de ingenol (Ingenol 3-angelate) es un ingrediente activo en Euphorbia peplus, actÚa como un potente modulador de PKC, con Kis de 0.3, 0.105, 0.162, 0.376 y 0.171 nM para PKC-α, PKC-β, PKC-β γ, PKC-δ y PKC-ε, respectivamente, y tiene actividad antiinflamatoria y antitumoral.
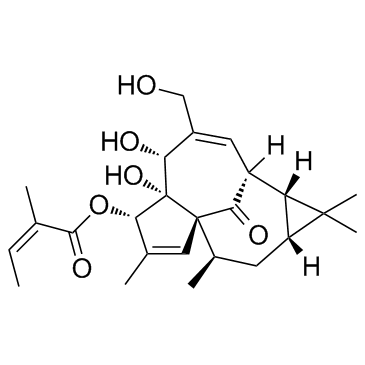
-
GC15148
Ionomycin calcium salt
Lonomycin es un ionóforo selectivo de calcio derivado de S. conglobatus que moviliza las reservas de calcio intracelular.
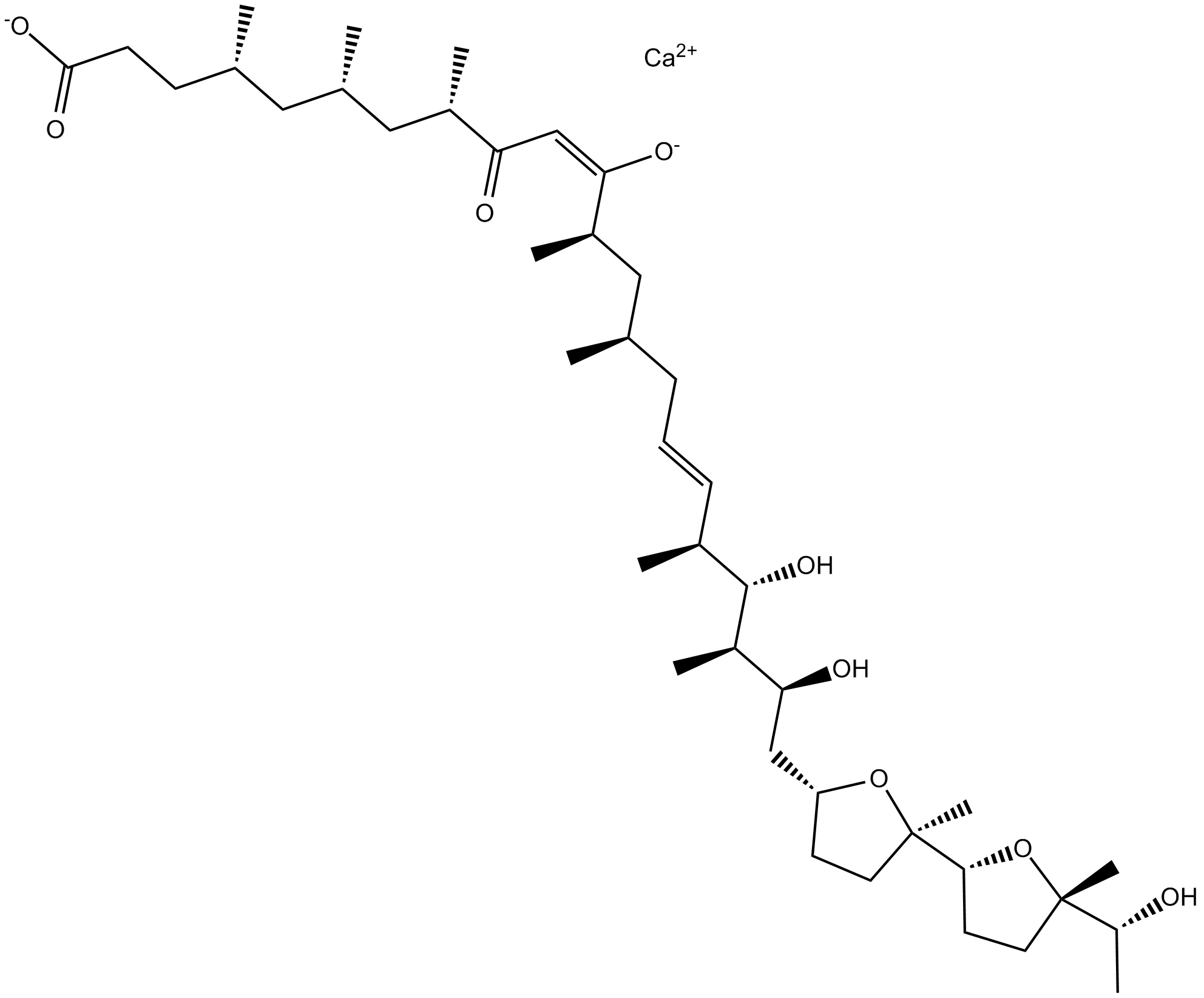
-
GC15446
Ionomycin free acid
El ácido libre de ionomicina es un portador de iones de calcio potente y selectivo que actúa como portador activo de Ca2+.
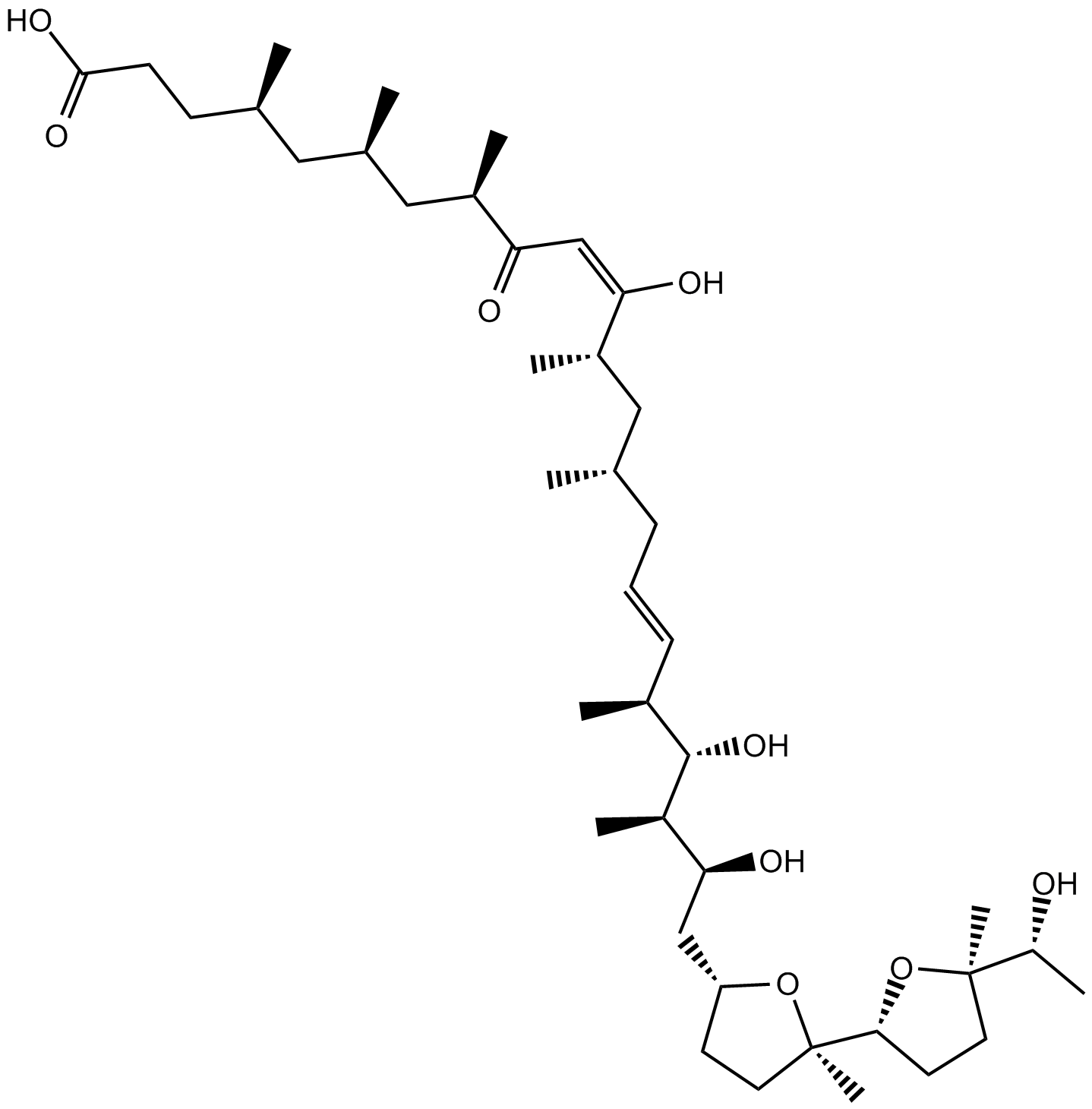
-
GC11362
K 252a
Un inhibidor de la proteína quinasa.
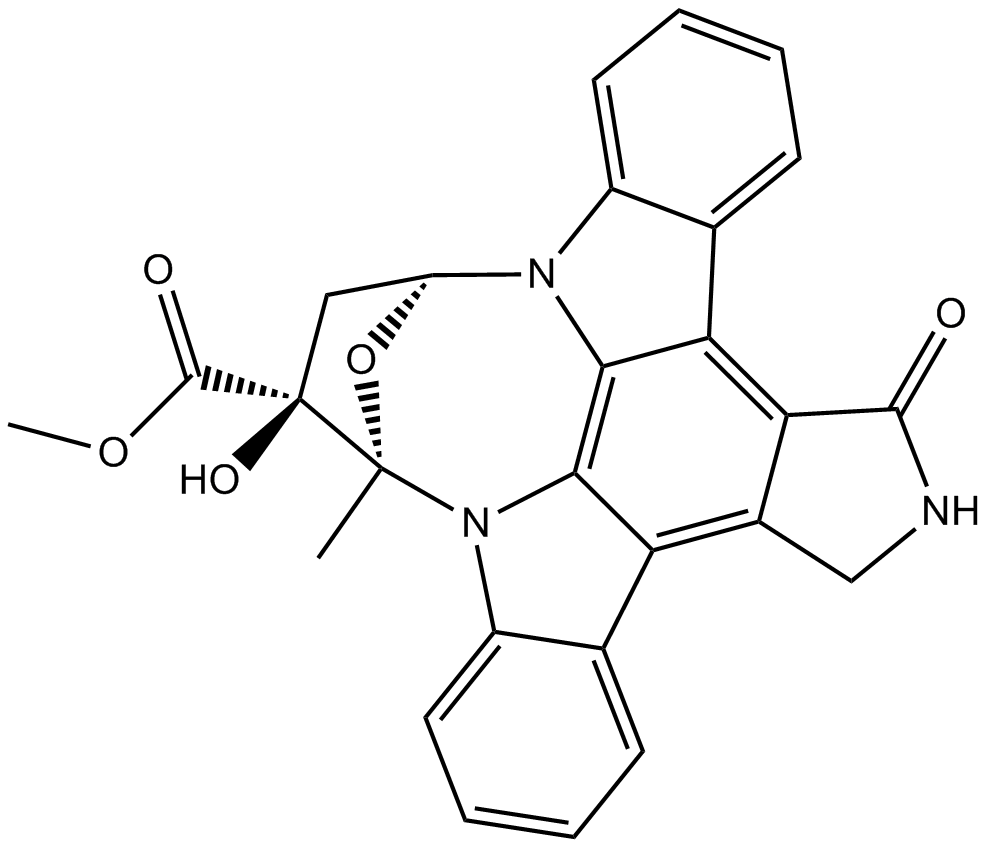
-
GC15281
K-252c
K-252c, un anÁlogo de estaurosporina aislado de Nocardiopsis sp.
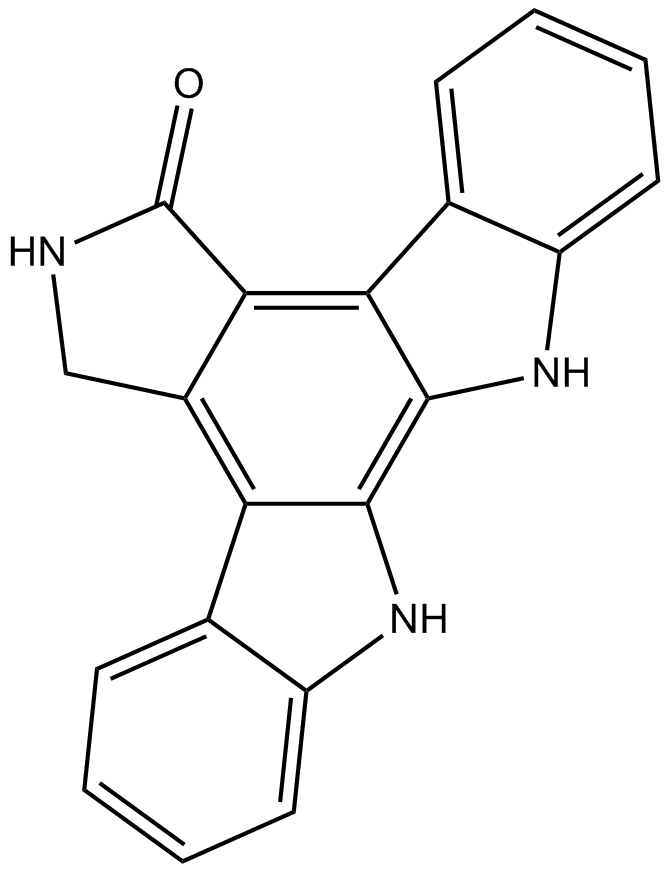
-
GC43993
K252b
K252b, un indolocarbazol aislado del actinomiceto Nocardiopsis, es un inhibidor de PKC.
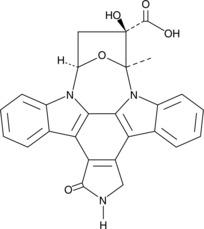
-
GC64263
Kobophenol A
El kobofenol A, un estilbeno oligomérico, bloquea la interacciÓn entre el receptor ACE2 y S1-RBD con una IC50 de 1,81 μM e inhibe la infecciÓn viral por SARS-CoV-2 en células con una EC50 de 71,6 μM.
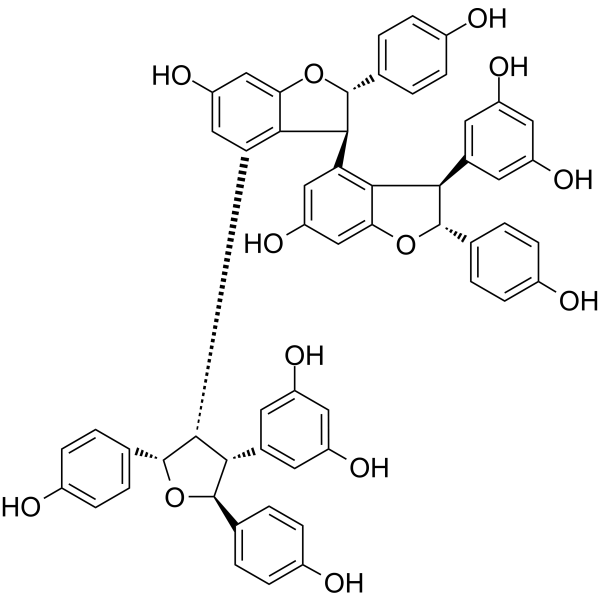
-
GC17346
KT 5823
KT 5823, un inhibidor selectivo de la proteína quinasa dependiente de cGMP (PKG) con un valor Ki de 0,23 μM, también inhibe PKA y PKC con valores Ki de 10 μM y 4 μM, respectivamente.
-
GC40770
L-erythro Sphingosine (d18:1)
L-erythro Sphingosine is a synthetic stereoisomer of sphingosine (d18:1).
-
GC17815
L-threo-Sphingosine C-18
L-threo-Sphingosine C-18 es un potente inhibidor de MAPK. L-threo-Sphingosine C-18 induce la apoptosis y la fragmentaciÓn clara del ADN. L-threo-Sphingosine C-18 muestra un efecto anticancerÍgeno.
-
GC32811
LXS196
LXS196 (LXS196) es un inhibidor de la proteÍna quinasa C (PKC) potente, selectivo y activo por vÍa oral, con valores IC50 de 1,9 nM, 0,4 nM y 3,1 μM para PKCα, PKCθ y GSK3β, respectivamente. LXS196 tiene potencial para la investigaciÓn del melanoma uveal.
-
GC17563
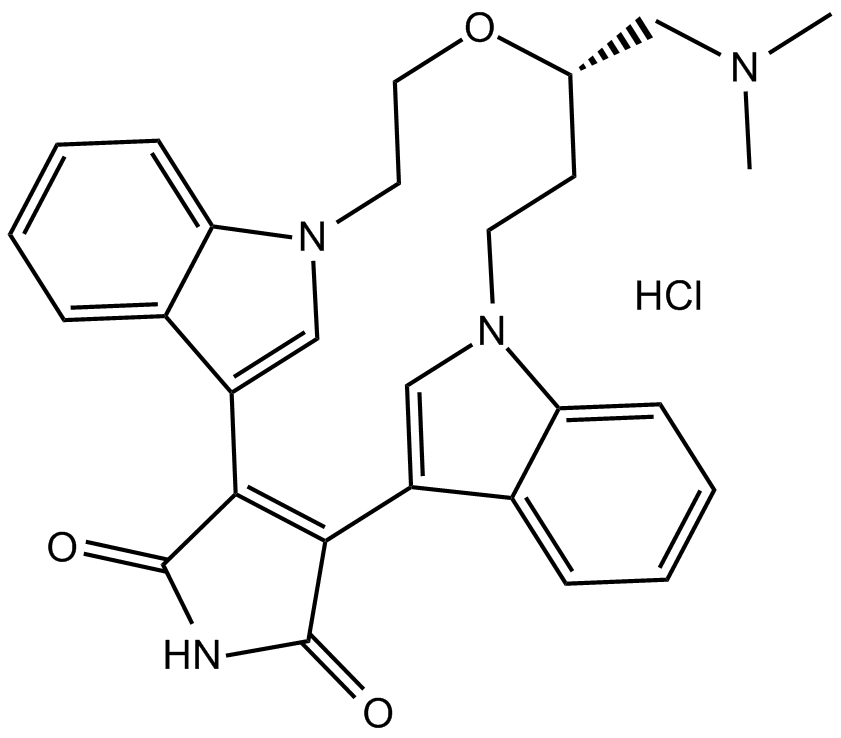
LY 333531 hydrochloride
El clorhidrato de ruboxistaurina (LY333531) es un inhibidor beta selectivo de la PKC activo por vÍa oral (Ki = 2 nM).
-
GC30545
Malantide
Malantide es un dodecapéptido sintético derivado del sitio fosforilado por la proteÍna quinasa dependiente de cAMP (PKA) en la subunidad β de la fosforilasa quinasa. Malantide es un sustrato altamente especÍfico para PKA con una Km de 15 μM y muestra una inhibiciÓn del inhibidor de proteÍnas (PKI) >90 % de la fosforilaciÓn del sustrato en varios extractos de tejido de rata. Malantide también es un sustrato eficiente para PKC con una Km de 16 μM.
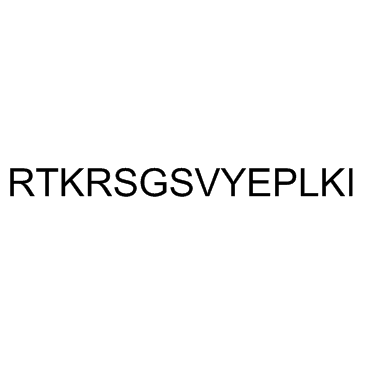
-
GC10496
Midostaurin (PKC412)
La midostaurina (PKC412) (PKC412; CGP 41251) es un inhibidor reversible de la proteÍna quinasa multidiana activo por vÍa oral. Midostaurina (PKC412) inhibe PKCα/β/γ Syk, Flk-1, Akt, PKA, c-Kit, c-Fgr, c-Src, FLT3, PDFRβ y VEGFR1/2 con IC50 que oscilan entre 22 y 500 nM. La midostaurina (PKC412) también aumenta la expresiÓn del gen de la Óxido nÍtrico sintasa endotelial (eNOS). La midostaurina (PKC412) muestra poderosos efectos anticancerÍgenos.
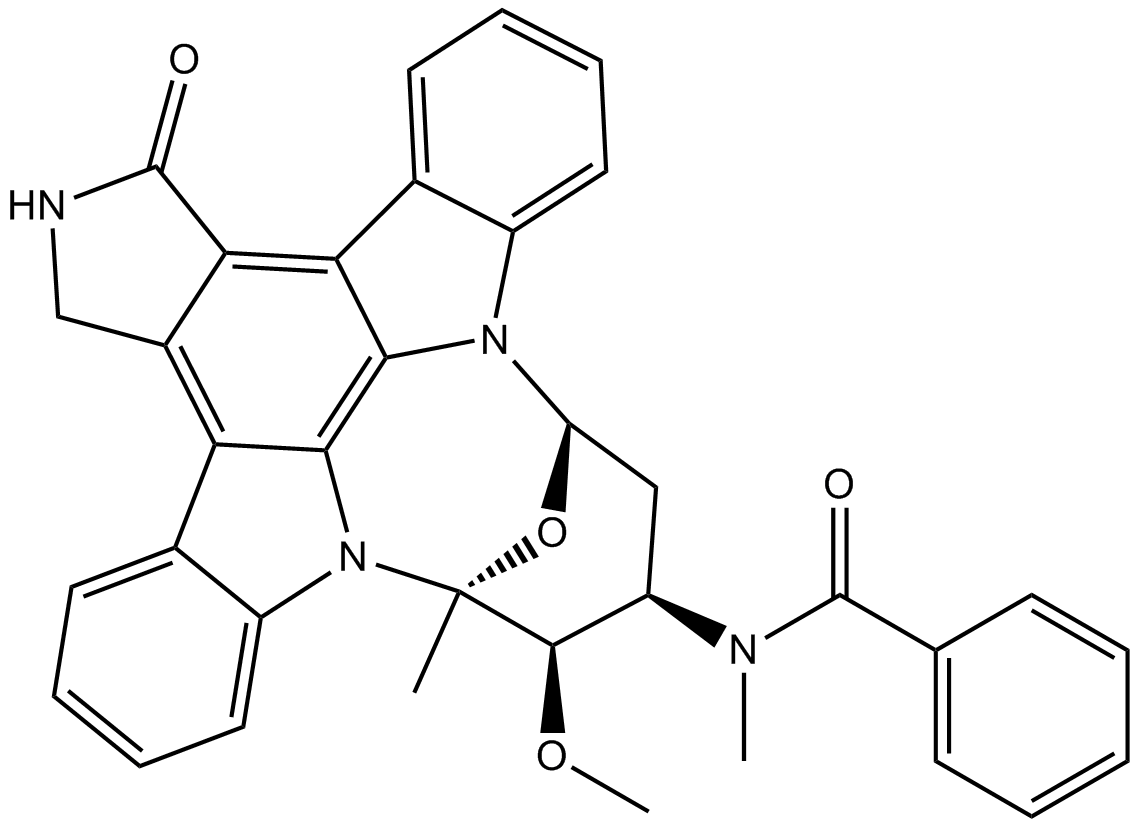
-
GC32714
Mitoxantrone (mitozantrone)
La mitoxantrona (mitozantrona) es un potente inhibidor de la topoisomerasa II.
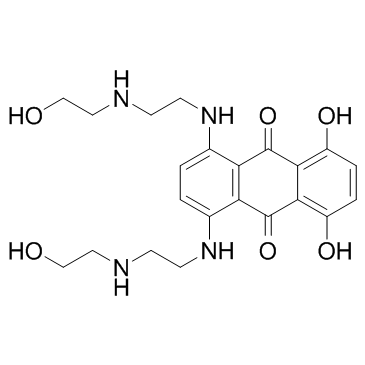
-
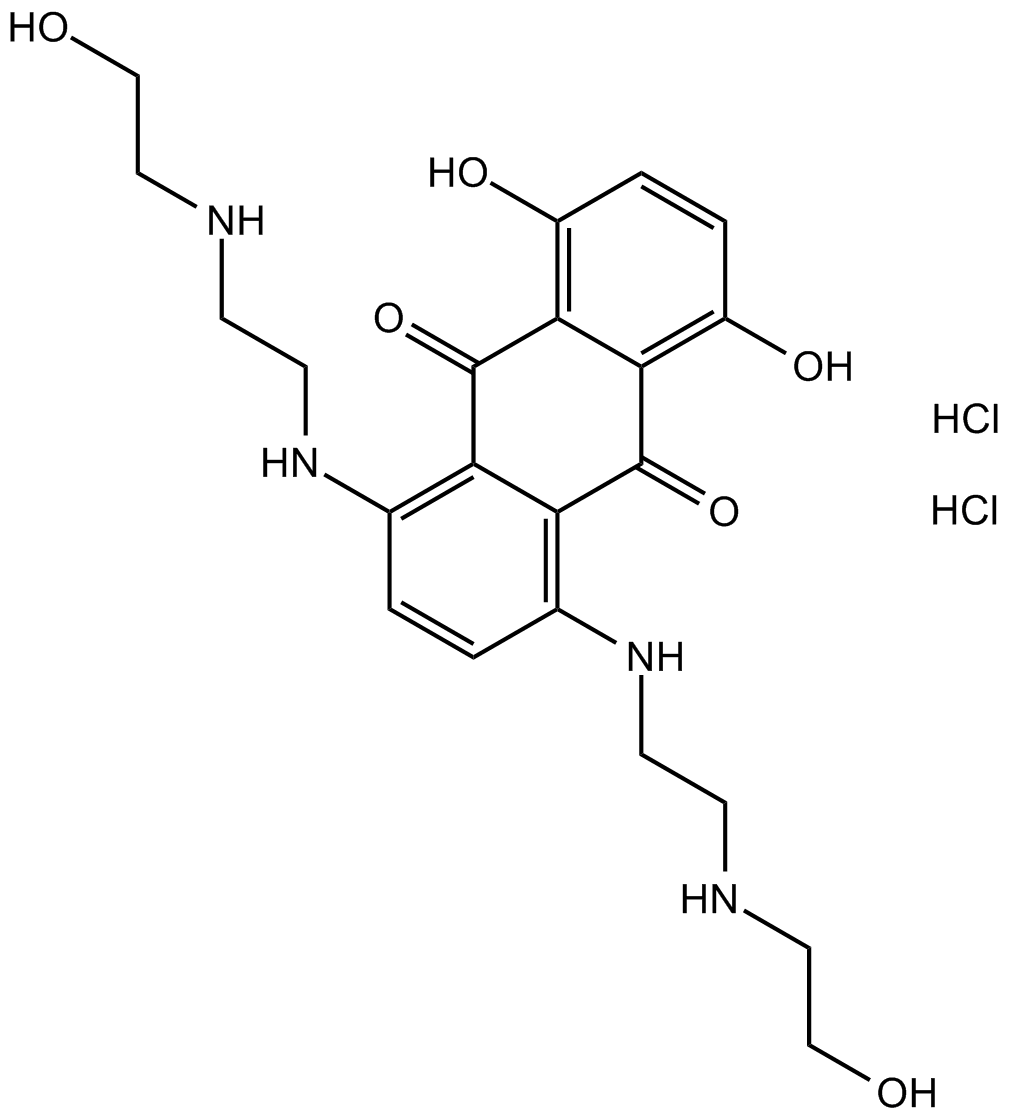
GC14363
Mitoxantrone HCl
Mitoxantrona HCl es un potente inhibidor de la topoisomerasa II.
-
GC63778
Myelin Basic Protein TFA
ProteÍna bÁsica de mielina (MHP4-14) TFA, un péptido sintético que comprende los residuos 4-14 de la proteÍna bÁsica de mielina, es un sustrato de PKC muy selectivo (Km = 7 μM).
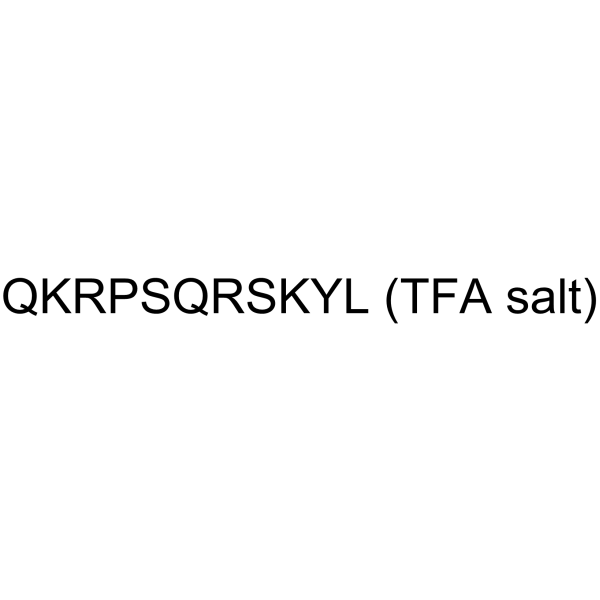
-
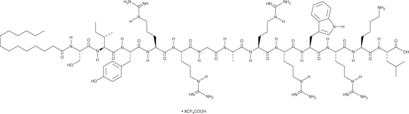
GC49269
Myr-ZIP
A PKMζ inhibitor
-
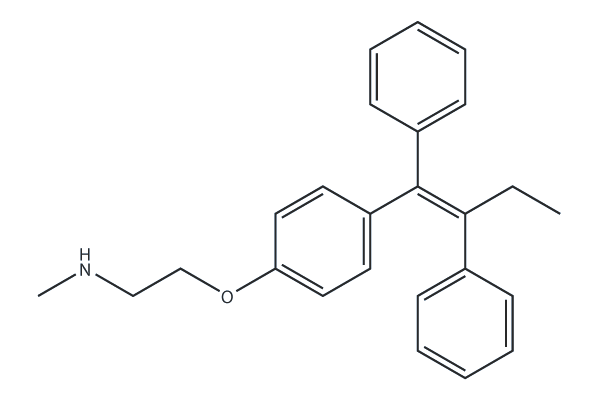
GC25662
N-Desmethyltamoxifen
N-Desmethyltamoxifen, the major metabolite of Tamoxifen in humans and a ten-fold more potent protein kinase C (PKC) inhibitor than Tamoxifen, also is a potent regulator of ceramide metabolism in human AML cells, limiting ceramide glycosylation, hydrolysis, and sphingosine phosphorylation.
-
GC38931
N-Desmethyltamoxifen hydrochloride
El clorhidrato de N-desmetiltamoxifeno es el principal metabolito del tamoxifeno en humanos. El N-desmetiltamoxifeno, un antiestrÓgeno pobre, es un inhibidor de la proteÍna quinasa C (PKC) diez veces mÁs potente que el tamoxifeno. El clorhidrato de N-desmetiltamoxifeno también es un potente regulador del metabolismo de la ceramida en las células de LMA humana, lo que limita la glicosilaciÓn, la hidrÓlisis y la fosforilaciÓn de la esfingosina de la ceramida.

-
GC60274
O-Desmethyl Midostaurin
La O-desmetil midostaurina (CGP62221; O-desmetil PKC412) es el metabolito activo de la midostaurina a través del metabolismo de las enzimas hepÁticas del citocromo P450. La O-desmetil midostaurina se puede utilizar como indicador del metabolismo de la midostaurina in vivo. La midostaurina es un inhibidor de la proteÍna quinasa multidirigidoconIC50que oscila entre 22 y 500 nM.

-
GC36833
p32 Inhibitor M36
El inhibidor de p32 M36 (M36) es un inhibidor de la proteÍna mitocondrial p32, que se une directamente a p32 e inhibe la asociaciÓn de p32 con LyP-1.
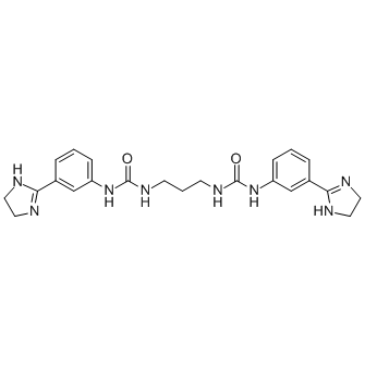
-
GC61585
Pep2m, myristoylated TFA
Pep2m, TFA miristoilado (Myr-Pep2m TFA) es un péptido permeable a las células.

-
GC17352
Phorbol 12,13-dibutyrate
El 12,13-dibutirato de forbol (dibutirato de forbol) es un activador de PKC y un potente promotor de tumores cutÁneos.
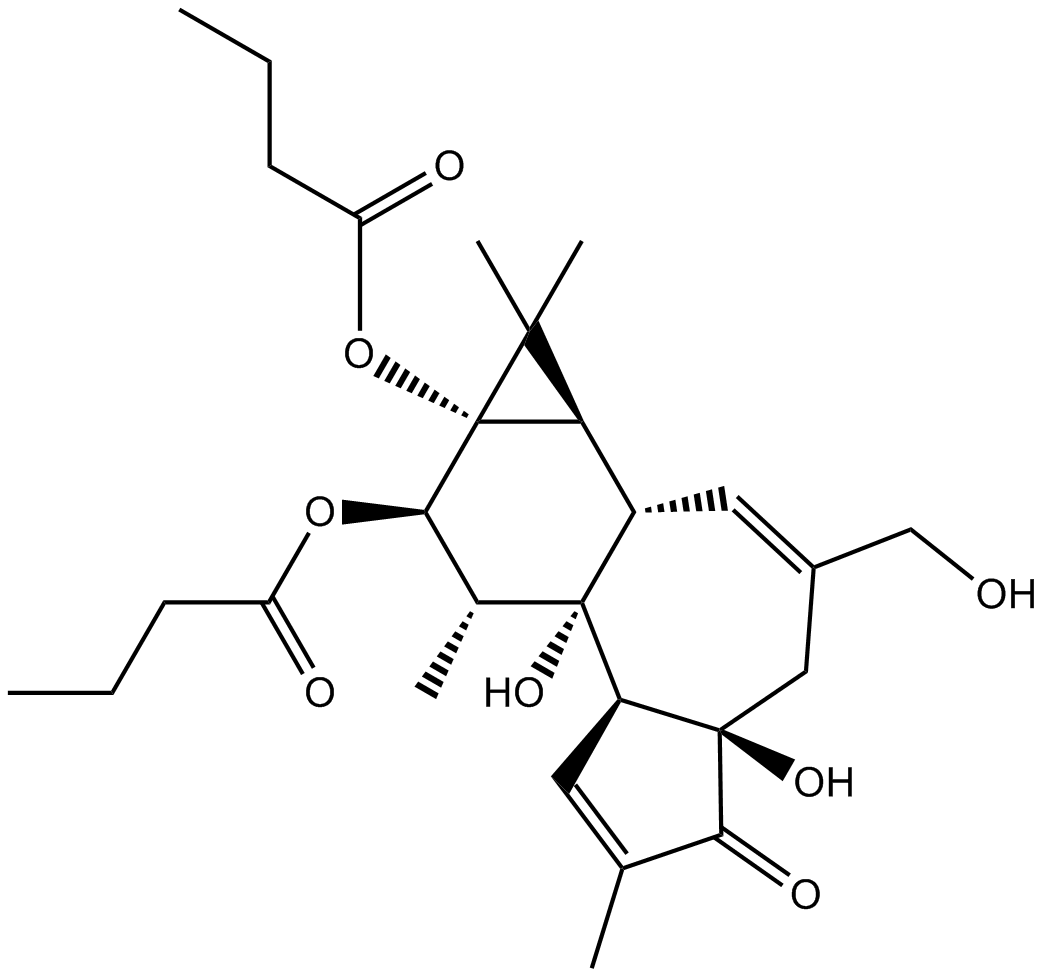
-
GC15471
PKC β pseudosubstrate
Selective cell-permeable peptide inhibitor of protein kinase C

-
GC10404
PKC ζ pseudosubstrate
PKCζ el pseudosustrato es un inhibidor selectivo de la PKC permeable a las células.

-
GC11671
PKC fragment (530-558)
Potent activator of protein kinase C

-
GC44655
PKCε Inhibitor Peptide
El péptido inhibidor PKCε, también llamado εV1-2, es un péptido derivado de la proteína quinasa C ε (PKCε), actúa como un inhibidor selectivo de PKCε e inhibe la translocación de PKCε.
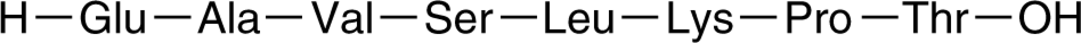
-
GC31711
PKC-IN-1
PKC-IN-1 es un inhibidor potente, competitivo con ATP y reversible de las enzimas PKC convencionales con Kis de 5,3 y 10,4 nM para PKCβ y PKCα humanas, y CI50 de 2,3, 8,1, 7,6, 25,6, 57,5, 314, 808 nM para PKCα, PKCβI, PKCβII, PKCθ, PKCγ, PKC mu y PKCε, respectivamente.
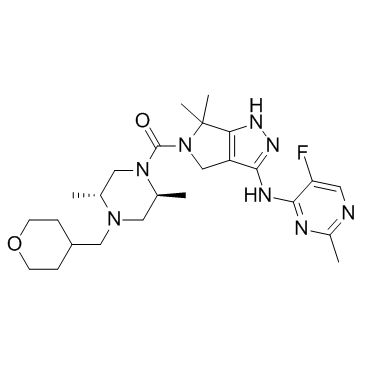
-
GC65126
PKC-iota inhibitor 1
El inhibidor 1 de PKC-iota (compuesto 19) es un inhibidor de la proteÍna quinasa C-iota (PKC-Ι ℩) con un valor IC50 de 0,34 μM.
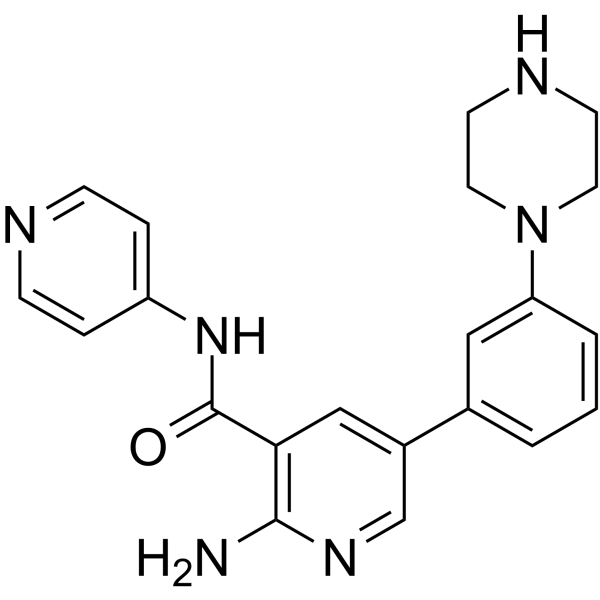
-
GC30200
PKC-theta inhibitor
El inhibidor de PKC-theta es un inhibidor selectivo de PKC-θ, con una IC50 de 12 nM.
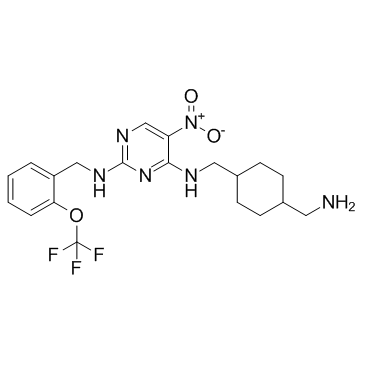
-
GC67686
PKCiota-IN-2 formic
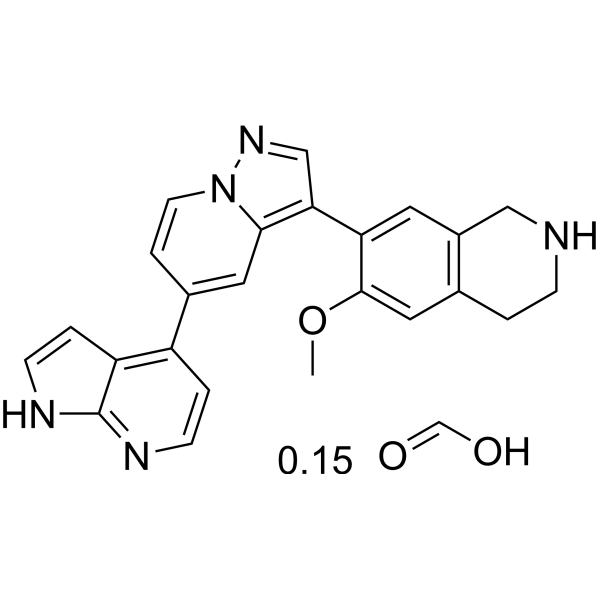
-
GC36974
Procyanidin A1
La procianidina A1 (proantocianidina A1) es un dÍmero de procianidina que inhibe la desgranulaciÓn aguas abajo de la activaciÓn de la proteÍna quinasa C o la entrada de Ca2+ desde un almacén interno en las células RBL-213.

-
GC44729
Prostratin
An HIV reactivator
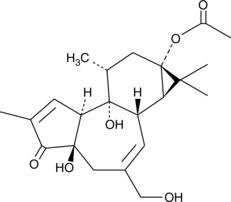
-
GC37014
Protein Kinase C 19-31
La proteína quinasa C 19-31, un inhibidor peptídico de la proteína quinasa C (PKC), derivado del dominio regulador de pseudosustrato de PKCa (residuos 19-31) con una serina en la posición 25 que reemplaza a la alanina de tipo salvaje, se usa como péptido sustrato de la proteína quinasa C para probar la actividad de la proteína quinasa C.

-
GC37015
Protein Kinase C 19-36
La proteína quinasa C 19-36 es un inhibidor peptídico de pseudosustrato de la proteína quinasa C (PKC), con una IC50 de 0,18 μM.

-
GC66006
Protein kinase inhibitor H-7
El inhibidor de la proteÍna quinasa H-7 es un potente inhibidor de la proteÍna quinasa C (PKC) y de la proteÍna quinasa dependiente de nucleÓtidos cÍclicos, con una Ki de 6 μM para PKC.
-
GC11803
Pseudo RACK1
Activator of protein kinase C
-
GC14583
R 59-022
R 59-022 (DKGI-I) es un inhibidor de diacilglicerol quinasa (IC50 = 2,8 μM).
-
GC69792
R 59-022 hydrochloride
R 59-022 (DKGI-I) hidrocloruro es un inhibidor de DGK (IC50: 2.8 µM). R 59-022 hidrocloruro inhibe la fosforilación de OAG a OAPA. R 59-022 hidrocloruro es un antagonista del receptor 5-HT y puede activar la proteína quinasa C (PKC). R 59-022 aumenta la producción de diacilglicerol inducida por trombina en plaquetas e inhibe la producción de ácido fosfatídico en neutrófilos.
-
GC18246
R-59-949
R-59-949 es un inhibidor de pan diacilglicerol quinasa (DGK) con una IC50 de 300 nM. El R-59-949 inhibe fuertemente la actividad de las DGK α y γ de tipo I y atenúa moderadamente la actividad de las DGK θ y κ de tipo II. El R-59-949 activa la proteína quinasa C (PKC) al aumentar los niveles del ligando endógeno diacilglicerol.
-
GC17322
Ro 31-8220
Ro 31-8220 es un potente inhibidor de PKC, con IC50 de 5, 24, 14, 27, 24 y 23 nM para PKCα, PKCβI, PKCβII, PKCγ, PKCε y PKC de cerebro de rata, respectivamente.