DNA/RNA Synthesis
RNA synthesis, which is also called DNA transcription, is a highly selective process. Transcription by RNA polymerase II extends beyond RNA synthesis, towards a more active role in mRNA maturation, surveillance and export to the cytoplasm.
Single-strand breaks are repaired by DNA ligase using the complementary strand of the double helix as a template, with DNA ligase creating the final phosphodiester bond to fully repair the DNA.DNA ligases discriminate against substrates containing RNA strands or mismatched base pairs at positions near the ends of the nickedDNA. Bleomycin (BLM) exerts its genotoxicity by generating free radicals, whichattack C-4′ in the deoxyribose backbone of DNA, leading to opening of the ribose ring and strand breakage; it is an S-independentradiomimetic agent that causes double-strand breaks in DNA.
First strand cDNA is synthesized using random hexamer primers and M-MuLV Reverse Transcriptase (RNase H). Second strand cDNA synthesis is subsequently performed using DNA Polymerase I and RNase H. The remaining overhangs are converted into blunt ends using exonuclease/polymerase activity. After adenylation of the 3′ ends of DNA fragments, NEBNext Adaptor with hairpin loop structure is ligated to prepare the samples for hybridization. Cell cycle and DNA replication are the top two pathways regulated by BET bromodomain inhibition. Cycloheximide blocks the translation of mRNA to protein.
Targets for DNA/RNA Synthesis
Products for DNA/RNA Synthesis
- Cat.No. Nom du produit Informations
-
GC52303
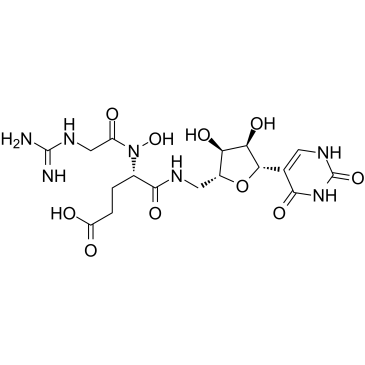
Ethyl Mycophenolate
A potential impurity found in commercial preparations of mycophenolate mofetil
-
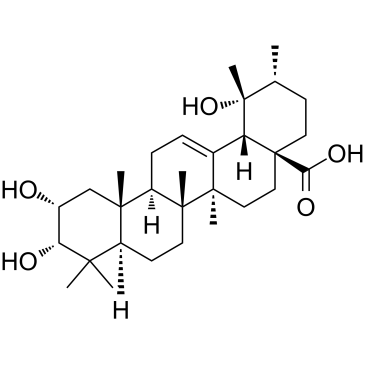
GC38392
Euscaphic acid
L'acide euscaphique, un inhibiteur de l'ADN polymérase, est un triterpène de la racine du R. alceaefolius Poir. Euscaphic inhibe l'ADN polymérase α de veau (pol α) et l'ADN polymérase β de rat (pol β) avec des valeurs IC50 de 61 et 108 μM. L'acide euscaphique induit l'apoptose.
-
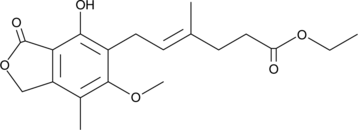
GC46146
FD-211
A δ lactone with anticancer activity
-
GC18014
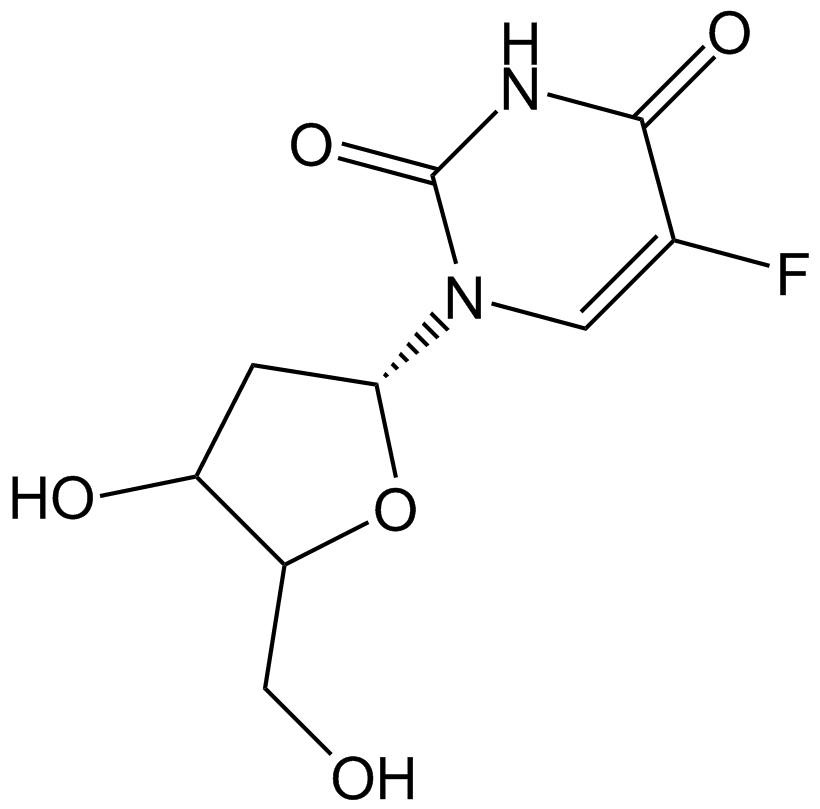
Floxuridine
La floxuridine (5-Fluorouracil 2'-désoxyriboside) est un analogue de la pyrimidine et est connu comme un antimétabolite oncologique.
-
GC14144
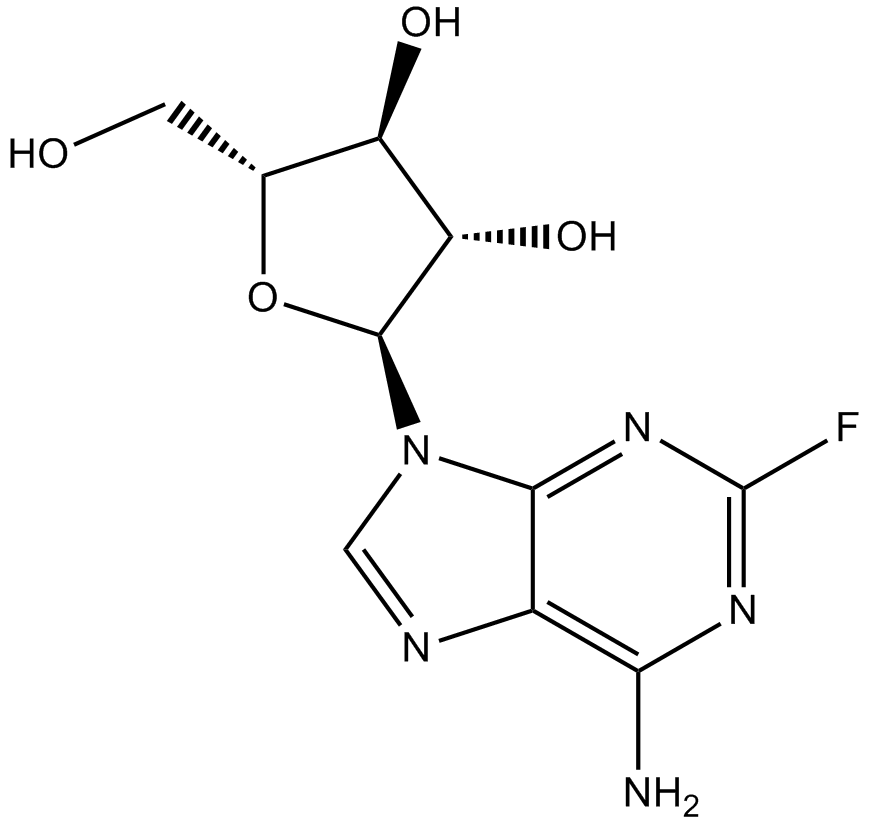
Fludarabine
La fludarabine (NSC 118218) est un inhibiteur de la synthèse de l'ADN et un analogue fluoré de la purine ayant une activité antinéoplasique dans les tumeurs malignes lymphoprolifératives. La fludarabine inhibe l'activation induite par les cytokines de la transcription des gènes STAT1 et STAT1-dépendante dans les lymphocytes normaux au repos ou activés.
-
GC15134
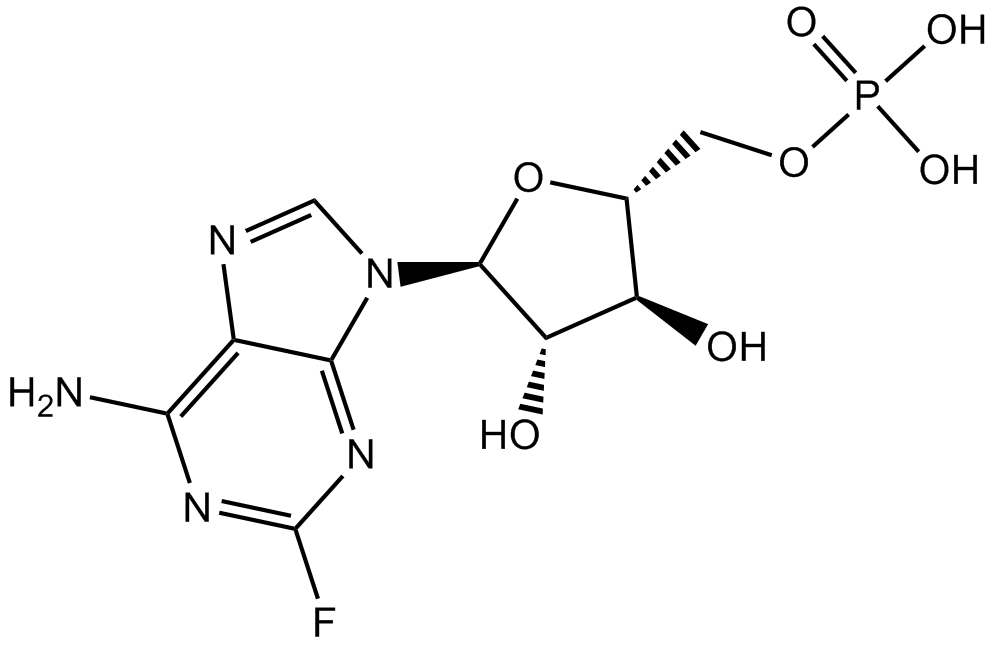
Fludarabine Phosphate (Fludara)
La fludarabine (phosphate) est un analogue de l'adénosine et de la désoxyadénosine, capable d'entrer en compétition avec le dATP pour l'incorporation dans l'ADN et d'inhiber la synthèse de l'ADN.
-
GC49134
Flumequine-13C3
An internal standard for the quantification of flumequine
-
GC49455
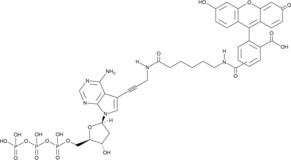
Fluorescein-12-dATP
A fluorescently labeled form of dATP
-
GC49746
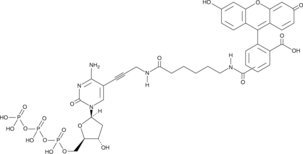
Fluorescein-12-dCTP
A fluorescently labeled form of dCTP
-
GC49456
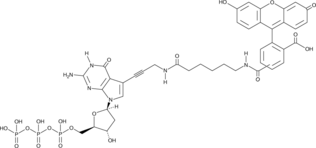
Fluorescein-12-dGTP
A fluorescently labeled form of dGTP
-
GC14466
Fluorouracil (Adrucil)
Une forme de promédicament de FdUMP
-
GN10306
Folic acid
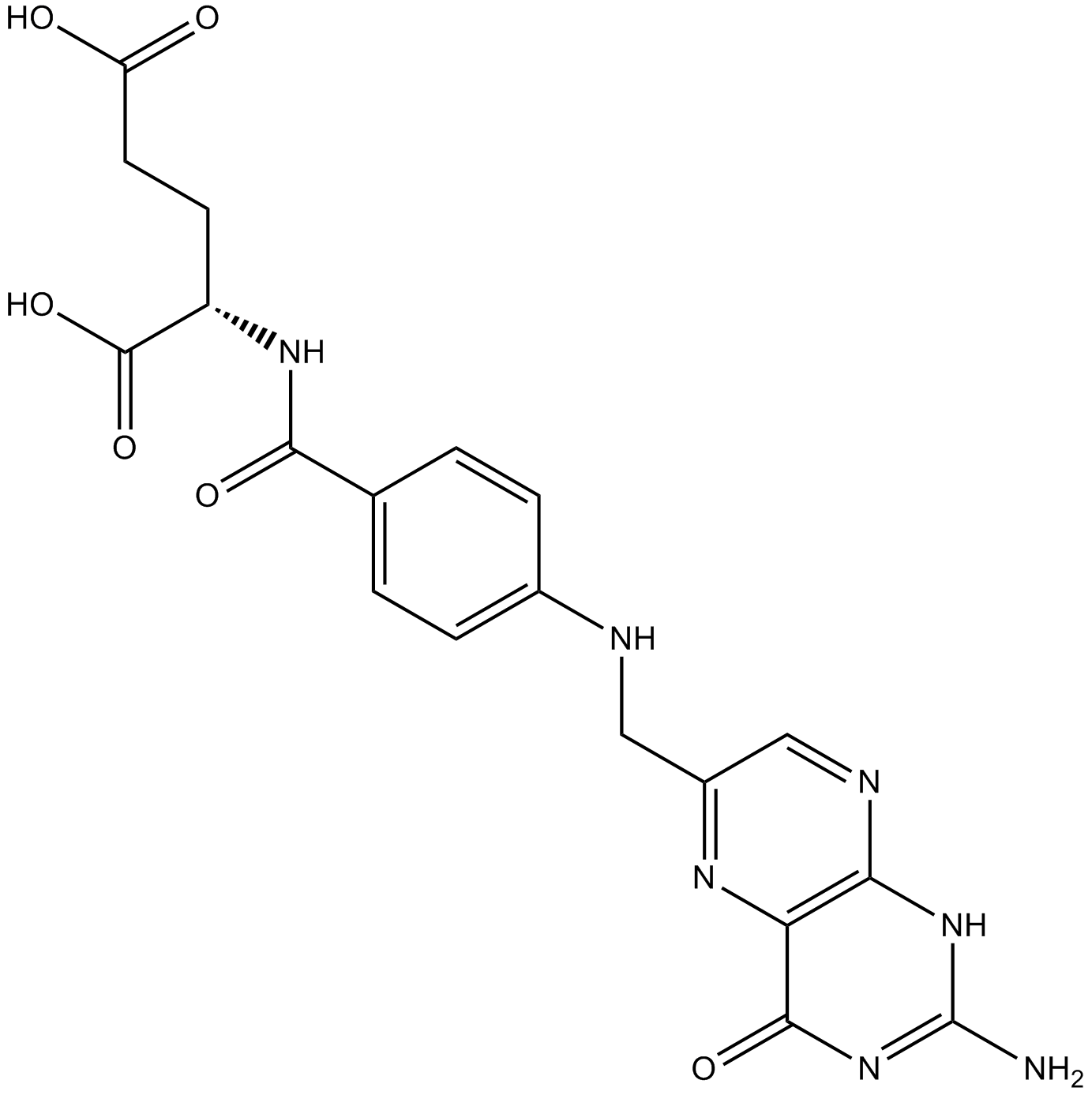
-
GC63952
Folic Acid-d2
L'acide folique-d2 est l'acide folique marqué au deutérium. L'acide folique (vitamine M ; vitamine B9) est une vitamine B ; est nécessaire À la production et au maintien de nouvelles cellules, À la synthèse d'ADN et À la synthèse d'ARN.
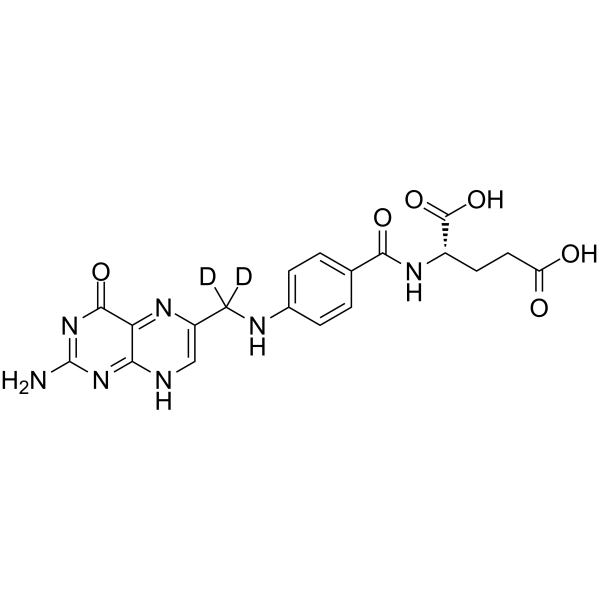
-
GC49126
Folitixorin
La folitixorine (5,10-méthylènetétrahydrofolate) est un cofacteur et un analogue de la leucovorine. La folitixorine est un agent prometteur pour la modulation de la cytotoxicité du 5-FU dans la recherche adjuvante sur le cancer.
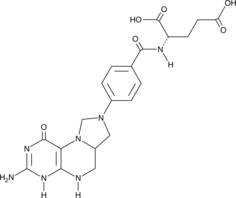
-
GC14885
Foscarnet Sodium
Le foscarnet sodique (phosphonoformate trisodique) est un inhibiteur de l'activité de l'ADN polymérase virale, entraÎnant une suppression réversible de la réplication virale.
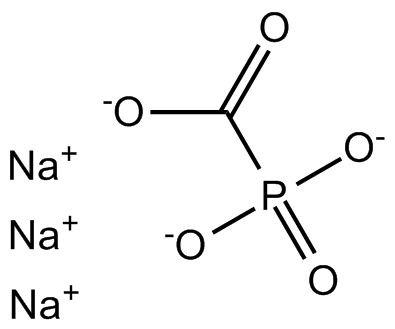
-
GC33094
GC7 Sulfate
GC7 Sulfate est un inhibiteur de la désoxyhypusine synthase (DHPS).
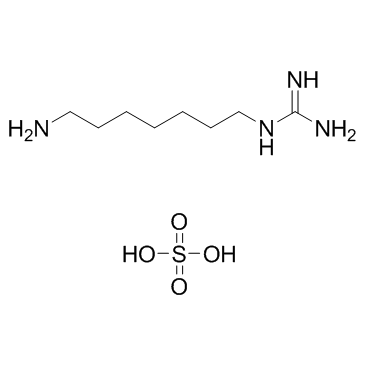
-
GC16805
Gemcitabine
La gemcitabine (LY 188011) est un antimétabolite analogue du nucléoside pyrimidique et un agent antinéoplasique. La gemcitabine inhibe la synthèse et la réparation de l'ADN, entraÎnant l'autophagie et l'apoptose.
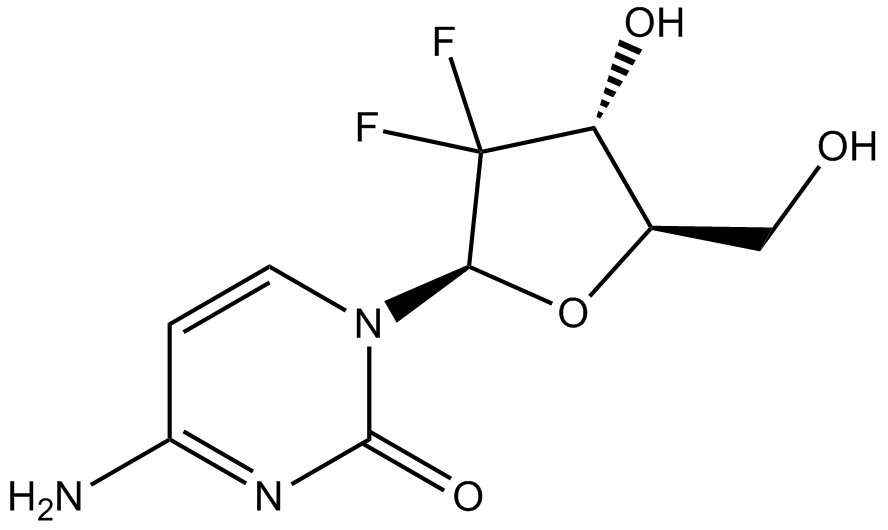
-
GC14447
Gemcitabine HCl
Le chlorhydrate de gemcitabine (chlorhydrate de LY 188011) est un antimétabolite analogue du nucléoside de la pyrimidine et un agent antinéoplasique. Le chlorhydrate de gemcitabine inhibe la synthèse et la réparation de l'ADN, entraÎnant l'autophagie et l'apoptose.
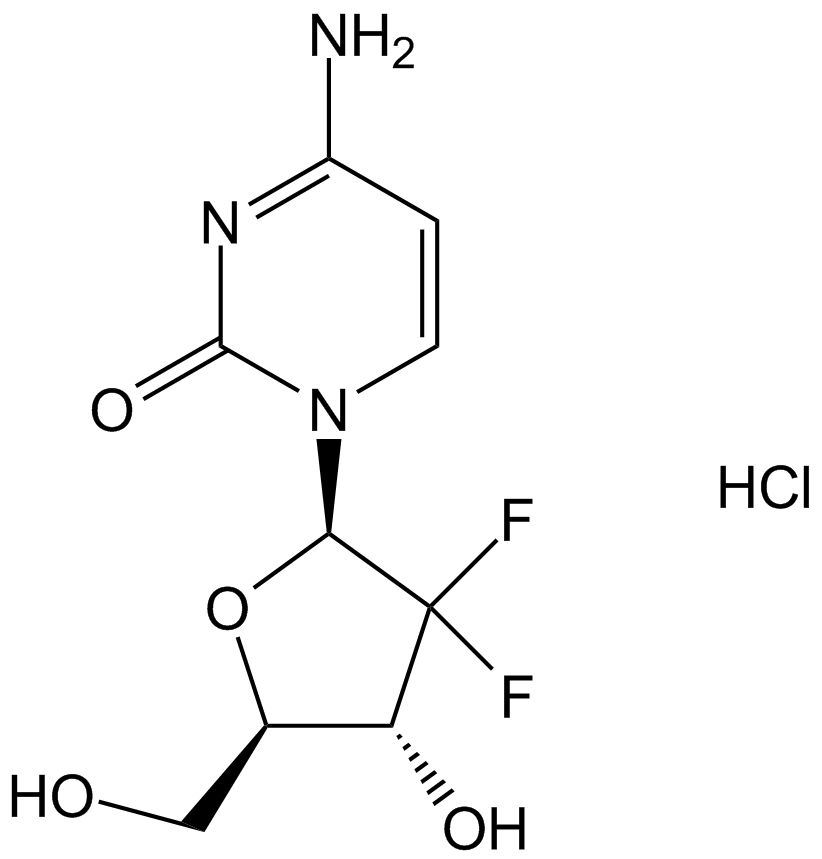
-
GC18840
Gilvocarcin V
Gilvocarcin V is an antitumor antibiotic with a coumarin-based aromatic structure that was orignally isolated from the culture broth of S.
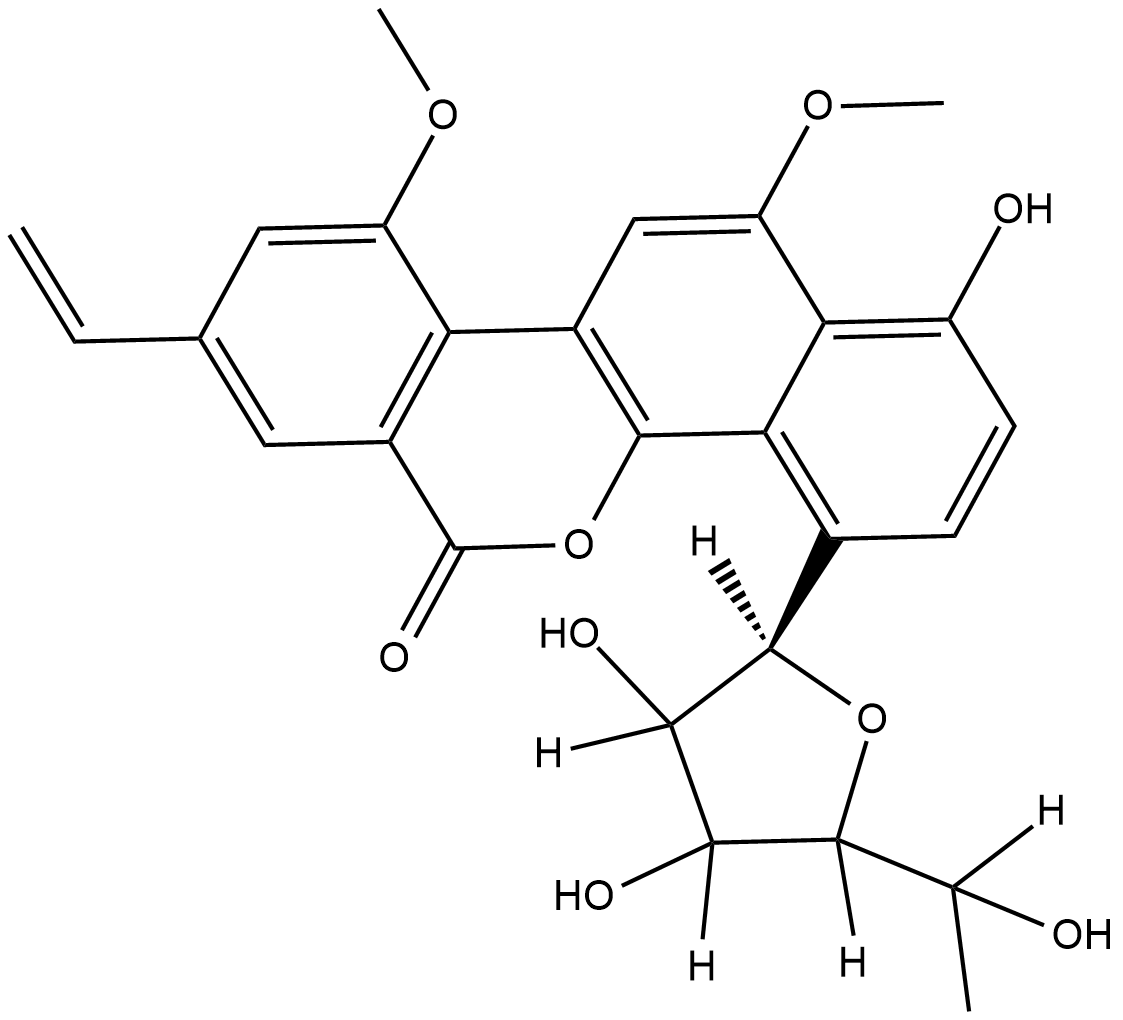
-
GC30529
GNE-371
GNE-371 est une sonde chimique puissante et sélective pour les deuxièmes bromodomaines de la sous-unité 1 TFIID du facteur d'initiation de la transcription humaine et de la sous-unité 1 du facteur d'initiation de la transcription TFIID, avec une IC50 de 10 nM pour TAF1(2).
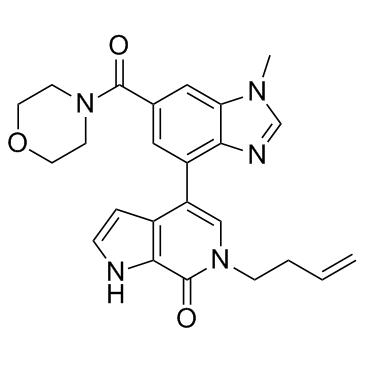
-
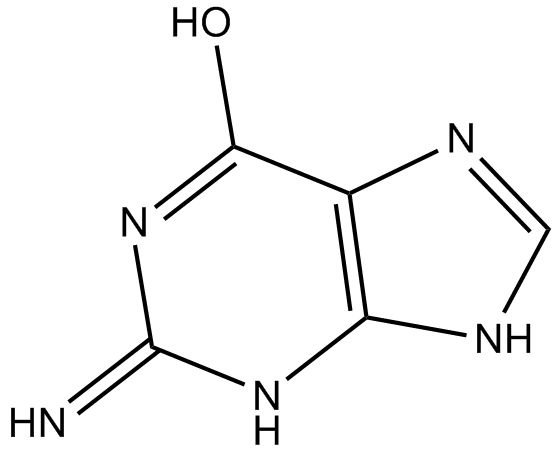
GC16949
Guanine
La guanine est l'un des composants fondamentaux des acides nucléiques (ADN et ARN). La guanine est un dérivé de la purine, constitué d'un système cyclique fusionné pyrimidine-imidazole avec des doubles liaisons conjuguées.
-
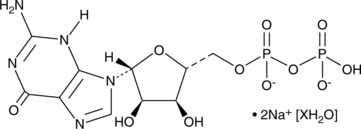
GC49033
Guanosine 5’-diphosphate (sodium salt hydrate)
A purine nucleotide
-
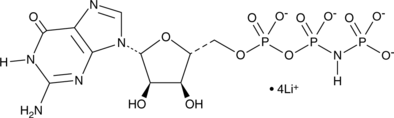
GC43798
Guanylyl Imidodiphosphate (lithium salt)
Le guanylyl imidodiphosphate (Gpp(NH)p) lithium, un analogue non hydrolysable du GTP, augmente l'activité de l'adénylate cyclase.
-
GC31650
Halofuginone (RU-19110)
L'halofuginone (RU-19110) (RU-19110), un dérivé de la fébrifugine, est un inhibiteur compétitif de la prolyl-ARNt synthétase avec un Ki de 18,3 nM.
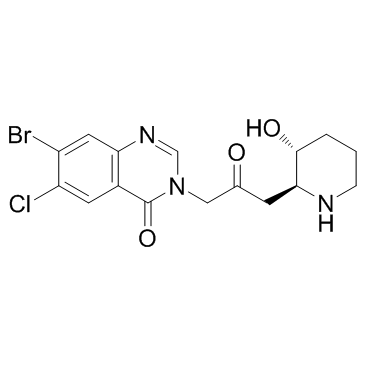
-
GC31950
Halofuginone hydrobromide (RU-19110 (hydrobromide))
Le bromhydrate d'halofuginone (RU-19110), un dérivé de la fébrifugine, est un inhibiteur compétitif de la prolyl-ARNt synthétase avec un Ki de 18,3 nM.
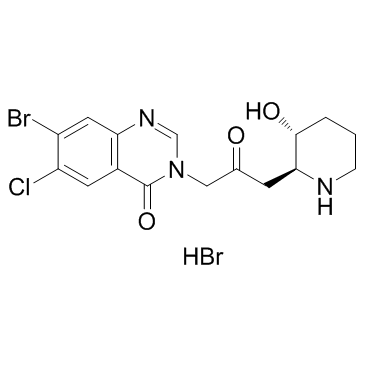
-
GC62328
HBV-IN-4
HBV-IN-4, un dérivé de la phtalazinone, est un inhibiteur de la réplication de l'ADN du VHB puissant et actif par voie orale avec une IC50 de 14 nM.
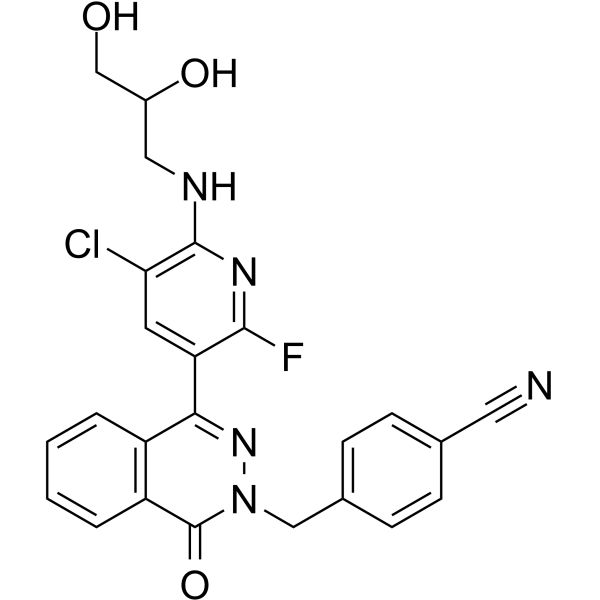
-
GC39279
hDHODH-IN-1
hDHODH-IN-1 est un inhibiteur de la dihydroorotate déshydrogénase humaine (hDHODH).
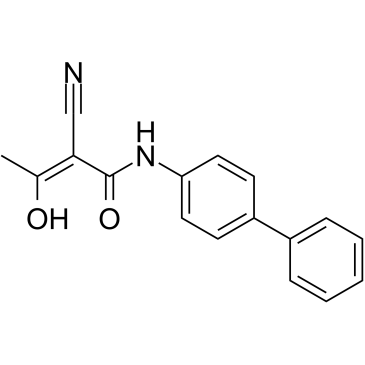
-
GC48388
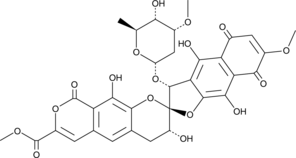
Heliquinomycin
A bacterial metabolite with diverse biological activities
-
GC40103
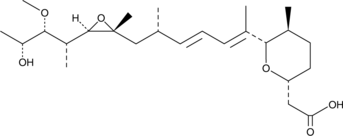
Herboxidiene
Herboxidiene, en tant qu'agent antitumoral puissant, cible l'unité SF3B du spliceosome. Herboxidiene induit également l'arrêt du cycle cellulaire en G1 et G2/M dans une ligne cellulaire de fibroblastes humains normaux WI-38.
-
GC16843
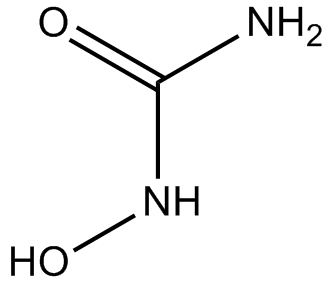
Hydroxyurea
L'hydroxyurée est un inducteur de l'apoptose cellulaire qui inhibe la synthèse de l'ADN par inhibition de la ribonucléotide réductase. L'hydroxyurée présente une activité anti-orthopoxvirus.
-
GC49694
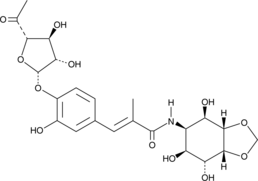
Hygromycin A
An antibiotic
-
GC48445
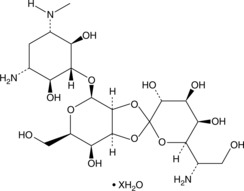
Hygromycin B (hydrate)
An aminoglycoside antibiotic
-
GC49267
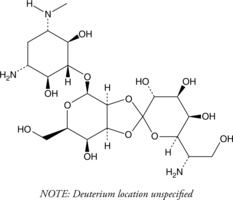
Hygromycin B-d4
An internal standard for the quantification of hygromycin
-
GC16003
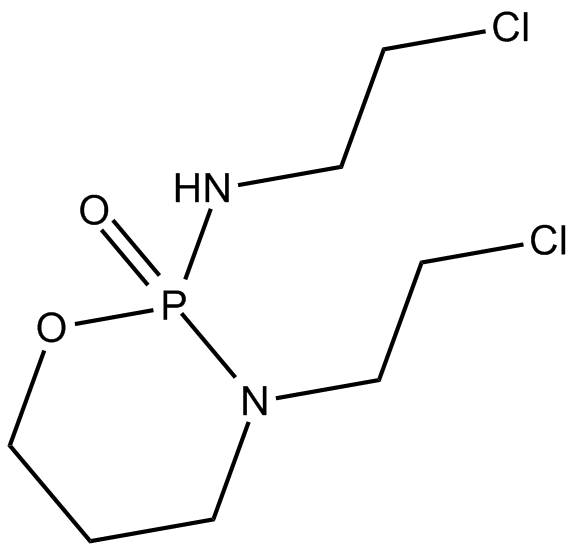
Ifosfamide
L'ifosfamide est un agent chimiothérapeutique alkylant ayant une activité contre un large éventail de tumeurs.
-
GC62137
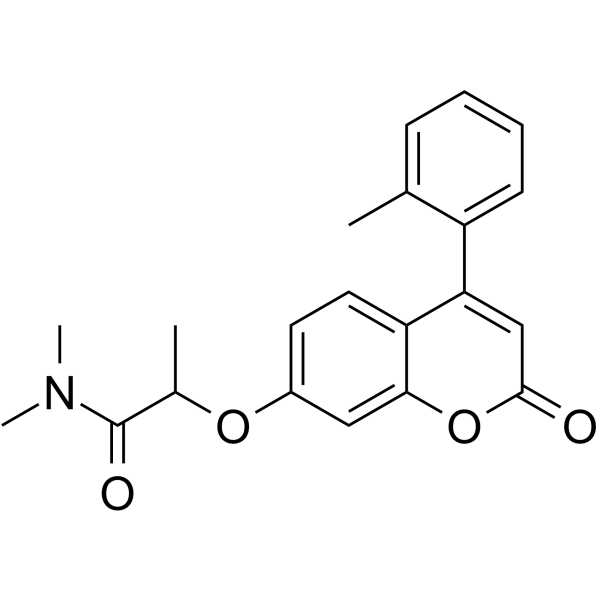
IMT1
IMT1 est un inhibiteur spécifique et non compétitif de l'ARN polymérase mitochondriale humaine (POLRMT) de première classe.
-
GC39140
Isopimpinellin
Isopimpinellin, un composé oralement actif isolé des racines de Pimpinella saxifrage.
-
GC10834
Isoxanthopterin
interferes with RNA and DNA synthesis
-
GC36367
JH-RE-06
JH-RE-06, un puissant inhibiteur de l'interface REV1-REV7 (IC50=0,78 μM ; Kd=0,42 μM), cible REV1 qui interagit avec la sous-unité REV7 de POLζ. JH-RE-06 perturbe la synthèse de la translésion mutagène (TLS) en empêchant le recrutement de POLζ mutagène. JH-RE-06 améliore la chimiothérapie.
-
GC50117
JTE 607 dihydrochloride
Le dichlorhydrate de JTE 607, un inhibiteur hautement sélectif de la synthèse des cytokines inflammatoires, protège du choc endotoxinique chez la souris.
-
GC47521
K22
An antiviral agent
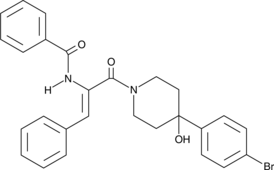
-
GC49381
L-Leucine-d10
An internal standard for the quantification of L-leucine
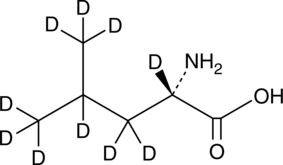
-
GC49368
L-Methionine-d3
La L-méthionine-d3 est la L-méthionine marquée au deutérium.
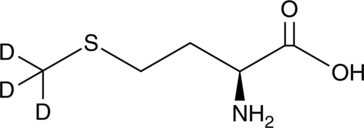
-
GC49384
L-Proline-d3
An internal standard for the quantification of L-proline
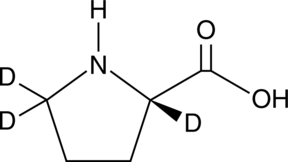
-
GC12131
L189
L189 est un inhibiteur de l'ADN ligase. L189 a un effet inhibiteur pour l'ADN Ligase I, III et IV avec des valeurs IC50 de 5 μM, 9 μM et 5 μM, respectivement. L189 n'a pas de cytotoxicité et augmente individuellement la mort cellulaire. L189 peut être utilisé pour la recherche sur le cancer.
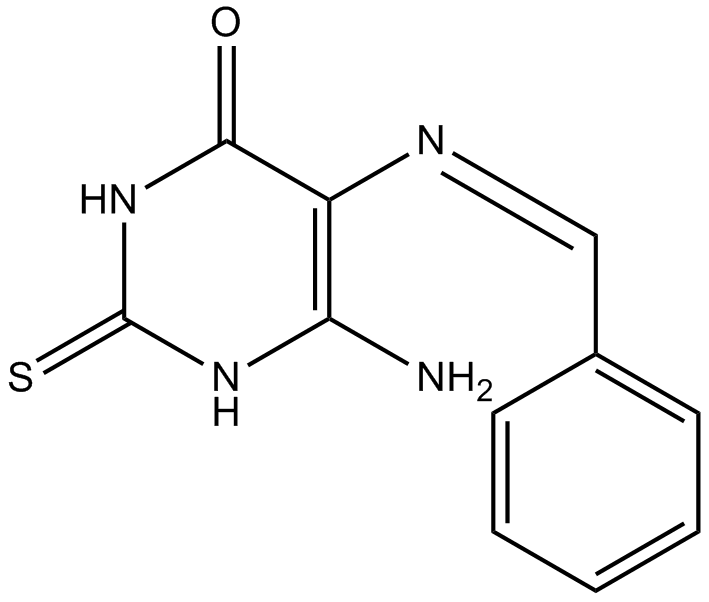
-
GC18186
L67
L67 (DNA Ligase Inhibitor) est un inhibiteur compétitif de l'ADN ligase qui inhibe efficacement les ADN ligases I/III (les deux IC50 sont de 10 μM). L67 (inhibiteur d'ADN ligase) peut causer des dommages À l'ADN nucléaire en réduisant les niveaux d'ADN mitochondrial et en augmentant les niveaux de ROS générés par les mitochondries. L67 (DNA Ligase Inhibitor) active également la voie de l'apoptose dépendante de la caspase 1 dans les cellules cancéreuses, peut être utilisé dans la recherche sur le cancer.
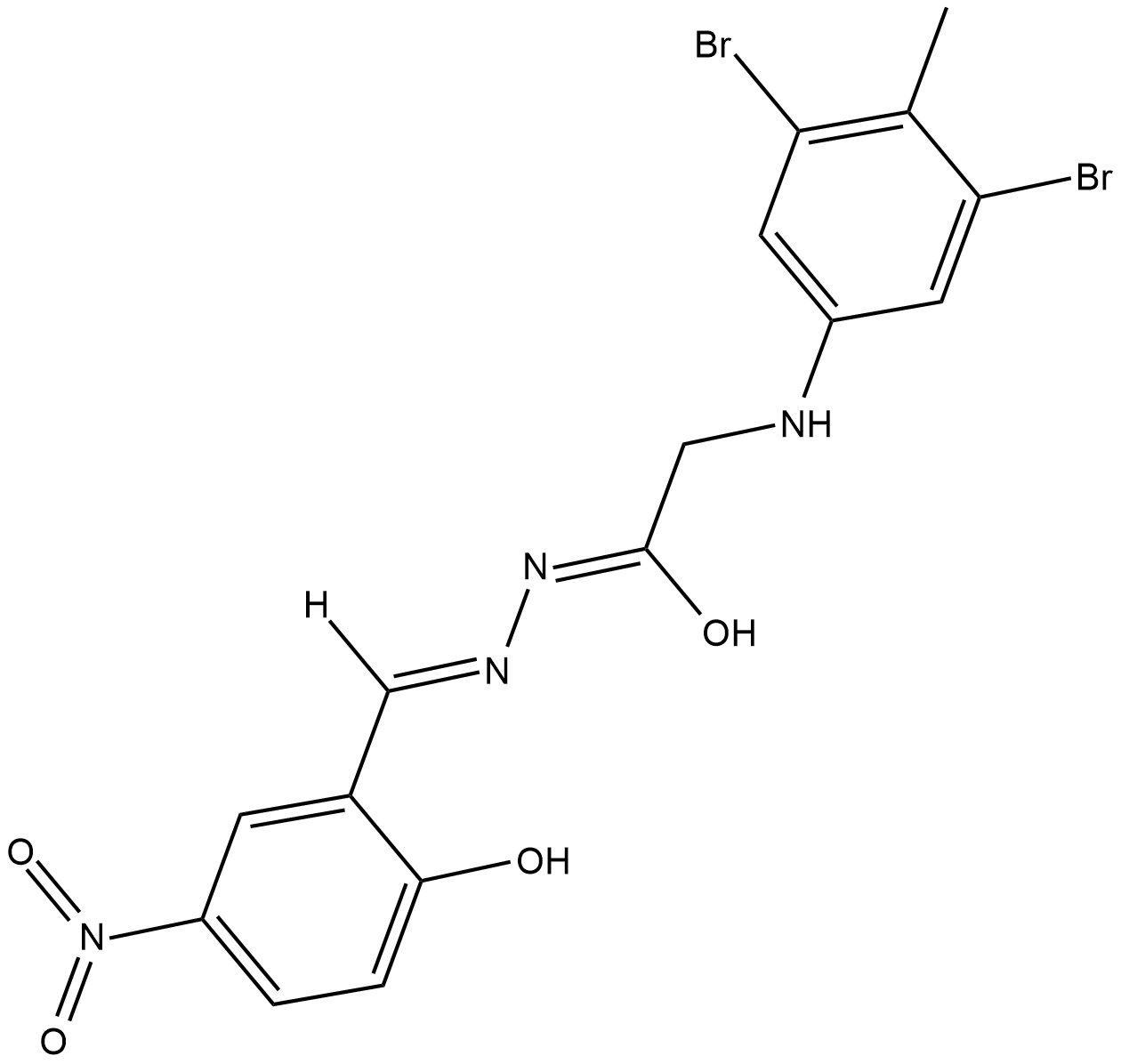
-
GC67785
L82

-
GC68436
L82-G17

-
GC39632
Laflunimus
Le laflunimus (HR325) est un agent immunosuppresseur et un analogue du métabolite actif du léflunomide A77 1726.
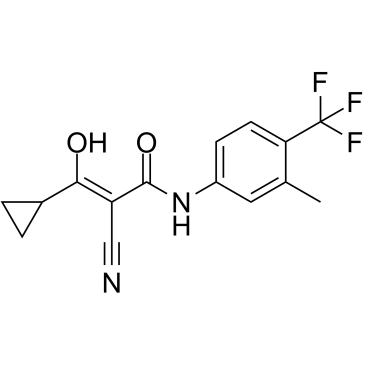
-
GC47552
Leflunomide-d4
Le léflunomide-d4 (HWA486-d4) est le léflunomide marqué au deutérium. Le léflunomide est un inhibiteur de la synthèse des pyrimidines, inhibant la dihydroorotate déshydrogénase (DHODH) et agit comme un médicament antirhumatismal modificateur de la maladie.
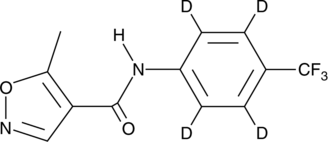
-
GC46166
Leoidin
A depsidone
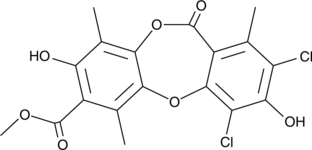
-
GC66659
LNA-A(Bz) amidite
L'amidite LNA-A(Bz) peut être utilisé pour la synthèse d'ASO (oligonucléotides antisens).
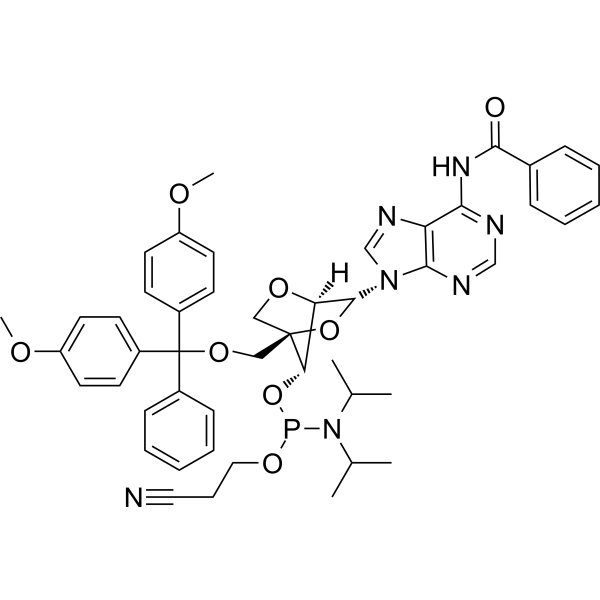
-
GC19238
Madrasin
La madrasine (DDD00107587) est un inhibiteur d'épissage qui empêche la formation d'intermédiaires et de produits d'épissage in vitro et interfère avec une ou plusieurs étapes précoces de la voie d'assemblage des splicéosomes. La madrasine peut également inhiber l'épissage du pré-ARNm in vitro et modifier l'épissage du pré-ARNm endogène dans les cellules.
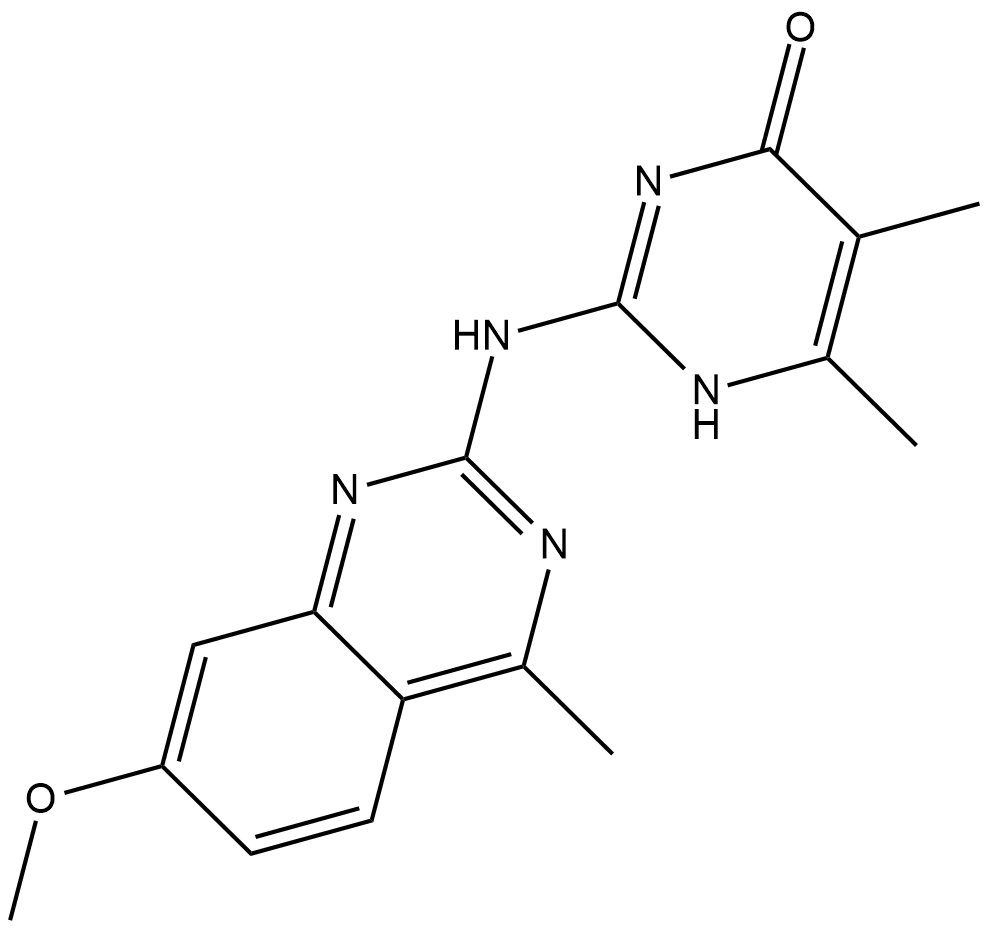
-
GC60234
Maleic hydrazide
L'hydrazide maléique est largement utilisé comme régulateur systémique de la croissance des plantes et comme herbicide. L'hydrazide maléique agit comme un inhibiteur de la synthèse des acides nucléiques et des protéines.
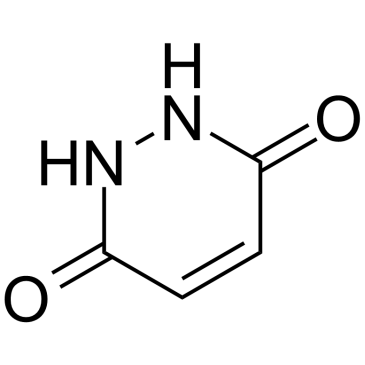
-
GC33411
MB-7133
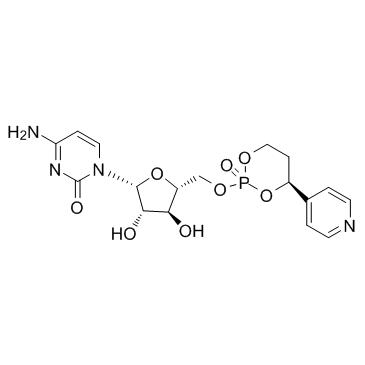
-
GC13704
Mercaptopurine (6-MP)
La mercaptopurine (6-MP) est un analogue de la purine qui agit comme un antagoniste des purines endogènes et a été largement utilisé comme agent antileucémique et médicament immunosuppresseur.
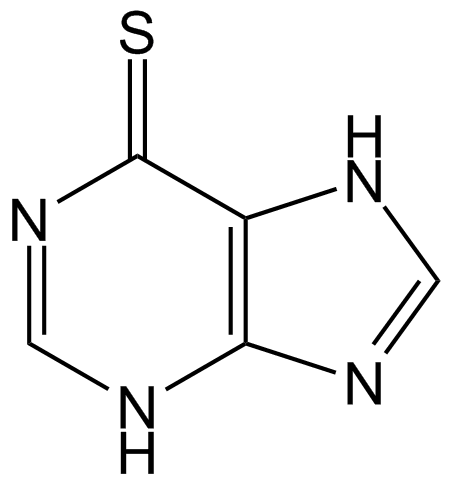
-
GC38201
Metarrestin
La métarestine (ML246) est un inhibiteur du compartiment périnucléolaire actif par voie orale, de première classe et spécifique. La métarestine perturbe la structure nucléolaire et inhibe la transcription de l'ARN polymérase (Pol) I, au moins en partie en interagissant avec le facteur d'élongation de la traduction eEF1A2. La métarestine bloque le développement métastatique et prolonge la survie dans les modèles de cancer de la souris.
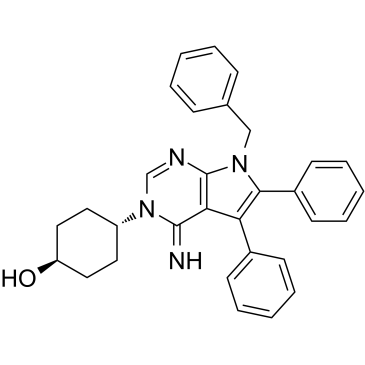
-
GC61047
Methotrexate disodium
Le méthotrexate (améthoptérine) disodique, un agent antimétabolite et antifolate, inhibe l'enzyme dihydrofolate réductase, empêchant ainsi la conversion de l'acide folique en tétrahydrofolate et inhibant la synthèse de l'ADN.
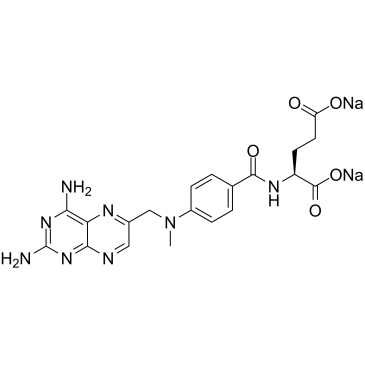
-
GC52165
Minosaminomycin
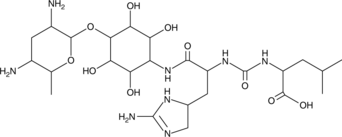
-
GC15060
Mithramycin A
A DNA-binding antitumor antibiotic
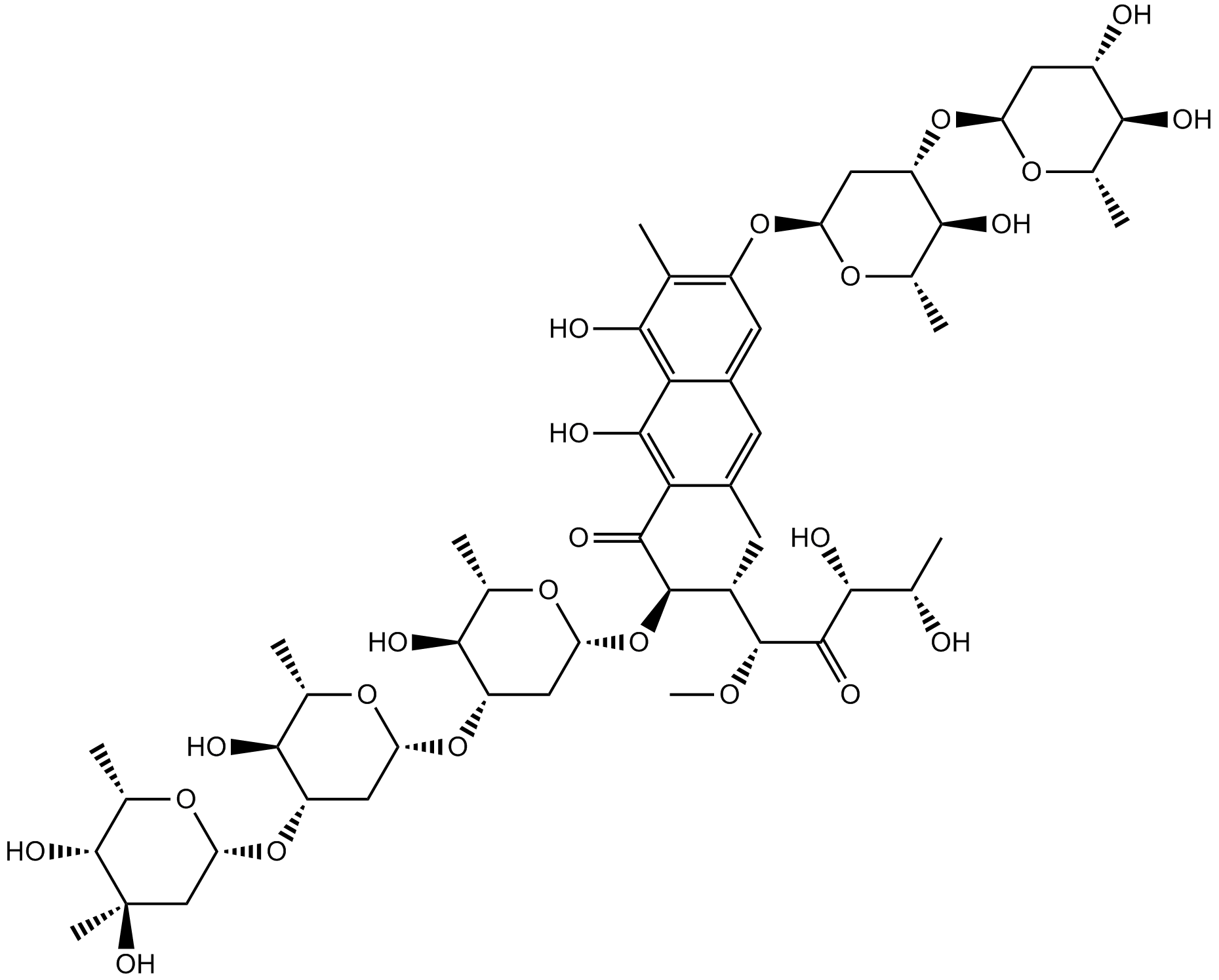
-
GC12353
Mitomycin C
La mitomycine C, un type d'antibiotique isolé à partir de Streptomyces caespitosus ou Streptomyces lavendulae, inhibe la synthèse de l'ADN grâce à un adduit covalent mitomycine C-ADN avec des valeurs EC50 de 0,14 μM dans les cellules PC3.
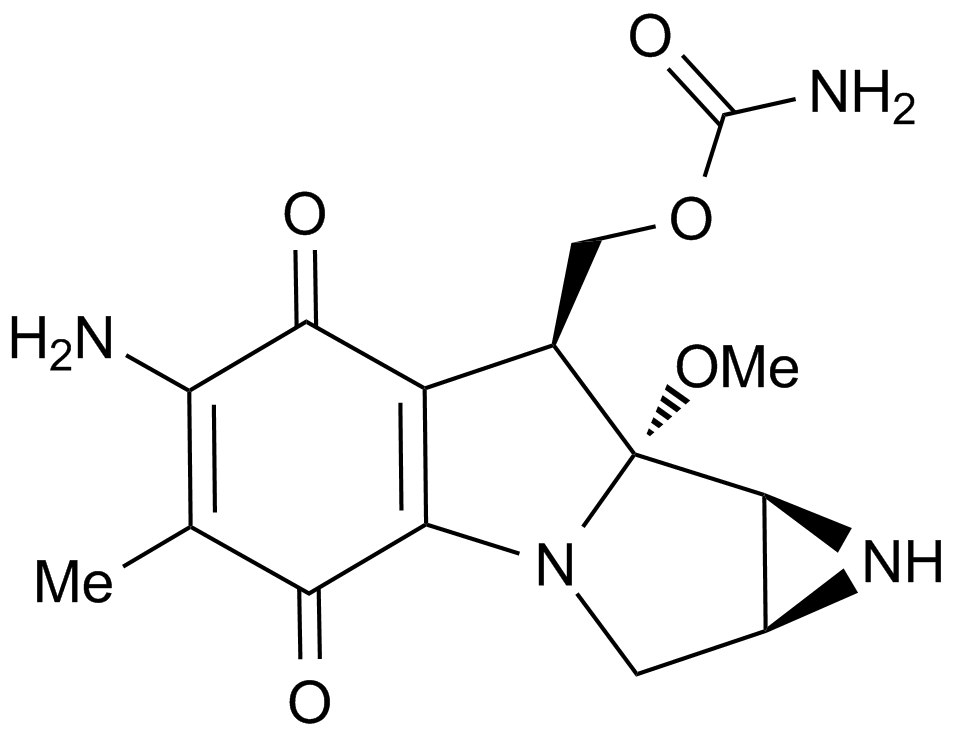
-
GC67716
Mitonafide
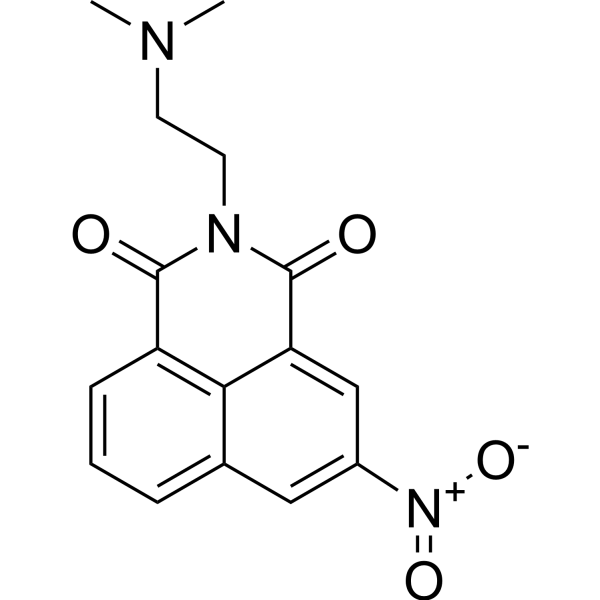
-
GC12786
ML216
ML216 (CID-49852229) est un inhibiteur puissant, sélectif et perméable aux cellules de l'activité de déroulement de l'ADN de l'hélicase BLM avec des IC50 de 2,98 μM et 0,97 μM pour BLM pleine longueur et BLM636-1298, respectivement. ML216 inhibe l'activité ATPase dépendante de l'ADNss de BLM avec un Ki de 1,76 μM. Activité antitumorale.
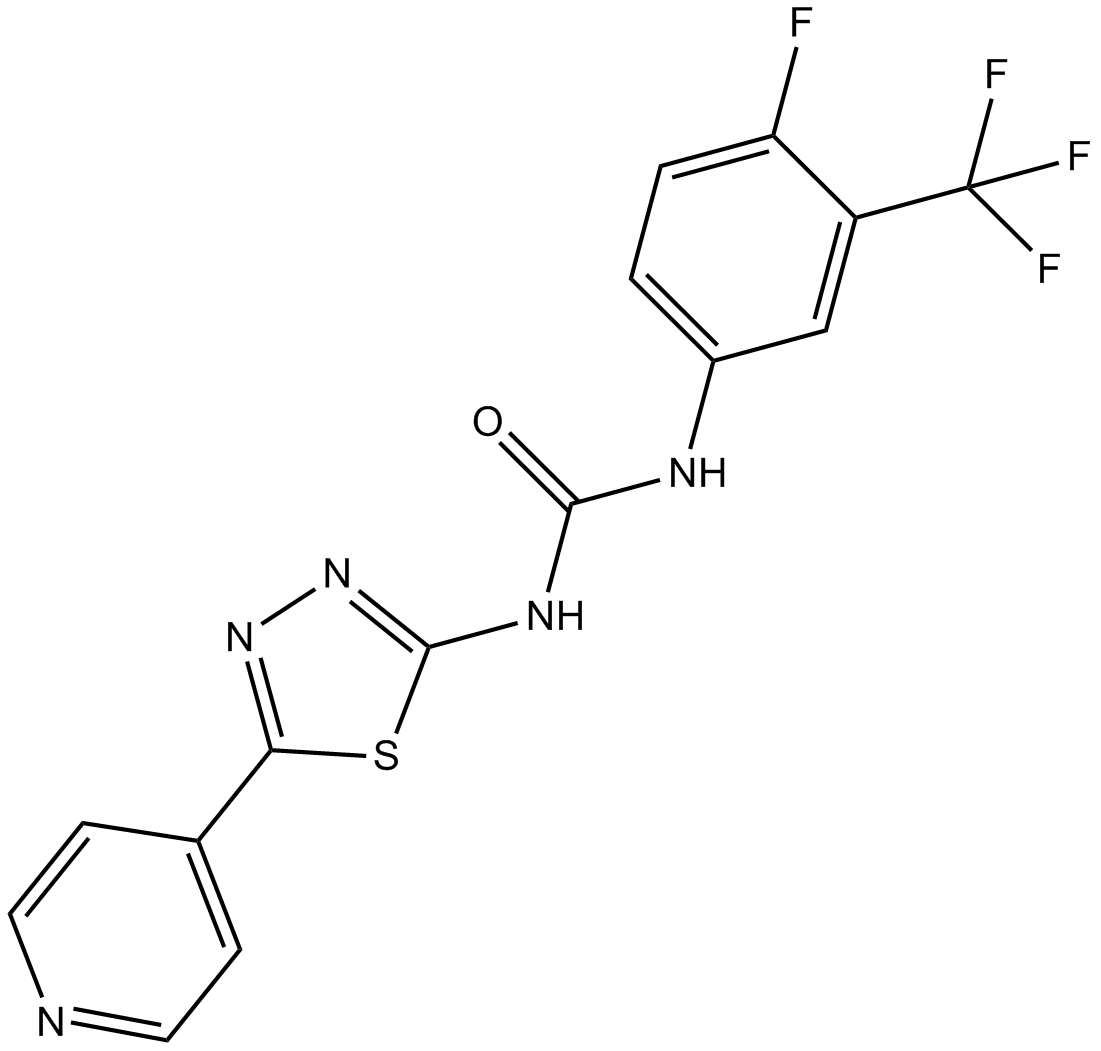
-
GC64666
ML372
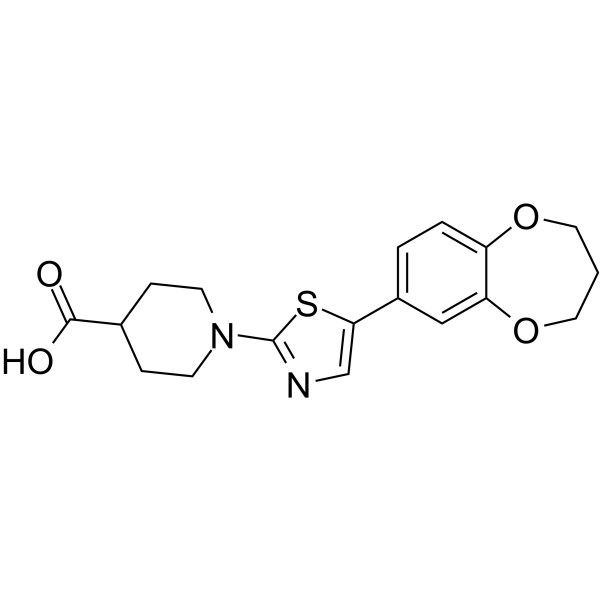
-
GC15238
Mupirocin
La mupirocine (BRL-4910A, acide pseudomonique) est un antibiotique actif par voie orale isolé de Pseudomonas fluorescens.
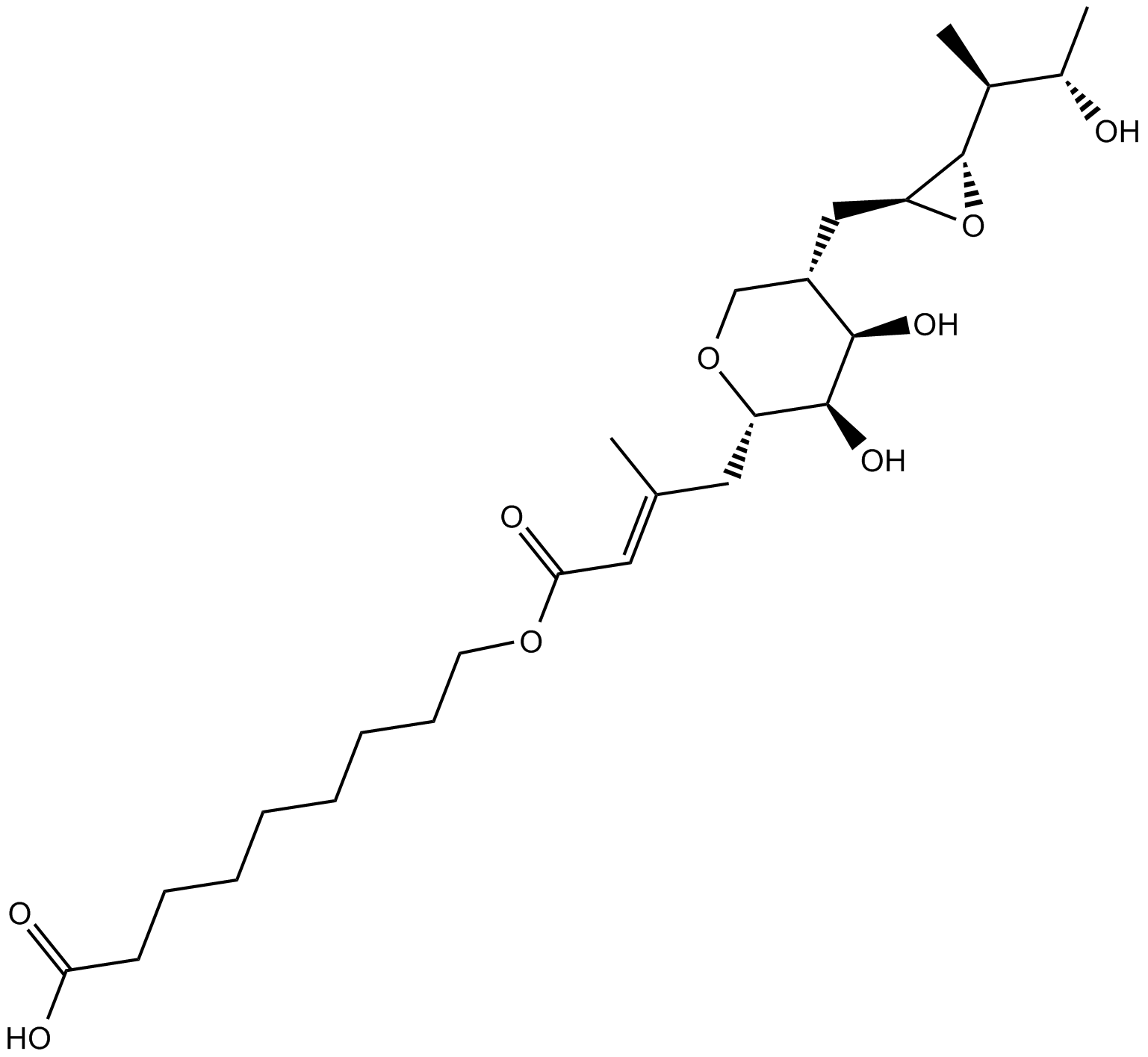
-
GC12423
Mycophenolate mofetil hydrochloride
Le chlorhydrate de mycophénolate mofétil (RS 61443) est un immunosuppresseur, un inhibiteur non compétitif, sélectif et réversible de l'inosine monophosphate déshydrogénase (IMPD) de type I/II avec des CI50 de 39 nM et 27 nM, respectivement.
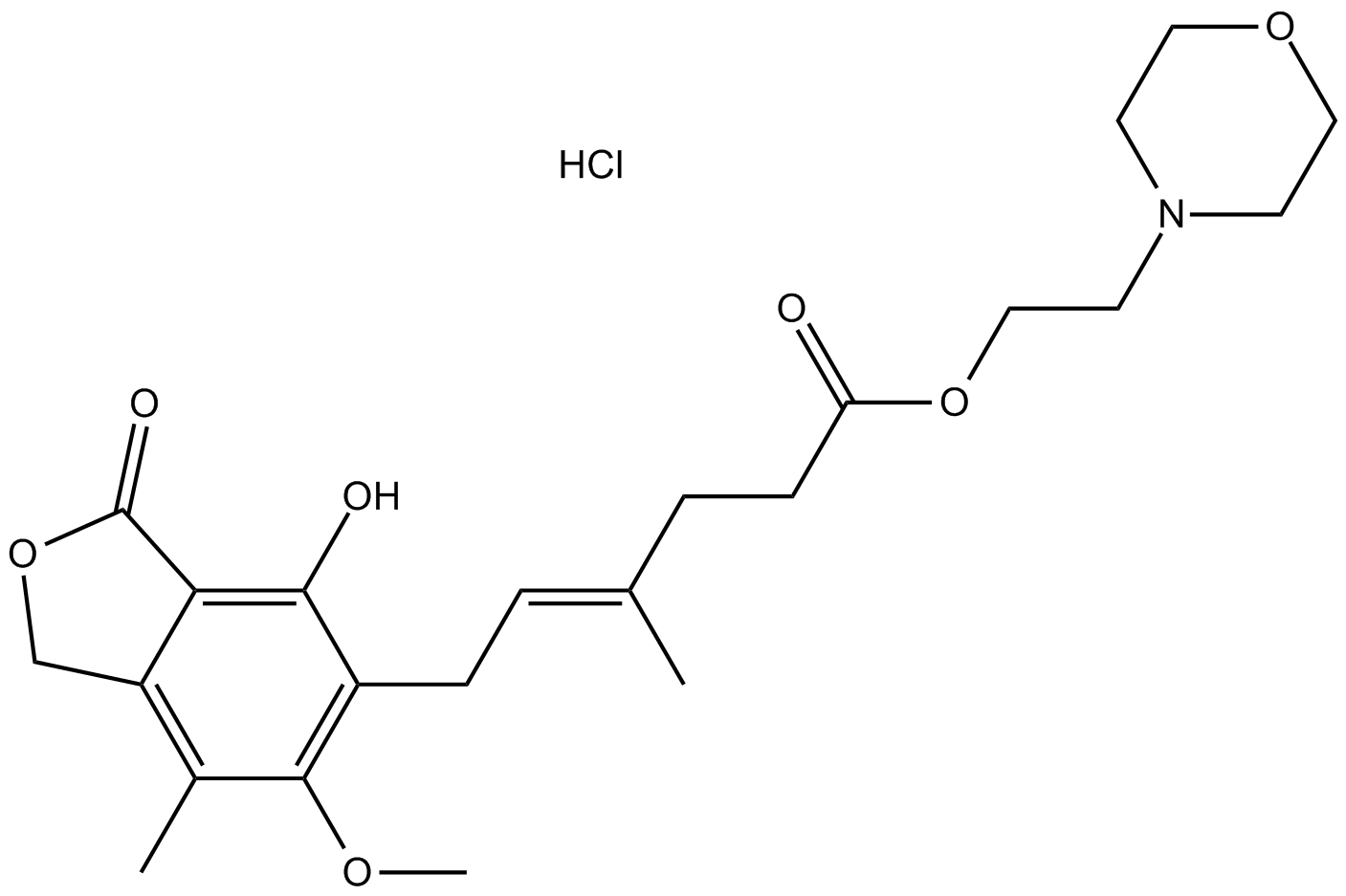
-
GC40683
N-(2-hydroxyethyl)-Naphthalimide
N-(2-hydroxyethyl)-Naphthalimide is an N-substituted 1,8-naphthalimide used as a fluorescent probe and as a precursor for protection of amine groups.
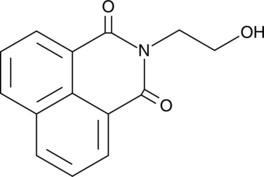
-
GC19566
N-Nitrosodiethylamine (950mg/mL)
La N-Nitrosodiéthylamine (Diéthylnitrosamine) est une dialkylnitrosoamine hépatocancérigène puissante. La N-nitrosodiéthylamine est principalement présente dans la fumée de tabac, l'eau, le fromage cheddar, les plats salés et frits et de nombreuses boissons alcoolisées. La N-nitrosodiéthylamine est responsable des modifications des enzymes nucléaires associées à la réparation/réplication de l'ADN. La N-nitrosodiéthylamine entraîne diverses tumeurs chez toutes les espèces animales. Les principaux organes cibles sont la cavité nasale, la trachée, les poumons, l'œsophage et le foie.
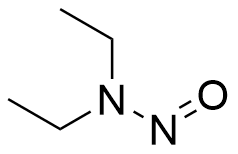
-
GC69577
N-Nitrosodiethylamine-d10
N-Nitrosodiethylamine-d10 est le déutérium de N-Nitrosodiethylamine. N-Nitrosodiethylamine (Diéthylnitrosamine) est un cancérigène efficace du groupe des nitrosamines dialkylées. N-Nitrosodiethylamine se trouve principalement dans la fumée de tabac, l'eau, le fromage cheddar sec, les aliments marinés, les aliments frits et de nombreuses boissons alcoolisées. N-Nitrosodiethylamine est responsable des changements dans les enzymes liées à la réparation / réplication d'ADN. Il peut causer divers types de tumeurs chez tous les animaux. Les organes cibles principaux sont le nez, la trachée, les poumons, l'œsophage et le foie.
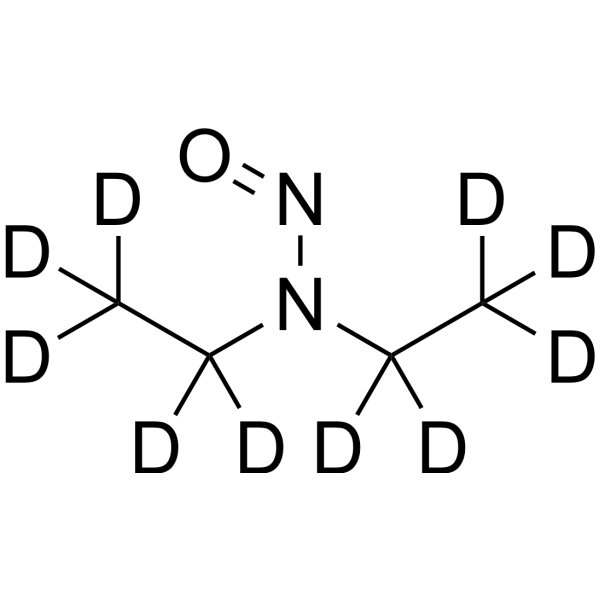
-
GC66325
N2-Acetylguanine
La N2-acétylguanine est une guanine modifiée en C2. La N2-acétylguanine se lie au GR (riboswitch guanine-guanine) avec une valeur Kd de 300 nM. La N2-acétylguanine module la terminaison transcriptionnelle. La N2-acétylguanine a le potentiel pour la recherche d'agent antimicrobien.
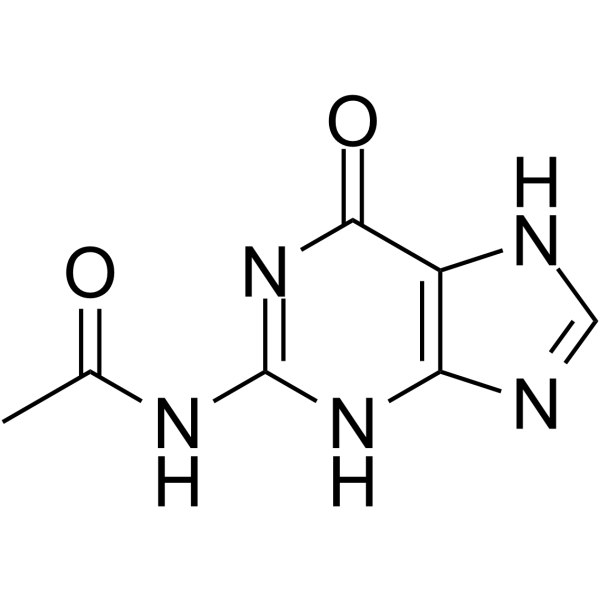
-
GC65561
N6-Methyl-dA phosphoramidite
Le N6-méthyl-dA phosphoramidite peut être utilisé dans la synthèse d'oligodésoxyribonucléotides.
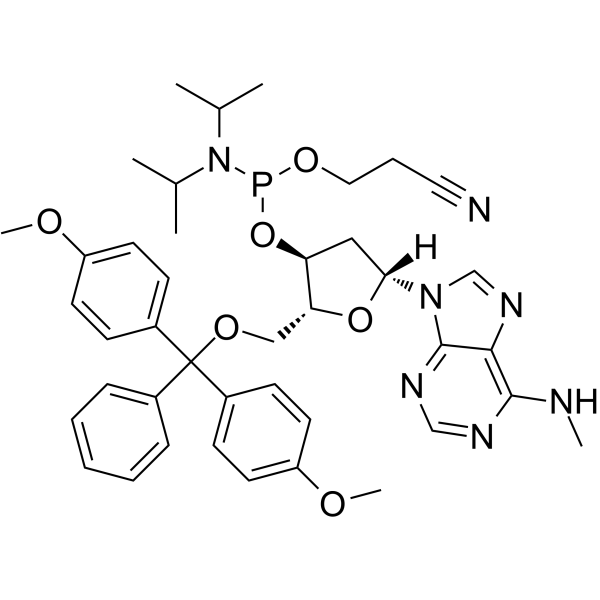
-
GC63653
NCGC00029283
NCGC00029283 est un inhibiteur d'hélicase hélicase-nucléase (WRN) du syndrome de Werner avec des CI50 de 2,3 μM, 12,5 μM et 3,4 μM pour l'hélicase WRN, BLM et FANCJ, respectivement.
-
GC15786
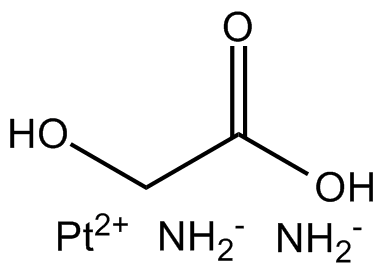
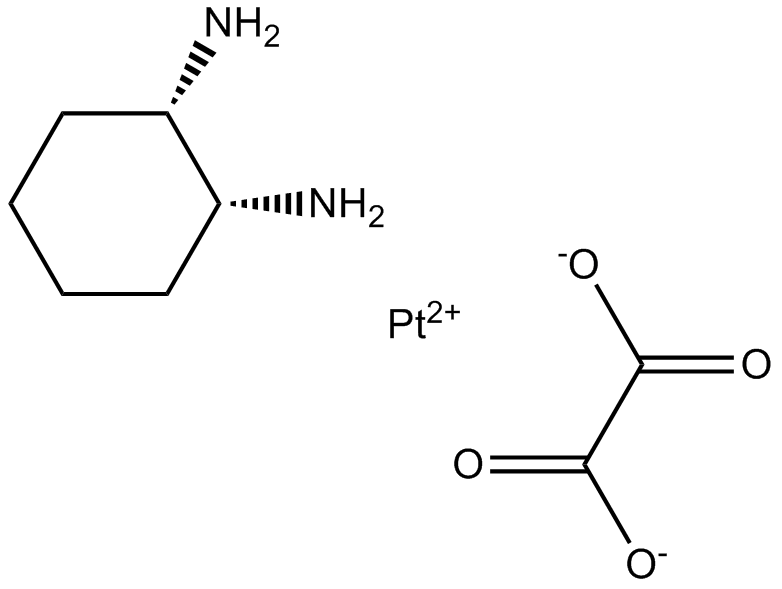
Nedaplatin
Le nédaplatine (NSC 375101D) est un dérivé du cisplatine et un agent endommageant l'ADN.
-
GC10591
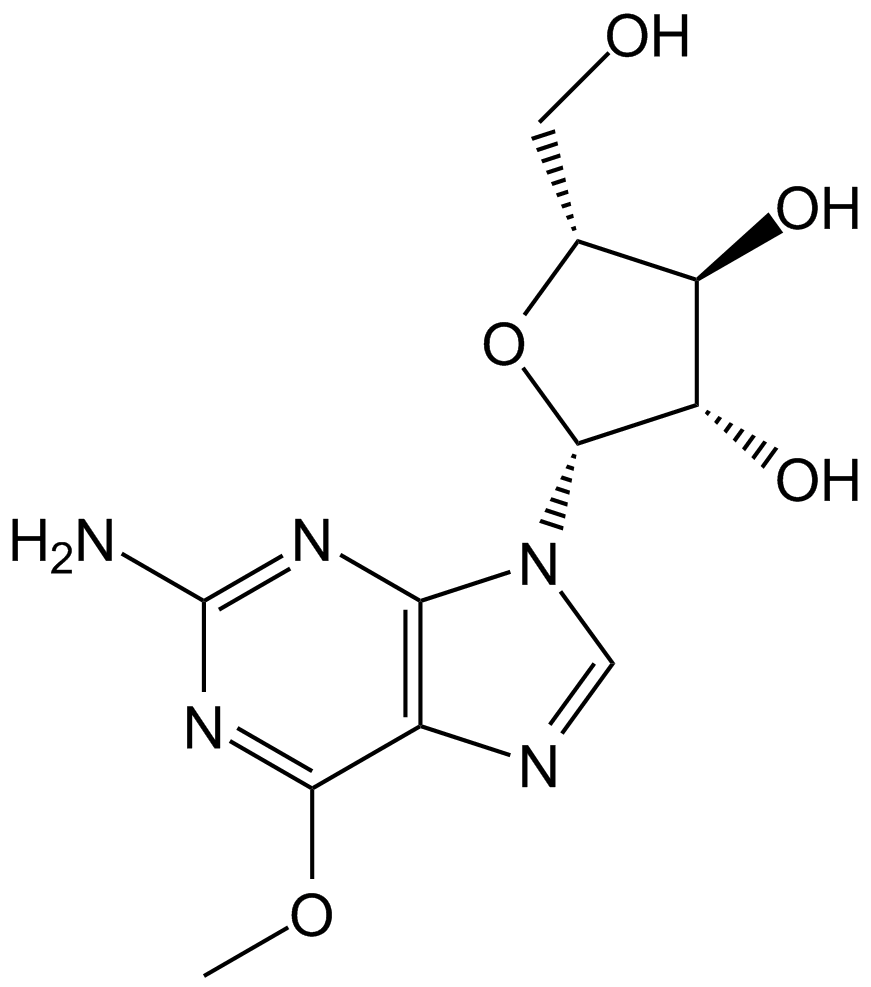
Nelarabine
La nélarabine (506U78) est un analogue nucléosidique et peut être utilisée pour la recherche sur la leucémie aiguë lymphoblastique À cellules T (T-ALL).
-
GC64493
Neocarzinostatin
La néocarzinostatine, un puissant antibiotique antitumoral endommageant l'ADN, reconnaÎt le renflement de l'ADN double brin et induit des cassures double brin de l'ADN (DSB).

-
GC60268
Neoxanthin
La néoxanthine est un caroténoÏde majeur de la xanthophylle et un précurseur de l'acide abscissique, une hormone végétale, dans les légumes À feuilles vert foncé. La néoxanthine est un puissant antioxydant et un pigment collecteur de lumière. La néoxanthine induit l'apoptose et a des actions anticancéreuses.
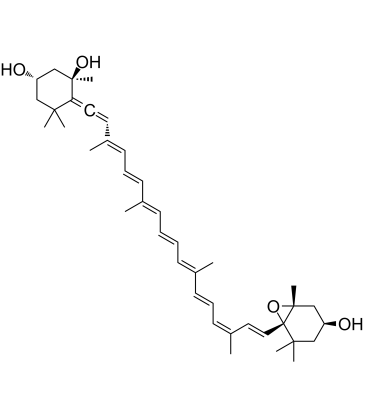
-
GC36743
Nimustine hydrochloride
Le chlorhydrate de nimustine (ACNU) est un agent de réticulation et d'alkylation de l'ADN, qui induit des lésions bloquant la réplication de l'ADN et des cassures double brin de l'ADN et inhibe la synthèse de l'ADN, couramment utilisé en chimiothérapie pour les glioblastomes.
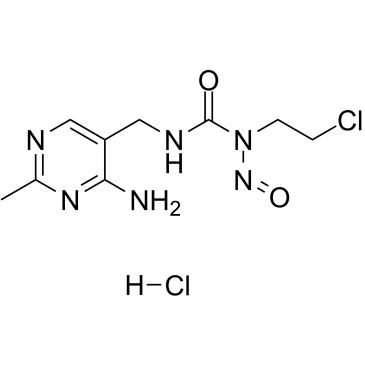
-
GC64559
NITD-2
NITD-2, un inhibiteur de la polymérase du virus de la dengue (DENV), inhibe l'élongation de l'ARN médiée par le RdRp du DENV.
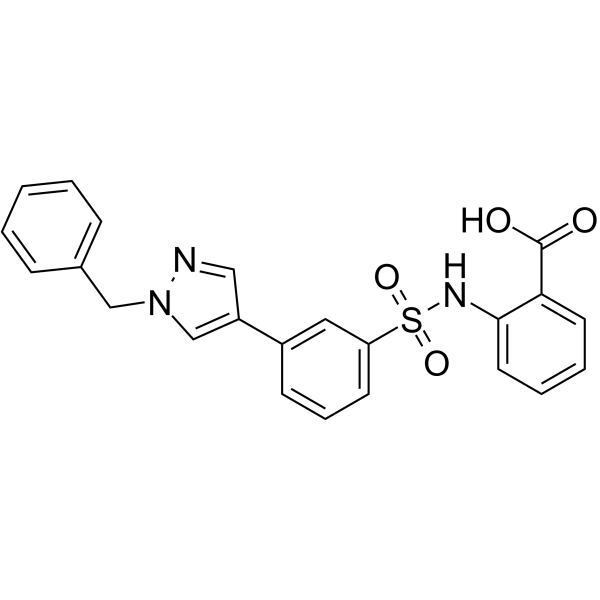
-
GC19506
NITD008
NITD008 est un inhibiteur de flavivirus puissant et sélectif qui peut inhiber le virus de la dengue de type 2 (DENV-2) avec une CE50 de 0,64 μM.
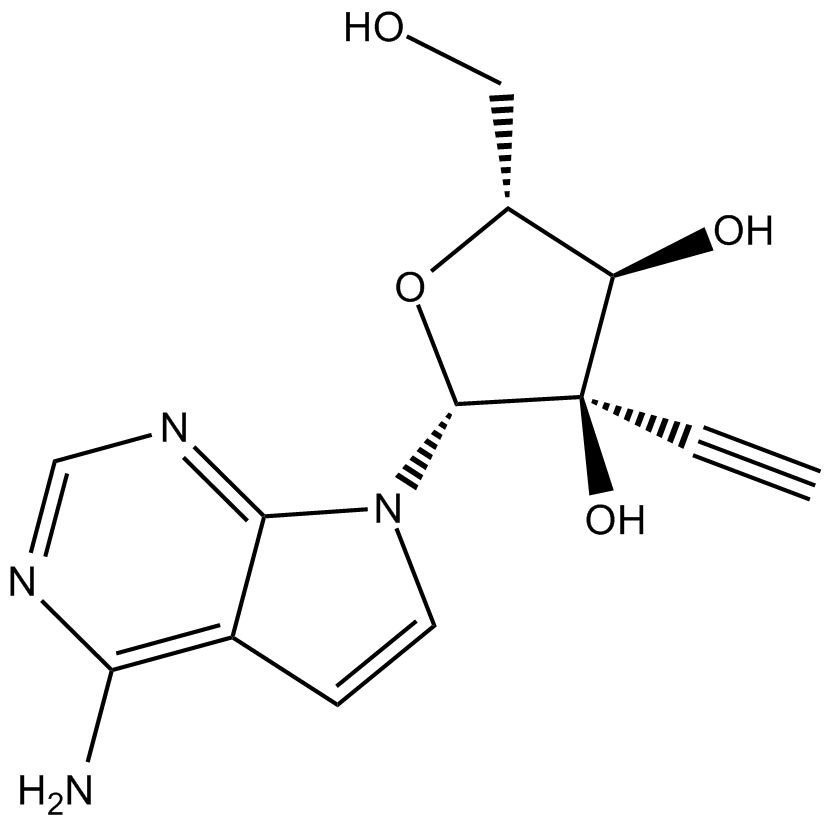
-
GC19263
NKP-1339
Le NKP-1339 (IT-139; KP-1339) est le premier agent anticancéreux À base de ruthénium en cours de développement contre le cancer solide avec des effets secondaires limités. Le NKP-1339 induit l'arrêt du cycle cellulaire G2/M, le blocage de la synthèse d'ADN et l'induction de l'apoptose via la voie mitochondriale. Le NKP-1339 a un potentiel de ciblage tumoral élevé, se lie fortement aux protéines sériques telles que l'albumine et la transferrine et s'active dans le milieu tumoral réducteur.
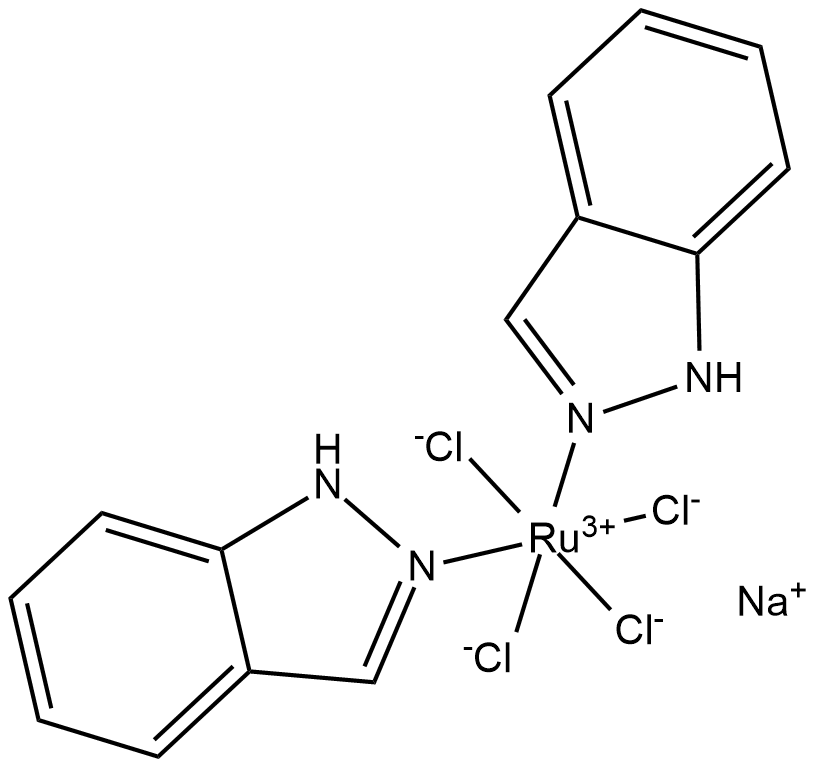
-
GC62679
NSAH
La NSAH est un inhibiteur réversible et compétitif de la ribonucléotide réductase (RR) non nucléosidique, avec une IC50 acellulaire de 32 μM et une IC50 cellulaire d'environ 250 nM, respectivement.
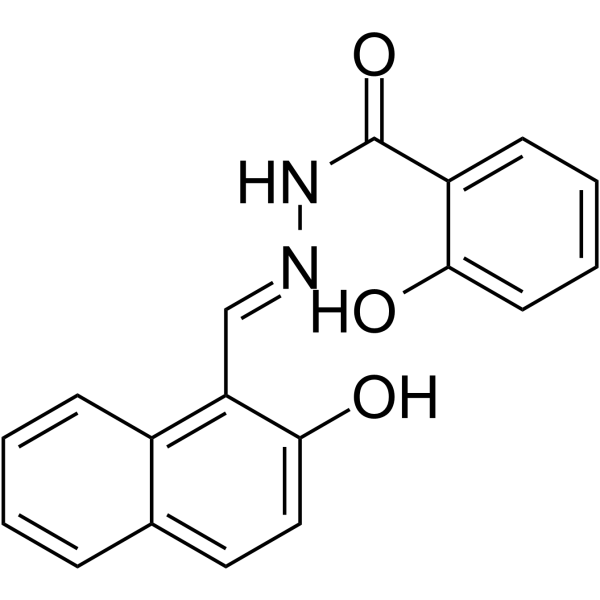
-
GC69598
NSC 80467
NSC 80467 est un agent de dommages à l'ADN qui inhibe sélectivement la survivine. NSC 80467 inhibe prioritairement la synthèse d'ADN, induisant ainsi deux marqueurs de dommages à l'ADN, γH2AX et pKAP1.
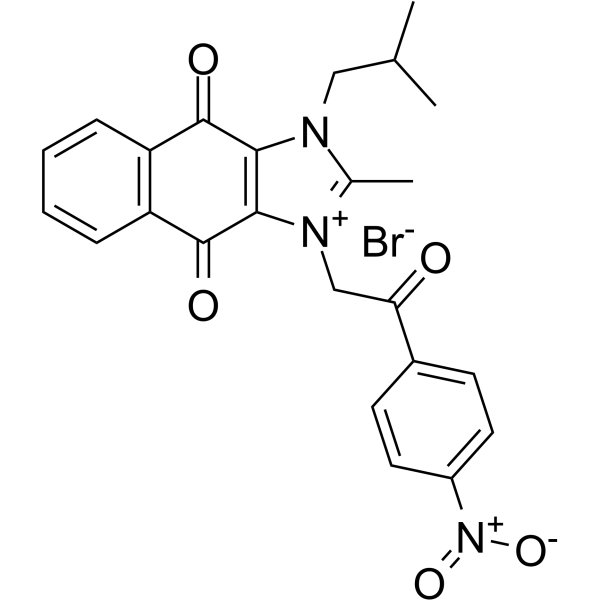
-
GC64103
NSC639828
NSC639828 est un puissant inhibiteur de l'ADN polymérase α avec une IC50 de 70 μM. NSC639828 a une activité antitumorale élevée. NSC639828 a le potentiel de rechercher des maladies cancéreuses.
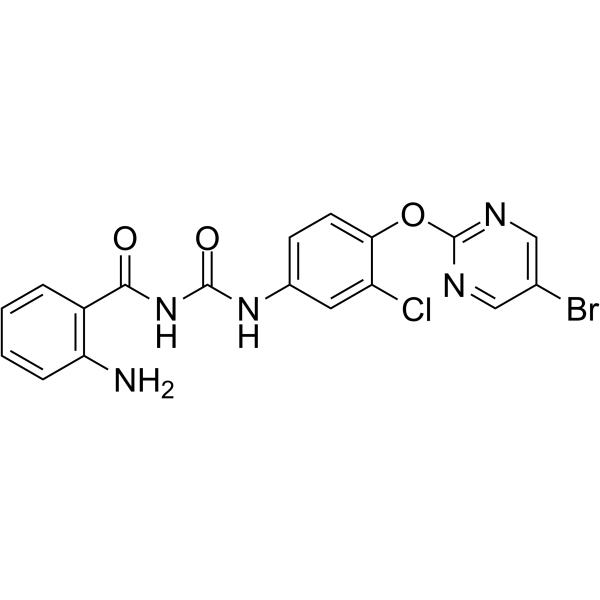
-
GC64293
Nusinersen
Nusinersen est un médicament oligonucléotidique antisens qui modifie l'épissage de l'ARN pré-messager du gène SMN2 et favorise ainsi une production accrue de protéine SMN pleine longueur .

-
GC63123
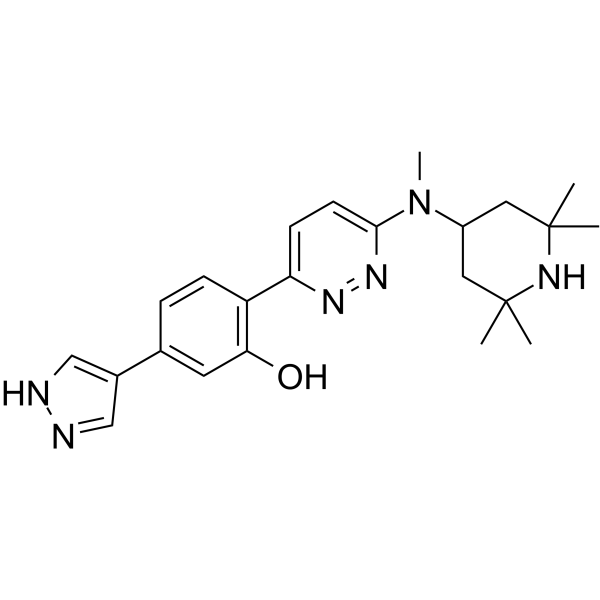
NVS-SM2
NVS-SM2 est un activateur d'épissage SMN2 puissant, actif par voie orale et pénétrant dans le cerveau avec une EC50 de 2 nM pour SMN.
-
GC49743
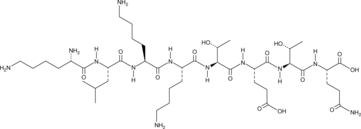
Octapeptide-2
A thymosin β4-derived peptide
-
GC64296
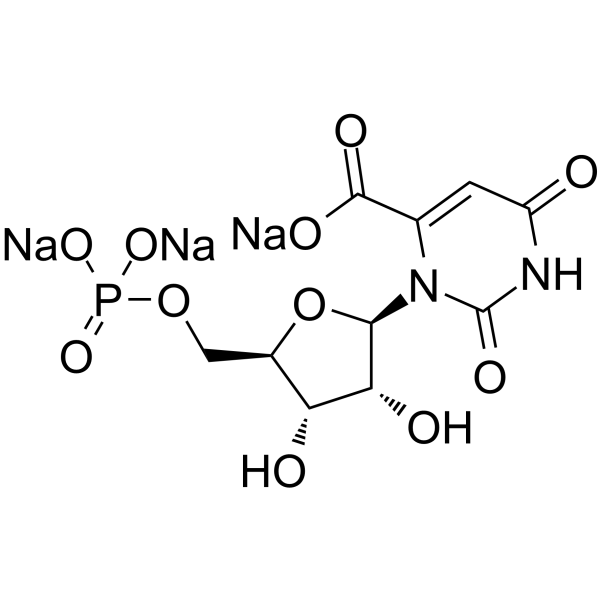
Orotidine 5′-monophosphate trisodium
L'orotidine 5'-monophosphate trisodique est un nucléotide pyrimidique.
-
GC17716
Oxaliplatin
Oxaliplatine est un médicament de chimiothérapie cytotoxique utilisé pour traiter le cancer.
-
GC49251
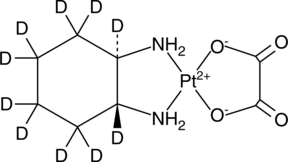
Oxaliplatin-d10
An internal standard for the quantification of oxaliplatin
-
GC14107
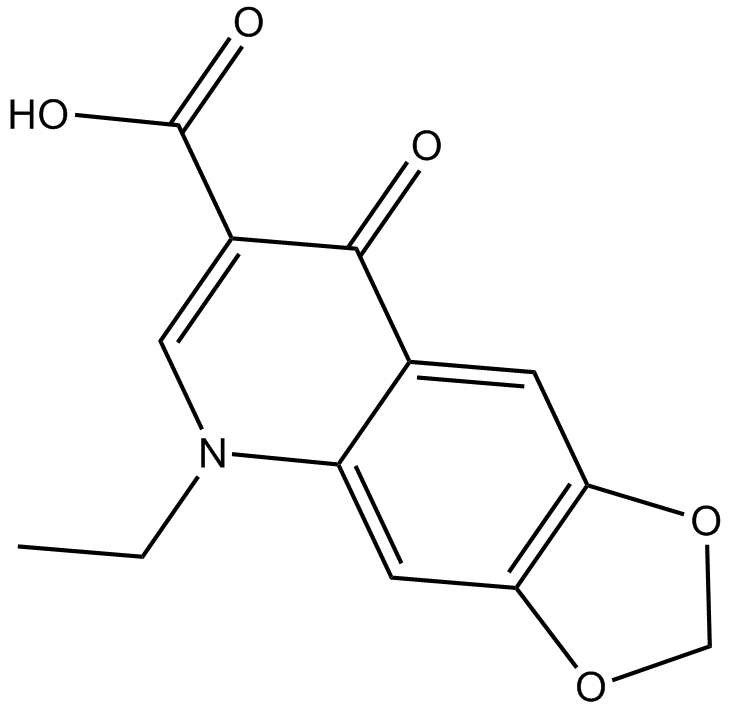
Oxolinic acid
L'acide oxolinique est un antibiotique contre les bactéries Gram-négatives et Gram-positives.
-
GC17232
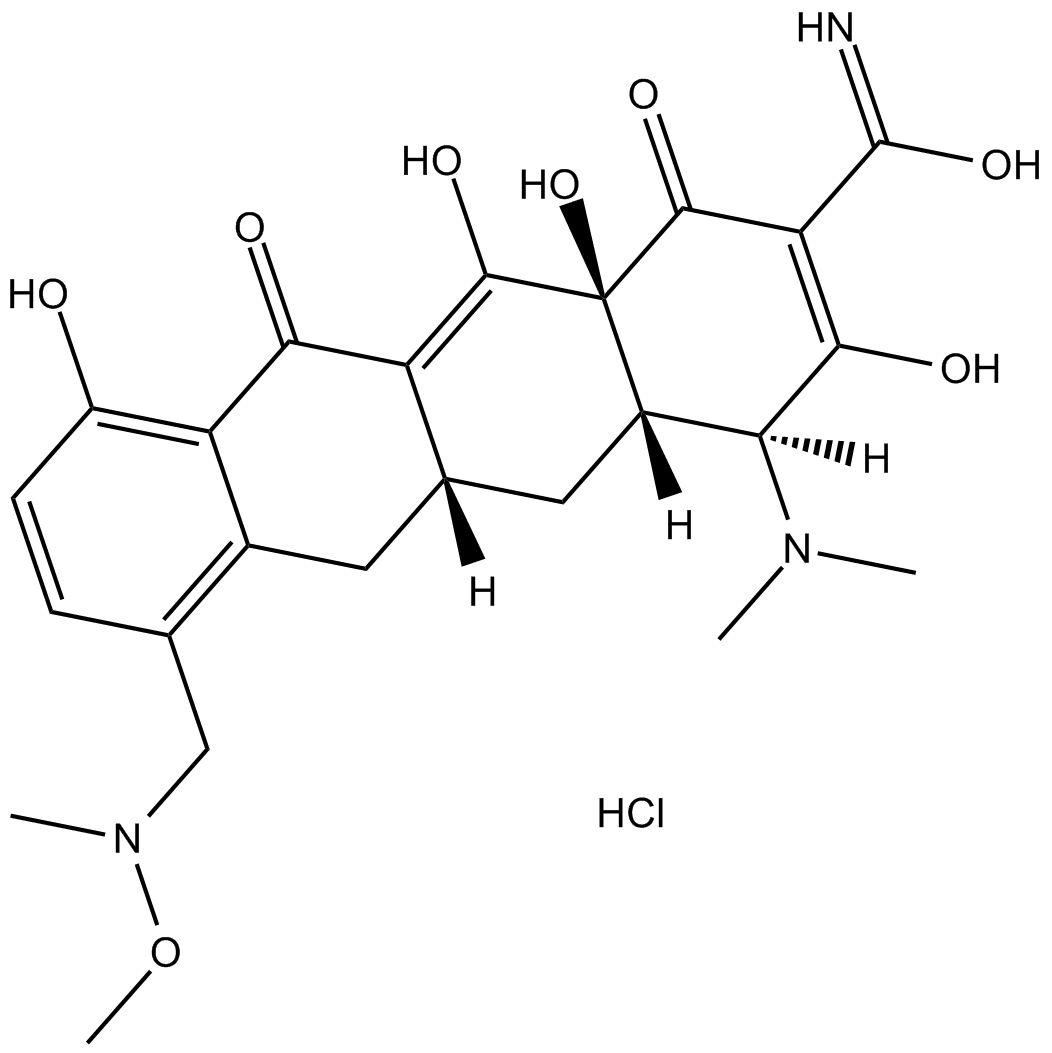
P005672 hydrochloride
Le chlorhydrate de P005672 est un antibiotique de la classe des tétracyclines À spectre étroit.
-
GC14849
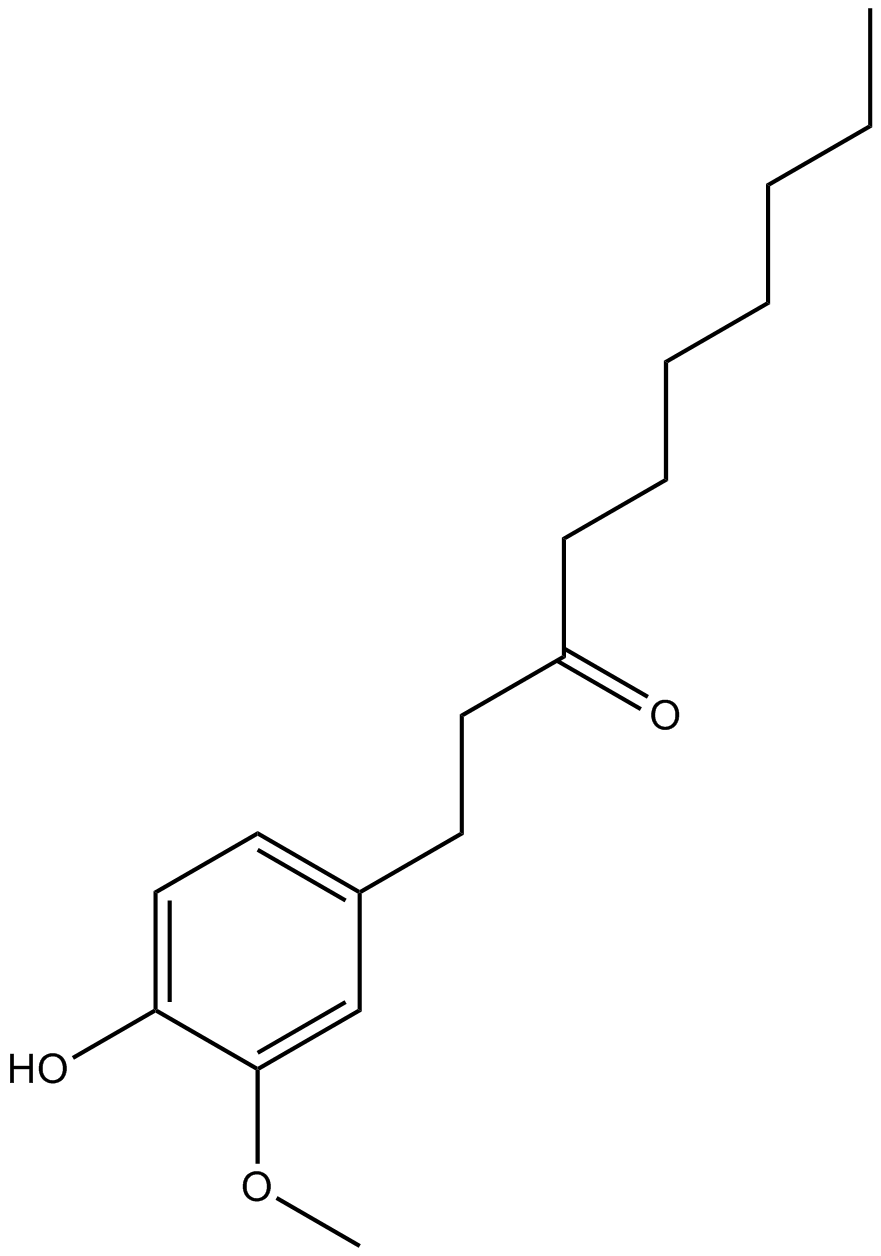
Paradol
A phenolic ketone with diverse biological activities
-
GC65891
PCLX-001
Le PCLX-001 est un inhibiteur double de la N-myristoyltransférase (NMT) actif par voie orale, À petite molécule, avec des CI50 de 5 nM (NMT1) et 8 nM (NMT2), respectivement. PCLX-001 présente une activité anti-tumorale et inhibe la signalisation précoce du récepteur des cellules B (BCR), peut être utilisé pour la recherche sur les tumeurs malignes des cellules B.
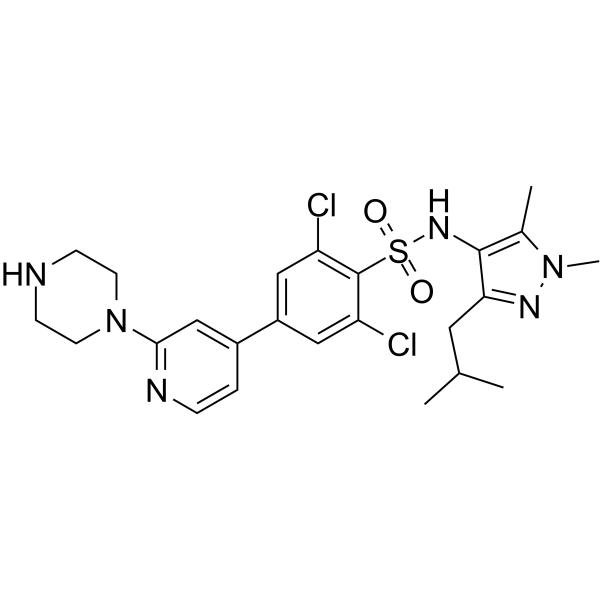
-
GC44591
Pemetrexed (sodium salt hydrate)
Le pemetrexed (sel de sodium hydraté) est un nouvel antifolate, les valeurs Ki du pentaglutamate de LY231514 sont de 1,3, 7,2 et 65 nM pour inhiber la thymidylate synthase (TS), la dihydrofolate réductase (DHFR) et la glycinamide ribonucléotide formyltransférase (GARFT), respectivement .
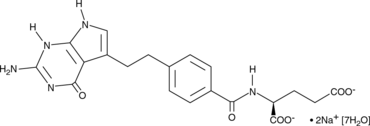
-
GC30695
Phen-DC3
Phen-DC3 est un ligand spécifique du G-quadruplex (G4) qui peut inhiber les hélicases FANCJ et DinG avec des IC50 de 65± 6 et 50± 10 nM, respectivement.
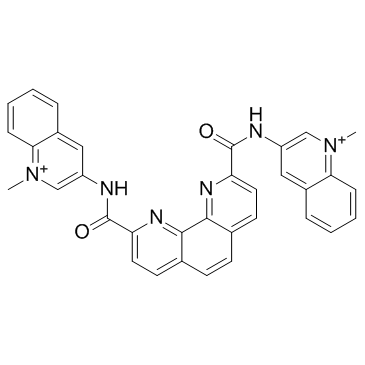
-
GC14066
Procaine
La procaÏne est un agent de déméthylation de l'ADN.
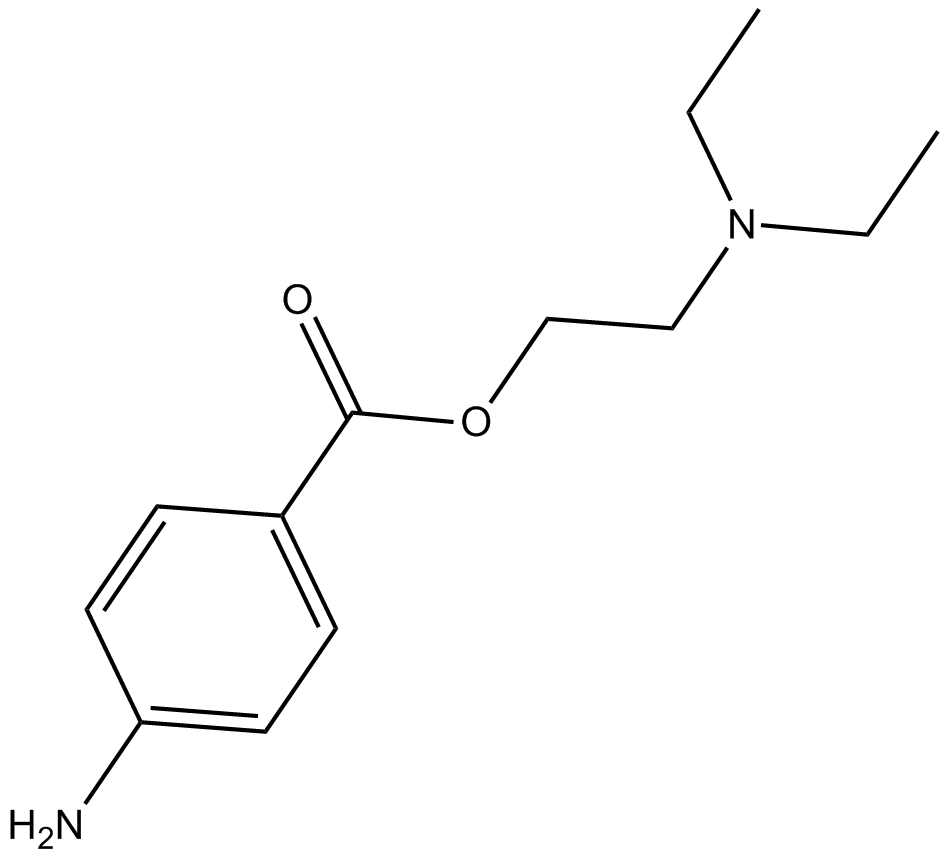
-
GC44685
Procaine (hydrochloride)
Procaine (hydrochloride) is an analytical reference standard categorized as a local anesthetic that is used as an adulterant.
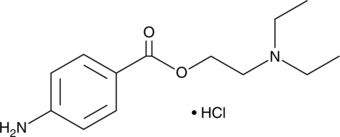
-
GC11793
Procarbazine HCl
Le chlorhydrate de procarbazine est un agent alkylant actif par voie orale, avec une activité anticancéreuse. Le chlorhydrate de procarbazine peut être utilisé dans la recherche sur la maladie de Hodgkin's.
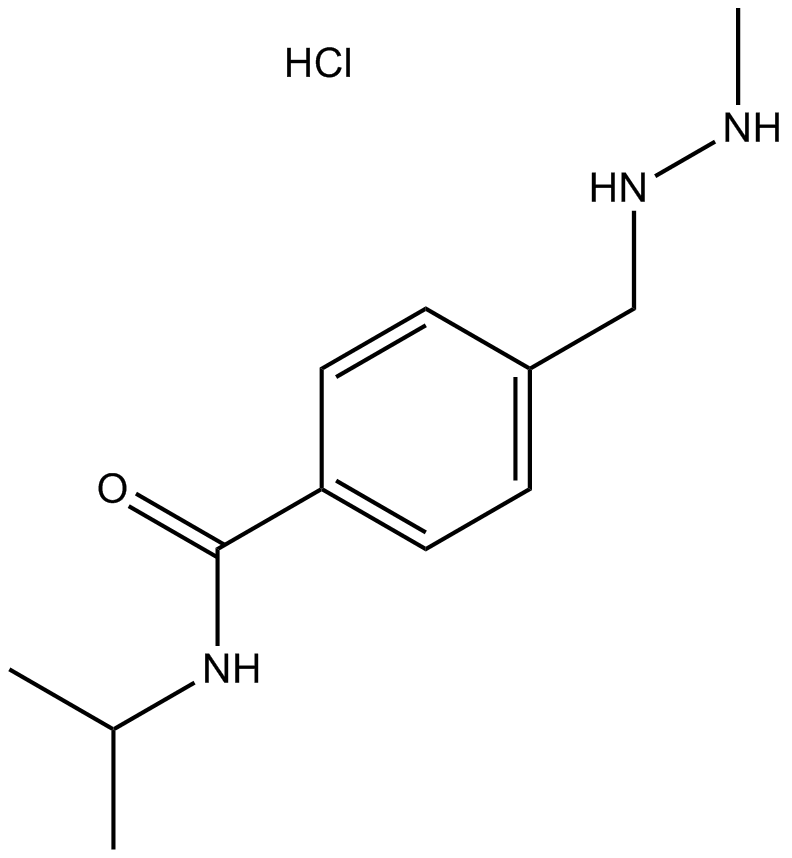
-
GC60310
Psammaplin A
La psammapline A, un métabolite marin, est un puissant inhibiteur des HDAC et des ADN méthyltransférases.
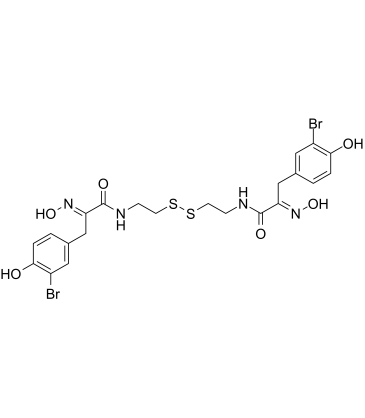
-
GC49053
Pseudomonic Acid (lithium salt)
An antibiotic
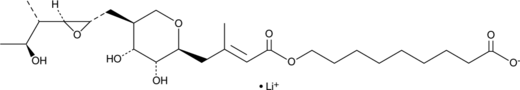
-
GC38466
Pseudouridimycin
La pseudouridimycine (PUM), un antibiotique, est un inhibiteur sélectif de l'ARN polymérase bactérienne (RNAP).