Mitochondrial Metabolism
Mitochondria use multiple carbon fuels to produce ATP and metabolites, including pyruvate, which is generated from glycolysis; amino acids such as glutamine; and fatty acids. These carbon fuels feed into the TCA cycle in the mitochondrial matrix to generate the reducing equivalents NADH and FADH2, which deliver their electrons to the electron transport chain. Mitochondria are complex organelles that play an important role in many facets of cellular function, from metabolism to immune regulation and cell death. Mitochondria are actively involved in a wide variety of cellular processes and molecular interactions, such as calcium buffering, lipid flux, and intracellular signaling. It is increasingly recognized that mitochondrial dysfunction is a hallmark of many diseases such as obesity/diabetes, cancer, cardiovascular and neurodegenerative diseases. Mitochondrial metabolism is a key determinant of tumor progression by impacting on functions such as epithelial-to-mesenchymal transition. Mitochondrial metabolism and derived oncometabolites shape the epigenetic landscape to alter aggressiveness features of cancer cells. Changes in mitochondrial metabolism are relevant for the survival of tumors in response to therapy.
Ziele für Mitochondrial Metabolism
Produkte für Mitochondrial Metabolism
- Bestell-Nr. Artikelname Informationen
-
GC62137
IMT1
IMT1 ist ein erstklassiger spezifischer und nicht kompetitiver Hemmer der humanen mitochondrialen RNA-Polymerase (POLRMT).
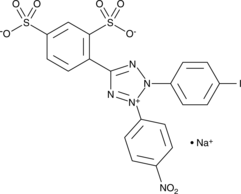
-
GC43909
Iromycin A
Iromycin A is a bacterial pyridone metabolite that inhibits nitric oxide synthase (NOS) activity, with selectivity for NOS III (endothelial NOS) over NOS I (neuronal NOS).
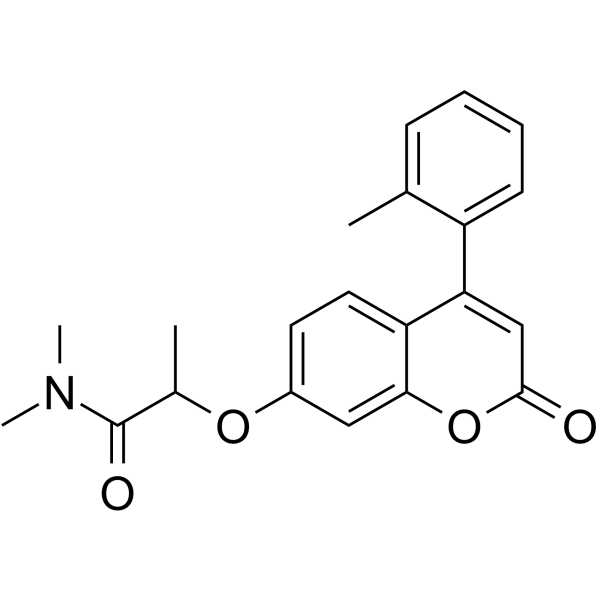
-
GC43912
Isoapoptolidin
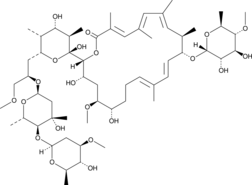
Isoapoptolidin is a stable derivative of apoptolidin that demonstrates 10-fold reduced F1FO-ATP synthase inhibitory activity compared to apoptolidin (IC50s = 17 and 0.7 μM, respectively).
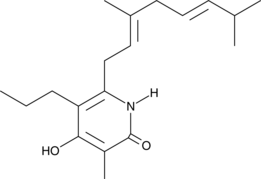
-
GC43922
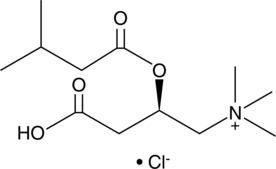
Isovaleryl-L-carnitine (chloride)
Isovaleryl-L-Carnitin (Chlorid), ein Produkt des Katabolismus von L-Leucin, ist ein starker Aktivator der Ca2+--abhÄngigen Proteinase (Calpain) menschlicher Neutrophiler.
-
GC47531
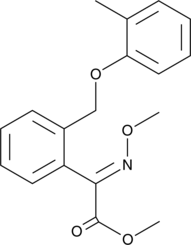
Kresoxim-methyl
Kresoxim-methyl (BAS 490 F), ein Fungizid auf Strobilurin-Basis, hemmt die Atmung am Komplex III (Cytochrom bc1-Komplex).
-
GC47548
L-Carnitine-d3 (chloride)
L-Carnitin-d3 ((R)-Carnitin-d3) Hydrochlorid ist das mit Deuterium markierte L-Carnitin.

-
GC52094
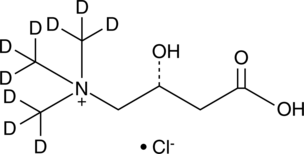
L-Carnitine-d9 (chloride)
L-Carnitin-d9 (Chlorid) ist das mit Deuterium bezeichnete L-Carnitin-Chlorid.
-
GC49382
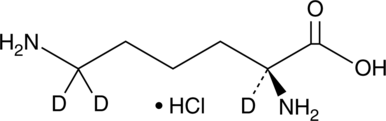
L-Lysine-d3 (hydrochloride)
An internal standard for the quantification of L-lysine
-
GC19221
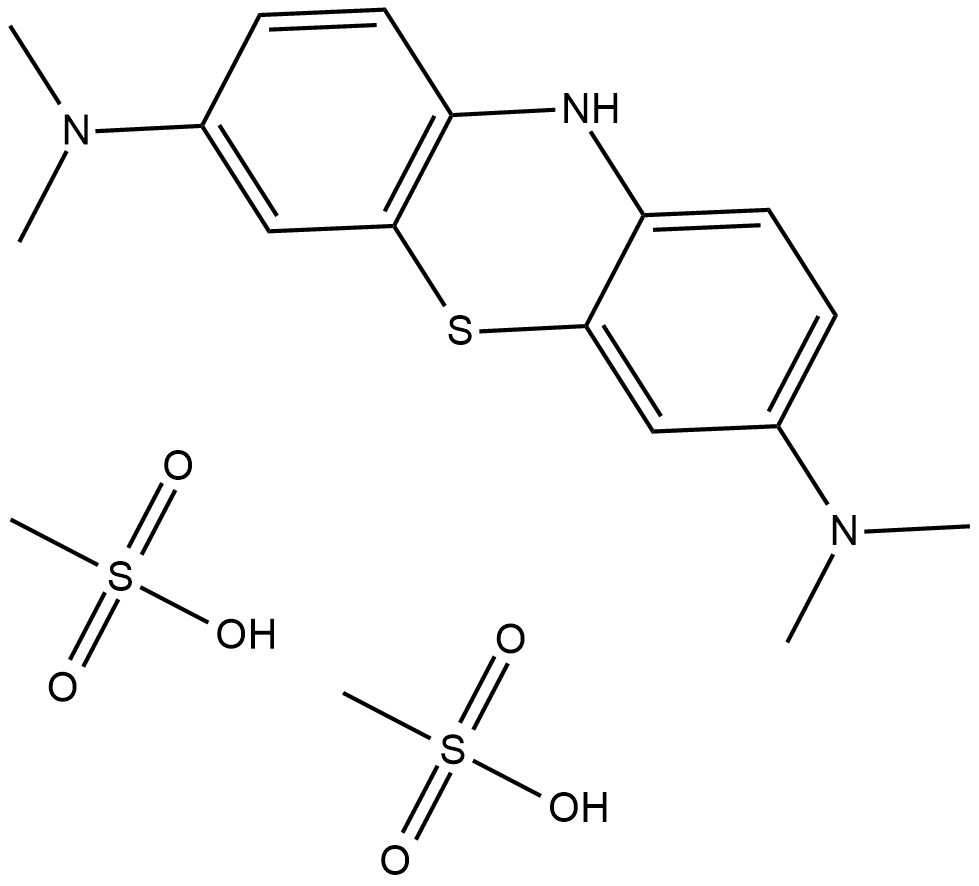
Leucomethylene blue Mesylate
Leucomethylenblau (TRx0237)-Mesylat, ein oral aktiver Tau-Protein-Aggregationsinhibitor der zweiten Generation (Ki von 0,12 μM), kÖnnte fÜr die Untersuchung der Alzheimer-Krankheit verwendet werden.
-
GC63973
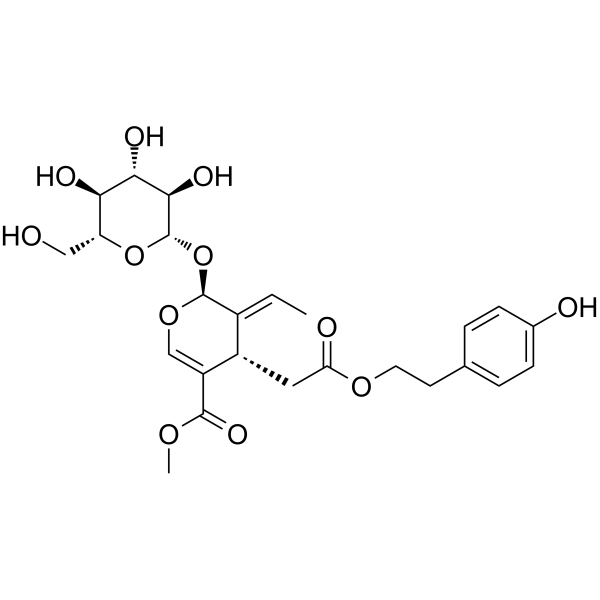
Ligustroside
Ligustrosid (Ligstrosid), ein Secoiridoid-Derivat, hat eine herausragende Leistung bei der mitochondrialen Bioenergetik in Modellen der frÜhen Alzheimer-Krankheit (AD) und der Gehirnalterung durch Mechanismen, die die Aβ-Produktion mÖglicherweise nicht beeintrÄchtigen.
-
GN10423
Lipoic acid

-
GC11532
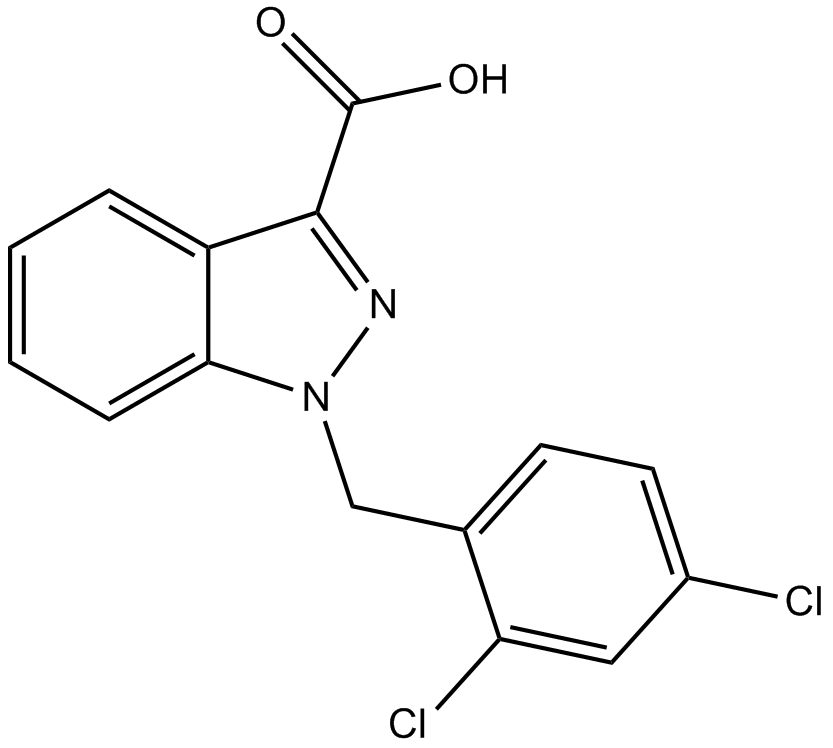
Lonidamine
Lonidamin (AF-1890) ist ein Hexokinase- und mitochondrialer PyruvattrÄger-Inhibitor (Ki: 2,5 μM).
-
GC48989
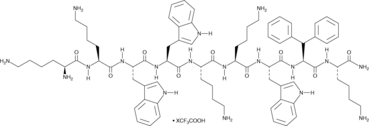
LTX-315 (trifluoroacetate salt)
A synthetic cationic amphiphilic peptide
-
GC44093
Lutein
Lutein is a natural yellow carotenoid, which can be found in plants, egg yolks, and in the human retina.
-
GC40007
Malformin A
Malformin A is a cyclopentapeptide fungal metabolite that has been found in A.
-
GC61790
MCU-i4
MCU-i4 blockiert die IP3-abhÄngige mitochondriale Ca2+--Aufnahme und behÄlt die Gatekeeping-Rolle ihres Ziels bei.
-
GC66184
Menadiol
Menadiol (Dihydrovitamin K3), ein Menaquinol-Analogon, ist ein Elektronendonor fÜr die umgekehrte oxidative Phosphorylierung in subchondrialen Partikeln.
-
GC47620
Mensacarcin
Mensacarcin, ein hochkomplexes Polyketid, hemmt universell das Zellwachstum in Krebszelllinien stark und induziert wirksam Apoptose in Melanomzellen. Mensacarcin zielt auf Mitochondrien ab, beeinflusst den Energiestoffwechsel in Mitochondrien und aktiviert Caspase-abhÄngige apoptotische Signalwege. Mensacarcin, ein Antibiotikum, kann als zytotoxische Komponente von AntikÖrper-Wirkstoff-Konjugaten (ADCs) verwendet werden.
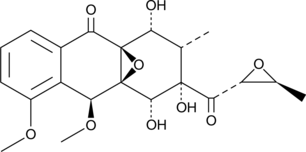
-
GC47663
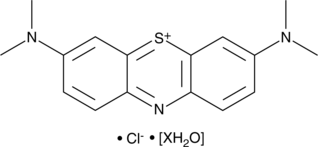
Methylene Blue (hydrate)
Methylenblau (Basic Blue 9) Hydrat ist ein Inhibitor der Guanylylcyclase (sGC), der Monoaminoxidase A (MAO-A) und der NO-Synthase (NOS).
-
GC49472
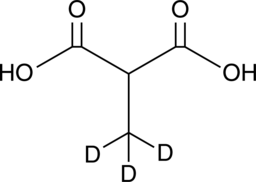
Methylmalonic Acid-d3
MethylmalonsÄure-d3 (MethylpropandisÄure-d3) ist die mit Deuterium bezeichnete MethylmalonsÄure. MethylmalonsÄure (Methylmalonat) ist ein Indikator fÜr Vitamin B-12-Mangel bei Krebs.
-
GC65989
MFI8
MFI8 ist ein niedermolekularer Inhibitor der mitochondrialen Fusion.
-
GC47685
MIT-PZR
A mitochondria-targeted fluorescent probe
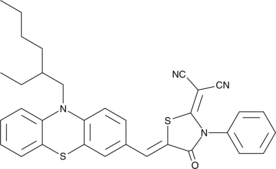
-
GC62564
Mito-LND
Mito-LND (Mito-Lonidamine) ist ein oral aktiver und auf die Mitochondrien gerichteter Inhibitor der oxidativen Phosphorylierung (OXPHOS). Mito-LND hemmt die mitochondriale Bioenergetik, stimuliert die Bildung reaktiver Sauerstoffspezies und induziert den autophagischen Zelltod in Lungenkrebszellen.
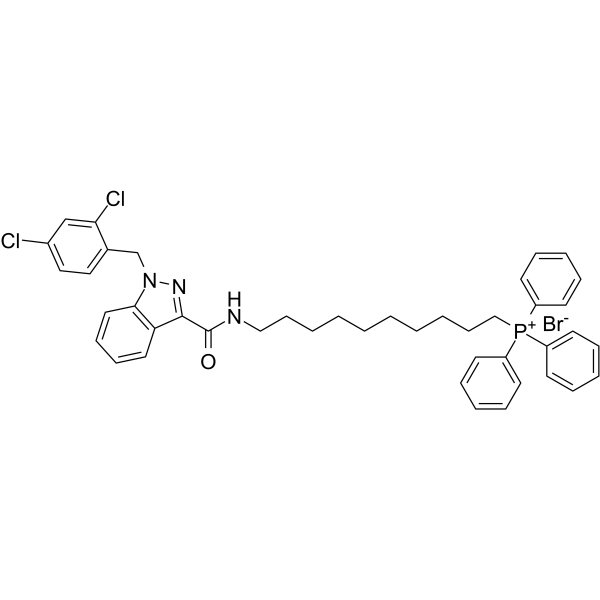
-
GC31682
Mito-TEMPO
Mito-TEMPO ist ein auf Mitochondrien gerichtetes Superoxid-Dismutase-Mimetikum mit Superoxid- und AlkylradikalfÄngereigenschaften.
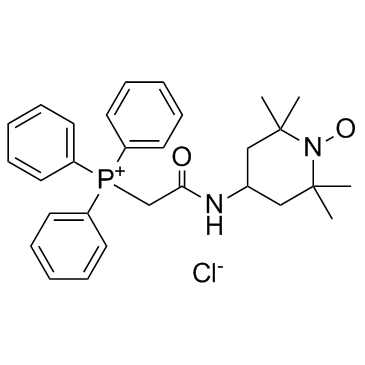
-
GC44198
MitoA
MitoA is a ratiometric mass spectrometry probe that can be used for assessing changes in H2S within mitochondria in vivo.
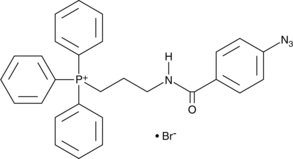
-
GC44199
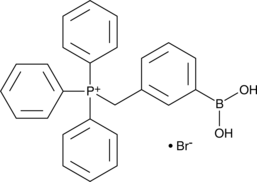
MitoB
MitoB is a ratiometric mass spectrometry probe that can be used for assessing changes in H2O2 within mitochondria in vivo.
-
GC61634
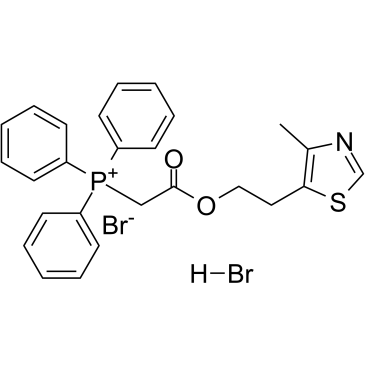
Mitochondrial respiration-IN-1 hydrobromide
Mitochondriale Atmung-IN-1-Hydrobromid (Verbindung 49) ist ein starker mitochondrialer Inhibitor (IC50 = 8,8 mg/ml), extrahiert aus dem Patent US20110301180A1, Verbindung 49.
-
GC31123
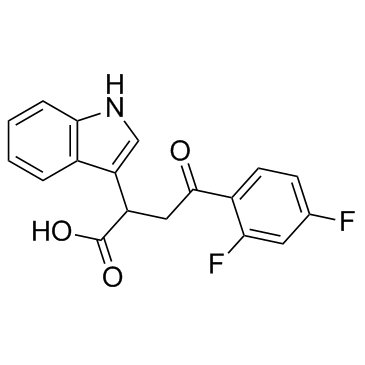
Mitochonic acid 5 (MA-5)
MitochonsÄure 5 (MA-5) bindet Mitochondrien und lindert renale tubulÄre und kardiale MyozytenschÄden.
-
GC44201
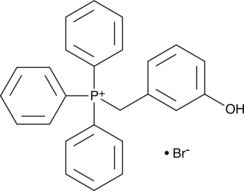
MitoP
MitoP is a phenol product produced by the reaction of H2O2 with the ratiometric mass spectrometry probe MitoB.
-
GC44202
MitoPBN
MitoPBN is a mitochondria-targeted antioxidant.
-
GC44203
MitoPerOx
MitoPerOx ist eine fluoreszierende Ratio-Sonde der Lipidperoxidation.
-
GC44204
MitoPQ
MitoPQ ist ein auf Mitochondrien ausgerichteter Redox-Cycler. MitoPQ produziert Superoxid durch Redoxzyklen an der Flavinstelle von Komplex I, wodurch die Superoxidproduktion in den Mitochondrien selektiv erhÖht wird. MitoPQ kann in Antioxidantienstudien verwendet werden.
-
GC44205
Mitoquinol
Mitoquinol ist eine Ubichinon-Derivat, das aufgrund der kovalenten Bindung des Kations Triphenylphosphonium spezifisch in Mitochondrien akkumuliert.
-
GC49222
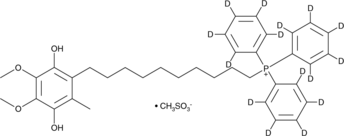
Mitoquinol-d15
An internal standard for the quantification of mitoquinol
-
GC36619
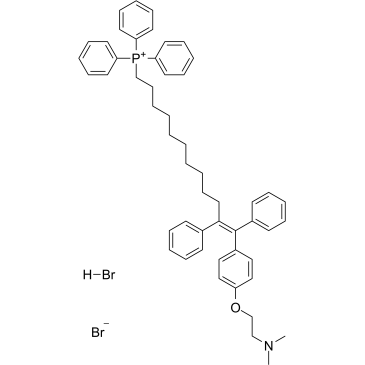
MitoTam bromide, hydrobromide
MitoTam-Bromid, Hydrobromid, ein Tamoxifen-Derivat, ist ein Inhibitor der Elektronentransportkette (ETC). MitoTam-Bromid, Hydrobromid reduziert das mitochondriale Membranpotential in seneszenten Zellen und beeinflusst die mitochondriale Morphologie. MitoTam-Bromid, Hydrobromid ist ein wirksames Antikrebsmittel, unterdrÜckt respiratorische Komplexe (CI-Atmung) und unterbricht die Bildung respiratorischer Superkomplexe (SCs) in Brustkrebszellen.
-
GC36620
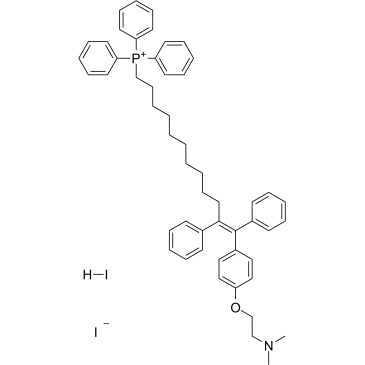
MitoTam iodide, hydriodide
MitoTami-Jodid, Hydriodid ist ein Tamoxifen-Derivat, ein Inhibitor der Elektronentransportkette (ETC), erhÖht das mitochondriale Membranpotential in seneszenten Zellen und beeinflusst die mitochondriale Morphologie Bildung von respiratorischen Superkomplexen (SCs) in Brustkrebszellen. MitoTami iodid, Hydriodid verursacht Apoptose.
-
GC44206
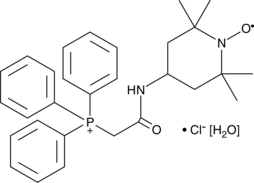
MitoTEMPO (hydrate)
MitoTEMPO is a mitochondria-targeted superoxide dismutase mimetic that possesses superoxide and alkyl radical scavenging properties.
-
GC44207
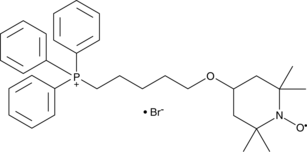
MitoTEMPOL
MitoTEMPOL ist ein mitochondrienzielgerichteter Superoxiddismutase-Mimetikum, das mitochondriales O2- zu H2O2 reduziert.
-
GC18188
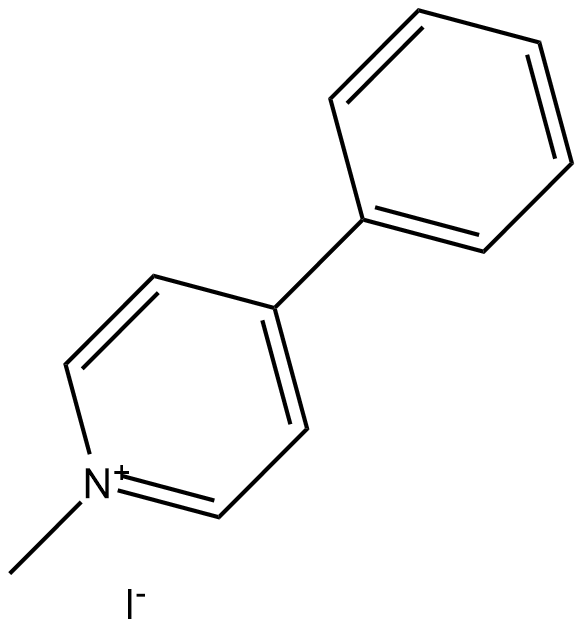
MPP+ Iodide
MPP+ Iodid (1-Methyl-4-phenylpyridiniumiodid) ist ein toxisches Metabolit des Neurotoxins MPTP und hat erfolgreich Parkinson-ähnliche Syndrome in einem In-vitro-Modell induziert, indem es selektiv dopaminerge Neuronen in der Substantia nigra zerstört hat.
-
GC66401
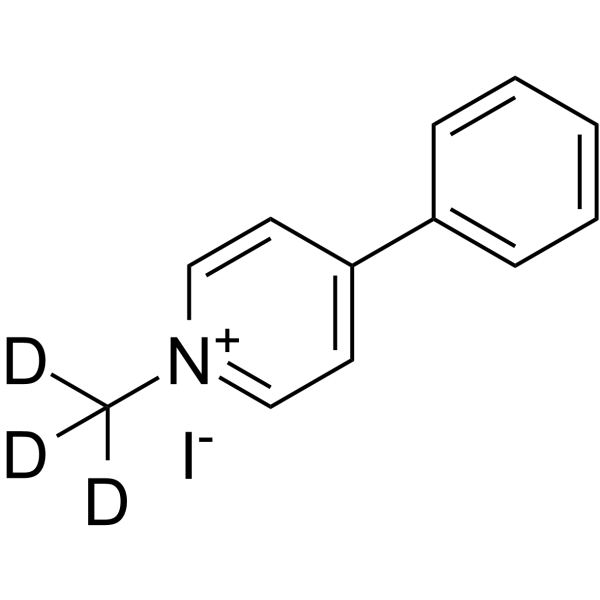
MPP+-d3(iodide)
MPP+-d3(Iodid) ist mit Deuterium markiertes MPP+ (Iodid). MPP+ Jodid, ein toxischer Metabolit des Neurotoxins MPTP, verursacht in Tiermodellen Symptome der Parkinson-Krankheit, indem es selektiv dopaminerge Neuronen in der Substantia nigra zerstÖrt. MPP+-Iodid wird vom Dopamintransporter in dopaminerge Neuronen aufgenommen, wo es seine neurotoxische Wirkung auf die Mitochondrien ausÜbt, indem es den Komplex I der Atmungskette beeinflusst. MPP+-Iodid ist auch ein hochaffines Substrat fÜr den Serotonin-Transporter (SERT).
-
GC36658
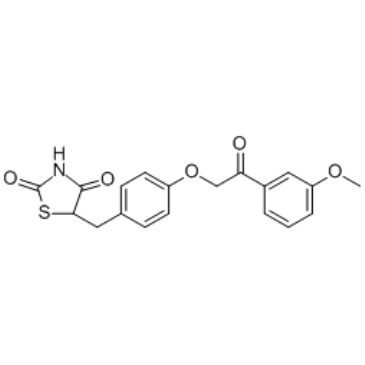
MSDC-0602
MSDC-0602 (MSDC-0602), ein PPARγ-sparendes Thiazolidindion (TZD), interagiert mit dem mitochondrialen PyruvattrÄger (MPC) und hemmt dessen AktivitÄt und hat das Potenzial fÜr Typ-2-Diabetes-Studien mit verringertem Risiko einer PPAR&7#947;-vermittelten Seite Auswirkungen.
-
GC48399
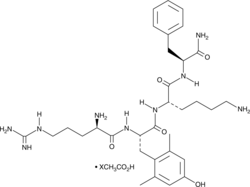
MTP 131 (acetate)
A mitochondria-targeted peptide antioxidant
-
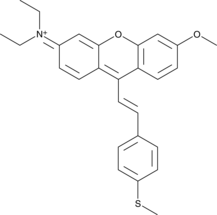
GC48526
MXS
A fluorescent probe for hypochlorous acid
-
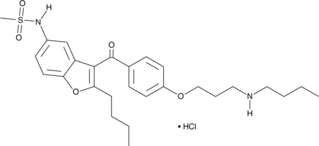
GC49399
N-Desbutyl Dronedarone (hydrochloride)
Debutyldronedaron (SR35021)-Hydrochlorid, der Hauptmetabolit von Dronedaron, ist ein selektiver Inhibitor des SchilddrÜsenhormonrezeptors α1 (TRα1).
-
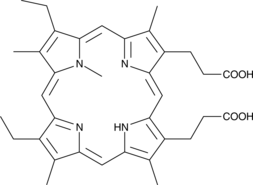
GC44416
N-methyl Mesoporphyrin IX
Ein Ferrochelatase-Inhibitor und Biosensor zur Aktivierung von DNA.
-
GC47789
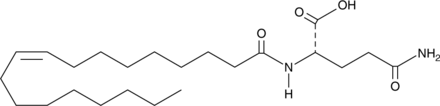
N-Oleoyl Glutamine
An endogenous N-acyl amine
-
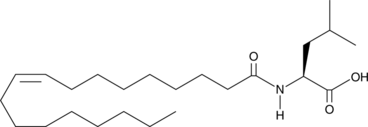
GC44441
N-Oleoyl Leucine
N-Oleoyl leucine is an N-acyl amide generated by PM20D1 that uncouples mitochondrial respiration independent of uncoupling protein 1 (UCP1) in vitro.
-
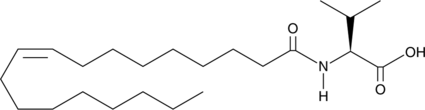
GC44444
N-Oleoyl Valine
N-Oleoyl valine is an endogenous N-acyl amine that acts as an antagonist at the transient receptor potential vanilloid type 3 (TRPV3) receptor, which is involved in thermoregulation.
-
GC47739
NADH (sodium salt hydrate)
A reduced form of NAD+
-
GC44308
NADP+ (sodium salt hydrate)
NADP+ (Natriumsalzhydrat), ein β-Nicotinamidadenindinukleotidphosphat-Natriumsalz, ist ein Redox-Cofaktor.
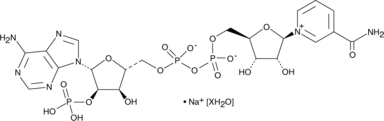
-
GC47747
Napyradiomycin A1
Napyradiomycin A1 ist eine enantioselektive Verbindung von Napyradiomycinen.
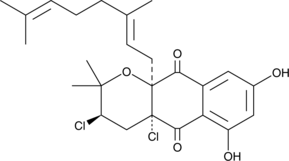
-
GC40669
Nerol
Nerol ist ein Bestandteil des NeroliÖls.
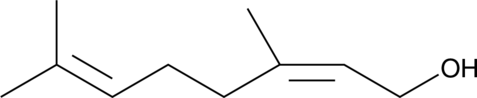
-
GC49511
Nicorandil N-oxide
A metabolite of nicorandil
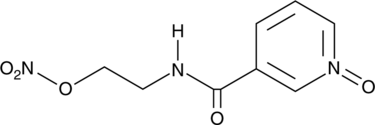
-
GC52214
Nicotinamide riboside-d4 (triflate)
An internal standard for the quantification of nicotinamide riboside
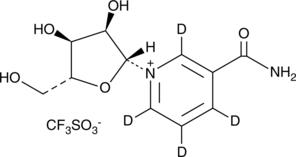
-
GC31646
NIM811 ((Melle-4)cyclosporin)
NIM811 ((Melle-4)Cyclosporin) ((Melle-4)Cyclosporin; SDZ NIM811 ((Melle-4)Cyclosporin)) ist ein oral bioverfÜgbarer mitochondrialer PermeabilitÄtsÜbergangs- und Cyclophilin-Dual-Inhibitor, der eine starke In-vitro-AktivitÄt gegen das Hepatitis-C-Virus aufweist (HCV) .
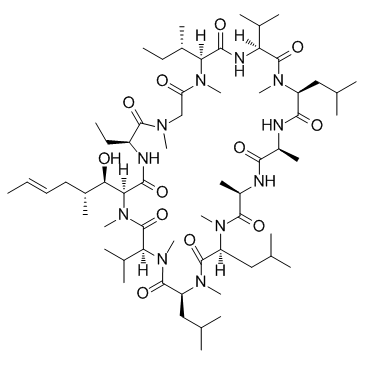
-
GC39411
NL-1
NL-1 ist ein MitoNEET-Hemmer mit antileukÄmischer Wirkung. NL-1 hemmt das Wachstum von REH- und REH/Ara-C-Zellen mit IC50-Werten von 47,35 μM bzw. 56,26 μM. Der NL-1-vermittelte Tod in LeukÄmiezellen erfordert die Aktivierung des autophagischen Signalwegs.
-
GC12090
Nonactin
Nonactin ist ein natÜrlich vorkommendes Makrotetrolid-Antibiotikum aus Streptomyces griseus.
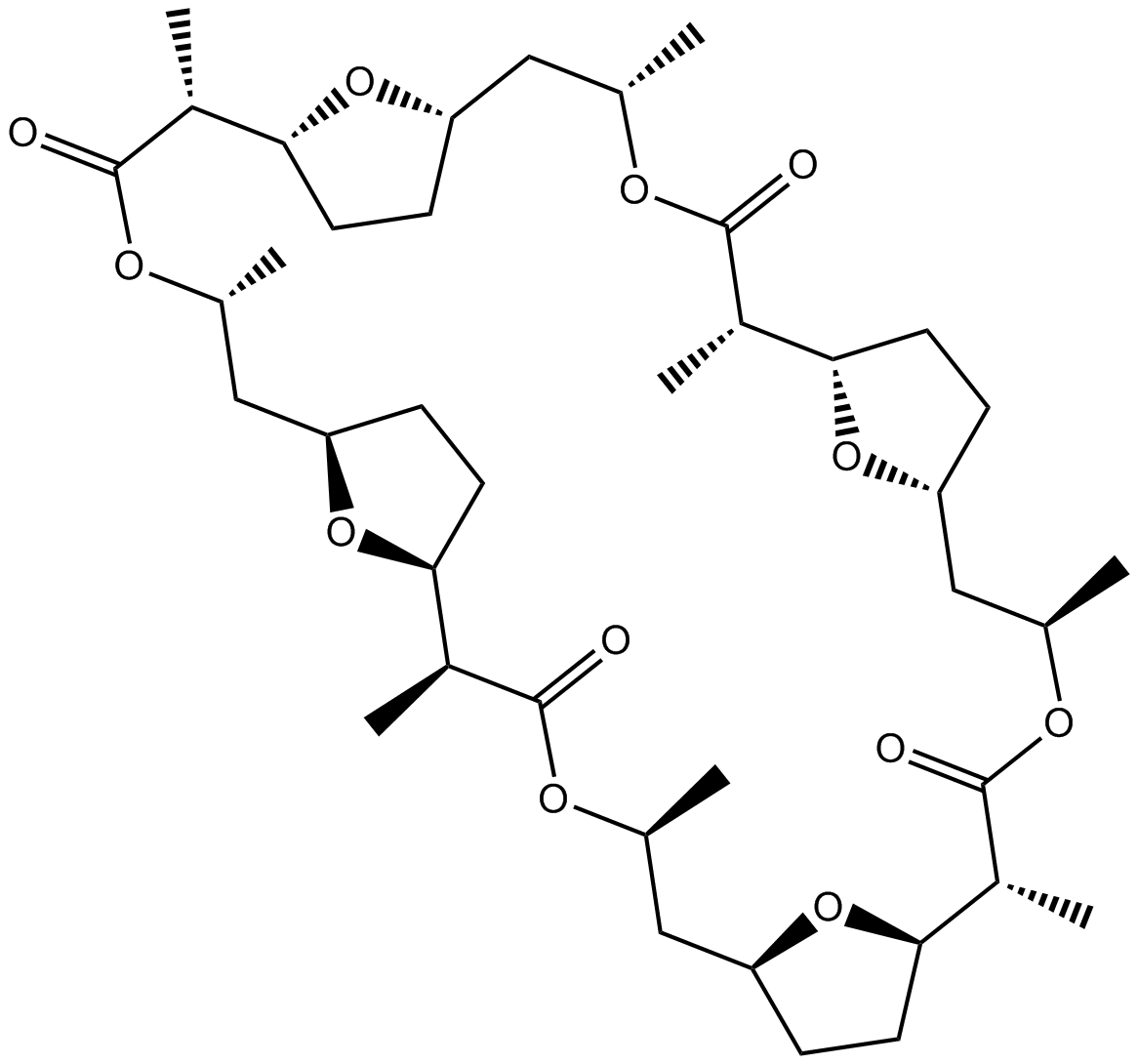
-
GC52223
Nonadecanoyl-L-carnitine (chloride)
A long-chain acylcarnitine
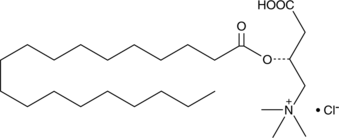
-
GC52236
Nonadecanoyl-L-carnitine-d3 (chloride)
An internal standard for the quantification of nonadecanoyl-L-carnitine
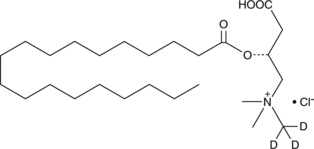
-
GC44464
NSC 109555
An inhibitor of Chk2
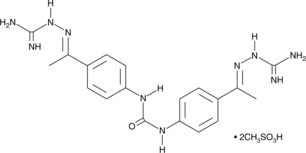
-
GC44488
Octanoyl Coenzyme A (sodium salt)
Octanoyl coenzyme A (octanoyl-CoA) is a medium-chain acyl CoA and a metabolic intermediate in mitochondrial fatty acid β-oxidation.

-
GC49457
Oleoyl-Coenzyme A (sodium salt)
An acyl-CoA thioester
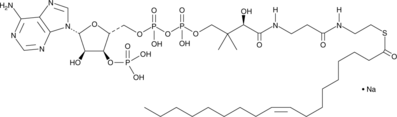
-
GC44506
Oligomycin D
Oligomycin D is a macrolide antibiotic produced by several species of Streptomyces that inhibits the mitochondrial F1FO-ATPase and is used to uncouple oxidative phosphorylation from electron transport.
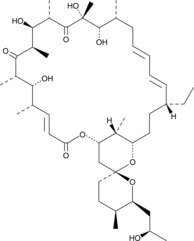
-
GC47868
Palmitoyl-L-carnitine-d3 (chloride)
An internal standard for the quantification of palmitoyl-L-carnitine
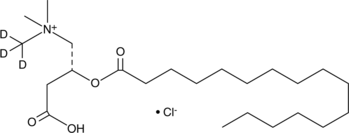
-
GC52306
Pentadecanoyl-L-carnitine (chloride)
A CB receptor agonist
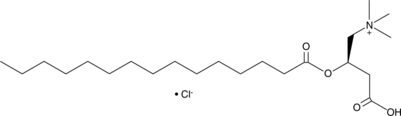
-
GC40252
Phosphatidic Acids (egg) (ammonium salt)
Phosphatidic acid is a phospholipid and an intermediate in glycerolipid biosynthesis.
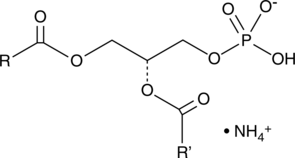
-
GC18522
Phosphatidylserines (sodium salt)
Phosphatidylserine is a naturally occurring phospholipid that comprises 2-10% of total phospholipids in mammals and is enriched in the central nervous system, particularly the retina.
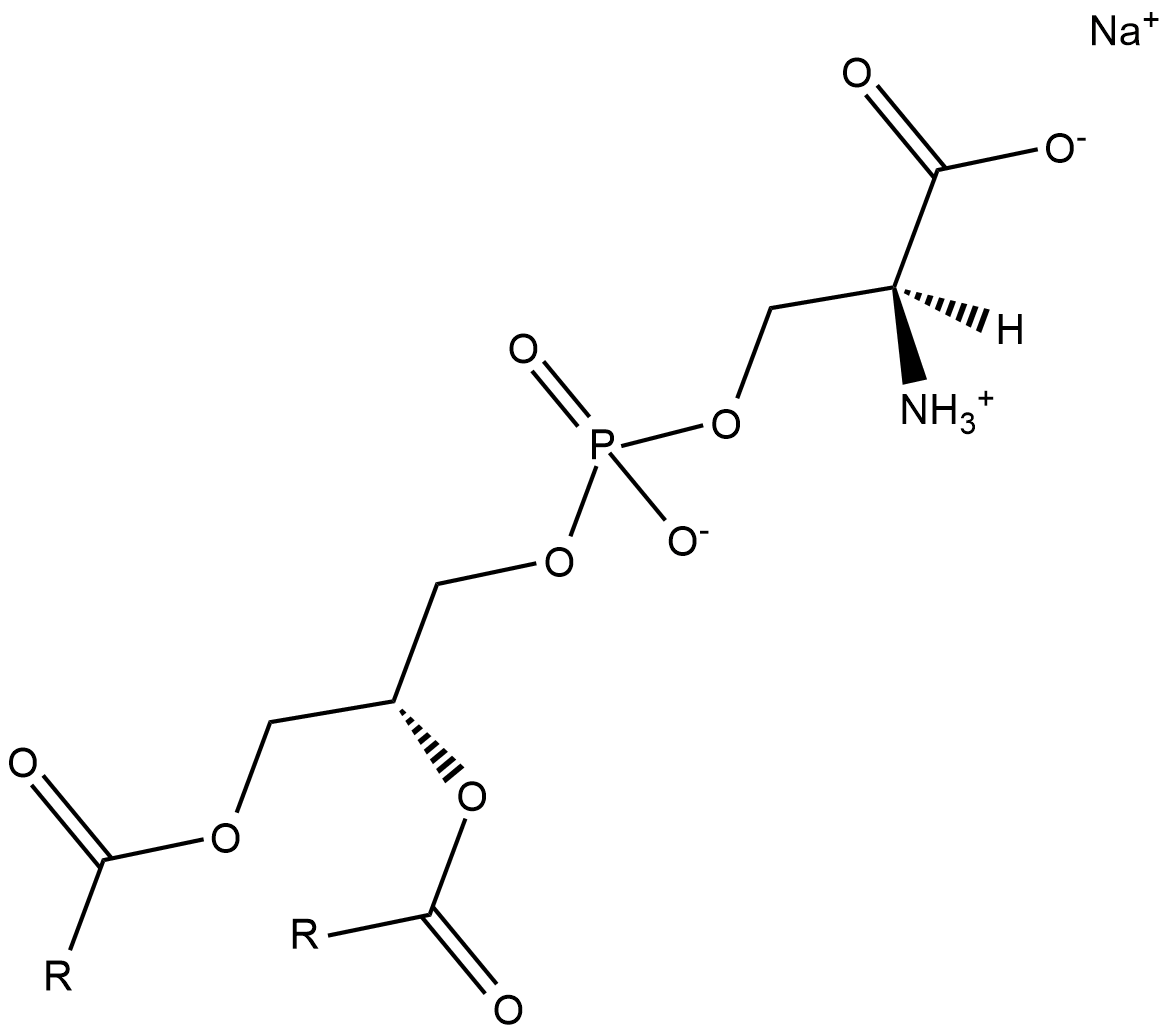
-
GC48528
Piericidin B
A bacterial metabolite with insecticidal and antimicrobial activities
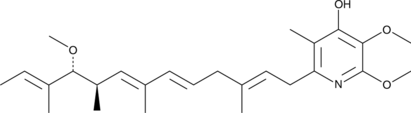
-
GC49192
Piracetam-d6
An internal standard for the quantification of piracetam
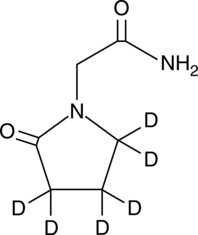
-
GC47980
Propionyl-L-carnitine-d3 (chloride)
An internal standard for the quantification of propionyl-L-carnitine
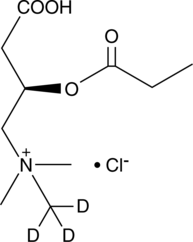
-
GC48030
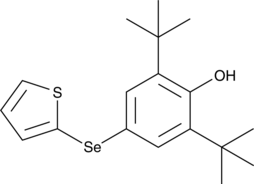
RC574
An inhibitor of ferroptosis
-
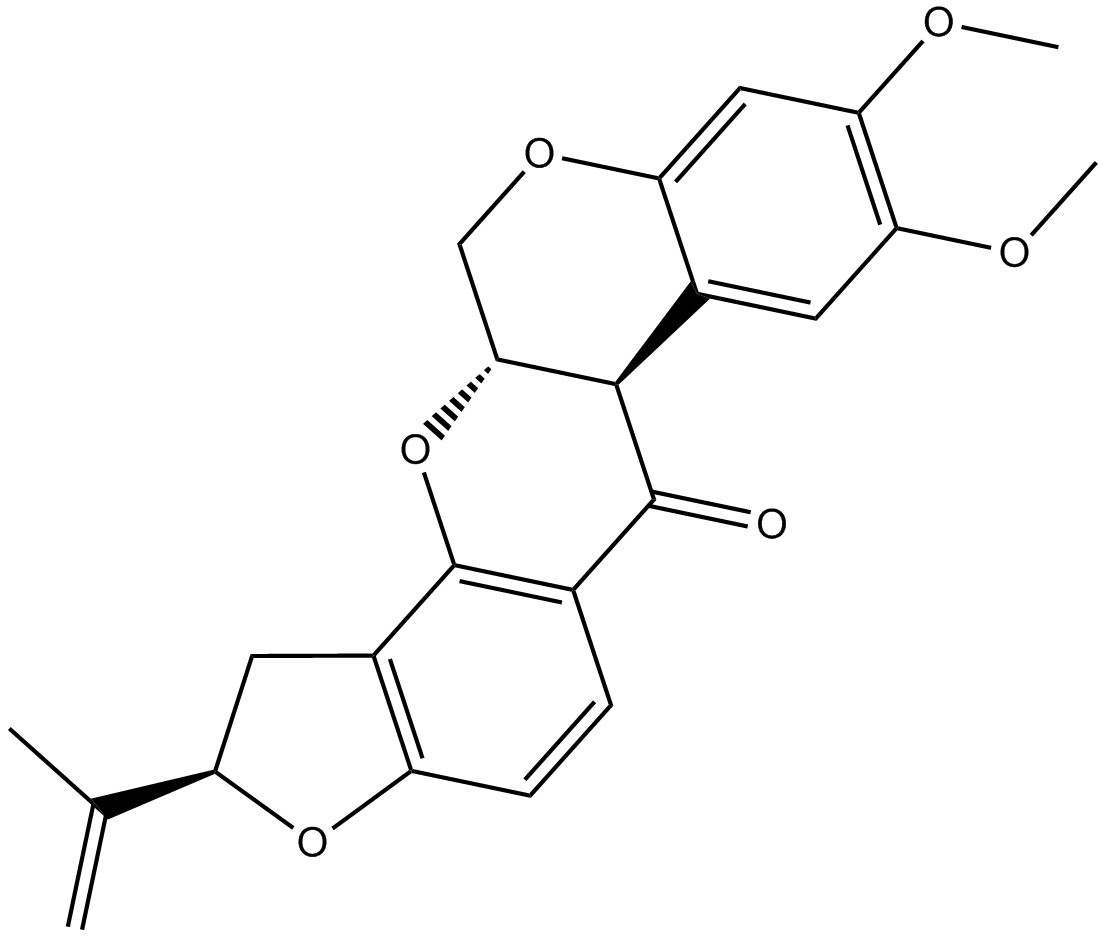
GC16775
Rotenone
Inhibitor der Elektronentransportkette des mitochondrialen Komplexes I
-
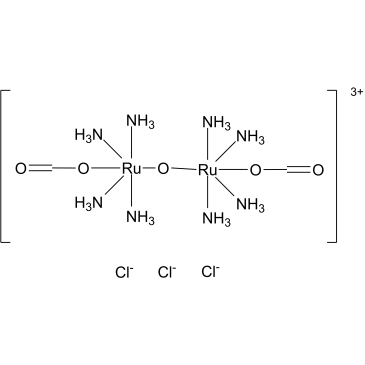
GC60330
Ru360
-
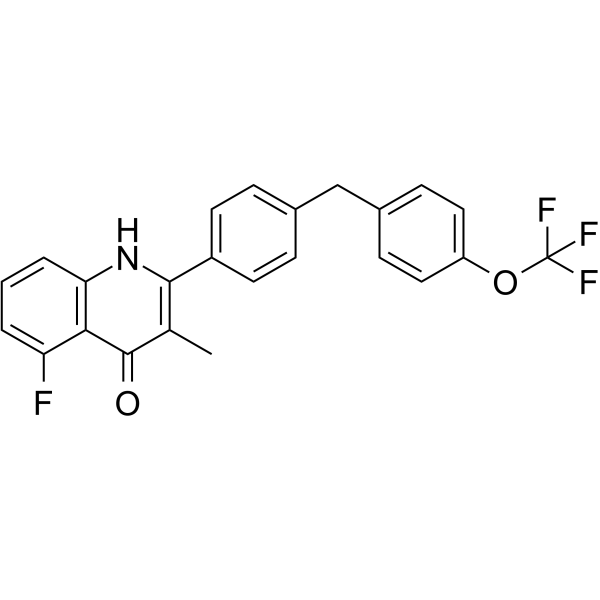
GC64669
RYL-552
RYL-552, ein Inhibitor der mitochondrialen Elektronentransportkette (ETC), ist ein P.
-
GC44860
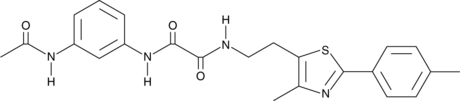
S1QEL1.1
S1QEL1.1 is a small molecule that suppresses superoxide production during reverse electron transport through the IQ site of the mitochondrial respiratory complex I (IC50 = 70 nM) without affecting oxidative phosphorylation.
-
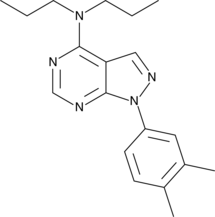
GC44862
S3QEL-2
S3QEL-2, ein Suppressor der Superoxidproduktion aus dem mitochondrialen Komplex III, unterdrÜckt wirksam und selektiv die Superoxidproduktion an der Stelle IIIQo (IC50 = 1,7 μM).
-
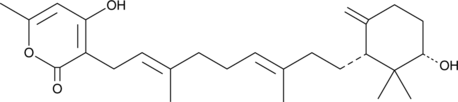
GC48547
Sartorypyrone D
A fungal metabolite
-
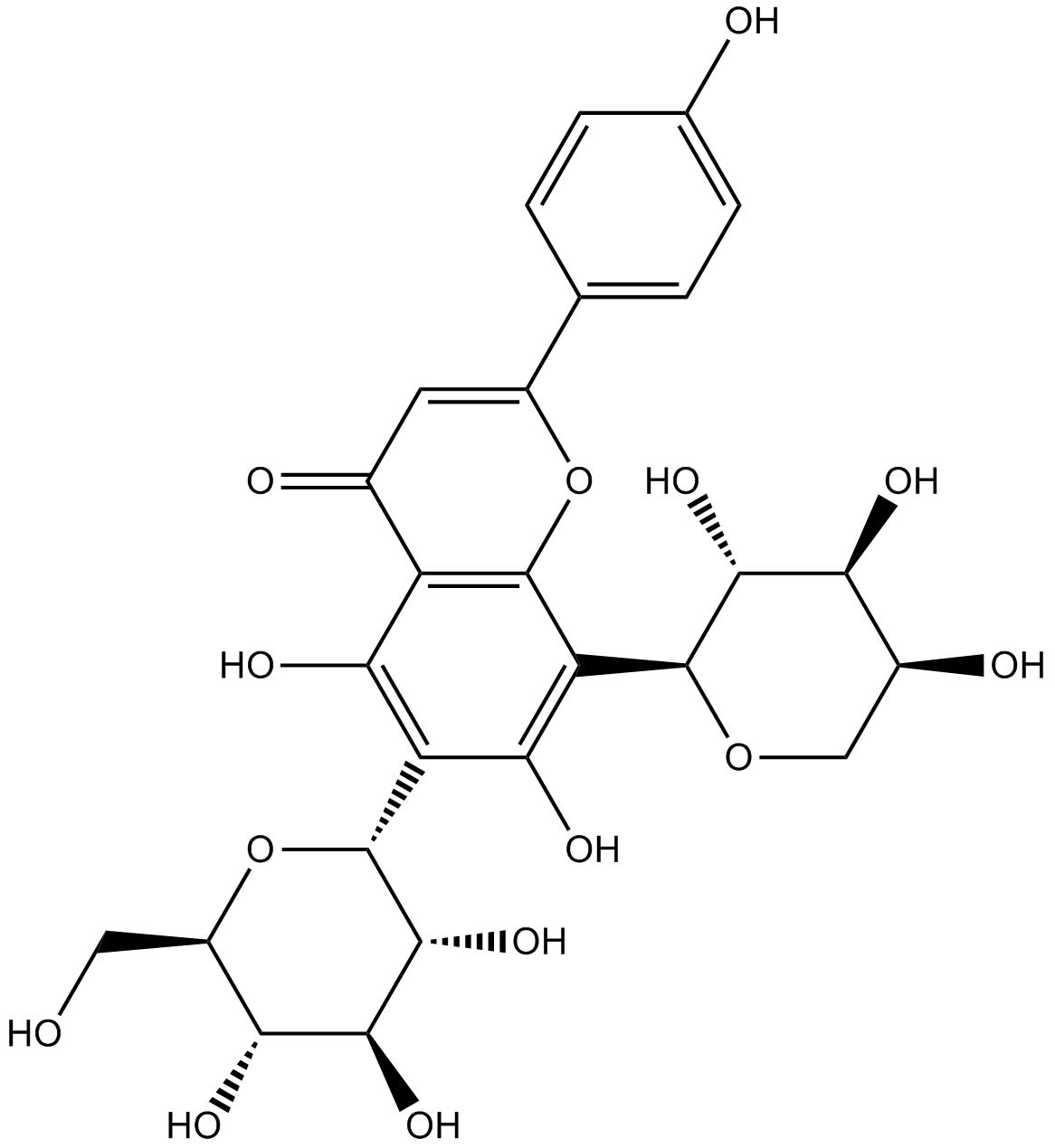
GN10091
Schaftoside
-
GC30459
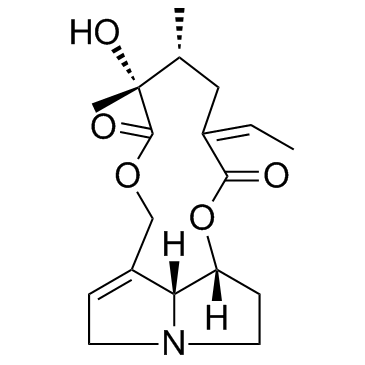
Senecionine (Senecionan-11,16-dione, 12-hydroxy-)
Senecionin (Senecionan-11,16-dion, 12-hydroxy-) (Senecionan-11,16-dion, 12-hydroxy-) ist ein aus Senecio vulgaris isoliertes Pyrrolizidinalkaloid.
-
GC61274
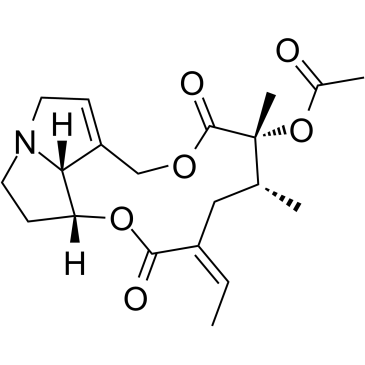
Senecionine acetate
Senecioninacetat (O-Acetylsenecionin) ist ein Pyrrolizidinalkaloid.
-
GC48077
SHS4121705
A mitochondrial uncoupler
-
GC48894
SIH
SIH ist eine antituberkulÖse Verbindung.
-
GC31491
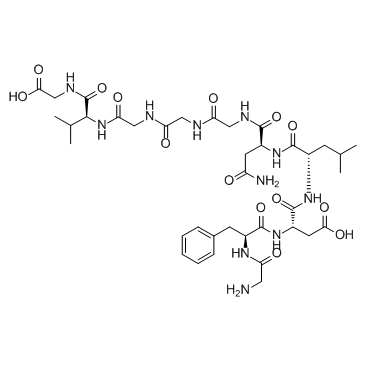
Speract
Speract, ein Seeigel-Ei-Peptid, das die Beweglichkeit der Spermien reguliert, stimuliert auch den mitochondrialen Stoffwechsel der Spermien.
-
GC63669
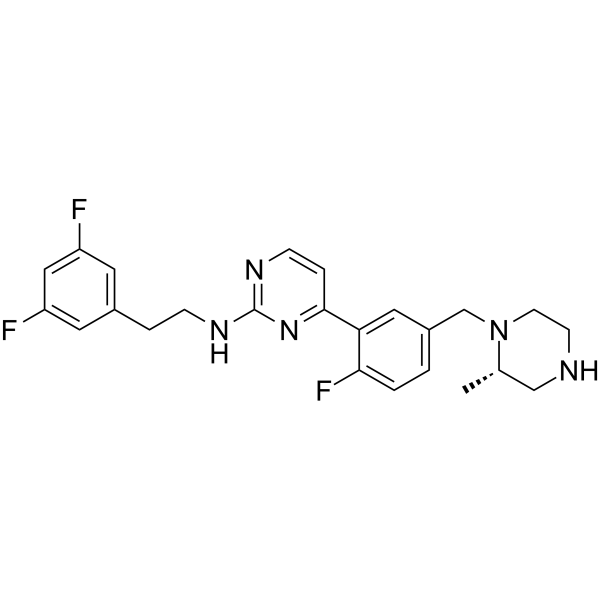
T0467
T0467 aktiviert die mitochondriale Translokation von Parkin PINK1-abhÄngig in vitro.
-
GC39512
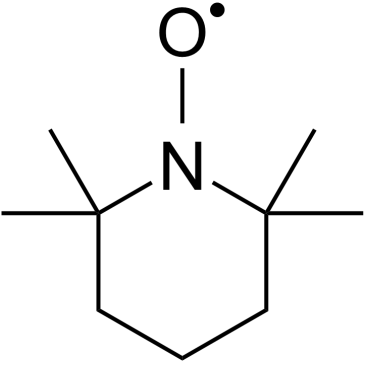
Tempo
Tempo ist ein klassisches Nitroxid-Radikal und ein selektiver ROS-FÄnger, der Superoxid im Katalysezyklus dismutiert. Tempo induziert einen DNA-Strangbruch. Tempo kann als Organokatalysator fÜr die Oxidation von primÄren Alkoholen zu Aldehyden eingesetzt werden. Tempo hat mutagene und antioxidative Wirkungen.
-
GC45020
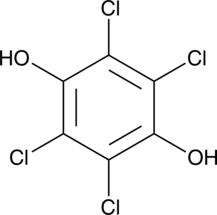
Tetrachlorohydroquinone
Tetrachlorohydroquinone (TCHQ) is a metabolite of the organochlorine biocide pentachlorophenol.
-
GC48924
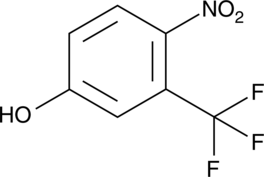
TFM
A piscicide
-
GC17228
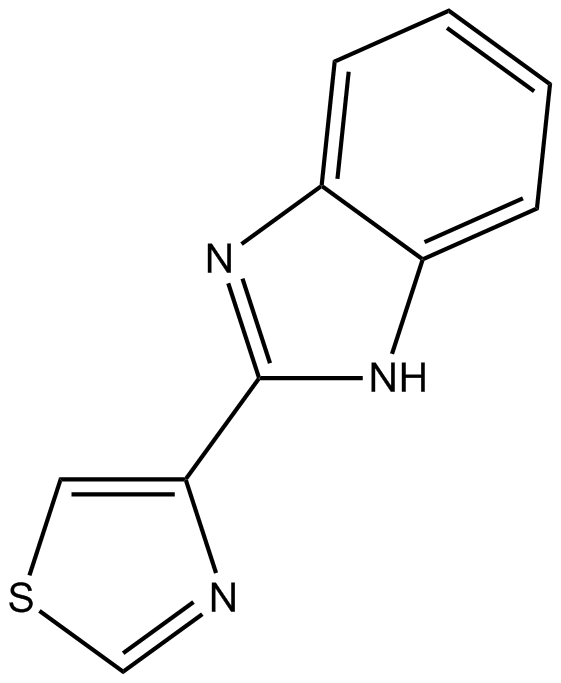
Thiabendazole
Thiabendazol hemmt das mitochondriale Helminthen-spezifische Enzym Fumaratreduktase mit anthelminthischen Eigenschaften.
-
GC64688
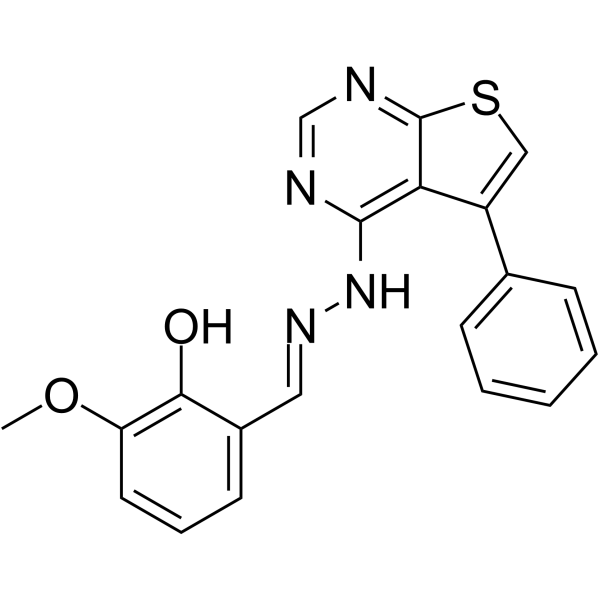
THP104c
THP104c ist ein Inhibitor der mitochondrialen Spaltung.
-
GC49529
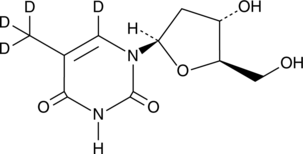
Thymidine-d4
An internal standard for the quantification of thymidine
-
GC48192
trans-2-Octenoyl-L-carnitine
A synthetic carnitine
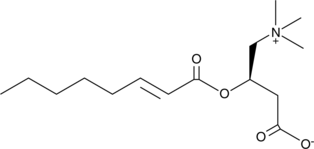
-
GC63238
Tricarballylic acid
TricarballylsÄure, eine konjugierte SÄure eines Tricarballylats, ist ein kompetitiver Inhibitor des Enzyms Aconitathydratase (Aconitase; EC 4.2.1.3) mit einem Ki-Wert von 0,52 mM.
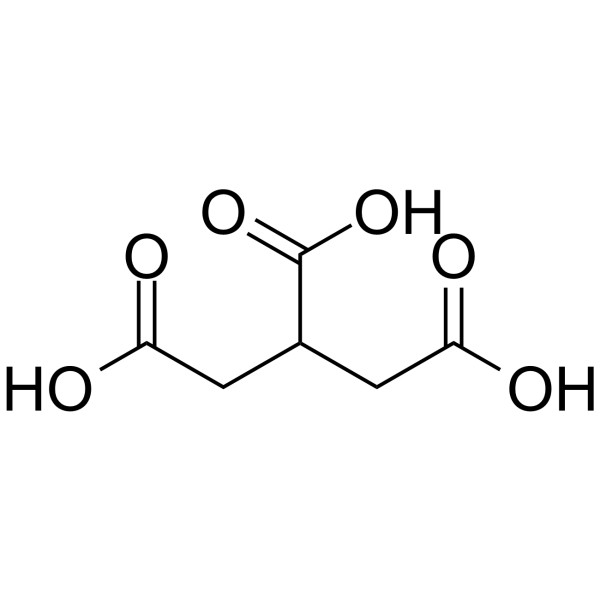
-
GC13726
TRO 19622
TRO 19622 (TRO 19622) ist eine mitochondriale zielgerichtete neuroprotektive Verbindung mit einem mittleren EC50-Wert zur Steigerung des ZellÜberlebens von 3,2±0,2 μM.
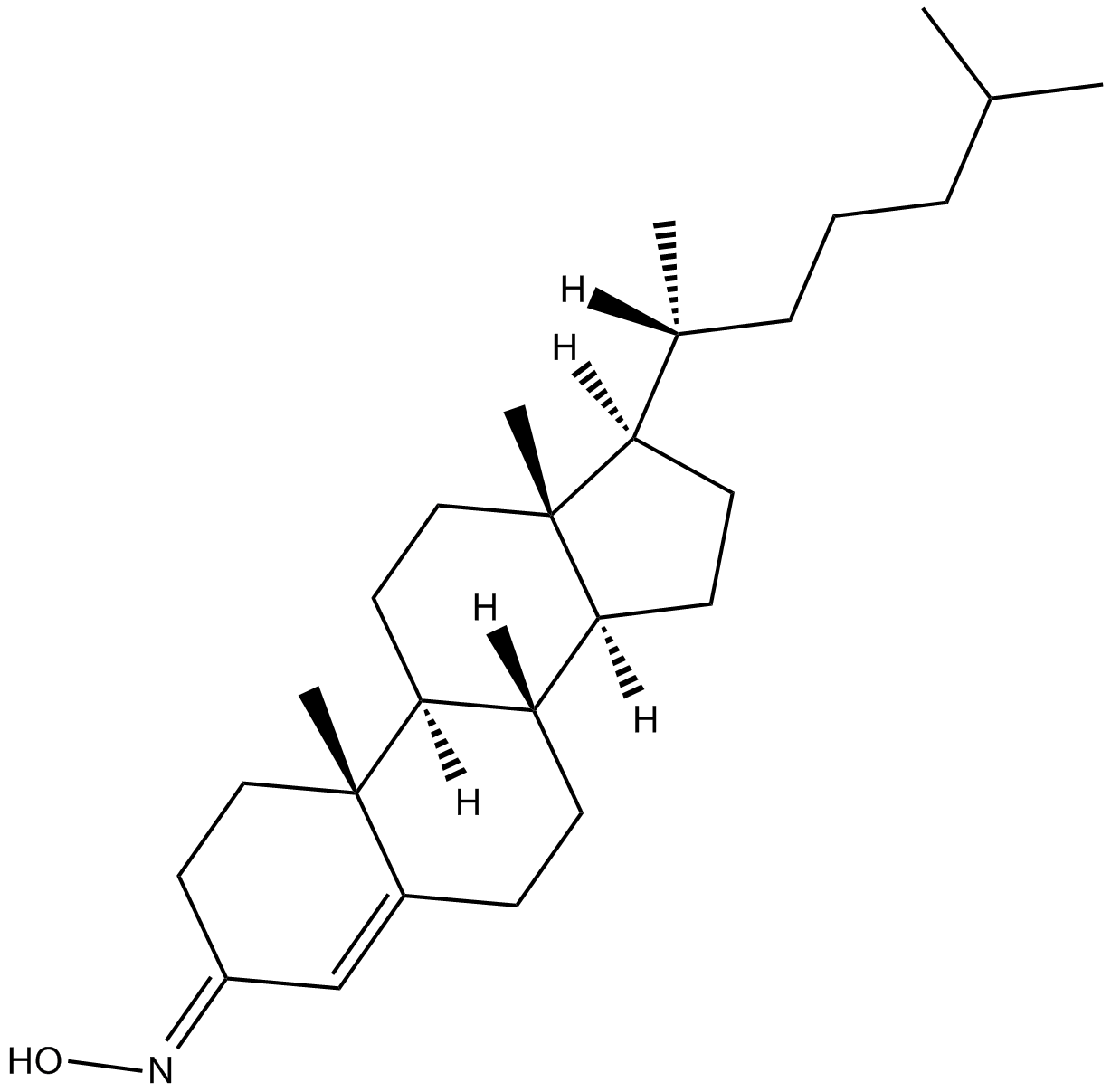
-
GC11865
UK-5099
An inhibitor of the mitochondrial pyruvate carrier
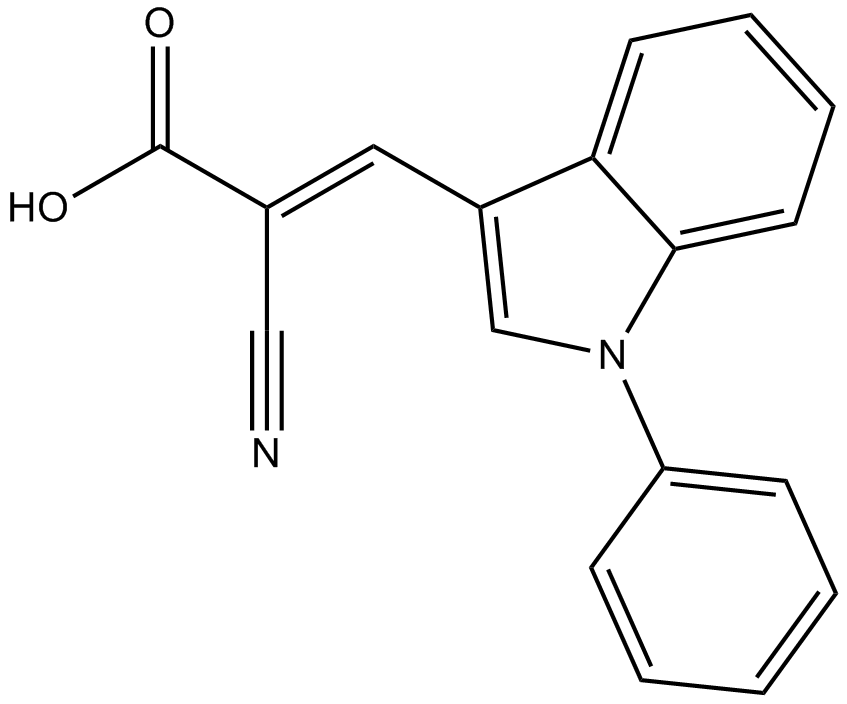
-
GC30512
Vatiquinone (EPI-743)
Vatiquinone (EPI-743) (Vatiquinone; α-Tocotrienolchinon; PTC-743; NCT04378075) ist ein wirksames zellulÄres Schutzmittel gegen oxidativen Stress, hemmt die Ferroptose in Zellen, was fÜr die Untersuchung von mitochondrialen Erkrankungen verwendet werden kÖnnte.
-
GC48398
WST-1
A water-soluble and cell-permeable tetrazolium dye