DNA Damage/DNA Repair
- MTH1(4)
- PARP(61)
- ATM/ATR(27)
- DNA Alkylating(19)
- DNA Ligases(3)
- DNA Methyltransferase(18)
- DNA-PK(27)
- HDAC(99)
- Nucleoside Antimetabolite/Analogue(146)
- Telomerase(14)
- Topoisomerase(134)
- tankyrase(5)
- Antifolate(32)
- CDK(234)
- Checkpoint Kinase (Chk)(29)
- CRISPR/Cas9(9)
- Deubiquitinase(64)
- DNA Alkylator/Crosslinker(65)
- DNA/RNA Synthesis(410)
- Eukaryotic Initiation Factor (eIF)(23)
- IRE1(22)
- LIM Kinase (LIMK)(10)
- TOPK(6)
- Casein Kinase(55)
- DNA Intercalating Agents(7)
- DNA/RNA Oxidative Damage(12)
Produkte für DNA Damage/DNA Repair
- Bestell-Nr. Artikelname Informationen
-
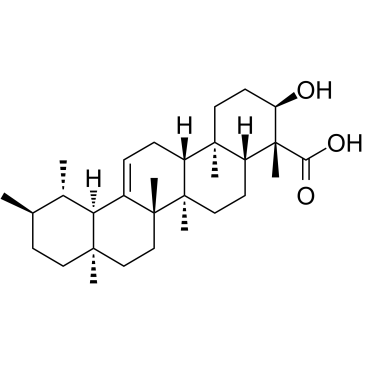
GC38000
β-Boswellic acid
- Boswelliasäure wird aus dem Gummiharz von Boswellia serrate isoliert . β-Boswelliasäure hemmt die Synthese von DNA, RNA und Protein in menschlichen Leukämie-HL-60-Zellen.
-
GC31365
γ-Oryzanol
γ-Oryzanol ist ein starker Inhibitor der DNA-Methyltransferasen (DNMTs) im Striatum von Mäusen.
-
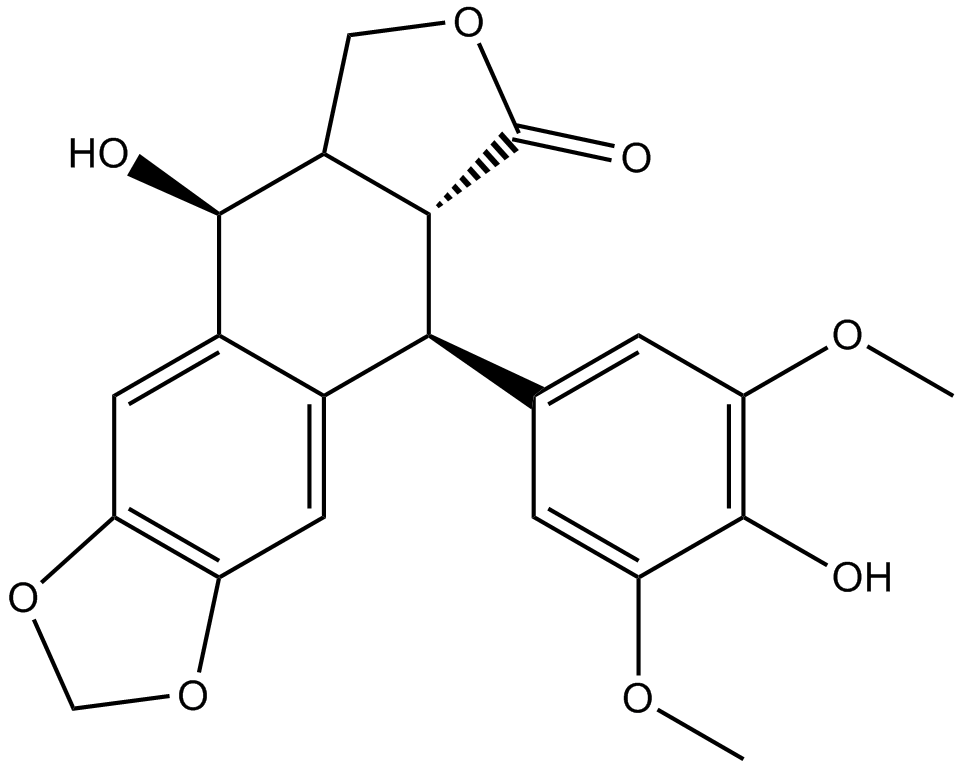
GC38194
(±)-10-Hydroxycamptothecin
(±)-10-Hydroxycamptothecin ist ein Indolalkaloid, das die Aktivität der Topoisomerase I hemmt und ein breites Spektrum an Antikrebsaktivität aufweist.
-
GC33107
(±)-BAY-1251152
(±)-BAY-1251152; (±)-VIP152
(±)-BAY-1251152 ((±)-BAY-1251152) ist eine racemische Mischung von BAY-1251152. BAY-1251152 ist ein potenter und hochselektiver PTEF/CDK9-Inhibitor.
-
GC10867
(+)-Aphidicolin
ICI 69653, NSC 234714
Aphidicolin ((+)-Aphidicolin), ein reversibler Inhibitor der nuklearen DNA-Replikation bei Eukaryoten, kann den Zellzyklus in der prä-S-Phase blockieren[1].
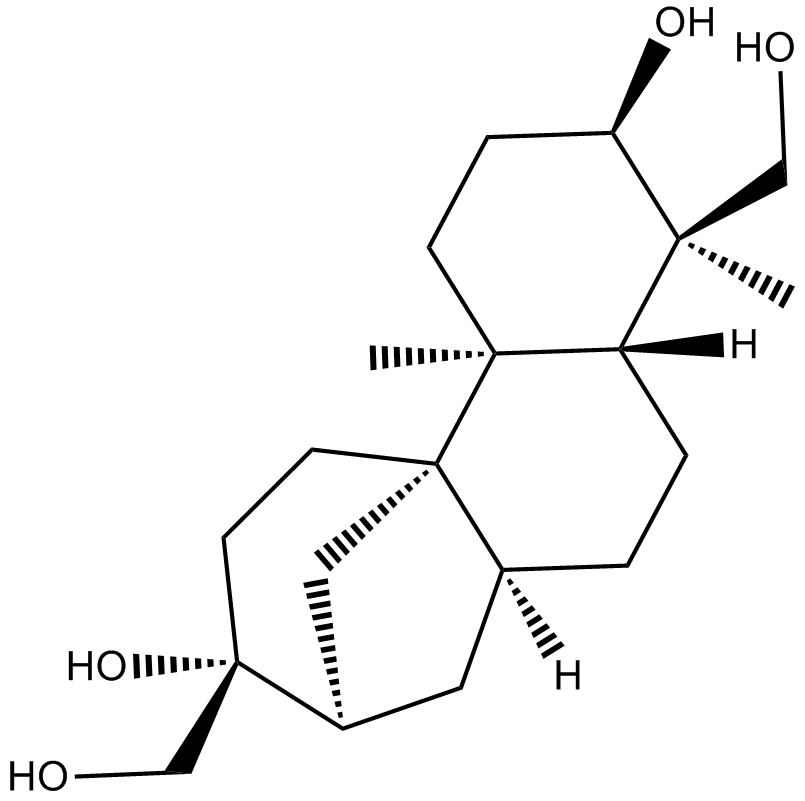
-
GC34955
(+)-CBI-CDPI1
(+)-CBI-CDPI1 ist ein erweitertes funktionelles Analogon von CC-1065. (+)-CBI-CDPI1 ist ein DNA-Alkylierungsmittel. (+)-CBI-CDPI1 ist ein AntikÖrper-Wirkstoff-Konjugat (ADCs)-Toxin.
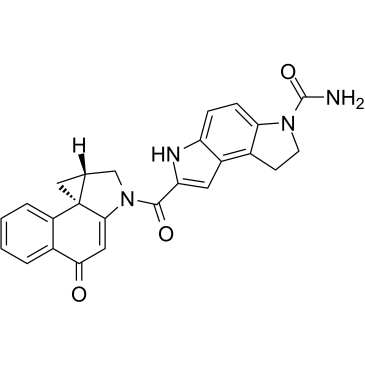
-
GC34956
(+)-CBI-CDPI2
(+)-CBI-CDPI2 ist ein erweitertes funktionelles Analogon von CC-1065. (+)-CBI-CDPI1 ist ein DNA-Alkylierungsmittel. (+)-CBI-CDPI2 ist ein AntikÖrper-Wirkstoff-Konjugat (ADCs)-Toxin.
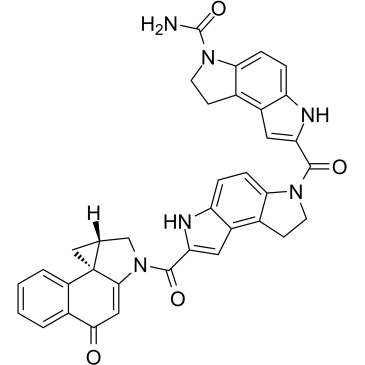
-
GC32429
(-)-BAY-1251152
(-)-BAY-1251152; (-)-VIP152
(-)-BAY-1251152 ((-)-BAY-1251152) ist ein Enanthiomer von BAY-1251152 mit Rotation (-). BAY-1251152 ist ein potenter und hochselektiver PTEF/CDK9-Inhibitor.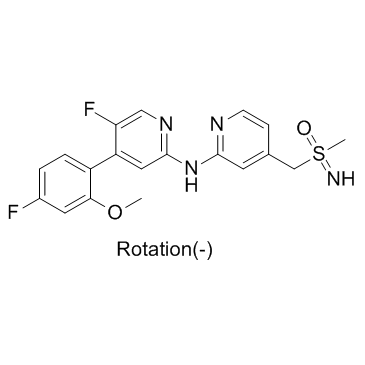
-
GC48635
(-)-Cryptopleurine
(R)-Cryptopleurine, NSC 19912
An alkaloid with diverse biological activities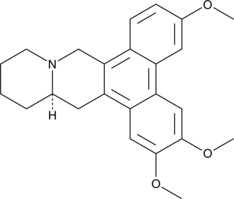
-
GC40076
(-)-Voacangarine
NSC 306219, (-)-Voacristine
(-)-Voacangarine is an indole alkaloid originally isolated from V.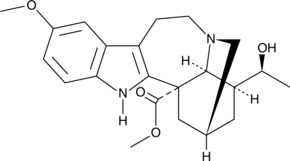
-
GC38377
(2S,3R)-Voruciclib hydrochloride
(2S,3R)-Voruciclib-Hydrochlorid ist das Enantiomer von Voruciclib-Hydrochlorid. (2S,3R)-Voruciclib ist ein oral aktiver CDK-Inhibitor.
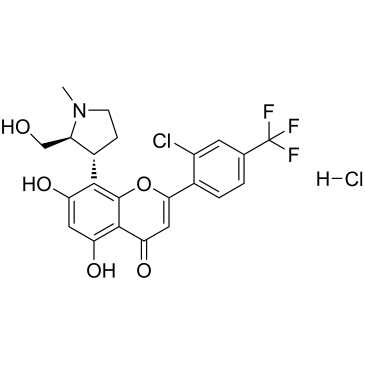
-
GC41695
(6R,S)-5,6,7,8-Tetrahydrofolic Acid (hydrochloride)
Tetrahydrofolate, THFA
(6R,S)-5,6,7,8-Tetrahydrofolic acid (THFA), the reduced form of folic acid, serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.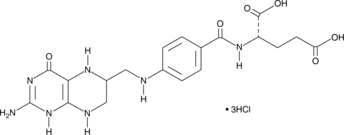
-
GC41088
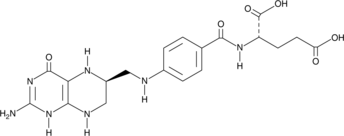
(6S)-Tetrahydrofolic Acid
(6S)-Tetrahydrofolic acid is a diastereomer of tetrahydrofolic acid, a reduced form of folic acid that serves as a cofactor in methyltransferase reactions and is the major one-carbon carrier in one carbon metabolism.
-
GC72591
(E,E)-RGFP966
(E,E)-RGFP966 is a selective and CNS permeable HDAC3 inhibitor that can be used for the research of Huntington’s disease.

-
GC62735
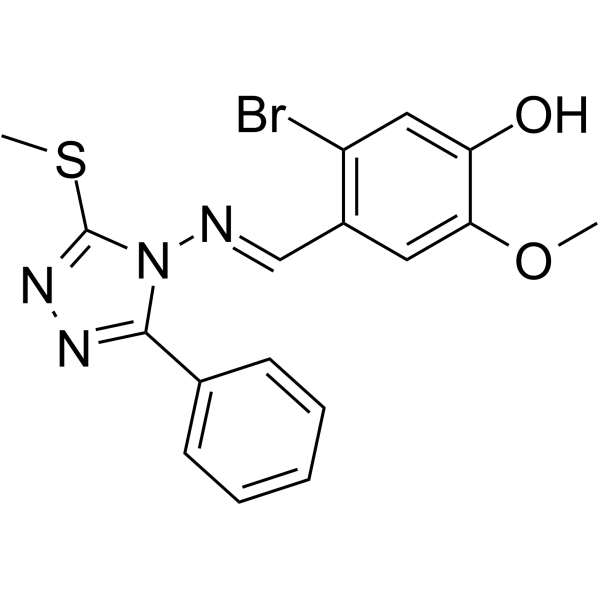
(E/Z)-GO289
(E/Z)-GO289 ist ein potenter und selektiver Caseinkinase 2 (CK2)-Inhibitor (IC50=7 nM). (E/Z)-GO289 verlÄngert die zirkadiane Periode stark. (E/Z)-GO289 zeigt eine zelltypabhÄngige Hemmung des Wachstums von Krebszellen, die mit der Funktion der Zelluhr korreliert.
-
GC64429
(E/Z)-Zotiraciclib citrate
(E/Z)-TG02 citrate; (E/Z)-SB1317 citrate
(E/Z)-Zotiraciclib-Citrat ist ein potenter CDK2-, JAK2- und FLT3-Inhibitor.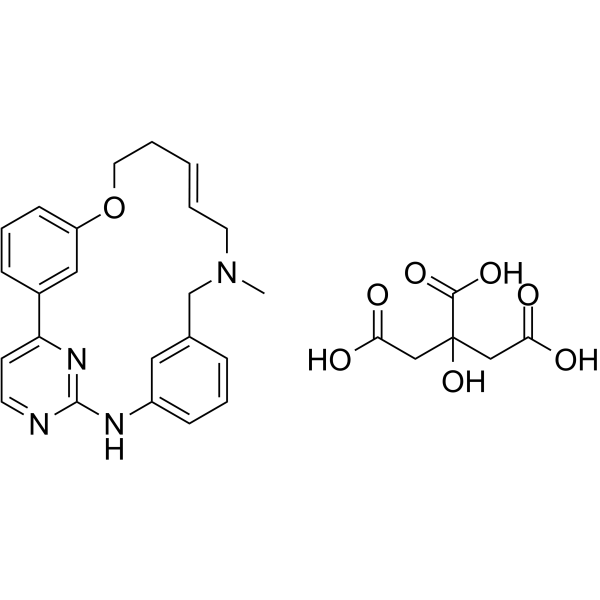
-
GC63864
(E/Z)-Zotiraciclib hydrochloride
(E/Z)-TG02 hydrochloride; (E/Z)-SB1317 hydrochloride
(E/Z)-Zotiraciclib ((E/Z)-TG02)-Hydrochlorid ist ein potenter CDK2-, JAK2- und FLT3-Inhibitor.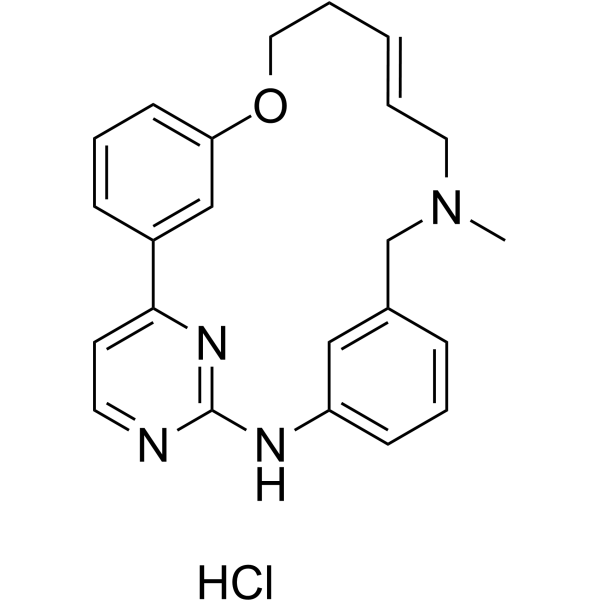
-
GC69793
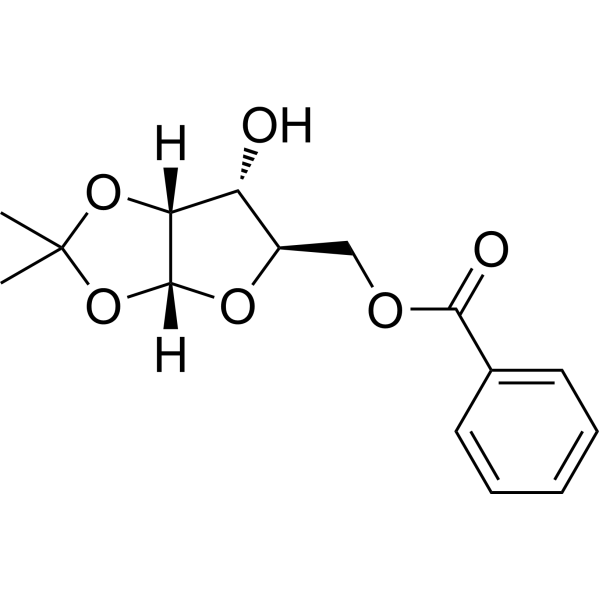
(R)-5-O-Benzoyl-1,2-di-O-isopropylidene-alpha-D-xylofuranose
(R)-5-O-Benzoyl-1,2-di-O-isopropylidene-alpha-D-xylofuranose ist ein Analogon von Purinnukleosiden. Purinnukleosidanaloge haben eine breite antitumorale Aktivität und zielen auf das inerte lymphatische System maligner Tumoren ab. Der Mechanismus der Antikrebswirkung beruht dabei auf der Hemmung der DNA-Synthese sowie der Induktion von Zellapoptose (programmierter Zelltod).
-
GC41716
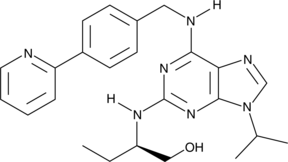
(R)-CR8
(R)-CR8 (CR8), ein Roscovitin-Analogon der zweiten Generation, ist ein potenter CDK1/2/5/7/9-Inhibitor.
-
GC39281
(R)-CR8 trihydrochloride
CR8, (R)-Isomer trihydrochloride
(R)-CR8 (CR8) Trihydrochlorid, ein Roscovitin-Analogon der zweiten Generation, ist ein potenter CDK1/2/5/7/9-Inhibitor.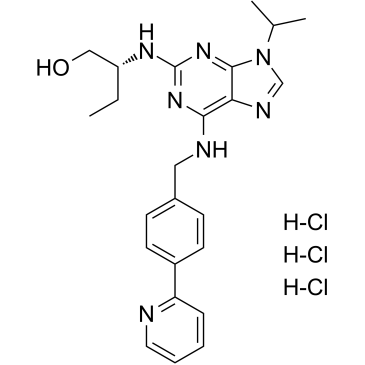
-
GC64210
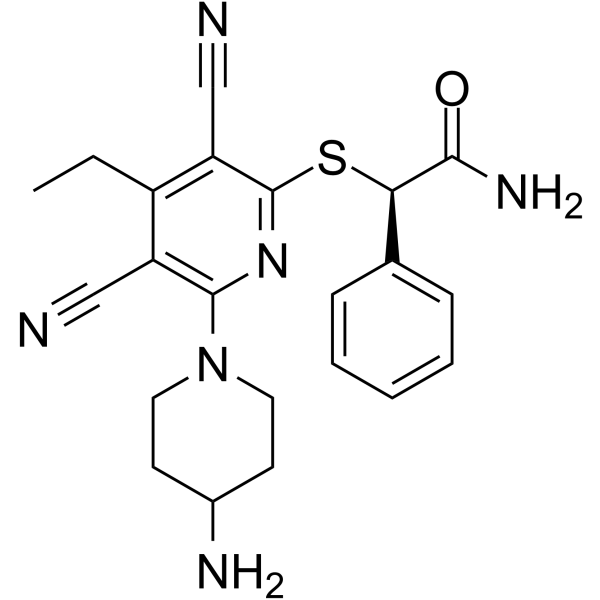
(R)-GSK-3685032
(R)-GSK-3685032 ist das R-Enantiomer von GSK-3685032. GSK-3685032 ist ein nicht zeitabhÄngiger, nicht kovalenter, erster reversibler DNMT1-selektiver Inhibitor seiner Klasse mit einem IC50-Wert von 0,036 μM. GSK-3685032 induziert einen starken Verlust der DNA-Methylierung, Transkriptionsaktivierung und Wachstumshemmung von Krebszellen.
-
GC41633
(R)-Prunasin
(R)-Prunasin ist ein Inhibitor der DNA-Polymerase β.
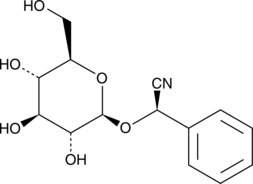
-
GC34124
(rel)-MC180295
(rel)-MC180295
(rel)-MC180295 ((rel)-(rel)-MC180295) ist ein potenter und selektiver CDK9-Cyclin-T1-Inhibitor mit einem IC50 von 5 nM, mindestens 22-mal selektiver fÜr CDK9 als fÜr andere CDKs. (rel)-MC180295 hemmt auch GSK-3α und GSK-3β. (rel)-MC180295 ((rel)-(rel)-MC180295) hat eine starke Antitumorwirkung.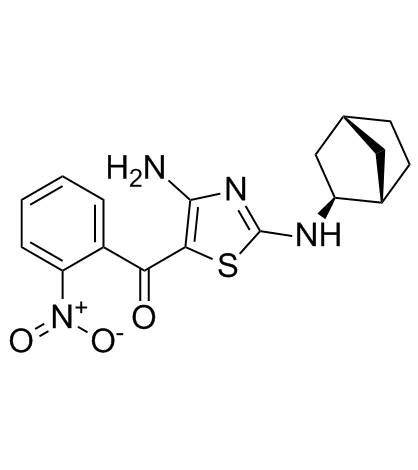
-
GC10098
(S)-10-Hydroxycamptothecin
ChEMBL 273862, NSC 107124
(S)-10-Hydroxycamptothecin (10-HCPT; 10-Hydroxycamptothecin) ist ein DNA-Topoisomerase-I-Inhibitor, der aus der chinesischen Pflanze Camptotheca accuminata isoliert wurde. (S)-10-Hydroxycamptothecin zeigt eine bemerkenswerte Apoptose-induzierende Wirkung. (S)-10-Hydroxycamptothecin hat das Potenzial zur Behandlung von Hepatom, Magenkarzinom, Dickdarmkrebs und LeukÄmie.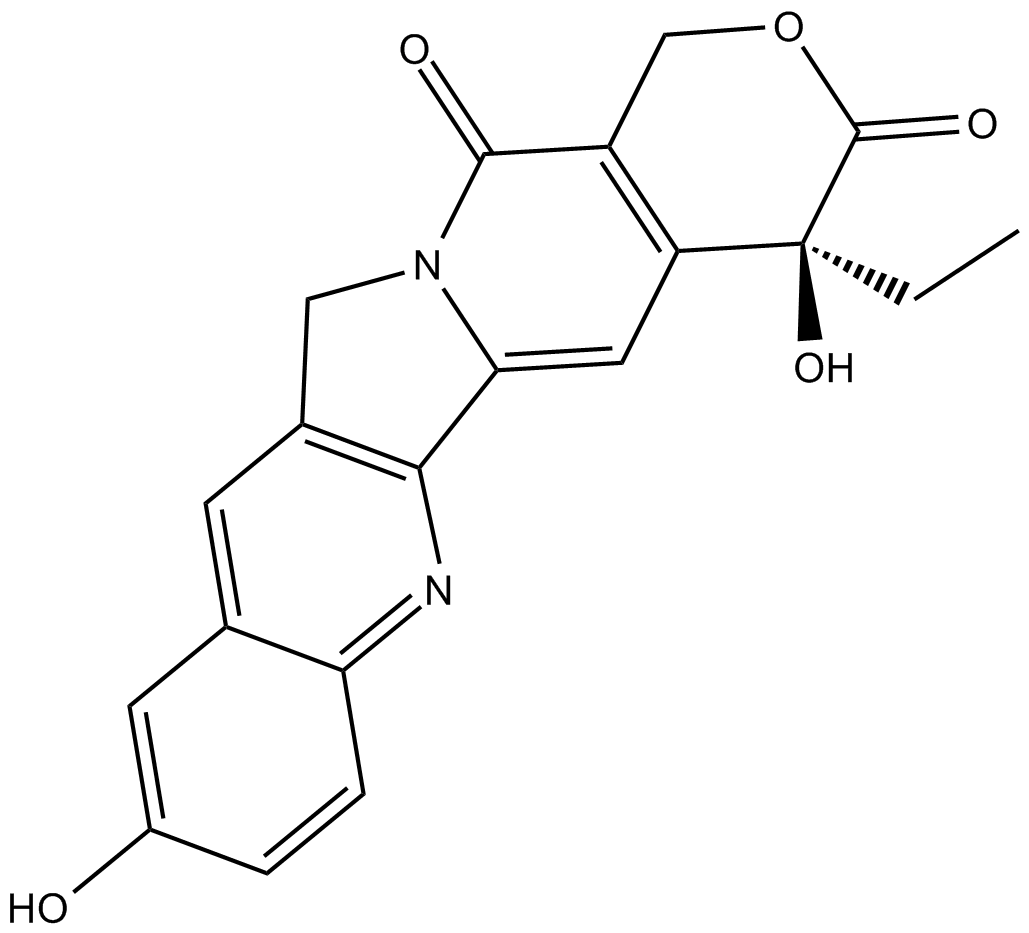
-
GC34999
(S)-Ceralasertib
(S)-AZD6738
(S)-Ceralasertib ((S)-AZD6738) wird aus dem Patent WO2011154737A1 extrahiert, Verbindung II, weist einen IC50 von 2,578 nM auf. (S)-Ceralasertib ist ein potenter und selektiver Sulfoximin-Morpholinopyrimidin-ATR-Inhibitor mit ausgezeichneter prÄklinischer physikalisch-chemischer und pharmakokinetischer (PK )-Eigenschaften.(S)-Ceralasertib wurde entwickelt, um die WasserlÖslichkeit zu verbessern und die zeitabhÄngige Hemmung von CYP3A4 zu eliminieren.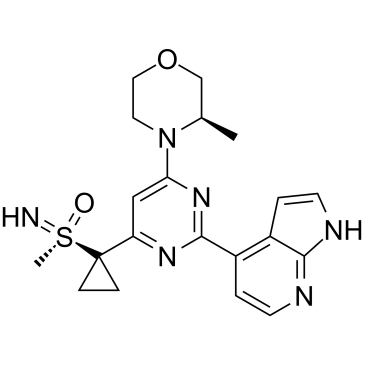
-
GC46351
(S)-CR8
(S)-CR8 ist das S-Isomer von CR8. (S)-CR8 ist ein potenter und selektiver CDK-Inhibitor mit IC50-Werten von 0,060, 0,080, 0,11, 0,12 und 0,15 μM fÜr CDK2/Cyclin E, CDK2/Cyclin A, CDK9/Cyclin T, CDK5/p25 und CDK1/Cyclin B bzw. (S)-CR8 reduziert das Überleben von SH-SY5Y-Zellen (IC50 0,40 μM).
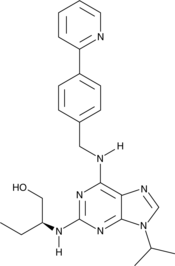
-
GC13136
(S)-Crizotinib
(S)-Crizotinib ist ein potenter und selektiver MTH1 (mutT-Homolog)-Inhibitor mit einem IC50 von 330 nM. (S)-Crizotinib stÖrt die Nukleotidpool-HomÖostase durch MTH1-Hemmung, induziert eine Zunahme von DNA-EinzelstrangbrÜchen, aktiviert die DNA-Reparatur in menschlichen Kolonkarzinomzellen und unterdrÜckt effektiv das Tumorwachstum in Tiermodellen.
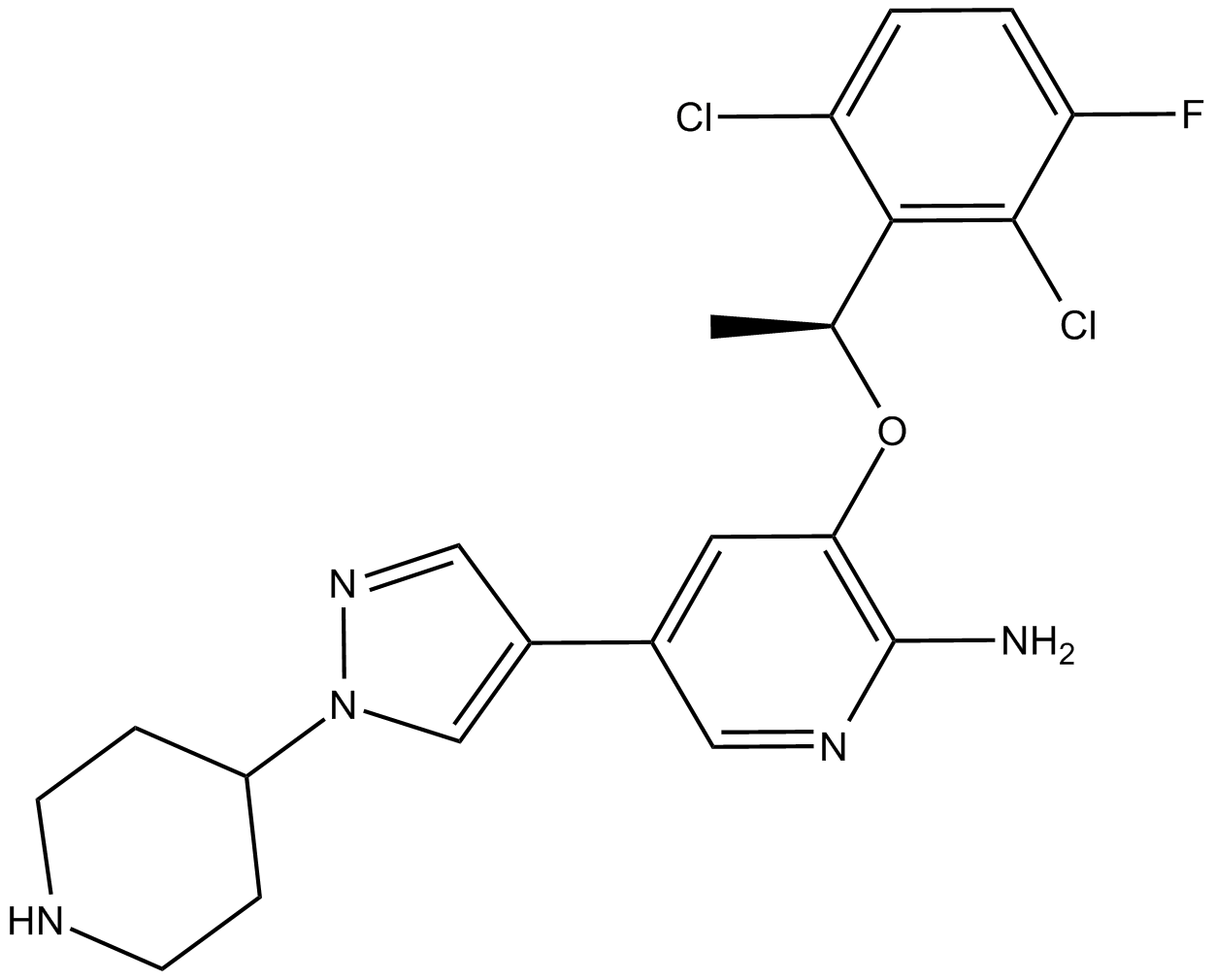
-
GC65877
(S)-GFB-12811
(S)-GFB-12811 (Verbindung 596) ist ein potenter und selektiver CDK5-Inhibitor mit einem IC50-Wert von weniger als 10 nM. (S)-GFB-12811 kann bei der Erforschung der Zellzyklusprogression, der neuronalen Entwicklung und der Tumorentstehung verwendet werden.
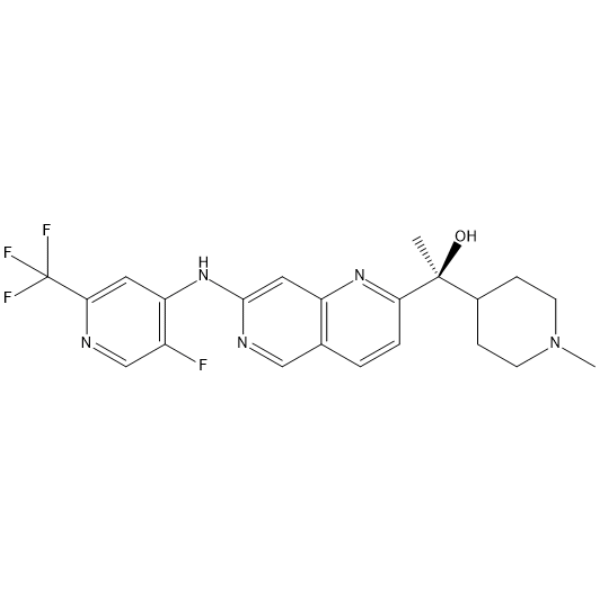
-
GC65997
(S)-LY3177833 hydrate
(S)-LY3177833 ((S)-Beispiel 2)-Hydrat ist ein oral aktiver CDC7-Kinase-Inhibitor. (S)-LY3177833-Hydrat zeigt eine breite In-vitro-AntikrebsaktivitÄt.
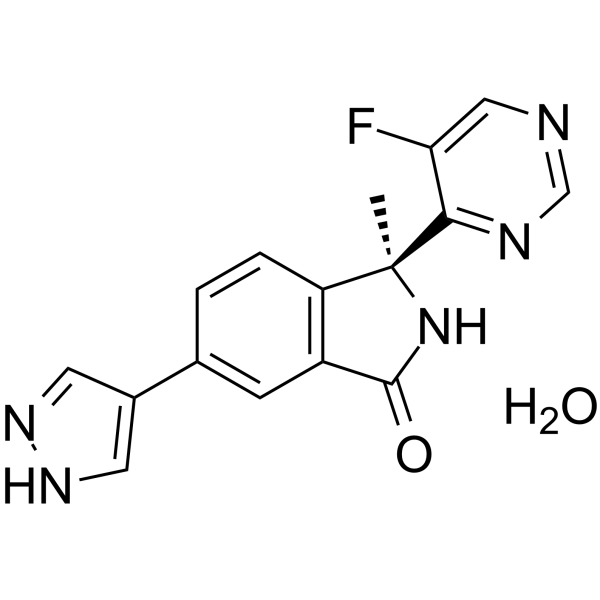
-
GC60421
(S)-Seco-Duocarmycin SA
(S)-Seco-Duocarmycin SA ist ein DNA-Alkylator, zytotoxisch fÜr Krebszellen und wirkt als ADC-Zytotoxin fÜr AntikÖrper-Wirkstoff-Konjugate.
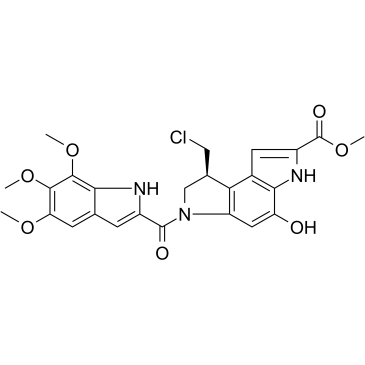
-
GC39842
(Z)-4EGI-1
(Z)-4EGI-1 ist das Z-Isomer von 4EGI-1 und ist ein Inhibitor der eIF4E/eIF4G-Interaktion und der Translationsinitiation. (Z)-4EGI-1 bindet effektiv an eIF4E mit einem IC50 von 43,5 μM und einem Kd-Wert von 8,74 μM. (Z)-4EGI-1 hat AntikrebsaktivitÄt.
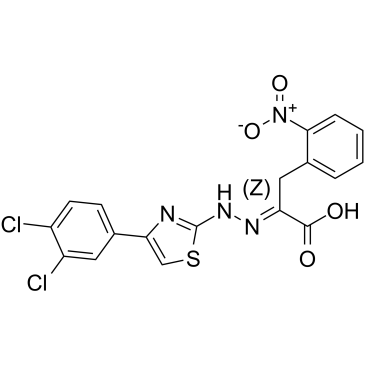
-
GC68484
1,2-O-Isopropylidene-5-O-p-toluoyl-a-D-xylofuranose
1,2-O-Isopropyliden-5-O-p-toluoyl-a-D-xylofuranose ist ein Analogon von Purinnukleosiden. Purinnukleosid-Analoga haben eine breite Palette an antitumoralen Aktivitäten und zielen auf maligne Lymphsystem-Tumoren ab. Der Mechanismus der Antikrebswirkung beruht dabei auf der Hemmung der DNA-Synthese, Induktion von Zellapoptose (Apoptose) usw.
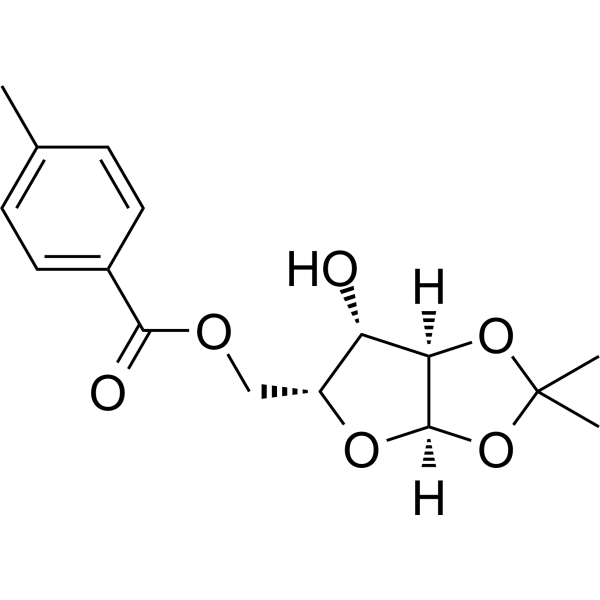
-
GC19528
1,4-Benzoquinone
p-Benzoquinone, NSC 36324, p-Quinone
A toxic metabolite of benzene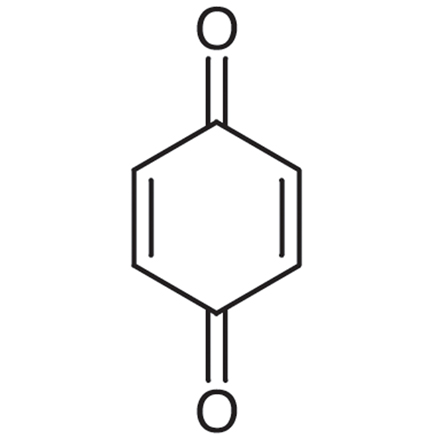
-
GC41859
1,5,7-Triazabicyclo[4.4.0]dec-5-ene
TBD, Triazabicyclodecene
1,5,7-Triazabicyclo[4.4.0]dec-5-ene is a bicyclic guanidine base that can be used as a catalyst for a variety of base-mediated organic transformations, including Michael additions, Wittig reactions, Henry reactions, and transesterification reactions.![1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure 1,5,7-Triazabicyclo[4.4.0]dec-5-ene Chemical Structure](/media/struct/GC4/GC41859.png)
-
GC65551
1-(2'-O-4-C-Methylene-beta-D-ribofuranosyl)thymine
1-(&2#39;-O-4-C-Methylen-beta-D-ribofuranosyl)thymin ist ein bicyclisches Nucleosid.
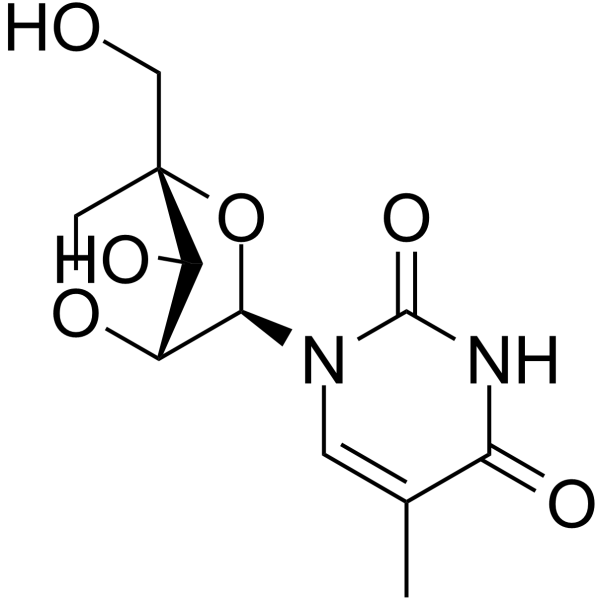
-
GC71294
1-(2-Deoxy-β-D-threo-pentofuranosyl)thymine
1-(2-Deoxy-β-D-threo-pentofuranosyl)tmine is a tmidine analog.

-
GC49470
1-Ethyl-1-nitrosourea (hydrate)
ENU, Ethylnitrosourea, N-Ethyl-N-nitrosourea, N-Nitroso-N-ethylurea
A DNA alkylating agent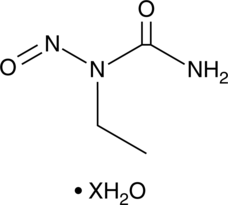
-
GC48909
1-Hydroxyanthraquinone
NSC 8640
1-Hydroxyanthrachinon, eine natÜrlich vorkommende Verbindung mit oraler Wirkung aus einigen Pflanzen wie Tabebuia avellanedae, weist eine krebserzeugende Wirkung auf.
-
GC65038
1-Methylinosine
1-methyl Inosine, N1-Methylinosine
1-Methylinosin ist ein modifiziertes Nukleotid, das an Position 37 in tRNA 3' zum Anticodon von eukaryotischer tRNA gefunden wird.
-
GC49872
10-Formyltetrahydrofolate (sodium salt) (technical grade)
10-CHO-FH4, 10-CHO-THF, N10-Formyltetrahydrofolate, 10-formyl H4PteGlu, 10-fTHF
10-Formyltetrahydrofolat (Natriumsalz) (technische QualitÄt) ist eine Form von TetrahydrofolsÄure, die als Spender von Formylgruppen im Anabolismus wirkt.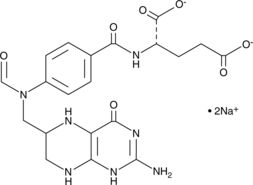
-
GC63796
116-9e
MAL2-11B
116-9e (MAL2-11B) ist ein Hsp70-Co-Chaperon-DNAJA1-Inhibitor.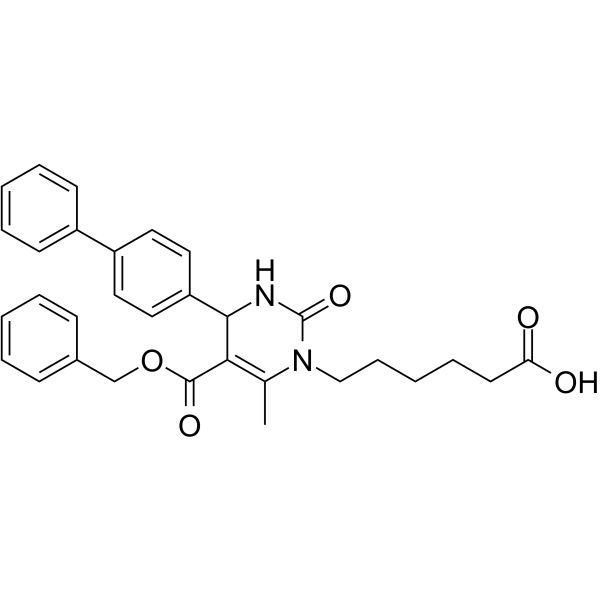
-
GC71326
12R-LOX-IN-2
12R-LOX-IN-2 (compound 7b) is an inhibitor of 12R-lipoxygenase (12R-LOX).

-
GC49759
13C17-Mycophenolic Acid
13C17-MPA
An internal standard for the quantification of mycophenolic acid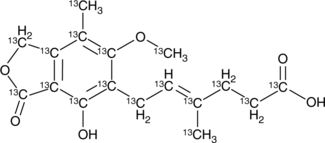
-
GC46474
18-Deoxyherboxidiene
RQN-18690A
18-Desoxyherboxidien (RQN-18690A) ist ein potenter Angiogenese-Inhibitor.
-
GC65489
2'-F-Bz-dC Phosphoramidite
&2#39;-F-Bz-dC Phosphoramidit kann bei der Synthese von Oligoribonukleotiden verwendet werden.
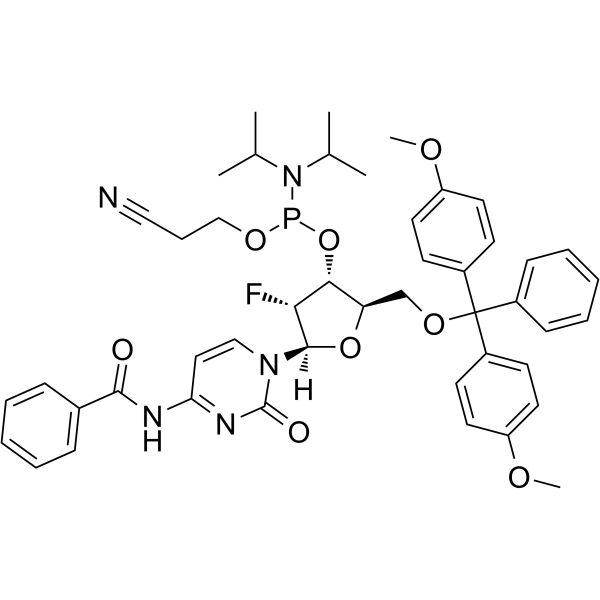
-
GC66651
2'-O,4'-C-Methyleneadenosine
LNA-A
2'-O,4'-C-Methylenadenosin (LNA-A) ist eine verschlossene Nukleinsäure (LNA) und ist auch ein Adenosin-Analogon.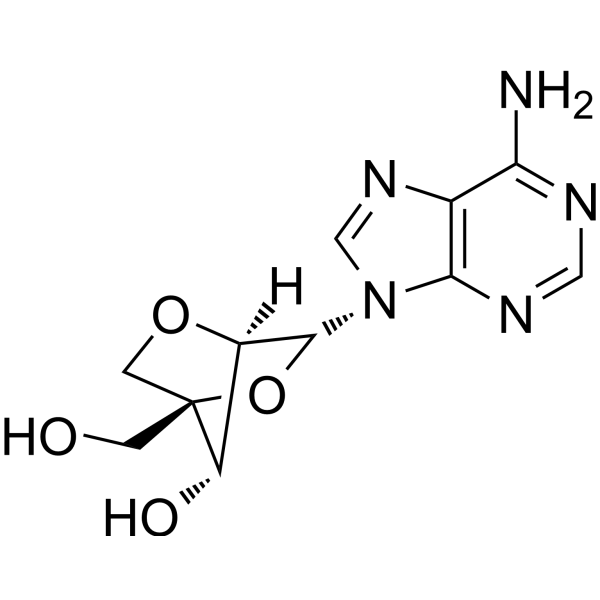
-
GC66654
2'-O,4'-C-Methylenecytidine
LNA-C(Bz)
2'-O,4'-C-Methylencytidin (LNA-C(Bz)) ist ein bicyclisches Nukleosid-Analogon mit fester N-Typ-Konformation. 2'-O,4'-C-Methylenecytidin kann zur Synthese von Oligonukleotiden verwendet werden. 2'-O,4'-C-Methylencytidin bildet Doppelstränge mit komplementären DNA- und RNA-Strängen.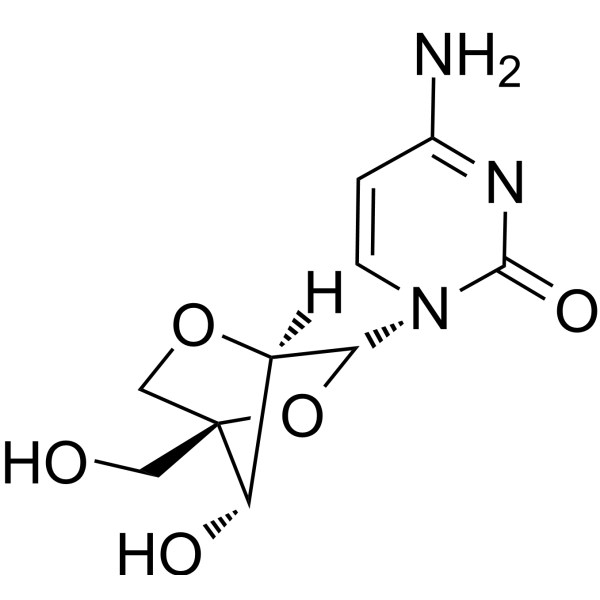
-
GC66655
2'-O,4'-C-Methyleneguanosine
LNA-G
2′-O,4′-C-Methylenguanosin (LNA-G) ist ein reverses Guanin-Analogon, wobei LNA (Locked Nucleinsäure) ein Nukleinsäure-Analogon ist. Die LNA-Modifikation kann in einer Vielzahl von Anwendungen verwendet werden, wie z. B. effektive Bindungsaffinität zu komplementären Sequenzen und größere Nukleaseresistenz als natürliche Nukleotide, was ein großes Potenzial für Anwendungen in der Krankheitsdiagnose und -forschung bietet. LNA-G ist auch über die KOD-DNA-Polymerase verfügbar, die die Integration von LNA-G-Nukleotiden in den DNA-Strang ermöglicht.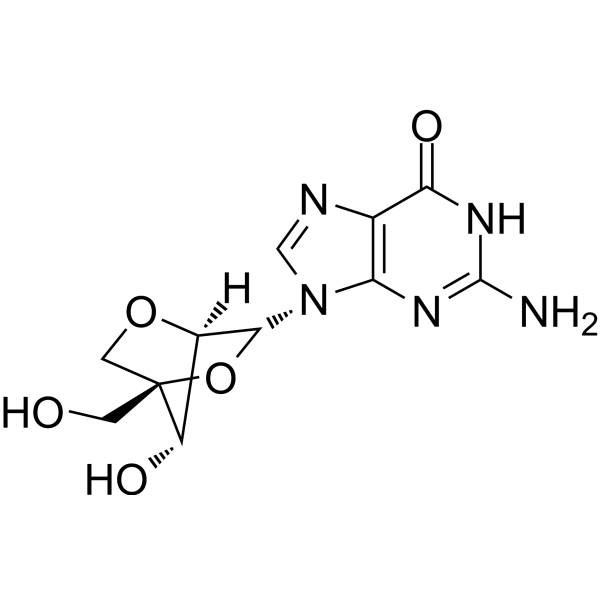
-
GC52183
2'-O-Methyl-5-methyluridine
2'-O-Methylribothymidine, 2'-O-Methylthymidine
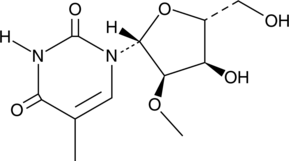
-
GC66713
2'-O-MOE-5-Me-C(Bz)
2'-O-MOE-5-Me-C (Bz) ist ein Nukleotid für die stereoselektive Synthese von Nukleosid-Alkylphosphonaten.
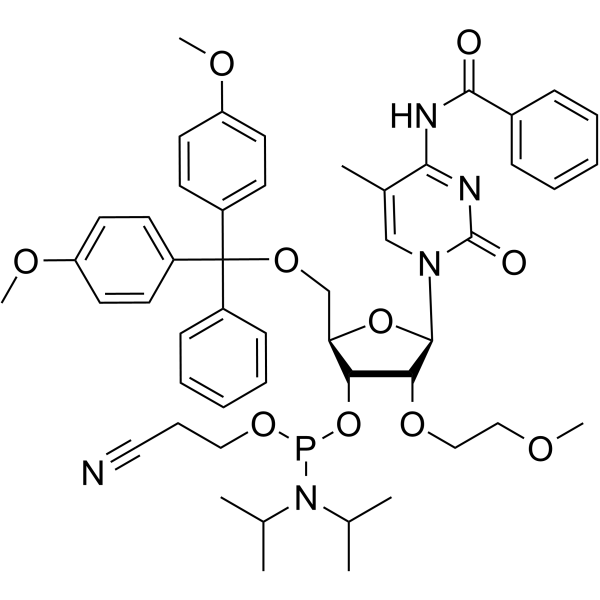
-
GC66657
2'-O-MOE-5-Me-rC
2'-O-MOE-5-Me-rC ist ein Wirkstoff. 2'-O-MOE-5-Me-rC kann für die Oligonukleotidsynthese verwendet werden.
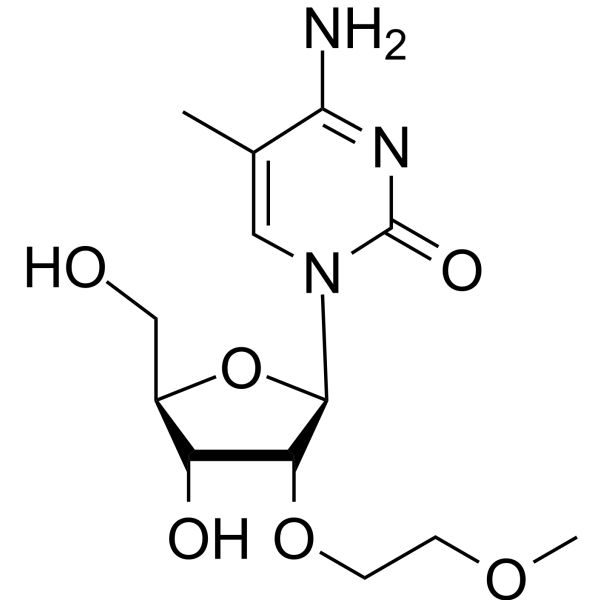
-
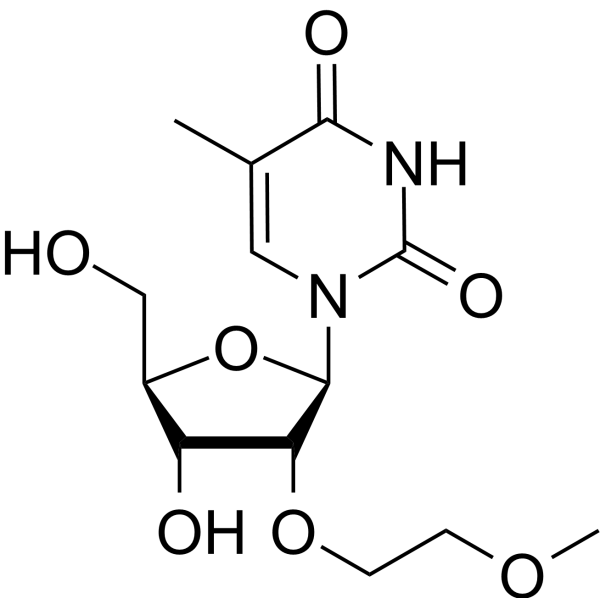
GC66091
2'-O-MOE-5-Me-rU
2'-O-MOE-5-Me-rU ist ein Wirkstoff. 2'-O-MOE-5-Me-rU kann zur Oligonukleotidsynthese verwendet werden.
-
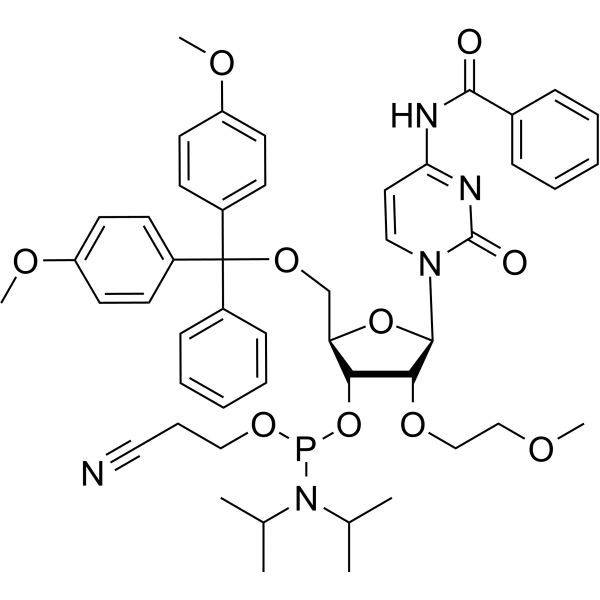
GC66656
2'-O-MOE-rC
2'-O-MOE-rC ist ein 2&7#39;-O-MOE-modifiziertes Nucleosid. 2'-O-MOE-rC kann zur Synthese von DNA verwendet werden.
-
GC66652
2'-O-MOE-U
2'-O-MOE-U ist ein Phosphoramidit, das zur Oligonukleotidsynthese verwendet werden kann.

-
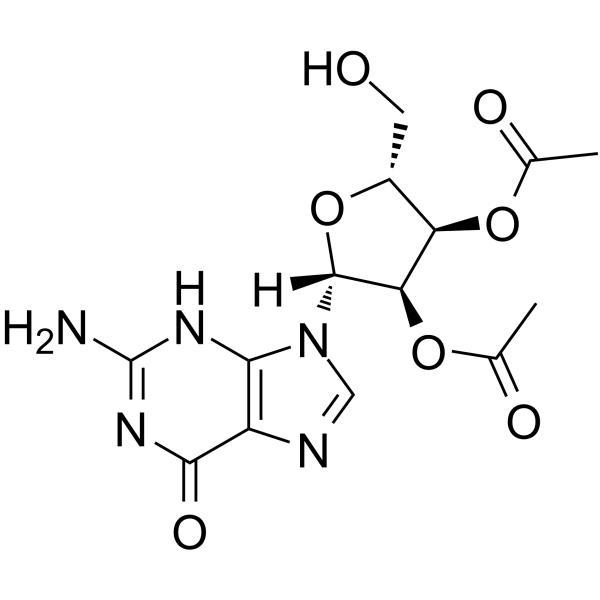
GC65170
2′,3′-Di-O-acetylguanosine
&2#8242;,3′-Di-O-Acetylguanosin ist ein Nukleosid-Analogon.
-
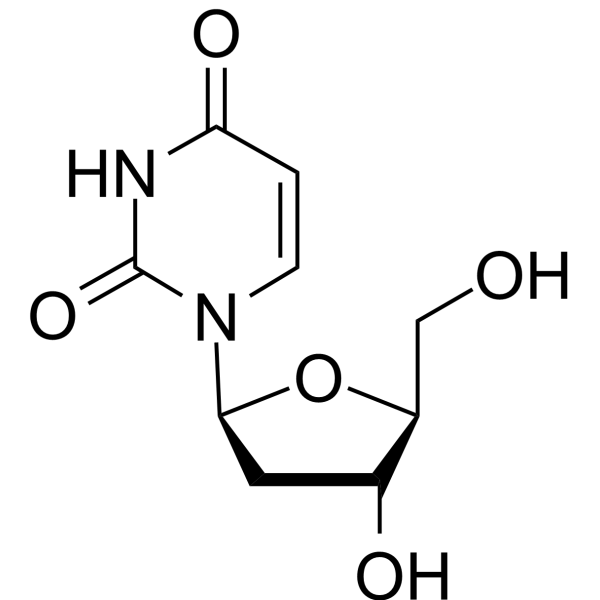
GC67384
2′-Deoxy-β-L-uridine
2'-Deoxy-β-L-Uridin ist ein Nucledside-Analogon und ein spezifisches Substrat für das virale Enzym, zeigt keine Stereospezifität gegen Herpes simplex 1 (HSV1) Thymidinkinase (TK). 2′-Deoxy-β-L-Uridin übt antivirale Aktivität über die Interaktion von 5&7#39;-Triphosphaten mit der viralen DNA-Polymerase aus.
-
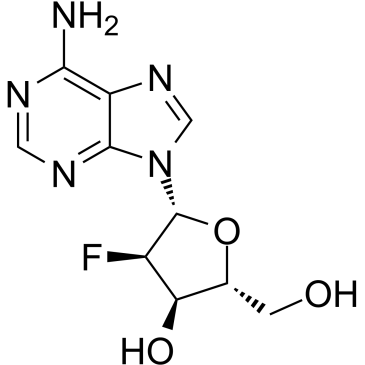
GC61667
2′-Deoxy-2′-fluoroadenosine
&2#8242;-Deoxy-&2#8242;-Fluoradenosin kann für die Synthese von 2′-Desoxy-2′-fluormodifizierten Oligonukleotiden verwendet werden, die mit RNA hybridisiert sind. &2#8242;-Desoxy-&2#8242;-Fluoradenosin kann effizient durch E. coli-Purinnukleosid-Phosphorylase (PNP) zum Giftstoff 2-Fluoradenin (FAde) gespalten werden. &2#8242;-Deoxy-&2#8242;-Fluoradenosin zeigt eine hervorragende in vivo-Aktivität gegen Tumore, die E. coli PNP exprimieren.
-
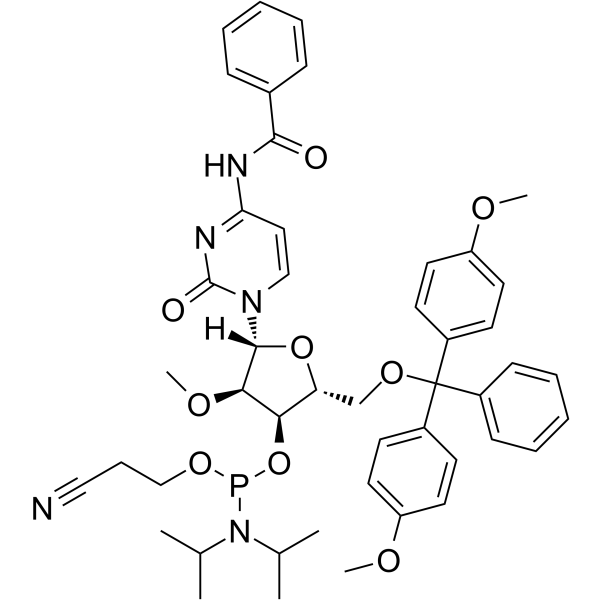
GC62530
2’-O-Me-C(Bz) Phosphoramidite
&2rsquo;-O-Me-C(Bz) Phosphoramidit ist ein modifiziertes Phosphoramidit-Monomer, das für die Oligonukleotidsynthese verwendet werden kann.
-
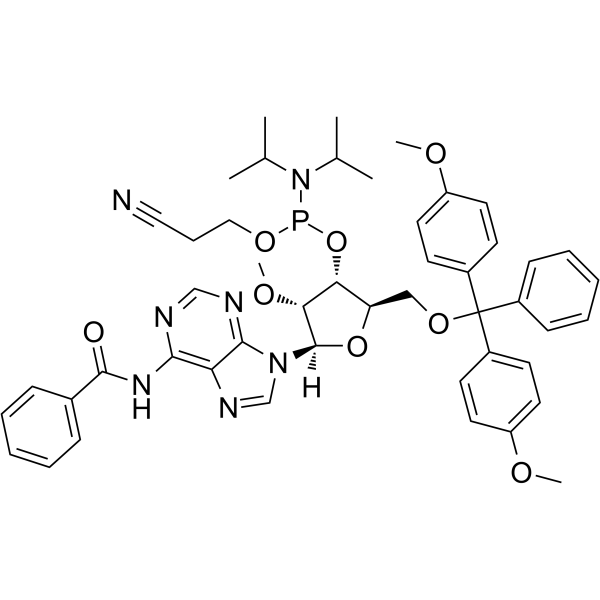
GC62529
2’-OMe-A(Bz) Phosphoramidite
&2rsquo;-OMe-A(Bz) Phosphoramidit ist ein modifiziertes Phosphoramidit-Monomer, das für die Oligonukleotidsynthese verwendet werden kann.
-
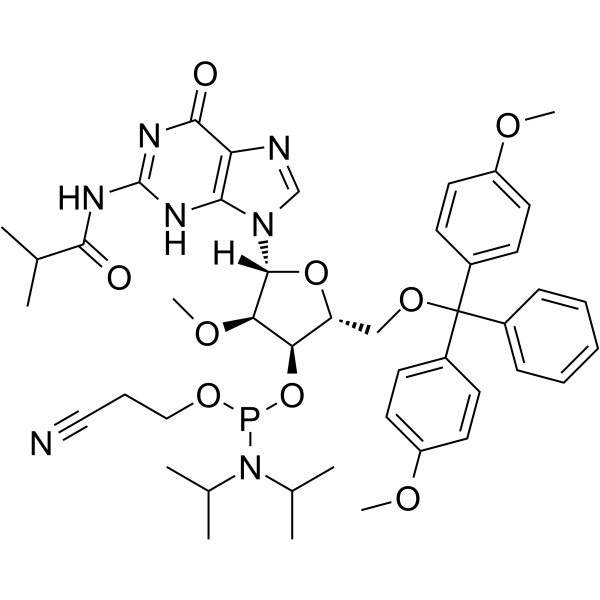
GC62531
2’-OMe-G(ibu) Phosphoramidite
&2rsquo;-OMe-G(ibu) Phosphoramidit ist ein modifiziertes Phosphoramidit-Monomer, das für die Oligonukleotidsynthese verwendet werden kann.
-
GC46508
2',2'-Difluoro-2'-deoxyuridine
dFdU
An active metabolite of gemcitabine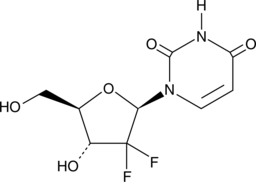
-
GC42061
2',3'-Dideoxyadenosine 5'-triphosphate
2',3'-Dideoxyadenosine 5'-triphosphate
2',3'-Dideoxyadenosine 5'-triphosphate (2',3'-Dideoxyadenosine 5'-triphosphate), an active metabolite of 2',3'-dideoxyinosine, is a chain-elongating inhibitor of DNA polymerase.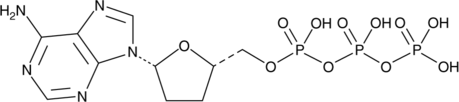
-
GC46519
2',3'-O-Isopropylideneguanosine
A building block
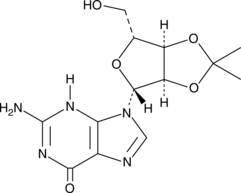
-
GC46540
2'-Deoxyadenosine-5'-triphosphate (sodium salt hydrate)
A purine nucleotide
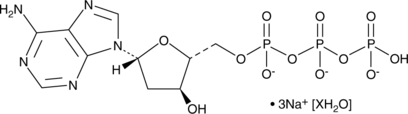
-
GC48440
2'-Deoxycytidine-5'-triphosphate (sodium salt)
dCTP
2'-Desoxycytidin-5'-Triphosphat (Natriumsalz) (dCTP-Trinatriumsalz) ist ein Nukleosidtriphosphat, das fÜr die DNA-Synthese verwendet werden kann.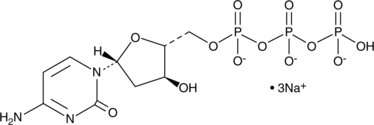
-
GC42151
2'-Deoxyguanosine 5'-monophosphate (sodium salt hydrate)
dGMP
2'-Deoxyguanosine 5'-monophosphate (dGMP) is used as a substrate of guanylate kinases to generate dGDP, which in turn is phosphorylated to dGTP, a nucleotide precursor used in DNA synthesis.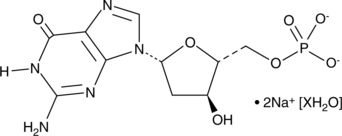
-
GC33430
2'-Deoxypseudoisocytidine
2'-Desoxypseudoisocytidin ist ein Nukleosid-Analogon.
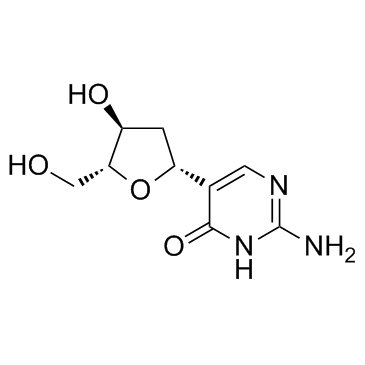
-
GC35072
2'-O,4'-C-Methyleneuridine
2'-O,4'-C-Methylenuridin (Verbindung 15a) ist ein bicyclisches Nucleosid.

-
GC42080
2'2'-cGAMP (sodium salt)
Adenosine-Guanosine 2’,2’-cyclic monophosphate, cGAMP(2’-5’), 2’,2’-Cyclic GMP-AMP
2'2'-cGAMP is a synthetic dinucleotide (CDN) that contains non-canonical 2'5'-phosphodiester bonds.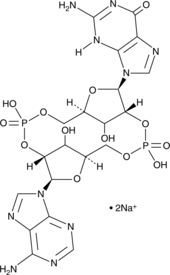
-
GC64399
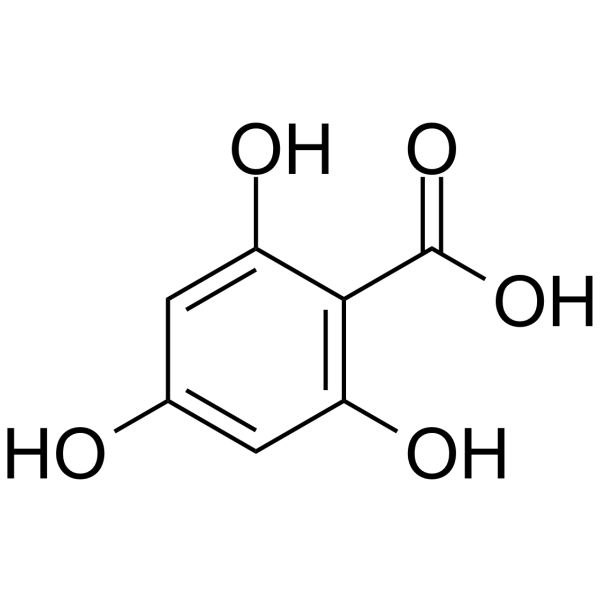
2,4,6-Trihydroxybenzoic acid
2,4,6-TrihydroxybenzoesÄure, der Flavonoid-Metabolit, ist ein CDK-Inhibitor. 2,4,6-TrihydroxybenzoesÄure kann fÜr die Krebsforschung eingesetzt werden.
-
GC60459
2,4-D (2,4-Dichlorophenoxyacetic acid)
2,4D, Diclordon, Vidon 638
2,4-D (2,4-DichlorphenoxyessigsÄure) (2,4-D (2,4-DichlorphenoxyessigsÄure)-ichlorphenoxyessigsÄure) ist ein selektives systemisches Herbizid zur BekÄmpfung von breitblÄttrigen UnkrÄutern.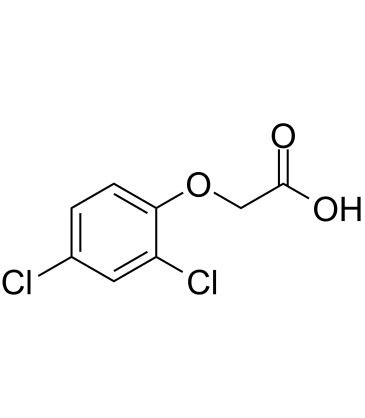
-
GC71530
2,4-D-13C6
2,4-D-13C6 is the 13C-labeled 2,4-D.

-
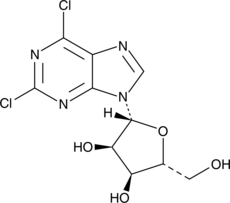
GC46524
2,6-Dichloropurine-9-β-D-riboside
2,6-Dichloropurine riboside
A building block
-
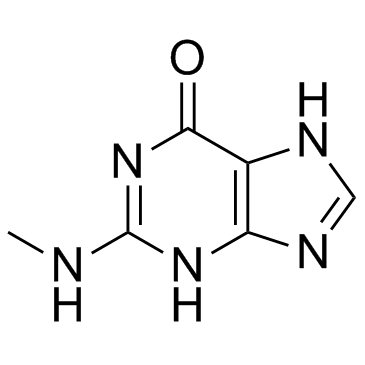
GC33496
2-(Methylamino)-1H-purin-6(7H)-one (N2-methylguanine)
2-(Methylamino)-1H-purin-6(7H)-on (N2-Methylguanin) (N2-Methylguanin) ist ein modifiziertes Nukleosid.
-
GC64983
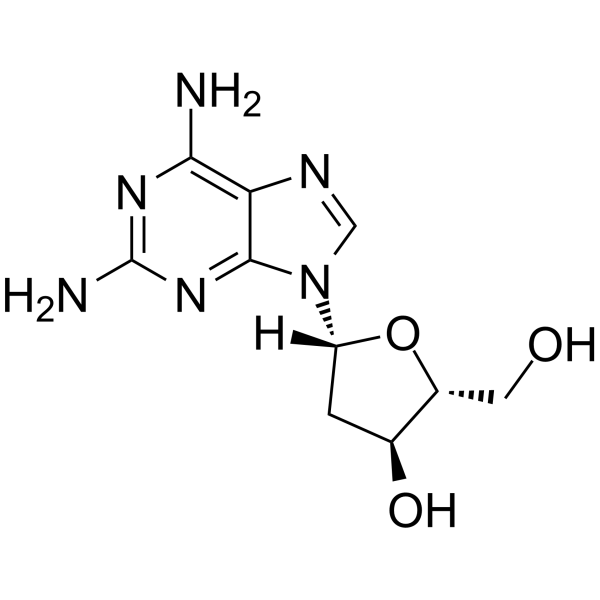
2-Amino-2'-deoxyadenosine
2-Amino-&2#39;-Desoxyadenosin ist ein Desoxyribonukleosid, das für die Oligonukleotidsynthese verwendet wird.
-
GC71560
2-Iodoacetamide-d4
2-Iodoacetamide-d4 is the deuterium labeled 2-Iodoacetamide.

-
GC71208
2-Methyladenosine
2-Metladenosine is an adenosine analogue.

-
GC39527
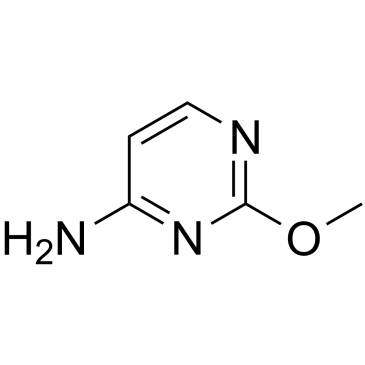
2-O-Methylcytosine
2-O-Methylcytosin, ein O-alkyliertes Analogon eines DNA-Addukts, ist die beschÄdigte Nukleobase.
-
GC49348
2-Thiocytidine
α-2-Thioribocytidine
A modified nucleobase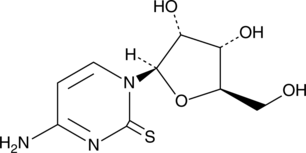
-
GC42197
2-Thiouridine
1-β-D-ribofuranosyl-2-thiouracil, s2U
2-Thiouridine (s2U) is a modified nucleobase found in tRNAs that is known to stabilize U:A pairs and modestly destabilize U:G wobble pairs.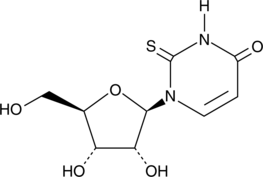
-
GC65083
3'-Azido-3'-deoxy-5-fluorocytidine
3'-Azido-3'-desoxy-5-fluorcytidin (Verbindung 12) ist ein Cytidin-Derivat.
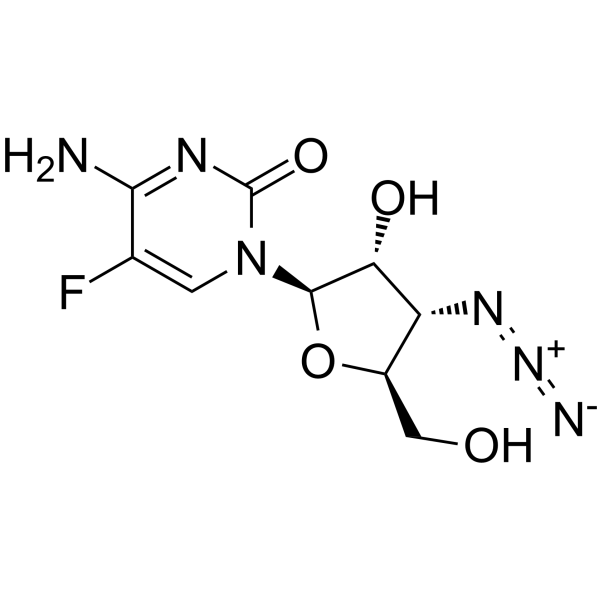
-
GC71204
3'-O-(2-Methoxyethyl)guanosine
3’-O-(2-Methoxyetl)guanosine is a guanosine analogue.

-
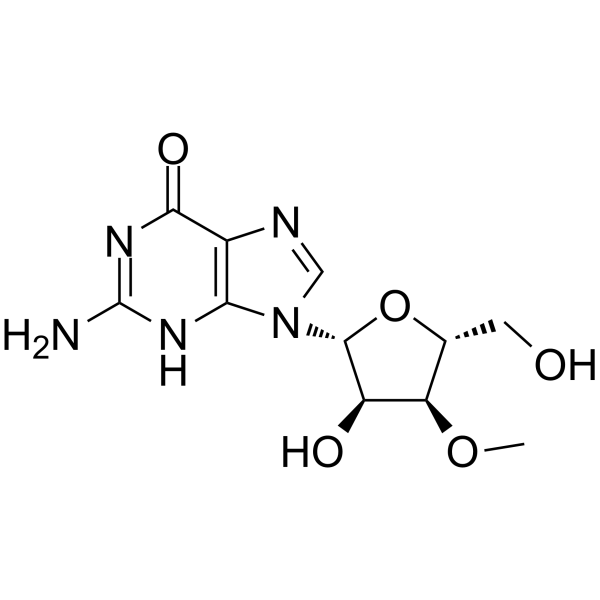
GC64985
3'-O-Methylguanosine
3'-O-Methylguanosin ist ein methyliertes Nukleosidanaloga und ein RNA-Kettenterminator.
-
GC68542
3'Ome-m7GpppAmpG ammonium
3'Ome-m7GpppAmpG-Ammonium ist ein Triphosphat-Cap-Analogon, das eine LNA-Molekül enthält. Es hat eine signifikante Translationseffizienz und kann als potenzielles Werkzeug in der molekularen Biologie für mRNA-Impfstoffe und mRNA-Transfektionsanwendungen wie Proteinproduktion, Gentherapie und Krebsimmuntherapie eingesetzt werden.

-
GC40618
3',4',7-Trihydroxyisoflavone
3'-hydroxy Daidzein, 3’,4’,7-THIF
3',4',7-Trihydroxyisoflavon, ein Hauptmetabolit von Daidzein, ist ein ATP-kompetitiver Inhibitor von Cot (Tpl2/MAP3K8) und MKK4. 3',4',7-Trihydroxyisoflavon hat krebshemmende, antiangiogene, chemoprotektive und RadikalfÄnger-AktivitÄten.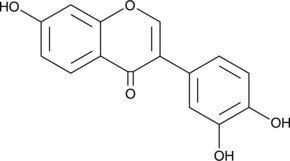
-
GC35105
3'-Azido-3'-deoxy-beta-L-uridine
3'-Azido-3'-desoxy-beta-L-uridin (Verbindung 25) ist ein Nukleosidderivat.
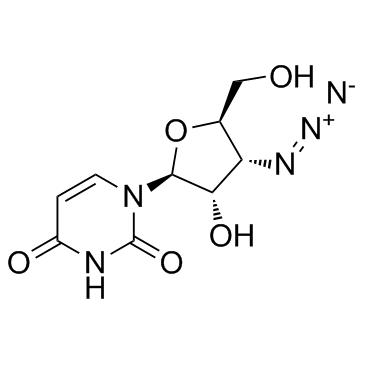
-
GC60023
3'-Deoxyuridine-5'-triphosphate
3'-dUTP
3'-Desoxyuridin-5'-Triphosphat (3'-dUTP) ist ein Nukleotidanalogon, das die DNA-abhÄngigen RNA-Polymerasen I und II hemmt.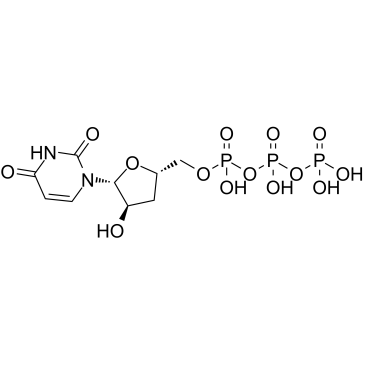
-
GC61862
3'-Deoxyuridine-5'-triphosphate trisodium
3'-dUTP trisodium
3'-Desoxyuridin-5'-Triphosphat-Trinatrium (3'-dUTP-Trinatrium) ist ein Nukleotidanalogon, das die DNA-abhÄngigen RNA-Polymerasen I und II hemmt.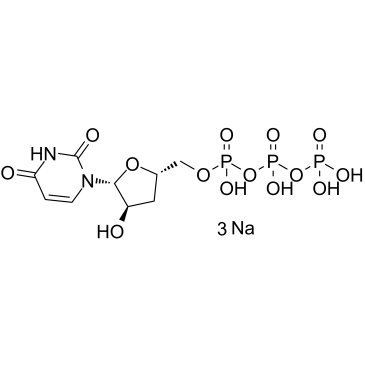
-
GC45332
3'-Dephosphocoenzyme A
depCoA, Dephospho-CoA
An intermediate in the biosynthesis of CoA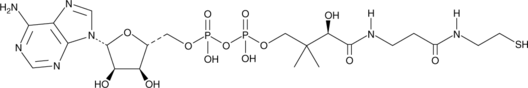
-
GC34384
3,6-DMAD hydrochloride
3,6-DMAD-Hydrochlorid, ein Acridin-Derivat, ist ein potenter Inhibitor des IRE1α-XBP1s-Signalwegs. 3,6-DMAD-Hydrochlorid fÖrdert die IL-6-Sekretion Über den IRE1α-XBP1s-Weg. 3,6-DMAD-Hydrochlorid hemmt die Oligomerisierung von IRE1α und die AktivitÄt von Endoribonuclease (RNase). 3,6-DMAD-Hydrochlorid kann fÜr die Krebsforschung verwendet werden.
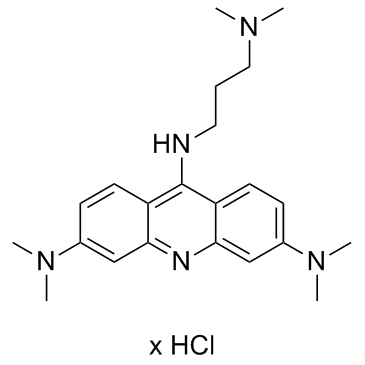
-
GC34452
3,7,4'-Trihydroxyflavone
3,7,4'-Trihydroxyflavon, isoliert aus Rhus javanica Var.
-
GC52129
3-Amino-5-hydroxybenzoic Acid
AHBA
-
GC13510
3-AP
3-Aminopyridine-2-Carboxyaldehyde Thiosemicarbazone,NSC 663249,Triapine™
3-AP (PAN-811) ist ein potenter Inhibitor der M2-Untereinheit der Ribonukleotidreduktase (RR) und ein potenter Radiosensibilisator.
-
GC48457
3-keto Fusidic Acid
3-keto FA, 3-Oxofusidic Acid
An active metabolite of fusidic acid
-
GC65084
3-Methylcytidine
3-Methylcytidin, ein Nukleosid im Urin, kann als Biomarker fÜr vier verschiedene Krebsarten verwendet werden: Lungenkrebs, Magenkrebs, Dickdarmkrebs und Brustkrebs.
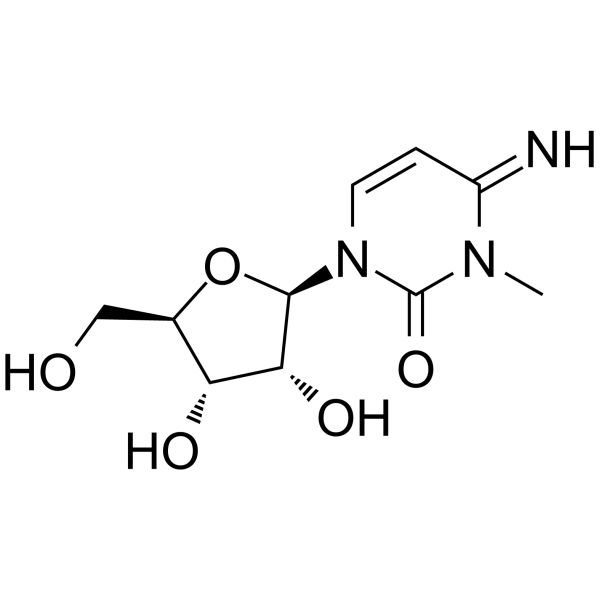
-
GC52391
306-O12B-3
An ionizable cationic lipidoid
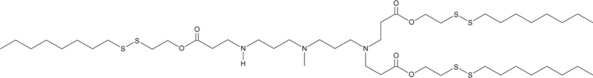
-
GC15389
360A
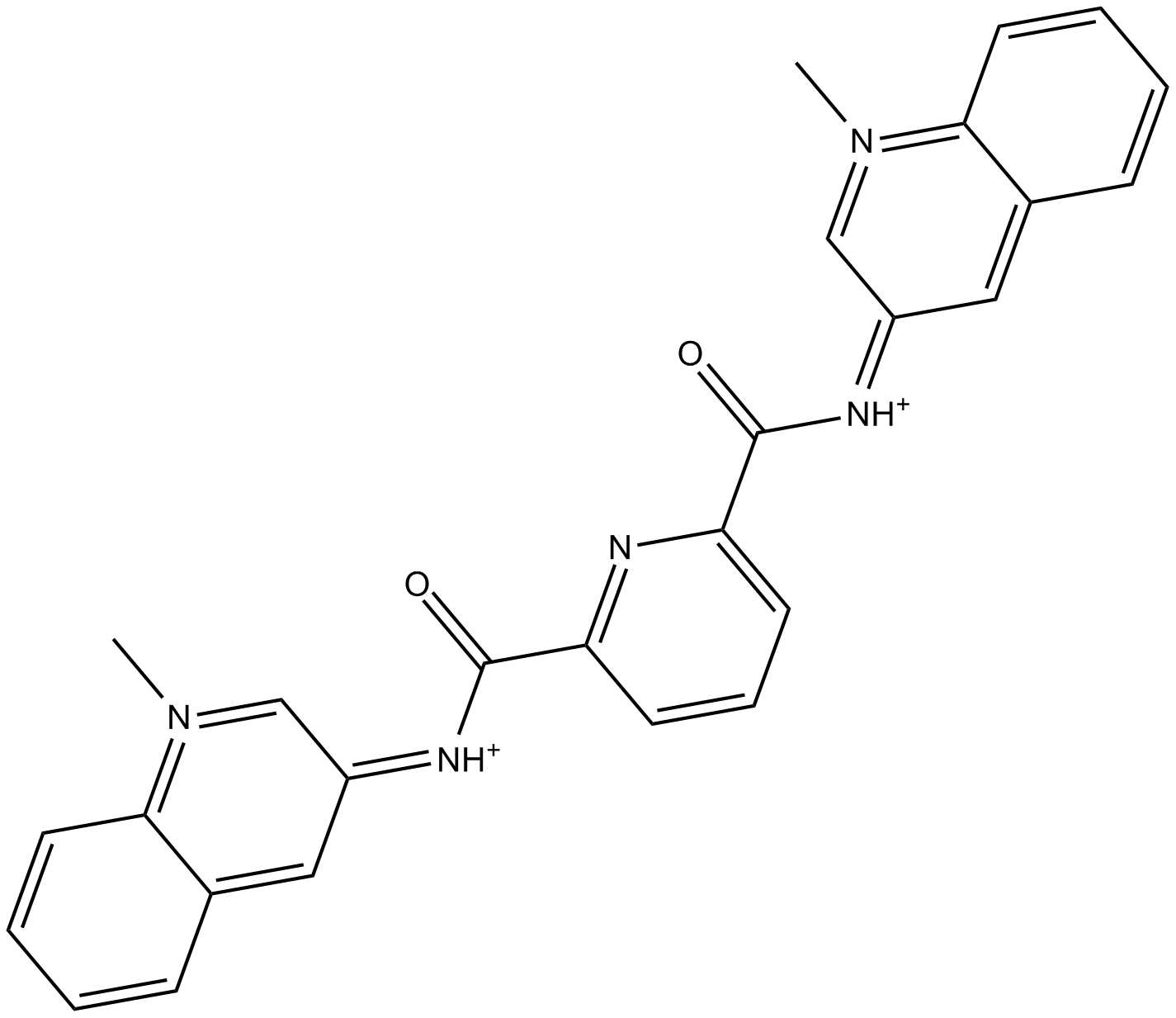
-
GC10115
360A iodide
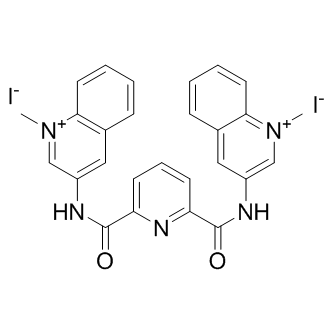
-
GC14493
4μ8C
4u8C
IRE1 Rnase-Inhibitor, potent und ungiftig
-
GC17271
4'-Demethylepipodophyllotoxin
(-)-4′-Demethylepipodophyllotoxin
An inhibitor of tubulin polymerization