Cell Cycle/Checkpoint
Cell Cycle
Cells undergo a complex cycle of growth and division that is referred to as the cell cycle. The cell cycle consists of four phases, G1 (GAP 1), S (synthesis), G2 (GAP 2) and M (mitosis). DNA replication occurs during S phase. When cells stop dividing temporarily or indefinitely, they enter a quiescent state called G0.
Ziele für Cell Cycle/Checkpoint
- ATM/ATR(26)
- Aurora Kinase(47)
- Cdc42(4)
- Cdc7(4)
- Chk(16)
- c-Myc(20)
- CRM1(8)
- Cyclin-Dependent Kinases(91)
- E1 enzyme(1)
- G-quadruplex(14)
- Haspin(7)
- HMTase(1)
- Kinesin(26)
- Ksp(6)
- Microtubule/Tubulin(243)
- Mps1(15)
- Mitotic(11)
- RAD51(18)
- ROCK(71)
- Rho(13)
- PERK(11)
- PLK(37)
- PTEN(8)
- Wee1(7)
- PAK(21)
- Arp2/3 Complex(8)
- Dynamin(12)
- ECM & Adhesion Molecules(40)
- Cholesterol Metabolism(3)
- Endomembrane System & Vesicular Trafficking(26)
- G1(38)
- G2/M(26)
- G2/S(10)
- Genotoxic Stress(18)
- Inositol Phosphates(18)
- Proteolysis(99)
- Cytoskeleton & Motor Proteins(53)
- Cellular Chaperones(8)
Produkte für Cell Cycle/Checkpoint
- Bestell-Nr. Artikelname Informationen
-
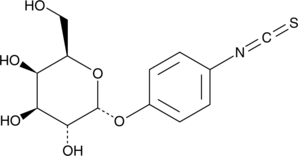
GC40702
α-D-Galactopyranosylphenyl isothiocyanate
α-D-Galactopyranosylphenyl isothiocyanate is a chemically activated form of galactose that has been used to prepare various neoglycoproteins, which consist of a glycosylated serum albumin substituted with either fluorescein or methotrexate.
-
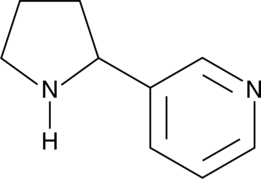
GC41676
(±)-Nornicotine
(±)-Nornicotine is a metabolite of nicotine that acts as a neuronal nicotinic acetylcholine receptor (nAChR) agonist.
-
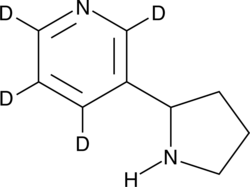
GC49482
(±)-Nornicotine-d4
An internal standard for the quantification of (±)-nornicotine
-
GC41684
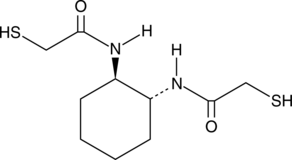
(±)-trans-1,2-Bis(2-mercaptoacetamido)cyclohexane
(±)-trans-1,2-Bis(2-mercaptoacetamido)cyclohexane (BMC) is a cyclohexane with two mercaptoacetamido groups.
-
GC40802
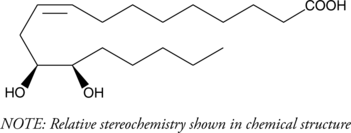
(±)12(13)-DiHOME
(±)12(13)-DiHOME is the diol form of (±)12(13)-EpOME, a cytochrome P450-derived epoxide of linoleic acid also known as isoleukotoxin.
-
GC41656
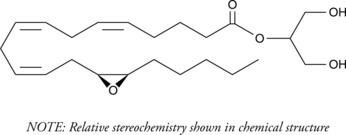
(±)2-(14,15-Epoxyeicosatrienoyl) Glycerol
2-Arachidonoyl glycerol (2-AG) is an endogenous central cannabinoid (CB1) receptor agonist that is present at relatively high levels in the central nervous system.
-
GC45890
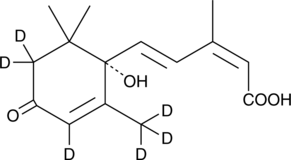
(+)-Abscisic Acid-d6
(+)-Abscisinsäure-d6 (ABA-d6) ist mit Deuterium markierte Abscisinsäure.
-
GC62728
(1E)-CFI-400437 dihydrochloride
(1E)-CFI-400437-Dihydrochlorid ist ein potenter PLK4-Inhibitor (IC50 = 0,6 nM) und selektiv gegenÜber anderen Mitgliedern der PLK-Familie (> 10 μM). (1E)-CFI-400437-Dihydrochlorid hemmt Aurora A, Aurora B, KDR und FLT-3 mit IC50-Werten von 0,37, 0,21, 0,48 bzw. 0,18 μM. Antiproliferative AktivitÄt.
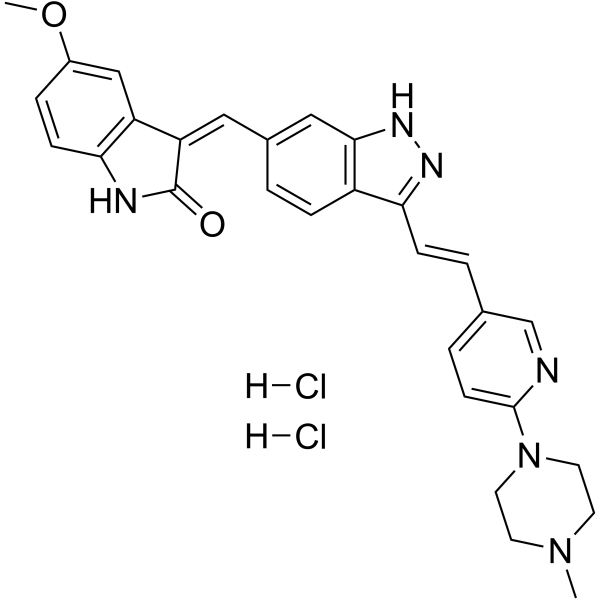
-
GC49690
(3R,5R)-Rosuvastatin (calcium salt)
A potential impurity found in bulk preparations of rosuvastatin
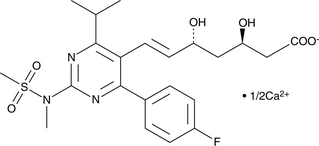
-
GC41268
(E)-2-Hexadecenal
Sphingosine-1-phosphate (S1P), a bioactive lipid involved in many signaling processes, is irreversibly degraded by the membrane-bound S1P lyase.
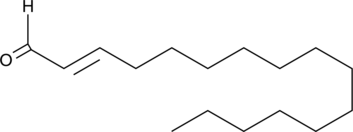
-
GC41701
(E)-2-Hexadecenal Alkyne
(E)-2-Hexadecenal alkyne is an alkyne version of the sphingolipid degradation product (E)-2-hexadecenal that can be used as a click chemistry probe.

-
GC46335
(E)-Fenpyroximate
(E)-Fenpyroximat ist ein starkes Akarizid.
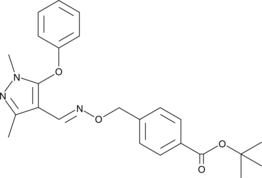
-
GC10419
(R)-CCG-1423
Rho inhibitor

-
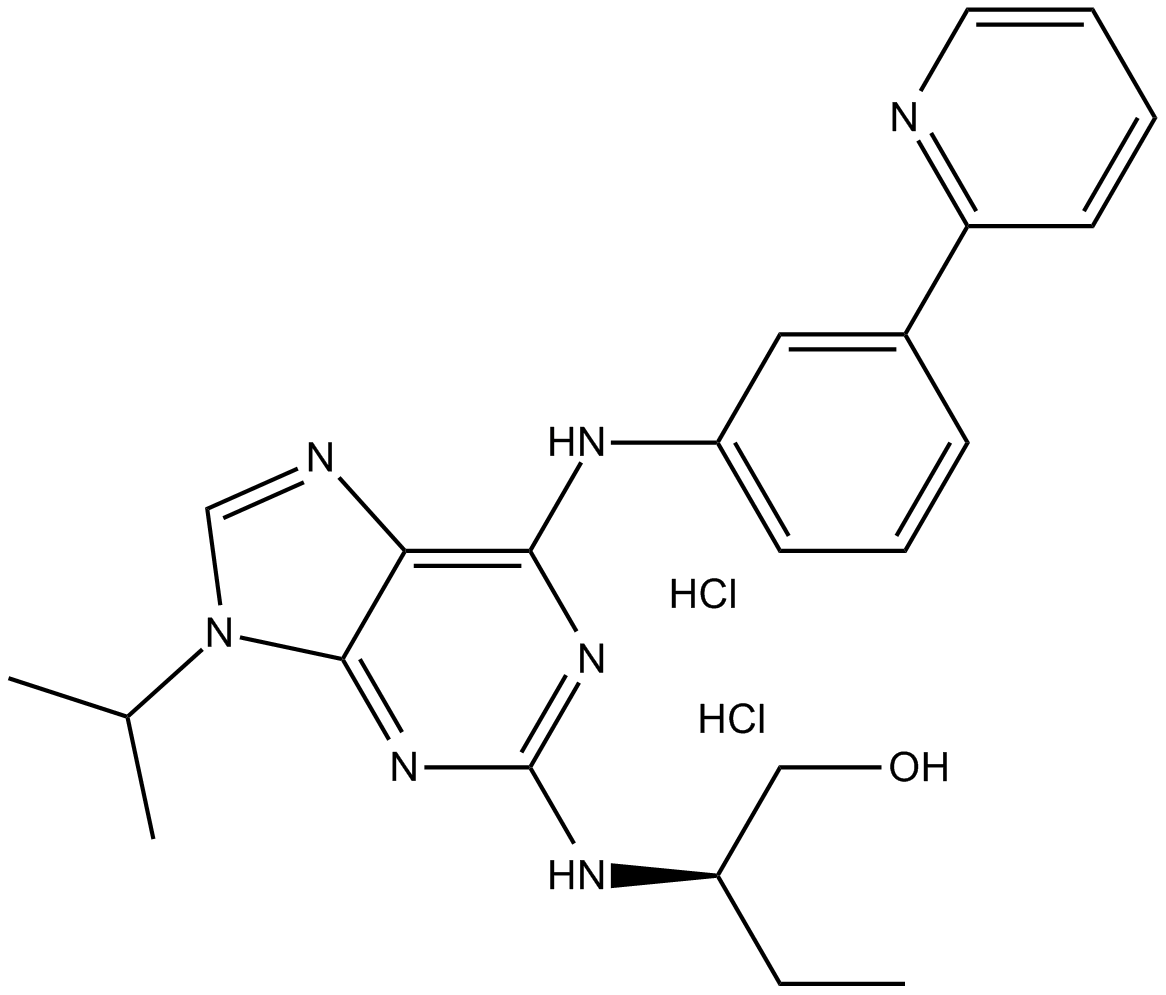
GC16429
(R)-DRF053 dihydrochloride
cdk/CK1 inhibitor,potent and ATP-competitive
-
GC41719
(R)-nitro-Blebbistatin
(R)-nitro-Blebbistatin is a more stable form of (+)-blebbistatin, which is the inactive form of (-)-blebbistatin.
-
GC46347
(S)-(+)-Methoprene
(S)-Methopren ist ein Juvenilhormon-Analogon, das die Fähigkeit des Insekts verhindert, sich von der Puppe zum Erwachsenen zu verändern, und als Insektizid verwendet wird.
-
GC41557
(S)-3'-amino Blebbistatin
(S)-3'-amino Blebbistatin is a more stable and less phototoxic form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
-
GC41484
(S)-3'-hydroxy Blebbistatin
(S)-3'-hydroxy Blebbistatin is a more stable and less phototoxic form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
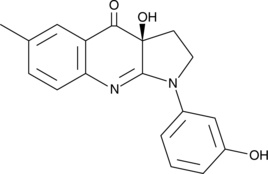
-
GC52192
(S)-4'-nitro-Blebbistatin
(S)-4'-Nitro-Blebbistatin ist ein nicht zytotoxischer, photostabiler, fluoreszierender und spezifischer Myosin-II-Inhibitor, der in der Untersuchung der spezifischen Rolle von Myosin II in physiologischen, entwicklungsbezogenen und zellbiologischen Studien verwendet wurde.
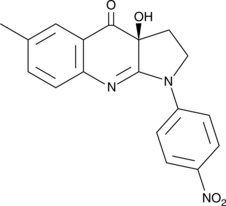
-
GC14497
(S)-CCG-1423
Rho inhibitor
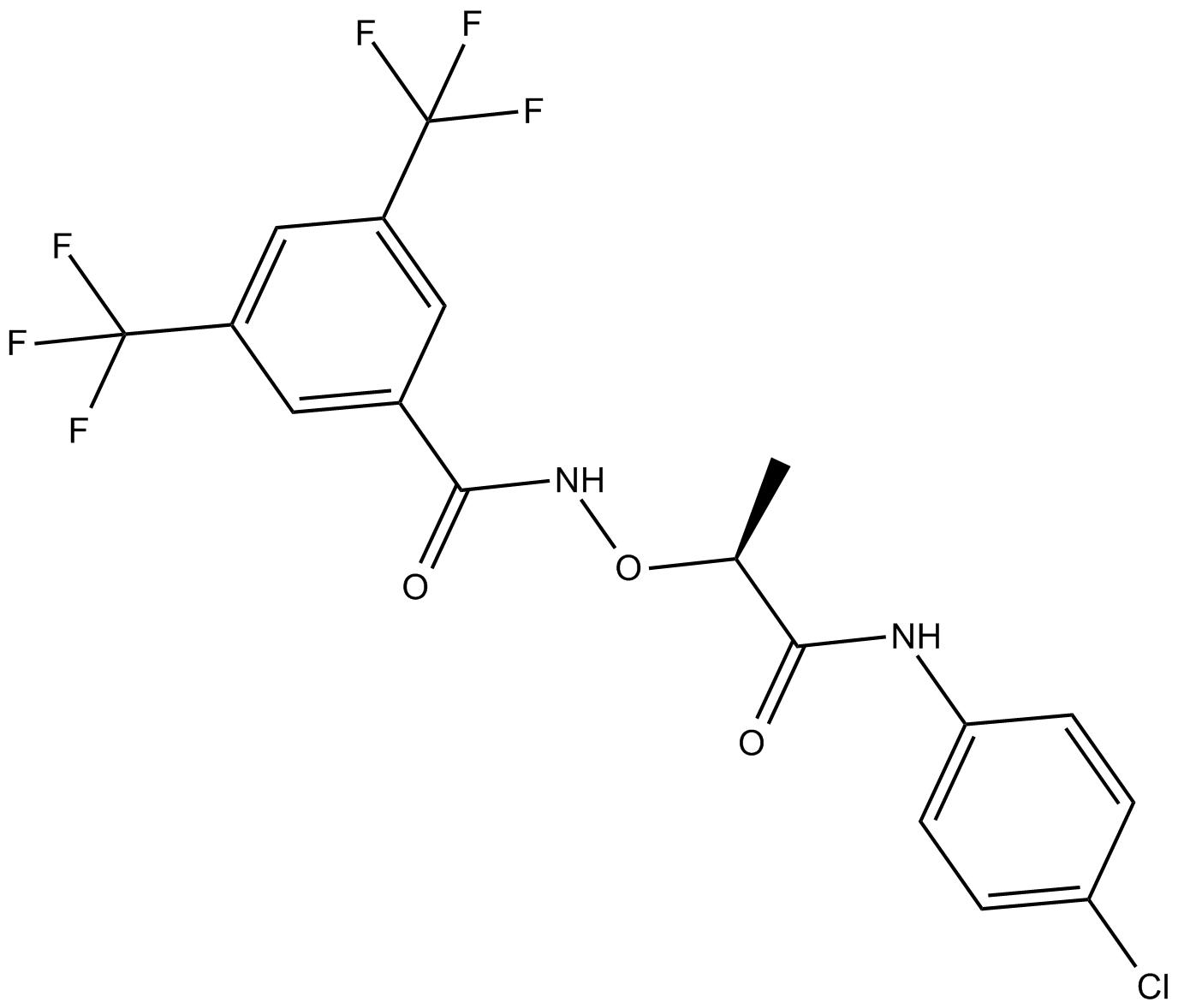
-
GC34999
(S)-Ceralasertib
(S)-Ceralasertib ((S)-AZD6738) wird aus dem Patent WO2011154737A1 extrahiert, Verbindung II, weist einen IC50 von 2,578 nM auf. (S)-Ceralasertib ist ein potenter und selektiver Sulfoximin-Morpholinopyrimidin-ATR-Inhibitor mit ausgezeichneter prÄklinischer physikalisch-chemischer und pharmakokinetischer (PK )-Eigenschaften.(S)-Ceralasertib wurde entwickelt, um die WasserlÖslichkeit zu verbessern und die zeitabhÄngige Hemmung von CYP3A4 zu eliminieren.
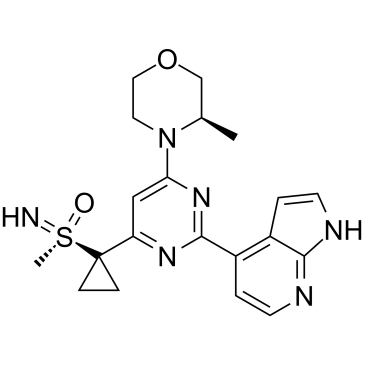
-
GC46351
(S)-CR8
(S)-CR8 ist das S-Isomer von CR8. (S)-CR8 ist ein potenter und selektiver CDK-Inhibitor mit IC50-Werten von 0,060, 0,080, 0,11, 0,12 und 0,15 μM fÜr CDK2/Cyclin E, CDK2/Cyclin A, CDK9/Cyclin T, CDK5/p25 und CDK1/Cyclin B bzw. (S)-CR8 reduziert das Überleben von SH-SY5Y-Zellen (IC50 0,40 μM).
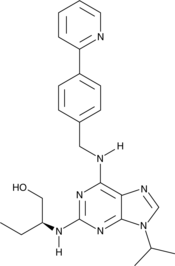
-
GC41737
(S)-Glycyl-H-1152 (hydrochloride)
Two Rho-associated kinases (ROCK), ROCK-I and ROCK-II, act downstream of the G protein Rho to regulate cytoskeletal stability.
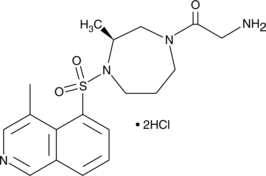
-
GC40145
(S)-Laudanosine
(S)-Laudanosine is the (S) enantiomer of laudanosine, a metabolite of the neuromuscular blocking agents atracurium and cisatracurium.
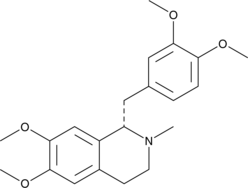
-
GC41739
(S)-nitro-Blebbistatin
(S)-nitro-Blebbistatin is a more stable form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
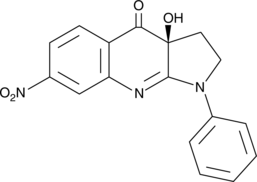
-
GC18275
1,2-Dihexadecyl-sn-glycero-3-PC
1,2-Dihexadecyl-sn-glycero-3-PC is a synthetic ether-linked phospholipid containing hexadecyl groups at the sn-1 and sn-2 positions.
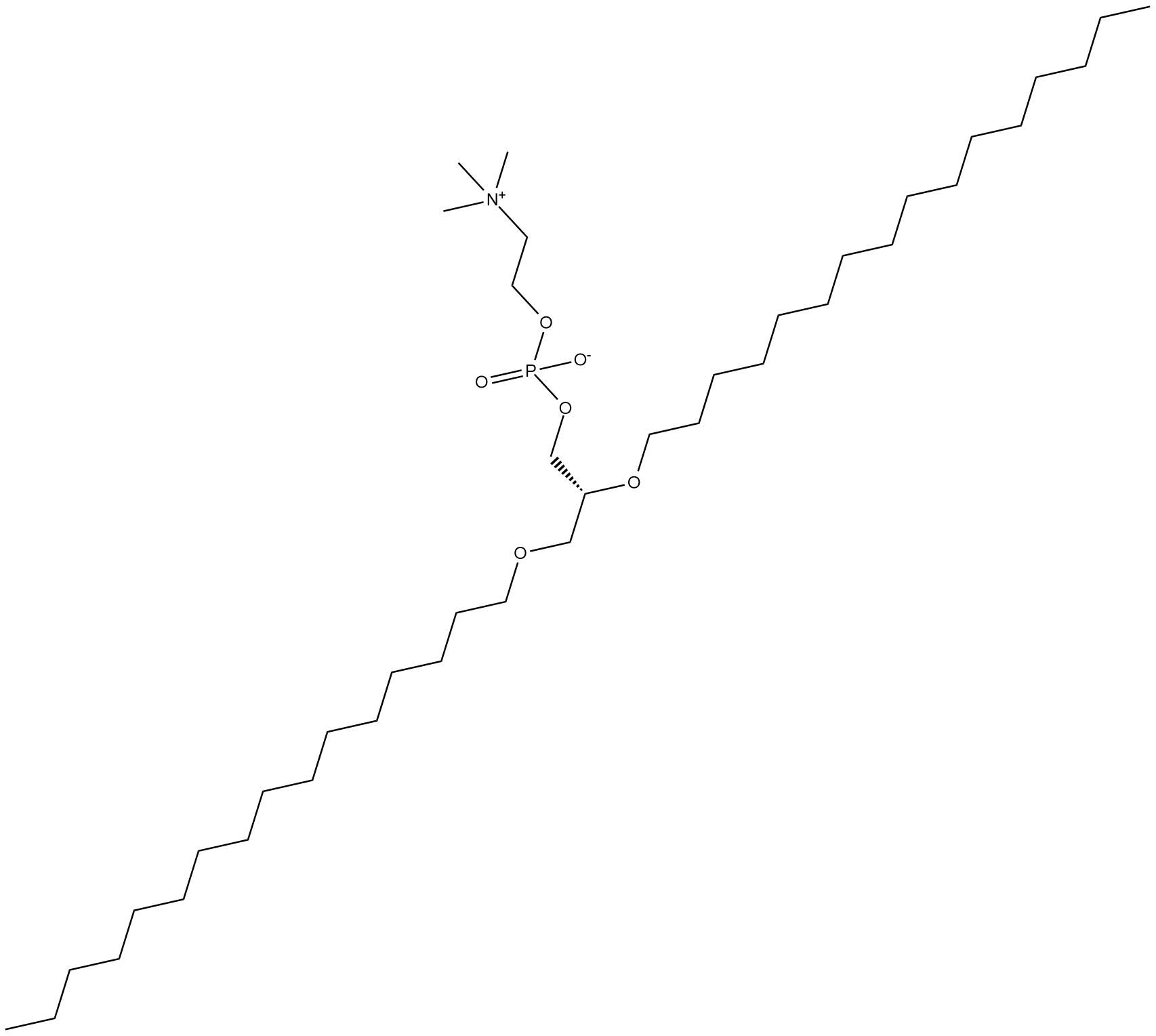
-
GC45783
1,2-Dioleoyl-rac-glycerol-13C3
An internal standard for the quantification of 1,2-dioleoyl-rac-glycerol
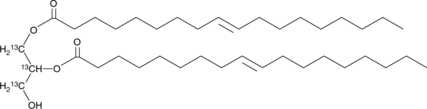
-
GC46379
1,2-Dioleoyl-sn-glycero-3-PS (sodium salt)
1,2-Dioleoyl-sn-glycero-3-PS (Natriumsalz) ist ein Ersatzstoff fÜr Phosphoserin/Phosphatidylserin.
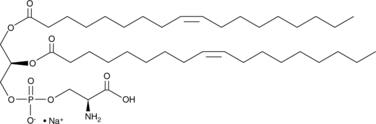
-
GC41823
1,2-Dipalmitoyl-sn-glycero-3-PE-N-(cap biotin) (sodium salt)
1,2-Dipalmitoyl-sn-glycero-3-PE-N-(cap biotin) is a biotinylated phospholipid.
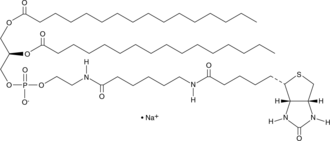
-
GC46385
1,3,4,6-Tetra-O-acetyl-2-azido-2-deoxy-α-D-Mannopyranose
A ManNAc analog and building block

-
GC19528
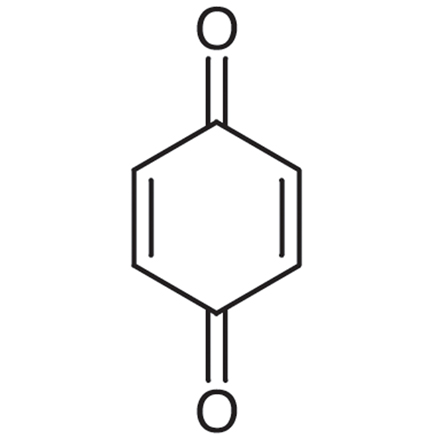
1,4-Benzoquinone
A toxic metabolite of benzene
-
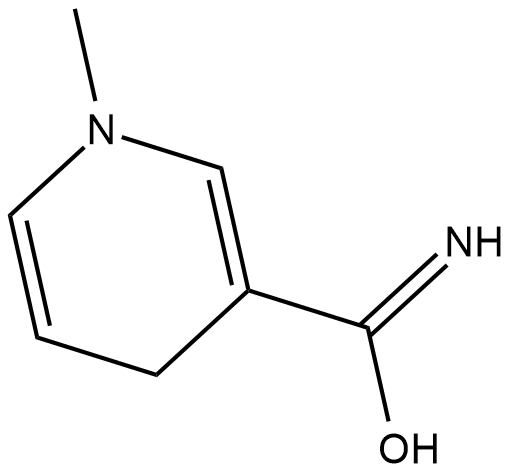
GC18726
1-Methyl-1,4-dihydronicotinamide
1-Methyl-1,4-dihydronicotinamid ist ein Derivat von Nicotinamid.
-
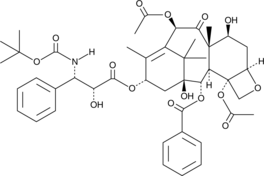
GC49736
10-acetyl Docetaxel
10-Acetyl-Docetaxel (10-Acetyl-Docetaxel) ist ein Analogon von Docetaxel mit AntikrebsaktivitÄt. Docetaxel ist ein Inhibitor der Mikrotubuli-Zerlegung mit antimitotischer AktivitÄt.
-
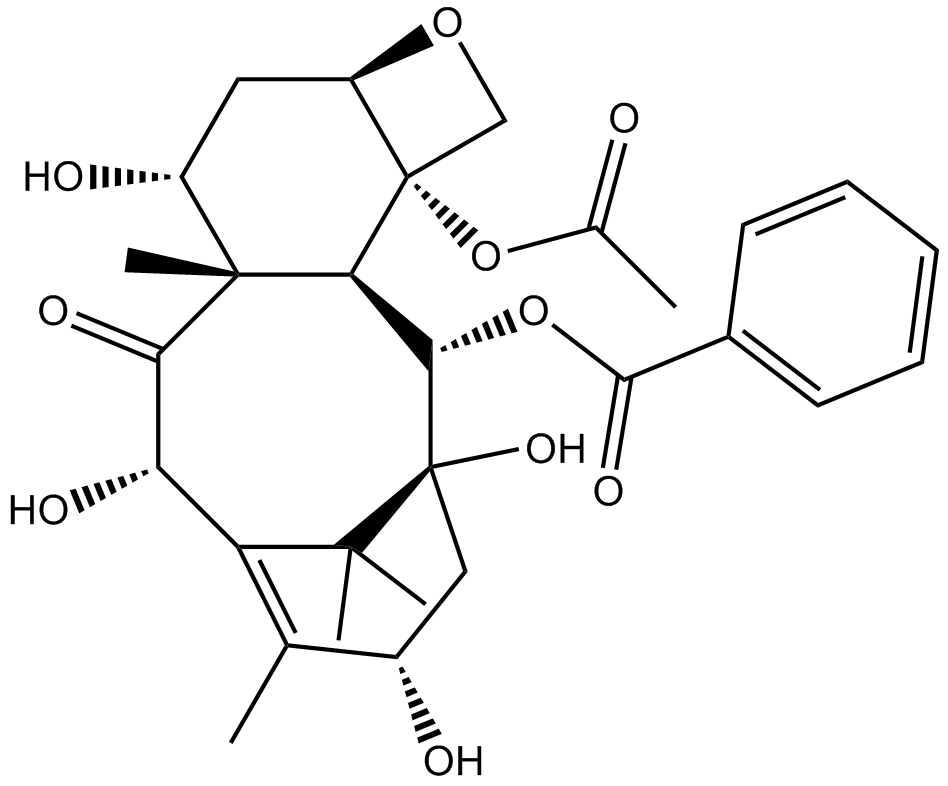
GC12954
10-DAB (10-Deacetylbaccatin)
An inhibitor of microtubule assembly
-
GC35044
10-Deacetyl-7-xylosyl paclitaxel
10-Deacetyl-7-xylosyl-Paclitaxel ist ein Paclitaxel-Derivat (ein Mikrotubulus-Stabilisierungsmittel; verstÄrkt die Tubulin-Polymerisation) mit verbesserten pharmakologischen Eigenschaften.
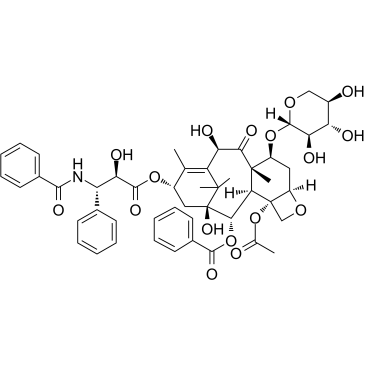
-
GC35045
10-Oxo Docetaxel
10-Oxo-Docetaxel (Docetaxel-Verunreinigung 1) ist ein neuartiges Taxoid mit bemerkenswerten Antitumoreigenschaften und ein Docetaxel-Zwischenprodukt.
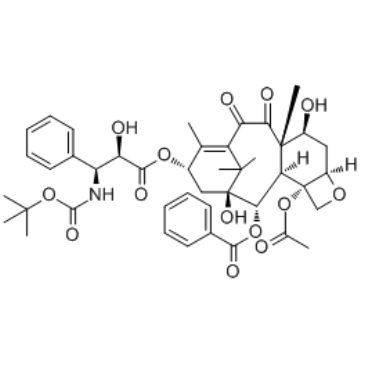
-
GC17295
10058-F4
10058-F4 ist ein c-Myc-Inhibitor, der die c-Myc-Max-Dimerisierung und Transaktivierung der c-Myc-Zielgenexpression verhindert.

-
GC63576
10074-A4
10074-A4 ist ein c-Myc-Inhibitor. 10074-A4 kÖnnte an verschiedenen Stellen entlang der Peptidkette an c-Myc370-409 binden. 10074-A4 hat Antikrebswirkungen.
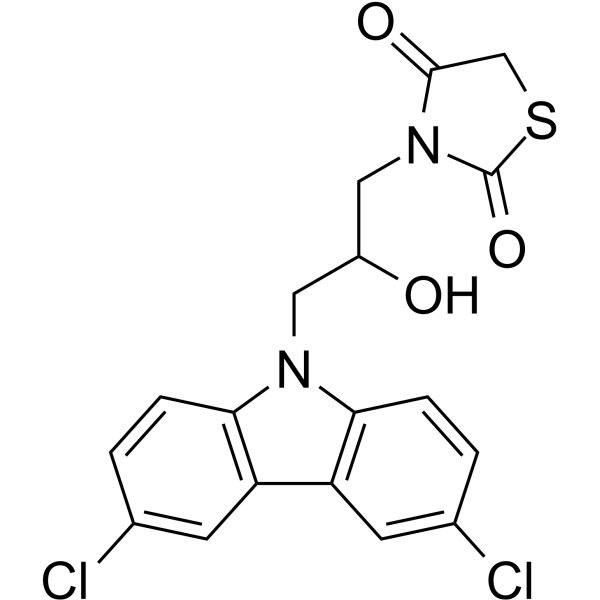
-
GC14918
10074-G5
10074-G5 ist ein Inhibitor der c-Myc-Max-Dimerisierung mit einem IC50 von 146 μM.
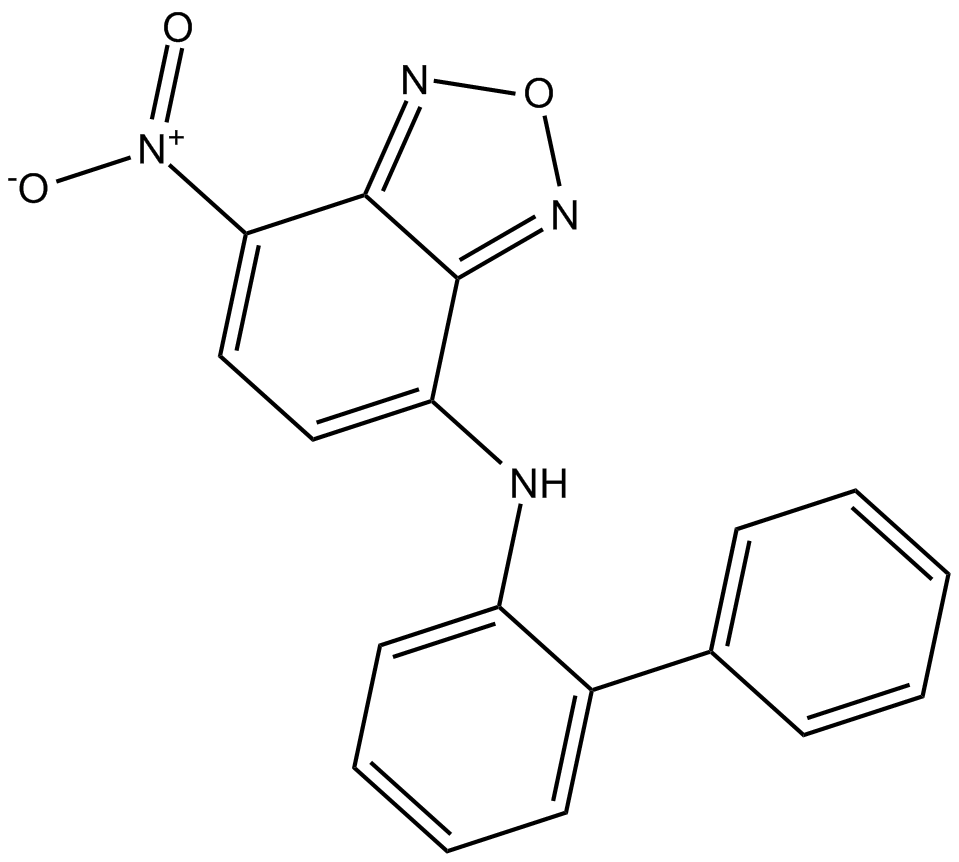
-
GC40448
12(S)-HETE
12(S)-HETE is the predominant lipoxygenase product of mammalian platelets.
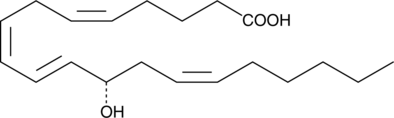
-
GC46434
13C15-Nivalenol
An internal standard for the quantification of nivalenol
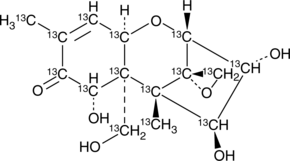
-
GC49390
13C6-4-Nitroaniline
An internal standard for the quantification of 4-nitroaniline
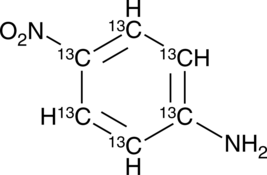
-
GC41110
16-epi Latrunculin B
16-epi Latrunculin B, first isolated from the Red Sea sponge N.
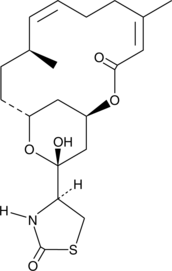
-
GC48423
19-O-Acetylchaetoglobosin A
19-O-Acetylchaetoglobosin A, ein Cytochalasan-Alkaloid, ist ein Pilzmetabolit, der ursprÜnglich aus C. globosum isoliert wurde und eine hemmende und zytotoxische Wirkung auf die Aktinpolymerisation hat. 19-O-Acetylchaetoglobosin A ist zytotoxisch fÜr HeLa-Zervixkrebszellen.
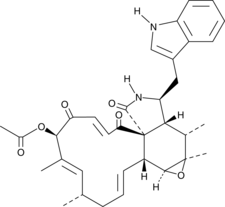
-
GC42151
2'-Deoxyguanosine 5'-monophosphate (sodium salt hydrate)
2'-Deoxyguanosine 5'-monophosphate (dGMP) is used as a substrate of guanylate kinases to generate dGDP, which in turn is phosphorylated to dGTP, a nucleotide precursor used in DNA synthesis.
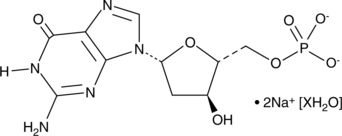
-
GC40947
2,3-Dimethoxy-5-methyl-p-benzoquinone
2,3-Dimethoxy-5-methyl-p-benzochinon (CoQ0) ist eine potente, oral aktive Ubichinonverbindung, die aus Antrodia cinnamomea gewonnen werden kann.
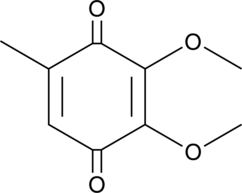
-
GC49671
2,3-Oxidosqualene
An intermediate in the biosynthesis of sterols
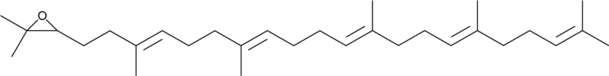
-
GC45324
2,5-dimethyl Celecoxib
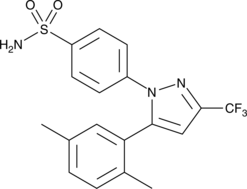
-
GC49362
2-Amino-3,8-dimethylimidazo-[4,5-f]-quinoxaline
A food-derived carcinogen
![2-Amino-3,8-dimethylimidazo-[4,5-f]-quinoxaline Chemical Structure 2-Amino-3,8-dimethylimidazo-[4,5-f]-quinoxaline Chemical Structure](/media/struct/GC4/GC49362.png)
-
GC52029
2-Aminoflubendazole
2-Aminoflubendazol ist der Metabolit von Benzimidazolen.
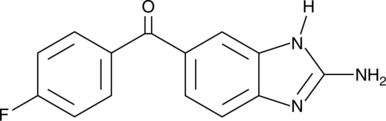
-
GC40675
2-deoxy-Artemisinin
2-deoxy-Artemisinin is an inactive metabolite of the antimalarial agent artemisinin.
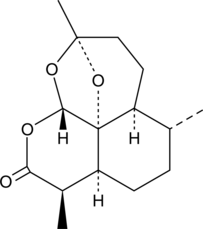
-
GC15084
2-Methoxyestradiol (2-MeOE2)
2-Methoxyestradiol (2-MeOE2/2-Me) is a HIF-1α inhibitor that inhibits HIF-1α accumulation and HIF transcriptional activity. 2-Methoxyestradiol can trigger p53-induced apoptosis and has potential antitumor activity..
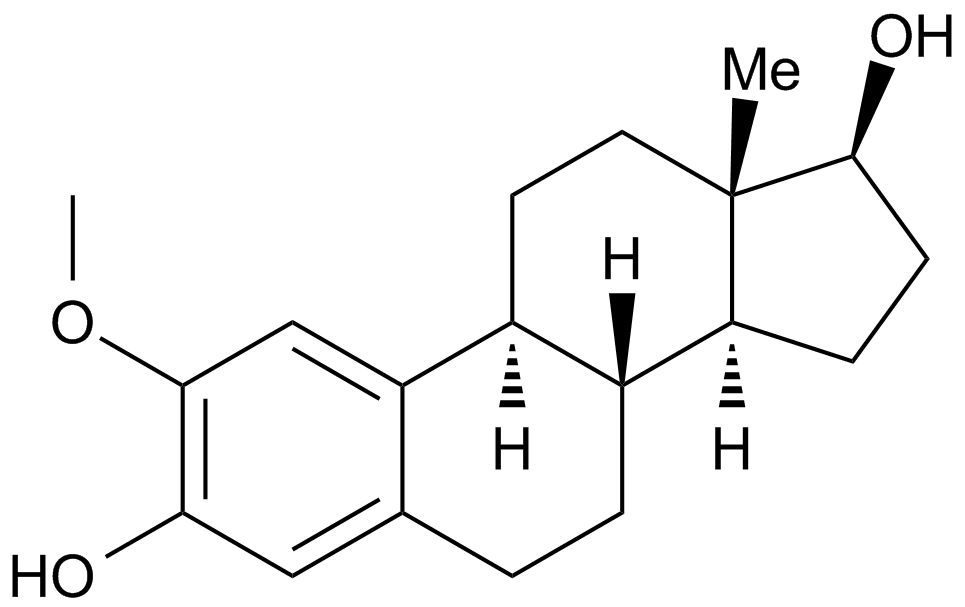
-
GC52140
2-Methoxyhydroquinone
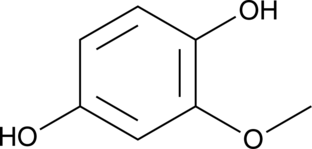
-
GC42161
2H-Cho-Arg (trifluoroacetate salt)
2H-Cho-Arg is a steroid-based cationic lipid that contains a 2H-cholesterol skeleton coupled to an L-arginine head group and can be used to facilitate gene transfection.
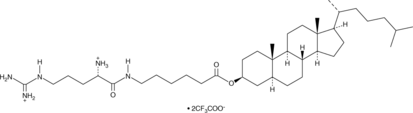
-
GC40618
3',4',7-Trihydroxyisoflavone
3',4',7-Trihydroxyisoflavon, ein Hauptmetabolit von Daidzein, ist ein ATP-kompetitiver Inhibitor von Cot (Tpl2/MAP3K8) und MKK4. 3',4',7-Trihydroxyisoflavon hat krebshemmende, antiangiogene, chemoprotektive und RadikalfÄnger-AktivitÄten.
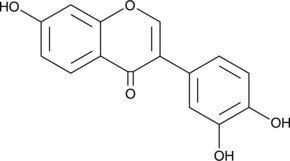
-
GC45332
3'-Dephosphocoenzyme A
An intermediate in the biosynthesis of CoA
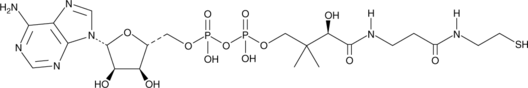
-
GC42239
3,6-diacetoxy Phthalonitrile
3,6-Diacetoxyphthalonitril ist eine zelldurchlÄssige Fluoreszenzsonde.
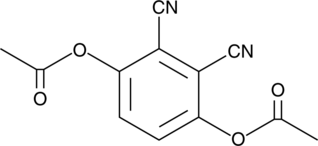
-
GC12314
3-(4-Pyridyl)indole
3-(4-Pyridyl)indol (Rockout) ist ein Rho-Kinase (ROCK)-Hemmer mit einem IC50 von 25 μM.
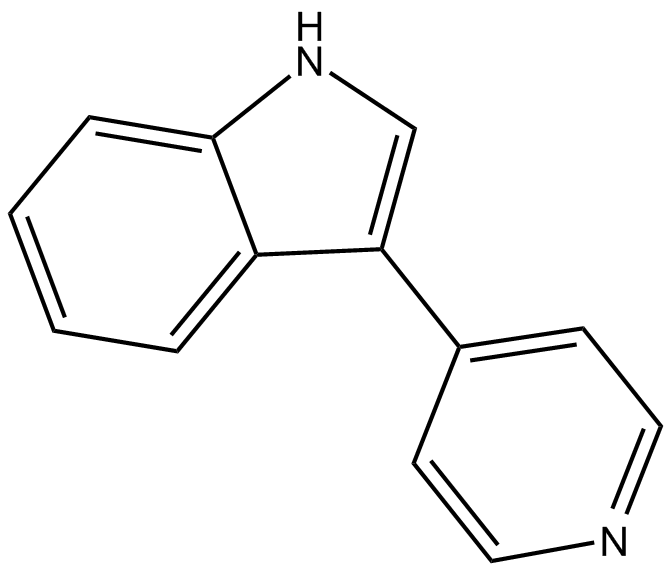
-
GC46582
3-Acetyldeoxy Nivalenol-13C17
An internal standard for the quantification of 3-acetyldeoxy nivalenol
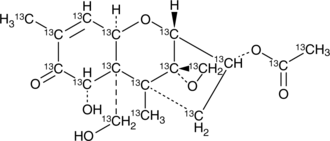
-
GC52129
3-Amino-5-hydroxybenzoic Acid
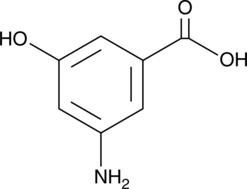
-
GC15389
360A
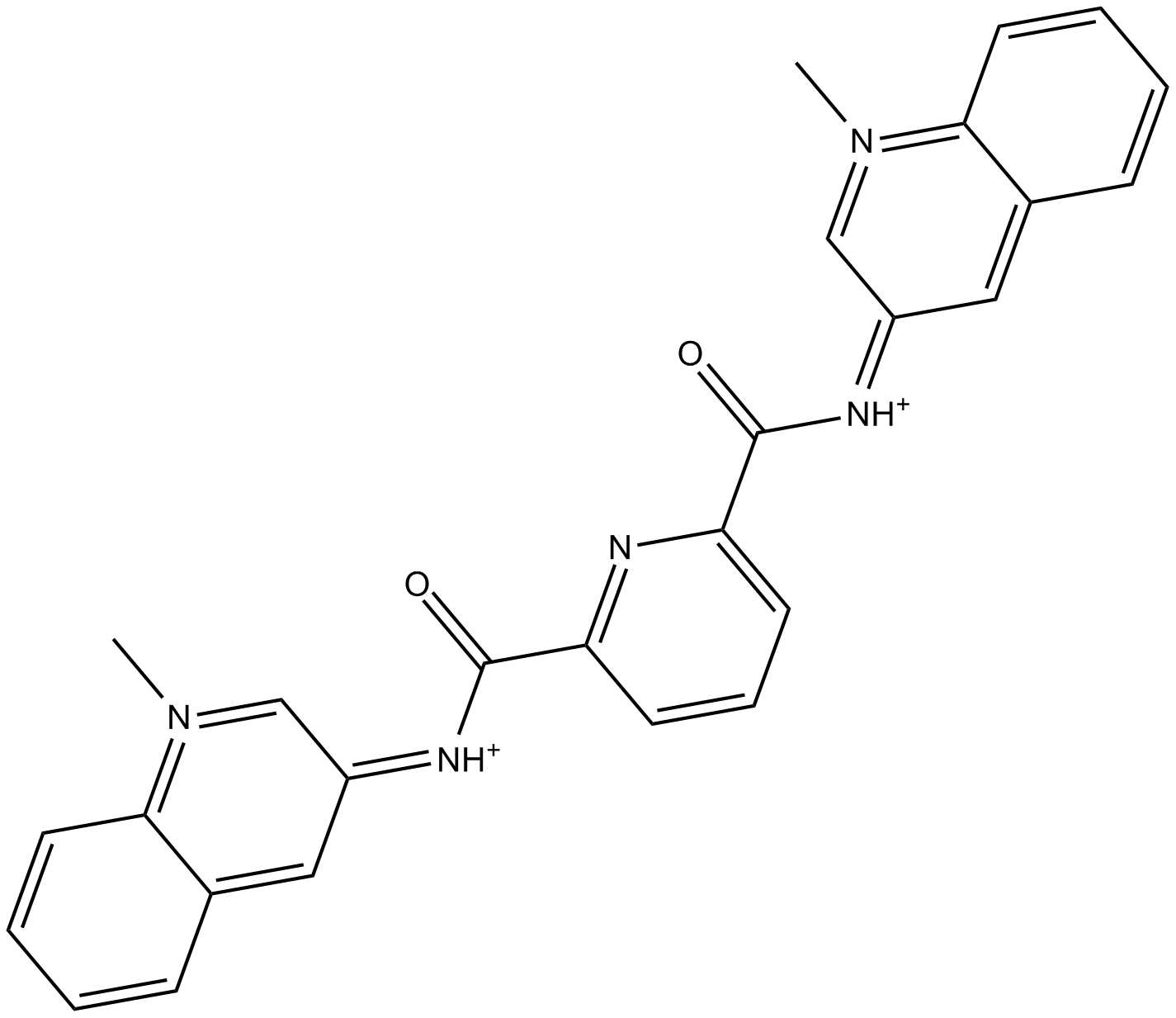
-
GC10115
360A iodide
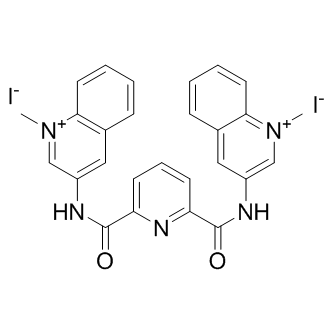
-
GC14186
3MB-PP1
3MB-PP1, ein voluminÖses Purin-Analogon, ist ein Inhibitor der Polo-Ähnlichen Kinase 1 (Plk1).
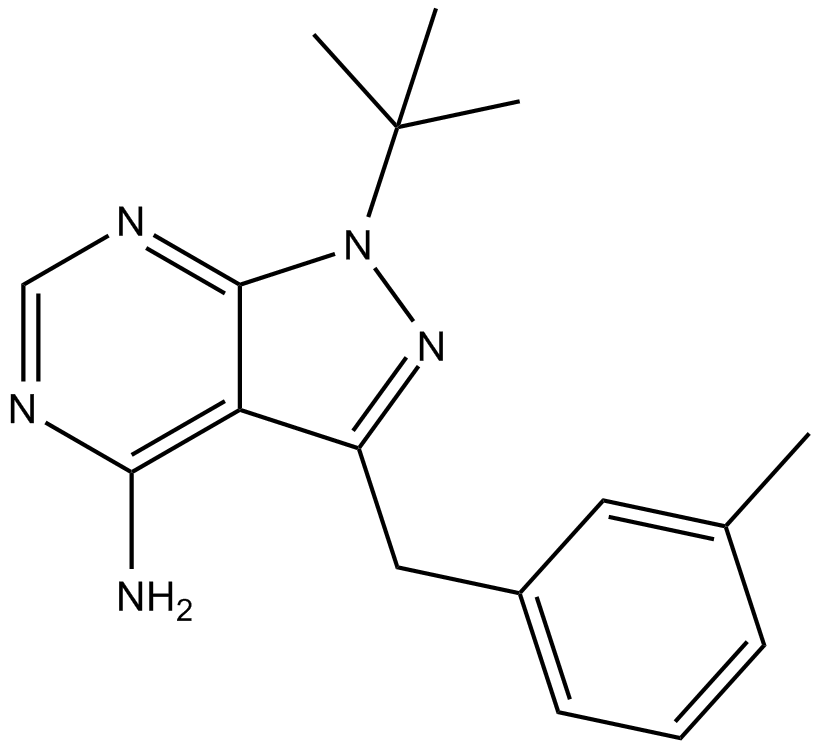
-
GC45354
4β-Hydroxywithanolide E
4β-Hydroxywithanolide E, isoliert aus Physalis peruviana L.
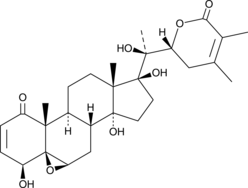
-
GC17271
4'-Demethylepipodophyllotoxin
An inhibitor of tubulin polymerization
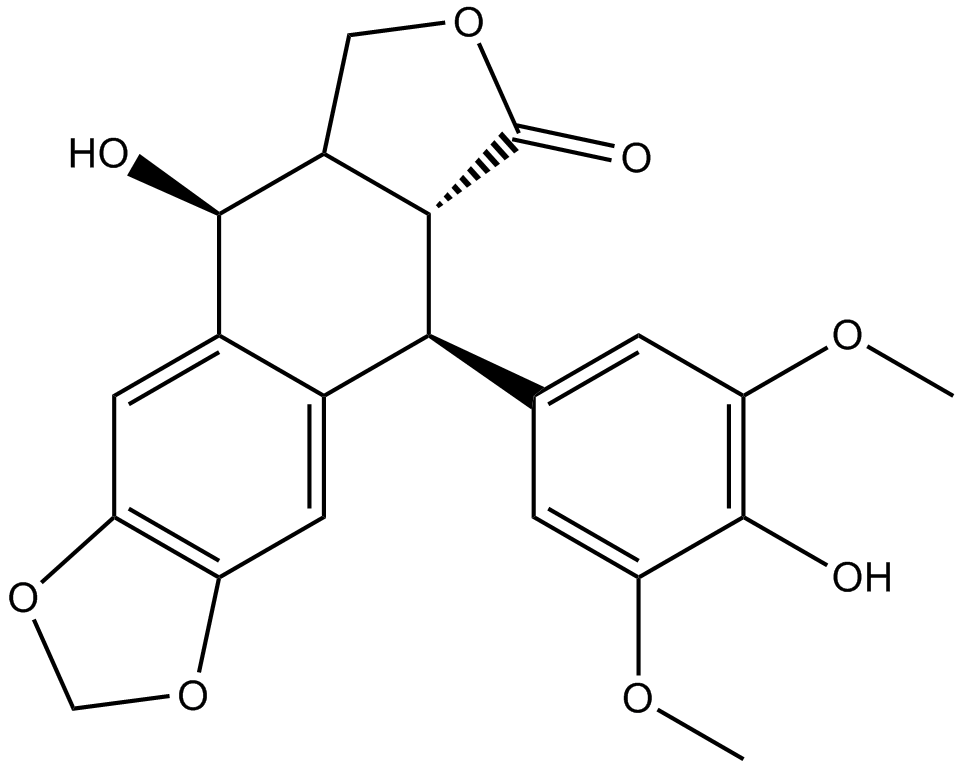
-
GC46606
4-(4,4,5,5-Tetramethyl-1,3,2-dioxaboran-2yl)aniline
A heterocyclic building block
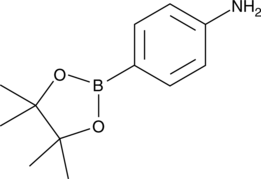
-
GC46635
4-deoxy Nivalenol-13C15
An internal standard for the quantification of 4-deoxy nivalenol
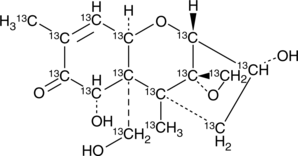
-
GC18359
4-Epianhydrochlortetracycline (hydrochloride)
4-Epianhydrochlortetracycline is a derivative of tetracycline .
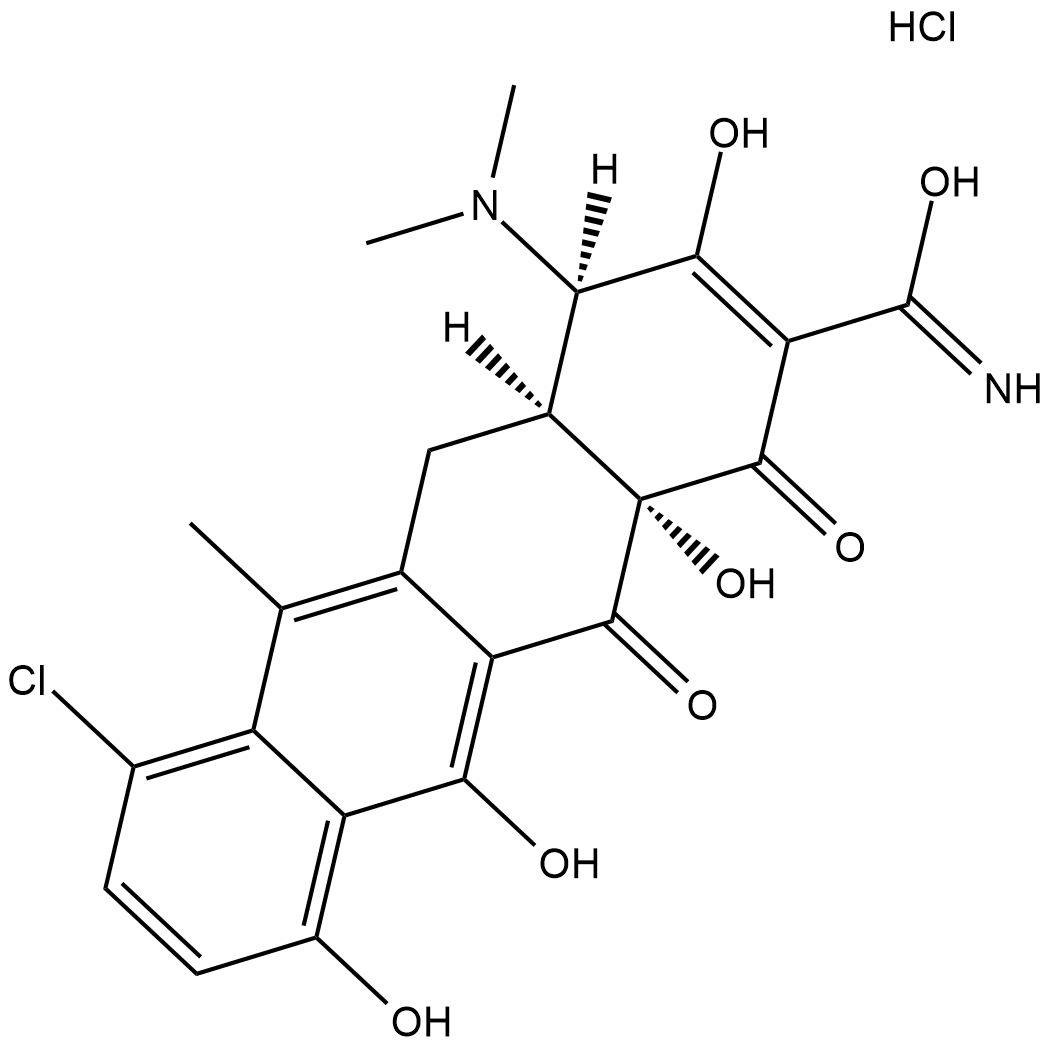
-
GC42449
4-Methylumbelliferyl-α-L-Iduronide (free acid)
4-Methylumbelliferyl-α-L-iduronide (free acid) is a fluorogenic substrate for α-L-iduronidase, an enzyme found in cell lysosomes that is involved in the degradation of glycosaminoglycans such as dermatan sulfate and heparin sulfate.
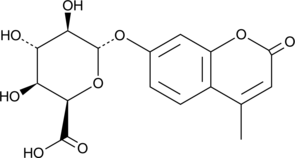
-
GC49244
4-oxo Isotretinoin
An active metabolite of isotretinoin
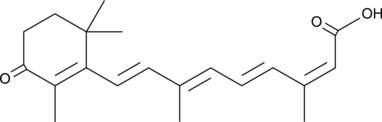
-
GC52365
4-tert-Octylphenol monoethoxylate
An alkylphenolethoxylate and a degradation product of non-ionic surfactants
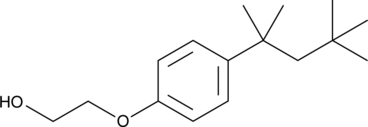
-
GC52227
5-(3',4'-Dihydroxyphenyl)-γ-Valerolactone
An active metabolite of various polyphenols
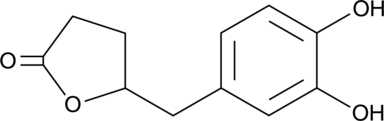
-
GC46681
5-Bromouridine
A brominated uridine analog
-
GC45357
5-Chlorouracil
-
GC41156
5-Octyl D-glutamate
5-Octyl D-glutamate, also known as 5-octyl ester D-glutamate, is a stable, cell-permeable molecule that generates free D-glutamate upon hydrolysis of the ester bond by cytoplasmic esterases.
-
GC41423
5-trans Prostaglandin E2
5-trans PGE2 occurs naturally in some gorgonian corals and is a common impurity in commercial lots of PGE1.
-
GC42586
6α-hydroxy Paclitaxel
6α-Hydroxy Paclitaxel ist ein primÄrer Metabolit von Paclitaxel. 6α-hydroxy Paclitaxel behÄlt eine zeitabhÄngige Wirkung auf die organischen Anionen-transportierenden Polypeptide 1B1/SLCO1B1 (OATP1B1) mit Ähnlicher Hemmkraft wie Paclitaxel, wÄhrend es keine zeitabhÄngige Hemmung von OATP1B3 mehr zeigte. 6α-Hydroxy-Paclitaxel kann fÜr die Krebsforschung verwendet werden.
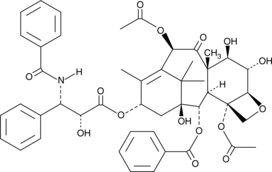
-
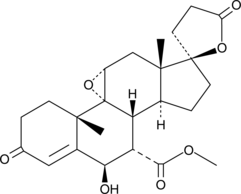
GC45969
6β-hydroxy Eplerenone
A major metabolite of eplerenone
-
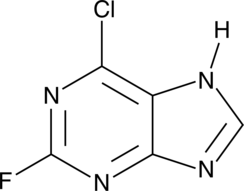
GC46721
6-Chloro-2-fluoropurine
A heterocyclic building block
-
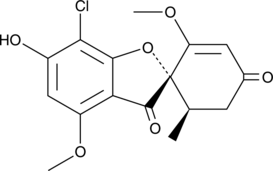
GC48721
6-O-Demethyl Griseofulvin
A metabolite of griseofulvin
-
GC15478
6H05
6H05 ist ein selektiver und allosterischer Inhibitor der onkogenen Mutante K-Ras(G12C).
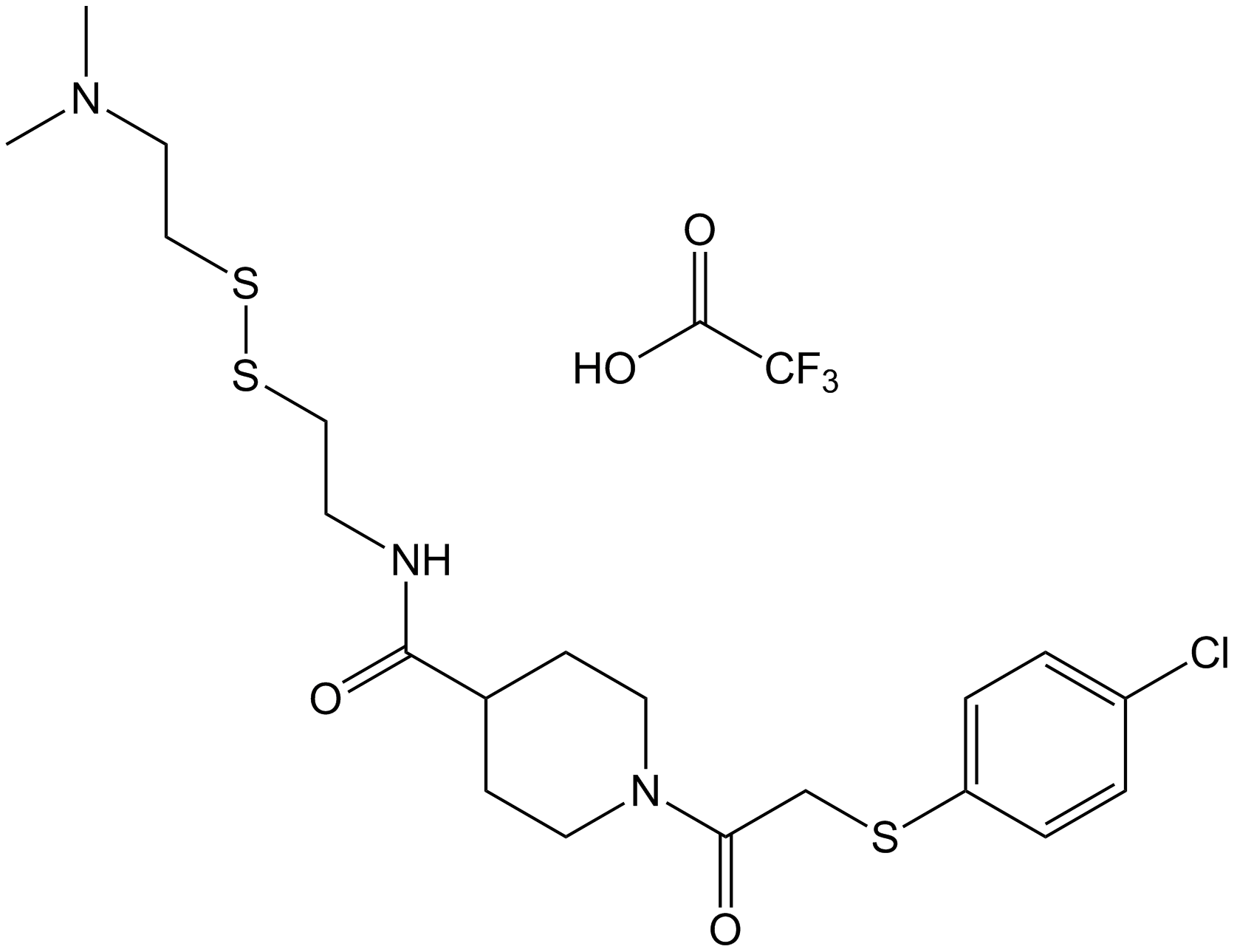
-
GC40202
7α-hydroxy Cholesterol-d7
7α-hydroxy Cholesterol-d7 is intended for use as an internal standard for the quantification of 7α-hydroxy cholesterol by GC- or LC-MS.
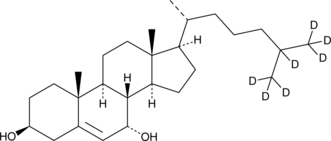
-
GC46733
7,12-Dimethylbenz[a]anthracene
7,12-Dimethylbenz[a]anthracen hat als polyzyklischer aromatischer Kohlenwasserstoff (PAK) krebserzeugende Wirkung. 7,12-Dimethylbenz[a]anthracen wird verwendet, um die Tumorbildung in verschiedenen Nagetiermodellen zu induzieren.
![7,12-Dimethylbenz[a]anthracene Chemical Structure 7,12-Dimethylbenz[a]anthracene Chemical Structure](/media/struct/GC4/GC46733.png)
-
GC35188
7-Epi-10-oxo-docetaxel
7-Epi-10-oxo-Docetaxel (Docetaxel-Verunreinigung 2) ist eine Verunreinigung von Docetaxel, die durch HochleistungsflÜssigkeitschromatographie (HPLC) nachgewiesen wird.
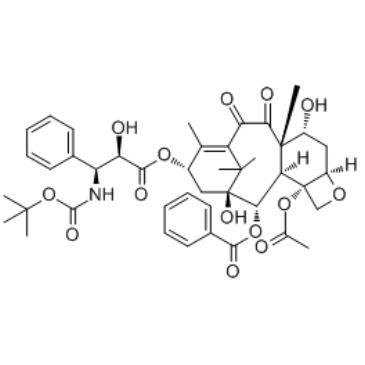
-
GC35189
7-Epi-docetaxel
7-Epi-10-oxo-Docetaxel (Docetaxel-Verunreinigung C; 7-Epitaxotere) ist eine Verunreinigung von Docetaxel.
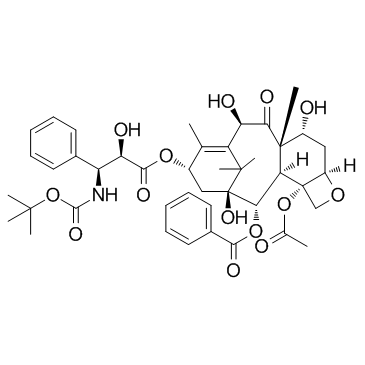
-
GN10625
7-Epitaxol
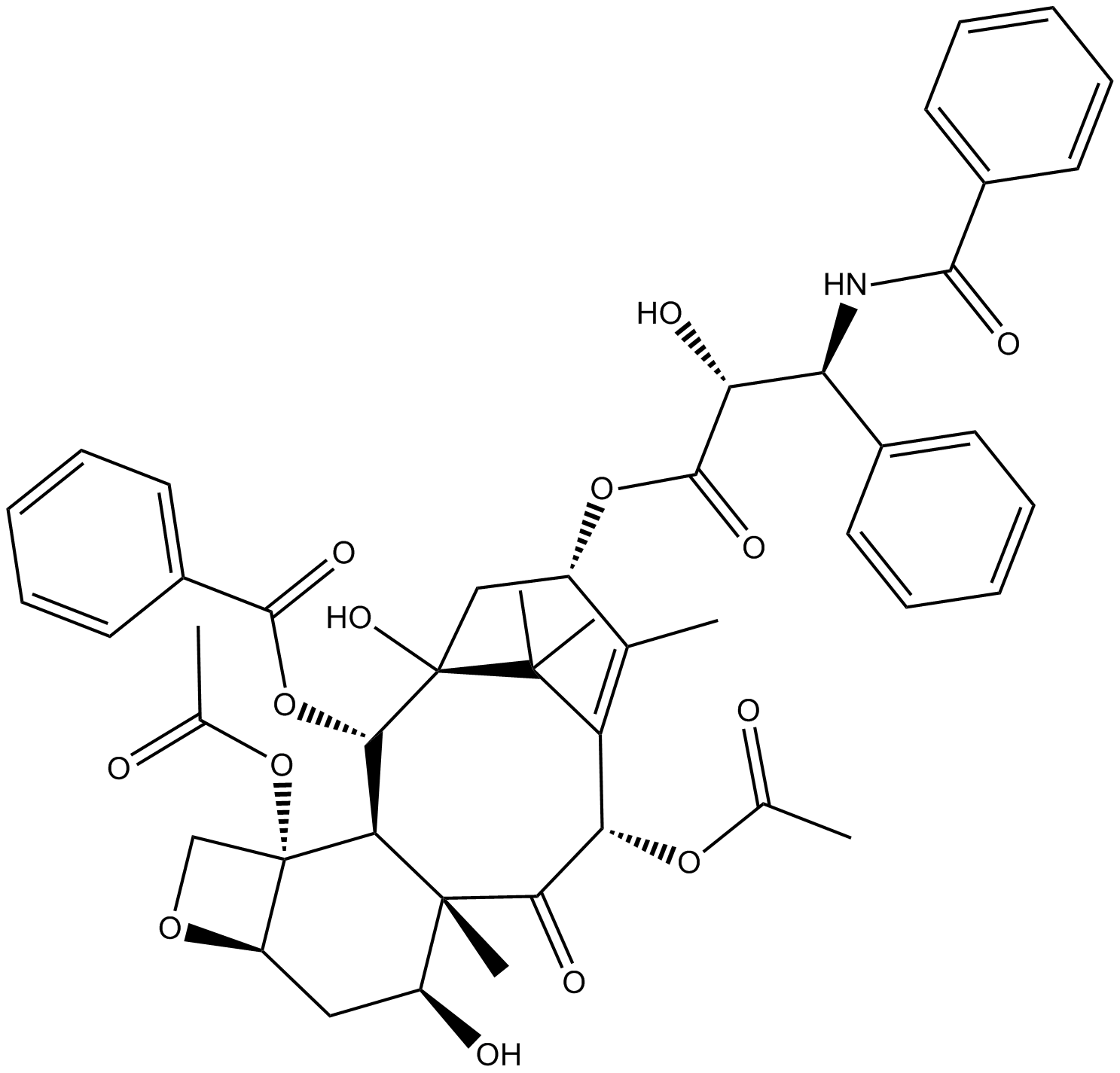
-
GC42610
7-hydroxy Pestalotin
7-Hydroxy-Pestalotin (LL-P880β) ist ein Pilzmetabolit.
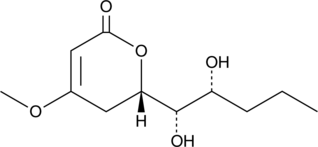
-
GC42616
7-oxo Staurosporine
7-oxo Staurosporine is an antibiotic originally isolated from S.
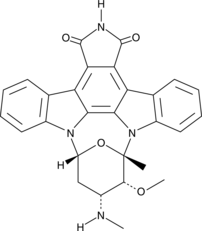
-
GC35197
7-xylosyltaxol
7-Xylosyltaxol (Taxol-7-xylosid) ist ein Taxol (Paclitaxel)-Derivat; Paclitaxel bindet an Tubulin und hemmt den Abbau von Mikrotubuli.

-
GC52126
8-chloro Caffeine
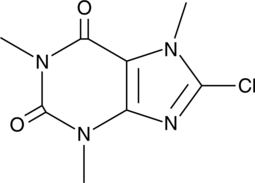
-
GC40844
9-(2,2-Dicyanovinyl)julolidine
9-(2,2-Dicyanovinyl)julolidine (9-(2,2-Dicyanovinyl)julolidine), ein molekularer Rotor und einzigartiger fluoreszierender Farbstoff, bindet an Tubulin und Aktin und erhÖht seine FluoreszenzintensitÄt bei der Polymerisation drastisch.
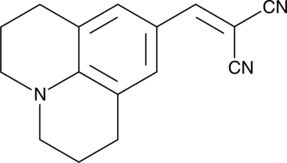
-
GC48986
9-hydroxy Stearic Acid
A hydroxy fatty acid
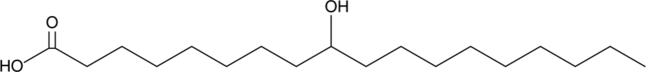
-
GC42665
AAF-CMK (trifluoroacetate salt)
Tripeptidyl peptidase II (TPPII) is a serine peptidase of the subtilisin-type which removes tripeptides from the free NH2 terminus of oligopeptides.

-
GC35216
AAPK-25
AAPK-25 ist ein potenter und selektiver Aurora/PLK-Doppelinhibitor mit Anti-Tumor-AktivitÄt, der eine mitotische VerzÖgerung verursachen und Zellen in einer Prometaphase anhalten kann, was durch die Phosphorylierung des Biomarkers Histon H3Ser10 widergespiegelt wird, gefolgt von einem Anstieg der Apoptose. AAPK-25 zielt auf Aurora-A, -B und -C mit Kd-Werten im Bereich von 23-289 nM sowie PLK-1, -2 und -3 mit Kd-Werten im Bereich von 55-456 nM ab.
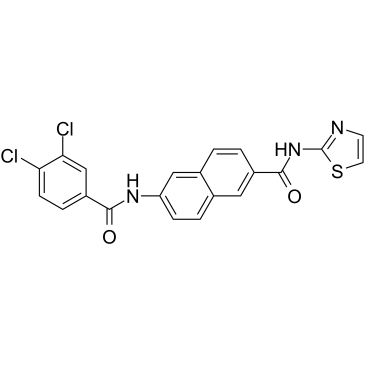
-
GC25025
Abraxane
Abraxane (Nab-Paclitaxel), a novel solvent-free taxane with the binding ratio of Paclitaxel to human serum albumin of 1:9, is an anti-microtubule drug that promotes microtubule aggregation in tubulin dimer and inhibits microtubule depolymerization to stabilize the microtubule system.

-
GC15875
ABT-751 (E7010)
ABT-751 (E7010)(E 7010) ist ein neuartiges bioverfÜgbares Tubulin-bindendes und antimitotisches Sulfonamid mit IC50-Werten von etwa 1,5 und 3,4 μM in Neuroblastom- bzw. Nicht-Neuroblastom-Zelllinien.
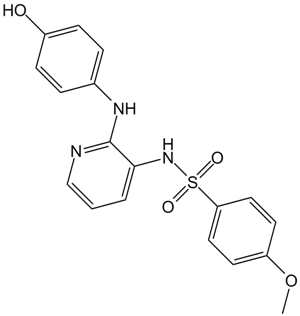
-
GC42683
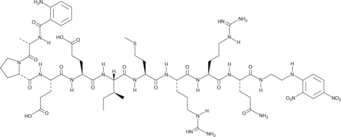
Abz-Ala-Pro-Glu-Glu-Ile-Met-Arg-Arg-Gln-EDDnp
Abz-Ala-Pro-Glu-Glu-Ile-Met-Arg-Arg-Gln-EDDnp is a fluorescence-quenched peptide substrate for human neutrophil elastase (kcat/Km = 531 mM-1s-1).
-
GC52499
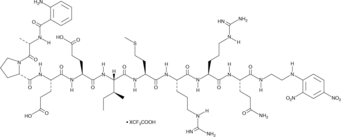
Abz-Ala-Pro-Glu-Glu-Ile-Met-Arg-Arg-Gln-EDDnp (trifluoroacetate salt)
A sensitive substrate for neutrophil elastase
-
GC42684
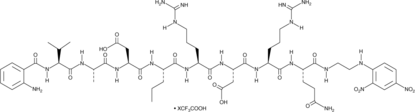
Abz-Val-Ala-Asp-Nva-Arg-Asp-Arg-Gln-EDDnp (trifluoroacetate salt)
Abz-Val-Ala-Asp-Nva-Arg-Asp-Arg-Gln-EDDnp is a fluorescence-quenched peptide substrate for human proteinase 3 (kcat/Km = 1,570 mM-1s-1).
-
GA20494
Ac-Asp-Glu-Val-Asp-pNA
The cleavage of the chromogenic caspase-3 substrate Ac-DEVD-pNA can be monitored at 405 nm.



