Apoptosis
As one of the cellular death mechanisms, apoptosis, also known as programmed cell death, can be defined as the process of a proper death of any cell under certain or necessary conditions. Apoptosis is controlled by the interactions between several molecules and responsible for the elimination of unwanted cells from the body.
Many biochemical events and a series of morphological changes occur at the early stage and increasingly continue till the end of apoptosis process. Morphological event cascade including cytoplasmic filament aggregation, nuclear condensation, cellular fragmentation, and plasma membrane blebbing finally results in the formation of apoptotic bodies. Several biochemical changes such as protein modifications/degradations, DNA and chromatin deteriorations, and synthesis of cell surface markers form morphological process during apoptosis.
Apoptosis can be stimulated by two different pathways: (1) intrinsic pathway (or mitochondria pathway) that mainly occurs via release of cytochrome c from the mitochondria and (2) extrinsic pathway when Fas death receptor is activated by a signal coming from the outside of the cell.
Different gene families such as caspases, inhibitor of apoptosis proteins, B cell lymphoma (Bcl)-2 family, tumor necrosis factor (TNF) receptor gene superfamily, or p53 gene are involved and/or collaborate in the process of apoptosis.
Caspase family comprises conserved cysteine aspartic-specific proteases, and members of caspase family are considerably crucial in the regulation of apoptosis. There are 14 different caspases in mammals, and they are basically classified as the initiators including caspase-2, -8, -9, and -10; and the effectors including caspase-3, -6, -7, and -14; and also the cytokine activators including caspase-1, -4, -5, -11, -12, and -13. In vertebrates, caspase-dependent apoptosis occurs through two main interconnected pathways which are intrinsic and extrinsic pathways. The intrinsic or mitochondrial apoptosis pathway can be activated through various cellular stresses that lead to cytochrome c release from the mitochondria and the formation of the apoptosome, comprised of APAF1, cytochrome c, ATP, and caspase-9, resulting in the activation of caspase-9. Active caspase-9 then initiates apoptosis by cleaving and thereby activating executioner caspases. The extrinsic apoptosis pathway is activated through the binding of a ligand to a death receptor, which in turn leads, with the help of the adapter proteins (FADD/TRADD), to recruitment, dimerization, and activation of caspase-8 (or 10). Active caspase-8 (or 10) then either initiates apoptosis directly by cleaving and thereby activating executioner caspase (-3, -6, -7), or activates the intrinsic apoptotic pathway through cleavage of BID to induce efficient cell death. In a heat shock-induced death, caspase-2 induces apoptosis via cleavage of Bid.
Bcl-2 family members are divided into three subfamilies including (i) pro-survival subfamily members (Bcl-2, Bcl-xl, Bcl-W, MCL1, and BFL1/A1), (ii) BH3-only subfamily members (Bad, Bim, Noxa, and Puma9), and (iii) pro-apoptotic mediator subfamily members (Bax and Bak). Following activation of the intrinsic pathway by cellular stress, pro‑apoptotic BCL‑2 homology 3 (BH3)‑only proteins inhibit the anti‑apoptotic proteins Bcl‑2, Bcl-xl, Bcl‑W and MCL1. The subsequent activation and oligomerization of the Bak and Bax result in mitochondrial outer membrane permeabilization (MOMP). This results in the release of cytochrome c and SMAC from the mitochondria. Cytochrome c forms a complex with caspase-9 and APAF1, which leads to the activation of caspase-9. Caspase-9 then activates caspase-3 and caspase-7, resulting in cell death. Inhibition of this process by anti‑apoptotic Bcl‑2 proteins occurs via sequestration of pro‑apoptotic proteins through binding to their BH3 motifs.
One of the most important ways of triggering apoptosis is mediated through death receptors (DRs), which are classified in TNF superfamily. There exist six DRs: DR1 (also called TNFR1); DR2 (also called Fas); DR3, to which VEGI binds; DR4 and DR5, to which TRAIL binds; and DR6, no ligand has yet been identified that binds to DR6. The induction of apoptosis by TNF ligands is initiated by binding to their specific DRs, such as TNFα/TNFR1, FasL /Fas (CD95, DR2), TRAIL (Apo2L)/DR4 (TRAIL-R1) or DR5 (TRAIL-R2). When TNF-α binds to TNFR1, it recruits a protein called TNFR-associated death domain (TRADD) through its death domain (DD). TRADD then recruits a protein called Fas-associated protein with death domain (FADD), which then sequentially activates caspase-8 and caspase-3, and thus apoptosis. Alternatively, TNF-α can activate mitochondria to sequentially release ROS, cytochrome c, and Bax, leading to activation of caspase-9 and caspase-3 and thus apoptosis. Some of the miRNAs can inhibit apoptosis by targeting the death-receptor pathway including miR-21, miR-24, and miR-200c.
p53 has the ability to activate intrinsic and extrinsic pathways of apoptosis by inducing transcription of several proteins like Puma, Bid, Bax, TRAIL-R2, and CD95.
Some inhibitors of apoptosis proteins (IAPs) can inhibit apoptosis indirectly (such as cIAP1/BIRC2, cIAP2/BIRC3) or inhibit caspase directly, such as XIAP/BIRC4 (inhibits caspase-3, -7, -9), and Bruce/BIRC6 (inhibits caspase-3, -6, -7, -8, -9).
Any alterations or abnormalities occurring in apoptotic processes contribute to development of human diseases and malignancies especially cancer.
References:
1.Yağmur Kiraz, Aysun Adan, Melis Kartal Yandim, et al. Major apoptotic mechanisms and genes involved in apoptosis[J]. Tumor Biology, 2016, 37(7):8471.
2.Aggarwal B B, Gupta S C, Kim J H. Historical perspectives on tumor necrosis factor and its superfamily: 25 years later, a golden journey.[J]. Blood, 2012, 119(3):651.
3.Ashkenazi A, Fairbrother W J, Leverson J D, et al. From basic apoptosis discoveries to advanced selective BCL-2 family inhibitors[J]. Nature Reviews Drug Discovery, 2017.
4.McIlwain D R, Berger T, Mak T W. Caspase functions in cell death and disease[J]. Cold Spring Harbor perspectives in biology, 2013, 5(4): a008656.
5.Ola M S, Nawaz M, Ahsan H. Role of Bcl-2 family proteins and caspases in the regulation of apoptosis[J]. Molecular and cellular biochemistry, 2011, 351(1-2): 41-58.
What is Apoptosis? The Apoptotic Pathways and the Caspase Cascade
Ziele für Apoptosis
- Pyroptosis(15)
- Caspase(77)
- 14.3.3 Proteins(3)
- Apoptosis Inducers(71)
- Bax(15)
- Bcl-2 Family(136)
- Bcl-xL(13)
- c-RET(15)
- IAP(32)
- KEAP1-Nrf2(73)
- MDM2(21)
- p53(137)
- PC-PLC(6)
- PKD(8)
- RasGAP (Ras- P21)(2)
- Survivin(8)
- Thymidylate Synthase(12)
- TNF-α(141)
- Other Apoptosis(1144)
- Apoptosis Detection(0)
- Caspase Substrate(0)
- APC(6)
- PD-1/PD-L1 interaction(60)
- ASK1(4)
- PAR4(2)
- RIP kinase(47)
- FKBP(22)
Produkte für Apoptosis
- Bestell-Nr. Artikelname Informationen
-
GC10350
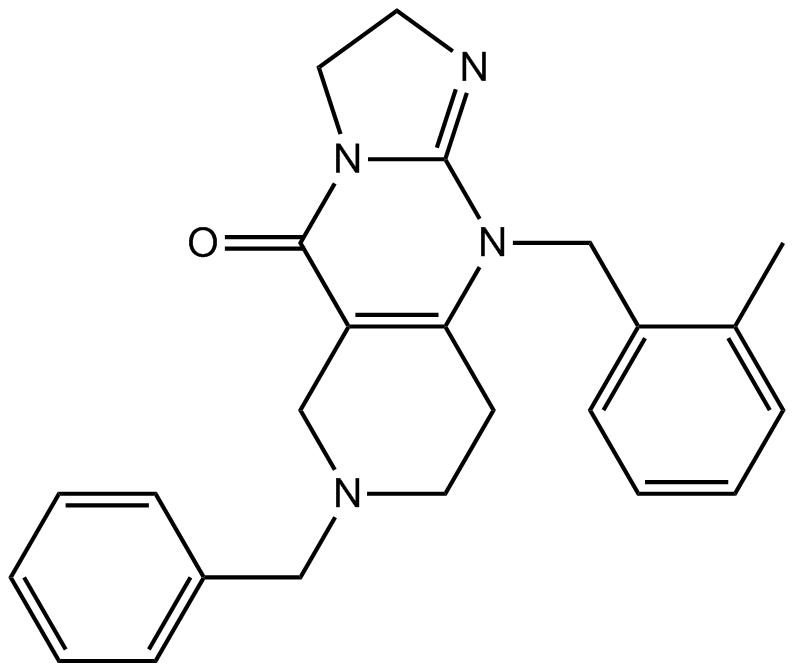
TIC10 isomer
TIC10-Isomer ist das Isomer von TIC10.
-
GC41183
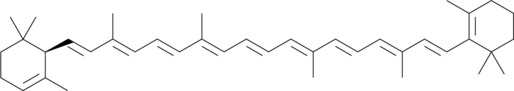
α-Carotene
α-Carotin, eine Vorstufe von Vitamin A, wird als antimetastatisches Mittel oder als Adjuvans fÜr Krebsmedikamente verwendet. α-Carotin wird aus gelb-orangen und dunkelgrÜnen GemÜsen isoliert.
-
GC48292
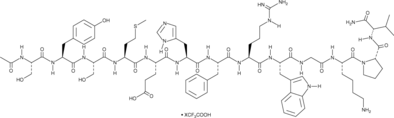
α-MSH (human, mouse, rat, porcine, bovine, ovine) (trifluoroacetate salt)
α-MSH (α-Melanozyten-stimulierendes Hormon) TFA, ein endogenes Neuropeptid, ist ein endogener Melanocortinrezeptor 4 (MC4R)-Agonist mit entzÜndungshemmenden und antipyretischen AktivitÄten.
-
GC45213
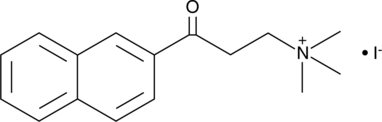
α-NETA
α-NETA ist ein potenter und nicht kompetitiver Hemmer der Cholinacetyltransferase (ChA; IC50\u003d76 μM) und der Cholinesterase (ChE; IC50\u003d40 μM). α-NETA hemmt schwach Acetylcholinesterase (AChE; IC50\u003d1 mM).
-
GC41499
α-Phellandrene
α-Phellandrene is a cyclic monoterpene that has been found in various plants, including Cannabis, and has diverse biological activities.
-
GC63941
α-Solanine
α-Solanin, eine bioaktive Komponente und eines der wichtigsten steroidalen Glykoalkaloide in Kartoffeln, hemmt nachweislich das Wachstum und induziert Apoptose in Krebszellen.
-
GC67618
α-Tocopherol phosphate disodium
α-Tocopherolphosphat (Alpha-Tocopherolphosphat)-Dinatrium, ein vielversprechendes Antioxidans, kann vor langwelligem UVA1-induziertem Zelltod schützen und UVA1-induzierte ROS in einem Hautzellmodell abfangen. α-Tocopherolphosphat-Dinatrium besitzt therapeutisches Potenzial bei der Hemmung der Apoptose und erhöht die Migrationskapazität von endothelialen Vorläuferzellen unter Bedingungen mit hohem Glukosegehalt/Hypoxie und fördert die Angiogenese.
-
GC48920
β-Carboline-1-carboxylic Acid
An alkaloid with diverse biological activities
-
GC41623
β-Elemonic Acid
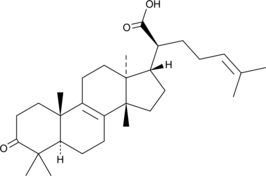
β-Elemonic Acid ist ein aus Boswellia papyrifera isoliertes Triterpen.
-
GC64619
β-Ionone
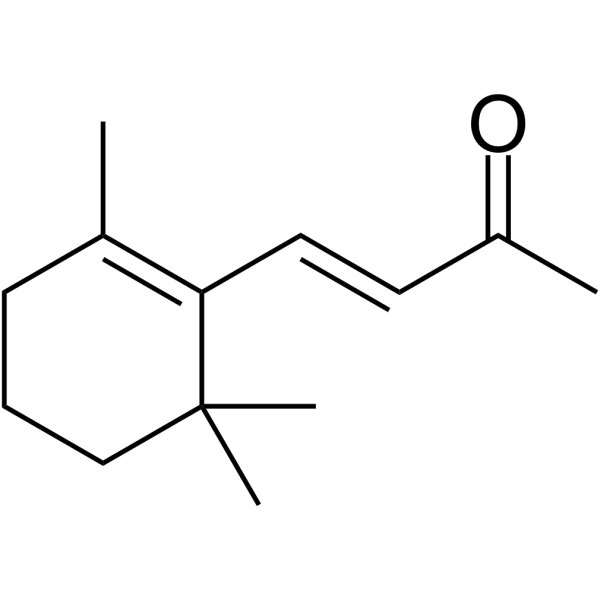
β-Ionone ist wirksam bei der Induktion von Apoptose in Adenokarzinomzellen des Magens SGC7901. Anti-Krebs-AktivitÄt.
-
GC66048
δ-Secretase inhibitor 11
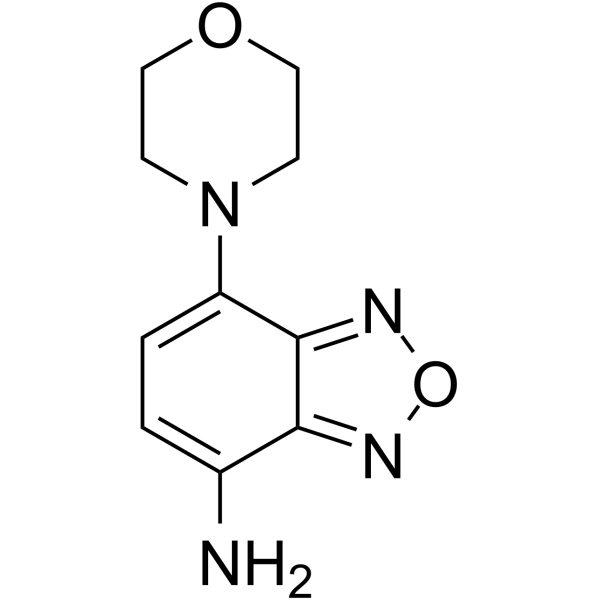
δ-Secretase-Inhibitor 11 (Verbindung 11) ist ein oral aktiver, potenter, BHS-penetrierter, nicht toxischer, selektiver und spezifischer ⋴-Secretase-Inhibitor mit einem IC50 von 0,7 μM. δ-Secretase-Inhibitor 11 interagiert sowohl mit dem aktiven Zentrum als auch mit dem allosterischen Zentrum von δ-Secretase. δ-Secretase Inhibitor 11 schwÄcht die Spaltung von Tau und APP (Amyloid Precursor Protein) ab. δ-Secretase-Inhibitor 11 verbessert synaptische Dysfunktion und kognitive BeeintrÄchtigungen in transgenen Mausmodellen mit Tau P301S und 5XFAD. δ-Secretase Inhibitor 11 kann fÜr die Erforschung der Alzheimer-Krankheit verwendet werden.
-
GC46008
(±)-Thalidomide-d4
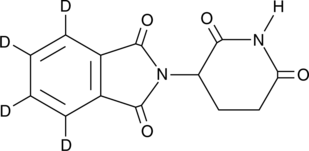
(±)-Thalidomid-d4 ist ein mit Deuterium bezeichnetes Thalidomid.
-
GC45618
(±)-trans-GK563
A GVIA iPLA2 inhibitor
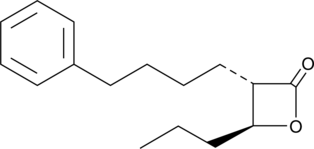
-
GC45270
(±)10(11)-EDP Ethanolamide
(±)10(11)-EDP ethanolamide is an ω-3 endocannabinoid epoxide and cannabinoid (CB) receptor agonist (EC50s = 0.43 and 22.5 nM for CB1 and CB2 receptors, respectively).
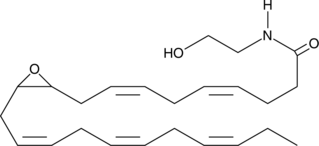
-
GC49268
(+)-δ-Cadinene
A sesquiterpene with antimicrobial and anticancer activities
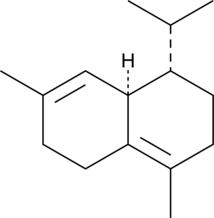
-
GC18516
(+)-Aeroplysinin-1
(+)-Aeroplysinin-1 ((+)-Aeroplysinin-1), ein aus Meeresschwämmen isolierter Sekundärmetabolit, zeigt starke antibiotische Wirkungen auf grampositive Bakterien und übt eine antivirale Aktivität gegen HIV-1 aus (IC50\u003d14,6 μM).
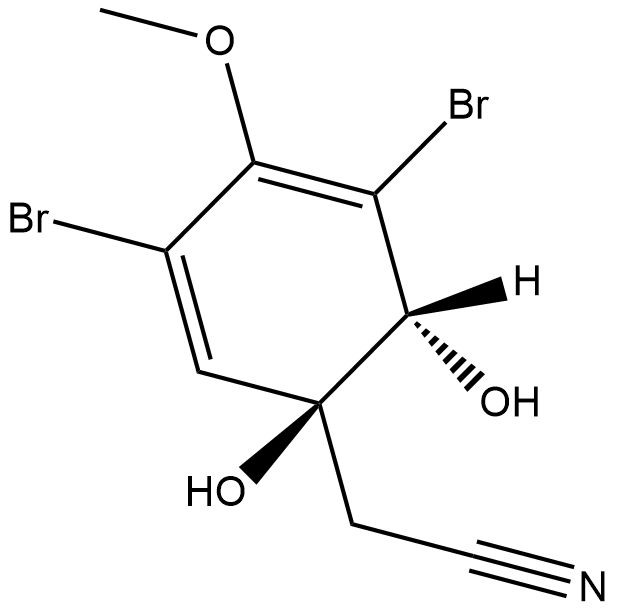
-
GC17008
(+)-Apogossypol
(+)-Apogossypol ist ein Pan-BCL-2-Antagonist. (+)-Apogossypol bindet an Mcl-1, Bcl-2 und Bcl-xL mit EC50-Werten von 2,6, 2,8 bzw. 3,69 μM.
-
GC45256
(+)-ar-Turmerone
(+)-ar-Turmerone ((+)-(+)-ar-Turmerone) ist eine wichtige bioaktive Verbindung des Krauts Curcuma longa mit Anti-Tumorgenese- und entzündungshemmenden Aktivitäten.
-
GN10654
(+)-Corynoline
-
GC31691
(+)-DHMEQ
(+)-DHMEQ ist ein Aktivator des antioxidativen Transkriptionsfaktors Nrf2.
-
GC45265
(+)-Goniothalesdiol
(+)-Goniothalesdiol, isolated from the bark of the Malaysian tree G.

-
GC45274
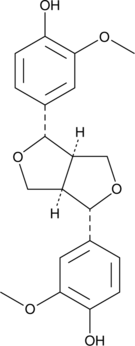
(+)-Pinoresinol
(+)-Pinoresinol ist ein Lignol pflanzlichen Ursprungs, das der Abwehr einer Raupe dient. (+)-Pinoresinol sensibilisiert Krebszellen drastisch gegen TNF-verwandte Apoptose-induzierende Liganden (TRAIL)-induzierte Apoptose.
-
GC18749
(+)-Rugulosin
(+)-Rugulosin ist ein kristalliner Farbstoff von Penicillium rugulosum Thom.
-
GC41345
(-)-α-Bisabolol
(-)-α-Bisabolol ((-)-α-Bisabolol), ein monozyklischer Sesquiterpenalkohol, Übt antioxidative, entzÜndungshemmende und antiapoptotische AktivitÄten aus.
-
GC49502
(-)-β-Sesquiphellandrene
A sesquiterpene with antiviral and anticancer activities
-
GC45244
(-)-(α)-Kainic Acid (hydrate)
Ein potentes Stimulans des zentralen Nervensystems zur Auslösung von Krampfanfällen.

-
GC45246
(-)-Chaetominine
(-)-Chaetominin ist ein Alkaloid-Metabolit. (-)-Chaetominin hat Zytotoxizität gegen humane Leukämie-K562- und Dickdarmkrebs-SW1116-Zelllinien. (-)-Chaetominin reduziert die MRP1-vermittelte Arzneimittelresistenz durch Hemmung des PI3K/Akt/Nrf2-Signalwegs in humanen K562/Adr-Leukämiezellen.
-
GC40698
(-)-Perillyl Alcohol
(-)-Perillylalkohol ist ein in Lavendel vorkommendes Monoterpen, das die Farnesylierung von Ras hemmt, den Mannose-6-Phosphat-Rezeptor hochreguliert und Apoptose induziert. Anti-Krebs-AktivitÄt.
-
GC40076
(-)-Voacangarine
(-)-Voacangarine is an indole alkaloid originally isolated from V.
-
GC62193
(1S,2S)-Bortezomib
(1S,2S)-Bortezomib ist ein Enantiomer von Bortezomib. Bortezomib ist ein zellgÄngiger, reversibler und selektiver Proteasom-Inhibitor und hemmt wirksam das 20S-Proteasom (Ki von 0,6 nM), indem es auf einen Threoninrest abzielt. Bortezomib unterbricht den Zellzyklus, induziert Apoptose und hemmt NF-κB. Bortezomib ist ein Krebsmittel und der erste therapeutische Proteasom-Inhibitor, der beim Menschen eingesetzt wird.
-
GC34965
(20S)-Protopanaxatriol
Ein aktiver Ginsenosid-Metabolit
-
GC60397
(5Z,2E)-CU-3
(5Z,2E)-CU-3 ist ein potenter und selektiver Inhibitor gegen das α-Isozym von DGK mit einem IC50-Wert von 0,6 μM, hemmt kompetitiv die AffinitÄt von DGKα fÜr ATP mit einem Km-Wert von 0,48 mM. (5Z,2E)-CU-3 zielt auf die katalytische Region ab, aber nicht auf die regulatorische Region von DGKα. (5Z,2E)-CU-3 hat antitumorale und proimmunogene Wirkungen, verstÄrkt die Apoptose von Krebszellen und die Aktivierung von T-Zellen.
-
GC60398
(6R)-FR054
(6R)-FR054 ist ein weniger aktives Isomer von FR054.
-
GC50482
(D)-PPA 1
PD-1/PD-L1 interaction inhibitor

-
GA20156
(D-Ser(tBu)⁶,Azagly¹⁰)-LHRH (free base)

-
GC41700
(E)-2-(2-Chlorostyryl)-3,5,6-trimethylpyrazine
(E)-2-(2-Chlorostyryl)-3,5,6-trimethylpyrazine (CSTMP) is a stilbene derivative with antioxidant and anticancer activities.
-
GC41268
(E)-2-Hexadecenal
Sphingosine-1-phosphate (S1P), a bioactive lipid involved in many signaling processes, is irreversibly degraded by the membrane-bound S1P lyase.
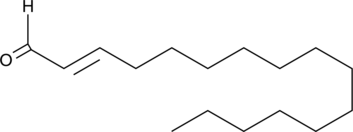
-
GC41701
(E)-2-Hexadecenal Alkyne
(E)-2-Hexadecenal alkyne is an alkyne version of the sphingolipid degradation product (E)-2-hexadecenal that can be used as a click chemistry probe.

-
GC34980
(E)-Ferulic acid
(E)-FerulasÄure ist ein Isomer von FerulasÄure, einer aromatischen Verbindung, die in PflanzenzellwÄnden reichlich vorhanden ist. (E)-FerulasÄure verursacht die Phosphorylierung von β-Catenin, was zu einem proteasomalen Abbau von β-Catenin fÜhrt und die Expression des pro-apoptotischen Faktors Bax erhÖht und die Expression des ÜberlebensfÖrdernden Faktors Survivin verringert. (E)-FerulasÄure zeigt eine starke FÄhigkeit, reaktive Sauerstoffspezies (ROS) zu entfernen und die Lipidperoxidation zu hemmen. (E)-FerulasÄure Übt sowohl Antiproliferations- als auch Antimigrationswirkungen in der menschlichen Lungenkrebszelllinie H1299 aus.
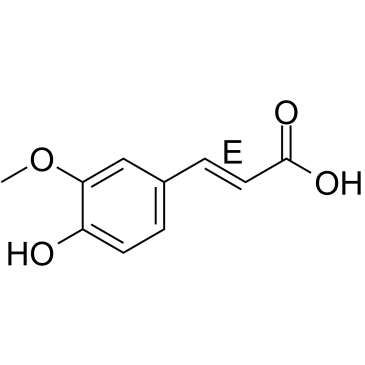
-
GC34981
(E)-Flavokawain A
(E)-Flavokawain A, ein aus Kava extrahiertes Chalcon, hat eine antikarzinogene Wirkung. (E)-Flavokawain A induziert Apoptose in Blasenkrebszellen durch Beteiligung des Bax-Protein-abhÄngigen und Mitochondrien-abhÄngigen apoptotischen Signalwegs und unterdrÜckt das Tumorwachstum in MÄusen.
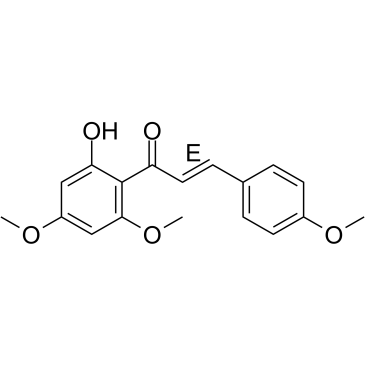
-
GC61437
(E)-Methyl 4-coumarate
(E)-Methyl 4-Coumarat (Methyl 4-hydroxycinnamate), gefunden in mehreren Pflanzen, wie z. B. FrÜhlingszwiebel (Allium cepa) oder Noni (Morinda citrifolia L.
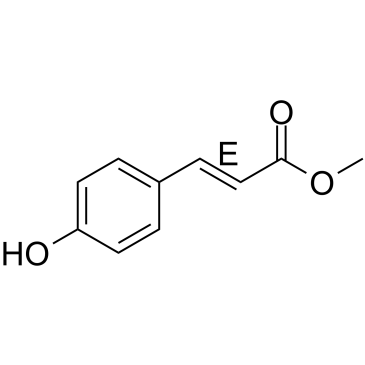
-
GC34125
(E)-[6]-Dehydroparadol
(E)-[6]-Dehydroparadol, ein oxidativer Metabolit von [6]-Shogaol, ist ein starker Nrf2-Aktivator. (E)-[6]-Dehydroparadol kann das Wachstum hemmen und die Apoptose menschlicher Krebszellen induzieren.
![(E)-[6]-Dehydroparadol Chemical Structure (E)-[6]-Dehydroparadol Chemical Structure](/media/struct/GC3/GC34125.png)
-
GC49189
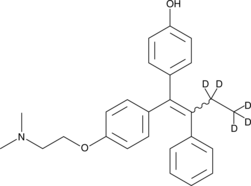
(E/Z)-4-hydroxy Tamoxifen-d5
An internal standard for the quantification of (E/Z)-4-hydroxy tamoxifen
-
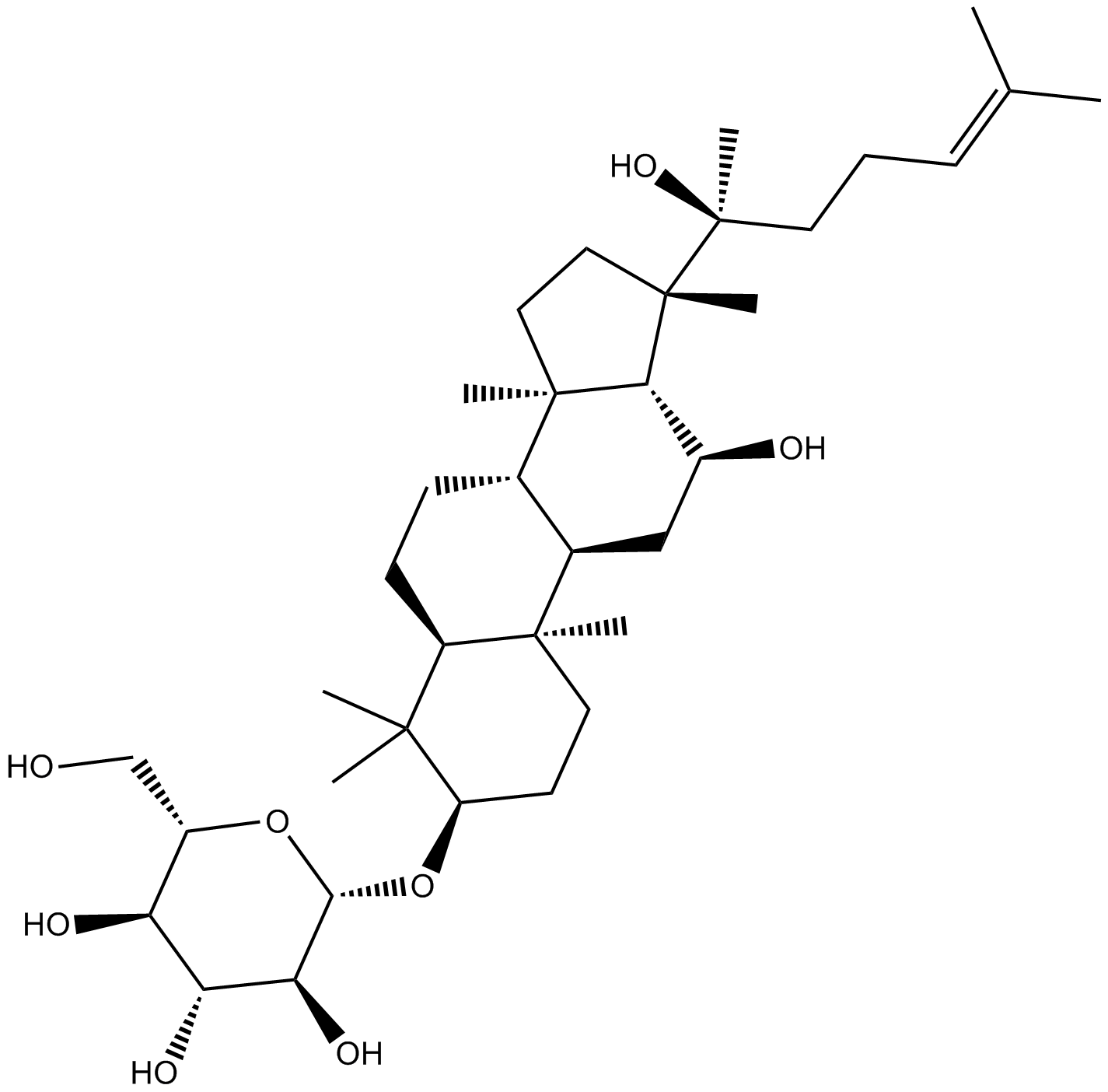
GN10783
(R) Ginsenoside Rh2
-
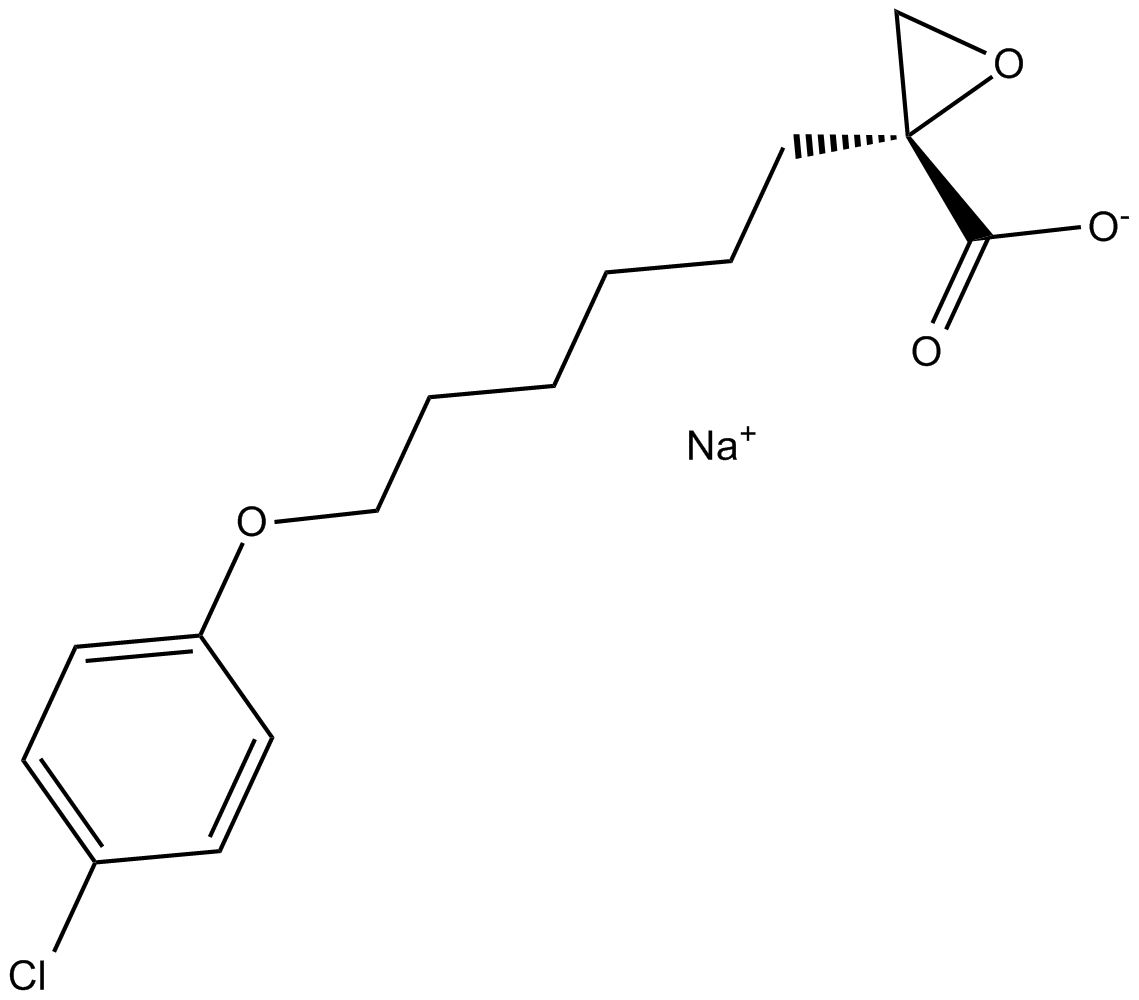
GC15104
(R)-(+)-Etomoxir sodium salt
Etomoxir((R)-(+)-Etomoxir-Natriumsalz ist ein irreversibler Inhibitor der Carnitin-Palmitoyltransferase 1a (CPT-1a), hemmt die FettsÄureoxidation (FAO) durch CPT-1a und hemmt die Palmitat-β-Oxidation bei Mensch und Ratte und Meerschweinchen.
-
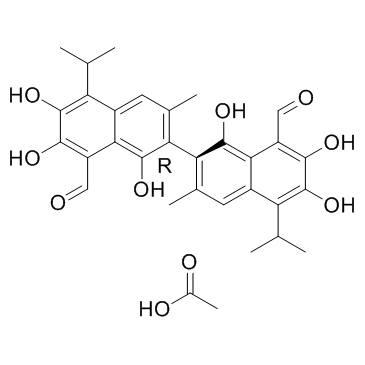
GC34096
(R)-(-)-Gossypol acetic acid (AT-101 (acetic acid))
(R)-(-)-Gossypol-EssigsÄure (AT-101 (EssigsÄure)) (AT-101 (EssigsÄure)) ist das linksdrehende Isomer des Naturprodukts Gossypol. Es wird bestimmt, dass AT-101 an Bcl-2-, Mcl-1- und Bcl-xL-Proteine mit Kis von 260&7#177;30 nM, 170&7#177;10 nM bzw. 480&7#177;40 nM bindet.
-
GC65610
(R)-5-Hydroxy-1,7-diphenyl-3-heptanone
(R)-5-Hydroxy-1,7-diphenyl-3-heptanon ist ein Diarylheptanoid, das in Alpinia officinarum vorkommt.

-
GC41716
(R)-CR8
(R)-CR8 (CR8), ein Roscovitin-Analogon der zweiten Generation, ist ein potenter CDK1/2/5/7/9-Inhibitor.
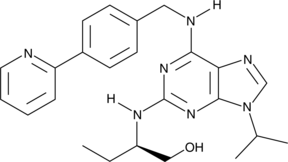
-
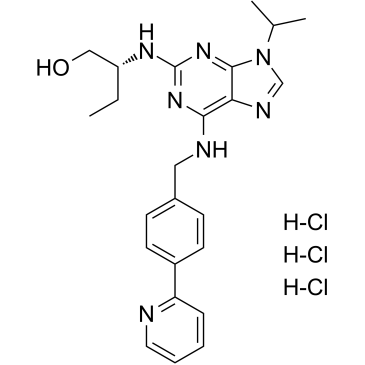
GC39281
(R)-CR8 trihydrochloride
(R)-CR8 (CR8) Trihydrochlorid, ein Roscovitin-Analogon der zweiten Generation, ist ein potenter CDK1/2/5/7/9-Inhibitor.
-
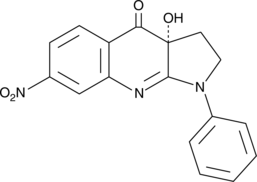
GC41719
(R)-nitro-Blebbistatin
(R)-nitro-Blebbistatin is a more stable form of (+)-blebbistatin, which is the inactive form of (-)-blebbistatin.
-
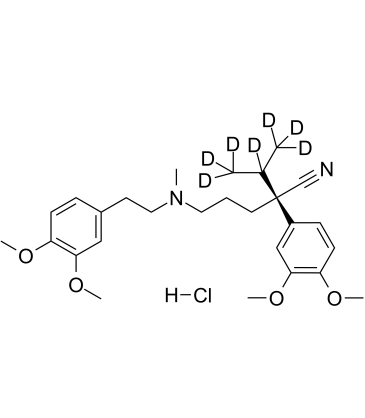
GC60407
(R)-Verapamil D7 hydrochloride
(R)-Verapamil D7-Hydrochlorid ((R)-(+)-Verapamil D7-Hydrochlorid) ist ein mit Deuterium markiertes (R)-Verapamil-Hydrochlorid. (R)-Verapamilhydrochlorid ((R)-(+)-Verapamilhydrochlorid) ist ein P-Glycoprotein-Inhibitor. (R)-Verapamilhydrochlorid blockiert den MRP1-vermittelten Transport, was zu einer Chemosensibilisierung von MRP1-Überexprimierenden Zellen gegenÜber Krebsmedikamenten fÜhrt.
-
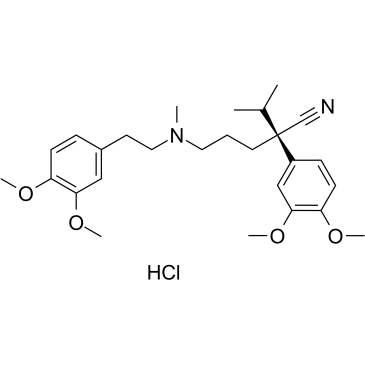
GC60408
(R)-Verapamil hydrochloride
(R)-Verapamilhydrochlorid ((R)-(+)-Verapamilhydrochlorid) ist ein P-Glycoprotein-Inhibitor. (R)-Verapamilhydrochlorid blockiert den MRP1-vermittelten Transport, was zu einer Chemosensibilisierung von MRP1-Überexprimierenden Zellen gegenÜber Krebsmedikamenten fÜhrt.
-
GC19541
(rac)-Antineoplaston A10
(rac)-Antineoplaston A10 ist das Racemat von Antineoplaston A10. Antineoplaston A10 ist ein Ras-Inhibitor, der potenziell zur Behandlung von Gliom, Lymphom, Astrozytom und Brustkrebs eingesetzt werden kann.

-
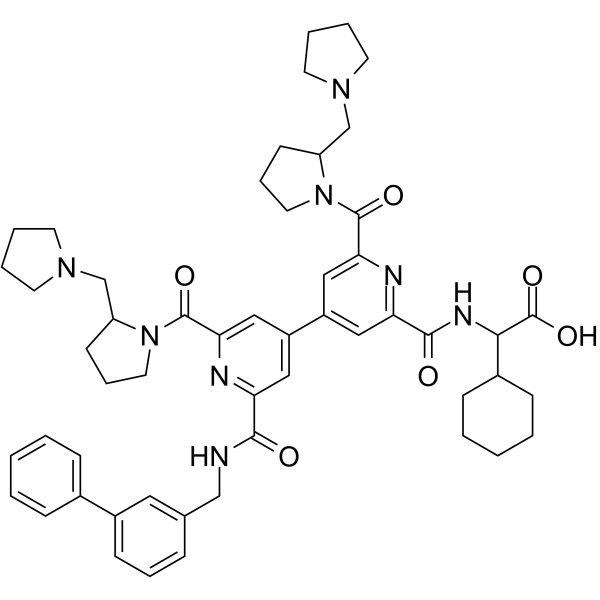
GC69795
(Rac)-BIO8898
(Rac)-BIO8898 ist ein Inhibitor der CD40-CD154-Ko-Stimulationsinteraktion. (Rac)-BIO8898 hemmt die Bindung von CD154 an CD40-Ig mit einer IC50 von 25 μM.
-
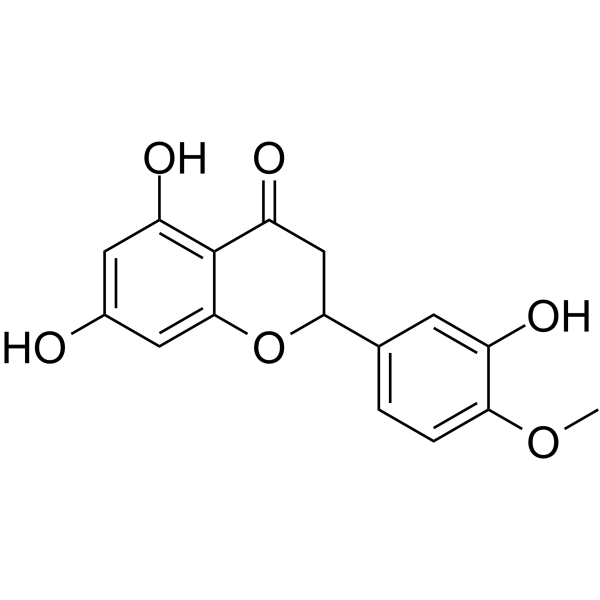
GC62528
(Rac)-Hesperetin
(Rac)-Hesperetin ist das Racemat von Hesperetin.
-
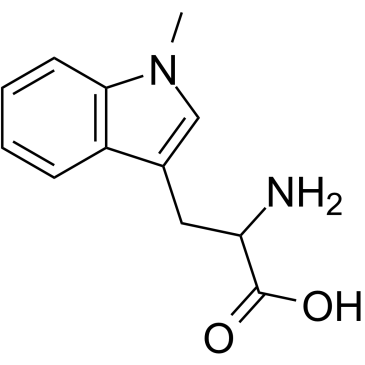
GC61750
(Rac)-Indoximod
(Rac)-Indoximod (1-Methyl-DL-Tryptophan) ist ein Indolamin-2,3-Dioxygenase (IDO)-Hemmer.
-
GC10098
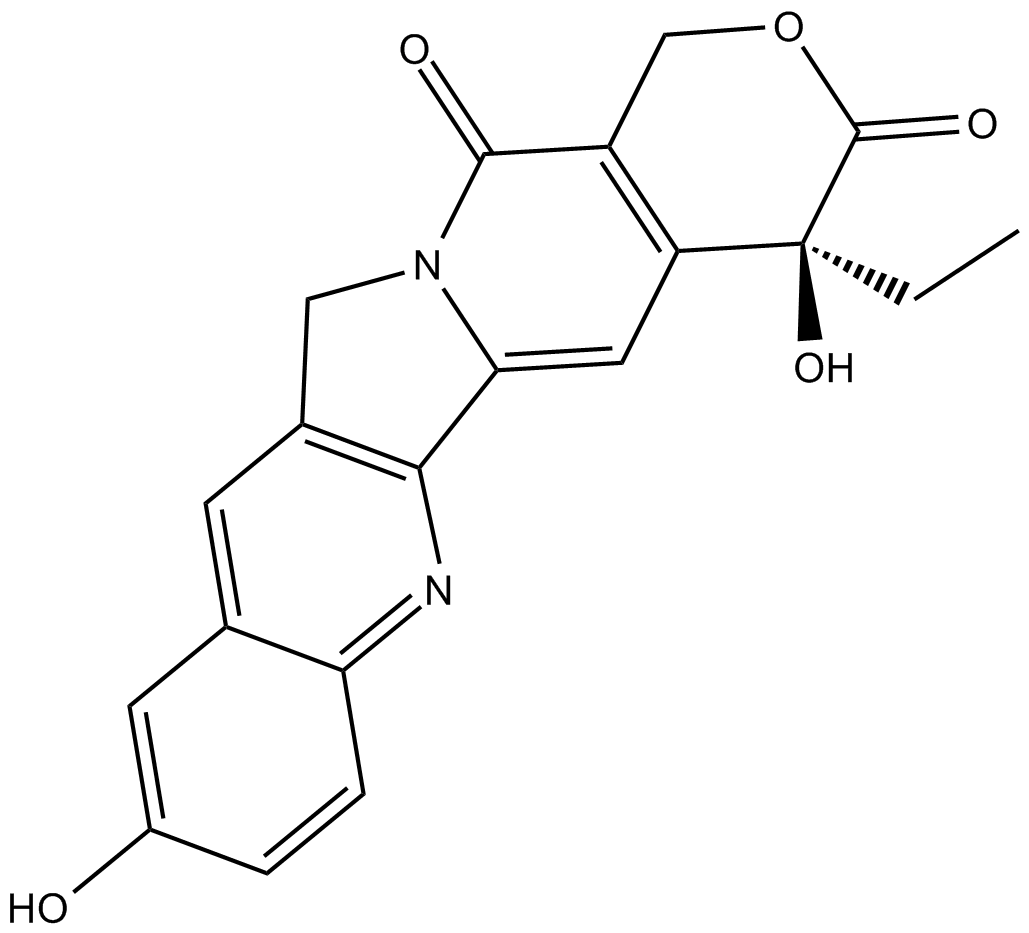
(S)-10-Hydroxycamptothecin
(S)-10-Hydroxycamptothecin (10-HCPT; 10-Hydroxycamptothecin) ist ein DNA-Topoisomerase-I-Inhibitor, der aus der chinesischen Pflanze Camptotheca accuminata isoliert wurde. (S)-10-Hydroxycamptothecin zeigt eine bemerkenswerte Apoptose-induzierende Wirkung. (S)-10-Hydroxycamptothecin hat das Potenzial zur Behandlung von Hepatom, Magenkarzinom, Dickdarmkrebs und LeukÄmie.
-
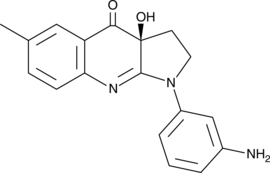
GC41557
(S)-3'-amino Blebbistatin
(S)-3'-amino Blebbistatin is a more stable and less phototoxic form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
-
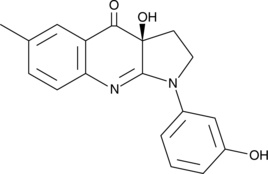
GC41484
(S)-3'-hydroxy Blebbistatin
(S)-3'-hydroxy Blebbistatin is a more stable and less phototoxic form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
-
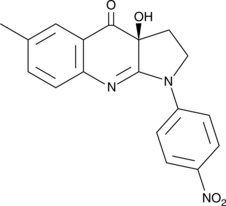
GC52192
(S)-4'-nitro-Blebbistatin
(S)-4'-Nitro-Blebbistatin ist ein nicht zytotoxischer, photostabiler, fluoreszierender und spezifischer Myosin-II-Inhibitor, der in der Untersuchung der spezifischen Rolle von Myosin II in physiologischen, entwicklungsbezogenen und zellbiologischen Studien verwendet wurde.
-
GC35001
(S)-Gossypol acetic acid
(S)-Gossypol ist das Isomer des Naturprodukts Gossypol. (S)-Gossypol bindet mit hoher Affinität an die BH3-Bindungsfurche von Bcl-xL- und Bcl-2-Proteinen.
-
GC41739
(S)-nitro-Blebbistatin
(S)-nitro-Blebbistatin is a more stable form of (-)-blebbistatin, which is a selective cell-permeable inhibitor of non-muscle myosin II ATPases.
-
GC60425
(S)-Verapamil D7 hydrochloride
(S)-Verapamil D7-Hydrochlorid ((S)-(-)-Verapamil D7-Hydrochlorid) ist ein mit Deuterium markiertes (S)-Verapamil-Hydrochlorid. (S)-Verapamilhydrochlorid (S(-)-Verapamilhydrochlorid) hemmt den Transport von Leukotrien C4 (LTC4) und Calcein durch MRP1. (S)-Verapamilhydrochlorid fÜhrt zum Absterben potenziell resistenter Tumorzellen.
-
GC60008
(S)-Verapamil hydrochloride
(S)-Verapamilhydrochlorid (S(-)-Verapamilhydrochlorid) hemmt den Transport von Leukotrien C4 (LTC4) und Calcein durch MRP1. (S)-Verapamilhydrochlorid fÜhrt zum Absterben potenziell resistenter Tumorzellen.
-
GC18787
(±)-Dunnione
(±)-Dunnione is a naturally occurring naphthoquinone with diverse biological activities.
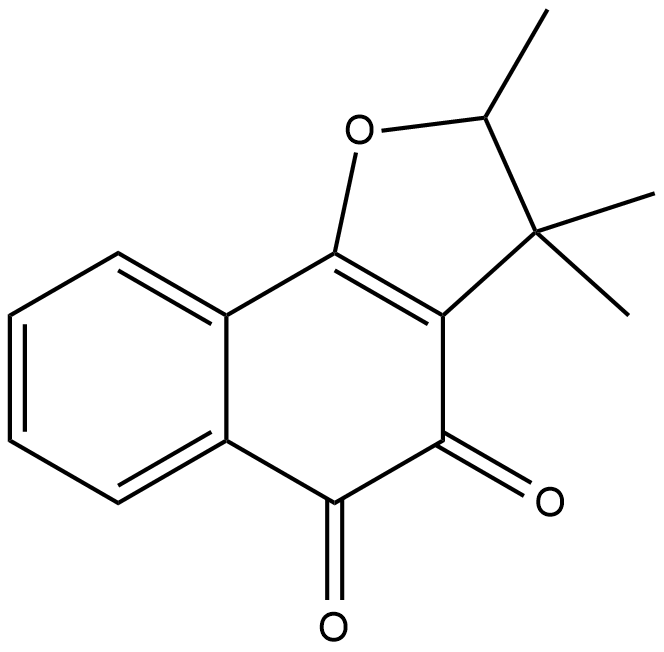
-
GC11965
(±)-Huperzine A
A neuroprotective AChE inhibitor
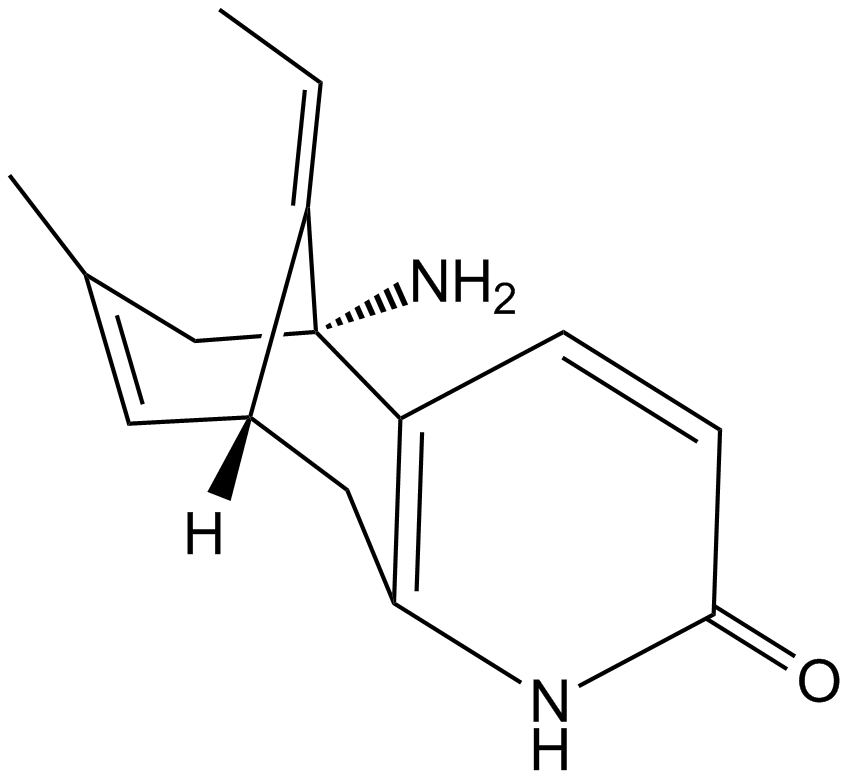
-
GC16375
(±)-Jasmonic Acid methyl ester
(±)-JasmonsÄuremethylester ist ein kÖrpereigener Metabolit.
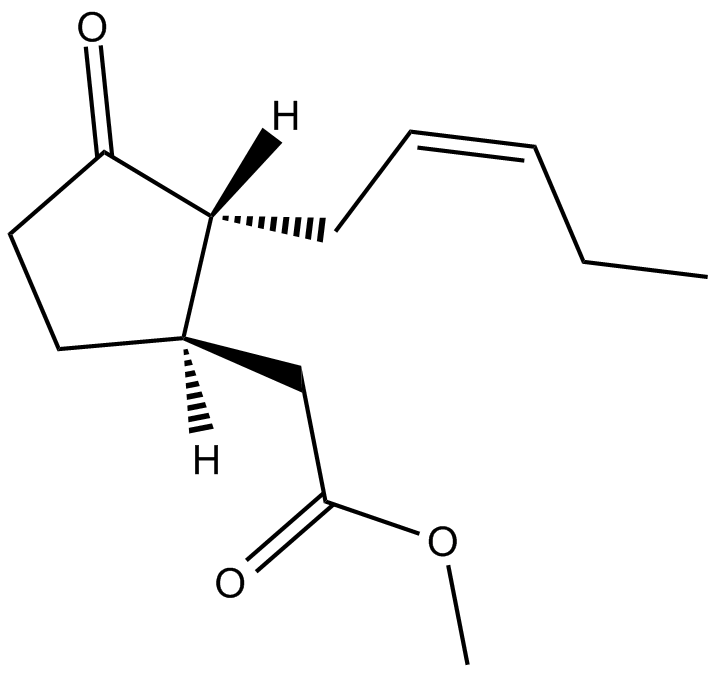
-
GC14154
(±)-Nutlin-3
MDM2 antagonist, potent and selective
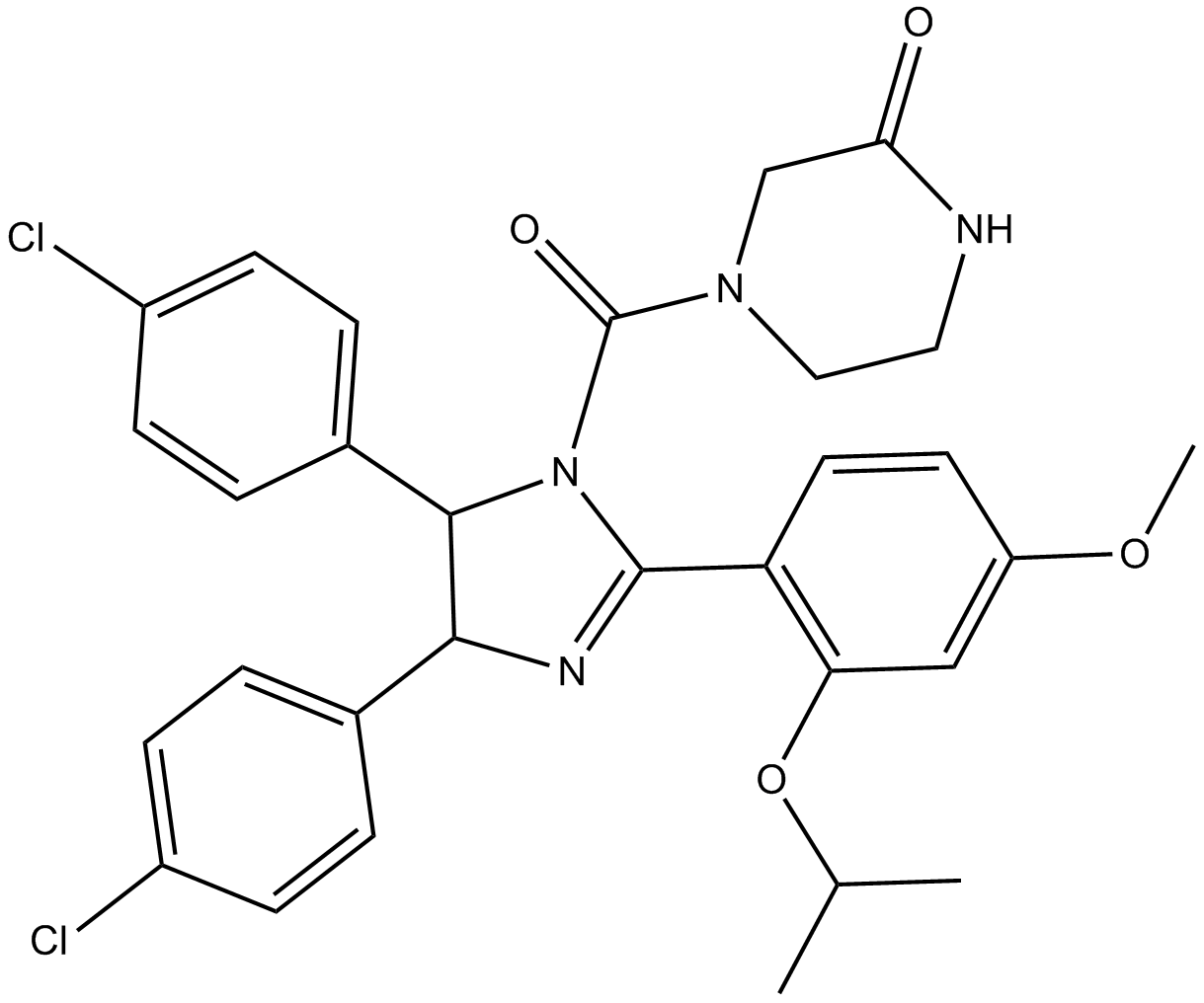
-
GC46379
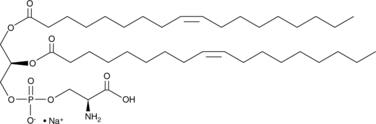
1,2-Dioleoyl-sn-glycero-3-PS (sodium salt)
1,2-Dioleoyl-sn-glycero-3-PS (Natriumsalz) ist ein Ersatzstoff fÜr Phosphoserin/Phosphatidylserin.
-
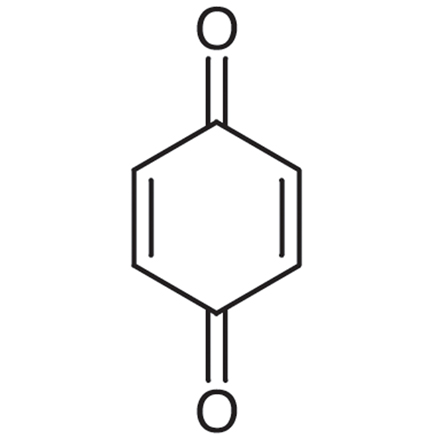
GC19528
1,4-Benzoquinone
A toxic metabolite of benzene
-
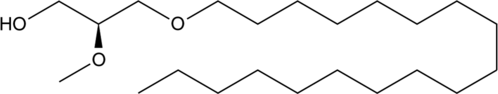
GC42018
1-O-Octadecyl-2-O-methyl-sn-glycerol
1-O-Octadecyl-2-O-methyl-sn-glycerol is a metabolite of a phosphotidylinositol ether lipid analog (PIA).
-
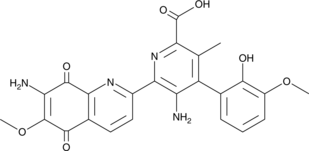
GC41865
10'-Desmethoxystreptonigrin
10'-Desmethoxystreptonigrin is an antibiotic originally isolated from Streptomyces and a derivative of the antibiotic streptonigrin.
-
GC49736
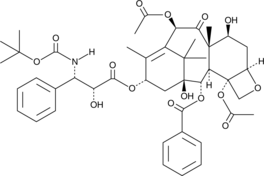
10-acetyl Docetaxel
10-Acetyl-Docetaxel (10-Acetyl-Docetaxel) ist ein Analogon von Docetaxel mit AntikrebsaktivitÄt. Docetaxel ist ein Inhibitor der Mikrotubuli-Zerlegung mit antimitotischer AktivitÄt.
-
GC64726
10-Formyl-5,8-dideazafolic acid
10-Formyl-5,8-dideazafolsÄure ist ein Thymidylat-Synthase-Hemmer.

-
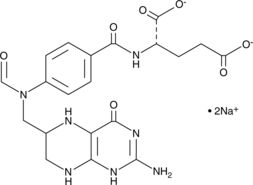
GC49872
10-Formyltetrahydrofolate (sodium salt) (technical grade)
10-Formyltetrahydrofolat (Natriumsalz) (technische QualitÄt) ist eine Form von TetrahydrofolsÄure, die als Spender von Formylgruppen im Anabolismus wirkt.
-
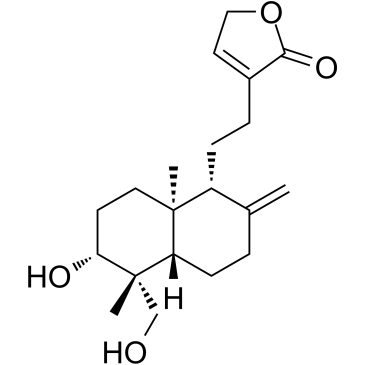
GC35057
14-Deoxyandrographolide
14-Desoxyandrographolid ist ein Labdan-Diterpen mit blockierender Wirkung auf CalciumkanÄle.
-
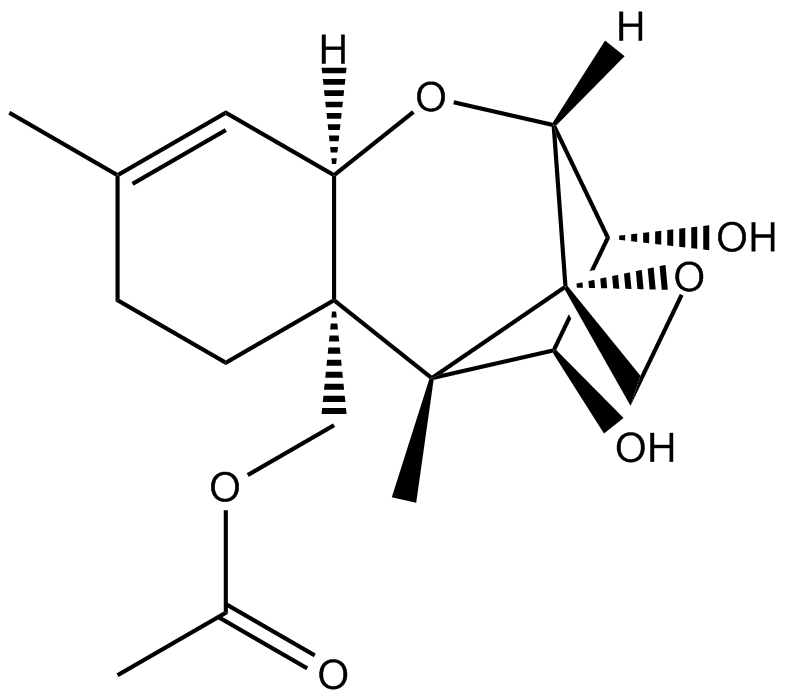
GC11988
15-acetoxy Scirpenol
15-Acetoxyscirpenol, eines der Mykotoxine der Acetoxyscirpenol-Einheit (ASMs), induziert stark Apoptose und hemmt das Wachstum von Jurkat-T-Zellen dosisabhängig, indem es andere Caspasen unabhängig von Caspase-3 aktiviert.
-
GC41938
15-Lipoxygenase Inhibitor 1
15-Lipoxygenase-Inhibitor 1 ist ein selektiver Inhibitor der 15-Lipoxygenase mit einem IC50-Wert von 18 μM. 15-Lipoxygenase-Inhibitor 1 hat IC50s von 19,5 μM und 19,1 ⋼M fÜr Sojabohnen-15-Lipoxygenase (SLO) bzw. menschliche 15-Lipoxygenase-1 (15-LOX-1). 15-Lipoxygenase Inhibitor 1 hat Potenzial fÜr die Erforschung von Prostatakrebs.
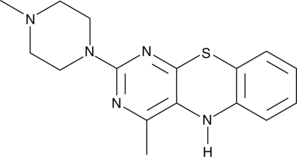
-
GC46451
16F16
A PDI inhibitor

-
GC11720
17-AAG (KOS953)
An inhibitor of Hsp90
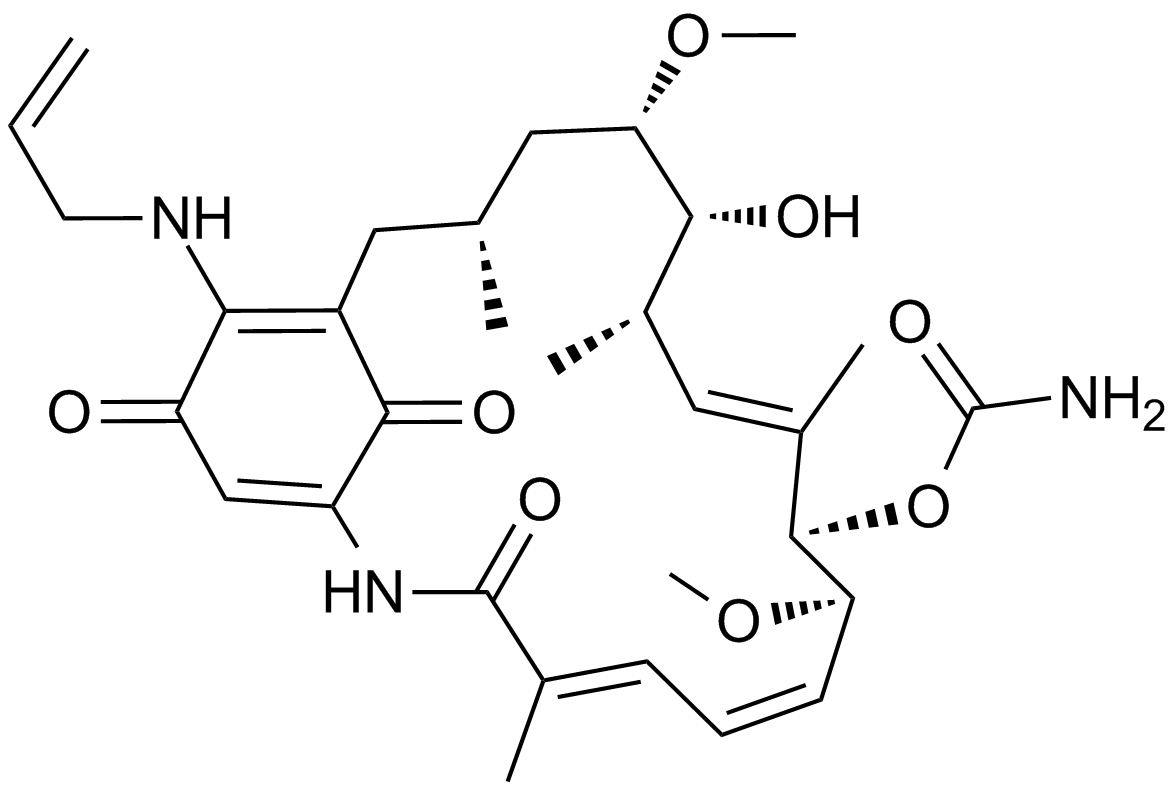
-
GC13044
17-DMAG (Alvespimycin) HCl
17-DMAG (Alvespimycin) HCl (17-DMAG Hydrochlorid; KOS-1022; BMS 826476) ist ein potenter Inhibitor von Hsp90, der an Hsp90 mit einem EC50 von 62±29 nM bindet.

-
GC41983
19,20-Epoxycytochalasin D
19,20-Epoxycytochalasin D, ein Cytochalasin, ist ein Pilzmetabolit von Nemania sp.
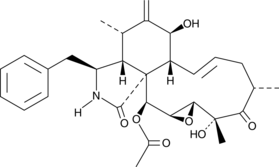
-
GC48423
19-O-Acetylchaetoglobosin A
19-O-Acetylchaetoglobosin A, ein Cytochalasan-Alkaloid, ist ein Pilzmetabolit, der ursprÜnglich aus C. globosum isoliert wurde und eine hemmende und zytotoxische Wirkung auf die Aktinpolymerisation hat. 19-O-Acetylchaetoglobosin A ist zytotoxisch fÜr HeLa-Zervixkrebszellen.
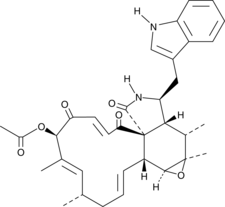
-
GC39296
1G244
1G244 ist ein potenter DPP8/9-Inhibitor mit IC50-Werten von 12 nM bzw. 84 nM. 1G244 hemmt DPPIV und DPPII nicht. 1G244 induziert Apoptose in multiplen Myelomzellen und hat Anti-Myelom-Wirkungen.
-
GC46508
2',2'-Difluoro-2'-deoxyuridine
An active metabolite of gemcitabine
-
GC41612
2'-O-Methylguanosine
2'-O-Methylguanosin ist ein modifiziertes Nukleosid, das in tRNAs durch die Wirkung von tRNA Guanosin-2'-O-Methyltransferase produziert wird.
-
GC12258
2,3-DCPE hydrochloride
-
GC40947
2,3-Dimethoxy-5-methyl-p-benzoquinone
2,3-Dimethoxy-5-methyl-p-benzochinon (CoQ0) ist eine potente, oral aktive Ubichinonverbindung, die aus Antrodia cinnamomea gewonnen werden kann.
-
GC68452
2,4,6-Triiodophenol

-
GC46057
2,5-Dihydroxycinnamic Acid phenethyl ester
An inhibitor of 5-LO
-
GC45324
2,5-dimethyl Celecoxib
-
GN10065
2-Atractylenolide
-
GC40675
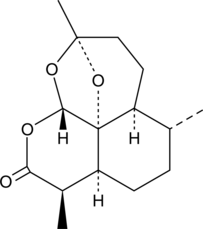
2-deoxy-Artemisinin
2-deoxy-Artemisinin is an inactive metabolite of the antimalarial agent artemisinin.
-
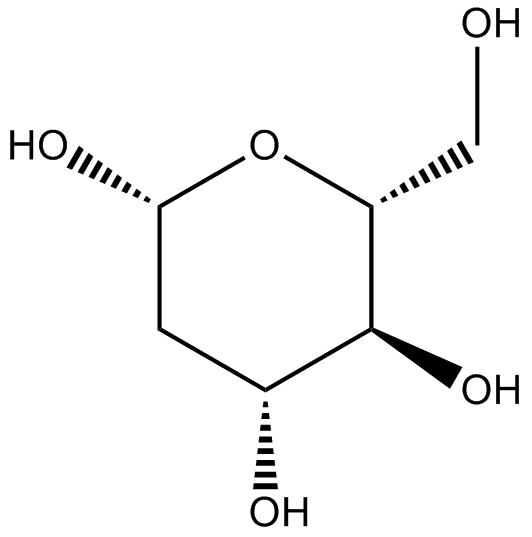
GC17430
2-Deoxy-D-glucose
2-Deoxy-D-Glukose (2DG) ist ein Glukose-Analogon, das als kompetitiver glykolytischer Inhibitor wirkt.
-
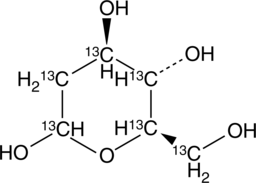
GC49223
2-deoxy-D-Glucose-13C6
An internal standard for the quantification of 2-deoxy-D-glucose
-
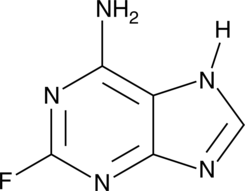
GC46545
2-Fluoroadenine
2-Fluoroadenin ist eine giftige Purinbase. 2-Fluoroadenin hat ToxizitÄt in nicht proliferierenden und proliferierenden Tumorzellen. 2-Fluoroadenin kann fÜr die Erforschung von Krebserkrankungen verwendet werden.
-
GC12545
2-HBA
2-HBA ist ein starker Induktor der NAD(P)H:Chinon-Akzeptor-Oxidoreduktase 1 (NQO1), die auch Caspase-3 und Caspase-10 aktivieren kann.
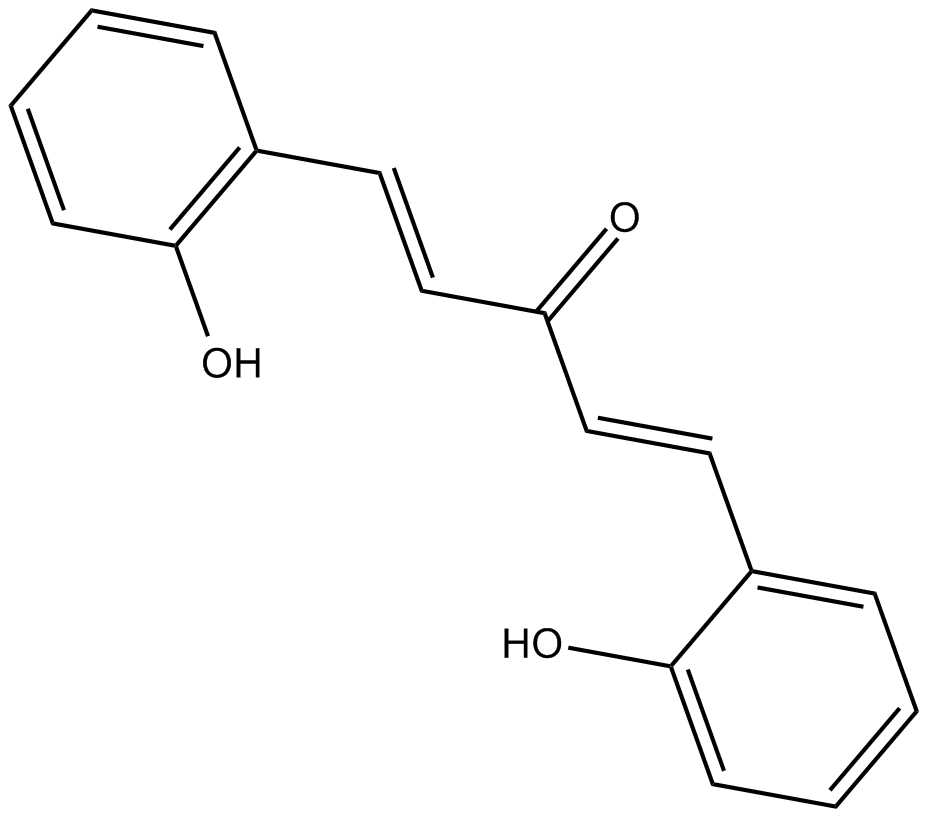
-
GC38318
2-Methoxycinnamaldehyde
2-Methoxyzimtaldehyd (o-Methoxyzimtaldehyd) ist eine natÜrliche Verbindung von Cinnamomum cassia mit AntitumoraktivitÄt. 2-Methoxyzimtaldehyd hemmt die Proliferation und induziert Apoptose durch Verlust des mitochondrialen Membranpotentials (δψm), Aktivierung von Caspase-3 und Caspase-9. 2-Methoxyzimtaldehyd hemmt wirksam die durch den BlutplÄttchen-Wachstumsfaktor (PDGF) induzierte HASMC-Migration.
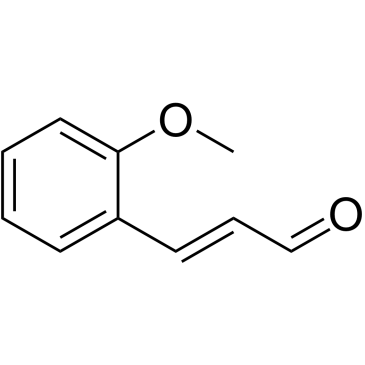
-
GC15084
2-Methoxyestradiol (2-MeOE2)
2-Methoxyestradiol (2-MeOE2/2-Me) is a HIF-1α inhibitor that inhibits HIF-1α accumulation and HIF transcriptional activity. 2-Methoxyestradiol can trigger p53-induced apoptosis and has potential antitumor activity..
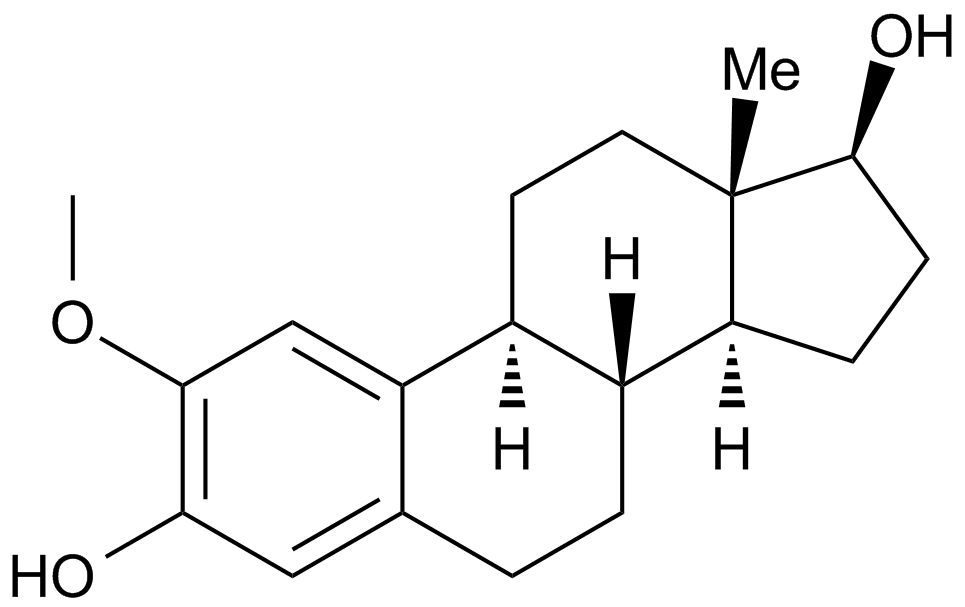
-
GC68043
2-tert-Butyl-1,4-benzoquinone



