Histone Methyltransferase
Histone methyltransferases are a group of enzymes that catalyze the methylation of histone lysine and arginine by adding methyl groups to specific histone arginine or lysine residues. Histone methyltransferaes can be classified into 3 classes, including SET domain lysine methyltransferases, non-SET domain lysine methyltransferases and arginine methyltransferases (PRMTs), all of which use S-adenosylmethionine as a cosubstrate for the transfer of the methyl group. Aberrant histone methylation has been associated with a wide range of human cancers (such as hematological malignancies), which leads to the development of novel cancer chemotherapies targeting cancer-associated histone methyltransferases (more than 20 lysine methyltransferases and 9 arginine methyltransferases in humans).
Products for Histone Methyltransferase
- Cat.No. Nom du produit Informations
-
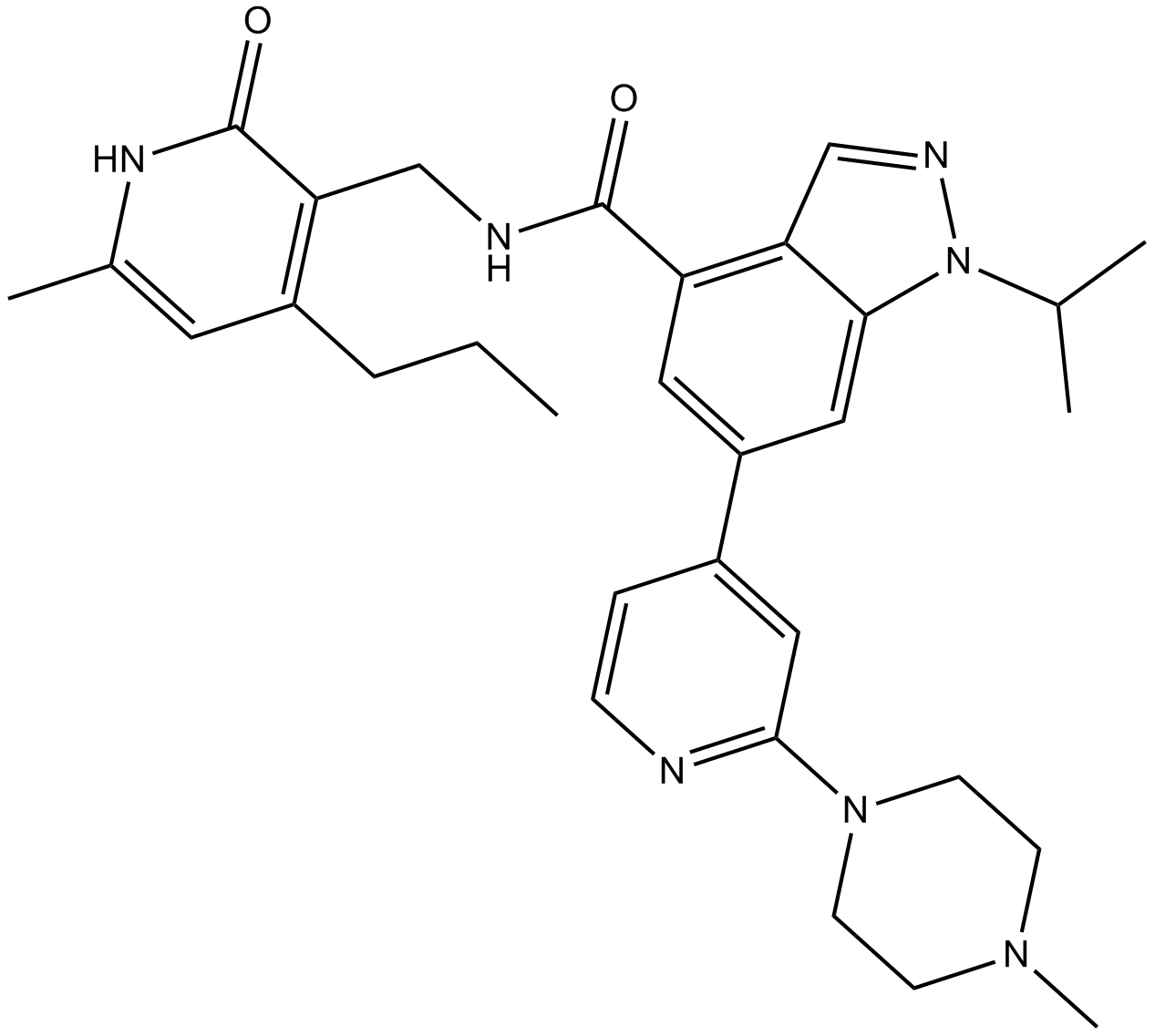
GC45908
(R)-BAY-598
An inhibitor of SMYD2
-
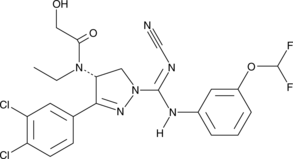
GC11340
(R)-PFI 2 hydrochloride
Le chlorhydrate de PFI-2 ((R)-(R)-PFI 2 hydrochloride) est un domaine SET puissant et sélectif contenant un inhibiteur de la lysine méthyltransférase 7 (SETD7).
-
GC65045
(S)-MRTX-1719
(S)-MRTX-1719 (exemple 16-7) est l'énantiomère S du MRTX-1719. (S)-MRTX-1719 est un inhibiteur du complexe PRMT5/MTA, avec une IC50 de 7070 nM.
-
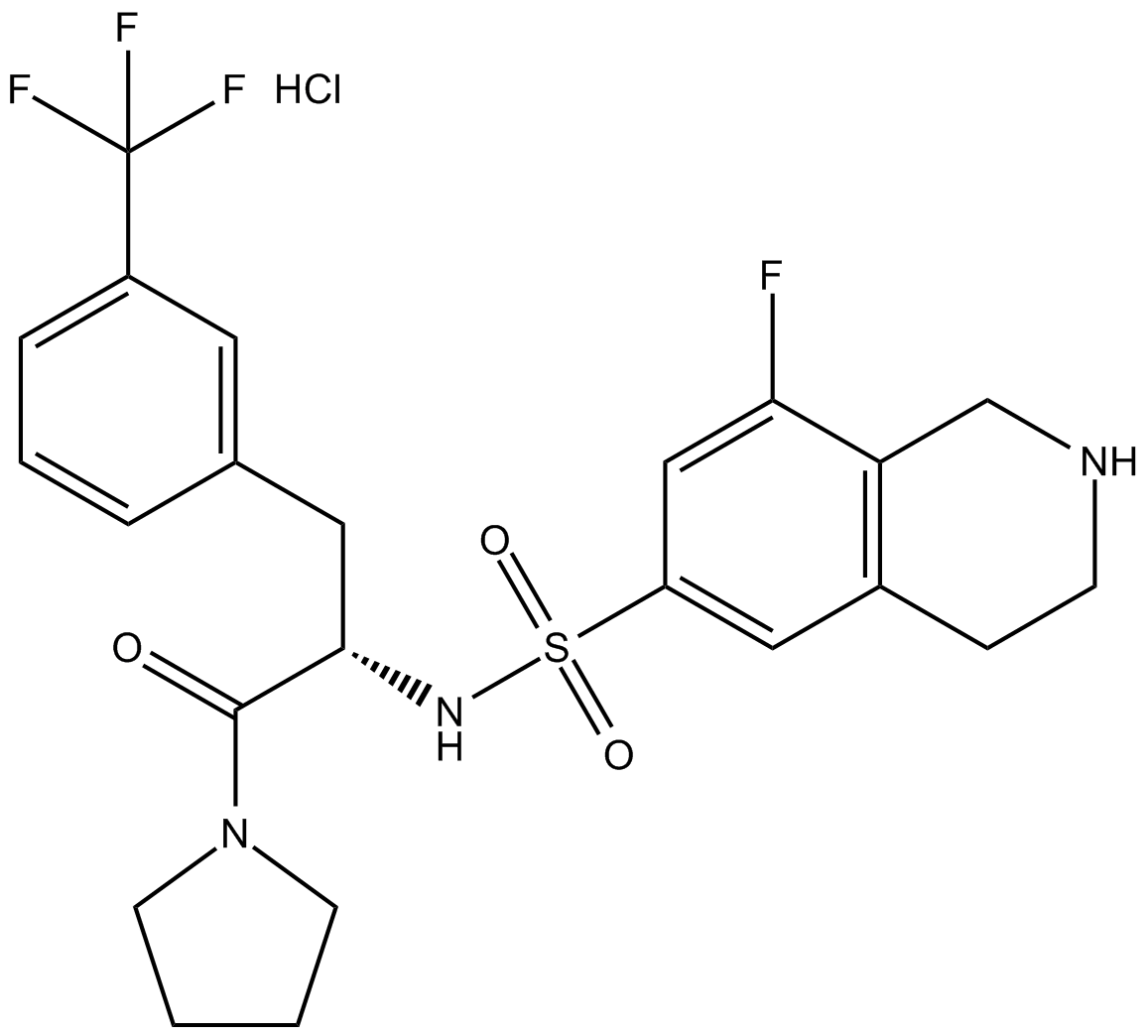
GC13634
(S)-PFI-2 (hydrochloride)
Negative control of (R)-PFI 2 hydrochloride
-
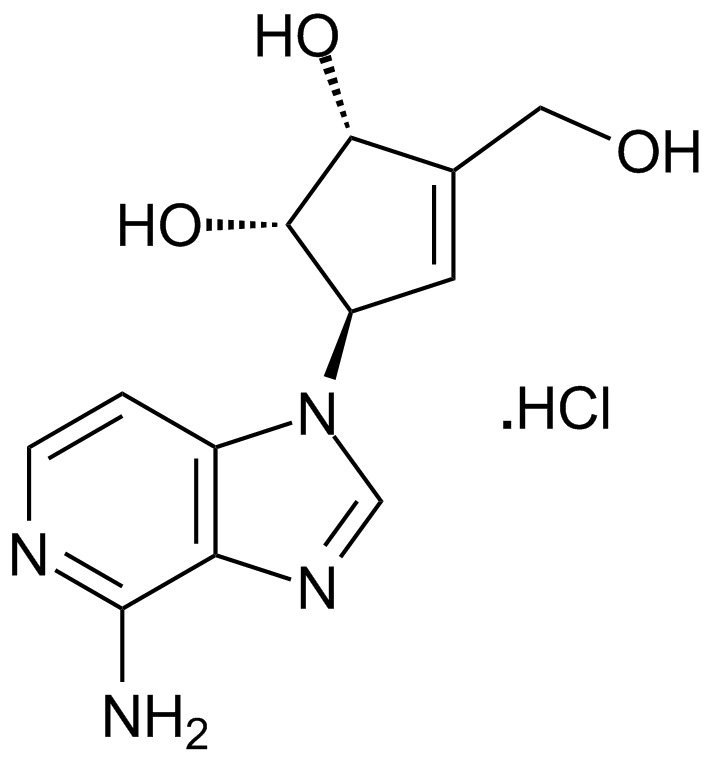
GC17907
3-Deazaneplanocin A (DZNep) hydrochloride
Un inhibiteur de la lysine méthyltransférase EZH2.
-
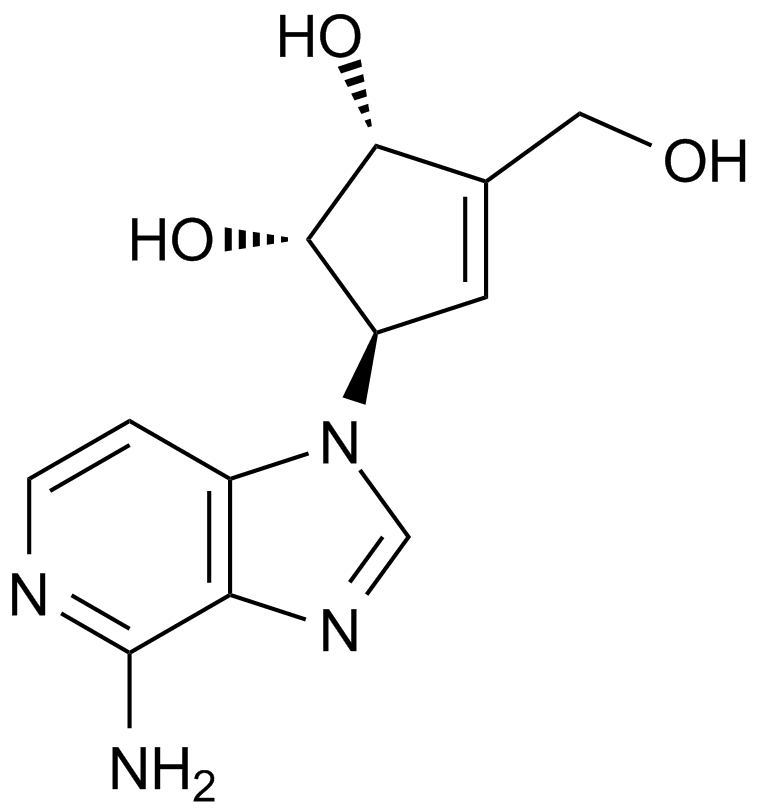
GC13145
3-Deazaneplanocin,DZNep
An inhibitor of lysine methyltransferase EZH2
-
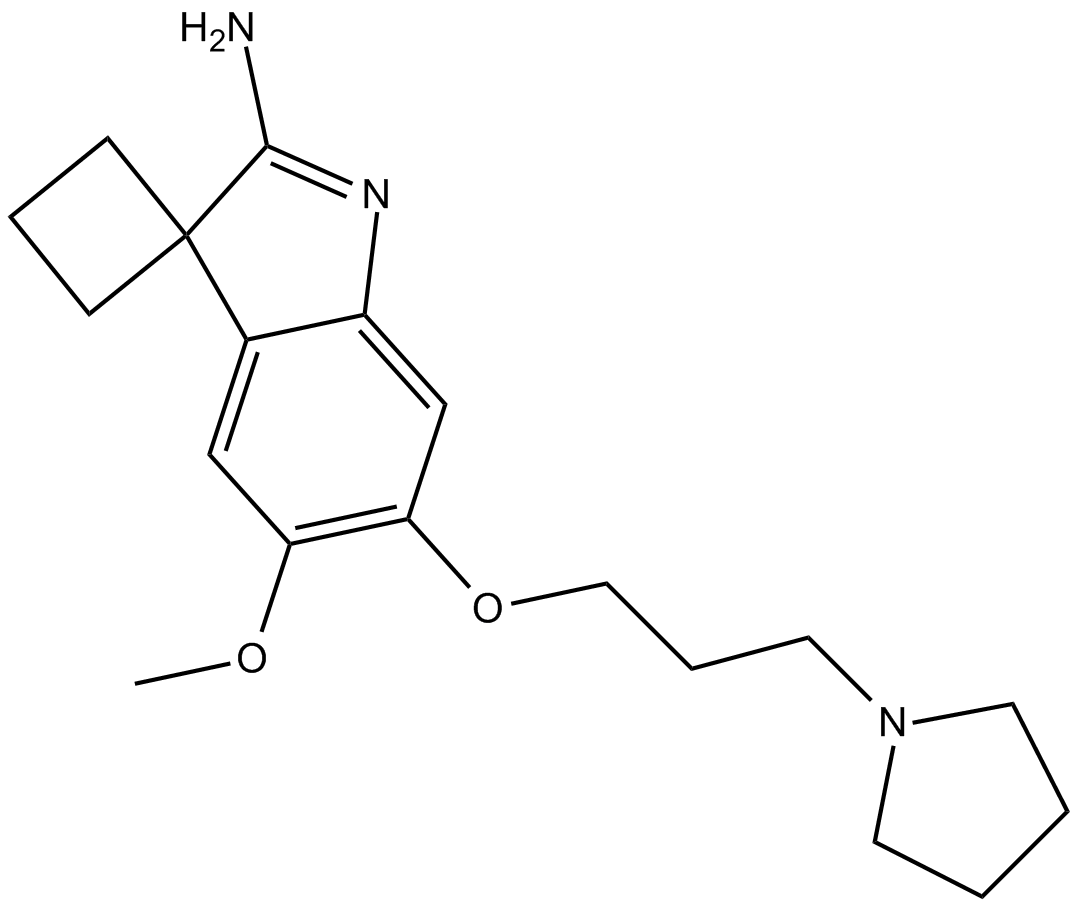
GC16015
A 366
A 366 est un puissant inhibiteur de l'histone méthyltransférase G9a, hautement sélectif et compétitif avec les peptides, avec des CI50 de 3,3 et 38 nM pour G9a et GLP (EHMT1), respectivement. A 366 montre une sélectivité \u003e 1000 fois supérieure à 21 autres méthyltransférases. A 366 est également un puissant inhibiteur nanomolaire de l'interaction Spindlin1-H3K4me3 (IC50 \u003d 182,6 nM). A 366 affiche une affinité élevée pour le récepteur de l'histamine H3 humain (Ki \u003d 17 nM) et présente une sélectivité de sous-type parmi les sous-ensembles des familles de récepteurs histaminergiques et dopaminergiques.
-
GC32861
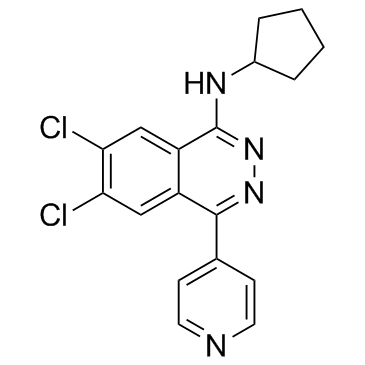
A-196
A-196 est un inhibiteur puissant et sélectif de SUV420H1 et SUV420H2 avec des valeurs IC50 de 25 nM et 144 nM, respectivement. A-196 inhibe biochimiquement SUV4-20 d'une manière compétitive avec le substrat. A-196 représente une sonde chimique de premier ordre de SUV4-20 pour étudier le rÔle des histones méthyltransférases dans l'intégrité génomique.
-
GC33187
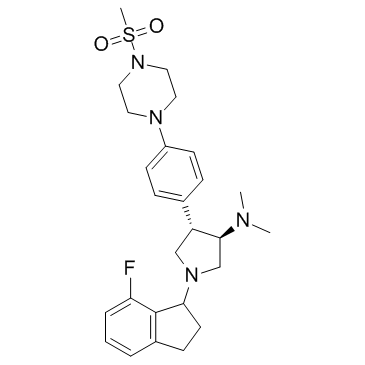
A-395 (A395)
A-395 (A395) est un antagoniste des interactions protéine-protéine du complexe répressif polycomb 2 (PRC2) qui inhibe puissamment le complexe trimérique PRC2 (EZH2-EED-SUZ12) avec une IC50 de 18 nM.
-
GC30503
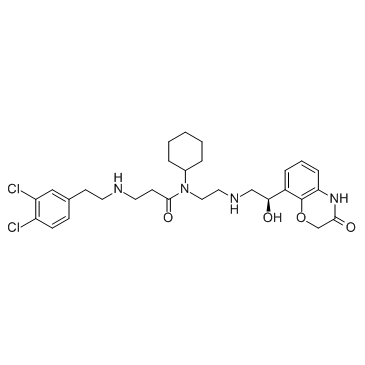
A-893
A-893 est un inhibiteur cellulaire actif de la méthyltransférase SMYD2, avec une IC50 de 2,8 nM.
-
GC16509
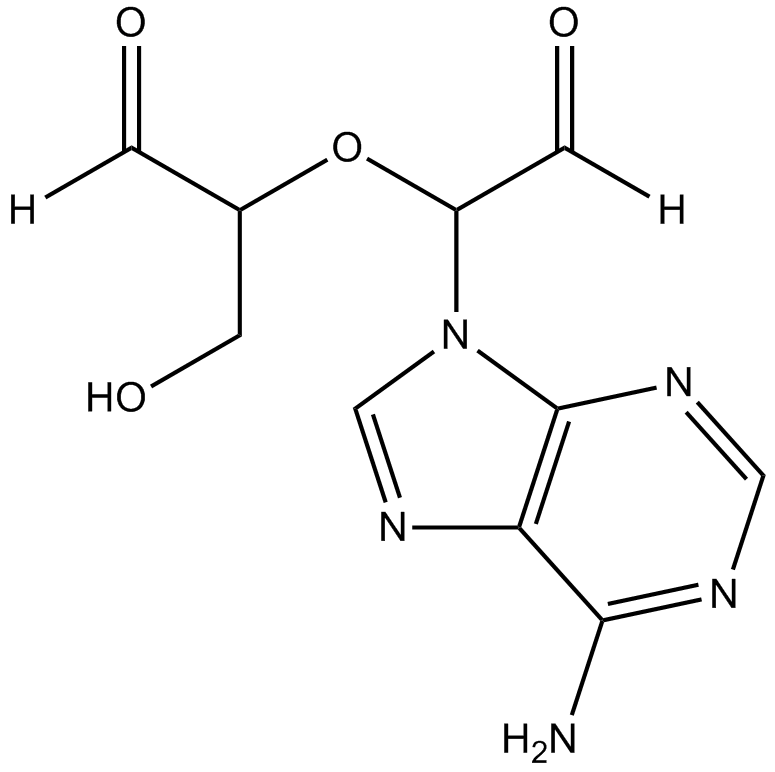
Adox
Adox, un analogue nucléosidique purique, est un puissant inhibiteur de la S-adénosylhomocystéine hydrolase (SAHH) (Ki = 3,3 nM). L'adénosine dialdéhyde présente une puissante activité anti-tumorale in vivo et peut être utilisée pour la recherche sur le cancer.
-
GC17275
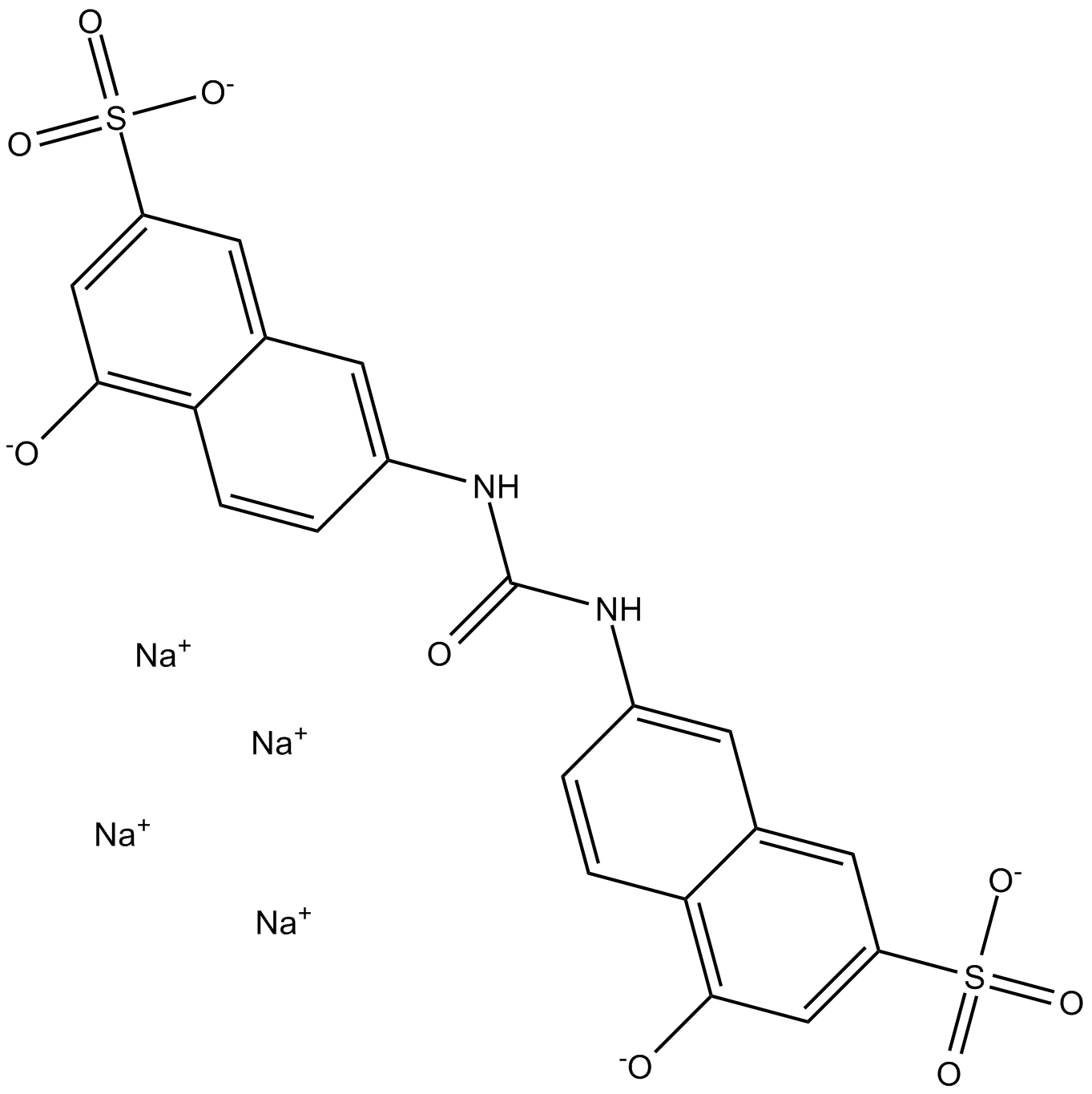
AMI-1
A cell permeable inhibitor of PRMTs
-
GC39840
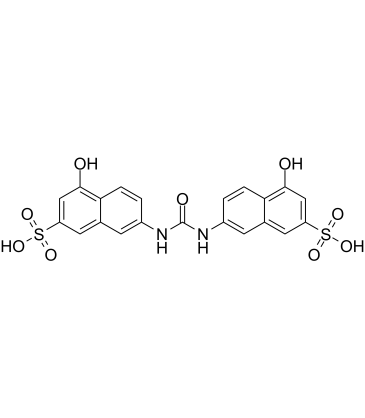
AMI-1 free acid
L'acide libre AMI-1 est un inhibiteur puissant, perméable aux cellules et réversible des protéines arginine N-méthyltransférases (PRMT), avec des IC50 de 8,8 μM et 3,0 μM pour le PRMT1 humain et la levure-Hmt1p, respectivement. L'acide libre AMI-1 exerce des effets inhibiteurs des PRMT en bloquant la liaison peptide-substrat.
-
GC17546
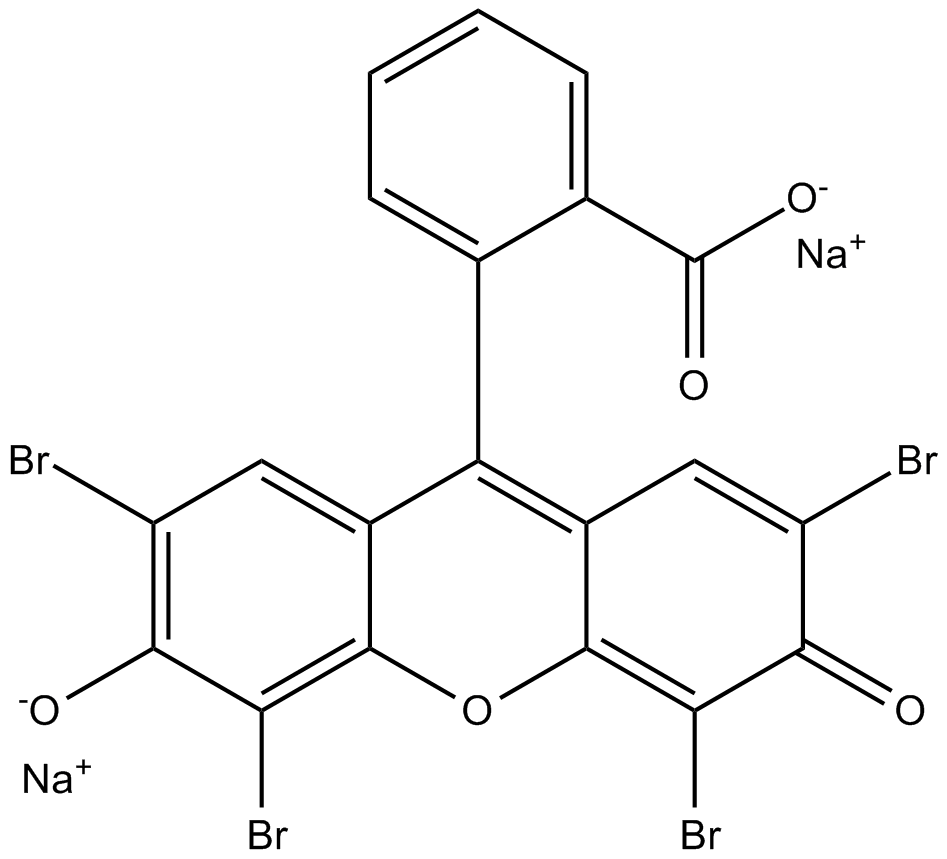
AMI5
L'éosine Y (disodique) est une molécule de colorant rouge acide soluble.
-
GC42790
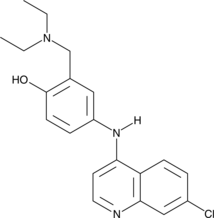
Amodiaquine
Amodiaquine est un composé antipaludique aminoquinoléine.
-
GC60579
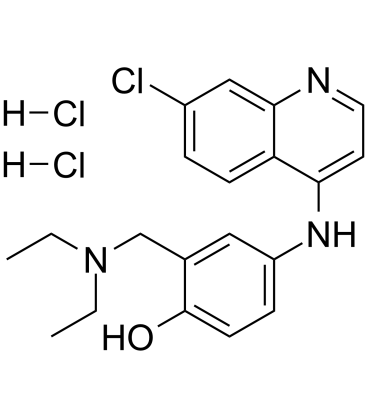
Amodiaquine dihydrochloride
Le dichlorhydrate d'amodiaquine (dichlorhydrate d'amodiaquine), une classe d'agents antipaludéens de la 4-aminoquinoléine, est un inhibiteur puissant et actif par voie orale de l'histamine N-méthyltransférase avec un Ki de 18,6 nM.
-
GC10905
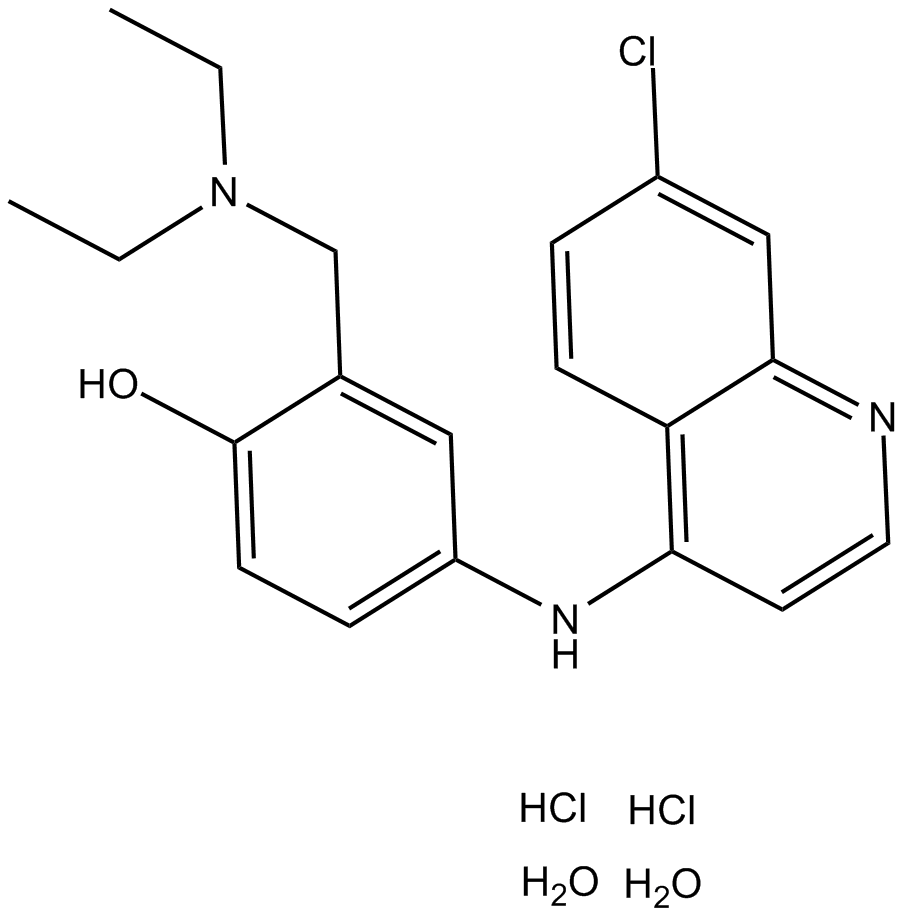
Amodiaquine dihydrochloride dihydrate
Le dichlorhydrate d'amodiaquine dihydraté (dichlorhydrate d'amodiaquine dihydraté), une classe d'agents antipaludiques de la 4-aminoquinoléine, est un puissant inhibiteur de l'histamine N-méthyltransférase actif par voie orale.
-
GC65918
AS-85
L'AS-85 est un puissant inhibiteur de l'histone méthyltransférase ASH1L (IC50=0,6 μM) avec une activité anti-leucémique. AS-85 se lie fortement au domaine ASH1L SET, avec la valeur Kd de 0,78μ ;M.
-
GC62615
AS-99
L'AS-99 est un inhibiteur puissant et sélectif de l'histone méthyltransférase ASH1L (IC50 = 0,79μ ; M, Kd = 0,89⋼ ; M) avec une activité anti-leucémique. L'AS-99 bloque la prolifération cellulaire, induit l'apoptose et la différenciation, régule À la baisse les gènes cibles de fusion MLL et réduit le fardeau de la leucémie in vivo.
-
GC62849
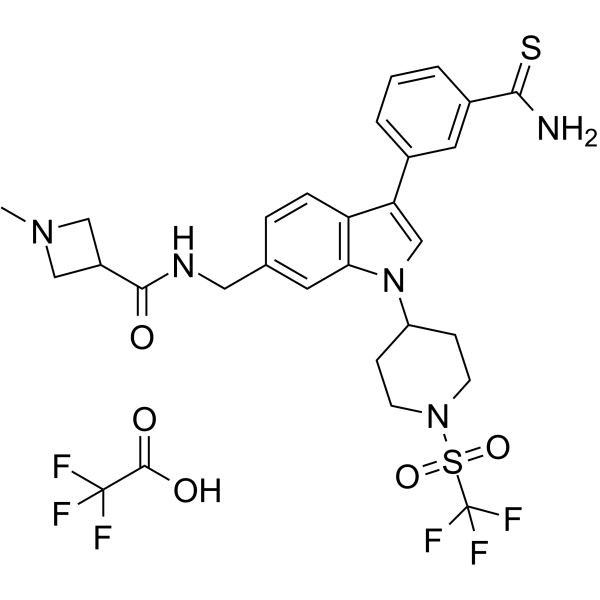
AS-99 TFA
L'AS-99 TFA est un inhibiteur puissant et sélectif de l'histone méthyltransférase ASH1L (IC50=0,79μM, Kd=0,89μM) avec une activité anti-leucémique. L'AS-99 TFA bloque la prolifération cellulaire, induit l'apoptose et la différenciation, régule À la baisse les gènes cibles de fusion MLL et réduit le fardeau de la leucémie in vivo.
-
GC13744
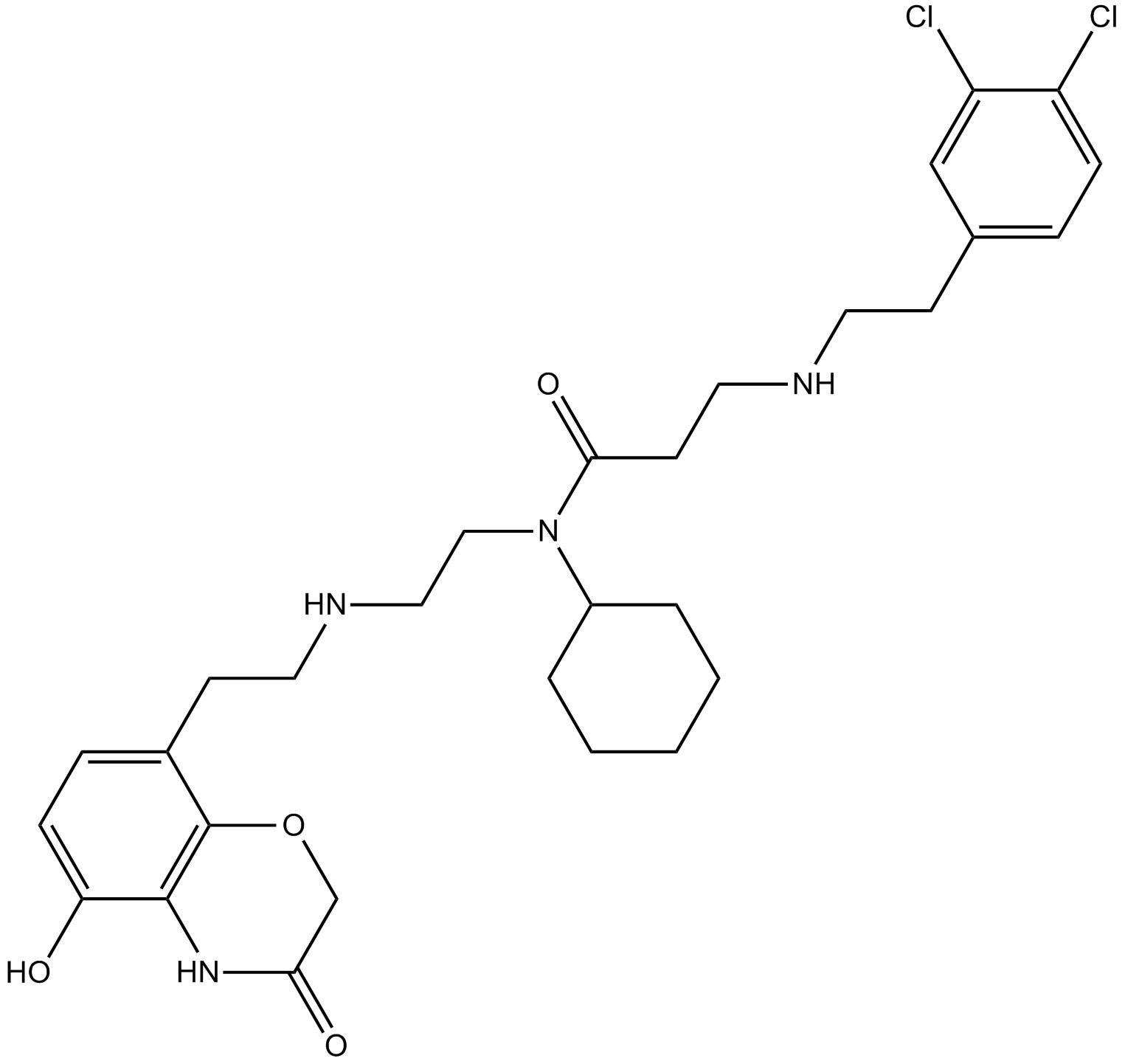
AZ505
AZ505 est un inhibiteur puissant et sélectif de SMYD2 avec une IC50 de 0,12 μM.
-
GC13103
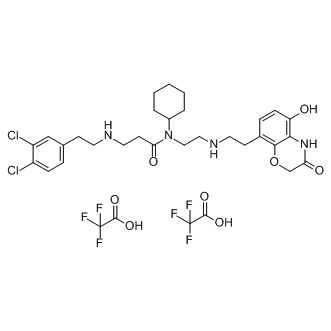
AZ505 ditrifluoroacetate
Le ditrifluoroacétate d'AZ505 est un inhibiteur puissant et sélectif de SMYD2 avec une IC50 de 0,12 μM.
-
GC64124
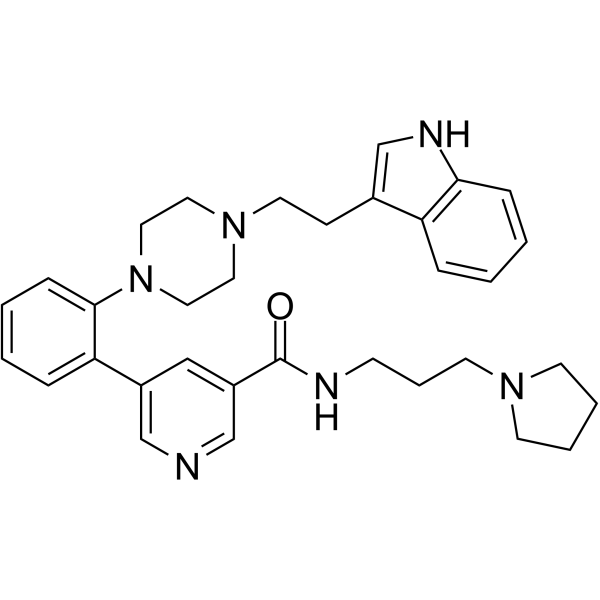
AZ506
AZ506 est un puissant inhibiteur de SMYD2 avec une IC50 de 17 nM. AZ506 inhibe l'activité de la méthyltransférase SMYD2 dans les cellules, entraÎnant une diminution du signal de méthylation médié par SMYD2.
-
GC18159
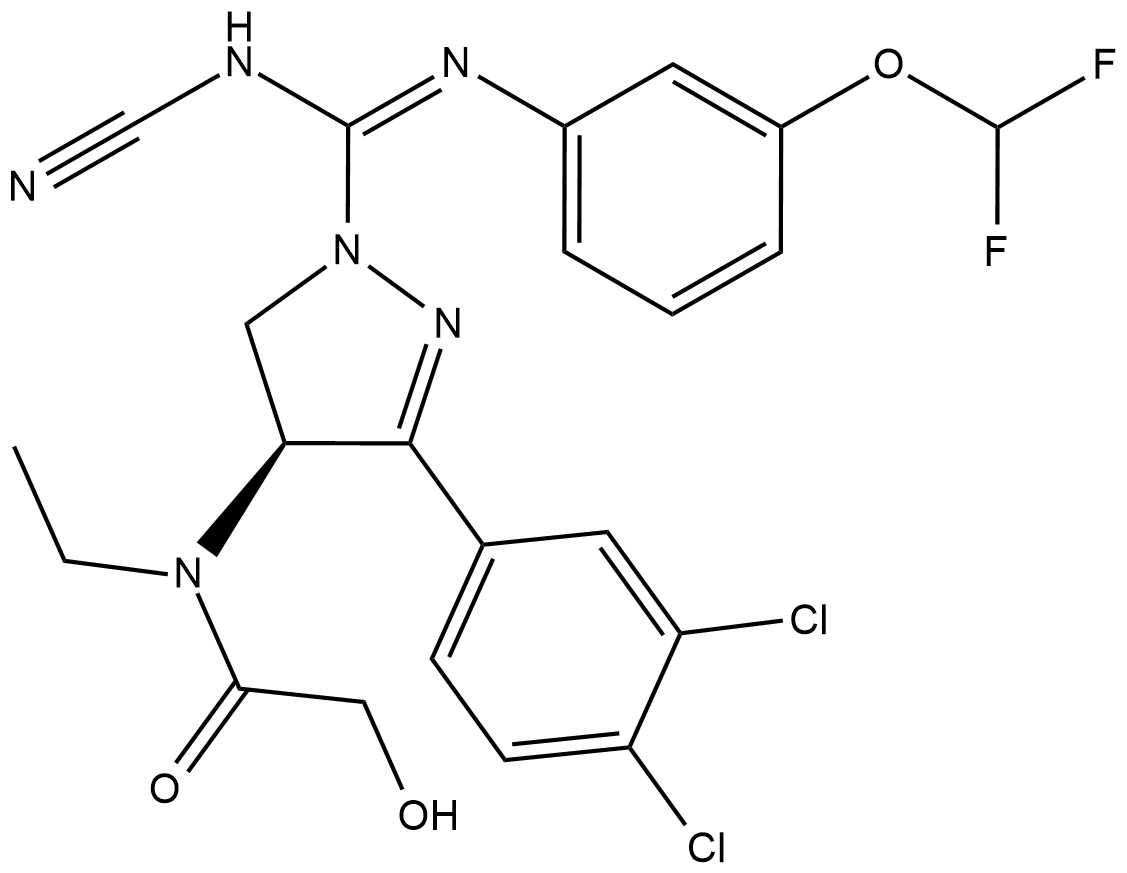
BAY-598
BAY-598 est un inhibiteur sélectif À petite molécule de SMYD2 avec une IC50 de 27 nM.
-
GC45389
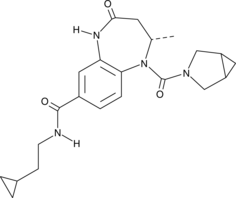
BAY-6035
BAY-6035 est un inhibiteur puissant, sélectif et compétitif du substrat de SMYD3. BAY-6035 inhibe la méthylation du peptide MEKK2 avec une IC50 de 88 nM.
-
GC18161
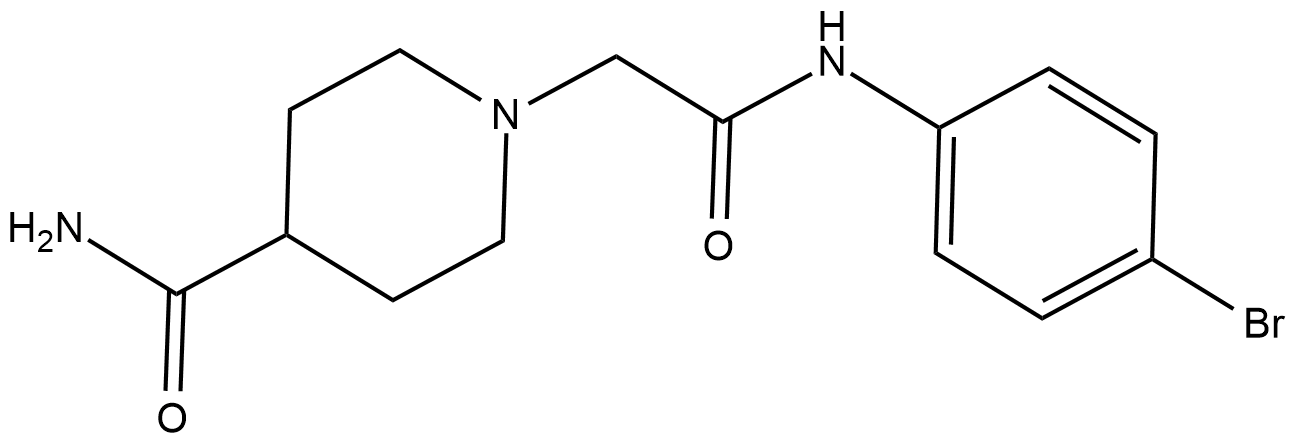
BCI-121
Le BCI-121 est un inhibiteur de SMYD3 qui entrave la prolifération des cellules cancéreuses.
-
GC50540
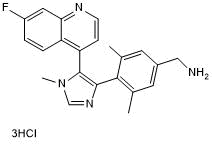
BI 9321
Nuclear receptor-binding SET domain (NSD) 3 antagonist; selectively binds PWWP1 domain
-
GC39663
BI-9321 trihydrochloride
Le trichlorhydrate de BI-9321 est un antagoniste du domaine SET 3 (NSD3)-PWWP1 puissant, sélectif et actif cellulaire avec une valeur Kd de 166 nM. Le trichlorhydrate de BI-9321 est inactif contre NSD2-PWWP1 et NSD3-PWWP2. Le trichlorhydrate de BI-9321 perturbe spécifiquement les interactions des histones du domaine NSD3-PWWP1 avec une IC50 de 1,2 μM dans les cellules U2OS.
-
GC48463
Bisubstrate Inhibitor 78
An inhibitor of NNMT
-
GC12171
BIX 01294
An inhibitor of G9a histone methyltransferase
-
GC33301
BIX-01338 hydrate (BIX01338 hydrate)
L'hydrate de BIX-01338 (hydrate de BIX01338) est un inhibiteur de l'histone lysine méthyltransférase.
-
GC42944
BIX01294 (hydrochloride hydrate)
The methylation of lysine residues on histones plays a central role in determining euchromatin structure and gene expression.
-
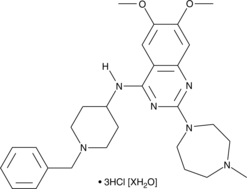
GC25159
BMF-219
BMF-219 est un nouvel inhibiteur de la menine puissant et irréversible, qui peut être utilisé dans le traitement de la leucémie.
-
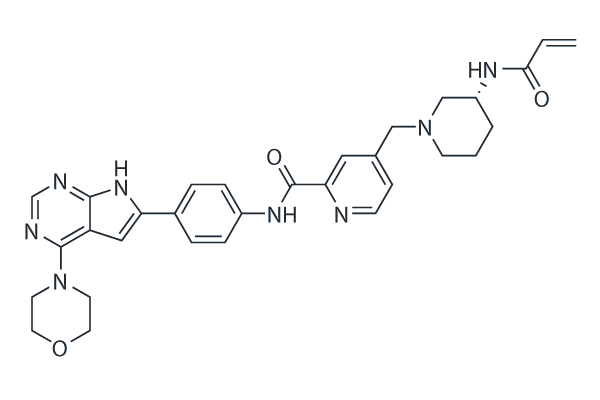
GC63731
BRD0639
BRD0639 est un inhibiteur de premier ordre de l'interaction adaptateur PRMT5-substrat. BRD0639 est un agent compétitif du motif de liaison PRMT5 (PBM) qui peut prendre en charge les études des activités PRMT5 dépendantes de PBM.
-
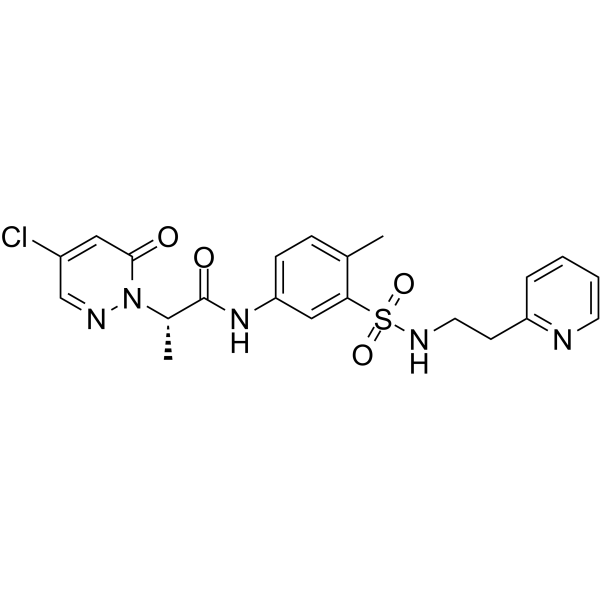
GC10259
BRD4770
BRD4770 est un inhibiteur de l'histone méthyltransférase G9a. BRD4770 réduit la di- et la triméthylation de la lysine 9 sur l'histone H3 (H3K9) avec une EC50 de 5 μM, et a moins ou peu d'effet sur H3K27me3, H3K36me3, H3K4me3 et H3K79me3. BRD4770 peut activer la voie de l'ataxie télangiectasie mutée (ATM) et induire la sénescence cellulaire.
-
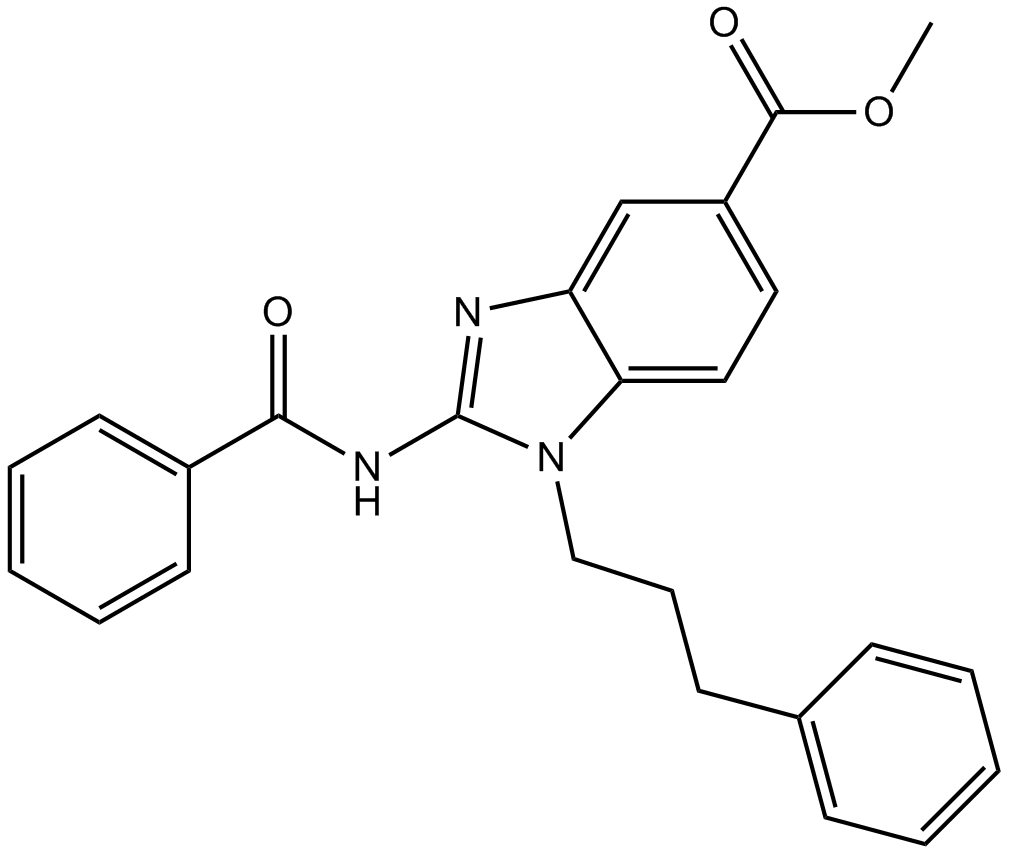
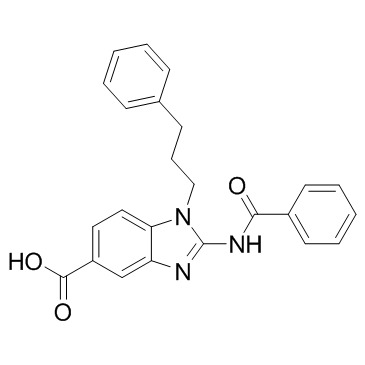
GC32988
BRD9539
BRD9539 est un inhibiteur de l'histone méthyltransférase G9a avec une IC50 de 6,3 μM. BRD9539 inhibe également l'activité de PRC2 et est inactif contre SUV39H1, NSD2 et DNMT1.
-
GC16130
C 21
Protein arginine methyltransferase 1 (PRMT1) inhibitor

-
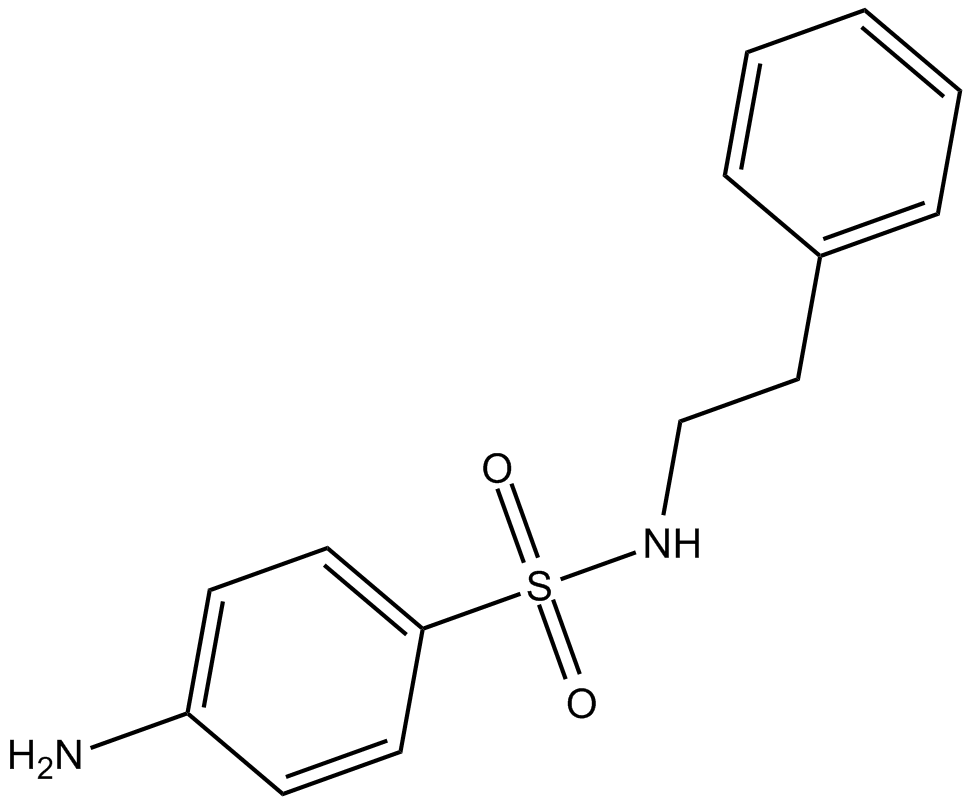
GC12005
C7280948
C7280948 est un inhibiteur sélectif et puissant de la protéine méthyltransférase1 (PRMT1) avec une valeur IC50 de 12,75 μM.
-
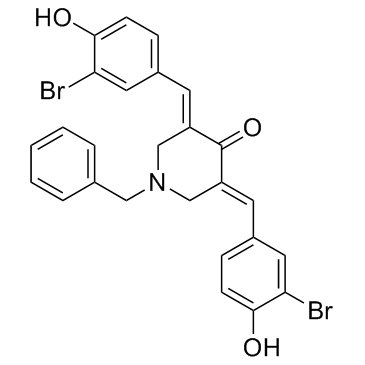
GC34108
CARM1-IN-1
CARM1-IN-1 est un inhibiteur puissant et spécifique de CARM1 (Coactivator-associated arginine methyltransferase 1) avec IC50 de 8,6 uM ; montre une très faible activité contre PRMT1 et SET7 (IC50 > 600 uM).
-
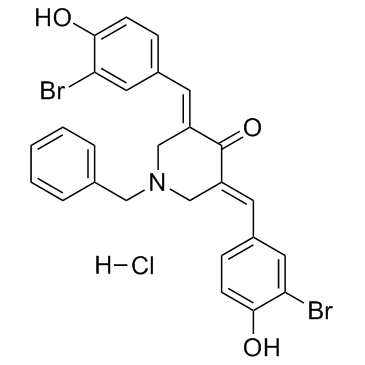
GC34424
CARM1-IN-1 hydrochloride
Le chlorhydrate de CARM1-IN-1 est un inhibiteur puissant et spécifique de CARM1 (coactivator-associated arginine methyltransferase 1) avec IC50 de 8,6 uM ; montre une très faible activité contre PRMT1 et SET7 (IC50 > 600 uM).
-
GC18949
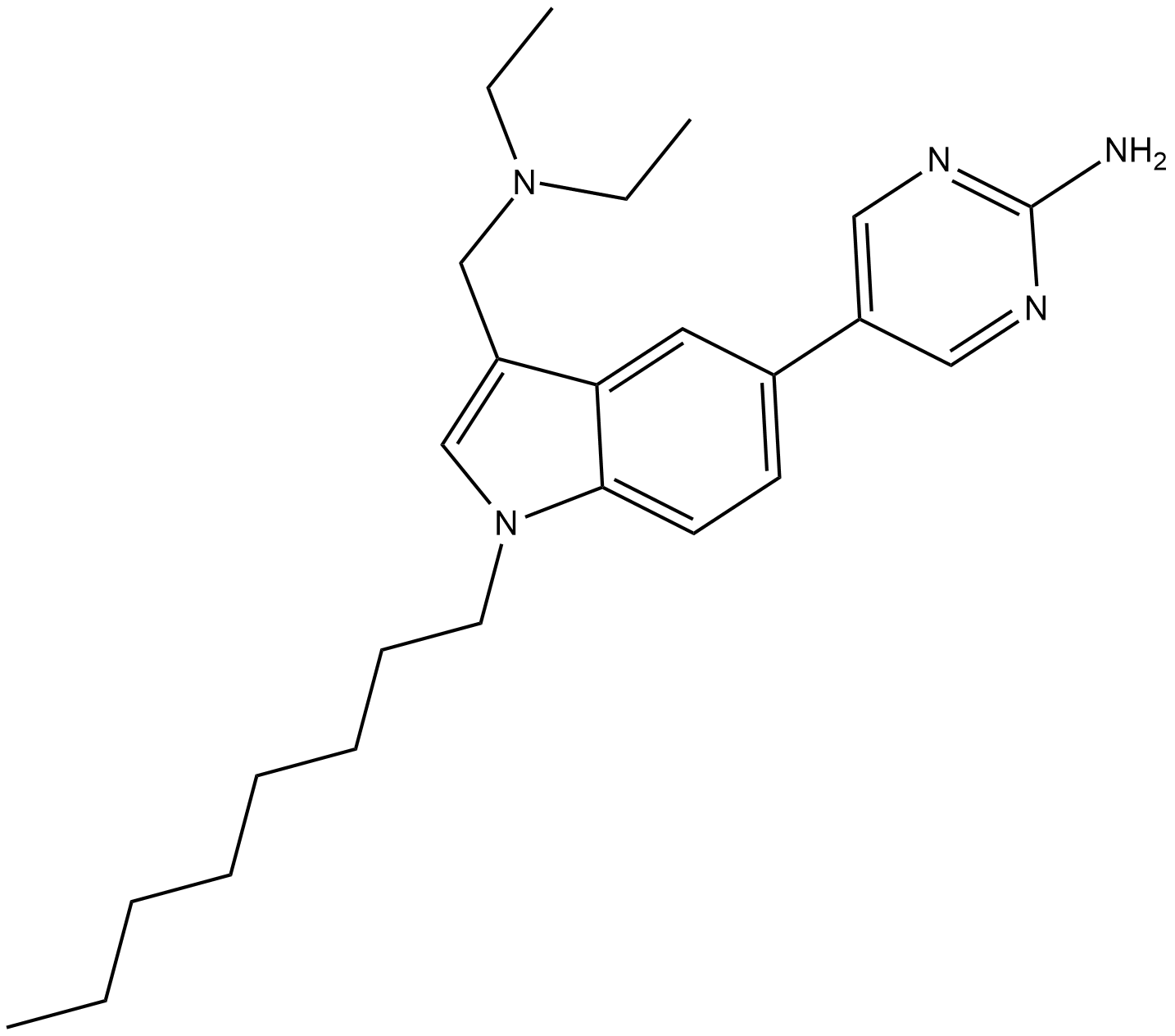
CAY10677
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.
-
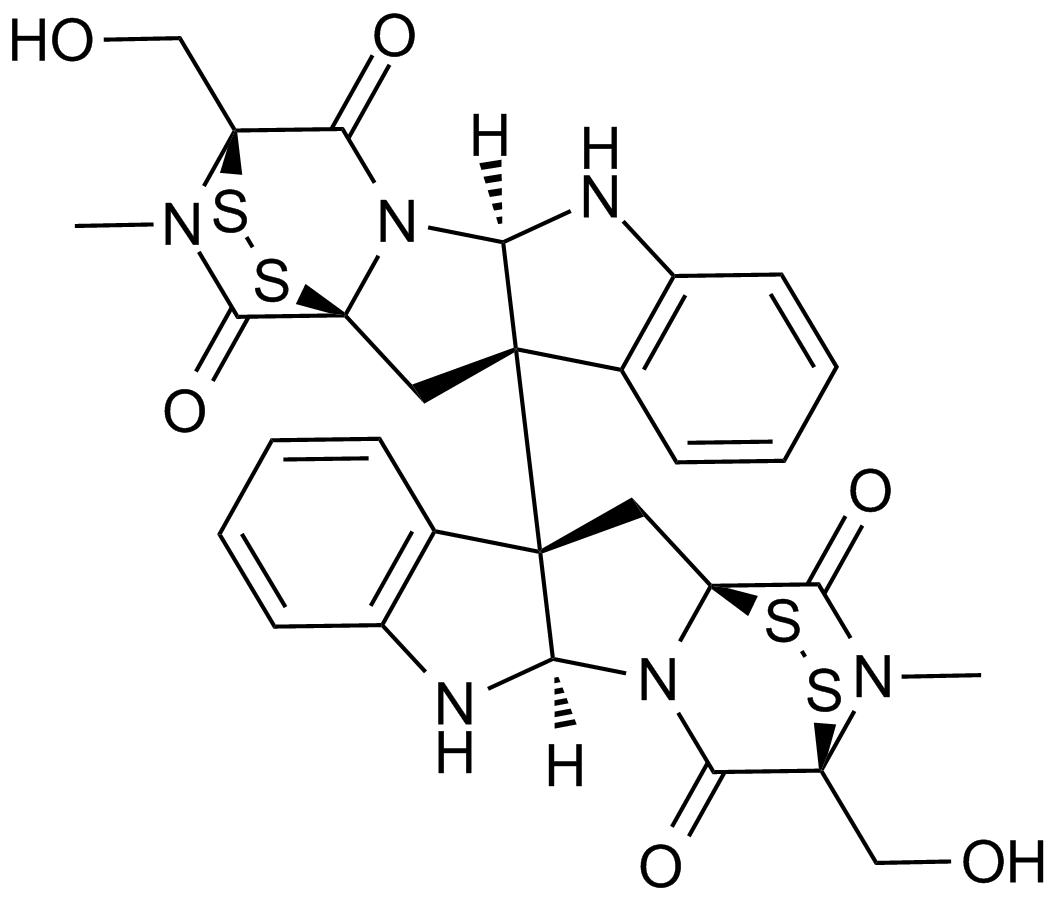
GC14936
Chaetocin
Inhibitor of lys9-specific HMTs
-
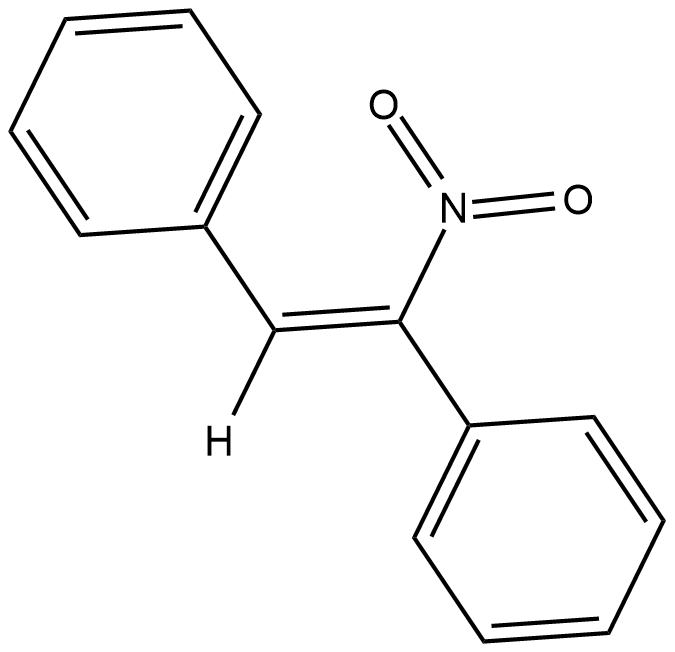
GC18577
CID-2818500
An inhibitor of PRMT1
-
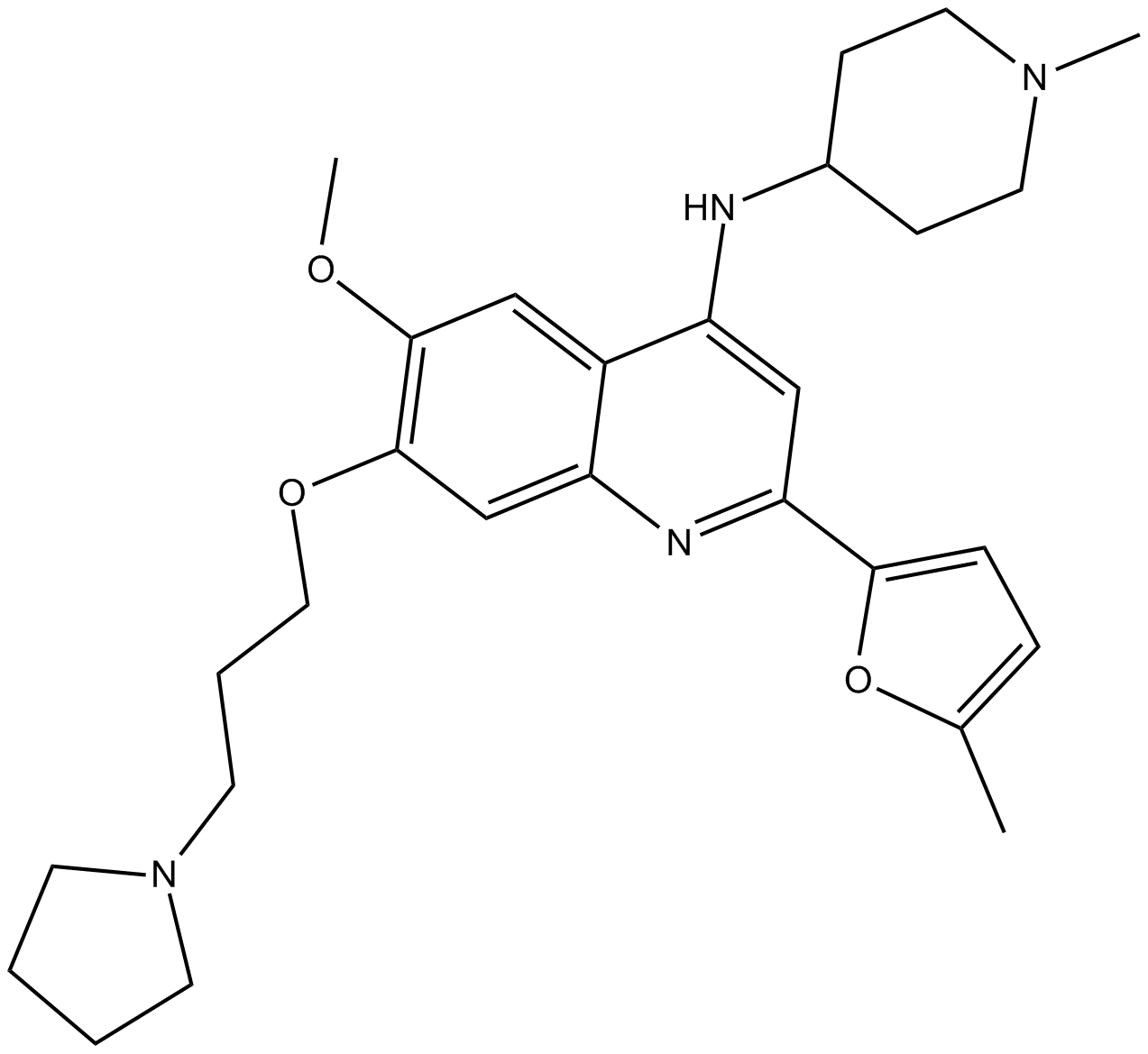
GC12367
CM-272
Le CM-272 est un inhibiteur double G9a/ADN méthyltransférases (DNMT) de premier ordre, puissant, sélectif, compétitif avec le substrat et réversible, doté d'activités antitumorales. CM-272 inhibe G9a, DNMT1, DNMT3A, DNMT3B et GLP avec des IC50 de 8 nM, 382 nM, 85 nM, 1200 nM et 2 nM, respectivement. Le CM-272 inhibe la prolifération cellulaire et favorise l'apoptose, induisant des gènes stimulés par l'IFN et la mort cellulaire immunogène.
-
GC33320
CM-579
Le CM-579 est un double inhibiteur réversible de premier ordre de G9a et de DNMT, avec des valeurs IC50 de 16 nM, 32 nM pour G9a et DNMT, respectivement. Possède une puissante activité cellulaire in vitro dans un large éventail de cellules cancéreuses.
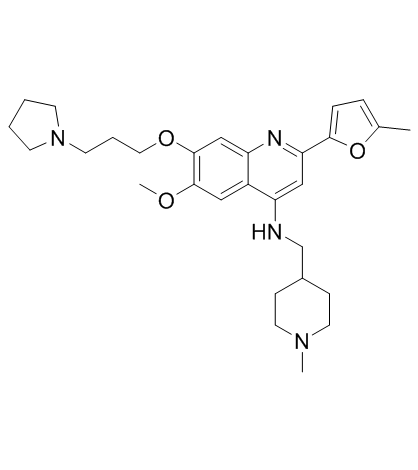
-
GC35714
CM-579 trihydrochloride
Le trichlorhydrate de CM-579 est un double inhibiteur réversible de premier ordre de G9a et de DNMT, avec des valeurs IC50 de 16 nM, 32 nM pour G9a et DNMT, respectivement. Possède une puissante activité cellulaire in vitro dans un large éventail de cellules cancéreuses.
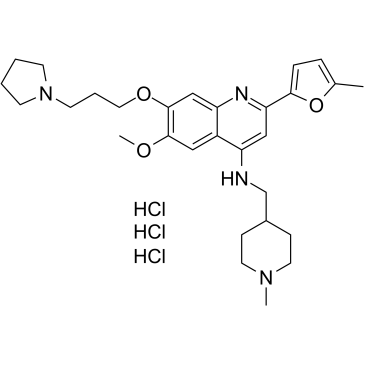
-
GC39665
CMP-5
Le CMP-5 est un inhibiteur puissant, spécifique et sélectif de PRMT5, alors qu'il n'affiche aucune activité contre les enzymes PRMT1, PRMT4 et PRMT7. Le CMP-5 bloque sélectivement S2Me-H4R3 en inhibant l'activité de PRMT5 méthyltransférase sur les préparations d'histones. Le CMP-5 empêche la transformation des lymphocytes B induite par le virus d'Epstein-Barr (EBV), mais laisse les cellules B normales inchangées.
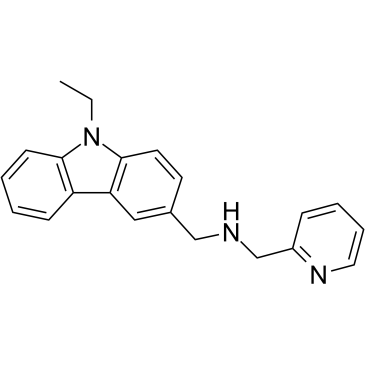
-
GC16298
CPI-1205
EZH2 inhibitor
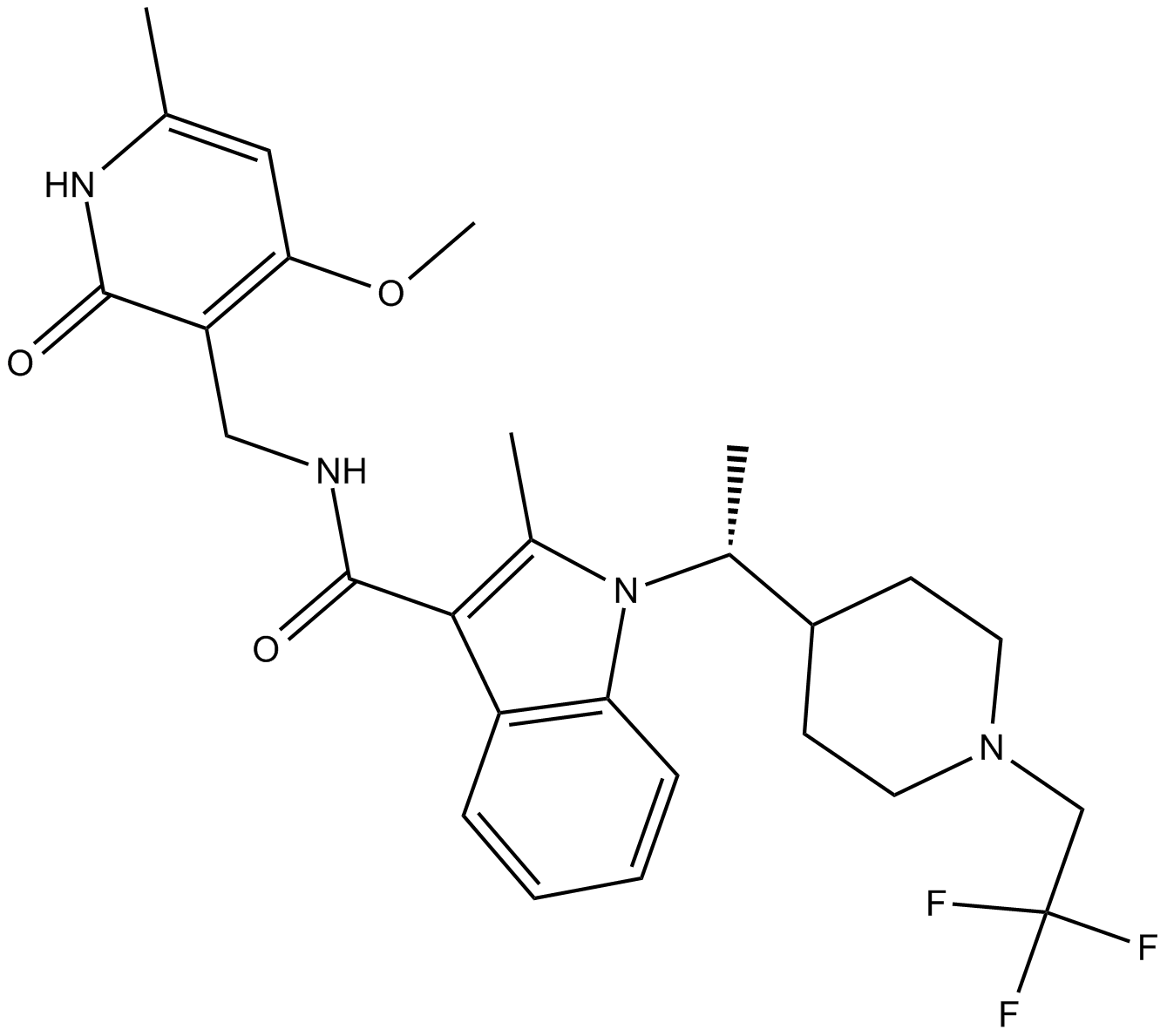
-
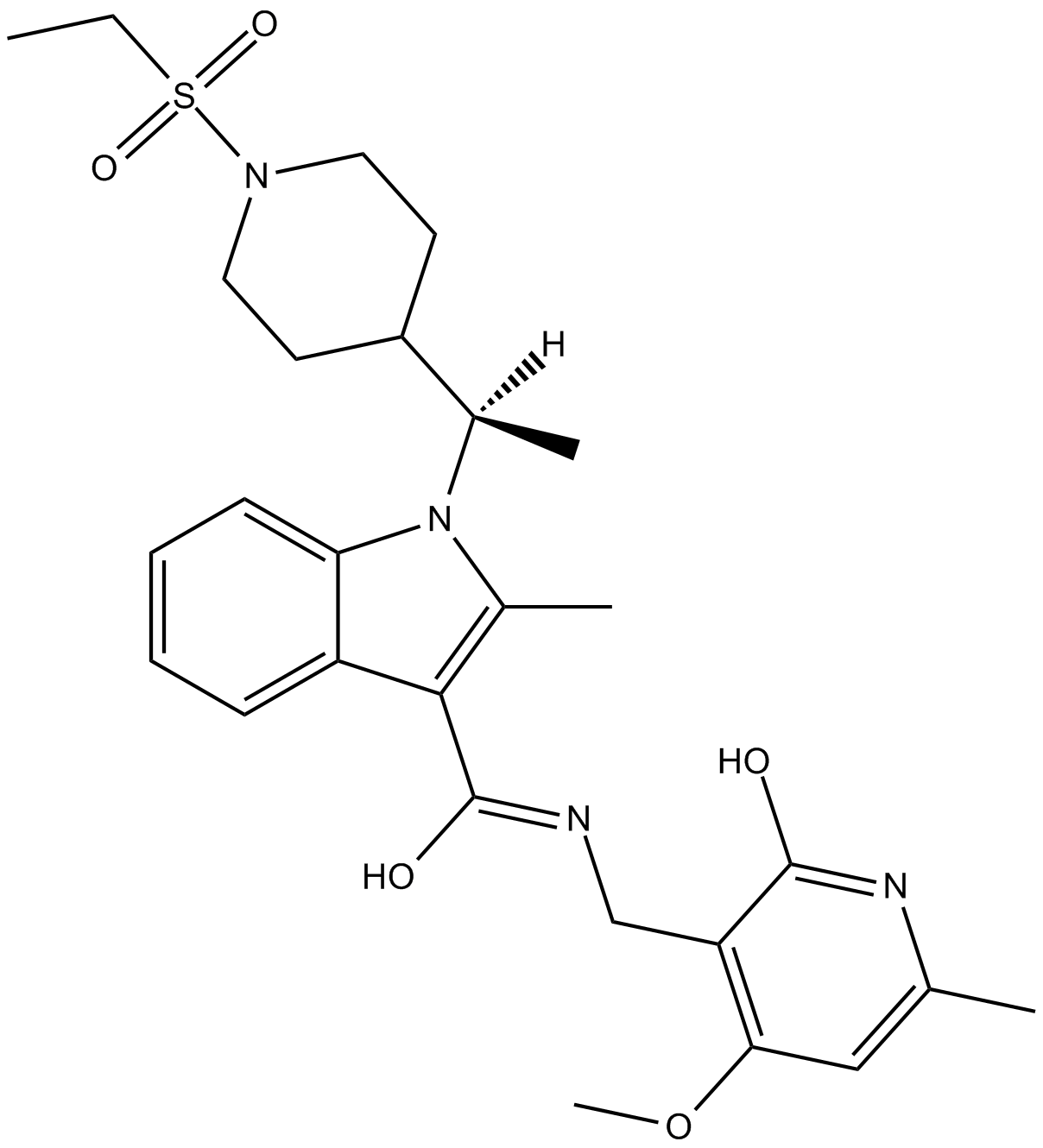
GC16599
CPI-169
EZH2 inhibitor
-
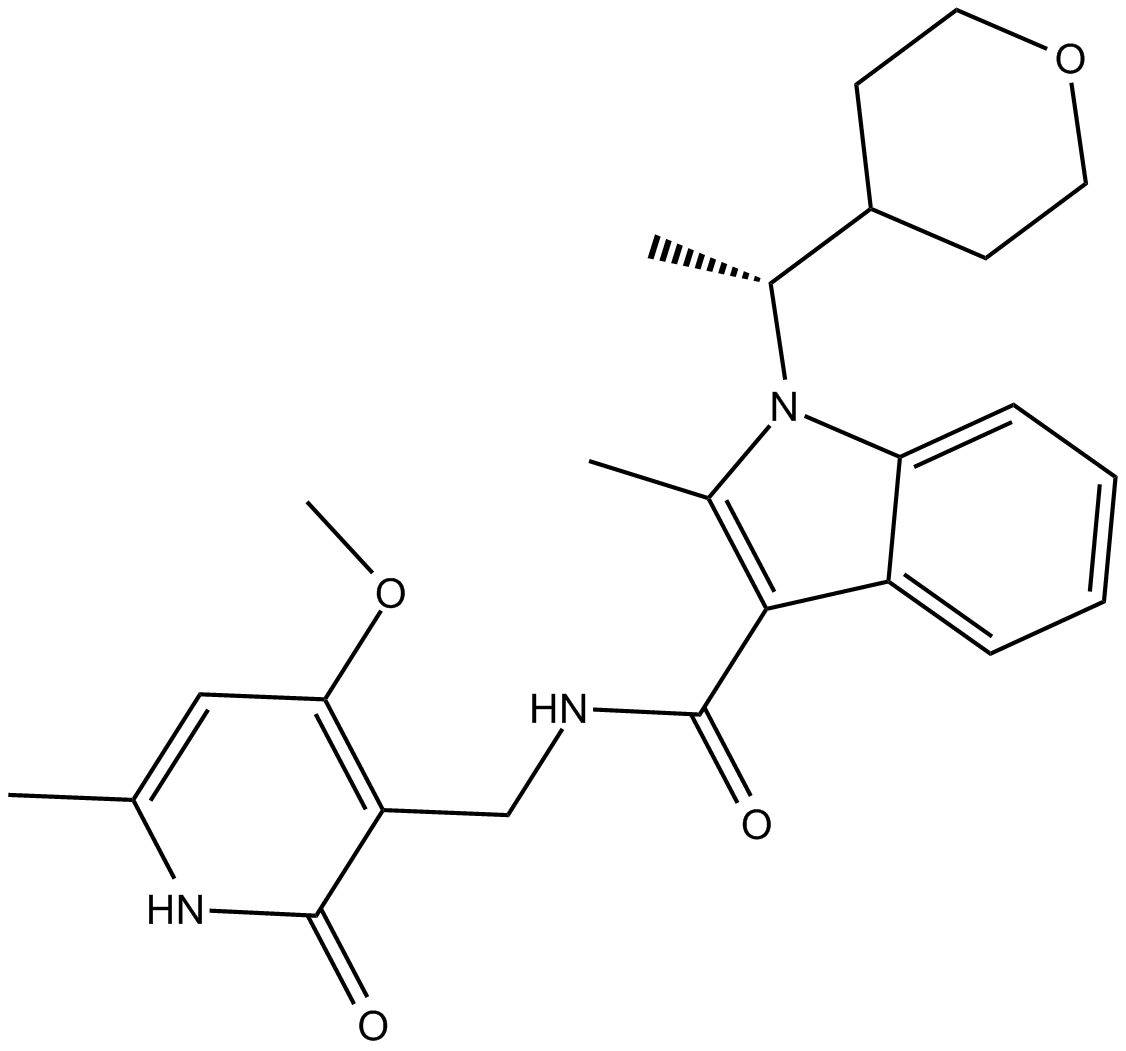
GC10021
CPI-360
EZH2 inhibitor
-
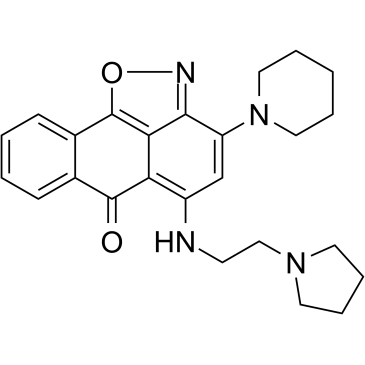
GC35742
CPUY074020
CPUY074020 est un inhibiteur biodisponible puissant et oral de l'histone méthyltransférase G9a, avec une IC50 de 2,18 μM. CPUY074020 possède une activité anti-proliférative.
-
GC43354
Cysmethynil
Post-translational protein prenylation is a 3-step process that occurs at the C-terminus of a number of proteins involved in cell growth control and oncogenesis.

-
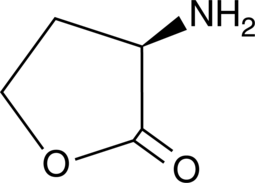
GC48967
D-Homoserine lactone
An enantiomer of L-homoserine lactone
-
GC65186
DC-S239
Le DC-S239 est un inhibiteur sélectif de l'histone méthyltransférase SET7 avec une valeur IC50 de 4,59 μM. DC-S239 affiche également la sélectivité pour DNMT1, DOT1L, EZH2, NSD1, SETD8 et G9a. DC-S239 a une activité anticancéreuse.
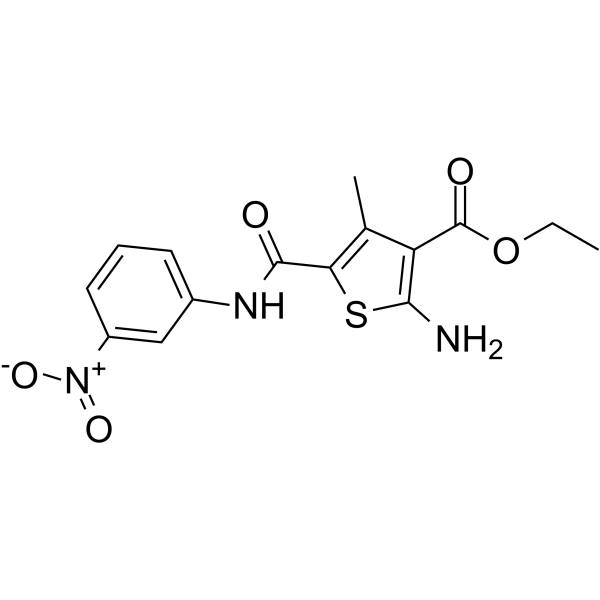
-
GC63662
DCLX069
DCLX069 est un inhibiteur sélectif de la protéine arginine méthyltransférase 1 (PRMT1) avec une valeur IC50 de 17,9 μM. DCLX069 se montre moins actif contre PRMT4 et PRMT6. DCLX069 a des effets anticancéreux.
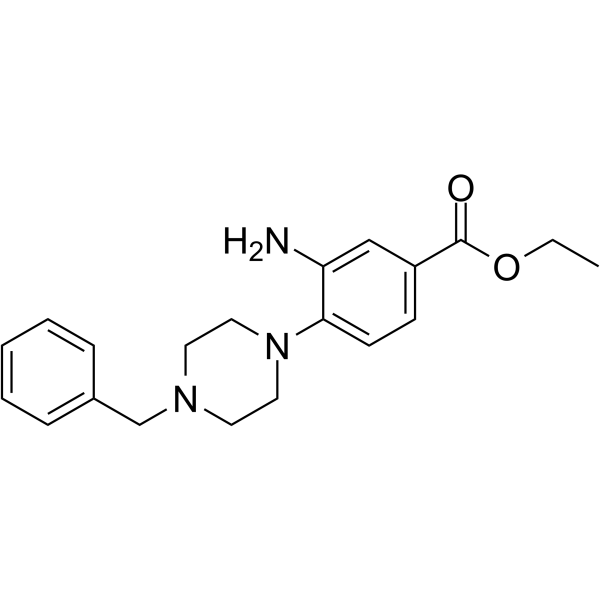
-
GC35816
DC_C66
DC_C66 est un inhibiteur sélectif de l'arginine méthyltransférase 1 (CARM1) associé À un coactivateur sélectif et perméable aux cellules avec une IC50 de 1,8 μM. DC_C66 a une bonne sélectivité pour CARM1 contre PRMT1 (IC50 = 21 μM), PRMT6 (IC50 = 47 μM) et PRMT5.
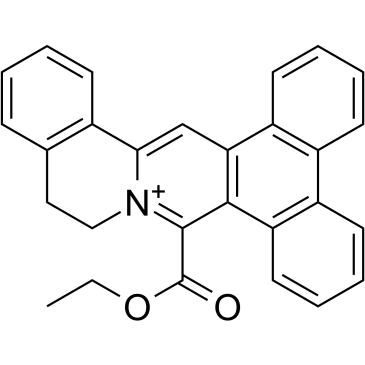
-
GC67863
DDO-2093 dihydrochloride

-
GC33208
Dot1L-IN-1
Dot1L-IN-1 est un inhibiteur Dot1L très puissant, sélectif et structurellement nouveau avec un Ki de 2 pM.
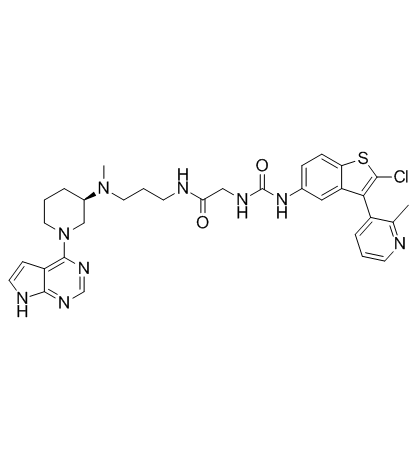
-
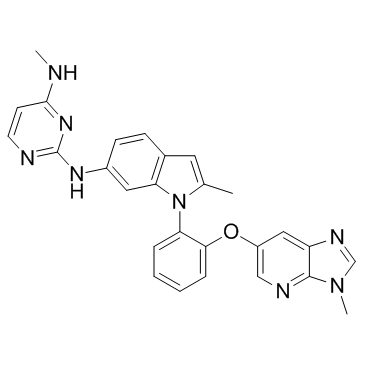
GC30530
Dot1L-IN-2
Dot1L-IN-2 est un inhibiteur puissant, sélectif et biodisponible par voie orale de Dot1L (une histone méthyltransférase), avec une IC50 et un Ki de 0,4 nM et 0,08 nM, respectivement.
-
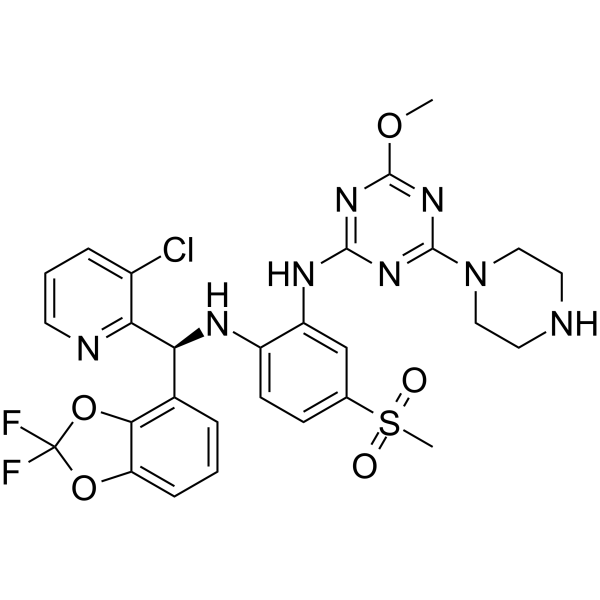
GC62613
Dot1L-IN-4
Dot1L-IN-4 est un puissant perturbateur de l'inhibiteur de la protéine de type 1 de silenÇage télomérique (DOT1L) avec une IC50 SPA DOT1L de 0,11 nM.
-
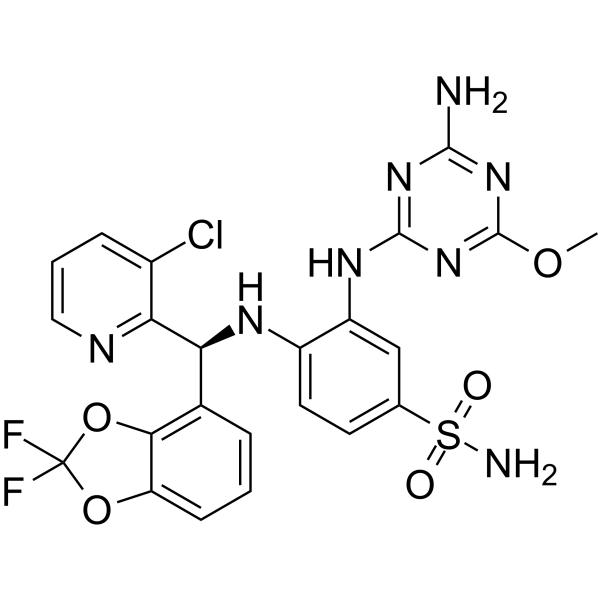
GC65962
Dot1L-IN-5
Dot1L-IN-5 est un puissant perturbateur de l'inhibiteur de la protéine de type 1 de silenÇage télomérique (DOT1L) avec une IC50 SPA DOT1L de 0,17 nM.
-
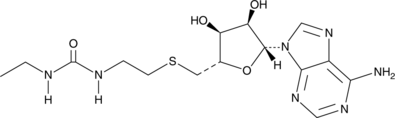
GC45927
DS-437
Le DS-437 est un double inhibiteur de PRMT5/7 (CI50 de PRMT5/7 = 6 μM).
-
GC35914
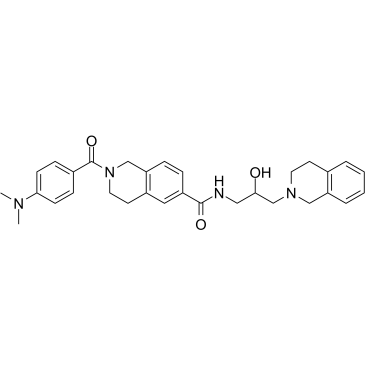
DW14800
DW14800 est un inhibiteur de la protéine arginine méthyltransférase 5 (PRMT5), avec une IC50 de 17 nM. DW14800 réduit les niveaux de H4R3me2s et améliore la transcription de HNF4α, mais ne modifie pas l'expression de PRMT5. Activité anticancéreuse.
-
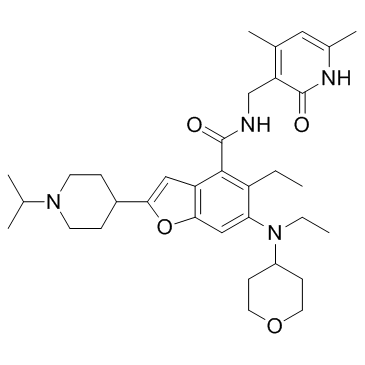
GC33220
EBI-2511
EBI-2511 est un inhibiteur EZH2 très puissant et actif par voie orale, avec une IC50 de 6 nM dans les lignées cellulaires Pfeffiera, respectivement.
-
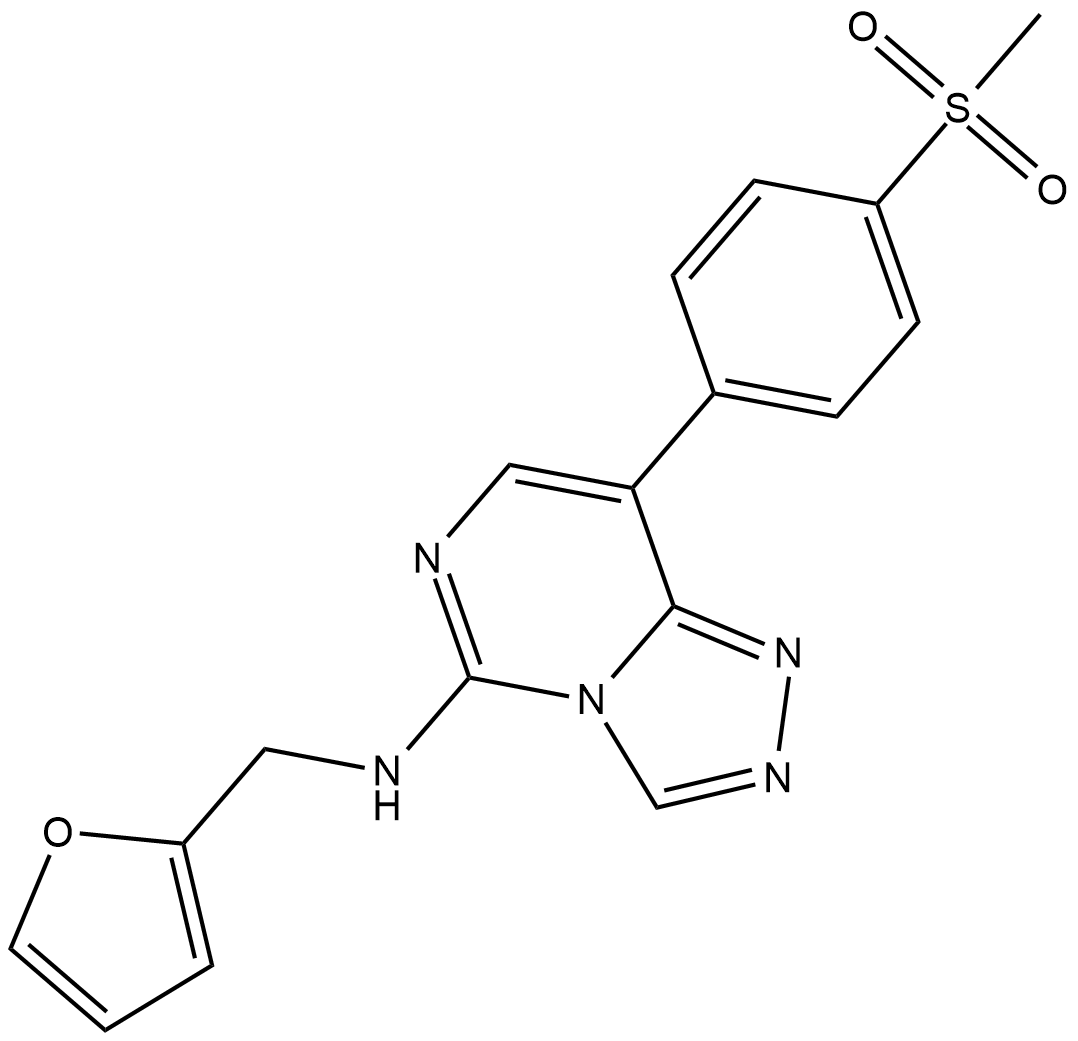
GC19130
EED226
EED226 est un inhibiteur du complexe répressif polycomb 2 (PRC2), qui se lie À la poche K27me3 lors du développement de l'ectoderme embryonnaire (EED) et présente une forte activité antitumorale dans le modèle de souris xénogreffe. L'EED226 est un inhibiteur de l'EED puissant, sélectif et biodisponible par voie orale. EED226 inhibe PRC2 avec une IC50 de 23,4 nM lorsque le peptide H3K27me0 est utilisé comme substrat dans les essais enzymatiques in vitro.
-
GC67934
EEDi-5285
-
GC34567
EHMT2-IN-1
EHMT2-IN-1 est un puissant inhibiteur de l'EHMT, avec des IC50 de tous <100 nM pour le peptide EHMT1, le peptide EHMT2 et l'EHMT2 cellulaire. Utilisé dans la recherche de troubles sanguins ou de cancer.
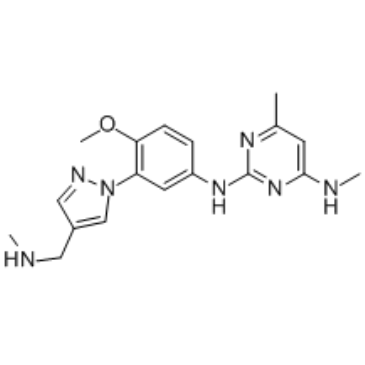
-
GC34568
EHMT2-IN-2
EHMT2-IN-2 est un puissant inhibiteur de l'EHMT, avec des IC50 de tous <100 nM pour le peptide EHMT1, le peptide EHMT2 et l'EHMT2 cellulaire. Utilisé dans la recherche d'une maladie du sang ou d'un cancer.
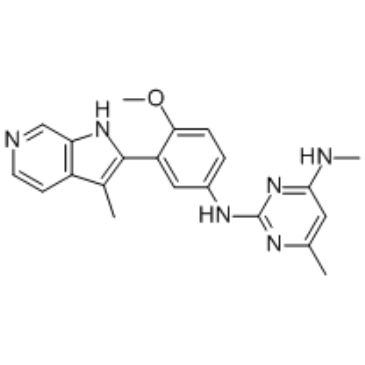
-
GC14756
EI1
EI1 (KB-145943) est un inhibiteur EZH2 puissant et sélectif avecIC50de 15nM et 13nM pour EZH2 (WT) et EZH2 (Y641F), respectivement.
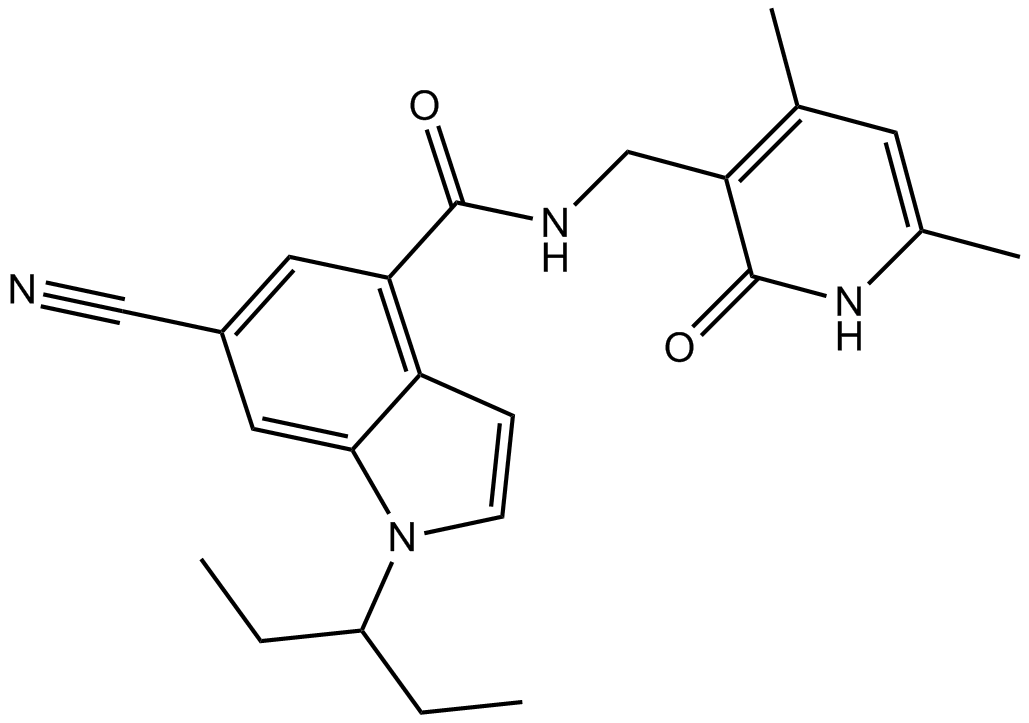
-
GC17334
Entacapone
A reversible COMT inhibitor
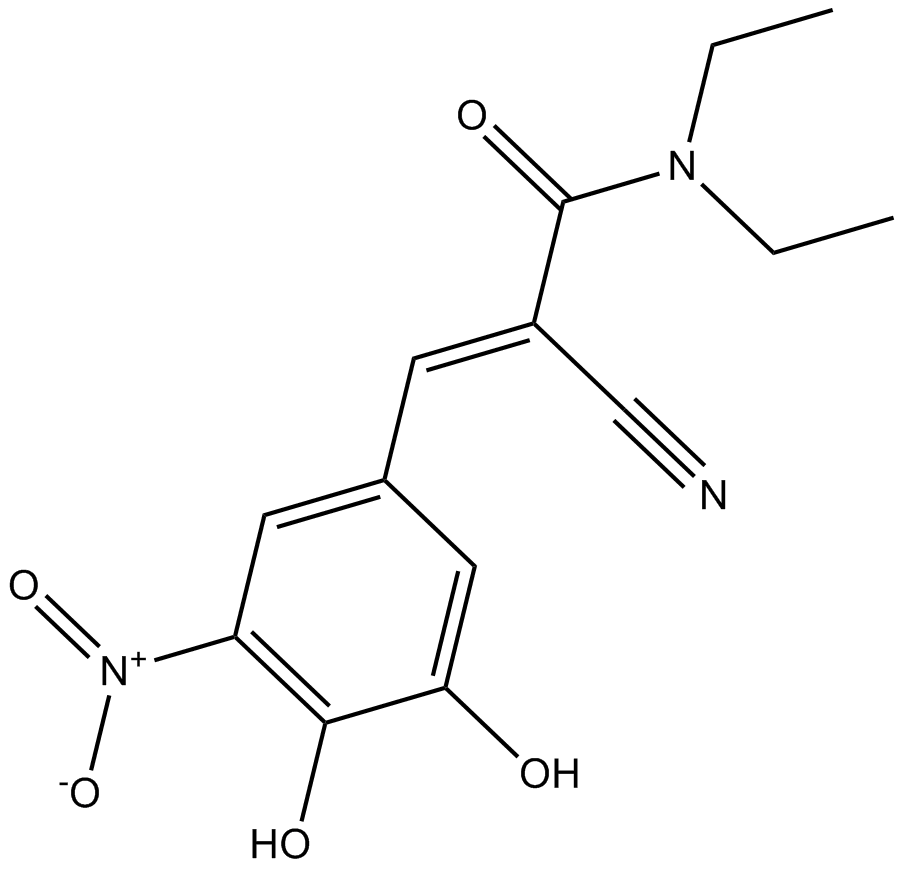
-
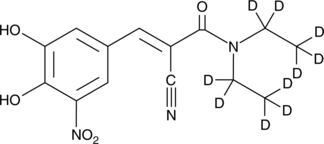
GC47294
Entacapone-d10
Entacapone-d10 est le deutérium marqué Entacapone.
-
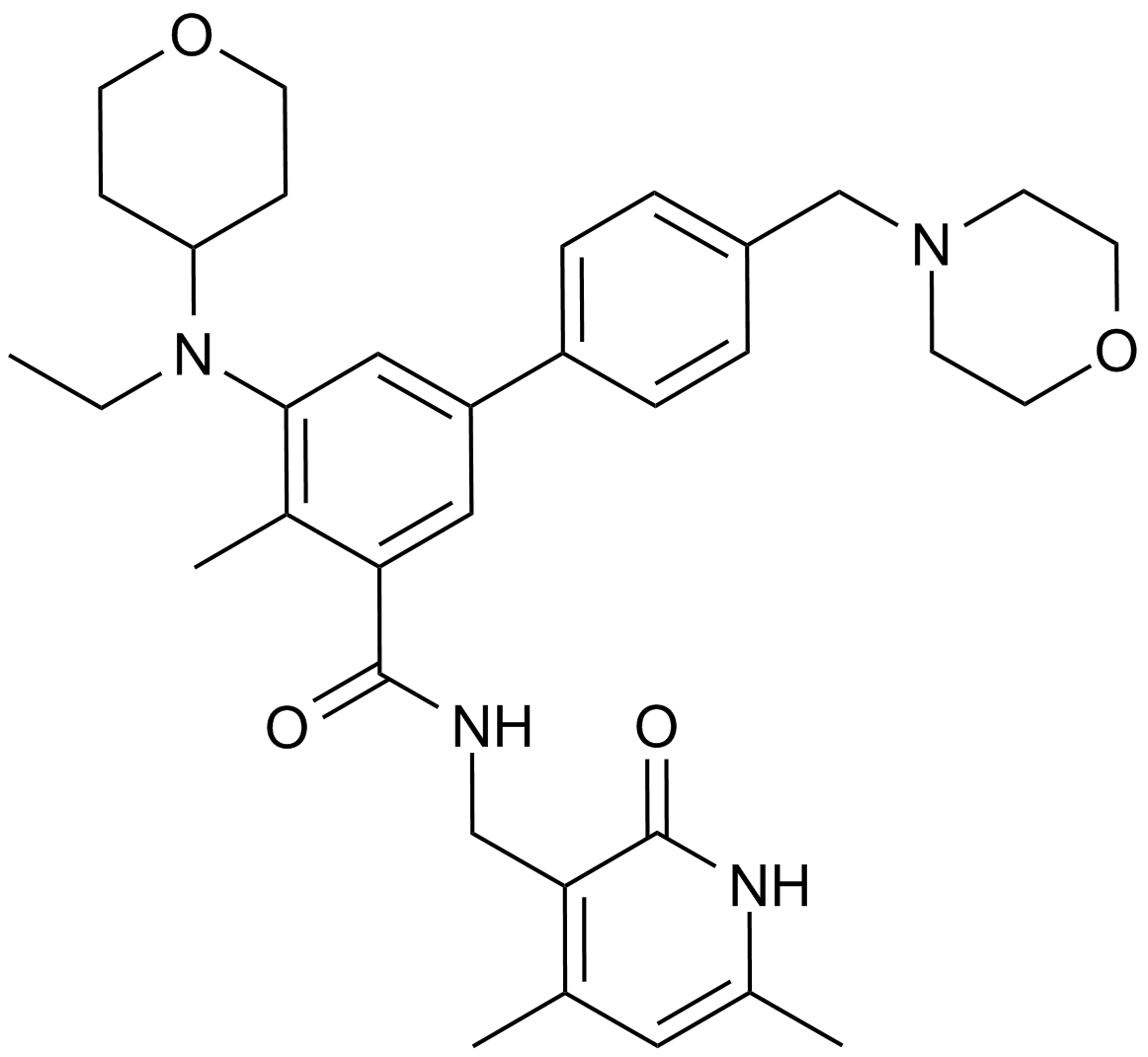
GC14062
EPZ-6438
A EZH2 inhibitor,potent and selective
-
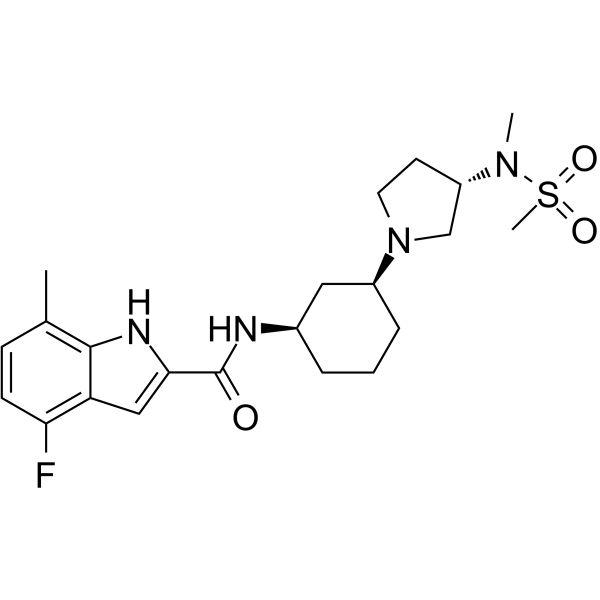
GC64343
EPZ-719
EPZ-719 est un nouvel inhibiteur puissant de SETD2 (IC50 = 0,005 μM) avec une sélectivité élevée par rapport aux autres histones méthyltransférases.
-
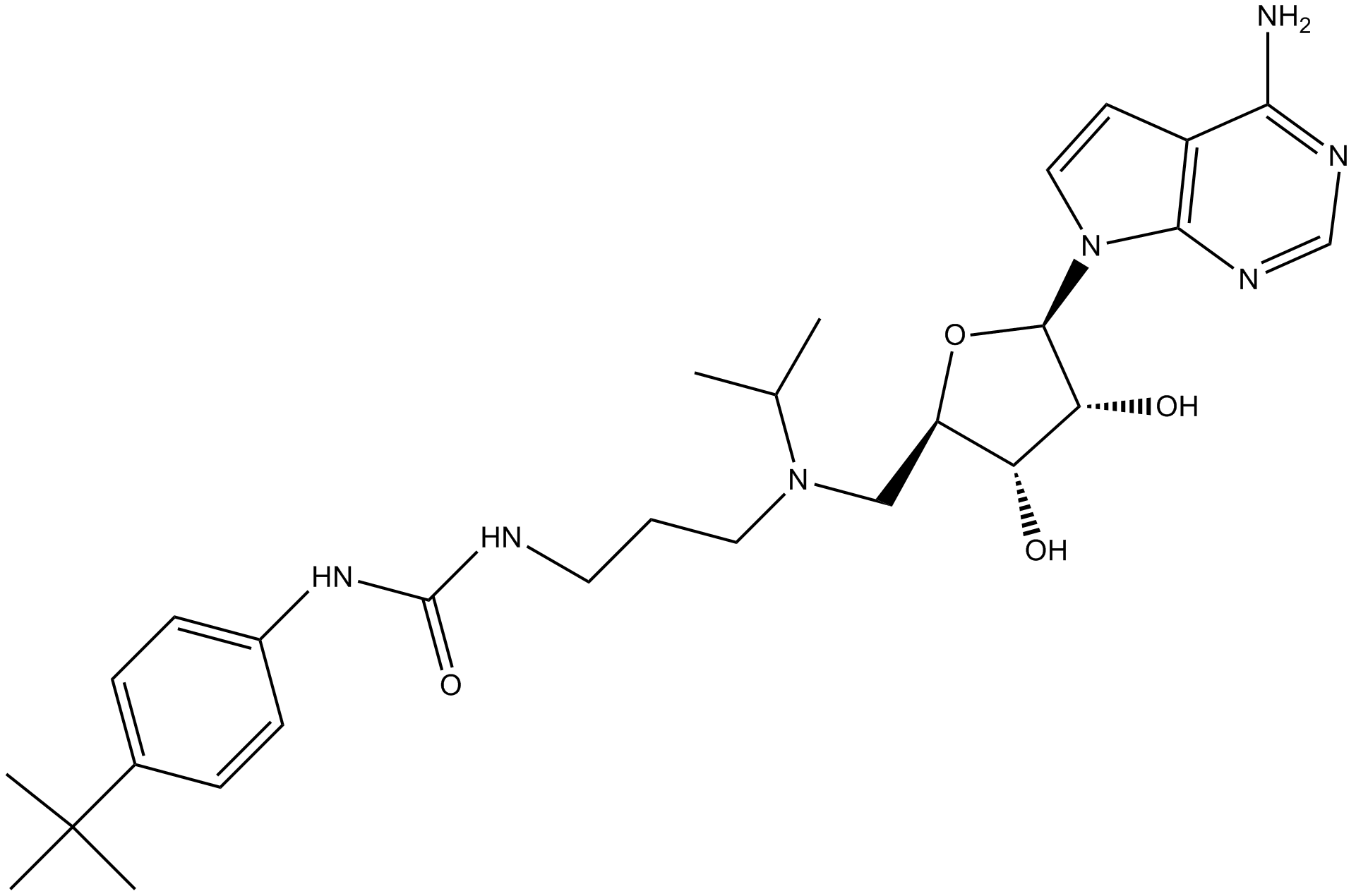
GC13383
EPZ004777
A potent inhibitor of DOT1L
-
GC48980
EPZ004777 (formate)
A potent inhibitor of DOT1L

-
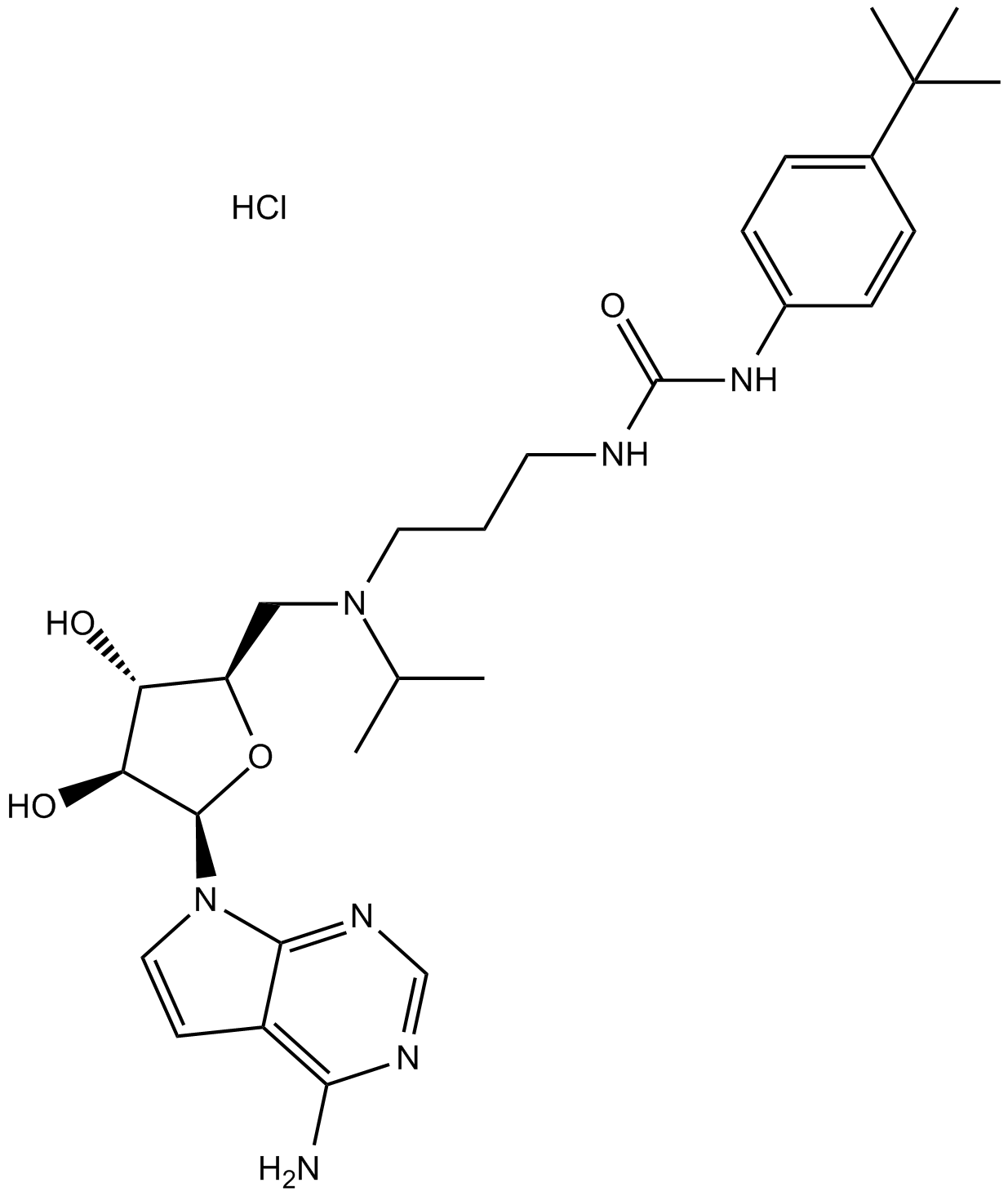
GC15259
EPZ004777 HCl
EPZ004777 HCl est un puissant inhibiteur sélectif de DOT1L avec une IC50 de 0,4 nM.
-
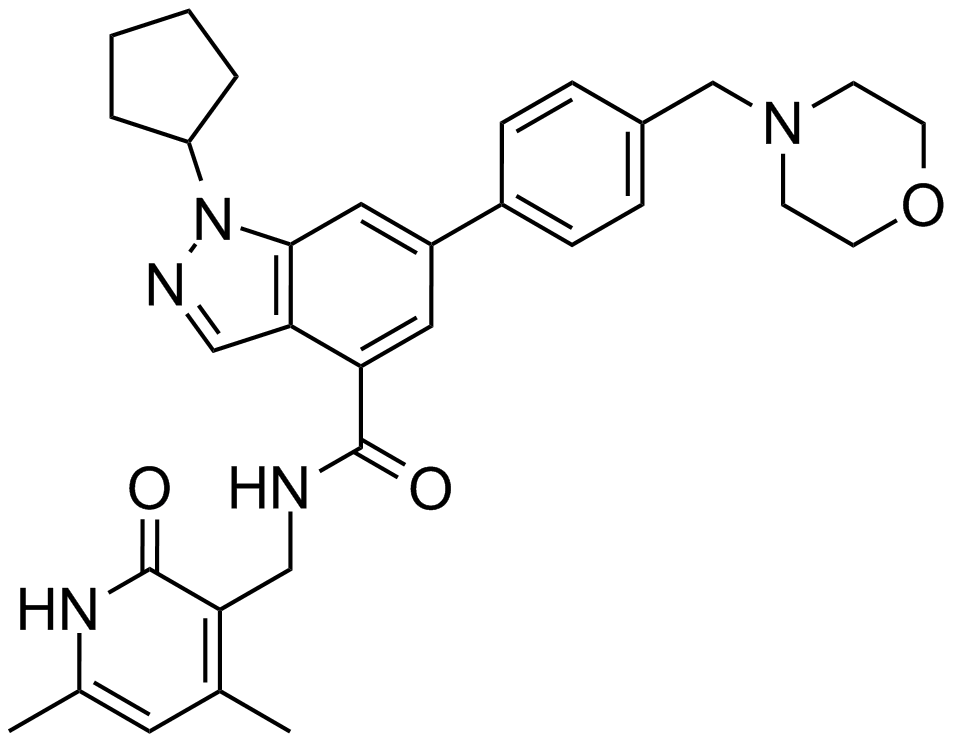
GC13878
EPZ005687
A potent, selective inhibitor of EZH2
-
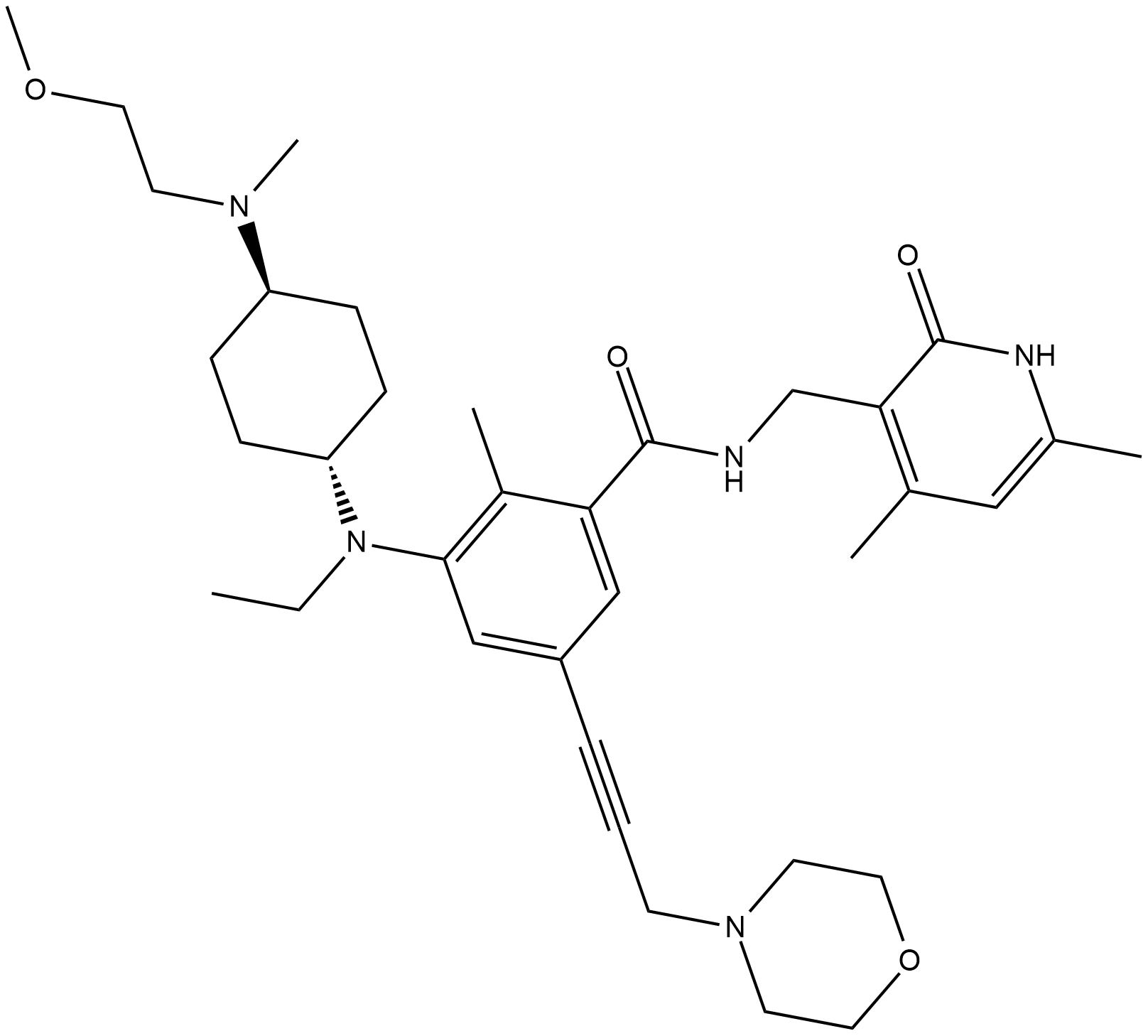
GC19141
EPZ011989
EPZ011989 est un inhibiteur Zeste Homolog 2 (EZH2) puissant et actif par voie orale avec une stabilité métabolique. EPZ011989 a une inhibition inhibitrice pour EZH2 avec une valeur Ki <3 nM. EPZ011989 montre une inhibition robuste de la marque méthyle et une activité anti-tumorale. EPZ011989 peut être utilisé pour la recherche de divers cancers.
-
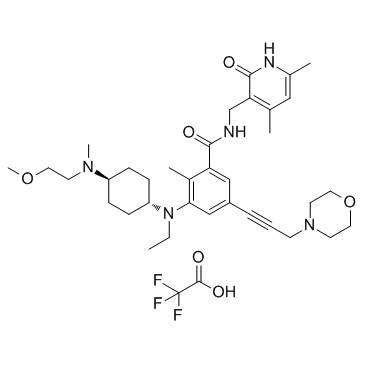
GC34136
EPZ011989 trifluoroacetate (EPZ-011989 trifluoroacetate)
Le trifluoroacétate EPZ-011989 est un inhibiteur de Zeste Homolog 2 (EZH2) puissant et actif par voie orale avec une stabilité métabolique. Le trifluoroacétate EPZ-011989 a une inhibition inhibitrice pour EZH2 avec une valeur Ki <3 nM. Le trifluoroacétate EPZ-011989 présente une inhibition robuste de la marque méthyle et une activité anti-tumorale. Le trifluoroacétate EPZ-011989 peut être utilisé pour la recherche de divers cancers.
-
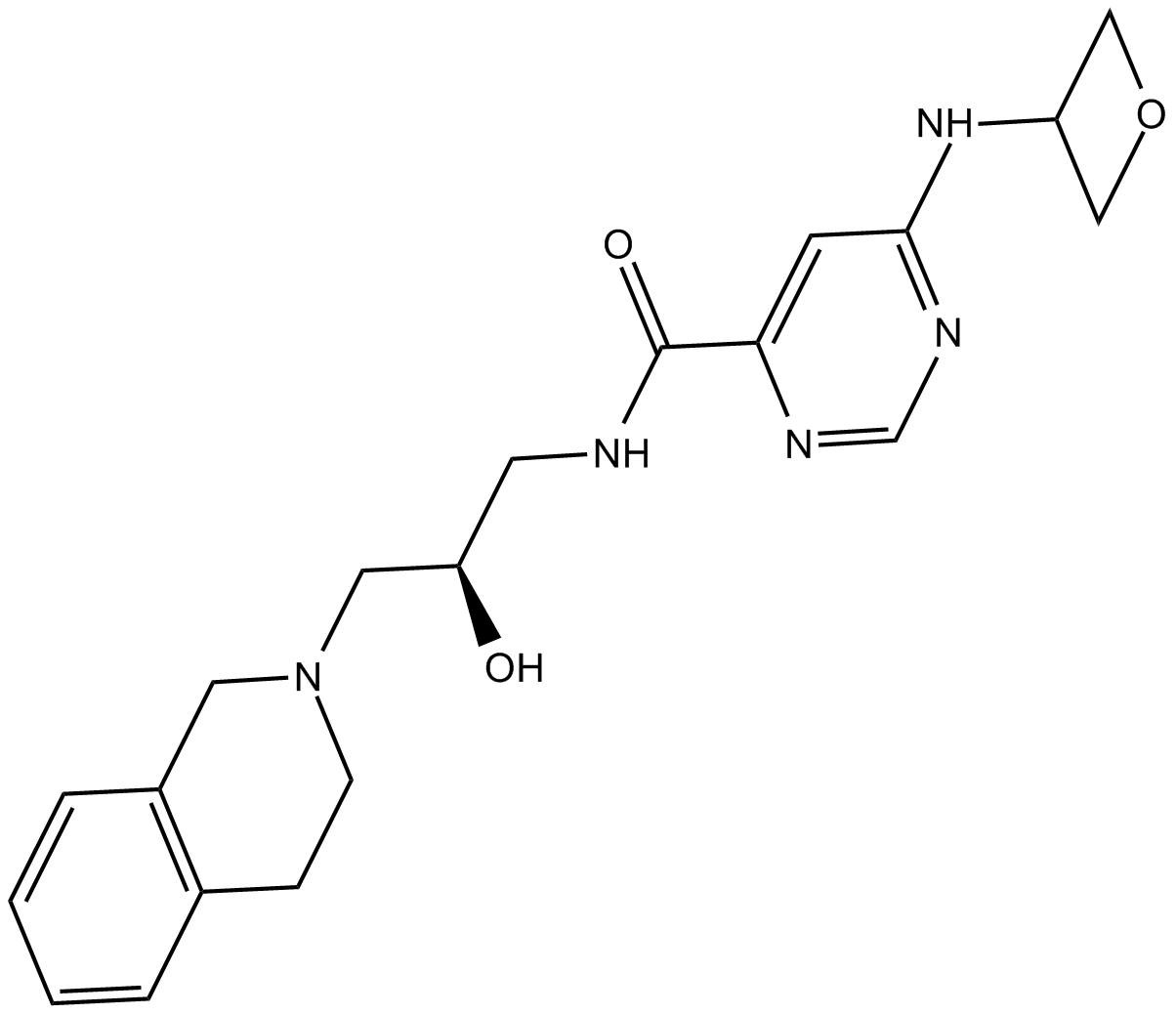
GC15302
EPZ015666
EPZ015666 (GSK3235025) est un inhibiteur de PRMT5 disponible par voie orale avec une IC50 de 22 nM.
-
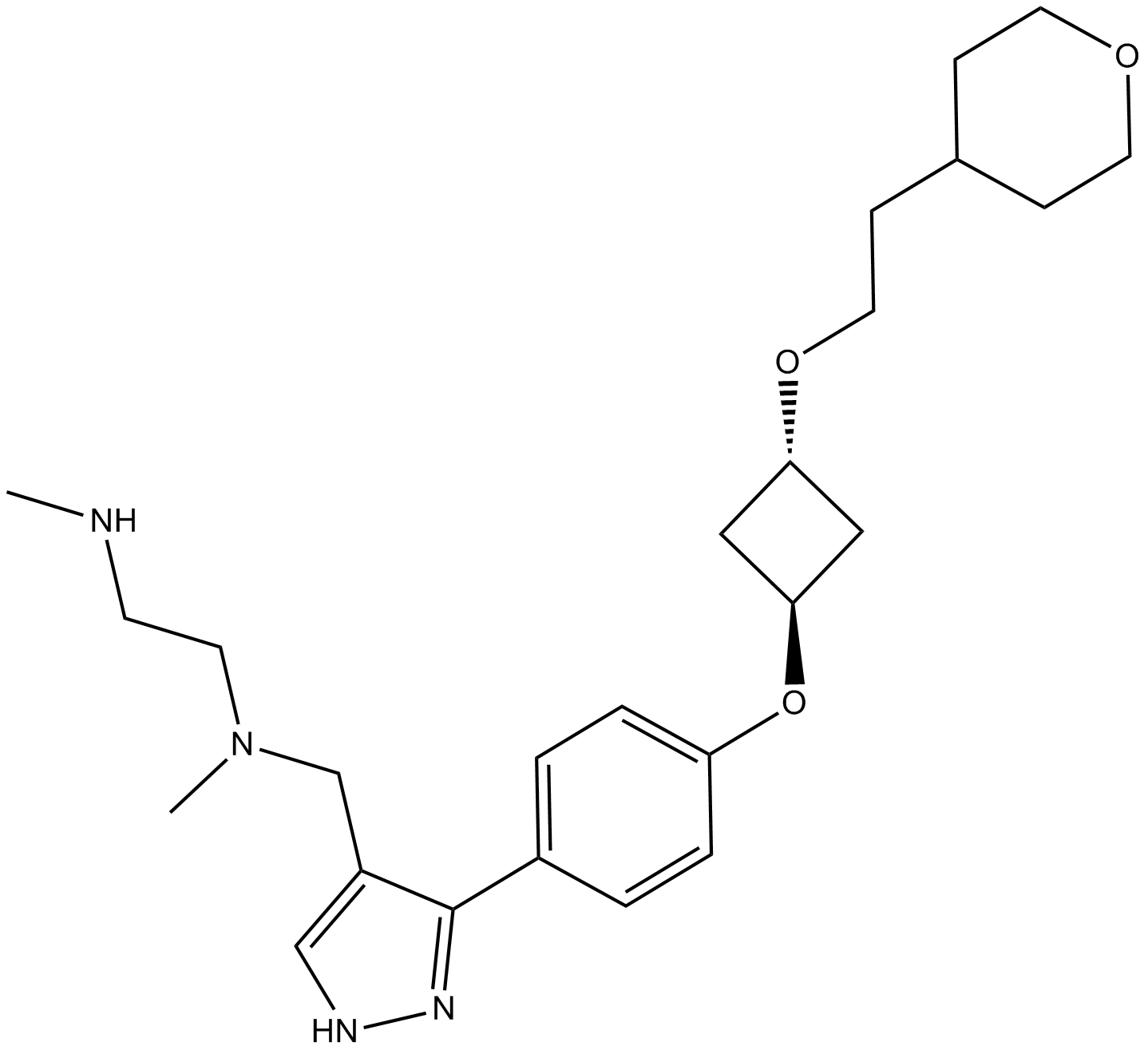
GC15102
EPZ020411
EPZ020411 est un inhibiteur sélectif de PRMT6 avec une IC50 de 10 nM, a une sélectivité >10 fois pour PRMT6 par rapport À PRMT1 et PRMT8. EPZ020411 peut être utilisé pour la recherche sur le cancer.
-
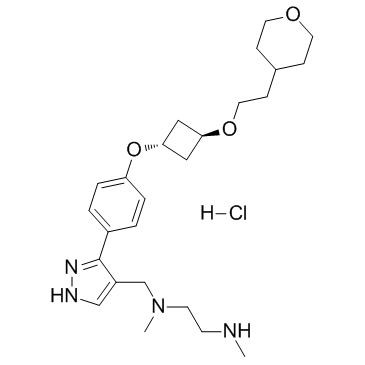
GC36000
EPZ020411 hydrochloride
Le chlorhydrate d'EPZ020411 est un inhibiteur sélectif de PRMT6 avec une IC50 de 10 nM, il a une sélectivité >10 fois pour PRMT6 par rapport À PRMT1 et PRMT8. Le chlorhydrate d'EPZ020411 peut être utilisé pour la recherche sur le cancer.
-
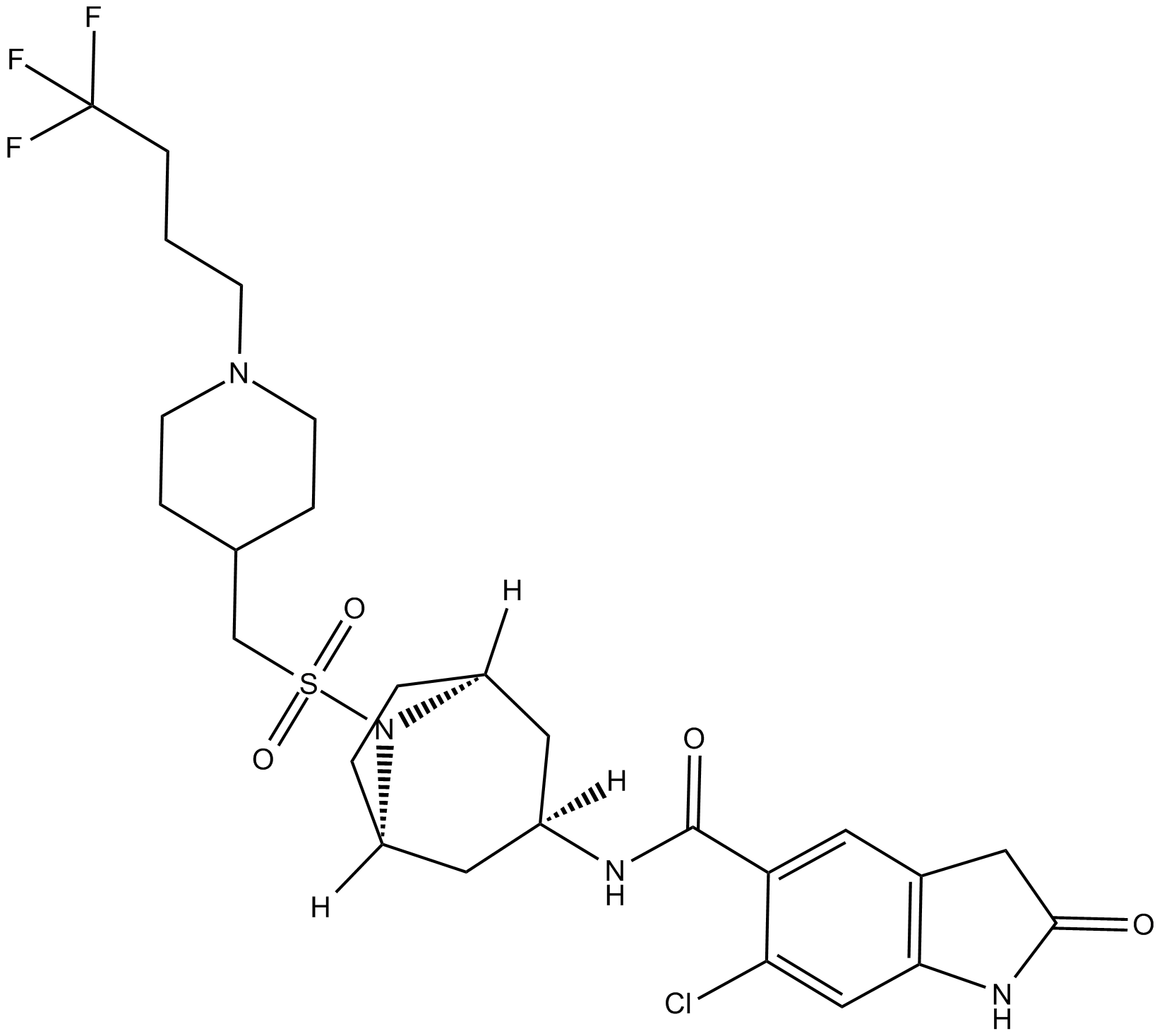
GC16224
EPZ031686
EPZ031686 est un inhibiteur SMYD3 puissant et actif par voie orale et avec une valeur IC50 de 3 nM. EPZ031686 peut être utilisé pour la recherche sur le cancer.
-
GC12932
EPZ5676
A highly potent DOT1L inhibitor
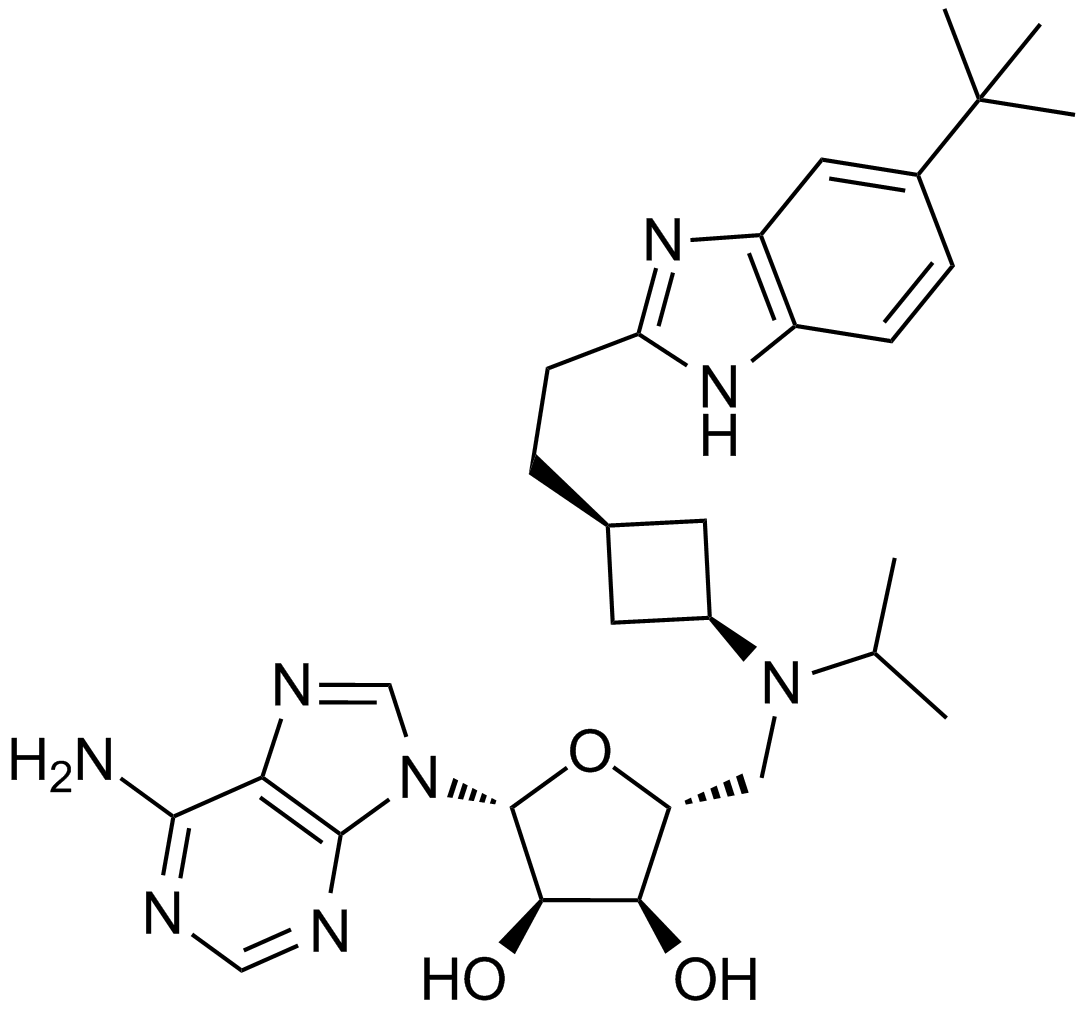
-
GC65980
EZH2-IN-13
EZH2-IN-13 est un puissant inhibiteur d'EZH2, pour plus de détails, veuillez vous référer au composé 73 dans le brevet WO2017139404. EZH2-IN-13 peut être utilisé pour étudier les cancers ou les lésions précancéreuses associés À l'activité EZH2.
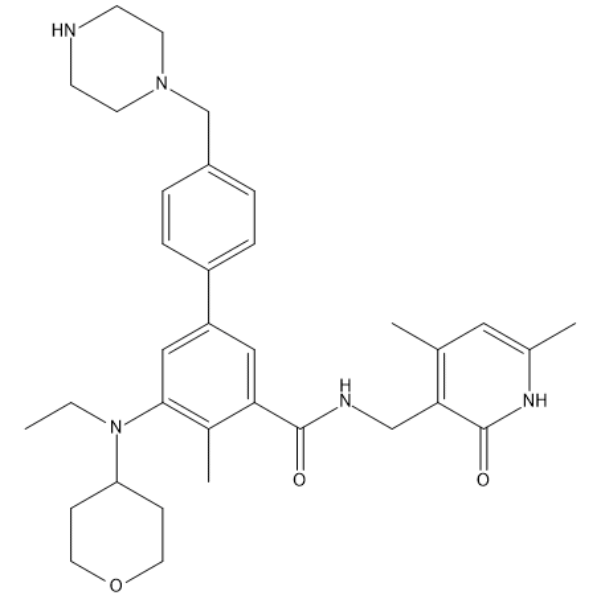
-
GC19149
EZM 2302
EZM 2302 est un inhibiteur de l'arginine méthyltransférase 1 associée au coactivateur (CARM1) avec une IC50 de 6nM.
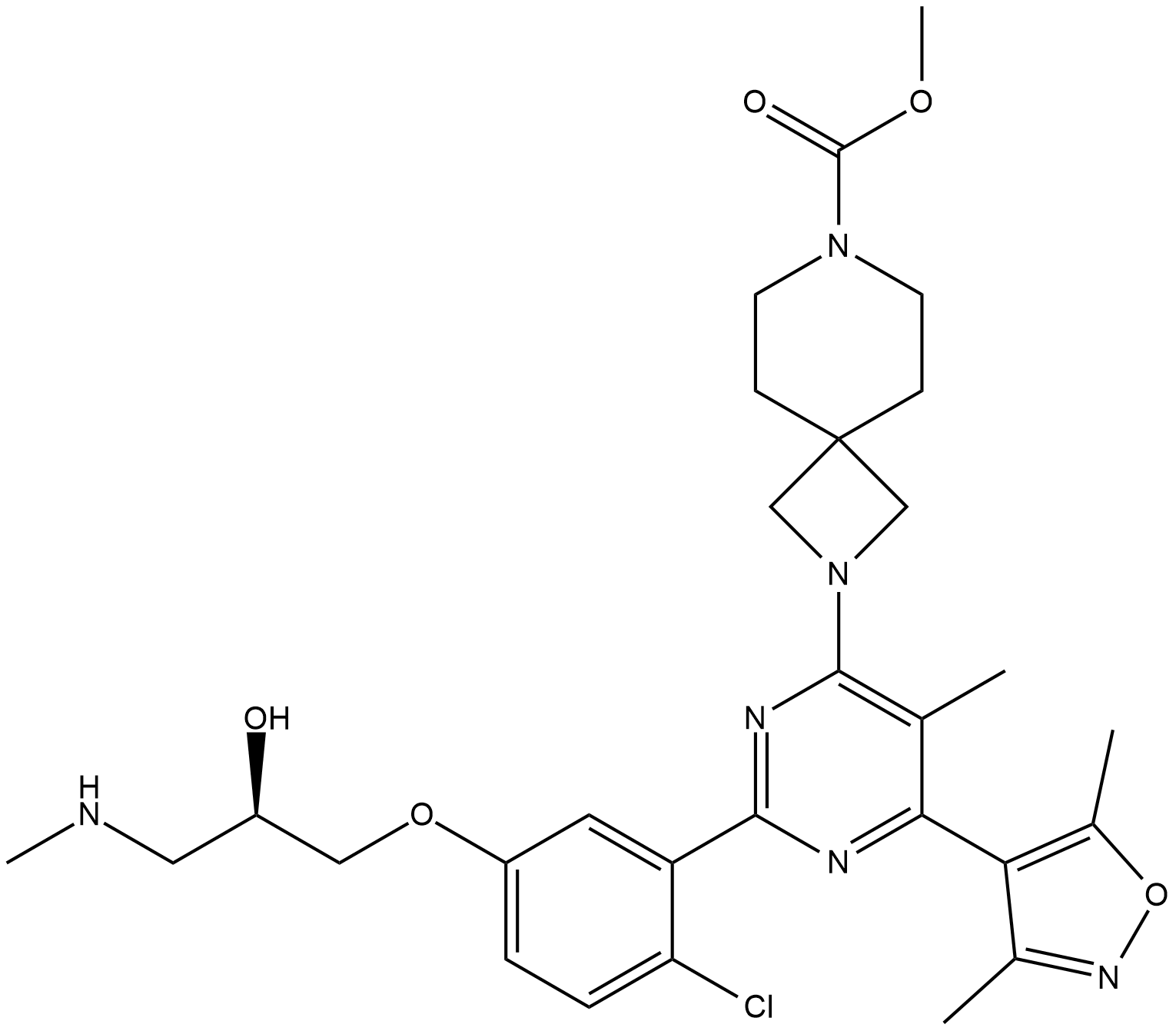
-
GC64900
EZM0414
EZM0414 est un inhibiteur puissant, sélectif et biodisponible par voie orale de SETD2 (IC50 = 18 nM dans le test biochimique SETD2; IC50 = 34 nM dans le test cellulaire). EZM0414 peut être utilisé pour la recherche du myélome multiple récidivant ou réfractaire et du lymphome diffus À grandes cellules B.
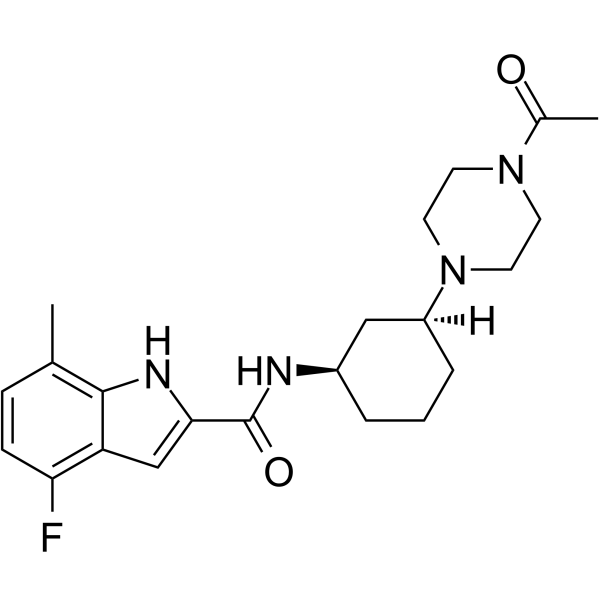
-
GC63545
FTX-6058
Le FTX-6058 ((S)-FTX-6058) est un inhibiteur puissant et oralement actif du développement de l'ectoderme embryonnaire (EED).
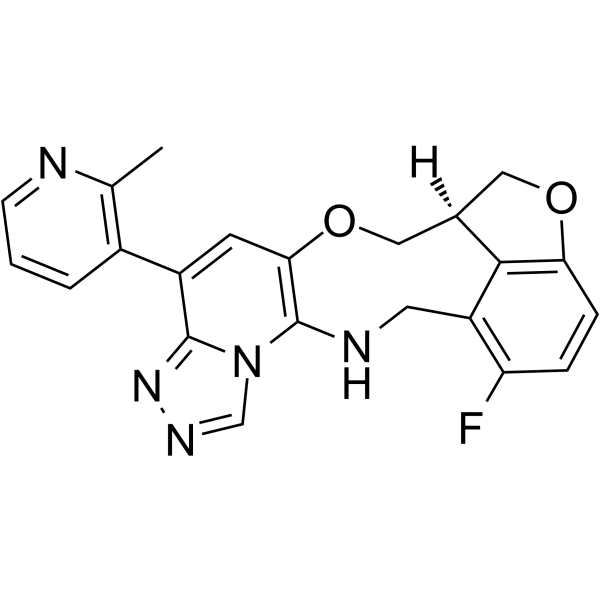
-
GC63546
FTX-6058 hydrochloride
Le chlorhydrate de (S)-Pociredir ((S)-FTX-6058) est un inhibiteur puissant et oralement actif du développement de l'ectoderme embryonnaire (EED).
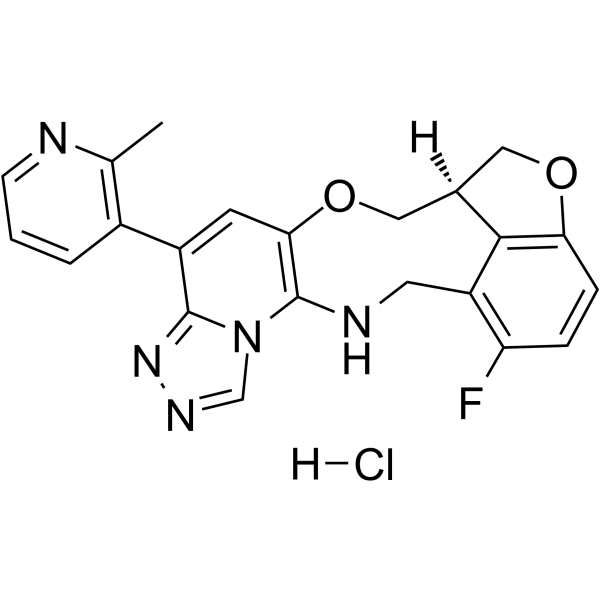
-
GC43715
Furamidine (hydrochloride)
La furamidine (chlorhydrate) (dichlorhydrate DB75) est un inhibiteur sélectif de la protéine arginine méthyltransférase 1 (PRMT1) avec une IC50 de 9,4 μM.
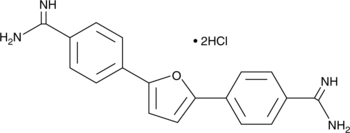
-
GC38062
Gambogenic acid
L'acide gambogénique est un ingrédient actif du gamboge, avec une activité anticancéreuse. L'acide gambogénique agit comme un inhibiteur efficace d'EZH2, se lie spécifiquement et de manière covalente À Cys668 dans le domaine EZH2-SET et induit l'ubiquitination d'EZH2.
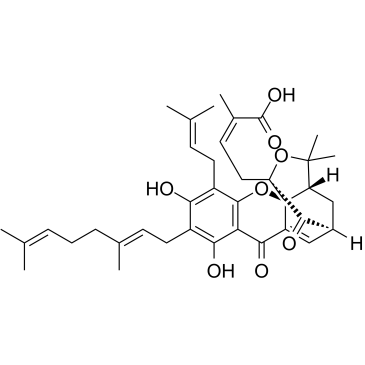
-
GC36168
GNA002
GNA002 est un inhibiteur EZH2 (Enhancer of zeste homolog 2) très puissant, spécifique et covalent avec une IC50 de 1,1 μM. GNA002 peut se lier spécifiquement et de manière covalente À Cys668 dans le domaine EZH2-SET, déclenchant la dégradation de EZH2 par l'extrémité COOH de l'ubiquitination médiée par la protéine interagissant avec Hsp70 (CHIP). GNA002 réduit efficacement la triméthylation H3K27 médiée par EZH2, réactive les gènes suppresseurs de tumeurs silencieux du complexe répresseur polycomb 2 (PRC2).
-
GC50697
GSK 591 dihydrochloride
-
GC15783
GSK126
Un inhibiteur sélectif d'EZH2
-
GC19181
GSK2807 Trifluoroacetate
Le trifluoroacétate de GSK2807 est un inhibiteur puissant, sélectif et compétitif de SAM de SMYD3, avec un Ki de 14 nM et une IC50 de 130 nM.
-
GC32693
GSK3326595 (EPZ015938)
GSK3326595 (EPZ015938) (EPZ015938) est un inhibiteur puissant, sélectif et réversible de la protéine arginine méthyltransférase 5 (PRMT5) avec une IC50 de 6,2 nM.
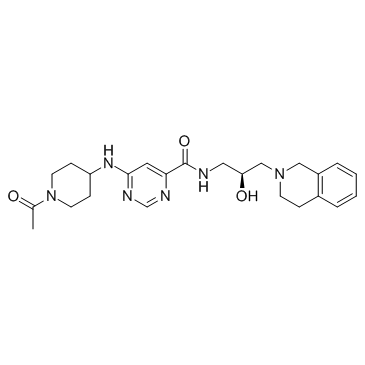
-
GC36191
GSK3368715
GSK3368715 (EPZ019997) est un inhibiteur de la protéine arginine méthyltransférase (PRMT) de type I actif par voie orale, réversible et S-adénosyl-L-méthionine (SAM) non compétitif (IC50 = 3,1 nM (PRMT1), 48 nM (PRMT3), 1148 nM ( PRMT4), 5,7 nM (PRMT6), 1,7 nM (PRMT8)). GSK3368715 (EPZ019997) produit un changement dans les états de méthylation de l'arginine, modifie l'utilisation des exons et possède une forte activité anticancéreuse.
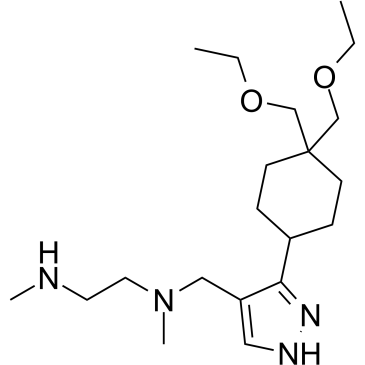
-
GC49398
GSK3368715 (hydrochloride)
An inhibitor of type I PRMTs
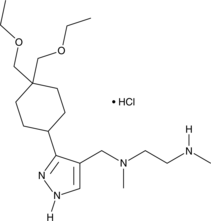
-
GC36192
GSK3368715 dihydrochloride
Le dichlorhydrate de GSK3368715 (dichlorhydrate EPZ019997) est un inhibiteur de la protéine arginine méthyltransférase (PRMT) de type I actif par voie orale, réversible et non compétitif de la S-adénosyl-L-méthionine (SAM) (IC50 = 3,1 nM (PRMT1), 48 nM (PRMT3), 1148 nM (PRMT4), 5,7 nM (PRMT6), 1,7 nM (PRMT8)). Le dichlorhydrate GSK3368715 (dichlorhydrate EPZ019997) produit un changement dans les états de méthylation de l'arginine, modifie l'utilisation des exons et a une forte activité anticancéreuse.
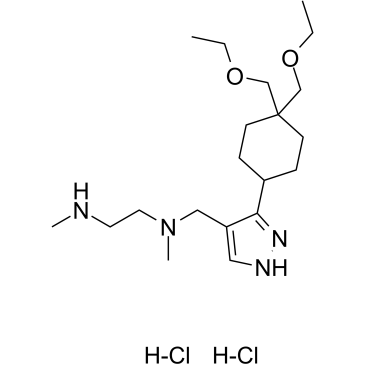
-
GC17534
GSK343
A selective, cell-permeable EZH2 inhibitor