Histone Methyltransferase
Histone methyltransferases are a group of enzymes that catalyze the methylation of histone lysine and arginine by adding methyl groups to specific histone arginine or lysine residues. Histone methyltransferaes can be classified into 3 classes, including SET domain lysine methyltransferases, non-SET domain lysine methyltransferases and arginine methyltransferases (PRMTs), all of which use S-adenosylmethionine as a cosubstrate for the transfer of the methyl group. Aberrant histone methylation has been associated with a wide range of human cancers (such as hematological malignancies), which leads to the development of novel cancer chemotherapies targeting cancer-associated histone methyltransferases (more than 20 lysine methyltransferases and 9 arginine methyltransferases in humans).
Products for Histone Methyltransferase
- Cat.No. Nombre del producto Información
-
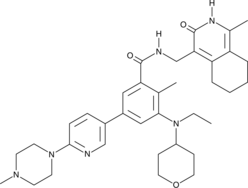
GC14585
GSK591
GSK591 (EPZ015866) es un inhibidor potente y selectivo de la proteÍna metiltransferasa 5 (PRMT5) con una IC50 de 4 nM.
-
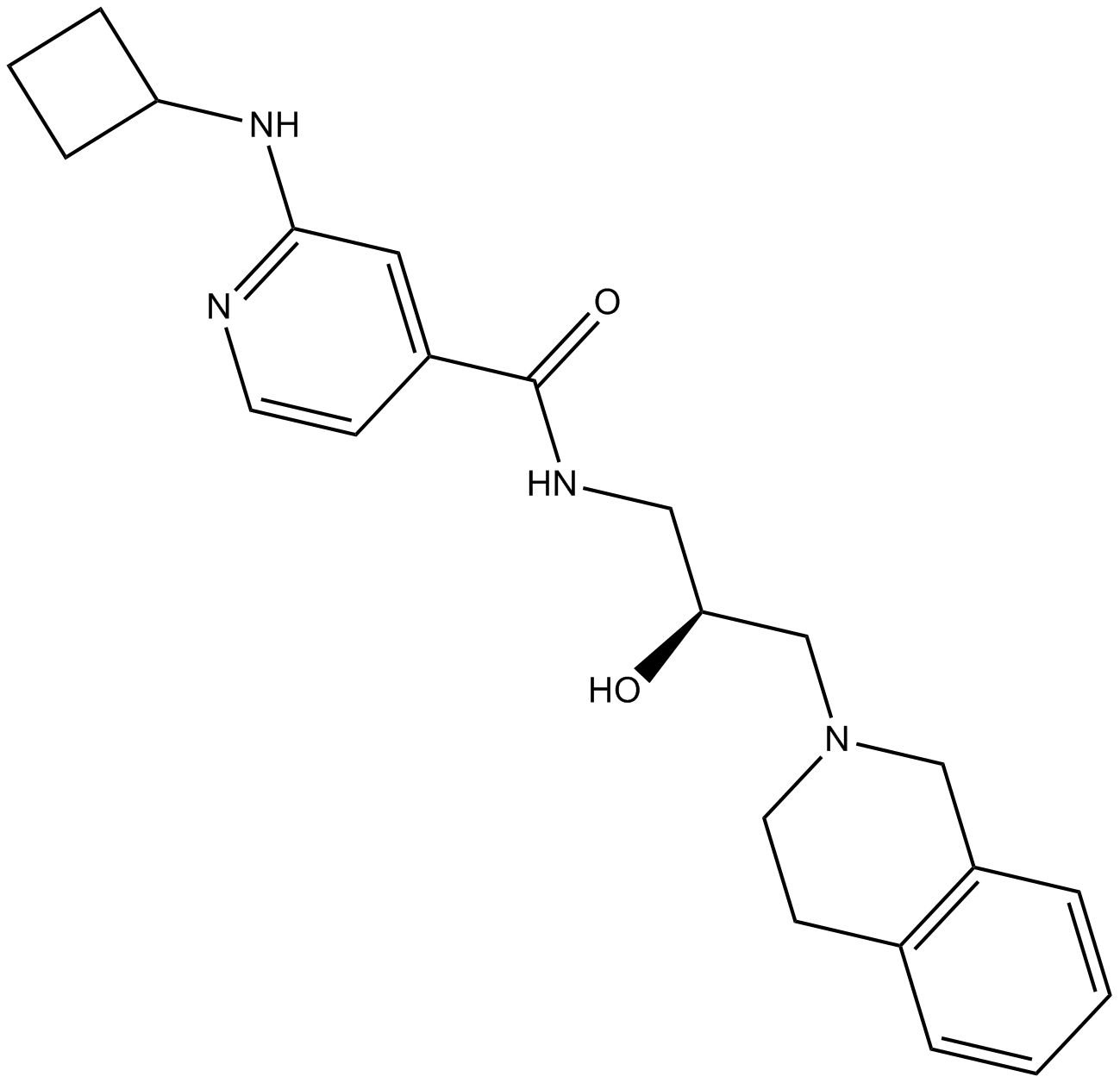
GP10020
Histone-H2A-(107-122)-Ac-OH
Histone-H2A peptide
-
GC17023
HLCL-61
HLCL-61 es el primer inhibidor de su clase de la proteÍna arginina metiltransferasa 5 (PRMT5).
-
GC19465
JNJ-64619178
JNJ-64619178 (Onametostat) es un inhibidor de la proteína arginina metiltransferasa 5 (PRMT5) selectivo, activo por vía oral y pseudoirreversible con una IC50 de 0,14 nM. JNJ-64619178 tiene una potente actividad en el cáncer de pulmón.

-
GC19211
JQEZ5
JQEZ5 es un inhibidor potente y selectivo de la lisina metiltransferasa EZH2. JQEZ5 InhibiciÓn competitiva de SAM del complejo represivo polycomb 2 (PRC2) con una IC50 de 80 nM. JQEZ5 tiene efectos antitumorales.
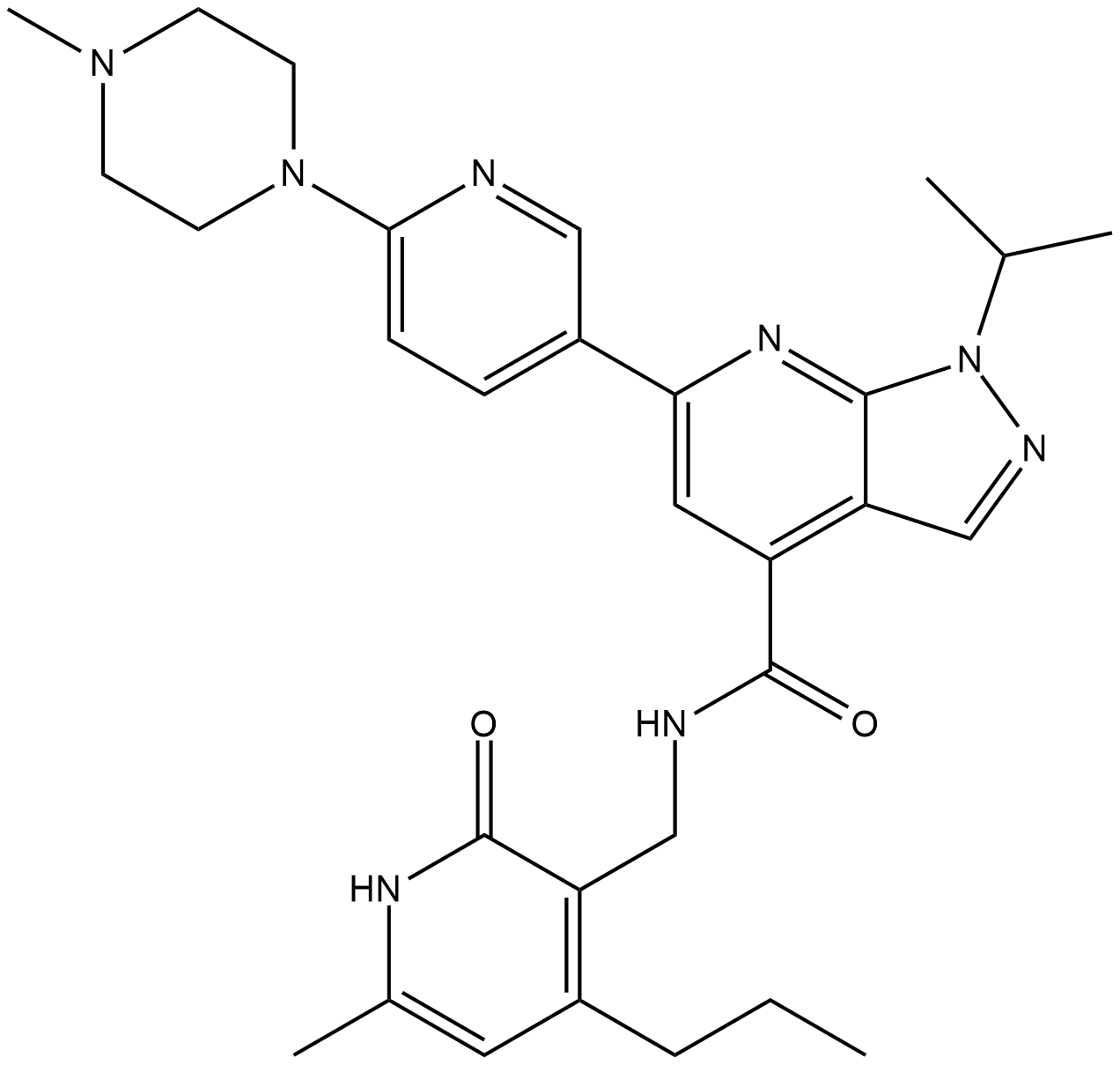
-
GC33184
LLY-283
LLY-283 es un inhibidor potente, selectivo y oral de la proteÍna arginina metiltransferasa 5 (PRMT5), con una IC50 de 22 nM y una Kd de 6 nM para el complejo PRMT5:MEP50, y muestra actividad antitumoral.
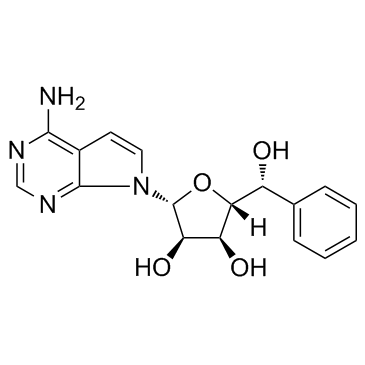
-
GC16261
LLY507
LLY507 es un inhibidor potente y selectivo de la proteína-lisina metiltransferasa SMYD2. LLY507 inhibe potentemente la capacidad de SMYD2 para metilar el péptido p53 con una IC50 \u003c15 nM. LLY507 sirve como una valiosa sonda química para ayudar en la disección de la función SMYD2 en el cáncer y otros procesos biológicos.
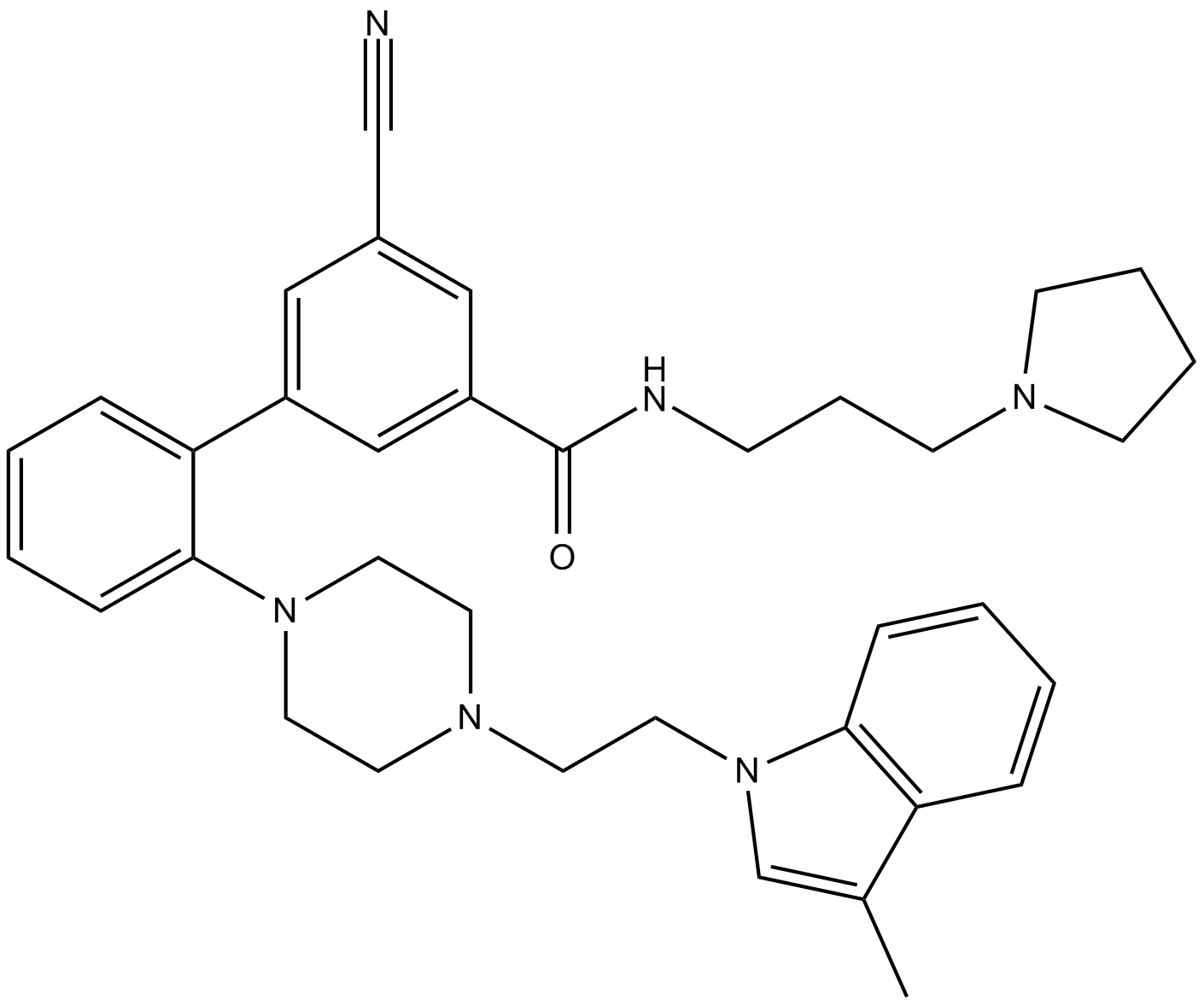
-
GC63056
MAK683-CH2CH2COOH
MAK683-CH2CH2COOH se une a EED (proteÍna de desarrollo del ectodermo embrionario). MAK683-CH2CH2COOH y un ligando VHL para la ligasa de ubiquitina E3 se han utilizado para diseÑar el degradador-1 de PROTAC EED y el degradador-2 de PROTAC EED.
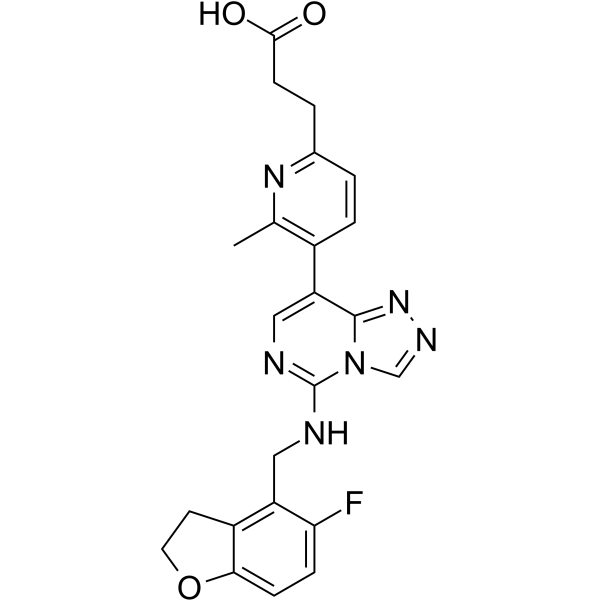
-
GC65254
MC4355
MC4355 es un inhibidor dual de EZH2 e histona desacetilasa (HDAC).
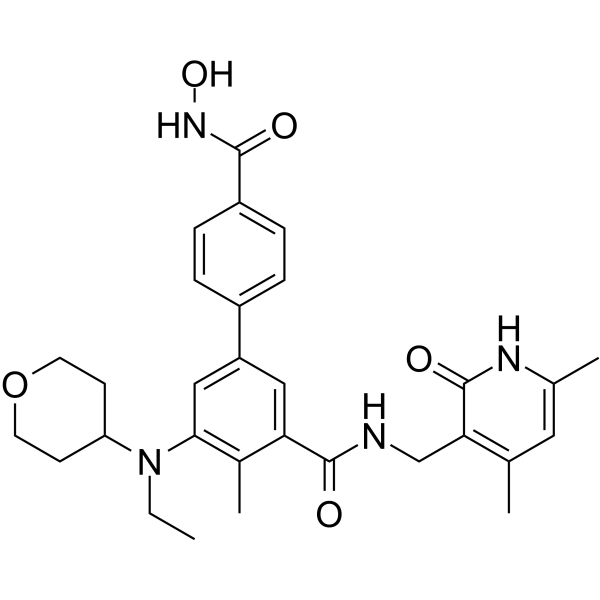
-
GC61058
Metoprine
La metoprina (BW 197U) es un potente inhibidor de la histamina N-metiltransferasa (HMT).
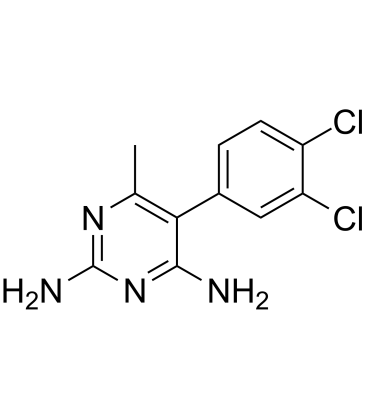
-
GC14652
MM-102
MM-102 (HMTase Inhibitor IX) es un potente inhibidor de la interacciÓn WDR5/MLL, alcanza IC50= 2,4 nM con una Ki estimada en 200 veces mÁs potente que el péptido ARA.
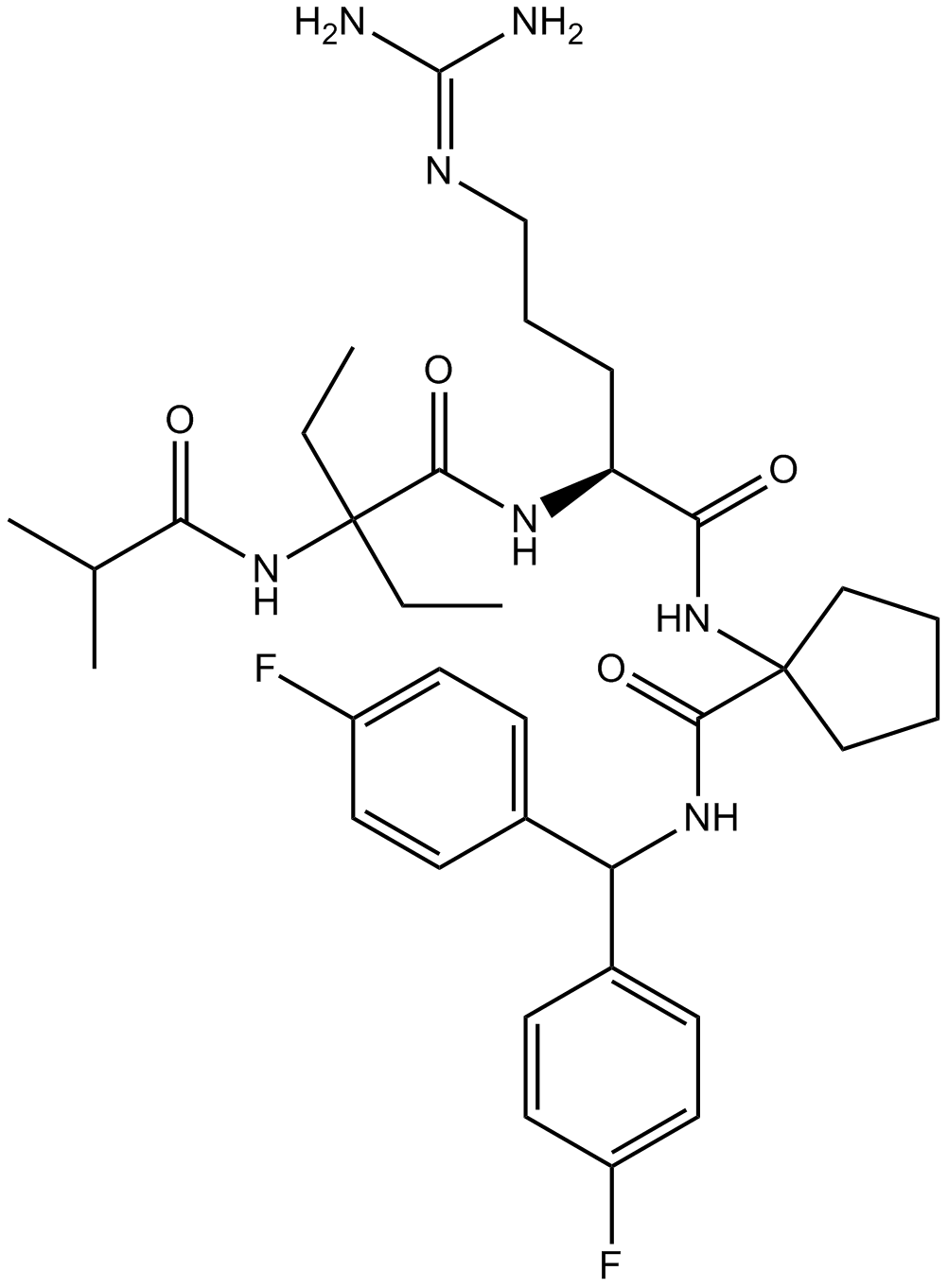
-
GC36632
MM-102 TFA
MM-102 TFA (HMTase Inhibitor IX TFA) es un potente inhibidor de la interacciÓn WDR5/MLL, alcanza IC50 = 2,4 nM con una Ki estimada 200 veces mÁs potente que el péptido ARA.
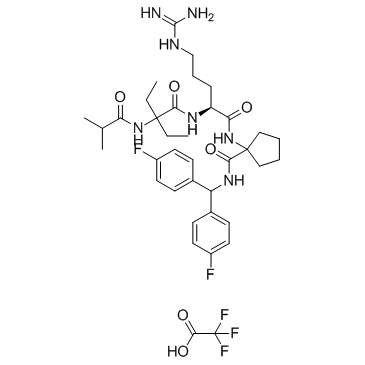
-
GC67758
MM-401 TFA
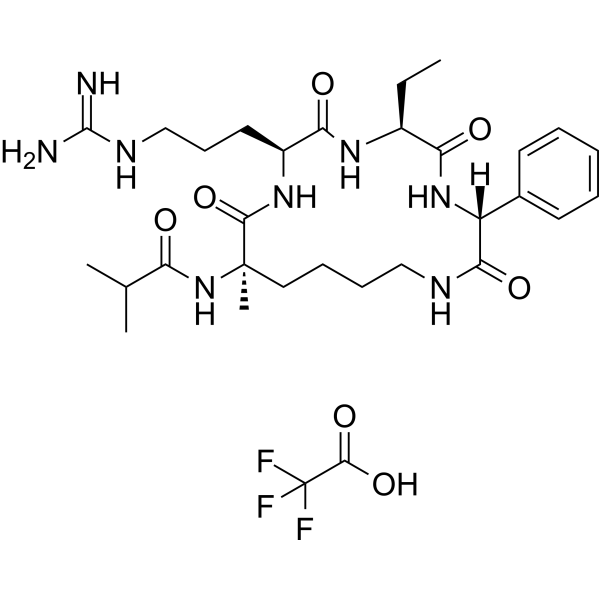
-
GC33278
MM-589
MM-589 es un potente inhibidor de la interacciÓn proteÍna-proteÍna del dominio de repeticiÓn 5 de WD (WDR5) y la leucemia de linaje mixto (MLL). MM-589 se une a WDR5 con una IC50 de 0,90 nM e inhibe la actividad de la metiltransferasa MLL H3K4 con una IC50 de 12,7 nM.
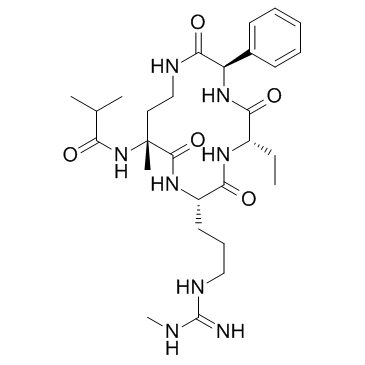
-
GC65037
MM-589 TFA
MM-589 TFA es un potente inhibidor de la interacciÓn proteÍna-proteÍna del dominio de repeticiÓn 5 de WD (WDR5) y la leucemia de linaje mixto (MLL). MM-589 se une a WDR5 con una IC50 de 0,90 nM e inhibe la actividad de la metiltransferasa MLL H3K4 con una IC50 de 12,7 nM.
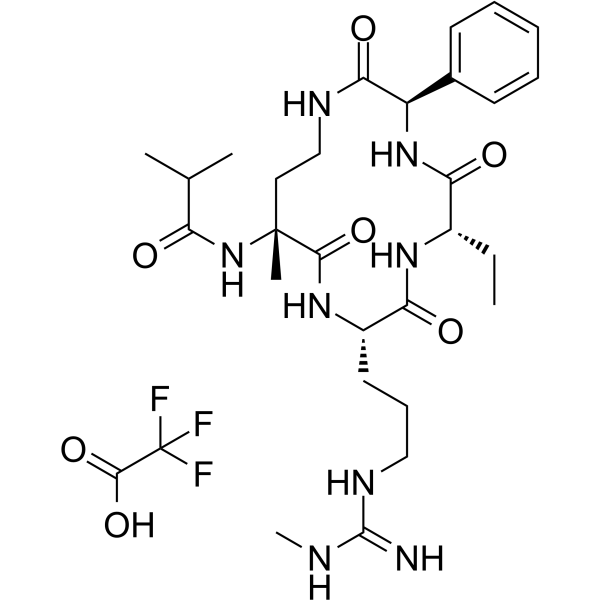
-
GC61468
MR837
MR837 es un inhibidor de NSD2-PWWP1. MR837 puede unirse a la proteÍna 2 del dominio SET de uniÓn al receptor nuclear humano (dominio PWWP).
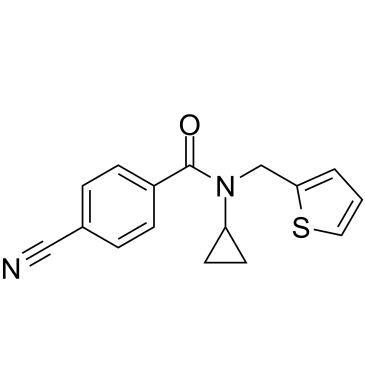
-
GC50576
MRK 740
Potent PRDM9 inhibitor
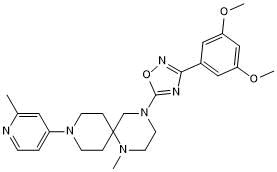
-
GC63558
MRTX-1719
MRTX-1719 es un potente inhibidor selectivo de primera clase del complejo PRMT5/MTA, con una IC50 de menos de 10 nM en células PRMT5/MTA MTAPDEL SDMA.
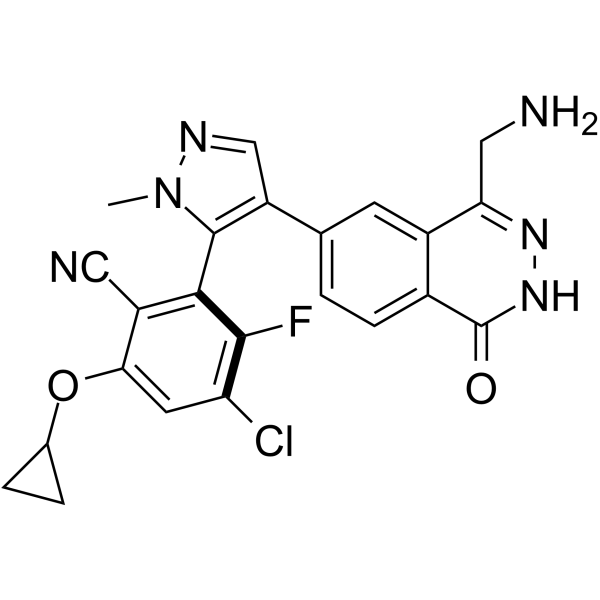
-
GC69499
MRTX-1719 hydrochloride
MRTX-1719 clorhidrato es un inhibidor efectivo, innovador y selectivo del complejo PRMT5/MTA. Su IC50 para las líneas celulares PRMT5/MTA MTAPDEL SDMA es <10 nM.
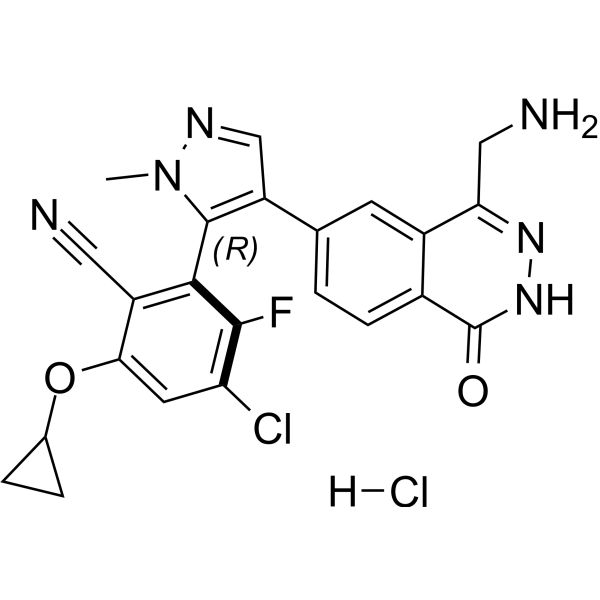
-
GC62715
MRTX9768
MRTX9768 es un inhibidor del complejo PRMT5-MTA potente, selectivo, activo por vÍa oral, primero en su clase.
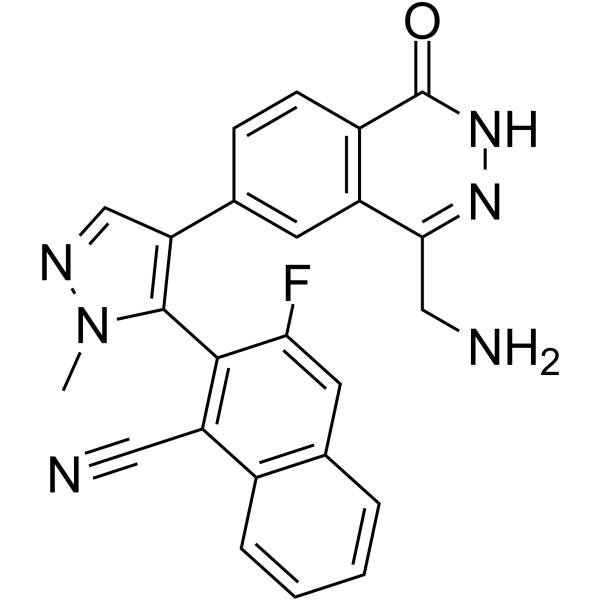
-
GC63080
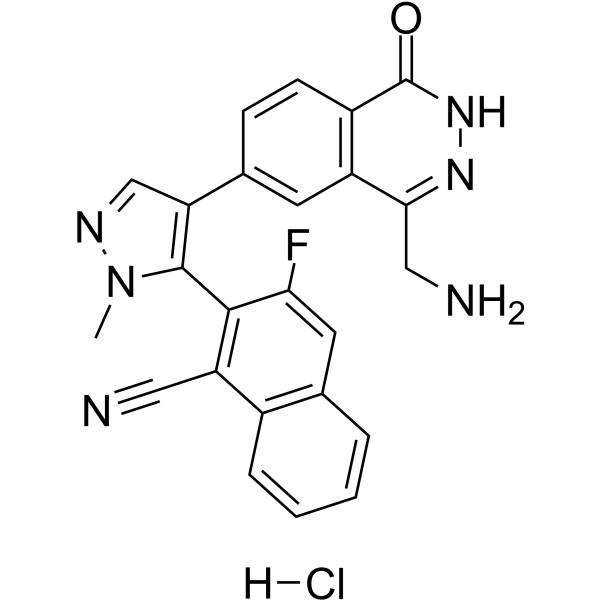
MRTX9768 hydrochloride
El clorhidrato de MRTX9768 es un inhibidor del complejo PRMT5-MTA potente, selectivo, activo por vÍa oral, primero en su clase.
-
GC10099
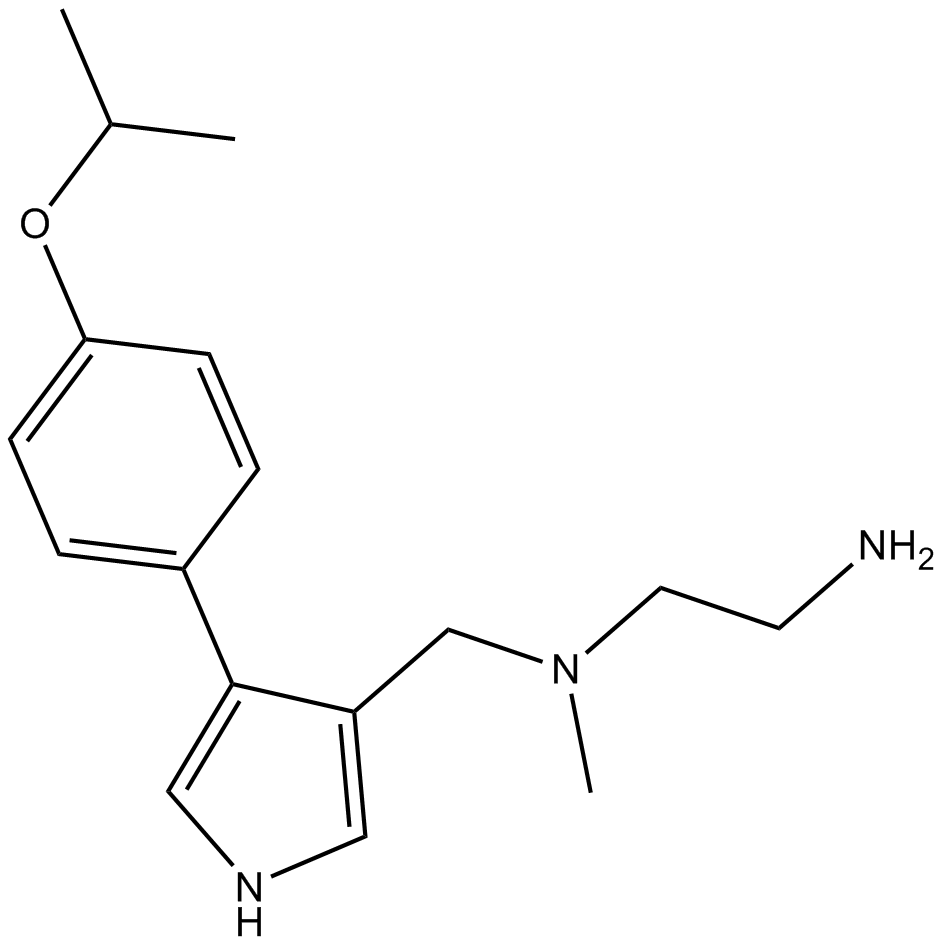
MS023
MS023 es un inhibidor potente, selectivo y activo en las células de las proteínas arginina metiltransferasas (PRMT) de tipo I.
-
GC16432
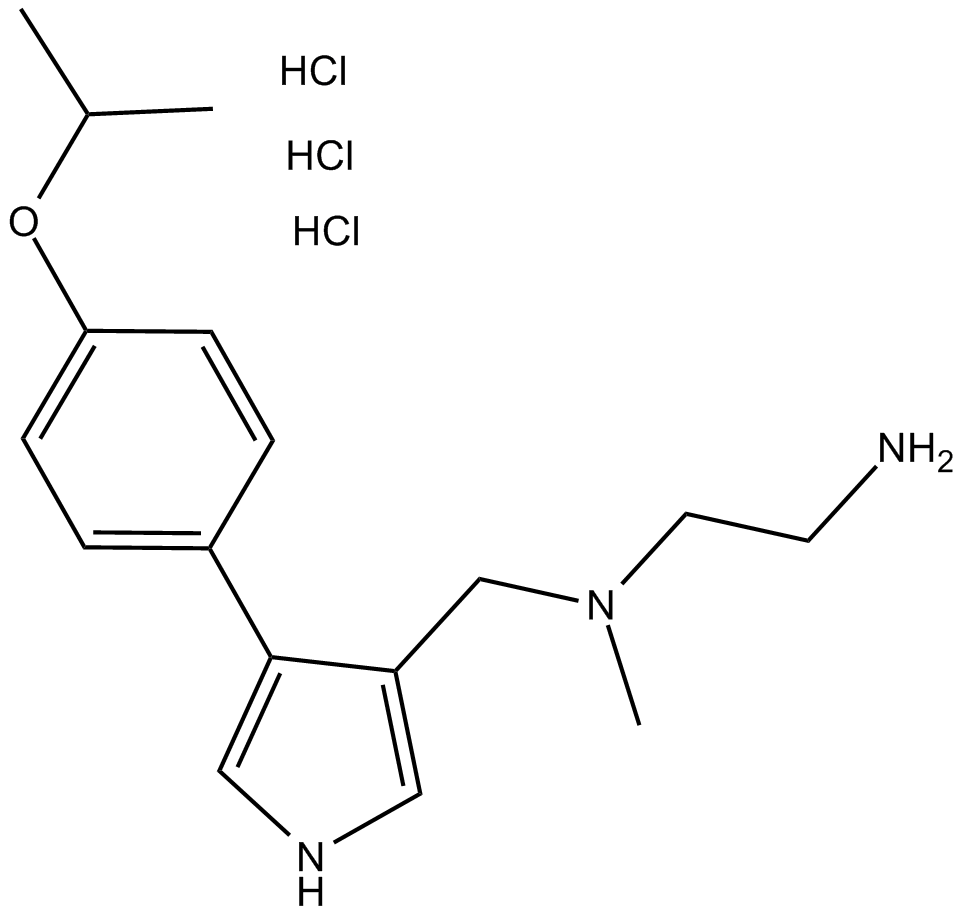
MS023 (hydrochloride)
type I PRMTs inhibitor
-
GC36655
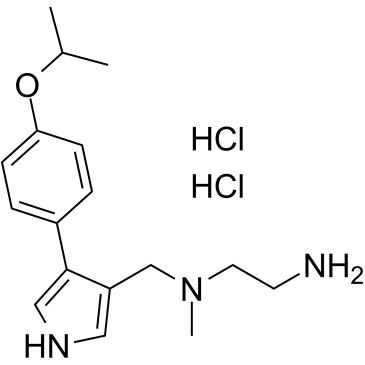
MS023 dihydrochloride
El diclorhidrato de MS023 es un inhibidor potente, selectivo y activo celular del inhibidor de las arginina metiltransferasas (PRMT) de la proteÍna tipo I humana, con IC50 de 30, 119, 83, 4 y 5 nM para PRMT1, PRMT3, PRMT4, PRMT6 y PRMT8. respectivamente.
-
GC36656
MS049
MS049 es un inhibidor dual potente, selectivo y celularmente activo de PRMT4 y PRMT6 con IC50 de 34 nM y 43 nM, respectivamente. MS049 reduce los niveles de Med12me2a y H3R2me2a en las células HEK293. MS049 no es tÓxico y no afecta el crecimiento de las células HEK293.
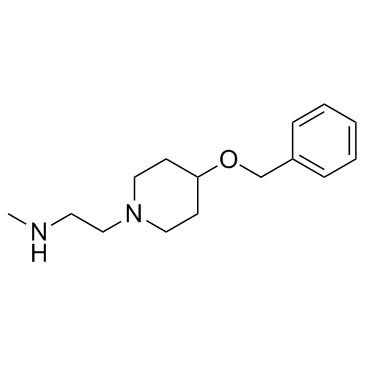
-
GC14240
MS049 (hydrochloride)
MS049 (clorhidrato) es un inhibidor dual potente, selectivo y celularmente activo de PRMT4 y PRMT6 con IC50 de 34 nM y 43 nM, respectivamente. MS049 (clorhidrato) reduce los niveles de Med12me2a y H3R2me2a en las células HEK293. MS049 (clorhidrato) no es tÓxico y no afecta el crecimiento de las células HEK293.
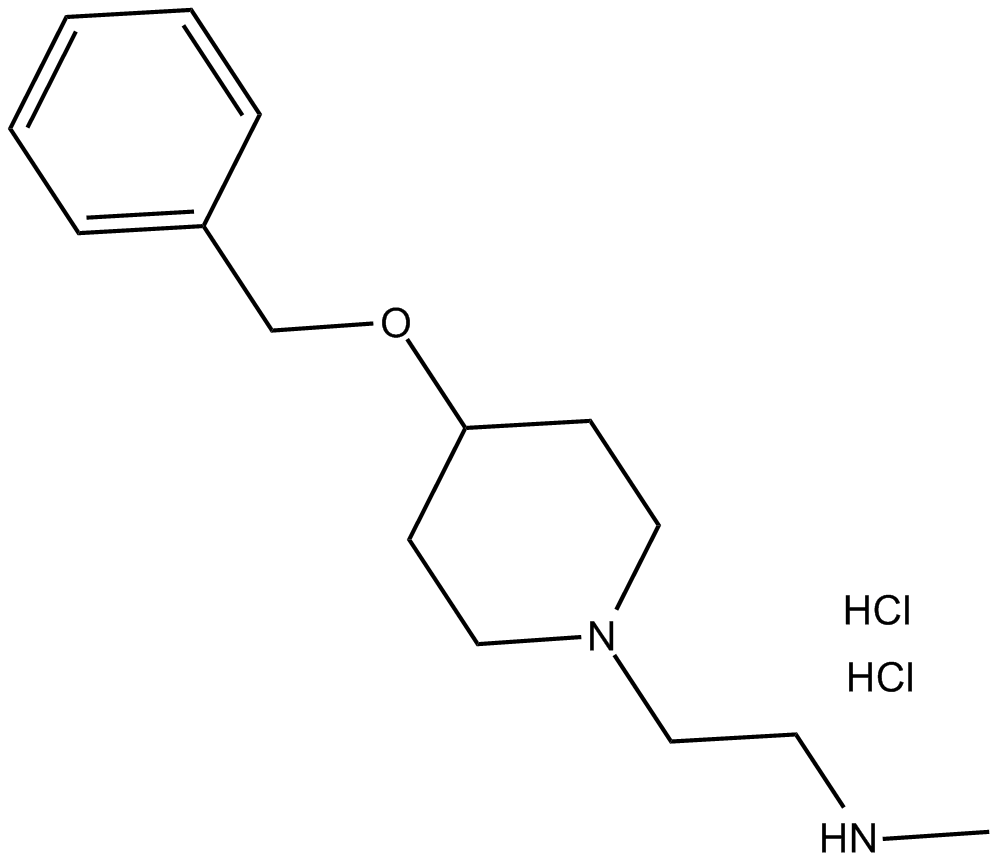
-
GC64295
MS67
MS67 es un degradador potente y selectivo del dominio repetido WD40 proteína 5 (WDR5) con una Kd de 63 nM. MS67 no es activo contra otras metiltransferasas de proteínas, quinasas, GPCR, canales iónicos ni transportadores. MS67 muestra efectos antitumorales potentes.

-
GC69504
MS8815
MS8815 es un degradador PROTAC selectivo del homólogo 2 (EZH2) de zeste. MS8815 tiene actividad inhibitoria sobre EZH2, con un valor IC50 de 8.6 nM. MS8815 se puede utilizar en la investigación del cáncer de mama triple negativo (TNBC).
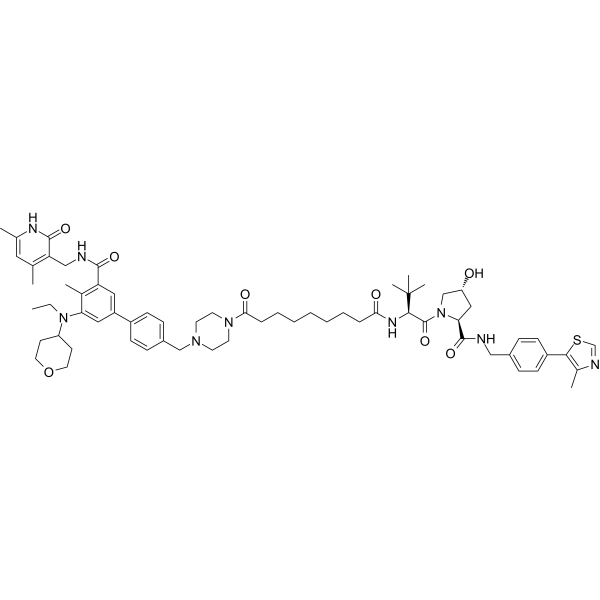
-
GC61142
NSC745885
NSC745885, un agente antitumoral eficaz, muestra toxicidad selectiva contra mÚltiples lÍneas celulares de cÁncer, pero no contra células normales. NSC745885 es un regulador a la baja eficaz de EZH2 a través de la degradaciÓn mediada por proteasoma. NSC745885 ofrece posibilidades para el estudio de cÁnceres avanzados de carcinoma de células escamosas de vejiga y oral (OSCC).
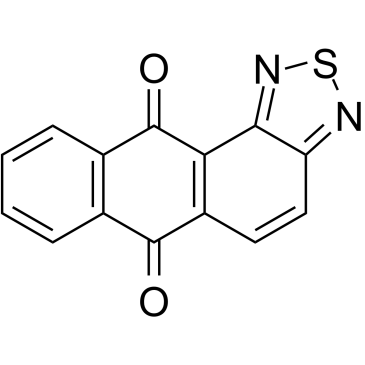
-
GC64772
NV03
NV03 es un antagonista potente y selectivo de la interacciÓn UHRF1 (similar a la ubiquitina con PHD y dominios de dedo RING 1)-H3K9me3 mediante la uniÓn al dominio tudor en tÁndem UHRF1, con una Kd de 2,4 μM. NV03 tiene actividad anticancerÍgena.
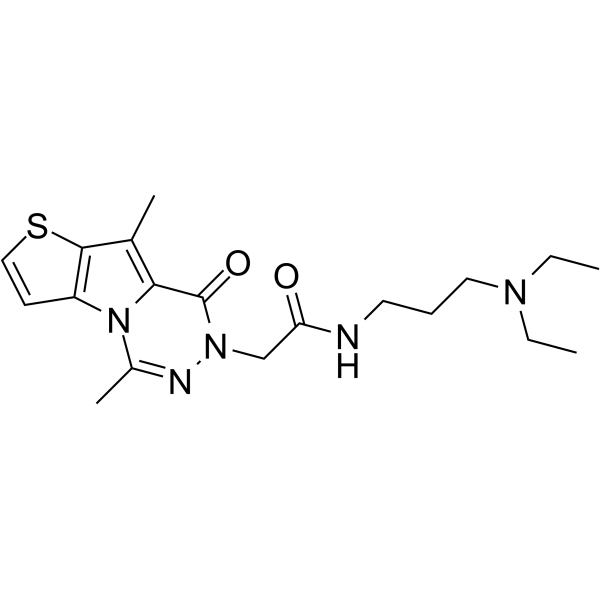
-
GC16397
OICR-9429
OICR-9429 es un inhibidor del dominio 5 de repeticiÓn de WD de alta afinidad (WDR5), bloquea de forma competitiva la interacciÓn de WDR5 con la proteÍna MLL mediante la uniÓn al bolsillo central de uniÓn de péptidos de WDR5. OICR-9429 puede suprimir la trimetilaciÓn de la histona H3K4 y puede usarse para la investigaciÓn de varios tipos de cÁncer, incluida la leucemia sin reordenamiento MLL, cÁncer de colon, pÁncreas, prÓstata y cÁncer de vejiga (BCa).
-
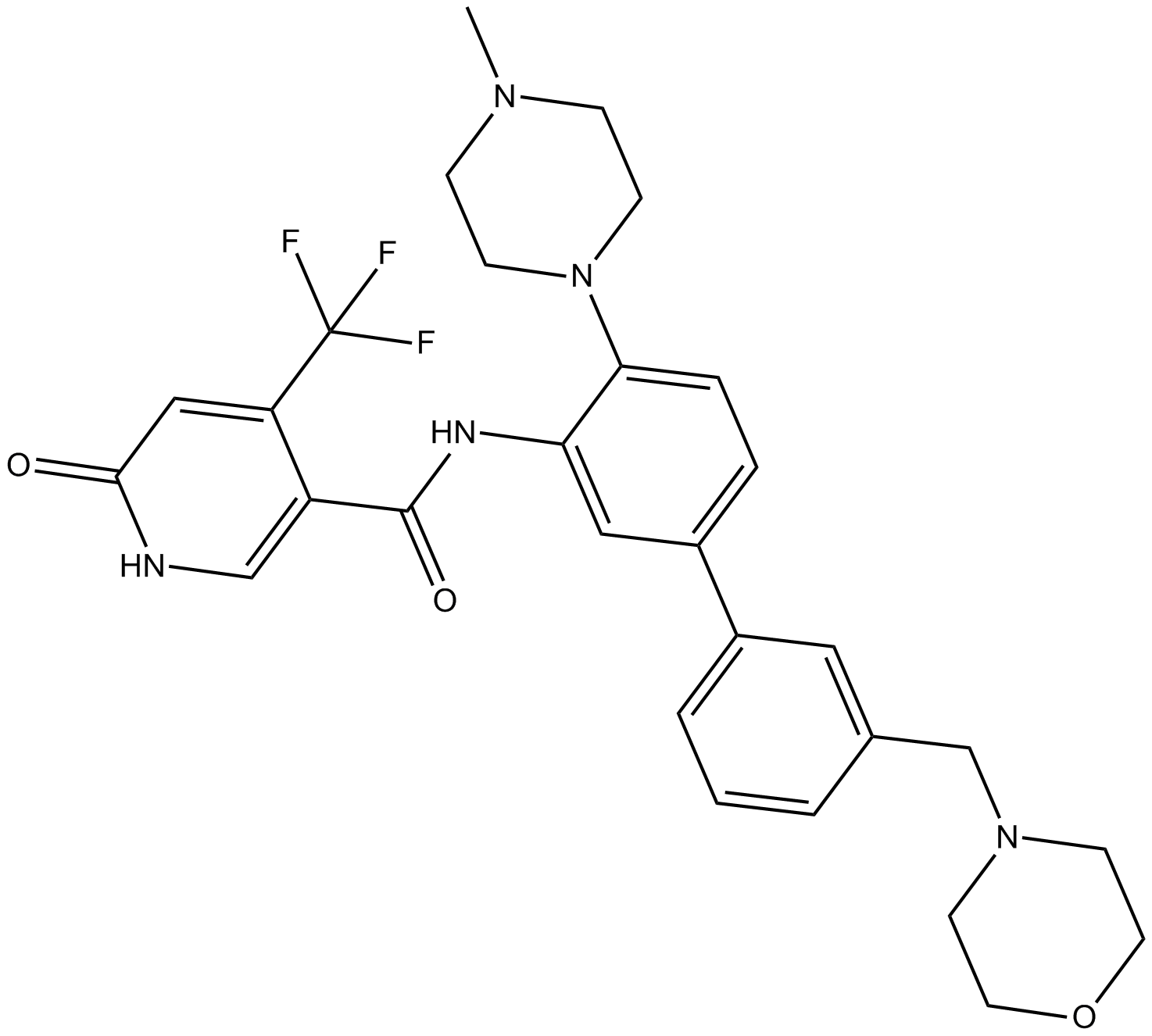
GC69636
OTS193320
OTS193320 es un compuesto de imidazopiridina [1,2-a], un inhibidor de la actividad metiltransferasa SUV39H2. OTS193320 reduce los niveles de trimetilación global del residuo lisina 9 en la histona H3 y provoca la muerte celular por apoptosis en células de cáncer de mama. En comparación con el tratamiento con OTS193320 o DOX solos, la combinación de OTS193320 y doxorrubicina (DOX; A) puede reducir los niveles de γ-H2AX y disminuir la viabilidad celular del cáncer.
-
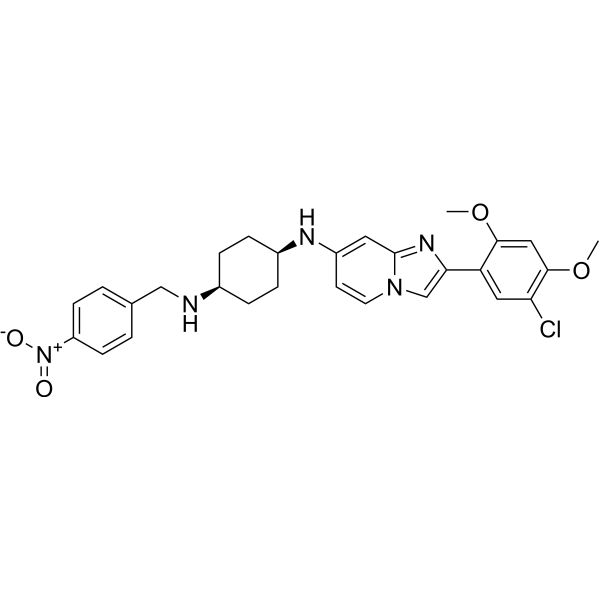
GC50367
PF 06726304 acetate
Highly potent and SAM-competitive EZH2 inhibitor
-
GC32977
PF-06726304
PF-06726304 es un inhibidor potente y selectivo de EZH2. PF-06726304 inhibe EZH2 mutante Y641N y de tipo salvaje con Kis de 0,7 y 3,0 nM, respectivamente. PF-06726304 muestra una sÓlida actividad de crecimiento antitumoral.
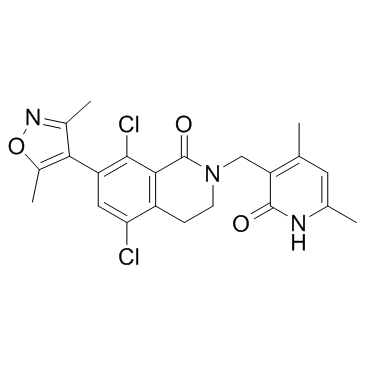
-
GC17956
PFI-2
El clorhidrato de PFI-2 ((R)-PFI-2 clorhidrato) es un dominio SET potente y selectivo que contiene un inhibidor de la lisina metiltransferasa 7 (SETD7).
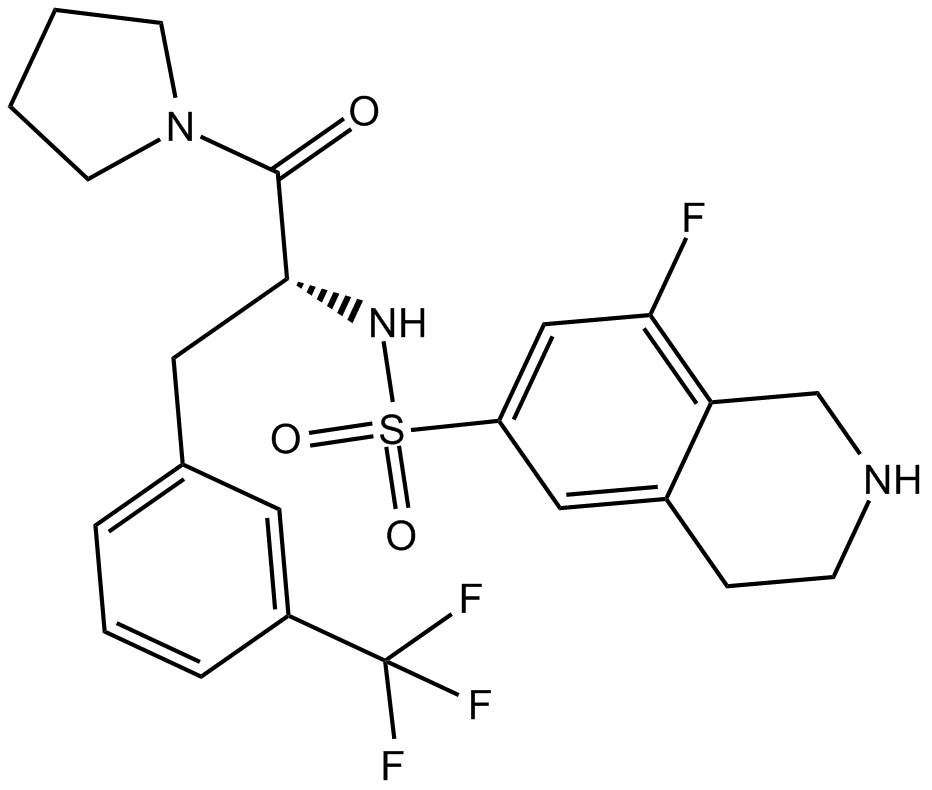
-
GC12530
PFI-2 (hydrochloride)

-
GC64941
PR5-LL-CM01
PR5-LL-CM01 es un potente inhibidor de la proteÍna arginina metiltransferasa 5 (PRMT5) (IC50= 7,5 μM). Actividades antitumorales.
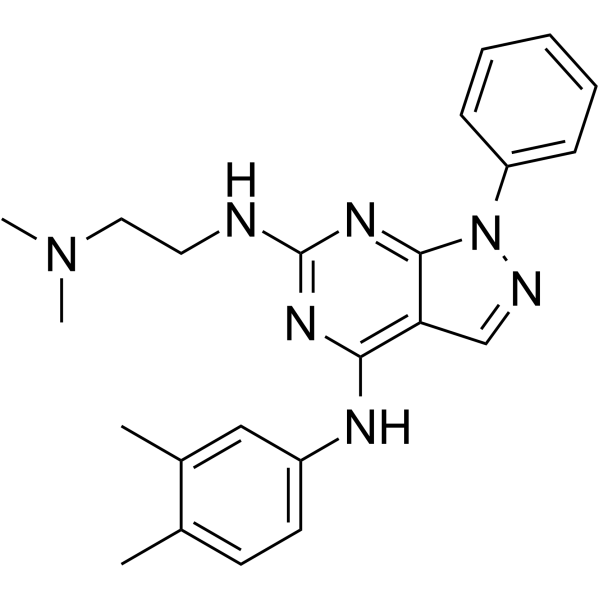
-
GC36969
PRMT5-IN-1
PRMT5 IN-1, un hemiaminal, es un potente inhibidor selectivo de la proteÍna arginina metiltransferasa 5 (PRMT5) con una IC50 de 11 nM para PRMT5/MEP50. PRMT5 IN-1 se puede convertir en aldehÍdos y reaccionar con C449 para formar aductos covalentes en condiciones fisiolÓgicas.
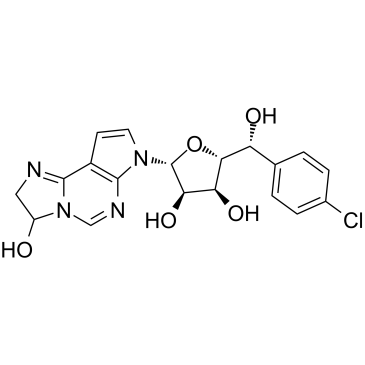
-
GC65027
PRMT5-IN-20
PRMT5-IN-20 es un inhibidor selectivo de la proteÍna arginina metiltransferasa 5 (PRMT5) con actividad antitumoral.
-
GC62187
PROTAC EED degrader-1
PROTAC EED degradador-1 es un PROTAC basado en von Hippel-Lindau dirigido a EED con un pKD de 9,02. PROTAC EED degradador-1 es un inhibidor del complejo represivo 2 de Polycomb (PRC2) (pIC50 = 8,17) que se dirige a la subunidad EED.
-
GC62717
PROTAC EED degrader-2
PROTAC EED degradador-2 es un PROTAC basado en von Hippel-Lindau dirigido a EED con un pKD de 9,27. PROTAC EED degradador-2 es un inhibidor del complejo represivo 2 de Polycomb (PRC2) (pIC50 = 8.11) que se dirige a la subunidad EED.
-
GC65509
PROTAC EZH2 Degrader-1
PROTAC EZH2 Degrader-1 (Compuesto 150d), un potente PROTAC EZH2 Degrader, ejerce un efecto inhibidor sobre la actividad de la metiltransferasa de EZH2 con una IC50 de 2,7 nM. EZH2 juega un papel importante en muchos procesos de tumorigénesis y desarrollo.
-
GC46213
Ro 41-0960
Ro 41-0960 es un inhibidor selectivo de la catecol-O-metiltransferasa (COMT).
-
GC18650
S-(5'-Adenosyl)-L-methionine (tosylate)
S-(5'-Adenosyl)-L-methionine (SAM) is a ubiquitous methyl donor involved in a wide variety of biological reactions, including those mediated by DNA and protein methyltransferases.
-
GC44859
S-(5'-Adenosyl)-L-methionine chloride (hydrochloride)
S-(5'-Adenosyl)-L-methionine chloride (SAM) is a ubiquitous methyl donor involved in a wide variety of biological reactions, including those mediated by DNA and protein methyltransferases.
-
GC44886
S-Farnesyl Thioacetic Acid
S-Farnesyl thioacetic acid is an analog of S-farnesyl cysteine which behaves as a competitive inhibitor of isoprenylated protein methyltransferase (also known as S-adenosylmethionine-dependent methyltransferase).
-
GC62555
SETD2-IN-1 TFA
SETD2-IN-1 TFA es un inhibidor potente, selectivo y activo por vÍa oral de SETD2, que es una histona metiltransferasa humana. SETD2-IN-1 TFA tiene efectos antiproliferativos.
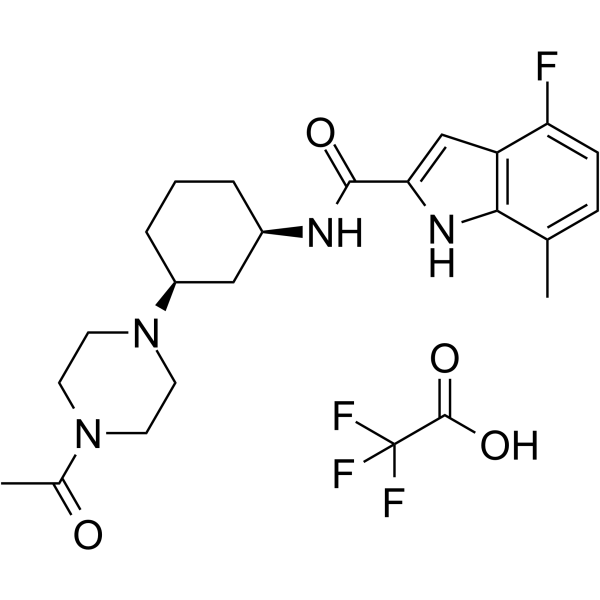
-
GC64893
SETDB1-TTD-IN-1
SETDB1-TTD-IN-1 es un inhibidor competitivo del aglutinante potente, selectivo y endÓgeno del dominio tudor en tÁndem de la proteÍna 1 bifurcada del dominio SET (SETDB1-TTD), con una Kd de 88 nM. SETDB1-TTD-IN-1 se puede utilizar para la investigaciÓn de funciones biolÓgicas y asociaciones de enfermedades de SETDB1-TTD.
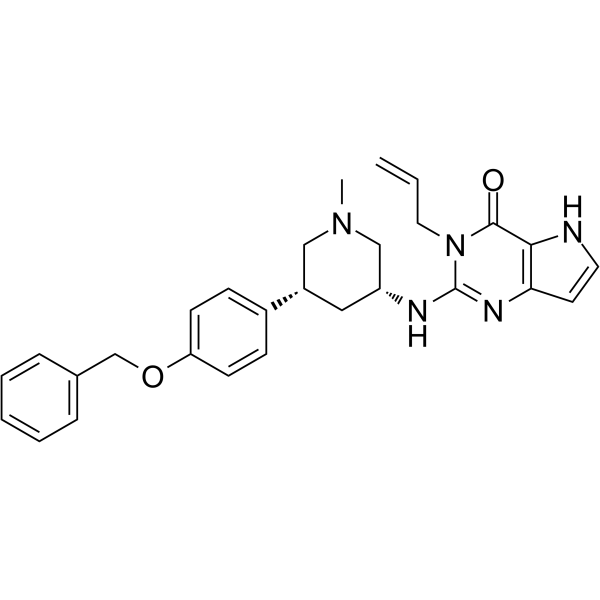
-
GC64894
SETDB1-TTD-IN-1 TFA
SETDB1-TTD-IN-1 TFA es un inhibidor competitivo del aglutinante potente, selectivo y endÓgeno del dominio tudor en tÁndem de la proteÍna 1 bifurcada del dominio SET (SETDB1-TTD), con una Kd de 88 nM. SETDB1-TTD-IN-1 TFA se puede utilizar para la investigaciÓn de funciones biolÓgicas y asociaciones de enfermedades de SETDB1-TTD.
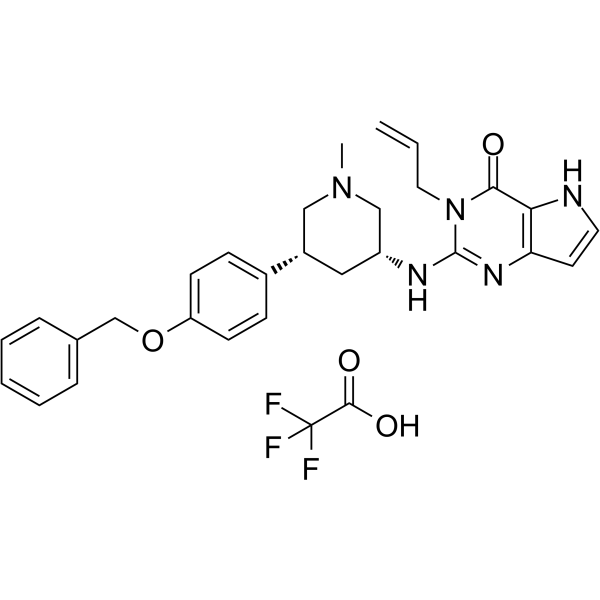
-
GC11491
SGC 0946
A potent inhibitor of DOT1L
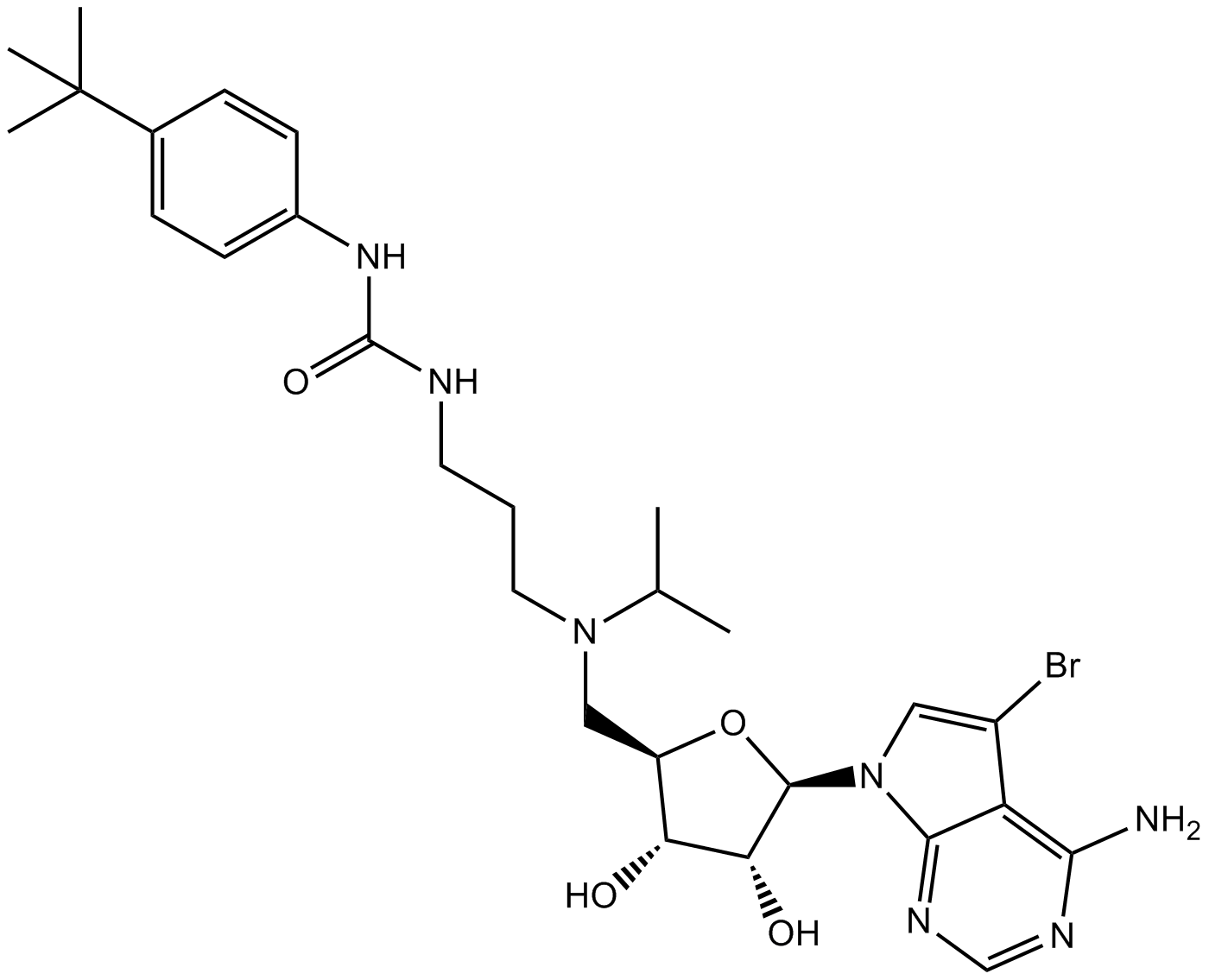
-
GC50699
SGC 6870
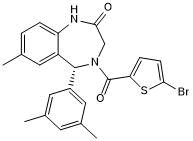
-
GC19329
SGC2085
SGC2085 es un inhibidor de la arginina metiltransferasa 1 (CARM1) asociado a un coactivador potente y selectivo con una IC50 de 50 nM.
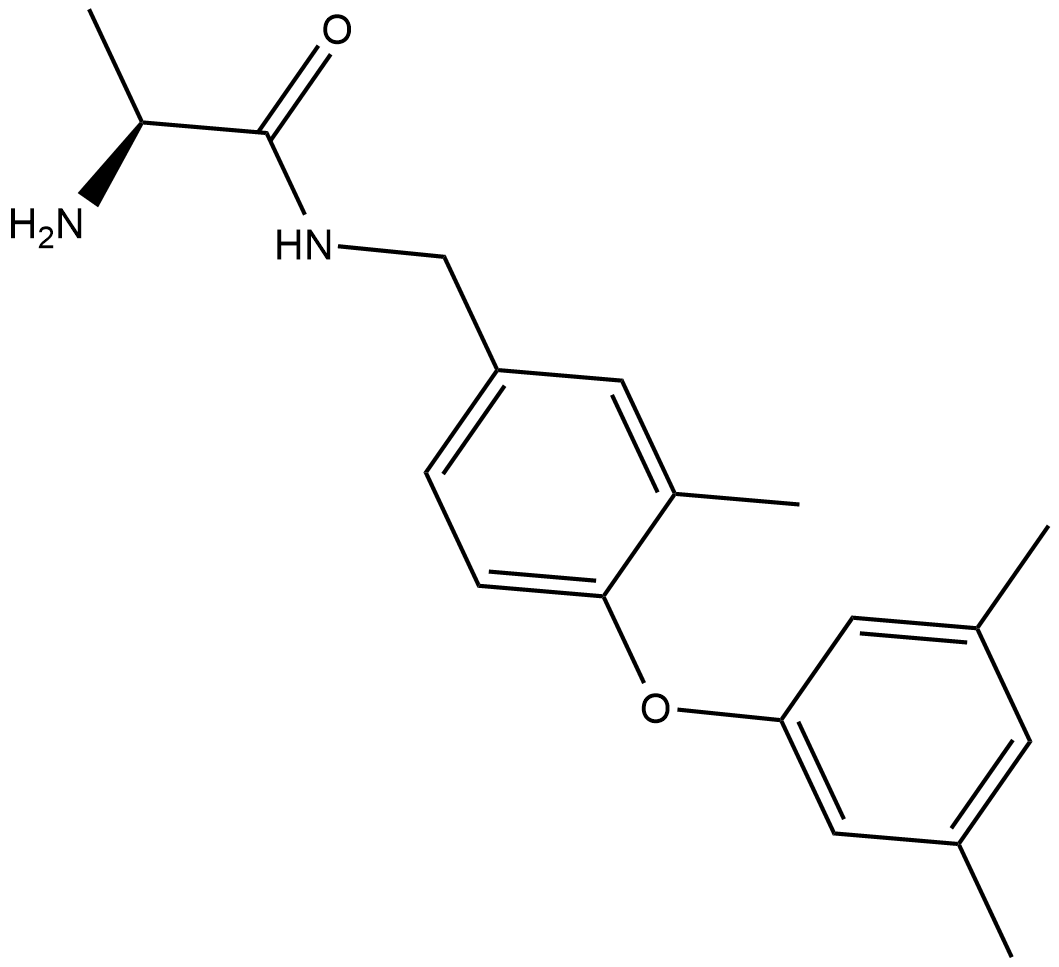
-
GC18428
SGC2085 (hydrochloride)
SGC2085 is an inhibitor of protein arginine methyltransferase 4 (PRMT4/CARM1; IC50 = 50 nM).
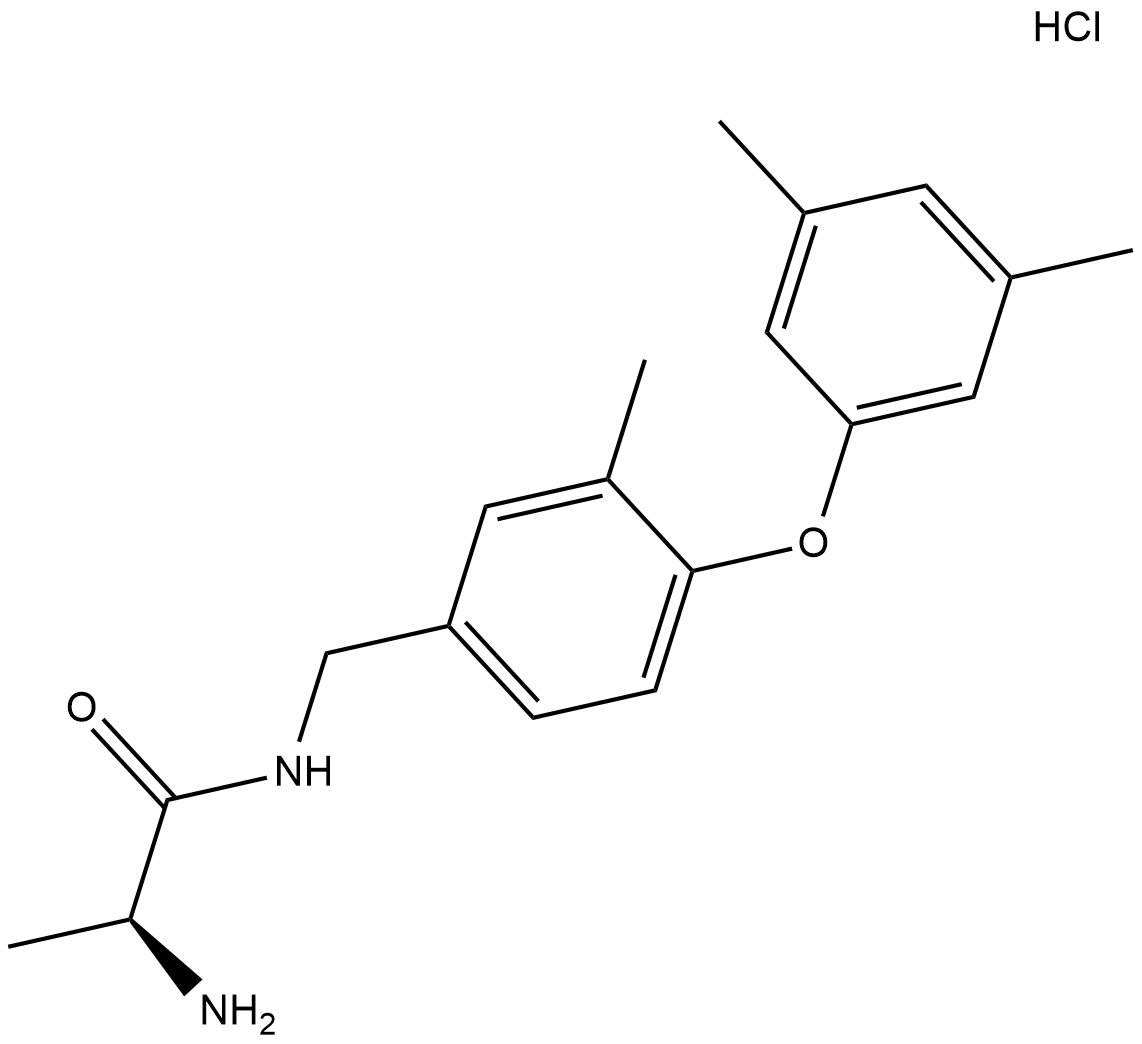
-
GC12874
SGC707
SGC707 es un inhibidor potente, selectivo y no competitivo de PRMT3 (proteÍna arginina metiltransferasa 3) (IC50 = 31 nM, Kd = 53 nM).
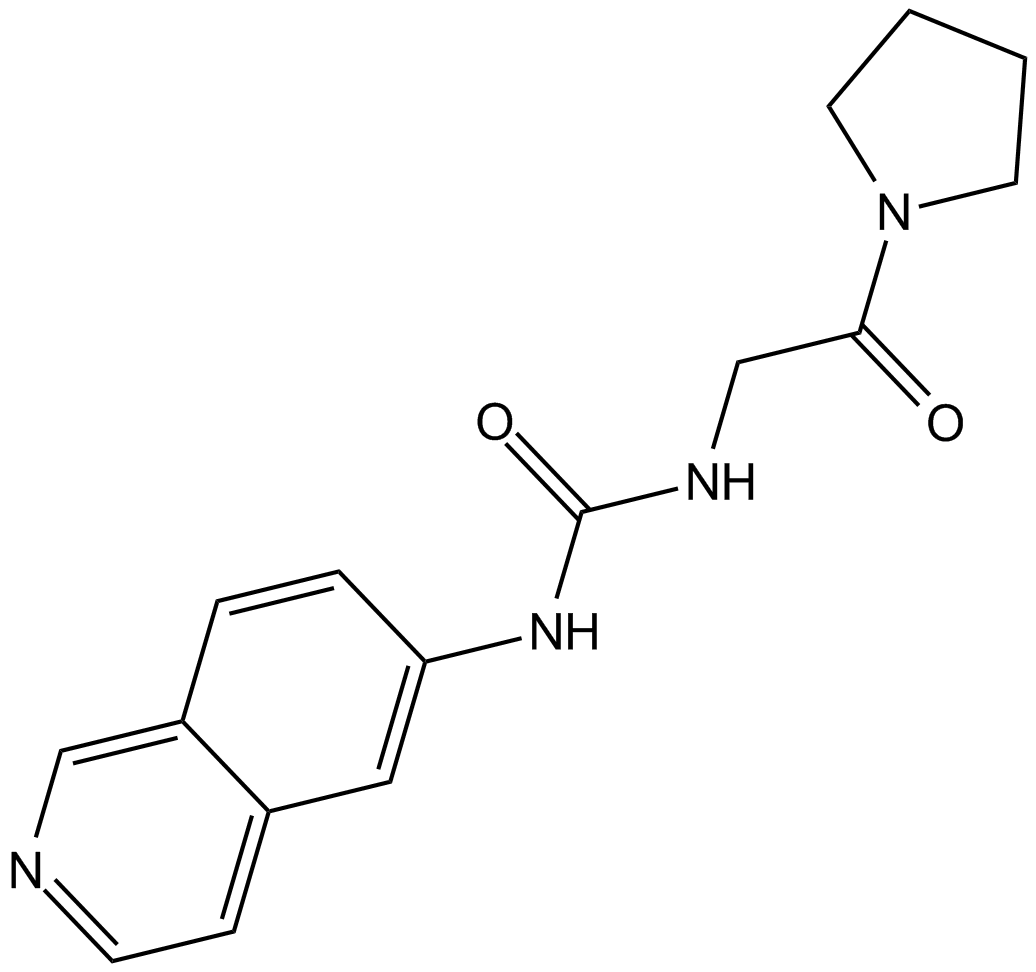
-
GC14364
SGI-1027
SGI-1027 es un inhibidor de la ADN metiltransferasa (DNMT), con IC50 de 7,5 μM, 8 μM y 12,5 μM para DNMT3B, DNMT3A y DNMT1 con poli(dI-dC) como sustrato.
-
GC34784
Sinefungin
La sinefungina es un potente inhibidor del ARNm (guanina-7-)-metiltransferasa del viriÓn, el ARNm (nucleÓsido-2'-)-metiltransferasa y la multiplicaciÓn viral.
-
GC37654
SMYD3-IN-1
SMYD3-IN-1 (compuesto 29) es un inhibidor irreversible y selectivo de SMYD3 (dominio SET y MYND que contiene 3), con una IC50 de 11,7 nM.
-
GC18642
SW155246
SW155246 es un inhibidor selectivo de la ADN metiltransferasa (DNMT1) con IC50 de 1,2 y 38 μM para hDNMT1 y mDNMT3A, respectivamente. SW155246 se puede utilizar para la investigaciÓn del cÁncer y otras enfermedades.
-
GC62390
SW2_110A
SW2_110A es un inhibidor selectivo del cromodominio chromobox 8 (CBX8 ChD) con una Kd de 800 nM. SW2_110A muestra una selectividad mÍnima de 5 veces para CBX8 ChD sobre todos los demÁs parÁlogos de CBX in vitro.
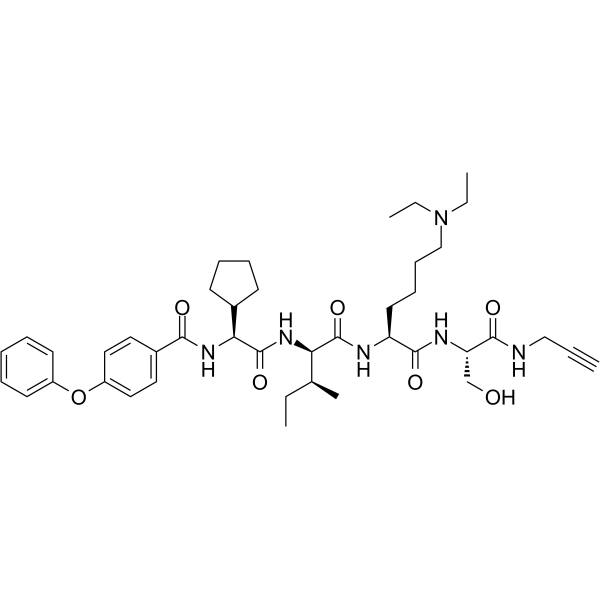
-
GC69980
SW2_152F
SW2_152F es un inhibidor efectivo selectivo del dominio cromodomo 2 de la caja chromobox 2 (CBX2 ChD), con una Kd de 80 nM. En comparación con otros parálogos CBX, SW2_152F tiene una selectividad en vitro hacia el CBX2 ChD que es entre 24 y 1000 veces mayor.
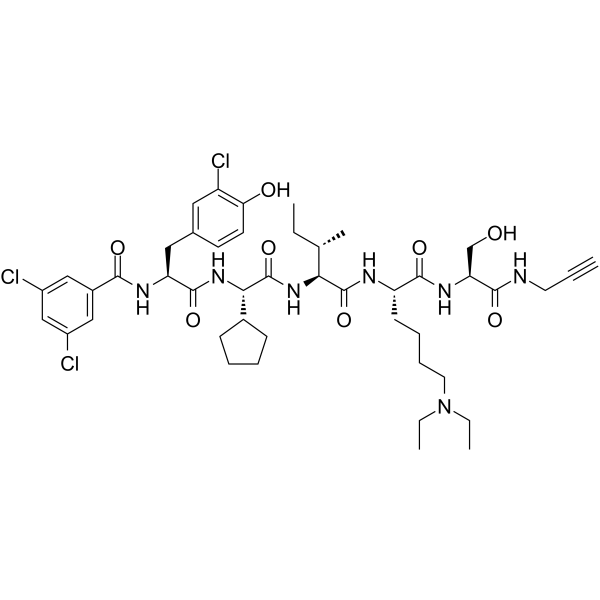
-
GC67676
Tazemetostat de(methylene morpholine)-O-C3-O-C-COOH
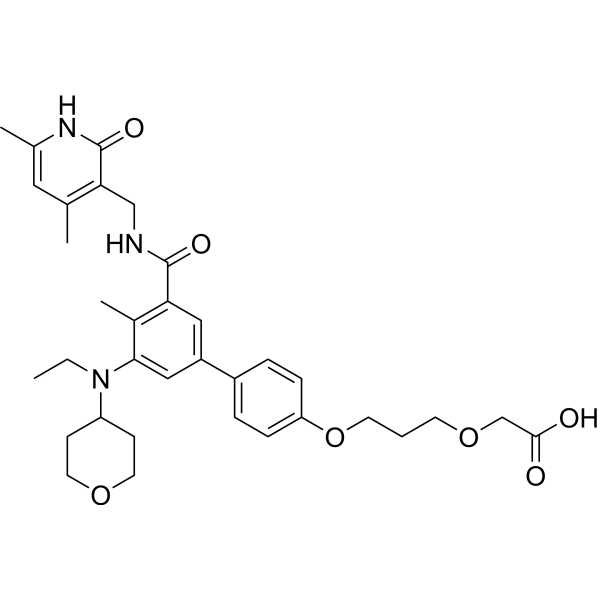
-
GC38163
Tazemetostat hydrobromide
El bromhidrato de tazemetostat (bromhidrato de EPZ-6438) es un inhibidor de EZH2 potente, selectivo y disponible por vÍa oral. El bromhidrato de tazemetostat inhibe la actividad del EZH2 de tipo salvaje que contiene el complejo represivo humano Polycomb 2 (PRC2) con un valor de Ki de 2,5 nM. El bromhidrato de tazemetostat inhibe EZH2 con IC50 de 11 y 16 nM en el ensayo de péptidos y el ensayo de nucleosomas, respectivamente. El bromhidrato de tazemetostat inhibe Rat EZH2 con una IC50 de 4 nM. El bromhidrato de tazemetostat también inhibe EZH1 con una IC50 de 392 nM.
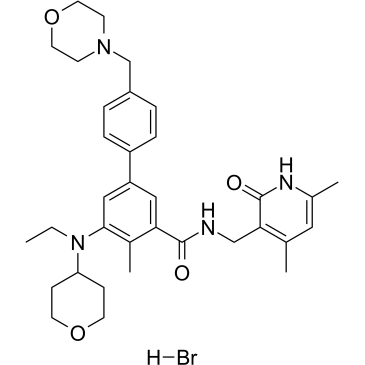
-
GC17672
TC-E 5003
TC-E 5003 es un inhibidor selectivo de PRMT1 con una IC50 de 1,5 μM frente a hPRMT1.
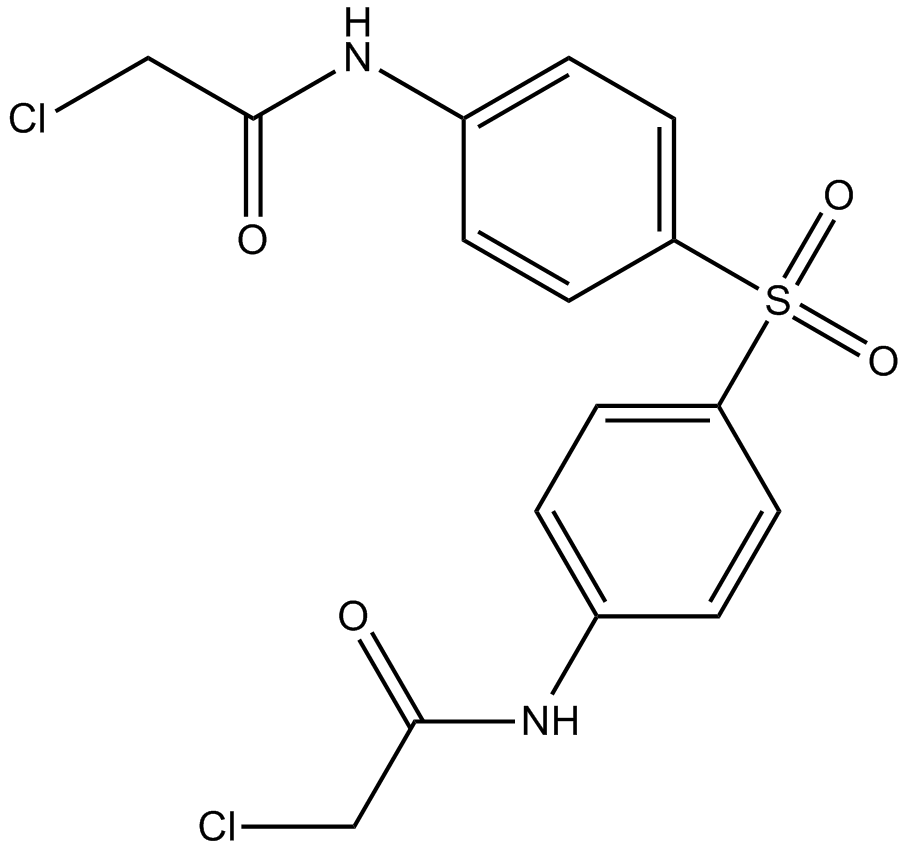
-
GC70041
TNG908
TNG908 es un inhibidor de PRMT5 sinérgico con MTAP que puede atravesar la barrera hematoencefálica. TNG908 tiene una selectividad 15 veces mayor para las células MTAPnull en comparación con las células MTAPWT y se puede utilizar en investigaciones sobre el cáncer.
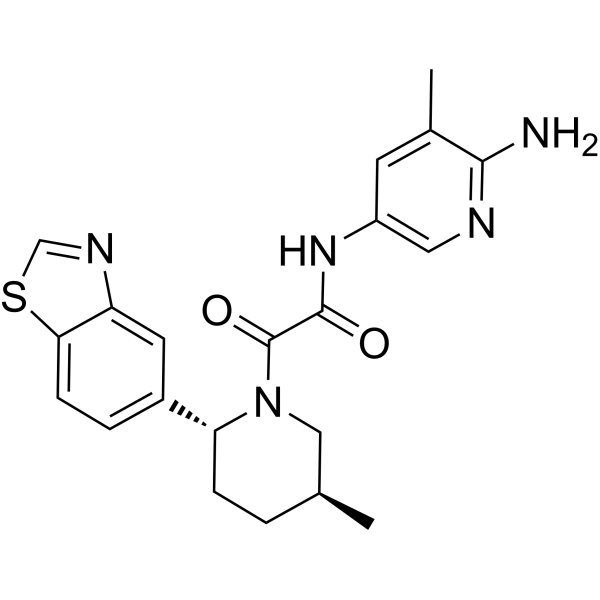
-
GC45763
Tolcapone-d4
An internal standard for the quantification of tolcapone
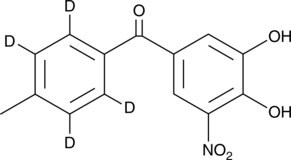
-
GC41263
TP-064
TP-064 es un inhibidor potente y selectivo de proteinarginina metiltransferasa 4 (PRMT4; CARM1) (IC50 <10 nM).
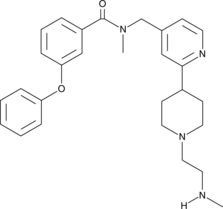
-
GC15688
UNC 0224
A potent inhibitor of G9a histone methyltransferase
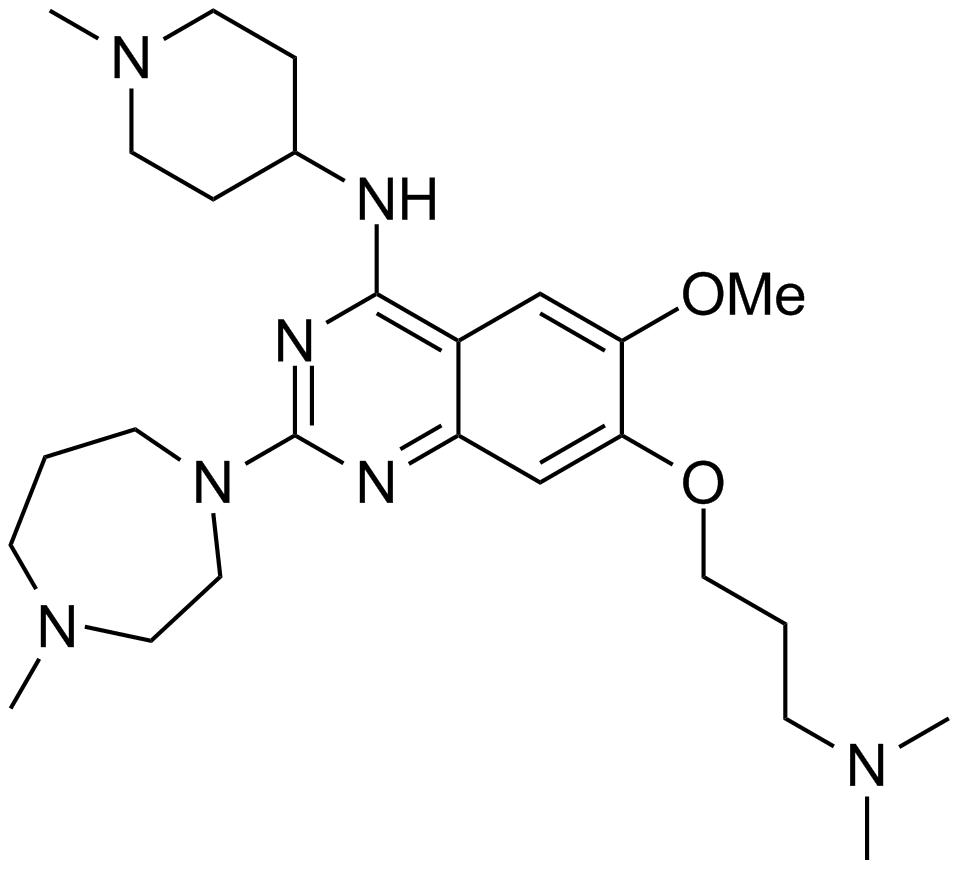
-
GC12548
UNC 0631
UNC 0631 es un potente inhibidor de la histona metiltransferasa G9a con una IC50 de 4 nM. UNC 0631 reduce de forma potente los niveles de H3K9me2 en células MDA-MB-231 con una IC50 de 25 nM.
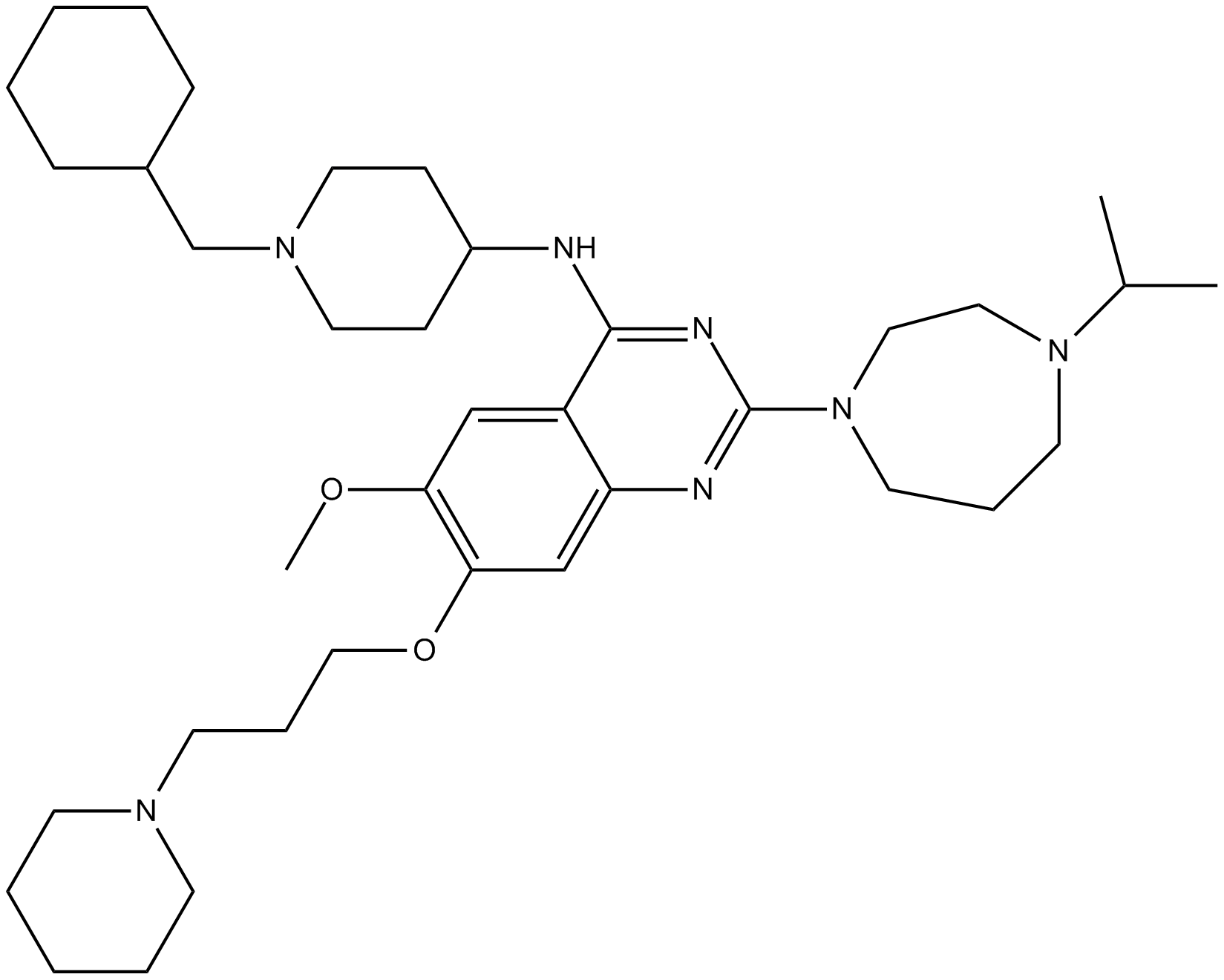
-
GC14249
UNC 0642
UNC 0642 es un inhibidor potente y selectivo de las lisina metiltransferasas G9a y GLP, con una IC50 de \u003c2,5 nM para G9a.
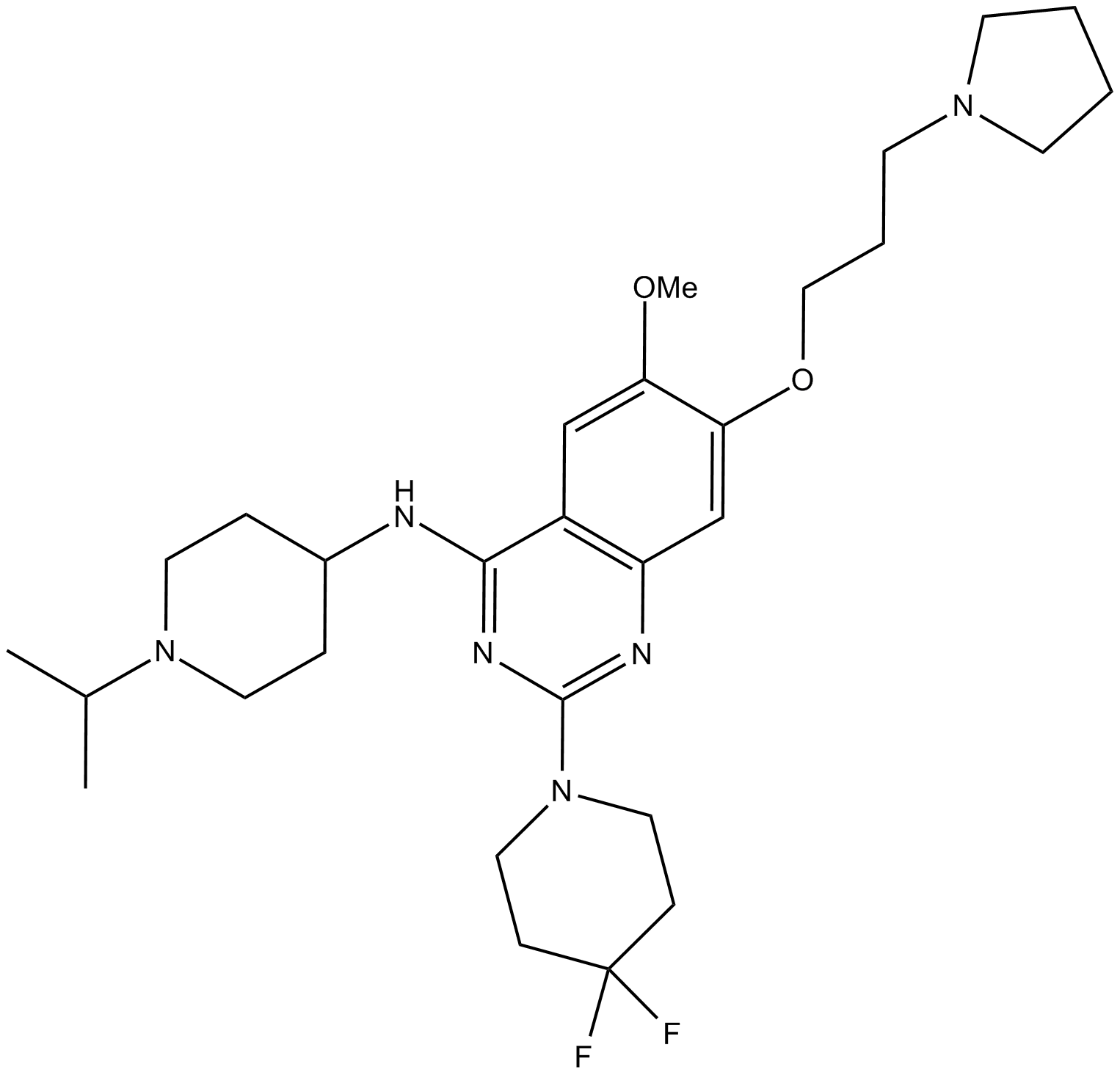
-
GC16518
UNC 0646
An inhibitor of G9a/GLP methyltransferases
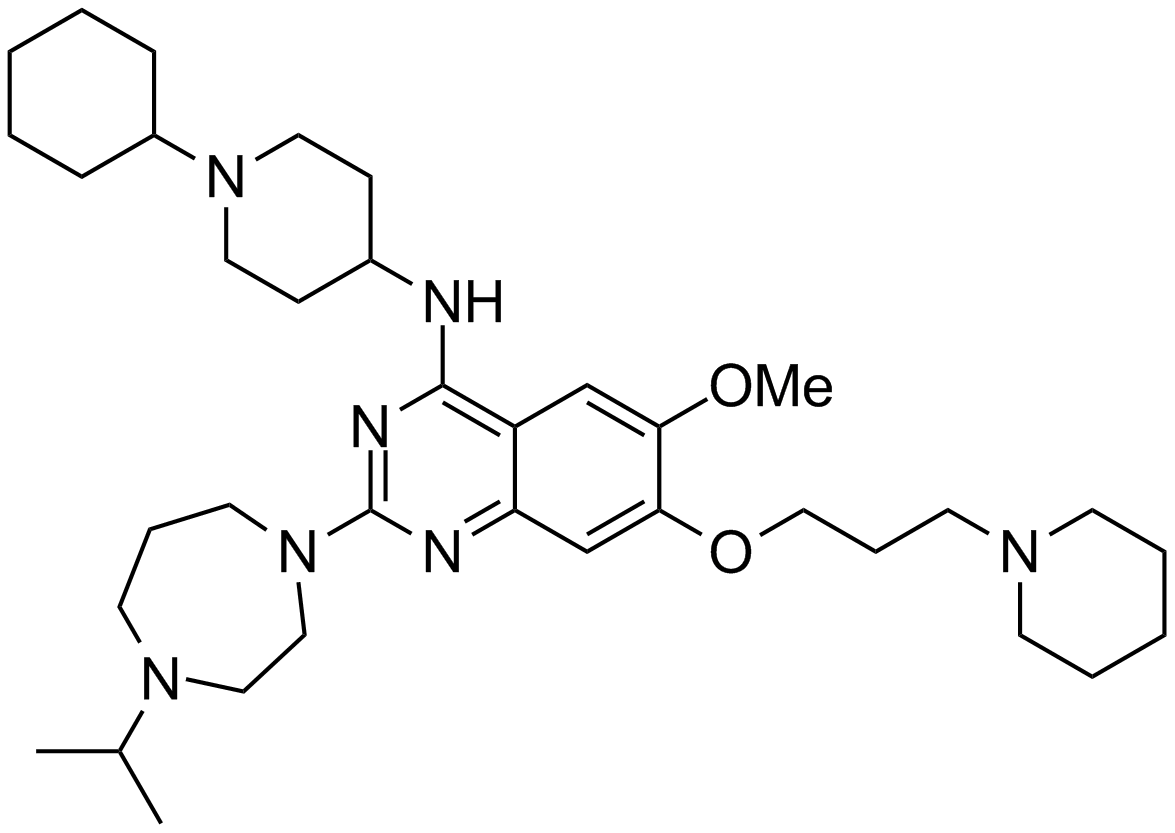
-
GC50082
UNC 2399
UNC 2399, un UNC1999 biotinilado, es un degradador selectivo de EZH2 que mantiene una alta potencia in vitro para EZH2, con una IC50 de 17 nM.
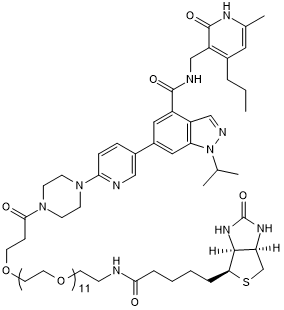
-
GC15652
UNC 2400
UNC 2400 es un análogo cercano de UNC1999 con una potencia \u003e1000 veces menor que UNC1999 como control negativo para estudios basados \u200b\u200ben células.
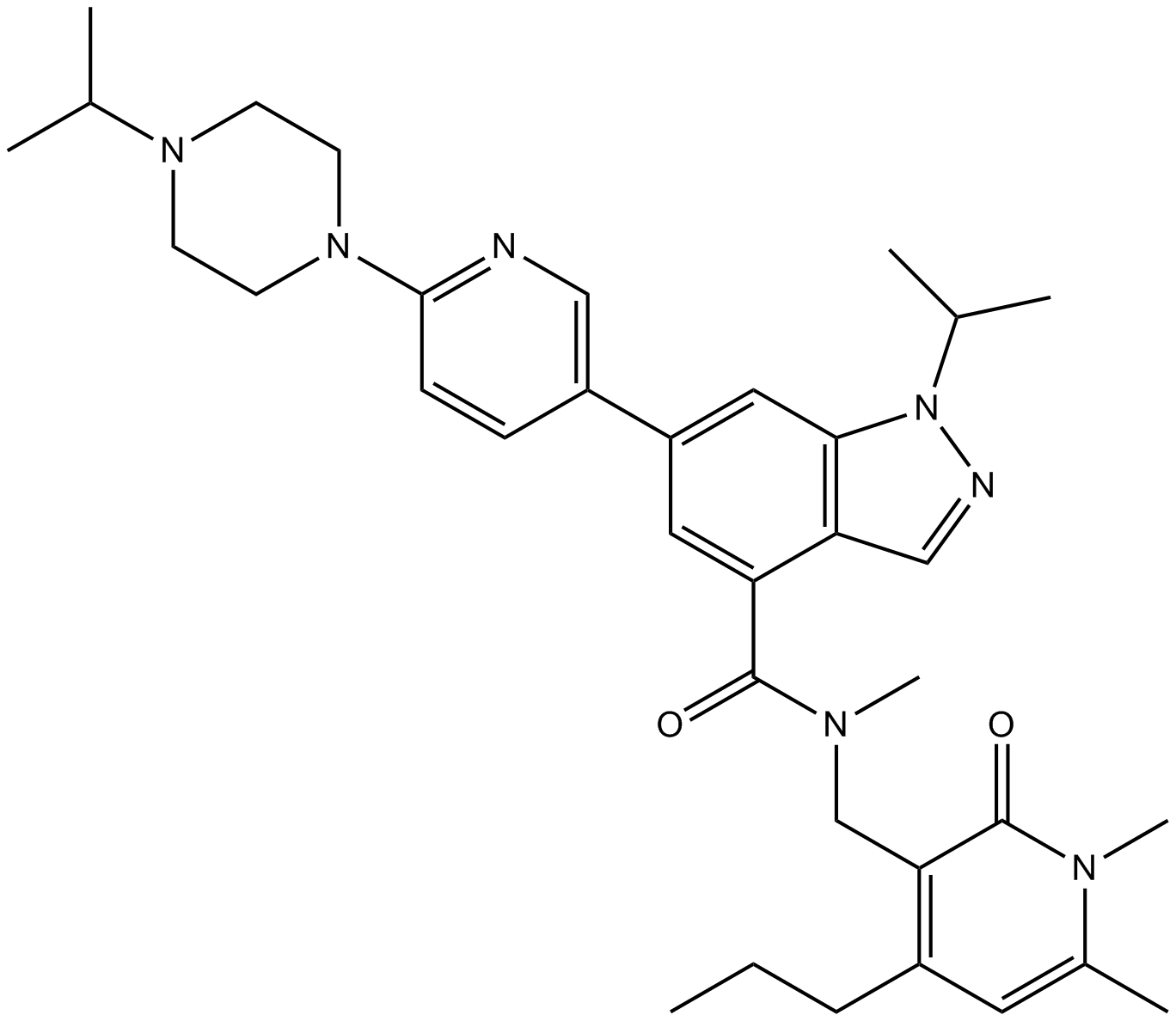
-
GC37856
UNC0321
UNC0321 es un inhibidor potente y selectivo de la histona metiltransferasa G9a con una Ki de 63 pM y con valores IC50 dependientes del ensayo de 6-9 nM. UNC0321 también inhibe GLP con valores IC50 dependientes del ensayo de 15-23 nM. UNC0321 estÁ inactivo frente a SET7/9, SET8/PreSET7, PRMT3 y JMJD2E.
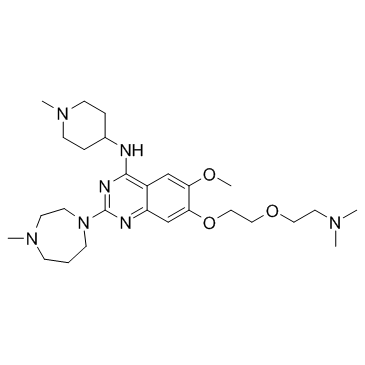
-
GC16741
UNC0379
UNC0379 es un inhibidor selectivo de sustrato competitivo de la lisina metiltransferasa SETD8 (KMT5A) con una IC50 de 7,3 μM, valor KD de 18,3 μM.
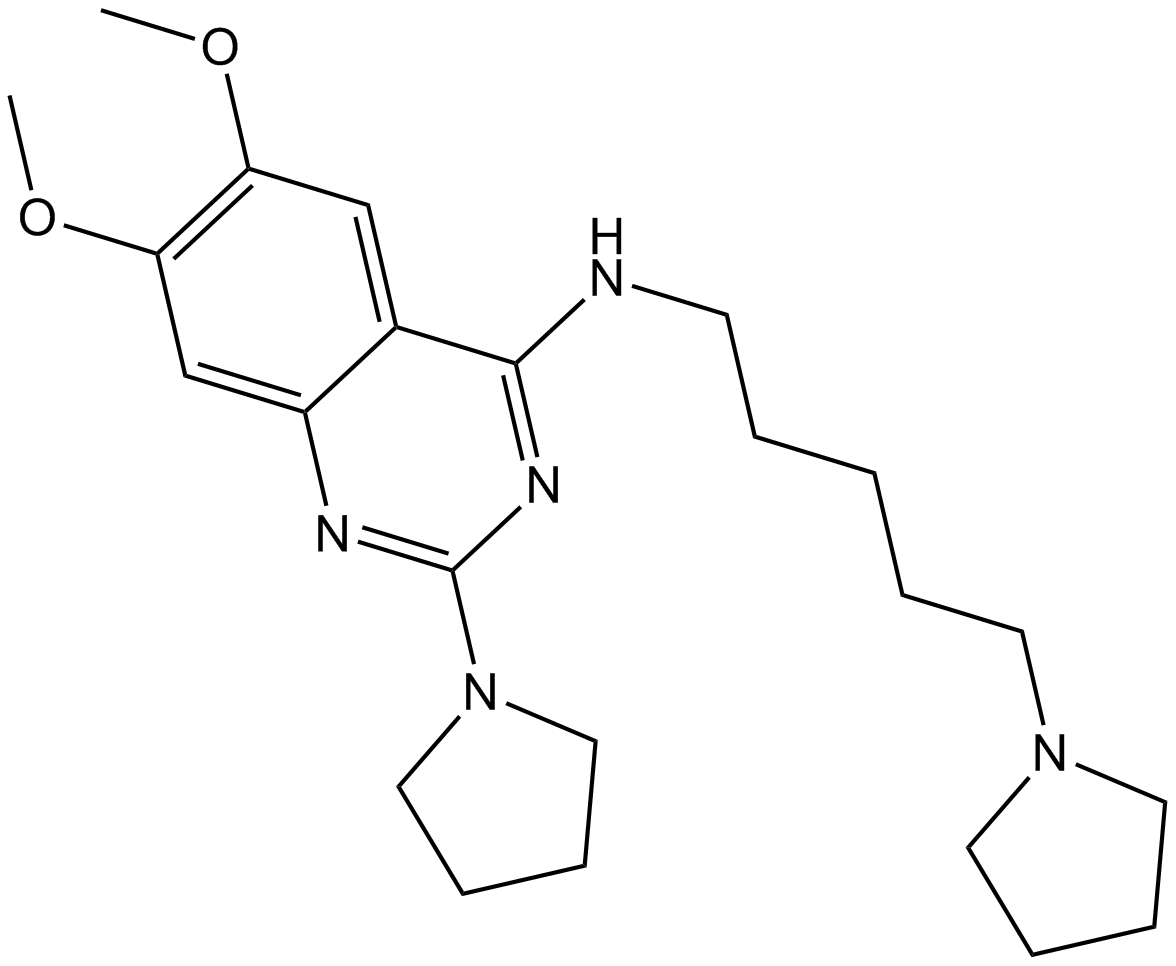
-
GC34160
UNC0379 trifluoroacetate (UNC-0379 trifluoroacetate)
El trifluoroacetato UNC0379 (trifluoroacetato UNC-0379) es un inhibidor selectivo competitivo de sustrato de la lisina metiltransferasa SETD8 (KMT5A) con un IC50 de 7,3 μM, valor KD de 18,3 μM.
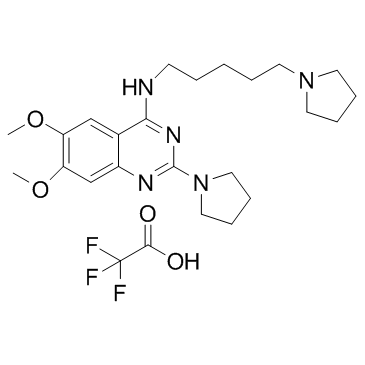
-
GC15610
UNC0638
A G9a and GLP histone methyltransferase inhibitor
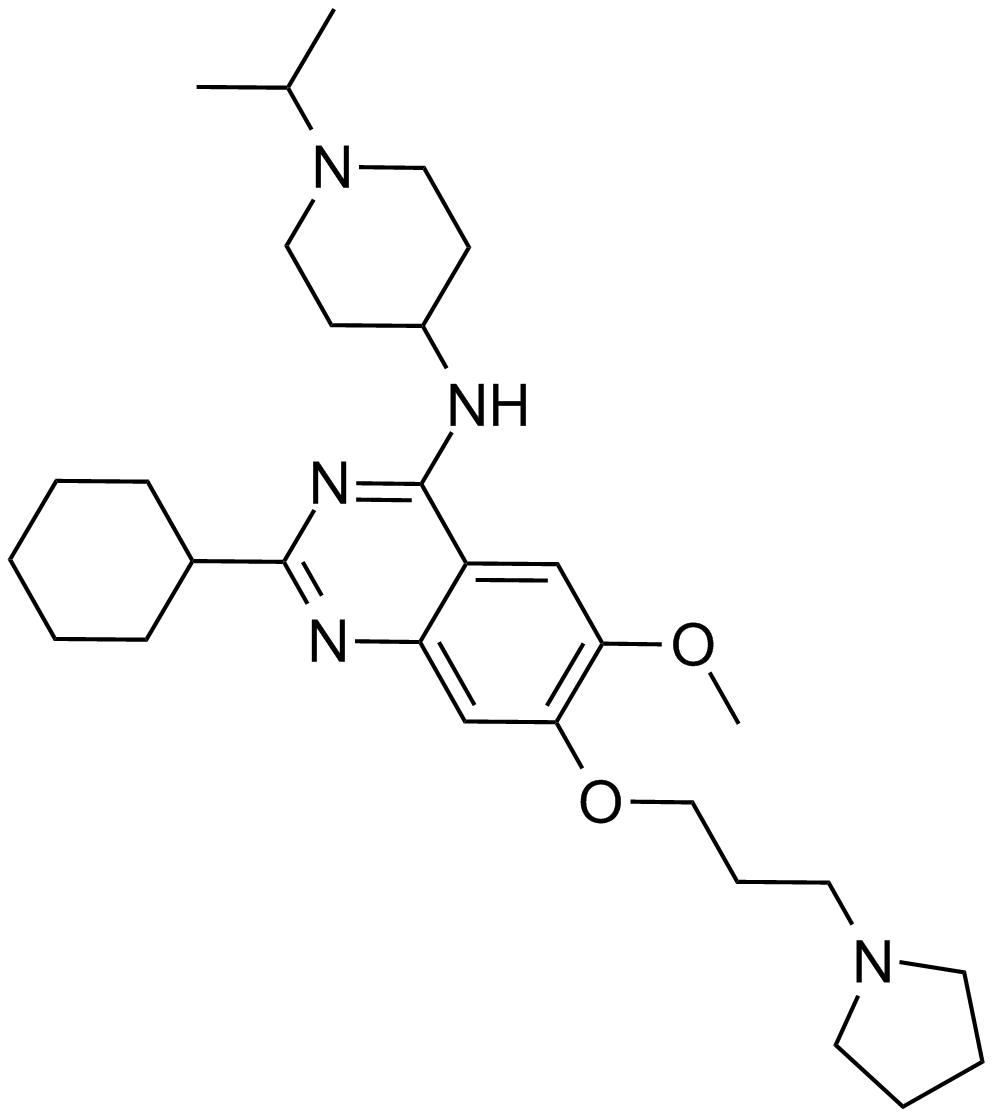
-
GC16794
UNC1215
Potent L3MBTL3 domain inhibitor
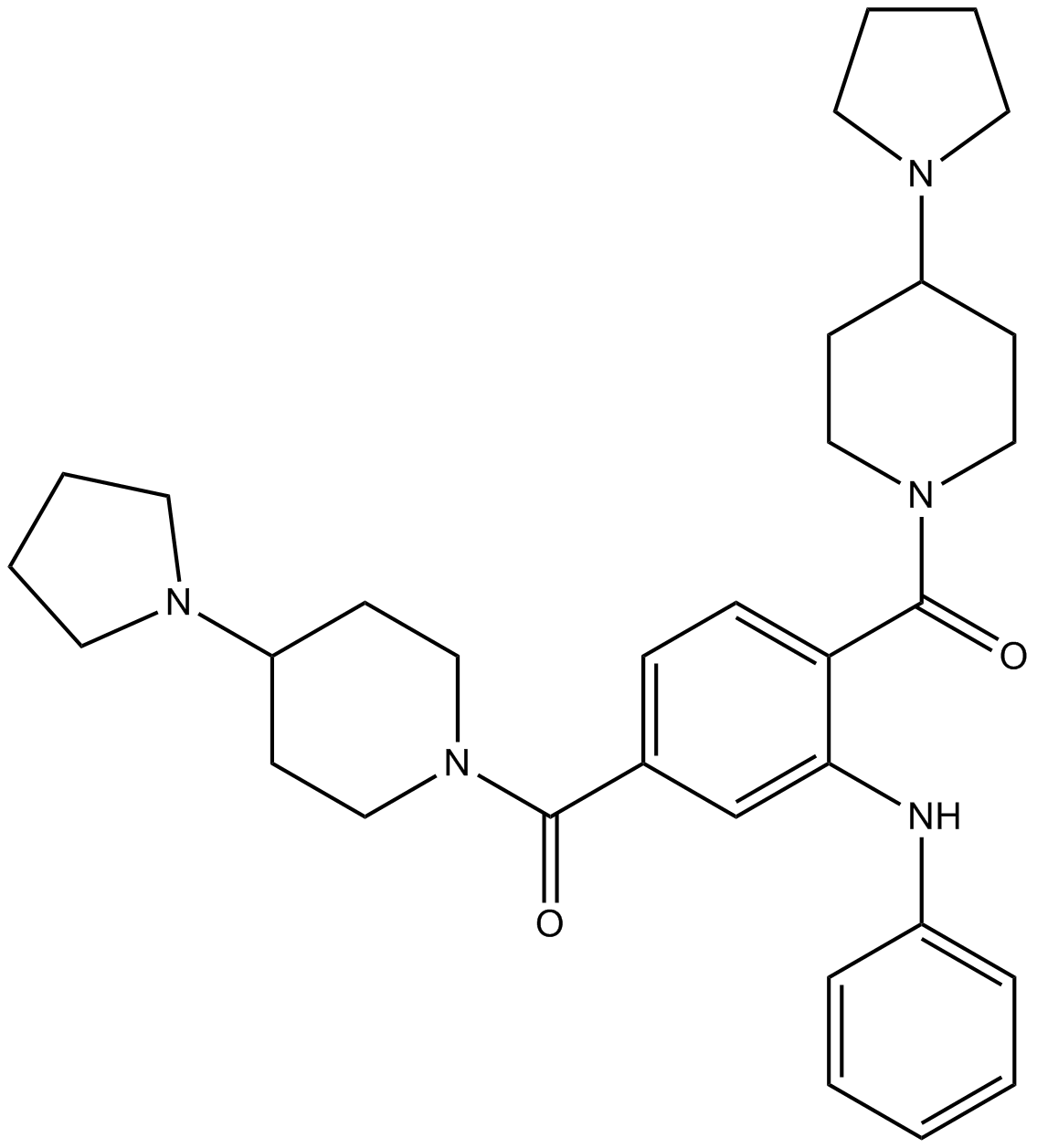
-
GC11375
UNC1999
UNC1999 es un inhibidor selectivo, potente y competitivo de SAM de EZH2/1 con IC50 de <10 nM y 45 nM, respectivamente.
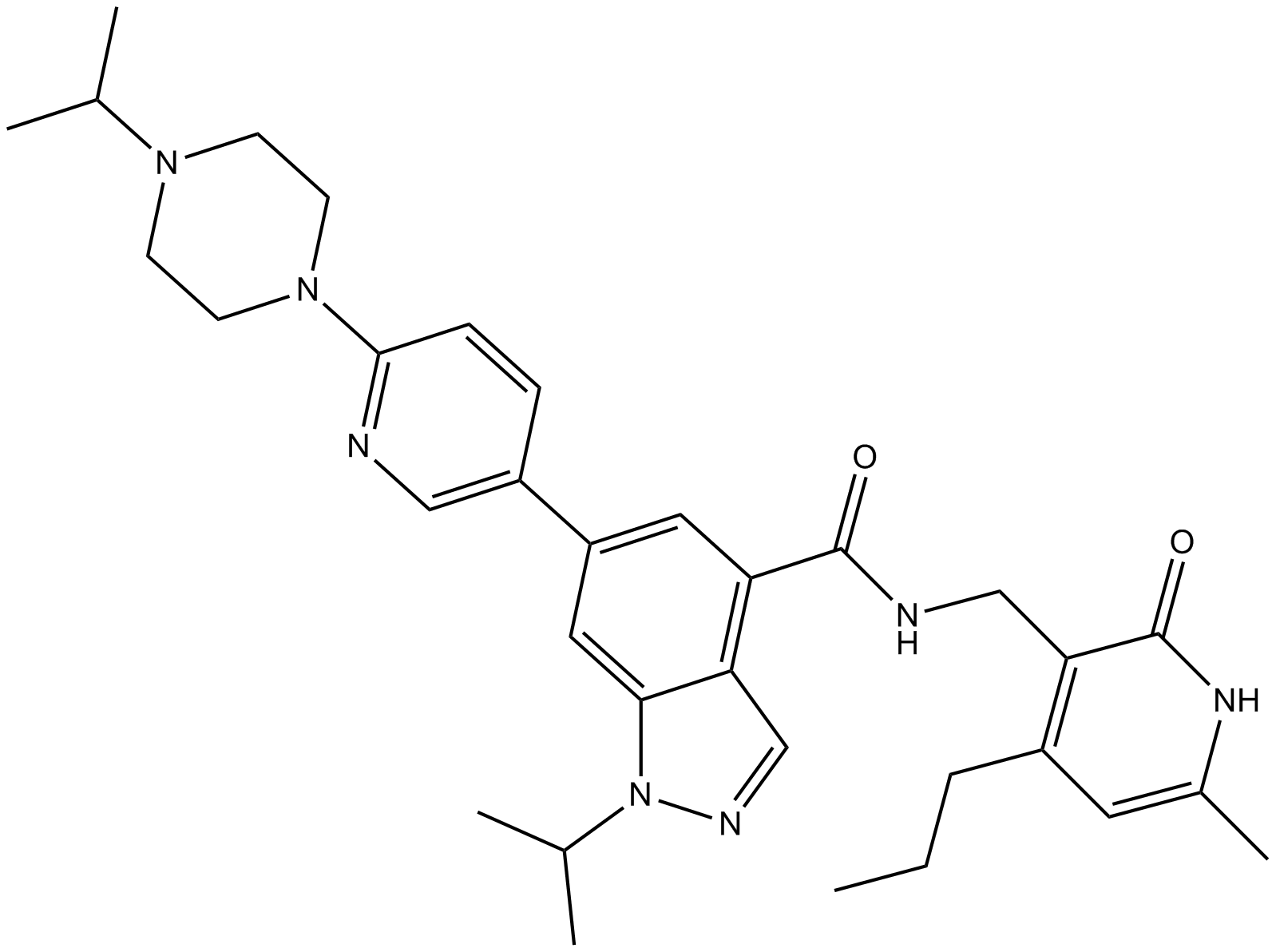
-
GC45117
UNC2327
UNC2327 es un inhibidor alostérico de la proteÍna arginina metiltransferasa 3 (PRMT3).
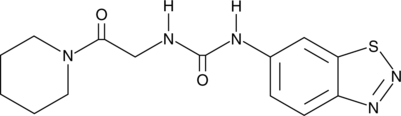
-
GC17207
UNC3866
UNC3866 es un potente antagonista de la interacciÓn CBX7-H3 segÚn lo determinado por AlphaScreen (IC50=66±1,2 nM) y es mÁs de 100 veces mÁs selectivo para CBX7 que para los otros nueve miembros de este panel de lectores de metil-lisina (Kme).
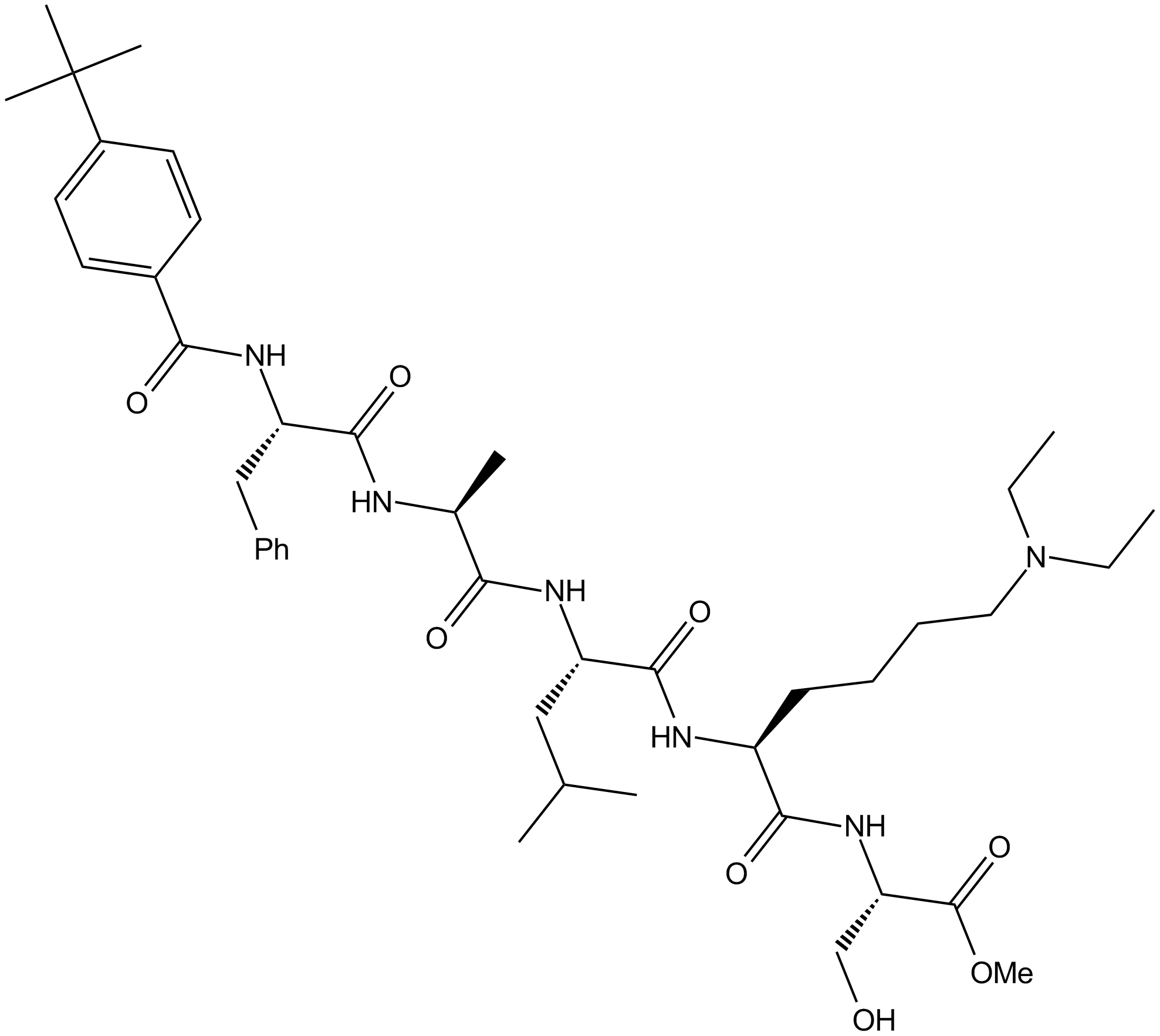
-
GC70091
UNC4976 TFA
UNC4976 TFA es un péptido mimético de modulación alósterica positiva (PAM) que se une al dominio estructural de cromatina CBX7 y a los ácidos nucleicos. UNC4976 TFA antagoniza la reclutación específica de CBX7 por H3K27me3 en genes diana, mientras aumenta la unión no específica con el ADN y ARN.
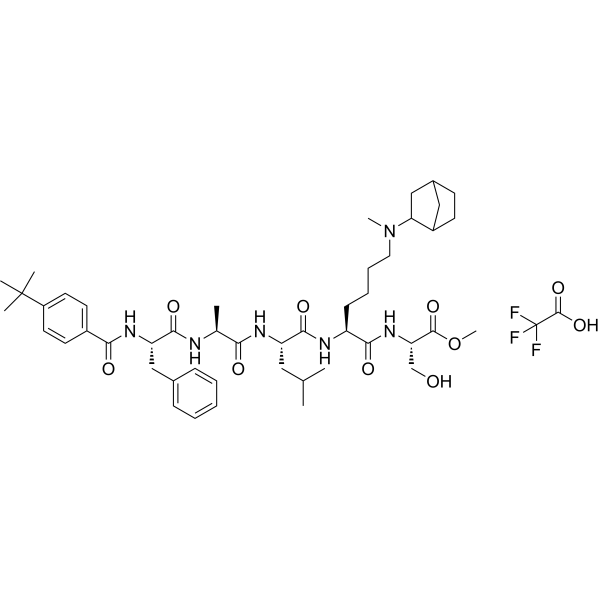
-
GC18096
UNC669
UNC669, un ligando para un dominio de unión de metil-lisina, es un potente inhibidor de L3MBTL1 (IC50 \u003d 4,2 uM) y L3MBTL3 (3,1 uM).
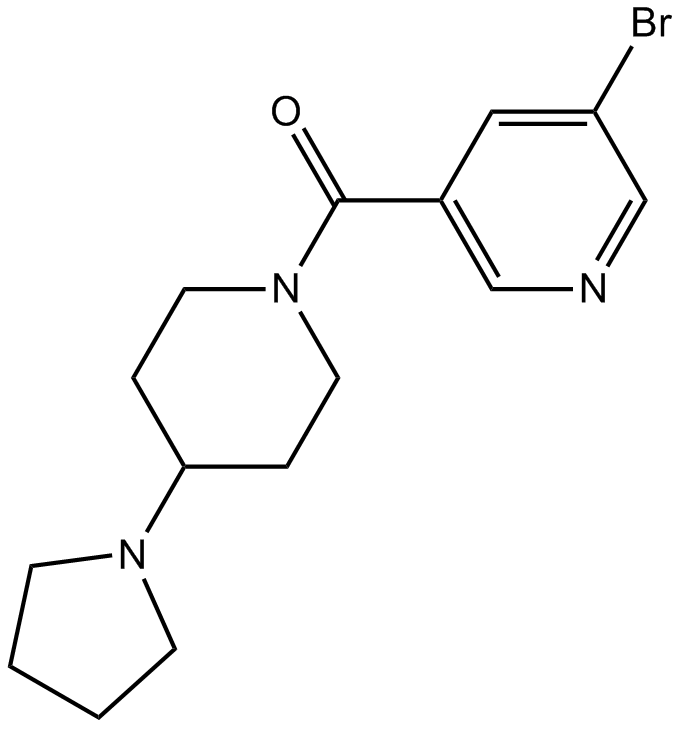
-
GC64083
UNC6934
UNC6934, una sonda quÍmica dirigida al dominio PWWP, altera la localizaciÓn nucleolar de NSD2.
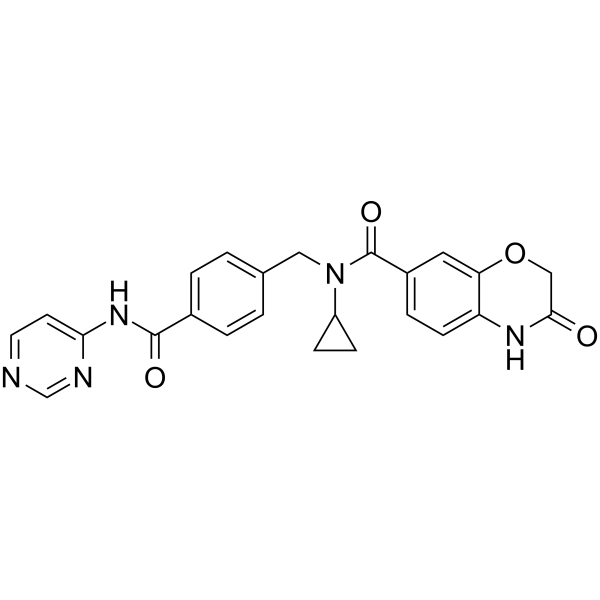
-
GC33397
Valemetostat
Valemetostat (DS-3201) es un inhibidor dual EZH1/2 de primera clase, utilizado en la investigaciÓn del linfoma de células T periférico en recaÍda/refractario.
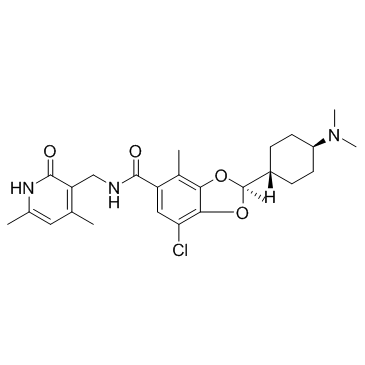
-
GC37881
Valemetostat tosylate
Tosilato de valemetostat (tosilato DS-3201), un inhibidor dual EZH1/2 de primera clase. El tosilato de valemetostat se puede utilizar para la investigaciÓn del linfoma periférico de células T en recaÍda o refractario.
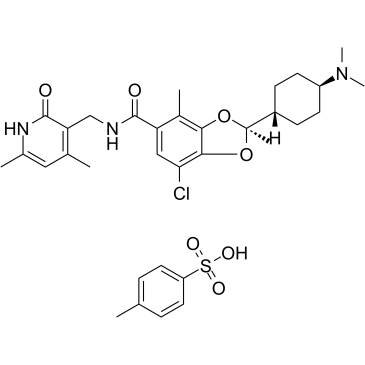
-
GC14763
WDR5 0103
WDR5 0103 es un potente y selectivo antagonista de la proteína 5 que contiene repeticiones de WD (WDR5) con Kd de 450 nM.
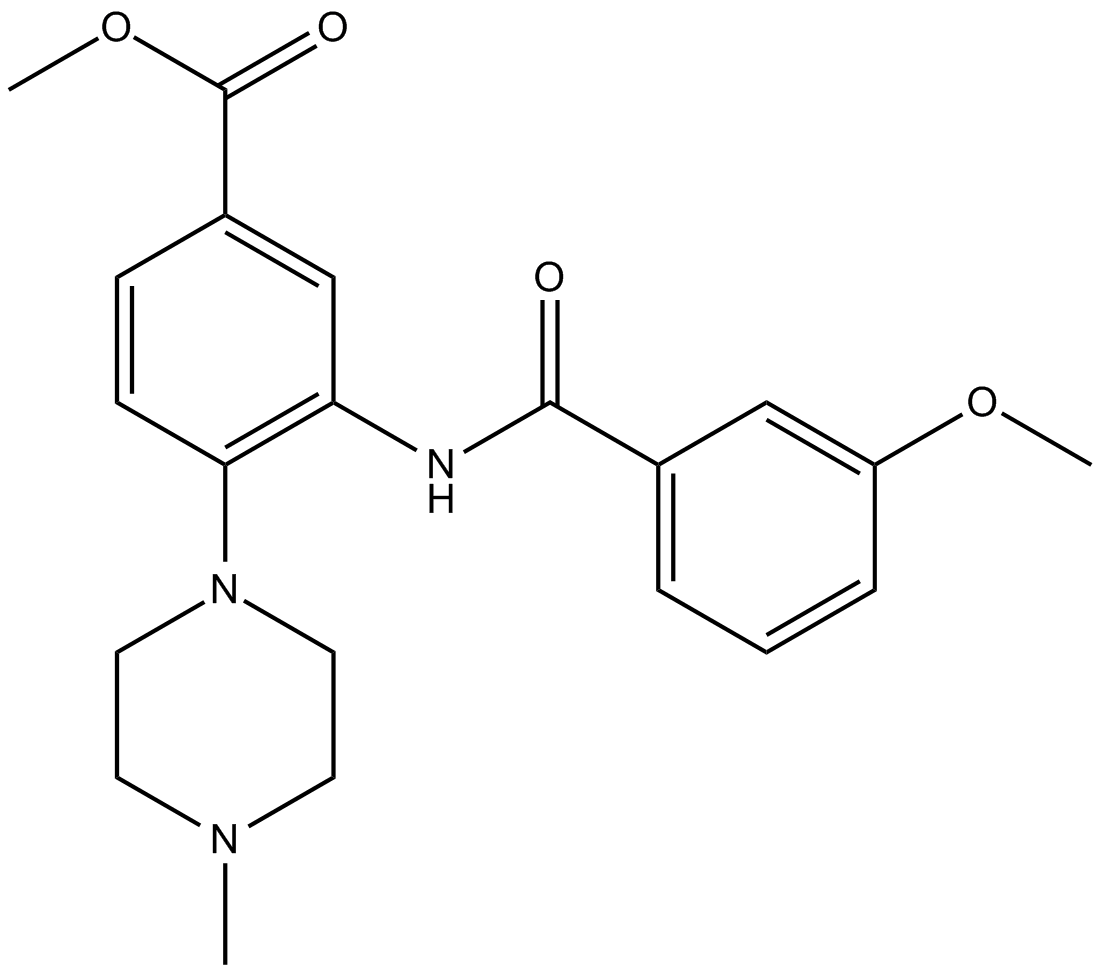
-
GC62558
WDR5-IN-1
WDR5-IN-1 es un inhibidor potente y selectivo del dominio 5 de repeticiÓn de WD (WDR5), con una Kd de <0,02 nM. WDR5-IN-1 inhibe la actividad de la histona metiltransferasa (HMT) de MLL1 con una IC50 de 2,2 nM. WDR5-IN-1 disminuye el reclutamiento de MYC en los genes desplazados por WDR5 y muestra potentes efectos antiproliferativos en las lÍneas CHP-134 (neuroblastoma) y Ramos (linfoma de Burkitt).
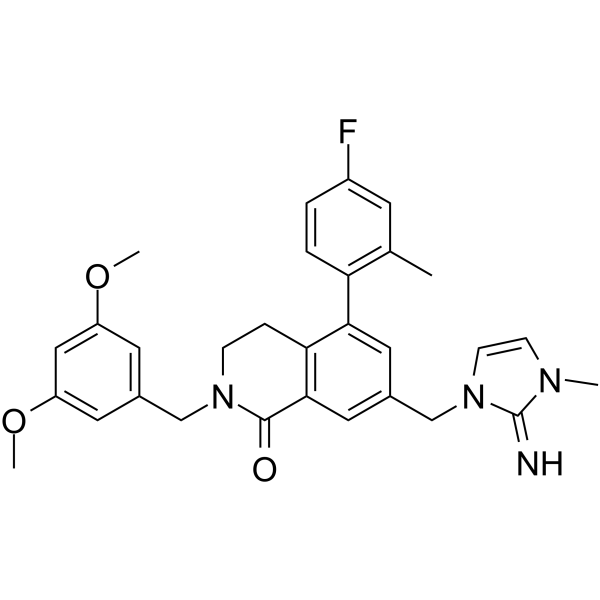
-
GC19388
XY1
XY1 es un anÁlogo muy cercano de SGC707 (un inhibidor potente, selectivo y no competitivo de PRMT3 con IC50 de 31 nM), pero XY1 es completamente inactivo.

-
GC45609
ZLD1039
ZLD1039 es un inhibidor de EZH2 potente, altamente selectivo y biodisponible por vÍa oral. ZLD1039 muestra una inhibiciÓn potente y dependiente de la concentraciÓn de la actividad enzimÁtica de PRC2 contra EZH2 de tipo salvaje, asÍ como enzimas mutantes Y641F y A677G con valores IC50 de 5,6, 15 y 4,0nM, respectivamente. ZLD1039 inhibe el crecimiento y la metÁstasis de tumores de mama.