Histone Methyltransferase
Histone methyltransferases are a group of enzymes that catalyze the methylation of histone lysine and arginine by adding methyl groups to specific histone arginine or lysine residues. Histone methyltransferaes can be classified into 3 classes, including SET domain lysine methyltransferases, non-SET domain lysine methyltransferases and arginine methyltransferases (PRMTs), all of which use S-adenosylmethionine as a cosubstrate for the transfer of the methyl group. Aberrant histone methylation has been associated with a wide range of human cancers (such as hematological malignancies), which leads to the development of novel cancer chemotherapies targeting cancer-associated histone methyltransferases (more than 20 lysine methyltransferases and 9 arginine methyltransferases in humans).
Products for Histone Methyltransferase
- Cat.No. Nom du produit Informations
-
GC14585
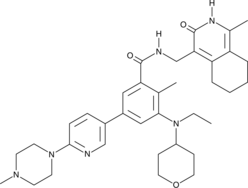
GSK591
GSK591 (EPZ015866) est un inhibiteur puissant et sélectif de la protéine méthyltransférase 5 (PRMT5) avec une IC50 de 4 nM.
-
GP10020
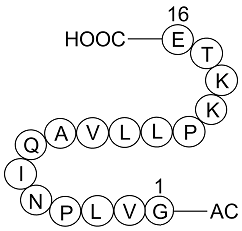
Histone-H2A-(107-122)-Ac-OH
Histone-H2A peptide
-
GC17023
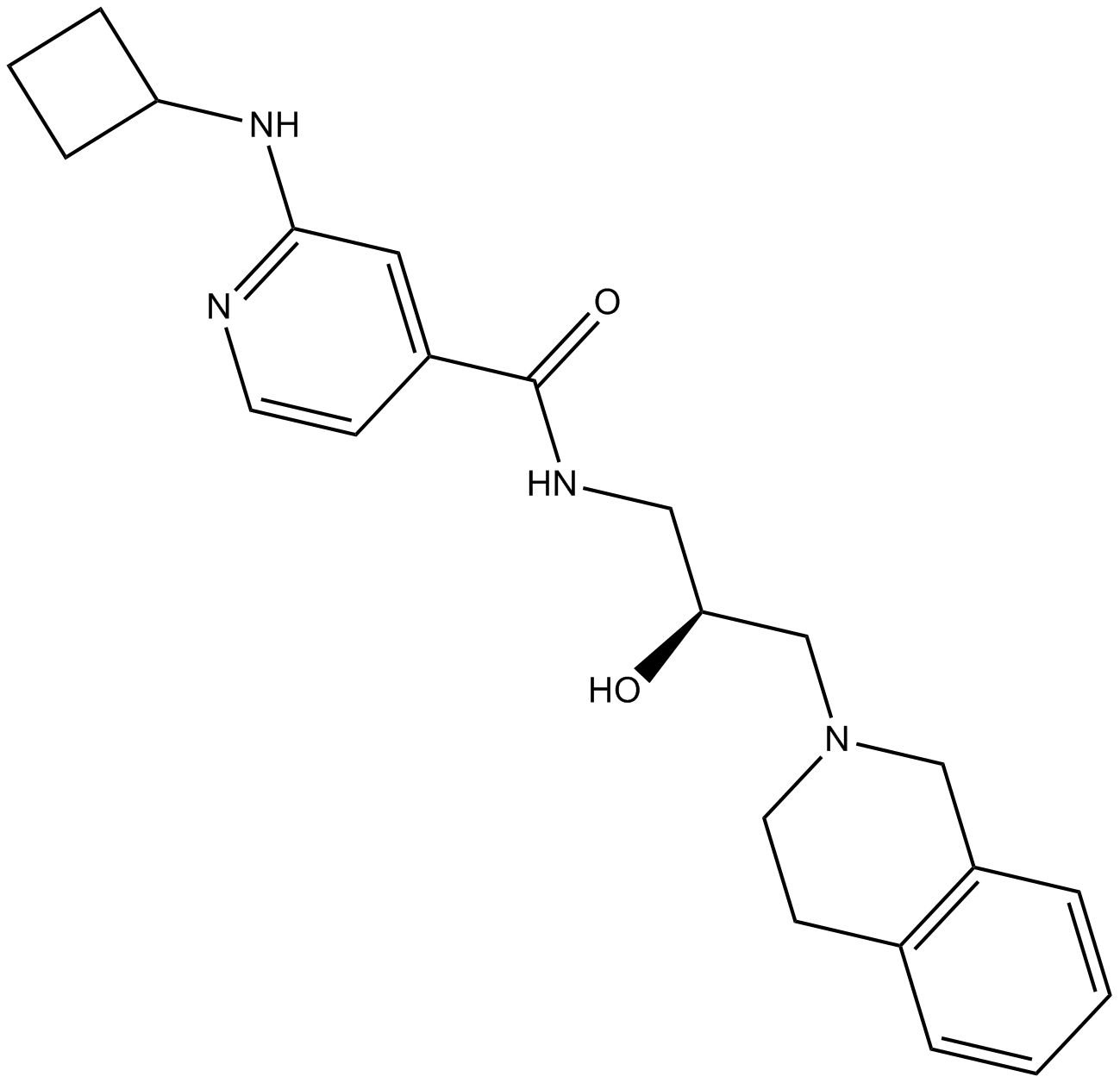
HLCL-61
HLCL-61 est un inhibiteur de premier ordre de la protéine arginine méthyltransférase 5 (PRMT5).
-
GC19465
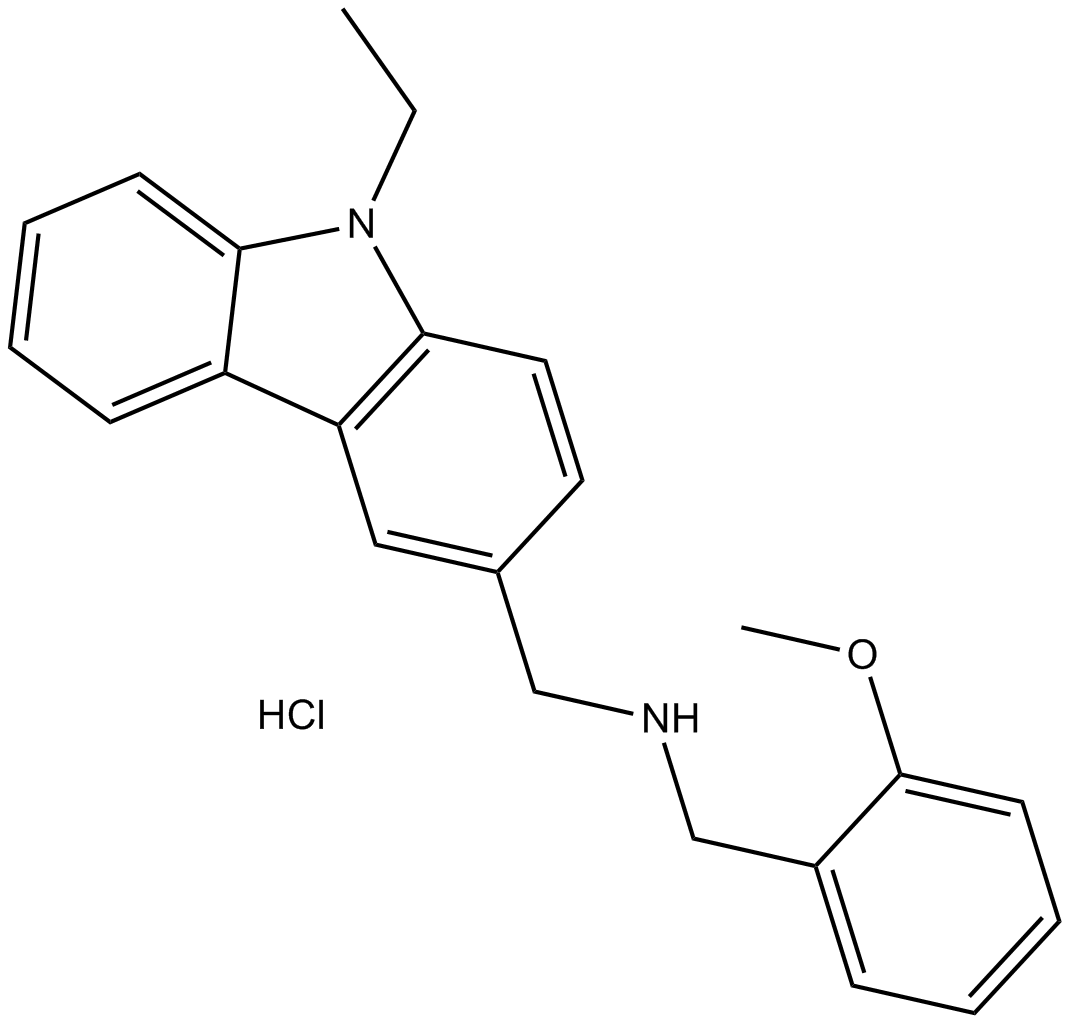
JNJ-64619178
JNJ-64619178 (Onametostat) est un inhibiteur sélectif, actif par voie orale et pseudo-irréversible de la protéine arginine méthyltransférase 5 (PRMT5) avec une IC50 de 0,14 nM. JNJ-64619178 a une activité puissante dans le cancer du poumon.

-
GC19211
JQEZ5
JQEZ5 est un inhibiteur puissant et sélectif de la lysine méthyltransférase EZH2. JQEZ5 SAM-inhibition compétitive du complexe répressif polycomb 2 (PRC2) avec une IC50 de 80 nM. JQEZ5 a des effets anti-tumoraux.
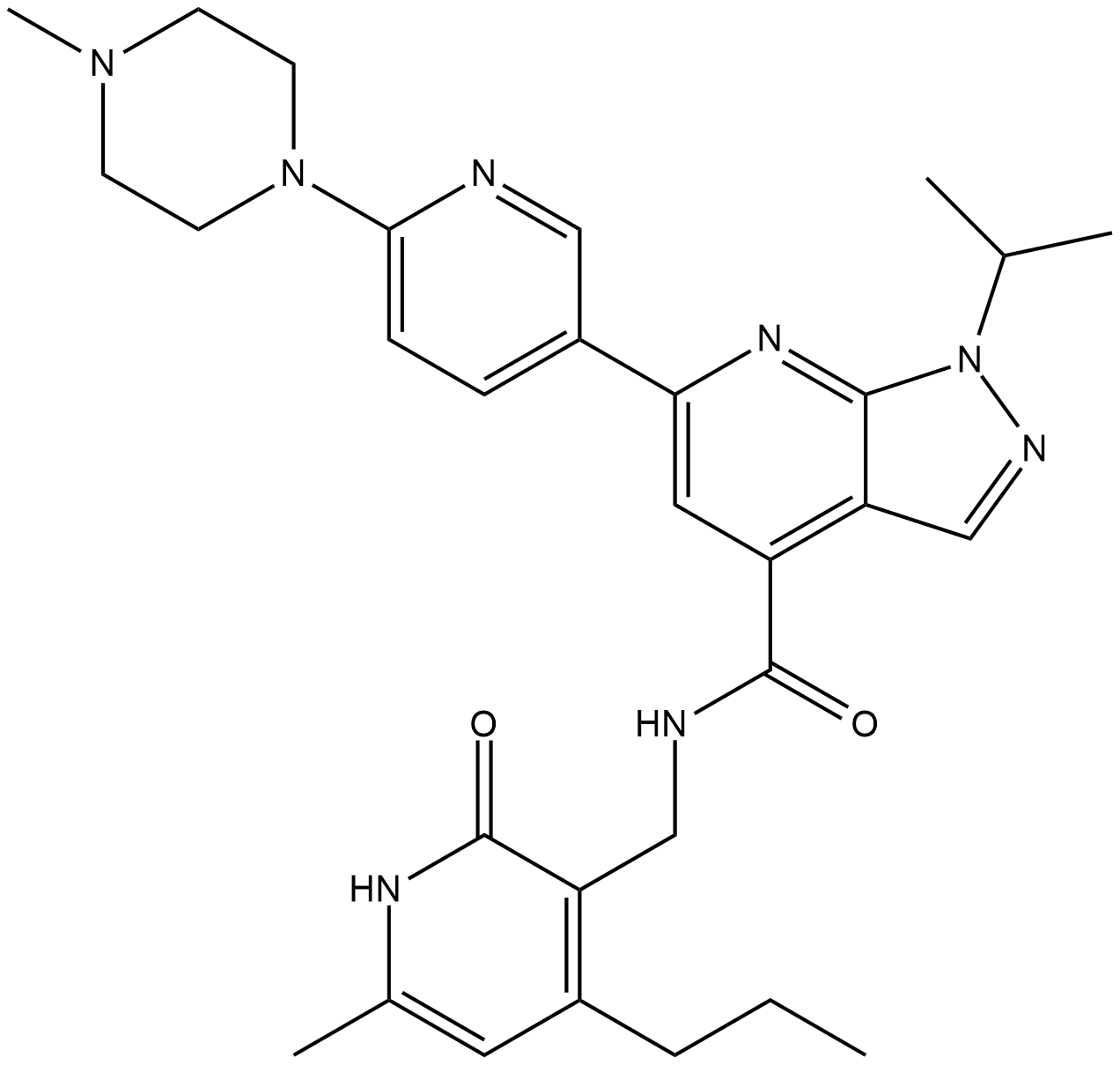
-
GC33184
LLY-283
LLY-283 est un inhibiteur puissant, sélectif et oral de la protéine arginine méthyltransférase 5 (PRMT5), avec une IC50 de 22 nM et un Kd de 6 nM pour le complexe PRMT5:MEP50, et présente une activité antitumorale.
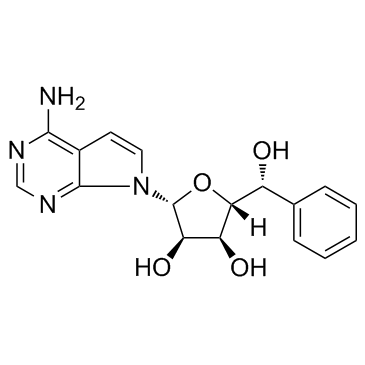
-
GC16261
LLY507
LLY507 est un inhibiteur puissant et sélectif de la protéine-lysine méthyltransférase SMYD2. LLY507 inhibe puissamment la capacité de SMYD2 à méthyler le peptide p53 avec une IC50 \u003c15 nM. LLY507 sert de sonde chimique précieuse pour aider à la dissection de la fonction SMYD2 dans le cancer et d'autres processus biologiques.
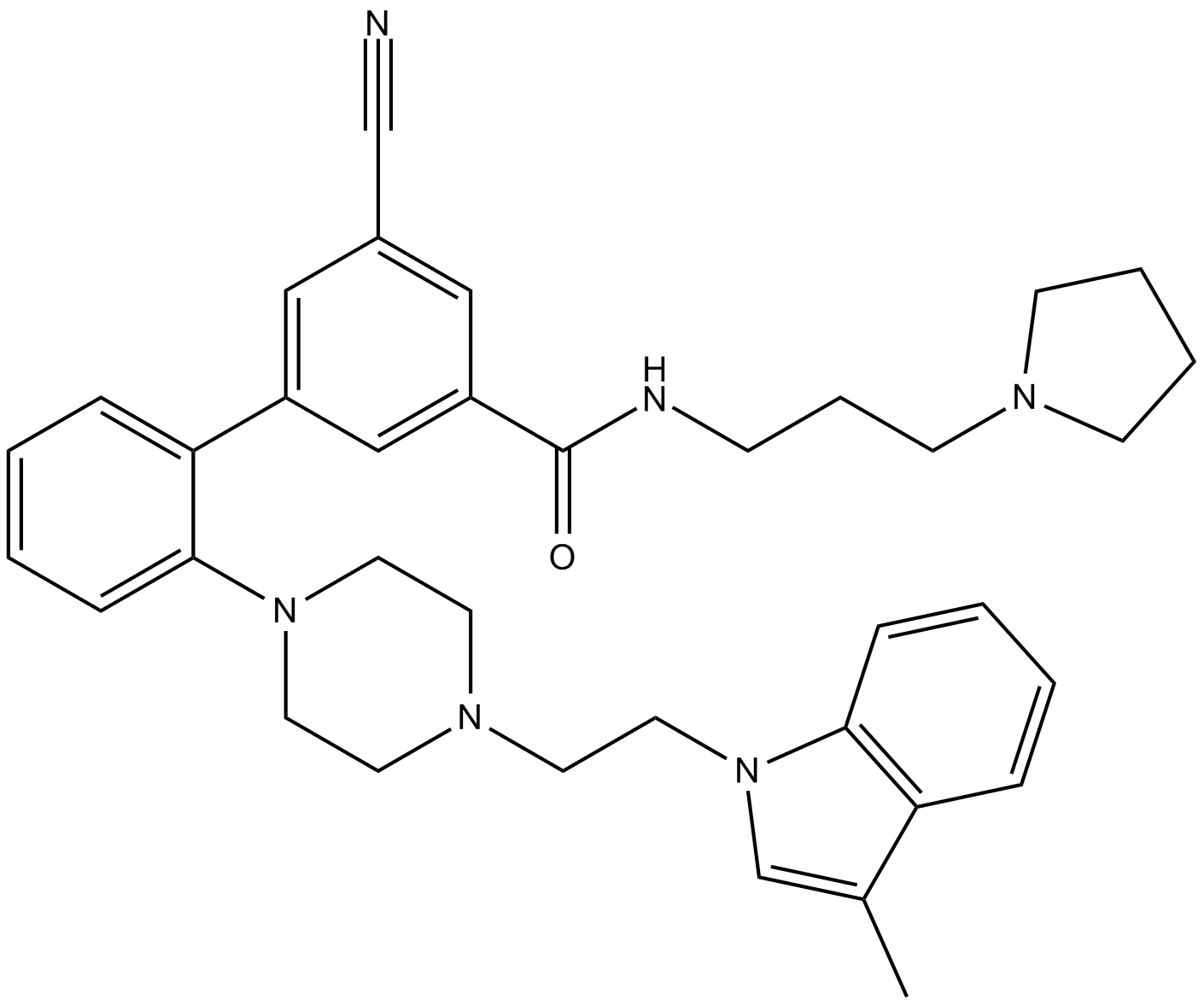
-
GC63056
MAK683-CH2CH2COOH
MAK683-CH2CH2COOH se lie À l'EED (protéine de développement de l'ectoderme embryonnaire). MAK683-CH2CH2COOH et un ligand VHL pour l'ubiquitine ligase E3 ont été utilisés pour concevoir PROTAC EED degrader-1 et PROTAC EED degrader-2.
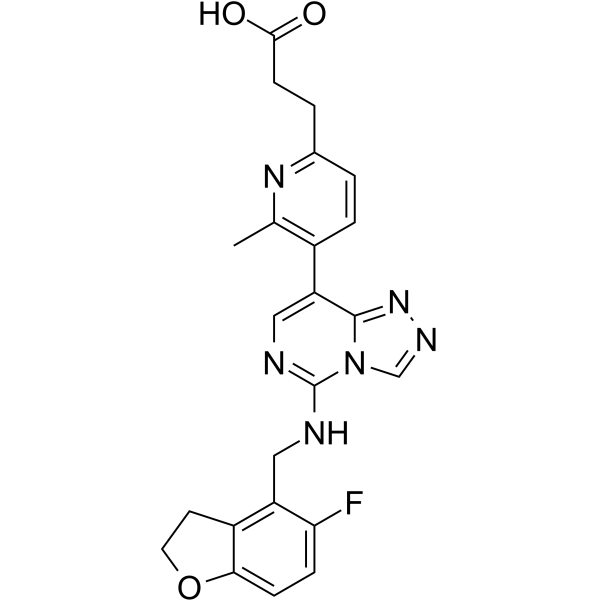
-
GC65254
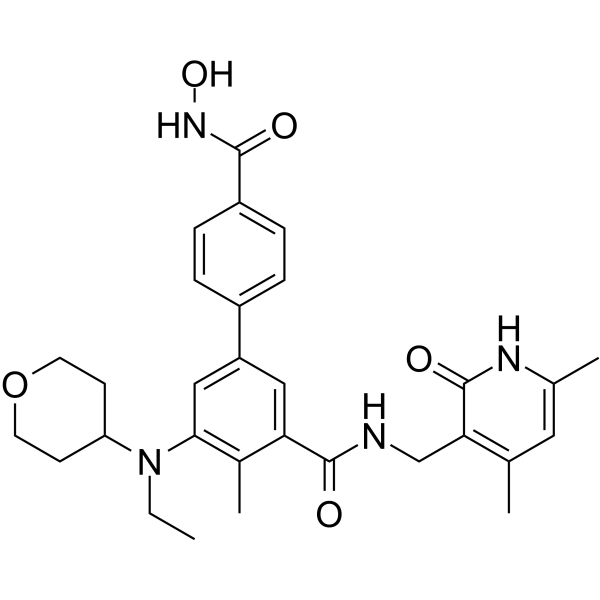
MC4355
Le MC4355 est un double inhibiteur d'EZH2 et d'histone désacétylase (HDAC).
-
GC61058
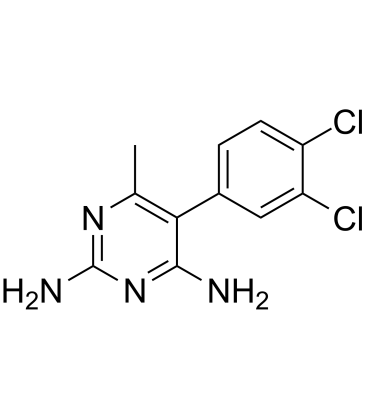
Metoprine
La métoprine (BW 197U) est un puissant inhibiteur de l'histamine N-méthyltransférase (HMT).
-
GC14652
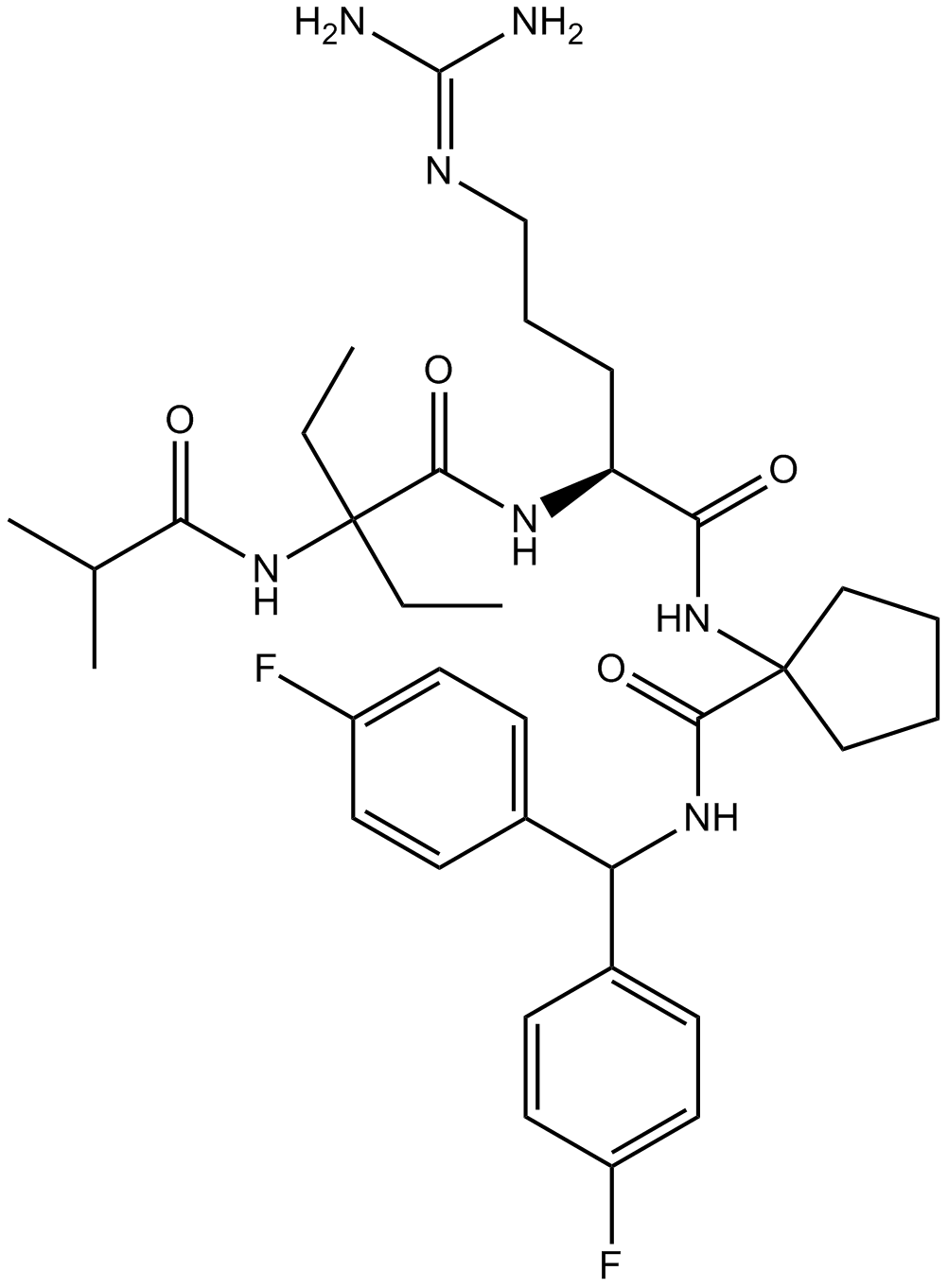
MM-102
MM-102 (HMTase Inhibitor IX) est un puissant inhibiteur de l'interaction WDR5/MLL, atteint IC50 = 2,4 nM avec un Ki200 fois plus puissant que le peptide ARA.
-
GC36632
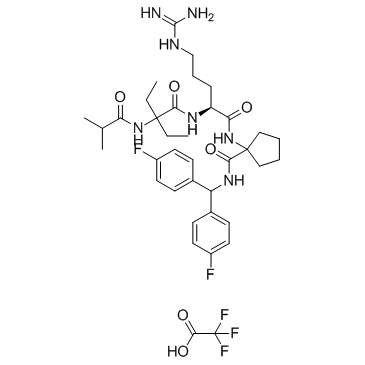
MM-102 TFA
Le MM-102 TFA (HMTase Inhibitor IX TFA) est un puissant inhibiteur de l'interaction WDR5/MLL, atteint IC50 = 2,4 nM avec un Ki estimé 200 fois plus puissant que le peptide ARA.
-
GC67758
MM-401 TFA
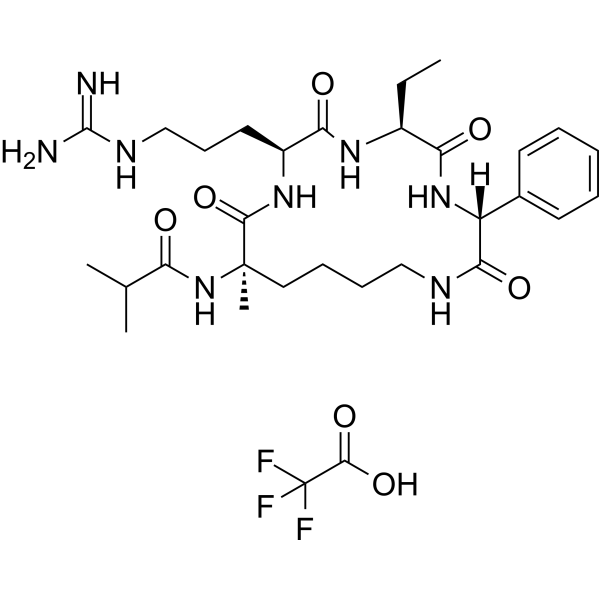
-
GC33278
MM-589
Le MM-589 est un puissant inhibiteur de l'interaction protéine-protéine du domaine de répétition WD (WDR5) et de la leucémie de lignée mixte (MLL). Le MM-589 se lie au WDR5 avec une IC50 de 0,90 nM et inhibe l'activité de la méthyltransférase MLL H3K4 avec une IC50 de 12,7 nM.
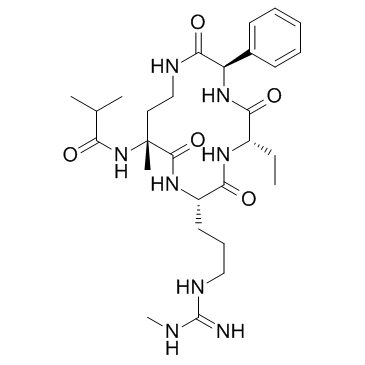
-
GC65037
MM-589 TFA
Le MM-589 TFA est un puissant inhibiteur de l'interaction protéine-protéine du domaine de répétition WD (WDR5) et de la leucémie de lignée mixte (MLL). Le MM-589 se lie au WDR5 avec une IC50 de 0,90 nM et inhibe l'activité de la méthyltransférase MLL H3K4 avec une IC50 de 12,7 nM.
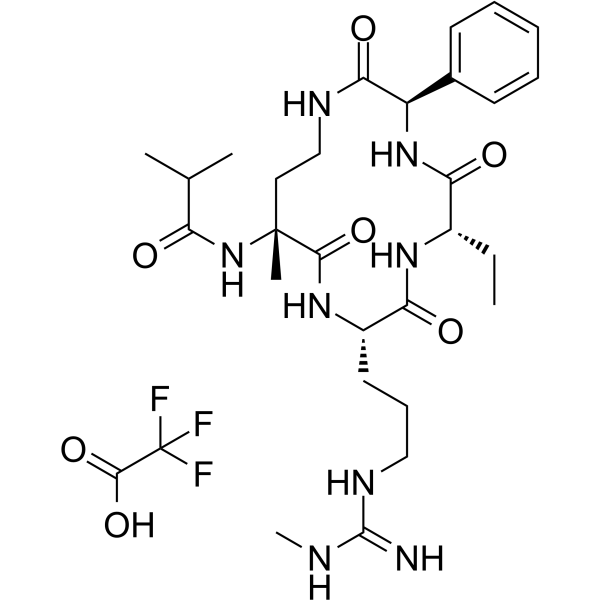
-
GC61468
MR837
MR837 est un inhibiteur de NSD2-PWWP1. MR837 peut se lier À la protéine 2 du domaine SET de liaison au récepteur nucléaire humain (domaine PWWP).
-
GC50576
MRK 740
Potent PRDM9 inhibitor
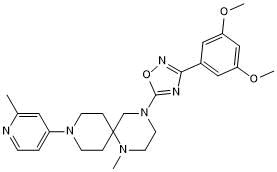
-
GC63558
MRTX-1719
MRTX-1719 est un inhibiteur sélectif de première classe puissant du complexe PRMT5/MTA, avec une IC50 de moins de 10 nM dans les cellules PRMT5/MTA MTAPDEL SDMA.
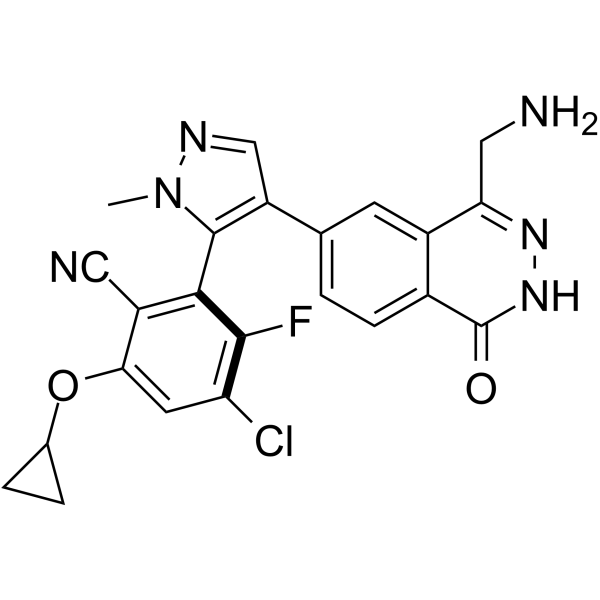
-
GC69499
MRTX-1719 hydrochloride
MRTX-1719 hydrochloride est un inhibiteur efficace, novateur et sélectif du complexe PRMT5/MTA. Son IC50 pour les cellules MTAPDEL SDMA de PRMT5/MTA est inférieur à 10 nM.
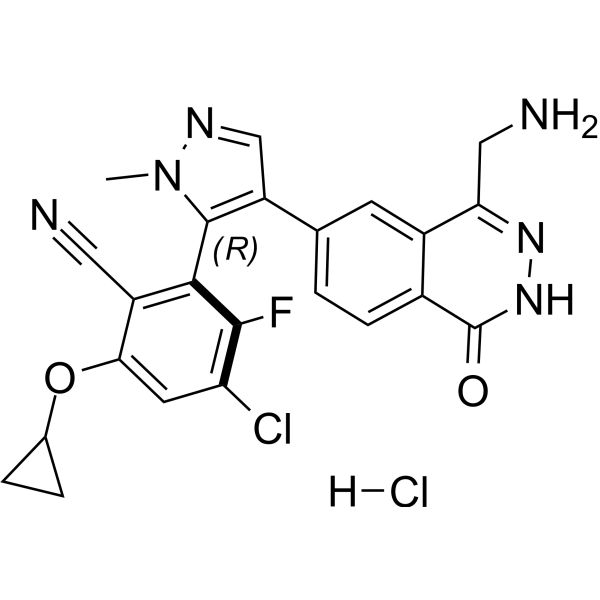
-
GC62715
MRTX9768
MRTX9768 est un inhibiteur du complexe PRMT5-MTA puissant, sélectif, actif par voie orale et de première classe.
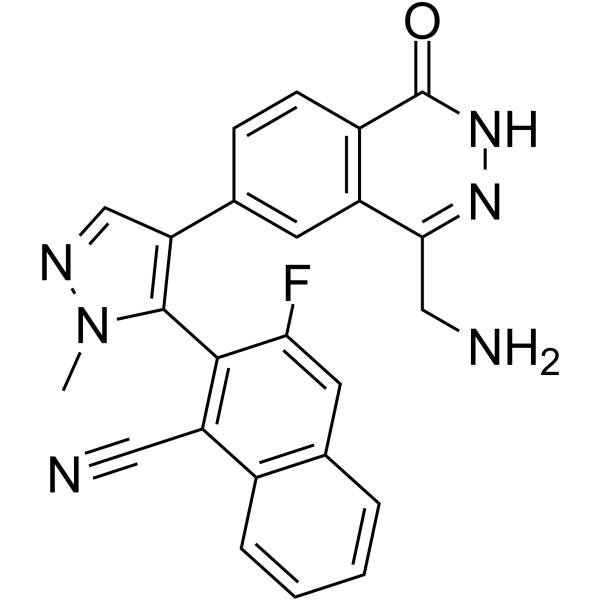
-
GC63080
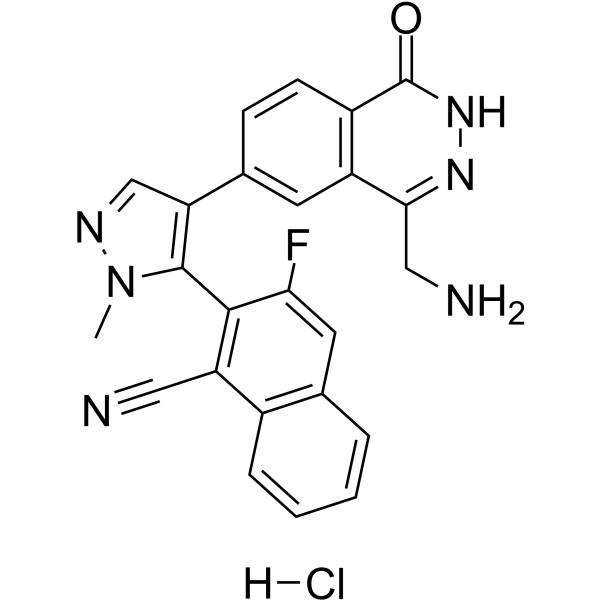
MRTX9768 hydrochloride
Le chlorhydrate de MRTX9768 est un inhibiteur du complexe PRMT5-MTA puissant, sélectif, actif par voie orale et de première classe.
-
GC10099
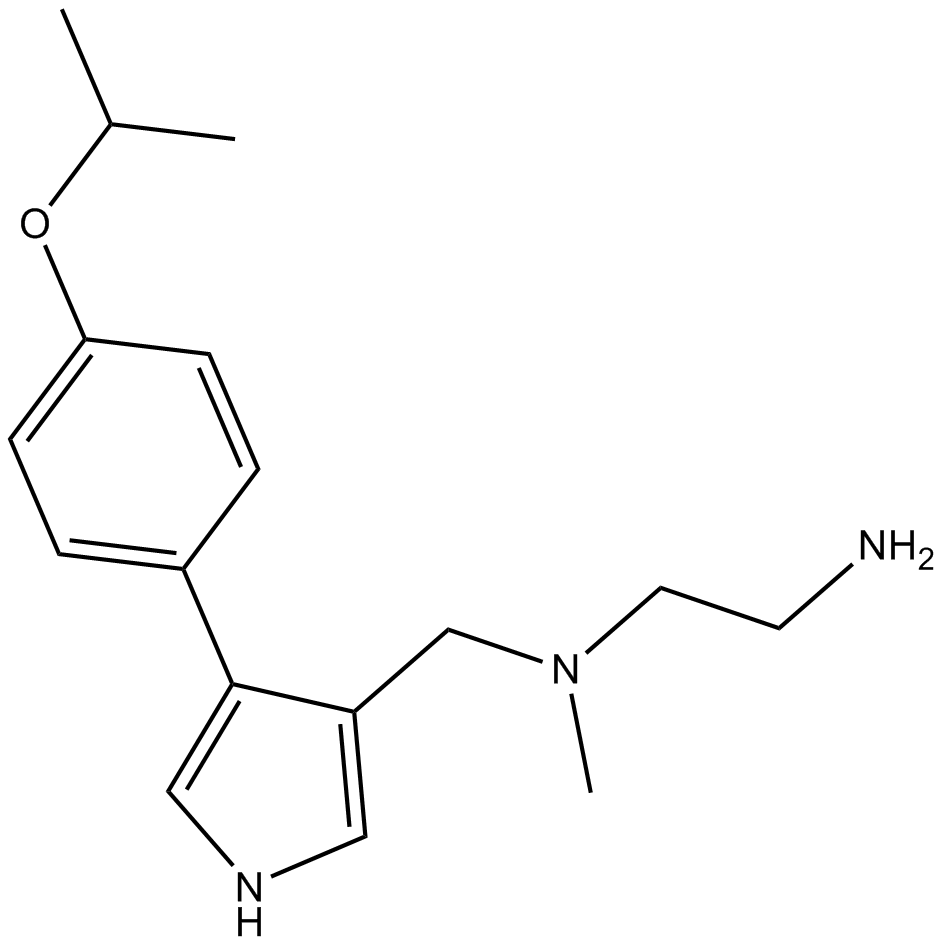
MS023
MS023 est un inhibiteur puissant, sélectif et actif sur les cellules des protéines arginine méthyltransférases de type I (PRMTs).
-
GC16432
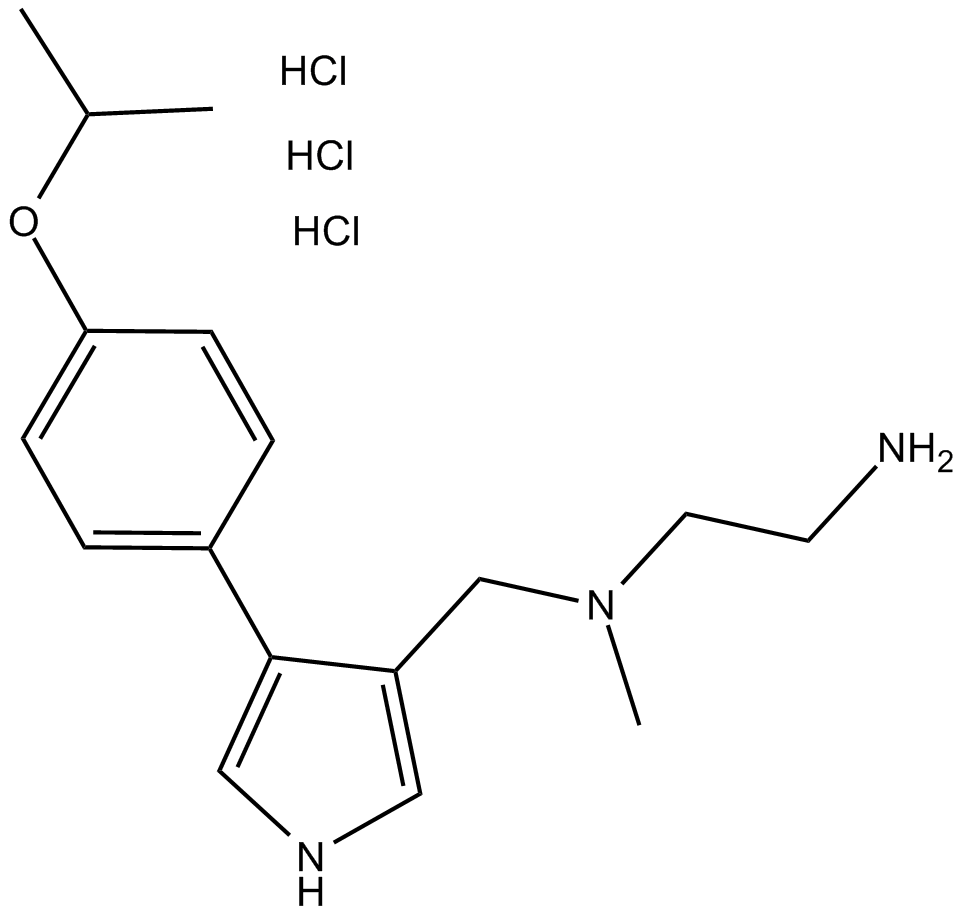
MS023 (hydrochloride)
type I PRMTs inhibitor
-
GC36655
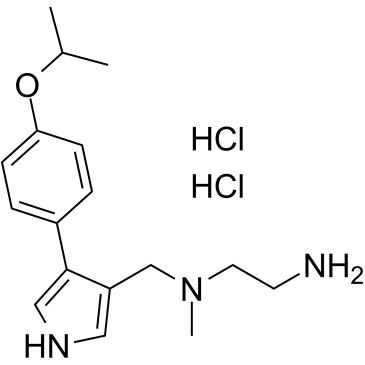
MS023 dihydrochloride
Le dichlorhydrate de MS023 est un inhibiteur puissant, sélectif et actif sur les cellules de l'inhibiteur de la protéine arginine méthyltransférase (PRMT) humaine de type I, avec des IC50 de 30, 119, 83, 4 et 5 nM pour PRMT1, PRMT3, PRMT4, PRMT6 et PRMT8, respectivement.
-
GC36656
MS049
MS049 est un double inhibiteur puissant, sélectif et actif sur les cellules de PRMT4 et PRMT6 avec des IC50 de 34 nM et 43 nM, respectivement. MS049 réduit les niveaux de Med12me2a et H3R2me2a dans les cellules HEK293. MS049 n'est pas toxique et n'affecte pas la croissance des cellules HEK293.
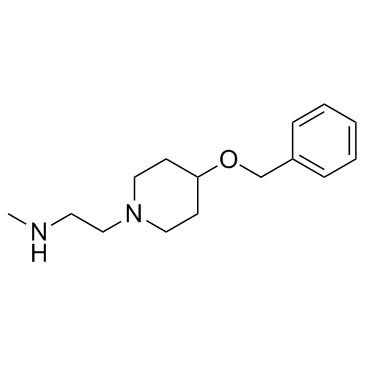
-
GC14240
MS049 (hydrochloride)
MS049 (chlorhydrate) est un double inhibiteur puissant, sélectif et actif sur les cellules de PRMT4 et PRMT6 avec des IC50 de 34 nM et 43 nM, respectivement. MS049 (chlorhydrate) réduit les niveaux de Med12me2a et H3R2me2a dans les cellules HEK293. MS049 (chlorhydrate) n'est pas toxique et n'affecte pas la croissance des cellules HEK293.
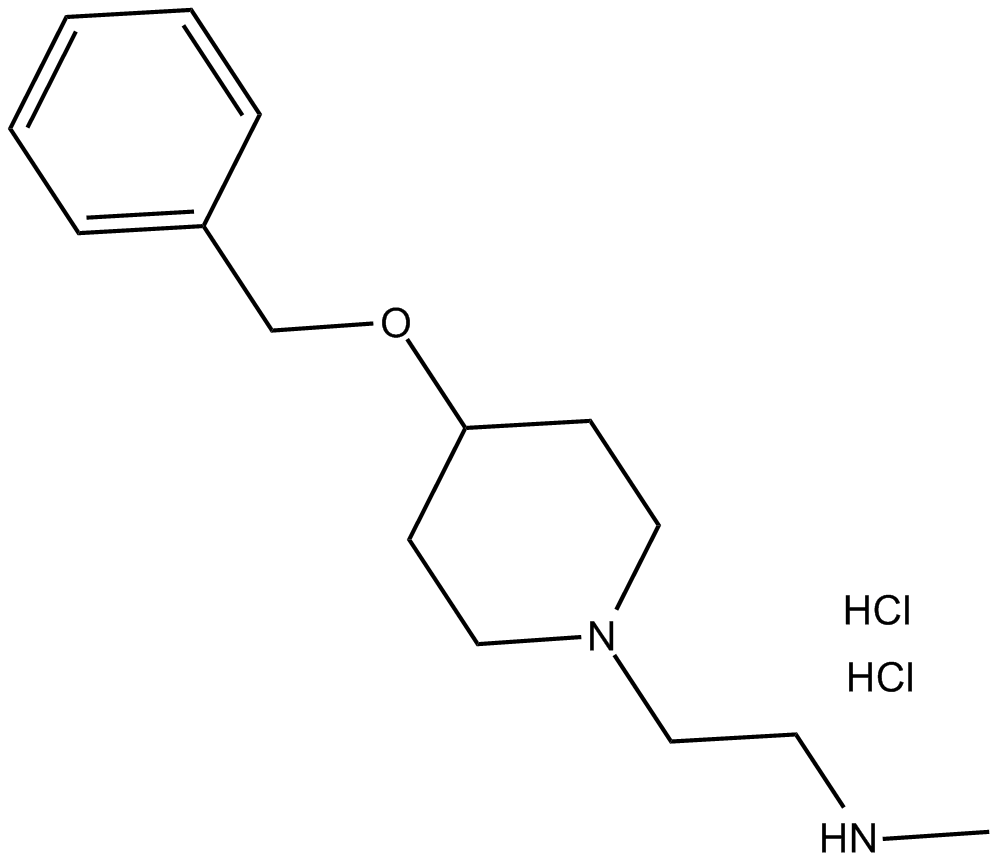
-
GC64295
MS67
MS67 est un dégradeur puissant et sélectif de la protéine 5 à répétition WD40 (WDR5) avec une Kd de 63 nM. MS67 est inactif contre d'autres méthyltransférases, kinases, RCPG, canaux ioniques et transporteurs. MS67 montre des effets anticancéreux puissants.

-
GC69504
MS8815
MS8815 est un dégradeur PROTAC sélectif de la protéine homologue 2 de zeste (EZH2). MS8815 présente une activité inhibitrice sur EZH2 avec une valeur IC50 de 8,6 nM. MS8815 peut être utilisé dans la recherche sur le cancer du sein triple négatif (TNBC).
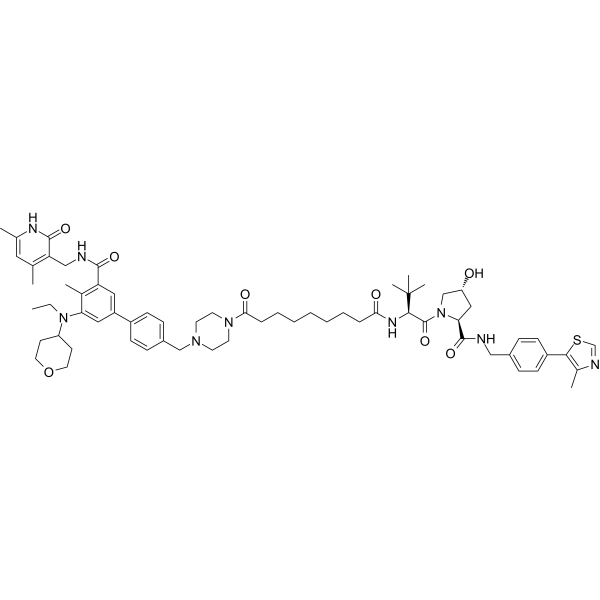
-
GC61142
NSC745885
NSC745885, un agent antitumoral efficace, présente une toxicité sélective contre plusieurs lignées de cellules cancéreuses, mais pas contre les cellules normales. NSC745885 est un régulateur À la baisse efficace d'EZH2 via la dégradation médiée par le protéasome. NSC745885 offre des possibilités pour l'étude des cancers avancés de la vessie et du carcinome épidermoÏde de la bouche (OSCC).
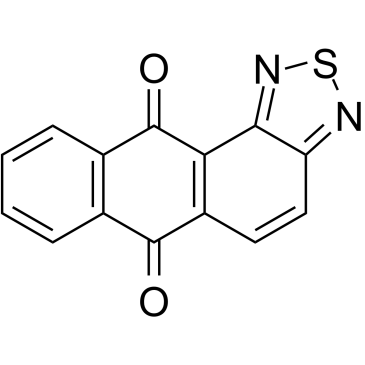
-
GC64772
NV03
NV03 est un antagoniste puissant et sélectif de l'interaction UHRF1 (Ubiquitin-like with PHD and RING finger domains 1)-H3K9me3 en se liant au domaine tudor tandem UHRF1, avec un Kd de 2,4 μM. NV03 a une activité anticancéreuse.
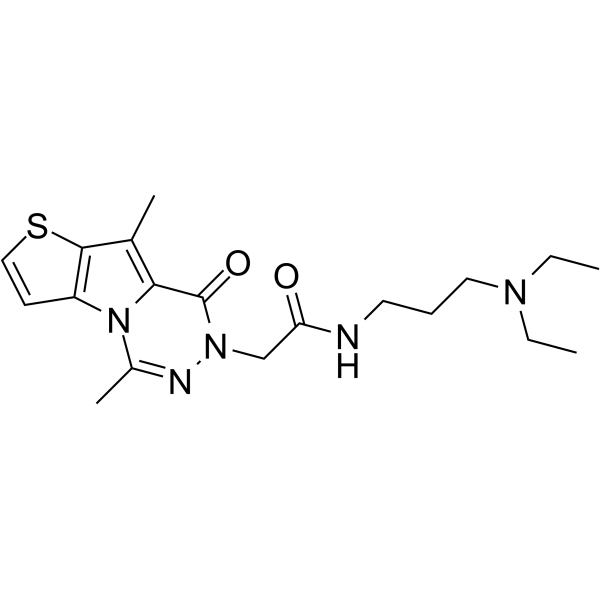
-
GC16397
OICR-9429
OICR-9429 est un inhibiteur À haute affinité du domaine 5 de répétition WD (WDR5), qui bloque de manière compétitive l'interaction de WDR5 avec la protéine MLL via la liaison de la poche centrale de liaison peptidique de WDR5. L'IORC-9429 peut supprimer la triméthylation de l'histone H3K4 et peut être utilisé pour la recherche de divers cancers, notamment la leucémie non réarrangée par la MLL, le cancer du cÔlon, du pancréas, de la prostate et le cancer de la vessie (BCa).
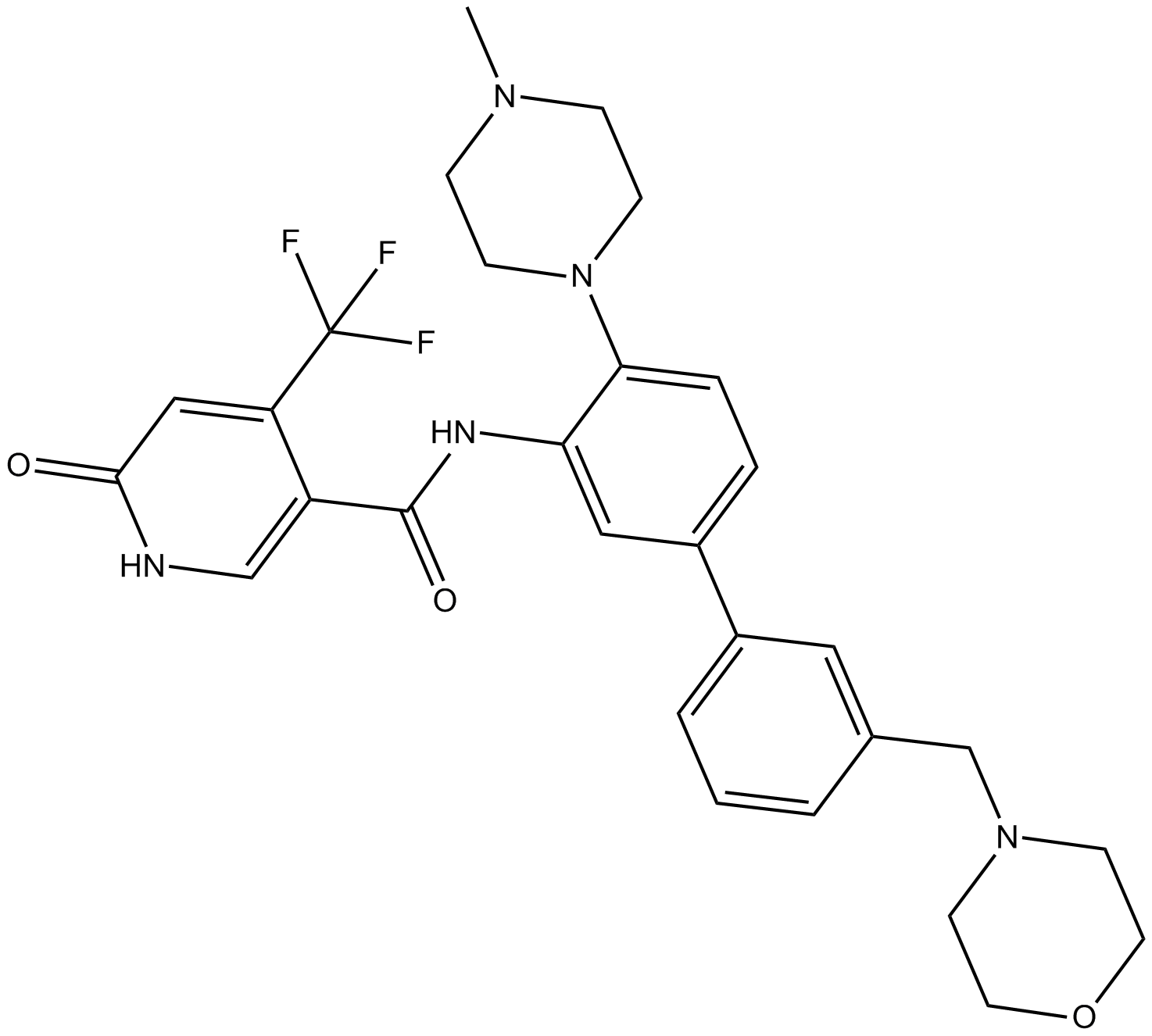
-
GC69636
OTS193320
OTS193320 est un composé d'imidazole et de [1,2-a] pyridine, un inhibiteur d'activité de la méthyltransférase SUV39H2. OTS193320 réduit le niveau de triméthylation globale de l'histone H3 lysine 9 dans les cellules cancéreuses du sein et induit la mort des cellules apoptotiques. Comparé à une seule dose d'OTS193320 ou DOX, la combinaison d'OTS193320 avec Doxorubicin (DOX; A) peut entraîner une diminution du niveau γ-H2AX ainsi que de la viabilité des cellules cancéreuses.
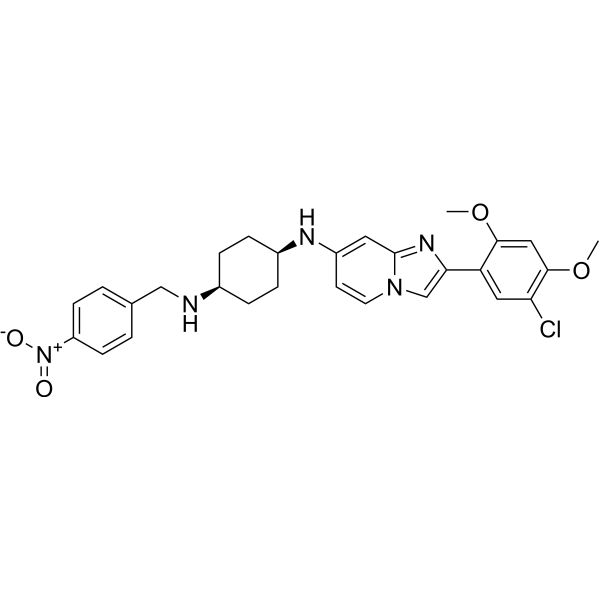
-
GC50367
PF 06726304 acetate
Highly potent and SAM-competitive EZH2 inhibitor
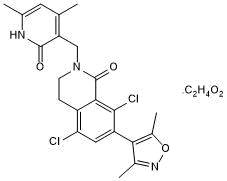
-
GC32977
PF-06726304
PF-06726304 est un inhibiteur EZH2 puissant et sélectif. PF-06726304 inhibe EZH2 de type sauvage et mutant Y641N avec Kis de 0,7 et 3,0 nM, respectivement. PF-06726304 affiche une activité de croissance antitumorale robuste.
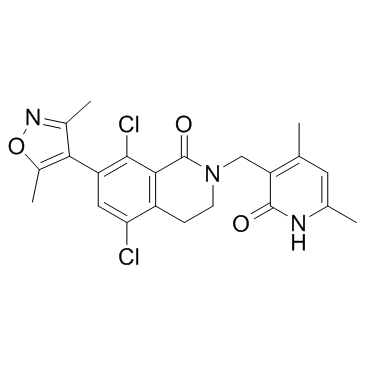
-
GC17956
PFI-2
Le chlorhydrate de PFI-2 ((R)-PFI-2 hydrochloride) est un domaine SET puissant et sélectif contenant un inhibiteur de la lysine méthyltransférase 7 (SETD7).
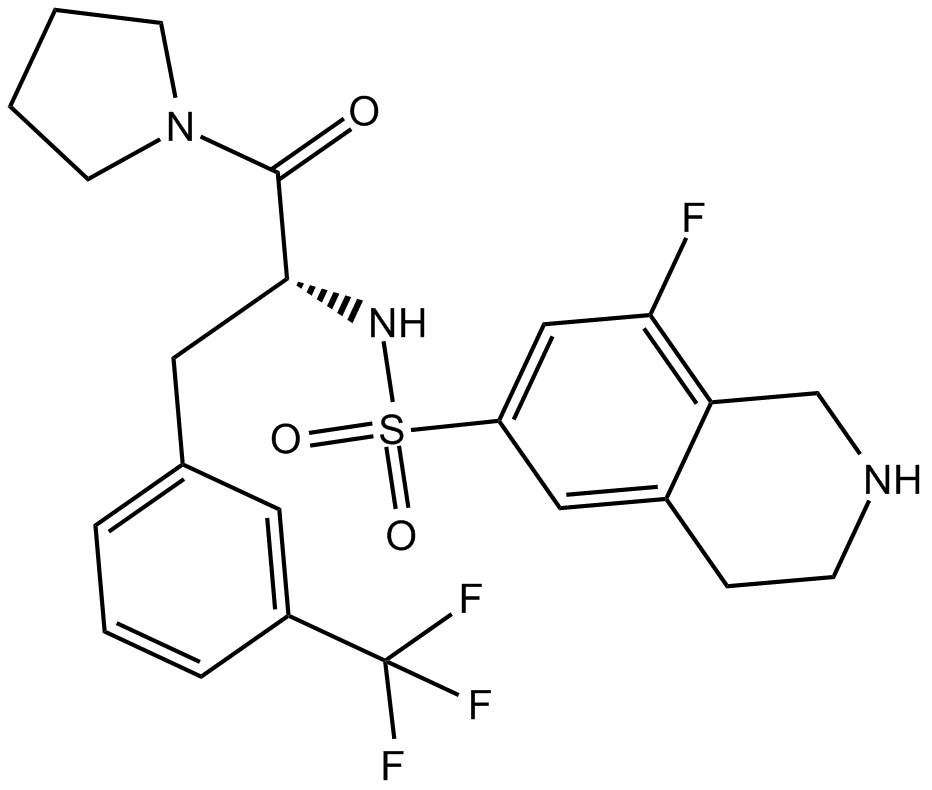
-
GC12530
PFI-2 (hydrochloride)

-
GC64941
PR5-LL-CM01
PR5-LL-CM01 est un puissant inhibiteur de la protéine arginine méthyltransférase 5 (PRMT5) (IC50 = 7,5 μM). Activités anti-tumorales.
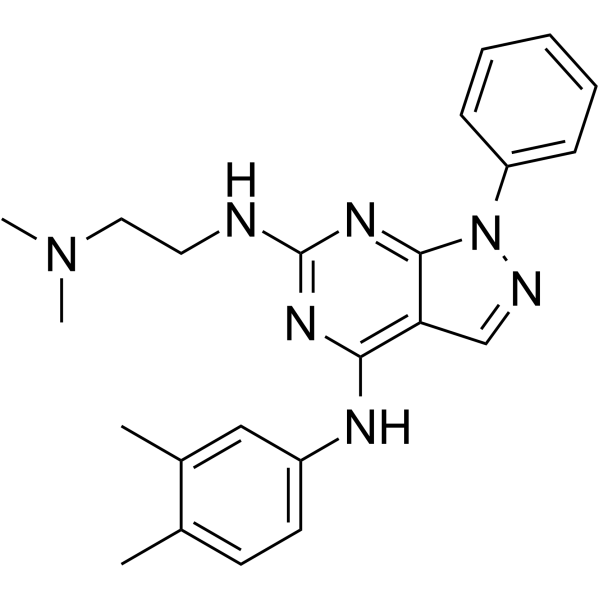
-
GC36969
PRMT5-IN-1
PRMT5 IN-1, un hémiaminal, est un puissant inhibiteur sélectif de la protéine arginine méthyltransférase 5 (PRMT5) avec une IC50 de 11 nM pour PRMT5/MEP50. PRMT5 IN-1 peut être converti en aldéhydes et réagir avec C449 pour former des adduits covalents dans des conditions physiologiques.
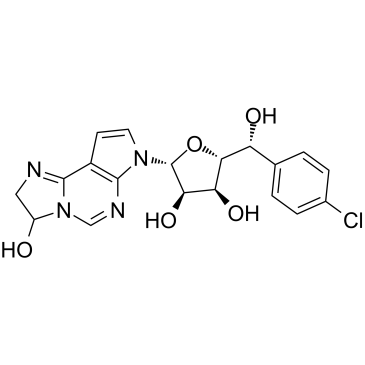
-
GC65027
PRMT5-IN-20
PRMT5-IN-20 est un inhibiteur sélectif de la protéine arginine méthyltransférase 5 (PRMT5) avec une activité anti-tumorale.
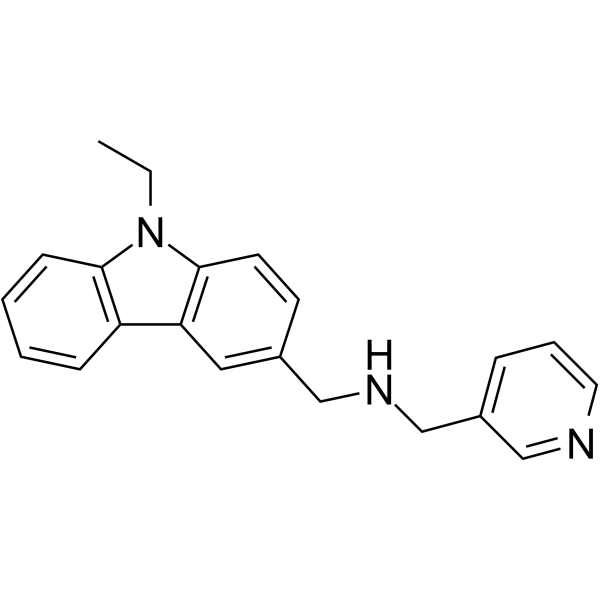
-
GC62187
PROTAC EED degrader-1
PROTAC EED degrader-1 est un PROTAC basé sur von Hippel-Lindau ciblant l'EED avec un pKD de 9,02. PROTAC EED degrader-1 est un inhibiteur du complexe répressif polycomb 2 (PRC2) (pIC50 = 8,17) ciblant la sous-unité EED.
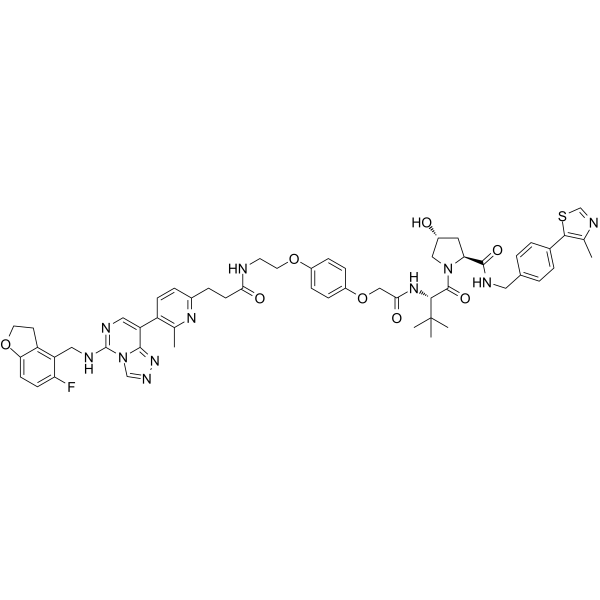
-
GC62717
PROTAC EED degrader-2
PROTAC EED degrader-2 est un PROTAC basé sur von Hippel-Lindau ciblant l'EED avec un pKD de 9,27. PROTAC EED degrader-2 est un inhibiteur du complexe répressif polycomb 2 (PRC2) (pIC50 = 8,11) ciblant la sous-unité EED.
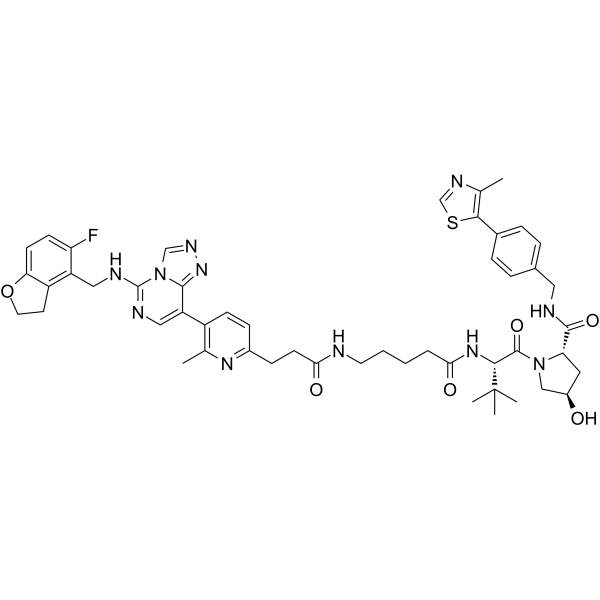
-
GC65509
PROTAC EZH2 Degrader-1
PROTAC EZH2 Degrader-1 (composé 150d), un puissant dégradeur PROTAC EZH2, exerce un effet inhibiteur sur l'activité de la méthyltransférase EZH2 avec une IC50 de 2,7 nM. EZH2 joue un rÔle important dans de nombreux processus de tumorigenèse et de développement.
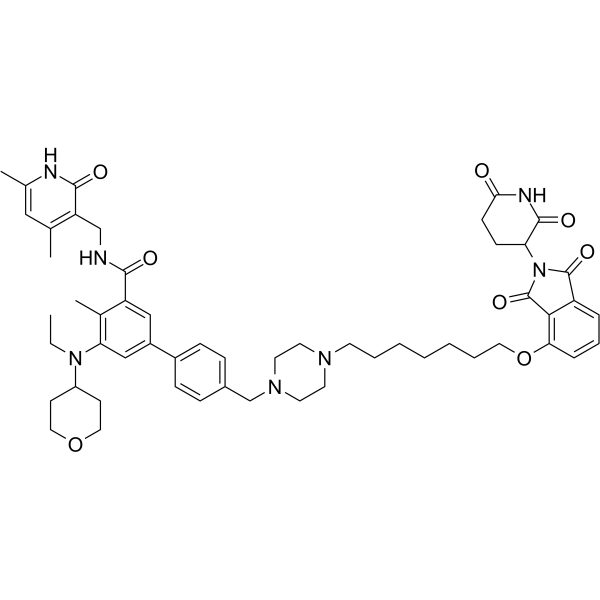
-
GC46213
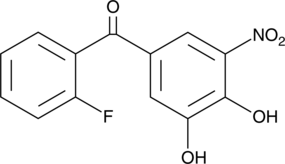
Ro 41-0960
Le Ro 41-0960 est un inhibiteur sélectif de la catéchol-O-méthyltransférase (COMT).
-
GC18650
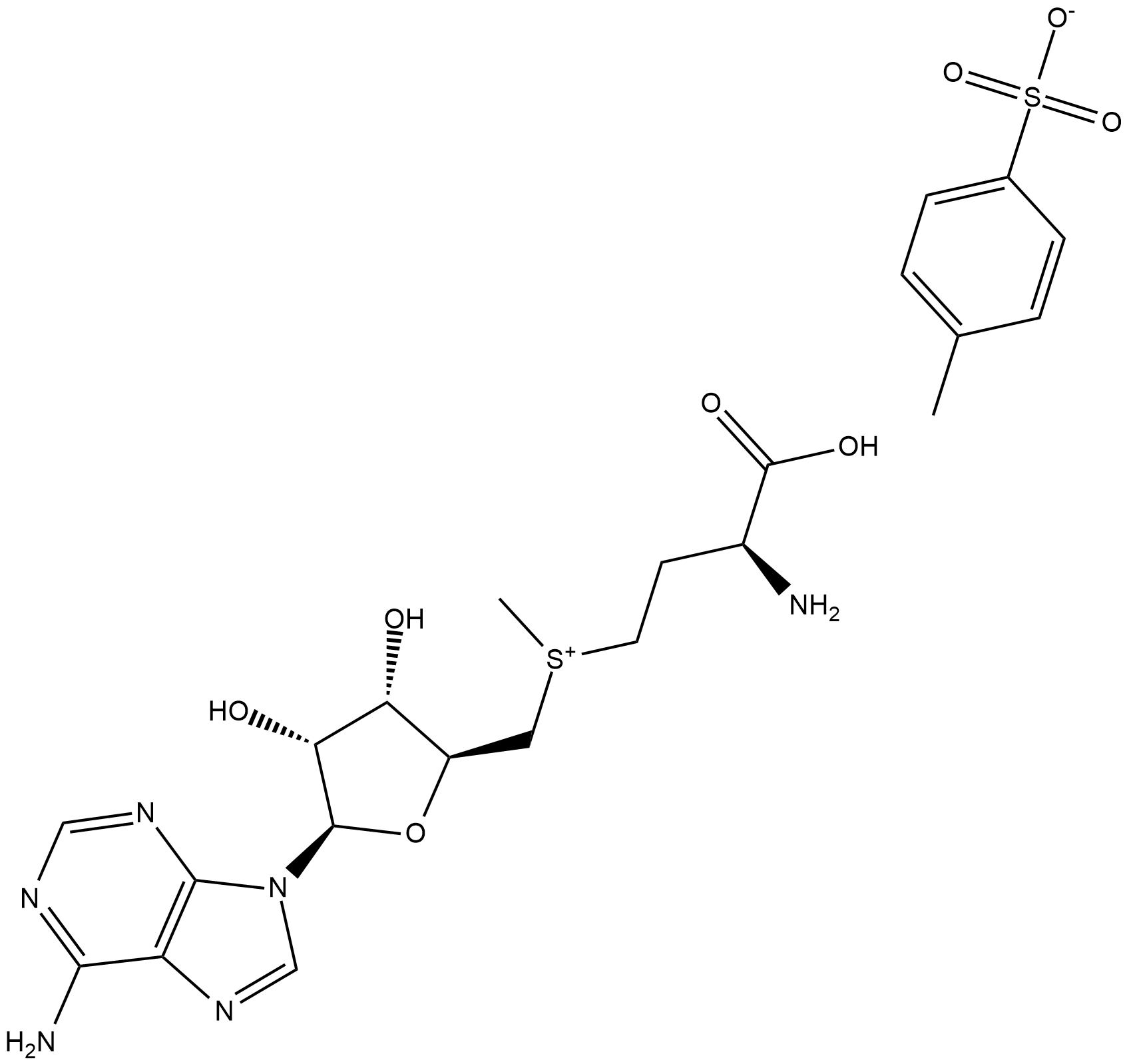
S-(5'-Adenosyl)-L-methionine (tosylate)
S-(5'-Adenosyl)-L-methionine (SAM) is a ubiquitous methyl donor involved in a wide variety of biological reactions, including those mediated by DNA and protein methyltransferases.
-
GC44859
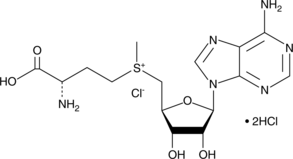
S-(5'-Adenosyl)-L-methionine chloride (hydrochloride)
S-(5'-Adenosyl)-L-methionine chloride (SAM) is a ubiquitous methyl donor involved in a wide variety of biological reactions, including those mediated by DNA and protein methyltransferases.
-
GC44886
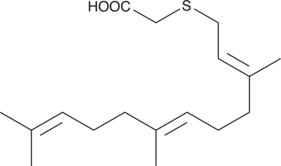
S-Farnesyl Thioacetic Acid
S-Farnesyl thioacetic acid is an analog of S-farnesyl cysteine which behaves as a competitive inhibitor of isoprenylated protein methyltransferase (also known as S-adenosylmethionine-dependent methyltransferase).
-
GC62555
SETD2-IN-1 TFA
SETD2-IN-1 TFA est un inhibiteur puissant, sélectif et oralement actif de SETD2 qui est une histone méthyltransférase humaine. SETD2-IN-1 TFA a des effets anti-prolifératifs.
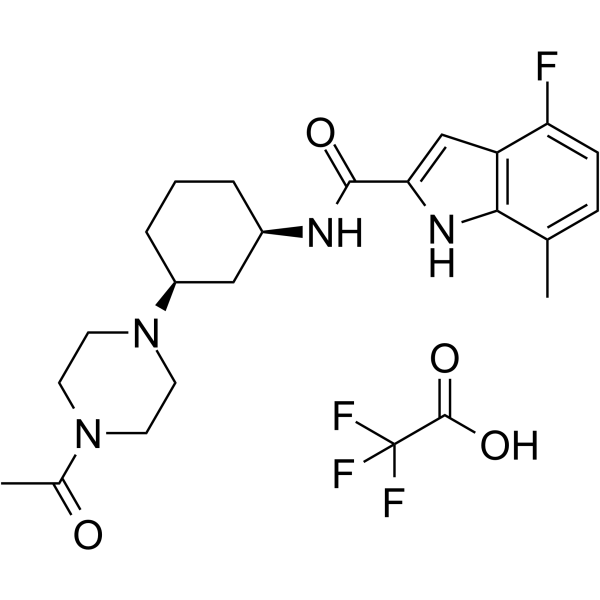
-
GC64893
SETDB1-TTD-IN-1
SETDB1-TTD-IN-1 est un inhibiteur compétitif puissant, sélectif et endogène du domaine tudor bifurqué de la protéine 1 bifurquée du domaine SET (SETDB1-TTD), avec un Kd de 88 nM. SETDB1-TTD-IN-1 peut être utilisé pour la recherche des fonctions biologiques et des associations de maladies de SETDB1-TTD.
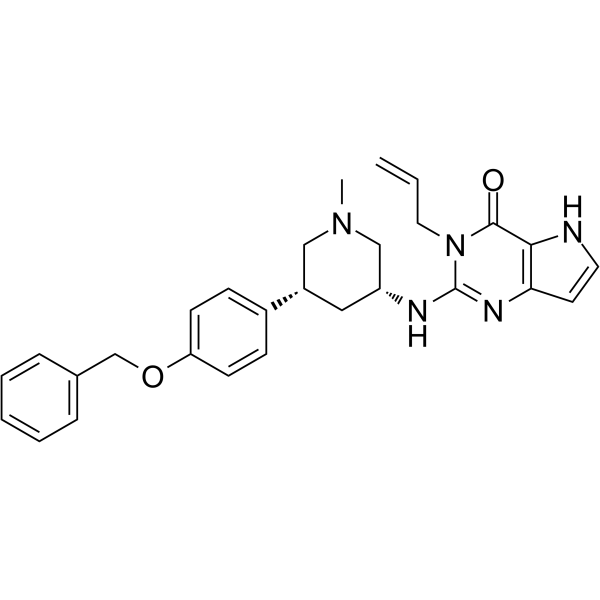
-
GC64894
SETDB1-TTD-IN-1 TFA
SETDB1-TTD-IN-1 TFA est un inhibiteur compétitif de liant puissant, sélectif et endogène du domaine tudor en tandem de la protéine 1 bifurquée du domaine SET (SETDB1-TTD), avec un Kd de 88 nM. SETDB1-TTD-IN-1 TFA peut être utilisé pour la recherche des fonctions biologiques et des associations de maladies de SETDB1-TTD.
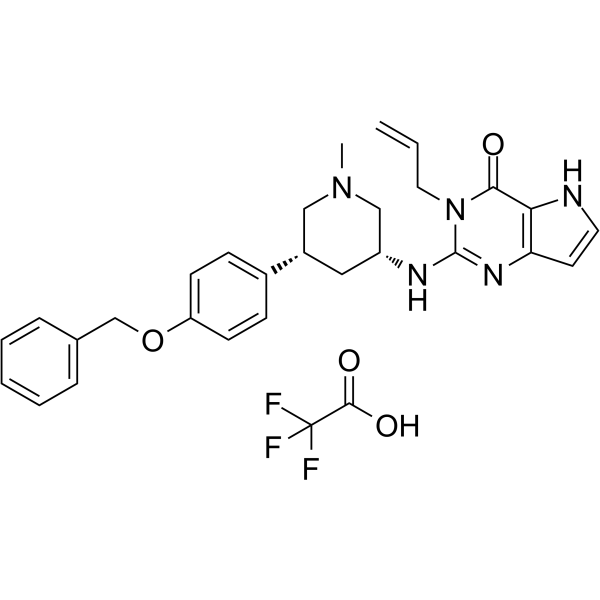
-
GC11491
SGC 0946
A potent inhibitor of DOT1L
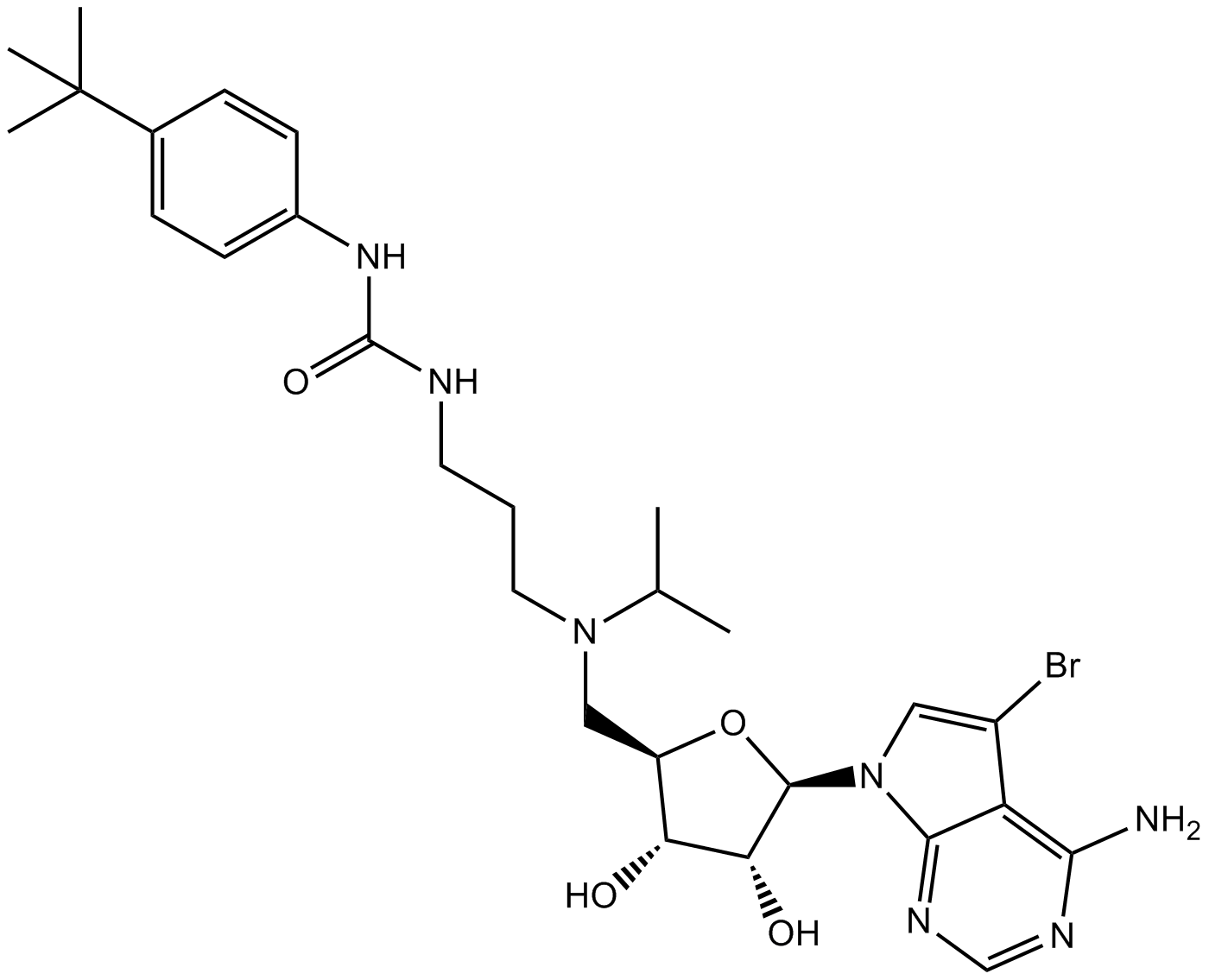
-
GC50699
SGC 6870
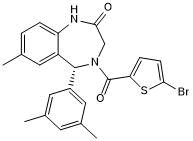
-
GC19329
SGC2085
SGC2085 est un inhibiteur puissant et sélectif de l'arginine méthyltransférase 1 (CARM1) associé À un coactivateur avec une IC50 de 50 nM.
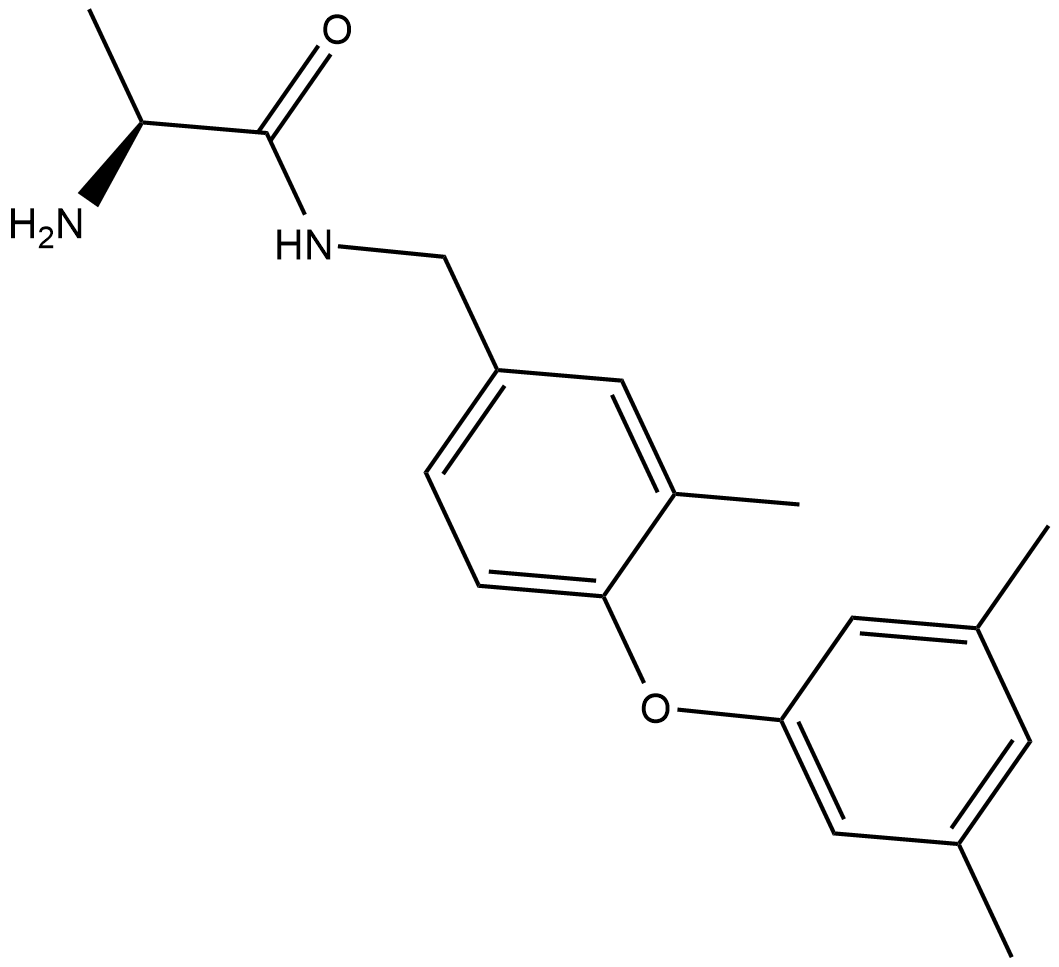
-
GC18428
SGC2085 (hydrochloride)
SGC2085 is an inhibitor of protein arginine methyltransferase 4 (PRMT4/CARM1; IC50 = 50 nM).
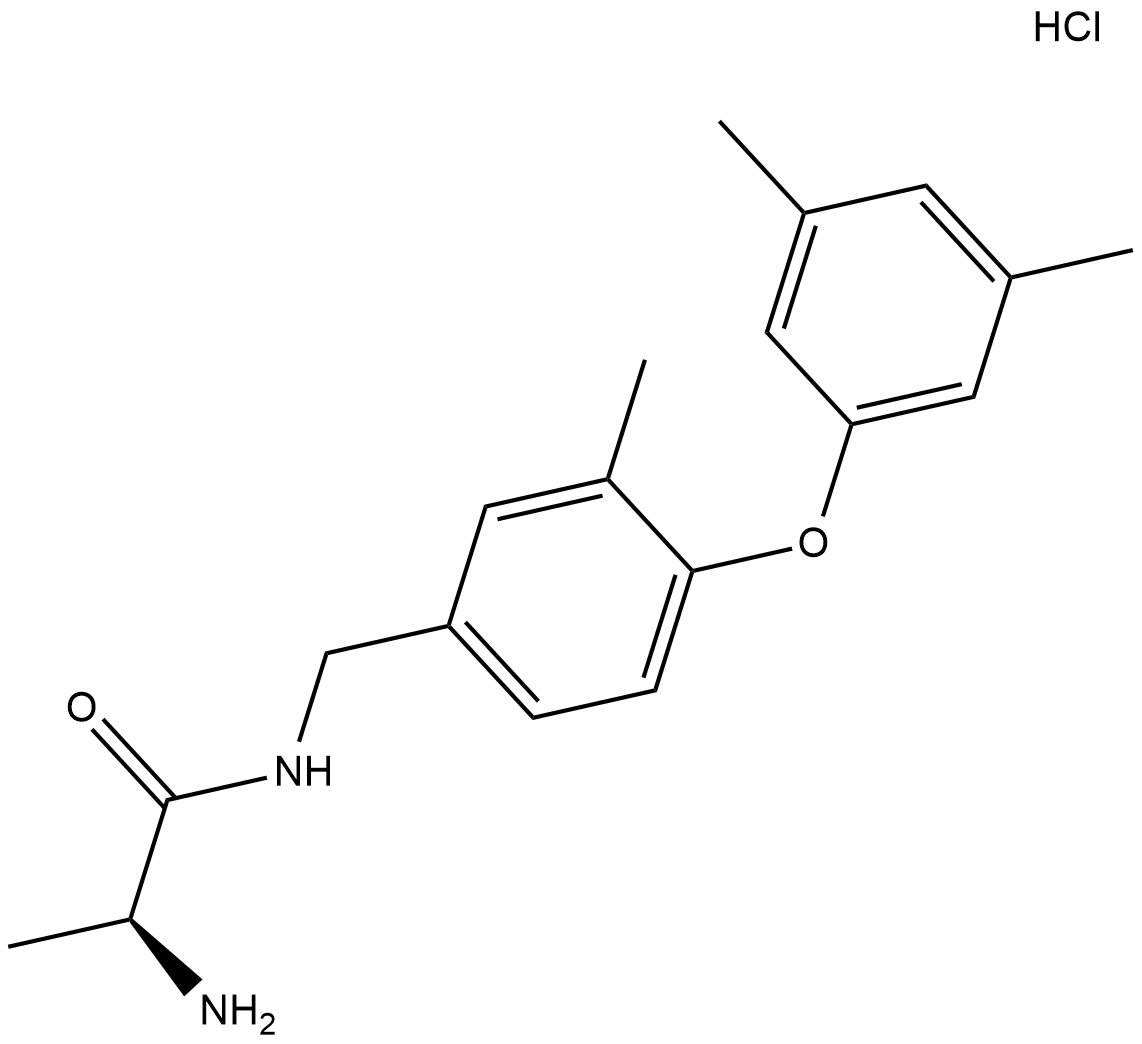
-
GC12874
SGC707
SGC707 est un inhibiteur puissant, sélectif et non compétitif de PRMT3 (protéine arginine méthyltransférase 3) (IC50 = 31 nM, Kd = 53 nM).
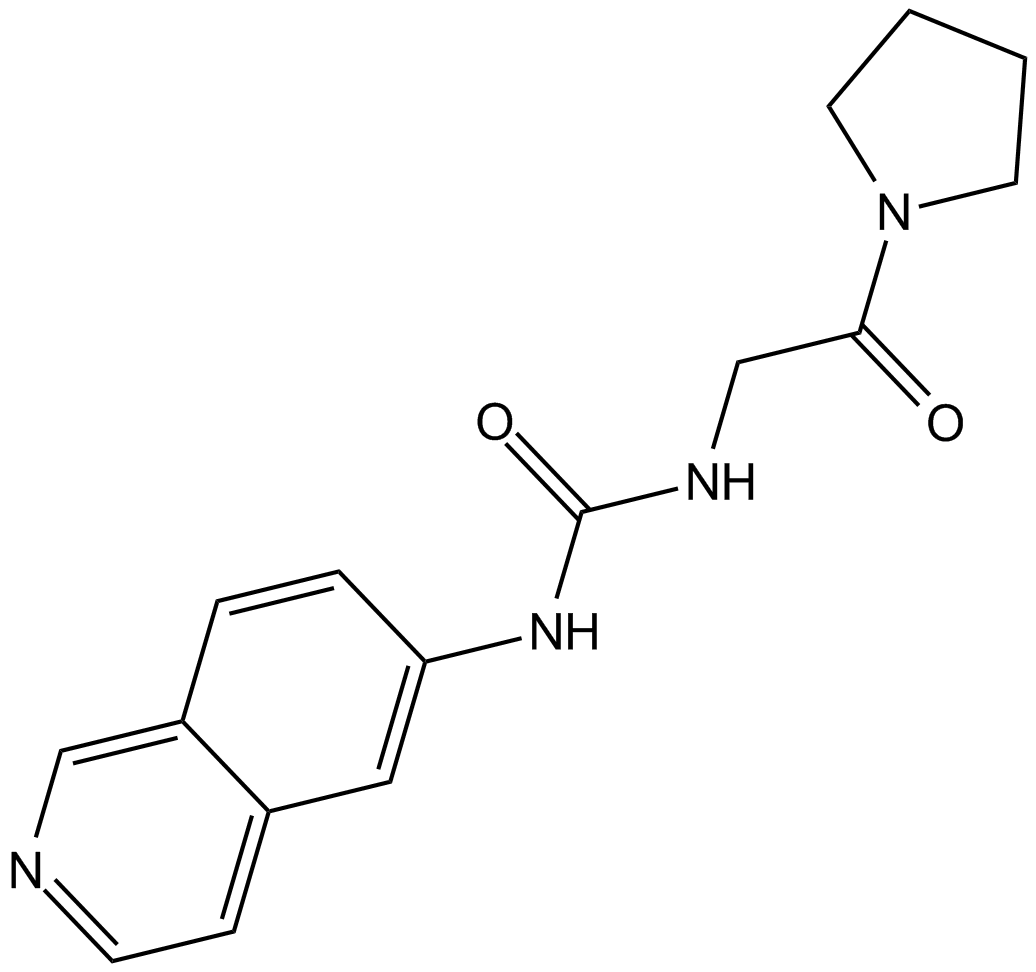
-
GC14364
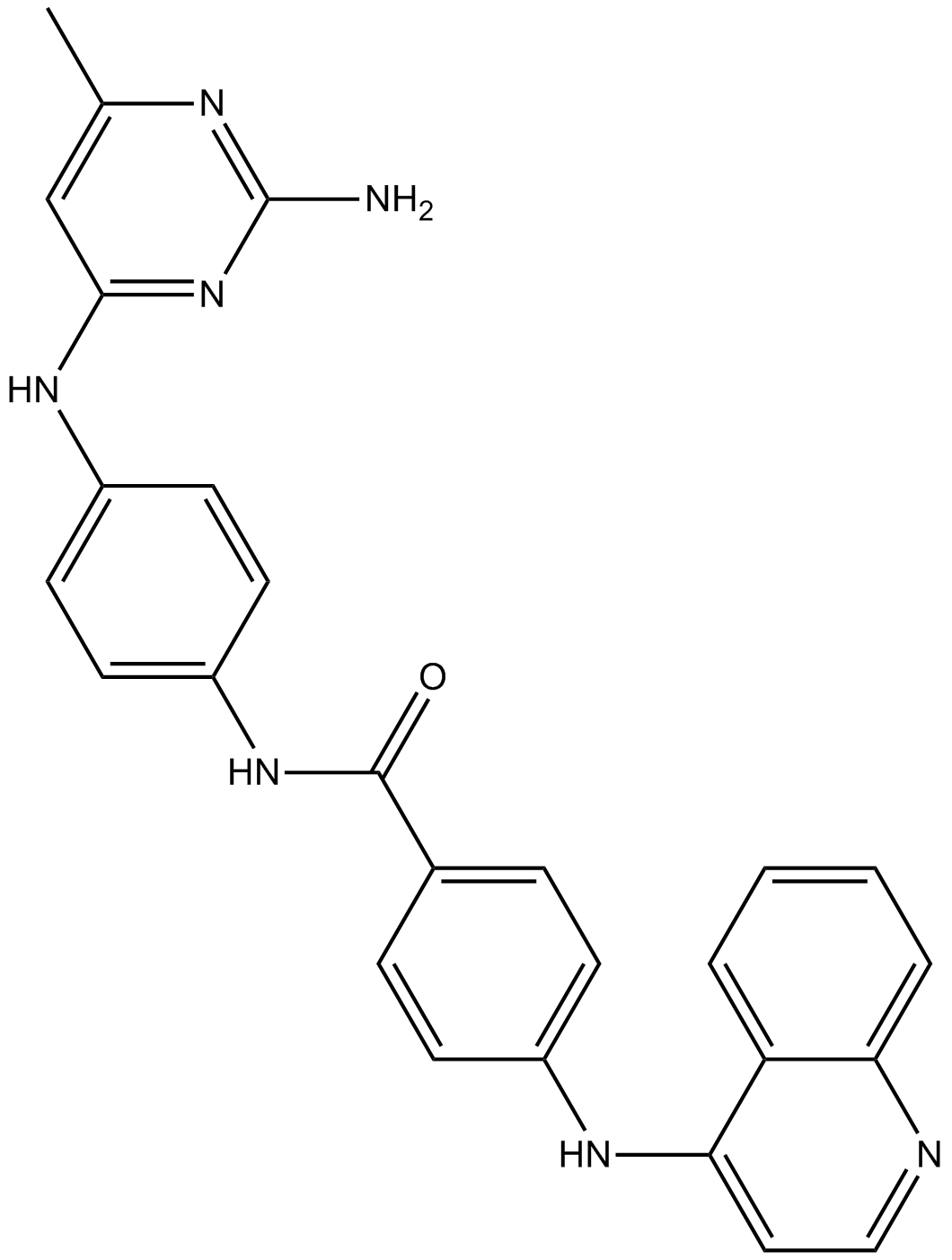
SGI-1027
Le SGI-1027 est un inhibiteur de l'ADN méthyltransférase (DNMT), avec des IC50 de 7,5 μM, 8 μM et 12,5 μM pour DNMT3B, DNMT3A et DNMT1 avec poly(dI-dC) comme substrat.
-
GC34784
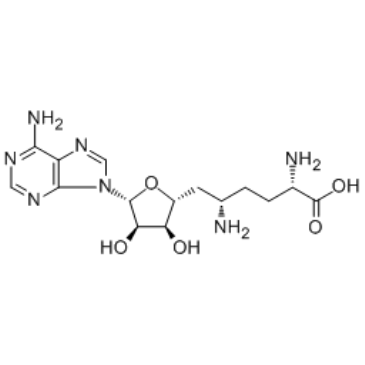
Sinefungin
La sinefungine est un puissant inhibiteur de l'ARNm du virion (guanine-7-)-méthyltransférase, de l'ARNm (nucléoside-2'-)-méthyltransférase et de la multiplication virale .
-
GC37654
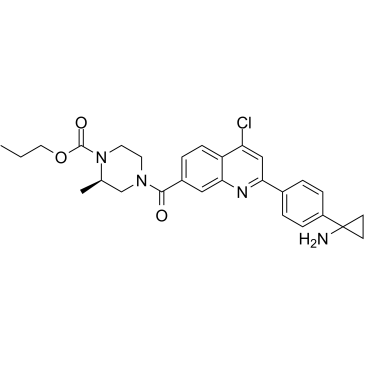
SMYD3-IN-1
SMYD3-IN-1 (composé 29) est un inhibiteur irréversible et sélectif de SMYD3 (domaine SET et MYND contenant 3), avec une IC50 de 11,7 nM.
-
GC18642
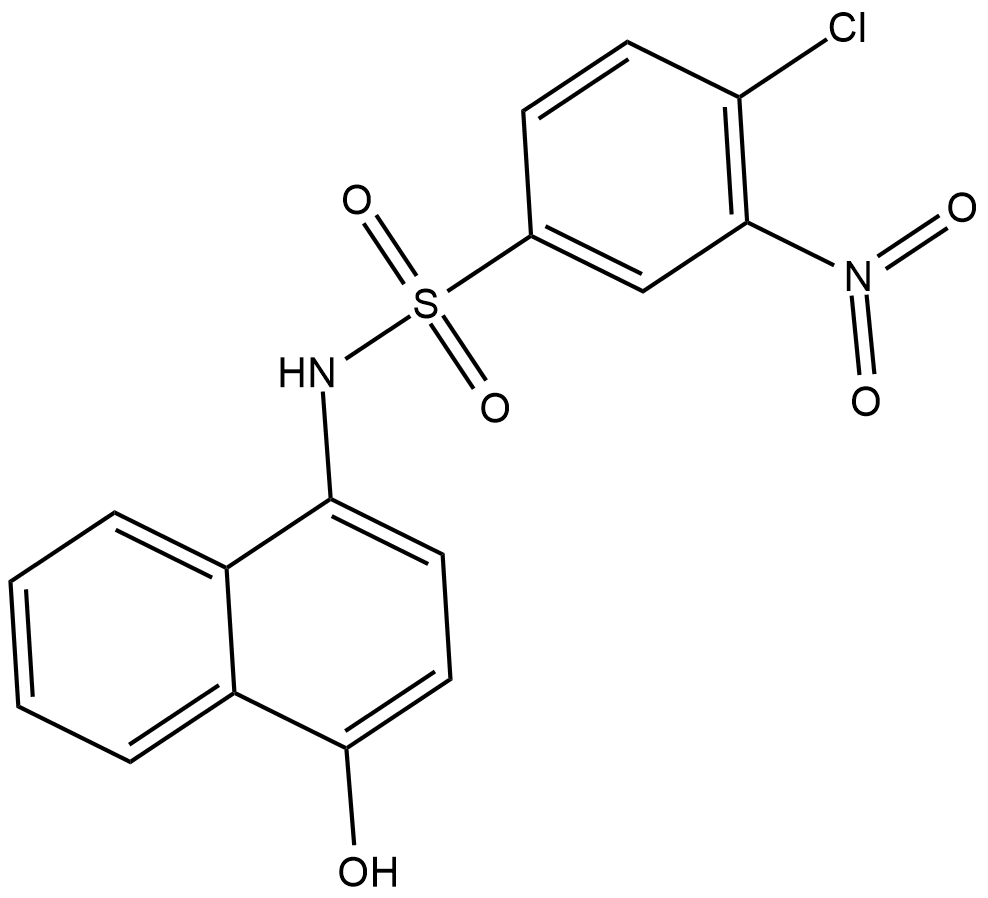
SW155246
SW155246 est un inhibiteur sélectif de l'ADN méthyltransférase (DNMT1) avec des IC50 de 1,2 et 38 μM pour hDNMT1 et mDNMT3A, respectivement. SW155246 peut être utilisé pour la recherche sur le cancer et d'autres maladies.
-
GC62390
SW2_110A
SW2_110A est un inhibiteur sélectif du chromodomaine chromobox 8 (CBX8 ChD) avec un Kd de 800 nM. SW2_110A montre une sélectivité minimale de 5 fois pour CBX8 ChD par rapport À tous les autres paralogues CBX in vitro.
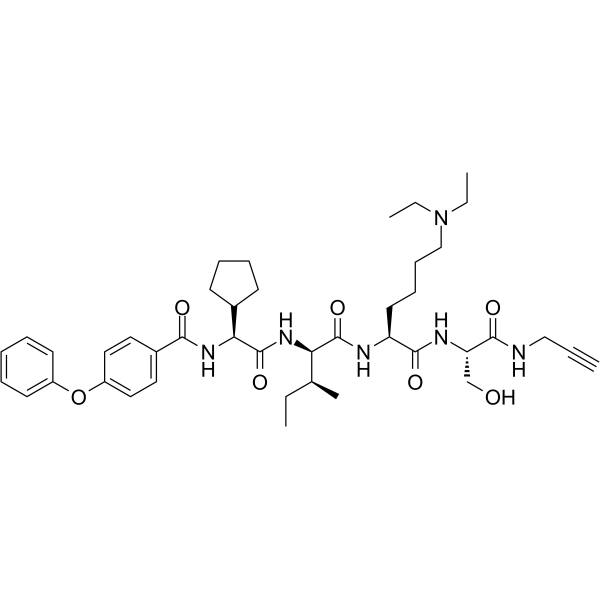
-
GC69980
SW2_152F
SW2_152F est un inhibiteur efficace sélectif du domaine chromodomaine 2 de la protéine chromobox 2 (CBX2 ChD), avec une valeur Kd de 80 nM. En vitro, SW2_152F présente une sélectivité pour CBX2 ChD qui est de 24 à 1000 fois supérieure à celle des autres paralogues CBX.
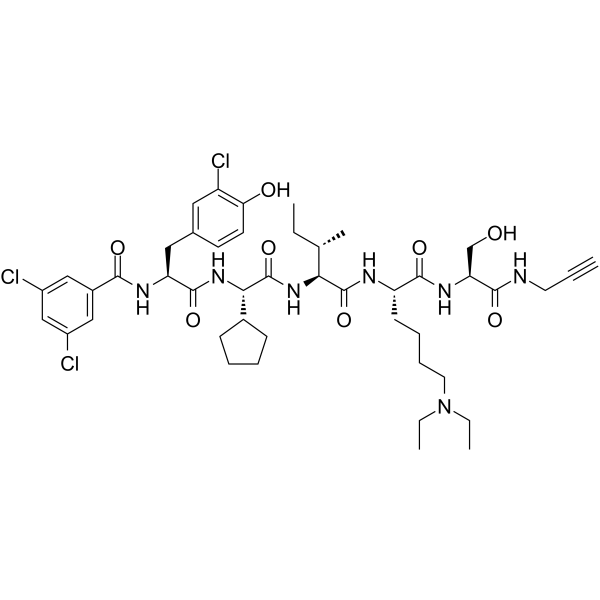
-
GC67676
Tazemetostat de(methylene morpholine)-O-C3-O-C-COOH
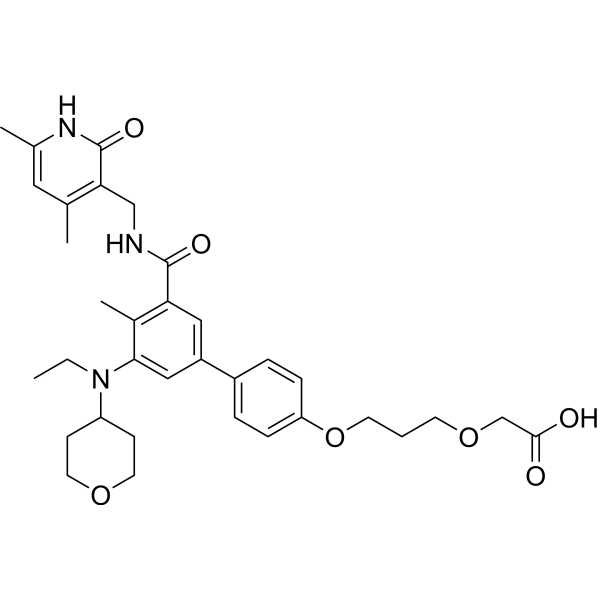
-
GC38163
Tazemetostat hydrobromide
Le bromhydrate de tazémétostat (bromhydrate EPZ-6438) est un inhibiteur EZH2 puissant, sélectif et disponible par voie orale. Le bromhydrate de tazémétostat inhibe l'activité de l'EZH2 de type sauvage contenant le complexe répressif polycomb humain 2 (PRC2) avec une valeur Ki de 2,5 nM. Le bromhydrate de tazémétostat inhibe EZH2 avec des CI50 de 11 et 16 nM dans le dosage des peptides et le dosage des nucléosomes, respectivement. Le bromhydrate de tazémétostat inhibe l'EZH2 du rat avec une CI50 de 4 nM. Le bromhydrate de tazémétostat inhibe également EZH1 avec une CI50 de 392 nM.
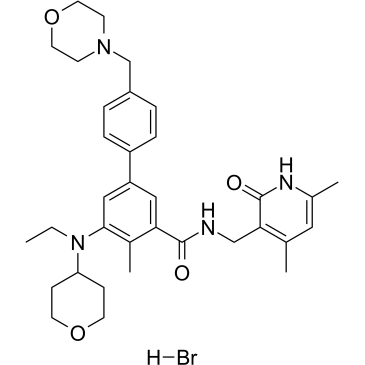
-
GC17672
TC-E 5003
TC-E 5003 est un inhibiteur sélectif de PRMT1 avec une IC50 de 1,5 μM contre hPRMT1.
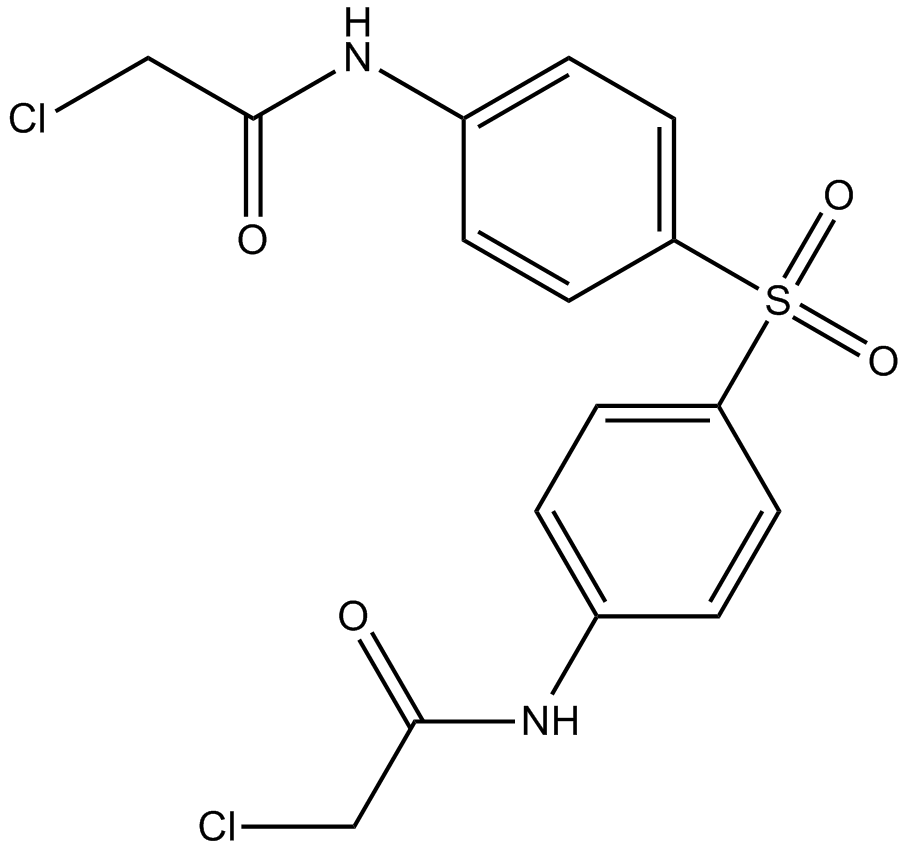
-
GC70041
TNG908
TNG908 est un inhibiteur de PRMT5 synergique avec MTAP qui peut traverser la barrière hémato-encéphalique. TNG908 présente une sélectivité 15 fois plus élevée pour les cellules MTAPnull que pour les cellules MTAPWT et peut être utilisé dans la recherche sur le cancer.
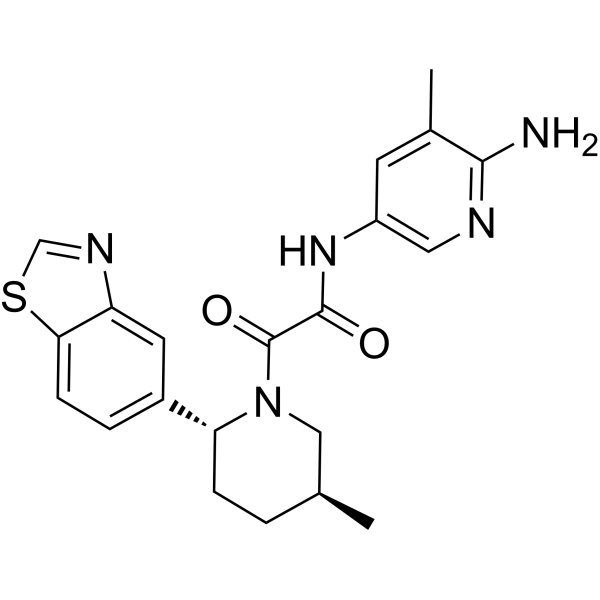
-
GC45763
Tolcapone-d4
An internal standard for the quantification of tolcapone
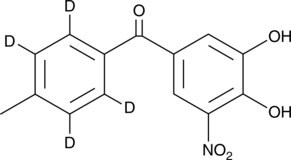
-
GC41263
TP-064
TP-064 est un inhibiteur puissant et sélectif de la protéinarginine méthyltransférase 4 (PRMT4; CARM1) (IC50 <10 nM).
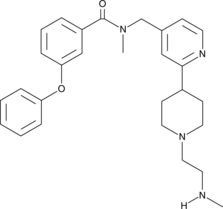
-
GC15688
UNC 0224
A potent inhibitor of G9a histone methyltransferase
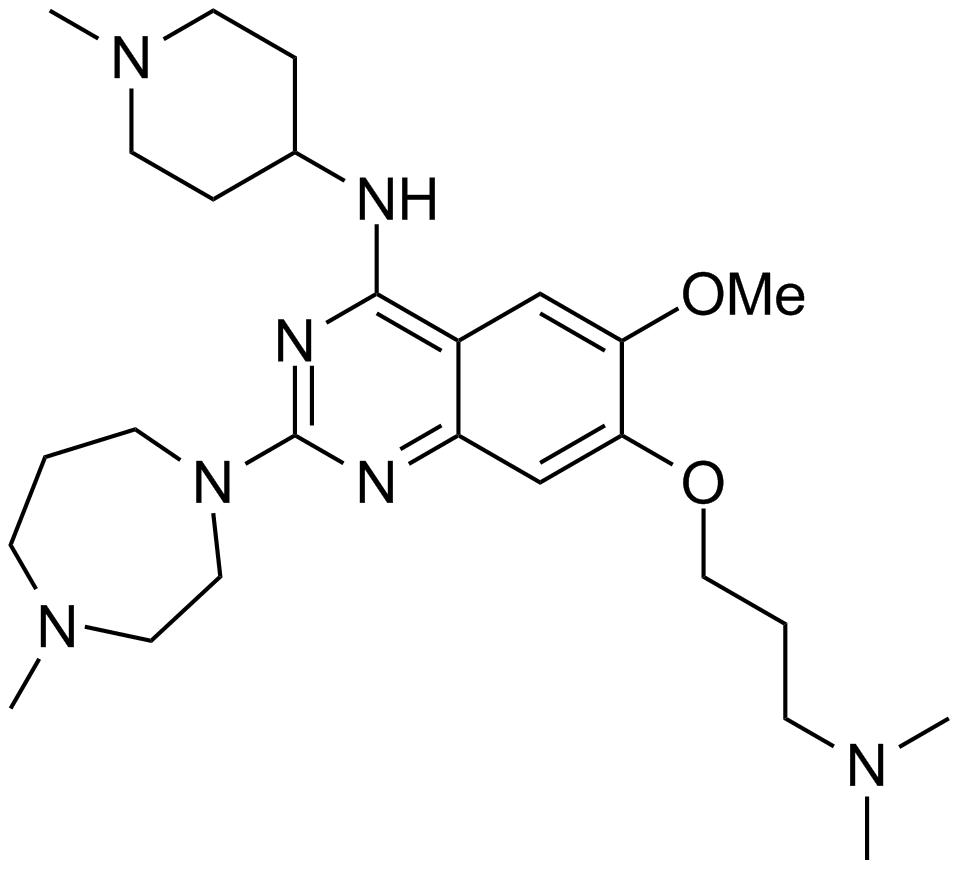
-
GC12548
UNC 0631
L'UNC 0631 est un puissant inhibiteur de l'histone méthyltransférase G9a avec une IC50 de 4 nM. L'UNC 0631 réduit puissamment les niveaux de H3K9me2 dans les cellules MDA-MB-231 avec une IC50 de 25 nM.
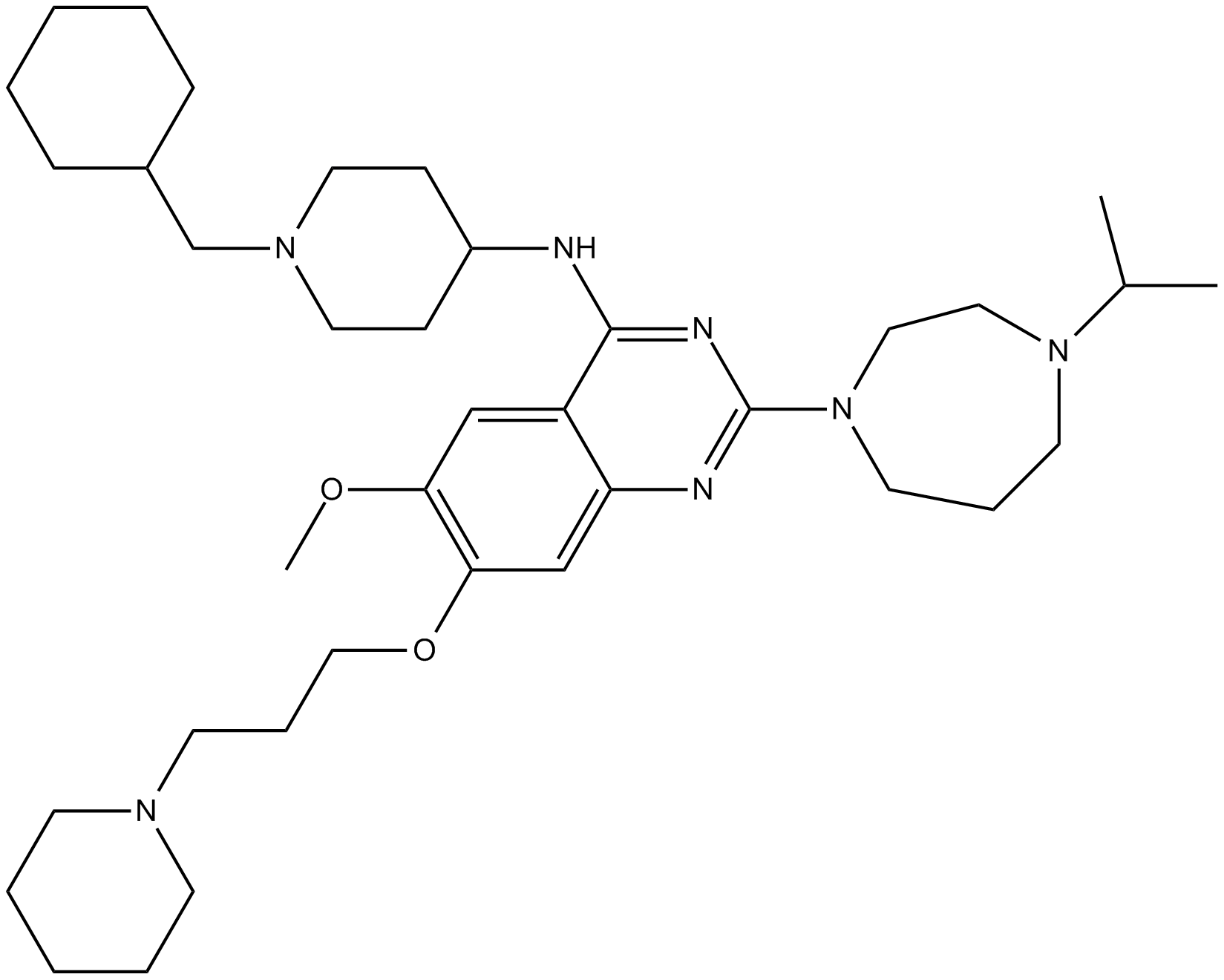
-
GC14249
UNC 0642
L'UNC 0642 est un inhibiteur puissant et sélectif des lysine méthyltransférases G9a et GLP, avec une IC50 \u003c2,5 nM pour G9a.
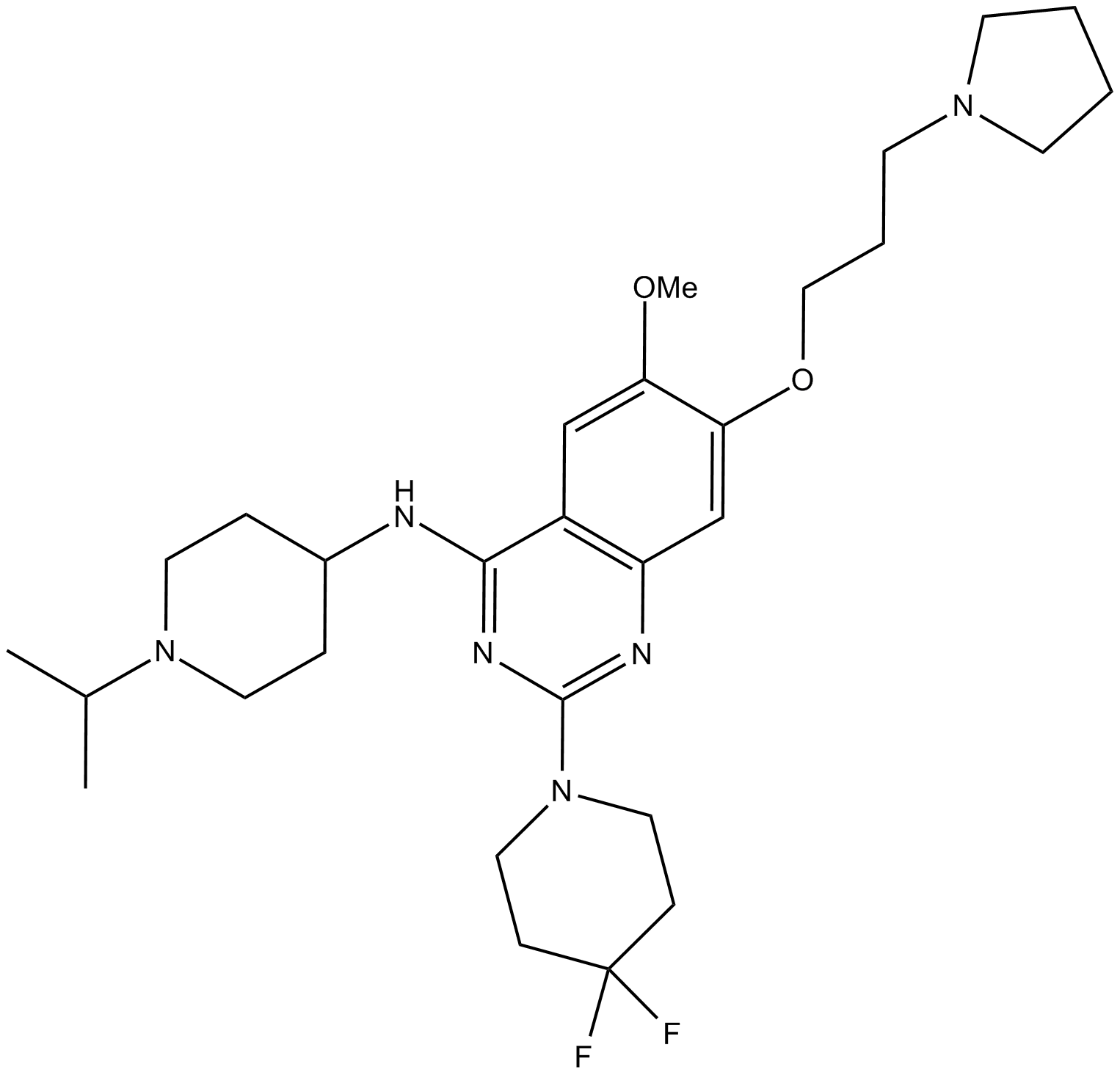
-
GC16518
UNC 0646
An inhibitor of G9a/GLP methyltransferases
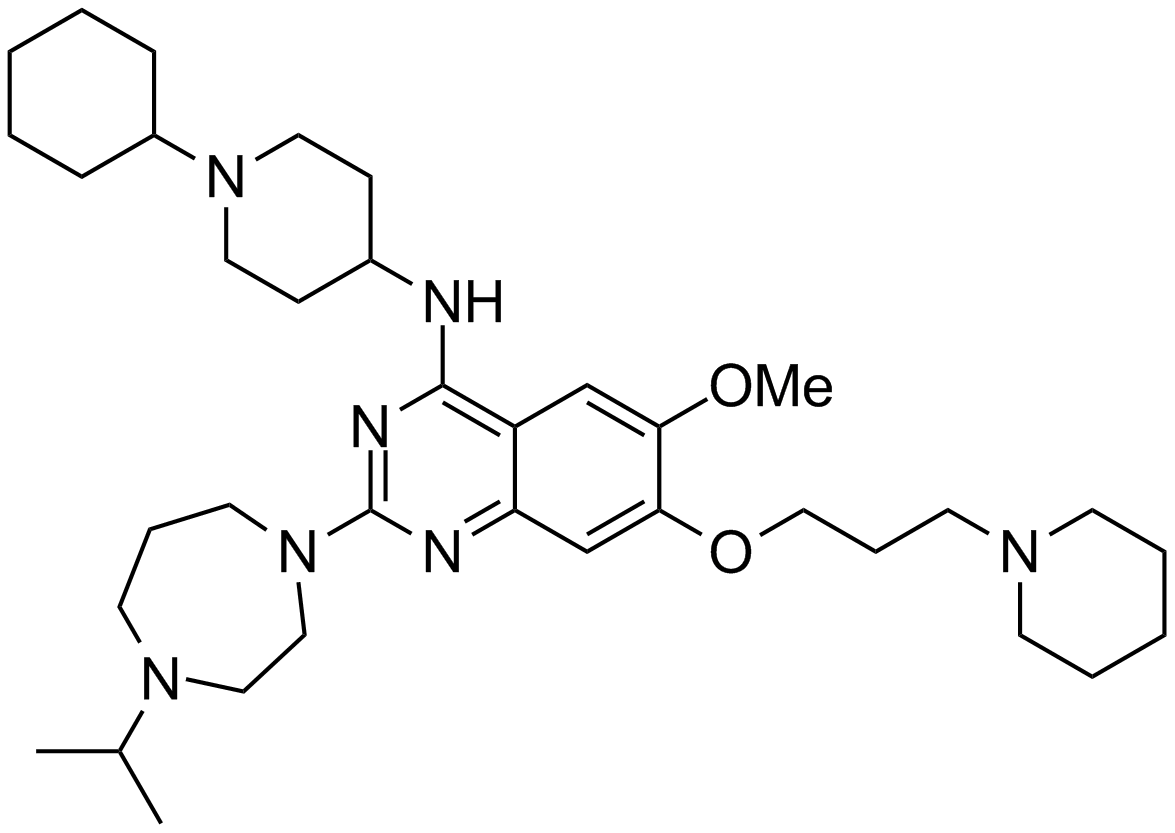
-
GC50082
UNC 2399
UNC 2399, un UNC1999 biotinylé, est un dégradeur sélectif d'EZH2, maintenant une puissance in vitro élevée pour EZH2, avec une IC50 de 17 nM.
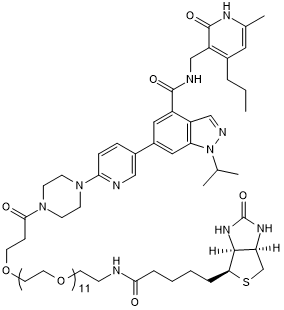
-
GC15652
UNC 2400
L'UNC 2400 est un analogue proche de l'UNC1999 avec une puissance \u003e 1 000 fois inférieure à celle de l'UNC1999 en tant que contrôle négatif pour les études cellulaires.
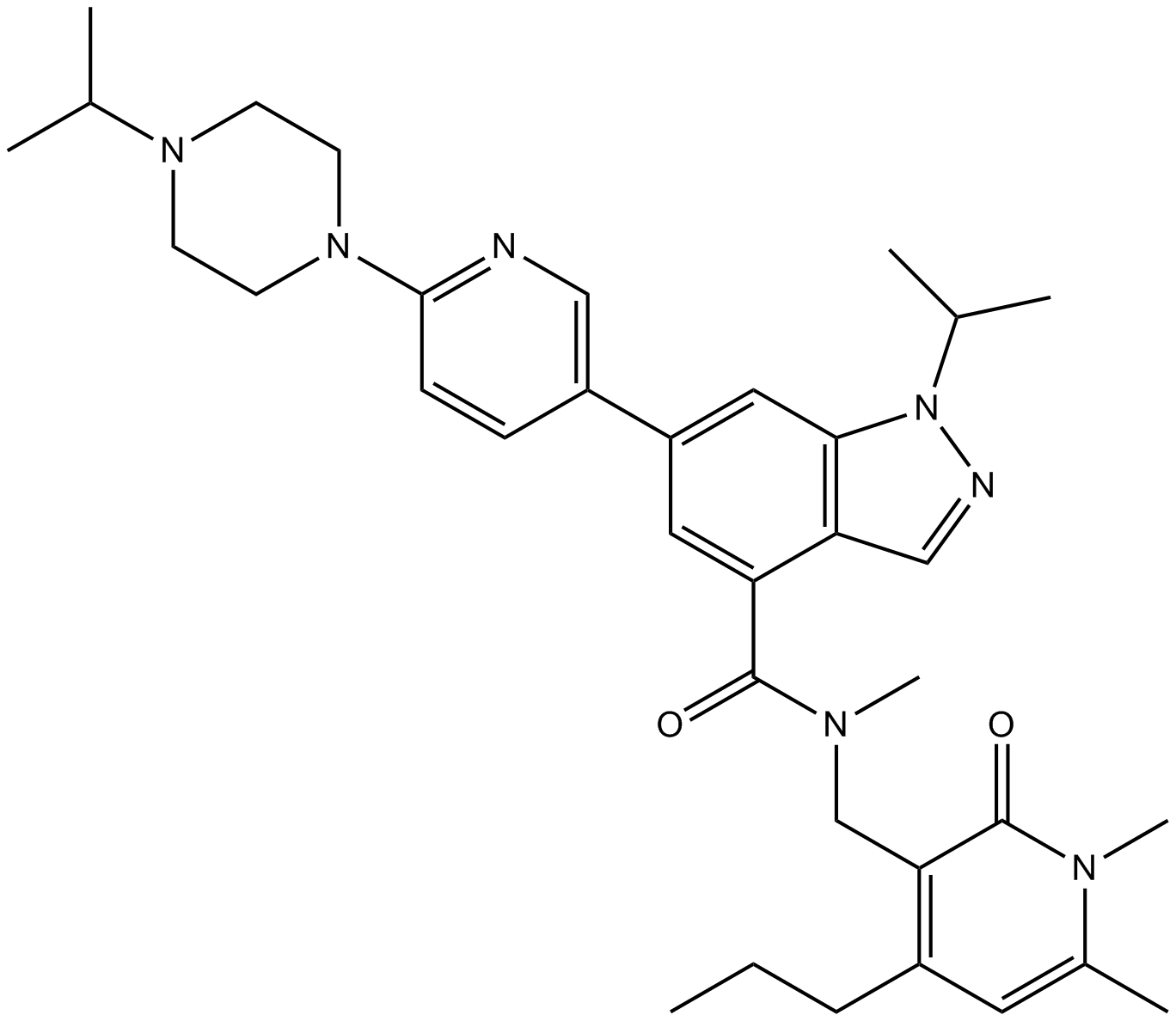
-
GC37856
UNC0321
UNC0321 est un inhibiteur puissant et sélectif de l'histone méthyltransférase G9a avec un Ki de 63 pM et des valeurs IC50 dépendantes du dosage de 6-9 nM. UNC0321 inhibe également le GLP avec des valeurs IC50 dépendantes du dosage de 15-23 nM. UNC0321 est inactif contre SET7/9, SET8/PreSET7, PRMT3 et JMJD2E.
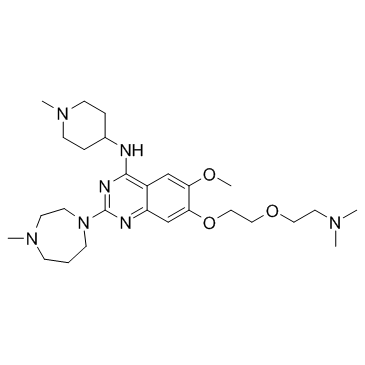
-
GC16741
UNC0379
UNC0379 est un inhibiteur sélectif et compétitif du substrat de la lysine méthyltransférase SETD8 (KMT5A) avec une IC50 de 7,3 μM, une valeur KD de 18,3 μM.
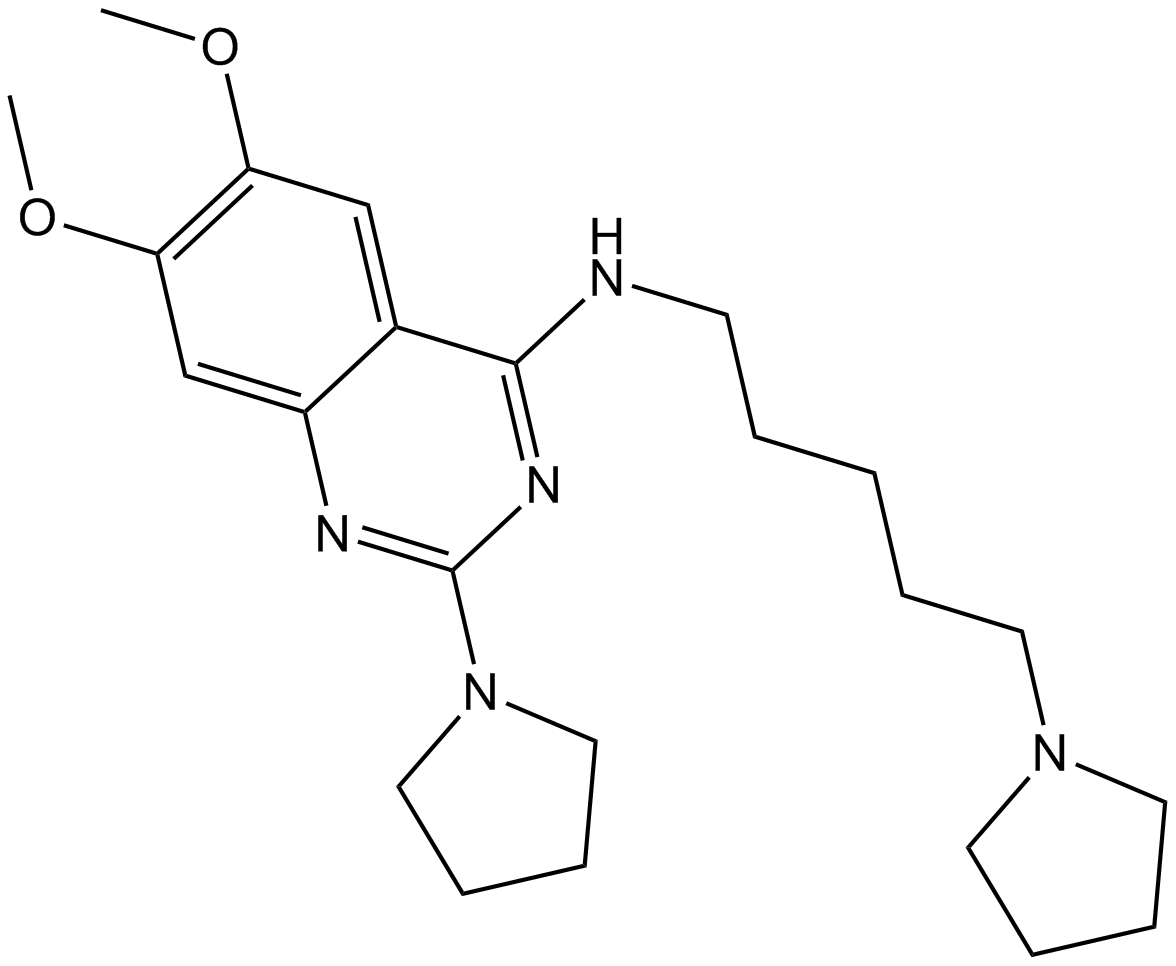
-
GC34160
UNC0379 trifluoroacetate (UNC-0379 trifluoroacetate)
Le trifluoroacétate UNC0379 (trifluoroacétate UNC-0379) est un inhibiteur sélectif et compétitif du substrat de la lysine méthyltransférase SETD8 (KMT5A) avec un IC50 de 7,3 μM, valeur KD de 18,3 μM.
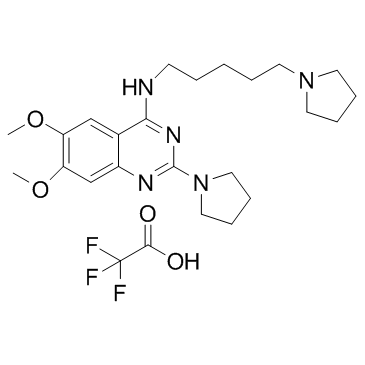
-
GC15610
UNC0638
A G9a and GLP histone methyltransferase inhibitor
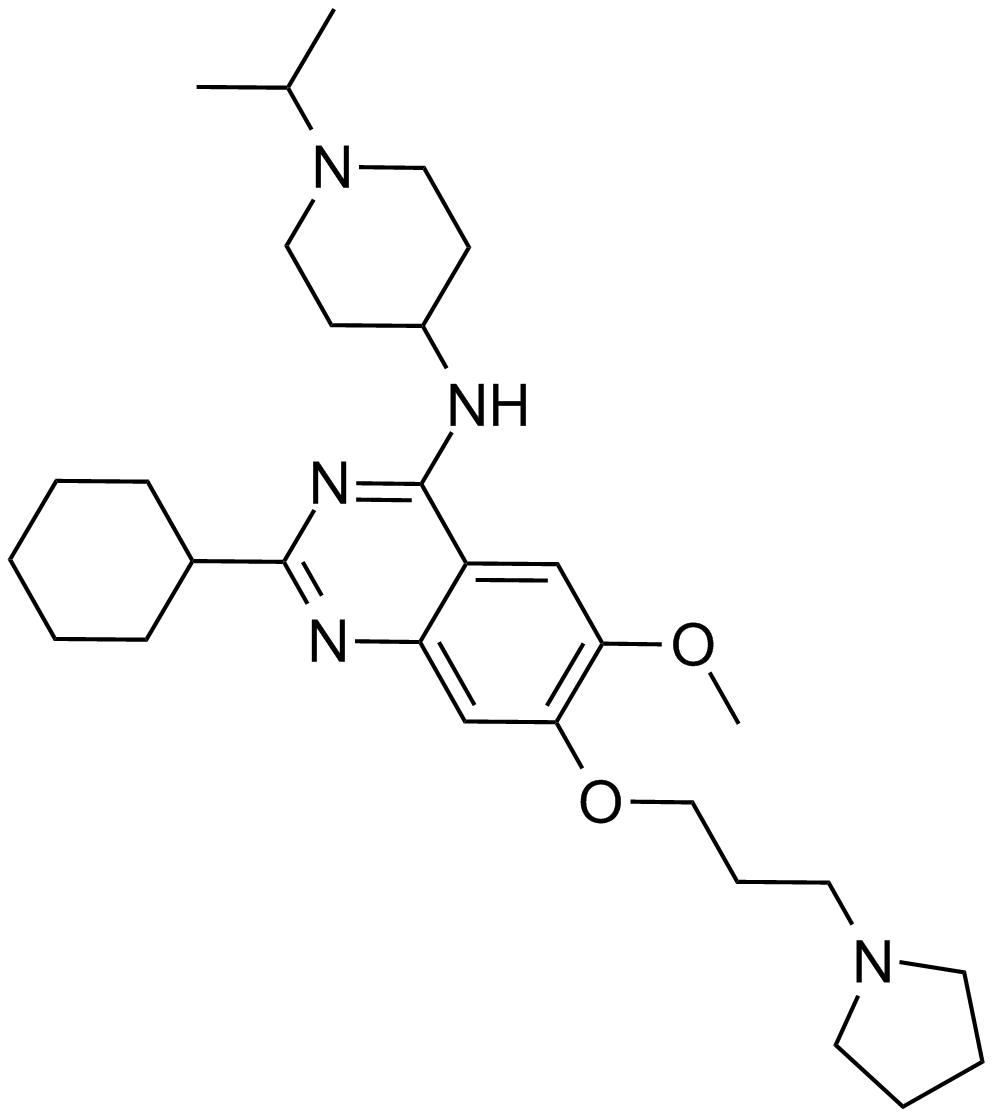
-
GC16794
UNC1215
Potent L3MBTL3 domain inhibitor
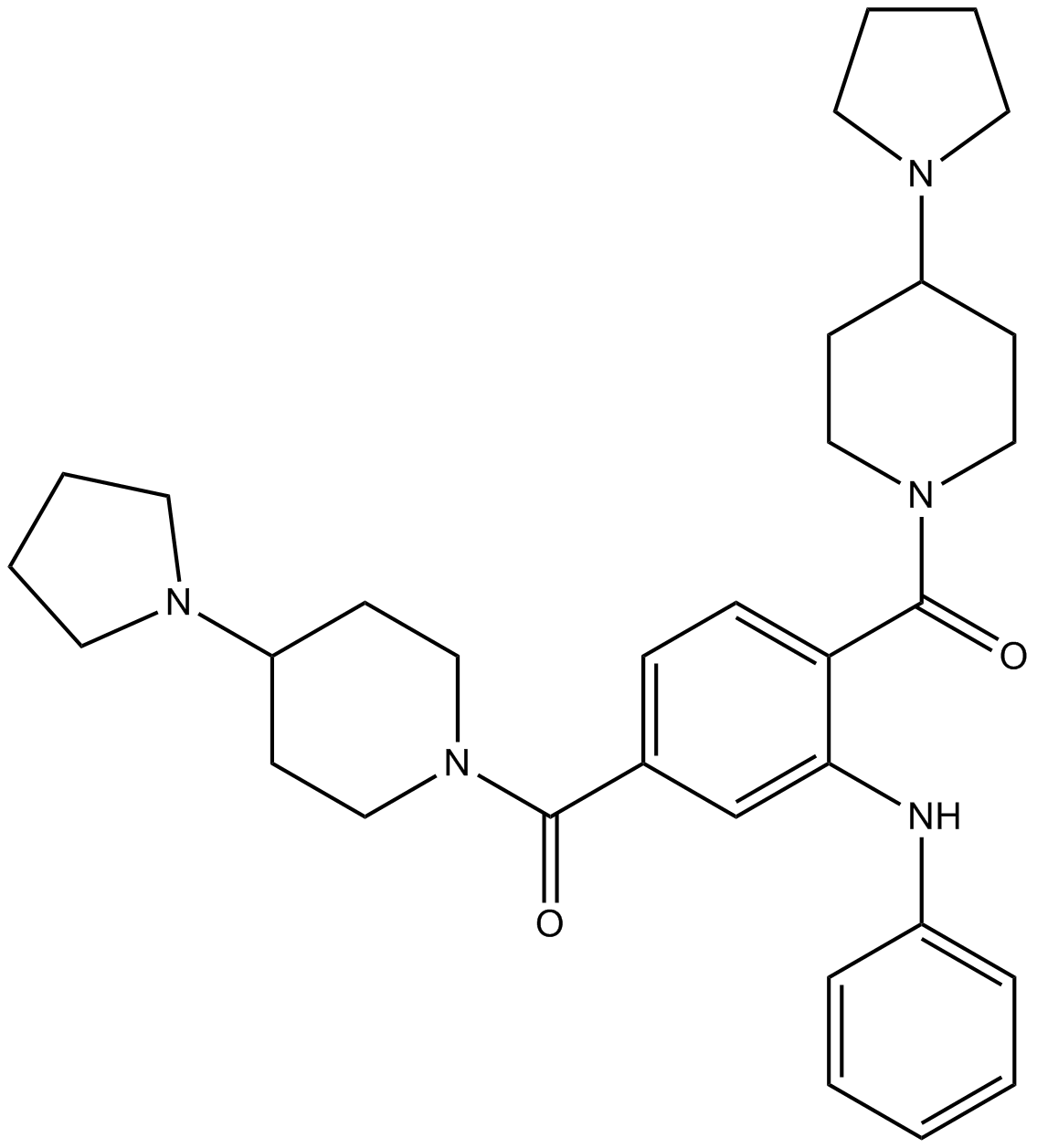
-
GC11375
UNC1999
UNC1999 est un inhibiteur SAM-compétitif, puissant et sélectif d'EZH2/1 avec des IC50 <10 nM et 45 nM, respectivement.
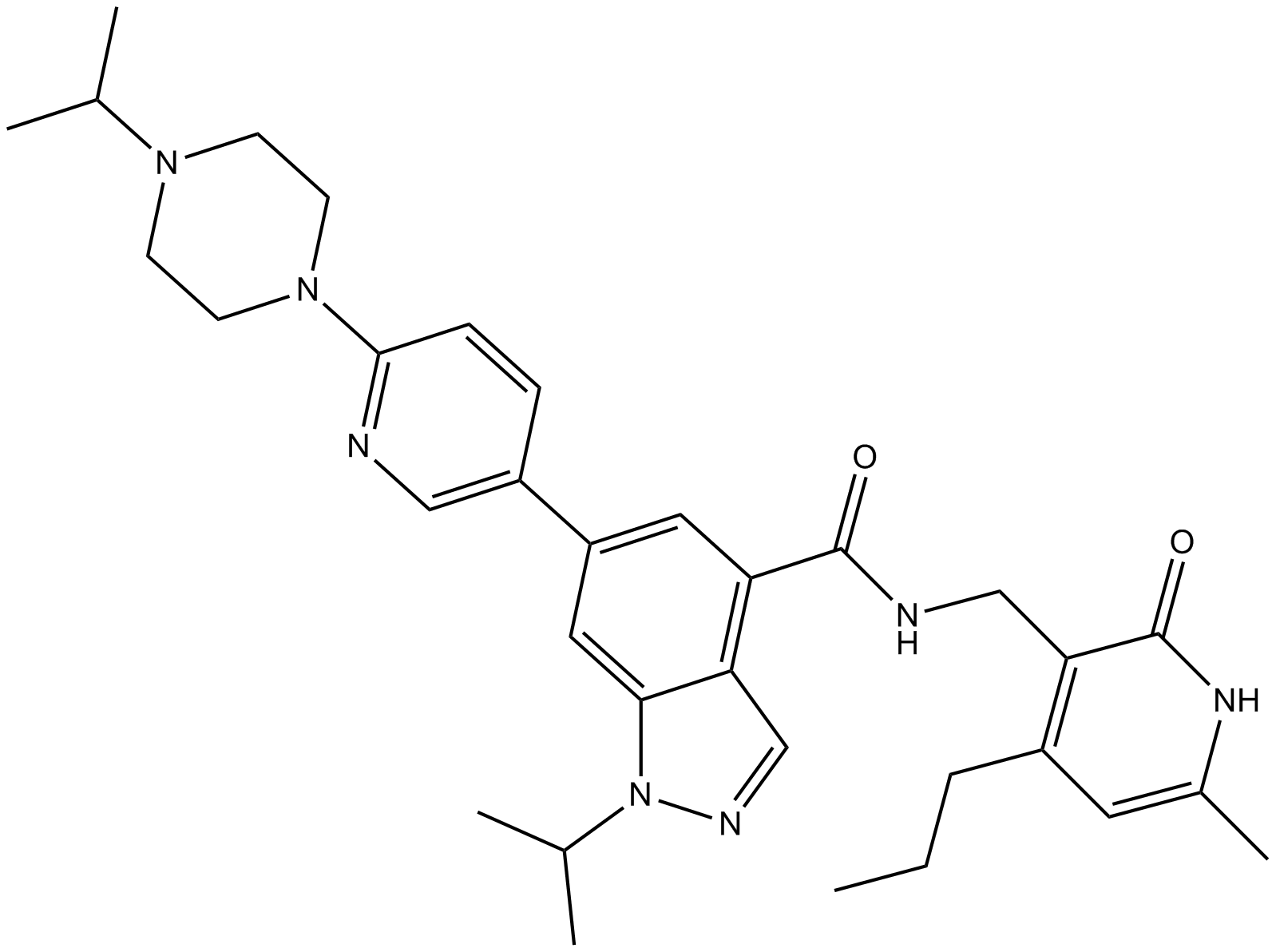
-
GC45117
UNC2327
UNC2327 est un inhibiteur allostérique de la protéine arginine méthyltransférase 3 (PRMT3).
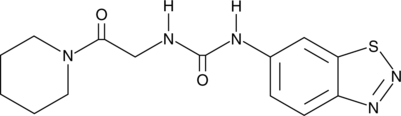
-
GC17207
UNC3866
UNC3866 est un puissant antagoniste de l'interaction CBX7-H3 tel que déterminé par AlphaScreen (IC50 = 66 ± 1,2 nM) et est plus de 100 fois sélectif pour CBX7 par rapport aux neuf autres membres de ce panel de lecteurs de méthyl-lysine (Kme).
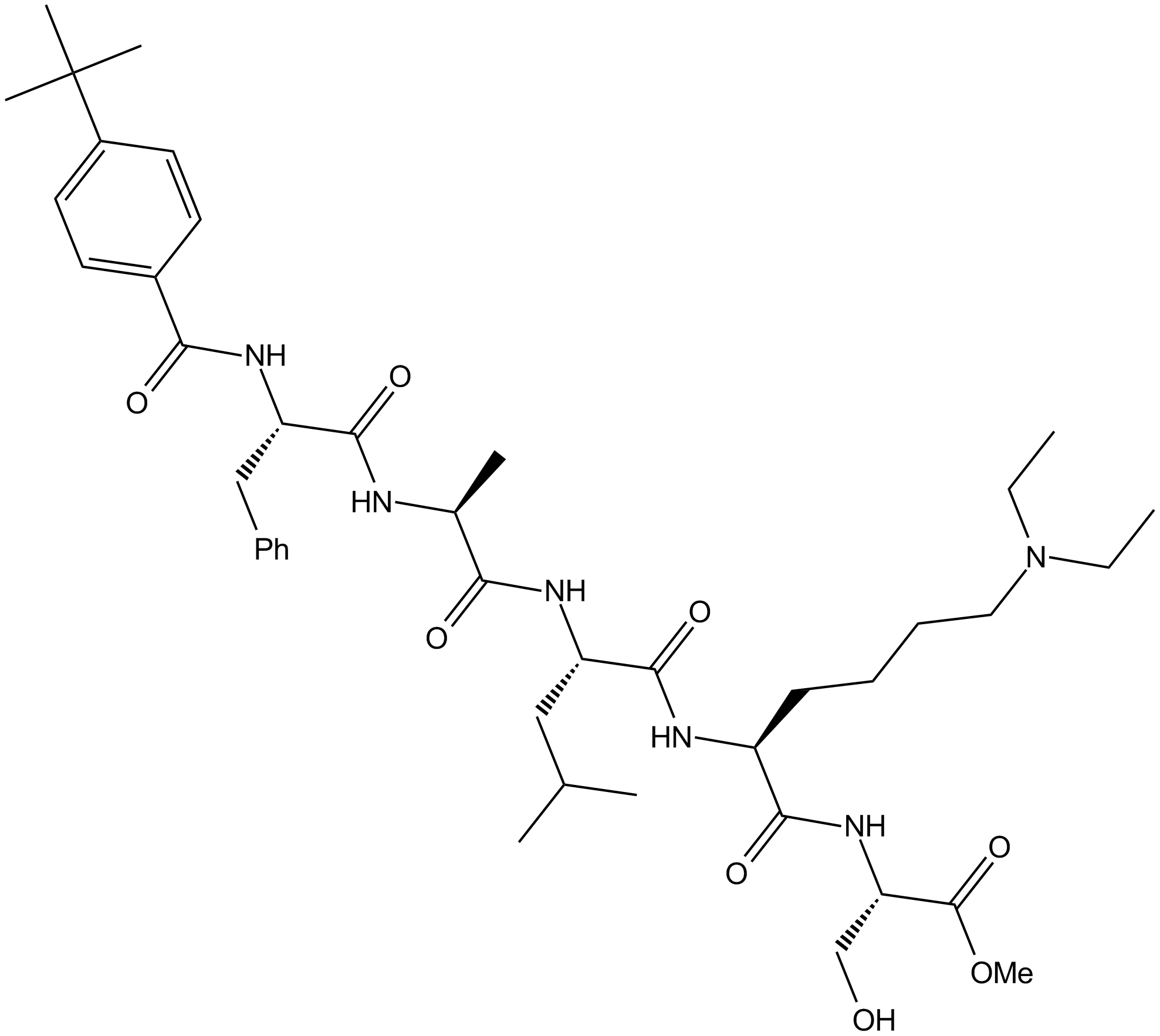
-
GC70091
UNC4976 TFA
UNC4976 TFA est un peptide mimétique régulateur de la conformation positive (PAM) qui se lie au domaine de structure chromatinienne CBX7 et à l'acide nucléique. UNC4976 TFA antagonise le recrutement spécifique de CBX7 sur H3K27me3 aux gènes cibles, tout en augmentant la liaison non-spécifique avec l'ADN et l'ARN.
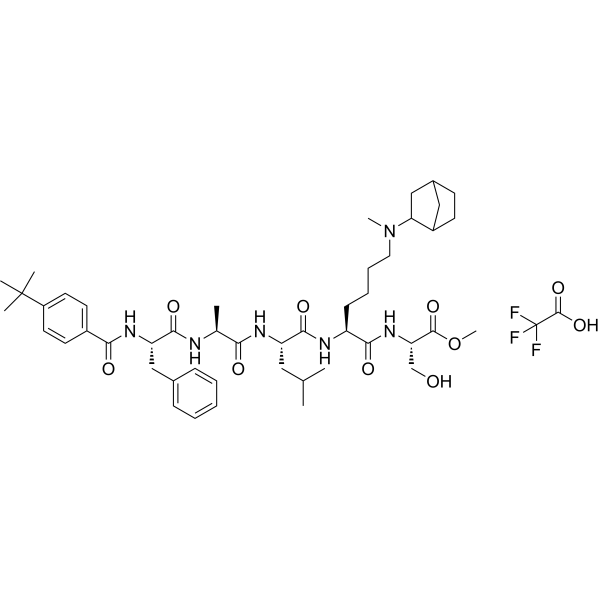
-
GC18096
UNC669
UNC669, un ligand pour un domaine de liaison à la méthyl-lysine, est un puissant inhibiteur de L3MBTL1 (IC50 \u003d 4,2 uM) et de L3MBTL3 (3,1 uM).
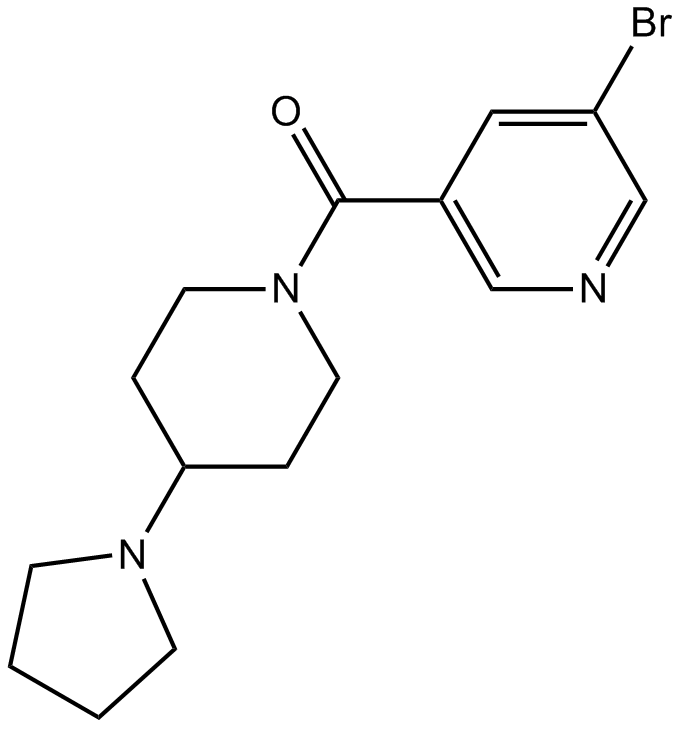
-
GC64083
UNC6934
UNC6934, une sonde chimique ciblant le domaine PWWP, modifie la localisation nucléolaire NSD2.
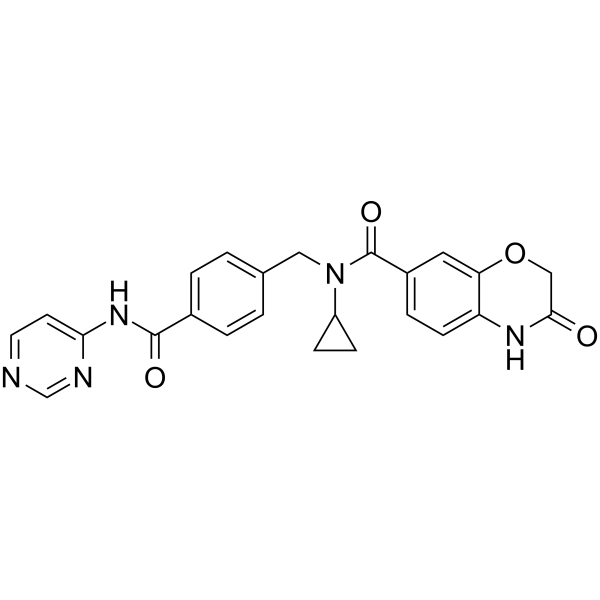
-
GC33397
Valemetostat
Le valemetostat (DS-3201) est un double inhibiteur EZH1/2 de premier ordre, utilisé dans la recherche sur le lymphome T périphérique récidivant/réfractaire.
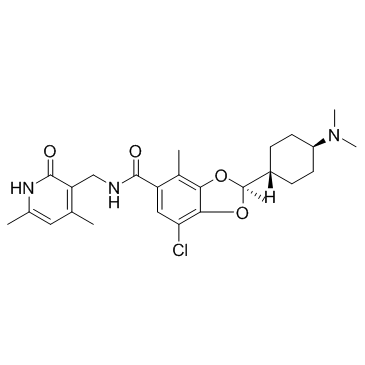
-
GC37881
Valemetostat tosylate
Tosylate de valemetostat (tosylate DS-3201), un double inhibiteur EZH1/2 de premier ordre. Le tosylate de valemetostat peut être utilisé pour la recherche du lymphome T périphérique récidivant/réfractaire.
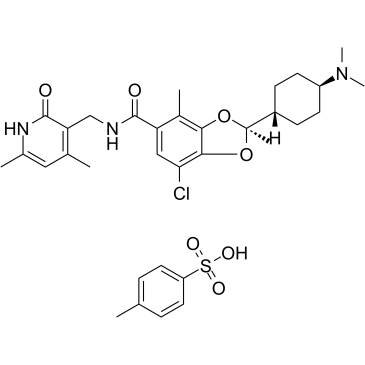
-
GC14763
WDR5 0103
WDR5 0103 est un antagoniste puissant et sélectif de la protéine 5 contenant des répétitions WD (WDR5) avec un Kd de 450 nM.
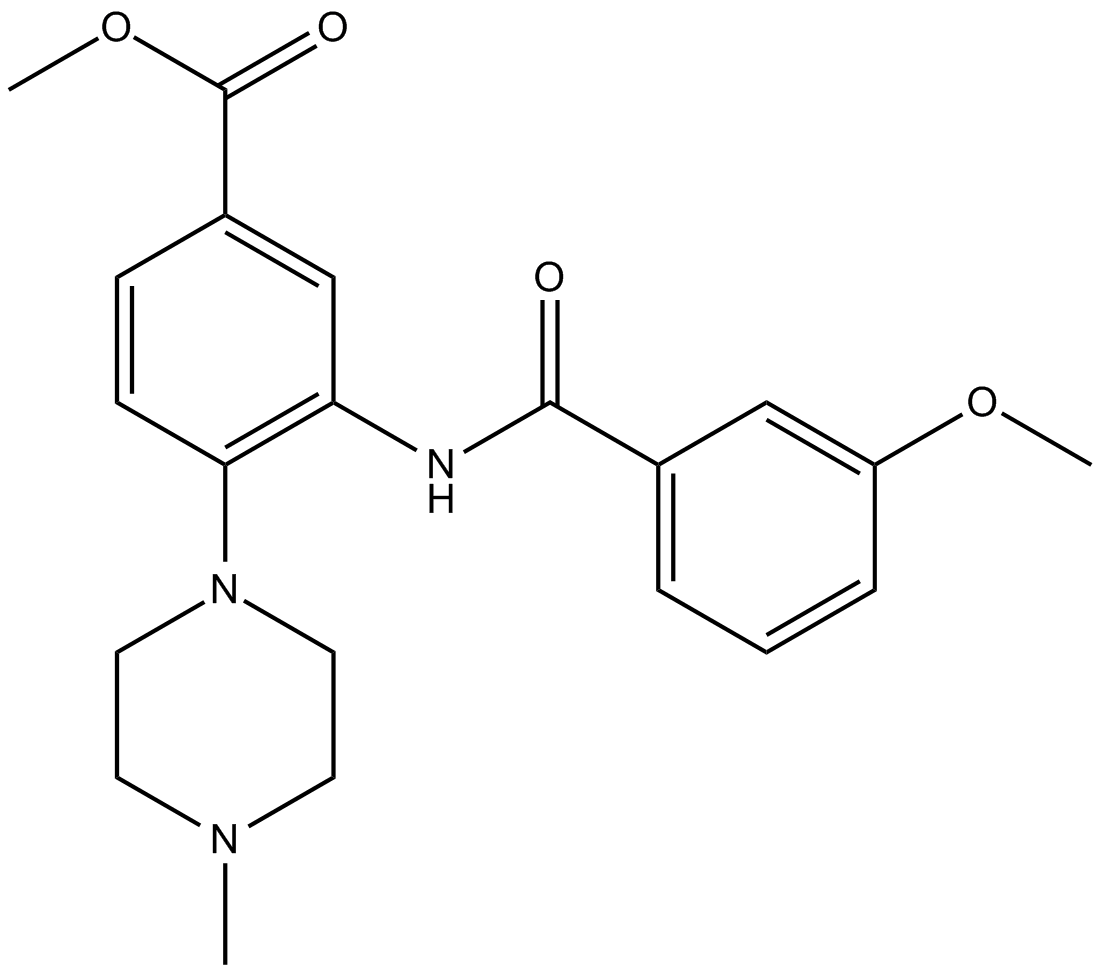
-
GC62558
WDR5-IN-1
WDR5-IN-1 est un inhibiteur puissant et sélectif du domaine 5 de répétition WD (WDR5), avec un Kd <0,02 nM. WDR5-IN-1 inhibe l'activité de l'histone méthyltransférase (HMT) MLL1 avec une IC50 de 2,2 nM. WDR5-IN-1 diminue le recrutement de MYC au niveau des gènes déplacés par WDR5 et présente de puissants effets anti-prolifératifs dans les lignées CHP-134 (neuroblastome) et Ramos (lymphome de Burkitt).
-
GC19388
XY1
XY1 est un analogue très proche de SGC707 (un inhibiteur puissant, sélectif et non compétitif de PRMT3 avec IC50 de 31 nM), mais XY1 est complètement inactif.

-
GC45609
ZLD1039
ZLD1039 est un inhibiteur EZH2 puissant, hautement sélectif et biodisponible par voie orale. ZLD1039 montre une inhibition puissante et dépendante de la concentration de l'activité enzymatique de PRC2 contre le type sauvage EZH2 ainsi que les enzymes mutantes Y641F et A677G avec des valeurs IC50 de 5, 6, 15 et 4, 0 nM, respectivement. Le ZLD1039 inhibe la croissance et les métastases des tumeurs mammaires.