DNA/RNA Synthesis
RNA synthesis, which is also called DNA transcription, is a highly selective process. Transcription by RNA polymerase II extends beyond RNA synthesis, towards a more active role in mRNA maturation, surveillance and export to the cytoplasm.
Single-strand breaks are repaired by DNA ligase using the complementary strand of the double helix as a template, with DNA ligase creating the final phosphodiester bond to fully repair the DNA.DNA ligases discriminate against substrates containing RNA strands or mismatched base pairs at positions near the ends of the nickedDNA. Bleomycin (BLM) exerts its genotoxicity by generating free radicals, whichattack C-4′ in the deoxyribose backbone of DNA, leading to opening of the ribose ring and strand breakage; it is an S-independentradiomimetic agent that causes double-strand breaks in DNA.
First strand cDNA is synthesized using random hexamer primers and M-MuLV Reverse Transcriptase (RNase H). Second strand cDNA synthesis is subsequently performed using DNA Polymerase I and RNase H. The remaining overhangs are converted into blunt ends using exonuclease/polymerase activity. After adenylation of the 3′ ends of DNA fragments, NEBNext Adaptor with hairpin loop structure is ligated to prepare the samples for hybridization. Cell cycle and DNA replication are the top two pathways regulated by BET bromodomain inhibition. Cycloheximide blocks the translation of mRNA to protein.
Ziele für DNA/RNA Synthesis
Produkte für DNA/RNA Synthesis
- Bestell-Nr. Artikelname Informationen
-
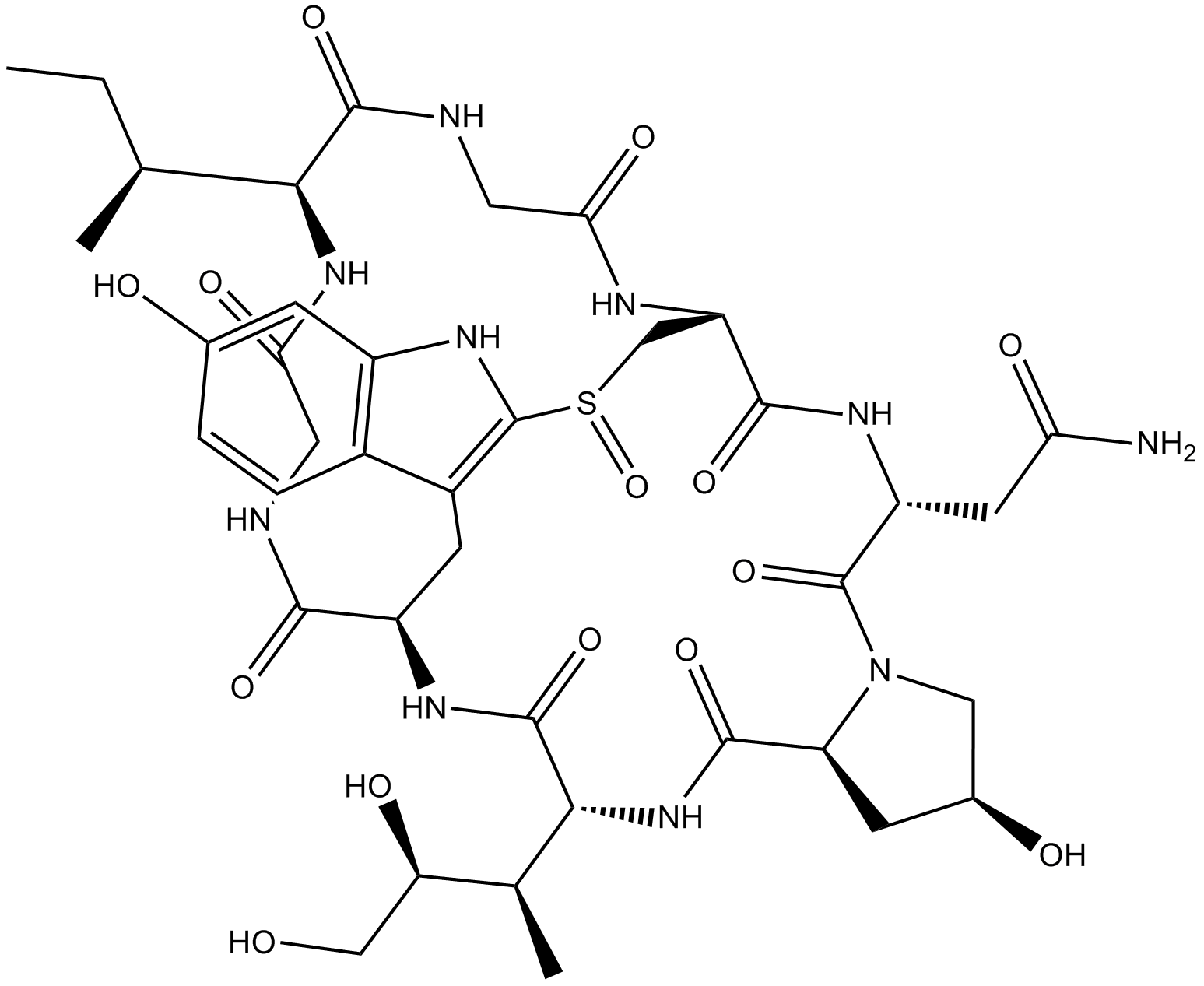
GC65372
Pseudouridine 5'-triphosphate trisodium
-
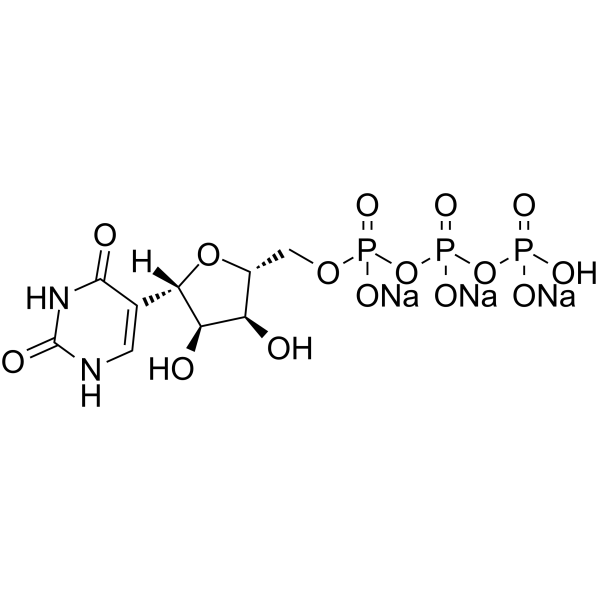
GC16367
PSI-7977
A prodrug form of PSI-7411
-
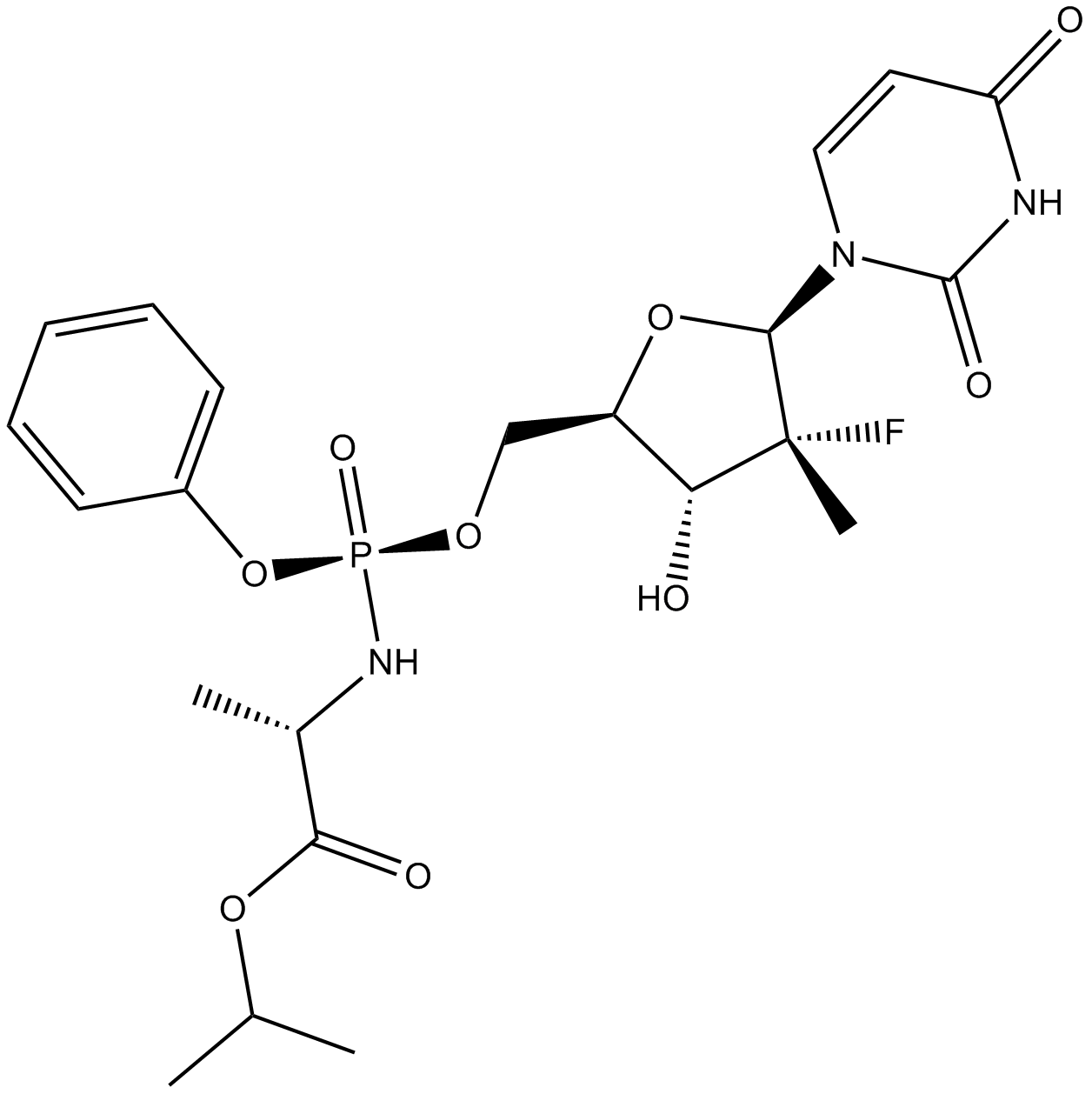
GC65335
PTC299
PTC299 ist ein oral aktiver Inhibitor der VEGFA-mRNA-Translation, der selektiv die VEGF-Proteinsynthese auf posttranskriptioneller Ebene hemmt. PTC299 ist auch ein starker Inhibitor der Dihydroorotat-Dehydrogenase (DHODH). PTC299 zeigt eine gute orale BioverfÜgbarkeit und das Fehlen einer Off-Target-Kinasehemmung und Myelosuppression. PTC299 kann fÜr die Erforschung hÄmatologischer Malignome nÜtzlich sein.
-
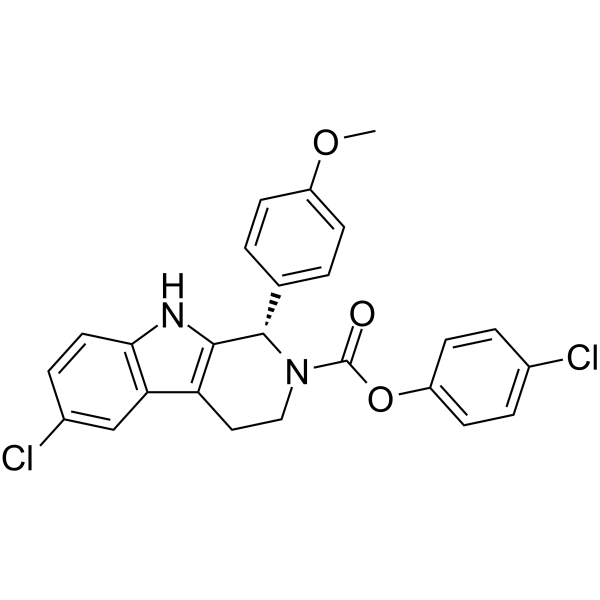
GC16384
Puromycin dihydrochloride
Puromycin-Dihydrochlorid wird von Streptomyces alboniger, einem grampositiven Actinomyceten, durch eine Reihe von enzymatischen Reaktionen produziert.
-
GC37042
Pyrazofurin
Pyrazofurin, ein Pyrimidin-Nucleosid-Analogon mit antineoplastischer AktivitÄt, hemmt die Zellproliferation und die DNA-Synthese in Zellen durch Hemmung der Uridin-5'-Phosphat (UMP)-Synthase. Pyrazofurin ist ein aktiver, sensitiver Orotat-Phosphoribosyltransferase-Inhibitor mit IC50-Werten zwischen 0,06 und 0,37 μM in den drei Plattenepithelkarzinom (SCC)-Zelllinien Hep-2, HNSCC-14B und HNSCC-14C.
-
GC45552
Pyrenophorol
Pyrenophorol ist eine C9H12O3-Verbindung, die in geringer Ausbeute aus Kulturfiltraten des phytopathogenen Pilzes Stemphylium radicinum, C.
-
GC45553
Pyridoxine-d3 (hydrochloride)
-
GC37044
Pyrindamycin A
Pyrindamycin A ist ein Antibiotikum, das die DNA-Synthese hemmt.
-
GC37051
Quarfloxin
Quarfloxin (CX-3543), ein Fluorchinolon-Derivat mit antineoplastischer AktivitÄt, zielt auf und hemmt die RNA-pol-I-AktivitÄt mit IC50-Werten im nanomolaren Bereich in Neuroblastomzellen. Quarfloxin unterbricht die Wechselwirkung zwischen dem Nucleolin-Protein und einer G-Quadruplex-DNA-Struktur in der ribosomalen DNA (rDNA)-Matrize.
-
GC61666
Quinizarin
Chinizarin (1,4-Dihydroxyanthrachinon), ein Bestandteil der Antikrebsmittel wie Doxorubicin, Daunorubicin und Adriamycin, interkaliert mit DNA durch Interkalationsmodus (Kd = 86,1 μM).
-
GC17750
Raltitrexed
Raltitrexed ist ein Antimetabolit-Medikament, das in der Chemotherapie verwendet wird und durch Hemmung der Thymidylat-Synthase wirkt.
-
GC18751
Reticulol
Reticulol is an isocoumarin derivative produced by certain species of Streptomyces that inhibits cAMP phosphodiesterase (IC50 = 41 uM).
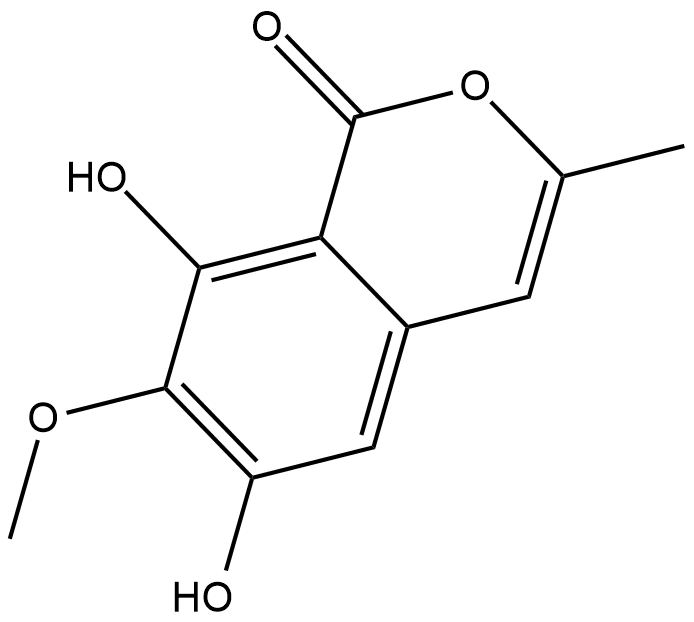
-
GC19309
RG7800
RG7800 ist ein SMN2-Spleißmodifikator.
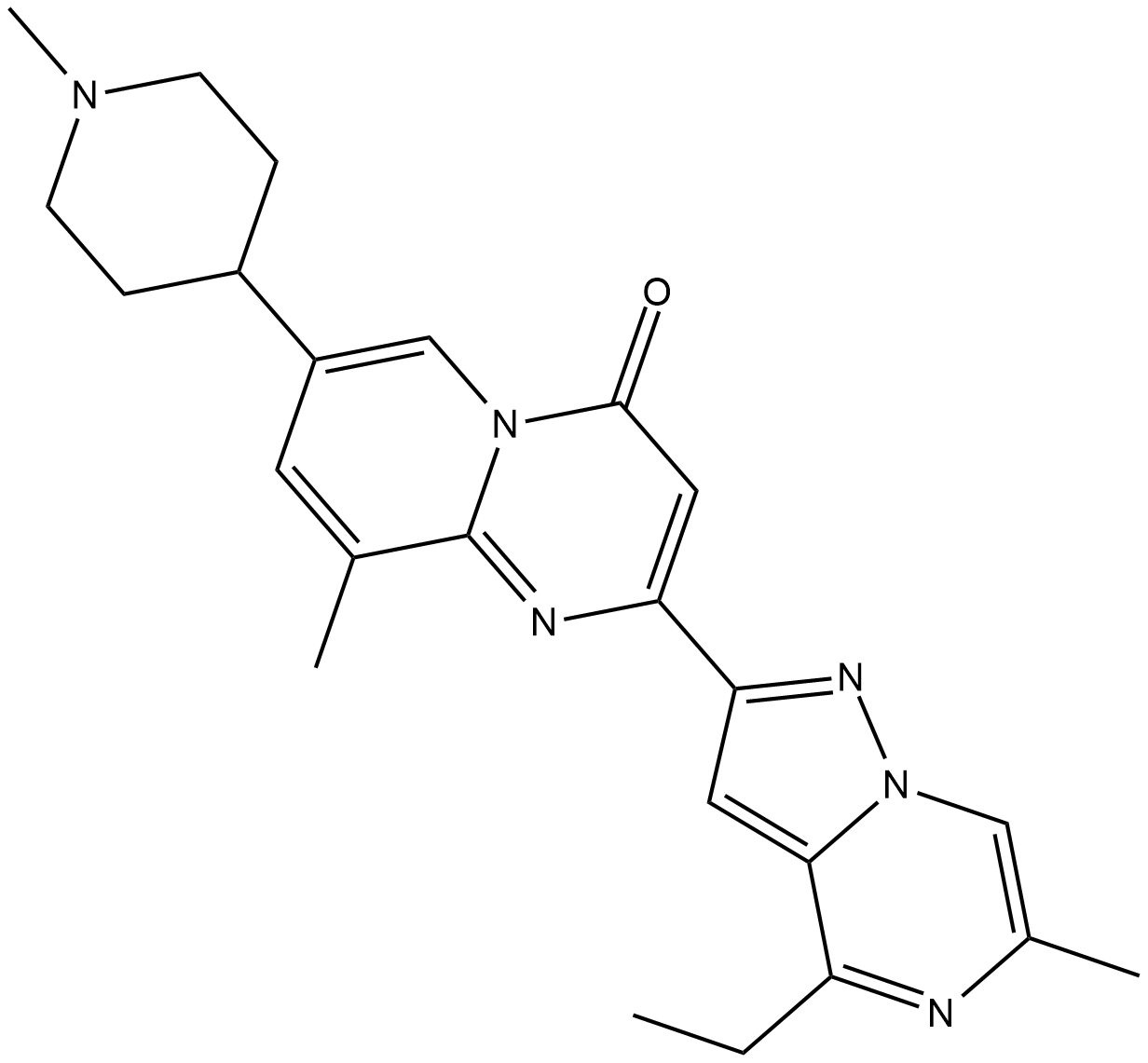
-
GC37521
RG7800 hydrochloride
RG7800-Hydrochlorid ist ein oral aktiver SMN2-Splicing-Modulator mit EC1.5xs von 23 nM und 87 nM fÜr SMN2-Splicing und SMN-Protein; RG7800-Hydrochlorid hat das Potenzial, spinale Muskelatrophie zu behandeln.
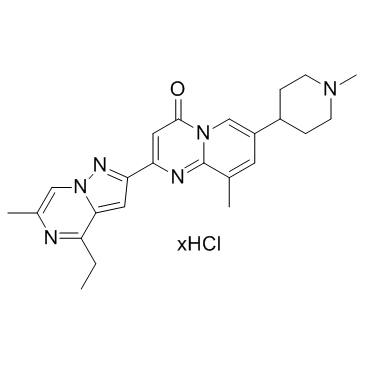
-
GC61246
Rifalazil
Rifalazil (KRM-1648; ABI-1648), ein Rifamycin-Derivat, hemmt die bakterielle DNA-abhÄngige RNA-Polymerase und tÖtet Bakterienzellen ab, indem es die β-Untereinheit in der RNA-Polymerase blockiert.
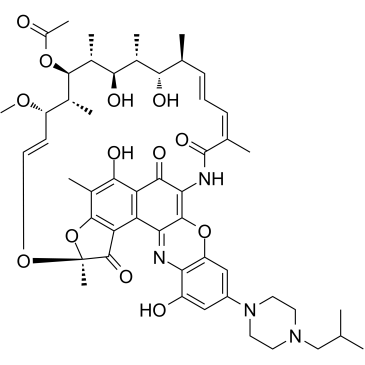
-
GC16970
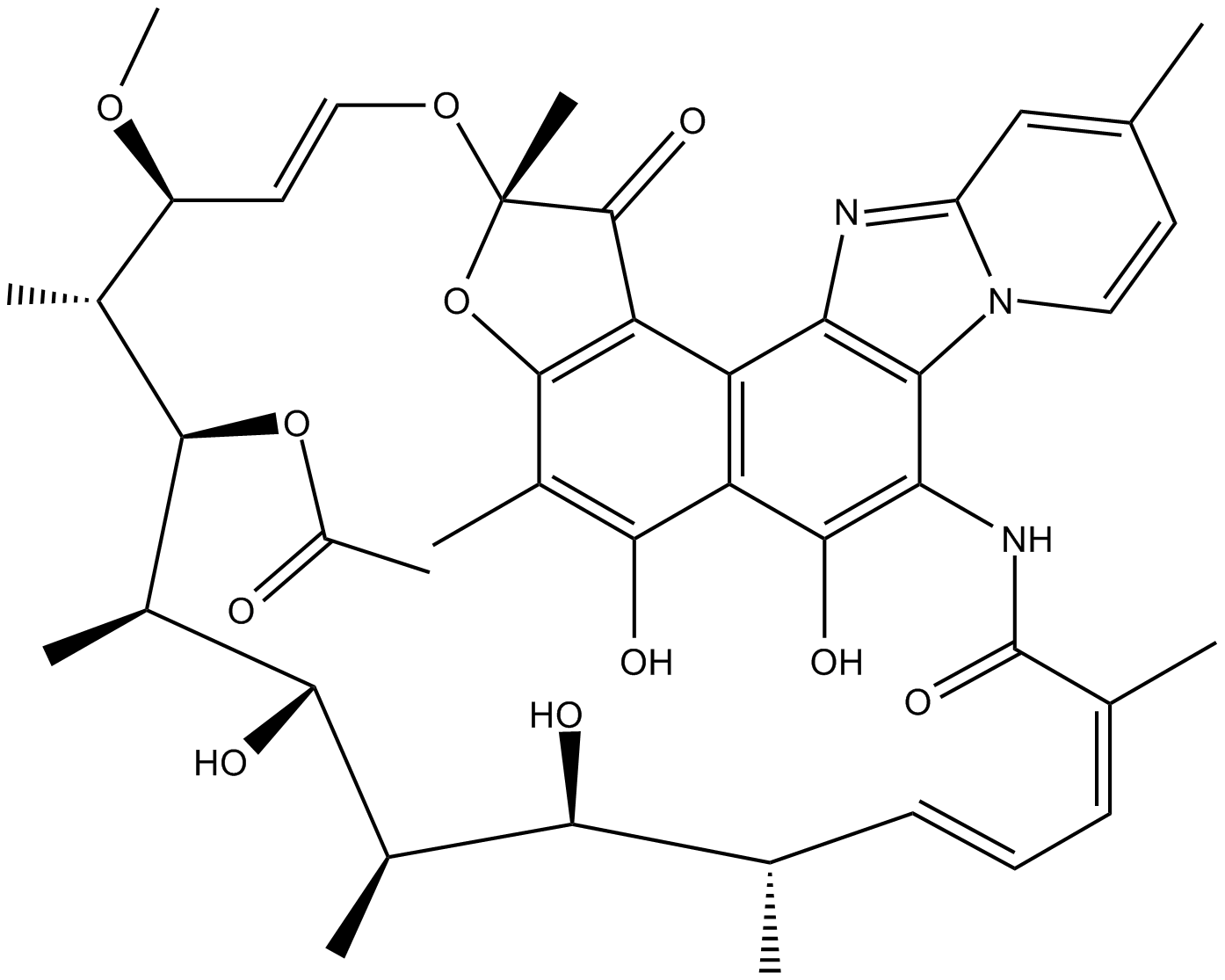
Rifaximin (Xifaxan)
Rifaximin (Xifaxan), ein gastrointestinal-selektives Antibiotikum, bindet die ⋲-Untereinheit der bakteriellen DNA-abhÄngigen RNA-Polymerase, was zu einer Hemmung der bakteriellen RNA-Synthese fÜhrt.
-
GC30845
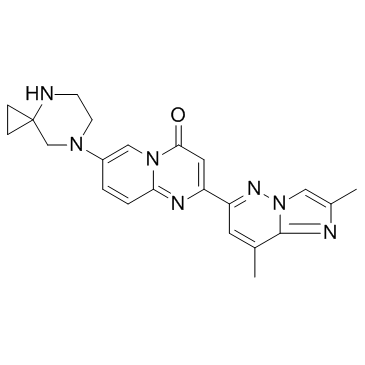
Risdiplam (RG7916)
Risdiplam (RG7916) (RG7916) ist ein oral verabreichter, zentral und peripher verteilter SMN2-PrÄ-mRNA-Splicing-Modifikator, der die Überlebensproteinspiegel von Motoneuronen (SMN) erhÖht.
-
GC67865
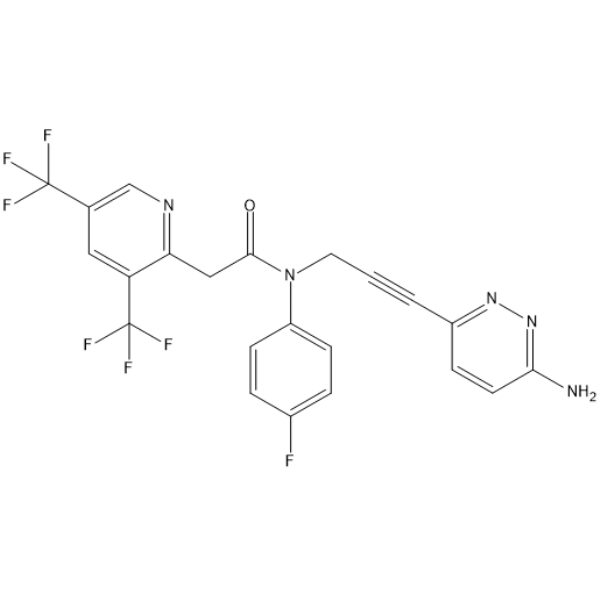
RP-6685
-
GC14808
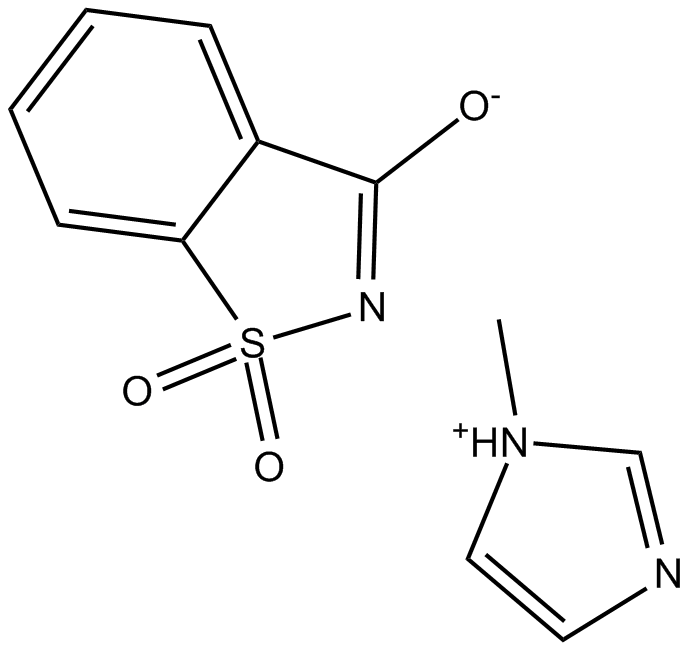
Saccharin 1-methylimidazole (SMI)
Saccharin-1-Methylimidazol (SMI) ist ein Aktivator fÜr die DNA/RNA-Synthese.
-
GC62693
SCR130
SCR130 ist ein SCR7-basierter DNA-Nonhomologe-End-Joining (NHEJ)-Inhibitor. SCR130 hemmt die Endverbindung von DNA auf Ligase IV-abhÄngige Weise. SCR130 ist spezifisch fÜr Ligase IV und zeigt minimale oder keine Wirkung auf die durch Ligase III und Ligase I vermittelte Verbindung. SCR130 induziert Zellapoptose und wirkt gegen Krebs.
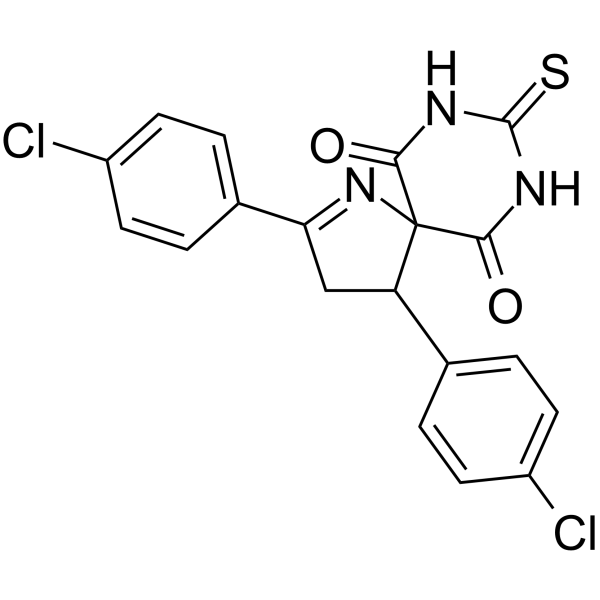
-
GC12106
SCR7
SCR7 ist eine instabile Form, die in eine stabile Form von SCR7-Pyrazin autocyclisiert werden kann. SCR7-Pyrazin ist ein DNA-Ligase-IV-Inhibitor, der die nicht-homologe EndverknÜpfung (NHEJ) auf Ligase-IV-abhÄngige Weise blockiert. SCR7-Pyrazin ist auch ein CRISPR/Cas9-Enhancer, der die Effizienz der Cas9-vermittelten homologiegesteuerten Reparatur (HDR) erhÖht. SCR7-Pyrazin induziert Zellapoptose und wirkt gegen Krebs.
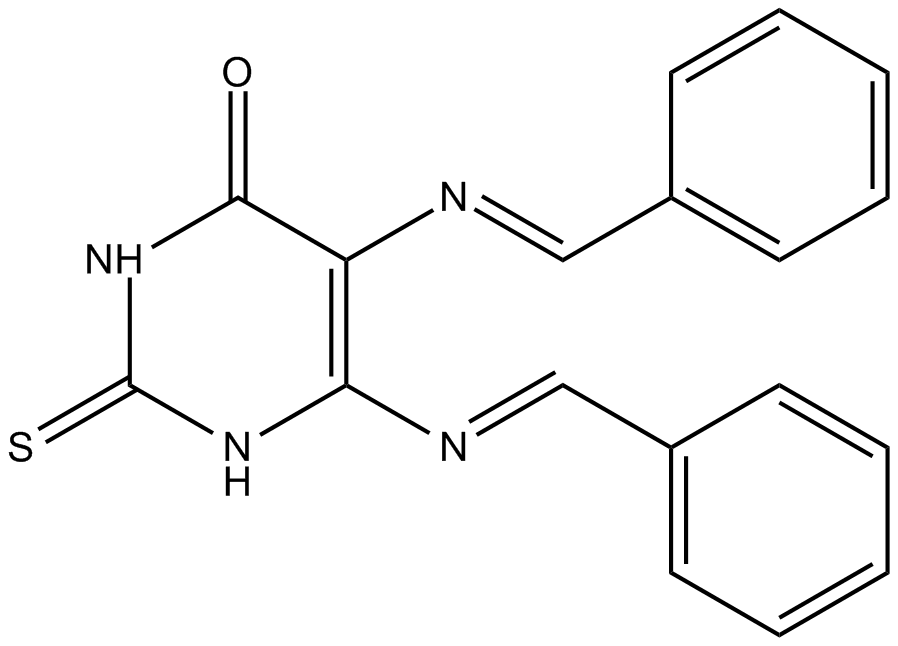
-
GC61276
Silver sulfadiazine
Silbersulfadiazin (AgSD), ein Sulfonamid-Antibiotikum, bewirkt durch seine Sulfa-Einheit (SD-SDZ), die die bakterielle Folatabsorption und die anschließende DNA-Synthese verhindert, eine doppelte Hemmwirkung auf das Bakterienwachstum.
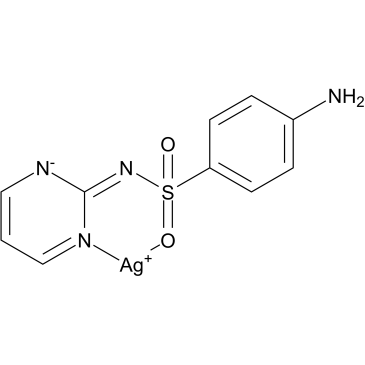
-
GC30185
SMN-C3
SMN-C3 ist ein oral aktiver SMN2-Spleißmodulator und hat das Potenzial zur Behandlung von spinaler Muskelatrophie (SMA).
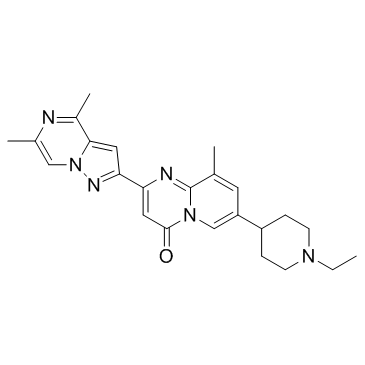
-
GC39552
Sorivudine
Sorivudin (BV-araU) ist ein oral aktiver synthetischer Pyrimidinnukleosid-Antimetabolit.
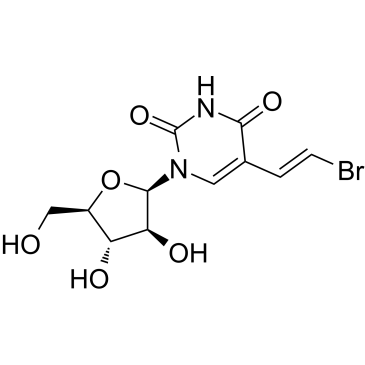
-
GC46223
Sparsomycin
A bacterial metabolite with diverse biological activities
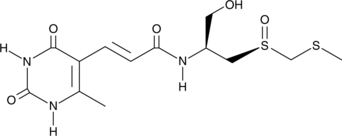
-
GC44953
Sterigmatocystin
Sterigmatocystin ist ein VorlÄufer von Aflatoxinen und ein Mykotoxin, das von gewÖhnlichen SchimmelpilzstÄmmen von Aspergillus versicolor produziert wird.
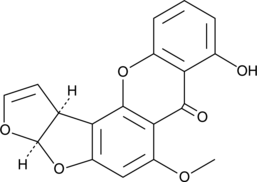
-
GC46228
Streptimidone
A bacterial metabolite with diverse biological activities
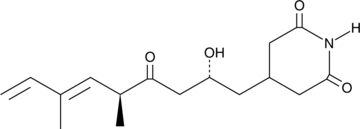
-
GC17131
Streptozocin
Ein diabetogenes Mittel, das auf Betazellen abzielt.
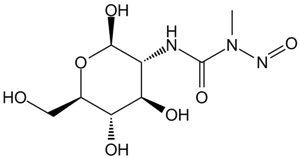
-
GC39510
Synucleozid
Synucleozid (NSC 377363) ist ein potenter Inhibitor des SNCA-mRNA, die das α-Synuclein-Protein kodiert (IC50=1,5 μM).
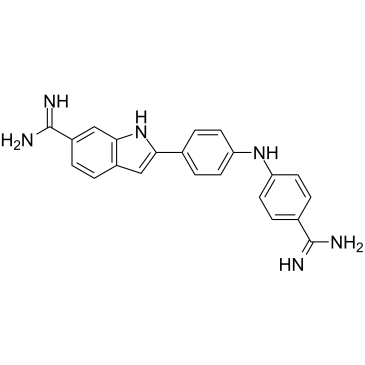
-
GC39511
Synucleozid hydrochloride
Synucleozid-Hydrochlorid (NSC 377363-Hydrochlorid) ist ein starker Inhibitor der SNCA-mRNA, die fÜr das α-Synuclein-Protein (IC50 = 1,5 μM) kodiert.
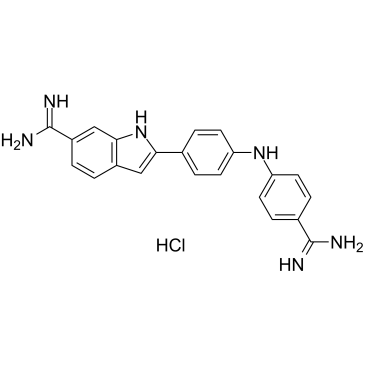
-
GC12350
T 705
T 705 (T-705) ist ein potenter viraler RNA-Polymerase-Inhibitor, er wird durch zellulÄre Enzyme zu seiner aktiven Form, T 705-Ribofuranosyl-5′-Triphosphat (RTP), phosphoribosyliert.
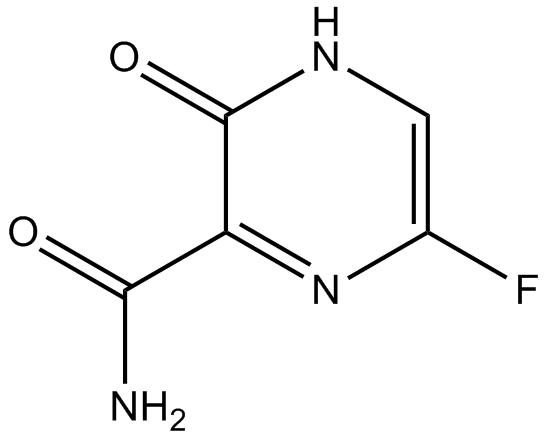
-
GC44981
T-2 Toxin
T-2-Toxin ist ein häufiges Trichothecen-Mykotoxin, das von Fusarium-Arten produziert wird und Getreidepflanzen infizieren kann. Dies führt bei Menschen und Tieren zu alimentärer toxischer Aleukie.
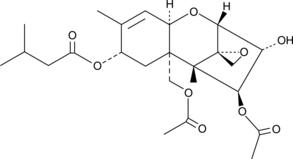
-
GC61500
T-705RTP sodium
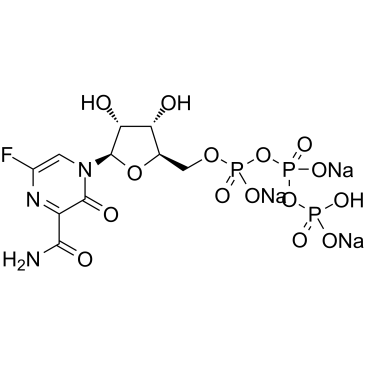
-
GC44984
TAF 10 Peptide
TAF10 is one of many protein factors or coactivators associated with RNA polymerase II activity.

-
GC39512
Tempo
Tempo ist ein klassisches Nitroxid-Radikal und ein selektiver ROS-FÄnger, der Superoxid im Katalysezyklus dismutiert. Tempo induziert einen DNA-Strangbruch. Tempo kann als Organokatalysator fÜr die Oxidation von primÄren Alkoholen zu Aldehyden eingesetzt werden. Tempo hat mutagene und antioxidative Wirkungen.
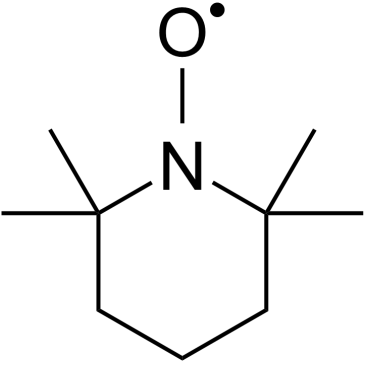
-
GC64373
Tezacitabine
Tezacitabin ist ein zytostatischer und zytotoxischer Antimetabolit und ein Nukleosid-Analogon. Tezacitabin hemmt irreversibel die Ribonukleotid-Reduktase und greift in die DNA-Replikation und -Reparatur ein. Tezacitabin induziert effektiv Zellen apoptotisch. Tezacitabin hat das Potenzial zur Behandlung von LeukÄmien und soliden Tumoren (Karzinomen).
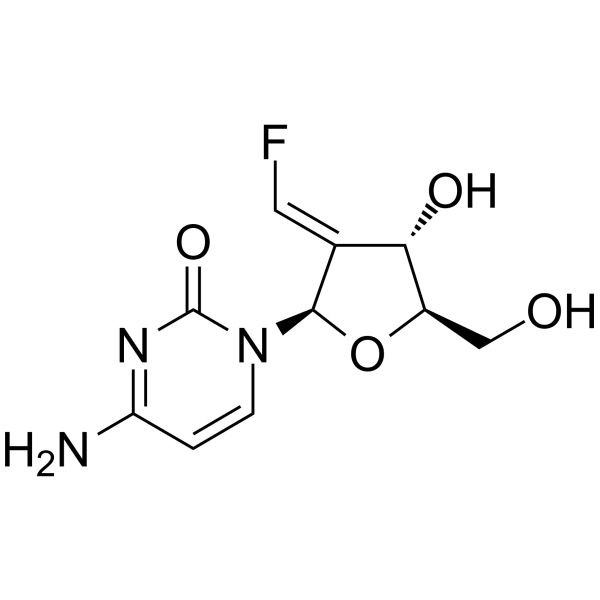
-
GC17601
TH287
TH287 ist ein potenter und selektiver Inhibitor von MTH1 mit einem IC50 von 0,8 nM. TH287 ist hochselektiv gegenÜber MTH1, ohne relevante Hemmung von MTH2, NUDT5, NUDT12, NUDT14, NUDT16, dCTPase, dUTPase und ITPA bei 100 μM. TH287 kÖnnte als Chemotherapeutikum fÜr die Krebsforschung fungieren.
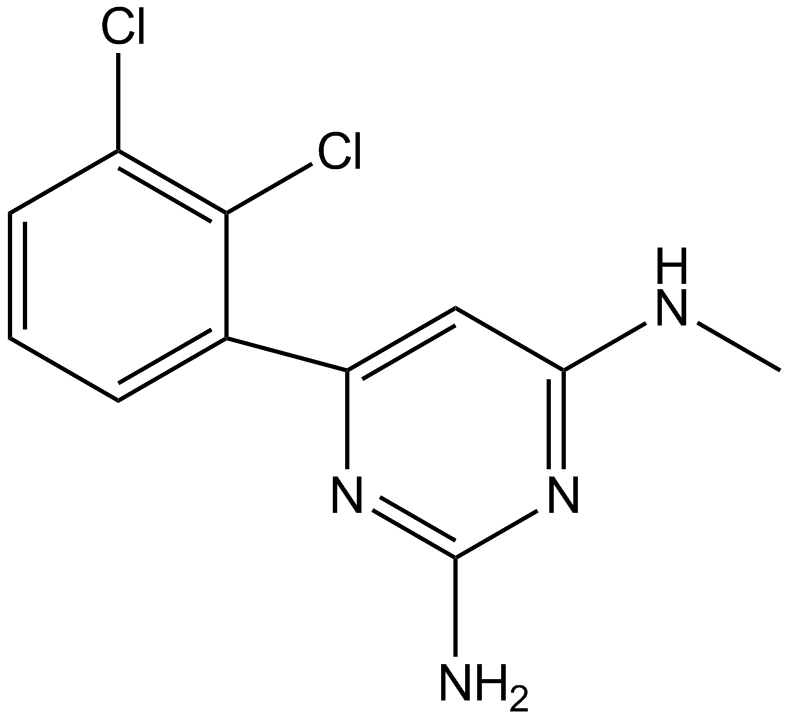
-
GC37775
TH287 hydrochloride
TH287-Hydrochlorid ist ein potenter und selektiver Inhibitor von MTH1 mit einem IC50-Wert von 0,8 nM. TH287-Hydrochlorid ist hochselektiv gegenÜber MTH1, ohne relevante Hemmung von MTH2, NUDT5, NUDT12, NUDT14, NUDT16, dCTPase, dUTPase und ITPA bei 100 μM. TH287-Hydrochlorid kÖnnte als Chemotherapeutikum fÜr die Krebsforschung dienen.
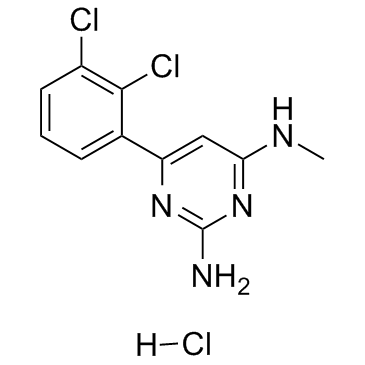
-
GC12716
TH588
TH588 ist der erste Inhibitor seiner Klasse der Nudix-Hydrolase-Familie, der MTH1 (IC50 = 5 nM) wirksam und selektiv angreift und hemmt.
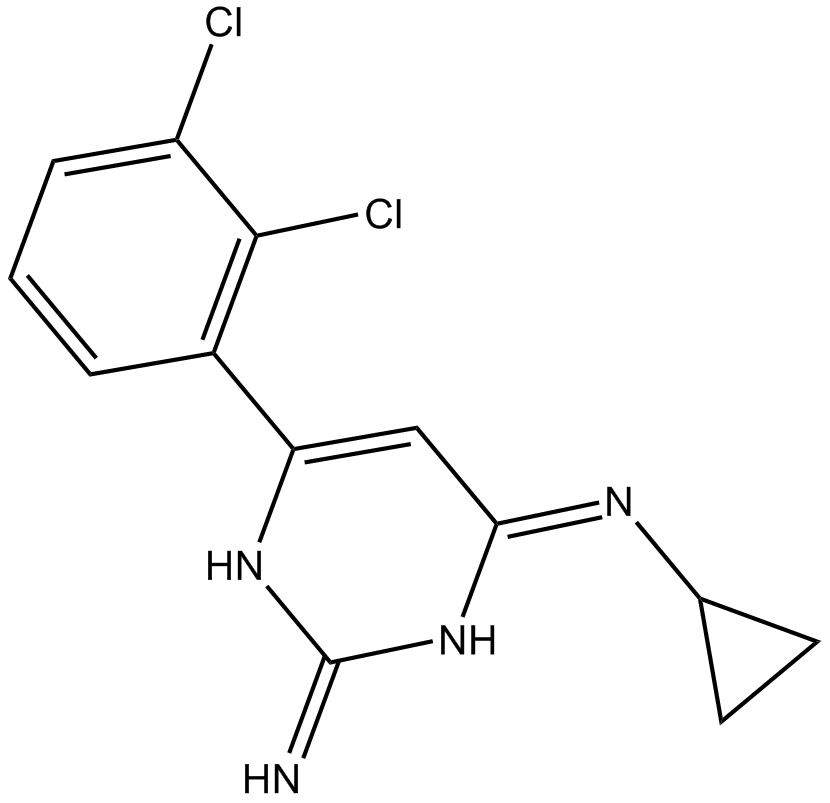
-
GC37777
TH588 hydrochloride
TH588-Hydrochlorid ist der erste Inhibitor seiner Klasse der Nudix-Hydrolase-Familie, der MTH1 (IC50 = 5 nM) wirksam und selektiv angreift und hemmt.
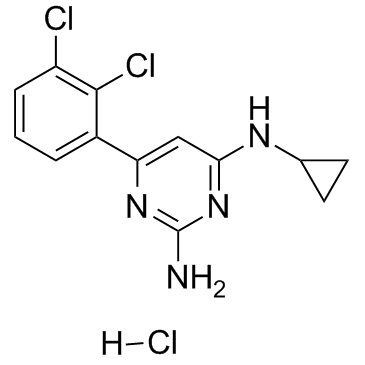
-
GC31868
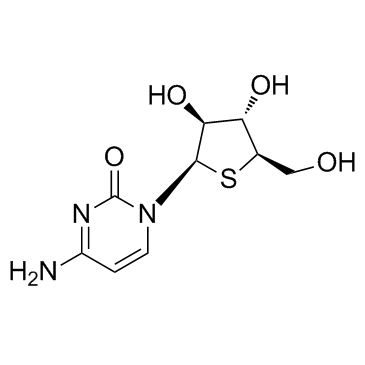
Thiarabine (OSI-7836)
Thiarabin (OSI-7836) (OSI-7836) zeigt starke Anti-Tumor-AktivitÄt und Hemmung der DNA-Synthese.
-
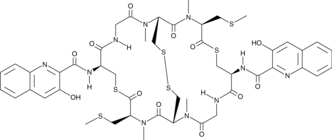
GC48165
Thiocoraline
A depsipeptide and DNA bis-intercalator with antibacterial and anticancer activities
-
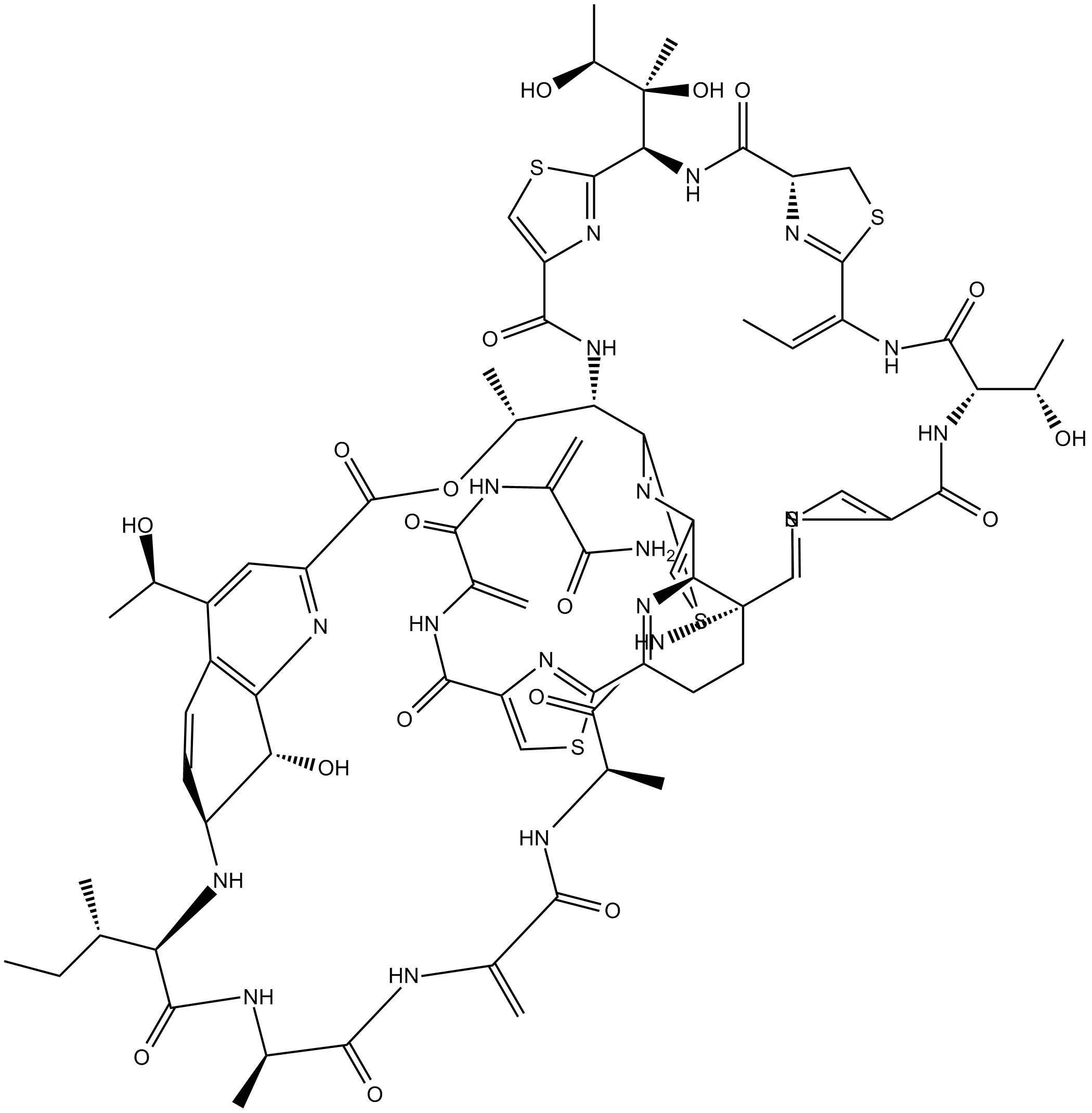
GC13002
Thiostrepton
Thiostrepton ist ein Thiazol-Antibiotikum, das FOXM1 selektiv hemmt.
-
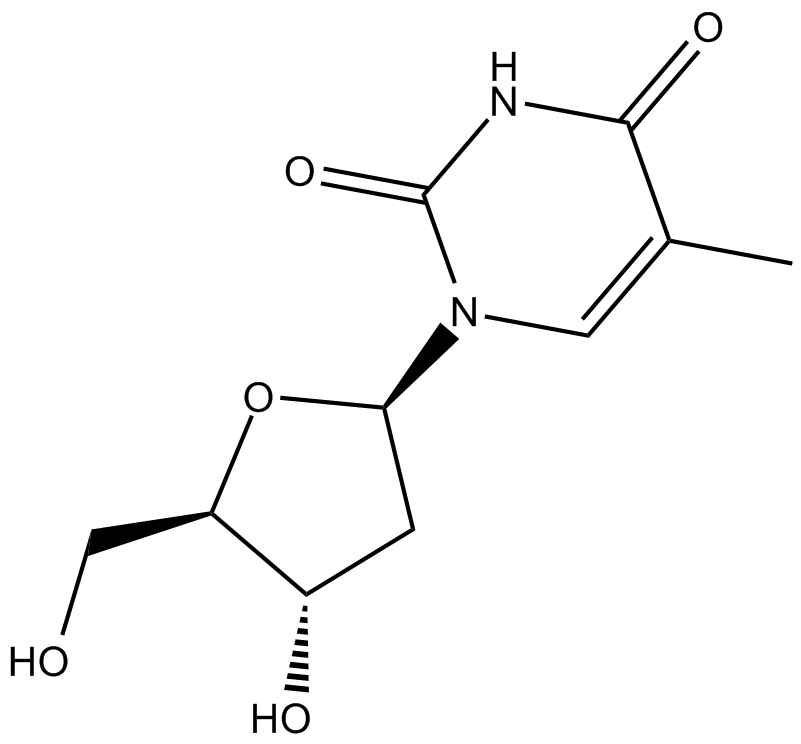
GC15815
Thymidine
Pyrimidin-Nukleosid
-
GC49529
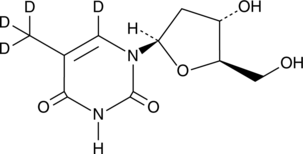
Thymidine-d4
An internal standard for the quantification of thymidine
-
GC49474
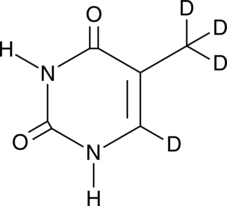
Thymine-d4
An internal standard for the quantification of thymine
-
GC49076
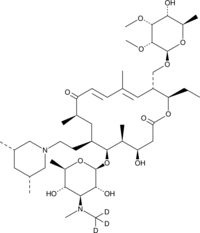
Tilmicosin-d3
An internal standard for the quantification of tilmicosin
-
GC64897
Tofersen
Tofersen (BIIB067) ist eine Antisense-Oligonukleotid, das den RNase H-abhängigen Abbau von Superoxiddismutase 1 (SOD1)-mRNA vermittelt, um die Synthese des SOD1-Proteins zu reduzieren.

-
GC48190
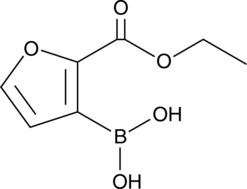
TP-4748
A heterocyclic building block
-
GC17897
Triapine
Ribonucleotide reductase inhibitor
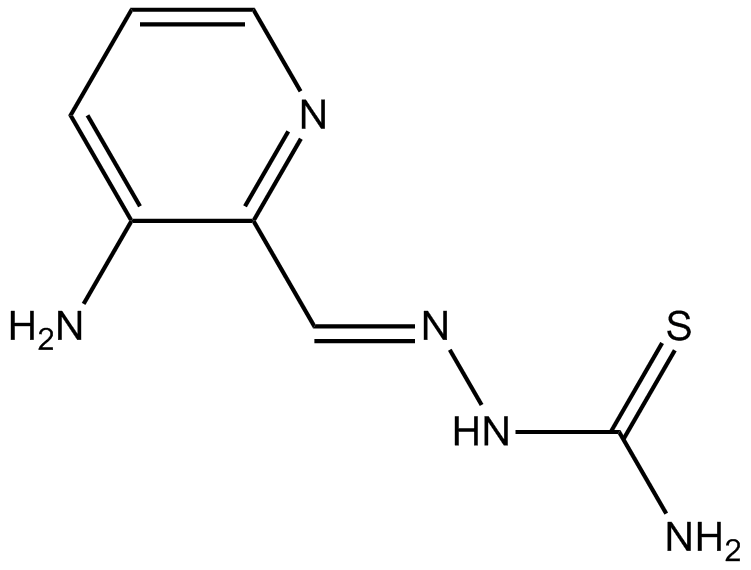
-
GC61348
Triazavirin
Triazavirin ist ein Nukleosid-Analogon der NukleinsÄure und ein antivirales Mittel.
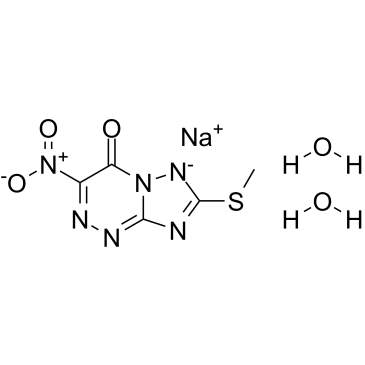
-
GC15392
Triciribine
Triciribin ist ein DNA-Synthesehemmer, der auch Akt und HIV-1/2 mit IC50 von 130 nM bzw. 0,02-0,46 μM hemmt.
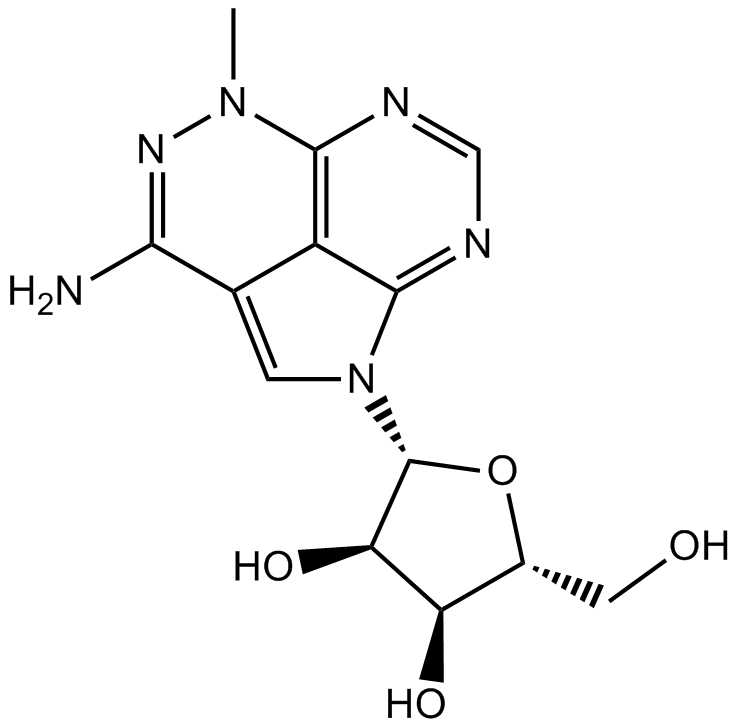
-
GC13399
Trimidox
specific ribonucleotide reductase inhibitor
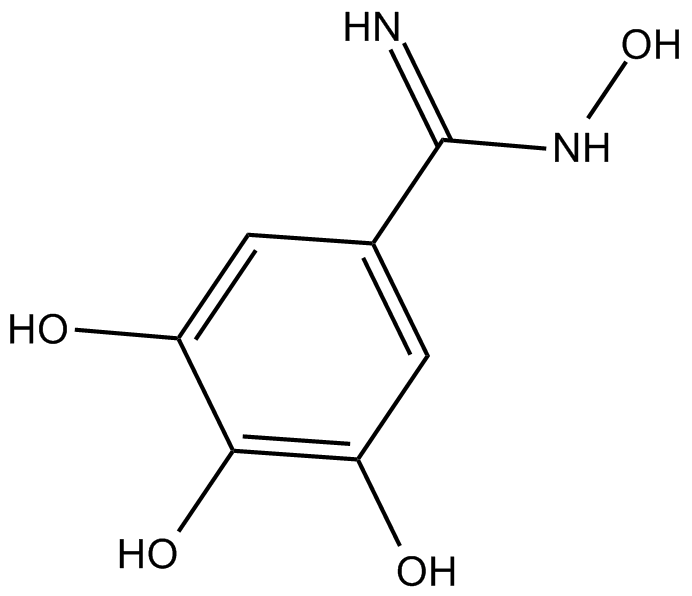
-
GC12044
Trovafloxacin mesylate
Trovafloxacin-Mesylat ist ein Breitspektrum-Chinolon-Antibiotikum mit starker AktivitÄt gegen grampositive, gramnegative und anaerobe Organismen.
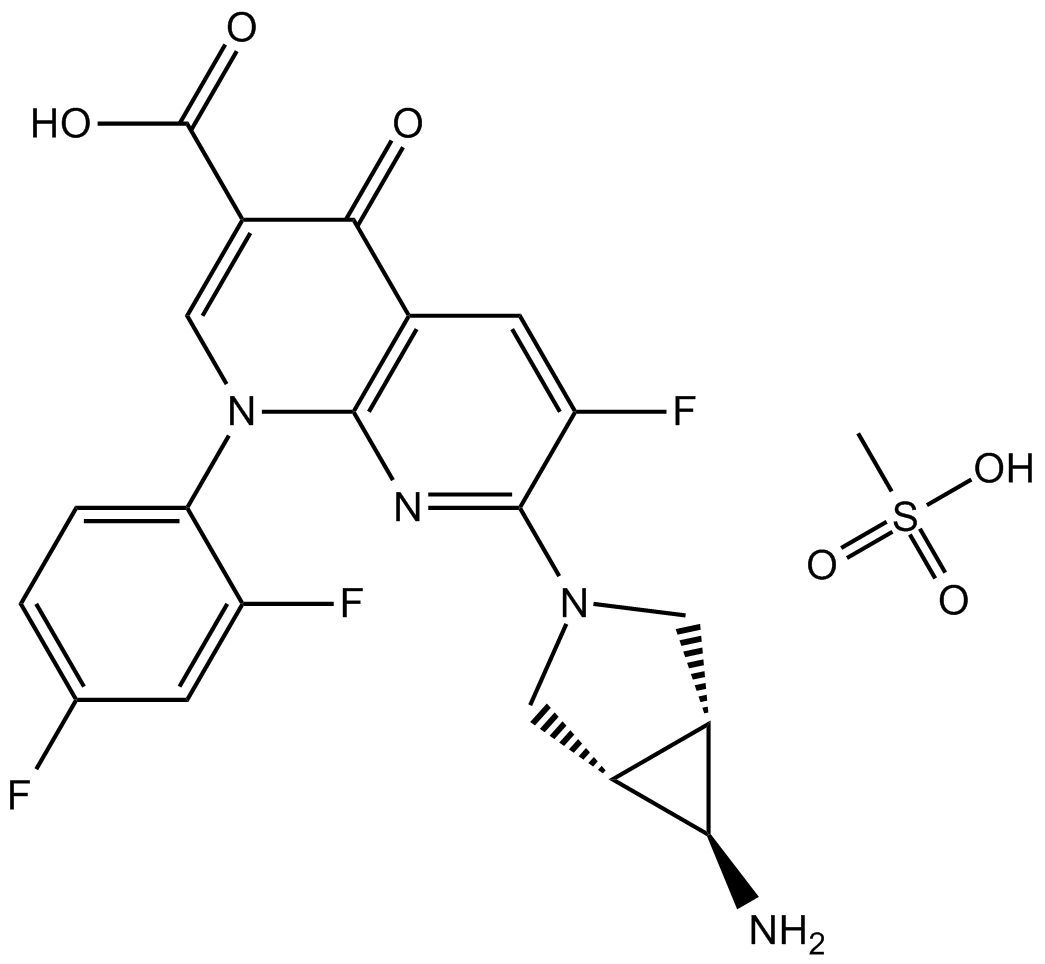
-
GC61359
TTP-8307
TTP-8307 ist ein potenter Inhibitor der Replikation mehrerer Rhino- und Enteroviren.
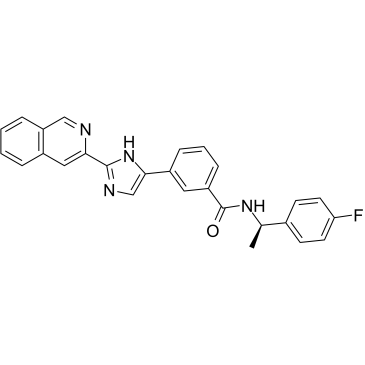
-
GC16879
Tubercidin
Tubercidin ist ein Pyrrolopyrimidin-Nukleosidanalogon mit signifikanter Aktivität gegen Schistosomeninfektionen.
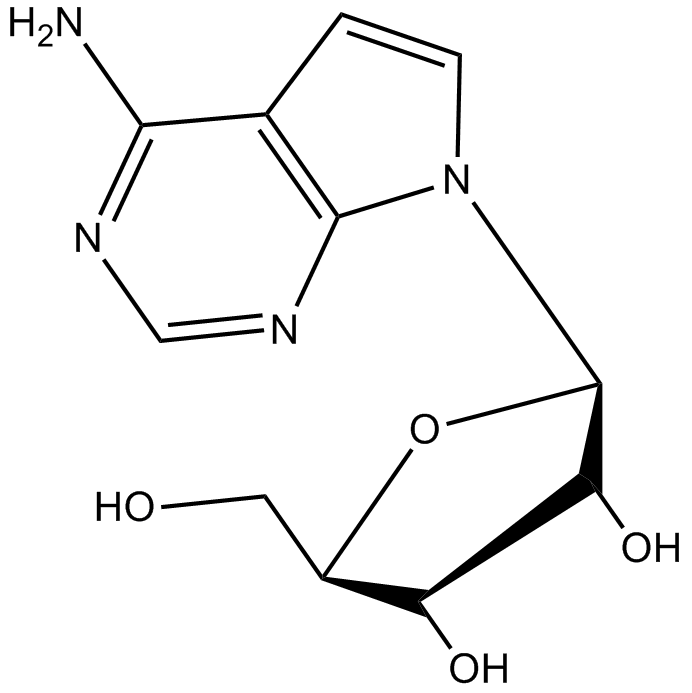
-
GC49835
Tumulosic Acid
TumulosinsÄure, ein Triterpenoid, hemmt die KLK5-ProteaseaktivitÄt (IC50 = 14,84 μ M).
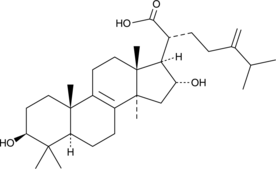
-
GC41598
Tyrocidine Complex
Tyrocidine complex is a mixture of cyclic decapeptides originally isolated from B.
-
GC48987
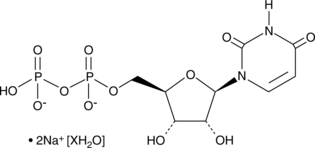
UDP (sodium salt hydrate)
An agonist of P2Y6
-
GC49132
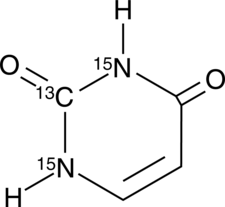
Uracil-13C,15N2
An internal standard for the quantification of uracil
-
GC49800
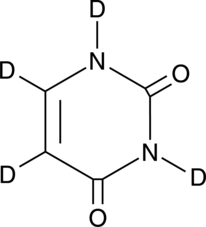
Uracil-d4
An internal standard for the quantification of uracil
-
GC13742
Uridine
Uridin (β-Uridin) ist ein glykosyliertes Pyrimidin-Analogon, das Uracil enthÄlt, das Über eine β-N1-glykosidische Bindung an einen Ribosering (oder genauer gesagt Aribofuranose) gebunden ist.
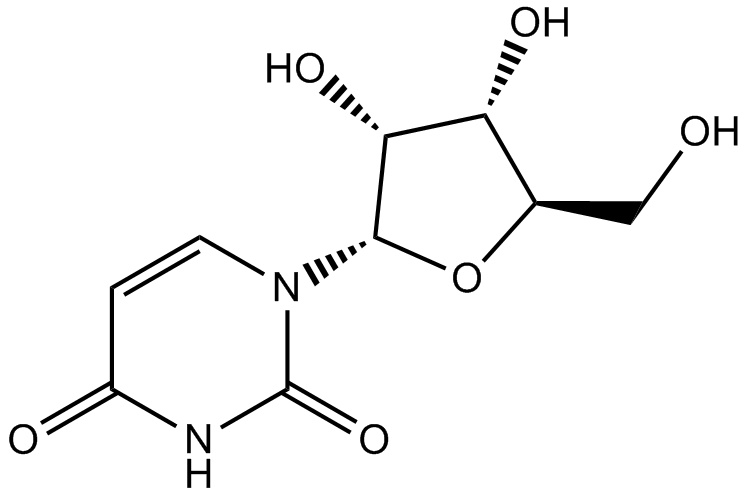
-
GC62546
Uridine triphosphate 13C9,15N2 sodium
Uridintriphosphat 13C9,15N2 (UTP 13C9,15N2) Natrium ist ein markiertes Uridintriphosphat-Natrium.
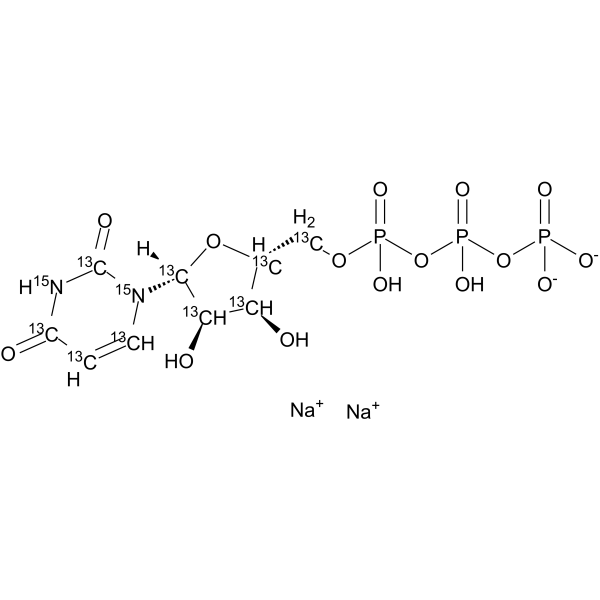
-
GC49297
Uridine-5’-monophosphate (sodium salt hydrate)
A ribonucleotide
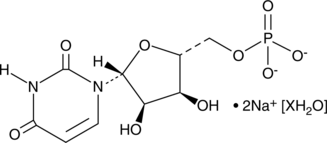
-
GC49528
Uridine-d2
Uridin-d2 ist das mit Uridin bezeichnete Deuterium.
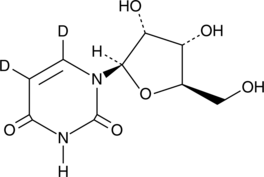
-
GC15168
Urolithin A
Ein Darmmikroben-Metabolit von Ellagsäure.
-
GC10339
VAL-083
-
GC48533
Vanoxonin
A bacterial metabolite
-
GC14851
Vidofludimus
Vidofludimus ist ein oral aktiver Hemmer der Dihydroorotat-Dehydrogenase (DHODH) und auch ein neuartiger Modulator des Farnesoid-X-Rezeptors (FXR).
-
GC14663
Viomycin
Viomycin ist ein starkes Antibiotikum gegen Mykobakterien. Viomycin hemmt schnell die PolypeptidkettenverlÄngerung, wenn es zu gereinigten endogenen Escherichia coli-Polysomen hinzugefÜgt wird, die aktiv an der Polypeptidsynthese beteiligt sind.
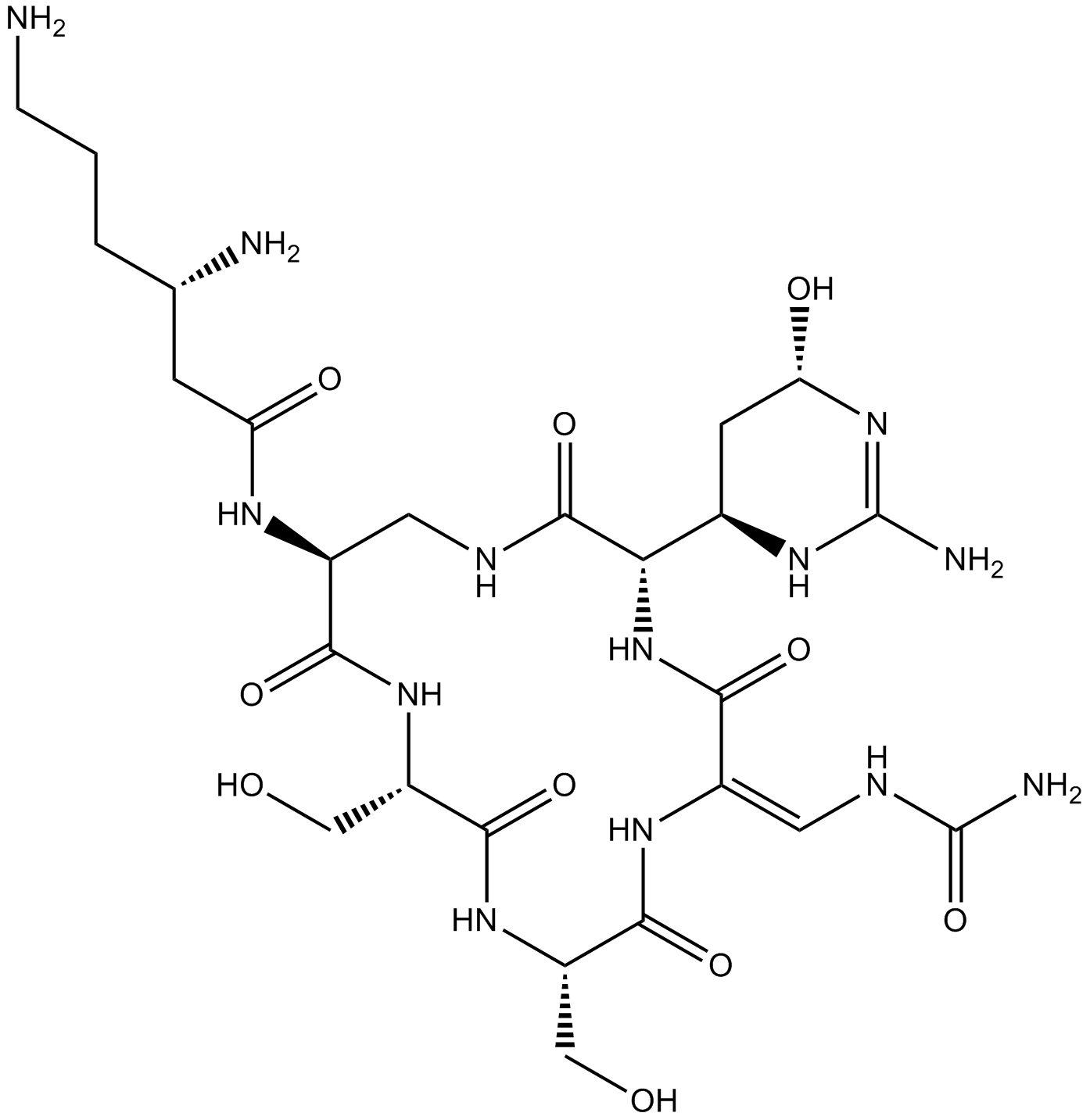
-
GC70142
Werner syndrome RecQ helicase-IN-3
Werner-Syndrom RecQ-Helicase-IN-3 ist ein wirksamer, oral aktiver Inhibitor der Werner-Syndrom RecQ-DNA-Helikase WRN mit einer IC50 von 0,06 µM. Werner-Syndrom RecQ-Helicase-IN-3 zeigt antiproliferative Aktivität und hat eine anti-karzinogene Wirkung.
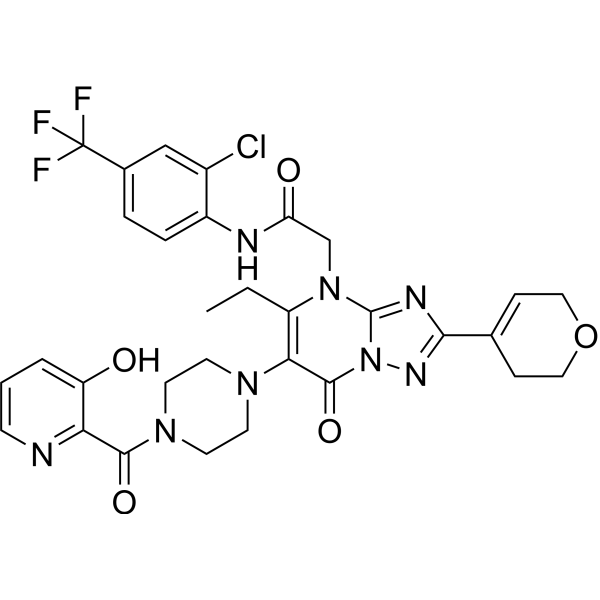
-
GC37940
Xanthopterin
Xanthopterin, eine unkonjugierte Pteridinverbindung, ist der Hauptbestandteil des gelben Granulats in den BÄrenflÜgeln der orientalischen Hornisse und erzeugt ein charakteristisches Anregungs-/Emissionsmaximum bei 386/456 nm.
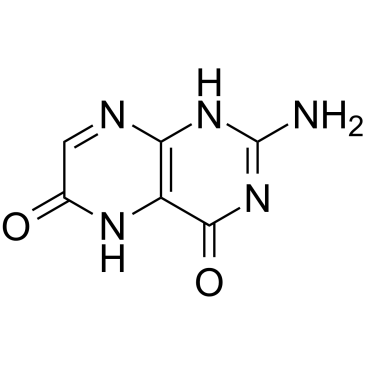
-
GC37941
Xanthopterin (hydrate)
Xanthopterin-Hydrat, eine unkonjugierte Pteridinverbindung, ist der Hauptbestandteil der gelben KÖrnchen in den BÄrenflÜgeln der orientalischen Hornisse und erzeugt ein charakteristisches Anregungs-/Emissionsmaximum bei 386/456 nm.
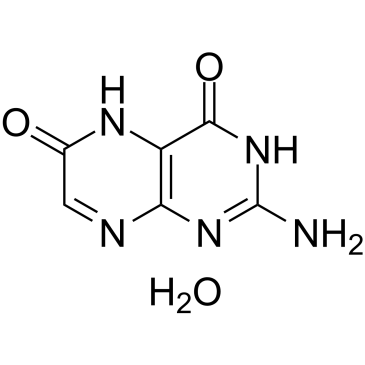
-
GC17386
YK-4-279
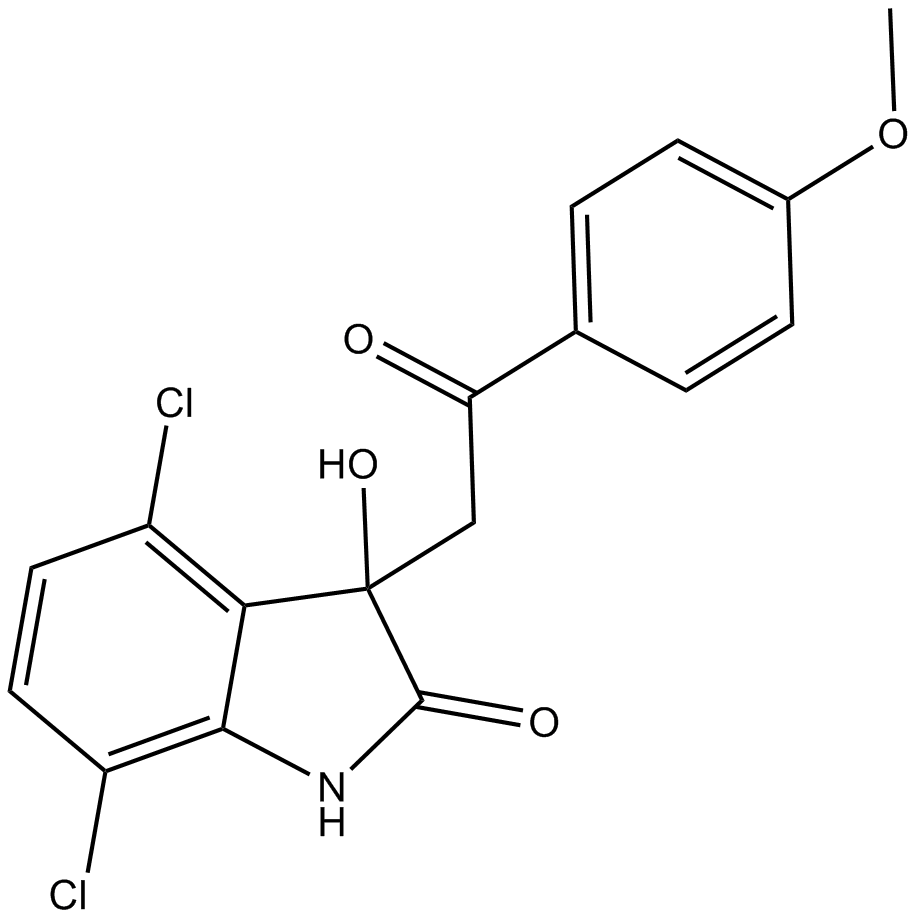
-
GC70172
ZIM
ZIM, ein Derivat von 4-Aminoantipyrin, ist ein wirksamer Induktor von DNA-Schäden, der genomische und chromosomale Schäden verursachen sowie Zelltod und Aktivierung der Phagozytose induzieren kann. ZIM hat das Potenzial zur Chemotherapie und kann in der Krebsforschung eingesetzt werden.
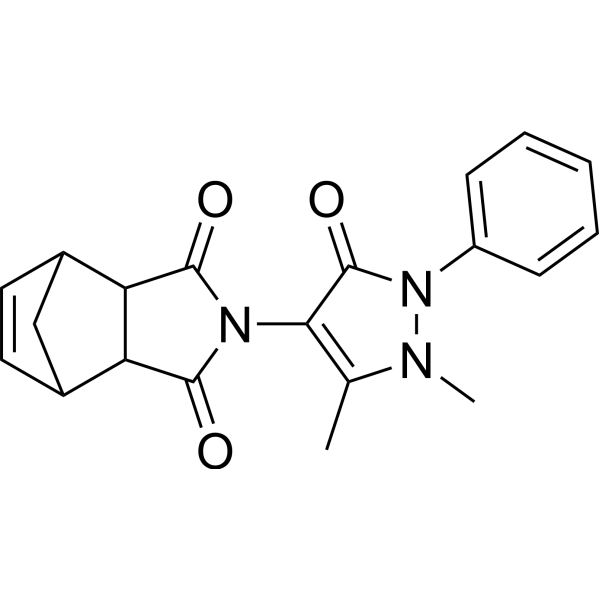
-
GC19391
Zoliflodacin
Zoliflodacin (ETX0914;AZD0914) ist ein neuartiger Spiropyrimidintrion-Gyrase/Topoisomerase-Hemmer aus bakterieller DNA.
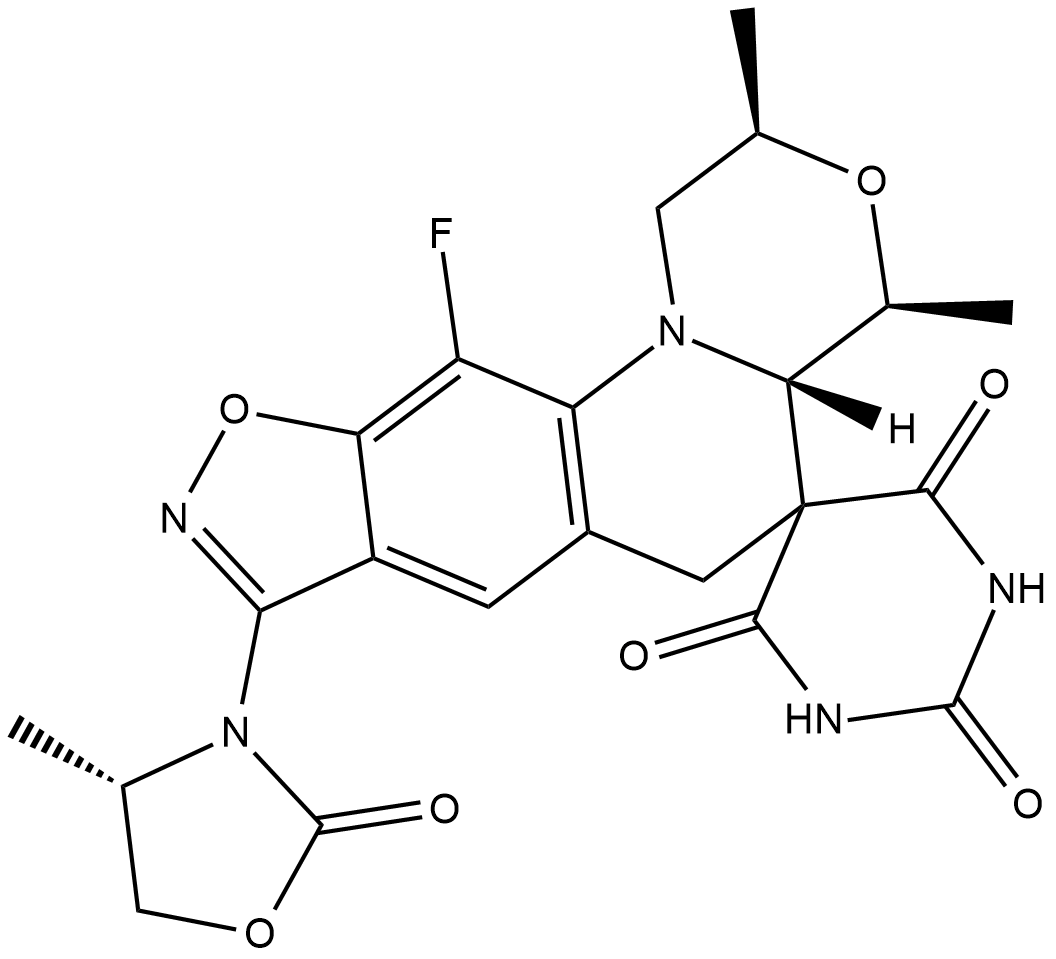
-
GC16277
α-Amanitin
A selective RNA polymerase II inhibitor