Transcription Factors
Produkte für Transcription Factors
- Bestell-Nr. Artikelname Informationen
-
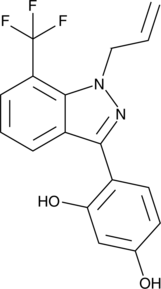
GC48292
α-MSH (human, mouse, rat, porcine, bovine, ovine) (trifluoroacetate salt)
α-MSH (α-Melanozyten-stimulierendes Hormon) TFA, ein endogenes Neuropeptid, ist ein endogener Melanocortinrezeptor 4 (MC4R)-Agonist mit entzÜndungshemmenden und antipyretischen AktivitÄten.
-
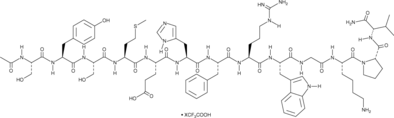
GC52010
(±)-10-hydroxy-12(Z),15(Z)-Octadecadienoic Acid
An oxylipin gut microbiota metabolite
-
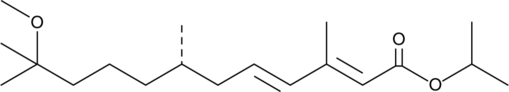
GC46347
(S)-(+)-Methoprene
(S)-Methopren ist ein Juvenilhormon-Analogon, das die Fähigkeit des Insekts verhindert, sich von der Puppe zum Erwachsenen zu verändern, und als Insektizid verwendet wird.
-
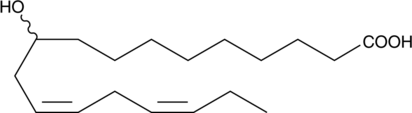
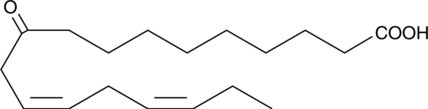
GC52026
10-oxo-12(Z),15(Z)-Octadecadienoic Acid
An oxylipin gut microbiota metabolite
-
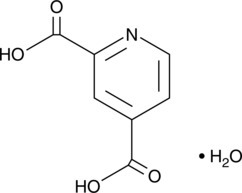
GC42075
2,4-Pyridinedicarboxylic Acid (hydrate)
2,4-Pyridinedicarboxylic Acid (2,4-PDCA) is a compound that structurally mimics 2-oxoglutarate (2-OG, also known as α-ketoglutarate) and chelates zinc, thus affecting a range of enzymes.
-
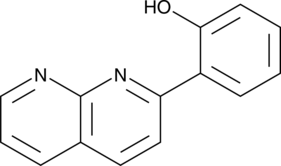
GC46503
2-(1,8-Naphthyridin-2-yl)phenol
2-(1,8-Naphthyridin-2-yl)phenol ist ein selektiver VerstÄrker der STAT1-Transkription. 2-(1,8-Naphthyridin-2-yl)phenol kann die FÄhigkeit von IFN-γ verstÄrken; um die Proliferation menschlicher Brustkrebs- und Fibrosarkomzellen zu hemmen.
-
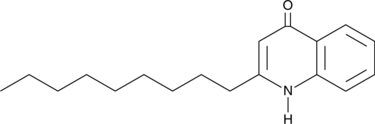
GC46553
2-Nonylquinolin-4(1H)-one
A quinolone alkaloid with diverse biological activities
-
GC52446
2-Nonylquinolin-4(1H)-one-d4
An internal standard for the quantification of 2-nonylquinolin-4(1H)-one

-
GC52129
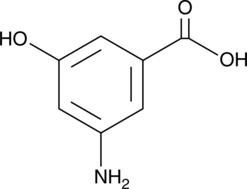
3-Amino-5-hydroxybenzoic Acid
-
GC18194
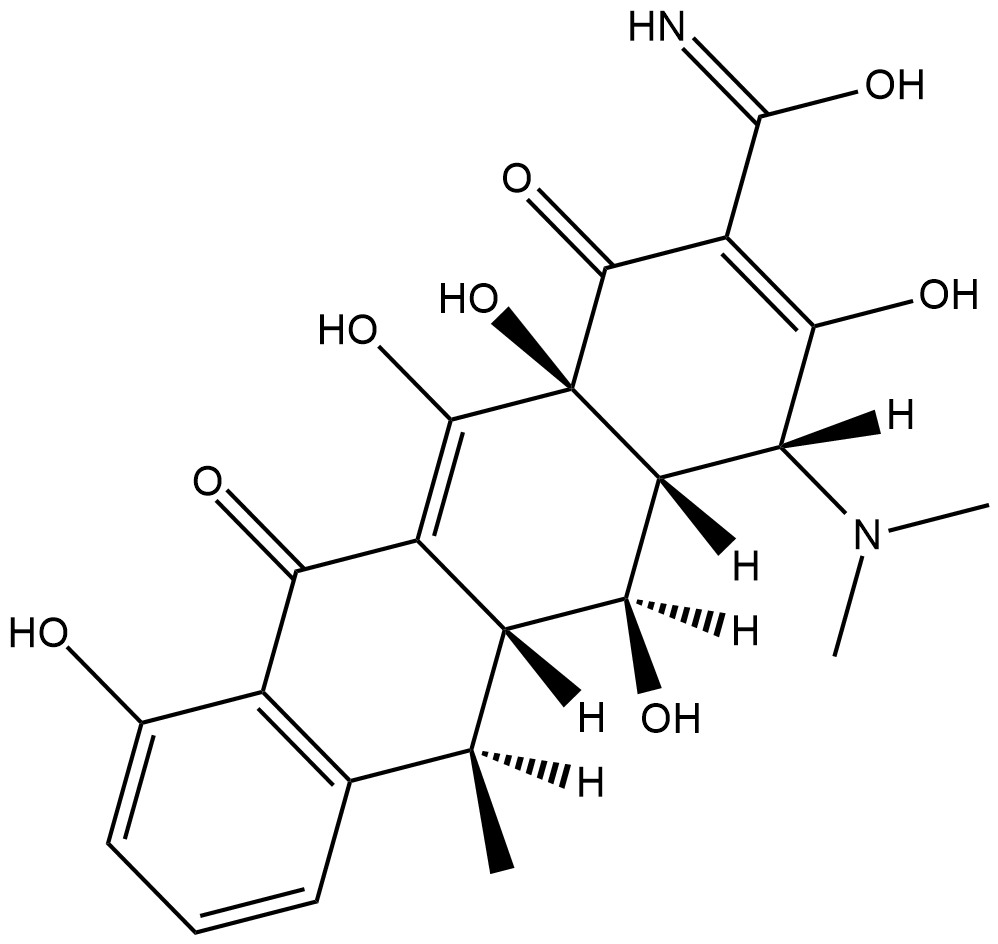
4-Epidoxycycline
4-Epidoxycycline is the 4-epimer hepatic metabolite of the antibiotic doxycycline .
-
GC48824
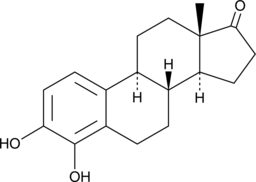
4-hydroxy Estrone
4-Hydroxy-Estron (4-OHE1), ein Estron-Metabolit, hat eine starke neuroprotektive Wirkung gegen oxidative Neurotoxizität.
-
GC46673
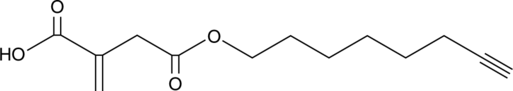
4-octynyl Itaconate
4-Octinyl-Itaconat (ITalk) ist eine spezifische bioorthogonale Sonde fÜr die quantitative und ortsspezifische chemoproteomische Profilierung von Itaconation in lebenden Zellen.
-
GC52413
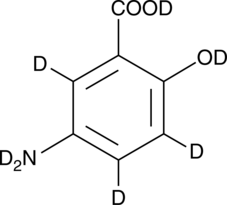
5-Aminosalicylic Acid-d7
An internal standard for the quantification of 5-aminosalicylic acid
-
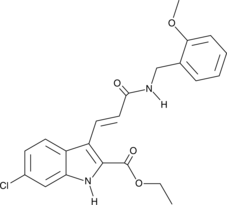
GC45960
9c(i472)
9c(i472) ist ein potenter Inhibitor von 15-LOX-1 (15-Lipoxygenase-1) mit einem IC50-Wert von 0,19 μM.
-
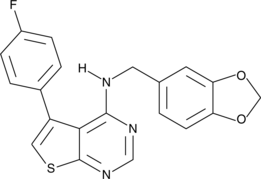
GC42743
AEM1
AEM1 ist ein Nrf2-Inhibitor. AEM1 reduziert die Expression von Nrf2-abhÄngigen Genen in A549-Zellen und hemmt das Wachstum von A549-Zellen in vitro und in vivo.
-
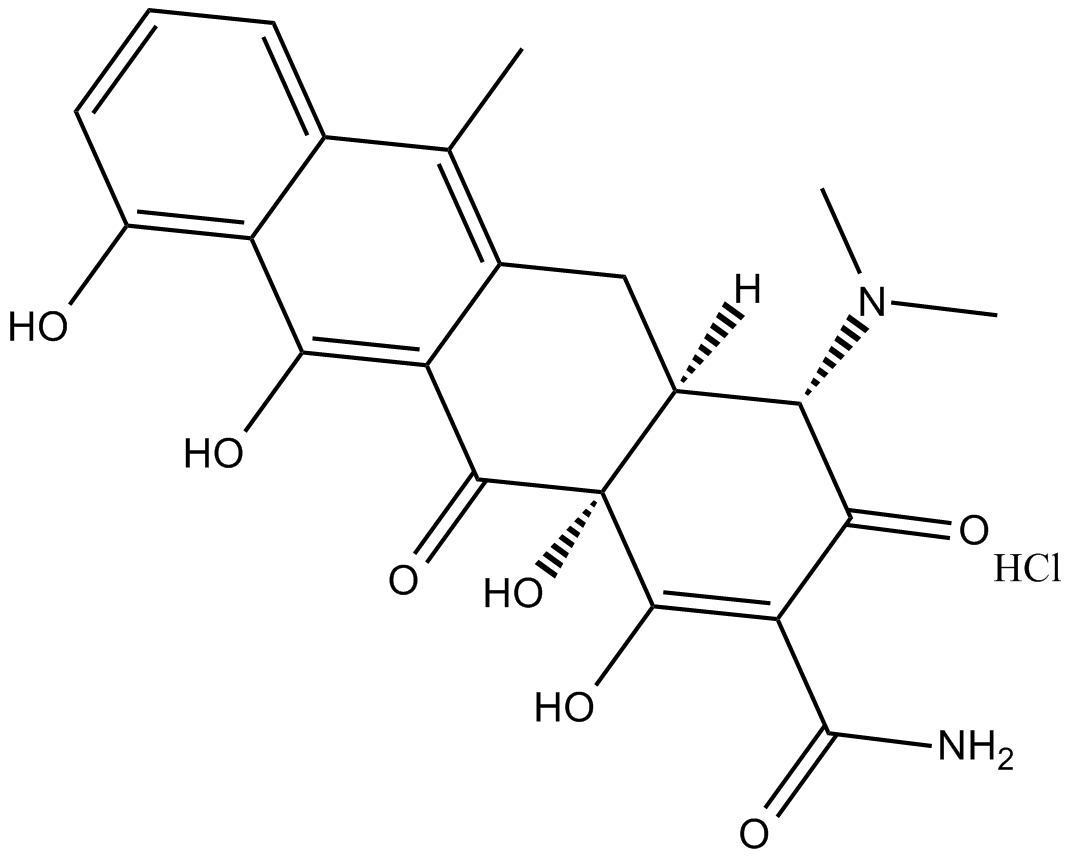
GC16227
Anhydrotetracycline (hydrochloride)
Leistungsstarker Effektor in den Tetracyclin-Repressor (TetR) und Reverse TetR (revTetR) Systemen
-
GC46862
Apigenin-d5
An internal standard for the quantification of apigenin
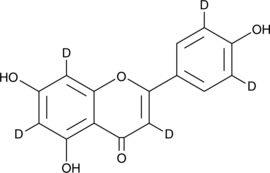
-
GC42880
Avenanthramide-C methyl ester
Avenanthramide-C methyl ester is an inhibitor of NF-κB activation that acts by blocking the phosphorylation of IKK and IκB (IC50 ~ 40 μM).
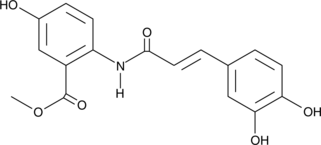
-
GC42925
Berteroin
Berteroin, ein natÜrlich vorkommendes Sulforaphan-Analogon, ist ein antimetastatisches Mittel.
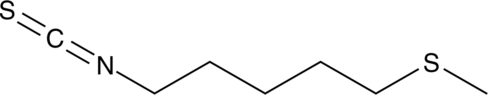
-
GC13231
BI 6015
BI 6015 ist ein Antagonist des Hepatozyten-Kernfaktors 4α (HNF4α), der die Expression bekannter HNF4α-Zielgene hemmen kann.
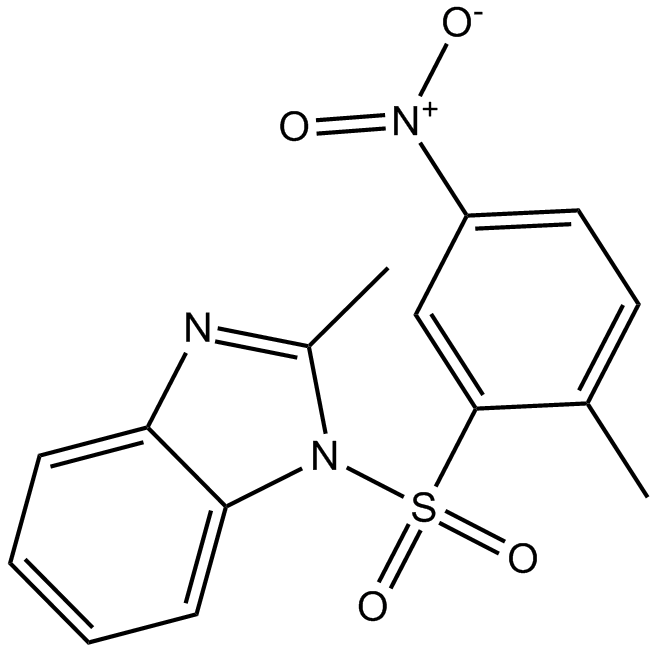
-
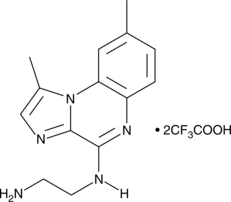
GC42953
BMS 345541 (trifluoroacetate salt)
BMS 345541 is a cell permeable inhibitor of the IκB kinases IKKα and IKKβ (IC50s = 4 and 0.3 μM).
-
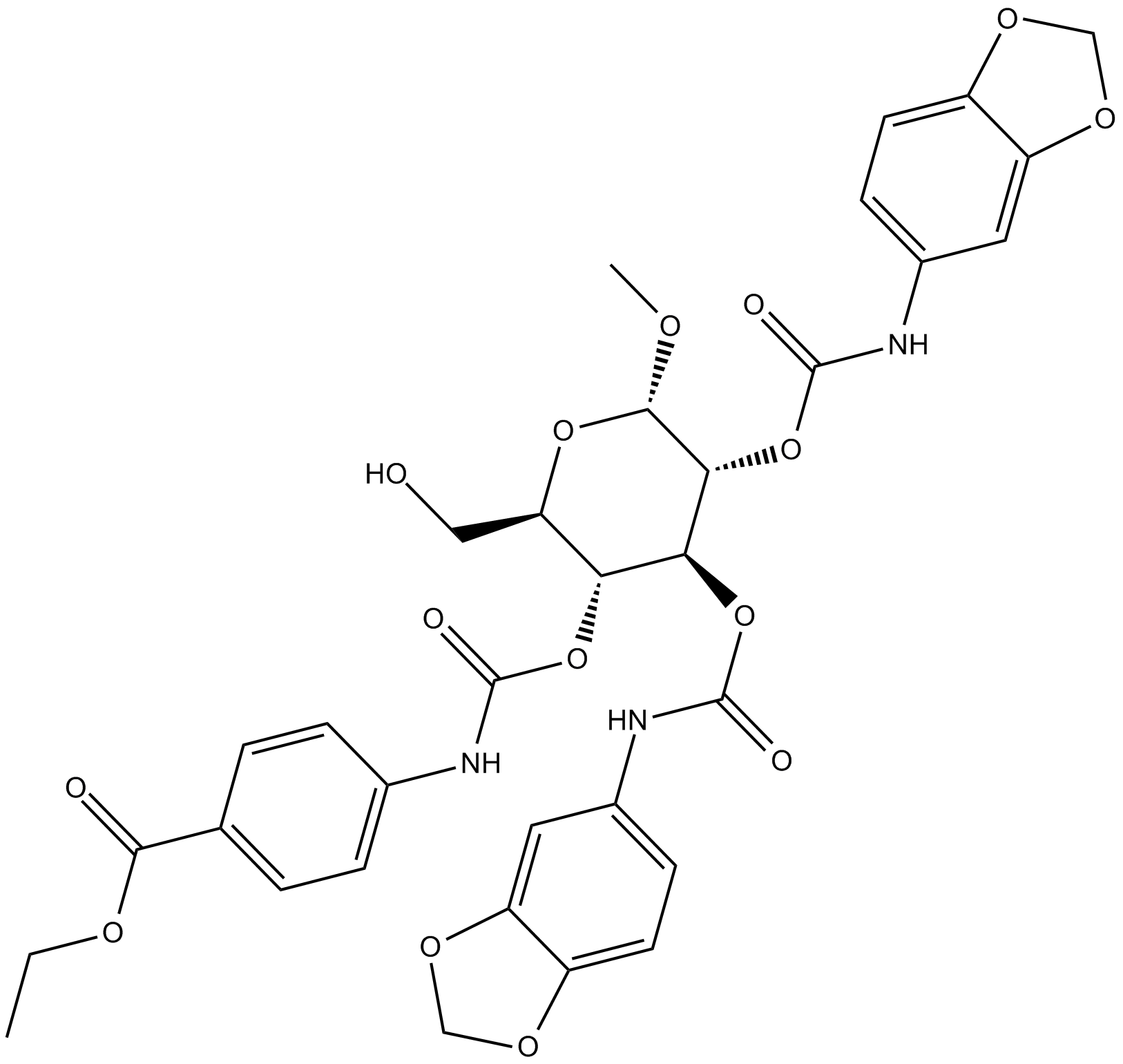
GC10233
BRD 7552
BRD 7552, ein potenter Induktor des PDX1-Transkriptionsfaktors, reguliert die PDX1-Expression sowohl in primären menschlichen Inseln als auch in Gangzellen hoch und induziert epigenetische Veränderungen im PDX1-Promotor, die mit der Transkriptionsaktivierung übereinstimmen.
-
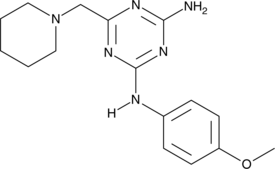
GC42975
BRD32048
BRD32048 ist ein direkter Binder von ETV1 mit einer KD von 17,1 μM. BRD32048 moduliert sowohl die ETV1-vermittelte TranskriptionsaktivitÄt als auch die Invasion von ETV1-gesteuerten Krebszellen. BRD32048 hemmt die ETV1-Acetylierung und fÖrdert deren Abbau. BRD32048 fungiert als Spitzenkandidat fÜr ETV1-StÖrungen.
-
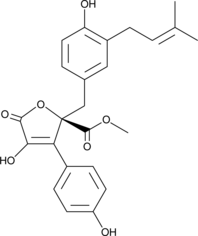
GC42997
Butyrolactone I
Butyrolacton I ist ein ATP-kompetitiver Inhibitor von CDK1 als sekundÄrer Metabolit von A. terreus. Butyrolacton I hat Antitumorwirkungen in nicht-kleinzelligen Lungen-, kleinzelligen Lungen- und Prostatakrebs-Zelllinien.
-
GC43110
C8 Galactosylceramide (d18:1/8:0)
C8 Galactosylceramide is a synthetic C8 short-chain derivative of known membrane microdomain-forming sphingolipids.

-
GC43176
CAY10575
CAY10575 (Verbindung 8) ist ein IKK2-Inhibitor mit einem IC50 von 0,075 μM.
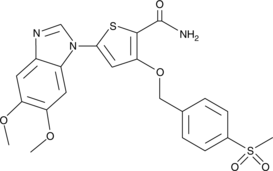
-
GC43189
CAY10681
Inactivation of the tumor suppressor p53 commonly coincides with increased signaling through NF-κB in cancer.
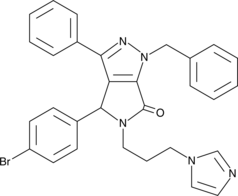
-
GC43190
CAY10682
(±)-Nutlin-3 blocks the interaction of p53 with its negative regulator Mdm2 (IC50 = 90 nM), inducing the expression of p53-regulated genes and blocking the growth of tumor xenografts in vivo.
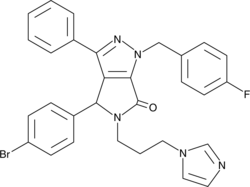
-
GC15474
CBFβ Inhibitor
CBFβ inhibitor
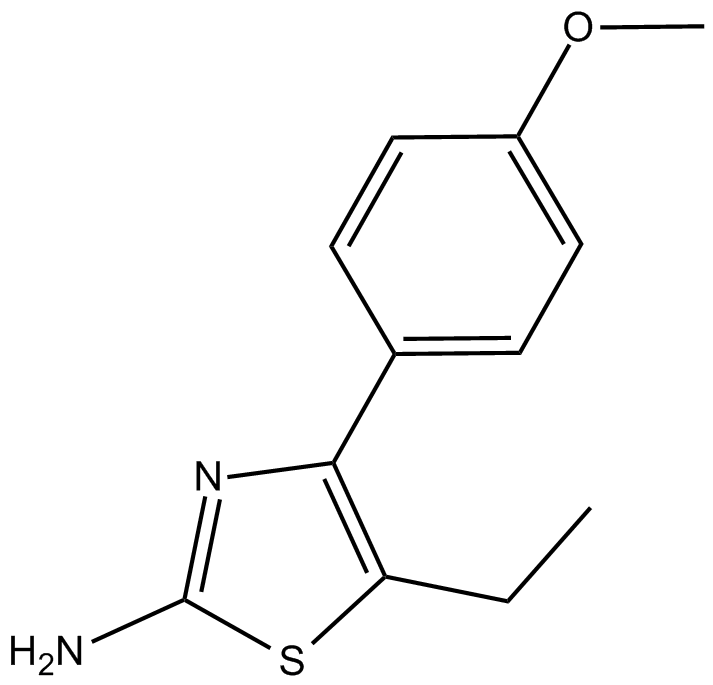
-
GC14266
CCG 203971
CCG 203971 ist ein Inhibitor des Rho/MRTF/SRF-Signalwegs der zweiten Generation.
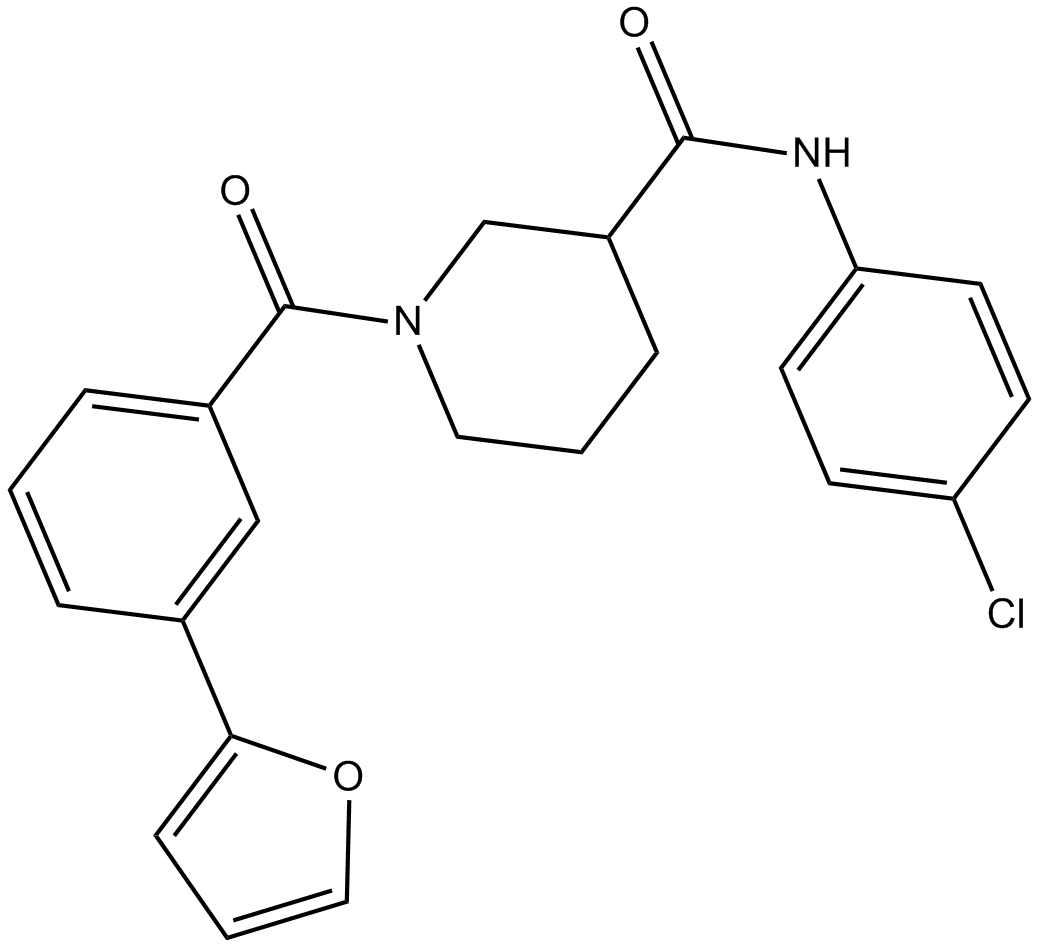
-
GC43212
CCG-232601
CCG-232601 (Verbindung 8f) ist ein potenter und oral aktiver Inhibitor des Rho/MRTF/SRF-Transkriptionswegs.
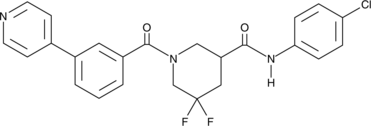
-
GC48982
CD532
CD532 ist ein potenter Aurora-A-Kinase-Inhibitor mit einem IC50 von 45 nM. CD532 hat die doppelte Wirkung, die AktivitÄt der Aurora-A-Kinase zu blockieren und den Abbau von MYCN voranzutreiben. CD532 kann auch direkt mit AURKA interagieren und induziert eine globale KonformationsÄnderung. CD532 kann fÜr die Krebsforschung verwendet werden.
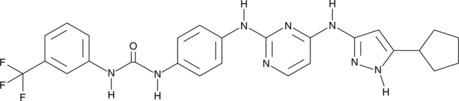
-
GC18392
Cellocidin
Cellocidin is an antibiotic originally isolated from S.
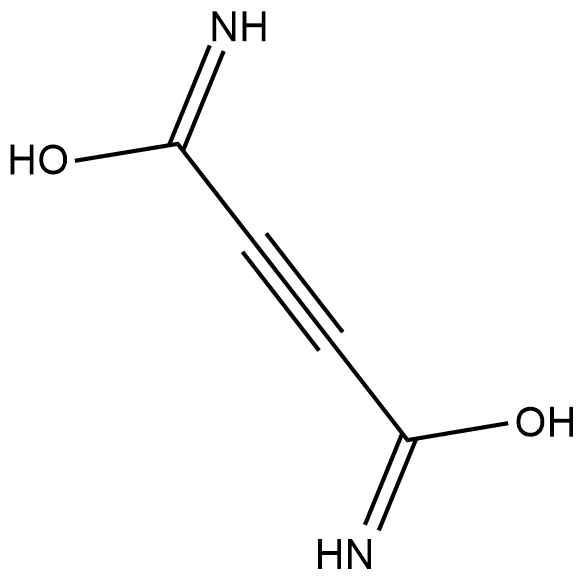
-
GC52081
Chamazulene
Chamazulen, eine natÜrliche Verbindung, ist ein antioxidativer Inhibitor der Leukotrien-B4-Bildung.
-
GC14685
CID 5951923
CID 5951923 ist ein potenter Inhibitor des KrÜppel-like Factor 5 (KLF5) mit einem IC50 von 603 nM. CID 5951923 kann die Proliferation von Krebszellen in vitro hemmen.
-
GC43368
D,L-1′-Acetoxychavicol Acetate
D,L-1′-Acetoxychavicol acetate is a natural compound first isolated from the rhizomes of ginger-like plants.
-
GC52194
Dimethylamino Parthenolide
-
GC43493
DL-Sulforaphane N-acetyl-L-cysteine
Nrf2 activation of the antioxidant response element (ARE) is central to cytoprotective gene expression against oxidative and/or electrophilic stress.
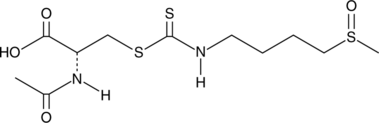
-
GC46148
Filgotinib-d4
Filgotinib-d4 (GLPG0634-d4) ist das mit Deuterium gekennzeichnete Filgotinib. Filgotinib (GLPG0634) ist ein selektiver JAK1-Inhibitor mit IC50-Werten von 10 nM, 28 nM, 810 nM und 116 nM fÜr JAK1, JAK2, JAK3 bzw. TYK2.
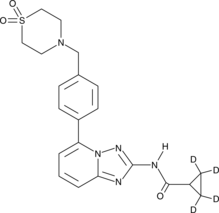
-
GC49344
Fisetin-d5
An internal standard for the quantification of fisetin
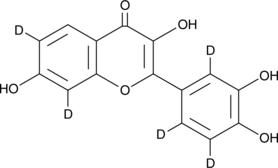
-
GC18652
FQI 1
An inhibitor of Late SV40 Factor
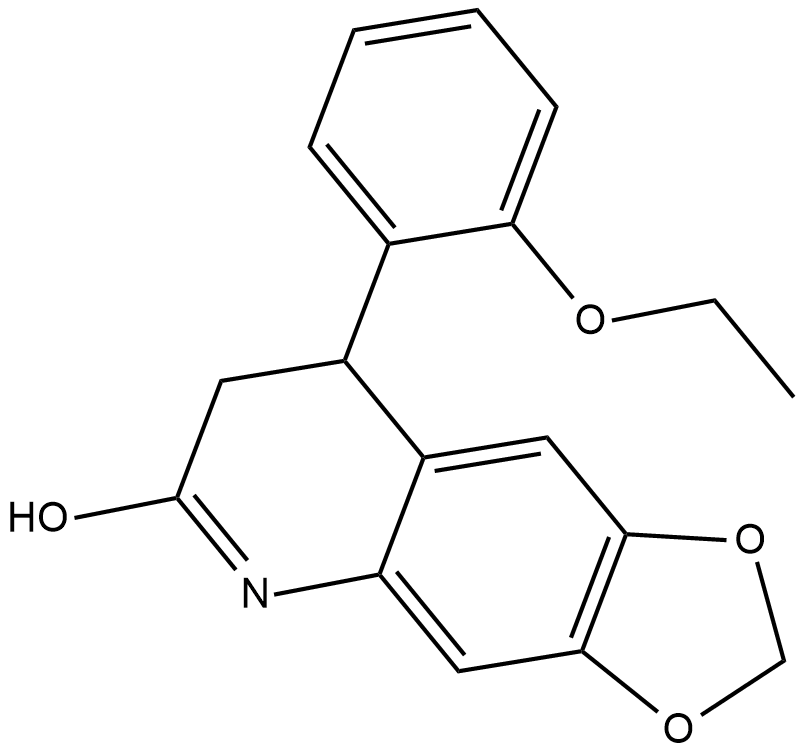
-
GC18796
Herbicidin A
Herbicidin A is an adenine nucleoside antibiotic originally isolated from S.
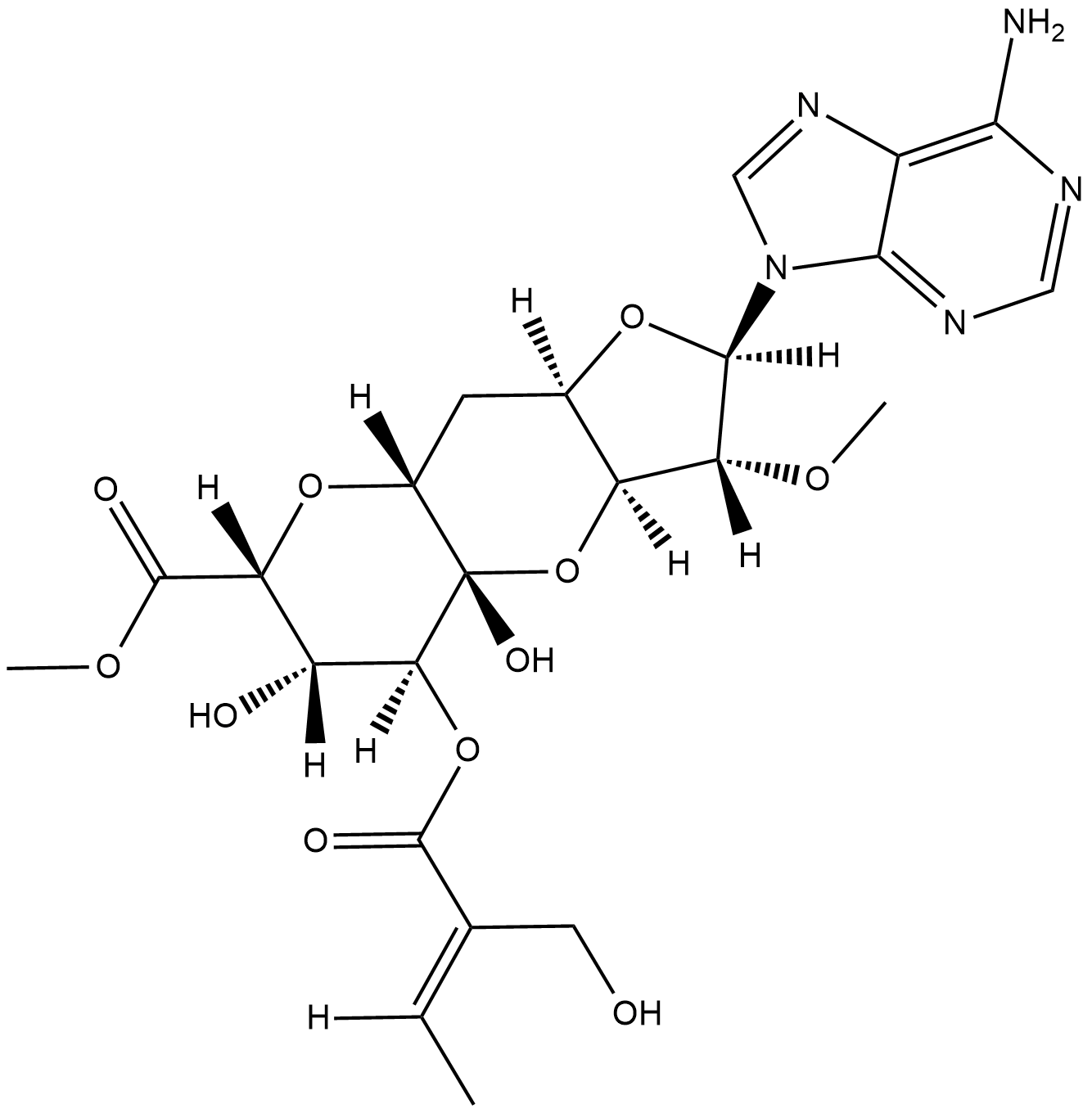
-
GC47435
HPI-1 (hydrate)
A Hedgehog pathway inhibitor
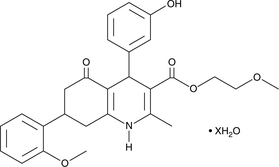
-
GC49742
Hsf1 Monoclonal Antibody (Clone 10H8)
For immunochemical analysis of Hsf1

-
GC43893
iKIX1
iKIX1 ist ein Antimykotikum und sensibilisiert arzneimittelresistente C.
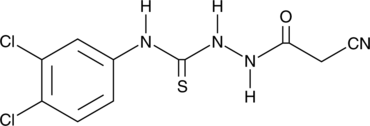
-
GC43894
IKK2 Inhibitor VI
Inhibitor of NF-κB kinase 2 (IKK2, also known as IKKβ) acts as part of an IKK complex in the canonical NF-κB pathway, phosphorylating inhibitors of NF-κB (IκBs) to initiate signaling.
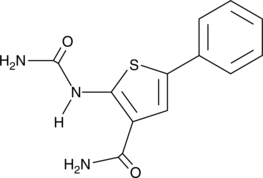
-
GC47456
Indole-3-pyruvic Acid
Indol-3-BrenztraubensÄure, ein Keto-Analogon von Tryptophan, ist ein oral aktiver AHR-Agonist.
-
GC49290
Indoxyl Sulfate-d5 (potassium salt)
An internal standard for the quantification of indoxyl sulfate
-
GC48618
Isonanangenine B
A drimane sesquiterpene lactone
-
GC11197
KL 001
KL 001 ist ein erstklassiger Cryptochrom-Stabilisator (CRY, ein Flavoprotein, das für blaues Licht empfindlich ist und am zirkadianen Rhythmus von Pflanzen und Tieren beteiligt ist), der spezifisch mit CRY1 und CRY2 interagiert.
-
GC52283
L-Cysteine-15N-d3
An internal standard for the quantification of L-cysteine
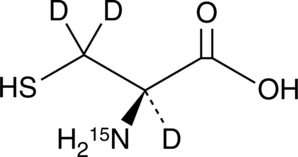
-
GC48907
Metaxalone-d6
An internal standard for the quantification of metaxalone
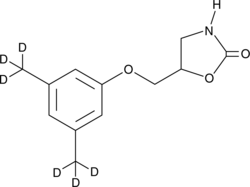
-
GC49241
Methyl Diethyldithiocarbamate
An active metabolite of disulfiram
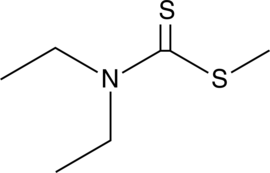
-
GC44216
ML-180
ML-180 (SR1848) ist ein potenter inverser Agonist des Kernrezeptor-Leberrezeptor-Homologs 1 (LRH-1; NR5A2) mit einem IC50-Wert von 3,7 μM. ML-180 ist fÜr den steroidogenen Faktor-1 (SF-1; NR5A1; IC50 > 10 μM) inaktiv. ML-180 hat das Potenzial fÜr LRH-1-abhÄngige Krebsarten.
-
GC11307
ML-264
ML-264 ist ein Antitumormittel, das die Expression des Krüppel-ähnlichen Faktors fünf (KLF5) wirksam und selektiv hemmt.
-
GC14832
N-3-oxo-octanoyl-L-Homoserine lactone
N-3-Oxo-Octanoyl-L-Homoserinlacton, ein Quorum-Sensing-Signal, ist ein Agrobacterium-Autoinducer.
-
GC44459
N-pentadecanoyl-L-Homoserine lactone
Quorum sensing is a regulatory system used by bacteria for controlling gene expression in response to increasing cell density.
-
GC44388
NF-κB Control
NF-κB inhibitor is a synthetic peptide corresponding to the nuclear localization sequence (NLS) of NF-κB p105 subunit (also known as p50) appended to a hydrophobic sequence to facilitate import into living cells.
-
GC48985
NF-κB Inhibitor (trifluoroacetate salt)
A cell-permeable peptide that blocks nuclear import of NF-κB

-
GC12499
O4I1
O4I1 ist ein potenter Oct3/4-Induktor.
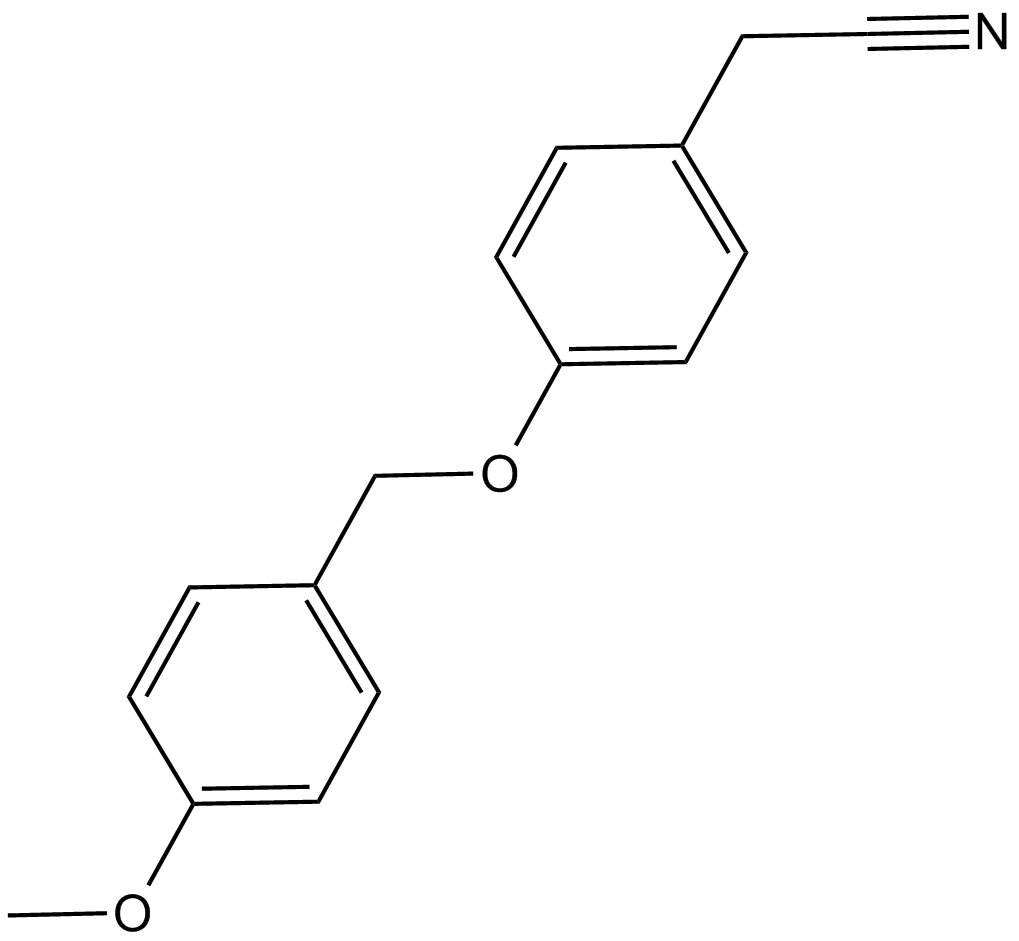
-
GC10151
OAC1
OAC1 ist ein potenter Oct4-Aktivator. OAC1 aktiviert Oct4- und Nanog-Promotoren und verstÄrkt die Bildung induzierter pluripotenter Stammzellen (iPSC). OAC1 aktiviert OCT4 durch Hochregulierung der HOXB4-Expression. OAC1 erhÖht die Transkription der Triade Oct4-Nanog-Sox2 und Tet1. OAC1 erleichtert die Umprogrammierung von Zellen, indem es die Effizienz erhÖht und die Umprogrammierungszeit verkÜrzt.
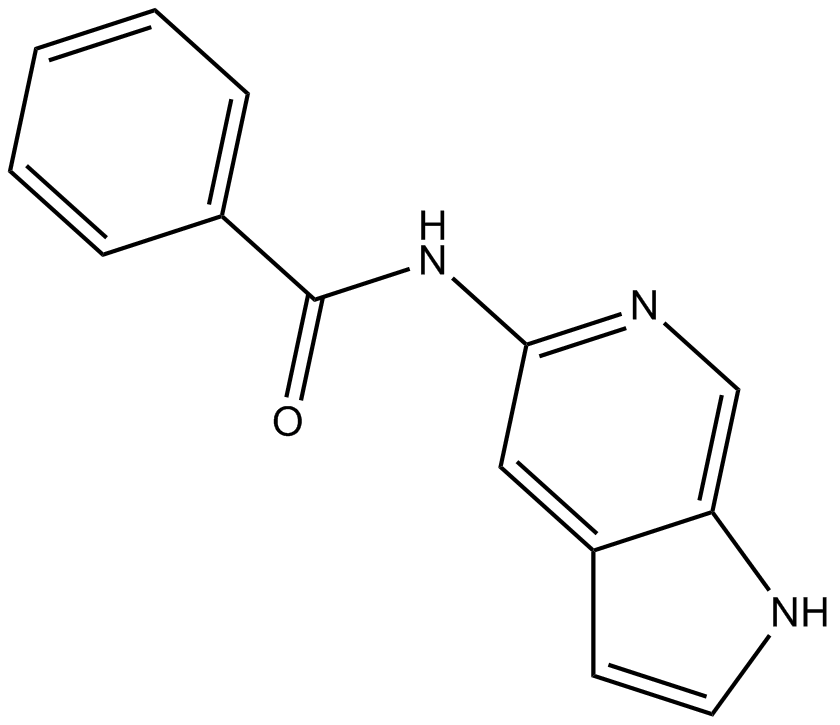
-
GC17327
OAC2
OAC2 ist eine Oct4-aktivierende Verbindung, die die Expression durch den Oct4-Genpromotor aktiviert.
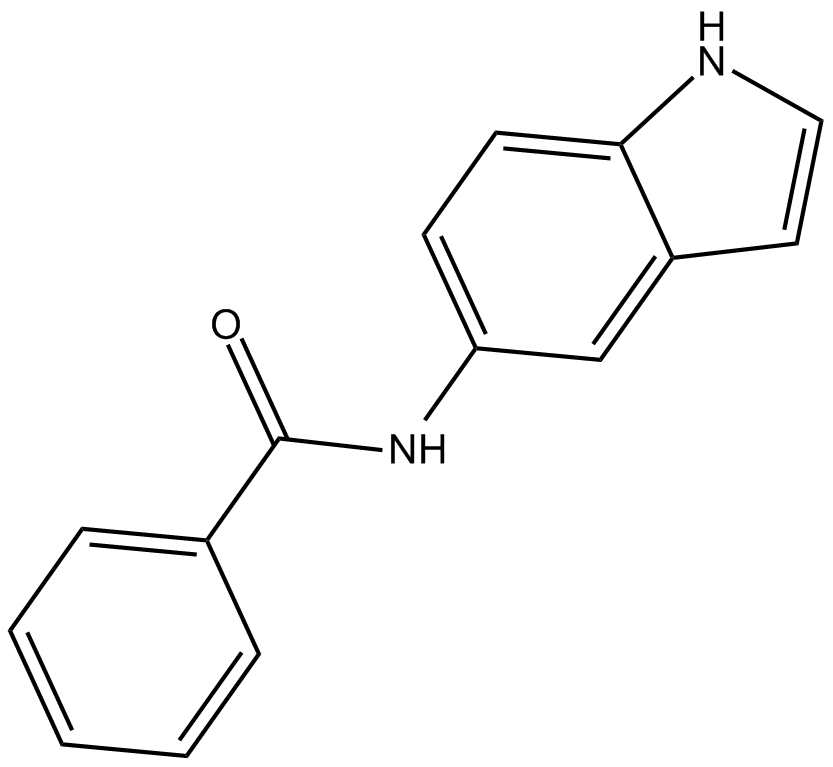
-
GC10724
OAC3
Oct4 activator
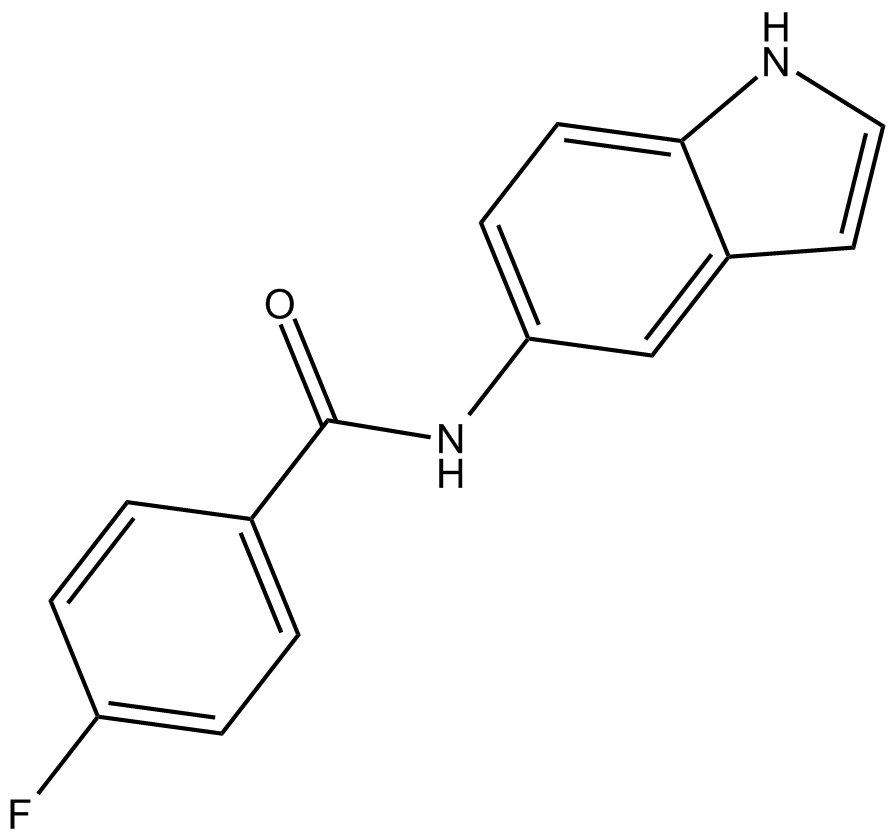
-
GC44652
PK7242 (maleate)
The protein p53, often called the 'guardian of the genome,' is a transcription factor that is activated in response to cellular stress (low oxygen levels, heat shock, DNA damage, etc.) and acts to prevent further proliferation of the stressed cell by promoting cell cycle arrest or apoptosis.
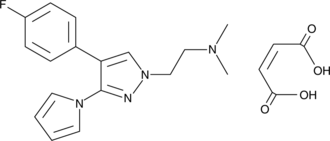
-
GC49860
Pyropheophorbide a methyl ester
Pyropheophorbide a methyl ester (Pyropheophorbide-a methyl ester), ein Chlorophyll-a-Derivat, ist ein starker Photosensibilisator, der in der photodynamischen Therapie (PDT) von Krebs eingesetzt werden kann. Pyropheophorbid, ein Methylester, hat eine photodynamische AktivitÄt und kann Apoptose induzieren und das Tumorwachstum hemmen.

-
GC50256
RG 102240
Gene switch ligand for use in inducible gene expression systems
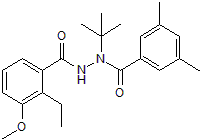
-
GC12727
Ro 5-3335
Ro 5-3335, ein Benzodiazepin, wirkt als Inhibitor der Core-Binding-Factor (CBF)-Leukämie. Ro 5-3335 ist ein Inhibitor der RUNX1-CBFβ-Interaktion, der die RUNX1/CBFB-abhängige Transaktivierung unterdrückt.
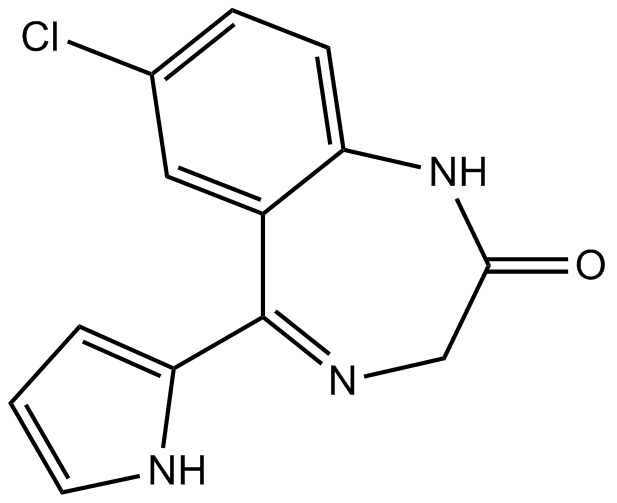
-
GC18624
Roslin-2
Roslin-2 (Benzylhexamethylentetraminbromid) ist ein p53-Reaktivator mit Antikrebswirkung. Roslin-2 bindet FAK, unterbricht die Bindung von FAK und p53.
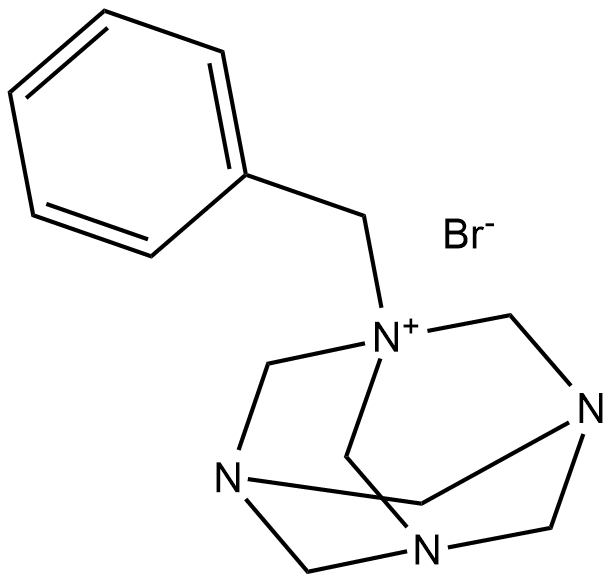
-
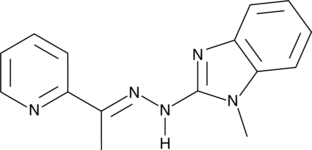
GC44888
SI-2
SI-2 is an inhibitor of steroid receptor coactivator 3 (SRC-3).
-
GC49002
Sinigrin (hydrate)
Sinigrin (Hydrat) ist ein natürliches aliphatisches Glucosinolat, das in Pflanzen der Familie Brassicaceae vorkommt.
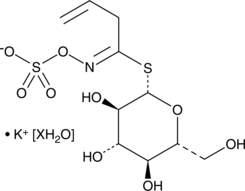
-
GC49049
SMU127
An agonist of TLR1/2
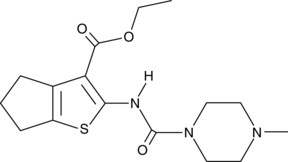
-
GC44943
sPLA2 Inhibitor
Der sPLA2-Inhibitor, ein D-Tyrosin-Derivat, ist ein oral wirksamer, potenter Hemmer der sekretorischen Phospholipase A2 (sPLA2) mit einem IC50-Wert von 29 nM fÜr die humane nichtpankreatische sekretorische PLA2-Isoform IIa (hnpsPLA2-IIa).
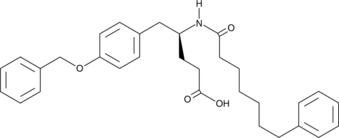
-
GC48098
SR 12343
An IKKβ NBD mimetic
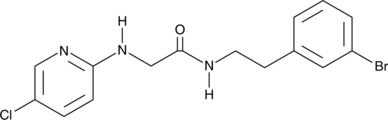
-
GC48099
SR 12460
An IKKβ NBD mimetic
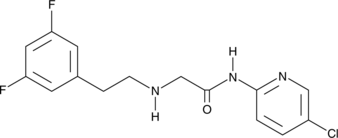
-
GC17839
Stauprimide
Stauprimid ist ein Staurosporin-Analogon, das die Differenzierung embryonaler Stammzellen (ESC) fÖrdert. Stauprimid ist ein Nicht-Breitspektrum-Inhibitor, der an den MYC-Transkriptionsfaktor NME2 bindet und dessen Kernlokalisierung in ESCs blockiert, was zu einer Herunterregulierung der MYC-Transkription fÜhrt.
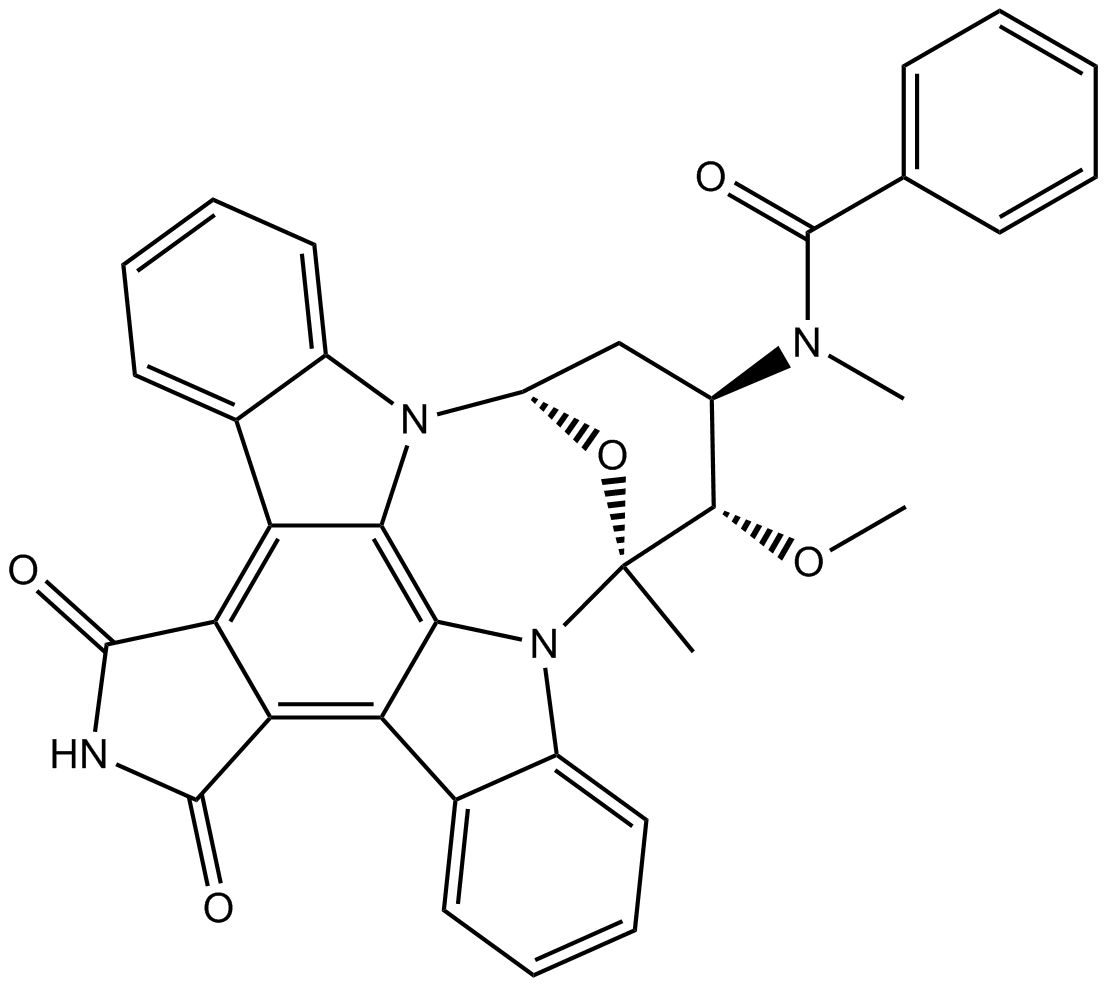
-
GC16165
T-5224
T-5224 ist ein nicht-peptidisches kleines Molekül-AP-1-Inhibitor.

-
GC44984
TAF 10 Peptide
TAF10 is one of many protein factors or coactivators associated with RNA polymerase II activity.

-
GC49692
Tofacitinib-d3 (citrate)
An internal standard for the quantification of tofacitinib
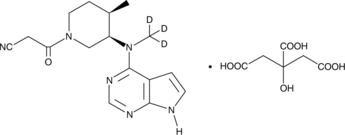
-
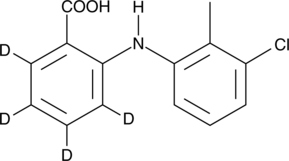
GC45965
Tolfenamic Acid-d4
An internal standard for the quantification of tolfenamic acid
-
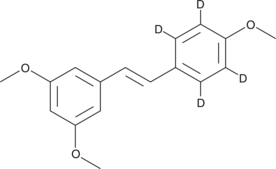
GC48195
trans-trismethoxy Resveratrol-d4
An internal standard for the quantification of trans-trismethoxy resveratrol
-
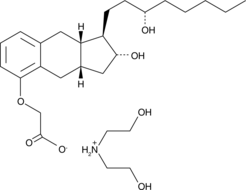
GC48202
Treprostinil (diethanolamine salt)
Treprostinil (UT-15C) Diethanolamin ist ein potenter EP2-, DP1- und IP-Agonist mit Ki-Werten von 3,6, 4,4, 32,1, 212, 826, 2505 und 4680 nM fÜr EP2, DP1, IP, EP1, EP4, EP3 bzw. FP .
-
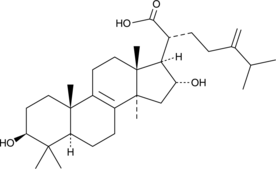
GC49835
Tumulosic Acid
TumulosinsÄure, ein Triterpenoid, hemmt die KLK5-ProteaseaktivitÄt (IC50 = 14,84 μ M).
-
GC52180
WAY-169916
WAY-169916 ist ein Signalweg-selektiver Ligand des ER (Östrogenrezeptor), der durch Hemmung der TranskriptionsaktivitÄt von NF-kB wirkt.